
Date: 2019-12-25 21:23:31 CET, cola version: 1.3.2
Document is loading... 
All available functions which can be applied to this res_list object:
res_list
#> A 'ConsensusPartitionList' object with 24 methods.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows are extracted by 'SD, CV, MAD, ATC' methods.
#> Subgroups are detected by 'hclust, kmeans, skmeans, pam, mclust, NMF' method.
#> Number of partitions are tried for k = 2, 3, 4, 5, 6.
#> Performed in total 30000 partitions by row resampling.
#>
#> Following methods can be applied to this 'ConsensusPartitionList' object:
#> [1] "cola_report" "collect_classes" "collect_plots" "collect_stats"
#> [5] "colnames" "functional_enrichment" "get_anno_col" "get_anno"
#> [9] "get_classes" "get_matrix" "get_membership" "get_stats"
#> [13] "is_best_k" "is_stable_k" "ncol" "nrow"
#> [17] "rownames" "show" "suggest_best_k" "test_to_known_factors"
#> [21] "top_rows_heatmap" "top_rows_overlap"
#>
#> You can get result for a single method by, e.g. object["SD", "hclust"] or object["SD:hclust"]
#> or a subset of methods by object[c("SD", "CV")], c("hclust", "kmeans")]
The call of run_all_consensus_partition_methods() was:
#> run_all_consensus_partition_methods(data = mat, mc.cores = 4, anno = anno)
Dimension of the input matrix:
mat = get_matrix(res_list)
dim(mat)
#> [1] 51941 154
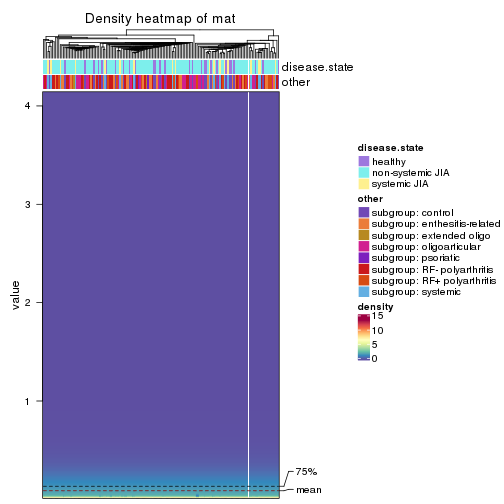
The density distribution for each sample is visualized as in one column in the following heatmap. The clustering is based on the distance which is the Kolmogorov-Smirnov statistic between two distributions.
library(ComplexHeatmap)
densityHeatmap(mat, top_annotation = HeatmapAnnotation(df = get_anno(res_list),
col = get_anno_col(res_list)), ylab = "value", cluster_columns = TRUE, show_column_names = FALSE,
mc.cores = 4)
Folowing table shows the best k (number of partitions) for each combination
of top-value methods and partition methods. Clicking on the method name in
the table goes to the section for a single combination of methods.
The cola vignette explains the definition of the metrics used for determining the best number of partitions.
suggest_best_k(res_list)
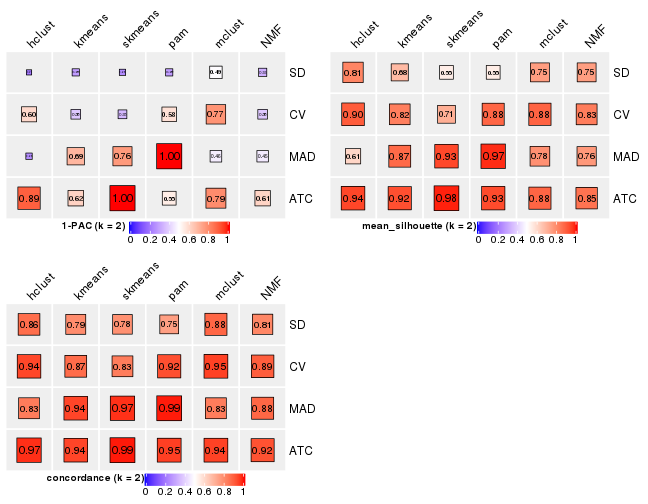
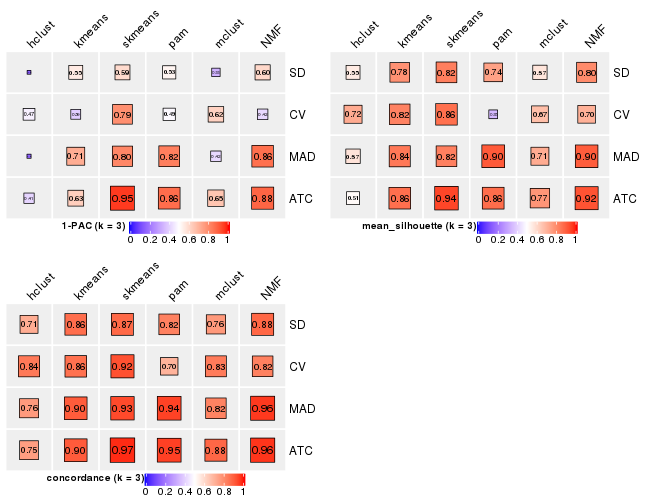
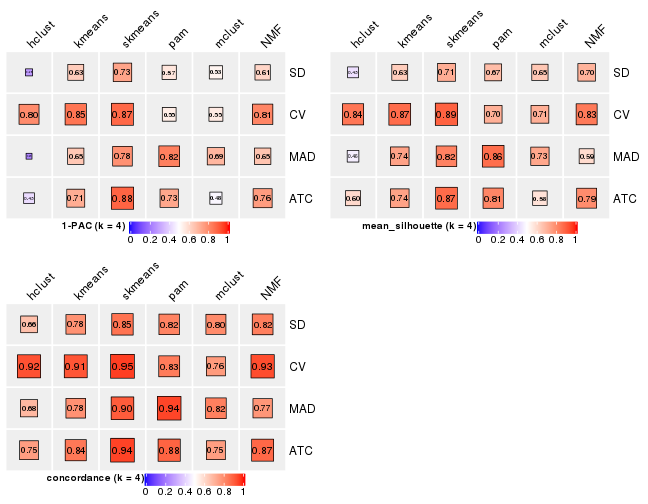
| The best k | 1-PAC | Mean silhouette | Concordance | Optional k | ||
|---|---|---|---|---|---|---|
| MAD:pam | 2 | 1.000 | 0.972 | 0.987 | ** | |
| ATC:skmeans | 3 | 0.955 | 0.937 | 0.974 | ** | 2 |
| ATC:NMF | 3 | 0.880 | 0.916 | 0.962 | ||
| CV:skmeans | 4 | 0.873 | 0.887 | 0.949 | ||
| ATC:pam | 3 | 0.863 | 0.864 | 0.946 | ||
| MAD:NMF | 3 | 0.858 | 0.897 | 0.956 | ||
| CV:kmeans | 4 | 0.847 | 0.865 | 0.912 | ||
| CV:NMF | 4 | 0.810 | 0.835 | 0.926 | ||
| CV:hclust | 4 | 0.795 | 0.843 | 0.918 | ||
| ATC:mclust | 2 | 0.793 | 0.880 | 0.944 | ||
| CV:mclust | 2 | 0.775 | 0.884 | 0.945 | ||
| MAD:skmeans | 2 | 0.760 | 0.933 | 0.966 | ||
| SD:pam | 6 | 0.703 | 0.688 | 0.825 | ||
| MAD:kmeans | 2 | 0.689 | 0.870 | 0.938 | ||
| MAD:mclust | 4 | 0.689 | 0.728 | 0.822 | ||
| CV:pam | 5 | 0.660 | 0.685 | 0.854 | ||
| ATC:kmeans | 3 | 0.629 | 0.859 | 0.900 | ||
| SD:NMF | 3 | 0.603 | 0.797 | 0.877 | ||
| SD:skmeans | 3 | 0.594 | 0.823 | 0.873 | ||
| SD:kmeans | 3 | 0.554 | 0.781 | 0.861 | ||
| SD:mclust | 2 | 0.494 | 0.752 | 0.877 | ||
| ATC:hclust | 5 | 0.476 | 0.560 | 0.763 | ||
| MAD:hclust | 3 | 0.164 | 0.574 | 0.758 | ||
| SD:hclust | 3 | 0.151 | 0.554 | 0.714 |
**: 1-PAC > 0.95, *: 1-PAC > 0.9
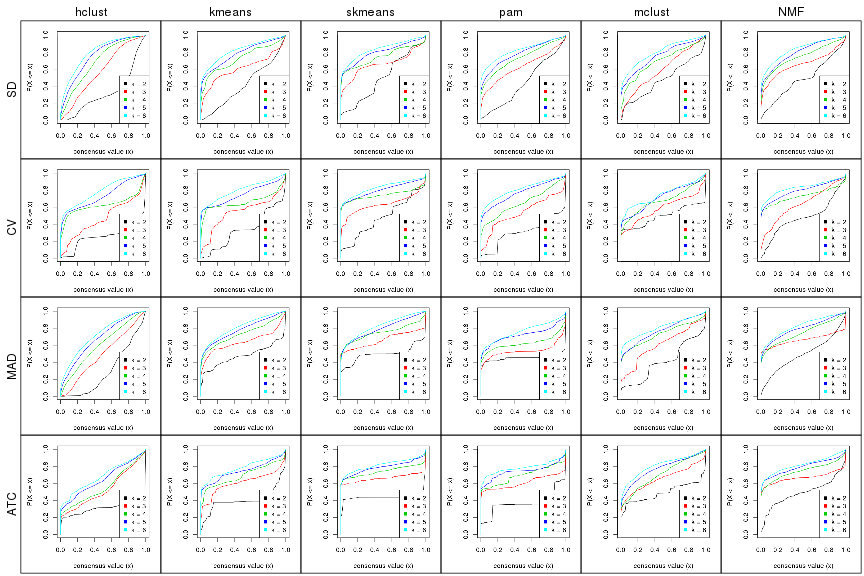
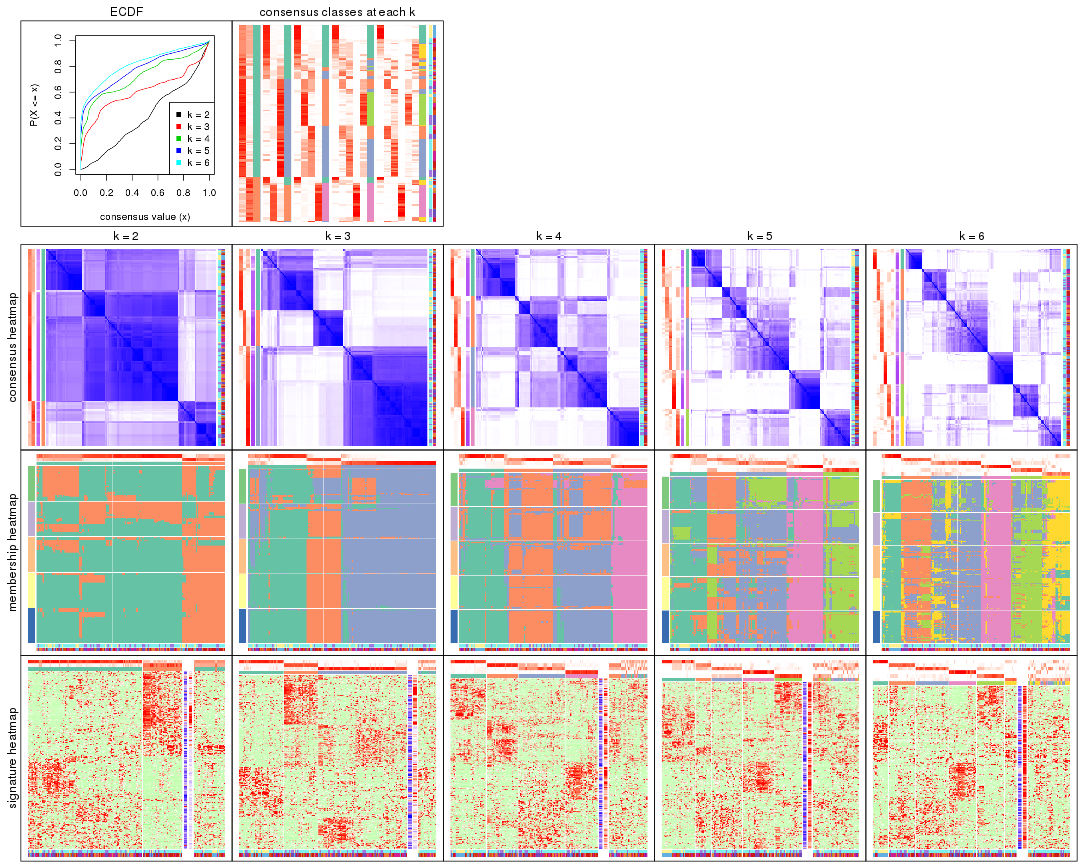
Cumulative distribution function curves of consensus matrix for all methods.
collect_plots(res_list, fun = plot_ecdf)
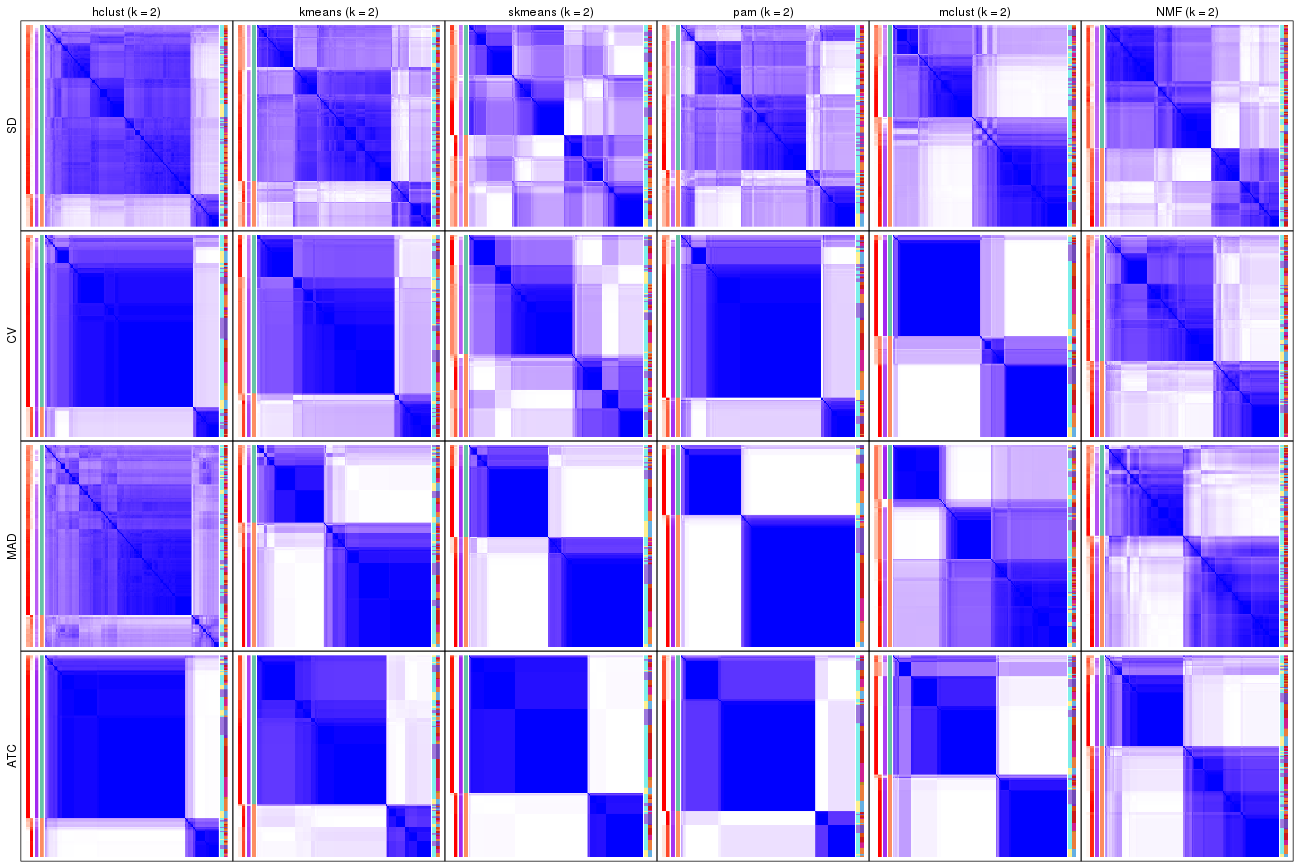
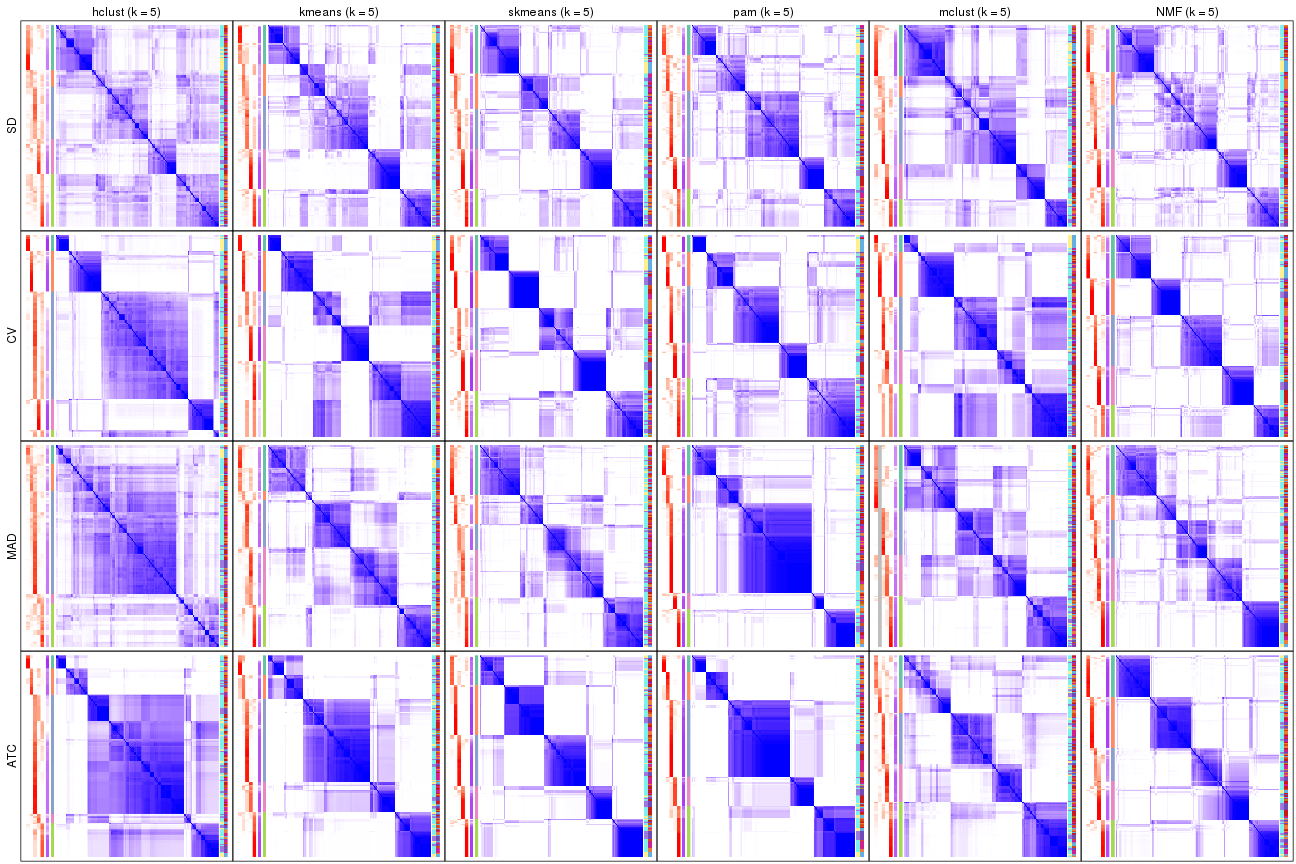
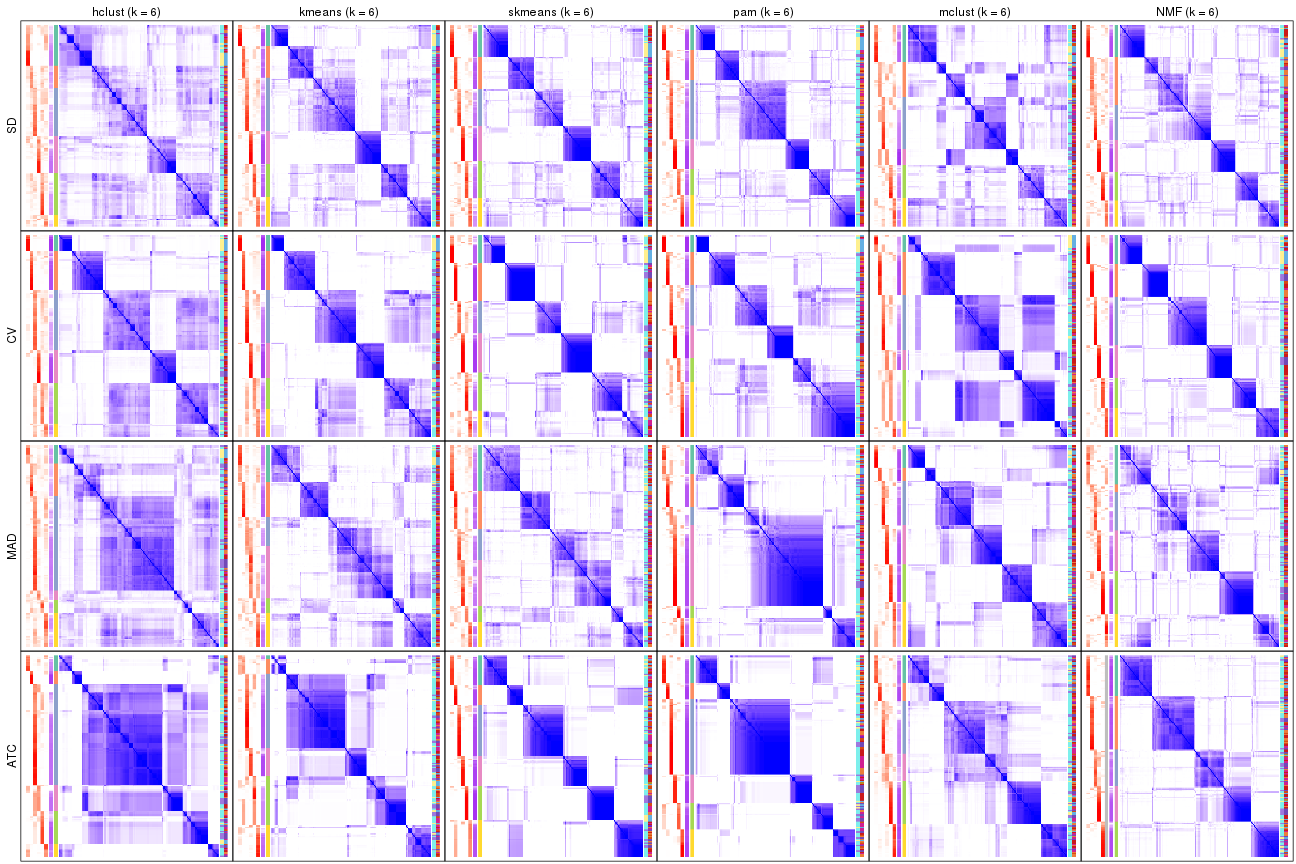
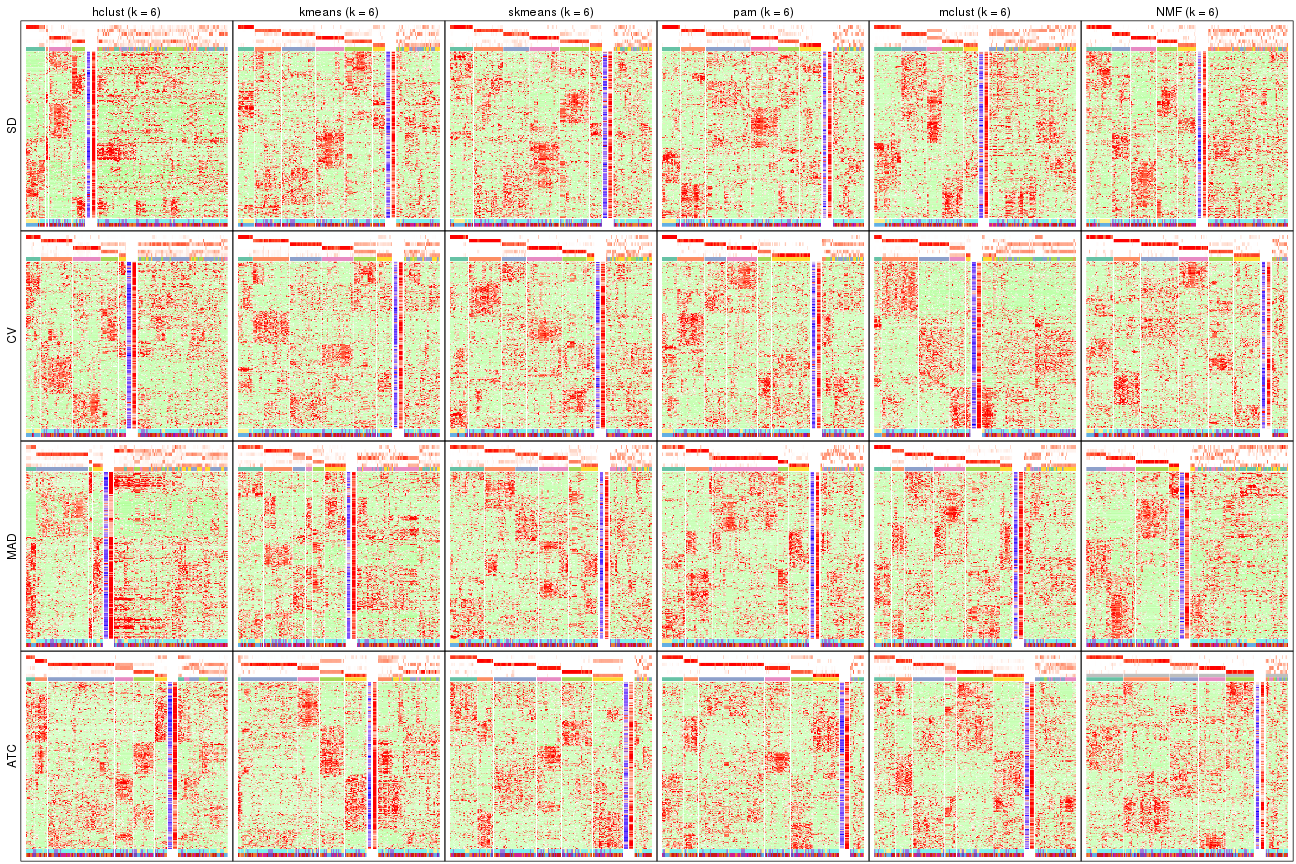
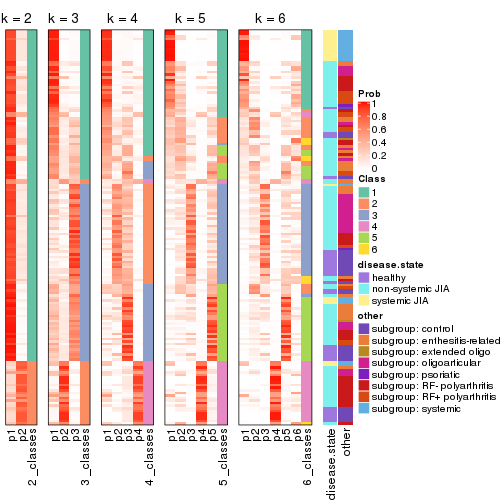
Consensus heatmaps for all methods. (What is a consensus heatmap?)
collect_plots(res_list, k = 2, fun = consensus_heatmap, mc.cores = 4)
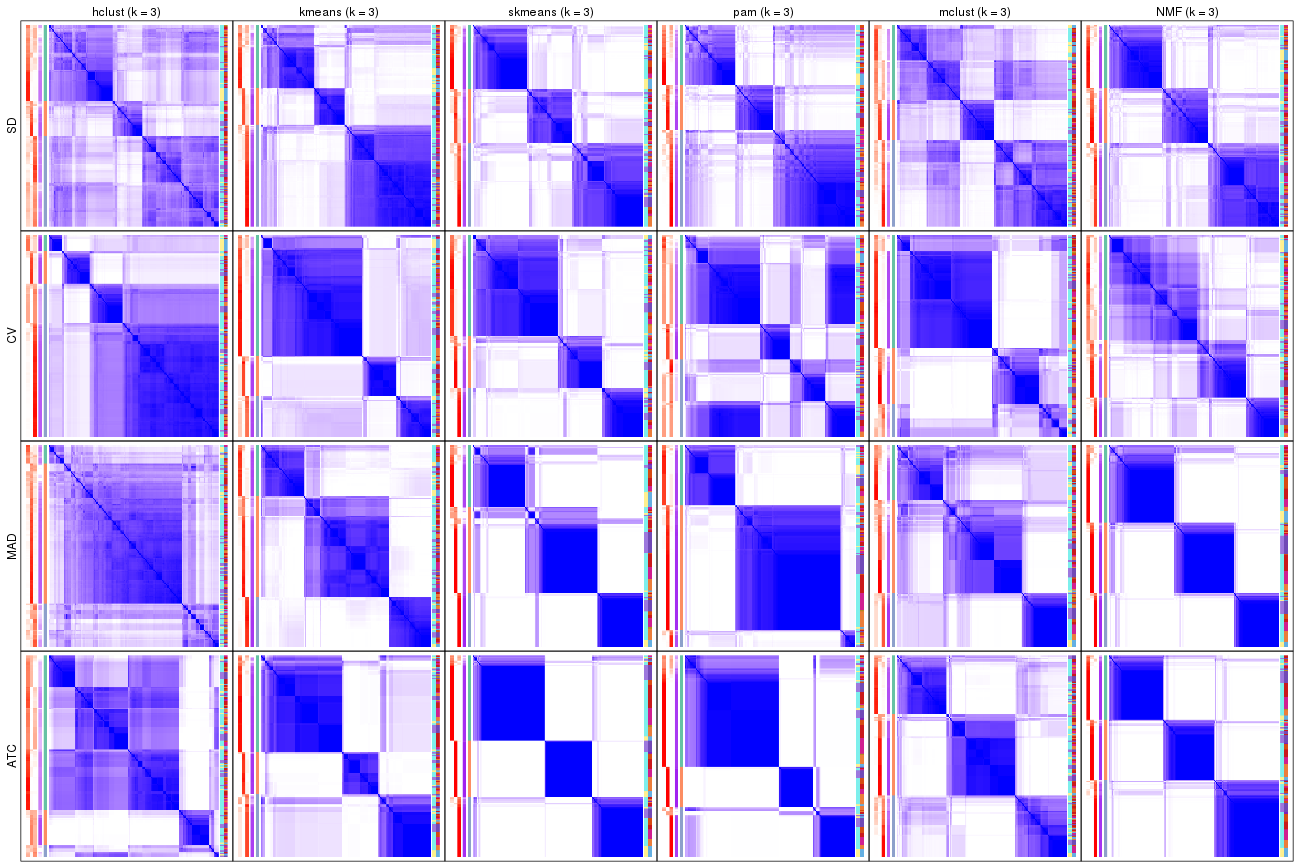
collect_plots(res_list, k = 3, fun = consensus_heatmap, mc.cores = 4)
collect_plots(res_list, k = 4, fun = consensus_heatmap, mc.cores = 4)
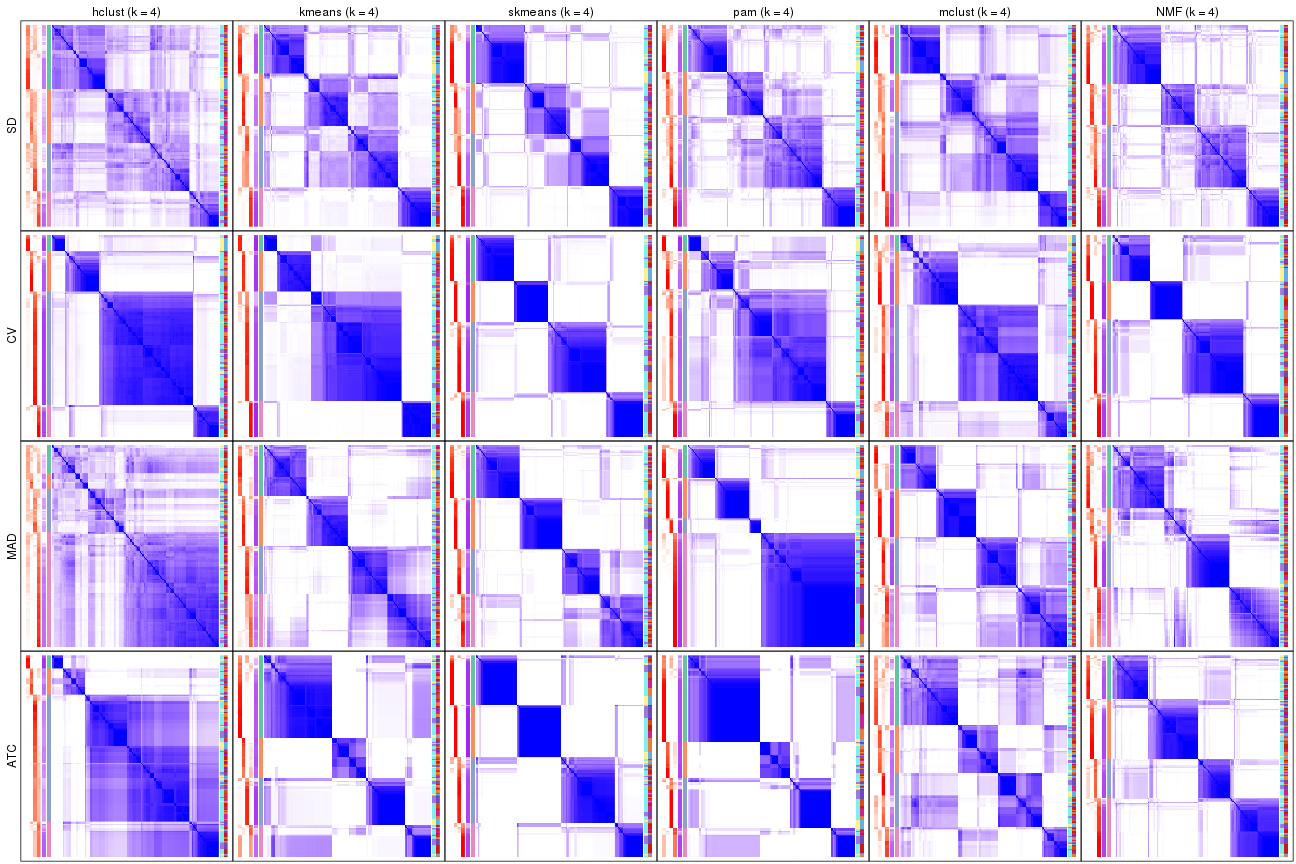
collect_plots(res_list, k = 5, fun = consensus_heatmap, mc.cores = 4)
collect_plots(res_list, k = 6, fun = consensus_heatmap, mc.cores = 4)
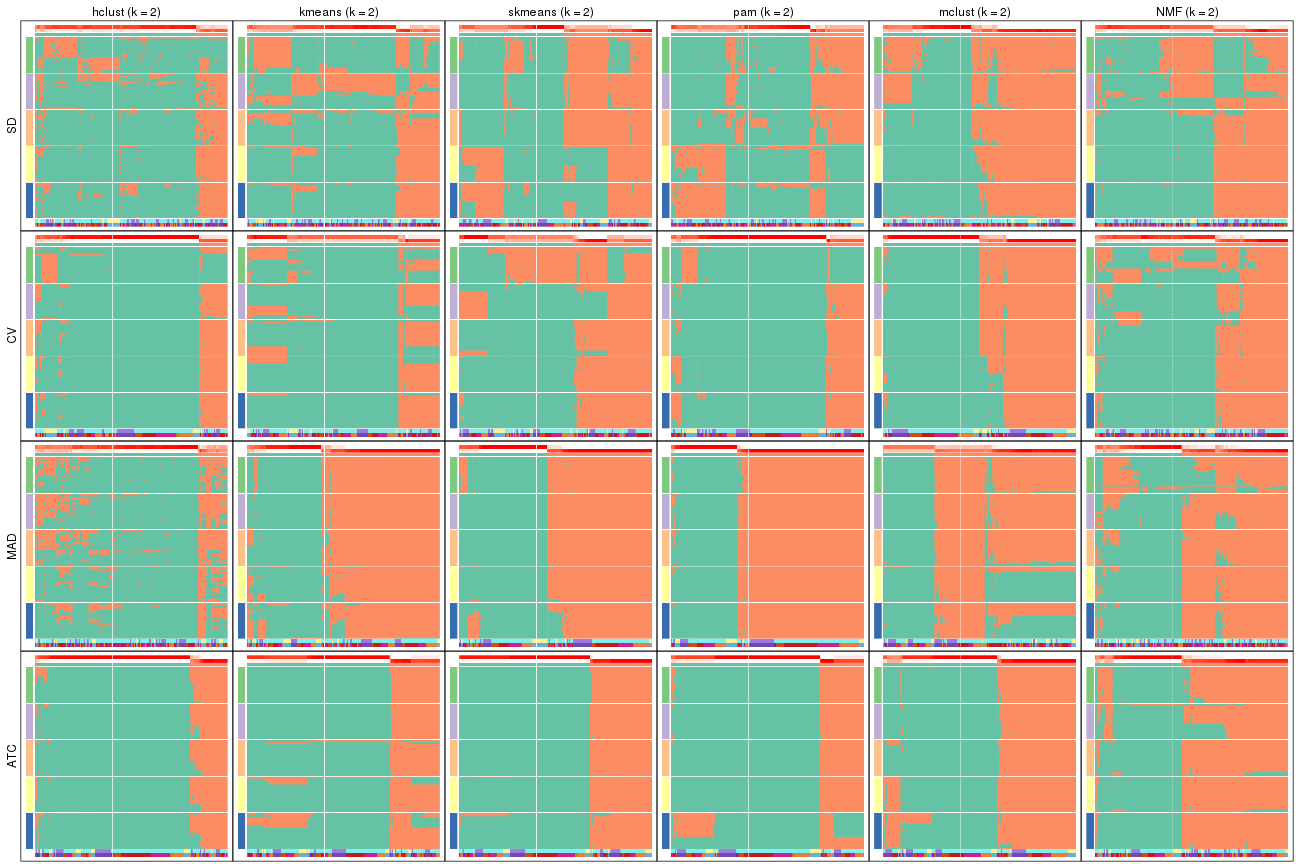
Membership heatmaps for all methods. (What is a membership heatmap?)
collect_plots(res_list, k = 2, fun = membership_heatmap, mc.cores = 4)
collect_plots(res_list, k = 3, fun = membership_heatmap, mc.cores = 4)
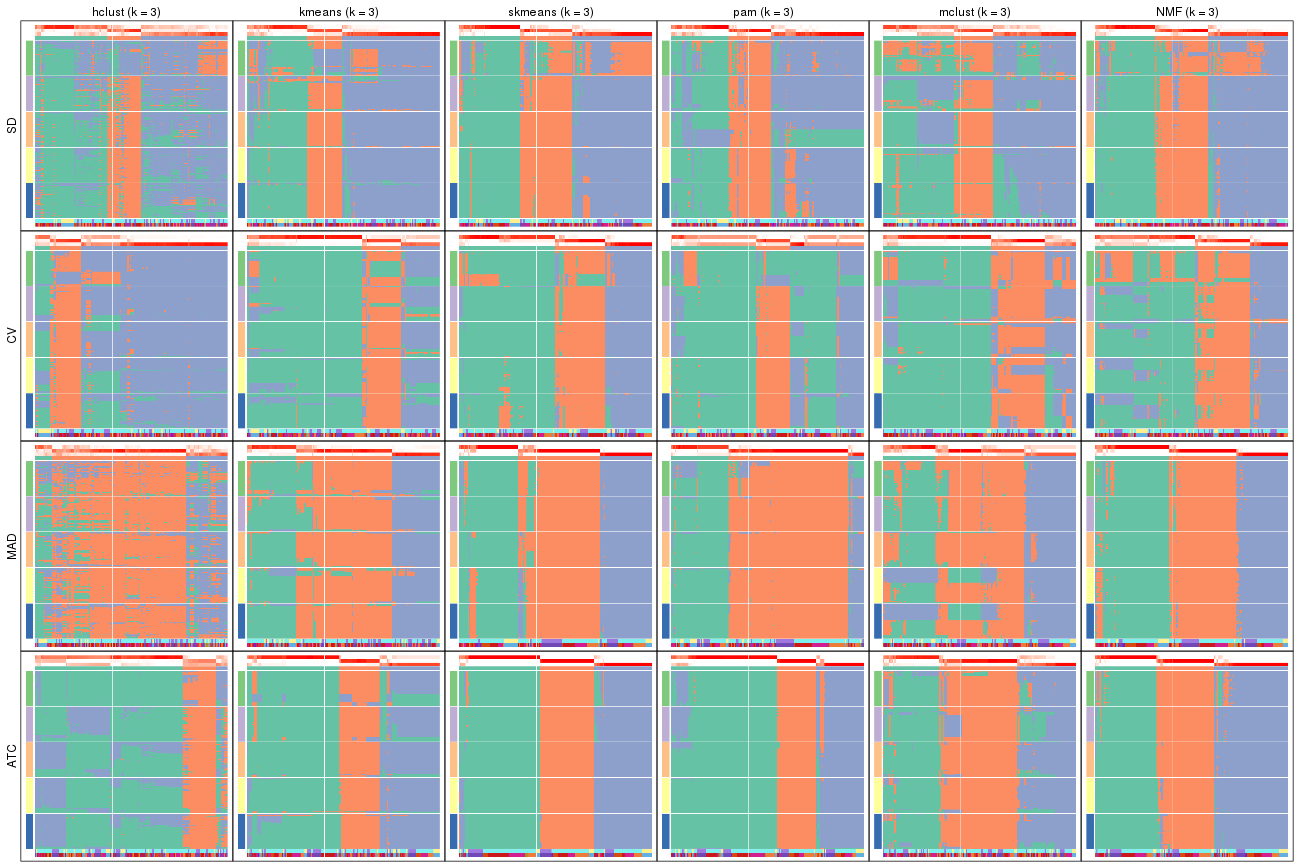
collect_plots(res_list, k = 4, fun = membership_heatmap, mc.cores = 4)
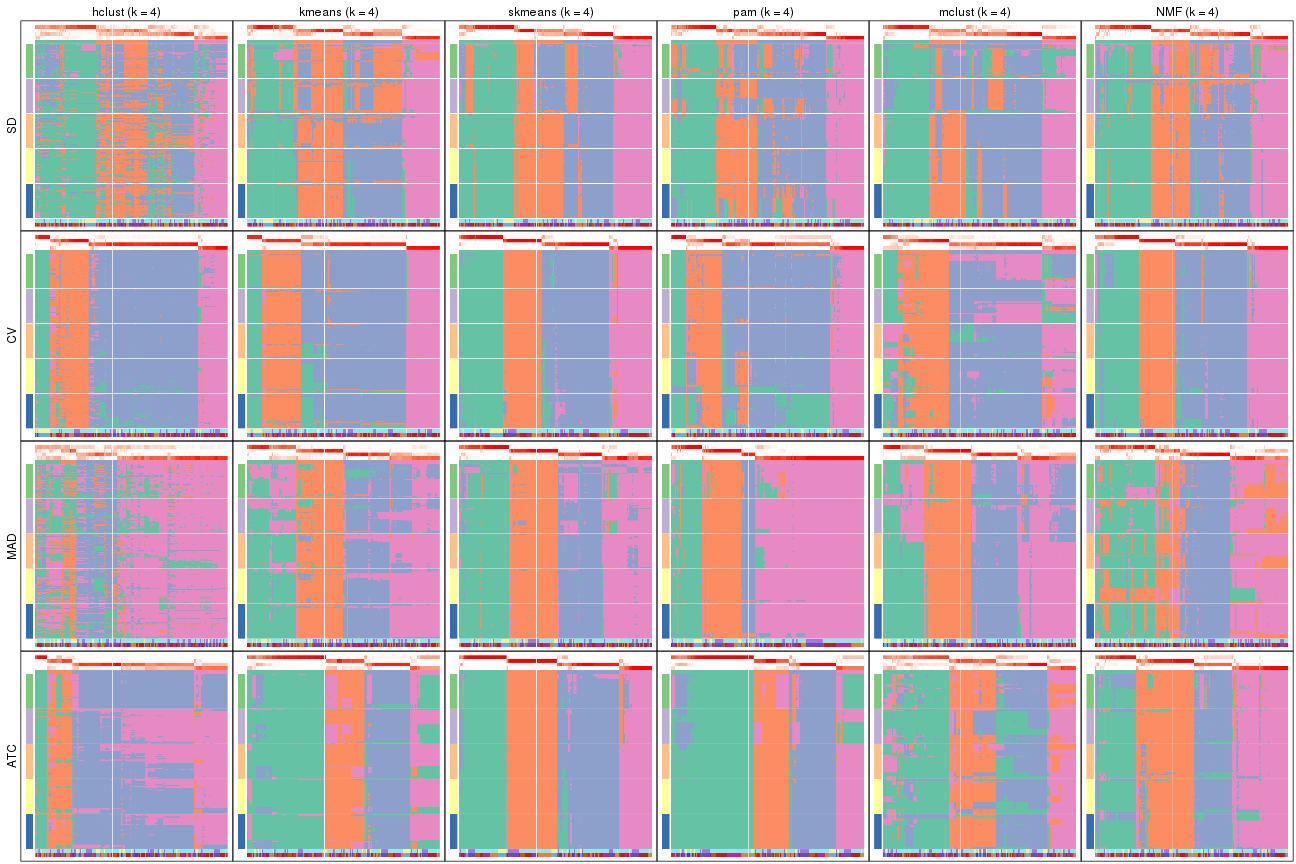
collect_plots(res_list, k = 5, fun = membership_heatmap, mc.cores = 4)
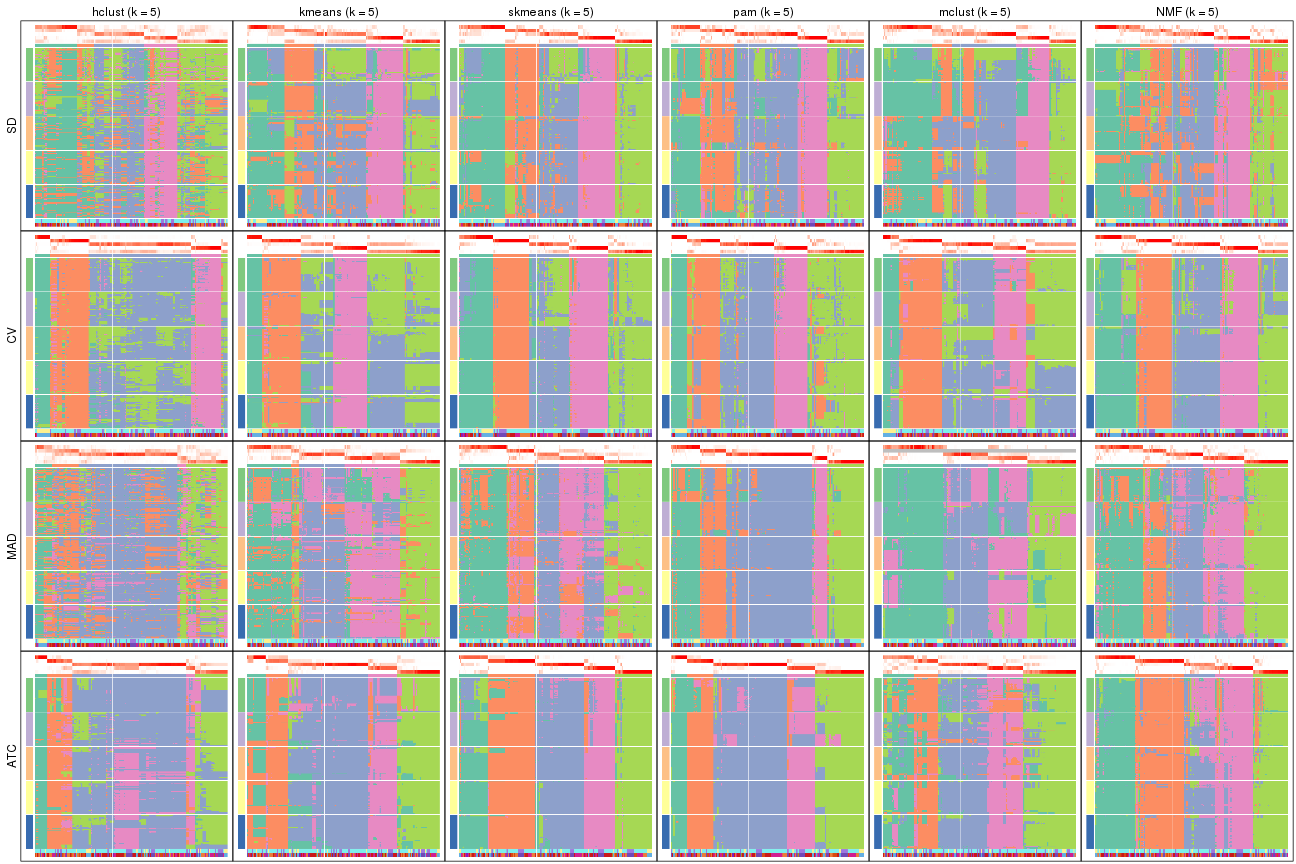
collect_plots(res_list, k = 6, fun = membership_heatmap, mc.cores = 4)
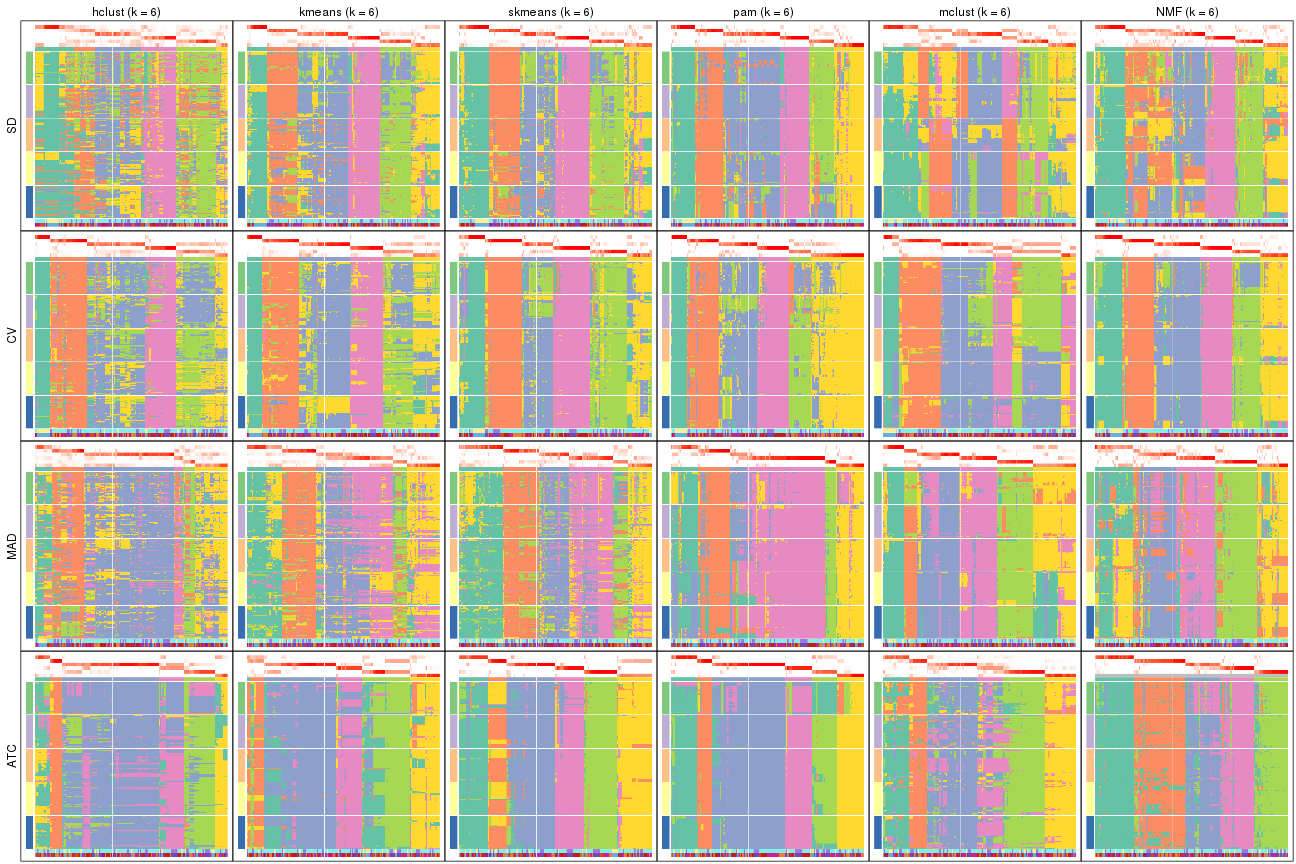
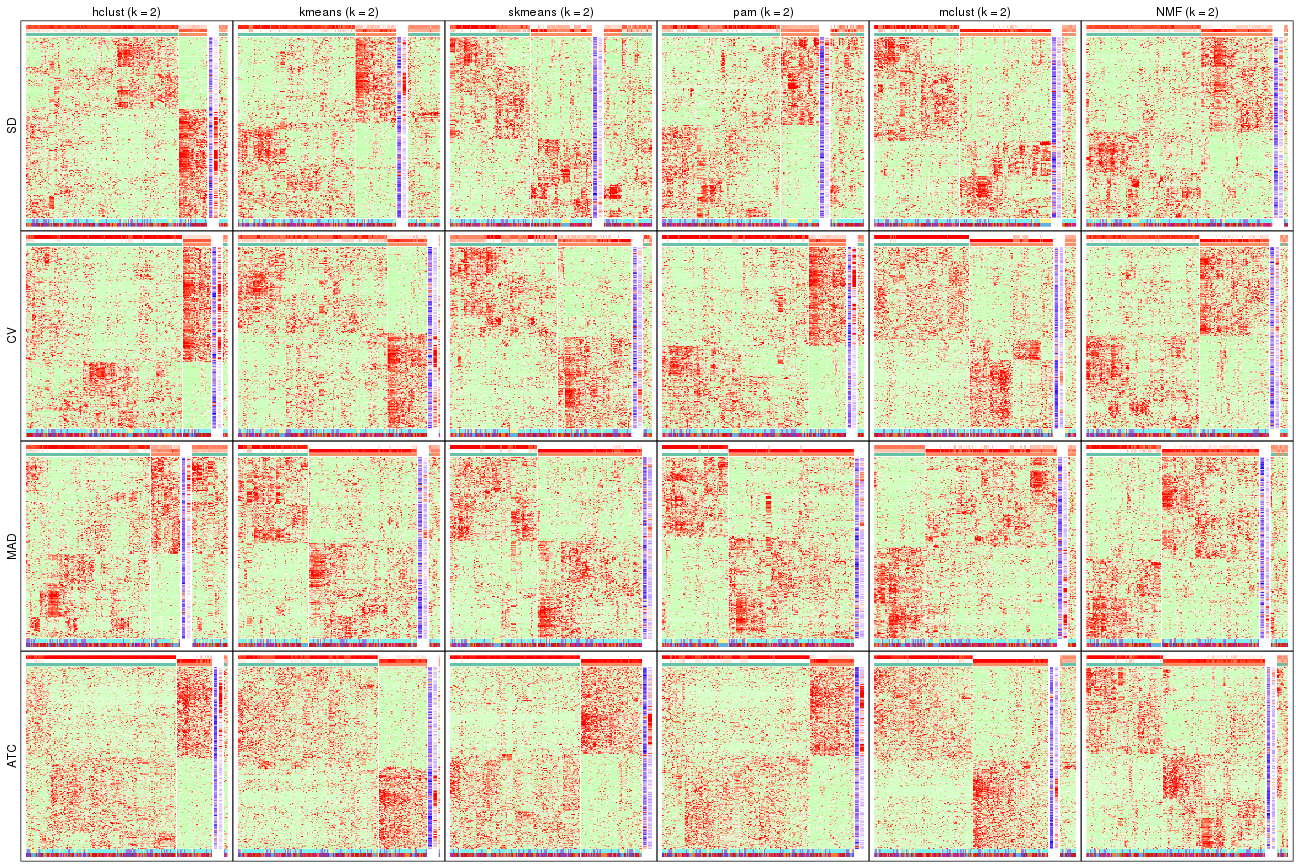
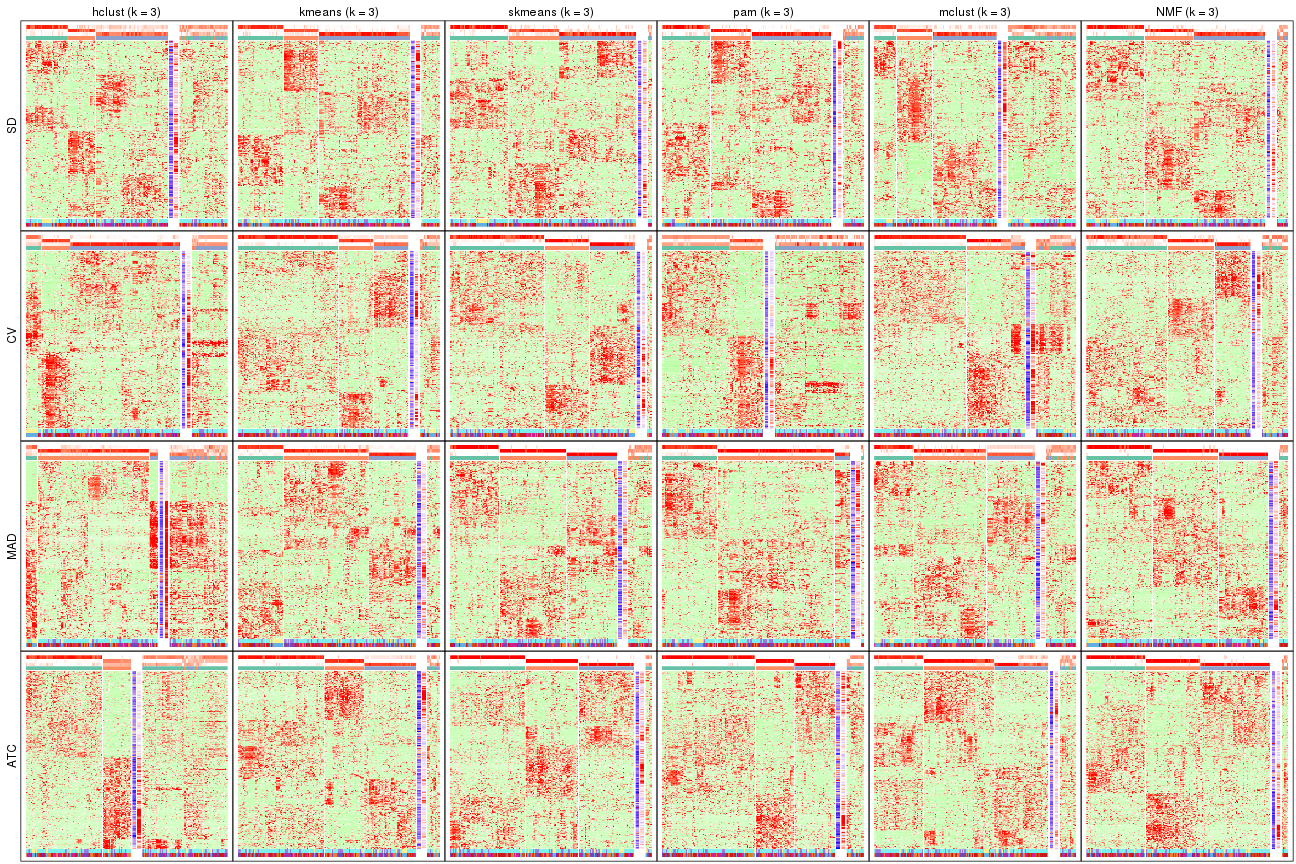
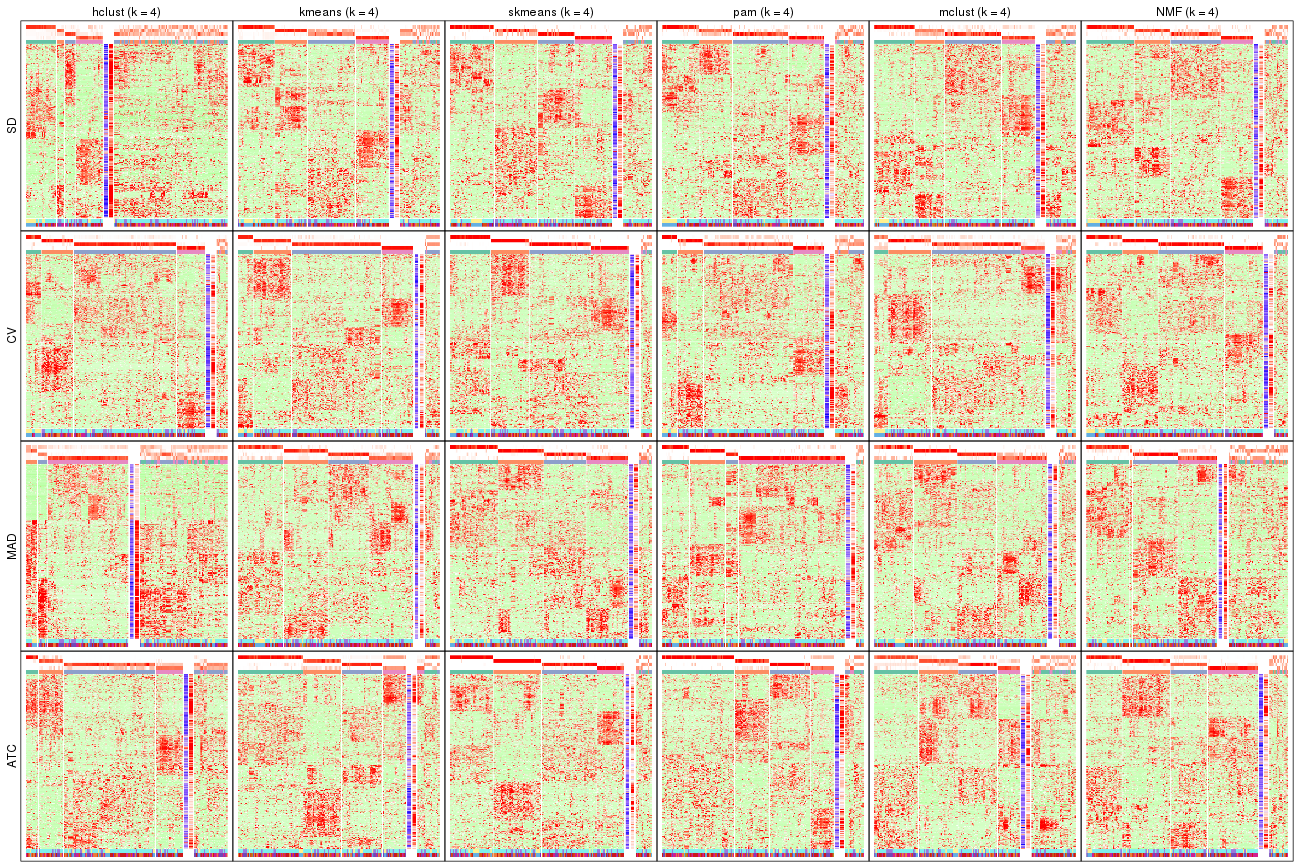
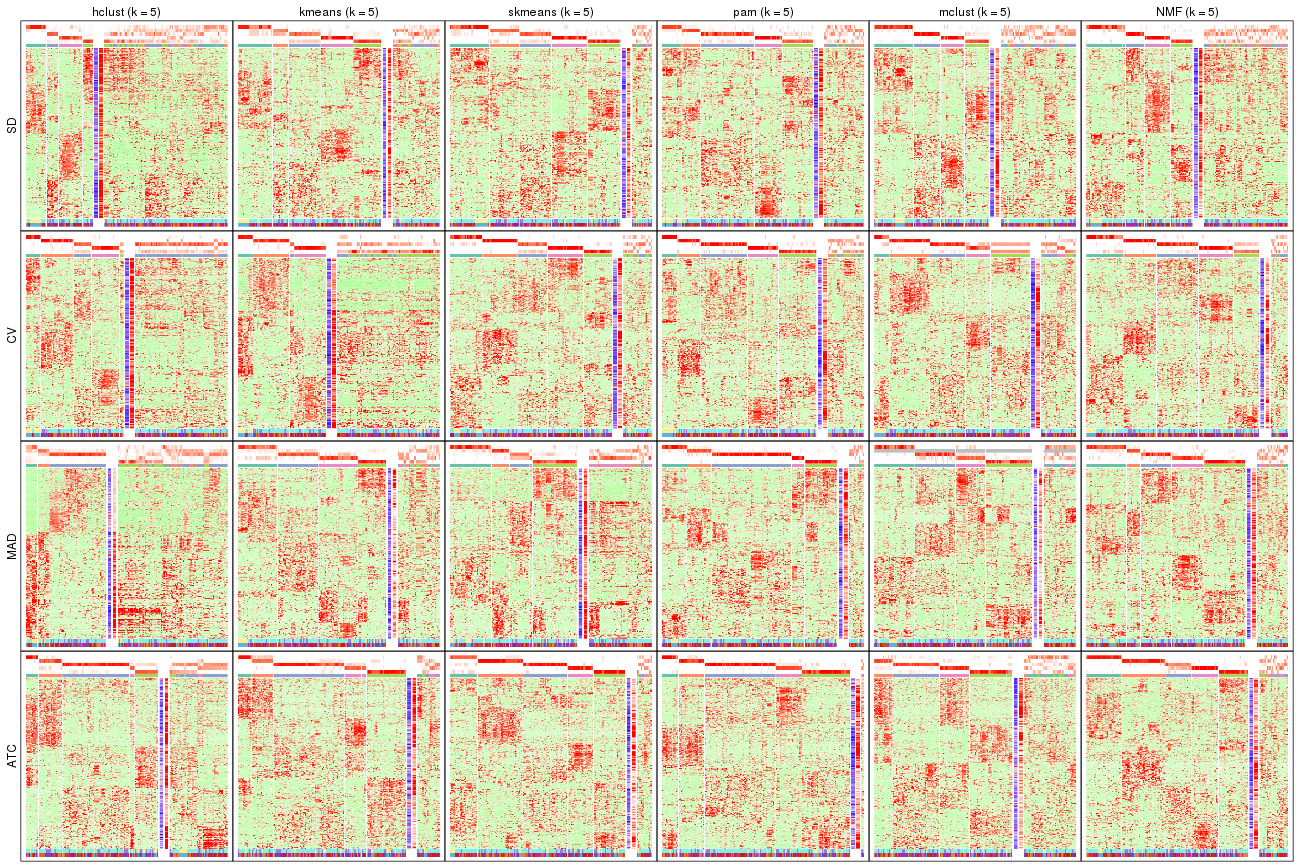
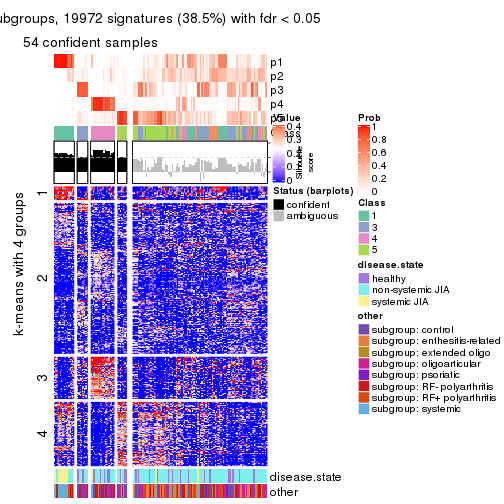
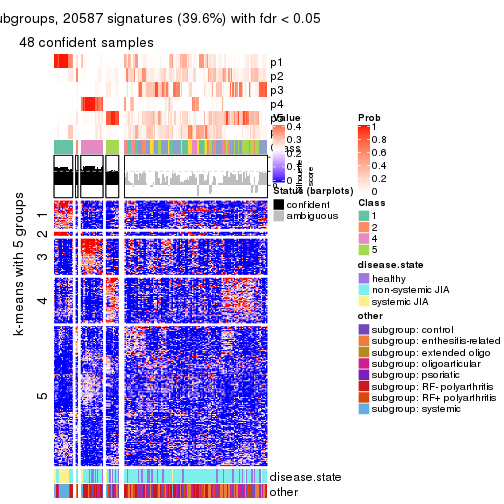
Signature heatmaps for all methods. (What is a signature heatmap?)
Note in following heatmaps, rows are scaled.
collect_plots(res_list, k = 2, fun = get_signatures, mc.cores = 4)
collect_plots(res_list, k = 3, fun = get_signatures, mc.cores = 4)
collect_plots(res_list, k = 4, fun = get_signatures, mc.cores = 4)
collect_plots(res_list, k = 5, fun = get_signatures, mc.cores = 4)
collect_plots(res_list, k = 6, fun = get_signatures, mc.cores = 4)
The statistics used for measuring the stability of consensus partitioning. (How are they defined?)
get_stats(res_list, k = 2)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> SD:NMF 2 0.328 0.751 0.806 0.486 0.521 0.521
#> CV:NMF 2 0.383 0.829 0.893 0.480 0.527 0.527
#> MAD:NMF 2 0.455 0.764 0.877 0.493 0.502 0.502
#> ATC:NMF 2 0.612 0.851 0.917 0.490 0.502 0.502
#> SD:skmeans 2 0.249 0.546 0.781 0.498 0.501 0.501
#> CV:skmeans 2 0.348 0.708 0.828 0.478 0.513 0.513
#> MAD:skmeans 2 0.760 0.933 0.966 0.498 0.501 0.501
#> ATC:skmeans 2 1.000 0.983 0.989 0.438 0.563 0.563
#> SD:mclust 2 0.494 0.752 0.877 0.477 0.501 0.501
#> CV:mclust 2 0.775 0.884 0.945 0.495 0.497 0.497
#> MAD:mclust 2 0.462 0.783 0.829 0.410 0.607 0.607
#> ATC:mclust 2 0.793 0.880 0.944 0.490 0.513 0.513
#> SD:kmeans 2 0.287 0.679 0.793 0.413 0.646 0.646
#> CV:kmeans 2 0.382 0.824 0.866 0.334 0.661 0.661
#> MAD:kmeans 2 0.689 0.870 0.938 0.477 0.524 0.524
#> ATC:kmeans 2 0.616 0.922 0.938 0.397 0.613 0.613
#> SD:pam 2 0.287 0.548 0.747 0.440 0.595 0.595
#> CV:pam 2 0.578 0.877 0.922 0.308 0.684 0.684
#> MAD:pam 2 1.000 0.972 0.987 0.455 0.546 0.546
#> ATC:pam 2 0.548 0.929 0.948 0.365 0.646 0.646
#> SD:hclust 2 0.208 0.811 0.862 0.324 0.726 0.726
#> CV:hclust 2 0.600 0.899 0.936 0.266 0.744 0.744
#> MAD:hclust 2 0.254 0.608 0.831 0.298 0.735 0.735
#> ATC:hclust 2 0.890 0.937 0.969 0.309 0.684 0.684
get_stats(res_list, k = 3)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> SD:NMF 3 0.603 0.797 0.877 0.354 0.673 0.449
#> CV:NMF 3 0.418 0.703 0.819 0.330 0.728 0.541
#> MAD:NMF 3 0.858 0.897 0.956 0.347 0.687 0.456
#> ATC:NMF 3 0.880 0.916 0.962 0.360 0.743 0.529
#> SD:skmeans 3 0.594 0.823 0.873 0.324 0.719 0.500
#> CV:skmeans 3 0.785 0.857 0.919 0.326 0.637 0.418
#> MAD:skmeans 3 0.804 0.821 0.931 0.327 0.749 0.542
#> ATC:skmeans 3 0.955 0.937 0.974 0.507 0.728 0.537
#> SD:mclust 3 0.335 0.568 0.763 0.249 0.765 0.569
#> CV:mclust 3 0.618 0.668 0.832 0.190 0.745 0.561
#> MAD:mclust 3 0.417 0.715 0.816 0.488 0.733 0.565
#> ATC:mclust 3 0.652 0.772 0.876 0.337 0.750 0.545
#> SD:kmeans 3 0.554 0.781 0.861 0.479 0.680 0.526
#> CV:kmeans 3 0.393 0.816 0.855 0.641 0.753 0.632
#> MAD:kmeans 3 0.713 0.839 0.898 0.346 0.777 0.601
#> ATC:kmeans 3 0.629 0.859 0.900 0.533 0.677 0.504
#> SD:pam 3 0.526 0.741 0.820 0.428 0.646 0.461
#> CV:pam 3 0.487 0.348 0.697 0.751 0.653 0.505
#> MAD:pam 3 0.824 0.902 0.943 0.179 0.921 0.856
#> ATC:pam 3 0.863 0.864 0.946 0.672 0.733 0.594
#> SD:hclust 3 0.151 0.554 0.714 0.797 0.634 0.500
#> CV:hclust 3 0.468 0.717 0.837 0.772 0.855 0.806
#> MAD:hclust 3 0.164 0.574 0.758 0.664 0.760 0.682
#> ATC:hclust 3 0.410 0.514 0.751 0.718 0.931 0.900
get_stats(res_list, k = 4)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> SD:NMF 4 0.609 0.697 0.824 0.1348 0.817 0.531
#> CV:NMF 4 0.810 0.835 0.926 0.1475 0.776 0.488
#> MAD:NMF 4 0.654 0.594 0.769 0.1238 0.891 0.693
#> ATC:NMF 4 0.755 0.792 0.874 0.1177 0.866 0.627
#> SD:skmeans 4 0.734 0.712 0.851 0.1321 0.832 0.556
#> CV:skmeans 4 0.873 0.887 0.949 0.1712 0.829 0.573
#> MAD:skmeans 4 0.781 0.816 0.898 0.1370 0.852 0.598
#> ATC:skmeans 4 0.883 0.866 0.942 0.1235 0.848 0.594
#> SD:mclust 4 0.527 0.646 0.796 0.1653 0.746 0.427
#> CV:mclust 4 0.554 0.708 0.758 0.1525 0.887 0.737
#> MAD:mclust 4 0.689 0.728 0.822 0.2033 0.813 0.527
#> ATC:mclust 4 0.482 0.576 0.751 0.0788 0.844 0.588
#> SD:kmeans 4 0.633 0.631 0.780 0.1922 0.799 0.518
#> CV:kmeans 4 0.847 0.865 0.912 0.2108 0.916 0.812
#> MAD:kmeans 4 0.647 0.739 0.781 0.1443 0.874 0.661
#> ATC:kmeans 4 0.714 0.741 0.839 0.1375 0.807 0.542
#> SD:pam 4 0.566 0.671 0.816 0.1692 0.789 0.496
#> CV:pam 4 0.548 0.705 0.831 0.2142 0.674 0.393
#> MAD:pam 4 0.822 0.862 0.938 0.1664 0.848 0.699
#> ATC:pam 4 0.728 0.807 0.883 0.1403 0.882 0.709
#> SD:hclust 4 0.281 0.432 0.664 0.1895 0.838 0.588
#> CV:hclust 4 0.795 0.843 0.918 0.3312 0.774 0.629
#> MAD:hclust 4 0.235 0.464 0.684 0.2246 0.829 0.689
#> ATC:hclust 4 0.426 0.598 0.751 0.0575 0.746 0.614
get_stats(res_list, k = 5)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> SD:NMF 5 0.594 0.466 0.708 0.0690 0.893 0.618
#> CV:NMF 5 0.739 0.750 0.845 0.0870 0.882 0.595
#> MAD:NMF 5 0.672 0.677 0.812 0.0639 0.844 0.502
#> ATC:NMF 5 0.776 0.721 0.850 0.0636 0.897 0.628
#> SD:skmeans 5 0.686 0.685 0.806 0.0690 0.888 0.600
#> CV:skmeans 5 0.776 0.728 0.849 0.0771 0.871 0.557
#> MAD:skmeans 5 0.677 0.583 0.746 0.0588 0.895 0.625
#> ATC:skmeans 5 0.809 0.813 0.894 0.0639 0.911 0.677
#> SD:mclust 5 0.575 0.501 0.742 0.0864 0.906 0.687
#> CV:mclust 5 0.594 0.566 0.749 0.0521 0.842 0.555
#> MAD:mclust 5 0.603 0.614 0.738 0.0499 0.903 0.676
#> ATC:mclust 5 0.615 0.641 0.792 0.0934 0.859 0.542
#> SD:kmeans 5 0.623 0.598 0.714 0.0768 0.869 0.550
#> CV:kmeans 5 0.713 0.569 0.744 0.1344 0.872 0.655
#> MAD:kmeans 5 0.630 0.560 0.726 0.0661 0.942 0.783
#> ATC:kmeans 5 0.681 0.737 0.830 0.0756 0.950 0.831
#> SD:pam 5 0.606 0.621 0.785 0.0658 0.914 0.696
#> CV:pam 5 0.660 0.685 0.854 0.1772 0.805 0.512
#> MAD:pam 5 0.726 0.785 0.895 0.1567 0.871 0.676
#> ATC:pam 5 0.786 0.861 0.912 0.0692 0.914 0.727
#> SD:hclust 5 0.391 0.374 0.614 0.0943 0.924 0.723
#> CV:hclust 5 0.619 0.537 0.763 0.1674 0.965 0.910
#> MAD:hclust 5 0.291 0.412 0.634 0.1245 0.893 0.744
#> ATC:hclust 5 0.476 0.560 0.763 0.1567 0.711 0.489
get_stats(res_list, k = 6)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> SD:NMF 6 0.661 0.535 0.699 0.0438 0.869 0.474
#> CV:NMF 6 0.793 0.779 0.873 0.0454 0.920 0.643
#> MAD:NMF 6 0.675 0.430 0.703 0.0398 0.903 0.599
#> ATC:NMF 6 0.743 0.742 0.822 0.0322 0.971 0.864
#> SD:skmeans 6 0.699 0.635 0.790 0.0451 0.931 0.677
#> CV:skmeans 6 0.775 0.640 0.811 0.0383 0.934 0.690
#> MAD:skmeans 6 0.660 0.567 0.725 0.0390 0.920 0.659
#> ATC:skmeans 6 0.838 0.807 0.870 0.0339 0.943 0.737
#> SD:mclust 6 0.593 0.422 0.651 0.0644 0.873 0.525
#> CV:mclust 6 0.662 0.465 0.709 0.0492 0.916 0.665
#> MAD:mclust 6 0.713 0.647 0.797 0.0528 0.877 0.559
#> ATC:mclust 6 0.679 0.646 0.776 0.0467 0.875 0.515
#> SD:kmeans 6 0.650 0.618 0.755 0.0493 0.922 0.656
#> CV:kmeans 6 0.689 0.654 0.747 0.0684 0.886 0.575
#> MAD:kmeans 6 0.628 0.468 0.650 0.0405 0.853 0.472
#> ATC:kmeans 6 0.681 0.541 0.761 0.0502 0.919 0.722
#> SD:pam 6 0.703 0.688 0.825 0.0492 0.924 0.681
#> CV:pam 6 0.715 0.678 0.829 0.0510 0.899 0.602
#> MAD:pam 6 0.699 0.631 0.794 0.0562 0.921 0.735
#> ATC:pam 6 0.847 0.824 0.885 0.0541 0.958 0.838
#> SD:hclust 6 0.481 0.361 0.591 0.0565 0.936 0.725
#> CV:hclust 6 0.613 0.550 0.695 0.0742 0.840 0.549
#> MAD:hclust 6 0.375 0.423 0.659 0.0634 0.920 0.771
#> ATC:hclust 6 0.474 0.552 0.740 0.0528 0.888 0.724
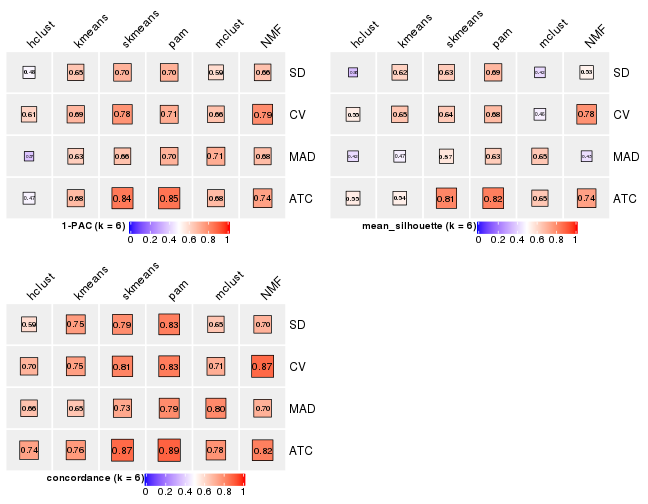
Following heatmap plots the partition for each combination of methods and the lightness correspond to the silhouette scores for samples in each method. On top the consensus subgroup is inferred from all methods by taking the mean silhouette scores as weight.
collect_stats(res_list, k = 2)
collect_stats(res_list, k = 3)
collect_stats(res_list, k = 4)
collect_stats(res_list, k = 5)
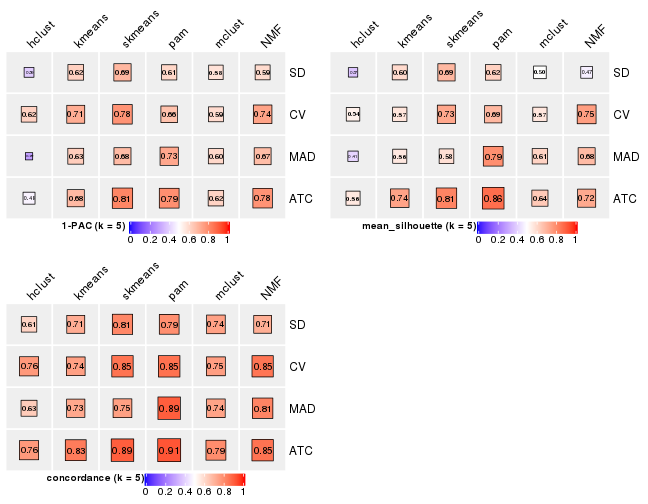
collect_stats(res_list, k = 6)
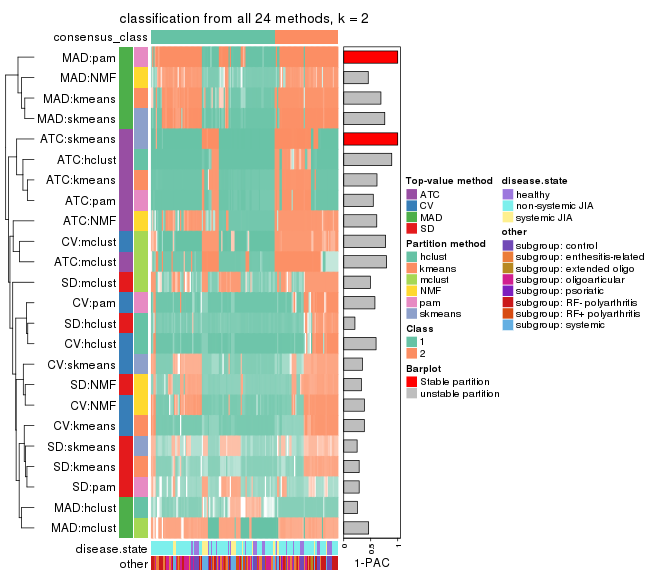
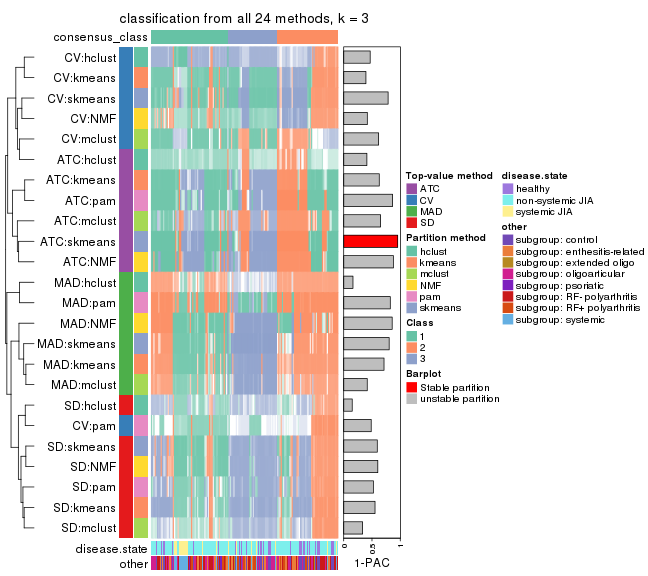
Collect partitions from all methods:
collect_classes(res_list, k = 2)
collect_classes(res_list, k = 3)
collect_classes(res_list, k = 4)
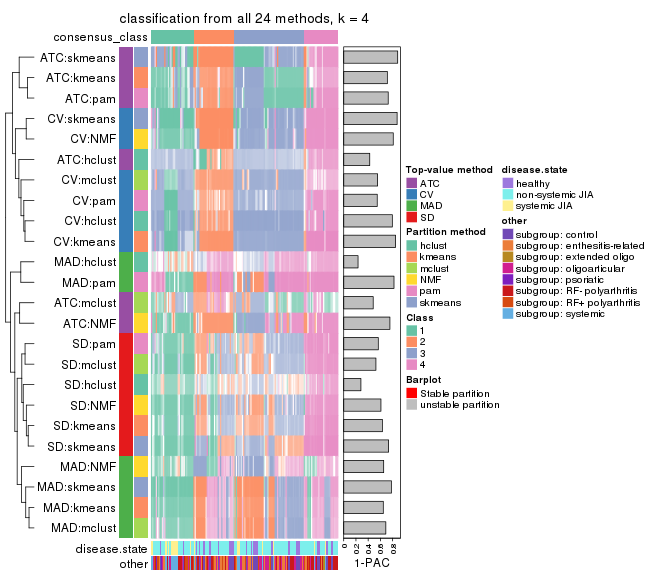
collect_classes(res_list, k = 5)
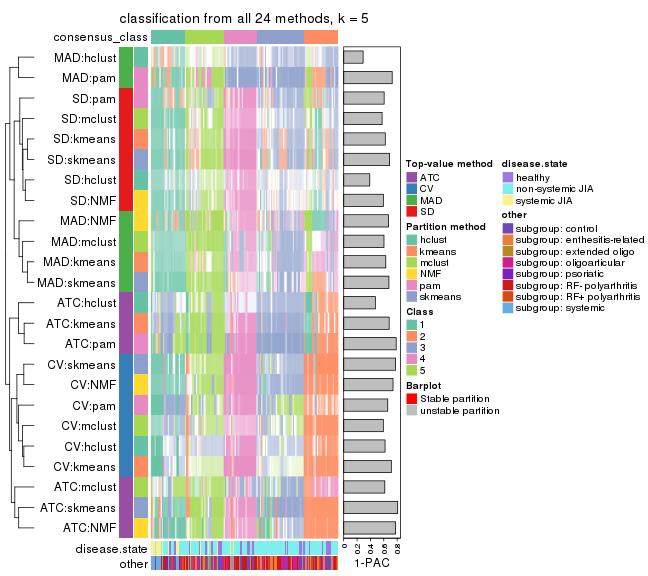
collect_classes(res_list, k = 6)
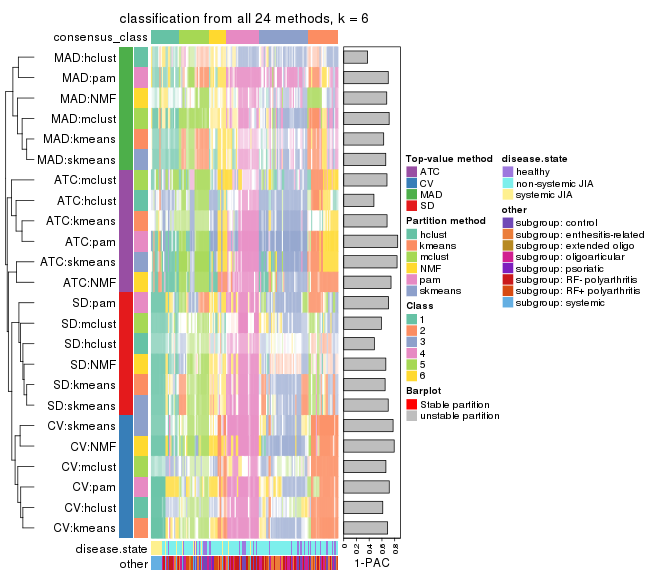
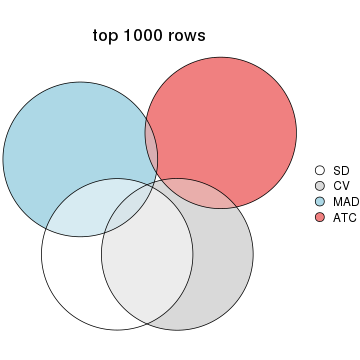
Overlap of top rows from different top-row methods:
top_rows_overlap(res_list, top_n = 1000, method = "euler")
top_rows_overlap(res_list, top_n = 2000, method = "euler")
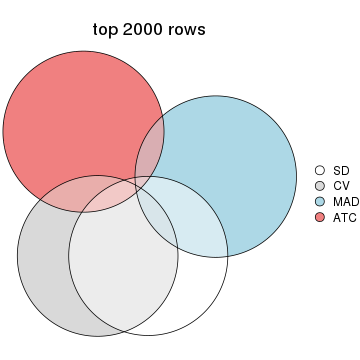
top_rows_overlap(res_list, top_n = 3000, method = "euler")
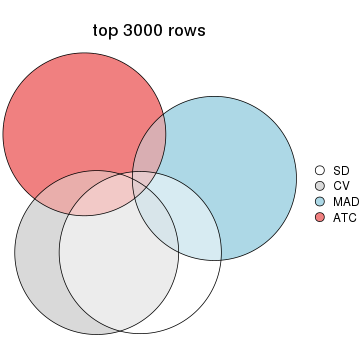
top_rows_overlap(res_list, top_n = 4000, method = "euler")
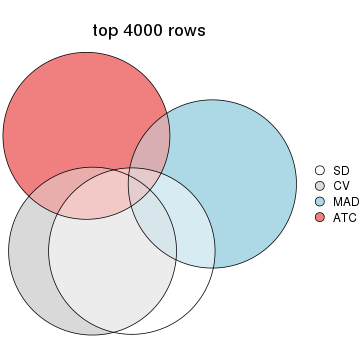
top_rows_overlap(res_list, top_n = 5000, method = "euler")
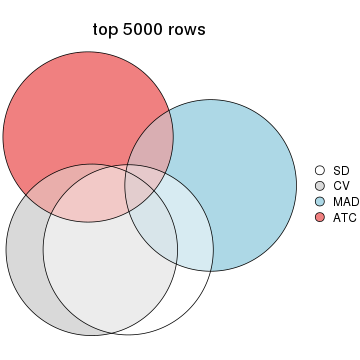
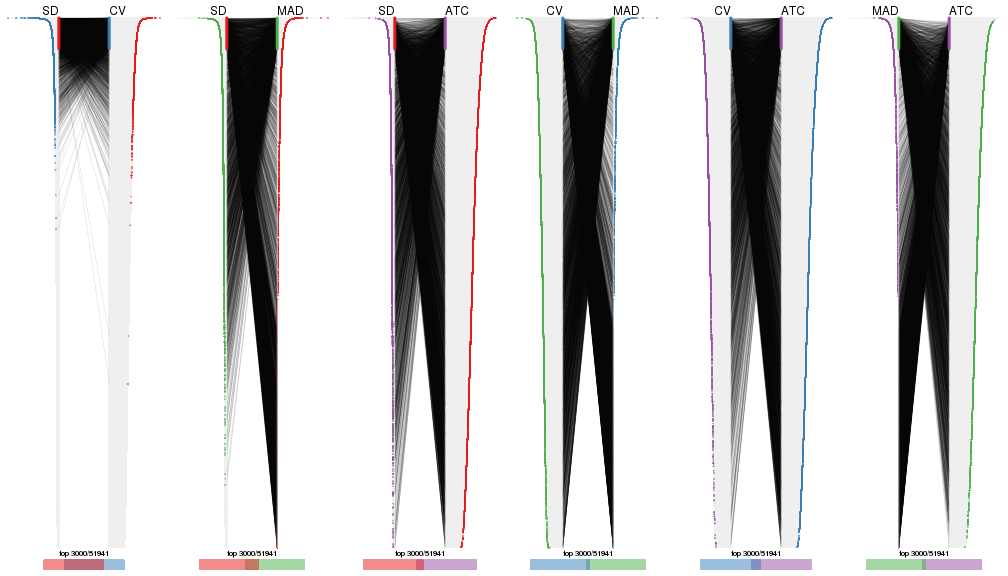
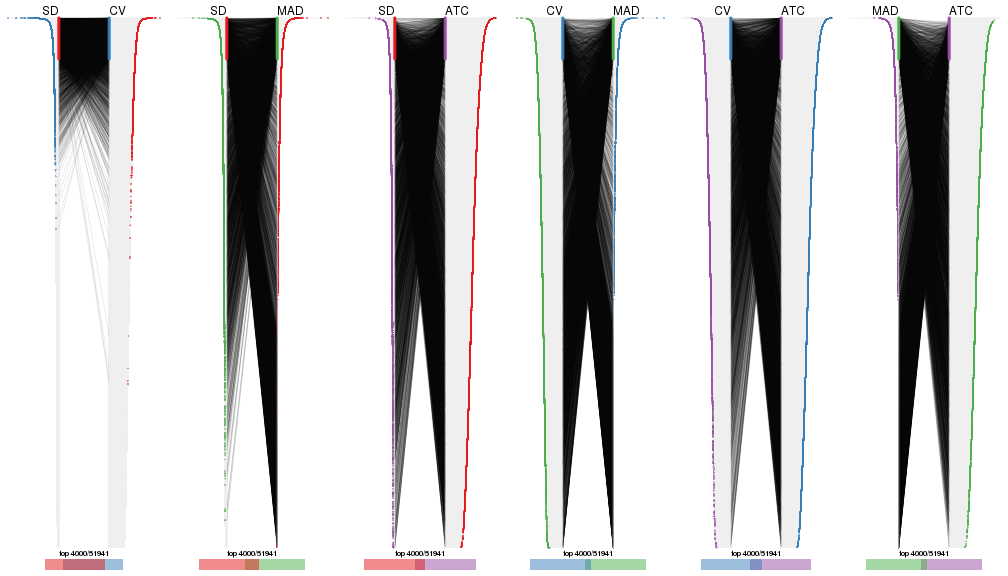
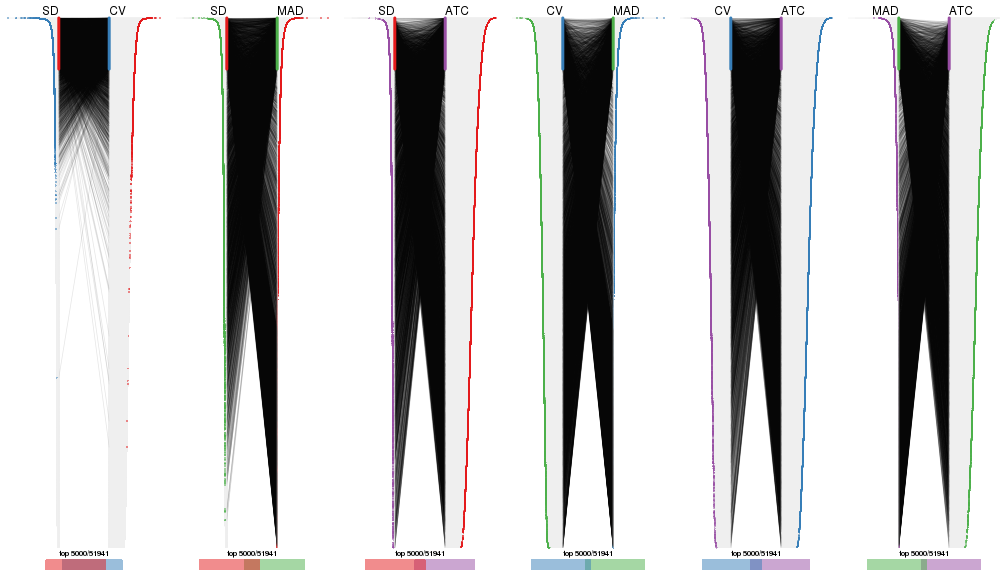
Also visualize the correspondance of rankings between different top-row methods:
top_rows_overlap(res_list, top_n = 1000, method = "correspondance")

top_rows_overlap(res_list, top_n = 2000, method = "correspondance")

top_rows_overlap(res_list, top_n = 3000, method = "correspondance")
top_rows_overlap(res_list, top_n = 4000, method = "correspondance")
top_rows_overlap(res_list, top_n = 5000, method = "correspondance")
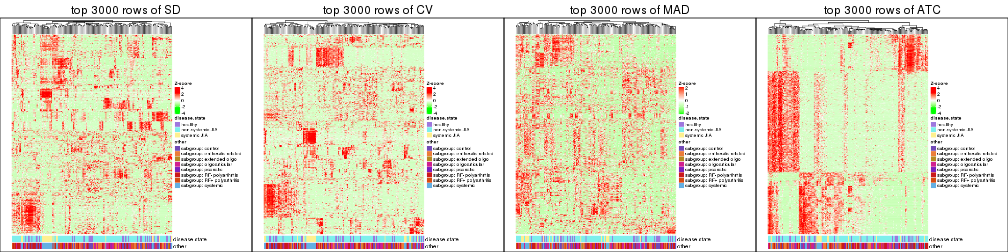
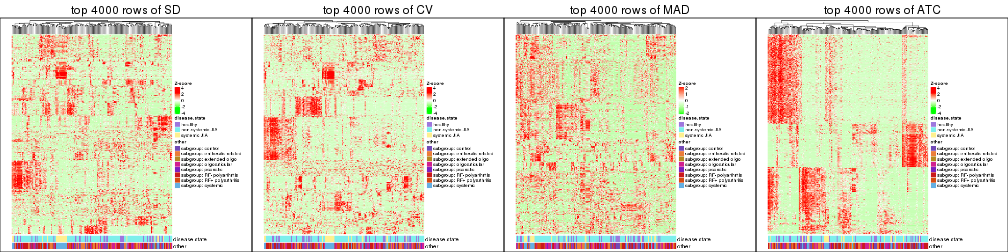
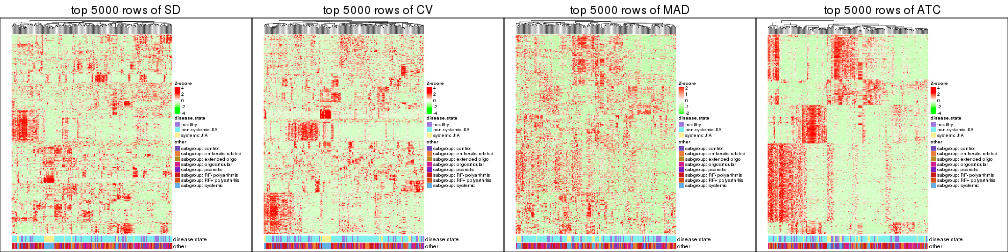
Heatmaps of the top rows:
top_rows_heatmap(res_list, top_n = 1000)
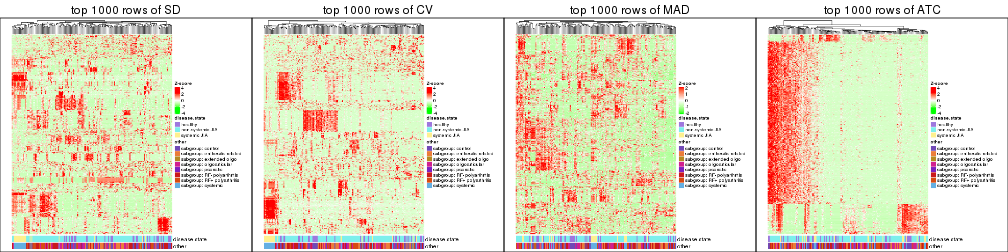
top_rows_heatmap(res_list, top_n = 2000)
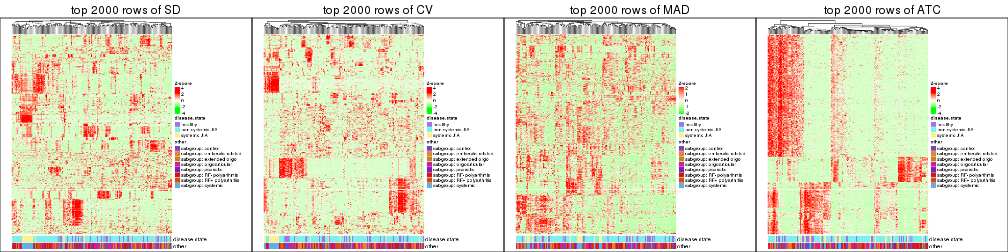
top_rows_heatmap(res_list, top_n = 3000)
top_rows_heatmap(res_list, top_n = 4000)
top_rows_heatmap(res_list, top_n = 5000)
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res_list, k = 2)
#> n disease.state(p) other(p) k
#> SD:NMF 151 7.41e-02 1.41e-02 2
#> CV:NMF 148 1.41e-01 7.12e-03 2
#> MAD:NMF 140 1.41e-02 2.47e-02 2
#> ATC:NMF 145 1.47e-01 1.41e-01 2
#> SD:skmeans 115 7.89e-05 1.22e-05 2
#> CV:skmeans 147 2.29e-01 2.69e-02 2
#> MAD:skmeans 154 1.26e-02 1.18e-01 2
#> ATC:skmeans 154 7.08e-01 5.13e-01 2
#> SD:mclust 143 6.94e-03 1.44e-03 2
#> CV:mclust 145 5.55e-01 3.18e-01 2
#> MAD:mclust 148 2.75e-01 2.51e-01 2
#> ATC:mclust 141 8.66e-01 5.76e-01 2
#> SD:kmeans 128 8.26e-01 2.79e-03 2
#> CV:kmeans 153 5.39e-01 3.06e-03 2
#> MAD:kmeans 145 9.33e-03 6.33e-02 2
#> ATC:kmeans 153 6.36e-01 6.76e-01 2
#> SD:pam 127 5.17e-08 4.34e-08 2
#> CV:pam 149 5.45e-01 8.17e-02 2
#> MAD:pam 154 1.54e-03 8.37e-03 2
#> ATC:pam 154 8.05e-01 5.10e-01 2
#> SD:hclust 147 6.47e-01 7.14e-03 2
#> CV:hclust 150 6.27e-01 8.62e-03 2
#> MAD:hclust 125 1.43e-03 8.22e-03 2
#> ATC:hclust 151 2.93e-01 2.48e-01 2
test_to_known_factors(res_list, k = 3)
#> n disease.state(p) other(p) k
#> SD:NMF 145 1.87e-04 5.09e-05 3
#> CV:NMF 133 5.11e-01 6.63e-02 3
#> MAD:NMF 147 1.34e-03 1.51e-03 3
#> ATC:NMF 149 6.58e-01 1.61e-01 3
#> SD:skmeans 151 1.26e-03 8.13e-04 3
#> CV:skmeans 150 7.34e-01 3.65e-02 3
#> MAD:skmeans 135 7.16e-04 3.17e-04 3
#> ATC:skmeans 149 2.83e-01 2.09e-01 3
#> SD:mclust 99 7.39e-02 6.46e-04 3
#> CV:mclust 122 2.68e-03 3.34e-02 3
#> MAD:mclust 130 2.00e-04 9.30e-05 3
#> ATC:mclust 141 6.44e-01 3.06e-01 3
#> SD:kmeans 139 2.34e-04 1.38e-06 3
#> CV:kmeans 138 9.11e-01 4.81e-02 3
#> MAD:kmeans 144 5.33e-04 3.77e-05 3
#> ATC:kmeans 144 1.54e-01 9.12e-02 3
#> SD:pam 137 5.81e-06 3.16e-07 3
#> CV:pam 82 1.83e-01 1.72e-02 3
#> MAD:pam 152 1.10e-03 1.09e-03 3
#> ATC:pam 140 8.79e-02 1.08e-01 3
#> SD:hclust 115 6.62e-04 1.20e-05 3
#> CV:hclust 125 1.32e-10 2.23e-09 3
#> MAD:hclust 107 1.06e-04 1.13e-02 3
#> ATC:hclust 85 4.22e-01 6.16e-01 3
test_to_known_factors(res_list, k = 4)
#> n disease.state(p) other(p) k
#> SD:NMF 135 7.85e-03 2.62e-05 4
#> CV:NMF 143 2.11e-06 1.85e-07 4
#> MAD:NMF 106 8.33e-03 1.62e-02 4
#> ATC:NMF 139 4.46e-01 4.05e-02 4
#> SD:skmeans 131 4.85e-04 5.69e-05 4
#> CV:skmeans 145 7.36e-06 2.64e-07 4
#> MAD:skmeans 144 3.18e-03 4.02e-03 4
#> ATC:skmeans 141 1.37e-01 3.84e-02 4
#> SD:mclust 130 5.91e-04 1.16e-06 4
#> CV:mclust 138 4.89e-13 2.88e-12 4
#> MAD:mclust 140 4.61e-03 3.05e-03 4
#> ATC:mclust 118 6.96e-01 6.98e-01 4
#> SD:kmeans 122 3.84e-05 1.47e-06 4
#> CV:kmeans 142 4.96e-14 1.05e-12 4
#> MAD:kmeans 142 1.84e-03 6.96e-04 4
#> ATC:kmeans 136 1.90e-01 6.79e-02 4
#> SD:pam 131 4.86e-06 4.06e-07 4
#> CV:pam 131 2.26e-13 2.90e-10 4
#> MAD:pam 148 1.79e-03 1.68e-03 4
#> ATC:pam 139 5.77e-02 3.20e-02 4
#> SD:hclust 62 3.10e-03 3.52e-04 4
#> CV:hclust 145 1.27e-11 5.99e-10 4
#> MAD:hclust 83 1.07e-04 1.09e-03 4
#> ATC:hclust 127 3.30e-01 7.50e-02 4
test_to_known_factors(res_list, k = 5)
#> n disease.state(p) other(p) k
#> SD:NMF 86 6.91e-03 8.15e-03 5
#> CV:NMF 140 8.07e-06 1.72e-07 5
#> MAD:NMF 129 1.18e-02 3.48e-03 5
#> ATC:NMF 131 5.21e-01 2.72e-02 5
#> SD:skmeans 138 2.34e-04 1.34e-06 5
#> CV:skmeans 131 2.44e-05 1.85e-06 5
#> MAD:skmeans 103 2.78e-02 5.40e-02 5
#> ATC:skmeans 142 3.87e-01 1.40e-01 5
#> SD:mclust 93 1.09e-03 2.48e-04 5
#> CV:mclust 126 2.66e-13 1.92e-11 5
#> MAD:mclust 128 3.78e-03 5.96e-04 5
#> ATC:mclust 112 5.57e-01 4.00e-01 5
#> SD:kmeans 116 3.25e-05 1.58e-05 5
#> CV:kmeans 71 1.73e-09 1.14e-08 5
#> MAD:kmeans 120 1.26e-03 5.25e-04 5
#> ATC:kmeans 136 6.20e-01 4.18e-01 5
#> SD:pam 122 1.43e-10 7.63e-12 5
#> CV:pam 125 1.18e-11 2.44e-09 5
#> MAD:pam 142 1.34e-03 9.55e-05 5
#> ATC:pam 152 1.67e-01 1.34e-01 5
#> SD:hclust 54 3.23e-04 2.08e-03 5
#> CV:hclust 79 8.52e-09 7.72e-09 5
#> MAD:hclust 65 1.16e-04 5.73e-04 5
#> ATC:hclust 107 2.99e-02 2.79e-01 5
test_to_known_factors(res_list, k = 6)
#> n disease.state(p) other(p) k
#> SD:NMF 89 8.83e-05 2.72e-04 6
#> CV:NMF 141 3.11e-09 1.78e-10 6
#> MAD:NMF 75 2.11e-01 4.16e-02 6
#> ATC:NMF 136 3.08e-01 2.61e-02 6
#> SD:skmeans 123 2.73e-06 2.27e-08 6
#> CV:skmeans 117 1.08e-09 8.66e-09 6
#> MAD:skmeans 120 7.32e-03 5.29e-04 6
#> ATC:skmeans 140 2.63e-01 2.42e-01 6
#> SD:mclust 84 2.67e-07 6.13e-08 6
#> CV:mclust 78 2.15e-08 1.05e-06 6
#> MAD:mclust 112 4.38e-04 2.57e-04 6
#> ATC:mclust 121 4.24e-01 1.09e-01 6
#> SD:kmeans 119 1.46e-10 3.73e-12 6
#> CV:kmeans 125 3.47e-12 9.34e-14 6
#> MAD:kmeans 87 3.67e-04 5.17e-04 6
#> ATC:kmeans 104 1.82e-01 1.80e-01 6
#> SD:pam 129 2.62e-09 1.01e-09 6
#> CV:pam 120 3.16e-10 3.42e-08 6
#> MAD:pam 119 2.01e-03 6.29e-03 6
#> ATC:pam 143 6.79e-02 7.83e-02 6
#> SD:hclust 48 3.29e-03 2.78e-03 6
#> CV:hclust 81 5.98e-08 5.69e-08 6
#> MAD:hclust 63 1.35e-03 8.56e-04 6
#> ATC:hclust 114 1.11e-01 3.32e-01 6
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["SD", "hclust"]
# you can also extract it by
# res = res_list["SD:hclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'SD' method.
#> Subgroups are detected by 'hclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
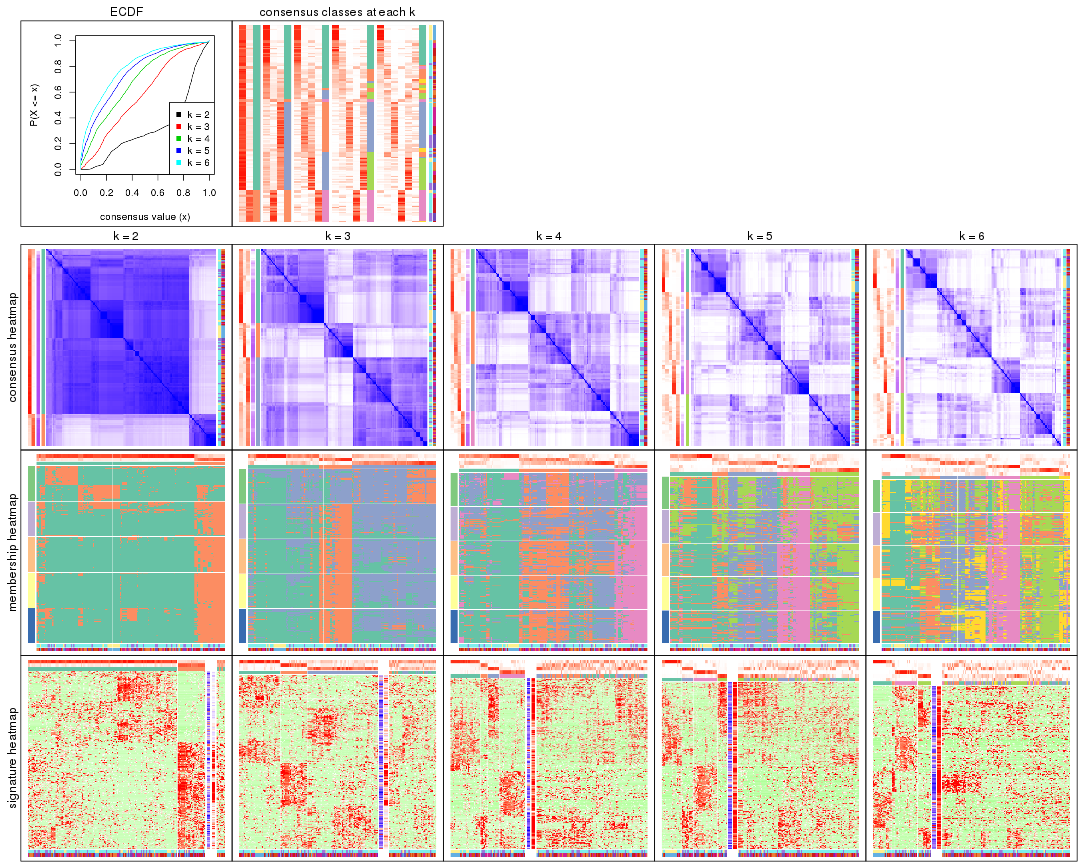
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
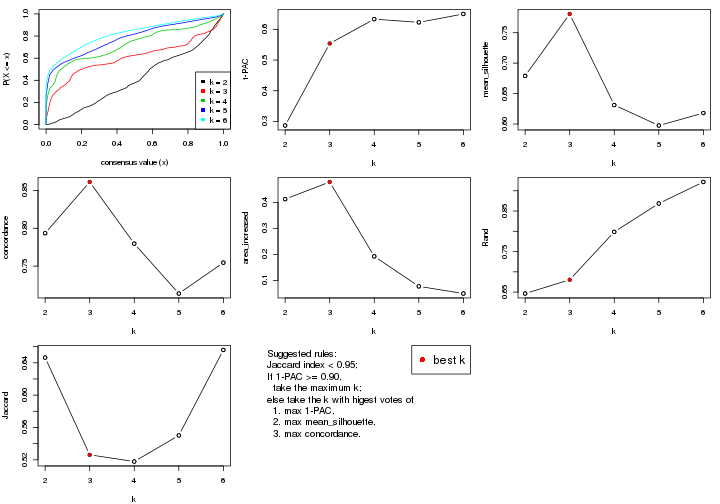
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
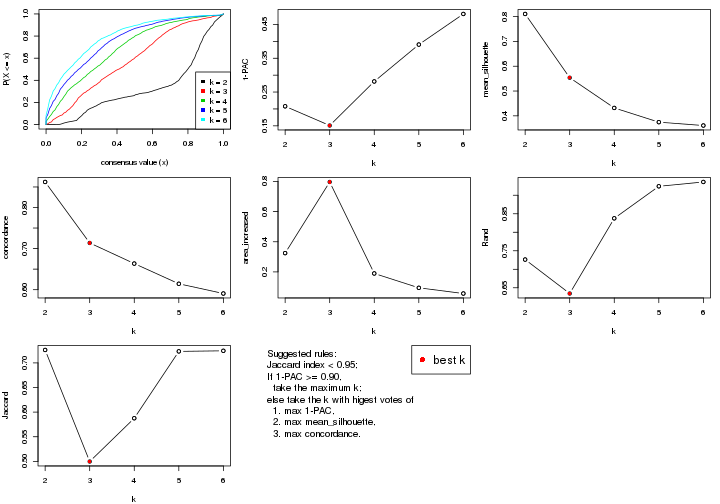
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.208 0.811 0.862 0.3245 0.726 0.726
#> 3 3 0.151 0.554 0.714 0.7965 0.634 0.500
#> 4 4 0.281 0.432 0.664 0.1895 0.838 0.588
#> 5 5 0.391 0.374 0.614 0.0943 0.924 0.723
#> 6 6 0.481 0.361 0.591 0.0565 0.936 0.725
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 1 0.5629 0.8317 0.868 0.132
#> GSM340359 1 0.5629 0.8317 0.868 0.132
#> GSM340361 1 0.5629 0.8308 0.868 0.132
#> GSM340362 1 0.0938 0.8686 0.988 0.012
#> GSM340363 1 0.7815 0.7206 0.768 0.232
#> GSM340364 1 0.5629 0.8317 0.868 0.132
#> GSM340365 1 0.4690 0.8431 0.900 0.100
#> GSM340366 1 0.5408 0.8162 0.876 0.124
#> GSM340367 1 0.5629 0.8317 0.868 0.132
#> GSM340368 2 0.7453 0.8469 0.212 0.788
#> GSM340369 2 0.7745 0.8361 0.228 0.772
#> GSM340370 1 0.5629 0.8317 0.868 0.132
#> GSM340371 1 0.2043 0.8699 0.968 0.032
#> GSM340372 1 0.5629 0.8317 0.868 0.132
#> GSM340373 1 0.5629 0.8317 0.868 0.132
#> GSM340375 1 0.5629 0.8317 0.868 0.132
#> GSM340376 1 0.5629 0.8317 0.868 0.132
#> GSM340378 1 0.5629 0.8317 0.868 0.132
#> GSM340243 1 0.5842 0.8223 0.860 0.140
#> GSM340244 1 0.2603 0.8727 0.956 0.044
#> GSM340246 1 0.2603 0.8721 0.956 0.044
#> GSM340247 2 0.6801 0.8798 0.180 0.820
#> GSM340248 1 0.4431 0.8617 0.908 0.092
#> GSM340249 1 0.9710 0.0297 0.600 0.400
#> GSM340250 1 0.8016 0.7292 0.756 0.244
#> GSM340251 1 0.6887 0.7573 0.816 0.184
#> GSM340252 1 0.9833 -0.0599 0.576 0.424
#> GSM340253 1 0.1184 0.8676 0.984 0.016
#> GSM340254 1 0.2043 0.8687 0.968 0.032
#> GSM340256 1 0.2603 0.8701 0.956 0.044
#> GSM340258 1 0.3431 0.8653 0.936 0.064
#> GSM340259 1 0.2423 0.8689 0.960 0.040
#> GSM340260 1 0.2948 0.8732 0.948 0.052
#> GSM340261 1 0.4939 0.8614 0.892 0.108
#> GSM340262 1 0.1633 0.8709 0.976 0.024
#> GSM340263 1 0.2423 0.8690 0.960 0.040
#> GSM340264 1 0.1414 0.8701 0.980 0.020
#> GSM340265 1 0.2236 0.8657 0.964 0.036
#> GSM340266 1 0.3879 0.8631 0.924 0.076
#> GSM340267 1 0.4161 0.8583 0.916 0.084
#> GSM340268 1 0.5178 0.8247 0.884 0.116
#> GSM340269 1 0.2043 0.8706 0.968 0.032
#> GSM340270 1 0.2948 0.8700 0.948 0.052
#> GSM537574 1 0.2603 0.8686 0.956 0.044
#> GSM537580 1 0.2043 0.8699 0.968 0.032
#> GSM537581 1 0.8081 0.7198 0.752 0.248
#> GSM340272 1 0.5059 0.8179 0.888 0.112
#> GSM340273 1 0.6148 0.8167 0.848 0.152
#> GSM340275 2 0.8555 0.8476 0.280 0.720
#> GSM340276 1 0.2423 0.8689 0.960 0.040
#> GSM340277 1 0.5178 0.8175 0.884 0.116
#> GSM340278 1 0.4298 0.8622 0.912 0.088
#> GSM340279 1 0.5059 0.8594 0.888 0.112
#> GSM340282 1 0.5059 0.8179 0.888 0.112
#> GSM340284 1 0.5294 0.8304 0.880 0.120
#> GSM340285 1 0.5059 0.8492 0.888 0.112
#> GSM340286 1 0.1633 0.8707 0.976 0.024
#> GSM340287 1 0.4939 0.8614 0.892 0.108
#> GSM340288 1 0.6148 0.8167 0.848 0.152
#> GSM340289 1 0.2778 0.8704 0.952 0.048
#> GSM340290 2 0.9983 0.4792 0.476 0.524
#> GSM340291 1 0.2043 0.8738 0.968 0.032
#> GSM340293 1 0.8861 0.6223 0.696 0.304
#> GSM340294 1 0.4939 0.8614 0.892 0.108
#> GSM340296 1 0.5519 0.8157 0.872 0.128
#> GSM340297 1 0.8386 0.7003 0.732 0.268
#> GSM340298 1 0.5519 0.8157 0.872 0.128
#> GSM340299 1 0.1414 0.8688 0.980 0.020
#> GSM340301 1 0.1414 0.8692 0.980 0.020
#> GSM340303 1 0.4939 0.8274 0.892 0.108
#> GSM340304 1 0.4298 0.8459 0.912 0.088
#> GSM340306 2 0.6801 0.8486 0.180 0.820
#> GSM340307 1 0.6148 0.8049 0.848 0.152
#> GSM340310 1 0.5737 0.8309 0.864 0.136
#> GSM340314 1 0.8327 0.5814 0.736 0.264
#> GSM340315 1 0.8081 0.7198 0.752 0.248
#> GSM340317 1 0.9393 0.3975 0.644 0.356
#> GSM340318 2 0.6973 0.8810 0.188 0.812
#> GSM340319 2 0.6973 0.8810 0.188 0.812
#> GSM340320 2 0.8713 0.7593 0.292 0.708
#> GSM340321 1 0.3733 0.8716 0.928 0.072
#> GSM340322 2 0.6973 0.8810 0.188 0.812
#> GSM340324 1 0.3431 0.8733 0.936 0.064
#> GSM340328 1 0.5737 0.8309 0.864 0.136
#> GSM340330 1 0.5519 0.8240 0.872 0.128
#> GSM340332 2 0.8608 0.8447 0.284 0.716
#> GSM340333 1 0.1184 0.8676 0.984 0.016
#> GSM340336 2 0.6973 0.8810 0.188 0.812
#> GSM340337 2 0.6531 0.8679 0.168 0.832
#> GSM340338 1 0.3584 0.8636 0.932 0.068
#> GSM340339 2 0.8661 0.7611 0.288 0.712
#> GSM340340 2 0.9710 0.5500 0.400 0.600
#> GSM340341 2 0.7453 0.8708 0.212 0.788
#> GSM340343 2 0.9977 0.4941 0.472 0.528
#> GSM340344 1 0.0938 0.8686 0.988 0.012
#> GSM340346 1 0.5408 0.8165 0.876 0.124
#> GSM340347 2 0.6801 0.8798 0.180 0.820
#> GSM340348 2 0.7376 0.8658 0.208 0.792
#> GSM340349 1 0.4939 0.8310 0.892 0.108
#> GSM340350 1 0.5059 0.8179 0.888 0.112
#> GSM340351 1 0.5629 0.8308 0.868 0.132
#> GSM340354 1 0.2948 0.8706 0.948 0.052
#> GSM340356 1 0.6148 0.8167 0.848 0.152
#> GSM340357 1 0.7453 0.8027 0.788 0.212
#> GSM348183 1 0.6623 0.7788 0.828 0.172
#> GSM348191 1 0.2603 0.8709 0.956 0.044
#> GSM348193 1 0.6531 0.8047 0.832 0.168
#> GSM537578 1 0.6048 0.8285 0.852 0.148
#> GSM348181 1 0.2948 0.8689 0.948 0.052
#> GSM348182 1 0.2043 0.8713 0.968 0.032
#> GSM348184 1 0.6343 0.8044 0.840 0.160
#> GSM348185 1 0.9635 0.2950 0.612 0.388
#> GSM348186 1 0.2603 0.8722 0.956 0.044
#> GSM348187 1 0.6148 0.8167 0.848 0.152
#> GSM348188 1 0.9608 0.2897 0.616 0.384
#> GSM348189 1 0.2043 0.8699 0.968 0.032
#> GSM348190 1 0.4431 0.8598 0.908 0.092
#> GSM348194 1 0.6148 0.8167 0.848 0.152
#> GSM348195 1 0.5737 0.8277 0.864 0.136
#> GSM348196 1 0.6148 0.8167 0.848 0.152
#> GSM537585 1 0.5519 0.8515 0.872 0.128
#> GSM537594 1 0.4815 0.8549 0.896 0.104
#> GSM537596 1 0.4939 0.8541 0.892 0.108
#> GSM537597 1 0.4690 0.8510 0.900 0.100
#> GSM537602 1 0.4939 0.8310 0.892 0.108
#> GSM340184 1 0.5519 0.8240 0.872 0.128
#> GSM340185 1 0.6531 0.7940 0.832 0.168
#> GSM340186 1 0.4939 0.8344 0.892 0.108
#> GSM340187 2 0.8861 0.8238 0.304 0.696
#> GSM340189 2 0.8081 0.8664 0.248 0.752
#> GSM340190 1 0.6887 0.7810 0.816 0.184
#> GSM340191 1 0.5294 0.8212 0.880 0.120
#> GSM340192 1 0.5519 0.8157 0.872 0.128
#> GSM340193 1 0.4431 0.8598 0.908 0.092
#> GSM340194 1 0.0938 0.8686 0.988 0.012
#> GSM340195 1 0.1414 0.8693 0.980 0.020
#> GSM340196 2 0.6973 0.8810 0.188 0.812
#> GSM340197 1 0.1184 0.8689 0.984 0.016
#> GSM340198 1 0.5178 0.8390 0.884 0.116
#> GSM340199 1 0.5408 0.8165 0.876 0.124
#> GSM340200 1 0.6148 0.8049 0.848 0.152
#> GSM340201 2 0.6801 0.8798 0.180 0.820
#> GSM340202 2 0.6973 0.8810 0.188 0.812
#> GSM340203 2 0.7219 0.8825 0.200 0.800
#> GSM340204 1 0.3114 0.8684 0.944 0.056
#> GSM340205 1 0.3114 0.8691 0.944 0.056
#> GSM340206 1 0.5842 0.8160 0.860 0.140
#> GSM340207 1 0.3584 0.8636 0.932 0.068
#> GSM340237 1 0.4161 0.8536 0.916 0.084
#> GSM340238 1 0.5629 0.8206 0.868 0.132
#> GSM340239 1 0.1414 0.8701 0.980 0.020
#> GSM340240 1 0.5178 0.8175 0.884 0.116
#> GSM340241 1 0.3114 0.8654 0.944 0.056
#> GSM340242 1 0.2043 0.8710 0.968 0.032
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.127 0.6882 0.972 0.004 0.024
#> GSM340359 1 0.127 0.6882 0.972 0.004 0.024
#> GSM340361 1 0.134 0.6850 0.972 0.012 0.016
#> GSM340362 3 0.835 0.6128 0.288 0.116 0.596
#> GSM340363 1 0.384 0.6070 0.872 0.116 0.012
#> GSM340364 1 0.127 0.6882 0.972 0.004 0.024
#> GSM340365 3 0.724 0.4342 0.392 0.032 0.576
#> GSM340366 3 0.504 0.6378 0.172 0.020 0.808
#> GSM340367 1 0.127 0.6882 0.972 0.004 0.024
#> GSM340368 2 0.571 0.6593 0.320 0.680 0.000
#> GSM340369 2 0.603 0.6383 0.336 0.660 0.004
#> GSM340370 1 0.127 0.6882 0.972 0.004 0.024
#> GSM340371 3 0.787 0.6642 0.216 0.124 0.660
#> GSM340372 1 0.127 0.6882 0.972 0.004 0.024
#> GSM340373 1 0.127 0.6882 0.972 0.004 0.024
#> GSM340375 1 0.127 0.6882 0.972 0.004 0.024
#> GSM340376 1 0.127 0.6882 0.972 0.004 0.024
#> GSM340378 1 0.127 0.6882 0.972 0.004 0.024
#> GSM340243 3 0.560 0.6140 0.216 0.020 0.764
#> GSM340244 1 0.796 0.4775 0.648 0.120 0.232
#> GSM340246 1 0.890 -0.0767 0.480 0.124 0.396
#> GSM340247 2 0.238 0.7927 0.044 0.940 0.016
#> GSM340248 1 0.800 0.3874 0.620 0.096 0.284
#> GSM340249 2 0.923 0.1485 0.176 0.508 0.316
#> GSM340250 1 0.917 0.0362 0.460 0.148 0.392
#> GSM340251 3 0.792 0.5864 0.108 0.248 0.644
#> GSM340252 2 0.921 0.1844 0.188 0.524 0.288
#> GSM340253 3 0.807 0.6230 0.284 0.100 0.616
#> GSM340254 3 0.856 0.2741 0.420 0.096 0.484
#> GSM340256 1 0.838 0.3424 0.600 0.124 0.276
#> GSM340258 3 0.846 0.6285 0.264 0.136 0.600
#> GSM340259 1 0.796 0.4353 0.648 0.120 0.232
#> GSM340260 1 0.857 0.1924 0.548 0.112 0.340
#> GSM340261 3 0.835 0.6113 0.280 0.120 0.600
#> GSM340262 3 0.855 0.5909 0.312 0.120 0.568
#> GSM340263 1 0.657 0.5733 0.756 0.104 0.140
#> GSM340264 3 0.855 0.5615 0.336 0.112 0.552
#> GSM340265 3 0.668 0.6901 0.168 0.088 0.744
#> GSM340266 1 0.856 0.3683 0.596 0.148 0.256
#> GSM340267 3 0.858 0.2583 0.444 0.096 0.460
#> GSM340268 3 0.816 0.6338 0.196 0.160 0.644
#> GSM340269 3 0.882 0.4895 0.364 0.124 0.512
#> GSM340270 1 0.696 0.5673 0.728 0.100 0.172
#> GSM537574 1 0.893 -0.0505 0.488 0.128 0.384
#> GSM537580 3 0.787 0.6642 0.216 0.124 0.660
#> GSM537581 1 0.755 0.4755 0.684 0.112 0.204
#> GSM340272 3 0.421 0.6425 0.128 0.016 0.856
#> GSM340273 1 0.200 0.6675 0.952 0.036 0.012
#> GSM340275 2 0.533 0.7452 0.076 0.824 0.100
#> GSM340276 1 0.796 0.4353 0.648 0.120 0.232
#> GSM340277 3 0.353 0.6424 0.092 0.016 0.892
#> GSM340278 3 0.912 0.5072 0.328 0.160 0.512
#> GSM340279 3 0.841 0.6092 0.280 0.124 0.596
#> GSM340282 3 0.435 0.6411 0.128 0.020 0.852
#> GSM340284 3 0.777 0.6515 0.172 0.152 0.676
#> GSM340285 3 0.716 0.5249 0.316 0.044 0.640
#> GSM340286 3 0.876 0.5161 0.360 0.120 0.520
#> GSM340287 3 0.835 0.6113 0.280 0.120 0.600
#> GSM340288 1 0.200 0.6675 0.952 0.036 0.012
#> GSM340289 1 0.664 0.5734 0.752 0.108 0.140
#> GSM340290 2 0.839 0.4237 0.316 0.576 0.108
#> GSM340291 1 0.816 0.3219 0.588 0.092 0.320
#> GSM340293 3 0.563 0.5220 0.044 0.164 0.792
#> GSM340294 3 0.835 0.6113 0.280 0.120 0.600
#> GSM340296 3 0.468 0.6405 0.148 0.020 0.832
#> GSM340297 1 0.902 0.0984 0.480 0.136 0.384
#> GSM340298 3 0.468 0.6405 0.148 0.020 0.832
#> GSM340299 3 0.826 0.6201 0.284 0.112 0.604
#> GSM340301 3 0.877 0.5345 0.348 0.124 0.528
#> GSM340303 3 0.398 0.6595 0.144 0.004 0.852
#> GSM340304 3 0.690 0.5597 0.292 0.040 0.668
#> GSM340306 2 0.550 0.6511 0.292 0.708 0.000
#> GSM340307 3 0.217 0.6340 0.048 0.008 0.944
#> GSM340310 1 0.117 0.6856 0.976 0.008 0.016
#> GSM340314 3 0.881 0.5119 0.140 0.312 0.548
#> GSM340315 1 0.755 0.4755 0.684 0.112 0.204
#> GSM340317 1 0.679 0.3401 0.648 0.324 0.028
#> GSM340318 2 0.230 0.7927 0.036 0.944 0.020
#> GSM340319 2 0.230 0.7927 0.036 0.944 0.020
#> GSM340320 2 0.775 0.5697 0.340 0.596 0.064
#> GSM340321 1 0.865 0.0856 0.512 0.108 0.380
#> GSM340322 2 0.230 0.7927 0.036 0.944 0.020
#> GSM340324 1 0.706 0.5587 0.708 0.080 0.212
#> GSM340328 1 0.145 0.6877 0.968 0.008 0.024
#> GSM340330 3 0.788 0.6455 0.160 0.172 0.668
#> GSM340332 2 0.636 0.7412 0.096 0.768 0.136
#> GSM340333 3 0.807 0.6230 0.284 0.100 0.616
#> GSM340336 2 0.230 0.7927 0.036 0.944 0.020
#> GSM340337 2 0.411 0.7668 0.152 0.844 0.004
#> GSM340338 3 0.819 0.6477 0.232 0.136 0.632
#> GSM340339 2 0.783 0.5753 0.340 0.592 0.068
#> GSM340340 2 0.683 0.3441 0.492 0.496 0.012
#> GSM340341 2 0.527 0.7442 0.200 0.784 0.016
#> GSM340343 2 0.795 0.4604 0.308 0.608 0.084
#> GSM340344 3 0.831 0.6105 0.292 0.112 0.596
#> GSM340346 3 0.353 0.6345 0.092 0.016 0.892
#> GSM340347 2 0.238 0.7927 0.044 0.940 0.016
#> GSM340348 2 0.574 0.7108 0.256 0.732 0.012
#> GSM340349 3 0.613 0.5786 0.268 0.020 0.712
#> GSM340350 3 0.421 0.6430 0.128 0.016 0.856
#> GSM340351 1 0.134 0.6850 0.972 0.012 0.016
#> GSM340354 3 0.898 0.4668 0.368 0.136 0.496
#> GSM340356 1 0.200 0.6675 0.952 0.036 0.012
#> GSM340357 1 0.823 0.1210 0.512 0.076 0.412
#> GSM348183 1 0.737 0.5658 0.688 0.220 0.092
#> GSM348191 1 0.860 0.2532 0.576 0.132 0.292
#> GSM348193 1 0.245 0.6736 0.936 0.052 0.012
#> GSM537578 3 0.561 0.6193 0.196 0.028 0.776
#> GSM348181 1 0.653 0.5832 0.760 0.112 0.128
#> GSM348182 1 0.884 -0.1821 0.488 0.120 0.392
#> GSM348184 3 0.542 0.6514 0.080 0.100 0.820
#> GSM348185 1 0.665 0.2610 0.640 0.340 0.020
#> GSM348186 3 0.869 0.1590 0.444 0.104 0.452
#> GSM348187 1 0.200 0.6675 0.952 0.036 0.012
#> GSM348188 1 0.714 0.2030 0.576 0.396 0.028
#> GSM348189 3 0.787 0.6642 0.216 0.124 0.660
#> GSM348190 1 0.804 0.3146 0.600 0.088 0.312
#> GSM348194 1 0.200 0.6675 0.952 0.036 0.012
#> GSM348195 1 0.164 0.6801 0.964 0.020 0.016
#> GSM348196 1 0.200 0.6675 0.952 0.036 0.012
#> GSM537585 3 0.728 0.1920 0.460 0.028 0.512
#> GSM537594 1 0.678 0.5998 0.732 0.080 0.188
#> GSM537596 1 0.694 0.5797 0.724 0.088 0.188
#> GSM537597 1 0.355 0.6750 0.896 0.080 0.024
#> GSM537602 3 0.629 0.5491 0.288 0.020 0.692
#> GSM340184 3 0.783 0.6454 0.156 0.172 0.672
#> GSM340185 3 0.257 0.6174 0.032 0.032 0.936
#> GSM340186 3 0.704 0.6579 0.128 0.144 0.728
#> GSM340187 2 0.576 0.7061 0.056 0.792 0.152
#> GSM340189 2 0.434 0.7679 0.048 0.868 0.084
#> GSM340190 3 0.288 0.6176 0.024 0.052 0.924
#> GSM340191 3 0.825 0.6313 0.200 0.164 0.636
#> GSM340192 3 0.440 0.6500 0.116 0.028 0.856
#> GSM340193 1 0.698 0.5751 0.720 0.088 0.192
#> GSM340194 3 0.835 0.6128 0.288 0.116 0.596
#> GSM340195 3 0.880 0.4450 0.396 0.116 0.488
#> GSM340196 2 0.230 0.7927 0.036 0.944 0.020
#> GSM340197 3 0.851 0.5889 0.316 0.116 0.568
#> GSM340198 3 0.933 0.2662 0.372 0.168 0.460
#> GSM340199 3 0.344 0.6342 0.088 0.016 0.896
#> GSM340200 3 0.253 0.6243 0.044 0.020 0.936
#> GSM340201 2 0.238 0.7927 0.044 0.940 0.016
#> GSM340202 2 0.230 0.7927 0.036 0.944 0.020
#> GSM340203 2 0.374 0.7915 0.072 0.892 0.036
#> GSM340204 1 0.835 0.3675 0.608 0.128 0.264
#> GSM340205 1 0.666 0.5727 0.752 0.116 0.132
#> GSM340206 3 0.792 0.6431 0.156 0.180 0.664
#> GSM340207 3 0.819 0.6477 0.232 0.136 0.632
#> GSM340237 1 0.760 0.2145 0.540 0.044 0.416
#> GSM340238 3 0.793 0.6436 0.160 0.176 0.664
#> GSM340239 3 0.846 0.6121 0.288 0.124 0.588
#> GSM340240 3 0.361 0.6419 0.096 0.016 0.888
#> GSM340241 3 0.845 0.2044 0.428 0.088 0.484
#> GSM340242 3 0.782 0.6651 0.224 0.116 0.660
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.196 0.6813 0.944 0.024 0.024 0.008
#> GSM340359 1 0.236 0.6784 0.928 0.028 0.036 0.008
#> GSM340361 1 0.250 0.6810 0.920 0.004 0.040 0.036
#> GSM340362 3 0.219 0.5059 0.032 0.020 0.936 0.012
#> GSM340363 1 0.436 0.6272 0.816 0.012 0.032 0.140
#> GSM340364 1 0.207 0.6814 0.940 0.028 0.024 0.008
#> GSM340365 3 0.698 0.3057 0.212 0.156 0.620 0.012
#> GSM340366 2 0.662 0.4537 0.096 0.596 0.304 0.004
#> GSM340367 1 0.207 0.6814 0.940 0.028 0.024 0.008
#> GSM340368 4 0.505 0.6194 0.304 0.004 0.012 0.680
#> GSM340369 4 0.527 0.5928 0.320 0.008 0.012 0.660
#> GSM340370 1 0.196 0.6813 0.944 0.024 0.024 0.008
#> GSM340371 3 0.357 0.4064 0.004 0.132 0.848 0.016
#> GSM340372 1 0.207 0.6814 0.940 0.028 0.024 0.008
#> GSM340373 1 0.207 0.6814 0.940 0.028 0.024 0.008
#> GSM340375 1 0.196 0.6813 0.944 0.024 0.024 0.008
#> GSM340376 1 0.196 0.6813 0.944 0.024 0.024 0.008
#> GSM340378 1 0.207 0.6814 0.940 0.028 0.024 0.008
#> GSM340243 2 0.694 0.4561 0.164 0.616 0.212 0.008
#> GSM340244 1 0.755 0.3065 0.536 0.136 0.308 0.020
#> GSM340246 3 0.797 0.1664 0.388 0.216 0.388 0.008
#> GSM340247 4 0.259 0.7667 0.004 0.000 0.104 0.892
#> GSM340248 1 0.770 0.2441 0.528 0.144 0.304 0.024
#> GSM340249 4 0.939 0.1496 0.120 0.200 0.288 0.392
#> GSM340250 2 0.901 0.0450 0.364 0.376 0.176 0.084
#> GSM340251 2 0.786 0.3012 0.044 0.524 0.316 0.116
#> GSM340252 4 0.880 0.1509 0.100 0.124 0.352 0.424
#> GSM340253 3 0.325 0.4885 0.044 0.060 0.888 0.008
#> GSM340254 3 0.759 0.3148 0.280 0.212 0.504 0.004
#> GSM340256 3 0.715 0.0127 0.428 0.084 0.472 0.016
#> GSM340258 3 0.655 0.1313 0.080 0.308 0.604 0.008
#> GSM340259 1 0.683 0.1410 0.500 0.068 0.420 0.012
#> GSM340260 3 0.740 0.1097 0.416 0.116 0.456 0.012
#> GSM340261 2 0.793 0.2174 0.172 0.436 0.376 0.016
#> GSM340262 3 0.302 0.5089 0.056 0.044 0.896 0.004
#> GSM340263 1 0.543 0.4853 0.668 0.028 0.300 0.004
#> GSM340264 3 0.564 0.4391 0.112 0.140 0.740 0.008
#> GSM340265 3 0.602 -0.1672 0.032 0.384 0.576 0.008
#> GSM340266 1 0.752 0.0398 0.448 0.108 0.424 0.020
#> GSM340267 3 0.759 0.3107 0.288 0.140 0.548 0.024
#> GSM340268 2 0.697 0.2424 0.064 0.492 0.424 0.020
#> GSM340269 3 0.719 0.2984 0.192 0.212 0.588 0.008
#> GSM340270 1 0.595 0.4654 0.652 0.036 0.296 0.016
#> GSM537574 3 0.738 0.2555 0.368 0.132 0.492 0.008
#> GSM537580 3 0.357 0.4064 0.004 0.132 0.848 0.016
#> GSM537581 1 0.786 0.4260 0.576 0.248 0.076 0.100
#> GSM340272 2 0.606 0.3807 0.024 0.536 0.428 0.012
#> GSM340273 1 0.433 0.6576 0.844 0.060 0.036 0.060
#> GSM340275 4 0.617 0.6783 0.032 0.064 0.200 0.704
#> GSM340276 1 0.683 0.1281 0.496 0.068 0.424 0.012
#> GSM340277 2 0.517 0.4609 0.012 0.656 0.328 0.004
#> GSM340278 2 0.879 0.0839 0.228 0.364 0.360 0.048
#> GSM340279 2 0.801 0.2218 0.172 0.436 0.372 0.020
#> GSM340282 2 0.627 0.3719 0.024 0.524 0.432 0.020
#> GSM340284 2 0.692 0.3063 0.072 0.532 0.380 0.016
#> GSM340285 2 0.805 0.3036 0.256 0.476 0.252 0.016
#> GSM340286 3 0.298 0.5191 0.108 0.004 0.880 0.008
#> GSM340287 2 0.793 0.2174 0.172 0.436 0.376 0.016
#> GSM340288 1 0.441 0.6567 0.840 0.060 0.036 0.064
#> GSM340289 1 0.557 0.4860 0.668 0.024 0.296 0.012
#> GSM340290 4 0.889 0.4013 0.232 0.080 0.224 0.464
#> GSM340291 1 0.773 0.2097 0.512 0.196 0.280 0.012
#> GSM340293 2 0.493 0.4650 0.008 0.792 0.100 0.100
#> GSM340294 2 0.793 0.2174 0.172 0.436 0.376 0.016
#> GSM340296 2 0.600 0.5037 0.084 0.680 0.232 0.004
#> GSM340297 2 0.863 0.0250 0.372 0.424 0.092 0.112
#> GSM340298 2 0.600 0.5037 0.084 0.680 0.232 0.004
#> GSM340299 3 0.240 0.5011 0.032 0.028 0.928 0.012
#> GSM340301 3 0.390 0.5224 0.136 0.016 0.836 0.012
#> GSM340303 2 0.624 0.3732 0.044 0.504 0.448 0.004
#> GSM340304 2 0.779 0.3248 0.232 0.492 0.268 0.008
#> GSM340306 4 0.566 0.6366 0.204 0.036 0.032 0.728
#> GSM340307 2 0.402 0.5161 0.004 0.772 0.224 0.000
#> GSM340310 1 0.175 0.6805 0.952 0.012 0.024 0.012
#> GSM340314 2 0.830 0.2127 0.040 0.452 0.344 0.164
#> GSM340315 1 0.786 0.4260 0.576 0.248 0.076 0.100
#> GSM340317 1 0.745 0.3801 0.576 0.044 0.092 0.288
#> GSM340318 4 0.253 0.7660 0.000 0.000 0.112 0.888
#> GSM340319 4 0.253 0.7660 0.000 0.000 0.112 0.888
#> GSM340320 4 0.755 0.5382 0.252 0.116 0.044 0.588
#> GSM340321 1 0.821 -0.0788 0.408 0.208 0.364 0.020
#> GSM340322 4 0.253 0.7649 0.000 0.000 0.112 0.888
#> GSM340324 1 0.721 0.4441 0.600 0.120 0.256 0.024
#> GSM340328 1 0.209 0.6806 0.940 0.024 0.024 0.012
#> GSM340330 2 0.695 0.2922 0.048 0.516 0.404 0.032
#> GSM340332 4 0.660 0.6907 0.040 0.124 0.140 0.696
#> GSM340333 3 0.325 0.4885 0.044 0.060 0.888 0.008
#> GSM340336 4 0.253 0.7660 0.000 0.000 0.112 0.888
#> GSM340337 4 0.462 0.7408 0.128 0.000 0.076 0.796
#> GSM340338 3 0.665 0.0017 0.072 0.364 0.556 0.008
#> GSM340339 4 0.757 0.5448 0.248 0.120 0.044 0.588
#> GSM340340 4 0.599 0.2659 0.476 0.008 0.024 0.492
#> GSM340341 4 0.578 0.7184 0.156 0.016 0.092 0.736
#> GSM340343 4 0.843 0.4593 0.228 0.056 0.208 0.508
#> GSM340344 3 0.248 0.5071 0.040 0.024 0.924 0.012
#> GSM340346 2 0.545 0.4978 0.020 0.676 0.292 0.012
#> GSM340347 4 0.259 0.7667 0.004 0.000 0.104 0.892
#> GSM340348 4 0.641 0.6913 0.176 0.036 0.088 0.700
#> GSM340349 2 0.744 0.3925 0.212 0.536 0.248 0.004
#> GSM340350 2 0.619 0.4053 0.032 0.548 0.408 0.012
#> GSM340351 1 0.250 0.6810 0.920 0.004 0.040 0.036
#> GSM340354 3 0.804 0.1784 0.228 0.284 0.472 0.016
#> GSM340356 1 0.433 0.6576 0.844 0.060 0.036 0.060
#> GSM340357 1 0.828 0.1461 0.468 0.348 0.128 0.056
#> GSM348183 1 0.695 0.4941 0.620 0.016 0.240 0.124
#> GSM348191 1 0.752 -0.0739 0.444 0.108 0.428 0.020
#> GSM348193 1 0.352 0.6731 0.876 0.016 0.032 0.076
#> GSM537578 2 0.682 0.4914 0.120 0.636 0.228 0.016
#> GSM348181 1 0.552 0.4980 0.676 0.024 0.288 0.012
#> GSM348182 3 0.494 0.4612 0.284 0.008 0.700 0.008
#> GSM348184 2 0.557 0.4712 0.036 0.708 0.240 0.016
#> GSM348185 1 0.714 0.3174 0.568 0.036 0.068 0.328
#> GSM348186 3 0.814 0.2568 0.320 0.248 0.420 0.012
#> GSM348187 1 0.441 0.6567 0.840 0.060 0.036 0.064
#> GSM348188 1 0.714 0.2480 0.536 0.012 0.104 0.348
#> GSM348189 3 0.357 0.4064 0.004 0.132 0.848 0.016
#> GSM348190 3 0.723 0.0857 0.400 0.040 0.504 0.056
#> GSM348194 1 0.441 0.6567 0.840 0.060 0.036 0.064
#> GSM348195 1 0.231 0.6806 0.928 0.004 0.028 0.040
#> GSM348196 1 0.441 0.6567 0.840 0.060 0.036 0.064
#> GSM537585 3 0.840 0.1564 0.312 0.200 0.452 0.036
#> GSM537594 1 0.729 0.5130 0.632 0.128 0.196 0.044
#> GSM537596 1 0.742 0.3982 0.568 0.060 0.308 0.064
#> GSM537597 1 0.496 0.6426 0.796 0.060 0.124 0.020
#> GSM537602 2 0.754 0.3502 0.236 0.520 0.240 0.004
#> GSM340184 2 0.696 0.2854 0.048 0.512 0.408 0.032
#> GSM340185 2 0.376 0.5085 0.004 0.812 0.180 0.004
#> GSM340186 2 0.626 0.3562 0.044 0.592 0.352 0.012
#> GSM340187 4 0.624 0.6535 0.004 0.124 0.196 0.676
#> GSM340189 4 0.489 0.7275 0.000 0.096 0.124 0.780
#> GSM340190 2 0.381 0.5063 0.000 0.812 0.176 0.012
#> GSM340191 2 0.704 0.2317 0.068 0.484 0.428 0.020
#> GSM340192 2 0.603 0.3825 0.028 0.540 0.424 0.008
#> GSM340193 1 0.737 0.3696 0.556 0.060 0.328 0.056
#> GSM340194 3 0.219 0.5059 0.032 0.020 0.936 0.012
#> GSM340195 3 0.358 0.5107 0.152 0.008 0.836 0.004
#> GSM340196 4 0.253 0.7660 0.000 0.000 0.112 0.888
#> GSM340197 3 0.275 0.5113 0.056 0.032 0.908 0.004
#> GSM340198 3 0.911 0.2189 0.252 0.256 0.412 0.080
#> GSM340199 2 0.545 0.4978 0.020 0.676 0.292 0.012
#> GSM340200 2 0.409 0.5139 0.004 0.780 0.212 0.004
#> GSM340201 4 0.259 0.7667 0.004 0.000 0.104 0.892
#> GSM340202 4 0.253 0.7660 0.000 0.000 0.112 0.888
#> GSM340203 4 0.431 0.7565 0.028 0.012 0.144 0.816
#> GSM340204 3 0.688 -0.0509 0.456 0.056 0.468 0.020
#> GSM340205 1 0.575 0.4798 0.660 0.032 0.296 0.012
#> GSM340206 2 0.707 0.3050 0.048 0.524 0.388 0.040
#> GSM340207 3 0.665 0.0017 0.072 0.364 0.556 0.008
#> GSM340237 1 0.781 0.2018 0.492 0.300 0.196 0.012
#> GSM340238 2 0.709 0.2961 0.048 0.516 0.396 0.040
#> GSM340239 3 0.281 0.4996 0.036 0.036 0.912 0.016
#> GSM340240 2 0.552 0.4029 0.004 0.556 0.428 0.012
#> GSM340241 3 0.823 0.2127 0.304 0.288 0.396 0.012
#> GSM340242 3 0.352 0.4094 0.008 0.136 0.848 0.008
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.0579 0.64109 0.984 0.008 0.000 0.000 0.008
#> GSM340359 1 0.0932 0.63416 0.972 0.004 0.000 0.004 0.020
#> GSM340361 1 0.4173 0.60293 0.760 0.204 0.000 0.008 0.028
#> GSM340362 5 0.1805 0.51369 0.012 0.008 0.020 0.016 0.944
#> GSM340363 1 0.5906 0.54865 0.652 0.216 0.004 0.108 0.020
#> GSM340364 1 0.0613 0.63945 0.984 0.004 0.000 0.004 0.008
#> GSM340365 5 0.7173 0.30103 0.148 0.076 0.180 0.012 0.584
#> GSM340366 3 0.5638 0.49001 0.072 0.052 0.716 0.008 0.152
#> GSM340367 1 0.0613 0.63945 0.984 0.004 0.000 0.004 0.008
#> GSM340368 4 0.4220 0.59125 0.300 0.008 0.000 0.688 0.004
#> GSM340369 4 0.4270 0.55899 0.336 0.004 0.000 0.656 0.004
#> GSM340370 1 0.0451 0.63975 0.988 0.000 0.000 0.004 0.008
#> GSM340371 5 0.4262 0.44064 0.004 0.020 0.140 0.040 0.796
#> GSM340372 1 0.0693 0.64123 0.980 0.012 0.000 0.000 0.008
#> GSM340373 1 0.0613 0.63945 0.984 0.004 0.000 0.004 0.008
#> GSM340375 1 0.0579 0.64109 0.984 0.008 0.000 0.000 0.008
#> GSM340376 1 0.0451 0.63975 0.988 0.000 0.000 0.004 0.008
#> GSM340378 1 0.0613 0.63945 0.984 0.004 0.000 0.004 0.008
#> GSM340243 3 0.5937 0.45548 0.136 0.084 0.704 0.012 0.064
#> GSM340244 2 0.8126 0.34146 0.268 0.400 0.056 0.020 0.256
#> GSM340246 2 0.8611 0.11278 0.168 0.324 0.172 0.012 0.324
#> GSM340247 4 0.1430 0.75241 0.000 0.004 0.000 0.944 0.052
#> GSM340248 2 0.8286 0.34269 0.220 0.428 0.100 0.016 0.236
#> GSM340249 4 0.9007 0.16568 0.056 0.148 0.168 0.396 0.232
#> GSM340250 2 0.8311 0.18189 0.156 0.428 0.280 0.020 0.116
#> GSM340251 3 0.7764 0.29030 0.000 0.172 0.476 0.120 0.232
#> GSM340252 4 0.8145 0.17364 0.012 0.156 0.108 0.416 0.308
#> GSM340253 5 0.2860 0.49839 0.016 0.036 0.044 0.008 0.896
#> GSM340254 5 0.7601 0.11057 0.076 0.288 0.144 0.008 0.484
#> GSM340256 5 0.7496 -0.09891 0.204 0.320 0.024 0.016 0.436
#> GSM340258 5 0.6956 0.16767 0.024 0.128 0.284 0.020 0.544
#> GSM340259 5 0.7844 -0.16654 0.268 0.328 0.032 0.016 0.356
#> GSM340260 5 0.8059 -0.04158 0.220 0.284 0.080 0.008 0.408
#> GSM340261 3 0.8190 0.21216 0.088 0.204 0.400 0.012 0.296
#> GSM340262 5 0.3884 0.51750 0.016 0.060 0.060 0.020 0.844
#> GSM340263 1 0.6732 0.20692 0.556 0.168 0.004 0.024 0.248
#> GSM340264 5 0.6314 0.43850 0.052 0.100 0.164 0.016 0.668
#> GSM340265 5 0.5630 -0.18245 0.004 0.032 0.472 0.016 0.476
#> GSM340266 5 0.8297 -0.15333 0.240 0.316 0.068 0.020 0.356
#> GSM340267 5 0.8109 0.18299 0.132 0.204 0.152 0.020 0.492
#> GSM340268 3 0.7435 0.19555 0.012 0.200 0.416 0.024 0.348
#> GSM340269 5 0.7915 0.26669 0.104 0.172 0.204 0.016 0.504
#> GSM340270 1 0.7346 0.17562 0.528 0.168 0.024 0.032 0.248
#> GSM537574 5 0.8243 0.02847 0.172 0.284 0.096 0.020 0.428
#> GSM537580 5 0.4262 0.44064 0.004 0.020 0.140 0.040 0.796
#> GSM537581 2 0.6964 0.08482 0.328 0.504 0.132 0.012 0.024
#> GSM340272 3 0.5049 0.39500 0.012 0.012 0.636 0.012 0.328
#> GSM340273 1 0.4734 0.47410 0.632 0.344 0.000 0.008 0.016
#> GSM340275 4 0.5649 0.64621 0.012 0.060 0.048 0.712 0.168
#> GSM340276 5 0.7837 -0.16046 0.264 0.328 0.032 0.016 0.360
#> GSM340277 3 0.4766 0.46801 0.000 0.072 0.708 0.000 0.220
#> GSM340278 3 0.8917 0.14098 0.136 0.168 0.420 0.060 0.216
#> GSM340279 3 0.8199 0.21608 0.088 0.208 0.400 0.012 0.292
#> GSM340282 3 0.5249 0.38687 0.012 0.020 0.624 0.012 0.332
#> GSM340284 3 0.7281 0.27895 0.024 0.160 0.492 0.020 0.304
#> GSM340285 3 0.7628 0.31425 0.172 0.140 0.560 0.024 0.104
#> GSM340286 5 0.3974 0.50053 0.072 0.044 0.016 0.028 0.840
#> GSM340287 3 0.8190 0.21216 0.088 0.204 0.400 0.012 0.296
#> GSM340288 1 0.4763 0.46783 0.624 0.352 0.000 0.008 0.016
#> GSM340289 1 0.6869 0.24901 0.556 0.160 0.004 0.036 0.244
#> GSM340290 4 0.8598 0.28995 0.120 0.204 0.044 0.452 0.180
#> GSM340291 2 0.8688 0.34190 0.248 0.372 0.136 0.020 0.224
#> GSM340293 3 0.4354 0.46066 0.000 0.108 0.788 0.092 0.012
#> GSM340294 3 0.8190 0.21216 0.088 0.204 0.400 0.012 0.296
#> GSM340296 3 0.4687 0.51131 0.072 0.060 0.796 0.008 0.064
#> GSM340297 2 0.7349 0.14671 0.148 0.492 0.308 0.020 0.032
#> GSM340298 3 0.4687 0.51131 0.072 0.060 0.796 0.008 0.064
#> GSM340299 5 0.1988 0.51008 0.012 0.008 0.028 0.016 0.936
#> GSM340301 5 0.4357 0.49857 0.072 0.072 0.020 0.020 0.816
#> GSM340303 3 0.5362 0.38753 0.016 0.032 0.616 0.004 0.332
#> GSM340304 3 0.7317 0.34298 0.148 0.136 0.588 0.016 0.112
#> GSM340306 4 0.5188 0.58393 0.048 0.308 0.000 0.636 0.008
#> GSM340307 3 0.3970 0.52752 0.000 0.104 0.800 0.000 0.096
#> GSM340310 1 0.1704 0.63189 0.928 0.068 0.000 0.000 0.004
#> GSM340314 3 0.8422 0.17966 0.004 0.172 0.372 0.180 0.272
#> GSM340315 2 0.6964 0.08482 0.328 0.504 0.132 0.012 0.024
#> GSM340317 1 0.7978 0.24886 0.432 0.216 0.004 0.256 0.092
#> GSM340318 4 0.1341 0.75309 0.000 0.000 0.000 0.944 0.056
#> GSM340319 4 0.1341 0.75309 0.000 0.000 0.000 0.944 0.056
#> GSM340320 4 0.6504 0.43801 0.064 0.400 0.020 0.496 0.020
#> GSM340321 2 0.8324 0.21165 0.176 0.372 0.148 0.004 0.300
#> GSM340322 4 0.1571 0.75260 0.000 0.004 0.000 0.936 0.060
#> GSM340324 1 0.7989 -0.33761 0.360 0.344 0.048 0.016 0.232
#> GSM340328 1 0.0693 0.64088 0.980 0.012 0.000 0.000 0.008
#> GSM340330 3 0.7627 0.26087 0.008 0.176 0.452 0.056 0.308
#> GSM340332 4 0.5960 0.66361 0.004 0.124 0.076 0.696 0.100
#> GSM340333 5 0.2860 0.49839 0.016 0.036 0.044 0.008 0.896
#> GSM340336 4 0.1341 0.75309 0.000 0.000 0.000 0.944 0.056
#> GSM340337 4 0.4060 0.70803 0.104 0.016 0.000 0.812 0.068
#> GSM340338 5 0.7107 0.05298 0.024 0.124 0.344 0.020 0.488
#> GSM340339 4 0.6446 0.44592 0.060 0.396 0.020 0.504 0.020
#> GSM340340 4 0.5444 0.24066 0.464 0.032 0.004 0.492 0.008
#> GSM340341 4 0.5691 0.68359 0.084 0.104 0.004 0.720 0.088
#> GSM340343 4 0.7810 0.41399 0.116 0.204 0.020 0.528 0.132
#> GSM340344 5 0.2225 0.51793 0.016 0.020 0.020 0.016 0.928
#> GSM340346 3 0.4166 0.50892 0.020 0.016 0.788 0.008 0.168
#> GSM340347 4 0.1430 0.75241 0.000 0.004 0.000 0.944 0.052
#> GSM340348 4 0.6066 0.63843 0.080 0.208 0.000 0.652 0.060
#> GSM340349 3 0.6540 0.40554 0.136 0.116 0.652 0.008 0.088
#> GSM340350 3 0.5242 0.41376 0.020 0.016 0.644 0.012 0.308
#> GSM340351 1 0.4173 0.60293 0.760 0.204 0.000 0.008 0.028
#> GSM340354 5 0.8454 0.16123 0.148 0.128 0.292 0.024 0.408
#> GSM340356 1 0.4749 0.47015 0.628 0.348 0.000 0.008 0.016
#> GSM340357 3 0.7922 -0.23578 0.336 0.248 0.360 0.016 0.040
#> GSM348183 1 0.7710 0.31304 0.512 0.148 0.004 0.136 0.200
#> GSM348191 5 0.8475 -0.07972 0.244 0.272 0.092 0.020 0.372
#> GSM348193 1 0.4925 0.59253 0.712 0.224 0.000 0.044 0.020
#> GSM537578 3 0.5259 0.50665 0.060 0.092 0.756 0.008 0.084
#> GSM348181 1 0.6818 0.25667 0.560 0.168 0.004 0.032 0.236
#> GSM348182 5 0.6221 0.32245 0.212 0.096 0.016 0.028 0.648
#> GSM348184 3 0.5872 0.45326 0.000 0.184 0.648 0.016 0.152
#> GSM348185 1 0.7795 0.24283 0.412 0.232 0.004 0.292 0.060
#> GSM348186 5 0.8119 -0.04962 0.108 0.324 0.184 0.004 0.380
#> GSM348187 1 0.4777 0.46473 0.620 0.356 0.000 0.008 0.016
#> GSM348188 1 0.7419 0.20846 0.448 0.096 0.004 0.360 0.092
#> GSM348189 5 0.4262 0.44064 0.004 0.020 0.140 0.040 0.796
#> GSM348190 5 0.6961 0.01553 0.180 0.328 0.008 0.012 0.472
#> GSM348194 1 0.4777 0.46473 0.620 0.356 0.000 0.008 0.016
#> GSM348195 1 0.3912 0.59753 0.752 0.228 0.000 0.000 0.020
#> GSM348196 1 0.4777 0.46473 0.620 0.356 0.000 0.008 0.016
#> GSM537585 5 0.8431 0.06903 0.136 0.240 0.204 0.012 0.408
#> GSM537594 2 0.7746 0.32870 0.340 0.432 0.048 0.020 0.160
#> GSM537596 2 0.7954 0.28444 0.284 0.404 0.024 0.036 0.252
#> GSM537597 1 0.5903 0.39847 0.608 0.280 0.000 0.016 0.096
#> GSM537602 3 0.6898 0.35926 0.152 0.132 0.616 0.008 0.092
#> GSM340184 3 0.7636 0.25402 0.008 0.176 0.448 0.056 0.312
#> GSM340185 3 0.3719 0.51455 0.000 0.116 0.816 0.000 0.068
#> GSM340186 3 0.6531 0.33517 0.000 0.168 0.544 0.016 0.272
#> GSM340187 4 0.5647 0.64339 0.000 0.072 0.076 0.708 0.144
#> GSM340189 4 0.4090 0.71715 0.000 0.056 0.056 0.824 0.064
#> GSM340190 3 0.4031 0.51306 0.000 0.124 0.804 0.008 0.064
#> GSM340191 3 0.7457 0.18565 0.012 0.204 0.408 0.024 0.352
#> GSM340192 3 0.4921 0.39673 0.000 0.036 0.640 0.004 0.320
#> GSM340193 2 0.7526 0.25652 0.288 0.404 0.020 0.012 0.276
#> GSM340194 5 0.1805 0.51369 0.012 0.008 0.020 0.016 0.944
#> GSM340195 5 0.4304 0.47288 0.092 0.064 0.008 0.024 0.812
#> GSM340196 4 0.1341 0.75309 0.000 0.000 0.000 0.944 0.056
#> GSM340197 5 0.3682 0.51942 0.016 0.052 0.056 0.020 0.856
#> GSM340198 5 0.8666 0.00587 0.056 0.304 0.188 0.072 0.380
#> GSM340199 3 0.4312 0.50716 0.020 0.016 0.780 0.012 0.172
#> GSM340200 3 0.3476 0.52765 0.000 0.076 0.836 0.000 0.088
#> GSM340201 4 0.1430 0.75241 0.000 0.004 0.000 0.944 0.052
#> GSM340202 4 0.1341 0.75309 0.000 0.000 0.000 0.944 0.056
#> GSM340203 4 0.3338 0.73269 0.020 0.008 0.008 0.852 0.112
#> GSM340204 5 0.7622 -0.07962 0.248 0.296 0.016 0.024 0.416
#> GSM340205 1 0.7065 0.17565 0.536 0.184 0.008 0.032 0.240
#> GSM340206 3 0.7709 0.27389 0.008 0.180 0.456 0.064 0.292
#> GSM340207 5 0.7107 0.05298 0.024 0.124 0.344 0.020 0.488
#> GSM340237 2 0.8309 0.32043 0.244 0.368 0.240 0.000 0.148
#> GSM340238 3 0.7707 0.26487 0.008 0.176 0.452 0.064 0.300
#> GSM340239 5 0.3545 0.51130 0.012 0.032 0.056 0.036 0.864
#> GSM340240 3 0.4235 0.40173 0.000 0.000 0.656 0.008 0.336
#> GSM340241 5 0.8085 -0.07373 0.088 0.340 0.216 0.004 0.352
#> GSM340242 5 0.4159 0.44562 0.004 0.024 0.144 0.028 0.800
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.0862 0.63585 0.972 0.004 0.000 0.000 0.008 0.016
#> GSM340359 1 0.0692 0.62936 0.976 0.004 0.000 0.000 0.020 0.000
#> GSM340361 1 0.5465 0.56847 0.600 0.108 0.000 0.000 0.020 0.272
#> GSM340362 5 0.1121 0.52825 0.000 0.016 0.008 0.008 0.964 0.004
#> GSM340363 1 0.6893 0.48970 0.492 0.100 0.000 0.100 0.016 0.292
#> GSM340364 1 0.0405 0.63522 0.988 0.004 0.000 0.000 0.008 0.000
#> GSM340365 5 0.7037 0.26342 0.084 0.156 0.172 0.004 0.552 0.032
#> GSM340366 3 0.4942 0.47093 0.044 0.100 0.744 0.000 0.092 0.020
#> GSM340367 1 0.0405 0.63522 0.988 0.004 0.000 0.000 0.008 0.000
#> GSM340368 4 0.4153 0.57735 0.292 0.020 0.000 0.680 0.004 0.004
#> GSM340369 4 0.3850 0.53977 0.340 0.000 0.000 0.652 0.004 0.004
#> GSM340370 1 0.0551 0.63517 0.984 0.004 0.000 0.000 0.008 0.004
#> GSM340371 5 0.4425 0.50551 0.004 0.044 0.104 0.028 0.788 0.032
#> GSM340372 1 0.0767 0.63630 0.976 0.004 0.000 0.000 0.008 0.012
#> GSM340373 1 0.0405 0.63522 0.988 0.004 0.000 0.000 0.008 0.000
#> GSM340375 1 0.0862 0.63585 0.972 0.004 0.000 0.000 0.008 0.016
#> GSM340376 1 0.0551 0.63517 0.984 0.004 0.000 0.000 0.008 0.004
#> GSM340378 1 0.0405 0.63522 0.988 0.004 0.000 0.000 0.008 0.000
#> GSM340243 3 0.4945 0.44320 0.072 0.156 0.724 0.004 0.004 0.040
#> GSM340244 2 0.6448 0.54014 0.156 0.608 0.016 0.016 0.160 0.044
#> GSM340246 2 0.7415 0.43425 0.080 0.516 0.116 0.008 0.224 0.056
#> GSM340247 4 0.0806 0.69998 0.000 0.000 0.000 0.972 0.020 0.008
#> GSM340248 2 0.7761 0.46283 0.088 0.472 0.052 0.008 0.176 0.204
#> GSM340249 4 0.8608 0.08028 0.044 0.252 0.080 0.392 0.152 0.080
#> GSM340250 6 0.7075 0.32887 0.104 0.048 0.192 0.008 0.084 0.564
#> GSM340251 3 0.8645 0.12376 0.000 0.252 0.308 0.116 0.152 0.172
#> GSM340252 4 0.7493 0.08460 0.004 0.272 0.052 0.412 0.228 0.032
#> GSM340253 5 0.2504 0.49392 0.000 0.104 0.012 0.004 0.876 0.004
#> GSM340254 2 0.6054 0.29044 0.024 0.500 0.072 0.008 0.384 0.012
#> GSM340256 2 0.7012 0.32653 0.120 0.428 0.012 0.004 0.360 0.076
#> GSM340258 5 0.7230 0.26622 0.012 0.080 0.252 0.016 0.496 0.144
#> GSM340259 2 0.8030 0.26754 0.156 0.356 0.024 0.012 0.312 0.140
#> GSM340260 5 0.7952 -0.25769 0.132 0.340 0.068 0.000 0.352 0.108
#> GSM340261 3 0.8511 0.09256 0.056 0.168 0.304 0.004 0.232 0.236
#> GSM340262 5 0.3805 0.51328 0.004 0.080 0.048 0.016 0.828 0.024
#> GSM340263 1 0.7498 0.14053 0.416 0.232 0.000 0.008 0.200 0.144
#> GSM340264 5 0.6666 0.36614 0.048 0.136 0.180 0.008 0.592 0.036
#> GSM340265 3 0.5347 0.17296 0.000 0.072 0.516 0.008 0.400 0.004
#> GSM340266 5 0.8390 -0.29479 0.152 0.312 0.044 0.012 0.312 0.168
#> GSM340267 5 0.8146 0.15179 0.084 0.088 0.172 0.008 0.428 0.220
#> GSM340268 6 0.8030 -0.07450 0.012 0.112 0.288 0.016 0.280 0.292
#> GSM340269 5 0.7828 0.13576 0.048 0.208 0.152 0.008 0.468 0.116
#> GSM340270 1 0.8156 0.12180 0.384 0.200 0.020 0.016 0.216 0.164
#> GSM537574 5 0.7975 -0.22593 0.096 0.344 0.064 0.008 0.368 0.120
#> GSM537580 5 0.4425 0.50551 0.004 0.044 0.104 0.028 0.788 0.032
#> GSM537581 6 0.4776 0.19227 0.240 0.024 0.056 0.000 0.000 0.680
#> GSM340272 3 0.4426 0.40935 0.008 0.036 0.684 0.004 0.268 0.000
#> GSM340273 1 0.4767 0.40172 0.528 0.032 0.000 0.004 0.004 0.432
#> GSM340275 4 0.5620 0.57143 0.008 0.088 0.028 0.700 0.132 0.044
#> GSM340276 2 0.8033 0.26601 0.156 0.352 0.024 0.012 0.316 0.140
#> GSM340277 3 0.4706 0.46554 0.000 0.156 0.696 0.000 0.144 0.004
#> GSM340278 3 0.8487 0.10829 0.068 0.216 0.428 0.048 0.156 0.084
#> GSM340279 3 0.8501 0.09023 0.056 0.164 0.304 0.004 0.228 0.244
#> GSM340282 3 0.4679 0.40125 0.008 0.044 0.668 0.008 0.272 0.000
#> GSM340284 3 0.8203 0.09040 0.012 0.180 0.356 0.020 0.216 0.216
#> GSM340285 3 0.7033 0.33885 0.096 0.196 0.572 0.012 0.052 0.072
#> GSM340286 5 0.3631 0.49820 0.036 0.068 0.008 0.016 0.844 0.028
#> GSM340287 3 0.8511 0.09256 0.056 0.168 0.304 0.004 0.232 0.236
#> GSM340288 1 0.4711 0.39697 0.524 0.028 0.000 0.004 0.004 0.440
#> GSM340289 1 0.7643 0.19443 0.412 0.228 0.000 0.016 0.200 0.144
#> GSM340290 4 0.8419 0.24366 0.056 0.204 0.032 0.428 0.152 0.128
#> GSM340291 2 0.6862 0.54222 0.156 0.584 0.092 0.008 0.132 0.028
#> GSM340293 3 0.5557 0.31615 0.004 0.048 0.652 0.076 0.004 0.216
#> GSM340294 3 0.8511 0.09256 0.056 0.168 0.304 0.004 0.232 0.236
#> GSM340296 3 0.3876 0.46803 0.044 0.100 0.816 0.004 0.008 0.028
#> GSM340297 6 0.5319 0.34611 0.084 0.032 0.208 0.000 0.008 0.668
#> GSM340298 3 0.3876 0.46803 0.044 0.100 0.816 0.004 0.008 0.028
#> GSM340299 5 0.1406 0.52869 0.000 0.020 0.016 0.008 0.952 0.004
#> GSM340301 5 0.3561 0.48975 0.028 0.100 0.008 0.012 0.836 0.016
#> GSM340303 3 0.5344 0.39882 0.012 0.080 0.632 0.004 0.264 0.008
#> GSM340304 3 0.6610 0.35291 0.088 0.196 0.600 0.004 0.052 0.060
#> GSM340306 4 0.4384 0.48860 0.000 0.036 0.000 0.616 0.000 0.348
#> GSM340307 3 0.5095 0.44784 0.004 0.164 0.700 0.000 0.036 0.096
#> GSM340310 1 0.3337 0.59956 0.824 0.064 0.000 0.000 0.004 0.108
#> GSM340314 6 0.8671 -0.04136 0.004 0.072 0.260 0.172 0.232 0.260
#> GSM340315 6 0.4776 0.19227 0.240 0.024 0.056 0.000 0.000 0.680
#> GSM340317 1 0.8421 0.07603 0.288 0.212 0.000 0.248 0.052 0.200
#> GSM340318 4 0.0632 0.70047 0.000 0.000 0.000 0.976 0.024 0.000
#> GSM340319 4 0.0632 0.70047 0.000 0.000 0.000 0.976 0.024 0.000
#> GSM340320 6 0.4586 -0.37368 0.004 0.008 0.008 0.484 0.004 0.492
#> GSM340321 2 0.7166 0.48205 0.084 0.552 0.084 0.004 0.200 0.076
#> GSM340322 4 0.1003 0.70020 0.000 0.004 0.000 0.964 0.028 0.004
#> GSM340324 2 0.6148 0.43953 0.244 0.580 0.028 0.000 0.128 0.020
#> GSM340328 1 0.1078 0.63470 0.964 0.012 0.000 0.000 0.008 0.016
#> GSM340330 3 0.8062 0.00826 0.008 0.080 0.316 0.040 0.264 0.292
#> GSM340332 4 0.5389 0.58652 0.000 0.016 0.056 0.696 0.076 0.156
#> GSM340333 5 0.2504 0.49392 0.000 0.104 0.012 0.004 0.876 0.004
#> GSM340336 4 0.0632 0.70047 0.000 0.000 0.000 0.976 0.024 0.000
#> GSM340337 4 0.4364 0.66561 0.072 0.068 0.000 0.788 0.060 0.012
#> GSM340338 5 0.7419 0.13375 0.012 0.080 0.324 0.016 0.424 0.144
#> GSM340339 4 0.4771 0.28088 0.008 0.012 0.008 0.488 0.004 0.480
#> GSM340340 4 0.5691 0.29440 0.420 0.024 0.000 0.484 0.008 0.064
#> GSM340341 4 0.5648 0.63299 0.052 0.076 0.000 0.704 0.076 0.092
#> GSM340343 4 0.7446 0.41242 0.056 0.188 0.008 0.528 0.092 0.128
#> GSM340344 5 0.1621 0.52349 0.000 0.048 0.008 0.004 0.936 0.004
#> GSM340346 3 0.3435 0.48297 0.016 0.016 0.836 0.004 0.112 0.016
#> GSM340347 4 0.0806 0.69998 0.000 0.000 0.000 0.972 0.020 0.008
#> GSM340348 4 0.6040 0.56384 0.040 0.060 0.000 0.628 0.056 0.216
#> GSM340349 3 0.5954 0.41203 0.084 0.160 0.664 0.004 0.032 0.056
#> GSM340350 3 0.4568 0.42796 0.016 0.032 0.696 0.004 0.248 0.004
#> GSM340351 1 0.5465 0.56847 0.600 0.108 0.000 0.000 0.020 0.272
#> GSM340354 5 0.8538 0.04437 0.084 0.140 0.260 0.012 0.364 0.140
#> GSM340356 1 0.4771 0.39753 0.524 0.032 0.000 0.004 0.004 0.436
#> GSM340357 6 0.7769 0.15405 0.292 0.092 0.292 0.008 0.012 0.304
#> GSM348183 1 0.8524 0.24682 0.368 0.176 0.000 0.120 0.172 0.164
#> GSM348191 5 0.8520 -0.22591 0.132 0.268 0.076 0.012 0.356 0.156
#> GSM348193 1 0.6301 0.54010 0.544 0.120 0.000 0.036 0.016 0.284
#> GSM537578 3 0.4542 0.44087 0.048 0.056 0.788 0.008 0.020 0.080
#> GSM348181 1 0.7640 0.19226 0.412 0.232 0.000 0.016 0.196 0.144
#> GSM348182 5 0.6063 0.31321 0.124 0.140 0.004 0.016 0.648 0.068
#> GSM348184 3 0.7057 0.30210 0.000 0.260 0.476 0.012 0.084 0.168
#> GSM348185 4 0.8219 -0.06385 0.272 0.192 0.000 0.280 0.032 0.224
#> GSM348186 2 0.6258 0.42652 0.036 0.584 0.104 0.008 0.252 0.016
#> GSM348187 1 0.4715 0.39349 0.520 0.028 0.000 0.004 0.004 0.444
#> GSM348188 4 0.8266 0.01161 0.296 0.144 0.000 0.340 0.076 0.144
#> GSM348189 5 0.4425 0.50551 0.004 0.044 0.104 0.028 0.788 0.032
#> GSM348190 5 0.7238 -0.07721 0.076 0.272 0.008 0.004 0.436 0.204
#> GSM348194 1 0.4715 0.39349 0.520 0.028 0.000 0.004 0.004 0.444
#> GSM348195 1 0.5329 0.55959 0.584 0.096 0.000 0.000 0.012 0.308
#> GSM348196 1 0.4715 0.39349 0.520 0.028 0.000 0.004 0.004 0.444
#> GSM537585 5 0.8439 0.08988 0.092 0.104 0.208 0.004 0.356 0.236
#> GSM537594 2 0.6390 0.48770 0.200 0.608 0.012 0.012 0.072 0.096
#> GSM537596 2 0.8297 0.31898 0.164 0.332 0.020 0.016 0.228 0.240
#> GSM537597 1 0.6147 0.35402 0.520 0.052 0.000 0.004 0.092 0.332
#> GSM537602 3 0.6296 0.37571 0.088 0.184 0.628 0.004 0.036 0.060
#> GSM340184 3 0.8064 0.00800 0.008 0.080 0.316 0.040 0.268 0.288
#> GSM340185 3 0.5098 0.41632 0.000 0.172 0.680 0.000 0.024 0.124
#> GSM340186 3 0.7626 0.20316 0.000 0.256 0.384 0.008 0.180 0.172
#> GSM340187 4 0.5167 0.57699 0.000 0.012 0.056 0.720 0.116 0.096
#> GSM340189 4 0.3307 0.65470 0.000 0.008 0.028 0.848 0.028 0.088
#> GSM340190 3 0.5417 0.40000 0.000 0.168 0.656 0.004 0.024 0.148
#> GSM340191 6 0.8051 -0.07287 0.012 0.116 0.284 0.016 0.284 0.288
#> GSM340192 3 0.4602 0.40881 0.000 0.068 0.668 0.000 0.260 0.004
#> GSM340193 2 0.7993 0.29959 0.152 0.332 0.016 0.004 0.244 0.252
#> GSM340194 5 0.1121 0.52825 0.000 0.016 0.008 0.008 0.964 0.004
#> GSM340195 5 0.3984 0.46829 0.036 0.100 0.004 0.012 0.812 0.036
#> GSM340196 4 0.0632 0.70047 0.000 0.000 0.000 0.976 0.024 0.000
#> GSM340197 5 0.3574 0.51484 0.004 0.080 0.044 0.016 0.840 0.016
#> GSM340198 2 0.7143 0.36320 0.012 0.520 0.116 0.072 0.252 0.028
#> GSM340199 3 0.3646 0.48148 0.016 0.024 0.824 0.004 0.116 0.016
#> GSM340200 3 0.4516 0.45965 0.000 0.116 0.756 0.000 0.048 0.080
#> GSM340201 4 0.0806 0.69998 0.000 0.000 0.000 0.972 0.020 0.008
#> GSM340202 4 0.0632 0.70047 0.000 0.000 0.000 0.976 0.024 0.000
#> GSM340203 4 0.3153 0.66957 0.012 0.008 0.004 0.848 0.112 0.016
#> GSM340204 5 0.7655 -0.27058 0.148 0.336 0.016 0.008 0.376 0.116
#> GSM340205 1 0.7693 0.11160 0.408 0.244 0.004 0.012 0.188 0.144
#> GSM340206 3 0.8133 0.01205 0.008 0.080 0.320 0.048 0.248 0.296
#> GSM340207 5 0.7419 0.13375 0.012 0.080 0.324 0.016 0.424 0.144
#> GSM340237 2 0.6357 0.46916 0.148 0.596 0.176 0.000 0.064 0.016
#> GSM340238 3 0.8141 0.00794 0.008 0.080 0.316 0.048 0.256 0.292
#> GSM340239 5 0.2899 0.53560 0.004 0.036 0.044 0.020 0.884 0.012
#> GSM340240 3 0.3808 0.41274 0.000 0.012 0.700 0.004 0.284 0.000
#> GSM340241 2 0.6156 0.43062 0.028 0.592 0.132 0.004 0.228 0.016
#> GSM340242 5 0.4324 0.50558 0.004 0.056 0.108 0.012 0.788 0.032
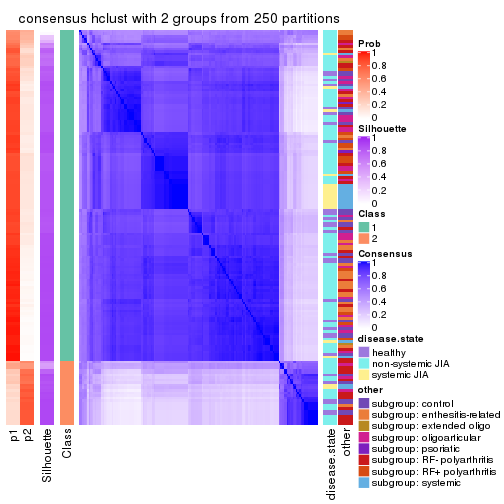
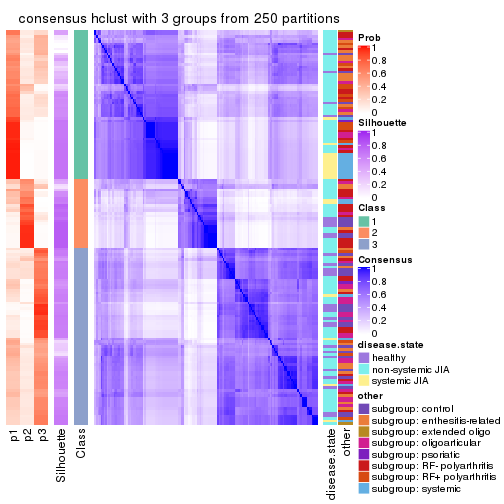
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
consensus_heatmap(res, k = 3)
consensus_heatmap(res, k = 4)
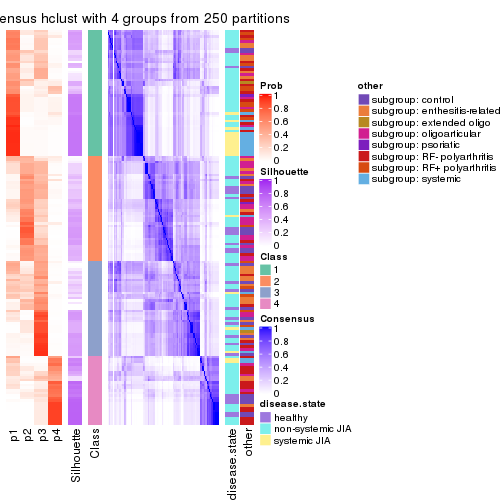
consensus_heatmap(res, k = 5)
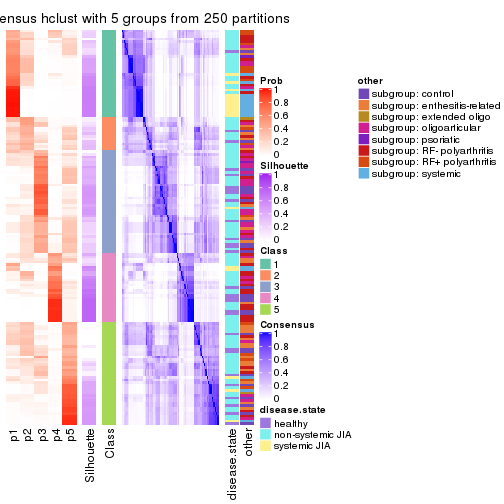
consensus_heatmap(res, k = 6)
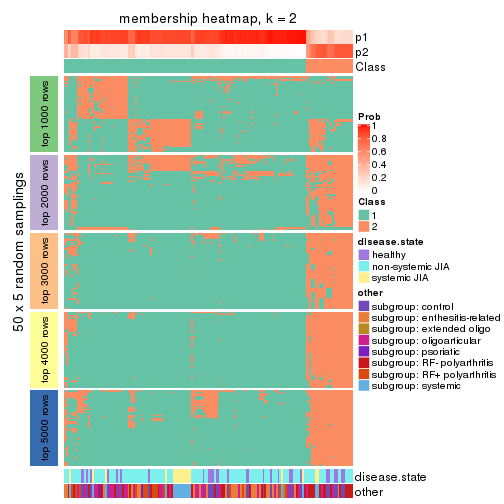
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
membership_heatmap(res, k = 3)
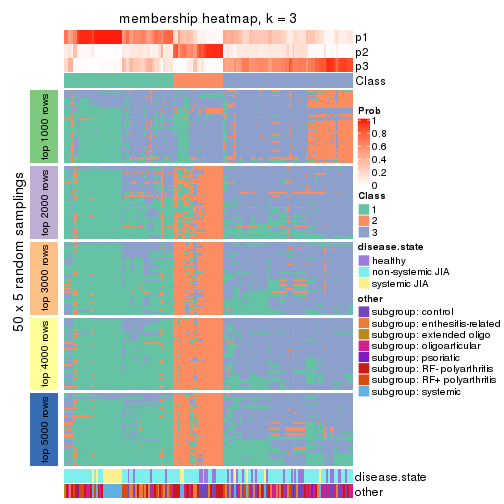
membership_heatmap(res, k = 4)
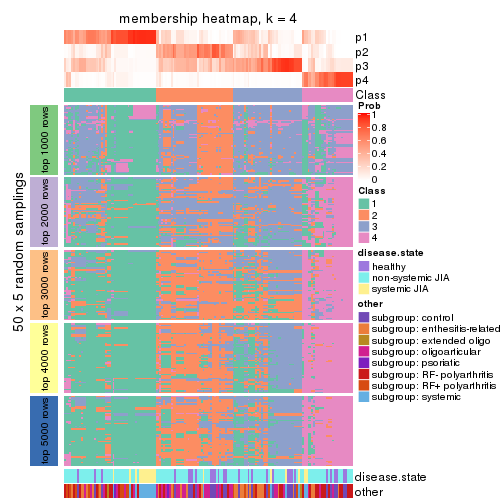
membership_heatmap(res, k = 5)
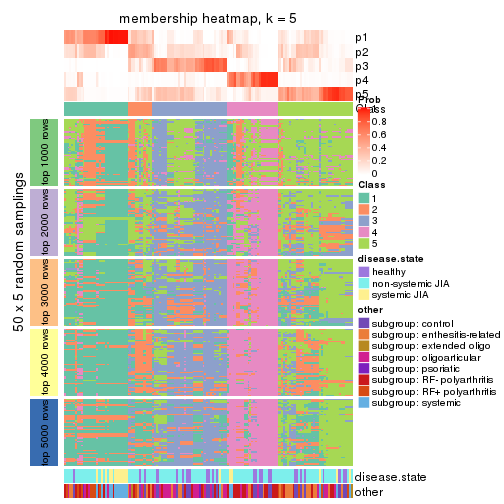
membership_heatmap(res, k = 6)
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
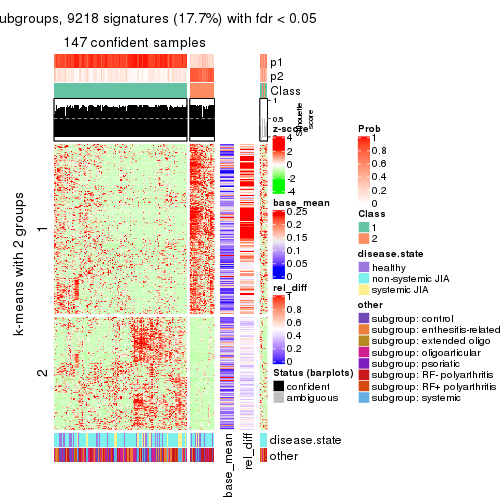
get_signatures(res, k = 3)
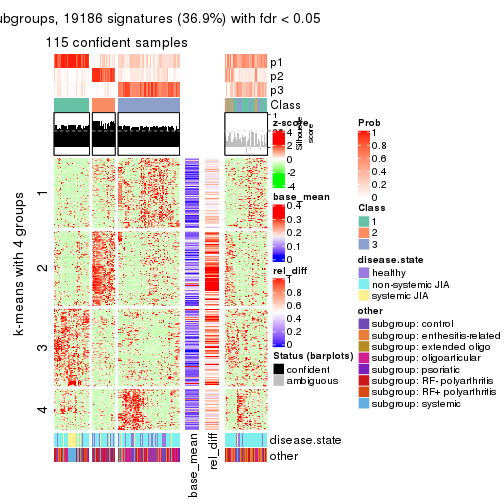
get_signatures(res, k = 4)
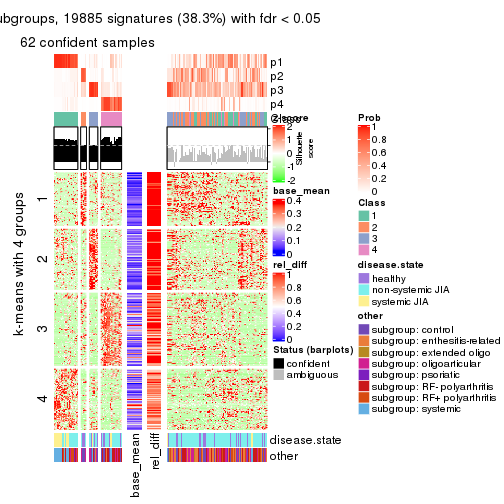
get_signatures(res, k = 5)
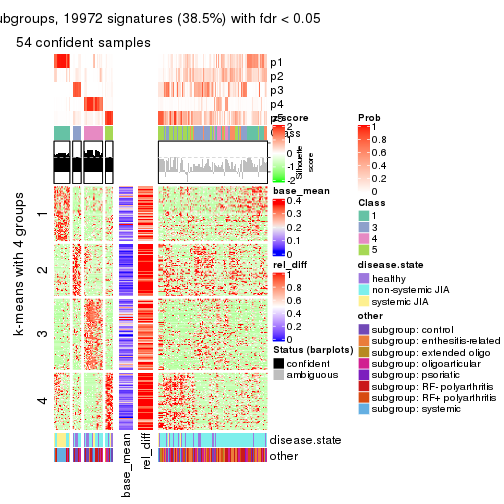
get_signatures(res, k = 6)
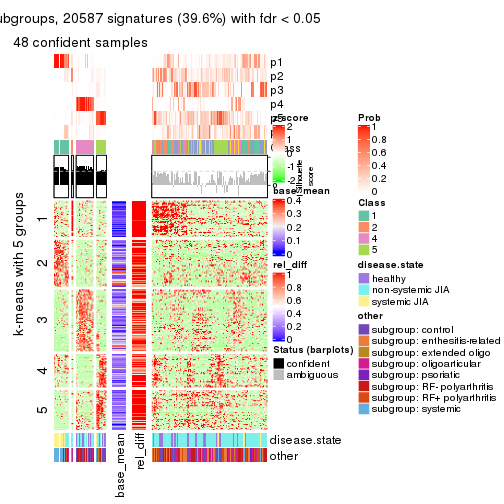
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
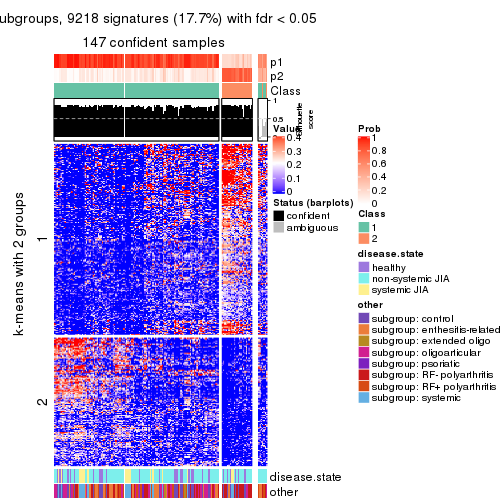
get_signatures(res, k = 3, scale_rows = FALSE)
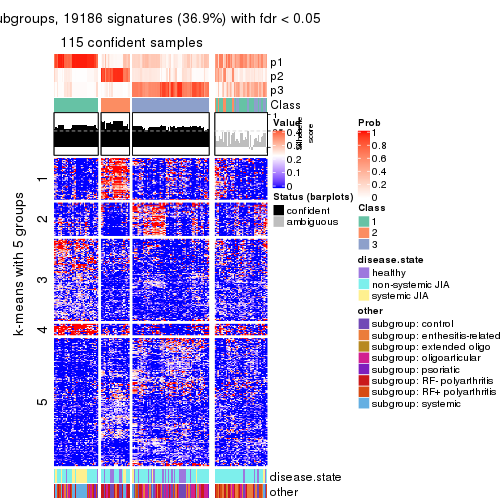
get_signatures(res, k = 4, scale_rows = FALSE)
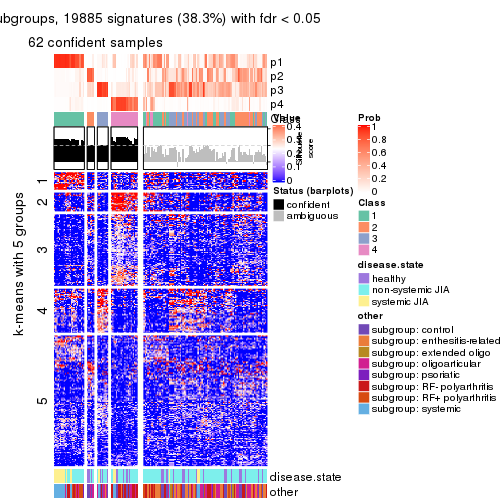
get_signatures(res, k = 5, scale_rows = FALSE)
get_signatures(res, k = 6, scale_rows = FALSE)
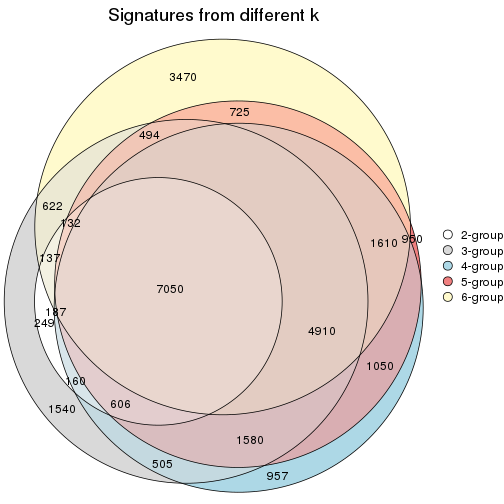
Compare the overlap of signatures from different k:
compare_signatures(res)
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
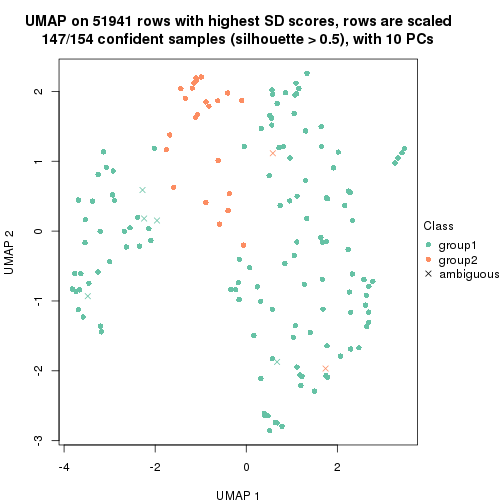
dimension_reduction(res, k = 3, method = "UMAP")
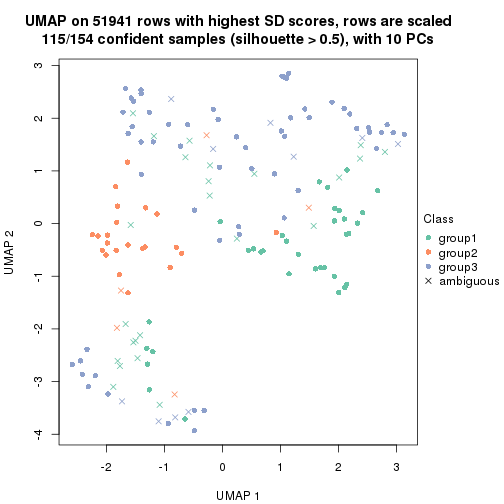
dimension_reduction(res, k = 4, method = "UMAP")
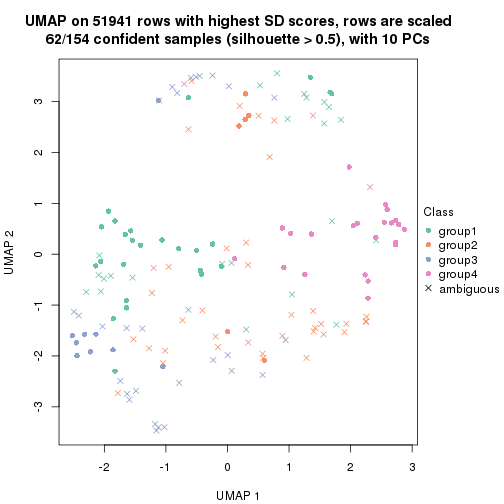
dimension_reduction(res, k = 5, method = "UMAP")
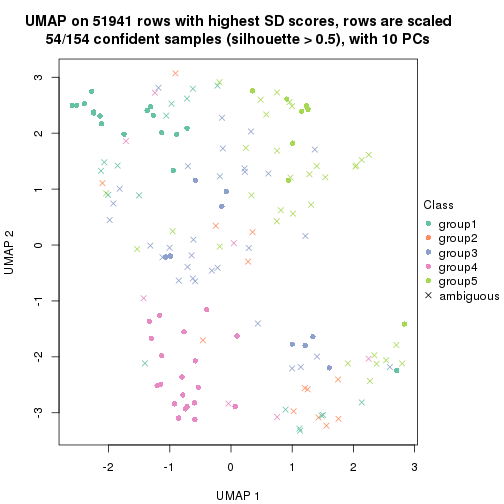
dimension_reduction(res, k = 6, method = "UMAP")
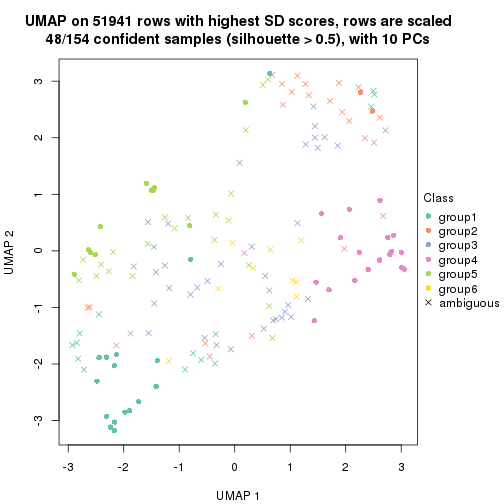
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> SD:hclust 147 0.646927 0.007140 2
#> SD:hclust 115 0.000662 0.000012 3
#> SD:hclust 62 0.003100 0.000352 4
#> SD:hclust 54 0.000323 0.002080 5
#> SD:hclust 48 0.003285 0.002778 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["SD", "kmeans"]
# you can also extract it by
# res = res_list["SD:kmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'SD' method.
#> Subgroups are detected by 'kmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.287 0.679 0.793 0.4125 0.646 0.646
#> 3 3 0.554 0.781 0.861 0.4786 0.680 0.526
#> 4 4 0.633 0.631 0.780 0.1922 0.799 0.518
#> 5 5 0.623 0.598 0.714 0.0768 0.869 0.550
#> 6 6 0.650 0.618 0.755 0.0493 0.922 0.656
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 2 0.8386 0.411 0.268 0.732
#> GSM340359 1 0.9323 0.550 0.652 0.348
#> GSM340361 1 0.9795 0.478 0.584 0.416
#> GSM340362 1 0.6531 0.696 0.832 0.168
#> GSM340363 2 0.6148 0.619 0.152 0.848
#> GSM340364 1 0.9358 0.560 0.648 0.352
#> GSM340365 1 0.4815 0.753 0.896 0.104
#> GSM340366 1 0.2423 0.772 0.960 0.040
#> GSM340367 1 0.9580 0.535 0.620 0.380
#> GSM340368 2 0.2043 0.706 0.032 0.968
#> GSM340369 2 0.2423 0.718 0.040 0.960
#> GSM340370 1 0.9881 0.451 0.564 0.436
#> GSM340371 1 0.2236 0.769 0.964 0.036
#> GSM340372 1 0.9954 0.441 0.540 0.460
#> GSM340373 1 0.9580 0.535 0.620 0.380
#> GSM340375 1 0.9881 0.451 0.564 0.436
#> GSM340376 1 0.9881 0.451 0.564 0.436
#> GSM340378 1 0.9580 0.535 0.620 0.380
#> GSM340243 1 0.3431 0.773 0.936 0.064
#> GSM340244 1 0.6712 0.696 0.824 0.176
#> GSM340246 1 0.1843 0.772 0.972 0.028
#> GSM340247 2 0.8813 0.791 0.300 0.700
#> GSM340248 1 0.6712 0.711 0.824 0.176
#> GSM340249 2 0.9087 0.770 0.324 0.676
#> GSM340250 1 0.9732 0.498 0.596 0.404
#> GSM340251 1 0.9754 -0.168 0.592 0.408
#> GSM340252 2 0.9248 0.751 0.340 0.660
#> GSM340253 1 0.0672 0.779 0.992 0.008
#> GSM340254 1 0.1184 0.778 0.984 0.016
#> GSM340256 1 0.0938 0.779 0.988 0.012
#> GSM340258 1 0.2236 0.769 0.964 0.036
#> GSM340259 1 0.1414 0.778 0.980 0.020
#> GSM340260 1 0.0672 0.779 0.992 0.008
#> GSM340261 1 0.2236 0.769 0.964 0.036
#> GSM340262 1 0.2043 0.771 0.968 0.032
#> GSM340263 1 0.7745 0.657 0.772 0.228
#> GSM340264 1 0.1184 0.776 0.984 0.016
#> GSM340265 1 0.2236 0.773 0.964 0.036
#> GSM340266 1 0.7299 0.633 0.796 0.204
#> GSM340267 1 0.2423 0.780 0.960 0.040
#> GSM340268 1 0.4939 0.711 0.892 0.108
#> GSM340269 1 0.2236 0.769 0.964 0.036
#> GSM340270 1 0.8861 0.606 0.696 0.304
#> GSM537574 1 0.1633 0.776 0.976 0.024
#> GSM537580 1 0.4815 0.715 0.896 0.104
#> GSM537581 2 0.2423 0.707 0.040 0.960
#> GSM340272 1 0.2778 0.772 0.952 0.048
#> GSM340273 1 0.9866 0.452 0.568 0.432
#> GSM340275 2 0.9087 0.770 0.324 0.676
#> GSM340276 1 0.3431 0.766 0.936 0.064
#> GSM340277 1 0.2236 0.772 0.964 0.036
#> GSM340278 1 0.6247 0.695 0.844 0.156
#> GSM340279 1 0.5737 0.698 0.864 0.136
#> GSM340282 1 0.2236 0.772 0.964 0.036
#> GSM340284 1 0.5408 0.700 0.876 0.124
#> GSM340285 1 0.7745 0.571 0.772 0.228
#> GSM340286 1 0.1843 0.775 0.972 0.028
#> GSM340287 1 0.1414 0.775 0.980 0.020
#> GSM340288 1 0.9866 0.452 0.568 0.432
#> GSM340289 1 0.2603 0.773 0.956 0.044
#> GSM340290 1 0.5294 0.705 0.880 0.120
#> GSM340291 1 0.5629 0.730 0.868 0.132
#> GSM340293 2 0.9944 0.472 0.456 0.544
#> GSM340294 1 0.2236 0.769 0.964 0.036
#> GSM340296 1 0.2778 0.772 0.952 0.048
#> GSM340297 1 0.9970 0.416 0.532 0.468
#> GSM340298 1 0.3274 0.771 0.940 0.060
#> GSM340299 1 0.2236 0.774 0.964 0.036
#> GSM340301 1 0.0938 0.778 0.988 0.012
#> GSM340303 1 0.2778 0.772 0.952 0.048
#> GSM340304 1 0.4022 0.769 0.920 0.080
#> GSM340306 2 0.2948 0.704 0.052 0.948
#> GSM340307 1 0.2948 0.771 0.948 0.052
#> GSM340310 1 0.9881 0.451 0.564 0.436
#> GSM340314 1 0.9732 -0.134 0.596 0.404
#> GSM340315 1 0.9795 0.496 0.584 0.416
#> GSM340317 2 0.7219 0.772 0.200 0.800
#> GSM340318 2 0.9000 0.778 0.316 0.684
#> GSM340319 2 0.8813 0.791 0.300 0.700
#> GSM340320 2 0.2778 0.714 0.048 0.952
#> GSM340321 1 0.1843 0.772 0.972 0.028
#> GSM340322 2 0.9248 0.751 0.340 0.660
#> GSM340324 1 0.9358 0.547 0.648 0.352
#> GSM340328 2 0.5629 0.617 0.132 0.868
#> GSM340330 1 0.4939 0.711 0.892 0.108
#> GSM340332 2 0.8813 0.791 0.300 0.700
#> GSM340333 1 0.0672 0.779 0.992 0.008
#> GSM340336 2 0.8813 0.791 0.300 0.700
#> GSM340337 2 0.6623 0.777 0.172 0.828
#> GSM340338 1 0.4939 0.711 0.892 0.108
#> GSM340339 2 0.8386 0.792 0.268 0.732
#> GSM340340 2 0.6247 0.772 0.156 0.844
#> GSM340341 2 0.8713 0.792 0.292 0.708
#> GSM340343 2 0.9087 0.770 0.324 0.676
#> GSM340344 1 0.0938 0.779 0.988 0.012
#> GSM340346 1 0.3114 0.770 0.944 0.056
#> GSM340347 2 0.8813 0.791 0.300 0.700
#> GSM340348 2 0.7376 0.787 0.208 0.792
#> GSM340349 1 0.3431 0.769 0.936 0.064
#> GSM340350 1 0.4022 0.764 0.920 0.080
#> GSM340351 1 0.9881 0.451 0.564 0.436
#> GSM340354 1 0.2236 0.769 0.964 0.036
#> GSM340356 1 0.9866 0.452 0.568 0.432
#> GSM340357 1 0.9850 0.486 0.572 0.428
#> GSM348183 1 0.9896 0.436 0.560 0.440
#> GSM348191 1 0.4939 0.711 0.892 0.108
#> GSM348193 2 0.3584 0.690 0.068 0.932
#> GSM537578 1 0.3584 0.768 0.932 0.068
#> GSM348181 1 0.4298 0.759 0.912 0.088
#> GSM348182 1 0.1633 0.777 0.976 0.024
#> GSM348184 1 0.5059 0.706 0.888 0.112
#> GSM348185 2 0.3114 0.696 0.056 0.944
#> GSM348186 1 0.0672 0.779 0.992 0.008
#> GSM348187 1 0.9881 0.451 0.564 0.436
#> GSM348188 2 0.6887 0.778 0.184 0.816
#> GSM348189 1 0.2236 0.769 0.964 0.036
#> GSM348190 1 0.2423 0.776 0.960 0.040
#> GSM348194 1 0.9866 0.452 0.568 0.432
#> GSM348195 1 0.9881 0.451 0.564 0.436
#> GSM348196 1 0.9881 0.451 0.564 0.436
#> GSM537585 1 0.8909 0.590 0.692 0.308
#> GSM537594 1 0.9795 0.472 0.584 0.416
#> GSM537596 1 0.9881 0.448 0.564 0.436
#> GSM537597 1 0.9608 0.509 0.616 0.384
#> GSM537602 1 0.4562 0.757 0.904 0.096
#> GSM340184 1 0.4939 0.711 0.892 0.108
#> GSM340185 1 0.3733 0.766 0.928 0.072
#> GSM340186 1 0.4939 0.711 0.892 0.108
#> GSM340187 2 0.9522 0.701 0.372 0.628
#> GSM340189 2 0.9552 0.694 0.376 0.624
#> GSM340190 1 0.5946 0.706 0.856 0.144
#> GSM340191 1 0.5294 0.705 0.880 0.120
#> GSM340192 1 0.2236 0.772 0.964 0.036
#> GSM340193 1 0.9427 0.534 0.640 0.360
#> GSM340194 1 0.0376 0.778 0.996 0.004
#> GSM340195 1 0.0672 0.779 0.992 0.008
#> GSM340196 2 0.8813 0.791 0.300 0.700
#> GSM340197 1 0.1414 0.778 0.980 0.020
#> GSM340198 1 0.2236 0.769 0.964 0.036
#> GSM340199 1 0.5519 0.720 0.872 0.128
#> GSM340200 1 0.3114 0.770 0.944 0.056
#> GSM340201 2 0.4690 0.743 0.100 0.900
#> GSM340202 2 0.8813 0.791 0.300 0.700
#> GSM340203 2 0.9286 0.745 0.344 0.656
#> GSM340204 1 0.0938 0.779 0.988 0.012
#> GSM340205 1 0.9815 0.479 0.580 0.420
#> GSM340206 1 0.4939 0.711 0.892 0.108
#> GSM340207 1 0.2236 0.769 0.964 0.036
#> GSM340237 1 0.6048 0.727 0.852 0.148
#> GSM340238 1 0.5408 0.700 0.876 0.124
#> GSM340239 1 0.0376 0.778 0.996 0.004
#> GSM340240 1 0.2778 0.772 0.952 0.048
#> GSM340241 1 0.1843 0.772 0.972 0.028
#> GSM340242 1 0.2236 0.769 0.964 0.036
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.1711 0.7776 0.960 0.032 0.008
#> GSM340359 1 0.1482 0.7823 0.968 0.012 0.020
#> GSM340361 1 0.1482 0.7853 0.968 0.012 0.020
#> GSM340362 3 0.4059 0.8183 0.128 0.012 0.860
#> GSM340363 1 0.2584 0.7727 0.928 0.064 0.008
#> GSM340364 1 0.1289 0.7709 0.968 0.032 0.000
#> GSM340365 1 0.9014 0.2119 0.484 0.136 0.380
#> GSM340366 3 0.5571 0.8162 0.056 0.140 0.804
#> GSM340367 1 0.1267 0.7733 0.972 0.024 0.004
#> GSM340368 2 0.4555 0.7650 0.200 0.800 0.000
#> GSM340369 2 0.4555 0.7488 0.200 0.800 0.000
#> GSM340370 1 0.1636 0.7851 0.964 0.016 0.020
#> GSM340371 3 0.1182 0.8995 0.012 0.012 0.976
#> GSM340372 1 0.1015 0.7813 0.980 0.012 0.008
#> GSM340373 1 0.1267 0.7733 0.972 0.024 0.004
#> GSM340375 1 0.1636 0.7851 0.964 0.016 0.020
#> GSM340376 1 0.1636 0.7851 0.964 0.016 0.020
#> GSM340378 1 0.1163 0.7717 0.972 0.028 0.000
#> GSM340243 1 0.9043 0.1934 0.468 0.136 0.396
#> GSM340244 1 0.6647 0.4835 0.592 0.012 0.396
#> GSM340246 3 0.1170 0.8981 0.008 0.016 0.976
#> GSM340247 2 0.4345 0.9248 0.016 0.848 0.136
#> GSM340248 3 0.6730 0.4381 0.284 0.036 0.680
#> GSM340249 2 0.4531 0.8995 0.008 0.824 0.168
#> GSM340250 1 0.7601 0.2795 0.540 0.044 0.416
#> GSM340251 3 0.0983 0.8976 0.004 0.016 0.980
#> GSM340252 2 0.4409 0.8895 0.004 0.824 0.172
#> GSM340253 3 0.2031 0.8918 0.032 0.016 0.952
#> GSM340254 3 0.1337 0.8968 0.016 0.012 0.972
#> GSM340256 3 0.2152 0.8920 0.036 0.016 0.948
#> GSM340258 3 0.0747 0.8992 0.000 0.016 0.984
#> GSM340259 3 0.4531 0.7377 0.168 0.008 0.824
#> GSM340260 3 0.0592 0.8987 0.012 0.000 0.988
#> GSM340261 3 0.0237 0.8980 0.004 0.000 0.996
#> GSM340262 3 0.0848 0.8976 0.008 0.008 0.984
#> GSM340263 1 0.6155 0.5850 0.664 0.008 0.328
#> GSM340264 3 0.1015 0.8991 0.008 0.012 0.980
#> GSM340265 3 0.5047 0.8297 0.036 0.140 0.824
#> GSM340266 3 0.1182 0.8929 0.012 0.012 0.976
#> GSM340267 3 0.3554 0.8608 0.064 0.036 0.900
#> GSM340268 3 0.0661 0.8981 0.004 0.008 0.988
#> GSM340269 3 0.0237 0.8980 0.004 0.000 0.996
#> GSM340270 1 0.5109 0.6895 0.780 0.008 0.212
#> GSM537574 3 0.0237 0.8980 0.004 0.000 0.996
#> GSM537580 3 0.0661 0.8981 0.008 0.004 0.988
#> GSM537581 2 0.5859 0.5274 0.344 0.656 0.000
#> GSM340272 3 0.5730 0.8148 0.060 0.144 0.796
#> GSM340273 1 0.2636 0.7810 0.932 0.048 0.020
#> GSM340275 2 0.4326 0.9209 0.012 0.844 0.144
#> GSM340276 3 0.5285 0.5696 0.244 0.004 0.752
#> GSM340277 3 0.5497 0.8229 0.048 0.148 0.804
#> GSM340278 3 0.3765 0.8622 0.028 0.084 0.888
#> GSM340279 3 0.2564 0.8734 0.028 0.036 0.936
#> GSM340282 3 0.5330 0.8247 0.044 0.144 0.812
#> GSM340284 3 0.1015 0.8978 0.008 0.012 0.980
#> GSM340285 3 0.6899 0.1812 0.364 0.024 0.612
#> GSM340286 3 0.1315 0.8950 0.020 0.008 0.972
#> GSM340287 3 0.1453 0.8898 0.008 0.024 0.968
#> GSM340288 1 0.2636 0.7819 0.932 0.048 0.020
#> GSM340289 1 0.6359 0.4590 0.592 0.004 0.404
#> GSM340290 3 0.0661 0.8981 0.004 0.008 0.988
#> GSM340291 1 0.6506 0.6607 0.720 0.044 0.236
#> GSM340293 3 0.6108 0.7559 0.028 0.240 0.732
#> GSM340294 3 0.0237 0.8980 0.004 0.000 0.996
#> GSM340296 3 0.5307 0.8180 0.048 0.136 0.816
#> GSM340297 3 0.7489 -0.0622 0.468 0.036 0.496
#> GSM340298 3 0.5307 0.8180 0.048 0.136 0.816
#> GSM340299 3 0.5202 0.8285 0.044 0.136 0.820
#> GSM340301 3 0.1015 0.8970 0.012 0.008 0.980
#> GSM340303 3 0.5094 0.8277 0.040 0.136 0.824
#> GSM340304 1 0.9102 0.1406 0.452 0.140 0.408
#> GSM340306 2 0.4399 0.7556 0.188 0.812 0.000
#> GSM340307 3 0.4931 0.8273 0.032 0.140 0.828
#> GSM340310 1 0.1636 0.7851 0.964 0.016 0.020
#> GSM340314 3 0.0983 0.8972 0.004 0.016 0.980
#> GSM340315 1 0.7378 0.3226 0.560 0.036 0.404
#> GSM340317 1 0.8890 0.2503 0.532 0.328 0.140
#> GSM340318 2 0.4345 0.9248 0.016 0.848 0.136
#> GSM340319 2 0.4345 0.9248 0.016 0.848 0.136
#> GSM340320 2 0.4399 0.7556 0.188 0.812 0.000
#> GSM340321 3 0.0848 0.8984 0.008 0.008 0.984
#> GSM340322 2 0.4164 0.9207 0.008 0.848 0.144
#> GSM340324 1 0.1620 0.7847 0.964 0.012 0.024
#> GSM340328 1 0.1529 0.7714 0.960 0.040 0.000
#> GSM340330 3 0.0424 0.8984 0.000 0.008 0.992
#> GSM340332 2 0.4475 0.9214 0.016 0.840 0.144
#> GSM340333 3 0.1774 0.8952 0.024 0.016 0.960
#> GSM340336 2 0.4345 0.9248 0.016 0.848 0.136
#> GSM340337 2 0.4636 0.9132 0.036 0.848 0.116
#> GSM340338 3 0.0424 0.8984 0.000 0.008 0.992
#> GSM340339 2 0.4413 0.9112 0.036 0.860 0.104
#> GSM340340 2 0.5492 0.8722 0.104 0.816 0.080
#> GSM340341 2 0.4591 0.9155 0.032 0.848 0.120
#> GSM340343 2 0.4261 0.9230 0.012 0.848 0.140
#> GSM340344 3 0.2176 0.8904 0.032 0.020 0.948
#> GSM340346 3 0.4744 0.8286 0.028 0.136 0.836
#> GSM340347 2 0.4345 0.9248 0.016 0.848 0.136
#> GSM340348 2 0.4563 0.9102 0.036 0.852 0.112
#> GSM340349 3 0.7785 0.6485 0.192 0.136 0.672
#> GSM340350 1 0.9065 0.0716 0.448 0.136 0.416
#> GSM340351 1 0.2527 0.7825 0.936 0.044 0.020
#> GSM340354 3 0.0000 0.8984 0.000 0.000 1.000
#> GSM340356 1 0.2636 0.7810 0.932 0.048 0.020
#> GSM340357 1 0.1999 0.7774 0.952 0.036 0.012
#> GSM348183 1 0.3683 0.7699 0.896 0.044 0.060
#> GSM348191 3 0.0237 0.8980 0.004 0.000 0.996
#> GSM348193 1 0.2448 0.7627 0.924 0.076 0.000
#> GSM537578 3 0.5119 0.8204 0.032 0.152 0.816
#> GSM348181 1 0.6468 0.3919 0.552 0.004 0.444
#> GSM348182 3 0.6205 0.3556 0.336 0.008 0.656
#> GSM348184 3 0.1337 0.8980 0.012 0.016 0.972
#> GSM348185 1 0.6204 0.2082 0.576 0.424 0.000
#> GSM348186 3 0.1491 0.8973 0.016 0.016 0.968
#> GSM348187 1 0.2663 0.7824 0.932 0.044 0.024
#> GSM348188 2 0.4662 0.9175 0.032 0.844 0.124
#> GSM348189 3 0.0661 0.8981 0.008 0.004 0.988
#> GSM348190 3 0.3155 0.8693 0.044 0.040 0.916
#> GSM348194 1 0.2636 0.7819 0.932 0.048 0.020
#> GSM348195 1 0.2050 0.7851 0.952 0.028 0.020
#> GSM348196 1 0.2773 0.7817 0.928 0.048 0.024
#> GSM537585 1 0.4295 0.7546 0.864 0.032 0.104
#> GSM537594 1 0.3009 0.7819 0.920 0.052 0.028
#> GSM537596 1 0.4423 0.7560 0.864 0.048 0.088
#> GSM537597 1 0.2773 0.7817 0.928 0.048 0.024
#> GSM537602 1 0.8349 0.5063 0.608 0.128 0.264
#> GSM340184 3 0.0661 0.8981 0.004 0.008 0.988
#> GSM340185 3 0.5047 0.8269 0.036 0.140 0.824
#> GSM340186 3 0.0983 0.8985 0.004 0.016 0.980
#> GSM340187 2 0.4399 0.8838 0.000 0.812 0.188
#> GSM340189 2 0.4346 0.8883 0.000 0.816 0.184
#> GSM340190 3 0.4931 0.8294 0.032 0.140 0.828
#> GSM340191 3 0.0237 0.8980 0.004 0.000 0.996
#> GSM340192 3 0.5267 0.8265 0.044 0.140 0.816
#> GSM340193 1 0.6142 0.6728 0.748 0.040 0.212
#> GSM340194 3 0.1315 0.8950 0.020 0.008 0.972
#> GSM340195 3 0.1585 0.8917 0.028 0.008 0.964
#> GSM340196 2 0.4345 0.9248 0.016 0.848 0.136
#> GSM340197 3 0.1315 0.8950 0.020 0.008 0.972
#> GSM340198 3 0.1774 0.8980 0.016 0.024 0.960
#> GSM340199 3 0.4865 0.8282 0.032 0.136 0.832
#> GSM340200 3 0.5047 0.8269 0.036 0.140 0.824
#> GSM340201 2 0.4540 0.9195 0.028 0.848 0.124
#> GSM340202 2 0.4345 0.9248 0.016 0.848 0.136
#> GSM340203 2 0.4121 0.9087 0.000 0.832 0.168
#> GSM340204 3 0.1585 0.8917 0.028 0.008 0.964
#> GSM340205 1 0.2945 0.7706 0.908 0.004 0.088
#> GSM340206 3 0.0661 0.8981 0.004 0.008 0.988
#> GSM340207 3 0.1170 0.8987 0.008 0.016 0.976
#> GSM340237 1 0.8176 0.5637 0.636 0.140 0.224
#> GSM340238 3 0.0661 0.8981 0.004 0.008 0.988
#> GSM340239 3 0.1015 0.8970 0.012 0.008 0.980
#> GSM340240 3 0.5111 0.8296 0.036 0.144 0.820
#> GSM340241 3 0.1129 0.8992 0.004 0.020 0.976
#> GSM340242 3 0.1015 0.8991 0.008 0.012 0.980
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.0336 0.8350 0.992 0.000 0.008 0.000
#> GSM340359 1 0.1388 0.8225 0.960 0.028 0.012 0.000
#> GSM340361 1 0.0000 0.8359 1.000 0.000 0.000 0.000
#> GSM340362 2 0.1471 0.7015 0.024 0.960 0.012 0.004
#> GSM340363 1 0.3508 0.8262 0.872 0.004 0.060 0.064
#> GSM340364 1 0.0469 0.8344 0.988 0.000 0.012 0.000
#> GSM340365 2 0.5746 0.4720 0.040 0.612 0.348 0.000
#> GSM340366 2 0.5125 0.4610 0.008 0.604 0.388 0.000
#> GSM340367 1 0.0469 0.8344 0.988 0.000 0.012 0.000
#> GSM340368 4 0.2921 0.8330 0.140 0.000 0.000 0.860
#> GSM340369 4 0.3528 0.7880 0.192 0.000 0.000 0.808
#> GSM340370 1 0.0469 0.8344 0.988 0.000 0.012 0.000
#> GSM340371 2 0.2179 0.6793 0.000 0.924 0.064 0.012
#> GSM340372 1 0.0469 0.8344 0.988 0.000 0.012 0.000
#> GSM340373 1 0.0469 0.8344 0.988 0.000 0.012 0.000
#> GSM340375 1 0.0188 0.8354 0.996 0.000 0.004 0.000
#> GSM340376 1 0.0469 0.8344 0.988 0.000 0.012 0.000
#> GSM340378 1 0.0469 0.8344 0.988 0.000 0.012 0.000
#> GSM340243 3 0.4989 0.4166 0.164 0.072 0.764 0.000
#> GSM340244 2 0.8130 -0.1692 0.356 0.368 0.268 0.008
#> GSM340246 3 0.5365 0.6271 0.004 0.412 0.576 0.008
#> GSM340247 4 0.1389 0.9124 0.000 0.048 0.000 0.952
#> GSM340248 3 0.6582 0.5611 0.024 0.320 0.604 0.052
#> GSM340249 4 0.1824 0.9027 0.000 0.060 0.004 0.936
#> GSM340250 3 0.7401 0.2492 0.356 0.060 0.532 0.052
#> GSM340251 3 0.5476 0.6270 0.000 0.396 0.584 0.020
#> GSM340252 4 0.2053 0.8961 0.000 0.072 0.004 0.924
#> GSM340253 2 0.1767 0.6940 0.012 0.944 0.044 0.000
#> GSM340254 2 0.1389 0.6928 0.000 0.952 0.048 0.000
#> GSM340256 2 0.3219 0.6440 0.020 0.868 0.112 0.000
#> GSM340258 2 0.5575 -0.5624 0.004 0.504 0.480 0.012
#> GSM340259 2 0.4475 0.5210 0.100 0.816 0.080 0.004
#> GSM340260 2 0.3764 0.3299 0.000 0.784 0.216 0.000
#> GSM340261 3 0.5112 0.6286 0.000 0.436 0.560 0.004
#> GSM340262 2 0.1124 0.6969 0.004 0.972 0.012 0.012
#> GSM340263 1 0.7561 0.1196 0.468 0.372 0.152 0.008
#> GSM340264 2 0.1484 0.6926 0.004 0.960 0.020 0.016
#> GSM340265 2 0.4804 0.4697 0.000 0.616 0.384 0.000
#> GSM340266 3 0.5515 0.5904 0.008 0.420 0.564 0.008
#> GSM340267 3 0.6591 0.6131 0.020 0.328 0.596 0.056
#> GSM340268 3 0.5630 0.6283 0.004 0.432 0.548 0.016
#> GSM340269 3 0.5155 0.5966 0.000 0.468 0.528 0.004
#> GSM340270 1 0.7505 0.1795 0.524 0.296 0.172 0.008
#> GSM537574 3 0.5155 0.5966 0.000 0.468 0.528 0.004
#> GSM537580 2 0.1174 0.6934 0.000 0.968 0.020 0.012
#> GSM537581 4 0.5517 0.4705 0.316 0.000 0.036 0.648
#> GSM340272 2 0.5231 0.4618 0.012 0.604 0.384 0.000
#> GSM340273 1 0.3728 0.8242 0.864 0.008 0.064 0.064
#> GSM340275 4 0.1576 0.9106 0.000 0.048 0.004 0.948
#> GSM340276 2 0.7477 -0.4406 0.152 0.428 0.416 0.004
#> GSM340277 2 0.4877 0.4580 0.000 0.592 0.408 0.000
#> GSM340278 3 0.2999 0.5523 0.000 0.132 0.864 0.004
#> GSM340279 3 0.6123 0.6039 0.000 0.336 0.600 0.064
#> GSM340282 2 0.4817 0.4677 0.000 0.612 0.388 0.000
#> GSM340284 3 0.5398 0.6312 0.000 0.404 0.580 0.016
#> GSM340285 3 0.7424 0.5321 0.108 0.360 0.512 0.020
#> GSM340286 2 0.1007 0.6981 0.008 0.976 0.008 0.008
#> GSM340287 3 0.6014 0.6141 0.000 0.360 0.588 0.052
#> GSM340288 1 0.3584 0.8248 0.868 0.004 0.064 0.064
#> GSM340289 1 0.6188 0.2177 0.532 0.424 0.036 0.008
#> GSM340290 3 0.5112 0.6286 0.000 0.436 0.560 0.004
#> GSM340291 1 0.8277 0.2062 0.472 0.168 0.320 0.040
#> GSM340293 3 0.2924 0.5268 0.000 0.100 0.884 0.016
#> GSM340294 3 0.5112 0.6286 0.000 0.436 0.560 0.004
#> GSM340296 3 0.2737 0.5212 0.008 0.104 0.888 0.000
#> GSM340297 3 0.7346 0.4248 0.272 0.080 0.596 0.052
#> GSM340298 3 0.2737 0.5212 0.008 0.104 0.888 0.000
#> GSM340299 2 0.4792 0.5141 0.008 0.680 0.312 0.000
#> GSM340301 2 0.0469 0.6968 0.000 0.988 0.012 0.000
#> GSM340303 2 0.4830 0.4622 0.000 0.608 0.392 0.000
#> GSM340304 3 0.6413 0.1231 0.320 0.076 0.600 0.004
#> GSM340306 4 0.3266 0.8186 0.064 0.004 0.048 0.884
#> GSM340307 3 0.2216 0.5205 0.000 0.092 0.908 0.000
#> GSM340310 1 0.0188 0.8354 0.996 0.000 0.004 0.000
#> GSM340314 3 0.5800 0.6303 0.000 0.420 0.548 0.032
#> GSM340315 3 0.7130 0.2503 0.356 0.044 0.548 0.052
#> GSM340317 4 0.7389 0.2839 0.352 0.060 0.052 0.536
#> GSM340318 4 0.1389 0.9124 0.000 0.048 0.000 0.952
#> GSM340319 4 0.1389 0.9124 0.000 0.048 0.000 0.952
#> GSM340320 4 0.3274 0.8199 0.056 0.004 0.056 0.884
#> GSM340321 3 0.4888 0.6279 0.000 0.412 0.588 0.000
#> GSM340322 4 0.1389 0.9124 0.000 0.048 0.000 0.952
#> GSM340324 1 0.1489 0.8333 0.952 0.004 0.044 0.000
#> GSM340328 1 0.0188 0.8354 0.996 0.000 0.004 0.000
#> GSM340330 3 0.5636 0.6265 0.004 0.436 0.544 0.016
#> GSM340332 4 0.1389 0.9124 0.000 0.048 0.000 0.952
#> GSM340333 2 0.1767 0.6940 0.012 0.944 0.044 0.000
#> GSM340336 4 0.1389 0.9124 0.000 0.048 0.000 0.952
#> GSM340337 4 0.1389 0.9088 0.000 0.048 0.000 0.952
#> GSM340338 3 0.5524 0.6217 0.004 0.432 0.552 0.012
#> GSM340339 4 0.0188 0.8805 0.000 0.000 0.004 0.996
#> GSM340340 4 0.2589 0.8510 0.116 0.000 0.000 0.884
#> GSM340341 4 0.2214 0.8722 0.000 0.028 0.044 0.928
#> GSM340343 4 0.1389 0.9124 0.000 0.048 0.000 0.952
#> GSM340344 2 0.1998 0.6946 0.016 0.944 0.020 0.020
#> GSM340346 3 0.2647 0.5224 0.000 0.120 0.880 0.000
#> GSM340347 4 0.1389 0.9124 0.000 0.048 0.000 0.952
#> GSM340348 4 0.2485 0.8474 0.004 0.016 0.064 0.916
#> GSM340349 3 0.5325 0.4055 0.160 0.096 0.744 0.000
#> GSM340350 3 0.6453 0.0553 0.360 0.080 0.560 0.000
#> GSM340351 1 0.2670 0.8321 0.908 0.000 0.040 0.052
#> GSM340354 3 0.4967 0.6263 0.000 0.452 0.548 0.000
#> GSM340356 1 0.3728 0.8242 0.864 0.008 0.064 0.064
#> GSM340357 1 0.2408 0.8366 0.920 0.000 0.044 0.036
#> GSM348183 1 0.3900 0.7864 0.860 0.060 0.016 0.064
#> GSM348191 3 0.5119 0.6257 0.000 0.440 0.556 0.004
#> GSM348193 1 0.3349 0.8280 0.880 0.004 0.052 0.064
#> GSM537578 3 0.1978 0.5213 0.000 0.068 0.928 0.004
#> GSM348181 2 0.7833 -0.1414 0.368 0.432 0.192 0.008
#> GSM348182 2 0.1356 0.6952 0.032 0.960 0.000 0.008
#> GSM348184 3 0.5300 0.6213 0.000 0.408 0.580 0.012
#> GSM348185 4 0.6763 0.2349 0.348 0.016 0.068 0.568
#> GSM348186 2 0.1762 0.6926 0.004 0.944 0.048 0.004
#> GSM348187 1 0.2928 0.8296 0.896 0.000 0.052 0.052
#> GSM348188 4 0.1557 0.9099 0.000 0.056 0.000 0.944
#> GSM348189 2 0.0992 0.6991 0.004 0.976 0.008 0.012
#> GSM348190 2 0.3888 0.6136 0.016 0.860 0.072 0.052
#> GSM348194 1 0.3584 0.8248 0.868 0.004 0.064 0.064
#> GSM348195 1 0.1913 0.8354 0.940 0.000 0.020 0.040
#> GSM348196 1 0.3584 0.8248 0.868 0.004 0.064 0.064
#> GSM537585 1 0.7463 0.2743 0.504 0.384 0.064 0.048
#> GSM537594 1 0.5058 0.8027 0.804 0.044 0.092 0.060
#> GSM537596 1 0.6124 0.7454 0.740 0.120 0.076 0.064
#> GSM537597 1 0.3728 0.8241 0.864 0.008 0.064 0.064
#> GSM537602 1 0.6206 0.4130 0.540 0.056 0.404 0.000
#> GSM340184 3 0.5643 0.6230 0.004 0.440 0.540 0.016
#> GSM340185 3 0.2281 0.5192 0.000 0.096 0.904 0.000
#> GSM340186 3 0.5473 0.6204 0.004 0.408 0.576 0.012
#> GSM340187 4 0.1716 0.9021 0.000 0.064 0.000 0.936
#> GSM340189 4 0.1890 0.9052 0.000 0.056 0.008 0.936
#> GSM340190 3 0.2216 0.5205 0.000 0.092 0.908 0.000
#> GSM340191 3 0.5648 0.6149 0.004 0.444 0.536 0.016
#> GSM340192 2 0.4830 0.4677 0.000 0.608 0.392 0.000
#> GSM340193 1 0.7781 0.3994 0.520 0.340 0.076 0.064
#> GSM340194 2 0.1042 0.7010 0.008 0.972 0.020 0.000
#> GSM340195 2 0.0779 0.6977 0.016 0.980 0.004 0.000
#> GSM340196 4 0.1389 0.9124 0.000 0.048 0.000 0.952
#> GSM340197 2 0.1139 0.6991 0.012 0.972 0.008 0.008
#> GSM340198 2 0.1909 0.6916 0.004 0.940 0.048 0.008
#> GSM340199 3 0.2647 0.5246 0.000 0.120 0.880 0.000
#> GSM340200 3 0.2216 0.5205 0.000 0.092 0.908 0.000
#> GSM340201 4 0.1302 0.9113 0.000 0.044 0.000 0.956
#> GSM340202 4 0.1389 0.9124 0.000 0.048 0.000 0.952
#> GSM340203 4 0.1743 0.9091 0.000 0.056 0.004 0.940
#> GSM340204 2 0.1388 0.6847 0.012 0.960 0.028 0.000
#> GSM340205 1 0.3638 0.7473 0.848 0.120 0.032 0.000
#> GSM340206 3 0.5459 0.6294 0.000 0.432 0.552 0.016
#> GSM340207 2 0.2125 0.6820 0.004 0.932 0.052 0.012
#> GSM340237 3 0.5649 0.0689 0.344 0.036 0.620 0.000
#> GSM340238 3 0.5630 0.6283 0.004 0.432 0.548 0.016
#> GSM340239 2 0.0992 0.6978 0.004 0.976 0.012 0.008
#> GSM340240 2 0.4804 0.4697 0.000 0.616 0.384 0.000
#> GSM340241 3 0.5303 0.5882 0.004 0.448 0.544 0.004
#> GSM340242 2 0.1593 0.6916 0.004 0.956 0.024 0.016
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.0290 0.73555 0.992 0.008 0.000 0.000 0.000
#> GSM340359 1 0.1310 0.71652 0.956 0.024 0.000 0.000 0.020
#> GSM340361 1 0.1209 0.74019 0.964 0.012 0.012 0.000 0.012
#> GSM340362 5 0.1488 0.75488 0.016 0.008 0.012 0.008 0.956
#> GSM340363 1 0.6673 0.69226 0.528 0.232 0.228 0.004 0.008
#> GSM340364 1 0.0771 0.72939 0.976 0.020 0.000 0.000 0.004
#> GSM340365 5 0.4610 -0.03314 0.016 0.388 0.000 0.000 0.596
#> GSM340366 2 0.4937 0.45373 0.004 0.580 0.024 0.000 0.392
#> GSM340367 1 0.0771 0.72939 0.976 0.020 0.000 0.000 0.004
#> GSM340368 4 0.2848 0.78048 0.156 0.000 0.004 0.840 0.000
#> GSM340369 4 0.3300 0.73461 0.204 0.004 0.000 0.792 0.000
#> GSM340370 1 0.0671 0.73100 0.980 0.016 0.000 0.000 0.004
#> GSM340371 5 0.3959 0.69622 0.000 0.068 0.104 0.012 0.816
#> GSM340372 1 0.0671 0.73100 0.980 0.016 0.000 0.000 0.004
#> GSM340373 1 0.0771 0.72939 0.976 0.020 0.000 0.000 0.004
#> GSM340375 1 0.0324 0.73397 0.992 0.004 0.000 0.000 0.004
#> GSM340376 1 0.0671 0.73100 0.980 0.016 0.000 0.000 0.004
#> GSM340378 1 0.0771 0.72939 0.976 0.020 0.000 0.000 0.004
#> GSM340243 2 0.5597 0.45287 0.056 0.616 0.308 0.000 0.020
#> GSM340244 3 0.8063 0.33425 0.084 0.344 0.348 0.004 0.220
#> GSM340246 3 0.5691 0.59814 0.000 0.128 0.632 0.004 0.236
#> GSM340247 4 0.0000 0.87058 0.000 0.000 0.000 1.000 0.000
#> GSM340248 3 0.4168 0.47744 0.000 0.184 0.764 0.000 0.052
#> GSM340249 4 0.1668 0.84406 0.000 0.000 0.032 0.940 0.028
#> GSM340250 3 0.4986 0.36095 0.180 0.064 0.732 0.000 0.024
#> GSM340251 3 0.6276 0.53515 0.000 0.204 0.560 0.004 0.232
#> GSM340252 4 0.2972 0.80258 0.000 0.004 0.040 0.872 0.084
#> GSM340253 5 0.2125 0.73694 0.004 0.024 0.052 0.000 0.920
#> GSM340254 5 0.3051 0.71593 0.000 0.076 0.060 0.000 0.864
#> GSM340256 5 0.6557 -0.00929 0.000 0.340 0.212 0.000 0.448
#> GSM340258 3 0.4520 0.61562 0.000 0.008 0.644 0.008 0.340
#> GSM340259 5 0.6342 -0.15519 0.000 0.128 0.380 0.008 0.484
#> GSM340260 5 0.5710 -0.27416 0.000 0.060 0.464 0.008 0.468
#> GSM340261 3 0.4083 0.66245 0.000 0.008 0.728 0.008 0.256
#> GSM340262 5 0.2305 0.73776 0.000 0.000 0.092 0.012 0.896
#> GSM340263 3 0.8596 0.32473 0.216 0.196 0.340 0.004 0.244
#> GSM340264 5 0.4048 0.58397 0.000 0.016 0.208 0.012 0.764
#> GSM340265 2 0.5083 0.39135 0.000 0.532 0.036 0.000 0.432
#> GSM340266 3 0.6355 0.58759 0.000 0.164 0.564 0.012 0.260
#> GSM340267 3 0.4122 0.40504 0.008 0.132 0.796 0.000 0.064
#> GSM340268 3 0.4444 0.65830 0.000 0.016 0.708 0.012 0.264
#> GSM340269 3 0.4870 0.64765 0.000 0.040 0.680 0.008 0.272
#> GSM340270 3 0.8437 0.41233 0.180 0.140 0.396 0.012 0.272
#> GSM537574 3 0.5047 0.63673 0.000 0.064 0.652 0.000 0.284
#> GSM537580 5 0.4036 0.67510 0.000 0.052 0.132 0.012 0.804
#> GSM537581 4 0.6504 0.43238 0.196 0.024 0.200 0.580 0.000
#> GSM340272 2 0.4814 0.43506 0.004 0.568 0.016 0.000 0.412
#> GSM340273 1 0.6785 0.67970 0.504 0.256 0.228 0.004 0.008
#> GSM340275 4 0.0451 0.86740 0.000 0.000 0.004 0.988 0.008
#> GSM340276 3 0.7184 0.47043 0.016 0.256 0.468 0.008 0.252
#> GSM340277 2 0.4638 0.46977 0.000 0.648 0.028 0.000 0.324
#> GSM340278 3 0.5088 0.10819 0.004 0.392 0.572 0.000 0.032
#> GSM340279 3 0.1788 0.52709 0.000 0.008 0.932 0.004 0.056
#> GSM340282 2 0.4833 0.44236 0.000 0.564 0.024 0.000 0.412
#> GSM340284 3 0.4479 0.65856 0.000 0.036 0.720 0.004 0.240
#> GSM340285 3 0.7084 0.47593 0.016 0.284 0.484 0.008 0.208
#> GSM340286 5 0.1444 0.75592 0.000 0.000 0.040 0.012 0.948
#> GSM340287 3 0.2971 0.62811 0.000 0.008 0.836 0.000 0.156
#> GSM340288 1 0.6609 0.69573 0.540 0.220 0.228 0.004 0.008
#> GSM340289 5 0.8633 -0.33275 0.228 0.156 0.300 0.008 0.308
#> GSM340290 3 0.4751 0.65618 0.000 0.036 0.692 0.008 0.264
#> GSM340291 3 0.7487 0.35616 0.080 0.360 0.424 0.000 0.136
#> GSM340293 2 0.4995 0.53783 0.000 0.552 0.420 0.004 0.024
#> GSM340294 3 0.4183 0.66126 0.000 0.008 0.712 0.008 0.272
#> GSM340296 2 0.4726 0.56181 0.000 0.580 0.400 0.000 0.020
#> GSM340297 3 0.3182 0.45338 0.096 0.028 0.864 0.004 0.008
#> GSM340298 2 0.4726 0.56181 0.000 0.580 0.400 0.000 0.020
#> GSM340299 5 0.3675 0.40321 0.004 0.216 0.008 0.000 0.772
#> GSM340301 5 0.1168 0.75632 0.000 0.000 0.032 0.008 0.960
#> GSM340303 2 0.4909 0.43988 0.000 0.560 0.028 0.000 0.412
#> GSM340304 2 0.5005 0.40285 0.060 0.720 0.200 0.000 0.020
#> GSM340306 4 0.6451 0.53415 0.056 0.104 0.232 0.608 0.000
#> GSM340307 2 0.4356 0.57517 0.000 0.648 0.340 0.000 0.012
#> GSM340310 1 0.0290 0.73713 0.992 0.008 0.000 0.000 0.000
#> GSM340314 3 0.4469 0.65989 0.000 0.016 0.704 0.012 0.268
#> GSM340315 3 0.4462 0.31952 0.184 0.044 0.760 0.004 0.008
#> GSM340317 4 0.7034 0.41246 0.084 0.288 0.068 0.548 0.012
#> GSM340318 4 0.0000 0.87058 0.000 0.000 0.000 1.000 0.000
#> GSM340319 4 0.0000 0.87058 0.000 0.000 0.000 1.000 0.000
#> GSM340320 4 0.6814 0.48161 0.052 0.148 0.232 0.568 0.000
#> GSM340321 3 0.5450 0.61633 0.000 0.124 0.648 0.000 0.228
#> GSM340322 4 0.0000 0.87058 0.000 0.000 0.000 1.000 0.000
#> GSM340324 1 0.5027 0.69488 0.724 0.196 0.048 0.000 0.032
#> GSM340328 1 0.1043 0.73743 0.960 0.040 0.000 0.000 0.000
#> GSM340330 3 0.4493 0.65902 0.000 0.016 0.700 0.012 0.272
#> GSM340332 4 0.0290 0.86874 0.000 0.000 0.008 0.992 0.000
#> GSM340333 5 0.2125 0.73694 0.004 0.024 0.052 0.000 0.920
#> GSM340336 4 0.0000 0.87058 0.000 0.000 0.000 1.000 0.000
#> GSM340337 4 0.0771 0.86557 0.000 0.000 0.020 0.976 0.004
#> GSM340338 3 0.5923 0.60398 0.000 0.108 0.604 0.012 0.276
#> GSM340339 4 0.0404 0.86627 0.000 0.000 0.012 0.988 0.000
#> GSM340340 4 0.2629 0.79554 0.136 0.000 0.004 0.860 0.000
#> GSM340341 4 0.4201 0.74421 0.004 0.136 0.064 0.792 0.004
#> GSM340343 4 0.0000 0.87058 0.000 0.000 0.000 1.000 0.000
#> GSM340344 5 0.1644 0.74642 0.004 0.008 0.048 0.000 0.940
#> GSM340346 2 0.4982 0.55285 0.000 0.556 0.412 0.000 0.032
#> GSM340347 4 0.0000 0.87058 0.000 0.000 0.000 1.000 0.000
#> GSM340348 4 0.6437 0.52614 0.020 0.152 0.232 0.592 0.004
#> GSM340349 2 0.4660 0.56794 0.024 0.724 0.228 0.000 0.024
#> GSM340350 2 0.5799 0.55270 0.168 0.676 0.124 0.000 0.032
#> GSM340351 1 0.5759 0.71957 0.648 0.116 0.224 0.004 0.008
#> GSM340354 3 0.5295 0.64011 0.000 0.064 0.648 0.008 0.280
#> GSM340356 1 0.6801 0.67780 0.500 0.260 0.228 0.004 0.008
#> GSM340357 1 0.5913 0.71465 0.636 0.164 0.188 0.000 0.012
#> GSM348183 1 0.6837 0.63880 0.652 0.116 0.036 0.080 0.116
#> GSM348191 3 0.4524 0.65705 0.000 0.020 0.692 0.008 0.280
#> GSM348193 1 0.6463 0.70551 0.564 0.196 0.228 0.004 0.008
#> GSM537578 2 0.4707 0.56307 0.000 0.588 0.392 0.000 0.020
#> GSM348181 3 0.8386 0.40451 0.148 0.152 0.392 0.012 0.296
#> GSM348182 5 0.1812 0.75495 0.004 0.008 0.036 0.012 0.940
#> GSM348184 3 0.6231 0.53113 0.000 0.204 0.568 0.004 0.224
#> GSM348185 4 0.8053 0.10781 0.104 0.268 0.236 0.392 0.000
#> GSM348186 5 0.3439 0.70319 0.004 0.092 0.060 0.000 0.844
#> GSM348187 1 0.6587 0.69707 0.544 0.220 0.224 0.004 0.008
#> GSM348188 4 0.0771 0.86557 0.000 0.000 0.020 0.976 0.004
#> GSM348189 5 0.3576 0.70928 0.000 0.048 0.100 0.012 0.840
#> GSM348190 5 0.3815 0.52201 0.004 0.012 0.220 0.000 0.764
#> GSM348194 1 0.6609 0.69573 0.540 0.220 0.228 0.004 0.008
#> GSM348195 1 0.4519 0.73141 0.752 0.048 0.188 0.000 0.012
#> GSM348196 1 0.6609 0.69573 0.540 0.220 0.228 0.004 0.008
#> GSM537585 1 0.7938 0.32332 0.352 0.084 0.224 0.000 0.340
#> GSM537594 1 0.7483 0.58247 0.388 0.320 0.260 0.004 0.028
#> GSM537596 3 0.7301 -0.08914 0.156 0.280 0.504 0.004 0.056
#> GSM537597 1 0.7000 0.65652 0.472 0.268 0.244 0.004 0.012
#> GSM537602 2 0.4993 0.28554 0.184 0.728 0.068 0.000 0.020
#> GSM340184 3 0.4194 0.65945 0.000 0.004 0.708 0.012 0.276
#> GSM340185 2 0.4356 0.57329 0.000 0.648 0.340 0.000 0.012
#> GSM340186 3 0.6267 0.54080 0.000 0.196 0.560 0.004 0.240
#> GSM340187 4 0.0451 0.86671 0.000 0.000 0.008 0.988 0.004
#> GSM340189 4 0.0290 0.86868 0.000 0.000 0.008 0.992 0.000
#> GSM340190 2 0.4327 0.55603 0.000 0.632 0.360 0.000 0.008
#> GSM340191 3 0.4419 0.65792 0.000 0.012 0.700 0.012 0.276
#> GSM340192 2 0.4757 0.45661 0.000 0.596 0.024 0.000 0.380
#> GSM340193 3 0.7042 0.13456 0.072 0.276 0.544 0.004 0.104
#> GSM340194 5 0.1143 0.75591 0.004 0.008 0.012 0.008 0.968
#> GSM340195 5 0.1059 0.75630 0.004 0.000 0.020 0.008 0.968
#> GSM340196 4 0.0000 0.87058 0.000 0.000 0.000 1.000 0.000
#> GSM340197 5 0.1597 0.75611 0.000 0.000 0.048 0.012 0.940
#> GSM340198 5 0.3559 0.69897 0.000 0.096 0.064 0.004 0.836
#> GSM340199 2 0.4909 0.54927 0.000 0.560 0.412 0.000 0.028
#> GSM340200 2 0.4339 0.57819 0.000 0.652 0.336 0.000 0.012
#> GSM340201 4 0.0000 0.87058 0.000 0.000 0.000 1.000 0.000
#> GSM340202 4 0.0000 0.87058 0.000 0.000 0.000 1.000 0.000
#> GSM340203 4 0.1117 0.85830 0.000 0.000 0.020 0.964 0.016
#> GSM340204 5 0.2300 0.73431 0.000 0.052 0.040 0.000 0.908
#> GSM340205 1 0.7600 0.04952 0.476 0.144 0.276 0.000 0.104
#> GSM340206 3 0.4493 0.65902 0.000 0.016 0.700 0.012 0.272
#> GSM340207 5 0.3658 0.69980 0.000 0.044 0.112 0.012 0.832
#> GSM340237 2 0.4334 0.54371 0.140 0.768 0.092 0.000 0.000
#> GSM340238 3 0.4493 0.65902 0.000 0.016 0.700 0.012 0.272
#> GSM340239 5 0.1522 0.75548 0.000 0.000 0.044 0.012 0.944
#> GSM340240 2 0.4774 0.42067 0.000 0.556 0.020 0.000 0.424
#> GSM340241 3 0.6335 0.52225 0.000 0.176 0.536 0.004 0.284
#> GSM340242 5 0.4208 0.67627 0.000 0.052 0.148 0.012 0.788
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.0260 0.8499 0.992 0.000 0.000 0.000 0.000 0.008
#> GSM340359 1 0.0937 0.8143 0.960 0.000 0.000 0.000 0.040 0.000
#> GSM340361 1 0.2357 0.7426 0.872 0.012 0.000 0.000 0.000 0.116
#> GSM340362 5 0.2631 0.7872 0.008 0.000 0.152 0.000 0.840 0.000
#> GSM340363 6 0.4479 0.5342 0.260 0.020 0.012 0.000 0.016 0.692
#> GSM340364 1 0.0260 0.8458 0.992 0.008 0.000 0.000 0.000 0.000
#> GSM340365 5 0.4241 0.2893 0.012 0.316 0.000 0.000 0.656 0.016
#> GSM340366 2 0.3698 0.7161 0.008 0.764 0.008 0.000 0.208 0.012
#> GSM340367 1 0.0000 0.8523 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340368 4 0.2562 0.7854 0.172 0.000 0.000 0.828 0.000 0.000
#> GSM340369 4 0.3265 0.6969 0.248 0.004 0.000 0.748 0.000 0.000
#> GSM340370 1 0.0000 0.8523 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340371 5 0.5790 0.6091 0.000 0.068 0.324 0.000 0.552 0.056
#> GSM340372 1 0.0000 0.8523 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.8523 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340375 1 0.0363 0.8478 0.988 0.000 0.000 0.000 0.000 0.012
#> GSM340376 1 0.0000 0.8523 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340378 1 0.0000 0.8523 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340243 2 0.5536 0.5891 0.008 0.632 0.224 0.000 0.020 0.116
#> GSM340244 6 0.6951 -0.1044 0.012 0.080 0.308 0.000 0.136 0.464
#> GSM340246 3 0.5526 0.5648 0.000 0.108 0.672 0.000 0.128 0.092
#> GSM340247 4 0.0000 0.9005 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340248 3 0.5764 0.4326 0.000 0.044 0.540 0.000 0.076 0.340
#> GSM340249 4 0.3420 0.7913 0.000 0.004 0.032 0.844 0.064 0.056
#> GSM340250 3 0.5347 0.3427 0.088 0.008 0.600 0.000 0.008 0.296
#> GSM340251 3 0.6188 0.5118 0.000 0.196 0.588 0.000 0.128 0.088
#> GSM340252 4 0.4662 0.6362 0.000 0.004 0.032 0.696 0.236 0.032
#> GSM340253 5 0.2044 0.7629 0.004 0.008 0.068 0.000 0.912 0.008
#> GSM340254 5 0.3113 0.7378 0.000 0.048 0.072 0.000 0.856 0.024
#> GSM340256 6 0.7042 0.0170 0.000 0.092 0.180 0.000 0.336 0.392
#> GSM340258 3 0.2051 0.6520 0.000 0.004 0.896 0.000 0.096 0.004
#> GSM340259 3 0.6069 0.4280 0.000 0.052 0.584 0.000 0.172 0.192
#> GSM340260 3 0.4920 0.5461 0.000 0.048 0.720 0.000 0.124 0.108
#> GSM340261 3 0.2007 0.6815 0.000 0.032 0.920 0.000 0.012 0.036
#> GSM340262 5 0.3601 0.6949 0.000 0.004 0.312 0.000 0.684 0.000
#> GSM340263 3 0.7690 0.3215 0.080 0.076 0.432 0.000 0.124 0.288
#> GSM340264 5 0.5008 0.3498 0.000 0.028 0.468 0.000 0.480 0.024
#> GSM340265 2 0.3807 0.6844 0.000 0.740 0.028 0.000 0.228 0.004
#> GSM340266 3 0.4069 0.6423 0.000 0.032 0.780 0.000 0.052 0.136
#> GSM340267 3 0.6189 0.3623 0.012 0.168 0.512 0.000 0.012 0.296
#> GSM340268 3 0.1995 0.6666 0.000 0.036 0.912 0.000 0.052 0.000
#> GSM340269 3 0.2332 0.6736 0.000 0.036 0.904 0.000 0.020 0.040
#> GSM340270 3 0.6436 0.5365 0.088 0.052 0.620 0.000 0.072 0.168
#> GSM537574 3 0.3833 0.6461 0.000 0.044 0.804 0.000 0.040 0.112
#> GSM537580 5 0.5945 0.5920 0.000 0.072 0.328 0.000 0.536 0.064
#> GSM537581 4 0.5852 0.1539 0.112 0.008 0.004 0.528 0.008 0.340
#> GSM340272 2 0.3678 0.7014 0.000 0.748 0.008 0.000 0.228 0.016
#> GSM340273 6 0.3136 0.5614 0.228 0.004 0.000 0.000 0.000 0.768
#> GSM340275 4 0.0893 0.8960 0.000 0.004 0.004 0.972 0.004 0.016
#> GSM340276 3 0.5334 0.2922 0.000 0.032 0.512 0.000 0.044 0.412
#> GSM340277 2 0.3492 0.7034 0.000 0.788 0.004 0.000 0.176 0.032
#> GSM340278 3 0.6419 0.1538 0.000 0.324 0.444 0.000 0.028 0.204
#> GSM340279 3 0.4111 0.5910 0.000 0.048 0.736 0.000 0.008 0.208
#> GSM340282 2 0.3398 0.7027 0.000 0.768 0.012 0.000 0.216 0.004
#> GSM340284 3 0.3219 0.6620 0.000 0.040 0.840 0.000 0.104 0.016
#> GSM340285 3 0.5860 0.2512 0.000 0.060 0.472 0.000 0.056 0.412
#> GSM340286 5 0.2845 0.7833 0.000 0.004 0.172 0.000 0.820 0.004
#> GSM340287 3 0.3031 0.6775 0.000 0.032 0.852 0.000 0.016 0.100
#> GSM340288 6 0.3508 0.5268 0.292 0.004 0.000 0.000 0.000 0.704
#> GSM340289 3 0.7284 0.4064 0.100 0.052 0.520 0.000 0.120 0.208
#> GSM340290 3 0.3064 0.6738 0.000 0.036 0.860 0.000 0.032 0.072
#> GSM340291 6 0.7603 -0.1014 0.020 0.176 0.260 0.000 0.128 0.416
#> GSM340293 2 0.3390 0.6473 0.000 0.704 0.296 0.000 0.000 0.000
#> GSM340294 3 0.2271 0.6797 0.000 0.032 0.908 0.000 0.024 0.036
#> GSM340296 2 0.3579 0.7579 0.008 0.788 0.180 0.000 0.012 0.012
#> GSM340297 3 0.5312 0.4738 0.056 0.040 0.652 0.000 0.008 0.244
#> GSM340298 2 0.3611 0.7566 0.008 0.784 0.184 0.000 0.012 0.012
#> GSM340299 5 0.3213 0.6875 0.004 0.124 0.036 0.000 0.832 0.004
#> GSM340301 5 0.2378 0.7874 0.000 0.000 0.152 0.000 0.848 0.000
#> GSM340303 2 0.3837 0.7135 0.000 0.744 0.016 0.000 0.224 0.016
#> GSM340304 2 0.5825 0.3019 0.008 0.492 0.072 0.000 0.028 0.400
#> GSM340306 6 0.4561 0.2122 0.024 0.008 0.000 0.404 0.000 0.564
#> GSM340307 2 0.2488 0.7588 0.000 0.864 0.124 0.000 0.004 0.008
#> GSM340310 1 0.0777 0.8408 0.972 0.004 0.000 0.000 0.000 0.024
#> GSM340314 3 0.2152 0.6743 0.000 0.040 0.912 0.012 0.036 0.000
#> GSM340315 3 0.5980 0.1649 0.092 0.028 0.504 0.000 0.008 0.368
#> GSM340317 6 0.7316 0.3175 0.016 0.068 0.044 0.272 0.096 0.504
#> GSM340318 4 0.0000 0.9005 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340319 4 0.0000 0.9005 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340320 6 0.4711 0.3620 0.024 0.012 0.000 0.336 0.008 0.620
#> GSM340321 3 0.5703 0.5802 0.000 0.088 0.648 0.000 0.100 0.164
#> GSM340322 4 0.0146 0.9000 0.000 0.004 0.000 0.996 0.000 0.000
#> GSM340324 1 0.7155 0.1803 0.460 0.076 0.024 0.000 0.144 0.296
#> GSM340328 1 0.1501 0.8023 0.924 0.000 0.000 0.000 0.000 0.076
#> GSM340330 3 0.2058 0.6672 0.000 0.036 0.908 0.000 0.056 0.000
#> GSM340332 4 0.0777 0.8934 0.000 0.004 0.024 0.972 0.000 0.000
#> GSM340333 5 0.2144 0.7617 0.004 0.008 0.068 0.000 0.908 0.012
#> GSM340336 4 0.0000 0.9005 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340337 4 0.1282 0.8878 0.000 0.004 0.004 0.956 0.012 0.024
#> GSM340338 3 0.4115 0.5950 0.000 0.148 0.764 0.000 0.076 0.012
#> GSM340339 4 0.0551 0.8985 0.000 0.004 0.004 0.984 0.000 0.008
#> GSM340340 4 0.2833 0.8006 0.148 0.000 0.004 0.836 0.012 0.000
#> GSM340341 4 0.5281 0.3437 0.000 0.008 0.040 0.588 0.028 0.336
#> GSM340343 4 0.0458 0.8978 0.000 0.000 0.000 0.984 0.000 0.016
#> GSM340344 5 0.1897 0.7690 0.004 0.004 0.084 0.000 0.908 0.000
#> GSM340346 2 0.2631 0.7587 0.000 0.820 0.180 0.000 0.000 0.000
#> GSM340347 4 0.0000 0.9005 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340348 6 0.4796 0.3507 0.000 0.012 0.036 0.332 0.004 0.616
#> GSM340349 2 0.4239 0.7547 0.012 0.784 0.104 0.000 0.020 0.080
#> GSM340350 2 0.4026 0.7665 0.032 0.816 0.072 0.000 0.032 0.048
#> GSM340351 6 0.4488 0.1811 0.468 0.016 0.000 0.000 0.008 0.508
#> GSM340354 3 0.2791 0.6622 0.000 0.096 0.864 0.000 0.032 0.008
#> GSM340356 6 0.3545 0.5573 0.236 0.008 0.000 0.000 0.008 0.748
#> GSM340357 6 0.5813 0.2600 0.412 0.024 0.076 0.000 0.008 0.480
#> GSM348183 1 0.8263 0.0547 0.416 0.052 0.092 0.052 0.100 0.288
#> GSM348191 3 0.2038 0.6777 0.000 0.028 0.920 0.000 0.032 0.020
#> GSM348193 6 0.4130 0.4911 0.300 0.024 0.000 0.004 0.000 0.672
#> GSM537578 2 0.3582 0.7595 0.004 0.792 0.172 0.000 0.016 0.016
#> GSM348181 3 0.6090 0.5277 0.056 0.052 0.636 0.000 0.060 0.196
#> GSM348182 5 0.4130 0.7600 0.000 0.036 0.180 0.000 0.756 0.028
#> GSM348184 3 0.6239 0.5045 0.000 0.212 0.576 0.000 0.128 0.084
#> GSM348185 6 0.3475 0.5576 0.032 0.012 0.000 0.136 0.004 0.816
#> GSM348186 5 0.3903 0.6911 0.000 0.072 0.060 0.000 0.808 0.060
#> GSM348187 6 0.3897 0.5165 0.300 0.008 0.000 0.000 0.008 0.684
#> GSM348188 4 0.1963 0.8740 0.000 0.012 0.012 0.928 0.016 0.032
#> GSM348189 5 0.5803 0.6206 0.000 0.068 0.312 0.000 0.560 0.060
#> GSM348190 5 0.4068 0.6477 0.000 0.004 0.080 0.000 0.756 0.160
#> GSM348194 6 0.3508 0.5268 0.292 0.004 0.000 0.000 0.000 0.704
#> GSM348195 1 0.4336 0.0598 0.572 0.012 0.000 0.000 0.008 0.408
#> GSM348196 6 0.3508 0.5268 0.292 0.004 0.000 0.000 0.000 0.704
#> GSM537585 6 0.6850 0.2931 0.176 0.056 0.008 0.000 0.308 0.452
#> GSM537594 6 0.5523 0.4832 0.092 0.056 0.040 0.000 0.104 0.708
#> GSM537596 6 0.4108 0.5149 0.020 0.020 0.160 0.000 0.024 0.776
#> GSM537597 6 0.3698 0.5672 0.208 0.012 0.008 0.000 0.008 0.764
#> GSM537602 2 0.5827 0.3751 0.048 0.544 0.032 0.000 0.024 0.352
#> GSM340184 3 0.1563 0.6700 0.000 0.012 0.932 0.000 0.056 0.000
#> GSM340185 2 0.3720 0.7177 0.000 0.788 0.152 0.000 0.008 0.052
#> GSM340186 3 0.6243 0.5007 0.000 0.196 0.580 0.000 0.140 0.084
#> GSM340187 4 0.0777 0.8925 0.000 0.004 0.024 0.972 0.000 0.000
#> GSM340189 4 0.0508 0.8969 0.000 0.004 0.012 0.984 0.000 0.000
#> GSM340190 2 0.4178 0.6165 0.000 0.708 0.248 0.000 0.008 0.036
#> GSM340191 3 0.1655 0.6723 0.000 0.008 0.932 0.000 0.052 0.008
#> GSM340192 2 0.3161 0.7041 0.000 0.776 0.008 0.000 0.216 0.000
#> GSM340193 6 0.4346 0.4508 0.004 0.024 0.200 0.000 0.036 0.736
#> GSM340194 5 0.2340 0.7879 0.000 0.000 0.148 0.000 0.852 0.000
#> GSM340195 5 0.2482 0.7872 0.004 0.000 0.148 0.000 0.848 0.000
#> GSM340196 4 0.0000 0.9005 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340197 5 0.2772 0.7846 0.000 0.004 0.180 0.000 0.816 0.000
#> GSM340198 5 0.4070 0.6948 0.000 0.072 0.072 0.000 0.796 0.060
#> GSM340199 2 0.3426 0.7532 0.000 0.784 0.192 0.000 0.012 0.012
#> GSM340200 2 0.2333 0.7614 0.000 0.872 0.120 0.000 0.004 0.004
#> GSM340201 4 0.0000 0.9005 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340202 4 0.0000 0.9005 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340203 4 0.2183 0.8627 0.000 0.004 0.052 0.912 0.020 0.012
#> GSM340204 5 0.4367 0.6932 0.000 0.024 0.180 0.000 0.740 0.056
#> GSM340205 3 0.7006 0.1681 0.352 0.052 0.396 0.000 0.012 0.188
#> GSM340206 3 0.2058 0.6672 0.000 0.036 0.908 0.000 0.056 0.000
#> GSM340207 5 0.4943 0.5369 0.000 0.044 0.392 0.000 0.552 0.012
#> GSM340237 2 0.3945 0.6999 0.024 0.800 0.048 0.000 0.008 0.120
#> GSM340238 3 0.2129 0.6675 0.000 0.040 0.904 0.000 0.056 0.000
#> GSM340239 5 0.2738 0.7828 0.000 0.000 0.176 0.000 0.820 0.004
#> GSM340240 2 0.3329 0.6997 0.000 0.768 0.008 0.000 0.220 0.004
#> GSM340241 3 0.6445 0.4571 0.000 0.152 0.552 0.000 0.208 0.088
#> GSM340242 5 0.5938 0.5849 0.000 0.072 0.344 0.000 0.524 0.060
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
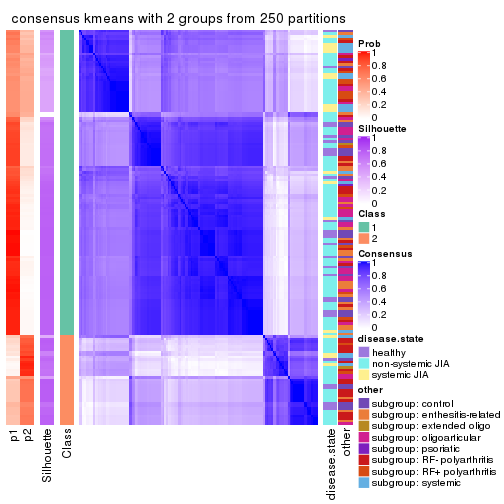
consensus_heatmap(res, k = 3)
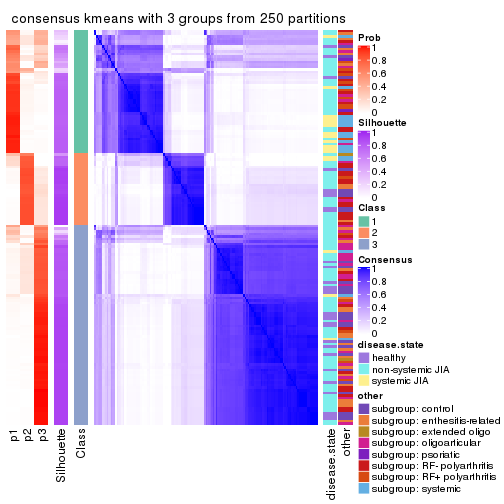
consensus_heatmap(res, k = 4)
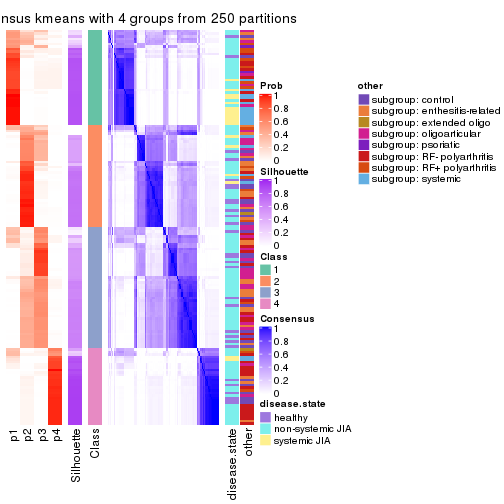
consensus_heatmap(res, k = 5)
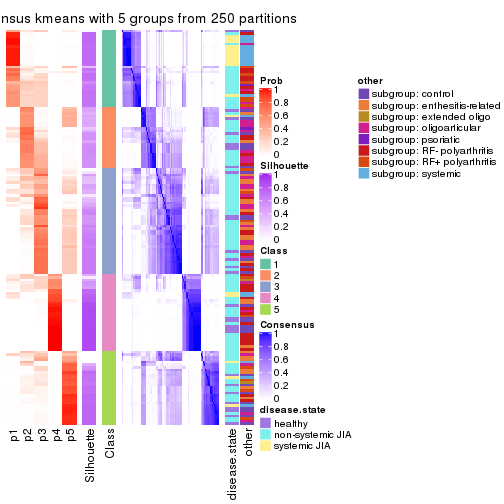
consensus_heatmap(res, k = 6)
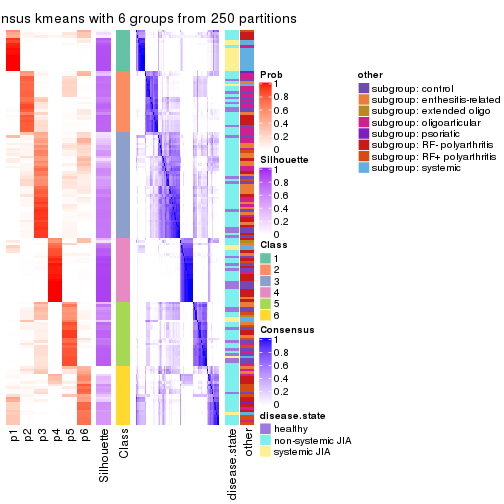
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
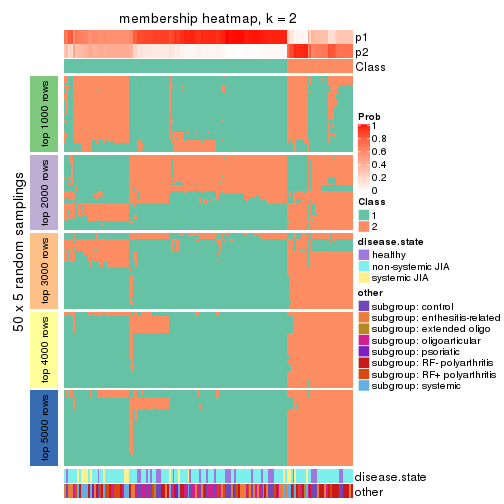
membership_heatmap(res, k = 3)
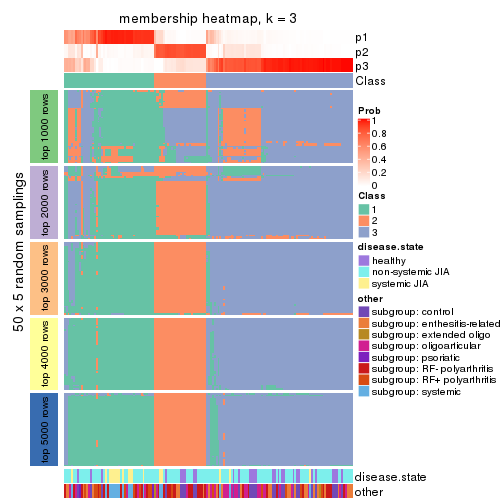
membership_heatmap(res, k = 4)
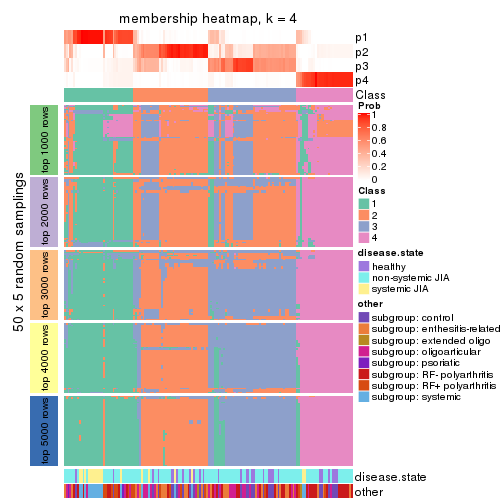
membership_heatmap(res, k = 5)
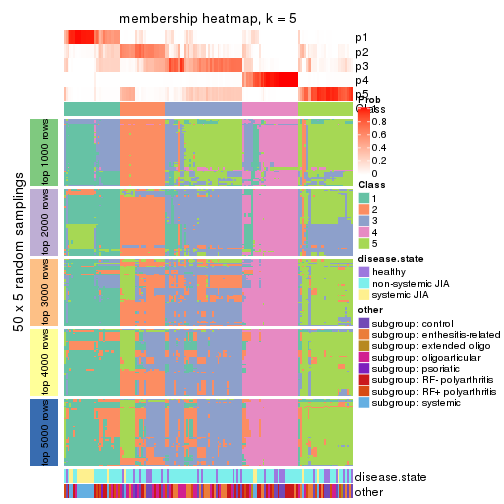
membership_heatmap(res, k = 6)
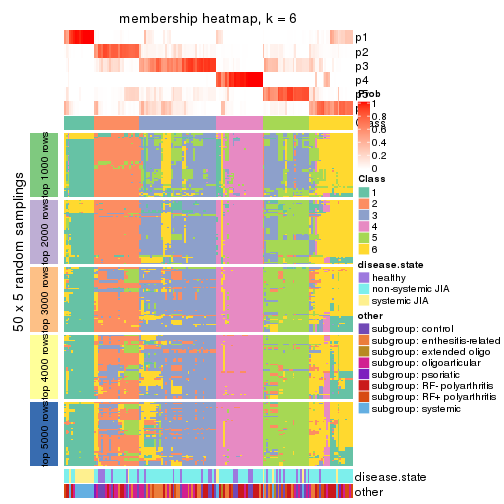
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
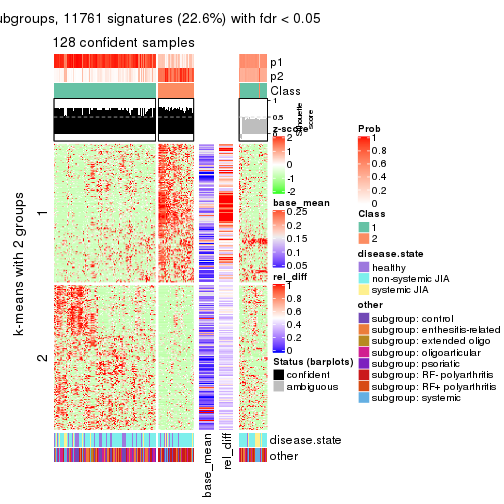
get_signatures(res, k = 3)
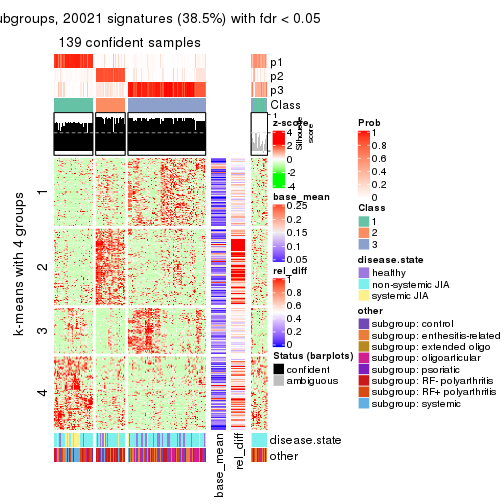
get_signatures(res, k = 4)
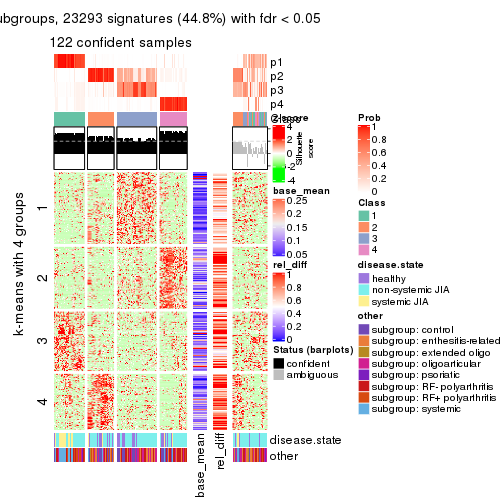
get_signatures(res, k = 5)
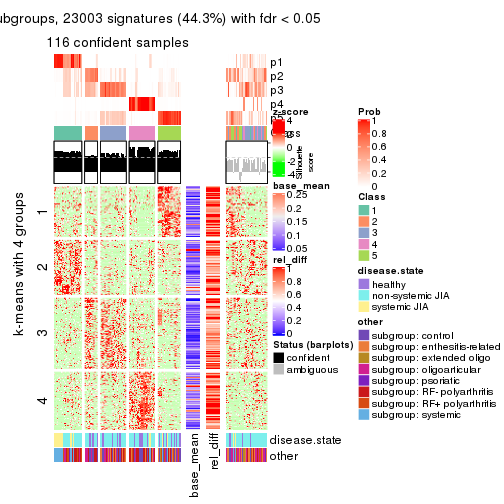
get_signatures(res, k = 6)
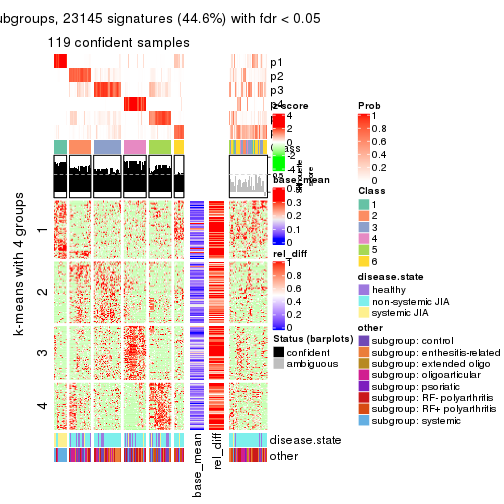
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
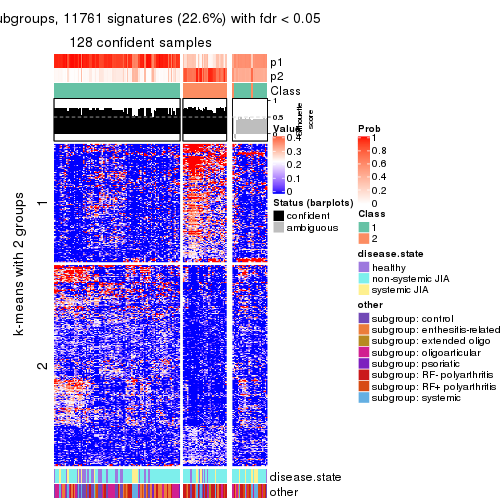
get_signatures(res, k = 3, scale_rows = FALSE)
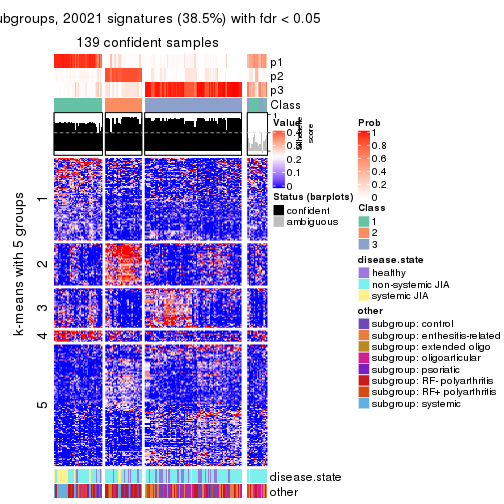
get_signatures(res, k = 4, scale_rows = FALSE)
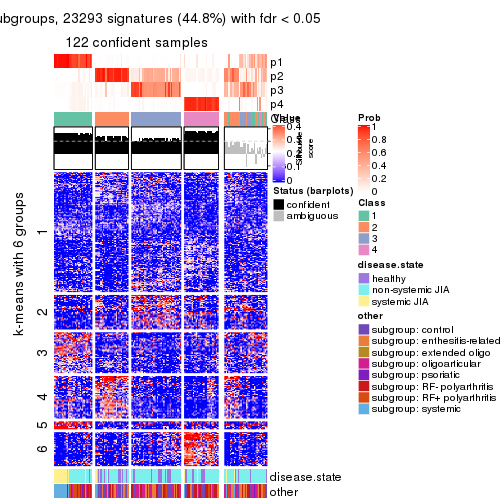
get_signatures(res, k = 5, scale_rows = FALSE)
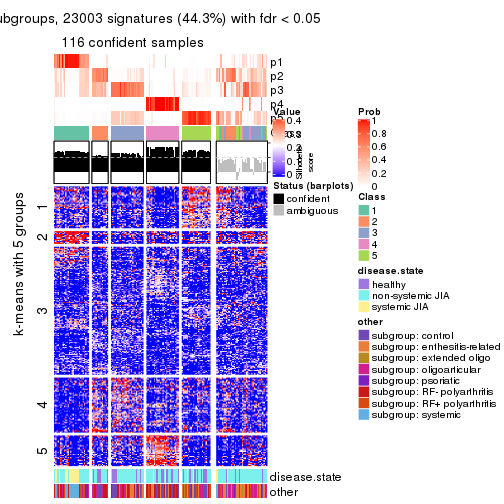
get_signatures(res, k = 6, scale_rows = FALSE)
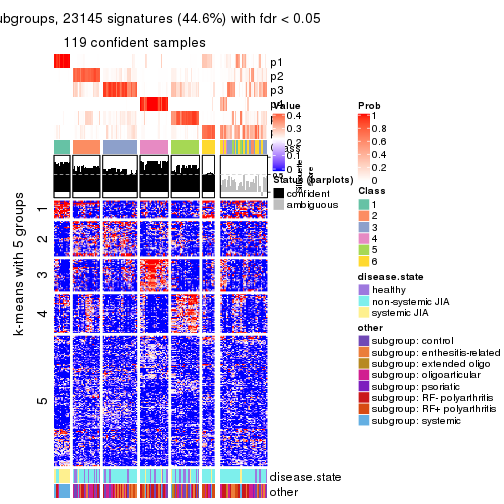
Compare the overlap of signatures from different k:
compare_signatures(res)
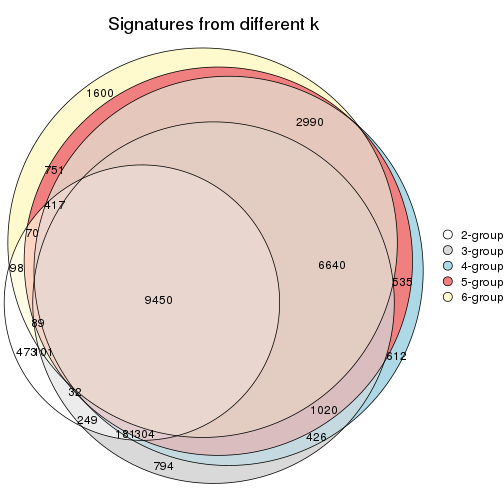
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
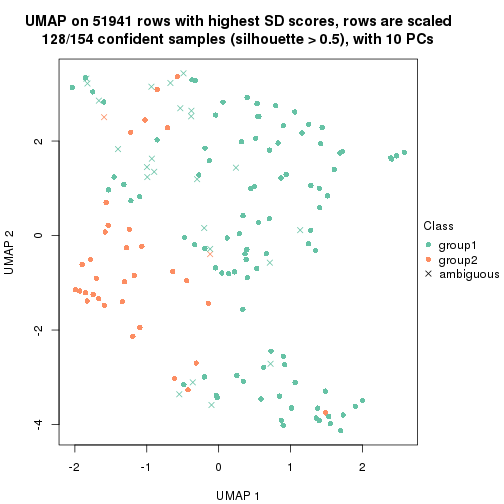
dimension_reduction(res, k = 3, method = "UMAP")
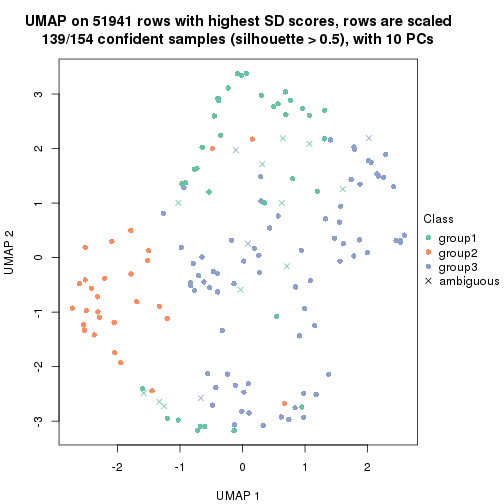
dimension_reduction(res, k = 4, method = "UMAP")
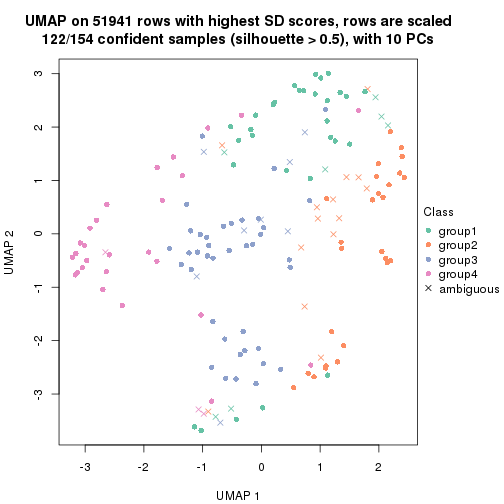
dimension_reduction(res, k = 5, method = "UMAP")
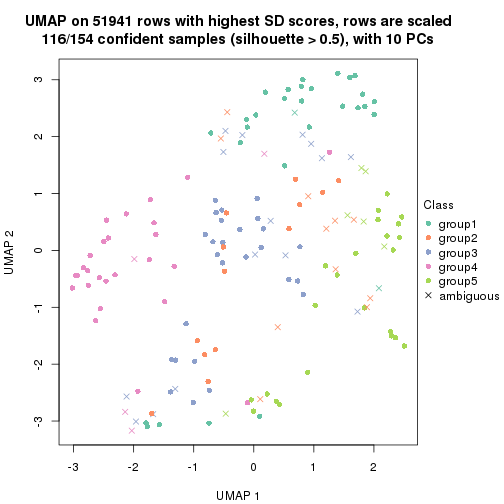
dimension_reduction(res, k = 6, method = "UMAP")
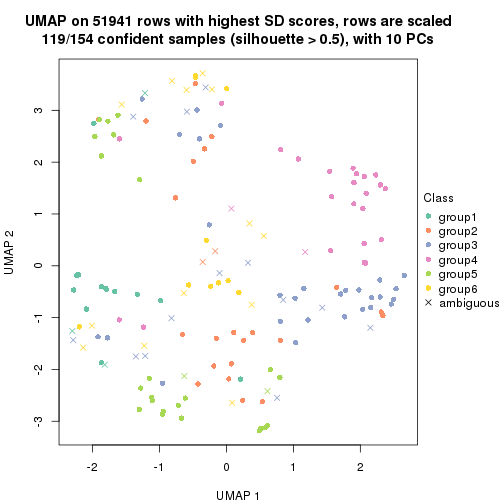
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
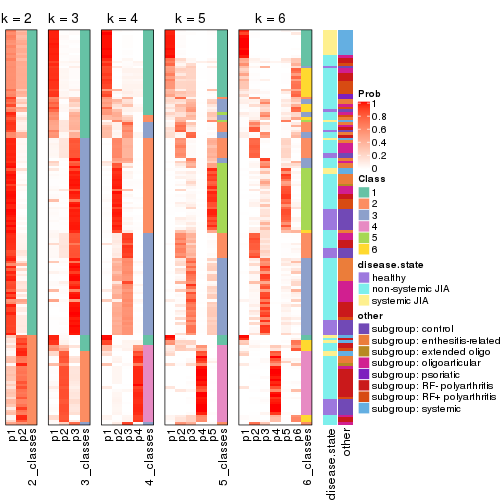
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> SD:kmeans 128 8.26e-01 2.79e-03 2
#> SD:kmeans 139 2.34e-04 1.38e-06 3
#> SD:kmeans 122 3.84e-05 1.47e-06 4
#> SD:kmeans 116 3.25e-05 1.58e-05 5
#> SD:kmeans 119 1.46e-10 3.73e-12 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["SD", "skmeans"]
# you can also extract it by
# res = res_list["SD:skmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'SD' method.
#> Subgroups are detected by 'skmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
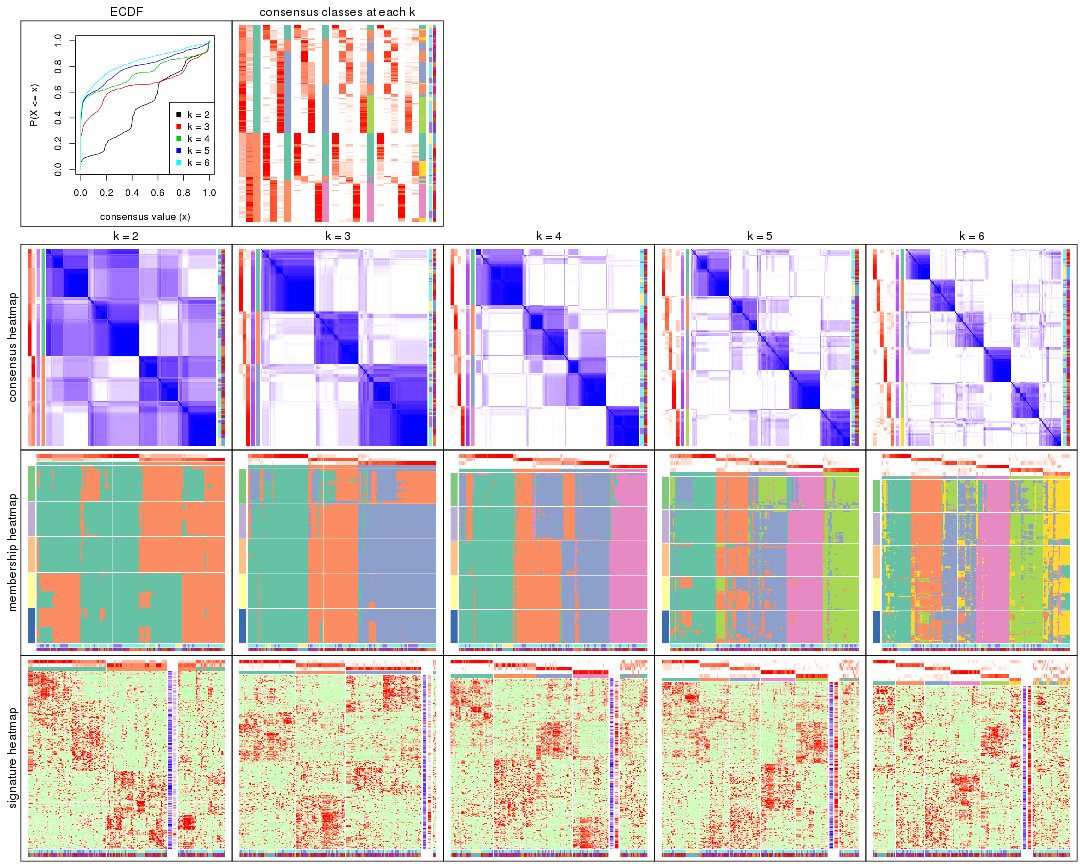
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
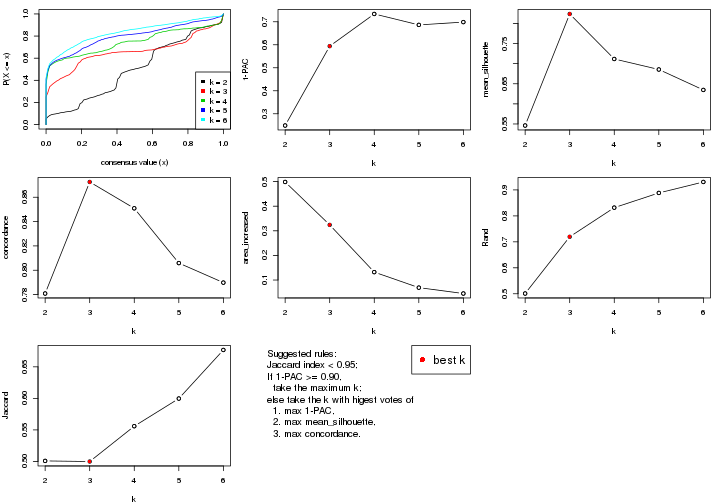
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.249 0.546 0.781 0.4985 0.501 0.501
#> 3 3 0.594 0.823 0.873 0.3240 0.719 0.500
#> 4 4 0.734 0.712 0.851 0.1321 0.832 0.556
#> 5 5 0.686 0.685 0.806 0.0690 0.888 0.600
#> 6 6 0.699 0.635 0.790 0.0451 0.931 0.677
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 2 0.7602 0.60897 0.220 0.780
#> GSM340359 1 0.9815 -0.13821 0.580 0.420
#> GSM340361 2 0.9686 0.55275 0.396 0.604
#> GSM340362 1 0.7376 0.46054 0.792 0.208
#> GSM340363 2 0.7453 0.61025 0.212 0.788
#> GSM340364 1 0.9427 0.07797 0.640 0.360
#> GSM340365 1 0.6973 0.49057 0.812 0.188
#> GSM340366 1 0.1843 0.66163 0.972 0.028
#> GSM340367 2 0.9686 0.55275 0.396 0.604
#> GSM340368 2 0.0000 0.61712 0.000 1.000
#> GSM340369 2 0.0000 0.61712 0.000 1.000
#> GSM340370 2 0.9686 0.55275 0.396 0.604
#> GSM340371 1 0.7453 0.63273 0.788 0.212
#> GSM340372 2 0.9686 0.55275 0.396 0.604
#> GSM340373 2 0.9710 0.54734 0.400 0.600
#> GSM340375 2 0.9686 0.55275 0.396 0.604
#> GSM340376 2 0.9686 0.55275 0.396 0.604
#> GSM340378 2 0.9710 0.54734 0.400 0.600
#> GSM340243 2 0.9944 0.30427 0.456 0.544
#> GSM340244 2 0.9608 0.50498 0.384 0.616
#> GSM340246 1 0.3431 0.67664 0.936 0.064
#> GSM340247 2 0.6973 0.49526 0.188 0.812
#> GSM340248 1 0.9833 -0.22433 0.576 0.424
#> GSM340249 2 0.7299 0.47315 0.204 0.796
#> GSM340250 2 0.9552 0.56181 0.376 0.624
#> GSM340251 1 0.9686 0.50487 0.604 0.396
#> GSM340252 2 0.7453 0.46121 0.212 0.788
#> GSM340253 1 0.0000 0.68047 1.000 0.000
#> GSM340254 1 0.0000 0.68047 1.000 0.000
#> GSM340256 1 0.0000 0.68047 1.000 0.000
#> GSM340258 1 0.4939 0.66858 0.892 0.108
#> GSM340259 1 0.0000 0.68047 1.000 0.000
#> GSM340260 1 0.3733 0.62443 0.928 0.072
#> GSM340261 1 0.9635 0.51149 0.612 0.388
#> GSM340262 1 0.0000 0.68047 1.000 0.000
#> GSM340263 2 0.9608 0.52545 0.384 0.616
#> GSM340264 1 0.0000 0.68047 1.000 0.000
#> GSM340265 1 0.0000 0.68047 1.000 0.000
#> GSM340266 2 0.6973 0.49526 0.188 0.812
#> GSM340267 1 0.6531 0.60913 0.832 0.168
#> GSM340268 1 0.9686 0.50487 0.604 0.396
#> GSM340269 1 0.4298 0.67308 0.912 0.088
#> GSM340270 2 0.8661 0.59272 0.288 0.712
#> GSM537574 1 0.0000 0.68047 1.000 0.000
#> GSM537580 1 0.9686 0.50487 0.604 0.396
#> GSM537581 2 0.0000 0.61712 0.000 1.000
#> GSM340272 1 0.4939 0.58759 0.892 0.108
#> GSM340273 2 0.9686 0.55275 0.396 0.604
#> GSM340275 2 0.6973 0.49526 0.188 0.812
#> GSM340276 1 0.8813 0.28375 0.700 0.300
#> GSM340277 1 0.0000 0.68047 1.000 0.000
#> GSM340278 1 0.9686 0.50487 0.604 0.396
#> GSM340279 2 0.6712 0.41955 0.176 0.824
#> GSM340282 1 0.0376 0.67831 0.996 0.004
#> GSM340284 1 0.9686 0.50487 0.604 0.396
#> GSM340285 2 0.7056 0.44068 0.192 0.808
#> GSM340286 1 0.0000 0.68047 1.000 0.000
#> GSM340287 1 0.8207 0.51937 0.744 0.256
#> GSM340288 2 0.9686 0.55275 0.396 0.604
#> GSM340289 1 0.7139 0.48047 0.804 0.196
#> GSM340290 1 0.9732 0.49375 0.596 0.404
#> GSM340291 1 0.8327 0.35006 0.736 0.264
#> GSM340293 1 0.9686 0.50487 0.604 0.396
#> GSM340294 1 0.9580 0.51786 0.620 0.380
#> GSM340296 1 0.8909 0.58798 0.692 0.308
#> GSM340297 2 0.0000 0.61712 0.000 1.000
#> GSM340298 1 0.9866 0.49512 0.568 0.432
#> GSM340299 1 0.0000 0.68047 1.000 0.000
#> GSM340301 1 0.0000 0.68047 1.000 0.000
#> GSM340303 1 0.0376 0.67843 0.996 0.004
#> GSM340304 1 0.9732 0.00616 0.596 0.404
#> GSM340306 2 0.0000 0.61712 0.000 1.000
#> GSM340307 1 0.7376 0.63466 0.792 0.208
#> GSM340310 2 0.9686 0.55275 0.396 0.604
#> GSM340314 1 0.9710 0.49939 0.600 0.400
#> GSM340315 2 0.1633 0.61947 0.024 0.976
#> GSM340317 2 0.1184 0.61263 0.016 0.984
#> GSM340318 2 0.6973 0.49526 0.188 0.812
#> GSM340319 2 0.6973 0.49526 0.188 0.812
#> GSM340320 2 0.0000 0.61712 0.000 1.000
#> GSM340321 1 0.3733 0.67560 0.928 0.072
#> GSM340322 2 0.7453 0.46121 0.212 0.788
#> GSM340324 2 0.9732 0.54156 0.404 0.596
#> GSM340328 2 0.7453 0.61025 0.212 0.788
#> GSM340330 1 0.9686 0.50487 0.604 0.396
#> GSM340332 2 0.6048 0.53073 0.148 0.852
#> GSM340333 1 0.0000 0.68047 1.000 0.000
#> GSM340336 2 0.6973 0.49526 0.188 0.812
#> GSM340337 2 0.0000 0.61712 0.000 1.000
#> GSM340338 1 0.9686 0.50487 0.604 0.396
#> GSM340339 2 0.2603 0.60069 0.044 0.956
#> GSM340340 2 0.0000 0.61712 0.000 1.000
#> GSM340341 2 0.1843 0.60811 0.028 0.972
#> GSM340343 2 0.6973 0.49526 0.188 0.812
#> GSM340344 1 0.1184 0.67051 0.984 0.016
#> GSM340346 1 0.7453 0.63273 0.788 0.212
#> GSM340347 2 0.6973 0.49526 0.188 0.812
#> GSM340348 2 0.0000 0.61712 0.000 1.000
#> GSM340349 1 0.6973 0.49057 0.812 0.188
#> GSM340350 1 0.6973 0.49057 0.812 0.188
#> GSM340351 2 0.9686 0.55275 0.396 0.604
#> GSM340354 1 0.7376 0.63466 0.792 0.208
#> GSM340356 2 0.9686 0.55275 0.396 0.604
#> GSM340357 2 0.9686 0.55275 0.396 0.604
#> GSM348183 2 0.9635 0.55653 0.388 0.612
#> GSM348191 1 0.9686 0.50487 0.604 0.396
#> GSM348193 2 0.7376 0.61094 0.208 0.792
#> GSM537578 1 0.7139 0.47928 0.804 0.196
#> GSM348181 2 0.9933 0.40289 0.452 0.548
#> GSM348182 1 0.0672 0.67582 0.992 0.008
#> GSM348184 1 0.9686 0.50487 0.604 0.396
#> GSM348185 2 0.0000 0.61712 0.000 1.000
#> GSM348186 1 0.0000 0.68047 1.000 0.000
#> GSM348187 2 0.9686 0.55275 0.396 0.604
#> GSM348188 2 0.3584 0.62249 0.068 0.932
#> GSM348189 1 0.7453 0.63273 0.788 0.212
#> GSM348190 1 0.7376 0.46054 0.792 0.208
#> GSM348194 2 0.9686 0.55275 0.396 0.604
#> GSM348195 2 0.9686 0.55275 0.396 0.604
#> GSM348196 2 0.9686 0.55275 0.396 0.604
#> GSM537585 1 0.7453 0.45372 0.788 0.212
#> GSM537594 2 0.9686 0.55275 0.396 0.604
#> GSM537596 2 0.9286 0.57387 0.344 0.656
#> GSM537597 2 0.9686 0.55275 0.396 0.604
#> GSM537602 1 0.6973 0.49057 0.812 0.188
#> GSM340184 1 0.9686 0.50487 0.604 0.396
#> GSM340185 1 0.9580 0.51778 0.620 0.380
#> GSM340186 1 0.9686 0.50487 0.604 0.396
#> GSM340187 2 0.8555 0.32803 0.280 0.720
#> GSM340189 2 0.9087 0.21568 0.324 0.676
#> GSM340190 1 0.9686 0.50487 0.604 0.396
#> GSM340191 1 0.9686 0.50487 0.604 0.396
#> GSM340192 1 0.0000 0.68047 1.000 0.000
#> GSM340193 2 0.9686 0.55275 0.396 0.604
#> GSM340194 1 0.0000 0.68047 1.000 0.000
#> GSM340195 1 0.0000 0.68047 1.000 0.000
#> GSM340196 2 0.6887 0.49919 0.184 0.816
#> GSM340197 1 0.0000 0.68047 1.000 0.000
#> GSM340198 1 0.7453 0.63273 0.788 0.212
#> GSM340199 1 0.9686 0.50487 0.604 0.396
#> GSM340200 1 0.7950 0.61616 0.760 0.240
#> GSM340201 2 0.2423 0.60299 0.040 0.960
#> GSM340202 2 0.6973 0.49526 0.188 0.812
#> GSM340203 2 0.7453 0.46121 0.212 0.788
#> GSM340204 1 0.0000 0.68047 1.000 0.000
#> GSM340205 2 0.9686 0.55275 0.396 0.604
#> GSM340206 1 0.9686 0.50487 0.604 0.396
#> GSM340207 1 0.7376 0.63466 0.792 0.208
#> GSM340237 1 0.7056 0.48522 0.808 0.192
#> GSM340238 1 0.9686 0.50487 0.604 0.396
#> GSM340239 1 0.0000 0.68047 1.000 0.000
#> GSM340240 1 0.0000 0.68047 1.000 0.000
#> GSM340241 1 0.6343 0.65295 0.840 0.160
#> GSM340242 1 0.7453 0.63273 0.788 0.212
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340359 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340361 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340362 3 0.4504 0.762 0.196 0.000 0.804
#> GSM340363 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340364 1 0.2537 0.875 0.920 0.080 0.000
#> GSM340365 3 0.8896 0.512 0.264 0.172 0.564
#> GSM340366 3 0.4589 0.830 0.008 0.172 0.820
#> GSM340367 1 0.2066 0.886 0.940 0.060 0.000
#> GSM340368 2 0.4452 0.798 0.192 0.808 0.000
#> GSM340369 2 0.3551 0.804 0.132 0.868 0.000
#> GSM340370 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340371 3 0.0424 0.877 0.000 0.008 0.992
#> GSM340372 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340373 1 0.2066 0.886 0.940 0.060 0.000
#> GSM340375 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340376 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340378 1 0.2066 0.886 0.940 0.060 0.000
#> GSM340243 1 0.4953 0.797 0.808 0.176 0.016
#> GSM340244 1 0.8263 0.519 0.636 0.188 0.176
#> GSM340246 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340247 2 0.4575 0.874 0.012 0.828 0.160
#> GSM340248 1 0.3619 0.812 0.864 0.000 0.136
#> GSM340249 2 0.4178 0.868 0.000 0.828 0.172
#> GSM340250 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340251 2 0.4605 0.847 0.000 0.796 0.204
#> GSM340252 2 0.4178 0.868 0.000 0.828 0.172
#> GSM340253 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340254 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340256 3 0.0892 0.876 0.000 0.020 0.980
#> GSM340258 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340259 3 0.5070 0.670 0.224 0.004 0.772
#> GSM340260 3 0.2682 0.866 0.004 0.076 0.920
#> GSM340261 3 0.2165 0.871 0.000 0.064 0.936
#> GSM340262 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340263 1 0.5521 0.728 0.788 0.032 0.180
#> GSM340264 3 0.3038 0.861 0.000 0.104 0.896
#> GSM340265 3 0.4178 0.833 0.000 0.172 0.828
#> GSM340266 2 0.4178 0.868 0.000 0.828 0.172
#> GSM340267 3 0.7082 0.748 0.156 0.120 0.724
#> GSM340268 3 0.4235 0.701 0.000 0.176 0.824
#> GSM340269 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340270 1 0.1411 0.897 0.964 0.036 0.000
#> GSM537574 3 0.0237 0.876 0.000 0.004 0.996
#> GSM537580 3 0.0237 0.876 0.000 0.004 0.996
#> GSM537581 2 0.6008 0.545 0.372 0.628 0.000
#> GSM340272 3 0.4749 0.828 0.012 0.172 0.816
#> GSM340273 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340275 2 0.4575 0.874 0.012 0.828 0.160
#> GSM340276 1 0.9045 0.335 0.552 0.256 0.192
#> GSM340277 3 0.4178 0.833 0.000 0.172 0.828
#> GSM340278 2 0.1860 0.779 0.000 0.948 0.052
#> GSM340279 2 0.4228 0.812 0.148 0.844 0.008
#> GSM340282 3 0.4178 0.833 0.000 0.172 0.828
#> GSM340284 2 0.5835 0.675 0.000 0.660 0.340
#> GSM340285 2 0.0983 0.802 0.016 0.980 0.004
#> GSM340286 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340287 3 0.6126 0.670 0.268 0.020 0.712
#> GSM340288 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340289 1 0.4228 0.789 0.844 0.008 0.148
#> GSM340290 2 0.2165 0.798 0.000 0.936 0.064
#> GSM340291 1 0.3619 0.838 0.864 0.136 0.000
#> GSM340293 2 0.0892 0.791 0.000 0.980 0.020
#> GSM340294 3 0.2066 0.870 0.000 0.060 0.940
#> GSM340296 3 0.4749 0.828 0.012 0.172 0.816
#> GSM340297 1 0.6111 0.170 0.604 0.396 0.000
#> GSM340298 3 0.4749 0.828 0.012 0.172 0.816
#> GSM340299 3 0.4178 0.833 0.000 0.172 0.828
#> GSM340301 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340303 3 0.4178 0.833 0.000 0.172 0.828
#> GSM340304 1 0.7328 0.591 0.612 0.344 0.044
#> GSM340306 2 0.4555 0.790 0.200 0.800 0.000
#> GSM340307 3 0.4178 0.833 0.000 0.172 0.828
#> GSM340310 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340314 2 0.4178 0.868 0.000 0.828 0.172
#> GSM340315 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340317 2 0.7624 0.725 0.224 0.672 0.104
#> GSM340318 2 0.4575 0.874 0.012 0.828 0.160
#> GSM340319 2 0.4575 0.874 0.012 0.828 0.160
#> GSM340320 2 0.4504 0.793 0.196 0.804 0.000
#> GSM340321 3 0.1860 0.872 0.000 0.052 0.948
#> GSM340322 2 0.4473 0.873 0.008 0.828 0.164
#> GSM340324 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340328 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340330 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340332 2 0.4931 0.874 0.032 0.828 0.140
#> GSM340333 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340336 2 0.4575 0.874 0.012 0.828 0.160
#> GSM340337 2 0.4473 0.818 0.164 0.828 0.008
#> GSM340338 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340339 2 0.4741 0.828 0.152 0.828 0.020
#> GSM340340 2 0.4749 0.817 0.172 0.816 0.012
#> GSM340341 2 0.5069 0.843 0.128 0.828 0.044
#> GSM340343 2 0.4473 0.873 0.008 0.828 0.164
#> GSM340344 3 0.2356 0.854 0.072 0.000 0.928
#> GSM340346 3 0.4178 0.833 0.000 0.172 0.828
#> GSM340347 2 0.4575 0.874 0.012 0.828 0.160
#> GSM340348 2 0.4121 0.811 0.168 0.832 0.000
#> GSM340349 1 0.5847 0.775 0.780 0.172 0.048
#> GSM340350 1 0.7661 0.663 0.684 0.172 0.144
#> GSM340351 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340354 3 0.0747 0.876 0.000 0.016 0.984
#> GSM340356 1 0.0000 0.908 1.000 0.000 0.000
#> GSM340357 1 0.2165 0.884 0.936 0.064 0.000
#> GSM348183 1 0.0892 0.899 0.980 0.020 0.000
#> GSM348191 3 0.3941 0.731 0.000 0.156 0.844
#> GSM348193 1 0.0424 0.905 0.992 0.008 0.000
#> GSM537578 3 0.8976 0.490 0.276 0.172 0.552
#> GSM348181 1 0.6646 0.679 0.740 0.076 0.184
#> GSM348182 3 0.4963 0.695 0.200 0.008 0.792
#> GSM348184 3 0.4452 0.675 0.000 0.192 0.808
#> GSM348185 2 0.5650 0.642 0.312 0.688 0.000
#> GSM348186 3 0.0237 0.876 0.000 0.004 0.996
#> GSM348187 1 0.0000 0.908 1.000 0.000 0.000
#> GSM348188 2 0.4811 0.831 0.148 0.828 0.024
#> GSM348189 3 0.0237 0.876 0.000 0.004 0.996
#> GSM348190 3 0.5443 0.683 0.260 0.004 0.736
#> GSM348194 1 0.0000 0.908 1.000 0.000 0.000
#> GSM348195 1 0.0000 0.908 1.000 0.000 0.000
#> GSM348196 1 0.0000 0.908 1.000 0.000 0.000
#> GSM537585 1 0.2031 0.894 0.952 0.032 0.016
#> GSM537594 1 0.0000 0.908 1.000 0.000 0.000
#> GSM537596 1 0.0592 0.903 0.988 0.012 0.000
#> GSM537597 1 0.0000 0.908 1.000 0.000 0.000
#> GSM537602 1 0.5412 0.787 0.796 0.172 0.032
#> GSM340184 3 0.1643 0.851 0.000 0.044 0.956
#> GSM340185 3 0.4178 0.833 0.000 0.172 0.828
#> GSM340186 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340187 2 0.4178 0.868 0.000 0.828 0.172
#> GSM340189 2 0.3038 0.860 0.000 0.896 0.104
#> GSM340190 3 0.5882 0.685 0.000 0.348 0.652
#> GSM340191 2 0.5988 0.623 0.000 0.632 0.368
#> GSM340192 3 0.4178 0.833 0.000 0.172 0.828
#> GSM340193 1 0.0829 0.901 0.984 0.012 0.004
#> GSM340194 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340195 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340196 2 0.4874 0.875 0.028 0.828 0.144
#> GSM340197 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340198 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340199 3 0.5882 0.685 0.000 0.348 0.652
#> GSM340200 3 0.4178 0.833 0.000 0.172 0.828
#> GSM340201 2 0.5260 0.858 0.092 0.828 0.080
#> GSM340202 2 0.4575 0.874 0.012 0.828 0.160
#> GSM340203 2 0.4178 0.868 0.000 0.828 0.172
#> GSM340204 3 0.0892 0.876 0.000 0.020 0.980
#> GSM340205 1 0.0237 0.907 0.996 0.000 0.004
#> GSM340206 3 0.4002 0.724 0.000 0.160 0.840
#> GSM340207 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340237 1 0.4589 0.805 0.820 0.172 0.008
#> GSM340238 2 0.5706 0.708 0.000 0.680 0.320
#> GSM340239 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340240 3 0.4178 0.833 0.000 0.172 0.828
#> GSM340241 3 0.0237 0.876 0.000 0.004 0.996
#> GSM340242 3 0.0592 0.877 0.000 0.012 0.988
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM340359 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM340361 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM340362 3 0.3751 0.605 0.196 0.004 0.800 0.000
#> GSM340363 1 0.0469 0.875 0.988 0.000 0.000 0.012
#> GSM340364 1 0.0592 0.870 0.984 0.016 0.000 0.000
#> GSM340365 3 0.4830 0.513 0.000 0.392 0.608 0.000
#> GSM340366 3 0.4907 0.495 0.000 0.420 0.580 0.000
#> GSM340367 1 0.0336 0.874 0.992 0.008 0.000 0.000
#> GSM340368 4 0.2281 0.892 0.096 0.000 0.000 0.904
#> GSM340369 4 0.3672 0.813 0.164 0.012 0.000 0.824
#> GSM340370 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM340371 3 0.1118 0.751 0.000 0.036 0.964 0.000
#> GSM340372 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM340375 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM340376 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM340378 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM340243 2 0.4961 -0.249 0.448 0.552 0.000 0.000
#> GSM340244 1 0.6439 0.566 0.656 0.008 0.228 0.108
#> GSM340246 2 0.4790 0.654 0.000 0.620 0.380 0.000
#> GSM340247 4 0.0336 0.956 0.000 0.000 0.008 0.992
#> GSM340248 2 0.7195 0.611 0.132 0.572 0.284 0.012
#> GSM340249 4 0.0707 0.949 0.000 0.000 0.020 0.980
#> GSM340250 1 0.4713 0.306 0.640 0.360 0.000 0.000
#> GSM340251 2 0.6785 0.606 0.000 0.608 0.208 0.184
#> GSM340252 4 0.0707 0.949 0.000 0.000 0.020 0.980
#> GSM340253 3 0.0188 0.765 0.000 0.004 0.996 0.000
#> GSM340254 3 0.0188 0.765 0.000 0.004 0.996 0.000
#> GSM340256 3 0.0524 0.763 0.000 0.004 0.988 0.008
#> GSM340258 3 0.4406 0.158 0.000 0.300 0.700 0.000
#> GSM340259 3 0.0000 0.764 0.000 0.000 1.000 0.000
#> GSM340260 3 0.0844 0.760 0.004 0.012 0.980 0.004
#> GSM340261 2 0.4905 0.661 0.000 0.632 0.364 0.004
#> GSM340262 3 0.0000 0.764 0.000 0.000 1.000 0.000
#> GSM340263 1 0.4220 0.632 0.748 0.000 0.248 0.004
#> GSM340264 3 0.3024 0.683 0.000 0.148 0.852 0.000
#> GSM340265 3 0.4907 0.495 0.000 0.420 0.580 0.000
#> GSM340266 3 0.7879 -0.339 0.000 0.332 0.380 0.288
#> GSM340267 2 0.3157 0.602 0.144 0.852 0.000 0.004
#> GSM340268 2 0.4830 0.646 0.000 0.608 0.392 0.000
#> GSM340269 3 0.4454 0.129 0.000 0.308 0.692 0.000
#> GSM340270 1 0.0188 0.875 0.996 0.000 0.000 0.004
#> GSM537574 2 0.4948 0.585 0.000 0.560 0.440 0.000
#> GSM537580 3 0.0188 0.763 0.000 0.000 0.996 0.004
#> GSM537581 4 0.4830 0.426 0.392 0.000 0.000 0.608
#> GSM340272 3 0.4888 0.501 0.000 0.412 0.588 0.000
#> GSM340273 1 0.0469 0.875 0.988 0.000 0.000 0.012
#> GSM340275 4 0.0336 0.956 0.000 0.000 0.008 0.992
#> GSM340276 3 0.7886 -0.137 0.376 0.200 0.416 0.008
#> GSM340277 3 0.4907 0.495 0.000 0.420 0.580 0.000
#> GSM340278 2 0.0188 0.620 0.000 0.996 0.004 0.000
#> GSM340279 2 0.6496 0.581 0.088 0.680 0.028 0.204
#> GSM340282 3 0.4907 0.495 0.000 0.420 0.580 0.000
#> GSM340284 2 0.5189 0.658 0.000 0.616 0.372 0.012
#> GSM340285 4 0.3982 0.733 0.000 0.220 0.004 0.776
#> GSM340286 3 0.0188 0.763 0.000 0.000 0.996 0.004
#> GSM340287 2 0.6127 0.658 0.044 0.624 0.320 0.012
#> GSM340288 1 0.0469 0.875 0.988 0.000 0.000 0.012
#> GSM340289 1 0.4661 0.475 0.652 0.000 0.348 0.000
#> GSM340290 2 0.3037 0.635 0.000 0.880 0.100 0.020
#> GSM340291 1 0.2777 0.813 0.888 0.104 0.004 0.004
#> GSM340293 2 0.0188 0.619 0.000 0.996 0.000 0.004
#> GSM340294 2 0.5127 0.663 0.000 0.632 0.356 0.012
#> GSM340296 2 0.0000 0.619 0.000 1.000 0.000 0.000
#> GSM340297 2 0.4843 0.373 0.396 0.604 0.000 0.000
#> GSM340298 2 0.0000 0.619 0.000 1.000 0.000 0.000
#> GSM340299 3 0.4522 0.565 0.000 0.320 0.680 0.000
#> GSM340301 3 0.0000 0.764 0.000 0.000 1.000 0.000
#> GSM340303 3 0.4907 0.495 0.000 0.420 0.580 0.000
#> GSM340304 1 0.5408 0.490 0.576 0.408 0.000 0.016
#> GSM340306 4 0.2345 0.882 0.100 0.000 0.000 0.900
#> GSM340307 2 0.0000 0.619 0.000 1.000 0.000 0.000
#> GSM340310 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM340314 2 0.6215 0.487 0.000 0.600 0.072 0.328
#> GSM340315 2 0.4977 0.229 0.460 0.540 0.000 0.000
#> GSM340317 4 0.0000 0.954 0.000 0.000 0.000 1.000
#> GSM340318 4 0.0336 0.956 0.000 0.000 0.008 0.992
#> GSM340319 4 0.0336 0.956 0.000 0.000 0.008 0.992
#> GSM340320 4 0.1716 0.913 0.064 0.000 0.000 0.936
#> GSM340321 2 0.4950 0.656 0.000 0.620 0.376 0.004
#> GSM340322 4 0.0336 0.956 0.000 0.000 0.008 0.992
#> GSM340324 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM340328 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM340330 2 0.4991 0.648 0.000 0.608 0.388 0.004
#> GSM340332 4 0.0336 0.956 0.000 0.000 0.008 0.992
#> GSM340333 3 0.0188 0.765 0.000 0.004 0.996 0.000
#> GSM340336 4 0.0336 0.956 0.000 0.000 0.008 0.992
#> GSM340337 4 0.0000 0.954 0.000 0.000 0.000 1.000
#> GSM340338 2 0.4661 0.664 0.000 0.652 0.348 0.000
#> GSM340339 4 0.0000 0.954 0.000 0.000 0.000 1.000
#> GSM340340 4 0.1302 0.933 0.044 0.000 0.000 0.956
#> GSM340341 4 0.0000 0.954 0.000 0.000 0.000 1.000
#> GSM340343 4 0.0336 0.956 0.000 0.000 0.008 0.992
#> GSM340344 3 0.0524 0.763 0.000 0.004 0.988 0.008
#> GSM340346 2 0.0000 0.619 0.000 1.000 0.000 0.000
#> GSM340347 4 0.0336 0.956 0.000 0.000 0.008 0.992
#> GSM340348 4 0.0000 0.954 0.000 0.000 0.000 1.000
#> GSM340349 1 0.5105 0.463 0.564 0.432 0.004 0.000
#> GSM340350 1 0.5212 0.477 0.572 0.420 0.008 0.000
#> GSM340351 1 0.0188 0.876 0.996 0.000 0.000 0.004
#> GSM340354 2 0.4817 0.650 0.000 0.612 0.388 0.000
#> GSM340356 1 0.0469 0.875 0.988 0.000 0.000 0.012
#> GSM340357 1 0.0336 0.874 0.992 0.008 0.000 0.000
#> GSM348183 1 0.2345 0.812 0.900 0.000 0.000 0.100
#> GSM348191 2 0.5004 0.644 0.000 0.604 0.392 0.004
#> GSM348193 1 0.0817 0.869 0.976 0.000 0.000 0.024
#> GSM537578 2 0.0000 0.619 0.000 1.000 0.000 0.000
#> GSM348181 1 0.5428 0.388 0.600 0.000 0.380 0.020
#> GSM348182 3 0.0188 0.763 0.000 0.000 0.996 0.004
#> GSM348184 2 0.4483 0.666 0.000 0.712 0.284 0.004
#> GSM348185 4 0.1302 0.928 0.044 0.000 0.000 0.956
#> GSM348186 3 0.0188 0.765 0.000 0.004 0.996 0.000
#> GSM348187 1 0.0188 0.876 0.996 0.000 0.000 0.004
#> GSM348188 4 0.0000 0.954 0.000 0.000 0.000 1.000
#> GSM348189 3 0.0188 0.763 0.000 0.000 0.996 0.004
#> GSM348190 3 0.1938 0.726 0.052 0.000 0.936 0.012
#> GSM348194 1 0.0469 0.875 0.988 0.000 0.000 0.012
#> GSM348195 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM348196 1 0.0469 0.875 0.988 0.000 0.000 0.012
#> GSM537585 1 0.6179 0.395 0.612 0.060 0.324 0.004
#> GSM537594 1 0.0469 0.875 0.988 0.000 0.000 0.012
#> GSM537596 1 0.0707 0.872 0.980 0.000 0.000 0.020
#> GSM537597 1 0.0469 0.875 0.988 0.000 0.000 0.012
#> GSM537602 1 0.4843 0.520 0.604 0.396 0.000 0.000
#> GSM340184 2 0.4991 0.648 0.000 0.608 0.388 0.004
#> GSM340185 2 0.0000 0.619 0.000 1.000 0.000 0.000
#> GSM340186 2 0.4804 0.651 0.000 0.616 0.384 0.000
#> GSM340187 4 0.0469 0.953 0.000 0.000 0.012 0.988
#> GSM340189 4 0.0524 0.954 0.000 0.004 0.008 0.988
#> GSM340190 2 0.0000 0.619 0.000 1.000 0.000 0.000
#> GSM340191 2 0.4843 0.642 0.000 0.604 0.396 0.000
#> GSM340192 3 0.4888 0.501 0.000 0.412 0.588 0.000
#> GSM340193 1 0.4420 0.648 0.748 0.000 0.240 0.012
#> GSM340194 3 0.0188 0.765 0.000 0.004 0.996 0.000
#> GSM340195 3 0.0000 0.764 0.000 0.000 1.000 0.000
#> GSM340196 4 0.0336 0.956 0.000 0.000 0.008 0.992
#> GSM340197 3 0.0000 0.764 0.000 0.000 1.000 0.000
#> GSM340198 3 0.0188 0.765 0.000 0.004 0.996 0.000
#> GSM340199 2 0.0000 0.619 0.000 1.000 0.000 0.000
#> GSM340200 2 0.0000 0.619 0.000 1.000 0.000 0.000
#> GSM340201 4 0.0336 0.956 0.000 0.000 0.008 0.992
#> GSM340202 4 0.0336 0.956 0.000 0.000 0.008 0.992
#> GSM340203 4 0.0188 0.955 0.000 0.000 0.004 0.996
#> GSM340204 3 0.0376 0.763 0.000 0.004 0.992 0.004
#> GSM340205 1 0.0000 0.876 1.000 0.000 0.000 0.000
#> GSM340206 2 0.4920 0.661 0.000 0.628 0.368 0.004
#> GSM340207 3 0.0469 0.761 0.000 0.012 0.988 0.000
#> GSM340237 1 0.4898 0.493 0.584 0.416 0.000 0.000
#> GSM340238 2 0.5371 0.659 0.000 0.616 0.364 0.020
#> GSM340239 3 0.0000 0.764 0.000 0.000 1.000 0.000
#> GSM340240 3 0.4907 0.495 0.000 0.420 0.580 0.000
#> GSM340241 2 0.4967 0.559 0.000 0.548 0.452 0.000
#> GSM340242 3 0.0000 0.764 0.000 0.000 1.000 0.000
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.0162 0.7938 0.996 0.004 0.000 0.000 0.000
#> GSM340359 1 0.0566 0.7899 0.984 0.004 0.000 0.000 0.012
#> GSM340361 1 0.0000 0.7939 1.000 0.000 0.000 0.000 0.000
#> GSM340362 5 0.3039 0.6753 0.192 0.000 0.000 0.000 0.808
#> GSM340363 1 0.5084 0.7244 0.712 0.144 0.140 0.004 0.000
#> GSM340364 1 0.0162 0.7938 0.996 0.004 0.000 0.000 0.000
#> GSM340365 5 0.4640 0.1379 0.016 0.400 0.000 0.000 0.584
#> GSM340366 2 0.3790 0.6255 0.004 0.724 0.000 0.000 0.272
#> GSM340367 1 0.0162 0.7938 0.996 0.004 0.000 0.000 0.000
#> GSM340368 4 0.2966 0.8012 0.184 0.000 0.000 0.816 0.000
#> GSM340369 4 0.3109 0.7846 0.200 0.000 0.000 0.800 0.000
#> GSM340370 1 0.0162 0.7938 0.996 0.004 0.000 0.000 0.000
#> GSM340371 5 0.2166 0.8212 0.000 0.012 0.072 0.004 0.912
#> GSM340372 1 0.0162 0.7938 0.996 0.004 0.000 0.000 0.000
#> GSM340373 1 0.0162 0.7938 0.996 0.004 0.000 0.000 0.000
#> GSM340375 1 0.0162 0.7938 0.996 0.004 0.000 0.000 0.000
#> GSM340376 1 0.0162 0.7938 0.996 0.004 0.000 0.000 0.000
#> GSM340378 1 0.0162 0.7938 0.996 0.004 0.000 0.000 0.000
#> GSM340243 2 0.4303 0.6407 0.192 0.752 0.056 0.000 0.000
#> GSM340244 1 0.8804 -0.0595 0.308 0.236 0.284 0.016 0.156
#> GSM340246 3 0.5016 0.7224 0.000 0.120 0.704 0.000 0.176
#> GSM340247 4 0.0000 0.9240 0.000 0.000 0.000 1.000 0.000
#> GSM340248 3 0.2707 0.6155 0.000 0.132 0.860 0.000 0.008
#> GSM340249 4 0.1106 0.9071 0.000 0.000 0.024 0.964 0.012
#> GSM340250 1 0.5313 0.3142 0.556 0.056 0.388 0.000 0.000
#> GSM340251 3 0.6719 0.6367 0.000 0.148 0.616 0.096 0.140
#> GSM340252 4 0.1310 0.9031 0.000 0.000 0.024 0.956 0.020
#> GSM340253 5 0.0880 0.8417 0.000 0.000 0.032 0.000 0.968
#> GSM340254 5 0.1579 0.8358 0.000 0.024 0.032 0.000 0.944
#> GSM340256 5 0.4879 0.6133 0.004 0.200 0.080 0.000 0.716
#> GSM340258 5 0.4278 -0.1187 0.000 0.000 0.452 0.000 0.548
#> GSM340259 5 0.4294 0.6590 0.004 0.072 0.148 0.000 0.776
#> GSM340260 5 0.3992 0.5611 0.000 0.012 0.268 0.000 0.720
#> GSM340261 3 0.3246 0.7512 0.000 0.008 0.808 0.000 0.184
#> GSM340262 5 0.1410 0.8306 0.000 0.000 0.060 0.000 0.940
#> GSM340263 1 0.7304 0.2122 0.516 0.068 0.228 0.000 0.188
#> GSM340264 5 0.3115 0.7720 0.000 0.112 0.036 0.000 0.852
#> GSM340265 2 0.3707 0.6151 0.000 0.716 0.000 0.000 0.284
#> GSM340266 3 0.6669 0.6110 0.000 0.184 0.592 0.048 0.176
#> GSM340267 3 0.5540 -0.1673 0.044 0.412 0.532 0.000 0.012
#> GSM340268 3 0.3530 0.7499 0.000 0.012 0.784 0.000 0.204
#> GSM340269 3 0.4651 0.5443 0.000 0.020 0.608 0.000 0.372
#> GSM340270 1 0.1117 0.7864 0.964 0.020 0.016 0.000 0.000
#> GSM537574 3 0.5192 0.6767 0.000 0.092 0.664 0.000 0.244
#> GSM537580 5 0.2349 0.8144 0.000 0.004 0.084 0.012 0.900
#> GSM537581 4 0.5505 0.4627 0.304 0.000 0.092 0.604 0.000
#> GSM340272 2 0.3684 0.6194 0.000 0.720 0.000 0.000 0.280
#> GSM340273 1 0.5693 0.6953 0.644 0.188 0.164 0.004 0.000
#> GSM340275 4 0.0162 0.9225 0.000 0.000 0.004 0.996 0.000
#> GSM340276 3 0.6964 0.5359 0.032 0.252 0.516 0.000 0.200
#> GSM340277 2 0.3480 0.6340 0.000 0.752 0.000 0.000 0.248
#> GSM340278 2 0.2773 0.6325 0.000 0.836 0.164 0.000 0.000
#> GSM340279 3 0.1299 0.6623 0.000 0.008 0.960 0.012 0.020
#> GSM340282 2 0.3684 0.6194 0.000 0.720 0.000 0.000 0.280
#> GSM340284 3 0.3280 0.7497 0.000 0.012 0.812 0.000 0.176
#> GSM340285 2 0.6512 0.0589 0.000 0.484 0.180 0.332 0.004
#> GSM340286 5 0.0451 0.8468 0.000 0.000 0.008 0.004 0.988
#> GSM340287 3 0.1168 0.6685 0.000 0.008 0.960 0.000 0.032
#> GSM340288 1 0.5630 0.6976 0.652 0.180 0.164 0.004 0.000
#> GSM340289 1 0.6016 0.3933 0.592 0.028 0.076 0.000 0.304
#> GSM340290 3 0.4268 0.5303 0.000 0.244 0.728 0.004 0.024
#> GSM340291 2 0.6532 -0.2982 0.388 0.452 0.152 0.000 0.008
#> GSM340293 2 0.3636 0.6482 0.000 0.728 0.272 0.000 0.000
#> GSM340294 3 0.3132 0.7537 0.000 0.008 0.820 0.000 0.172
#> GSM340296 2 0.3586 0.6522 0.000 0.736 0.264 0.000 0.000
#> GSM340297 3 0.4329 0.3905 0.312 0.016 0.672 0.000 0.000
#> GSM340298 2 0.3636 0.6482 0.000 0.728 0.272 0.000 0.000
#> GSM340299 5 0.3109 0.6193 0.000 0.200 0.000 0.000 0.800
#> GSM340301 5 0.0510 0.8467 0.000 0.000 0.016 0.000 0.984
#> GSM340303 2 0.3707 0.6151 0.000 0.716 0.000 0.000 0.284
#> GSM340304 2 0.2843 0.5785 0.144 0.848 0.008 0.000 0.000
#> GSM340306 4 0.3403 0.8157 0.008 0.012 0.160 0.820 0.000
#> GSM340307 2 0.3424 0.6537 0.000 0.760 0.240 0.000 0.000
#> GSM340310 1 0.0000 0.7939 1.000 0.000 0.000 0.000 0.000
#> GSM340314 3 0.4181 0.6683 0.000 0.008 0.784 0.156 0.052
#> GSM340315 3 0.4787 0.1502 0.324 0.036 0.640 0.000 0.000
#> GSM340317 4 0.2158 0.8888 0.008 0.052 0.020 0.920 0.000
#> GSM340318 4 0.0000 0.9240 0.000 0.000 0.000 1.000 0.000
#> GSM340319 4 0.0000 0.9240 0.000 0.000 0.000 1.000 0.000
#> GSM340320 4 0.3558 0.8126 0.008 0.020 0.156 0.816 0.000
#> GSM340321 3 0.4159 0.7419 0.000 0.068 0.776 0.000 0.156
#> GSM340322 4 0.0000 0.9240 0.000 0.000 0.000 1.000 0.000
#> GSM340324 1 0.1492 0.7785 0.948 0.040 0.008 0.000 0.004
#> GSM340328 1 0.0290 0.7942 0.992 0.008 0.000 0.000 0.000
#> GSM340330 3 0.3618 0.7504 0.000 0.012 0.788 0.004 0.196
#> GSM340332 4 0.0000 0.9240 0.000 0.000 0.000 1.000 0.000
#> GSM340333 5 0.0880 0.8417 0.000 0.000 0.032 0.000 0.968
#> GSM340336 4 0.0000 0.9240 0.000 0.000 0.000 1.000 0.000
#> GSM340337 4 0.0162 0.9232 0.000 0.000 0.004 0.996 0.000
#> GSM340338 3 0.5811 0.6450 0.000 0.140 0.596 0.000 0.264
#> GSM340339 4 0.0404 0.9205 0.000 0.000 0.012 0.988 0.000
#> GSM340340 4 0.2891 0.8089 0.176 0.000 0.000 0.824 0.000
#> GSM340341 4 0.2054 0.8896 0.000 0.028 0.052 0.920 0.000
#> GSM340343 4 0.0000 0.9240 0.000 0.000 0.000 1.000 0.000
#> GSM340344 5 0.2329 0.7880 0.000 0.000 0.124 0.000 0.876
#> GSM340346 2 0.3636 0.6482 0.000 0.728 0.272 0.000 0.000
#> GSM340347 4 0.0000 0.9240 0.000 0.000 0.000 1.000 0.000
#> GSM340348 4 0.4772 0.7338 0.000 0.108 0.164 0.728 0.000
#> GSM340349 2 0.2818 0.6605 0.132 0.856 0.012 0.000 0.000
#> GSM340350 2 0.3636 0.6133 0.272 0.728 0.000 0.000 0.000
#> GSM340351 1 0.2886 0.7603 0.844 0.008 0.148 0.000 0.000
#> GSM340354 3 0.4757 0.7340 0.000 0.080 0.716 0.000 0.204
#> GSM340356 1 0.5810 0.6870 0.628 0.204 0.164 0.004 0.000
#> GSM340357 1 0.0794 0.7918 0.972 0.028 0.000 0.000 0.000
#> GSM348183 1 0.1530 0.7828 0.952 0.008 0.004 0.028 0.008
#> GSM348191 3 0.4160 0.7467 0.000 0.036 0.772 0.008 0.184
#> GSM348193 1 0.4370 0.7487 0.784 0.032 0.148 0.036 0.000
#> GSM537578 2 0.3395 0.6618 0.000 0.764 0.236 0.000 0.000
#> GSM348181 1 0.7886 -0.2653 0.364 0.052 0.364 0.012 0.208
#> GSM348182 5 0.0451 0.8468 0.000 0.000 0.008 0.004 0.988
#> GSM348184 3 0.5939 0.6511 0.000 0.188 0.640 0.016 0.156
#> GSM348185 4 0.5496 0.6635 0.004 0.164 0.164 0.668 0.000
#> GSM348186 5 0.1753 0.8321 0.000 0.032 0.032 0.000 0.936
#> GSM348187 1 0.5408 0.7028 0.664 0.180 0.156 0.000 0.000
#> GSM348188 4 0.0162 0.9232 0.000 0.000 0.004 0.996 0.000
#> GSM348189 5 0.1502 0.8342 0.000 0.000 0.056 0.004 0.940
#> GSM348190 5 0.3086 0.7054 0.000 0.000 0.180 0.004 0.816
#> GSM348194 1 0.5630 0.6976 0.652 0.180 0.164 0.004 0.000
#> GSM348195 1 0.1121 0.7899 0.956 0.000 0.044 0.000 0.000
#> GSM348196 1 0.5630 0.6976 0.652 0.180 0.164 0.004 0.000
#> GSM537585 1 0.7142 0.5031 0.536 0.068 0.156 0.000 0.240
#> GSM537594 1 0.5928 0.6700 0.596 0.212 0.192 0.000 0.000
#> GSM537596 1 0.6883 0.6052 0.528 0.220 0.228 0.020 0.004
#> GSM537597 1 0.5630 0.6976 0.652 0.180 0.164 0.004 0.000
#> GSM537602 2 0.3300 0.5426 0.204 0.792 0.004 0.000 0.000
#> GSM340184 3 0.3670 0.7524 0.000 0.008 0.792 0.012 0.188
#> GSM340185 2 0.3480 0.6473 0.000 0.752 0.248 0.000 0.000
#> GSM340186 3 0.5854 0.6356 0.000 0.152 0.596 0.000 0.252
#> GSM340187 4 0.0162 0.9225 0.000 0.000 0.000 0.996 0.004
#> GSM340189 4 0.0162 0.9225 0.000 0.000 0.000 0.996 0.004
#> GSM340190 2 0.4101 0.4696 0.000 0.628 0.372 0.000 0.000
#> GSM340191 3 0.3897 0.7473 0.000 0.028 0.768 0.000 0.204
#> GSM340192 2 0.3837 0.5800 0.000 0.692 0.000 0.000 0.308
#> GSM340193 3 0.8698 -0.2544 0.260 0.228 0.292 0.004 0.216
#> GSM340194 5 0.0510 0.8467 0.000 0.000 0.016 0.000 0.984
#> GSM340195 5 0.0404 0.8470 0.000 0.000 0.012 0.000 0.988
#> GSM340196 4 0.0000 0.9240 0.000 0.000 0.000 1.000 0.000
#> GSM340197 5 0.0000 0.8467 0.000 0.000 0.000 0.000 1.000
#> GSM340198 5 0.1753 0.8321 0.000 0.032 0.032 0.000 0.936
#> GSM340199 2 0.3636 0.6482 0.000 0.728 0.272 0.000 0.000
#> GSM340200 2 0.3395 0.6560 0.000 0.764 0.236 0.000 0.000
#> GSM340201 4 0.0000 0.9240 0.000 0.000 0.000 1.000 0.000
#> GSM340202 4 0.0000 0.9240 0.000 0.000 0.000 1.000 0.000
#> GSM340203 4 0.0324 0.9211 0.000 0.000 0.004 0.992 0.004
#> GSM340204 5 0.1818 0.8371 0.000 0.024 0.044 0.000 0.932
#> GSM340205 1 0.0771 0.7897 0.976 0.020 0.004 0.000 0.000
#> GSM340206 3 0.3778 0.7525 0.000 0.012 0.788 0.012 0.188
#> GSM340207 5 0.2574 0.7814 0.000 0.012 0.112 0.000 0.876
#> GSM340237 2 0.3336 0.6356 0.228 0.772 0.000 0.000 0.000
#> GSM340238 3 0.3900 0.7537 0.000 0.012 0.788 0.020 0.180
#> GSM340239 5 0.0290 0.8468 0.000 0.000 0.008 0.000 0.992
#> GSM340240 2 0.3796 0.5946 0.000 0.700 0.000 0.000 0.300
#> GSM340241 3 0.6047 0.4803 0.000 0.124 0.500 0.000 0.376
#> GSM340242 5 0.1892 0.8153 0.000 0.004 0.080 0.000 0.916
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.0000 0.8265 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340359 1 0.0363 0.8208 0.988 0.000 0.000 0.000 0.012 0.000
#> GSM340361 1 0.0508 0.8219 0.984 0.004 0.000 0.000 0.000 0.012
#> GSM340362 5 0.3124 0.6791 0.164 0.004 0.012 0.000 0.816 0.004
#> GSM340363 6 0.4039 0.2733 0.424 0.008 0.000 0.000 0.000 0.568
#> GSM340364 1 0.0000 0.8265 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340365 5 0.5112 -0.0491 0.080 0.444 0.000 0.000 0.476 0.000
#> GSM340366 2 0.1863 0.8292 0.000 0.896 0.000 0.000 0.104 0.000
#> GSM340367 1 0.0000 0.8265 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340368 4 0.2730 0.7513 0.192 0.000 0.000 0.808 0.000 0.000
#> GSM340369 4 0.2969 0.7141 0.224 0.000 0.000 0.776 0.000 0.000
#> GSM340370 1 0.0000 0.8265 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340371 5 0.4401 0.6425 0.000 0.040 0.300 0.004 0.656 0.000
#> GSM340372 1 0.0000 0.8265 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.8265 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340375 1 0.0000 0.8265 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340376 1 0.0000 0.8265 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340378 1 0.0000 0.8265 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340243 2 0.3622 0.7820 0.100 0.820 0.032 0.000 0.000 0.048
#> GSM340244 6 0.6414 0.2420 0.028 0.060 0.120 0.012 0.148 0.632
#> GSM340246 3 0.6609 0.5468 0.000 0.120 0.544 0.000 0.156 0.180
#> GSM340247 4 0.0000 0.9063 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340248 6 0.5725 0.0628 0.000 0.068 0.304 0.000 0.056 0.572
#> GSM340249 4 0.2592 0.8266 0.000 0.004 0.020 0.884 0.080 0.012
#> GSM340250 1 0.6180 -0.0344 0.420 0.004 0.356 0.000 0.004 0.216
#> GSM340251 3 0.7786 0.5033 0.000 0.164 0.480 0.076 0.144 0.136
#> GSM340252 4 0.2815 0.8052 0.000 0.000 0.028 0.864 0.096 0.012
#> GSM340253 5 0.1442 0.7209 0.000 0.004 0.040 0.000 0.944 0.012
#> GSM340254 5 0.3160 0.6693 0.000 0.036 0.044 0.000 0.856 0.064
#> GSM340256 5 0.6295 0.1652 0.000 0.068 0.092 0.000 0.456 0.384
#> GSM340258 3 0.4366 0.1751 0.000 0.004 0.540 0.000 0.440 0.016
#> GSM340259 5 0.5563 0.4826 0.000 0.016 0.288 0.000 0.576 0.120
#> GSM340260 5 0.5410 0.3936 0.004 0.020 0.404 0.000 0.516 0.056
#> GSM340261 3 0.1616 0.6932 0.000 0.012 0.940 0.000 0.028 0.020
#> GSM340262 5 0.2793 0.7318 0.000 0.000 0.200 0.000 0.800 0.000
#> GSM340263 1 0.7546 0.1805 0.436 0.024 0.136 0.000 0.144 0.260
#> GSM340264 5 0.4467 0.7039 0.000 0.112 0.164 0.000 0.720 0.004
#> GSM340265 2 0.2520 0.7991 0.000 0.844 0.004 0.000 0.152 0.000
#> GSM340266 3 0.5310 0.4517 0.000 0.004 0.596 0.004 0.108 0.288
#> GSM340267 6 0.6525 0.2120 0.020 0.344 0.200 0.000 0.008 0.428
#> GSM340268 3 0.2152 0.6897 0.000 0.024 0.904 0.000 0.068 0.004
#> GSM340269 3 0.4136 0.4753 0.000 0.004 0.708 0.000 0.248 0.040
#> GSM340270 1 0.2208 0.7880 0.912 0.008 0.016 0.000 0.012 0.052
#> GSM537574 3 0.4733 0.6052 0.000 0.012 0.704 0.000 0.172 0.112
#> GSM537580 5 0.4078 0.6525 0.000 0.016 0.300 0.008 0.676 0.000
#> GSM537581 4 0.5051 0.3443 0.104 0.000 0.000 0.596 0.000 0.300
#> GSM340272 2 0.2135 0.8165 0.000 0.872 0.000 0.000 0.128 0.000
#> GSM340273 6 0.2996 0.5655 0.228 0.000 0.000 0.000 0.000 0.772
#> GSM340275 4 0.0146 0.9045 0.000 0.000 0.004 0.996 0.000 0.000
#> GSM340276 6 0.5523 -0.0259 0.000 0.012 0.384 0.000 0.096 0.508
#> GSM340277 2 0.3017 0.7986 0.000 0.844 0.000 0.000 0.072 0.084
#> GSM340278 2 0.4500 0.6681 0.000 0.688 0.088 0.000 0.000 0.224
#> GSM340279 3 0.4139 0.3342 0.000 0.024 0.640 0.000 0.000 0.336
#> GSM340282 2 0.1957 0.8261 0.000 0.888 0.000 0.000 0.112 0.000
#> GSM340284 3 0.4084 0.6614 0.000 0.032 0.776 0.000 0.144 0.048
#> GSM340285 6 0.7589 0.0376 0.000 0.288 0.200 0.156 0.004 0.352
#> GSM340286 5 0.2442 0.7521 0.000 0.000 0.144 0.004 0.852 0.000
#> GSM340287 3 0.3259 0.5538 0.000 0.012 0.772 0.000 0.000 0.216
#> GSM340288 6 0.3330 0.5350 0.284 0.000 0.000 0.000 0.000 0.716
#> GSM340289 1 0.6855 0.3404 0.528 0.008 0.208 0.000 0.136 0.120
#> GSM340290 3 0.4601 0.5856 0.000 0.176 0.728 0.008 0.012 0.076
#> GSM340291 6 0.6974 0.2596 0.060 0.188 0.060 0.000 0.136 0.556
#> GSM340293 2 0.2841 0.7663 0.000 0.824 0.164 0.012 0.000 0.000
#> GSM340294 3 0.2016 0.6844 0.000 0.016 0.920 0.000 0.040 0.024
#> GSM340296 2 0.1501 0.8317 0.000 0.924 0.076 0.000 0.000 0.000
#> GSM340297 3 0.6060 0.2402 0.180 0.028 0.544 0.000 0.000 0.248
#> GSM340298 2 0.1556 0.8307 0.000 0.920 0.080 0.000 0.000 0.000
#> GSM340299 5 0.2706 0.6874 0.000 0.160 0.008 0.000 0.832 0.000
#> GSM340301 5 0.2219 0.7548 0.000 0.000 0.136 0.000 0.864 0.000
#> GSM340303 2 0.2135 0.8169 0.000 0.872 0.000 0.000 0.128 0.000
#> GSM340304 2 0.4375 0.6175 0.028 0.680 0.016 0.000 0.000 0.276
#> GSM340306 4 0.3955 0.1926 0.004 0.000 0.000 0.560 0.000 0.436
#> GSM340307 2 0.1498 0.8265 0.000 0.940 0.028 0.000 0.000 0.032
#> GSM340310 1 0.0260 0.8240 0.992 0.000 0.000 0.000 0.000 0.008
#> GSM340314 3 0.2507 0.6828 0.000 0.020 0.892 0.060 0.028 0.000
#> GSM340315 6 0.5866 0.1617 0.136 0.012 0.412 0.000 0.000 0.440
#> GSM340317 4 0.4166 0.6845 0.012 0.020 0.000 0.756 0.024 0.188
#> GSM340318 4 0.0000 0.9063 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340319 4 0.0000 0.9063 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340320 6 0.4091 0.0228 0.008 0.000 0.000 0.472 0.000 0.520
#> GSM340321 3 0.6448 0.5476 0.000 0.096 0.552 0.000 0.136 0.216
#> GSM340322 4 0.0000 0.9063 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340324 1 0.3888 0.6531 0.788 0.036 0.000 0.000 0.032 0.144
#> GSM340328 1 0.0937 0.8041 0.960 0.000 0.000 0.000 0.000 0.040
#> GSM340330 3 0.2094 0.6793 0.000 0.024 0.908 0.004 0.064 0.000
#> GSM340332 4 0.0146 0.9046 0.000 0.000 0.004 0.996 0.000 0.000
#> GSM340333 5 0.1464 0.7202 0.000 0.004 0.036 0.000 0.944 0.016
#> GSM340336 4 0.0000 0.9063 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340337 4 0.0146 0.9052 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM340338 3 0.5279 0.5774 0.000 0.180 0.648 0.000 0.156 0.016
#> GSM340339 4 0.0260 0.9023 0.000 0.000 0.000 0.992 0.000 0.008
#> GSM340340 4 0.2838 0.7534 0.188 0.000 0.000 0.808 0.004 0.000
#> GSM340341 4 0.2278 0.8026 0.000 0.000 0.004 0.868 0.000 0.128
#> GSM340343 4 0.0000 0.9063 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340344 5 0.1565 0.7279 0.000 0.004 0.028 0.000 0.940 0.028
#> GSM340346 2 0.1501 0.8317 0.000 0.924 0.076 0.000 0.000 0.000
#> GSM340347 4 0.0000 0.9063 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340348 6 0.3950 0.1301 0.000 0.000 0.004 0.432 0.000 0.564
#> GSM340349 2 0.2645 0.8173 0.020 0.880 0.012 0.000 0.004 0.084
#> GSM340350 2 0.2163 0.8138 0.096 0.892 0.000 0.000 0.008 0.004
#> GSM340351 1 0.3841 0.2148 0.616 0.004 0.000 0.000 0.000 0.380
#> GSM340354 3 0.4062 0.6418 0.000 0.144 0.772 0.000 0.068 0.016
#> GSM340356 6 0.3394 0.5622 0.236 0.012 0.000 0.000 0.000 0.752
#> GSM340357 1 0.1411 0.7913 0.936 0.004 0.000 0.000 0.000 0.060
#> GSM348183 1 0.2379 0.7880 0.904 0.008 0.000 0.024 0.012 0.052
#> GSM348191 3 0.2394 0.6758 0.000 0.008 0.900 0.004 0.052 0.036
#> GSM348193 1 0.4300 0.0292 0.540 0.008 0.000 0.008 0.000 0.444
#> GSM537578 2 0.1745 0.8360 0.000 0.924 0.056 0.000 0.000 0.020
#> GSM348181 3 0.7032 -0.0199 0.388 0.012 0.404 0.008 0.064 0.124
#> GSM348182 5 0.2837 0.7517 0.000 0.004 0.144 0.008 0.840 0.004
#> GSM348184 3 0.6705 0.5422 0.000 0.184 0.532 0.000 0.148 0.136
#> GSM348185 6 0.3468 0.4421 0.004 0.000 0.000 0.284 0.000 0.712
#> GSM348186 5 0.3470 0.6378 0.000 0.048 0.024 0.000 0.828 0.100
#> GSM348187 6 0.3351 0.5308 0.288 0.000 0.000 0.000 0.000 0.712
#> GSM348188 4 0.0146 0.9052 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM348189 5 0.4002 0.6687 0.000 0.016 0.284 0.008 0.692 0.000
#> GSM348190 5 0.3589 0.6109 0.000 0.008 0.012 0.000 0.752 0.228
#> GSM348194 6 0.3330 0.5350 0.284 0.000 0.000 0.000 0.000 0.716
#> GSM348195 1 0.2703 0.6528 0.824 0.004 0.000 0.000 0.000 0.172
#> GSM348196 6 0.3330 0.5350 0.284 0.000 0.000 0.000 0.000 0.716
#> GSM537585 6 0.6968 0.3834 0.240 0.100 0.000 0.000 0.196 0.464
#> GSM537594 6 0.3869 0.5430 0.104 0.040 0.000 0.000 0.052 0.804
#> GSM537596 6 0.3377 0.5788 0.136 0.000 0.056 0.000 0.000 0.808
#> GSM537597 6 0.3151 0.5558 0.252 0.000 0.000 0.000 0.000 0.748
#> GSM537602 2 0.4280 0.6689 0.044 0.716 0.012 0.000 0.000 0.228
#> GSM340184 3 0.2014 0.6820 0.000 0.012 0.920 0.008 0.052 0.008
#> GSM340185 2 0.3141 0.7571 0.000 0.836 0.048 0.000 0.004 0.112
#> GSM340186 3 0.6953 0.5117 0.000 0.168 0.492 0.000 0.200 0.140
#> GSM340187 4 0.0405 0.9013 0.000 0.000 0.008 0.988 0.004 0.000
#> GSM340189 4 0.0000 0.9063 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340190 2 0.4799 0.3335 0.000 0.592 0.340 0.000 0.000 0.068
#> GSM340191 3 0.2573 0.6889 0.000 0.008 0.884 0.000 0.064 0.044
#> GSM340192 2 0.3231 0.7469 0.000 0.784 0.000 0.000 0.200 0.016
#> GSM340193 6 0.4258 0.5265 0.024 0.008 0.076 0.000 0.112 0.780
#> GSM340194 5 0.2362 0.7545 0.000 0.004 0.136 0.000 0.860 0.000
#> GSM340195 5 0.2504 0.7539 0.000 0.004 0.136 0.000 0.856 0.004
#> GSM340196 4 0.0000 0.9063 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340197 5 0.2092 0.7549 0.000 0.000 0.124 0.000 0.876 0.000
#> GSM340198 5 0.3953 0.6221 0.000 0.052 0.048 0.000 0.800 0.100
#> GSM340199 2 0.1610 0.8292 0.000 0.916 0.084 0.000 0.000 0.000
#> GSM340200 2 0.1074 0.8321 0.000 0.960 0.028 0.000 0.000 0.012
#> GSM340201 4 0.0000 0.9063 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340202 4 0.0000 0.9063 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340203 4 0.0603 0.8970 0.000 0.000 0.016 0.980 0.004 0.000
#> GSM340204 5 0.3646 0.7185 0.000 0.008 0.132 0.000 0.800 0.060
#> GSM340205 1 0.1845 0.7849 0.916 0.008 0.004 0.000 0.000 0.072
#> GSM340206 3 0.2101 0.6836 0.000 0.028 0.912 0.008 0.052 0.000
#> GSM340207 5 0.4234 0.6091 0.000 0.044 0.280 0.000 0.676 0.000
#> GSM340237 2 0.3885 0.7255 0.100 0.780 0.004 0.000 0.000 0.116
#> GSM340238 3 0.1938 0.6829 0.000 0.020 0.920 0.008 0.052 0.000
#> GSM340239 5 0.2491 0.7509 0.000 0.000 0.164 0.000 0.836 0.000
#> GSM340240 2 0.2491 0.7895 0.000 0.836 0.000 0.000 0.164 0.000
#> GSM340241 3 0.7260 0.3090 0.000 0.128 0.352 0.000 0.348 0.172
#> GSM340242 5 0.3888 0.6525 0.000 0.016 0.312 0.000 0.672 0.000
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
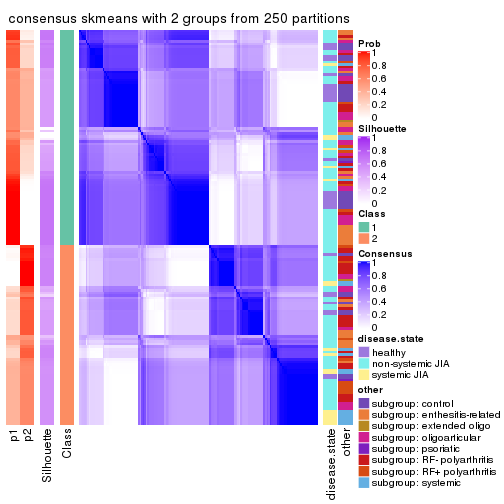
consensus_heatmap(res, k = 3)
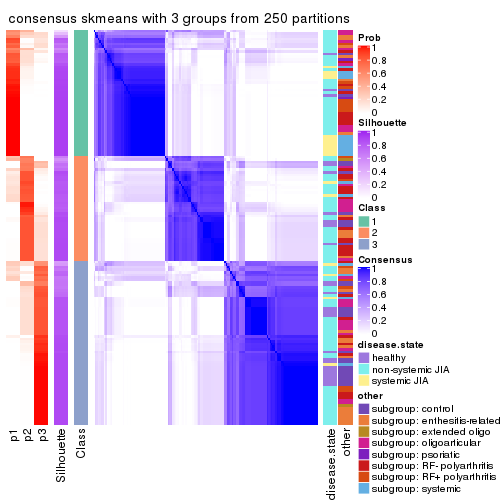
consensus_heatmap(res, k = 4)
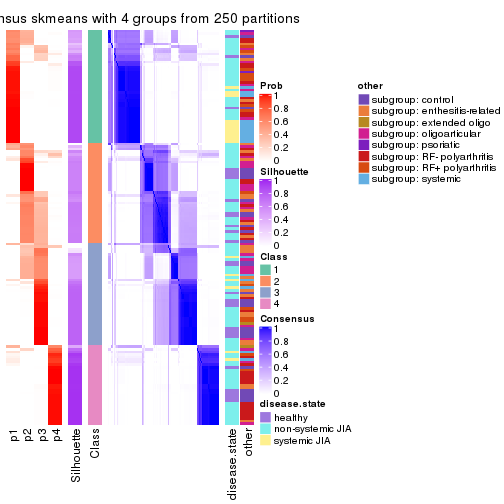
consensus_heatmap(res, k = 5)
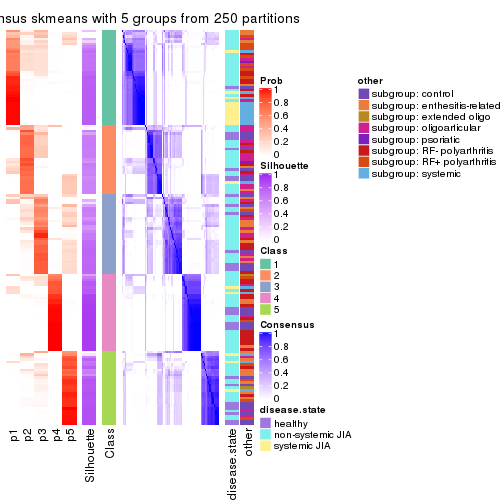
consensus_heatmap(res, k = 6)
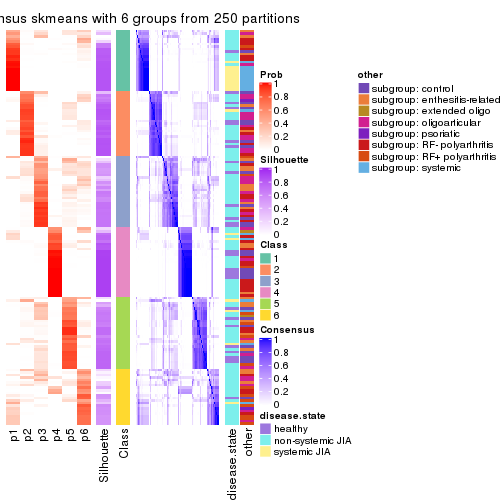
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
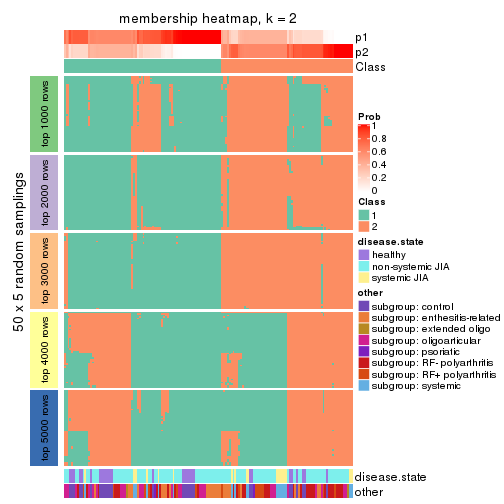
membership_heatmap(res, k = 3)
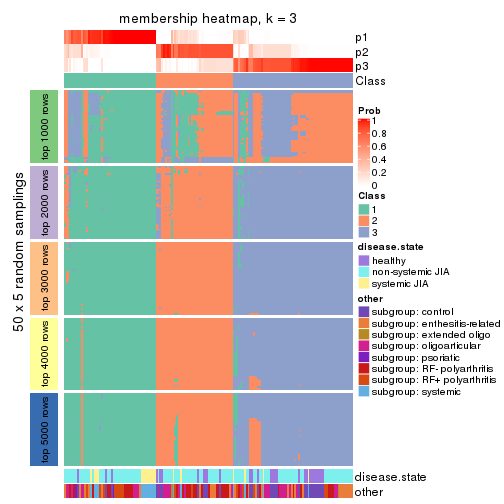
membership_heatmap(res, k = 4)
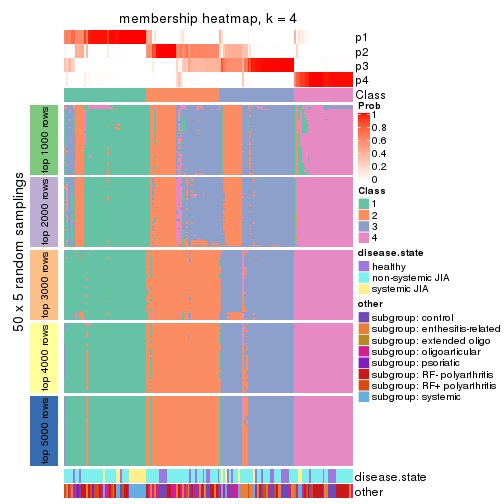
membership_heatmap(res, k = 5)
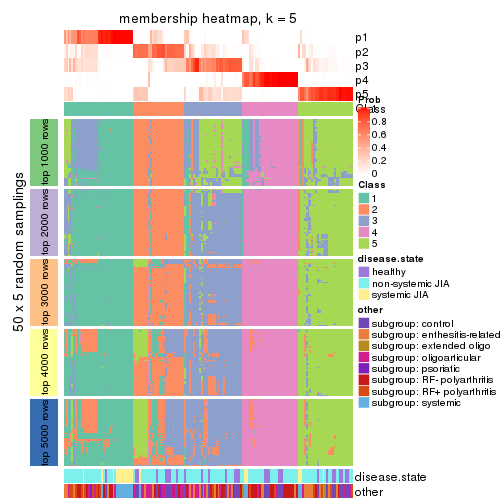
membership_heatmap(res, k = 6)
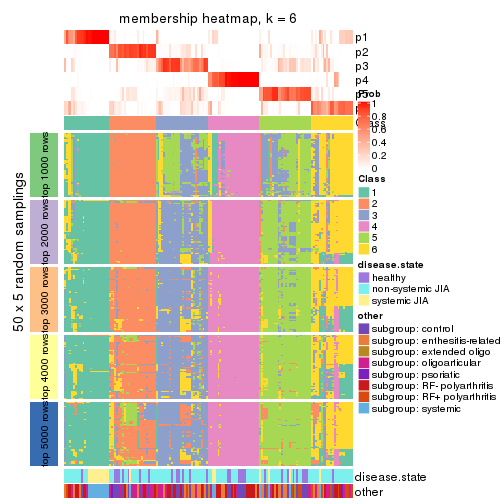
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
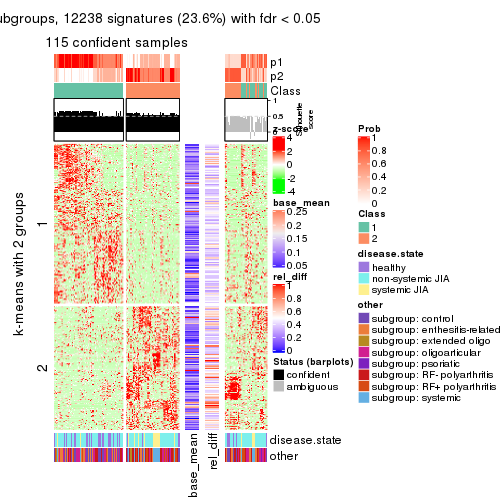
get_signatures(res, k = 3)
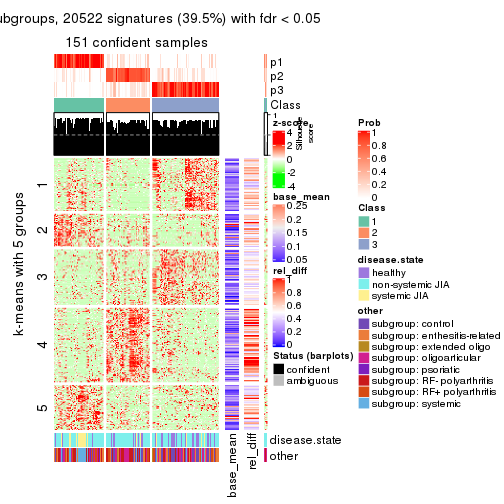
get_signatures(res, k = 4)
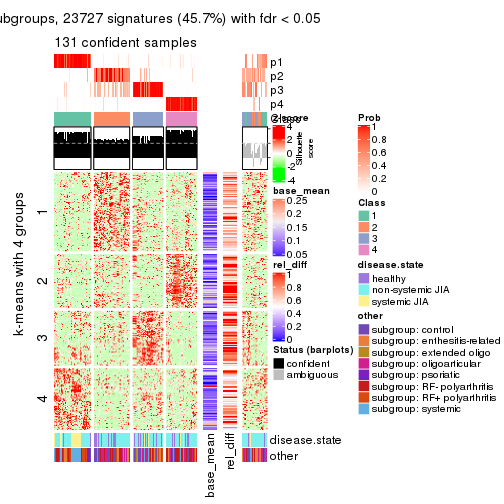
get_signatures(res, k = 5)
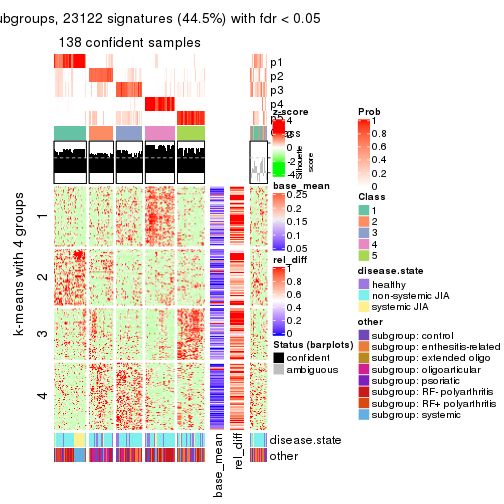
get_signatures(res, k = 6)
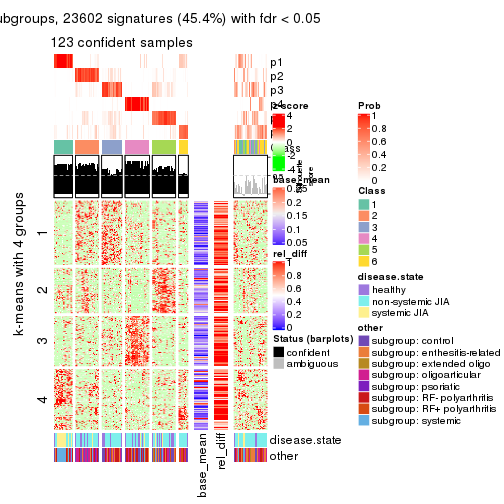
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
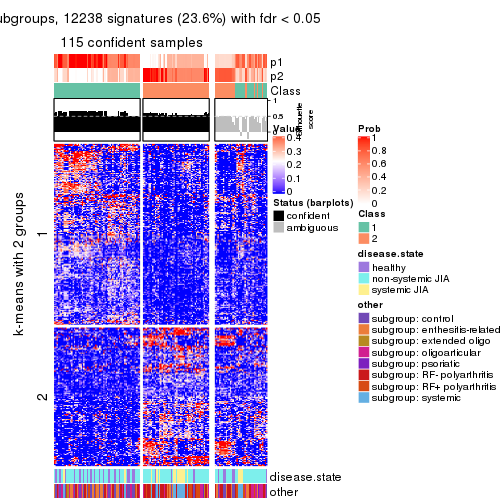
get_signatures(res, k = 3, scale_rows = FALSE)
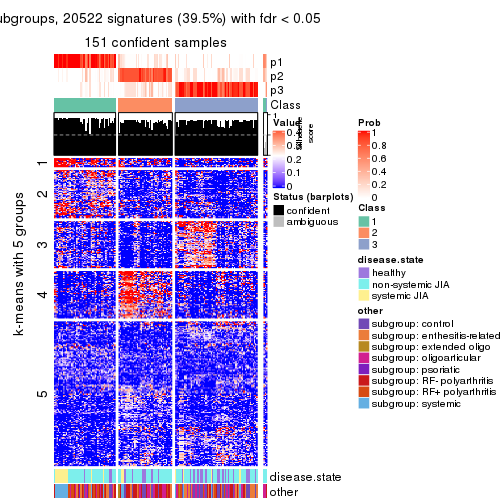
get_signatures(res, k = 4, scale_rows = FALSE)
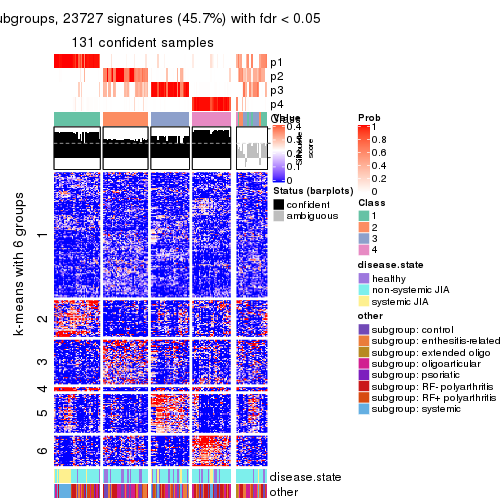
get_signatures(res, k = 5, scale_rows = FALSE)
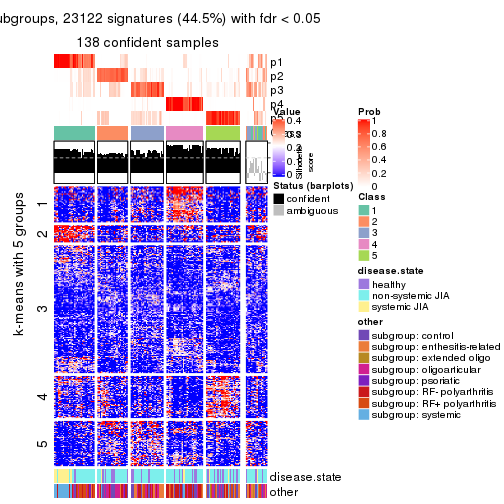
get_signatures(res, k = 6, scale_rows = FALSE)
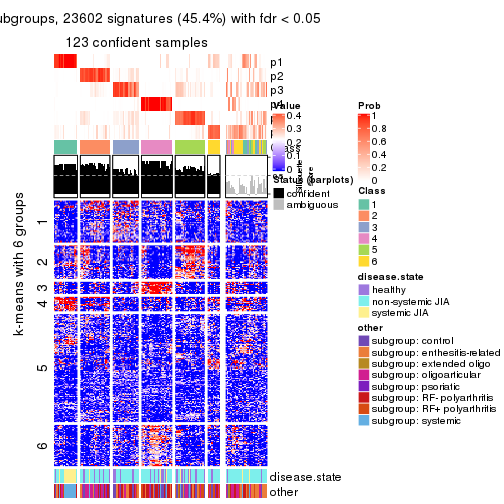
Compare the overlap of signatures from different k:
compare_signatures(res)
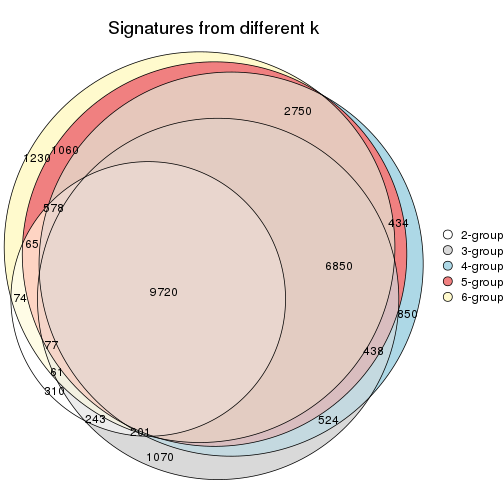
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
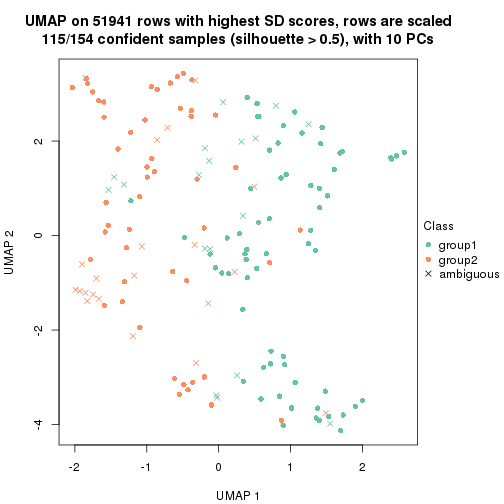
dimension_reduction(res, k = 3, method = "UMAP")
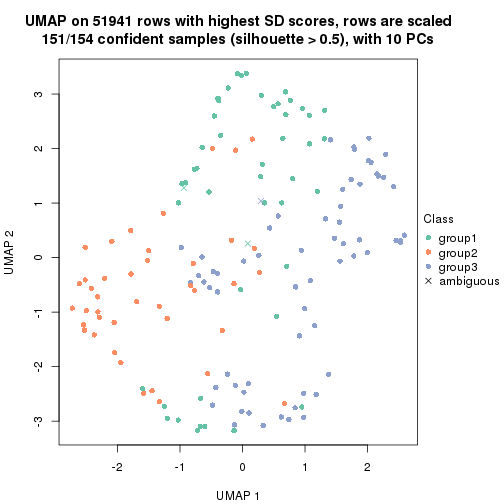
dimension_reduction(res, k = 4, method = "UMAP")
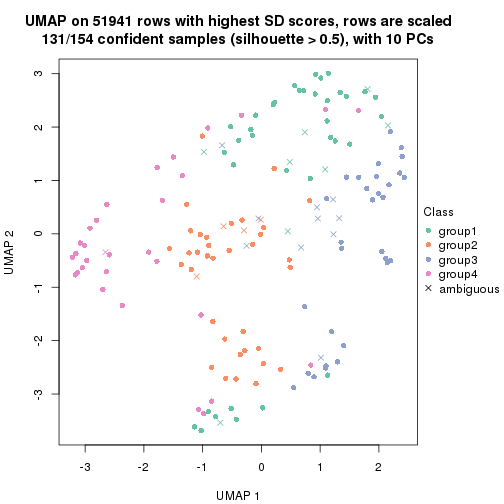
dimension_reduction(res, k = 5, method = "UMAP")
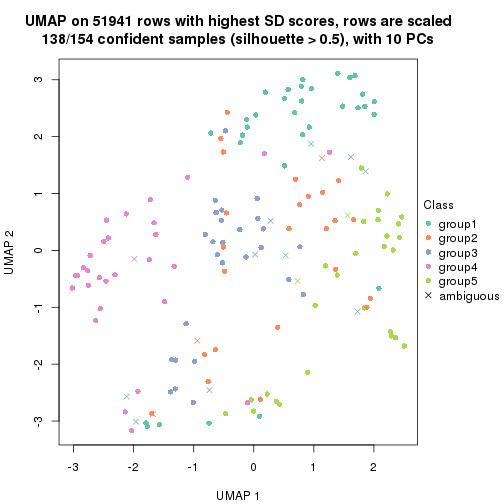
dimension_reduction(res, k = 6, method = "UMAP")
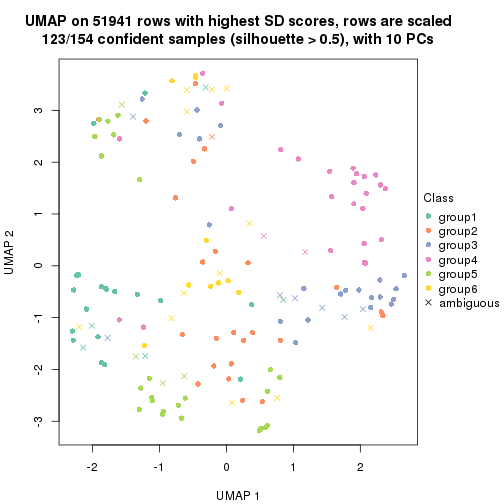
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
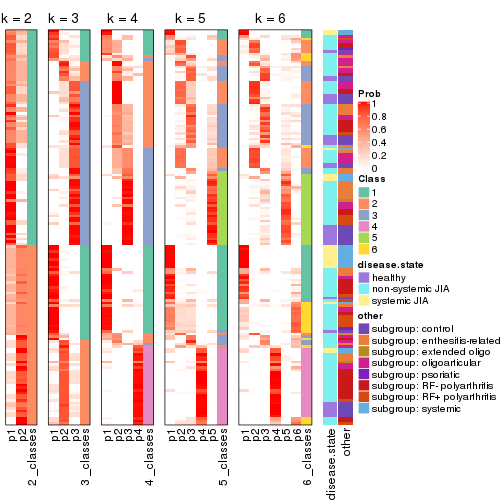
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> SD:skmeans 115 7.89e-05 1.22e-05 2
#> SD:skmeans 151 1.26e-03 8.13e-04 3
#> SD:skmeans 131 4.85e-04 5.69e-05 4
#> SD:skmeans 138 2.34e-04 1.34e-06 5
#> SD:skmeans 123 2.73e-06 2.27e-08 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["SD", "pam"]
# you can also extract it by
# res = res_list["SD:pam"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'SD' method.
#> Subgroups are detected by 'pam' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 6.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
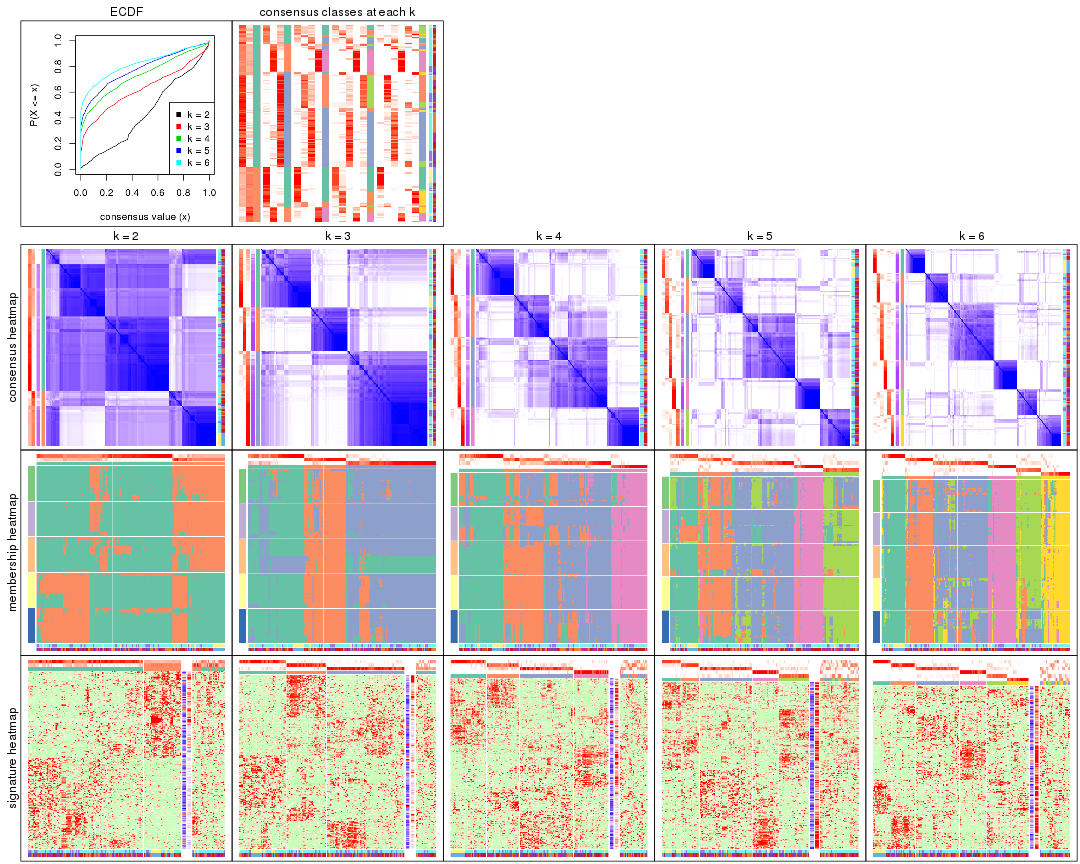
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
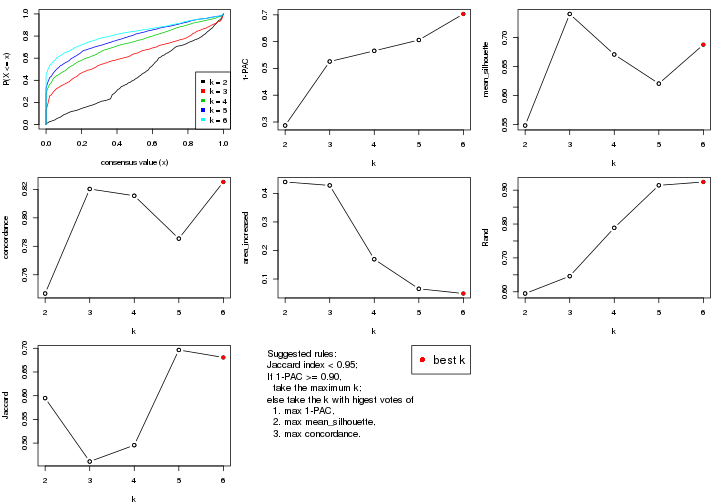
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.287 0.548 0.747 0.4400 0.595 0.595
#> 3 3 0.526 0.741 0.820 0.4279 0.646 0.461
#> 4 4 0.566 0.671 0.816 0.1692 0.789 0.496
#> 5 5 0.606 0.621 0.785 0.0658 0.914 0.696
#> 6 6 0.703 0.688 0.825 0.0492 0.924 0.681
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 6
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 2 0.8813 0.726531 0.300 0.700
#> GSM340359 2 0.9460 0.763424 0.364 0.636
#> GSM340361 2 0.9460 0.763424 0.364 0.636
#> GSM340362 1 0.5294 0.568988 0.880 0.120
#> GSM340363 2 0.9580 0.742864 0.380 0.620
#> GSM340364 2 0.9460 0.763424 0.364 0.636
#> GSM340365 2 1.0000 0.554535 0.496 0.504
#> GSM340366 1 0.6048 0.504981 0.852 0.148
#> GSM340367 2 0.9460 0.763424 0.364 0.636
#> GSM340368 2 0.0000 0.494008 0.000 1.000
#> GSM340369 2 0.3274 0.457620 0.060 0.940
#> GSM340370 2 0.9460 0.763424 0.364 0.636
#> GSM340371 1 0.7815 0.636855 0.768 0.232
#> GSM340372 2 0.9460 0.763424 0.364 0.636
#> GSM340373 2 0.9460 0.763424 0.364 0.636
#> GSM340375 2 0.9460 0.763424 0.364 0.636
#> GSM340376 2 0.9460 0.763424 0.364 0.636
#> GSM340378 2 0.9460 0.763424 0.364 0.636
#> GSM340243 2 0.9944 0.673518 0.456 0.544
#> GSM340244 1 0.1843 0.666193 0.972 0.028
#> GSM340246 1 0.0000 0.677386 1.000 0.000
#> GSM340247 1 0.9580 0.555236 0.620 0.380
#> GSM340248 1 0.2423 0.658629 0.960 0.040
#> GSM340249 1 0.9580 0.555236 0.620 0.380
#> GSM340250 1 0.9977 -0.549381 0.528 0.472
#> GSM340251 1 0.9460 0.563628 0.636 0.364
#> GSM340252 1 0.9815 0.538606 0.580 0.420
#> GSM340253 1 0.3274 0.637078 0.940 0.060
#> GSM340254 1 0.0376 0.676447 0.996 0.004
#> GSM340256 1 0.3274 0.638056 0.940 0.060
#> GSM340258 1 0.0000 0.677386 1.000 0.000
#> GSM340259 1 0.2603 0.653948 0.956 0.044
#> GSM340260 1 0.0376 0.676569 0.996 0.004
#> GSM340261 1 0.0000 0.677386 1.000 0.000
#> GSM340262 1 0.0376 0.676447 0.996 0.004
#> GSM340263 1 0.5737 0.550889 0.864 0.136
#> GSM340264 1 0.1843 0.665917 0.972 0.028
#> GSM340265 1 0.0000 0.677386 1.000 0.000
#> GSM340266 1 0.8763 0.616638 0.704 0.296
#> GSM340267 1 0.0376 0.676666 0.996 0.004
#> GSM340268 1 0.8499 0.622380 0.724 0.276
#> GSM340269 1 0.0000 0.677386 1.000 0.000
#> GSM340270 1 0.9963 -0.515716 0.536 0.464
#> GSM537574 1 0.0000 0.677386 1.000 0.000
#> GSM537580 1 0.8499 0.622380 0.724 0.276
#> GSM537581 2 0.3274 0.457620 0.060 0.940
#> GSM340272 1 0.7528 0.374839 0.784 0.216
#> GSM340273 2 0.9710 0.740011 0.400 0.600
#> GSM340275 1 0.9754 0.539291 0.592 0.408
#> GSM340276 1 0.6531 0.654169 0.832 0.168
#> GSM340277 1 0.2043 0.659489 0.968 0.032
#> GSM340278 1 0.5408 0.665473 0.876 0.124
#> GSM340279 1 0.8499 0.622380 0.724 0.276
#> GSM340282 1 0.2043 0.659489 0.968 0.032
#> GSM340284 1 0.8763 0.611752 0.704 0.296
#> GSM340285 1 0.9044 0.607712 0.680 0.320
#> GSM340286 1 0.1633 0.668411 0.976 0.024
#> GSM340287 1 0.0938 0.673651 0.988 0.012
#> GSM340288 2 0.9732 0.735887 0.404 0.596
#> GSM340289 1 0.5408 0.568544 0.876 0.124
#> GSM340290 1 0.9087 0.601138 0.676 0.324
#> GSM340291 1 0.5178 0.577202 0.884 0.116
#> GSM340293 1 0.9460 0.563628 0.636 0.364
#> GSM340294 1 0.0000 0.677386 1.000 0.000
#> GSM340296 1 0.8661 0.137061 0.712 0.288
#> GSM340297 1 0.9686 -0.257241 0.604 0.396
#> GSM340298 1 0.2778 0.669221 0.952 0.048
#> GSM340299 1 0.3274 0.637078 0.940 0.060
#> GSM340301 1 0.0376 0.676447 0.996 0.004
#> GSM340303 1 0.1633 0.666652 0.976 0.024
#> GSM340304 1 0.2948 0.648003 0.948 0.052
#> GSM340306 2 0.4562 0.437463 0.096 0.904
#> GSM340307 1 0.0000 0.677386 1.000 0.000
#> GSM340310 2 0.9460 0.763424 0.364 0.636
#> GSM340314 1 0.9358 0.573383 0.648 0.352
#> GSM340315 1 0.9833 -0.424669 0.576 0.424
#> GSM340317 2 0.6887 0.324365 0.184 0.816
#> GSM340318 1 0.9522 0.559795 0.628 0.372
#> GSM340319 1 0.9580 0.555236 0.620 0.380
#> GSM340320 2 0.8144 0.205693 0.252 0.748
#> GSM340321 1 0.0000 0.677386 1.000 0.000
#> GSM340322 1 0.9460 0.563628 0.636 0.364
#> GSM340324 2 0.9460 0.763424 0.364 0.636
#> GSM340328 2 0.8499 0.708335 0.276 0.724
#> GSM340330 1 0.8499 0.622380 0.724 0.276
#> GSM340332 1 0.9635 0.550117 0.612 0.388
#> GSM340333 1 0.3274 0.637078 0.940 0.060
#> GSM340336 1 0.9580 0.555236 0.620 0.380
#> GSM340337 1 0.9996 0.473515 0.512 0.488
#> GSM340338 1 0.8327 0.627273 0.736 0.264
#> GSM340339 1 0.9460 0.563628 0.636 0.364
#> GSM340340 2 0.8386 0.094026 0.268 0.732
#> GSM340341 1 0.9286 0.579350 0.656 0.344
#> GSM340343 1 0.9983 0.459373 0.524 0.476
#> GSM340344 1 0.4562 0.597064 0.904 0.096
#> GSM340346 1 0.0000 0.677386 1.000 0.000
#> GSM340347 1 0.9795 0.527648 0.584 0.416
#> GSM340348 2 0.7219 0.296812 0.200 0.800
#> GSM340349 1 0.9881 -0.419099 0.564 0.436
#> GSM340350 1 0.9323 -0.078025 0.652 0.348
#> GSM340351 2 0.9491 0.761968 0.368 0.632
#> GSM340354 1 0.0000 0.677386 1.000 0.000
#> GSM340356 2 0.9491 0.761985 0.368 0.632
#> GSM340357 2 0.9460 0.763424 0.364 0.636
#> GSM348183 2 0.9686 0.743476 0.396 0.604
#> GSM348191 1 0.8499 0.622380 0.724 0.276
#> GSM348193 2 0.2948 0.499213 0.052 0.948
#> GSM537578 1 0.3584 0.628927 0.932 0.068
#> GSM348181 1 0.3879 0.627559 0.924 0.076
#> GSM348182 1 0.5059 0.585437 0.888 0.112
#> GSM348184 1 0.8713 0.614454 0.708 0.292
#> GSM348185 2 0.5737 0.394323 0.136 0.864
#> GSM348186 1 0.2778 0.648386 0.952 0.048
#> GSM348187 2 0.9460 0.763424 0.364 0.636
#> GSM348188 2 0.8608 0.133453 0.284 0.716
#> GSM348189 1 0.7528 0.642306 0.784 0.216
#> GSM348190 1 0.9775 -0.365319 0.588 0.412
#> GSM348194 2 0.9686 0.743476 0.396 0.604
#> GSM348195 2 0.9460 0.763424 0.364 0.636
#> GSM348196 2 0.9491 0.761968 0.368 0.632
#> GSM537585 2 0.9710 0.736271 0.400 0.600
#> GSM537594 2 0.9795 0.717696 0.416 0.584
#> GSM537596 1 0.9866 -0.421626 0.568 0.432
#> GSM537597 1 0.9970 -0.511112 0.532 0.468
#> GSM537602 1 0.9087 0.000927 0.676 0.324
#> GSM340184 1 0.8386 0.625803 0.732 0.268
#> GSM340185 1 0.2778 0.669215 0.952 0.048
#> GSM340186 1 0.6973 0.650358 0.812 0.188
#> GSM340187 1 0.9460 0.563628 0.636 0.364
#> GSM340189 1 0.9460 0.563628 0.636 0.364
#> GSM340190 1 0.8813 0.609298 0.700 0.300
#> GSM340191 1 0.8499 0.622380 0.724 0.276
#> GSM340192 1 0.1414 0.668103 0.980 0.020
#> GSM340193 1 0.9881 -0.430357 0.564 0.436
#> GSM340194 1 0.3274 0.637078 0.940 0.060
#> GSM340195 1 0.4431 0.606052 0.908 0.092
#> GSM340196 1 0.9710 0.544147 0.600 0.400
#> GSM340197 1 0.2948 0.644960 0.948 0.052
#> GSM340198 1 0.0376 0.676447 0.996 0.004
#> GSM340199 1 0.3114 0.677589 0.944 0.056
#> GSM340200 1 0.2236 0.675620 0.964 0.036
#> GSM340201 2 0.7883 0.241315 0.236 0.764
#> GSM340202 1 0.9580 0.555236 0.620 0.380
#> GSM340203 1 0.9460 0.563628 0.636 0.364
#> GSM340204 1 0.5294 0.567887 0.880 0.120
#> GSM340205 2 0.9963 0.630977 0.464 0.536
#> GSM340206 1 0.8443 0.624229 0.728 0.272
#> GSM340207 1 0.0376 0.678475 0.996 0.004
#> GSM340237 2 0.9710 0.735584 0.400 0.600
#> GSM340238 1 0.8555 0.620395 0.720 0.280
#> GSM340239 1 0.1184 0.672035 0.984 0.016
#> GSM340240 1 0.0376 0.676313 0.996 0.004
#> GSM340241 1 0.0000 0.677386 1.000 0.000
#> GSM340242 1 0.1414 0.680378 0.980 0.020
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.0000 0.7859 1.000 0.000 0.000
#> GSM340359 1 0.1411 0.7789 0.964 0.000 0.036
#> GSM340361 1 0.0592 0.7843 0.988 0.012 0.000
#> GSM340362 3 0.3941 0.7368 0.156 0.000 0.844
#> GSM340363 1 0.5667 0.7250 0.800 0.060 0.140
#> GSM340364 1 0.0000 0.7859 1.000 0.000 0.000
#> GSM340365 1 0.3983 0.7494 0.852 0.004 0.144
#> GSM340366 1 0.6008 0.5756 0.664 0.004 0.332
#> GSM340367 1 0.0000 0.7859 1.000 0.000 0.000
#> GSM340368 2 0.2356 0.8589 0.072 0.928 0.000
#> GSM340369 2 0.4842 0.7141 0.224 0.776 0.000
#> GSM340370 1 0.0000 0.7859 1.000 0.000 0.000
#> GSM340371 3 0.0237 0.8650 0.000 0.004 0.996
#> GSM340372 1 0.0000 0.7859 1.000 0.000 0.000
#> GSM340373 1 0.0000 0.7859 1.000 0.000 0.000
#> GSM340375 1 0.0000 0.7859 1.000 0.000 0.000
#> GSM340376 1 0.0000 0.7859 1.000 0.000 0.000
#> GSM340378 1 0.0000 0.7859 1.000 0.000 0.000
#> GSM340243 1 0.1878 0.7874 0.952 0.004 0.044
#> GSM340244 3 0.4094 0.8008 0.100 0.028 0.872
#> GSM340246 3 0.0000 0.8649 0.000 0.000 1.000
#> GSM340247 2 0.0747 0.8898 0.000 0.984 0.016
#> GSM340248 3 0.4346 0.7047 0.184 0.000 0.816
#> GSM340249 2 0.2356 0.8628 0.000 0.928 0.072
#> GSM340250 1 0.5397 0.6182 0.720 0.000 0.280
#> GSM340251 2 0.5363 0.6038 0.000 0.724 0.276
#> GSM340252 2 0.5859 0.4762 0.000 0.656 0.344
#> GSM340253 3 0.0424 0.8651 0.008 0.000 0.992
#> GSM340254 3 0.0000 0.8649 0.000 0.000 1.000
#> GSM340256 3 0.0237 0.8653 0.004 0.000 0.996
#> GSM340258 3 0.0000 0.8649 0.000 0.000 1.000
#> GSM340259 3 0.1529 0.8528 0.040 0.000 0.960
#> GSM340260 3 0.0237 0.8654 0.004 0.000 0.996
#> GSM340261 3 0.2711 0.8207 0.088 0.000 0.912
#> GSM340262 3 0.0237 0.8653 0.004 0.000 0.996
#> GSM340263 3 0.5178 0.6070 0.256 0.000 0.744
#> GSM340264 3 0.0424 0.8651 0.008 0.000 0.992
#> GSM340265 3 0.0237 0.8644 0.000 0.004 0.996
#> GSM340266 3 0.7391 0.5036 0.056 0.308 0.636
#> GSM340267 3 0.3340 0.7978 0.000 0.120 0.880
#> GSM340268 3 0.3816 0.7828 0.000 0.148 0.852
#> GSM340269 3 0.0000 0.8649 0.000 0.000 1.000
#> GSM340270 1 0.5431 0.6051 0.716 0.000 0.284
#> GSM537574 3 0.0237 0.8653 0.004 0.000 0.996
#> GSM537580 3 0.6154 0.3624 0.000 0.408 0.592
#> GSM537581 2 0.5058 0.6955 0.244 0.756 0.000
#> GSM340272 1 0.5480 0.6683 0.732 0.004 0.264
#> GSM340273 1 0.5159 0.7365 0.820 0.040 0.140
#> GSM340275 2 0.0747 0.8898 0.000 0.984 0.016
#> GSM340276 3 0.2383 0.8486 0.044 0.016 0.940
#> GSM340277 3 0.4834 0.6511 0.204 0.004 0.792
#> GSM340278 3 0.6026 0.4318 0.000 0.376 0.624
#> GSM340279 3 0.5968 0.4569 0.000 0.364 0.636
#> GSM340282 3 0.3983 0.7431 0.144 0.004 0.852
#> GSM340284 3 0.5591 0.5752 0.000 0.304 0.696
#> GSM340285 3 0.6286 0.1815 0.000 0.464 0.536
#> GSM340286 3 0.0424 0.8651 0.008 0.000 0.992
#> GSM340287 3 0.2711 0.8207 0.088 0.000 0.912
#> GSM340288 1 0.4261 0.7504 0.848 0.012 0.140
#> GSM340289 1 0.6302 0.1745 0.520 0.000 0.480
#> GSM340290 3 0.7159 0.1848 0.024 0.448 0.528
#> GSM340291 3 0.4291 0.7695 0.152 0.008 0.840
#> GSM340293 2 0.4399 0.7471 0.000 0.812 0.188
#> GSM340294 3 0.1411 0.8523 0.036 0.000 0.964
#> GSM340296 1 0.4629 0.7330 0.808 0.004 0.188
#> GSM340297 1 0.7318 0.5866 0.668 0.068 0.264
#> GSM340298 1 0.6345 0.4212 0.596 0.004 0.400
#> GSM340299 3 0.0424 0.8651 0.008 0.000 0.992
#> GSM340301 3 0.0424 0.8651 0.008 0.000 0.992
#> GSM340303 3 0.0475 0.8643 0.004 0.004 0.992
#> GSM340304 3 0.6386 0.1381 0.412 0.004 0.584
#> GSM340306 2 0.1031 0.8782 0.024 0.976 0.000
#> GSM340307 3 0.0237 0.8644 0.000 0.004 0.996
#> GSM340310 1 0.0000 0.7859 1.000 0.000 0.000
#> GSM340314 2 0.5591 0.5456 0.000 0.696 0.304
#> GSM340315 1 0.6126 0.4916 0.644 0.004 0.352
#> GSM340317 2 0.3771 0.8217 0.112 0.876 0.012
#> GSM340318 2 0.0747 0.8898 0.000 0.984 0.016
#> GSM340319 2 0.0747 0.8898 0.000 0.984 0.016
#> GSM340320 2 0.1411 0.8730 0.036 0.964 0.000
#> GSM340321 3 0.0000 0.8649 0.000 0.000 1.000
#> GSM340322 2 0.0747 0.8898 0.000 0.984 0.016
#> GSM340324 1 0.4842 0.6893 0.776 0.000 0.224
#> GSM340328 1 0.0000 0.7859 1.000 0.000 0.000
#> GSM340330 3 0.0237 0.8653 0.000 0.004 0.996
#> GSM340332 2 0.4654 0.7150 0.000 0.792 0.208
#> GSM340333 3 0.0424 0.8651 0.008 0.000 0.992
#> GSM340336 2 0.0747 0.8898 0.000 0.984 0.016
#> GSM340337 2 0.0237 0.8835 0.000 0.996 0.004
#> GSM340338 3 0.2711 0.8222 0.000 0.088 0.912
#> GSM340339 2 0.0747 0.8898 0.000 0.984 0.016
#> GSM340340 2 0.4110 0.7940 0.152 0.844 0.004
#> GSM340341 2 0.4178 0.7674 0.000 0.828 0.172
#> GSM340343 2 0.0747 0.8898 0.000 0.984 0.016
#> GSM340344 3 0.1015 0.8608 0.008 0.012 0.980
#> GSM340346 3 0.0237 0.8644 0.000 0.004 0.996
#> GSM340347 2 0.0747 0.8898 0.000 0.984 0.016
#> GSM340348 2 0.1765 0.8765 0.004 0.956 0.040
#> GSM340349 1 0.4521 0.7387 0.816 0.004 0.180
#> GSM340350 1 0.4351 0.7390 0.828 0.004 0.168
#> GSM340351 1 0.0829 0.7853 0.984 0.012 0.004
#> GSM340354 3 0.0000 0.8649 0.000 0.000 1.000
#> GSM340356 1 0.6979 0.6627 0.732 0.140 0.128
#> GSM340357 1 0.0000 0.7859 1.000 0.000 0.000
#> GSM348183 1 0.5722 0.7122 0.804 0.112 0.084
#> GSM348191 3 0.2301 0.8437 0.004 0.060 0.936
#> GSM348193 2 0.5397 0.5907 0.280 0.720 0.000
#> GSM537578 1 0.6451 0.3702 0.560 0.004 0.436
#> GSM348181 1 0.6302 0.1745 0.520 0.000 0.480
#> GSM348182 3 0.4805 0.7178 0.176 0.012 0.812
#> GSM348184 3 0.4121 0.7776 0.000 0.168 0.832
#> GSM348185 2 0.3116 0.8226 0.108 0.892 0.000
#> GSM348186 3 0.0000 0.8649 0.000 0.000 1.000
#> GSM348187 1 0.0592 0.7843 0.988 0.012 0.000
#> GSM348188 2 0.0475 0.8841 0.004 0.992 0.004
#> GSM348189 3 0.0237 0.8653 0.004 0.000 0.996
#> GSM348190 3 0.3031 0.8184 0.076 0.012 0.912
#> GSM348194 1 0.2446 0.7818 0.936 0.012 0.052
#> GSM348195 1 0.0592 0.7843 0.988 0.012 0.000
#> GSM348196 1 0.0829 0.7853 0.984 0.012 0.004
#> GSM537585 1 0.5986 0.6576 0.704 0.012 0.284
#> GSM537594 1 0.7759 0.1532 0.480 0.048 0.472
#> GSM537596 1 0.6825 0.1377 0.496 0.012 0.492
#> GSM537597 3 0.6799 0.0098 0.456 0.012 0.532
#> GSM537602 1 0.6062 0.6607 0.708 0.016 0.276
#> GSM340184 3 0.2636 0.8458 0.020 0.048 0.932
#> GSM340185 3 0.2689 0.8394 0.032 0.036 0.932
#> GSM340186 3 0.1411 0.8544 0.000 0.036 0.964
#> GSM340187 2 0.1860 0.8766 0.000 0.948 0.052
#> GSM340189 2 0.0592 0.8883 0.000 0.988 0.012
#> GSM340190 3 0.4291 0.7637 0.000 0.180 0.820
#> GSM340191 3 0.4178 0.7630 0.000 0.172 0.828
#> GSM340192 3 0.3644 0.7671 0.124 0.004 0.872
#> GSM340193 3 0.5171 0.6672 0.204 0.012 0.784
#> GSM340194 3 0.0424 0.8651 0.008 0.000 0.992
#> GSM340195 3 0.0424 0.8651 0.008 0.000 0.992
#> GSM340196 2 0.0747 0.8898 0.000 0.984 0.016
#> GSM340197 3 0.0424 0.8651 0.008 0.000 0.992
#> GSM340198 3 0.0000 0.8649 0.000 0.000 1.000
#> GSM340199 3 0.5968 0.4541 0.000 0.364 0.636
#> GSM340200 3 0.2564 0.8427 0.028 0.036 0.936
#> GSM340201 2 0.0829 0.8884 0.004 0.984 0.012
#> GSM340202 2 0.0747 0.8898 0.000 0.984 0.016
#> GSM340203 2 0.3551 0.8187 0.000 0.868 0.132
#> GSM340204 3 0.0424 0.8651 0.008 0.000 0.992
#> GSM340205 1 0.4796 0.6906 0.780 0.000 0.220
#> GSM340206 3 0.2711 0.8274 0.000 0.088 0.912
#> GSM340207 3 0.0424 0.8644 0.000 0.008 0.992
#> GSM340237 1 0.0983 0.7853 0.980 0.004 0.016
#> GSM340238 3 0.6291 0.1810 0.000 0.468 0.532
#> GSM340239 3 0.0424 0.8651 0.008 0.000 0.992
#> GSM340240 3 0.1399 0.8536 0.028 0.004 0.968
#> GSM340241 3 0.0237 0.8644 0.000 0.004 0.996
#> GSM340242 3 0.0000 0.8649 0.000 0.000 1.000
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.0000 0.8256 1.000 0.000 0.000 0.000
#> GSM340359 1 0.2530 0.7378 0.888 0.112 0.000 0.000
#> GSM340361 1 0.0188 0.8249 0.996 0.004 0.000 0.000
#> GSM340362 2 0.5897 0.6959 0.136 0.700 0.164 0.000
#> GSM340363 1 0.2224 0.8037 0.928 0.032 0.040 0.000
#> GSM340364 1 0.0000 0.8256 1.000 0.000 0.000 0.000
#> GSM340365 1 0.5288 0.5942 0.720 0.224 0.056 0.000
#> GSM340366 1 0.7390 0.4414 0.512 0.284 0.204 0.000
#> GSM340367 1 0.0000 0.8256 1.000 0.000 0.000 0.000
#> GSM340368 4 0.3356 0.7946 0.176 0.000 0.000 0.824
#> GSM340369 4 0.3649 0.7679 0.204 0.000 0.000 0.796
#> GSM340370 1 0.0000 0.8256 1.000 0.000 0.000 0.000
#> GSM340371 3 0.3908 0.6509 0.000 0.212 0.784 0.004
#> GSM340372 1 0.0000 0.8256 1.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.8256 1.000 0.000 0.000 0.000
#> GSM340375 1 0.0000 0.8256 1.000 0.000 0.000 0.000
#> GSM340376 1 0.0000 0.8256 1.000 0.000 0.000 0.000
#> GSM340378 1 0.0000 0.8256 1.000 0.000 0.000 0.000
#> GSM340243 1 0.5939 0.6324 0.668 0.248 0.084 0.000
#> GSM340244 3 0.6452 0.4202 0.112 0.208 0.668 0.012
#> GSM340246 3 0.3610 0.6301 0.000 0.200 0.800 0.000
#> GSM340247 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340248 3 0.3962 0.6449 0.124 0.044 0.832 0.000
#> GSM340249 4 0.1389 0.8832 0.000 0.000 0.048 0.952
#> GSM340250 3 0.5229 0.3207 0.428 0.008 0.564 0.000
#> GSM340251 3 0.5929 0.5243 0.000 0.064 0.640 0.296
#> GSM340252 2 0.5152 0.4873 0.000 0.664 0.020 0.316
#> GSM340253 2 0.4304 0.7314 0.000 0.716 0.284 0.000
#> GSM340254 2 0.4331 0.7305 0.000 0.712 0.288 0.000
#> GSM340256 2 0.4382 0.7291 0.000 0.704 0.296 0.000
#> GSM340258 3 0.3726 0.5622 0.000 0.212 0.788 0.000
#> GSM340259 3 0.5792 -0.2059 0.032 0.416 0.552 0.000
#> GSM340260 3 0.4304 0.3722 0.000 0.284 0.716 0.000
#> GSM340261 3 0.0188 0.7254 0.000 0.004 0.996 0.000
#> GSM340262 3 0.3528 0.5770 0.000 0.192 0.808 0.000
#> GSM340263 3 0.3946 0.6323 0.168 0.020 0.812 0.000
#> GSM340264 3 0.3873 0.5359 0.000 0.228 0.772 0.000
#> GSM340265 2 0.3837 0.4668 0.000 0.776 0.224 0.000
#> GSM340266 3 0.2224 0.7122 0.040 0.000 0.928 0.032
#> GSM340267 3 0.4728 0.6402 0.000 0.216 0.752 0.032
#> GSM340268 3 0.0188 0.7254 0.000 0.004 0.996 0.000
#> GSM340269 3 0.0921 0.7166 0.000 0.028 0.972 0.000
#> GSM340270 3 0.4730 0.4573 0.364 0.000 0.636 0.000
#> GSM537574 3 0.2345 0.6678 0.000 0.100 0.900 0.000
#> GSM537580 3 0.3710 0.6440 0.000 0.004 0.804 0.192
#> GSM537581 4 0.4692 0.7306 0.212 0.032 0.000 0.756
#> GSM340272 2 0.5280 0.4710 0.124 0.752 0.124 0.000
#> GSM340273 1 0.1488 0.8171 0.956 0.032 0.012 0.000
#> GSM340275 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340276 3 0.2654 0.6787 0.108 0.004 0.888 0.000
#> GSM340277 2 0.1022 0.6269 0.032 0.968 0.000 0.000
#> GSM340278 3 0.3494 0.6745 0.000 0.172 0.824 0.004
#> GSM340279 3 0.1488 0.7273 0.000 0.012 0.956 0.032
#> GSM340282 2 0.3991 0.5040 0.020 0.808 0.172 0.000
#> GSM340284 3 0.2480 0.7146 0.000 0.088 0.904 0.008
#> GSM340285 3 0.1545 0.7265 0.000 0.008 0.952 0.040
#> GSM340286 2 0.4855 0.6125 0.000 0.600 0.400 0.000
#> GSM340287 3 0.0000 0.7245 0.000 0.000 1.000 0.000
#> GSM340288 1 0.1936 0.8106 0.940 0.032 0.028 0.000
#> GSM340289 1 0.4830 0.3311 0.608 0.000 0.392 0.000
#> GSM340290 3 0.7037 0.0721 0.000 0.120 0.464 0.416
#> GSM340291 2 0.4401 0.5989 0.112 0.812 0.076 0.000
#> GSM340293 3 0.7147 0.4827 0.000 0.224 0.560 0.216
#> GSM340294 3 0.0188 0.7254 0.000 0.004 0.996 0.000
#> GSM340296 1 0.7868 0.1049 0.372 0.276 0.352 0.000
#> GSM340297 3 0.4277 0.5511 0.280 0.000 0.720 0.000
#> GSM340298 3 0.5393 0.5825 0.044 0.268 0.688 0.000
#> GSM340299 2 0.4134 0.7332 0.000 0.740 0.260 0.000
#> GSM340301 2 0.4477 0.7194 0.000 0.688 0.312 0.000
#> GSM340303 2 0.3486 0.5314 0.000 0.812 0.188 0.000
#> GSM340304 3 0.7359 0.1687 0.312 0.184 0.504 0.000
#> GSM340306 4 0.2224 0.8849 0.040 0.032 0.000 0.928
#> GSM340307 3 0.4304 0.6033 0.000 0.284 0.716 0.000
#> GSM340310 1 0.0000 0.8256 1.000 0.000 0.000 0.000
#> GSM340314 3 0.3306 0.6788 0.000 0.004 0.840 0.156
#> GSM340315 3 0.5431 0.5237 0.300 0.028 0.668 0.004
#> GSM340317 4 0.3160 0.8250 0.120 0.004 0.008 0.868
#> GSM340318 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340319 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340320 4 0.0336 0.9097 0.008 0.000 0.000 0.992
#> GSM340321 3 0.4776 0.2858 0.000 0.376 0.624 0.000
#> GSM340322 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340324 2 0.4643 0.4770 0.344 0.656 0.000 0.000
#> GSM340328 1 0.0000 0.8256 1.000 0.000 0.000 0.000
#> GSM340330 3 0.0336 0.7256 0.000 0.008 0.992 0.000
#> GSM340332 4 0.4500 0.4455 0.000 0.000 0.316 0.684
#> GSM340333 2 0.4304 0.7314 0.000 0.716 0.284 0.000
#> GSM340336 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340337 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340338 3 0.3966 0.6969 0.000 0.072 0.840 0.088
#> GSM340339 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340340 4 0.3448 0.8021 0.168 0.004 0.000 0.828
#> GSM340341 4 0.3402 0.7556 0.000 0.004 0.164 0.832
#> GSM340343 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340344 2 0.4331 0.7317 0.000 0.712 0.288 0.000
#> GSM340346 3 0.4304 0.6033 0.000 0.284 0.716 0.000
#> GSM340347 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340348 4 0.2036 0.8888 0.000 0.032 0.032 0.936
#> GSM340349 1 0.6833 0.5477 0.584 0.272 0.144 0.000
#> GSM340350 1 0.6461 0.5734 0.632 0.240 0.128 0.000
#> GSM340351 1 0.1209 0.8187 0.964 0.032 0.004 0.000
#> GSM340354 3 0.0592 0.7267 0.000 0.016 0.984 0.000
#> GSM340356 2 0.4950 0.3606 0.376 0.620 0.004 0.000
#> GSM340357 1 0.0000 0.8256 1.000 0.000 0.000 0.000
#> GSM348183 1 0.2635 0.7851 0.908 0.004 0.072 0.016
#> GSM348191 3 0.0000 0.7245 0.000 0.000 1.000 0.000
#> GSM348193 4 0.5271 0.5333 0.320 0.024 0.000 0.656
#> GSM537578 1 0.7671 0.3786 0.456 0.300 0.244 0.000
#> GSM348181 3 0.2704 0.6662 0.124 0.000 0.876 0.000
#> GSM348182 2 0.6818 0.5343 0.076 0.504 0.412 0.008
#> GSM348184 3 0.4638 0.6625 0.000 0.180 0.776 0.044
#> GSM348185 4 0.4057 0.7842 0.152 0.032 0.000 0.816
#> GSM348186 2 0.4331 0.7303 0.000 0.712 0.288 0.000
#> GSM348187 1 0.1022 0.8196 0.968 0.032 0.000 0.000
#> GSM348188 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM348189 3 0.4328 0.4971 0.000 0.244 0.748 0.008
#> GSM348190 2 0.5025 0.7216 0.032 0.716 0.252 0.000
#> GSM348194 1 0.1022 0.8196 0.968 0.032 0.000 0.000
#> GSM348195 1 0.1022 0.8196 0.968 0.032 0.000 0.000
#> GSM348196 1 0.1022 0.8196 0.968 0.032 0.000 0.000
#> GSM537585 2 0.6158 0.5202 0.292 0.628 0.080 0.000
#> GSM537594 2 0.6587 0.5465 0.292 0.596 0.112 0.000
#> GSM537596 1 0.5792 0.2898 0.552 0.032 0.416 0.000
#> GSM537597 1 0.5746 0.4301 0.612 0.040 0.348 0.000
#> GSM537602 1 0.5050 0.6716 0.704 0.268 0.028 0.000
#> GSM340184 3 0.0188 0.7254 0.000 0.004 0.996 0.000
#> GSM340185 3 0.5284 0.5340 0.016 0.368 0.616 0.000
#> GSM340186 3 0.5411 0.5699 0.000 0.312 0.656 0.032
#> GSM340187 4 0.1389 0.8861 0.000 0.000 0.048 0.952
#> GSM340189 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340190 3 0.4193 0.6152 0.000 0.268 0.732 0.000
#> GSM340191 3 0.0188 0.7254 0.000 0.004 0.996 0.000
#> GSM340192 2 0.1388 0.6322 0.028 0.960 0.012 0.000
#> GSM340193 2 0.7301 0.4600 0.152 0.452 0.396 0.000
#> GSM340194 2 0.4477 0.7194 0.000 0.688 0.312 0.000
#> GSM340195 2 0.4477 0.7194 0.000 0.688 0.312 0.000
#> GSM340196 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340197 2 0.4898 0.5813 0.000 0.584 0.416 0.000
#> GSM340198 2 0.4331 0.7303 0.000 0.712 0.288 0.000
#> GSM340199 3 0.4313 0.6152 0.004 0.260 0.736 0.000
#> GSM340200 3 0.4744 0.5954 0.012 0.284 0.704 0.000
#> GSM340201 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340202 4 0.0000 0.9121 0.000 0.000 0.000 1.000
#> GSM340203 4 0.2345 0.8386 0.000 0.000 0.100 0.900
#> GSM340204 2 0.4406 0.7261 0.000 0.700 0.300 0.000
#> GSM340205 1 0.3074 0.7152 0.848 0.000 0.152 0.000
#> GSM340206 3 0.0469 0.7265 0.000 0.012 0.988 0.000
#> GSM340207 3 0.4516 0.5035 0.000 0.252 0.736 0.012
#> GSM340237 1 0.4372 0.6634 0.728 0.268 0.004 0.000
#> GSM340238 3 0.1902 0.7211 0.000 0.004 0.932 0.064
#> GSM340239 2 0.4866 0.6032 0.000 0.596 0.404 0.000
#> GSM340240 2 0.1211 0.6354 0.000 0.960 0.040 0.000
#> GSM340241 2 0.3400 0.7102 0.000 0.820 0.180 0.000
#> GSM340242 3 0.1474 0.7119 0.000 0.052 0.948 0.000
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.2127 0.7701 0.892 0.108 0.000 0.000 0.000
#> GSM340359 1 0.2569 0.7255 0.892 0.040 0.000 0.000 0.068
#> GSM340361 1 0.2648 0.7359 0.848 0.152 0.000 0.000 0.000
#> GSM340362 5 0.3885 0.6542 0.176 0.000 0.040 0.000 0.784
#> GSM340363 2 0.2891 0.6170 0.176 0.824 0.000 0.000 0.000
#> GSM340364 1 0.2127 0.7701 0.892 0.108 0.000 0.000 0.000
#> GSM340365 1 0.3943 0.6143 0.796 0.020 0.020 0.000 0.164
#> GSM340366 1 0.7284 0.3686 0.536 0.088 0.160 0.000 0.216
#> GSM340367 1 0.2127 0.7701 0.892 0.108 0.000 0.000 0.000
#> GSM340368 4 0.3438 0.7254 0.172 0.020 0.000 0.808 0.000
#> GSM340369 4 0.4029 0.6539 0.232 0.024 0.000 0.744 0.000
#> GSM340370 1 0.2127 0.7701 0.892 0.108 0.000 0.000 0.000
#> GSM340371 3 0.3691 0.6997 0.040 0.000 0.804 0.000 0.156
#> GSM340372 1 0.2127 0.7701 0.892 0.108 0.000 0.000 0.000
#> GSM340373 1 0.2127 0.7701 0.892 0.108 0.000 0.000 0.000
#> GSM340375 1 0.2127 0.7701 0.892 0.108 0.000 0.000 0.000
#> GSM340376 1 0.2127 0.7701 0.892 0.108 0.000 0.000 0.000
#> GSM340378 1 0.2127 0.7701 0.892 0.108 0.000 0.000 0.000
#> GSM340243 1 0.6561 0.4847 0.616 0.132 0.064 0.000 0.188
#> GSM340244 3 0.5497 0.5084 0.000 0.196 0.664 0.004 0.136
#> GSM340246 3 0.3849 0.6849 0.004 0.052 0.808 0.000 0.136
#> GSM340247 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340248 2 0.4659 0.0341 0.000 0.500 0.488 0.000 0.012
#> GSM340249 4 0.1764 0.8330 0.000 0.008 0.064 0.928 0.000
#> GSM340250 3 0.7081 0.0282 0.364 0.168 0.436 0.000 0.032
#> GSM340251 3 0.5089 0.6245 0.004 0.040 0.716 0.212 0.028
#> GSM340252 5 0.3671 0.6062 0.000 0.000 0.008 0.236 0.756
#> GSM340253 5 0.3003 0.7610 0.000 0.000 0.188 0.000 0.812
#> GSM340254 5 0.3039 0.7597 0.000 0.000 0.192 0.000 0.808
#> GSM340256 5 0.3954 0.7520 0.000 0.036 0.192 0.000 0.772
#> GSM340258 3 0.3196 0.6335 0.000 0.004 0.804 0.000 0.192
#> GSM340259 3 0.4781 -0.0625 0.000 0.020 0.552 0.000 0.428
#> GSM340260 3 0.4435 0.2985 0.016 0.000 0.648 0.000 0.336
#> GSM340261 3 0.0162 0.7364 0.000 0.000 0.996 0.000 0.004
#> GSM340262 3 0.2852 0.6347 0.000 0.000 0.828 0.000 0.172
#> GSM340263 3 0.5201 0.6174 0.088 0.176 0.716 0.000 0.020
#> GSM340264 3 0.3455 0.5904 0.008 0.000 0.784 0.000 0.208
#> GSM340265 5 0.6014 0.4510 0.064 0.088 0.180 0.000 0.668
#> GSM340266 3 0.2075 0.7304 0.000 0.032 0.924 0.040 0.004
#> GSM340267 3 0.4947 0.6568 0.012 0.068 0.756 0.016 0.148
#> GSM340268 3 0.0290 0.7370 0.000 0.000 0.992 0.000 0.008
#> GSM340269 3 0.1121 0.7282 0.000 0.000 0.956 0.000 0.044
#> GSM340270 3 0.5840 0.3603 0.296 0.112 0.588 0.000 0.004
#> GSM537574 3 0.2722 0.6853 0.000 0.020 0.872 0.000 0.108
#> GSM537580 3 0.3590 0.6865 0.036 0.000 0.828 0.128 0.008
#> GSM537581 2 0.5840 0.1618 0.096 0.488 0.000 0.416 0.000
#> GSM340272 5 0.5799 0.4738 0.132 0.044 0.136 0.000 0.688
#> GSM340273 2 0.1851 0.6668 0.088 0.912 0.000 0.000 0.000
#> GSM340275 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340276 2 0.4420 0.1797 0.000 0.548 0.448 0.000 0.004
#> GSM340277 5 0.1830 0.6603 0.028 0.040 0.000 0.000 0.932
#> GSM340278 3 0.3536 0.6851 0.008 0.016 0.824 0.004 0.148
#> GSM340279 3 0.2890 0.6898 0.000 0.160 0.836 0.000 0.004
#> GSM340282 5 0.6012 0.4618 0.076 0.088 0.160 0.000 0.676
#> GSM340284 3 0.2036 0.7343 0.000 0.036 0.928 0.008 0.028
#> GSM340285 3 0.3962 0.5568 0.000 0.240 0.744 0.012 0.004
#> GSM340286 5 0.3949 0.6319 0.000 0.000 0.332 0.000 0.668
#> GSM340287 3 0.2763 0.6860 0.000 0.148 0.848 0.000 0.004
#> GSM340288 2 0.1965 0.6649 0.096 0.904 0.000 0.000 0.000
#> GSM340289 1 0.5234 0.0604 0.496 0.044 0.460 0.000 0.000
#> GSM340290 3 0.7570 0.2001 0.032 0.036 0.456 0.348 0.128
#> GSM340291 5 0.6001 0.4686 0.052 0.224 0.076 0.000 0.648
#> GSM340293 3 0.7428 0.5005 0.024 0.068 0.568 0.180 0.160
#> GSM340294 3 0.0324 0.7373 0.000 0.004 0.992 0.000 0.004
#> GSM340296 1 0.7858 0.1198 0.404 0.088 0.308 0.000 0.200
#> GSM340297 3 0.4732 0.5570 0.208 0.076 0.716 0.000 0.000
#> GSM340298 3 0.7024 0.4690 0.140 0.088 0.572 0.000 0.200
#> GSM340299 5 0.2970 0.7642 0.004 0.000 0.168 0.000 0.828
#> GSM340301 5 0.3242 0.7518 0.000 0.000 0.216 0.000 0.784
#> GSM340303 5 0.5858 0.5043 0.072 0.092 0.144 0.000 0.692
#> GSM340304 2 0.6550 0.4790 0.064 0.620 0.144 0.000 0.172
#> GSM340306 2 0.3796 0.4821 0.000 0.700 0.000 0.300 0.000
#> GSM340307 3 0.6413 0.5331 0.072 0.092 0.624 0.000 0.212
#> GSM340310 1 0.2127 0.7701 0.892 0.108 0.000 0.000 0.000
#> GSM340314 3 0.2233 0.7197 0.000 0.004 0.892 0.104 0.000
#> GSM340315 2 0.3496 0.6057 0.012 0.788 0.200 0.000 0.000
#> GSM340317 4 0.4432 0.5343 0.008 0.268 0.008 0.708 0.008
#> GSM340318 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340319 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340320 4 0.4307 -0.1042 0.000 0.500 0.000 0.500 0.000
#> GSM340321 3 0.6203 0.2941 0.004 0.152 0.548 0.000 0.296
#> GSM340322 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340324 5 0.4169 0.5609 0.240 0.028 0.000 0.000 0.732
#> GSM340328 1 0.2127 0.7701 0.892 0.108 0.000 0.000 0.000
#> GSM340330 3 0.0404 0.7373 0.000 0.000 0.988 0.000 0.012
#> GSM340332 4 0.4114 0.2809 0.000 0.000 0.376 0.624 0.000
#> GSM340333 5 0.2966 0.7625 0.000 0.000 0.184 0.000 0.816
#> GSM340336 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340337 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340338 3 0.2675 0.7344 0.004 0.020 0.904 0.040 0.032
#> GSM340339 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340340 4 0.3086 0.7361 0.180 0.004 0.000 0.816 0.000
#> GSM340341 2 0.5751 0.2277 0.000 0.516 0.076 0.404 0.004
#> GSM340343 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340344 5 0.3906 0.7433 0.000 0.068 0.132 0.000 0.800
#> GSM340346 3 0.6337 0.5347 0.072 0.088 0.632 0.000 0.208
#> GSM340347 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340348 2 0.3966 0.4256 0.000 0.664 0.000 0.336 0.000
#> GSM340349 2 0.7938 0.2052 0.228 0.452 0.132 0.000 0.188
#> GSM340350 1 0.5926 0.4730 0.664 0.032 0.140 0.000 0.164
#> GSM340351 2 0.3932 0.3748 0.328 0.672 0.000 0.000 0.000
#> GSM340354 3 0.0613 0.7384 0.004 0.004 0.984 0.000 0.008
#> GSM340356 2 0.2208 0.6697 0.020 0.908 0.000 0.000 0.072
#> GSM340357 2 0.4161 0.2987 0.392 0.608 0.000 0.000 0.000
#> GSM348183 1 0.3837 0.7183 0.808 0.156 0.008 0.008 0.020
#> GSM348191 3 0.0162 0.7364 0.000 0.000 0.996 0.000 0.004
#> GSM348193 2 0.3691 0.6438 0.104 0.820 0.000 0.076 0.000
#> GSM537578 2 0.7561 0.3043 0.172 0.520 0.136 0.000 0.172
#> GSM348181 3 0.3497 0.6824 0.108 0.044 0.840 0.000 0.008
#> GSM348182 5 0.5291 0.5168 0.024 0.012 0.388 0.004 0.572
#> GSM348184 3 0.3534 0.7089 0.004 0.040 0.848 0.012 0.096
#> GSM348185 2 0.1851 0.6683 0.000 0.912 0.000 0.088 0.000
#> GSM348186 5 0.2966 0.7626 0.000 0.000 0.184 0.000 0.816
#> GSM348187 2 0.1965 0.6649 0.096 0.904 0.000 0.000 0.000
#> GSM348188 4 0.0162 0.8895 0.000 0.004 0.000 0.996 0.000
#> GSM348189 3 0.3883 0.6081 0.036 0.000 0.780 0.000 0.184
#> GSM348190 5 0.3958 0.6411 0.000 0.184 0.040 0.000 0.776
#> GSM348194 2 0.1965 0.6649 0.096 0.904 0.000 0.000 0.000
#> GSM348195 1 0.4015 0.4870 0.652 0.348 0.000 0.000 0.000
#> GSM348196 2 0.1965 0.6649 0.096 0.904 0.000 0.000 0.000
#> GSM537585 2 0.5864 0.2771 0.092 0.536 0.004 0.000 0.368
#> GSM537594 2 0.5444 0.5778 0.048 0.700 0.056 0.000 0.196
#> GSM537596 2 0.2484 0.6720 0.028 0.900 0.068 0.000 0.004
#> GSM537597 2 0.1907 0.6764 0.028 0.928 0.044 0.000 0.000
#> GSM537602 2 0.5327 0.5380 0.120 0.712 0.020 0.000 0.148
#> GSM340184 3 0.0740 0.7379 0.008 0.004 0.980 0.000 0.008
#> GSM340185 3 0.6563 0.5170 0.072 0.092 0.600 0.000 0.236
#> GSM340186 3 0.4085 0.6765 0.004 0.036 0.784 0.004 0.172
#> GSM340187 4 0.1197 0.8557 0.000 0.000 0.048 0.952 0.000
#> GSM340189 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340190 3 0.5229 0.6092 0.028 0.068 0.712 0.000 0.192
#> GSM340191 3 0.0451 0.7374 0.000 0.004 0.988 0.000 0.008
#> GSM340192 5 0.3479 0.6113 0.080 0.084 0.000 0.000 0.836
#> GSM340193 2 0.3387 0.6492 0.004 0.836 0.128 0.000 0.032
#> GSM340194 5 0.3210 0.7535 0.000 0.000 0.212 0.000 0.788
#> GSM340195 5 0.3242 0.7534 0.000 0.000 0.216 0.000 0.784
#> GSM340196 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340197 5 0.4114 0.5560 0.000 0.000 0.376 0.000 0.624
#> GSM340198 5 0.3003 0.7610 0.000 0.000 0.188 0.000 0.812
#> GSM340199 3 0.6251 0.5400 0.072 0.088 0.644 0.000 0.196
#> GSM340200 3 0.6413 0.5331 0.072 0.092 0.624 0.000 0.212
#> GSM340201 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340202 4 0.0000 0.8919 0.000 0.000 0.000 1.000 0.000
#> GSM340203 4 0.1410 0.8436 0.000 0.000 0.060 0.940 0.000
#> GSM340204 5 0.3003 0.7625 0.000 0.000 0.188 0.000 0.812
#> GSM340205 1 0.4410 0.6577 0.764 0.112 0.124 0.000 0.000
#> GSM340206 3 0.0324 0.7373 0.000 0.004 0.992 0.000 0.004
#> GSM340207 3 0.3424 0.5740 0.000 0.000 0.760 0.000 0.240
#> GSM340237 1 0.5998 0.4462 0.604 0.192 0.004 0.000 0.200
#> GSM340238 3 0.1168 0.7382 0.000 0.000 0.960 0.032 0.008
#> GSM340239 5 0.3999 0.5987 0.000 0.000 0.344 0.000 0.656
#> GSM340240 5 0.3574 0.6109 0.072 0.088 0.004 0.000 0.836
#> GSM340241 5 0.2127 0.7476 0.000 0.000 0.108 0.000 0.892
#> GSM340242 3 0.2291 0.7164 0.036 0.000 0.908 0.000 0.056
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.0000 0.8675 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340359 1 0.0000 0.8675 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340361 1 0.1327 0.8234 0.936 0.000 0.000 0.000 0.000 0.064
#> GSM340362 2 0.1863 0.7865 0.104 0.896 0.000 0.000 0.000 0.000
#> GSM340363 6 0.1858 0.7520 0.092 0.000 0.004 0.000 0.000 0.904
#> GSM340364 1 0.0000 0.8675 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340365 1 0.5071 0.1675 0.540 0.084 0.000 0.000 0.376 0.000
#> GSM340366 5 0.3650 0.7785 0.092 0.000 0.116 0.000 0.792 0.000
#> GSM340367 1 0.0000 0.8675 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340368 4 0.2996 0.6984 0.228 0.000 0.000 0.772 0.000 0.000
#> GSM340369 4 0.2996 0.6987 0.228 0.000 0.000 0.772 0.000 0.000
#> GSM340370 1 0.0000 0.8675 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340371 3 0.4220 0.7343 0.000 0.096 0.732 0.000 0.172 0.000
#> GSM340372 1 0.0000 0.8675 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.8675 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340375 1 0.0000 0.8675 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340376 1 0.0000 0.8675 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340378 1 0.0000 0.8675 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340243 5 0.3655 0.7252 0.136 0.000 0.076 0.000 0.788 0.000
#> GSM340244 3 0.5907 0.6525 0.000 0.084 0.640 0.004 0.136 0.136
#> GSM340246 3 0.4297 0.7306 0.000 0.100 0.724 0.000 0.176 0.000
#> GSM340247 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340248 3 0.5590 0.1580 0.000 0.032 0.484 0.000 0.064 0.420
#> GSM340249 4 0.2164 0.8418 0.000 0.000 0.032 0.900 0.068 0.000
#> GSM340250 1 0.6118 0.1323 0.480 0.048 0.372 0.000 0.000 0.100
#> GSM340251 3 0.4447 0.6531 0.000 0.012 0.736 0.148 0.104 0.000
#> GSM340252 2 0.1204 0.8148 0.000 0.944 0.000 0.056 0.000 0.000
#> GSM340253 2 0.0146 0.8418 0.000 0.996 0.004 0.000 0.000 0.000
#> GSM340254 2 0.0547 0.8417 0.000 0.980 0.020 0.000 0.000 0.000
#> GSM340256 2 0.0713 0.8419 0.000 0.972 0.028 0.000 0.000 0.000
#> GSM340258 3 0.3037 0.7231 0.000 0.176 0.808 0.000 0.016 0.000
#> GSM340259 3 0.4687 0.5377 0.000 0.296 0.632 0.000 0.072 0.000
#> GSM340260 3 0.4507 0.5650 0.004 0.284 0.660 0.000 0.052 0.000
#> GSM340261 3 0.1176 0.7604 0.000 0.024 0.956 0.000 0.020 0.000
#> GSM340262 3 0.2823 0.7011 0.000 0.204 0.796 0.000 0.000 0.000
#> GSM340263 3 0.7499 0.3559 0.248 0.032 0.460 0.000 0.132 0.128
#> GSM340264 3 0.3076 0.6657 0.000 0.240 0.760 0.000 0.000 0.000
#> GSM340265 5 0.4990 0.6987 0.000 0.204 0.152 0.000 0.644 0.000
#> GSM340266 3 0.2842 0.7636 0.000 0.028 0.880 0.040 0.048 0.004
#> GSM340267 3 0.4073 0.5795 0.000 0.012 0.724 0.020 0.240 0.004
#> GSM340268 3 0.0547 0.7635 0.000 0.020 0.980 0.000 0.000 0.000
#> GSM340269 3 0.1501 0.7687 0.000 0.076 0.924 0.000 0.000 0.000
#> GSM340270 3 0.5364 0.3211 0.360 0.004 0.532 0.000 0.104 0.000
#> GSM537574 3 0.3023 0.7423 0.000 0.120 0.836 0.000 0.044 0.000
#> GSM537580 3 0.3904 0.7494 0.000 0.044 0.792 0.032 0.132 0.000
#> GSM537581 6 0.5077 0.3466 0.088 0.000 0.000 0.360 0.000 0.552
#> GSM340272 5 0.5915 0.6344 0.036 0.264 0.132 0.000 0.568 0.000
#> GSM340273 6 0.0000 0.7881 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM340275 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340276 6 0.4343 0.2062 0.000 0.028 0.380 0.000 0.000 0.592
#> GSM340277 2 0.3547 0.4834 0.000 0.696 0.004 0.000 0.300 0.000
#> GSM340278 3 0.2362 0.6827 0.000 0.004 0.860 0.000 0.136 0.000
#> GSM340279 3 0.3358 0.7085 0.000 0.024 0.824 0.000 0.024 0.128
#> GSM340282 5 0.3508 0.7943 0.000 0.068 0.132 0.000 0.800 0.000
#> GSM340284 3 0.1151 0.7634 0.000 0.012 0.956 0.000 0.032 0.000
#> GSM340285 3 0.5296 0.5611 0.000 0.028 0.680 0.008 0.112 0.172
#> GSM340286 2 0.2664 0.7132 0.000 0.816 0.184 0.000 0.000 0.000
#> GSM340287 3 0.3318 0.7120 0.000 0.032 0.796 0.000 0.000 0.172
#> GSM340288 6 0.0000 0.7881 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM340289 1 0.5080 0.1221 0.544 0.016 0.392 0.000 0.048 0.000
#> GSM340290 3 0.6636 -0.1703 0.000 0.028 0.372 0.320 0.280 0.000
#> GSM340291 2 0.7023 0.0835 0.060 0.416 0.024 0.000 0.368 0.132
#> GSM340293 3 0.5784 -0.0667 0.000 0.000 0.460 0.184 0.356 0.000
#> GSM340294 3 0.1713 0.7568 0.000 0.028 0.928 0.000 0.044 0.000
#> GSM340296 5 0.2706 0.7962 0.008 0.000 0.160 0.000 0.832 0.000
#> GSM340297 3 0.4112 0.5881 0.224 0.000 0.724 0.000 0.048 0.004
#> GSM340298 5 0.2631 0.7915 0.000 0.000 0.180 0.000 0.820 0.000
#> GSM340299 2 0.0806 0.8352 0.000 0.972 0.008 0.000 0.020 0.000
#> GSM340301 2 0.0865 0.8381 0.000 0.964 0.036 0.000 0.000 0.000
#> GSM340303 5 0.4810 0.6860 0.000 0.220 0.120 0.000 0.660 0.000
#> GSM340304 6 0.4455 0.2026 0.000 0.008 0.020 0.000 0.388 0.584
#> GSM340306 6 0.2527 0.7074 0.000 0.000 0.000 0.168 0.000 0.832
#> GSM340307 5 0.2340 0.7947 0.000 0.000 0.148 0.000 0.852 0.000
#> GSM340310 1 0.0000 0.8675 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340314 3 0.1053 0.7610 0.000 0.004 0.964 0.020 0.012 0.000
#> GSM340315 6 0.2730 0.6858 0.012 0.000 0.152 0.000 0.000 0.836
#> GSM340317 4 0.5423 0.5206 0.024 0.000 0.012 0.664 0.108 0.192
#> GSM340318 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340319 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340320 6 0.3789 0.3352 0.000 0.000 0.000 0.416 0.000 0.584
#> GSM340321 3 0.6908 0.4672 0.000 0.224 0.492 0.000 0.152 0.132
#> GSM340322 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340324 2 0.3076 0.6537 0.240 0.760 0.000 0.000 0.000 0.000
#> GSM340328 1 0.0000 0.8675 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340330 3 0.1088 0.7627 0.000 0.024 0.960 0.000 0.016 0.000
#> GSM340332 4 0.3737 0.2226 0.000 0.000 0.392 0.608 0.000 0.000
#> GSM340333 2 0.0458 0.8415 0.000 0.984 0.016 0.000 0.000 0.000
#> GSM340336 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340337 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340338 3 0.2052 0.7579 0.000 0.028 0.912 0.004 0.056 0.000
#> GSM340339 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340340 4 0.3023 0.6927 0.232 0.000 0.000 0.768 0.000 0.000
#> GSM340341 6 0.4680 0.5280 0.000 0.028 0.028 0.292 0.000 0.652
#> GSM340343 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340344 2 0.0632 0.8407 0.000 0.976 0.024 0.000 0.000 0.000
#> GSM340346 5 0.2941 0.7674 0.000 0.000 0.220 0.000 0.780 0.000
#> GSM340347 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340348 6 0.2416 0.7180 0.000 0.000 0.000 0.156 0.000 0.844
#> GSM340349 5 0.4059 0.7280 0.000 0.000 0.100 0.000 0.752 0.148
#> GSM340350 5 0.4354 0.7368 0.144 0.000 0.132 0.000 0.724 0.000
#> GSM340351 6 0.3409 0.4947 0.300 0.000 0.000 0.000 0.000 0.700
#> GSM340354 3 0.2309 0.7529 0.000 0.028 0.888 0.000 0.084 0.000
#> GSM340356 6 0.0000 0.7881 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM340357 6 0.3371 0.5472 0.292 0.000 0.000 0.000 0.000 0.708
#> GSM348183 1 0.2279 0.8142 0.900 0.004 0.000 0.000 0.048 0.048
#> GSM348191 3 0.0713 0.7614 0.000 0.028 0.972 0.000 0.000 0.000
#> GSM348193 6 0.3065 0.7370 0.088 0.000 0.000 0.012 0.048 0.852
#> GSM537578 5 0.5472 0.4122 0.000 0.000 0.132 0.000 0.504 0.364
#> GSM348181 3 0.3565 0.7292 0.096 0.004 0.808 0.000 0.092 0.000
#> GSM348182 2 0.5404 0.4702 0.004 0.588 0.284 0.004 0.120 0.000
#> GSM348184 3 0.2218 0.7419 0.000 0.012 0.884 0.000 0.104 0.000
#> GSM348185 6 0.0000 0.7881 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM348186 2 0.0146 0.8418 0.000 0.996 0.004 0.000 0.000 0.000
#> GSM348187 6 0.0000 0.7881 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM348188 4 0.0713 0.8938 0.000 0.028 0.000 0.972 0.000 0.000
#> GSM348189 3 0.5314 0.3756 0.000 0.336 0.544 0.000 0.120 0.000
#> GSM348190 2 0.3349 0.6429 0.000 0.748 0.008 0.000 0.000 0.244
#> GSM348194 6 0.0000 0.7881 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM348195 1 0.3446 0.4893 0.692 0.000 0.000 0.000 0.000 0.308
#> GSM348196 6 0.0000 0.7881 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM537585 6 0.3629 0.5480 0.016 0.260 0.000 0.000 0.000 0.724
#> GSM537594 6 0.7139 0.3102 0.104 0.292 0.024 0.000 0.108 0.472
#> GSM537596 6 0.0000 0.7881 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM537597 6 0.0000 0.7881 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM537602 5 0.3979 0.1906 0.004 0.000 0.000 0.000 0.540 0.456
#> GSM340184 3 0.1549 0.7681 0.000 0.020 0.936 0.000 0.044 0.000
#> GSM340185 5 0.3037 0.7792 0.000 0.016 0.176 0.000 0.808 0.000
#> GSM340186 3 0.3928 0.7319 0.000 0.160 0.760 0.000 0.080 0.000
#> GSM340187 4 0.1267 0.8633 0.000 0.000 0.060 0.940 0.000 0.000
#> GSM340189 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340190 3 0.3898 0.3928 0.000 0.012 0.652 0.000 0.336 0.000
#> GSM340191 3 0.1461 0.7678 0.000 0.016 0.940 0.000 0.044 0.000
#> GSM340192 5 0.3727 0.3801 0.000 0.388 0.000 0.000 0.612 0.000
#> GSM340193 6 0.1421 0.7685 0.000 0.028 0.028 0.000 0.000 0.944
#> GSM340194 2 0.0363 0.8425 0.000 0.988 0.012 0.000 0.000 0.000
#> GSM340195 2 0.0363 0.8425 0.000 0.988 0.012 0.000 0.000 0.000
#> GSM340196 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340197 2 0.3409 0.5390 0.000 0.700 0.300 0.000 0.000 0.000
#> GSM340198 2 0.0547 0.8362 0.000 0.980 0.020 0.000 0.000 0.000
#> GSM340199 5 0.3023 0.7626 0.000 0.000 0.232 0.000 0.768 0.000
#> GSM340200 5 0.2300 0.7951 0.000 0.000 0.144 0.000 0.856 0.000
#> GSM340201 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340202 4 0.0000 0.9096 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340203 4 0.1320 0.8764 0.000 0.016 0.036 0.948 0.000 0.000
#> GSM340204 2 0.0713 0.8402 0.000 0.972 0.028 0.000 0.000 0.000
#> GSM340205 1 0.3563 0.7177 0.800 0.000 0.092 0.000 0.108 0.000
#> GSM340206 3 0.0725 0.7609 0.000 0.012 0.976 0.000 0.012 0.000
#> GSM340207 3 0.3566 0.6886 0.000 0.224 0.752 0.000 0.024 0.000
#> GSM340237 5 0.4143 0.5746 0.196 0.000 0.008 0.000 0.740 0.056
#> GSM340238 3 0.0692 0.7640 0.000 0.020 0.976 0.004 0.000 0.000
#> GSM340239 2 0.3409 0.5379 0.000 0.700 0.300 0.000 0.000 0.000
#> GSM340240 5 0.3508 0.5592 0.000 0.292 0.004 0.000 0.704 0.000
#> GSM340241 2 0.1418 0.8251 0.000 0.944 0.024 0.000 0.032 0.000
#> GSM340242 3 0.3595 0.7459 0.000 0.084 0.796 0.000 0.120 0.000
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
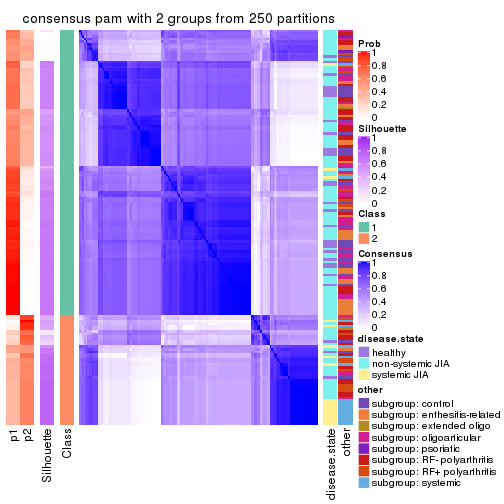
consensus_heatmap(res, k = 3)
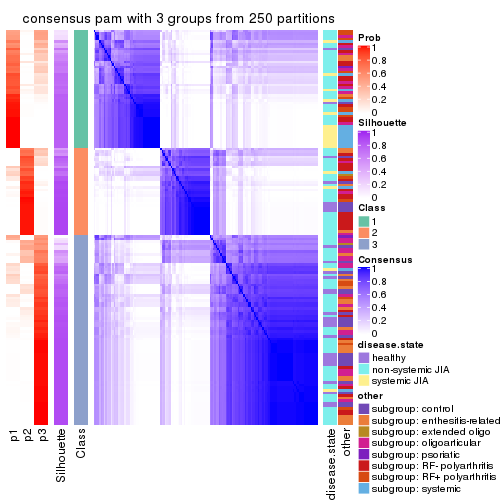
consensus_heatmap(res, k = 4)
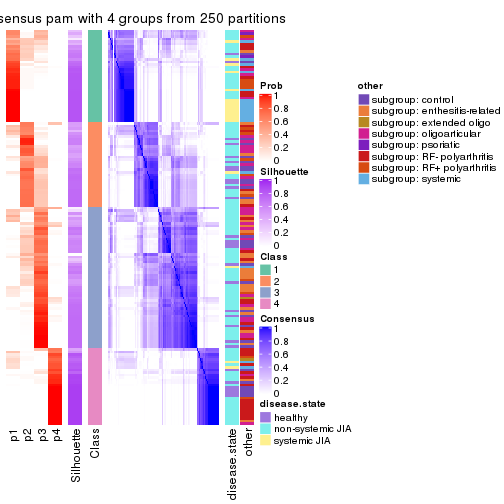
consensus_heatmap(res, k = 5)
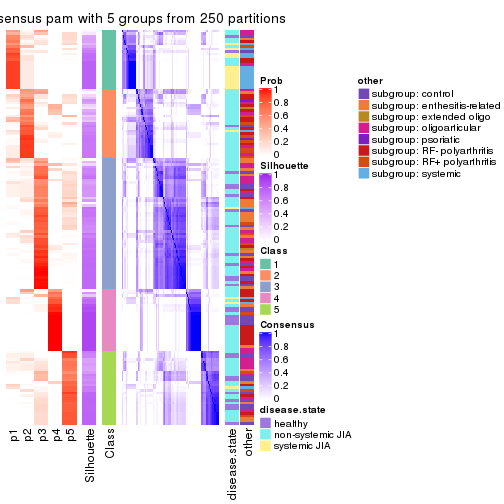
consensus_heatmap(res, k = 6)
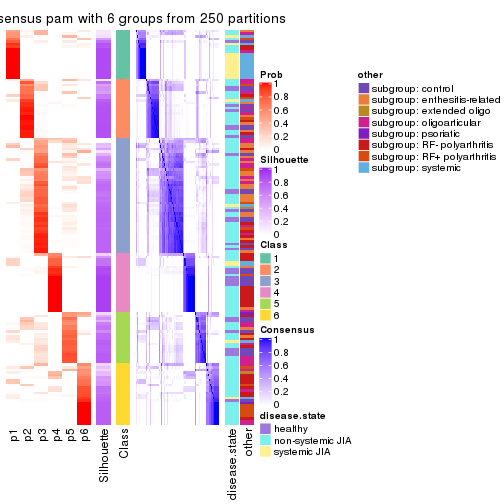
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
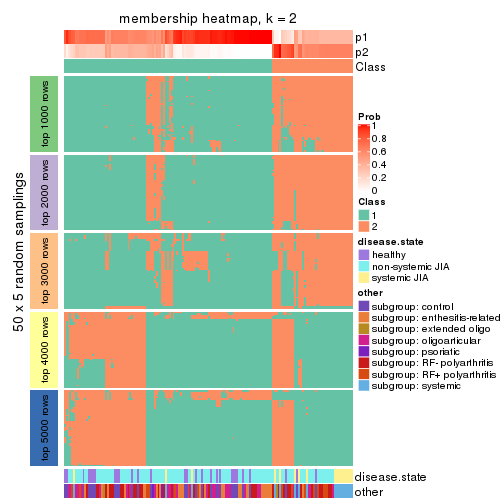
membership_heatmap(res, k = 3)
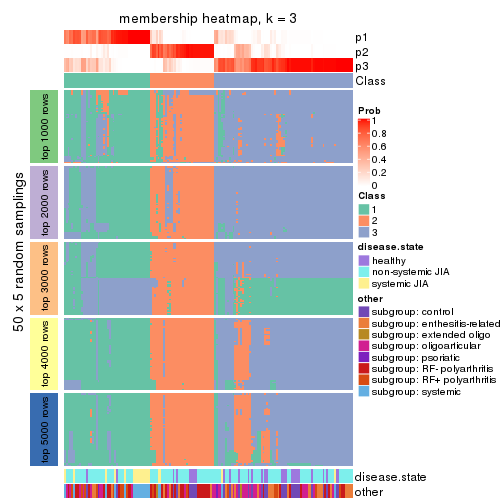
membership_heatmap(res, k = 4)
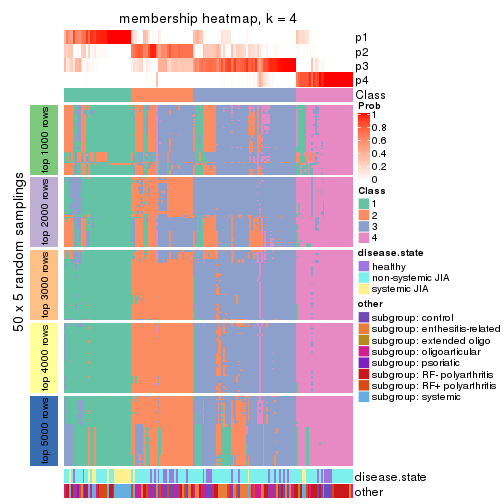
membership_heatmap(res, k = 5)
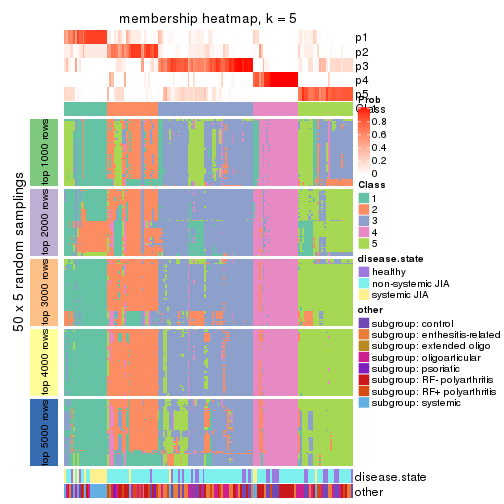
membership_heatmap(res, k = 6)
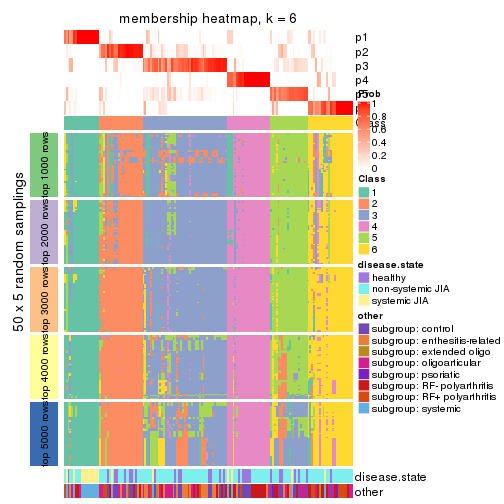
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
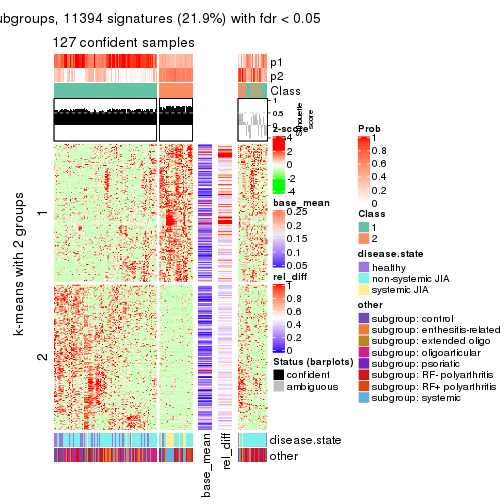
get_signatures(res, k = 3)
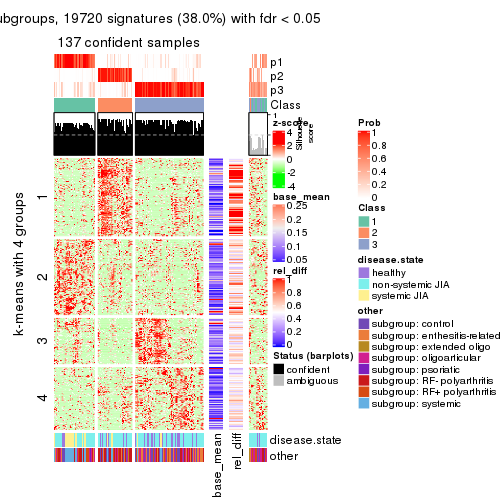
get_signatures(res, k = 4)
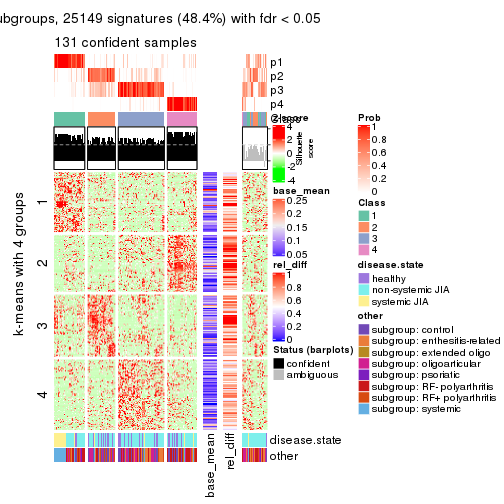
get_signatures(res, k = 5)
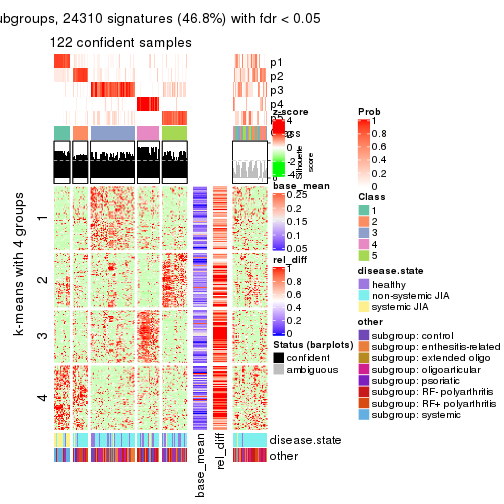
get_signatures(res, k = 6)
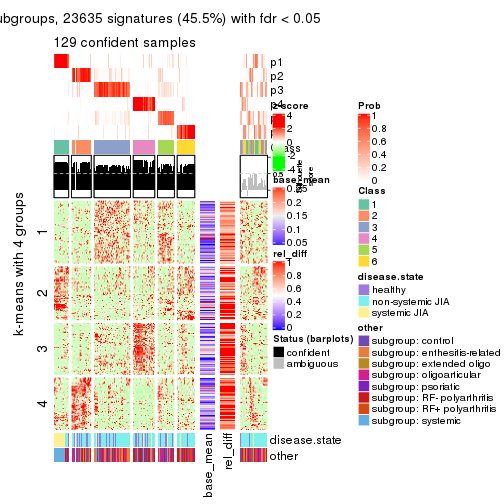
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
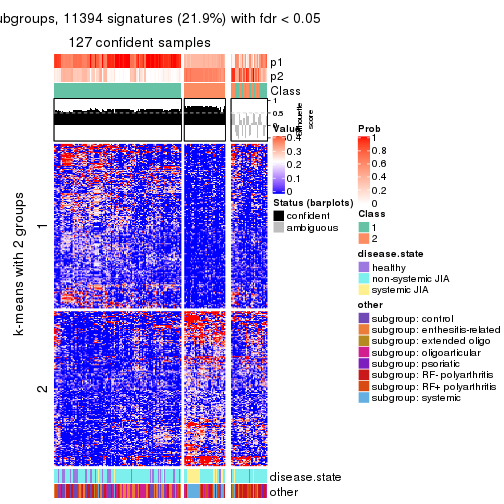
get_signatures(res, k = 3, scale_rows = FALSE)
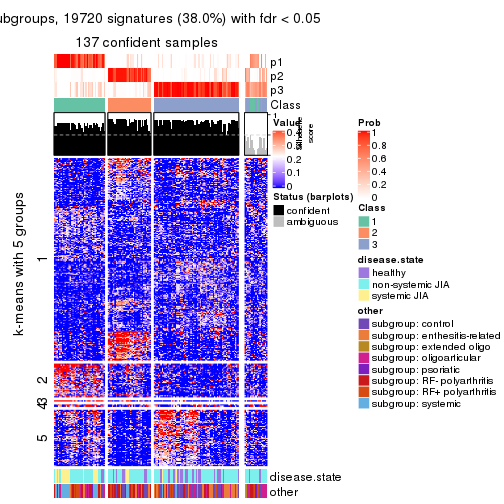
get_signatures(res, k = 4, scale_rows = FALSE)
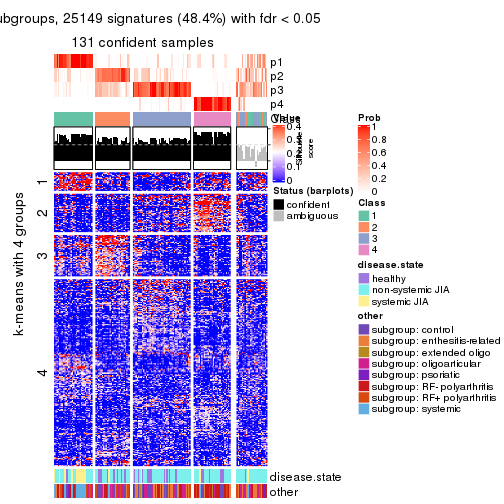
get_signatures(res, k = 5, scale_rows = FALSE)
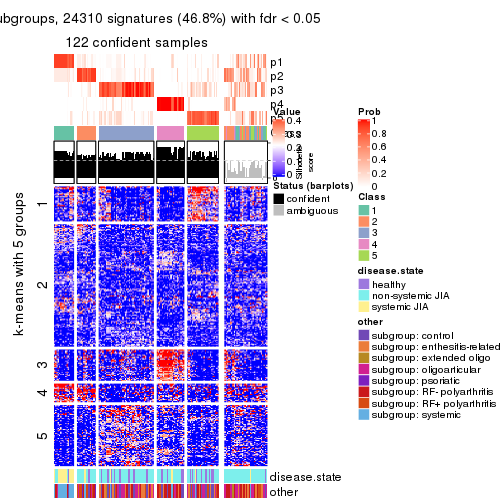
get_signatures(res, k = 6, scale_rows = FALSE)
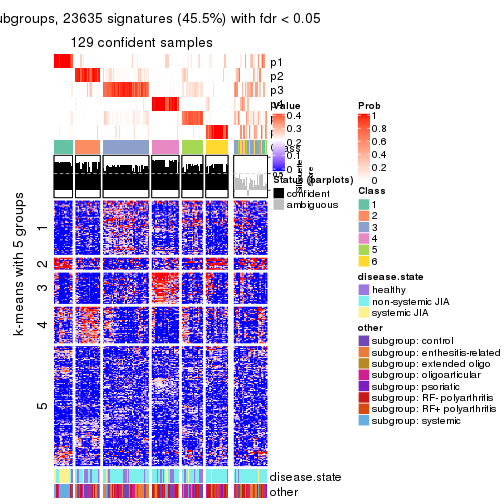
Compare the overlap of signatures from different k:
compare_signatures(res)
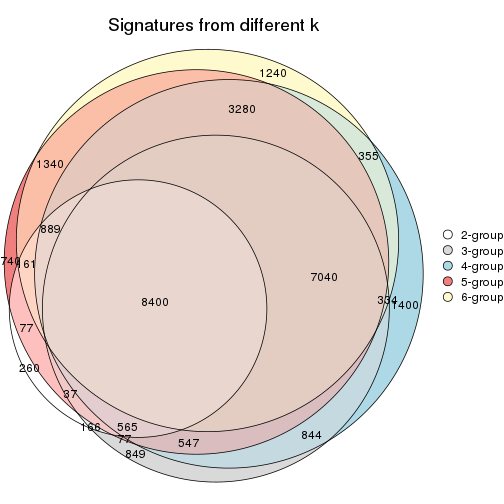
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
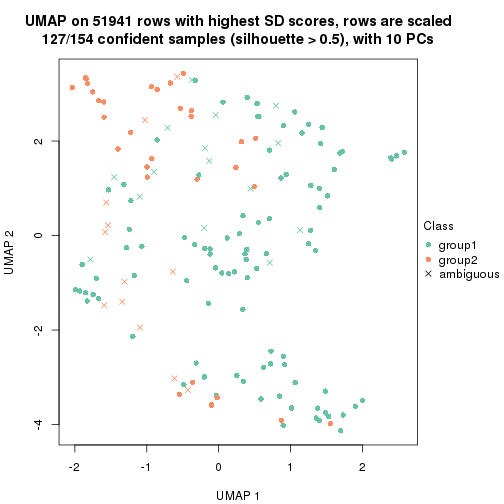
dimension_reduction(res, k = 3, method = "UMAP")
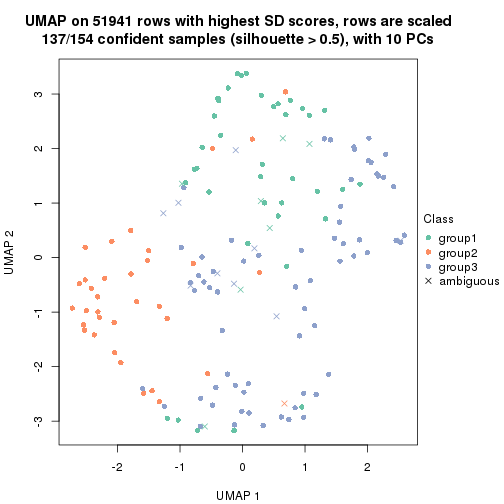
dimension_reduction(res, k = 4, method = "UMAP")
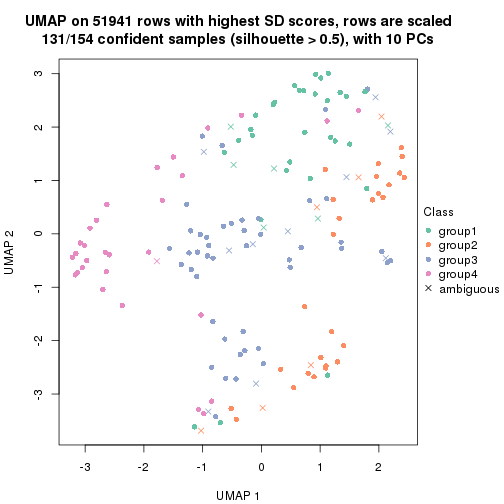
dimension_reduction(res, k = 5, method = "UMAP")
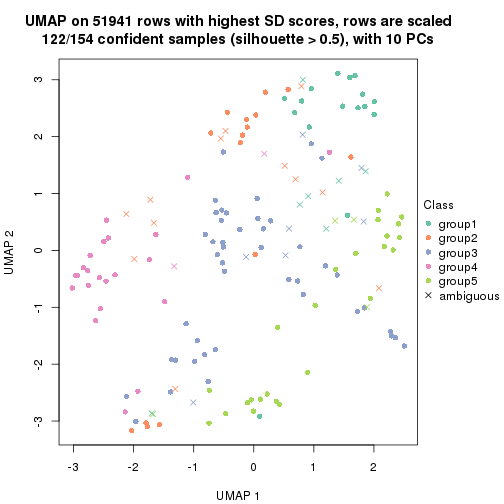
dimension_reduction(res, k = 6, method = "UMAP")
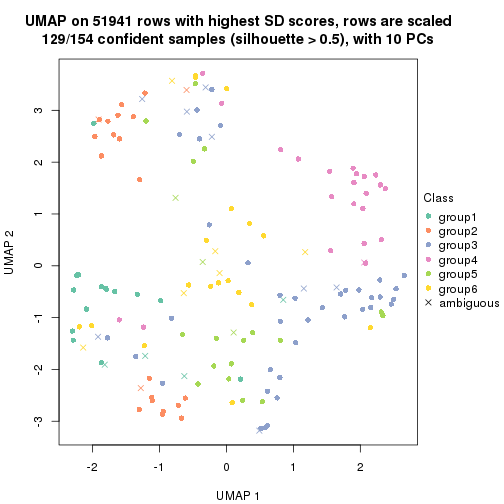
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
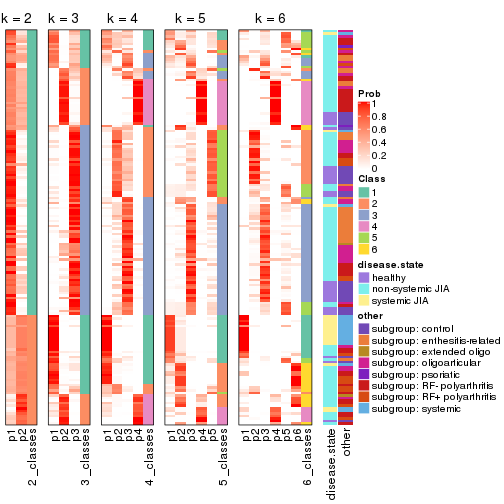
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> SD:pam 127 5.17e-08 4.34e-08 2
#> SD:pam 137 5.81e-06 3.16e-07 3
#> SD:pam 131 4.86e-06 4.06e-07 4
#> SD:pam 122 1.43e-10 7.63e-12 5
#> SD:pam 129 2.62e-09 1.01e-09 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["SD", "mclust"]
# you can also extract it by
# res = res_list["SD:mclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'SD' method.
#> Subgroups are detected by 'mclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
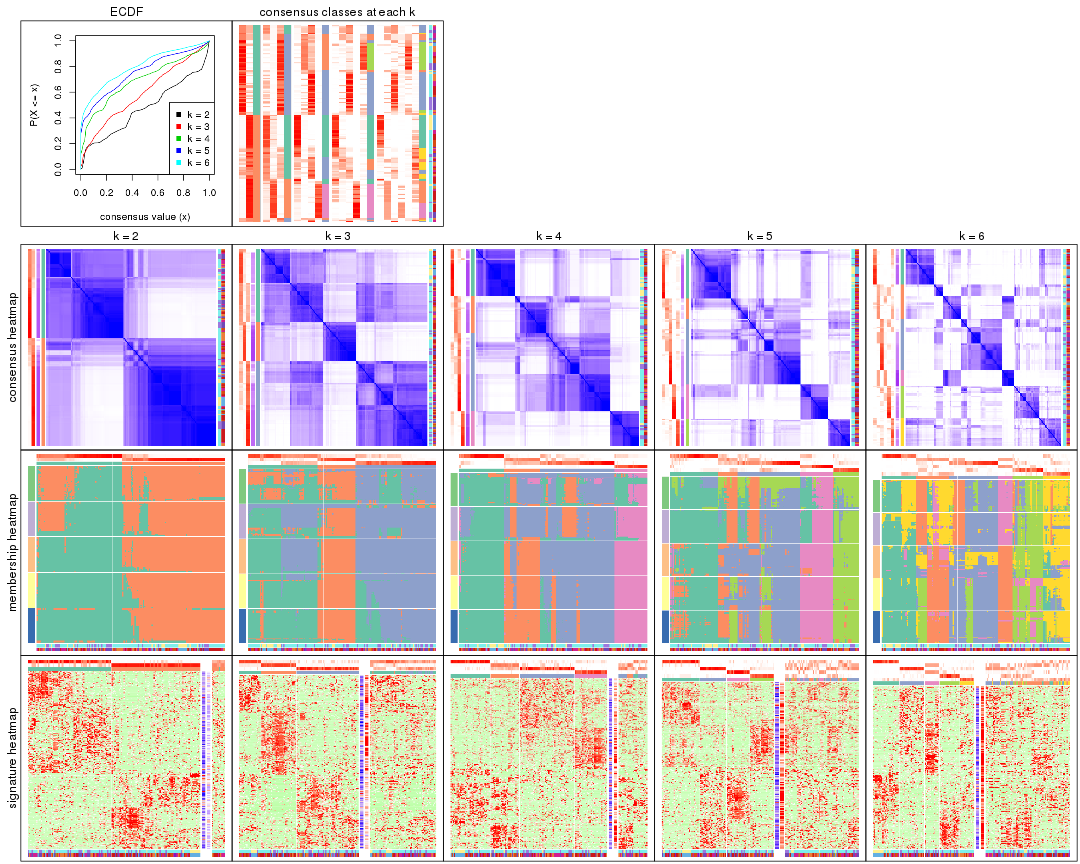
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
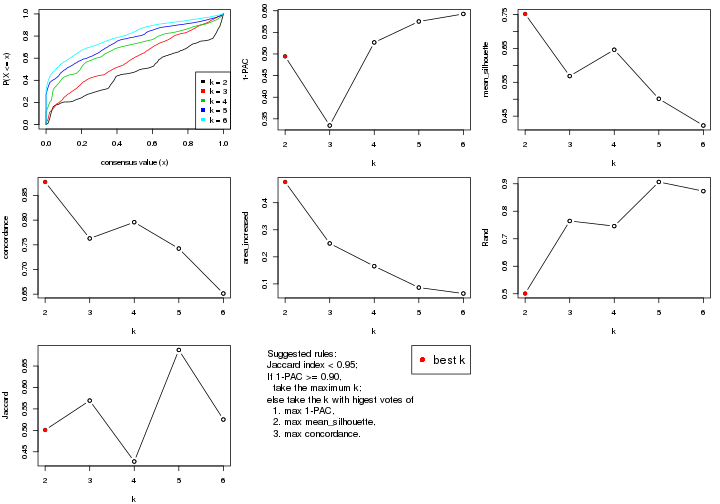
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.494 0.752 0.877 0.4766 0.501 0.501
#> 3 3 0.335 0.568 0.763 0.2494 0.765 0.569
#> 4 4 0.527 0.646 0.796 0.1653 0.746 0.427
#> 5 5 0.575 0.501 0.742 0.0864 0.906 0.687
#> 6 6 0.593 0.422 0.651 0.0644 0.873 0.525
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 2 0.1414 0.88185 0.020 0.980
#> GSM340359 2 0.2948 0.86795 0.052 0.948
#> GSM340361 2 0.1414 0.88185 0.020 0.980
#> GSM340362 1 0.6973 0.75827 0.812 0.188
#> GSM340363 2 0.0672 0.88134 0.008 0.992
#> GSM340364 2 0.2043 0.87910 0.032 0.968
#> GSM340365 1 0.9460 0.58998 0.636 0.364
#> GSM340366 1 0.9522 0.58568 0.628 0.372
#> GSM340367 2 0.1843 0.88049 0.028 0.972
#> GSM340368 2 0.0672 0.87977 0.008 0.992
#> GSM340369 2 0.1414 0.88183 0.020 0.980
#> GSM340370 2 0.1414 0.88185 0.020 0.980
#> GSM340371 1 0.5737 0.78552 0.864 0.136
#> GSM340372 2 0.1414 0.88185 0.020 0.980
#> GSM340373 2 0.1843 0.88049 0.028 0.972
#> GSM340375 2 0.1414 0.88185 0.020 0.980
#> GSM340376 2 0.1414 0.88185 0.020 0.980
#> GSM340378 2 0.1843 0.88049 0.028 0.972
#> GSM340243 2 0.6048 0.77134 0.148 0.852
#> GSM340244 2 0.8763 0.57897 0.296 0.704
#> GSM340246 1 0.1184 0.82170 0.984 0.016
#> GSM340247 2 0.1633 0.88054 0.024 0.976
#> GSM340248 2 1.0000 0.00678 0.496 0.504
#> GSM340249 2 0.1633 0.88054 0.024 0.976
#> GSM340250 2 0.6531 0.74455 0.168 0.832
#> GSM340251 1 0.8386 0.70474 0.732 0.268
#> GSM340252 2 0.9795 0.29842 0.416 0.584
#> GSM340253 1 0.1184 0.82482 0.984 0.016
#> GSM340254 1 0.0000 0.82265 1.000 0.000
#> GSM340256 1 0.3584 0.81269 0.932 0.068
#> GSM340258 1 0.0000 0.82265 1.000 0.000
#> GSM340259 1 0.0000 0.82265 1.000 0.000
#> GSM340260 1 0.7376 0.74547 0.792 0.208
#> GSM340261 1 0.2423 0.82286 0.960 0.040
#> GSM340262 1 0.0000 0.82265 1.000 0.000
#> GSM340263 2 0.7376 0.71932 0.208 0.792
#> GSM340264 1 0.0672 0.82410 0.992 0.008
#> GSM340265 1 0.9358 0.60729 0.648 0.352
#> GSM340266 2 0.7453 0.69910 0.212 0.788
#> GSM340267 1 0.9323 0.61830 0.652 0.348
#> GSM340268 1 0.1414 0.82064 0.980 0.020
#> GSM340269 1 0.0376 0.82371 0.996 0.004
#> GSM340270 2 0.2043 0.87910 0.032 0.968
#> GSM537574 1 0.0938 0.82316 0.988 0.012
#> GSM537580 1 0.0000 0.82265 1.000 0.000
#> GSM537581 2 0.0672 0.87977 0.008 0.992
#> GSM340272 1 0.9460 0.58998 0.636 0.364
#> GSM340273 2 0.1633 0.88255 0.024 0.976
#> GSM340275 2 0.1633 0.88054 0.024 0.976
#> GSM340276 2 0.9608 0.42841 0.384 0.616
#> GSM340277 1 0.9460 0.58998 0.636 0.364
#> GSM340278 2 0.9970 0.01051 0.468 0.532
#> GSM340279 2 0.9977 -0.09627 0.472 0.528
#> GSM340282 1 0.9460 0.58998 0.636 0.364
#> GSM340284 1 0.1633 0.81913 0.976 0.024
#> GSM340285 2 0.5842 0.79028 0.140 0.860
#> GSM340286 1 0.0000 0.82265 1.000 0.000
#> GSM340287 1 0.8081 0.69028 0.752 0.248
#> GSM340288 2 0.1633 0.88255 0.024 0.976
#> GSM340289 2 0.9286 0.50899 0.344 0.656
#> GSM340290 1 0.8499 0.54069 0.724 0.276
#> GSM340291 2 0.7674 0.65660 0.224 0.776
#> GSM340293 1 0.9661 0.57798 0.608 0.392
#> GSM340294 1 0.2236 0.82346 0.964 0.036
#> GSM340296 1 0.9608 0.58399 0.616 0.384
#> GSM340297 2 0.9608 0.20458 0.384 0.616
#> GSM340298 1 0.9608 0.58399 0.616 0.384
#> GSM340299 1 0.6887 0.76346 0.816 0.184
#> GSM340301 1 0.0376 0.82371 0.996 0.004
#> GSM340303 1 0.9286 0.61617 0.656 0.344
#> GSM340304 2 0.3114 0.86560 0.056 0.944
#> GSM340306 2 0.0938 0.88064 0.012 0.988
#> GSM340307 1 0.9608 0.58399 0.616 0.384
#> GSM340310 2 0.1414 0.88185 0.020 0.980
#> GSM340314 1 0.9754 0.47312 0.592 0.408
#> GSM340315 2 0.7674 0.63362 0.224 0.776
#> GSM340317 2 0.1184 0.88024 0.016 0.984
#> GSM340318 2 0.1633 0.88054 0.024 0.976
#> GSM340319 2 0.1633 0.88054 0.024 0.976
#> GSM340320 2 0.1414 0.88091 0.020 0.980
#> GSM340321 1 0.2043 0.82338 0.968 0.032
#> GSM340322 2 0.1633 0.88054 0.024 0.976
#> GSM340324 2 0.1843 0.88049 0.028 0.972
#> GSM340328 2 0.0376 0.88027 0.004 0.996
#> GSM340330 1 0.0938 0.82221 0.988 0.012
#> GSM340332 2 0.1633 0.88054 0.024 0.976
#> GSM340333 1 0.0938 0.82475 0.988 0.012
#> GSM340336 2 0.1633 0.88054 0.024 0.976
#> GSM340337 2 0.1633 0.88054 0.024 0.976
#> GSM340338 1 0.7219 0.75766 0.800 0.200
#> GSM340339 2 0.1633 0.88054 0.024 0.976
#> GSM340340 2 0.0672 0.87977 0.008 0.992
#> GSM340341 2 0.1633 0.88054 0.024 0.976
#> GSM340343 2 0.1633 0.88054 0.024 0.976
#> GSM340344 1 0.5842 0.78289 0.860 0.140
#> GSM340346 1 0.9608 0.58399 0.616 0.384
#> GSM340347 2 0.1633 0.88054 0.024 0.976
#> GSM340348 2 0.1184 0.88111 0.016 0.984
#> GSM340349 2 0.9427 0.33205 0.360 0.640
#> GSM340350 2 0.9552 0.28000 0.376 0.624
#> GSM340351 2 0.1414 0.88185 0.020 0.980
#> GSM340354 1 0.0672 0.82191 0.992 0.008
#> GSM340356 2 0.1633 0.88255 0.024 0.976
#> GSM340357 2 0.1843 0.88049 0.028 0.972
#> GSM348183 2 0.1414 0.88269 0.020 0.980
#> GSM348191 1 0.4022 0.81446 0.920 0.080
#> GSM348193 2 0.0672 0.88134 0.008 0.992
#> GSM537578 2 0.9998 -0.20750 0.492 0.508
#> GSM348181 2 0.9000 0.54815 0.316 0.684
#> GSM348182 1 0.0000 0.82265 1.000 0.000
#> GSM348184 1 0.8861 0.66659 0.696 0.304
#> GSM348185 2 0.0376 0.88027 0.004 0.996
#> GSM348186 1 0.0376 0.82371 0.996 0.004
#> GSM348187 2 0.1414 0.88185 0.020 0.980
#> GSM348188 2 0.1414 0.88103 0.020 0.980
#> GSM348189 1 0.0000 0.82265 1.000 0.000
#> GSM348190 1 0.6712 0.76385 0.824 0.176
#> GSM348194 2 0.1633 0.88255 0.024 0.976
#> GSM348195 2 0.1414 0.88185 0.020 0.980
#> GSM348196 2 0.1414 0.88185 0.020 0.980
#> GSM537585 2 0.9608 0.24795 0.384 0.616
#> GSM537594 2 0.2423 0.87911 0.040 0.960
#> GSM537596 2 0.2236 0.88174 0.036 0.964
#> GSM537597 2 0.2236 0.87979 0.036 0.964
#> GSM537602 2 0.3733 0.85224 0.072 0.928
#> GSM340184 1 0.1414 0.82064 0.980 0.020
#> GSM340185 1 0.9608 0.58399 0.616 0.384
#> GSM340186 1 0.1633 0.82209 0.976 0.024
#> GSM340187 2 0.1633 0.88054 0.024 0.976
#> GSM340189 2 0.2236 0.87734 0.036 0.964
#> GSM340190 1 0.9608 0.58399 0.616 0.384
#> GSM340191 1 0.1633 0.81913 0.976 0.024
#> GSM340192 1 0.9460 0.58998 0.636 0.364
#> GSM340193 2 0.7453 0.70599 0.212 0.788
#> GSM340194 1 0.1414 0.82409 0.980 0.020
#> GSM340195 1 0.0672 0.82431 0.992 0.008
#> GSM340196 2 0.1633 0.88054 0.024 0.976
#> GSM340197 1 0.0000 0.82265 1.000 0.000
#> GSM340198 1 0.1414 0.82478 0.980 0.020
#> GSM340199 1 0.9608 0.58399 0.616 0.384
#> GSM340200 1 0.9608 0.58399 0.616 0.384
#> GSM340201 2 0.1633 0.88054 0.024 0.976
#> GSM340202 2 0.1633 0.88054 0.024 0.976
#> GSM340203 2 0.1843 0.88026 0.028 0.972
#> GSM340204 1 0.2948 0.81727 0.948 0.052
#> GSM340205 2 0.1843 0.88049 0.028 0.972
#> GSM340206 1 0.1414 0.82064 0.980 0.020
#> GSM340207 1 0.0000 0.82265 1.000 0.000
#> GSM340237 2 0.7528 0.66921 0.216 0.784
#> GSM340238 1 0.4161 0.81040 0.916 0.084
#> GSM340239 1 0.0000 0.82265 1.000 0.000
#> GSM340240 1 0.9460 0.58998 0.636 0.364
#> GSM340241 1 0.0000 0.82265 1.000 0.000
#> GSM340242 1 0.0000 0.82265 1.000 0.000
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.6274 -0.349 0.544 0.456 0.000
#> GSM340359 1 0.4636 0.499 0.848 0.116 0.036
#> GSM340361 1 0.4862 0.484 0.820 0.160 0.020
#> GSM340362 3 0.5588 0.669 0.124 0.068 0.808
#> GSM340363 1 0.6513 -0.463 0.520 0.476 0.004
#> GSM340364 1 0.3112 0.523 0.900 0.096 0.004
#> GSM340365 3 0.5831 0.567 0.284 0.008 0.708
#> GSM340366 3 0.6522 0.581 0.272 0.032 0.696
#> GSM340367 1 0.2878 0.522 0.904 0.096 0.000
#> GSM340368 2 0.3340 0.894 0.120 0.880 0.000
#> GSM340369 2 0.4099 0.877 0.140 0.852 0.008
#> GSM340370 1 0.3879 0.499 0.848 0.152 0.000
#> GSM340371 3 0.1999 0.743 0.036 0.012 0.952
#> GSM340372 1 0.3038 0.519 0.896 0.104 0.000
#> GSM340373 1 0.2878 0.522 0.904 0.096 0.000
#> GSM340375 1 0.3879 0.499 0.848 0.152 0.000
#> GSM340376 1 0.3879 0.499 0.848 0.152 0.000
#> GSM340378 1 0.2878 0.522 0.904 0.096 0.000
#> GSM340243 1 0.6521 0.413 0.644 0.016 0.340
#> GSM340244 1 0.7578 0.190 0.500 0.040 0.460
#> GSM340246 3 0.3349 0.706 0.108 0.004 0.888
#> GSM340247 2 0.2955 0.907 0.080 0.912 0.008
#> GSM340248 1 0.7940 0.312 0.524 0.060 0.416
#> GSM340249 2 0.3377 0.907 0.092 0.896 0.012
#> GSM340250 1 0.8924 0.465 0.524 0.140 0.336
#> GSM340251 3 0.6247 0.266 0.376 0.004 0.620
#> GSM340252 3 0.6677 0.573 0.080 0.180 0.740
#> GSM340253 3 0.2492 0.742 0.016 0.048 0.936
#> GSM340254 3 0.1753 0.740 0.000 0.048 0.952
#> GSM340256 3 0.3375 0.744 0.048 0.044 0.908
#> GSM340258 3 0.1453 0.743 0.024 0.008 0.968
#> GSM340259 3 0.4399 0.716 0.092 0.044 0.864
#> GSM340260 3 0.4963 0.661 0.200 0.008 0.792
#> GSM340261 3 0.7050 0.243 0.372 0.028 0.600
#> GSM340262 3 0.1289 0.741 0.000 0.032 0.968
#> GSM340263 1 0.8125 0.443 0.576 0.084 0.340
#> GSM340264 3 0.1711 0.746 0.032 0.008 0.960
#> GSM340265 3 0.6303 0.597 0.248 0.032 0.720
#> GSM340266 1 0.8643 0.380 0.516 0.108 0.376
#> GSM340267 1 0.7169 0.345 0.568 0.028 0.404
#> GSM340268 3 0.5335 0.564 0.232 0.008 0.760
#> GSM340269 3 0.2313 0.745 0.032 0.024 0.944
#> GSM340270 1 0.8666 0.461 0.544 0.120 0.336
#> GSM537574 3 0.5581 0.651 0.168 0.040 0.792
#> GSM537580 3 0.0747 0.740 0.000 0.016 0.984
#> GSM537581 2 0.4887 0.816 0.228 0.772 0.000
#> GSM340272 3 0.6341 0.590 0.252 0.032 0.716
#> GSM340273 1 0.3500 0.528 0.880 0.116 0.004
#> GSM340275 2 0.3207 0.908 0.084 0.904 0.012
#> GSM340276 3 0.7389 0.111 0.408 0.036 0.556
#> GSM340277 3 0.6452 0.589 0.252 0.036 0.712
#> GSM340278 3 0.6676 -0.078 0.476 0.008 0.516
#> GSM340279 1 0.7901 0.340 0.540 0.060 0.400
#> GSM340282 3 0.6562 0.584 0.264 0.036 0.700
#> GSM340284 3 0.6298 0.226 0.388 0.004 0.608
#> GSM340285 1 0.8173 0.277 0.508 0.072 0.420
#> GSM340286 3 0.1163 0.741 0.000 0.028 0.972
#> GSM340287 1 0.7377 0.204 0.516 0.032 0.452
#> GSM340288 1 0.3784 0.512 0.864 0.132 0.004
#> GSM340289 1 0.7159 0.252 0.528 0.024 0.448
#> GSM340290 3 0.6298 0.211 0.388 0.004 0.608
#> GSM340291 1 0.6566 0.414 0.636 0.016 0.348
#> GSM340293 1 0.7828 0.202 0.500 0.052 0.448
#> GSM340294 3 0.6601 0.445 0.296 0.028 0.676
#> GSM340296 1 0.7023 0.350 0.624 0.032 0.344
#> GSM340297 1 0.8971 0.456 0.520 0.144 0.336
#> GSM340298 1 0.7044 0.341 0.620 0.032 0.348
#> GSM340299 3 0.5384 0.663 0.188 0.024 0.788
#> GSM340301 3 0.1753 0.740 0.000 0.048 0.952
#> GSM340303 3 0.6066 0.601 0.248 0.024 0.728
#> GSM340304 1 0.6717 0.406 0.628 0.020 0.352
#> GSM340306 2 0.4692 0.869 0.168 0.820 0.012
#> GSM340307 3 0.6653 0.571 0.288 0.032 0.680
#> GSM340310 1 0.4178 0.474 0.828 0.172 0.000
#> GSM340314 3 0.7030 0.158 0.396 0.024 0.580
#> GSM340315 1 0.8756 0.466 0.540 0.128 0.332
#> GSM340317 2 0.6673 0.777 0.200 0.732 0.068
#> GSM340318 2 0.2955 0.907 0.080 0.912 0.008
#> GSM340319 2 0.2955 0.907 0.080 0.912 0.008
#> GSM340320 2 0.3918 0.899 0.120 0.868 0.012
#> GSM340321 3 0.6226 0.533 0.252 0.028 0.720
#> GSM340322 2 0.3183 0.903 0.076 0.908 0.016
#> GSM340324 1 0.3967 0.571 0.884 0.072 0.044
#> GSM340328 2 0.6309 0.439 0.500 0.500 0.000
#> GSM340330 3 0.1585 0.742 0.028 0.008 0.964
#> GSM340332 2 0.3129 0.908 0.088 0.904 0.008
#> GSM340333 3 0.1989 0.741 0.004 0.048 0.948
#> GSM340336 2 0.2955 0.907 0.080 0.912 0.008
#> GSM340337 2 0.3618 0.903 0.104 0.884 0.012
#> GSM340338 3 0.4228 0.703 0.148 0.008 0.844
#> GSM340339 2 0.3213 0.908 0.092 0.900 0.008
#> GSM340340 2 0.6031 0.789 0.116 0.788 0.096
#> GSM340341 2 0.6754 0.751 0.168 0.740 0.092
#> GSM340343 2 0.3293 0.908 0.088 0.900 0.012
#> GSM340344 3 0.3356 0.734 0.056 0.036 0.908
#> GSM340346 3 0.6653 0.571 0.288 0.032 0.680
#> GSM340347 2 0.2955 0.907 0.080 0.912 0.008
#> GSM340348 2 0.4575 0.868 0.160 0.828 0.012
#> GSM340349 1 0.6229 0.403 0.652 0.008 0.340
#> GSM340350 1 0.6229 0.403 0.652 0.008 0.340
#> GSM340351 1 0.3500 0.526 0.880 0.116 0.004
#> GSM340354 3 0.2318 0.744 0.028 0.028 0.944
#> GSM340356 1 0.3112 0.542 0.900 0.096 0.004
#> GSM340357 1 0.3272 0.527 0.892 0.104 0.004
#> GSM348183 1 0.6783 -0.251 0.588 0.396 0.016
#> GSM348191 3 0.6082 0.460 0.296 0.012 0.692
#> GSM348193 2 0.6819 0.494 0.476 0.512 0.012
#> GSM537578 1 0.6381 0.405 0.648 0.012 0.340
#> GSM348181 1 0.7647 0.258 0.516 0.044 0.440
#> GSM348182 3 0.1411 0.741 0.000 0.036 0.964
#> GSM348184 3 0.6018 0.436 0.308 0.008 0.684
#> GSM348185 2 0.6754 0.585 0.432 0.556 0.012
#> GSM348186 3 0.1950 0.743 0.008 0.040 0.952
#> GSM348187 1 0.3619 0.514 0.864 0.136 0.000
#> GSM348188 2 0.4411 0.883 0.140 0.844 0.016
#> GSM348189 3 0.0747 0.740 0.000 0.016 0.984
#> GSM348190 3 0.5191 0.690 0.112 0.060 0.828
#> GSM348194 1 0.3500 0.526 0.880 0.116 0.004
#> GSM348195 1 0.4062 0.488 0.836 0.164 0.000
#> GSM348196 1 0.3644 0.518 0.872 0.124 0.004
#> GSM537585 1 0.7400 0.308 0.552 0.036 0.412
#> GSM537594 1 0.4056 0.555 0.876 0.092 0.032
#> GSM537596 1 0.5889 0.579 0.796 0.108 0.096
#> GSM537597 1 0.4316 0.572 0.868 0.088 0.044
#> GSM537602 1 0.6255 0.462 0.684 0.016 0.300
#> GSM340184 3 0.4353 0.664 0.156 0.008 0.836
#> GSM340185 3 0.7141 0.453 0.368 0.032 0.600
#> GSM340186 3 0.1950 0.740 0.040 0.008 0.952
#> GSM340187 2 0.3129 0.908 0.088 0.904 0.008
#> GSM340189 2 0.4925 0.859 0.080 0.844 0.076
#> GSM340190 1 0.7382 -0.037 0.512 0.032 0.456
#> GSM340191 3 0.6359 0.273 0.364 0.008 0.628
#> GSM340192 3 0.6452 0.586 0.264 0.032 0.704
#> GSM340193 1 0.8716 0.424 0.532 0.120 0.348
#> GSM340194 3 0.1989 0.741 0.004 0.048 0.948
#> GSM340195 3 0.1753 0.740 0.000 0.048 0.952
#> GSM340196 2 0.2955 0.907 0.080 0.912 0.008
#> GSM340197 3 0.1411 0.741 0.000 0.036 0.964
#> GSM340198 3 0.1751 0.744 0.028 0.012 0.960
#> GSM340199 1 0.7190 0.328 0.608 0.036 0.356
#> GSM340200 3 0.7353 0.285 0.436 0.032 0.532
#> GSM340201 2 0.2955 0.907 0.080 0.912 0.008
#> GSM340202 2 0.2955 0.907 0.080 0.912 0.008
#> GSM340203 2 0.4418 0.885 0.132 0.848 0.020
#> GSM340204 3 0.2173 0.742 0.008 0.048 0.944
#> GSM340205 1 0.8600 0.513 0.580 0.136 0.284
#> GSM340206 3 0.5244 0.562 0.240 0.004 0.756
#> GSM340207 3 0.0424 0.740 0.000 0.008 0.992
#> GSM340237 1 0.6229 0.403 0.652 0.008 0.340
#> GSM340238 3 0.6416 0.259 0.376 0.008 0.616
#> GSM340239 3 0.1031 0.741 0.000 0.024 0.976
#> GSM340240 3 0.6452 0.589 0.252 0.036 0.712
#> GSM340241 3 0.1453 0.743 0.024 0.008 0.968
#> GSM340242 3 0.0661 0.742 0.004 0.008 0.988
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.0707 0.8842 0.980 0.020 0.000 0.000
#> GSM340359 1 0.0707 0.8842 0.980 0.020 0.000 0.000
#> GSM340361 1 0.0188 0.8848 0.996 0.004 0.000 0.000
#> GSM340362 2 0.6745 0.6217 0.176 0.612 0.212 0.000
#> GSM340363 1 0.0188 0.8854 0.996 0.000 0.004 0.000
#> GSM340364 1 0.1118 0.8791 0.964 0.036 0.000 0.000
#> GSM340365 2 0.6723 0.5993 0.188 0.616 0.196 0.000
#> GSM340366 3 0.6581 0.5118 0.036 0.312 0.612 0.040
#> GSM340367 1 0.1022 0.8809 0.968 0.032 0.000 0.000
#> GSM340368 4 0.4284 0.8452 0.200 0.020 0.000 0.780
#> GSM340369 4 0.4446 0.8451 0.196 0.028 0.000 0.776
#> GSM340370 1 0.0707 0.8842 0.980 0.020 0.000 0.000
#> GSM340371 3 0.4843 -0.3457 0.000 0.396 0.604 0.000
#> GSM340372 1 0.0921 0.8828 0.972 0.028 0.000 0.000
#> GSM340373 1 0.1022 0.8809 0.968 0.032 0.000 0.000
#> GSM340375 1 0.0707 0.8842 0.980 0.020 0.000 0.000
#> GSM340376 1 0.0707 0.8842 0.980 0.020 0.000 0.000
#> GSM340378 1 0.1022 0.8809 0.968 0.032 0.000 0.000
#> GSM340243 3 0.6669 0.4480 0.332 0.104 0.564 0.000
#> GSM340244 3 0.5256 0.2825 0.392 0.012 0.596 0.000
#> GSM340246 3 0.0336 0.6538 0.000 0.008 0.992 0.000
#> GSM340247 4 0.1211 0.8774 0.040 0.000 0.000 0.960
#> GSM340248 3 0.3591 0.6130 0.168 0.008 0.824 0.000
#> GSM340249 4 0.2882 0.8865 0.084 0.000 0.024 0.892
#> GSM340250 3 0.4188 0.5660 0.244 0.004 0.752 0.000
#> GSM340251 3 0.0469 0.6588 0.012 0.000 0.988 0.000
#> GSM340252 2 0.8448 0.5781 0.044 0.428 0.356 0.172
#> GSM340253 2 0.4522 0.7566 0.000 0.680 0.320 0.000
#> GSM340254 2 0.4543 0.7569 0.000 0.676 0.324 0.000
#> GSM340256 2 0.4564 0.7538 0.000 0.672 0.328 0.000
#> GSM340258 3 0.0000 0.6540 0.000 0.000 1.000 0.000
#> GSM340259 2 0.5619 0.7323 0.040 0.640 0.320 0.000
#> GSM340260 3 0.5397 0.3525 0.064 0.220 0.716 0.000
#> GSM340261 3 0.1637 0.6479 0.000 0.060 0.940 0.000
#> GSM340262 2 0.4746 0.7497 0.000 0.632 0.368 0.000
#> GSM340263 1 0.4605 0.4538 0.664 0.000 0.336 0.000
#> GSM340264 3 0.4866 -0.3690 0.000 0.404 0.596 0.000
#> GSM340265 3 0.6383 0.3417 0.036 0.276 0.648 0.040
#> GSM340266 3 0.2149 0.6505 0.088 0.000 0.912 0.000
#> GSM340267 3 0.2408 0.6472 0.104 0.000 0.896 0.000
#> GSM340268 3 0.0000 0.6540 0.000 0.000 1.000 0.000
#> GSM340269 3 0.1637 0.6479 0.000 0.060 0.940 0.000
#> GSM340270 3 0.4898 0.3670 0.416 0.000 0.584 0.000
#> GSM537574 3 0.2198 0.6464 0.008 0.072 0.920 0.000
#> GSM537580 2 0.4994 0.6290 0.000 0.520 0.480 0.000
#> GSM537581 4 0.5323 0.6501 0.352 0.020 0.000 0.628
#> GSM340272 2 0.6043 0.1798 0.036 0.696 0.228 0.040
#> GSM340273 1 0.0188 0.8854 0.996 0.000 0.004 0.000
#> GSM340275 4 0.3881 0.8633 0.172 0.000 0.016 0.812
#> GSM340276 3 0.5636 0.4100 0.260 0.060 0.680 0.000
#> GSM340277 2 0.6823 -0.1900 0.036 0.548 0.376 0.040
#> GSM340278 3 0.1174 0.6623 0.020 0.012 0.968 0.000
#> GSM340279 3 0.2542 0.6550 0.084 0.012 0.904 0.000
#> GSM340282 2 0.6921 -0.2984 0.036 0.496 0.428 0.040
#> GSM340284 3 0.0000 0.6540 0.000 0.000 1.000 0.000
#> GSM340285 3 0.4072 0.6004 0.120 0.052 0.828 0.000
#> GSM340286 2 0.4790 0.7441 0.000 0.620 0.380 0.000
#> GSM340287 3 0.2483 0.6592 0.052 0.032 0.916 0.000
#> GSM340288 1 0.0188 0.8854 0.996 0.000 0.004 0.000
#> GSM340289 1 0.6023 0.3609 0.612 0.060 0.328 0.000
#> GSM340290 3 0.0817 0.6542 0.000 0.024 0.976 0.000
#> GSM340291 3 0.4072 0.5571 0.252 0.000 0.748 0.000
#> GSM340293 3 0.6515 0.5348 0.128 0.248 0.624 0.000
#> GSM340294 3 0.1637 0.6479 0.000 0.060 0.940 0.000
#> GSM340296 3 0.6520 0.5191 0.036 0.300 0.624 0.040
#> GSM340297 3 0.4323 0.5905 0.204 0.020 0.776 0.000
#> GSM340298 3 0.6520 0.5191 0.036 0.300 0.624 0.040
#> GSM340299 2 0.5993 0.7183 0.064 0.628 0.308 0.000
#> GSM340301 2 0.4522 0.7566 0.000 0.680 0.320 0.000
#> GSM340303 3 0.6302 0.1219 0.068 0.368 0.564 0.000
#> GSM340304 3 0.5404 0.1277 0.476 0.012 0.512 0.000
#> GSM340306 4 0.3801 0.8440 0.220 0.000 0.000 0.780
#> GSM340307 3 0.6520 0.5191 0.036 0.300 0.624 0.040
#> GSM340310 1 0.0707 0.8842 0.980 0.020 0.000 0.000
#> GSM340314 3 0.0817 0.6617 0.024 0.000 0.976 0.000
#> GSM340315 3 0.4164 0.5604 0.264 0.000 0.736 0.000
#> GSM340317 1 0.6766 0.1202 0.520 0.000 0.100 0.380
#> GSM340318 4 0.1211 0.8774 0.040 0.000 0.000 0.960
#> GSM340319 4 0.1211 0.8774 0.040 0.000 0.000 0.960
#> GSM340320 4 0.3801 0.8440 0.220 0.000 0.000 0.780
#> GSM340321 3 0.1637 0.6479 0.000 0.060 0.940 0.000
#> GSM340322 4 0.1792 0.8884 0.068 0.000 0.000 0.932
#> GSM340324 1 0.0469 0.8828 0.988 0.000 0.012 0.000
#> GSM340328 1 0.0707 0.8842 0.980 0.020 0.000 0.000
#> GSM340330 3 0.0000 0.6540 0.000 0.000 1.000 0.000
#> GSM340332 4 0.2149 0.8905 0.088 0.000 0.000 0.912
#> GSM340333 2 0.4522 0.7566 0.000 0.680 0.320 0.000
#> GSM340336 4 0.1211 0.8774 0.040 0.000 0.000 0.960
#> GSM340337 4 0.3908 0.8490 0.212 0.000 0.004 0.784
#> GSM340338 3 0.1807 0.6629 0.052 0.008 0.940 0.000
#> GSM340339 4 0.2593 0.8890 0.104 0.000 0.004 0.892
#> GSM340340 4 0.4584 0.8450 0.196 0.016 0.012 0.776
#> GSM340341 4 0.6595 0.6274 0.212 0.000 0.160 0.628
#> GSM340343 4 0.2412 0.8904 0.084 0.000 0.008 0.908
#> GSM340344 2 0.6714 0.6239 0.176 0.616 0.208 0.000
#> GSM340346 3 0.6520 0.5191 0.036 0.300 0.624 0.040
#> GSM340347 4 0.1211 0.8774 0.040 0.000 0.000 0.960
#> GSM340348 4 0.4175 0.8436 0.212 0.000 0.012 0.776
#> GSM340349 3 0.6719 0.5294 0.180 0.204 0.616 0.000
#> GSM340350 3 0.7882 0.3987 0.264 0.288 0.444 0.004
#> GSM340351 1 0.0188 0.8854 0.996 0.000 0.004 0.000
#> GSM340354 3 0.1557 0.6490 0.000 0.056 0.944 0.000
#> GSM340356 1 0.0188 0.8854 0.996 0.000 0.004 0.000
#> GSM340357 1 0.0921 0.8818 0.972 0.028 0.000 0.000
#> GSM348183 1 0.2281 0.8221 0.904 0.000 0.096 0.000
#> GSM348191 3 0.1837 0.6600 0.028 0.028 0.944 0.000
#> GSM348193 1 0.0188 0.8854 0.996 0.000 0.004 0.000
#> GSM537578 3 0.6621 0.5332 0.184 0.188 0.628 0.000
#> GSM348181 3 0.4981 0.1628 0.464 0.000 0.536 0.000
#> GSM348182 2 0.5269 0.7476 0.016 0.620 0.364 0.000
#> GSM348184 3 0.2522 0.6460 0.016 0.076 0.908 0.000
#> GSM348185 1 0.4677 0.4187 0.680 0.000 0.004 0.316
#> GSM348186 2 0.4564 0.7568 0.000 0.672 0.328 0.000
#> GSM348187 1 0.0000 0.8843 1.000 0.000 0.000 0.000
#> GSM348188 4 0.4327 0.8386 0.216 0.000 0.016 0.768
#> GSM348189 2 0.4866 0.7326 0.000 0.596 0.404 0.000
#> GSM348190 2 0.6790 0.6081 0.196 0.608 0.196 0.000
#> GSM348194 1 0.0188 0.8854 0.996 0.000 0.004 0.000
#> GSM348195 1 0.0188 0.8848 0.996 0.004 0.000 0.000
#> GSM348196 1 0.0000 0.8843 1.000 0.000 0.000 0.000
#> GSM537585 1 0.4485 0.6771 0.772 0.028 0.200 0.000
#> GSM537594 1 0.2081 0.8326 0.916 0.000 0.084 0.000
#> GSM537596 1 0.3444 0.7249 0.816 0.000 0.184 0.000
#> GSM537597 1 0.1302 0.8634 0.956 0.000 0.044 0.000
#> GSM537602 1 0.4406 0.7032 0.780 0.028 0.192 0.000
#> GSM340184 3 0.0000 0.6540 0.000 0.000 1.000 0.000
#> GSM340185 3 0.6520 0.5191 0.036 0.300 0.624 0.040
#> GSM340186 3 0.0000 0.6540 0.000 0.000 1.000 0.000
#> GSM340187 4 0.1940 0.8899 0.076 0.000 0.000 0.924
#> GSM340189 4 0.2670 0.8868 0.072 0.000 0.024 0.904
#> GSM340190 3 0.6520 0.5191 0.036 0.300 0.624 0.040
#> GSM340191 3 0.0000 0.6540 0.000 0.000 1.000 0.000
#> GSM340192 2 0.6911 -0.2832 0.036 0.504 0.420 0.040
#> GSM340193 1 0.4459 0.6898 0.780 0.032 0.188 0.000
#> GSM340194 2 0.4522 0.7566 0.000 0.680 0.320 0.000
#> GSM340195 2 0.4522 0.7566 0.000 0.680 0.320 0.000
#> GSM340196 4 0.1211 0.8774 0.040 0.000 0.000 0.960
#> GSM340197 2 0.4713 0.7516 0.000 0.640 0.360 0.000
#> GSM340198 2 0.4888 0.7265 0.000 0.588 0.412 0.000
#> GSM340199 3 0.6520 0.5191 0.036 0.300 0.624 0.040
#> GSM340200 3 0.6520 0.5191 0.036 0.300 0.624 0.040
#> GSM340201 4 0.1474 0.8831 0.052 0.000 0.000 0.948
#> GSM340202 4 0.1211 0.8774 0.040 0.000 0.000 0.960
#> GSM340203 4 0.4591 0.8047 0.084 0.000 0.116 0.800
#> GSM340204 2 0.4522 0.7566 0.000 0.680 0.320 0.000
#> GSM340205 1 0.3852 0.7152 0.800 0.008 0.192 0.000
#> GSM340206 3 0.0188 0.6550 0.000 0.004 0.996 0.000
#> GSM340207 3 0.4222 0.1148 0.000 0.272 0.728 0.000
#> GSM340237 3 0.7617 0.4495 0.208 0.292 0.496 0.004
#> GSM340238 3 0.0469 0.6591 0.012 0.000 0.988 0.000
#> GSM340239 2 0.4776 0.7461 0.000 0.624 0.376 0.000
#> GSM340240 2 0.6306 0.0986 0.036 0.660 0.264 0.040
#> GSM340241 3 0.0000 0.6540 0.000 0.000 1.000 0.000
#> GSM340242 3 0.4933 -0.4481 0.000 0.432 0.568 0.000
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.1851 0.7983 0.912 0.000 0.000 0.000 0.088
#> GSM340359 1 0.2712 0.7998 0.880 0.032 0.000 0.000 0.088
#> GSM340361 1 0.0963 0.8038 0.964 0.000 0.000 0.000 0.036
#> GSM340362 5 0.4685 0.7320 0.140 0.028 0.064 0.000 0.768
#> GSM340363 1 0.0963 0.7922 0.964 0.036 0.000 0.000 0.000
#> GSM340364 1 0.3019 0.7968 0.864 0.048 0.000 0.000 0.088
#> GSM340365 5 0.5887 0.1466 0.404 0.088 0.004 0.000 0.504
#> GSM340366 2 0.6059 0.0507 0.000 0.468 0.412 0.000 0.120
#> GSM340367 1 0.2871 0.7979 0.872 0.040 0.000 0.000 0.088
#> GSM340368 4 0.6498 0.4806 0.316 0.048 0.000 0.552 0.084
#> GSM340369 4 0.5691 0.5067 0.308 0.008 0.000 0.600 0.084
#> GSM340370 1 0.1851 0.7983 0.912 0.000 0.000 0.000 0.088
#> GSM340371 3 0.4779 0.1474 0.000 0.032 0.628 0.000 0.340
#> GSM340372 1 0.2011 0.7961 0.908 0.004 0.000 0.000 0.088
#> GSM340373 1 0.2248 0.7979 0.900 0.012 0.000 0.000 0.088
#> GSM340375 1 0.1851 0.7983 0.912 0.000 0.000 0.000 0.088
#> GSM340376 1 0.1851 0.7983 0.912 0.000 0.000 0.000 0.088
#> GSM340378 1 0.3226 0.7940 0.852 0.060 0.000 0.000 0.088
#> GSM340243 2 0.6826 0.2387 0.328 0.336 0.336 0.000 0.000
#> GSM340244 2 0.7631 0.2702 0.236 0.440 0.260 0.000 0.064
#> GSM340246 3 0.0880 0.5669 0.000 0.000 0.968 0.000 0.032
#> GSM340247 4 0.0000 0.7690 0.000 0.000 0.000 1.000 0.000
#> GSM340248 3 0.7585 -0.2966 0.308 0.304 0.348 0.000 0.040
#> GSM340249 4 0.1200 0.7682 0.008 0.012 0.016 0.964 0.000
#> GSM340250 1 0.6596 -0.0350 0.416 0.372 0.212 0.000 0.000
#> GSM340251 3 0.0609 0.5639 0.000 0.020 0.980 0.000 0.000
#> GSM340252 3 0.8319 -0.2460 0.072 0.016 0.332 0.304 0.276
#> GSM340253 5 0.2729 0.8435 0.004 0.028 0.084 0.000 0.884
#> GSM340254 5 0.1851 0.8459 0.000 0.000 0.088 0.000 0.912
#> GSM340256 5 0.4655 0.7349 0.016 0.080 0.140 0.000 0.764
#> GSM340258 3 0.0963 0.5614 0.000 0.000 0.964 0.000 0.036
#> GSM340259 5 0.4296 0.7860 0.028 0.080 0.088 0.000 0.804
#> GSM340260 3 0.8287 -0.1712 0.180 0.168 0.368 0.000 0.284
#> GSM340261 3 0.1952 0.5449 0.004 0.000 0.912 0.000 0.084
#> GSM340262 5 0.3535 0.8059 0.000 0.028 0.164 0.000 0.808
#> GSM340263 1 0.6661 0.2754 0.468 0.396 0.100 0.000 0.036
#> GSM340264 3 0.5091 0.0423 0.000 0.044 0.584 0.000 0.372
#> GSM340265 2 0.6796 0.1730 0.000 0.360 0.352 0.000 0.288
#> GSM340266 3 0.7544 -0.2412 0.280 0.272 0.404 0.000 0.044
#> GSM340267 3 0.4674 0.2452 0.244 0.016 0.712 0.000 0.028
#> GSM340268 3 0.0162 0.5696 0.000 0.004 0.996 0.000 0.000
#> GSM340269 3 0.3760 0.4596 0.000 0.028 0.784 0.000 0.188
#> GSM340270 2 0.6405 -0.0797 0.384 0.444 0.172 0.000 0.000
#> GSM537574 3 0.5656 0.2791 0.000 0.140 0.624 0.000 0.236
#> GSM537580 3 0.4718 -0.1647 0.000 0.016 0.540 0.000 0.444
#> GSM537581 4 0.6510 0.4698 0.328 0.056 0.000 0.544 0.072
#> GSM340272 2 0.6564 0.2483 0.000 0.444 0.212 0.000 0.344
#> GSM340273 1 0.0162 0.8018 0.996 0.004 0.000 0.000 0.000
#> GSM340275 4 0.3735 0.7186 0.100 0.008 0.064 0.828 0.000
#> GSM340276 2 0.7855 0.2429 0.160 0.424 0.304 0.000 0.112
#> GSM340277 2 0.6680 0.2675 0.000 0.436 0.268 0.000 0.296
#> GSM340278 3 0.0727 0.5684 0.004 0.004 0.980 0.000 0.012
#> GSM340279 3 0.4396 0.3755 0.160 0.012 0.772 0.000 0.056
#> GSM340282 2 0.6690 0.2402 0.000 0.432 0.300 0.000 0.268
#> GSM340284 3 0.0000 0.5698 0.000 0.000 1.000 0.000 0.000
#> GSM340285 3 0.7829 -0.1466 0.172 0.284 0.460 0.012 0.072
#> GSM340286 5 0.3561 0.7416 0.000 0.000 0.260 0.000 0.740
#> GSM340287 3 0.5450 0.3518 0.112 0.096 0.728 0.000 0.064
#> GSM340288 1 0.0000 0.8012 1.000 0.000 0.000 0.000 0.000
#> GSM340289 1 0.6729 0.4598 0.592 0.224 0.104 0.000 0.080
#> GSM340290 3 0.0510 0.5693 0.000 0.000 0.984 0.000 0.016
#> GSM340291 1 0.6779 -0.2299 0.364 0.276 0.360 0.000 0.000
#> GSM340293 3 0.4599 0.2543 0.020 0.356 0.624 0.000 0.000
#> GSM340294 3 0.2416 0.5337 0.012 0.000 0.888 0.000 0.100
#> GSM340296 3 0.4256 0.1932 0.000 0.436 0.564 0.000 0.000
#> GSM340297 3 0.5356 0.0627 0.328 0.020 0.616 0.000 0.036
#> GSM340298 3 0.4256 0.1940 0.000 0.436 0.564 0.000 0.000
#> GSM340299 5 0.4507 0.8005 0.028 0.104 0.080 0.000 0.788
#> GSM340301 5 0.1851 0.8459 0.000 0.000 0.088 0.000 0.912
#> GSM340303 3 0.7410 -0.1180 0.036 0.236 0.400 0.000 0.328
#> GSM340304 2 0.6826 -0.0121 0.356 0.452 0.176 0.000 0.016
#> GSM340306 4 0.5250 0.4548 0.416 0.048 0.000 0.536 0.000
#> GSM340307 3 0.4256 0.1940 0.000 0.436 0.564 0.000 0.000
#> GSM340310 1 0.1851 0.7983 0.912 0.000 0.000 0.000 0.088
#> GSM340314 3 0.0162 0.5696 0.000 0.004 0.996 0.000 0.000
#> GSM340315 3 0.6939 -0.2654 0.376 0.240 0.376 0.000 0.008
#> GSM340317 1 0.7136 0.0707 0.476 0.112 0.068 0.344 0.000
#> GSM340318 4 0.0000 0.7690 0.000 0.000 0.000 1.000 0.000
#> GSM340319 4 0.0000 0.7690 0.000 0.000 0.000 1.000 0.000
#> GSM340320 4 0.5338 0.4670 0.400 0.056 0.000 0.544 0.000
#> GSM340321 3 0.2462 0.5290 0.008 0.000 0.880 0.000 0.112
#> GSM340322 4 0.0404 0.7728 0.012 0.000 0.000 0.988 0.000
#> GSM340324 1 0.1121 0.7988 0.956 0.044 0.000 0.000 0.000
#> GSM340328 1 0.1792 0.7992 0.916 0.000 0.000 0.000 0.084
#> GSM340330 3 0.0000 0.5698 0.000 0.000 1.000 0.000 0.000
#> GSM340332 4 0.0794 0.7732 0.028 0.000 0.000 0.972 0.000
#> GSM340333 5 0.2795 0.8431 0.008 0.028 0.080 0.000 0.884
#> GSM340336 4 0.0000 0.7690 0.000 0.000 0.000 1.000 0.000
#> GSM340337 4 0.4990 0.5862 0.324 0.048 0.000 0.628 0.000
#> GSM340338 3 0.0162 0.5694 0.004 0.000 0.996 0.000 0.000
#> GSM340339 4 0.1809 0.7645 0.060 0.000 0.012 0.928 0.000
#> GSM340340 4 0.6540 0.4807 0.324 0.056 0.004 0.552 0.064
#> GSM340341 4 0.5774 0.4744 0.380 0.052 0.020 0.548 0.000
#> GSM340343 4 0.0981 0.7714 0.008 0.012 0.008 0.972 0.000
#> GSM340344 5 0.4390 0.7746 0.104 0.028 0.072 0.000 0.796
#> GSM340346 3 0.4256 0.1940 0.000 0.436 0.564 0.000 0.000
#> GSM340347 4 0.0000 0.7690 0.000 0.000 0.000 1.000 0.000
#> GSM340348 4 0.5338 0.4670 0.400 0.056 0.000 0.544 0.000
#> GSM340349 2 0.6779 0.2712 0.308 0.392 0.300 0.000 0.000
#> GSM340350 2 0.5096 0.2783 0.072 0.656 0.272 0.000 0.000
#> GSM340351 1 0.0000 0.8012 1.000 0.000 0.000 0.000 0.000
#> GSM340354 3 0.1341 0.5593 0.000 0.000 0.944 0.000 0.056
#> GSM340356 1 0.2127 0.7706 0.892 0.108 0.000 0.000 0.000
#> GSM340357 1 0.5260 0.6506 0.648 0.264 0.000 0.000 0.088
#> GSM348183 1 0.0963 0.7922 0.964 0.036 0.000 0.000 0.000
#> GSM348191 3 0.1197 0.5619 0.000 0.000 0.952 0.000 0.048
#> GSM348193 1 0.1800 0.7754 0.932 0.048 0.000 0.020 0.000
#> GSM537578 3 0.6410 -0.0688 0.200 0.304 0.496 0.000 0.000
#> GSM348181 2 0.7218 0.2605 0.240 0.444 0.288 0.000 0.028
#> GSM348182 5 0.3757 0.7822 0.020 0.000 0.208 0.000 0.772
#> GSM348184 3 0.1043 0.5532 0.000 0.040 0.960 0.000 0.000
#> GSM348185 1 0.5131 0.0528 0.588 0.048 0.000 0.364 0.000
#> GSM348186 5 0.1908 0.8460 0.000 0.000 0.092 0.000 0.908
#> GSM348187 1 0.1281 0.8080 0.956 0.012 0.000 0.000 0.032
#> GSM348188 4 0.5360 0.4804 0.396 0.048 0.004 0.552 0.000
#> GSM348189 5 0.4138 0.5547 0.000 0.000 0.384 0.000 0.616
#> GSM348190 5 0.3649 0.7329 0.152 0.000 0.040 0.000 0.808
#> GSM348194 1 0.0000 0.8012 1.000 0.000 0.000 0.000 0.000
#> GSM348195 1 0.1270 0.8054 0.948 0.000 0.000 0.000 0.052
#> GSM348196 1 0.0000 0.8012 1.000 0.000 0.000 0.000 0.000
#> GSM537585 1 0.6595 0.4666 0.600 0.232 0.088 0.000 0.080
#> GSM537594 1 0.0794 0.8018 0.972 0.028 0.000 0.000 0.000
#> GSM537596 1 0.3424 0.6871 0.760 0.240 0.000 0.000 0.000
#> GSM537597 1 0.3561 0.6519 0.740 0.260 0.000 0.000 0.000
#> GSM537602 1 0.4433 0.6709 0.740 0.200 0.060 0.000 0.000
#> GSM340184 3 0.0000 0.5698 0.000 0.000 1.000 0.000 0.000
#> GSM340185 3 0.4256 0.1940 0.000 0.436 0.564 0.000 0.000
#> GSM340186 3 0.0000 0.5698 0.000 0.000 1.000 0.000 0.000
#> GSM340187 4 0.0162 0.7704 0.004 0.000 0.000 0.996 0.000
#> GSM340189 4 0.1800 0.7539 0.020 0.000 0.048 0.932 0.000
#> GSM340190 3 0.4256 0.1940 0.000 0.436 0.564 0.000 0.000
#> GSM340191 3 0.0000 0.5698 0.000 0.000 1.000 0.000 0.000
#> GSM340192 2 0.6703 0.2644 0.000 0.428 0.276 0.000 0.296
#> GSM340193 1 0.3954 0.6963 0.772 0.192 0.000 0.000 0.036
#> GSM340194 5 0.2354 0.8455 0.012 0.008 0.076 0.000 0.904
#> GSM340195 5 0.2795 0.8431 0.008 0.028 0.080 0.000 0.884
#> GSM340196 4 0.0000 0.7690 0.000 0.000 0.000 1.000 0.000
#> GSM340197 5 0.2230 0.8414 0.000 0.000 0.116 0.000 0.884
#> GSM340198 3 0.4561 -0.2900 0.000 0.008 0.504 0.000 0.488
#> GSM340199 3 0.4242 0.2006 0.000 0.428 0.572 0.000 0.000
#> GSM340200 3 0.4249 0.1963 0.000 0.432 0.568 0.000 0.000
#> GSM340201 4 0.0404 0.7728 0.012 0.000 0.000 0.988 0.000
#> GSM340202 4 0.0000 0.7690 0.000 0.000 0.000 1.000 0.000
#> GSM340203 4 0.3301 0.7192 0.080 0.000 0.072 0.848 0.000
#> GSM340204 5 0.2069 0.8452 0.012 0.000 0.076 0.000 0.912
#> GSM340205 1 0.5412 0.5724 0.632 0.300 0.052 0.000 0.016
#> GSM340206 3 0.0162 0.5696 0.000 0.004 0.996 0.000 0.000
#> GSM340207 3 0.4718 0.2042 0.000 0.028 0.628 0.000 0.344
#> GSM340237 2 0.5290 0.2719 0.072 0.644 0.280 0.000 0.004
#> GSM340238 3 0.0162 0.5696 0.000 0.004 0.996 0.000 0.000
#> GSM340239 5 0.2891 0.8113 0.000 0.000 0.176 0.000 0.824
#> GSM340240 2 0.6646 0.2742 0.000 0.436 0.240 0.000 0.324
#> GSM340241 3 0.0404 0.5699 0.000 0.000 0.988 0.000 0.012
#> GSM340242 3 0.4835 0.0325 0.000 0.028 0.592 0.000 0.380
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.0713 0.75861 0.972 0.000 0.000 0.000 0.000 0.028
#> GSM340359 1 0.1845 0.76152 0.916 0.004 0.000 0.008 0.000 0.072
#> GSM340361 1 0.2260 0.75525 0.860 0.000 0.000 0.000 0.000 0.140
#> GSM340362 5 0.5646 0.41998 0.000 0.000 0.000 0.264 0.532 0.204
#> GSM340363 1 0.4905 0.44252 0.528 0.064 0.000 0.000 0.000 0.408
#> GSM340364 1 0.1453 0.73893 0.944 0.008 0.000 0.008 0.000 0.040
#> GSM340365 5 0.7763 0.33901 0.164 0.184 0.044 0.020 0.496 0.092
#> GSM340366 2 0.7563 -0.16501 0.000 0.420 0.172 0.068 0.292 0.048
#> GSM340367 1 0.1065 0.74341 0.964 0.008 0.000 0.008 0.000 0.020
#> GSM340368 2 0.5860 -0.12165 0.252 0.560 0.000 0.020 0.000 0.168
#> GSM340369 2 0.6848 -0.30863 0.272 0.476 0.000 0.144 0.000 0.108
#> GSM340370 1 0.0632 0.75713 0.976 0.000 0.000 0.000 0.000 0.024
#> GSM340371 3 0.5686 0.01642 0.000 0.004 0.552 0.080 0.336 0.028
#> GSM340372 1 0.0891 0.75677 0.968 0.008 0.000 0.000 0.000 0.024
#> GSM340373 1 0.0881 0.74265 0.972 0.008 0.000 0.008 0.000 0.012
#> GSM340375 1 0.0632 0.75713 0.976 0.000 0.000 0.000 0.000 0.024
#> GSM340376 1 0.0632 0.75713 0.976 0.000 0.000 0.000 0.000 0.024
#> GSM340378 1 0.1307 0.74080 0.952 0.008 0.000 0.008 0.000 0.032
#> GSM340243 6 0.6844 0.38974 0.052 0.228 0.240 0.000 0.008 0.472
#> GSM340244 6 0.3031 0.63178 0.020 0.000 0.072 0.000 0.048 0.860
#> GSM340246 3 0.1708 0.65062 0.000 0.004 0.932 0.000 0.040 0.024
#> GSM340247 4 0.3847 0.96297 0.000 0.456 0.000 0.544 0.000 0.000
#> GSM340248 6 0.3798 0.63569 0.004 0.000 0.216 0.000 0.032 0.748
#> GSM340249 2 0.5054 -0.81370 0.000 0.504 0.000 0.420 0.000 0.076
#> GSM340250 6 0.2706 0.64136 0.036 0.000 0.104 0.000 0.000 0.860
#> GSM340251 3 0.1942 0.63861 0.000 0.012 0.916 0.064 0.000 0.008
#> GSM340252 2 0.7382 -0.02779 0.000 0.444 0.212 0.008 0.140 0.196
#> GSM340253 5 0.3606 0.58547 0.000 0.000 0.004 0.264 0.724 0.008
#> GSM340254 5 0.0951 0.69676 0.000 0.000 0.020 0.008 0.968 0.004
#> GSM340256 5 0.3579 0.61806 0.000 0.000 0.060 0.016 0.816 0.108
#> GSM340258 3 0.5264 0.24187 0.000 0.008 0.644 0.068 0.256 0.024
#> GSM340259 5 0.2515 0.66214 0.012 0.000 0.012 0.012 0.892 0.072
#> GSM340260 6 0.6432 0.16919 0.004 0.012 0.148 0.016 0.376 0.444
#> GSM340261 3 0.2831 0.61947 0.000 0.000 0.840 0.000 0.136 0.024
#> GSM340262 5 0.3621 0.64779 0.000 0.000 0.160 0.048 0.788 0.004
#> GSM340263 6 0.2915 0.63132 0.044 0.000 0.048 0.000 0.036 0.872
#> GSM340264 3 0.5303 -0.12380 0.000 0.008 0.508 0.016 0.424 0.044
#> GSM340265 2 0.7572 -0.21802 0.000 0.384 0.160 0.100 0.328 0.028
#> GSM340266 6 0.4853 0.59995 0.000 0.032 0.272 0.000 0.040 0.656
#> GSM340267 3 0.4993 0.09302 0.004 0.016 0.592 0.000 0.040 0.348
#> GSM340268 3 0.1267 0.64437 0.000 0.000 0.940 0.060 0.000 0.000
#> GSM340269 5 0.5895 0.01152 0.000 0.004 0.436 0.044 0.452 0.064
#> GSM340270 6 0.2401 0.62578 0.044 0.000 0.060 0.000 0.004 0.892
#> GSM537574 5 0.6143 0.26788 0.000 0.004 0.264 0.020 0.528 0.184
#> GSM537580 3 0.4596 -0.21011 0.000 0.000 0.508 0.028 0.460 0.004
#> GSM537581 2 0.5461 0.00258 0.168 0.560 0.000 0.000 0.000 0.272
#> GSM340272 2 0.7406 -0.20667 0.000 0.416 0.116 0.084 0.340 0.044
#> GSM340273 1 0.3428 0.65483 0.696 0.000 0.000 0.000 0.000 0.304
#> GSM340275 2 0.5643 -0.60639 0.000 0.496 0.000 0.340 0.000 0.164
#> GSM340276 6 0.4659 0.52185 0.008 0.000 0.108 0.000 0.180 0.704
#> GSM340277 2 0.7302 -0.18751 0.000 0.428 0.120 0.096 0.328 0.028
#> GSM340278 3 0.1500 0.64388 0.000 0.000 0.936 0.000 0.012 0.052
#> GSM340279 3 0.4384 0.13558 0.000 0.000 0.616 0.000 0.036 0.348
#> GSM340282 2 0.7302 -0.18751 0.000 0.428 0.120 0.096 0.328 0.028
#> GSM340284 3 0.1411 0.64565 0.000 0.000 0.936 0.060 0.000 0.004
#> GSM340285 6 0.5762 0.61749 0.028 0.040 0.224 0.000 0.068 0.640
#> GSM340286 5 0.3190 0.62611 0.000 0.000 0.220 0.008 0.772 0.000
#> GSM340287 6 0.4774 0.35627 0.000 0.000 0.420 0.000 0.052 0.528
#> GSM340288 1 0.3266 0.68684 0.728 0.000 0.000 0.000 0.000 0.272
#> GSM340289 6 0.5996 0.19661 0.372 0.000 0.064 0.000 0.068 0.496
#> GSM340290 3 0.1890 0.64663 0.000 0.000 0.916 0.000 0.060 0.024
#> GSM340291 6 0.5839 0.59775 0.104 0.016 0.272 0.000 0.020 0.588
#> GSM340293 3 0.5194 0.48077 0.000 0.384 0.544 0.052 0.000 0.020
#> GSM340294 3 0.3202 0.59592 0.000 0.000 0.800 0.000 0.176 0.024
#> GSM340296 3 0.4692 0.47183 0.000 0.428 0.532 0.036 0.000 0.004
#> GSM340297 3 0.5553 -0.01878 0.068 0.000 0.540 0.024 0.004 0.364
#> GSM340298 3 0.4565 0.47146 0.000 0.432 0.532 0.036 0.000 0.000
#> GSM340299 5 0.5885 0.54050 0.000 0.168 0.012 0.264 0.552 0.004
#> GSM340301 5 0.0146 0.69379 0.000 0.000 0.004 0.000 0.996 0.000
#> GSM340303 5 0.7902 0.12792 0.000 0.308 0.200 0.080 0.356 0.056
#> GSM340304 6 0.3577 0.62441 0.012 0.048 0.064 0.000 0.036 0.840
#> GSM340306 2 0.4136 0.13546 0.012 0.560 0.000 0.000 0.000 0.428
#> GSM340307 3 0.4561 0.47231 0.000 0.428 0.536 0.036 0.000 0.000
#> GSM340310 1 0.1075 0.76353 0.952 0.000 0.000 0.000 0.000 0.048
#> GSM340314 3 0.2231 0.63635 0.000 0.004 0.900 0.068 0.000 0.028
#> GSM340315 6 0.4146 0.60061 0.028 0.000 0.288 0.000 0.004 0.680
#> GSM340317 2 0.4521 0.15646 0.024 0.524 0.004 0.000 0.000 0.448
#> GSM340318 4 0.3847 0.96297 0.000 0.456 0.000 0.544 0.000 0.000
#> GSM340319 4 0.3847 0.96297 0.000 0.456 0.000 0.544 0.000 0.000
#> GSM340320 2 0.4357 0.12801 0.012 0.560 0.000 0.008 0.000 0.420
#> GSM340321 3 0.3333 0.58548 0.000 0.000 0.784 0.000 0.192 0.024
#> GSM340322 4 0.4401 0.95220 0.000 0.464 0.000 0.512 0.000 0.024
#> GSM340324 1 0.3668 0.62475 0.668 0.000 0.000 0.000 0.004 0.328
#> GSM340328 1 0.1327 0.76511 0.936 0.000 0.000 0.000 0.000 0.064
#> GSM340330 3 0.0806 0.65349 0.000 0.000 0.972 0.008 0.000 0.020
#> GSM340332 4 0.4884 0.91159 0.000 0.460 0.004 0.488 0.000 0.048
#> GSM340333 5 0.3476 0.58918 0.000 0.000 0.004 0.260 0.732 0.004
#> GSM340336 4 0.3847 0.96297 0.000 0.456 0.000 0.544 0.000 0.000
#> GSM340337 2 0.4468 0.09301 0.000 0.560 0.000 0.032 0.000 0.408
#> GSM340338 3 0.1364 0.65714 0.000 0.012 0.952 0.016 0.000 0.020
#> GSM340339 2 0.5153 -0.83558 0.000 0.464 0.000 0.452 0.000 0.084
#> GSM340340 2 0.5763 0.05270 0.108 0.560 0.016 0.008 0.000 0.308
#> GSM340341 2 0.4262 0.13562 0.004 0.560 0.012 0.000 0.000 0.424
#> GSM340343 2 0.4529 -0.89657 0.000 0.508 0.000 0.460 0.000 0.032
#> GSM340344 5 0.5561 0.45825 0.000 0.000 0.004 0.264 0.564 0.168
#> GSM340346 3 0.4565 0.47146 0.000 0.432 0.532 0.036 0.000 0.000
#> GSM340347 4 0.4256 0.95913 0.000 0.464 0.000 0.520 0.000 0.016
#> GSM340348 2 0.4178 0.13232 0.008 0.560 0.000 0.004 0.000 0.428
#> GSM340349 6 0.6671 0.19258 0.028 0.344 0.232 0.000 0.004 0.392
#> GSM340350 2 0.6914 -0.29002 0.072 0.412 0.196 0.000 0.000 0.320
#> GSM340351 1 0.3151 0.70170 0.748 0.000 0.000 0.000 0.000 0.252
#> GSM340354 3 0.2622 0.63315 0.000 0.000 0.868 0.004 0.104 0.024
#> GSM340356 1 0.3810 0.48434 0.572 0.000 0.000 0.000 0.000 0.428
#> GSM340357 1 0.4109 0.30170 0.596 0.008 0.000 0.000 0.004 0.392
#> GSM348183 1 0.4787 0.41085 0.516 0.052 0.000 0.000 0.000 0.432
#> GSM348191 3 0.2106 0.64149 0.000 0.000 0.904 0.000 0.064 0.032
#> GSM348193 1 0.5372 0.37341 0.484 0.112 0.000 0.000 0.000 0.404
#> GSM537578 3 0.6827 -0.07565 0.020 0.288 0.348 0.000 0.012 0.332
#> GSM348181 6 0.3251 0.61931 0.008 0.000 0.124 0.000 0.040 0.828
#> GSM348182 5 0.4309 0.62568 0.000 0.000 0.176 0.008 0.736 0.080
#> GSM348184 3 0.2249 0.64107 0.000 0.032 0.900 0.064 0.000 0.004
#> GSM348185 2 0.4565 0.14965 0.036 0.532 0.000 0.000 0.000 0.432
#> GSM348186 5 0.0777 0.69770 0.000 0.000 0.024 0.000 0.972 0.004
#> GSM348187 1 0.1863 0.76397 0.896 0.000 0.000 0.000 0.000 0.104
#> GSM348188 2 0.4051 0.13572 0.008 0.560 0.000 0.000 0.000 0.432
#> GSM348189 5 0.4015 0.42763 0.000 0.000 0.372 0.012 0.616 0.000
#> GSM348190 5 0.3012 0.56416 0.000 0.000 0.000 0.008 0.796 0.196
#> GSM348194 1 0.3221 0.69416 0.736 0.000 0.000 0.000 0.000 0.264
#> GSM348195 1 0.1814 0.76507 0.900 0.000 0.000 0.000 0.000 0.100
#> GSM348196 1 0.3101 0.71117 0.756 0.000 0.000 0.000 0.000 0.244
#> GSM537585 6 0.6753 0.24169 0.300 0.000 0.060 0.000 0.196 0.444
#> GSM537594 1 0.3756 0.51919 0.600 0.000 0.000 0.000 0.000 0.400
#> GSM537596 6 0.3499 0.19447 0.320 0.000 0.000 0.000 0.000 0.680
#> GSM537597 6 0.3607 0.14004 0.348 0.000 0.000 0.000 0.000 0.652
#> GSM537602 1 0.5768 0.45032 0.576 0.108 0.020 0.000 0.008 0.288
#> GSM340184 3 0.0891 0.65249 0.000 0.000 0.968 0.008 0.000 0.024
#> GSM340185 3 0.4620 0.47409 0.000 0.428 0.532 0.040 0.000 0.000
#> GSM340186 3 0.1036 0.65385 0.000 0.004 0.964 0.024 0.000 0.008
#> GSM340187 4 0.4529 0.95209 0.000 0.460 0.004 0.512 0.000 0.024
#> GSM340189 4 0.5134 0.91583 0.000 0.464 0.032 0.476 0.000 0.028
#> GSM340190 3 0.4731 0.47385 0.000 0.428 0.524 0.048 0.000 0.000
#> GSM340191 3 0.0777 0.65198 0.000 0.000 0.972 0.004 0.000 0.024
#> GSM340192 2 0.7389 -0.19317 0.000 0.416 0.128 0.100 0.328 0.028
#> GSM340193 6 0.4863 -0.15310 0.412 0.000 0.000 0.000 0.060 0.528
#> GSM340194 5 0.1753 0.68093 0.000 0.000 0.004 0.084 0.912 0.000
#> GSM340195 5 0.3499 0.58633 0.000 0.000 0.004 0.264 0.728 0.004
#> GSM340196 4 0.3847 0.96297 0.000 0.456 0.000 0.544 0.000 0.000
#> GSM340197 5 0.1858 0.69592 0.000 0.000 0.076 0.012 0.912 0.000
#> GSM340198 5 0.4602 0.22144 0.000 0.000 0.484 0.028 0.484 0.004
#> GSM340199 3 0.4561 0.47604 0.000 0.428 0.536 0.036 0.000 0.000
#> GSM340200 3 0.4565 0.47146 0.000 0.432 0.532 0.036 0.000 0.000
#> GSM340201 4 0.4256 0.95913 0.000 0.464 0.000 0.520 0.000 0.016
#> GSM340202 4 0.3847 0.96297 0.000 0.456 0.000 0.544 0.000 0.000
#> GSM340203 2 0.6061 -0.54777 0.000 0.464 0.004 0.332 0.004 0.196
#> GSM340204 5 0.0405 0.69352 0.000 0.000 0.004 0.008 0.988 0.000
#> GSM340205 6 0.3627 0.46092 0.224 0.000 0.020 0.000 0.004 0.752
#> GSM340206 3 0.1411 0.64277 0.000 0.004 0.936 0.060 0.000 0.000
#> GSM340207 3 0.5894 -0.02723 0.000 0.008 0.516 0.084 0.364 0.028
#> GSM340237 2 0.7237 -0.27199 0.120 0.412 0.208 0.000 0.000 0.260
#> GSM340238 3 0.1841 0.64046 0.000 0.008 0.920 0.064 0.000 0.008
#> GSM340239 5 0.2738 0.65134 0.000 0.000 0.176 0.000 0.820 0.004
#> GSM340240 2 0.7306 -0.18962 0.000 0.428 0.116 0.100 0.328 0.028
#> GSM340241 3 0.3891 0.53897 0.000 0.008 0.808 0.056 0.104 0.024
#> GSM340242 3 0.5137 -0.08972 0.000 0.004 0.536 0.036 0.404 0.020
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
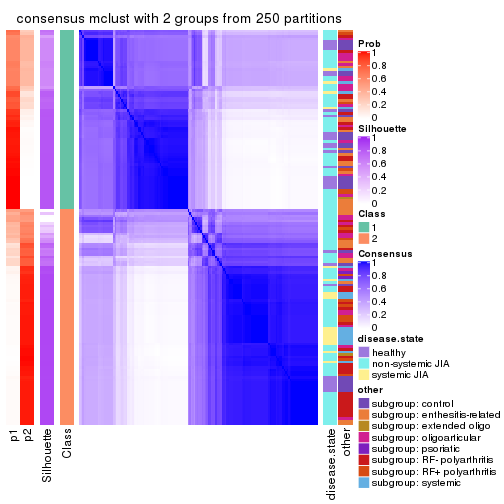
consensus_heatmap(res, k = 3)
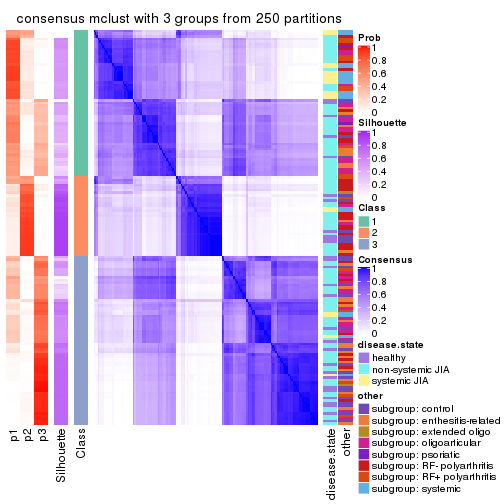
consensus_heatmap(res, k = 4)
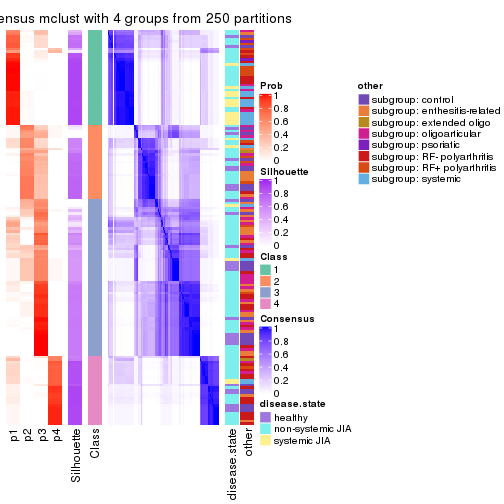
consensus_heatmap(res, k = 5)
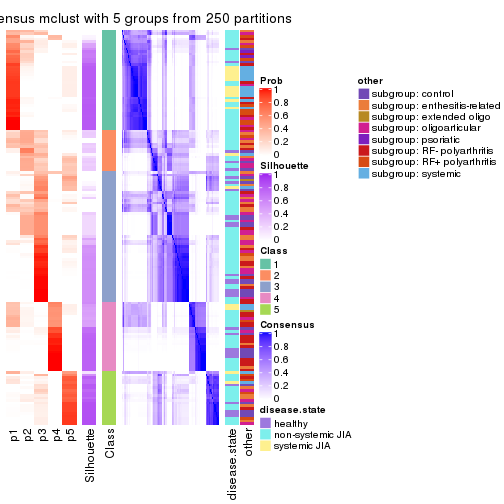
consensus_heatmap(res, k = 6)
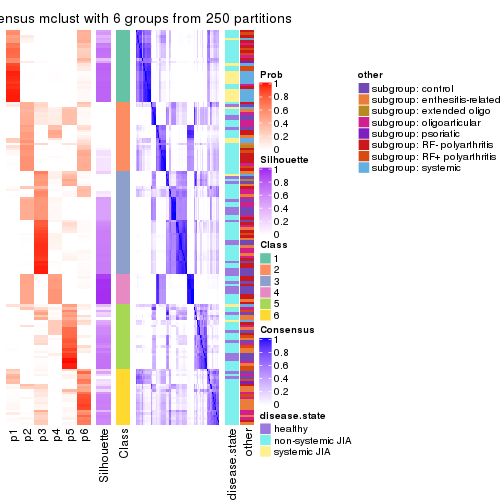
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
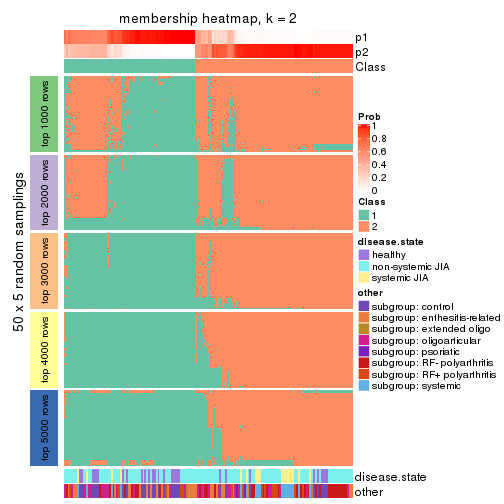
membership_heatmap(res, k = 3)
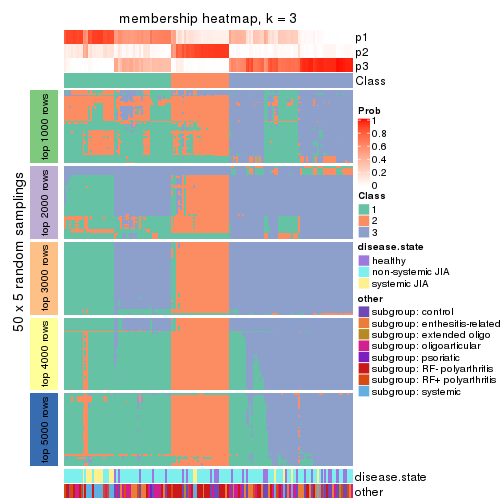
membership_heatmap(res, k = 4)
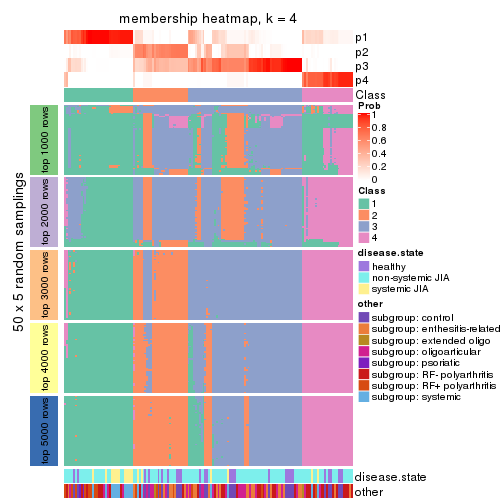
membership_heatmap(res, k = 5)
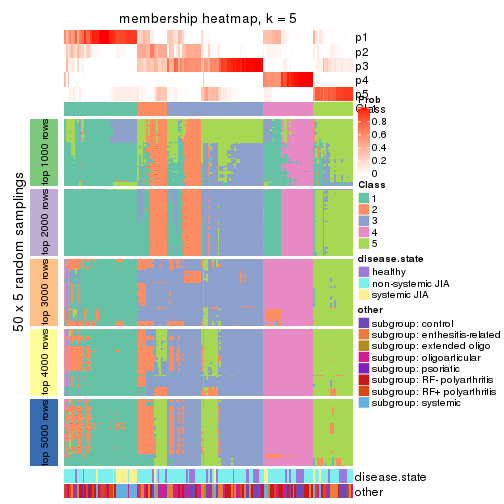
membership_heatmap(res, k = 6)
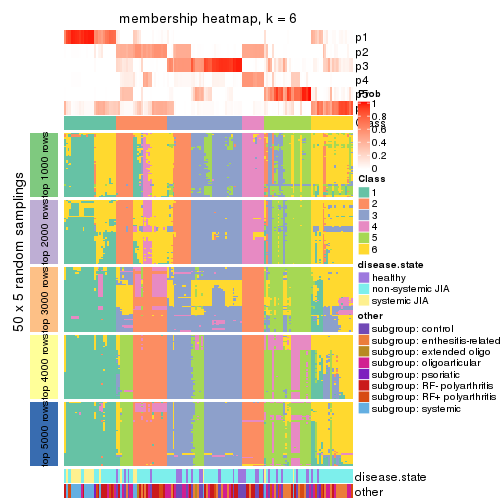
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
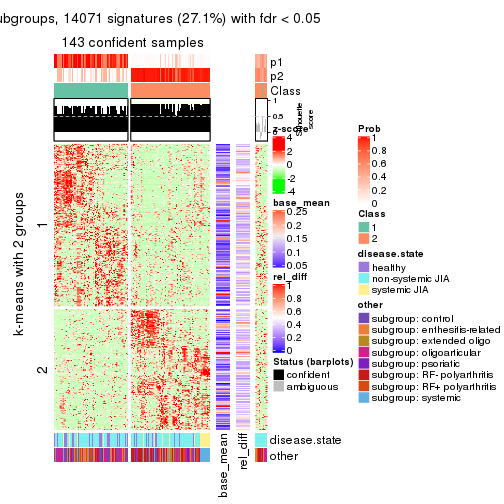
get_signatures(res, k = 3)
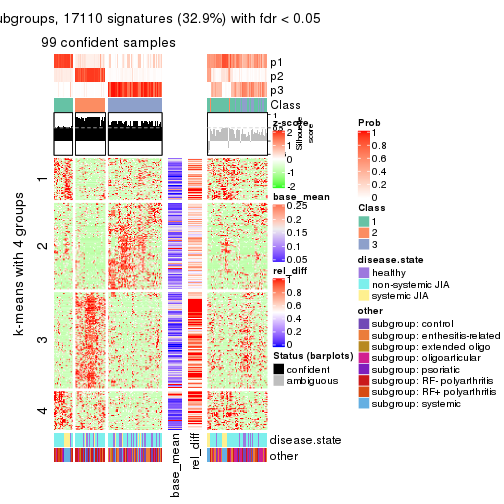
get_signatures(res, k = 4)
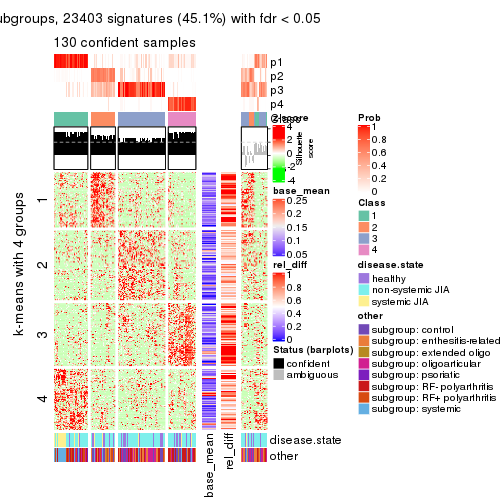
get_signatures(res, k = 5)
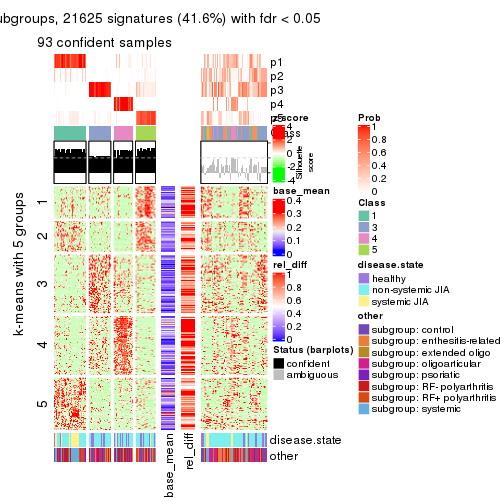
get_signatures(res, k = 6)
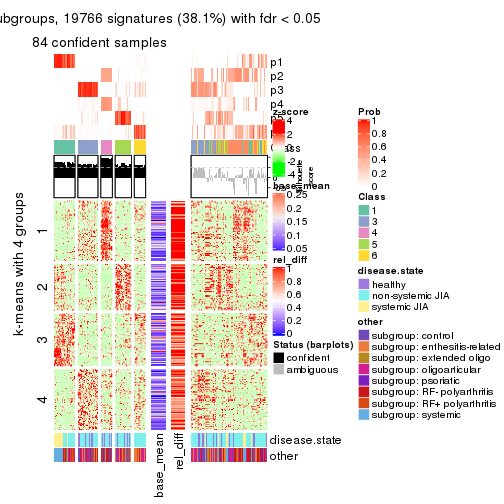
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
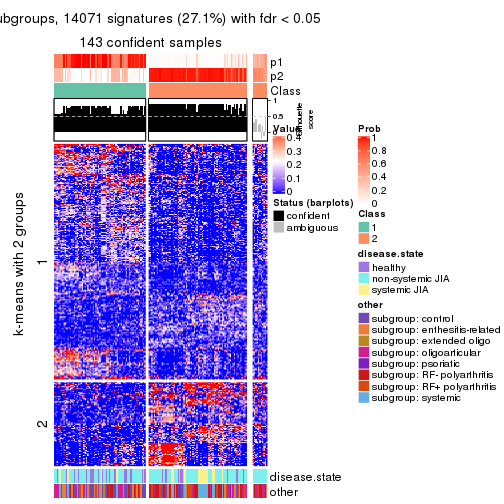
get_signatures(res, k = 3, scale_rows = FALSE)
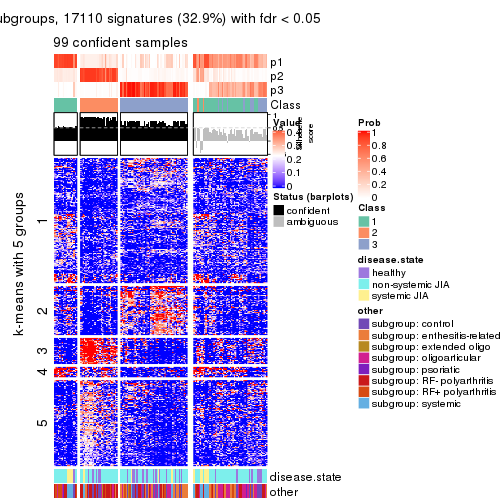
get_signatures(res, k = 4, scale_rows = FALSE)
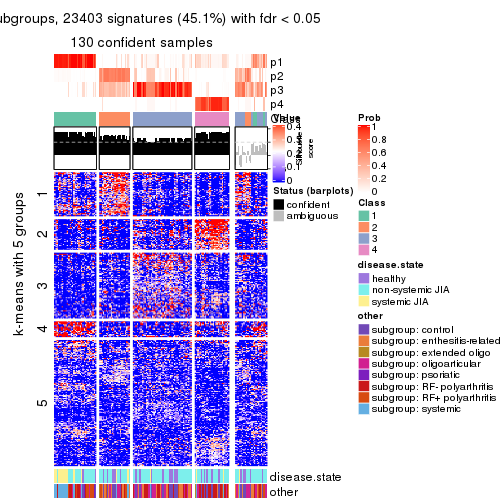
get_signatures(res, k = 5, scale_rows = FALSE)
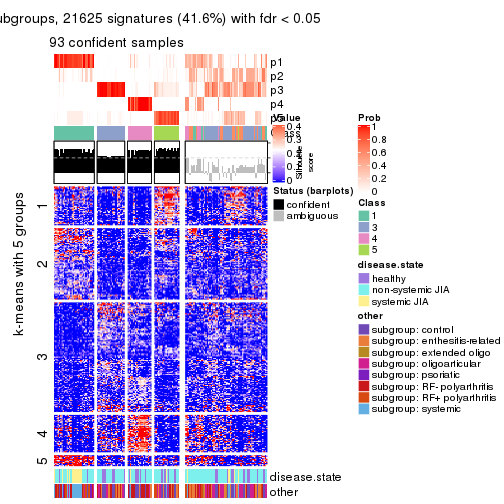
get_signatures(res, k = 6, scale_rows = FALSE)
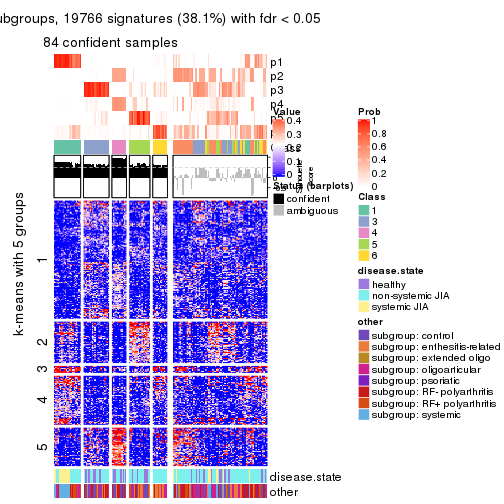
Compare the overlap of signatures from different k:
compare_signatures(res)
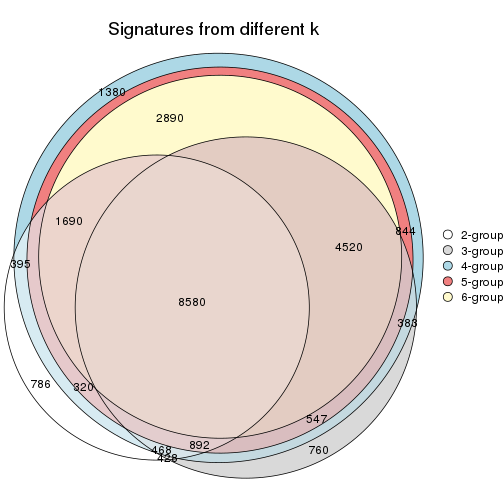
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
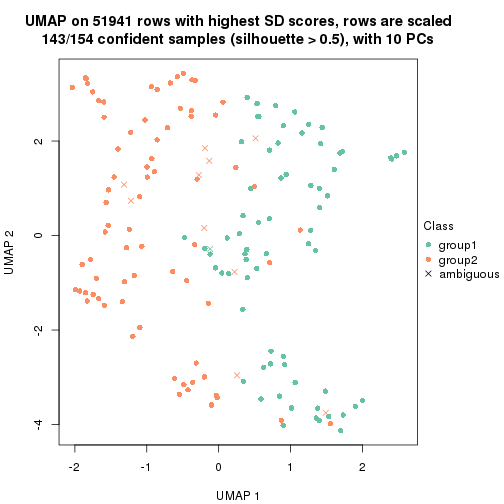
dimension_reduction(res, k = 3, method = "UMAP")
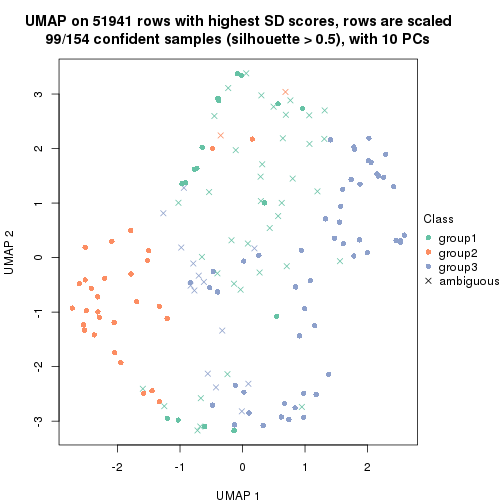
dimension_reduction(res, k = 4, method = "UMAP")
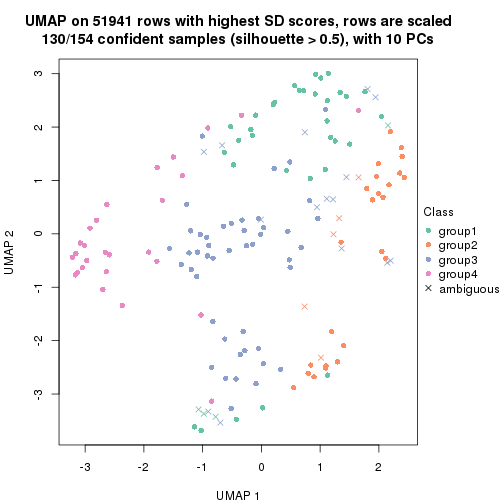
dimension_reduction(res, k = 5, method = "UMAP")
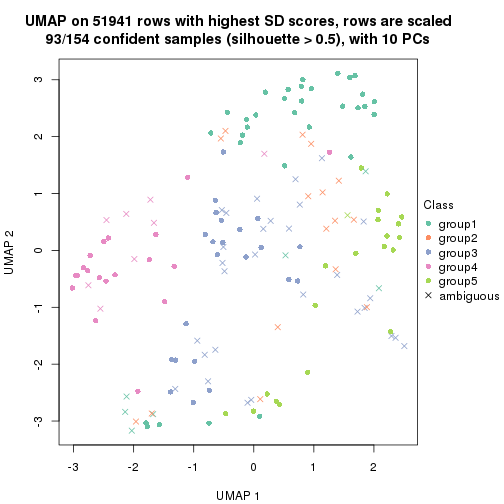
dimension_reduction(res, k = 6, method = "UMAP")
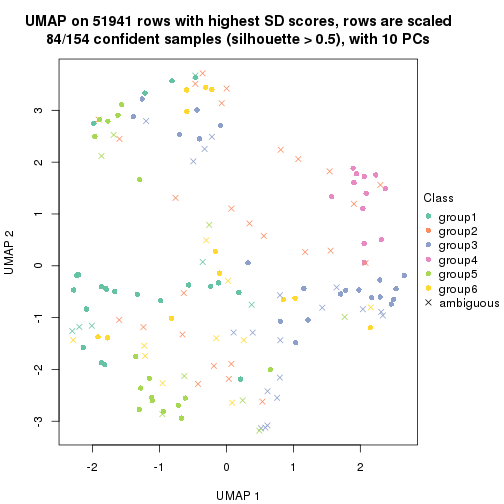
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
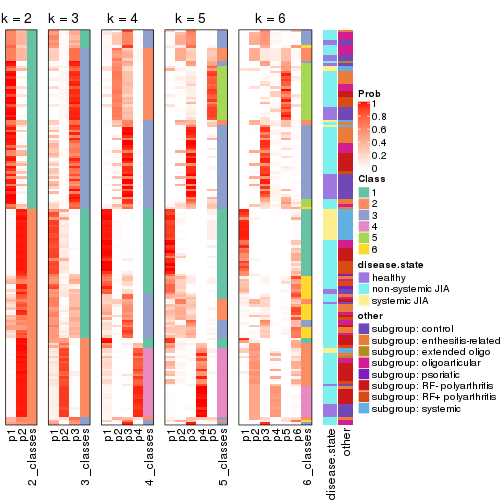
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> SD:mclust 143 6.94e-03 1.44e-03 2
#> SD:mclust 99 7.39e-02 6.46e-04 3
#> SD:mclust 130 5.91e-04 1.16e-06 4
#> SD:mclust 93 1.09e-03 2.48e-04 5
#> SD:mclust 84 2.67e-07 6.13e-08 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["SD", "NMF"]
# you can also extract it by
# res = res_list["SD:NMF"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'SD' method.
#> Subgroups are detected by 'NMF' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
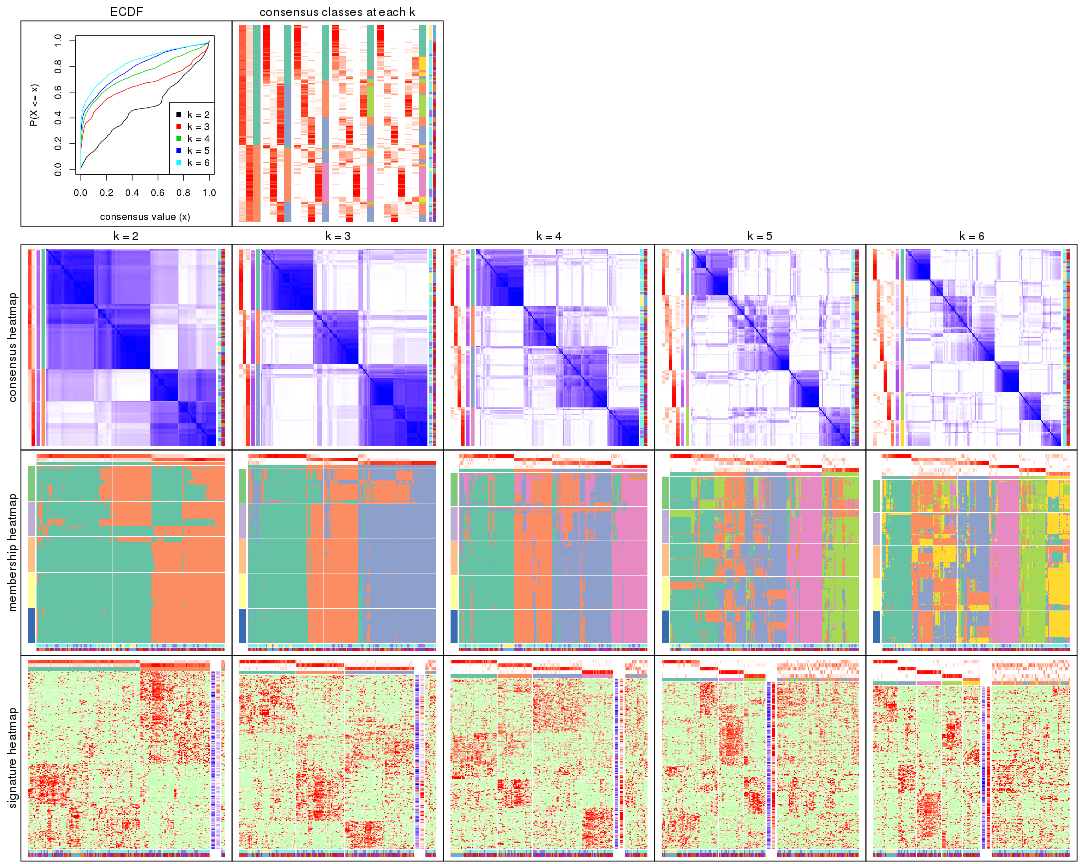
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
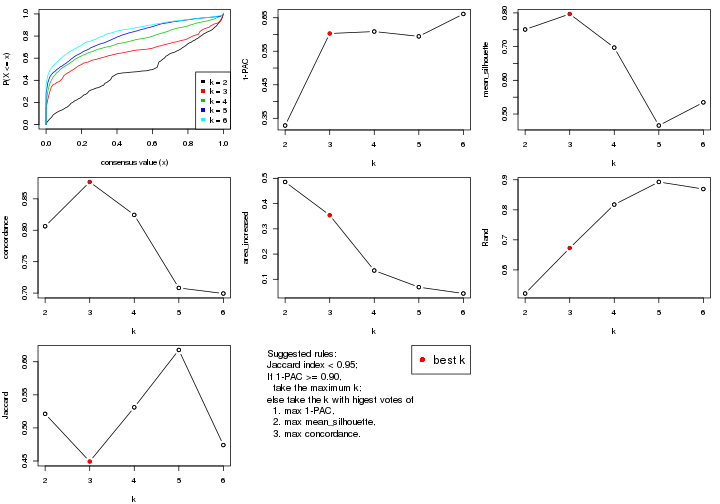
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.328 0.751 0.806 0.4856 0.521 0.521
#> 3 3 0.603 0.797 0.877 0.3539 0.673 0.449
#> 4 4 0.609 0.697 0.824 0.1348 0.817 0.531
#> 5 5 0.594 0.466 0.708 0.0690 0.893 0.618
#> 6 6 0.661 0.535 0.699 0.0438 0.869 0.474
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 1 0.7883 0.734 0.764 0.236
#> GSM340359 1 0.6973 0.753 0.812 0.188
#> GSM340361 1 0.7815 0.736 0.768 0.232
#> GSM340362 1 0.5059 0.773 0.888 0.112
#> GSM340363 1 0.7950 0.734 0.760 0.240
#> GSM340364 1 0.4161 0.778 0.916 0.084
#> GSM340365 1 0.2603 0.783 0.956 0.044
#> GSM340366 1 0.5519 0.767 0.872 0.128
#> GSM340367 1 0.7815 0.736 0.768 0.232
#> GSM340368 2 0.5629 0.717 0.132 0.868
#> GSM340369 2 0.4431 0.752 0.092 0.908
#> GSM340370 1 0.7883 0.734 0.764 0.236
#> GSM340371 2 0.8443 0.738 0.272 0.728
#> GSM340372 1 0.7883 0.734 0.764 0.236
#> GSM340373 1 0.7453 0.744 0.788 0.212
#> GSM340375 1 0.7883 0.734 0.764 0.236
#> GSM340376 1 0.7883 0.734 0.764 0.236
#> GSM340378 1 0.7376 0.746 0.792 0.208
#> GSM340243 1 0.4815 0.788 0.896 0.104
#> GSM340244 1 0.8813 0.709 0.700 0.300
#> GSM340246 1 0.5737 0.764 0.864 0.136
#> GSM340247 2 0.0376 0.802 0.004 0.996
#> GSM340248 1 0.7139 0.772 0.804 0.196
#> GSM340249 2 0.4690 0.803 0.100 0.900
#> GSM340250 1 0.7950 0.734 0.760 0.240
#> GSM340251 2 0.7815 0.768 0.232 0.768
#> GSM340252 2 0.5294 0.802 0.120 0.880
#> GSM340253 1 0.4815 0.783 0.896 0.104
#> GSM340254 1 0.5629 0.766 0.868 0.132
#> GSM340256 1 0.4815 0.782 0.896 0.104
#> GSM340258 1 0.8144 0.611 0.748 0.252
#> GSM340259 1 0.5408 0.778 0.876 0.124
#> GSM340260 1 0.2778 0.788 0.952 0.048
#> GSM340261 2 0.8016 0.762 0.244 0.756
#> GSM340262 1 0.6048 0.764 0.852 0.148
#> GSM340263 1 0.7674 0.763 0.776 0.224
#> GSM340264 1 0.5737 0.764 0.864 0.136
#> GSM340265 1 0.5629 0.764 0.868 0.132
#> GSM340266 2 0.4298 0.804 0.088 0.912
#> GSM340267 1 0.5842 0.772 0.860 0.140
#> GSM340268 2 0.7745 0.770 0.228 0.772
#> GSM340269 1 0.6973 0.731 0.812 0.188
#> GSM340270 1 0.7815 0.737 0.768 0.232
#> GSM537574 1 0.5842 0.772 0.860 0.140
#> GSM537580 2 0.7883 0.766 0.236 0.764
#> GSM537581 2 0.5629 0.715 0.132 0.868
#> GSM340272 1 0.4562 0.779 0.904 0.096
#> GSM340273 1 0.7950 0.734 0.760 0.240
#> GSM340275 2 0.3114 0.807 0.056 0.944
#> GSM340276 1 0.8081 0.753 0.752 0.248
#> GSM340277 1 0.5519 0.767 0.872 0.128
#> GSM340278 2 0.7528 0.777 0.216 0.784
#> GSM340279 2 0.4022 0.780 0.080 0.920
#> GSM340282 1 0.5519 0.767 0.872 0.128
#> GSM340284 2 0.7674 0.771 0.224 0.776
#> GSM340285 2 0.4815 0.760 0.104 0.896
#> GSM340286 1 0.5946 0.758 0.856 0.144
#> GSM340287 1 0.7056 0.752 0.808 0.192
#> GSM340288 1 0.7950 0.734 0.760 0.240
#> GSM340289 1 0.6712 0.759 0.824 0.176
#> GSM340290 2 0.7528 0.777 0.216 0.784
#> GSM340291 1 0.4298 0.785 0.912 0.088
#> GSM340293 2 0.7602 0.777 0.220 0.780
#> GSM340294 2 0.8861 0.709 0.304 0.696
#> GSM340296 1 0.5519 0.767 0.872 0.128
#> GSM340297 2 0.6148 0.706 0.152 0.848
#> GSM340298 2 0.9286 0.653 0.344 0.656
#> GSM340299 1 0.5519 0.767 0.872 0.128
#> GSM340301 1 0.5629 0.766 0.868 0.132
#> GSM340303 1 0.5519 0.767 0.872 0.128
#> GSM340304 1 0.4690 0.790 0.900 0.100
#> GSM340306 2 0.5737 0.713 0.136 0.864
#> GSM340307 1 0.5629 0.764 0.868 0.132
#> GSM340310 1 0.7883 0.734 0.764 0.236
#> GSM340314 2 0.7376 0.780 0.208 0.792
#> GSM340315 2 0.8713 0.488 0.292 0.708
#> GSM340317 2 0.4431 0.751 0.092 0.908
#> GSM340318 2 0.0000 0.801 0.000 1.000
#> GSM340319 2 0.0000 0.801 0.000 1.000
#> GSM340320 2 0.5519 0.719 0.128 0.872
#> GSM340321 1 0.5737 0.764 0.864 0.136
#> GSM340322 2 0.1843 0.806 0.028 0.972
#> GSM340324 1 0.7376 0.746 0.792 0.208
#> GSM340328 1 0.7883 0.734 0.764 0.236
#> GSM340330 2 0.7883 0.766 0.236 0.764
#> GSM340332 2 0.0000 0.801 0.000 1.000
#> GSM340333 1 0.4939 0.776 0.892 0.108
#> GSM340336 2 0.0000 0.801 0.000 1.000
#> GSM340337 2 0.4298 0.753 0.088 0.912
#> GSM340338 2 0.7950 0.765 0.240 0.760
#> GSM340339 2 0.1843 0.789 0.028 0.972
#> GSM340340 2 0.4562 0.748 0.096 0.904
#> GSM340341 2 0.4298 0.753 0.088 0.912
#> GSM340343 2 0.2603 0.807 0.044 0.956
#> GSM340344 1 0.4431 0.785 0.908 0.092
#> GSM340346 1 0.9732 0.161 0.596 0.404
#> GSM340347 2 0.0376 0.800 0.004 0.996
#> GSM340348 2 0.5294 0.726 0.120 0.880
#> GSM340349 1 0.3584 0.780 0.932 0.068
#> GSM340350 1 0.0938 0.784 0.988 0.012
#> GSM340351 1 0.7950 0.734 0.760 0.240
#> GSM340354 1 0.6247 0.748 0.844 0.156
#> GSM340356 1 0.7950 0.734 0.760 0.240
#> GSM340357 1 0.7815 0.735 0.768 0.232
#> GSM348183 1 0.7950 0.734 0.760 0.240
#> GSM348191 2 0.7883 0.766 0.236 0.764
#> GSM348193 1 0.8443 0.704 0.728 0.272
#> GSM537578 1 0.2423 0.786 0.960 0.040
#> GSM348181 1 0.8499 0.728 0.724 0.276
#> GSM348182 1 0.5519 0.782 0.872 0.128
#> GSM348184 2 0.7883 0.766 0.236 0.764
#> GSM348185 2 0.5946 0.706 0.144 0.856
#> GSM348186 1 0.5629 0.766 0.868 0.132
#> GSM348187 1 0.7883 0.734 0.764 0.236
#> GSM348188 2 0.5946 0.707 0.144 0.856
#> GSM348189 1 0.7056 0.709 0.808 0.192
#> GSM348190 1 0.4815 0.775 0.896 0.104
#> GSM348194 1 0.7950 0.734 0.760 0.240
#> GSM348195 1 0.7883 0.734 0.764 0.236
#> GSM348196 1 0.7950 0.734 0.760 0.240
#> GSM537585 1 0.4562 0.774 0.904 0.096
#> GSM537594 1 0.7950 0.734 0.760 0.240
#> GSM537596 1 0.7950 0.734 0.760 0.240
#> GSM537597 1 0.7950 0.734 0.760 0.240
#> GSM537602 1 0.3733 0.779 0.928 0.072
#> GSM340184 2 0.7883 0.766 0.236 0.764
#> GSM340185 2 0.8207 0.755 0.256 0.744
#> GSM340186 2 0.7883 0.766 0.236 0.764
#> GSM340187 2 0.4815 0.803 0.104 0.896
#> GSM340189 2 0.5059 0.802 0.112 0.888
#> GSM340190 2 0.7950 0.765 0.240 0.760
#> GSM340191 2 0.7376 0.781 0.208 0.792
#> GSM340192 1 0.5519 0.767 0.872 0.128
#> GSM340193 1 0.7950 0.734 0.760 0.240
#> GSM340194 1 0.5629 0.766 0.868 0.132
#> GSM340195 1 0.4431 0.780 0.908 0.092
#> GSM340196 2 0.0000 0.801 0.000 1.000
#> GSM340197 1 0.5737 0.771 0.864 0.136
#> GSM340198 1 0.6048 0.755 0.852 0.148
#> GSM340199 2 0.8327 0.748 0.264 0.736
#> GSM340200 2 0.9922 0.403 0.448 0.552
#> GSM340201 2 0.3431 0.769 0.064 0.936
#> GSM340202 2 0.0000 0.801 0.000 1.000
#> GSM340203 2 0.1843 0.806 0.028 0.972
#> GSM340204 1 0.4562 0.780 0.904 0.096
#> GSM340205 1 0.7815 0.736 0.768 0.232
#> GSM340206 2 0.7883 0.766 0.236 0.764
#> GSM340207 1 0.6148 0.751 0.848 0.152
#> GSM340237 1 0.4161 0.781 0.916 0.084
#> GSM340238 2 0.7376 0.780 0.208 0.792
#> GSM340239 1 0.5629 0.766 0.868 0.132
#> GSM340240 1 0.5629 0.764 0.868 0.132
#> GSM340241 1 0.5842 0.761 0.860 0.140
#> GSM340242 1 0.7056 0.709 0.808 0.192
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.0424 0.8992 0.992 0.008 0.000
#> GSM340359 1 0.0000 0.8979 1.000 0.000 0.000
#> GSM340361 1 0.0592 0.8991 0.988 0.012 0.000
#> GSM340362 3 0.4654 0.7417 0.208 0.000 0.792
#> GSM340363 1 0.0892 0.8987 0.980 0.020 0.000
#> GSM340364 1 0.0592 0.8947 0.988 0.000 0.012
#> GSM340365 1 0.5835 0.5514 0.660 0.000 0.340
#> GSM340366 3 0.1620 0.8591 0.012 0.024 0.964
#> GSM340367 1 0.0237 0.8968 0.996 0.000 0.004
#> GSM340368 2 0.4452 0.7604 0.192 0.808 0.000
#> GSM340369 2 0.4575 0.7775 0.184 0.812 0.004
#> GSM340370 1 0.0000 0.8979 1.000 0.000 0.000
#> GSM340371 3 0.1529 0.8658 0.000 0.040 0.960
#> GSM340372 1 0.0000 0.8979 1.000 0.000 0.000
#> GSM340373 1 0.0237 0.8968 0.996 0.000 0.004
#> GSM340375 1 0.0592 0.8991 0.988 0.012 0.000
#> GSM340376 1 0.0000 0.8979 1.000 0.000 0.000
#> GSM340378 1 0.0237 0.8968 0.996 0.000 0.004
#> GSM340243 1 0.5951 0.7349 0.764 0.040 0.196
#> GSM340244 1 0.7205 0.6438 0.708 0.100 0.192
#> GSM340246 3 0.3213 0.8705 0.008 0.092 0.900
#> GSM340247 2 0.0237 0.8689 0.000 0.996 0.004
#> GSM340248 3 0.7199 0.7003 0.204 0.092 0.704
#> GSM340249 2 0.2486 0.8493 0.008 0.932 0.060
#> GSM340250 1 0.1337 0.8934 0.972 0.012 0.016
#> GSM340251 2 0.4121 0.7728 0.000 0.832 0.168
#> GSM340252 2 0.3213 0.8311 0.008 0.900 0.092
#> GSM340253 3 0.3377 0.8697 0.012 0.092 0.896
#> GSM340254 3 0.3112 0.8701 0.004 0.096 0.900
#> GSM340256 3 0.3377 0.8697 0.012 0.092 0.896
#> GSM340258 3 0.2878 0.8703 0.000 0.096 0.904
#> GSM340259 3 0.8720 0.1918 0.412 0.108 0.480
#> GSM340260 3 0.3832 0.8575 0.076 0.036 0.888
#> GSM340261 3 0.3500 0.8700 0.004 0.116 0.880
#> GSM340262 3 0.3375 0.8682 0.008 0.100 0.892
#> GSM340263 1 0.5505 0.7598 0.816 0.096 0.088
#> GSM340264 3 0.2878 0.8715 0.000 0.096 0.904
#> GSM340265 3 0.0237 0.8631 0.000 0.004 0.996
#> GSM340266 2 0.1643 0.8572 0.000 0.956 0.044
#> GSM340267 3 0.2689 0.8669 0.036 0.032 0.932
#> GSM340268 2 0.5465 0.5954 0.000 0.712 0.288
#> GSM340269 3 0.4110 0.8497 0.004 0.152 0.844
#> GSM340270 1 0.1015 0.8962 0.980 0.008 0.012
#> GSM537574 3 0.3112 0.8701 0.004 0.096 0.900
#> GSM537580 3 0.4291 0.8391 0.000 0.180 0.820
#> GSM537581 2 0.6026 0.5061 0.376 0.624 0.000
#> GSM340272 3 0.1620 0.8591 0.012 0.024 0.964
#> GSM340273 1 0.0892 0.8987 0.980 0.020 0.000
#> GSM340275 2 0.1399 0.8615 0.004 0.968 0.028
#> GSM340276 1 0.8532 0.5109 0.608 0.168 0.224
#> GSM340277 3 0.1337 0.8592 0.012 0.016 0.972
#> GSM340278 2 0.5873 0.5598 0.004 0.684 0.312
#> GSM340279 2 0.5884 0.7845 0.064 0.788 0.148
#> GSM340282 3 0.1620 0.8591 0.012 0.024 0.964
#> GSM340284 2 0.5431 0.6026 0.000 0.716 0.284
#> GSM340285 2 0.3295 0.8232 0.008 0.896 0.096
#> GSM340286 3 0.4062 0.8494 0.000 0.164 0.836
#> GSM340287 1 0.8070 -0.0388 0.472 0.064 0.464
#> GSM340288 1 0.0892 0.8987 0.980 0.020 0.000
#> GSM340289 1 0.0661 0.8996 0.988 0.008 0.004
#> GSM340290 2 0.5497 0.5572 0.000 0.708 0.292
#> GSM340291 3 0.6879 0.3963 0.360 0.024 0.616
#> GSM340293 2 0.5122 0.7658 0.012 0.788 0.200
#> GSM340294 3 0.3030 0.8413 0.004 0.092 0.904
#> GSM340296 3 0.1620 0.8591 0.012 0.024 0.964
#> GSM340297 2 0.7069 0.2569 0.472 0.508 0.020
#> GSM340298 3 0.1620 0.8591 0.012 0.024 0.964
#> GSM340299 3 0.1620 0.8591 0.012 0.024 0.964
#> GSM340301 3 0.3619 0.8578 0.000 0.136 0.864
#> GSM340303 3 0.2116 0.8567 0.012 0.040 0.948
#> GSM340304 1 0.6441 0.6480 0.696 0.028 0.276
#> GSM340306 2 0.5678 0.6050 0.316 0.684 0.000
#> GSM340307 3 0.1620 0.8591 0.012 0.024 0.964
#> GSM340310 1 0.0592 0.8991 0.988 0.012 0.000
#> GSM340314 2 0.1031 0.8628 0.000 0.976 0.024
#> GSM340315 1 0.0892 0.8987 0.980 0.020 0.000
#> GSM340317 2 0.5760 0.4628 0.328 0.672 0.000
#> GSM340318 2 0.0237 0.8708 0.004 0.996 0.000
#> GSM340319 2 0.0237 0.8708 0.004 0.996 0.000
#> GSM340320 2 0.3752 0.7955 0.144 0.856 0.000
#> GSM340321 3 0.1529 0.8669 0.000 0.040 0.960
#> GSM340322 2 0.0237 0.8708 0.004 0.996 0.000
#> GSM340324 1 0.0424 0.8992 0.992 0.008 0.000
#> GSM340328 1 0.0892 0.8987 0.980 0.020 0.000
#> GSM340330 3 0.3941 0.8557 0.000 0.156 0.844
#> GSM340332 2 0.0237 0.8708 0.004 0.996 0.000
#> GSM340333 3 0.3377 0.8697 0.012 0.092 0.896
#> GSM340336 2 0.0237 0.8708 0.004 0.996 0.000
#> GSM340337 2 0.2625 0.8355 0.084 0.916 0.000
#> GSM340338 3 0.2878 0.8703 0.000 0.096 0.904
#> GSM340339 2 0.0424 0.8699 0.008 0.992 0.000
#> GSM340340 2 0.4235 0.7845 0.176 0.824 0.000
#> GSM340341 2 0.2711 0.8336 0.088 0.912 0.000
#> GSM340343 2 0.0237 0.8696 0.000 0.996 0.004
#> GSM340344 3 0.3445 0.8700 0.016 0.088 0.896
#> GSM340346 3 0.1620 0.8591 0.012 0.024 0.964
#> GSM340347 2 0.0000 0.8699 0.000 1.000 0.000
#> GSM340348 2 0.3116 0.8169 0.108 0.892 0.000
#> GSM340349 1 0.6570 0.5808 0.668 0.024 0.308
#> GSM340350 3 0.7152 0.0654 0.444 0.024 0.532
#> GSM340351 1 0.0592 0.8991 0.988 0.012 0.000
#> GSM340354 3 0.3412 0.8548 0.000 0.124 0.876
#> GSM340356 1 0.0892 0.8987 0.980 0.020 0.000
#> GSM340357 1 0.2261 0.8641 0.932 0.000 0.068
#> GSM348183 1 0.1163 0.8958 0.972 0.028 0.000
#> GSM348191 3 0.6274 0.3501 0.000 0.456 0.544
#> GSM348193 1 0.0892 0.8987 0.980 0.020 0.000
#> GSM537578 3 0.1620 0.8591 0.012 0.024 0.964
#> GSM348181 1 0.5200 0.7453 0.796 0.184 0.020
#> GSM348182 1 0.8229 0.5317 0.620 0.256 0.124
#> GSM348184 3 0.5138 0.7242 0.000 0.252 0.748
#> GSM348185 1 0.3340 0.8250 0.880 0.120 0.000
#> GSM348186 3 0.3030 0.8710 0.004 0.092 0.904
#> GSM348187 1 0.0892 0.8987 0.980 0.020 0.000
#> GSM348188 2 0.5058 0.6852 0.244 0.756 0.000
#> GSM348189 3 0.4178 0.8483 0.000 0.172 0.828
#> GSM348190 3 0.8196 0.5221 0.284 0.108 0.608
#> GSM348194 1 0.0892 0.8987 0.980 0.020 0.000
#> GSM348195 1 0.0747 0.8990 0.984 0.016 0.000
#> GSM348196 1 0.0892 0.8987 0.980 0.020 0.000
#> GSM537585 1 0.5365 0.7060 0.744 0.004 0.252
#> GSM537594 1 0.0892 0.8987 0.980 0.020 0.000
#> GSM537596 1 0.2356 0.8666 0.928 0.072 0.000
#> GSM537597 1 0.0892 0.8987 0.980 0.020 0.000
#> GSM537602 1 0.3921 0.8223 0.872 0.016 0.112
#> GSM340184 3 0.5497 0.7149 0.000 0.292 0.708
#> GSM340185 3 0.1620 0.8591 0.012 0.024 0.964
#> GSM340186 3 0.3213 0.8705 0.008 0.092 0.900
#> GSM340187 2 0.0592 0.8658 0.000 0.988 0.012
#> GSM340189 2 0.0747 0.8671 0.000 0.984 0.016
#> GSM340190 3 0.1585 0.8601 0.008 0.028 0.964
#> GSM340191 2 0.3682 0.8159 0.008 0.876 0.116
#> GSM340192 3 0.1620 0.8591 0.012 0.024 0.964
#> GSM340193 1 0.2537 0.8613 0.920 0.080 0.000
#> GSM340194 3 0.3879 0.8504 0.000 0.152 0.848
#> GSM340195 3 0.3816 0.8531 0.000 0.148 0.852
#> GSM340196 2 0.0237 0.8708 0.004 0.996 0.000
#> GSM340197 3 0.3295 0.8695 0.008 0.096 0.896
#> GSM340198 3 0.3295 0.8695 0.008 0.096 0.896
#> GSM340199 3 0.1620 0.8591 0.012 0.024 0.964
#> GSM340200 3 0.1453 0.8602 0.008 0.024 0.968
#> GSM340201 2 0.1163 0.8633 0.028 0.972 0.000
#> GSM340202 2 0.0237 0.8708 0.004 0.996 0.000
#> GSM340203 2 0.0237 0.8708 0.004 0.996 0.000
#> GSM340204 3 0.4110 0.8501 0.004 0.152 0.844
#> GSM340205 1 0.0661 0.8960 0.988 0.004 0.008
#> GSM340206 3 0.6095 0.4983 0.000 0.392 0.608
#> GSM340207 3 0.2878 0.8703 0.000 0.096 0.904
#> GSM340237 3 0.6675 0.2141 0.404 0.012 0.584
#> GSM340238 2 0.1031 0.8628 0.000 0.976 0.024
#> GSM340239 3 0.2959 0.8711 0.000 0.100 0.900
#> GSM340240 3 0.1620 0.8591 0.012 0.024 0.964
#> GSM340241 3 0.2796 0.8710 0.000 0.092 0.908
#> GSM340242 3 0.2796 0.8719 0.000 0.092 0.908
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.0188 0.8518 0.996 0.004 0.000 0.000
#> GSM340359 1 0.4776 0.3403 0.624 0.376 0.000 0.000
#> GSM340361 1 0.0336 0.8513 0.992 0.008 0.000 0.000
#> GSM340362 2 0.3328 0.7811 0.100 0.872 0.024 0.004
#> GSM340363 1 0.1510 0.8486 0.956 0.028 0.000 0.016
#> GSM340364 1 0.0188 0.8518 0.996 0.004 0.000 0.000
#> GSM340365 2 0.5274 0.6475 0.036 0.724 0.232 0.008
#> GSM340366 3 0.1305 0.7330 0.004 0.036 0.960 0.000
#> GSM340367 1 0.0188 0.8518 0.996 0.004 0.000 0.000
#> GSM340368 4 0.4222 0.6616 0.272 0.000 0.000 0.728
#> GSM340369 4 0.4378 0.7623 0.164 0.000 0.040 0.796
#> GSM340370 1 0.0188 0.8518 0.996 0.004 0.000 0.000
#> GSM340371 2 0.5478 0.5517 0.000 0.628 0.344 0.028
#> GSM340372 1 0.0188 0.8518 0.996 0.004 0.000 0.000
#> GSM340373 1 0.0188 0.8518 0.996 0.004 0.000 0.000
#> GSM340375 1 0.0188 0.8518 0.996 0.004 0.000 0.000
#> GSM340376 1 0.0188 0.8518 0.996 0.004 0.000 0.000
#> GSM340378 1 0.0188 0.8518 0.996 0.004 0.000 0.000
#> GSM340243 1 0.6158 0.3750 0.548 0.036 0.408 0.008
#> GSM340244 1 0.7310 0.4455 0.568 0.292 0.120 0.020
#> GSM340246 3 0.4797 0.6508 0.000 0.260 0.720 0.020
#> GSM340247 4 0.0564 0.8702 0.004 0.004 0.004 0.988
#> GSM340248 3 0.7160 0.5673 0.104 0.300 0.576 0.020
#> GSM340249 4 0.3172 0.7369 0.000 0.160 0.000 0.840
#> GSM340250 1 0.5809 0.5543 0.692 0.092 0.216 0.000
#> GSM340251 3 0.6469 0.6281 0.000 0.164 0.644 0.192
#> GSM340252 2 0.4644 0.6544 0.000 0.748 0.024 0.228
#> GSM340253 2 0.1968 0.8122 0.008 0.940 0.044 0.008
#> GSM340254 2 0.2021 0.8086 0.000 0.932 0.056 0.012
#> GSM340256 2 0.3123 0.7053 0.000 0.844 0.156 0.000
#> GSM340258 3 0.5371 0.5254 0.000 0.364 0.616 0.020
#> GSM340259 2 0.4017 0.7247 0.128 0.828 0.044 0.000
#> GSM340260 2 0.4980 0.5082 0.012 0.708 0.272 0.008
#> GSM340261 3 0.3166 0.7455 0.000 0.116 0.868 0.016
#> GSM340262 2 0.2413 0.8030 0.000 0.916 0.064 0.020
#> GSM340263 1 0.5512 0.5843 0.692 0.268 0.020 0.020
#> GSM340264 2 0.2256 0.8091 0.000 0.924 0.056 0.020
#> GSM340265 3 0.3444 0.7206 0.000 0.184 0.816 0.000
#> GSM340266 4 0.6083 0.4593 0.028 0.336 0.020 0.616
#> GSM340267 3 0.3584 0.7269 0.004 0.152 0.836 0.008
#> GSM340268 3 0.6236 0.6498 0.000 0.180 0.668 0.152
#> GSM340269 2 0.5384 0.3673 0.000 0.648 0.324 0.028
#> GSM340270 1 0.0672 0.8515 0.984 0.008 0.008 0.000
#> GSM537574 3 0.5055 0.5426 0.000 0.368 0.624 0.008
#> GSM537580 2 0.4644 0.7010 0.000 0.748 0.024 0.228
#> GSM537581 4 0.5386 0.4538 0.368 0.020 0.000 0.612
#> GSM340272 2 0.5412 0.5148 0.016 0.624 0.356 0.004
#> GSM340273 1 0.1488 0.8464 0.956 0.032 0.000 0.012
#> GSM340275 4 0.0817 0.8618 0.000 0.024 0.000 0.976
#> GSM340276 1 0.6455 0.5019 0.596 0.336 0.016 0.052
#> GSM340277 3 0.4072 0.4972 0.000 0.252 0.748 0.000
#> GSM340278 3 0.1296 0.7449 0.004 0.004 0.964 0.028
#> GSM340279 3 0.4990 0.7137 0.032 0.064 0.804 0.100
#> GSM340282 3 0.4431 0.3622 0.000 0.304 0.696 0.000
#> GSM340284 3 0.5417 0.6887 0.000 0.180 0.732 0.088
#> GSM340285 4 0.8522 -0.1009 0.392 0.056 0.152 0.400
#> GSM340286 2 0.3789 0.7832 0.004 0.836 0.020 0.140
#> GSM340287 3 0.6334 0.6331 0.192 0.068 0.700 0.040
#> GSM340288 1 0.2256 0.8411 0.924 0.056 0.000 0.020
#> GSM340289 1 0.3401 0.7687 0.840 0.152 0.000 0.008
#> GSM340290 3 0.5265 0.6890 0.000 0.092 0.748 0.160
#> GSM340291 3 0.6855 0.5961 0.152 0.216 0.624 0.008
#> GSM340293 3 0.1211 0.7402 0.000 0.000 0.960 0.040
#> GSM340294 3 0.3320 0.7344 0.000 0.056 0.876 0.068
#> GSM340296 3 0.0524 0.7379 0.004 0.008 0.988 0.000
#> GSM340297 3 0.5114 0.5905 0.280 0.020 0.696 0.004
#> GSM340298 3 0.0672 0.7375 0.008 0.008 0.984 0.000
#> GSM340299 2 0.3810 0.7393 0.000 0.804 0.188 0.008
#> GSM340301 2 0.3037 0.8141 0.000 0.888 0.036 0.076
#> GSM340303 3 0.5105 -0.0186 0.000 0.432 0.564 0.004
#> GSM340304 1 0.6280 0.5269 0.604 0.056 0.332 0.008
#> GSM340306 4 0.5169 0.6156 0.272 0.032 0.000 0.696
#> GSM340307 3 0.0469 0.7377 0.000 0.012 0.988 0.000
#> GSM340310 1 0.0188 0.8518 0.996 0.004 0.000 0.000
#> GSM340314 4 0.5300 0.0888 0.000 0.012 0.408 0.580
#> GSM340315 3 0.6186 0.1558 0.468 0.028 0.492 0.012
#> GSM340317 1 0.5682 0.2066 0.520 0.024 0.000 0.456
#> GSM340318 4 0.0564 0.8702 0.004 0.004 0.004 0.988
#> GSM340319 4 0.0564 0.8702 0.004 0.004 0.004 0.988
#> GSM340320 4 0.3803 0.7872 0.132 0.032 0.000 0.836
#> GSM340321 3 0.4576 0.6759 0.000 0.260 0.728 0.012
#> GSM340322 4 0.0524 0.8678 0.000 0.008 0.004 0.988
#> GSM340324 1 0.1743 0.8375 0.940 0.056 0.000 0.004
#> GSM340328 1 0.0376 0.8516 0.992 0.004 0.000 0.004
#> GSM340330 3 0.5204 0.6893 0.000 0.088 0.752 0.160
#> GSM340332 4 0.0712 0.8682 0.004 0.008 0.004 0.984
#> GSM340333 2 0.1489 0.8127 0.000 0.952 0.044 0.004
#> GSM340336 4 0.0564 0.8702 0.004 0.004 0.004 0.988
#> GSM340337 4 0.0707 0.8584 0.000 0.020 0.000 0.980
#> GSM340338 3 0.3946 0.7174 0.000 0.168 0.812 0.020
#> GSM340339 4 0.0376 0.8693 0.004 0.000 0.004 0.992
#> GSM340340 4 0.2760 0.8100 0.128 0.000 0.000 0.872
#> GSM340341 4 0.2101 0.8397 0.012 0.060 0.000 0.928
#> GSM340343 4 0.0564 0.8702 0.004 0.004 0.004 0.988
#> GSM340344 2 0.0817 0.8144 0.000 0.976 0.024 0.000
#> GSM340346 3 0.0336 0.7381 0.000 0.008 0.992 0.000
#> GSM340347 4 0.0564 0.8702 0.004 0.004 0.004 0.988
#> GSM340348 4 0.1902 0.8396 0.004 0.064 0.000 0.932
#> GSM340349 1 0.5946 0.2324 0.496 0.028 0.472 0.004
#> GSM340350 3 0.5279 0.1238 0.400 0.012 0.588 0.000
#> GSM340351 1 0.1004 0.8492 0.972 0.024 0.000 0.004
#> GSM340354 3 0.4621 0.7081 0.000 0.128 0.796 0.076
#> GSM340356 1 0.1488 0.8464 0.956 0.032 0.000 0.012
#> GSM340357 1 0.2011 0.8245 0.920 0.000 0.080 0.000
#> GSM348183 1 0.2256 0.8361 0.924 0.056 0.000 0.020
#> GSM348191 3 0.6620 0.5822 0.000 0.180 0.628 0.192
#> GSM348193 1 0.1256 0.8477 0.964 0.028 0.000 0.008
#> GSM537578 3 0.0707 0.7379 0.000 0.020 0.980 0.000
#> GSM348181 1 0.6435 0.6114 0.672 0.164 0.008 0.156
#> GSM348182 2 0.3945 0.7711 0.024 0.828 0.004 0.144
#> GSM348184 3 0.3625 0.7232 0.000 0.160 0.828 0.012
#> GSM348185 1 0.2840 0.8286 0.900 0.044 0.000 0.056
#> GSM348186 2 0.1305 0.8138 0.000 0.960 0.036 0.004
#> GSM348187 1 0.1575 0.8465 0.956 0.028 0.004 0.012
#> GSM348188 4 0.1211 0.8489 0.000 0.040 0.000 0.960
#> GSM348189 2 0.3940 0.7740 0.004 0.824 0.020 0.152
#> GSM348190 2 0.3853 0.7740 0.040 0.852 0.008 0.100
#> GSM348194 1 0.1584 0.8466 0.952 0.036 0.000 0.012
#> GSM348195 1 0.0524 0.8516 0.988 0.004 0.000 0.008
#> GSM348196 1 0.1488 0.8464 0.956 0.032 0.000 0.012
#> GSM537585 1 0.7472 0.1081 0.444 0.412 0.136 0.008
#> GSM537594 1 0.1888 0.8461 0.940 0.044 0.000 0.016
#> GSM537596 1 0.4068 0.7899 0.840 0.064 0.004 0.092
#> GSM537597 1 0.2207 0.8424 0.928 0.056 0.004 0.012
#> GSM537602 1 0.5670 0.6581 0.704 0.056 0.232 0.008
#> GSM340184 3 0.5783 0.6468 0.000 0.088 0.692 0.220
#> GSM340185 3 0.0336 0.7405 0.000 0.008 0.992 0.000
#> GSM340186 3 0.5062 0.6141 0.000 0.300 0.680 0.020
#> GSM340187 4 0.0804 0.8645 0.000 0.012 0.008 0.980
#> GSM340189 4 0.0804 0.8652 0.000 0.008 0.012 0.980
#> GSM340190 3 0.0336 0.7410 0.000 0.008 0.992 0.000
#> GSM340191 3 0.6160 0.5674 0.000 0.316 0.612 0.072
#> GSM340192 3 0.1118 0.7345 0.000 0.036 0.964 0.000
#> GSM340193 1 0.5888 0.6770 0.704 0.192 0.004 0.100
#> GSM340194 2 0.3080 0.8055 0.000 0.880 0.024 0.096
#> GSM340195 2 0.3060 0.8077 0.008 0.888 0.016 0.088
#> GSM340196 4 0.0564 0.8702 0.004 0.004 0.004 0.988
#> GSM340197 2 0.1584 0.8140 0.000 0.952 0.036 0.012
#> GSM340198 2 0.2256 0.8058 0.000 0.924 0.056 0.020
#> GSM340199 3 0.0336 0.7381 0.000 0.008 0.992 0.000
#> GSM340200 3 0.0469 0.7377 0.000 0.012 0.988 0.000
#> GSM340201 4 0.0657 0.8689 0.012 0.004 0.000 0.984
#> GSM340202 4 0.0564 0.8702 0.004 0.004 0.004 0.988
#> GSM340203 4 0.0469 0.8650 0.000 0.012 0.000 0.988
#> GSM340204 2 0.3027 0.8072 0.004 0.888 0.020 0.088
#> GSM340205 1 0.0188 0.8518 0.996 0.004 0.000 0.000
#> GSM340206 3 0.3764 0.7072 0.000 0.012 0.816 0.172
#> GSM340207 2 0.5440 0.1663 0.000 0.596 0.384 0.020
#> GSM340237 3 0.4059 0.6033 0.200 0.012 0.788 0.000
#> GSM340238 3 0.5294 0.2313 0.000 0.008 0.508 0.484
#> GSM340239 2 0.3168 0.8183 0.000 0.884 0.056 0.060
#> GSM340240 3 0.4008 0.4954 0.000 0.244 0.756 0.000
#> GSM340241 3 0.4699 0.5999 0.000 0.320 0.676 0.004
#> GSM340242 2 0.2413 0.8059 0.000 0.916 0.064 0.020
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.0162 0.758309 0.996 0.004 0.000 0.000 0.000
#> GSM340359 1 0.2930 0.630474 0.832 0.004 0.000 0.000 0.164
#> GSM340361 1 0.0162 0.758253 0.996 0.000 0.000 0.000 0.004
#> GSM340362 5 0.3023 0.637127 0.112 0.024 0.004 0.000 0.860
#> GSM340363 1 0.4442 0.635001 0.676 0.304 0.000 0.004 0.016
#> GSM340364 1 0.0162 0.758309 0.996 0.004 0.000 0.000 0.000
#> GSM340365 5 0.5026 0.468539 0.244 0.004 0.068 0.000 0.684
#> GSM340366 3 0.0000 0.608915 0.000 0.000 1.000 0.000 0.000
#> GSM340367 1 0.0162 0.758309 0.996 0.004 0.000 0.000 0.000
#> GSM340368 4 0.3796 0.585632 0.300 0.000 0.000 0.700 0.000
#> GSM340369 4 0.3845 0.677254 0.208 0.000 0.024 0.768 0.000
#> GSM340370 1 0.0162 0.758309 0.996 0.004 0.000 0.000 0.000
#> GSM340371 5 0.4895 0.363575 0.000 0.004 0.376 0.024 0.596
#> GSM340372 1 0.0162 0.758309 0.996 0.004 0.000 0.000 0.000
#> GSM340373 1 0.0162 0.758309 0.996 0.004 0.000 0.000 0.000
#> GSM340375 1 0.0324 0.758256 0.992 0.004 0.000 0.004 0.000
#> GSM340376 1 0.0162 0.758309 0.996 0.004 0.000 0.000 0.000
#> GSM340378 1 0.0162 0.758309 0.996 0.004 0.000 0.000 0.000
#> GSM340243 3 0.5456 0.137086 0.428 0.032 0.524 0.000 0.016
#> GSM340244 2 0.7779 0.285149 0.204 0.512 0.144 0.008 0.132
#> GSM340246 2 0.6413 0.119518 0.004 0.444 0.420 0.004 0.128
#> GSM340247 4 0.0000 0.805462 0.000 0.000 0.000 1.000 0.000
#> GSM340248 2 0.2264 0.409987 0.004 0.912 0.024 0.000 0.060
#> GSM340249 4 0.4946 0.456764 0.000 0.300 0.000 0.648 0.052
#> GSM340250 2 0.6432 0.349157 0.188 0.632 0.128 0.004 0.048
#> GSM340251 2 0.7469 0.123398 0.000 0.416 0.372 0.132 0.080
#> GSM340252 5 0.6160 0.131332 0.000 0.132 0.000 0.420 0.448
#> GSM340253 5 0.2629 0.649708 0.004 0.136 0.000 0.000 0.860
#> GSM340254 5 0.4210 0.375439 0.000 0.412 0.000 0.000 0.588
#> GSM340256 2 0.6380 0.166988 0.004 0.524 0.176 0.000 0.296
#> GSM340258 2 0.6322 0.197645 0.000 0.508 0.352 0.008 0.132
#> GSM340259 2 0.4930 0.107374 0.032 0.580 0.000 0.000 0.388
#> GSM340260 5 0.6617 -0.113818 0.004 0.328 0.200 0.000 0.468
#> GSM340261 3 0.6220 0.132714 0.000 0.308 0.524 0.000 0.168
#> GSM340262 5 0.4907 0.206135 0.000 0.468 0.012 0.008 0.512
#> GSM340263 1 0.6169 -0.071188 0.464 0.436 0.004 0.008 0.088
#> GSM340264 5 0.3734 0.634404 0.000 0.184 0.016 0.008 0.792
#> GSM340265 3 0.4054 0.506166 0.000 0.072 0.788 0.000 0.140
#> GSM340266 2 0.4464 0.399012 0.004 0.800 0.096 0.032 0.068
#> GSM340267 3 0.4358 0.400360 0.000 0.284 0.696 0.012 0.008
#> GSM340268 2 0.6411 0.126867 0.000 0.484 0.400 0.028 0.088
#> GSM340269 5 0.6443 -0.180268 0.000 0.376 0.180 0.000 0.444
#> GSM340270 1 0.1041 0.744084 0.964 0.032 0.000 0.004 0.000
#> GSM537574 2 0.5394 0.346715 0.000 0.660 0.208 0.000 0.132
#> GSM537580 5 0.2787 0.626372 0.000 0.004 0.004 0.136 0.856
#> GSM537581 4 0.5556 0.456424 0.276 0.108 0.000 0.616 0.000
#> GSM340272 5 0.4697 0.342789 0.020 0.000 0.388 0.000 0.592
#> GSM340273 1 0.4430 0.592630 0.628 0.360 0.000 0.012 0.000
#> GSM340275 4 0.1329 0.782430 0.004 0.032 0.000 0.956 0.008
#> GSM340276 2 0.5519 0.333236 0.148 0.648 0.000 0.000 0.204
#> GSM340277 3 0.4746 0.418793 0.004 0.120 0.744 0.000 0.132
#> GSM340278 3 0.4392 0.296432 0.000 0.380 0.612 0.008 0.000
#> GSM340279 2 0.6516 0.028852 0.000 0.460 0.400 0.016 0.124
#> GSM340282 3 0.0162 0.608234 0.000 0.000 0.996 0.000 0.004
#> GSM340284 2 0.6077 0.151699 0.000 0.508 0.392 0.012 0.088
#> GSM340285 2 0.8922 -0.139171 0.280 0.380 0.052 0.140 0.148
#> GSM340286 5 0.1518 0.669941 0.000 0.004 0.004 0.048 0.944
#> GSM340287 2 0.6691 0.237645 0.028 0.580 0.256 0.012 0.124
#> GSM340288 1 0.5455 0.549472 0.576 0.364 0.000 0.008 0.052
#> GSM340289 1 0.3863 0.561716 0.740 0.012 0.000 0.000 0.248
#> GSM340290 2 0.7002 0.150660 0.000 0.460 0.340 0.028 0.172
#> GSM340291 2 0.7334 0.144202 0.056 0.416 0.376 0.000 0.152
#> GSM340293 3 0.1965 0.572837 0.000 0.000 0.904 0.096 0.000
#> GSM340294 3 0.6326 0.153850 0.000 0.328 0.524 0.008 0.140
#> GSM340296 3 0.0000 0.608915 0.000 0.000 1.000 0.000 0.000
#> GSM340297 3 0.6598 0.149117 0.256 0.220 0.516 0.008 0.000
#> GSM340298 3 0.0000 0.608915 0.000 0.000 1.000 0.000 0.000
#> GSM340299 5 0.1671 0.657570 0.000 0.000 0.076 0.000 0.924
#> GSM340301 5 0.1408 0.670057 0.000 0.044 0.000 0.008 0.948
#> GSM340303 3 0.4196 0.268606 0.000 0.004 0.640 0.000 0.356
#> GSM340304 1 0.8231 0.195519 0.356 0.284 0.236 0.000 0.124
#> GSM340306 4 0.6709 0.129419 0.248 0.352 0.000 0.400 0.000
#> GSM340307 3 0.0000 0.608915 0.000 0.000 1.000 0.000 0.000
#> GSM340310 1 0.0162 0.758522 0.996 0.004 0.000 0.000 0.000
#> GSM340314 4 0.7001 -0.172993 0.000 0.220 0.328 0.436 0.016
#> GSM340315 2 0.6933 0.056280 0.328 0.436 0.224 0.012 0.000
#> GSM340317 4 0.6381 0.155887 0.376 0.112 0.000 0.496 0.016
#> GSM340318 4 0.0000 0.805462 0.000 0.000 0.000 1.000 0.000
#> GSM340319 4 0.0000 0.805462 0.000 0.000 0.000 1.000 0.000
#> GSM340320 4 0.6194 0.357828 0.148 0.352 0.000 0.500 0.000
#> GSM340321 3 0.6756 -0.192759 0.000 0.368 0.368 0.000 0.264
#> GSM340322 4 0.0290 0.802243 0.000 0.000 0.000 0.992 0.008
#> GSM340324 1 0.3437 0.654383 0.832 0.120 0.000 0.000 0.048
#> GSM340328 1 0.0324 0.758449 0.992 0.004 0.000 0.004 0.000
#> GSM340330 3 0.7254 0.176345 0.000 0.284 0.504 0.140 0.072
#> GSM340332 4 0.0000 0.805462 0.000 0.000 0.000 1.000 0.000
#> GSM340333 5 0.2329 0.653742 0.000 0.124 0.000 0.000 0.876
#> GSM340336 4 0.0000 0.805462 0.000 0.000 0.000 1.000 0.000
#> GSM340337 4 0.0510 0.799353 0.000 0.000 0.000 0.984 0.016
#> GSM340338 3 0.5589 0.248141 0.000 0.296 0.616 0.008 0.080
#> GSM340339 4 0.0290 0.803082 0.000 0.008 0.000 0.992 0.000
#> GSM340340 4 0.2813 0.717948 0.168 0.000 0.000 0.832 0.000
#> GSM340341 4 0.5901 0.395567 0.000 0.344 0.000 0.540 0.116
#> GSM340343 4 0.0000 0.805462 0.000 0.000 0.000 1.000 0.000
#> GSM340344 5 0.2338 0.664937 0.000 0.112 0.004 0.000 0.884
#> GSM340346 3 0.0000 0.608915 0.000 0.000 1.000 0.000 0.000
#> GSM340347 4 0.0000 0.805462 0.000 0.000 0.000 1.000 0.000
#> GSM340348 4 0.6020 0.369078 0.004 0.364 0.000 0.524 0.108
#> GSM340349 3 0.3310 0.521376 0.136 0.024 0.836 0.000 0.004
#> GSM340350 3 0.3730 0.370342 0.288 0.000 0.712 0.000 0.000
#> GSM340351 1 0.2389 0.734335 0.880 0.116 0.000 0.004 0.000
#> GSM340354 3 0.6678 0.201546 0.000 0.244 0.548 0.024 0.184
#> GSM340356 1 0.4604 0.538240 0.560 0.428 0.000 0.012 0.000
#> GSM340357 1 0.5027 0.641195 0.700 0.188 0.112 0.000 0.000
#> GSM348183 1 0.3366 0.601045 0.784 0.004 0.000 0.000 0.212
#> GSM348191 2 0.7467 0.232517 0.000 0.484 0.248 0.068 0.200
#> GSM348193 1 0.2470 0.735599 0.884 0.104 0.000 0.012 0.000
#> GSM537578 3 0.1341 0.584052 0.000 0.056 0.944 0.000 0.000
#> GSM348181 1 0.7164 0.012175 0.460 0.320 0.000 0.036 0.184
#> GSM348182 5 0.2284 0.657003 0.028 0.004 0.000 0.056 0.912
#> GSM348184 3 0.5911 0.081607 0.000 0.360 0.552 0.016 0.072
#> GSM348185 1 0.5247 0.566111 0.592 0.364 0.000 0.028 0.016
#> GSM348186 5 0.2471 0.638507 0.000 0.136 0.000 0.000 0.864
#> GSM348187 1 0.4402 0.597456 0.636 0.352 0.000 0.012 0.000
#> GSM348188 4 0.3160 0.666735 0.000 0.004 0.000 0.808 0.188
#> GSM348189 5 0.1662 0.666212 0.000 0.004 0.004 0.056 0.936
#> GSM348190 5 0.4288 0.292656 0.000 0.384 0.000 0.004 0.612
#> GSM348194 1 0.4567 0.592765 0.628 0.356 0.000 0.012 0.004
#> GSM348195 1 0.0451 0.758315 0.988 0.008 0.000 0.004 0.000
#> GSM348196 1 0.4402 0.597456 0.636 0.352 0.000 0.012 0.000
#> GSM537585 5 0.7611 0.186840 0.244 0.204 0.080 0.000 0.472
#> GSM537594 1 0.4775 0.625992 0.660 0.304 0.000 0.004 0.032
#> GSM537596 2 0.6557 -0.253804 0.376 0.468 0.000 0.012 0.144
#> GSM537597 1 0.4402 0.597456 0.636 0.352 0.000 0.012 0.000
#> GSM537602 1 0.7038 0.402103 0.560 0.076 0.220 0.000 0.144
#> GSM340184 3 0.7638 0.009160 0.000 0.336 0.408 0.188 0.068
#> GSM340185 3 0.2583 0.516324 0.000 0.132 0.864 0.000 0.004
#> GSM340186 2 0.6676 0.138607 0.000 0.404 0.396 0.004 0.196
#> GSM340187 4 0.0290 0.801948 0.000 0.000 0.000 0.992 0.008
#> GSM340189 4 0.0451 0.800364 0.000 0.000 0.008 0.988 0.004
#> GSM340190 3 0.0000 0.608915 0.000 0.000 1.000 0.000 0.000
#> GSM340191 2 0.6340 0.195692 0.000 0.516 0.356 0.016 0.112
#> GSM340192 3 0.1197 0.592083 0.000 0.000 0.952 0.000 0.048
#> GSM340193 2 0.6519 -0.089406 0.304 0.512 0.000 0.008 0.176
#> GSM340194 5 0.1179 0.675328 0.000 0.016 0.004 0.016 0.964
#> GSM340195 5 0.1016 0.675092 0.004 0.008 0.004 0.012 0.972
#> GSM340196 4 0.0000 0.805462 0.000 0.000 0.000 1.000 0.000
#> GSM340197 5 0.2377 0.652838 0.000 0.128 0.000 0.000 0.872
#> GSM340198 5 0.4655 0.420917 0.004 0.368 0.004 0.008 0.616
#> GSM340199 3 0.0609 0.604278 0.000 0.020 0.980 0.000 0.000
#> GSM340200 3 0.0000 0.608915 0.000 0.000 1.000 0.000 0.000
#> GSM340201 4 0.0000 0.805462 0.000 0.000 0.000 1.000 0.000
#> GSM340202 4 0.0000 0.805462 0.000 0.000 0.000 1.000 0.000
#> GSM340203 4 0.0404 0.801181 0.000 0.000 0.000 0.988 0.012
#> GSM340204 5 0.1267 0.672407 0.004 0.024 0.000 0.012 0.960
#> GSM340205 1 0.0290 0.756895 0.992 0.008 0.000 0.000 0.000
#> GSM340206 3 0.6447 0.307458 0.000 0.232 0.588 0.152 0.028
#> GSM340207 5 0.6922 0.000572 0.000 0.284 0.276 0.008 0.432
#> GSM340237 3 0.5968 0.252834 0.268 0.156 0.576 0.000 0.000
#> GSM340238 3 0.7106 0.071282 0.000 0.244 0.400 0.340 0.016
#> GSM340239 5 0.1484 0.679526 0.000 0.048 0.000 0.008 0.944
#> GSM340240 3 0.3366 0.459154 0.000 0.000 0.768 0.000 0.232
#> GSM340241 2 0.6630 0.151440 0.000 0.404 0.376 0.000 0.220
#> GSM340242 5 0.4199 0.617935 0.000 0.180 0.040 0.008 0.772
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.0146 0.85304 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM340359 1 0.0790 0.83495 0.968 0.000 0.000 0.000 0.032 0.000
#> GSM340361 1 0.0000 0.85262 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340362 5 0.2462 0.76460 0.096 0.028 0.000 0.000 0.876 0.000
#> GSM340363 1 0.4491 0.25817 0.604 0.016 0.000 0.000 0.016 0.364
#> GSM340364 1 0.0146 0.85149 0.996 0.000 0.004 0.000 0.000 0.000
#> GSM340365 5 0.5217 0.28506 0.380 0.000 0.028 0.000 0.548 0.044
#> GSM340366 3 0.1088 0.65315 0.024 0.000 0.960 0.000 0.016 0.000
#> GSM340367 1 0.0146 0.85149 0.996 0.000 0.004 0.000 0.000 0.000
#> GSM340368 4 0.3563 0.56201 0.336 0.000 0.000 0.664 0.000 0.000
#> GSM340369 4 0.3748 0.60836 0.300 0.000 0.012 0.688 0.000 0.000
#> GSM340370 1 0.0146 0.85304 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM340371 5 0.2838 0.67283 0.000 0.000 0.188 0.000 0.808 0.004
#> GSM340372 1 0.0000 0.85262 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340373 1 0.0146 0.85304 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM340375 1 0.0146 0.85304 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM340376 1 0.0146 0.85304 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM340378 1 0.0146 0.85304 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM340243 3 0.5846 0.18592 0.388 0.004 0.492 0.000 0.024 0.092
#> GSM340244 2 0.5984 0.27674 0.004 0.564 0.192 0.004 0.012 0.224
#> GSM340246 2 0.6091 0.28163 0.000 0.564 0.204 0.004 0.028 0.200
#> GSM340247 4 0.0000 0.87989 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340248 2 0.2553 0.37349 0.000 0.848 0.000 0.000 0.008 0.144
#> GSM340249 2 0.5742 0.06135 0.000 0.484 0.000 0.332 0.000 0.184
#> GSM340250 2 0.6858 0.19184 0.116 0.524 0.104 0.004 0.008 0.244
#> GSM340251 2 0.5999 0.27234 0.000 0.568 0.208 0.032 0.000 0.192
#> GSM340252 4 0.5717 0.47506 0.000 0.184 0.000 0.600 0.192 0.024
#> GSM340253 5 0.2673 0.73546 0.000 0.132 0.000 0.004 0.852 0.012
#> GSM340254 2 0.5767 0.06833 0.000 0.484 0.000 0.000 0.324 0.192
#> GSM340256 2 0.6957 0.27066 0.004 0.496 0.164 0.004 0.088 0.244
#> GSM340258 2 0.4171 0.40892 0.000 0.732 0.208 0.000 0.052 0.008
#> GSM340259 2 0.6160 0.28971 0.044 0.556 0.000 0.000 0.228 0.172
#> GSM340260 2 0.7439 0.28435 0.024 0.424 0.120 0.000 0.304 0.128
#> GSM340261 2 0.6121 0.29116 0.000 0.532 0.312 0.000 0.072 0.084
#> GSM340262 2 0.3717 0.15113 0.000 0.616 0.000 0.000 0.384 0.000
#> GSM340263 2 0.5572 0.01549 0.388 0.504 0.000 0.004 0.008 0.096
#> GSM340264 5 0.3650 0.64010 0.000 0.272 0.008 0.004 0.716 0.000
#> GSM340265 3 0.3158 0.57733 0.000 0.020 0.812 0.000 0.164 0.004
#> GSM340266 2 0.3713 0.25696 0.000 0.704 0.000 0.004 0.008 0.284
#> GSM340267 3 0.5585 0.28106 0.024 0.076 0.556 0.004 0.000 0.340
#> GSM340268 2 0.3795 0.37959 0.000 0.724 0.256 0.004 0.012 0.004
#> GSM340269 2 0.5619 0.41868 0.000 0.640 0.108 0.000 0.196 0.056
#> GSM340270 1 0.1649 0.81049 0.932 0.036 0.000 0.000 0.032 0.000
#> GSM537574 2 0.4231 0.37951 0.000 0.752 0.028 0.004 0.032 0.184
#> GSM537580 5 0.1713 0.78971 0.000 0.000 0.000 0.044 0.928 0.028
#> GSM537581 4 0.5423 0.31457 0.184 0.000 0.000 0.576 0.000 0.240
#> GSM340272 3 0.4264 -0.00499 0.016 0.000 0.496 0.000 0.488 0.000
#> GSM340273 6 0.3330 0.62950 0.284 0.000 0.000 0.000 0.000 0.716
#> GSM340275 4 0.1267 0.84484 0.000 0.060 0.000 0.940 0.000 0.000
#> GSM340276 6 0.5044 0.34997 0.000 0.320 0.000 0.000 0.096 0.584
#> GSM340277 3 0.5681 0.34646 0.000 0.188 0.636 0.000 0.056 0.120
#> GSM340278 3 0.6082 0.14468 0.000 0.232 0.432 0.004 0.000 0.332
#> GSM340279 2 0.7178 0.20995 0.000 0.344 0.284 0.000 0.080 0.292
#> GSM340282 3 0.0000 0.65364 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340284 2 0.3679 0.40791 0.000 0.764 0.208 0.004 0.016 0.008
#> GSM340285 6 0.6236 0.62317 0.080 0.048 0.008 0.112 0.088 0.664
#> GSM340286 5 0.0146 0.80407 0.000 0.000 0.000 0.000 0.996 0.004
#> GSM340287 2 0.7018 0.22653 0.000 0.428 0.172 0.000 0.100 0.300
#> GSM340288 6 0.3630 0.66111 0.196 0.012 0.000 0.000 0.020 0.772
#> GSM340289 1 0.4677 0.50979 0.664 0.008 0.000 0.000 0.264 0.064
#> GSM340290 2 0.6251 0.29678 0.000 0.528 0.300 0.000 0.092 0.080
#> GSM340291 2 0.6499 0.25398 0.004 0.524 0.216 0.004 0.036 0.216
#> GSM340293 3 0.2605 0.59287 0.000 0.028 0.864 0.108 0.000 0.000
#> GSM340294 2 0.6814 0.24475 0.000 0.444 0.328 0.000 0.108 0.120
#> GSM340296 3 0.0146 0.65332 0.000 0.004 0.996 0.000 0.000 0.000
#> GSM340297 3 0.7184 -0.11442 0.160 0.348 0.368 0.000 0.000 0.124
#> GSM340298 3 0.0865 0.64251 0.000 0.036 0.964 0.000 0.000 0.000
#> GSM340299 5 0.0603 0.80316 0.000 0.000 0.016 0.000 0.980 0.004
#> GSM340301 5 0.0603 0.80217 0.000 0.016 0.000 0.000 0.980 0.004
#> GSM340303 3 0.3885 0.45539 0.000 0.004 0.684 0.000 0.300 0.012
#> GSM340304 6 0.6408 0.58105 0.124 0.028 0.148 0.000 0.084 0.616
#> GSM340306 6 0.4890 0.60887 0.160 0.000 0.000 0.180 0.000 0.660
#> GSM340307 3 0.0000 0.65364 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340310 1 0.0146 0.85304 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM340314 2 0.6502 0.23254 0.000 0.420 0.268 0.292 0.016 0.004
#> GSM340315 6 0.7586 0.17458 0.208 0.280 0.152 0.004 0.000 0.356
#> GSM340317 4 0.6643 0.31391 0.056 0.252 0.000 0.524 0.012 0.156
#> GSM340318 4 0.0000 0.87989 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340319 4 0.0000 0.87989 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340320 6 0.4586 0.55020 0.076 0.000 0.000 0.264 0.000 0.660
#> GSM340321 2 0.6934 0.25839 0.000 0.484 0.208 0.000 0.112 0.196
#> GSM340322 4 0.0146 0.87883 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM340324 1 0.2976 0.73700 0.844 0.016 0.004 0.000 0.008 0.128
#> GSM340328 1 0.0000 0.85262 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340330 2 0.6607 0.24602 0.000 0.460 0.348 0.068 0.120 0.004
#> GSM340332 4 0.1036 0.86013 0.000 0.024 0.000 0.964 0.004 0.008
#> GSM340333 5 0.2199 0.76283 0.000 0.088 0.000 0.000 0.892 0.020
#> GSM340336 4 0.0000 0.87989 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340337 4 0.0291 0.87733 0.000 0.004 0.000 0.992 0.004 0.000
#> GSM340338 3 0.5156 0.02253 0.000 0.400 0.528 0.004 0.064 0.004
#> GSM340339 4 0.0260 0.87711 0.000 0.000 0.000 0.992 0.000 0.008
#> GSM340340 4 0.2969 0.70220 0.224 0.000 0.000 0.776 0.000 0.000
#> GSM340341 6 0.4888 0.56892 0.000 0.020 0.000 0.228 0.076 0.676
#> GSM340343 4 0.0000 0.87989 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340344 5 0.1075 0.79761 0.000 0.048 0.000 0.000 0.952 0.000
#> GSM340346 3 0.0000 0.65364 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340347 4 0.0000 0.87989 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340348 6 0.4754 0.58302 0.000 0.020 0.000 0.200 0.080 0.700
#> GSM340349 3 0.2575 0.62116 0.024 0.004 0.872 0.000 0.000 0.100
#> GSM340350 3 0.3547 0.40659 0.332 0.000 0.668 0.000 0.000 0.000
#> GSM340351 1 0.2912 0.62242 0.784 0.000 0.000 0.000 0.000 0.216
#> GSM340354 3 0.6106 -0.20592 0.000 0.388 0.404 0.000 0.200 0.008
#> GSM340356 6 0.3699 0.57891 0.336 0.000 0.000 0.004 0.000 0.660
#> GSM340357 1 0.4895 0.42584 0.648 0.000 0.124 0.000 0.000 0.228
#> GSM348183 1 0.3328 0.69758 0.816 0.000 0.000 0.000 0.120 0.064
#> GSM348191 2 0.6864 0.31650 0.000 0.472 0.260 0.000 0.176 0.092
#> GSM348193 1 0.3081 0.61150 0.776 0.000 0.000 0.004 0.000 0.220
#> GSM537578 3 0.1556 0.63383 0.000 0.000 0.920 0.000 0.000 0.080
#> GSM348181 1 0.6630 0.21822 0.500 0.268 0.000 0.000 0.152 0.080
#> GSM348182 5 0.1261 0.79478 0.024 0.000 0.000 0.000 0.952 0.024
#> GSM348184 2 0.6568 0.19112 0.000 0.452 0.296 0.000 0.040 0.212
#> GSM348185 6 0.3998 0.66407 0.200 0.016 0.000 0.008 0.020 0.756
#> GSM348186 5 0.5046 0.44790 0.000 0.256 0.000 0.000 0.620 0.124
#> GSM348187 6 0.3699 0.57891 0.336 0.000 0.000 0.004 0.000 0.660
#> GSM348188 4 0.3495 0.72511 0.000 0.004 0.000 0.808 0.128 0.060
#> GSM348189 5 0.0260 0.80419 0.000 0.000 0.000 0.000 0.992 0.008
#> GSM348190 5 0.4933 0.15122 0.000 0.068 0.000 0.000 0.536 0.396
#> GSM348194 6 0.3713 0.62790 0.284 0.008 0.000 0.000 0.004 0.704
#> GSM348195 1 0.1387 0.80851 0.932 0.000 0.000 0.000 0.000 0.068
#> GSM348196 6 0.3652 0.59310 0.324 0.000 0.000 0.004 0.000 0.672
#> GSM537585 5 0.6502 0.15300 0.160 0.000 0.056 0.000 0.488 0.296
#> GSM537594 6 0.5335 0.29027 0.080 0.276 0.000 0.000 0.028 0.616
#> GSM537596 6 0.5082 0.56896 0.052 0.160 0.000 0.000 0.088 0.700
#> GSM537597 6 0.3972 0.59424 0.320 0.012 0.000 0.004 0.000 0.664
#> GSM537602 6 0.7529 0.19794 0.340 0.020 0.212 0.000 0.084 0.344
#> GSM340184 2 0.6672 0.31582 0.000 0.508 0.288 0.088 0.108 0.008
#> GSM340185 3 0.4977 0.33917 0.000 0.188 0.648 0.000 0.000 0.164
#> GSM340186 2 0.6687 0.25286 0.000 0.504 0.220 0.004 0.060 0.212
#> GSM340187 4 0.0260 0.87719 0.000 0.000 0.000 0.992 0.008 0.000
#> GSM340189 4 0.0146 0.87883 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM340190 3 0.0146 0.65293 0.000 0.000 0.996 0.000 0.000 0.004
#> GSM340191 2 0.3327 0.41785 0.000 0.792 0.188 0.004 0.012 0.004
#> GSM340192 3 0.2378 0.59329 0.000 0.000 0.848 0.000 0.152 0.000
#> GSM340193 6 0.4792 0.48787 0.004 0.208 0.000 0.000 0.112 0.676
#> GSM340194 5 0.0000 0.80417 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340195 5 0.0713 0.79855 0.000 0.000 0.000 0.000 0.972 0.028
#> GSM340196 4 0.0146 0.87850 0.000 0.000 0.000 0.996 0.000 0.004
#> GSM340197 5 0.2048 0.76215 0.000 0.120 0.000 0.000 0.880 0.000
#> GSM340198 2 0.5918 0.07894 0.000 0.484 0.000 0.004 0.308 0.204
#> GSM340199 3 0.0632 0.64789 0.000 0.024 0.976 0.000 0.000 0.000
#> GSM340200 3 0.0000 0.65364 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340201 4 0.0000 0.87989 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340202 4 0.0000 0.87989 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340203 4 0.0363 0.87586 0.000 0.000 0.000 0.988 0.012 0.000
#> GSM340204 5 0.2526 0.74429 0.004 0.024 0.000 0.000 0.876 0.096
#> GSM340205 1 0.1007 0.82653 0.956 0.044 0.000 0.000 0.000 0.000
#> GSM340206 2 0.6611 0.17043 0.000 0.420 0.396 0.076 0.104 0.004
#> GSM340207 2 0.6037 0.28663 0.000 0.444 0.220 0.000 0.332 0.004
#> GSM340237 3 0.6633 0.18841 0.332 0.080 0.460 0.000 0.000 0.128
#> GSM340238 2 0.6772 0.22757 0.000 0.428 0.316 0.208 0.044 0.004
#> GSM340239 5 0.0935 0.79969 0.000 0.032 0.000 0.000 0.964 0.004
#> GSM340240 3 0.3081 0.55070 0.000 0.000 0.776 0.000 0.220 0.004
#> GSM340241 2 0.6828 0.25250 0.000 0.496 0.216 0.004 0.076 0.208
#> GSM340242 5 0.3819 0.48213 0.000 0.340 0.008 0.000 0.652 0.000
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
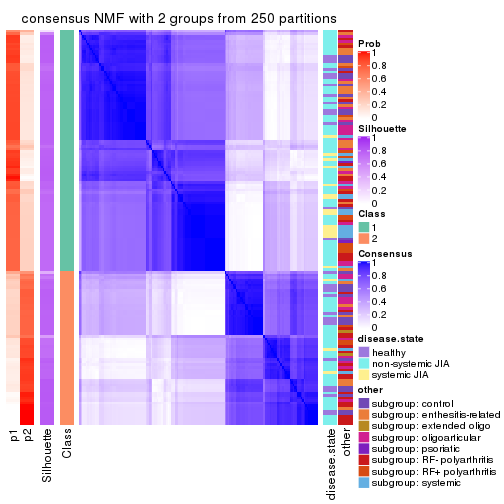
consensus_heatmap(res, k = 3)
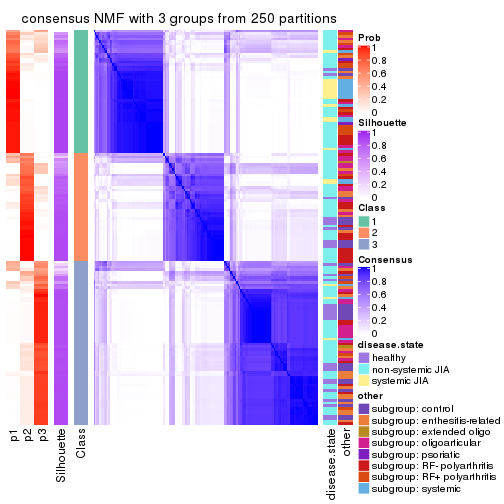
consensus_heatmap(res, k = 4)
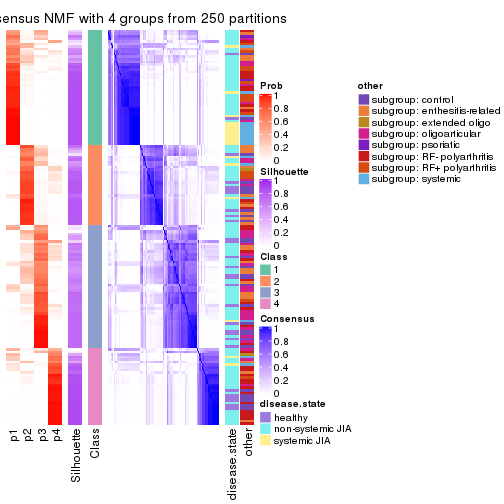
consensus_heatmap(res, k = 5)
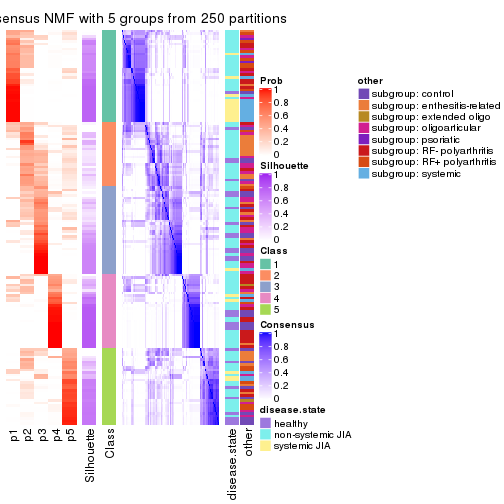
consensus_heatmap(res, k = 6)
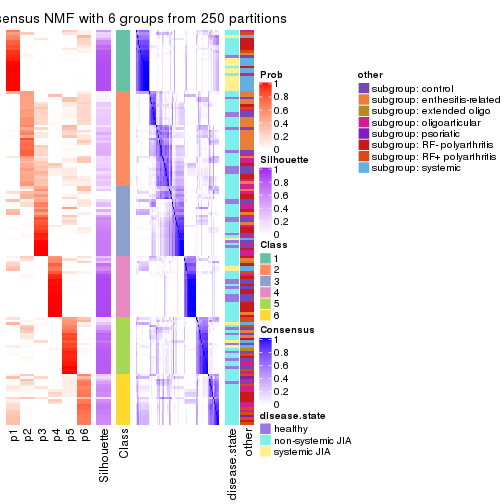
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
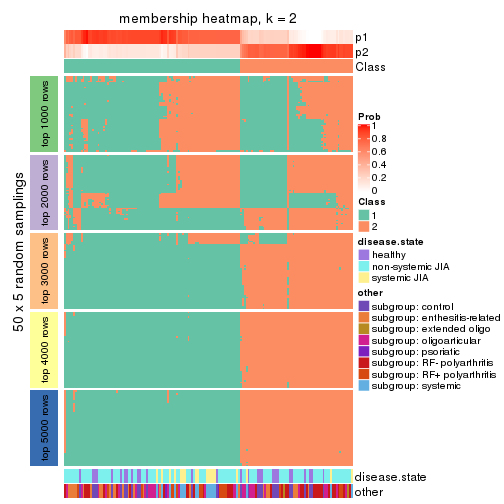
membership_heatmap(res, k = 3)
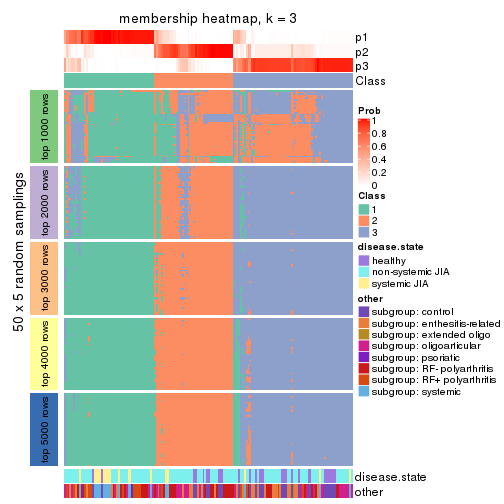
membership_heatmap(res, k = 4)
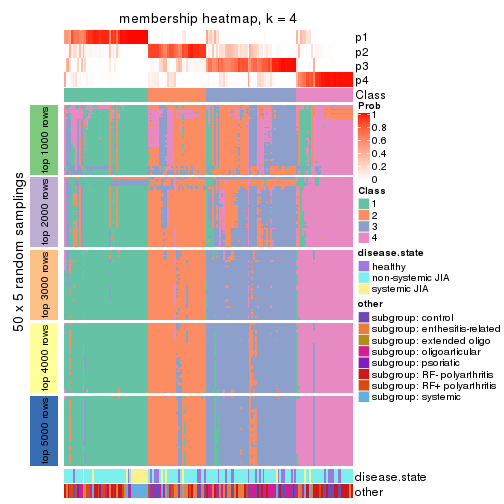
membership_heatmap(res, k = 5)
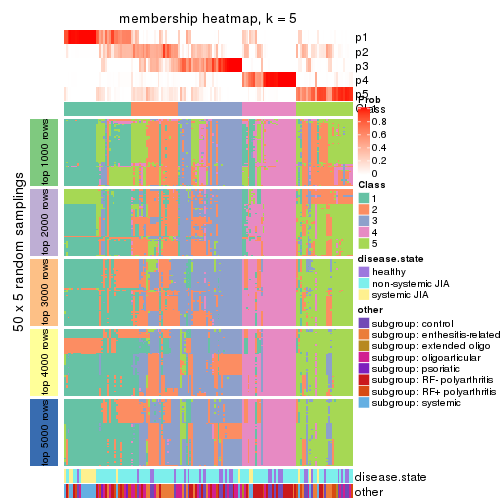
membership_heatmap(res, k = 6)
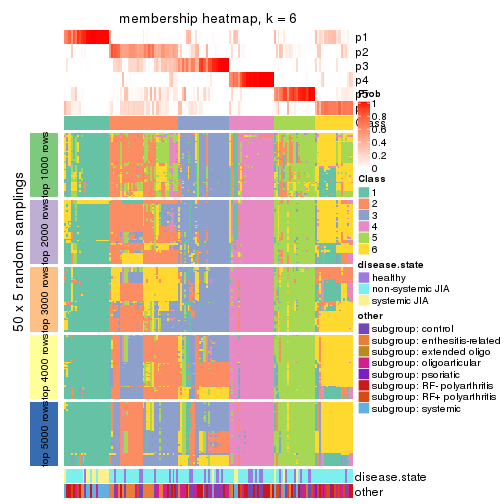
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
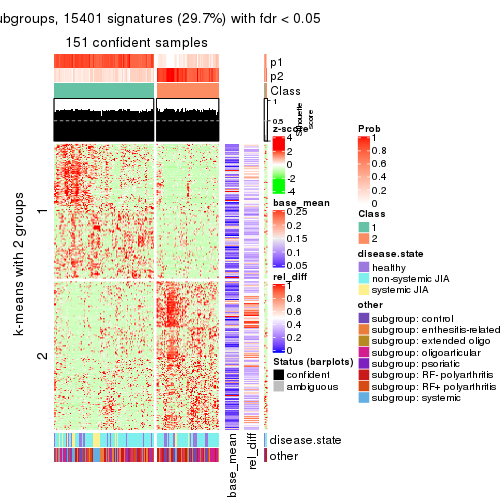
get_signatures(res, k = 3)
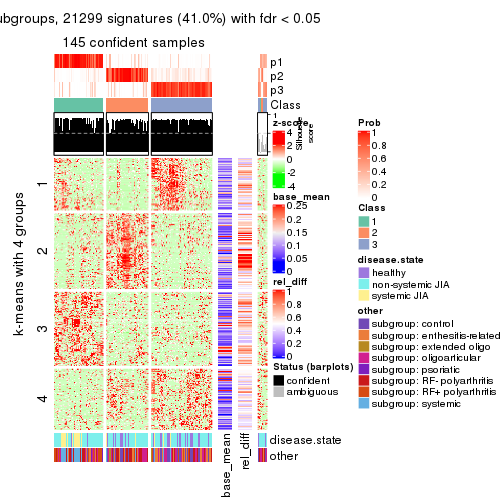
get_signatures(res, k = 4)
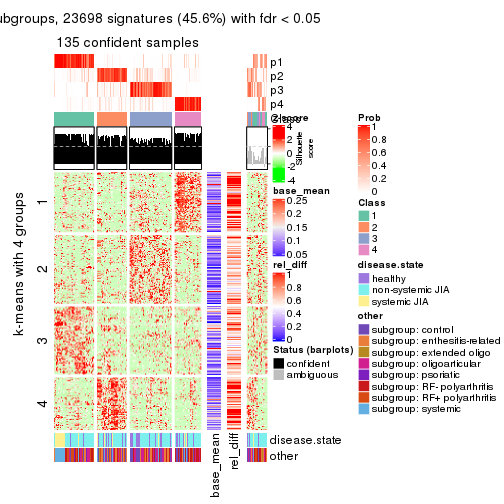
get_signatures(res, k = 5)
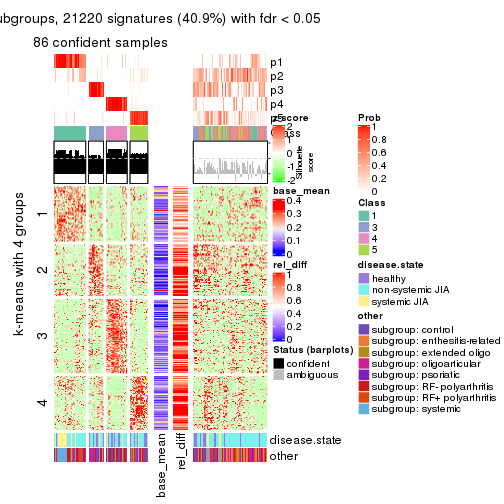
get_signatures(res, k = 6)
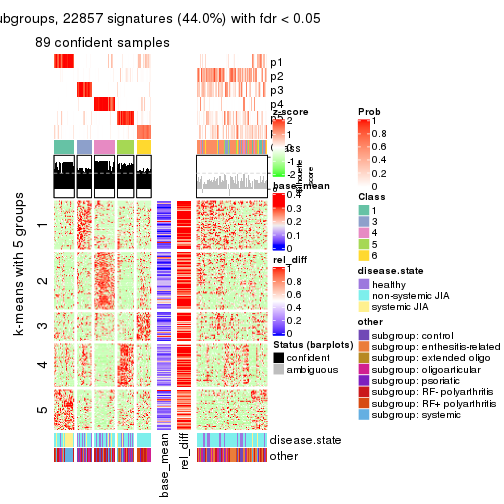
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
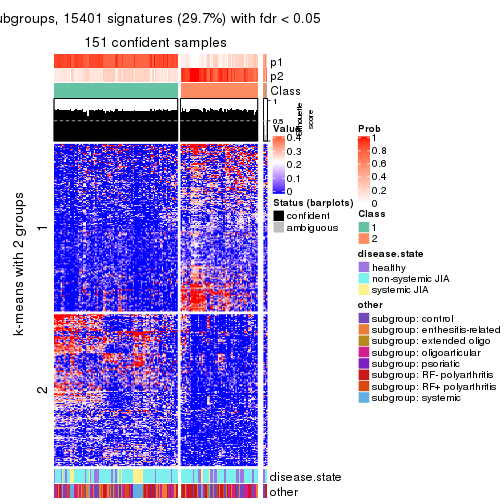
get_signatures(res, k = 3, scale_rows = FALSE)
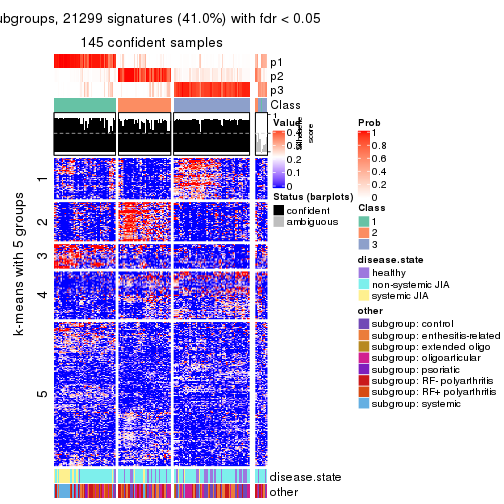
get_signatures(res, k = 4, scale_rows = FALSE)
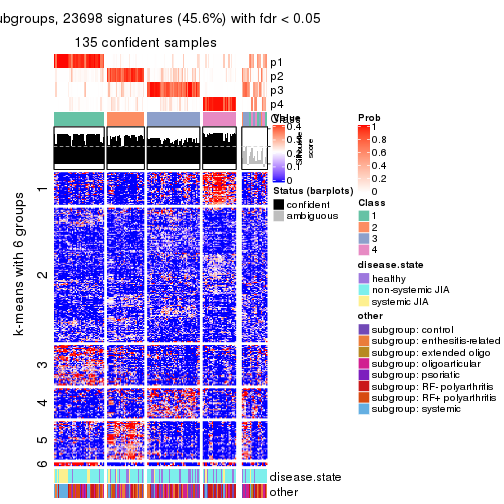
get_signatures(res, k = 5, scale_rows = FALSE)
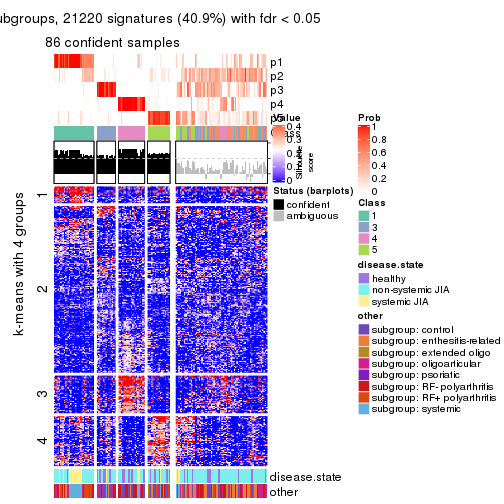
get_signatures(res, k = 6, scale_rows = FALSE)
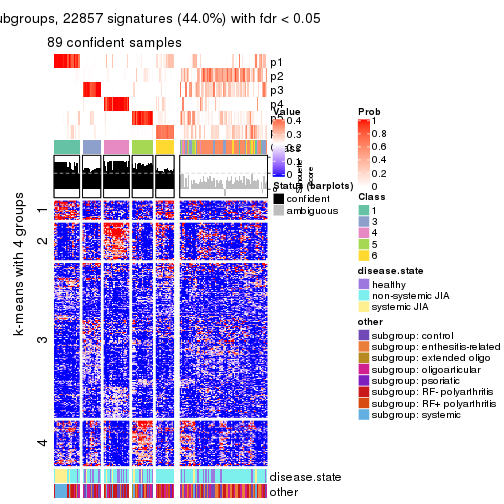
Compare the overlap of signatures from different k:
compare_signatures(res)
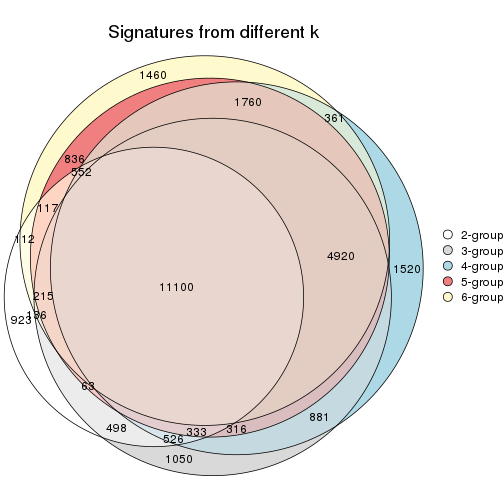
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
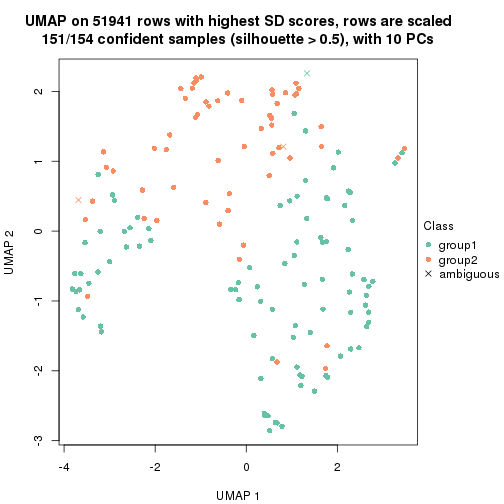
dimension_reduction(res, k = 3, method = "UMAP")
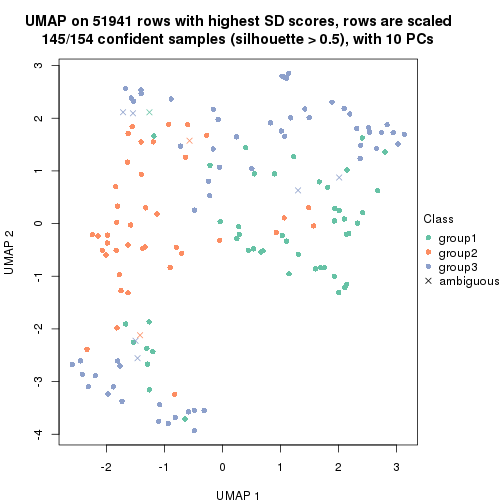
dimension_reduction(res, k = 4, method = "UMAP")
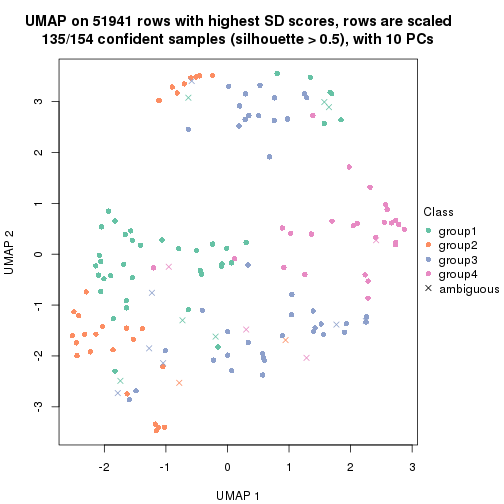
dimension_reduction(res, k = 5, method = "UMAP")
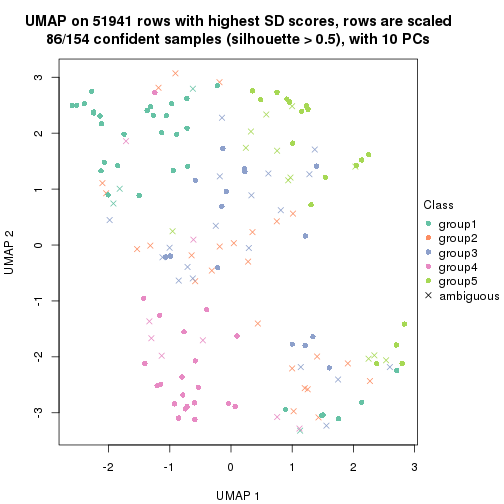
dimension_reduction(res, k = 6, method = "UMAP")
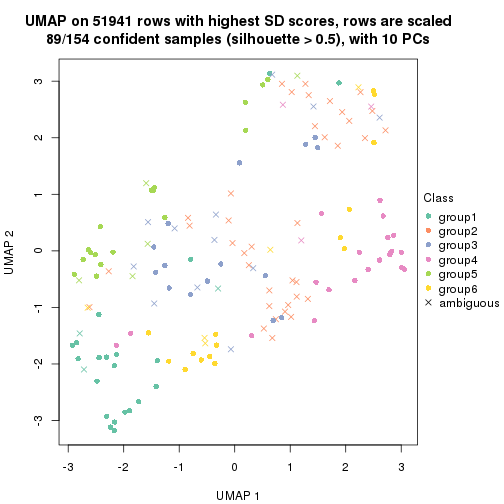
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
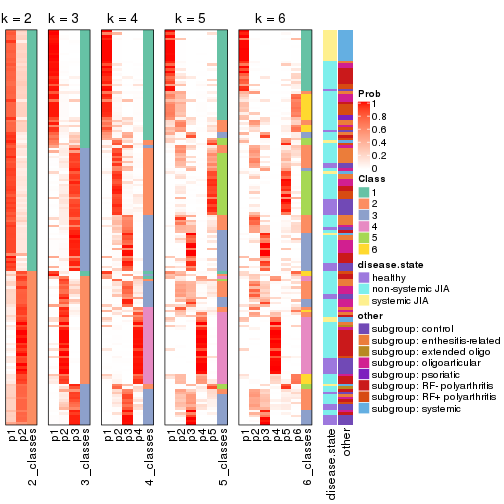
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> SD:NMF 151 7.41e-02 1.41e-02 2
#> SD:NMF 145 1.87e-04 5.09e-05 3
#> SD:NMF 135 7.85e-03 2.62e-05 4
#> SD:NMF 86 6.91e-03 8.15e-03 5
#> SD:NMF 89 8.83e-05 2.72e-04 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["CV", "hclust"]
# you can also extract it by
# res = res_list["CV:hclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'CV' method.
#> Subgroups are detected by 'hclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 4.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
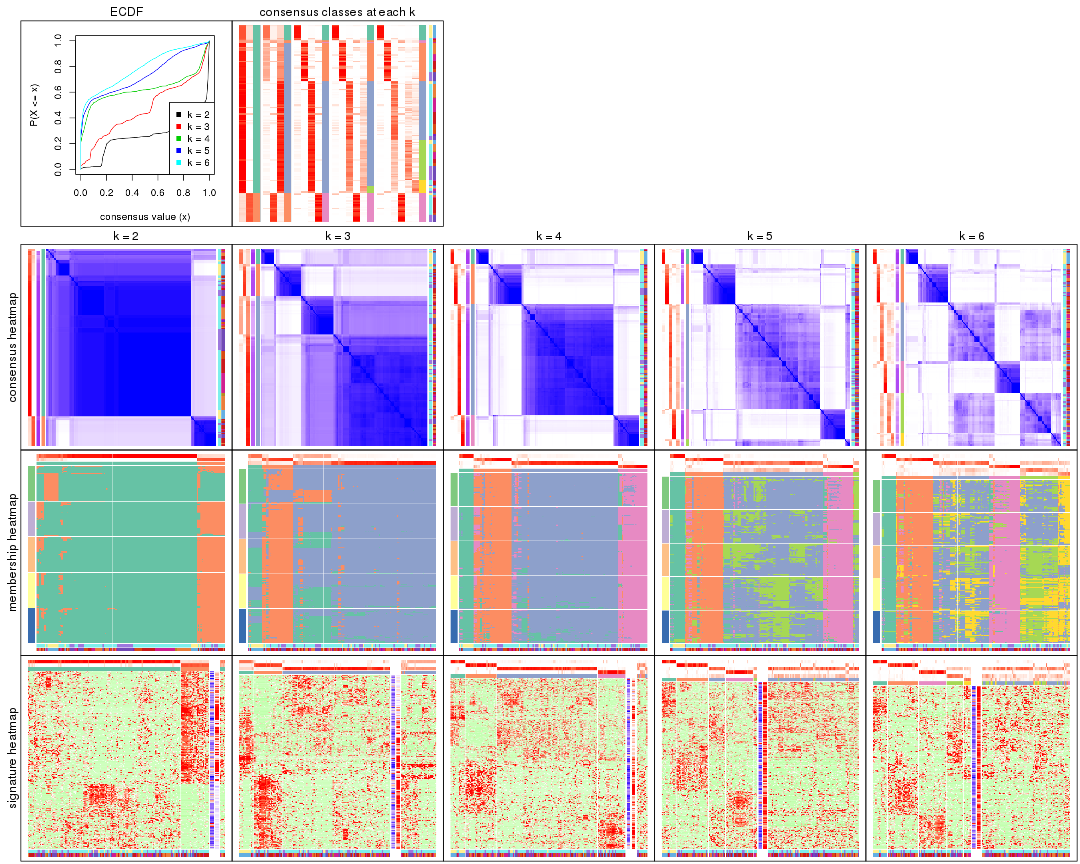
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
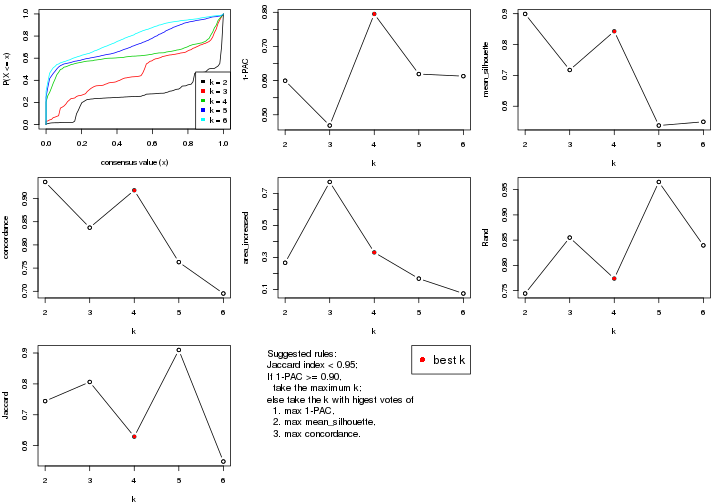
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.600 0.899 0.936 0.2665 0.744 0.744
#> 3 3 0.468 0.717 0.837 0.7715 0.855 0.806
#> 4 4 0.795 0.843 0.918 0.3312 0.774 0.629
#> 5 5 0.619 0.537 0.763 0.1674 0.965 0.910
#> 6 6 0.613 0.550 0.695 0.0742 0.840 0.549
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 4
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 1 0.7950 0.6938 0.760 0.240
#> GSM340359 1 0.6531 0.7791 0.832 0.168
#> GSM340361 1 0.0000 0.9467 1.000 0.000
#> GSM340362 1 0.0000 0.9467 1.000 0.000
#> GSM340363 1 0.4562 0.8565 0.904 0.096
#> GSM340364 1 0.6531 0.7791 0.832 0.168
#> GSM340365 1 0.0000 0.9467 1.000 0.000
#> GSM340366 1 0.0000 0.9467 1.000 0.000
#> GSM340367 1 0.6531 0.7791 0.832 0.168
#> GSM340368 2 0.7299 0.8577 0.204 0.796
#> GSM340369 2 0.6148 0.8039 0.152 0.848
#> GSM340370 1 0.6531 0.7791 0.832 0.168
#> GSM340371 1 0.0000 0.9467 1.000 0.000
#> GSM340372 1 0.6531 0.7791 0.832 0.168
#> GSM340373 1 0.6531 0.7791 0.832 0.168
#> GSM340375 1 0.6531 0.7791 0.832 0.168
#> GSM340376 1 0.6531 0.7791 0.832 0.168
#> GSM340378 1 0.6531 0.7791 0.832 0.168
#> GSM340243 1 0.0376 0.9451 0.996 0.004
#> GSM340244 1 0.1184 0.9413 0.984 0.016
#> GSM340246 1 0.1184 0.9413 0.984 0.016
#> GSM340247 2 0.6531 0.9637 0.168 0.832
#> GSM340248 1 0.1184 0.9413 0.984 0.016
#> GSM340249 1 0.9087 0.4201 0.676 0.324
#> GSM340250 1 0.0672 0.9428 0.992 0.008
#> GSM340251 1 0.6531 0.7618 0.832 0.168
#> GSM340252 1 0.7453 0.6875 0.788 0.212
#> GSM340253 1 0.1184 0.9413 0.984 0.016
#> GSM340254 1 0.1184 0.9413 0.984 0.016
#> GSM340256 1 0.1184 0.9413 0.984 0.016
#> GSM340258 1 0.0000 0.9467 1.000 0.000
#> GSM340259 1 0.0000 0.9467 1.000 0.000
#> GSM340260 1 0.0000 0.9467 1.000 0.000
#> GSM340261 1 0.0000 0.9467 1.000 0.000
#> GSM340262 1 0.0000 0.9467 1.000 0.000
#> GSM340263 1 0.2603 0.9190 0.956 0.044
#> GSM340264 1 0.0000 0.9467 1.000 0.000
#> GSM340265 1 0.0000 0.9467 1.000 0.000
#> GSM340266 1 0.0672 0.9427 0.992 0.008
#> GSM340267 1 0.0000 0.9467 1.000 0.000
#> GSM340268 1 0.0000 0.9467 1.000 0.000
#> GSM340269 1 0.0000 0.9467 1.000 0.000
#> GSM340270 1 0.0672 0.9430 0.992 0.008
#> GSM537574 1 0.0000 0.9467 1.000 0.000
#> GSM537580 1 0.0000 0.9467 1.000 0.000
#> GSM537581 1 0.0000 0.9467 1.000 0.000
#> GSM340272 1 0.0000 0.9467 1.000 0.000
#> GSM340273 1 0.1414 0.9397 0.980 0.020
#> GSM340275 1 0.9954 -0.1436 0.540 0.460
#> GSM340276 1 0.0000 0.9467 1.000 0.000
#> GSM340277 1 0.1184 0.9413 0.984 0.016
#> GSM340278 1 0.0000 0.9467 1.000 0.000
#> GSM340279 1 0.0000 0.9467 1.000 0.000
#> GSM340282 1 0.0000 0.9467 1.000 0.000
#> GSM340284 1 0.1414 0.9394 0.980 0.020
#> GSM340285 1 0.0000 0.9467 1.000 0.000
#> GSM340286 1 0.0000 0.9467 1.000 0.000
#> GSM340287 1 0.0000 0.9467 1.000 0.000
#> GSM340288 1 0.0376 0.9451 0.996 0.004
#> GSM340289 1 0.0376 0.9451 0.996 0.004
#> GSM340290 1 0.8081 0.5915 0.752 0.248
#> GSM340291 1 0.1184 0.9413 0.984 0.016
#> GSM340293 1 0.6531 0.7450 0.832 0.168
#> GSM340294 1 0.0000 0.9467 1.000 0.000
#> GSM340296 1 0.0000 0.9467 1.000 0.000
#> GSM340297 1 0.0000 0.9467 1.000 0.000
#> GSM340298 1 0.0000 0.9467 1.000 0.000
#> GSM340299 1 0.0000 0.9467 1.000 0.000
#> GSM340301 1 0.0000 0.9467 1.000 0.000
#> GSM340303 1 0.0000 0.9467 1.000 0.000
#> GSM340304 1 0.0000 0.9467 1.000 0.000
#> GSM340306 2 0.6973 0.9615 0.188 0.812
#> GSM340307 1 0.1184 0.9413 0.984 0.016
#> GSM340310 1 0.6712 0.7757 0.824 0.176
#> GSM340314 1 0.4161 0.8668 0.916 0.084
#> GSM340315 1 0.0000 0.9467 1.000 0.000
#> GSM340317 1 0.8861 0.4768 0.696 0.304
#> GSM340318 2 0.6531 0.9637 0.168 0.832
#> GSM340319 2 0.6531 0.9637 0.168 0.832
#> GSM340320 2 0.7056 0.9590 0.192 0.808
#> GSM340321 1 0.1184 0.9413 0.984 0.016
#> GSM340322 2 0.6531 0.9637 0.168 0.832
#> GSM340324 1 0.1184 0.9413 0.984 0.016
#> GSM340328 1 0.7883 0.6999 0.764 0.236
#> GSM340330 1 0.0000 0.9467 1.000 0.000
#> GSM340332 2 0.6973 0.9620 0.188 0.812
#> GSM340333 1 0.1184 0.9413 0.984 0.016
#> GSM340336 2 0.6531 0.9637 0.168 0.832
#> GSM340337 2 0.6623 0.9641 0.172 0.828
#> GSM340338 1 0.0000 0.9467 1.000 0.000
#> GSM340339 2 0.6973 0.9615 0.188 0.812
#> GSM340340 2 0.7815 0.8307 0.232 0.768
#> GSM340341 2 0.6973 0.9614 0.188 0.812
#> GSM340343 1 0.8499 0.5304 0.724 0.276
#> GSM340344 1 0.0672 0.9443 0.992 0.008
#> GSM340346 1 0.0000 0.9467 1.000 0.000
#> GSM340347 2 0.6531 0.9637 0.168 0.832
#> GSM340348 2 0.7056 0.9590 0.192 0.808
#> GSM340349 1 0.0000 0.9467 1.000 0.000
#> GSM340350 1 0.0000 0.9467 1.000 0.000
#> GSM340351 1 0.0000 0.9467 1.000 0.000
#> GSM340354 1 0.0000 0.9467 1.000 0.000
#> GSM340356 1 0.1414 0.9397 0.980 0.020
#> GSM340357 1 0.0000 0.9467 1.000 0.000
#> GSM348183 1 0.4562 0.8565 0.904 0.096
#> GSM348191 1 0.0000 0.9467 1.000 0.000
#> GSM348193 1 0.4161 0.8713 0.916 0.084
#> GSM537578 1 0.0000 0.9467 1.000 0.000
#> GSM348181 1 0.1843 0.9275 0.972 0.028
#> GSM348182 1 0.0000 0.9467 1.000 0.000
#> GSM348184 1 0.1184 0.9413 0.984 0.016
#> GSM348185 1 0.9909 -0.0711 0.556 0.444
#> GSM348186 1 0.1184 0.9413 0.984 0.016
#> GSM348187 1 0.0376 0.9451 0.996 0.004
#> GSM348188 2 0.7528 0.9322 0.216 0.784
#> GSM348189 1 0.0000 0.9467 1.000 0.000
#> GSM348190 1 0.0000 0.9467 1.000 0.000
#> GSM348194 1 0.0376 0.9451 0.996 0.004
#> GSM348195 1 0.0000 0.9467 1.000 0.000
#> GSM348196 1 0.0376 0.9451 0.996 0.004
#> GSM537585 1 0.0000 0.9467 1.000 0.000
#> GSM537594 1 0.1184 0.9413 0.984 0.016
#> GSM537596 1 0.0000 0.9467 1.000 0.000
#> GSM537597 1 0.0376 0.9451 0.996 0.004
#> GSM537602 1 0.0000 0.9467 1.000 0.000
#> GSM340184 1 0.0000 0.9467 1.000 0.000
#> GSM340185 1 0.1184 0.9413 0.984 0.016
#> GSM340186 1 0.1184 0.9413 0.984 0.016
#> GSM340187 2 0.6973 0.9620 0.188 0.812
#> GSM340189 2 0.6712 0.9640 0.176 0.824
#> GSM340190 1 0.1184 0.9413 0.984 0.016
#> GSM340191 1 0.0000 0.9467 1.000 0.000
#> GSM340192 1 0.0672 0.9435 0.992 0.008
#> GSM340193 1 0.0000 0.9467 1.000 0.000
#> GSM340194 1 0.0000 0.9467 1.000 0.000
#> GSM340195 1 0.0000 0.9467 1.000 0.000
#> GSM340196 2 0.6531 0.9637 0.168 0.832
#> GSM340197 1 0.0000 0.9467 1.000 0.000
#> GSM340198 1 0.2236 0.9285 0.964 0.036
#> GSM340199 1 0.0000 0.9467 1.000 0.000
#> GSM340200 1 0.0672 0.9435 0.992 0.008
#> GSM340201 2 0.6531 0.9637 0.168 0.832
#> GSM340202 2 0.6531 0.9637 0.168 0.832
#> GSM340203 2 0.7139 0.9569 0.196 0.804
#> GSM340204 1 0.0376 0.9453 0.996 0.004
#> GSM340205 1 0.1843 0.9275 0.972 0.028
#> GSM340206 1 0.0000 0.9467 1.000 0.000
#> GSM340207 1 0.0000 0.9467 1.000 0.000
#> GSM340237 1 0.1184 0.9413 0.984 0.016
#> GSM340238 1 0.0000 0.9467 1.000 0.000
#> GSM340239 1 0.0000 0.9467 1.000 0.000
#> GSM340240 1 0.0000 0.9467 1.000 0.000
#> GSM340241 1 0.1184 0.9413 0.984 0.016
#> GSM340242 1 0.0000 0.9467 1.000 0.000
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.8304 0.8765 0.624 0.144 0.232
#> GSM340359 1 0.6895 0.9453 0.716 0.072 0.212
#> GSM340361 3 0.1860 0.7936 0.052 0.000 0.948
#> GSM340362 3 0.1753 0.7937 0.048 0.000 0.952
#> GSM340363 3 0.5153 0.6768 0.068 0.100 0.832
#> GSM340364 1 0.6939 0.9515 0.712 0.072 0.216
#> GSM340365 3 0.1163 0.7992 0.028 0.000 0.972
#> GSM340366 3 0.1031 0.8014 0.024 0.000 0.976
#> GSM340367 1 0.6939 0.9515 0.712 0.072 0.216
#> GSM340368 2 0.6062 0.6941 0.148 0.780 0.072
#> GSM340369 2 0.6096 0.5595 0.208 0.752 0.040
#> GSM340370 1 0.6939 0.9515 0.712 0.072 0.216
#> GSM340371 3 0.1529 0.7953 0.040 0.000 0.960
#> GSM340372 1 0.6939 0.9515 0.712 0.072 0.216
#> GSM340373 1 0.6939 0.9515 0.712 0.072 0.216
#> GSM340375 1 0.6982 0.9478 0.708 0.072 0.220
#> GSM340376 1 0.6939 0.9515 0.712 0.072 0.216
#> GSM340378 1 0.6939 0.9515 0.712 0.072 0.216
#> GSM340243 3 0.1399 0.8036 0.028 0.004 0.968
#> GSM340244 3 0.6260 0.4349 0.448 0.000 0.552
#> GSM340246 3 0.6252 0.4395 0.444 0.000 0.556
#> GSM340247 2 0.2356 0.8707 0.000 0.928 0.072
#> GSM340248 3 0.6235 0.4454 0.436 0.000 0.564
#> GSM340249 3 0.9886 0.0392 0.276 0.320 0.404
#> GSM340250 3 0.0829 0.8013 0.012 0.004 0.984
#> GSM340251 3 0.9293 0.1905 0.400 0.160 0.440
#> GSM340252 3 0.9621 0.1396 0.360 0.208 0.432
#> GSM340253 3 0.6204 0.4654 0.424 0.000 0.576
#> GSM340254 3 0.6235 0.4516 0.436 0.000 0.564
#> GSM340256 3 0.6235 0.4494 0.436 0.000 0.564
#> GSM340258 3 0.0592 0.8020 0.012 0.000 0.988
#> GSM340259 3 0.1031 0.8012 0.024 0.000 0.976
#> GSM340260 3 0.0747 0.8025 0.016 0.000 0.984
#> GSM340261 3 0.0747 0.8026 0.016 0.000 0.984
#> GSM340262 3 0.1643 0.7949 0.044 0.000 0.956
#> GSM340263 3 0.5467 0.6921 0.176 0.032 0.792
#> GSM340264 3 0.1529 0.7989 0.040 0.000 0.960
#> GSM340265 3 0.1643 0.7984 0.044 0.000 0.956
#> GSM340266 3 0.1015 0.8029 0.012 0.008 0.980
#> GSM340267 3 0.2066 0.7979 0.060 0.000 0.940
#> GSM340268 3 0.0424 0.8008 0.008 0.000 0.992
#> GSM340269 3 0.0892 0.8035 0.020 0.000 0.980
#> GSM340270 3 0.1832 0.7979 0.036 0.008 0.956
#> GSM537574 3 0.0747 0.8025 0.016 0.000 0.984
#> GSM537580 3 0.1529 0.7953 0.040 0.000 0.960
#> GSM537581 3 0.1163 0.8020 0.028 0.000 0.972
#> GSM340272 3 0.0892 0.8005 0.020 0.000 0.980
#> GSM340273 3 0.6228 0.5121 0.372 0.004 0.624
#> GSM340275 2 0.9350 0.1802 0.184 0.488 0.328
#> GSM340276 3 0.0892 0.8002 0.020 0.000 0.980
#> GSM340277 3 0.6260 0.4349 0.448 0.000 0.552
#> GSM340278 3 0.1163 0.7996 0.028 0.000 0.972
#> GSM340279 3 0.0747 0.8026 0.016 0.000 0.984
#> GSM340282 3 0.0892 0.8005 0.020 0.000 0.980
#> GSM340284 3 0.6386 0.4657 0.412 0.004 0.584
#> GSM340285 3 0.1163 0.7996 0.028 0.000 0.972
#> GSM340286 3 0.2063 0.7908 0.044 0.008 0.948
#> GSM340287 3 0.0747 0.8026 0.016 0.000 0.984
#> GSM340288 3 0.3851 0.7380 0.136 0.004 0.860
#> GSM340289 3 0.1765 0.7978 0.040 0.004 0.956
#> GSM340290 3 0.5541 0.5178 0.008 0.252 0.740
#> GSM340291 3 0.6260 0.4349 0.448 0.000 0.552
#> GSM340293 3 0.4178 0.6563 0.000 0.172 0.828
#> GSM340294 3 0.0747 0.8026 0.016 0.000 0.984
#> GSM340296 3 0.0892 0.8025 0.020 0.000 0.980
#> GSM340297 3 0.0424 0.8011 0.008 0.000 0.992
#> GSM340298 3 0.0892 0.8025 0.020 0.000 0.980
#> GSM340299 3 0.1753 0.7937 0.048 0.000 0.952
#> GSM340301 3 0.1289 0.7997 0.032 0.000 0.968
#> GSM340303 3 0.1860 0.7963 0.052 0.000 0.948
#> GSM340304 3 0.1411 0.7988 0.036 0.000 0.964
#> GSM340306 2 0.3267 0.8650 0.000 0.884 0.116
#> GSM340307 3 0.6192 0.4668 0.420 0.000 0.580
#> GSM340310 1 0.8271 0.7188 0.520 0.080 0.400
#> GSM340314 3 0.3043 0.7525 0.008 0.084 0.908
#> GSM340315 3 0.1163 0.8020 0.028 0.000 0.972
#> GSM340317 3 0.9901 0.0470 0.296 0.300 0.404
#> GSM340318 2 0.2356 0.8707 0.000 0.928 0.072
#> GSM340319 2 0.2356 0.8707 0.000 0.928 0.072
#> GSM340320 2 0.3619 0.8420 0.000 0.864 0.136
#> GSM340321 3 0.6260 0.4349 0.448 0.000 0.552
#> GSM340322 2 0.2356 0.8707 0.000 0.928 0.072
#> GSM340324 3 0.6260 0.4349 0.448 0.000 0.552
#> GSM340328 1 0.8513 0.8662 0.596 0.140 0.264
#> GSM340330 3 0.0424 0.8008 0.008 0.000 0.992
#> GSM340332 2 0.3116 0.8687 0.000 0.892 0.108
#> GSM340333 3 0.6204 0.4654 0.424 0.000 0.576
#> GSM340336 2 0.2356 0.8707 0.000 0.928 0.072
#> GSM340337 2 0.2796 0.8726 0.000 0.908 0.092
#> GSM340338 3 0.0592 0.8020 0.012 0.000 0.988
#> GSM340339 2 0.3267 0.8650 0.000 0.884 0.116
#> GSM340340 2 0.6518 0.6730 0.168 0.752 0.080
#> GSM340341 2 0.3267 0.8641 0.000 0.884 0.116
#> GSM340343 3 0.6252 0.3062 0.008 0.344 0.648
#> GSM340344 3 0.5859 0.5581 0.344 0.000 0.656
#> GSM340346 3 0.0592 0.8018 0.012 0.000 0.988
#> GSM340347 2 0.2356 0.8707 0.000 0.928 0.072
#> GSM340348 2 0.3412 0.8574 0.000 0.876 0.124
#> GSM340349 3 0.0747 0.8015 0.016 0.000 0.984
#> GSM340350 3 0.1163 0.8005 0.028 0.000 0.972
#> GSM340351 3 0.1860 0.7936 0.052 0.000 0.948
#> GSM340354 3 0.0747 0.8026 0.016 0.000 0.984
#> GSM340356 3 0.6228 0.5121 0.372 0.004 0.624
#> GSM340357 3 0.1643 0.7987 0.044 0.000 0.956
#> GSM348183 3 0.5153 0.6768 0.068 0.100 0.832
#> GSM348191 3 0.0747 0.8013 0.016 0.000 0.984
#> GSM348193 3 0.5744 0.6189 0.072 0.128 0.800
#> GSM537578 3 0.1031 0.8041 0.024 0.000 0.976
#> GSM348181 3 0.2793 0.7787 0.044 0.028 0.928
#> GSM348182 3 0.1950 0.7959 0.040 0.008 0.952
#> GSM348184 3 0.6260 0.4349 0.448 0.000 0.552
#> GSM348185 2 0.9560 0.1213 0.204 0.452 0.344
#> GSM348186 3 0.6260 0.4349 0.448 0.000 0.552
#> GSM348187 3 0.3851 0.7380 0.136 0.004 0.860
#> GSM348188 2 0.3896 0.8482 0.008 0.864 0.128
#> GSM348189 3 0.1529 0.7953 0.040 0.000 0.960
#> GSM348190 3 0.1031 0.8025 0.024 0.000 0.976
#> GSM348194 3 0.3851 0.7380 0.136 0.004 0.860
#> GSM348195 3 0.1753 0.7951 0.048 0.000 0.952
#> GSM348196 3 0.3851 0.7380 0.136 0.004 0.860
#> GSM537585 3 0.1964 0.7966 0.056 0.000 0.944
#> GSM537594 3 0.6260 0.4349 0.448 0.000 0.552
#> GSM537596 3 0.0892 0.8002 0.020 0.000 0.980
#> GSM537597 3 0.3715 0.7449 0.128 0.004 0.868
#> GSM537602 3 0.1411 0.7988 0.036 0.000 0.964
#> GSM340184 3 0.0424 0.8008 0.008 0.000 0.992
#> GSM340185 3 0.6260 0.4349 0.448 0.000 0.552
#> GSM340186 3 0.6260 0.4349 0.448 0.000 0.552
#> GSM340187 2 0.3116 0.8687 0.000 0.892 0.108
#> GSM340189 2 0.2711 0.8728 0.000 0.912 0.088
#> GSM340190 3 0.6260 0.4349 0.448 0.000 0.552
#> GSM340191 3 0.0424 0.8008 0.008 0.000 0.992
#> GSM340192 3 0.4654 0.6914 0.208 0.000 0.792
#> GSM340193 3 0.1031 0.8012 0.024 0.000 0.976
#> GSM340194 3 0.1753 0.7937 0.048 0.000 0.952
#> GSM340195 3 0.1753 0.7937 0.048 0.000 0.952
#> GSM340196 2 0.2356 0.8707 0.000 0.928 0.072
#> GSM340197 3 0.1643 0.7949 0.044 0.000 0.956
#> GSM340198 3 0.6973 0.4470 0.416 0.020 0.564
#> GSM340199 3 0.0592 0.8018 0.012 0.000 0.988
#> GSM340200 3 0.5678 0.5786 0.316 0.000 0.684
#> GSM340201 2 0.2356 0.8707 0.000 0.928 0.072
#> GSM340202 2 0.2356 0.8707 0.000 0.928 0.072
#> GSM340203 2 0.3619 0.8417 0.000 0.864 0.136
#> GSM340204 3 0.1411 0.8026 0.036 0.000 0.964
#> GSM340205 3 0.2793 0.7787 0.044 0.028 0.928
#> GSM340206 3 0.0424 0.8008 0.008 0.000 0.992
#> GSM340207 3 0.0592 0.8020 0.012 0.000 0.988
#> GSM340237 3 0.6260 0.4349 0.448 0.000 0.552
#> GSM340238 3 0.0424 0.8008 0.008 0.000 0.992
#> GSM340239 3 0.1411 0.7984 0.036 0.000 0.964
#> GSM340240 3 0.1031 0.8009 0.024 0.000 0.976
#> GSM340241 3 0.6260 0.4349 0.448 0.000 0.552
#> GSM340242 3 0.1529 0.7953 0.040 0.000 0.960
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.2965 0.8651 0.892 0.000 0.036 0.072
#> GSM340359 1 0.0524 0.9288 0.988 0.004 0.008 0.000
#> GSM340361 3 0.1637 0.9210 0.060 0.000 0.940 0.000
#> GSM340362 3 0.1637 0.9214 0.060 0.000 0.940 0.000
#> GSM340363 3 0.5012 0.7606 0.116 0.000 0.772 0.112
#> GSM340364 1 0.0469 0.9342 0.988 0.000 0.012 0.000
#> GSM340365 3 0.1022 0.9309 0.032 0.000 0.968 0.000
#> GSM340366 3 0.0921 0.9326 0.028 0.000 0.972 0.000
#> GSM340367 1 0.0469 0.9342 0.988 0.000 0.012 0.000
#> GSM340368 4 0.3945 0.7111 0.216 0.000 0.004 0.780
#> GSM340369 4 0.4677 0.5700 0.316 0.000 0.004 0.680
#> GSM340370 1 0.0469 0.9342 0.988 0.000 0.012 0.000
#> GSM340371 3 0.1302 0.9267 0.044 0.000 0.956 0.000
#> GSM340372 1 0.0469 0.9342 0.988 0.000 0.012 0.000
#> GSM340373 1 0.0469 0.9342 0.988 0.000 0.012 0.000
#> GSM340375 1 0.0592 0.9310 0.984 0.000 0.016 0.000
#> GSM340376 1 0.0469 0.9342 0.988 0.000 0.012 0.000
#> GSM340378 1 0.0469 0.9342 0.988 0.000 0.012 0.000
#> GSM340243 3 0.1109 0.9347 0.028 0.000 0.968 0.004
#> GSM340244 2 0.0336 0.8364 0.000 0.992 0.008 0.000
#> GSM340246 2 0.0592 0.8343 0.000 0.984 0.016 0.000
#> GSM340247 4 0.0000 0.8886 0.000 0.000 0.000 1.000
#> GSM340248 2 0.1022 0.8276 0.000 0.968 0.032 0.000
#> GSM340249 2 0.6292 0.4382 0.016 0.608 0.044 0.332
#> GSM340250 3 0.0592 0.9348 0.016 0.000 0.984 0.000
#> GSM340251 2 0.4149 0.7173 0.000 0.804 0.028 0.168
#> GSM340252 2 0.5496 0.6475 0.016 0.724 0.040 0.220
#> GSM340253 2 0.2830 0.7961 0.060 0.900 0.040 0.000
#> GSM340254 2 0.2408 0.8079 0.044 0.920 0.036 0.000
#> GSM340256 2 0.0524 0.8355 0.004 0.988 0.008 0.000
#> GSM340258 3 0.0707 0.9330 0.020 0.000 0.980 0.000
#> GSM340259 3 0.1022 0.9318 0.032 0.000 0.968 0.000
#> GSM340260 3 0.0707 0.9335 0.020 0.000 0.980 0.000
#> GSM340261 3 0.0707 0.9341 0.020 0.000 0.980 0.000
#> GSM340262 3 0.1557 0.9224 0.056 0.000 0.944 0.000
#> GSM340263 3 0.7172 0.3725 0.076 0.308 0.580 0.036
#> GSM340264 3 0.1302 0.9295 0.044 0.000 0.956 0.000
#> GSM340265 3 0.1389 0.9297 0.048 0.000 0.952 0.000
#> GSM340266 3 0.0927 0.9341 0.016 0.000 0.976 0.008
#> GSM340267 3 0.1792 0.9279 0.068 0.000 0.932 0.000
#> GSM340268 3 0.0336 0.9313 0.008 0.000 0.992 0.000
#> GSM340269 3 0.0921 0.9350 0.028 0.000 0.972 0.000
#> GSM340270 3 0.1970 0.9178 0.060 0.000 0.932 0.008
#> GSM537574 3 0.0707 0.9335 0.020 0.000 0.980 0.000
#> GSM537580 3 0.1302 0.9267 0.044 0.000 0.956 0.000
#> GSM537581 3 0.1302 0.9327 0.044 0.000 0.956 0.000
#> GSM340272 3 0.0817 0.9310 0.024 0.000 0.976 0.000
#> GSM340273 2 0.6370 0.5185 0.180 0.668 0.148 0.004
#> GSM340275 4 0.6353 0.0436 0.008 0.436 0.044 0.512
#> GSM340276 3 0.0921 0.9313 0.028 0.000 0.972 0.000
#> GSM340277 2 0.0000 0.8367 0.000 1.000 0.000 0.000
#> GSM340278 3 0.1118 0.9304 0.036 0.000 0.964 0.000
#> GSM340279 3 0.0707 0.9341 0.020 0.000 0.980 0.000
#> GSM340282 3 0.0817 0.9310 0.024 0.000 0.976 0.000
#> GSM340284 2 0.2999 0.7405 0.000 0.864 0.132 0.004
#> GSM340285 3 0.1305 0.9308 0.036 0.000 0.960 0.004
#> GSM340286 3 0.1970 0.9190 0.060 0.000 0.932 0.008
#> GSM340287 3 0.0707 0.9341 0.020 0.000 0.980 0.000
#> GSM340288 3 0.3710 0.7969 0.192 0.000 0.804 0.004
#> GSM340289 3 0.1743 0.9225 0.056 0.000 0.940 0.004
#> GSM340290 3 0.4663 0.6358 0.012 0.000 0.716 0.272
#> GSM340291 2 0.0000 0.8367 0.000 1.000 0.000 0.000
#> GSM340293 3 0.3311 0.7915 0.000 0.000 0.828 0.172
#> GSM340294 3 0.0707 0.9341 0.020 0.000 0.980 0.000
#> GSM340296 3 0.0707 0.9343 0.020 0.000 0.980 0.000
#> GSM340297 3 0.0336 0.9332 0.008 0.000 0.992 0.000
#> GSM340298 3 0.0707 0.9343 0.020 0.000 0.980 0.000
#> GSM340299 3 0.1637 0.9214 0.060 0.000 0.940 0.000
#> GSM340301 3 0.1211 0.9299 0.040 0.000 0.960 0.000
#> GSM340303 3 0.1661 0.9275 0.052 0.004 0.944 0.000
#> GSM340304 3 0.1302 0.9290 0.044 0.000 0.956 0.000
#> GSM340306 4 0.1637 0.8793 0.000 0.000 0.060 0.940
#> GSM340307 2 0.2281 0.7751 0.000 0.904 0.096 0.000
#> GSM340310 1 0.4663 0.5880 0.716 0.000 0.272 0.012
#> GSM340314 3 0.2480 0.8810 0.008 0.000 0.904 0.088
#> GSM340315 3 0.1302 0.9327 0.044 0.000 0.956 0.000
#> GSM340317 2 0.6471 0.4663 0.024 0.616 0.048 0.312
#> GSM340318 4 0.0000 0.8886 0.000 0.000 0.000 1.000
#> GSM340319 4 0.0000 0.8886 0.000 0.000 0.000 1.000
#> GSM340320 4 0.2345 0.8307 0.000 0.000 0.100 0.900
#> GSM340321 2 0.0000 0.8367 0.000 1.000 0.000 0.000
#> GSM340322 4 0.0000 0.8886 0.000 0.000 0.000 1.000
#> GSM340324 2 0.0000 0.8367 0.000 1.000 0.000 0.000
#> GSM340328 1 0.4215 0.7975 0.824 0.000 0.104 0.072
#> GSM340330 3 0.0336 0.9313 0.008 0.000 0.992 0.000
#> GSM340332 4 0.1792 0.8699 0.000 0.000 0.068 0.932
#> GSM340333 2 0.2830 0.7961 0.060 0.900 0.040 0.000
#> GSM340336 4 0.0000 0.8886 0.000 0.000 0.000 1.000
#> GSM340337 4 0.1022 0.8893 0.000 0.000 0.032 0.968
#> GSM340338 3 0.0707 0.9330 0.020 0.000 0.980 0.000
#> GSM340339 4 0.1637 0.8793 0.000 0.000 0.060 0.940
#> GSM340340 4 0.4815 0.6946 0.216 0.028 0.004 0.752
#> GSM340341 4 0.1661 0.8832 0.000 0.004 0.052 0.944
#> GSM340343 3 0.5268 0.3642 0.012 0.000 0.592 0.396
#> GSM340344 2 0.5690 0.4700 0.060 0.672 0.268 0.000
#> GSM340346 3 0.0469 0.9331 0.012 0.000 0.988 0.000
#> GSM340347 4 0.0000 0.8886 0.000 0.000 0.000 1.000
#> GSM340348 4 0.1940 0.8645 0.000 0.000 0.076 0.924
#> GSM340349 3 0.0592 0.9347 0.016 0.000 0.984 0.000
#> GSM340350 3 0.0921 0.9330 0.028 0.000 0.972 0.000
#> GSM340351 3 0.1637 0.9210 0.060 0.000 0.940 0.000
#> GSM340354 3 0.0592 0.9354 0.016 0.000 0.984 0.000
#> GSM340356 2 0.6370 0.5185 0.180 0.668 0.148 0.004
#> GSM340357 3 0.1389 0.9285 0.048 0.000 0.952 0.000
#> GSM348183 3 0.5012 0.7606 0.116 0.000 0.772 0.112
#> GSM348191 3 0.0817 0.9325 0.024 0.000 0.976 0.000
#> GSM348193 3 0.5664 0.6819 0.124 0.000 0.720 0.156
#> GSM537578 3 0.0817 0.9348 0.024 0.000 0.976 0.000
#> GSM348181 3 0.2773 0.8950 0.072 0.000 0.900 0.028
#> GSM348182 3 0.1970 0.9232 0.060 0.000 0.932 0.008
#> GSM348184 2 0.0000 0.8367 0.000 1.000 0.000 0.000
#> GSM348185 2 0.6375 0.0509 0.008 0.480 0.044 0.468
#> GSM348186 2 0.0000 0.8367 0.000 1.000 0.000 0.000
#> GSM348187 3 0.3710 0.7969 0.192 0.000 0.804 0.004
#> GSM348188 4 0.2179 0.8679 0.012 0.000 0.064 0.924
#> GSM348189 3 0.1302 0.9267 0.044 0.000 0.956 0.000
#> GSM348190 3 0.1022 0.9331 0.032 0.000 0.968 0.000
#> GSM348194 3 0.3710 0.7969 0.192 0.000 0.804 0.004
#> GSM348195 3 0.1557 0.9225 0.056 0.000 0.944 0.000
#> GSM348196 3 0.3710 0.7969 0.192 0.000 0.804 0.004
#> GSM537585 3 0.1716 0.9254 0.064 0.000 0.936 0.000
#> GSM537594 2 0.0000 0.8367 0.000 1.000 0.000 0.000
#> GSM537596 3 0.1022 0.9316 0.032 0.000 0.968 0.000
#> GSM537597 3 0.3626 0.8063 0.184 0.000 0.812 0.004
#> GSM537602 3 0.1302 0.9290 0.044 0.000 0.956 0.000
#> GSM340184 3 0.0336 0.9313 0.008 0.000 0.992 0.000
#> GSM340185 2 0.0000 0.8367 0.000 1.000 0.000 0.000
#> GSM340186 2 0.0000 0.8367 0.000 1.000 0.000 0.000
#> GSM340187 4 0.1792 0.8699 0.000 0.000 0.068 0.932
#> GSM340189 4 0.0817 0.8900 0.000 0.000 0.024 0.976
#> GSM340190 2 0.0000 0.8367 0.000 1.000 0.000 0.000
#> GSM340191 3 0.0336 0.9313 0.008 0.000 0.992 0.000
#> GSM340192 3 0.5713 0.4159 0.040 0.340 0.620 0.000
#> GSM340193 3 0.1022 0.9318 0.032 0.000 0.968 0.000
#> GSM340194 3 0.1637 0.9214 0.060 0.000 0.940 0.000
#> GSM340195 3 0.1637 0.9214 0.060 0.000 0.940 0.000
#> GSM340196 4 0.0000 0.8886 0.000 0.000 0.000 1.000
#> GSM340197 3 0.1557 0.9224 0.056 0.000 0.944 0.000
#> GSM340198 2 0.2107 0.8247 0.016 0.940 0.024 0.020
#> GSM340199 3 0.0469 0.9331 0.012 0.000 0.988 0.000
#> GSM340200 2 0.5268 0.1610 0.008 0.540 0.452 0.000
#> GSM340201 4 0.0000 0.8886 0.000 0.000 0.000 1.000
#> GSM340202 4 0.0000 0.8886 0.000 0.000 0.000 1.000
#> GSM340203 4 0.1940 0.8645 0.000 0.000 0.076 0.924
#> GSM340204 3 0.1724 0.9283 0.032 0.020 0.948 0.000
#> GSM340205 3 0.2699 0.8982 0.068 0.000 0.904 0.028
#> GSM340206 3 0.0336 0.9313 0.008 0.000 0.992 0.000
#> GSM340207 3 0.0707 0.9330 0.020 0.000 0.980 0.000
#> GSM340237 2 0.0000 0.8367 0.000 1.000 0.000 0.000
#> GSM340238 3 0.0336 0.9313 0.008 0.000 0.992 0.000
#> GSM340239 3 0.1389 0.9269 0.048 0.000 0.952 0.000
#> GSM340240 3 0.0921 0.9309 0.028 0.000 0.972 0.000
#> GSM340241 2 0.0000 0.8367 0.000 1.000 0.000 0.000
#> GSM340242 3 0.1302 0.9267 0.044 0.000 0.956 0.000
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.2672 0.88325 0.896 0.000 0.016 0.064 0.024
#> GSM340359 1 0.0324 0.94457 0.992 0.004 0.004 0.000 0.000
#> GSM340361 3 0.3691 0.39545 0.040 0.000 0.804 0.000 0.156
#> GSM340362 3 0.4675 0.43745 0.020 0.000 0.600 0.000 0.380
#> GSM340363 3 0.6487 -0.06526 0.076 0.000 0.600 0.076 0.248
#> GSM340364 1 0.0162 0.94887 0.996 0.000 0.004 0.000 0.000
#> GSM340365 3 0.4415 0.28769 0.008 0.000 0.604 0.000 0.388
#> GSM340366 5 0.4574 0.38518 0.012 0.000 0.412 0.000 0.576
#> GSM340367 1 0.0162 0.94887 0.996 0.000 0.004 0.000 0.000
#> GSM340368 4 0.3628 0.70472 0.216 0.000 0.000 0.772 0.012
#> GSM340369 4 0.4251 0.55710 0.316 0.000 0.000 0.672 0.012
#> GSM340370 1 0.0162 0.94887 0.996 0.000 0.004 0.000 0.000
#> GSM340371 3 0.4455 0.43857 0.008 0.000 0.588 0.000 0.404
#> GSM340372 1 0.0162 0.94887 0.996 0.000 0.004 0.000 0.000
#> GSM340373 1 0.0162 0.94887 0.996 0.000 0.004 0.000 0.000
#> GSM340375 1 0.0290 0.94651 0.992 0.000 0.008 0.000 0.000
#> GSM340376 1 0.0162 0.94887 0.996 0.000 0.004 0.000 0.000
#> GSM340378 1 0.0162 0.94887 0.996 0.000 0.004 0.000 0.000
#> GSM340243 5 0.4659 0.57258 0.012 0.000 0.492 0.000 0.496
#> GSM340244 2 0.0324 0.85704 0.000 0.992 0.004 0.000 0.004
#> GSM340246 2 0.0693 0.85448 0.000 0.980 0.012 0.000 0.008
#> GSM340247 4 0.0000 0.89286 0.000 0.000 0.000 1.000 0.000
#> GSM340248 2 0.0955 0.84877 0.000 0.968 0.028 0.000 0.004
#> GSM340249 2 0.6054 0.47275 0.008 0.596 0.012 0.296 0.088
#> GSM340250 3 0.4138 0.49823 0.016 0.000 0.708 0.000 0.276
#> GSM340251 2 0.3936 0.74227 0.000 0.800 0.004 0.144 0.052
#> GSM340252 2 0.5141 0.67316 0.004 0.712 0.008 0.192 0.084
#> GSM340253 2 0.2547 0.81950 0.016 0.900 0.016 0.000 0.068
#> GSM340254 2 0.2095 0.83034 0.008 0.920 0.012 0.000 0.060
#> GSM340256 2 0.0451 0.85584 0.000 0.988 0.004 0.000 0.008
#> GSM340258 3 0.3861 0.51269 0.008 0.000 0.728 0.000 0.264
#> GSM340259 3 0.1764 0.46189 0.008 0.000 0.928 0.000 0.064
#> GSM340260 3 0.1764 0.49800 0.008 0.000 0.928 0.000 0.064
#> GSM340261 3 0.3160 0.51806 0.004 0.000 0.808 0.000 0.188
#> GSM340262 3 0.4639 0.44562 0.020 0.000 0.612 0.000 0.368
#> GSM340263 3 0.7485 -0.33221 0.044 0.308 0.444 0.004 0.200
#> GSM340264 3 0.4232 0.49451 0.012 0.000 0.676 0.000 0.312
#> GSM340265 3 0.4213 0.48903 0.012 0.000 0.680 0.000 0.308
#> GSM340266 3 0.2408 0.49305 0.004 0.000 0.892 0.008 0.096
#> GSM340267 3 0.4000 0.51056 0.024 0.000 0.748 0.000 0.228
#> GSM340268 3 0.3790 0.51110 0.004 0.000 0.724 0.000 0.272
#> GSM340269 3 0.2411 0.51669 0.008 0.000 0.884 0.000 0.108
#> GSM340270 3 0.3988 0.44577 0.036 0.000 0.768 0.000 0.196
#> GSM537574 3 0.1764 0.49307 0.008 0.000 0.928 0.000 0.064
#> GSM537580 3 0.4455 0.43857 0.008 0.000 0.588 0.000 0.404
#> GSM537581 3 0.4252 0.47280 0.020 0.000 0.700 0.000 0.280
#> GSM340272 3 0.4331 0.29231 0.004 0.000 0.596 0.000 0.400
#> GSM340273 2 0.6129 0.57125 0.140 0.668 0.124 0.000 0.068
#> GSM340275 4 0.5869 0.00471 0.000 0.428 0.004 0.484 0.084
#> GSM340276 3 0.1831 0.44708 0.004 0.000 0.920 0.000 0.076
#> GSM340277 2 0.0000 0.85741 0.000 1.000 0.000 0.000 0.000
#> GSM340278 3 0.2389 0.41764 0.004 0.000 0.880 0.000 0.116
#> GSM340279 3 0.3160 0.51806 0.004 0.000 0.808 0.000 0.188
#> GSM340282 3 0.4276 0.34815 0.004 0.000 0.616 0.000 0.380
#> GSM340284 2 0.3161 0.77184 0.000 0.860 0.092 0.004 0.044
#> GSM340285 3 0.2877 0.36991 0.004 0.000 0.848 0.004 0.144
#> GSM340286 3 0.4817 0.43640 0.016 0.000 0.608 0.008 0.368
#> GSM340287 3 0.3123 0.51974 0.004 0.000 0.812 0.000 0.184
#> GSM340288 3 0.4929 0.24150 0.148 0.000 0.716 0.000 0.136
#> GSM340289 3 0.3988 0.36746 0.036 0.000 0.768 0.000 0.196
#> GSM340290 3 0.6587 -0.20003 0.004 0.000 0.500 0.244 0.252
#> GSM340291 2 0.0000 0.85741 0.000 1.000 0.000 0.000 0.000
#> GSM340293 3 0.6401 -0.47867 0.000 0.000 0.448 0.172 0.380
#> GSM340294 3 0.3160 0.51806 0.004 0.000 0.808 0.000 0.188
#> GSM340296 5 0.4562 0.61603 0.008 0.000 0.496 0.000 0.496
#> GSM340297 3 0.3934 0.50228 0.008 0.000 0.716 0.000 0.276
#> GSM340298 5 0.4562 0.61415 0.008 0.000 0.492 0.000 0.500
#> GSM340299 3 0.4675 0.43745 0.020 0.000 0.600 0.000 0.380
#> GSM340301 3 0.4540 0.47216 0.020 0.000 0.640 0.000 0.340
#> GSM340303 3 0.4794 0.09192 0.012 0.004 0.520 0.000 0.464
#> GSM340304 3 0.3835 0.00814 0.008 0.000 0.732 0.000 0.260
#> GSM340306 4 0.2054 0.88039 0.000 0.000 0.052 0.920 0.028
#> GSM340307 2 0.2438 0.81189 0.000 0.900 0.060 0.000 0.040
#> GSM340310 1 0.4918 0.60046 0.708 0.000 0.192 0.000 0.100
#> GSM340314 3 0.5412 0.45043 0.004 0.000 0.644 0.088 0.264
#> GSM340315 3 0.4252 0.47280 0.020 0.000 0.700 0.000 0.280
#> GSM340317 2 0.6055 0.50121 0.008 0.608 0.012 0.276 0.096
#> GSM340318 4 0.0000 0.89286 0.000 0.000 0.000 1.000 0.000
#> GSM340319 4 0.0000 0.89286 0.000 0.000 0.000 1.000 0.000
#> GSM340320 4 0.2685 0.83830 0.000 0.000 0.092 0.880 0.028
#> GSM340321 2 0.0000 0.85741 0.000 1.000 0.000 0.000 0.000
#> GSM340322 4 0.0162 0.89222 0.000 0.000 0.000 0.996 0.004
#> GSM340324 2 0.0000 0.85741 0.000 1.000 0.000 0.000 0.000
#> GSM340328 1 0.4045 0.84270 0.824 0.000 0.036 0.064 0.076
#> GSM340330 3 0.3949 0.49905 0.004 0.000 0.696 0.000 0.300
#> GSM340332 4 0.1697 0.87369 0.000 0.000 0.060 0.932 0.008
#> GSM340333 2 0.2547 0.81950 0.016 0.900 0.016 0.000 0.068
#> GSM340336 4 0.0000 0.89286 0.000 0.000 0.000 1.000 0.000
#> GSM340337 4 0.1082 0.89233 0.000 0.000 0.028 0.964 0.008
#> GSM340338 3 0.3957 0.51069 0.008 0.000 0.712 0.000 0.280
#> GSM340339 4 0.2054 0.88066 0.000 0.000 0.052 0.920 0.028
#> GSM340340 4 0.4308 0.69420 0.212 0.024 0.008 0.752 0.004
#> GSM340341 4 0.1978 0.88461 0.000 0.004 0.044 0.928 0.024
#> GSM340343 3 0.6821 -0.26187 0.004 0.000 0.392 0.368 0.236
#> GSM340344 2 0.5482 0.43505 0.016 0.672 0.224 0.000 0.088
#> GSM340346 3 0.4350 -0.28347 0.004 0.000 0.588 0.000 0.408
#> GSM340347 4 0.0000 0.89286 0.000 0.000 0.000 1.000 0.000
#> GSM340348 4 0.2344 0.87013 0.000 0.000 0.064 0.904 0.032
#> GSM340349 3 0.4182 -0.07996 0.004 0.000 0.644 0.000 0.352
#> GSM340350 3 0.3890 0.31153 0.012 0.000 0.736 0.000 0.252
#> GSM340351 3 0.3691 0.39545 0.040 0.000 0.804 0.000 0.156
#> GSM340354 3 0.2462 0.51203 0.008 0.000 0.880 0.000 0.112
#> GSM340356 2 0.6129 0.57125 0.140 0.668 0.124 0.000 0.068
#> GSM340357 3 0.4193 0.21325 0.024 0.000 0.720 0.000 0.256
#> GSM348183 3 0.6487 -0.06526 0.076 0.000 0.600 0.076 0.248
#> GSM348191 3 0.1952 0.47401 0.004 0.000 0.912 0.000 0.084
#> GSM348193 3 0.7090 -0.17994 0.084 0.000 0.544 0.120 0.252
#> GSM537578 3 0.4367 -0.42206 0.004 0.000 0.580 0.000 0.416
#> GSM348181 3 0.4201 0.23793 0.044 0.000 0.752 0.000 0.204
#> GSM348182 3 0.4804 0.44320 0.016 0.000 0.612 0.008 0.364
#> GSM348184 2 0.0162 0.85711 0.000 0.996 0.000 0.000 0.004
#> GSM348185 2 0.5956 0.10467 0.000 0.472 0.004 0.432 0.092
#> GSM348186 2 0.0000 0.85741 0.000 1.000 0.000 0.000 0.000
#> GSM348187 3 0.4970 0.24520 0.148 0.000 0.712 0.000 0.140
#> GSM348188 4 0.2433 0.87081 0.012 0.000 0.056 0.908 0.024
#> GSM348189 3 0.4455 0.43857 0.008 0.000 0.588 0.000 0.404
#> GSM348190 3 0.2006 0.48308 0.012 0.000 0.916 0.000 0.072
#> GSM348194 3 0.5010 0.23727 0.148 0.000 0.708 0.000 0.144
#> GSM348195 3 0.3531 0.40921 0.036 0.000 0.816 0.000 0.148
#> GSM348196 3 0.5010 0.23727 0.148 0.000 0.708 0.000 0.144
#> GSM537585 3 0.3769 0.44699 0.032 0.000 0.788 0.000 0.180
#> GSM537594 2 0.0000 0.85741 0.000 1.000 0.000 0.000 0.000
#> GSM537596 3 0.1956 0.44427 0.008 0.000 0.916 0.000 0.076
#> GSM537597 3 0.4805 0.27093 0.144 0.000 0.728 0.000 0.128
#> GSM537602 3 0.3835 0.00814 0.008 0.000 0.732 0.000 0.260
#> GSM340184 3 0.3884 0.50357 0.004 0.000 0.708 0.000 0.288
#> GSM340185 2 0.0162 0.85711 0.000 0.996 0.000 0.000 0.004
#> GSM340186 2 0.0162 0.85711 0.000 0.996 0.000 0.000 0.004
#> GSM340187 4 0.1697 0.87369 0.000 0.000 0.060 0.932 0.008
#> GSM340189 4 0.0798 0.89307 0.000 0.000 0.016 0.976 0.008
#> GSM340190 2 0.0162 0.85711 0.000 0.996 0.000 0.000 0.004
#> GSM340191 3 0.3838 0.50686 0.004 0.000 0.716 0.000 0.280
#> GSM340192 5 0.7047 0.35964 0.008 0.340 0.300 0.000 0.352
#> GSM340193 3 0.1830 0.46883 0.008 0.000 0.924 0.000 0.068
#> GSM340194 3 0.4675 0.43745 0.020 0.000 0.600 0.000 0.380
#> GSM340195 3 0.4686 0.43457 0.020 0.000 0.596 0.000 0.384
#> GSM340196 4 0.0000 0.89286 0.000 0.000 0.000 1.000 0.000
#> GSM340197 3 0.4639 0.44562 0.020 0.000 0.612 0.000 0.368
#> GSM340198 2 0.1948 0.84297 0.004 0.928 0.004 0.008 0.056
#> GSM340199 3 0.4084 0.11967 0.004 0.000 0.668 0.000 0.328
#> GSM340200 2 0.6402 0.12696 0.004 0.536 0.208 0.000 0.252
#> GSM340201 4 0.0000 0.89286 0.000 0.000 0.000 1.000 0.000
#> GSM340202 4 0.0000 0.89286 0.000 0.000 0.000 1.000 0.000
#> GSM340203 4 0.2110 0.86785 0.000 0.000 0.072 0.912 0.016
#> GSM340204 3 0.2554 0.44722 0.008 0.020 0.896 0.000 0.076
#> GSM340205 3 0.4193 0.24216 0.040 0.000 0.748 0.000 0.212
#> GSM340206 3 0.3949 0.49905 0.004 0.000 0.696 0.000 0.300
#> GSM340207 3 0.3910 0.51054 0.008 0.000 0.720 0.000 0.272
#> GSM340237 2 0.0000 0.85741 0.000 1.000 0.000 0.000 0.000
#> GSM340238 3 0.3949 0.49905 0.004 0.000 0.696 0.000 0.300
#> GSM340239 3 0.4227 0.49634 0.016 0.000 0.692 0.000 0.292
#> GSM340240 3 0.4517 0.16832 0.008 0.000 0.556 0.000 0.436
#> GSM340241 2 0.0000 0.85741 0.000 1.000 0.000 0.000 0.000
#> GSM340242 3 0.4455 0.43857 0.008 0.000 0.588 0.000 0.404
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.2559 0.8846 0.896 0.000 0.020 0.052 0.008 0.024
#> GSM340359 1 0.0146 0.9434 0.996 0.000 0.000 0.000 0.004 0.000
#> GSM340361 3 0.3757 0.4868 0.032 0.000 0.804 0.000 0.124 0.040
#> GSM340362 5 0.3653 0.6046 0.008 0.000 0.300 0.000 0.692 0.000
#> GSM340363 3 0.6005 0.3110 0.052 0.000 0.672 0.072 0.100 0.104
#> GSM340364 1 0.0291 0.9473 0.992 0.000 0.004 0.000 0.004 0.000
#> GSM340365 5 0.6197 0.2486 0.004 0.000 0.352 0.000 0.376 0.268
#> GSM340366 6 0.5807 0.4587 0.004 0.000 0.204 0.000 0.272 0.520
#> GSM340367 1 0.0291 0.9473 0.992 0.000 0.004 0.000 0.004 0.000
#> GSM340368 4 0.3859 0.6867 0.204 0.000 0.000 0.756 0.016 0.024
#> GSM340369 4 0.4347 0.5479 0.304 0.000 0.000 0.660 0.012 0.024
#> GSM340370 1 0.0291 0.9473 0.992 0.000 0.004 0.000 0.004 0.000
#> GSM340371 5 0.3404 0.6039 0.000 0.000 0.224 0.000 0.760 0.016
#> GSM340372 1 0.0291 0.9473 0.992 0.000 0.004 0.000 0.004 0.000
#> GSM340373 1 0.0291 0.9473 0.992 0.000 0.004 0.000 0.004 0.000
#> GSM340375 1 0.0405 0.9445 0.988 0.000 0.008 0.000 0.004 0.000
#> GSM340376 1 0.0291 0.9473 0.992 0.000 0.004 0.000 0.004 0.000
#> GSM340378 1 0.0291 0.9473 0.992 0.000 0.004 0.000 0.004 0.000
#> GSM340243 6 0.4287 0.5702 0.008 0.000 0.312 0.000 0.024 0.656
#> GSM340244 2 0.0291 0.8562 0.000 0.992 0.004 0.000 0.004 0.000
#> GSM340246 2 0.0870 0.8550 0.000 0.972 0.012 0.000 0.004 0.012
#> GSM340247 4 0.0000 0.8732 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340248 2 0.1053 0.8501 0.000 0.964 0.020 0.000 0.004 0.012
#> GSM340249 2 0.6970 0.3552 0.000 0.484 0.020 0.272 0.060 0.164
#> GSM340250 3 0.6235 -0.2576 0.012 0.000 0.412 0.000 0.360 0.216
#> GSM340251 2 0.5104 0.7122 0.000 0.724 0.012 0.120 0.052 0.092
#> GSM340252 2 0.6529 0.5967 0.004 0.596 0.016 0.168 0.080 0.136
#> GSM340253 2 0.2370 0.8171 0.008 0.896 0.012 0.000 0.076 0.008
#> GSM340254 2 0.1900 0.8283 0.000 0.916 0.008 0.000 0.068 0.008
#> GSM340256 2 0.0551 0.8547 0.000 0.984 0.004 0.000 0.008 0.004
#> GSM340258 5 0.5686 0.4431 0.004 0.000 0.416 0.000 0.444 0.136
#> GSM340259 3 0.3481 0.4435 0.000 0.000 0.792 0.000 0.160 0.048
#> GSM340260 3 0.4392 0.3682 0.004 0.000 0.708 0.000 0.216 0.072
#> GSM340261 3 0.5492 -0.0549 0.000 0.000 0.536 0.000 0.312 0.152
#> GSM340262 5 0.3967 0.6046 0.008 0.000 0.316 0.000 0.668 0.008
#> GSM340263 3 0.6925 0.0196 0.036 0.308 0.500 0.004 0.064 0.088
#> GSM340264 5 0.5063 0.4717 0.004 0.000 0.432 0.000 0.500 0.064
#> GSM340265 5 0.5345 0.4591 0.004 0.000 0.424 0.000 0.480 0.092
#> GSM340266 3 0.4771 0.3619 0.000 0.000 0.692 0.008 0.184 0.116
#> GSM340267 3 0.4927 0.1611 0.020 0.000 0.640 0.000 0.284 0.056
#> GSM340268 5 0.5812 0.4322 0.000 0.000 0.348 0.000 0.460 0.192
#> GSM340269 3 0.4796 0.2279 0.004 0.000 0.652 0.000 0.260 0.084
#> GSM340270 3 0.5254 0.3551 0.024 0.000 0.628 0.000 0.264 0.084
#> GSM537574 3 0.4416 0.3466 0.004 0.000 0.708 0.000 0.212 0.076
#> GSM537580 5 0.3404 0.6039 0.000 0.000 0.224 0.000 0.760 0.016
#> GSM537581 3 0.6003 -0.0245 0.016 0.000 0.496 0.000 0.324 0.164
#> GSM340272 5 0.6104 0.2326 0.000 0.000 0.336 0.000 0.372 0.292
#> GSM340273 2 0.5347 0.5816 0.140 0.668 0.164 0.000 0.008 0.020
#> GSM340275 4 0.6879 0.1794 0.000 0.324 0.016 0.460 0.060 0.140
#> GSM340276 3 0.3285 0.4722 0.000 0.000 0.820 0.000 0.116 0.064
#> GSM340277 2 0.0000 0.8552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340278 3 0.4352 0.4378 0.000 0.000 0.724 0.000 0.128 0.148
#> GSM340279 3 0.5492 -0.0549 0.000 0.000 0.536 0.000 0.312 0.152
#> GSM340282 5 0.6044 0.3660 0.000 0.000 0.328 0.000 0.408 0.264
#> GSM340284 2 0.4030 0.7738 0.000 0.804 0.060 0.004 0.052 0.080
#> GSM340285 3 0.4513 0.4388 0.000 0.000 0.704 0.000 0.124 0.172
#> GSM340286 5 0.4187 0.5285 0.004 0.000 0.356 0.000 0.624 0.016
#> GSM340287 3 0.5463 -0.0579 0.000 0.000 0.540 0.000 0.312 0.148
#> GSM340288 3 0.3806 0.4616 0.144 0.000 0.792 0.000 0.024 0.040
#> GSM340289 3 0.5388 0.4184 0.028 0.000 0.648 0.000 0.192 0.132
#> GSM340290 3 0.6622 0.0740 0.000 0.000 0.524 0.224 0.092 0.160
#> GSM340291 2 0.0260 0.8559 0.000 0.992 0.000 0.000 0.008 0.000
#> GSM340293 6 0.6235 0.5409 0.000 0.000 0.192 0.160 0.072 0.576
#> GSM340294 3 0.5492 -0.0549 0.000 0.000 0.536 0.000 0.312 0.152
#> GSM340296 6 0.4013 0.6540 0.008 0.000 0.212 0.000 0.040 0.740
#> GSM340297 3 0.6082 -0.2894 0.004 0.000 0.392 0.000 0.384 0.220
#> GSM340298 6 0.4077 0.6555 0.008 0.000 0.212 0.000 0.044 0.736
#> GSM340299 5 0.3653 0.6046 0.008 0.000 0.300 0.000 0.692 0.000
#> GSM340301 5 0.4079 0.5623 0.004 0.000 0.380 0.000 0.608 0.008
#> GSM340303 5 0.6324 0.1339 0.004 0.004 0.344 0.000 0.376 0.272
#> GSM340304 3 0.4928 0.1784 0.004 0.000 0.624 0.000 0.084 0.288
#> GSM340306 4 0.2994 0.8533 0.004 0.000 0.044 0.872 0.040 0.040
#> GSM340307 2 0.2883 0.8134 0.000 0.864 0.016 0.000 0.032 0.088
#> GSM340310 1 0.4847 0.6355 0.680 0.000 0.236 0.000 0.036 0.048
#> GSM340314 5 0.6673 0.3870 0.000 0.000 0.248 0.084 0.504 0.164
#> GSM340315 3 0.6003 -0.0245 0.016 0.000 0.496 0.000 0.324 0.164
#> GSM340317 2 0.6961 0.3868 0.000 0.496 0.024 0.256 0.056 0.168
#> GSM340318 4 0.0000 0.8732 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340319 4 0.0000 0.8732 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340320 4 0.3591 0.8248 0.004 0.000 0.080 0.832 0.044 0.040
#> GSM340321 2 0.0000 0.8552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340322 4 0.1245 0.8701 0.000 0.000 0.000 0.952 0.032 0.016
#> GSM340324 2 0.0146 0.8556 0.004 0.996 0.000 0.000 0.000 0.000
#> GSM340328 1 0.4175 0.8326 0.808 0.000 0.068 0.052 0.036 0.036
#> GSM340330 5 0.5492 0.4856 0.000 0.000 0.244 0.000 0.564 0.192
#> GSM340332 4 0.1682 0.8588 0.000 0.000 0.020 0.928 0.052 0.000
#> GSM340333 2 0.2370 0.8171 0.008 0.896 0.012 0.000 0.076 0.008
#> GSM340336 4 0.0000 0.8732 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340337 4 0.2016 0.8679 0.000 0.000 0.024 0.920 0.040 0.016
#> GSM340338 5 0.5712 0.4188 0.004 0.000 0.416 0.000 0.440 0.140
#> GSM340339 4 0.3062 0.8530 0.004 0.000 0.044 0.868 0.044 0.040
#> GSM340340 4 0.4347 0.6865 0.192 0.012 0.008 0.748 0.012 0.028
#> GSM340341 4 0.2776 0.8576 0.000 0.004 0.040 0.884 0.044 0.028
#> GSM340343 3 0.6638 -0.0655 0.000 0.000 0.444 0.344 0.068 0.144
#> GSM340344 2 0.5178 0.4775 0.008 0.668 0.140 0.000 0.176 0.008
#> GSM340346 6 0.5190 0.5811 0.000 0.000 0.280 0.000 0.128 0.592
#> GSM340347 4 0.0000 0.8732 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340348 4 0.3253 0.8465 0.004 0.000 0.056 0.856 0.040 0.044
#> GSM340349 6 0.5657 0.3162 0.004 0.000 0.412 0.000 0.132 0.452
#> GSM340350 3 0.5869 0.1288 0.012 0.000 0.528 0.000 0.172 0.288
#> GSM340351 3 0.3757 0.4868 0.032 0.000 0.804 0.000 0.124 0.040
#> GSM340354 3 0.5176 0.1855 0.004 0.000 0.620 0.000 0.248 0.128
#> GSM340356 2 0.5347 0.5816 0.140 0.668 0.164 0.000 0.008 0.020
#> GSM340357 3 0.5842 0.1482 0.024 0.000 0.548 0.000 0.132 0.296
#> GSM348183 3 0.6047 0.3156 0.052 0.000 0.668 0.072 0.104 0.104
#> GSM348191 3 0.3786 0.4381 0.000 0.000 0.768 0.000 0.168 0.064
#> GSM348193 3 0.6536 0.2284 0.056 0.000 0.624 0.112 0.096 0.112
#> GSM537578 6 0.4976 0.5908 0.004 0.000 0.324 0.000 0.076 0.596
#> GSM348181 3 0.4757 0.4662 0.036 0.000 0.728 0.000 0.136 0.100
#> GSM348182 5 0.4167 0.5119 0.000 0.000 0.368 0.000 0.612 0.020
#> GSM348184 2 0.1524 0.8472 0.000 0.932 0.000 0.000 0.008 0.060
#> GSM348185 4 0.7046 0.0156 0.000 0.364 0.016 0.404 0.064 0.152
#> GSM348186 2 0.0000 0.8552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM348187 3 0.3888 0.4562 0.144 0.000 0.788 0.000 0.036 0.032
#> GSM348188 4 0.3155 0.8458 0.008 0.000 0.056 0.864 0.040 0.032
#> GSM348189 5 0.3404 0.6039 0.000 0.000 0.224 0.000 0.760 0.016
#> GSM348190 3 0.3810 0.3994 0.004 0.000 0.752 0.000 0.208 0.036
#> GSM348194 3 0.3815 0.4570 0.144 0.000 0.792 0.000 0.028 0.036
#> GSM348195 3 0.3583 0.4834 0.032 0.000 0.820 0.000 0.108 0.040
#> GSM348196 3 0.3815 0.4570 0.144 0.000 0.792 0.000 0.028 0.036
#> GSM537585 3 0.3813 0.4439 0.024 0.000 0.788 0.000 0.152 0.036
#> GSM537594 2 0.0000 0.8552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM537596 3 0.3244 0.4873 0.004 0.000 0.832 0.000 0.100 0.064
#> GSM537597 3 0.4361 0.4733 0.140 0.000 0.760 0.000 0.060 0.040
#> GSM537602 3 0.4882 0.1740 0.004 0.000 0.628 0.000 0.080 0.288
#> GSM340184 5 0.5682 0.4315 0.000 0.000 0.316 0.000 0.504 0.180
#> GSM340185 2 0.1265 0.8500 0.000 0.948 0.000 0.000 0.008 0.044
#> GSM340186 2 0.1462 0.8480 0.000 0.936 0.000 0.000 0.008 0.056
#> GSM340187 4 0.1682 0.8588 0.000 0.000 0.020 0.928 0.052 0.000
#> GSM340189 4 0.0820 0.8723 0.000 0.000 0.016 0.972 0.012 0.000
#> GSM340190 2 0.1524 0.8472 0.000 0.932 0.000 0.000 0.008 0.060
#> GSM340191 5 0.5841 0.3775 0.000 0.000 0.372 0.000 0.436 0.192
#> GSM340192 6 0.6533 0.2597 0.000 0.340 0.108 0.000 0.084 0.468
#> GSM340193 3 0.3455 0.4269 0.000 0.000 0.784 0.000 0.180 0.036
#> GSM340194 5 0.3653 0.6046 0.008 0.000 0.300 0.000 0.692 0.000
#> GSM340195 5 0.3753 0.6054 0.008 0.000 0.292 0.000 0.696 0.004
#> GSM340196 4 0.0000 0.8732 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340197 5 0.3967 0.6046 0.008 0.000 0.316 0.000 0.668 0.008
#> GSM340198 2 0.3668 0.8084 0.004 0.816 0.008 0.008 0.044 0.120
#> GSM340199 6 0.5487 0.4048 0.000 0.000 0.356 0.000 0.136 0.508
#> GSM340200 2 0.5352 0.2326 0.000 0.500 0.084 0.000 0.008 0.408
#> GSM340201 4 0.0000 0.8732 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340202 4 0.0000 0.8732 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340203 4 0.2880 0.8530 0.000 0.000 0.056 0.872 0.048 0.024
#> GSM340204 3 0.3733 0.4654 0.000 0.020 0.792 0.000 0.152 0.036
#> GSM340205 3 0.4743 0.4695 0.032 0.000 0.724 0.000 0.152 0.092
#> GSM340206 5 0.5492 0.4856 0.000 0.000 0.244 0.000 0.564 0.192
#> GSM340207 5 0.5395 0.4961 0.004 0.000 0.400 0.000 0.496 0.100
#> GSM340237 2 0.0260 0.8559 0.000 0.992 0.000 0.000 0.008 0.000
#> GSM340238 5 0.5473 0.4838 0.000 0.000 0.240 0.000 0.568 0.192
#> GSM340239 5 0.5000 0.5282 0.008 0.000 0.416 0.000 0.524 0.052
#> GSM340240 5 0.6183 0.1270 0.004 0.000 0.268 0.000 0.396 0.332
#> GSM340241 2 0.0146 0.8557 0.000 0.996 0.000 0.000 0.004 0.000
#> GSM340242 5 0.3404 0.6039 0.000 0.000 0.224 0.000 0.760 0.016
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
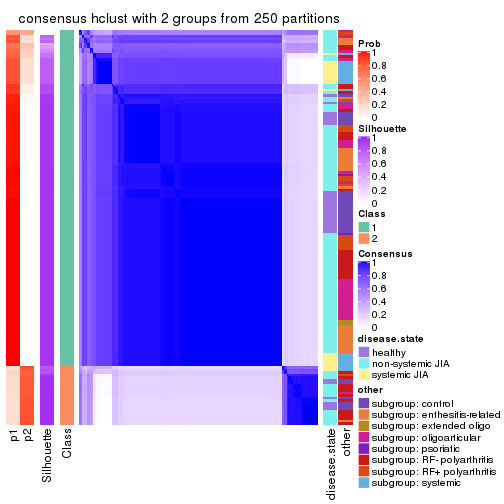
consensus_heatmap(res, k = 3)
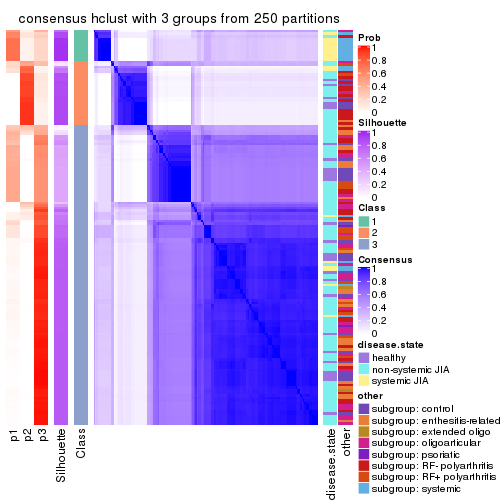
consensus_heatmap(res, k = 4)
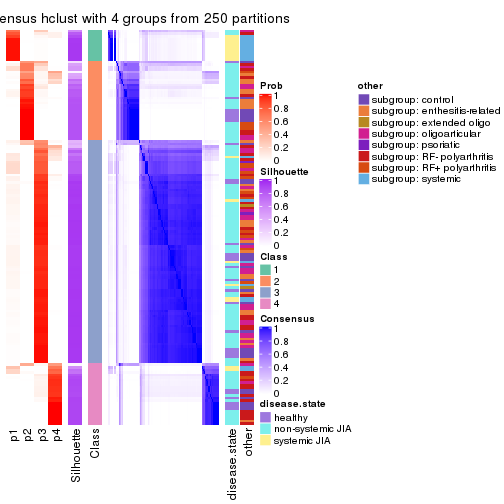
consensus_heatmap(res, k = 5)
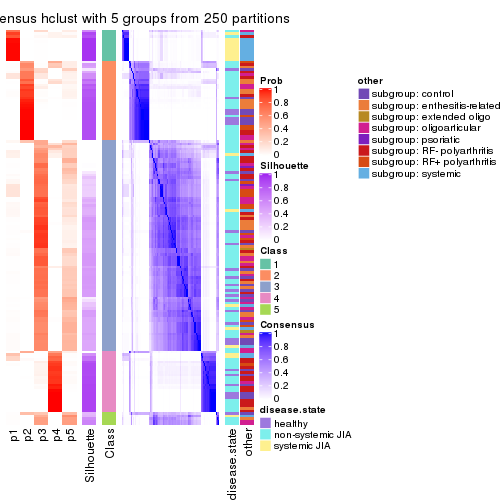
consensus_heatmap(res, k = 6)
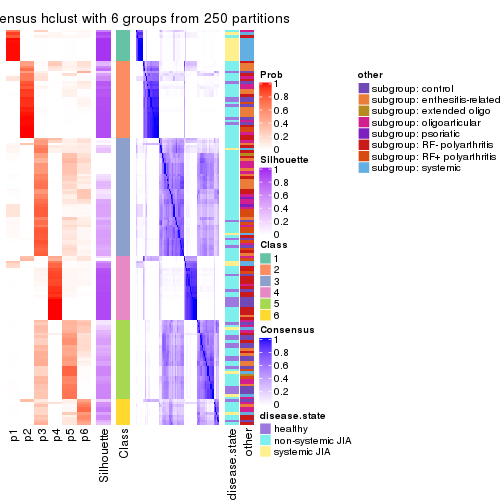
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
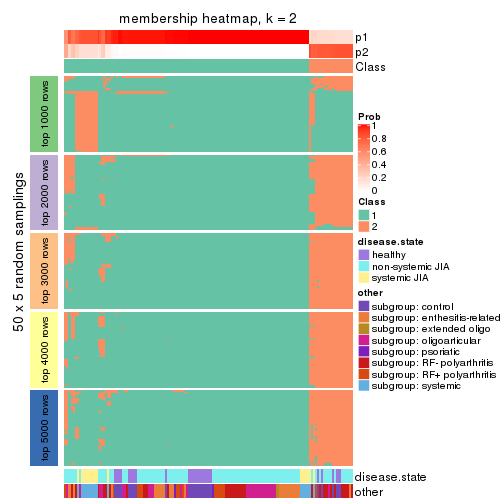
membership_heatmap(res, k = 3)
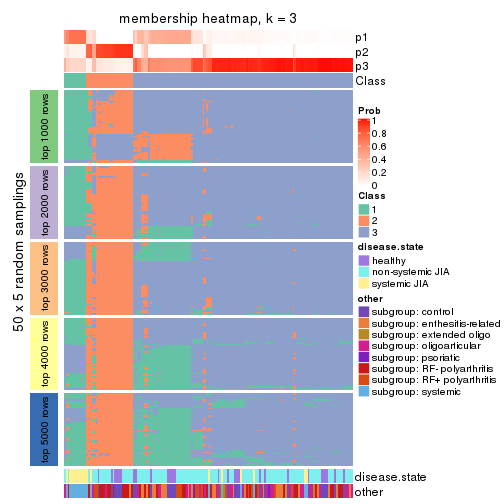
membership_heatmap(res, k = 4)
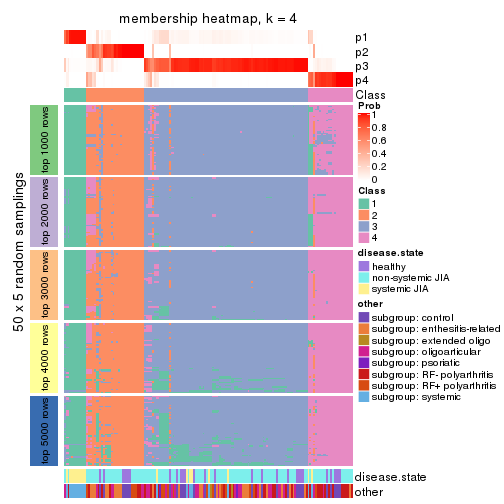
membership_heatmap(res, k = 5)
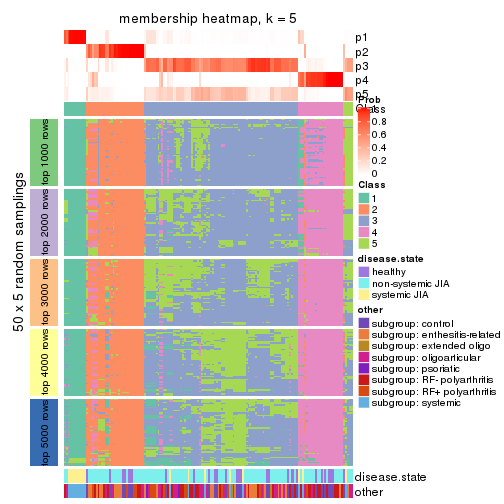
membership_heatmap(res, k = 6)
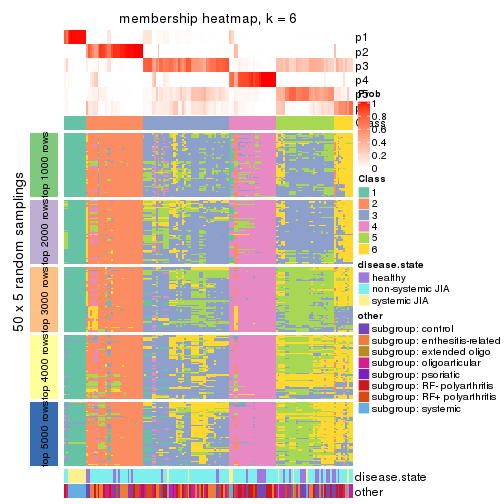
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
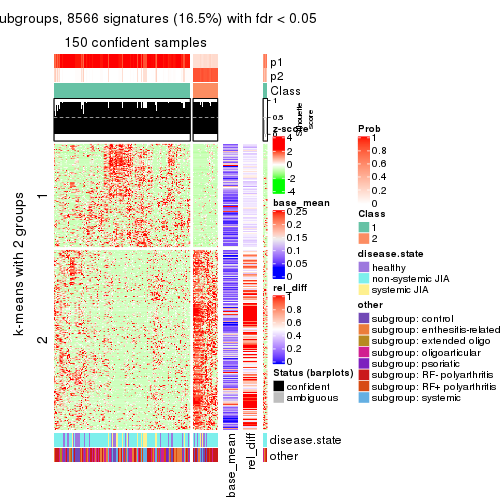
get_signatures(res, k = 3)
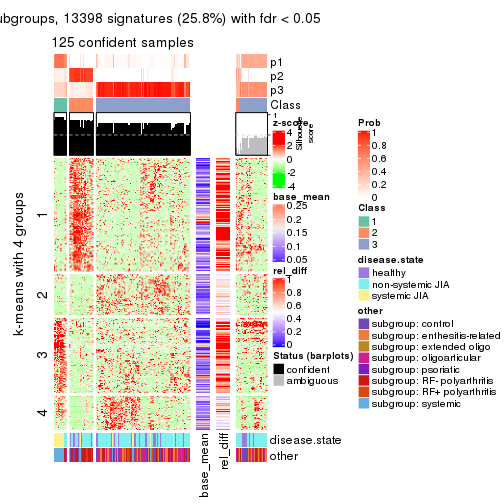
get_signatures(res, k = 4)
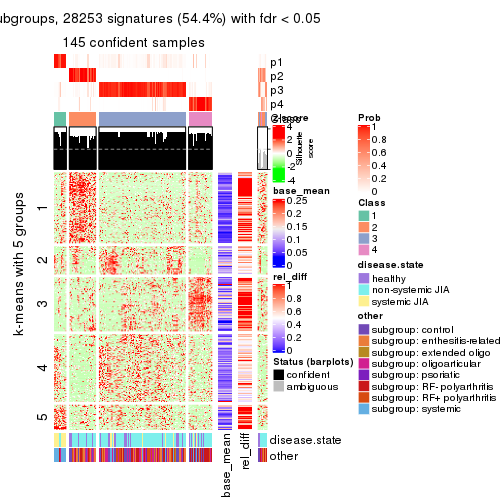
get_signatures(res, k = 5)
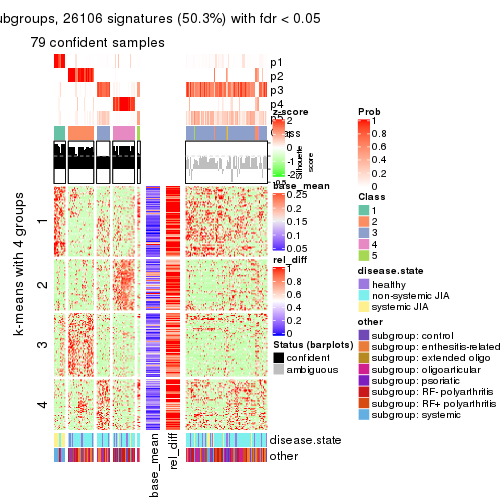
get_signatures(res, k = 6)
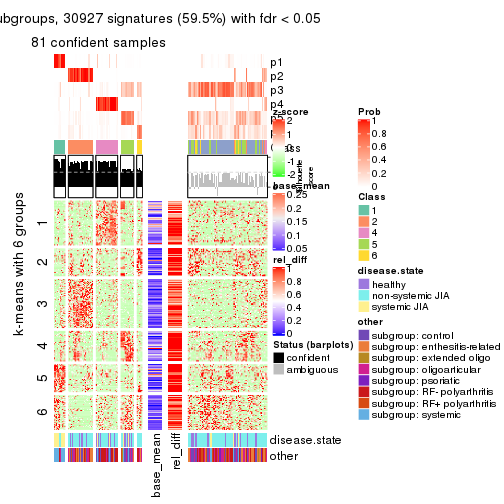
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
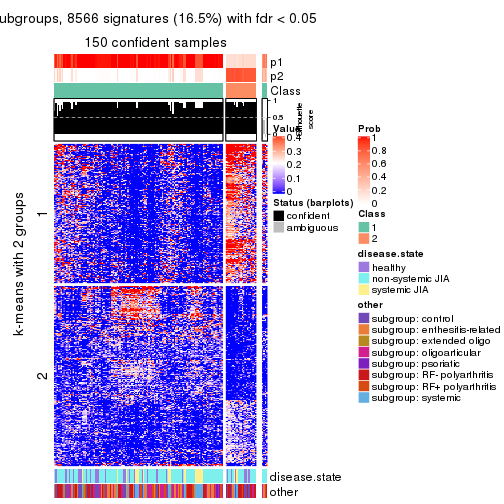
get_signatures(res, k = 3, scale_rows = FALSE)
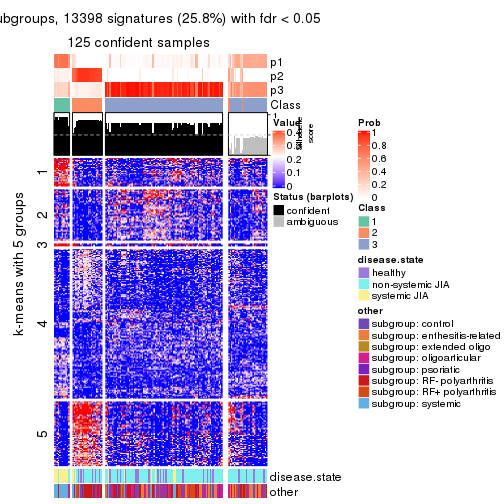
get_signatures(res, k = 4, scale_rows = FALSE)
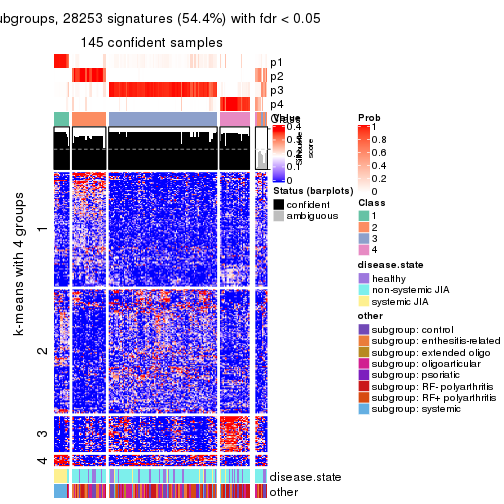
get_signatures(res, k = 5, scale_rows = FALSE)
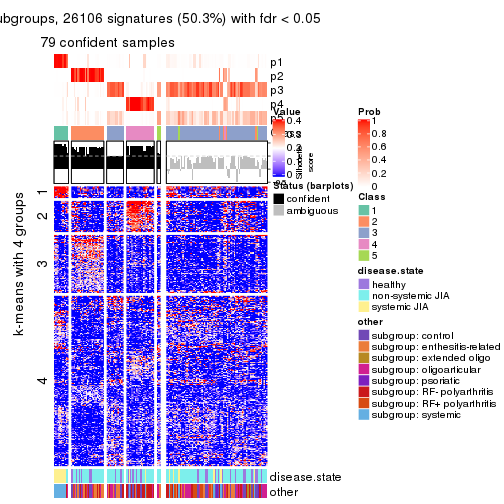
get_signatures(res, k = 6, scale_rows = FALSE)
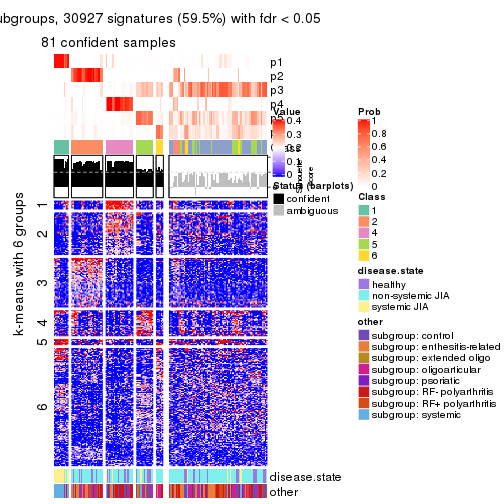
Compare the overlap of signatures from different k:
compare_signatures(res)
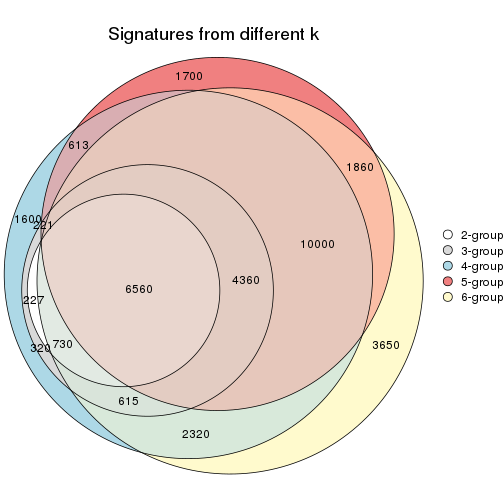
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
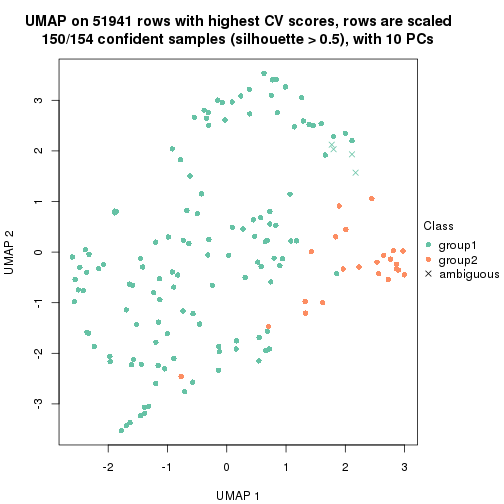
dimension_reduction(res, k = 3, method = "UMAP")
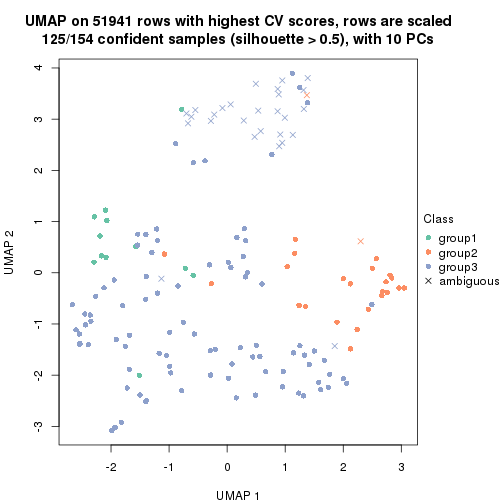
dimension_reduction(res, k = 4, method = "UMAP")
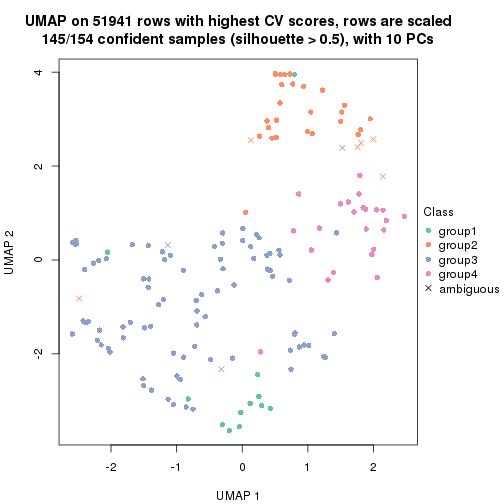
dimension_reduction(res, k = 5, method = "UMAP")
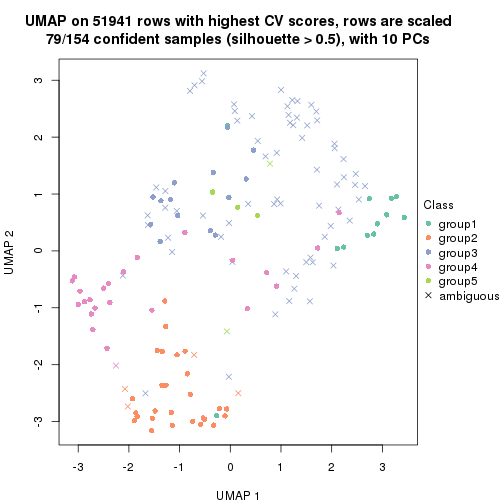
dimension_reduction(res, k = 6, method = "UMAP")
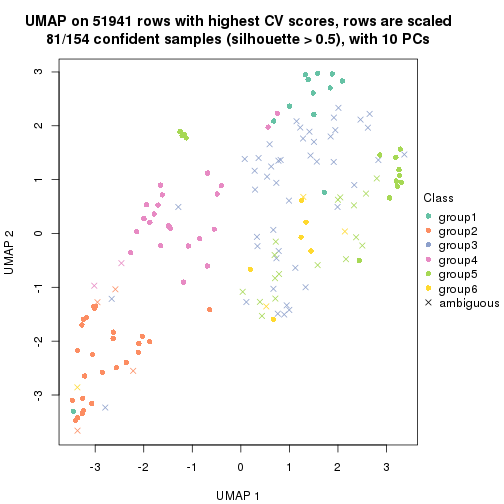
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
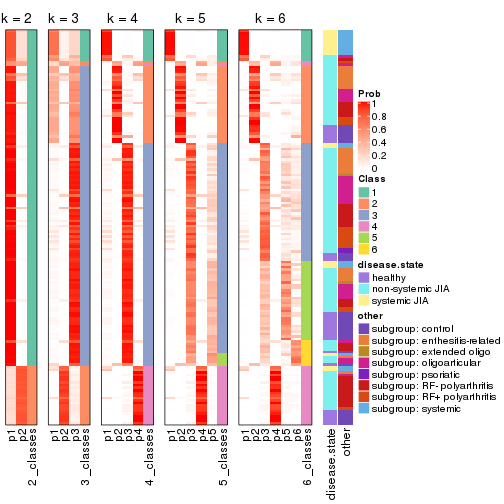
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> CV:hclust 150 6.27e-01 8.62e-03 2
#> CV:hclust 125 1.32e-10 2.23e-09 3
#> CV:hclust 145 1.27e-11 5.99e-10 4
#> CV:hclust 79 8.52e-09 7.72e-09 5
#> CV:hclust 81 5.98e-08 5.69e-08 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["CV", "kmeans"]
# you can also extract it by
# res = res_list["CV:kmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'CV' method.
#> Subgroups are detected by 'kmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 4.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
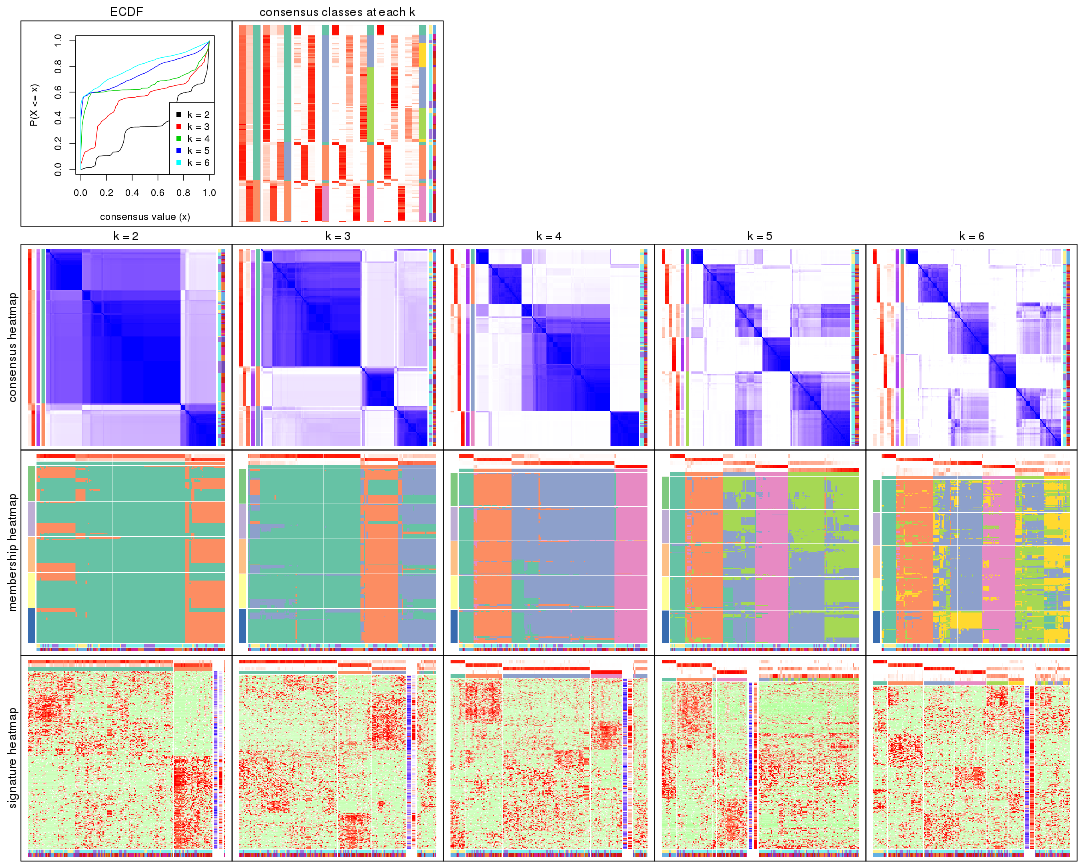
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
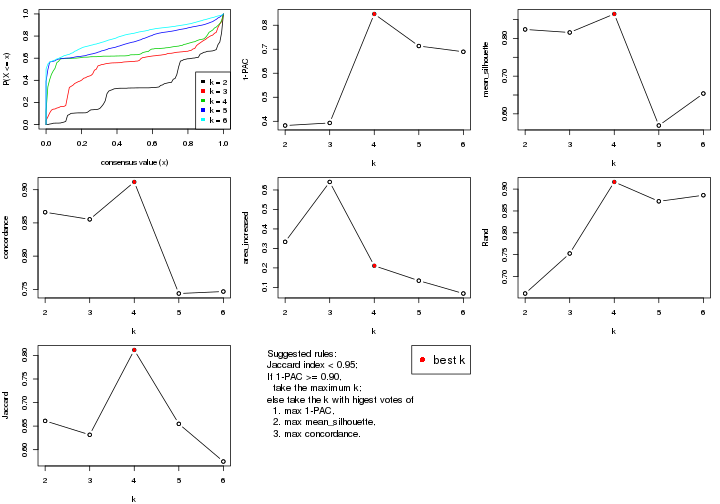
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.382 0.824 0.866 0.3337 0.661 0.661
#> 3 3 0.393 0.816 0.855 0.6411 0.753 0.632
#> 4 4 0.847 0.865 0.912 0.2108 0.916 0.812
#> 5 5 0.713 0.569 0.744 0.1344 0.872 0.655
#> 6 6 0.689 0.654 0.747 0.0684 0.886 0.575
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 4
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 1 0.9661 0.716 0.608 0.392
#> GSM340359 1 0.5408 0.604 0.876 0.124
#> GSM340361 1 0.8207 0.867 0.744 0.256
#> GSM340362 1 0.7815 0.877 0.768 0.232
#> GSM340363 1 0.8713 0.813 0.708 0.292
#> GSM340364 1 0.5842 0.623 0.860 0.140
#> GSM340365 1 0.8267 0.864 0.740 0.260
#> GSM340366 1 0.8267 0.864 0.740 0.260
#> GSM340367 1 0.9286 0.778 0.656 0.344
#> GSM340368 2 0.0938 0.832 0.012 0.988
#> GSM340369 2 0.0672 0.822 0.008 0.992
#> GSM340370 1 0.9286 0.778 0.656 0.344
#> GSM340371 1 0.7815 0.877 0.768 0.232
#> GSM340372 1 0.9286 0.778 0.656 0.344
#> GSM340373 1 0.9286 0.778 0.656 0.344
#> GSM340375 1 0.9286 0.778 0.656 0.344
#> GSM340376 1 0.9286 0.778 0.656 0.344
#> GSM340378 1 0.5842 0.623 0.860 0.140
#> GSM340243 1 0.8267 0.864 0.740 0.260
#> GSM340244 1 0.1633 0.705 0.976 0.024
#> GSM340246 1 0.1633 0.705 0.976 0.024
#> GSM340247 2 0.4815 0.914 0.104 0.896
#> GSM340248 1 0.1633 0.705 0.976 0.024
#> GSM340249 2 0.9286 0.689 0.344 0.656
#> GSM340250 1 0.8267 0.864 0.740 0.260
#> GSM340251 1 0.2603 0.687 0.956 0.044
#> GSM340252 2 0.9286 0.689 0.344 0.656
#> GSM340253 1 0.1414 0.708 0.980 0.020
#> GSM340254 1 0.1633 0.705 0.976 0.024
#> GSM340256 1 0.1633 0.705 0.976 0.024
#> GSM340258 1 0.7815 0.877 0.768 0.232
#> GSM340259 1 0.7745 0.877 0.772 0.228
#> GSM340260 1 0.7950 0.875 0.760 0.240
#> GSM340261 1 0.7745 0.877 0.772 0.228
#> GSM340262 1 0.7815 0.877 0.768 0.232
#> GSM340263 1 0.1633 0.705 0.976 0.024
#> GSM340264 1 0.7815 0.877 0.768 0.232
#> GSM340265 1 0.7745 0.877 0.772 0.228
#> GSM340266 1 0.7815 0.877 0.768 0.232
#> GSM340267 1 0.7745 0.877 0.772 0.228
#> GSM340268 1 0.7815 0.877 0.768 0.232
#> GSM340269 1 0.7815 0.877 0.768 0.232
#> GSM340270 1 0.7815 0.877 0.768 0.232
#> GSM537574 1 0.7745 0.877 0.772 0.228
#> GSM537580 1 0.7815 0.877 0.768 0.232
#> GSM537581 2 0.5059 0.910 0.112 0.888
#> GSM340272 1 0.8016 0.873 0.756 0.244
#> GSM340273 1 0.1633 0.705 0.976 0.024
#> GSM340275 2 0.5059 0.903 0.112 0.888
#> GSM340276 1 0.7815 0.877 0.768 0.232
#> GSM340277 1 0.1633 0.705 0.976 0.024
#> GSM340278 1 0.7815 0.877 0.768 0.232
#> GSM340279 1 0.7815 0.877 0.768 0.232
#> GSM340282 1 0.7950 0.875 0.760 0.240
#> GSM340284 1 0.3274 0.735 0.940 0.060
#> GSM340285 1 0.7815 0.877 0.768 0.232
#> GSM340286 1 0.7815 0.877 0.768 0.232
#> GSM340287 1 0.7745 0.877 0.772 0.228
#> GSM340288 1 0.7950 0.875 0.760 0.240
#> GSM340289 1 0.7950 0.875 0.760 0.240
#> GSM340290 1 0.7815 0.877 0.768 0.232
#> GSM340291 1 0.1633 0.705 0.976 0.024
#> GSM340293 2 0.9993 -0.228 0.484 0.516
#> GSM340294 1 0.7745 0.877 0.772 0.228
#> GSM340296 1 0.8267 0.864 0.740 0.260
#> GSM340297 1 0.7815 0.877 0.768 0.232
#> GSM340298 1 0.8267 0.864 0.740 0.260
#> GSM340299 1 0.7745 0.877 0.772 0.228
#> GSM340301 1 0.7745 0.877 0.772 0.228
#> GSM340303 1 0.7745 0.877 0.772 0.228
#> GSM340304 1 0.7745 0.877 0.772 0.228
#> GSM340306 2 0.4939 0.913 0.108 0.892
#> GSM340307 1 0.1184 0.711 0.984 0.016
#> GSM340310 1 0.9358 0.780 0.648 0.352
#> GSM340314 1 0.7815 0.877 0.768 0.232
#> GSM340315 1 0.7815 0.877 0.768 0.232
#> GSM340317 2 0.9358 0.686 0.352 0.648
#> GSM340318 2 0.4815 0.914 0.104 0.896
#> GSM340319 2 0.4815 0.914 0.104 0.896
#> GSM340320 2 0.5408 0.901 0.124 0.876
#> GSM340321 1 0.1633 0.705 0.976 0.024
#> GSM340322 2 0.4815 0.914 0.104 0.896
#> GSM340324 1 0.3114 0.685 0.944 0.056
#> GSM340328 2 0.6148 0.712 0.152 0.848
#> GSM340330 1 0.7815 0.877 0.768 0.232
#> GSM340332 2 0.4939 0.913 0.108 0.892
#> GSM340333 1 0.1414 0.708 0.980 0.020
#> GSM340336 2 0.4815 0.914 0.104 0.896
#> GSM340337 2 0.4939 0.913 0.108 0.892
#> GSM340338 1 0.7815 0.877 0.768 0.232
#> GSM340339 2 0.4939 0.913 0.108 0.892
#> GSM340340 2 0.4562 0.899 0.096 0.904
#> GSM340341 2 0.5294 0.902 0.120 0.880
#> GSM340343 2 0.4939 0.913 0.108 0.892
#> GSM340344 1 0.0000 0.722 1.000 0.000
#> GSM340346 1 0.7745 0.877 0.772 0.228
#> GSM340347 2 0.4815 0.914 0.104 0.896
#> GSM340348 2 0.5294 0.904 0.120 0.880
#> GSM340349 1 0.8081 0.871 0.752 0.248
#> GSM340350 1 0.8081 0.871 0.752 0.248
#> GSM340351 1 0.7950 0.875 0.760 0.240
#> GSM340354 1 0.7745 0.877 0.772 0.228
#> GSM340356 1 0.1633 0.705 0.976 0.024
#> GSM340357 1 0.7950 0.875 0.760 0.240
#> GSM348183 1 0.8207 0.860 0.744 0.256
#> GSM348191 1 0.7815 0.877 0.768 0.232
#> GSM348193 2 0.5059 0.910 0.112 0.888
#> GSM537578 1 0.8016 0.873 0.756 0.244
#> GSM348181 1 0.7815 0.877 0.768 0.232
#> GSM348182 1 0.7815 0.877 0.768 0.232
#> GSM348184 1 0.1633 0.705 0.976 0.024
#> GSM348185 2 0.9358 0.686 0.352 0.648
#> GSM348186 1 0.1633 0.705 0.976 0.024
#> GSM348187 1 0.8016 0.873 0.756 0.244
#> GSM348188 2 0.4939 0.913 0.108 0.892
#> GSM348189 1 0.7815 0.877 0.768 0.232
#> GSM348190 1 0.7745 0.877 0.772 0.228
#> GSM348194 1 0.7950 0.875 0.760 0.240
#> GSM348195 1 0.8267 0.864 0.740 0.260
#> GSM348196 1 0.7950 0.875 0.760 0.240
#> GSM537585 1 0.7950 0.875 0.760 0.240
#> GSM537594 1 0.1633 0.705 0.976 0.024
#> GSM537596 1 0.7745 0.877 0.772 0.228
#> GSM537597 1 0.7883 0.876 0.764 0.236
#> GSM537602 1 0.8081 0.871 0.752 0.248
#> GSM340184 1 0.7815 0.877 0.768 0.232
#> GSM340185 1 0.1633 0.705 0.976 0.024
#> GSM340186 1 0.1633 0.705 0.976 0.024
#> GSM340187 2 0.5178 0.906 0.116 0.884
#> GSM340189 2 0.4939 0.913 0.108 0.892
#> GSM340190 1 0.1633 0.705 0.976 0.024
#> GSM340191 1 0.7815 0.877 0.768 0.232
#> GSM340192 1 0.0376 0.719 0.996 0.004
#> GSM340193 1 0.7745 0.877 0.772 0.228
#> GSM340194 1 0.7815 0.877 0.768 0.232
#> GSM340195 1 0.7745 0.877 0.772 0.228
#> GSM340196 2 0.4815 0.914 0.104 0.896
#> GSM340197 1 0.7815 0.877 0.768 0.232
#> GSM340198 1 0.1633 0.705 0.976 0.024
#> GSM340199 1 0.7745 0.877 0.772 0.228
#> GSM340200 1 0.1843 0.703 0.972 0.028
#> GSM340201 2 0.4815 0.914 0.104 0.896
#> GSM340202 2 0.4815 0.914 0.104 0.896
#> GSM340203 2 0.5408 0.900 0.124 0.876
#> GSM340204 1 0.7219 0.867 0.800 0.200
#> GSM340205 1 0.8386 0.859 0.732 0.268
#> GSM340206 1 0.7815 0.877 0.768 0.232
#> GSM340207 1 0.7815 0.877 0.768 0.232
#> GSM340237 1 0.3114 0.685 0.944 0.056
#> GSM340238 1 0.7815 0.877 0.768 0.232
#> GSM340239 1 0.7815 0.877 0.768 0.232
#> GSM340240 1 0.7745 0.877 0.772 0.228
#> GSM340241 1 0.1633 0.705 0.976 0.024
#> GSM340242 1 0.7815 0.877 0.768 0.232
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.8719 0.4362 0.548 0.128 0.324
#> GSM340359 3 0.4483 0.5063 0.024 0.128 0.848
#> GSM340361 1 0.4409 0.7989 0.824 0.004 0.172
#> GSM340362 1 0.0747 0.8812 0.984 0.000 0.016
#> GSM340363 1 0.4277 0.8245 0.852 0.016 0.132
#> GSM340364 3 0.7424 0.4449 0.172 0.128 0.700
#> GSM340365 1 0.4172 0.8171 0.840 0.004 0.156
#> GSM340366 1 0.3784 0.8329 0.864 0.004 0.132
#> GSM340367 1 0.8719 0.4362 0.548 0.128 0.324
#> GSM340368 2 0.0237 0.7791 0.000 0.996 0.004
#> GSM340369 2 0.2878 0.7197 0.000 0.904 0.096
#> GSM340370 1 0.8719 0.4362 0.548 0.128 0.324
#> GSM340371 1 0.0747 0.8802 0.984 0.000 0.016
#> GSM340372 1 0.8719 0.4362 0.548 0.128 0.324
#> GSM340373 1 0.8719 0.4362 0.548 0.128 0.324
#> GSM340375 1 0.8719 0.4362 0.548 0.128 0.324
#> GSM340376 1 0.8719 0.4362 0.548 0.128 0.324
#> GSM340378 3 0.7424 0.4449 0.172 0.128 0.700
#> GSM340243 1 0.4233 0.8206 0.836 0.004 0.160
#> GSM340244 3 0.5938 0.8926 0.248 0.020 0.732
#> GSM340246 3 0.6062 0.8961 0.276 0.016 0.708
#> GSM340247 2 0.3682 0.9118 0.116 0.876 0.008
#> GSM340248 3 0.6062 0.8961 0.276 0.016 0.708
#> GSM340249 2 0.6168 0.3285 0.000 0.588 0.412
#> GSM340250 1 0.1647 0.8806 0.960 0.004 0.036
#> GSM340251 3 0.6161 0.8973 0.272 0.020 0.708
#> GSM340252 2 0.5733 0.5097 0.000 0.676 0.324
#> GSM340253 3 0.6062 0.8929 0.276 0.016 0.708
#> GSM340254 3 0.6096 0.8932 0.280 0.016 0.704
#> GSM340256 3 0.6229 0.8960 0.280 0.020 0.700
#> GSM340258 1 0.1031 0.8795 0.976 0.000 0.024
#> GSM340259 1 0.0237 0.8822 0.996 0.000 0.004
#> GSM340260 1 0.0237 0.8822 0.996 0.000 0.004
#> GSM340261 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340262 1 0.1289 0.8774 0.968 0.000 0.032
#> GSM340263 3 0.5858 0.8885 0.240 0.020 0.740
#> GSM340264 1 0.0592 0.8811 0.988 0.000 0.012
#> GSM340265 1 0.0747 0.8802 0.984 0.000 0.016
#> GSM340266 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340267 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340268 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340269 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340270 1 0.0892 0.8816 0.980 0.000 0.020
#> GSM537574 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM537580 1 0.0747 0.8802 0.984 0.000 0.016
#> GSM537581 2 0.3412 0.9112 0.124 0.876 0.000
#> GSM340272 1 0.1529 0.8784 0.960 0.000 0.040
#> GSM340273 3 0.5597 0.8564 0.216 0.020 0.764
#> GSM340275 2 0.4146 0.8713 0.080 0.876 0.044
#> GSM340276 1 0.1643 0.8796 0.956 0.000 0.044
#> GSM340277 3 0.6096 0.8932 0.280 0.016 0.704
#> GSM340278 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340279 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340282 1 0.0592 0.8811 0.988 0.000 0.012
#> GSM340284 3 0.6527 0.8494 0.320 0.020 0.660
#> GSM340285 1 0.2356 0.8712 0.928 0.000 0.072
#> GSM340286 1 0.0592 0.8811 0.988 0.000 0.012
#> GSM340287 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340288 1 0.3619 0.8264 0.864 0.000 0.136
#> GSM340289 1 0.2625 0.8570 0.916 0.000 0.084
#> GSM340290 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340291 3 0.5938 0.8926 0.248 0.020 0.732
#> GSM340293 1 0.5356 0.6675 0.784 0.196 0.020
#> GSM340294 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340296 1 0.3784 0.8376 0.864 0.004 0.132
#> GSM340297 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340298 1 0.3715 0.8428 0.868 0.004 0.128
#> GSM340299 1 0.0747 0.8812 0.984 0.000 0.016
#> GSM340301 1 0.0592 0.8811 0.988 0.000 0.012
#> GSM340303 1 0.0892 0.8808 0.980 0.000 0.020
#> GSM340304 1 0.2711 0.8552 0.912 0.000 0.088
#> GSM340306 2 0.3412 0.9112 0.124 0.876 0.000
#> GSM340307 3 0.5497 0.8835 0.292 0.000 0.708
#> GSM340310 1 0.8491 0.4988 0.588 0.128 0.284
#> GSM340314 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340315 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340317 3 0.6195 0.4603 0.020 0.276 0.704
#> GSM340318 2 0.3682 0.9118 0.116 0.876 0.008
#> GSM340319 2 0.3682 0.9118 0.116 0.876 0.008
#> GSM340320 2 0.3482 0.9086 0.128 0.872 0.000
#> GSM340321 3 0.6161 0.8973 0.272 0.020 0.708
#> GSM340322 2 0.3573 0.9120 0.120 0.876 0.004
#> GSM340324 3 0.5092 0.8217 0.176 0.020 0.804
#> GSM340328 2 0.9920 0.0261 0.332 0.388 0.280
#> GSM340330 1 0.1031 0.8795 0.976 0.000 0.024
#> GSM340332 2 0.3412 0.9112 0.124 0.876 0.000
#> GSM340333 3 0.6062 0.8929 0.276 0.016 0.708
#> GSM340336 2 0.3846 0.9042 0.108 0.876 0.016
#> GSM340337 2 0.3412 0.9112 0.124 0.876 0.000
#> GSM340338 1 0.1031 0.8795 0.976 0.000 0.024
#> GSM340339 2 0.3412 0.9112 0.124 0.876 0.000
#> GSM340340 2 0.3682 0.9118 0.116 0.876 0.008
#> GSM340341 2 0.3755 0.9114 0.120 0.872 0.008
#> GSM340343 2 0.3482 0.9086 0.128 0.872 0.000
#> GSM340344 1 0.5178 0.4709 0.744 0.000 0.256
#> GSM340346 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340347 2 0.3682 0.9118 0.116 0.876 0.008
#> GSM340348 2 0.3482 0.9086 0.128 0.872 0.000
#> GSM340349 1 0.3038 0.8466 0.896 0.000 0.104
#> GSM340350 1 0.3340 0.8372 0.880 0.000 0.120
#> GSM340351 1 0.3619 0.8264 0.864 0.000 0.136
#> GSM340354 1 0.1031 0.8795 0.976 0.000 0.024
#> GSM340356 3 0.5597 0.8564 0.216 0.020 0.764
#> GSM340357 1 0.3340 0.8372 0.880 0.000 0.120
#> GSM348183 1 0.4677 0.8082 0.840 0.028 0.132
#> GSM348191 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM348193 2 0.3644 0.9097 0.124 0.872 0.004
#> GSM537578 1 0.3116 0.8552 0.892 0.000 0.108
#> GSM348181 1 0.1643 0.8791 0.956 0.000 0.044
#> GSM348182 1 0.0747 0.8812 0.984 0.000 0.016
#> GSM348184 3 0.6161 0.8973 0.272 0.020 0.708
#> GSM348185 2 0.6229 0.4753 0.008 0.652 0.340
#> GSM348186 3 0.6195 0.8936 0.276 0.020 0.704
#> GSM348187 1 0.3619 0.8264 0.864 0.000 0.136
#> GSM348188 2 0.3412 0.9112 0.124 0.876 0.000
#> GSM348189 1 0.0747 0.8802 0.984 0.000 0.016
#> GSM348190 1 0.0747 0.8812 0.984 0.000 0.016
#> GSM348194 1 0.3619 0.8264 0.864 0.000 0.136
#> GSM348195 1 0.4293 0.7996 0.832 0.004 0.164
#> GSM348196 1 0.3619 0.8264 0.864 0.000 0.136
#> GSM537585 1 0.1964 0.8687 0.944 0.000 0.056
#> GSM537594 3 0.5597 0.8564 0.216 0.020 0.764
#> GSM537596 1 0.2261 0.8635 0.932 0.000 0.068
#> GSM537597 1 0.2448 0.8597 0.924 0.000 0.076
#> GSM537602 1 0.3551 0.8292 0.868 0.000 0.132
#> GSM340184 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340185 3 0.6161 0.8973 0.272 0.020 0.708
#> GSM340186 3 0.6090 0.8955 0.264 0.020 0.716
#> GSM340187 2 0.3755 0.9114 0.120 0.872 0.008
#> GSM340189 2 0.3482 0.9086 0.128 0.872 0.000
#> GSM340190 3 0.6161 0.8973 0.272 0.020 0.708
#> GSM340191 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340192 1 0.6305 -0.4297 0.516 0.000 0.484
#> GSM340193 1 0.1163 0.8785 0.972 0.000 0.028
#> GSM340194 1 0.0747 0.8812 0.984 0.000 0.016
#> GSM340195 1 0.0747 0.8812 0.984 0.000 0.016
#> GSM340196 2 0.3682 0.9118 0.116 0.876 0.008
#> GSM340197 1 0.0592 0.8811 0.988 0.000 0.012
#> GSM340198 3 0.6195 0.8936 0.276 0.020 0.704
#> GSM340199 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340200 3 0.6161 0.8973 0.272 0.020 0.708
#> GSM340201 2 0.3682 0.9118 0.116 0.876 0.008
#> GSM340202 2 0.3682 0.9118 0.116 0.876 0.008
#> GSM340203 2 0.3482 0.9086 0.128 0.872 0.000
#> GSM340204 1 0.0747 0.8812 0.984 0.000 0.016
#> GSM340205 1 0.4172 0.8070 0.840 0.004 0.156
#> GSM340206 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340207 1 0.1289 0.8774 0.968 0.000 0.032
#> GSM340237 3 0.5036 0.8358 0.172 0.020 0.808
#> GSM340238 1 0.0892 0.8802 0.980 0.000 0.020
#> GSM340239 1 0.0592 0.8811 0.988 0.000 0.012
#> GSM340240 1 0.0747 0.8802 0.984 0.000 0.016
#> GSM340241 3 0.6090 0.8955 0.264 0.020 0.716
#> GSM340242 1 0.0747 0.8802 0.984 0.000 0.016
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.1109 0.986 0.968 0.004 0.028 0.000
#> GSM340359 1 0.1716 0.911 0.936 0.064 0.000 0.000
#> GSM340361 3 0.5548 0.375 0.448 0.012 0.536 0.004
#> GSM340362 3 0.1296 0.873 0.004 0.028 0.964 0.004
#> GSM340363 3 0.5290 0.395 0.440 0.004 0.552 0.004
#> GSM340364 1 0.1284 0.969 0.964 0.024 0.012 0.000
#> GSM340365 3 0.4998 0.748 0.176 0.048 0.768 0.008
#> GSM340366 3 0.4371 0.800 0.124 0.048 0.820 0.008
#> GSM340367 1 0.1109 0.986 0.968 0.004 0.028 0.000
#> GSM340368 4 0.0376 0.977 0.004 0.004 0.000 0.992
#> GSM340369 4 0.4817 0.415 0.388 0.000 0.000 0.612
#> GSM340370 1 0.1109 0.986 0.968 0.004 0.028 0.000
#> GSM340371 3 0.1510 0.869 0.016 0.028 0.956 0.000
#> GSM340372 1 0.1109 0.986 0.968 0.004 0.028 0.000
#> GSM340373 1 0.1109 0.986 0.968 0.004 0.028 0.000
#> GSM340375 1 0.1109 0.986 0.968 0.004 0.028 0.000
#> GSM340376 1 0.1109 0.986 0.968 0.004 0.028 0.000
#> GSM340378 1 0.1284 0.969 0.964 0.024 0.012 0.000
#> GSM340243 3 0.4520 0.803 0.156 0.036 0.800 0.008
#> GSM340244 2 0.1913 0.941 0.020 0.940 0.040 0.000
#> GSM340246 2 0.1661 0.940 0.004 0.944 0.052 0.000
#> GSM340247 4 0.0376 0.981 0.000 0.004 0.004 0.992
#> GSM340248 2 0.1661 0.940 0.004 0.944 0.052 0.000
#> GSM340249 2 0.4136 0.762 0.016 0.788 0.000 0.196
#> GSM340250 3 0.1706 0.884 0.036 0.016 0.948 0.000
#> GSM340251 2 0.2060 0.939 0.016 0.932 0.052 0.000
#> GSM340252 2 0.5003 0.580 0.016 0.676 0.000 0.308
#> GSM340253 2 0.1576 0.914 0.004 0.948 0.048 0.000
#> GSM340254 2 0.1302 0.920 0.000 0.956 0.044 0.000
#> GSM340256 2 0.1545 0.942 0.008 0.952 0.040 0.000
#> GSM340258 3 0.0707 0.881 0.000 0.020 0.980 0.000
#> GSM340259 3 0.1911 0.885 0.032 0.020 0.944 0.004
#> GSM340260 3 0.1229 0.884 0.020 0.008 0.968 0.004
#> GSM340261 3 0.1833 0.884 0.032 0.024 0.944 0.000
#> GSM340262 3 0.1109 0.873 0.004 0.028 0.968 0.000
#> GSM340263 2 0.2124 0.940 0.028 0.932 0.040 0.000
#> GSM340264 3 0.1109 0.873 0.004 0.028 0.968 0.000
#> GSM340265 3 0.1398 0.872 0.004 0.040 0.956 0.000
#> GSM340266 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM340267 3 0.1297 0.883 0.016 0.020 0.964 0.000
#> GSM340268 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM340269 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM340270 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM537574 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM537580 3 0.1510 0.869 0.016 0.028 0.956 0.000
#> GSM537581 4 0.0524 0.979 0.000 0.004 0.008 0.988
#> GSM340272 3 0.1822 0.870 0.004 0.044 0.944 0.008
#> GSM340273 2 0.2036 0.935 0.032 0.936 0.032 0.000
#> GSM340275 4 0.0712 0.979 0.004 0.008 0.004 0.984
#> GSM340276 3 0.2019 0.883 0.032 0.024 0.940 0.004
#> GSM340277 2 0.1022 0.916 0.000 0.968 0.032 0.000
#> GSM340278 3 0.2019 0.883 0.032 0.024 0.940 0.004
#> GSM340279 3 0.1833 0.884 0.032 0.024 0.944 0.000
#> GSM340282 3 0.1822 0.870 0.004 0.044 0.944 0.008
#> GSM340284 2 0.2222 0.934 0.016 0.924 0.060 0.000
#> GSM340285 3 0.2123 0.883 0.032 0.028 0.936 0.004
#> GSM340286 3 0.1109 0.873 0.004 0.028 0.968 0.000
#> GSM340287 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM340288 3 0.5297 0.386 0.444 0.004 0.548 0.004
#> GSM340289 3 0.2010 0.881 0.060 0.004 0.932 0.004
#> GSM340290 3 0.1833 0.884 0.032 0.024 0.944 0.000
#> GSM340291 2 0.1677 0.940 0.012 0.948 0.040 0.000
#> GSM340293 3 0.3103 0.868 0.020 0.036 0.900 0.044
#> GSM340294 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM340296 3 0.4561 0.805 0.152 0.040 0.800 0.008
#> GSM340297 3 0.1833 0.884 0.032 0.024 0.944 0.000
#> GSM340298 3 0.4561 0.805 0.152 0.040 0.800 0.008
#> GSM340299 3 0.1492 0.872 0.004 0.036 0.956 0.004
#> GSM340301 3 0.1109 0.873 0.004 0.028 0.968 0.000
#> GSM340303 3 0.1822 0.870 0.004 0.044 0.944 0.008
#> GSM340304 3 0.3908 0.840 0.116 0.032 0.844 0.008
#> GSM340306 4 0.0376 0.979 0.000 0.004 0.004 0.992
#> GSM340307 2 0.1305 0.937 0.004 0.960 0.036 0.000
#> GSM340310 1 0.1109 0.986 0.968 0.004 0.028 0.000
#> GSM340314 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM340315 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM340317 2 0.2310 0.912 0.028 0.928 0.004 0.040
#> GSM340318 4 0.0376 0.981 0.000 0.004 0.004 0.992
#> GSM340319 4 0.0376 0.981 0.000 0.004 0.004 0.992
#> GSM340320 4 0.0376 0.979 0.000 0.004 0.004 0.992
#> GSM340321 2 0.1474 0.939 0.000 0.948 0.052 0.000
#> GSM340322 4 0.0376 0.981 0.000 0.004 0.004 0.992
#> GSM340324 2 0.1624 0.932 0.028 0.952 0.020 0.000
#> GSM340328 1 0.1109 0.986 0.968 0.004 0.028 0.000
#> GSM340330 3 0.0895 0.882 0.004 0.020 0.976 0.000
#> GSM340332 4 0.0336 0.979 0.000 0.000 0.008 0.992
#> GSM340333 2 0.1489 0.917 0.004 0.952 0.044 0.000
#> GSM340336 4 0.0376 0.981 0.000 0.004 0.004 0.992
#> GSM340337 4 0.0524 0.981 0.000 0.008 0.004 0.988
#> GSM340338 3 0.0707 0.881 0.000 0.020 0.980 0.000
#> GSM340339 4 0.0524 0.981 0.000 0.008 0.004 0.988
#> GSM340340 4 0.0524 0.981 0.000 0.008 0.004 0.988
#> GSM340341 4 0.0859 0.977 0.008 0.008 0.004 0.980
#> GSM340343 4 0.0524 0.981 0.000 0.008 0.004 0.988
#> GSM340344 3 0.2715 0.825 0.004 0.100 0.892 0.004
#> GSM340346 3 0.1492 0.881 0.004 0.036 0.956 0.004
#> GSM340347 4 0.0376 0.981 0.000 0.004 0.004 0.992
#> GSM340348 4 0.0376 0.979 0.000 0.004 0.004 0.992
#> GSM340349 3 0.4427 0.800 0.164 0.028 0.800 0.008
#> GSM340350 3 0.4193 0.793 0.184 0.016 0.796 0.004
#> GSM340351 3 0.5126 0.393 0.444 0.000 0.552 0.004
#> GSM340354 3 0.0707 0.881 0.000 0.020 0.980 0.000
#> GSM340356 2 0.2036 0.935 0.032 0.936 0.032 0.000
#> GSM340357 3 0.3908 0.774 0.212 0.000 0.784 0.004
#> GSM348183 3 0.5408 0.406 0.432 0.004 0.556 0.008
#> GSM348191 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM348193 4 0.0564 0.976 0.004 0.004 0.004 0.988
#> GSM537578 3 0.3884 0.842 0.108 0.036 0.848 0.008
#> GSM348181 3 0.1911 0.884 0.032 0.020 0.944 0.004
#> GSM348182 3 0.1109 0.873 0.004 0.028 0.968 0.000
#> GSM348184 2 0.1938 0.939 0.012 0.936 0.052 0.000
#> GSM348185 2 0.4840 0.694 0.028 0.732 0.000 0.240
#> GSM348186 2 0.1302 0.920 0.000 0.956 0.044 0.000
#> GSM348187 3 0.5126 0.393 0.444 0.000 0.552 0.004
#> GSM348188 4 0.0524 0.981 0.000 0.008 0.004 0.988
#> GSM348189 3 0.1510 0.869 0.016 0.028 0.956 0.000
#> GSM348190 3 0.1296 0.873 0.004 0.028 0.964 0.004
#> GSM348194 3 0.5126 0.393 0.444 0.000 0.552 0.004
#> GSM348195 3 0.5303 0.376 0.448 0.004 0.544 0.004
#> GSM348196 3 0.5290 0.395 0.440 0.004 0.552 0.004
#> GSM537585 3 0.1585 0.883 0.040 0.004 0.952 0.004
#> GSM537594 2 0.2036 0.935 0.032 0.936 0.032 0.000
#> GSM537596 3 0.2076 0.881 0.056 0.008 0.932 0.004
#> GSM537597 3 0.3680 0.821 0.160 0.008 0.828 0.004
#> GSM537602 3 0.5085 0.706 0.256 0.020 0.716 0.008
#> GSM340184 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM340185 2 0.1545 0.939 0.008 0.952 0.040 0.000
#> GSM340186 2 0.1722 0.941 0.008 0.944 0.048 0.000
#> GSM340187 4 0.0376 0.981 0.000 0.004 0.004 0.992
#> GSM340189 4 0.0336 0.979 0.000 0.000 0.008 0.992
#> GSM340190 2 0.1677 0.938 0.012 0.948 0.040 0.000
#> GSM340191 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM340192 3 0.5266 0.458 0.008 0.344 0.640 0.008
#> GSM340193 3 0.1994 0.882 0.052 0.008 0.936 0.004
#> GSM340194 3 0.1109 0.873 0.004 0.028 0.968 0.000
#> GSM340195 3 0.1296 0.873 0.004 0.028 0.964 0.004
#> GSM340196 4 0.0376 0.981 0.000 0.004 0.004 0.992
#> GSM340197 3 0.1109 0.873 0.004 0.028 0.968 0.000
#> GSM340198 2 0.1635 0.920 0.008 0.948 0.044 0.000
#> GSM340199 3 0.2463 0.880 0.032 0.036 0.924 0.008
#> GSM340200 2 0.1543 0.933 0.008 0.956 0.032 0.004
#> GSM340201 4 0.0376 0.981 0.000 0.004 0.004 0.992
#> GSM340202 4 0.0376 0.981 0.000 0.004 0.004 0.992
#> GSM340203 4 0.0524 0.979 0.000 0.004 0.008 0.988
#> GSM340204 3 0.1296 0.873 0.004 0.028 0.964 0.004
#> GSM340205 3 0.5119 0.403 0.440 0.000 0.556 0.004
#> GSM340206 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM340207 3 0.1109 0.873 0.004 0.028 0.968 0.000
#> GSM340237 2 0.1406 0.937 0.016 0.960 0.024 0.000
#> GSM340238 3 0.1724 0.884 0.032 0.020 0.948 0.000
#> GSM340239 3 0.1109 0.873 0.004 0.028 0.968 0.000
#> GSM340240 3 0.1675 0.870 0.004 0.044 0.948 0.004
#> GSM340241 2 0.1389 0.940 0.000 0.952 0.048 0.000
#> GSM340242 3 0.1510 0.869 0.016 0.028 0.956 0.000
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.0510 0.9863 0.984 0.000 0.016 0.000 0.000
#> GSM340359 1 0.1331 0.9424 0.952 0.008 0.040 0.000 0.000
#> GSM340361 5 0.6852 -0.3040 0.324 0.004 0.260 0.000 0.412
#> GSM340362 5 0.1502 0.4040 0.004 0.000 0.056 0.000 0.940
#> GSM340363 3 0.6874 0.4785 0.280 0.008 0.448 0.000 0.264
#> GSM340364 1 0.0451 0.9858 0.988 0.004 0.008 0.000 0.000
#> GSM340365 5 0.5080 0.0776 0.056 0.000 0.316 0.000 0.628
#> GSM340366 5 0.4990 0.0812 0.048 0.000 0.324 0.000 0.628
#> GSM340367 1 0.0404 0.9884 0.988 0.000 0.012 0.000 0.000
#> GSM340368 4 0.0162 0.9551 0.000 0.000 0.004 0.996 0.000
#> GSM340369 4 0.4331 0.3622 0.400 0.000 0.004 0.596 0.000
#> GSM340370 1 0.0404 0.9884 0.988 0.000 0.012 0.000 0.000
#> GSM340371 5 0.1282 0.4219 0.000 0.004 0.044 0.000 0.952
#> GSM340372 1 0.0404 0.9884 0.988 0.000 0.012 0.000 0.000
#> GSM340373 1 0.0404 0.9884 0.988 0.000 0.012 0.000 0.000
#> GSM340375 1 0.0404 0.9884 0.988 0.000 0.012 0.000 0.000
#> GSM340376 1 0.0404 0.9884 0.988 0.000 0.012 0.000 0.000
#> GSM340378 1 0.0451 0.9858 0.988 0.004 0.008 0.000 0.000
#> GSM340243 3 0.4635 0.4979 0.048 0.004 0.716 0.000 0.232
#> GSM340244 2 0.0794 0.9153 0.000 0.972 0.028 0.000 0.000
#> GSM340246 2 0.0671 0.9179 0.004 0.980 0.016 0.000 0.000
#> GSM340247 4 0.0000 0.9552 0.000 0.000 0.000 1.000 0.000
#> GSM340248 2 0.0963 0.9159 0.000 0.964 0.036 0.000 0.000
#> GSM340249 2 0.4124 0.7776 0.008 0.792 0.056 0.144 0.000
#> GSM340250 5 0.4278 0.1725 0.000 0.000 0.452 0.000 0.548
#> GSM340251 2 0.1430 0.9135 0.004 0.944 0.052 0.000 0.000
#> GSM340252 2 0.5702 0.6361 0.008 0.640 0.120 0.232 0.000
#> GSM340253 2 0.4293 0.7551 0.004 0.772 0.064 0.000 0.160
#> GSM340254 2 0.2037 0.8978 0.004 0.920 0.064 0.000 0.012
#> GSM340256 2 0.1041 0.9142 0.004 0.964 0.032 0.000 0.000
#> GSM340258 5 0.4161 0.2391 0.000 0.000 0.392 0.000 0.608
#> GSM340259 5 0.4375 0.1355 0.000 0.004 0.420 0.000 0.576
#> GSM340260 5 0.4283 0.0222 0.000 0.000 0.456 0.000 0.544
#> GSM340261 5 0.4307 0.0107 0.000 0.000 0.496 0.000 0.504
#> GSM340262 5 0.0880 0.4311 0.000 0.000 0.032 0.000 0.968
#> GSM340263 2 0.1270 0.9121 0.000 0.948 0.052 0.000 0.000
#> GSM340264 5 0.0703 0.4329 0.000 0.000 0.024 0.000 0.976
#> GSM340265 5 0.1608 0.3979 0.000 0.000 0.072 0.000 0.928
#> GSM340266 5 0.4249 0.2087 0.000 0.000 0.432 0.000 0.568
#> GSM340267 5 0.4283 0.1617 0.000 0.000 0.456 0.000 0.544
#> GSM340268 5 0.4242 0.2163 0.000 0.000 0.428 0.000 0.572
#> GSM340269 5 0.4227 0.2120 0.000 0.000 0.420 0.000 0.580
#> GSM340270 5 0.4397 0.1962 0.000 0.004 0.432 0.000 0.564
#> GSM537574 5 0.4227 0.2107 0.000 0.000 0.420 0.000 0.580
#> GSM537580 5 0.1205 0.4226 0.000 0.004 0.040 0.000 0.956
#> GSM537581 4 0.1341 0.9412 0.000 0.000 0.056 0.944 0.000
#> GSM340272 5 0.3521 0.2372 0.004 0.000 0.232 0.000 0.764
#> GSM340273 2 0.0865 0.9145 0.004 0.972 0.024 0.000 0.000
#> GSM340275 4 0.1282 0.9452 0.004 0.000 0.044 0.952 0.000
#> GSM340276 3 0.4533 0.2242 0.000 0.008 0.544 0.000 0.448
#> GSM340277 2 0.2037 0.8978 0.004 0.920 0.064 0.000 0.012
#> GSM340278 3 0.4455 0.3143 0.000 0.008 0.588 0.000 0.404
#> GSM340279 5 0.4283 0.1507 0.000 0.000 0.456 0.000 0.544
#> GSM340282 5 0.3461 0.2437 0.004 0.000 0.224 0.000 0.772
#> GSM340284 2 0.5219 0.4516 0.004 0.616 0.328 0.000 0.052
#> GSM340285 3 0.4298 0.4275 0.000 0.008 0.640 0.000 0.352
#> GSM340286 5 0.0000 0.4325 0.000 0.000 0.000 0.000 1.000
#> GSM340287 5 0.4304 0.0690 0.000 0.000 0.484 0.000 0.516
#> GSM340288 3 0.6931 0.4879 0.292 0.008 0.428 0.000 0.272
#> GSM340289 5 0.4705 -0.2202 0.008 0.004 0.484 0.000 0.504
#> GSM340290 5 0.4249 0.2115 0.000 0.000 0.432 0.000 0.568
#> GSM340291 2 0.0290 0.9179 0.000 0.992 0.008 0.000 0.000
#> GSM340293 3 0.4251 0.3220 0.000 0.000 0.672 0.012 0.316
#> GSM340294 5 0.4262 0.1893 0.000 0.000 0.440 0.000 0.560
#> GSM340296 3 0.4536 0.4863 0.048 0.000 0.712 0.000 0.240
#> GSM340297 5 0.4268 0.1911 0.000 0.000 0.444 0.000 0.556
#> GSM340298 3 0.4563 0.4824 0.048 0.000 0.708 0.000 0.244
#> GSM340299 5 0.2068 0.3837 0.004 0.000 0.092 0.000 0.904
#> GSM340301 5 0.0000 0.4325 0.000 0.000 0.000 0.000 1.000
#> GSM340303 5 0.4047 0.1482 0.004 0.000 0.320 0.000 0.676
#> GSM340304 3 0.3855 0.5136 0.032 0.008 0.800 0.000 0.160
#> GSM340306 4 0.1952 0.9199 0.004 0.000 0.084 0.912 0.000
#> GSM340307 2 0.0865 0.9176 0.004 0.972 0.024 0.000 0.000
#> GSM340310 1 0.0609 0.9844 0.980 0.000 0.020 0.000 0.000
#> GSM340314 5 0.4256 0.2086 0.000 0.000 0.436 0.000 0.564
#> GSM340315 5 0.4305 0.0736 0.000 0.000 0.488 0.000 0.512
#> GSM340317 2 0.1638 0.9029 0.004 0.932 0.064 0.000 0.000
#> GSM340318 4 0.0000 0.9552 0.000 0.000 0.000 1.000 0.000
#> GSM340319 4 0.0000 0.9552 0.000 0.000 0.000 1.000 0.000
#> GSM340320 4 0.2942 0.8804 0.004 0.004 0.116 0.864 0.012
#> GSM340321 2 0.0510 0.9176 0.000 0.984 0.016 0.000 0.000
#> GSM340322 4 0.0000 0.9552 0.000 0.000 0.000 1.000 0.000
#> GSM340324 2 0.1518 0.9062 0.004 0.944 0.048 0.000 0.004
#> GSM340328 1 0.1270 0.9533 0.948 0.000 0.052 0.000 0.000
#> GSM340330 5 0.4227 0.2238 0.000 0.000 0.420 0.000 0.580
#> GSM340332 4 0.0510 0.9520 0.000 0.000 0.016 0.984 0.000
#> GSM340333 2 0.3087 0.8603 0.004 0.868 0.064 0.000 0.064
#> GSM340336 4 0.0000 0.9552 0.000 0.000 0.000 1.000 0.000
#> GSM340337 4 0.0510 0.9539 0.000 0.000 0.016 0.984 0.000
#> GSM340338 5 0.4171 0.2370 0.000 0.000 0.396 0.000 0.604
#> GSM340339 4 0.0510 0.9539 0.000 0.000 0.016 0.984 0.000
#> GSM340340 4 0.1124 0.9492 0.004 0.000 0.036 0.960 0.000
#> GSM340341 4 0.2787 0.8834 0.004 0.004 0.136 0.856 0.000
#> GSM340343 4 0.0771 0.9520 0.004 0.000 0.020 0.976 0.000
#> GSM340344 5 0.3669 0.3149 0.008 0.048 0.116 0.000 0.828
#> GSM340346 3 0.4060 0.2790 0.000 0.000 0.640 0.000 0.360
#> GSM340347 4 0.0000 0.9552 0.000 0.000 0.000 1.000 0.000
#> GSM340348 4 0.2497 0.8955 0.004 0.004 0.112 0.880 0.000
#> GSM340349 3 0.4644 0.5043 0.052 0.004 0.720 0.000 0.224
#> GSM340350 3 0.5353 0.4276 0.052 0.004 0.576 0.000 0.368
#> GSM340351 3 0.7015 0.4533 0.296 0.008 0.388 0.000 0.308
#> GSM340354 5 0.4201 0.2252 0.000 0.000 0.408 0.000 0.592
#> GSM340356 2 0.0865 0.9145 0.004 0.972 0.024 0.000 0.000
#> GSM340357 3 0.5468 0.4207 0.060 0.004 0.568 0.000 0.368
#> GSM348183 3 0.7051 0.4315 0.280 0.004 0.376 0.004 0.336
#> GSM348191 5 0.4242 0.2141 0.000 0.000 0.428 0.000 0.572
#> GSM348193 4 0.2284 0.9093 0.004 0.004 0.096 0.896 0.000
#> GSM537578 3 0.4284 0.5001 0.040 0.000 0.736 0.000 0.224
#> GSM348181 5 0.4425 0.1483 0.000 0.004 0.452 0.000 0.544
#> GSM348182 5 0.0510 0.4271 0.000 0.000 0.016 0.000 0.984
#> GSM348184 2 0.1041 0.9173 0.004 0.964 0.032 0.000 0.000
#> GSM348185 2 0.5227 0.6725 0.004 0.696 0.132 0.168 0.000
#> GSM348186 2 0.2037 0.8978 0.004 0.920 0.064 0.000 0.012
#> GSM348187 3 0.6931 0.4879 0.292 0.008 0.428 0.000 0.272
#> GSM348188 4 0.0955 0.9502 0.004 0.000 0.028 0.968 0.000
#> GSM348189 5 0.1205 0.4226 0.000 0.004 0.040 0.000 0.956
#> GSM348190 5 0.1851 0.3964 0.000 0.000 0.088 0.000 0.912
#> GSM348194 3 0.6931 0.4879 0.292 0.008 0.428 0.000 0.272
#> GSM348195 3 0.6945 0.4281 0.324 0.004 0.364 0.000 0.308
#> GSM348196 3 0.6897 0.4848 0.288 0.008 0.440 0.000 0.264
#> GSM537585 5 0.3989 0.1649 0.008 0.004 0.260 0.000 0.728
#> GSM537594 2 0.0290 0.9171 0.000 0.992 0.008 0.000 0.000
#> GSM537596 3 0.4637 0.2932 0.004 0.008 0.568 0.000 0.420
#> GSM537597 3 0.5124 0.4673 0.040 0.008 0.628 0.000 0.324
#> GSM537602 3 0.5507 0.4906 0.156 0.008 0.676 0.000 0.160
#> GSM340184 5 0.4242 0.2141 0.000 0.000 0.428 0.000 0.572
#> GSM340185 2 0.0955 0.9174 0.004 0.968 0.028 0.000 0.000
#> GSM340186 2 0.0955 0.9174 0.004 0.968 0.028 0.000 0.000
#> GSM340187 4 0.0290 0.9530 0.000 0.000 0.008 0.992 0.000
#> GSM340189 4 0.0162 0.9542 0.000 0.000 0.004 0.996 0.000
#> GSM340190 2 0.1124 0.9173 0.004 0.960 0.036 0.000 0.000
#> GSM340191 5 0.4242 0.2141 0.000 0.000 0.428 0.000 0.572
#> GSM340192 5 0.6632 0.0583 0.008 0.220 0.264 0.000 0.508
#> GSM340193 3 0.4559 0.1249 0.000 0.008 0.512 0.000 0.480
#> GSM340194 5 0.0794 0.4241 0.000 0.000 0.028 0.000 0.972
#> GSM340195 5 0.1357 0.4085 0.004 0.000 0.048 0.000 0.948
#> GSM340196 4 0.0000 0.9552 0.000 0.000 0.000 1.000 0.000
#> GSM340197 5 0.0404 0.4335 0.000 0.000 0.012 0.000 0.988
#> GSM340198 2 0.2166 0.8983 0.004 0.912 0.072 0.000 0.012
#> GSM340199 3 0.4114 0.2709 0.000 0.000 0.624 0.000 0.376
#> GSM340200 2 0.3196 0.7886 0.004 0.804 0.192 0.000 0.000
#> GSM340201 4 0.0000 0.9552 0.000 0.000 0.000 1.000 0.000
#> GSM340202 4 0.0000 0.9552 0.000 0.000 0.000 1.000 0.000
#> GSM340203 4 0.1282 0.9466 0.004 0.000 0.044 0.952 0.000
#> GSM340204 5 0.2536 0.3745 0.004 0.000 0.128 0.000 0.868
#> GSM340205 5 0.6913 -0.4538 0.280 0.004 0.336 0.000 0.380
#> GSM340206 5 0.4242 0.2163 0.000 0.000 0.428 0.000 0.572
#> GSM340207 5 0.0794 0.4316 0.000 0.000 0.028 0.000 0.972
#> GSM340237 2 0.0290 0.9179 0.000 0.992 0.008 0.000 0.000
#> GSM340238 5 0.4249 0.2130 0.000 0.000 0.432 0.000 0.568
#> GSM340239 5 0.0290 0.4334 0.000 0.000 0.008 0.000 0.992
#> GSM340240 5 0.2806 0.3194 0.004 0.000 0.152 0.000 0.844
#> GSM340241 2 0.0794 0.9173 0.000 0.972 0.028 0.000 0.000
#> GSM340242 5 0.1205 0.4226 0.000 0.004 0.040 0.000 0.956
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.0000 0.9711 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340359 1 0.0622 0.9608 0.980 0.000 0.000 0.000 0.012 0.008
#> GSM340361 6 0.6161 0.5352 0.176 0.000 0.072 0.000 0.164 0.588
#> GSM340362 5 0.5184 0.6929 0.000 0.000 0.316 0.000 0.572 0.112
#> GSM340363 6 0.5526 0.6462 0.140 0.000 0.156 0.004 0.040 0.660
#> GSM340364 1 0.0146 0.9742 0.996 0.000 0.000 0.000 0.004 0.000
#> GSM340365 5 0.4787 0.2434 0.008 0.000 0.056 0.000 0.624 0.312
#> GSM340366 5 0.5129 0.1663 0.008 0.000 0.076 0.000 0.576 0.340
#> GSM340367 1 0.0146 0.9742 0.996 0.000 0.000 0.000 0.004 0.000
#> GSM340368 4 0.1196 0.9028 0.000 0.000 0.000 0.952 0.040 0.008
#> GSM340369 4 0.4426 0.4022 0.376 0.000 0.000 0.596 0.020 0.008
#> GSM340370 1 0.0146 0.9742 0.996 0.000 0.000 0.000 0.004 0.000
#> GSM340371 5 0.5071 0.6571 0.000 0.000 0.400 0.000 0.520 0.080
#> GSM340372 1 0.0146 0.9742 0.996 0.000 0.000 0.000 0.004 0.000
#> GSM340373 1 0.0146 0.9742 0.996 0.000 0.000 0.000 0.004 0.000
#> GSM340375 1 0.0146 0.9742 0.996 0.000 0.000 0.000 0.004 0.000
#> GSM340376 1 0.0146 0.9742 0.996 0.000 0.000 0.000 0.004 0.000
#> GSM340378 1 0.0146 0.9742 0.996 0.000 0.000 0.000 0.004 0.000
#> GSM340243 6 0.6179 0.3605 0.008 0.000 0.324 0.000 0.236 0.432
#> GSM340244 2 0.1088 0.8966 0.000 0.960 0.000 0.000 0.024 0.016
#> GSM340246 2 0.0260 0.8997 0.000 0.992 0.000 0.000 0.008 0.000
#> GSM340247 4 0.0146 0.9091 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM340248 2 0.0717 0.8983 0.000 0.976 0.000 0.000 0.016 0.008
#> GSM340249 2 0.4381 0.7805 0.000 0.772 0.000 0.092 0.068 0.068
#> GSM340250 3 0.2301 0.6332 0.000 0.000 0.884 0.000 0.020 0.096
#> GSM340251 2 0.2568 0.8883 0.000 0.876 0.000 0.000 0.056 0.068
#> GSM340252 2 0.6458 0.6615 0.000 0.568 0.000 0.152 0.152 0.128
#> GSM340253 2 0.4065 0.7650 0.000 0.724 0.000 0.000 0.220 0.056
#> GSM340254 2 0.2937 0.8688 0.000 0.848 0.000 0.000 0.096 0.056
#> GSM340256 2 0.1418 0.8961 0.000 0.944 0.000 0.000 0.032 0.024
#> GSM340258 3 0.1225 0.6582 0.000 0.000 0.952 0.000 0.036 0.012
#> GSM340259 3 0.3982 0.4724 0.000 0.000 0.740 0.000 0.060 0.200
#> GSM340260 3 0.4104 0.5052 0.000 0.000 0.748 0.000 0.104 0.148
#> GSM340261 3 0.1958 0.6464 0.000 0.000 0.896 0.000 0.004 0.100
#> GSM340262 3 0.4002 -0.4027 0.000 0.000 0.588 0.000 0.404 0.008
#> GSM340263 2 0.2070 0.8809 0.000 0.908 0.000 0.000 0.044 0.048
#> GSM340264 3 0.4639 -0.5692 0.000 0.000 0.512 0.000 0.448 0.040
#> GSM340265 5 0.5176 0.6637 0.000 0.000 0.352 0.000 0.548 0.100
#> GSM340266 3 0.0972 0.6905 0.000 0.000 0.964 0.000 0.008 0.028
#> GSM340267 3 0.2706 0.5909 0.000 0.000 0.832 0.000 0.008 0.160
#> GSM340268 3 0.0146 0.6899 0.000 0.000 0.996 0.000 0.004 0.000
#> GSM340269 3 0.1088 0.6867 0.000 0.000 0.960 0.000 0.016 0.024
#> GSM340270 3 0.1007 0.6836 0.000 0.000 0.956 0.000 0.000 0.044
#> GSM537574 3 0.1257 0.6822 0.000 0.000 0.952 0.000 0.020 0.028
#> GSM537580 5 0.5106 0.6600 0.000 0.000 0.396 0.000 0.520 0.084
#> GSM537581 4 0.3785 0.8451 0.004 0.000 0.012 0.800 0.056 0.128
#> GSM340272 5 0.5394 0.4451 0.000 0.000 0.156 0.000 0.568 0.276
#> GSM340273 2 0.1003 0.8973 0.000 0.964 0.000 0.000 0.016 0.020
#> GSM340275 4 0.2714 0.8810 0.004 0.000 0.000 0.872 0.060 0.064
#> GSM340276 6 0.4264 0.2530 0.000 0.000 0.488 0.000 0.016 0.496
#> GSM340277 2 0.2888 0.8715 0.000 0.852 0.000 0.000 0.092 0.056
#> GSM340278 3 0.4258 -0.2705 0.000 0.000 0.516 0.000 0.016 0.468
#> GSM340279 3 0.1700 0.6679 0.000 0.000 0.916 0.000 0.004 0.080
#> GSM340282 5 0.5357 0.4938 0.000 0.000 0.180 0.000 0.588 0.232
#> GSM340284 3 0.5262 0.1878 0.000 0.348 0.572 0.000 0.048 0.032
#> GSM340285 6 0.4219 0.5168 0.000 0.000 0.388 0.000 0.020 0.592
#> GSM340286 5 0.4735 0.6603 0.000 0.000 0.432 0.000 0.520 0.048
#> GSM340287 3 0.2416 0.5892 0.000 0.000 0.844 0.000 0.000 0.156
#> GSM340288 6 0.5102 0.6812 0.152 0.000 0.172 0.000 0.012 0.664
#> GSM340289 3 0.5019 -0.1979 0.000 0.000 0.500 0.000 0.072 0.428
#> GSM340290 3 0.0603 0.6914 0.000 0.000 0.980 0.000 0.004 0.016
#> GSM340291 2 0.0146 0.8994 0.000 0.996 0.000 0.000 0.004 0.000
#> GSM340293 3 0.5308 0.2117 0.000 0.000 0.616 0.004 0.180 0.200
#> GSM340294 3 0.1010 0.6930 0.000 0.000 0.960 0.000 0.004 0.036
#> GSM340296 3 0.6201 -0.2254 0.008 0.000 0.416 0.000 0.236 0.340
#> GSM340297 3 0.1219 0.6829 0.000 0.000 0.948 0.000 0.004 0.048
#> GSM340298 3 0.6201 -0.2254 0.008 0.000 0.416 0.000 0.236 0.340
#> GSM340299 5 0.4905 0.7033 0.000 0.000 0.344 0.000 0.580 0.076
#> GSM340301 5 0.4742 0.6531 0.000 0.000 0.440 0.000 0.512 0.048
#> GSM340303 5 0.4767 0.3515 0.000 0.000 0.076 0.000 0.620 0.304
#> GSM340304 6 0.4787 0.5818 0.000 0.000 0.184 0.000 0.144 0.672
#> GSM340306 4 0.3389 0.8306 0.004 0.000 0.000 0.800 0.032 0.164
#> GSM340307 2 0.2474 0.8758 0.000 0.880 0.000 0.000 0.040 0.080
#> GSM340310 1 0.1745 0.9044 0.920 0.000 0.000 0.000 0.012 0.068
#> GSM340314 3 0.0146 0.6899 0.000 0.000 0.996 0.000 0.004 0.000
#> GSM340315 3 0.2595 0.5754 0.000 0.000 0.836 0.000 0.004 0.160
#> GSM340317 2 0.2688 0.8629 0.000 0.868 0.000 0.000 0.064 0.068
#> GSM340318 4 0.0146 0.9091 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM340319 4 0.0146 0.9091 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM340320 4 0.4440 0.7063 0.004 0.000 0.008 0.672 0.032 0.284
#> GSM340321 2 0.0146 0.8994 0.000 0.996 0.000 0.000 0.004 0.000
#> GSM340322 4 0.0146 0.9091 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM340324 2 0.1564 0.8934 0.000 0.936 0.000 0.000 0.040 0.024
#> GSM340328 1 0.2692 0.8113 0.840 0.000 0.000 0.000 0.012 0.148
#> GSM340330 3 0.0146 0.6899 0.000 0.000 0.996 0.000 0.004 0.000
#> GSM340332 4 0.1148 0.9016 0.000 0.000 0.020 0.960 0.016 0.004
#> GSM340333 2 0.3530 0.8313 0.000 0.792 0.000 0.000 0.152 0.056
#> GSM340336 4 0.0146 0.9091 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM340337 4 0.1793 0.9003 0.004 0.000 0.000 0.928 0.036 0.032
#> GSM340338 3 0.0458 0.6770 0.000 0.000 0.984 0.000 0.016 0.000
#> GSM340339 4 0.1562 0.9021 0.004 0.000 0.000 0.940 0.032 0.024
#> GSM340340 4 0.2575 0.8896 0.004 0.000 0.000 0.880 0.072 0.044
#> GSM340341 4 0.4361 0.7679 0.004 0.000 0.000 0.700 0.060 0.236
#> GSM340343 4 0.1237 0.9049 0.004 0.000 0.000 0.956 0.020 0.020
#> GSM340344 5 0.5815 0.5993 0.000 0.056 0.192 0.000 0.620 0.132
#> GSM340346 3 0.5434 0.1574 0.000 0.000 0.576 0.000 0.192 0.232
#> GSM340347 4 0.0146 0.9091 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM340348 4 0.3904 0.7745 0.004 0.000 0.000 0.732 0.032 0.232
#> GSM340349 6 0.6059 0.4422 0.008 0.000 0.264 0.000 0.244 0.484
#> GSM340350 6 0.5826 0.4298 0.008 0.000 0.320 0.000 0.164 0.508
#> GSM340351 6 0.5393 0.6787 0.156 0.000 0.156 0.000 0.032 0.656
#> GSM340354 3 0.0777 0.6799 0.000 0.000 0.972 0.000 0.024 0.004
#> GSM340356 2 0.1003 0.8973 0.000 0.964 0.000 0.000 0.016 0.020
#> GSM340357 6 0.4533 0.4398 0.020 0.000 0.432 0.000 0.008 0.540
#> GSM348183 6 0.6236 0.6347 0.136 0.000 0.184 0.008 0.072 0.600
#> GSM348191 3 0.0725 0.6867 0.000 0.000 0.976 0.000 0.012 0.012
#> GSM348193 4 0.4136 0.7528 0.004 0.000 0.000 0.708 0.040 0.248
#> GSM537578 6 0.5988 0.3193 0.000 0.000 0.348 0.000 0.236 0.416
#> GSM348181 3 0.1957 0.6431 0.000 0.000 0.888 0.000 0.000 0.112
#> GSM348182 5 0.5045 0.6643 0.000 0.000 0.412 0.000 0.512 0.076
#> GSM348184 2 0.1865 0.8966 0.000 0.920 0.000 0.000 0.040 0.040
#> GSM348185 2 0.5834 0.6234 0.004 0.624 0.000 0.076 0.080 0.216
#> GSM348186 2 0.2937 0.8688 0.000 0.848 0.000 0.000 0.096 0.056
#> GSM348187 6 0.4990 0.6779 0.152 0.000 0.184 0.000 0.004 0.660
#> GSM348188 4 0.2078 0.8965 0.004 0.000 0.000 0.912 0.040 0.044
#> GSM348189 5 0.5106 0.6600 0.000 0.000 0.396 0.000 0.520 0.084
#> GSM348190 5 0.5501 0.6072 0.000 0.000 0.412 0.000 0.460 0.128
#> GSM348194 6 0.5102 0.6812 0.152 0.000 0.172 0.000 0.012 0.664
#> GSM348195 6 0.5384 0.6724 0.172 0.000 0.140 0.000 0.032 0.656
#> GSM348196 6 0.4946 0.6783 0.152 0.000 0.164 0.000 0.008 0.676
#> GSM537585 6 0.5510 0.3331 0.004 0.000 0.156 0.000 0.272 0.568
#> GSM537594 2 0.0291 0.8990 0.000 0.992 0.000 0.000 0.004 0.004
#> GSM537596 6 0.3899 0.4497 0.000 0.000 0.404 0.000 0.004 0.592
#> GSM537597 6 0.4092 0.5913 0.012 0.004 0.316 0.000 0.004 0.664
#> GSM537602 6 0.5930 0.5344 0.040 0.000 0.140 0.000 0.240 0.580
#> GSM340184 3 0.0291 0.6903 0.000 0.000 0.992 0.000 0.004 0.004
#> GSM340185 2 0.1794 0.8965 0.000 0.924 0.000 0.000 0.036 0.040
#> GSM340186 2 0.1794 0.8970 0.000 0.924 0.000 0.000 0.036 0.040
#> GSM340187 4 0.0146 0.9089 0.000 0.000 0.004 0.996 0.000 0.000
#> GSM340189 4 0.0000 0.9091 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340190 2 0.2078 0.8959 0.000 0.912 0.004 0.000 0.040 0.044
#> GSM340191 3 0.0291 0.6903 0.000 0.000 0.992 0.000 0.004 0.004
#> GSM340192 5 0.5505 0.2275 0.000 0.148 0.024 0.000 0.628 0.200
#> GSM340193 3 0.4437 0.0476 0.000 0.000 0.576 0.000 0.032 0.392
#> GSM340194 5 0.4928 0.6991 0.000 0.000 0.352 0.000 0.572 0.076
#> GSM340195 5 0.5057 0.6980 0.000 0.000 0.352 0.000 0.560 0.088
#> GSM340196 4 0.0146 0.9091 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM340197 5 0.4742 0.6548 0.000 0.000 0.440 0.000 0.512 0.048
#> GSM340198 2 0.3206 0.8699 0.000 0.828 0.000 0.000 0.104 0.068
#> GSM340199 3 0.5289 0.1320 0.000 0.000 0.580 0.000 0.140 0.280
#> GSM340200 2 0.5303 0.5767 0.000 0.596 0.000 0.000 0.232 0.172
#> GSM340201 4 0.0146 0.9091 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM340202 4 0.0146 0.9091 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM340203 4 0.2697 0.8878 0.004 0.000 0.016 0.884 0.032 0.064
#> GSM340204 5 0.5643 0.6044 0.000 0.000 0.340 0.000 0.496 0.164
#> GSM340205 3 0.5695 -0.3081 0.104 0.000 0.480 0.000 0.016 0.400
#> GSM340206 3 0.0146 0.6899 0.000 0.000 0.996 0.000 0.004 0.000
#> GSM340207 3 0.3993 -0.5608 0.000 0.000 0.520 0.000 0.476 0.004
#> GSM340237 2 0.0146 0.8997 0.000 0.996 0.000 0.000 0.004 0.000
#> GSM340238 3 0.0146 0.6899 0.000 0.000 0.996 0.000 0.004 0.000
#> GSM340239 5 0.4742 0.6548 0.000 0.000 0.440 0.000 0.512 0.048
#> GSM340240 5 0.4810 0.6320 0.000 0.000 0.292 0.000 0.624 0.084
#> GSM340241 2 0.1649 0.8971 0.000 0.932 0.000 0.000 0.032 0.036
#> GSM340242 5 0.5106 0.6600 0.000 0.000 0.396 0.000 0.520 0.084
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
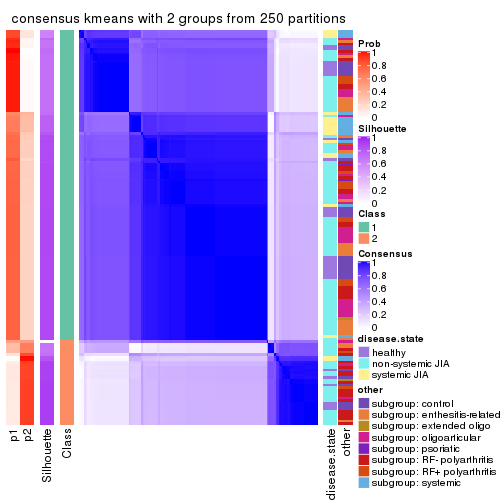
consensus_heatmap(res, k = 3)
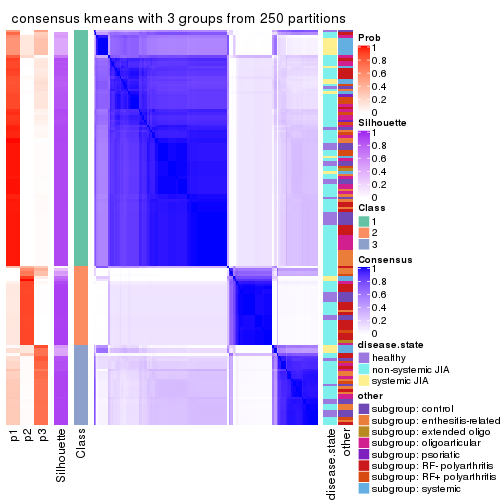
consensus_heatmap(res, k = 4)
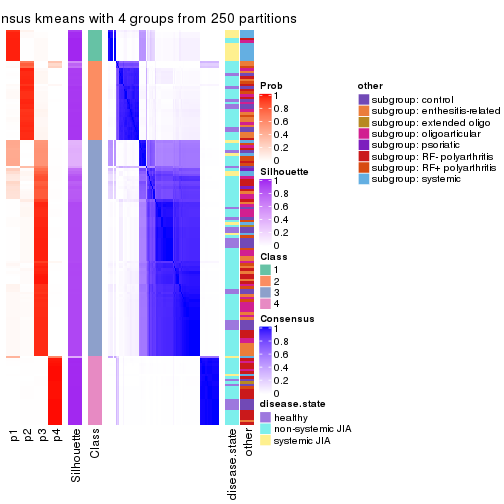
consensus_heatmap(res, k = 5)
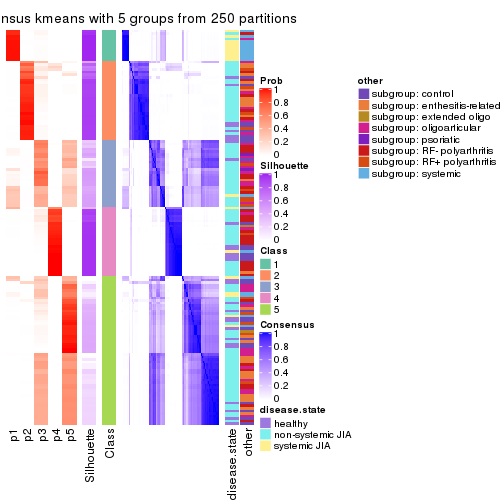
consensus_heatmap(res, k = 6)
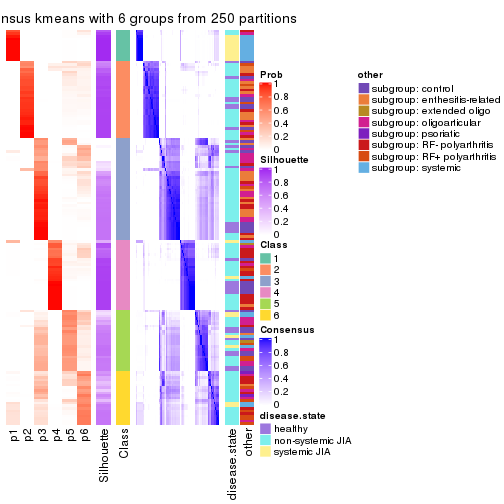
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
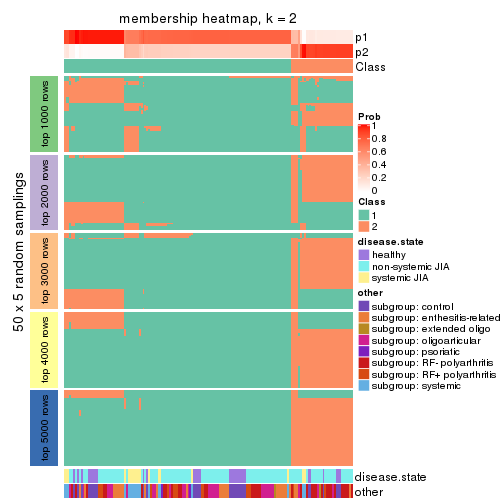
membership_heatmap(res, k = 3)
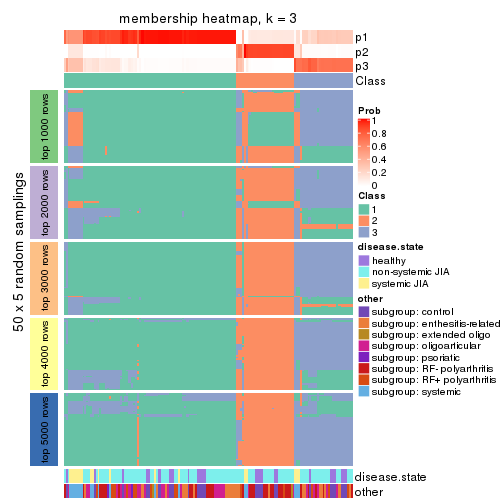
membership_heatmap(res, k = 4)
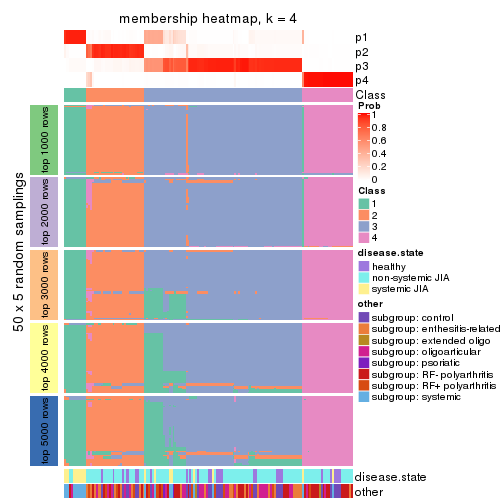
membership_heatmap(res, k = 5)
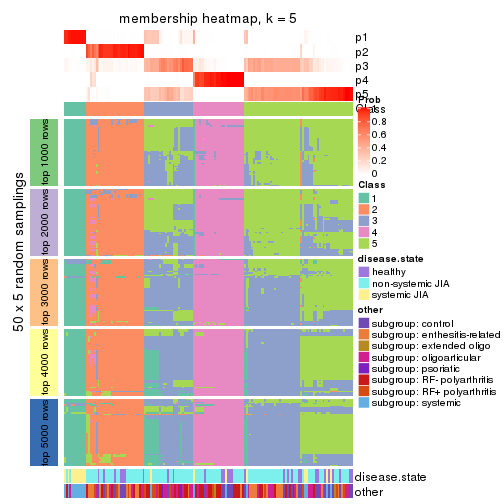
membership_heatmap(res, k = 6)
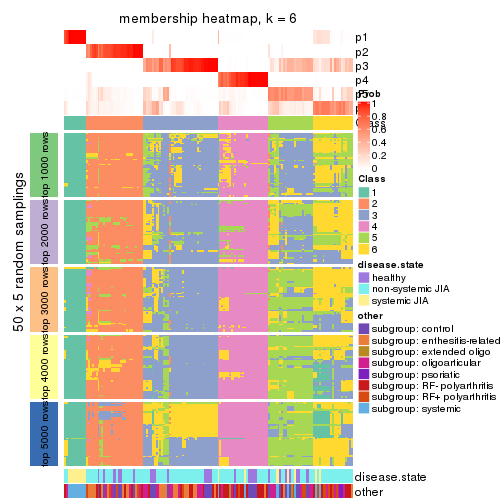
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
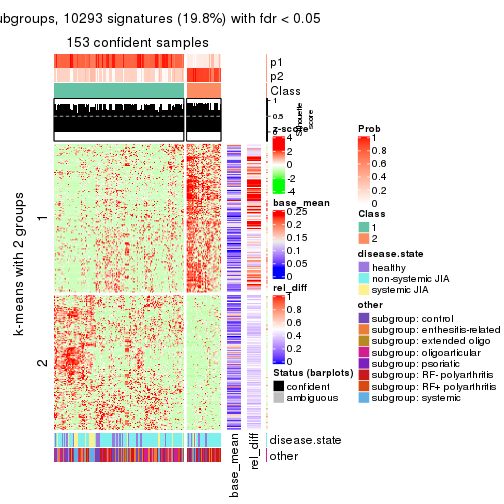
get_signatures(res, k = 3)
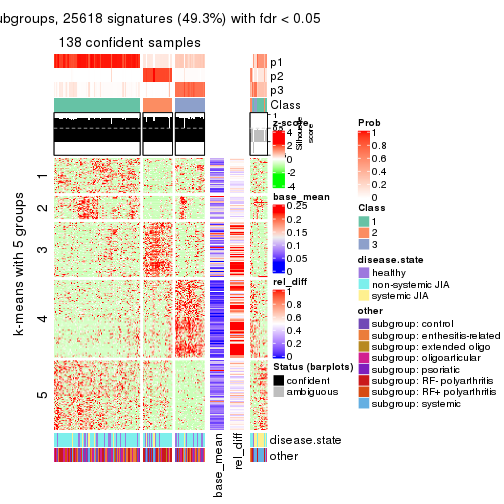
get_signatures(res, k = 4)
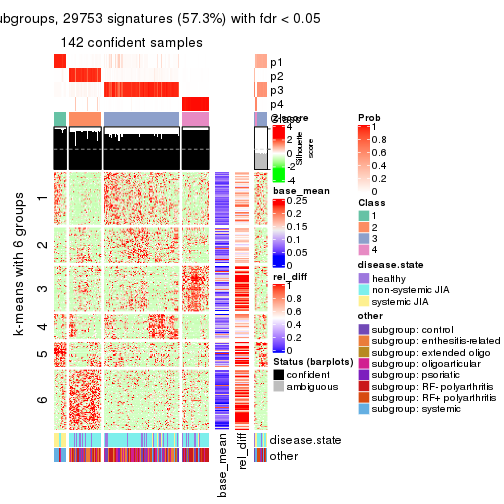
get_signatures(res, k = 5)
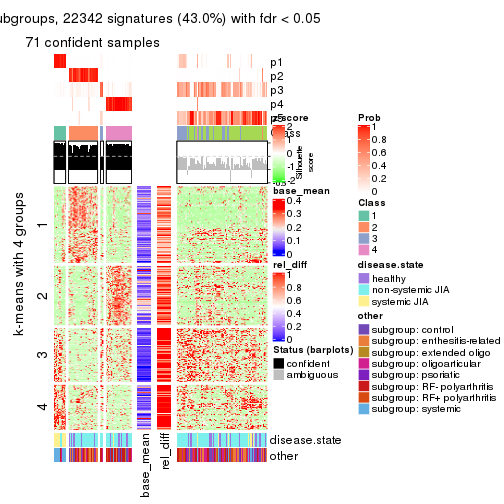
get_signatures(res, k = 6)
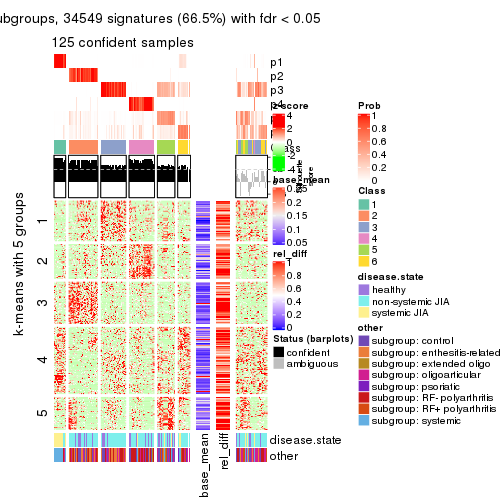
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
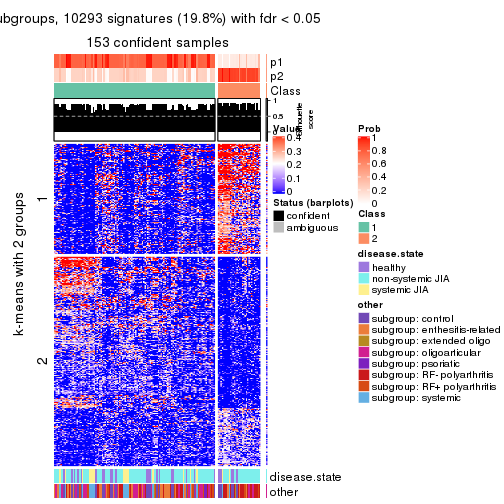
get_signatures(res, k = 3, scale_rows = FALSE)
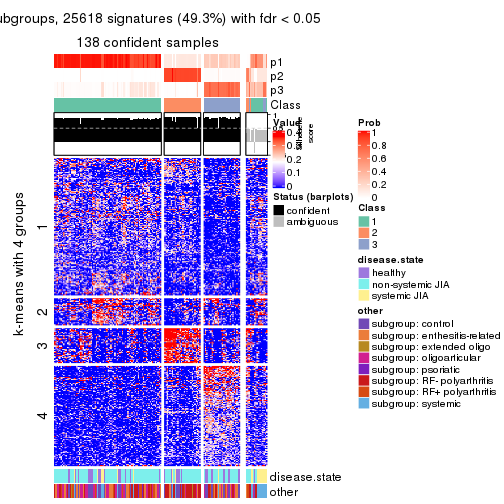
get_signatures(res, k = 4, scale_rows = FALSE)
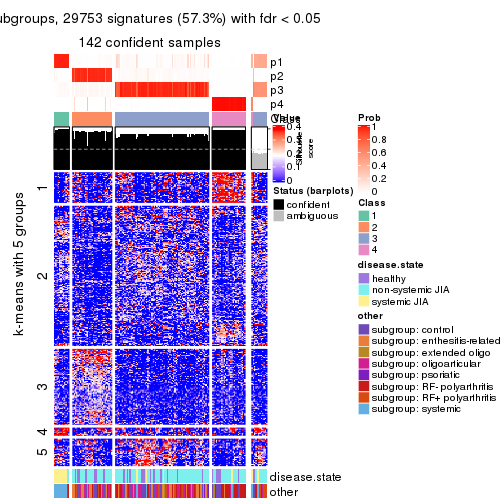
get_signatures(res, k = 5, scale_rows = FALSE)
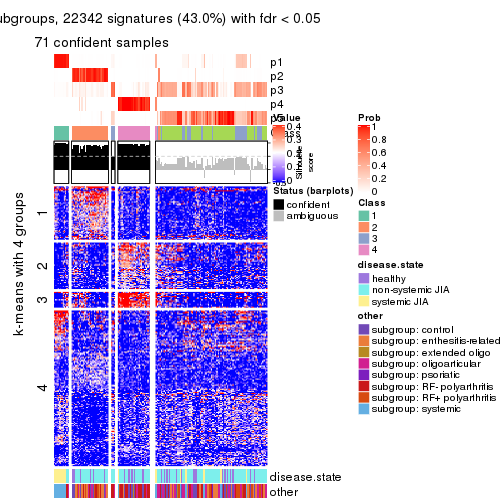
get_signatures(res, k = 6, scale_rows = FALSE)
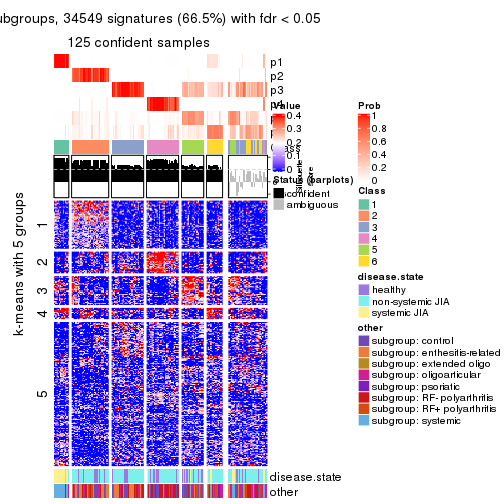
Compare the overlap of signatures from different k:
compare_signatures(res)
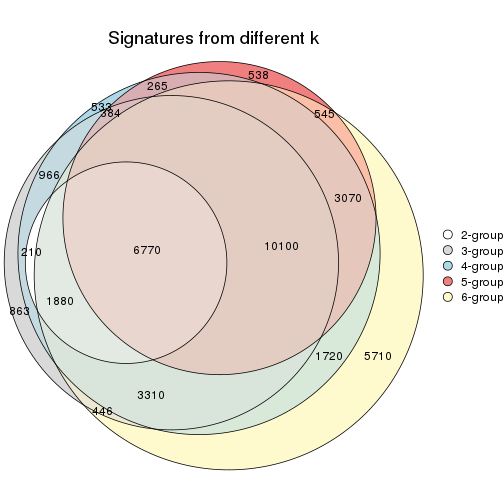
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
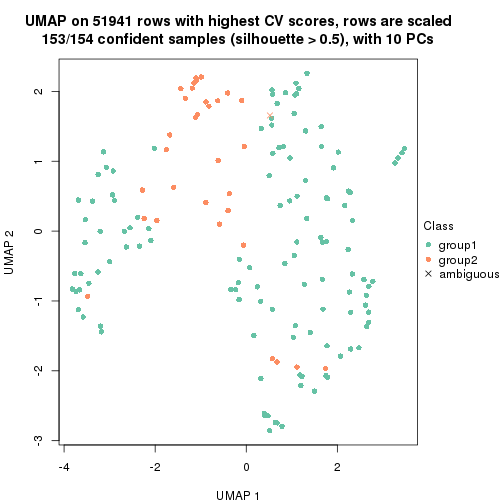
dimension_reduction(res, k = 3, method = "UMAP")
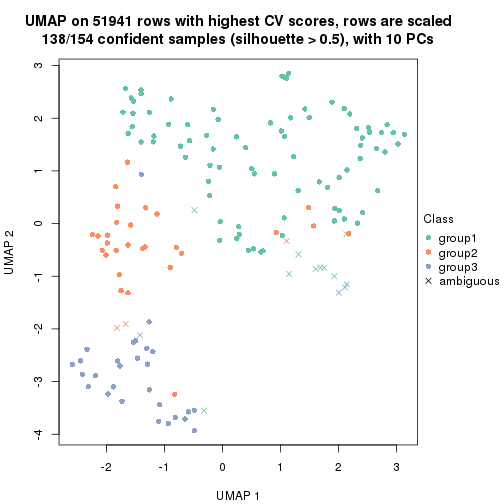
dimension_reduction(res, k = 4, method = "UMAP")
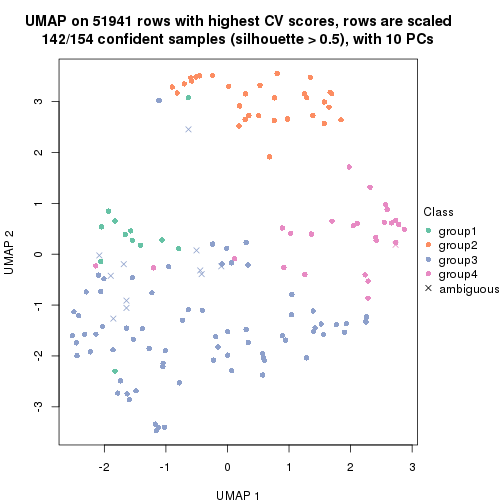
dimension_reduction(res, k = 5, method = "UMAP")
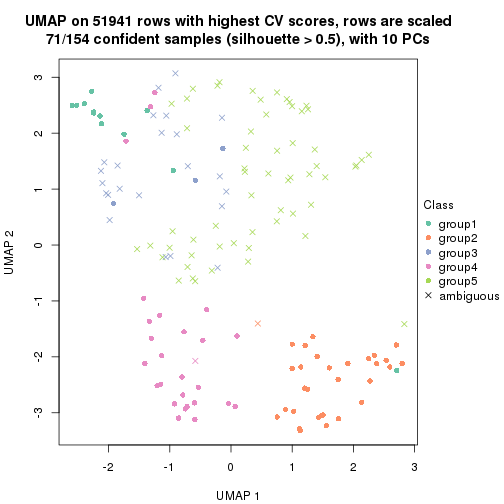
dimension_reduction(res, k = 6, method = "UMAP")
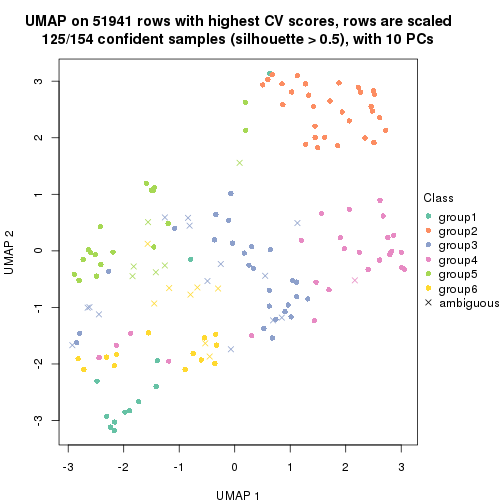
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
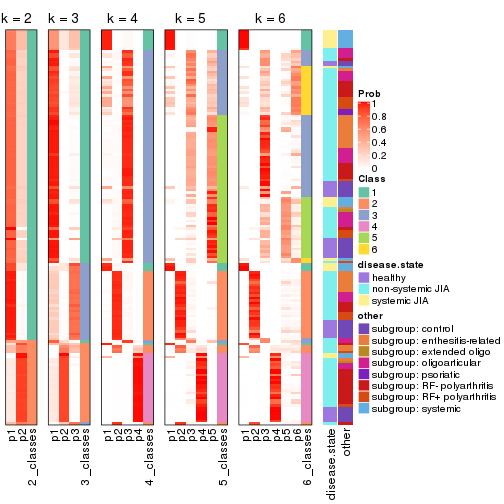
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> CV:kmeans 153 5.39e-01 3.06e-03 2
#> CV:kmeans 138 9.11e-01 4.81e-02 3
#> CV:kmeans 142 4.96e-14 1.05e-12 4
#> CV:kmeans 71 1.73e-09 1.14e-08 5
#> CV:kmeans 125 3.47e-12 9.34e-14 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["CV", "skmeans"]
# you can also extract it by
# res = res_list["CV:skmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'CV' method.
#> Subgroups are detected by 'skmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 4.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
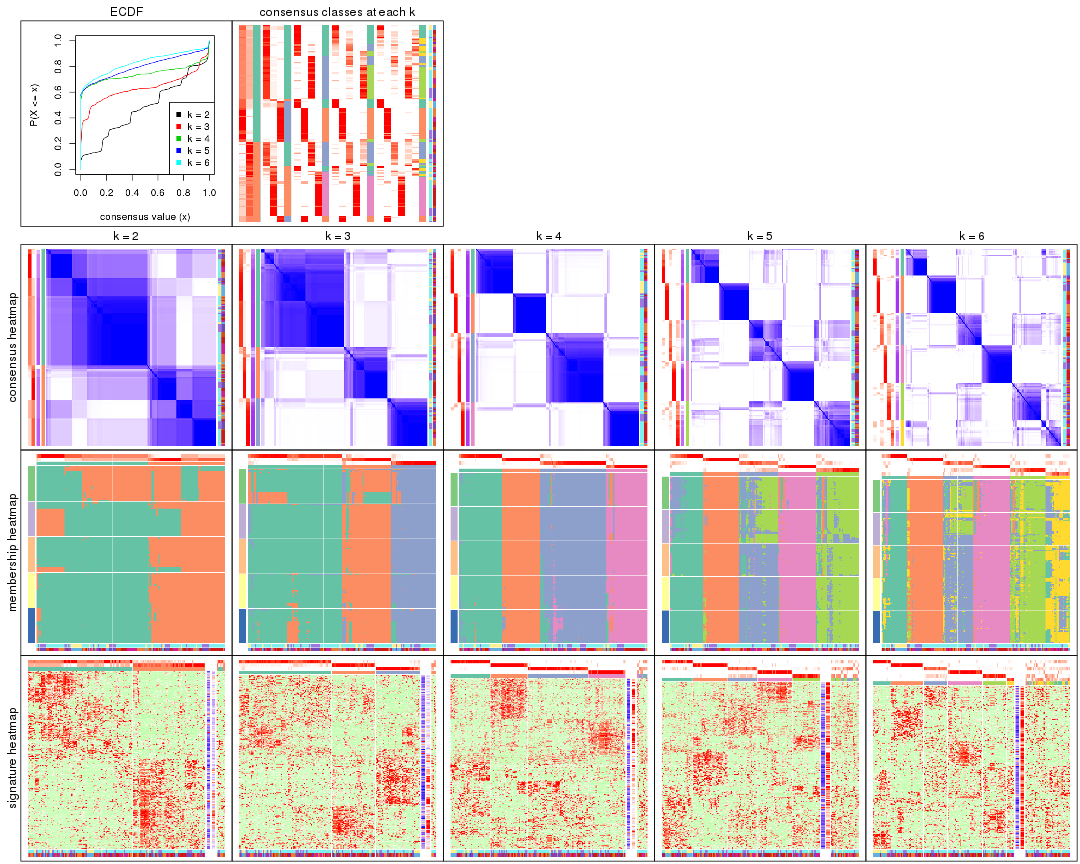
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
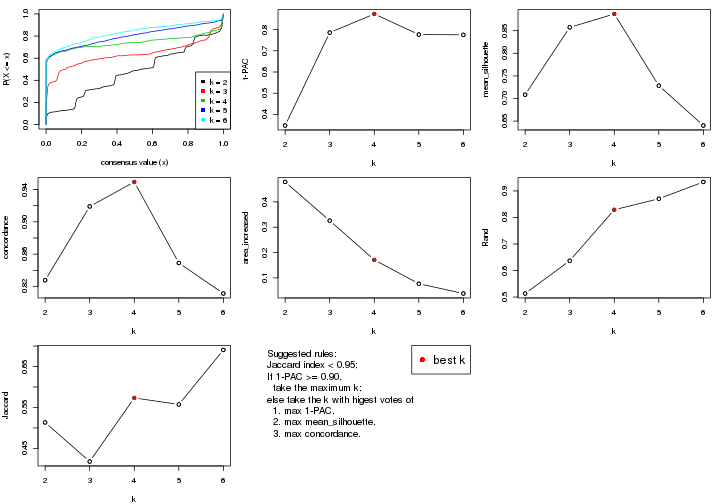
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.348 0.708 0.828 0.4785 0.513 0.513
#> 3 3 0.785 0.857 0.919 0.3259 0.637 0.418
#> 4 4 0.873 0.887 0.949 0.1712 0.829 0.573
#> 5 5 0.776 0.728 0.849 0.0771 0.871 0.557
#> 6 6 0.775 0.640 0.811 0.0383 0.934 0.690
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 4
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 2 0.7219 0.628 0.200 0.800
#> GSM340359 1 0.7602 0.757 0.780 0.220
#> GSM340361 1 0.9608 0.818 0.616 0.384
#> GSM340362 1 0.9635 0.818 0.612 0.388
#> GSM340363 2 0.0376 0.699 0.004 0.996
#> GSM340364 1 0.7602 0.757 0.780 0.220
#> GSM340365 1 0.9608 0.818 0.616 0.384
#> GSM340366 1 0.9522 0.816 0.628 0.372
#> GSM340367 1 0.7602 0.757 0.780 0.220
#> GSM340368 2 0.9635 0.695 0.388 0.612
#> GSM340369 2 0.9580 0.698 0.380 0.620
#> GSM340370 1 0.7602 0.757 0.780 0.220
#> GSM340371 1 0.9710 0.808 0.600 0.400
#> GSM340372 1 0.7602 0.757 0.780 0.220
#> GSM340373 1 0.7602 0.757 0.780 0.220
#> GSM340375 1 0.7602 0.757 0.780 0.220
#> GSM340376 1 0.7602 0.757 0.780 0.220
#> GSM340378 1 0.7602 0.757 0.780 0.220
#> GSM340243 1 0.9580 0.814 0.620 0.380
#> GSM340244 1 0.5408 0.382 0.876 0.124
#> GSM340246 1 0.0000 0.596 1.000 0.000
#> GSM340247 2 0.7602 0.783 0.220 0.780
#> GSM340248 1 0.0000 0.596 1.000 0.000
#> GSM340249 2 0.9635 0.695 0.388 0.612
#> GSM340250 1 0.9358 0.809 0.648 0.352
#> GSM340251 2 0.9635 0.695 0.388 0.612
#> GSM340252 2 0.9635 0.695 0.388 0.612
#> GSM340253 1 0.0000 0.596 1.000 0.000
#> GSM340254 1 0.0000 0.596 1.000 0.000
#> GSM340256 1 0.0000 0.596 1.000 0.000
#> GSM340258 1 0.9635 0.818 0.612 0.388
#> GSM340259 1 0.9635 0.818 0.612 0.388
#> GSM340260 1 0.9635 0.818 0.612 0.388
#> GSM340261 1 0.9635 0.818 0.612 0.388
#> GSM340262 1 0.9635 0.818 0.612 0.388
#> GSM340263 1 0.8861 -0.184 0.696 0.304
#> GSM340264 1 0.9909 0.757 0.556 0.444
#> GSM340265 1 0.9635 0.818 0.612 0.388
#> GSM340266 2 0.0000 0.696 0.000 1.000
#> GSM340267 1 0.9635 0.818 0.612 0.388
#> GSM340268 2 0.0000 0.696 0.000 1.000
#> GSM340269 1 0.9635 0.818 0.612 0.388
#> GSM340270 2 0.8861 -0.139 0.304 0.696
#> GSM537574 1 0.9522 0.816 0.628 0.372
#> GSM537580 2 0.3733 0.599 0.072 0.928
#> GSM537581 2 0.8386 0.762 0.268 0.732
#> GSM340272 1 0.9635 0.818 0.612 0.388
#> GSM340273 1 0.0000 0.596 1.000 0.000
#> GSM340275 2 0.9635 0.695 0.388 0.612
#> GSM340276 2 0.4298 0.569 0.088 0.912
#> GSM340277 1 0.0000 0.596 1.000 0.000
#> GSM340278 2 0.0000 0.696 0.000 1.000
#> GSM340279 2 0.0000 0.696 0.000 1.000
#> GSM340282 1 0.9635 0.818 0.612 0.388
#> GSM340284 2 0.9635 0.695 0.388 0.612
#> GSM340285 2 0.0000 0.696 0.000 1.000
#> GSM340286 1 0.9710 0.808 0.600 0.400
#> GSM340287 1 0.9635 0.818 0.612 0.388
#> GSM340288 1 0.9635 0.818 0.612 0.388
#> GSM340289 1 0.9635 0.818 0.612 0.388
#> GSM340290 2 0.0000 0.696 0.000 1.000
#> GSM340291 1 0.0000 0.596 1.000 0.000
#> GSM340293 2 0.6973 0.776 0.188 0.812
#> GSM340294 1 0.9635 0.818 0.612 0.388
#> GSM340296 1 0.9522 0.816 0.628 0.372
#> GSM340297 2 0.0000 0.696 0.000 1.000
#> GSM340298 1 0.9522 0.816 0.628 0.372
#> GSM340299 1 0.9635 0.818 0.612 0.388
#> GSM340301 1 0.9635 0.818 0.612 0.388
#> GSM340303 1 0.9608 0.818 0.616 0.384
#> GSM340304 2 0.4161 0.576 0.084 0.916
#> GSM340306 2 0.7602 0.783 0.220 0.780
#> GSM340307 1 0.0000 0.596 1.000 0.000
#> GSM340310 1 0.7602 0.757 0.780 0.220
#> GSM340314 2 0.3584 0.732 0.068 0.932
#> GSM340315 1 0.9775 0.796 0.588 0.412
#> GSM340317 2 0.9635 0.695 0.388 0.612
#> GSM340318 2 0.7602 0.783 0.220 0.780
#> GSM340319 2 0.7602 0.783 0.220 0.780
#> GSM340320 2 0.7528 0.783 0.216 0.784
#> GSM340321 1 0.0000 0.596 1.000 0.000
#> GSM340322 2 0.7602 0.783 0.220 0.780
#> GSM340324 1 0.0000 0.596 1.000 0.000
#> GSM340328 2 0.6531 0.663 0.168 0.832
#> GSM340330 2 0.0000 0.696 0.000 1.000
#> GSM340332 2 0.7602 0.783 0.220 0.780
#> GSM340333 1 0.0000 0.596 1.000 0.000
#> GSM340336 2 0.9608 0.697 0.384 0.616
#> GSM340337 2 0.7602 0.783 0.220 0.780
#> GSM340338 2 0.3114 0.626 0.056 0.944
#> GSM340339 2 0.7602 0.783 0.220 0.780
#> GSM340340 2 0.9635 0.695 0.388 0.612
#> GSM340341 2 0.9580 0.700 0.380 0.620
#> GSM340343 2 0.7602 0.783 0.220 0.780
#> GSM340344 1 0.7453 0.753 0.788 0.212
#> GSM340346 1 0.9635 0.818 0.612 0.388
#> GSM340347 2 0.7602 0.783 0.220 0.780
#> GSM340348 2 0.7602 0.783 0.220 0.780
#> GSM340349 1 0.9608 0.818 0.616 0.384
#> GSM340350 1 0.9608 0.818 0.616 0.384
#> GSM340351 1 0.9635 0.818 0.612 0.388
#> GSM340354 1 0.9635 0.818 0.612 0.388
#> GSM340356 1 0.0000 0.596 1.000 0.000
#> GSM340357 1 0.9635 0.818 0.612 0.388
#> GSM348183 2 0.0376 0.699 0.004 0.996
#> GSM348191 2 0.0000 0.696 0.000 1.000
#> GSM348193 2 0.7602 0.783 0.220 0.780
#> GSM537578 1 0.9635 0.818 0.612 0.388
#> GSM348181 2 0.0376 0.692 0.004 0.996
#> GSM348182 1 0.9866 0.772 0.568 0.432
#> GSM348184 2 0.9661 0.693 0.392 0.608
#> GSM348185 2 0.9635 0.695 0.388 0.612
#> GSM348186 1 0.0000 0.596 1.000 0.000
#> GSM348187 1 0.9635 0.818 0.612 0.388
#> GSM348188 2 0.7602 0.783 0.220 0.780
#> GSM348189 1 0.9710 0.808 0.600 0.400
#> GSM348190 1 0.9635 0.818 0.612 0.388
#> GSM348194 1 0.9635 0.818 0.612 0.388
#> GSM348195 1 0.9608 0.818 0.616 0.384
#> GSM348196 2 0.9248 -0.265 0.340 0.660
#> GSM537585 1 0.9635 0.818 0.612 0.388
#> GSM537594 1 0.0000 0.596 1.000 0.000
#> GSM537596 2 0.9944 -0.586 0.456 0.544
#> GSM537597 1 0.9608 0.818 0.616 0.384
#> GSM537602 1 0.9608 0.818 0.616 0.384
#> GSM340184 2 0.0376 0.692 0.004 0.996
#> GSM340185 1 0.4022 0.472 0.920 0.080
#> GSM340186 1 0.5629 0.364 0.868 0.132
#> GSM340187 2 0.7602 0.783 0.220 0.780
#> GSM340189 2 0.7602 0.783 0.220 0.780
#> GSM340190 2 0.9635 0.695 0.388 0.612
#> GSM340191 2 0.0000 0.696 0.000 1.000
#> GSM340192 1 0.7453 0.753 0.788 0.212
#> GSM340193 1 0.9635 0.818 0.612 0.388
#> GSM340194 1 0.9635 0.818 0.612 0.388
#> GSM340195 1 0.9635 0.818 0.612 0.388
#> GSM340196 2 0.7602 0.783 0.220 0.780
#> GSM340197 1 0.9635 0.818 0.612 0.388
#> GSM340198 1 0.0000 0.596 1.000 0.000
#> GSM340199 2 0.0000 0.696 0.000 1.000
#> GSM340200 1 0.0000 0.596 1.000 0.000
#> GSM340201 2 0.7602 0.783 0.220 0.780
#> GSM340202 2 0.7602 0.783 0.220 0.780
#> GSM340203 2 0.7602 0.783 0.220 0.780
#> GSM340204 1 0.7453 0.753 0.788 0.212
#> GSM340205 1 0.8016 0.768 0.756 0.244
#> GSM340206 2 0.0000 0.696 0.000 1.000
#> GSM340207 1 0.9661 0.815 0.608 0.392
#> GSM340237 1 0.0000 0.596 1.000 0.000
#> GSM340238 2 0.0000 0.696 0.000 1.000
#> GSM340239 1 0.9635 0.818 0.612 0.388
#> GSM340240 1 0.9635 0.818 0.612 0.388
#> GSM340241 1 0.0000 0.596 1.000 0.000
#> GSM340242 1 0.9710 0.808 0.600 0.400
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 2 0.3325 0.844 0.076 0.904 0.020
#> GSM340359 3 0.2866 0.890 0.076 0.008 0.916
#> GSM340361 1 0.0661 0.897 0.988 0.004 0.008
#> GSM340362 1 0.0237 0.901 0.996 0.000 0.004
#> GSM340363 2 0.2165 0.862 0.064 0.936 0.000
#> GSM340364 3 0.2866 0.890 0.076 0.008 0.916
#> GSM340365 1 0.0237 0.900 0.996 0.004 0.000
#> GSM340366 1 0.0237 0.900 0.996 0.004 0.000
#> GSM340367 1 0.7762 0.520 0.668 0.120 0.212
#> GSM340368 2 0.2261 0.857 0.068 0.932 0.000
#> GSM340369 2 0.2261 0.857 0.068 0.932 0.000
#> GSM340370 3 0.8033 0.588 0.240 0.120 0.640
#> GSM340371 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340372 1 0.7762 0.520 0.668 0.120 0.212
#> GSM340373 1 0.7762 0.520 0.668 0.120 0.212
#> GSM340375 3 0.8153 0.578 0.240 0.128 0.632
#> GSM340376 3 0.8033 0.588 0.240 0.120 0.640
#> GSM340378 3 0.4469 0.848 0.076 0.060 0.864
#> GSM340243 1 0.0237 0.900 0.996 0.004 0.000
#> GSM340244 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340246 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340247 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340248 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340249 2 0.6095 0.394 0.000 0.608 0.392
#> GSM340250 1 0.3340 0.807 0.880 0.120 0.000
#> GSM340251 3 0.0592 0.943 0.000 0.012 0.988
#> GSM340252 2 0.6126 0.374 0.000 0.600 0.400
#> GSM340253 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340254 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340256 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340258 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340259 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340260 1 0.0237 0.901 0.996 0.000 0.004
#> GSM340261 1 0.2496 0.915 0.928 0.068 0.004
#> GSM340262 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340263 3 0.0237 0.949 0.004 0.000 0.996
#> GSM340264 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340265 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340266 1 0.5363 0.713 0.724 0.276 0.000
#> GSM340267 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340268 1 0.5156 0.789 0.776 0.216 0.008
#> GSM340269 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340270 1 0.2261 0.915 0.932 0.068 0.000
#> GSM537574 1 0.4399 0.787 0.812 0.000 0.188
#> GSM537580 1 0.2680 0.916 0.924 0.068 0.008
#> GSM537581 2 0.2261 0.857 0.068 0.932 0.000
#> GSM340272 1 0.0237 0.901 0.996 0.000 0.004
#> GSM340273 3 0.0237 0.949 0.004 0.000 0.996
#> GSM340275 2 0.2356 0.846 0.000 0.928 0.072
#> GSM340276 1 0.2261 0.915 0.932 0.068 0.000
#> GSM340277 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340278 1 0.5968 0.553 0.636 0.364 0.000
#> GSM340279 1 0.5254 0.731 0.736 0.264 0.000
#> GSM340282 1 0.1015 0.906 0.980 0.012 0.008
#> GSM340284 3 0.0592 0.943 0.000 0.012 0.988
#> GSM340285 2 0.6045 0.289 0.380 0.620 0.000
#> GSM340286 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340287 1 0.2261 0.915 0.932 0.068 0.000
#> GSM340288 1 0.0424 0.898 0.992 0.000 0.008
#> GSM340289 1 0.0424 0.904 0.992 0.008 0.000
#> GSM340290 1 0.5465 0.695 0.712 0.288 0.000
#> GSM340291 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340293 2 0.2537 0.832 0.080 0.920 0.000
#> GSM340294 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340296 1 0.0237 0.900 0.996 0.004 0.000
#> GSM340297 1 0.2356 0.874 0.928 0.072 0.000
#> GSM340298 1 0.0237 0.900 0.996 0.004 0.000
#> GSM340299 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340301 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340303 1 0.2448 0.886 0.924 0.000 0.076
#> GSM340304 2 0.6280 0.264 0.460 0.540 0.000
#> GSM340306 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340307 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340310 2 0.5858 0.691 0.240 0.740 0.020
#> GSM340314 2 0.5178 0.591 0.256 0.744 0.000
#> GSM340315 1 0.1031 0.907 0.976 0.024 0.000
#> GSM340317 3 0.1860 0.909 0.000 0.052 0.948
#> GSM340318 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340319 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340320 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340321 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340322 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340324 3 0.0237 0.949 0.004 0.000 0.996
#> GSM340328 2 0.2448 0.853 0.076 0.924 0.000
#> GSM340330 1 0.3965 0.875 0.860 0.132 0.008
#> GSM340332 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340333 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340336 2 0.0592 0.891 0.000 0.988 0.012
#> GSM340337 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340338 1 0.3043 0.909 0.908 0.084 0.008
#> GSM340339 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340340 2 0.2550 0.860 0.056 0.932 0.012
#> GSM340341 2 0.2356 0.846 0.000 0.928 0.072
#> GSM340343 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340344 3 0.3340 0.833 0.120 0.000 0.880
#> GSM340346 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340347 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340348 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340349 1 0.0237 0.900 0.996 0.004 0.000
#> GSM340350 1 0.0237 0.900 0.996 0.004 0.000
#> GSM340351 1 0.0424 0.898 0.992 0.000 0.008
#> GSM340354 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340356 3 0.0237 0.949 0.004 0.000 0.996
#> GSM340357 1 0.0237 0.900 0.996 0.004 0.000
#> GSM348183 2 0.1289 0.881 0.032 0.968 0.000
#> GSM348191 1 0.3295 0.902 0.896 0.096 0.008
#> GSM348193 2 0.0237 0.894 0.000 0.996 0.004
#> GSM537578 1 0.0000 0.901 1.000 0.000 0.000
#> GSM348181 1 0.2711 0.908 0.912 0.088 0.000
#> GSM348182 1 0.2680 0.916 0.924 0.068 0.008
#> GSM348184 3 0.0000 0.950 0.000 0.000 1.000
#> GSM348185 2 0.5591 0.571 0.000 0.696 0.304
#> GSM348186 3 0.0000 0.950 0.000 0.000 1.000
#> GSM348187 1 0.0848 0.895 0.984 0.008 0.008
#> GSM348188 2 0.0237 0.894 0.000 0.996 0.004
#> GSM348189 1 0.2680 0.916 0.924 0.068 0.008
#> GSM348190 1 0.2680 0.916 0.924 0.068 0.008
#> GSM348194 1 0.0424 0.898 0.992 0.000 0.008
#> GSM348195 1 0.0848 0.895 0.984 0.008 0.008
#> GSM348196 2 0.6416 0.522 0.376 0.616 0.008
#> GSM537585 1 0.0000 0.901 1.000 0.000 0.000
#> GSM537594 3 0.0237 0.949 0.004 0.000 0.996
#> GSM537596 1 0.2261 0.915 0.932 0.068 0.000
#> GSM537597 1 0.4121 0.756 0.832 0.000 0.168
#> GSM537602 1 0.0424 0.898 0.992 0.000 0.008
#> GSM340184 1 0.3043 0.909 0.908 0.084 0.008
#> GSM340185 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340186 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340187 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340189 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340190 3 0.0424 0.945 0.000 0.008 0.992
#> GSM340191 1 0.5115 0.777 0.768 0.228 0.004
#> GSM340192 3 0.0592 0.942 0.012 0.000 0.988
#> GSM340193 1 0.2261 0.915 0.932 0.068 0.000
#> GSM340194 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340195 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340196 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340197 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340198 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340199 1 0.4702 0.790 0.788 0.212 0.000
#> GSM340200 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340201 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340202 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340203 2 0.0237 0.894 0.000 0.996 0.004
#> GSM340204 3 0.1643 0.916 0.044 0.000 0.956
#> GSM340205 1 0.3607 0.810 0.880 0.112 0.008
#> GSM340206 1 0.5156 0.789 0.776 0.216 0.008
#> GSM340207 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340237 3 0.0475 0.947 0.004 0.004 0.992
#> GSM340238 1 0.5733 0.633 0.676 0.324 0.000
#> GSM340239 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340240 1 0.2680 0.916 0.924 0.068 0.008
#> GSM340241 3 0.0000 0.950 0.000 0.000 1.000
#> GSM340242 1 0.2680 0.916 0.924 0.068 0.008
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340359 1 0.2760 0.835 0.872 0.128 0.000 0.000
#> GSM340361 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340362 3 0.1637 0.893 0.060 0.000 0.940 0.000
#> GSM340363 4 0.3688 0.708 0.208 0.000 0.000 0.792
#> GSM340364 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340365 1 0.0817 0.943 0.976 0.000 0.024 0.000
#> GSM340366 1 0.1792 0.907 0.932 0.000 0.068 0.000
#> GSM340367 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340368 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340369 4 0.3311 0.763 0.172 0.000 0.000 0.828
#> GSM340370 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340371 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340372 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340375 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340376 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340378 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340243 1 0.0188 0.954 0.996 0.000 0.004 0.000
#> GSM340244 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340246 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340247 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340248 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340249 4 0.4933 0.289 0.000 0.432 0.000 0.568
#> GSM340250 1 0.4730 0.412 0.636 0.000 0.364 0.000
#> GSM340251 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340252 4 0.4933 0.288 0.000 0.432 0.000 0.568
#> GSM340253 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340254 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340256 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340258 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340259 3 0.0592 0.922 0.016 0.000 0.984 0.000
#> GSM340260 3 0.3172 0.794 0.160 0.000 0.840 0.000
#> GSM340261 3 0.0188 0.926 0.004 0.000 0.996 0.000
#> GSM340262 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340263 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340264 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340265 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340266 3 0.2530 0.855 0.000 0.000 0.888 0.112
#> GSM340267 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340268 3 0.0188 0.926 0.000 0.000 0.996 0.004
#> GSM340269 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340270 3 0.3266 0.807 0.168 0.000 0.832 0.000
#> GSM537574 3 0.0469 0.922 0.000 0.012 0.988 0.000
#> GSM537580 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM537581 4 0.0336 0.921 0.008 0.000 0.000 0.992
#> GSM340272 1 0.4877 0.333 0.592 0.000 0.408 0.000
#> GSM340273 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340275 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340276 3 0.3172 0.814 0.160 0.000 0.840 0.000
#> GSM340277 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340278 3 0.4422 0.668 0.008 0.000 0.736 0.256
#> GSM340279 3 0.1940 0.884 0.000 0.000 0.924 0.076
#> GSM340282 3 0.0336 0.925 0.008 0.000 0.992 0.000
#> GSM340284 2 0.0188 0.984 0.000 0.996 0.004 0.000
#> GSM340285 4 0.5417 0.206 0.016 0.000 0.412 0.572
#> GSM340286 3 0.0336 0.925 0.008 0.000 0.992 0.000
#> GSM340287 3 0.0817 0.918 0.024 0.000 0.976 0.000
#> GSM340288 1 0.0336 0.953 0.992 0.000 0.008 0.000
#> GSM340289 3 0.4967 0.255 0.452 0.000 0.548 0.000
#> GSM340290 3 0.2469 0.859 0.000 0.000 0.892 0.108
#> GSM340291 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340293 4 0.0469 0.917 0.000 0.000 0.012 0.988
#> GSM340294 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340296 1 0.1211 0.932 0.960 0.000 0.040 0.000
#> GSM340297 3 0.3547 0.823 0.144 0.000 0.840 0.016
#> GSM340298 1 0.1716 0.913 0.936 0.000 0.064 0.000
#> GSM340299 3 0.0336 0.925 0.008 0.000 0.992 0.000
#> GSM340301 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340303 3 0.4655 0.689 0.032 0.208 0.760 0.000
#> GSM340304 1 0.0804 0.948 0.980 0.000 0.012 0.008
#> GSM340306 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340307 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340310 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340314 3 0.4730 0.466 0.000 0.000 0.636 0.364
#> GSM340315 3 0.4877 0.359 0.408 0.000 0.592 0.000
#> GSM340317 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340318 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340319 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340320 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340321 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340322 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340324 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340328 1 0.0336 0.951 0.992 0.000 0.000 0.008
#> GSM340330 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340332 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340333 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340336 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340337 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340338 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340339 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340340 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340341 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340343 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340344 2 0.2944 0.850 0.004 0.868 0.128 0.000
#> GSM340346 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340347 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340348 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340349 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340350 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340351 1 0.0469 0.951 0.988 0.000 0.012 0.000
#> GSM340354 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340356 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340357 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM348183 4 0.4564 0.500 0.328 0.000 0.000 0.672
#> GSM348191 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM348193 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM537578 1 0.0592 0.949 0.984 0.000 0.016 0.000
#> GSM348181 3 0.3311 0.801 0.172 0.000 0.828 0.000
#> GSM348182 3 0.0592 0.922 0.016 0.000 0.984 0.000
#> GSM348184 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM348185 4 0.4431 0.569 0.000 0.304 0.000 0.696
#> GSM348186 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM348187 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM348188 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM348189 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM348190 3 0.0336 0.925 0.008 0.000 0.992 0.000
#> GSM348194 1 0.0336 0.953 0.992 0.000 0.008 0.000
#> GSM348195 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM348196 1 0.0336 0.953 0.992 0.000 0.008 0.000
#> GSM537585 1 0.2760 0.844 0.872 0.000 0.128 0.000
#> GSM537594 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM537596 3 0.4981 0.216 0.464 0.000 0.536 0.000
#> GSM537597 1 0.0779 0.947 0.980 0.004 0.016 0.000
#> GSM537602 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340184 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340185 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340186 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340187 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340189 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340190 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340191 3 0.1474 0.901 0.000 0.000 0.948 0.052
#> GSM340192 2 0.1792 0.920 0.000 0.932 0.068 0.000
#> GSM340193 3 0.2469 0.863 0.108 0.000 0.892 0.000
#> GSM340194 3 0.0336 0.925 0.008 0.000 0.992 0.000
#> GSM340195 3 0.0336 0.925 0.008 0.000 0.992 0.000
#> GSM340196 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340197 3 0.0336 0.925 0.008 0.000 0.992 0.000
#> GSM340198 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340199 3 0.1557 0.898 0.000 0.000 0.944 0.056
#> GSM340200 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340201 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340202 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340203 4 0.0000 0.926 0.000 0.000 0.000 1.000
#> GSM340204 2 0.2760 0.853 0.000 0.872 0.128 0.000
#> GSM340205 1 0.0000 0.956 1.000 0.000 0.000 0.000
#> GSM340206 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340207 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340237 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340238 3 0.2149 0.875 0.000 0.000 0.912 0.088
#> GSM340239 3 0.0336 0.925 0.008 0.000 0.992 0.000
#> GSM340240 3 0.0000 0.927 0.000 0.000 1.000 0.000
#> GSM340241 2 0.0000 0.987 0.000 1.000 0.000 0.000
#> GSM340242 3 0.0000 0.927 0.000 0.000 1.000 0.000
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.0000 0.8707 1.000 0.000 0.000 0.000 0.000
#> GSM340359 1 0.0162 0.8685 0.996 0.004 0.000 0.000 0.000
#> GSM340361 1 0.1270 0.8637 0.948 0.000 0.052 0.000 0.000
#> GSM340362 5 0.1117 0.7192 0.020 0.000 0.016 0.000 0.964
#> GSM340363 1 0.6024 0.2259 0.472 0.000 0.116 0.412 0.000
#> GSM340364 1 0.0000 0.8707 1.000 0.000 0.000 0.000 0.000
#> GSM340365 5 0.6244 0.3640 0.200 0.000 0.260 0.000 0.540
#> GSM340366 5 0.6526 0.2487 0.256 0.000 0.260 0.000 0.484
#> GSM340367 1 0.0000 0.8707 1.000 0.000 0.000 0.000 0.000
#> GSM340368 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340369 4 0.3305 0.7206 0.224 0.000 0.000 0.776 0.000
#> GSM340370 1 0.0000 0.8707 1.000 0.000 0.000 0.000 0.000
#> GSM340371 5 0.0000 0.7263 0.000 0.000 0.000 0.000 1.000
#> GSM340372 1 0.0000 0.8707 1.000 0.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.8707 1.000 0.000 0.000 0.000 0.000
#> GSM340375 1 0.0000 0.8707 1.000 0.000 0.000 0.000 0.000
#> GSM340376 1 0.0000 0.8707 1.000 0.000 0.000 0.000 0.000
#> GSM340378 1 0.0000 0.8707 1.000 0.000 0.000 0.000 0.000
#> GSM340243 3 0.4475 0.3216 0.276 0.000 0.692 0.000 0.032
#> GSM340244 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340246 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340247 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340248 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340249 4 0.4235 0.3103 0.000 0.424 0.000 0.576 0.000
#> GSM340250 3 0.4138 0.4538 0.384 0.000 0.616 0.000 0.000
#> GSM340251 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340252 4 0.3661 0.6271 0.000 0.276 0.000 0.724 0.000
#> GSM340253 2 0.2516 0.8245 0.000 0.860 0.000 0.000 0.140
#> GSM340254 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340256 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340258 5 0.3876 0.2500 0.000 0.000 0.316 0.000 0.684
#> GSM340259 5 0.3895 0.2616 0.000 0.000 0.320 0.000 0.680
#> GSM340260 5 0.5929 0.3905 0.156 0.000 0.260 0.000 0.584
#> GSM340261 3 0.2074 0.6775 0.000 0.000 0.896 0.000 0.104
#> GSM340262 5 0.1608 0.6895 0.000 0.000 0.072 0.000 0.928
#> GSM340263 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340264 5 0.0510 0.7232 0.000 0.000 0.016 0.000 0.984
#> GSM340265 5 0.1197 0.7101 0.000 0.000 0.048 0.000 0.952
#> GSM340266 3 0.3895 0.6721 0.000 0.000 0.680 0.000 0.320
#> GSM340267 5 0.2690 0.6383 0.000 0.000 0.156 0.000 0.844
#> GSM340268 3 0.3966 0.6657 0.000 0.000 0.664 0.000 0.336
#> GSM340269 3 0.4227 0.5136 0.000 0.000 0.580 0.000 0.420
#> GSM340270 3 0.5976 0.4890 0.112 0.000 0.488 0.000 0.400
#> GSM537574 3 0.4909 0.5271 0.000 0.028 0.560 0.000 0.412
#> GSM537580 5 0.0000 0.7263 0.000 0.000 0.000 0.000 1.000
#> GSM537581 4 0.2424 0.8239 0.132 0.000 0.000 0.868 0.000
#> GSM340272 5 0.4563 0.5366 0.048 0.000 0.244 0.000 0.708
#> GSM340273 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340275 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340276 3 0.2970 0.6979 0.004 0.000 0.828 0.000 0.168
#> GSM340277 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340278 3 0.1478 0.6370 0.000 0.000 0.936 0.000 0.064
#> GSM340279 3 0.3242 0.7013 0.000 0.000 0.784 0.000 0.216
#> GSM340282 5 0.2929 0.6235 0.000 0.000 0.180 0.000 0.820
#> GSM340284 2 0.1043 0.9405 0.000 0.960 0.040 0.000 0.000
#> GSM340285 3 0.2152 0.5965 0.004 0.000 0.920 0.044 0.032
#> GSM340286 5 0.0609 0.7220 0.000 0.000 0.020 0.000 0.980
#> GSM340287 3 0.3003 0.7005 0.000 0.000 0.812 0.000 0.188
#> GSM340288 1 0.3452 0.7941 0.756 0.000 0.244 0.000 0.000
#> GSM340289 5 0.6184 0.1331 0.380 0.000 0.140 0.000 0.480
#> GSM340290 3 0.3074 0.6977 0.000 0.000 0.804 0.000 0.196
#> GSM340291 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340293 3 0.3910 0.4857 0.000 0.000 0.720 0.272 0.008
#> GSM340294 3 0.3534 0.6951 0.000 0.000 0.744 0.000 0.256
#> GSM340296 3 0.4302 0.3858 0.248 0.000 0.720 0.000 0.032
#> GSM340297 3 0.5104 0.6463 0.192 0.000 0.692 0.000 0.116
#> GSM340298 3 0.4276 0.3938 0.244 0.000 0.724 0.000 0.032
#> GSM340299 5 0.1197 0.7102 0.000 0.000 0.048 0.000 0.952
#> GSM340301 5 0.0880 0.7163 0.000 0.000 0.032 0.000 0.968
#> GSM340303 5 0.3913 0.5006 0.000 0.000 0.324 0.000 0.676
#> GSM340304 1 0.4949 0.6171 0.572 0.000 0.396 0.000 0.032
#> GSM340306 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340307 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340310 1 0.0000 0.8707 1.000 0.000 0.000 0.000 0.000
#> GSM340314 3 0.4777 0.6729 0.000 0.000 0.664 0.044 0.292
#> GSM340315 3 0.4723 0.6726 0.128 0.000 0.736 0.000 0.136
#> GSM340317 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340318 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340319 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340320 4 0.0880 0.9106 0.000 0.000 0.032 0.968 0.000
#> GSM340321 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340322 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340324 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340328 1 0.0000 0.8707 1.000 0.000 0.000 0.000 0.000
#> GSM340330 3 0.3966 0.6657 0.000 0.000 0.664 0.000 0.336
#> GSM340332 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340333 2 0.1043 0.9413 0.000 0.960 0.000 0.000 0.040
#> GSM340336 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340337 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340338 5 0.4397 -0.2059 0.004 0.000 0.432 0.000 0.564
#> GSM340339 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340340 4 0.0290 0.9277 0.008 0.000 0.000 0.992 0.000
#> GSM340341 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340343 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340344 5 0.4074 0.3324 0.000 0.364 0.000 0.000 0.636
#> GSM340346 3 0.3074 0.5779 0.000 0.000 0.804 0.000 0.196
#> GSM340347 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340348 4 0.1197 0.8978 0.000 0.000 0.048 0.952 0.000
#> GSM340349 1 0.4930 0.6125 0.580 0.000 0.388 0.000 0.032
#> GSM340350 1 0.2654 0.8267 0.884 0.000 0.084 0.000 0.032
#> GSM340351 1 0.3366 0.8008 0.768 0.000 0.232 0.000 0.000
#> GSM340354 5 0.4307 -0.4039 0.000 0.000 0.500 0.000 0.500
#> GSM340356 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340357 1 0.2561 0.8392 0.856 0.000 0.144 0.000 0.000
#> GSM348183 4 0.4294 0.0144 0.468 0.000 0.000 0.532 0.000
#> GSM348191 3 0.3932 0.6695 0.000 0.000 0.672 0.000 0.328
#> GSM348193 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM537578 3 0.3687 0.4163 0.180 0.000 0.792 0.000 0.028
#> GSM348181 3 0.3932 0.6695 0.000 0.000 0.672 0.000 0.328
#> GSM348182 5 0.0609 0.7220 0.000 0.000 0.020 0.000 0.980
#> GSM348184 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM348185 4 0.3561 0.6546 0.000 0.260 0.000 0.740 0.000
#> GSM348186 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM348187 1 0.2280 0.8433 0.880 0.000 0.120 0.000 0.000
#> GSM348188 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM348189 5 0.0000 0.7263 0.000 0.000 0.000 0.000 1.000
#> GSM348190 5 0.1608 0.7018 0.000 0.000 0.072 0.000 0.928
#> GSM348194 1 0.3424 0.7965 0.760 0.000 0.240 0.000 0.000
#> GSM348195 1 0.1965 0.8539 0.904 0.000 0.096 0.000 0.000
#> GSM348196 1 0.3424 0.7965 0.760 0.000 0.240 0.000 0.000
#> GSM537585 5 0.6023 0.3871 0.248 0.000 0.176 0.000 0.576
#> GSM537594 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM537596 3 0.5831 0.3644 0.292 0.000 0.580 0.000 0.128
#> GSM537597 1 0.3612 0.7771 0.732 0.000 0.268 0.000 0.000
#> GSM537602 1 0.4822 0.6755 0.616 0.000 0.352 0.000 0.032
#> GSM340184 3 0.3966 0.6657 0.000 0.000 0.664 0.000 0.336
#> GSM340185 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340186 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340187 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340189 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340190 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340191 3 0.3966 0.6657 0.000 0.000 0.664 0.000 0.336
#> GSM340192 2 0.5140 0.5406 0.000 0.664 0.084 0.000 0.252
#> GSM340193 5 0.5476 -0.0970 0.068 0.000 0.388 0.000 0.544
#> GSM340194 5 0.0000 0.7263 0.000 0.000 0.000 0.000 1.000
#> GSM340195 5 0.0000 0.7263 0.000 0.000 0.000 0.000 1.000
#> GSM340196 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340197 5 0.0794 0.7182 0.000 0.000 0.028 0.000 0.972
#> GSM340198 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340199 3 0.1478 0.6276 0.000 0.000 0.936 0.000 0.064
#> GSM340200 2 0.1544 0.9177 0.000 0.932 0.068 0.000 0.000
#> GSM340201 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340202 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340203 4 0.0000 0.9325 0.000 0.000 0.000 1.000 0.000
#> GSM340204 5 0.4306 -0.0140 0.000 0.492 0.000 0.000 0.508
#> GSM340205 1 0.0000 0.8707 1.000 0.000 0.000 0.000 0.000
#> GSM340206 3 0.3966 0.6657 0.000 0.000 0.664 0.000 0.336
#> GSM340207 5 0.1043 0.7121 0.000 0.000 0.040 0.000 0.960
#> GSM340237 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340238 3 0.3966 0.6657 0.000 0.000 0.664 0.000 0.336
#> GSM340239 5 0.0880 0.7169 0.000 0.000 0.032 0.000 0.968
#> GSM340240 5 0.1908 0.6843 0.000 0.000 0.092 0.000 0.908
#> GSM340241 2 0.0000 0.9769 0.000 1.000 0.000 0.000 0.000
#> GSM340242 5 0.0000 0.7263 0.000 0.000 0.000 0.000 1.000
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.0000 0.7851 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340359 1 0.0000 0.7851 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340361 1 0.2762 0.7016 0.860 0.000 0.092 0.000 0.000 0.048
#> GSM340362 5 0.1218 0.7206 0.012 0.000 0.028 0.000 0.956 0.004
#> GSM340363 1 0.7670 0.1362 0.312 0.000 0.204 0.236 0.000 0.248
#> GSM340364 1 0.0000 0.7851 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340365 6 0.4936 0.0396 0.064 0.000 0.000 0.000 0.436 0.500
#> GSM340366 6 0.4972 0.3683 0.116 0.000 0.000 0.000 0.256 0.628
#> GSM340367 1 0.0000 0.7851 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340368 4 0.0260 0.9274 0.008 0.000 0.000 0.992 0.000 0.000
#> GSM340369 4 0.3464 0.5624 0.312 0.000 0.000 0.688 0.000 0.000
#> GSM340370 1 0.0000 0.7851 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340371 5 0.0458 0.7287 0.000 0.000 0.016 0.000 0.984 0.000
#> GSM340372 1 0.0000 0.7851 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.7851 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340375 1 0.0000 0.7851 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340376 1 0.0000 0.7851 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340378 1 0.0000 0.7851 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340243 6 0.4502 0.5360 0.116 0.000 0.140 0.000 0.012 0.732
#> GSM340244 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340246 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340247 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340248 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340249 4 0.3864 0.1452 0.000 0.480 0.000 0.520 0.000 0.000
#> GSM340250 3 0.4168 0.3369 0.400 0.000 0.584 0.000 0.000 0.016
#> GSM340251 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340252 4 0.3050 0.6894 0.000 0.236 0.000 0.764 0.000 0.000
#> GSM340253 2 0.2092 0.8182 0.000 0.876 0.000 0.000 0.124 0.000
#> GSM340254 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340256 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340258 5 0.3864 -0.3468 0.000 0.000 0.480 0.000 0.520 0.000
#> GSM340259 5 0.5398 0.1689 0.000 0.000 0.240 0.000 0.580 0.180
#> GSM340260 5 0.5982 0.4474 0.144 0.000 0.172 0.000 0.612 0.072
#> GSM340261 3 0.3221 0.6467 0.000 0.000 0.828 0.000 0.076 0.096
#> GSM340262 5 0.2883 0.5114 0.000 0.000 0.212 0.000 0.788 0.000
#> GSM340263 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340264 5 0.0865 0.7232 0.000 0.000 0.036 0.000 0.964 0.000
#> GSM340265 5 0.2146 0.6803 0.000 0.000 0.004 0.000 0.880 0.116
#> GSM340266 3 0.4834 0.6980 0.000 0.000 0.656 0.000 0.224 0.120
#> GSM340267 3 0.5654 -0.1511 0.000 0.000 0.444 0.000 0.404 0.152
#> GSM340268 3 0.3050 0.7407 0.000 0.000 0.764 0.000 0.236 0.000
#> GSM340269 3 0.4453 0.5511 0.000 0.000 0.568 0.000 0.400 0.032
#> GSM340270 3 0.5046 0.6700 0.100 0.000 0.620 0.000 0.276 0.004
#> GSM537574 3 0.6033 0.4270 0.000 0.076 0.452 0.000 0.416 0.056
#> GSM537580 5 0.0547 0.7282 0.000 0.000 0.020 0.000 0.980 0.000
#> GSM537581 4 0.2631 0.7903 0.152 0.000 0.008 0.840 0.000 0.000
#> GSM340272 5 0.3986 0.0972 0.000 0.000 0.004 0.000 0.532 0.464
#> GSM340273 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340275 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340276 6 0.5327 0.1067 0.000 0.000 0.372 0.000 0.112 0.516
#> GSM340277 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340278 6 0.3717 0.4792 0.000 0.000 0.148 0.000 0.072 0.780
#> GSM340279 3 0.2972 0.7029 0.000 0.000 0.836 0.000 0.128 0.036
#> GSM340282 5 0.3838 0.1507 0.000 0.000 0.000 0.000 0.552 0.448
#> GSM340284 2 0.2793 0.7196 0.000 0.800 0.200 0.000 0.000 0.000
#> GSM340285 6 0.2994 0.5190 0.000 0.000 0.164 0.008 0.008 0.820
#> GSM340286 5 0.0458 0.7288 0.000 0.000 0.016 0.000 0.984 0.000
#> GSM340287 3 0.1921 0.5691 0.000 0.000 0.916 0.000 0.032 0.052
#> GSM340288 6 0.6114 -0.1051 0.348 0.000 0.236 0.000 0.004 0.412
#> GSM340289 5 0.7602 -0.0151 0.208 0.000 0.224 0.000 0.348 0.220
#> GSM340290 3 0.4613 0.5206 0.000 0.000 0.660 0.000 0.080 0.260
#> GSM340291 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340293 6 0.5398 0.3262 0.000 0.000 0.320 0.136 0.000 0.544
#> GSM340294 3 0.3290 0.7425 0.000 0.000 0.776 0.000 0.208 0.016
#> GSM340296 6 0.4954 0.4982 0.100 0.000 0.208 0.000 0.016 0.676
#> GSM340297 3 0.3821 0.6324 0.156 0.000 0.776 0.000 0.064 0.004
#> GSM340298 6 0.5062 0.4748 0.092 0.000 0.240 0.000 0.016 0.652
#> GSM340299 5 0.0865 0.7200 0.000 0.000 0.000 0.000 0.964 0.036
#> GSM340301 5 0.0632 0.7210 0.000 0.000 0.024 0.000 0.976 0.000
#> GSM340303 6 0.4097 -0.0787 0.000 0.000 0.008 0.000 0.492 0.500
#> GSM340304 6 0.1010 0.5547 0.004 0.000 0.036 0.000 0.000 0.960
#> GSM340306 4 0.0146 0.9297 0.000 0.000 0.000 0.996 0.000 0.004
#> GSM340307 2 0.0146 0.9460 0.000 0.996 0.000 0.000 0.000 0.004
#> GSM340310 1 0.0000 0.7851 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340314 3 0.3744 0.7311 0.000 0.000 0.764 0.052 0.184 0.000
#> GSM340315 3 0.1606 0.5379 0.008 0.000 0.932 0.000 0.004 0.056
#> GSM340317 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340318 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340319 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340320 4 0.2868 0.7963 0.000 0.000 0.132 0.840 0.000 0.028
#> GSM340321 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340322 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340324 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340328 1 0.0146 0.7828 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM340330 3 0.3050 0.7407 0.000 0.000 0.764 0.000 0.236 0.000
#> GSM340332 4 0.0547 0.9189 0.000 0.000 0.020 0.980 0.000 0.000
#> GSM340333 2 0.0790 0.9195 0.000 0.968 0.000 0.000 0.032 0.000
#> GSM340336 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340337 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340338 3 0.4015 0.6153 0.012 0.000 0.616 0.000 0.372 0.000
#> GSM340339 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340340 4 0.0547 0.9197 0.020 0.000 0.000 0.980 0.000 0.000
#> GSM340341 4 0.0146 0.9297 0.000 0.000 0.000 0.996 0.000 0.004
#> GSM340343 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340344 5 0.3380 0.5044 0.000 0.244 0.004 0.000 0.748 0.004
#> GSM340346 6 0.4683 0.3774 0.000 0.000 0.320 0.000 0.064 0.616
#> GSM340347 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340348 4 0.1616 0.8832 0.000 0.000 0.020 0.932 0.000 0.048
#> GSM340349 6 0.2763 0.5576 0.072 0.000 0.024 0.000 0.028 0.876
#> GSM340350 1 0.4631 0.0452 0.536 0.000 0.004 0.000 0.032 0.428
#> GSM340351 1 0.5932 0.3613 0.532 0.000 0.232 0.000 0.012 0.224
#> GSM340354 3 0.4264 0.3830 0.000 0.000 0.492 0.000 0.492 0.016
#> GSM340356 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340357 1 0.5595 0.3248 0.516 0.000 0.164 0.000 0.000 0.320
#> GSM348183 1 0.4787 0.1038 0.484 0.000 0.012 0.476 0.000 0.028
#> GSM348191 3 0.4478 0.7269 0.000 0.000 0.680 0.000 0.244 0.076
#> GSM348193 4 0.0146 0.9297 0.000 0.000 0.000 0.996 0.000 0.004
#> GSM537578 6 0.2313 0.5675 0.012 0.000 0.100 0.000 0.004 0.884
#> GSM348181 3 0.4643 0.7337 0.028 0.000 0.704 0.000 0.216 0.052
#> GSM348182 5 0.0632 0.7271 0.000 0.000 0.024 0.000 0.976 0.000
#> GSM348184 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM348185 4 0.3608 0.6354 0.000 0.272 0.000 0.716 0.000 0.012
#> GSM348186 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM348187 1 0.5979 0.1595 0.416 0.000 0.232 0.000 0.000 0.352
#> GSM348188 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM348189 5 0.0547 0.7282 0.000 0.000 0.020 0.000 0.980 0.000
#> GSM348190 5 0.2852 0.6643 0.000 0.000 0.064 0.000 0.856 0.080
#> GSM348194 6 0.6128 -0.1517 0.372 0.000 0.236 0.000 0.004 0.388
#> GSM348195 1 0.4809 0.5282 0.664 0.000 0.208 0.000 0.000 0.128
#> GSM348196 6 0.6125 -0.1353 0.364 0.000 0.236 0.000 0.004 0.396
#> GSM537585 5 0.7252 0.0594 0.160 0.000 0.236 0.000 0.432 0.172
#> GSM537594 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM537596 3 0.6064 -0.2429 0.124 0.000 0.428 0.000 0.028 0.420
#> GSM537597 6 0.6199 -0.0584 0.320 0.000 0.244 0.000 0.008 0.428
#> GSM537602 6 0.0964 0.5583 0.016 0.000 0.012 0.000 0.004 0.968
#> GSM340184 3 0.3050 0.7407 0.000 0.000 0.764 0.000 0.236 0.000
#> GSM340185 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340186 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340187 4 0.0146 0.9295 0.000 0.000 0.004 0.996 0.000 0.000
#> GSM340189 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340190 2 0.0260 0.9423 0.000 0.992 0.008 0.000 0.000 0.000
#> GSM340191 3 0.3189 0.7404 0.000 0.000 0.760 0.000 0.236 0.004
#> GSM340192 2 0.5980 -0.0763 0.000 0.400 0.000 0.000 0.228 0.372
#> GSM340193 5 0.6454 -0.0999 0.016 0.000 0.276 0.000 0.364 0.344
#> GSM340194 5 0.0291 0.7278 0.000 0.000 0.004 0.000 0.992 0.004
#> GSM340195 5 0.0291 0.7278 0.000 0.000 0.004 0.000 0.992 0.004
#> GSM340196 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340197 5 0.0547 0.7273 0.000 0.000 0.020 0.000 0.980 0.000
#> GSM340198 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340199 6 0.4026 0.3622 0.000 0.000 0.348 0.000 0.016 0.636
#> GSM340200 2 0.3634 0.4483 0.000 0.644 0.000 0.000 0.000 0.356
#> GSM340201 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340202 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340203 4 0.0000 0.9313 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340204 5 0.4836 0.2963 0.000 0.380 0.004 0.000 0.564 0.052
#> GSM340205 1 0.0146 0.7831 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM340206 3 0.3050 0.7407 0.000 0.000 0.764 0.000 0.236 0.000
#> GSM340207 5 0.1814 0.6625 0.000 0.000 0.100 0.000 0.900 0.000
#> GSM340237 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340238 3 0.3050 0.7407 0.000 0.000 0.764 0.000 0.236 0.000
#> GSM340239 5 0.0547 0.7261 0.000 0.000 0.020 0.000 0.980 0.000
#> GSM340240 5 0.3076 0.5336 0.000 0.000 0.000 0.000 0.760 0.240
#> GSM340241 2 0.0000 0.9490 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340242 5 0.0547 0.7282 0.000 0.000 0.020 0.000 0.980 0.000
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
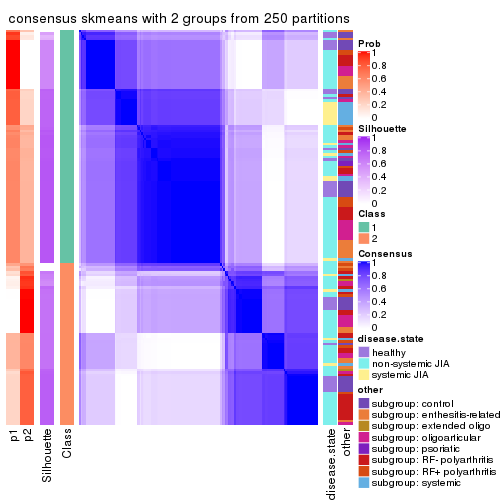
consensus_heatmap(res, k = 3)
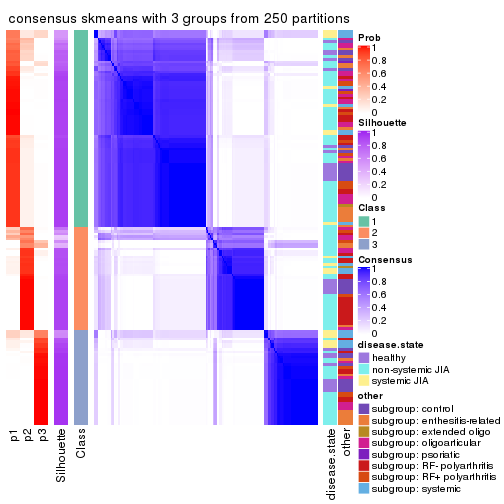
consensus_heatmap(res, k = 4)
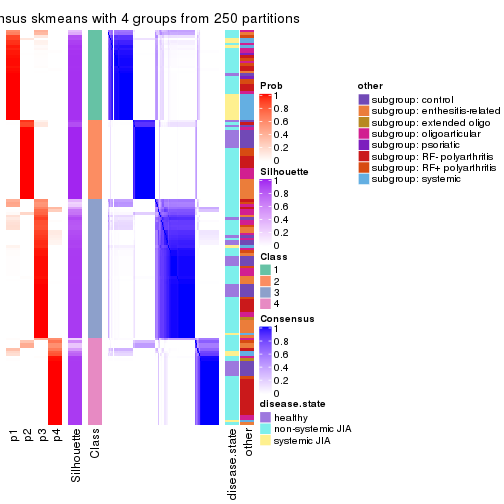
consensus_heatmap(res, k = 5)
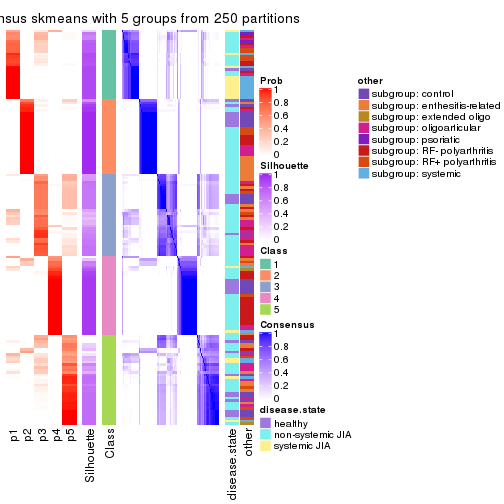
consensus_heatmap(res, k = 6)
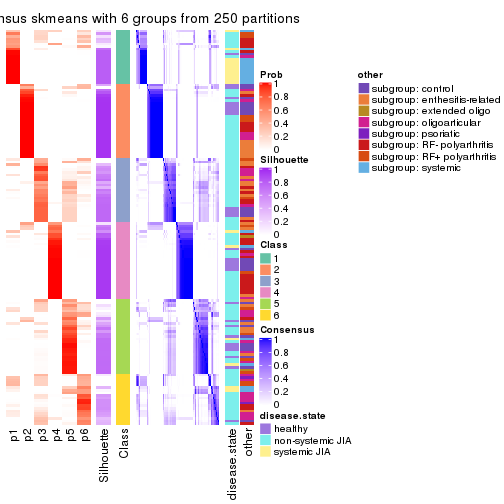
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
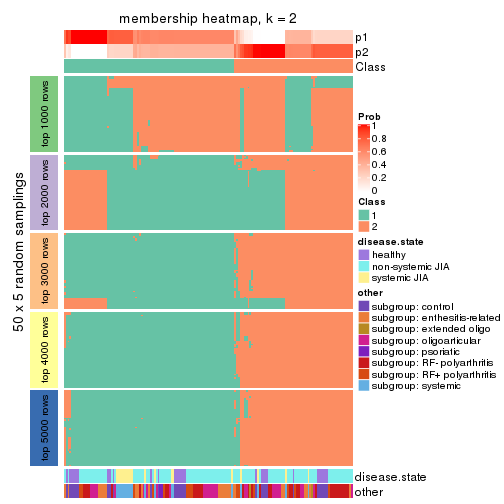
membership_heatmap(res, k = 3)
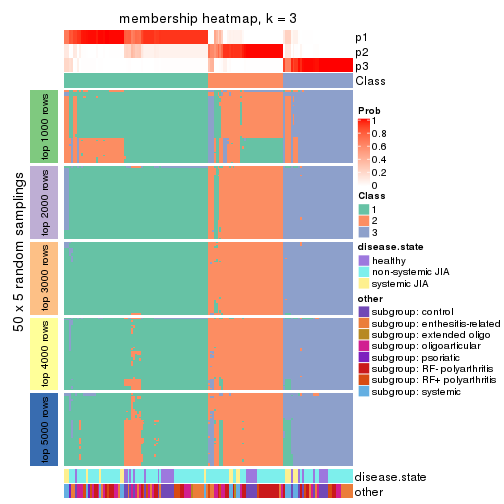
membership_heatmap(res, k = 4)
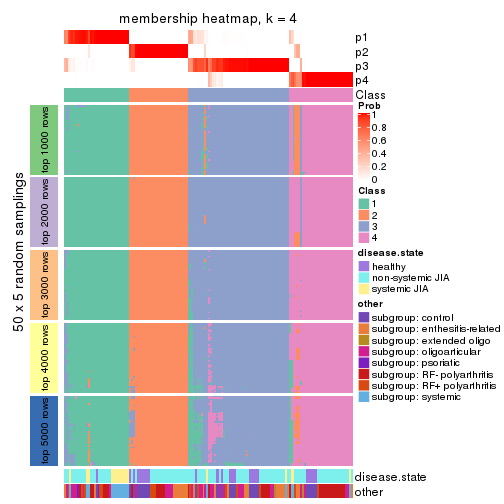
membership_heatmap(res, k = 5)
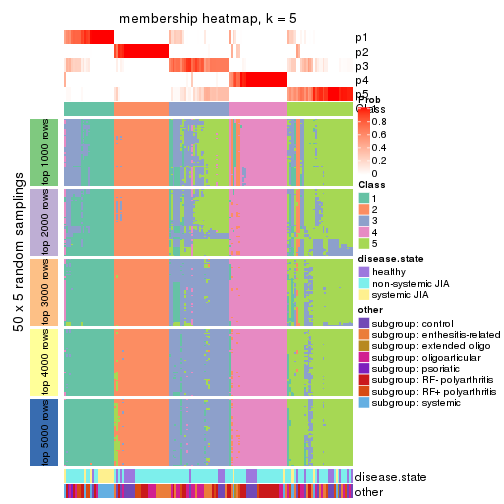
membership_heatmap(res, k = 6)
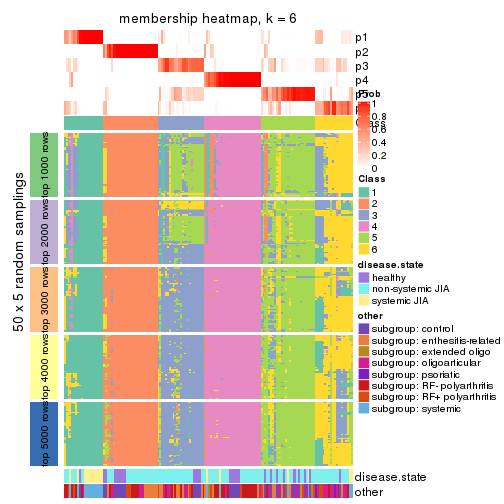
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
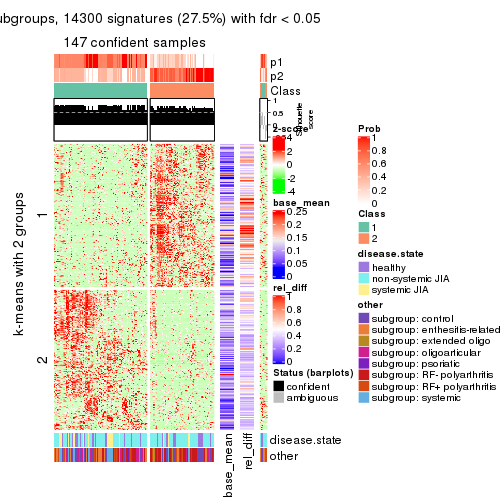
get_signatures(res, k = 3)
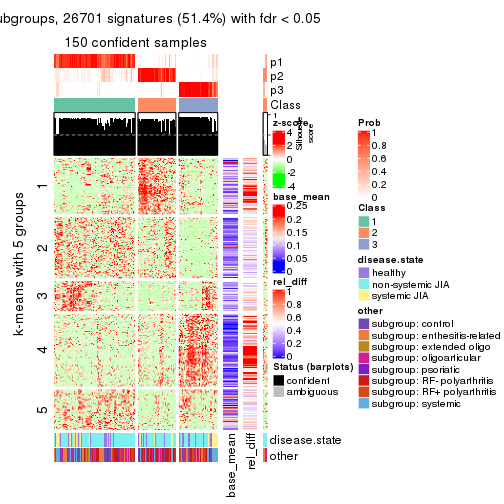
get_signatures(res, k = 4)
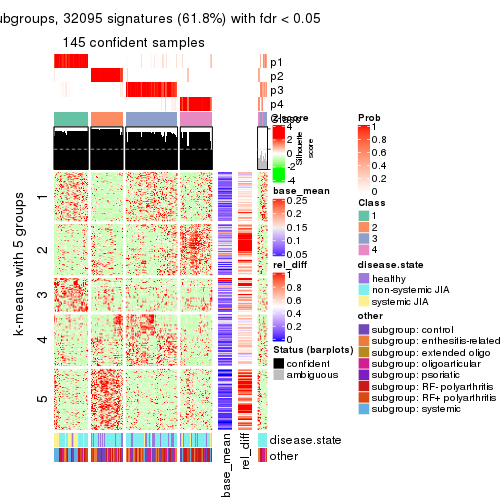
get_signatures(res, k = 5)
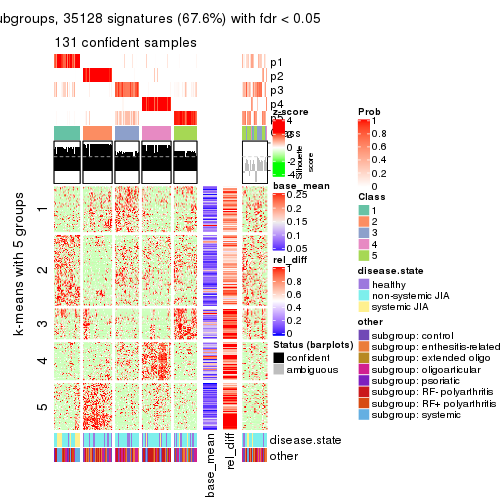
get_signatures(res, k = 6)
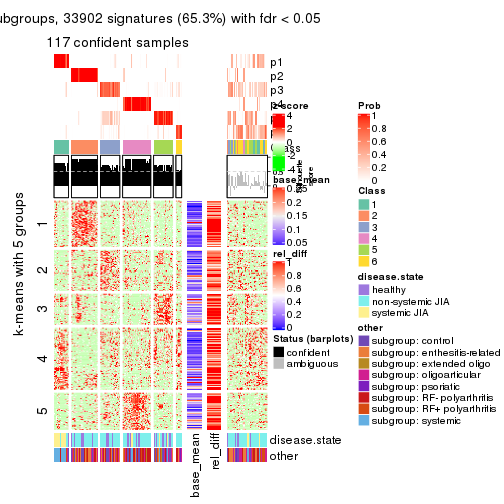
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
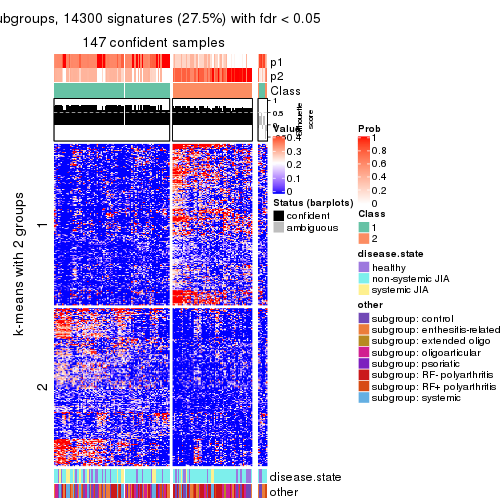
get_signatures(res, k = 3, scale_rows = FALSE)
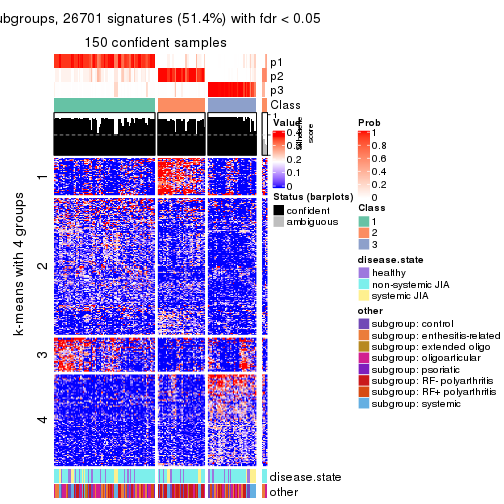
get_signatures(res, k = 4, scale_rows = FALSE)
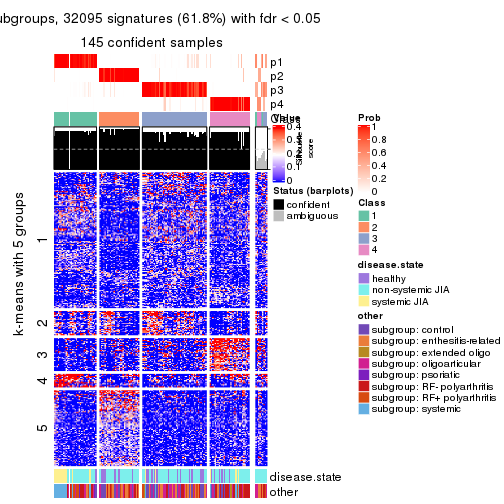
get_signatures(res, k = 5, scale_rows = FALSE)
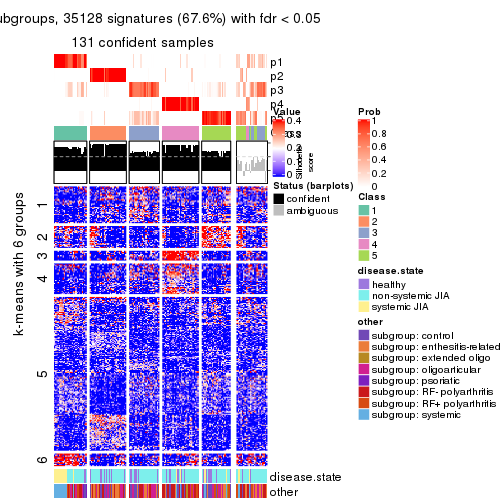
get_signatures(res, k = 6, scale_rows = FALSE)
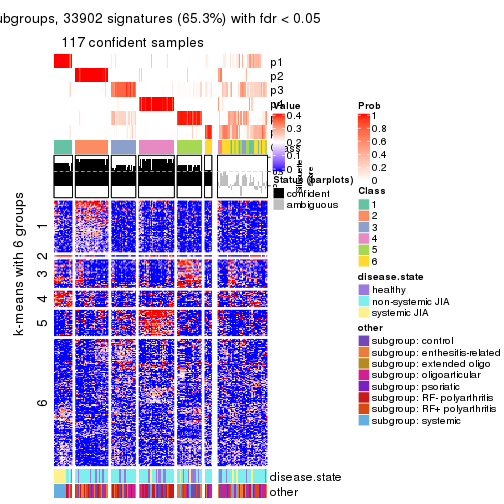
Compare the overlap of signatures from different k:
compare_signatures(res)
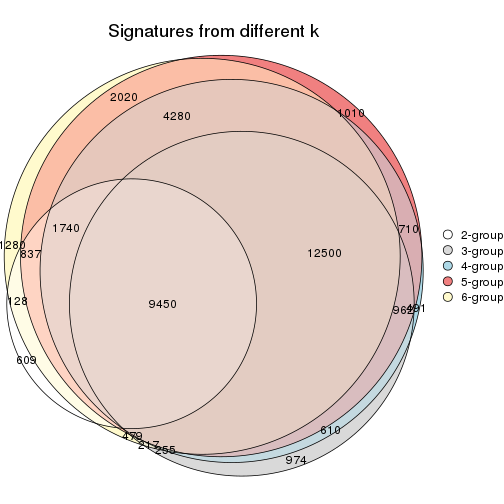
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
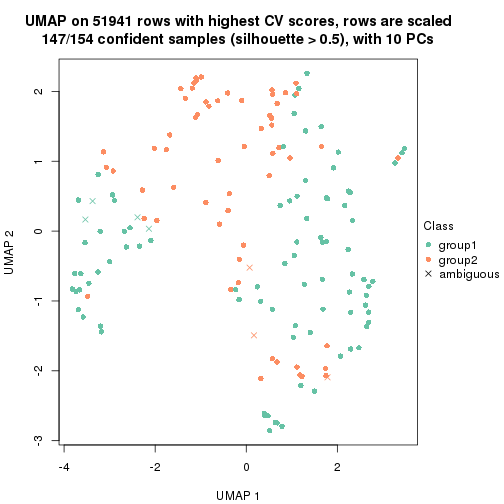
dimension_reduction(res, k = 3, method = "UMAP")
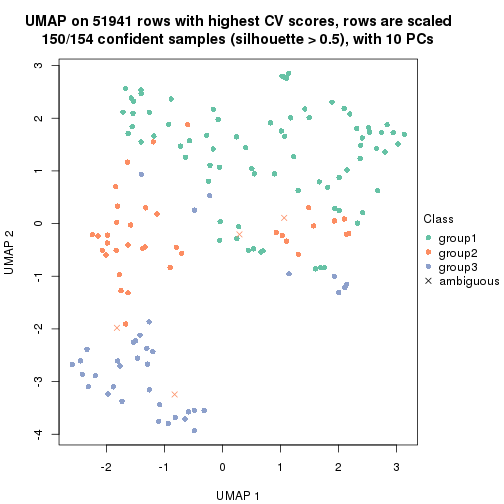
dimension_reduction(res, k = 4, method = "UMAP")
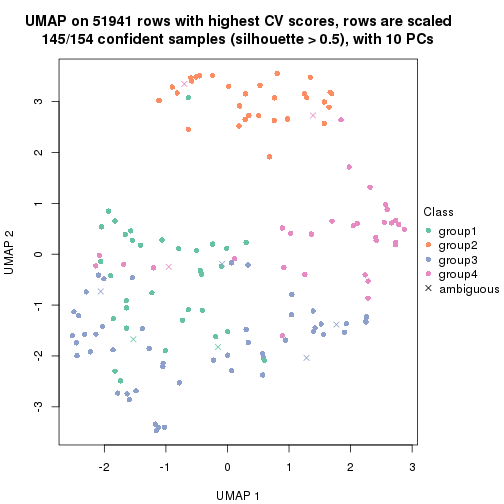
dimension_reduction(res, k = 5, method = "UMAP")
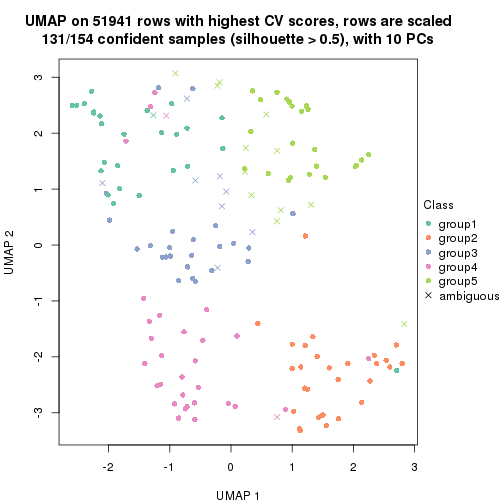
dimension_reduction(res, k = 6, method = "UMAP")
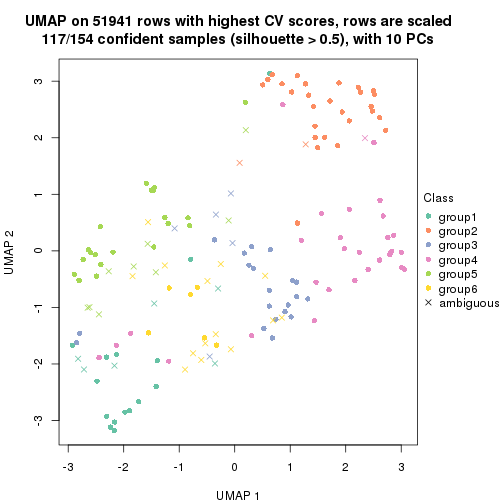
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
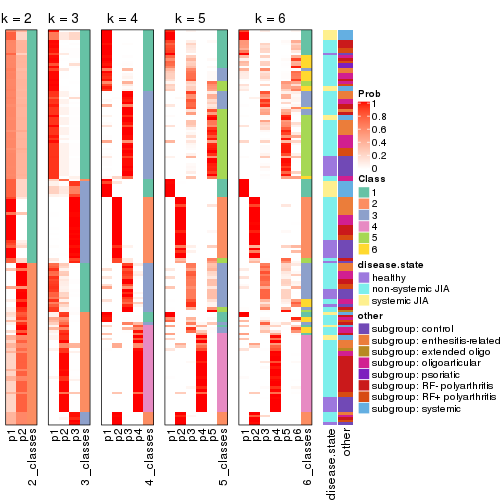
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> CV:skmeans 147 2.29e-01 2.69e-02 2
#> CV:skmeans 150 7.34e-01 3.65e-02 3
#> CV:skmeans 145 7.36e-06 2.64e-07 4
#> CV:skmeans 131 2.44e-05 1.85e-06 5
#> CV:skmeans 117 1.08e-09 8.66e-09 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["CV", "pam"]
# you can also extract it by
# res = res_list["CV:pam"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'CV' method.
#> Subgroups are detected by 'pam' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 5.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
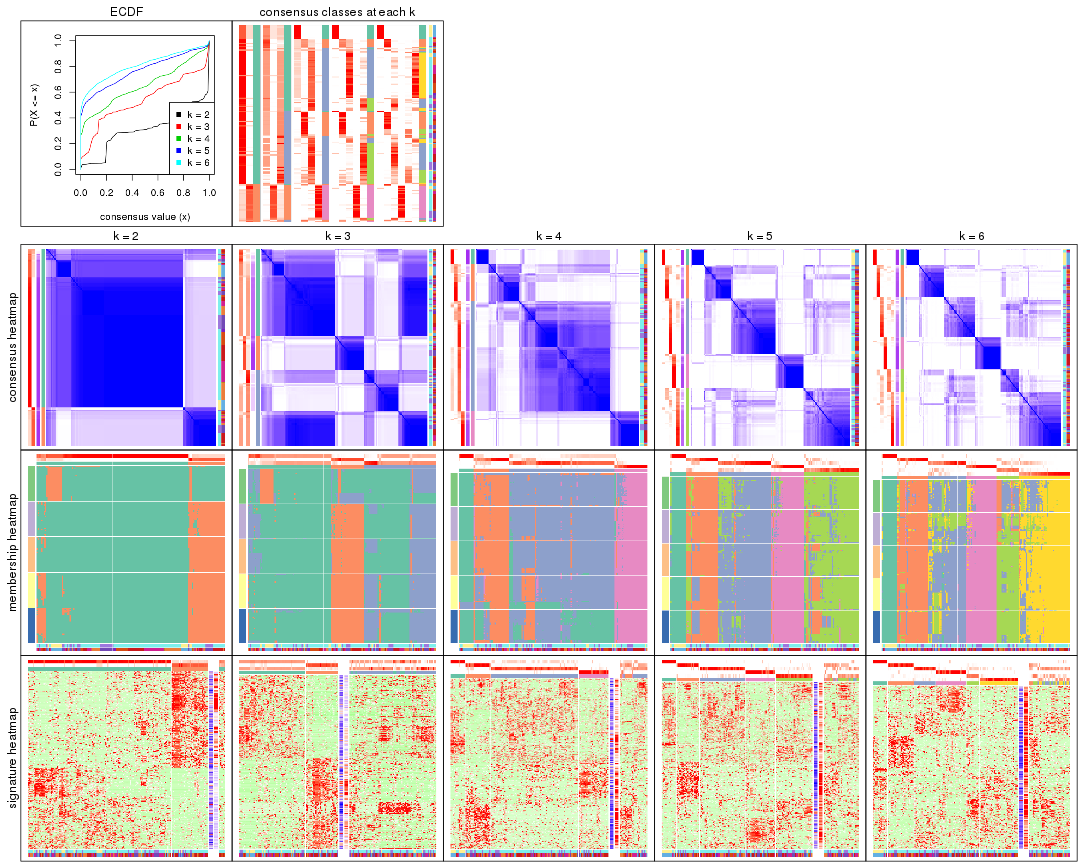
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
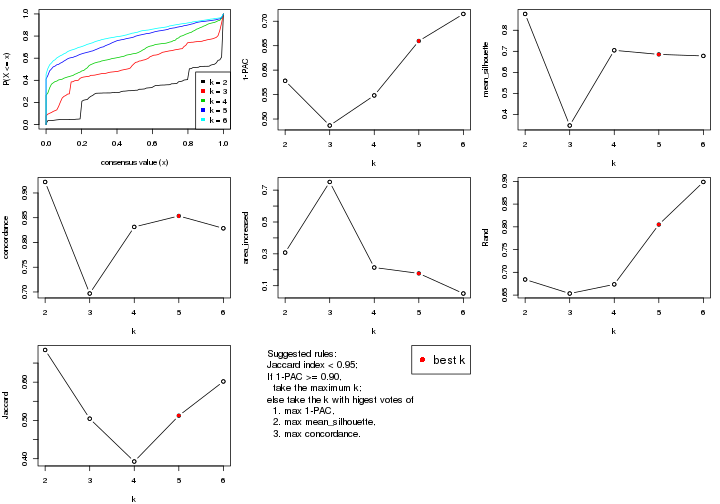
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.578 0.877 0.922 0.308 0.684 0.684
#> 3 3 0.487 0.348 0.697 0.751 0.653 0.505
#> 4 4 0.548 0.705 0.831 0.214 0.674 0.393
#> 5 5 0.660 0.685 0.854 0.177 0.805 0.512
#> 6 6 0.715 0.678 0.829 0.051 0.899 0.602
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 5
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 1 0.7139 0.7352 0.804 0.196
#> GSM340359 1 0.7219 0.7324 0.800 0.200
#> GSM340361 1 0.0000 0.9389 1.000 0.000
#> GSM340362 1 0.0000 0.9389 1.000 0.000
#> GSM340363 1 0.0000 0.9389 1.000 0.000
#> GSM340364 1 0.7139 0.7352 0.804 0.196
#> GSM340365 1 0.0000 0.9389 1.000 0.000
#> GSM340366 1 0.0000 0.9389 1.000 0.000
#> GSM340367 1 0.7139 0.7352 0.804 0.196
#> GSM340368 2 0.0376 0.7613 0.004 0.996
#> GSM340369 2 0.0376 0.7613 0.004 0.996
#> GSM340370 1 0.7139 0.7352 0.804 0.196
#> GSM340371 1 0.0000 0.9389 1.000 0.000
#> GSM340372 1 0.7139 0.7352 0.804 0.196
#> GSM340373 1 0.7139 0.7352 0.804 0.196
#> GSM340375 1 0.7139 0.7352 0.804 0.196
#> GSM340376 1 0.7139 0.7352 0.804 0.196
#> GSM340378 1 0.7139 0.7352 0.804 0.196
#> GSM340243 1 0.0000 0.9389 1.000 0.000
#> GSM340244 1 0.0376 0.9370 0.996 0.004
#> GSM340246 1 0.0376 0.9370 0.996 0.004
#> GSM340247 2 0.7219 0.9545 0.200 0.800
#> GSM340248 1 0.0376 0.9370 0.996 0.004
#> GSM340249 2 0.7139 0.9519 0.196 0.804
#> GSM340250 1 0.1184 0.9268 0.984 0.016
#> GSM340251 2 0.7950 0.9194 0.240 0.760
#> GSM340252 2 0.7139 0.9519 0.196 0.804
#> GSM340253 1 0.0376 0.9370 0.996 0.004
#> GSM340254 1 0.0376 0.9370 0.996 0.004
#> GSM340256 1 0.0376 0.9370 0.996 0.004
#> GSM340258 1 0.0000 0.9389 1.000 0.000
#> GSM340259 1 0.0000 0.9389 1.000 0.000
#> GSM340260 1 0.0000 0.9389 1.000 0.000
#> GSM340261 1 0.0000 0.9389 1.000 0.000
#> GSM340262 1 0.0000 0.9389 1.000 0.000
#> GSM340263 1 0.0376 0.9370 0.996 0.004
#> GSM340264 1 0.0000 0.9389 1.000 0.000
#> GSM340265 1 0.0000 0.9389 1.000 0.000
#> GSM340266 1 0.0000 0.9389 1.000 0.000
#> GSM340267 1 0.0000 0.9389 1.000 0.000
#> GSM340268 1 0.2423 0.9020 0.960 0.040
#> GSM340269 1 0.0000 0.9389 1.000 0.000
#> GSM340270 1 0.0000 0.9389 1.000 0.000
#> GSM537574 1 0.0000 0.9389 1.000 0.000
#> GSM537580 1 0.0000 0.9389 1.000 0.000
#> GSM537581 2 0.8386 0.8894 0.268 0.732
#> GSM340272 1 0.0000 0.9389 1.000 0.000
#> GSM340273 1 0.0376 0.9370 0.996 0.004
#> GSM340275 2 0.7139 0.9519 0.196 0.804
#> GSM340276 1 0.0000 0.9389 1.000 0.000
#> GSM340277 1 0.0376 0.9370 0.996 0.004
#> GSM340278 1 0.1414 0.9222 0.980 0.020
#> GSM340279 1 0.0000 0.9389 1.000 0.000
#> GSM340282 1 0.0000 0.9389 1.000 0.000
#> GSM340284 1 0.8267 0.5364 0.740 0.260
#> GSM340285 1 0.0000 0.9389 1.000 0.000
#> GSM340286 1 0.0000 0.9389 1.000 0.000
#> GSM340287 1 0.0000 0.9389 1.000 0.000
#> GSM340288 1 0.0000 0.9389 1.000 0.000
#> GSM340289 1 0.0000 0.9389 1.000 0.000
#> GSM340290 1 0.8207 0.5382 0.744 0.256
#> GSM340291 1 0.0376 0.9370 0.996 0.004
#> GSM340293 2 0.8081 0.9140 0.248 0.752
#> GSM340294 1 0.0000 0.9389 1.000 0.000
#> GSM340296 1 0.0672 0.9335 0.992 0.008
#> GSM340297 1 0.0000 0.9389 1.000 0.000
#> GSM340298 1 0.0000 0.9389 1.000 0.000
#> GSM340299 1 0.0000 0.9389 1.000 0.000
#> GSM340301 1 0.0000 0.9389 1.000 0.000
#> GSM340303 1 0.0000 0.9389 1.000 0.000
#> GSM340304 1 0.0000 0.9389 1.000 0.000
#> GSM340306 2 0.7950 0.9226 0.240 0.760
#> GSM340307 1 0.0376 0.9370 0.996 0.004
#> GSM340310 1 0.7139 0.7352 0.804 0.196
#> GSM340314 1 0.9686 0.0640 0.604 0.396
#> GSM340315 1 0.0000 0.9389 1.000 0.000
#> GSM340317 1 0.8813 0.4302 0.700 0.300
#> GSM340318 2 0.7219 0.9545 0.200 0.800
#> GSM340319 2 0.7219 0.9545 0.200 0.800
#> GSM340320 1 0.9896 -0.1440 0.560 0.440
#> GSM340321 1 0.0376 0.9370 0.996 0.004
#> GSM340322 2 0.7219 0.9545 0.200 0.800
#> GSM340324 1 0.0672 0.9348 0.992 0.008
#> GSM340328 1 0.7139 0.7352 0.804 0.196
#> GSM340330 1 0.1414 0.9222 0.980 0.020
#> GSM340332 2 0.7219 0.9545 0.200 0.800
#> GSM340333 1 0.0376 0.9370 0.996 0.004
#> GSM340336 2 0.7139 0.9519 0.196 0.804
#> GSM340337 2 0.7219 0.9545 0.200 0.800
#> GSM340338 1 0.0000 0.9389 1.000 0.000
#> GSM340339 2 0.7219 0.9545 0.200 0.800
#> GSM340340 2 0.7219 0.9545 0.200 0.800
#> GSM340341 2 0.8267 0.8966 0.260 0.740
#> GSM340343 2 0.7219 0.9545 0.200 0.800
#> GSM340344 1 0.0000 0.9389 1.000 0.000
#> GSM340346 1 0.0000 0.9389 1.000 0.000
#> GSM340347 2 0.7219 0.9545 0.200 0.800
#> GSM340348 2 0.7453 0.9466 0.212 0.788
#> GSM340349 1 0.0000 0.9389 1.000 0.000
#> GSM340350 1 0.0000 0.9389 1.000 0.000
#> GSM340351 1 0.0000 0.9389 1.000 0.000
#> GSM340354 1 0.0000 0.9389 1.000 0.000
#> GSM340356 1 0.0376 0.9370 0.996 0.004
#> GSM340357 1 0.0000 0.9389 1.000 0.000
#> GSM348183 1 0.0000 0.9389 1.000 0.000
#> GSM348191 1 0.0000 0.9389 1.000 0.000
#> GSM348193 1 0.0000 0.9389 1.000 0.000
#> GSM537578 1 0.0000 0.9389 1.000 0.000
#> GSM348181 1 0.0000 0.9389 1.000 0.000
#> GSM348182 1 0.0000 0.9389 1.000 0.000
#> GSM348184 1 0.9993 -0.3096 0.516 0.484
#> GSM348185 1 0.9661 0.0941 0.608 0.392
#> GSM348186 1 0.0376 0.9370 0.996 0.004
#> GSM348187 1 0.0000 0.9389 1.000 0.000
#> GSM348188 2 0.8499 0.8762 0.276 0.724
#> GSM348189 1 0.0000 0.9389 1.000 0.000
#> GSM348190 1 0.0000 0.9389 1.000 0.000
#> GSM348194 1 0.0000 0.9389 1.000 0.000
#> GSM348195 1 0.0000 0.9389 1.000 0.000
#> GSM348196 1 0.0000 0.9389 1.000 0.000
#> GSM537585 1 0.0000 0.9389 1.000 0.000
#> GSM537594 1 0.0376 0.9370 0.996 0.004
#> GSM537596 1 0.0000 0.9389 1.000 0.000
#> GSM537597 1 0.0000 0.9389 1.000 0.000
#> GSM537602 1 0.0000 0.9389 1.000 0.000
#> GSM340184 1 0.0000 0.9389 1.000 0.000
#> GSM340185 1 0.6712 0.7144 0.824 0.176
#> GSM340186 1 0.4022 0.8594 0.920 0.080
#> GSM340187 2 0.7219 0.9545 0.200 0.800
#> GSM340189 2 0.7219 0.9545 0.200 0.800
#> GSM340190 2 0.9833 0.5752 0.424 0.576
#> GSM340191 1 0.0672 0.9329 0.992 0.008
#> GSM340192 1 0.0000 0.9389 1.000 0.000
#> GSM340193 1 0.0000 0.9389 1.000 0.000
#> GSM340194 1 0.0000 0.9389 1.000 0.000
#> GSM340195 1 0.0000 0.9389 1.000 0.000
#> GSM340196 2 0.7219 0.9545 0.200 0.800
#> GSM340197 1 0.0000 0.9389 1.000 0.000
#> GSM340198 1 0.0376 0.9370 0.996 0.004
#> GSM340199 1 0.2778 0.8933 0.952 0.048
#> GSM340200 1 0.0376 0.9370 0.996 0.004
#> GSM340201 2 0.7219 0.9545 0.200 0.800
#> GSM340202 2 0.7219 0.9545 0.200 0.800
#> GSM340203 2 0.7219 0.9545 0.200 0.800
#> GSM340204 1 0.0000 0.9389 1.000 0.000
#> GSM340205 1 0.2043 0.9121 0.968 0.032
#> GSM340206 1 0.2236 0.9062 0.964 0.036
#> GSM340207 1 0.0000 0.9389 1.000 0.000
#> GSM340237 1 0.5842 0.8018 0.860 0.140
#> GSM340238 1 0.7815 0.5961 0.768 0.232
#> GSM340239 1 0.0000 0.9389 1.000 0.000
#> GSM340240 1 0.0000 0.9389 1.000 0.000
#> GSM340241 1 0.0376 0.9370 0.996 0.004
#> GSM340242 1 0.0000 0.9389 1.000 0.000
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.8599 0.5620 0.584 0.140 0.276
#> GSM340359 3 0.8969 0.1292 0.348 0.140 0.512
#> GSM340361 1 0.6299 0.7323 0.524 0.000 0.476
#> GSM340362 3 0.0592 0.1984 0.012 0.000 0.988
#> GSM340363 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM340364 1 0.7797 0.4586 0.672 0.140 0.188
#> GSM340365 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM340366 1 0.6309 0.7124 0.500 0.000 0.500
#> GSM340367 1 0.8599 0.5620 0.584 0.140 0.276
#> GSM340368 2 0.0000 0.7929 0.000 1.000 0.000
#> GSM340369 2 0.2165 0.7649 0.064 0.936 0.000
#> GSM340370 1 0.8599 0.5620 0.584 0.140 0.276
#> GSM340371 3 0.0000 0.2007 0.000 0.000 1.000
#> GSM340372 1 0.8599 0.5620 0.584 0.140 0.276
#> GSM340373 1 0.8599 0.5620 0.584 0.140 0.276
#> GSM340375 1 0.8599 0.5620 0.584 0.140 0.276
#> GSM340376 1 0.8599 0.5620 0.584 0.140 0.276
#> GSM340378 1 0.7932 0.4746 0.660 0.140 0.200
#> GSM340243 1 0.6305 0.7313 0.516 0.000 0.484
#> GSM340244 1 0.4842 -0.1086 0.776 0.000 0.224
#> GSM340246 3 0.6180 0.2538 0.416 0.000 0.584
#> GSM340247 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340248 1 0.2625 0.1736 0.916 0.000 0.084
#> GSM340249 2 0.6081 0.6110 0.344 0.652 0.004
#> GSM340250 1 0.6286 0.7205 0.536 0.000 0.464
#> GSM340251 1 0.9914 -0.4454 0.392 0.328 0.280
#> GSM340252 3 0.8731 -0.2387 0.120 0.352 0.528
#> GSM340253 3 0.6168 0.2581 0.412 0.000 0.588
#> GSM340254 3 0.6180 0.2538 0.416 0.000 0.584
#> GSM340256 3 0.6192 0.2539 0.420 0.000 0.580
#> GSM340258 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM340259 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM340260 1 0.6308 0.7220 0.508 0.000 0.492
#> GSM340261 1 0.6309 0.7200 0.504 0.000 0.496
#> GSM340262 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM340263 1 0.3619 0.2741 0.864 0.000 0.136
#> GSM340264 3 0.6309 -0.7214 0.500 0.000 0.500
#> GSM340265 3 0.6126 -0.5877 0.400 0.000 0.600
#> GSM340266 1 0.6308 0.7249 0.508 0.000 0.492
#> GSM340267 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM340268 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM340269 1 0.6309 0.7200 0.504 0.000 0.496
#> GSM340270 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM537574 3 0.6307 -0.6954 0.488 0.000 0.512
#> GSM537580 3 0.6307 -0.7114 0.488 0.000 0.512
#> GSM537581 2 0.6096 0.8093 0.040 0.752 0.208
#> GSM340272 1 0.6308 0.7220 0.508 0.000 0.492
#> GSM340273 1 0.2165 0.1665 0.936 0.000 0.064
#> GSM340275 2 0.4556 0.8778 0.060 0.860 0.080
#> GSM340276 1 0.6309 0.7188 0.504 0.000 0.496
#> GSM340277 3 0.6180 0.2538 0.416 0.000 0.584
#> GSM340278 3 0.6309 -0.7214 0.500 0.000 0.500
#> GSM340279 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM340282 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM340284 1 0.5455 0.2867 0.776 0.020 0.204
#> GSM340285 1 0.6307 0.7283 0.512 0.000 0.488
#> GSM340286 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM340287 1 0.6308 0.7249 0.508 0.000 0.492
#> GSM340288 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM340289 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM340290 3 0.9378 -0.2357 0.184 0.336 0.480
#> GSM340291 3 0.6225 0.2505 0.432 0.000 0.568
#> GSM340293 2 0.6388 0.7142 0.024 0.692 0.284
#> GSM340294 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM340296 1 0.6682 0.7131 0.504 0.008 0.488
#> GSM340297 1 0.6308 0.7249 0.508 0.000 0.492
#> GSM340298 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM340299 3 0.0592 0.1984 0.012 0.000 0.988
#> GSM340301 3 0.2796 0.0570 0.092 0.000 0.908
#> GSM340303 3 0.2165 0.1151 0.064 0.000 0.936
#> GSM340304 1 0.6307 0.7283 0.512 0.000 0.488
#> GSM340306 2 0.4291 0.9203 0.008 0.840 0.152
#> GSM340307 3 0.6180 0.2538 0.416 0.000 0.584
#> GSM340310 1 0.8599 0.5620 0.584 0.140 0.276
#> GSM340314 3 0.9291 -0.2177 0.168 0.356 0.476
#> GSM340315 1 0.6308 0.7249 0.508 0.000 0.492
#> GSM340317 1 0.3272 0.0931 0.904 0.080 0.016
#> GSM340318 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340319 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340320 1 0.9717 0.4635 0.392 0.220 0.388
#> GSM340321 3 0.6180 0.2538 0.416 0.000 0.584
#> GSM340322 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340324 3 0.6244 0.2480 0.440 0.000 0.560
#> GSM340328 1 0.8599 0.5620 0.584 0.140 0.276
#> GSM340330 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM340332 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340333 3 0.6168 0.2581 0.412 0.000 0.588
#> GSM340336 2 0.4209 0.9202 0.020 0.860 0.120
#> GSM340337 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340338 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM340339 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340340 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340341 2 0.6458 0.8257 0.072 0.752 0.176
#> GSM340343 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340344 3 0.1411 0.2154 0.036 0.000 0.964
#> GSM340346 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM340347 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340348 2 0.4629 0.8869 0.004 0.808 0.188
#> GSM340349 1 0.6307 0.7282 0.512 0.000 0.488
#> GSM340350 1 0.6308 0.7220 0.508 0.000 0.492
#> GSM340351 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM340354 3 0.6308 -0.7142 0.492 0.000 0.508
#> GSM340356 1 0.3686 0.0452 0.860 0.000 0.140
#> GSM340357 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM348183 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM348191 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM348193 1 0.8018 0.6671 0.520 0.064 0.416
#> GSM537578 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM348181 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM348182 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM348184 3 0.6180 0.2538 0.416 0.000 0.584
#> GSM348185 1 0.7447 0.2160 0.696 0.120 0.184
#> GSM348186 3 0.6192 0.2539 0.420 0.000 0.580
#> GSM348187 1 0.6299 0.7323 0.524 0.000 0.476
#> GSM348188 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM348189 3 0.5016 -0.2721 0.240 0.000 0.760
#> GSM348190 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM348194 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM348195 1 0.6299 0.7323 0.524 0.000 0.476
#> GSM348196 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM537585 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM537594 3 0.6244 0.2480 0.440 0.000 0.560
#> GSM537596 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM537597 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM537602 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM340184 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM340185 3 0.6180 0.2538 0.416 0.000 0.584
#> GSM340186 3 0.6180 0.2538 0.416 0.000 0.584
#> GSM340187 2 0.3965 0.9305 0.008 0.860 0.132
#> GSM340189 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340190 3 0.7156 0.2287 0.400 0.028 0.572
#> GSM340191 3 0.6309 -0.7161 0.496 0.000 0.504
#> GSM340192 3 0.5216 0.2529 0.260 0.000 0.740
#> GSM340193 1 0.6302 0.7337 0.520 0.000 0.480
#> GSM340194 3 0.0237 0.2001 0.004 0.000 0.996
#> GSM340195 3 0.2165 0.1371 0.064 0.000 0.936
#> GSM340196 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340197 1 0.6305 0.7309 0.516 0.000 0.484
#> GSM340198 3 0.6192 0.2539 0.420 0.000 0.580
#> GSM340199 3 0.6521 -0.7164 0.496 0.004 0.500
#> GSM340200 3 0.6180 0.2538 0.416 0.000 0.584
#> GSM340201 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340202 2 0.3686 0.9363 0.000 0.860 0.140
#> GSM340203 2 0.4399 0.8882 0.000 0.812 0.188
#> GSM340204 3 0.1031 0.2076 0.024 0.000 0.976
#> GSM340205 1 0.6495 0.7215 0.536 0.004 0.460
#> GSM340206 1 0.6307 0.7022 0.512 0.000 0.488
#> GSM340207 3 0.6126 -0.5859 0.400 0.000 0.600
#> GSM340237 1 0.4235 -0.0594 0.824 0.000 0.176
#> GSM340238 1 0.6955 0.7025 0.496 0.016 0.488
#> GSM340239 1 0.6307 0.7286 0.512 0.000 0.488
#> GSM340240 3 0.0000 0.2007 0.000 0.000 1.000
#> GSM340241 3 0.6180 0.2538 0.416 0.000 0.584
#> GSM340242 3 0.6309 -0.7161 0.496 0.000 0.504
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.0000 0.96569 1.000 0.000 0.000 0.000
#> GSM340359 1 0.3801 0.57893 0.780 0.220 0.000 0.000
#> GSM340361 3 0.4134 0.76573 0.260 0.000 0.740 0.000
#> GSM340362 3 0.5581 0.36596 0.020 0.448 0.532 0.000
#> GSM340363 3 0.3975 0.77580 0.240 0.000 0.760 0.000
#> GSM340364 1 0.0000 0.96569 1.000 0.000 0.000 0.000
#> GSM340365 3 0.4134 0.76573 0.260 0.000 0.740 0.000
#> GSM340366 3 0.5250 0.45021 0.316 0.024 0.660 0.000
#> GSM340367 1 0.0000 0.96569 1.000 0.000 0.000 0.000
#> GSM340368 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340369 4 0.4454 0.57122 0.308 0.000 0.000 0.692
#> GSM340370 1 0.0000 0.96569 1.000 0.000 0.000 0.000
#> GSM340371 3 0.4477 0.49606 0.000 0.312 0.688 0.000
#> GSM340372 1 0.0000 0.96569 1.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.96569 1.000 0.000 0.000 0.000
#> GSM340375 1 0.0000 0.96569 1.000 0.000 0.000 0.000
#> GSM340376 1 0.0000 0.96569 1.000 0.000 0.000 0.000
#> GSM340378 1 0.0000 0.96569 1.000 0.000 0.000 0.000
#> GSM340243 3 0.3668 0.78641 0.188 0.004 0.808 0.000
#> GSM340244 2 0.6404 0.43394 0.220 0.644 0.136 0.000
#> GSM340246 2 0.0592 0.73470 0.000 0.984 0.016 0.000
#> GSM340247 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340248 2 0.7362 0.29772 0.220 0.524 0.256 0.000
#> GSM340249 4 0.4655 0.47381 0.000 0.312 0.004 0.684
#> GSM340250 3 0.2814 0.78729 0.132 0.000 0.868 0.000
#> GSM340251 2 0.6672 0.52464 0.000 0.620 0.212 0.168
#> GSM340252 2 0.5383 0.00961 0.000 0.536 0.012 0.452
#> GSM340253 2 0.0707 0.72800 0.020 0.980 0.000 0.000
#> GSM340254 2 0.0000 0.73834 0.000 1.000 0.000 0.000
#> GSM340256 2 0.0000 0.73834 0.000 1.000 0.000 0.000
#> GSM340258 3 0.3015 0.78688 0.092 0.024 0.884 0.000
#> GSM340259 3 0.4535 0.78045 0.240 0.016 0.744 0.000
#> GSM340260 3 0.3205 0.78886 0.104 0.024 0.872 0.000
#> GSM340261 3 0.2408 0.79035 0.104 0.000 0.896 0.000
#> GSM340262 3 0.2909 0.78764 0.092 0.020 0.888 0.000
#> GSM340263 2 0.7647 0.14595 0.220 0.444 0.336 0.000
#> GSM340264 3 0.4574 0.78823 0.220 0.024 0.756 0.000
#> GSM340265 3 0.1867 0.73264 0.000 0.072 0.928 0.000
#> GSM340266 3 0.4436 0.78850 0.216 0.020 0.764 0.000
#> GSM340267 3 0.3401 0.79516 0.152 0.008 0.840 0.000
#> GSM340268 3 0.0817 0.76184 0.000 0.024 0.976 0.000
#> GSM340269 3 0.3790 0.79477 0.164 0.016 0.820 0.000
#> GSM340270 3 0.4155 0.77719 0.240 0.004 0.756 0.000
#> GSM537574 3 0.4679 0.79276 0.184 0.044 0.772 0.000
#> GSM537580 3 0.2949 0.78644 0.088 0.024 0.888 0.000
#> GSM537581 4 0.4040 0.60662 0.000 0.000 0.248 0.752
#> GSM340272 3 0.1520 0.76813 0.020 0.024 0.956 0.000
#> GSM340273 2 0.7200 0.32914 0.220 0.552 0.228 0.000
#> GSM340275 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340276 3 0.4542 0.78574 0.228 0.020 0.752 0.000
#> GSM340277 2 0.0000 0.73834 0.000 1.000 0.000 0.000
#> GSM340278 3 0.3552 0.79049 0.128 0.024 0.848 0.000
#> GSM340279 3 0.0817 0.76184 0.000 0.024 0.976 0.000
#> GSM340282 3 0.0817 0.76184 0.000 0.024 0.976 0.000
#> GSM340284 3 0.5372 -0.19273 0.000 0.444 0.544 0.012
#> GSM340285 3 0.3074 0.78665 0.152 0.000 0.848 0.000
#> GSM340286 3 0.4642 0.78171 0.240 0.020 0.740 0.000
#> GSM340287 3 0.3801 0.78091 0.220 0.000 0.780 0.000
#> GSM340288 3 0.4072 0.77030 0.252 0.000 0.748 0.000
#> GSM340289 3 0.3975 0.77580 0.240 0.000 0.760 0.000
#> GSM340290 3 0.3552 0.66638 0.000 0.024 0.848 0.128
#> GSM340291 2 0.0000 0.73834 0.000 1.000 0.000 0.000
#> GSM340293 4 0.5650 0.31480 0.000 0.024 0.432 0.544
#> GSM340294 3 0.0592 0.76368 0.000 0.016 0.984 0.000
#> GSM340296 3 0.5695 -0.18217 0.476 0.024 0.500 0.000
#> GSM340297 3 0.0707 0.77274 0.020 0.000 0.980 0.000
#> GSM340298 3 0.5560 0.11751 0.392 0.024 0.584 0.000
#> GSM340299 3 0.5581 0.36596 0.020 0.448 0.532 0.000
#> GSM340301 3 0.4677 0.52673 0.004 0.316 0.680 0.000
#> GSM340303 3 0.5476 0.44667 0.020 0.396 0.584 0.000
#> GSM340304 3 0.4088 0.78048 0.232 0.004 0.764 0.000
#> GSM340306 4 0.2530 0.79779 0.000 0.000 0.112 0.888
#> GSM340307 2 0.4040 0.62263 0.000 0.752 0.248 0.000
#> GSM340310 1 0.0336 0.95355 0.992 0.000 0.008 0.000
#> GSM340314 3 0.3606 0.66424 0.000 0.024 0.844 0.132
#> GSM340315 3 0.3801 0.78091 0.220 0.000 0.780 0.000
#> GSM340317 2 0.8183 0.33733 0.216 0.540 0.192 0.052
#> GSM340318 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340319 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340320 3 0.6626 0.61700 0.160 0.000 0.624 0.216
#> GSM340321 2 0.0000 0.73834 0.000 1.000 0.000 0.000
#> GSM340322 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340324 2 0.0592 0.72752 0.000 0.984 0.016 0.000
#> GSM340328 1 0.0000 0.96569 1.000 0.000 0.000 0.000
#> GSM340330 3 0.0817 0.76184 0.000 0.024 0.976 0.000
#> GSM340332 4 0.1716 0.83930 0.000 0.000 0.064 0.936
#> GSM340333 2 0.0707 0.72800 0.020 0.980 0.000 0.000
#> GSM340336 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340337 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340338 3 0.0817 0.76184 0.000 0.024 0.976 0.000
#> GSM340339 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340340 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340341 4 0.5091 0.65930 0.000 0.068 0.180 0.752
#> GSM340343 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340344 3 0.5607 0.26156 0.020 0.488 0.492 0.000
#> GSM340346 3 0.1004 0.76068 0.004 0.024 0.972 0.000
#> GSM340347 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340348 4 0.3528 0.69964 0.000 0.000 0.192 0.808
#> GSM340349 3 0.3172 0.78122 0.160 0.000 0.840 0.000
#> GSM340350 3 0.3443 0.78673 0.136 0.016 0.848 0.000
#> GSM340351 3 0.3975 0.77580 0.240 0.000 0.760 0.000
#> GSM340354 3 0.0817 0.76184 0.000 0.024 0.976 0.000
#> GSM340356 2 0.6973 0.37047 0.220 0.584 0.196 0.000
#> GSM340357 3 0.3975 0.77580 0.240 0.000 0.760 0.000
#> GSM348183 3 0.3975 0.77580 0.240 0.000 0.760 0.000
#> GSM348191 3 0.0817 0.76184 0.000 0.024 0.976 0.000
#> GSM348193 3 0.5881 0.71215 0.240 0.000 0.676 0.084
#> GSM537578 3 0.2737 0.78770 0.104 0.008 0.888 0.000
#> GSM348181 3 0.3975 0.77580 0.240 0.000 0.760 0.000
#> GSM348182 3 0.4642 0.78121 0.240 0.020 0.740 0.000
#> GSM348184 2 0.3649 0.64615 0.000 0.796 0.204 0.000
#> GSM348185 3 0.9248 -0.02085 0.192 0.344 0.364 0.100
#> GSM348186 2 0.0000 0.73834 0.000 1.000 0.000 0.000
#> GSM348187 3 0.4134 0.76573 0.260 0.000 0.740 0.000
#> GSM348188 4 0.1302 0.85637 0.000 0.000 0.044 0.956
#> GSM348189 3 0.4019 0.69224 0.012 0.196 0.792 0.000
#> GSM348190 3 0.4542 0.78615 0.228 0.020 0.752 0.000
#> GSM348194 3 0.3975 0.77580 0.240 0.000 0.760 0.000
#> GSM348195 3 0.4134 0.76573 0.260 0.000 0.740 0.000
#> GSM348196 3 0.4134 0.76573 0.260 0.000 0.740 0.000
#> GSM537585 3 0.3975 0.77580 0.240 0.000 0.760 0.000
#> GSM537594 2 0.0188 0.73625 0.000 0.996 0.004 0.000
#> GSM537596 3 0.3975 0.77580 0.240 0.000 0.760 0.000
#> GSM537597 3 0.3975 0.77580 0.240 0.000 0.760 0.000
#> GSM537602 3 0.3975 0.77580 0.240 0.000 0.760 0.000
#> GSM340184 3 0.0817 0.76184 0.000 0.024 0.976 0.000
#> GSM340185 2 0.0000 0.73834 0.000 1.000 0.000 0.000
#> GSM340186 2 0.0188 0.73789 0.000 0.996 0.004 0.000
#> GSM340187 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340189 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340190 2 0.4993 0.60140 0.000 0.712 0.260 0.028
#> GSM340191 3 0.1151 0.76575 0.008 0.024 0.968 0.000
#> GSM340192 2 0.4103 0.51482 0.000 0.744 0.256 0.000
#> GSM340193 3 0.4535 0.78066 0.240 0.016 0.744 0.000
#> GSM340194 3 0.5132 0.36420 0.004 0.448 0.548 0.000
#> GSM340195 3 0.5564 0.39253 0.020 0.436 0.544 0.000
#> GSM340196 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340197 3 0.4163 0.79363 0.188 0.020 0.792 0.000
#> GSM340198 2 0.0000 0.73834 0.000 1.000 0.000 0.000
#> GSM340199 3 0.0817 0.76184 0.000 0.024 0.976 0.000
#> GSM340200 2 0.4123 0.64035 0.008 0.772 0.220 0.000
#> GSM340201 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340202 4 0.0000 0.88614 0.000 0.000 0.000 1.000
#> GSM340203 4 0.3597 0.73413 0.000 0.016 0.148 0.836
#> GSM340204 3 0.5600 0.31684 0.020 0.468 0.512 0.000
#> GSM340205 3 0.4008 0.77413 0.244 0.000 0.756 0.000
#> GSM340206 3 0.0817 0.76184 0.000 0.024 0.976 0.000
#> GSM340207 3 0.1022 0.75819 0.000 0.032 0.968 0.000
#> GSM340237 2 0.6651 0.40454 0.236 0.616 0.148 0.000
#> GSM340238 3 0.0817 0.76184 0.000 0.024 0.976 0.000
#> GSM340239 3 0.3266 0.78965 0.108 0.024 0.868 0.000
#> GSM340240 3 0.3907 0.54674 0.000 0.232 0.768 0.000
#> GSM340241 2 0.0000 0.73834 0.000 1.000 0.000 0.000
#> GSM340242 3 0.3325 0.79123 0.112 0.024 0.864 0.000
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.0000 0.94794 1.000 0.000 0.000 0.000 0.000
#> GSM340359 1 0.0000 0.94794 1.000 0.000 0.000 0.000 0.000
#> GSM340361 3 0.0000 0.84224 0.000 0.000 1.000 0.000 0.000
#> GSM340362 5 0.4908 0.46035 0.000 0.356 0.036 0.000 0.608
#> GSM340363 3 0.0000 0.84224 0.000 0.000 1.000 0.000 0.000
#> GSM340364 1 0.0000 0.94794 1.000 0.000 0.000 0.000 0.000
#> GSM340365 3 0.0162 0.84229 0.000 0.000 0.996 0.000 0.004
#> GSM340366 1 0.6451 -0.04963 0.452 0.000 0.184 0.000 0.364
#> GSM340367 1 0.0000 0.94794 1.000 0.000 0.000 0.000 0.000
#> GSM340368 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340369 4 0.3774 0.59470 0.296 0.000 0.000 0.704 0.000
#> GSM340370 1 0.0000 0.94794 1.000 0.000 0.000 0.000 0.000
#> GSM340371 5 0.1671 0.73113 0.000 0.076 0.000 0.000 0.924
#> GSM340372 1 0.0000 0.94794 1.000 0.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.94794 1.000 0.000 0.000 0.000 0.000
#> GSM340375 1 0.0000 0.94794 1.000 0.000 0.000 0.000 0.000
#> GSM340376 1 0.0000 0.94794 1.000 0.000 0.000 0.000 0.000
#> GSM340378 1 0.0000 0.94794 1.000 0.000 0.000 0.000 0.000
#> GSM340243 3 0.3800 0.75368 0.080 0.000 0.812 0.000 0.108
#> GSM340244 2 0.3661 0.58355 0.000 0.724 0.276 0.000 0.000
#> GSM340246 2 0.0404 0.76631 0.000 0.988 0.000 0.000 0.012
#> GSM340247 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340248 2 0.5645 0.32515 0.000 0.540 0.376 0.000 0.084
#> GSM340249 4 0.4768 0.29340 0.000 0.384 0.000 0.592 0.024
#> GSM340250 5 0.4440 0.18909 0.004 0.000 0.468 0.000 0.528
#> GSM340251 2 0.4794 0.44307 0.000 0.624 0.000 0.032 0.344
#> GSM340252 4 0.4906 0.16690 0.000 0.480 0.000 0.496 0.024
#> GSM340253 2 0.1012 0.75643 0.000 0.968 0.012 0.000 0.020
#> GSM340254 2 0.0609 0.76136 0.000 0.980 0.000 0.000 0.020
#> GSM340256 2 0.0000 0.76938 0.000 1.000 0.000 0.000 0.000
#> GSM340258 5 0.2280 0.73516 0.000 0.000 0.120 0.000 0.880
#> GSM340259 3 0.2561 0.77372 0.000 0.000 0.856 0.000 0.144
#> GSM340260 5 0.3160 0.69868 0.004 0.000 0.188 0.000 0.808
#> GSM340261 3 0.2813 0.74746 0.000 0.000 0.832 0.000 0.168
#> GSM340262 5 0.3109 0.68702 0.000 0.000 0.200 0.000 0.800
#> GSM340263 3 0.4517 0.09329 0.000 0.436 0.556 0.000 0.008
#> GSM340264 3 0.4045 0.44044 0.000 0.000 0.644 0.000 0.356
#> GSM340265 5 0.0404 0.75805 0.000 0.000 0.012 0.000 0.988
#> GSM340266 3 0.3857 0.56602 0.000 0.000 0.688 0.000 0.312
#> GSM340267 3 0.3586 0.60853 0.000 0.000 0.736 0.000 0.264
#> GSM340268 5 0.0609 0.75979 0.000 0.000 0.020 0.000 0.980
#> GSM340269 3 0.3336 0.70285 0.000 0.000 0.772 0.000 0.228
#> GSM340270 3 0.1544 0.82452 0.000 0.000 0.932 0.000 0.068
#> GSM537574 5 0.4451 -0.07058 0.000 0.004 0.492 0.000 0.504
#> GSM537580 5 0.2389 0.73840 0.000 0.004 0.116 0.000 0.880
#> GSM537581 4 0.4067 0.60558 0.000 0.000 0.300 0.692 0.008
#> GSM340272 5 0.3730 0.55542 0.000 0.000 0.288 0.000 0.712
#> GSM340273 2 0.4074 0.44256 0.000 0.636 0.364 0.000 0.000
#> GSM340275 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340276 3 0.3534 0.64736 0.000 0.000 0.744 0.000 0.256
#> GSM340277 2 0.0000 0.76938 0.000 1.000 0.000 0.000 0.000
#> GSM340278 3 0.4294 0.21154 0.000 0.000 0.532 0.000 0.468
#> GSM340279 5 0.2732 0.70498 0.000 0.000 0.160 0.000 0.840
#> GSM340282 5 0.2127 0.74567 0.000 0.000 0.108 0.000 0.892
#> GSM340284 5 0.4126 0.24109 0.000 0.380 0.000 0.000 0.620
#> GSM340285 3 0.2648 0.77450 0.000 0.000 0.848 0.000 0.152
#> GSM340286 3 0.3395 0.68461 0.000 0.000 0.764 0.000 0.236
#> GSM340287 3 0.0510 0.83986 0.000 0.000 0.984 0.000 0.016
#> GSM340288 3 0.0000 0.84224 0.000 0.000 1.000 0.000 0.000
#> GSM340289 3 0.0162 0.84229 0.000 0.000 0.996 0.000 0.004
#> GSM340290 5 0.0693 0.75798 0.000 0.000 0.012 0.008 0.980
#> GSM340291 2 0.0000 0.76938 0.000 1.000 0.000 0.000 0.000
#> GSM340293 5 0.3274 0.62280 0.000 0.000 0.000 0.220 0.780
#> GSM340294 5 0.2852 0.69339 0.000 0.000 0.172 0.000 0.828
#> GSM340296 5 0.4758 0.50930 0.276 0.000 0.048 0.000 0.676
#> GSM340297 5 0.4045 0.41822 0.000 0.000 0.356 0.000 0.644
#> GSM340298 5 0.5188 0.41403 0.328 0.000 0.060 0.000 0.612
#> GSM340299 5 0.5783 0.40605 0.000 0.360 0.100 0.000 0.540
#> GSM340301 5 0.3327 0.69259 0.000 0.144 0.028 0.000 0.828
#> GSM340303 5 0.6275 0.42202 0.000 0.308 0.176 0.000 0.516
#> GSM340304 3 0.1671 0.81968 0.000 0.000 0.924 0.000 0.076
#> GSM340306 4 0.2690 0.78034 0.000 0.000 0.156 0.844 0.000
#> GSM340307 2 0.4235 0.31549 0.000 0.576 0.000 0.000 0.424
#> GSM340310 1 0.0162 0.94335 0.996 0.000 0.004 0.000 0.000
#> GSM340314 5 0.0807 0.75768 0.000 0.000 0.012 0.012 0.976
#> GSM340315 3 0.0510 0.83946 0.000 0.000 0.984 0.000 0.016
#> GSM340317 2 0.5071 0.45793 0.000 0.616 0.340 0.040 0.004
#> GSM340318 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340319 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340320 3 0.2329 0.76215 0.000 0.000 0.876 0.124 0.000
#> GSM340321 2 0.0000 0.76938 0.000 1.000 0.000 0.000 0.000
#> GSM340322 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340324 2 0.0000 0.76938 0.000 1.000 0.000 0.000 0.000
#> GSM340328 1 0.0000 0.94794 1.000 0.000 0.000 0.000 0.000
#> GSM340330 5 0.0609 0.75979 0.000 0.000 0.020 0.000 0.980
#> GSM340332 4 0.1270 0.86096 0.000 0.000 0.000 0.948 0.052
#> GSM340333 2 0.1106 0.75452 0.000 0.964 0.012 0.000 0.024
#> GSM340336 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340337 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340338 5 0.0404 0.75810 0.000 0.000 0.012 0.000 0.988
#> GSM340339 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340340 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340341 4 0.5307 0.70438 0.000 0.052 0.096 0.736 0.116
#> GSM340343 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340344 5 0.6118 0.29853 0.000 0.404 0.128 0.000 0.468
#> GSM340346 5 0.0609 0.75979 0.000 0.000 0.020 0.000 0.980
#> GSM340347 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340348 4 0.3919 0.72668 0.000 0.000 0.188 0.776 0.036
#> GSM340349 3 0.3476 0.74997 0.076 0.000 0.836 0.000 0.088
#> GSM340350 3 0.3895 0.54896 0.000 0.000 0.680 0.000 0.320
#> GSM340351 3 0.0000 0.84224 0.000 0.000 1.000 0.000 0.000
#> GSM340354 5 0.0404 0.75810 0.000 0.000 0.012 0.000 0.988
#> GSM340356 2 0.3949 0.50283 0.000 0.668 0.332 0.000 0.000
#> GSM340357 3 0.0000 0.84224 0.000 0.000 1.000 0.000 0.000
#> GSM348183 3 0.0162 0.84229 0.000 0.000 0.996 0.000 0.004
#> GSM348191 5 0.0703 0.76050 0.000 0.000 0.024 0.000 0.976
#> GSM348193 3 0.0000 0.84224 0.000 0.000 1.000 0.000 0.000
#> GSM537578 3 0.5475 0.41127 0.084 0.000 0.596 0.000 0.320
#> GSM348181 3 0.0794 0.83619 0.000 0.000 0.972 0.000 0.028
#> GSM348182 3 0.3210 0.70269 0.000 0.000 0.788 0.000 0.212
#> GSM348184 2 0.3857 0.50596 0.000 0.688 0.000 0.000 0.312
#> GSM348185 3 0.6078 0.01255 0.000 0.400 0.508 0.072 0.020
#> GSM348186 2 0.0000 0.76938 0.000 1.000 0.000 0.000 0.000
#> GSM348187 3 0.0404 0.83855 0.012 0.000 0.988 0.000 0.000
#> GSM348188 4 0.1364 0.87056 0.000 0.000 0.036 0.952 0.012
#> GSM348189 5 0.4221 0.69474 0.000 0.112 0.108 0.000 0.780
#> GSM348190 3 0.3480 0.67728 0.000 0.000 0.752 0.000 0.248
#> GSM348194 3 0.0000 0.84224 0.000 0.000 1.000 0.000 0.000
#> GSM348195 3 0.0000 0.84224 0.000 0.000 1.000 0.000 0.000
#> GSM348196 3 0.0000 0.84224 0.000 0.000 1.000 0.000 0.000
#> GSM537585 3 0.0000 0.84224 0.000 0.000 1.000 0.000 0.000
#> GSM537594 2 0.0000 0.76938 0.000 1.000 0.000 0.000 0.000
#> GSM537596 3 0.0880 0.83513 0.000 0.000 0.968 0.000 0.032
#> GSM537597 3 0.0000 0.84224 0.000 0.000 1.000 0.000 0.000
#> GSM537602 3 0.0000 0.84224 0.000 0.000 1.000 0.000 0.000
#> GSM340184 5 0.0963 0.76109 0.000 0.000 0.036 0.000 0.964
#> GSM340185 2 0.0000 0.76938 0.000 1.000 0.000 0.000 0.000
#> GSM340186 2 0.0162 0.76856 0.000 0.996 0.000 0.000 0.004
#> GSM340187 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340189 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340190 2 0.4278 0.25365 0.000 0.548 0.000 0.000 0.452
#> GSM340191 5 0.1671 0.75752 0.000 0.000 0.076 0.000 0.924
#> GSM340192 5 0.4138 0.43595 0.000 0.384 0.000 0.000 0.616
#> GSM340193 3 0.2966 0.73285 0.000 0.000 0.816 0.000 0.184
#> GSM340194 5 0.4774 0.45979 0.000 0.360 0.028 0.000 0.612
#> GSM340195 5 0.6798 0.21891 0.000 0.340 0.292 0.000 0.368
#> GSM340196 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340197 5 0.4307 0.00197 0.000 0.000 0.496 0.000 0.504
#> GSM340198 2 0.0510 0.76323 0.000 0.984 0.000 0.000 0.016
#> GSM340199 5 0.0880 0.75946 0.000 0.000 0.032 0.000 0.968
#> GSM340200 5 0.4450 -0.03131 0.004 0.488 0.000 0.000 0.508
#> GSM340201 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340202 4 0.0000 0.89535 0.000 0.000 0.000 1.000 0.000
#> GSM340203 4 0.3442 0.77527 0.000 0.000 0.060 0.836 0.104
#> GSM340204 2 0.6756 -0.23943 0.000 0.368 0.264 0.000 0.368
#> GSM340205 3 0.0162 0.84229 0.000 0.000 0.996 0.000 0.004
#> GSM340206 5 0.0609 0.75979 0.000 0.000 0.020 0.000 0.980
#> GSM340207 5 0.0000 0.75331 0.000 0.000 0.000 0.000 1.000
#> GSM340237 2 0.3949 0.54895 0.004 0.696 0.300 0.000 0.000
#> GSM340238 5 0.0609 0.75979 0.000 0.000 0.020 0.000 0.980
#> GSM340239 5 0.2773 0.71242 0.000 0.000 0.164 0.000 0.836
#> GSM340240 5 0.0404 0.75281 0.000 0.012 0.000 0.000 0.988
#> GSM340241 2 0.0000 0.76938 0.000 1.000 0.000 0.000 0.000
#> GSM340242 5 0.3612 0.57188 0.000 0.000 0.268 0.000 0.732
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.0000 0.9589 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340359 1 0.0000 0.9589 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340361 6 0.0146 0.7919 0.004 0.000 0.000 0.000 0.000 0.996
#> GSM340362 5 0.4304 0.6969 0.000 0.020 0.248 0.000 0.704 0.028
#> GSM340363 6 0.0000 0.7921 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM340364 1 0.0000 0.9589 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340365 6 0.2941 0.6920 0.000 0.000 0.000 0.000 0.220 0.780
#> GSM340366 1 0.7063 0.3352 0.460 0.000 0.172 0.000 0.240 0.128
#> GSM340367 1 0.0000 0.9589 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340368 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340369 4 0.3428 0.5950 0.304 0.000 0.000 0.696 0.000 0.000
#> GSM340370 1 0.0000 0.9589 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340371 3 0.3765 0.0966 0.000 0.000 0.596 0.000 0.404 0.000
#> GSM340372 1 0.0000 0.9589 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.9589 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340375 1 0.0000 0.9589 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340376 1 0.0000 0.9589 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340378 1 0.0000 0.9589 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340243 6 0.6495 0.3576 0.048 0.000 0.240 0.000 0.212 0.500
#> GSM340244 2 0.0146 0.8663 0.000 0.996 0.000 0.000 0.000 0.004
#> GSM340246 2 0.0260 0.8656 0.000 0.992 0.008 0.000 0.000 0.000
#> GSM340247 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340248 2 0.2375 0.8075 0.000 0.896 0.060 0.000 0.008 0.036
#> GSM340249 2 0.3309 0.6056 0.000 0.720 0.000 0.280 0.000 0.000
#> GSM340250 6 0.4517 0.1385 0.004 0.000 0.444 0.000 0.024 0.528
#> GSM340251 2 0.1434 0.8399 0.000 0.940 0.048 0.000 0.012 0.000
#> GSM340252 5 0.5230 0.5444 0.000 0.132 0.012 0.216 0.640 0.000
#> GSM340253 5 0.3515 0.4958 0.000 0.324 0.000 0.000 0.676 0.000
#> GSM340254 5 0.3515 0.4958 0.000 0.324 0.000 0.000 0.676 0.000
#> GSM340256 2 0.1327 0.8366 0.000 0.936 0.000 0.000 0.064 0.000
#> GSM340258 3 0.4151 0.4546 0.000 0.000 0.684 0.000 0.040 0.276
#> GSM340259 6 0.3345 0.6958 0.000 0.000 0.184 0.000 0.028 0.788
#> GSM340260 3 0.5012 0.2459 0.000 0.000 0.600 0.000 0.300 0.100
#> GSM340261 6 0.3695 0.4018 0.000 0.000 0.376 0.000 0.000 0.624
#> GSM340262 3 0.4332 0.3270 0.000 0.000 0.616 0.000 0.032 0.352
#> GSM340263 6 0.3860 0.1054 0.000 0.472 0.000 0.000 0.000 0.528
#> GSM340264 6 0.3907 0.5820 0.000 0.000 0.268 0.000 0.028 0.704
#> GSM340265 5 0.3950 0.4615 0.000 0.000 0.432 0.000 0.564 0.004
#> GSM340266 6 0.4283 0.3974 0.000 0.000 0.384 0.000 0.024 0.592
#> GSM340267 6 0.2841 0.6998 0.000 0.000 0.164 0.000 0.012 0.824
#> GSM340268 3 0.0458 0.6857 0.000 0.000 0.984 0.000 0.016 0.000
#> GSM340269 6 0.3619 0.6577 0.000 0.000 0.232 0.000 0.024 0.744
#> GSM340270 6 0.2358 0.7550 0.000 0.000 0.108 0.000 0.016 0.876
#> GSM537574 6 0.5602 0.1118 0.000 0.000 0.400 0.000 0.144 0.456
#> GSM537580 5 0.4294 0.3970 0.000 0.000 0.428 0.000 0.552 0.020
#> GSM537581 4 0.3690 0.6113 0.000 0.000 0.008 0.684 0.000 0.308
#> GSM340272 3 0.4972 0.4638 0.000 0.000 0.620 0.000 0.108 0.272
#> GSM340273 2 0.0858 0.8576 0.000 0.968 0.000 0.000 0.004 0.028
#> GSM340275 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340276 6 0.4009 0.5833 0.000 0.000 0.288 0.000 0.028 0.684
#> GSM340277 2 0.1610 0.8223 0.000 0.916 0.000 0.000 0.084 0.000
#> GSM340278 3 0.3967 0.2484 0.000 0.000 0.632 0.000 0.012 0.356
#> GSM340279 3 0.2446 0.6493 0.000 0.000 0.864 0.000 0.012 0.124
#> GSM340282 3 0.2985 0.6489 0.000 0.000 0.844 0.000 0.056 0.100
#> GSM340284 2 0.4246 0.1285 0.000 0.532 0.452 0.000 0.016 0.000
#> GSM340285 6 0.3907 0.3622 0.000 0.000 0.408 0.000 0.004 0.588
#> GSM340286 6 0.4405 0.5927 0.000 0.000 0.240 0.000 0.072 0.688
#> GSM340287 6 0.0363 0.7928 0.000 0.000 0.012 0.000 0.000 0.988
#> GSM340288 6 0.0000 0.7921 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM340289 6 0.0458 0.7916 0.000 0.000 0.016 0.000 0.000 0.984
#> GSM340290 3 0.0260 0.6868 0.000 0.000 0.992 0.000 0.008 0.000
#> GSM340291 2 0.0000 0.8664 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340293 3 0.2581 0.6063 0.000 0.000 0.860 0.120 0.020 0.000
#> GSM340294 3 0.2932 0.6241 0.000 0.000 0.820 0.000 0.016 0.164
#> GSM340296 3 0.5644 0.3854 0.168 0.000 0.580 0.000 0.240 0.012
#> GSM340297 3 0.2996 0.5702 0.000 0.000 0.772 0.000 0.000 0.228
#> GSM340298 3 0.5866 0.3236 0.204 0.000 0.544 0.000 0.240 0.012
#> GSM340299 5 0.4436 0.6999 0.000 0.020 0.236 0.000 0.704 0.040
#> GSM340301 5 0.4103 0.4337 0.000 0.004 0.448 0.000 0.544 0.004
#> GSM340303 5 0.4454 0.6737 0.000 0.004 0.252 0.000 0.684 0.060
#> GSM340304 6 0.2006 0.7625 0.000 0.000 0.104 0.000 0.004 0.892
#> GSM340306 4 0.2562 0.7923 0.000 0.000 0.000 0.828 0.000 0.172
#> GSM340307 2 0.1806 0.8083 0.000 0.908 0.088 0.000 0.004 0.000
#> GSM340310 1 0.0000 0.9589 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340314 3 0.0000 0.6872 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340315 6 0.0458 0.7920 0.000 0.000 0.016 0.000 0.000 0.984
#> GSM340317 2 0.1116 0.8555 0.000 0.960 0.004 0.028 0.000 0.008
#> GSM340318 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340319 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340320 6 0.2003 0.7227 0.000 0.000 0.000 0.116 0.000 0.884
#> GSM340321 2 0.0000 0.8664 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340322 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340324 2 0.1556 0.8255 0.000 0.920 0.000 0.000 0.080 0.000
#> GSM340328 1 0.0000 0.9589 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340330 3 0.0713 0.6861 0.000 0.000 0.972 0.000 0.028 0.000
#> GSM340332 4 0.2340 0.8008 0.000 0.000 0.148 0.852 0.000 0.000
#> GSM340333 5 0.3515 0.4958 0.000 0.324 0.000 0.000 0.676 0.000
#> GSM340336 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340337 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340338 3 0.0458 0.6854 0.000 0.000 0.984 0.000 0.016 0.000
#> GSM340339 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340340 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340341 4 0.5278 0.6847 0.000 0.052 0.152 0.708 0.020 0.068
#> GSM340343 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340344 5 0.4937 0.6968 0.000 0.056 0.172 0.000 0.708 0.064
#> GSM340346 3 0.0935 0.6820 0.000 0.000 0.964 0.000 0.032 0.004
#> GSM340347 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340348 4 0.4151 0.7385 0.000 0.000 0.084 0.748 0.004 0.164
#> GSM340349 6 0.6254 0.3669 0.036 0.000 0.228 0.000 0.208 0.528
#> GSM340350 3 0.4493 -0.0578 0.008 0.000 0.492 0.000 0.016 0.484
#> GSM340351 6 0.0000 0.7921 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM340354 3 0.0865 0.6825 0.000 0.000 0.964 0.000 0.036 0.000
#> GSM340356 2 0.0508 0.8650 0.000 0.984 0.000 0.000 0.004 0.012
#> GSM340357 6 0.0000 0.7921 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM348183 6 0.0508 0.7917 0.000 0.000 0.012 0.000 0.004 0.984
#> GSM348191 3 0.0935 0.6818 0.000 0.000 0.964 0.000 0.032 0.004
#> GSM348193 6 0.0000 0.7921 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM537578 6 0.4801 0.1329 0.036 0.000 0.436 0.000 0.008 0.520
#> GSM348181 6 0.1890 0.7729 0.000 0.000 0.060 0.000 0.024 0.916
#> GSM348182 6 0.3558 0.6619 0.000 0.000 0.212 0.000 0.028 0.760
#> GSM348184 2 0.0260 0.8657 0.000 0.992 0.008 0.000 0.000 0.000
#> GSM348185 2 0.4339 0.6141 0.000 0.724 0.032 0.020 0.004 0.220
#> GSM348186 2 0.2454 0.7418 0.000 0.840 0.000 0.000 0.160 0.000
#> GSM348187 6 0.0000 0.7921 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM348188 4 0.1418 0.8926 0.000 0.000 0.024 0.944 0.000 0.032
#> GSM348189 5 0.4378 0.5719 0.000 0.000 0.328 0.000 0.632 0.040
#> GSM348190 5 0.5270 0.5280 0.000 0.000 0.216 0.000 0.604 0.180
#> GSM348194 6 0.0000 0.7921 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM348195 6 0.0146 0.7919 0.004 0.000 0.000 0.000 0.000 0.996
#> GSM348196 6 0.0000 0.7921 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM537585 6 0.0000 0.7921 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM537594 2 0.0000 0.8664 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM537596 6 0.1657 0.7773 0.000 0.000 0.056 0.000 0.016 0.928
#> GSM537597 6 0.0000 0.7921 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM537602 6 0.0260 0.7917 0.000 0.000 0.000 0.000 0.008 0.992
#> GSM340184 3 0.1225 0.6798 0.000 0.000 0.952 0.000 0.036 0.012
#> GSM340185 2 0.0000 0.8664 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340186 2 0.0000 0.8664 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340187 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340189 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340190 2 0.3833 0.4891 0.000 0.648 0.344 0.000 0.008 0.000
#> GSM340191 3 0.2726 0.6309 0.000 0.000 0.856 0.000 0.032 0.112
#> GSM340192 5 0.3385 0.6765 0.000 0.032 0.180 0.000 0.788 0.000
#> GSM340193 6 0.3202 0.7036 0.000 0.000 0.176 0.000 0.024 0.800
#> GSM340194 5 0.4278 0.7030 0.000 0.020 0.232 0.000 0.716 0.032
#> GSM340195 5 0.3812 0.6780 0.000 0.004 0.268 0.000 0.712 0.016
#> GSM340196 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340197 6 0.5112 0.2218 0.000 0.000 0.400 0.000 0.084 0.516
#> GSM340198 5 0.3717 0.3892 0.000 0.384 0.000 0.000 0.616 0.000
#> GSM340199 3 0.1049 0.6828 0.000 0.000 0.960 0.000 0.032 0.008
#> GSM340200 2 0.5859 0.3152 0.000 0.480 0.288 0.000 0.232 0.000
#> GSM340201 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340202 4 0.0000 0.9233 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340203 4 0.3248 0.7904 0.000 0.000 0.116 0.828 0.004 0.052
#> GSM340204 5 0.4225 0.6765 0.000 0.012 0.264 0.000 0.696 0.028
#> GSM340205 6 0.0603 0.7916 0.004 0.000 0.016 0.000 0.000 0.980
#> GSM340206 3 0.0260 0.6872 0.000 0.000 0.992 0.000 0.008 0.000
#> GSM340207 3 0.3592 0.2196 0.000 0.000 0.656 0.000 0.344 0.000
#> GSM340237 2 0.0146 0.8663 0.000 0.996 0.000 0.000 0.000 0.004
#> GSM340238 3 0.0632 0.6858 0.000 0.000 0.976 0.000 0.024 0.000
#> GSM340239 3 0.4736 0.4128 0.000 0.000 0.620 0.000 0.072 0.308
#> GSM340240 3 0.3717 0.0682 0.000 0.000 0.616 0.000 0.384 0.000
#> GSM340241 2 0.0260 0.8648 0.000 0.992 0.000 0.000 0.008 0.000
#> GSM340242 3 0.5629 -0.0421 0.000 0.000 0.448 0.000 0.404 0.148
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
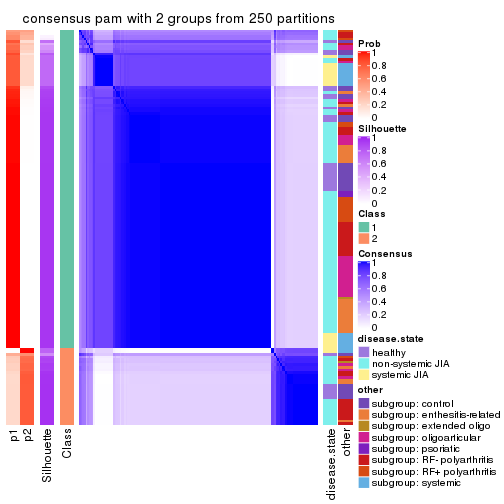
consensus_heatmap(res, k = 3)
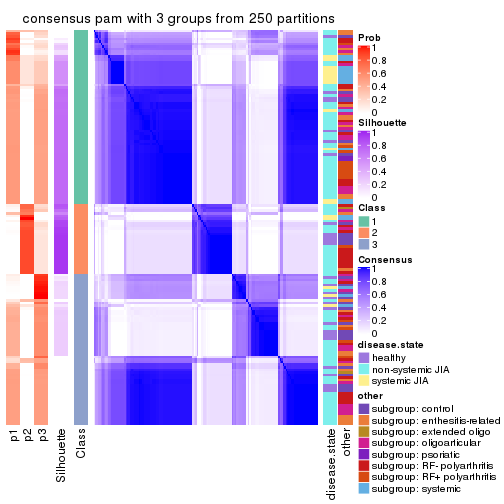
consensus_heatmap(res, k = 4)
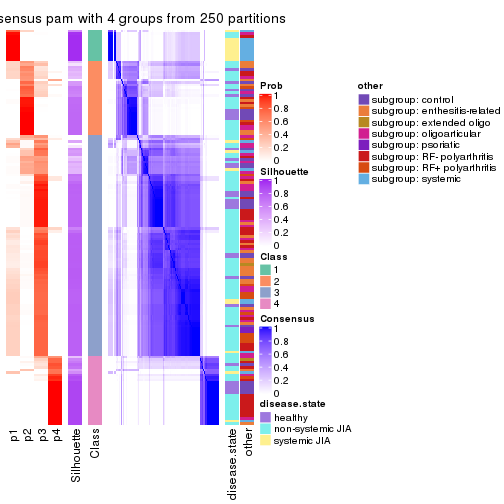
consensus_heatmap(res, k = 5)
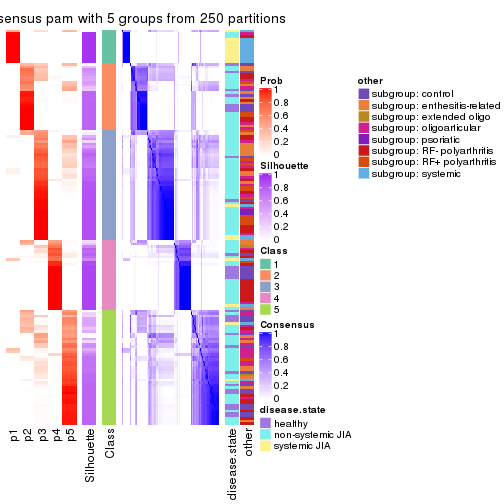
consensus_heatmap(res, k = 6)
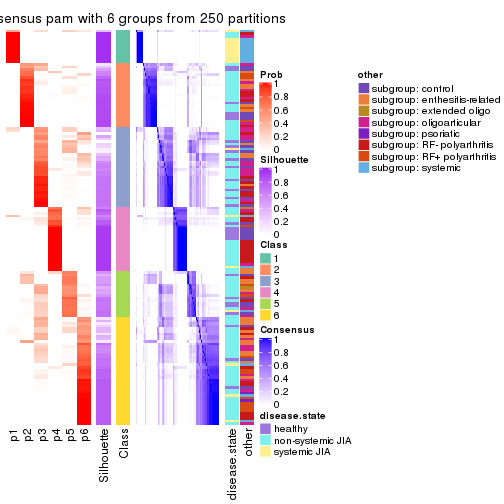
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
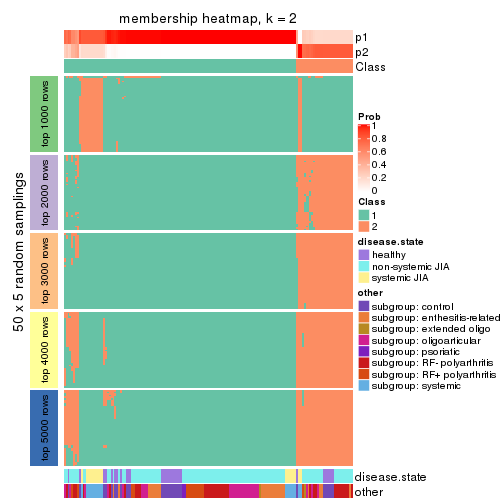
membership_heatmap(res, k = 3)
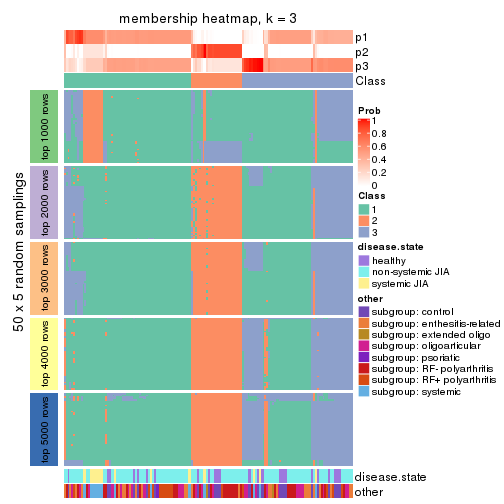
membership_heatmap(res, k = 4)
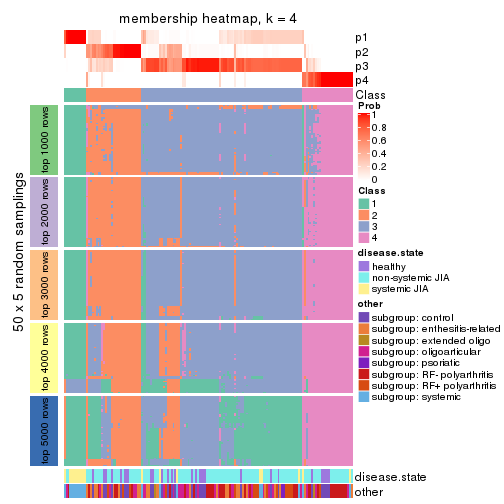
membership_heatmap(res, k = 5)
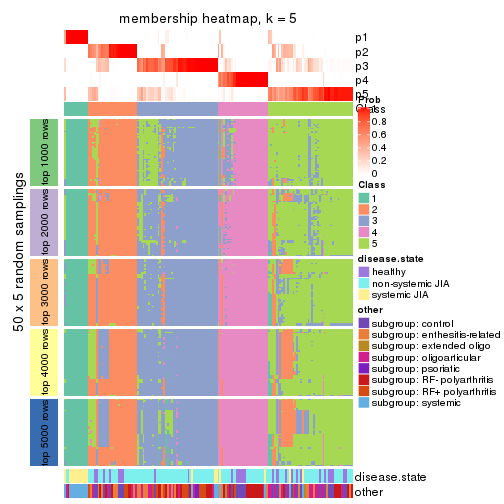
membership_heatmap(res, k = 6)
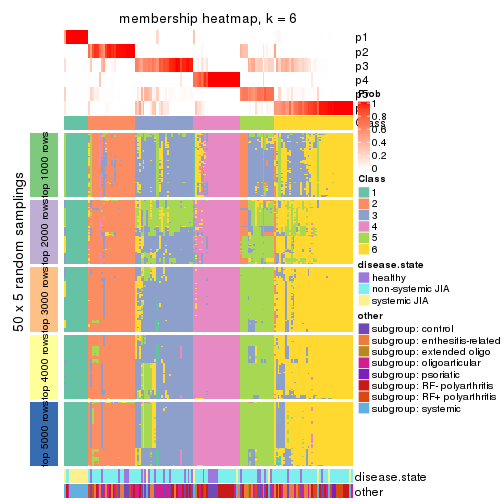
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
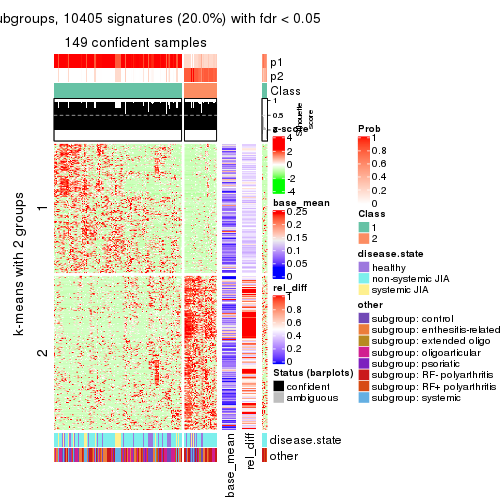
get_signatures(res, k = 3)
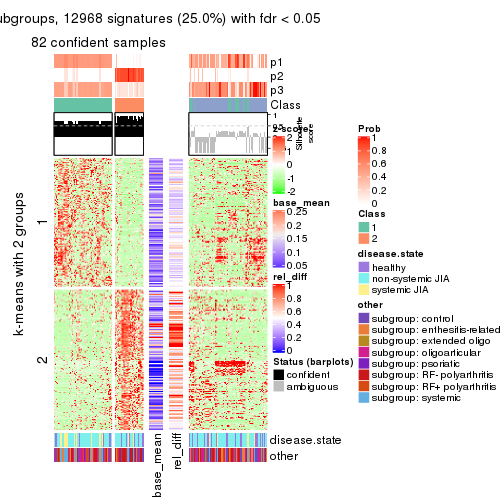
get_signatures(res, k = 4)
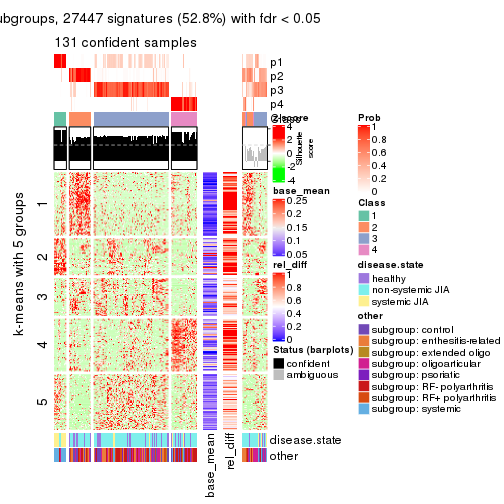
get_signatures(res, k = 5)
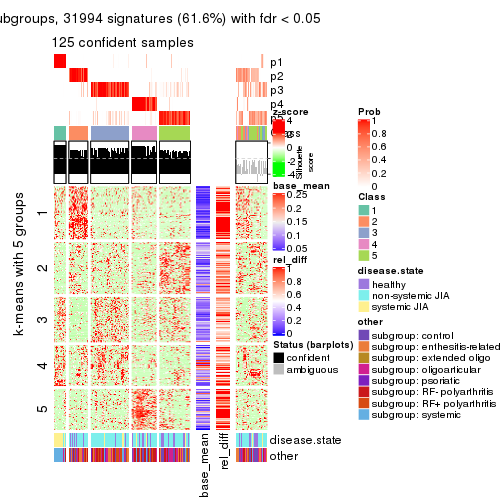
get_signatures(res, k = 6)
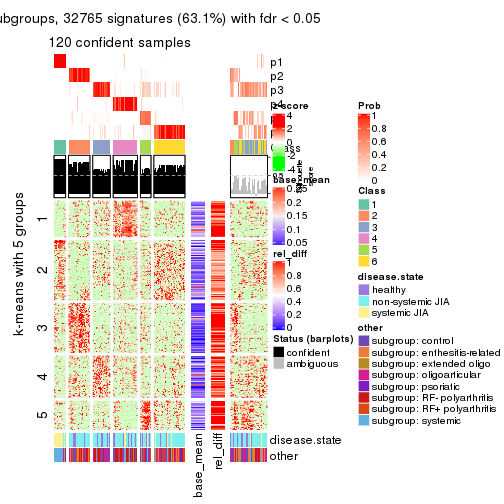
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
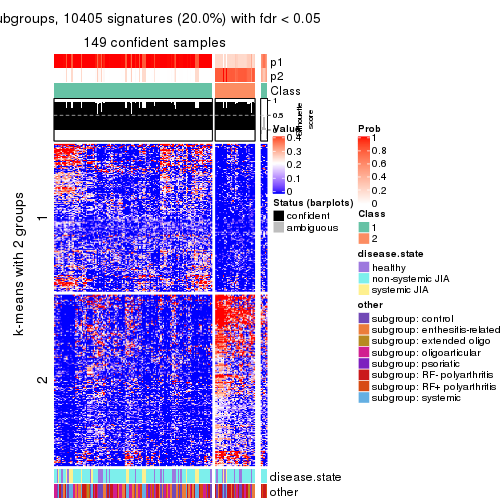
get_signatures(res, k = 3, scale_rows = FALSE)
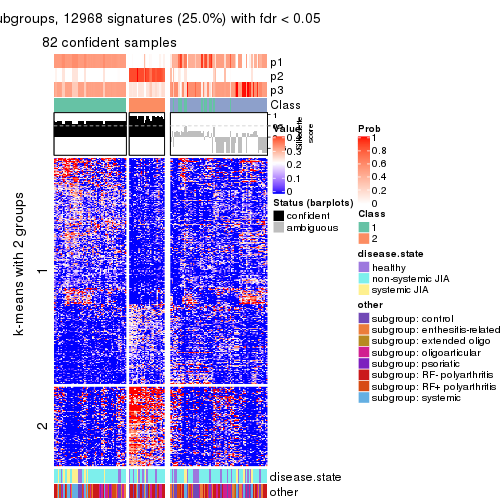
get_signatures(res, k = 4, scale_rows = FALSE)
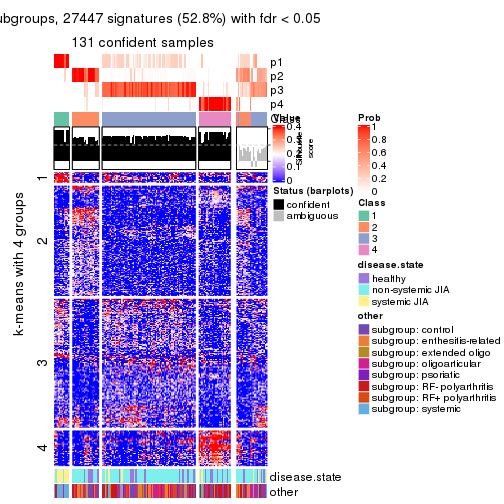
get_signatures(res, k = 5, scale_rows = FALSE)
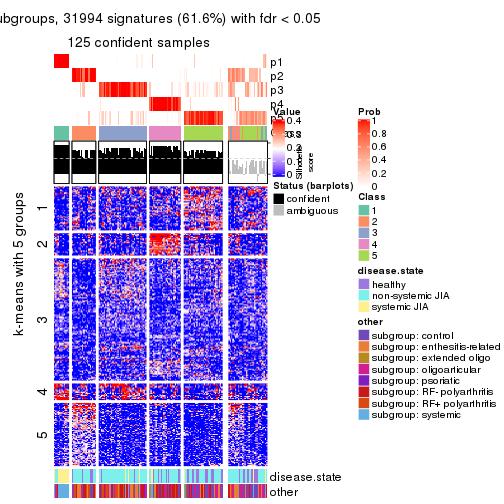
get_signatures(res, k = 6, scale_rows = FALSE)
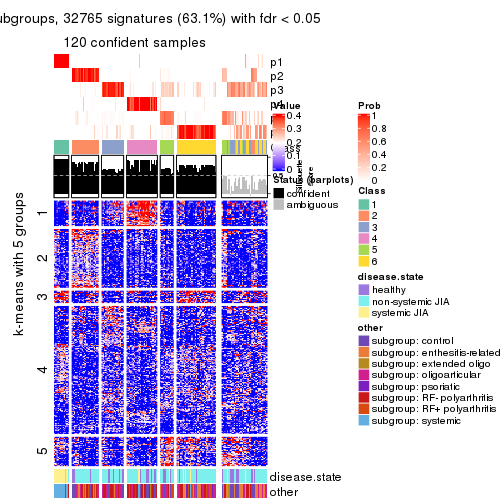
Compare the overlap of signatures from different k:
compare_signatures(res)
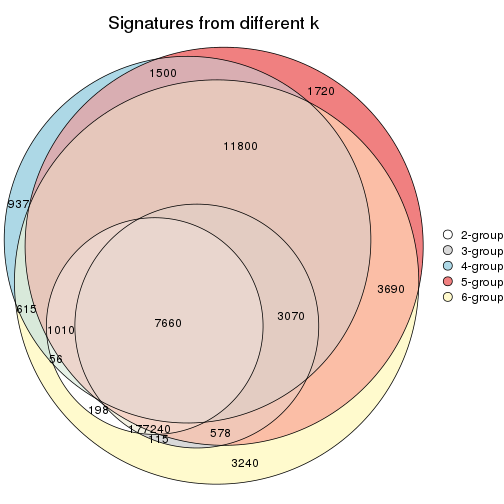
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
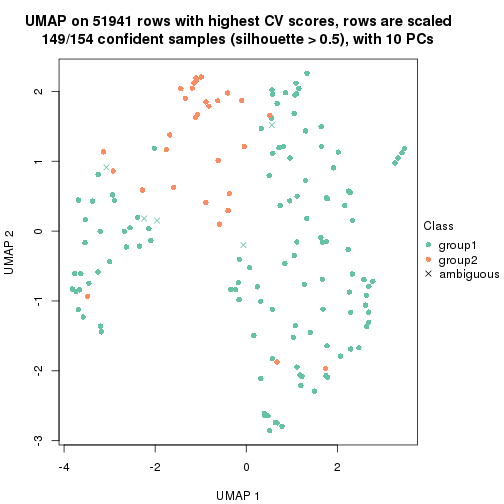
dimension_reduction(res, k = 3, method = "UMAP")
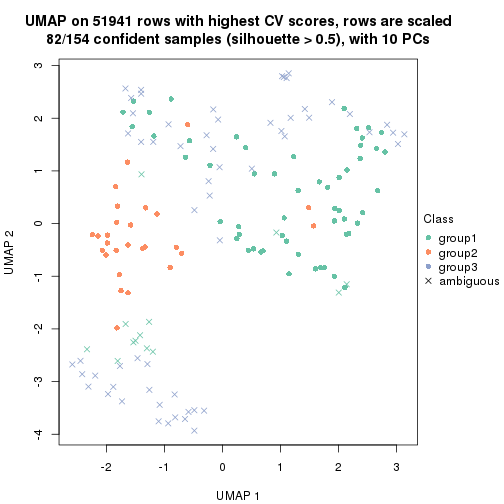
dimension_reduction(res, k = 4, method = "UMAP")
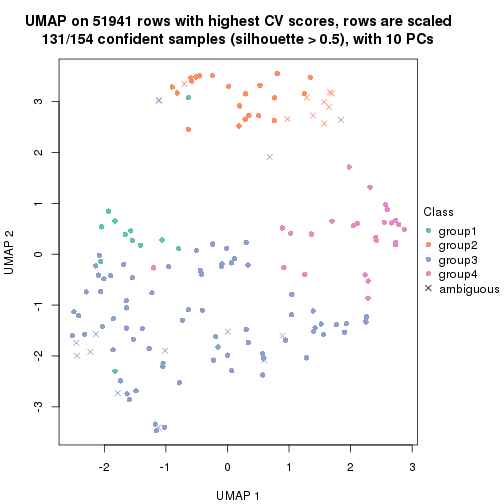
dimension_reduction(res, k = 5, method = "UMAP")
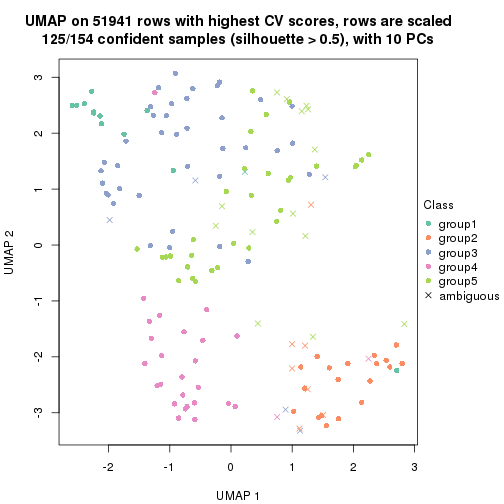
dimension_reduction(res, k = 6, method = "UMAP")
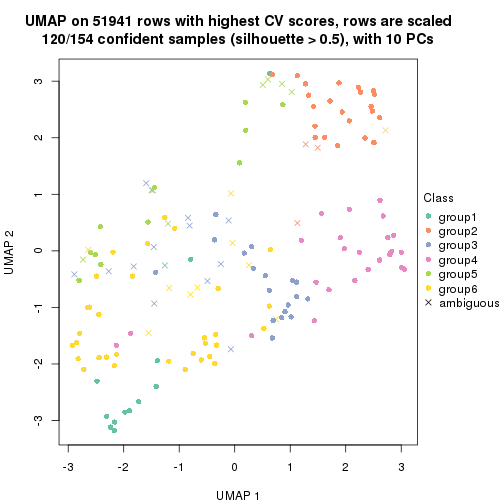
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
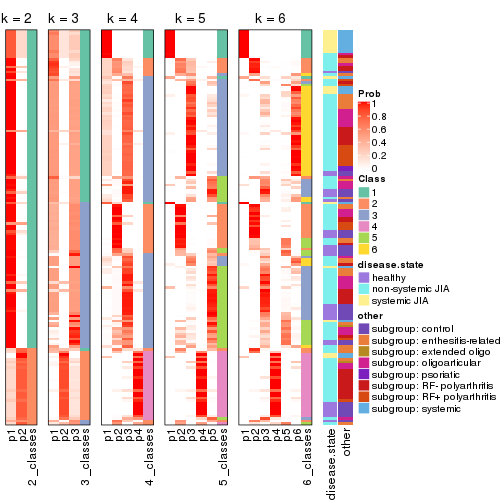
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> CV:pam 149 5.45e-01 8.17e-02 2
#> CV:pam 82 1.83e-01 1.72e-02 3
#> CV:pam 131 2.26e-13 2.90e-10 4
#> CV:pam 125 1.18e-11 2.44e-09 5
#> CV:pam 120 3.16e-10 3.42e-08 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["CV", "mclust"]
# you can also extract it by
# res = res_list["CV:mclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'CV' method.
#> Subgroups are detected by 'mclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
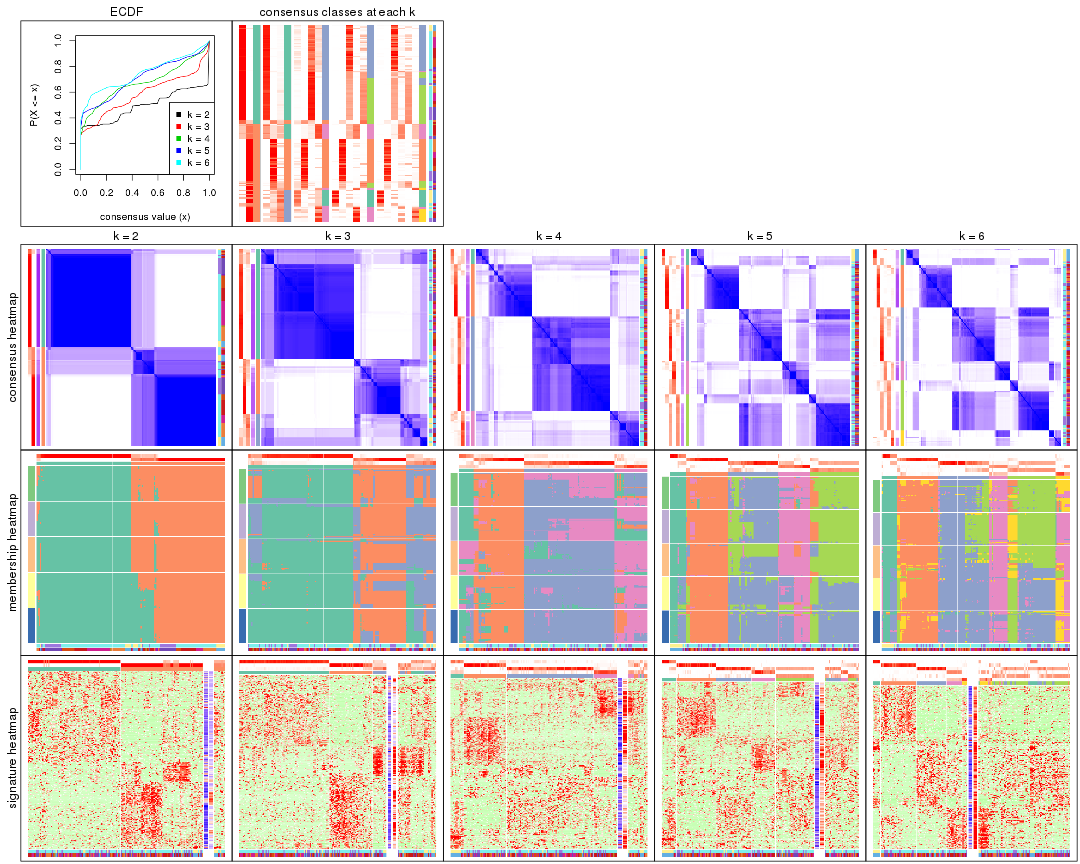
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
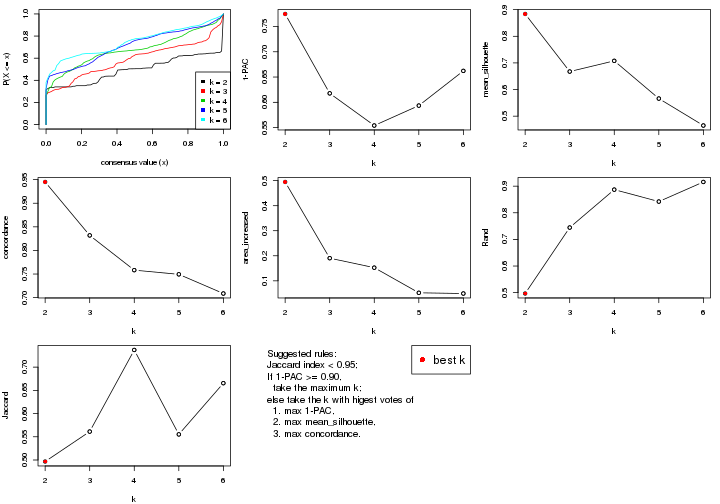
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.775 0.884 0.945 0.4947 0.497 0.497
#> 3 3 0.618 0.668 0.832 0.1903 0.745 0.561
#> 4 4 0.554 0.708 0.758 0.1525 0.887 0.737
#> 5 5 0.594 0.566 0.749 0.0521 0.842 0.555
#> 6 6 0.662 0.465 0.709 0.0492 0.916 0.665
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 2 0.9710 0.475 0.400 0.600
#> GSM340359 2 0.0000 0.896 0.000 1.000
#> GSM340361 1 0.0000 0.985 1.000 0.000
#> GSM340362 1 0.0000 0.985 1.000 0.000
#> GSM340363 1 0.6531 0.758 0.832 0.168
#> GSM340364 2 0.0000 0.896 0.000 1.000
#> GSM340365 1 0.0000 0.985 1.000 0.000
#> GSM340366 1 0.0000 0.985 1.000 0.000
#> GSM340367 2 0.9710 0.475 0.400 0.600
#> GSM340368 2 0.0000 0.896 0.000 1.000
#> GSM340369 2 0.7745 0.725 0.228 0.772
#> GSM340370 2 0.0000 0.896 0.000 1.000
#> GSM340371 1 0.0000 0.985 1.000 0.000
#> GSM340372 2 0.9710 0.475 0.400 0.600
#> GSM340373 2 0.9710 0.475 0.400 0.600
#> GSM340375 2 0.0000 0.896 0.000 1.000
#> GSM340376 2 0.0000 0.896 0.000 1.000
#> GSM340378 2 0.0000 0.896 0.000 1.000
#> GSM340243 1 0.0000 0.985 1.000 0.000
#> GSM340244 2 0.0000 0.896 0.000 1.000
#> GSM340246 2 0.0000 0.896 0.000 1.000
#> GSM340247 2 0.0000 0.896 0.000 1.000
#> GSM340248 2 0.0000 0.896 0.000 1.000
#> GSM340249 2 0.0000 0.896 0.000 1.000
#> GSM340250 1 0.0000 0.985 1.000 0.000
#> GSM340251 2 0.0000 0.896 0.000 1.000
#> GSM340252 2 0.0000 0.896 0.000 1.000
#> GSM340253 2 0.0000 0.896 0.000 1.000
#> GSM340254 2 0.0000 0.896 0.000 1.000
#> GSM340256 2 0.0000 0.896 0.000 1.000
#> GSM340258 2 0.9944 0.205 0.456 0.544
#> GSM340259 1 0.0000 0.985 1.000 0.000
#> GSM340260 1 0.0000 0.985 1.000 0.000
#> GSM340261 1 0.0000 0.985 1.000 0.000
#> GSM340262 1 0.0000 0.985 1.000 0.000
#> GSM340263 2 0.0000 0.896 0.000 1.000
#> GSM340264 1 0.0000 0.985 1.000 0.000
#> GSM340265 1 0.8207 0.603 0.744 0.256
#> GSM340266 1 0.0000 0.985 1.000 0.000
#> GSM340267 1 0.8207 0.624 0.744 0.256
#> GSM340268 1 0.0000 0.985 1.000 0.000
#> GSM340269 1 0.0000 0.985 1.000 0.000
#> GSM340270 1 0.0000 0.985 1.000 0.000
#> GSM537574 2 0.2603 0.867 0.044 0.956
#> GSM537580 1 0.0000 0.985 1.000 0.000
#> GSM537581 2 0.8955 0.631 0.312 0.688
#> GSM340272 1 0.0000 0.985 1.000 0.000
#> GSM340273 2 0.0000 0.896 0.000 1.000
#> GSM340275 2 0.0000 0.896 0.000 1.000
#> GSM340276 1 0.0000 0.985 1.000 0.000
#> GSM340277 2 0.0000 0.896 0.000 1.000
#> GSM340278 1 0.0000 0.985 1.000 0.000
#> GSM340279 1 0.0000 0.985 1.000 0.000
#> GSM340282 1 0.0000 0.985 1.000 0.000
#> GSM340284 2 0.0000 0.896 0.000 1.000
#> GSM340285 1 0.0000 0.985 1.000 0.000
#> GSM340286 1 0.0000 0.985 1.000 0.000
#> GSM340287 1 0.0000 0.985 1.000 0.000
#> GSM340288 1 0.0000 0.985 1.000 0.000
#> GSM340289 1 0.0000 0.985 1.000 0.000
#> GSM340290 1 0.0000 0.985 1.000 0.000
#> GSM340291 2 0.0000 0.896 0.000 1.000
#> GSM340293 1 0.0000 0.985 1.000 0.000
#> GSM340294 1 0.0000 0.985 1.000 0.000
#> GSM340296 1 0.0000 0.985 1.000 0.000
#> GSM340297 1 0.0000 0.985 1.000 0.000
#> GSM340298 1 0.0000 0.985 1.000 0.000
#> GSM340299 1 0.0000 0.985 1.000 0.000
#> GSM340301 1 0.0000 0.985 1.000 0.000
#> GSM340303 2 0.9732 0.380 0.404 0.596
#> GSM340304 1 0.0000 0.985 1.000 0.000
#> GSM340306 2 0.0672 0.891 0.008 0.992
#> GSM340307 2 0.0000 0.896 0.000 1.000
#> GSM340310 2 0.9710 0.475 0.400 0.600
#> GSM340314 1 0.0000 0.985 1.000 0.000
#> GSM340315 1 0.0376 0.981 0.996 0.004
#> GSM340317 2 0.0000 0.896 0.000 1.000
#> GSM340318 2 0.0000 0.896 0.000 1.000
#> GSM340319 2 0.0000 0.896 0.000 1.000
#> GSM340320 2 0.9635 0.503 0.388 0.612
#> GSM340321 2 0.0000 0.896 0.000 1.000
#> GSM340322 2 0.8661 0.662 0.288 0.712
#> GSM340324 2 0.0000 0.896 0.000 1.000
#> GSM340328 2 0.9661 0.492 0.392 0.608
#> GSM340330 1 0.0000 0.985 1.000 0.000
#> GSM340332 2 0.8813 0.647 0.300 0.700
#> GSM340333 2 0.0000 0.896 0.000 1.000
#> GSM340336 2 0.0000 0.896 0.000 1.000
#> GSM340337 2 0.8813 0.647 0.300 0.700
#> GSM340338 1 0.0000 0.985 1.000 0.000
#> GSM340339 2 0.8661 0.662 0.288 0.712
#> GSM340340 2 0.0000 0.896 0.000 1.000
#> GSM340341 2 0.0000 0.896 0.000 1.000
#> GSM340343 2 0.8813 0.647 0.300 0.700
#> GSM340344 2 0.0000 0.896 0.000 1.000
#> GSM340346 1 0.0000 0.985 1.000 0.000
#> GSM340347 2 0.0000 0.896 0.000 1.000
#> GSM340348 2 0.9661 0.494 0.392 0.608
#> GSM340349 1 0.0000 0.985 1.000 0.000
#> GSM340350 1 0.0000 0.985 1.000 0.000
#> GSM340351 1 0.0000 0.985 1.000 0.000
#> GSM340354 1 0.8386 0.603 0.732 0.268
#> GSM340356 2 0.0000 0.896 0.000 1.000
#> GSM340357 1 0.0000 0.985 1.000 0.000
#> GSM348183 1 0.0938 0.973 0.988 0.012
#> GSM348191 1 0.0000 0.985 1.000 0.000
#> GSM348193 2 0.8861 0.642 0.304 0.696
#> GSM537578 1 0.0000 0.985 1.000 0.000
#> GSM348181 1 0.0000 0.985 1.000 0.000
#> GSM348182 1 0.0000 0.985 1.000 0.000
#> GSM348184 2 0.0000 0.896 0.000 1.000
#> GSM348185 2 0.0000 0.896 0.000 1.000
#> GSM348186 2 0.0000 0.896 0.000 1.000
#> GSM348187 1 0.0000 0.985 1.000 0.000
#> GSM348188 2 0.8861 0.642 0.304 0.696
#> GSM348189 1 0.0000 0.985 1.000 0.000
#> GSM348190 1 0.0000 0.985 1.000 0.000
#> GSM348194 1 0.0000 0.985 1.000 0.000
#> GSM348195 1 0.0000 0.985 1.000 0.000
#> GSM348196 1 0.0000 0.985 1.000 0.000
#> GSM537585 1 0.0000 0.985 1.000 0.000
#> GSM537594 2 0.0000 0.896 0.000 1.000
#> GSM537596 1 0.0000 0.985 1.000 0.000
#> GSM537597 2 0.5946 0.783 0.144 0.856
#> GSM537602 1 0.0000 0.985 1.000 0.000
#> GSM340184 1 0.0000 0.985 1.000 0.000
#> GSM340185 2 0.0000 0.896 0.000 1.000
#> GSM340186 2 0.0000 0.896 0.000 1.000
#> GSM340187 2 0.0000 0.896 0.000 1.000
#> GSM340189 2 0.8813 0.647 0.300 0.700
#> GSM340190 2 0.0000 0.896 0.000 1.000
#> GSM340191 1 0.0000 0.985 1.000 0.000
#> GSM340192 2 0.0000 0.896 0.000 1.000
#> GSM340193 1 0.0000 0.985 1.000 0.000
#> GSM340194 1 0.0000 0.985 1.000 0.000
#> GSM340195 1 0.0000 0.985 1.000 0.000
#> GSM340196 2 0.0000 0.896 0.000 1.000
#> GSM340197 1 0.0000 0.985 1.000 0.000
#> GSM340198 2 0.0000 0.896 0.000 1.000
#> GSM340199 1 0.0000 0.985 1.000 0.000
#> GSM340200 2 0.0000 0.896 0.000 1.000
#> GSM340201 2 0.0000 0.896 0.000 1.000
#> GSM340202 2 0.0000 0.896 0.000 1.000
#> GSM340203 2 0.9087 0.614 0.324 0.676
#> GSM340204 2 0.0000 0.896 0.000 1.000
#> GSM340205 1 0.1414 0.964 0.980 0.020
#> GSM340206 1 0.0000 0.985 1.000 0.000
#> GSM340207 1 0.0000 0.985 1.000 0.000
#> GSM340237 2 0.0000 0.896 0.000 1.000
#> GSM340238 1 0.0000 0.985 1.000 0.000
#> GSM340239 1 0.0000 0.985 1.000 0.000
#> GSM340240 1 0.0000 0.985 1.000 0.000
#> GSM340241 2 0.0000 0.896 0.000 1.000
#> GSM340242 1 0.0000 0.985 1.000 0.000
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 3 0.9030 0.4772 0.388 0.136 0.476
#> GSM340359 3 0.5948 0.3251 0.000 0.360 0.640
#> GSM340361 1 0.1129 0.8719 0.976 0.004 0.020
#> GSM340362 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340363 1 0.1453 0.8647 0.968 0.008 0.024
#> GSM340364 3 0.5905 0.3415 0.000 0.352 0.648
#> GSM340365 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340366 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340367 3 0.8951 0.4567 0.396 0.128 0.476
#> GSM340368 3 0.5678 0.4866 0.000 0.316 0.684
#> GSM340369 3 0.8939 0.5517 0.340 0.140 0.520
#> GSM340370 3 0.4555 0.5182 0.000 0.200 0.800
#> GSM340371 1 0.2066 0.8701 0.940 0.000 0.060
#> GSM340372 3 0.9014 0.4920 0.380 0.136 0.484
#> GSM340373 3 0.9030 0.4772 0.388 0.136 0.476
#> GSM340375 3 0.4555 0.5182 0.000 0.200 0.800
#> GSM340376 3 0.4555 0.5182 0.000 0.200 0.800
#> GSM340378 3 0.5497 0.4394 0.000 0.292 0.708
#> GSM340243 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340244 2 0.0237 0.8039 0.000 0.996 0.004
#> GSM340246 2 0.0000 0.8050 0.000 1.000 0.000
#> GSM340247 3 0.6267 0.3685 0.000 0.452 0.548
#> GSM340248 2 0.0000 0.8050 0.000 1.000 0.000
#> GSM340249 2 0.5926 0.0940 0.000 0.644 0.356
#> GSM340250 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340251 2 0.0237 0.8030 0.000 0.996 0.004
#> GSM340252 2 0.5178 0.3724 0.000 0.744 0.256
#> GSM340253 2 0.2261 0.7761 0.000 0.932 0.068
#> GSM340254 2 0.0000 0.8050 0.000 1.000 0.000
#> GSM340256 2 0.0237 0.8039 0.000 0.996 0.004
#> GSM340258 2 0.6867 0.5909 0.040 0.672 0.288
#> GSM340259 1 0.1753 0.8714 0.952 0.000 0.048
#> GSM340260 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340261 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM340262 1 0.2261 0.8664 0.932 0.000 0.068
#> GSM340263 2 0.0237 0.8039 0.000 0.996 0.004
#> GSM340264 1 0.1753 0.8735 0.952 0.000 0.048
#> GSM340265 2 0.8996 0.3303 0.196 0.560 0.244
#> GSM340266 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM340267 2 0.7742 0.5373 0.080 0.632 0.288
#> GSM340268 1 0.1860 0.8718 0.948 0.000 0.052
#> GSM340269 1 0.3879 0.7859 0.848 0.000 0.152
#> GSM340270 1 0.0592 0.8742 0.988 0.000 0.012
#> GSM537574 2 0.5623 0.6349 0.004 0.716 0.280
#> GSM537580 1 0.3482 0.8191 0.872 0.000 0.128
#> GSM537581 1 0.7396 -0.2357 0.488 0.032 0.480
#> GSM340272 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340273 2 0.0237 0.8039 0.000 0.996 0.004
#> GSM340275 3 0.6274 0.3657 0.000 0.456 0.544
#> GSM340276 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM340277 2 0.0000 0.8050 0.000 1.000 0.000
#> GSM340278 1 0.1529 0.8744 0.960 0.000 0.040
#> GSM340279 1 0.1860 0.8726 0.948 0.000 0.052
#> GSM340282 1 0.1411 0.8745 0.964 0.000 0.036
#> GSM340284 2 0.0000 0.8050 0.000 1.000 0.000
#> GSM340285 1 0.0237 0.8738 0.996 0.000 0.004
#> GSM340286 1 0.2165 0.8683 0.936 0.000 0.064
#> GSM340287 1 0.2066 0.8675 0.940 0.000 0.060
#> GSM340288 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340289 1 0.0747 0.8726 0.984 0.000 0.016
#> GSM340290 1 0.0592 0.8737 0.988 0.000 0.012
#> GSM340291 2 0.0237 0.8039 0.000 0.996 0.004
#> GSM340293 1 0.3502 0.7871 0.896 0.084 0.020
#> GSM340294 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM340296 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340297 1 0.0747 0.8727 0.984 0.000 0.016
#> GSM340298 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340299 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM340301 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM340303 2 0.7188 0.5797 0.056 0.664 0.280
#> GSM340304 1 0.0747 0.8727 0.984 0.000 0.016
#> GSM340306 3 0.9141 0.5945 0.244 0.212 0.544
#> GSM340307 2 0.2537 0.7694 0.000 0.920 0.080
#> GSM340310 3 0.9030 0.4772 0.388 0.136 0.476
#> GSM340314 1 0.1129 0.8738 0.976 0.004 0.020
#> GSM340315 1 0.2313 0.8473 0.944 0.032 0.024
#> GSM340317 2 0.5835 0.1594 0.000 0.660 0.340
#> GSM340318 3 0.8948 0.6099 0.188 0.248 0.564
#> GSM340319 3 0.8948 0.6099 0.188 0.248 0.564
#> GSM340320 1 0.7004 0.5113 0.728 0.160 0.112
#> GSM340321 2 0.0000 0.8050 0.000 1.000 0.000
#> GSM340322 1 0.9213 -0.2426 0.484 0.160 0.356
#> GSM340324 2 0.3816 0.6590 0.000 0.852 0.148
#> GSM340328 3 0.9030 0.4772 0.388 0.136 0.476
#> GSM340330 1 0.2165 0.8683 0.936 0.000 0.064
#> GSM340332 1 0.9148 -0.1815 0.504 0.160 0.336
#> GSM340333 2 0.2261 0.7761 0.000 0.932 0.068
#> GSM340336 3 0.6274 0.3657 0.000 0.456 0.544
#> GSM340337 1 0.9201 -0.2303 0.488 0.160 0.352
#> GSM340338 1 0.0237 0.8733 0.996 0.000 0.004
#> GSM340339 1 0.9201 -0.2303 0.488 0.160 0.352
#> GSM340340 3 0.5678 0.4866 0.000 0.316 0.684
#> GSM340341 2 0.6168 -0.0986 0.000 0.588 0.412
#> GSM340343 1 0.9189 -0.2185 0.492 0.160 0.348
#> GSM340344 2 0.4931 0.6789 0.000 0.768 0.232
#> GSM340346 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM340347 3 0.8948 0.6099 0.188 0.248 0.564
#> GSM340348 1 0.8199 0.2923 0.640 0.160 0.200
#> GSM340349 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340350 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340351 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340354 2 0.7889 0.5267 0.088 0.624 0.288
#> GSM340356 2 0.0237 0.8039 0.000 0.996 0.004
#> GSM340357 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM348183 1 0.1919 0.8587 0.956 0.024 0.020
#> GSM348191 1 0.2066 0.8701 0.940 0.000 0.060
#> GSM348193 1 0.9411 -0.3564 0.444 0.176 0.380
#> GSM537578 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM348181 1 0.0592 0.8724 0.988 0.000 0.012
#> GSM348182 1 0.1529 0.8744 0.960 0.000 0.040
#> GSM348184 2 0.0000 0.8050 0.000 1.000 0.000
#> GSM348185 2 0.6309 -0.2972 0.000 0.504 0.496
#> GSM348186 2 0.0000 0.8050 0.000 1.000 0.000
#> GSM348187 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM348188 1 0.9189 -0.2185 0.492 0.160 0.348
#> GSM348189 1 0.3340 0.8268 0.880 0.000 0.120
#> GSM348190 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM348194 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM348195 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM348196 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM537585 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM537594 2 0.0237 0.8039 0.000 0.996 0.004
#> GSM537596 1 0.0424 0.8737 0.992 0.000 0.008
#> GSM537597 2 0.4629 0.7059 0.004 0.808 0.188
#> GSM537602 1 0.1031 0.8722 0.976 0.000 0.024
#> GSM340184 1 0.2448 0.8592 0.924 0.000 0.076
#> GSM340185 2 0.0000 0.8050 0.000 1.000 0.000
#> GSM340186 2 0.0000 0.8050 0.000 1.000 0.000
#> GSM340187 2 0.6204 -0.1302 0.000 0.576 0.424
#> GSM340189 1 0.9201 -0.2303 0.488 0.160 0.352
#> GSM340190 2 0.0000 0.8050 0.000 1.000 0.000
#> GSM340191 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM340192 2 0.4887 0.6814 0.000 0.772 0.228
#> GSM340193 1 0.1753 0.8714 0.952 0.000 0.048
#> GSM340194 1 0.2356 0.8643 0.928 0.000 0.072
#> GSM340195 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM340196 3 0.8948 0.6099 0.188 0.248 0.564
#> GSM340197 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM340198 2 0.0000 0.8050 0.000 1.000 0.000
#> GSM340199 1 0.0592 0.8721 0.988 0.000 0.012
#> GSM340200 2 0.0237 0.8039 0.000 0.996 0.004
#> GSM340201 3 0.8917 0.6120 0.188 0.244 0.568
#> GSM340202 3 0.8821 0.6163 0.188 0.232 0.580
#> GSM340203 1 0.8371 0.2374 0.624 0.164 0.212
#> GSM340204 2 0.5254 0.6538 0.000 0.736 0.264
#> GSM340205 1 0.1315 0.8706 0.972 0.008 0.020
#> GSM340206 1 0.1860 0.8703 0.948 0.000 0.052
#> GSM340207 1 0.2261 0.8666 0.932 0.000 0.068
#> GSM340237 2 0.3941 0.6524 0.000 0.844 0.156
#> GSM340238 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM340239 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM340240 1 0.1964 0.8715 0.944 0.000 0.056
#> GSM340241 2 0.0000 0.8050 0.000 1.000 0.000
#> GSM340242 1 0.2711 0.8540 0.912 0.000 0.088
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.6992 0.6187 0.564 0.000 0.156 0.280
#> GSM340359 1 0.5235 0.6513 0.716 0.048 0.000 0.236
#> GSM340361 3 0.6457 0.7280 0.156 0.000 0.644 0.200
#> GSM340362 3 0.4544 0.8160 0.048 0.000 0.788 0.164
#> GSM340363 3 0.5408 0.4551 0.008 0.004 0.556 0.432
#> GSM340364 1 0.5156 0.6540 0.720 0.044 0.000 0.236
#> GSM340365 3 0.5637 0.7842 0.112 0.000 0.720 0.168
#> GSM340366 3 0.5637 0.7842 0.112 0.000 0.720 0.168
#> GSM340367 1 0.7660 0.4836 0.464 0.000 0.276 0.260
#> GSM340368 4 0.7663 -0.1863 0.380 0.212 0.000 0.408
#> GSM340369 4 0.6539 -0.0642 0.348 0.008 0.068 0.576
#> GSM340370 1 0.4295 0.6658 0.752 0.008 0.000 0.240
#> GSM340371 3 0.0524 0.8424 0.004 0.000 0.988 0.008
#> GSM340372 1 0.7442 0.5653 0.504 0.000 0.212 0.284
#> GSM340373 1 0.7645 0.5005 0.468 0.000 0.268 0.264
#> GSM340375 1 0.4295 0.6658 0.752 0.008 0.000 0.240
#> GSM340376 1 0.4295 0.6658 0.752 0.008 0.000 0.240
#> GSM340378 1 0.5156 0.6540 0.720 0.044 0.000 0.236
#> GSM340243 3 0.6149 0.7572 0.144 0.000 0.676 0.180
#> GSM340244 2 0.0592 0.8444 0.016 0.984 0.000 0.000
#> GSM340246 2 0.0000 0.8460 0.000 1.000 0.000 0.000
#> GSM340247 4 0.7315 0.2139 0.216 0.252 0.000 0.532
#> GSM340248 2 0.0000 0.8460 0.000 1.000 0.000 0.000
#> GSM340249 2 0.6050 0.4684 0.112 0.676 0.000 0.212
#> GSM340250 3 0.5690 0.7824 0.116 0.000 0.716 0.168
#> GSM340251 2 0.1888 0.8244 0.044 0.940 0.000 0.016
#> GSM340252 2 0.5593 0.5138 0.080 0.708 0.000 0.212
#> GSM340253 2 0.1302 0.8341 0.044 0.956 0.000 0.000
#> GSM340254 2 0.0000 0.8460 0.000 1.000 0.000 0.000
#> GSM340256 2 0.0469 0.8452 0.012 0.988 0.000 0.000
#> GSM340258 2 0.6955 0.5435 0.204 0.616 0.172 0.008
#> GSM340259 3 0.0524 0.8431 0.004 0.000 0.988 0.008
#> GSM340260 3 0.4944 0.8119 0.072 0.000 0.768 0.160
#> GSM340261 3 0.0188 0.8440 0.000 0.000 0.996 0.004
#> GSM340262 3 0.0672 0.8429 0.008 0.000 0.984 0.008
#> GSM340263 2 0.1209 0.8385 0.032 0.964 0.000 0.004
#> GSM340264 3 0.0188 0.8440 0.004 0.000 0.996 0.000
#> GSM340265 2 0.7308 0.2456 0.124 0.496 0.372 0.008
#> GSM340266 3 0.0336 0.8435 0.008 0.000 0.992 0.000
#> GSM340267 2 0.7455 0.3607 0.168 0.532 0.292 0.008
#> GSM340268 3 0.1256 0.8359 0.028 0.000 0.964 0.008
#> GSM340269 3 0.1584 0.8239 0.036 0.000 0.952 0.012
#> GSM340270 3 0.3948 0.8296 0.036 0.000 0.828 0.136
#> GSM537574 2 0.5152 0.7062 0.204 0.748 0.036 0.012
#> GSM537580 3 0.1059 0.8367 0.016 0.000 0.972 0.012
#> GSM537581 4 0.4524 0.5618 0.052 0.012 0.120 0.816
#> GSM340272 3 0.5412 0.7928 0.096 0.000 0.736 0.168
#> GSM340273 2 0.1389 0.8304 0.048 0.952 0.000 0.000
#> GSM340275 4 0.7385 0.2029 0.196 0.296 0.000 0.508
#> GSM340276 3 0.0657 0.8417 0.004 0.000 0.984 0.012
#> GSM340277 2 0.0000 0.8460 0.000 1.000 0.000 0.000
#> GSM340278 3 0.1510 0.8456 0.028 0.000 0.956 0.016
#> GSM340279 3 0.0672 0.8419 0.008 0.000 0.984 0.008
#> GSM340282 3 0.1256 0.8468 0.008 0.000 0.964 0.028
#> GSM340284 2 0.0524 0.8454 0.004 0.988 0.000 0.008
#> GSM340285 3 0.4379 0.8103 0.036 0.000 0.792 0.172
#> GSM340286 3 0.0804 0.8400 0.008 0.000 0.980 0.012
#> GSM340287 3 0.1004 0.8352 0.024 0.000 0.972 0.004
#> GSM340288 3 0.6924 0.6669 0.180 0.000 0.588 0.232
#> GSM340289 3 0.4578 0.8145 0.052 0.000 0.788 0.160
#> GSM340290 3 0.3015 0.8386 0.024 0.000 0.884 0.092
#> GSM340291 2 0.0592 0.8444 0.016 0.984 0.000 0.000
#> GSM340293 3 0.4872 0.7899 0.024 0.012 0.760 0.204
#> GSM340294 3 0.0804 0.8400 0.008 0.000 0.980 0.012
#> GSM340296 3 0.6245 0.7511 0.164 0.000 0.668 0.168
#> GSM340297 3 0.4105 0.8197 0.032 0.000 0.812 0.156
#> GSM340298 3 0.6119 0.7601 0.152 0.000 0.680 0.168
#> GSM340299 3 0.0672 0.8429 0.008 0.000 0.984 0.008
#> GSM340301 3 0.0804 0.8422 0.008 0.000 0.980 0.012
#> GSM340303 2 0.6857 0.5338 0.204 0.616 0.176 0.004
#> GSM340304 3 0.4008 0.8229 0.032 0.000 0.820 0.148
#> GSM340306 4 0.5052 0.5664 0.040 0.132 0.036 0.792
#> GSM340307 2 0.1389 0.8323 0.048 0.952 0.000 0.000
#> GSM340310 1 0.7015 0.6229 0.568 0.000 0.168 0.264
#> GSM340314 3 0.2401 0.8306 0.004 0.000 0.904 0.092
#> GSM340315 3 0.5065 0.8109 0.072 0.016 0.788 0.124
#> GSM340317 2 0.5964 0.4841 0.108 0.684 0.000 0.208
#> GSM340318 4 0.2587 0.6149 0.028 0.040 0.012 0.920
#> GSM340319 4 0.2587 0.6149 0.028 0.040 0.012 0.920
#> GSM340320 4 0.4244 0.5368 0.000 0.036 0.160 0.804
#> GSM340321 2 0.0000 0.8460 0.000 1.000 0.000 0.000
#> GSM340322 4 0.3463 0.6309 0.000 0.040 0.096 0.864
#> GSM340324 2 0.3710 0.7040 0.192 0.804 0.000 0.004
#> GSM340328 1 0.7178 0.6270 0.572 0.008 0.148 0.272
#> GSM340330 3 0.0804 0.8400 0.008 0.000 0.980 0.012
#> GSM340332 4 0.3758 0.6270 0.000 0.048 0.104 0.848
#> GSM340333 2 0.1302 0.8341 0.044 0.956 0.000 0.000
#> GSM340336 4 0.7434 0.1829 0.232 0.256 0.000 0.512
#> GSM340337 4 0.3399 0.6331 0.000 0.040 0.092 0.868
#> GSM340338 3 0.3088 0.8340 0.008 0.000 0.864 0.128
#> GSM340339 4 0.3128 0.6351 0.000 0.040 0.076 0.884
#> GSM340340 4 0.7433 0.0804 0.276 0.216 0.000 0.508
#> GSM340341 4 0.6906 0.2123 0.108 0.408 0.000 0.484
#> GSM340343 4 0.3525 0.6278 0.000 0.040 0.100 0.860
#> GSM340344 2 0.3636 0.7585 0.172 0.820 0.000 0.008
#> GSM340346 3 0.0524 0.8424 0.004 0.000 0.988 0.008
#> GSM340347 4 0.2376 0.6173 0.020 0.040 0.012 0.928
#> GSM340348 4 0.4139 0.5652 0.000 0.040 0.144 0.816
#> GSM340349 3 0.6245 0.7511 0.164 0.000 0.668 0.168
#> GSM340350 3 0.5938 0.7705 0.136 0.000 0.696 0.168
#> GSM340351 3 0.6719 0.7004 0.180 0.000 0.616 0.204
#> GSM340354 2 0.7425 0.4215 0.192 0.552 0.248 0.008
#> GSM340356 2 0.1118 0.8368 0.036 0.964 0.000 0.000
#> GSM340357 3 0.5985 0.7678 0.140 0.000 0.692 0.168
#> GSM348183 3 0.5133 0.7374 0.024 0.004 0.704 0.268
#> GSM348191 3 0.0376 0.8434 0.004 0.000 0.992 0.004
#> GSM348193 4 0.3587 0.6082 0.004 0.032 0.104 0.860
#> GSM537578 3 0.6245 0.7511 0.164 0.000 0.668 0.168
#> GSM348181 3 0.4008 0.8219 0.032 0.000 0.820 0.148
#> GSM348182 3 0.0804 0.8450 0.008 0.000 0.980 0.012
#> GSM348184 2 0.0188 0.8460 0.004 0.996 0.000 0.000
#> GSM348185 2 0.6634 0.2252 0.108 0.580 0.000 0.312
#> GSM348186 2 0.0000 0.8460 0.000 1.000 0.000 0.000
#> GSM348187 3 0.6750 0.6957 0.180 0.000 0.612 0.208
#> GSM348188 4 0.3647 0.6200 0.000 0.040 0.108 0.852
#> GSM348189 3 0.0804 0.8401 0.012 0.000 0.980 0.008
#> GSM348190 3 0.0804 0.8422 0.008 0.000 0.980 0.012
#> GSM348194 3 0.6950 0.6614 0.180 0.000 0.584 0.236
#> GSM348195 3 0.6931 0.6682 0.184 0.000 0.588 0.228
#> GSM348196 3 0.6788 0.6600 0.144 0.000 0.592 0.264
#> GSM537585 3 0.5096 0.8098 0.084 0.000 0.760 0.156
#> GSM537594 2 0.0707 0.8433 0.020 0.980 0.000 0.000
#> GSM537596 3 0.4387 0.8218 0.052 0.000 0.804 0.144
#> GSM537597 2 0.5096 0.7289 0.176 0.768 0.036 0.020
#> GSM537602 3 0.5938 0.7718 0.136 0.000 0.696 0.168
#> GSM340184 3 0.1296 0.8335 0.028 0.004 0.964 0.004
#> GSM340185 2 0.0188 0.8460 0.004 0.996 0.000 0.000
#> GSM340186 2 0.0188 0.8460 0.004 0.996 0.000 0.000
#> GSM340187 4 0.6850 0.2354 0.108 0.376 0.000 0.516
#> GSM340189 4 0.3399 0.6332 0.000 0.040 0.092 0.868
#> GSM340190 2 0.0188 0.8460 0.004 0.996 0.000 0.000
#> GSM340191 3 0.0336 0.8435 0.008 0.000 0.992 0.000
#> GSM340192 2 0.3591 0.7608 0.168 0.824 0.000 0.008
#> GSM340193 3 0.1247 0.8457 0.016 0.004 0.968 0.012
#> GSM340194 3 0.0804 0.8422 0.008 0.000 0.980 0.012
#> GSM340195 3 0.0804 0.8422 0.008 0.000 0.980 0.012
#> GSM340196 4 0.2587 0.6149 0.028 0.040 0.012 0.920
#> GSM340197 3 0.0804 0.8422 0.008 0.000 0.980 0.012
#> GSM340198 2 0.0188 0.8460 0.004 0.996 0.000 0.000
#> GSM340199 3 0.3552 0.8301 0.024 0.000 0.848 0.128
#> GSM340200 2 0.1118 0.8371 0.036 0.964 0.000 0.000
#> GSM340201 4 0.2587 0.6149 0.028 0.040 0.012 0.920
#> GSM340202 4 0.2587 0.6149 0.028 0.040 0.012 0.920
#> GSM340203 4 0.5522 0.5344 0.000 0.120 0.148 0.732
#> GSM340204 2 0.4255 0.7356 0.200 0.784 0.008 0.008
#> GSM340205 3 0.5979 0.7713 0.136 0.000 0.692 0.172
#> GSM340206 3 0.1109 0.8338 0.028 0.000 0.968 0.004
#> GSM340207 3 0.0524 0.8424 0.004 0.000 0.988 0.008
#> GSM340237 2 0.4011 0.6799 0.208 0.784 0.000 0.008
#> GSM340238 3 0.0524 0.8428 0.008 0.000 0.988 0.004
#> GSM340239 3 0.0804 0.8422 0.008 0.000 0.980 0.012
#> GSM340240 3 0.0804 0.8422 0.008 0.000 0.980 0.012
#> GSM340241 2 0.0000 0.8460 0.000 1.000 0.000 0.000
#> GSM340242 3 0.0672 0.8416 0.008 0.000 0.984 0.008
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.4880 0.55568 0.616 0.000 0.348 0.036 0.000
#> GSM340359 1 0.0404 0.64471 0.988 0.012 0.000 0.000 0.000
#> GSM340361 3 0.0955 0.68143 0.000 0.000 0.968 0.028 0.004
#> GSM340362 3 0.3074 0.56586 0.000 0.000 0.804 0.000 0.196
#> GSM340363 3 0.4491 0.25921 0.000 0.000 0.652 0.328 0.020
#> GSM340364 1 0.0290 0.64636 0.992 0.008 0.000 0.000 0.000
#> GSM340365 3 0.1121 0.69402 0.000 0.000 0.956 0.000 0.044
#> GSM340366 3 0.1478 0.68199 0.000 0.000 0.936 0.000 0.064
#> GSM340367 1 0.4787 0.58680 0.640 0.000 0.324 0.036 0.000
#> GSM340368 4 0.6588 -0.00227 0.372 0.096 0.000 0.496 0.036
#> GSM340369 1 0.6881 0.35321 0.460 0.000 0.304 0.224 0.012
#> GSM340370 1 0.0000 0.64656 1.000 0.000 0.000 0.000 0.000
#> GSM340371 5 0.4283 0.60413 0.000 0.000 0.456 0.000 0.544
#> GSM340372 1 0.4378 0.62710 0.716 0.000 0.248 0.036 0.000
#> GSM340373 1 0.4503 0.62106 0.696 0.000 0.268 0.036 0.000
#> GSM340375 1 0.0162 0.64620 0.996 0.000 0.000 0.000 0.004
#> GSM340376 1 0.0000 0.64656 1.000 0.000 0.000 0.000 0.000
#> GSM340378 1 0.0290 0.64636 0.992 0.008 0.000 0.000 0.000
#> GSM340243 3 0.0451 0.69081 0.000 0.000 0.988 0.004 0.008
#> GSM340244 2 0.0404 0.91864 0.000 0.988 0.000 0.012 0.000
#> GSM340246 2 0.0290 0.91925 0.000 0.992 0.000 0.000 0.008
#> GSM340247 4 0.3110 0.53573 0.004 0.112 0.000 0.856 0.028
#> GSM340248 2 0.0290 0.91925 0.000 0.992 0.000 0.000 0.008
#> GSM340249 2 0.3989 0.72724 0.012 0.784 0.000 0.180 0.024
#> GSM340250 3 0.0290 0.69296 0.000 0.000 0.992 0.000 0.008
#> GSM340251 2 0.1357 0.89673 0.000 0.948 0.000 0.048 0.004
#> GSM340252 2 0.3365 0.74891 0.004 0.808 0.000 0.180 0.008
#> GSM340253 2 0.0290 0.91925 0.000 0.992 0.000 0.000 0.008
#> GSM340254 2 0.0162 0.91984 0.000 0.996 0.000 0.000 0.004
#> GSM340256 2 0.0000 0.91983 0.000 1.000 0.000 0.000 0.000
#> GSM340258 5 0.4526 -0.01597 0.004 0.260 0.024 0.004 0.708
#> GSM340259 5 0.4242 0.61369 0.000 0.000 0.428 0.000 0.572
#> GSM340260 3 0.2929 0.61805 0.000 0.000 0.820 0.000 0.180
#> GSM340261 5 0.4294 0.58559 0.000 0.000 0.468 0.000 0.532
#> GSM340262 5 0.4268 0.61428 0.000 0.000 0.444 0.000 0.556
#> GSM340263 2 0.0609 0.91635 0.000 0.980 0.000 0.020 0.000
#> GSM340264 5 0.4304 0.55798 0.000 0.000 0.484 0.000 0.516
#> GSM340265 5 0.4821 -0.01594 0.000 0.272 0.044 0.004 0.680
#> GSM340266 3 0.4307 -0.51762 0.000 0.000 0.500 0.000 0.500
#> GSM340267 5 0.4711 0.01219 0.004 0.252 0.036 0.004 0.704
#> GSM340268 5 0.4249 0.57759 0.000 0.000 0.432 0.000 0.568
#> GSM340269 5 0.4161 0.57628 0.000 0.000 0.392 0.000 0.608
#> GSM340270 3 0.4480 0.27846 0.004 0.000 0.592 0.004 0.400
#> GSM537574 5 0.4770 -0.36597 0.004 0.412 0.004 0.008 0.572
#> GSM537580 5 0.4262 0.61117 0.000 0.000 0.440 0.000 0.560
#> GSM537581 4 0.5036 0.50949 0.032 0.000 0.372 0.592 0.004
#> GSM340272 3 0.1851 0.68119 0.000 0.000 0.912 0.000 0.088
#> GSM340273 2 0.0451 0.91833 0.000 0.988 0.000 0.008 0.004
#> GSM340275 4 0.4706 0.47951 0.048 0.152 0.000 0.764 0.036
#> GSM340276 5 0.4305 0.51848 0.000 0.000 0.488 0.000 0.512
#> GSM340277 2 0.0162 0.91984 0.000 0.996 0.000 0.000 0.004
#> GSM340278 3 0.4304 -0.48438 0.000 0.000 0.516 0.000 0.484
#> GSM340279 3 0.4305 -0.49428 0.000 0.000 0.512 0.000 0.488
#> GSM340282 5 0.4304 0.53555 0.000 0.000 0.484 0.000 0.516
#> GSM340284 2 0.1211 0.91119 0.000 0.960 0.000 0.024 0.016
#> GSM340285 3 0.4808 0.10215 0.000 0.000 0.620 0.032 0.348
#> GSM340286 5 0.4273 0.61035 0.000 0.000 0.448 0.000 0.552
#> GSM340287 5 0.4182 0.55781 0.000 0.000 0.400 0.000 0.600
#> GSM340288 3 0.0963 0.67533 0.000 0.000 0.964 0.036 0.000
#> GSM340289 3 0.2966 0.58366 0.000 0.000 0.816 0.000 0.184
#> GSM340290 3 0.4138 -0.17986 0.000 0.000 0.616 0.000 0.384
#> GSM340291 2 0.0290 0.91929 0.000 0.992 0.000 0.008 0.000
#> GSM340293 3 0.6350 0.23956 0.000 0.000 0.524 0.236 0.240
#> GSM340294 5 0.4256 0.61761 0.000 0.000 0.436 0.000 0.564
#> GSM340296 3 0.1121 0.69209 0.000 0.000 0.956 0.000 0.044
#> GSM340297 3 0.2230 0.64589 0.000 0.000 0.884 0.000 0.116
#> GSM340298 3 0.1197 0.69012 0.000 0.000 0.952 0.000 0.048
#> GSM340299 5 0.4278 0.60788 0.000 0.000 0.452 0.000 0.548
#> GSM340301 5 0.4268 0.61375 0.000 0.000 0.444 0.000 0.556
#> GSM340303 5 0.5188 -0.15570 0.004 0.320 0.036 0.008 0.632
#> GSM340304 3 0.2848 0.62677 0.004 0.000 0.840 0.000 0.156
#> GSM340306 4 0.3716 0.59852 0.000 0.012 0.152 0.812 0.024
#> GSM340307 2 0.0404 0.91821 0.000 0.988 0.000 0.000 0.012
#> GSM340310 1 0.4866 0.56325 0.620 0.000 0.344 0.036 0.000
#> GSM340314 3 0.4743 -0.40953 0.000 0.000 0.512 0.016 0.472
#> GSM340315 3 0.5140 0.26777 0.004 0.012 0.520 0.012 0.452
#> GSM340317 2 0.3381 0.75089 0.000 0.808 0.000 0.176 0.016
#> GSM340318 4 0.0510 0.59597 0.000 0.000 0.016 0.984 0.000
#> GSM340319 4 0.0510 0.59597 0.000 0.000 0.016 0.984 0.000
#> GSM340320 4 0.4264 0.51824 0.000 0.000 0.376 0.620 0.004
#> GSM340321 2 0.0290 0.91925 0.000 0.992 0.000 0.000 0.008
#> GSM340322 4 0.4047 0.57391 0.000 0.000 0.320 0.676 0.004
#> GSM340324 2 0.2312 0.86714 0.060 0.912 0.000 0.016 0.012
#> GSM340328 1 0.4880 0.55568 0.616 0.000 0.348 0.036 0.000
#> GSM340330 5 0.4305 0.53062 0.000 0.000 0.488 0.000 0.512
#> GSM340332 4 0.4211 0.54838 0.000 0.000 0.360 0.636 0.004
#> GSM340333 2 0.0290 0.91925 0.000 0.992 0.000 0.000 0.008
#> GSM340336 4 0.4559 0.49346 0.060 0.120 0.000 0.784 0.036
#> GSM340337 4 0.4066 0.57299 0.000 0.000 0.324 0.672 0.004
#> GSM340338 3 0.4225 0.10453 0.000 0.000 0.632 0.004 0.364
#> GSM340339 4 0.3790 0.58411 0.000 0.000 0.272 0.724 0.004
#> GSM340340 4 0.4729 0.48601 0.068 0.124 0.000 0.772 0.036
#> GSM340341 4 0.4223 0.44250 0.000 0.248 0.000 0.724 0.028
#> GSM340343 4 0.4182 0.55522 0.000 0.000 0.352 0.644 0.004
#> GSM340344 2 0.4414 0.56626 0.004 0.616 0.000 0.004 0.376
#> GSM340346 5 0.4273 0.61060 0.000 0.000 0.448 0.000 0.552
#> GSM340347 4 0.0510 0.59597 0.000 0.000 0.016 0.984 0.000
#> GSM340348 4 0.4402 0.52819 0.000 0.004 0.372 0.620 0.004
#> GSM340349 3 0.1205 0.69457 0.000 0.000 0.956 0.004 0.040
#> GSM340350 3 0.1197 0.69277 0.000 0.000 0.952 0.000 0.048
#> GSM340351 3 0.0880 0.67726 0.000 0.000 0.968 0.032 0.000
#> GSM340354 5 0.4636 0.00684 0.004 0.252 0.032 0.004 0.708
#> GSM340356 2 0.0290 0.91929 0.000 0.992 0.000 0.008 0.000
#> GSM340357 3 0.0162 0.69194 0.000 0.000 0.996 0.000 0.004
#> GSM348183 3 0.4364 0.52711 0.000 0.000 0.736 0.216 0.048
#> GSM348191 5 0.4297 0.56601 0.000 0.000 0.472 0.000 0.528
#> GSM348193 4 0.4101 0.53085 0.000 0.000 0.372 0.628 0.000
#> GSM537578 3 0.0963 0.69431 0.000 0.000 0.964 0.000 0.036
#> GSM348181 3 0.2690 0.60517 0.000 0.000 0.844 0.000 0.156
#> GSM348182 5 0.4287 0.58855 0.000 0.000 0.460 0.000 0.540
#> GSM348184 2 0.0162 0.92006 0.000 0.996 0.000 0.004 0.000
#> GSM348185 2 0.3829 0.71778 0.000 0.776 0.000 0.196 0.028
#> GSM348186 2 0.0290 0.91925 0.000 0.992 0.000 0.000 0.008
#> GSM348187 3 0.0880 0.67726 0.000 0.000 0.968 0.032 0.000
#> GSM348188 4 0.4238 0.53682 0.000 0.000 0.368 0.628 0.004
#> GSM348189 5 0.4256 0.61238 0.000 0.000 0.436 0.000 0.564
#> GSM348190 5 0.4262 0.61666 0.000 0.000 0.440 0.000 0.560
#> GSM348194 3 0.0963 0.67533 0.000 0.000 0.964 0.036 0.000
#> GSM348195 3 0.0963 0.67533 0.000 0.000 0.964 0.036 0.000
#> GSM348196 3 0.0963 0.67533 0.000 0.000 0.964 0.036 0.000
#> GSM537585 3 0.3508 0.56774 0.000 0.000 0.748 0.000 0.252
#> GSM537594 2 0.0290 0.91929 0.000 0.992 0.000 0.008 0.000
#> GSM537596 3 0.3656 0.53580 0.000 0.000 0.784 0.020 0.196
#> GSM537597 2 0.6836 0.28418 0.008 0.424 0.132 0.016 0.420
#> GSM537602 3 0.1341 0.68874 0.000 0.000 0.944 0.000 0.056
#> GSM340184 5 0.4341 0.57445 0.000 0.004 0.404 0.000 0.592
#> GSM340185 2 0.0451 0.91960 0.000 0.988 0.000 0.004 0.008
#> GSM340186 2 0.0324 0.92017 0.000 0.992 0.000 0.004 0.004
#> GSM340187 4 0.3687 0.49474 0.000 0.180 0.000 0.792 0.028
#> GSM340189 4 0.4084 0.57104 0.000 0.000 0.328 0.668 0.004
#> GSM340190 2 0.0451 0.91960 0.000 0.988 0.000 0.004 0.008
#> GSM340191 5 0.4302 0.55612 0.000 0.000 0.480 0.000 0.520
#> GSM340192 2 0.4362 0.58666 0.004 0.632 0.000 0.004 0.360
#> GSM340193 5 0.4446 0.56646 0.000 0.000 0.476 0.004 0.520
#> GSM340194 5 0.4262 0.61666 0.000 0.000 0.440 0.000 0.560
#> GSM340195 5 0.4268 0.61094 0.000 0.000 0.444 0.000 0.556
#> GSM340196 4 0.0510 0.59597 0.000 0.000 0.016 0.984 0.000
#> GSM340197 5 0.4256 0.61761 0.000 0.000 0.436 0.000 0.564
#> GSM340198 2 0.0807 0.91853 0.000 0.976 0.000 0.012 0.012
#> GSM340199 3 0.3895 0.16240 0.000 0.000 0.680 0.000 0.320
#> GSM340200 2 0.0807 0.91415 0.000 0.976 0.000 0.012 0.012
#> GSM340201 4 0.0510 0.59597 0.000 0.000 0.016 0.984 0.000
#> GSM340202 4 0.0510 0.59597 0.000 0.000 0.016 0.984 0.000
#> GSM340203 4 0.4457 0.53969 0.000 0.012 0.368 0.620 0.000
#> GSM340204 5 0.4675 -0.42201 0.004 0.444 0.000 0.008 0.544
#> GSM340205 3 0.2011 0.66630 0.004 0.000 0.908 0.000 0.088
#> GSM340206 5 0.4375 0.57540 0.000 0.004 0.420 0.000 0.576
#> GSM340207 5 0.4278 0.60788 0.000 0.000 0.452 0.000 0.548
#> GSM340237 2 0.2312 0.86714 0.060 0.912 0.000 0.016 0.012
#> GSM340238 5 0.4307 0.47936 0.000 0.000 0.500 0.000 0.500
#> GSM340239 5 0.4256 0.61761 0.000 0.000 0.436 0.000 0.564
#> GSM340240 5 0.4256 0.61761 0.000 0.000 0.436 0.000 0.564
#> GSM340241 2 0.0290 0.91925 0.000 0.992 0.000 0.000 0.008
#> GSM340242 5 0.4268 0.61516 0.000 0.000 0.444 0.000 0.556
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.4246 0.3837 0.580 0.000 0.400 0.020 0.000 0.000
#> GSM340359 1 0.0622 0.5065 0.980 0.012 0.000 0.000 0.000 0.008
#> GSM340361 3 0.1088 0.6556 0.016 0.000 0.960 0.024 0.000 0.000
#> GSM340362 3 0.3161 0.5377 0.000 0.000 0.776 0.008 0.216 0.000
#> GSM340363 3 0.6073 0.2403 0.016 0.000 0.552 0.280 0.016 0.136
#> GSM340364 1 0.0622 0.5065 0.980 0.012 0.000 0.000 0.000 0.008
#> GSM340365 3 0.0603 0.6659 0.000 0.000 0.980 0.004 0.016 0.000
#> GSM340366 3 0.0909 0.6656 0.000 0.000 0.968 0.012 0.020 0.000
#> GSM340367 1 0.4116 0.3512 0.572 0.000 0.416 0.012 0.000 0.000
#> GSM340368 6 0.4495 0.1120 0.388 0.028 0.000 0.004 0.000 0.580
#> GSM340369 1 0.6177 -0.1705 0.448 0.000 0.056 0.404 0.000 0.092
#> GSM340370 1 0.0146 0.5132 0.996 0.000 0.000 0.004 0.000 0.000
#> GSM340371 5 0.3955 0.4225 0.000 0.000 0.436 0.004 0.560 0.000
#> GSM340372 1 0.4176 0.3808 0.580 0.000 0.404 0.016 0.000 0.000
#> GSM340373 1 0.4057 0.4116 0.600 0.000 0.388 0.012 0.000 0.000
#> GSM340375 1 0.0603 0.5110 0.980 0.000 0.000 0.004 0.016 0.000
#> GSM340376 1 0.0146 0.5132 0.996 0.000 0.000 0.004 0.000 0.000
#> GSM340378 1 0.0622 0.5065 0.980 0.012 0.000 0.000 0.000 0.008
#> GSM340243 3 0.0363 0.6668 0.000 0.000 0.988 0.012 0.000 0.000
#> GSM340244 2 0.2201 0.8668 0.004 0.904 0.000 0.036 0.000 0.056
#> GSM340246 2 0.0964 0.8672 0.000 0.968 0.000 0.004 0.012 0.016
#> GSM340247 6 0.2221 0.4957 0.000 0.032 0.000 0.072 0.000 0.896
#> GSM340248 2 0.1930 0.8727 0.000 0.924 0.000 0.036 0.012 0.028
#> GSM340249 2 0.4201 0.6738 0.000 0.664 0.000 0.036 0.000 0.300
#> GSM340250 3 0.0260 0.6668 0.000 0.000 0.992 0.008 0.000 0.000
#> GSM340251 2 0.1501 0.8664 0.000 0.924 0.000 0.000 0.000 0.076
#> GSM340252 2 0.4087 0.7014 0.000 0.688 0.000 0.036 0.000 0.276
#> GSM340253 2 0.0909 0.8668 0.000 0.968 0.000 0.000 0.012 0.020
#> GSM340254 2 0.0862 0.8763 0.000 0.972 0.000 0.004 0.008 0.016
#> GSM340256 2 0.1370 0.8761 0.000 0.948 0.000 0.036 0.004 0.012
#> GSM340258 5 0.5948 -0.1134 0.004 0.104 0.012 0.228 0.616 0.036
#> GSM340259 5 0.3993 0.3671 0.000 0.000 0.476 0.004 0.520 0.000
#> GSM340260 3 0.2980 0.5595 0.000 0.000 0.800 0.008 0.192 0.000
#> GSM340261 5 0.3999 0.3054 0.000 0.000 0.496 0.004 0.500 0.000
#> GSM340262 5 0.3955 0.4225 0.000 0.000 0.436 0.004 0.560 0.000
#> GSM340263 2 0.2711 0.8590 0.000 0.872 0.000 0.036 0.008 0.084
#> GSM340264 3 0.3868 -0.3304 0.000 0.000 0.504 0.000 0.496 0.000
#> GSM340265 5 0.6169 -0.1246 0.004 0.128 0.012 0.228 0.592 0.036
#> GSM340266 5 0.5488 0.2431 0.000 0.000 0.396 0.128 0.476 0.000
#> GSM340267 5 0.5948 -0.1131 0.004 0.104 0.012 0.228 0.616 0.036
#> GSM340268 3 0.4830 -0.3041 0.000 0.004 0.484 0.028 0.476 0.008
#> GSM340269 5 0.4736 0.3346 0.000 0.000 0.432 0.032 0.528 0.008
#> GSM340270 3 0.5270 0.3999 0.004 0.000 0.604 0.092 0.292 0.008
#> GSM537574 5 0.6648 -0.2230 0.004 0.192 0.004 0.252 0.504 0.044
#> GSM537580 5 0.4098 0.4133 0.000 0.000 0.444 0.004 0.548 0.004
#> GSM537581 4 0.5524 0.6440 0.024 0.000 0.072 0.508 0.000 0.396
#> GSM340272 3 0.2100 0.6297 0.000 0.000 0.884 0.004 0.112 0.000
#> GSM340273 2 0.2217 0.8696 0.004 0.908 0.000 0.036 0.004 0.048
#> GSM340275 6 0.2264 0.5226 0.012 0.096 0.000 0.004 0.000 0.888
#> GSM340276 3 0.4532 -0.2902 0.000 0.000 0.500 0.032 0.468 0.000
#> GSM340277 2 0.0820 0.8771 0.000 0.972 0.000 0.000 0.012 0.016
#> GSM340278 5 0.5392 0.2103 0.000 0.000 0.440 0.112 0.448 0.000
#> GSM340279 5 0.4722 0.3167 0.000 0.000 0.468 0.036 0.492 0.004
#> GSM340282 5 0.4179 0.3034 0.000 0.000 0.472 0.012 0.516 0.000
#> GSM340284 2 0.4261 0.6670 0.000 0.692 0.000 0.000 0.056 0.252
#> GSM340285 3 0.4798 0.2745 0.000 0.000 0.612 0.076 0.312 0.000
#> GSM340286 5 0.3989 0.3740 0.000 0.000 0.468 0.004 0.528 0.000
#> GSM340287 5 0.4325 0.3187 0.000 0.000 0.456 0.020 0.524 0.000
#> GSM340288 3 0.1148 0.6573 0.016 0.000 0.960 0.020 0.004 0.000
#> GSM340289 3 0.2980 0.5550 0.000 0.000 0.800 0.008 0.192 0.000
#> GSM340290 3 0.4955 -0.0256 0.000 0.000 0.548 0.060 0.388 0.004
#> GSM340291 2 0.1860 0.8727 0.004 0.928 0.000 0.036 0.004 0.028
#> GSM340293 4 0.6368 0.2509 0.000 0.000 0.168 0.532 0.244 0.056
#> GSM340294 5 0.4051 0.4209 0.000 0.000 0.432 0.008 0.560 0.000
#> GSM340296 3 0.0405 0.6664 0.000 0.000 0.988 0.008 0.004 0.000
#> GSM340297 3 0.3023 0.5945 0.000 0.000 0.828 0.032 0.140 0.000
#> GSM340298 3 0.0260 0.6668 0.000 0.000 0.992 0.008 0.000 0.000
#> GSM340299 5 0.3833 0.4136 0.000 0.000 0.444 0.000 0.556 0.000
#> GSM340301 5 0.3955 0.4225 0.000 0.000 0.436 0.004 0.560 0.000
#> GSM340303 5 0.6655 -0.1841 0.004 0.188 0.012 0.228 0.528 0.040
#> GSM340304 3 0.4574 0.4658 0.000 0.000 0.688 0.068 0.236 0.008
#> GSM340306 4 0.4524 0.6827 0.000 0.000 0.040 0.584 0.000 0.376
#> GSM340307 2 0.2288 0.8331 0.000 0.896 0.000 0.004 0.072 0.028
#> GSM340310 1 0.4254 0.3729 0.576 0.000 0.404 0.020 0.000 0.000
#> GSM340314 5 0.5961 0.0812 0.000 0.000 0.192 0.364 0.440 0.004
#> GSM340315 3 0.5484 0.3556 0.004 0.008 0.556 0.072 0.352 0.008
#> GSM340317 2 0.4024 0.7181 0.000 0.700 0.000 0.036 0.000 0.264
#> GSM340318 6 0.3991 -0.2573 0.000 0.000 0.004 0.472 0.000 0.524
#> GSM340319 6 0.3996 -0.2768 0.000 0.000 0.004 0.484 0.000 0.512
#> GSM340320 4 0.4720 0.8082 0.000 0.000 0.072 0.624 0.000 0.304
#> GSM340321 2 0.0405 0.8733 0.000 0.988 0.000 0.000 0.008 0.004
#> GSM340322 4 0.4674 0.7933 0.000 0.000 0.060 0.608 0.000 0.332
#> GSM340324 2 0.2911 0.8523 0.032 0.876 0.000 0.036 0.004 0.052
#> GSM340328 1 0.4246 0.3837 0.580 0.000 0.400 0.020 0.000 0.000
#> GSM340330 5 0.4093 0.4188 0.000 0.000 0.440 0.004 0.552 0.004
#> GSM340332 4 0.4637 0.8130 0.000 0.000 0.064 0.628 0.000 0.308
#> GSM340333 2 0.1053 0.8652 0.000 0.964 0.000 0.004 0.012 0.020
#> GSM340336 6 0.1881 0.5180 0.004 0.040 0.000 0.020 0.008 0.928
#> GSM340337 4 0.4601 0.8124 0.000 0.000 0.060 0.628 0.000 0.312
#> GSM340338 3 0.4788 0.3401 0.000 0.000 0.636 0.072 0.288 0.004
#> GSM340339 4 0.4563 0.8045 0.000 0.000 0.056 0.628 0.000 0.316
#> GSM340340 6 0.2594 0.5190 0.056 0.060 0.000 0.004 0.000 0.880
#> GSM340341 6 0.2848 0.4806 0.000 0.160 0.000 0.004 0.008 0.828
#> GSM340343 4 0.4601 0.8124 0.000 0.000 0.060 0.628 0.000 0.312
#> GSM340344 2 0.6610 0.2536 0.004 0.400 0.000 0.168 0.388 0.040
#> GSM340346 5 0.3955 0.4225 0.000 0.000 0.436 0.004 0.560 0.000
#> GSM340347 4 0.3997 0.2407 0.000 0.000 0.004 0.508 0.000 0.488
#> GSM340348 4 0.4767 0.8048 0.000 0.000 0.076 0.620 0.000 0.304
#> GSM340349 3 0.0000 0.6671 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340350 3 0.0363 0.6668 0.000 0.000 0.988 0.012 0.000 0.000
#> GSM340351 3 0.0964 0.6593 0.016 0.000 0.968 0.012 0.004 0.000
#> GSM340354 5 0.5948 -0.1131 0.004 0.104 0.012 0.228 0.616 0.036
#> GSM340356 2 0.2050 0.8724 0.004 0.920 0.000 0.036 0.008 0.032
#> GSM340357 3 0.0000 0.6671 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM348183 3 0.5883 0.3902 0.016 0.000 0.608 0.256 0.068 0.052
#> GSM348191 5 0.4127 0.3391 0.000 0.000 0.484 0.004 0.508 0.004
#> GSM348193 4 0.5924 0.5807 0.016 0.000 0.160 0.520 0.000 0.304
#> GSM537578 3 0.0260 0.6663 0.000 0.000 0.992 0.000 0.008 0.000
#> GSM348181 3 0.3947 0.4938 0.000 0.000 0.732 0.048 0.220 0.000
#> GSM348182 3 0.4797 -0.2260 0.000 0.000 0.504 0.052 0.444 0.000
#> GSM348184 2 0.1007 0.8746 0.000 0.956 0.000 0.000 0.000 0.044
#> GSM348185 2 0.4574 0.4661 0.000 0.524 0.000 0.036 0.000 0.440
#> GSM348186 2 0.0405 0.8733 0.000 0.988 0.000 0.000 0.008 0.004
#> GSM348187 3 0.1003 0.6566 0.016 0.000 0.964 0.020 0.000 0.000
#> GSM348188 4 0.4652 0.8124 0.000 0.000 0.064 0.624 0.000 0.312
#> GSM348189 5 0.3950 0.4212 0.000 0.000 0.432 0.004 0.564 0.000
#> GSM348190 5 0.3971 0.4093 0.000 0.000 0.448 0.004 0.548 0.000
#> GSM348194 3 0.1148 0.6573 0.016 0.000 0.960 0.020 0.004 0.000
#> GSM348195 3 0.1003 0.6566 0.016 0.000 0.964 0.020 0.000 0.000
#> GSM348196 3 0.1672 0.6398 0.016 0.000 0.932 0.048 0.004 0.000
#> GSM537585 3 0.3758 0.5093 0.000 0.000 0.700 0.016 0.284 0.000
#> GSM537594 2 0.1860 0.8727 0.004 0.928 0.000 0.036 0.004 0.028
#> GSM537596 3 0.3884 0.4849 0.000 0.000 0.724 0.036 0.240 0.000
#> GSM537597 5 0.8144 -0.2564 0.020 0.192 0.092 0.224 0.424 0.048
#> GSM537602 3 0.0665 0.6679 0.008 0.000 0.980 0.004 0.008 0.000
#> GSM340184 5 0.4598 0.3172 0.000 0.000 0.464 0.028 0.504 0.004
#> GSM340185 2 0.0291 0.8741 0.000 0.992 0.000 0.000 0.004 0.004
#> GSM340186 2 0.0865 0.8753 0.000 0.964 0.000 0.000 0.000 0.036
#> GSM340187 6 0.2734 0.5232 0.000 0.104 0.000 0.024 0.008 0.864
#> GSM340189 4 0.4601 0.8124 0.000 0.000 0.060 0.628 0.000 0.312
#> GSM340190 2 0.1007 0.8751 0.000 0.956 0.000 0.000 0.000 0.044
#> GSM340191 5 0.5188 0.2904 0.000 0.000 0.424 0.076 0.496 0.004
#> GSM340192 2 0.6342 0.3246 0.004 0.452 0.000 0.124 0.380 0.040
#> GSM340193 3 0.4331 -0.2712 0.000 0.000 0.516 0.020 0.464 0.000
#> GSM340194 5 0.3955 0.4225 0.000 0.000 0.436 0.004 0.560 0.000
#> GSM340195 5 0.3961 0.4207 0.000 0.000 0.440 0.004 0.556 0.000
#> GSM340196 6 0.3996 -0.2768 0.000 0.000 0.004 0.484 0.000 0.512
#> GSM340197 5 0.3961 0.4207 0.000 0.000 0.440 0.004 0.556 0.000
#> GSM340198 2 0.1007 0.8733 0.000 0.956 0.000 0.000 0.000 0.044
#> GSM340199 3 0.4544 0.3313 0.000 0.000 0.652 0.052 0.292 0.004
#> GSM340200 2 0.1692 0.8771 0.000 0.932 0.000 0.008 0.012 0.048
#> GSM340201 6 0.3997 -0.2884 0.000 0.000 0.004 0.488 0.000 0.508
#> GSM340202 6 0.3996 -0.2768 0.000 0.000 0.004 0.484 0.000 0.512
#> GSM340203 4 0.4751 0.8089 0.000 0.000 0.072 0.616 0.000 0.312
#> GSM340204 5 0.6761 -0.3239 0.004 0.256 0.000 0.244 0.452 0.044
#> GSM340205 3 0.2334 0.6311 0.012 0.000 0.908 0.032 0.040 0.008
#> GSM340206 3 0.4830 -0.3045 0.000 0.004 0.484 0.028 0.476 0.008
#> GSM340207 5 0.3955 0.4225 0.000 0.000 0.436 0.004 0.560 0.000
#> GSM340237 2 0.2911 0.8523 0.032 0.876 0.000 0.036 0.004 0.052
#> GSM340238 5 0.5097 0.3196 0.000 0.000 0.420 0.068 0.508 0.004
#> GSM340239 5 0.3961 0.4207 0.000 0.000 0.440 0.004 0.556 0.000
#> GSM340240 5 0.3961 0.4176 0.000 0.000 0.440 0.004 0.556 0.000
#> GSM340241 2 0.0405 0.8733 0.000 0.988 0.000 0.000 0.008 0.004
#> GSM340242 5 0.3955 0.4225 0.000 0.000 0.436 0.004 0.560 0.000
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
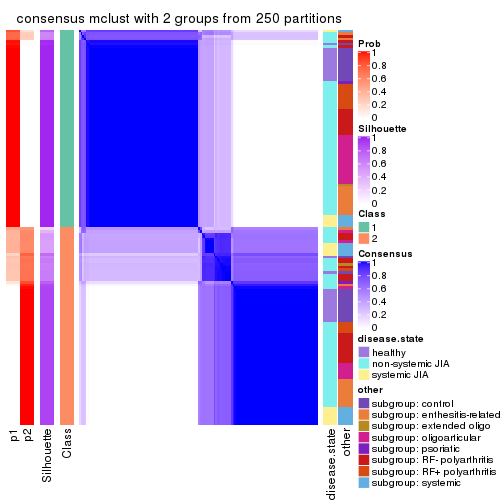
consensus_heatmap(res, k = 3)
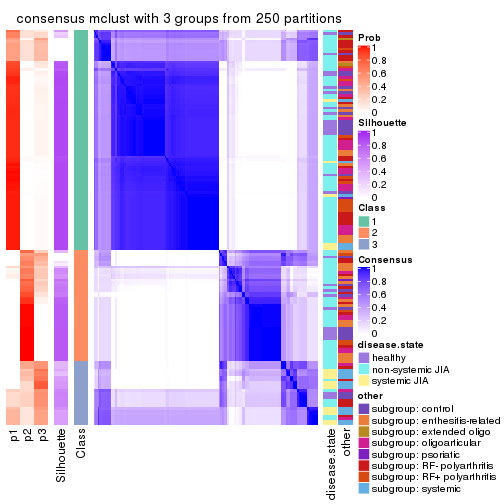
consensus_heatmap(res, k = 4)
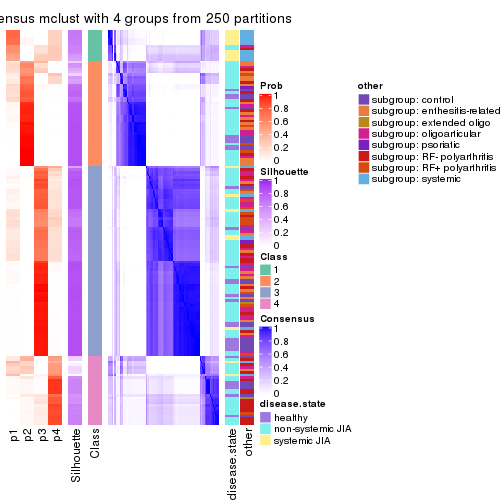
consensus_heatmap(res, k = 5)
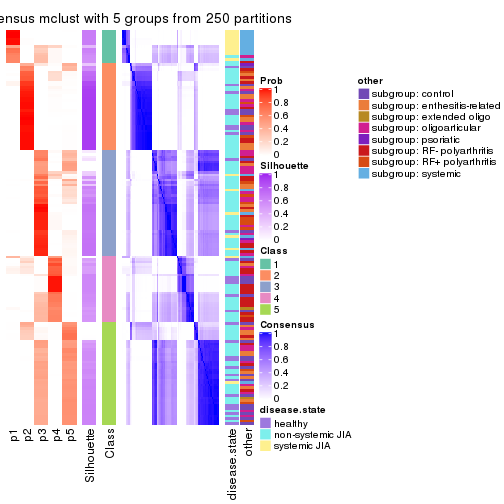
consensus_heatmap(res, k = 6)
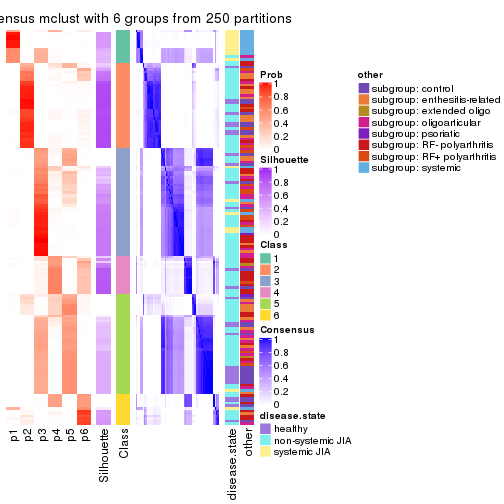
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
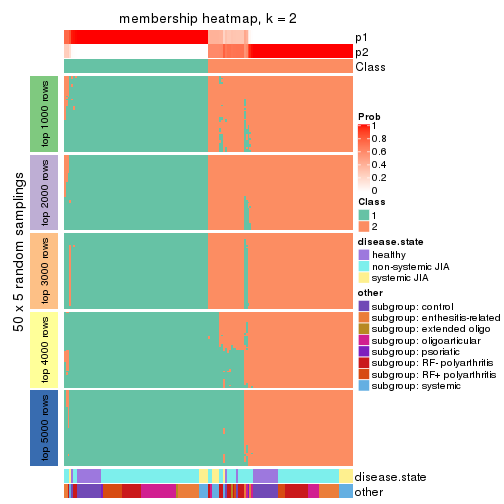
membership_heatmap(res, k = 3)
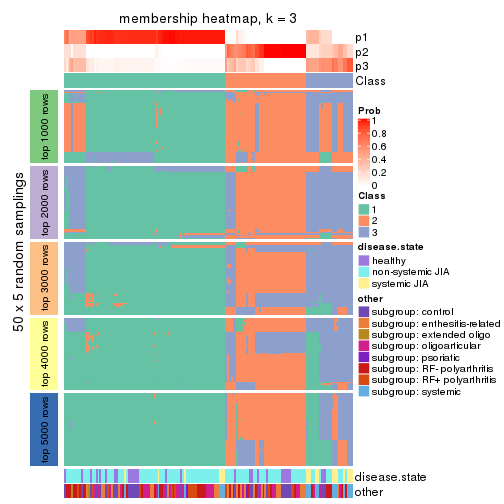
membership_heatmap(res, k = 4)
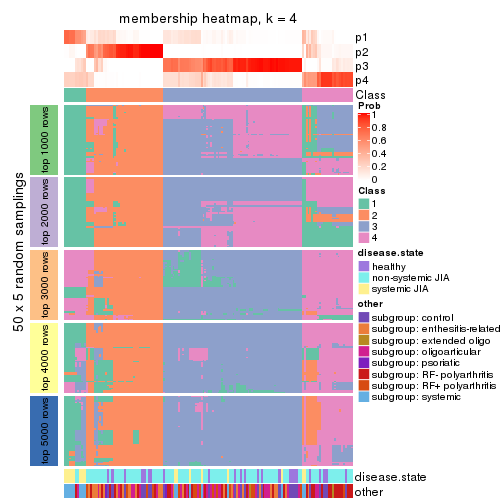
membership_heatmap(res, k = 5)
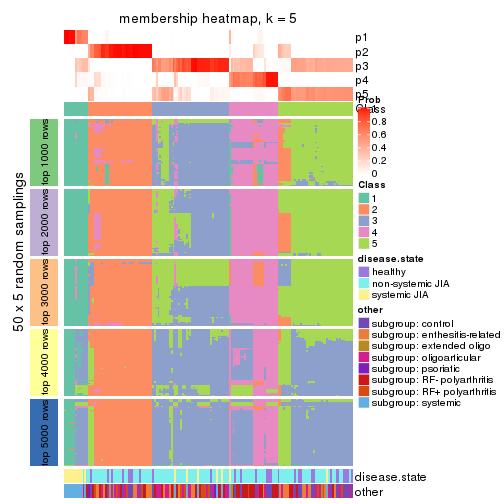
membership_heatmap(res, k = 6)
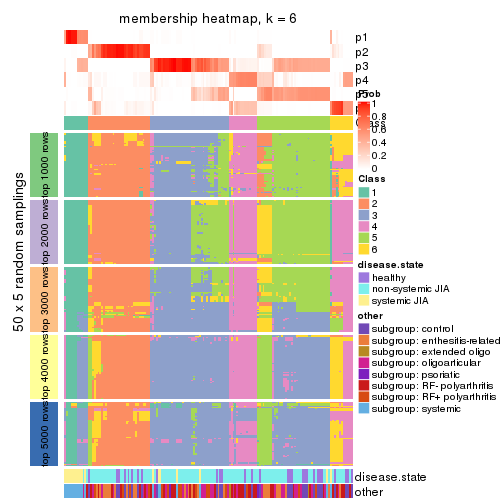
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
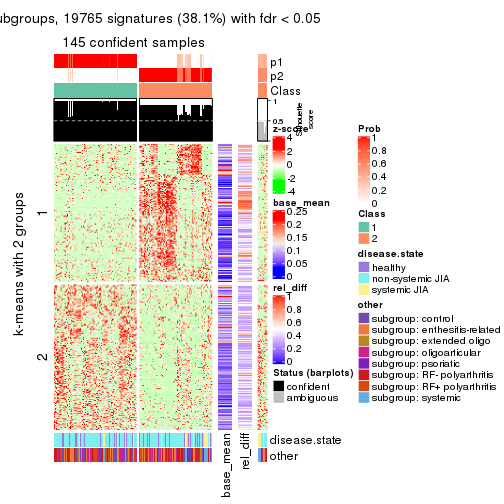
get_signatures(res, k = 3)
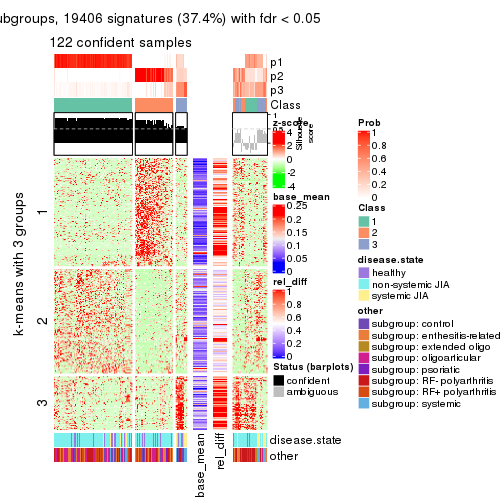
get_signatures(res, k = 4)
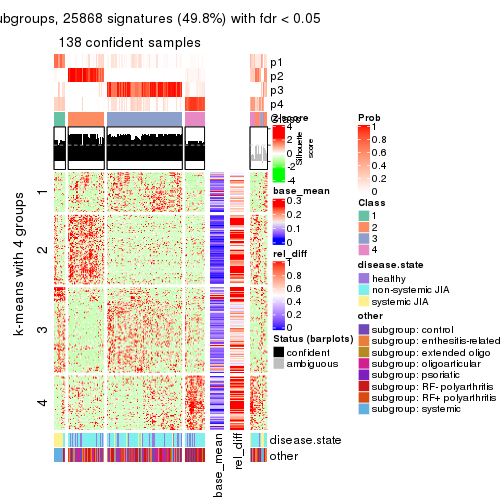
get_signatures(res, k = 5)
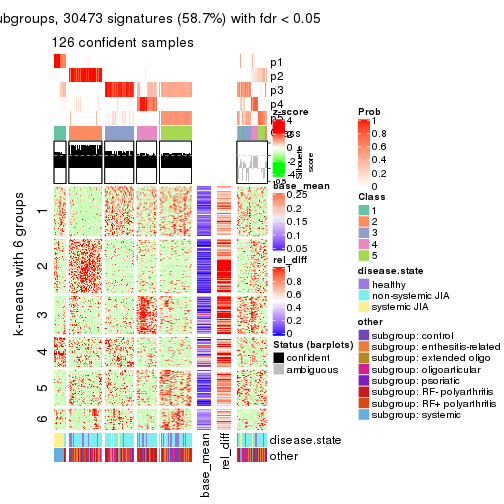
get_signatures(res, k = 6)
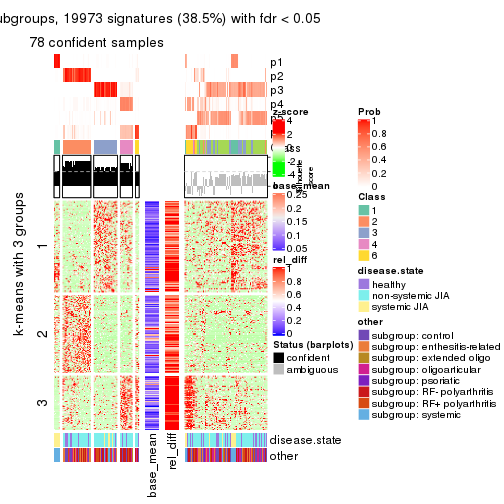
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
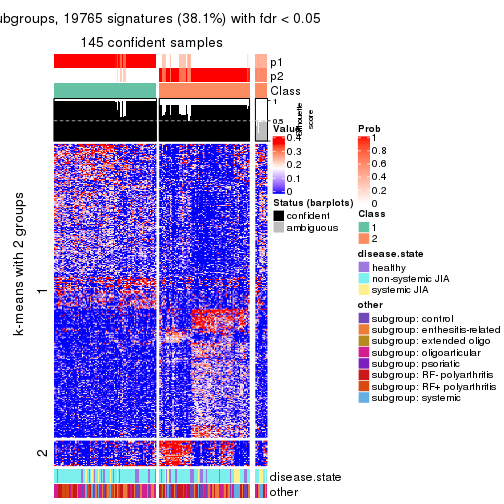
get_signatures(res, k = 3, scale_rows = FALSE)
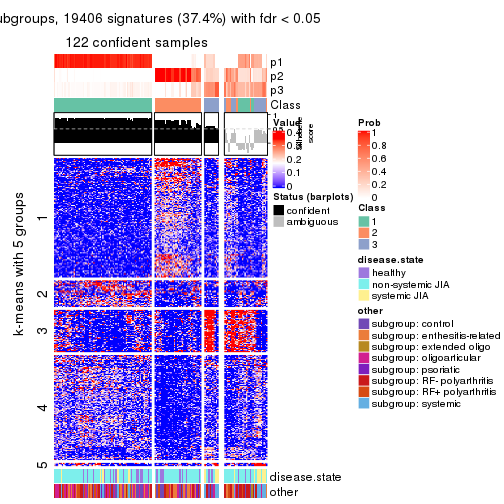
get_signatures(res, k = 4, scale_rows = FALSE)
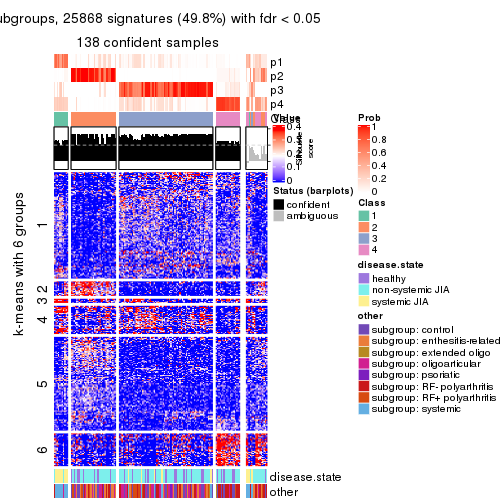
get_signatures(res, k = 5, scale_rows = FALSE)
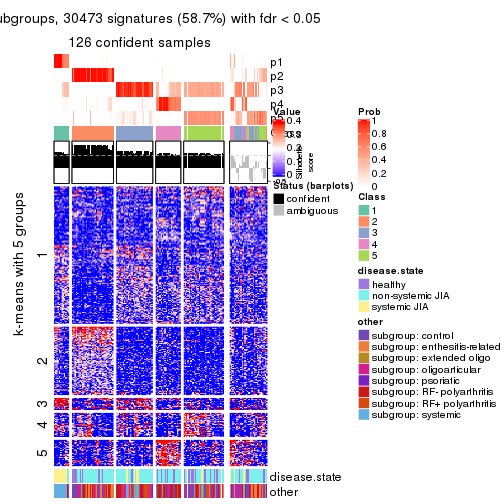
get_signatures(res, k = 6, scale_rows = FALSE)
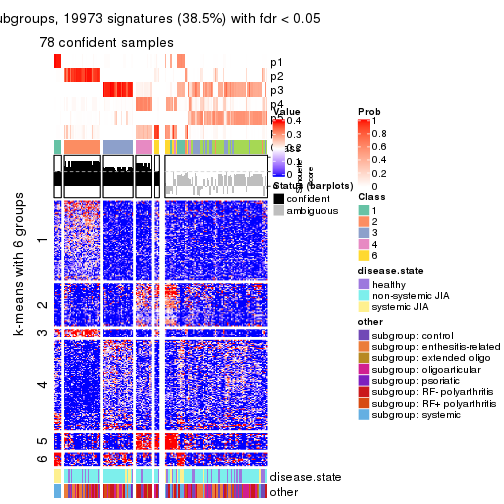
Compare the overlap of signatures from different k:
compare_signatures(res)
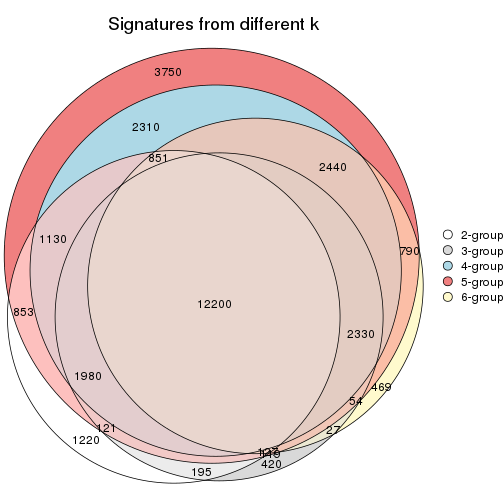
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
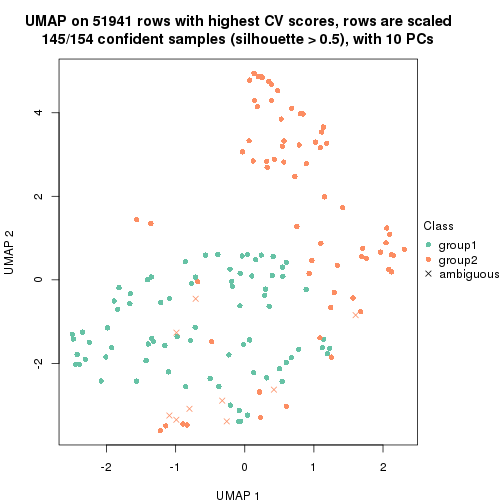
dimension_reduction(res, k = 3, method = "UMAP")
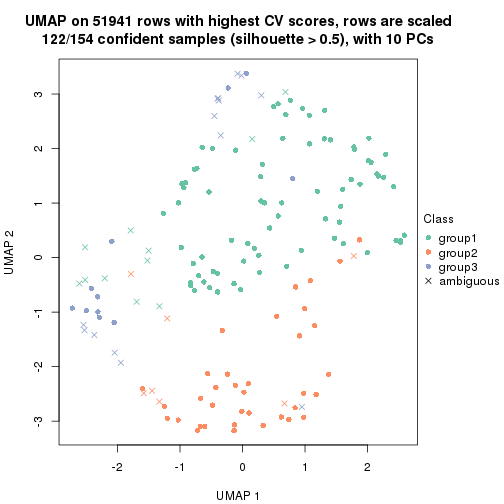
dimension_reduction(res, k = 4, method = "UMAP")
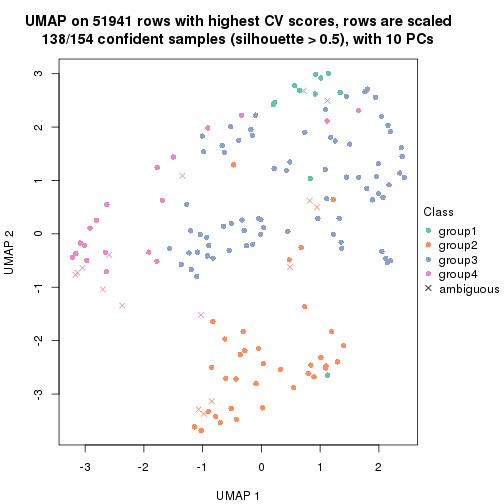
dimension_reduction(res, k = 5, method = "UMAP")
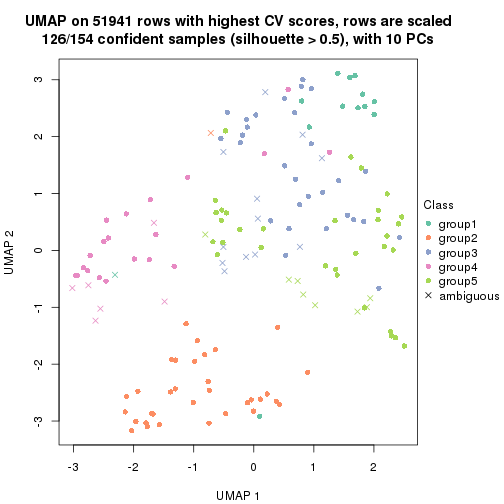
dimension_reduction(res, k = 6, method = "UMAP")
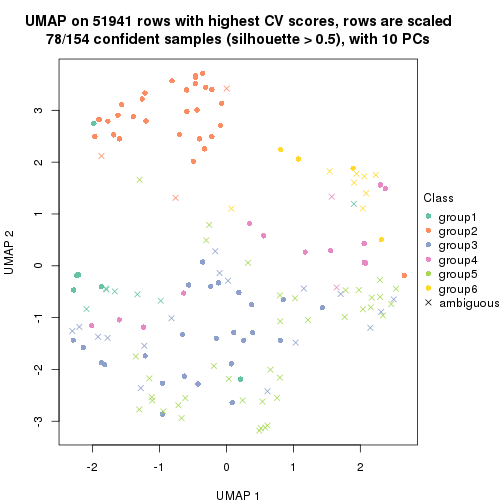
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
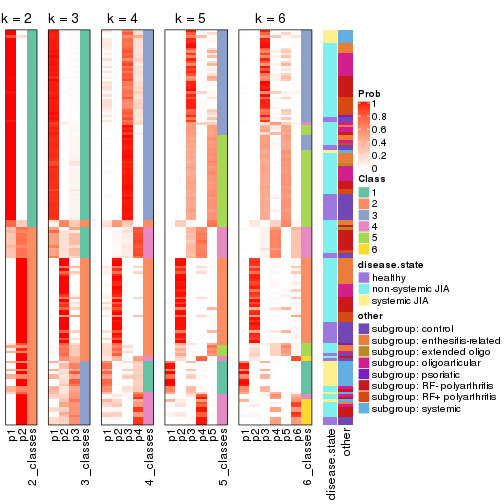
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> CV:mclust 145 5.55e-01 3.18e-01 2
#> CV:mclust 122 2.68e-03 3.34e-02 3
#> CV:mclust 138 4.89e-13 2.88e-12 4
#> CV:mclust 126 2.66e-13 1.92e-11 5
#> CV:mclust 78 2.15e-08 1.05e-06 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["CV", "NMF"]
# you can also extract it by
# res = res_list["CV:NMF"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'CV' method.
#> Subgroups are detected by 'NMF' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 4.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
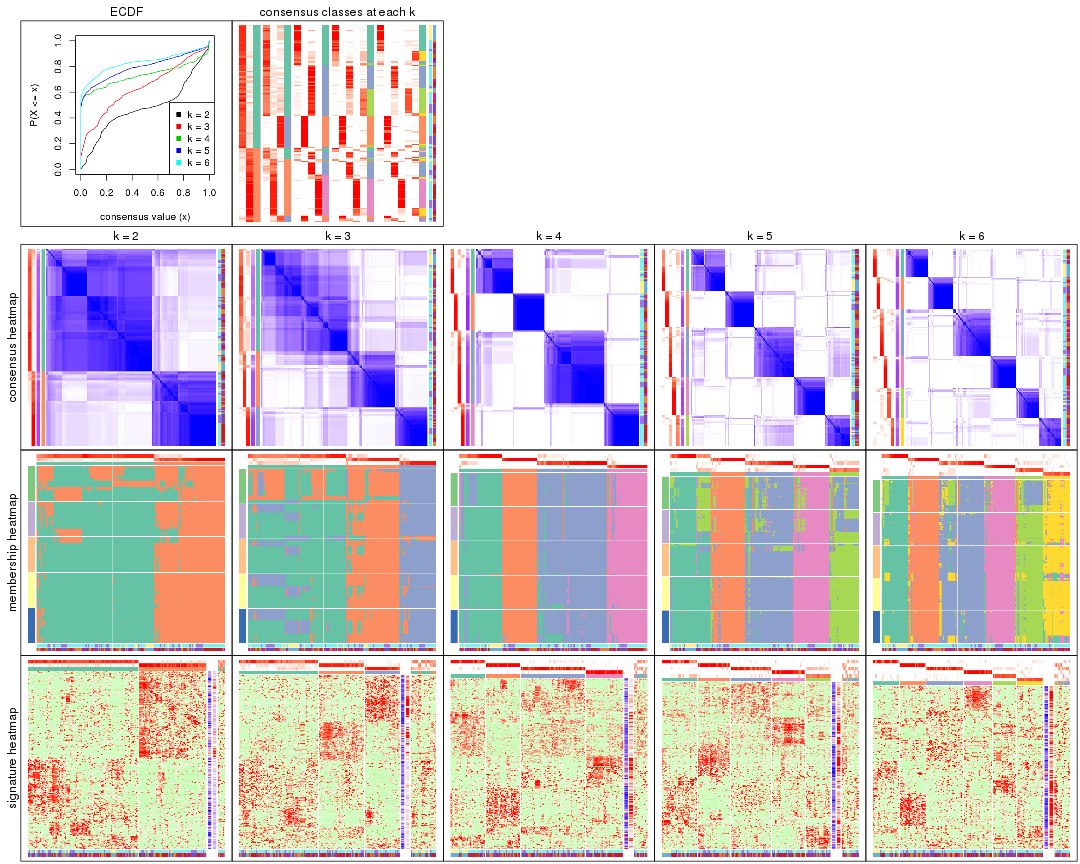
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
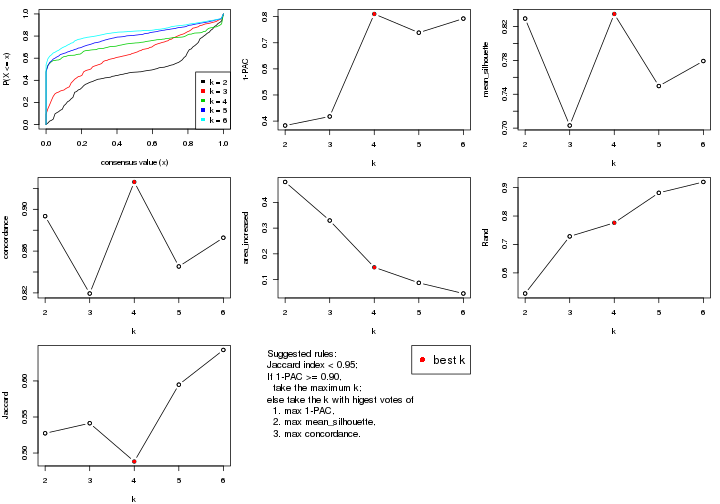
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.383 0.829 0.893 0.4798 0.527 0.527
#> 3 3 0.418 0.703 0.819 0.3297 0.728 0.541
#> 4 4 0.810 0.835 0.926 0.1475 0.776 0.488
#> 5 5 0.739 0.750 0.845 0.0870 0.882 0.595
#> 6 6 0.793 0.779 0.873 0.0454 0.920 0.643
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 4
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 1 0.5294 0.8400 0.880 0.120
#> GSM340359 1 0.2236 0.8727 0.964 0.036
#> GSM340361 1 0.2778 0.8739 0.952 0.048
#> GSM340362 1 0.4431 0.8695 0.908 0.092
#> GSM340363 2 0.5737 0.8398 0.136 0.864
#> GSM340364 1 0.2603 0.8741 0.956 0.044
#> GSM340365 1 0.2423 0.8742 0.960 0.040
#> GSM340366 1 0.2603 0.8741 0.956 0.044
#> GSM340367 1 0.2778 0.8739 0.952 0.048
#> GSM340368 2 0.2423 0.8919 0.040 0.960
#> GSM340369 2 0.6247 0.8367 0.156 0.844
#> GSM340370 1 0.2778 0.8739 0.952 0.048
#> GSM340371 1 0.7219 0.7838 0.800 0.200
#> GSM340372 1 0.2778 0.8739 0.952 0.048
#> GSM340373 1 0.2778 0.8739 0.952 0.048
#> GSM340375 1 0.2778 0.8739 0.952 0.048
#> GSM340376 1 0.2778 0.8739 0.952 0.048
#> GSM340378 1 0.2603 0.8741 0.956 0.044
#> GSM340243 1 0.2778 0.8739 0.952 0.048
#> GSM340244 1 0.6247 0.8086 0.844 0.156
#> GSM340246 1 0.6247 0.8086 0.844 0.156
#> GSM340247 2 0.0000 0.9063 0.000 1.000
#> GSM340248 1 0.6247 0.8086 0.844 0.156
#> GSM340249 2 0.2603 0.8830 0.044 0.956
#> GSM340250 1 0.4562 0.8683 0.904 0.096
#> GSM340251 2 0.4939 0.8429 0.108 0.892
#> GSM340252 2 0.2948 0.8799 0.052 0.948
#> GSM340253 1 0.4562 0.8454 0.904 0.096
#> GSM340254 1 0.6247 0.8086 0.844 0.156
#> GSM340256 1 0.6247 0.8086 0.844 0.156
#> GSM340258 1 0.4815 0.8633 0.896 0.104
#> GSM340259 1 0.4690 0.8641 0.900 0.100
#> GSM340260 1 0.2603 0.8741 0.956 0.044
#> GSM340261 1 0.9977 0.0785 0.528 0.472
#> GSM340262 1 0.5059 0.8594 0.888 0.112
#> GSM340263 1 0.6247 0.8086 0.844 0.156
#> GSM340264 1 0.8713 0.6868 0.708 0.292
#> GSM340265 1 0.3114 0.8734 0.944 0.056
#> GSM340266 2 0.5737 0.8452 0.136 0.864
#> GSM340267 1 0.4815 0.8633 0.896 0.104
#> GSM340268 2 0.2423 0.8859 0.040 0.960
#> GSM340269 1 0.5842 0.8412 0.860 0.140
#> GSM340270 1 0.9775 0.3944 0.588 0.412
#> GSM537574 1 0.4298 0.8687 0.912 0.088
#> GSM537580 2 0.6343 0.8328 0.160 0.840
#> GSM537581 2 0.3879 0.8786 0.076 0.924
#> GSM340272 1 0.2423 0.8742 0.960 0.040
#> GSM340273 1 0.6247 0.8086 0.844 0.156
#> GSM340275 2 0.0000 0.9063 0.000 1.000
#> GSM340276 2 0.9393 0.4910 0.356 0.644
#> GSM340277 1 0.5629 0.8248 0.868 0.132
#> GSM340278 2 0.4690 0.8619 0.100 0.900
#> GSM340279 2 0.5946 0.8340 0.144 0.856
#> GSM340282 1 0.0672 0.8690 0.992 0.008
#> GSM340284 2 0.2948 0.8804 0.052 0.948
#> GSM340285 2 0.5294 0.8508 0.120 0.880
#> GSM340286 1 0.7453 0.7694 0.788 0.212
#> GSM340287 1 0.4562 0.8653 0.904 0.096
#> GSM340288 1 0.5408 0.8553 0.876 0.124
#> GSM340289 1 0.4690 0.8666 0.900 0.100
#> GSM340290 2 0.5737 0.8331 0.136 0.864
#> GSM340291 1 0.6247 0.8086 0.844 0.156
#> GSM340293 2 0.1184 0.9025 0.016 0.984
#> GSM340294 1 0.8499 0.6694 0.724 0.276
#> GSM340296 1 0.2603 0.8741 0.956 0.044
#> GSM340297 2 0.6623 0.8100 0.172 0.828
#> GSM340298 1 0.2778 0.8746 0.952 0.048
#> GSM340299 1 0.4022 0.8673 0.920 0.080
#> GSM340301 1 0.4690 0.8641 0.900 0.100
#> GSM340303 1 0.0376 0.8673 0.996 0.004
#> GSM340304 2 0.9491 0.4716 0.368 0.632
#> GSM340306 2 0.0000 0.9063 0.000 1.000
#> GSM340307 1 0.6048 0.8144 0.852 0.148
#> GSM340310 1 0.2778 0.8739 0.952 0.048
#> GSM340314 2 0.0000 0.9063 0.000 1.000
#> GSM340315 1 0.9129 0.6175 0.672 0.328
#> GSM340317 2 0.7453 0.7415 0.212 0.788
#> GSM340318 2 0.0000 0.9063 0.000 1.000
#> GSM340319 2 0.0000 0.9063 0.000 1.000
#> GSM340320 2 0.0000 0.9063 0.000 1.000
#> GSM340321 1 0.6247 0.8086 0.844 0.156
#> GSM340322 2 0.0000 0.9063 0.000 1.000
#> GSM340324 1 0.5842 0.8185 0.860 0.140
#> GSM340328 2 0.9522 0.5594 0.372 0.628
#> GSM340330 2 0.6712 0.8296 0.176 0.824
#> GSM340332 2 0.0000 0.9063 0.000 1.000
#> GSM340333 1 0.4562 0.8454 0.904 0.096
#> GSM340336 2 0.0000 0.9063 0.000 1.000
#> GSM340337 2 0.0000 0.9063 0.000 1.000
#> GSM340338 2 0.8386 0.6671 0.268 0.732
#> GSM340339 2 0.0000 0.9063 0.000 1.000
#> GSM340340 2 0.3274 0.8642 0.060 0.940
#> GSM340341 2 0.0000 0.9063 0.000 1.000
#> GSM340343 2 0.0000 0.9063 0.000 1.000
#> GSM340344 1 0.1414 0.8681 0.980 0.020
#> GSM340346 1 0.3431 0.8721 0.936 0.064
#> GSM340347 2 0.0000 0.9063 0.000 1.000
#> GSM340348 2 0.0000 0.9063 0.000 1.000
#> GSM340349 1 0.2778 0.8739 0.952 0.048
#> GSM340350 1 0.2778 0.8739 0.952 0.048
#> GSM340351 1 0.5294 0.8577 0.880 0.120
#> GSM340354 1 0.4815 0.8633 0.896 0.104
#> GSM340356 1 0.6247 0.8086 0.844 0.156
#> GSM340357 1 0.3584 0.8736 0.932 0.068
#> GSM348183 2 0.7883 0.7217 0.236 0.764
#> GSM348191 2 0.5946 0.8545 0.144 0.856
#> GSM348193 2 0.0000 0.9063 0.000 1.000
#> GSM537578 1 0.2603 0.8741 0.956 0.044
#> GSM348181 2 0.6247 0.8207 0.156 0.844
#> GSM348182 1 0.9881 0.3622 0.564 0.436
#> GSM348184 1 0.9661 0.4118 0.608 0.392
#> GSM348185 2 0.1633 0.8950 0.024 0.976
#> GSM348186 1 0.6247 0.8086 0.844 0.156
#> GSM348187 1 0.4431 0.8692 0.908 0.092
#> GSM348188 2 0.0000 0.9063 0.000 1.000
#> GSM348189 1 0.7453 0.7691 0.788 0.212
#> GSM348190 1 0.4690 0.8641 0.900 0.100
#> GSM348194 1 0.5408 0.8553 0.876 0.124
#> GSM348195 1 0.2778 0.8739 0.952 0.048
#> GSM348196 1 0.8443 0.6958 0.728 0.272
#> GSM537585 1 0.4562 0.8681 0.904 0.096
#> GSM537594 1 0.6247 0.8086 0.844 0.156
#> GSM537596 1 0.7602 0.7727 0.780 0.220
#> GSM537597 1 0.1184 0.8712 0.984 0.016
#> GSM537602 1 0.2603 0.8741 0.956 0.044
#> GSM340184 2 0.6438 0.8399 0.164 0.836
#> GSM340185 1 0.6247 0.8086 0.844 0.156
#> GSM340186 1 0.6247 0.8086 0.844 0.156
#> GSM340187 2 0.0376 0.9047 0.004 0.996
#> GSM340189 2 0.0000 0.9063 0.000 1.000
#> GSM340190 2 0.8861 0.5914 0.304 0.696
#> GSM340191 2 0.4939 0.8760 0.108 0.892
#> GSM340192 1 0.0938 0.8681 0.988 0.012
#> GSM340193 1 0.5178 0.8579 0.884 0.116
#> GSM340194 1 0.5059 0.8594 0.888 0.112
#> GSM340195 1 0.4431 0.8654 0.908 0.092
#> GSM340196 2 0.0000 0.9063 0.000 1.000
#> GSM340197 1 0.4815 0.8633 0.896 0.104
#> GSM340198 1 0.6247 0.8086 0.844 0.156
#> GSM340199 2 0.6531 0.8127 0.168 0.832
#> GSM340200 1 0.6247 0.8086 0.844 0.156
#> GSM340201 2 0.0000 0.9063 0.000 1.000
#> GSM340202 2 0.0000 0.9063 0.000 1.000
#> GSM340203 2 0.0000 0.9063 0.000 1.000
#> GSM340204 1 0.1843 0.8705 0.972 0.028
#> GSM340205 1 0.2778 0.8739 0.952 0.048
#> GSM340206 2 0.2423 0.8859 0.040 0.960
#> GSM340207 1 0.6801 0.8057 0.820 0.180
#> GSM340237 1 0.4431 0.8420 0.908 0.092
#> GSM340238 2 0.2236 0.8877 0.036 0.964
#> GSM340239 1 0.4815 0.8633 0.896 0.104
#> GSM340240 1 0.4431 0.8665 0.908 0.092
#> GSM340241 1 0.6247 0.8086 0.844 0.156
#> GSM340242 1 0.6801 0.8055 0.820 0.180
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.6441 0.4946 0.696 0.028 0.276
#> GSM340359 1 0.7030 0.3938 0.580 0.024 0.396
#> GSM340361 1 0.3918 0.6812 0.868 0.012 0.120
#> GSM340362 1 0.5053 0.7046 0.812 0.024 0.164
#> GSM340363 2 0.5763 0.6067 0.244 0.740 0.016
#> GSM340364 1 0.6441 0.4946 0.696 0.028 0.276
#> GSM340365 1 0.0983 0.7298 0.980 0.004 0.016
#> GSM340366 1 0.1015 0.7302 0.980 0.008 0.012
#> GSM340367 1 0.6441 0.4946 0.696 0.028 0.276
#> GSM340368 2 0.4345 0.7256 0.136 0.848 0.016
#> GSM340369 2 0.4994 0.6877 0.160 0.816 0.024
#> GSM340370 1 0.6441 0.4946 0.696 0.028 0.276
#> GSM340371 1 0.8371 0.6564 0.624 0.164 0.212
#> GSM340372 1 0.6441 0.4946 0.696 0.028 0.276
#> GSM340373 1 0.6441 0.4946 0.696 0.028 0.276
#> GSM340375 1 0.6441 0.4946 0.696 0.028 0.276
#> GSM340376 1 0.6441 0.4946 0.696 0.028 0.276
#> GSM340378 1 0.6441 0.4946 0.696 0.028 0.276
#> GSM340243 1 0.1170 0.7292 0.976 0.008 0.016
#> GSM340244 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM340246 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM340247 2 0.1163 0.8459 0.000 0.972 0.028
#> GSM340248 3 0.1182 0.9214 0.012 0.012 0.976
#> GSM340249 2 0.6252 0.1065 0.000 0.556 0.444
#> GSM340250 1 0.1289 0.7337 0.968 0.032 0.000
#> GSM340251 3 0.3941 0.8013 0.000 0.156 0.844
#> GSM340252 3 0.5706 0.5487 0.000 0.320 0.680
#> GSM340253 3 0.0747 0.9175 0.016 0.000 0.984
#> GSM340254 3 0.0848 0.9235 0.008 0.008 0.984
#> GSM340256 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM340258 1 0.8371 0.6564 0.624 0.164 0.212
#> GSM340259 1 0.5178 0.7033 0.808 0.164 0.028
#> GSM340260 1 0.0747 0.7329 0.984 0.016 0.000
#> GSM340261 1 0.4002 0.7071 0.840 0.160 0.000
#> GSM340262 1 0.8371 0.6564 0.624 0.164 0.212
#> GSM340263 3 0.2599 0.8817 0.052 0.016 0.932
#> GSM340264 1 0.8109 0.5916 0.620 0.272 0.108
#> GSM340265 1 0.6934 0.5565 0.624 0.028 0.348
#> GSM340266 2 0.4796 0.6524 0.220 0.780 0.000
#> GSM340267 1 0.8371 0.6564 0.624 0.164 0.212
#> GSM340268 2 0.3983 0.7475 0.144 0.852 0.004
#> GSM340269 1 0.8206 0.6640 0.640 0.164 0.196
#> GSM340270 1 0.4178 0.6995 0.828 0.172 0.000
#> GSM537574 1 0.6859 0.5457 0.620 0.024 0.356
#> GSM537580 1 0.8749 0.5570 0.572 0.276 0.152
#> GSM537581 2 0.1620 0.8367 0.024 0.964 0.012
#> GSM340272 1 0.3695 0.7270 0.880 0.012 0.108
#> GSM340273 3 0.3851 0.7615 0.136 0.004 0.860
#> GSM340275 2 0.1163 0.8459 0.000 0.972 0.028
#> GSM340276 1 0.4293 0.7035 0.832 0.164 0.004
#> GSM340277 3 0.0829 0.9259 0.004 0.012 0.984
#> GSM340278 2 0.4346 0.7071 0.184 0.816 0.000
#> GSM340279 2 0.5465 0.5410 0.288 0.712 0.000
#> GSM340282 1 0.5486 0.6870 0.780 0.024 0.196
#> GSM340284 3 0.5291 0.6449 0.000 0.268 0.732
#> GSM340285 2 0.4453 0.7409 0.152 0.836 0.012
#> GSM340286 1 0.8371 0.6564 0.624 0.164 0.212
#> GSM340287 1 0.3941 0.7070 0.844 0.156 0.000
#> GSM340288 1 0.2550 0.7267 0.932 0.012 0.056
#> GSM340289 1 0.1182 0.7319 0.976 0.012 0.012
#> GSM340290 2 0.6308 -0.0766 0.492 0.508 0.000
#> GSM340291 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM340293 2 0.1182 0.8438 0.012 0.976 0.012
#> GSM340294 1 0.5746 0.6951 0.780 0.180 0.040
#> GSM340296 1 0.0848 0.7310 0.984 0.008 0.008
#> GSM340297 1 0.4452 0.6409 0.808 0.192 0.000
#> GSM340298 1 0.1015 0.7302 0.980 0.008 0.012
#> GSM340299 1 0.7431 0.6764 0.688 0.100 0.212
#> GSM340301 1 0.8371 0.6564 0.624 0.164 0.212
#> GSM340303 1 0.6742 0.5939 0.656 0.028 0.316
#> GSM340304 1 0.2998 0.7203 0.916 0.068 0.016
#> GSM340306 2 0.0829 0.8454 0.004 0.984 0.012
#> GSM340307 3 0.2651 0.8621 0.060 0.012 0.928
#> GSM340310 1 0.6441 0.4946 0.696 0.028 0.276
#> GSM340314 2 0.1643 0.8266 0.044 0.956 0.000
#> GSM340315 1 0.4293 0.7043 0.832 0.164 0.004
#> GSM340317 3 0.4452 0.7158 0.000 0.192 0.808
#> GSM340318 2 0.1163 0.8459 0.000 0.972 0.028
#> GSM340319 2 0.1163 0.8459 0.000 0.972 0.028
#> GSM340320 2 0.0983 0.8457 0.004 0.980 0.016
#> GSM340321 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM340322 2 0.1163 0.8459 0.000 0.972 0.028
#> GSM340324 3 0.0592 0.9079 0.012 0.000 0.988
#> GSM340328 1 0.7610 0.5199 0.688 0.168 0.144
#> GSM340330 2 0.5902 0.4832 0.316 0.680 0.004
#> GSM340332 2 0.1031 0.8465 0.000 0.976 0.024
#> GSM340333 3 0.1170 0.9099 0.016 0.008 0.976
#> GSM340336 2 0.1163 0.8459 0.000 0.972 0.028
#> GSM340337 2 0.1129 0.8466 0.004 0.976 0.020
#> GSM340338 1 0.8000 0.4852 0.580 0.344 0.076
#> GSM340339 2 0.1031 0.8465 0.000 0.976 0.024
#> GSM340340 2 0.2793 0.8213 0.028 0.928 0.044
#> GSM340341 2 0.1399 0.8459 0.004 0.968 0.028
#> GSM340343 2 0.1163 0.8459 0.000 0.972 0.028
#> GSM340344 1 0.6985 0.4987 0.592 0.024 0.384
#> GSM340346 1 0.6699 0.7093 0.744 0.092 0.164
#> GSM340347 2 0.1163 0.8459 0.000 0.972 0.028
#> GSM340348 2 0.1620 0.8401 0.024 0.964 0.012
#> GSM340349 1 0.1170 0.7292 0.976 0.008 0.016
#> GSM340350 1 0.1491 0.7274 0.968 0.016 0.016
#> GSM340351 1 0.3918 0.6944 0.868 0.012 0.120
#> GSM340354 1 0.8371 0.6564 0.624 0.164 0.212
#> GSM340356 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM340357 1 0.0983 0.7298 0.980 0.004 0.016
#> GSM348183 1 0.7075 0.1439 0.496 0.484 0.020
#> GSM348191 2 0.6625 0.1024 0.440 0.552 0.008
#> GSM348193 2 0.2318 0.8349 0.028 0.944 0.028
#> GSM537578 1 0.0592 0.7310 0.988 0.000 0.012
#> GSM348181 1 0.5244 0.6463 0.756 0.240 0.004
#> GSM348182 1 0.6956 0.5788 0.660 0.300 0.040
#> GSM348184 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM348185 2 0.5905 0.3993 0.000 0.648 0.352
#> GSM348186 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM348187 1 0.3644 0.6825 0.872 0.004 0.124
#> GSM348188 2 0.0892 0.8465 0.000 0.980 0.020
#> GSM348189 1 0.8371 0.6564 0.624 0.164 0.212
#> GSM348190 1 0.4473 0.7035 0.828 0.164 0.008
#> GSM348194 1 0.3610 0.7106 0.888 0.016 0.096
#> GSM348195 1 0.3272 0.6960 0.892 0.004 0.104
#> GSM348196 1 0.4636 0.6993 0.852 0.044 0.104
#> GSM537585 1 0.1182 0.7319 0.976 0.012 0.012
#> GSM537594 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM537596 1 0.4634 0.7047 0.824 0.164 0.012
#> GSM537597 1 0.4059 0.6889 0.860 0.012 0.128
#> GSM537602 1 0.1643 0.7264 0.956 0.000 0.044
#> GSM340184 2 0.7319 0.1217 0.420 0.548 0.032
#> GSM340185 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM340186 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM340187 2 0.1163 0.8459 0.000 0.972 0.028
#> GSM340189 2 0.1163 0.8459 0.000 0.972 0.028
#> GSM340190 3 0.3412 0.8345 0.000 0.124 0.876
#> GSM340191 2 0.4974 0.6323 0.236 0.764 0.000
#> GSM340192 3 0.5953 0.4802 0.280 0.012 0.708
#> GSM340193 1 0.4293 0.7035 0.832 0.164 0.004
#> GSM340194 1 0.8371 0.6564 0.624 0.164 0.212
#> GSM340195 1 0.8282 0.6613 0.632 0.160 0.208
#> GSM340196 2 0.1163 0.8459 0.000 0.972 0.028
#> GSM340197 1 0.8371 0.6564 0.624 0.164 0.212
#> GSM340198 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM340199 1 0.5465 0.5773 0.712 0.288 0.000
#> GSM340200 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM340201 2 0.1163 0.8459 0.000 0.972 0.028
#> GSM340202 2 0.1163 0.8459 0.000 0.972 0.028
#> GSM340203 2 0.1751 0.8384 0.028 0.960 0.012
#> GSM340204 1 0.6839 0.5518 0.624 0.024 0.352
#> GSM340205 1 0.5292 0.6244 0.800 0.028 0.172
#> GSM340206 2 0.6126 0.5610 0.268 0.712 0.020
#> GSM340207 1 0.8371 0.6564 0.624 0.164 0.212
#> GSM340237 3 0.0892 0.9026 0.020 0.000 0.980
#> GSM340238 2 0.2066 0.8187 0.060 0.940 0.000
#> GSM340239 1 0.8371 0.6564 0.624 0.164 0.212
#> GSM340240 1 0.8322 0.6590 0.628 0.160 0.212
#> GSM340241 3 0.0747 0.9278 0.000 0.016 0.984
#> GSM340242 1 0.8371 0.6564 0.624 0.164 0.212
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.0188 0.848 0.996 0.004 0.000 0.000
#> GSM340359 1 0.0188 0.848 0.996 0.004 0.000 0.000
#> GSM340361 1 0.0000 0.848 1.000 0.000 0.000 0.000
#> GSM340362 3 0.2216 0.865 0.092 0.000 0.908 0.000
#> GSM340363 1 0.5630 0.429 0.608 0.000 0.032 0.360
#> GSM340364 1 0.0188 0.848 0.996 0.004 0.000 0.000
#> GSM340365 1 0.0592 0.844 0.984 0.000 0.016 0.000
#> GSM340366 1 0.4941 0.215 0.564 0.000 0.436 0.000
#> GSM340367 1 0.0188 0.848 0.996 0.004 0.000 0.000
#> GSM340368 4 0.0817 0.907 0.024 0.000 0.000 0.976
#> GSM340369 4 0.3610 0.734 0.200 0.000 0.000 0.800
#> GSM340370 1 0.0188 0.848 0.996 0.004 0.000 0.000
#> GSM340371 3 0.0188 0.903 0.004 0.000 0.996 0.000
#> GSM340372 1 0.0188 0.848 0.996 0.004 0.000 0.000
#> GSM340373 1 0.0188 0.848 0.996 0.004 0.000 0.000
#> GSM340375 1 0.0188 0.848 0.996 0.004 0.000 0.000
#> GSM340376 1 0.0188 0.848 0.996 0.004 0.000 0.000
#> GSM340378 1 0.0188 0.848 0.996 0.004 0.000 0.000
#> GSM340243 1 0.2149 0.799 0.912 0.000 0.088 0.000
#> GSM340244 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340246 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340247 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340248 2 0.0188 0.984 0.000 0.996 0.004 0.000
#> GSM340249 2 0.3400 0.773 0.000 0.820 0.000 0.180
#> GSM340250 3 0.2647 0.808 0.120 0.000 0.880 0.000
#> GSM340251 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340252 4 0.4985 0.135 0.000 0.468 0.000 0.532
#> GSM340253 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340254 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340256 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340258 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340259 3 0.1557 0.885 0.056 0.000 0.944 0.000
#> GSM340260 3 0.0469 0.902 0.012 0.000 0.988 0.000
#> GSM340261 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340262 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340263 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340264 3 0.1474 0.888 0.052 0.000 0.948 0.000
#> GSM340265 3 0.0188 0.903 0.004 0.000 0.996 0.000
#> GSM340266 3 0.3726 0.702 0.000 0.000 0.788 0.212
#> GSM340267 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340268 3 0.0336 0.901 0.000 0.000 0.992 0.008
#> GSM340269 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340270 3 0.2589 0.820 0.116 0.000 0.884 0.000
#> GSM537574 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM537580 3 0.0188 0.903 0.004 0.000 0.996 0.000
#> GSM537581 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340272 3 0.3311 0.796 0.172 0.000 0.828 0.000
#> GSM340273 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340275 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340276 3 0.3837 0.667 0.224 0.000 0.776 0.000
#> GSM340277 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340278 4 0.4961 0.201 0.000 0.000 0.448 0.552
#> GSM340279 3 0.0921 0.891 0.000 0.000 0.972 0.028
#> GSM340282 3 0.0188 0.903 0.004 0.000 0.996 0.000
#> GSM340284 2 0.1940 0.908 0.000 0.924 0.076 0.000
#> GSM340285 4 0.3764 0.678 0.000 0.000 0.216 0.784
#> GSM340286 3 0.2216 0.865 0.092 0.000 0.908 0.000
#> GSM340287 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340288 1 0.3649 0.741 0.796 0.000 0.204 0.000
#> GSM340289 1 0.4624 0.508 0.660 0.000 0.340 0.000
#> GSM340290 3 0.0469 0.900 0.000 0.000 0.988 0.012
#> GSM340291 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340293 4 0.2216 0.844 0.000 0.000 0.092 0.908
#> GSM340294 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340296 1 0.4877 0.346 0.592 0.000 0.408 0.000
#> GSM340297 3 0.2342 0.848 0.080 0.000 0.912 0.008
#> GSM340298 1 0.4661 0.486 0.652 0.000 0.348 0.000
#> GSM340299 3 0.2216 0.865 0.092 0.000 0.908 0.000
#> GSM340301 3 0.0188 0.903 0.004 0.000 0.996 0.000
#> GSM340303 3 0.2216 0.865 0.092 0.000 0.908 0.000
#> GSM340304 1 0.3610 0.745 0.800 0.000 0.200 0.000
#> GSM340306 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340307 2 0.1022 0.957 0.000 0.968 0.032 0.000
#> GSM340310 1 0.0188 0.848 0.996 0.000 0.004 0.000
#> GSM340314 4 0.4564 0.520 0.000 0.000 0.328 0.672
#> GSM340315 3 0.3219 0.764 0.164 0.000 0.836 0.000
#> GSM340317 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340318 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340319 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340320 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340321 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340322 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340324 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340328 1 0.0188 0.847 0.996 0.000 0.000 0.004
#> GSM340330 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340332 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340333 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340336 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340337 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340338 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340339 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340340 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340341 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340343 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340344 3 0.3015 0.854 0.092 0.024 0.884 0.000
#> GSM340346 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340347 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340348 4 0.0592 0.910 0.000 0.000 0.016 0.984
#> GSM340349 1 0.0592 0.842 0.984 0.000 0.016 0.000
#> GSM340350 1 0.0000 0.848 1.000 0.000 0.000 0.000
#> GSM340351 1 0.3610 0.745 0.800 0.000 0.200 0.000
#> GSM340354 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340356 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340357 1 0.0336 0.847 0.992 0.000 0.008 0.000
#> GSM348183 1 0.4855 0.355 0.600 0.000 0.000 0.400
#> GSM348191 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM348193 4 0.2760 0.792 0.128 0.000 0.000 0.872
#> GSM537578 3 0.4916 0.125 0.424 0.000 0.576 0.000
#> GSM348181 3 0.5420 0.333 0.352 0.000 0.624 0.024
#> GSM348182 3 0.2281 0.862 0.096 0.000 0.904 0.000
#> GSM348184 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM348185 4 0.3764 0.702 0.000 0.216 0.000 0.784
#> GSM348186 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM348187 1 0.3219 0.775 0.836 0.000 0.164 0.000
#> GSM348188 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM348189 3 0.0336 0.903 0.008 0.000 0.992 0.000
#> GSM348190 3 0.2149 0.866 0.088 0.000 0.912 0.000
#> GSM348194 1 0.3649 0.741 0.796 0.000 0.204 0.000
#> GSM348195 1 0.0000 0.848 1.000 0.000 0.000 0.000
#> GSM348196 1 0.3649 0.741 0.796 0.000 0.204 0.000
#> GSM537585 3 0.4543 0.515 0.324 0.000 0.676 0.000
#> GSM537594 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM537596 1 0.4877 0.344 0.592 0.000 0.408 0.000
#> GSM537597 1 0.3649 0.741 0.796 0.000 0.204 0.000
#> GSM537602 1 0.2530 0.808 0.888 0.000 0.112 0.000
#> GSM340184 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340185 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340186 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340187 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340189 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340190 2 0.0817 0.966 0.000 0.976 0.024 0.000
#> GSM340191 3 0.1118 0.886 0.000 0.000 0.964 0.036
#> GSM340192 3 0.5105 0.208 0.004 0.432 0.564 0.000
#> GSM340193 3 0.4406 0.573 0.300 0.000 0.700 0.000
#> GSM340194 3 0.2216 0.865 0.092 0.000 0.908 0.000
#> GSM340195 3 0.2216 0.865 0.092 0.000 0.908 0.000
#> GSM340196 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340197 3 0.1716 0.882 0.064 0.000 0.936 0.000
#> GSM340198 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340199 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340200 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340201 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340202 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340203 4 0.0000 0.923 0.000 0.000 0.000 1.000
#> GSM340204 3 0.2216 0.865 0.092 0.000 0.908 0.000
#> GSM340205 1 0.0188 0.848 0.996 0.000 0.004 0.000
#> GSM340206 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340207 3 0.0000 0.904 0.000 0.000 1.000 0.000
#> GSM340237 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340238 3 0.4331 0.571 0.000 0.000 0.712 0.288
#> GSM340239 3 0.0592 0.901 0.016 0.000 0.984 0.000
#> GSM340240 3 0.0188 0.903 0.004 0.000 0.996 0.000
#> GSM340241 2 0.0000 0.988 0.000 1.000 0.000 0.000
#> GSM340242 3 0.0188 0.903 0.004 0.000 0.996 0.000
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.0000 0.7958 1.000 0.000 0.000 0.000 0.000
#> GSM340359 1 0.0451 0.7908 0.988 0.008 0.000 0.000 0.004
#> GSM340361 1 0.1341 0.7912 0.944 0.000 0.000 0.000 0.056
#> GSM340362 5 0.4706 0.7970 0.052 0.000 0.256 0.000 0.692
#> GSM340363 1 0.6183 0.6586 0.584 0.000 0.020 0.112 0.284
#> GSM340364 1 0.0000 0.7958 1.000 0.000 0.000 0.000 0.000
#> GSM340365 5 0.4045 0.3576 0.356 0.000 0.000 0.000 0.644
#> GSM340366 1 0.5786 0.0674 0.524 0.000 0.380 0.000 0.096
#> GSM340367 1 0.0000 0.7958 1.000 0.000 0.000 0.000 0.000
#> GSM340368 4 0.1608 0.8739 0.072 0.000 0.000 0.928 0.000
#> GSM340369 4 0.3612 0.6621 0.268 0.000 0.000 0.732 0.000
#> GSM340370 1 0.0000 0.7958 1.000 0.000 0.000 0.000 0.000
#> GSM340371 5 0.4150 0.7750 0.000 0.000 0.388 0.000 0.612
#> GSM340372 1 0.0000 0.7958 1.000 0.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.7958 1.000 0.000 0.000 0.000 0.000
#> GSM340375 1 0.0000 0.7958 1.000 0.000 0.000 0.000 0.000
#> GSM340376 1 0.0000 0.7958 1.000 0.000 0.000 0.000 0.000
#> GSM340378 1 0.0000 0.7958 1.000 0.000 0.000 0.000 0.000
#> GSM340243 1 0.2482 0.7710 0.892 0.000 0.024 0.000 0.084
#> GSM340244 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340246 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340247 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340248 2 0.1043 0.8901 0.000 0.960 0.040 0.000 0.000
#> GSM340249 2 0.3143 0.7139 0.000 0.796 0.000 0.204 0.000
#> GSM340250 3 0.3649 0.7285 0.152 0.000 0.808 0.000 0.040
#> GSM340251 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340252 4 0.3561 0.6464 0.000 0.260 0.000 0.740 0.000
#> GSM340253 5 0.4415 0.1313 0.000 0.444 0.004 0.000 0.552
#> GSM340254 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340256 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340258 3 0.0290 0.7912 0.000 0.000 0.992 0.000 0.008
#> GSM340259 3 0.2966 0.7429 0.016 0.000 0.848 0.000 0.136
#> GSM340260 3 0.3476 0.6999 0.176 0.000 0.804 0.000 0.020
#> GSM340261 3 0.2230 0.7819 0.000 0.000 0.884 0.000 0.116
#> GSM340262 3 0.0290 0.7912 0.000 0.000 0.992 0.000 0.008
#> GSM340263 2 0.0162 0.9167 0.000 0.996 0.000 0.000 0.004
#> GSM340264 5 0.3983 0.8141 0.000 0.000 0.340 0.000 0.660
#> GSM340265 3 0.4138 -0.2389 0.000 0.000 0.616 0.000 0.384
#> GSM340266 3 0.2329 0.7660 0.000 0.000 0.876 0.000 0.124
#> GSM340267 3 0.2230 0.7746 0.000 0.000 0.884 0.000 0.116
#> GSM340268 3 0.0324 0.7951 0.000 0.000 0.992 0.004 0.004
#> GSM340269 3 0.0000 0.7951 0.000 0.000 1.000 0.000 0.000
#> GSM340270 3 0.0579 0.7965 0.008 0.000 0.984 0.000 0.008
#> GSM537574 3 0.0880 0.7981 0.000 0.000 0.968 0.000 0.032
#> GSM537580 5 0.3949 0.8174 0.000 0.000 0.332 0.000 0.668
#> GSM537581 4 0.0794 0.9021 0.028 0.000 0.000 0.972 0.000
#> GSM340272 5 0.4805 0.7092 0.144 0.000 0.128 0.000 0.728
#> GSM340273 2 0.3563 0.7195 0.012 0.780 0.000 0.000 0.208
#> GSM340275 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340276 3 0.6090 0.3592 0.136 0.000 0.516 0.000 0.348
#> GSM340277 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340278 3 0.4029 0.6242 0.000 0.000 0.680 0.004 0.316
#> GSM340279 3 0.3086 0.7385 0.000 0.000 0.816 0.004 0.180
#> GSM340282 5 0.4702 0.6313 0.016 0.000 0.432 0.000 0.552
#> GSM340284 3 0.4030 0.4392 0.000 0.352 0.648 0.000 0.000
#> GSM340285 4 0.5819 0.4729 0.004 0.000 0.088 0.540 0.368
#> GSM340286 5 0.4084 0.8186 0.004 0.000 0.328 0.000 0.668
#> GSM340287 3 0.3707 0.6450 0.000 0.000 0.716 0.000 0.284
#> GSM340288 1 0.5168 0.6746 0.592 0.000 0.052 0.000 0.356
#> GSM340289 1 0.5240 0.5921 0.676 0.000 0.204 0.000 0.120
#> GSM340290 3 0.2011 0.7950 0.000 0.000 0.908 0.004 0.088
#> GSM340291 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340293 3 0.4857 0.5199 0.000 0.000 0.636 0.324 0.040
#> GSM340294 3 0.0290 0.7981 0.000 0.000 0.992 0.000 0.008
#> GSM340296 3 0.5589 0.3817 0.372 0.000 0.548 0.000 0.080
#> GSM340297 3 0.2153 0.7957 0.040 0.000 0.916 0.000 0.044
#> GSM340298 3 0.5579 0.3904 0.368 0.000 0.552 0.000 0.080
#> GSM340299 5 0.4251 0.8189 0.012 0.000 0.316 0.000 0.672
#> GSM340301 5 0.3966 0.8159 0.000 0.000 0.336 0.000 0.664
#> GSM340303 5 0.2624 0.7019 0.012 0.000 0.116 0.000 0.872
#> GSM340304 1 0.5000 0.6727 0.576 0.000 0.036 0.000 0.388
#> GSM340306 4 0.2773 0.8126 0.000 0.000 0.000 0.836 0.164
#> GSM340307 2 0.3612 0.6688 0.000 0.764 0.228 0.000 0.008
#> GSM340310 1 0.0404 0.7964 0.988 0.000 0.000 0.000 0.012
#> GSM340314 3 0.2424 0.7376 0.000 0.000 0.868 0.132 0.000
#> GSM340315 3 0.3039 0.7263 0.000 0.000 0.808 0.000 0.192
#> GSM340317 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340318 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340319 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340320 4 0.3048 0.8016 0.000 0.000 0.004 0.820 0.176
#> GSM340321 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340322 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340324 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340328 1 0.0290 0.7964 0.992 0.000 0.000 0.000 0.008
#> GSM340330 3 0.0162 0.7933 0.000 0.000 0.996 0.000 0.004
#> GSM340332 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340333 2 0.3612 0.6032 0.000 0.732 0.000 0.000 0.268
#> GSM340336 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340337 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340338 3 0.0510 0.7854 0.000 0.000 0.984 0.000 0.016
#> GSM340339 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340340 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340341 4 0.3266 0.7789 0.000 0.000 0.004 0.796 0.200
#> GSM340343 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340344 5 0.4793 0.7013 0.012 0.112 0.124 0.000 0.752
#> GSM340346 3 0.1357 0.7840 0.004 0.000 0.948 0.000 0.048
#> GSM340347 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340348 4 0.3906 0.6827 0.000 0.000 0.004 0.704 0.292
#> GSM340349 1 0.2293 0.7749 0.900 0.000 0.016 0.000 0.084
#> GSM340350 1 0.0162 0.7948 0.996 0.000 0.000 0.000 0.004
#> GSM340351 1 0.4779 0.6914 0.628 0.000 0.032 0.000 0.340
#> GSM340354 3 0.0404 0.7897 0.000 0.000 0.988 0.000 0.012
#> GSM340356 2 0.1732 0.8605 0.000 0.920 0.000 0.000 0.080
#> GSM340357 1 0.2946 0.7752 0.868 0.000 0.044 0.000 0.088
#> GSM348183 1 0.5601 0.1873 0.480 0.000 0.000 0.448 0.072
#> GSM348191 3 0.1043 0.7974 0.000 0.000 0.960 0.000 0.040
#> GSM348193 4 0.1608 0.8802 0.000 0.000 0.000 0.928 0.072
#> GSM537578 3 0.5756 0.5847 0.176 0.000 0.620 0.000 0.204
#> GSM348181 3 0.3454 0.7305 0.100 0.000 0.836 0.000 0.064
#> GSM348182 5 0.4270 0.8194 0.012 0.000 0.320 0.000 0.668
#> GSM348184 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM348185 4 0.6302 0.4827 0.004 0.252 0.000 0.552 0.192
#> GSM348186 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM348187 1 0.5006 0.6929 0.624 0.000 0.048 0.000 0.328
#> GSM348188 4 0.0404 0.9116 0.000 0.000 0.000 0.988 0.012
#> GSM348189 5 0.3949 0.8174 0.000 0.000 0.332 0.000 0.668
#> GSM348190 5 0.3636 0.6738 0.000 0.000 0.272 0.000 0.728
#> GSM348194 1 0.5370 0.6654 0.584 0.000 0.068 0.000 0.348
#> GSM348195 1 0.2561 0.7771 0.856 0.000 0.000 0.000 0.144
#> GSM348196 1 0.5316 0.6683 0.588 0.000 0.064 0.000 0.348
#> GSM537585 5 0.4649 0.1123 0.220 0.000 0.064 0.000 0.716
#> GSM537594 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM537596 1 0.6235 0.5817 0.500 0.000 0.156 0.000 0.344
#> GSM537597 1 0.5435 0.6613 0.576 0.000 0.072 0.000 0.352
#> GSM537602 1 0.4074 0.7033 0.636 0.000 0.000 0.000 0.364
#> GSM340184 3 0.0000 0.7951 0.000 0.000 1.000 0.000 0.000
#> GSM340185 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340186 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340187 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340189 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340190 2 0.3816 0.5373 0.000 0.696 0.304 0.000 0.000
#> GSM340191 3 0.0671 0.7993 0.000 0.000 0.980 0.004 0.016
#> GSM340192 2 0.6338 0.0874 0.000 0.448 0.392 0.000 0.160
#> GSM340193 1 0.6790 0.3198 0.384 0.000 0.316 0.000 0.300
#> GSM340194 5 0.4270 0.8194 0.012 0.000 0.320 0.000 0.668
#> GSM340195 5 0.4270 0.8194 0.012 0.000 0.320 0.000 0.668
#> GSM340196 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340197 5 0.4101 0.7923 0.000 0.000 0.372 0.000 0.628
#> GSM340198 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340199 3 0.1205 0.7883 0.004 0.000 0.956 0.000 0.040
#> GSM340200 2 0.1270 0.8861 0.000 0.948 0.000 0.000 0.052
#> GSM340201 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340202 4 0.0000 0.9164 0.000 0.000 0.000 1.000 0.000
#> GSM340203 4 0.0162 0.9145 0.000 0.000 0.004 0.996 0.000
#> GSM340204 5 0.3086 0.7309 0.000 0.004 0.180 0.000 0.816
#> GSM340205 1 0.0162 0.7961 0.996 0.000 0.000 0.000 0.004
#> GSM340206 3 0.0324 0.7951 0.000 0.000 0.992 0.004 0.004
#> GSM340207 3 0.1121 0.7581 0.000 0.000 0.956 0.000 0.044
#> GSM340237 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340238 3 0.0404 0.7965 0.000 0.000 0.988 0.012 0.000
#> GSM340239 5 0.4150 0.7767 0.000 0.000 0.388 0.000 0.612
#> GSM340240 5 0.4045 0.7692 0.000 0.000 0.356 0.000 0.644
#> GSM340241 2 0.0000 0.9190 0.000 1.000 0.000 0.000 0.000
#> GSM340242 5 0.4150 0.7750 0.000 0.000 0.388 0.000 0.612
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.0000 0.90320 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340359 1 0.0000 0.90320 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340361 1 0.0508 0.89575 0.984 0.000 0.000 0.000 0.004 0.012
#> GSM340362 5 0.2965 0.80559 0.080 0.000 0.072 0.000 0.848 0.000
#> GSM340363 6 0.2651 0.83411 0.112 0.000 0.000 0.028 0.000 0.860
#> GSM340364 1 0.0000 0.90320 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340365 1 0.4144 0.60710 0.620 0.000 0.000 0.000 0.360 0.020
#> GSM340366 1 0.4799 0.71743 0.700 0.000 0.012 0.000 0.156 0.132
#> GSM340367 1 0.0000 0.90320 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340368 4 0.1957 0.85401 0.112 0.000 0.000 0.888 0.000 0.000
#> GSM340369 4 0.3515 0.56278 0.324 0.000 0.000 0.676 0.000 0.000
#> GSM340370 1 0.0000 0.90320 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340371 5 0.2454 0.85691 0.000 0.000 0.160 0.000 0.840 0.000
#> GSM340372 1 0.0000 0.90320 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.90320 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340375 1 0.0000 0.90320 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340376 1 0.0000 0.90320 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340378 1 0.0000 0.90320 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340243 1 0.4743 0.71749 0.700 0.000 0.008 0.000 0.152 0.140
#> GSM340244 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340246 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340247 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340248 2 0.1610 0.83954 0.000 0.916 0.084 0.000 0.000 0.000
#> GSM340249 2 0.3592 0.45578 0.000 0.656 0.000 0.344 0.000 0.000
#> GSM340250 3 0.2362 0.76835 0.136 0.000 0.860 0.000 0.000 0.004
#> GSM340251 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340252 4 0.2994 0.71310 0.000 0.208 0.000 0.788 0.004 0.000
#> GSM340253 5 0.3515 0.47563 0.000 0.324 0.000 0.000 0.676 0.000
#> GSM340254 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340256 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340258 3 0.0260 0.81430 0.000 0.000 0.992 0.000 0.008 0.000
#> GSM340259 3 0.4155 0.30746 0.000 0.000 0.616 0.000 0.020 0.364
#> GSM340260 3 0.4923 0.60043 0.264 0.000 0.656 0.000 0.052 0.028
#> GSM340261 3 0.1967 0.79678 0.000 0.000 0.904 0.000 0.084 0.012
#> GSM340262 3 0.0363 0.81294 0.000 0.000 0.988 0.000 0.012 0.000
#> GSM340263 2 0.0405 0.90743 0.004 0.988 0.000 0.000 0.000 0.008
#> GSM340264 5 0.2378 0.85871 0.000 0.000 0.152 0.000 0.848 0.000
#> GSM340265 5 0.4793 0.66595 0.000 0.000 0.288 0.000 0.628 0.084
#> GSM340266 3 0.3765 0.21950 0.000 0.000 0.596 0.000 0.000 0.404
#> GSM340267 3 0.2454 0.76836 0.000 0.000 0.840 0.000 0.000 0.160
#> GSM340268 3 0.0000 0.81633 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340269 3 0.0508 0.81417 0.000 0.000 0.984 0.000 0.012 0.004
#> GSM340270 3 0.2473 0.72894 0.136 0.000 0.856 0.000 0.008 0.000
#> GSM537574 3 0.1152 0.80265 0.000 0.000 0.952 0.000 0.004 0.044
#> GSM537580 5 0.2378 0.85871 0.000 0.000 0.152 0.000 0.848 0.000
#> GSM537581 4 0.1663 0.87442 0.088 0.000 0.000 0.912 0.000 0.000
#> GSM340272 5 0.1049 0.74202 0.008 0.000 0.000 0.000 0.960 0.032
#> GSM340273 6 0.2520 0.78395 0.004 0.152 0.000 0.000 0.000 0.844
#> GSM340275 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340276 6 0.1918 0.82672 0.000 0.000 0.088 0.000 0.008 0.904
#> GSM340277 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340278 6 0.2362 0.75156 0.000 0.000 0.136 0.004 0.000 0.860
#> GSM340279 3 0.2092 0.77547 0.000 0.000 0.876 0.000 0.000 0.124
#> GSM340282 5 0.5271 -0.02954 0.000 0.000 0.380 0.000 0.516 0.104
#> GSM340284 3 0.2454 0.73211 0.000 0.160 0.840 0.000 0.000 0.000
#> GSM340285 6 0.1843 0.83699 0.000 0.000 0.004 0.080 0.004 0.912
#> GSM340286 5 0.2378 0.85871 0.000 0.000 0.152 0.000 0.848 0.000
#> GSM340287 3 0.3151 0.64835 0.000 0.000 0.748 0.000 0.000 0.252
#> GSM340288 6 0.2062 0.84183 0.088 0.000 0.004 0.000 0.008 0.900
#> GSM340289 1 0.5090 0.60829 0.680 0.000 0.104 0.000 0.188 0.028
#> GSM340290 3 0.0937 0.81382 0.000 0.000 0.960 0.000 0.040 0.000
#> GSM340291 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340293 3 0.4558 0.67994 0.000 0.000 0.720 0.008 0.148 0.124
#> GSM340294 3 0.0000 0.81633 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340296 3 0.7186 0.15298 0.312 0.000 0.400 0.000 0.152 0.136
#> GSM340297 3 0.1285 0.80982 0.052 0.000 0.944 0.000 0.004 0.000
#> GSM340298 3 0.7192 0.13872 0.316 0.000 0.396 0.000 0.152 0.136
#> GSM340299 5 0.2300 0.85657 0.000 0.000 0.144 0.000 0.856 0.000
#> GSM340301 5 0.2491 0.85495 0.000 0.000 0.164 0.000 0.836 0.000
#> GSM340303 5 0.2613 0.65624 0.000 0.000 0.012 0.000 0.848 0.140
#> GSM340304 6 0.2568 0.82133 0.068 0.000 0.000 0.000 0.056 0.876
#> GSM340306 6 0.3409 0.63470 0.000 0.000 0.000 0.300 0.000 0.700
#> GSM340307 2 0.5430 0.21596 0.000 0.540 0.356 0.000 0.012 0.092
#> GSM340310 1 0.0000 0.90320 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340314 3 0.0790 0.81305 0.000 0.000 0.968 0.032 0.000 0.000
#> GSM340315 3 0.1863 0.79016 0.000 0.000 0.896 0.000 0.000 0.104
#> GSM340317 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340318 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340319 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340320 6 0.2883 0.75413 0.000 0.000 0.000 0.212 0.000 0.788
#> GSM340321 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340322 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340324 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340328 1 0.0000 0.90320 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340330 3 0.0146 0.81552 0.000 0.000 0.996 0.000 0.004 0.000
#> GSM340332 4 0.0865 0.90625 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340333 2 0.3547 0.45231 0.000 0.668 0.000 0.000 0.332 0.000
#> GSM340336 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340337 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340338 3 0.0717 0.81346 0.000 0.000 0.976 0.000 0.008 0.016
#> GSM340339 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340340 4 0.0146 0.93261 0.004 0.000 0.000 0.996 0.000 0.000
#> GSM340341 6 0.3023 0.72994 0.000 0.000 0.000 0.232 0.000 0.768
#> GSM340343 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340344 5 0.3005 0.74134 0.000 0.036 0.008 0.000 0.848 0.108
#> GSM340346 3 0.4267 0.68690 0.000 0.000 0.732 0.000 0.152 0.116
#> GSM340347 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340348 6 0.2092 0.82074 0.000 0.000 0.000 0.124 0.000 0.876
#> GSM340349 1 0.4961 0.70754 0.688 0.000 0.016 0.000 0.152 0.144
#> GSM340350 1 0.1957 0.83862 0.888 0.000 0.000 0.000 0.000 0.112
#> GSM340351 6 0.3489 0.64183 0.288 0.000 0.000 0.000 0.004 0.708
#> GSM340354 3 0.0632 0.81328 0.000 0.000 0.976 0.000 0.000 0.024
#> GSM340356 2 0.3428 0.50332 0.000 0.696 0.000 0.000 0.000 0.304
#> GSM340357 1 0.1966 0.86881 0.924 0.000 0.024 0.000 0.028 0.024
#> GSM348183 4 0.5624 -0.00202 0.440 0.000 0.000 0.464 0.056 0.040
#> GSM348191 3 0.0260 0.81598 0.000 0.000 0.992 0.000 0.000 0.008
#> GSM348193 4 0.2070 0.86639 0.044 0.000 0.000 0.908 0.000 0.048
#> GSM537578 3 0.6927 0.44346 0.148 0.000 0.496 0.000 0.152 0.204
#> GSM348181 3 0.3245 0.68281 0.172 0.000 0.800 0.000 0.000 0.028
#> GSM348182 5 0.2378 0.85871 0.000 0.000 0.152 0.000 0.848 0.000
#> GSM348184 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM348185 6 0.3481 0.79339 0.000 0.124 0.000 0.072 0.000 0.804
#> GSM348186 2 0.0146 0.91265 0.000 0.996 0.000 0.000 0.004 0.000
#> GSM348187 6 0.2003 0.83308 0.116 0.000 0.000 0.000 0.000 0.884
#> GSM348188 4 0.1257 0.90608 0.000 0.000 0.000 0.952 0.020 0.028
#> GSM348189 5 0.2378 0.85871 0.000 0.000 0.152 0.000 0.848 0.000
#> GSM348190 6 0.4358 0.65016 0.000 0.000 0.196 0.000 0.092 0.712
#> GSM348194 6 0.2313 0.83896 0.100 0.000 0.004 0.000 0.012 0.884
#> GSM348195 1 0.3542 0.72674 0.788 0.000 0.000 0.000 0.052 0.160
#> GSM348196 6 0.2163 0.84021 0.096 0.000 0.004 0.000 0.008 0.892
#> GSM537585 6 0.4439 0.65026 0.064 0.000 0.004 0.000 0.240 0.692
#> GSM537594 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM537596 6 0.2247 0.83872 0.024 0.000 0.060 0.000 0.012 0.904
#> GSM537597 6 0.2009 0.84213 0.084 0.000 0.004 0.000 0.008 0.904
#> GSM537602 6 0.4462 0.67578 0.136 0.000 0.000 0.000 0.152 0.712
#> GSM340184 3 0.0146 0.81552 0.000 0.000 0.996 0.000 0.004 0.000
#> GSM340185 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340186 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340187 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340189 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340190 3 0.4718 0.47692 0.000 0.316 0.616 0.000 0.000 0.068
#> GSM340191 3 0.0260 0.81430 0.000 0.000 0.992 0.000 0.008 0.000
#> GSM340192 5 0.6857 0.01255 0.000 0.340 0.104 0.000 0.428 0.128
#> GSM340193 6 0.2946 0.77982 0.004 0.000 0.160 0.000 0.012 0.824
#> GSM340194 5 0.2378 0.85871 0.000 0.000 0.152 0.000 0.848 0.000
#> GSM340195 5 0.2378 0.85871 0.000 0.000 0.152 0.000 0.848 0.000
#> GSM340196 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340197 5 0.2491 0.85568 0.000 0.000 0.164 0.000 0.836 0.000
#> GSM340198 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340199 3 0.4159 0.69598 0.000 0.000 0.744 0.000 0.140 0.116
#> GSM340200 2 0.4765 0.63826 0.000 0.704 0.012 0.000 0.152 0.132
#> GSM340201 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340202 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340203 4 0.0000 0.93465 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340204 5 0.2908 0.82895 0.000 0.000 0.104 0.000 0.848 0.048
#> GSM340205 1 0.0260 0.89857 0.992 0.000 0.000 0.000 0.000 0.008
#> GSM340206 3 0.0000 0.81633 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340207 3 0.0458 0.81163 0.000 0.000 0.984 0.000 0.016 0.000
#> GSM340237 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340238 3 0.0000 0.81633 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340239 5 0.2941 0.81284 0.000 0.000 0.220 0.000 0.780 0.000
#> GSM340240 5 0.4269 0.76341 0.000 0.000 0.184 0.000 0.724 0.092
#> GSM340241 2 0.0000 0.91552 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340242 5 0.2527 0.85415 0.000 0.000 0.168 0.000 0.832 0.000
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
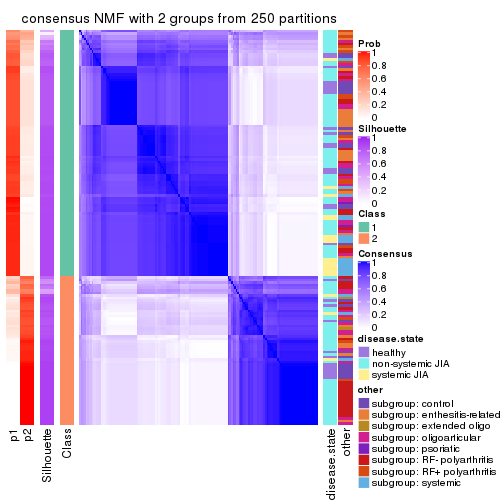
consensus_heatmap(res, k = 3)
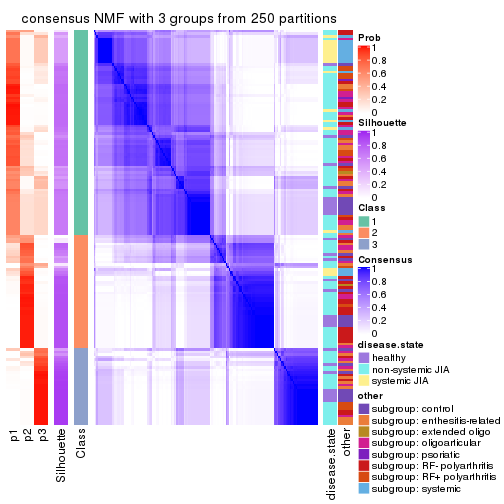
consensus_heatmap(res, k = 4)
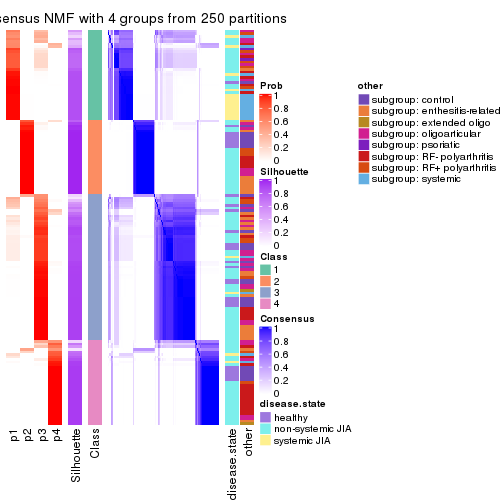
consensus_heatmap(res, k = 5)
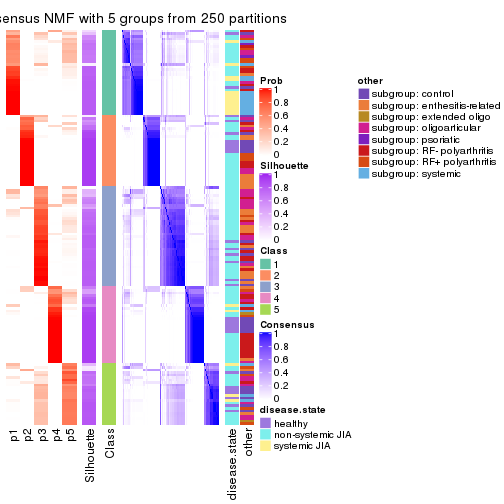
consensus_heatmap(res, k = 6)
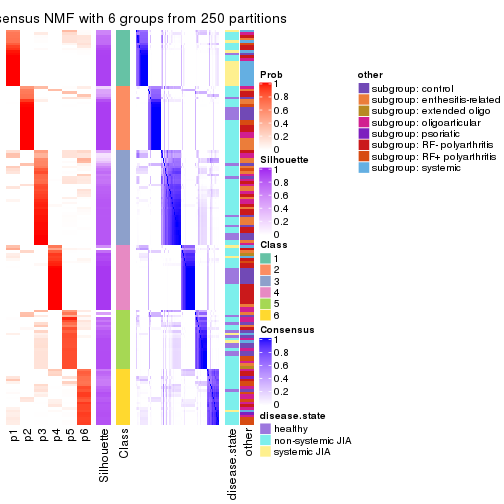
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
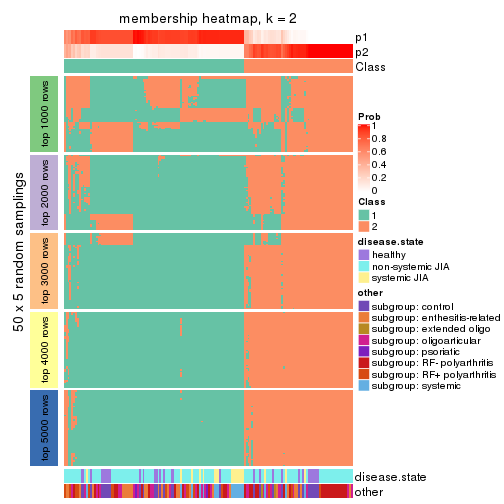
membership_heatmap(res, k = 3)
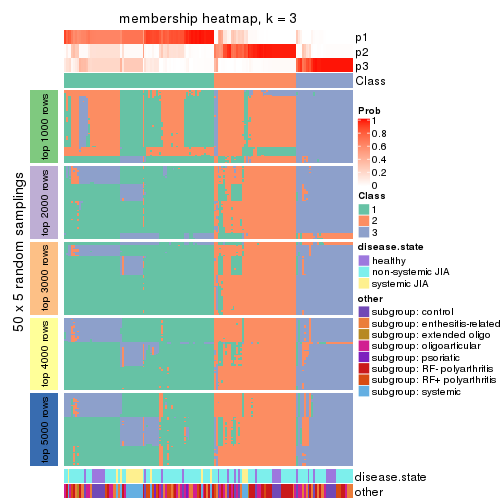
membership_heatmap(res, k = 4)
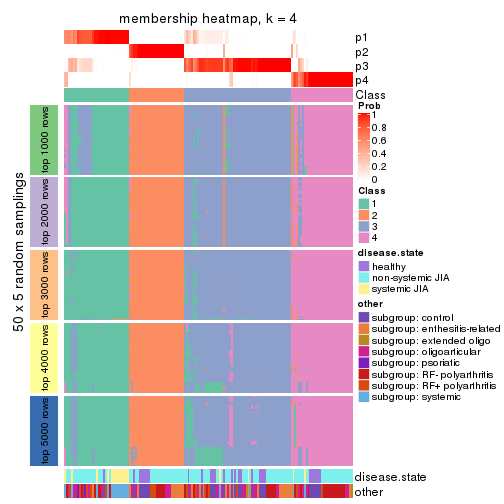
membership_heatmap(res, k = 5)
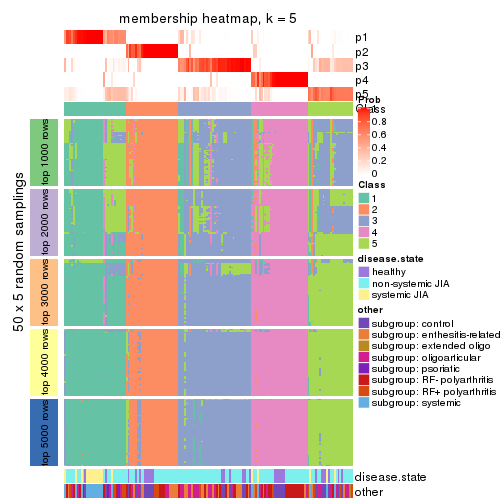
membership_heatmap(res, k = 6)
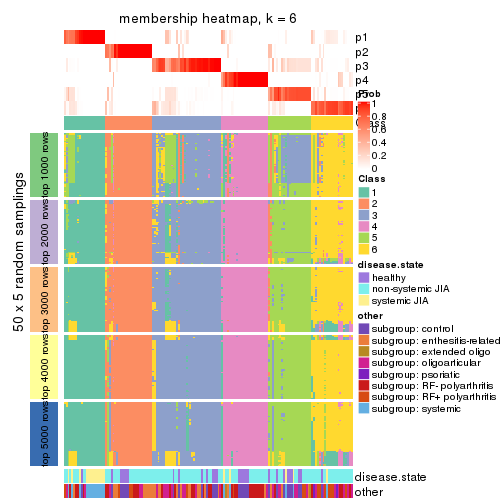
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
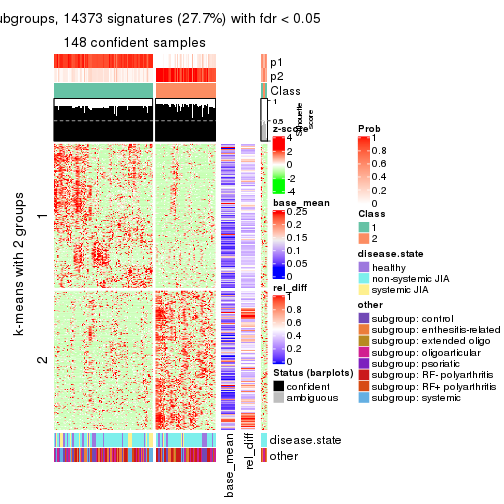
get_signatures(res, k = 3)
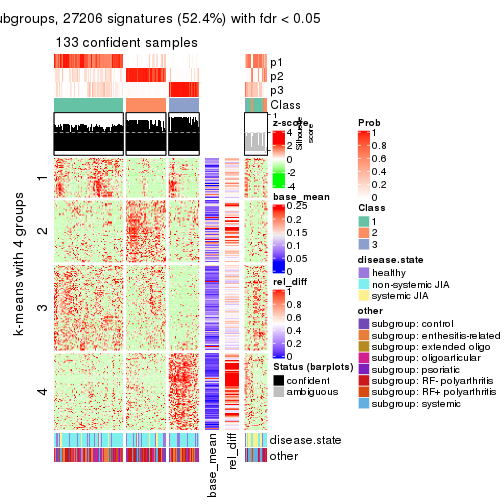
get_signatures(res, k = 4)
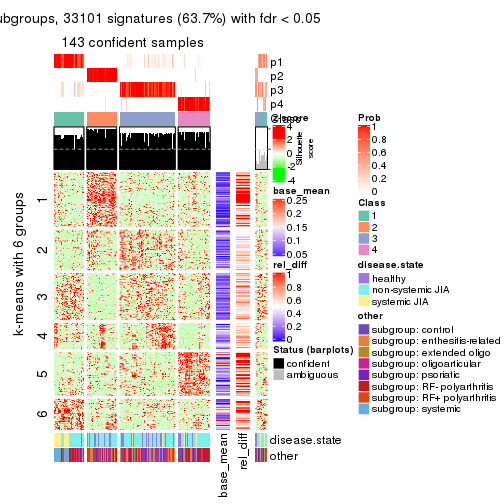
get_signatures(res, k = 5)
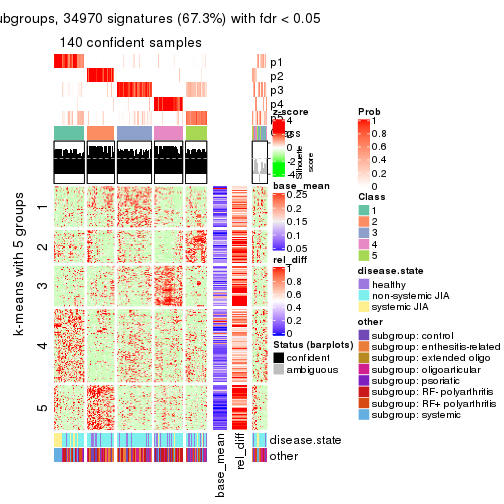
get_signatures(res, k = 6)
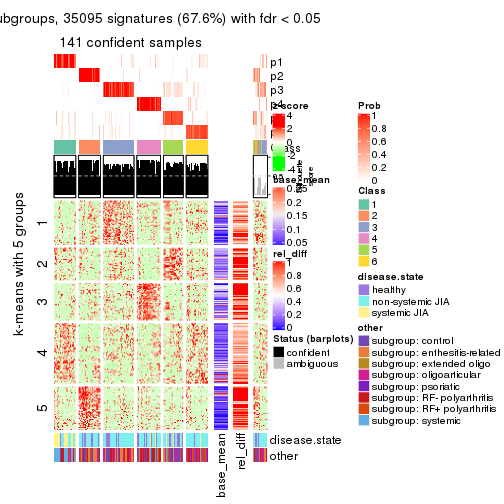
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
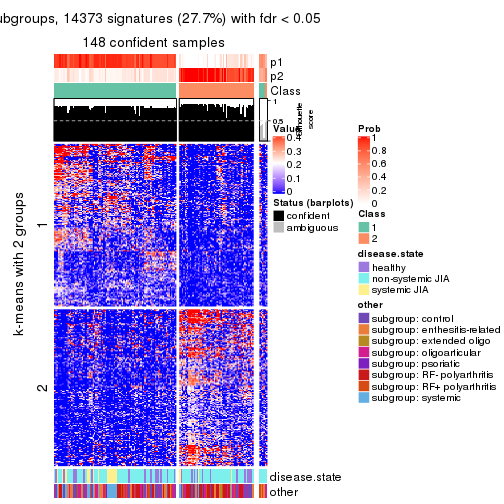
get_signatures(res, k = 3, scale_rows = FALSE)
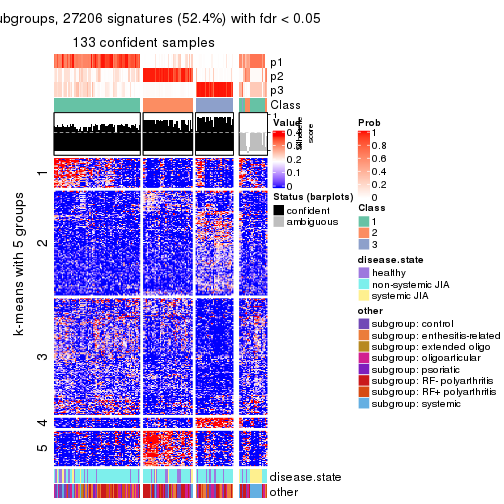
get_signatures(res, k = 4, scale_rows = FALSE)
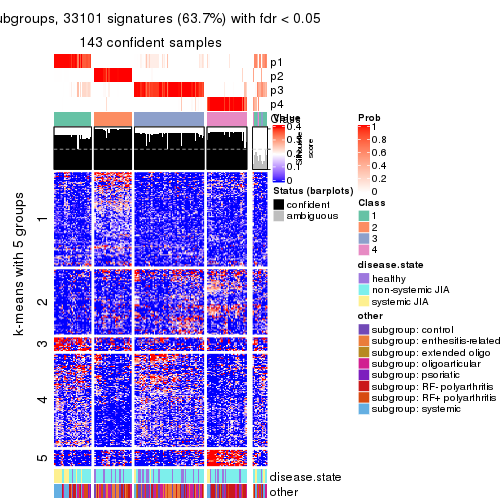
get_signatures(res, k = 5, scale_rows = FALSE)
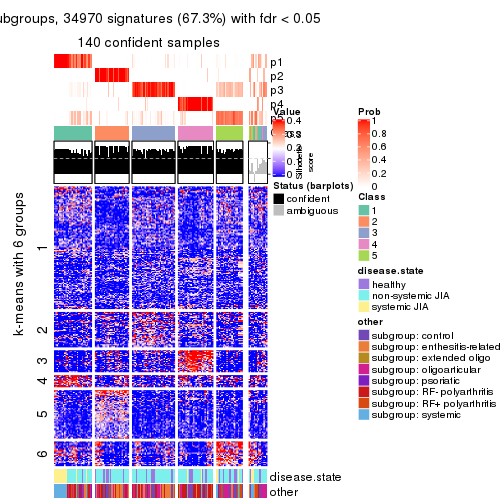
get_signatures(res, k = 6, scale_rows = FALSE)
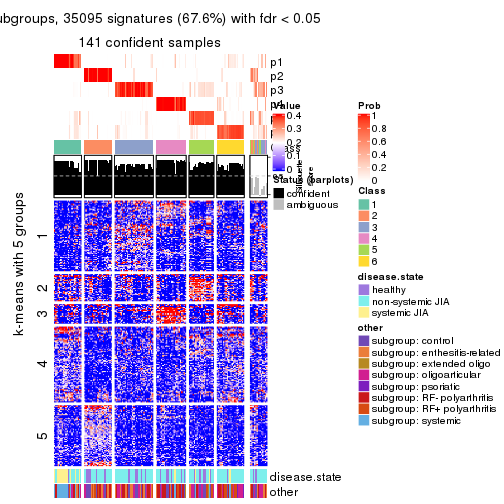
Compare the overlap of signatures from different k:
compare_signatures(res)
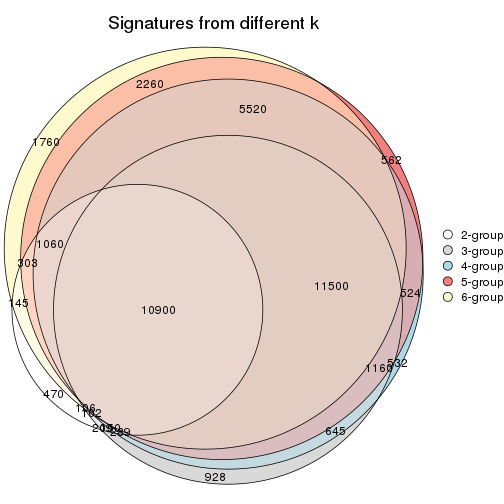
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
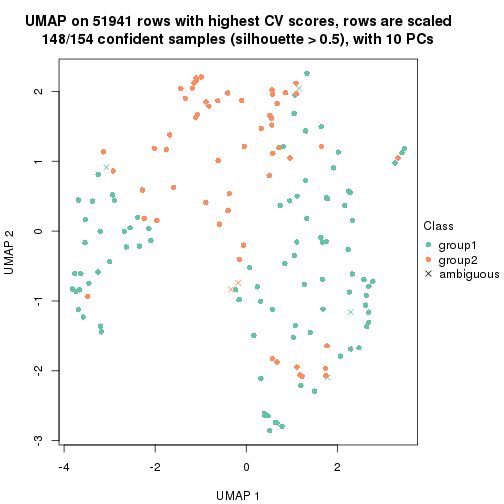
dimension_reduction(res, k = 3, method = "UMAP")
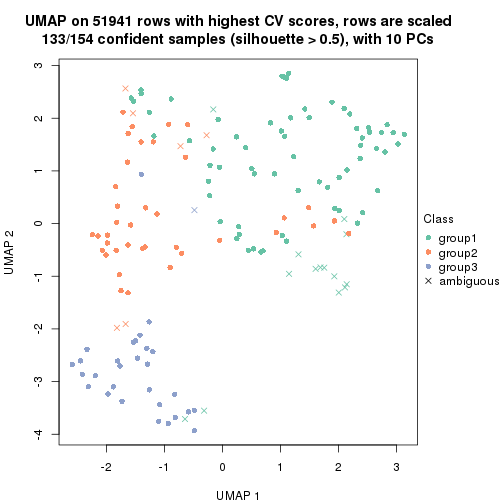
dimension_reduction(res, k = 4, method = "UMAP")
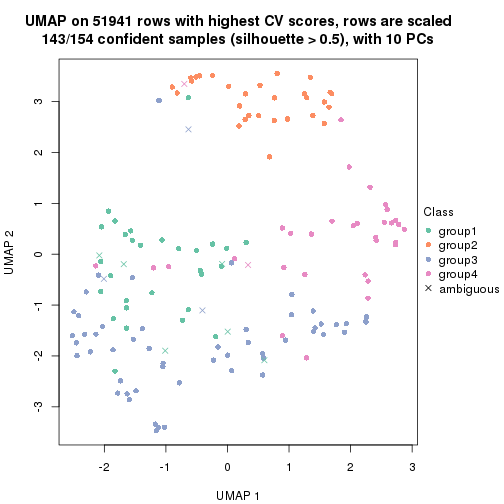
dimension_reduction(res, k = 5, method = "UMAP")
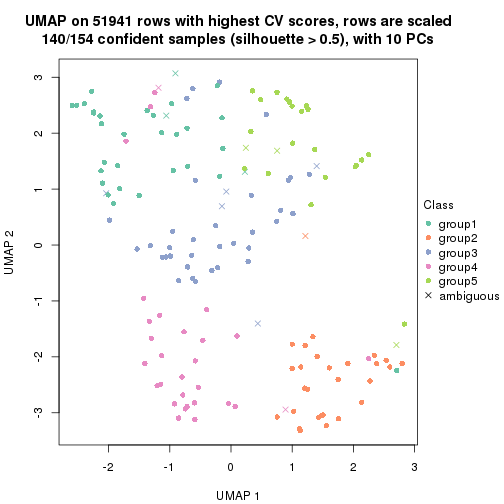
dimension_reduction(res, k = 6, method = "UMAP")
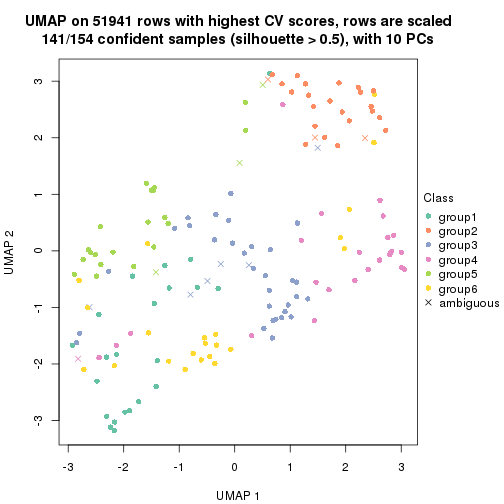
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
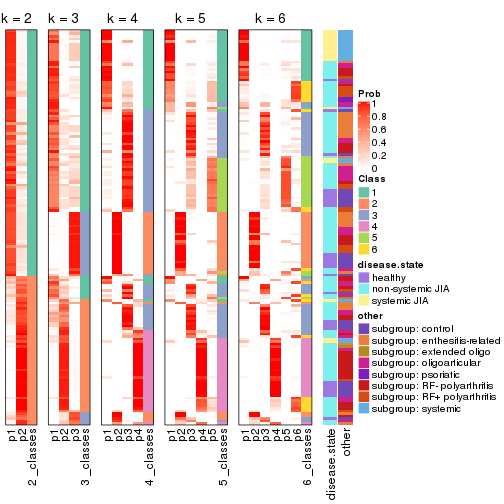
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> CV:NMF 148 1.41e-01 7.12e-03 2
#> CV:NMF 133 5.11e-01 6.63e-02 3
#> CV:NMF 143 2.11e-06 1.85e-07 4
#> CV:NMF 140 8.07e-06 1.72e-07 5
#> CV:NMF 141 3.11e-09 1.78e-10 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["MAD", "hclust"]
# you can also extract it by
# res = res_list["MAD:hclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'MAD' method.
#> Subgroups are detected by 'hclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
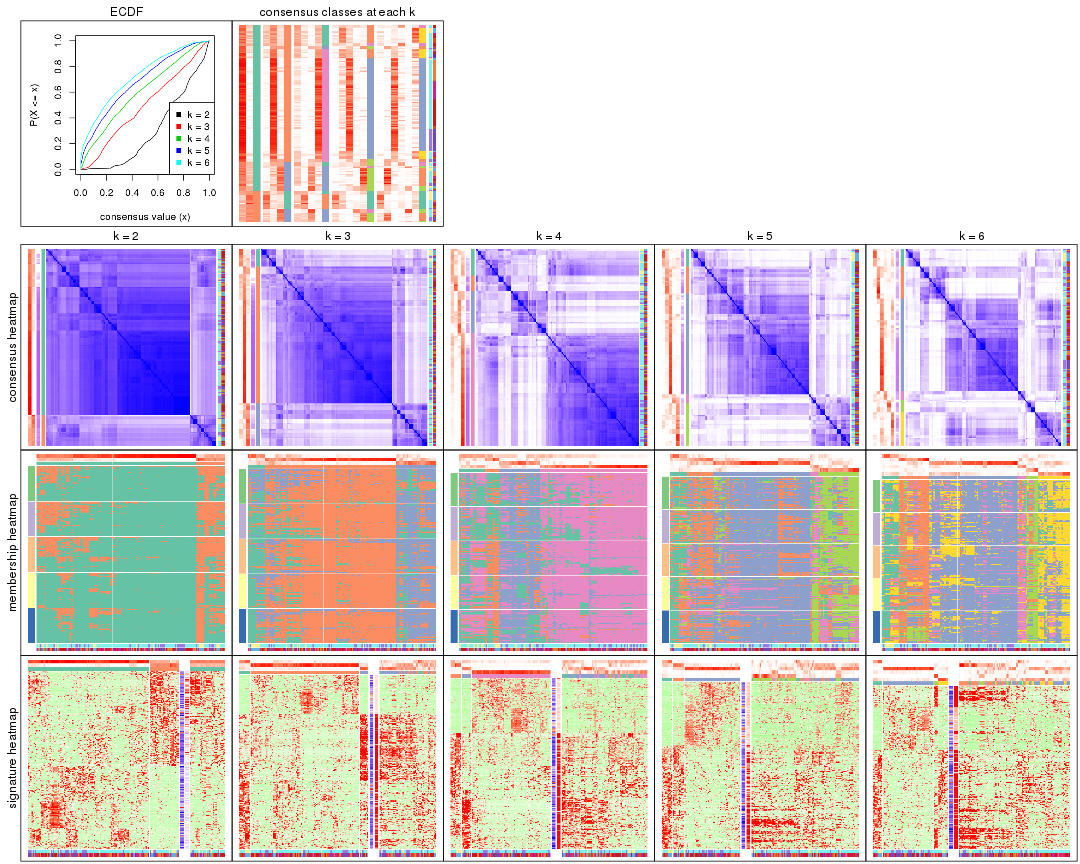
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
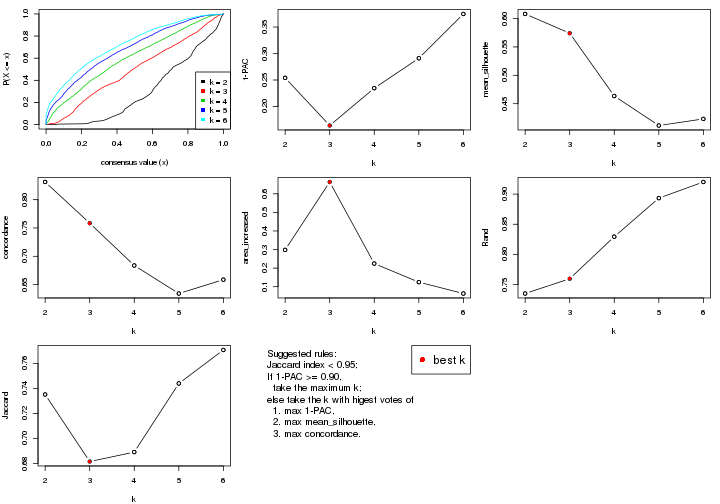
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.254 0.608 0.831 0.2983 0.735 0.735
#> 3 3 0.164 0.574 0.758 0.6637 0.760 0.682
#> 4 4 0.235 0.464 0.684 0.2246 0.829 0.689
#> 5 5 0.291 0.412 0.634 0.1245 0.893 0.744
#> 6 6 0.375 0.423 0.659 0.0634 0.920 0.771
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 1 0.6973 0.64541 0.812 0.188
#> GSM340359 1 0.8661 0.35571 0.712 0.288
#> GSM340361 2 0.9977 0.59374 0.472 0.528
#> GSM340362 1 0.9460 0.05366 0.636 0.364
#> GSM340363 1 0.6801 0.65321 0.820 0.180
#> GSM340364 2 0.9996 0.54572 0.488 0.512
#> GSM340365 2 0.9922 0.64183 0.448 0.552
#> GSM340366 1 0.9358 0.19797 0.648 0.352
#> GSM340367 2 0.7883 0.65231 0.236 0.764
#> GSM340368 1 0.2948 0.78571 0.948 0.052
#> GSM340369 1 0.1414 0.78710 0.980 0.020
#> GSM340370 2 0.7950 0.65122 0.240 0.760
#> GSM340371 1 0.9754 -0.19119 0.592 0.408
#> GSM340372 2 0.7883 0.65231 0.236 0.764
#> GSM340373 2 0.7883 0.65231 0.236 0.764
#> GSM340375 1 0.8861 0.28991 0.696 0.304
#> GSM340376 1 0.6973 0.65206 0.812 0.188
#> GSM340378 1 0.9710 0.00101 0.600 0.400
#> GSM340243 1 0.0938 0.79119 0.988 0.012
#> GSM340244 1 0.1414 0.78947 0.980 0.020
#> GSM340246 1 0.0938 0.79111 0.988 0.012
#> GSM340247 1 0.0000 0.78796 1.000 0.000
#> GSM340248 1 0.0000 0.78796 1.000 0.000
#> GSM340249 1 0.0672 0.79079 0.992 0.008
#> GSM340250 1 0.2778 0.78436 0.952 0.048
#> GSM340251 1 0.0672 0.78969 0.992 0.008
#> GSM340252 1 0.6623 0.63754 0.828 0.172
#> GSM340253 1 0.9286 0.13765 0.656 0.344
#> GSM340254 1 0.6801 0.63041 0.820 0.180
#> GSM340256 1 0.4298 0.75082 0.912 0.088
#> GSM340258 1 0.5408 0.71141 0.876 0.124
#> GSM340259 2 0.9358 0.57994 0.352 0.648
#> GSM340260 2 0.9286 0.68044 0.344 0.656
#> GSM340261 1 0.4431 0.73807 0.908 0.092
#> GSM340262 1 0.9170 0.18717 0.668 0.332
#> GSM340263 1 0.1633 0.78913 0.976 0.024
#> GSM340264 2 0.9896 0.56321 0.440 0.560
#> GSM340265 2 0.9896 0.56321 0.440 0.560
#> GSM340266 1 0.0938 0.79101 0.988 0.012
#> GSM340267 1 0.9608 -0.11224 0.616 0.384
#> GSM340268 1 0.1633 0.78607 0.976 0.024
#> GSM340269 1 0.0938 0.79009 0.988 0.012
#> GSM340270 1 0.7139 0.63699 0.804 0.196
#> GSM537574 1 0.0938 0.79012 0.988 0.012
#> GSM537580 1 0.9754 -0.19119 0.592 0.408
#> GSM537581 1 0.0672 0.78944 0.992 0.008
#> GSM340272 1 0.9427 0.15845 0.640 0.360
#> GSM340273 1 0.1843 0.78944 0.972 0.028
#> GSM340275 1 0.1414 0.78947 0.980 0.020
#> GSM340276 1 0.5408 0.73166 0.876 0.124
#> GSM340277 1 0.6148 0.66825 0.848 0.152
#> GSM340278 1 0.3114 0.77491 0.944 0.056
#> GSM340279 1 0.1184 0.79201 0.984 0.016
#> GSM340282 1 0.9358 0.19797 0.648 0.352
#> GSM340284 1 0.0000 0.78796 1.000 0.000
#> GSM340285 1 0.6623 0.66573 0.828 0.172
#> GSM340286 1 0.8955 0.26355 0.688 0.312
#> GSM340287 1 0.4562 0.73394 0.904 0.096
#> GSM340288 2 0.9881 0.63788 0.436 0.564
#> GSM340289 2 0.2778 0.53897 0.048 0.952
#> GSM340290 1 0.1414 0.79273 0.980 0.020
#> GSM340291 1 0.1843 0.79085 0.972 0.028
#> GSM340293 1 0.1633 0.78568 0.976 0.024
#> GSM340294 1 0.4022 0.74924 0.920 0.080
#> GSM340296 1 0.0938 0.79119 0.988 0.012
#> GSM340297 1 0.4690 0.73015 0.900 0.100
#> GSM340298 1 0.2043 0.78947 0.968 0.032
#> GSM340299 1 0.9460 0.05366 0.636 0.364
#> GSM340301 2 0.9983 0.55546 0.476 0.524
#> GSM340303 2 0.9896 0.56321 0.440 0.560
#> GSM340304 1 0.6712 0.68452 0.824 0.176
#> GSM340306 1 0.7950 0.50682 0.760 0.240
#> GSM340307 1 0.2236 0.78969 0.964 0.036
#> GSM340310 1 0.7745 0.58452 0.772 0.228
#> GSM340314 1 0.4690 0.73015 0.900 0.100
#> GSM340315 1 0.2043 0.78522 0.968 0.032
#> GSM340317 1 0.1633 0.78913 0.976 0.024
#> GSM340318 1 0.0000 0.78796 1.000 0.000
#> GSM340319 1 0.0000 0.78796 1.000 0.000
#> GSM340320 1 0.4690 0.73015 0.900 0.100
#> GSM340321 1 0.0000 0.78796 1.000 0.000
#> GSM340322 1 0.0938 0.79009 0.988 0.012
#> GSM340324 1 0.4298 0.75082 0.912 0.088
#> GSM340328 1 0.6973 0.64541 0.812 0.188
#> GSM340330 1 0.1633 0.79196 0.976 0.024
#> GSM340332 1 0.0938 0.78899 0.988 0.012
#> GSM340333 1 0.8713 0.34133 0.708 0.292
#> GSM340336 1 0.0000 0.78796 1.000 0.000
#> GSM340337 1 0.0938 0.79009 0.988 0.012
#> GSM340338 1 0.5842 0.71687 0.860 0.140
#> GSM340339 1 0.0938 0.78923 0.988 0.012
#> GSM340340 1 0.2948 0.78454 0.948 0.052
#> GSM340341 1 0.3733 0.76909 0.928 0.072
#> GSM340343 1 0.8813 0.36518 0.700 0.300
#> GSM340344 1 0.9286 0.13765 0.656 0.344
#> GSM340346 1 0.3431 0.78081 0.936 0.064
#> GSM340347 1 0.0000 0.78796 1.000 0.000
#> GSM340348 1 0.1843 0.79158 0.972 0.028
#> GSM340349 1 0.8909 0.37678 0.692 0.308
#> GSM340350 1 0.8813 0.41726 0.700 0.300
#> GSM340351 2 0.9850 0.64898 0.428 0.572
#> GSM340354 1 0.0938 0.79120 0.988 0.012
#> GSM340356 1 0.2236 0.78782 0.964 0.036
#> GSM340357 1 0.9552 0.09394 0.624 0.376
#> GSM348183 1 0.9896 -0.18746 0.560 0.440
#> GSM348191 1 0.4161 0.75401 0.916 0.084
#> GSM348193 1 0.7745 0.58452 0.772 0.228
#> GSM537578 1 0.9732 -0.05314 0.596 0.404
#> GSM348181 1 0.9732 0.02259 0.596 0.404
#> GSM348182 2 0.9044 0.67762 0.320 0.680
#> GSM348184 1 0.0000 0.78796 1.000 0.000
#> GSM348185 1 0.2236 0.78861 0.964 0.036
#> GSM348186 1 0.6247 0.66835 0.844 0.156
#> GSM348187 1 0.6048 0.69970 0.852 0.148
#> GSM348188 1 0.9248 0.29175 0.660 0.340
#> GSM348189 1 0.9754 -0.19119 0.592 0.408
#> GSM348190 1 0.9732 -0.14084 0.596 0.404
#> GSM348194 2 0.9881 0.63788 0.436 0.564
#> GSM348195 2 0.9850 0.64898 0.428 0.572
#> GSM348196 1 0.6048 0.69970 0.852 0.148
#> GSM537585 2 0.9427 0.68250 0.360 0.640
#> GSM537594 1 0.1633 0.78901 0.976 0.024
#> GSM537596 1 0.4298 0.76732 0.912 0.088
#> GSM537597 1 0.3431 0.78090 0.936 0.064
#> GSM537602 1 0.6712 0.68452 0.824 0.176
#> GSM340184 1 0.1184 0.79002 0.984 0.016
#> GSM340185 1 0.0672 0.78962 0.992 0.008
#> GSM340186 1 0.0672 0.78969 0.992 0.008
#> GSM340187 1 0.0000 0.78796 1.000 0.000
#> GSM340189 1 0.1633 0.78568 0.976 0.024
#> GSM340190 1 0.0000 0.78796 1.000 0.000
#> GSM340191 1 0.1633 0.78663 0.976 0.024
#> GSM340192 1 0.5294 0.71065 0.880 0.120
#> GSM340193 2 0.9552 0.56064 0.376 0.624
#> GSM340194 1 0.9460 0.05366 0.636 0.364
#> GSM340195 2 0.9998 0.51488 0.492 0.508
#> GSM340196 1 0.0000 0.78796 1.000 0.000
#> GSM340197 2 0.9970 0.57343 0.468 0.532
#> GSM340198 1 0.6801 0.63041 0.820 0.180
#> GSM340199 1 0.1843 0.78515 0.972 0.028
#> GSM340200 1 0.0938 0.79135 0.988 0.012
#> GSM340201 1 0.0000 0.78796 1.000 0.000
#> GSM340202 1 0.0000 0.78796 1.000 0.000
#> GSM340203 1 0.0376 0.78889 0.996 0.004
#> GSM340204 1 0.6531 0.65072 0.832 0.168
#> GSM340205 1 0.8955 0.36498 0.688 0.312
#> GSM340206 1 0.0672 0.79045 0.992 0.008
#> GSM340207 1 0.6531 0.64173 0.832 0.168
#> GSM340237 1 0.1843 0.79085 0.972 0.028
#> GSM340238 1 0.1414 0.78759 0.980 0.020
#> GSM340239 2 0.9754 0.66193 0.408 0.592
#> GSM340240 1 0.6148 0.66825 0.848 0.152
#> GSM340241 1 0.1843 0.78897 0.972 0.028
#> GSM340242 1 0.9754 -0.19119 0.592 0.408
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 2 0.7906 0.5860 0.220 0.656 0.124
#> GSM340359 3 0.5812 0.5091 0.012 0.264 0.724
#> GSM340361 1 0.8938 0.5328 0.528 0.144 0.328
#> GSM340362 3 0.5178 0.5158 0.028 0.164 0.808
#> GSM340363 2 0.7906 0.5947 0.220 0.656 0.124
#> GSM340364 1 0.9553 0.4477 0.484 0.272 0.244
#> GSM340365 1 0.9289 0.3868 0.444 0.160 0.396
#> GSM340366 3 0.8984 0.1610 0.128 0.432 0.440
#> GSM340367 1 0.5915 0.6698 0.792 0.080 0.128
#> GSM340368 2 0.4914 0.7711 0.088 0.844 0.068
#> GSM340369 2 0.1751 0.7938 0.012 0.960 0.028
#> GSM340370 1 0.6091 0.6697 0.784 0.092 0.124
#> GSM340371 3 0.7421 0.4889 0.084 0.240 0.676
#> GSM340372 1 0.5915 0.6698 0.792 0.080 0.128
#> GSM340373 1 0.5915 0.6698 0.792 0.080 0.128
#> GSM340375 3 0.9385 0.1083 0.248 0.240 0.512
#> GSM340376 2 0.7884 0.5876 0.224 0.656 0.120
#> GSM340378 2 0.9398 -0.1718 0.400 0.428 0.172
#> GSM340243 2 0.1919 0.7974 0.020 0.956 0.024
#> GSM340244 2 0.3481 0.7835 0.044 0.904 0.052
#> GSM340246 2 0.2443 0.7974 0.028 0.940 0.032
#> GSM340247 2 0.0892 0.7938 0.000 0.980 0.020
#> GSM340248 2 0.1585 0.7918 0.008 0.964 0.028
#> GSM340249 2 0.2527 0.7961 0.020 0.936 0.044
#> GSM340250 2 0.5803 0.6505 0.028 0.760 0.212
#> GSM340251 2 0.1289 0.7921 0.000 0.968 0.032
#> GSM340252 3 0.7069 0.2389 0.020 0.472 0.508
#> GSM340253 3 0.4473 0.5244 0.008 0.164 0.828
#> GSM340254 3 0.7174 0.2678 0.024 0.460 0.516
#> GSM340256 2 0.5826 0.6615 0.032 0.764 0.204
#> GSM340258 2 0.6651 0.4237 0.024 0.656 0.320
#> GSM340259 1 0.7979 0.4903 0.640 0.248 0.112
#> GSM340260 3 0.7874 0.2000 0.320 0.076 0.604
#> GSM340261 2 0.3888 0.7676 0.064 0.888 0.048
#> GSM340262 3 0.6954 0.4431 0.028 0.352 0.620
#> GSM340263 2 0.3694 0.7811 0.052 0.896 0.052
#> GSM340264 3 0.5988 0.3510 0.168 0.056 0.776
#> GSM340265 3 0.5875 0.3559 0.160 0.056 0.784
#> GSM340266 2 0.2947 0.7973 0.020 0.920 0.060
#> GSM340267 3 0.7930 0.3948 0.168 0.168 0.664
#> GSM340268 2 0.1711 0.7938 0.008 0.960 0.032
#> GSM340269 2 0.2584 0.7905 0.008 0.928 0.064
#> GSM340270 2 0.7317 0.6297 0.208 0.696 0.096
#> GSM537574 2 0.2173 0.7953 0.008 0.944 0.048
#> GSM537580 3 0.7421 0.4889 0.084 0.240 0.676
#> GSM537581 2 0.3337 0.7941 0.032 0.908 0.060
#> GSM340272 3 0.9070 0.1514 0.136 0.428 0.436
#> GSM340273 2 0.4921 0.7643 0.072 0.844 0.084
#> GSM340275 2 0.3481 0.7835 0.044 0.904 0.052
#> GSM340276 2 0.5965 0.7453 0.108 0.792 0.100
#> GSM340277 2 0.6819 -0.1499 0.012 0.512 0.476
#> GSM340278 2 0.3042 0.7907 0.040 0.920 0.040
#> GSM340279 2 0.1832 0.7953 0.008 0.956 0.036
#> GSM340282 3 0.8984 0.1610 0.128 0.432 0.440
#> GSM340284 2 0.1163 0.7891 0.000 0.972 0.028
#> GSM340285 2 0.5875 0.7214 0.136 0.792 0.072
#> GSM340286 3 0.6326 0.4928 0.020 0.292 0.688
#> GSM340287 2 0.4189 0.7627 0.068 0.876 0.056
#> GSM340288 1 0.8494 0.6487 0.608 0.156 0.236
#> GSM340289 1 0.3995 0.4979 0.868 0.016 0.116
#> GSM340290 2 0.2527 0.8007 0.020 0.936 0.044
#> GSM340291 2 0.3481 0.7915 0.052 0.904 0.044
#> GSM340293 2 0.1877 0.7926 0.012 0.956 0.032
#> GSM340294 2 0.3583 0.7748 0.056 0.900 0.044
#> GSM340296 2 0.1919 0.7974 0.020 0.956 0.024
#> GSM340297 2 0.4189 0.7645 0.068 0.876 0.056
#> GSM340298 2 0.3183 0.7911 0.016 0.908 0.076
#> GSM340299 3 0.5178 0.5158 0.028 0.164 0.808
#> GSM340301 3 0.7699 0.3152 0.212 0.116 0.672
#> GSM340303 3 0.5875 0.3559 0.160 0.056 0.784
#> GSM340304 2 0.7223 0.6679 0.144 0.716 0.140
#> GSM340306 2 0.9213 0.2487 0.228 0.536 0.236
#> GSM340307 2 0.3528 0.7849 0.016 0.892 0.092
#> GSM340310 2 0.8255 0.5226 0.252 0.620 0.128
#> GSM340314 2 0.3583 0.7668 0.056 0.900 0.044
#> GSM340315 2 0.3896 0.7784 0.060 0.888 0.052
#> GSM340317 2 0.3694 0.7811 0.052 0.896 0.052
#> GSM340318 2 0.0747 0.7932 0.000 0.984 0.016
#> GSM340319 2 0.0747 0.7932 0.000 0.984 0.016
#> GSM340320 2 0.4189 0.7645 0.068 0.876 0.056
#> GSM340321 2 0.1163 0.7891 0.000 0.972 0.028
#> GSM340322 2 0.2550 0.7959 0.012 0.932 0.056
#> GSM340324 2 0.5826 0.6615 0.032 0.764 0.204
#> GSM340328 2 0.7844 0.5918 0.220 0.660 0.120
#> GSM340330 2 0.3129 0.7851 0.008 0.904 0.088
#> GSM340332 2 0.1877 0.7985 0.012 0.956 0.032
#> GSM340333 3 0.5775 0.5112 0.012 0.260 0.728
#> GSM340336 2 0.0747 0.7932 0.000 0.984 0.016
#> GSM340337 2 0.2902 0.7931 0.016 0.920 0.064
#> GSM340338 2 0.5919 0.6234 0.016 0.724 0.260
#> GSM340339 2 0.2229 0.7997 0.012 0.944 0.044
#> GSM340340 2 0.4544 0.7723 0.084 0.860 0.056
#> GSM340341 2 0.4782 0.7356 0.016 0.820 0.164
#> GSM340343 2 0.7424 0.5121 0.288 0.648 0.064
#> GSM340344 3 0.4473 0.5244 0.008 0.164 0.828
#> GSM340346 2 0.4209 0.7727 0.020 0.860 0.120
#> GSM340347 2 0.1765 0.7961 0.004 0.956 0.040
#> GSM340348 2 0.3325 0.7906 0.020 0.904 0.076
#> GSM340349 2 0.8994 0.3278 0.260 0.556 0.184
#> GSM340350 2 0.9181 0.3107 0.236 0.540 0.224
#> GSM340351 1 0.8494 0.6497 0.608 0.156 0.236
#> GSM340354 2 0.1643 0.7898 0.000 0.956 0.044
#> GSM340356 2 0.6111 0.7217 0.112 0.784 0.104
#> GSM340357 2 0.9621 -0.1323 0.360 0.432 0.208
#> GSM348183 2 0.9576 -0.2246 0.396 0.408 0.196
#> GSM348191 2 0.4179 0.7740 0.052 0.876 0.072
#> GSM348193 2 0.8255 0.5226 0.252 0.620 0.128
#> GSM537578 2 0.9616 -0.1704 0.376 0.420 0.204
#> GSM348181 2 0.8714 0.0731 0.408 0.484 0.108
#> GSM348182 3 0.7600 0.1483 0.344 0.056 0.600
#> GSM348184 2 0.1163 0.7891 0.000 0.972 0.028
#> GSM348185 2 0.5093 0.7587 0.088 0.836 0.076
#> GSM348186 2 0.7187 -0.1797 0.024 0.496 0.480
#> GSM348187 2 0.8392 0.5446 0.200 0.624 0.176
#> GSM348188 2 0.9133 0.2083 0.332 0.508 0.160
#> GSM348189 3 0.7421 0.4889 0.084 0.240 0.676
#> GSM348190 3 0.8608 0.3235 0.204 0.192 0.604
#> GSM348194 1 0.8494 0.6487 0.608 0.156 0.236
#> GSM348195 1 0.8494 0.6497 0.608 0.156 0.236
#> GSM348196 2 0.8392 0.5446 0.200 0.624 0.176
#> GSM537585 3 0.8902 -0.1205 0.396 0.124 0.480
#> GSM537594 2 0.4925 0.7618 0.076 0.844 0.080
#> GSM537596 2 0.6313 0.7352 0.084 0.768 0.148
#> GSM537597 2 0.6850 0.6927 0.120 0.740 0.140
#> GSM537602 2 0.7223 0.6679 0.144 0.716 0.140
#> GSM340184 2 0.2749 0.7902 0.012 0.924 0.064
#> GSM340185 2 0.1647 0.7894 0.004 0.960 0.036
#> GSM340186 2 0.1529 0.7910 0.000 0.960 0.040
#> GSM340187 2 0.1031 0.7895 0.000 0.976 0.024
#> GSM340189 2 0.1711 0.7924 0.008 0.960 0.032
#> GSM340190 2 0.1399 0.7891 0.004 0.968 0.028
#> GSM340191 2 0.2414 0.8008 0.020 0.940 0.040
#> GSM340192 2 0.6745 0.0414 0.012 0.560 0.428
#> GSM340193 1 0.8103 0.4913 0.632 0.248 0.120
#> GSM340194 3 0.5178 0.5158 0.028 0.164 0.808
#> GSM340195 3 0.7113 0.4170 0.168 0.112 0.720
#> GSM340196 2 0.0892 0.7938 0.000 0.980 0.020
#> GSM340197 3 0.7757 0.2934 0.224 0.112 0.664
#> GSM340198 3 0.7174 0.2678 0.024 0.460 0.516
#> GSM340199 2 0.2063 0.7946 0.008 0.948 0.044
#> GSM340200 2 0.1989 0.7930 0.004 0.948 0.048
#> GSM340201 2 0.1711 0.7963 0.008 0.960 0.032
#> GSM340202 2 0.1031 0.7949 0.000 0.976 0.024
#> GSM340203 2 0.1620 0.7942 0.012 0.964 0.024
#> GSM340204 3 0.7584 0.2211 0.040 0.472 0.488
#> GSM340205 2 0.8137 0.4193 0.316 0.592 0.092
#> GSM340206 2 0.2492 0.7940 0.016 0.936 0.048
#> GSM340207 2 0.6994 0.0486 0.020 0.556 0.424
#> GSM340237 2 0.3481 0.7915 0.052 0.904 0.044
#> GSM340238 2 0.1751 0.7950 0.012 0.960 0.028
#> GSM340239 3 0.8345 0.0390 0.344 0.096 0.560
#> GSM340240 2 0.6825 -0.1850 0.012 0.500 0.488
#> GSM340241 2 0.3459 0.7843 0.012 0.892 0.096
#> GSM340242 3 0.7421 0.4889 0.084 0.240 0.676
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.6655 0.271357 0.476 0.084 0.000 0.440
#> GSM340359 3 0.6829 0.530009 0.256 0.004 0.604 0.136
#> GSM340361 1 0.7944 -0.582029 0.436 0.412 0.112 0.040
#> GSM340362 3 0.5544 0.503477 0.332 0.008 0.640 0.020
#> GSM340363 1 0.6658 0.257443 0.472 0.084 0.000 0.444
#> GSM340364 2 0.8818 0.373019 0.352 0.408 0.072 0.168
#> GSM340365 1 0.8694 -0.507253 0.384 0.380 0.180 0.056
#> GSM340366 1 0.8627 -0.005863 0.464 0.056 0.280 0.200
#> GSM340367 2 0.5080 0.681535 0.216 0.744 0.012 0.028
#> GSM340368 4 0.5090 0.491833 0.312 0.012 0.004 0.672
#> GSM340369 4 0.1792 0.726983 0.068 0.000 0.000 0.932
#> GSM340370 2 0.5242 0.675801 0.224 0.732 0.012 0.032
#> GSM340371 3 0.4612 0.480478 0.020 0.012 0.780 0.188
#> GSM340372 2 0.5080 0.681535 0.216 0.744 0.012 0.028
#> GSM340373 2 0.5080 0.681535 0.216 0.744 0.012 0.028
#> GSM340375 3 0.9250 0.000662 0.256 0.144 0.440 0.160
#> GSM340376 1 0.6826 0.251228 0.460 0.084 0.004 0.452
#> GSM340378 1 0.8331 0.270293 0.388 0.304 0.016 0.292
#> GSM340243 4 0.1938 0.733560 0.052 0.012 0.000 0.936
#> GSM340244 4 0.3764 0.631389 0.216 0.000 0.000 0.784
#> GSM340246 4 0.2599 0.730238 0.064 0.020 0.004 0.912
#> GSM340247 4 0.1824 0.728040 0.060 0.000 0.004 0.936
#> GSM340248 4 0.2234 0.732742 0.064 0.008 0.004 0.924
#> GSM340249 4 0.3024 0.694857 0.148 0.000 0.000 0.852
#> GSM340250 4 0.5977 0.462461 0.192 0.000 0.120 0.688
#> GSM340251 4 0.0817 0.729727 0.024 0.000 0.000 0.976
#> GSM340252 3 0.7911 0.211563 0.236 0.004 0.404 0.356
#> GSM340253 3 0.5471 0.521852 0.284 0.008 0.680 0.028
#> GSM340254 3 0.7908 0.238276 0.240 0.004 0.416 0.340
#> GSM340256 4 0.6441 0.408809 0.220 0.004 0.124 0.652
#> GSM340258 4 0.7156 0.144904 0.184 0.004 0.236 0.576
#> GSM340259 2 0.7193 0.338636 0.268 0.580 0.012 0.140
#> GSM340260 3 0.7621 0.268997 0.200 0.252 0.536 0.012
#> GSM340261 4 0.4611 0.641893 0.172 0.032 0.008 0.788
#> GSM340262 3 0.7908 0.434392 0.232 0.016 0.504 0.248
#> GSM340263 4 0.4194 0.607969 0.228 0.008 0.000 0.764
#> GSM340264 3 0.4098 0.402406 0.024 0.104 0.844 0.028
#> GSM340265 3 0.3975 0.407937 0.024 0.096 0.852 0.028
#> GSM340266 4 0.3577 0.699469 0.156 0.000 0.012 0.832
#> GSM340267 3 0.7524 0.377078 0.144 0.124 0.640 0.092
#> GSM340268 4 0.2149 0.724703 0.088 0.000 0.000 0.912
#> GSM340269 4 0.2730 0.724906 0.088 0.000 0.016 0.896
#> GSM340270 4 0.7187 -0.042619 0.364 0.104 0.012 0.520
#> GSM537574 4 0.2329 0.725308 0.072 0.000 0.012 0.916
#> GSM537580 3 0.4612 0.480478 0.020 0.012 0.780 0.188
#> GSM537581 4 0.3808 0.673606 0.184 0.004 0.004 0.808
#> GSM340272 1 0.8691 -0.009905 0.456 0.060 0.288 0.196
#> GSM340273 4 0.4957 0.433306 0.336 0.004 0.004 0.656
#> GSM340275 4 0.3764 0.634600 0.216 0.000 0.000 0.784
#> GSM340276 4 0.5646 0.497395 0.272 0.056 0.000 0.672
#> GSM340277 3 0.8099 0.221386 0.256 0.008 0.380 0.356
#> GSM340278 4 0.3672 0.691851 0.164 0.012 0.000 0.824
#> GSM340279 4 0.2862 0.726174 0.076 0.012 0.012 0.900
#> GSM340282 1 0.8627 -0.005863 0.464 0.056 0.280 0.200
#> GSM340284 4 0.0707 0.727433 0.020 0.000 0.000 0.980
#> GSM340285 4 0.6333 0.441057 0.272 0.088 0.004 0.636
#> GSM340286 3 0.7532 0.520586 0.256 0.016 0.556 0.172
#> GSM340287 4 0.4640 0.633519 0.188 0.032 0.004 0.776
#> GSM340288 2 0.7588 0.603355 0.436 0.448 0.060 0.056
#> GSM340289 2 0.2111 0.534490 0.024 0.932 0.044 0.000
#> GSM340290 4 0.2197 0.735912 0.080 0.004 0.000 0.916
#> GSM340291 4 0.4139 0.659396 0.176 0.024 0.000 0.800
#> GSM340293 4 0.2053 0.725325 0.072 0.004 0.000 0.924
#> GSM340294 4 0.4381 0.664172 0.152 0.032 0.008 0.808
#> GSM340296 4 0.1938 0.733560 0.052 0.012 0.000 0.936
#> GSM340297 4 0.4644 0.628570 0.208 0.024 0.004 0.764
#> GSM340298 4 0.5118 0.561463 0.224 0.008 0.032 0.736
#> GSM340299 3 0.5544 0.503477 0.332 0.008 0.640 0.020
#> GSM340301 3 0.7623 0.361363 0.288 0.192 0.512 0.008
#> GSM340303 3 0.3975 0.407937 0.024 0.096 0.852 0.028
#> GSM340304 4 0.6863 0.040548 0.380 0.056 0.024 0.540
#> GSM340306 4 0.8983 -0.235404 0.284 0.140 0.120 0.456
#> GSM340307 4 0.5408 0.536953 0.228 0.008 0.044 0.720
#> GSM340310 1 0.7030 0.349340 0.472 0.120 0.000 0.408
#> GSM340314 4 0.4232 0.651908 0.168 0.024 0.004 0.804
#> GSM340315 4 0.3845 0.691848 0.068 0.048 0.020 0.864
#> GSM340317 4 0.4194 0.607969 0.228 0.008 0.000 0.764
#> GSM340318 4 0.1661 0.728308 0.052 0.000 0.004 0.944
#> GSM340319 4 0.1661 0.728308 0.052 0.000 0.004 0.944
#> GSM340320 4 0.4785 0.621424 0.208 0.024 0.008 0.760
#> GSM340321 4 0.0707 0.727433 0.020 0.000 0.000 0.980
#> GSM340322 4 0.3032 0.709570 0.124 0.000 0.008 0.868
#> GSM340324 4 0.6441 0.408809 0.220 0.004 0.124 0.652
#> GSM340328 1 0.6658 0.253039 0.472 0.084 0.000 0.444
#> GSM340330 4 0.3170 0.713988 0.056 0.008 0.044 0.892
#> GSM340332 4 0.1940 0.734797 0.076 0.000 0.000 0.924
#> GSM340333 3 0.6786 0.531235 0.256 0.004 0.608 0.132
#> GSM340336 4 0.1661 0.728308 0.052 0.000 0.004 0.944
#> GSM340337 4 0.3529 0.691728 0.152 0.000 0.012 0.836
#> GSM340338 4 0.7114 0.253862 0.236 0.008 0.164 0.592
#> GSM340339 4 0.2466 0.732747 0.096 0.000 0.004 0.900
#> GSM340340 4 0.4826 0.547240 0.264 0.020 0.000 0.716
#> GSM340341 4 0.5427 0.602307 0.148 0.004 0.100 0.748
#> GSM340343 4 0.7540 0.022927 0.244 0.232 0.004 0.520
#> GSM340344 3 0.5471 0.521852 0.284 0.008 0.680 0.028
#> GSM340346 4 0.5778 0.469074 0.256 0.008 0.052 0.684
#> GSM340347 4 0.2466 0.721713 0.096 0.000 0.004 0.900
#> GSM340348 4 0.4289 0.668216 0.172 0.000 0.032 0.796
#> GSM340349 1 0.8151 0.311745 0.484 0.140 0.044 0.332
#> GSM340350 1 0.7957 0.344222 0.520 0.112 0.052 0.316
#> GSM340351 2 0.7585 0.605569 0.428 0.456 0.060 0.056
#> GSM340354 4 0.2360 0.728070 0.052 0.004 0.020 0.924
#> GSM340356 4 0.5664 0.305666 0.360 0.020 0.008 0.612
#> GSM340357 1 0.8347 0.076030 0.516 0.216 0.052 0.216
#> GSM348183 1 0.8841 0.192110 0.416 0.296 0.056 0.232
#> GSM348191 4 0.4374 0.652290 0.168 0.024 0.008 0.800
#> GSM348193 1 0.7030 0.349340 0.472 0.120 0.000 0.408
#> GSM537578 1 0.8421 0.026670 0.508 0.240 0.056 0.196
#> GSM348181 1 0.7877 0.336220 0.388 0.300 0.000 0.312
#> GSM348182 3 0.7363 0.214878 0.200 0.284 0.516 0.000
#> GSM348184 4 0.0817 0.724328 0.024 0.000 0.000 0.976
#> GSM348185 4 0.4978 0.449030 0.324 0.012 0.000 0.664
#> GSM348186 3 0.7922 0.159035 0.236 0.004 0.384 0.376
#> GSM348187 1 0.6650 0.313736 0.500 0.072 0.004 0.424
#> GSM348188 1 0.8441 0.402068 0.444 0.208 0.036 0.312
#> GSM348189 3 0.4612 0.480478 0.020 0.012 0.780 0.188
#> GSM348190 3 0.8138 0.321509 0.384 0.144 0.436 0.036
#> GSM348194 2 0.7588 0.603355 0.436 0.448 0.060 0.056
#> GSM348195 2 0.7585 0.605569 0.428 0.456 0.060 0.056
#> GSM348196 1 0.6650 0.313736 0.500 0.072 0.004 0.424
#> GSM537585 3 0.8868 -0.112741 0.284 0.288 0.380 0.048
#> GSM537594 4 0.5060 0.512114 0.288 0.016 0.004 0.692
#> GSM537596 4 0.5409 0.439986 0.332 0.020 0.004 0.644
#> GSM537597 4 0.5728 0.081197 0.432 0.020 0.004 0.544
#> GSM537602 4 0.6863 0.040548 0.380 0.056 0.024 0.540
#> GSM340184 4 0.2956 0.715776 0.036 0.012 0.048 0.904
#> GSM340185 4 0.2443 0.717600 0.044 0.008 0.024 0.924
#> GSM340186 4 0.1388 0.728489 0.028 0.000 0.012 0.960
#> GSM340187 4 0.0592 0.728897 0.016 0.000 0.000 0.984
#> GSM340189 4 0.1792 0.725173 0.068 0.000 0.000 0.932
#> GSM340190 4 0.2221 0.717514 0.044 0.008 0.016 0.932
#> GSM340191 4 0.2773 0.732034 0.116 0.004 0.000 0.880
#> GSM340192 4 0.8013 -0.318740 0.232 0.008 0.352 0.408
#> GSM340193 2 0.7206 0.347905 0.312 0.548 0.008 0.132
#> GSM340194 3 0.5544 0.503477 0.332 0.008 0.640 0.020
#> GSM340195 3 0.6879 0.425572 0.264 0.124 0.604 0.008
#> GSM340196 4 0.1824 0.728040 0.060 0.000 0.004 0.936
#> GSM340197 3 0.7688 0.347744 0.284 0.204 0.504 0.008
#> GSM340198 3 0.7908 0.238276 0.240 0.004 0.416 0.340
#> GSM340199 4 0.3067 0.720996 0.084 0.004 0.024 0.888
#> GSM340200 4 0.2954 0.716874 0.064 0.008 0.028 0.900
#> GSM340201 4 0.1902 0.735018 0.064 0.000 0.004 0.932
#> GSM340202 4 0.2053 0.725985 0.072 0.000 0.004 0.924
#> GSM340203 4 0.1584 0.734990 0.036 0.012 0.000 0.952
#> GSM340204 3 0.8308 0.160286 0.260 0.016 0.372 0.352
#> GSM340205 4 0.7661 -0.345592 0.376 0.212 0.000 0.412
#> GSM340206 4 0.2761 0.712576 0.064 0.012 0.016 0.908
#> GSM340207 4 0.7850 -0.193100 0.204 0.008 0.336 0.452
#> GSM340237 4 0.4139 0.659396 0.176 0.024 0.000 0.800
#> GSM340238 4 0.2310 0.725149 0.068 0.008 0.004 0.920
#> GSM340239 3 0.8209 0.188859 0.268 0.324 0.396 0.012
#> GSM340240 3 0.7993 0.238418 0.264 0.004 0.388 0.344
#> GSM340241 4 0.3717 0.702158 0.080 0.004 0.056 0.860
#> GSM340242 3 0.4612 0.480478 0.020 0.012 0.780 0.188
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 2 0.4570 0.6373 0.008 0.720 0.236 0.000 0.036
#> GSM340359 5 0.3912 0.3955 0.000 0.084 0.076 0.016 0.824
#> GSM340361 1 0.8329 0.5049 0.376 0.292 0.016 0.084 0.232
#> GSM340362 5 0.1770 0.3601 0.012 0.008 0.008 0.028 0.944
#> GSM340363 2 0.4671 0.6344 0.008 0.712 0.240 0.000 0.040
#> GSM340364 1 0.8864 0.4727 0.404 0.268 0.096 0.072 0.160
#> GSM340365 1 0.8365 0.3901 0.368 0.192 0.016 0.100 0.324
#> GSM340366 5 0.8704 0.0771 0.064 0.164 0.120 0.192 0.460
#> GSM340367 1 0.4600 0.5916 0.752 0.184 0.008 0.004 0.052
#> GSM340368 3 0.4796 0.0792 0.000 0.468 0.516 0.004 0.012
#> GSM340369 3 0.2582 0.6617 0.004 0.080 0.892 0.024 0.000
#> GSM340370 1 0.4654 0.5901 0.740 0.196 0.012 0.000 0.052
#> GSM340371 5 0.6868 0.0248 0.008 0.012 0.160 0.344 0.476
#> GSM340372 1 0.4600 0.5916 0.752 0.184 0.008 0.004 0.052
#> GSM340373 1 0.4600 0.5916 0.752 0.184 0.008 0.004 0.052
#> GSM340375 4 0.9699 0.0856 0.144 0.216 0.120 0.292 0.228
#> GSM340376 2 0.4779 0.6302 0.016 0.704 0.248 0.000 0.032
#> GSM340378 2 0.7229 0.2170 0.264 0.512 0.160 0.000 0.064
#> GSM340243 3 0.2824 0.6718 0.016 0.068 0.888 0.028 0.000
#> GSM340244 3 0.4298 0.3972 0.000 0.352 0.640 0.000 0.008
#> GSM340246 3 0.3139 0.6668 0.024 0.056 0.880 0.036 0.004
#> GSM340247 3 0.2488 0.6518 0.000 0.124 0.872 0.004 0.000
#> GSM340248 3 0.2597 0.6718 0.004 0.060 0.900 0.032 0.004
#> GSM340249 3 0.3838 0.5254 0.000 0.280 0.716 0.004 0.000
#> GSM340250 3 0.6756 0.3219 0.000 0.184 0.580 0.048 0.188
#> GSM340251 3 0.1117 0.6717 0.000 0.016 0.964 0.020 0.000
#> GSM340252 5 0.6515 0.3329 0.000 0.132 0.280 0.028 0.560
#> GSM340253 5 0.1580 0.3639 0.004 0.016 0.016 0.012 0.952
#> GSM340254 5 0.6461 0.3417 0.000 0.132 0.268 0.028 0.572
#> GSM340256 3 0.6543 0.2126 0.000 0.244 0.528 0.008 0.220
#> GSM340258 3 0.6699 0.1252 0.004 0.108 0.496 0.028 0.364
#> GSM340259 1 0.7494 0.2913 0.488 0.332 0.088 0.028 0.064
#> GSM340260 5 0.7095 0.0992 0.224 0.044 0.008 0.172 0.552
#> GSM340261 3 0.5808 0.4444 0.020 0.212 0.668 0.092 0.008
#> GSM340262 5 0.5015 0.3826 0.012 0.040 0.220 0.012 0.716
#> GSM340263 3 0.4380 0.3424 0.000 0.376 0.616 0.000 0.008
#> GSM340264 4 0.4973 0.3945 0.028 0.000 0.004 0.592 0.376
#> GSM340265 4 0.4835 0.3902 0.020 0.000 0.004 0.592 0.384
#> GSM340266 3 0.4521 0.5562 0.000 0.248 0.716 0.012 0.024
#> GSM340267 4 0.7306 0.3419 0.100 0.036 0.032 0.492 0.340
#> GSM340268 3 0.2325 0.6573 0.000 0.068 0.904 0.028 0.000
#> GSM340269 3 0.3604 0.6532 0.000 0.100 0.836 0.008 0.056
#> GSM340270 2 0.5894 0.4783 0.044 0.568 0.356 0.004 0.028
#> GSM537574 3 0.3031 0.6473 0.000 0.128 0.852 0.004 0.016
#> GSM537580 5 0.6868 0.0248 0.008 0.012 0.160 0.344 0.476
#> GSM537581 3 0.4667 0.5184 0.012 0.272 0.696 0.012 0.008
#> GSM340272 5 0.8878 0.0560 0.072 0.164 0.120 0.208 0.436
#> GSM340273 3 0.5305 -0.0804 0.008 0.480 0.484 0.004 0.024
#> GSM340275 3 0.4298 0.4030 0.000 0.352 0.640 0.000 0.008
#> GSM340276 3 0.6511 0.0544 0.048 0.348 0.544 0.044 0.016
#> GSM340277 5 0.7273 0.3027 0.024 0.060 0.288 0.092 0.536
#> GSM340278 3 0.4966 0.5660 0.020 0.192 0.732 0.052 0.004
#> GSM340279 3 0.3704 0.6677 0.008 0.092 0.844 0.040 0.016
#> GSM340282 5 0.8704 0.0771 0.064 0.164 0.120 0.192 0.460
#> GSM340284 3 0.1012 0.6688 0.000 0.020 0.968 0.012 0.000
#> GSM340285 3 0.6852 -0.0866 0.056 0.404 0.460 0.076 0.004
#> GSM340286 5 0.4402 0.4038 0.008 0.052 0.140 0.012 0.788
#> GSM340287 3 0.5986 0.4055 0.020 0.228 0.640 0.108 0.004
#> GSM340288 1 0.8076 0.5811 0.420 0.336 0.024 0.084 0.136
#> GSM340289 1 0.3758 0.4239 0.836 0.060 0.000 0.084 0.020
#> GSM340290 3 0.3400 0.6664 0.012 0.104 0.852 0.028 0.004
#> GSM340291 3 0.4602 0.4363 0.028 0.316 0.656 0.000 0.000
#> GSM340293 3 0.2845 0.6605 0.008 0.096 0.876 0.020 0.000
#> GSM340294 3 0.5669 0.4768 0.020 0.200 0.684 0.088 0.008
#> GSM340296 3 0.2824 0.6718 0.016 0.068 0.888 0.028 0.000
#> GSM340297 3 0.5683 0.3359 0.008 0.332 0.584 0.076 0.000
#> GSM340298 3 0.6870 0.3842 0.020 0.120 0.636 0.136 0.088
#> GSM340299 5 0.1770 0.3601 0.012 0.008 0.008 0.028 0.944
#> GSM340301 5 0.4609 0.2756 0.172 0.016 0.000 0.056 0.756
#> GSM340303 4 0.4835 0.3902 0.020 0.000 0.004 0.592 0.384
#> GSM340304 2 0.7633 0.3714 0.036 0.420 0.400 0.080 0.064
#> GSM340306 3 0.9002 -0.3482 0.136 0.300 0.372 0.108 0.084
#> GSM340307 3 0.7132 0.3558 0.024 0.120 0.616 0.140 0.100
#> GSM340310 2 0.4910 0.6241 0.032 0.724 0.208 0.000 0.036
#> GSM340314 3 0.5204 0.4498 0.008 0.236 0.680 0.076 0.000
#> GSM340315 3 0.4280 0.6278 0.052 0.052 0.824 0.060 0.012
#> GSM340317 3 0.4380 0.3424 0.000 0.376 0.616 0.000 0.008
#> GSM340318 3 0.2389 0.6541 0.000 0.116 0.880 0.004 0.000
#> GSM340319 3 0.2389 0.6541 0.000 0.116 0.880 0.004 0.000
#> GSM340320 3 0.5747 0.3195 0.008 0.336 0.576 0.080 0.000
#> GSM340321 3 0.1012 0.6688 0.000 0.020 0.968 0.012 0.000
#> GSM340322 3 0.4124 0.6054 0.000 0.180 0.776 0.008 0.036
#> GSM340324 3 0.6543 0.2126 0.000 0.244 0.528 0.008 0.220
#> GSM340328 2 0.4598 0.6323 0.008 0.716 0.240 0.000 0.036
#> GSM340330 3 0.3067 0.6525 0.004 0.032 0.880 0.016 0.068
#> GSM340332 3 0.2561 0.6721 0.000 0.096 0.884 0.020 0.000
#> GSM340333 5 0.3854 0.3955 0.000 0.080 0.076 0.016 0.828
#> GSM340336 3 0.2389 0.6541 0.000 0.116 0.880 0.004 0.000
#> GSM340337 3 0.4539 0.5707 0.000 0.212 0.736 0.008 0.044
#> GSM340338 3 0.7600 0.1522 0.016 0.108 0.504 0.088 0.284
#> GSM340339 3 0.3197 0.6539 0.000 0.140 0.836 0.024 0.000
#> GSM340340 3 0.4833 0.2128 0.000 0.412 0.564 0.000 0.024
#> GSM340341 3 0.6394 0.4494 0.012 0.216 0.632 0.036 0.104
#> GSM340343 2 0.7330 0.3525 0.180 0.408 0.368 0.044 0.000
#> GSM340344 5 0.1475 0.3636 0.004 0.012 0.016 0.012 0.956
#> GSM340346 3 0.7629 0.2589 0.024 0.144 0.564 0.152 0.116
#> GSM340347 3 0.3243 0.6263 0.000 0.180 0.812 0.004 0.004
#> GSM340348 3 0.5082 0.4991 0.004 0.260 0.684 0.016 0.036
#> GSM340349 2 0.9684 -0.0670 0.156 0.276 0.204 0.256 0.108
#> GSM340350 2 0.9520 0.0176 0.120 0.356 0.192 0.188 0.144
#> GSM340351 1 0.8063 0.5826 0.428 0.328 0.024 0.084 0.136
#> GSM340354 3 0.3096 0.6659 0.000 0.044 0.880 0.040 0.036
#> GSM340356 2 0.6063 0.1602 0.032 0.472 0.456 0.012 0.028
#> GSM340357 4 0.9474 -0.1058 0.272 0.248 0.104 0.272 0.104
#> GSM348183 2 0.7127 0.1649 0.188 0.604 0.096 0.024 0.088
#> GSM348191 3 0.5879 0.4440 0.020 0.196 0.668 0.108 0.008
#> GSM348193 2 0.4910 0.6241 0.032 0.724 0.208 0.000 0.036
#> GSM537578 4 0.9328 -0.0224 0.264 0.212 0.100 0.328 0.096
#> GSM348181 2 0.6673 0.4387 0.196 0.580 0.196 0.012 0.016
#> GSM348182 5 0.7043 0.0588 0.260 0.044 0.000 0.176 0.520
#> GSM348184 3 0.0798 0.6678 0.000 0.008 0.976 0.016 0.000
#> GSM348185 3 0.4913 -0.0385 0.008 0.484 0.496 0.000 0.012
#> GSM348186 5 0.6629 0.3146 0.000 0.136 0.300 0.028 0.536
#> GSM348187 2 0.7640 0.5522 0.084 0.528 0.272 0.060 0.056
#> GSM348188 2 0.6736 0.4517 0.096 0.636 0.160 0.012 0.096
#> GSM348189 5 0.6868 0.0248 0.008 0.012 0.160 0.344 0.476
#> GSM348190 5 0.6215 0.2463 0.136 0.112 0.020 0.048 0.684
#> GSM348194 1 0.8076 0.5811 0.420 0.336 0.024 0.084 0.136
#> GSM348195 1 0.8063 0.5826 0.428 0.328 0.024 0.084 0.136
#> GSM348196 2 0.7640 0.5522 0.084 0.528 0.272 0.060 0.056
#> GSM537585 5 0.8822 -0.3033 0.248 0.140 0.020 0.288 0.304
#> GSM537594 3 0.4870 0.1233 0.004 0.448 0.532 0.000 0.016
#> GSM537596 3 0.6721 0.0691 0.020 0.340 0.536 0.068 0.036
#> GSM537597 2 0.6634 0.4148 0.032 0.516 0.376 0.040 0.036
#> GSM537602 2 0.7633 0.3714 0.036 0.420 0.400 0.080 0.064
#> GSM340184 3 0.3318 0.6484 0.004 0.024 0.868 0.072 0.032
#> GSM340185 3 0.3250 0.6427 0.008 0.032 0.876 0.060 0.024
#> GSM340186 3 0.1461 0.6685 0.000 0.004 0.952 0.028 0.016
#> GSM340187 3 0.0992 0.6706 0.000 0.024 0.968 0.008 0.000
#> GSM340189 3 0.2582 0.6599 0.004 0.080 0.892 0.024 0.000
#> GSM340190 3 0.2791 0.6504 0.008 0.028 0.900 0.044 0.020
#> GSM340191 3 0.2773 0.6640 0.000 0.112 0.868 0.020 0.000
#> GSM340192 5 0.7511 0.2743 0.020 0.076 0.324 0.092 0.488
#> GSM340193 1 0.7786 0.2873 0.468 0.328 0.088 0.032 0.084
#> GSM340194 5 0.1770 0.3601 0.012 0.008 0.008 0.028 0.944
#> GSM340195 5 0.5045 0.2371 0.120 0.032 0.000 0.100 0.748
#> GSM340196 3 0.2488 0.6518 0.000 0.124 0.872 0.004 0.000
#> GSM340197 5 0.4774 0.2687 0.180 0.020 0.000 0.056 0.744
#> GSM340198 5 0.6461 0.3417 0.000 0.132 0.268 0.028 0.572
#> GSM340199 3 0.4106 0.6408 0.008 0.092 0.824 0.052 0.024
#> GSM340200 3 0.3966 0.6292 0.012 0.048 0.840 0.068 0.032
#> GSM340201 3 0.2488 0.6617 0.000 0.124 0.872 0.004 0.000
#> GSM340202 3 0.2833 0.6427 0.000 0.140 0.852 0.004 0.004
#> GSM340203 3 0.2011 0.6783 0.008 0.044 0.928 0.020 0.000
#> GSM340204 5 0.7000 0.3065 0.008 0.164 0.272 0.028 0.528
#> GSM340205 2 0.6096 0.5759 0.116 0.604 0.264 0.004 0.012
#> GSM340206 3 0.3359 0.6374 0.012 0.040 0.868 0.068 0.012
#> GSM340207 5 0.6726 0.2619 0.012 0.056 0.380 0.052 0.500
#> GSM340237 3 0.4602 0.4363 0.028 0.316 0.656 0.000 0.000
#> GSM340238 3 0.2427 0.6596 0.004 0.048 0.912 0.028 0.008
#> GSM340239 5 0.5896 0.1916 0.292 0.028 0.004 0.060 0.616
#> GSM340240 5 0.7145 0.3080 0.020 0.060 0.276 0.092 0.552
#> GSM340241 3 0.4181 0.6317 0.000 0.056 0.816 0.044 0.084
#> GSM340242 5 0.6868 0.0248 0.008 0.012 0.160 0.344 0.476
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 6 0.2499 0.5530 0.004 0.000 0.096 0.016 0.004 0.880
#> GSM340359 2 0.4370 0.4571 0.000 0.788 0.056 0.016 0.052 0.088
#> GSM340361 1 0.7714 0.4547 0.396 0.216 0.000 0.104 0.028 0.256
#> GSM340362 2 0.0260 0.4500 0.000 0.992 0.000 0.000 0.000 0.008
#> GSM340363 6 0.2996 0.5623 0.012 0.004 0.108 0.016 0.004 0.856
#> GSM340364 1 0.7938 0.4123 0.456 0.116 0.048 0.100 0.024 0.256
#> GSM340365 1 0.7631 0.3124 0.392 0.300 0.000 0.116 0.024 0.168
#> GSM340366 4 0.6237 0.4998 0.028 0.424 0.052 0.456 0.004 0.036
#> GSM340367 1 0.3709 0.5423 0.808 0.020 0.004 0.040 0.000 0.128
#> GSM340368 6 0.3944 0.1880 0.000 0.000 0.428 0.000 0.004 0.568
#> GSM340369 3 0.2706 0.6518 0.000 0.000 0.876 0.060 0.008 0.056
#> GSM340370 1 0.3766 0.5411 0.796 0.020 0.004 0.032 0.000 0.148
#> GSM340371 5 0.6794 0.4146 0.000 0.356 0.152 0.056 0.428 0.008
#> GSM340372 1 0.3709 0.5423 0.808 0.020 0.004 0.040 0.000 0.128
#> GSM340373 1 0.3709 0.5423 0.808 0.020 0.004 0.040 0.000 0.128
#> GSM340375 5 0.8817 0.0719 0.172 0.096 0.088 0.044 0.380 0.220
#> GSM340376 6 0.3008 0.5657 0.012 0.000 0.120 0.008 0.012 0.848
#> GSM340378 6 0.6397 0.0200 0.300 0.020 0.088 0.032 0.012 0.548
#> GSM340243 3 0.2803 0.6621 0.012 0.000 0.872 0.052 0.000 0.064
#> GSM340244 3 0.3982 0.1246 0.000 0.000 0.536 0.004 0.000 0.460
#> GSM340246 3 0.2973 0.6563 0.016 0.000 0.860 0.084 0.000 0.040
#> GSM340247 3 0.2416 0.6253 0.000 0.000 0.844 0.000 0.000 0.156
#> GSM340248 3 0.2376 0.6633 0.000 0.000 0.888 0.068 0.000 0.044
#> GSM340249 3 0.3983 0.3915 0.000 0.000 0.640 0.008 0.004 0.348
#> GSM340250 3 0.7161 0.2272 0.004 0.140 0.512 0.056 0.044 0.244
#> GSM340251 3 0.1780 0.6649 0.000 0.000 0.924 0.048 0.000 0.028
#> GSM340252 2 0.7047 0.3578 0.000 0.516 0.220 0.036 0.060 0.168
#> GSM340253 2 0.1836 0.4480 0.000 0.928 0.008 0.004 0.048 0.012
#> GSM340254 2 0.7008 0.3649 0.000 0.524 0.212 0.036 0.060 0.168
#> GSM340256 3 0.6577 0.0170 0.000 0.216 0.428 0.036 0.000 0.320
#> GSM340258 3 0.7032 0.0849 0.004 0.328 0.464 0.044 0.036 0.124
#> GSM340259 1 0.7514 0.2536 0.472 0.052 0.064 0.072 0.032 0.308
#> GSM340260 2 0.6894 0.1182 0.168 0.560 0.004 0.064 0.172 0.032
#> GSM340261 3 0.6337 0.3688 0.004 0.004 0.584 0.180 0.060 0.168
#> GSM340262 2 0.5149 0.4070 0.008 0.696 0.208 0.016 0.040 0.032
#> GSM340263 3 0.4126 0.0424 0.004 0.000 0.512 0.004 0.000 0.480
#> GSM340264 5 0.2278 0.5270 0.000 0.128 0.000 0.004 0.868 0.000
#> GSM340265 5 0.2219 0.5310 0.000 0.136 0.000 0.000 0.864 0.000
#> GSM340266 3 0.4499 0.4405 0.000 0.012 0.636 0.028 0.000 0.324
#> GSM340267 5 0.6487 0.3977 0.056 0.136 0.028 0.172 0.604 0.004
#> GSM340268 3 0.2649 0.6452 0.000 0.000 0.876 0.068 0.004 0.052
#> GSM340269 3 0.3875 0.6290 0.000 0.060 0.796 0.024 0.000 0.120
#> GSM340270 6 0.5333 0.5716 0.064 0.004 0.236 0.024 0.012 0.660
#> GSM537574 3 0.3226 0.6091 0.000 0.012 0.808 0.012 0.000 0.168
#> GSM537580 5 0.6794 0.4146 0.000 0.356 0.152 0.056 0.428 0.008
#> GSM537581 3 0.4430 0.3840 0.016 0.000 0.624 0.016 0.000 0.344
#> GSM340272 4 0.6483 0.5100 0.028 0.400 0.052 0.468 0.016 0.036
#> GSM340273 6 0.4479 0.3569 0.012 0.004 0.372 0.012 0.000 0.600
#> GSM340275 3 0.3982 0.1338 0.000 0.000 0.536 0.004 0.000 0.460
#> GSM340276 3 0.6624 -0.1398 0.048 0.004 0.456 0.120 0.008 0.364
#> GSM340277 2 0.7211 0.2719 0.000 0.468 0.248 0.196 0.056 0.032
#> GSM340278 3 0.5216 0.5274 0.008 0.008 0.692 0.128 0.012 0.152
#> GSM340279 3 0.3393 0.6604 0.004 0.008 0.840 0.076 0.004 0.068
#> GSM340282 4 0.6237 0.4998 0.028 0.424 0.052 0.456 0.004 0.036
#> GSM340284 3 0.0603 0.6603 0.000 0.000 0.980 0.004 0.000 0.016
#> GSM340285 6 0.7643 0.1876 0.060 0.000 0.344 0.156 0.064 0.376
#> GSM340286 2 0.4750 0.4552 0.000 0.752 0.128 0.016 0.052 0.052
#> GSM340287 3 0.6504 0.3147 0.004 0.000 0.544 0.200 0.064 0.188
#> GSM340288 1 0.7205 0.5229 0.452 0.076 0.000 0.152 0.024 0.296
#> GSM340289 1 0.3755 0.3775 0.812 0.008 0.000 0.112 0.052 0.016
#> GSM340290 3 0.3237 0.6524 0.008 0.000 0.836 0.056 0.000 0.100
#> GSM340291 3 0.4994 0.1959 0.032 0.000 0.552 0.024 0.000 0.392
#> GSM340293 3 0.3304 0.6439 0.004 0.000 0.840 0.068 0.008 0.080
#> GSM340294 3 0.6120 0.4044 0.004 0.004 0.608 0.168 0.052 0.164
#> GSM340296 3 0.2865 0.6620 0.012 0.000 0.868 0.056 0.000 0.064
#> GSM340297 3 0.6598 0.1865 0.000 0.000 0.484 0.164 0.064 0.288
#> GSM340298 3 0.4923 0.3889 0.000 0.052 0.596 0.340 0.000 0.012
#> GSM340299 2 0.0260 0.4500 0.000 0.992 0.000 0.000 0.000 0.008
#> GSM340301 2 0.3818 0.3589 0.132 0.804 0.000 0.020 0.032 0.012
#> GSM340303 5 0.2219 0.5310 0.000 0.136 0.000 0.000 0.864 0.000
#> GSM340304 6 0.7645 0.4009 0.060 0.040 0.312 0.196 0.004 0.388
#> GSM340306 6 0.8198 0.3444 0.164 0.012 0.324 0.044 0.116 0.340
#> GSM340307 3 0.5084 0.3582 0.000 0.060 0.568 0.360 0.000 0.012
#> GSM340310 6 0.2881 0.5257 0.040 0.000 0.084 0.012 0.000 0.864
#> GSM340314 3 0.6008 0.3500 0.000 0.000 0.596 0.156 0.056 0.192
#> GSM340315 3 0.3783 0.6256 0.032 0.000 0.816 0.112 0.020 0.020
#> GSM340317 3 0.4126 0.0424 0.004 0.000 0.512 0.004 0.000 0.480
#> GSM340318 3 0.2340 0.6286 0.000 0.000 0.852 0.000 0.000 0.148
#> GSM340319 3 0.2340 0.6286 0.000 0.000 0.852 0.000 0.000 0.148
#> GSM340320 3 0.6739 0.1760 0.004 0.000 0.480 0.168 0.064 0.284
#> GSM340321 3 0.0603 0.6603 0.000 0.000 0.980 0.004 0.000 0.016
#> GSM340322 3 0.4513 0.5232 0.000 0.024 0.700 0.040 0.000 0.236
#> GSM340324 3 0.6577 0.0170 0.000 0.216 0.428 0.036 0.000 0.320
#> GSM340328 6 0.2548 0.5572 0.004 0.000 0.100 0.016 0.004 0.876
#> GSM340330 3 0.2875 0.6533 0.004 0.060 0.880 0.028 0.004 0.024
#> GSM340332 3 0.2776 0.6573 0.000 0.000 0.860 0.052 0.000 0.088
#> GSM340333 2 0.4320 0.4579 0.000 0.792 0.056 0.016 0.052 0.084
#> GSM340336 3 0.2340 0.6286 0.000 0.000 0.852 0.000 0.000 0.148
#> GSM340337 3 0.4823 0.4583 0.000 0.028 0.652 0.040 0.000 0.280
#> GSM340338 3 0.6882 0.1667 0.004 0.256 0.460 0.228 0.004 0.048
#> GSM340339 3 0.3672 0.6286 0.004 0.000 0.792 0.064 0.000 0.140
#> GSM340340 6 0.4443 0.0243 0.012 0.004 0.480 0.004 0.000 0.500
#> GSM340341 3 0.6386 0.3448 0.008 0.080 0.568 0.044 0.024 0.276
#> GSM340343 6 0.7854 0.3634 0.192 0.000 0.276 0.100 0.044 0.388
#> GSM340344 2 0.1692 0.4477 0.000 0.932 0.008 0.000 0.048 0.012
#> GSM340346 3 0.5425 0.2157 0.000 0.064 0.516 0.400 0.004 0.016
#> GSM340347 3 0.3136 0.5805 0.000 0.000 0.768 0.000 0.004 0.228
#> GSM340348 3 0.4902 0.3654 0.008 0.012 0.612 0.012 0.016 0.340
#> GSM340349 4 0.7121 0.4545 0.104 0.048 0.120 0.580 0.012 0.136
#> GSM340350 4 0.8005 0.3983 0.112 0.112 0.108 0.460 0.004 0.204
#> GSM340351 1 0.7185 0.5241 0.460 0.076 0.000 0.152 0.024 0.288
#> GSM340354 3 0.3162 0.6610 0.000 0.032 0.860 0.064 0.004 0.040
#> GSM340356 6 0.5489 0.3869 0.052 0.004 0.364 0.032 0.000 0.548
#> GSM340357 4 0.6119 0.3976 0.208 0.036 0.036 0.636 0.012 0.072
#> GSM348183 6 0.6292 0.0712 0.216 0.040 0.048 0.056 0.016 0.624
#> GSM348191 3 0.5968 0.3973 0.004 0.000 0.608 0.212 0.056 0.120
#> GSM348193 6 0.2881 0.5257 0.040 0.000 0.084 0.012 0.000 0.864
#> GSM537578 4 0.5742 0.4179 0.188 0.020 0.032 0.676 0.056 0.028
#> GSM348181 6 0.6601 0.2958 0.200 0.000 0.124 0.064 0.032 0.580
#> GSM348182 2 0.6951 0.0813 0.208 0.528 0.000 0.060 0.172 0.032
#> GSM348184 3 0.1088 0.6612 0.000 0.000 0.960 0.024 0.000 0.016
#> GSM348185 6 0.3841 0.3390 0.004 0.000 0.380 0.000 0.000 0.616
#> GSM348186 2 0.7216 0.3331 0.000 0.488 0.240 0.040 0.060 0.172
#> GSM348187 6 0.6897 0.4410 0.108 0.008 0.180 0.160 0.004 0.540
#> GSM348188 6 0.6089 0.3295 0.108 0.052 0.080 0.060 0.016 0.684
#> GSM348189 5 0.6794 0.4146 0.000 0.356 0.152 0.056 0.428 0.008
#> GSM348190 2 0.5272 0.3319 0.100 0.728 0.008 0.036 0.024 0.104
#> GSM348194 1 0.7205 0.5229 0.452 0.076 0.000 0.152 0.024 0.296
#> GSM348195 1 0.7185 0.5241 0.460 0.076 0.000 0.152 0.024 0.288
#> GSM348196 6 0.6897 0.4410 0.108 0.008 0.180 0.160 0.004 0.540
#> GSM537585 5 0.8619 -0.2295 0.228 0.172 0.000 0.140 0.332 0.128
#> GSM537594 6 0.4045 0.1945 0.008 0.000 0.428 0.000 0.000 0.564
#> GSM537596 3 0.6681 -0.0792 0.028 0.008 0.460 0.164 0.008 0.332
#> GSM537597 6 0.6096 0.5173 0.052 0.004 0.280 0.104 0.000 0.560
#> GSM537602 6 0.7645 0.4009 0.060 0.040 0.312 0.196 0.004 0.388
#> GSM340184 3 0.3211 0.6432 0.000 0.016 0.852 0.092 0.028 0.012
#> GSM340185 3 0.2544 0.6392 0.000 0.012 0.864 0.120 0.000 0.004
#> GSM340186 3 0.1426 0.6634 0.000 0.016 0.948 0.028 0.000 0.008
#> GSM340187 3 0.1333 0.6611 0.000 0.000 0.944 0.008 0.000 0.048
#> GSM340189 3 0.2744 0.6503 0.000 0.000 0.876 0.060 0.012 0.052
#> GSM340190 3 0.2308 0.6456 0.000 0.008 0.880 0.108 0.000 0.004
#> GSM340191 3 0.2984 0.6473 0.000 0.000 0.848 0.044 0.004 0.104
#> GSM340192 2 0.7502 0.2328 0.000 0.420 0.288 0.188 0.056 0.048
#> GSM340193 1 0.7748 0.2427 0.444 0.068 0.060 0.080 0.032 0.316
#> GSM340194 2 0.0260 0.4500 0.000 0.992 0.000 0.000 0.000 0.008
#> GSM340195 2 0.4873 0.3000 0.092 0.752 0.000 0.036 0.092 0.028
#> GSM340196 3 0.2416 0.6253 0.000 0.000 0.844 0.000 0.000 0.156
#> GSM340197 2 0.4009 0.3493 0.136 0.792 0.000 0.024 0.036 0.012
#> GSM340198 2 0.7008 0.3649 0.000 0.524 0.212 0.036 0.060 0.168
#> GSM340199 3 0.3807 0.6300 0.000 0.008 0.792 0.148 0.008 0.044
#> GSM340200 3 0.3310 0.6255 0.000 0.016 0.816 0.148 0.000 0.020
#> GSM340201 3 0.2595 0.6327 0.000 0.000 0.836 0.004 0.000 0.160
#> GSM340202 3 0.2631 0.6108 0.000 0.000 0.820 0.000 0.000 0.180
#> GSM340203 3 0.2487 0.6699 0.004 0.000 0.892 0.048 0.004 0.052
#> GSM340204 2 0.7463 0.3238 0.004 0.480 0.212 0.048 0.060 0.196
#> GSM340205 6 0.6019 0.4912 0.132 0.000 0.192 0.036 0.020 0.620
#> GSM340206 3 0.2807 0.6386 0.004 0.000 0.864 0.104 0.012 0.016
#> GSM340207 2 0.7010 0.2219 0.004 0.448 0.356 0.108 0.040 0.044
#> GSM340237 3 0.4994 0.1959 0.032 0.000 0.552 0.024 0.000 0.392
#> GSM340238 3 0.2321 0.6526 0.004 0.004 0.904 0.060 0.004 0.024
#> GSM340239 2 0.5404 0.2529 0.232 0.664 0.004 0.036 0.044 0.020
#> GSM340240 2 0.7146 0.2763 0.000 0.484 0.236 0.192 0.056 0.032
#> GSM340241 3 0.4007 0.6158 0.000 0.080 0.800 0.064 0.000 0.056
#> GSM340242 5 0.6794 0.4146 0.000 0.356 0.152 0.056 0.428 0.008
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
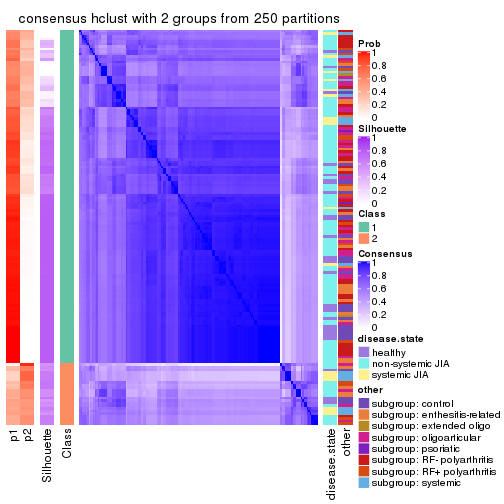
consensus_heatmap(res, k = 3)
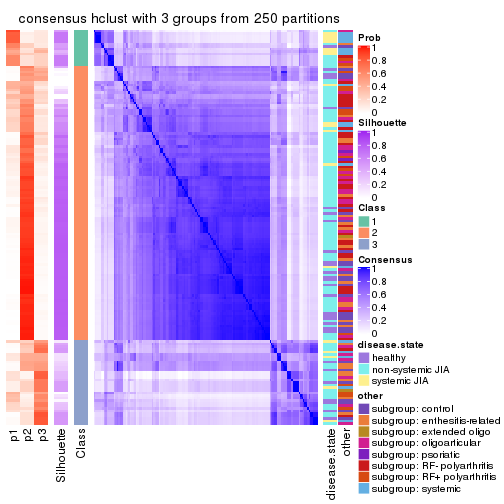
consensus_heatmap(res, k = 4)
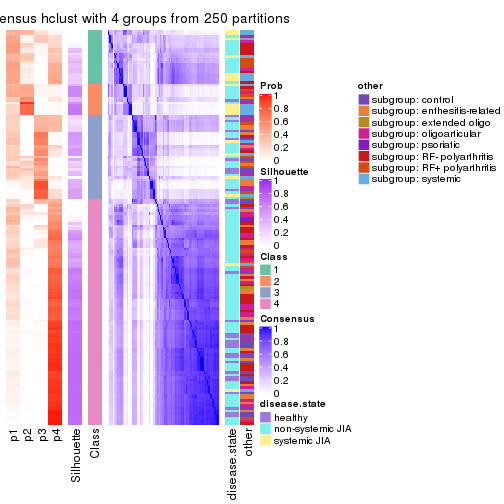
consensus_heatmap(res, k = 5)
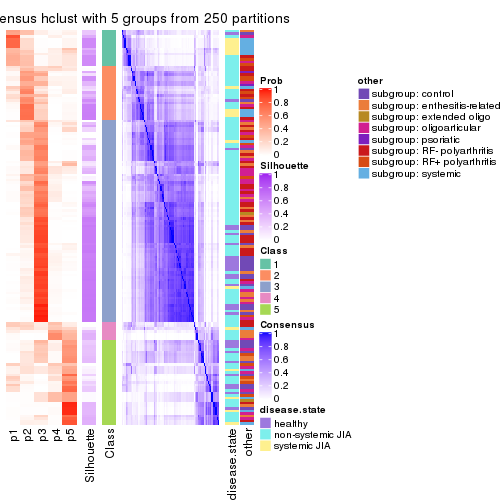
consensus_heatmap(res, k = 6)
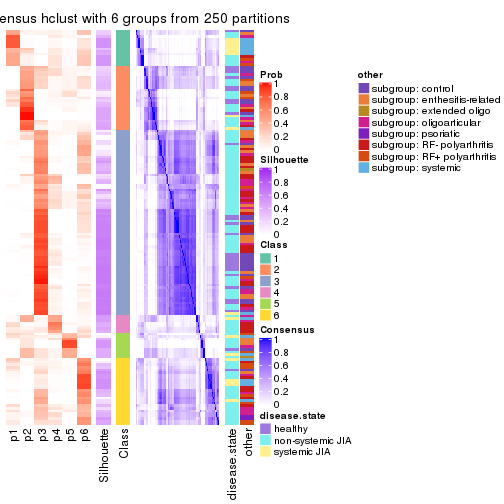
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
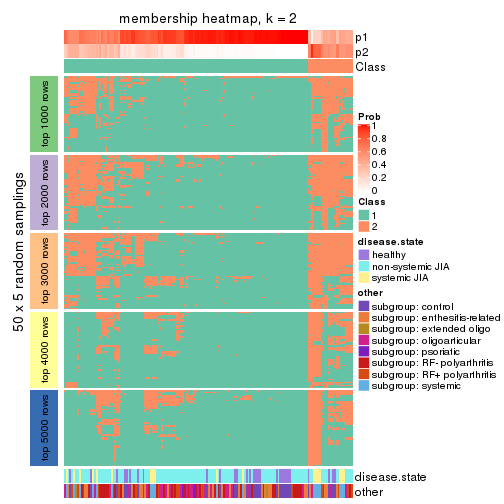
membership_heatmap(res, k = 3)
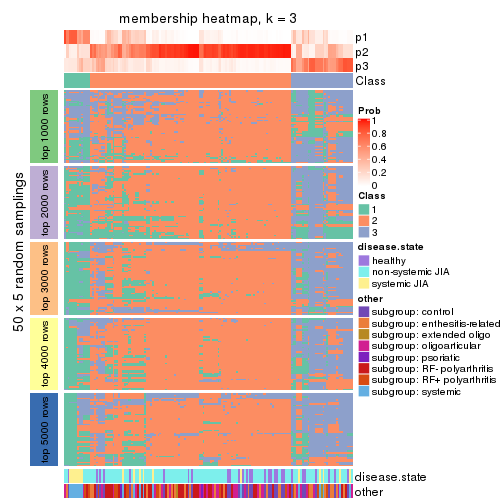
membership_heatmap(res, k = 4)
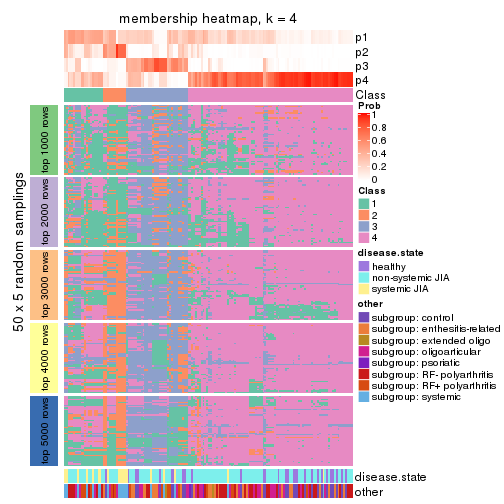
membership_heatmap(res, k = 5)
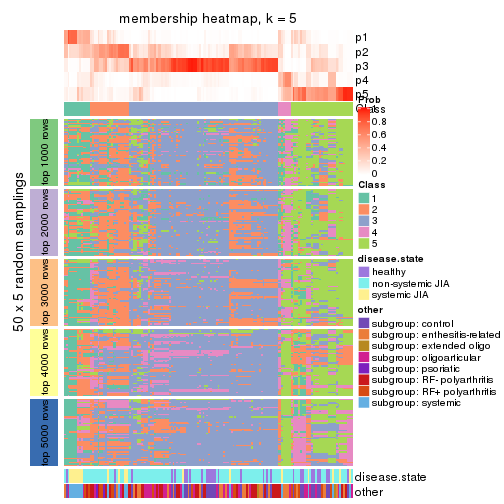
membership_heatmap(res, k = 6)
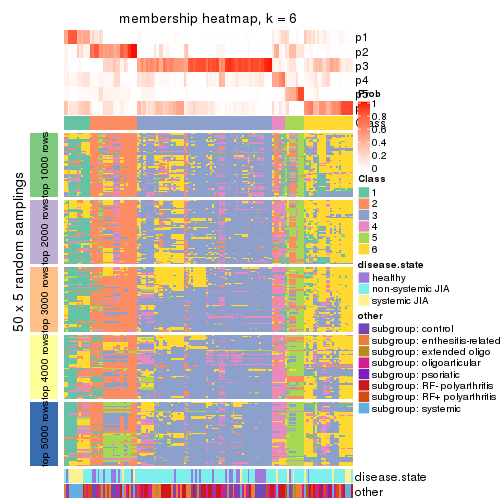
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
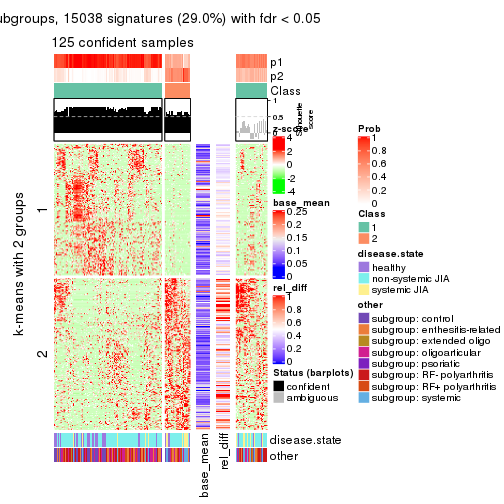
get_signatures(res, k = 3)
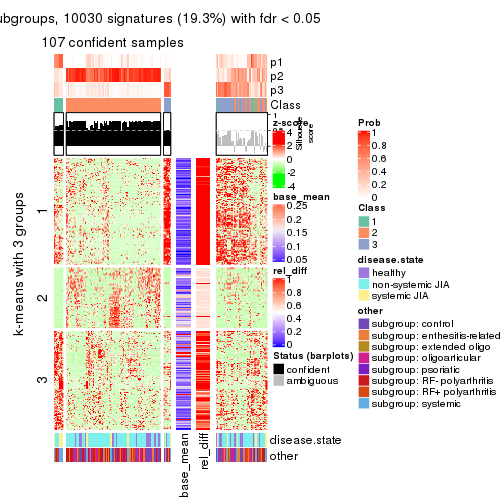
get_signatures(res, k = 4)
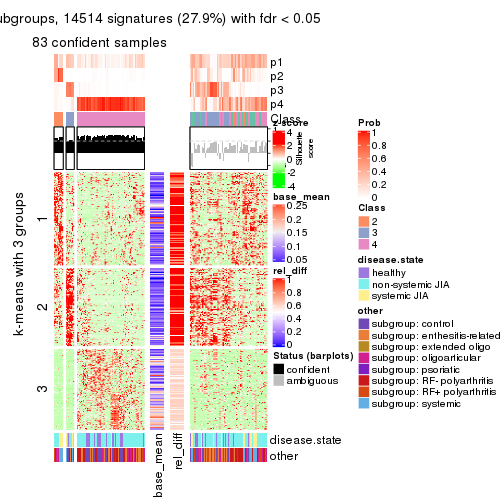
get_signatures(res, k = 5)
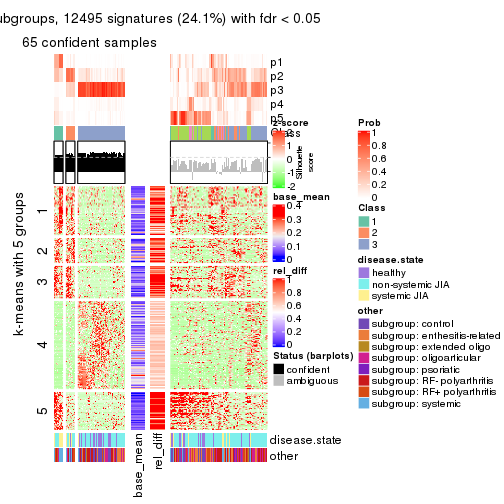
get_signatures(res, k = 6)
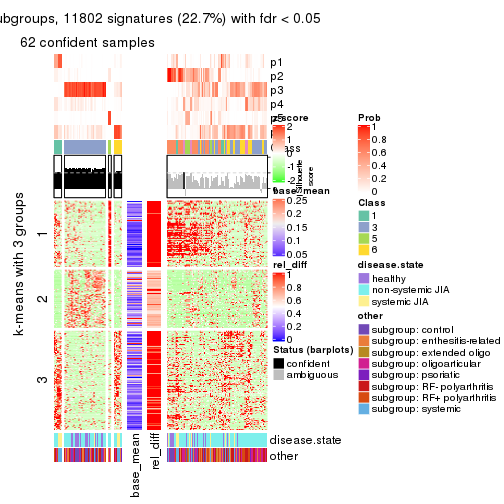
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
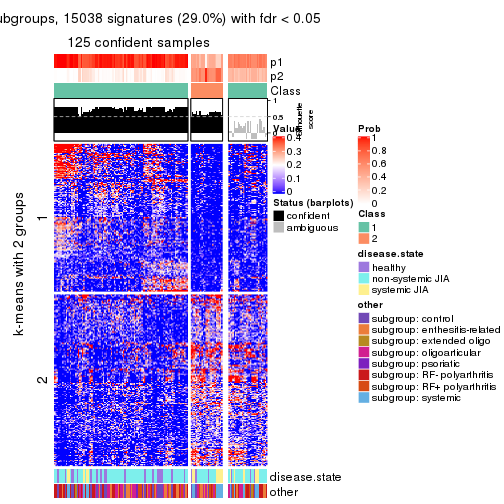
get_signatures(res, k = 3, scale_rows = FALSE)
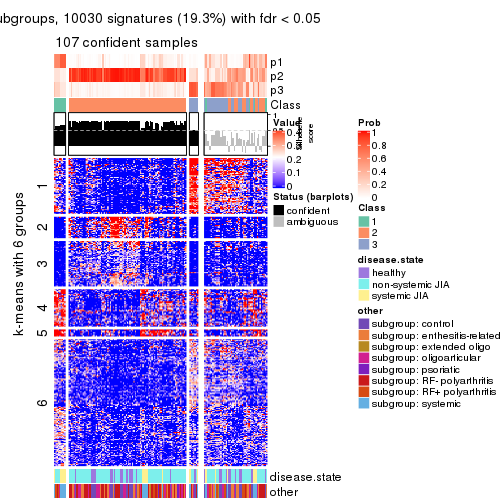
get_signatures(res, k = 4, scale_rows = FALSE)
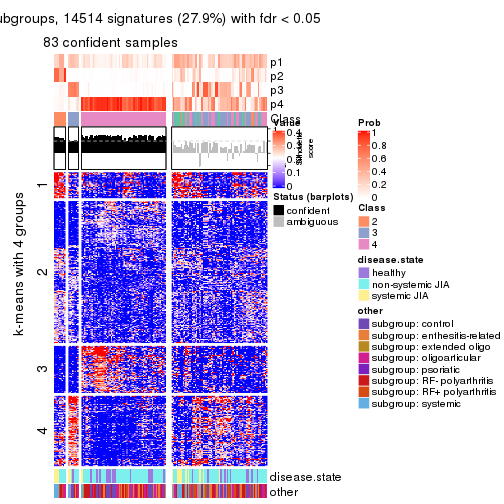
get_signatures(res, k = 5, scale_rows = FALSE)
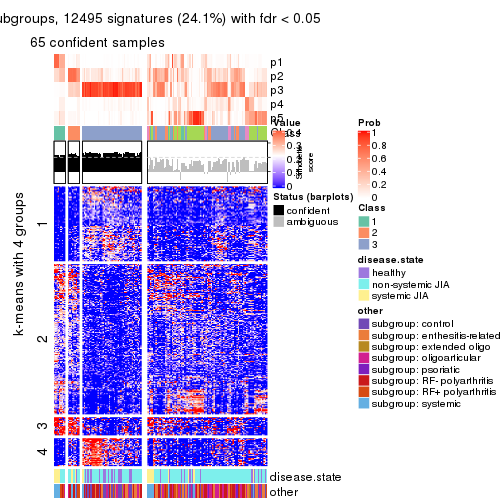
get_signatures(res, k = 6, scale_rows = FALSE)
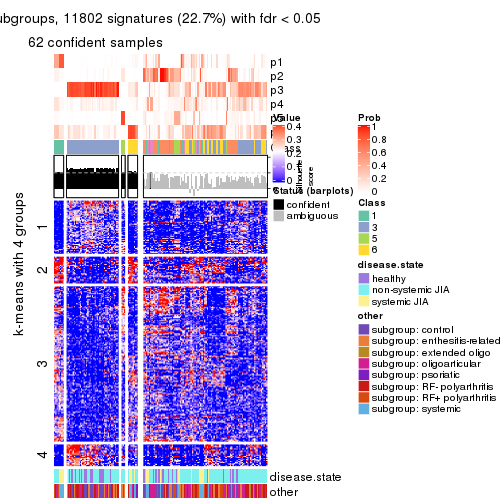
Compare the overlap of signatures from different k:
compare_signatures(res)
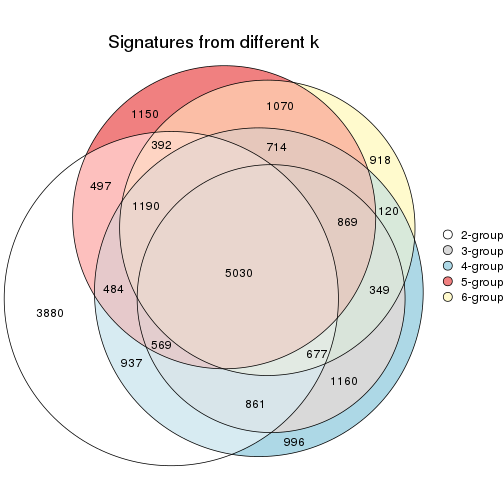
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
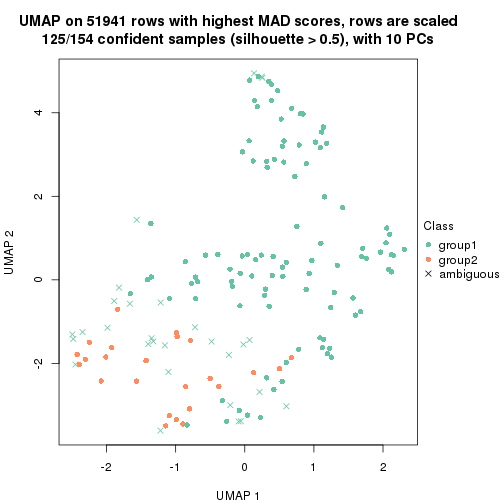
dimension_reduction(res, k = 3, method = "UMAP")
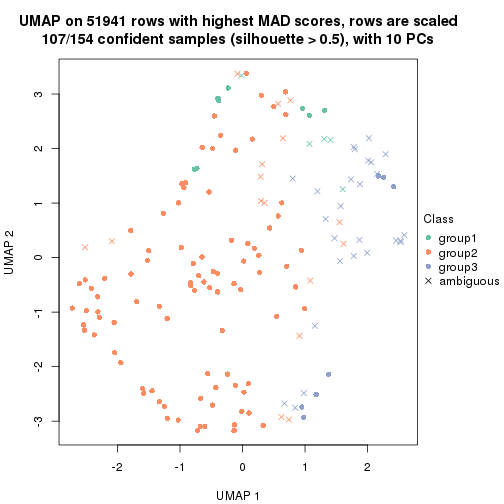
dimension_reduction(res, k = 4, method = "UMAP")
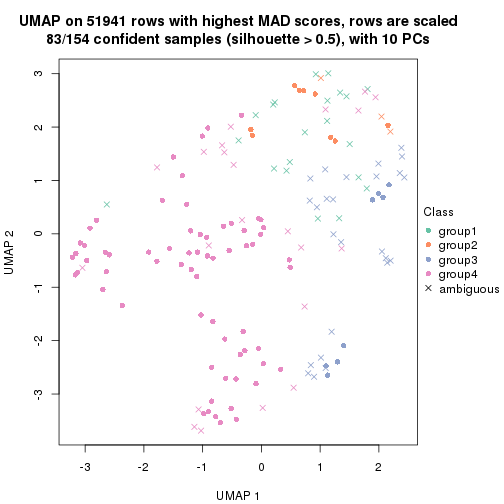
dimension_reduction(res, k = 5, method = "UMAP")
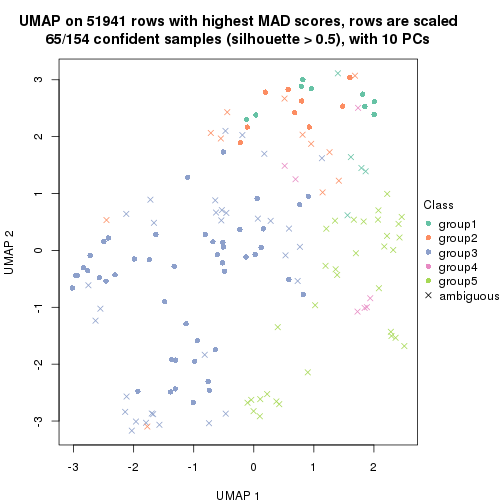
dimension_reduction(res, k = 6, method = "UMAP")
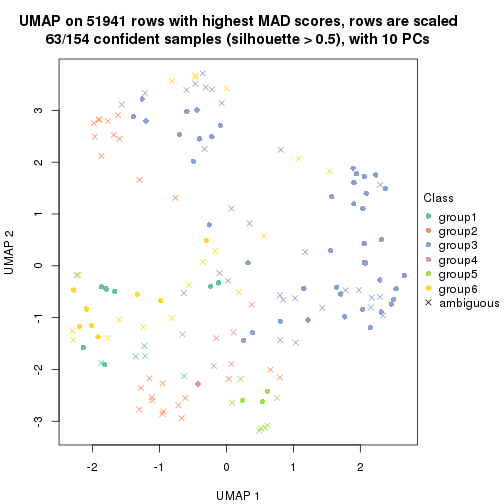
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
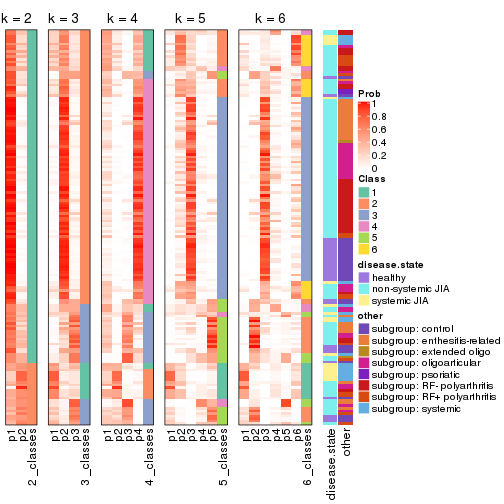
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> MAD:hclust 125 0.001426 0.008218 2
#> MAD:hclust 107 0.000106 0.011315 3
#> MAD:hclust 83 0.000107 0.001090 4
#> MAD:hclust 65 0.000116 0.000573 5
#> MAD:hclust 63 0.001345 0.000856 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["MAD", "kmeans"]
# you can also extract it by
# res = res_list["MAD:kmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'MAD' method.
#> Subgroups are detected by 'kmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
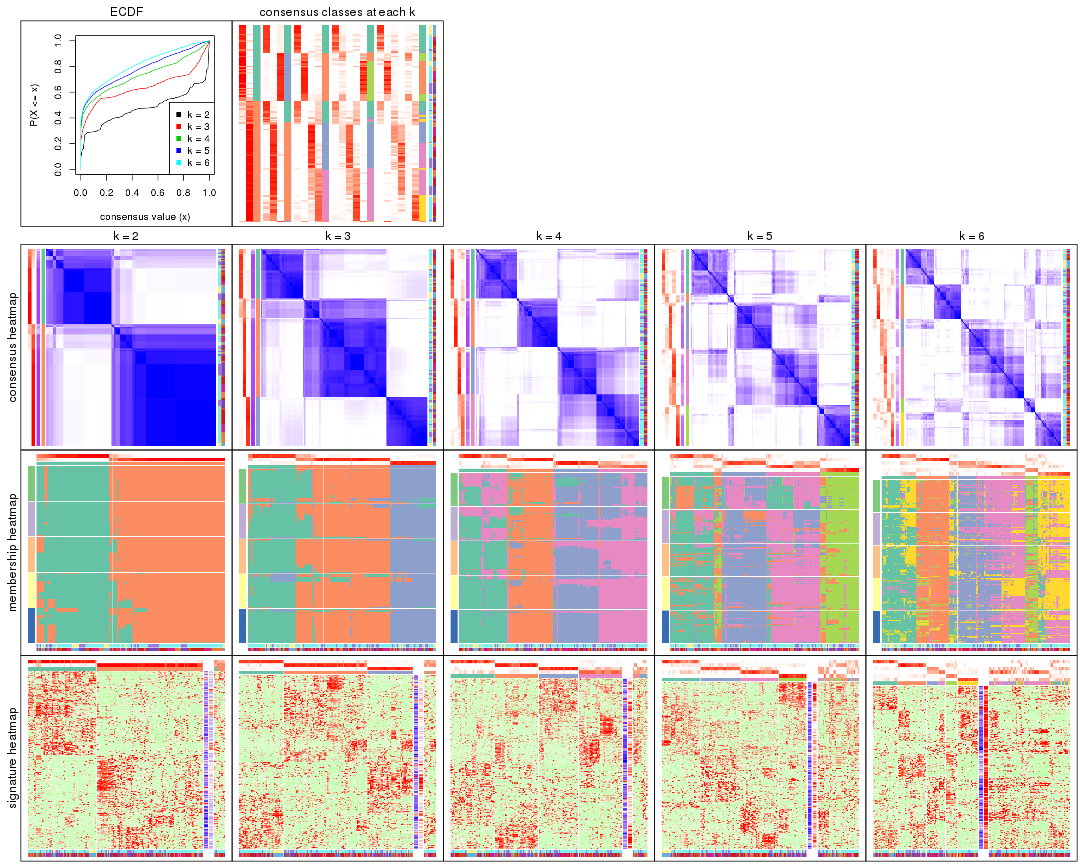
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
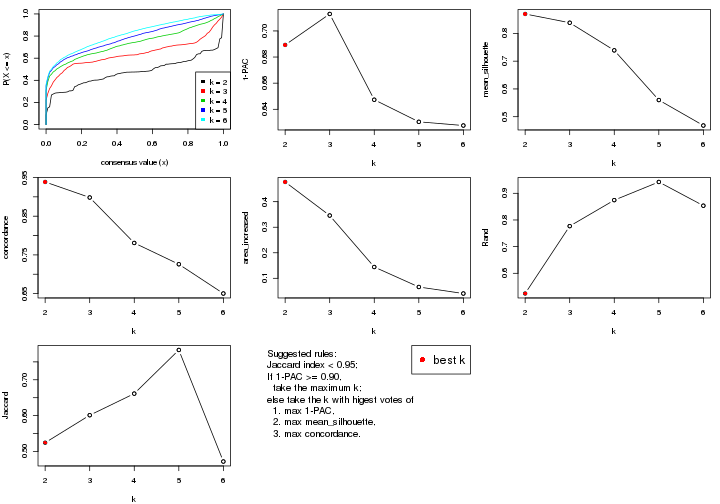
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.689 0.870 0.938 0.4771 0.524 0.524
#> 3 3 0.713 0.839 0.898 0.3457 0.777 0.601
#> 4 4 0.647 0.739 0.781 0.1443 0.874 0.661
#> 5 5 0.630 0.560 0.726 0.0661 0.942 0.783
#> 6 6 0.628 0.468 0.650 0.0405 0.853 0.472
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 2 0.6438 0.797 0.164 0.836
#> GSM340359 1 0.0000 0.917 1.000 0.000
#> GSM340361 1 0.0376 0.916 0.996 0.004
#> GSM340362 1 0.0000 0.917 1.000 0.000
#> GSM340363 2 0.5178 0.848 0.116 0.884
#> GSM340364 1 0.1414 0.915 0.980 0.020
#> GSM340365 1 0.0000 0.917 1.000 0.000
#> GSM340366 1 0.0000 0.917 1.000 0.000
#> GSM340367 1 0.1843 0.914 0.972 0.028
#> GSM340368 2 0.0000 0.941 0.000 1.000
#> GSM340369 2 0.0000 0.941 0.000 1.000
#> GSM340370 1 0.1843 0.914 0.972 0.028
#> GSM340371 1 0.9170 0.567 0.668 0.332
#> GSM340372 1 0.1843 0.914 0.972 0.028
#> GSM340373 1 0.1843 0.914 0.972 0.028
#> GSM340375 1 0.1843 0.914 0.972 0.028
#> GSM340376 2 0.6438 0.798 0.164 0.836
#> GSM340378 1 0.7883 0.687 0.764 0.236
#> GSM340243 2 0.0000 0.941 0.000 1.000
#> GSM340244 2 0.0000 0.941 0.000 1.000
#> GSM340246 2 0.0376 0.941 0.004 0.996
#> GSM340247 2 0.0000 0.941 0.000 1.000
#> GSM340248 2 0.0000 0.941 0.000 1.000
#> GSM340249 2 0.0000 0.941 0.000 1.000
#> GSM340250 2 0.9491 0.376 0.368 0.632
#> GSM340251 2 0.0376 0.941 0.004 0.996
#> GSM340252 1 0.9909 0.294 0.556 0.444
#> GSM340253 1 0.0000 0.917 1.000 0.000
#> GSM340254 1 0.6801 0.784 0.820 0.180
#> GSM340256 2 0.2043 0.924 0.032 0.968
#> GSM340258 2 0.1843 0.925 0.028 0.972
#> GSM340259 1 0.1843 0.914 0.972 0.028
#> GSM340260 1 0.0000 0.917 1.000 0.000
#> GSM340261 2 0.0000 0.941 0.000 1.000
#> GSM340262 1 0.2603 0.897 0.956 0.044
#> GSM340263 2 0.0000 0.941 0.000 1.000
#> GSM340264 1 0.0000 0.917 1.000 0.000
#> GSM340265 1 0.0000 0.917 1.000 0.000
#> GSM340266 2 0.0000 0.941 0.000 1.000
#> GSM340267 1 0.6148 0.811 0.848 0.152
#> GSM340268 2 0.0376 0.941 0.004 0.996
#> GSM340269 2 0.0376 0.941 0.004 0.996
#> GSM340270 2 0.2603 0.911 0.044 0.956
#> GSM537574 2 0.0376 0.941 0.004 0.996
#> GSM537580 1 0.9170 0.567 0.668 0.332
#> GSM537581 2 0.0000 0.941 0.000 1.000
#> GSM340272 1 0.0000 0.917 1.000 0.000
#> GSM340273 2 0.0000 0.941 0.000 1.000
#> GSM340275 2 0.0000 0.941 0.000 1.000
#> GSM340276 2 0.5408 0.841 0.124 0.876
#> GSM340277 1 0.6148 0.811 0.848 0.152
#> GSM340278 2 0.0000 0.941 0.000 1.000
#> GSM340279 2 0.0376 0.941 0.004 0.996
#> GSM340282 1 0.0000 0.917 1.000 0.000
#> GSM340284 2 0.0376 0.941 0.004 0.996
#> GSM340285 2 0.5946 0.819 0.144 0.856
#> GSM340286 1 0.0000 0.917 1.000 0.000
#> GSM340287 2 0.5408 0.841 0.124 0.876
#> GSM340288 1 0.1843 0.914 0.972 0.028
#> GSM340289 1 0.1843 0.914 0.972 0.028
#> GSM340290 2 0.0376 0.941 0.004 0.996
#> GSM340291 2 0.0000 0.941 0.000 1.000
#> GSM340293 2 0.0376 0.941 0.004 0.996
#> GSM340294 2 0.0376 0.941 0.004 0.996
#> GSM340296 2 0.0000 0.941 0.000 1.000
#> GSM340297 2 0.0000 0.941 0.000 1.000
#> GSM340298 2 0.0376 0.941 0.004 0.996
#> GSM340299 1 0.0000 0.917 1.000 0.000
#> GSM340301 1 0.0000 0.917 1.000 0.000
#> GSM340303 1 0.0000 0.917 1.000 0.000
#> GSM340304 2 0.5408 0.841 0.124 0.876
#> GSM340306 1 0.1843 0.914 0.972 0.028
#> GSM340307 2 0.1843 0.925 0.028 0.972
#> GSM340310 2 0.9686 0.384 0.396 0.604
#> GSM340314 2 0.0000 0.941 0.000 1.000
#> GSM340315 2 0.0000 0.941 0.000 1.000
#> GSM340317 2 0.0000 0.941 0.000 1.000
#> GSM340318 2 0.0376 0.941 0.004 0.996
#> GSM340319 2 0.0000 0.941 0.000 1.000
#> GSM340320 2 0.0000 0.941 0.000 1.000
#> GSM340321 2 0.0376 0.941 0.004 0.996
#> GSM340322 2 0.1843 0.925 0.028 0.972
#> GSM340324 2 0.0376 0.940 0.004 0.996
#> GSM340328 2 0.9661 0.394 0.392 0.608
#> GSM340330 2 0.1843 0.925 0.028 0.972
#> GSM340332 2 0.0000 0.941 0.000 1.000
#> GSM340333 1 0.0000 0.917 1.000 0.000
#> GSM340336 2 0.0376 0.941 0.004 0.996
#> GSM340337 2 0.0376 0.941 0.004 0.996
#> GSM340338 2 0.3274 0.903 0.060 0.940
#> GSM340339 2 0.0000 0.941 0.000 1.000
#> GSM340340 2 0.0000 0.941 0.000 1.000
#> GSM340341 2 0.0376 0.941 0.004 0.996
#> GSM340343 2 0.6148 0.811 0.152 0.848
#> GSM340344 1 0.0000 0.917 1.000 0.000
#> GSM340346 2 0.1843 0.925 0.028 0.972
#> GSM340347 2 0.0000 0.941 0.000 1.000
#> GSM340348 2 0.0000 0.941 0.000 1.000
#> GSM340349 1 0.2043 0.912 0.968 0.032
#> GSM340350 1 0.7883 0.687 0.764 0.236
#> GSM340351 1 0.1843 0.914 0.972 0.028
#> GSM340354 2 0.1843 0.925 0.028 0.972
#> GSM340356 2 0.0000 0.941 0.000 1.000
#> GSM340357 1 0.1843 0.914 0.972 0.028
#> GSM348183 1 0.1843 0.914 0.972 0.028
#> GSM348191 2 0.0376 0.941 0.004 0.996
#> GSM348193 2 0.9732 0.363 0.404 0.596
#> GSM537578 1 0.1843 0.914 0.972 0.028
#> GSM348181 2 0.6343 0.802 0.160 0.840
#> GSM348182 1 0.0000 0.917 1.000 0.000
#> GSM348184 2 0.0376 0.941 0.004 0.996
#> GSM348185 2 0.0000 0.941 0.000 1.000
#> GSM348186 2 0.9129 0.468 0.328 0.672
#> GSM348187 2 0.8386 0.649 0.268 0.732
#> GSM348188 1 0.1843 0.914 0.972 0.028
#> GSM348189 1 0.8955 0.603 0.688 0.312
#> GSM348190 1 0.0000 0.917 1.000 0.000
#> GSM348194 1 0.1843 0.914 0.972 0.028
#> GSM348195 1 0.1843 0.914 0.972 0.028
#> GSM348196 2 0.9661 0.394 0.392 0.608
#> GSM537585 1 0.0000 0.917 1.000 0.000
#> GSM537594 2 0.0000 0.941 0.000 1.000
#> GSM537596 2 0.5408 0.841 0.124 0.876
#> GSM537597 2 0.0000 0.941 0.000 1.000
#> GSM537602 2 0.9393 0.479 0.356 0.644
#> GSM340184 2 0.0376 0.941 0.004 0.996
#> GSM340185 2 0.0376 0.941 0.004 0.996
#> GSM340186 2 0.1843 0.925 0.028 0.972
#> GSM340187 2 0.0376 0.941 0.004 0.996
#> GSM340189 2 0.0000 0.941 0.000 1.000
#> GSM340190 2 0.0376 0.941 0.004 0.996
#> GSM340191 2 0.0000 0.941 0.000 1.000
#> GSM340192 1 0.7219 0.762 0.800 0.200
#> GSM340193 1 0.8267 0.645 0.740 0.260
#> GSM340194 1 0.0000 0.917 1.000 0.000
#> GSM340195 1 0.0000 0.917 1.000 0.000
#> GSM340196 2 0.0000 0.941 0.000 1.000
#> GSM340197 1 0.0000 0.917 1.000 0.000
#> GSM340198 1 0.9608 0.454 0.616 0.384
#> GSM340199 2 0.0376 0.941 0.004 0.996
#> GSM340200 2 0.1184 0.934 0.016 0.984
#> GSM340201 2 0.0000 0.941 0.000 1.000
#> GSM340202 2 0.0376 0.941 0.004 0.996
#> GSM340203 2 0.0376 0.941 0.004 0.996
#> GSM340204 1 0.0000 0.917 1.000 0.000
#> GSM340205 2 0.6148 0.811 0.152 0.848
#> GSM340206 2 0.0376 0.941 0.004 0.996
#> GSM340207 1 0.6148 0.811 0.848 0.152
#> GSM340237 2 0.0000 0.941 0.000 1.000
#> GSM340238 2 0.0376 0.941 0.004 0.996
#> GSM340239 1 0.0000 0.917 1.000 0.000
#> GSM340240 1 0.6148 0.811 0.848 0.152
#> GSM340241 2 0.1843 0.925 0.028 0.972
#> GSM340242 1 0.9170 0.567 0.668 0.332
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.2400 0.872 0.932 0.064 0.004
#> GSM340359 3 0.2165 0.943 0.064 0.000 0.936
#> GSM340361 1 0.2625 0.886 0.916 0.000 0.084
#> GSM340362 3 0.2165 0.943 0.064 0.000 0.936
#> GSM340363 1 0.2356 0.863 0.928 0.072 0.000
#> GSM340364 1 0.2625 0.886 0.916 0.000 0.084
#> GSM340365 3 0.2261 0.941 0.068 0.000 0.932
#> GSM340366 3 0.0661 0.943 0.004 0.008 0.988
#> GSM340367 1 0.2537 0.888 0.920 0.000 0.080
#> GSM340368 2 0.5835 0.569 0.340 0.660 0.000
#> GSM340369 2 0.1643 0.873 0.044 0.956 0.000
#> GSM340370 1 0.2356 0.890 0.928 0.000 0.072
#> GSM340371 3 0.2200 0.917 0.004 0.056 0.940
#> GSM340372 1 0.2625 0.886 0.916 0.000 0.084
#> GSM340373 1 0.2625 0.886 0.916 0.000 0.084
#> GSM340375 1 0.2625 0.886 0.916 0.000 0.084
#> GSM340376 1 0.2537 0.864 0.920 0.080 0.000
#> GSM340378 1 0.2200 0.893 0.940 0.004 0.056
#> GSM340243 2 0.4178 0.791 0.172 0.828 0.000
#> GSM340244 2 0.2165 0.865 0.064 0.936 0.000
#> GSM340246 2 0.2297 0.866 0.020 0.944 0.036
#> GSM340247 2 0.1860 0.869 0.052 0.948 0.000
#> GSM340248 2 0.1964 0.870 0.056 0.944 0.000
#> GSM340249 2 0.2165 0.865 0.064 0.936 0.000
#> GSM340250 2 0.7112 0.354 0.424 0.552 0.024
#> GSM340251 2 0.0592 0.875 0.012 0.988 0.000
#> GSM340252 3 0.4189 0.876 0.068 0.056 0.876
#> GSM340253 3 0.2165 0.943 0.064 0.000 0.936
#> GSM340254 3 0.2063 0.927 0.008 0.044 0.948
#> GSM340256 2 0.8275 0.506 0.108 0.596 0.296
#> GSM340258 2 0.1905 0.873 0.016 0.956 0.028
#> GSM340259 1 0.2959 0.878 0.900 0.000 0.100
#> GSM340260 3 0.1289 0.948 0.032 0.000 0.968
#> GSM340261 2 0.2297 0.866 0.020 0.944 0.036
#> GSM340262 3 0.1399 0.936 0.004 0.028 0.968
#> GSM340263 2 0.5810 0.574 0.336 0.664 0.000
#> GSM340264 3 0.1411 0.947 0.036 0.000 0.964
#> GSM340265 3 0.0661 0.942 0.008 0.004 0.988
#> GSM340266 2 0.2356 0.866 0.072 0.928 0.000
#> GSM340267 3 0.1832 0.930 0.008 0.036 0.956
#> GSM340268 2 0.0747 0.875 0.016 0.984 0.000
#> GSM340269 2 0.0747 0.875 0.016 0.984 0.000
#> GSM340270 1 0.5926 0.363 0.644 0.356 0.000
#> GSM537574 2 0.0892 0.875 0.020 0.980 0.000
#> GSM537580 3 0.2200 0.917 0.004 0.056 0.940
#> GSM537581 2 0.2261 0.864 0.068 0.932 0.000
#> GSM340272 3 0.1643 0.947 0.044 0.000 0.956
#> GSM340273 2 0.6126 0.452 0.400 0.600 0.000
#> GSM340275 2 0.2165 0.865 0.064 0.936 0.000
#> GSM340276 1 0.5254 0.644 0.736 0.264 0.000
#> GSM340277 3 0.1399 0.936 0.004 0.028 0.968
#> GSM340278 2 0.1585 0.875 0.028 0.964 0.008
#> GSM340279 2 0.2947 0.856 0.020 0.920 0.060
#> GSM340282 3 0.0424 0.944 0.008 0.000 0.992
#> GSM340284 2 0.0592 0.875 0.012 0.988 0.000
#> GSM340285 1 0.6079 0.378 0.612 0.388 0.000
#> GSM340286 3 0.1860 0.946 0.052 0.000 0.948
#> GSM340287 2 0.7577 0.424 0.324 0.616 0.060
#> GSM340288 1 0.2625 0.886 0.916 0.000 0.084
#> GSM340289 1 0.2878 0.882 0.904 0.000 0.096
#> GSM340290 2 0.2947 0.856 0.020 0.920 0.060
#> GSM340291 2 0.5926 0.538 0.356 0.644 0.000
#> GSM340293 2 0.1482 0.873 0.020 0.968 0.012
#> GSM340294 2 0.2947 0.856 0.020 0.920 0.060
#> GSM340296 2 0.2947 0.856 0.020 0.920 0.060
#> GSM340297 2 0.2066 0.869 0.060 0.940 0.000
#> GSM340298 2 0.2947 0.856 0.020 0.920 0.060
#> GSM340299 3 0.2165 0.943 0.064 0.000 0.936
#> GSM340301 3 0.1289 0.948 0.032 0.000 0.968
#> GSM340303 3 0.0747 0.944 0.016 0.000 0.984
#> GSM340304 1 0.3267 0.822 0.884 0.116 0.000
#> GSM340306 1 0.1525 0.891 0.964 0.004 0.032
#> GSM340307 2 0.2947 0.856 0.020 0.920 0.060
#> GSM340310 1 0.2269 0.885 0.944 0.040 0.016
#> GSM340314 2 0.0747 0.875 0.016 0.984 0.000
#> GSM340315 2 0.2947 0.856 0.020 0.920 0.060
#> GSM340317 2 0.5291 0.675 0.268 0.732 0.000
#> GSM340318 2 0.1753 0.870 0.048 0.952 0.000
#> GSM340319 2 0.1860 0.869 0.052 0.948 0.000
#> GSM340320 2 0.4346 0.777 0.184 0.816 0.000
#> GSM340321 2 0.0592 0.875 0.012 0.988 0.000
#> GSM340322 2 0.1636 0.875 0.020 0.964 0.016
#> GSM340324 2 0.6140 0.445 0.404 0.596 0.000
#> GSM340328 1 0.2527 0.886 0.936 0.044 0.020
#> GSM340330 2 0.3045 0.854 0.020 0.916 0.064
#> GSM340332 2 0.0747 0.875 0.016 0.984 0.000
#> GSM340333 3 0.2165 0.943 0.064 0.000 0.936
#> GSM340336 2 0.1860 0.869 0.052 0.948 0.000
#> GSM340337 2 0.2625 0.857 0.084 0.916 0.000
#> GSM340338 2 0.5455 0.722 0.020 0.776 0.204
#> GSM340339 2 0.1753 0.872 0.048 0.952 0.000
#> GSM340340 2 0.2959 0.847 0.100 0.900 0.000
#> GSM340341 2 0.2448 0.860 0.076 0.924 0.000
#> GSM340343 1 0.5678 0.545 0.684 0.316 0.000
#> GSM340344 3 0.2165 0.943 0.064 0.000 0.936
#> GSM340346 2 0.2947 0.856 0.020 0.920 0.060
#> GSM340347 2 0.2165 0.865 0.064 0.936 0.000
#> GSM340348 2 0.5785 0.581 0.332 0.668 0.000
#> GSM340349 1 0.4128 0.868 0.856 0.012 0.132
#> GSM340350 1 0.3670 0.890 0.888 0.020 0.092
#> GSM340351 1 0.2625 0.886 0.916 0.000 0.084
#> GSM340354 2 0.3045 0.854 0.020 0.916 0.064
#> GSM340356 2 0.6126 0.452 0.400 0.600 0.000
#> GSM340357 1 0.2537 0.888 0.920 0.000 0.080
#> GSM348183 1 0.2448 0.889 0.924 0.000 0.076
#> GSM348191 2 0.2947 0.856 0.020 0.920 0.060
#> GSM348193 1 0.1765 0.883 0.956 0.040 0.004
#> GSM537578 1 0.3965 0.857 0.860 0.008 0.132
#> GSM348181 1 0.2448 0.874 0.924 0.076 0.000
#> GSM348182 3 0.2165 0.942 0.064 0.000 0.936
#> GSM348184 2 0.0592 0.876 0.000 0.988 0.012
#> GSM348185 2 0.6192 0.405 0.420 0.580 0.000
#> GSM348186 3 0.3805 0.884 0.024 0.092 0.884
#> GSM348187 1 0.2356 0.875 0.928 0.072 0.000
#> GSM348188 1 0.2165 0.892 0.936 0.000 0.064
#> GSM348189 3 0.2200 0.917 0.004 0.056 0.940
#> GSM348190 3 0.2165 0.943 0.064 0.000 0.936
#> GSM348194 1 0.2448 0.889 0.924 0.000 0.076
#> GSM348195 1 0.2625 0.886 0.916 0.000 0.084
#> GSM348196 1 0.2527 0.886 0.936 0.044 0.020
#> GSM537585 3 0.2165 0.942 0.064 0.000 0.936
#> GSM537594 2 0.6126 0.452 0.400 0.600 0.000
#> GSM537596 1 0.2625 0.868 0.916 0.084 0.000
#> GSM537597 2 0.6154 0.450 0.408 0.592 0.000
#> GSM537602 1 0.2947 0.881 0.920 0.060 0.020
#> GSM340184 2 0.2947 0.856 0.020 0.920 0.060
#> GSM340185 2 0.1774 0.873 0.016 0.960 0.024
#> GSM340186 2 0.1620 0.873 0.012 0.964 0.024
#> GSM340187 2 0.0592 0.875 0.012 0.988 0.000
#> GSM340189 2 0.0747 0.875 0.016 0.984 0.000
#> GSM340190 2 0.1999 0.867 0.012 0.952 0.036
#> GSM340191 2 0.0747 0.875 0.016 0.984 0.000
#> GSM340192 3 0.1950 0.928 0.008 0.040 0.952
#> GSM340193 1 0.3434 0.894 0.904 0.032 0.064
#> GSM340194 3 0.1529 0.947 0.040 0.000 0.960
#> GSM340195 3 0.2165 0.943 0.064 0.000 0.936
#> GSM340196 2 0.1860 0.869 0.052 0.948 0.000
#> GSM340197 3 0.2165 0.943 0.064 0.000 0.936
#> GSM340198 3 0.2846 0.912 0.020 0.056 0.924
#> GSM340199 2 0.2414 0.865 0.020 0.940 0.040
#> GSM340200 2 0.2804 0.860 0.016 0.924 0.060
#> GSM340201 2 0.2165 0.865 0.064 0.936 0.000
#> GSM340202 2 0.1860 0.869 0.052 0.948 0.000
#> GSM340203 2 0.0747 0.875 0.016 0.984 0.000
#> GSM340204 3 0.2261 0.941 0.068 0.000 0.932
#> GSM340205 1 0.2448 0.866 0.924 0.076 0.000
#> GSM340206 2 0.2947 0.856 0.020 0.920 0.060
#> GSM340207 3 0.1711 0.933 0.008 0.032 0.960
#> GSM340237 2 0.5785 0.581 0.332 0.668 0.000
#> GSM340238 2 0.1636 0.872 0.020 0.964 0.016
#> GSM340239 3 0.2261 0.941 0.068 0.000 0.932
#> GSM340240 3 0.1399 0.936 0.004 0.028 0.968
#> GSM340241 2 0.2031 0.872 0.016 0.952 0.032
#> GSM340242 3 0.2200 0.917 0.004 0.056 0.940
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.4008 0.81415 0.756 0.000 0.000 0.244
#> GSM340359 2 0.1174 0.89832 0.012 0.968 0.000 0.020
#> GSM340361 1 0.1411 0.82176 0.960 0.020 0.000 0.020
#> GSM340362 2 0.0657 0.89902 0.012 0.984 0.000 0.004
#> GSM340363 1 0.4164 0.80067 0.736 0.000 0.000 0.264
#> GSM340364 1 0.0927 0.81987 0.976 0.016 0.000 0.008
#> GSM340365 2 0.3893 0.77919 0.196 0.796 0.000 0.008
#> GSM340366 2 0.1920 0.89439 0.004 0.944 0.028 0.024
#> GSM340367 1 0.0336 0.82171 0.992 0.008 0.000 0.000
#> GSM340368 4 0.4389 0.70734 0.072 0.000 0.116 0.812
#> GSM340369 3 0.2868 0.77550 0.000 0.000 0.864 0.136
#> GSM340370 1 0.1209 0.82953 0.964 0.004 0.000 0.032
#> GSM340371 2 0.5121 0.82800 0.004 0.772 0.096 0.128
#> GSM340372 1 0.0937 0.82131 0.976 0.012 0.000 0.012
#> GSM340373 1 0.0592 0.81888 0.984 0.016 0.000 0.000
#> GSM340375 1 0.1722 0.82055 0.944 0.008 0.000 0.048
#> GSM340376 1 0.4898 0.59137 0.584 0.000 0.000 0.416
#> GSM340378 1 0.3052 0.83531 0.860 0.004 0.000 0.136
#> GSM340243 3 0.5905 0.32917 0.060 0.000 0.636 0.304
#> GSM340244 4 0.3751 0.72887 0.004 0.000 0.196 0.800
#> GSM340246 3 0.1822 0.82977 0.008 0.004 0.944 0.044
#> GSM340247 4 0.4843 0.63260 0.000 0.000 0.396 0.604
#> GSM340248 4 0.4866 0.62565 0.000 0.000 0.404 0.596
#> GSM340249 4 0.3764 0.72906 0.000 0.000 0.216 0.784
#> GSM340250 3 0.8080 -0.21448 0.324 0.004 0.340 0.332
#> GSM340251 4 0.4994 0.48153 0.000 0.000 0.480 0.520
#> GSM340252 2 0.5137 0.21825 0.004 0.544 0.000 0.452
#> GSM340253 2 0.1174 0.89832 0.012 0.968 0.000 0.020
#> GSM340254 2 0.1004 0.89914 0.004 0.972 0.000 0.024
#> GSM340256 4 0.4356 0.55951 0.004 0.200 0.016 0.780
#> GSM340258 4 0.6009 0.59107 0.000 0.048 0.380 0.572
#> GSM340259 1 0.4010 0.82161 0.836 0.064 0.000 0.100
#> GSM340260 2 0.4166 0.85813 0.008 0.836 0.052 0.104
#> GSM340261 3 0.0469 0.83261 0.000 0.000 0.988 0.012
#> GSM340262 2 0.0712 0.90003 0.004 0.984 0.004 0.008
#> GSM340263 4 0.4948 0.69815 0.100 0.000 0.124 0.776
#> GSM340264 2 0.6854 0.75204 0.144 0.680 0.048 0.128
#> GSM340265 2 0.4937 0.83758 0.020 0.796 0.056 0.128
#> GSM340266 4 0.4008 0.72400 0.000 0.000 0.244 0.756
#> GSM340267 2 0.6838 0.75727 0.052 0.684 0.136 0.128
#> GSM340268 3 0.2921 0.77444 0.000 0.000 0.860 0.140
#> GSM340269 3 0.2868 0.78240 0.000 0.000 0.864 0.136
#> GSM340270 1 0.6392 0.33909 0.484 0.000 0.064 0.452
#> GSM537574 4 0.4866 0.62195 0.000 0.000 0.404 0.596
#> GSM537580 2 0.5121 0.82800 0.004 0.772 0.096 0.128
#> GSM537581 4 0.4343 0.71582 0.004 0.000 0.264 0.732
#> GSM340272 2 0.1598 0.89836 0.020 0.956 0.004 0.020
#> GSM340273 4 0.4969 0.63095 0.140 0.000 0.088 0.772
#> GSM340275 4 0.3870 0.72896 0.004 0.000 0.208 0.788
#> GSM340276 1 0.6987 0.65937 0.568 0.000 0.160 0.272
#> GSM340277 2 0.1543 0.89857 0.004 0.956 0.008 0.032
#> GSM340278 3 0.1174 0.83245 0.012 0.000 0.968 0.020
#> GSM340279 3 0.0524 0.83025 0.000 0.008 0.988 0.004
#> GSM340282 2 0.1739 0.89738 0.016 0.952 0.008 0.024
#> GSM340284 4 0.4992 0.49128 0.000 0.000 0.476 0.524
#> GSM340285 1 0.7822 0.27780 0.380 0.000 0.364 0.256
#> GSM340286 2 0.1059 0.89856 0.012 0.972 0.000 0.016
#> GSM340287 3 0.2353 0.77658 0.056 0.008 0.924 0.012
#> GSM340288 1 0.1722 0.82055 0.944 0.008 0.000 0.048
#> GSM340289 1 0.1733 0.80254 0.948 0.024 0.000 0.028
#> GSM340290 3 0.1139 0.82337 0.012 0.008 0.972 0.008
#> GSM340291 4 0.5480 0.67103 0.140 0.000 0.124 0.736
#> GSM340293 3 0.1637 0.82365 0.000 0.000 0.940 0.060
#> GSM340294 3 0.0336 0.83036 0.000 0.008 0.992 0.000
#> GSM340296 3 0.1271 0.82040 0.012 0.008 0.968 0.012
#> GSM340297 3 0.4214 0.68863 0.016 0.000 0.780 0.204
#> GSM340298 3 0.1394 0.81754 0.012 0.008 0.964 0.016
#> GSM340299 2 0.0804 0.89877 0.012 0.980 0.000 0.008
#> GSM340301 2 0.0376 0.90004 0.004 0.992 0.000 0.004
#> GSM340303 2 0.4937 0.83758 0.020 0.796 0.056 0.128
#> GSM340304 1 0.5859 0.74518 0.652 0.000 0.064 0.284
#> GSM340306 1 0.3726 0.82829 0.788 0.000 0.000 0.212
#> GSM340307 3 0.1042 0.82237 0.000 0.008 0.972 0.020
#> GSM340310 1 0.3873 0.81466 0.772 0.000 0.000 0.228
#> GSM340314 3 0.2408 0.80204 0.000 0.000 0.896 0.104
#> GSM340315 3 0.1516 0.81675 0.016 0.008 0.960 0.016
#> GSM340317 4 0.4312 0.71411 0.056 0.000 0.132 0.812
#> GSM340318 4 0.4843 0.63260 0.000 0.000 0.396 0.604
#> GSM340319 4 0.4843 0.63260 0.000 0.000 0.396 0.604
#> GSM340320 3 0.6840 0.00981 0.100 0.000 0.468 0.432
#> GSM340321 4 0.4989 0.50855 0.000 0.000 0.472 0.528
#> GSM340322 4 0.5730 0.63457 0.000 0.040 0.344 0.616
#> GSM340324 4 0.5259 0.55752 0.136 0.052 0.032 0.780
#> GSM340328 1 0.3942 0.81406 0.764 0.000 0.000 0.236
#> GSM340330 3 0.1661 0.82922 0.000 0.004 0.944 0.052
#> GSM340332 3 0.2868 0.77550 0.000 0.000 0.864 0.136
#> GSM340333 2 0.1174 0.89832 0.012 0.968 0.000 0.020
#> GSM340336 4 0.4843 0.63260 0.000 0.000 0.396 0.604
#> GSM340337 4 0.3591 0.72658 0.008 0.000 0.168 0.824
#> GSM340338 3 0.3370 0.76806 0.000 0.080 0.872 0.048
#> GSM340339 3 0.3172 0.74732 0.000 0.000 0.840 0.160
#> GSM340340 4 0.3978 0.72906 0.012 0.000 0.192 0.796
#> GSM340341 4 0.3725 0.72562 0.008 0.000 0.180 0.812
#> GSM340343 1 0.6121 0.66603 0.620 0.000 0.072 0.308
#> GSM340344 2 0.1174 0.89832 0.012 0.968 0.000 0.020
#> GSM340346 3 0.1936 0.79391 0.000 0.028 0.940 0.032
#> GSM340347 4 0.3837 0.72819 0.000 0.000 0.224 0.776
#> GSM340348 4 0.4389 0.69827 0.072 0.000 0.116 0.812
#> GSM340349 1 0.5435 0.67972 0.748 0.016 0.180 0.056
#> GSM340350 1 0.3486 0.82951 0.864 0.000 0.044 0.092
#> GSM340351 1 0.1452 0.82114 0.956 0.008 0.000 0.036
#> GSM340354 3 0.0927 0.82487 0.000 0.008 0.976 0.016
#> GSM340356 4 0.4906 0.62728 0.140 0.000 0.084 0.776
#> GSM340357 1 0.1339 0.81958 0.964 0.004 0.008 0.024
#> GSM348183 1 0.1635 0.83045 0.948 0.008 0.000 0.044
#> GSM348191 3 0.0524 0.82952 0.000 0.008 0.988 0.004
#> GSM348193 1 0.3975 0.80913 0.760 0.000 0.000 0.240
#> GSM537578 1 0.6140 0.56761 0.672 0.024 0.256 0.048
#> GSM348181 1 0.3743 0.83077 0.824 0.000 0.016 0.160
#> GSM348182 2 0.4599 0.83118 0.112 0.800 0.000 0.088
#> GSM348184 3 0.3219 0.73974 0.000 0.000 0.836 0.164
#> GSM348185 4 0.5066 0.62352 0.148 0.000 0.088 0.764
#> GSM348186 2 0.5268 0.20059 0.000 0.540 0.008 0.452
#> GSM348187 1 0.3908 0.82656 0.784 0.000 0.004 0.212
#> GSM348188 1 0.4807 0.81083 0.728 0.024 0.000 0.248
#> GSM348189 2 0.5121 0.82800 0.004 0.772 0.096 0.128
#> GSM348190 2 0.0895 0.89798 0.020 0.976 0.000 0.004
#> GSM348194 1 0.1557 0.82485 0.944 0.000 0.000 0.056
#> GSM348195 1 0.1452 0.82114 0.956 0.008 0.000 0.036
#> GSM348196 1 0.4040 0.81402 0.752 0.000 0.000 0.248
#> GSM537585 2 0.6545 0.70531 0.216 0.652 0.008 0.124
#> GSM537594 4 0.5066 0.62922 0.148 0.000 0.088 0.764
#> GSM537596 1 0.5546 0.79510 0.680 0.000 0.052 0.268
#> GSM537597 4 0.4920 0.63209 0.136 0.000 0.088 0.776
#> GSM537602 1 0.5085 0.79515 0.708 0.000 0.032 0.260
#> GSM340184 3 0.0712 0.82849 0.004 0.008 0.984 0.004
#> GSM340185 4 0.4961 0.56590 0.000 0.000 0.448 0.552
#> GSM340186 4 0.4994 0.48153 0.000 0.000 0.480 0.520
#> GSM340187 4 0.4994 0.48153 0.000 0.000 0.480 0.520
#> GSM340189 3 0.2868 0.77550 0.000 0.000 0.864 0.136
#> GSM340190 3 0.1716 0.82332 0.000 0.000 0.936 0.064
#> GSM340191 3 0.2814 0.77961 0.000 0.000 0.868 0.132
#> GSM340192 2 0.1985 0.89722 0.004 0.940 0.016 0.040
#> GSM340193 1 0.3710 0.83044 0.804 0.004 0.000 0.192
#> GSM340194 2 0.0927 0.89936 0.008 0.976 0.000 0.016
#> GSM340195 2 0.0804 0.89921 0.012 0.980 0.000 0.008
#> GSM340196 4 0.4843 0.63260 0.000 0.000 0.396 0.604
#> GSM340197 2 0.0895 0.89829 0.020 0.976 0.000 0.004
#> GSM340198 2 0.1209 0.89816 0.004 0.964 0.000 0.032
#> GSM340199 3 0.0817 0.83225 0.000 0.000 0.976 0.024
#> GSM340200 3 0.4857 0.14775 0.000 0.008 0.668 0.324
#> GSM340201 4 0.4103 0.72058 0.000 0.000 0.256 0.744
#> GSM340202 4 0.4843 0.63260 0.000 0.000 0.396 0.604
#> GSM340203 3 0.2921 0.77444 0.000 0.000 0.860 0.140
#> GSM340204 2 0.1284 0.89813 0.012 0.964 0.000 0.024
#> GSM340205 1 0.4248 0.81214 0.768 0.000 0.012 0.220
#> GSM340206 3 0.0524 0.82952 0.000 0.008 0.988 0.004
#> GSM340207 2 0.1297 0.89635 0.000 0.964 0.016 0.020
#> GSM340237 4 0.5452 0.68367 0.108 0.000 0.156 0.736
#> GSM340238 3 0.2281 0.80699 0.000 0.000 0.904 0.096
#> GSM340239 2 0.2053 0.87583 0.072 0.924 0.000 0.004
#> GSM340240 2 0.1339 0.89886 0.004 0.964 0.008 0.024
#> GSM340241 4 0.4898 0.58976 0.000 0.000 0.416 0.584
#> GSM340242 2 0.5121 0.82800 0.004 0.772 0.096 0.128
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.5026 0.6853 0.656 0.064 0.000 0.280 0.000
#> GSM340359 5 0.0290 0.7396 0.000 0.000 0.000 0.008 0.992
#> GSM340361 1 0.2026 0.6361 0.924 0.056 0.000 0.012 0.008
#> GSM340362 5 0.1608 0.7252 0.000 0.072 0.000 0.000 0.928
#> GSM340363 1 0.5542 0.6172 0.532 0.072 0.000 0.396 0.000
#> GSM340364 1 0.2006 0.6340 0.916 0.072 0.000 0.012 0.000
#> GSM340365 5 0.5507 0.3191 0.188 0.160 0.000 0.000 0.652
#> GSM340366 5 0.3086 0.6812 0.004 0.180 0.000 0.000 0.816
#> GSM340367 1 0.1430 0.6402 0.944 0.052 0.000 0.004 0.000
#> GSM340368 4 0.0865 0.6286 0.000 0.004 0.024 0.972 0.000
#> GSM340369 3 0.2411 0.6712 0.000 0.008 0.884 0.108 0.000
#> GSM340370 1 0.1082 0.6658 0.964 0.008 0.000 0.028 0.000
#> GSM340371 5 0.5045 -0.1752 0.000 0.464 0.024 0.004 0.508
#> GSM340372 1 0.1124 0.6450 0.960 0.036 0.000 0.004 0.000
#> GSM340373 1 0.1121 0.6415 0.956 0.044 0.000 0.000 0.000
#> GSM340375 1 0.3023 0.6406 0.860 0.112 0.000 0.024 0.004
#> GSM340376 4 0.4781 -0.3968 0.428 0.020 0.000 0.552 0.000
#> GSM340378 1 0.3795 0.7042 0.780 0.028 0.000 0.192 0.000
#> GSM340243 3 0.6898 0.1705 0.032 0.136 0.436 0.396 0.000
#> GSM340244 4 0.1121 0.6434 0.000 0.000 0.044 0.956 0.000
#> GSM340246 3 0.3807 0.7651 0.004 0.176 0.792 0.028 0.000
#> GSM340247 4 0.4651 0.5863 0.000 0.020 0.372 0.608 0.000
#> GSM340248 4 0.4135 0.5922 0.000 0.004 0.340 0.656 0.000
#> GSM340249 4 0.3710 0.6665 0.000 0.024 0.192 0.784 0.000
#> GSM340250 1 0.8391 0.3672 0.320 0.164 0.208 0.308 0.000
#> GSM340251 4 0.4827 0.4463 0.000 0.020 0.476 0.504 0.000
#> GSM340252 5 0.4990 0.2223 0.000 0.048 0.000 0.324 0.628
#> GSM340253 5 0.0290 0.7396 0.000 0.000 0.000 0.008 0.992
#> GSM340254 5 0.1331 0.7268 0.000 0.040 0.000 0.008 0.952
#> GSM340256 4 0.3596 0.5077 0.000 0.016 0.000 0.784 0.200
#> GSM340258 4 0.7379 0.4629 0.000 0.052 0.312 0.452 0.184
#> GSM340259 1 0.6766 0.5755 0.620 0.164 0.004 0.120 0.092
#> GSM340260 5 0.4865 -0.0190 0.016 0.444 0.004 0.000 0.536
#> GSM340261 3 0.3300 0.7581 0.004 0.204 0.792 0.000 0.000
#> GSM340262 5 0.1478 0.7366 0.000 0.064 0.000 0.000 0.936
#> GSM340263 4 0.1251 0.6390 0.000 0.008 0.036 0.956 0.000
#> GSM340264 2 0.6412 0.4611 0.148 0.508 0.008 0.000 0.336
#> GSM340265 2 0.5057 0.3009 0.020 0.532 0.008 0.000 0.440
#> GSM340266 4 0.4839 0.6411 0.004 0.040 0.288 0.668 0.000
#> GSM340267 2 0.5585 0.4238 0.036 0.656 0.052 0.000 0.256
#> GSM340268 3 0.2286 0.6697 0.000 0.004 0.888 0.108 0.000
#> GSM340269 3 0.3839 0.6992 0.004 0.072 0.816 0.108 0.000
#> GSM340270 4 0.5840 -0.2071 0.320 0.084 0.012 0.584 0.000
#> GSM537574 4 0.4538 0.5786 0.000 0.016 0.364 0.620 0.000
#> GSM537580 5 0.5045 -0.1752 0.000 0.464 0.024 0.004 0.508
#> GSM537581 4 0.4513 0.6409 0.004 0.024 0.284 0.688 0.000
#> GSM340272 5 0.3242 0.6695 0.012 0.172 0.000 0.000 0.816
#> GSM340273 4 0.2727 0.5400 0.080 0.020 0.012 0.888 0.000
#> GSM340275 4 0.2464 0.6700 0.000 0.016 0.096 0.888 0.000
#> GSM340276 1 0.8251 0.4754 0.340 0.176 0.156 0.328 0.000
#> GSM340277 5 0.2017 0.7183 0.000 0.080 0.000 0.008 0.912
#> GSM340278 3 0.3616 0.7531 0.004 0.224 0.768 0.004 0.000
#> GSM340279 3 0.3398 0.7567 0.000 0.216 0.780 0.004 0.000
#> GSM340282 5 0.3318 0.6639 0.008 0.192 0.000 0.000 0.800
#> GSM340284 4 0.4560 0.4395 0.000 0.008 0.484 0.508 0.000
#> GSM340285 3 0.8499 -0.2863 0.244 0.180 0.312 0.264 0.000
#> GSM340286 5 0.0162 0.7400 0.000 0.000 0.000 0.004 0.996
#> GSM340287 3 0.3906 0.7360 0.016 0.240 0.744 0.000 0.000
#> GSM340288 1 0.2824 0.6338 0.864 0.116 0.000 0.020 0.000
#> GSM340289 1 0.3612 0.5363 0.784 0.204 0.004 0.004 0.004
#> GSM340290 3 0.3607 0.7386 0.004 0.244 0.752 0.000 0.000
#> GSM340291 4 0.3146 0.5698 0.056 0.028 0.040 0.876 0.000
#> GSM340293 3 0.1364 0.7300 0.000 0.012 0.952 0.036 0.000
#> GSM340294 3 0.3398 0.7518 0.004 0.216 0.780 0.000 0.000
#> GSM340296 3 0.3741 0.7287 0.004 0.264 0.732 0.000 0.000
#> GSM340297 3 0.4703 0.6289 0.016 0.072 0.756 0.156 0.000
#> GSM340298 3 0.3728 0.7364 0.008 0.244 0.748 0.000 0.000
#> GSM340299 5 0.1792 0.7222 0.000 0.084 0.000 0.000 0.916
#> GSM340301 5 0.1792 0.7212 0.000 0.084 0.000 0.000 0.916
#> GSM340303 2 0.5065 0.2868 0.020 0.524 0.008 0.000 0.448
#> GSM340304 4 0.7351 -0.5320 0.396 0.132 0.068 0.404 0.000
#> GSM340306 1 0.5305 0.6836 0.672 0.132 0.000 0.196 0.000
#> GSM340307 3 0.3895 0.7239 0.004 0.264 0.728 0.004 0.000
#> GSM340310 1 0.4622 0.6904 0.684 0.040 0.000 0.276 0.000
#> GSM340314 3 0.2664 0.7225 0.004 0.040 0.892 0.064 0.000
#> GSM340315 3 0.3607 0.7422 0.004 0.244 0.752 0.000 0.000
#> GSM340317 4 0.0794 0.6348 0.000 0.000 0.028 0.972 0.000
#> GSM340318 4 0.4651 0.5863 0.000 0.020 0.372 0.608 0.000
#> GSM340319 4 0.4639 0.5900 0.000 0.020 0.368 0.612 0.000
#> GSM340320 4 0.7335 0.1560 0.100 0.100 0.328 0.472 0.000
#> GSM340321 4 0.4555 0.4462 0.000 0.008 0.472 0.520 0.000
#> GSM340322 4 0.6820 0.5304 0.000 0.044 0.308 0.524 0.124
#> GSM340324 4 0.2331 0.5466 0.064 0.004 0.000 0.908 0.024
#> GSM340328 1 0.4967 0.6877 0.660 0.060 0.000 0.280 0.000
#> GSM340330 3 0.3413 0.7565 0.000 0.124 0.832 0.044 0.000
#> GSM340332 3 0.2389 0.6590 0.000 0.004 0.880 0.116 0.000
#> GSM340333 5 0.0290 0.7396 0.000 0.000 0.000 0.008 0.992
#> GSM340336 4 0.4651 0.5863 0.000 0.020 0.372 0.608 0.000
#> GSM340337 4 0.3450 0.6744 0.004 0.032 0.120 0.840 0.004
#> GSM340338 3 0.5091 0.7145 0.004 0.172 0.736 0.028 0.060
#> GSM340339 3 0.2997 0.6231 0.000 0.012 0.840 0.148 0.000
#> GSM340340 4 0.2519 0.6719 0.000 0.016 0.100 0.884 0.000
#> GSM340341 4 0.2308 0.6378 0.004 0.036 0.048 0.912 0.000
#> GSM340343 1 0.7352 0.5190 0.420 0.140 0.064 0.376 0.000
#> GSM340344 5 0.0451 0.7403 0.000 0.008 0.000 0.004 0.988
#> GSM340346 3 0.4111 0.7116 0.008 0.280 0.708 0.000 0.004
#> GSM340347 4 0.4219 0.6526 0.000 0.024 0.260 0.716 0.000
#> GSM340348 4 0.3201 0.5518 0.028 0.064 0.036 0.872 0.000
#> GSM340349 2 0.6618 -0.2532 0.388 0.456 0.144 0.008 0.004
#> GSM340350 1 0.6404 0.6244 0.628 0.176 0.052 0.144 0.000
#> GSM340351 1 0.2470 0.6198 0.884 0.104 0.000 0.012 0.000
#> GSM340354 3 0.3521 0.7496 0.000 0.232 0.764 0.004 0.000
#> GSM340356 4 0.2429 0.5526 0.068 0.020 0.008 0.904 0.000
#> GSM340357 1 0.2233 0.6476 0.892 0.104 0.000 0.004 0.000
#> GSM348183 1 0.2770 0.6541 0.880 0.076 0.000 0.044 0.000
#> GSM348191 3 0.3398 0.7518 0.004 0.216 0.780 0.000 0.000
#> GSM348193 1 0.5260 0.6680 0.604 0.064 0.000 0.332 0.000
#> GSM537578 1 0.6733 0.0172 0.416 0.368 0.212 0.000 0.004
#> GSM348181 1 0.6005 0.6627 0.620 0.128 0.016 0.236 0.000
#> GSM348182 2 0.6739 0.2913 0.256 0.372 0.000 0.000 0.372
#> GSM348184 3 0.3194 0.5975 0.000 0.020 0.832 0.148 0.000
#> GSM348185 4 0.2707 0.5255 0.080 0.024 0.008 0.888 0.000
#> GSM348186 5 0.3991 0.4800 0.000 0.048 0.000 0.172 0.780
#> GSM348187 1 0.5024 0.6960 0.692 0.096 0.000 0.212 0.000
#> GSM348188 1 0.6085 0.6678 0.572 0.100 0.000 0.312 0.016
#> GSM348189 5 0.5045 -0.1752 0.000 0.464 0.024 0.004 0.508
#> GSM348190 5 0.2563 0.6929 0.008 0.120 0.000 0.000 0.872
#> GSM348194 1 0.2761 0.6469 0.872 0.104 0.000 0.024 0.000
#> GSM348195 1 0.2470 0.6198 0.884 0.104 0.000 0.012 0.000
#> GSM348196 1 0.4960 0.6894 0.668 0.064 0.000 0.268 0.000
#> GSM537585 2 0.6439 0.4501 0.260 0.504 0.000 0.000 0.236
#> GSM537594 4 0.2340 0.5500 0.068 0.012 0.012 0.908 0.000
#> GSM537596 1 0.7709 0.5827 0.424 0.184 0.080 0.312 0.000
#> GSM537597 4 0.3900 0.4471 0.108 0.068 0.008 0.816 0.000
#> GSM537602 1 0.6960 0.6110 0.480 0.128 0.044 0.348 0.000
#> GSM340184 3 0.3366 0.7505 0.000 0.232 0.768 0.000 0.000
#> GSM340185 4 0.5242 0.4912 0.000 0.036 0.444 0.516 0.004
#> GSM340186 4 0.5491 0.4481 0.000 0.052 0.452 0.492 0.004
#> GSM340187 4 0.4829 0.4391 0.000 0.020 0.480 0.500 0.000
#> GSM340189 3 0.2233 0.6737 0.000 0.004 0.892 0.104 0.000
#> GSM340190 3 0.1981 0.7313 0.000 0.028 0.924 0.048 0.000
#> GSM340191 3 0.2470 0.6783 0.000 0.012 0.884 0.104 0.000
#> GSM340192 5 0.2136 0.7142 0.000 0.088 0.000 0.008 0.904
#> GSM340193 1 0.5223 0.6919 0.672 0.108 0.000 0.220 0.000
#> GSM340194 5 0.0162 0.7404 0.000 0.004 0.000 0.000 0.996
#> GSM340195 5 0.2377 0.6915 0.000 0.128 0.000 0.000 0.872
#> GSM340196 4 0.4651 0.5863 0.000 0.020 0.372 0.608 0.000
#> GSM340197 5 0.2753 0.6818 0.008 0.136 0.000 0.000 0.856
#> GSM340198 5 0.1331 0.7268 0.000 0.040 0.000 0.008 0.952
#> GSM340199 3 0.2771 0.7715 0.000 0.128 0.860 0.012 0.000
#> GSM340200 3 0.6733 0.3323 0.004 0.224 0.492 0.276 0.004
#> GSM340201 4 0.4268 0.6495 0.000 0.024 0.268 0.708 0.000
#> GSM340202 4 0.4709 0.5917 0.000 0.024 0.364 0.612 0.000
#> GSM340203 3 0.2179 0.6772 0.000 0.004 0.896 0.100 0.000
#> GSM340204 5 0.0671 0.7368 0.000 0.004 0.000 0.016 0.980
#> GSM340205 1 0.6359 0.6236 0.520 0.116 0.016 0.348 0.000
#> GSM340206 3 0.3210 0.7561 0.000 0.212 0.788 0.000 0.000
#> GSM340207 5 0.2338 0.7136 0.000 0.112 0.004 0.000 0.884
#> GSM340237 4 0.3367 0.5865 0.016 0.040 0.088 0.856 0.000
#> GSM340238 3 0.1914 0.7217 0.000 0.016 0.924 0.060 0.000
#> GSM340239 5 0.4948 0.4674 0.108 0.184 0.000 0.000 0.708
#> GSM340240 5 0.2077 0.7162 0.000 0.084 0.000 0.008 0.908
#> GSM340241 4 0.6027 0.5161 0.000 0.048 0.388 0.528 0.036
#> GSM340242 5 0.5045 -0.1752 0.000 0.464 0.024 0.004 0.508
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.5775 0.5138 0.528 0.000 0.100 0.000 0.028 0.344
#> GSM340359 2 0.0146 0.7941 0.000 0.996 0.000 0.000 0.000 0.004
#> GSM340361 1 0.1408 0.6668 0.952 0.008 0.008 0.000 0.008 0.024
#> GSM340362 2 0.2462 0.7714 0.000 0.876 0.028 0.000 0.096 0.000
#> GSM340363 6 0.5642 -0.1014 0.308 0.000 0.112 0.000 0.020 0.560
#> GSM340364 1 0.2095 0.6478 0.916 0.000 0.028 0.000 0.040 0.016
#> GSM340365 2 0.5706 0.4035 0.228 0.608 0.036 0.000 0.128 0.000
#> GSM340366 2 0.5433 0.6010 0.012 0.652 0.124 0.004 0.200 0.008
#> GSM340367 1 0.1760 0.6587 0.928 0.000 0.048 0.000 0.020 0.004
#> GSM340368 6 0.2631 0.6663 0.008 0.000 0.004 0.124 0.004 0.860
#> GSM340369 4 0.1536 0.4724 0.000 0.000 0.040 0.940 0.004 0.016
#> GSM340370 1 0.1780 0.6768 0.932 0.000 0.028 0.000 0.012 0.028
#> GSM340371 5 0.5248 0.6744 0.000 0.308 0.076 0.012 0.600 0.004
#> GSM340372 1 0.1296 0.6636 0.952 0.000 0.032 0.000 0.012 0.004
#> GSM340373 1 0.1552 0.6558 0.940 0.000 0.036 0.000 0.020 0.004
#> GSM340375 1 0.4295 0.6305 0.776 0.000 0.080 0.000 0.096 0.048
#> GSM340376 6 0.3888 0.1675 0.312 0.000 0.016 0.000 0.000 0.672
#> GSM340378 1 0.3739 0.6746 0.776 0.000 0.040 0.000 0.008 0.176
#> GSM340243 3 0.6927 0.1783 0.024 0.000 0.396 0.192 0.028 0.360
#> GSM340244 6 0.2566 0.6646 0.000 0.000 0.012 0.112 0.008 0.868
#> GSM340246 3 0.5409 0.4375 0.000 0.000 0.468 0.448 0.020 0.064
#> GSM340247 4 0.4181 0.3920 0.000 0.000 0.004 0.600 0.012 0.384
#> GSM340248 6 0.5037 -0.1152 0.000 0.000 0.036 0.428 0.020 0.516
#> GSM340249 6 0.4514 0.2711 0.000 0.000 0.028 0.328 0.012 0.632
#> GSM340250 3 0.8205 -0.0769 0.188 0.004 0.328 0.104 0.060 0.316
#> GSM340251 4 0.3483 0.5343 0.000 0.000 0.000 0.748 0.016 0.236
#> GSM340252 2 0.4827 0.4201 0.000 0.712 0.024 0.024 0.036 0.204
#> GSM340253 2 0.0146 0.7941 0.000 0.996 0.000 0.000 0.000 0.004
#> GSM340254 2 0.1363 0.7809 0.000 0.952 0.028 0.004 0.012 0.004
#> GSM340256 6 0.4593 0.5338 0.000 0.224 0.020 0.020 0.024 0.712
#> GSM340258 4 0.7145 0.3183 0.000 0.224 0.052 0.480 0.032 0.212
#> GSM340259 1 0.7599 0.4791 0.500 0.076 0.228 0.008 0.104 0.084
#> GSM340260 5 0.5615 0.5243 0.012 0.308 0.112 0.000 0.564 0.004
#> GSM340261 3 0.4450 0.4945 0.000 0.000 0.528 0.448 0.020 0.004
#> GSM340262 2 0.2221 0.7887 0.000 0.896 0.032 0.000 0.072 0.000
#> GSM340263 6 0.3081 0.6672 0.004 0.000 0.032 0.100 0.012 0.852
#> GSM340264 5 0.5349 0.6717 0.088 0.180 0.060 0.000 0.672 0.000
#> GSM340265 5 0.4738 0.6831 0.008 0.224 0.076 0.000 0.688 0.004
#> GSM340266 4 0.5472 0.2457 0.000 0.000 0.048 0.524 0.040 0.388
#> GSM340267 5 0.5928 0.5398 0.024 0.116 0.292 0.000 0.560 0.008
#> GSM340268 4 0.1500 0.4581 0.000 0.000 0.052 0.936 0.000 0.012
#> GSM340269 4 0.4584 0.1867 0.000 0.000 0.248 0.688 0.024 0.040
#> GSM340270 6 0.6899 0.2906 0.208 0.000 0.148 0.064 0.036 0.544
#> GSM537574 4 0.5147 0.2625 0.000 0.000 0.048 0.480 0.016 0.456
#> GSM537580 5 0.5365 0.6738 0.000 0.312 0.076 0.012 0.592 0.008
#> GSM537581 4 0.4631 0.2116 0.000 0.000 0.008 0.504 0.024 0.464
#> GSM340272 2 0.4946 0.6417 0.020 0.688 0.080 0.000 0.208 0.004
#> GSM340273 6 0.1691 0.6876 0.028 0.000 0.012 0.012 0.008 0.940
#> GSM340275 6 0.4168 0.5431 0.000 0.000 0.032 0.204 0.024 0.740
#> GSM340276 3 0.7478 -0.0736 0.120 0.000 0.448 0.064 0.072 0.296
#> GSM340277 2 0.3140 0.7302 0.000 0.848 0.068 0.004 0.076 0.004
#> GSM340278 3 0.4611 0.5017 0.000 0.000 0.584 0.380 0.020 0.016
#> GSM340279 4 0.4834 -0.4528 0.000 0.000 0.472 0.480 0.044 0.004
#> GSM340282 2 0.5193 0.6278 0.012 0.664 0.100 0.004 0.216 0.004
#> GSM340284 4 0.3828 0.5324 0.000 0.000 0.016 0.724 0.008 0.252
#> GSM340285 3 0.7707 0.1457 0.096 0.000 0.480 0.136 0.084 0.204
#> GSM340286 2 0.0508 0.7969 0.000 0.984 0.000 0.000 0.012 0.004
#> GSM340287 3 0.4749 0.5433 0.004 0.000 0.588 0.368 0.032 0.008
#> GSM340288 1 0.3931 0.6472 0.804 0.000 0.080 0.000 0.072 0.044
#> GSM340289 1 0.4141 0.5613 0.756 0.000 0.128 0.000 0.112 0.004
#> GSM340290 3 0.4607 0.5452 0.000 0.000 0.604 0.356 0.028 0.012
#> GSM340291 6 0.3627 0.6759 0.020 0.000 0.068 0.068 0.012 0.832
#> GSM340293 4 0.2488 0.3575 0.000 0.000 0.124 0.864 0.004 0.008
#> GSM340294 3 0.4555 0.5126 0.000 0.000 0.548 0.420 0.028 0.004
#> GSM340296 3 0.5109 0.5400 0.008 0.000 0.592 0.336 0.056 0.008
#> GSM340297 4 0.5656 0.0758 0.008 0.000 0.260 0.612 0.032 0.088
#> GSM340298 3 0.4897 0.5280 0.000 0.000 0.556 0.384 0.056 0.004
#> GSM340299 2 0.2412 0.7723 0.000 0.880 0.028 0.000 0.092 0.000
#> GSM340301 2 0.2843 0.7598 0.000 0.848 0.036 0.000 0.116 0.000
#> GSM340303 5 0.4763 0.6827 0.008 0.228 0.076 0.000 0.684 0.004
#> GSM340304 6 0.6513 0.1329 0.132 0.000 0.304 0.008 0.052 0.504
#> GSM340306 1 0.6429 0.5553 0.524 0.000 0.116 0.000 0.084 0.276
#> GSM340307 3 0.5519 0.3923 0.000 0.000 0.456 0.436 0.100 0.008
#> GSM340310 1 0.5185 0.5350 0.572 0.000 0.064 0.000 0.016 0.348
#> GSM340314 4 0.3240 0.1555 0.000 0.000 0.244 0.752 0.004 0.000
#> GSM340315 3 0.4808 0.5352 0.000 0.000 0.568 0.384 0.036 0.012
#> GSM340317 6 0.2101 0.6749 0.004 0.000 0.000 0.100 0.004 0.892
#> GSM340318 4 0.4181 0.3920 0.000 0.000 0.004 0.600 0.012 0.384
#> GSM340319 4 0.4181 0.3920 0.000 0.000 0.004 0.600 0.012 0.384
#> GSM340320 6 0.7555 0.2012 0.044 0.000 0.288 0.244 0.048 0.376
#> GSM340321 4 0.4504 0.4945 0.000 0.000 0.032 0.648 0.012 0.308
#> GSM340322 4 0.6384 0.3655 0.000 0.084 0.032 0.536 0.040 0.308
#> GSM340324 6 0.2451 0.6900 0.028 0.036 0.008 0.024 0.000 0.904
#> GSM340328 1 0.5770 0.5310 0.540 0.000 0.104 0.000 0.028 0.328
#> GSM340330 4 0.4441 -0.1074 0.000 0.004 0.344 0.620 0.032 0.000
#> GSM340332 4 0.1151 0.4707 0.000 0.000 0.032 0.956 0.000 0.012
#> GSM340333 2 0.0146 0.7941 0.000 0.996 0.000 0.000 0.000 0.004
#> GSM340336 4 0.4181 0.3920 0.000 0.000 0.004 0.600 0.012 0.384
#> GSM340337 6 0.4725 0.3775 0.004 0.004 0.012 0.296 0.032 0.652
#> GSM340338 4 0.5898 -0.2349 0.000 0.044 0.360 0.520 0.072 0.004
#> GSM340339 4 0.2680 0.4780 0.000 0.000 0.048 0.880 0.012 0.060
#> GSM340340 6 0.3930 0.5358 0.000 0.000 0.028 0.216 0.012 0.744
#> GSM340341 6 0.3005 0.6856 0.000 0.000 0.024 0.060 0.052 0.864
#> GSM340343 3 0.8174 -0.2859 0.228 0.000 0.332 0.064 0.100 0.276
#> GSM340344 2 0.0405 0.7963 0.000 0.988 0.004 0.000 0.008 0.000
#> GSM340346 3 0.5322 0.5037 0.000 0.000 0.524 0.376 0.096 0.004
#> GSM340347 4 0.4617 0.2376 0.000 0.000 0.008 0.524 0.024 0.444
#> GSM340348 6 0.3451 0.6467 0.004 0.000 0.092 0.024 0.044 0.836
#> GSM340349 3 0.5427 0.1746 0.144 0.000 0.664 0.008 0.160 0.024
#> GSM340350 1 0.6406 0.4931 0.504 0.000 0.312 0.000 0.092 0.092
#> GSM340351 1 0.2366 0.6459 0.900 0.000 0.024 0.000 0.056 0.020
#> GSM340354 3 0.4903 0.4081 0.000 0.000 0.472 0.468 0.060 0.000
#> GSM340356 6 0.1691 0.6890 0.028 0.000 0.008 0.012 0.012 0.940
#> GSM340357 1 0.3578 0.6559 0.800 0.000 0.152 0.000 0.032 0.016
#> GSM348183 1 0.2201 0.6729 0.912 0.000 0.028 0.000 0.028 0.032
#> GSM348191 3 0.4777 0.5180 0.000 0.000 0.540 0.416 0.036 0.008
#> GSM348193 1 0.4980 0.3874 0.492 0.000 0.048 0.000 0.008 0.452
#> GSM537578 3 0.6257 0.0837 0.264 0.000 0.536 0.028 0.164 0.008
#> GSM348181 1 0.7295 0.4412 0.456 0.000 0.248 0.020 0.084 0.192
#> GSM348182 5 0.6600 0.4558 0.240 0.224 0.052 0.000 0.484 0.000
#> GSM348184 4 0.1666 0.4969 0.000 0.000 0.020 0.936 0.008 0.036
#> GSM348185 6 0.1599 0.6789 0.028 0.000 0.024 0.008 0.000 0.940
#> GSM348186 2 0.3495 0.6912 0.000 0.848 0.036 0.020 0.036 0.060
#> GSM348187 1 0.6400 0.5409 0.508 0.000 0.172 0.000 0.048 0.272
#> GSM348188 1 0.7074 0.4263 0.436 0.040 0.092 0.000 0.072 0.360
#> GSM348189 5 0.5365 0.6738 0.000 0.312 0.076 0.012 0.592 0.008
#> GSM348190 2 0.3307 0.7456 0.012 0.828 0.040 0.000 0.120 0.000
#> GSM348194 1 0.4222 0.6530 0.784 0.000 0.088 0.000 0.064 0.064
#> GSM348195 1 0.2279 0.6445 0.904 0.000 0.024 0.000 0.056 0.016
#> GSM348196 1 0.6030 0.5013 0.508 0.000 0.116 0.000 0.036 0.340
#> GSM537585 5 0.6655 0.5003 0.288 0.128 0.080 0.000 0.500 0.004
#> GSM537594 6 0.1575 0.6969 0.032 0.000 0.000 0.032 0.000 0.936
#> GSM537596 3 0.7560 -0.2313 0.164 0.000 0.388 0.040 0.076 0.332
#> GSM537597 6 0.4201 0.5763 0.048 0.000 0.108 0.012 0.040 0.792
#> GSM537602 6 0.6802 -0.1512 0.240 0.000 0.268 0.000 0.056 0.436
#> GSM340184 3 0.4780 0.5191 0.000 0.000 0.544 0.408 0.044 0.004
#> GSM340185 4 0.4924 0.5027 0.000 0.000 0.048 0.648 0.028 0.276
#> GSM340186 4 0.4815 0.5427 0.000 0.012 0.048 0.712 0.028 0.200
#> GSM340187 4 0.3596 0.5388 0.000 0.000 0.004 0.748 0.016 0.232
#> GSM340189 4 0.1367 0.4668 0.000 0.000 0.044 0.944 0.000 0.012
#> GSM340190 4 0.3020 0.3242 0.000 0.000 0.156 0.824 0.012 0.008
#> GSM340191 4 0.2883 0.3757 0.000 0.000 0.132 0.844 0.012 0.012
#> GSM340192 2 0.3195 0.7335 0.000 0.852 0.068 0.008 0.064 0.008
#> GSM340193 1 0.7296 0.4999 0.432 0.000 0.200 0.008 0.104 0.256
#> GSM340194 2 0.0603 0.7958 0.000 0.980 0.004 0.000 0.016 0.000
#> GSM340195 2 0.3398 0.7430 0.016 0.824 0.040 0.000 0.120 0.000
#> GSM340196 4 0.4181 0.3920 0.000 0.000 0.004 0.600 0.012 0.384
#> GSM340197 2 0.3934 0.7181 0.036 0.792 0.044 0.000 0.128 0.000
#> GSM340198 2 0.2127 0.7650 0.000 0.920 0.032 0.016 0.024 0.008
#> GSM340199 4 0.4302 -0.2212 0.000 0.000 0.368 0.608 0.020 0.004
#> GSM340200 4 0.7132 -0.0273 0.000 0.008 0.368 0.380 0.088 0.156
#> GSM340201 4 0.4553 0.2396 0.000 0.000 0.008 0.520 0.020 0.452
#> GSM340202 4 0.4181 0.3920 0.000 0.000 0.004 0.600 0.012 0.384
#> GSM340203 4 0.2070 0.4167 0.000 0.000 0.100 0.892 0.000 0.008
#> GSM340204 2 0.1078 0.7865 0.000 0.964 0.012 0.000 0.016 0.008
#> GSM340205 1 0.7339 0.2291 0.356 0.000 0.220 0.020 0.060 0.344
#> GSM340206 4 0.4642 -0.4180 0.000 0.000 0.452 0.508 0.040 0.000
#> GSM340207 2 0.3998 0.7059 0.000 0.776 0.084 0.004 0.132 0.004
#> GSM340237 6 0.4047 0.6553 0.012 0.000 0.092 0.076 0.020 0.800
#> GSM340238 4 0.2854 0.2352 0.000 0.000 0.208 0.792 0.000 0.000
#> GSM340239 2 0.5668 0.4919 0.156 0.640 0.052 0.000 0.152 0.000
#> GSM340240 2 0.3296 0.7307 0.000 0.836 0.068 0.004 0.088 0.004
#> GSM340241 4 0.6335 0.4469 0.000 0.076 0.052 0.568 0.032 0.272
#> GSM340242 5 0.5365 0.6738 0.000 0.312 0.076 0.012 0.592 0.008
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
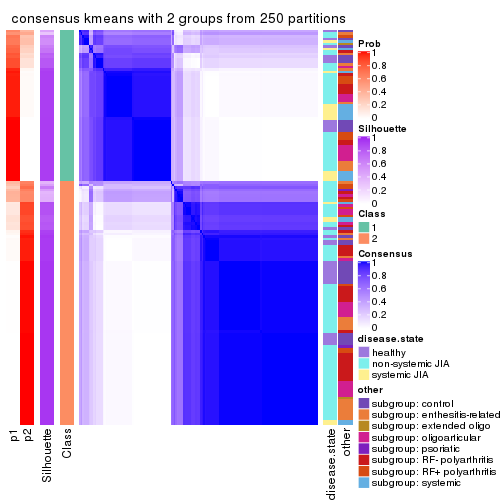
consensus_heatmap(res, k = 3)
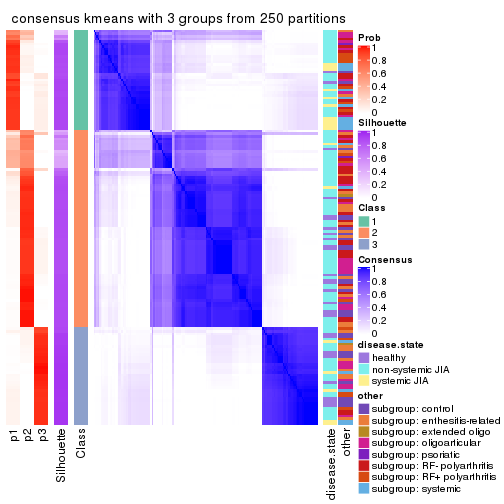
consensus_heatmap(res, k = 4)
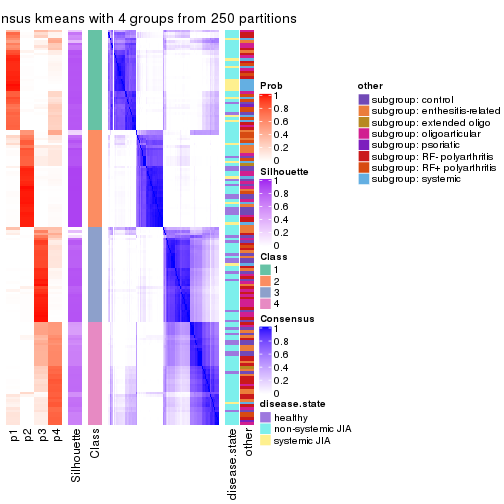
consensus_heatmap(res, k = 5)
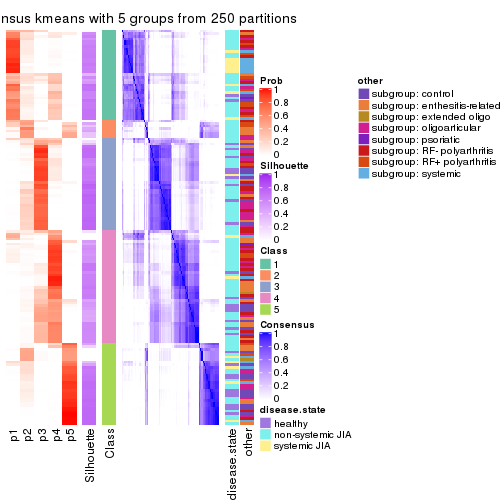
consensus_heatmap(res, k = 6)
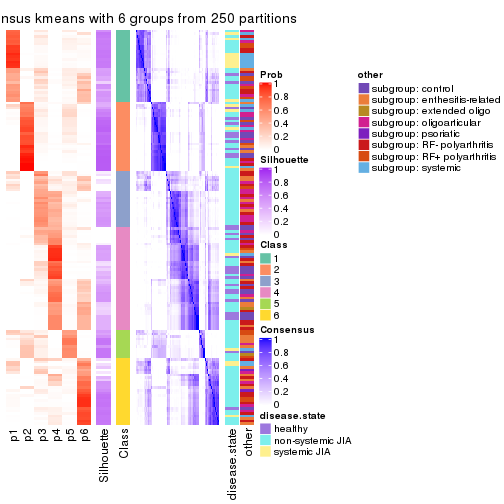
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
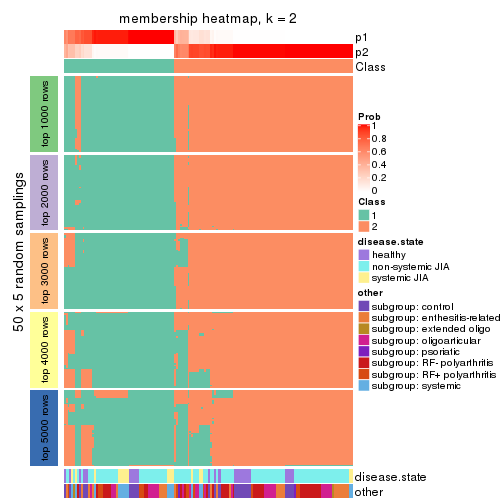
membership_heatmap(res, k = 3)
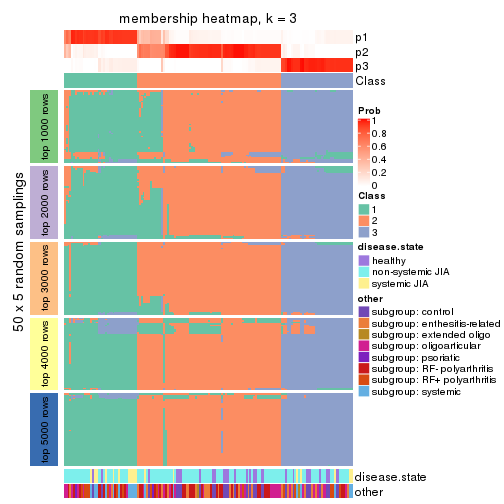
membership_heatmap(res, k = 4)
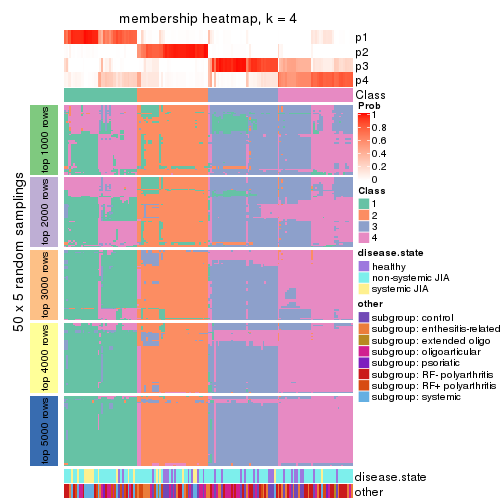
membership_heatmap(res, k = 5)
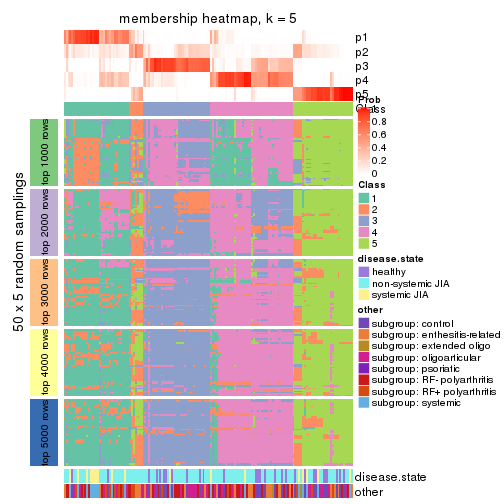
membership_heatmap(res, k = 6)
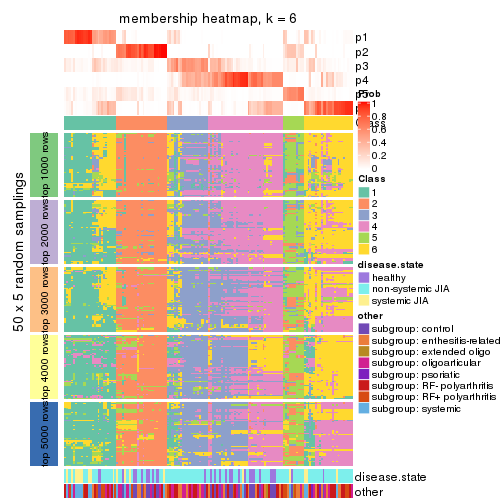
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
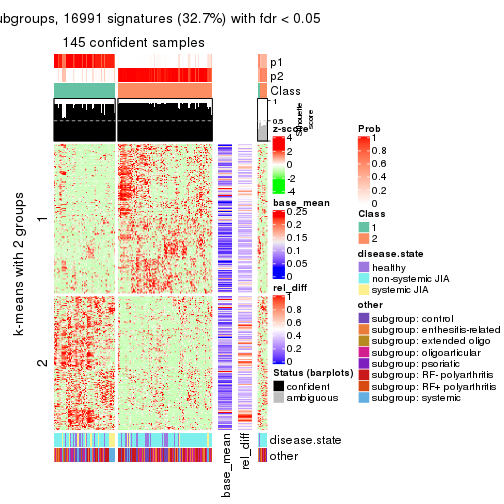
get_signatures(res, k = 3)
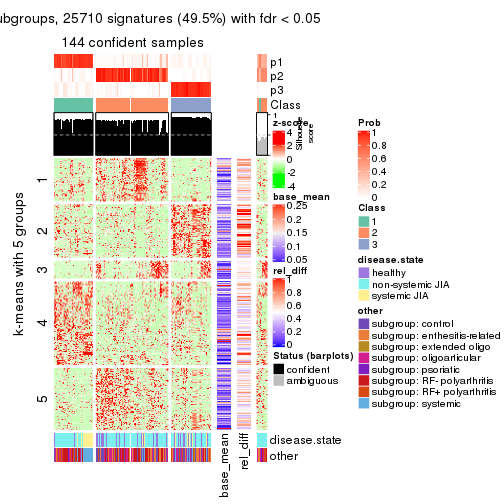
get_signatures(res, k = 4)
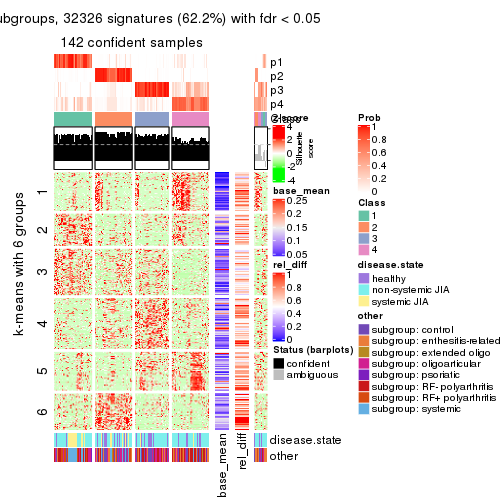
get_signatures(res, k = 5)
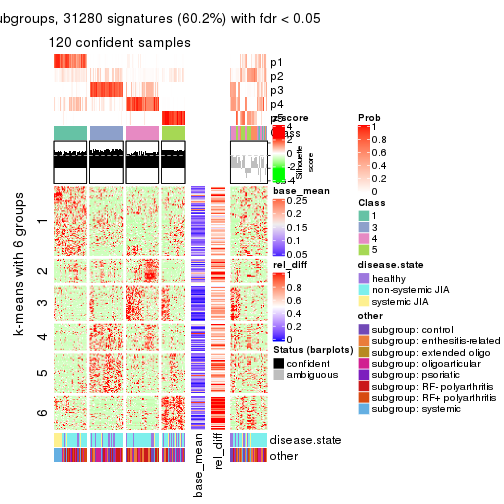
get_signatures(res, k = 6)
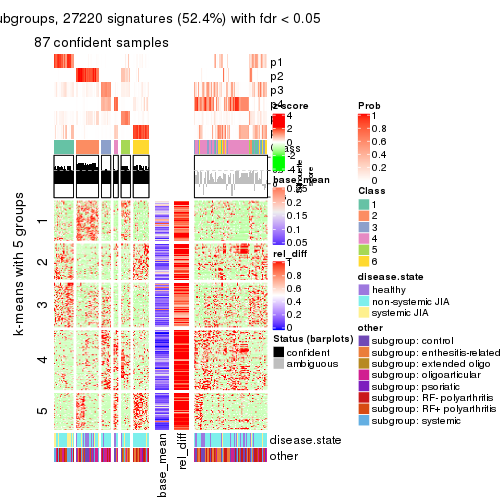
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
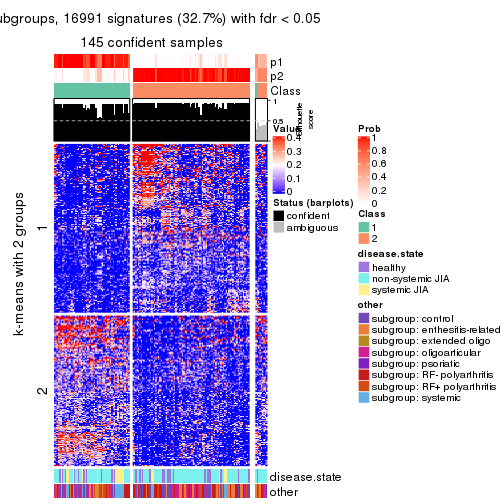
get_signatures(res, k = 3, scale_rows = FALSE)
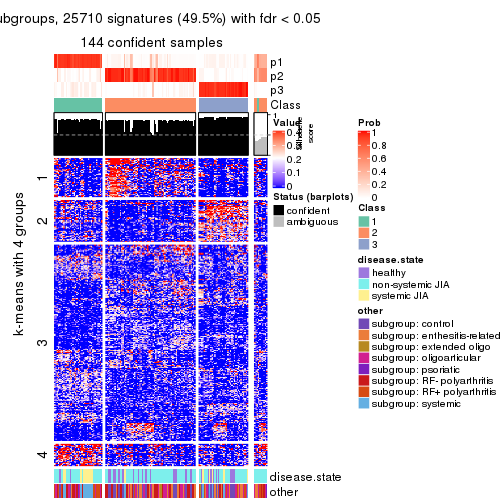
get_signatures(res, k = 4, scale_rows = FALSE)
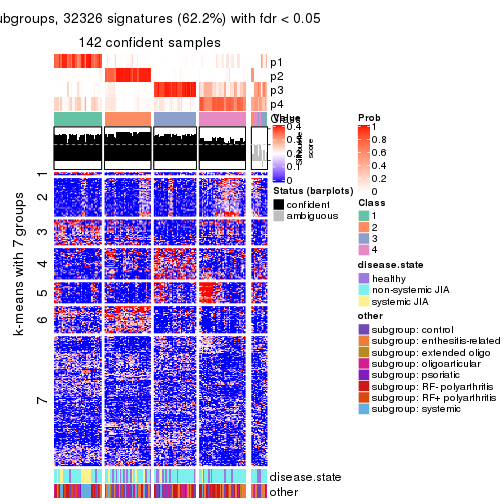
get_signatures(res, k = 5, scale_rows = FALSE)
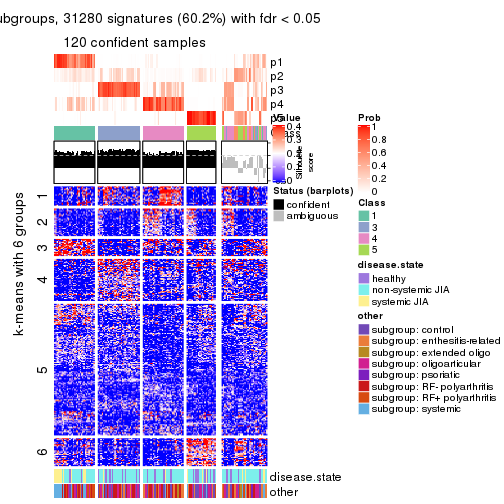
get_signatures(res, k = 6, scale_rows = FALSE)
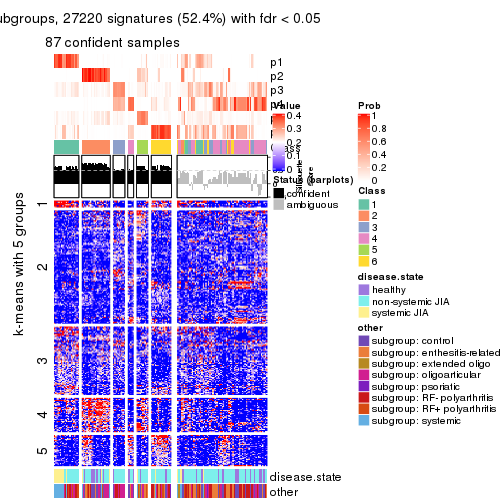
Compare the overlap of signatures from different k:
compare_signatures(res)
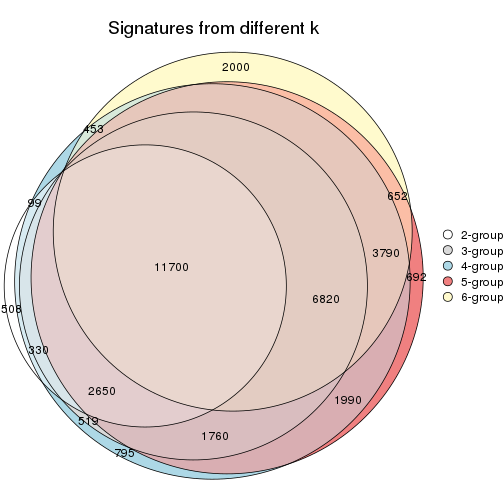
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
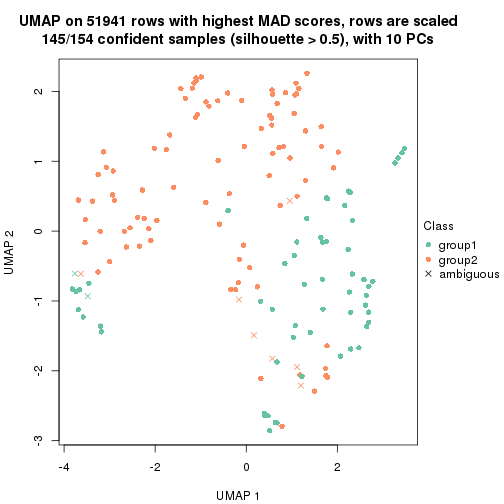
dimension_reduction(res, k = 3, method = "UMAP")
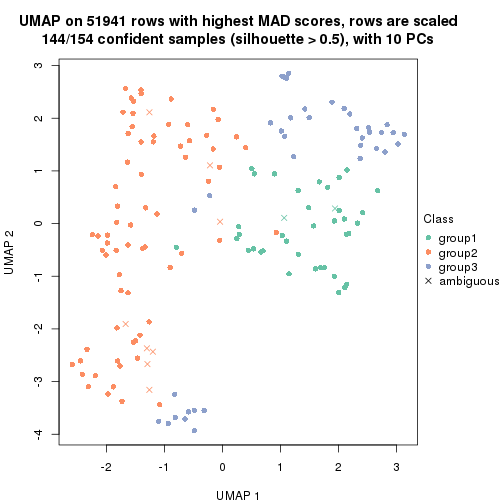
dimension_reduction(res, k = 4, method = "UMAP")
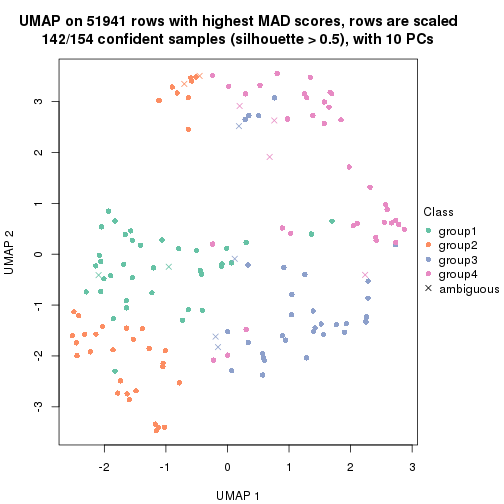
dimension_reduction(res, k = 5, method = "UMAP")
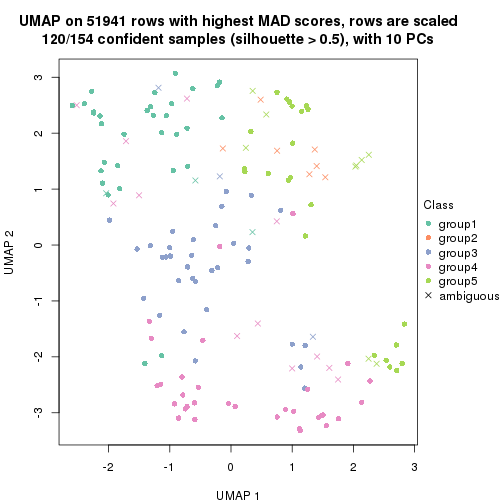
dimension_reduction(res, k = 6, method = "UMAP")
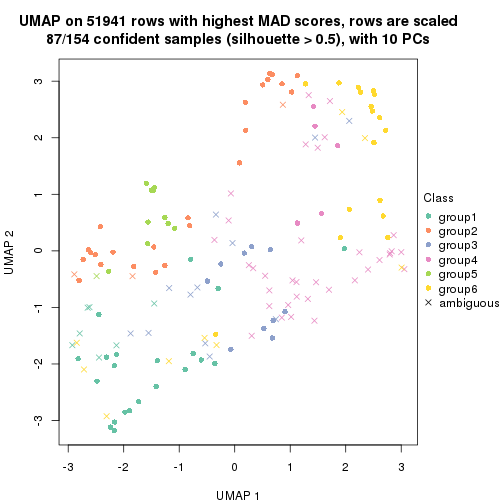
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
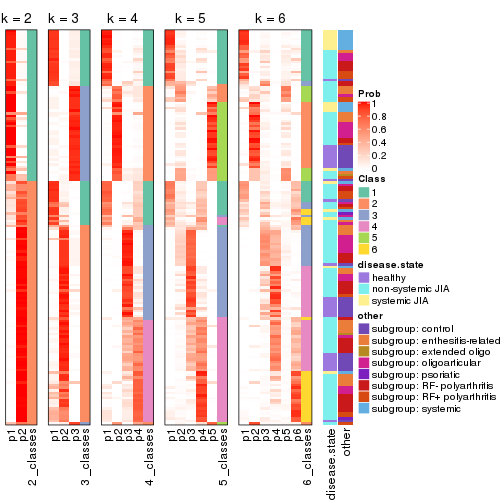
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> MAD:kmeans 145 0.009335 6.33e-02 2
#> MAD:kmeans 144 0.000533 3.77e-05 3
#> MAD:kmeans 142 0.001836 6.96e-04 4
#> MAD:kmeans 120 0.001262 5.25e-04 5
#> MAD:kmeans 87 0.000367 5.17e-04 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["MAD", "skmeans"]
# you can also extract it by
# res = res_list["MAD:skmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'MAD' method.
#> Subgroups are detected by 'skmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
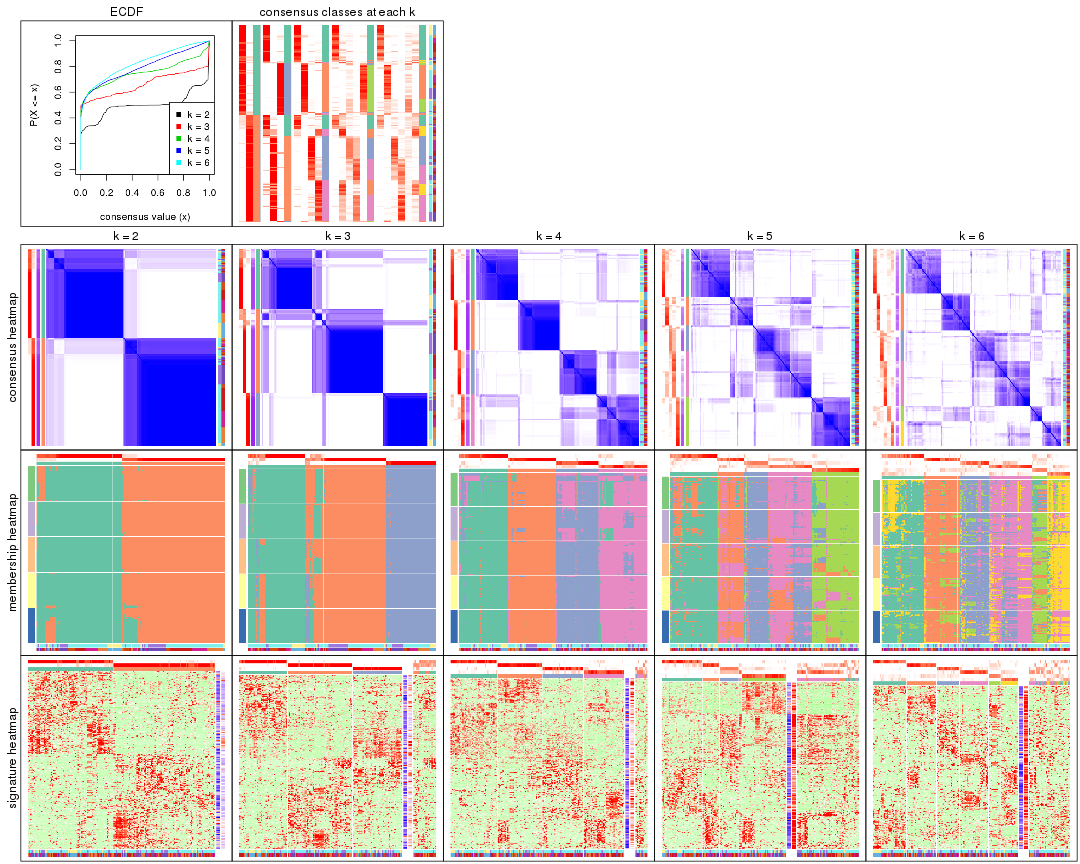
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
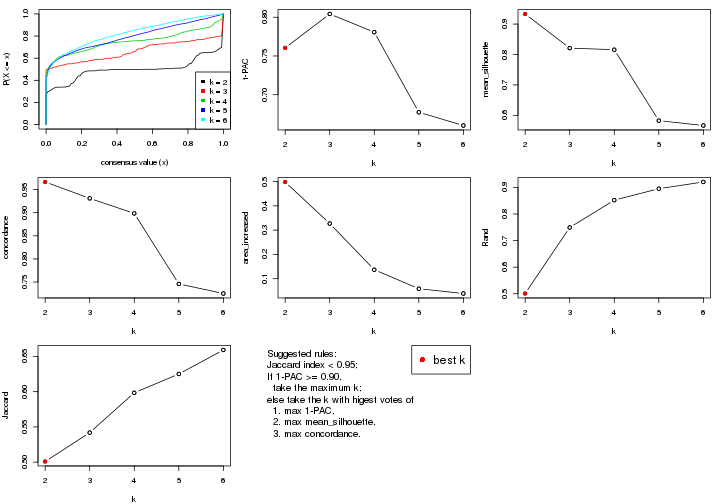
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.760 0.933 0.966 0.4983 0.501 0.501
#> 3 3 0.804 0.821 0.931 0.3271 0.749 0.542
#> 4 4 0.781 0.816 0.898 0.1370 0.852 0.598
#> 5 5 0.677 0.583 0.746 0.0588 0.895 0.625
#> 6 6 0.660 0.567 0.725 0.0390 0.920 0.659
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 2 0.6343 0.825 0.160 0.840
#> GSM340359 1 0.0000 0.955 1.000 0.000
#> GSM340361 1 0.0000 0.955 1.000 0.000
#> GSM340362 1 0.0000 0.955 1.000 0.000
#> GSM340363 2 0.1843 0.951 0.028 0.972
#> GSM340364 1 0.0000 0.955 1.000 0.000
#> GSM340365 1 0.0000 0.955 1.000 0.000
#> GSM340366 1 0.0000 0.955 1.000 0.000
#> GSM340367 1 0.0000 0.955 1.000 0.000
#> GSM340368 2 0.0000 0.971 0.000 1.000
#> GSM340369 2 0.0000 0.971 0.000 1.000
#> GSM340370 1 0.0000 0.955 1.000 0.000
#> GSM340371 1 0.5842 0.847 0.860 0.140
#> GSM340372 1 0.0000 0.955 1.000 0.000
#> GSM340373 1 0.0000 0.955 1.000 0.000
#> GSM340375 1 0.0000 0.955 1.000 0.000
#> GSM340376 1 0.7674 0.729 0.776 0.224
#> GSM340378 1 0.0000 0.955 1.000 0.000
#> GSM340243 2 0.0000 0.971 0.000 1.000
#> GSM340244 2 0.0000 0.971 0.000 1.000
#> GSM340246 2 0.0000 0.971 0.000 1.000
#> GSM340247 2 0.0000 0.971 0.000 1.000
#> GSM340248 2 0.0000 0.971 0.000 1.000
#> GSM340249 2 0.0000 0.971 0.000 1.000
#> GSM340250 1 0.0000 0.955 1.000 0.000
#> GSM340251 2 0.0000 0.971 0.000 1.000
#> GSM340252 1 0.6531 0.818 0.832 0.168
#> GSM340253 1 0.0000 0.955 1.000 0.000
#> GSM340254 1 0.5737 0.851 0.864 0.136
#> GSM340256 1 0.6801 0.807 0.820 0.180
#> GSM340258 2 0.7528 0.715 0.216 0.784
#> GSM340259 1 0.0000 0.955 1.000 0.000
#> GSM340260 1 0.0000 0.955 1.000 0.000
#> GSM340261 2 0.0000 0.971 0.000 1.000
#> GSM340262 1 0.0000 0.955 1.000 0.000
#> GSM340263 2 0.0000 0.971 0.000 1.000
#> GSM340264 1 0.0000 0.955 1.000 0.000
#> GSM340265 1 0.0000 0.955 1.000 0.000
#> GSM340266 2 0.0000 0.971 0.000 1.000
#> GSM340267 1 0.0000 0.955 1.000 0.000
#> GSM340268 2 0.0000 0.971 0.000 1.000
#> GSM340269 2 0.3114 0.928 0.056 0.944
#> GSM340270 2 0.6247 0.830 0.156 0.844
#> GSM537574 2 0.0000 0.971 0.000 1.000
#> GSM537580 1 0.5842 0.847 0.860 0.140
#> GSM537581 2 0.0000 0.971 0.000 1.000
#> GSM340272 1 0.0000 0.955 1.000 0.000
#> GSM340273 2 0.0000 0.971 0.000 1.000
#> GSM340275 2 0.0000 0.971 0.000 1.000
#> GSM340276 2 0.6048 0.838 0.148 0.852
#> GSM340277 1 0.0000 0.955 1.000 0.000
#> GSM340278 2 0.0000 0.971 0.000 1.000
#> GSM340279 2 0.0000 0.971 0.000 1.000
#> GSM340282 1 0.0000 0.955 1.000 0.000
#> GSM340284 2 0.0000 0.971 0.000 1.000
#> GSM340285 2 0.6148 0.834 0.152 0.848
#> GSM340286 1 0.0000 0.955 1.000 0.000
#> GSM340287 2 0.6623 0.812 0.172 0.828
#> GSM340288 1 0.0000 0.955 1.000 0.000
#> GSM340289 1 0.0000 0.955 1.000 0.000
#> GSM340290 2 0.0376 0.969 0.004 0.996
#> GSM340291 2 0.0000 0.971 0.000 1.000
#> GSM340293 2 0.0000 0.971 0.000 1.000
#> GSM340294 2 0.0000 0.971 0.000 1.000
#> GSM340296 2 0.0000 0.971 0.000 1.000
#> GSM340297 2 0.0000 0.971 0.000 1.000
#> GSM340298 2 0.0000 0.971 0.000 1.000
#> GSM340299 1 0.0000 0.955 1.000 0.000
#> GSM340301 1 0.0000 0.955 1.000 0.000
#> GSM340303 1 0.0000 0.955 1.000 0.000
#> GSM340304 2 0.6247 0.830 0.156 0.844
#> GSM340306 1 0.0000 0.955 1.000 0.000
#> GSM340307 2 0.0000 0.971 0.000 1.000
#> GSM340310 1 0.7139 0.767 0.804 0.196
#> GSM340314 2 0.0000 0.971 0.000 1.000
#> GSM340315 2 0.0000 0.971 0.000 1.000
#> GSM340317 2 0.0000 0.971 0.000 1.000
#> GSM340318 2 0.0000 0.971 0.000 1.000
#> GSM340319 2 0.0000 0.971 0.000 1.000
#> GSM340320 2 0.0000 0.971 0.000 1.000
#> GSM340321 2 0.0000 0.971 0.000 1.000
#> GSM340322 2 0.0000 0.971 0.000 1.000
#> GSM340324 1 0.7139 0.791 0.804 0.196
#> GSM340328 1 0.7219 0.762 0.800 0.200
#> GSM340330 2 0.0000 0.971 0.000 1.000
#> GSM340332 2 0.0000 0.971 0.000 1.000
#> GSM340333 1 0.0000 0.955 1.000 0.000
#> GSM340336 2 0.0000 0.971 0.000 1.000
#> GSM340337 2 0.0000 0.971 0.000 1.000
#> GSM340338 1 0.8713 0.609 0.708 0.292
#> GSM340339 2 0.0000 0.971 0.000 1.000
#> GSM340340 2 0.0000 0.971 0.000 1.000
#> GSM340341 2 0.0376 0.969 0.004 0.996
#> GSM340343 2 0.6438 0.821 0.164 0.836
#> GSM340344 1 0.0000 0.955 1.000 0.000
#> GSM340346 2 0.2603 0.937 0.044 0.956
#> GSM340347 2 0.0000 0.971 0.000 1.000
#> GSM340348 2 0.0000 0.971 0.000 1.000
#> GSM340349 1 0.0000 0.955 1.000 0.000
#> GSM340350 1 0.0000 0.955 1.000 0.000
#> GSM340351 1 0.0000 0.955 1.000 0.000
#> GSM340354 2 0.0000 0.971 0.000 1.000
#> GSM340356 2 0.0000 0.971 0.000 1.000
#> GSM340357 1 0.0000 0.955 1.000 0.000
#> GSM348183 1 0.0000 0.955 1.000 0.000
#> GSM348191 2 0.0376 0.969 0.004 0.996
#> GSM348193 1 0.2236 0.931 0.964 0.036
#> GSM537578 1 0.0000 0.955 1.000 0.000
#> GSM348181 2 0.6623 0.812 0.172 0.828
#> GSM348182 1 0.0000 0.955 1.000 0.000
#> GSM348184 2 0.0000 0.971 0.000 1.000
#> GSM348185 2 0.0000 0.971 0.000 1.000
#> GSM348186 1 0.6531 0.818 0.832 0.168
#> GSM348187 2 0.7453 0.758 0.212 0.788
#> GSM348188 1 0.0000 0.955 1.000 0.000
#> GSM348189 1 0.0376 0.953 0.996 0.004
#> GSM348190 1 0.0000 0.955 1.000 0.000
#> GSM348194 1 0.0000 0.955 1.000 0.000
#> GSM348195 1 0.0000 0.955 1.000 0.000
#> GSM348196 1 0.7219 0.762 0.800 0.200
#> GSM537585 1 0.0000 0.955 1.000 0.000
#> GSM537594 2 0.0000 0.971 0.000 1.000
#> GSM537596 2 0.6531 0.817 0.168 0.832
#> GSM537597 2 0.1843 0.951 0.028 0.972
#> GSM537602 1 0.7299 0.756 0.796 0.204
#> GSM340184 2 0.0376 0.969 0.004 0.996
#> GSM340185 2 0.0000 0.971 0.000 1.000
#> GSM340186 2 0.0000 0.971 0.000 1.000
#> GSM340187 2 0.0000 0.971 0.000 1.000
#> GSM340189 2 0.0000 0.971 0.000 1.000
#> GSM340190 2 0.0000 0.971 0.000 1.000
#> GSM340191 2 0.0000 0.971 0.000 1.000
#> GSM340192 1 0.2043 0.934 0.968 0.032
#> GSM340193 1 0.5059 0.866 0.888 0.112
#> GSM340194 1 0.0000 0.955 1.000 0.000
#> GSM340195 1 0.0000 0.955 1.000 0.000
#> GSM340196 2 0.0000 0.971 0.000 1.000
#> GSM340197 1 0.0000 0.955 1.000 0.000
#> GSM340198 1 0.6531 0.818 0.832 0.168
#> GSM340199 2 0.0000 0.971 0.000 1.000
#> GSM340200 2 0.0938 0.962 0.012 0.988
#> GSM340201 2 0.0000 0.971 0.000 1.000
#> GSM340202 2 0.0000 0.971 0.000 1.000
#> GSM340203 2 0.0000 0.971 0.000 1.000
#> GSM340204 1 0.0000 0.955 1.000 0.000
#> GSM340205 2 0.6623 0.814 0.172 0.828
#> GSM340206 2 0.0000 0.971 0.000 1.000
#> GSM340207 1 0.0000 0.955 1.000 0.000
#> GSM340237 2 0.0000 0.971 0.000 1.000
#> GSM340238 2 0.0000 0.971 0.000 1.000
#> GSM340239 1 0.0000 0.955 1.000 0.000
#> GSM340240 1 0.0000 0.955 1.000 0.000
#> GSM340241 2 0.0000 0.971 0.000 1.000
#> GSM340242 1 0.5842 0.847 0.860 0.140
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340359 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340361 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340362 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340363 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340364 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340365 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340366 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340367 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340368 2 0.6308 0.00209 0.492 0.508 0.000
#> GSM340369 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340370 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340371 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340372 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340373 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340375 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340376 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340378 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340243 2 0.5760 0.44569 0.328 0.672 0.000
#> GSM340244 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340246 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340247 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340248 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340249 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340250 1 0.4002 0.76470 0.840 0.000 0.160
#> GSM340251 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340252 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340253 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340254 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340256 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340258 3 0.3116 0.87318 0.000 0.108 0.892
#> GSM340259 1 0.4291 0.73544 0.820 0.000 0.180
#> GSM340260 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340261 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340262 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340263 2 0.6307 0.01761 0.488 0.512 0.000
#> GSM340264 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340265 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340266 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340267 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340268 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340269 2 0.5968 0.40357 0.000 0.636 0.364
#> GSM340270 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM537574 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM537580 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM537581 2 0.1643 0.85656 0.044 0.956 0.000
#> GSM340272 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340273 1 0.6225 0.23287 0.568 0.432 0.000
#> GSM340275 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340276 1 0.3340 0.79415 0.880 0.120 0.000
#> GSM340277 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340278 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340279 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340282 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340284 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340285 1 0.5760 0.46785 0.672 0.328 0.000
#> GSM340286 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340287 2 0.6260 0.16179 0.448 0.552 0.000
#> GSM340288 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340289 1 0.2959 0.82122 0.900 0.000 0.100
#> GSM340290 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340291 2 0.6307 0.01761 0.488 0.512 0.000
#> GSM340293 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340294 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340296 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340297 2 0.5327 0.56079 0.272 0.728 0.000
#> GSM340298 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340299 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340301 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340303 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340304 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340306 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340307 2 0.0237 0.88888 0.000 0.996 0.004
#> GSM340310 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340314 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340315 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340317 2 0.6299 0.05968 0.476 0.524 0.000
#> GSM340318 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340319 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340320 2 0.6267 0.14491 0.452 0.548 0.000
#> GSM340321 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340322 2 0.6286 0.13681 0.000 0.536 0.464
#> GSM340324 1 0.9457 0.37190 0.500 0.236 0.264
#> GSM340328 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340330 2 0.0237 0.88884 0.000 0.996 0.004
#> GSM340332 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340333 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340336 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340337 2 0.4346 0.70561 0.184 0.816 0.000
#> GSM340338 3 0.2165 0.92440 0.000 0.064 0.936
#> GSM340339 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340340 2 0.3879 0.74600 0.152 0.848 0.000
#> GSM340341 2 0.3554 0.81607 0.064 0.900 0.036
#> GSM340343 1 0.4452 0.70643 0.808 0.192 0.000
#> GSM340344 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340346 2 0.6204 0.25313 0.000 0.576 0.424
#> GSM340347 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340348 1 0.6215 0.24424 0.572 0.428 0.000
#> GSM340349 1 0.5016 0.64988 0.760 0.000 0.240
#> GSM340350 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340351 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340354 2 0.6244 0.20799 0.000 0.560 0.440
#> GSM340356 1 0.6225 0.23287 0.568 0.432 0.000
#> GSM340357 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM348183 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM348191 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM348193 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM537578 1 0.5254 0.61366 0.736 0.000 0.264
#> GSM348181 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM348182 3 0.1031 0.97062 0.024 0.000 0.976
#> GSM348184 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM348185 1 0.6111 0.32772 0.604 0.396 0.000
#> GSM348186 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM348187 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM348188 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM348189 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM348190 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM348194 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM348195 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM348196 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM537585 3 0.0237 0.98967 0.004 0.000 0.996
#> GSM537594 1 0.6225 0.23287 0.568 0.432 0.000
#> GSM537596 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM537597 1 0.2356 0.84108 0.928 0.072 0.000
#> GSM537602 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340184 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340185 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340186 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340187 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340189 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340190 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340191 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340192 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340193 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340194 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340195 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340196 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340197 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340198 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340199 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340200 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340201 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340202 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340203 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340204 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340205 1 0.0000 0.89663 1.000 0.000 0.000
#> GSM340206 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340207 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340237 2 0.6307 0.01761 0.488 0.512 0.000
#> GSM340238 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340239 3 0.1643 0.94952 0.044 0.000 0.956
#> GSM340240 3 0.0000 0.99320 0.000 0.000 1.000
#> GSM340241 2 0.0000 0.89176 0.000 1.000 0.000
#> GSM340242 3 0.0000 0.99320 0.000 0.000 1.000
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.1557 0.903 0.944 0.000 0.000 0.056
#> GSM340359 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340361 1 0.0000 0.917 1.000 0.000 0.000 0.000
#> GSM340362 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340363 1 0.1716 0.900 0.936 0.000 0.000 0.064
#> GSM340364 1 0.0000 0.917 1.000 0.000 0.000 0.000
#> GSM340365 2 0.1302 0.934 0.044 0.956 0.000 0.000
#> GSM340366 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340367 1 0.0000 0.917 1.000 0.000 0.000 0.000
#> GSM340368 4 0.0336 0.803 0.008 0.000 0.000 0.992
#> GSM340369 3 0.3975 0.757 0.000 0.000 0.760 0.240
#> GSM340370 1 0.0000 0.917 1.000 0.000 0.000 0.000
#> GSM340371 2 0.1474 0.935 0.000 0.948 0.052 0.000
#> GSM340372 1 0.0000 0.917 1.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.917 1.000 0.000 0.000 0.000
#> GSM340375 1 0.0336 0.914 0.992 0.008 0.000 0.000
#> GSM340376 1 0.4072 0.686 0.748 0.000 0.000 0.252
#> GSM340378 1 0.0188 0.917 0.996 0.000 0.000 0.004
#> GSM340243 3 0.5292 0.657 0.088 0.000 0.744 0.168
#> GSM340244 4 0.0000 0.804 0.000 0.000 0.000 1.000
#> GSM340246 3 0.0524 0.859 0.004 0.000 0.988 0.008
#> GSM340247 4 0.1716 0.797 0.000 0.000 0.064 0.936
#> GSM340248 4 0.3907 0.646 0.000 0.000 0.232 0.768
#> GSM340249 4 0.0817 0.806 0.000 0.000 0.024 0.976
#> GSM340250 1 0.5169 0.579 0.696 0.272 0.032 0.000
#> GSM340251 4 0.4543 0.479 0.000 0.000 0.324 0.676
#> GSM340252 2 0.4624 0.445 0.000 0.660 0.000 0.340
#> GSM340253 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340254 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340256 4 0.4222 0.568 0.000 0.272 0.000 0.728
#> GSM340258 2 0.5482 0.345 0.000 0.608 0.024 0.368
#> GSM340259 1 0.2814 0.814 0.868 0.132 0.000 0.000
#> GSM340260 2 0.1118 0.944 0.000 0.964 0.036 0.000
#> GSM340261 3 0.0188 0.859 0.000 0.000 0.996 0.004
#> GSM340262 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340263 4 0.1792 0.784 0.068 0.000 0.000 0.932
#> GSM340264 2 0.2926 0.902 0.056 0.896 0.048 0.000
#> GSM340265 2 0.1389 0.938 0.000 0.952 0.048 0.000
#> GSM340266 4 0.1557 0.802 0.000 0.000 0.056 0.944
#> GSM340267 2 0.4274 0.810 0.044 0.808 0.148 0.000
#> GSM340268 3 0.4040 0.748 0.000 0.000 0.752 0.248
#> GSM340269 3 0.4122 0.761 0.000 0.004 0.760 0.236
#> GSM340270 1 0.4585 0.579 0.668 0.000 0.000 0.332
#> GSM537574 4 0.3486 0.701 0.000 0.000 0.188 0.812
#> GSM537580 2 0.1474 0.935 0.000 0.948 0.052 0.000
#> GSM537581 4 0.0707 0.806 0.000 0.000 0.020 0.980
#> GSM340272 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340273 4 0.3801 0.667 0.220 0.000 0.000 0.780
#> GSM340275 4 0.0336 0.806 0.000 0.000 0.008 0.992
#> GSM340276 1 0.4336 0.826 0.812 0.000 0.128 0.060
#> GSM340277 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340278 3 0.0336 0.859 0.000 0.000 0.992 0.008
#> GSM340279 3 0.0000 0.859 0.000 0.000 1.000 0.000
#> GSM340282 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340284 4 0.4454 0.516 0.000 0.000 0.308 0.692
#> GSM340285 1 0.6536 0.441 0.560 0.000 0.352 0.088
#> GSM340286 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340287 3 0.0817 0.843 0.024 0.000 0.976 0.000
#> GSM340288 1 0.0000 0.917 1.000 0.000 0.000 0.000
#> GSM340289 1 0.0336 0.915 0.992 0.008 0.000 0.000
#> GSM340290 3 0.0000 0.859 0.000 0.000 1.000 0.000
#> GSM340291 4 0.4103 0.656 0.256 0.000 0.000 0.744
#> GSM340293 3 0.1867 0.849 0.000 0.000 0.928 0.072
#> GSM340294 3 0.0000 0.859 0.000 0.000 1.000 0.000
#> GSM340296 3 0.0188 0.856 0.004 0.000 0.996 0.000
#> GSM340297 3 0.4164 0.740 0.000 0.000 0.736 0.264
#> GSM340298 3 0.0000 0.859 0.000 0.000 1.000 0.000
#> GSM340299 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340301 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340303 2 0.1389 0.938 0.000 0.952 0.048 0.000
#> GSM340304 1 0.3239 0.881 0.880 0.000 0.052 0.068
#> GSM340306 1 0.0469 0.916 0.988 0.000 0.000 0.012
#> GSM340307 3 0.0000 0.859 0.000 0.000 1.000 0.000
#> GSM340310 1 0.1557 0.903 0.944 0.000 0.000 0.056
#> GSM340314 3 0.3649 0.785 0.000 0.000 0.796 0.204
#> GSM340315 3 0.1474 0.820 0.052 0.000 0.948 0.000
#> GSM340317 4 0.0000 0.804 0.000 0.000 0.000 1.000
#> GSM340318 4 0.1792 0.795 0.000 0.000 0.068 0.932
#> GSM340319 4 0.1637 0.799 0.000 0.000 0.060 0.940
#> GSM340320 4 0.7082 0.279 0.308 0.000 0.152 0.540
#> GSM340321 4 0.4522 0.496 0.000 0.000 0.320 0.680
#> GSM340322 4 0.2500 0.790 0.000 0.044 0.040 0.916
#> GSM340324 4 0.4387 0.675 0.200 0.024 0.000 0.776
#> GSM340328 1 0.1557 0.903 0.944 0.000 0.000 0.056
#> GSM340330 3 0.1716 0.850 0.000 0.000 0.936 0.064
#> GSM340332 3 0.4040 0.748 0.000 0.000 0.752 0.248
#> GSM340333 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340336 4 0.1716 0.797 0.000 0.000 0.064 0.936
#> GSM340337 4 0.0000 0.804 0.000 0.000 0.000 1.000
#> GSM340338 3 0.4647 0.575 0.000 0.288 0.704 0.008
#> GSM340339 3 0.4454 0.669 0.000 0.000 0.692 0.308
#> GSM340340 4 0.0188 0.805 0.000 0.000 0.004 0.996
#> GSM340341 4 0.1631 0.796 0.008 0.016 0.020 0.956
#> GSM340343 1 0.3726 0.756 0.788 0.000 0.000 0.212
#> GSM340344 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340346 3 0.0000 0.859 0.000 0.000 1.000 0.000
#> GSM340347 4 0.1118 0.805 0.000 0.000 0.036 0.964
#> GSM340348 4 0.3306 0.733 0.156 0.000 0.004 0.840
#> GSM340349 1 0.4040 0.705 0.752 0.000 0.248 0.000
#> GSM340350 1 0.1792 0.882 0.932 0.000 0.068 0.000
#> GSM340351 1 0.0000 0.917 1.000 0.000 0.000 0.000
#> GSM340354 3 0.0000 0.859 0.000 0.000 1.000 0.000
#> GSM340356 4 0.3726 0.676 0.212 0.000 0.000 0.788
#> GSM340357 1 0.0000 0.917 1.000 0.000 0.000 0.000
#> GSM348183 1 0.0000 0.917 1.000 0.000 0.000 0.000
#> GSM348191 3 0.0000 0.859 0.000 0.000 1.000 0.000
#> GSM348193 1 0.1716 0.900 0.936 0.000 0.000 0.064
#> GSM537578 1 0.4277 0.660 0.720 0.000 0.280 0.000
#> GSM348181 1 0.0336 0.917 0.992 0.000 0.000 0.008
#> GSM348182 2 0.0707 0.950 0.020 0.980 0.000 0.000
#> GSM348184 3 0.4040 0.748 0.000 0.000 0.752 0.248
#> GSM348185 4 0.3942 0.645 0.236 0.000 0.000 0.764
#> GSM348186 2 0.0921 0.940 0.000 0.972 0.000 0.028
#> GSM348187 1 0.0336 0.917 0.992 0.000 0.000 0.008
#> GSM348188 1 0.2179 0.898 0.924 0.012 0.000 0.064
#> GSM348189 2 0.1474 0.935 0.000 0.948 0.052 0.000
#> GSM348190 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM348194 1 0.0000 0.917 1.000 0.000 0.000 0.000
#> GSM348195 1 0.0000 0.917 1.000 0.000 0.000 0.000
#> GSM348196 1 0.1557 0.903 0.944 0.000 0.000 0.056
#> GSM537585 2 0.1824 0.921 0.060 0.936 0.004 0.000
#> GSM537594 4 0.3801 0.667 0.220 0.000 0.000 0.780
#> GSM537596 1 0.0817 0.914 0.976 0.000 0.000 0.024
#> GSM537597 4 0.4776 0.376 0.376 0.000 0.000 0.624
#> GSM537602 1 0.2300 0.898 0.920 0.000 0.016 0.064
#> GSM340184 3 0.0000 0.859 0.000 0.000 1.000 0.000
#> GSM340185 4 0.4250 0.607 0.000 0.000 0.276 0.724
#> GSM340186 4 0.4564 0.472 0.000 0.000 0.328 0.672
#> GSM340187 4 0.4661 0.424 0.000 0.000 0.348 0.652
#> GSM340189 3 0.3975 0.757 0.000 0.000 0.760 0.240
#> GSM340190 3 0.1940 0.848 0.000 0.000 0.924 0.076
#> GSM340191 3 0.4008 0.753 0.000 0.000 0.756 0.244
#> GSM340192 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340193 1 0.1557 0.903 0.944 0.000 0.000 0.056
#> GSM340194 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340195 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340196 4 0.1716 0.797 0.000 0.000 0.064 0.936
#> GSM340197 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340198 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340199 3 0.1118 0.855 0.000 0.000 0.964 0.036
#> GSM340200 3 0.4250 0.492 0.000 0.000 0.724 0.276
#> GSM340201 4 0.1118 0.805 0.000 0.000 0.036 0.964
#> GSM340202 4 0.1716 0.797 0.000 0.000 0.064 0.936
#> GSM340203 3 0.3907 0.764 0.000 0.000 0.768 0.232
#> GSM340204 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340205 1 0.0000 0.917 1.000 0.000 0.000 0.000
#> GSM340206 3 0.0000 0.859 0.000 0.000 1.000 0.000
#> GSM340207 2 0.0188 0.958 0.000 0.996 0.004 0.000
#> GSM340237 4 0.5033 0.708 0.168 0.000 0.072 0.760
#> GSM340238 3 0.2868 0.823 0.000 0.000 0.864 0.136
#> GSM340239 2 0.0592 0.951 0.016 0.984 0.000 0.000
#> GSM340240 2 0.0000 0.959 0.000 1.000 0.000 0.000
#> GSM340241 4 0.3400 0.706 0.000 0.000 0.180 0.820
#> GSM340242 2 0.1474 0.935 0.000 0.948 0.052 0.000
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.4167 0.7289 0.724 0.252 0.024 0.000 0.000
#> GSM340359 5 0.2179 0.8236 0.000 0.000 0.112 0.000 0.888
#> GSM340361 1 0.0451 0.8097 0.988 0.008 0.004 0.000 0.000
#> GSM340362 5 0.0290 0.8388 0.000 0.000 0.008 0.000 0.992
#> GSM340363 1 0.4808 0.5746 0.576 0.400 0.024 0.000 0.000
#> GSM340364 1 0.0566 0.8089 0.984 0.012 0.004 0.000 0.000
#> GSM340365 5 0.3074 0.7370 0.196 0.000 0.000 0.000 0.804
#> GSM340366 5 0.1831 0.8395 0.000 0.004 0.076 0.000 0.920
#> GSM340367 1 0.0324 0.8080 0.992 0.004 0.004 0.000 0.000
#> GSM340368 2 0.3010 0.6368 0.004 0.824 0.000 0.172 0.000
#> GSM340369 4 0.2338 0.4448 0.000 0.004 0.112 0.884 0.000
#> GSM340370 1 0.0162 0.8097 0.996 0.004 0.000 0.000 0.000
#> GSM340371 5 0.4029 0.6825 0.000 0.004 0.316 0.000 0.680
#> GSM340372 1 0.0000 0.8089 1.000 0.000 0.000 0.000 0.000
#> GSM340373 1 0.0324 0.8080 0.992 0.004 0.004 0.000 0.000
#> GSM340375 1 0.2237 0.7778 0.904 0.008 0.084 0.000 0.004
#> GSM340376 2 0.4383 -0.1925 0.424 0.572 0.004 0.000 0.000
#> GSM340378 1 0.1357 0.8118 0.948 0.048 0.004 0.000 0.000
#> GSM340243 2 0.7345 -0.1019 0.048 0.428 0.348 0.176 0.000
#> GSM340244 2 0.2011 0.6783 0.000 0.908 0.004 0.088 0.000
#> GSM340246 3 0.5681 0.6998 0.004 0.072 0.532 0.392 0.000
#> GSM340247 4 0.4030 0.3854 0.000 0.352 0.000 0.648 0.000
#> GSM340248 2 0.5334 -0.0838 0.000 0.512 0.052 0.436 0.000
#> GSM340249 2 0.4437 -0.0254 0.000 0.532 0.004 0.464 0.000
#> GSM340250 1 0.6584 0.5339 0.636 0.064 0.120 0.008 0.172
#> GSM340251 4 0.3790 0.4692 0.000 0.272 0.004 0.724 0.000
#> GSM340252 5 0.5491 0.6814 0.000 0.092 0.120 0.064 0.724
#> GSM340253 5 0.2179 0.8236 0.000 0.000 0.112 0.000 0.888
#> GSM340254 5 0.2280 0.8207 0.000 0.000 0.120 0.000 0.880
#> GSM340256 2 0.6027 0.3098 0.000 0.572 0.116 0.008 0.304
#> GSM340258 5 0.7345 0.0429 0.000 0.076 0.120 0.384 0.420
#> GSM340259 1 0.1830 0.7932 0.932 0.004 0.012 0.000 0.052
#> GSM340260 5 0.4311 0.7189 0.020 0.004 0.264 0.000 0.712
#> GSM340261 3 0.4074 0.7861 0.000 0.000 0.636 0.364 0.000
#> GSM340262 5 0.0510 0.8400 0.000 0.000 0.016 0.000 0.984
#> GSM340263 2 0.1717 0.6971 0.008 0.936 0.004 0.052 0.000
#> GSM340264 5 0.6214 0.5842 0.188 0.004 0.240 0.000 0.568
#> GSM340265 5 0.4365 0.6764 0.012 0.004 0.308 0.000 0.676
#> GSM340266 4 0.4419 0.4098 0.000 0.312 0.020 0.668 0.000
#> GSM340267 3 0.5652 -0.3811 0.064 0.004 0.468 0.000 0.464
#> GSM340268 4 0.2516 0.4089 0.000 0.000 0.140 0.860 0.000
#> GSM340269 4 0.4185 0.3224 0.000 0.008 0.216 0.752 0.024
#> GSM340270 1 0.6378 0.3706 0.512 0.356 0.016 0.116 0.000
#> GSM537574 4 0.5360 0.3126 0.000 0.384 0.060 0.556 0.000
#> GSM537580 5 0.4029 0.6825 0.000 0.004 0.316 0.000 0.680
#> GSM537581 4 0.4331 0.2973 0.004 0.400 0.000 0.596 0.000
#> GSM340272 5 0.1872 0.8319 0.020 0.000 0.052 0.000 0.928
#> GSM340273 2 0.0865 0.7040 0.024 0.972 0.004 0.000 0.000
#> GSM340275 2 0.3838 0.4588 0.000 0.716 0.004 0.280 0.000
#> GSM340276 1 0.6590 0.5926 0.552 0.248 0.180 0.020 0.000
#> GSM340277 5 0.2280 0.8256 0.000 0.000 0.120 0.000 0.880
#> GSM340278 3 0.4470 0.7538 0.000 0.012 0.616 0.372 0.000
#> GSM340279 3 0.4060 0.7960 0.000 0.000 0.640 0.360 0.000
#> GSM340282 5 0.1965 0.8333 0.024 0.000 0.052 0.000 0.924
#> GSM340284 4 0.4086 0.5065 0.000 0.240 0.024 0.736 0.000
#> GSM340285 1 0.8016 0.3490 0.416 0.176 0.280 0.128 0.000
#> GSM340286 5 0.1410 0.8363 0.000 0.000 0.060 0.000 0.940
#> GSM340287 3 0.4201 0.8016 0.008 0.000 0.664 0.328 0.000
#> GSM340288 1 0.1106 0.8100 0.964 0.012 0.024 0.000 0.000
#> GSM340289 1 0.0912 0.8066 0.972 0.012 0.016 0.000 0.000
#> GSM340290 3 0.4047 0.8010 0.000 0.004 0.676 0.320 0.000
#> GSM340291 2 0.3004 0.6707 0.108 0.864 0.020 0.008 0.000
#> GSM340293 4 0.3814 0.0814 0.000 0.004 0.276 0.720 0.000
#> GSM340294 3 0.3983 0.8016 0.000 0.000 0.660 0.340 0.000
#> GSM340296 3 0.4306 0.7991 0.000 0.012 0.660 0.328 0.000
#> GSM340297 4 0.4240 0.3010 0.000 0.036 0.228 0.736 0.000
#> GSM340298 3 0.4135 0.8010 0.000 0.004 0.656 0.340 0.000
#> GSM340299 5 0.0290 0.8394 0.000 0.000 0.008 0.000 0.992
#> GSM340301 5 0.0609 0.8375 0.000 0.000 0.020 0.000 0.980
#> GSM340303 5 0.4134 0.7021 0.008 0.004 0.284 0.000 0.704
#> GSM340304 1 0.5793 0.3944 0.464 0.456 0.076 0.004 0.000
#> GSM340306 1 0.4037 0.7793 0.812 0.084 0.092 0.012 0.000
#> GSM340307 3 0.4166 0.7907 0.000 0.004 0.648 0.348 0.000
#> GSM340310 1 0.3961 0.7331 0.736 0.248 0.016 0.000 0.000
#> GSM340314 4 0.3661 0.1385 0.000 0.000 0.276 0.724 0.000
#> GSM340315 3 0.4852 0.7942 0.016 0.016 0.644 0.324 0.000
#> GSM340317 2 0.1205 0.7014 0.000 0.956 0.004 0.040 0.000
#> GSM340318 4 0.4030 0.3854 0.000 0.352 0.000 0.648 0.000
#> GSM340319 4 0.4030 0.3854 0.000 0.352 0.000 0.648 0.000
#> GSM340320 4 0.7881 -0.1105 0.184 0.324 0.096 0.396 0.000
#> GSM340321 4 0.4938 0.4444 0.000 0.312 0.048 0.640 0.000
#> GSM340322 4 0.6059 0.3763 0.000 0.224 0.116 0.632 0.028
#> GSM340324 2 0.3811 0.6447 0.028 0.836 0.104 0.008 0.024
#> GSM340328 1 0.4141 0.7315 0.728 0.248 0.024 0.000 0.000
#> GSM340330 4 0.4430 -0.4810 0.000 0.000 0.456 0.540 0.004
#> GSM340332 4 0.2389 0.4417 0.000 0.004 0.116 0.880 0.000
#> GSM340333 5 0.2179 0.8236 0.000 0.000 0.112 0.000 0.888
#> GSM340336 4 0.4030 0.3854 0.000 0.352 0.000 0.648 0.000
#> GSM340337 2 0.5026 0.0552 0.004 0.516 0.016 0.460 0.004
#> GSM340338 3 0.6793 0.3134 0.000 0.000 0.376 0.292 0.332
#> GSM340339 4 0.2959 0.4913 0.000 0.036 0.100 0.864 0.000
#> GSM340340 2 0.4118 0.3640 0.000 0.660 0.004 0.336 0.000
#> GSM340341 2 0.4012 0.6377 0.008 0.796 0.032 0.160 0.004
#> GSM340343 1 0.6615 0.6166 0.592 0.244 0.080 0.084 0.000
#> GSM340344 5 0.0880 0.8393 0.000 0.000 0.032 0.000 0.968
#> GSM340346 3 0.4029 0.8015 0.000 0.000 0.680 0.316 0.004
#> GSM340347 4 0.4313 0.3719 0.000 0.356 0.008 0.636 0.000
#> GSM340348 2 0.2721 0.6912 0.012 0.892 0.028 0.068 0.000
#> GSM340349 3 0.6038 -0.1626 0.392 0.012 0.520 0.004 0.072
#> GSM340350 1 0.2149 0.8014 0.916 0.036 0.048 0.000 0.000
#> GSM340351 1 0.0290 0.8086 0.992 0.000 0.008 0.000 0.000
#> GSM340354 3 0.4182 0.7933 0.000 0.000 0.644 0.352 0.004
#> GSM340356 2 0.1124 0.7033 0.036 0.960 0.000 0.004 0.000
#> GSM340357 1 0.0566 0.8079 0.984 0.004 0.012 0.000 0.000
#> GSM348183 1 0.0771 0.8118 0.976 0.020 0.004 0.000 0.000
#> GSM348191 3 0.3966 0.8023 0.000 0.000 0.664 0.336 0.000
#> GSM348193 1 0.4418 0.6604 0.652 0.332 0.016 0.000 0.000
#> GSM537578 1 0.5306 0.4285 0.596 0.004 0.360 0.024 0.016
#> GSM348181 1 0.2388 0.8034 0.900 0.072 0.028 0.000 0.000
#> GSM348182 5 0.5435 0.6749 0.204 0.004 0.124 0.000 0.668
#> GSM348184 4 0.1956 0.4787 0.000 0.008 0.076 0.916 0.000
#> GSM348185 2 0.0898 0.7031 0.020 0.972 0.008 0.000 0.000
#> GSM348186 5 0.3878 0.7804 0.000 0.020 0.120 0.040 0.820
#> GSM348187 1 0.3292 0.7936 0.844 0.120 0.032 0.004 0.000
#> GSM348188 1 0.5052 0.7392 0.724 0.200 0.024 0.004 0.048
#> GSM348189 5 0.4029 0.6825 0.000 0.004 0.316 0.000 0.680
#> GSM348190 5 0.1251 0.8368 0.036 0.000 0.008 0.000 0.956
#> GSM348194 1 0.2171 0.8061 0.912 0.064 0.024 0.000 0.000
#> GSM348195 1 0.0290 0.8086 0.992 0.000 0.008 0.000 0.000
#> GSM348196 1 0.4295 0.7297 0.724 0.248 0.024 0.004 0.000
#> GSM537585 5 0.6250 0.5203 0.292 0.004 0.160 0.000 0.544
#> GSM537594 2 0.0898 0.7048 0.020 0.972 0.000 0.008 0.000
#> GSM537596 1 0.4575 0.7589 0.744 0.184 0.068 0.004 0.000
#> GSM537597 2 0.3567 0.6184 0.112 0.832 0.052 0.004 0.000
#> GSM537602 1 0.5376 0.6099 0.584 0.356 0.056 0.004 0.000
#> GSM340184 3 0.3861 0.7741 0.000 0.004 0.712 0.284 0.000
#> GSM340185 4 0.5659 0.4111 0.000 0.280 0.116 0.604 0.000
#> GSM340186 4 0.4718 0.4863 0.000 0.180 0.092 0.728 0.000
#> GSM340187 4 0.3491 0.5015 0.000 0.228 0.004 0.768 0.000
#> GSM340189 4 0.2329 0.4279 0.000 0.000 0.124 0.876 0.000
#> GSM340190 4 0.3857 -0.0343 0.000 0.000 0.312 0.688 0.000
#> GSM340191 4 0.3086 0.3577 0.000 0.004 0.180 0.816 0.000
#> GSM340192 5 0.2329 0.8212 0.000 0.000 0.124 0.000 0.876
#> GSM340193 1 0.3722 0.7715 0.796 0.176 0.024 0.004 0.000
#> GSM340194 5 0.0880 0.8393 0.000 0.000 0.032 0.000 0.968
#> GSM340195 5 0.0451 0.8391 0.004 0.000 0.008 0.000 0.988
#> GSM340196 4 0.4030 0.3854 0.000 0.352 0.000 0.648 0.000
#> GSM340197 5 0.1117 0.8371 0.016 0.000 0.020 0.000 0.964
#> GSM340198 5 0.2563 0.8172 0.000 0.000 0.120 0.008 0.872
#> GSM340199 4 0.4449 -0.5393 0.000 0.004 0.484 0.512 0.000
#> GSM340200 3 0.5864 0.3795 0.000 0.128 0.572 0.300 0.000
#> GSM340201 4 0.4264 0.3439 0.000 0.376 0.004 0.620 0.000
#> GSM340202 4 0.4166 0.3862 0.000 0.348 0.004 0.648 0.000
#> GSM340203 4 0.2732 0.3710 0.000 0.000 0.160 0.840 0.000
#> GSM340204 5 0.2179 0.8236 0.000 0.000 0.112 0.000 0.888
#> GSM340205 1 0.3197 0.7764 0.836 0.140 0.024 0.000 0.000
#> GSM340206 3 0.4030 0.7969 0.000 0.000 0.648 0.352 0.000
#> GSM340207 5 0.1851 0.8263 0.000 0.000 0.088 0.000 0.912
#> GSM340237 2 0.3674 0.6590 0.064 0.844 0.068 0.024 0.000
#> GSM340238 4 0.3636 0.1348 0.000 0.000 0.272 0.728 0.000
#> GSM340239 5 0.2561 0.8093 0.096 0.000 0.020 0.000 0.884
#> GSM340240 5 0.1908 0.8339 0.000 0.000 0.092 0.000 0.908
#> GSM340241 4 0.6743 0.3147 0.000 0.264 0.128 0.560 0.048
#> GSM340242 5 0.4029 0.6825 0.000 0.004 0.316 0.000 0.680
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.4078 0.58939 0.656 0.000 0.000 0.000 0.024 0.320
#> GSM340359 2 0.0291 0.75609 0.000 0.992 0.000 0.000 0.004 0.004
#> GSM340361 1 0.0862 0.73476 0.972 0.004 0.000 0.000 0.008 0.016
#> GSM340362 2 0.2933 0.72768 0.000 0.796 0.000 0.000 0.200 0.004
#> GSM340363 6 0.4338 -0.36780 0.484 0.000 0.000 0.000 0.020 0.496
#> GSM340364 1 0.2008 0.72955 0.920 0.004 0.004 0.000 0.032 0.040
#> GSM340365 2 0.5372 0.43381 0.220 0.604 0.000 0.000 0.172 0.004
#> GSM340366 2 0.4121 0.66853 0.004 0.716 0.020 0.000 0.248 0.012
#> GSM340367 1 0.1320 0.73212 0.948 0.000 0.000 0.000 0.016 0.036
#> GSM340368 6 0.3706 0.51317 0.000 0.000 0.000 0.380 0.000 0.620
#> GSM340369 4 0.3101 0.61879 0.000 0.000 0.244 0.756 0.000 0.000
#> GSM340370 1 0.0547 0.73676 0.980 0.000 0.000 0.000 0.000 0.020
#> GSM340371 5 0.5169 0.71730 0.000 0.212 0.128 0.000 0.648 0.012
#> GSM340372 1 0.0260 0.73502 0.992 0.000 0.000 0.000 0.008 0.000
#> GSM340373 1 0.0922 0.73497 0.968 0.000 0.004 0.000 0.024 0.004
#> GSM340375 1 0.3504 0.60122 0.776 0.000 0.004 0.000 0.196 0.024
#> GSM340376 6 0.4189 0.09687 0.376 0.000 0.000 0.020 0.000 0.604
#> GSM340378 1 0.1958 0.72809 0.896 0.000 0.000 0.000 0.004 0.100
#> GSM340243 3 0.6747 0.00623 0.036 0.000 0.408 0.016 0.156 0.384
#> GSM340244 6 0.4203 0.65814 0.000 0.000 0.000 0.216 0.068 0.716
#> GSM340246 3 0.6128 0.58084 0.004 0.000 0.620 0.120 0.116 0.140
#> GSM340247 4 0.1204 0.67581 0.000 0.000 0.000 0.944 0.000 0.056
#> GSM340248 4 0.5914 0.20775 0.000 0.000 0.112 0.500 0.028 0.360
#> GSM340249 4 0.4795 0.14915 0.000 0.000 0.000 0.604 0.072 0.324
#> GSM340250 1 0.7735 0.29174 0.504 0.132 0.072 0.012 0.192 0.088
#> GSM340251 4 0.1515 0.69781 0.000 0.000 0.028 0.944 0.008 0.020
#> GSM340252 2 0.2245 0.69061 0.000 0.908 0.000 0.040 0.016 0.036
#> GSM340253 2 0.0291 0.75609 0.000 0.992 0.000 0.000 0.004 0.004
#> GSM340254 2 0.0405 0.75174 0.000 0.988 0.000 0.000 0.004 0.008
#> GSM340256 2 0.4693 0.05521 0.000 0.540 0.000 0.020 0.016 0.424
#> GSM340258 2 0.4424 0.41811 0.000 0.708 0.000 0.232 0.024 0.036
#> GSM340259 1 0.4797 0.68097 0.756 0.068 0.016 0.000 0.100 0.060
#> GSM340260 5 0.5958 0.65400 0.040 0.260 0.088 0.000 0.596 0.016
#> GSM340261 3 0.1367 0.70831 0.000 0.000 0.944 0.044 0.012 0.000
#> GSM340262 2 0.3052 0.72648 0.000 0.780 0.000 0.000 0.216 0.004
#> GSM340263 6 0.4298 0.67919 0.004 0.000 0.000 0.176 0.088 0.732
#> GSM340264 5 0.5505 0.65223 0.184 0.144 0.032 0.000 0.640 0.000
#> GSM340265 5 0.5286 0.71049 0.024 0.204 0.120 0.000 0.652 0.000
#> GSM340266 4 0.2818 0.67208 0.000 0.000 0.028 0.872 0.024 0.076
#> GSM340267 5 0.5571 0.64272 0.056 0.088 0.200 0.004 0.652 0.000
#> GSM340268 4 0.3547 0.55869 0.000 0.000 0.300 0.696 0.004 0.000
#> GSM340269 4 0.7035 0.11294 0.004 0.064 0.388 0.416 0.100 0.028
#> GSM340270 1 0.6785 0.22877 0.436 0.000 0.008 0.080 0.112 0.364
#> GSM537574 4 0.6217 0.39873 0.000 0.028 0.104 0.564 0.028 0.276
#> GSM537580 5 0.5157 0.71647 0.000 0.216 0.124 0.000 0.648 0.012
#> GSM537581 4 0.2772 0.55417 0.004 0.000 0.000 0.816 0.000 0.180
#> GSM340272 2 0.5150 0.49743 0.048 0.600 0.016 0.000 0.328 0.008
#> GSM340273 6 0.2450 0.71228 0.016 0.000 0.000 0.116 0.000 0.868
#> GSM340275 6 0.5353 0.27621 0.000 0.000 0.004 0.440 0.092 0.464
#> GSM340276 1 0.7768 0.33185 0.352 0.000 0.228 0.008 0.228 0.184
#> GSM340277 2 0.2162 0.74575 0.000 0.896 0.004 0.000 0.088 0.012
#> GSM340278 3 0.3428 0.65405 0.000 0.000 0.820 0.032 0.128 0.020
#> GSM340279 3 0.3006 0.70139 0.000 0.000 0.844 0.064 0.092 0.000
#> GSM340282 2 0.4707 0.62041 0.020 0.660 0.016 0.000 0.288 0.016
#> GSM340284 4 0.2806 0.69680 0.000 0.000 0.060 0.872 0.012 0.056
#> GSM340285 3 0.7822 -0.12616 0.268 0.000 0.380 0.024 0.180 0.148
#> GSM340286 2 0.1814 0.76895 0.000 0.900 0.000 0.000 0.100 0.000
#> GSM340287 3 0.1542 0.70365 0.000 0.000 0.936 0.008 0.052 0.004
#> GSM340288 1 0.2209 0.72745 0.904 0.000 0.004 0.000 0.052 0.040
#> GSM340289 1 0.2392 0.72296 0.896 0.000 0.008 0.000 0.048 0.048
#> GSM340290 3 0.2243 0.68908 0.000 0.000 0.880 0.004 0.112 0.004
#> GSM340291 6 0.5367 0.66898 0.052 0.000 0.008 0.144 0.104 0.692
#> GSM340293 4 0.4072 0.27057 0.000 0.000 0.448 0.544 0.008 0.000
#> GSM340294 3 0.1682 0.70821 0.000 0.000 0.928 0.020 0.052 0.000
#> GSM340296 3 0.3226 0.67888 0.000 0.000 0.836 0.020 0.116 0.028
#> GSM340297 4 0.5776 0.36777 0.008 0.000 0.368 0.528 0.044 0.052
#> GSM340298 3 0.1644 0.70435 0.000 0.000 0.932 0.012 0.052 0.004
#> GSM340299 2 0.2632 0.74944 0.000 0.832 0.000 0.000 0.164 0.004
#> GSM340301 2 0.3023 0.71841 0.000 0.784 0.000 0.000 0.212 0.004
#> GSM340303 5 0.5322 0.69404 0.024 0.236 0.104 0.000 0.636 0.000
#> GSM340304 6 0.7102 -0.06949 0.288 0.000 0.104 0.000 0.188 0.420
#> GSM340306 1 0.5059 0.63287 0.652 0.000 0.004 0.000 0.168 0.176
#> GSM340307 3 0.4688 0.66886 0.000 0.016 0.740 0.080 0.148 0.016
#> GSM340310 1 0.3672 0.60336 0.688 0.000 0.000 0.000 0.008 0.304
#> GSM340314 3 0.4181 -0.17328 0.000 0.000 0.512 0.476 0.012 0.000
#> GSM340315 3 0.4218 0.68559 0.032 0.000 0.800 0.032 0.092 0.044
#> GSM340317 6 0.3588 0.69166 0.000 0.000 0.000 0.180 0.044 0.776
#> GSM340318 4 0.1204 0.67581 0.000 0.000 0.000 0.944 0.000 0.056
#> GSM340319 4 0.1204 0.67581 0.000 0.000 0.000 0.944 0.000 0.056
#> GSM340320 4 0.7660 0.27420 0.084 0.000 0.152 0.464 0.068 0.232
#> GSM340321 4 0.5332 0.54715 0.000 0.000 0.144 0.628 0.012 0.216
#> GSM340322 4 0.4254 0.62481 0.000 0.112 0.000 0.776 0.044 0.068
#> GSM340324 6 0.4037 0.64325 0.012 0.168 0.000 0.048 0.004 0.768
#> GSM340328 1 0.4094 0.59214 0.652 0.000 0.000 0.000 0.024 0.324
#> GSM340330 3 0.5016 0.50297 0.000 0.004 0.636 0.268 0.088 0.004
#> GSM340332 4 0.3221 0.60048 0.000 0.000 0.264 0.736 0.000 0.000
#> GSM340333 2 0.0405 0.75722 0.000 0.988 0.000 0.000 0.008 0.004
#> GSM340336 4 0.1204 0.67581 0.000 0.000 0.000 0.944 0.000 0.056
#> GSM340337 4 0.5354 0.12392 0.004 0.036 0.000 0.584 0.044 0.332
#> GSM340338 3 0.7468 -0.11349 0.000 0.184 0.340 0.136 0.336 0.004
#> GSM340339 4 0.3329 0.63488 0.000 0.000 0.220 0.768 0.008 0.004
#> GSM340340 6 0.4808 0.25383 0.000 0.000 0.000 0.472 0.052 0.476
#> GSM340341 6 0.5875 0.55248 0.000 0.016 0.012 0.260 0.132 0.580
#> GSM340343 1 0.7766 0.43639 0.460 0.000 0.108 0.060 0.196 0.176
#> GSM340344 2 0.1663 0.76928 0.000 0.912 0.000 0.000 0.088 0.000
#> GSM340346 3 0.3390 0.67914 0.000 0.000 0.808 0.032 0.152 0.008
#> GSM340347 4 0.1644 0.66421 0.000 0.000 0.000 0.920 0.004 0.076
#> GSM340348 6 0.5108 0.65490 0.016 0.000 0.016 0.176 0.096 0.696
#> GSM340349 5 0.7027 0.07461 0.240 0.004 0.272 0.000 0.420 0.064
#> GSM340350 1 0.5172 0.64724 0.692 0.000 0.068 0.000 0.168 0.072
#> GSM340351 1 0.1268 0.73018 0.952 0.000 0.004 0.000 0.036 0.008
#> GSM340354 3 0.3977 0.66948 0.000 0.008 0.776 0.060 0.152 0.004
#> GSM340356 6 0.2790 0.70873 0.024 0.000 0.000 0.132 0.000 0.844
#> GSM340357 1 0.2085 0.73178 0.912 0.000 0.008 0.000 0.056 0.024
#> GSM348183 1 0.1285 0.73757 0.944 0.000 0.000 0.000 0.004 0.052
#> GSM348191 3 0.1584 0.70321 0.000 0.000 0.928 0.008 0.064 0.000
#> GSM348193 1 0.3923 0.45979 0.580 0.000 0.000 0.000 0.004 0.416
#> GSM537578 1 0.6424 0.20982 0.460 0.004 0.356 0.000 0.140 0.040
#> GSM348181 1 0.5162 0.64065 0.692 0.000 0.044 0.000 0.148 0.116
#> GSM348182 5 0.6250 0.46871 0.244 0.272 0.000 0.000 0.468 0.016
#> GSM348184 4 0.3394 0.62223 0.000 0.000 0.200 0.776 0.024 0.000
#> GSM348185 6 0.2312 0.71256 0.012 0.000 0.000 0.112 0.000 0.876
#> GSM348186 2 0.0951 0.74167 0.000 0.968 0.000 0.004 0.020 0.008
#> GSM348187 1 0.4489 0.67173 0.712 0.000 0.012 0.000 0.068 0.208
#> GSM348188 1 0.5601 0.57143 0.600 0.104 0.000 0.000 0.032 0.264
#> GSM348189 5 0.5157 0.71647 0.000 0.216 0.124 0.000 0.648 0.012
#> GSM348190 2 0.3263 0.73645 0.020 0.800 0.000 0.000 0.176 0.004
#> GSM348194 1 0.2954 0.72314 0.852 0.000 0.004 0.000 0.048 0.096
#> GSM348195 1 0.1340 0.72994 0.948 0.000 0.004 0.000 0.040 0.008
#> GSM348196 1 0.4306 0.60147 0.656 0.000 0.004 0.000 0.032 0.308
#> GSM537585 5 0.5548 0.53019 0.316 0.140 0.004 0.000 0.540 0.000
#> GSM537594 6 0.2799 0.71315 0.012 0.000 0.000 0.124 0.012 0.852
#> GSM537596 1 0.6604 0.58441 0.544 0.000 0.108 0.004 0.120 0.224
#> GSM537597 6 0.3804 0.54173 0.152 0.004 0.000 0.020 0.032 0.792
#> GSM537602 1 0.6739 0.43835 0.460 0.000 0.072 0.000 0.168 0.300
#> GSM340184 3 0.3656 0.55820 0.000 0.000 0.728 0.012 0.256 0.004
#> GSM340185 4 0.5795 0.59442 0.000 0.036 0.092 0.672 0.048 0.152
#> GSM340186 4 0.3674 0.67713 0.000 0.060 0.056 0.836 0.028 0.020
#> GSM340187 4 0.1686 0.70102 0.000 0.000 0.064 0.924 0.000 0.012
#> GSM340189 4 0.3221 0.60147 0.000 0.000 0.264 0.736 0.000 0.000
#> GSM340190 4 0.4760 0.14539 0.000 0.004 0.436 0.520 0.040 0.000
#> GSM340191 4 0.4473 0.44261 0.000 0.000 0.380 0.584 0.036 0.000
#> GSM340192 2 0.1882 0.73295 0.000 0.920 0.008 0.000 0.060 0.012
#> GSM340193 1 0.4765 0.65247 0.672 0.000 0.012 0.000 0.072 0.244
#> GSM340194 2 0.2135 0.76178 0.000 0.872 0.000 0.000 0.128 0.000
#> GSM340195 2 0.3074 0.72693 0.004 0.792 0.000 0.000 0.200 0.004
#> GSM340196 4 0.1204 0.67581 0.000 0.000 0.000 0.944 0.000 0.056
#> GSM340197 2 0.3798 0.69252 0.032 0.748 0.000 0.000 0.216 0.004
#> GSM340198 2 0.0717 0.74666 0.000 0.976 0.000 0.000 0.016 0.008
#> GSM340199 3 0.3950 0.50344 0.000 0.000 0.720 0.240 0.040 0.000
#> GSM340200 3 0.7449 0.50128 0.000 0.080 0.524 0.168 0.124 0.104
#> GSM340201 4 0.2163 0.64389 0.000 0.000 0.000 0.892 0.016 0.092
#> GSM340202 4 0.1349 0.67607 0.000 0.000 0.000 0.940 0.004 0.056
#> GSM340203 4 0.3634 0.56336 0.000 0.000 0.296 0.696 0.008 0.000
#> GSM340204 2 0.0622 0.75491 0.000 0.980 0.000 0.000 0.012 0.008
#> GSM340205 1 0.5380 0.57892 0.640 0.000 0.028 0.000 0.112 0.220
#> GSM340206 3 0.3268 0.70041 0.000 0.000 0.824 0.076 0.100 0.000
#> GSM340207 2 0.4227 0.50943 0.000 0.632 0.020 0.000 0.344 0.004
#> GSM340237 6 0.5877 0.63873 0.032 0.000 0.036 0.140 0.136 0.656
#> GSM340238 3 0.3860 -0.12405 0.000 0.000 0.528 0.472 0.000 0.000
#> GSM340239 2 0.4919 0.61849 0.092 0.676 0.000 0.000 0.216 0.016
#> GSM340240 2 0.2573 0.75356 0.000 0.856 0.004 0.000 0.132 0.008
#> GSM340241 4 0.6702 0.42184 0.000 0.260 0.032 0.532 0.044 0.132
#> GSM340242 5 0.5169 0.71730 0.000 0.212 0.128 0.000 0.648 0.012
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
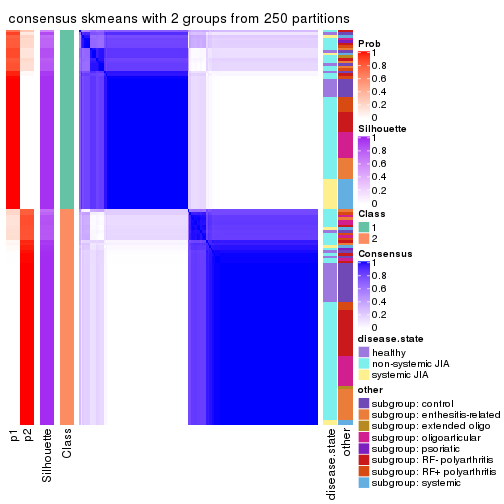
consensus_heatmap(res, k = 3)
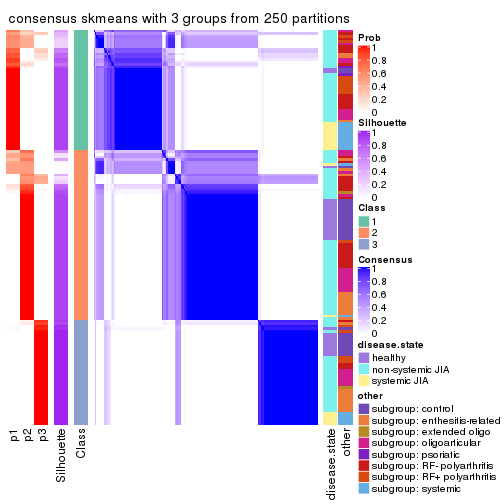
consensus_heatmap(res, k = 4)
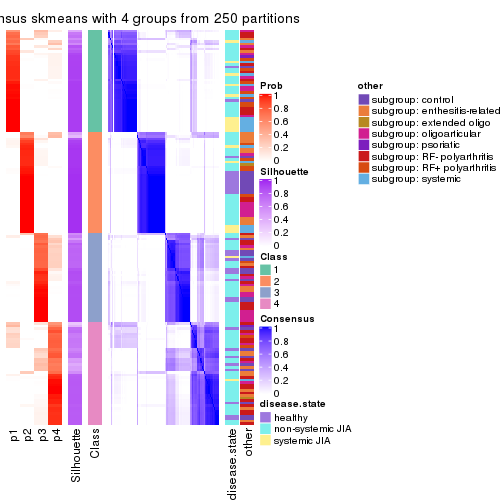
consensus_heatmap(res, k = 5)
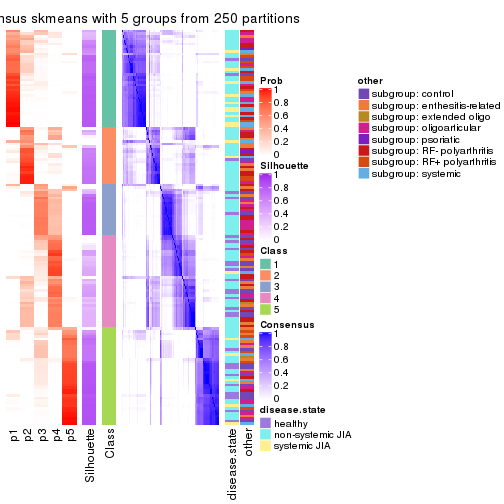
consensus_heatmap(res, k = 6)
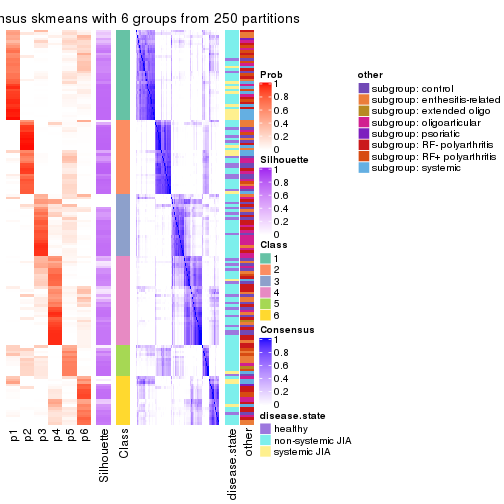
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
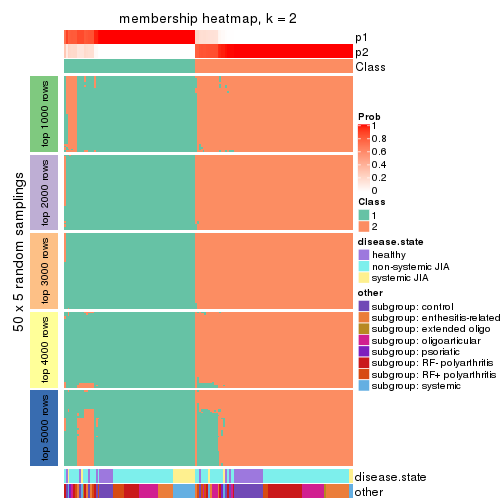
membership_heatmap(res, k = 3)
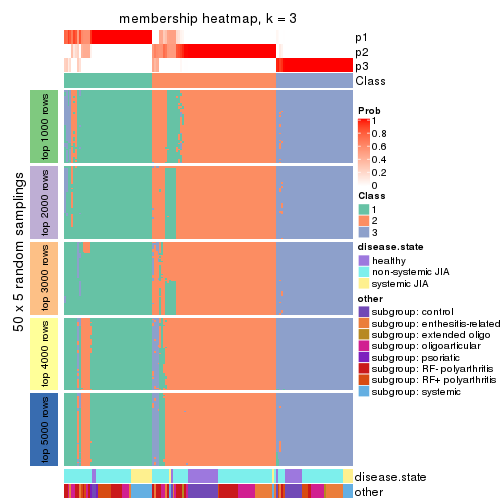
membership_heatmap(res, k = 4)
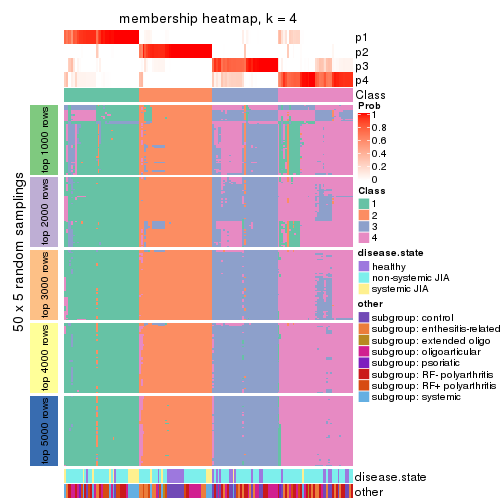
membership_heatmap(res, k = 5)
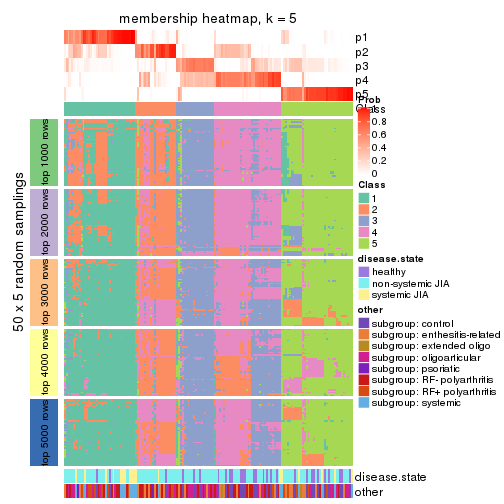
membership_heatmap(res, k = 6)
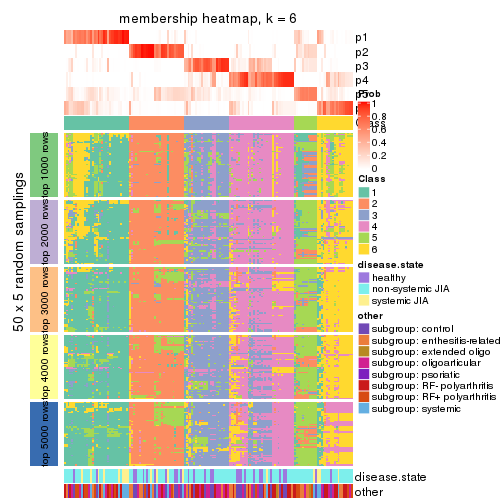
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
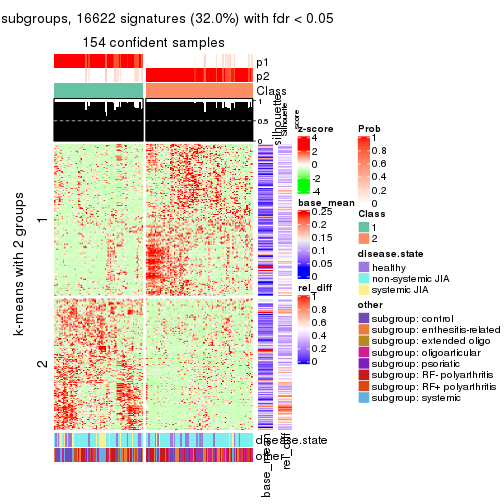
get_signatures(res, k = 3)
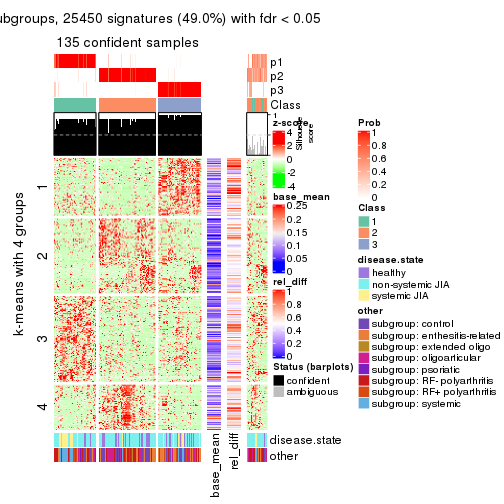
get_signatures(res, k = 4)
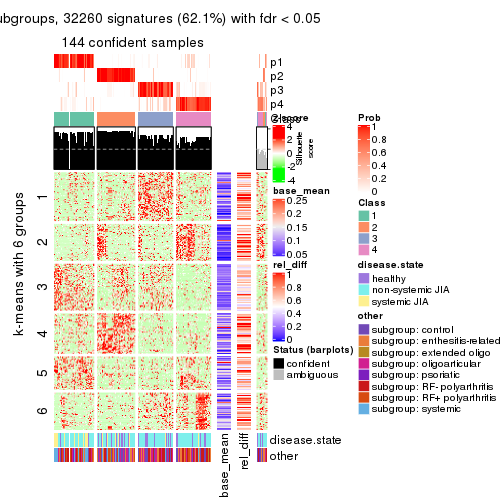
get_signatures(res, k = 5)
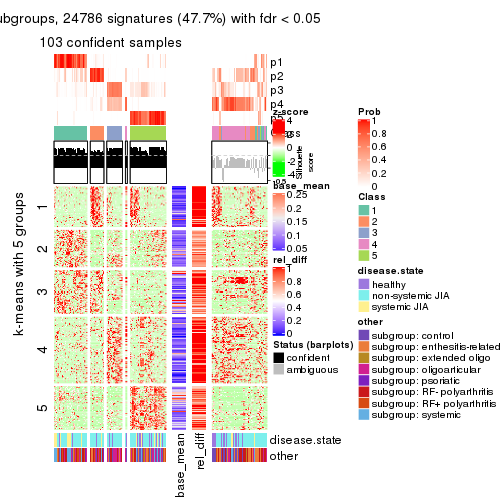
get_signatures(res, k = 6)
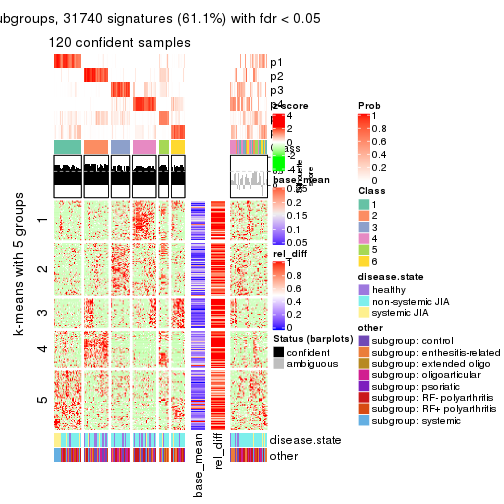
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
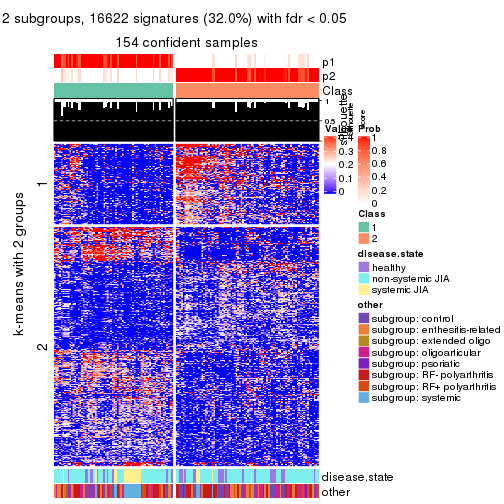
get_signatures(res, k = 3, scale_rows = FALSE)
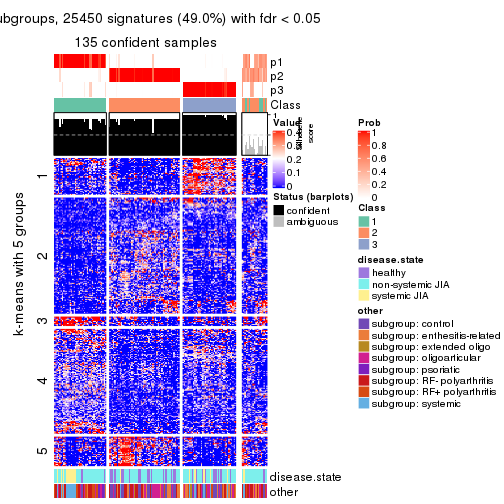
get_signatures(res, k = 4, scale_rows = FALSE)
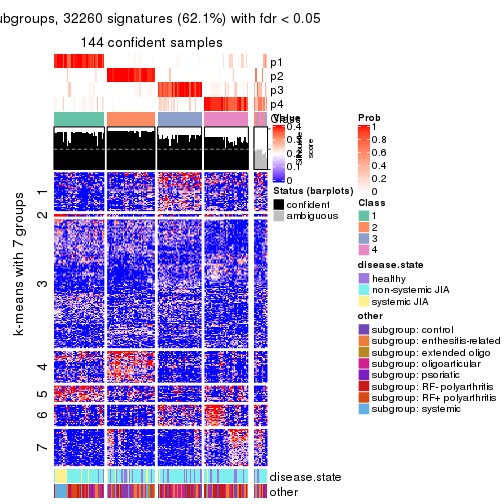
get_signatures(res, k = 5, scale_rows = FALSE)
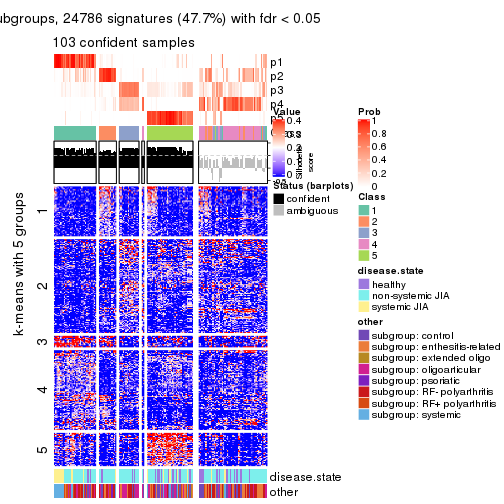
get_signatures(res, k = 6, scale_rows = FALSE)
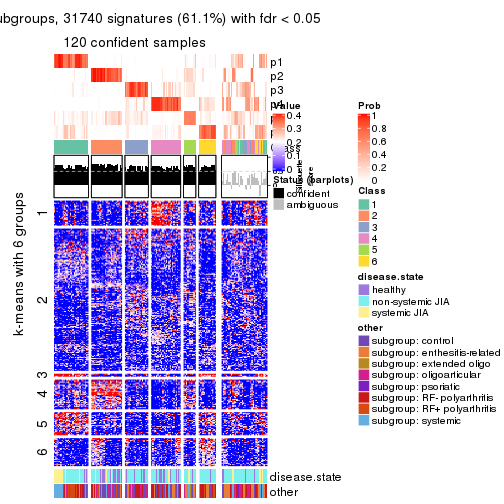
Compare the overlap of signatures from different k:
compare_signatures(res)
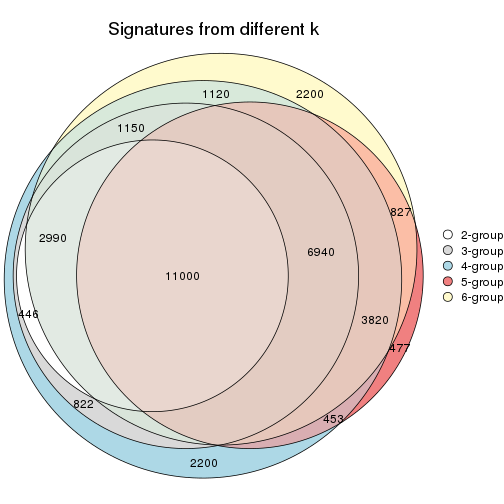
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
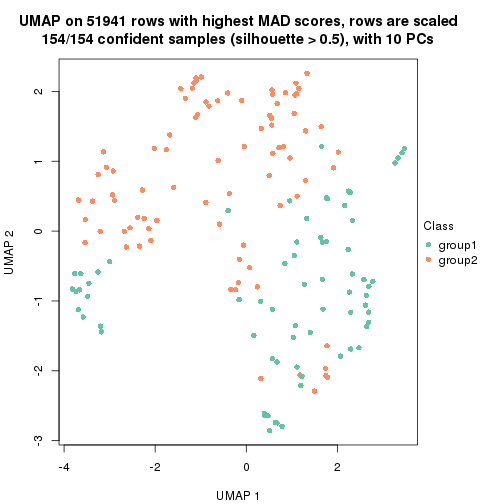
dimension_reduction(res, k = 3, method = "UMAP")
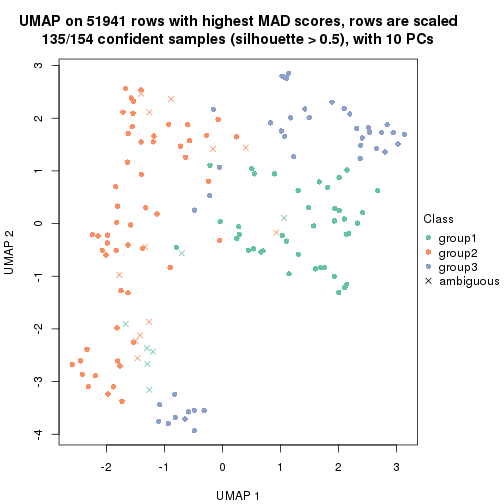
dimension_reduction(res, k = 4, method = "UMAP")
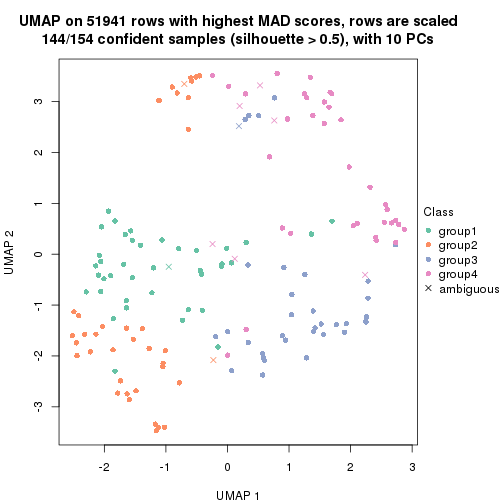
dimension_reduction(res, k = 5, method = "UMAP")
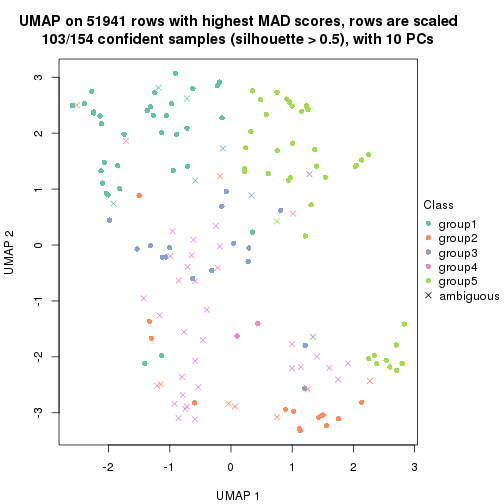
dimension_reduction(res, k = 6, method = "UMAP")
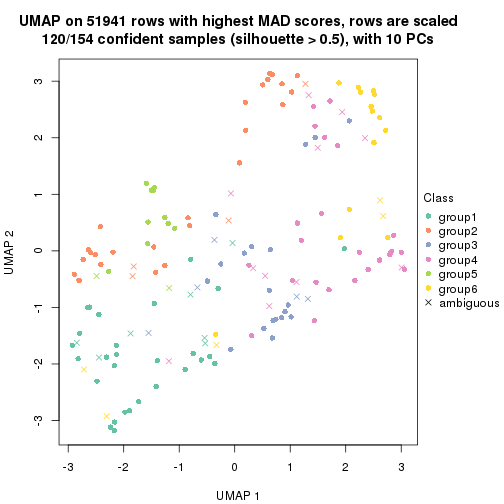
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
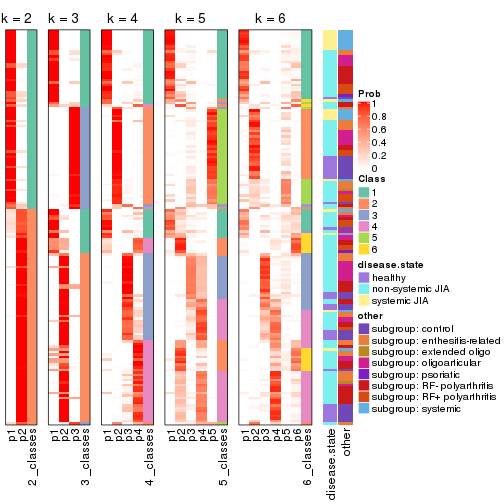
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> MAD:skmeans 154 0.012615 0.117873 2
#> MAD:skmeans 135 0.000716 0.000317 3
#> MAD:skmeans 144 0.003175 0.004020 4
#> MAD:skmeans 103 0.027770 0.054039 5
#> MAD:skmeans 120 0.007322 0.000529 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["MAD", "pam"]
# you can also extract it by
# res = res_list["MAD:pam"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'MAD' method.
#> Subgroups are detected by 'pam' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
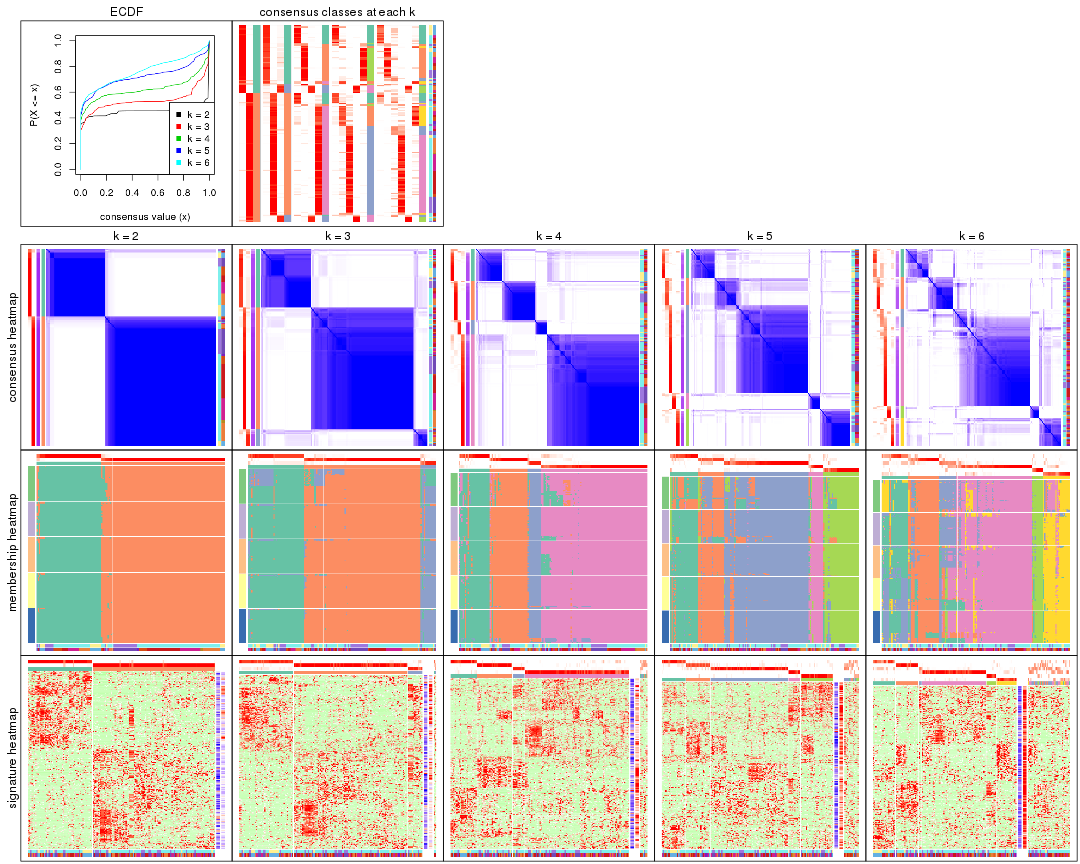
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
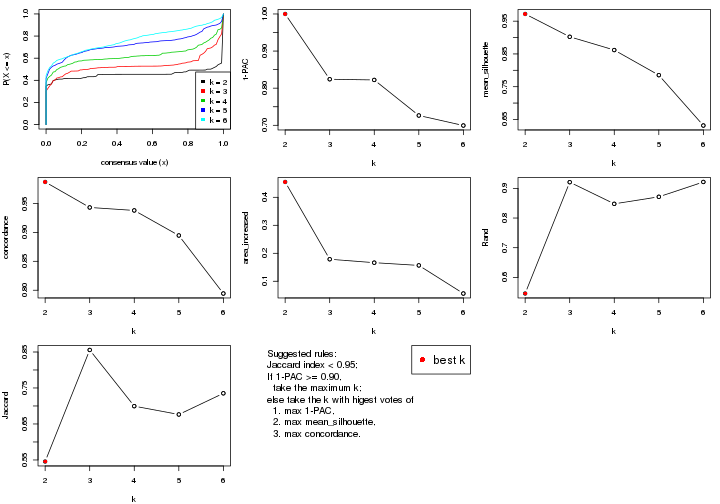
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 1.000 0.972 0.987 0.4548 0.546 0.546
#> 3 3 0.824 0.902 0.943 0.1787 0.921 0.856
#> 4 4 0.822 0.862 0.938 0.1664 0.848 0.699
#> 5 5 0.726 0.785 0.895 0.1567 0.871 0.676
#> 6 6 0.699 0.631 0.794 0.0562 0.921 0.735
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 2 0.0000 0.990 0.000 1.000
#> GSM340359 1 0.0000 0.981 1.000 0.000
#> GSM340361 1 0.0000 0.981 1.000 0.000
#> GSM340362 1 0.0000 0.981 1.000 0.000
#> GSM340363 2 0.0000 0.990 0.000 1.000
#> GSM340364 1 0.0000 0.981 1.000 0.000
#> GSM340365 1 0.0000 0.981 1.000 0.000
#> GSM340366 1 0.0000 0.981 1.000 0.000
#> GSM340367 1 0.2423 0.949 0.960 0.040
#> GSM340368 2 0.0000 0.990 0.000 1.000
#> GSM340369 2 0.0000 0.990 0.000 1.000
#> GSM340370 1 0.2236 0.952 0.964 0.036
#> GSM340371 2 0.3584 0.924 0.068 0.932
#> GSM340372 1 0.0000 0.981 1.000 0.000
#> GSM340373 1 0.0000 0.981 1.000 0.000
#> GSM340375 1 0.0000 0.981 1.000 0.000
#> GSM340376 1 0.2423 0.949 0.960 0.040
#> GSM340378 1 0.2423 0.949 0.960 0.040
#> GSM340243 2 0.0000 0.990 0.000 1.000
#> GSM340244 2 0.0000 0.990 0.000 1.000
#> GSM340246 2 0.0000 0.990 0.000 1.000
#> GSM340247 2 0.0000 0.990 0.000 1.000
#> GSM340248 2 0.0000 0.990 0.000 1.000
#> GSM340249 2 0.0000 0.990 0.000 1.000
#> GSM340250 2 0.1184 0.976 0.016 0.984
#> GSM340251 2 0.0000 0.990 0.000 1.000
#> GSM340252 1 0.0000 0.981 1.000 0.000
#> GSM340253 1 0.0000 0.981 1.000 0.000
#> GSM340254 1 0.0000 0.981 1.000 0.000
#> GSM340256 1 0.0000 0.981 1.000 0.000
#> GSM340258 2 0.1843 0.965 0.028 0.972
#> GSM340259 1 0.0000 0.981 1.000 0.000
#> GSM340260 1 0.0000 0.981 1.000 0.000
#> GSM340261 2 0.0000 0.990 0.000 1.000
#> GSM340262 1 0.0000 0.981 1.000 0.000
#> GSM340263 2 0.0000 0.990 0.000 1.000
#> GSM340264 1 0.0000 0.981 1.000 0.000
#> GSM340265 1 0.0000 0.981 1.000 0.000
#> GSM340266 2 0.0000 0.990 0.000 1.000
#> GSM340267 2 0.2603 0.950 0.044 0.956
#> GSM340268 2 0.0000 0.990 0.000 1.000
#> GSM340269 2 0.0000 0.990 0.000 1.000
#> GSM340270 2 0.0000 0.990 0.000 1.000
#> GSM537574 2 0.0000 0.990 0.000 1.000
#> GSM537580 2 0.0000 0.990 0.000 1.000
#> GSM537581 2 0.0000 0.990 0.000 1.000
#> GSM340272 1 0.0000 0.981 1.000 0.000
#> GSM340273 2 0.0000 0.990 0.000 1.000
#> GSM340275 2 0.0000 0.990 0.000 1.000
#> GSM340276 2 0.0000 0.990 0.000 1.000
#> GSM340277 1 0.0000 0.981 1.000 0.000
#> GSM340278 2 0.0000 0.990 0.000 1.000
#> GSM340279 2 0.0000 0.990 0.000 1.000
#> GSM340282 1 0.0000 0.981 1.000 0.000
#> GSM340284 2 0.0000 0.990 0.000 1.000
#> GSM340285 2 0.0000 0.990 0.000 1.000
#> GSM340286 1 0.0000 0.981 1.000 0.000
#> GSM340287 2 0.0000 0.990 0.000 1.000
#> GSM340288 1 0.0000 0.981 1.000 0.000
#> GSM340289 1 0.0000 0.981 1.000 0.000
#> GSM340290 2 0.0000 0.990 0.000 1.000
#> GSM340291 2 0.0000 0.990 0.000 1.000
#> GSM340293 2 0.0000 0.990 0.000 1.000
#> GSM340294 2 0.0000 0.990 0.000 1.000
#> GSM340296 2 0.0000 0.990 0.000 1.000
#> GSM340297 2 0.0000 0.990 0.000 1.000
#> GSM340298 2 0.0000 0.990 0.000 1.000
#> GSM340299 1 0.0000 0.981 1.000 0.000
#> GSM340301 1 0.0000 0.981 1.000 0.000
#> GSM340303 1 0.0000 0.981 1.000 0.000
#> GSM340304 2 0.0000 0.990 0.000 1.000
#> GSM340306 2 0.7219 0.752 0.200 0.800
#> GSM340307 2 0.0000 0.990 0.000 1.000
#> GSM340310 2 0.2948 0.940 0.052 0.948
#> GSM340314 2 0.0000 0.990 0.000 1.000
#> GSM340315 2 0.0000 0.990 0.000 1.000
#> GSM340317 2 0.0000 0.990 0.000 1.000
#> GSM340318 2 0.0000 0.990 0.000 1.000
#> GSM340319 2 0.0000 0.990 0.000 1.000
#> GSM340320 2 0.0000 0.990 0.000 1.000
#> GSM340321 2 0.0000 0.990 0.000 1.000
#> GSM340322 2 0.0000 0.990 0.000 1.000
#> GSM340324 1 0.0000 0.981 1.000 0.000
#> GSM340328 2 0.0000 0.990 0.000 1.000
#> GSM340330 2 0.0000 0.990 0.000 1.000
#> GSM340332 2 0.0000 0.990 0.000 1.000
#> GSM340333 1 0.0000 0.981 1.000 0.000
#> GSM340336 2 0.0000 0.990 0.000 1.000
#> GSM340337 2 0.0000 0.990 0.000 1.000
#> GSM340338 2 0.0000 0.990 0.000 1.000
#> GSM340339 2 0.0000 0.990 0.000 1.000
#> GSM340340 2 0.0000 0.990 0.000 1.000
#> GSM340341 2 0.0000 0.990 0.000 1.000
#> GSM340343 2 0.0000 0.990 0.000 1.000
#> GSM340344 1 0.0000 0.981 1.000 0.000
#> GSM340346 2 0.0000 0.990 0.000 1.000
#> GSM340347 2 0.0000 0.990 0.000 1.000
#> GSM340348 2 0.0000 0.990 0.000 1.000
#> GSM340349 2 0.0000 0.990 0.000 1.000
#> GSM340350 2 0.0000 0.990 0.000 1.000
#> GSM340351 1 0.0000 0.981 1.000 0.000
#> GSM340354 2 0.0000 0.990 0.000 1.000
#> GSM340356 2 0.0000 0.990 0.000 1.000
#> GSM340357 2 0.0000 0.990 0.000 1.000
#> GSM348183 1 0.0000 0.981 1.000 0.000
#> GSM348191 2 0.0000 0.990 0.000 1.000
#> GSM348193 1 0.8713 0.605 0.708 0.292
#> GSM537578 2 0.7376 0.741 0.208 0.792
#> GSM348181 2 0.0000 0.990 0.000 1.000
#> GSM348182 1 0.0000 0.981 1.000 0.000
#> GSM348184 2 0.0000 0.990 0.000 1.000
#> GSM348185 2 0.0000 0.990 0.000 1.000
#> GSM348186 1 0.0000 0.981 1.000 0.000
#> GSM348187 2 0.0000 0.990 0.000 1.000
#> GSM348188 1 0.0000 0.981 1.000 0.000
#> GSM348189 2 0.8555 0.614 0.280 0.720
#> GSM348190 1 0.0000 0.981 1.000 0.000
#> GSM348194 1 0.0000 0.981 1.000 0.000
#> GSM348195 1 0.0000 0.981 1.000 0.000
#> GSM348196 2 0.0000 0.990 0.000 1.000
#> GSM537585 1 0.0000 0.981 1.000 0.000
#> GSM537594 2 0.0376 0.986 0.004 0.996
#> GSM537596 2 0.0000 0.990 0.000 1.000
#> GSM537597 2 0.0000 0.990 0.000 1.000
#> GSM537602 2 0.0000 0.990 0.000 1.000
#> GSM340184 2 0.0000 0.990 0.000 1.000
#> GSM340185 2 0.0000 0.990 0.000 1.000
#> GSM340186 2 0.0000 0.990 0.000 1.000
#> GSM340187 2 0.0000 0.990 0.000 1.000
#> GSM340189 2 0.0000 0.990 0.000 1.000
#> GSM340190 2 0.0000 0.990 0.000 1.000
#> GSM340191 2 0.0000 0.990 0.000 1.000
#> GSM340192 1 0.8608 0.618 0.716 0.284
#> GSM340193 2 0.0000 0.990 0.000 1.000
#> GSM340194 1 0.0000 0.981 1.000 0.000
#> GSM340195 1 0.0000 0.981 1.000 0.000
#> GSM340196 2 0.0000 0.990 0.000 1.000
#> GSM340197 1 0.0000 0.981 1.000 0.000
#> GSM340198 1 0.0000 0.981 1.000 0.000
#> GSM340199 2 0.0000 0.990 0.000 1.000
#> GSM340200 2 0.0000 0.990 0.000 1.000
#> GSM340201 2 0.0000 0.990 0.000 1.000
#> GSM340202 2 0.0000 0.990 0.000 1.000
#> GSM340203 2 0.0000 0.990 0.000 1.000
#> GSM340204 1 0.0000 0.981 1.000 0.000
#> GSM340205 2 0.0000 0.990 0.000 1.000
#> GSM340206 2 0.0000 0.990 0.000 1.000
#> GSM340207 1 0.7745 0.708 0.772 0.228
#> GSM340237 2 0.0000 0.990 0.000 1.000
#> GSM340238 2 0.0000 0.990 0.000 1.000
#> GSM340239 1 0.0000 0.981 1.000 0.000
#> GSM340240 1 0.0376 0.978 0.996 0.004
#> GSM340241 2 0.0000 0.990 0.000 1.000
#> GSM340242 2 0.4431 0.896 0.092 0.908
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 2 0.4295 0.864 0.104 0.864 0.032
#> GSM340359 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340361 1 0.1289 0.869 0.968 0.000 0.032
#> GSM340362 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340363 2 0.3213 0.899 0.092 0.900 0.008
#> GSM340364 1 0.1163 0.880 0.972 0.000 0.028
#> GSM340365 1 0.1529 0.891 0.960 0.000 0.040
#> GSM340366 1 0.3295 0.900 0.896 0.008 0.096
#> GSM340367 1 0.1289 0.869 0.968 0.000 0.032
#> GSM340368 2 0.1163 0.955 0.028 0.972 0.000
#> GSM340369 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340370 1 0.1289 0.869 0.968 0.000 0.032
#> GSM340371 3 0.1399 0.853 0.004 0.028 0.968
#> GSM340372 1 0.1289 0.869 0.968 0.000 0.032
#> GSM340373 1 0.1289 0.869 0.968 0.000 0.032
#> GSM340375 1 0.1163 0.871 0.972 0.000 0.028
#> GSM340376 1 0.2486 0.841 0.932 0.060 0.008
#> GSM340378 1 0.1289 0.869 0.968 0.000 0.032
#> GSM340243 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340244 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340246 2 0.1289 0.949 0.000 0.968 0.032
#> GSM340247 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340248 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340249 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340250 2 0.0592 0.962 0.012 0.988 0.000
#> GSM340251 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340252 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340253 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340254 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340256 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340258 2 0.3590 0.883 0.028 0.896 0.076
#> GSM340259 1 0.3528 0.897 0.892 0.016 0.092
#> GSM340260 3 0.1289 0.844 0.032 0.000 0.968
#> GSM340261 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340262 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340263 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340264 3 0.2448 0.832 0.076 0.000 0.924
#> GSM340265 3 0.1289 0.844 0.032 0.000 0.968
#> GSM340266 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340267 3 0.1482 0.852 0.012 0.020 0.968
#> GSM340268 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340269 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340270 2 0.0000 0.968 0.000 1.000 0.000
#> GSM537574 2 0.0000 0.968 0.000 1.000 0.000
#> GSM537580 3 0.1529 0.846 0.000 0.040 0.960
#> GSM537581 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340272 1 0.5291 0.628 0.732 0.000 0.268
#> GSM340273 2 0.1289 0.953 0.032 0.968 0.000
#> GSM340275 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340276 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340277 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340278 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340279 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340282 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340284 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340285 2 0.1163 0.955 0.028 0.972 0.000
#> GSM340286 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340287 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340288 1 0.1289 0.869 0.968 0.000 0.032
#> GSM340289 1 0.2625 0.841 0.916 0.000 0.084
#> GSM340290 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340291 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340293 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340294 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340296 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340297 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340298 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340299 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340301 1 0.5706 0.627 0.680 0.000 0.320
#> GSM340303 3 0.1289 0.844 0.032 0.000 0.968
#> GSM340304 2 0.0424 0.965 0.008 0.992 0.000
#> GSM340306 2 0.4912 0.764 0.196 0.796 0.008
#> GSM340307 2 0.1163 0.951 0.000 0.972 0.028
#> GSM340310 2 0.3805 0.881 0.092 0.884 0.024
#> GSM340314 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340315 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340317 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340318 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340319 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340320 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340321 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340322 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340324 1 0.2947 0.857 0.920 0.060 0.020
#> GSM340328 2 0.4295 0.864 0.104 0.864 0.032
#> GSM340330 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340332 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340333 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340336 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340337 2 0.1163 0.955 0.028 0.972 0.000
#> GSM340338 2 0.2066 0.926 0.000 0.940 0.060
#> GSM340339 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340340 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340341 2 0.0892 0.959 0.020 0.980 0.000
#> GSM340343 2 0.0237 0.966 0.000 0.996 0.004
#> GSM340344 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340346 2 0.5216 0.641 0.000 0.740 0.260
#> GSM340347 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340348 2 0.1163 0.955 0.028 0.972 0.000
#> GSM340349 3 0.8457 0.277 0.092 0.396 0.512
#> GSM340350 2 0.4137 0.872 0.096 0.872 0.032
#> GSM340351 1 0.1289 0.869 0.968 0.000 0.032
#> GSM340354 2 0.4178 0.801 0.000 0.828 0.172
#> GSM340356 2 0.1289 0.953 0.032 0.968 0.000
#> GSM340357 2 0.3499 0.894 0.072 0.900 0.028
#> GSM348183 1 0.1289 0.869 0.968 0.000 0.032
#> GSM348191 2 0.1643 0.939 0.000 0.956 0.044
#> GSM348193 1 0.5502 0.464 0.744 0.248 0.008
#> GSM537578 2 0.5803 0.713 0.212 0.760 0.028
#> GSM348181 2 0.1015 0.960 0.012 0.980 0.008
#> GSM348182 3 0.5621 0.575 0.308 0.000 0.692
#> GSM348184 2 0.0000 0.968 0.000 1.000 0.000
#> GSM348185 2 0.1289 0.953 0.032 0.968 0.000
#> GSM348186 1 0.3038 0.904 0.896 0.000 0.104
#> GSM348187 2 0.4172 0.868 0.104 0.868 0.028
#> GSM348188 1 0.0424 0.881 0.992 0.008 0.000
#> GSM348189 3 0.1289 0.852 0.000 0.032 0.968
#> GSM348190 1 0.3038 0.904 0.896 0.000 0.104
#> GSM348194 1 0.1289 0.869 0.968 0.000 0.032
#> GSM348195 1 0.1289 0.869 0.968 0.000 0.032
#> GSM348196 2 0.4295 0.864 0.104 0.864 0.032
#> GSM537585 3 0.5178 0.703 0.256 0.000 0.744
#> GSM537594 2 0.1411 0.951 0.036 0.964 0.000
#> GSM537596 2 0.1289 0.953 0.032 0.968 0.000
#> GSM537597 2 0.1015 0.960 0.012 0.980 0.008
#> GSM537602 2 0.1711 0.948 0.032 0.960 0.008
#> GSM340184 2 0.2066 0.923 0.000 0.940 0.060
#> GSM340185 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340186 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340187 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340189 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340190 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340191 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340192 1 0.7228 0.587 0.708 0.188 0.104
#> GSM340193 2 0.2689 0.928 0.036 0.932 0.032
#> GSM340194 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340195 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340196 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340197 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340198 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340199 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340200 2 0.2878 0.889 0.000 0.904 0.096
#> GSM340201 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340202 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340203 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340204 1 0.3038 0.904 0.896 0.000 0.104
#> GSM340205 2 0.0424 0.965 0.000 0.992 0.008
#> GSM340206 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340207 3 0.8153 0.533 0.240 0.128 0.632
#> GSM340237 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340238 2 0.0000 0.968 0.000 1.000 0.000
#> GSM340239 1 0.3038 0.902 0.896 0.000 0.104
#> GSM340240 1 0.3116 0.901 0.892 0.000 0.108
#> GSM340241 2 0.2878 0.889 0.000 0.904 0.096
#> GSM340242 3 0.1289 0.852 0.000 0.032 0.968
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.0000 0.8596 1.000 0.000 0.000 0.000
#> GSM340359 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340361 1 0.0000 0.8596 1.000 0.000 0.000 0.000
#> GSM340362 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340363 4 0.5432 0.5677 0.316 0.000 0.032 0.652
#> GSM340364 1 0.5861 0.0275 0.492 0.476 0.032 0.000
#> GSM340365 2 0.2281 0.8326 0.096 0.904 0.000 0.000
#> GSM340366 2 0.0921 0.8953 0.000 0.972 0.000 0.028
#> GSM340367 1 0.0000 0.8596 1.000 0.000 0.000 0.000
#> GSM340368 4 0.1474 0.9227 0.052 0.000 0.000 0.948
#> GSM340369 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340370 1 0.0000 0.8596 1.000 0.000 0.000 0.000
#> GSM340371 3 0.1022 0.9265 0.000 0.032 0.968 0.000
#> GSM340372 1 0.0000 0.8596 1.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.8596 1.000 0.000 0.000 0.000
#> GSM340375 1 0.2382 0.7939 0.912 0.080 0.004 0.004
#> GSM340376 4 0.5453 0.5596 0.320 0.000 0.032 0.648
#> GSM340378 1 0.0000 0.8596 1.000 0.000 0.000 0.000
#> GSM340243 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340244 4 0.1022 0.9363 0.000 0.000 0.032 0.968
#> GSM340246 4 0.1833 0.9276 0.000 0.024 0.032 0.944
#> GSM340247 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340248 4 0.0592 0.9426 0.000 0.000 0.016 0.984
#> GSM340249 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340250 4 0.3400 0.8282 0.004 0.128 0.012 0.856
#> GSM340251 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340252 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340253 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340254 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340256 2 0.1022 0.8993 0.000 0.968 0.032 0.000
#> GSM340258 2 0.4989 0.0872 0.000 0.528 0.000 0.472
#> GSM340259 1 0.5461 0.0733 0.508 0.480 0.004 0.008
#> GSM340260 3 0.1022 0.9265 0.000 0.032 0.968 0.000
#> GSM340261 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340262 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340263 4 0.1022 0.9363 0.000 0.000 0.032 0.968
#> GSM340264 3 0.1022 0.9265 0.000 0.032 0.968 0.000
#> GSM340265 3 0.1022 0.9265 0.000 0.032 0.968 0.000
#> GSM340266 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340267 3 0.3257 0.8168 0.000 0.152 0.844 0.004
#> GSM340268 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340269 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340270 4 0.0469 0.9434 0.012 0.000 0.000 0.988
#> GSM537574 4 0.1022 0.9363 0.000 0.000 0.032 0.968
#> GSM537580 3 0.1182 0.9050 0.000 0.016 0.968 0.016
#> GSM537581 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340272 2 0.1637 0.8775 0.060 0.940 0.000 0.000
#> GSM340273 4 0.3581 0.8551 0.116 0.000 0.032 0.852
#> GSM340275 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340276 4 0.0188 0.9463 0.004 0.000 0.000 0.996
#> GSM340277 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340278 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340279 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340282 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340284 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340285 4 0.1867 0.9087 0.072 0.000 0.000 0.928
#> GSM340286 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340287 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340288 1 0.0000 0.8596 1.000 0.000 0.000 0.000
#> GSM340289 1 0.2053 0.8018 0.924 0.004 0.072 0.000
#> GSM340290 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340291 4 0.1356 0.9333 0.008 0.000 0.032 0.960
#> GSM340293 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340294 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340296 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340297 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340298 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340299 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340301 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340303 3 0.1022 0.9265 0.000 0.032 0.968 0.000
#> GSM340304 4 0.1610 0.9307 0.016 0.000 0.032 0.952
#> GSM340306 4 0.4643 0.5466 0.344 0.000 0.000 0.656
#> GSM340307 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340310 1 0.4617 0.5906 0.764 0.000 0.032 0.204
#> GSM340314 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340315 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340317 4 0.1022 0.9363 0.000 0.000 0.032 0.968
#> GSM340318 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340319 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340320 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340321 4 0.1022 0.9363 0.000 0.000 0.032 0.968
#> GSM340322 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340324 2 0.4441 0.7475 0.052 0.836 0.032 0.080
#> GSM340328 1 0.0000 0.8596 1.000 0.000 0.000 0.000
#> GSM340330 4 0.0336 0.9445 0.000 0.008 0.000 0.992
#> GSM340332 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340333 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340336 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340337 4 0.1792 0.9115 0.068 0.000 0.000 0.932
#> GSM340338 4 0.2149 0.8900 0.000 0.088 0.000 0.912
#> GSM340339 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340340 4 0.1022 0.9363 0.000 0.000 0.032 0.968
#> GSM340341 4 0.1474 0.9228 0.052 0.000 0.000 0.948
#> GSM340343 4 0.1211 0.9287 0.040 0.000 0.000 0.960
#> GSM340344 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340346 4 0.1302 0.9256 0.000 0.044 0.000 0.956
#> GSM340347 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340348 4 0.1867 0.9087 0.072 0.000 0.000 0.928
#> GSM340349 1 0.7472 0.0173 0.428 0.000 0.396 0.176
#> GSM340350 1 0.1118 0.8379 0.964 0.000 0.000 0.036
#> GSM340351 1 0.0000 0.8596 1.000 0.000 0.000 0.000
#> GSM340354 4 0.2281 0.8832 0.000 0.096 0.000 0.904
#> GSM340356 4 0.3803 0.8388 0.132 0.000 0.032 0.836
#> GSM340357 1 0.3024 0.7085 0.852 0.000 0.000 0.148
#> GSM348183 1 0.0707 0.8510 0.980 0.000 0.020 0.000
#> GSM348191 4 0.1474 0.9200 0.000 0.052 0.000 0.948
#> GSM348193 4 0.5492 0.5435 0.328 0.000 0.032 0.640
#> GSM537578 1 0.2483 0.8053 0.916 0.032 0.000 0.052
#> GSM348181 4 0.3649 0.7650 0.204 0.000 0.000 0.796
#> GSM348182 3 0.1151 0.9188 0.008 0.024 0.968 0.000
#> GSM348184 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM348185 4 0.3523 0.8590 0.112 0.000 0.032 0.856
#> GSM348186 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM348187 1 0.1792 0.8020 0.932 0.000 0.000 0.068
#> GSM348188 2 0.4187 0.7357 0.152 0.816 0.024 0.008
#> GSM348189 3 0.1022 0.9265 0.000 0.032 0.968 0.000
#> GSM348190 2 0.0336 0.9192 0.008 0.992 0.000 0.000
#> GSM348194 1 0.0592 0.8533 0.984 0.000 0.016 0.000
#> GSM348195 1 0.0000 0.8596 1.000 0.000 0.000 0.000
#> GSM348196 1 0.0188 0.8584 0.996 0.000 0.000 0.004
#> GSM537585 3 0.7314 0.1527 0.152 0.424 0.424 0.000
#> GSM537594 4 0.3581 0.8551 0.116 0.000 0.032 0.852
#> GSM537596 4 0.2843 0.8876 0.088 0.000 0.020 0.892
#> GSM537597 4 0.3638 0.8485 0.120 0.000 0.032 0.848
#> GSM537602 4 0.5113 0.6588 0.264 0.000 0.032 0.704
#> GSM340184 4 0.2011 0.8987 0.000 0.000 0.080 0.920
#> GSM340185 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340186 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340187 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340189 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340190 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340191 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340192 2 0.1557 0.8539 0.000 0.944 0.000 0.056
#> GSM340193 1 0.3074 0.6953 0.848 0.000 0.000 0.152
#> GSM340194 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340195 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340196 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340197 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340198 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340199 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340200 4 0.2647 0.8593 0.000 0.120 0.000 0.880
#> GSM340201 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340202 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340203 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340204 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340205 4 0.4332 0.7722 0.176 0.000 0.032 0.792
#> GSM340206 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340207 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340237 4 0.1022 0.9363 0.000 0.000 0.032 0.968
#> GSM340238 4 0.0000 0.9476 0.000 0.000 0.000 1.000
#> GSM340239 2 0.4948 0.1525 0.440 0.560 0.000 0.000
#> GSM340240 2 0.0000 0.9255 0.000 1.000 0.000 0.000
#> GSM340241 4 0.2647 0.8593 0.000 0.120 0.000 0.880
#> GSM340242 3 0.1022 0.9265 0.000 0.032 0.968 0.000
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.2377 0.794174 0.872 0.128 0.000 0.000 0.000
#> GSM340359 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340361 1 0.0404 0.826904 0.988 0.000 0.000 0.000 0.012
#> GSM340362 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340363 2 0.2286 0.738278 0.108 0.888 0.004 0.000 0.000
#> GSM340364 1 0.4744 0.065503 0.508 0.476 0.000 0.000 0.016
#> GSM340365 5 0.2377 0.782366 0.128 0.000 0.000 0.000 0.872
#> GSM340366 5 0.4221 0.689881 0.000 0.112 0.108 0.000 0.780
#> GSM340367 1 0.0162 0.832490 0.996 0.004 0.000 0.000 0.000
#> GSM340368 3 0.2464 0.844763 0.016 0.096 0.888 0.000 0.000
#> GSM340369 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340370 1 0.0162 0.832490 0.996 0.004 0.000 0.000 0.000
#> GSM340371 4 0.0000 0.867347 0.000 0.000 0.000 1.000 0.000
#> GSM340372 1 0.0162 0.832490 0.996 0.004 0.000 0.000 0.000
#> GSM340373 1 0.0162 0.832490 0.996 0.004 0.000 0.000 0.000
#> GSM340375 1 0.0703 0.830764 0.976 0.024 0.000 0.000 0.000
#> GSM340376 2 0.2358 0.743796 0.104 0.888 0.008 0.000 0.000
#> GSM340378 1 0.0162 0.832490 0.996 0.004 0.000 0.000 0.000
#> GSM340243 3 0.0404 0.914198 0.000 0.012 0.988 0.000 0.000
#> GSM340244 2 0.3480 0.717781 0.000 0.752 0.248 0.000 0.000
#> GSM340246 2 0.2813 0.713553 0.000 0.868 0.108 0.000 0.024
#> GSM340247 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340248 3 0.2648 0.765012 0.000 0.152 0.848 0.000 0.000
#> GSM340249 3 0.0703 0.909161 0.000 0.024 0.976 0.000 0.000
#> GSM340250 3 0.3656 0.778474 0.008 0.056 0.832 0.000 0.104
#> GSM340251 3 0.0162 0.917851 0.000 0.004 0.996 0.000 0.000
#> GSM340252 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340253 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340254 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340256 2 0.3684 0.544670 0.000 0.720 0.000 0.000 0.280
#> GSM340258 5 0.4452 0.000254 0.000 0.004 0.496 0.000 0.500
#> GSM340259 5 0.7055 -0.056953 0.328 0.260 0.012 0.000 0.400
#> GSM340260 4 0.0000 0.867347 0.000 0.000 0.000 1.000 0.000
#> GSM340261 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340262 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340263 2 0.3424 0.724289 0.000 0.760 0.240 0.000 0.000
#> GSM340264 4 0.0000 0.867347 0.000 0.000 0.000 1.000 0.000
#> GSM340265 4 0.0404 0.862084 0.000 0.012 0.000 0.988 0.000
#> GSM340266 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340267 4 0.4069 0.737227 0.000 0.112 0.000 0.792 0.096
#> GSM340268 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340269 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340270 3 0.0290 0.916769 0.008 0.000 0.992 0.000 0.000
#> GSM537574 3 0.4227 0.081179 0.000 0.420 0.580 0.000 0.000
#> GSM537580 4 0.0000 0.867347 0.000 0.000 0.000 1.000 0.000
#> GSM537581 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340272 5 0.3132 0.739738 0.172 0.008 0.000 0.000 0.820
#> GSM340273 2 0.2505 0.754351 0.092 0.888 0.020 0.000 0.000
#> GSM340275 3 0.0404 0.914919 0.000 0.012 0.988 0.000 0.000
#> GSM340276 3 0.0609 0.911379 0.000 0.020 0.980 0.000 0.000
#> GSM340277 5 0.2074 0.828371 0.000 0.104 0.000 0.000 0.896
#> GSM340278 3 0.0290 0.916013 0.000 0.008 0.992 0.000 0.000
#> GSM340279 3 0.0880 0.908415 0.000 0.032 0.968 0.000 0.000
#> GSM340282 5 0.2179 0.822487 0.000 0.112 0.000 0.000 0.888
#> GSM340284 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340285 3 0.2921 0.808902 0.020 0.124 0.856 0.000 0.000
#> GSM340286 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340287 3 0.0794 0.912270 0.000 0.028 0.972 0.000 0.000
#> GSM340288 1 0.1197 0.826599 0.952 0.048 0.000 0.000 0.000
#> GSM340289 1 0.1197 0.802188 0.952 0.000 0.000 0.048 0.000
#> GSM340290 3 0.1043 0.905928 0.000 0.040 0.960 0.000 0.000
#> GSM340291 2 0.3586 0.746619 0.020 0.792 0.188 0.000 0.000
#> GSM340293 3 0.0162 0.918020 0.000 0.004 0.996 0.000 0.000
#> GSM340294 3 0.0290 0.917726 0.000 0.008 0.992 0.000 0.000
#> GSM340296 3 0.2230 0.858597 0.000 0.116 0.884 0.000 0.000
#> GSM340297 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340298 3 0.2127 0.861027 0.000 0.108 0.892 0.000 0.000
#> GSM340299 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340301 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340303 4 0.2616 0.798439 0.000 0.020 0.000 0.880 0.100
#> GSM340304 2 0.3487 0.724433 0.008 0.780 0.212 0.000 0.000
#> GSM340306 3 0.4772 0.649465 0.108 0.164 0.728 0.000 0.000
#> GSM340307 3 0.2179 0.858806 0.000 0.112 0.888 0.000 0.000
#> GSM340310 2 0.2574 0.739876 0.112 0.876 0.012 0.000 0.000
#> GSM340314 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340315 3 0.0404 0.916217 0.000 0.012 0.988 0.000 0.000
#> GSM340317 2 0.3452 0.722287 0.000 0.756 0.244 0.000 0.000
#> GSM340318 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340319 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340320 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340321 3 0.4060 0.286947 0.000 0.360 0.640 0.000 0.000
#> GSM340322 3 0.0162 0.917851 0.000 0.004 0.996 0.000 0.000
#> GSM340324 2 0.4087 0.735159 0.028 0.816 0.056 0.000 0.100
#> GSM340328 1 0.2377 0.794174 0.872 0.128 0.000 0.000 0.000
#> GSM340330 3 0.0162 0.917851 0.000 0.004 0.996 0.000 0.000
#> GSM340332 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340333 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340336 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340337 3 0.2773 0.818367 0.020 0.112 0.868 0.000 0.000
#> GSM340338 3 0.1764 0.877774 0.000 0.008 0.928 0.000 0.064
#> GSM340339 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340340 3 0.4306 -0.220528 0.000 0.492 0.508 0.000 0.000
#> GSM340341 3 0.2470 0.845260 0.012 0.104 0.884 0.000 0.000
#> GSM340343 3 0.0162 0.917797 0.004 0.000 0.996 0.000 0.000
#> GSM340344 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340346 3 0.2773 0.844941 0.000 0.112 0.868 0.000 0.020
#> GSM340347 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340348 3 0.2921 0.808902 0.020 0.124 0.856 0.000 0.000
#> GSM340349 4 0.8081 0.103056 0.288 0.232 0.104 0.376 0.000
#> GSM340350 1 0.3430 0.745879 0.776 0.220 0.004 0.000 0.000
#> GSM340351 1 0.0880 0.826482 0.968 0.032 0.000 0.000 0.000
#> GSM340354 3 0.3754 0.791972 0.000 0.100 0.816 0.000 0.084
#> GSM340356 2 0.2464 0.751831 0.096 0.888 0.016 0.000 0.000
#> GSM340357 1 0.2304 0.744608 0.892 0.008 0.100 0.000 0.000
#> GSM348183 1 0.3039 0.684742 0.808 0.192 0.000 0.000 0.000
#> GSM348191 3 0.3085 0.834644 0.000 0.116 0.852 0.000 0.032
#> GSM348193 2 0.2338 0.738595 0.112 0.884 0.004 0.000 0.000
#> GSM537578 1 0.2179 0.750375 0.888 0.112 0.000 0.000 0.000
#> GSM348181 3 0.5162 0.374370 0.064 0.308 0.628 0.000 0.000
#> GSM348182 4 0.0000 0.867347 0.000 0.000 0.000 1.000 0.000
#> GSM348184 3 0.0162 0.917851 0.000 0.004 0.996 0.000 0.000
#> GSM348185 2 0.2505 0.754352 0.092 0.888 0.020 0.000 0.000
#> GSM348186 5 0.0162 0.889075 0.000 0.004 0.000 0.000 0.996
#> GSM348187 1 0.5025 0.629894 0.704 0.172 0.124 0.000 0.000
#> GSM348188 5 0.4658 0.646074 0.032 0.196 0.028 0.000 0.744
#> GSM348189 4 0.0000 0.867347 0.000 0.000 0.000 1.000 0.000
#> GSM348190 5 0.0162 0.888733 0.004 0.000 0.000 0.000 0.996
#> GSM348194 1 0.4227 0.369714 0.580 0.420 0.000 0.000 0.000
#> GSM348195 1 0.0000 0.831440 1.000 0.000 0.000 0.000 0.000
#> GSM348196 1 0.2707 0.791024 0.860 0.132 0.008 0.000 0.000
#> GSM537585 4 0.6428 0.198594 0.176 0.000 0.000 0.440 0.384
#> GSM537594 2 0.2505 0.754351 0.092 0.888 0.020 0.000 0.000
#> GSM537596 2 0.5109 0.180998 0.036 0.504 0.460 0.000 0.000
#> GSM537597 2 0.5441 0.580275 0.080 0.596 0.324 0.000 0.000
#> GSM537602 2 0.2416 0.748320 0.100 0.888 0.012 0.000 0.000
#> GSM340184 3 0.1831 0.877762 0.000 0.004 0.920 0.076 0.000
#> GSM340185 3 0.1908 0.872772 0.000 0.092 0.908 0.000 0.000
#> GSM340186 3 0.0162 0.917851 0.000 0.004 0.996 0.000 0.000
#> GSM340187 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340189 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340190 3 0.2179 0.858806 0.000 0.112 0.888 0.000 0.000
#> GSM340191 3 0.0162 0.917391 0.000 0.004 0.996 0.000 0.000
#> GSM340192 5 0.2864 0.800597 0.000 0.112 0.024 0.000 0.864
#> GSM340193 1 0.3622 0.764744 0.820 0.124 0.056 0.000 0.000
#> GSM340194 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340195 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340196 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340197 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340198 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340199 3 0.1544 0.889232 0.000 0.068 0.932 0.000 0.000
#> GSM340200 3 0.4069 0.764498 0.000 0.112 0.792 0.000 0.096
#> GSM340201 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340202 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340203 3 0.0162 0.917851 0.000 0.004 0.996 0.000 0.000
#> GSM340204 5 0.0000 0.891011 0.000 0.000 0.000 0.000 1.000
#> GSM340205 2 0.4190 0.755593 0.060 0.768 0.172 0.000 0.000
#> GSM340206 3 0.2179 0.858806 0.000 0.112 0.888 0.000 0.000
#> GSM340207 5 0.1851 0.841047 0.000 0.088 0.000 0.000 0.912
#> GSM340237 2 0.3177 0.735572 0.000 0.792 0.208 0.000 0.000
#> GSM340238 3 0.0000 0.918392 0.000 0.000 1.000 0.000 0.000
#> GSM340239 1 0.4278 0.181190 0.548 0.000 0.000 0.000 0.452
#> GSM340240 5 0.2020 0.832759 0.000 0.100 0.000 0.000 0.900
#> GSM340241 3 0.2561 0.842046 0.000 0.020 0.884 0.000 0.096
#> GSM340242 4 0.0000 0.867347 0.000 0.000 0.000 1.000 0.000
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 1 0.5778 0.62035 0.464 0.000 0.352 0.000 0.000 0.184
#> GSM340359 2 0.0000 0.81930 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340361 1 0.1074 0.63770 0.960 0.028 0.012 0.000 0.000 0.000
#> GSM340362 2 0.0146 0.81804 0.004 0.996 0.000 0.000 0.000 0.000
#> GSM340363 6 0.0865 0.70606 0.000 0.000 0.036 0.000 0.000 0.964
#> GSM340364 1 0.3253 0.54205 0.788 0.000 0.020 0.000 0.000 0.192
#> GSM340365 2 0.4116 0.36846 0.416 0.572 0.012 0.000 0.000 0.000
#> GSM340366 3 0.5150 0.49528 0.000 0.160 0.620 0.220 0.000 0.000
#> GSM340367 1 0.3922 0.67612 0.664 0.000 0.320 0.000 0.000 0.016
#> GSM340368 4 0.2214 0.78905 0.000 0.000 0.016 0.888 0.000 0.096
#> GSM340369 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340370 1 0.3922 0.67612 0.664 0.000 0.320 0.000 0.000 0.016
#> GSM340371 5 0.0000 0.87599 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340372 1 0.3922 0.67612 0.664 0.000 0.320 0.000 0.000 0.016
#> GSM340373 1 0.3922 0.67612 0.664 0.000 0.320 0.000 0.000 0.016
#> GSM340375 1 0.2039 0.66591 0.904 0.000 0.076 0.000 0.000 0.020
#> GSM340376 6 0.1219 0.69706 0.004 0.000 0.048 0.000 0.000 0.948
#> GSM340378 1 0.3922 0.67612 0.664 0.000 0.320 0.000 0.000 0.016
#> GSM340243 4 0.0713 0.85619 0.000 0.000 0.000 0.972 0.000 0.028
#> GSM340244 6 0.2730 0.70854 0.000 0.000 0.000 0.192 0.000 0.808
#> GSM340246 6 0.3874 0.44288 0.000 0.000 0.356 0.008 0.000 0.636
#> GSM340247 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340248 4 0.2983 0.71277 0.000 0.000 0.032 0.832 0.000 0.136
#> GSM340249 4 0.1327 0.83725 0.000 0.000 0.000 0.936 0.000 0.064
#> GSM340250 4 0.3422 0.74515 0.000 0.032 0.036 0.832 0.000 0.100
#> GSM340251 4 0.0458 0.85818 0.000 0.000 0.016 0.984 0.000 0.000
#> GSM340252 2 0.0937 0.81076 0.000 0.960 0.040 0.000 0.000 0.000
#> GSM340253 2 0.0000 0.81930 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340254 2 0.0937 0.81076 0.000 0.960 0.040 0.000 0.000 0.000
#> GSM340256 6 0.3691 0.54808 0.000 0.260 0.008 0.008 0.000 0.724
#> GSM340258 4 0.5705 -0.00874 0.000 0.280 0.204 0.516 0.000 0.000
#> GSM340259 1 0.6360 -0.05523 0.392 0.352 0.008 0.004 0.000 0.244
#> GSM340260 5 0.0000 0.87599 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340261 4 0.0717 0.85947 0.000 0.000 0.008 0.976 0.000 0.016
#> GSM340262 2 0.1957 0.77350 0.000 0.888 0.112 0.000 0.000 0.000
#> GSM340263 6 0.2664 0.71323 0.000 0.000 0.000 0.184 0.000 0.816
#> GSM340264 5 0.0725 0.86697 0.012 0.012 0.000 0.000 0.976 0.000
#> GSM340265 5 0.2872 0.77827 0.000 0.024 0.140 0.000 0.836 0.000
#> GSM340266 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340267 3 0.4570 -0.06143 0.008 0.028 0.588 0.000 0.376 0.000
#> GSM340268 4 0.0146 0.86228 0.000 0.000 0.004 0.996 0.000 0.000
#> GSM340269 4 0.0260 0.86129 0.000 0.000 0.008 0.992 0.000 0.000
#> GSM340270 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM537574 4 0.4555 0.03758 0.000 0.000 0.036 0.540 0.000 0.424
#> GSM537580 5 0.0000 0.87599 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM537581 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340272 2 0.5393 0.45430 0.256 0.576 0.168 0.000 0.000 0.000
#> GSM340273 6 0.0458 0.71392 0.000 0.000 0.016 0.000 0.000 0.984
#> GSM340275 4 0.1204 0.84104 0.000 0.000 0.000 0.944 0.000 0.056
#> GSM340276 4 0.1387 0.83426 0.000 0.000 0.000 0.932 0.000 0.068
#> GSM340277 2 0.3864 0.22525 0.000 0.520 0.480 0.000 0.000 0.000
#> GSM340278 4 0.1219 0.84299 0.000 0.000 0.004 0.948 0.000 0.048
#> GSM340279 4 0.1806 0.80547 0.000 0.000 0.088 0.908 0.000 0.004
#> GSM340282 3 0.3789 -0.06175 0.000 0.416 0.584 0.000 0.000 0.000
#> GSM340284 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340285 4 0.2778 0.72878 0.000 0.000 0.008 0.824 0.000 0.168
#> GSM340286 2 0.0865 0.81221 0.000 0.964 0.036 0.000 0.000 0.000
#> GSM340287 4 0.1633 0.83997 0.000 0.000 0.024 0.932 0.000 0.044
#> GSM340288 1 0.2614 0.63877 0.884 0.012 0.044 0.000 0.000 0.060
#> GSM340289 1 0.3446 0.67396 0.692 0.000 0.308 0.000 0.000 0.000
#> GSM340290 4 0.1970 0.82165 0.000 0.000 0.060 0.912 0.000 0.028
#> GSM340291 6 0.2454 0.72376 0.000 0.000 0.000 0.160 0.000 0.840
#> GSM340293 4 0.0260 0.86194 0.000 0.000 0.000 0.992 0.000 0.008
#> GSM340294 4 0.1480 0.84781 0.000 0.000 0.020 0.940 0.000 0.040
#> GSM340296 3 0.4698 0.35578 0.000 0.000 0.504 0.452 0.000 0.044
#> GSM340297 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340298 4 0.3868 -0.32725 0.000 0.000 0.492 0.508 0.000 0.000
#> GSM340299 2 0.0000 0.81930 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340301 2 0.0260 0.81831 0.000 0.992 0.008 0.000 0.000 0.000
#> GSM340303 5 0.5624 0.40265 0.000 0.296 0.180 0.000 0.524 0.000
#> GSM340304 6 0.4175 0.67710 0.000 0.000 0.104 0.156 0.000 0.740
#> GSM340306 4 0.5060 0.49568 0.052 0.000 0.060 0.684 0.000 0.204
#> GSM340307 3 0.4090 0.51076 0.000 0.008 0.604 0.384 0.004 0.000
#> GSM340310 6 0.4294 0.34142 0.280 0.000 0.048 0.000 0.000 0.672
#> GSM340314 4 0.0146 0.86253 0.000 0.000 0.004 0.996 0.000 0.000
#> GSM340315 4 0.1141 0.83917 0.000 0.000 0.052 0.948 0.000 0.000
#> GSM340317 6 0.2941 0.69480 0.000 0.000 0.000 0.220 0.000 0.780
#> GSM340318 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340319 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340320 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340321 4 0.5005 0.37381 0.000 0.000 0.124 0.628 0.000 0.248
#> GSM340322 4 0.0458 0.85818 0.000 0.000 0.016 0.984 0.000 0.000
#> GSM340324 6 0.3051 0.68980 0.000 0.112 0.008 0.036 0.000 0.844
#> GSM340328 1 0.5778 0.62035 0.464 0.000 0.352 0.000 0.000 0.184
#> GSM340330 4 0.1556 0.81916 0.000 0.000 0.080 0.920 0.000 0.000
#> GSM340332 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340333 2 0.0000 0.81930 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340336 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340337 4 0.2494 0.75208 0.000 0.000 0.016 0.864 0.000 0.120
#> GSM340338 4 0.1421 0.83966 0.000 0.028 0.028 0.944 0.000 0.000
#> GSM340339 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340340 6 0.3998 0.18488 0.000 0.000 0.004 0.492 0.000 0.504
#> GSM340341 4 0.3351 0.72602 0.000 0.000 0.040 0.800 0.000 0.160
#> GSM340343 4 0.0508 0.86041 0.004 0.000 0.000 0.984 0.000 0.012
#> GSM340344 2 0.0000 0.81930 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340346 3 0.4624 0.54779 0.000 0.012 0.616 0.340 0.032 0.000
#> GSM340347 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340348 4 0.2814 0.72105 0.000 0.000 0.008 0.820 0.000 0.172
#> GSM340349 3 0.6421 -0.07148 0.068 0.000 0.524 0.032 0.324 0.052
#> GSM340350 3 0.4721 -0.53787 0.420 0.000 0.540 0.008 0.000 0.032
#> GSM340351 1 0.1995 0.64205 0.912 0.000 0.036 0.000 0.000 0.052
#> GSM340354 3 0.4833 0.49159 0.000 0.028 0.568 0.388 0.008 0.008
#> GSM340356 6 0.0790 0.70813 0.000 0.000 0.032 0.000 0.000 0.968
#> GSM340357 1 0.1575 0.64800 0.936 0.000 0.032 0.032 0.000 0.000
#> GSM348183 1 0.1983 0.62783 0.908 0.000 0.020 0.000 0.000 0.072
#> GSM348191 4 0.4731 -0.18489 0.000 0.000 0.428 0.524 0.000 0.048
#> GSM348193 6 0.1649 0.69680 0.032 0.000 0.036 0.000 0.000 0.932
#> GSM537578 3 0.3445 -0.22206 0.260 0.000 0.732 0.000 0.000 0.008
#> GSM348181 4 0.3862 0.34381 0.004 0.000 0.000 0.608 0.000 0.388
#> GSM348182 5 0.3351 0.57086 0.288 0.000 0.000 0.000 0.712 0.000
#> GSM348184 4 0.1765 0.80210 0.000 0.000 0.096 0.904 0.000 0.000
#> GSM348185 6 0.0000 0.71741 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM348186 2 0.1007 0.80958 0.000 0.956 0.044 0.000 0.000 0.000
#> GSM348187 1 0.7228 0.51494 0.348 0.000 0.336 0.100 0.000 0.216
#> GSM348188 2 0.7411 0.12138 0.280 0.388 0.024 0.060 0.000 0.248
#> GSM348189 5 0.0000 0.87599 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM348190 2 0.0458 0.81315 0.016 0.984 0.000 0.000 0.000 0.000
#> GSM348194 1 0.4481 0.49262 0.656 0.000 0.060 0.000 0.000 0.284
#> GSM348195 1 0.0713 0.64195 0.972 0.000 0.028 0.000 0.000 0.000
#> GSM348196 1 0.5932 0.61426 0.452 0.000 0.356 0.004 0.000 0.188
#> GSM537585 1 0.6094 0.03781 0.536 0.180 0.028 0.000 0.256 0.000
#> GSM537594 6 0.0146 0.71933 0.000 0.000 0.000 0.004 0.000 0.996
#> GSM537596 6 0.5208 0.16724 0.020 0.000 0.048 0.432 0.000 0.500
#> GSM537597 6 0.4331 0.49736 0.004 0.000 0.028 0.332 0.000 0.636
#> GSM537602 6 0.0260 0.71744 0.000 0.000 0.008 0.000 0.000 0.992
#> GSM340184 4 0.2136 0.82025 0.000 0.000 0.012 0.908 0.064 0.016
#> GSM340185 4 0.3854 -0.19625 0.000 0.000 0.464 0.536 0.000 0.000
#> GSM340186 4 0.2553 0.74102 0.000 0.008 0.144 0.848 0.000 0.000
#> GSM340187 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340189 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340190 3 0.3866 0.32128 0.000 0.000 0.516 0.484 0.000 0.000
#> GSM340191 4 0.0260 0.86169 0.000 0.000 0.000 0.992 0.000 0.008
#> GSM340192 3 0.3737 -0.00328 0.000 0.392 0.608 0.000 0.000 0.000
#> GSM340193 1 0.6588 0.59758 0.432 0.000 0.352 0.052 0.000 0.164
#> GSM340194 2 0.0000 0.81930 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340195 2 0.0260 0.81635 0.008 0.992 0.000 0.000 0.000 0.000
#> GSM340196 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340197 2 0.3288 0.57647 0.276 0.724 0.000 0.000 0.000 0.000
#> GSM340198 2 0.1007 0.80951 0.000 0.956 0.044 0.000 0.000 0.000
#> GSM340199 4 0.2996 0.56425 0.000 0.000 0.228 0.772 0.000 0.000
#> GSM340200 3 0.4289 0.44542 0.000 0.020 0.556 0.424 0.000 0.000
#> GSM340201 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340202 4 0.0000 0.86308 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM340203 4 0.0458 0.85818 0.000 0.000 0.016 0.984 0.000 0.000
#> GSM340204 2 0.0146 0.81909 0.000 0.996 0.004 0.000 0.000 0.000
#> GSM340205 6 0.2300 0.72694 0.000 0.000 0.000 0.144 0.000 0.856
#> GSM340206 3 0.4141 0.42450 0.000 0.000 0.556 0.432 0.000 0.012
#> GSM340207 2 0.4356 0.44896 0.000 0.608 0.360 0.000 0.032 0.000
#> GSM340237 6 0.2805 0.71298 0.000 0.000 0.004 0.184 0.000 0.812
#> GSM340238 4 0.0146 0.86228 0.000 0.000 0.004 0.996 0.000 0.000
#> GSM340239 2 0.5961 -0.08412 0.244 0.444 0.312 0.000 0.000 0.000
#> GSM340240 2 0.3607 0.50259 0.000 0.652 0.348 0.000 0.000 0.000
#> GSM340241 4 0.3773 0.59949 0.000 0.044 0.204 0.752 0.000 0.000
#> GSM340242 5 0.0000 0.87599 0.000 0.000 0.000 0.000 1.000 0.000
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
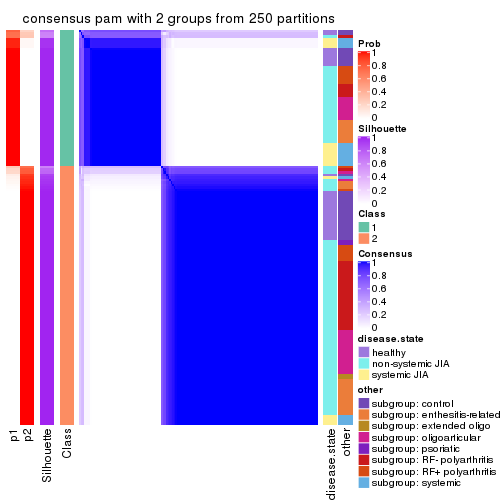
consensus_heatmap(res, k = 3)
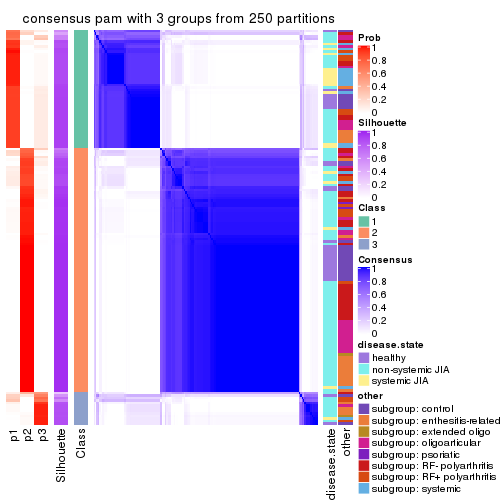
consensus_heatmap(res, k = 4)
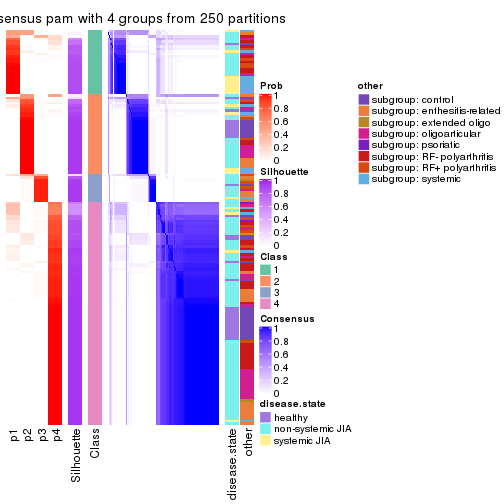
consensus_heatmap(res, k = 5)
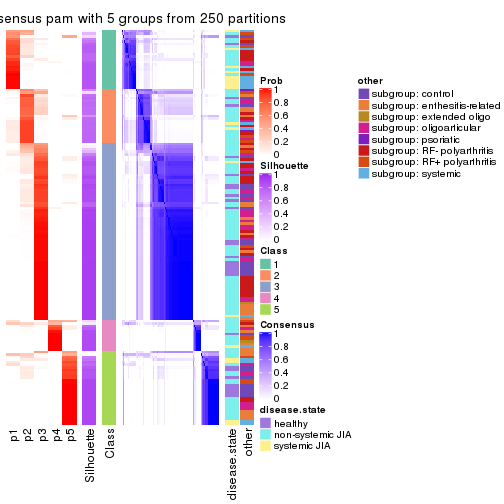
consensus_heatmap(res, k = 6)
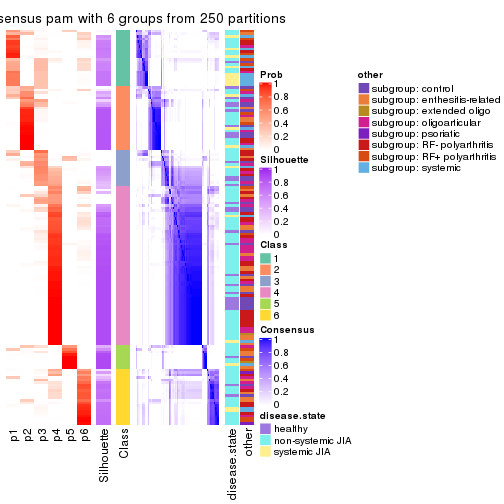
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
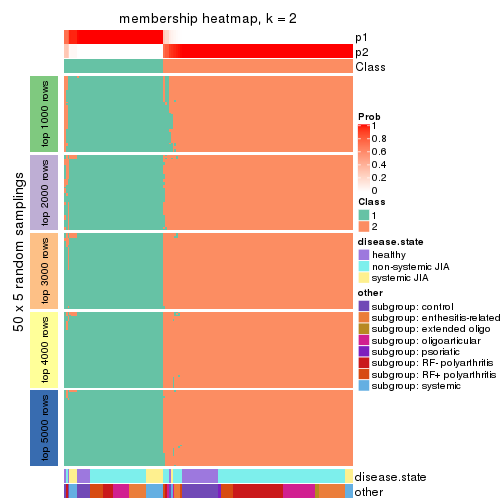
membership_heatmap(res, k = 3)
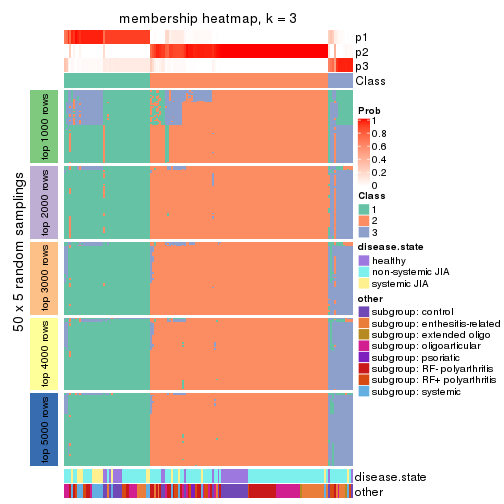
membership_heatmap(res, k = 4)
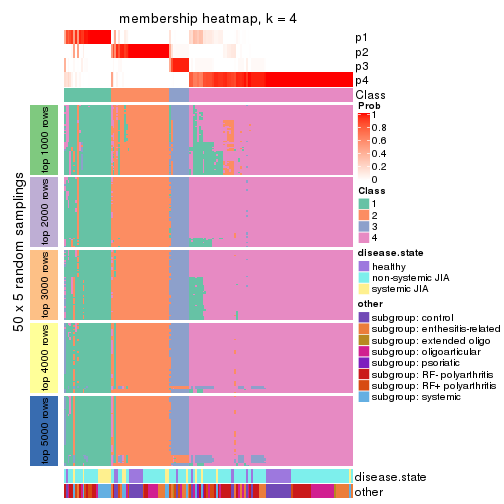
membership_heatmap(res, k = 5)
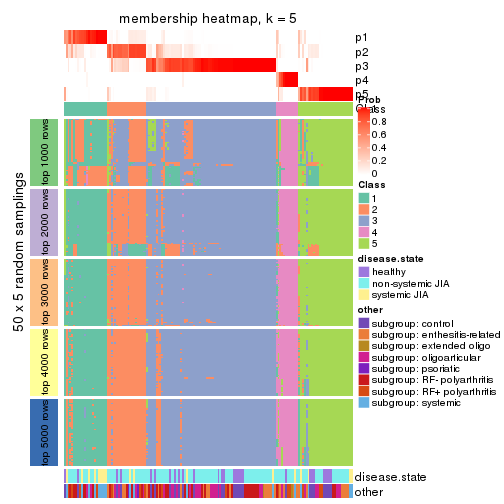
membership_heatmap(res, k = 6)
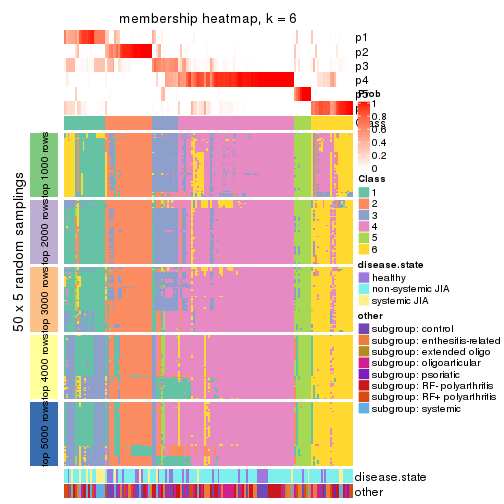
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
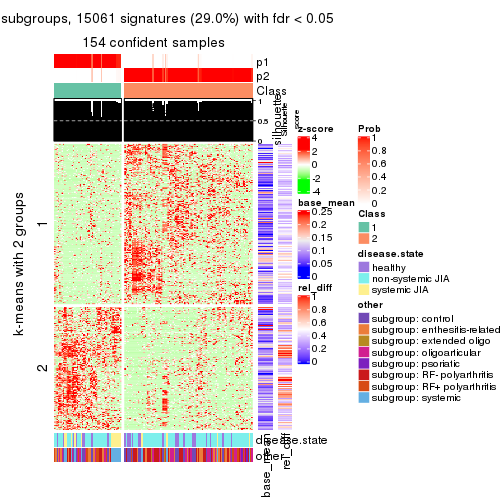
get_signatures(res, k = 3)
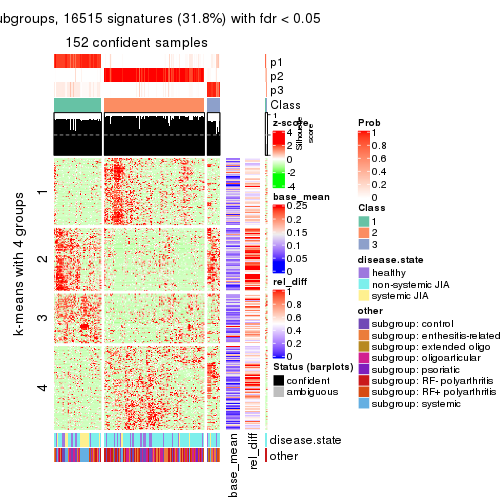
get_signatures(res, k = 4)
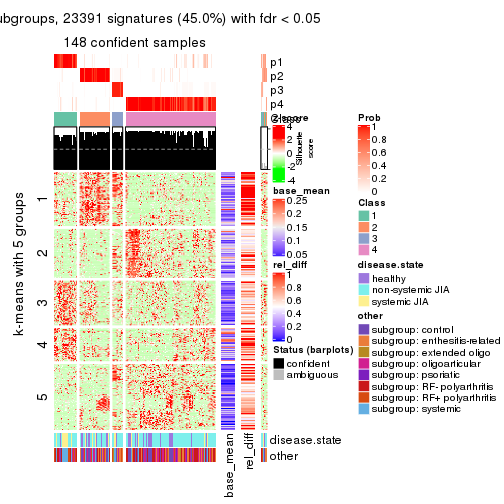
get_signatures(res, k = 5)
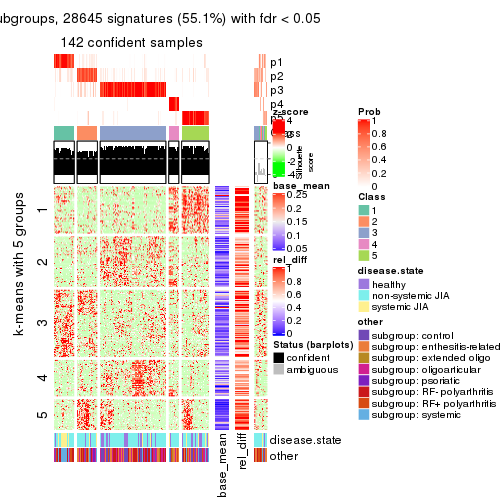
get_signatures(res, k = 6)
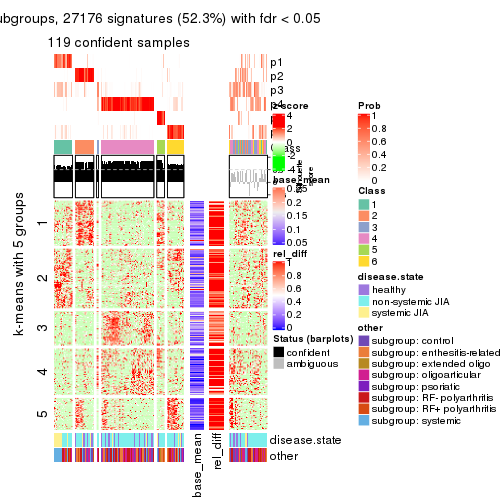
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
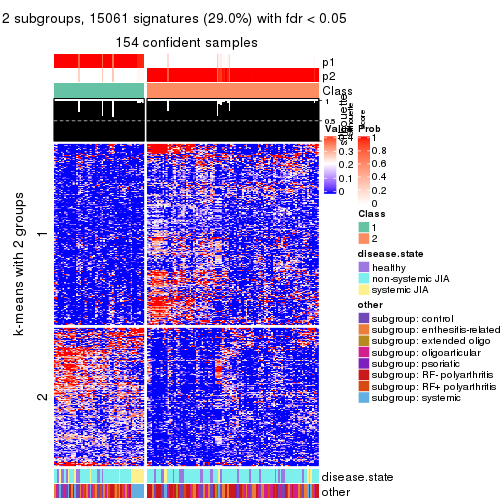
get_signatures(res, k = 3, scale_rows = FALSE)
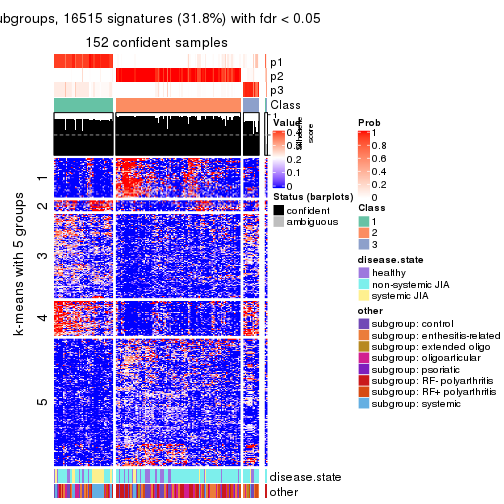
get_signatures(res, k = 4, scale_rows = FALSE)
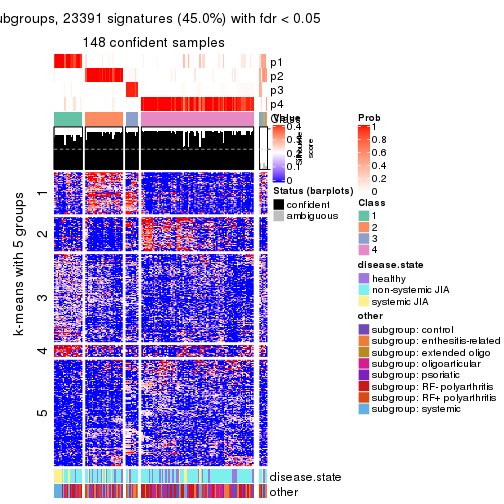
get_signatures(res, k = 5, scale_rows = FALSE)
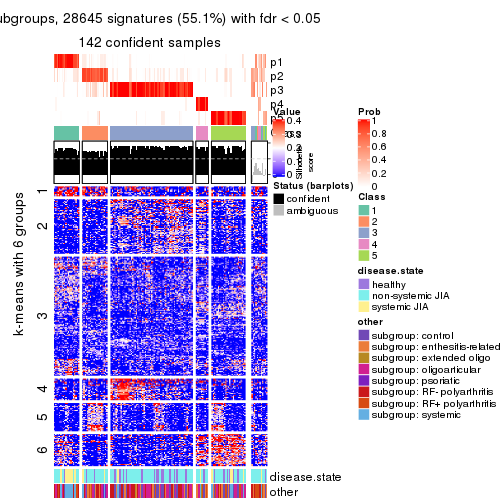
get_signatures(res, k = 6, scale_rows = FALSE)
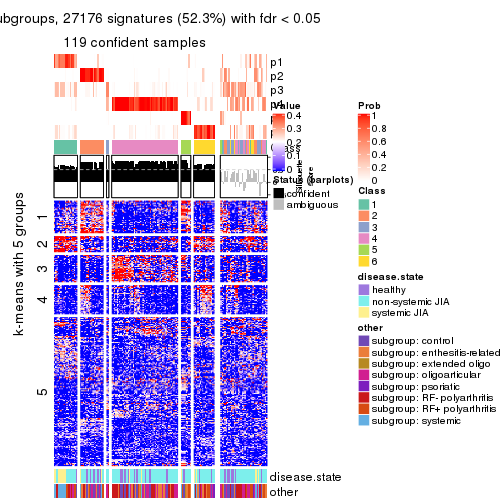
Compare the overlap of signatures from different k:
compare_signatures(res)
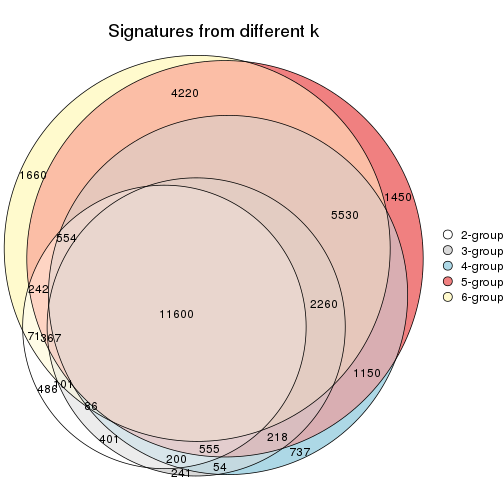
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
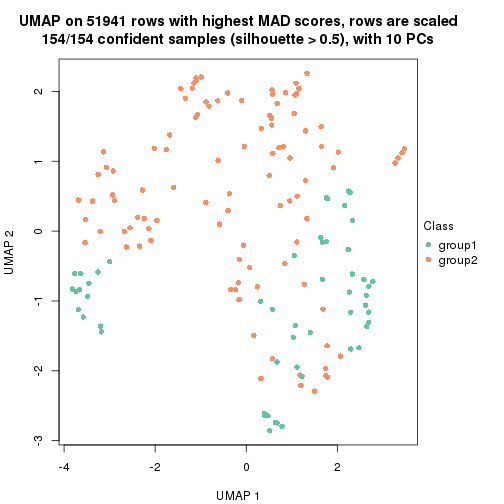
dimension_reduction(res, k = 3, method = "UMAP")
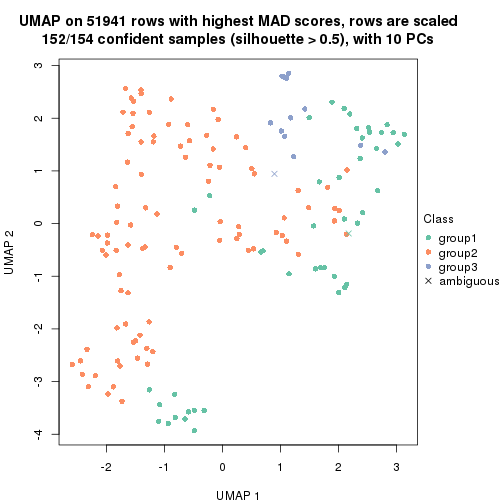
dimension_reduction(res, k = 4, method = "UMAP")
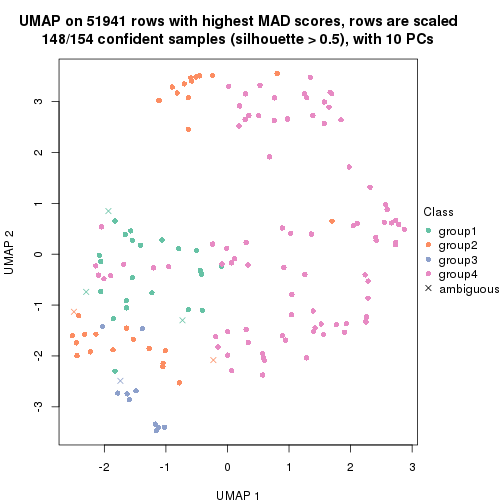
dimension_reduction(res, k = 5, method = "UMAP")
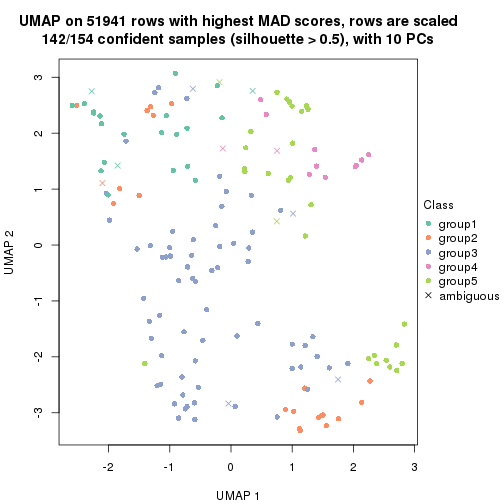
dimension_reduction(res, k = 6, method = "UMAP")
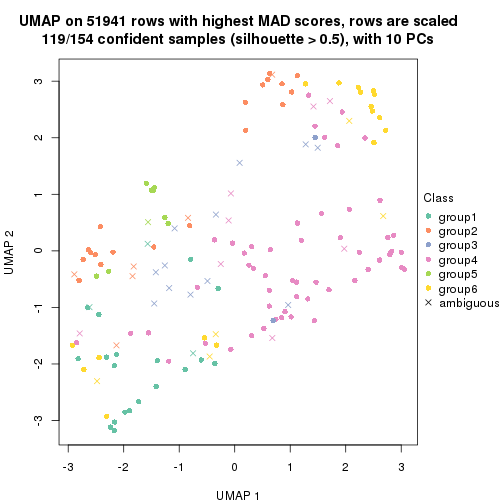
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
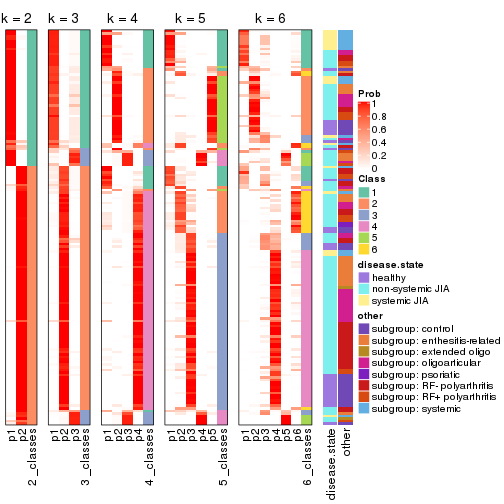
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> MAD:pam 154 0.00154 8.37e-03 2
#> MAD:pam 152 0.00110 1.09e-03 3
#> MAD:pam 148 0.00179 1.68e-03 4
#> MAD:pam 142 0.00134 9.55e-05 5
#> MAD:pam 119 0.00201 6.29e-03 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["MAD", "mclust"]
# you can also extract it by
# res = res_list["MAD:mclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'MAD' method.
#> Subgroups are detected by 'mclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 4.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
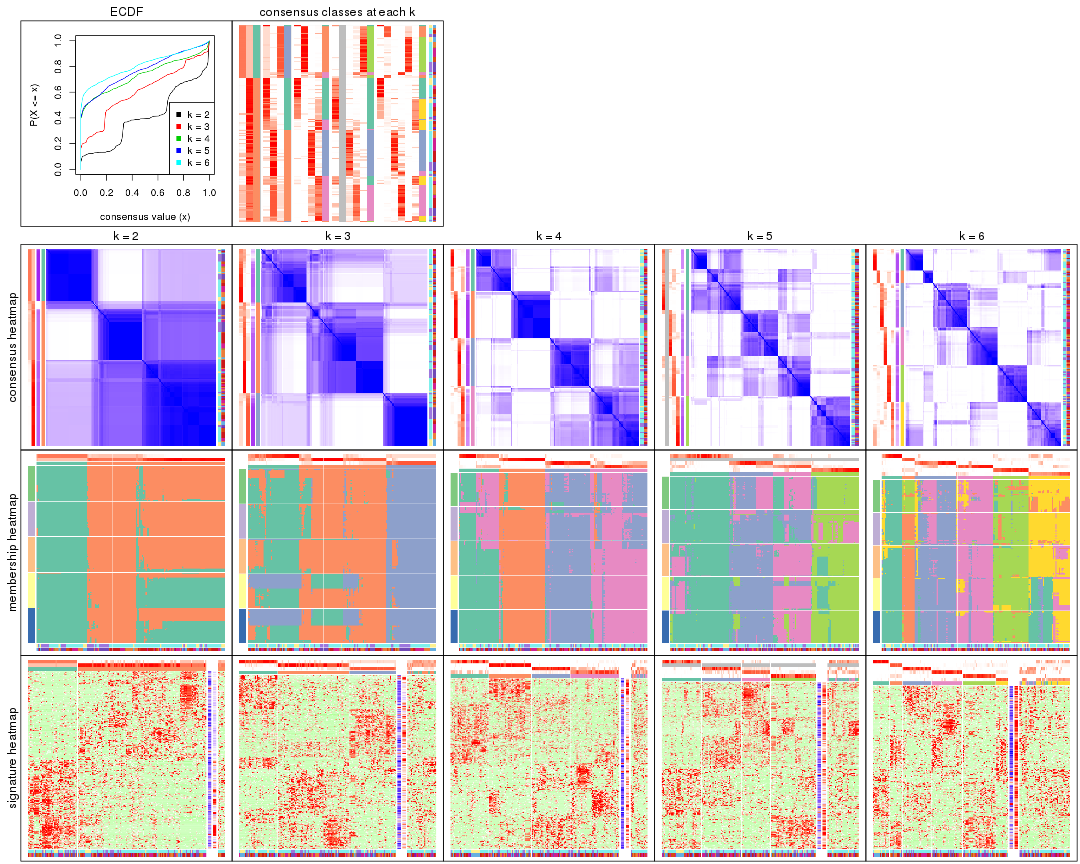
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
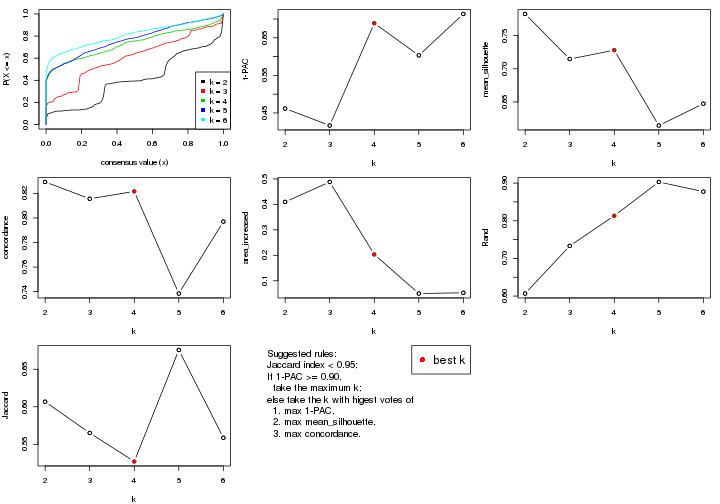
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.462 0.783 0.829 0.4097 0.607 0.607
#> 3 3 0.417 0.715 0.816 0.4881 0.733 0.565
#> 4 4 0.689 0.728 0.822 0.2033 0.813 0.527
#> 5 5 0.603 0.614 0.738 0.0499 0.903 0.676
#> 6 6 0.713 0.647 0.797 0.0528 0.877 0.559
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 4
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 2 0.0672 0.7895 0.008 0.992
#> GSM340359 1 0.9000 0.9925 0.684 0.316
#> GSM340361 1 0.9775 0.8392 0.588 0.412
#> GSM340362 1 0.9000 0.9925 0.684 0.316
#> GSM340363 2 0.0376 0.7909 0.004 0.996
#> GSM340364 2 0.1633 0.7782 0.024 0.976
#> GSM340365 1 0.9000 0.9925 0.684 0.316
#> GSM340366 1 0.9087 0.9876 0.676 0.324
#> GSM340367 2 0.0938 0.7881 0.012 0.988
#> GSM340368 2 0.2778 0.7927 0.048 0.952
#> GSM340369 2 0.9000 0.7215 0.316 0.684
#> GSM340370 2 0.0938 0.7881 0.012 0.988
#> GSM340371 1 0.9044 0.9921 0.680 0.320
#> GSM340372 2 0.1184 0.7854 0.016 0.984
#> GSM340373 2 0.1184 0.7854 0.016 0.984
#> GSM340375 2 0.1184 0.7854 0.016 0.984
#> GSM340376 2 0.0672 0.7895 0.008 0.992
#> GSM340378 2 0.0672 0.7895 0.008 0.992
#> GSM340243 2 0.1414 0.7949 0.020 0.980
#> GSM340244 2 0.9000 0.7215 0.316 0.684
#> GSM340246 2 0.8763 0.7289 0.296 0.704
#> GSM340247 2 0.9000 0.7215 0.316 0.684
#> GSM340248 2 0.9000 0.7215 0.316 0.684
#> GSM340249 2 0.9000 0.7215 0.316 0.684
#> GSM340250 2 0.0672 0.7895 0.008 0.992
#> GSM340251 2 0.9000 0.7215 0.316 0.684
#> GSM340252 1 0.9044 0.9921 0.680 0.320
#> GSM340253 1 0.9000 0.9925 0.684 0.316
#> GSM340254 1 0.9044 0.9921 0.680 0.320
#> GSM340256 1 0.9044 0.9921 0.680 0.320
#> GSM340258 2 0.9933 -0.5031 0.452 0.548
#> GSM340259 2 0.2778 0.7511 0.048 0.952
#> GSM340260 1 0.9000 0.9925 0.684 0.316
#> GSM340261 2 0.0376 0.7934 0.004 0.996
#> GSM340262 1 0.9044 0.9921 0.680 0.320
#> GSM340263 2 0.3114 0.7921 0.056 0.944
#> GSM340264 1 0.9000 0.9925 0.684 0.316
#> GSM340265 1 0.9000 0.9925 0.684 0.316
#> GSM340266 2 0.9000 0.7215 0.316 0.684
#> GSM340267 2 0.9909 -0.4849 0.444 0.556
#> GSM340268 2 0.9000 0.7215 0.316 0.684
#> GSM340269 2 0.0376 0.7934 0.004 0.996
#> GSM340270 2 0.0000 0.7923 0.000 1.000
#> GSM537574 2 0.8499 0.7304 0.276 0.724
#> GSM537580 1 0.9044 0.9921 0.680 0.320
#> GSM537581 2 0.9000 0.7215 0.316 0.684
#> GSM340272 1 0.9000 0.9925 0.684 0.316
#> GSM340273 2 0.2948 0.7925 0.052 0.948
#> GSM340275 2 0.9000 0.7215 0.316 0.684
#> GSM340276 2 0.0000 0.7923 0.000 1.000
#> GSM340277 1 0.9044 0.9921 0.680 0.320
#> GSM340278 2 0.0376 0.7934 0.004 0.996
#> GSM340279 2 0.8608 0.7320 0.284 0.716
#> GSM340282 1 0.9000 0.9925 0.684 0.316
#> GSM340284 2 0.9000 0.7215 0.316 0.684
#> GSM340285 2 0.0000 0.7923 0.000 1.000
#> GSM340286 1 0.9044 0.9921 0.680 0.320
#> GSM340287 2 0.0672 0.7895 0.008 0.992
#> GSM340288 2 0.1184 0.7854 0.016 0.984
#> GSM340289 2 0.1184 0.7854 0.016 0.984
#> GSM340290 2 0.0000 0.7923 0.000 1.000
#> GSM340291 2 0.9000 0.7215 0.316 0.684
#> GSM340293 2 0.9000 0.7215 0.316 0.684
#> GSM340294 2 0.2778 0.7927 0.048 0.952
#> GSM340296 2 0.3114 0.7927 0.056 0.944
#> GSM340297 2 0.0376 0.7934 0.004 0.996
#> GSM340298 2 0.0938 0.7933 0.012 0.988
#> GSM340299 1 0.9000 0.9925 0.684 0.316
#> GSM340301 1 0.9000 0.9925 0.684 0.316
#> GSM340303 1 0.9000 0.9925 0.684 0.316
#> GSM340304 2 0.0000 0.7923 0.000 1.000
#> GSM340306 2 0.0938 0.7869 0.012 0.988
#> GSM340307 2 0.3879 0.7161 0.076 0.924
#> GSM340310 2 0.0672 0.7895 0.008 0.992
#> GSM340314 2 0.1184 0.7949 0.016 0.984
#> GSM340315 2 0.4161 0.7892 0.084 0.916
#> GSM340317 2 0.3584 0.7907 0.068 0.932
#> GSM340318 2 0.9000 0.7215 0.316 0.684
#> GSM340319 2 0.9000 0.7215 0.316 0.684
#> GSM340320 2 0.0672 0.7941 0.008 0.992
#> GSM340321 2 0.9000 0.7215 0.316 0.684
#> GSM340322 1 0.9044 0.9838 0.680 0.320
#> GSM340324 2 0.5946 0.5918 0.144 0.856
#> GSM340328 2 0.0672 0.7895 0.008 0.992
#> GSM340330 2 0.9044 0.0182 0.320 0.680
#> GSM340332 2 0.9000 0.7215 0.316 0.684
#> GSM340333 1 0.9000 0.9925 0.684 0.316
#> GSM340336 2 0.9000 0.7215 0.316 0.684
#> GSM340337 2 0.6531 0.7509 0.168 0.832
#> GSM340338 2 0.9286 -0.0893 0.344 0.656
#> GSM340339 2 0.9000 0.7215 0.316 0.684
#> GSM340340 2 0.9000 0.7215 0.316 0.684
#> GSM340341 2 0.5408 0.7780 0.124 0.876
#> GSM340343 2 0.0000 0.7923 0.000 1.000
#> GSM340344 1 0.9000 0.9925 0.684 0.316
#> GSM340346 2 0.3584 0.7227 0.068 0.932
#> GSM340347 2 0.9000 0.7215 0.316 0.684
#> GSM340348 2 0.3431 0.7913 0.064 0.936
#> GSM340349 2 0.0672 0.7895 0.008 0.992
#> GSM340350 2 0.0672 0.7895 0.008 0.992
#> GSM340351 2 0.1184 0.7854 0.016 0.984
#> GSM340354 2 0.9286 -0.1050 0.344 0.656
#> GSM340356 2 0.2948 0.7926 0.052 0.948
#> GSM340357 2 0.0672 0.7895 0.008 0.992
#> GSM348183 2 0.1184 0.7854 0.016 0.984
#> GSM348191 2 0.0376 0.7909 0.004 0.996
#> GSM348193 2 0.0672 0.7895 0.008 0.992
#> GSM537578 2 0.1184 0.7854 0.016 0.984
#> GSM348181 2 0.0672 0.7895 0.008 0.992
#> GSM348182 1 0.9000 0.9925 0.684 0.316
#> GSM348184 2 0.9000 0.7215 0.316 0.684
#> GSM348185 2 0.2778 0.7930 0.048 0.952
#> GSM348186 1 0.9044 0.9921 0.680 0.320
#> GSM348187 2 0.0672 0.7895 0.008 0.992
#> GSM348188 2 0.9286 -0.0954 0.344 0.656
#> GSM348189 1 0.9044 0.9921 0.680 0.320
#> GSM348190 1 0.9000 0.9925 0.684 0.316
#> GSM348194 2 0.1184 0.7854 0.016 0.984
#> GSM348195 2 0.1184 0.7854 0.016 0.984
#> GSM348196 2 0.0672 0.7895 0.008 0.992
#> GSM537585 1 0.9000 0.9925 0.684 0.316
#> GSM537594 2 0.2948 0.7926 0.052 0.948
#> GSM537596 2 0.0376 0.7909 0.004 0.996
#> GSM537597 2 0.0000 0.7923 0.000 1.000
#> GSM537602 2 0.0672 0.7895 0.008 0.992
#> GSM340184 2 0.0376 0.7909 0.004 0.996
#> GSM340185 2 0.8443 0.7305 0.272 0.728
#> GSM340186 2 0.9922 0.5501 0.448 0.552
#> GSM340187 2 0.9000 0.7215 0.316 0.684
#> GSM340189 2 0.9000 0.7215 0.316 0.684
#> GSM340190 2 0.8763 0.7266 0.296 0.704
#> GSM340191 2 0.9000 0.7215 0.316 0.684
#> GSM340192 1 0.9044 0.9921 0.680 0.320
#> GSM340193 2 0.0938 0.7881 0.012 0.988
#> GSM340194 1 0.9000 0.9925 0.684 0.316
#> GSM340195 1 0.9000 0.9925 0.684 0.316
#> GSM340196 2 0.9000 0.7215 0.316 0.684
#> GSM340197 1 0.9000 0.9925 0.684 0.316
#> GSM340198 1 0.9044 0.9921 0.680 0.320
#> GSM340199 2 0.7528 0.7539 0.216 0.784
#> GSM340200 2 0.8608 0.7110 0.284 0.716
#> GSM340201 2 0.9000 0.7215 0.316 0.684
#> GSM340202 2 0.9000 0.7215 0.316 0.684
#> GSM340203 2 0.9000 0.7215 0.316 0.684
#> GSM340204 1 0.9044 0.9921 0.680 0.320
#> GSM340205 2 0.0672 0.7895 0.008 0.992
#> GSM340206 2 0.7299 0.7595 0.204 0.796
#> GSM340207 1 0.9044 0.9921 0.680 0.320
#> GSM340237 2 0.9000 0.7215 0.316 0.684
#> GSM340238 2 0.8386 0.7382 0.268 0.732
#> GSM340239 1 0.9000 0.9925 0.684 0.316
#> GSM340240 1 0.9044 0.9921 0.680 0.320
#> GSM340241 1 0.9087 0.9619 0.676 0.324
#> GSM340242 1 0.9044 0.9921 0.680 0.320
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.1753 0.7873 0.952 0.048 0.000
#> GSM340359 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340361 1 0.0237 0.7649 0.996 0.000 0.004
#> GSM340362 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340363 1 0.4796 0.6749 0.780 0.220 0.000
#> GSM340364 1 0.0237 0.7649 0.996 0.000 0.004
#> GSM340365 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340366 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340367 1 0.0475 0.7675 0.992 0.004 0.004
#> GSM340368 2 0.9485 0.1404 0.388 0.428 0.184
#> GSM340369 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340370 1 0.0475 0.7675 0.992 0.004 0.004
#> GSM340371 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340372 1 0.0237 0.7649 0.996 0.000 0.004
#> GSM340373 1 0.0237 0.7649 0.996 0.000 0.004
#> GSM340375 1 0.3112 0.7511 0.900 0.096 0.004
#> GSM340376 1 0.5024 0.6805 0.776 0.220 0.004
#> GSM340378 1 0.1989 0.7871 0.948 0.048 0.004
#> GSM340243 2 0.5138 0.6032 0.252 0.748 0.000
#> GSM340244 2 0.7862 0.6617 0.148 0.668 0.184
#> GSM340246 2 0.0237 0.7938 0.004 0.996 0.000
#> GSM340247 2 0.4346 0.7863 0.000 0.816 0.184
#> GSM340248 2 0.4346 0.7863 0.000 0.816 0.184
#> GSM340249 2 0.5331 0.7769 0.024 0.792 0.184
#> GSM340250 2 0.5443 0.5950 0.260 0.736 0.004
#> GSM340251 2 0.3752 0.7918 0.000 0.856 0.144
#> GSM340252 3 0.4915 0.9276 0.184 0.012 0.804
#> GSM340253 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340254 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340256 3 0.8482 0.6397 0.184 0.200 0.616
#> GSM340258 2 0.8746 0.3646 0.184 0.588 0.228
#> GSM340259 1 0.3295 0.7683 0.896 0.096 0.008
#> GSM340260 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340261 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340262 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340263 2 0.9485 0.1404 0.388 0.428 0.184
#> GSM340264 3 0.4887 0.9052 0.228 0.000 0.772
#> GSM340265 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340266 2 0.5428 0.7744 0.064 0.816 0.120
#> GSM340267 3 0.9400 0.4968 0.228 0.264 0.508
#> GSM340268 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340269 2 0.4346 0.6742 0.184 0.816 0.000
#> GSM340270 2 0.6295 0.0257 0.472 0.528 0.000
#> GSM537574 2 0.4346 0.7863 0.000 0.816 0.184
#> GSM537580 3 0.6962 0.8240 0.184 0.092 0.724
#> GSM537581 2 0.5331 0.7772 0.024 0.792 0.184
#> GSM340272 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340273 2 0.9492 0.1068 0.400 0.416 0.184
#> GSM340275 2 0.4915 0.7827 0.012 0.804 0.184
#> GSM340276 1 0.6252 0.2519 0.556 0.444 0.000
#> GSM340277 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340278 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340279 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340282 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340284 2 0.4346 0.7863 0.000 0.816 0.184
#> GSM340285 1 0.6111 0.3844 0.604 0.396 0.000
#> GSM340286 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340287 2 0.5443 0.5855 0.260 0.736 0.004
#> GSM340288 1 0.0237 0.7649 0.996 0.000 0.004
#> GSM340289 1 0.0237 0.7649 0.996 0.000 0.004
#> GSM340290 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340291 2 0.9433 0.2358 0.356 0.460 0.184
#> GSM340293 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340294 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340296 2 0.0237 0.7928 0.000 0.996 0.004
#> GSM340297 2 0.5882 0.4430 0.348 0.652 0.000
#> GSM340298 2 0.0237 0.7928 0.000 0.996 0.004
#> GSM340299 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340301 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340303 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340304 2 0.6308 -0.0452 0.492 0.508 0.000
#> GSM340306 1 0.3425 0.7529 0.884 0.112 0.004
#> GSM340307 2 0.4413 0.7202 0.124 0.852 0.024
#> GSM340310 1 0.1753 0.7873 0.952 0.048 0.000
#> GSM340314 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340315 2 0.0237 0.7928 0.000 0.996 0.004
#> GSM340317 2 0.9485 0.1404 0.388 0.428 0.184
#> GSM340318 2 0.4346 0.7863 0.000 0.816 0.184
#> GSM340319 2 0.4346 0.7863 0.000 0.816 0.184
#> GSM340320 1 0.6215 0.2951 0.572 0.428 0.000
#> GSM340321 2 0.4346 0.7863 0.000 0.816 0.184
#> GSM340322 3 0.8659 0.5799 0.176 0.228 0.596
#> GSM340324 1 0.6713 0.3070 0.572 0.416 0.012
#> GSM340328 1 0.1753 0.7873 0.952 0.048 0.000
#> GSM340330 2 0.6192 0.6181 0.060 0.764 0.176
#> GSM340332 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340333 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340336 2 0.4346 0.7863 0.000 0.816 0.184
#> GSM340337 2 0.4629 0.7853 0.004 0.808 0.188
#> GSM340338 2 0.8953 0.3283 0.180 0.560 0.260
#> GSM340339 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340340 2 0.4346 0.7863 0.000 0.816 0.184
#> GSM340341 2 0.5235 0.7827 0.036 0.812 0.152
#> GSM340343 1 0.5650 0.5607 0.688 0.312 0.000
#> GSM340344 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340346 2 0.6388 0.6153 0.184 0.752 0.064
#> GSM340347 2 0.4575 0.7854 0.004 0.812 0.184
#> GSM340348 2 0.8007 0.5805 0.244 0.640 0.116
#> GSM340349 2 0.6033 0.4483 0.336 0.660 0.004
#> GSM340350 1 0.5365 0.6268 0.744 0.252 0.004
#> GSM340351 1 0.0237 0.7649 0.996 0.000 0.004
#> GSM340354 2 0.7875 0.5012 0.176 0.668 0.156
#> GSM340356 1 0.8039 0.1517 0.508 0.428 0.064
#> GSM340357 1 0.1989 0.7871 0.948 0.048 0.004
#> GSM348183 1 0.0237 0.7649 0.996 0.000 0.004
#> GSM348191 2 0.0237 0.7928 0.000 0.996 0.004
#> GSM348193 1 0.1989 0.7871 0.948 0.048 0.004
#> GSM537578 1 0.6264 0.2964 0.616 0.380 0.004
#> GSM348181 1 0.1860 0.7873 0.948 0.052 0.000
#> GSM348182 3 0.5529 0.8169 0.296 0.000 0.704
#> GSM348184 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM348185 1 0.8527 0.1570 0.504 0.400 0.096
#> GSM348186 3 0.7316 0.7933 0.184 0.112 0.704
#> GSM348187 1 0.1989 0.7871 0.948 0.048 0.004
#> GSM348188 1 0.3193 0.6934 0.896 0.004 0.100
#> GSM348189 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM348190 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM348194 1 0.0237 0.7649 0.996 0.000 0.004
#> GSM348195 1 0.0237 0.7649 0.996 0.000 0.004
#> GSM348196 1 0.1753 0.7873 0.952 0.048 0.000
#> GSM537585 3 0.4931 0.9017 0.232 0.000 0.768
#> GSM537594 1 0.6565 0.2962 0.576 0.416 0.008
#> GSM537596 1 0.6451 0.2601 0.560 0.436 0.004
#> GSM537597 1 0.6305 0.1038 0.516 0.484 0.000
#> GSM537602 1 0.3349 0.7703 0.888 0.108 0.004
#> GSM340184 2 0.0237 0.7928 0.000 0.996 0.004
#> GSM340185 2 0.4346 0.7863 0.000 0.816 0.184
#> GSM340186 2 0.4953 0.6779 0.176 0.808 0.016
#> GSM340187 2 0.4346 0.7863 0.000 0.816 0.184
#> GSM340189 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340190 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340191 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340192 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340193 1 0.1989 0.7871 0.948 0.048 0.004
#> GSM340194 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340195 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340196 2 0.4346 0.7863 0.000 0.816 0.184
#> GSM340197 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340198 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340199 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340200 2 0.4953 0.6712 0.176 0.808 0.016
#> GSM340201 2 0.4755 0.7842 0.008 0.808 0.184
#> GSM340202 2 0.4346 0.7863 0.000 0.816 0.184
#> GSM340203 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340204 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340205 1 0.4733 0.7037 0.800 0.196 0.004
#> GSM340206 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340207 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340237 2 0.9145 0.4198 0.284 0.532 0.184
#> GSM340238 2 0.0000 0.7939 0.000 1.000 0.000
#> GSM340239 3 0.5529 0.8158 0.296 0.000 0.704
#> GSM340240 3 0.4346 0.9394 0.184 0.000 0.816
#> GSM340241 3 0.9383 0.2851 0.176 0.364 0.460
#> GSM340242 3 0.4346 0.9394 0.184 0.000 0.816
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.4964 0.7582 0.616 0.000 0.004 0.380
#> GSM340359 2 0.0707 0.9443 0.000 0.980 0.000 0.020
#> GSM340361 1 0.0779 0.7460 0.980 0.004 0.000 0.016
#> GSM340362 2 0.0707 0.9443 0.000 0.980 0.000 0.020
#> GSM340363 1 0.5028 0.7399 0.596 0.000 0.004 0.400
#> GSM340364 1 0.0000 0.7544 1.000 0.000 0.000 0.000
#> GSM340365 2 0.2796 0.8814 0.092 0.892 0.000 0.016
#> GSM340366 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM340367 1 0.1637 0.7674 0.940 0.000 0.000 0.060
#> GSM340368 4 0.1824 0.5692 0.060 0.000 0.004 0.936
#> GSM340369 3 0.1867 0.8734 0.000 0.000 0.928 0.072
#> GSM340370 1 0.1557 0.7643 0.944 0.000 0.000 0.056
#> GSM340371 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM340372 1 0.0000 0.7544 1.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.7544 1.000 0.000 0.000 0.000
#> GSM340375 1 0.0707 0.7595 0.980 0.000 0.000 0.020
#> GSM340376 4 0.1716 0.5646 0.064 0.000 0.000 0.936
#> GSM340378 1 0.4790 0.7603 0.620 0.000 0.000 0.380
#> GSM340243 3 0.4817 0.4317 0.000 0.000 0.612 0.388
#> GSM340244 4 0.1118 0.6226 0.000 0.000 0.036 0.964
#> GSM340246 3 0.1557 0.8796 0.000 0.000 0.944 0.056
#> GSM340247 4 0.4790 0.5921 0.000 0.000 0.380 0.620
#> GSM340248 4 0.4543 0.6293 0.000 0.000 0.324 0.676
#> GSM340249 4 0.4164 0.6572 0.000 0.000 0.264 0.736
#> GSM340250 4 0.4420 0.4559 0.012 0.000 0.240 0.748
#> GSM340251 4 0.4877 0.5746 0.000 0.000 0.408 0.592
#> GSM340252 4 0.4985 0.0854 0.000 0.468 0.000 0.532
#> GSM340253 2 0.0707 0.9443 0.000 0.980 0.000 0.020
#> GSM340254 2 0.0469 0.9458 0.000 0.988 0.000 0.012
#> GSM340256 4 0.6192 0.5484 0.000 0.244 0.104 0.652
#> GSM340258 4 0.6042 0.5669 0.000 0.048 0.392 0.560
#> GSM340259 1 0.5247 0.7734 0.684 0.032 0.000 0.284
#> GSM340260 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM340261 3 0.1557 0.8782 0.000 0.000 0.944 0.056
#> GSM340262 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM340263 4 0.1890 0.5735 0.056 0.000 0.008 0.936
#> GSM340264 2 0.3123 0.8235 0.156 0.844 0.000 0.000
#> GSM340265 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM340266 4 0.4746 0.6027 0.000 0.000 0.368 0.632
#> GSM340267 2 0.3088 0.8340 0.000 0.864 0.128 0.008
#> GSM340268 3 0.1211 0.8790 0.000 0.000 0.960 0.040
#> GSM340269 3 0.2345 0.8234 0.000 0.000 0.900 0.100
#> GSM340270 4 0.3856 0.4563 0.136 0.000 0.032 0.832
#> GSM537574 4 0.4830 0.5818 0.000 0.000 0.392 0.608
#> GSM537580 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM537581 4 0.3311 0.6730 0.000 0.000 0.172 0.828
#> GSM340272 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM340273 4 0.1824 0.5692 0.060 0.000 0.004 0.936
#> GSM340275 4 0.4008 0.6605 0.000 0.000 0.244 0.756
#> GSM340276 1 0.7772 0.4405 0.392 0.000 0.240 0.368
#> GSM340277 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM340278 3 0.2647 0.8347 0.000 0.000 0.880 0.120
#> GSM340279 3 0.1389 0.8784 0.000 0.000 0.952 0.048
#> GSM340282 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM340284 4 0.4855 0.5716 0.000 0.000 0.400 0.600
#> GSM340285 3 0.6508 0.3538 0.084 0.000 0.556 0.360
#> GSM340286 2 0.0336 0.9463 0.000 0.992 0.000 0.008
#> GSM340287 3 0.2921 0.8081 0.000 0.000 0.860 0.140
#> GSM340288 1 0.0000 0.7544 1.000 0.000 0.000 0.000
#> GSM340289 1 0.0000 0.7544 1.000 0.000 0.000 0.000
#> GSM340290 3 0.1557 0.8795 0.000 0.000 0.944 0.056
#> GSM340291 4 0.1488 0.6147 0.012 0.000 0.032 0.956
#> GSM340293 3 0.1474 0.8781 0.000 0.000 0.948 0.052
#> GSM340294 3 0.1302 0.8788 0.000 0.000 0.956 0.044
#> GSM340296 3 0.2647 0.8328 0.000 0.000 0.880 0.120
#> GSM340297 3 0.4643 0.4952 0.000 0.000 0.656 0.344
#> GSM340298 3 0.0469 0.8716 0.000 0.000 0.988 0.012
#> GSM340299 2 0.0707 0.9443 0.000 0.980 0.000 0.020
#> GSM340301 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM340303 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM340304 4 0.4793 0.2640 0.204 0.000 0.040 0.756
#> GSM340306 1 0.4522 0.7757 0.680 0.000 0.000 0.320
#> GSM340307 3 0.0188 0.8640 0.000 0.000 0.996 0.004
#> GSM340310 1 0.4790 0.7603 0.620 0.000 0.000 0.380
#> GSM340314 3 0.1716 0.8762 0.000 0.000 0.936 0.064
#> GSM340315 3 0.3400 0.7597 0.000 0.000 0.820 0.180
#> GSM340317 4 0.1576 0.5806 0.048 0.000 0.004 0.948
#> GSM340318 4 0.4817 0.5859 0.000 0.000 0.388 0.612
#> GSM340319 4 0.4790 0.5921 0.000 0.000 0.380 0.620
#> GSM340320 4 0.5000 -0.2205 0.000 0.000 0.496 0.504
#> GSM340321 4 0.4830 0.5818 0.000 0.000 0.392 0.608
#> GSM340322 4 0.7173 0.5318 0.000 0.228 0.216 0.556
#> GSM340324 4 0.2222 0.5650 0.056 0.008 0.008 0.928
#> GSM340328 1 0.4790 0.7603 0.620 0.000 0.000 0.380
#> GSM340330 3 0.0188 0.8640 0.000 0.000 0.996 0.004
#> GSM340332 3 0.1716 0.8762 0.000 0.000 0.936 0.064
#> GSM340333 2 0.0707 0.9443 0.000 0.980 0.000 0.020
#> GSM340336 4 0.4790 0.5921 0.000 0.000 0.380 0.620
#> GSM340337 4 0.4739 0.6599 0.012 0.008 0.240 0.740
#> GSM340338 3 0.2530 0.7797 0.000 0.100 0.896 0.004
#> GSM340339 3 0.2216 0.8585 0.000 0.000 0.908 0.092
#> GSM340340 4 0.3356 0.6734 0.000 0.000 0.176 0.824
#> GSM340341 4 0.4643 0.6256 0.000 0.000 0.344 0.656
#> GSM340343 1 0.6101 0.6871 0.560 0.000 0.052 0.388
#> GSM340344 2 0.0707 0.9443 0.000 0.980 0.000 0.020
#> GSM340346 3 0.0188 0.8640 0.000 0.000 0.996 0.004
#> GSM340347 4 0.4543 0.6331 0.000 0.000 0.324 0.676
#> GSM340348 4 0.0817 0.6151 0.000 0.000 0.024 0.976
#> GSM340349 3 0.8369 0.2373 0.288 0.060 0.500 0.152
#> GSM340350 1 0.6549 0.6564 0.556 0.000 0.088 0.356
#> GSM340351 1 0.0000 0.7544 1.000 0.000 0.000 0.000
#> GSM340354 3 0.0188 0.8640 0.000 0.000 0.996 0.004
#> GSM340356 4 0.1824 0.5692 0.060 0.000 0.004 0.936
#> GSM340357 1 0.4456 0.7787 0.716 0.000 0.004 0.280
#> GSM348183 1 0.0707 0.7584 0.980 0.000 0.000 0.020
#> GSM348191 3 0.0000 0.8662 0.000 0.000 1.000 0.000
#> GSM348193 1 0.4761 0.7637 0.628 0.000 0.000 0.372
#> GSM537578 1 0.5730 0.6403 0.756 0.036 0.132 0.076
#> GSM348181 1 0.4964 0.7582 0.616 0.000 0.004 0.380
#> GSM348182 2 0.5167 0.4364 0.340 0.644 0.000 0.016
#> GSM348184 3 0.1302 0.8636 0.000 0.000 0.956 0.044
#> GSM348185 4 0.1824 0.5692 0.060 0.000 0.004 0.936
#> GSM348186 2 0.5080 0.2133 0.000 0.576 0.004 0.420
#> GSM348187 1 0.5217 0.7544 0.608 0.000 0.012 0.380
#> GSM348188 1 0.6655 0.7201 0.640 0.128 0.008 0.224
#> GSM348189 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM348190 2 0.0592 0.9449 0.000 0.984 0.000 0.016
#> GSM348194 1 0.0000 0.7544 1.000 0.000 0.000 0.000
#> GSM348195 1 0.0000 0.7544 1.000 0.000 0.000 0.000
#> GSM348196 1 0.4964 0.7582 0.616 0.000 0.004 0.380
#> GSM537585 2 0.3219 0.8153 0.164 0.836 0.000 0.000
#> GSM537594 4 0.1824 0.5692 0.060 0.000 0.004 0.936
#> GSM537596 4 0.7702 -0.2533 0.224 0.000 0.360 0.416
#> GSM537597 4 0.1042 0.6012 0.020 0.000 0.008 0.972
#> GSM537602 1 0.4991 0.7544 0.608 0.000 0.004 0.388
#> GSM340184 3 0.0000 0.8662 0.000 0.000 1.000 0.000
#> GSM340185 4 0.4948 0.5628 0.000 0.000 0.440 0.560
#> GSM340186 4 0.4948 0.5591 0.000 0.000 0.440 0.560
#> GSM340187 4 0.4855 0.5801 0.000 0.000 0.400 0.600
#> GSM340189 3 0.1716 0.8762 0.000 0.000 0.936 0.064
#> GSM340190 3 0.0469 0.8684 0.000 0.000 0.988 0.012
#> GSM340191 3 0.1792 0.8746 0.000 0.000 0.932 0.068
#> GSM340192 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM340193 1 0.4585 0.7734 0.668 0.000 0.000 0.332
#> GSM340194 2 0.0707 0.9443 0.000 0.980 0.000 0.020
#> GSM340195 2 0.0707 0.9443 0.000 0.980 0.000 0.020
#> GSM340196 4 0.4790 0.5921 0.000 0.000 0.380 0.620
#> GSM340197 2 0.0592 0.9449 0.000 0.984 0.000 0.016
#> GSM340198 2 0.0336 0.9463 0.000 0.992 0.000 0.008
#> GSM340199 3 0.1302 0.8788 0.000 0.000 0.956 0.044
#> GSM340200 3 0.3907 0.4722 0.000 0.000 0.768 0.232
#> GSM340201 4 0.3688 0.6677 0.000 0.000 0.208 0.792
#> GSM340202 4 0.4817 0.5859 0.000 0.000 0.388 0.612
#> GSM340203 3 0.1940 0.8652 0.000 0.000 0.924 0.076
#> GSM340204 2 0.0336 0.9463 0.000 0.992 0.000 0.008
#> GSM340205 1 0.5217 0.7528 0.608 0.000 0.012 0.380
#> GSM340206 3 0.0188 0.8640 0.000 0.000 0.996 0.004
#> GSM340207 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM340237 4 0.1940 0.6443 0.000 0.000 0.076 0.924
#> GSM340238 3 0.1389 0.8790 0.000 0.000 0.952 0.048
#> GSM340239 2 0.4250 0.5825 0.276 0.724 0.000 0.000
#> GSM340240 2 0.0000 0.9471 0.000 1.000 0.000 0.000
#> GSM340241 4 0.6101 0.5662 0.000 0.052 0.388 0.560
#> GSM340242 2 0.0000 0.9471 0.000 1.000 0.000 0.000
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.0000 0.64845 1.000 NA 0.000 0.000 0.000
#> GSM340359 5 0.5655 0.74248 0.000 NA 0.000 0.112 0.600
#> GSM340361 1 0.4294 0.57564 0.532 NA 0.000 0.000 0.000
#> GSM340362 5 0.2574 0.83910 0.000 NA 0.000 0.112 0.876
#> GSM340363 1 0.0566 0.64256 0.984 NA 0.000 0.012 0.000
#> GSM340364 1 0.4291 0.57733 0.536 NA 0.000 0.000 0.000
#> GSM340365 5 0.5303 0.72175 0.000 NA 0.000 0.108 0.660
#> GSM340366 5 0.1493 0.83937 0.000 NA 0.028 0.024 0.948
#> GSM340367 1 0.4249 0.58722 0.568 NA 0.000 0.000 0.000
#> GSM340368 1 0.6576 -0.09655 0.436 NA 0.000 0.352 0.000
#> GSM340369 3 0.3561 0.70996 0.000 NA 0.740 0.260 0.000
#> GSM340370 1 0.4088 0.60404 0.632 NA 0.000 0.000 0.000
#> GSM340371 5 0.0404 0.84933 0.000 NA 0.000 0.012 0.988
#> GSM340372 1 0.4291 0.57733 0.536 NA 0.000 0.000 0.000
#> GSM340373 1 0.4291 0.57733 0.536 NA 0.000 0.000 0.000
#> GSM340375 1 0.5231 0.57396 0.536 NA 0.020 0.000 0.016
#> GSM340376 1 0.3838 0.31322 0.716 NA 0.000 0.280 0.000
#> GSM340378 1 0.1043 0.65259 0.960 NA 0.000 0.000 0.000
#> GSM340243 3 0.5509 0.28783 0.360 NA 0.564 0.076 0.000
#> GSM340244 4 0.6443 0.38812 0.368 NA 0.028 0.508 0.000
#> GSM340246 3 0.0609 0.75850 0.000 NA 0.980 0.020 0.000
#> GSM340247 4 0.3003 0.69257 0.000 NA 0.188 0.812 0.000
#> GSM340248 4 0.4078 0.70850 0.068 NA 0.148 0.784 0.000
#> GSM340249 4 0.4951 0.69794 0.144 NA 0.092 0.744 0.000
#> GSM340250 1 0.7062 0.17803 0.508 NA 0.284 0.032 0.172
#> GSM340251 4 0.3074 0.68667 0.000 NA 0.196 0.804 0.000
#> GSM340252 5 0.4485 0.73101 0.000 NA 0.000 0.028 0.680
#> GSM340253 5 0.5655 0.74248 0.000 NA 0.000 0.112 0.600
#> GSM340254 5 0.4003 0.74838 0.000 NA 0.000 0.008 0.704
#> GSM340256 5 0.8599 0.00313 0.216 NA 0.016 0.264 0.372
#> GSM340258 4 0.5066 0.40524 0.000 NA 0.048 0.608 0.344
#> GSM340259 1 0.2915 0.65297 0.860 NA 0.000 0.000 0.024
#> GSM340260 5 0.1568 0.84696 0.000 NA 0.000 0.020 0.944
#> GSM340261 3 0.1544 0.76161 0.000 NA 0.932 0.068 0.000
#> GSM340262 5 0.0290 0.84952 0.000 NA 0.000 0.008 0.992
#> GSM340263 1 0.6570 -0.08448 0.440 NA 0.000 0.348 0.000
#> GSM340264 5 0.5257 0.73261 0.000 NA 0.004 0.116 0.688
#> GSM340265 5 0.0912 0.84915 0.000 NA 0.016 0.012 0.972
#> GSM340266 4 0.4424 0.69776 0.084 NA 0.144 0.768 0.000
#> GSM340267 5 0.4622 0.57747 0.000 NA 0.276 0.040 0.684
#> GSM340268 3 0.3508 0.71465 0.000 NA 0.748 0.252 0.000
#> GSM340269 3 0.3969 0.66122 0.004 NA 0.692 0.304 0.000
#> GSM340270 1 0.4605 0.29614 0.692 NA 0.032 0.272 0.000
#> GSM537574 4 0.3160 0.69227 0.000 NA 0.188 0.808 0.004
#> GSM537580 5 0.1356 0.84086 0.000 NA 0.028 0.012 0.956
#> GSM537581 4 0.5840 0.58741 0.280 NA 0.108 0.604 0.000
#> GSM340272 5 0.2864 0.83693 0.000 NA 0.000 0.112 0.864
#> GSM340273 1 0.6543 -0.05150 0.456 NA 0.000 0.332 0.000
#> GSM340275 4 0.3728 0.70218 0.024 NA 0.164 0.804 0.000
#> GSM340276 1 0.2540 0.60921 0.888 NA 0.088 0.024 0.000
#> GSM340277 5 0.0162 0.84981 0.000 NA 0.000 0.004 0.996
#> GSM340278 3 0.0703 0.75924 0.000 NA 0.976 0.024 0.000
#> GSM340279 3 0.1121 0.76241 0.000 NA 0.956 0.044 0.000
#> GSM340282 5 0.0451 0.85015 0.000 NA 0.000 0.008 0.988
#> GSM340284 4 0.3143 0.67790 0.000 NA 0.204 0.796 0.000
#> GSM340285 1 0.4555 0.29311 0.636 NA 0.344 0.020 0.000
#> GSM340286 5 0.1469 0.85224 0.000 NA 0.000 0.036 0.948
#> GSM340287 3 0.1117 0.75178 0.020 NA 0.964 0.016 0.000
#> GSM340288 1 0.4291 0.57733 0.536 NA 0.000 0.000 0.000
#> GSM340289 1 0.4291 0.57733 0.536 NA 0.000 0.000 0.000
#> GSM340290 3 0.0162 0.75612 0.000 NA 0.996 0.004 0.000
#> GSM340291 4 0.6255 0.34701 0.420 NA 0.044 0.484 0.000
#> GSM340293 3 0.3305 0.73023 0.000 NA 0.776 0.224 0.000
#> GSM340294 3 0.0703 0.76028 0.000 NA 0.976 0.024 0.000
#> GSM340296 3 0.0510 0.75693 0.000 NA 0.984 0.016 0.000
#> GSM340297 3 0.5541 0.39524 0.372 NA 0.552 0.076 0.000
#> GSM340298 3 0.0290 0.75675 0.000 NA 0.992 0.008 0.000
#> GSM340299 5 0.2574 0.83910 0.000 NA 0.000 0.112 0.876
#> GSM340301 5 0.1124 0.85119 0.000 NA 0.000 0.036 0.960
#> GSM340303 5 0.0566 0.85041 0.000 NA 0.004 0.012 0.984
#> GSM340304 1 0.4205 0.45827 0.776 NA 0.056 0.164 0.000
#> GSM340306 1 0.1478 0.65629 0.936 NA 0.000 0.000 0.000
#> GSM340307 3 0.3794 0.64874 0.000 NA 0.800 0.048 0.152
#> GSM340310 1 0.1965 0.65339 0.904 NA 0.000 0.000 0.000
#> GSM340314 3 0.3424 0.72257 0.000 NA 0.760 0.240 0.000
#> GSM340315 3 0.1211 0.74455 0.024 NA 0.960 0.016 0.000
#> GSM340317 1 0.6600 -0.15610 0.408 NA 0.000 0.380 0.000
#> GSM340318 4 0.3003 0.69257 0.000 NA 0.188 0.812 0.000
#> GSM340319 4 0.3003 0.69257 0.000 NA 0.188 0.812 0.000
#> GSM340320 1 0.5059 0.40605 0.700 NA 0.176 0.124 0.000
#> GSM340321 4 0.3039 0.68940 0.000 NA 0.192 0.808 0.000
#> GSM340322 4 0.4874 0.43764 0.000 NA 0.040 0.632 0.328
#> GSM340324 1 0.8201 -0.21651 0.392 NA 0.052 0.320 0.204
#> GSM340328 1 0.0609 0.65275 0.980 NA 0.000 0.000 0.000
#> GSM340330 3 0.5810 0.61245 0.000 NA 0.604 0.244 0.152
#> GSM340332 3 0.3661 0.69265 0.000 NA 0.724 0.276 0.000
#> GSM340333 5 0.5655 0.74248 0.000 NA 0.000 0.112 0.600
#> GSM340336 4 0.3003 0.69257 0.000 NA 0.188 0.812 0.000
#> GSM340337 4 0.5509 0.52496 0.328 NA 0.064 0.600 0.000
#> GSM340338 3 0.5827 0.57104 0.000 NA 0.596 0.144 0.260
#> GSM340339 3 0.3857 0.64723 0.000 NA 0.688 0.312 0.000
#> GSM340340 4 0.5593 0.51411 0.336 NA 0.068 0.588 0.000
#> GSM340341 4 0.5872 0.55874 0.304 NA 0.088 0.596 0.008
#> GSM340343 1 0.0566 0.64428 0.984 NA 0.004 0.012 0.000
#> GSM340344 5 0.5493 0.76090 0.000 NA 0.000 0.112 0.632
#> GSM340346 3 0.3695 0.62669 0.000 NA 0.800 0.036 0.164
#> GSM340347 4 0.4796 0.69459 0.164 NA 0.088 0.740 0.000
#> GSM340348 4 0.5675 0.44381 0.384 NA 0.064 0.544 0.000
#> GSM340349 3 0.5328 0.16302 0.360 NA 0.596 0.012 0.008
#> GSM340350 1 0.4253 0.59677 0.756 NA 0.204 0.008 0.000
#> GSM340351 1 0.4291 0.57733 0.536 NA 0.000 0.000 0.000
#> GSM340354 3 0.5958 0.57353 0.000 NA 0.592 0.200 0.208
#> GSM340356 4 0.6374 0.38820 0.404 NA 0.048 0.500 0.008
#> GSM340357 1 0.5293 0.60758 0.668 NA 0.092 0.004 0.000
#> GSM348183 1 0.4283 0.58046 0.544 NA 0.000 0.000 0.000
#> GSM348191 3 0.0404 0.75122 0.000 NA 0.988 0.012 0.000
#> GSM348193 1 0.2020 0.65294 0.900 NA 0.000 0.000 0.000
#> GSM537578 1 0.6281 0.39669 0.548 NA 0.344 0.012 0.012
#> GSM348181 1 0.0162 0.64769 0.996 NA 0.000 0.004 0.000
#> GSM348182 5 0.6164 0.65694 0.184 NA 0.000 0.108 0.652
#> GSM348184 3 0.4074 0.56571 0.000 NA 0.636 0.364 0.000
#> GSM348185 1 0.6407 0.02421 0.500 NA 0.000 0.296 0.000
#> GSM348186 5 0.4541 0.73324 0.000 NA 0.000 0.032 0.680
#> GSM348187 1 0.0324 0.64772 0.992 NA 0.004 0.004 0.000
#> GSM348188 1 0.3051 0.65246 0.852 NA 0.000 0.028 0.000
#> GSM348189 5 0.0324 0.85009 0.000 NA 0.000 0.004 0.992
#> GSM348190 5 0.3425 0.83059 0.004 NA 0.000 0.112 0.840
#> GSM348194 1 0.4291 0.57733 0.536 NA 0.000 0.000 0.000
#> GSM348195 1 0.4291 0.57733 0.536 NA 0.000 0.000 0.000
#> GSM348196 1 0.0000 0.64845 1.000 NA 0.000 0.000 0.000
#> GSM537585 5 0.5442 0.70243 0.000 NA 0.000 0.116 0.644
#> GSM537594 1 0.6685 -0.06654 0.452 NA 0.004 0.332 0.000
#> GSM537596 1 0.3760 0.52823 0.784 NA 0.188 0.028 0.000
#> GSM537597 1 0.5409 0.07494 0.600 NA 0.064 0.332 0.000
#> GSM537602 1 0.0693 0.64506 0.980 NA 0.012 0.008 0.000
#> GSM340184 3 0.0609 0.74757 0.000 NA 0.980 0.020 0.000
#> GSM340185 4 0.5264 0.56738 0.000 NA 0.196 0.676 0.128
#> GSM340186 4 0.5123 0.53645 0.000 NA 0.144 0.696 0.160
#> GSM340187 4 0.3039 0.68940 0.000 NA 0.192 0.808 0.000
#> GSM340189 3 0.3508 0.71475 0.000 NA 0.748 0.252 0.000
#> GSM340190 3 0.3988 0.70981 0.000 NA 0.732 0.252 0.016
#> GSM340191 3 0.3480 0.71860 0.000 NA 0.752 0.248 0.000
#> GSM340192 5 0.0162 0.84981 0.000 NA 0.000 0.004 0.996
#> GSM340193 1 0.2020 0.65517 0.900 NA 0.000 0.000 0.000
#> GSM340194 5 0.2519 0.84287 0.000 NA 0.000 0.100 0.884
#> GSM340195 5 0.2773 0.83869 0.000 NA 0.000 0.112 0.868
#> GSM340196 4 0.3003 0.69257 0.000 NA 0.188 0.812 0.000
#> GSM340197 5 0.3192 0.83183 0.000 NA 0.000 0.112 0.848
#> GSM340198 5 0.4003 0.74838 0.000 NA 0.000 0.008 0.704
#> GSM340199 3 0.3274 0.73172 0.000 NA 0.780 0.220 0.000
#> GSM340200 3 0.5048 0.56045 0.000 NA 0.704 0.144 0.152
#> GSM340201 4 0.4712 0.70193 0.100 NA 0.168 0.732 0.000
#> GSM340202 4 0.3003 0.69257 0.000 NA 0.188 0.812 0.000
#> GSM340203 3 0.3857 0.64744 0.000 NA 0.688 0.312 0.000
#> GSM340204 5 0.5618 0.74735 0.000 NA 0.000 0.112 0.608
#> GSM340205 1 0.0162 0.64769 0.996 NA 0.000 0.004 0.000
#> GSM340206 3 0.0162 0.75534 0.000 NA 0.996 0.000 0.004
#> GSM340207 5 0.0404 0.84933 0.000 NA 0.000 0.012 0.988
#> GSM340237 4 0.6720 0.37966 0.372 NA 0.076 0.492 0.000
#> GSM340238 3 0.3424 0.72254 0.000 NA 0.760 0.240 0.000
#> GSM340239 5 0.5593 0.70114 0.144 NA 0.000 0.112 0.704
#> GSM340240 5 0.0162 0.84981 0.000 NA 0.000 0.004 0.996
#> GSM340241 4 0.4847 0.51974 0.000 NA 0.068 0.692 0.240
#> GSM340242 5 0.0404 0.84920 0.000 NA 0.000 0.012 0.988
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 6 0.4921 0.36497 0.420 0.064 0.000 0.000 0.000 0.516
#> GSM340359 2 0.2697 0.93514 0.000 0.812 0.000 0.000 0.188 0.000
#> GSM340361 1 0.0363 0.84770 0.988 0.000 0.000 0.000 0.012 0.000
#> GSM340362 5 0.2608 0.83054 0.000 0.080 0.000 0.000 0.872 0.048
#> GSM340363 6 0.4621 0.47600 0.304 0.064 0.000 0.000 0.000 0.632
#> GSM340364 1 0.0146 0.85218 0.996 0.000 0.000 0.000 0.004 0.000
#> GSM340365 5 0.4120 0.69793 0.196 0.012 0.000 0.000 0.744 0.048
#> GSM340366 5 0.1485 0.85841 0.000 0.028 0.024 0.000 0.944 0.004
#> GSM340367 1 0.0363 0.85079 0.988 0.000 0.000 0.000 0.000 0.012
#> GSM340368 6 0.3229 0.55948 0.000 0.044 0.000 0.140 0.000 0.816
#> GSM340369 3 0.4118 0.48566 0.000 0.004 0.592 0.396 0.000 0.008
#> GSM340370 1 0.0909 0.83774 0.968 0.012 0.000 0.000 0.000 0.020
#> GSM340371 5 0.0858 0.86270 0.000 0.028 0.000 0.000 0.968 0.004
#> GSM340372 1 0.0000 0.85483 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.85483 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340375 1 0.1802 0.80347 0.916 0.000 0.000 0.000 0.012 0.072
#> GSM340376 6 0.2451 0.56710 0.000 0.056 0.000 0.060 0.000 0.884
#> GSM340378 6 0.4205 0.38131 0.420 0.016 0.000 0.000 0.000 0.564
#> GSM340243 3 0.5361 0.02155 0.000 0.060 0.480 0.020 0.000 0.440
#> GSM340244 6 0.3810 -0.01516 0.000 0.000 0.000 0.428 0.000 0.572
#> GSM340246 3 0.1753 0.74532 0.000 0.004 0.912 0.084 0.000 0.000
#> GSM340247 4 0.0520 0.84002 0.000 0.000 0.008 0.984 0.000 0.008
#> GSM340248 4 0.1910 0.79044 0.000 0.000 0.000 0.892 0.000 0.108
#> GSM340249 4 0.2762 0.73963 0.000 0.000 0.000 0.804 0.000 0.196
#> GSM340250 6 0.5743 0.19071 0.008 0.052 0.392 0.024 0.008 0.516
#> GSM340251 4 0.0603 0.83710 0.000 0.000 0.016 0.980 0.000 0.004
#> GSM340252 2 0.2664 0.93409 0.000 0.816 0.000 0.000 0.184 0.000
#> GSM340253 2 0.2697 0.93514 0.000 0.812 0.000 0.000 0.188 0.000
#> GSM340254 2 0.2730 0.93569 0.000 0.808 0.000 0.000 0.192 0.000
#> GSM340256 2 0.4742 0.74240 0.000 0.744 0.004 0.044 0.096 0.112
#> GSM340258 4 0.1794 0.82371 0.000 0.016 0.028 0.932 0.024 0.000
#> GSM340259 1 0.4993 0.18548 0.608 0.024 0.000 0.000 0.044 0.324
#> GSM340260 5 0.1700 0.85728 0.000 0.024 0.000 0.000 0.928 0.048
#> GSM340261 3 0.1462 0.77316 0.000 0.000 0.936 0.056 0.000 0.008
#> GSM340262 5 0.0692 0.86379 0.000 0.020 0.000 0.000 0.976 0.004
#> GSM340263 6 0.2613 0.56243 0.000 0.012 0.000 0.140 0.000 0.848
#> GSM340264 5 0.4185 0.76420 0.116 0.060 0.000 0.000 0.780 0.044
#> GSM340265 5 0.1760 0.84865 0.000 0.048 0.020 0.000 0.928 0.004
#> GSM340266 4 0.2679 0.79833 0.000 0.000 0.040 0.864 0.000 0.096
#> GSM340267 5 0.3585 0.70449 0.000 0.048 0.156 0.000 0.792 0.004
#> GSM340268 3 0.3955 0.41915 0.000 0.004 0.560 0.436 0.000 0.000
#> GSM340269 3 0.4221 0.46909 0.000 0.008 0.588 0.396 0.000 0.008
#> GSM340270 6 0.4463 0.54817 0.192 0.016 0.000 0.068 0.000 0.724
#> GSM537574 4 0.0363 0.83876 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM537580 5 0.1477 0.86078 0.000 0.048 0.008 0.000 0.940 0.004
#> GSM537581 4 0.2793 0.68008 0.000 0.000 0.000 0.800 0.000 0.200
#> GSM340272 5 0.1333 0.85846 0.000 0.008 0.000 0.000 0.944 0.048
#> GSM340273 6 0.3252 0.56032 0.000 0.068 0.000 0.108 0.000 0.824
#> GSM340275 4 0.2260 0.78735 0.000 0.000 0.000 0.860 0.000 0.140
#> GSM340276 6 0.6712 0.39544 0.256 0.056 0.164 0.012 0.000 0.512
#> GSM340277 5 0.1644 0.84202 0.000 0.076 0.000 0.000 0.920 0.004
#> GSM340278 3 0.1053 0.77042 0.000 0.004 0.964 0.020 0.000 0.012
#> GSM340279 3 0.1082 0.77280 0.000 0.004 0.956 0.040 0.000 0.000
#> GSM340282 5 0.0665 0.86592 0.000 0.008 0.004 0.000 0.980 0.008
#> GSM340284 4 0.0458 0.83579 0.000 0.000 0.016 0.984 0.000 0.000
#> GSM340285 3 0.5300 0.08757 0.008 0.056 0.492 0.008 0.000 0.436
#> GSM340286 5 0.3151 0.62512 0.000 0.252 0.000 0.000 0.748 0.000
#> GSM340287 3 0.1231 0.75978 0.000 0.012 0.960 0.012 0.004 0.012
#> GSM340288 1 0.0000 0.85483 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340289 1 0.0000 0.85483 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340290 3 0.0363 0.76752 0.000 0.000 0.988 0.012 0.000 0.000
#> GSM340291 6 0.4423 0.46963 0.000 0.060 0.000 0.272 0.000 0.668
#> GSM340293 3 0.2964 0.70983 0.000 0.004 0.792 0.204 0.000 0.000
#> GSM340294 3 0.0363 0.76935 0.000 0.000 0.988 0.012 0.000 0.000
#> GSM340296 3 0.0405 0.76717 0.000 0.000 0.988 0.008 0.000 0.004
#> GSM340297 3 0.5288 0.52398 0.000 0.048 0.640 0.060 0.000 0.252
#> GSM340298 3 0.0520 0.76615 0.000 0.000 0.984 0.008 0.000 0.008
#> GSM340299 5 0.2325 0.84535 0.000 0.060 0.000 0.000 0.892 0.048
#> GSM340301 5 0.1075 0.86050 0.000 0.000 0.000 0.000 0.952 0.048
#> GSM340303 5 0.1075 0.85990 0.000 0.048 0.000 0.000 0.952 0.000
#> GSM340304 6 0.5670 0.44897 0.336 0.064 0.008 0.032 0.000 0.560
#> GSM340306 6 0.4689 0.34325 0.440 0.044 0.000 0.000 0.000 0.516
#> GSM340307 3 0.0547 0.76955 0.000 0.000 0.980 0.020 0.000 0.000
#> GSM340310 6 0.4921 0.36497 0.420 0.064 0.000 0.000 0.000 0.516
#> GSM340314 3 0.3789 0.60271 0.000 0.004 0.668 0.324 0.000 0.004
#> GSM340315 3 0.1078 0.76390 0.000 0.008 0.964 0.016 0.000 0.012
#> GSM340317 6 0.3422 0.54808 0.000 0.040 0.000 0.168 0.000 0.792
#> GSM340318 4 0.0520 0.84002 0.000 0.000 0.008 0.984 0.000 0.008
#> GSM340319 4 0.0520 0.84002 0.000 0.000 0.008 0.984 0.000 0.008
#> GSM340320 6 0.5288 0.21148 0.008 0.028 0.388 0.032 0.000 0.544
#> GSM340321 4 0.0458 0.83579 0.000 0.000 0.016 0.984 0.000 0.000
#> GSM340322 4 0.1767 0.81921 0.000 0.020 0.012 0.932 0.036 0.000
#> GSM340324 6 0.4039 0.53638 0.000 0.116 0.000 0.104 0.008 0.772
#> GSM340328 6 0.4921 0.36497 0.420 0.064 0.000 0.000 0.000 0.516
#> GSM340330 3 0.3296 0.71458 0.000 0.012 0.792 0.188 0.008 0.000
#> GSM340332 3 0.3996 0.29452 0.000 0.004 0.512 0.484 0.000 0.000
#> GSM340333 2 0.2697 0.93514 0.000 0.812 0.000 0.000 0.188 0.000
#> GSM340336 4 0.0520 0.84002 0.000 0.000 0.008 0.984 0.000 0.008
#> GSM340337 4 0.4612 0.51758 0.000 0.052 0.000 0.656 0.008 0.284
#> GSM340338 3 0.3676 0.72208 0.000 0.012 0.808 0.092 0.088 0.000
#> GSM340339 4 0.4181 -0.22116 0.000 0.000 0.476 0.512 0.000 0.012
#> GSM340340 4 0.4129 0.22473 0.000 0.012 0.000 0.564 0.000 0.424
#> GSM340341 4 0.3850 0.59332 0.000 0.020 0.000 0.716 0.004 0.260
#> GSM340343 6 0.4842 0.37241 0.420 0.040 0.000 0.008 0.000 0.532
#> GSM340344 2 0.3601 0.75189 0.000 0.684 0.000 0.000 0.312 0.004
#> GSM340346 3 0.0405 0.76417 0.000 0.000 0.988 0.008 0.000 0.004
#> GSM340347 4 0.2697 0.74537 0.000 0.000 0.000 0.812 0.000 0.188
#> GSM340348 6 0.4047 0.32022 0.000 0.012 0.000 0.384 0.000 0.604
#> GSM340349 3 0.3853 0.63605 0.128 0.012 0.808 0.008 0.032 0.012
#> GSM340350 1 0.7040 0.00877 0.448 0.052 0.180 0.008 0.008 0.304
#> GSM340351 1 0.0000 0.85483 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM340354 3 0.2213 0.75876 0.000 0.008 0.888 0.100 0.004 0.000
#> GSM340356 6 0.3213 0.56088 0.000 0.048 0.000 0.132 0.000 0.820
#> GSM340357 1 0.4780 0.62690 0.744 0.052 0.044 0.000 0.016 0.144
#> GSM348183 1 0.0665 0.84906 0.980 0.008 0.000 0.000 0.004 0.008
#> GSM348191 3 0.0260 0.76333 0.000 0.000 0.992 0.000 0.000 0.008
#> GSM348193 6 0.4443 0.47285 0.276 0.060 0.000 0.000 0.000 0.664
#> GSM537578 3 0.5580 0.27852 0.308 0.040 0.600 0.008 0.032 0.012
#> GSM348181 6 0.4726 0.35799 0.424 0.048 0.000 0.000 0.000 0.528
#> GSM348182 5 0.5259 0.43451 0.280 0.048 0.000 0.000 0.624 0.048
#> GSM348184 4 0.1663 0.76446 0.000 0.000 0.088 0.912 0.000 0.000
#> GSM348185 6 0.2571 0.56444 0.000 0.064 0.000 0.060 0.000 0.876
#> GSM348186 2 0.2664 0.93409 0.000 0.816 0.000 0.000 0.184 0.000
#> GSM348187 6 0.4878 0.34683 0.424 0.060 0.000 0.000 0.000 0.516
#> GSM348188 6 0.5197 0.35717 0.420 0.068 0.000 0.000 0.008 0.504
#> GSM348189 5 0.1471 0.85573 0.000 0.064 0.000 0.000 0.932 0.004
#> GSM348190 5 0.2608 0.83079 0.000 0.080 0.000 0.000 0.872 0.048
#> GSM348194 1 0.0000 0.85483 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM348195 1 0.0000 0.85483 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM348196 6 0.4921 0.36497 0.420 0.064 0.000 0.000 0.000 0.516
#> GSM537585 5 0.4183 0.73823 0.140 0.040 0.000 0.000 0.772 0.048
#> GSM537594 6 0.3426 0.55632 0.000 0.068 0.000 0.124 0.000 0.808
#> GSM537596 6 0.6526 0.37726 0.124 0.060 0.296 0.008 0.000 0.512
#> GSM537597 6 0.3784 0.45352 0.000 0.012 0.000 0.308 0.000 0.680
#> GSM537602 6 0.4423 0.37712 0.420 0.028 0.000 0.000 0.000 0.552
#> GSM340184 3 0.0146 0.76545 0.000 0.000 0.996 0.004 0.000 0.000
#> GSM340185 4 0.0937 0.83051 0.000 0.000 0.040 0.960 0.000 0.000
#> GSM340186 4 0.1333 0.82867 0.000 0.008 0.048 0.944 0.000 0.000
#> GSM340187 4 0.0458 0.83579 0.000 0.000 0.016 0.984 0.000 0.000
#> GSM340189 3 0.3950 0.42755 0.000 0.004 0.564 0.432 0.000 0.000
#> GSM340190 4 0.3528 0.41187 0.000 0.004 0.296 0.700 0.000 0.000
#> GSM340191 3 0.3899 0.48538 0.000 0.004 0.592 0.404 0.000 0.000
#> GSM340192 5 0.1010 0.86092 0.000 0.036 0.000 0.000 0.960 0.004
#> GSM340193 1 0.4654 -0.11547 0.544 0.044 0.000 0.000 0.000 0.412
#> GSM340194 5 0.3126 0.63879 0.000 0.248 0.000 0.000 0.752 0.000
#> GSM340195 5 0.3776 0.69258 0.000 0.196 0.000 0.000 0.756 0.048
#> GSM340196 4 0.0520 0.84002 0.000 0.000 0.008 0.984 0.000 0.008
#> GSM340197 5 0.1700 0.85522 0.000 0.024 0.000 0.000 0.928 0.048
#> GSM340198 2 0.2730 0.93569 0.000 0.808 0.000 0.000 0.192 0.000
#> GSM340199 3 0.2838 0.71859 0.000 0.004 0.808 0.188 0.000 0.000
#> GSM340200 3 0.3841 0.24203 0.000 0.004 0.616 0.380 0.000 0.000
#> GSM340201 4 0.1910 0.79026 0.000 0.000 0.000 0.892 0.000 0.108
#> GSM340202 4 0.0520 0.84002 0.000 0.000 0.008 0.984 0.000 0.008
#> GSM340203 4 0.3847 -0.16015 0.000 0.000 0.456 0.544 0.000 0.000
#> GSM340204 2 0.2664 0.93704 0.000 0.816 0.000 0.000 0.184 0.000
#> GSM340205 6 0.4873 0.35410 0.420 0.060 0.000 0.000 0.000 0.520
#> GSM340206 3 0.0458 0.76768 0.000 0.000 0.984 0.016 0.000 0.000
#> GSM340207 5 0.0777 0.86326 0.000 0.024 0.000 0.000 0.972 0.004
#> GSM340237 6 0.4649 0.49204 0.000 0.060 0.016 0.236 0.000 0.688
#> GSM340238 3 0.3468 0.63888 0.000 0.004 0.712 0.284 0.000 0.000
#> GSM340239 5 0.2394 0.84803 0.020 0.032 0.000 0.000 0.900 0.048
#> GSM340240 5 0.0858 0.86270 0.000 0.028 0.000 0.000 0.968 0.004
#> GSM340241 4 0.1585 0.82908 0.000 0.012 0.036 0.940 0.012 0.000
#> GSM340242 5 0.1471 0.84993 0.000 0.064 0.000 0.000 0.932 0.004
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
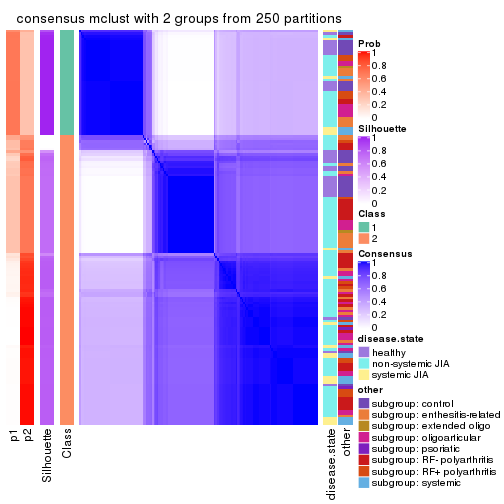
consensus_heatmap(res, k = 3)
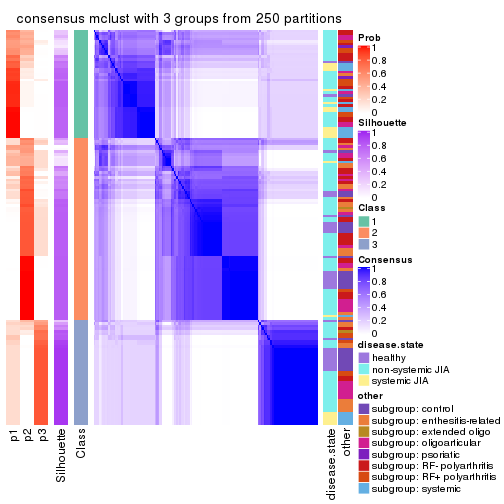
consensus_heatmap(res, k = 4)
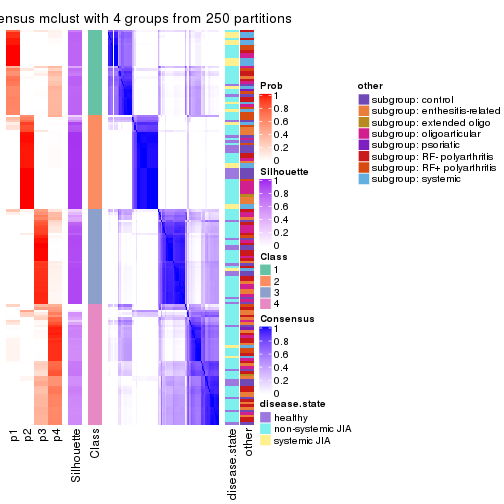
consensus_heatmap(res, k = 5)
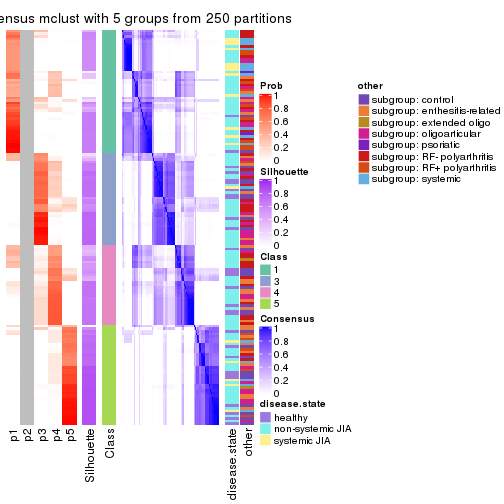
consensus_heatmap(res, k = 6)
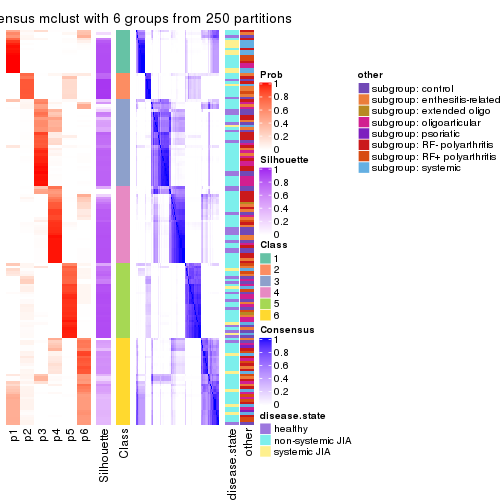
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
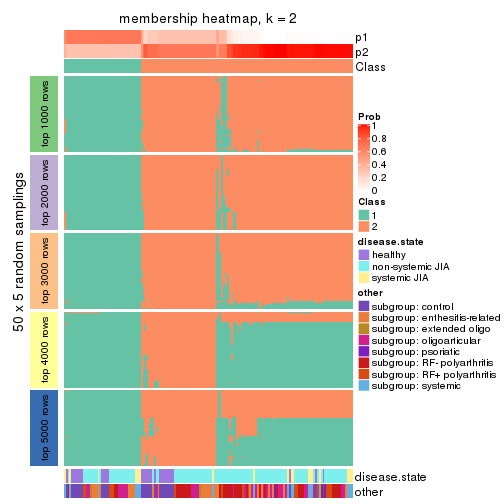
membership_heatmap(res, k = 3)
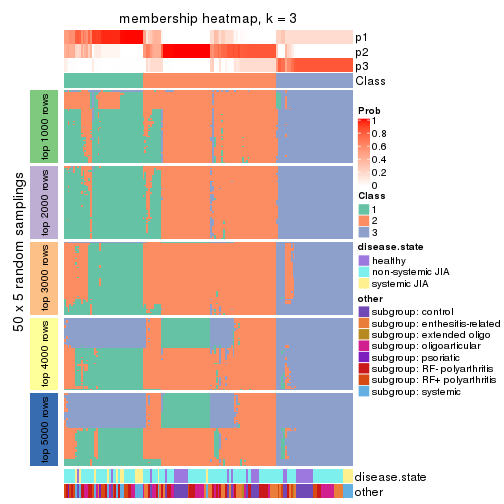
membership_heatmap(res, k = 4)
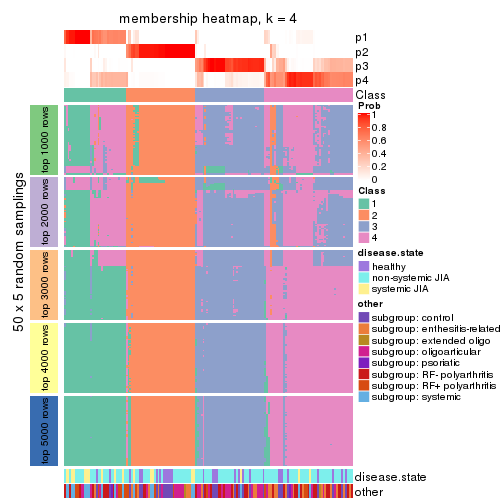
membership_heatmap(res, k = 5)
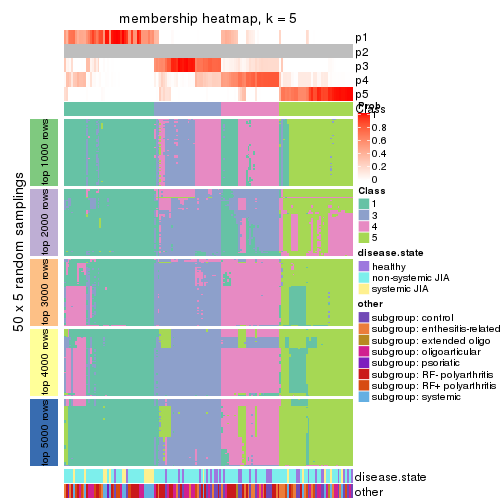
membership_heatmap(res, k = 6)
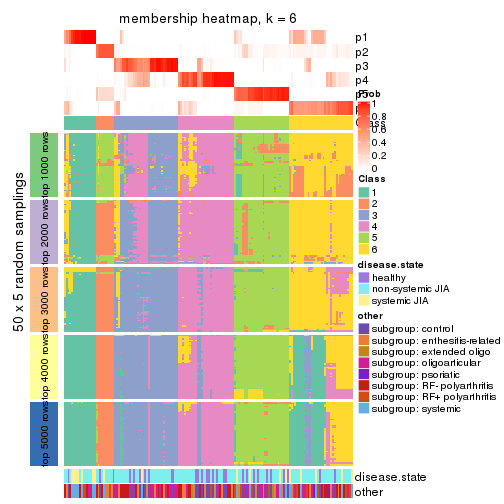
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
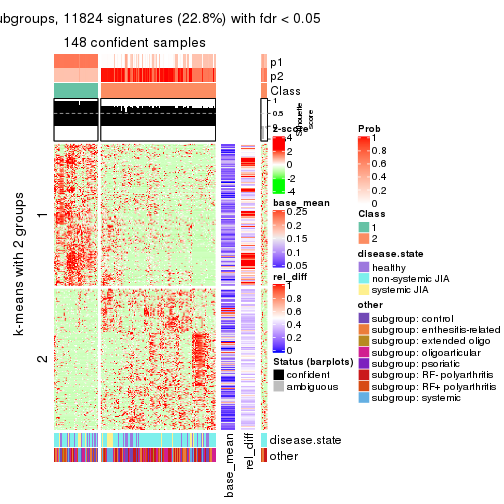
get_signatures(res, k = 3)
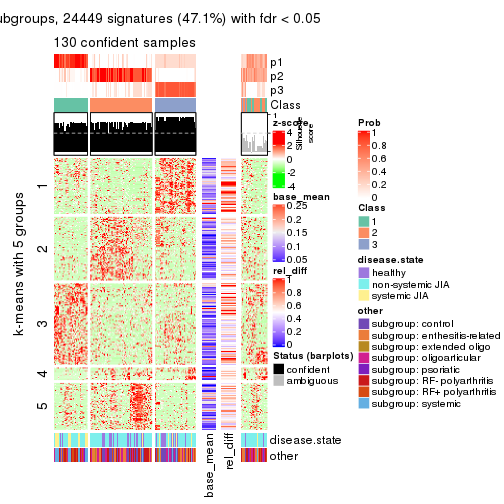
get_signatures(res, k = 4)
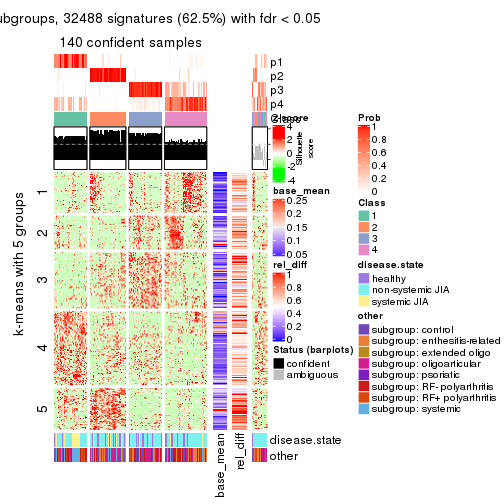
get_signatures(res, k = 5)
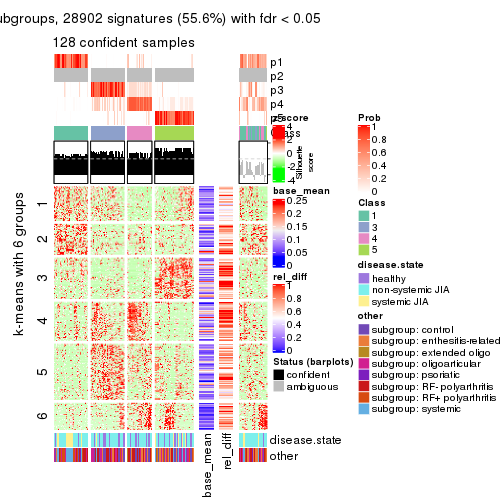
get_signatures(res, k = 6)
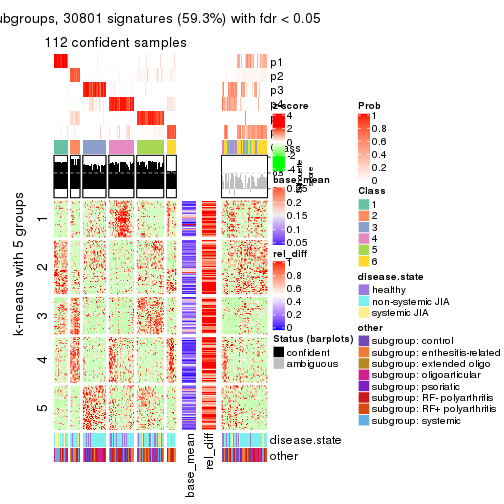
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
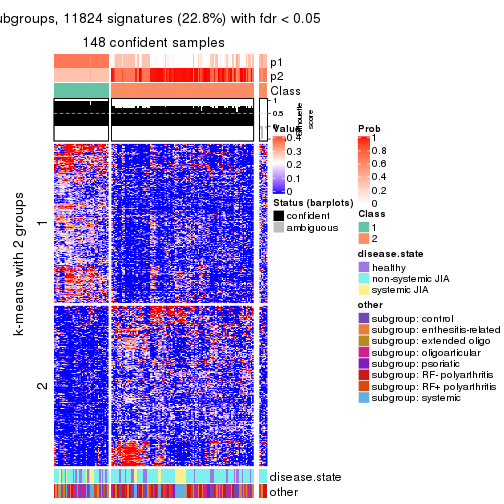
get_signatures(res, k = 3, scale_rows = FALSE)
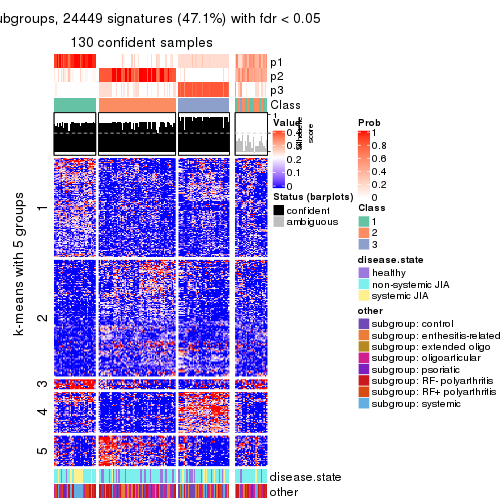
get_signatures(res, k = 4, scale_rows = FALSE)
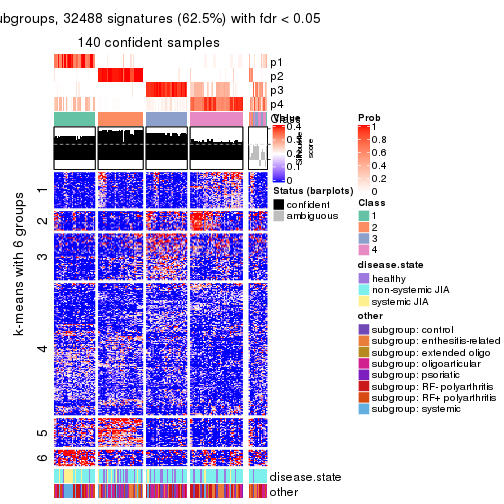
get_signatures(res, k = 5, scale_rows = FALSE)
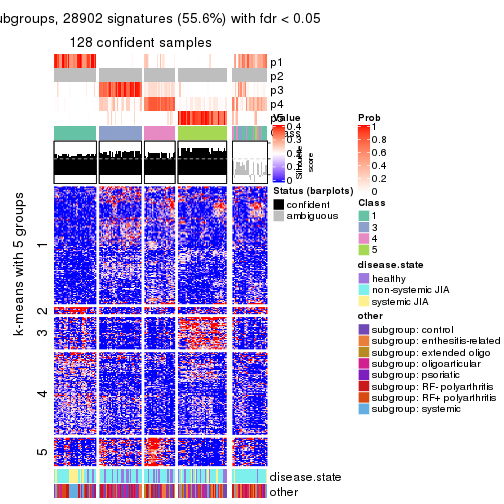
get_signatures(res, k = 6, scale_rows = FALSE)
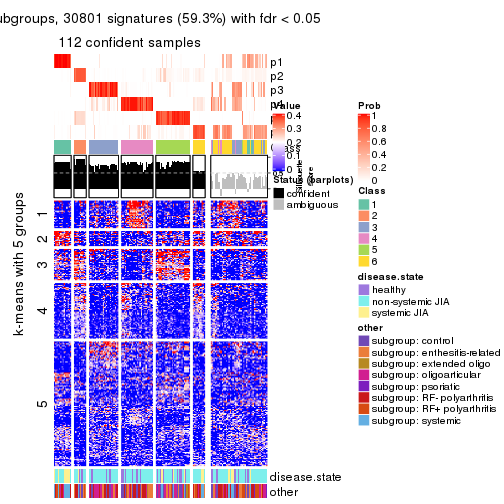
Compare the overlap of signatures from different k:
compare_signatures(res)
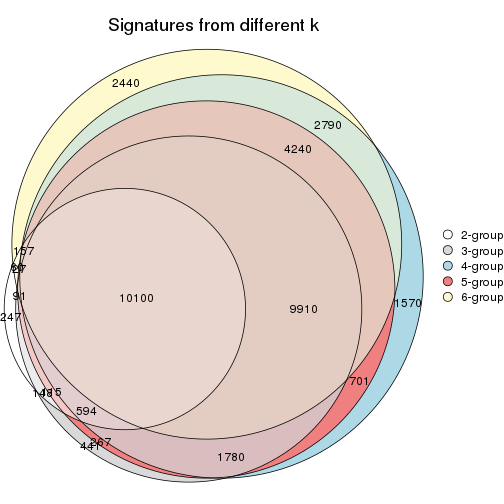
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
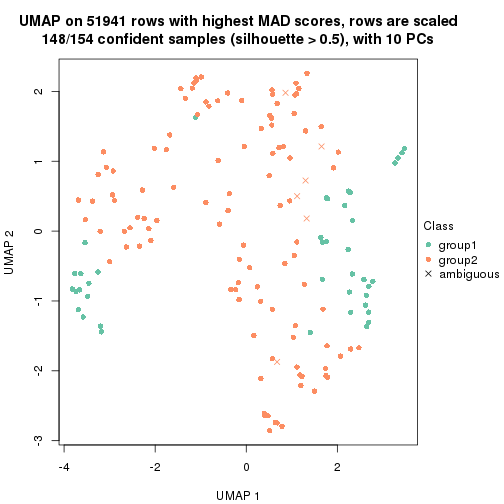
dimension_reduction(res, k = 3, method = "UMAP")
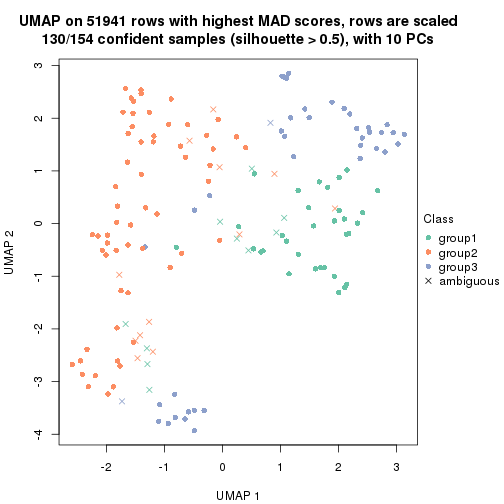
dimension_reduction(res, k = 4, method = "UMAP")
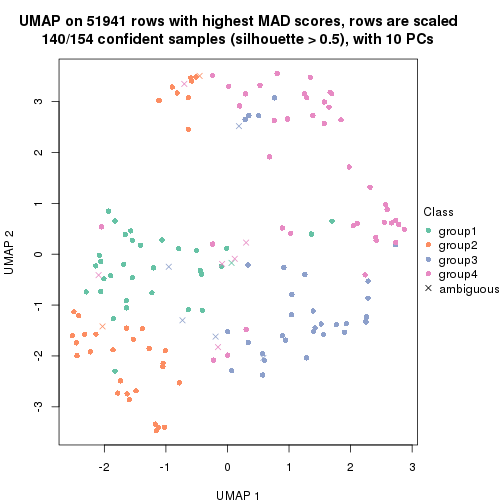
dimension_reduction(res, k = 5, method = "UMAP")
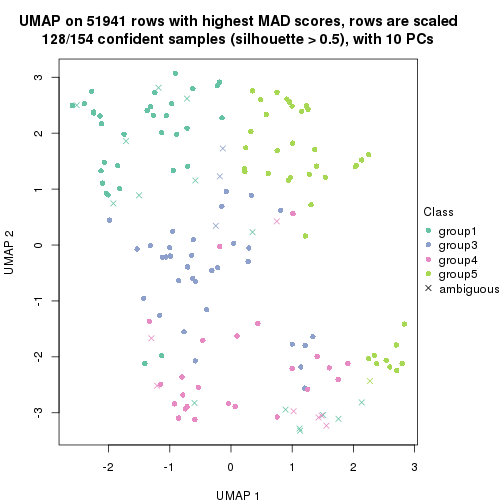
dimension_reduction(res, k = 6, method = "UMAP")
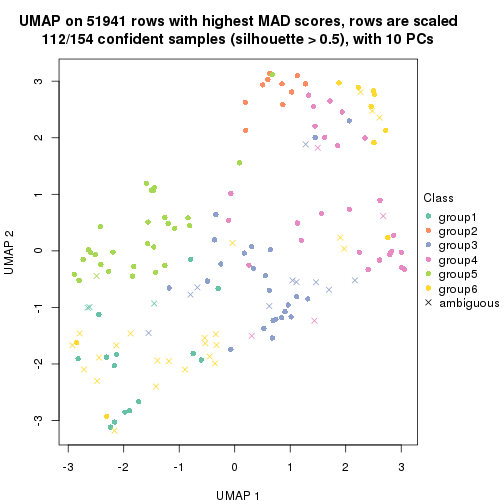
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
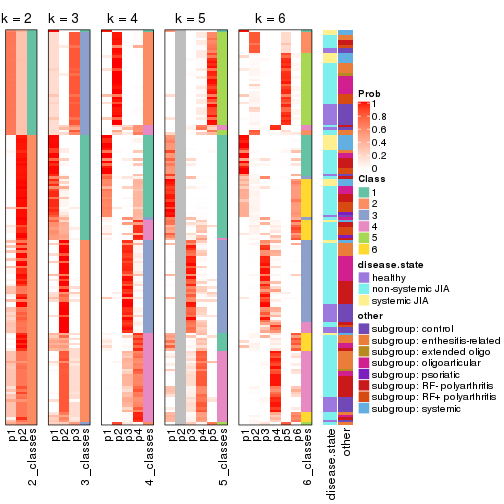
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> MAD:mclust 148 0.274896 0.251061 2
#> MAD:mclust 130 0.000200 0.000093 3
#> MAD:mclust 140 0.004613 0.003050 4
#> MAD:mclust 128 0.003778 0.000596 5
#> MAD:mclust 112 0.000438 0.000257 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["MAD", "NMF"]
# you can also extract it by
# res = res_list["MAD:NMF"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'MAD' method.
#> Subgroups are detected by 'NMF' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
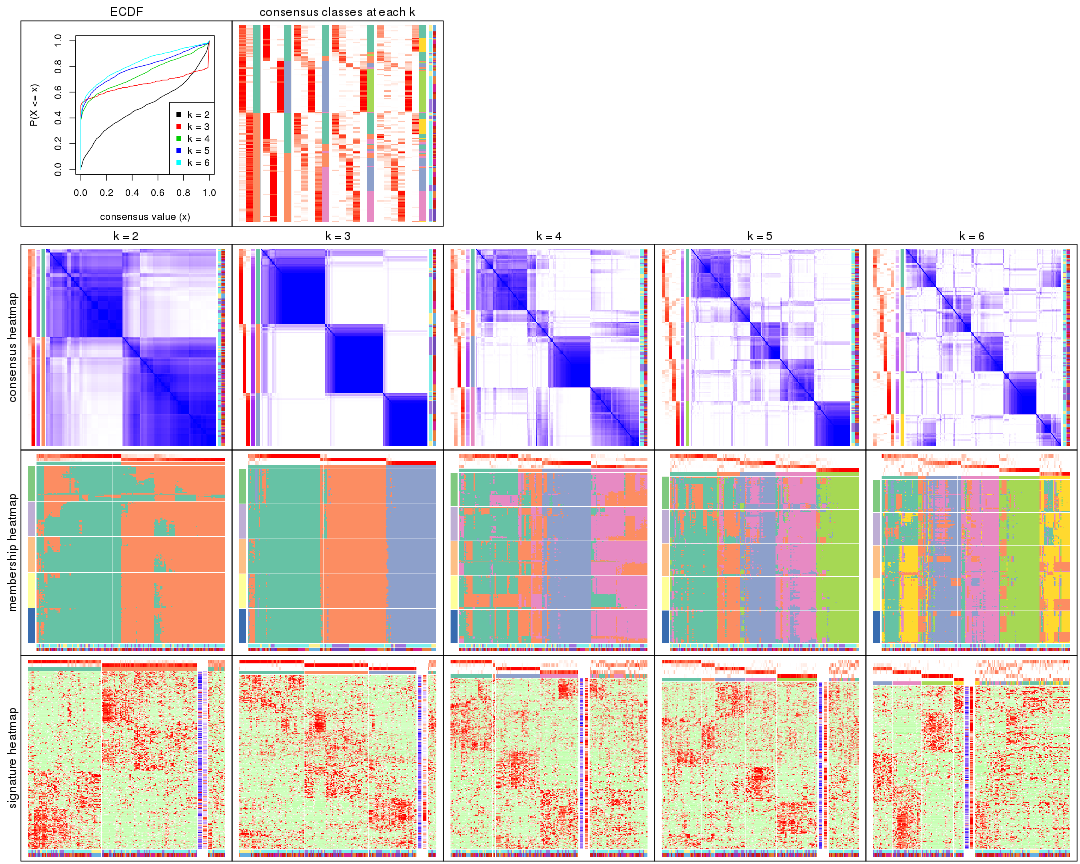
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
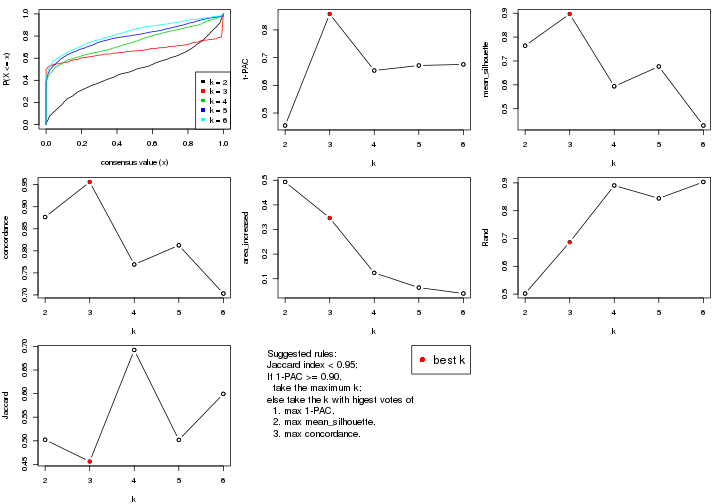
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.455 0.764 0.877 0.4931 0.502 0.502
#> 3 3 0.858 0.897 0.956 0.3468 0.687 0.456
#> 4 4 0.654 0.594 0.769 0.1238 0.891 0.693
#> 5 5 0.672 0.677 0.812 0.0639 0.844 0.502
#> 6 6 0.675 0.430 0.703 0.0398 0.903 0.599
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 2 0.6623 0.7323 0.172 0.828
#> GSM340359 1 0.1184 0.8419 0.984 0.016
#> GSM340361 1 0.4815 0.8127 0.896 0.104
#> GSM340362 1 0.0000 0.8446 1.000 0.000
#> GSM340363 2 0.4562 0.8198 0.096 0.904
#> GSM340364 1 0.3274 0.8304 0.940 0.060
#> GSM340365 1 0.1184 0.8419 0.984 0.016
#> GSM340366 1 0.1414 0.8390 0.980 0.020
#> GSM340367 1 0.9608 0.5119 0.616 0.384
#> GSM340368 2 0.7219 0.6901 0.200 0.800
#> GSM340369 2 0.1184 0.8783 0.016 0.984
#> GSM340370 1 0.8813 0.6490 0.700 0.300
#> GSM340371 1 0.9000 0.4651 0.684 0.316
#> GSM340372 1 0.6438 0.7820 0.836 0.164
#> GSM340373 1 0.3114 0.8323 0.944 0.056
#> GSM340375 1 0.5946 0.7948 0.856 0.144
#> GSM340376 1 0.7883 0.7221 0.764 0.236
#> GSM340378 1 0.6801 0.7711 0.820 0.180
#> GSM340243 2 0.0000 0.8762 0.000 1.000
#> GSM340244 2 0.1184 0.8720 0.016 0.984
#> GSM340246 2 0.4690 0.8532 0.100 0.900
#> GSM340247 2 0.0000 0.8762 0.000 1.000
#> GSM340248 2 0.0376 0.8752 0.004 0.996
#> GSM340249 2 0.0672 0.8743 0.008 0.992
#> GSM340250 1 0.9996 0.2408 0.512 0.488
#> GSM340251 2 0.4022 0.8639 0.080 0.920
#> GSM340252 1 0.0000 0.8446 1.000 0.000
#> GSM340253 1 0.0000 0.8446 1.000 0.000
#> GSM340254 1 0.0672 0.8435 0.992 0.008
#> GSM340256 1 0.2236 0.8379 0.964 0.036
#> GSM340258 1 0.7815 0.6254 0.768 0.232
#> GSM340259 1 0.3431 0.8317 0.936 0.064
#> GSM340260 1 0.0376 0.8443 0.996 0.004
#> GSM340261 2 0.2603 0.8751 0.044 0.956
#> GSM340262 1 0.0938 0.8421 0.988 0.012
#> GSM340263 2 0.5294 0.7962 0.120 0.880
#> GSM340264 1 0.0672 0.8435 0.992 0.008
#> GSM340265 1 0.2948 0.8205 0.948 0.052
#> GSM340266 2 0.0672 0.8743 0.008 0.992
#> GSM340267 2 0.8955 0.5925 0.312 0.688
#> GSM340268 2 0.4431 0.8578 0.092 0.908
#> GSM340269 2 0.6343 0.8133 0.160 0.840
#> GSM340270 2 0.9754 0.1632 0.408 0.592
#> GSM537574 2 0.1414 0.8783 0.020 0.980
#> GSM537580 1 0.9993 -0.0621 0.516 0.484
#> GSM537581 2 0.1414 0.8702 0.020 0.980
#> GSM340272 1 0.0672 0.8435 0.992 0.008
#> GSM340273 2 0.6623 0.7319 0.172 0.828
#> GSM340275 2 0.0376 0.8752 0.004 0.996
#> GSM340276 2 0.0000 0.8762 0.000 1.000
#> GSM340277 1 0.0938 0.8421 0.988 0.012
#> GSM340278 2 0.1184 0.8783 0.016 0.984
#> GSM340279 2 0.2778 0.8743 0.048 0.952
#> GSM340282 1 0.1414 0.8390 0.980 0.020
#> GSM340284 2 0.1414 0.8783 0.020 0.980
#> GSM340285 2 0.0000 0.8762 0.000 1.000
#> GSM340286 1 0.0376 0.8443 0.996 0.004
#> GSM340287 2 0.3879 0.8660 0.076 0.924
#> GSM340288 1 0.7139 0.7590 0.804 0.196
#> GSM340289 1 0.3584 0.8328 0.932 0.068
#> GSM340290 2 0.4562 0.8555 0.096 0.904
#> GSM340291 2 0.1184 0.8720 0.016 0.984
#> GSM340293 2 0.2778 0.8743 0.048 0.952
#> GSM340294 2 0.4161 0.8620 0.084 0.916
#> GSM340296 2 0.3274 0.8715 0.060 0.940
#> GSM340297 2 0.0000 0.8762 0.000 1.000
#> GSM340298 2 0.4939 0.8478 0.108 0.892
#> GSM340299 1 0.0376 0.8443 0.996 0.004
#> GSM340301 1 0.0672 0.8435 0.992 0.008
#> GSM340303 1 0.1414 0.8390 0.980 0.020
#> GSM340304 2 0.9323 0.3594 0.348 0.652
#> GSM340306 1 0.9323 0.5782 0.652 0.348
#> GSM340307 2 0.6148 0.8123 0.152 0.848
#> GSM340310 1 0.9608 0.5114 0.616 0.384
#> GSM340314 2 0.1184 0.8783 0.016 0.984
#> GSM340315 2 0.3274 0.8714 0.060 0.940
#> GSM340317 2 0.4562 0.8196 0.096 0.904
#> GSM340318 2 0.1184 0.8783 0.016 0.984
#> GSM340319 2 0.0000 0.8762 0.000 1.000
#> GSM340320 2 0.0938 0.8732 0.012 0.988
#> GSM340321 2 0.1414 0.8783 0.020 0.980
#> GSM340322 1 0.9608 0.2769 0.616 0.384
#> GSM340324 1 0.4815 0.8127 0.896 0.104
#> GSM340328 1 0.9996 0.2310 0.512 0.488
#> GSM340330 2 0.7453 0.7456 0.212 0.788
#> GSM340332 2 0.1184 0.8783 0.016 0.984
#> GSM340333 1 0.0000 0.8446 1.000 0.000
#> GSM340336 2 0.0376 0.8768 0.004 0.996
#> GSM340337 2 0.8267 0.5862 0.260 0.740
#> GSM340338 2 0.8267 0.6790 0.260 0.740
#> GSM340339 2 0.0000 0.8762 0.000 1.000
#> GSM340340 2 0.4298 0.8273 0.088 0.912
#> GSM340341 2 0.8861 0.4845 0.304 0.696
#> GSM340343 2 0.1184 0.8720 0.016 0.984
#> GSM340344 1 0.0000 0.8446 1.000 0.000
#> GSM340346 2 0.6623 0.7921 0.172 0.828
#> GSM340347 2 0.1184 0.8721 0.016 0.984
#> GSM340348 2 0.2948 0.8545 0.052 0.948
#> GSM340349 2 0.5946 0.8342 0.144 0.856
#> GSM340350 2 0.9795 0.1977 0.416 0.584
#> GSM340351 1 0.7219 0.7552 0.800 0.200
#> GSM340354 2 0.7219 0.7607 0.200 0.800
#> GSM340356 1 0.9427 0.5576 0.640 0.360
#> GSM340357 2 0.9833 0.0903 0.424 0.576
#> GSM348183 1 0.6148 0.7896 0.848 0.152
#> GSM348191 2 0.4939 0.8478 0.108 0.892
#> GSM348193 1 0.9170 0.6034 0.668 0.332
#> GSM537578 2 0.6531 0.7992 0.168 0.832
#> GSM348181 2 0.2043 0.8652 0.032 0.968
#> GSM348182 1 0.0672 0.8437 0.992 0.008
#> GSM348184 2 0.4562 0.8555 0.096 0.904
#> GSM348185 2 0.5178 0.8001 0.116 0.884
#> GSM348186 1 0.0376 0.8443 0.996 0.004
#> GSM348187 2 0.1633 0.8690 0.024 0.976
#> GSM348188 1 0.5059 0.8095 0.888 0.112
#> GSM348189 1 0.1414 0.8390 0.980 0.020
#> GSM348190 1 0.0376 0.8442 0.996 0.004
#> GSM348194 1 0.8661 0.6643 0.712 0.288
#> GSM348195 1 0.5946 0.7944 0.856 0.144
#> GSM348196 1 0.9922 0.3547 0.552 0.448
#> GSM537585 1 0.0000 0.8446 1.000 0.000
#> GSM537594 1 0.9661 0.4948 0.608 0.392
#> GSM537596 2 0.0938 0.8732 0.012 0.988
#> GSM537597 2 1.0000 -0.2089 0.500 0.500
#> GSM537602 1 0.9491 0.5433 0.632 0.368
#> GSM340184 2 0.6247 0.8087 0.156 0.844
#> GSM340185 2 0.6048 0.8158 0.148 0.852
#> GSM340186 2 0.8081 0.6964 0.248 0.752
#> GSM340187 2 0.4298 0.8599 0.088 0.912
#> GSM340189 2 0.2043 0.8772 0.032 0.968
#> GSM340190 2 0.4939 0.8478 0.108 0.892
#> GSM340191 2 0.1184 0.8783 0.016 0.984
#> GSM340192 1 0.1633 0.8373 0.976 0.024
#> GSM340193 1 0.9710 0.4768 0.600 0.400
#> GSM340194 1 0.0376 0.8443 0.996 0.004
#> GSM340195 1 0.0000 0.8446 1.000 0.000
#> GSM340196 2 0.0000 0.8762 0.000 1.000
#> GSM340197 1 0.0376 0.8443 0.996 0.004
#> GSM340198 1 0.0672 0.8435 0.992 0.008
#> GSM340199 2 0.4022 0.8640 0.080 0.920
#> GSM340200 2 0.7950 0.7082 0.240 0.760
#> GSM340201 2 0.0938 0.8732 0.012 0.988
#> GSM340202 2 0.1184 0.8783 0.016 0.984
#> GSM340203 2 0.3274 0.8715 0.060 0.940
#> GSM340204 1 0.0938 0.8429 0.988 0.012
#> GSM340205 2 0.5059 0.8048 0.112 0.888
#> GSM340206 2 0.4815 0.8504 0.104 0.896
#> GSM340207 1 0.3879 0.8022 0.924 0.076
#> GSM340237 2 0.0672 0.8743 0.008 0.992
#> GSM340238 2 0.4022 0.8639 0.080 0.920
#> GSM340239 1 0.0000 0.8446 1.000 0.000
#> GSM340240 1 0.1414 0.8390 0.980 0.020
#> GSM340241 1 0.8016 0.6053 0.756 0.244
#> GSM340242 1 0.6247 0.7275 0.844 0.156
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340359 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340361 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340362 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340363 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340364 1 0.0892 0.9130 0.980 0.000 0.020
#> GSM340365 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340366 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340367 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340368 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340369 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340370 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340371 3 0.0592 0.9664 0.000 0.012 0.988
#> GSM340372 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340373 1 0.1411 0.9010 0.964 0.000 0.036
#> GSM340375 1 0.0424 0.9210 0.992 0.000 0.008
#> GSM340376 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340378 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340243 1 0.4452 0.7498 0.808 0.192 0.000
#> GSM340244 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340246 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340247 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340248 2 0.4887 0.6914 0.228 0.772 0.000
#> GSM340249 1 0.3551 0.8166 0.868 0.132 0.000
#> GSM340250 1 0.6556 0.5860 0.692 0.032 0.276
#> GSM340251 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340252 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340253 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340254 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340256 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340258 3 0.3551 0.8452 0.000 0.132 0.868
#> GSM340259 1 0.4605 0.7221 0.796 0.000 0.204
#> GSM340260 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340261 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340262 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340263 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340264 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340265 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340266 2 0.4291 0.7672 0.180 0.820 0.000
#> GSM340267 2 0.3941 0.7949 0.000 0.844 0.156
#> GSM340268 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340269 2 0.1860 0.9189 0.000 0.948 0.052
#> GSM340270 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM537574 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM537580 3 0.5988 0.4127 0.000 0.368 0.632
#> GSM537581 2 0.5678 0.5269 0.316 0.684 0.000
#> GSM340272 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340273 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340275 1 0.6267 0.2169 0.548 0.452 0.000
#> GSM340276 1 0.3116 0.8426 0.892 0.108 0.000
#> GSM340277 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340278 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340279 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340282 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340284 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340285 1 0.5678 0.5555 0.684 0.316 0.000
#> GSM340286 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340287 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340288 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340289 1 0.4399 0.7440 0.812 0.000 0.188
#> GSM340290 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340291 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340293 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340294 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340296 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340297 2 0.0237 0.9607 0.004 0.996 0.000
#> GSM340298 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340299 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340301 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340303 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340304 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340306 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340307 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340310 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340314 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340315 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340317 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340318 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340319 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340320 1 0.0424 0.9211 0.992 0.008 0.000
#> GSM340321 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340322 3 0.5098 0.6684 0.000 0.248 0.752
#> GSM340324 1 0.3412 0.8224 0.876 0.000 0.124
#> GSM340328 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340330 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340332 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340333 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340336 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340337 1 0.6701 0.3035 0.576 0.412 0.012
#> GSM340338 2 0.1529 0.9300 0.000 0.960 0.040
#> GSM340339 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340340 1 0.6244 0.2368 0.560 0.440 0.000
#> GSM340341 1 0.8487 0.4515 0.584 0.292 0.124
#> GSM340343 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340344 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340346 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340347 1 0.6302 0.0995 0.520 0.480 0.000
#> GSM340348 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340349 2 0.3551 0.8260 0.132 0.868 0.000
#> GSM340350 1 0.4399 0.7547 0.812 0.188 0.000
#> GSM340351 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340354 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340356 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340357 1 0.1411 0.9029 0.964 0.036 0.000
#> GSM348183 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM348191 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM348193 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM537578 2 0.5254 0.6157 0.264 0.736 0.000
#> GSM348181 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM348182 3 0.2448 0.9003 0.076 0.000 0.924
#> GSM348184 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM348185 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM348186 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM348187 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM348188 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM348189 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM348190 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM348194 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM348195 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM348196 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM537585 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM537594 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM537596 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM537597 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM537602 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340184 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340185 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340186 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340187 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340189 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340190 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340191 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340192 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340193 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340194 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340195 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340196 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340197 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340198 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340199 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340200 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340201 2 0.6026 0.3792 0.376 0.624 0.000
#> GSM340202 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340203 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340204 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340205 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340206 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340207 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340237 1 0.0000 0.9257 1.000 0.000 0.000
#> GSM340238 2 0.0000 0.9639 0.000 1.000 0.000
#> GSM340239 3 0.0237 0.9723 0.004 0.000 0.996
#> GSM340240 3 0.0000 0.9756 0.000 0.000 1.000
#> GSM340241 3 0.2625 0.8983 0.000 0.084 0.916
#> GSM340242 3 0.0237 0.9726 0.000 0.004 0.996
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.0817 0.7592 0.976 0.024 0.000 0.000
#> GSM340359 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340361 1 0.3128 0.7423 0.884 0.076 0.040 0.000
#> GSM340362 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340363 1 0.0524 0.7599 0.988 0.008 0.000 0.004
#> GSM340364 1 0.5964 0.5637 0.676 0.228 0.096 0.000
#> GSM340365 3 0.2867 0.8248 0.012 0.104 0.884 0.000
#> GSM340366 3 0.4989 0.2552 0.000 0.472 0.528 0.000
#> GSM340367 2 0.4941 -0.0727 0.436 0.564 0.000 0.000
#> GSM340368 1 0.1902 0.7300 0.932 0.004 0.000 0.064
#> GSM340369 2 0.4907 0.1267 0.000 0.580 0.000 0.420
#> GSM340370 1 0.2921 0.7278 0.860 0.140 0.000 0.000
#> GSM340371 3 0.2494 0.8692 0.000 0.048 0.916 0.036
#> GSM340372 1 0.4998 0.2204 0.512 0.488 0.000 0.000
#> GSM340373 2 0.5311 0.1830 0.328 0.648 0.024 0.000
#> GSM340375 1 0.1557 0.7471 0.944 0.000 0.056 0.000
#> GSM340376 1 0.1022 0.7606 0.968 0.032 0.000 0.000
#> GSM340378 1 0.3311 0.7078 0.828 0.172 0.000 0.000
#> GSM340243 2 0.3760 0.5002 0.136 0.836 0.000 0.028
#> GSM340244 1 0.4420 0.5645 0.748 0.012 0.000 0.240
#> GSM340246 4 0.4985 0.4555 0.000 0.468 0.000 0.532
#> GSM340247 4 0.1940 0.6170 0.076 0.000 0.000 0.924
#> GSM340248 4 0.7093 0.4600 0.220 0.212 0.000 0.568
#> GSM340249 1 0.6503 0.2768 0.480 0.072 0.000 0.448
#> GSM340250 1 0.6638 0.2874 0.560 0.016 0.368 0.056
#> GSM340251 4 0.0336 0.6569 0.000 0.008 0.000 0.992
#> GSM340252 3 0.0336 0.9109 0.000 0.008 0.992 0.000
#> GSM340253 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340254 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340256 3 0.0188 0.9123 0.000 0.004 0.996 0.000
#> GSM340258 3 0.2647 0.8137 0.000 0.000 0.880 0.120
#> GSM340259 2 0.7314 0.0397 0.348 0.488 0.164 0.000
#> GSM340260 3 0.3801 0.7324 0.000 0.220 0.780 0.000
#> GSM340261 2 0.4382 0.2785 0.000 0.704 0.000 0.296
#> GSM340262 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340263 1 0.2197 0.7499 0.916 0.080 0.000 0.004
#> GSM340264 3 0.0469 0.9107 0.000 0.012 0.988 0.000
#> GSM340265 3 0.4477 0.5774 0.000 0.312 0.688 0.000
#> GSM340266 4 0.3367 0.5740 0.108 0.028 0.000 0.864
#> GSM340267 4 0.6071 0.4165 0.000 0.452 0.044 0.504
#> GSM340268 4 0.3172 0.6621 0.000 0.160 0.000 0.840
#> GSM340269 2 0.5898 0.2557 0.000 0.604 0.048 0.348
#> GSM340270 1 0.4817 0.4321 0.612 0.388 0.000 0.000
#> GSM537574 4 0.1557 0.6505 0.000 0.056 0.000 0.944
#> GSM537580 3 0.5453 0.5114 0.000 0.032 0.648 0.320
#> GSM537581 4 0.3791 0.5039 0.200 0.004 0.000 0.796
#> GSM340272 3 0.0817 0.9053 0.000 0.024 0.976 0.000
#> GSM340273 1 0.0524 0.7561 0.988 0.004 0.000 0.008
#> GSM340275 1 0.6134 0.2905 0.508 0.048 0.000 0.444
#> GSM340276 2 0.7049 0.0205 0.392 0.484 0.000 0.124
#> GSM340277 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340278 4 0.4817 0.5469 0.000 0.388 0.000 0.612
#> GSM340279 4 0.4103 0.6392 0.000 0.256 0.000 0.744
#> GSM340282 3 0.4981 0.2693 0.000 0.464 0.536 0.000
#> GSM340284 4 0.1118 0.6645 0.000 0.036 0.000 0.964
#> GSM340285 2 0.7399 0.3466 0.208 0.512 0.000 0.280
#> GSM340286 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340287 2 0.3219 0.4117 0.000 0.836 0.000 0.164
#> GSM340288 1 0.2704 0.7356 0.876 0.124 0.000 0.000
#> GSM340289 2 0.6354 -0.0531 0.416 0.520 0.064 0.000
#> GSM340290 2 0.4250 0.2136 0.000 0.724 0.000 0.276
#> GSM340291 1 0.2593 0.7478 0.892 0.104 0.000 0.004
#> GSM340293 4 0.2921 0.6690 0.000 0.140 0.000 0.860
#> GSM340294 4 0.4713 0.5809 0.000 0.360 0.000 0.640
#> GSM340296 2 0.5000 -0.4328 0.000 0.504 0.000 0.496
#> GSM340297 2 0.5755 0.1277 0.028 0.528 0.000 0.444
#> GSM340298 2 0.3726 0.3450 0.000 0.788 0.000 0.212
#> GSM340299 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340301 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340303 3 0.2216 0.8635 0.000 0.092 0.908 0.000
#> GSM340304 1 0.4877 0.4158 0.592 0.408 0.000 0.000
#> GSM340306 1 0.1174 0.7519 0.968 0.020 0.000 0.012
#> GSM340307 4 0.4992 0.4435 0.000 0.476 0.000 0.524
#> GSM340310 1 0.0921 0.7614 0.972 0.028 0.000 0.000
#> GSM340314 4 0.4522 0.4896 0.000 0.320 0.000 0.680
#> GSM340315 4 0.4992 0.4435 0.000 0.476 0.000 0.524
#> GSM340317 1 0.0779 0.7571 0.980 0.004 0.000 0.016
#> GSM340318 4 0.1706 0.6335 0.016 0.036 0.000 0.948
#> GSM340319 4 0.3117 0.5888 0.092 0.028 0.000 0.880
#> GSM340320 1 0.6972 0.1891 0.520 0.356 0.000 0.124
#> GSM340321 4 0.4164 0.6341 0.000 0.264 0.000 0.736
#> GSM340322 4 0.5894 -0.0184 0.000 0.036 0.428 0.536
#> GSM340324 1 0.2926 0.7466 0.896 0.056 0.048 0.000
#> GSM340328 1 0.0817 0.7592 0.976 0.024 0.000 0.000
#> GSM340330 4 0.4250 0.6271 0.000 0.276 0.000 0.724
#> GSM340332 4 0.0817 0.6618 0.000 0.024 0.000 0.976
#> GSM340333 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340336 4 0.3182 0.5852 0.096 0.028 0.000 0.876
#> GSM340337 4 0.5924 0.1047 0.404 0.040 0.000 0.556
#> GSM340338 2 0.6875 0.0664 0.000 0.504 0.108 0.388
#> GSM340339 4 0.4070 0.6613 0.044 0.132 0.000 0.824
#> GSM340340 1 0.6351 0.4453 0.588 0.080 0.000 0.332
#> GSM340341 1 0.6926 0.2828 0.532 0.036 0.044 0.388
#> GSM340343 1 0.5168 0.2102 0.504 0.492 0.000 0.004
#> GSM340344 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340346 4 0.4994 0.4366 0.000 0.480 0.000 0.520
#> GSM340347 4 0.4655 0.4532 0.208 0.032 0.000 0.760
#> GSM340348 1 0.4677 0.5903 0.768 0.040 0.000 0.192
#> GSM340349 2 0.1940 0.4816 0.000 0.924 0.000 0.076
#> GSM340350 2 0.3427 0.5219 0.112 0.860 0.000 0.028
#> GSM340351 1 0.2408 0.7414 0.896 0.104 0.000 0.000
#> GSM340354 4 0.4331 0.6269 0.000 0.288 0.000 0.712
#> GSM340356 1 0.0657 0.7554 0.984 0.004 0.000 0.012
#> GSM340357 2 0.5671 0.0629 0.400 0.572 0.000 0.028
#> GSM348183 1 0.2647 0.7364 0.880 0.120 0.000 0.000
#> GSM348191 4 0.4605 0.5914 0.000 0.336 0.000 0.664
#> GSM348193 1 0.0000 0.7588 1.000 0.000 0.000 0.000
#> GSM537578 2 0.2589 0.4590 0.000 0.884 0.000 0.116
#> GSM348181 1 0.4996 0.2305 0.516 0.484 0.000 0.000
#> GSM348182 3 0.4203 0.7720 0.068 0.108 0.824 0.000
#> GSM348184 4 0.3569 0.6534 0.000 0.196 0.000 0.804
#> GSM348185 1 0.0672 0.7596 0.984 0.008 0.000 0.008
#> GSM348186 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM348187 1 0.2814 0.7021 0.868 0.132 0.000 0.000
#> GSM348188 1 0.1749 0.7591 0.952 0.024 0.012 0.012
#> GSM348189 3 0.1610 0.8927 0.000 0.032 0.952 0.016
#> GSM348190 3 0.0336 0.9106 0.000 0.008 0.992 0.000
#> GSM348194 1 0.0188 0.7580 0.996 0.004 0.000 0.000
#> GSM348195 1 0.2469 0.7403 0.892 0.108 0.000 0.000
#> GSM348196 1 0.4164 0.5337 0.736 0.264 0.000 0.000
#> GSM537585 3 0.1109 0.8983 0.004 0.028 0.968 0.000
#> GSM537594 1 0.1389 0.7571 0.952 0.048 0.000 0.000
#> GSM537596 1 0.2888 0.6835 0.872 0.004 0.000 0.124
#> GSM537597 1 0.0524 0.7561 0.988 0.004 0.000 0.008
#> GSM537602 1 0.4981 0.2840 0.536 0.464 0.000 0.000
#> GSM340184 4 0.4761 0.5653 0.000 0.372 0.000 0.628
#> GSM340185 4 0.4977 0.4634 0.000 0.460 0.000 0.540
#> GSM340186 4 0.2647 0.6694 0.000 0.120 0.000 0.880
#> GSM340187 4 0.0921 0.6631 0.000 0.028 0.000 0.972
#> GSM340189 4 0.1474 0.6667 0.000 0.052 0.000 0.948
#> GSM340190 4 0.4746 0.5693 0.000 0.368 0.000 0.632
#> GSM340191 4 0.0921 0.6632 0.000 0.028 0.000 0.972
#> GSM340192 3 0.0469 0.9092 0.000 0.012 0.988 0.000
#> GSM340193 1 0.5543 0.2385 0.556 0.424 0.000 0.020
#> GSM340194 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340195 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340196 4 0.1302 0.6368 0.044 0.000 0.000 0.956
#> GSM340197 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340198 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340199 4 0.4985 0.4459 0.000 0.468 0.000 0.532
#> GSM340200 4 0.4989 0.4499 0.000 0.472 0.000 0.528
#> GSM340201 4 0.3870 0.4909 0.208 0.004 0.000 0.788
#> GSM340202 4 0.3243 0.5865 0.088 0.036 0.000 0.876
#> GSM340203 4 0.1389 0.6459 0.000 0.048 0.000 0.952
#> GSM340204 3 0.1109 0.9006 0.004 0.028 0.968 0.000
#> GSM340205 1 0.2589 0.7376 0.884 0.116 0.000 0.000
#> GSM340206 4 0.4866 0.5334 0.000 0.404 0.000 0.596
#> GSM340207 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340237 1 0.4406 0.5585 0.700 0.300 0.000 0.000
#> GSM340238 4 0.3688 0.6488 0.000 0.208 0.000 0.792
#> GSM340239 3 0.5016 0.3682 0.004 0.396 0.600 0.000
#> GSM340240 3 0.0000 0.9133 0.000 0.000 1.000 0.000
#> GSM340241 3 0.2522 0.8477 0.000 0.016 0.908 0.076
#> GSM340242 3 0.1356 0.8968 0.000 0.032 0.960 0.008
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 1 0.0324 0.7602 0.992 0.000 0.004 0.004 0.000
#> GSM340359 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340361 1 0.4238 0.7569 0.756 0.192 0.000 0.000 0.052
#> GSM340362 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340363 1 0.2997 0.7857 0.840 0.148 0.000 0.012 0.000
#> GSM340364 1 0.5520 0.7073 0.664 0.244 0.068 0.000 0.024
#> GSM340365 5 0.2798 0.7945 0.008 0.140 0.000 0.000 0.852
#> GSM340366 3 0.3508 0.5256 0.000 0.000 0.748 0.000 0.252
#> GSM340367 2 0.2813 0.6806 0.084 0.876 0.040 0.000 0.000
#> GSM340368 1 0.5972 0.5231 0.572 0.152 0.000 0.276 0.000
#> GSM340369 4 0.6080 0.4658 0.000 0.168 0.272 0.560 0.000
#> GSM340370 1 0.3816 0.7010 0.696 0.304 0.000 0.000 0.000
#> GSM340371 5 0.4104 0.7804 0.000 0.088 0.000 0.124 0.788
#> GSM340372 2 0.2377 0.6419 0.128 0.872 0.000 0.000 0.000
#> GSM340373 2 0.4184 0.6471 0.048 0.772 0.176 0.000 0.004
#> GSM340375 1 0.1443 0.7585 0.948 0.000 0.004 0.004 0.044
#> GSM340376 1 0.2516 0.7882 0.860 0.140 0.000 0.000 0.000
#> GSM340378 1 0.4487 0.7354 0.756 0.140 0.104 0.000 0.000
#> GSM340243 3 0.6235 -0.0125 0.156 0.344 0.500 0.000 0.000
#> GSM340244 1 0.4498 0.7488 0.756 0.132 0.000 0.112 0.000
#> GSM340246 3 0.1124 0.7602 0.000 0.004 0.960 0.036 0.000
#> GSM340247 4 0.2046 0.7635 0.016 0.000 0.068 0.916 0.000
#> GSM340248 3 0.6122 0.4516 0.284 0.004 0.564 0.148 0.000
#> GSM340249 4 0.4294 0.6033 0.080 0.152 0.000 0.768 0.000
#> GSM340250 1 0.6326 0.3056 0.592 0.000 0.096 0.040 0.272
#> GSM340251 4 0.1908 0.7580 0.000 0.000 0.092 0.908 0.000
#> GSM340252 5 0.0404 0.9191 0.000 0.000 0.000 0.012 0.988
#> GSM340253 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340254 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340256 5 0.0324 0.9207 0.000 0.004 0.000 0.004 0.992
#> GSM340258 5 0.2782 0.8280 0.000 0.000 0.072 0.048 0.880
#> GSM340259 2 0.2331 0.6843 0.020 0.900 0.000 0.000 0.080
#> GSM340260 2 0.5490 0.1716 0.000 0.556 0.000 0.072 0.372
#> GSM340261 3 0.6287 0.4682 0.000 0.276 0.528 0.196 0.000
#> GSM340262 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340263 1 0.3039 0.7716 0.808 0.192 0.000 0.000 0.000
#> GSM340264 5 0.2588 0.8743 0.004 0.068 0.008 0.020 0.900
#> GSM340265 3 0.4114 0.6128 0.000 0.044 0.776 0.004 0.176
#> GSM340266 4 0.1281 0.7631 0.032 0.000 0.012 0.956 0.000
#> GSM340267 3 0.0162 0.7601 0.000 0.000 0.996 0.000 0.004
#> GSM340268 4 0.2848 0.7277 0.000 0.004 0.156 0.840 0.000
#> GSM340269 2 0.4270 0.4396 0.000 0.668 0.012 0.320 0.000
#> GSM340270 2 0.4201 -0.0841 0.408 0.592 0.000 0.000 0.000
#> GSM537574 4 0.3928 0.5212 0.000 0.004 0.296 0.700 0.000
#> GSM537580 4 0.5345 0.3998 0.000 0.088 0.000 0.632 0.280
#> GSM537581 4 0.4928 0.5877 0.284 0.000 0.056 0.660 0.000
#> GSM340272 5 0.2079 0.8783 0.000 0.020 0.064 0.000 0.916
#> GSM340273 1 0.0566 0.7577 0.984 0.000 0.004 0.012 0.000
#> GSM340275 4 0.5531 0.4336 0.248 0.120 0.000 0.632 0.000
#> GSM340276 2 0.3073 0.6963 0.052 0.868 0.004 0.076 0.000
#> GSM340277 5 0.0794 0.9099 0.000 0.000 0.028 0.000 0.972
#> GSM340278 3 0.3928 0.5673 0.000 0.004 0.700 0.296 0.000
#> GSM340279 3 0.4292 0.5956 0.000 0.024 0.704 0.272 0.000
#> GSM340282 3 0.3461 0.5571 0.000 0.004 0.772 0.000 0.224
#> GSM340284 4 0.4262 0.1963 0.000 0.000 0.440 0.560 0.000
#> GSM340285 2 0.2871 0.6971 0.040 0.872 0.000 0.088 0.000
#> GSM340286 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340287 3 0.4866 0.5133 0.000 0.284 0.664 0.052 0.000
#> GSM340288 1 0.2497 0.7735 0.880 0.112 0.004 0.004 0.000
#> GSM340289 2 0.2474 0.6781 0.084 0.896 0.008 0.000 0.012
#> GSM340290 3 0.4333 0.6745 0.000 0.188 0.752 0.060 0.000
#> GSM340291 1 0.3622 0.7546 0.820 0.056 0.124 0.000 0.000
#> GSM340293 4 0.3561 0.6253 0.000 0.000 0.260 0.740 0.000
#> GSM340294 3 0.4725 0.6618 0.000 0.080 0.720 0.200 0.000
#> GSM340296 3 0.0162 0.7621 0.000 0.000 0.996 0.004 0.000
#> GSM340297 2 0.5708 0.3239 0.028 0.584 0.044 0.344 0.000
#> GSM340298 3 0.0162 0.7621 0.000 0.000 0.996 0.004 0.000
#> GSM340299 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340301 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340303 5 0.4474 0.7956 0.000 0.088 0.076 0.040 0.796
#> GSM340304 1 0.5551 0.6314 0.600 0.304 0.096 0.000 0.000
#> GSM340306 1 0.2806 0.6737 0.844 0.000 0.004 0.152 0.000
#> GSM340307 3 0.0324 0.7621 0.000 0.004 0.992 0.004 0.000
#> GSM340310 1 0.1282 0.7736 0.952 0.044 0.000 0.004 0.000
#> GSM340314 4 0.6011 0.3313 0.000 0.344 0.128 0.528 0.000
#> GSM340315 3 0.0162 0.7606 0.000 0.004 0.996 0.000 0.000
#> GSM340317 1 0.3039 0.7841 0.836 0.152 0.000 0.012 0.000
#> GSM340318 4 0.0566 0.7608 0.004 0.000 0.012 0.984 0.000
#> GSM340319 4 0.1153 0.7640 0.008 0.004 0.024 0.964 0.000
#> GSM340320 2 0.6907 0.3137 0.264 0.436 0.008 0.292 0.000
#> GSM340321 3 0.3774 0.5723 0.000 0.000 0.704 0.296 0.000
#> GSM340322 4 0.3226 0.6771 0.000 0.088 0.000 0.852 0.060
#> GSM340324 1 0.3495 0.7807 0.812 0.160 0.000 0.000 0.028
#> GSM340328 1 0.2929 0.7224 0.840 0.152 0.000 0.008 0.000
#> GSM340330 3 0.4101 0.4134 0.000 0.000 0.628 0.372 0.000
#> GSM340332 4 0.2471 0.7396 0.000 0.000 0.136 0.864 0.000
#> GSM340333 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340336 4 0.1310 0.7651 0.020 0.000 0.024 0.956 0.000
#> GSM340337 4 0.2974 0.6923 0.080 0.052 0.000 0.868 0.000
#> GSM340338 2 0.7144 0.2365 0.000 0.496 0.104 0.320 0.080
#> GSM340339 4 0.4266 0.7292 0.104 0.000 0.120 0.776 0.000
#> GSM340340 4 0.6240 0.2925 0.256 0.180 0.004 0.560 0.000
#> GSM340341 4 0.5239 0.5795 0.072 0.152 0.004 0.736 0.036
#> GSM340343 2 0.1908 0.6707 0.092 0.908 0.000 0.000 0.000
#> GSM340344 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340346 3 0.0162 0.7621 0.000 0.000 0.996 0.004 0.000
#> GSM340347 4 0.0609 0.7599 0.020 0.000 0.000 0.980 0.000
#> GSM340348 1 0.5976 0.1800 0.492 0.096 0.004 0.408 0.000
#> GSM340349 2 0.3966 0.4381 0.000 0.664 0.336 0.000 0.000
#> GSM340350 2 0.3642 0.6075 0.008 0.760 0.232 0.000 0.000
#> GSM340351 1 0.3177 0.7658 0.792 0.208 0.000 0.000 0.000
#> GSM340354 3 0.4616 0.5594 0.000 0.036 0.676 0.288 0.000
#> GSM340356 1 0.0566 0.7577 0.984 0.000 0.004 0.012 0.000
#> GSM340357 1 0.5778 0.3671 0.528 0.096 0.376 0.000 0.000
#> GSM348183 1 0.3612 0.7315 0.732 0.268 0.000 0.000 0.000
#> GSM348191 3 0.4114 0.5994 0.000 0.016 0.712 0.272 0.000
#> GSM348193 1 0.2719 0.7862 0.852 0.144 0.000 0.004 0.000
#> GSM537578 3 0.2329 0.6570 0.000 0.124 0.876 0.000 0.000
#> GSM348181 2 0.2179 0.6584 0.112 0.888 0.000 0.000 0.000
#> GSM348182 5 0.4677 0.5952 0.000 0.300 0.000 0.036 0.664
#> GSM348184 4 0.4227 0.2711 0.000 0.000 0.420 0.580 0.000
#> GSM348185 1 0.3031 0.7867 0.852 0.128 0.004 0.016 0.000
#> GSM348186 5 0.0404 0.9191 0.000 0.000 0.000 0.012 0.988
#> GSM348187 1 0.1059 0.7556 0.968 0.020 0.004 0.008 0.000
#> GSM348188 5 0.7547 0.1871 0.144 0.272 0.000 0.100 0.484
#> GSM348189 5 0.4595 0.7247 0.000 0.088 0.000 0.172 0.740
#> GSM348190 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM348194 1 0.0451 0.7587 0.988 0.000 0.004 0.008 0.000
#> GSM348195 1 0.3336 0.7557 0.772 0.228 0.000 0.000 0.000
#> GSM348196 1 0.4220 0.3564 0.688 0.300 0.004 0.008 0.000
#> GSM537585 5 0.1153 0.9107 0.004 0.024 0.008 0.000 0.964
#> GSM537594 1 0.2806 0.7842 0.844 0.152 0.000 0.004 0.000
#> GSM537596 1 0.4442 0.5095 0.676 0.016 0.004 0.304 0.000
#> GSM537597 1 0.0566 0.7577 0.984 0.000 0.004 0.012 0.000
#> GSM537602 1 0.4443 0.4520 0.524 0.472 0.004 0.000 0.000
#> GSM340184 3 0.5322 0.6126 0.000 0.112 0.660 0.228 0.000
#> GSM340185 3 0.0162 0.7621 0.000 0.000 0.996 0.004 0.000
#> GSM340186 4 0.4003 0.5797 0.000 0.000 0.288 0.704 0.008
#> GSM340187 4 0.2280 0.7491 0.000 0.000 0.120 0.880 0.000
#> GSM340189 4 0.2389 0.7496 0.000 0.004 0.116 0.880 0.000
#> GSM340190 3 0.2471 0.7239 0.000 0.000 0.864 0.136 0.000
#> GSM340191 4 0.3123 0.7053 0.000 0.004 0.184 0.812 0.000
#> GSM340192 5 0.0290 0.9198 0.000 0.000 0.008 0.000 0.992
#> GSM340193 2 0.3670 0.6622 0.180 0.796 0.000 0.020 0.004
#> GSM340194 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340195 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340196 4 0.2144 0.7634 0.020 0.000 0.068 0.912 0.000
#> GSM340197 5 0.0290 0.9194 0.000 0.008 0.000 0.000 0.992
#> GSM340198 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340199 3 0.0162 0.7621 0.000 0.000 0.996 0.004 0.000
#> GSM340200 3 0.0162 0.7621 0.000 0.000 0.996 0.004 0.000
#> GSM340201 4 0.2628 0.7547 0.088 0.000 0.028 0.884 0.000
#> GSM340202 4 0.1197 0.7405 0.000 0.048 0.000 0.952 0.000
#> GSM340203 4 0.1845 0.7447 0.000 0.056 0.016 0.928 0.000
#> GSM340204 5 0.2130 0.8637 0.000 0.080 0.000 0.012 0.908
#> GSM340205 1 0.3796 0.7070 0.700 0.300 0.000 0.000 0.000
#> GSM340206 3 0.2930 0.7066 0.000 0.004 0.832 0.164 0.000
#> GSM340207 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340237 1 0.5268 0.6926 0.680 0.148 0.172 0.000 0.000
#> GSM340238 4 0.3684 0.5976 0.000 0.000 0.280 0.720 0.000
#> GSM340239 2 0.3999 0.4443 0.000 0.656 0.000 0.000 0.344
#> GSM340240 5 0.0000 0.9225 0.000 0.000 0.000 0.000 1.000
#> GSM340241 5 0.2629 0.8001 0.000 0.000 0.136 0.004 0.860
#> GSM340242 5 0.4149 0.7765 0.000 0.088 0.000 0.128 0.784
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 6 0.0146 0.56089 0.000 0.004 0.000 0.000 0.000 0.996
#> GSM340359 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340361 1 0.5525 0.08606 0.484 0.004 0.000 0.000 0.116 0.396
#> GSM340362 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340363 6 0.5023 0.11387 0.372 0.060 0.000 0.008 0.000 0.560
#> GSM340364 1 0.4914 0.17227 0.576 0.012 0.036 0.000 0.004 0.372
#> GSM340365 5 0.3163 0.56007 0.232 0.004 0.000 0.000 0.764 0.000
#> GSM340366 3 0.5083 0.39025 0.000 0.164 0.632 0.000 0.204 0.000
#> GSM340367 1 0.3361 0.38097 0.788 0.188 0.020 0.000 0.000 0.004
#> GSM340368 1 0.7131 -0.06564 0.324 0.072 0.000 0.300 0.000 0.304
#> GSM340369 4 0.4647 0.55817 0.008 0.104 0.184 0.704 0.000 0.000
#> GSM340370 1 0.3984 0.17794 0.596 0.008 0.000 0.000 0.000 0.396
#> GSM340371 5 0.4475 0.00584 0.000 0.448 0.016 0.008 0.528 0.000
#> GSM340372 1 0.3705 0.36120 0.740 0.236 0.000 0.004 0.000 0.020
#> GSM340373 1 0.6021 0.14149 0.524 0.280 0.180 0.004 0.000 0.012
#> GSM340375 6 0.1010 0.55165 0.004 0.000 0.000 0.000 0.036 0.960
#> GSM340376 1 0.3996 0.05112 0.512 0.004 0.000 0.000 0.000 0.484
#> GSM340378 1 0.6198 0.07112 0.444 0.040 0.120 0.000 0.000 0.396
#> GSM340243 3 0.6665 0.15898 0.316 0.104 0.472 0.000 0.000 0.108
#> GSM340244 6 0.6478 0.15663 0.180 0.044 0.000 0.300 0.000 0.476
#> GSM340246 3 0.2980 0.59971 0.040 0.024 0.864 0.072 0.000 0.000
#> GSM340247 4 0.0405 0.75381 0.000 0.000 0.004 0.988 0.000 0.008
#> GSM340248 3 0.6256 0.28197 0.000 0.028 0.492 0.184 0.000 0.296
#> GSM340249 4 0.3490 0.50853 0.268 0.000 0.000 0.724 0.000 0.008
#> GSM340250 6 0.6322 -0.05669 0.004 0.004 0.120 0.032 0.376 0.464
#> GSM340251 4 0.0363 0.75399 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340252 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340253 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340254 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340256 5 0.1471 0.78729 0.000 0.064 0.000 0.004 0.932 0.000
#> GSM340258 5 0.2420 0.72659 0.000 0.000 0.040 0.076 0.884 0.000
#> GSM340259 1 0.3673 0.35947 0.764 0.204 0.000 0.008 0.024 0.000
#> GSM340260 2 0.5223 0.39463 0.072 0.628 0.028 0.000 0.272 0.000
#> GSM340261 3 0.7056 0.30046 0.148 0.196 0.476 0.180 0.000 0.000
#> GSM340262 5 0.2048 0.72252 0.000 0.000 0.120 0.000 0.880 0.000
#> GSM340263 1 0.3851 0.08493 0.540 0.000 0.000 0.000 0.000 0.460
#> GSM340264 2 0.5855 0.06132 0.032 0.444 0.076 0.000 0.444 0.004
#> GSM340265 3 0.5751 -0.09077 0.000 0.320 0.508 0.000 0.168 0.004
#> GSM340266 4 0.0260 0.75316 0.000 0.000 0.000 0.992 0.000 0.008
#> GSM340267 3 0.1152 0.61115 0.000 0.044 0.952 0.000 0.000 0.004
#> GSM340268 4 0.2346 0.72077 0.000 0.008 0.124 0.868 0.000 0.000
#> GSM340269 1 0.6449 -0.02039 0.392 0.368 0.024 0.216 0.000 0.000
#> GSM340270 1 0.2320 0.33303 0.864 0.004 0.000 0.000 0.000 0.132
#> GSM537574 4 0.4924 0.47843 0.000 0.092 0.268 0.636 0.004 0.000
#> GSM537580 2 0.6193 0.32961 0.000 0.456 0.016 0.332 0.196 0.000
#> GSM537581 6 0.3847 -0.07432 0.000 0.000 0.000 0.456 0.000 0.544
#> GSM340272 5 0.4361 0.60410 0.032 0.104 0.100 0.000 0.764 0.000
#> GSM340273 6 0.0146 0.56072 0.004 0.000 0.000 0.000 0.000 0.996
#> GSM340275 4 0.4635 0.55337 0.088 0.068 0.000 0.752 0.000 0.092
#> GSM340276 1 0.5635 0.19343 0.544 0.364 0.040 0.040 0.000 0.012
#> GSM340277 5 0.2383 0.74049 0.000 0.096 0.024 0.000 0.880 0.000
#> GSM340278 3 0.4480 0.47280 0.000 0.044 0.648 0.304 0.000 0.004
#> GSM340279 3 0.4624 0.51330 0.000 0.096 0.692 0.208 0.000 0.004
#> GSM340282 3 0.4703 0.46349 0.000 0.164 0.684 0.000 0.152 0.000
#> GSM340284 4 0.3482 0.49882 0.000 0.000 0.316 0.684 0.000 0.000
#> GSM340285 1 0.4997 0.21636 0.572 0.372 0.016 0.036 0.000 0.004
#> GSM340286 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340287 3 0.5864 0.43541 0.148 0.152 0.628 0.072 0.000 0.000
#> GSM340288 6 0.2263 0.53559 0.048 0.056 0.000 0.000 0.000 0.896
#> GSM340289 1 0.3373 0.35472 0.744 0.248 0.000 0.000 0.000 0.008
#> GSM340290 3 0.3925 0.48859 0.024 0.220 0.744 0.008 0.000 0.004
#> GSM340291 6 0.5852 0.02074 0.356 0.008 0.156 0.000 0.000 0.480
#> GSM340293 4 0.1007 0.75256 0.000 0.000 0.044 0.956 0.000 0.000
#> GSM340294 3 0.4530 0.52150 0.000 0.160 0.704 0.136 0.000 0.000
#> GSM340296 3 0.2491 0.60169 0.000 0.164 0.836 0.000 0.000 0.000
#> GSM340297 4 0.7219 0.03917 0.328 0.272 0.020 0.340 0.000 0.040
#> GSM340298 3 0.2491 0.60214 0.000 0.164 0.836 0.000 0.000 0.000
#> GSM340299 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340301 5 0.0146 0.83462 0.000 0.000 0.004 0.000 0.996 0.000
#> GSM340303 5 0.4250 0.00507 0.000 0.456 0.016 0.000 0.528 0.000
#> GSM340304 6 0.6987 0.12173 0.268 0.160 0.088 0.008 0.000 0.476
#> GSM340306 6 0.3342 0.35386 0.000 0.228 0.000 0.012 0.000 0.760
#> GSM340307 3 0.2454 0.60298 0.000 0.160 0.840 0.000 0.000 0.000
#> GSM340310 6 0.3290 0.35038 0.252 0.004 0.000 0.000 0.000 0.744
#> GSM340314 4 0.5660 0.48412 0.184 0.140 0.044 0.632 0.000 0.000
#> GSM340315 3 0.0935 0.60694 0.000 0.032 0.964 0.000 0.000 0.004
#> GSM340317 6 0.4413 -0.06764 0.488 0.012 0.000 0.008 0.000 0.492
#> GSM340318 4 0.0146 0.75289 0.000 0.000 0.000 0.996 0.000 0.004
#> GSM340319 4 0.1296 0.72963 0.004 0.044 0.000 0.948 0.000 0.004
#> GSM340320 6 0.5971 0.13782 0.212 0.020 0.000 0.220 0.000 0.548
#> GSM340321 3 0.3725 0.46779 0.000 0.008 0.676 0.316 0.000 0.000
#> GSM340322 4 0.5226 -0.20488 0.000 0.448 0.000 0.460 0.092 0.000
#> GSM340324 6 0.5070 -0.04496 0.460 0.012 0.000 0.000 0.048 0.480
#> GSM340328 6 0.4319 0.41798 0.136 0.136 0.000 0.000 0.000 0.728
#> GSM340330 3 0.4246 0.25903 0.000 0.020 0.580 0.400 0.000 0.000
#> GSM340332 4 0.1007 0.75288 0.000 0.000 0.044 0.956 0.000 0.000
#> GSM340333 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340336 4 0.0260 0.75316 0.000 0.000 0.000 0.992 0.000 0.008
#> GSM340337 4 0.4201 0.48154 0.008 0.252 0.000 0.704 0.000 0.036
#> GSM340338 4 0.8080 -0.02455 0.296 0.240 0.056 0.316 0.092 0.000
#> GSM340339 4 0.3944 0.65883 0.000 0.044 0.032 0.788 0.000 0.136
#> GSM340340 1 0.4471 -0.01472 0.500 0.000 0.000 0.472 0.000 0.028
#> GSM340341 2 0.6465 0.28117 0.012 0.528 0.036 0.168 0.000 0.256
#> GSM340343 1 0.3646 0.31734 0.700 0.292 0.000 0.004 0.000 0.004
#> GSM340344 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340346 3 0.2178 0.60870 0.000 0.132 0.868 0.000 0.000 0.000
#> GSM340347 4 0.0363 0.75228 0.000 0.000 0.000 0.988 0.000 0.012
#> GSM340348 6 0.5552 -0.09769 0.008 0.436 0.000 0.104 0.000 0.452
#> GSM340349 2 0.5915 0.01711 0.156 0.508 0.324 0.008 0.000 0.004
#> GSM340350 1 0.6007 0.12366 0.464 0.308 0.224 0.004 0.000 0.000
#> GSM340351 1 0.3843 0.10321 0.548 0.000 0.000 0.000 0.000 0.452
#> GSM340354 3 0.4065 0.46898 0.000 0.028 0.672 0.300 0.000 0.000
#> GSM340356 6 0.0146 0.56046 0.004 0.000 0.000 0.000 0.000 0.996
#> GSM340357 3 0.6294 -0.15637 0.076 0.068 0.432 0.004 0.000 0.420
#> GSM348183 1 0.3881 0.17460 0.600 0.004 0.000 0.000 0.000 0.396
#> GSM348191 3 0.4142 0.52994 0.000 0.056 0.712 0.232 0.000 0.000
#> GSM348193 1 0.3860 0.06056 0.528 0.000 0.000 0.000 0.000 0.472
#> GSM537578 3 0.3025 0.59678 0.008 0.164 0.820 0.004 0.000 0.004
#> GSM348181 1 0.0405 0.37413 0.988 0.004 0.000 0.000 0.000 0.008
#> GSM348182 1 0.5982 -0.14259 0.436 0.316 0.000 0.000 0.248 0.000
#> GSM348184 4 0.3151 0.57218 0.000 0.000 0.252 0.748 0.000 0.000
#> GSM348185 6 0.3615 0.45768 0.140 0.060 0.000 0.004 0.000 0.796
#> GSM348186 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM348187 6 0.0146 0.56089 0.000 0.004 0.000 0.000 0.000 0.996
#> GSM348188 1 0.6568 0.12401 0.448 0.120 0.000 0.016 0.376 0.040
#> GSM348189 5 0.5080 -0.12630 0.000 0.452 0.008 0.056 0.484 0.000
#> GSM348190 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM348194 6 0.0146 0.56115 0.000 0.004 0.000 0.000 0.000 0.996
#> GSM348195 1 0.3828 0.12180 0.560 0.000 0.000 0.000 0.000 0.440
#> GSM348196 6 0.2805 0.44965 0.160 0.012 0.000 0.000 0.000 0.828
#> GSM537585 5 0.4097 0.43260 0.012 0.284 0.016 0.000 0.688 0.000
#> GSM537594 6 0.3868 -0.06022 0.496 0.000 0.000 0.000 0.000 0.504
#> GSM537596 6 0.6357 0.30350 0.004 0.080 0.140 0.200 0.000 0.576
#> GSM537597 6 0.0000 0.56087 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM537602 1 0.5870 0.13862 0.480 0.160 0.000 0.008 0.000 0.352
#> GSM340184 2 0.5916 0.02584 0.012 0.464 0.376 0.148 0.000 0.000
#> GSM340185 3 0.2454 0.60339 0.000 0.160 0.840 0.000 0.000 0.000
#> GSM340186 4 0.2170 0.73156 0.000 0.012 0.100 0.888 0.000 0.000
#> GSM340187 4 0.1910 0.73183 0.000 0.000 0.108 0.892 0.000 0.000
#> GSM340189 4 0.0547 0.75414 0.000 0.000 0.020 0.980 0.000 0.000
#> GSM340190 3 0.4613 0.59146 0.000 0.128 0.692 0.180 0.000 0.000
#> GSM340191 4 0.2980 0.67830 0.000 0.012 0.180 0.808 0.000 0.000
#> GSM340192 5 0.0260 0.83286 0.000 0.000 0.008 0.000 0.992 0.000
#> GSM340193 1 0.5476 0.21286 0.556 0.360 0.000 0.036 0.008 0.040
#> GSM340194 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340195 5 0.0146 0.83468 0.004 0.000 0.000 0.000 0.996 0.000
#> GSM340196 4 0.0405 0.75406 0.000 0.000 0.008 0.988 0.000 0.004
#> GSM340197 5 0.0363 0.83068 0.012 0.000 0.000 0.000 0.988 0.000
#> GSM340198 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340199 3 0.2527 0.60046 0.000 0.168 0.832 0.000 0.000 0.000
#> GSM340200 3 0.2491 0.60169 0.000 0.164 0.836 0.000 0.000 0.000
#> GSM340201 4 0.1327 0.73684 0.000 0.000 0.000 0.936 0.000 0.064
#> GSM340202 4 0.2730 0.60917 0.000 0.192 0.000 0.808 0.000 0.000
#> GSM340203 4 0.3878 0.66277 0.000 0.112 0.116 0.772 0.000 0.000
#> GSM340204 5 0.1867 0.78453 0.036 0.036 0.000 0.004 0.924 0.000
#> GSM340205 1 0.3830 0.19111 0.620 0.004 0.000 0.000 0.000 0.376
#> GSM340206 3 0.3210 0.57714 0.000 0.028 0.804 0.168 0.000 0.000
#> GSM340207 5 0.0458 0.82809 0.000 0.000 0.016 0.000 0.984 0.000
#> GSM340237 1 0.6538 0.06397 0.444 0.064 0.132 0.000 0.000 0.360
#> GSM340238 4 0.3564 0.58684 0.000 0.012 0.264 0.724 0.000 0.000
#> GSM340239 5 0.5956 -0.05957 0.356 0.224 0.000 0.000 0.420 0.000
#> GSM340240 5 0.0000 0.83635 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340241 5 0.1471 0.78582 0.000 0.000 0.064 0.004 0.932 0.000
#> GSM340242 5 0.4961 -0.10445 0.000 0.456 0.040 0.012 0.492 0.000
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
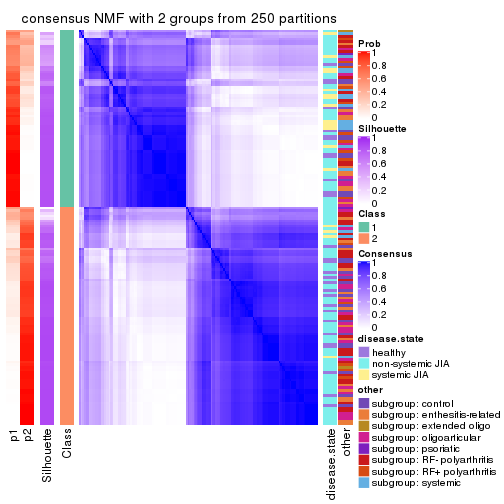
consensus_heatmap(res, k = 3)
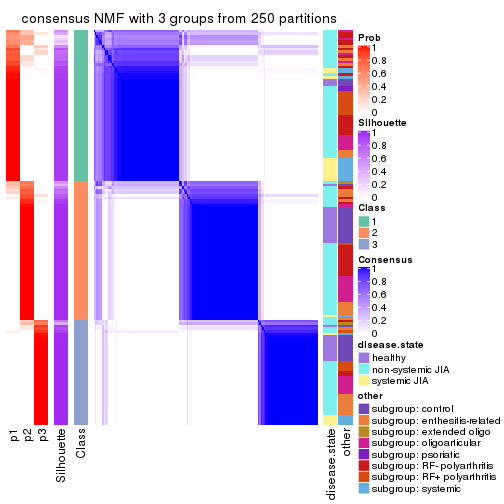
consensus_heatmap(res, k = 4)
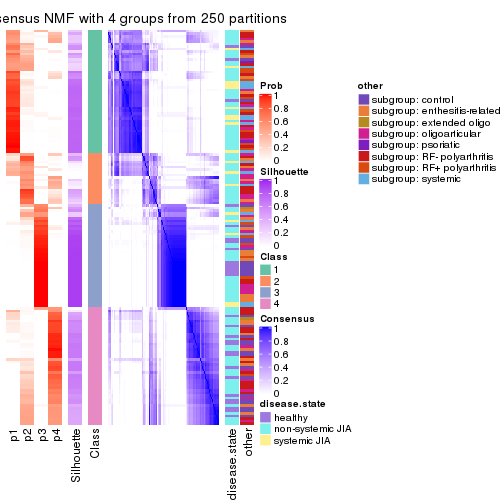
consensus_heatmap(res, k = 5)
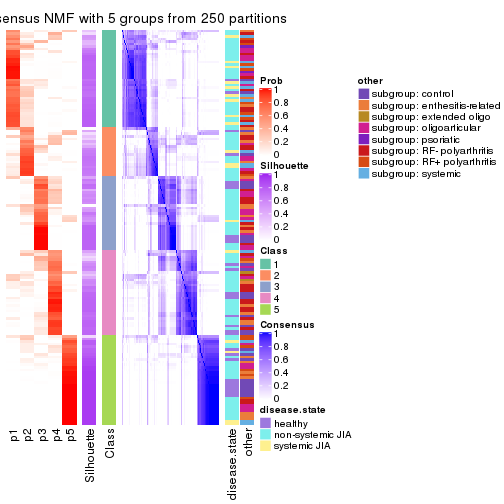
consensus_heatmap(res, k = 6)
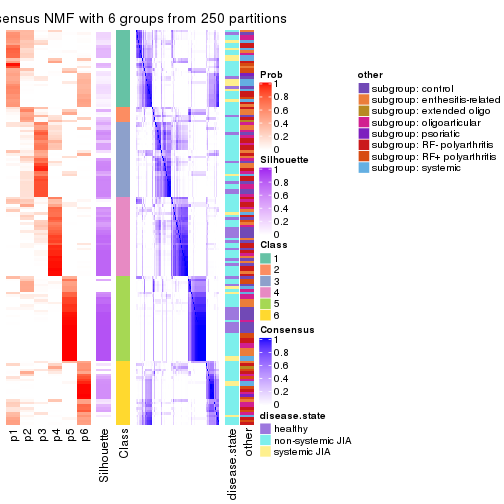
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
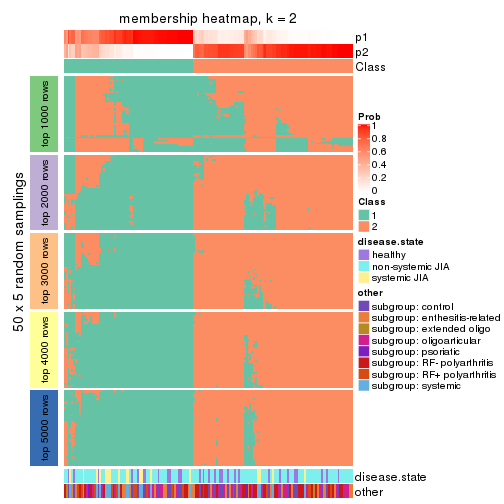
membership_heatmap(res, k = 3)
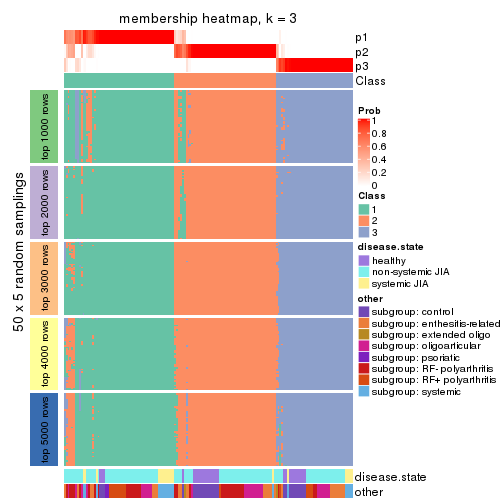
membership_heatmap(res, k = 4)
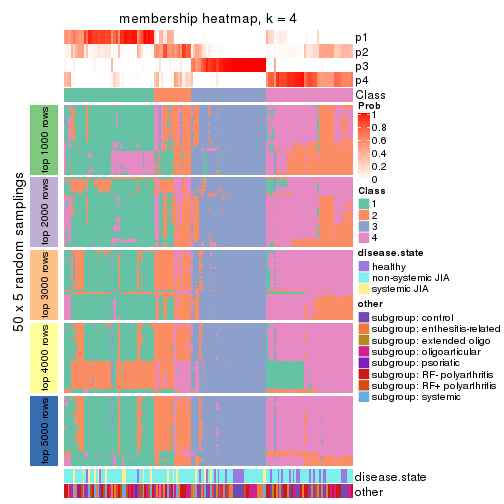
membership_heatmap(res, k = 5)
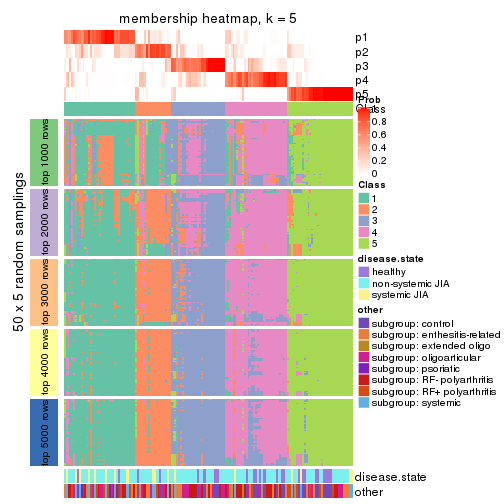
membership_heatmap(res, k = 6)
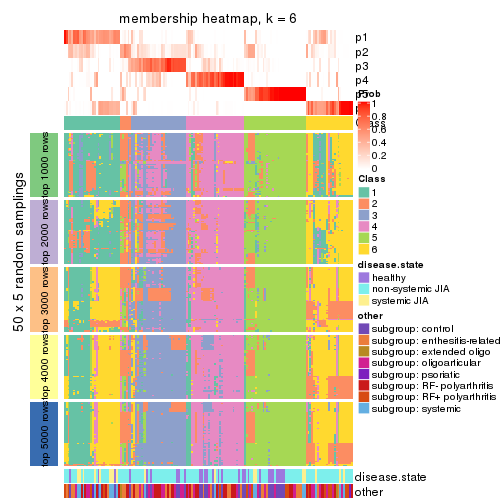
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
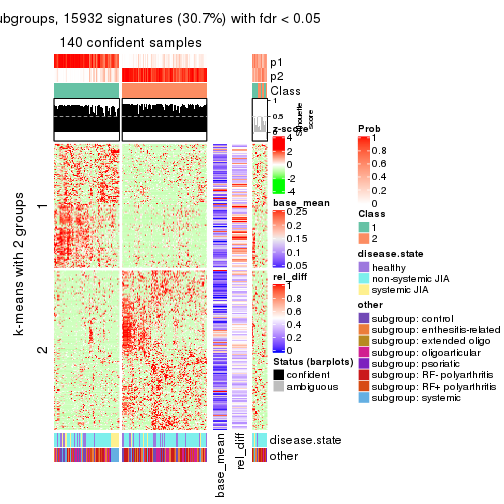
get_signatures(res, k = 3)
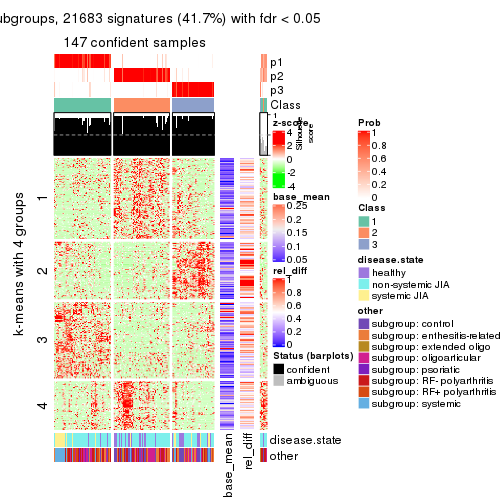
get_signatures(res, k = 4)
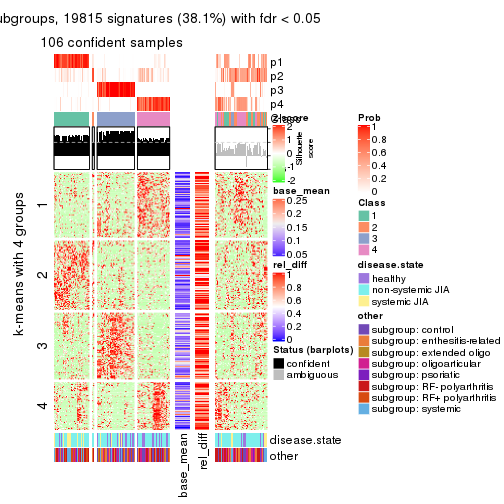
get_signatures(res, k = 5)
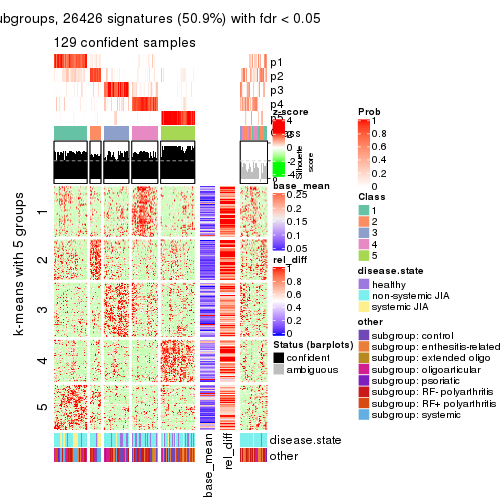
get_signatures(res, k = 6)
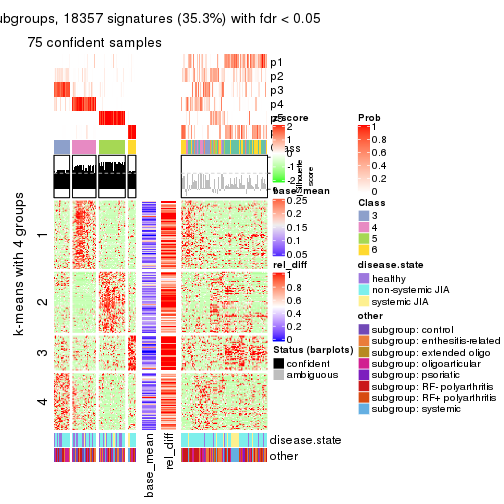
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
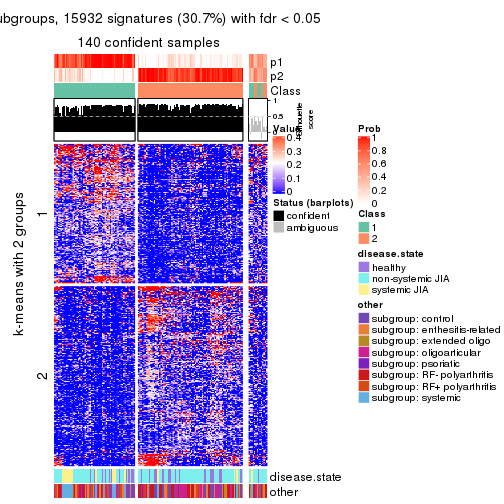
get_signatures(res, k = 3, scale_rows = FALSE)
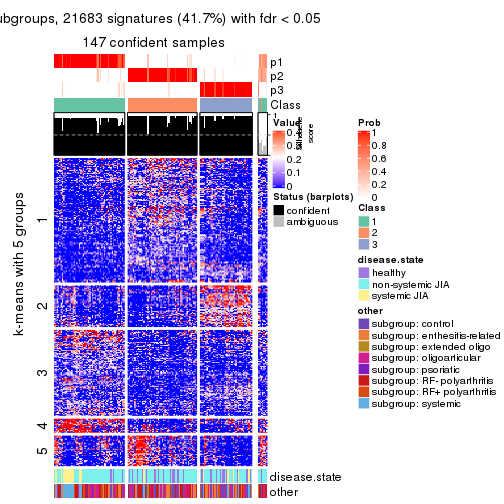
get_signatures(res, k = 4, scale_rows = FALSE)
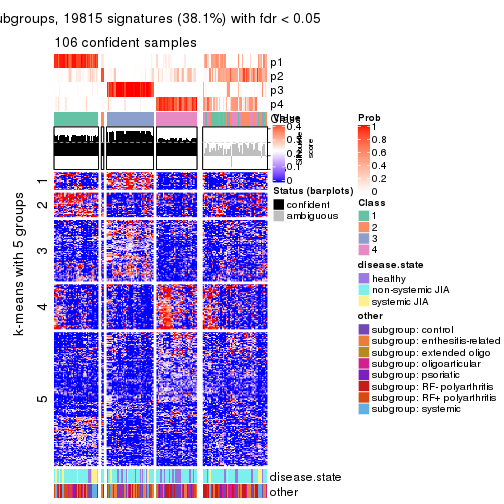
get_signatures(res, k = 5, scale_rows = FALSE)
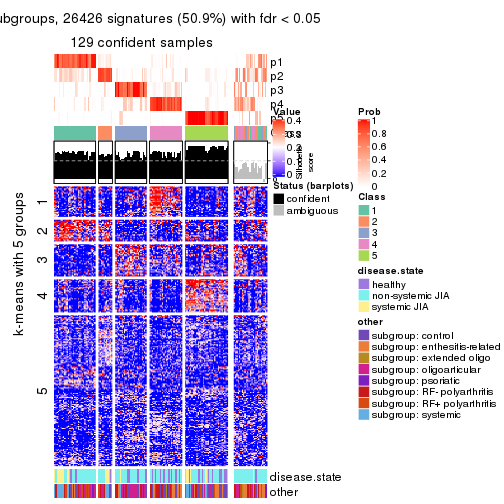
get_signatures(res, k = 6, scale_rows = FALSE)
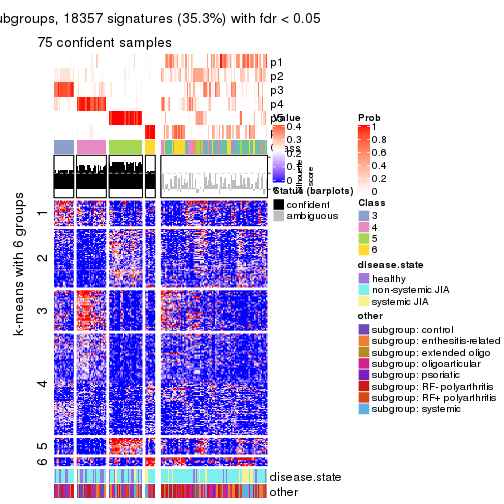
Compare the overlap of signatures from different k:
compare_signatures(res)
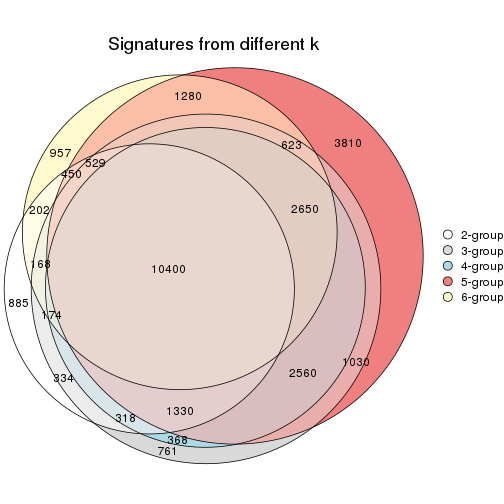
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
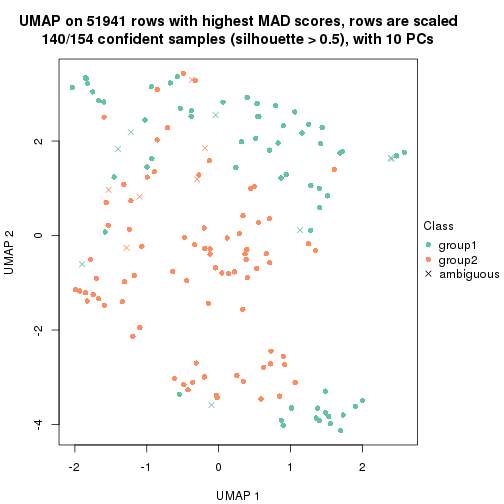
dimension_reduction(res, k = 3, method = "UMAP")
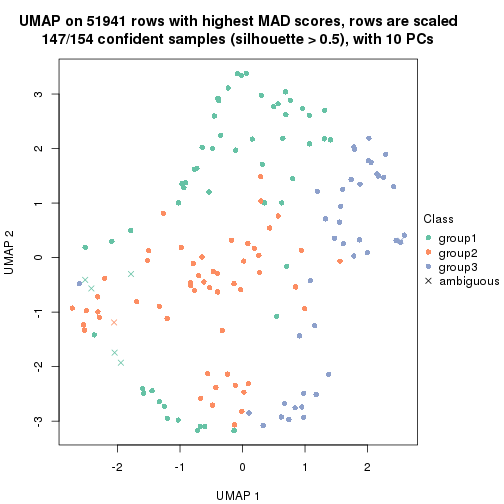
dimension_reduction(res, k = 4, method = "UMAP")
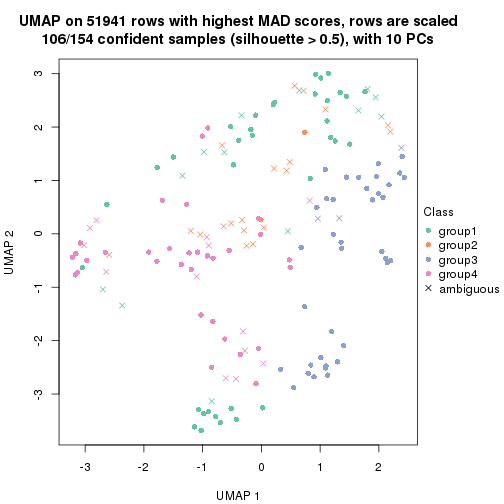
dimension_reduction(res, k = 5, method = "UMAP")
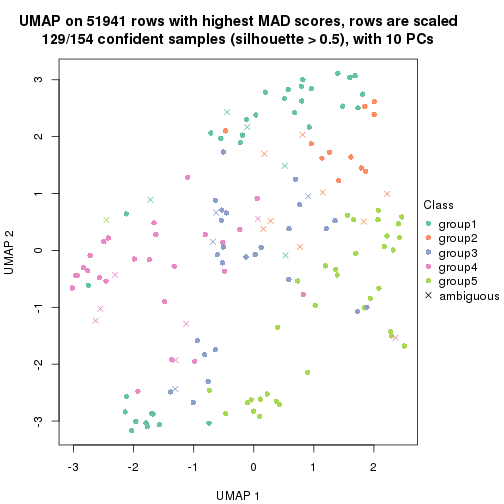
dimension_reduction(res, k = 6, method = "UMAP")
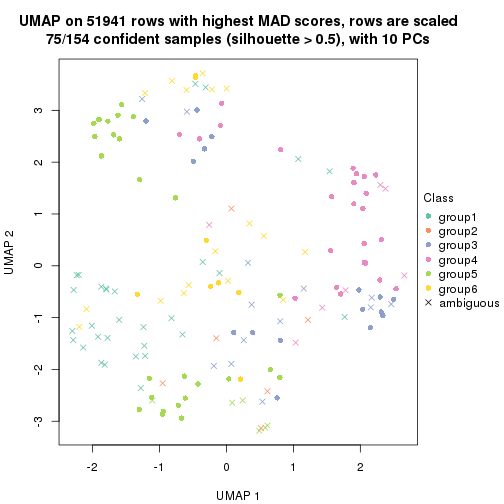
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
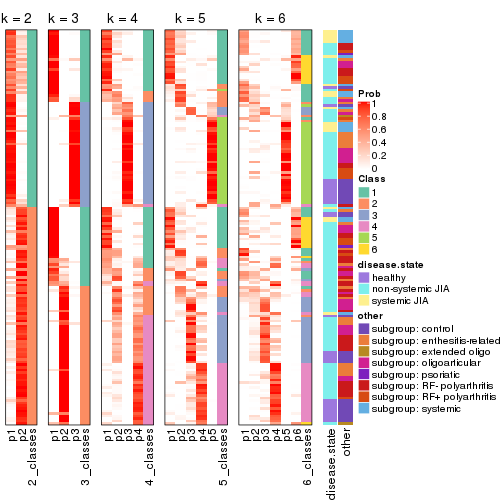
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> MAD:NMF 140 0.01413 0.02471 2
#> MAD:NMF 147 0.00134 0.00151 3
#> MAD:NMF 106 0.00833 0.01615 4
#> MAD:NMF 129 0.01184 0.00348 5
#> MAD:NMF 75 0.21090 0.04165 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["ATC", "hclust"]
# you can also extract it by
# res = res_list["ATC:hclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'ATC' method.
#> Subgroups are detected by 'hclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 5.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
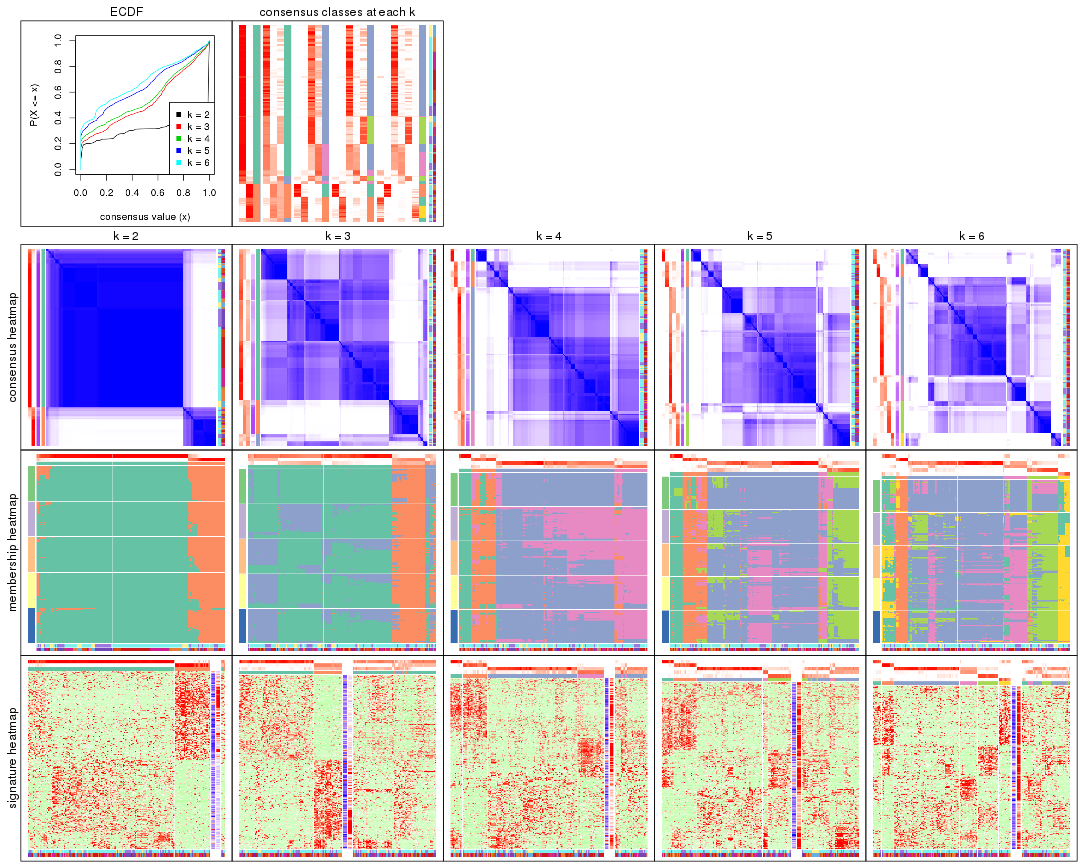
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
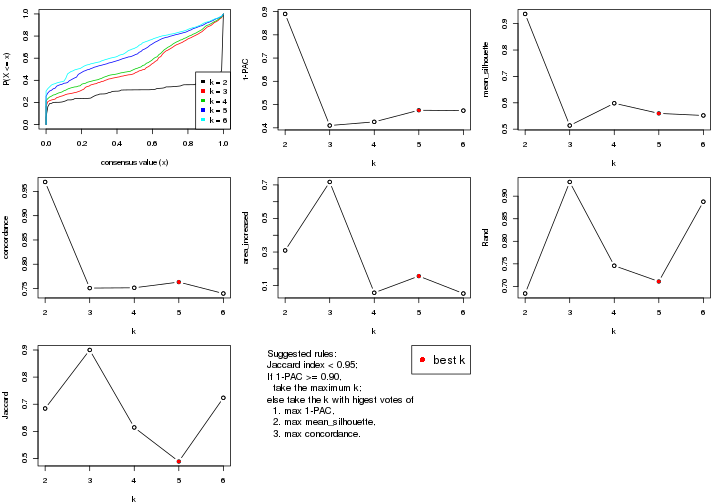
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.890 0.937 0.969 0.3095 0.684 0.684
#> 3 3 0.410 0.514 0.751 0.7176 0.931 0.900
#> 4 4 0.426 0.598 0.751 0.0575 0.746 0.614
#> 5 5 0.476 0.560 0.763 0.1567 0.711 0.489
#> 6 6 0.474 0.552 0.740 0.0528 0.888 0.724
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 5
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 1 0.0000 0.982 1.000 0.000
#> GSM340359 2 0.0000 0.907 0.000 1.000
#> GSM340361 1 0.0672 0.978 0.992 0.008
#> GSM340362 1 0.0672 0.978 0.992 0.008
#> GSM340363 1 0.0000 0.982 1.000 0.000
#> GSM340364 1 0.4939 0.877 0.892 0.108
#> GSM340365 1 0.0672 0.978 0.992 0.008
#> GSM340366 1 0.0672 0.978 0.992 0.008
#> GSM340367 1 0.0000 0.982 1.000 0.000
#> GSM340368 1 0.2236 0.950 0.964 0.036
#> GSM340369 1 0.0000 0.982 1.000 0.000
#> GSM340370 1 0.1414 0.969 0.980 0.020
#> GSM340371 1 0.0000 0.982 1.000 0.000
#> GSM340372 1 0.0376 0.980 0.996 0.004
#> GSM340373 1 0.0376 0.980 0.996 0.004
#> GSM340375 1 0.0376 0.980 0.996 0.004
#> GSM340376 1 0.1414 0.969 0.980 0.020
#> GSM340378 1 0.4690 0.887 0.900 0.100
#> GSM340243 1 0.0000 0.982 1.000 0.000
#> GSM340244 2 0.0938 0.908 0.012 0.988
#> GSM340246 2 0.0938 0.908 0.012 0.988
#> GSM340247 1 0.0000 0.982 1.000 0.000
#> GSM340248 2 0.2236 0.898 0.036 0.964
#> GSM340249 2 0.8327 0.710 0.264 0.736
#> GSM340250 1 0.0000 0.982 1.000 0.000
#> GSM340251 2 0.8267 0.715 0.260 0.740
#> GSM340252 2 0.0000 0.907 0.000 1.000
#> GSM340253 2 0.0000 0.907 0.000 1.000
#> GSM340254 2 0.0000 0.907 0.000 1.000
#> GSM340256 2 0.0672 0.909 0.008 0.992
#> GSM340258 1 0.0938 0.975 0.988 0.012
#> GSM340259 1 0.0000 0.982 1.000 0.000
#> GSM340260 1 0.0376 0.980 0.996 0.004
#> GSM340261 1 0.0000 0.982 1.000 0.000
#> GSM340262 1 0.0672 0.978 0.992 0.008
#> GSM340263 2 0.9815 0.394 0.420 0.580
#> GSM340264 1 0.0672 0.978 0.992 0.008
#> GSM340265 1 0.0672 0.978 0.992 0.008
#> GSM340266 1 0.0000 0.982 1.000 0.000
#> GSM340267 1 0.0376 0.980 0.996 0.004
#> GSM340268 1 0.0000 0.982 1.000 0.000
#> GSM340269 1 0.0000 0.982 1.000 0.000
#> GSM340270 1 0.0000 0.982 1.000 0.000
#> GSM537574 1 0.0376 0.979 0.996 0.004
#> GSM537580 1 0.0000 0.982 1.000 0.000
#> GSM537581 1 0.0000 0.982 1.000 0.000
#> GSM340272 1 0.0672 0.978 0.992 0.008
#> GSM340273 2 0.0672 0.909 0.008 0.992
#> GSM340275 1 0.4562 0.883 0.904 0.096
#> GSM340276 1 0.0000 0.982 1.000 0.000
#> GSM340277 2 0.0000 0.907 0.000 1.000
#> GSM340278 1 0.0000 0.982 1.000 0.000
#> GSM340279 1 0.0000 0.982 1.000 0.000
#> GSM340282 1 0.0672 0.978 0.992 0.008
#> GSM340284 1 0.9129 0.451 0.672 0.328
#> GSM340285 1 0.0000 0.982 1.000 0.000
#> GSM340286 1 0.0672 0.978 0.992 0.008
#> GSM340287 1 0.0000 0.982 1.000 0.000
#> GSM340288 1 0.0376 0.980 0.996 0.004
#> GSM340289 1 0.0000 0.982 1.000 0.000
#> GSM340290 1 0.0000 0.982 1.000 0.000
#> GSM340291 2 0.0672 0.909 0.008 0.992
#> GSM340293 1 0.0000 0.982 1.000 0.000
#> GSM340294 1 0.0000 0.982 1.000 0.000
#> GSM340296 1 0.0000 0.982 1.000 0.000
#> GSM340297 1 0.0000 0.982 1.000 0.000
#> GSM340298 1 0.0000 0.982 1.000 0.000
#> GSM340299 1 0.0672 0.978 0.992 0.008
#> GSM340301 1 0.0672 0.978 0.992 0.008
#> GSM340303 1 0.0672 0.978 0.992 0.008
#> GSM340304 1 0.0000 0.982 1.000 0.000
#> GSM340306 1 0.0000 0.982 1.000 0.000
#> GSM340307 2 0.9286 0.574 0.344 0.656
#> GSM340310 1 0.0000 0.982 1.000 0.000
#> GSM340314 1 0.0000 0.982 1.000 0.000
#> GSM340315 1 0.0000 0.982 1.000 0.000
#> GSM340317 2 0.8555 0.687 0.280 0.720
#> GSM340318 1 0.0000 0.982 1.000 0.000
#> GSM340319 1 0.0000 0.982 1.000 0.000
#> GSM340320 1 0.0000 0.982 1.000 0.000
#> GSM340321 2 0.0672 0.909 0.008 0.992
#> GSM340322 1 0.0000 0.982 1.000 0.000
#> GSM340324 2 0.0672 0.909 0.008 0.992
#> GSM340328 1 0.0000 0.982 1.000 0.000
#> GSM340330 1 0.0000 0.982 1.000 0.000
#> GSM340332 1 0.0000 0.982 1.000 0.000
#> GSM340333 2 0.0000 0.907 0.000 1.000
#> GSM340336 1 0.4562 0.883 0.904 0.096
#> GSM340337 1 0.0000 0.982 1.000 0.000
#> GSM340338 1 0.0000 0.982 1.000 0.000
#> GSM340339 1 0.0000 0.982 1.000 0.000
#> GSM340340 1 0.3879 0.907 0.924 0.076
#> GSM340341 1 0.3879 0.907 0.924 0.076
#> GSM340343 1 0.0000 0.982 1.000 0.000
#> GSM340344 1 0.7883 0.682 0.764 0.236
#> GSM340346 1 0.0000 0.982 1.000 0.000
#> GSM340347 1 0.0000 0.982 1.000 0.000
#> GSM340348 1 0.0000 0.982 1.000 0.000
#> GSM340349 1 0.0000 0.982 1.000 0.000
#> GSM340350 1 0.0000 0.982 1.000 0.000
#> GSM340351 1 0.0376 0.980 0.996 0.004
#> GSM340354 1 0.0000 0.982 1.000 0.000
#> GSM340356 2 0.1184 0.907 0.016 0.984
#> GSM340357 1 0.0000 0.982 1.000 0.000
#> GSM348183 1 0.0000 0.982 1.000 0.000
#> GSM348191 1 0.0000 0.982 1.000 0.000
#> GSM348193 1 0.0000 0.982 1.000 0.000
#> GSM537578 1 0.0000 0.982 1.000 0.000
#> GSM348181 1 0.0000 0.982 1.000 0.000
#> GSM348182 1 0.0376 0.980 0.996 0.004
#> GSM348184 2 0.6148 0.823 0.152 0.848
#> GSM348185 2 0.8763 0.663 0.296 0.704
#> GSM348186 2 0.0000 0.907 0.000 1.000
#> GSM348187 1 0.0000 0.982 1.000 0.000
#> GSM348188 1 0.0000 0.982 1.000 0.000
#> GSM348189 1 0.0000 0.982 1.000 0.000
#> GSM348190 1 0.0672 0.978 0.992 0.008
#> GSM348194 1 0.0376 0.980 0.996 0.004
#> GSM348195 1 0.0376 0.980 0.996 0.004
#> GSM348196 1 0.0000 0.982 1.000 0.000
#> GSM537585 1 0.0376 0.980 0.996 0.004
#> GSM537594 2 0.0672 0.909 0.008 0.992
#> GSM537596 1 0.0000 0.982 1.000 0.000
#> GSM537597 1 0.0000 0.982 1.000 0.000
#> GSM537602 1 0.0000 0.982 1.000 0.000
#> GSM340184 1 0.0000 0.982 1.000 0.000
#> GSM340185 2 0.0672 0.909 0.008 0.992
#> GSM340186 2 0.5178 0.850 0.116 0.884
#> GSM340187 1 0.0376 0.979 0.996 0.004
#> GSM340189 1 0.0000 0.982 1.000 0.000
#> GSM340190 1 0.9580 0.300 0.620 0.380
#> GSM340191 1 0.0000 0.982 1.000 0.000
#> GSM340192 1 0.6343 0.807 0.840 0.160
#> GSM340193 1 0.0000 0.982 1.000 0.000
#> GSM340194 1 0.0672 0.978 0.992 0.008
#> GSM340195 1 0.0672 0.978 0.992 0.008
#> GSM340196 1 0.0000 0.982 1.000 0.000
#> GSM340197 1 0.0672 0.978 0.992 0.008
#> GSM340198 2 0.0000 0.907 0.000 1.000
#> GSM340199 1 0.0000 0.982 1.000 0.000
#> GSM340200 2 0.9491 0.524 0.368 0.632
#> GSM340201 1 0.0000 0.982 1.000 0.000
#> GSM340202 1 0.0000 0.982 1.000 0.000
#> GSM340203 1 0.0000 0.982 1.000 0.000
#> GSM340204 1 0.4939 0.877 0.892 0.108
#> GSM340205 1 0.0000 0.982 1.000 0.000
#> GSM340206 1 0.0000 0.982 1.000 0.000
#> GSM340207 1 0.0672 0.978 0.992 0.008
#> GSM340237 2 0.0672 0.909 0.008 0.992
#> GSM340238 1 0.0000 0.982 1.000 0.000
#> GSM340239 1 0.0672 0.978 0.992 0.008
#> GSM340240 1 0.0672 0.978 0.992 0.008
#> GSM340241 2 0.0672 0.909 0.008 0.992
#> GSM340242 1 0.0000 0.982 1.000 0.000
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.3129 0.5979 0.904 0.088 0.008
#> GSM340359 2 0.5988 0.8793 0.000 0.632 0.368
#> GSM340361 1 0.6192 0.3557 0.580 0.000 0.420
#> GSM340362 1 0.6192 0.3557 0.580 0.000 0.420
#> GSM340363 1 0.0237 0.6404 0.996 0.000 0.004
#> GSM340364 3 0.7276 0.1238 0.404 0.032 0.564
#> GSM340365 1 0.6192 0.3557 0.580 0.000 0.420
#> GSM340366 1 0.6111 0.3922 0.604 0.000 0.396
#> GSM340367 1 0.4235 0.5979 0.824 0.000 0.176
#> GSM340368 1 0.8705 0.0822 0.524 0.360 0.116
#> GSM340369 1 0.5948 0.3170 0.640 0.360 0.000
#> GSM340370 1 0.5070 0.5623 0.772 0.004 0.224
#> GSM340371 1 0.3590 0.6390 0.896 0.028 0.076
#> GSM340372 1 0.5760 0.4801 0.672 0.000 0.328
#> GSM340373 1 0.5760 0.4801 0.672 0.000 0.328
#> GSM340375 1 0.5591 0.5048 0.696 0.000 0.304
#> GSM340376 1 0.5070 0.5623 0.772 0.004 0.224
#> GSM340378 1 0.7181 0.1331 0.564 0.028 0.408
#> GSM340243 1 0.0237 0.6404 0.996 0.000 0.004
#> GSM340244 2 0.6111 0.8687 0.000 0.604 0.396
#> GSM340246 2 0.6168 0.8626 0.000 0.588 0.412
#> GSM340247 1 0.6169 0.3104 0.636 0.360 0.004
#> GSM340248 2 0.7043 0.8244 0.020 0.532 0.448
#> GSM340249 2 0.9491 0.3982 0.184 0.420 0.396
#> GSM340250 1 0.3752 0.6142 0.856 0.000 0.144
#> GSM340251 2 0.9492 0.4032 0.184 0.416 0.400
#> GSM340252 2 0.5988 0.8793 0.000 0.632 0.368
#> GSM340253 2 0.5988 0.8793 0.000 0.632 0.368
#> GSM340254 2 0.5988 0.8793 0.000 0.632 0.368
#> GSM340256 2 0.5948 0.8805 0.000 0.640 0.360
#> GSM340258 1 0.6075 0.4837 0.676 0.008 0.316
#> GSM340259 1 0.5621 0.5002 0.692 0.000 0.308
#> GSM340260 1 0.5016 0.5569 0.760 0.000 0.240
#> GSM340261 1 0.0892 0.6345 0.980 0.020 0.000
#> GSM340262 1 0.6111 0.3922 0.604 0.000 0.396
#> GSM340263 3 0.9452 -0.1676 0.220 0.284 0.496
#> GSM340264 1 0.5988 0.4308 0.632 0.000 0.368
#> GSM340265 1 0.5926 0.4466 0.644 0.000 0.356
#> GSM340266 1 0.0892 0.6355 0.980 0.020 0.000
#> GSM340267 1 0.5678 0.4931 0.684 0.000 0.316
#> GSM340268 1 0.2261 0.6093 0.932 0.068 0.000
#> GSM340269 1 0.0747 0.6358 0.984 0.016 0.000
#> GSM340270 1 0.0892 0.6414 0.980 0.000 0.020
#> GSM537574 1 0.3425 0.6252 0.884 0.004 0.112
#> GSM537580 1 0.3590 0.6390 0.896 0.028 0.076
#> GSM537581 1 0.5859 0.3362 0.656 0.344 0.000
#> GSM340272 1 0.6180 0.3621 0.584 0.000 0.416
#> GSM340273 2 0.6192 0.8620 0.000 0.580 0.420
#> GSM340275 1 0.9672 -0.1469 0.424 0.360 0.216
#> GSM340276 1 0.0424 0.6410 0.992 0.000 0.008
#> GSM340277 2 0.5988 0.8793 0.000 0.632 0.368
#> GSM340278 1 0.0475 0.6399 0.992 0.004 0.004
#> GSM340279 1 0.1031 0.6330 0.976 0.024 0.000
#> GSM340282 1 0.6111 0.3922 0.604 0.000 0.396
#> GSM340284 3 0.9866 0.2469 0.356 0.256 0.388
#> GSM340285 1 0.0237 0.6404 0.996 0.000 0.004
#> GSM340286 1 0.6192 0.3557 0.580 0.000 0.420
#> GSM340287 1 0.0747 0.6358 0.984 0.016 0.000
#> GSM340288 1 0.5650 0.4970 0.688 0.000 0.312
#> GSM340289 1 0.4452 0.5886 0.808 0.000 0.192
#> GSM340290 1 0.1031 0.6330 0.976 0.024 0.000
#> GSM340291 2 0.5948 0.8805 0.000 0.640 0.360
#> GSM340293 1 0.5016 0.4488 0.760 0.240 0.000
#> GSM340294 1 0.0892 0.6345 0.980 0.020 0.000
#> GSM340296 1 0.0747 0.6362 0.984 0.016 0.000
#> GSM340297 1 0.0892 0.6345 0.980 0.020 0.000
#> GSM340298 1 0.0892 0.6345 0.980 0.020 0.000
#> GSM340299 1 0.6192 0.3557 0.580 0.000 0.420
#> GSM340301 1 0.6192 0.3557 0.580 0.000 0.420
#> GSM340303 1 0.5926 0.4466 0.644 0.000 0.356
#> GSM340304 1 0.1753 0.6404 0.952 0.000 0.048
#> GSM340306 1 0.6155 0.3521 0.664 0.328 0.008
#> GSM340307 3 0.9470 -0.3041 0.200 0.324 0.476
#> GSM340310 1 0.3340 0.6227 0.880 0.000 0.120
#> GSM340314 1 0.2625 0.5984 0.916 0.084 0.000
#> GSM340315 1 0.2339 0.6418 0.940 0.012 0.048
#> GSM340317 2 0.9544 0.3627 0.192 0.420 0.388
#> GSM340318 1 0.5948 0.3170 0.640 0.360 0.000
#> GSM340319 1 0.5948 0.3170 0.640 0.360 0.000
#> GSM340320 1 0.5905 0.3266 0.648 0.352 0.000
#> GSM340321 2 0.5948 0.8805 0.000 0.640 0.360
#> GSM340322 1 0.6427 0.3308 0.640 0.348 0.012
#> GSM340324 2 0.5948 0.8805 0.000 0.640 0.360
#> GSM340328 1 0.3129 0.5979 0.904 0.088 0.008
#> GSM340330 1 0.2564 0.6382 0.936 0.036 0.028
#> GSM340332 1 0.5948 0.3170 0.640 0.360 0.000
#> GSM340333 2 0.5988 0.8793 0.000 0.632 0.368
#> GSM340336 1 0.9672 -0.1469 0.424 0.360 0.216
#> GSM340337 1 0.6427 0.3308 0.640 0.348 0.012
#> GSM340338 1 0.2564 0.6382 0.936 0.036 0.028
#> GSM340339 1 0.5948 0.3170 0.640 0.360 0.000
#> GSM340340 1 0.9054 0.0120 0.496 0.360 0.144
#> GSM340341 1 0.6168 0.4856 0.780 0.096 0.124
#> GSM340343 1 0.5882 0.3313 0.652 0.348 0.000
#> GSM340344 3 0.7618 0.3250 0.304 0.068 0.628
#> GSM340346 1 0.0983 0.6374 0.980 0.016 0.004
#> GSM340347 1 0.5948 0.3170 0.640 0.360 0.000
#> GSM340348 1 0.5706 0.3609 0.680 0.320 0.000
#> GSM340349 1 0.3816 0.6119 0.852 0.000 0.148
#> GSM340350 1 0.3752 0.6138 0.856 0.000 0.144
#> GSM340351 1 0.5760 0.4801 0.672 0.000 0.328
#> GSM340354 1 0.2860 0.6349 0.912 0.004 0.084
#> GSM340356 2 0.6476 0.8429 0.004 0.548 0.448
#> GSM340357 1 0.4002 0.6061 0.840 0.000 0.160
#> GSM348183 1 0.5465 0.5178 0.712 0.000 0.288
#> GSM348191 1 0.0747 0.6358 0.984 0.016 0.000
#> GSM348193 1 0.4261 0.6189 0.848 0.012 0.140
#> GSM537578 1 0.3816 0.6119 0.852 0.000 0.148
#> GSM348181 1 0.0237 0.6404 0.996 0.000 0.004
#> GSM348182 1 0.5327 0.5320 0.728 0.000 0.272
#> GSM348184 2 0.8608 0.6781 0.104 0.512 0.384
#> GSM348185 2 0.9672 0.3050 0.212 0.404 0.384
#> GSM348186 2 0.5988 0.8793 0.000 0.632 0.368
#> GSM348187 1 0.0424 0.6410 0.992 0.000 0.008
#> GSM348188 1 0.5731 0.6096 0.804 0.088 0.108
#> GSM348189 1 0.3590 0.6390 0.896 0.028 0.076
#> GSM348190 1 0.6192 0.3557 0.580 0.000 0.420
#> GSM348194 1 0.5650 0.4970 0.688 0.000 0.312
#> GSM348195 1 0.5760 0.4801 0.672 0.000 0.328
#> GSM348196 1 0.0424 0.6410 0.992 0.000 0.008
#> GSM537585 1 0.5785 0.4756 0.668 0.000 0.332
#> GSM537594 2 0.5948 0.8805 0.000 0.640 0.360
#> GSM537596 1 0.0424 0.6410 0.992 0.000 0.008
#> GSM537597 1 0.2860 0.6349 0.912 0.004 0.084
#> GSM537602 1 0.3412 0.6214 0.876 0.000 0.124
#> GSM340184 1 0.1999 0.6426 0.952 0.012 0.036
#> GSM340185 2 0.5948 0.8805 0.000 0.640 0.360
#> GSM340186 2 0.8215 0.7441 0.080 0.540 0.380
#> GSM340187 1 0.7310 0.2458 0.600 0.360 0.040
#> GSM340189 1 0.5948 0.3170 0.640 0.360 0.000
#> GSM340190 3 0.9937 0.2107 0.328 0.288 0.384
#> GSM340191 1 0.2066 0.6146 0.940 0.060 0.000
#> GSM340192 3 0.7534 0.2221 0.368 0.048 0.584
#> GSM340193 1 0.4346 0.5933 0.816 0.000 0.184
#> GSM340194 1 0.6192 0.3557 0.580 0.000 0.420
#> GSM340195 1 0.6192 0.3557 0.580 0.000 0.420
#> GSM340196 1 0.5948 0.3170 0.640 0.360 0.000
#> GSM340197 1 0.6192 0.3557 0.580 0.000 0.420
#> GSM340198 2 0.5988 0.8793 0.000 0.632 0.368
#> GSM340199 1 0.2066 0.6146 0.940 0.060 0.000
#> GSM340200 3 0.9502 -0.2392 0.212 0.308 0.480
#> GSM340201 1 0.5948 0.3170 0.640 0.360 0.000
#> GSM340202 1 0.5948 0.3170 0.640 0.360 0.000
#> GSM340203 1 0.5948 0.3170 0.640 0.360 0.000
#> GSM340204 3 0.7389 0.1201 0.408 0.036 0.556
#> GSM340205 1 0.0237 0.6404 0.996 0.000 0.004
#> GSM340206 1 0.2165 0.6118 0.936 0.064 0.000
#> GSM340207 1 0.6111 0.3922 0.604 0.000 0.396
#> GSM340237 2 0.6192 0.8620 0.000 0.580 0.420
#> GSM340238 1 0.2448 0.6042 0.924 0.076 0.000
#> GSM340239 1 0.6192 0.3557 0.580 0.000 0.420
#> GSM340240 1 0.6192 0.3557 0.580 0.000 0.420
#> GSM340241 2 0.5948 0.8805 0.000 0.640 0.360
#> GSM340242 1 0.3590 0.6390 0.896 0.028 0.076
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 3 0.4916 0.31067 0.000 0.000 0.576 0.424
#> GSM340359 1 0.0188 0.95857 0.996 0.004 0.000 0.000
#> GSM340361 3 0.2586 0.56560 0.004 0.004 0.900 0.092
#> GSM340362 3 0.2586 0.56560 0.004 0.004 0.900 0.092
#> GSM340363 3 0.4624 0.53461 0.000 0.000 0.660 0.340
#> GSM340364 3 0.5751 0.30886 0.048 0.004 0.664 0.284
#> GSM340365 3 0.2586 0.56560 0.004 0.004 0.900 0.092
#> GSM340366 3 0.1902 0.58708 0.000 0.004 0.932 0.064
#> GSM340367 3 0.3219 0.63822 0.000 0.000 0.836 0.164
#> GSM340368 4 0.5361 0.63238 0.000 0.108 0.148 0.744
#> GSM340369 4 0.4382 0.79887 0.000 0.000 0.296 0.704
#> GSM340370 3 0.4134 0.62567 0.008 0.008 0.796 0.188
#> GSM340371 3 0.4382 0.57241 0.000 0.000 0.704 0.296
#> GSM340372 3 0.0188 0.62415 0.000 0.000 0.996 0.004
#> GSM340373 3 0.0188 0.62415 0.000 0.000 0.996 0.004
#> GSM340375 3 0.1211 0.63253 0.000 0.000 0.960 0.040
#> GSM340376 3 0.4134 0.62567 0.008 0.008 0.796 0.188
#> GSM340378 3 0.6185 0.33641 0.016 0.036 0.604 0.344
#> GSM340243 3 0.4624 0.53461 0.000 0.000 0.660 0.340
#> GSM340244 2 0.2999 0.71917 0.132 0.864 0.000 0.004
#> GSM340246 2 0.2714 0.72459 0.112 0.884 0.000 0.004
#> GSM340247 4 0.4356 0.79625 0.000 0.000 0.292 0.708
#> GSM340248 2 0.3463 0.73489 0.096 0.868 0.004 0.032
#> GSM340249 2 0.3486 0.67963 0.000 0.812 0.000 0.188
#> GSM340250 3 0.3569 0.63222 0.000 0.000 0.804 0.196
#> GSM340251 2 0.3444 0.68204 0.000 0.816 0.000 0.184
#> GSM340252 1 0.0469 0.95901 0.988 0.012 0.000 0.000
#> GSM340253 1 0.0188 0.95857 0.996 0.004 0.000 0.000
#> GSM340254 1 0.0336 0.96041 0.992 0.008 0.000 0.000
#> GSM340256 1 0.2814 0.83278 0.868 0.132 0.000 0.000
#> GSM340258 3 0.1635 0.62767 0.008 0.000 0.948 0.044
#> GSM340259 3 0.0817 0.63024 0.000 0.000 0.976 0.024
#> GSM340260 3 0.2345 0.63946 0.000 0.000 0.900 0.100
#> GSM340261 3 0.4730 0.49582 0.000 0.000 0.636 0.364
#> GSM340262 3 0.1902 0.58708 0.000 0.004 0.932 0.064
#> GSM340263 2 0.6058 0.56976 0.020 0.604 0.024 0.352
#> GSM340264 3 0.1305 0.60283 0.004 0.000 0.960 0.036
#> GSM340265 3 0.1474 0.61339 0.000 0.000 0.948 0.052
#> GSM340266 3 0.4730 0.49610 0.000 0.000 0.636 0.364
#> GSM340267 3 0.1118 0.62958 0.000 0.000 0.964 0.036
#> GSM340268 3 0.4888 0.37027 0.000 0.000 0.588 0.412
#> GSM340269 3 0.4713 0.50351 0.000 0.000 0.640 0.360
#> GSM340270 3 0.4543 0.55315 0.000 0.000 0.676 0.324
#> GSM537574 3 0.4697 0.58169 0.000 0.008 0.696 0.296
#> GSM537580 3 0.4382 0.57241 0.000 0.000 0.704 0.296
#> GSM537581 4 0.4477 0.77581 0.000 0.000 0.312 0.688
#> GSM340272 3 0.2334 0.57132 0.000 0.004 0.908 0.088
#> GSM340273 2 0.3443 0.72490 0.136 0.848 0.000 0.016
#> GSM340275 4 0.4286 0.42635 0.000 0.136 0.052 0.812
#> GSM340276 3 0.4605 0.54003 0.000 0.000 0.664 0.336
#> GSM340277 1 0.0336 0.96041 0.992 0.008 0.000 0.000
#> GSM340278 3 0.4643 0.52870 0.000 0.000 0.656 0.344
#> GSM340279 3 0.4746 0.48749 0.000 0.000 0.632 0.368
#> GSM340282 3 0.1978 0.58443 0.000 0.004 0.928 0.068
#> GSM340284 4 0.5936 -0.12333 0.000 0.380 0.044 0.576
#> GSM340285 3 0.4624 0.53461 0.000 0.000 0.660 0.340
#> GSM340286 3 0.2586 0.56560 0.004 0.004 0.900 0.092
#> GSM340287 3 0.4713 0.50351 0.000 0.000 0.640 0.360
#> GSM340288 3 0.0707 0.62966 0.000 0.000 0.980 0.020
#> GSM340289 3 0.3024 0.63926 0.000 0.000 0.852 0.148
#> GSM340290 3 0.4746 0.48749 0.000 0.000 0.632 0.368
#> GSM340291 2 0.4250 0.62910 0.276 0.724 0.000 0.000
#> GSM340293 4 0.4898 0.44966 0.000 0.000 0.416 0.584
#> GSM340294 3 0.4730 0.49582 0.000 0.000 0.636 0.364
#> GSM340296 3 0.4713 0.50348 0.000 0.000 0.640 0.360
#> GSM340297 3 0.4730 0.49582 0.000 0.000 0.636 0.364
#> GSM340298 3 0.4730 0.49582 0.000 0.000 0.636 0.364
#> GSM340299 3 0.2586 0.56560 0.004 0.004 0.900 0.092
#> GSM340301 3 0.2401 0.56856 0.000 0.004 0.904 0.092
#> GSM340303 3 0.1474 0.61339 0.000 0.000 0.948 0.052
#> GSM340304 3 0.4382 0.57826 0.000 0.000 0.704 0.296
#> GSM340306 4 0.4605 0.73941 0.000 0.000 0.336 0.664
#> GSM340307 2 0.6183 0.62393 0.020 0.680 0.064 0.236
#> GSM340310 3 0.3801 0.62346 0.000 0.000 0.780 0.220
#> GSM340314 3 0.4925 0.31605 0.000 0.000 0.572 0.428
#> GSM340315 3 0.4608 0.56724 0.000 0.004 0.692 0.304
#> GSM340317 2 0.4808 0.66468 0.020 0.768 0.016 0.196
#> GSM340318 4 0.4382 0.79887 0.000 0.000 0.296 0.704
#> GSM340319 4 0.4382 0.79887 0.000 0.000 0.296 0.704
#> GSM340320 4 0.4431 0.78910 0.000 0.000 0.304 0.696
#> GSM340321 2 0.4643 0.56065 0.344 0.656 0.000 0.000
#> GSM340322 4 0.4522 0.77545 0.000 0.000 0.320 0.680
#> GSM340324 1 0.2814 0.83278 0.868 0.132 0.000 0.000
#> GSM340328 3 0.4916 0.31067 0.000 0.000 0.576 0.424
#> GSM340330 3 0.4679 0.51035 0.000 0.000 0.648 0.352
#> GSM340332 4 0.4382 0.79887 0.000 0.000 0.296 0.704
#> GSM340333 1 0.0188 0.95857 0.996 0.004 0.000 0.000
#> GSM340336 4 0.4286 0.42635 0.000 0.136 0.052 0.812
#> GSM340337 4 0.4522 0.77545 0.000 0.000 0.320 0.680
#> GSM340338 3 0.4679 0.51035 0.000 0.000 0.648 0.352
#> GSM340339 4 0.4382 0.79887 0.000 0.000 0.296 0.704
#> GSM340340 4 0.5432 0.58443 0.000 0.136 0.124 0.740
#> GSM340341 3 0.7254 0.00598 0.004 0.128 0.476 0.392
#> GSM340343 4 0.4454 0.78314 0.000 0.000 0.308 0.692
#> GSM340344 3 0.7387 0.05287 0.180 0.004 0.532 0.284
#> GSM340346 3 0.4697 0.51085 0.000 0.000 0.644 0.356
#> GSM340347 4 0.4382 0.79887 0.000 0.000 0.296 0.704
#> GSM340348 4 0.4605 0.72301 0.000 0.000 0.336 0.664
#> GSM340349 3 0.3528 0.63285 0.000 0.000 0.808 0.192
#> GSM340350 3 0.3569 0.63180 0.000 0.000 0.804 0.196
#> GSM340351 3 0.0188 0.62415 0.000 0.000 0.996 0.004
#> GSM340354 3 0.4372 0.59981 0.000 0.004 0.728 0.268
#> GSM340356 2 0.3367 0.73163 0.108 0.864 0.000 0.028
#> GSM340357 3 0.3400 0.63535 0.000 0.000 0.820 0.180
#> GSM348183 3 0.1302 0.63382 0.000 0.000 0.956 0.044
#> GSM348191 3 0.4713 0.50351 0.000 0.000 0.640 0.360
#> GSM348193 3 0.3726 0.62595 0.000 0.000 0.788 0.212
#> GSM537578 3 0.3528 0.63285 0.000 0.000 0.808 0.192
#> GSM348181 3 0.4624 0.53461 0.000 0.000 0.660 0.340
#> GSM348182 3 0.1716 0.63687 0.000 0.000 0.936 0.064
#> GSM348184 2 0.2596 0.72443 0.024 0.908 0.000 0.068
#> GSM348185 2 0.4497 0.65225 0.008 0.776 0.016 0.200
#> GSM348186 1 0.0336 0.96041 0.992 0.008 0.000 0.000
#> GSM348187 3 0.4605 0.54003 0.000 0.000 0.664 0.336
#> GSM348188 3 0.4522 0.49243 0.000 0.000 0.680 0.320
#> GSM348189 3 0.4382 0.57241 0.000 0.000 0.704 0.296
#> GSM348190 3 0.2586 0.56560 0.004 0.004 0.900 0.092
#> GSM348194 3 0.0707 0.62966 0.000 0.000 0.980 0.020
#> GSM348195 3 0.0188 0.62415 0.000 0.000 0.996 0.004
#> GSM348196 3 0.4605 0.54003 0.000 0.000 0.664 0.336
#> GSM537585 3 0.0188 0.62431 0.000 0.000 0.996 0.004
#> GSM537594 2 0.4643 0.56065 0.344 0.656 0.000 0.000
#> GSM537596 3 0.4605 0.54003 0.000 0.000 0.664 0.336
#> GSM537597 3 0.4372 0.59981 0.000 0.004 0.728 0.268
#> GSM537602 3 0.3764 0.62516 0.000 0.000 0.784 0.216
#> GSM340184 3 0.4522 0.55598 0.000 0.000 0.680 0.320
#> GSM340185 2 0.4643 0.56065 0.344 0.656 0.000 0.000
#> GSM340186 2 0.3071 0.73402 0.068 0.888 0.000 0.044
#> GSM340187 4 0.5598 0.70824 0.000 0.076 0.220 0.704
#> GSM340189 4 0.4382 0.79887 0.000 0.000 0.296 0.704
#> GSM340190 4 0.5982 -0.24844 0.000 0.436 0.040 0.524
#> GSM340191 3 0.4866 0.39544 0.000 0.000 0.596 0.404
#> GSM340192 3 0.6669 0.24713 0.108 0.004 0.604 0.284
#> GSM340193 3 0.3123 0.63861 0.000 0.000 0.844 0.156
#> GSM340194 3 0.2586 0.56560 0.004 0.004 0.900 0.092
#> GSM340195 3 0.2586 0.56560 0.004 0.004 0.900 0.092
#> GSM340196 4 0.4382 0.79887 0.000 0.000 0.296 0.704
#> GSM340197 3 0.2586 0.56560 0.004 0.004 0.900 0.092
#> GSM340198 1 0.0469 0.95901 0.988 0.012 0.000 0.000
#> GSM340199 3 0.4866 0.39544 0.000 0.000 0.596 0.404
#> GSM340200 2 0.6263 0.60740 0.016 0.664 0.068 0.252
#> GSM340201 4 0.4382 0.79887 0.000 0.000 0.296 0.704
#> GSM340202 4 0.4382 0.79887 0.000 0.000 0.296 0.704
#> GSM340203 4 0.4382 0.79887 0.000 0.000 0.296 0.704
#> GSM340204 3 0.5825 0.30774 0.052 0.004 0.660 0.284
#> GSM340205 3 0.4624 0.53461 0.000 0.000 0.660 0.340
#> GSM340206 3 0.4877 0.38292 0.000 0.000 0.592 0.408
#> GSM340207 3 0.1902 0.58708 0.000 0.004 0.932 0.064
#> GSM340237 2 0.3443 0.72490 0.136 0.848 0.000 0.016
#> GSM340238 3 0.4907 0.34524 0.000 0.000 0.580 0.420
#> GSM340239 3 0.2586 0.56560 0.004 0.004 0.900 0.092
#> GSM340240 3 0.2586 0.56560 0.004 0.004 0.900 0.092
#> GSM340241 2 0.4661 0.55582 0.348 0.652 0.000 0.000
#> GSM340242 3 0.4382 0.57241 0.000 0.000 0.704 0.296
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 3 0.2361 0.6821 0.000 0.000 0.892 0.096 0.012
#> GSM340359 1 0.0162 0.9488 0.996 0.000 0.000 0.004 0.000
#> GSM340361 5 0.3132 0.8117 0.000 0.000 0.172 0.008 0.820
#> GSM340362 5 0.3132 0.8117 0.000 0.000 0.172 0.008 0.820
#> GSM340363 3 0.0290 0.7044 0.000 0.000 0.992 0.000 0.008
#> GSM340364 5 0.5406 0.5531 0.032 0.004 0.084 0.160 0.720
#> GSM340365 5 0.3132 0.8117 0.000 0.000 0.172 0.008 0.820
#> GSM340366 5 0.3586 0.7578 0.000 0.000 0.264 0.000 0.736
#> GSM340367 3 0.3123 0.5743 0.000 0.000 0.812 0.004 0.184
#> GSM340368 4 0.4564 0.6714 0.000 0.020 0.196 0.748 0.036
#> GSM340369 3 0.4300 0.1156 0.000 0.000 0.524 0.476 0.000
#> GSM340370 3 0.5707 0.1162 0.004 0.004 0.576 0.072 0.344
#> GSM340371 3 0.2511 0.6826 0.000 0.000 0.892 0.028 0.080
#> GSM340372 3 0.4397 -0.0673 0.000 0.000 0.564 0.004 0.432
#> GSM340373 3 0.4397 -0.0673 0.000 0.000 0.564 0.004 0.432
#> GSM340375 3 0.4030 0.2523 0.000 0.000 0.648 0.000 0.352
#> GSM340376 3 0.5707 0.1184 0.004 0.004 0.576 0.072 0.344
#> GSM340378 5 0.7441 0.3305 0.008 0.024 0.324 0.232 0.412
#> GSM340243 3 0.0290 0.7044 0.000 0.000 0.992 0.000 0.008
#> GSM340244 2 0.1996 0.7609 0.036 0.928 0.000 0.032 0.004
#> GSM340246 2 0.1808 0.7662 0.020 0.936 0.000 0.040 0.004
#> GSM340247 3 0.4446 0.1014 0.000 0.000 0.520 0.476 0.004
#> GSM340248 2 0.2364 0.7731 0.020 0.908 0.000 0.064 0.008
#> GSM340249 2 0.4533 0.6568 0.000 0.704 0.004 0.260 0.032
#> GSM340250 3 0.2806 0.6142 0.000 0.000 0.844 0.004 0.152
#> GSM340251 2 0.4508 0.6625 0.000 0.708 0.004 0.256 0.032
#> GSM340252 1 0.0162 0.9491 0.996 0.004 0.000 0.000 0.000
#> GSM340253 1 0.0162 0.9488 0.996 0.000 0.000 0.004 0.000
#> GSM340254 1 0.0000 0.9496 1.000 0.000 0.000 0.000 0.000
#> GSM340256 1 0.3551 0.8049 0.820 0.136 0.000 0.000 0.044
#> GSM340258 5 0.4787 0.4243 0.008 0.000 0.444 0.008 0.540
#> GSM340259 3 0.4211 0.1957 0.000 0.000 0.636 0.004 0.360
#> GSM340260 3 0.3715 0.4635 0.000 0.000 0.736 0.004 0.260
#> GSM340261 3 0.0510 0.7052 0.000 0.000 0.984 0.016 0.000
#> GSM340262 5 0.3661 0.7445 0.000 0.000 0.276 0.000 0.724
#> GSM340263 2 0.5590 0.4744 0.000 0.536 0.024 0.408 0.032
#> GSM340264 5 0.4256 0.4437 0.000 0.000 0.436 0.000 0.564
#> GSM340265 5 0.4305 0.2937 0.000 0.000 0.488 0.000 0.512
#> GSM340266 3 0.0510 0.7058 0.000 0.000 0.984 0.016 0.000
#> GSM340267 3 0.4210 0.0368 0.000 0.000 0.588 0.000 0.412
#> GSM340268 3 0.1544 0.6938 0.000 0.000 0.932 0.068 0.000
#> GSM340269 3 0.0404 0.7052 0.000 0.000 0.988 0.012 0.000
#> GSM340270 3 0.0703 0.7001 0.000 0.000 0.976 0.000 0.024
#> GSM537574 3 0.3749 0.6012 0.000 0.000 0.816 0.104 0.080
#> GSM537580 3 0.2511 0.6826 0.000 0.000 0.892 0.028 0.080
#> GSM537581 3 0.4242 0.2256 0.000 0.000 0.572 0.428 0.000
#> GSM340272 5 0.3300 0.8057 0.000 0.000 0.204 0.004 0.792
#> GSM340273 2 0.1251 0.7716 0.036 0.956 0.000 0.008 0.000
#> GSM340275 4 0.4195 0.6803 0.000 0.056 0.096 0.812 0.036
#> GSM340276 3 0.0404 0.7038 0.000 0.000 0.988 0.000 0.012
#> GSM340277 1 0.0000 0.9496 1.000 0.000 0.000 0.000 0.000
#> GSM340278 3 0.0451 0.7050 0.000 0.000 0.988 0.004 0.008
#> GSM340279 3 0.0609 0.7048 0.000 0.000 0.980 0.020 0.000
#> GSM340282 5 0.3534 0.7655 0.000 0.000 0.256 0.000 0.744
#> GSM340284 4 0.5969 0.2866 0.000 0.288 0.072 0.608 0.032
#> GSM340285 3 0.0290 0.7044 0.000 0.000 0.992 0.000 0.008
#> GSM340286 5 0.3048 0.8135 0.000 0.000 0.176 0.004 0.820
#> GSM340287 3 0.0404 0.7052 0.000 0.000 0.988 0.012 0.000
#> GSM340288 3 0.4151 0.2586 0.000 0.000 0.652 0.004 0.344
#> GSM340289 3 0.3231 0.5606 0.000 0.000 0.800 0.004 0.196
#> GSM340290 3 0.0609 0.7048 0.000 0.000 0.980 0.020 0.000
#> GSM340291 2 0.3758 0.7095 0.128 0.816 0.000 0.004 0.052
#> GSM340293 3 0.3774 0.4594 0.000 0.000 0.704 0.296 0.000
#> GSM340294 3 0.0510 0.7052 0.000 0.000 0.984 0.016 0.000
#> GSM340296 3 0.0671 0.7056 0.000 0.000 0.980 0.016 0.004
#> GSM340297 3 0.0510 0.7052 0.000 0.000 0.984 0.016 0.000
#> GSM340298 3 0.0510 0.7052 0.000 0.000 0.984 0.016 0.000
#> GSM340299 5 0.3132 0.8117 0.000 0.000 0.172 0.008 0.820
#> GSM340301 5 0.3231 0.8100 0.000 0.000 0.196 0.004 0.800
#> GSM340303 5 0.4305 0.2937 0.000 0.000 0.488 0.000 0.512
#> GSM340304 3 0.1270 0.6890 0.000 0.000 0.948 0.000 0.052
#> GSM340306 3 0.4403 0.3080 0.000 0.000 0.608 0.384 0.008
#> GSM340307 2 0.5854 0.5695 0.004 0.596 0.060 0.320 0.020
#> GSM340310 3 0.2488 0.6383 0.000 0.000 0.872 0.004 0.124
#> GSM340314 3 0.1851 0.6868 0.000 0.000 0.912 0.088 0.000
#> GSM340315 3 0.2153 0.6830 0.000 0.000 0.916 0.040 0.044
#> GSM340317 2 0.4905 0.6377 0.000 0.692 0.016 0.256 0.036
#> GSM340318 3 0.4300 0.1156 0.000 0.000 0.524 0.476 0.000
#> GSM340319 3 0.4300 0.1156 0.000 0.000 0.524 0.476 0.000
#> GSM340320 3 0.4235 0.2401 0.000 0.000 0.576 0.424 0.000
#> GSM340321 2 0.5113 0.6519 0.176 0.728 0.000 0.032 0.064
#> GSM340322 3 0.4617 0.1926 0.000 0.000 0.552 0.436 0.012
#> GSM340324 1 0.3551 0.8049 0.820 0.136 0.000 0.000 0.044
#> GSM340328 3 0.2361 0.6821 0.000 0.000 0.892 0.096 0.012
#> GSM340330 3 0.1668 0.7041 0.000 0.000 0.940 0.032 0.028
#> GSM340332 3 0.4300 0.1156 0.000 0.000 0.524 0.476 0.000
#> GSM340333 1 0.0162 0.9488 0.996 0.000 0.000 0.004 0.000
#> GSM340336 4 0.4195 0.6803 0.000 0.056 0.096 0.812 0.036
#> GSM340337 3 0.4617 0.1926 0.000 0.000 0.552 0.436 0.012
#> GSM340338 3 0.1668 0.7041 0.000 0.000 0.940 0.032 0.028
#> GSM340339 3 0.4256 0.2166 0.000 0.000 0.564 0.436 0.000
#> GSM340340 4 0.4993 0.6895 0.000 0.056 0.168 0.740 0.036
#> GSM340341 3 0.5968 0.3541 0.000 0.072 0.636 0.248 0.044
#> GSM340343 3 0.4219 0.2541 0.000 0.000 0.584 0.416 0.000
#> GSM340344 5 0.5541 0.3179 0.168 0.000 0.008 0.152 0.672
#> GSM340346 3 0.0566 0.7056 0.000 0.000 0.984 0.012 0.004
#> GSM340347 3 0.4300 0.1156 0.000 0.000 0.524 0.476 0.000
#> GSM340348 3 0.4101 0.3315 0.000 0.000 0.628 0.372 0.000
#> GSM340349 3 0.2848 0.6102 0.000 0.000 0.840 0.004 0.156
#> GSM340350 3 0.2763 0.6162 0.000 0.000 0.848 0.004 0.148
#> GSM340351 3 0.4397 -0.0673 0.000 0.000 0.564 0.004 0.432
#> GSM340354 3 0.3051 0.6426 0.000 0.000 0.864 0.060 0.076
#> GSM340356 2 0.1901 0.7748 0.024 0.932 0.000 0.040 0.004
#> GSM340357 3 0.3160 0.5722 0.000 0.000 0.808 0.004 0.188
#> GSM348183 3 0.4299 0.0925 0.000 0.000 0.608 0.004 0.388
#> GSM348191 3 0.0404 0.7052 0.000 0.000 0.988 0.012 0.000
#> GSM348193 3 0.3224 0.6153 0.000 0.000 0.824 0.016 0.160
#> GSM537578 3 0.2848 0.6102 0.000 0.000 0.840 0.004 0.156
#> GSM348181 3 0.0290 0.7044 0.000 0.000 0.992 0.000 0.008
#> GSM348182 3 0.4251 0.1709 0.000 0.000 0.624 0.004 0.372
#> GSM348184 2 0.3812 0.7407 0.000 0.800 0.004 0.160 0.036
#> GSM348185 2 0.5000 0.6198 0.000 0.676 0.016 0.272 0.036
#> GSM348186 1 0.1357 0.9270 0.948 0.004 0.000 0.000 0.048
#> GSM348187 3 0.0404 0.7038 0.000 0.000 0.988 0.000 0.012
#> GSM348188 3 0.5122 0.5145 0.000 0.000 0.688 0.112 0.200
#> GSM348189 3 0.2511 0.6826 0.000 0.000 0.892 0.028 0.080
#> GSM348190 5 0.3086 0.8133 0.000 0.000 0.180 0.004 0.816
#> GSM348194 3 0.4151 0.2586 0.000 0.000 0.652 0.004 0.344
#> GSM348195 3 0.4397 -0.0673 0.000 0.000 0.564 0.004 0.432
#> GSM348196 3 0.0404 0.7038 0.000 0.000 0.988 0.000 0.012
#> GSM537585 3 0.4375 -0.0140 0.000 0.000 0.576 0.004 0.420
#> GSM537594 2 0.5059 0.6481 0.184 0.728 0.000 0.032 0.056
#> GSM537596 3 0.0404 0.7038 0.000 0.000 0.988 0.000 0.012
#> GSM537597 3 0.3239 0.6330 0.000 0.000 0.852 0.068 0.080
#> GSM537602 3 0.2536 0.6347 0.000 0.000 0.868 0.004 0.128
#> GSM340184 3 0.1281 0.6993 0.000 0.000 0.956 0.012 0.032
#> GSM340185 2 0.5113 0.6519 0.176 0.728 0.000 0.032 0.064
#> GSM340186 2 0.3796 0.7618 0.028 0.824 0.004 0.128 0.016
#> GSM340187 4 0.5454 0.4056 0.000 0.020 0.356 0.588 0.036
#> GSM340189 3 0.4300 0.1156 0.000 0.000 0.524 0.476 0.000
#> GSM340190 4 0.5922 0.1395 0.000 0.328 0.052 0.584 0.036
#> GSM340191 3 0.1410 0.6969 0.000 0.000 0.940 0.060 0.000
#> GSM340192 5 0.5679 0.4645 0.092 0.000 0.048 0.164 0.696
#> GSM340193 3 0.3160 0.5705 0.000 0.000 0.808 0.004 0.188
#> GSM340194 5 0.3132 0.8117 0.000 0.000 0.172 0.008 0.820
#> GSM340195 5 0.3132 0.8117 0.000 0.000 0.172 0.008 0.820
#> GSM340196 3 0.4300 0.1156 0.000 0.000 0.524 0.476 0.000
#> GSM340197 5 0.3048 0.8135 0.000 0.000 0.176 0.004 0.820
#> GSM340198 1 0.0162 0.9491 0.996 0.004 0.000 0.000 0.000
#> GSM340199 3 0.1410 0.6969 0.000 0.000 0.940 0.060 0.000
#> GSM340200 2 0.5981 0.5387 0.004 0.572 0.064 0.340 0.020
#> GSM340201 3 0.4300 0.1156 0.000 0.000 0.524 0.476 0.000
#> GSM340202 3 0.4300 0.1156 0.000 0.000 0.524 0.476 0.000
#> GSM340203 3 0.4300 0.1156 0.000 0.000 0.524 0.476 0.000
#> GSM340204 5 0.4918 0.5411 0.036 0.000 0.056 0.160 0.748
#> GSM340205 3 0.0290 0.7044 0.000 0.000 0.992 0.000 0.008
#> GSM340206 3 0.1478 0.6953 0.000 0.000 0.936 0.064 0.000
#> GSM340207 5 0.3661 0.7445 0.000 0.000 0.276 0.000 0.724
#> GSM340237 2 0.1251 0.7716 0.036 0.956 0.000 0.008 0.000
#> GSM340238 3 0.1732 0.6903 0.000 0.000 0.920 0.080 0.000
#> GSM340239 5 0.3048 0.8135 0.000 0.000 0.176 0.004 0.820
#> GSM340240 5 0.3132 0.8117 0.000 0.000 0.172 0.008 0.820
#> GSM340241 2 0.5148 0.6489 0.180 0.724 0.000 0.032 0.064
#> GSM340242 3 0.2511 0.6826 0.000 0.000 0.892 0.028 0.080
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 3 0.2118 0.6229 0.000 0.000 0.888 0.104 0.008 0.000
#> GSM340359 2 0.0146 0.9290 0.000 0.996 0.000 0.004 0.000 0.000
#> GSM340361 5 0.2243 0.8041 0.000 0.000 0.112 0.004 0.880 0.004
#> GSM340362 5 0.2243 0.8041 0.000 0.000 0.112 0.004 0.880 0.004
#> GSM340363 3 0.0260 0.7143 0.000 0.000 0.992 0.000 0.008 0.000
#> GSM340364 5 0.6334 0.3773 0.020 0.024 0.036 0.220 0.612 0.088
#> GSM340365 5 0.2243 0.8041 0.000 0.000 0.112 0.004 0.880 0.004
#> GSM340366 5 0.3341 0.7573 0.000 0.000 0.208 0.012 0.776 0.004
#> GSM340367 3 0.3580 0.6693 0.000 0.000 0.808 0.036 0.136 0.020
#> GSM340368 4 0.5386 0.1374 0.316 0.000 0.136 0.548 0.000 0.000
#> GSM340369 4 0.3838 0.7135 0.000 0.000 0.448 0.552 0.000 0.000
#> GSM340370 3 0.7034 0.2411 0.020 0.000 0.520 0.176 0.192 0.092
#> GSM340371 3 0.2639 0.7026 0.000 0.000 0.876 0.032 0.084 0.008
#> GSM340372 3 0.5017 0.1097 0.000 0.000 0.532 0.036 0.412 0.020
#> GSM340373 3 0.5017 0.1097 0.000 0.000 0.532 0.036 0.412 0.020
#> GSM340375 3 0.4928 0.4210 0.000 0.000 0.628 0.040 0.304 0.028
#> GSM340376 3 0.7034 0.2412 0.020 0.000 0.520 0.176 0.192 0.092
#> GSM340378 4 0.8216 -0.2334 0.056 0.004 0.264 0.312 0.272 0.092
#> GSM340243 3 0.0260 0.7143 0.000 0.000 0.992 0.000 0.008 0.000
#> GSM340244 6 0.4885 0.6293 0.392 0.000 0.000 0.040 0.012 0.556
#> GSM340246 6 0.4991 0.5947 0.424 0.000 0.000 0.044 0.012 0.520
#> GSM340247 4 0.3828 0.7108 0.000 0.000 0.440 0.560 0.000 0.000
#> GSM340248 1 0.3847 -0.4772 0.544 0.000 0.000 0.000 0.000 0.456
#> GSM340249 1 0.2019 0.5974 0.900 0.000 0.000 0.012 0.000 0.088
#> GSM340250 3 0.3150 0.6896 0.000 0.000 0.844 0.036 0.104 0.016
#> GSM340251 1 0.2214 0.5946 0.888 0.000 0.000 0.016 0.000 0.096
#> GSM340252 2 0.0146 0.9283 0.004 0.996 0.000 0.000 0.000 0.000
#> GSM340253 2 0.0146 0.9290 0.000 0.996 0.000 0.004 0.000 0.000
#> GSM340254 2 0.0000 0.9290 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340256 2 0.3509 0.7418 0.016 0.744 0.000 0.000 0.000 0.240
#> GSM340258 5 0.6057 0.3678 0.004 0.004 0.384 0.076 0.492 0.040
#> GSM340259 3 0.4811 0.3636 0.000 0.000 0.616 0.036 0.328 0.020
#> GSM340260 3 0.4242 0.6009 0.000 0.000 0.728 0.036 0.216 0.020
#> GSM340261 3 0.0508 0.7063 0.000 0.000 0.984 0.012 0.000 0.004
#> GSM340262 5 0.3426 0.7439 0.000 0.000 0.220 0.012 0.764 0.004
#> GSM340263 1 0.4313 0.5863 0.728 0.000 0.000 0.184 0.004 0.084
#> GSM340264 5 0.4780 0.3709 0.000 0.000 0.392 0.028 0.564 0.016
#> GSM340265 5 0.4916 0.2182 0.000 0.000 0.444 0.032 0.508 0.016
#> GSM340266 3 0.0547 0.7044 0.000 0.000 0.980 0.020 0.000 0.000
#> GSM340267 3 0.5176 0.1599 0.000 0.000 0.544 0.040 0.388 0.028
#> GSM340268 3 0.1531 0.6623 0.000 0.000 0.928 0.068 0.000 0.004
#> GSM340269 3 0.0405 0.7084 0.000 0.000 0.988 0.008 0.000 0.004
#> GSM340270 3 0.0725 0.7164 0.000 0.000 0.976 0.012 0.012 0.000
#> GSM537574 3 0.4088 0.5654 0.020 0.000 0.768 0.172 0.008 0.032
#> GSM537580 3 0.2639 0.7026 0.000 0.000 0.876 0.032 0.084 0.008
#> GSM537581 3 0.3867 -0.6162 0.000 0.000 0.512 0.488 0.000 0.000
#> GSM340272 5 0.2442 0.8011 0.000 0.000 0.144 0.000 0.852 0.004
#> GSM340273 6 0.4209 0.6241 0.428 0.004 0.000 0.004 0.004 0.560
#> GSM340275 4 0.4514 -0.2066 0.372 0.000 0.040 0.588 0.000 0.000
#> GSM340276 3 0.0405 0.7152 0.000 0.000 0.988 0.004 0.008 0.000
#> GSM340277 2 0.0000 0.9290 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340278 3 0.0405 0.7131 0.000 0.000 0.988 0.004 0.008 0.000
#> GSM340279 3 0.0692 0.7027 0.000 0.000 0.976 0.020 0.000 0.004
#> GSM340282 5 0.3281 0.7646 0.000 0.000 0.200 0.012 0.784 0.004
#> GSM340284 1 0.4118 0.4417 0.628 0.000 0.020 0.352 0.000 0.000
#> GSM340285 3 0.0260 0.7143 0.000 0.000 0.992 0.000 0.008 0.000
#> GSM340286 5 0.2146 0.8060 0.000 0.000 0.116 0.000 0.880 0.004
#> GSM340287 3 0.0405 0.7084 0.000 0.000 0.988 0.008 0.000 0.004
#> GSM340288 3 0.4735 0.4291 0.000 0.000 0.636 0.036 0.308 0.020
#> GSM340289 3 0.3696 0.6617 0.000 0.000 0.796 0.036 0.148 0.020
#> GSM340290 3 0.0692 0.7027 0.000 0.000 0.976 0.020 0.000 0.004
#> GSM340291 6 0.3991 0.6764 0.272 0.004 0.000 0.008 0.012 0.704
#> GSM340293 3 0.3515 -0.0463 0.000 0.000 0.676 0.324 0.000 0.000
#> GSM340294 3 0.0508 0.7063 0.000 0.000 0.984 0.012 0.000 0.004
#> GSM340296 3 0.0653 0.7083 0.000 0.000 0.980 0.012 0.004 0.004
#> GSM340297 3 0.0508 0.7063 0.000 0.000 0.984 0.012 0.000 0.004
#> GSM340298 3 0.0508 0.7063 0.000 0.000 0.984 0.012 0.000 0.004
#> GSM340299 5 0.2243 0.8041 0.000 0.000 0.112 0.004 0.880 0.004
#> GSM340301 5 0.2362 0.8034 0.000 0.000 0.136 0.000 0.860 0.004
#> GSM340303 5 0.4916 0.2182 0.000 0.000 0.444 0.032 0.508 0.016
#> GSM340304 3 0.1334 0.7163 0.000 0.000 0.948 0.020 0.032 0.000
#> GSM340306 3 0.4364 -0.4719 0.000 0.000 0.556 0.424 0.012 0.008
#> GSM340307 1 0.5153 0.5570 0.692 0.000 0.028 0.132 0.004 0.144
#> GSM340310 3 0.2797 0.6992 0.000 0.000 0.872 0.036 0.076 0.016
#> GSM340314 3 0.1908 0.6315 0.000 0.000 0.900 0.096 0.000 0.004
#> GSM340315 3 0.2182 0.6910 0.004 0.000 0.900 0.076 0.000 0.020
#> GSM340317 1 0.2290 0.6079 0.892 0.000 0.000 0.020 0.004 0.084
#> GSM340318 4 0.3838 0.7135 0.000 0.000 0.448 0.552 0.000 0.000
#> GSM340319 4 0.3838 0.7135 0.000 0.000 0.448 0.552 0.000 0.000
#> GSM340320 3 0.3862 -0.5838 0.000 0.000 0.524 0.476 0.000 0.000
#> GSM340321 6 0.2473 0.6643 0.104 0.012 0.000 0.008 0.000 0.876
#> GSM340322 4 0.4413 0.6355 0.000 0.000 0.484 0.496 0.012 0.008
#> GSM340324 2 0.3606 0.7250 0.016 0.728 0.000 0.000 0.000 0.256
#> GSM340328 3 0.2118 0.6229 0.000 0.000 0.888 0.104 0.008 0.000
#> GSM340330 3 0.1562 0.7100 0.000 0.000 0.940 0.032 0.024 0.004
#> GSM340332 4 0.3838 0.7135 0.000 0.000 0.448 0.552 0.000 0.000
#> GSM340333 2 0.0146 0.9290 0.000 0.996 0.000 0.004 0.000 0.000
#> GSM340336 4 0.4514 -0.2066 0.372 0.000 0.040 0.588 0.000 0.000
#> GSM340337 4 0.4413 0.6355 0.000 0.000 0.484 0.496 0.012 0.008
#> GSM340338 3 0.1562 0.7100 0.000 0.000 0.940 0.032 0.024 0.004
#> GSM340339 4 0.3869 0.6106 0.000 0.000 0.500 0.500 0.000 0.000
#> GSM340340 4 0.5319 0.0193 0.368 0.000 0.112 0.520 0.000 0.000
#> GSM340341 3 0.6057 0.1272 0.220 0.000 0.568 0.180 0.004 0.028
#> GSM340343 3 0.3854 -0.5548 0.000 0.000 0.536 0.464 0.000 0.000
#> GSM340344 5 0.4905 0.2966 0.004 0.164 0.000 0.160 0.672 0.000
#> GSM340346 3 0.0508 0.7104 0.000 0.000 0.984 0.012 0.000 0.004
#> GSM340347 4 0.3838 0.7135 0.000 0.000 0.448 0.552 0.000 0.000
#> GSM340348 3 0.3804 -0.4422 0.000 0.000 0.576 0.424 0.000 0.000
#> GSM340349 3 0.3282 0.6851 0.000 0.000 0.836 0.036 0.108 0.020
#> GSM340350 3 0.3103 0.6903 0.000 0.000 0.848 0.036 0.100 0.016
#> GSM340351 3 0.5017 0.1097 0.000 0.000 0.532 0.036 0.412 0.020
#> GSM340354 3 0.3461 0.6365 0.012 0.000 0.824 0.124 0.008 0.032
#> GSM340356 6 0.3998 0.5140 0.492 0.004 0.000 0.000 0.000 0.504
#> GSM340357 3 0.3658 0.6642 0.000 0.000 0.800 0.036 0.144 0.020
#> GSM348183 3 0.4878 0.2405 0.000 0.000 0.576 0.032 0.372 0.020
#> GSM348191 3 0.0405 0.7084 0.000 0.000 0.988 0.008 0.000 0.004
#> GSM348193 3 0.3614 0.6814 0.000 0.000 0.812 0.048 0.120 0.020
#> GSM537578 3 0.3236 0.6871 0.000 0.000 0.840 0.036 0.104 0.020
#> GSM348181 3 0.0260 0.7143 0.000 0.000 0.992 0.000 0.008 0.000
#> GSM348182 3 0.4888 0.3082 0.000 0.000 0.592 0.036 0.352 0.020
#> GSM348184 1 0.4330 0.3084 0.708 0.000 0.000 0.044 0.012 0.236
#> GSM348185 1 0.1923 0.6146 0.916 0.000 0.000 0.016 0.004 0.064
#> GSM348186 2 0.2048 0.8699 0.000 0.880 0.000 0.000 0.000 0.120
#> GSM348187 3 0.0405 0.7152 0.000 0.000 0.988 0.004 0.008 0.000
#> GSM348188 3 0.5264 0.4751 0.000 0.000 0.636 0.136 0.216 0.012
#> GSM348189 3 0.2639 0.7026 0.000 0.000 0.876 0.032 0.084 0.008
#> GSM348190 5 0.2191 0.8059 0.000 0.000 0.120 0.000 0.876 0.004
#> GSM348194 3 0.4735 0.4291 0.000 0.000 0.636 0.036 0.308 0.020
#> GSM348195 3 0.5017 0.1097 0.000 0.000 0.532 0.036 0.412 0.020
#> GSM348196 3 0.0405 0.7152 0.000 0.000 0.988 0.004 0.008 0.000
#> GSM537585 3 0.5089 0.1198 0.000 0.000 0.532 0.032 0.408 0.028
#> GSM537594 6 0.2917 0.6565 0.104 0.040 0.000 0.004 0.000 0.852
#> GSM537596 3 0.0405 0.7152 0.000 0.000 0.988 0.004 0.008 0.000
#> GSM537597 3 0.3683 0.6215 0.012 0.000 0.808 0.136 0.012 0.032
#> GSM537602 3 0.2851 0.6981 0.000 0.000 0.868 0.036 0.080 0.016
#> GSM340184 3 0.1320 0.7093 0.000 0.000 0.948 0.036 0.000 0.016
#> GSM340185 6 0.2425 0.6622 0.100 0.012 0.000 0.008 0.000 0.880
#> GSM340186 1 0.4962 0.0127 0.608 0.012 0.000 0.040 0.008 0.332
#> GSM340187 4 0.5916 0.5302 0.236 0.000 0.304 0.460 0.000 0.000
#> GSM340189 4 0.3838 0.7135 0.000 0.000 0.448 0.552 0.000 0.000
#> GSM340190 1 0.3835 0.4757 0.684 0.000 0.016 0.300 0.000 0.000
#> GSM340191 3 0.1349 0.6730 0.000 0.000 0.940 0.056 0.000 0.004
#> GSM340192 5 0.5450 0.3810 0.016 0.084 0.012 0.208 0.668 0.012
#> GSM340193 3 0.3620 0.6668 0.000 0.000 0.804 0.036 0.140 0.020
#> GSM340194 5 0.2243 0.8041 0.000 0.000 0.112 0.004 0.880 0.004
#> GSM340195 5 0.2243 0.8041 0.000 0.000 0.112 0.004 0.880 0.004
#> GSM340196 4 0.3838 0.7135 0.000 0.000 0.448 0.552 0.000 0.000
#> GSM340197 5 0.2146 0.8060 0.000 0.000 0.116 0.000 0.880 0.004
#> GSM340198 2 0.0146 0.9283 0.004 0.996 0.000 0.000 0.000 0.000
#> GSM340199 3 0.1349 0.6730 0.000 0.000 0.940 0.056 0.000 0.004
#> GSM340200 1 0.5080 0.5644 0.700 0.000 0.028 0.140 0.004 0.128
#> GSM340201 4 0.3838 0.7135 0.000 0.000 0.448 0.552 0.000 0.000
#> GSM340202 4 0.3833 0.7124 0.000 0.000 0.444 0.556 0.000 0.000
#> GSM340203 4 0.3838 0.7135 0.000 0.000 0.448 0.552 0.000 0.000
#> GSM340204 5 0.5111 0.4321 0.016 0.028 0.016 0.216 0.696 0.028
#> GSM340205 3 0.0260 0.7143 0.000 0.000 0.992 0.000 0.008 0.000
#> GSM340206 3 0.1471 0.6672 0.000 0.000 0.932 0.064 0.000 0.004
#> GSM340207 5 0.3426 0.7439 0.000 0.000 0.220 0.012 0.764 0.004
#> GSM340237 6 0.4209 0.6241 0.428 0.004 0.000 0.004 0.004 0.560
#> GSM340238 3 0.1806 0.6428 0.000 0.000 0.908 0.088 0.000 0.004
#> GSM340239 5 0.2146 0.8060 0.000 0.000 0.116 0.000 0.880 0.004
#> GSM340240 5 0.2243 0.8041 0.000 0.000 0.112 0.004 0.880 0.004
#> GSM340241 6 0.2517 0.6599 0.100 0.016 0.000 0.008 0.000 0.876
#> GSM340242 3 0.2639 0.7026 0.000 0.000 0.876 0.032 0.084 0.008
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
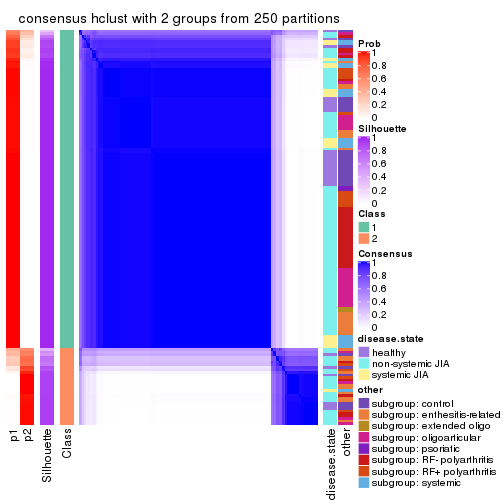
consensus_heatmap(res, k = 3)
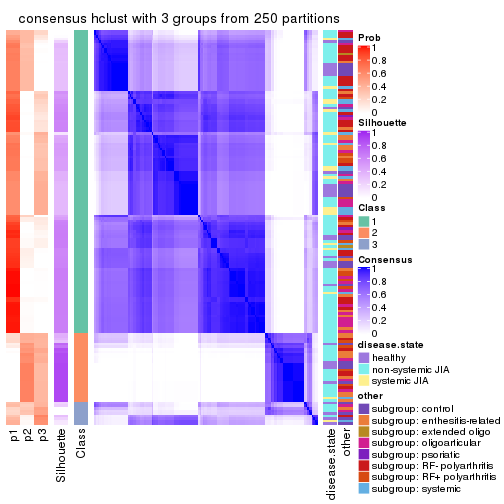
consensus_heatmap(res, k = 4)
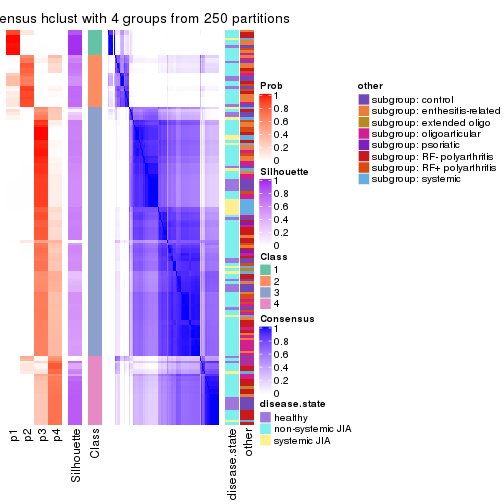
consensus_heatmap(res, k = 5)
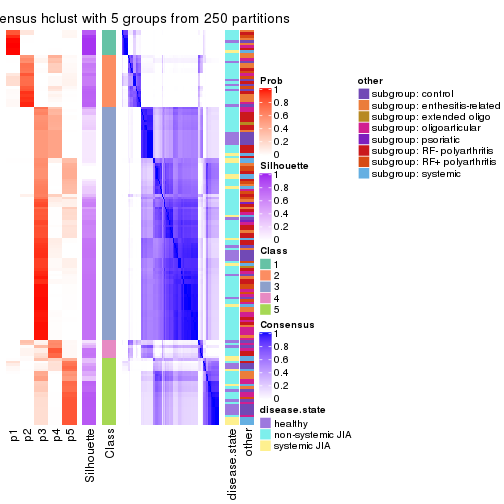
consensus_heatmap(res, k = 6)
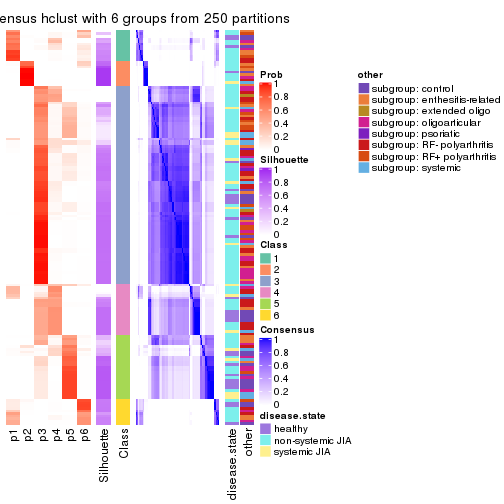
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
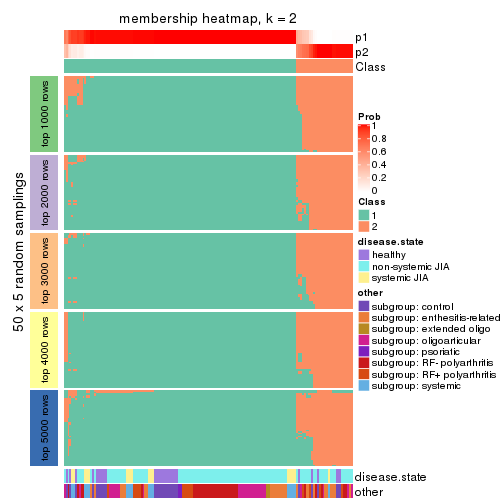
membership_heatmap(res, k = 3)
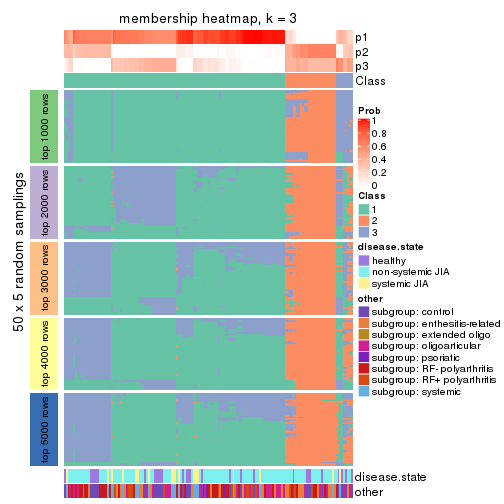
membership_heatmap(res, k = 4)
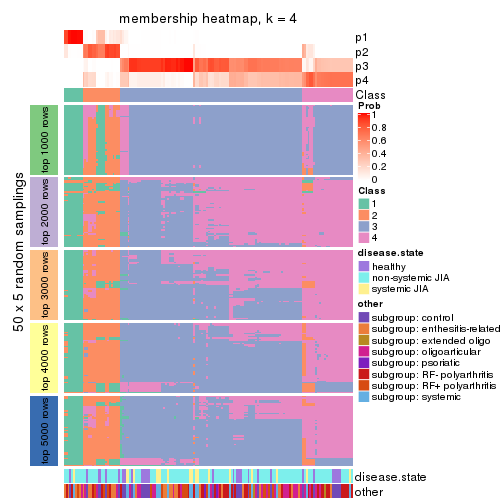
membership_heatmap(res, k = 5)
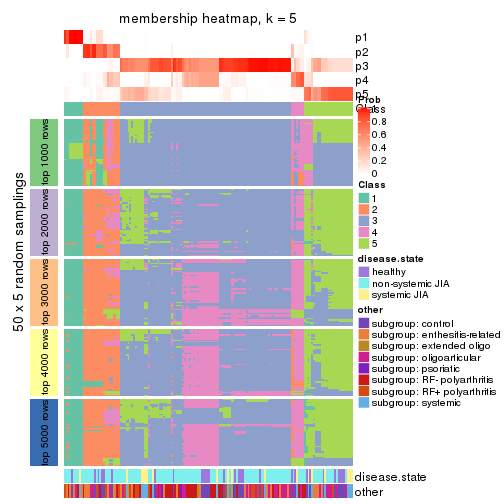
membership_heatmap(res, k = 6)
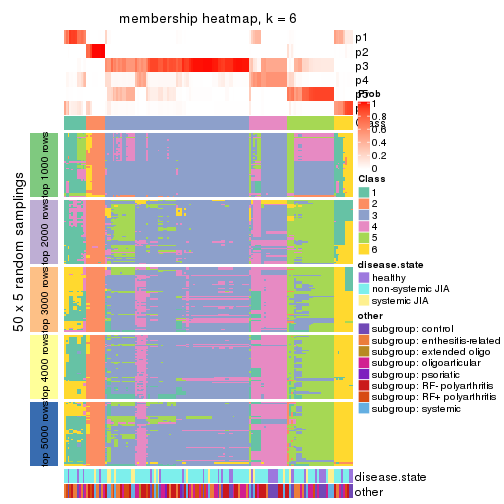
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
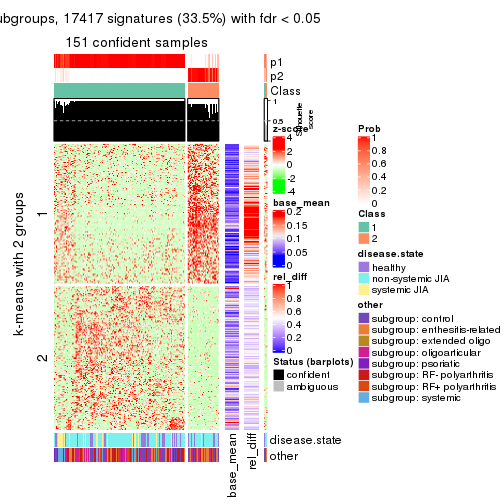
get_signatures(res, k = 3)
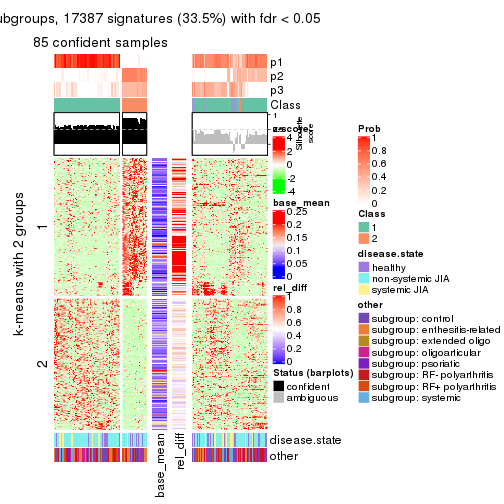
get_signatures(res, k = 4)
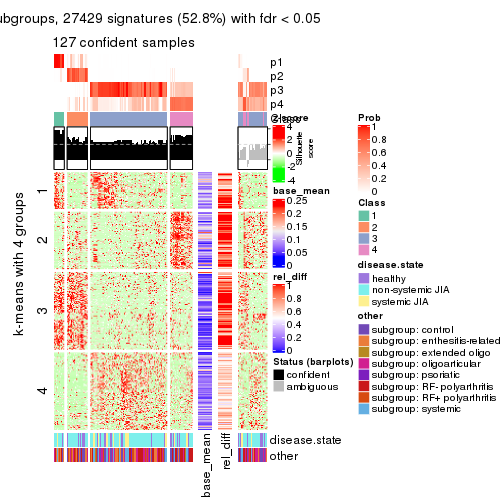
get_signatures(res, k = 5)
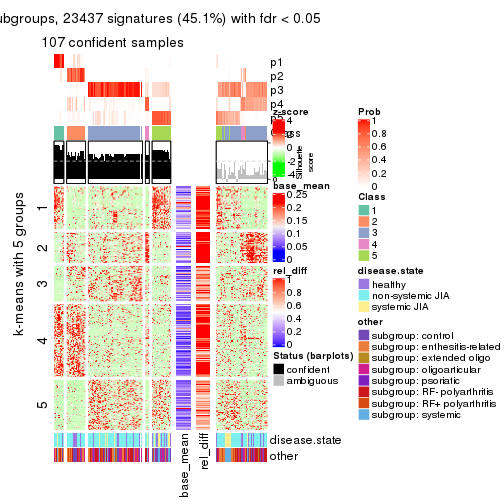
get_signatures(res, k = 6)
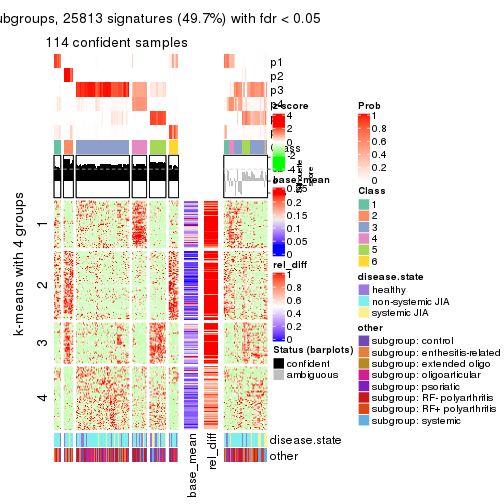
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
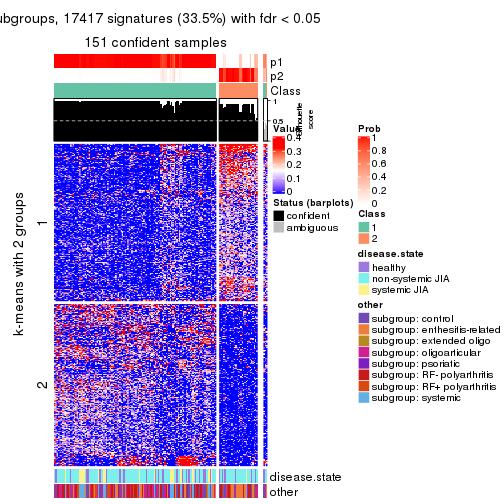
get_signatures(res, k = 3, scale_rows = FALSE)
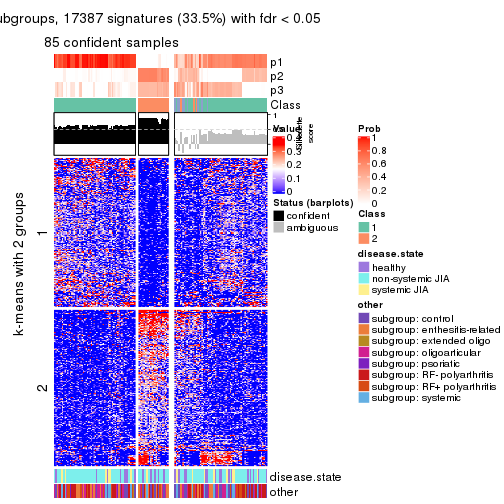
get_signatures(res, k = 4, scale_rows = FALSE)
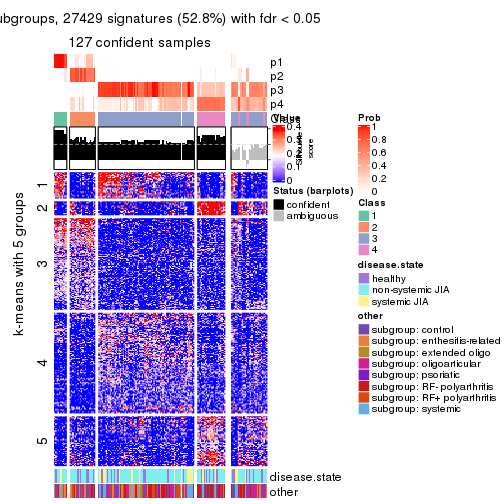
get_signatures(res, k = 5, scale_rows = FALSE)
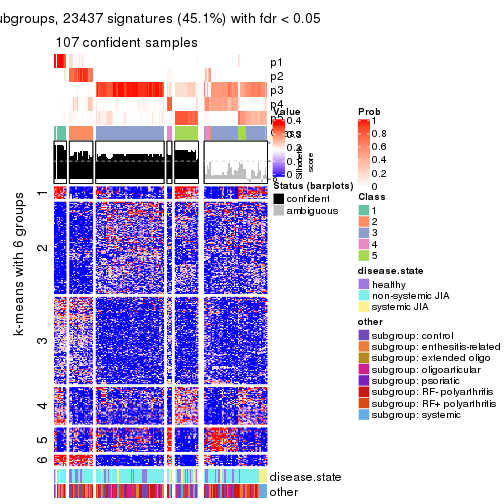
get_signatures(res, k = 6, scale_rows = FALSE)
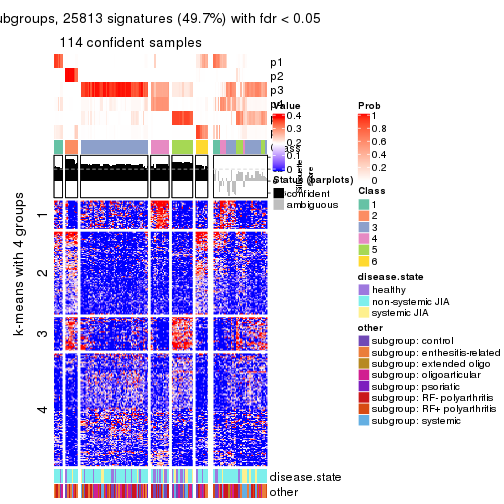
Compare the overlap of signatures from different k:
compare_signatures(res)
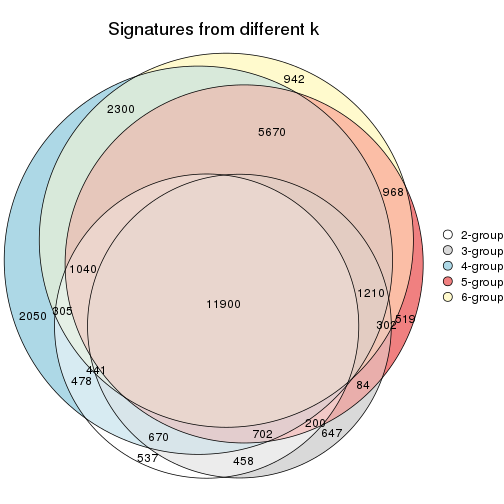
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
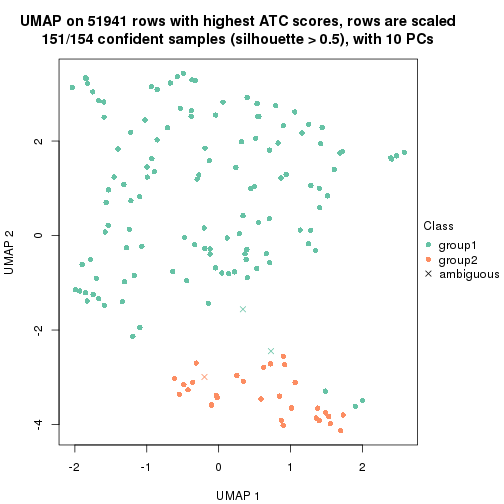
dimension_reduction(res, k = 3, method = "UMAP")
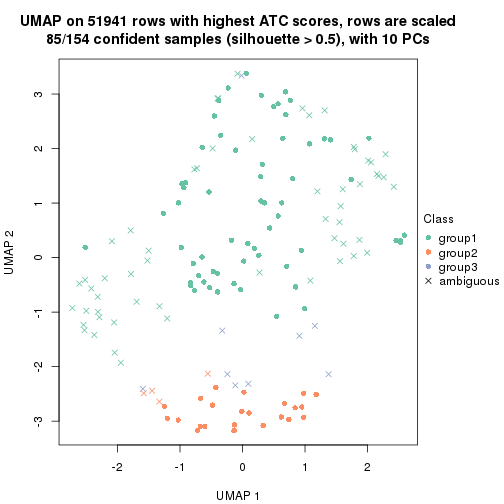
dimension_reduction(res, k = 4, method = "UMAP")
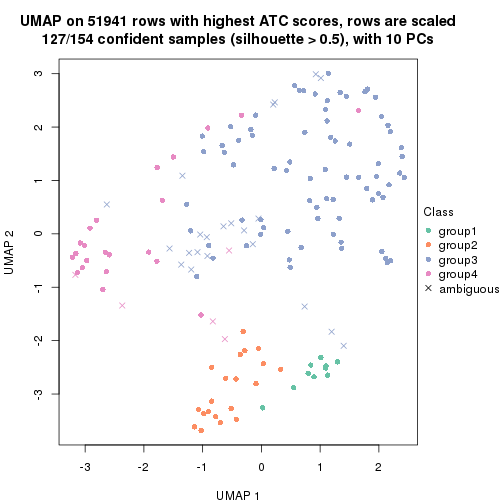
dimension_reduction(res, k = 5, method = "UMAP")
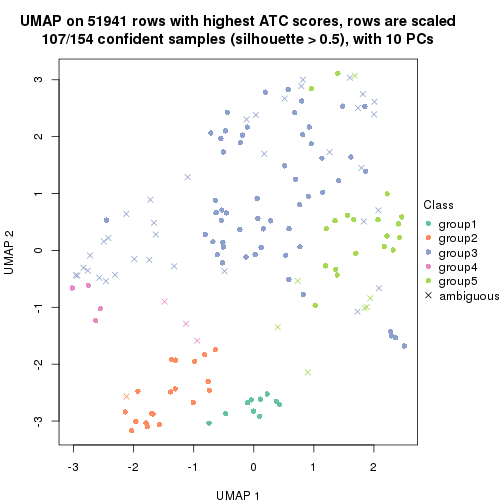
dimension_reduction(res, k = 6, method = "UMAP")
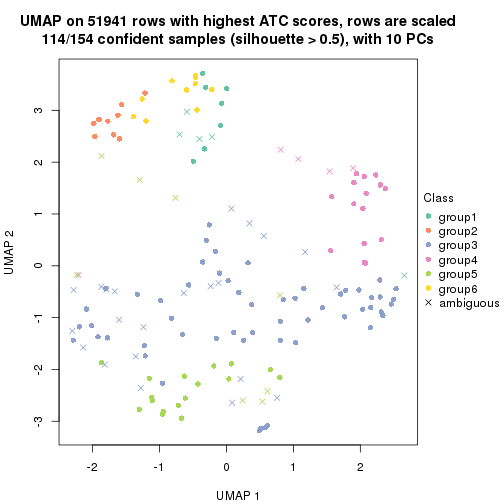
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
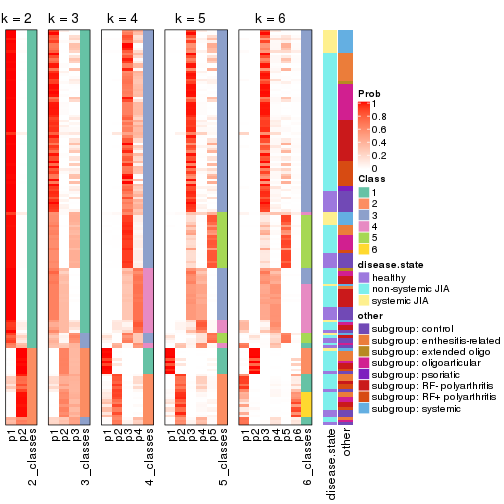
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> ATC:hclust 151 0.2927 0.248 2
#> ATC:hclust 85 0.4224 0.616 3
#> ATC:hclust 127 0.3295 0.075 4
#> ATC:hclust 107 0.0299 0.279 5
#> ATC:hclust 114 0.1110 0.332 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["ATC", "kmeans"]
# you can also extract it by
# res = res_list["ATC:kmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'ATC' method.
#> Subgroups are detected by 'kmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
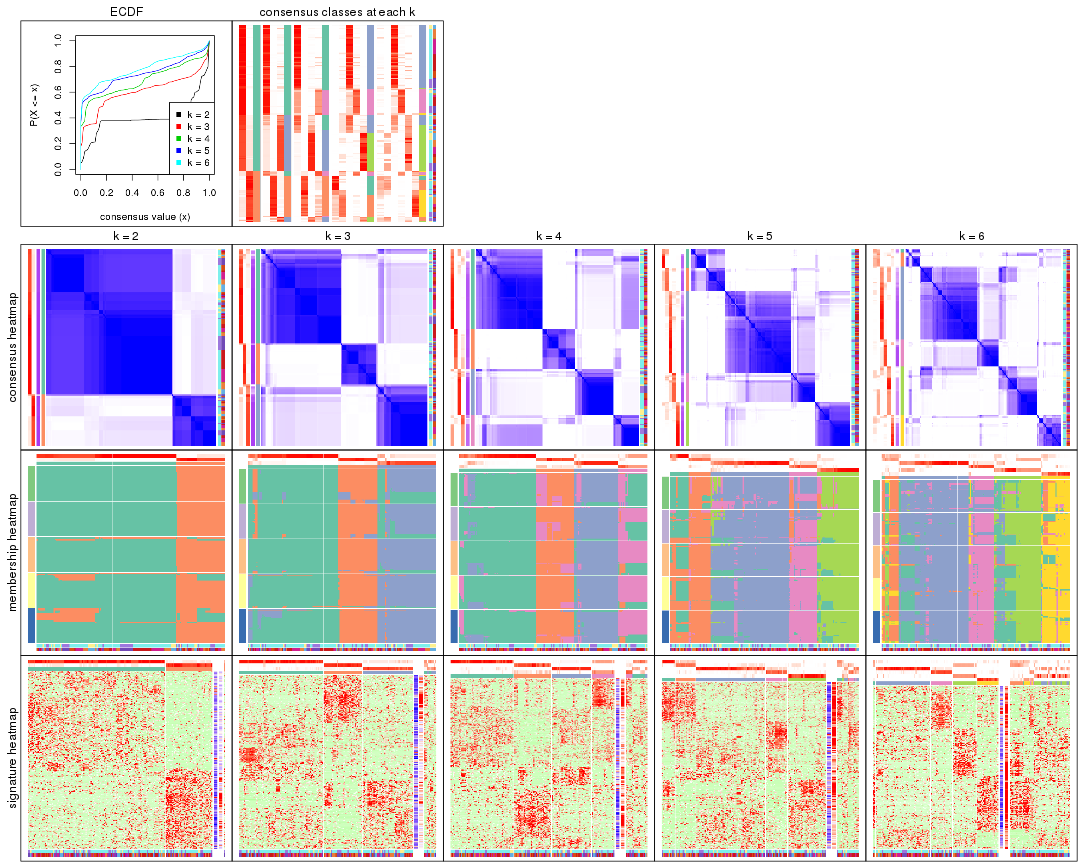
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
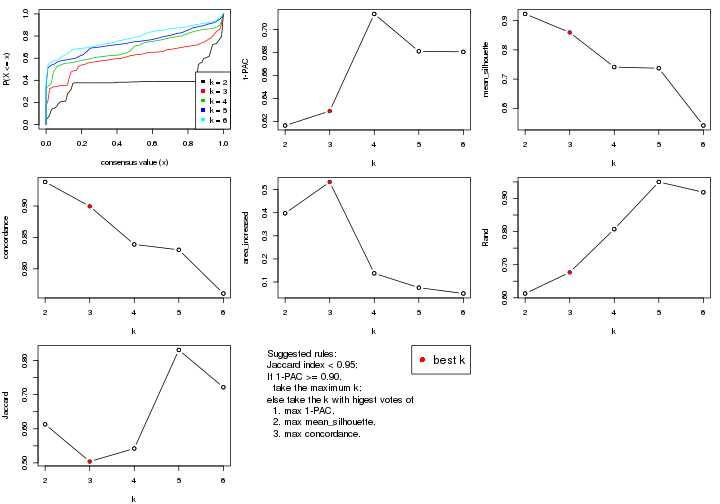
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.616 0.922 0.938 0.3974 0.613 0.613
#> 3 3 0.629 0.859 0.900 0.5330 0.677 0.504
#> 4 4 0.714 0.741 0.839 0.1375 0.807 0.542
#> 5 5 0.681 0.737 0.830 0.0756 0.950 0.831
#> 6 6 0.681 0.541 0.761 0.0502 0.919 0.722
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 1 0.0376 0.944 0.996 0.004
#> GSM340359 2 0.0376 0.901 0.004 0.996
#> GSM340361 1 0.5946 0.899 0.856 0.144
#> GSM340362 1 0.5946 0.899 0.856 0.144
#> GSM340363 1 0.0376 0.944 0.996 0.004
#> GSM340364 2 0.0376 0.901 0.004 0.996
#> GSM340365 1 0.5946 0.899 0.856 0.144
#> GSM340366 1 0.5946 0.899 0.856 0.144
#> GSM340367 1 0.3879 0.929 0.924 0.076
#> GSM340368 1 0.0376 0.944 0.996 0.004
#> GSM340369 1 0.0376 0.944 0.996 0.004
#> GSM340370 1 0.5059 0.915 0.888 0.112
#> GSM340371 1 0.3431 0.931 0.936 0.064
#> GSM340372 1 0.5946 0.899 0.856 0.144
#> GSM340373 1 0.5946 0.899 0.856 0.144
#> GSM340375 1 0.5842 0.901 0.860 0.140
#> GSM340376 1 0.1843 0.937 0.972 0.028
#> GSM340378 2 0.1633 0.912 0.024 0.976
#> GSM340243 1 0.0376 0.944 0.996 0.004
#> GSM340244 2 0.5178 0.935 0.116 0.884
#> GSM340246 2 0.5178 0.935 0.116 0.884
#> GSM340247 1 0.0376 0.944 0.996 0.004
#> GSM340248 2 0.5178 0.935 0.116 0.884
#> GSM340249 2 0.5946 0.921 0.144 0.856
#> GSM340250 1 0.3431 0.931 0.936 0.064
#> GSM340251 2 0.5946 0.921 0.144 0.856
#> GSM340252 2 0.1414 0.914 0.020 0.980
#> GSM340253 2 0.0376 0.901 0.004 0.996
#> GSM340254 2 0.0376 0.901 0.004 0.996
#> GSM340256 2 0.1414 0.914 0.020 0.980
#> GSM340258 1 0.5946 0.899 0.856 0.144
#> GSM340259 1 0.5629 0.904 0.868 0.132
#> GSM340260 1 0.3879 0.929 0.924 0.076
#> GSM340261 1 0.0376 0.944 0.996 0.004
#> GSM340262 1 0.5946 0.899 0.856 0.144
#> GSM340263 2 0.5629 0.928 0.132 0.868
#> GSM340264 1 0.5946 0.899 0.856 0.144
#> GSM340265 1 0.5946 0.899 0.856 0.144
#> GSM340266 1 0.0376 0.944 0.996 0.004
#> GSM340267 1 0.5842 0.901 0.860 0.140
#> GSM340268 1 0.0376 0.944 0.996 0.004
#> GSM340269 1 0.0000 0.944 1.000 0.000
#> GSM340270 1 0.0000 0.944 1.000 0.000
#> GSM537574 1 0.2043 0.936 0.968 0.032
#> GSM537580 1 0.0000 0.944 1.000 0.000
#> GSM537581 1 0.0376 0.944 0.996 0.004
#> GSM340272 1 0.5946 0.899 0.856 0.144
#> GSM340273 2 0.5178 0.935 0.116 0.884
#> GSM340275 2 0.5946 0.921 0.144 0.856
#> GSM340276 1 0.0000 0.944 1.000 0.000
#> GSM340277 2 0.0376 0.901 0.004 0.996
#> GSM340278 1 0.0376 0.944 0.996 0.004
#> GSM340279 1 0.0376 0.944 0.996 0.004
#> GSM340282 1 0.5946 0.899 0.856 0.144
#> GSM340284 2 0.5946 0.921 0.144 0.856
#> GSM340285 1 0.0376 0.944 0.996 0.004
#> GSM340286 1 0.5946 0.899 0.856 0.144
#> GSM340287 1 0.0000 0.944 1.000 0.000
#> GSM340288 1 0.5178 0.912 0.884 0.116
#> GSM340289 1 0.3879 0.929 0.924 0.076
#> GSM340290 1 0.0376 0.944 0.996 0.004
#> GSM340291 2 0.5178 0.935 0.116 0.884
#> GSM340293 1 0.0376 0.944 0.996 0.004
#> GSM340294 1 0.0376 0.944 0.996 0.004
#> GSM340296 1 0.0376 0.944 0.996 0.004
#> GSM340297 1 0.0376 0.944 0.996 0.004
#> GSM340298 1 0.0376 0.944 0.996 0.004
#> GSM340299 1 0.5946 0.899 0.856 0.144
#> GSM340301 1 0.5946 0.899 0.856 0.144
#> GSM340303 1 0.5946 0.899 0.856 0.144
#> GSM340304 1 0.0000 0.944 1.000 0.000
#> GSM340306 1 0.0376 0.944 0.996 0.004
#> GSM340307 2 0.5178 0.935 0.116 0.884
#> GSM340310 1 0.0672 0.943 0.992 0.008
#> GSM340314 1 0.0376 0.944 0.996 0.004
#> GSM340315 1 0.0376 0.944 0.996 0.004
#> GSM340317 2 0.5629 0.928 0.132 0.868
#> GSM340318 1 0.0376 0.944 0.996 0.004
#> GSM340319 1 0.0376 0.944 0.996 0.004
#> GSM340320 1 0.0376 0.944 0.996 0.004
#> GSM340321 2 0.5178 0.935 0.116 0.884
#> GSM340322 1 0.0376 0.944 0.996 0.004
#> GSM340324 2 0.1414 0.914 0.020 0.980
#> GSM340328 1 0.0376 0.944 0.996 0.004
#> GSM340330 1 0.0000 0.944 1.000 0.000
#> GSM340332 1 0.0376 0.944 0.996 0.004
#> GSM340333 2 0.0376 0.901 0.004 0.996
#> GSM340336 2 0.5946 0.921 0.144 0.856
#> GSM340337 1 0.0376 0.944 0.996 0.004
#> GSM340338 1 0.1414 0.941 0.980 0.020
#> GSM340339 1 0.0376 0.944 0.996 0.004
#> GSM340340 2 0.6048 0.919 0.148 0.852
#> GSM340341 1 0.0376 0.944 0.996 0.004
#> GSM340343 1 0.0376 0.944 0.996 0.004
#> GSM340344 2 0.0376 0.901 0.004 0.996
#> GSM340346 1 0.0000 0.944 1.000 0.000
#> GSM340347 1 0.0376 0.944 0.996 0.004
#> GSM340348 1 0.0376 0.944 0.996 0.004
#> GSM340349 1 0.3431 0.931 0.936 0.064
#> GSM340350 1 0.3431 0.931 0.936 0.064
#> GSM340351 1 0.5946 0.899 0.856 0.144
#> GSM340354 1 0.0938 0.942 0.988 0.012
#> GSM340356 2 0.5178 0.935 0.116 0.884
#> GSM340357 1 0.3879 0.929 0.924 0.076
#> GSM348183 1 0.5059 0.914 0.888 0.112
#> GSM348191 1 0.0376 0.944 0.996 0.004
#> GSM348193 1 0.0000 0.944 1.000 0.000
#> GSM537578 1 0.3733 0.930 0.928 0.072
#> GSM348181 1 0.0000 0.944 1.000 0.000
#> GSM348182 1 0.5946 0.899 0.856 0.144
#> GSM348184 2 0.5629 0.928 0.132 0.868
#> GSM348185 2 0.5629 0.928 0.132 0.868
#> GSM348186 2 0.1414 0.914 0.020 0.980
#> GSM348187 1 0.0000 0.944 1.000 0.000
#> GSM348188 1 0.3879 0.928 0.924 0.076
#> GSM348189 1 0.3274 0.932 0.940 0.060
#> GSM348190 1 0.5946 0.899 0.856 0.144
#> GSM348194 1 0.4939 0.916 0.892 0.108
#> GSM348195 1 0.5946 0.899 0.856 0.144
#> GSM348196 1 0.0000 0.944 1.000 0.000
#> GSM537585 1 0.5946 0.899 0.856 0.144
#> GSM537594 2 0.3431 0.924 0.064 0.936
#> GSM537596 1 0.0000 0.944 1.000 0.000
#> GSM537597 1 0.1843 0.937 0.972 0.028
#> GSM537602 1 0.3879 0.929 0.924 0.076
#> GSM340184 1 0.0376 0.944 0.996 0.004
#> GSM340185 2 0.5178 0.935 0.116 0.884
#> GSM340186 2 0.5178 0.935 0.116 0.884
#> GSM340187 1 0.0376 0.944 0.996 0.004
#> GSM340189 1 0.0376 0.944 0.996 0.004
#> GSM340190 2 0.5946 0.921 0.144 0.856
#> GSM340191 1 0.0376 0.944 0.996 0.004
#> GSM340192 2 0.0376 0.901 0.004 0.996
#> GSM340193 1 0.3431 0.931 0.936 0.064
#> GSM340194 1 0.5946 0.899 0.856 0.144
#> GSM340195 1 0.5946 0.899 0.856 0.144
#> GSM340196 1 0.0376 0.944 0.996 0.004
#> GSM340197 1 0.5946 0.899 0.856 0.144
#> GSM340198 2 0.1414 0.914 0.020 0.980
#> GSM340199 1 0.0376 0.944 0.996 0.004
#> GSM340200 2 0.5178 0.935 0.116 0.884
#> GSM340201 1 0.0376 0.944 0.996 0.004
#> GSM340202 1 0.0376 0.944 0.996 0.004
#> GSM340203 1 0.0376 0.944 0.996 0.004
#> GSM340204 2 0.9635 0.224 0.388 0.612
#> GSM340205 1 0.0000 0.944 1.000 0.000
#> GSM340206 1 0.0376 0.944 0.996 0.004
#> GSM340207 1 0.5946 0.899 0.856 0.144
#> GSM340237 2 0.5178 0.935 0.116 0.884
#> GSM340238 1 0.0376 0.944 0.996 0.004
#> GSM340239 1 0.5946 0.899 0.856 0.144
#> GSM340240 1 0.5946 0.899 0.856 0.144
#> GSM340241 2 0.3431 0.924 0.064 0.936
#> GSM340242 1 0.3274 0.932 0.940 0.060
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340359 2 0.2448 0.9446 0.000 0.924 0.076
#> GSM340361 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340362 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340363 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340364 3 0.5138 0.4891 0.000 0.252 0.748
#> GSM340365 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340366 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340367 3 0.4931 0.8253 0.232 0.000 0.768
#> GSM340368 1 0.3472 0.8478 0.904 0.040 0.056
#> GSM340369 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340370 3 0.4702 0.8464 0.212 0.000 0.788
#> GSM340371 1 0.5785 0.3745 0.668 0.000 0.332
#> GSM340372 3 0.3551 0.9114 0.132 0.000 0.868
#> GSM340373 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340375 3 0.3619 0.9096 0.136 0.000 0.864
#> GSM340376 1 0.4399 0.7892 0.812 0.000 0.188
#> GSM340378 2 0.2446 0.9409 0.012 0.936 0.052
#> GSM340243 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340244 2 0.1182 0.9515 0.012 0.976 0.012
#> GSM340246 2 0.0829 0.9517 0.012 0.984 0.004
#> GSM340247 1 0.1182 0.9139 0.976 0.012 0.012
#> GSM340248 2 0.2651 0.9391 0.012 0.928 0.060
#> GSM340249 2 0.2301 0.9372 0.004 0.936 0.060
#> GSM340250 3 0.6252 0.4517 0.444 0.000 0.556
#> GSM340251 2 0.2301 0.9372 0.004 0.936 0.060
#> GSM340252 2 0.2066 0.9474 0.000 0.940 0.060
#> GSM340253 2 0.2448 0.9446 0.000 0.924 0.076
#> GSM340254 2 0.2448 0.9446 0.000 0.924 0.076
#> GSM340256 2 0.2448 0.9446 0.000 0.924 0.076
#> GSM340258 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340259 3 0.3619 0.9096 0.136 0.000 0.864
#> GSM340260 3 0.6252 0.4517 0.444 0.000 0.556
#> GSM340261 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340262 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340263 2 0.2651 0.9391 0.012 0.928 0.060
#> GSM340264 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340265 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340266 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340267 3 0.3619 0.9096 0.136 0.000 0.864
#> GSM340268 1 0.0000 0.9228 1.000 0.000 0.000
#> GSM340269 1 0.1860 0.9026 0.948 0.000 0.052
#> GSM340270 1 0.1860 0.9026 0.948 0.000 0.052
#> GSM537574 1 0.2878 0.8870 0.904 0.000 0.096
#> GSM537580 1 0.1529 0.9099 0.960 0.000 0.040
#> GSM537581 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340272 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340273 2 0.1182 0.9506 0.012 0.976 0.012
#> GSM340275 1 0.7866 0.2243 0.552 0.388 0.060
#> GSM340276 1 0.1753 0.9053 0.952 0.000 0.048
#> GSM340277 2 0.2448 0.9446 0.000 0.924 0.076
#> GSM340278 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340279 1 0.0000 0.9228 1.000 0.000 0.000
#> GSM340282 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340284 1 0.6722 0.6004 0.720 0.220 0.060
#> GSM340285 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340286 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340287 1 0.1753 0.9053 0.952 0.000 0.048
#> GSM340288 3 0.3619 0.9096 0.136 0.000 0.864
#> GSM340289 3 0.5835 0.6725 0.340 0.000 0.660
#> GSM340290 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340291 2 0.1999 0.9501 0.012 0.952 0.036
#> GSM340293 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340294 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340296 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340297 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340298 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340299 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340301 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340303 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340304 1 0.1860 0.9026 0.948 0.000 0.052
#> GSM340306 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340307 2 0.2651 0.9391 0.012 0.928 0.060
#> GSM340310 1 0.2959 0.8517 0.900 0.000 0.100
#> GSM340314 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340315 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340317 2 0.2651 0.9391 0.012 0.928 0.060
#> GSM340318 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340319 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340320 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340321 2 0.1999 0.9501 0.012 0.952 0.036
#> GSM340322 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340324 2 0.2448 0.9446 0.000 0.924 0.076
#> GSM340328 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM340330 1 0.0592 0.9223 0.988 0.000 0.012
#> GSM340332 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340333 2 0.2448 0.9446 0.000 0.924 0.076
#> GSM340336 1 0.7839 0.2484 0.560 0.380 0.060
#> GSM340337 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340338 1 0.1860 0.9026 0.948 0.000 0.052
#> GSM340339 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340340 1 0.6633 0.6313 0.728 0.212 0.060
#> GSM340341 1 0.4097 0.8120 0.880 0.060 0.060
#> GSM340343 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340344 3 0.1289 0.7672 0.000 0.032 0.968
#> GSM340346 1 0.1860 0.9026 0.948 0.000 0.052
#> GSM340347 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340348 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340349 3 0.6305 0.3319 0.484 0.000 0.516
#> GSM340350 1 0.6252 -0.0773 0.556 0.000 0.444
#> GSM340351 3 0.3551 0.9114 0.132 0.000 0.868
#> GSM340354 1 0.1860 0.9026 0.948 0.000 0.052
#> GSM340356 2 0.0829 0.9517 0.012 0.984 0.004
#> GSM340357 3 0.4974 0.8206 0.236 0.000 0.764
#> GSM348183 3 0.3619 0.9096 0.136 0.000 0.864
#> GSM348191 1 0.0424 0.9234 0.992 0.000 0.008
#> GSM348193 1 0.1753 0.9053 0.952 0.000 0.048
#> GSM537578 3 0.6079 0.5816 0.388 0.000 0.612
#> GSM348181 1 0.1753 0.9053 0.952 0.000 0.048
#> GSM348182 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM348184 2 0.1337 0.9500 0.012 0.972 0.016
#> GSM348185 2 0.2651 0.9391 0.012 0.928 0.060
#> GSM348186 2 0.2448 0.9446 0.000 0.924 0.076
#> GSM348187 1 0.1860 0.9026 0.948 0.000 0.052
#> GSM348188 3 0.3989 0.9093 0.124 0.012 0.864
#> GSM348189 1 0.4974 0.6260 0.764 0.000 0.236
#> GSM348190 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM348194 3 0.3619 0.9096 0.136 0.000 0.864
#> GSM348195 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM348196 1 0.1860 0.9026 0.948 0.000 0.052
#> GSM537585 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM537594 2 0.2116 0.9498 0.012 0.948 0.040
#> GSM537596 1 0.1860 0.9026 0.948 0.000 0.052
#> GSM537597 1 0.3941 0.8287 0.844 0.000 0.156
#> GSM537602 3 0.6286 0.3950 0.464 0.000 0.536
#> GSM340184 1 0.1411 0.9120 0.964 0.000 0.036
#> GSM340185 2 0.1999 0.9501 0.012 0.952 0.036
#> GSM340186 2 0.1015 0.9511 0.012 0.980 0.008
#> GSM340187 1 0.3472 0.8478 0.904 0.040 0.056
#> GSM340189 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340190 2 0.7478 0.5221 0.308 0.632 0.060
#> GSM340191 1 0.0000 0.9228 1.000 0.000 0.000
#> GSM340192 3 0.6260 -0.0677 0.000 0.448 0.552
#> GSM340193 3 0.4702 0.8465 0.212 0.000 0.788
#> GSM340194 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340195 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340196 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340197 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340198 2 0.2448 0.9446 0.000 0.924 0.076
#> GSM340199 1 0.0000 0.9228 1.000 0.000 0.000
#> GSM340200 2 0.2651 0.9391 0.012 0.928 0.060
#> GSM340201 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340202 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340203 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340204 3 0.2318 0.8051 0.028 0.028 0.944
#> GSM340205 1 0.1753 0.9053 0.952 0.000 0.048
#> GSM340206 1 0.0000 0.9228 1.000 0.000 0.000
#> GSM340207 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340237 2 0.0592 0.9518 0.012 0.988 0.000
#> GSM340238 1 0.0592 0.9217 0.988 0.012 0.000
#> GSM340239 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340240 3 0.3412 0.9141 0.124 0.000 0.876
#> GSM340241 2 0.2496 0.9463 0.004 0.928 0.068
#> GSM340242 1 0.4974 0.6260 0.764 0.000 0.236
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.0000 0.8220 1.000 0.000 0.000 0.000
#> GSM340359 2 0.3196 0.8201 0.000 0.856 0.008 0.136
#> GSM340361 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340362 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340363 1 0.0000 0.8220 1.000 0.000 0.000 0.000
#> GSM340364 3 0.5478 0.5838 0.000 0.056 0.696 0.248
#> GSM340365 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340366 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340367 1 0.4978 0.3719 0.612 0.000 0.384 0.004
#> GSM340368 4 0.5022 0.6430 0.264 0.000 0.028 0.708
#> GSM340369 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340370 1 0.5558 0.2524 0.548 0.000 0.432 0.020
#> GSM340371 1 0.2546 0.7836 0.912 0.000 0.060 0.028
#> GSM340372 3 0.1398 0.9230 0.040 0.000 0.956 0.004
#> GSM340373 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340375 3 0.5550 0.1478 0.428 0.000 0.552 0.020
#> GSM340376 1 0.5220 0.5767 0.752 0.000 0.092 0.156
#> GSM340378 2 0.5161 0.7762 0.000 0.676 0.024 0.300
#> GSM340243 1 0.0188 0.8243 0.996 0.000 0.004 0.000
#> GSM340244 2 0.2814 0.8479 0.000 0.868 0.000 0.132
#> GSM340246 2 0.2888 0.8488 0.000 0.872 0.004 0.124
#> GSM340247 4 0.4994 0.7353 0.480 0.000 0.000 0.520
#> GSM340248 2 0.4655 0.7868 0.000 0.684 0.004 0.312
#> GSM340249 2 0.4564 0.7794 0.000 0.672 0.000 0.328
#> GSM340250 1 0.3583 0.6746 0.816 0.000 0.180 0.004
#> GSM340251 2 0.4500 0.7865 0.000 0.684 0.000 0.316
#> GSM340252 2 0.3933 0.8310 0.000 0.792 0.008 0.200
#> GSM340253 2 0.3196 0.8201 0.000 0.856 0.008 0.136
#> GSM340254 2 0.3196 0.8201 0.000 0.856 0.008 0.136
#> GSM340256 2 0.2868 0.8208 0.000 0.864 0.000 0.136
#> GSM340258 3 0.0927 0.9000 0.016 0.000 0.976 0.008
#> GSM340259 3 0.2197 0.8873 0.080 0.000 0.916 0.004
#> GSM340260 1 0.3583 0.6746 0.816 0.000 0.180 0.004
#> GSM340261 1 0.0188 0.8243 0.996 0.000 0.004 0.000
#> GSM340262 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340263 2 0.4836 0.7788 0.000 0.672 0.008 0.320
#> GSM340264 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340265 3 0.1209 0.9167 0.032 0.000 0.964 0.004
#> GSM340266 1 0.0000 0.8220 1.000 0.000 0.000 0.000
#> GSM340267 3 0.5281 0.0773 0.464 0.000 0.528 0.008
#> GSM340268 1 0.0188 0.8197 0.996 0.000 0.000 0.004
#> GSM340269 1 0.0592 0.8266 0.984 0.000 0.016 0.000
#> GSM340270 1 0.0592 0.8266 0.984 0.000 0.016 0.000
#> GSM537574 1 0.4405 0.6126 0.800 0.000 0.048 0.152
#> GSM537580 1 0.1109 0.8159 0.968 0.000 0.004 0.028
#> GSM537581 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340272 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340273 2 0.2999 0.8475 0.000 0.864 0.004 0.132
#> GSM340275 4 0.3994 0.2860 0.028 0.140 0.004 0.828
#> GSM340276 1 0.0592 0.8266 0.984 0.000 0.016 0.000
#> GSM340277 2 0.3196 0.8201 0.000 0.856 0.008 0.136
#> GSM340278 1 0.0000 0.8220 1.000 0.000 0.000 0.000
#> GSM340279 1 0.0000 0.8220 1.000 0.000 0.000 0.000
#> GSM340282 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340284 4 0.4596 0.3002 0.028 0.140 0.024 0.808
#> GSM340285 1 0.0000 0.8220 1.000 0.000 0.000 0.000
#> GSM340286 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340287 1 0.0592 0.8266 0.984 0.000 0.016 0.000
#> GSM340288 1 0.5167 0.0352 0.508 0.000 0.488 0.004
#> GSM340289 1 0.4188 0.5966 0.752 0.000 0.244 0.004
#> GSM340290 1 0.0000 0.8220 1.000 0.000 0.000 0.000
#> GSM340291 2 0.0817 0.8488 0.000 0.976 0.000 0.024
#> GSM340293 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340294 1 0.0188 0.8243 0.996 0.000 0.004 0.000
#> GSM340296 1 0.0188 0.8243 0.996 0.000 0.004 0.000
#> GSM340297 1 0.0000 0.8220 1.000 0.000 0.000 0.000
#> GSM340298 1 0.0188 0.8243 0.996 0.000 0.004 0.000
#> GSM340299 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340301 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340303 3 0.0779 0.9003 0.016 0.000 0.980 0.004
#> GSM340304 1 0.0779 0.8257 0.980 0.000 0.016 0.004
#> GSM340306 1 0.4564 -0.1821 0.672 0.000 0.000 0.328
#> GSM340307 2 0.4720 0.7799 0.000 0.672 0.004 0.324
#> GSM340310 1 0.1004 0.8226 0.972 0.000 0.024 0.004
#> GSM340314 1 0.0188 0.8197 0.996 0.000 0.000 0.004
#> GSM340315 1 0.0592 0.8174 0.984 0.000 0.016 0.000
#> GSM340317 2 0.4500 0.7865 0.000 0.684 0.000 0.316
#> GSM340318 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340319 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340320 1 0.4477 -0.0976 0.688 0.000 0.000 0.312
#> GSM340321 2 0.0592 0.8483 0.000 0.984 0.000 0.016
#> GSM340322 4 0.4989 0.7276 0.472 0.000 0.000 0.528
#> GSM340324 2 0.2868 0.8208 0.000 0.864 0.000 0.136
#> GSM340328 1 0.0000 0.8220 1.000 0.000 0.000 0.000
#> GSM340330 1 0.0524 0.8261 0.988 0.000 0.008 0.004
#> GSM340332 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340333 2 0.3196 0.8201 0.000 0.856 0.008 0.136
#> GSM340336 4 0.4113 0.3090 0.040 0.128 0.004 0.828
#> GSM340337 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340338 1 0.0779 0.8262 0.980 0.000 0.016 0.004
#> GSM340339 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340340 4 0.4846 0.3296 0.044 0.124 0.028 0.804
#> GSM340341 4 0.4446 0.5596 0.196 0.000 0.028 0.776
#> GSM340343 1 0.3569 0.4175 0.804 0.000 0.000 0.196
#> GSM340344 3 0.4331 0.6057 0.000 0.000 0.712 0.288
#> GSM340346 1 0.0592 0.8266 0.984 0.000 0.016 0.000
#> GSM340347 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340348 1 0.4277 0.0666 0.720 0.000 0.000 0.280
#> GSM340349 1 0.3208 0.7104 0.848 0.000 0.148 0.004
#> GSM340350 1 0.2888 0.7349 0.872 0.000 0.124 0.004
#> GSM340351 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340354 1 0.1151 0.8210 0.968 0.000 0.024 0.008
#> GSM340356 2 0.2888 0.8488 0.000 0.872 0.004 0.124
#> GSM340357 1 0.4936 0.4032 0.624 0.000 0.372 0.004
#> GSM348183 3 0.2266 0.8831 0.084 0.000 0.912 0.004
#> GSM348191 1 0.0188 0.8243 0.996 0.000 0.004 0.000
#> GSM348193 1 0.1004 0.8226 0.972 0.000 0.024 0.004
#> GSM537578 1 0.3982 0.6274 0.776 0.000 0.220 0.004
#> GSM348181 1 0.0592 0.8266 0.984 0.000 0.016 0.000
#> GSM348182 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM348184 2 0.3486 0.8344 0.000 0.812 0.000 0.188
#> GSM348185 2 0.4564 0.7794 0.000 0.672 0.000 0.328
#> GSM348186 2 0.2868 0.8208 0.000 0.864 0.000 0.136
#> GSM348187 1 0.0779 0.8257 0.980 0.000 0.016 0.004
#> GSM348188 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM348189 1 0.2032 0.8036 0.936 0.000 0.036 0.028
#> GSM348190 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM348194 1 0.5132 0.1752 0.548 0.000 0.448 0.004
#> GSM348195 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM348196 1 0.0592 0.8266 0.984 0.000 0.016 0.000
#> GSM537585 3 0.1305 0.9212 0.036 0.000 0.960 0.004
#> GSM537594 2 0.0000 0.8466 0.000 1.000 0.000 0.000
#> GSM537596 1 0.0592 0.8266 0.984 0.000 0.016 0.000
#> GSM537597 1 0.4849 0.5854 0.772 0.000 0.064 0.164
#> GSM537602 1 0.3355 0.6973 0.836 0.000 0.160 0.004
#> GSM340184 1 0.0469 0.8263 0.988 0.000 0.012 0.000
#> GSM340185 2 0.0592 0.8483 0.000 0.984 0.000 0.016
#> GSM340186 2 0.2814 0.8479 0.000 0.868 0.000 0.132
#> GSM340187 4 0.5558 0.6889 0.364 0.000 0.028 0.608
#> GSM340189 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340190 4 0.4248 0.1007 0.012 0.220 0.000 0.768
#> GSM340191 1 0.0188 0.8197 0.996 0.000 0.000 0.004
#> GSM340192 3 0.6732 0.3287 0.000 0.108 0.556 0.336
#> GSM340193 1 0.5016 0.3404 0.600 0.000 0.396 0.004
#> GSM340194 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340195 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340196 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340197 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340198 2 0.3196 0.8201 0.000 0.856 0.008 0.136
#> GSM340199 1 0.0188 0.8197 0.996 0.000 0.000 0.004
#> GSM340200 2 0.4720 0.7799 0.000 0.672 0.004 0.324
#> GSM340201 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340202 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340203 4 0.4999 0.7398 0.492 0.000 0.000 0.508
#> GSM340204 3 0.3311 0.7394 0.000 0.000 0.828 0.172
#> GSM340205 1 0.0592 0.8266 0.984 0.000 0.016 0.000
#> GSM340206 1 0.0188 0.8197 0.996 0.000 0.000 0.004
#> GSM340207 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340237 2 0.2704 0.8488 0.000 0.876 0.000 0.124
#> GSM340238 1 0.0336 0.8161 0.992 0.000 0.000 0.008
#> GSM340239 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340240 3 0.1211 0.9252 0.040 0.000 0.960 0.000
#> GSM340241 2 0.2281 0.8311 0.000 0.904 0.000 0.096
#> GSM340242 1 0.1929 0.8043 0.940 0.000 0.036 0.024
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 3 0.1117 0.8359 0.000 0.020 0.964 0.016 0.000
#> GSM340359 1 0.0290 0.7607 0.992 0.000 0.000 0.000 0.008
#> GSM340361 5 0.0162 0.8854 0.000 0.000 0.004 0.000 0.996
#> GSM340362 5 0.0324 0.8844 0.004 0.000 0.004 0.000 0.992
#> GSM340363 3 0.0609 0.8340 0.000 0.000 0.980 0.020 0.000
#> GSM340364 5 0.7879 0.4392 0.192 0.224 0.000 0.124 0.460
#> GSM340365 5 0.0162 0.8854 0.000 0.000 0.004 0.000 0.996
#> GSM340366 5 0.0566 0.8843 0.000 0.012 0.004 0.000 0.984
#> GSM340367 3 0.5884 0.6239 0.000 0.168 0.664 0.028 0.140
#> GSM340368 4 0.2304 0.6829 0.000 0.044 0.048 0.908 0.000
#> GSM340369 4 0.3274 0.8610 0.000 0.000 0.220 0.780 0.000
#> GSM340370 3 0.6730 0.5707 0.000 0.172 0.604 0.152 0.072
#> GSM340371 3 0.2726 0.7888 0.000 0.064 0.884 0.052 0.000
#> GSM340372 5 0.3360 0.8227 0.000 0.168 0.004 0.012 0.816
#> GSM340373 5 0.2970 0.8278 0.000 0.168 0.004 0.000 0.828
#> GSM340375 3 0.7965 0.3157 0.000 0.180 0.456 0.152 0.212
#> GSM340376 3 0.5670 0.5973 0.000 0.192 0.632 0.176 0.000
#> GSM340378 2 0.5879 0.6876 0.236 0.612 0.000 0.148 0.004
#> GSM340243 3 0.0162 0.8360 0.000 0.000 0.996 0.004 0.000
#> GSM340244 2 0.3876 0.7843 0.316 0.684 0.000 0.000 0.000
#> GSM340246 2 0.3837 0.7949 0.308 0.692 0.000 0.000 0.000
#> GSM340247 4 0.3300 0.8518 0.000 0.004 0.204 0.792 0.000
#> GSM340248 2 0.3579 0.8294 0.240 0.756 0.000 0.004 0.000
#> GSM340249 2 0.4496 0.8113 0.216 0.728 0.000 0.056 0.000
#> GSM340250 3 0.3997 0.7490 0.000 0.152 0.800 0.028 0.020
#> GSM340251 2 0.4413 0.8253 0.232 0.724 0.000 0.044 0.000
#> GSM340252 1 0.2763 0.6194 0.848 0.148 0.000 0.000 0.004
#> GSM340253 1 0.0290 0.7607 0.992 0.000 0.000 0.000 0.008
#> GSM340254 1 0.0162 0.7675 0.996 0.004 0.000 0.000 0.000
#> GSM340256 1 0.0290 0.7675 0.992 0.008 0.000 0.000 0.000
#> GSM340258 5 0.4468 0.8036 0.000 0.104 0.004 0.124 0.768
#> GSM340259 5 0.4537 0.7724 0.000 0.168 0.060 0.012 0.760
#> GSM340260 3 0.3236 0.7646 0.000 0.152 0.828 0.000 0.020
#> GSM340261 3 0.0609 0.8340 0.000 0.000 0.980 0.020 0.000
#> GSM340262 5 0.0162 0.8854 0.000 0.000 0.004 0.000 0.996
#> GSM340263 2 0.4701 0.8127 0.236 0.704 0.000 0.060 0.000
#> GSM340264 5 0.1356 0.8799 0.000 0.028 0.004 0.012 0.956
#> GSM340265 5 0.1356 0.8787 0.000 0.012 0.004 0.028 0.956
#> GSM340266 3 0.0771 0.8327 0.000 0.004 0.976 0.020 0.000
#> GSM340267 3 0.7603 0.3522 0.000 0.156 0.496 0.112 0.236
#> GSM340268 3 0.1282 0.8213 0.000 0.004 0.952 0.044 0.000
#> GSM340269 3 0.0000 0.8361 0.000 0.000 1.000 0.000 0.000
#> GSM340270 3 0.0880 0.8324 0.000 0.032 0.968 0.000 0.000
#> GSM537574 3 0.3695 0.7010 0.000 0.036 0.800 0.164 0.000
#> GSM537580 3 0.3055 0.7718 0.000 0.064 0.864 0.072 0.000
#> GSM537581 4 0.3430 0.8602 0.000 0.004 0.220 0.776 0.000
#> GSM340272 5 0.0162 0.8854 0.000 0.000 0.004 0.000 0.996
#> GSM340273 2 0.3837 0.7949 0.308 0.692 0.000 0.000 0.000
#> GSM340275 4 0.4331 0.2145 0.004 0.400 0.000 0.596 0.000
#> GSM340276 3 0.0000 0.8361 0.000 0.000 1.000 0.000 0.000
#> GSM340277 1 0.0162 0.7675 0.996 0.004 0.000 0.000 0.000
#> GSM340278 3 0.0609 0.8340 0.000 0.000 0.980 0.020 0.000
#> GSM340279 3 0.0771 0.8327 0.000 0.004 0.976 0.020 0.000
#> GSM340282 5 0.0162 0.8854 0.000 0.000 0.004 0.000 0.996
#> GSM340284 2 0.5289 0.4355 0.004 0.620 0.060 0.316 0.000
#> GSM340285 3 0.0609 0.8340 0.000 0.000 0.980 0.020 0.000
#> GSM340286 5 0.0162 0.8854 0.000 0.000 0.004 0.000 0.996
#> GSM340287 3 0.0000 0.8361 0.000 0.000 1.000 0.000 0.000
#> GSM340288 3 0.6702 0.1973 0.000 0.168 0.456 0.012 0.364
#> GSM340289 3 0.5137 0.6895 0.000 0.168 0.728 0.028 0.076
#> GSM340290 3 0.0771 0.8327 0.000 0.004 0.976 0.020 0.000
#> GSM340291 1 0.4300 -0.2204 0.524 0.476 0.000 0.000 0.000
#> GSM340293 4 0.4135 0.7204 0.000 0.004 0.340 0.656 0.000
#> GSM340294 3 0.0609 0.8340 0.000 0.000 0.980 0.020 0.000
#> GSM340296 3 0.0510 0.8348 0.000 0.000 0.984 0.016 0.000
#> GSM340297 3 0.0609 0.8340 0.000 0.000 0.980 0.020 0.000
#> GSM340298 3 0.0609 0.8340 0.000 0.000 0.980 0.020 0.000
#> GSM340299 5 0.0324 0.8844 0.004 0.000 0.004 0.000 0.992
#> GSM340301 5 0.0162 0.8854 0.000 0.000 0.004 0.000 0.996
#> GSM340303 5 0.2568 0.8439 0.004 0.008 0.004 0.096 0.888
#> GSM340304 3 0.1981 0.8178 0.000 0.048 0.924 0.028 0.000
#> GSM340306 4 0.5042 0.4219 0.000 0.032 0.460 0.508 0.000
#> GSM340307 2 0.4141 0.8309 0.236 0.736 0.000 0.028 0.000
#> GSM340310 3 0.3495 0.7556 0.000 0.160 0.812 0.028 0.000
#> GSM340314 3 0.1704 0.8035 0.000 0.004 0.928 0.068 0.000
#> GSM340315 3 0.0609 0.8349 0.000 0.000 0.980 0.020 0.000
#> GSM340317 2 0.4169 0.8317 0.240 0.732 0.000 0.028 0.000
#> GSM340318 4 0.3430 0.8614 0.000 0.004 0.220 0.776 0.000
#> GSM340319 4 0.3430 0.8614 0.000 0.004 0.220 0.776 0.000
#> GSM340320 3 0.4118 0.2419 0.000 0.004 0.660 0.336 0.000
#> GSM340321 1 0.4268 -0.0779 0.556 0.444 0.000 0.000 0.000
#> GSM340322 4 0.3876 0.8336 0.000 0.032 0.192 0.776 0.000
#> GSM340324 1 0.0290 0.7675 0.992 0.008 0.000 0.000 0.000
#> GSM340328 3 0.0794 0.8333 0.000 0.028 0.972 0.000 0.000
#> GSM340330 3 0.0771 0.8327 0.000 0.004 0.976 0.020 0.000
#> GSM340332 4 0.3430 0.8602 0.000 0.004 0.220 0.776 0.000
#> GSM340333 1 0.0290 0.7607 0.992 0.000 0.000 0.000 0.008
#> GSM340336 4 0.4029 0.4005 0.004 0.316 0.000 0.680 0.000
#> GSM340337 4 0.3877 0.8498 0.000 0.024 0.212 0.764 0.000
#> GSM340338 3 0.0566 0.8364 0.000 0.004 0.984 0.012 0.000
#> GSM340339 4 0.3430 0.8602 0.000 0.004 0.220 0.776 0.000
#> GSM340340 4 0.3550 0.4583 0.000 0.236 0.004 0.760 0.000
#> GSM340341 4 0.6282 0.3778 0.000 0.216 0.248 0.536 0.000
#> GSM340343 3 0.3861 0.4020 0.000 0.004 0.712 0.284 0.000
#> GSM340344 5 0.6794 0.5088 0.188 0.088 0.000 0.124 0.600
#> GSM340346 3 0.0404 0.8354 0.000 0.000 0.988 0.012 0.000
#> GSM340347 4 0.3430 0.8614 0.000 0.004 0.220 0.776 0.000
#> GSM340348 3 0.3861 0.4020 0.000 0.004 0.712 0.284 0.000
#> GSM340349 3 0.3626 0.7585 0.000 0.152 0.816 0.020 0.012
#> GSM340350 3 0.3692 0.7567 0.000 0.152 0.812 0.028 0.008
#> GSM340351 5 0.3360 0.8227 0.000 0.168 0.004 0.012 0.816
#> GSM340354 3 0.1408 0.8245 0.000 0.008 0.948 0.044 0.000
#> GSM340356 2 0.3837 0.7949 0.308 0.692 0.000 0.000 0.000
#> GSM340357 3 0.5722 0.6411 0.000 0.168 0.680 0.028 0.124
#> GSM348183 5 0.4473 0.7763 0.000 0.168 0.056 0.012 0.764
#> GSM348191 3 0.0609 0.8340 0.000 0.000 0.980 0.020 0.000
#> GSM348193 3 0.3574 0.7504 0.000 0.168 0.804 0.028 0.000
#> GSM537578 3 0.4786 0.7145 0.000 0.156 0.756 0.028 0.060
#> GSM348181 3 0.0671 0.8361 0.000 0.016 0.980 0.004 0.000
#> GSM348182 5 0.3320 0.8250 0.000 0.164 0.004 0.012 0.820
#> GSM348184 2 0.3969 0.8004 0.304 0.692 0.000 0.004 0.000
#> GSM348185 2 0.4169 0.8317 0.240 0.732 0.000 0.028 0.000
#> GSM348186 1 0.0290 0.7675 0.992 0.008 0.000 0.000 0.000
#> GSM348187 3 0.0510 0.8355 0.000 0.016 0.984 0.000 0.000
#> GSM348188 5 0.3951 0.8068 0.000 0.192 0.004 0.028 0.776
#> GSM348189 3 0.2654 0.7915 0.000 0.064 0.888 0.048 0.000
#> GSM348190 5 0.0162 0.8854 0.000 0.000 0.004 0.000 0.996
#> GSM348194 3 0.6291 0.5664 0.000 0.168 0.616 0.028 0.188
#> GSM348195 5 0.3360 0.8227 0.000 0.168 0.004 0.012 0.816
#> GSM348196 3 0.0794 0.8333 0.000 0.028 0.972 0.000 0.000
#> GSM537585 5 0.3569 0.8258 0.000 0.152 0.004 0.028 0.816
#> GSM537594 1 0.4210 0.0551 0.588 0.412 0.000 0.000 0.000
#> GSM537596 3 0.0000 0.8361 0.000 0.000 1.000 0.000 0.000
#> GSM537597 3 0.5413 0.6219 0.000 0.172 0.664 0.164 0.000
#> GSM537602 3 0.3883 0.7484 0.000 0.160 0.800 0.028 0.012
#> GSM340184 3 0.0162 0.8361 0.000 0.000 0.996 0.004 0.000
#> GSM340185 1 0.4268 -0.0779 0.556 0.444 0.000 0.000 0.000
#> GSM340186 2 0.3876 0.7843 0.316 0.684 0.000 0.000 0.000
#> GSM340187 4 0.1831 0.7259 0.000 0.004 0.076 0.920 0.000
#> GSM340189 4 0.3274 0.8610 0.000 0.000 0.220 0.780 0.000
#> GSM340190 2 0.4223 0.5430 0.028 0.724 0.000 0.248 0.000
#> GSM340191 3 0.0865 0.8313 0.000 0.004 0.972 0.024 0.000
#> GSM340192 5 0.7613 0.2931 0.276 0.112 0.000 0.136 0.476
#> GSM340193 3 0.6101 0.5960 0.000 0.168 0.640 0.028 0.164
#> GSM340194 5 0.0324 0.8844 0.004 0.000 0.004 0.000 0.992
#> GSM340195 5 0.0324 0.8844 0.004 0.000 0.004 0.000 0.992
#> GSM340196 4 0.3430 0.8614 0.000 0.004 0.220 0.776 0.000
#> GSM340197 5 0.0162 0.8854 0.000 0.000 0.004 0.000 0.996
#> GSM340198 1 0.0162 0.7675 0.996 0.004 0.000 0.000 0.000
#> GSM340199 3 0.0865 0.8313 0.000 0.004 0.972 0.024 0.000
#> GSM340200 2 0.4141 0.8309 0.236 0.736 0.000 0.028 0.000
#> GSM340201 4 0.3430 0.8614 0.000 0.004 0.220 0.776 0.000
#> GSM340202 4 0.3430 0.8614 0.000 0.004 0.220 0.776 0.000
#> GSM340203 4 0.3430 0.8602 0.000 0.004 0.220 0.776 0.000
#> GSM340204 5 0.5116 0.7136 0.016 0.132 0.000 0.124 0.728
#> GSM340205 3 0.0510 0.8355 0.000 0.016 0.984 0.000 0.000
#> GSM340206 3 0.1124 0.8261 0.000 0.004 0.960 0.036 0.000
#> GSM340207 5 0.0451 0.8838 0.000 0.008 0.004 0.000 0.988
#> GSM340237 2 0.3876 0.7843 0.316 0.684 0.000 0.000 0.000
#> GSM340238 3 0.2389 0.7543 0.000 0.004 0.880 0.116 0.000
#> GSM340239 5 0.0162 0.8854 0.000 0.000 0.004 0.000 0.996
#> GSM340240 5 0.0324 0.8844 0.004 0.000 0.004 0.000 0.992
#> GSM340241 1 0.3177 0.5625 0.792 0.208 0.000 0.000 0.000
#> GSM340242 3 0.2654 0.7915 0.000 0.064 0.888 0.048 0.000
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 3 0.1167 0.81723 0.020 0.000 0.960 0.008 0.012 0.000
#> GSM340359 2 0.5064 -0.35831 0.372 0.552 0.000 0.004 0.000 0.072
#> GSM340361 5 0.4062 0.58245 0.000 0.440 0.008 0.000 0.552 0.000
#> GSM340362 5 0.4076 0.57602 0.000 0.452 0.008 0.000 0.540 0.000
#> GSM340363 3 0.0692 0.81786 0.004 0.000 0.976 0.020 0.000 0.000
#> GSM340364 5 0.7093 -0.04878 0.240 0.256 0.000 0.040 0.440 0.024
#> GSM340365 5 0.4076 0.57602 0.000 0.452 0.008 0.000 0.540 0.000
#> GSM340366 5 0.3993 0.57859 0.000 0.400 0.008 0.000 0.592 0.000
#> GSM340367 3 0.3868 0.35600 0.000 0.000 0.508 0.000 0.492 0.000
#> GSM340368 4 0.1750 0.71075 0.056 0.000 0.004 0.928 0.004 0.008
#> GSM340369 4 0.2048 0.87488 0.000 0.000 0.120 0.880 0.000 0.000
#> GSM340370 5 0.6714 0.00406 0.236 0.000 0.276 0.048 0.440 0.000
#> GSM340371 3 0.3062 0.72660 0.160 0.000 0.816 0.024 0.000 0.000
#> GSM340372 5 0.0260 0.49604 0.000 0.000 0.008 0.000 0.992 0.000
#> GSM340373 5 0.1333 0.50451 0.000 0.048 0.008 0.000 0.944 0.000
#> GSM340375 5 0.6597 0.12470 0.248 0.000 0.228 0.048 0.476 0.000
#> GSM340376 5 0.6910 -0.01170 0.240 0.000 0.276 0.064 0.420 0.000
#> GSM340378 6 0.4816 0.55731 0.208 0.016 0.000 0.076 0.004 0.696
#> GSM340243 3 0.0000 0.81933 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340244 6 0.2491 0.69233 0.164 0.000 0.000 0.000 0.000 0.836
#> GSM340246 6 0.2454 0.69538 0.160 0.000 0.000 0.000 0.000 0.840
#> GSM340247 4 0.2053 0.86790 0.000 0.000 0.108 0.888 0.004 0.000
#> GSM340248 6 0.0865 0.73245 0.036 0.000 0.000 0.000 0.000 0.964
#> GSM340249 6 0.1333 0.72567 0.008 0.000 0.000 0.048 0.000 0.944
#> GSM340250 3 0.3774 0.49838 0.000 0.000 0.592 0.000 0.408 0.000
#> GSM340251 6 0.1049 0.73086 0.008 0.000 0.000 0.032 0.000 0.960
#> GSM340252 2 0.6317 -0.34069 0.320 0.412 0.000 0.012 0.000 0.256
#> GSM340253 2 0.5064 -0.35831 0.372 0.552 0.000 0.004 0.000 0.072
#> GSM340254 2 0.5310 -0.45458 0.392 0.512 0.000 0.004 0.000 0.092
#> GSM340256 1 0.5221 0.58738 0.476 0.432 0.000 0.000 0.000 0.092
#> GSM340258 5 0.5790 0.31554 0.232 0.128 0.000 0.040 0.600 0.000
#> GSM340259 5 0.0632 0.48819 0.000 0.000 0.024 0.000 0.976 0.000
#> GSM340260 3 0.3756 0.50932 0.000 0.000 0.600 0.000 0.400 0.000
#> GSM340261 3 0.0547 0.81814 0.000 0.000 0.980 0.020 0.000 0.000
#> GSM340262 5 0.4062 0.58245 0.000 0.440 0.008 0.000 0.552 0.000
#> GSM340263 6 0.1643 0.72189 0.008 0.000 0.000 0.068 0.000 0.924
#> GSM340264 5 0.4212 0.57619 0.008 0.392 0.008 0.000 0.592 0.000
#> GSM340265 5 0.4666 0.54927 0.048 0.388 0.000 0.000 0.564 0.000
#> GSM340266 3 0.0806 0.81724 0.008 0.000 0.972 0.020 0.000 0.000
#> GSM340267 5 0.6263 0.03658 0.208 0.000 0.304 0.020 0.468 0.000
#> GSM340268 3 0.1297 0.80752 0.012 0.000 0.948 0.040 0.000 0.000
#> GSM340269 3 0.0146 0.81936 0.004 0.000 0.996 0.000 0.000 0.000
#> GSM340270 3 0.1556 0.79416 0.000 0.000 0.920 0.000 0.080 0.000
#> GSM537574 3 0.4772 0.53901 0.248 0.000 0.672 0.064 0.016 0.000
#> GSM537580 3 0.3481 0.70364 0.160 0.000 0.792 0.048 0.000 0.000
#> GSM537581 4 0.2760 0.86922 0.024 0.000 0.116 0.856 0.004 0.000
#> GSM340272 5 0.4062 0.58245 0.000 0.440 0.008 0.000 0.552 0.000
#> GSM340273 6 0.2454 0.69538 0.160 0.000 0.000 0.000 0.000 0.840
#> GSM340275 6 0.4183 0.08657 0.012 0.000 0.000 0.480 0.000 0.508
#> GSM340276 3 0.0000 0.81933 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340277 2 0.5310 -0.45458 0.392 0.512 0.000 0.004 0.000 0.092
#> GSM340278 3 0.0692 0.81786 0.004 0.000 0.976 0.020 0.000 0.000
#> GSM340279 3 0.0909 0.81675 0.012 0.000 0.968 0.020 0.000 0.000
#> GSM340282 5 0.4062 0.58245 0.000 0.440 0.008 0.000 0.552 0.000
#> GSM340284 6 0.4240 0.62248 0.068 0.000 0.016 0.164 0.000 0.752
#> GSM340285 3 0.0692 0.81786 0.004 0.000 0.976 0.020 0.000 0.000
#> GSM340286 5 0.4062 0.58245 0.000 0.440 0.008 0.000 0.552 0.000
#> GSM340287 3 0.0000 0.81933 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340288 5 0.3428 0.16505 0.000 0.000 0.304 0.000 0.696 0.000
#> GSM340289 3 0.3854 0.41500 0.000 0.000 0.536 0.000 0.464 0.000
#> GSM340290 3 0.0692 0.81786 0.004 0.000 0.976 0.020 0.000 0.000
#> GSM340291 6 0.5364 0.27749 0.300 0.140 0.000 0.000 0.000 0.560
#> GSM340293 4 0.3923 0.53375 0.008 0.000 0.372 0.620 0.000 0.000
#> GSM340294 3 0.0547 0.81814 0.000 0.000 0.980 0.020 0.000 0.000
#> GSM340296 3 0.0146 0.81953 0.000 0.000 0.996 0.004 0.000 0.000
#> GSM340297 3 0.0547 0.81814 0.000 0.000 0.980 0.020 0.000 0.000
#> GSM340298 3 0.0547 0.81814 0.000 0.000 0.980 0.020 0.000 0.000
#> GSM340299 5 0.4076 0.57602 0.000 0.452 0.008 0.000 0.540 0.000
#> GSM340301 5 0.4062 0.58245 0.000 0.440 0.008 0.000 0.552 0.000
#> GSM340303 5 0.6119 0.44540 0.124 0.404 0.000 0.032 0.440 0.000
#> GSM340304 3 0.2219 0.76203 0.000 0.000 0.864 0.000 0.136 0.000
#> GSM340306 4 0.5083 0.38068 0.060 0.000 0.404 0.528 0.008 0.000
#> GSM340307 6 0.1713 0.73442 0.028 0.000 0.000 0.044 0.000 0.928
#> GSM340310 3 0.3828 0.45662 0.000 0.000 0.560 0.000 0.440 0.000
#> GSM340314 3 0.2302 0.73845 0.008 0.000 0.872 0.120 0.000 0.000
#> GSM340315 3 0.0964 0.81179 0.012 0.000 0.968 0.016 0.004 0.000
#> GSM340317 6 0.0603 0.73328 0.004 0.000 0.000 0.016 0.000 0.980
#> GSM340318 4 0.2048 0.87488 0.000 0.000 0.120 0.880 0.000 0.000
#> GSM340319 4 0.2048 0.87488 0.000 0.000 0.120 0.880 0.000 0.000
#> GSM340320 3 0.3876 0.47670 0.024 0.000 0.700 0.276 0.000 0.000
#> GSM340321 6 0.5636 0.16664 0.308 0.176 0.000 0.000 0.000 0.516
#> GSM340322 4 0.3123 0.85048 0.056 0.000 0.112 0.832 0.000 0.000
#> GSM340324 1 0.5209 0.62115 0.492 0.416 0.000 0.000 0.000 0.092
#> GSM340328 3 0.1983 0.79272 0.020 0.000 0.908 0.000 0.072 0.000
#> GSM340330 3 0.0692 0.81751 0.004 0.000 0.976 0.020 0.000 0.000
#> GSM340332 4 0.2191 0.87415 0.004 0.000 0.120 0.876 0.000 0.000
#> GSM340333 2 0.5064 -0.35831 0.372 0.552 0.000 0.004 0.000 0.072
#> GSM340336 4 0.4076 0.18931 0.012 0.000 0.000 0.592 0.000 0.396
#> GSM340337 4 0.2843 0.86125 0.036 0.000 0.116 0.848 0.000 0.000
#> GSM340338 3 0.0520 0.81929 0.008 0.000 0.984 0.000 0.008 0.000
#> GSM340339 4 0.2494 0.87169 0.016 0.000 0.120 0.864 0.000 0.000
#> GSM340340 4 0.4945 0.24052 0.092 0.000 0.000 0.604 0.000 0.304
#> GSM340341 6 0.7729 -0.00410 0.232 0.000 0.244 0.256 0.000 0.268
#> GSM340343 3 0.3592 0.55580 0.020 0.000 0.740 0.240 0.000 0.000
#> GSM340344 2 0.6057 -0.07461 0.176 0.612 0.000 0.036 0.160 0.016
#> GSM340346 3 0.0000 0.81933 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340347 4 0.2402 0.87261 0.012 0.000 0.120 0.868 0.000 0.000
#> GSM340348 3 0.3409 0.64254 0.024 0.000 0.788 0.184 0.004 0.000
#> GSM340349 3 0.3756 0.50816 0.000 0.000 0.600 0.000 0.400 0.000
#> GSM340350 3 0.3789 0.49104 0.000 0.000 0.584 0.000 0.416 0.000
#> GSM340351 5 0.0260 0.49604 0.000 0.000 0.008 0.000 0.992 0.000
#> GSM340354 3 0.2698 0.75819 0.092 0.000 0.872 0.020 0.016 0.000
#> GSM340356 6 0.2454 0.69538 0.160 0.000 0.000 0.000 0.000 0.840
#> GSM340357 3 0.3867 0.36456 0.000 0.000 0.512 0.000 0.488 0.000
#> GSM348183 5 0.0713 0.48576 0.000 0.000 0.028 0.000 0.972 0.000
#> GSM348191 3 0.0547 0.81814 0.000 0.000 0.980 0.020 0.000 0.000
#> GSM348193 3 0.4212 0.46390 0.016 0.000 0.560 0.000 0.424 0.000
#> GSM537578 3 0.3810 0.47028 0.000 0.000 0.572 0.000 0.428 0.000
#> GSM348181 3 0.0363 0.81864 0.000 0.000 0.988 0.000 0.012 0.000
#> GSM348182 5 0.0520 0.49878 0.000 0.008 0.008 0.000 0.984 0.000
#> GSM348184 6 0.0603 0.73419 0.016 0.000 0.000 0.004 0.000 0.980
#> GSM348185 6 0.0777 0.73233 0.004 0.000 0.000 0.024 0.000 0.972
#> GSM348186 2 0.5223 -0.60636 0.436 0.472 0.000 0.000 0.000 0.092
#> GSM348187 3 0.0458 0.81810 0.000 0.000 0.984 0.000 0.016 0.000
#> GSM348188 5 0.2107 0.48046 0.052 0.008 0.008 0.016 0.916 0.000
#> GSM348189 3 0.3062 0.72660 0.160 0.000 0.816 0.024 0.000 0.000
#> GSM348190 5 0.4062 0.58245 0.000 0.440 0.008 0.000 0.552 0.000
#> GSM348194 5 0.3851 -0.27148 0.000 0.000 0.460 0.000 0.540 0.000
#> GSM348195 5 0.0260 0.49604 0.000 0.000 0.008 0.000 0.992 0.000
#> GSM348196 3 0.1327 0.80119 0.000 0.000 0.936 0.000 0.064 0.000
#> GSM537585 5 0.0508 0.49810 0.000 0.012 0.004 0.000 0.984 0.000
#> GSM537594 6 0.5682 0.12411 0.316 0.180 0.000 0.000 0.000 0.504
#> GSM537596 3 0.0000 0.81933 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM537597 3 0.7119 0.13425 0.244 0.000 0.360 0.064 0.328 0.004
#> GSM537602 3 0.3823 0.46271 0.000 0.000 0.564 0.000 0.436 0.000
#> GSM340184 3 0.0291 0.81888 0.004 0.000 0.992 0.000 0.004 0.000
#> GSM340185 6 0.5636 0.16664 0.308 0.176 0.000 0.000 0.000 0.516
#> GSM340186 6 0.1957 0.71397 0.112 0.000 0.000 0.000 0.000 0.888
#> GSM340187 4 0.1338 0.72942 0.032 0.000 0.004 0.952 0.004 0.008
#> GSM340189 4 0.2048 0.87488 0.000 0.000 0.120 0.880 0.000 0.000
#> GSM340190 6 0.2572 0.67556 0.012 0.000 0.000 0.136 0.000 0.852
#> GSM340191 3 0.0806 0.81740 0.008 0.000 0.972 0.020 0.000 0.000
#> GSM340192 2 0.6833 0.16932 0.316 0.492 0.000 0.056 0.100 0.036
#> GSM340193 5 0.3866 -0.32763 0.000 0.000 0.484 0.000 0.516 0.000
#> GSM340194 5 0.4076 0.57602 0.000 0.452 0.008 0.000 0.540 0.000
#> GSM340195 5 0.4076 0.57602 0.000 0.452 0.008 0.000 0.540 0.000
#> GSM340196 4 0.2048 0.87488 0.000 0.000 0.120 0.880 0.000 0.000
#> GSM340197 5 0.4062 0.58245 0.000 0.440 0.008 0.000 0.552 0.000
#> GSM340198 2 0.5297 -0.43846 0.384 0.520 0.000 0.004 0.000 0.092
#> GSM340199 3 0.0806 0.81740 0.008 0.000 0.972 0.020 0.000 0.000
#> GSM340200 6 0.1411 0.72833 0.004 0.000 0.000 0.060 0.000 0.936
#> GSM340201 4 0.2048 0.87488 0.000 0.000 0.120 0.880 0.000 0.000
#> GSM340202 4 0.2146 0.87284 0.000 0.000 0.116 0.880 0.004 0.000
#> GSM340203 4 0.2494 0.87169 0.016 0.000 0.120 0.864 0.000 0.000
#> GSM340204 2 0.7126 -0.23541 0.244 0.392 0.000 0.044 0.304 0.016
#> GSM340205 3 0.0363 0.81864 0.000 0.000 0.988 0.000 0.012 0.000
#> GSM340206 3 0.1297 0.80764 0.012 0.000 0.948 0.040 0.000 0.000
#> GSM340207 5 0.4062 0.58245 0.000 0.440 0.008 0.000 0.552 0.000
#> GSM340237 6 0.2562 0.68685 0.172 0.000 0.000 0.000 0.000 0.828
#> GSM340238 3 0.2948 0.64982 0.008 0.000 0.804 0.188 0.000 0.000
#> GSM340239 5 0.4062 0.58245 0.000 0.440 0.008 0.000 0.552 0.000
#> GSM340240 5 0.4076 0.57602 0.000 0.452 0.008 0.000 0.540 0.000
#> GSM340241 1 0.5727 0.48926 0.516 0.264 0.000 0.000 0.000 0.220
#> GSM340242 3 0.3062 0.72660 0.160 0.000 0.816 0.024 0.000 0.000
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
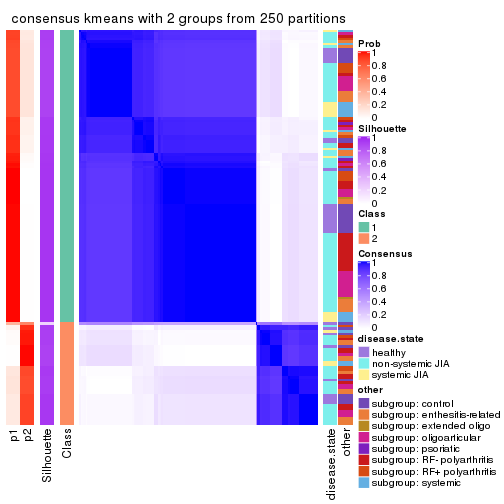
consensus_heatmap(res, k = 3)
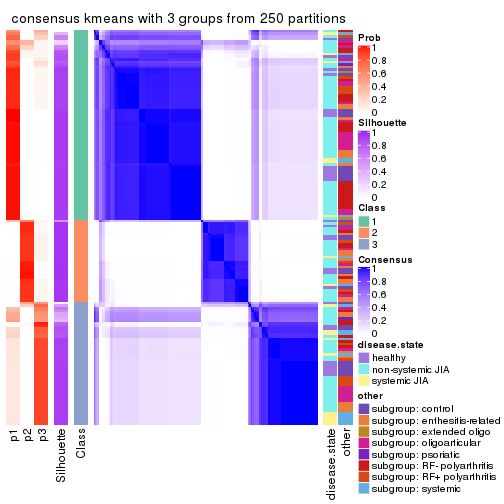
consensus_heatmap(res, k = 4)
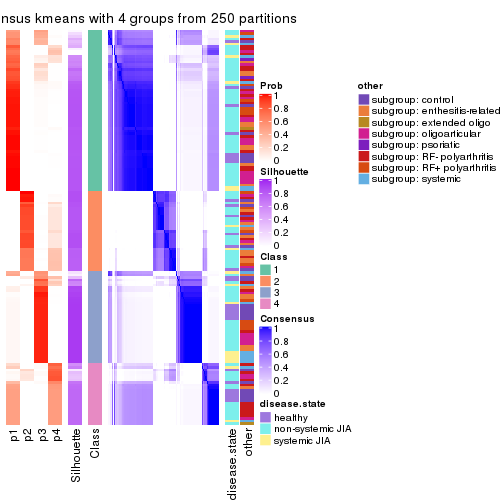
consensus_heatmap(res, k = 5)
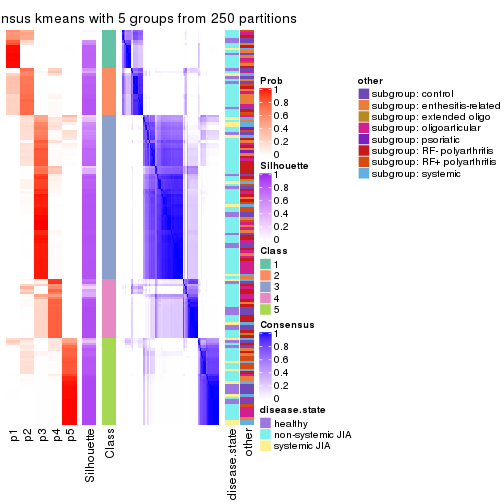
consensus_heatmap(res, k = 6)
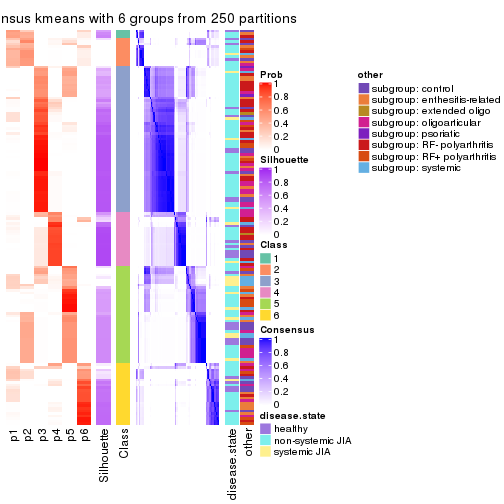
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
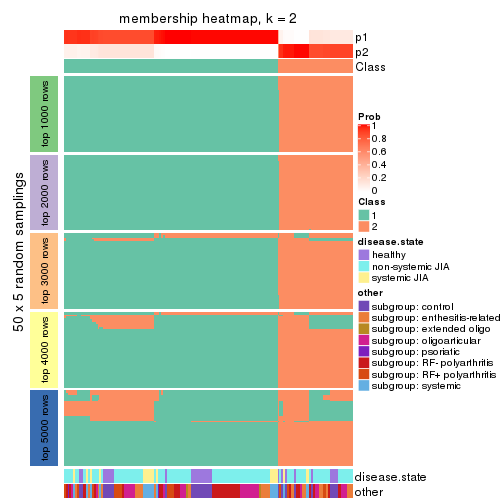
membership_heatmap(res, k = 3)
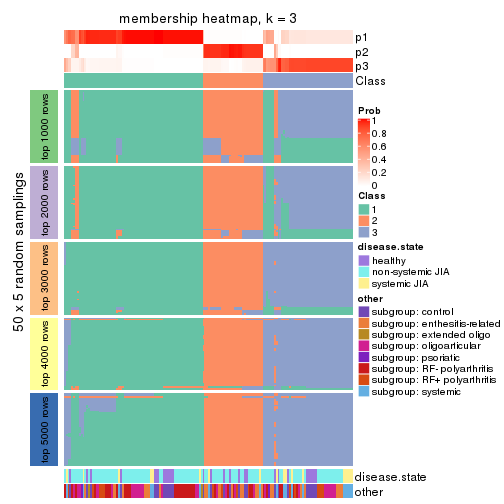
membership_heatmap(res, k = 4)
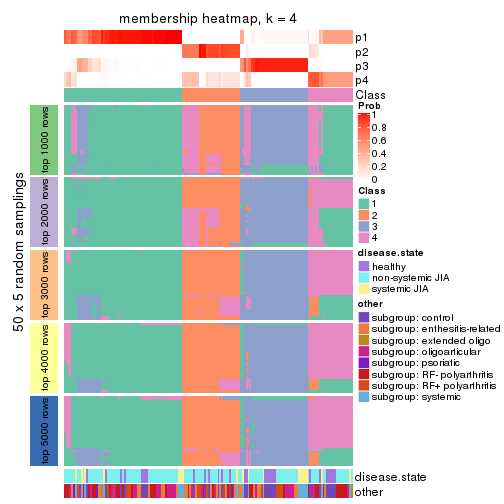
membership_heatmap(res, k = 5)
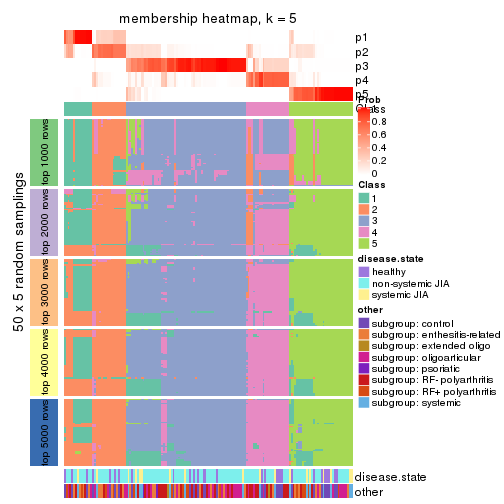
membership_heatmap(res, k = 6)
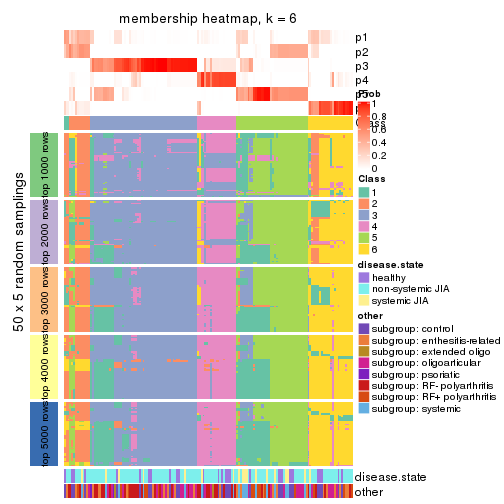
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
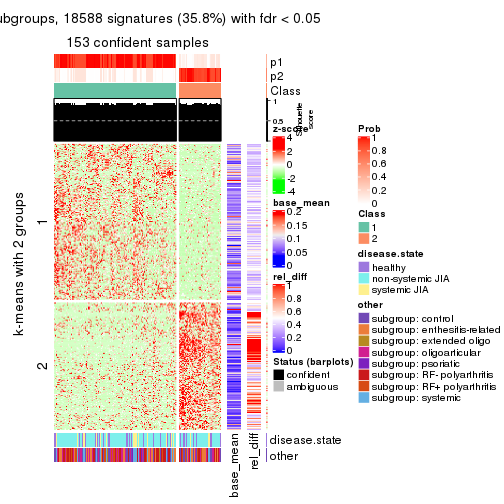
get_signatures(res, k = 3)
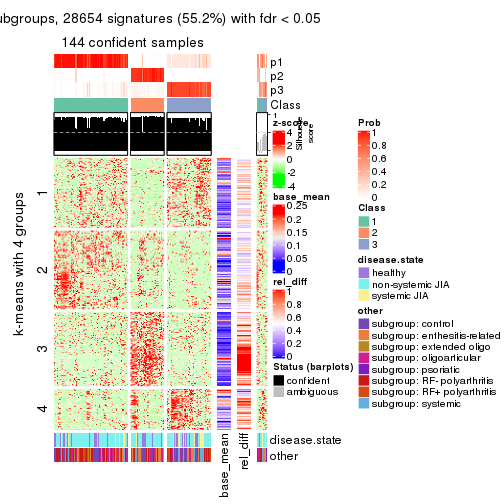
get_signatures(res, k = 4)
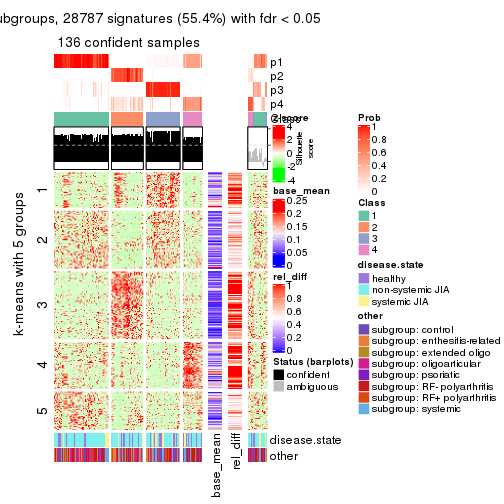
get_signatures(res, k = 5)
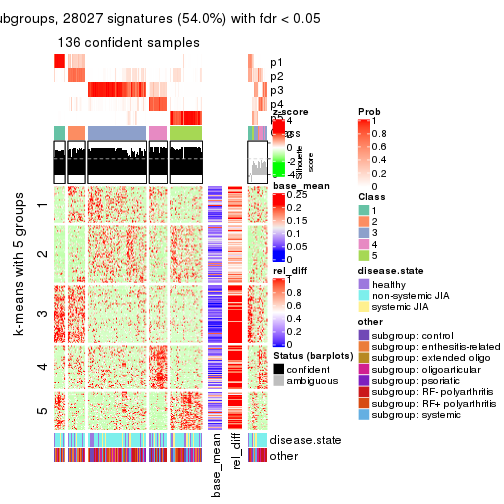
get_signatures(res, k = 6)
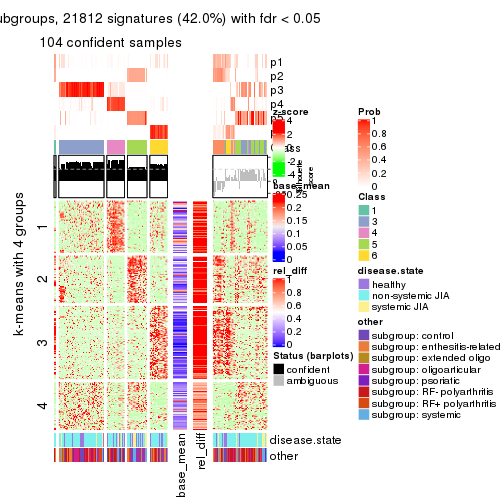
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
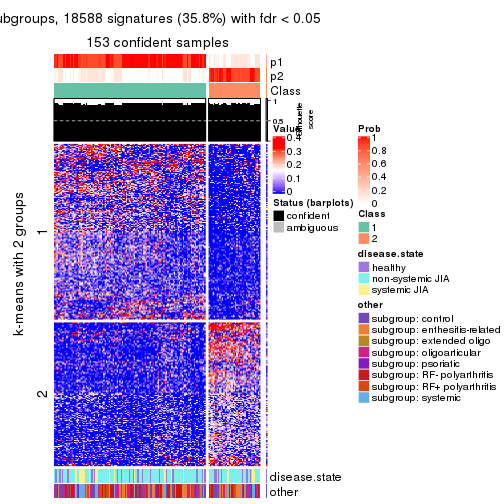
get_signatures(res, k = 3, scale_rows = FALSE)
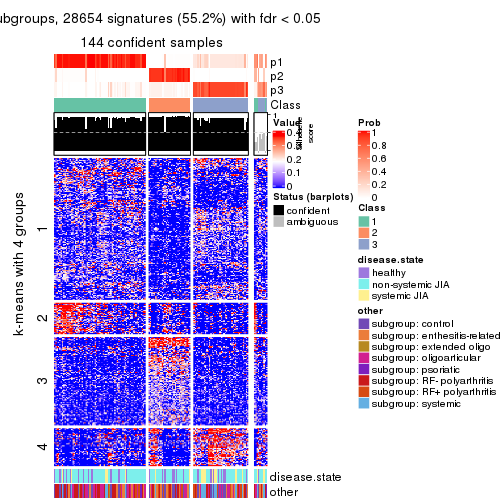
get_signatures(res, k = 4, scale_rows = FALSE)
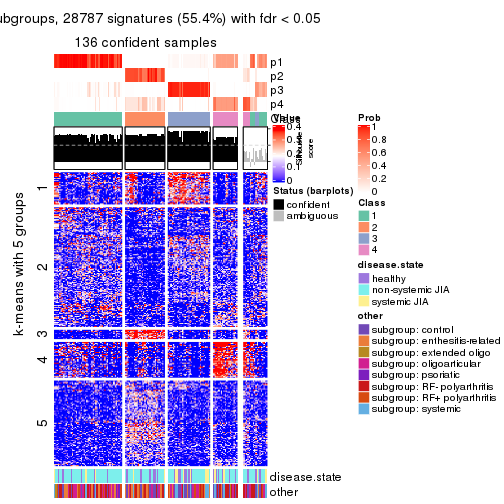
get_signatures(res, k = 5, scale_rows = FALSE)
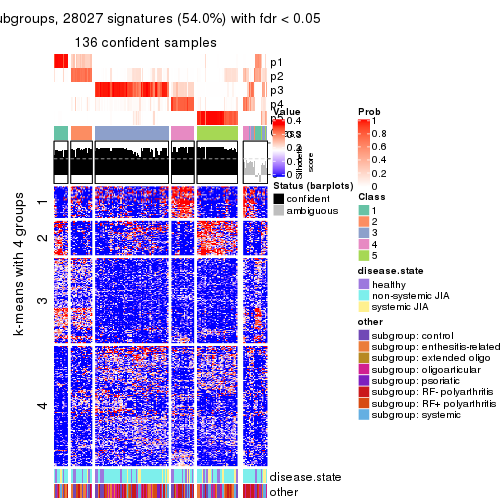
get_signatures(res, k = 6, scale_rows = FALSE)
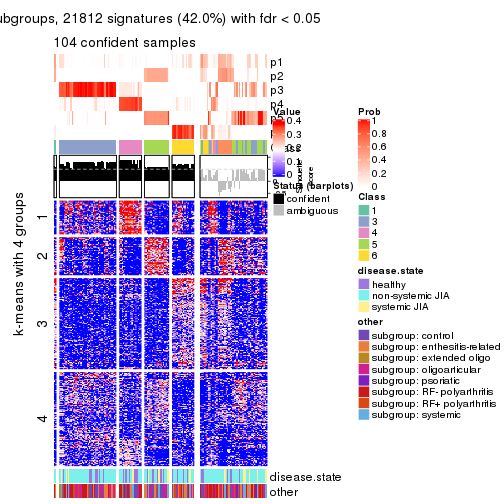
Compare the overlap of signatures from different k:
compare_signatures(res)
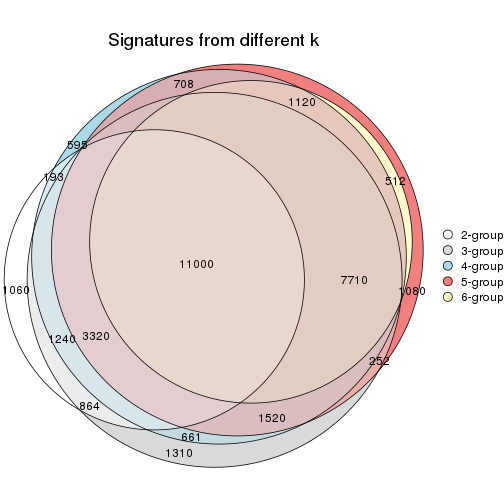
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
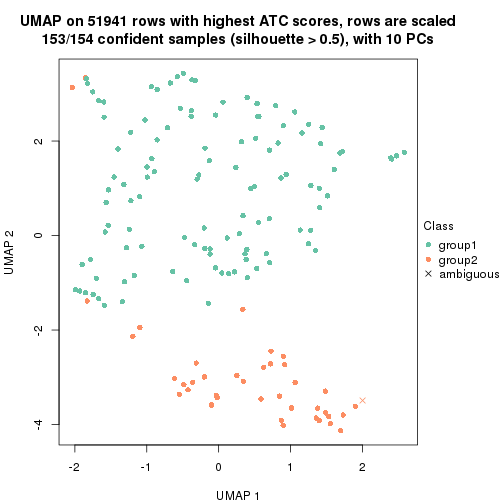
dimension_reduction(res, k = 3, method = "UMAP")
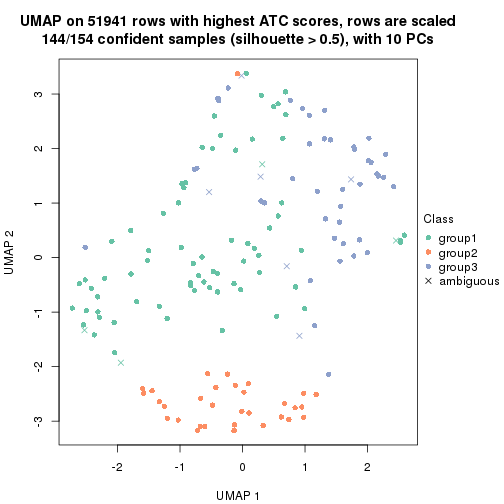
dimension_reduction(res, k = 4, method = "UMAP")
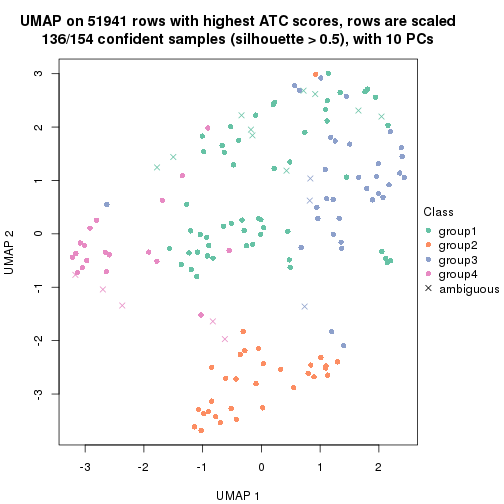
dimension_reduction(res, k = 5, method = "UMAP")
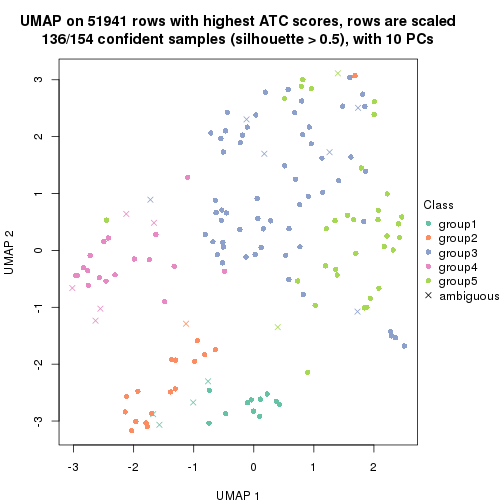
dimension_reduction(res, k = 6, method = "UMAP")
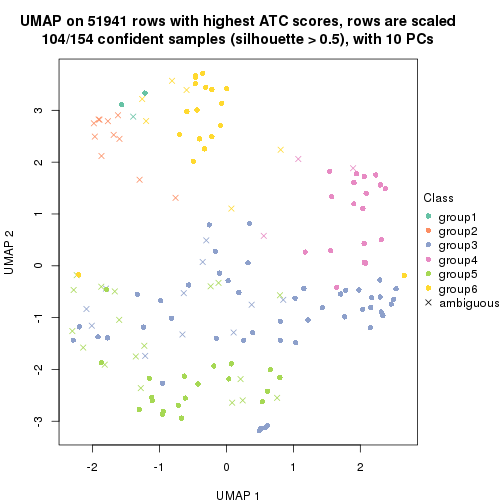
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
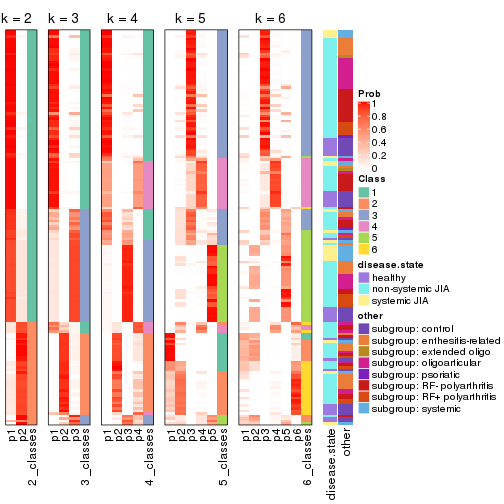
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> ATC:kmeans 153 0.636 0.6755 2
#> ATC:kmeans 144 0.154 0.0912 3
#> ATC:kmeans 136 0.190 0.0679 4
#> ATC:kmeans 136 0.620 0.4182 5
#> ATC:kmeans 104 0.182 0.1797 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["ATC", "skmeans"]
# you can also extract it by
# res = res_list["ATC:skmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'ATC' method.
#> Subgroups are detected by 'skmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
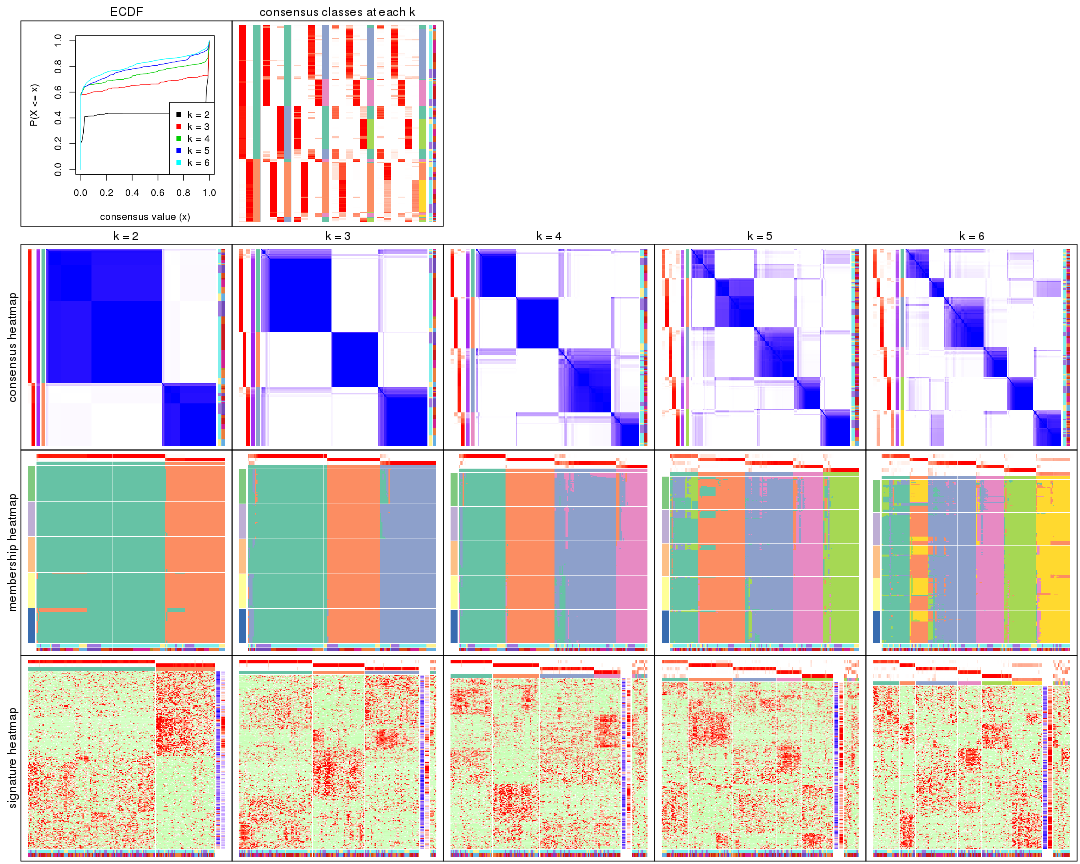
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
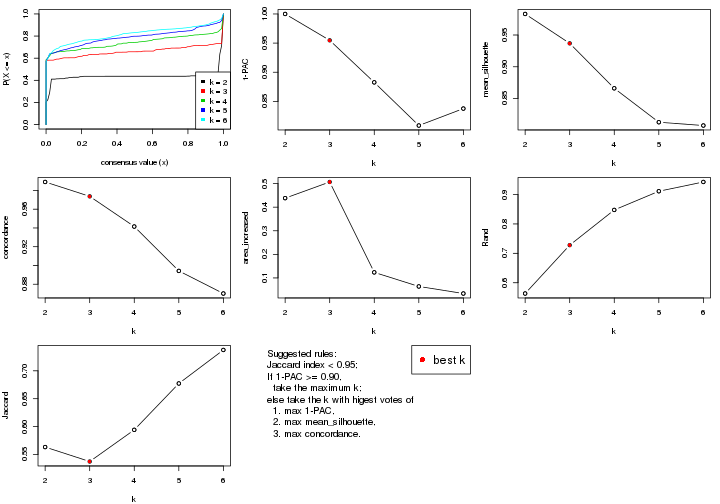
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 1.000 0.983 0.989 0.4382 0.563 0.563
#> 3 3 0.955 0.937 0.974 0.5068 0.728 0.537
#> 4 4 0.883 0.866 0.942 0.1235 0.848 0.594
#> 5 5 0.809 0.813 0.894 0.0639 0.911 0.677
#> 6 6 0.838 0.807 0.870 0.0339 0.943 0.737
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
#> attr(,"optional")
#> [1] 2
There is also optional best \(k\) = 2 that is worth to check.
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 1 0.000 0.990 1.000 0.000
#> GSM340359 2 0.000 0.987 0.000 1.000
#> GSM340361 1 0.163 0.983 0.976 0.024
#> GSM340362 1 0.163 0.983 0.976 0.024
#> GSM340363 1 0.000 0.990 1.000 0.000
#> GSM340364 2 0.000 0.987 0.000 1.000
#> GSM340365 1 0.163 0.983 0.976 0.024
#> GSM340366 1 0.163 0.983 0.976 0.024
#> GSM340367 1 0.141 0.984 0.980 0.020
#> GSM340368 2 0.163 0.977 0.024 0.976
#> GSM340369 1 0.000 0.990 1.000 0.000
#> GSM340370 2 0.482 0.883 0.104 0.896
#> GSM340371 1 0.000 0.990 1.000 0.000
#> GSM340372 1 0.163 0.983 0.976 0.024
#> GSM340373 1 0.163 0.983 0.976 0.024
#> GSM340375 2 0.000 0.987 0.000 1.000
#> GSM340376 2 0.000 0.987 0.000 1.000
#> GSM340378 2 0.000 0.987 0.000 1.000
#> GSM340243 1 0.000 0.990 1.000 0.000
#> GSM340244 2 0.000 0.987 0.000 1.000
#> GSM340246 2 0.000 0.987 0.000 1.000
#> GSM340247 1 0.295 0.944 0.948 0.052
#> GSM340248 2 0.000 0.987 0.000 1.000
#> GSM340249 2 0.141 0.979 0.020 0.980
#> GSM340250 1 0.118 0.986 0.984 0.016
#> GSM340251 2 0.141 0.979 0.020 0.980
#> GSM340252 2 0.000 0.987 0.000 1.000
#> GSM340253 2 0.000 0.987 0.000 1.000
#> GSM340254 2 0.000 0.987 0.000 1.000
#> GSM340256 2 0.000 0.987 0.000 1.000
#> GSM340258 2 0.689 0.773 0.184 0.816
#> GSM340259 1 0.163 0.983 0.976 0.024
#> GSM340260 1 0.141 0.984 0.980 0.020
#> GSM340261 1 0.000 0.990 1.000 0.000
#> GSM340262 1 0.163 0.983 0.976 0.024
#> GSM340263 2 0.141 0.979 0.020 0.980
#> GSM340264 1 0.163 0.983 0.976 0.024
#> GSM340265 1 0.163 0.983 0.976 0.024
#> GSM340266 1 0.000 0.990 1.000 0.000
#> GSM340267 1 0.163 0.983 0.976 0.024
#> GSM340268 1 0.000 0.990 1.000 0.000
#> GSM340269 1 0.000 0.990 1.000 0.000
#> GSM340270 1 0.000 0.990 1.000 0.000
#> GSM537574 2 0.000 0.987 0.000 1.000
#> GSM537580 1 0.000 0.990 1.000 0.000
#> GSM537581 1 0.000 0.990 1.000 0.000
#> GSM340272 1 0.163 0.983 0.976 0.024
#> GSM340273 2 0.000 0.987 0.000 1.000
#> GSM340275 2 0.163 0.977 0.024 0.976
#> GSM340276 1 0.000 0.990 1.000 0.000
#> GSM340277 2 0.000 0.987 0.000 1.000
#> GSM340278 1 0.000 0.990 1.000 0.000
#> GSM340279 1 0.000 0.990 1.000 0.000
#> GSM340282 1 0.163 0.983 0.976 0.024
#> GSM340284 2 0.163 0.977 0.024 0.976
#> GSM340285 1 0.000 0.990 1.000 0.000
#> GSM340286 1 0.163 0.983 0.976 0.024
#> GSM340287 1 0.000 0.990 1.000 0.000
#> GSM340288 1 0.141 0.984 0.980 0.020
#> GSM340289 1 0.141 0.984 0.980 0.020
#> GSM340290 1 0.000 0.990 1.000 0.000
#> GSM340291 2 0.000 0.987 0.000 1.000
#> GSM340293 1 0.000 0.990 1.000 0.000
#> GSM340294 1 0.000 0.990 1.000 0.000
#> GSM340296 1 0.000 0.990 1.000 0.000
#> GSM340297 1 0.000 0.990 1.000 0.000
#> GSM340298 1 0.000 0.990 1.000 0.000
#> GSM340299 1 0.163 0.983 0.976 0.024
#> GSM340301 1 0.163 0.983 0.976 0.024
#> GSM340303 1 0.163 0.983 0.976 0.024
#> GSM340304 1 0.000 0.990 1.000 0.000
#> GSM340306 1 0.000 0.990 1.000 0.000
#> GSM340307 2 0.000 0.987 0.000 1.000
#> GSM340310 1 0.000 0.990 1.000 0.000
#> GSM340314 1 0.000 0.990 1.000 0.000
#> GSM340315 1 0.000 0.990 1.000 0.000
#> GSM340317 2 0.141 0.979 0.020 0.980
#> GSM340318 1 0.000 0.990 1.000 0.000
#> GSM340319 1 0.000 0.990 1.000 0.000
#> GSM340320 1 0.000 0.990 1.000 0.000
#> GSM340321 2 0.000 0.987 0.000 1.000
#> GSM340322 1 0.000 0.990 1.000 0.000
#> GSM340324 2 0.000 0.987 0.000 1.000
#> GSM340328 1 0.000 0.990 1.000 0.000
#> GSM340330 1 0.000 0.990 1.000 0.000
#> GSM340332 1 0.000 0.990 1.000 0.000
#> GSM340333 2 0.000 0.987 0.000 1.000
#> GSM340336 2 0.163 0.977 0.024 0.976
#> GSM340337 1 0.000 0.990 1.000 0.000
#> GSM340338 1 0.000 0.990 1.000 0.000
#> GSM340339 1 0.000 0.990 1.000 0.000
#> GSM340340 2 0.163 0.977 0.024 0.976
#> GSM340341 2 0.163 0.977 0.024 0.976
#> GSM340343 1 0.000 0.990 1.000 0.000
#> GSM340344 2 0.000 0.987 0.000 1.000
#> GSM340346 1 0.000 0.990 1.000 0.000
#> GSM340347 1 0.000 0.990 1.000 0.000
#> GSM340348 1 0.000 0.990 1.000 0.000
#> GSM340349 1 0.000 0.990 1.000 0.000
#> GSM340350 1 0.000 0.990 1.000 0.000
#> GSM340351 1 0.163 0.983 0.976 0.024
#> GSM340354 1 0.605 0.846 0.852 0.148
#> GSM340356 2 0.000 0.987 0.000 1.000
#> GSM340357 1 0.141 0.984 0.980 0.020
#> GSM348183 1 0.141 0.984 0.980 0.020
#> GSM348191 1 0.000 0.990 1.000 0.000
#> GSM348193 1 0.000 0.990 1.000 0.000
#> GSM537578 1 0.141 0.984 0.980 0.020
#> GSM348181 1 0.000 0.990 1.000 0.000
#> GSM348182 1 0.163 0.983 0.976 0.024
#> GSM348184 2 0.141 0.979 0.020 0.980
#> GSM348185 2 0.141 0.979 0.020 0.980
#> GSM348186 2 0.000 0.987 0.000 1.000
#> GSM348187 1 0.000 0.990 1.000 0.000
#> GSM348188 1 0.000 0.990 1.000 0.000
#> GSM348189 1 0.000 0.990 1.000 0.000
#> GSM348190 1 0.163 0.983 0.976 0.024
#> GSM348194 1 0.141 0.984 0.980 0.020
#> GSM348195 1 0.163 0.983 0.976 0.024
#> GSM348196 1 0.000 0.990 1.000 0.000
#> GSM537585 1 0.163 0.983 0.976 0.024
#> GSM537594 2 0.000 0.987 0.000 1.000
#> GSM537596 1 0.000 0.990 1.000 0.000
#> GSM537597 2 0.000 0.987 0.000 1.000
#> GSM537602 1 0.141 0.984 0.980 0.020
#> GSM340184 1 0.000 0.990 1.000 0.000
#> GSM340185 2 0.000 0.987 0.000 1.000
#> GSM340186 2 0.000 0.987 0.000 1.000
#> GSM340187 2 0.163 0.977 0.024 0.976
#> GSM340189 1 0.000 0.990 1.000 0.000
#> GSM340190 2 0.163 0.977 0.024 0.976
#> GSM340191 1 0.000 0.990 1.000 0.000
#> GSM340192 2 0.000 0.987 0.000 1.000
#> GSM340193 1 0.141 0.984 0.980 0.020
#> GSM340194 1 0.163 0.983 0.976 0.024
#> GSM340195 1 0.163 0.983 0.976 0.024
#> GSM340196 1 0.000 0.990 1.000 0.000
#> GSM340197 1 0.163 0.983 0.976 0.024
#> GSM340198 2 0.000 0.987 0.000 1.000
#> GSM340199 1 0.000 0.990 1.000 0.000
#> GSM340200 2 0.000 0.987 0.000 1.000
#> GSM340201 1 0.000 0.990 1.000 0.000
#> GSM340202 1 0.000 0.990 1.000 0.000
#> GSM340203 1 0.000 0.990 1.000 0.000
#> GSM340204 2 0.000 0.987 0.000 1.000
#> GSM340205 1 0.000 0.990 1.000 0.000
#> GSM340206 1 0.000 0.990 1.000 0.000
#> GSM340207 1 0.163 0.983 0.976 0.024
#> GSM340237 2 0.000 0.987 0.000 1.000
#> GSM340238 1 0.000 0.990 1.000 0.000
#> GSM340239 1 0.163 0.983 0.976 0.024
#> GSM340240 1 0.163 0.983 0.976 0.024
#> GSM340241 2 0.000 0.987 0.000 1.000
#> GSM340242 1 0.000 0.990 1.000 0.000
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340359 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340361 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340362 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340363 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340364 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340365 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340366 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340367 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340368 1 0.4974 0.677 0.764 0.236 0.000
#> GSM340369 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340370 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340371 1 0.6286 0.107 0.536 0.000 0.464
#> GSM340372 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340373 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340375 3 0.0424 0.957 0.000 0.008 0.992
#> GSM340376 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340378 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340243 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340244 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340246 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340247 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340248 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340249 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340250 3 0.4291 0.785 0.180 0.000 0.820
#> GSM340251 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340252 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340253 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340254 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340256 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340258 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340259 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340260 3 0.4702 0.740 0.212 0.000 0.788
#> GSM340261 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340262 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340263 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340264 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340265 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340266 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340267 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340268 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340269 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340270 1 0.0000 0.960 1.000 0.000 0.000
#> GSM537574 2 0.0000 0.998 0.000 1.000 0.000
#> GSM537580 1 0.0000 0.960 1.000 0.000 0.000
#> GSM537581 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340272 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340273 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340275 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340276 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340277 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340278 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340279 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340282 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340284 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340285 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340286 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340287 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340288 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340289 3 0.2066 0.916 0.060 0.000 0.940
#> GSM340290 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340291 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340293 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340294 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340296 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340297 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340298 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340299 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340301 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340303 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340304 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340306 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340307 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340310 1 0.6045 0.399 0.620 0.000 0.380
#> GSM340314 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340315 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340317 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340318 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340319 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340320 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340321 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340322 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340324 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340328 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340330 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340332 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340333 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340336 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340337 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340338 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340339 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340340 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340341 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340343 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340344 3 0.4654 0.736 0.000 0.208 0.792
#> GSM340346 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340347 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340348 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340349 3 0.5529 0.597 0.296 0.000 0.704
#> GSM340350 3 0.5859 0.493 0.344 0.000 0.656
#> GSM340351 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340354 1 0.7569 0.586 0.668 0.240 0.092
#> GSM340356 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340357 3 0.0000 0.963 0.000 0.000 1.000
#> GSM348183 3 0.0000 0.963 0.000 0.000 1.000
#> GSM348191 1 0.0000 0.960 1.000 0.000 0.000
#> GSM348193 1 0.4346 0.760 0.816 0.000 0.184
#> GSM537578 3 0.1753 0.926 0.048 0.000 0.952
#> GSM348181 1 0.0000 0.960 1.000 0.000 0.000
#> GSM348182 3 0.0000 0.963 0.000 0.000 1.000
#> GSM348184 2 0.0000 0.998 0.000 1.000 0.000
#> GSM348185 2 0.0000 0.998 0.000 1.000 0.000
#> GSM348186 2 0.0000 0.998 0.000 1.000 0.000
#> GSM348187 1 0.0000 0.960 1.000 0.000 0.000
#> GSM348188 3 0.0000 0.963 0.000 0.000 1.000
#> GSM348189 1 0.6062 0.361 0.616 0.000 0.384
#> GSM348190 3 0.0000 0.963 0.000 0.000 1.000
#> GSM348194 3 0.0000 0.963 0.000 0.000 1.000
#> GSM348195 3 0.0000 0.963 0.000 0.000 1.000
#> GSM348196 1 0.0000 0.960 1.000 0.000 0.000
#> GSM537585 3 0.0000 0.963 0.000 0.000 1.000
#> GSM537594 2 0.0000 0.998 0.000 1.000 0.000
#> GSM537596 1 0.0000 0.960 1.000 0.000 0.000
#> GSM537597 2 0.0000 0.998 0.000 1.000 0.000
#> GSM537602 3 0.4062 0.806 0.164 0.000 0.836
#> GSM340184 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340185 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340186 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340187 1 0.2711 0.875 0.912 0.088 0.000
#> GSM340189 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340190 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340191 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340192 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340193 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340194 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340195 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340196 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340197 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340198 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340199 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340200 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340201 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340202 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340203 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340204 2 0.2537 0.909 0.000 0.920 0.080
#> GSM340205 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340206 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340207 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340237 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340238 1 0.0000 0.960 1.000 0.000 0.000
#> GSM340239 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340240 3 0.0000 0.963 0.000 0.000 1.000
#> GSM340241 2 0.0000 0.998 0.000 1.000 0.000
#> GSM340242 1 0.6062 0.361 0.616 0.000 0.384
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 3 0.0592 0.8955 0.000 0.000 0.984 0.016
#> GSM340359 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340361 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340362 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340363 3 0.0921 0.8961 0.000 0.000 0.972 0.028
#> GSM340364 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340365 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340366 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340367 3 0.4925 0.2907 0.428 0.000 0.572 0.000
#> GSM340368 4 0.1474 0.8601 0.000 0.052 0.000 0.948
#> GSM340369 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340370 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340371 3 0.6031 0.3918 0.388 0.000 0.564 0.048
#> GSM340372 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340373 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340375 1 0.0188 0.9498 0.996 0.004 0.000 0.000
#> GSM340376 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340378 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340243 3 0.0707 0.8975 0.000 0.000 0.980 0.020
#> GSM340244 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340246 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340247 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340248 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340249 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340250 3 0.4605 0.5171 0.336 0.000 0.664 0.000
#> GSM340251 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340252 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340253 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340254 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340256 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340258 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340259 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340260 3 0.4843 0.3873 0.396 0.000 0.604 0.000
#> GSM340261 3 0.0921 0.8961 0.000 0.000 0.972 0.028
#> GSM340262 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340263 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340264 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340265 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340266 3 0.0921 0.8961 0.000 0.000 0.972 0.028
#> GSM340267 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340268 3 0.2530 0.8356 0.000 0.000 0.888 0.112
#> GSM340269 3 0.0592 0.8975 0.000 0.000 0.984 0.016
#> GSM340270 3 0.0000 0.8947 0.000 0.000 1.000 0.000
#> GSM537574 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM537580 3 0.4567 0.6211 0.008 0.000 0.716 0.276
#> GSM537581 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340272 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340273 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340275 4 0.4933 0.2971 0.000 0.432 0.000 0.568
#> GSM340276 3 0.0000 0.8947 0.000 0.000 1.000 0.000
#> GSM340277 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340278 3 0.0817 0.8972 0.000 0.000 0.976 0.024
#> GSM340279 3 0.0921 0.8961 0.000 0.000 0.972 0.028
#> GSM340282 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340284 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340285 3 0.0817 0.8972 0.000 0.000 0.976 0.024
#> GSM340286 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340287 3 0.0707 0.8975 0.000 0.000 0.980 0.020
#> GSM340288 1 0.0707 0.9378 0.980 0.000 0.020 0.000
#> GSM340289 3 0.4972 0.2160 0.456 0.000 0.544 0.000
#> GSM340290 3 0.0921 0.8961 0.000 0.000 0.972 0.028
#> GSM340291 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340293 4 0.0707 0.8829 0.000 0.000 0.020 0.980
#> GSM340294 3 0.0817 0.8972 0.000 0.000 0.976 0.024
#> GSM340296 3 0.0707 0.8975 0.000 0.000 0.980 0.020
#> GSM340297 3 0.0817 0.8972 0.000 0.000 0.976 0.024
#> GSM340298 3 0.0817 0.8972 0.000 0.000 0.976 0.024
#> GSM340299 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340301 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340303 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340304 3 0.0000 0.8947 0.000 0.000 1.000 0.000
#> GSM340306 4 0.3311 0.7721 0.000 0.000 0.172 0.828
#> GSM340307 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340310 3 0.0336 0.8928 0.008 0.000 0.992 0.000
#> GSM340314 3 0.4543 0.5259 0.000 0.000 0.676 0.324
#> GSM340315 3 0.2011 0.8628 0.000 0.000 0.920 0.080
#> GSM340317 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340318 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340319 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340320 4 0.3356 0.7544 0.000 0.000 0.176 0.824
#> GSM340321 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340322 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340324 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340328 3 0.0592 0.8906 0.000 0.000 0.984 0.016
#> GSM340330 3 0.0817 0.8972 0.000 0.000 0.976 0.024
#> GSM340332 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340333 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340336 4 0.4855 0.3809 0.000 0.400 0.000 0.600
#> GSM340337 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340338 3 0.0000 0.8947 0.000 0.000 1.000 0.000
#> GSM340339 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340340 4 0.4855 0.3809 0.000 0.400 0.000 0.600
#> GSM340341 2 0.4605 0.4268 0.000 0.664 0.000 0.336
#> GSM340343 4 0.4040 0.6574 0.000 0.000 0.248 0.752
#> GSM340344 1 0.3610 0.7159 0.800 0.200 0.000 0.000
#> GSM340346 3 0.0469 0.8970 0.000 0.000 0.988 0.012
#> GSM340347 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340348 4 0.3266 0.7645 0.000 0.000 0.168 0.832
#> GSM340349 3 0.2921 0.7945 0.140 0.000 0.860 0.000
#> GSM340350 3 0.0188 0.8938 0.004 0.000 0.996 0.000
#> GSM340351 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340354 3 0.0592 0.8974 0.000 0.000 0.984 0.016
#> GSM340356 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340357 1 0.4996 -0.0279 0.516 0.000 0.484 0.000
#> GSM348183 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM348191 3 0.0817 0.8972 0.000 0.000 0.976 0.024
#> GSM348193 4 0.5150 0.3781 0.008 0.000 0.396 0.596
#> GSM537578 3 0.2469 0.8228 0.108 0.000 0.892 0.000
#> GSM348181 3 0.0000 0.8947 0.000 0.000 1.000 0.000
#> GSM348182 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM348184 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM348185 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM348186 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM348187 3 0.0000 0.8947 0.000 0.000 1.000 0.000
#> GSM348188 1 0.4605 0.4267 0.664 0.000 0.000 0.336
#> GSM348189 3 0.5219 0.6718 0.244 0.000 0.712 0.044
#> GSM348190 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM348194 1 0.1118 0.9239 0.964 0.000 0.036 0.000
#> GSM348195 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM348196 3 0.0000 0.8947 0.000 0.000 1.000 0.000
#> GSM537585 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM537594 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM537596 3 0.0000 0.8947 0.000 0.000 1.000 0.000
#> GSM537597 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM537602 3 0.0707 0.8868 0.020 0.000 0.980 0.000
#> GSM340184 3 0.0921 0.8961 0.000 0.000 0.972 0.028
#> GSM340185 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340186 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340187 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340189 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340190 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340191 3 0.0921 0.8961 0.000 0.000 0.972 0.028
#> GSM340192 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340193 1 0.4866 0.2498 0.596 0.000 0.404 0.000
#> GSM340194 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340195 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340196 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340197 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340198 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340199 3 0.0921 0.8961 0.000 0.000 0.972 0.028
#> GSM340200 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340201 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340202 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340203 4 0.0000 0.8946 0.000 0.000 0.000 1.000
#> GSM340204 2 0.1637 0.9186 0.060 0.940 0.000 0.000
#> GSM340205 3 0.0000 0.8947 0.000 0.000 1.000 0.000
#> GSM340206 3 0.2345 0.8462 0.000 0.000 0.900 0.100
#> GSM340207 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340237 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340238 3 0.4790 0.4141 0.000 0.000 0.620 0.380
#> GSM340239 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340240 1 0.0000 0.9534 1.000 0.000 0.000 0.000
#> GSM340241 2 0.0000 0.9886 0.000 1.000 0.000 0.000
#> GSM340242 3 0.4485 0.7375 0.200 0.000 0.772 0.028
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 3 0.1892 0.8483 0.080 0.000 0.916 0.004 0.000
#> GSM340359 2 0.2886 0.8894 0.148 0.844 0.000 0.008 0.000
#> GSM340361 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340362 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340363 3 0.0579 0.9011 0.008 0.000 0.984 0.008 0.000
#> GSM340364 2 0.3013 0.8818 0.160 0.832 0.000 0.008 0.000
#> GSM340365 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340366 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340367 1 0.4212 0.8019 0.776 0.000 0.144 0.000 0.080
#> GSM340368 4 0.2464 0.8117 0.016 0.096 0.000 0.888 0.000
#> GSM340369 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340370 1 0.2770 0.7550 0.864 0.000 0.008 0.004 0.124
#> GSM340371 5 0.5586 -0.0763 0.012 0.000 0.464 0.044 0.480
#> GSM340372 1 0.3534 0.7294 0.744 0.000 0.000 0.000 0.256
#> GSM340373 1 0.3895 0.6537 0.680 0.000 0.000 0.000 0.320
#> GSM340375 5 0.2722 0.8042 0.120 0.004 0.000 0.008 0.868
#> GSM340376 1 0.4560 -0.1457 0.508 0.484 0.000 0.008 0.000
#> GSM340378 2 0.2563 0.8996 0.120 0.872 0.000 0.008 0.000
#> GSM340243 3 0.0162 0.8994 0.004 0.000 0.996 0.000 0.000
#> GSM340244 2 0.0000 0.9347 0.000 1.000 0.000 0.000 0.000
#> GSM340246 2 0.0000 0.9347 0.000 1.000 0.000 0.000 0.000
#> GSM340247 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340248 2 0.0000 0.9347 0.000 1.000 0.000 0.000 0.000
#> GSM340249 2 0.0404 0.9312 0.012 0.988 0.000 0.000 0.000
#> GSM340250 1 0.3727 0.7672 0.768 0.000 0.216 0.000 0.016
#> GSM340251 2 0.0404 0.9312 0.012 0.988 0.000 0.000 0.000
#> GSM340252 2 0.2843 0.8915 0.144 0.848 0.000 0.008 0.000
#> GSM340253 2 0.2886 0.8894 0.148 0.844 0.000 0.008 0.000
#> GSM340254 2 0.2843 0.8915 0.144 0.848 0.000 0.008 0.000
#> GSM340256 2 0.2843 0.8915 0.144 0.848 0.000 0.008 0.000
#> GSM340258 5 0.0609 0.8987 0.020 0.000 0.000 0.000 0.980
#> GSM340259 1 0.3534 0.7294 0.744 0.000 0.000 0.000 0.256
#> GSM340260 1 0.4181 0.6995 0.712 0.000 0.268 0.000 0.020
#> GSM340261 3 0.0290 0.9023 0.000 0.000 0.992 0.008 0.000
#> GSM340262 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340263 2 0.0510 0.9296 0.016 0.984 0.000 0.000 0.000
#> GSM340264 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340265 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340266 3 0.0404 0.9010 0.000 0.000 0.988 0.012 0.000
#> GSM340267 5 0.0162 0.9106 0.004 0.000 0.000 0.000 0.996
#> GSM340268 3 0.1732 0.8542 0.000 0.000 0.920 0.080 0.000
#> GSM340269 3 0.0162 0.9017 0.000 0.000 0.996 0.004 0.000
#> GSM340270 3 0.4150 0.2736 0.388 0.000 0.612 0.000 0.000
#> GSM537574 2 0.0880 0.9226 0.032 0.968 0.000 0.000 0.000
#> GSM537580 3 0.5372 0.6415 0.012 0.000 0.692 0.112 0.184
#> GSM537581 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340272 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340273 2 0.0000 0.9347 0.000 1.000 0.000 0.000 0.000
#> GSM340275 4 0.4702 0.3239 0.016 0.432 0.000 0.552 0.000
#> GSM340276 3 0.0880 0.8855 0.032 0.000 0.968 0.000 0.000
#> GSM340277 2 0.2843 0.8915 0.144 0.848 0.000 0.008 0.000
#> GSM340278 3 0.0290 0.9023 0.000 0.000 0.992 0.008 0.000
#> GSM340279 3 0.0290 0.9023 0.000 0.000 0.992 0.008 0.000
#> GSM340282 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340284 2 0.0671 0.9277 0.016 0.980 0.000 0.004 0.000
#> GSM340285 3 0.0451 0.9021 0.004 0.000 0.988 0.008 0.000
#> GSM340286 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340287 3 0.0000 0.9004 0.000 0.000 1.000 0.000 0.000
#> GSM340288 1 0.3461 0.7505 0.772 0.000 0.004 0.000 0.224
#> GSM340289 1 0.4028 0.7897 0.776 0.000 0.176 0.000 0.048
#> GSM340290 3 0.0290 0.9023 0.000 0.000 0.992 0.008 0.000
#> GSM340291 2 0.0000 0.9347 0.000 1.000 0.000 0.000 0.000
#> GSM340293 4 0.1410 0.8499 0.000 0.000 0.060 0.940 0.000
#> GSM340294 3 0.0290 0.9023 0.000 0.000 0.992 0.008 0.000
#> GSM340296 3 0.0162 0.9018 0.000 0.000 0.996 0.004 0.000
#> GSM340297 3 0.0290 0.9023 0.000 0.000 0.992 0.008 0.000
#> GSM340298 3 0.0290 0.9023 0.000 0.000 0.992 0.008 0.000
#> GSM340299 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340301 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340303 5 0.0290 0.9081 0.008 0.000 0.000 0.000 0.992
#> GSM340304 1 0.4015 0.5619 0.652 0.000 0.348 0.000 0.000
#> GSM340306 4 0.3141 0.7758 0.040 0.000 0.108 0.852 0.000
#> GSM340307 2 0.0000 0.9347 0.000 1.000 0.000 0.000 0.000
#> GSM340310 1 0.3305 0.7585 0.776 0.000 0.224 0.000 0.000
#> GSM340314 3 0.2605 0.7893 0.000 0.000 0.852 0.148 0.000
#> GSM340315 3 0.1638 0.8649 0.004 0.000 0.932 0.064 0.000
#> GSM340317 2 0.0290 0.9326 0.008 0.992 0.000 0.000 0.000
#> GSM340318 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340319 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340320 4 0.3932 0.5158 0.000 0.000 0.328 0.672 0.000
#> GSM340321 2 0.0000 0.9347 0.000 1.000 0.000 0.000 0.000
#> GSM340322 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340324 2 0.2843 0.8915 0.144 0.848 0.000 0.008 0.000
#> GSM340328 3 0.4538 0.0342 0.452 0.000 0.540 0.008 0.000
#> GSM340330 3 0.0693 0.8984 0.012 0.000 0.980 0.008 0.000
#> GSM340332 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340333 2 0.2886 0.8894 0.148 0.844 0.000 0.008 0.000
#> GSM340336 4 0.4599 0.4278 0.016 0.384 0.000 0.600 0.000
#> GSM340337 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340338 3 0.1121 0.8849 0.044 0.000 0.956 0.000 0.000
#> GSM340339 4 0.0510 0.8773 0.000 0.000 0.016 0.984 0.000
#> GSM340340 4 0.4674 0.3646 0.016 0.416 0.000 0.568 0.000
#> GSM340341 2 0.4451 0.3595 0.016 0.644 0.000 0.340 0.000
#> GSM340343 4 0.4060 0.4546 0.000 0.000 0.360 0.640 0.000
#> GSM340344 5 0.5189 0.6214 0.160 0.124 0.000 0.008 0.708
#> GSM340346 3 0.0000 0.9004 0.000 0.000 1.000 0.000 0.000
#> GSM340347 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340348 4 0.2891 0.7473 0.000 0.000 0.176 0.824 0.000
#> GSM340349 1 0.3612 0.7562 0.764 0.000 0.228 0.000 0.008
#> GSM340350 1 0.3305 0.7585 0.776 0.000 0.224 0.000 0.000
#> GSM340351 1 0.3561 0.7250 0.740 0.000 0.000 0.000 0.260
#> GSM340354 3 0.0451 0.9005 0.008 0.000 0.988 0.004 0.000
#> GSM340356 2 0.0000 0.9347 0.000 1.000 0.000 0.000 0.000
#> GSM340357 1 0.4226 0.7950 0.776 0.000 0.084 0.000 0.140
#> GSM348183 1 0.3534 0.7294 0.744 0.000 0.000 0.000 0.256
#> GSM348191 3 0.0290 0.9023 0.000 0.000 0.992 0.008 0.000
#> GSM348193 1 0.4352 0.7695 0.772 0.000 0.160 0.060 0.008
#> GSM537578 1 0.3582 0.7599 0.768 0.000 0.224 0.000 0.008
#> GSM348181 3 0.1478 0.8639 0.064 0.000 0.936 0.000 0.000
#> GSM348182 5 0.3774 0.4680 0.296 0.000 0.000 0.000 0.704
#> GSM348184 2 0.0404 0.9312 0.012 0.988 0.000 0.000 0.000
#> GSM348185 2 0.0290 0.9326 0.008 0.992 0.000 0.000 0.000
#> GSM348186 2 0.2843 0.8915 0.144 0.848 0.000 0.008 0.000
#> GSM348187 3 0.1608 0.8575 0.072 0.000 0.928 0.000 0.000
#> GSM348188 5 0.4180 0.6256 0.036 0.000 0.000 0.220 0.744
#> GSM348189 3 0.4810 0.5222 0.012 0.000 0.652 0.020 0.316
#> GSM348190 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM348194 1 0.3596 0.7580 0.776 0.000 0.012 0.000 0.212
#> GSM348195 1 0.3612 0.7153 0.732 0.000 0.000 0.000 0.268
#> GSM348196 3 0.4150 0.2746 0.388 0.000 0.612 0.000 0.000
#> GSM537585 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM537594 2 0.0000 0.9347 0.000 1.000 0.000 0.000 0.000
#> GSM537596 3 0.0963 0.8836 0.036 0.000 0.964 0.000 0.000
#> GSM537597 2 0.0703 0.9257 0.024 0.976 0.000 0.000 0.000
#> GSM537602 1 0.3305 0.7585 0.776 0.000 0.224 0.000 0.000
#> GSM340184 3 0.0290 0.9023 0.000 0.000 0.992 0.008 0.000
#> GSM340185 2 0.0000 0.9347 0.000 1.000 0.000 0.000 0.000
#> GSM340186 2 0.0000 0.9347 0.000 1.000 0.000 0.000 0.000
#> GSM340187 4 0.1018 0.8647 0.016 0.016 0.000 0.968 0.000
#> GSM340189 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340190 2 0.0693 0.9273 0.012 0.980 0.000 0.008 0.000
#> GSM340191 3 0.0290 0.9023 0.000 0.000 0.992 0.008 0.000
#> GSM340192 2 0.2886 0.8894 0.148 0.844 0.000 0.008 0.000
#> GSM340193 1 0.4179 0.7912 0.776 0.000 0.072 0.000 0.152
#> GSM340194 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340195 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340196 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340197 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340198 2 0.2843 0.8915 0.144 0.848 0.000 0.008 0.000
#> GSM340199 3 0.0290 0.9023 0.000 0.000 0.992 0.008 0.000
#> GSM340200 2 0.0000 0.9347 0.000 1.000 0.000 0.000 0.000
#> GSM340201 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340202 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340203 4 0.0290 0.8811 0.000 0.000 0.008 0.992 0.000
#> GSM340204 5 0.6347 0.3104 0.156 0.296 0.000 0.008 0.540
#> GSM340205 3 0.1478 0.8639 0.064 0.000 0.936 0.000 0.000
#> GSM340206 3 0.1908 0.8436 0.000 0.000 0.908 0.092 0.000
#> GSM340207 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340237 2 0.0000 0.9347 0.000 1.000 0.000 0.000 0.000
#> GSM340238 3 0.2891 0.7559 0.000 0.000 0.824 0.176 0.000
#> GSM340239 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340240 5 0.0000 0.9131 0.000 0.000 0.000 0.000 1.000
#> GSM340241 2 0.0162 0.9343 0.004 0.996 0.000 0.000 0.000
#> GSM340242 3 0.3774 0.7005 0.012 0.000 0.780 0.008 0.200
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 3 0.2285 0.8427 0.064 0.000 0.900 0.028 0.000 0.008
#> GSM340359 2 0.0405 0.8818 0.004 0.988 0.000 0.000 0.008 0.000
#> GSM340361 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340362 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340363 3 0.0717 0.8815 0.016 0.000 0.976 0.000 0.000 0.008
#> GSM340364 2 0.0881 0.8694 0.012 0.972 0.000 0.000 0.008 0.008
#> GSM340365 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340366 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340367 1 0.1789 0.8702 0.924 0.000 0.044 0.000 0.032 0.000
#> GSM340368 4 0.2678 0.8150 0.004 0.020 0.000 0.860 0.000 0.116
#> GSM340369 4 0.0363 0.9061 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340370 1 0.3285 0.8131 0.844 0.008 0.000 0.012 0.040 0.096
#> GSM340371 3 0.6895 0.2419 0.024 0.000 0.412 0.020 0.312 0.232
#> GSM340372 1 0.2597 0.8035 0.824 0.000 0.000 0.000 0.176 0.000
#> GSM340373 1 0.3351 0.6650 0.712 0.000 0.000 0.000 0.288 0.000
#> GSM340375 5 0.4798 0.7272 0.044 0.116 0.000 0.012 0.748 0.080
#> GSM340376 6 0.6228 0.0945 0.372 0.212 0.000 0.012 0.000 0.404
#> GSM340378 2 0.2854 0.3765 0.000 0.792 0.000 0.000 0.000 0.208
#> GSM340243 3 0.0603 0.8819 0.016 0.000 0.980 0.000 0.000 0.004
#> GSM340244 6 0.3782 0.8441 0.000 0.412 0.000 0.000 0.000 0.588
#> GSM340246 6 0.3810 0.8422 0.000 0.428 0.000 0.000 0.000 0.572
#> GSM340247 4 0.0363 0.9061 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340248 6 0.3804 0.8432 0.000 0.424 0.000 0.000 0.000 0.576
#> GSM340249 6 0.3807 0.8313 0.004 0.368 0.000 0.000 0.000 0.628
#> GSM340250 1 0.1812 0.8653 0.912 0.000 0.080 0.000 0.008 0.000
#> GSM340251 6 0.3819 0.8339 0.004 0.372 0.000 0.000 0.000 0.624
#> GSM340252 2 0.0000 0.8825 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340253 2 0.0508 0.8793 0.004 0.984 0.000 0.000 0.012 0.000
#> GSM340254 2 0.0000 0.8825 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340256 2 0.0000 0.8825 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340258 5 0.2376 0.8581 0.012 0.096 0.000 0.000 0.884 0.008
#> GSM340259 1 0.2527 0.8101 0.832 0.000 0.000 0.000 0.168 0.000
#> GSM340260 1 0.4136 0.7553 0.760 0.000 0.172 0.000 0.036 0.032
#> GSM340261 3 0.0000 0.8871 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340262 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340263 6 0.3684 0.8356 0.000 0.372 0.000 0.000 0.000 0.628
#> GSM340264 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340265 5 0.0405 0.9484 0.008 0.000 0.000 0.000 0.988 0.004
#> GSM340266 3 0.0000 0.8871 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340267 5 0.1625 0.9081 0.012 0.000 0.000 0.000 0.928 0.060
#> GSM340268 3 0.0713 0.8759 0.000 0.000 0.972 0.028 0.000 0.000
#> GSM340269 3 0.0000 0.8871 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340270 3 0.3937 0.2353 0.424 0.000 0.572 0.000 0.000 0.004
#> GSM537574 6 0.3899 0.7938 0.008 0.364 0.000 0.000 0.000 0.628
#> GSM537580 3 0.6805 0.5169 0.024 0.000 0.532 0.060 0.152 0.232
#> GSM537581 4 0.0363 0.9061 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340272 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340273 6 0.3810 0.8422 0.000 0.428 0.000 0.000 0.000 0.572
#> GSM340275 6 0.5627 0.2462 0.004 0.132 0.000 0.380 0.000 0.484
#> GSM340276 3 0.1082 0.8704 0.040 0.000 0.956 0.000 0.000 0.004
#> GSM340277 2 0.0000 0.8825 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340278 3 0.0146 0.8867 0.000 0.000 0.996 0.000 0.000 0.004
#> GSM340279 3 0.0000 0.8871 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340282 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340284 6 0.3714 0.8076 0.004 0.340 0.000 0.000 0.000 0.656
#> GSM340285 3 0.0508 0.8834 0.012 0.000 0.984 0.000 0.000 0.004
#> GSM340286 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340287 3 0.0146 0.8867 0.000 0.000 0.996 0.000 0.000 0.004
#> GSM340288 1 0.1501 0.8579 0.924 0.000 0.000 0.000 0.076 0.000
#> GSM340289 1 0.1643 0.8681 0.924 0.000 0.068 0.000 0.008 0.000
#> GSM340290 3 0.0000 0.8871 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340291 6 0.3810 0.8422 0.000 0.428 0.000 0.000 0.000 0.572
#> GSM340293 4 0.1863 0.8495 0.000 0.000 0.104 0.896 0.000 0.000
#> GSM340294 3 0.0000 0.8871 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340296 3 0.0146 0.8867 0.000 0.000 0.996 0.000 0.000 0.004
#> GSM340297 3 0.0146 0.8867 0.000 0.000 0.996 0.000 0.000 0.004
#> GSM340298 3 0.0000 0.8871 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340299 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340301 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340303 5 0.0717 0.9410 0.016 0.000 0.000 0.000 0.976 0.008
#> GSM340304 1 0.3636 0.5333 0.676 0.000 0.320 0.000 0.000 0.004
#> GSM340306 4 0.3049 0.8151 0.104 0.000 0.048 0.844 0.000 0.004
#> GSM340307 6 0.3810 0.8422 0.000 0.428 0.000 0.000 0.000 0.572
#> GSM340310 1 0.1588 0.8670 0.924 0.000 0.072 0.000 0.004 0.000
#> GSM340314 3 0.1444 0.8474 0.000 0.000 0.928 0.072 0.000 0.000
#> GSM340315 3 0.0862 0.8790 0.004 0.000 0.972 0.016 0.000 0.008
#> GSM340317 6 0.3695 0.8369 0.000 0.376 0.000 0.000 0.000 0.624
#> GSM340318 4 0.0363 0.9061 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340319 4 0.0363 0.9061 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340320 4 0.3684 0.4435 0.000 0.000 0.372 0.628 0.000 0.000
#> GSM340321 6 0.3810 0.8422 0.000 0.428 0.000 0.000 0.000 0.572
#> GSM340322 4 0.0363 0.9061 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340324 2 0.0000 0.8825 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340328 1 0.4406 0.0765 0.516 0.000 0.464 0.012 0.000 0.008
#> GSM340330 3 0.3134 0.7789 0.024 0.000 0.808 0.000 0.000 0.168
#> GSM340332 4 0.0363 0.9061 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340333 2 0.0405 0.8818 0.004 0.988 0.000 0.000 0.008 0.000
#> GSM340336 4 0.5415 0.3073 0.004 0.128 0.000 0.564 0.000 0.304
#> GSM340337 4 0.0363 0.9061 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340338 3 0.5555 0.6089 0.116 0.000 0.620 0.000 0.032 0.232
#> GSM340339 4 0.1007 0.8917 0.000 0.000 0.044 0.956 0.000 0.000
#> GSM340340 6 0.5635 0.0894 0.004 0.128 0.000 0.428 0.000 0.440
#> GSM340341 6 0.5064 0.6075 0.000 0.216 0.000 0.152 0.000 0.632
#> GSM340343 4 0.3862 0.4058 0.000 0.000 0.388 0.608 0.000 0.004
#> GSM340344 2 0.3973 0.4715 0.012 0.684 0.000 0.000 0.296 0.008
#> GSM340346 3 0.0000 0.8871 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340347 4 0.0363 0.9061 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340348 4 0.2664 0.7739 0.000 0.000 0.184 0.816 0.000 0.000
#> GSM340349 1 0.1556 0.8639 0.920 0.000 0.080 0.000 0.000 0.000
#> GSM340350 1 0.1556 0.8639 0.920 0.000 0.080 0.000 0.000 0.000
#> GSM340351 1 0.2664 0.7955 0.816 0.000 0.000 0.000 0.184 0.000
#> GSM340354 3 0.2538 0.8193 0.016 0.000 0.860 0.000 0.000 0.124
#> GSM340356 6 0.3810 0.8422 0.000 0.428 0.000 0.000 0.000 0.572
#> GSM340357 1 0.1745 0.8659 0.924 0.000 0.020 0.000 0.056 0.000
#> GSM348183 1 0.2527 0.8099 0.832 0.000 0.000 0.000 0.168 0.000
#> GSM348191 3 0.0000 0.8871 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM348193 1 0.2118 0.8655 0.916 0.000 0.048 0.020 0.012 0.004
#> GSM537578 1 0.1501 0.8653 0.924 0.000 0.076 0.000 0.000 0.000
#> GSM348181 3 0.1644 0.8480 0.076 0.000 0.920 0.000 0.000 0.004
#> GSM348182 5 0.3563 0.4355 0.336 0.000 0.000 0.000 0.664 0.000
#> GSM348184 6 0.3684 0.8356 0.000 0.372 0.000 0.000 0.000 0.628
#> GSM348185 6 0.3684 0.8356 0.000 0.372 0.000 0.000 0.000 0.628
#> GSM348186 2 0.0000 0.8825 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM348187 3 0.1700 0.8454 0.080 0.000 0.916 0.000 0.000 0.004
#> GSM348188 5 0.3784 0.6416 0.024 0.000 0.000 0.236 0.736 0.004
#> GSM348189 3 0.6481 0.4864 0.024 0.000 0.520 0.016 0.208 0.232
#> GSM348190 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM348194 1 0.1501 0.8579 0.924 0.000 0.000 0.000 0.076 0.000
#> GSM348195 1 0.2854 0.7683 0.792 0.000 0.000 0.000 0.208 0.000
#> GSM348196 3 0.3890 0.3089 0.400 0.000 0.596 0.000 0.000 0.004
#> GSM537585 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM537594 6 0.3810 0.8422 0.000 0.428 0.000 0.000 0.000 0.572
#> GSM537596 3 0.1700 0.8444 0.080 0.000 0.916 0.000 0.000 0.004
#> GSM537597 6 0.4219 0.7535 0.012 0.360 0.000 0.008 0.000 0.620
#> GSM537602 1 0.1501 0.8653 0.924 0.000 0.076 0.000 0.000 0.000
#> GSM340184 3 0.0000 0.8871 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340185 6 0.3810 0.8422 0.000 0.428 0.000 0.000 0.000 0.572
#> GSM340186 6 0.3774 0.8438 0.000 0.408 0.000 0.000 0.000 0.592
#> GSM340187 4 0.1588 0.8661 0.004 0.000 0.000 0.924 0.000 0.072
#> GSM340189 4 0.0363 0.9061 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340190 6 0.3795 0.8283 0.004 0.364 0.000 0.000 0.000 0.632
#> GSM340191 3 0.0000 0.8871 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340192 2 0.0622 0.8770 0.008 0.980 0.000 0.000 0.012 0.000
#> GSM340193 1 0.1794 0.8695 0.924 0.000 0.036 0.000 0.040 0.000
#> GSM340194 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340195 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340196 4 0.0363 0.9061 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340197 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340198 2 0.0000 0.8825 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340199 3 0.0000 0.8871 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340200 6 0.3797 0.8438 0.000 0.420 0.000 0.000 0.000 0.580
#> GSM340201 4 0.0363 0.9061 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340202 4 0.0363 0.9061 0.000 0.000 0.012 0.988 0.000 0.000
#> GSM340203 4 0.0865 0.8962 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340204 2 0.3370 0.6151 0.012 0.772 0.000 0.000 0.212 0.004
#> GSM340205 3 0.1471 0.8558 0.064 0.000 0.932 0.000 0.000 0.004
#> GSM340206 3 0.0713 0.8759 0.000 0.000 0.972 0.028 0.000 0.000
#> GSM340207 5 0.0291 0.9506 0.004 0.000 0.000 0.000 0.992 0.004
#> GSM340237 6 0.3810 0.8422 0.000 0.428 0.000 0.000 0.000 0.572
#> GSM340238 3 0.1863 0.8182 0.000 0.000 0.896 0.104 0.000 0.000
#> GSM340239 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340240 5 0.0000 0.9551 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM340241 6 0.3838 0.8186 0.000 0.448 0.000 0.000 0.000 0.552
#> GSM340242 3 0.5864 0.5477 0.024 0.000 0.572 0.000 0.172 0.232
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
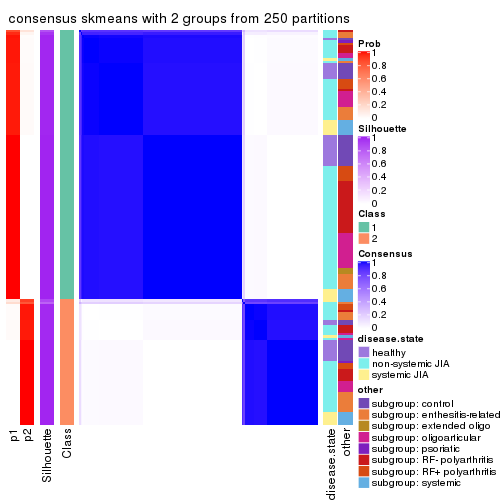
consensus_heatmap(res, k = 3)
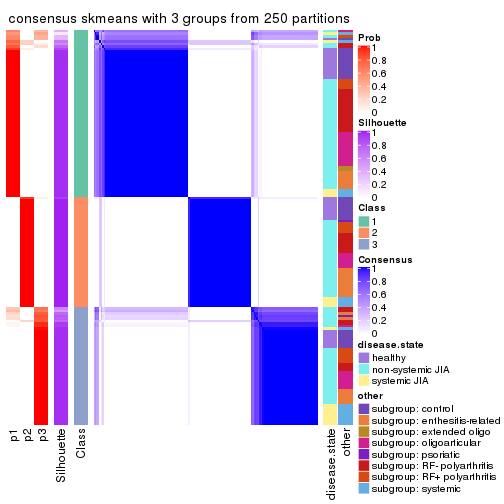
consensus_heatmap(res, k = 4)
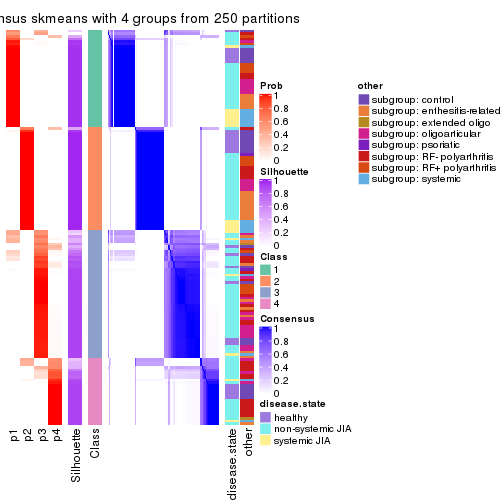
consensus_heatmap(res, k = 5)
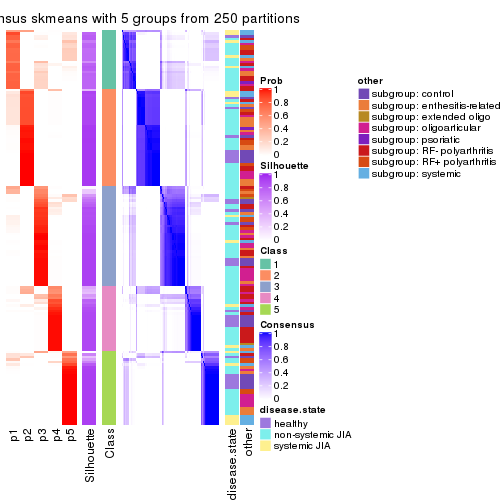
consensus_heatmap(res, k = 6)
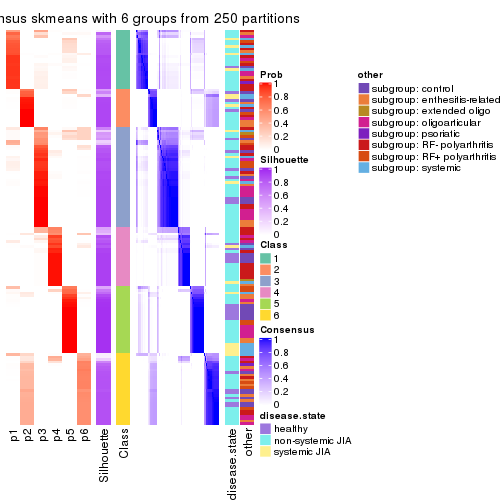
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
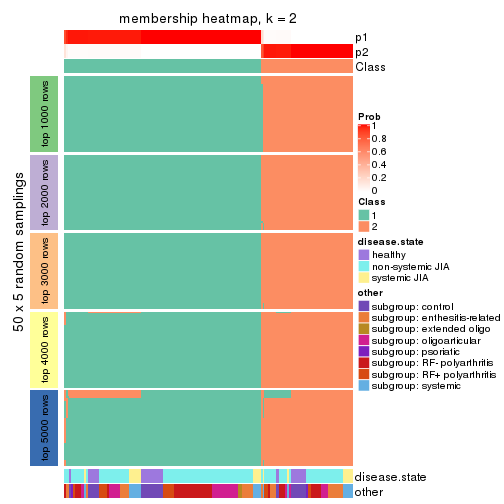
membership_heatmap(res, k = 3)
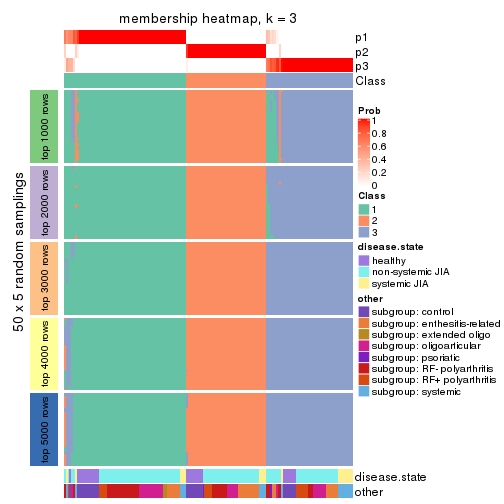
membership_heatmap(res, k = 4)
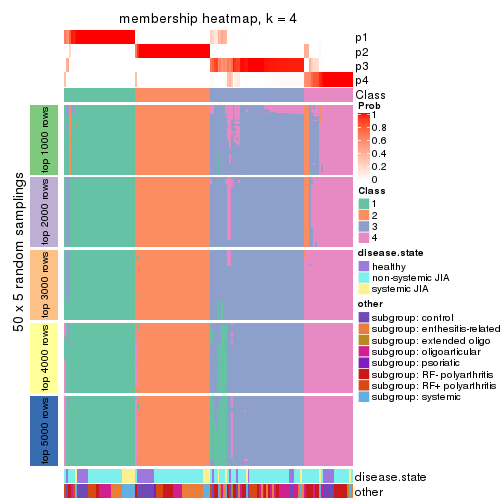
membership_heatmap(res, k = 5)
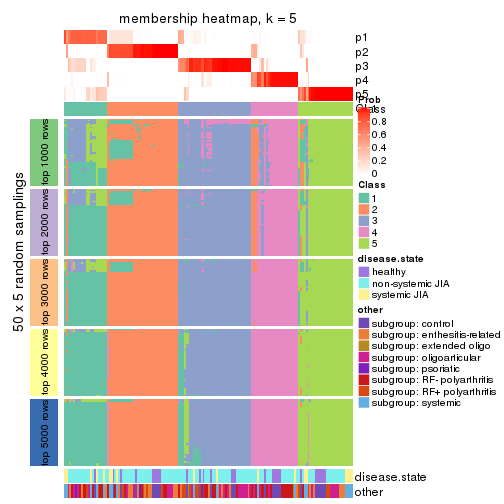
membership_heatmap(res, k = 6)
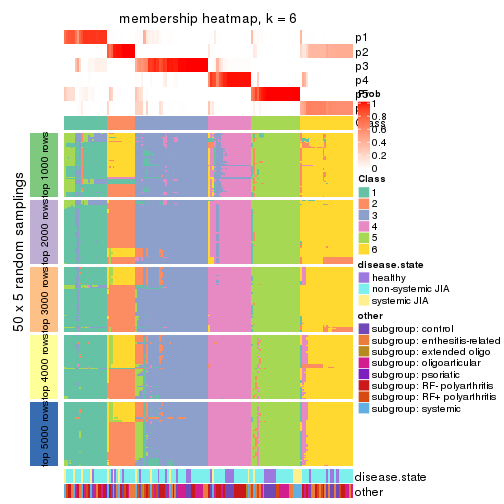
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
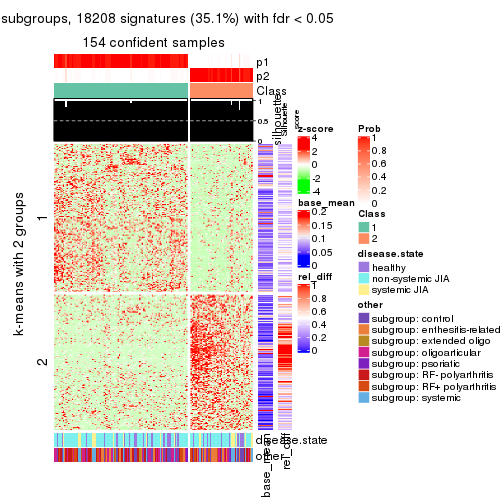
get_signatures(res, k = 3)
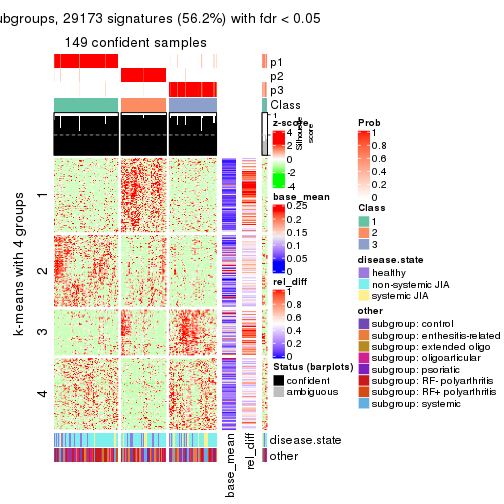
get_signatures(res, k = 4)
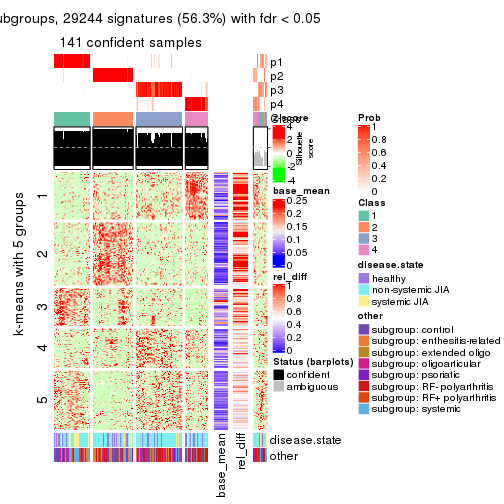
get_signatures(res, k = 5)
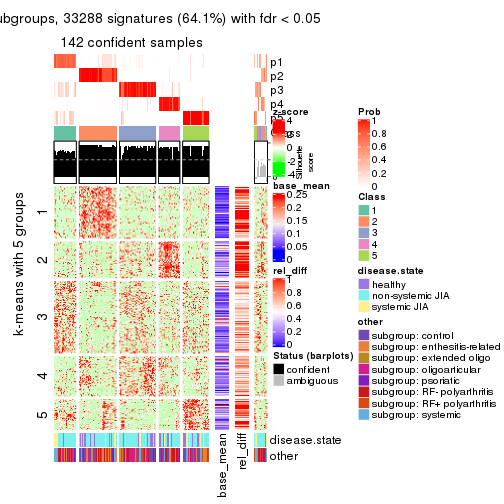
get_signatures(res, k = 6)
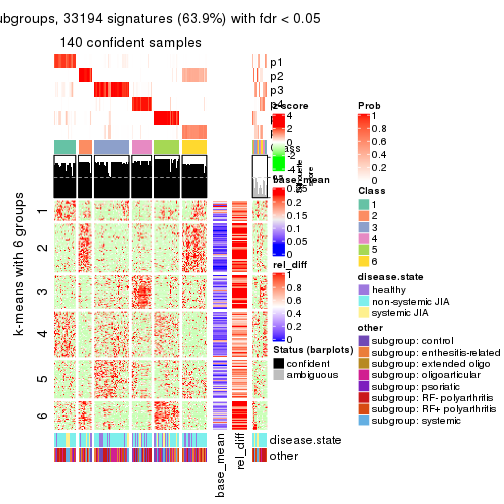
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
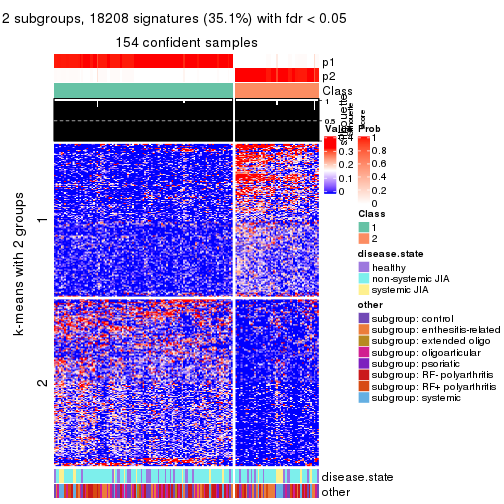
get_signatures(res, k = 3, scale_rows = FALSE)
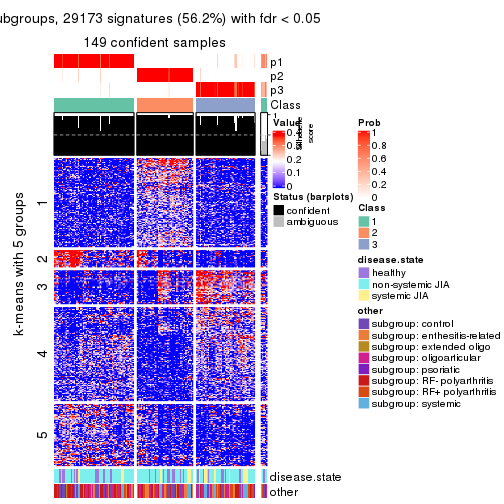
get_signatures(res, k = 4, scale_rows = FALSE)
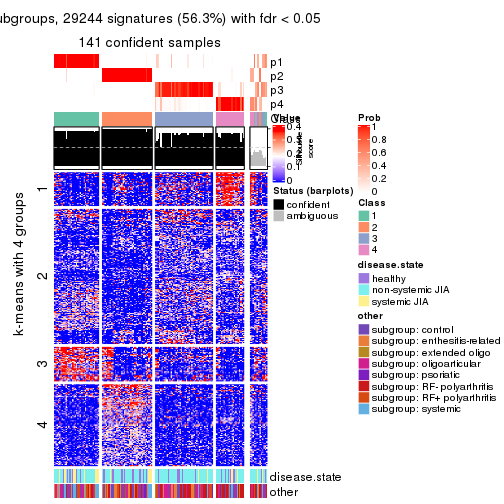
get_signatures(res, k = 5, scale_rows = FALSE)
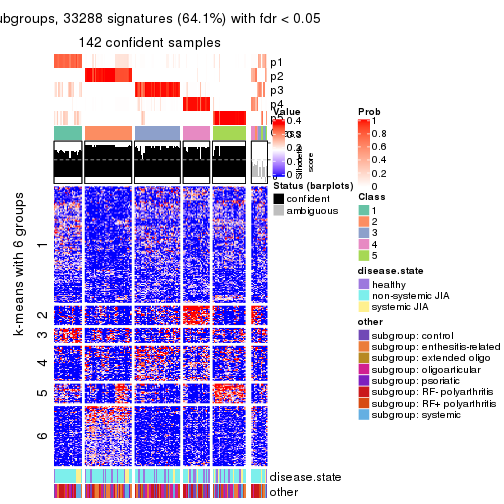
get_signatures(res, k = 6, scale_rows = FALSE)
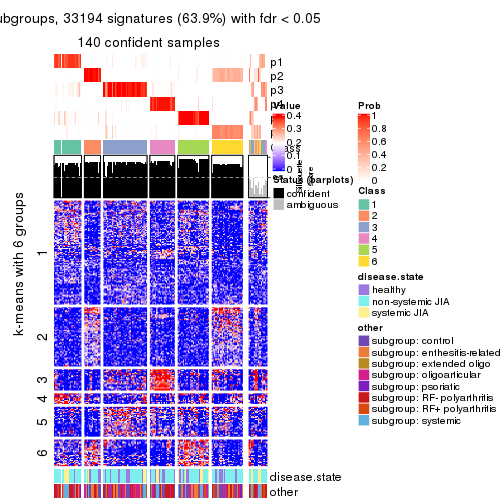
Compare the overlap of signatures from different k:
compare_signatures(res)
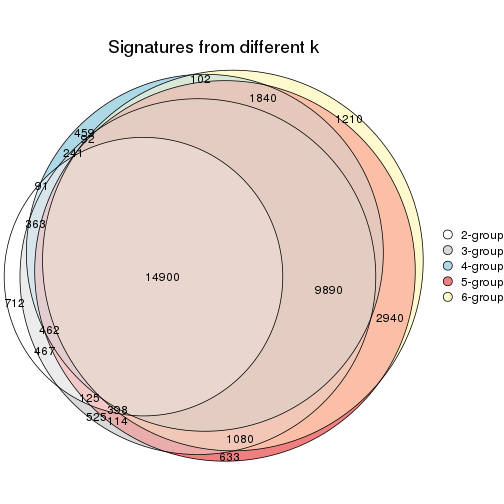
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
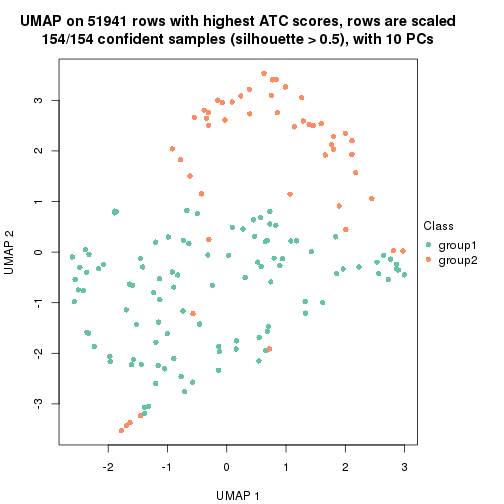
dimension_reduction(res, k = 3, method = "UMAP")
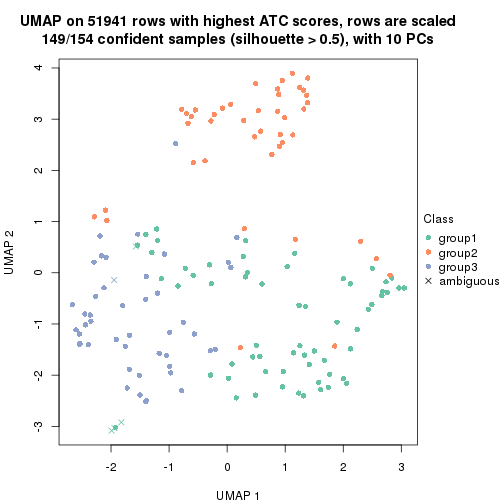
dimension_reduction(res, k = 4, method = "UMAP")
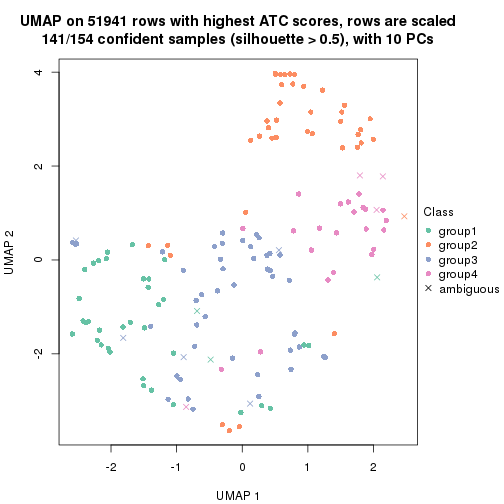
dimension_reduction(res, k = 5, method = "UMAP")
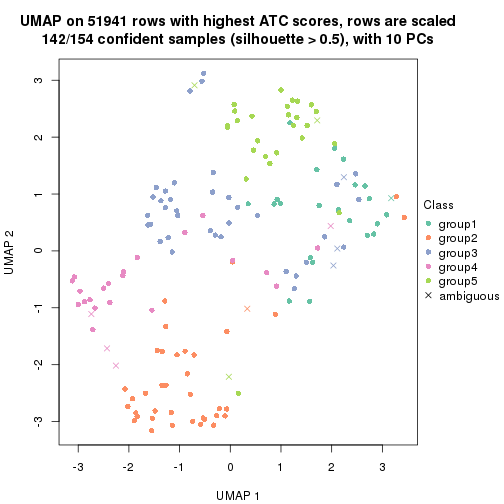
dimension_reduction(res, k = 6, method = "UMAP")
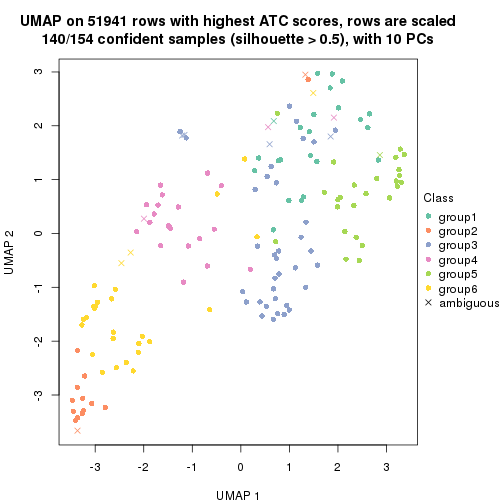
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
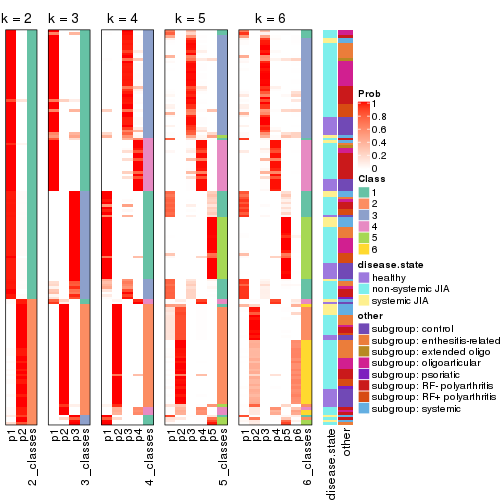
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> ATC:skmeans 154 0.708 0.5125 2
#> ATC:skmeans 149 0.283 0.2092 3
#> ATC:skmeans 141 0.137 0.0384 4
#> ATC:skmeans 142 0.387 0.1403 5
#> ATC:skmeans 140 0.263 0.2418 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["ATC", "pam"]
# you can also extract it by
# res = res_list["ATC:pam"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'ATC' method.
#> Subgroups are detected by 'pam' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
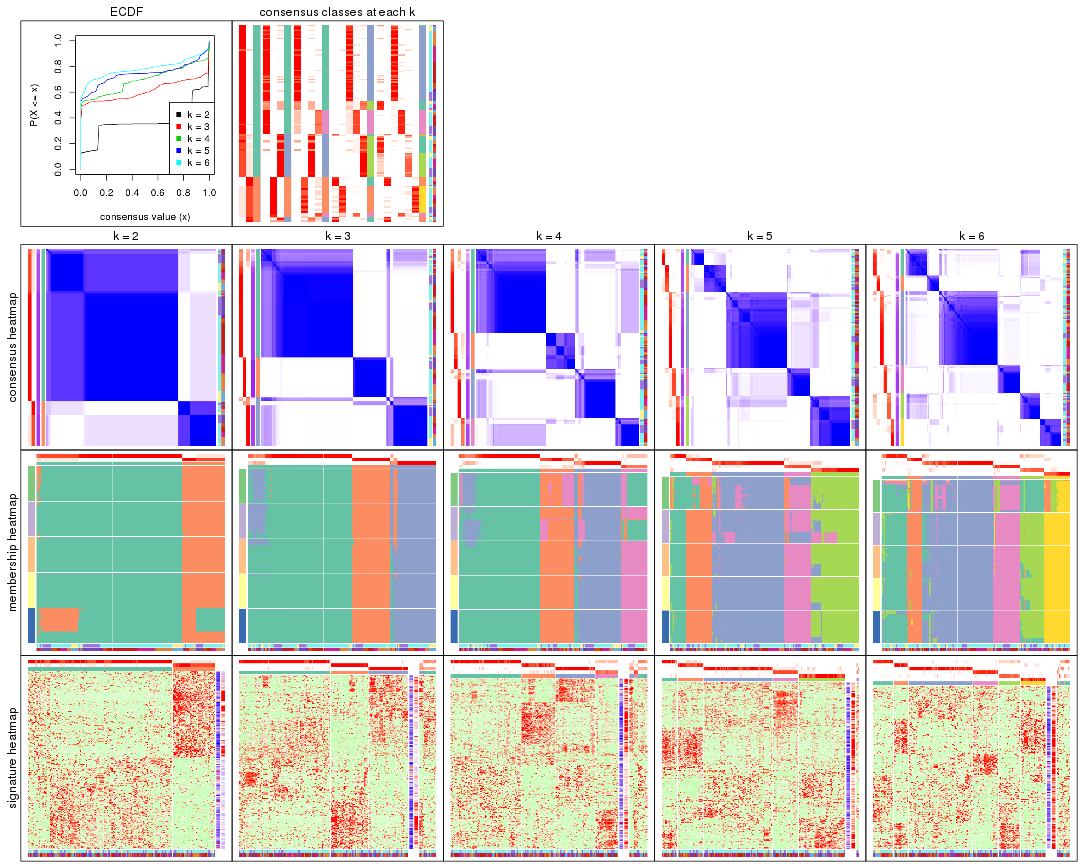
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
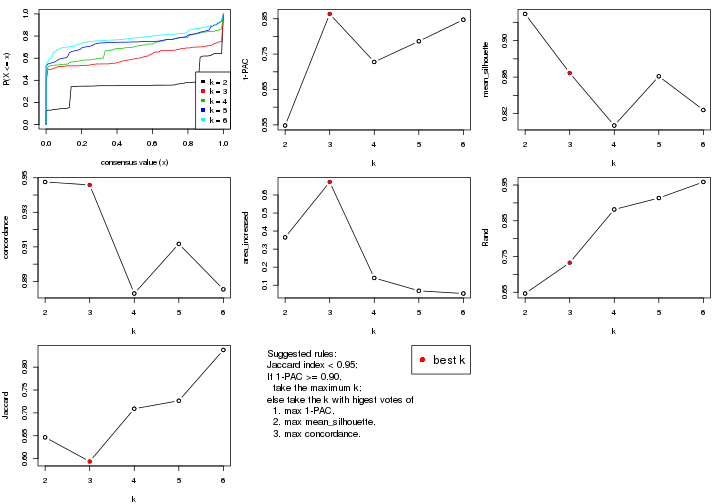
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.548 0.929 0.948 0.3650 0.646 0.646
#> 3 3 0.863 0.864 0.946 0.6719 0.733 0.594
#> 4 4 0.728 0.807 0.883 0.1403 0.882 0.709
#> 5 5 0.786 0.861 0.912 0.0692 0.914 0.727
#> 6 6 0.847 0.824 0.885 0.0541 0.958 0.838
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 1 0.0000 0.954 1.000 0.000
#> GSM340359 2 0.0000 0.895 0.000 1.000
#> GSM340361 1 0.5629 0.882 0.868 0.132
#> GSM340362 1 0.5629 0.882 0.868 0.132
#> GSM340363 1 0.0000 0.954 1.000 0.000
#> GSM340364 2 0.2423 0.881 0.040 0.960
#> GSM340365 1 0.5629 0.882 0.868 0.132
#> GSM340366 1 0.5629 0.882 0.868 0.132
#> GSM340367 1 0.0000 0.954 1.000 0.000
#> GSM340368 1 0.0000 0.954 1.000 0.000
#> GSM340369 1 0.0000 0.954 1.000 0.000
#> GSM340370 1 0.0000 0.954 1.000 0.000
#> GSM340371 1 0.0376 0.952 0.996 0.004
#> GSM340372 1 0.5629 0.882 0.868 0.132
#> GSM340373 1 0.5629 0.882 0.868 0.132
#> GSM340375 1 0.0000 0.954 1.000 0.000
#> GSM340376 1 0.0000 0.954 1.000 0.000
#> GSM340378 2 0.5629 0.950 0.132 0.868
#> GSM340243 1 0.0000 0.954 1.000 0.000
#> GSM340244 2 0.5629 0.950 0.132 0.868
#> GSM340246 2 0.5629 0.950 0.132 0.868
#> GSM340247 1 0.0000 0.954 1.000 0.000
#> GSM340248 2 0.5629 0.950 0.132 0.868
#> GSM340249 2 0.5629 0.950 0.132 0.868
#> GSM340250 1 0.0000 0.954 1.000 0.000
#> GSM340251 2 0.5629 0.950 0.132 0.868
#> GSM340252 2 0.0000 0.895 0.000 1.000
#> GSM340253 2 0.0000 0.895 0.000 1.000
#> GSM340254 2 0.0000 0.895 0.000 1.000
#> GSM340256 2 0.0000 0.895 0.000 1.000
#> GSM340258 1 0.0000 0.954 1.000 0.000
#> GSM340259 1 0.3274 0.923 0.940 0.060
#> GSM340260 1 0.0000 0.954 1.000 0.000
#> GSM340261 1 0.0000 0.954 1.000 0.000
#> GSM340262 1 0.5629 0.882 0.868 0.132
#> GSM340263 2 0.5629 0.950 0.132 0.868
#> GSM340264 1 0.5629 0.882 0.868 0.132
#> GSM340265 1 0.5629 0.882 0.868 0.132
#> GSM340266 1 0.0000 0.954 1.000 0.000
#> GSM340267 1 0.0000 0.954 1.000 0.000
#> GSM340268 1 0.0000 0.954 1.000 0.000
#> GSM340269 1 0.0000 0.954 1.000 0.000
#> GSM340270 1 0.0000 0.954 1.000 0.000
#> GSM537574 1 0.0000 0.954 1.000 0.000
#> GSM537580 1 0.0000 0.954 1.000 0.000
#> GSM537581 1 0.0000 0.954 1.000 0.000
#> GSM340272 1 0.5629 0.882 0.868 0.132
#> GSM340273 2 0.5629 0.950 0.132 0.868
#> GSM340275 2 0.5629 0.950 0.132 0.868
#> GSM340276 1 0.0000 0.954 1.000 0.000
#> GSM340277 2 0.0000 0.895 0.000 1.000
#> GSM340278 1 0.0000 0.954 1.000 0.000
#> GSM340279 1 0.0000 0.954 1.000 0.000
#> GSM340282 1 0.5629 0.882 0.868 0.132
#> GSM340284 1 0.8443 0.564 0.728 0.272
#> GSM340285 1 0.0000 0.954 1.000 0.000
#> GSM340286 1 0.5629 0.882 0.868 0.132
#> GSM340287 1 0.0000 0.954 1.000 0.000
#> GSM340288 1 0.0376 0.952 0.996 0.004
#> GSM340289 1 0.0000 0.954 1.000 0.000
#> GSM340290 1 0.0000 0.954 1.000 0.000
#> GSM340291 2 0.5629 0.950 0.132 0.868
#> GSM340293 1 0.0000 0.954 1.000 0.000
#> GSM340294 1 0.0000 0.954 1.000 0.000
#> GSM340296 1 0.0000 0.954 1.000 0.000
#> GSM340297 1 0.0000 0.954 1.000 0.000
#> GSM340298 1 0.0000 0.954 1.000 0.000
#> GSM340299 1 0.5629 0.882 0.868 0.132
#> GSM340301 1 0.5629 0.882 0.868 0.132
#> GSM340303 1 0.5629 0.882 0.868 0.132
#> GSM340304 1 0.0000 0.954 1.000 0.000
#> GSM340306 1 0.0000 0.954 1.000 0.000
#> GSM340307 2 0.5629 0.950 0.132 0.868
#> GSM340310 1 0.0000 0.954 1.000 0.000
#> GSM340314 1 0.0000 0.954 1.000 0.000
#> GSM340315 1 0.0000 0.954 1.000 0.000
#> GSM340317 2 0.5629 0.950 0.132 0.868
#> GSM340318 1 0.0000 0.954 1.000 0.000
#> GSM340319 1 0.0000 0.954 1.000 0.000
#> GSM340320 1 0.0000 0.954 1.000 0.000
#> GSM340321 2 0.5629 0.950 0.132 0.868
#> GSM340322 1 0.0000 0.954 1.000 0.000
#> GSM340324 2 0.0376 0.896 0.004 0.996
#> GSM340328 1 0.0000 0.954 1.000 0.000
#> GSM340330 1 0.0000 0.954 1.000 0.000
#> GSM340332 1 0.0000 0.954 1.000 0.000
#> GSM340333 2 0.0000 0.895 0.000 1.000
#> GSM340336 2 0.5629 0.950 0.132 0.868
#> GSM340337 1 0.0000 0.954 1.000 0.000
#> GSM340338 1 0.0000 0.954 1.000 0.000
#> GSM340339 1 0.0000 0.954 1.000 0.000
#> GSM340340 1 0.6343 0.774 0.840 0.160
#> GSM340341 1 0.0000 0.954 1.000 0.000
#> GSM340343 1 0.0000 0.954 1.000 0.000
#> GSM340344 1 0.7528 0.804 0.784 0.216
#> GSM340346 1 0.0000 0.954 1.000 0.000
#> GSM340347 1 0.0000 0.954 1.000 0.000
#> GSM340348 1 0.0000 0.954 1.000 0.000
#> GSM340349 1 0.0000 0.954 1.000 0.000
#> GSM340350 1 0.0000 0.954 1.000 0.000
#> GSM340351 1 0.5629 0.882 0.868 0.132
#> GSM340354 1 0.0000 0.954 1.000 0.000
#> GSM340356 2 0.5629 0.950 0.132 0.868
#> GSM340357 1 0.0000 0.954 1.000 0.000
#> GSM348183 1 0.5629 0.882 0.868 0.132
#> GSM348191 1 0.0000 0.954 1.000 0.000
#> GSM348193 1 0.0000 0.954 1.000 0.000
#> GSM537578 1 0.0000 0.954 1.000 0.000
#> GSM348181 1 0.0000 0.954 1.000 0.000
#> GSM348182 1 0.5629 0.882 0.868 0.132
#> GSM348184 2 0.5629 0.950 0.132 0.868
#> GSM348185 2 0.5629 0.950 0.132 0.868
#> GSM348186 2 0.0000 0.895 0.000 1.000
#> GSM348187 1 0.0000 0.954 1.000 0.000
#> GSM348188 1 0.5629 0.882 0.868 0.132
#> GSM348189 1 0.0376 0.952 0.996 0.004
#> GSM348190 1 0.5629 0.882 0.868 0.132
#> GSM348194 1 0.3584 0.919 0.932 0.068
#> GSM348195 1 0.5629 0.882 0.868 0.132
#> GSM348196 1 0.0000 0.954 1.000 0.000
#> GSM537585 1 0.5629 0.882 0.868 0.132
#> GSM537594 2 0.5629 0.950 0.132 0.868
#> GSM537596 1 0.0000 0.954 1.000 0.000
#> GSM537597 1 0.0000 0.954 1.000 0.000
#> GSM537602 1 0.0000 0.954 1.000 0.000
#> GSM340184 1 0.0000 0.954 1.000 0.000
#> GSM340185 2 0.5629 0.950 0.132 0.868
#> GSM340186 2 0.5629 0.950 0.132 0.868
#> GSM340187 1 0.0000 0.954 1.000 0.000
#> GSM340189 1 0.0000 0.954 1.000 0.000
#> GSM340190 2 0.5629 0.950 0.132 0.868
#> GSM340191 1 0.0000 0.954 1.000 0.000
#> GSM340192 1 0.8081 0.762 0.752 0.248
#> GSM340193 1 0.0000 0.954 1.000 0.000
#> GSM340194 1 0.5629 0.882 0.868 0.132
#> GSM340195 1 0.5629 0.882 0.868 0.132
#> GSM340196 1 0.0000 0.954 1.000 0.000
#> GSM340197 1 0.5629 0.882 0.868 0.132
#> GSM340198 2 0.0000 0.895 0.000 1.000
#> GSM340199 1 0.0000 0.954 1.000 0.000
#> GSM340200 2 0.5629 0.950 0.132 0.868
#> GSM340201 1 0.0000 0.954 1.000 0.000
#> GSM340202 1 0.0000 0.954 1.000 0.000
#> GSM340203 1 0.0000 0.954 1.000 0.000
#> GSM340204 1 0.5629 0.882 0.868 0.132
#> GSM340205 1 0.0000 0.954 1.000 0.000
#> GSM340206 1 0.0000 0.954 1.000 0.000
#> GSM340207 1 0.5629 0.882 0.868 0.132
#> GSM340237 2 0.5629 0.950 0.132 0.868
#> GSM340238 1 0.0000 0.954 1.000 0.000
#> GSM340239 1 0.5629 0.882 0.868 0.132
#> GSM340240 1 0.5629 0.882 0.868 0.132
#> GSM340241 2 0.5629 0.950 0.132 0.868
#> GSM340242 1 0.0000 0.954 1.000 0.000
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340359 3 0.6308 0.000767 0.000 0.492 0.508
#> GSM340361 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340362 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340363 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340364 3 0.0237 0.909518 0.000 0.004 0.996
#> GSM340365 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340366 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340367 1 0.6168 0.389041 0.588 0.000 0.412
#> GSM340368 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340369 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340370 1 0.6140 0.407881 0.596 0.000 0.404
#> GSM340371 1 0.1289 0.910169 0.968 0.000 0.032
#> GSM340372 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340373 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340375 1 0.6286 0.247790 0.536 0.000 0.464
#> GSM340376 1 0.5098 0.684454 0.752 0.000 0.248
#> GSM340378 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340243 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340244 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340246 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340247 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340248 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340249 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340250 1 0.2261 0.882546 0.932 0.000 0.068
#> GSM340251 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340252 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340253 3 0.6252 0.153921 0.000 0.444 0.556
#> GSM340254 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340256 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340258 3 0.6215 0.113068 0.428 0.000 0.572
#> GSM340259 3 0.5138 0.585011 0.252 0.000 0.748
#> GSM340260 1 0.3038 0.849937 0.896 0.000 0.104
#> GSM340261 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340262 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340263 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340264 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340265 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340266 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340267 1 0.5948 0.500768 0.640 0.000 0.360
#> GSM340268 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340269 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340270 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM537574 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM537580 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM537581 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340272 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340273 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340275 2 0.0237 0.992374 0.004 0.996 0.000
#> GSM340276 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340277 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340278 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340279 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340282 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340284 1 0.2066 0.885160 0.940 0.060 0.000
#> GSM340285 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340286 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340287 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340288 1 0.6267 0.283316 0.548 0.000 0.452
#> GSM340289 1 0.6140 0.407881 0.596 0.000 0.404
#> GSM340290 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340291 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340293 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340294 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340296 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340297 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340298 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340299 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340301 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340303 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340304 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340306 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340307 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340310 1 0.5968 0.492387 0.636 0.000 0.364
#> GSM340314 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340315 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340317 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340318 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340319 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340320 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340321 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340322 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340324 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340328 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340330 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340332 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340333 3 0.6309 -0.013312 0.000 0.496 0.504
#> GSM340336 2 0.1964 0.930055 0.056 0.944 0.000
#> GSM340337 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340338 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340339 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340340 1 0.1289 0.906683 0.968 0.032 0.000
#> GSM340341 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340343 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340344 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340346 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340347 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340348 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340349 1 0.2261 0.882648 0.932 0.000 0.068
#> GSM340350 1 0.4346 0.765993 0.816 0.000 0.184
#> GSM340351 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340354 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340356 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340357 1 0.6204 0.359367 0.576 0.000 0.424
#> GSM348183 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM348191 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM348193 1 0.6079 0.443011 0.612 0.000 0.388
#> GSM537578 1 0.5363 0.643580 0.724 0.000 0.276
#> GSM348181 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM348182 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM348184 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM348185 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM348186 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM348187 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM348188 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM348189 1 0.0424 0.928311 0.992 0.000 0.008
#> GSM348190 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM348194 3 0.6295 -0.053665 0.472 0.000 0.528
#> GSM348195 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM348196 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM537585 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM537594 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM537596 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM537597 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM537602 1 0.4796 0.721682 0.780 0.000 0.220
#> GSM340184 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340185 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340186 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340187 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340189 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340190 2 0.1643 0.945558 0.044 0.956 0.000
#> GSM340191 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340192 3 0.3038 0.810045 0.000 0.104 0.896
#> GSM340193 1 0.6192 0.369449 0.580 0.000 0.420
#> GSM340194 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340195 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340196 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340197 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340198 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340199 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340200 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340201 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340202 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340203 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340204 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340205 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340206 1 0.0237 0.930537 0.996 0.000 0.004
#> GSM340207 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340237 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340238 1 0.0000 0.929197 1.000 0.000 0.000
#> GSM340239 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340240 3 0.0000 0.912821 0.000 0.000 1.000
#> GSM340241 2 0.0000 0.995703 0.000 1.000 0.000
#> GSM340242 1 0.0237 0.930537 0.996 0.000 0.004
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340359 3 0.7013 0.2415 0.000 0.356 0.516 0.128
#> GSM340361 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340362 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340363 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340364 3 0.4164 0.6651 0.000 0.000 0.736 0.264
#> GSM340365 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340366 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340367 1 0.4817 0.4317 0.612 0.000 0.388 0.000
#> GSM340368 4 0.2760 0.6997 0.128 0.000 0.000 0.872
#> GSM340369 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340370 1 0.4817 0.4317 0.612 0.000 0.388 0.000
#> GSM340371 1 0.0921 0.8576 0.972 0.000 0.028 0.000
#> GSM340372 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340373 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340375 1 0.4972 0.2600 0.544 0.000 0.456 0.000
#> GSM340376 1 0.3219 0.7337 0.836 0.000 0.164 0.000
#> GSM340378 2 0.3751 0.8626 0.004 0.800 0.000 0.196
#> GSM340243 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340244 2 0.0188 0.8942 0.000 0.996 0.000 0.004
#> GSM340246 2 0.0000 0.8939 0.000 1.000 0.000 0.000
#> GSM340247 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340248 2 0.3569 0.8645 0.000 0.804 0.000 0.196
#> GSM340249 2 0.3610 0.8623 0.000 0.800 0.000 0.200
#> GSM340250 1 0.0469 0.8747 0.988 0.000 0.012 0.000
#> GSM340251 2 0.3610 0.8623 0.000 0.800 0.000 0.200
#> GSM340252 2 0.3356 0.8572 0.000 0.824 0.000 0.176
#> GSM340253 3 0.6845 0.3556 0.000 0.308 0.564 0.128
#> GSM340254 2 0.2760 0.8499 0.000 0.872 0.000 0.128
#> GSM340256 2 0.2760 0.8499 0.000 0.872 0.000 0.128
#> GSM340258 3 0.4941 0.0842 0.436 0.000 0.564 0.000
#> GSM340259 3 0.4103 0.5736 0.256 0.000 0.744 0.000
#> GSM340260 1 0.1302 0.8479 0.956 0.000 0.044 0.000
#> GSM340261 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340262 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340263 2 0.3751 0.8626 0.004 0.800 0.000 0.196
#> GSM340264 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340265 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340266 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340267 1 0.4250 0.6146 0.724 0.000 0.276 0.000
#> GSM340268 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340269 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340270 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM537574 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM537580 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM537581 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340272 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340273 2 0.2216 0.8900 0.000 0.908 0.000 0.092
#> GSM340275 4 0.2760 0.4810 0.000 0.128 0.000 0.872
#> GSM340276 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340277 2 0.2760 0.8499 0.000 0.872 0.000 0.128
#> GSM340278 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340279 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340282 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340284 1 0.3610 0.6631 0.800 0.000 0.000 0.200
#> GSM340285 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340286 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340287 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340288 1 0.4925 0.3364 0.572 0.000 0.428 0.000
#> GSM340289 1 0.4804 0.4408 0.616 0.000 0.384 0.000
#> GSM340290 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340291 2 0.0000 0.8939 0.000 1.000 0.000 0.000
#> GSM340293 1 0.3266 0.6307 0.832 0.000 0.000 0.168
#> GSM340294 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340296 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340297 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340298 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340299 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340301 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340303 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340304 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340306 4 0.4730 0.8408 0.364 0.000 0.000 0.636
#> GSM340307 2 0.3751 0.8626 0.004 0.800 0.000 0.196
#> GSM340310 1 0.4331 0.5998 0.712 0.000 0.288 0.000
#> GSM340314 1 0.0188 0.8793 0.996 0.000 0.000 0.004
#> GSM340315 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340317 2 0.3569 0.8645 0.000 0.804 0.000 0.196
#> GSM340318 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340319 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340320 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340321 2 0.0000 0.8939 0.000 1.000 0.000 0.000
#> GSM340322 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340324 2 0.2760 0.8499 0.000 0.872 0.000 0.128
#> GSM340328 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340330 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340332 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340333 3 0.7024 0.2308 0.000 0.360 0.512 0.128
#> GSM340336 4 0.2976 0.4977 0.008 0.120 0.000 0.872
#> GSM340337 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340338 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340339 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340340 4 0.2760 0.6997 0.128 0.000 0.000 0.872
#> GSM340341 1 0.3569 0.6670 0.804 0.000 0.000 0.196
#> GSM340343 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340344 3 0.2760 0.7971 0.000 0.000 0.872 0.128
#> GSM340346 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340347 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340348 1 0.0188 0.8797 0.996 0.000 0.000 0.004
#> GSM340349 1 0.0707 0.8685 0.980 0.000 0.020 0.000
#> GSM340350 1 0.2345 0.7972 0.900 0.000 0.100 0.000
#> GSM340351 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340354 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340356 2 0.1474 0.8947 0.000 0.948 0.000 0.052
#> GSM340357 1 0.4643 0.5143 0.656 0.000 0.344 0.000
#> GSM348183 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM348191 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM348193 1 0.4761 0.4662 0.628 0.000 0.372 0.000
#> GSM537578 1 0.3444 0.7124 0.816 0.000 0.184 0.000
#> GSM348181 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM348182 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM348184 2 0.3569 0.8645 0.000 0.804 0.000 0.196
#> GSM348185 2 0.3569 0.8645 0.000 0.804 0.000 0.196
#> GSM348186 2 0.2760 0.8499 0.000 0.872 0.000 0.128
#> GSM348187 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM348188 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM348189 1 0.0188 0.8798 0.996 0.000 0.004 0.000
#> GSM348190 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM348194 3 0.5000 -0.1477 0.500 0.000 0.500 0.000
#> GSM348195 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM348196 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM537585 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM537594 2 0.0000 0.8939 0.000 1.000 0.000 0.000
#> GSM537596 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM537597 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM537602 1 0.2921 0.7573 0.860 0.000 0.140 0.000
#> GSM340184 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340185 2 0.0000 0.8939 0.000 1.000 0.000 0.000
#> GSM340186 2 0.1940 0.8926 0.000 0.924 0.000 0.076
#> GSM340187 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340189 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340190 4 0.4222 0.2044 0.000 0.272 0.000 0.728
#> GSM340191 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340192 3 0.4543 0.5865 0.000 0.000 0.676 0.324
#> GSM340193 1 0.4804 0.4402 0.616 0.000 0.384 0.000
#> GSM340194 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340195 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340196 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340197 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340198 2 0.2760 0.8499 0.000 0.872 0.000 0.128
#> GSM340199 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340200 2 0.3751 0.8626 0.004 0.800 0.000 0.196
#> GSM340201 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340202 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340203 4 0.4543 0.8966 0.324 0.000 0.000 0.676
#> GSM340204 3 0.0817 0.8810 0.000 0.000 0.976 0.024
#> GSM340205 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340206 1 0.0000 0.8829 1.000 0.000 0.000 0.000
#> GSM340207 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340237 2 0.0000 0.8939 0.000 1.000 0.000 0.000
#> GSM340238 1 0.0188 0.8793 0.996 0.000 0.000 0.004
#> GSM340239 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340240 3 0.0000 0.8984 0.000 0.000 1.000 0.000
#> GSM340241 2 0.0000 0.8939 0.000 1.000 0.000 0.000
#> GSM340242 1 0.0000 0.8829 1.000 0.000 0.000 0.000
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 3 0.1671 0.888 0.000 0.000 0.924 0.076 0.000
#> GSM340359 1 0.0000 0.898 1.000 0.000 0.000 0.000 0.000
#> GSM340361 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340362 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340363 3 0.0963 0.918 0.000 0.000 0.964 0.036 0.000
#> GSM340364 1 0.5889 0.655 0.668 0.116 0.000 0.036 0.180
#> GSM340365 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340366 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340367 5 0.5074 0.613 0.000 0.000 0.268 0.072 0.660
#> GSM340368 4 0.2144 0.859 0.000 0.020 0.068 0.912 0.000
#> GSM340369 4 0.2329 0.921 0.000 0.000 0.124 0.876 0.000
#> GSM340370 5 0.5051 0.618 0.000 0.000 0.264 0.072 0.664
#> GSM340371 3 0.2179 0.848 0.000 0.000 0.896 0.004 0.100
#> GSM340372 5 0.1608 0.868 0.000 0.000 0.000 0.072 0.928
#> GSM340373 5 0.1608 0.868 0.000 0.000 0.000 0.072 0.928
#> GSM340375 5 0.4010 0.756 0.000 0.000 0.136 0.072 0.792
#> GSM340376 3 0.2189 0.880 0.000 0.000 0.904 0.084 0.012
#> GSM340378 2 0.1270 0.878 0.000 0.948 0.000 0.052 0.000
#> GSM340243 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340244 2 0.2561 0.879 0.144 0.856 0.000 0.000 0.000
#> GSM340246 2 0.2605 0.877 0.148 0.852 0.000 0.000 0.000
#> GSM340247 4 0.2179 0.913 0.000 0.000 0.112 0.888 0.000
#> GSM340248 2 0.0000 0.893 0.000 1.000 0.000 0.000 0.000
#> GSM340249 2 0.1043 0.884 0.000 0.960 0.000 0.040 0.000
#> GSM340250 3 0.1608 0.895 0.000 0.000 0.928 0.072 0.000
#> GSM340251 2 0.0880 0.887 0.000 0.968 0.000 0.032 0.000
#> GSM340252 1 0.1608 0.856 0.928 0.072 0.000 0.000 0.000
#> GSM340253 1 0.0000 0.898 1.000 0.000 0.000 0.000 0.000
#> GSM340254 1 0.0000 0.898 1.000 0.000 0.000 0.000 0.000
#> GSM340256 1 0.0290 0.894 0.992 0.008 0.000 0.000 0.000
#> GSM340258 5 0.3910 0.679 0.000 0.000 0.196 0.032 0.772
#> GSM340259 5 0.1768 0.867 0.000 0.000 0.004 0.072 0.924
#> GSM340260 3 0.1544 0.898 0.000 0.000 0.932 0.068 0.000
#> GSM340261 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340262 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340263 2 0.1270 0.878 0.000 0.948 0.000 0.052 0.000
#> GSM340264 5 0.1121 0.875 0.000 0.000 0.000 0.044 0.956
#> GSM340265 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340266 3 0.0404 0.932 0.000 0.000 0.988 0.012 0.000
#> GSM340267 3 0.4054 0.760 0.000 0.000 0.788 0.072 0.140
#> GSM340268 3 0.0404 0.931 0.000 0.000 0.988 0.012 0.000
#> GSM340269 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340270 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM537574 3 0.0162 0.935 0.000 0.000 0.996 0.004 0.000
#> GSM537580 3 0.2561 0.822 0.000 0.000 0.856 0.144 0.000
#> GSM537581 4 0.2377 0.917 0.000 0.000 0.128 0.872 0.000
#> GSM340272 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340273 2 0.1851 0.896 0.088 0.912 0.000 0.000 0.000
#> GSM340275 4 0.3242 0.663 0.000 0.216 0.000 0.784 0.000
#> GSM340276 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340277 1 0.0000 0.898 1.000 0.000 0.000 0.000 0.000
#> GSM340278 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340279 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340282 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340284 3 0.3888 0.765 0.000 0.148 0.796 0.056 0.000
#> GSM340285 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340286 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340287 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340288 5 0.4514 0.700 0.000 0.000 0.188 0.072 0.740
#> GSM340289 5 0.5237 0.571 0.000 0.000 0.300 0.072 0.628
#> GSM340290 3 0.0290 0.933 0.000 0.000 0.992 0.008 0.000
#> GSM340291 2 0.3039 0.862 0.192 0.808 0.000 0.000 0.000
#> GSM340293 3 0.4060 0.400 0.000 0.000 0.640 0.360 0.000
#> GSM340294 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340296 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340297 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340298 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340299 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340301 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340303 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340304 3 0.1608 0.895 0.000 0.000 0.928 0.072 0.000
#> GSM340306 4 0.3109 0.823 0.000 0.000 0.200 0.800 0.000
#> GSM340307 2 0.0404 0.891 0.000 0.988 0.000 0.012 0.000
#> GSM340310 3 0.4645 0.649 0.000 0.000 0.724 0.072 0.204
#> GSM340314 3 0.2605 0.817 0.000 0.000 0.852 0.148 0.000
#> GSM340315 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340317 2 0.0000 0.893 0.000 1.000 0.000 0.000 0.000
#> GSM340318 4 0.2329 0.921 0.000 0.000 0.124 0.876 0.000
#> GSM340319 4 0.2329 0.921 0.000 0.000 0.124 0.876 0.000
#> GSM340320 3 0.2424 0.835 0.000 0.000 0.868 0.132 0.000
#> GSM340321 2 0.3109 0.858 0.200 0.800 0.000 0.000 0.000
#> GSM340322 4 0.2329 0.921 0.000 0.000 0.124 0.876 0.000
#> GSM340324 1 0.0290 0.894 0.992 0.008 0.000 0.000 0.000
#> GSM340328 3 0.0162 0.935 0.000 0.000 0.996 0.004 0.000
#> GSM340330 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340332 4 0.2329 0.921 0.000 0.000 0.124 0.876 0.000
#> GSM340333 1 0.0000 0.898 1.000 0.000 0.000 0.000 0.000
#> GSM340336 4 0.3366 0.670 0.000 0.212 0.004 0.784 0.000
#> GSM340337 4 0.2329 0.921 0.000 0.000 0.124 0.876 0.000
#> GSM340338 3 0.0162 0.935 0.000 0.000 0.996 0.004 0.000
#> GSM340339 4 0.2329 0.921 0.000 0.000 0.124 0.876 0.000
#> GSM340340 4 0.4078 0.754 0.000 0.148 0.068 0.784 0.000
#> GSM340341 3 0.3821 0.769 0.000 0.148 0.800 0.052 0.000
#> GSM340343 3 0.2329 0.843 0.000 0.000 0.876 0.124 0.000
#> GSM340344 1 0.3109 0.721 0.800 0.000 0.000 0.000 0.200
#> GSM340346 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340347 4 0.2329 0.921 0.000 0.000 0.124 0.876 0.000
#> GSM340348 3 0.1043 0.916 0.000 0.000 0.960 0.040 0.000
#> GSM340349 3 0.1608 0.895 0.000 0.000 0.928 0.072 0.000
#> GSM340350 3 0.1768 0.892 0.000 0.000 0.924 0.072 0.004
#> GSM340351 5 0.1608 0.868 0.000 0.000 0.000 0.072 0.928
#> GSM340354 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340356 2 0.2230 0.890 0.116 0.884 0.000 0.000 0.000
#> GSM340357 3 0.4409 0.696 0.000 0.000 0.752 0.072 0.176
#> GSM348183 5 0.1894 0.865 0.000 0.000 0.008 0.072 0.920
#> GSM348191 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM348193 5 0.5096 0.608 0.000 0.000 0.272 0.072 0.656
#> GSM537578 3 0.1894 0.890 0.000 0.000 0.920 0.072 0.008
#> GSM348181 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM348182 5 0.1608 0.868 0.000 0.000 0.000 0.072 0.928
#> GSM348184 2 0.0000 0.893 0.000 1.000 0.000 0.000 0.000
#> GSM348185 2 0.0609 0.890 0.000 0.980 0.000 0.020 0.000
#> GSM348186 1 0.0000 0.898 1.000 0.000 0.000 0.000 0.000
#> GSM348187 3 0.0404 0.931 0.000 0.000 0.988 0.012 0.000
#> GSM348188 5 0.1341 0.873 0.000 0.000 0.000 0.056 0.944
#> GSM348189 3 0.0703 0.924 0.000 0.000 0.976 0.000 0.024
#> GSM348190 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM348194 5 0.5028 0.622 0.000 0.000 0.260 0.072 0.668
#> GSM348195 5 0.1608 0.868 0.000 0.000 0.000 0.072 0.928
#> GSM348196 3 0.0880 0.921 0.000 0.000 0.968 0.032 0.000
#> GSM537585 5 0.1478 0.870 0.000 0.000 0.000 0.064 0.936
#> GSM537594 2 0.3109 0.858 0.200 0.800 0.000 0.000 0.000
#> GSM537596 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM537597 3 0.1270 0.912 0.000 0.000 0.948 0.052 0.000
#> GSM537602 3 0.1608 0.895 0.000 0.000 0.928 0.072 0.000
#> GSM340184 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340185 2 0.3109 0.858 0.200 0.800 0.000 0.000 0.000
#> GSM340186 2 0.1965 0.895 0.096 0.904 0.000 0.000 0.000
#> GSM340187 4 0.2179 0.913 0.000 0.000 0.112 0.888 0.000
#> GSM340189 4 0.2329 0.921 0.000 0.000 0.124 0.876 0.000
#> GSM340190 4 0.4227 0.272 0.000 0.420 0.000 0.580 0.000
#> GSM340191 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340192 1 0.7024 0.517 0.524 0.148 0.000 0.052 0.276
#> GSM340193 5 0.5159 0.593 0.000 0.000 0.284 0.072 0.644
#> GSM340194 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340195 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340196 4 0.2329 0.921 0.000 0.000 0.124 0.876 0.000
#> GSM340197 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340198 1 0.0000 0.898 1.000 0.000 0.000 0.000 0.000
#> GSM340199 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340200 2 0.1043 0.884 0.000 0.960 0.000 0.040 0.000
#> GSM340201 4 0.2329 0.921 0.000 0.000 0.124 0.876 0.000
#> GSM340202 4 0.2329 0.921 0.000 0.000 0.124 0.876 0.000
#> GSM340203 4 0.2329 0.921 0.000 0.000 0.124 0.876 0.000
#> GSM340204 5 0.1117 0.862 0.000 0.016 0.000 0.020 0.964
#> GSM340205 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
#> GSM340206 3 0.0794 0.923 0.000 0.000 0.972 0.028 0.000
#> GSM340207 5 0.1270 0.849 0.000 0.000 0.052 0.000 0.948
#> GSM340237 2 0.2605 0.877 0.148 0.852 0.000 0.000 0.000
#> GSM340238 3 0.2648 0.812 0.000 0.000 0.848 0.152 0.000
#> GSM340239 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340240 5 0.0000 0.881 0.000 0.000 0.000 0.000 1.000
#> GSM340241 2 0.3109 0.858 0.200 0.800 0.000 0.000 0.000
#> GSM340242 3 0.0000 0.936 0.000 0.000 1.000 0.000 0.000
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 3 0.1556 0.8761 0.000 0.000 0.920 0.080 0.000 0.000
#> GSM340359 2 0.0146 0.9181 0.000 0.996 0.000 0.000 0.004 0.000
#> GSM340361 1 0.3737 -0.1768 0.608 0.000 0.000 0.000 0.392 0.000
#> GSM340362 5 0.2823 0.8809 0.204 0.000 0.000 0.000 0.796 0.000
#> GSM340363 3 0.0865 0.9049 0.000 0.000 0.964 0.036 0.000 0.000
#> GSM340364 2 0.5178 0.6878 0.160 0.684 0.000 0.024 0.128 0.004
#> GSM340365 5 0.3862 0.4650 0.476 0.000 0.000 0.000 0.524 0.000
#> GSM340366 5 0.3765 0.6978 0.404 0.000 0.000 0.000 0.596 0.000
#> GSM340367 1 0.1204 0.8165 0.944 0.000 0.056 0.000 0.000 0.000
#> GSM340368 4 0.0547 0.8854 0.000 0.000 0.000 0.980 0.020 0.000
#> GSM340369 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340370 1 0.1141 0.8197 0.948 0.000 0.052 0.000 0.000 0.000
#> GSM340371 3 0.3972 0.5684 0.016 0.000 0.680 0.004 0.300 0.000
#> GSM340372 1 0.0458 0.8280 0.984 0.000 0.000 0.000 0.016 0.000
#> GSM340373 1 0.0632 0.8244 0.976 0.000 0.000 0.000 0.024 0.000
#> GSM340375 1 0.0820 0.8329 0.972 0.000 0.012 0.000 0.016 0.000
#> GSM340376 3 0.1204 0.8889 0.056 0.000 0.944 0.000 0.000 0.000
#> GSM340378 6 0.3283 0.8465 0.000 0.000 0.000 0.036 0.160 0.804
#> GSM340243 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340244 6 0.0146 0.9017 0.000 0.004 0.000 0.000 0.000 0.996
#> GSM340246 6 0.0146 0.9017 0.000 0.004 0.000 0.000 0.000 0.996
#> GSM340247 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340248 6 0.0000 0.9019 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM340249 6 0.3283 0.8465 0.000 0.000 0.000 0.036 0.160 0.804
#> GSM340250 3 0.0790 0.9039 0.032 0.000 0.968 0.000 0.000 0.000
#> GSM340251 6 0.2983 0.8599 0.000 0.000 0.000 0.032 0.136 0.832
#> GSM340252 2 0.2734 0.8078 0.000 0.840 0.000 0.004 0.008 0.148
#> GSM340253 2 0.0146 0.9181 0.000 0.996 0.000 0.000 0.004 0.000
#> GSM340254 2 0.0146 0.9180 0.000 0.996 0.000 0.000 0.004 0.000
#> GSM340256 2 0.1010 0.8980 0.000 0.960 0.000 0.000 0.004 0.036
#> GSM340258 1 0.5562 0.0641 0.532 0.000 0.168 0.000 0.300 0.000
#> GSM340259 1 0.0458 0.8338 0.984 0.000 0.016 0.000 0.000 0.000
#> GSM340260 3 0.0363 0.9160 0.012 0.000 0.988 0.000 0.000 0.000
#> GSM340261 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340262 5 0.3464 0.8212 0.312 0.000 0.000 0.000 0.688 0.000
#> GSM340263 6 0.3283 0.8465 0.000 0.000 0.000 0.036 0.160 0.804
#> GSM340264 1 0.0632 0.8236 0.976 0.000 0.000 0.000 0.024 0.000
#> GSM340265 5 0.3515 0.8091 0.324 0.000 0.000 0.000 0.676 0.000
#> GSM340266 3 0.0363 0.9166 0.000 0.000 0.988 0.012 0.000 0.000
#> GSM340267 3 0.4205 0.2817 0.420 0.000 0.564 0.000 0.016 0.000
#> GSM340268 3 0.0363 0.9165 0.000 0.000 0.988 0.012 0.000 0.000
#> GSM340269 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340270 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM537574 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM537580 3 0.3721 0.7656 0.016 0.000 0.788 0.160 0.036 0.000
#> GSM537581 4 0.1007 0.9220 0.000 0.000 0.044 0.956 0.000 0.000
#> GSM340272 5 0.2823 0.8809 0.204 0.000 0.000 0.000 0.796 0.000
#> GSM340273 6 0.0146 0.9017 0.000 0.004 0.000 0.000 0.000 0.996
#> GSM340275 4 0.2595 0.7738 0.000 0.000 0.000 0.836 0.160 0.004
#> GSM340276 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340277 2 0.0146 0.9180 0.000 0.996 0.000 0.000 0.004 0.000
#> GSM340278 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340279 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340282 5 0.3482 0.8164 0.316 0.000 0.000 0.000 0.684 0.000
#> GSM340284 3 0.3699 0.7597 0.000 0.000 0.788 0.040 0.160 0.012
#> GSM340285 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340286 5 0.2823 0.8809 0.204 0.000 0.000 0.000 0.796 0.000
#> GSM340287 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340288 1 0.0547 0.8331 0.980 0.000 0.020 0.000 0.000 0.000
#> GSM340289 1 0.1663 0.7762 0.912 0.000 0.088 0.000 0.000 0.000
#> GSM340290 3 0.0260 0.9179 0.000 0.000 0.992 0.008 0.000 0.000
#> GSM340291 6 0.2668 0.8426 0.000 0.168 0.000 0.000 0.004 0.828
#> GSM340293 3 0.3756 0.3820 0.000 0.000 0.600 0.400 0.000 0.000
#> GSM340294 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340296 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340297 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340298 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340299 5 0.2823 0.8809 0.204 0.000 0.000 0.000 0.796 0.000
#> GSM340301 5 0.2823 0.8809 0.204 0.000 0.000 0.000 0.796 0.000
#> GSM340303 5 0.2883 0.8784 0.212 0.000 0.000 0.000 0.788 0.000
#> GSM340304 3 0.0790 0.9038 0.032 0.000 0.968 0.000 0.000 0.000
#> GSM340306 4 0.3883 0.7323 0.144 0.000 0.088 0.768 0.000 0.000
#> GSM340307 6 0.1285 0.8970 0.000 0.000 0.000 0.004 0.052 0.944
#> GSM340310 1 0.3851 0.0574 0.540 0.000 0.460 0.000 0.000 0.000
#> GSM340314 3 0.2527 0.7967 0.000 0.000 0.832 0.168 0.000 0.000
#> GSM340315 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340317 6 0.0000 0.9019 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM340318 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340319 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340320 3 0.2260 0.8252 0.000 0.000 0.860 0.140 0.000 0.000
#> GSM340321 6 0.2902 0.8255 0.000 0.196 0.000 0.000 0.004 0.800
#> GSM340322 4 0.1010 0.9283 0.000 0.000 0.036 0.960 0.004 0.000
#> GSM340324 2 0.1010 0.8980 0.000 0.960 0.000 0.000 0.004 0.036
#> GSM340328 3 0.0547 0.9122 0.020 0.000 0.980 0.000 0.000 0.000
#> GSM340330 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340332 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340333 2 0.0146 0.9181 0.000 0.996 0.000 0.000 0.004 0.000
#> GSM340336 4 0.2595 0.7738 0.000 0.000 0.000 0.836 0.160 0.004
#> GSM340337 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340338 3 0.0146 0.9193 0.000 0.000 0.996 0.004 0.000 0.000
#> GSM340339 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340340 4 0.2595 0.7738 0.000 0.000 0.000 0.836 0.160 0.004
#> GSM340341 3 0.3424 0.7713 0.000 0.000 0.800 0.036 0.160 0.004
#> GSM340343 3 0.2135 0.8365 0.000 0.000 0.872 0.128 0.000 0.000
#> GSM340344 2 0.4636 0.5529 0.160 0.692 0.000 0.000 0.148 0.000
#> GSM340346 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340347 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340348 3 0.0865 0.9052 0.000 0.000 0.964 0.036 0.000 0.000
#> GSM340349 3 0.2996 0.6993 0.228 0.000 0.772 0.000 0.000 0.000
#> GSM340350 3 0.2135 0.8253 0.128 0.000 0.872 0.000 0.000 0.000
#> GSM340351 1 0.0458 0.8280 0.984 0.000 0.000 0.000 0.016 0.000
#> GSM340354 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340356 6 0.0146 0.9017 0.000 0.004 0.000 0.000 0.000 0.996
#> GSM340357 3 0.3862 0.1290 0.476 0.000 0.524 0.000 0.000 0.000
#> GSM348183 1 0.0458 0.8338 0.984 0.000 0.016 0.000 0.000 0.000
#> GSM348191 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM348193 1 0.1204 0.8168 0.944 0.000 0.056 0.000 0.000 0.000
#> GSM537578 3 0.3684 0.4296 0.372 0.000 0.628 0.000 0.000 0.000
#> GSM348181 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM348182 1 0.1075 0.8000 0.952 0.000 0.000 0.000 0.048 0.000
#> GSM348184 6 0.0000 0.9019 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM348185 6 0.1265 0.8979 0.000 0.000 0.000 0.008 0.044 0.948
#> GSM348186 2 0.0146 0.9180 0.000 0.996 0.000 0.000 0.004 0.000
#> GSM348187 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM348188 1 0.2941 0.4777 0.780 0.000 0.000 0.000 0.220 0.000
#> GSM348189 3 0.2006 0.8647 0.016 0.000 0.904 0.000 0.080 0.000
#> GSM348190 5 0.2823 0.8809 0.204 0.000 0.000 0.000 0.796 0.000
#> GSM348194 1 0.1075 0.8223 0.952 0.000 0.048 0.000 0.000 0.000
#> GSM348195 1 0.0458 0.8280 0.984 0.000 0.000 0.000 0.016 0.000
#> GSM348196 3 0.0146 0.9187 0.004 0.000 0.996 0.000 0.000 0.000
#> GSM537585 1 0.0632 0.8236 0.976 0.000 0.000 0.000 0.024 0.000
#> GSM537594 6 0.2902 0.8255 0.000 0.196 0.000 0.000 0.004 0.800
#> GSM537596 3 0.0146 0.9187 0.004 0.000 0.996 0.000 0.000 0.000
#> GSM537597 3 0.0146 0.9187 0.004 0.000 0.996 0.000 0.000 0.000
#> GSM537602 3 0.2941 0.7115 0.220 0.000 0.780 0.000 0.000 0.000
#> GSM340184 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340185 6 0.2902 0.8255 0.000 0.196 0.000 0.000 0.004 0.800
#> GSM340186 6 0.0000 0.9019 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM340187 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340189 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340190 4 0.5422 0.3349 0.000 0.000 0.000 0.564 0.160 0.276
#> GSM340191 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340192 5 0.3735 0.4420 0.004 0.180 0.000 0.036 0.776 0.004
#> GSM340193 1 0.1444 0.7981 0.928 0.000 0.072 0.000 0.000 0.000
#> GSM340194 5 0.2823 0.8809 0.204 0.000 0.000 0.000 0.796 0.000
#> GSM340195 5 0.2823 0.8809 0.204 0.000 0.000 0.000 0.796 0.000
#> GSM340196 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340197 5 0.3050 0.8698 0.236 0.000 0.000 0.000 0.764 0.000
#> GSM340198 2 0.0000 0.9182 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM340199 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340200 6 0.3283 0.8465 0.000 0.000 0.000 0.036 0.160 0.804
#> GSM340201 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340202 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340203 4 0.0865 0.9304 0.000 0.000 0.036 0.964 0.000 0.000
#> GSM340204 5 0.3843 0.5727 0.452 0.000 0.000 0.000 0.548 0.000
#> GSM340205 3 0.0000 0.9201 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM340206 3 0.0790 0.9076 0.000 0.000 0.968 0.032 0.000 0.000
#> GSM340207 5 0.3235 0.7862 0.128 0.000 0.052 0.000 0.820 0.000
#> GSM340237 6 0.0146 0.9017 0.000 0.004 0.000 0.000 0.000 0.996
#> GSM340238 3 0.2527 0.7967 0.000 0.000 0.832 0.168 0.000 0.000
#> GSM340239 5 0.3371 0.8365 0.292 0.000 0.000 0.000 0.708 0.000
#> GSM340240 5 0.2823 0.8809 0.204 0.000 0.000 0.000 0.796 0.000
#> GSM340241 6 0.2902 0.8255 0.000 0.196 0.000 0.000 0.004 0.800
#> GSM340242 3 0.1320 0.8955 0.016 0.000 0.948 0.000 0.036 0.000
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
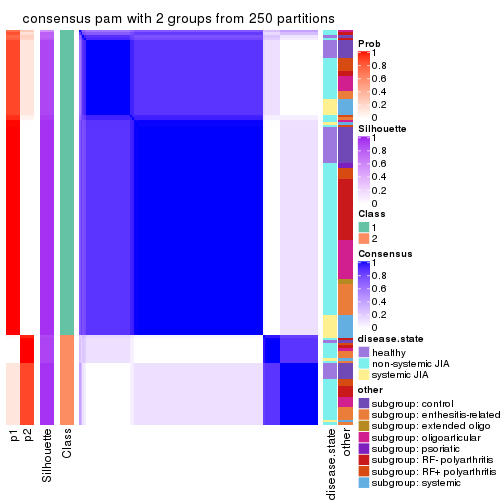
consensus_heatmap(res, k = 3)
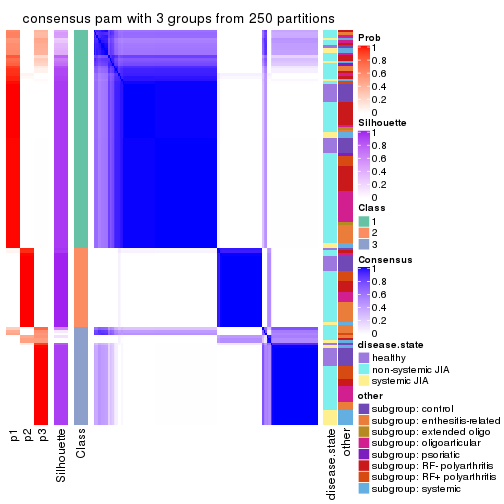
consensus_heatmap(res, k = 4)
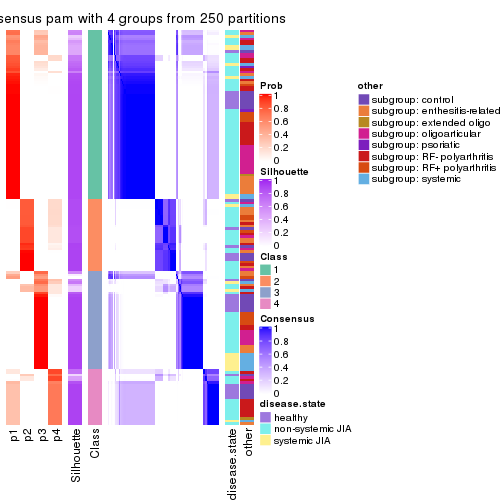
consensus_heatmap(res, k = 5)
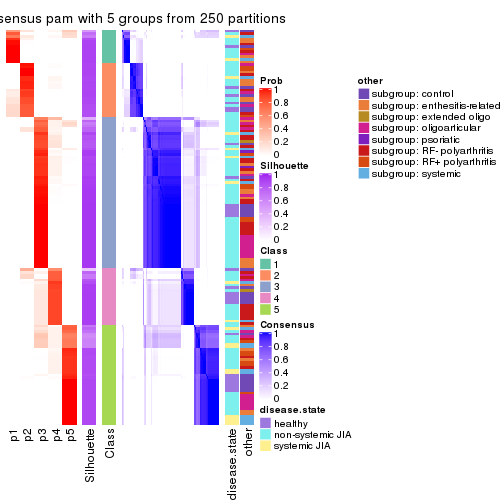
consensus_heatmap(res, k = 6)
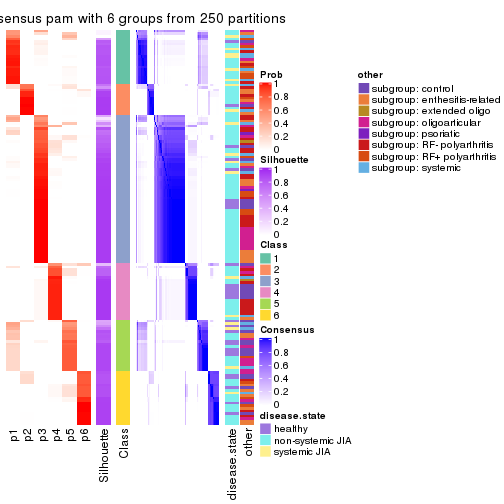
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
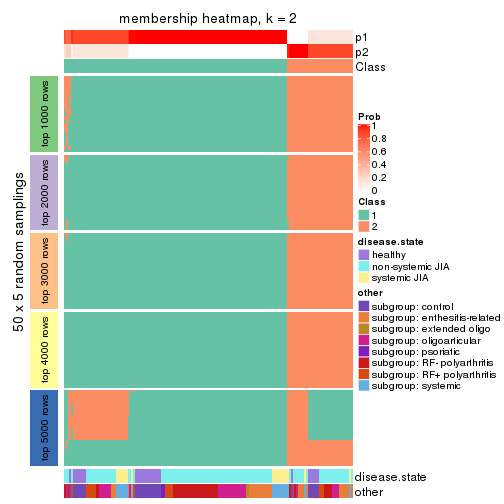
membership_heatmap(res, k = 3)
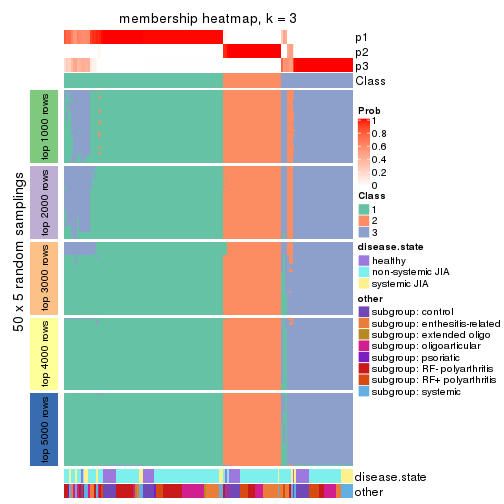
membership_heatmap(res, k = 4)
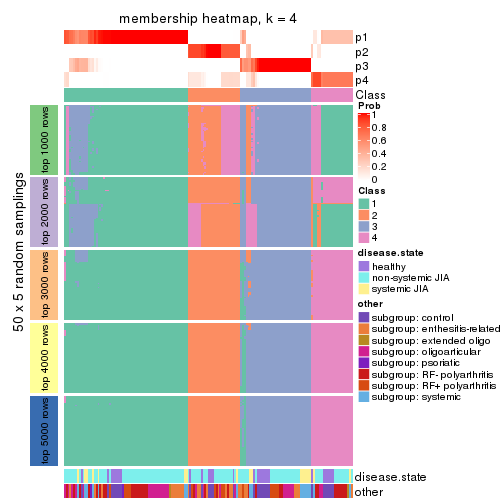
membership_heatmap(res, k = 5)
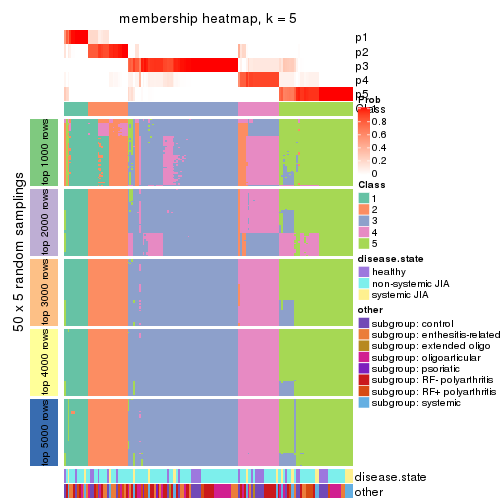
membership_heatmap(res, k = 6)
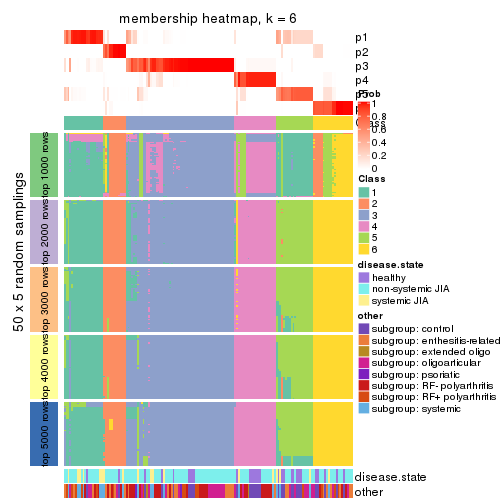
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
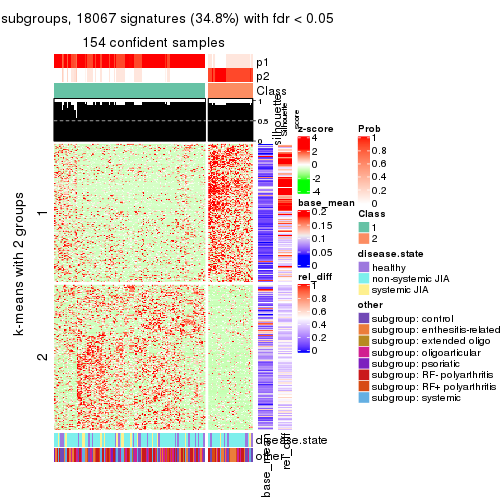
get_signatures(res, k = 3)
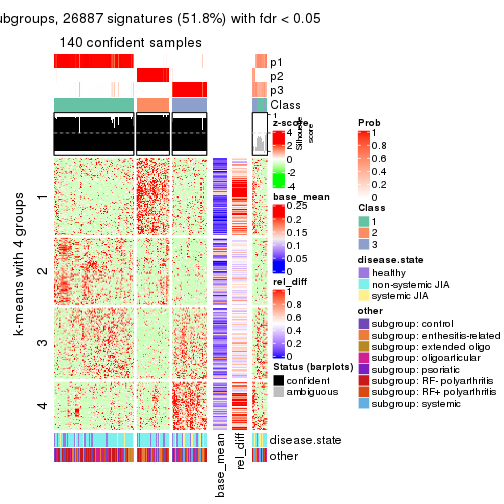
get_signatures(res, k = 4)
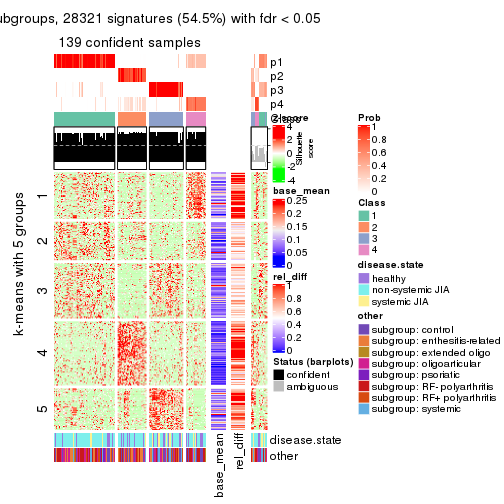
get_signatures(res, k = 5)
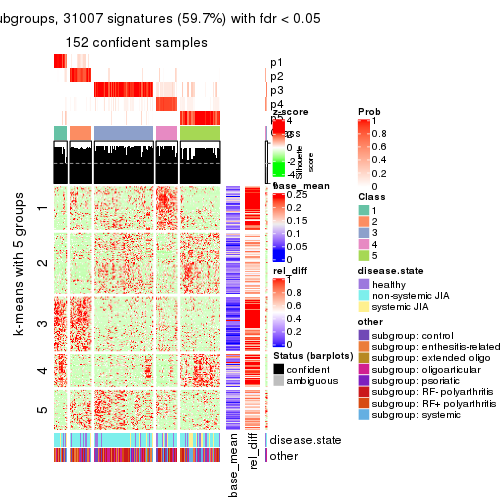
get_signatures(res, k = 6)
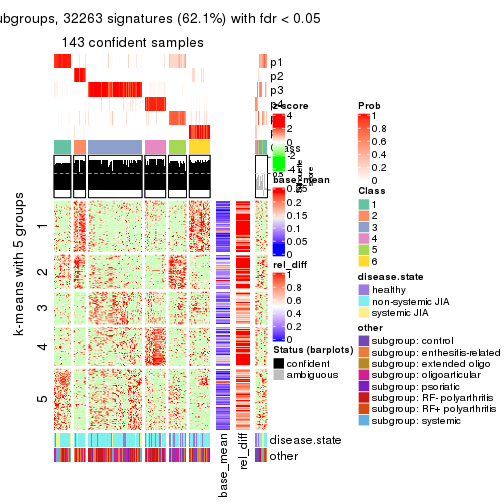
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
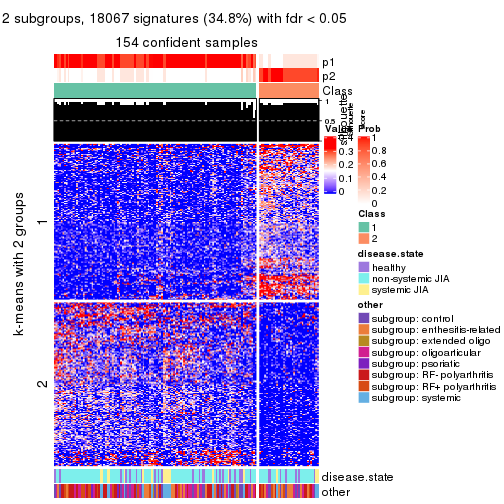
get_signatures(res, k = 3, scale_rows = FALSE)
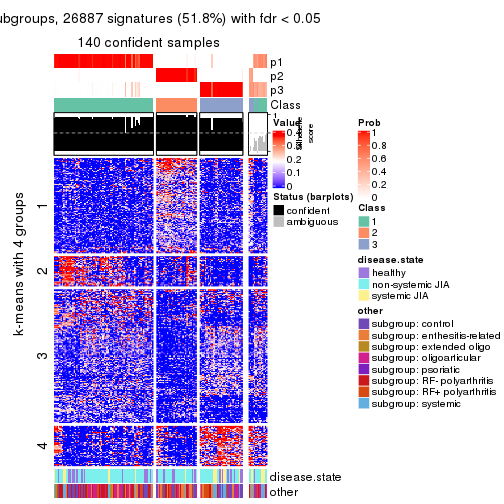
get_signatures(res, k = 4, scale_rows = FALSE)
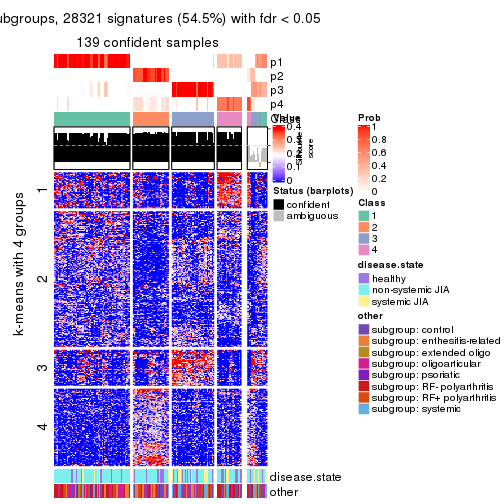
get_signatures(res, k = 5, scale_rows = FALSE)
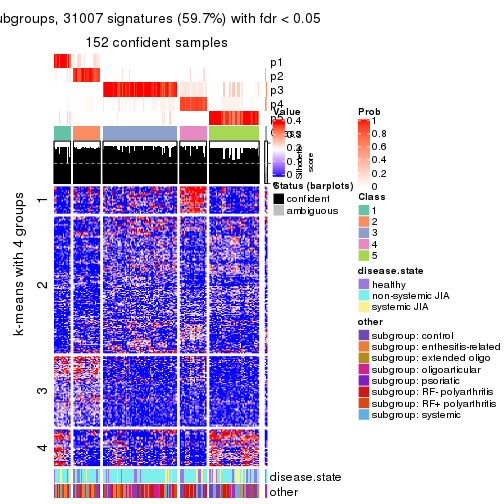
get_signatures(res, k = 6, scale_rows = FALSE)
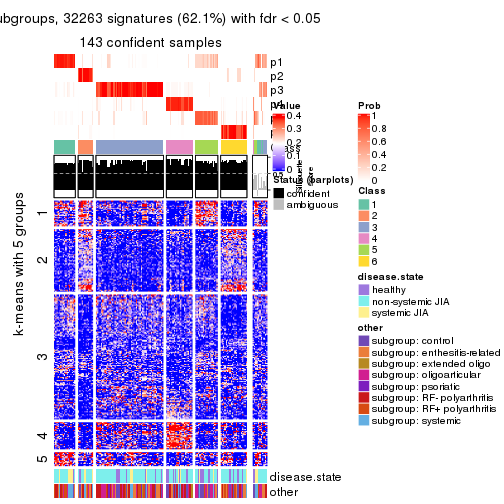
Compare the overlap of signatures from different k:
compare_signatures(res)
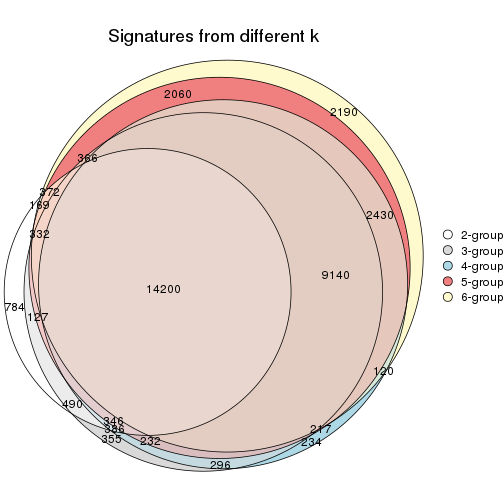
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
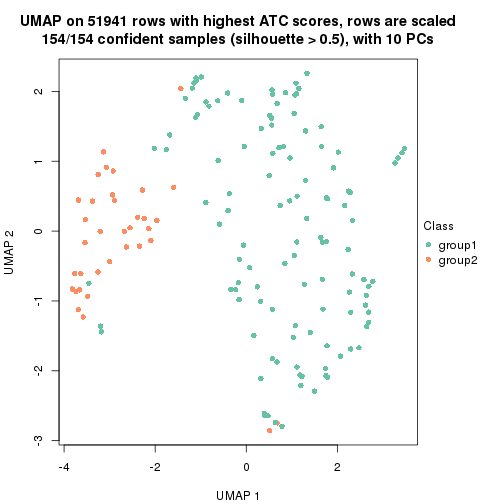
dimension_reduction(res, k = 3, method = "UMAP")
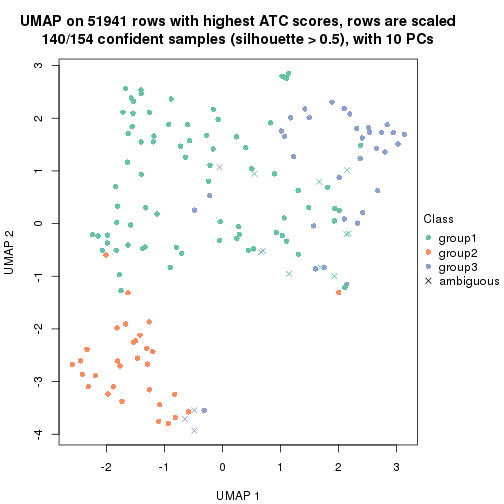
dimension_reduction(res, k = 4, method = "UMAP")
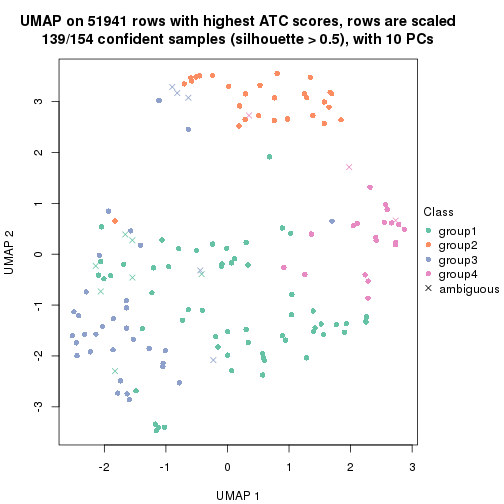
dimension_reduction(res, k = 5, method = "UMAP")
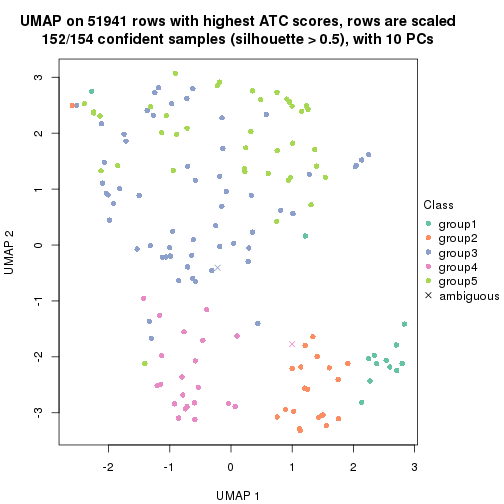
dimension_reduction(res, k = 6, method = "UMAP")
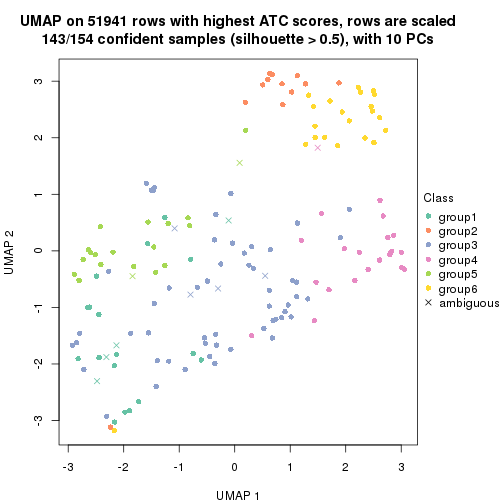
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
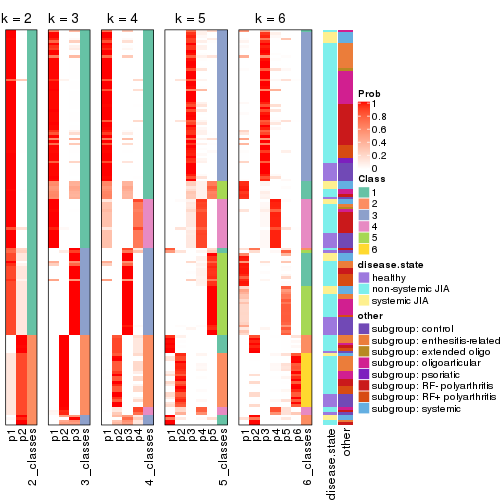
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> ATC:pam 154 0.8049 0.5096 2
#> ATC:pam 140 0.0879 0.1081 3
#> ATC:pam 139 0.0577 0.0320 4
#> ATC:pam 152 0.1671 0.1345 5
#> ATC:pam 143 0.0679 0.0783 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["ATC", "mclust"]
# you can also extract it by
# res = res_list["ATC:mclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'ATC' method.
#> Subgroups are detected by 'mclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
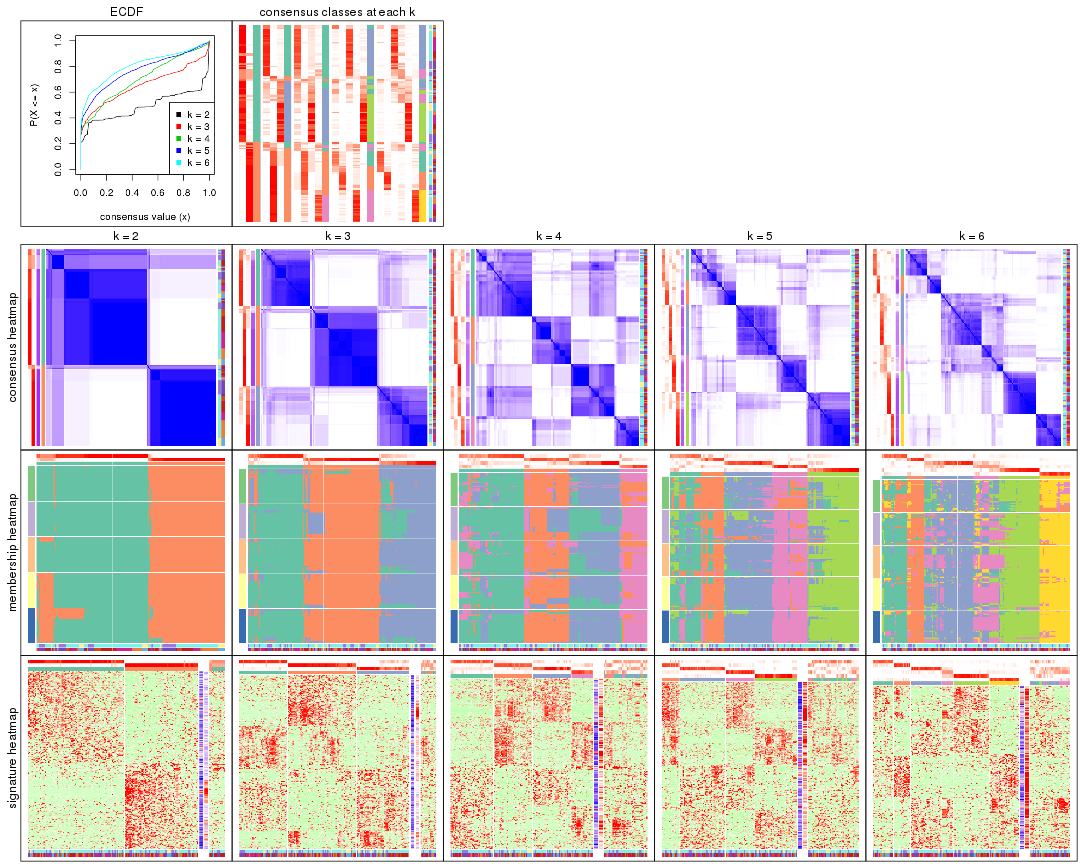
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
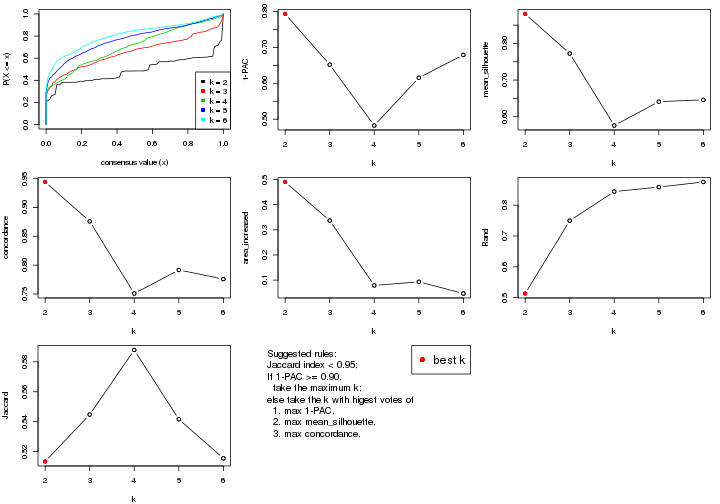
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.793 0.880 0.944 0.4902 0.513 0.513
#> 3 3 0.652 0.772 0.876 0.3367 0.750 0.545
#> 4 4 0.482 0.576 0.751 0.0788 0.844 0.588
#> 5 5 0.615 0.641 0.792 0.0934 0.859 0.542
#> 6 6 0.679 0.646 0.776 0.0467 0.875 0.515
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 1 0.0000 0.917 1.000 0.000
#> GSM340359 2 0.0000 0.975 0.000 1.000
#> GSM340361 1 0.3274 0.899 0.940 0.060
#> GSM340362 1 0.3274 0.899 0.940 0.060
#> GSM340363 1 0.0000 0.917 1.000 0.000
#> GSM340364 2 0.0000 0.975 0.000 1.000
#> GSM340365 1 0.3274 0.899 0.940 0.060
#> GSM340366 1 0.2603 0.905 0.956 0.044
#> GSM340367 1 0.0000 0.917 1.000 0.000
#> GSM340368 2 0.0000 0.975 0.000 1.000
#> GSM340369 1 0.9833 0.395 0.576 0.424
#> GSM340370 2 0.1843 0.953 0.028 0.972
#> GSM340371 1 0.3274 0.899 0.940 0.060
#> GSM340372 1 0.0000 0.917 1.000 0.000
#> GSM340373 1 0.0000 0.917 1.000 0.000
#> GSM340375 2 0.0000 0.975 0.000 1.000
#> GSM340376 2 0.0000 0.975 0.000 1.000
#> GSM340378 2 0.0000 0.975 0.000 1.000
#> GSM340243 1 0.0000 0.917 1.000 0.000
#> GSM340244 2 0.0000 0.975 0.000 1.000
#> GSM340246 2 0.0000 0.975 0.000 1.000
#> GSM340247 2 0.0000 0.975 0.000 1.000
#> GSM340248 2 0.0000 0.975 0.000 1.000
#> GSM340249 2 0.0000 0.975 0.000 1.000
#> GSM340250 1 0.0000 0.917 1.000 0.000
#> GSM340251 2 0.0000 0.975 0.000 1.000
#> GSM340252 2 0.0000 0.975 0.000 1.000
#> GSM340253 2 0.0000 0.975 0.000 1.000
#> GSM340254 2 0.0000 0.975 0.000 1.000
#> GSM340256 2 0.0000 0.975 0.000 1.000
#> GSM340258 2 0.0000 0.975 0.000 1.000
#> GSM340259 1 0.0000 0.917 1.000 0.000
#> GSM340260 1 0.0000 0.917 1.000 0.000
#> GSM340261 1 0.0000 0.917 1.000 0.000
#> GSM340262 1 0.3274 0.899 0.940 0.060
#> GSM340263 2 0.0000 0.975 0.000 1.000
#> GSM340264 1 0.8443 0.659 0.728 0.272
#> GSM340265 2 0.1843 0.953 0.028 0.972
#> GSM340266 1 0.0000 0.917 1.000 0.000
#> GSM340267 2 0.1843 0.953 0.028 0.972
#> GSM340268 1 0.0000 0.917 1.000 0.000
#> GSM340269 1 0.0000 0.917 1.000 0.000
#> GSM340270 1 0.0672 0.914 0.992 0.008
#> GSM537574 2 0.0000 0.975 0.000 1.000
#> GSM537580 1 0.3274 0.899 0.940 0.060
#> GSM537581 2 0.0000 0.975 0.000 1.000
#> GSM340272 1 0.3274 0.899 0.940 0.060
#> GSM340273 2 0.0000 0.975 0.000 1.000
#> GSM340275 2 0.0000 0.975 0.000 1.000
#> GSM340276 1 0.0000 0.917 1.000 0.000
#> GSM340277 2 0.0000 0.975 0.000 1.000
#> GSM340278 1 0.0000 0.917 1.000 0.000
#> GSM340279 1 0.0000 0.917 1.000 0.000
#> GSM340282 1 0.2236 0.907 0.964 0.036
#> GSM340284 2 0.0000 0.975 0.000 1.000
#> GSM340285 1 0.0000 0.917 1.000 0.000
#> GSM340286 1 0.3274 0.899 0.940 0.060
#> GSM340287 1 0.0000 0.917 1.000 0.000
#> GSM340288 1 0.0000 0.917 1.000 0.000
#> GSM340289 1 0.0000 0.917 1.000 0.000
#> GSM340290 1 0.0000 0.917 1.000 0.000
#> GSM340291 2 0.0000 0.975 0.000 1.000
#> GSM340293 1 0.3274 0.899 0.940 0.060
#> GSM340294 1 0.0000 0.917 1.000 0.000
#> GSM340296 1 0.0000 0.917 1.000 0.000
#> GSM340297 1 0.0000 0.917 1.000 0.000
#> GSM340298 1 0.0000 0.917 1.000 0.000
#> GSM340299 1 0.3274 0.899 0.940 0.060
#> GSM340301 1 0.3274 0.899 0.940 0.060
#> GSM340303 2 0.1843 0.953 0.028 0.972
#> GSM340304 2 0.8861 0.565 0.304 0.696
#> GSM340306 1 0.8443 0.676 0.728 0.272
#> GSM340307 2 0.0000 0.975 0.000 1.000
#> GSM340310 1 0.0000 0.917 1.000 0.000
#> GSM340314 1 0.0000 0.917 1.000 0.000
#> GSM340315 2 0.1843 0.953 0.028 0.972
#> GSM340317 2 0.0000 0.975 0.000 1.000
#> GSM340318 1 0.9833 0.395 0.576 0.424
#> GSM340319 1 0.9833 0.395 0.576 0.424
#> GSM340320 1 0.0000 0.917 1.000 0.000
#> GSM340321 2 0.0000 0.975 0.000 1.000
#> GSM340322 1 0.9833 0.395 0.576 0.424
#> GSM340324 2 0.0000 0.975 0.000 1.000
#> GSM340328 1 0.0000 0.917 1.000 0.000
#> GSM340330 1 0.0938 0.914 0.988 0.012
#> GSM340332 1 0.9833 0.395 0.576 0.424
#> GSM340333 2 0.0000 0.975 0.000 1.000
#> GSM340336 2 0.0000 0.975 0.000 1.000
#> GSM340337 1 0.9833 0.395 0.576 0.424
#> GSM340338 1 0.3274 0.899 0.940 0.060
#> GSM340339 1 0.9833 0.395 0.576 0.424
#> GSM340340 2 0.0000 0.975 0.000 1.000
#> GSM340341 2 0.0000 0.975 0.000 1.000
#> GSM340343 1 0.0000 0.917 1.000 0.000
#> GSM340344 2 0.0000 0.975 0.000 1.000
#> GSM340346 1 0.0000 0.917 1.000 0.000
#> GSM340347 2 0.9552 0.285 0.376 0.624
#> GSM340348 2 0.1843 0.953 0.028 0.972
#> GSM340349 1 0.0000 0.917 1.000 0.000
#> GSM340350 1 0.0000 0.917 1.000 0.000
#> GSM340351 1 0.0000 0.917 1.000 0.000
#> GSM340354 2 0.1414 0.959 0.020 0.980
#> GSM340356 2 0.0000 0.975 0.000 1.000
#> GSM340357 1 0.0000 0.917 1.000 0.000
#> GSM348183 1 0.0000 0.917 1.000 0.000
#> GSM348191 1 0.0000 0.917 1.000 0.000
#> GSM348193 2 0.9909 0.163 0.444 0.556
#> GSM537578 1 0.0000 0.917 1.000 0.000
#> GSM348181 1 0.0000 0.917 1.000 0.000
#> GSM348182 1 0.3274 0.899 0.940 0.060
#> GSM348184 2 0.0000 0.975 0.000 1.000
#> GSM348185 2 0.0000 0.975 0.000 1.000
#> GSM348186 2 0.0000 0.975 0.000 1.000
#> GSM348187 1 0.0000 0.917 1.000 0.000
#> GSM348188 1 0.6623 0.803 0.828 0.172
#> GSM348189 1 0.3274 0.899 0.940 0.060
#> GSM348190 1 0.3274 0.899 0.940 0.060
#> GSM348194 1 0.0000 0.917 1.000 0.000
#> GSM348195 1 0.0000 0.917 1.000 0.000
#> GSM348196 1 0.0000 0.917 1.000 0.000
#> GSM537585 2 0.1843 0.953 0.028 0.972
#> GSM537594 2 0.0000 0.975 0.000 1.000
#> GSM537596 1 0.0000 0.917 1.000 0.000
#> GSM537597 2 0.0000 0.975 0.000 1.000
#> GSM537602 1 0.0938 0.912 0.988 0.012
#> GSM340184 2 0.4161 0.896 0.084 0.916
#> GSM340185 2 0.0000 0.975 0.000 1.000
#> GSM340186 2 0.0000 0.975 0.000 1.000
#> GSM340187 2 0.0000 0.975 0.000 1.000
#> GSM340189 1 0.9833 0.395 0.576 0.424
#> GSM340190 2 0.0000 0.975 0.000 1.000
#> GSM340191 1 0.0000 0.917 1.000 0.000
#> GSM340192 2 0.0000 0.975 0.000 1.000
#> GSM340193 1 0.0000 0.917 1.000 0.000
#> GSM340194 1 0.3274 0.899 0.940 0.060
#> GSM340195 1 0.3274 0.899 0.940 0.060
#> GSM340196 1 0.9833 0.395 0.576 0.424
#> GSM340197 1 0.3274 0.899 0.940 0.060
#> GSM340198 2 0.0000 0.975 0.000 1.000
#> GSM340199 1 0.0000 0.917 1.000 0.000
#> GSM340200 2 0.0000 0.975 0.000 1.000
#> GSM340201 1 0.9833 0.395 0.576 0.424
#> GSM340202 2 0.0672 0.968 0.008 0.992
#> GSM340203 1 0.9833 0.395 0.576 0.424
#> GSM340204 2 0.0000 0.975 0.000 1.000
#> GSM340205 1 0.0000 0.917 1.000 0.000
#> GSM340206 1 0.6973 0.757 0.812 0.188
#> GSM340207 1 0.3274 0.899 0.940 0.060
#> GSM340237 2 0.0000 0.975 0.000 1.000
#> GSM340238 1 0.0000 0.917 1.000 0.000
#> GSM340239 1 0.3274 0.899 0.940 0.060
#> GSM340240 1 0.7602 0.746 0.780 0.220
#> GSM340241 2 0.0000 0.975 0.000 1.000
#> GSM340242 1 0.3274 0.899 0.940 0.060
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.1643 0.8568 0.956 0.000 0.044
#> GSM340359 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340361 3 0.0237 0.7950 0.004 0.000 0.996
#> GSM340362 3 0.0237 0.7950 0.004 0.000 0.996
#> GSM340363 1 0.1163 0.8606 0.972 0.000 0.028
#> GSM340364 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340365 3 0.0237 0.7950 0.004 0.000 0.996
#> GSM340366 3 0.0424 0.7957 0.008 0.000 0.992
#> GSM340367 3 0.5465 0.6994 0.288 0.000 0.712
#> GSM340368 2 0.0892 0.9140 0.020 0.980 0.000
#> GSM340369 1 0.1989 0.8404 0.948 0.004 0.048
#> GSM340370 2 0.4178 0.8164 0.000 0.828 0.172
#> GSM340371 3 0.8202 0.6104 0.260 0.120 0.620
#> GSM340372 3 0.3116 0.7938 0.108 0.000 0.892
#> GSM340373 3 0.2878 0.7961 0.096 0.000 0.904
#> GSM340375 2 0.4291 0.8125 0.000 0.820 0.180
#> GSM340376 2 0.4178 0.8164 0.000 0.828 0.172
#> GSM340378 2 0.0000 0.9157 0.000 1.000 0.000
#> GSM340243 1 0.1289 0.8608 0.968 0.000 0.032
#> GSM340244 2 0.0747 0.9146 0.016 0.984 0.000
#> GSM340246 2 0.0747 0.9146 0.016 0.984 0.000
#> GSM340247 2 0.1399 0.9098 0.028 0.968 0.004
#> GSM340248 2 0.0747 0.9146 0.016 0.984 0.000
#> GSM340249 2 0.0892 0.9139 0.020 0.980 0.000
#> GSM340250 3 0.5291 0.7170 0.268 0.000 0.732
#> GSM340251 2 0.1031 0.9128 0.024 0.976 0.000
#> GSM340252 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340253 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340254 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340256 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340258 2 0.4235 0.8157 0.000 0.824 0.176
#> GSM340259 3 0.3038 0.7938 0.104 0.000 0.896
#> GSM340260 3 0.5098 0.7346 0.248 0.000 0.752
#> GSM340261 1 0.1289 0.8609 0.968 0.000 0.032
#> GSM340262 3 0.0424 0.7957 0.008 0.000 0.992
#> GSM340263 2 0.0892 0.9139 0.020 0.980 0.000
#> GSM340264 2 0.6521 0.2343 0.004 0.504 0.492
#> GSM340265 2 0.4291 0.8125 0.000 0.820 0.180
#> GSM340266 1 0.2682 0.8230 0.920 0.004 0.076
#> GSM340267 2 0.4291 0.8125 0.000 0.820 0.180
#> GSM340268 1 0.0661 0.8545 0.988 0.004 0.008
#> GSM340269 1 0.6522 0.5240 0.696 0.032 0.272
#> GSM340270 1 0.6803 0.5124 0.680 0.040 0.280
#> GSM537574 2 0.4897 0.8154 0.016 0.812 0.172
#> GSM537580 3 0.9146 0.3434 0.380 0.148 0.472
#> GSM537581 2 0.6994 0.1045 0.424 0.556 0.020
#> GSM340272 3 0.0237 0.7950 0.004 0.000 0.996
#> GSM340273 2 0.0892 0.9139 0.020 0.980 0.000
#> GSM340275 2 0.1031 0.9128 0.024 0.976 0.000
#> GSM340276 1 0.2356 0.8345 0.928 0.000 0.072
#> GSM340277 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340278 1 0.1289 0.8609 0.968 0.000 0.032
#> GSM340279 1 0.2959 0.8102 0.900 0.000 0.100
#> GSM340282 3 0.0237 0.7950 0.004 0.000 0.996
#> GSM340284 2 0.1031 0.9128 0.024 0.976 0.000
#> GSM340285 1 0.1289 0.8609 0.968 0.000 0.032
#> GSM340286 3 0.0237 0.7950 0.004 0.000 0.996
#> GSM340287 1 0.4172 0.7366 0.840 0.004 0.156
#> GSM340288 3 0.3116 0.7938 0.108 0.000 0.892
#> GSM340289 3 0.4974 0.7427 0.236 0.000 0.764
#> GSM340290 1 0.3267 0.7926 0.884 0.000 0.116
#> GSM340291 2 0.0747 0.9146 0.016 0.984 0.000
#> GSM340293 1 0.2261 0.8457 0.932 0.000 0.068
#> GSM340294 1 0.1289 0.8609 0.968 0.000 0.032
#> GSM340296 1 0.1289 0.8609 0.968 0.000 0.032
#> GSM340297 1 0.1289 0.8609 0.968 0.000 0.032
#> GSM340298 1 0.1289 0.8609 0.968 0.000 0.032
#> GSM340299 3 0.0237 0.7950 0.004 0.000 0.996
#> GSM340301 3 0.0237 0.7950 0.004 0.000 0.996
#> GSM340303 2 0.4291 0.8125 0.000 0.820 0.180
#> GSM340304 1 0.7911 0.4934 0.632 0.096 0.272
#> GSM340306 3 0.9730 0.3051 0.228 0.352 0.420
#> GSM340307 2 0.0000 0.9157 0.000 1.000 0.000
#> GSM340310 3 0.5431 0.7035 0.284 0.000 0.716
#> GSM340314 1 0.1163 0.8610 0.972 0.000 0.028
#> GSM340315 2 0.5092 0.8110 0.020 0.804 0.176
#> GSM340317 2 0.0892 0.9139 0.020 0.980 0.000
#> GSM340318 1 0.1989 0.8404 0.948 0.004 0.048
#> GSM340319 1 0.1989 0.8404 0.948 0.004 0.048
#> GSM340320 1 0.1411 0.8604 0.964 0.000 0.036
#> GSM340321 2 0.0747 0.9146 0.016 0.984 0.000
#> GSM340322 2 0.4749 0.8064 0.076 0.852 0.072
#> GSM340324 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340328 3 0.6467 0.5723 0.388 0.008 0.604
#> GSM340330 3 0.7944 0.6117 0.296 0.088 0.616
#> GSM340332 1 0.2096 0.8419 0.944 0.004 0.052
#> GSM340333 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340336 2 0.1031 0.9128 0.024 0.976 0.000
#> GSM340337 2 0.8013 0.3297 0.332 0.588 0.080
#> GSM340338 3 0.8120 0.6320 0.224 0.136 0.640
#> GSM340339 1 0.2590 0.8441 0.924 0.004 0.072
#> GSM340340 2 0.0000 0.9157 0.000 1.000 0.000
#> GSM340341 2 0.0592 0.9152 0.012 0.988 0.000
#> GSM340343 1 0.1411 0.8613 0.964 0.000 0.036
#> GSM340344 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340346 3 0.6215 0.5009 0.428 0.000 0.572
#> GSM340347 1 0.7095 0.5388 0.660 0.292 0.048
#> GSM340348 1 0.6910 0.3796 0.584 0.396 0.020
#> GSM340349 3 0.5098 0.7339 0.248 0.000 0.752
#> GSM340350 3 0.5363 0.7098 0.276 0.000 0.724
#> GSM340351 3 0.2959 0.7951 0.100 0.000 0.900
#> GSM340354 2 0.4235 0.8131 0.000 0.824 0.176
#> GSM340356 2 0.0000 0.9157 0.000 1.000 0.000
#> GSM340357 3 0.6875 0.7187 0.244 0.056 0.700
#> GSM348183 3 0.2066 0.7984 0.060 0.000 0.940
#> GSM348191 1 0.1529 0.8582 0.960 0.000 0.040
#> GSM348193 2 0.9211 0.2493 0.224 0.536 0.240
#> GSM537578 3 0.4931 0.7440 0.232 0.000 0.768
#> GSM348181 1 0.2165 0.8453 0.936 0.000 0.064
#> GSM348182 3 0.0424 0.7957 0.008 0.000 0.992
#> GSM348184 2 0.0747 0.9146 0.016 0.984 0.000
#> GSM348185 2 0.0892 0.9139 0.020 0.980 0.000
#> GSM348186 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM348187 1 0.1163 0.8606 0.972 0.000 0.028
#> GSM348188 3 0.7913 0.1920 0.056 0.452 0.492
#> GSM348189 3 0.7727 0.5567 0.336 0.064 0.600
#> GSM348190 3 0.0237 0.7950 0.004 0.000 0.996
#> GSM348194 3 0.4121 0.7766 0.168 0.000 0.832
#> GSM348195 3 0.2537 0.7984 0.080 0.000 0.920
#> GSM348196 3 0.6154 0.5404 0.408 0.000 0.592
#> GSM537585 2 0.4291 0.8125 0.000 0.820 0.180
#> GSM537594 2 0.0000 0.9157 0.000 1.000 0.000
#> GSM537596 3 0.6154 0.5401 0.408 0.000 0.592
#> GSM537597 2 0.4178 0.8164 0.000 0.828 0.172
#> GSM537602 1 0.7230 0.3939 0.616 0.040 0.344
#> GSM340184 1 0.9431 0.0017 0.424 0.400 0.176
#> GSM340185 2 0.0000 0.9157 0.000 1.000 0.000
#> GSM340186 2 0.0000 0.9157 0.000 1.000 0.000
#> GSM340187 2 0.1031 0.9128 0.024 0.976 0.000
#> GSM340189 1 0.1989 0.8404 0.948 0.004 0.048
#> GSM340190 2 0.1031 0.9128 0.024 0.976 0.000
#> GSM340191 1 0.1163 0.8606 0.972 0.000 0.028
#> GSM340192 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340193 3 0.4931 0.7440 0.232 0.000 0.768
#> GSM340194 3 0.0237 0.7950 0.004 0.000 0.996
#> GSM340195 3 0.0237 0.7950 0.004 0.000 0.996
#> GSM340196 1 0.1989 0.8404 0.948 0.004 0.048
#> GSM340197 3 0.0237 0.7950 0.004 0.000 0.996
#> GSM340198 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340199 1 0.1031 0.8602 0.976 0.000 0.024
#> GSM340200 2 0.0000 0.9157 0.000 1.000 0.000
#> GSM340201 1 0.1989 0.8404 0.948 0.004 0.048
#> GSM340202 1 0.7292 0.1122 0.500 0.472 0.028
#> GSM340203 1 0.2096 0.8419 0.944 0.004 0.052
#> GSM340204 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340205 1 0.1411 0.8597 0.964 0.000 0.036
#> GSM340206 1 0.7053 0.5377 0.692 0.244 0.064
#> GSM340207 3 0.1751 0.7848 0.012 0.028 0.960
#> GSM340237 2 0.0747 0.9146 0.016 0.984 0.000
#> GSM340238 1 0.1643 0.8583 0.956 0.000 0.044
#> GSM340239 3 0.0237 0.7950 0.004 0.000 0.996
#> GSM340240 2 0.6520 0.2359 0.004 0.508 0.488
#> GSM340241 2 0.0237 0.9156 0.000 0.996 0.004
#> GSM340242 3 0.6814 0.5281 0.372 0.020 0.608
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 1 0.3208 0.69699 0.848 0.000 0.148 0.004
#> GSM340359 2 0.2830 0.71118 0.060 0.900 0.000 0.040
#> GSM340361 3 0.0000 0.70705 0.000 0.000 1.000 0.000
#> GSM340362 3 0.0000 0.70705 0.000 0.000 1.000 0.000
#> GSM340363 1 0.2589 0.71323 0.884 0.000 0.116 0.000
#> GSM340364 2 0.4467 0.73538 0.000 0.788 0.172 0.040
#> GSM340365 3 0.0000 0.70705 0.000 0.000 1.000 0.000
#> GSM340366 3 0.0707 0.71094 0.020 0.000 0.980 0.000
#> GSM340367 3 0.5620 0.31560 0.416 0.000 0.560 0.024
#> GSM340368 4 0.2081 0.75724 0.000 0.084 0.000 0.916
#> GSM340369 1 0.6476 0.59155 0.616 0.000 0.112 0.272
#> GSM340370 2 0.5050 0.70508 0.000 0.756 0.176 0.068
#> GSM340371 3 0.7259 0.17078 0.448 0.060 0.456 0.036
#> GSM340372 3 0.3810 0.70893 0.188 0.000 0.804 0.008
#> GSM340373 3 0.3810 0.70893 0.188 0.000 0.804 0.008
#> GSM340375 2 0.3356 0.73032 0.000 0.824 0.176 0.000
#> GSM340376 2 0.5185 0.69813 0.000 0.748 0.176 0.076
#> GSM340378 2 0.4507 0.73492 0.000 0.788 0.168 0.044
#> GSM340243 1 0.4054 0.67459 0.796 0.000 0.188 0.016
#> GSM340244 4 0.4522 0.60306 0.000 0.320 0.000 0.680
#> GSM340246 2 0.4996 -0.08892 0.000 0.516 0.000 0.484
#> GSM340247 4 0.1474 0.73189 0.000 0.052 0.000 0.948
#> GSM340248 4 0.4500 0.60860 0.000 0.316 0.000 0.684
#> GSM340249 4 0.2469 0.77405 0.000 0.108 0.000 0.892
#> GSM340250 3 0.4957 0.59732 0.300 0.000 0.684 0.016
#> GSM340251 4 0.2469 0.77405 0.000 0.108 0.000 0.892
#> GSM340252 2 0.3706 0.70146 0.112 0.848 0.000 0.040
#> GSM340253 2 0.2908 0.71071 0.064 0.896 0.000 0.040
#> GSM340254 2 0.3706 0.70146 0.112 0.848 0.000 0.040
#> GSM340256 2 0.3706 0.70146 0.112 0.848 0.000 0.040
#> GSM340258 2 0.3356 0.73032 0.000 0.824 0.176 0.000
#> GSM340259 3 0.3810 0.70893 0.188 0.000 0.804 0.008
#> GSM340260 3 0.5026 0.57852 0.312 0.000 0.672 0.016
#> GSM340261 1 0.2530 0.71323 0.888 0.000 0.112 0.000
#> GSM340262 3 0.0188 0.70767 0.004 0.000 0.996 0.000
#> GSM340263 4 0.4193 0.66811 0.000 0.268 0.000 0.732
#> GSM340264 2 0.4855 0.51413 0.000 0.600 0.400 0.000
#> GSM340265 2 0.3356 0.73032 0.000 0.824 0.176 0.000
#> GSM340266 1 0.4462 0.69079 0.792 0.000 0.164 0.044
#> GSM340267 2 0.3356 0.73032 0.000 0.824 0.176 0.000
#> GSM340268 1 0.4680 0.69171 0.788 0.004 0.160 0.048
#> GSM340269 1 0.7696 0.55306 0.616 0.108 0.188 0.088
#> GSM340270 1 0.7936 0.52870 0.604 0.152 0.144 0.100
#> GSM537574 2 0.5719 0.66869 0.000 0.712 0.176 0.112
#> GSM537580 1 0.7387 -0.17649 0.452 0.040 0.444 0.064
#> GSM537581 1 0.7640 0.02208 0.444 0.180 0.004 0.372
#> GSM340272 3 0.0000 0.70705 0.000 0.000 1.000 0.000
#> GSM340273 4 0.4522 0.60306 0.000 0.320 0.000 0.680
#> GSM340275 4 0.2469 0.77405 0.000 0.108 0.000 0.892
#> GSM340276 1 0.2918 0.71226 0.876 0.000 0.116 0.008
#> GSM340277 2 0.3706 0.70146 0.112 0.848 0.000 0.040
#> GSM340278 1 0.2530 0.71323 0.888 0.000 0.112 0.000
#> GSM340279 1 0.4082 0.70347 0.820 0.008 0.152 0.020
#> GSM340282 3 0.0188 0.70767 0.004 0.000 0.996 0.000
#> GSM340284 4 0.2469 0.77405 0.000 0.108 0.000 0.892
#> GSM340285 1 0.2589 0.71328 0.884 0.000 0.116 0.000
#> GSM340286 3 0.0000 0.70705 0.000 0.000 1.000 0.000
#> GSM340287 1 0.6149 0.61390 0.728 0.136 0.100 0.036
#> GSM340288 3 0.3937 0.70772 0.188 0.000 0.800 0.012
#> GSM340289 3 0.4327 0.69232 0.216 0.000 0.768 0.016
#> GSM340290 1 0.4059 0.69607 0.832 0.040 0.124 0.004
#> GSM340291 4 0.4790 0.48772 0.000 0.380 0.000 0.620
#> GSM340293 1 0.4245 0.70407 0.820 0.000 0.116 0.064
#> GSM340294 1 0.2714 0.71333 0.884 0.000 0.112 0.004
#> GSM340296 1 0.2714 0.71333 0.884 0.000 0.112 0.004
#> GSM340297 1 0.2530 0.71323 0.888 0.000 0.112 0.000
#> GSM340298 1 0.2714 0.71333 0.884 0.000 0.112 0.004
#> GSM340299 3 0.0000 0.70705 0.000 0.000 1.000 0.000
#> GSM340301 3 0.0000 0.70705 0.000 0.000 1.000 0.000
#> GSM340303 2 0.3356 0.73032 0.000 0.824 0.176 0.000
#> GSM340304 1 0.8229 0.44069 0.548 0.248 0.108 0.096
#> GSM340306 3 0.7075 0.28366 0.412 0.060 0.500 0.028
#> GSM340307 2 0.4961 0.06388 0.000 0.552 0.000 0.448
#> GSM340310 3 0.5488 0.21132 0.452 0.000 0.532 0.016
#> GSM340314 1 0.2918 0.71253 0.876 0.000 0.116 0.008
#> GSM340315 2 0.7039 0.44366 0.000 0.568 0.176 0.256
#> GSM340317 4 0.2814 0.76810 0.000 0.132 0.000 0.868
#> GSM340318 1 0.5112 0.38178 0.560 0.000 0.004 0.436
#> GSM340319 1 0.4948 0.37138 0.560 0.000 0.000 0.440
#> GSM340320 1 0.2918 0.71253 0.876 0.000 0.116 0.008
#> GSM340321 4 0.4624 0.57235 0.000 0.340 0.000 0.660
#> GSM340322 1 0.8157 0.00926 0.444 0.036 0.372 0.148
#> GSM340324 2 0.3706 0.70146 0.112 0.848 0.000 0.040
#> GSM340328 1 0.6586 -0.07125 0.512 0.040 0.428 0.020
#> GSM340330 3 0.7079 0.18578 0.452 0.040 0.464 0.044
#> GSM340332 1 0.5277 0.67628 0.752 0.000 0.116 0.132
#> GSM340333 2 0.3706 0.70146 0.112 0.848 0.000 0.040
#> GSM340336 4 0.2469 0.77405 0.000 0.108 0.000 0.892
#> GSM340337 1 0.7922 0.02005 0.452 0.020 0.368 0.160
#> GSM340338 3 0.7034 0.39824 0.364 0.064 0.544 0.028
#> GSM340339 1 0.4780 0.68905 0.788 0.000 0.116 0.096
#> GSM340340 4 0.4746 0.48630 0.000 0.368 0.000 0.632
#> GSM340341 4 0.4804 0.45839 0.000 0.384 0.000 0.616
#> GSM340343 1 0.3088 0.70876 0.864 0.000 0.128 0.008
#> GSM340344 2 0.4467 0.73538 0.000 0.788 0.172 0.040
#> GSM340346 1 0.5295 -0.20599 0.504 0.000 0.488 0.008
#> GSM340347 4 0.6174 -0.19990 0.460 0.040 0.004 0.496
#> GSM340348 1 0.7701 0.16803 0.460 0.200 0.004 0.336
#> GSM340349 3 0.4933 0.61579 0.296 0.000 0.688 0.016
#> GSM340350 3 0.4933 0.60229 0.296 0.000 0.688 0.016
#> GSM340351 3 0.3810 0.70893 0.188 0.000 0.804 0.008
#> GSM340354 2 0.5118 0.70068 0.000 0.752 0.176 0.072
#> GSM340356 2 0.4713 0.34391 0.000 0.640 0.000 0.360
#> GSM340357 1 0.8689 0.18175 0.408 0.264 0.288 0.040
#> GSM348183 3 0.4428 0.63210 0.276 0.000 0.720 0.004
#> GSM348191 1 0.3074 0.70227 0.848 0.000 0.152 0.000
#> GSM348193 1 0.8802 0.36520 0.464 0.260 0.204 0.072
#> GSM537578 3 0.4214 0.70006 0.204 0.000 0.780 0.016
#> GSM348181 1 0.3401 0.69205 0.840 0.000 0.152 0.008
#> GSM348182 3 0.2401 0.71804 0.092 0.000 0.904 0.004
#> GSM348184 4 0.2589 0.77292 0.000 0.116 0.000 0.884
#> GSM348185 4 0.3074 0.75949 0.000 0.152 0.000 0.848
#> GSM348186 2 0.3706 0.70146 0.112 0.848 0.000 0.040
#> GSM348187 1 0.3300 0.70558 0.848 0.000 0.144 0.008
#> GSM348188 3 0.7007 0.48053 0.312 0.104 0.572 0.012
#> GSM348189 3 0.7146 0.17808 0.452 0.040 0.460 0.048
#> GSM348190 3 0.0000 0.70705 0.000 0.000 1.000 0.000
#> GSM348194 3 0.3937 0.70772 0.188 0.000 0.800 0.012
#> GSM348195 3 0.3810 0.70893 0.188 0.000 0.804 0.008
#> GSM348196 1 0.5388 -0.10565 0.532 0.000 0.456 0.012
#> GSM537585 2 0.3486 0.72655 0.000 0.812 0.188 0.000
#> GSM537594 2 0.3706 0.70146 0.112 0.848 0.000 0.040
#> GSM537596 1 0.5093 0.32620 0.640 0.000 0.348 0.012
#> GSM537597 2 0.5250 0.69552 0.000 0.744 0.176 0.080
#> GSM537602 1 0.7873 0.51677 0.584 0.100 0.232 0.084
#> GSM340184 1 0.9226 0.03402 0.360 0.312 0.080 0.248
#> GSM340185 2 0.4261 0.68544 0.112 0.820 0.000 0.068
#> GSM340186 2 0.4843 0.66478 0.112 0.784 0.000 0.104
#> GSM340187 4 0.1940 0.75277 0.000 0.076 0.000 0.924
#> GSM340189 1 0.6377 0.60292 0.632 0.000 0.112 0.256
#> GSM340190 4 0.2469 0.77405 0.000 0.108 0.000 0.892
#> GSM340191 1 0.2589 0.71321 0.884 0.000 0.116 0.000
#> GSM340192 2 0.4467 0.73538 0.000 0.788 0.172 0.040
#> GSM340193 3 0.3937 0.70772 0.188 0.000 0.800 0.012
#> GSM340194 3 0.0000 0.70705 0.000 0.000 1.000 0.000
#> GSM340195 3 0.0000 0.70705 0.000 0.000 1.000 0.000
#> GSM340196 1 0.4948 0.37138 0.560 0.000 0.000 0.440
#> GSM340197 3 0.0000 0.70705 0.000 0.000 1.000 0.000
#> GSM340198 2 0.3706 0.70146 0.112 0.848 0.000 0.040
#> GSM340199 1 0.2918 0.71253 0.876 0.000 0.116 0.008
#> GSM340200 2 0.4898 0.17217 0.000 0.584 0.000 0.416
#> GSM340201 1 0.5112 0.38178 0.560 0.000 0.004 0.436
#> GSM340202 4 0.6143 0.07375 0.368 0.048 0.004 0.580
#> GSM340203 1 0.6965 0.57635 0.584 0.004 0.140 0.272
#> GSM340204 2 0.4467 0.73538 0.000 0.788 0.172 0.040
#> GSM340205 1 0.3893 0.66796 0.796 0.000 0.196 0.008
#> GSM340206 1 0.7298 0.62429 0.636 0.044 0.160 0.160
#> GSM340207 3 0.3271 0.71720 0.132 0.012 0.856 0.000
#> GSM340237 4 0.4679 0.54966 0.000 0.352 0.000 0.648
#> GSM340238 1 0.2918 0.71253 0.876 0.000 0.116 0.008
#> GSM340239 3 0.0000 0.70705 0.000 0.000 1.000 0.000
#> GSM340240 2 0.4898 0.50440 0.000 0.584 0.416 0.000
#> GSM340241 2 0.3706 0.70146 0.112 0.848 0.000 0.040
#> GSM340242 3 0.7080 0.17275 0.456 0.040 0.460 0.044
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 3 0.3459 0.8012 0.052 0.000 0.832 0.000 0.116
#> GSM340359 2 0.4057 0.7300 0.120 0.792 0.000 0.088 0.000
#> GSM340361 5 0.0404 0.8047 0.012 0.000 0.000 0.000 0.988
#> GSM340362 5 0.0404 0.8047 0.012 0.000 0.000 0.000 0.988
#> GSM340363 3 0.3243 0.8196 0.036 0.012 0.860 0.000 0.092
#> GSM340364 2 0.5559 0.5167 0.344 0.572 0.000 0.084 0.000
#> GSM340365 5 0.0404 0.8047 0.012 0.000 0.000 0.000 0.988
#> GSM340366 5 0.0000 0.8064 0.000 0.000 0.000 0.000 1.000
#> GSM340367 1 0.6054 0.0683 0.496 0.000 0.124 0.000 0.380
#> GSM340368 4 0.1484 0.7838 0.000 0.048 0.008 0.944 0.000
#> GSM340369 3 0.3896 0.7663 0.028 0.048 0.828 0.096 0.000
#> GSM340370 1 0.7153 0.2876 0.540 0.124 0.088 0.248 0.000
#> GSM340371 5 0.5863 0.5961 0.140 0.004 0.204 0.008 0.644
#> GSM340372 5 0.1205 0.8012 0.040 0.000 0.004 0.000 0.956
#> GSM340373 5 0.0865 0.8043 0.024 0.000 0.004 0.000 0.972
#> GSM340375 1 0.4835 0.3615 0.724 0.188 0.000 0.084 0.004
#> GSM340376 1 0.5927 0.1558 0.540 0.120 0.000 0.340 0.000
#> GSM340378 2 0.6724 0.3740 0.296 0.420 0.000 0.284 0.000
#> GSM340243 3 0.3238 0.8173 0.124 0.004 0.848 0.004 0.020
#> GSM340244 4 0.1628 0.8001 0.008 0.056 0.000 0.936 0.000
#> GSM340246 4 0.3209 0.7149 0.008 0.180 0.000 0.812 0.000
#> GSM340247 4 0.2747 0.7354 0.004 0.060 0.048 0.888 0.000
#> GSM340248 4 0.2358 0.7790 0.008 0.104 0.000 0.888 0.000
#> GSM340249 4 0.0566 0.8073 0.000 0.012 0.004 0.984 0.000
#> GSM340250 1 0.5341 -0.0490 0.504 0.000 0.052 0.000 0.444
#> GSM340251 4 0.0613 0.8065 0.004 0.008 0.004 0.984 0.000
#> GSM340252 2 0.2471 0.7665 0.000 0.864 0.000 0.136 0.000
#> GSM340253 2 0.2628 0.7780 0.028 0.884 0.000 0.088 0.000
#> GSM340254 2 0.2074 0.7927 0.000 0.896 0.000 0.104 0.000
#> GSM340256 2 0.2074 0.7927 0.000 0.896 0.000 0.104 0.000
#> GSM340258 1 0.4835 0.3615 0.724 0.188 0.000 0.084 0.004
#> GSM340259 5 0.1331 0.8011 0.040 0.000 0.008 0.000 0.952
#> GSM340260 5 0.5350 0.1318 0.460 0.000 0.052 0.000 0.488
#> GSM340261 3 0.3154 0.8099 0.024 0.012 0.860 0.000 0.104
#> GSM340262 5 0.0000 0.8064 0.000 0.000 0.000 0.000 1.000
#> GSM340263 4 0.1357 0.8041 0.004 0.048 0.000 0.948 0.000
#> GSM340264 1 0.7279 0.3937 0.528 0.160 0.000 0.080 0.232
#> GSM340265 1 0.5040 0.3614 0.716 0.192 0.000 0.080 0.012
#> GSM340266 3 0.3483 0.8110 0.104 0.012 0.848 0.032 0.004
#> GSM340267 1 0.4955 0.3638 0.720 0.188 0.000 0.084 0.008
#> GSM340268 3 0.2925 0.8125 0.056 0.012 0.888 0.040 0.004
#> GSM340269 1 0.5050 0.3035 0.652 0.000 0.300 0.012 0.036
#> GSM340270 1 0.4336 0.3684 0.700 0.000 0.280 0.008 0.012
#> GSM537574 1 0.5856 0.1186 0.504 0.100 0.000 0.396 0.000
#> GSM537580 5 0.6572 0.3898 0.140 0.008 0.324 0.008 0.520
#> GSM537581 4 0.6298 0.3589 0.100 0.028 0.304 0.568 0.000
#> GSM340272 5 0.0000 0.8064 0.000 0.000 0.000 0.000 1.000
#> GSM340273 4 0.2411 0.7767 0.008 0.108 0.000 0.884 0.000
#> GSM340275 4 0.0486 0.8048 0.004 0.004 0.004 0.988 0.000
#> GSM340276 3 0.3749 0.7960 0.080 0.000 0.816 0.000 0.104
#> GSM340277 2 0.2074 0.7927 0.000 0.896 0.000 0.104 0.000
#> GSM340278 3 0.2692 0.8251 0.016 0.008 0.884 0.000 0.092
#> GSM340279 3 0.2462 0.8278 0.080 0.008 0.900 0.004 0.008
#> GSM340282 5 0.0000 0.8064 0.000 0.000 0.000 0.000 1.000
#> GSM340284 4 0.0613 0.8065 0.004 0.008 0.004 0.984 0.000
#> GSM340285 3 0.3187 0.8205 0.036 0.012 0.864 0.000 0.088
#> GSM340286 5 0.0404 0.8047 0.012 0.000 0.000 0.000 0.988
#> GSM340287 3 0.4451 0.5966 0.316 0.004 0.668 0.004 0.008
#> GSM340288 5 0.2017 0.7861 0.080 0.000 0.008 0.000 0.912
#> GSM340289 5 0.3409 0.7511 0.112 0.000 0.052 0.000 0.836
#> GSM340290 3 0.3838 0.8015 0.108 0.008 0.820 0.000 0.064
#> GSM340291 4 0.3171 0.7199 0.008 0.176 0.000 0.816 0.000
#> GSM340293 3 0.1988 0.8261 0.028 0.012 0.936 0.016 0.008
#> GSM340294 3 0.3134 0.8159 0.028 0.012 0.864 0.000 0.096
#> GSM340296 3 0.3076 0.8211 0.036 0.008 0.868 0.000 0.088
#> GSM340297 3 0.2900 0.8191 0.020 0.012 0.876 0.000 0.092
#> GSM340298 3 0.3593 0.8082 0.052 0.012 0.840 0.000 0.096
#> GSM340299 5 0.0404 0.8047 0.012 0.000 0.000 0.000 0.988
#> GSM340301 5 0.0000 0.8064 0.000 0.000 0.000 0.000 1.000
#> GSM340303 1 0.5040 0.3614 0.716 0.192 0.000 0.080 0.012
#> GSM340304 1 0.3666 0.4855 0.788 0.004 0.196 0.008 0.004
#> GSM340306 5 0.6645 0.5229 0.124 0.004 0.236 0.044 0.592
#> GSM340307 4 0.3246 0.7093 0.008 0.184 0.000 0.808 0.000
#> GSM340310 1 0.6273 0.1641 0.512 0.000 0.172 0.000 0.316
#> GSM340314 3 0.0290 0.8296 0.000 0.000 0.992 0.000 0.008
#> GSM340315 1 0.5496 0.2978 0.624 0.064 0.012 0.300 0.000
#> GSM340317 4 0.0404 0.8078 0.000 0.012 0.000 0.988 0.000
#> GSM340318 3 0.4415 0.7382 0.028 0.064 0.792 0.116 0.000
#> GSM340319 3 0.4415 0.7382 0.028 0.064 0.792 0.116 0.000
#> GSM340320 3 0.0613 0.8292 0.004 0.000 0.984 0.004 0.008
#> GSM340321 4 0.2843 0.7520 0.008 0.144 0.000 0.848 0.000
#> GSM340322 5 0.7536 0.3184 0.044 0.016 0.316 0.156 0.468
#> GSM340324 2 0.2074 0.7927 0.000 0.896 0.000 0.104 0.000
#> GSM340328 5 0.6349 0.2714 0.168 0.000 0.360 0.000 0.472
#> GSM340330 5 0.6190 0.4200 0.140 0.000 0.320 0.004 0.536
#> GSM340332 3 0.3365 0.7977 0.028 0.028 0.872 0.060 0.012
#> GSM340333 2 0.1851 0.7853 0.000 0.912 0.000 0.088 0.000
#> GSM340336 4 0.0613 0.8039 0.004 0.004 0.008 0.984 0.000
#> GSM340337 5 0.7545 0.3085 0.044 0.016 0.320 0.156 0.464
#> GSM340338 5 0.5575 0.6020 0.168 0.004 0.152 0.004 0.672
#> GSM340339 3 0.2739 0.8194 0.024 0.012 0.904 0.040 0.020
#> GSM340340 4 0.2563 0.7715 0.000 0.120 0.008 0.872 0.000
#> GSM340341 4 0.2136 0.7883 0.008 0.088 0.000 0.904 0.000
#> GSM340343 3 0.2100 0.8386 0.016 0.012 0.924 0.000 0.048
#> GSM340344 2 0.5719 0.5038 0.348 0.564 0.000 0.084 0.004
#> GSM340346 5 0.6022 0.4258 0.136 0.000 0.324 0.000 0.540
#> GSM340347 4 0.6432 0.2940 0.060 0.056 0.356 0.528 0.000
#> GSM340348 4 0.6409 0.2577 0.152 0.008 0.324 0.516 0.000
#> GSM340349 5 0.3888 0.7310 0.148 0.000 0.056 0.000 0.796
#> GSM340350 1 0.6028 -0.0277 0.468 0.000 0.116 0.000 0.416
#> GSM340351 5 0.1205 0.8012 0.040 0.000 0.004 0.000 0.956
#> GSM340354 1 0.4513 0.4009 0.764 0.128 0.000 0.104 0.004
#> GSM340356 4 0.4183 0.4420 0.008 0.324 0.000 0.668 0.000
#> GSM340357 1 0.5606 0.3345 0.636 0.012 0.068 0.004 0.280
#> GSM348183 5 0.1942 0.7862 0.068 0.000 0.012 0.000 0.920
#> GSM348191 3 0.3012 0.8326 0.060 0.008 0.876 0.000 0.056
#> GSM348193 1 0.6300 0.4025 0.644 0.056 0.228 0.020 0.052
#> GSM537578 5 0.2984 0.7648 0.108 0.000 0.032 0.000 0.860
#> GSM348181 3 0.4334 0.7493 0.092 0.000 0.768 0.000 0.140
#> GSM348182 5 0.0162 0.8067 0.004 0.000 0.000 0.000 0.996
#> GSM348184 4 0.0854 0.8078 0.008 0.012 0.004 0.976 0.000
#> GSM348185 4 0.1557 0.8029 0.008 0.052 0.000 0.940 0.000
#> GSM348186 2 0.2074 0.7927 0.000 0.896 0.000 0.104 0.000
#> GSM348187 3 0.3119 0.8284 0.072 0.000 0.860 0.000 0.068
#> GSM348188 5 0.4669 0.6761 0.164 0.008 0.068 0.004 0.756
#> GSM348189 5 0.5922 0.5650 0.140 0.000 0.236 0.008 0.616
#> GSM348190 5 0.0404 0.8047 0.012 0.000 0.000 0.000 0.988
#> GSM348194 5 0.2193 0.7799 0.092 0.000 0.008 0.000 0.900
#> GSM348195 5 0.1205 0.8012 0.040 0.000 0.004 0.000 0.956
#> GSM348196 3 0.6752 0.0985 0.280 0.000 0.404 0.000 0.316
#> GSM537585 1 0.6580 0.3864 0.620 0.188 0.000 0.084 0.108
#> GSM537594 2 0.2773 0.7674 0.000 0.836 0.000 0.164 0.000
#> GSM537596 3 0.5872 0.5436 0.168 0.000 0.600 0.000 0.232
#> GSM537597 1 0.5868 0.1330 0.516 0.104 0.000 0.380 0.000
#> GSM537602 1 0.5005 0.3116 0.660 0.004 0.296 0.008 0.032
#> GSM340184 1 0.5040 0.4785 0.752 0.036 0.144 0.064 0.004
#> GSM340185 2 0.4268 0.2341 0.000 0.556 0.000 0.444 0.000
#> GSM340186 2 0.4403 0.2590 0.000 0.560 0.004 0.436 0.000
#> GSM340187 4 0.1644 0.7715 0.004 0.048 0.008 0.940 0.000
#> GSM340189 3 0.4024 0.7785 0.028 0.060 0.836 0.064 0.012
#> GSM340190 4 0.0613 0.8065 0.004 0.008 0.004 0.984 0.000
#> GSM340191 3 0.1952 0.8306 0.000 0.004 0.912 0.000 0.084
#> GSM340192 2 0.5694 0.5158 0.340 0.572 0.000 0.084 0.004
#> GSM340193 5 0.2519 0.7752 0.100 0.000 0.016 0.000 0.884
#> GSM340194 5 0.0404 0.8047 0.012 0.000 0.000 0.000 0.988
#> GSM340195 5 0.0404 0.8047 0.012 0.000 0.000 0.000 0.988
#> GSM340196 3 0.4415 0.7382 0.028 0.064 0.792 0.116 0.000
#> GSM340197 5 0.0000 0.8064 0.000 0.000 0.000 0.000 1.000
#> GSM340198 2 0.2074 0.7927 0.000 0.896 0.000 0.104 0.000
#> GSM340199 3 0.1205 0.8363 0.000 0.004 0.956 0.000 0.040
#> GSM340200 4 0.3662 0.5973 0.000 0.252 0.004 0.744 0.000
#> GSM340201 3 0.4415 0.7382 0.028 0.064 0.792 0.116 0.000
#> GSM340202 4 0.6055 0.4152 0.036 0.076 0.280 0.608 0.000
#> GSM340203 3 0.3507 0.7787 0.032 0.024 0.848 0.096 0.000
#> GSM340204 2 0.5753 0.4804 0.360 0.552 0.000 0.084 0.004
#> GSM340205 3 0.3366 0.8079 0.140 0.000 0.828 0.000 0.032
#> GSM340206 3 0.4187 0.7600 0.120 0.012 0.804 0.060 0.004
#> GSM340207 5 0.1243 0.7965 0.028 0.008 0.004 0.000 0.960
#> GSM340237 4 0.2886 0.7491 0.008 0.148 0.000 0.844 0.000
#> GSM340238 3 0.1173 0.8297 0.004 0.012 0.964 0.000 0.020
#> GSM340239 5 0.0000 0.8064 0.000 0.000 0.000 0.000 1.000
#> GSM340240 1 0.7377 0.3830 0.500 0.160 0.000 0.076 0.264
#> GSM340241 2 0.2074 0.7927 0.000 0.896 0.000 0.104 0.000
#> GSM340242 5 0.5853 0.5600 0.140 0.000 0.244 0.004 0.612
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 3 0.1666 0.66574 0.008 0.000 0.936 0.020 0.036 0.000
#> GSM340359 2 0.2627 0.83533 0.064 0.884 0.000 0.016 0.000 0.036
#> GSM340361 5 0.0363 0.88018 0.012 0.000 0.000 0.000 0.988 0.000
#> GSM340362 5 0.0713 0.87752 0.028 0.000 0.000 0.000 0.972 0.000
#> GSM340363 3 0.0665 0.66588 0.004 0.000 0.980 0.008 0.008 0.000
#> GSM340364 1 0.4176 0.69412 0.720 0.232 0.000 0.012 0.000 0.036
#> GSM340365 5 0.0363 0.88018 0.012 0.000 0.000 0.000 0.988 0.000
#> GSM340366 5 0.0260 0.88006 0.008 0.000 0.000 0.000 0.992 0.000
#> GSM340367 5 0.7566 -0.15870 0.108 0.008 0.292 0.264 0.328 0.000
#> GSM340368 6 0.1958 0.86896 0.000 0.004 0.000 0.100 0.000 0.896
#> GSM340369 4 0.5150 0.22509 0.000 0.004 0.436 0.496 0.004 0.060
#> GSM340370 1 0.4268 0.73857 0.788 0.036 0.012 0.060 0.000 0.104
#> GSM340371 4 0.6341 0.53519 0.040 0.004 0.164 0.528 0.264 0.000
#> GSM340372 5 0.1965 0.86576 0.024 0.008 0.004 0.040 0.924 0.000
#> GSM340373 5 0.1067 0.87582 0.004 0.004 0.004 0.024 0.964 0.000
#> GSM340375 1 0.2815 0.75867 0.848 0.120 0.000 0.000 0.000 0.032
#> GSM340376 1 0.3676 0.73376 0.808 0.020 0.000 0.052 0.000 0.120
#> GSM340378 1 0.5419 0.68336 0.668 0.160 0.000 0.052 0.000 0.120
#> GSM340243 3 0.3536 0.61655 0.056 0.004 0.812 0.124 0.004 0.000
#> GSM340244 6 0.0972 0.89348 0.000 0.008 0.000 0.028 0.000 0.964
#> GSM340246 6 0.3017 0.83197 0.000 0.052 0.000 0.108 0.000 0.840
#> GSM340247 6 0.3376 0.79146 0.004 0.024 0.000 0.180 0.000 0.792
#> GSM340248 6 0.0914 0.89088 0.000 0.016 0.000 0.016 0.000 0.968
#> GSM340249 6 0.1320 0.89197 0.000 0.016 0.000 0.036 0.000 0.948
#> GSM340250 5 0.5880 0.45712 0.132 0.008 0.020 0.264 0.576 0.000
#> GSM340251 6 0.2006 0.87851 0.000 0.016 0.000 0.080 0.000 0.904
#> GSM340252 2 0.2322 0.86471 0.004 0.896 0.000 0.064 0.000 0.036
#> GSM340253 2 0.2380 0.84845 0.048 0.900 0.000 0.016 0.000 0.036
#> GSM340254 2 0.1124 0.88140 0.008 0.956 0.000 0.000 0.000 0.036
#> GSM340256 2 0.1124 0.88140 0.008 0.956 0.000 0.000 0.000 0.036
#> GSM340258 1 0.2815 0.75867 0.848 0.120 0.000 0.000 0.000 0.032
#> GSM340259 5 0.2017 0.86526 0.020 0.008 0.004 0.048 0.920 0.000
#> GSM340260 5 0.5369 0.61931 0.112 0.008 0.024 0.192 0.664 0.000
#> GSM340261 3 0.2510 0.61531 0.008 0.000 0.884 0.028 0.080 0.000
#> GSM340262 5 0.0260 0.88006 0.008 0.000 0.000 0.000 0.992 0.000
#> GSM340263 6 0.0622 0.89207 0.000 0.008 0.000 0.012 0.000 0.980
#> GSM340264 1 0.5929 0.42095 0.536 0.100 0.000 0.004 0.328 0.032
#> GSM340265 1 0.3024 0.75850 0.844 0.116 0.000 0.008 0.000 0.032
#> GSM340266 3 0.4487 0.56559 0.056 0.004 0.732 0.192 0.004 0.012
#> GSM340267 1 0.2913 0.75900 0.848 0.116 0.000 0.004 0.000 0.032
#> GSM340268 3 0.4399 0.55339 0.044 0.004 0.752 0.172 0.004 0.024
#> GSM340269 3 0.6534 0.20401 0.284 0.004 0.432 0.260 0.020 0.000
#> GSM340270 3 0.6387 0.16476 0.324 0.000 0.408 0.252 0.016 0.000
#> GSM537574 1 0.3854 0.69520 0.760 0.004 0.000 0.048 0.000 0.188
#> GSM537580 4 0.6460 0.53658 0.052 0.004 0.188 0.536 0.220 0.000
#> GSM537581 4 0.7309 0.33991 0.028 0.036 0.220 0.372 0.004 0.340
#> GSM340272 5 0.0260 0.88006 0.008 0.000 0.000 0.000 0.992 0.000
#> GSM340273 6 0.1088 0.88864 0.000 0.024 0.000 0.016 0.000 0.960
#> GSM340275 6 0.2163 0.87288 0.000 0.016 0.000 0.092 0.000 0.892
#> GSM340276 3 0.2307 0.65049 0.016 0.000 0.904 0.032 0.048 0.000
#> GSM340277 2 0.1124 0.88140 0.008 0.956 0.000 0.000 0.000 0.036
#> GSM340278 3 0.0520 0.66532 0.000 0.000 0.984 0.008 0.008 0.000
#> GSM340279 3 0.3566 0.61019 0.028 0.004 0.796 0.164 0.008 0.000
#> GSM340282 5 0.0260 0.88006 0.008 0.000 0.000 0.000 0.992 0.000
#> GSM340284 6 0.2006 0.87851 0.000 0.016 0.000 0.080 0.000 0.904
#> GSM340285 3 0.1155 0.66113 0.004 0.000 0.956 0.004 0.036 0.000
#> GSM340286 5 0.0547 0.87884 0.020 0.000 0.000 0.000 0.980 0.000
#> GSM340287 3 0.5976 0.29536 0.292 0.000 0.508 0.188 0.012 0.000
#> GSM340288 5 0.2263 0.85863 0.036 0.008 0.004 0.044 0.908 0.000
#> GSM340289 5 0.3358 0.82940 0.060 0.012 0.024 0.052 0.852 0.000
#> GSM340290 3 0.2669 0.64305 0.024 0.000 0.864 0.108 0.004 0.000
#> GSM340291 6 0.1700 0.87903 0.000 0.048 0.000 0.024 0.000 0.928
#> GSM340293 3 0.3611 0.54859 0.000 0.004 0.780 0.188 0.008 0.020
#> GSM340294 3 0.1401 0.66268 0.004 0.000 0.948 0.028 0.020 0.000
#> GSM340296 3 0.0891 0.66530 0.000 0.000 0.968 0.024 0.008 0.000
#> GSM340297 3 0.1321 0.66345 0.004 0.000 0.952 0.024 0.020 0.000
#> GSM340298 3 0.1053 0.66680 0.004 0.000 0.964 0.020 0.012 0.000
#> GSM340299 5 0.0713 0.87752 0.028 0.000 0.000 0.000 0.972 0.000
#> GSM340301 5 0.0260 0.88006 0.008 0.000 0.000 0.000 0.992 0.000
#> GSM340303 1 0.2957 0.75814 0.844 0.120 0.000 0.004 0.000 0.032
#> GSM340304 1 0.5499 0.16461 0.616 0.000 0.244 0.120 0.016 0.004
#> GSM340306 4 0.6547 0.54424 0.060 0.012 0.096 0.544 0.280 0.008
#> GSM340307 6 0.3076 0.83149 0.004 0.044 0.000 0.112 0.000 0.840
#> GSM340310 3 0.7678 0.06708 0.140 0.008 0.348 0.260 0.244 0.000
#> GSM340314 3 0.1674 0.64622 0.000 0.000 0.924 0.068 0.004 0.004
#> GSM340315 1 0.3452 0.65317 0.736 0.004 0.000 0.004 0.000 0.256
#> GSM340317 6 0.1320 0.89183 0.000 0.016 0.000 0.036 0.000 0.948
#> GSM340318 4 0.5693 0.32521 0.004 0.024 0.368 0.532 0.004 0.068
#> GSM340319 4 0.5693 0.32521 0.004 0.024 0.368 0.532 0.004 0.068
#> GSM340320 3 0.2243 0.63039 0.000 0.000 0.880 0.112 0.004 0.004
#> GSM340321 6 0.2263 0.85298 0.000 0.100 0.000 0.016 0.000 0.884
#> GSM340322 4 0.6521 0.55142 0.008 0.008 0.148 0.576 0.200 0.060
#> GSM340324 2 0.1124 0.88140 0.008 0.956 0.000 0.000 0.000 0.036
#> GSM340328 3 0.6009 0.34208 0.060 0.000 0.600 0.184 0.156 0.000
#> GSM340330 4 0.6401 0.53610 0.044 0.004 0.172 0.528 0.252 0.000
#> GSM340332 3 0.4952 0.10123 0.000 0.004 0.568 0.376 0.008 0.044
#> GSM340333 2 0.1464 0.87808 0.004 0.944 0.000 0.016 0.000 0.036
#> GSM340336 6 0.2257 0.86047 0.000 0.008 0.000 0.116 0.000 0.876
#> GSM340337 4 0.6325 0.54412 0.008 0.008 0.180 0.576 0.196 0.032
#> GSM340338 4 0.6443 0.52342 0.076 0.012 0.080 0.520 0.312 0.000
#> GSM340339 3 0.5023 0.03324 0.000 0.000 0.560 0.372 0.008 0.060
#> GSM340340 6 0.3563 0.80566 0.000 0.092 0.000 0.108 0.000 0.800
#> GSM340341 6 0.2860 0.84586 0.000 0.048 0.000 0.100 0.000 0.852
#> GSM340343 3 0.1390 0.66237 0.004 0.000 0.948 0.032 0.016 0.000
#> GSM340344 1 0.4225 0.68301 0.712 0.240 0.000 0.012 0.000 0.036
#> GSM340346 3 0.7025 -0.06868 0.064 0.000 0.364 0.268 0.304 0.000
#> GSM340347 4 0.6291 0.40892 0.012 0.016 0.252 0.540 0.004 0.176
#> GSM340348 4 0.7944 0.34491 0.136 0.016 0.244 0.372 0.008 0.224
#> GSM340349 5 0.5554 0.55324 0.092 0.004 0.048 0.208 0.648 0.000
#> GSM340350 5 0.6594 0.41205 0.108 0.008 0.112 0.216 0.556 0.000
#> GSM340351 5 0.1819 0.86833 0.024 0.008 0.004 0.032 0.932 0.000
#> GSM340354 1 0.2918 0.74530 0.864 0.020 0.000 0.032 0.000 0.084
#> GSM340356 6 0.3196 0.82040 0.000 0.064 0.000 0.108 0.000 0.828
#> GSM340357 3 0.7727 0.00439 0.260 0.004 0.296 0.276 0.164 0.000
#> GSM348183 5 0.1623 0.86049 0.020 0.004 0.004 0.032 0.940 0.000
#> GSM348191 3 0.1549 0.66776 0.024 0.004 0.944 0.024 0.004 0.000
#> GSM348193 4 0.6873 0.29770 0.364 0.008 0.196 0.396 0.028 0.008
#> GSM537578 5 0.3593 0.80518 0.060 0.008 0.016 0.088 0.828 0.000
#> GSM348181 3 0.3023 0.63662 0.024 0.004 0.868 0.048 0.056 0.000
#> GSM348182 5 0.0405 0.88046 0.008 0.000 0.004 0.000 0.988 0.000
#> GSM348184 6 0.1461 0.89053 0.000 0.016 0.000 0.044 0.000 0.940
#> GSM348185 6 0.0632 0.89136 0.000 0.024 0.000 0.000 0.000 0.976
#> GSM348186 2 0.1124 0.88140 0.008 0.956 0.000 0.000 0.000 0.036
#> GSM348187 3 0.2001 0.66940 0.032 0.004 0.924 0.012 0.028 0.000
#> GSM348188 4 0.6216 0.41850 0.080 0.016 0.040 0.492 0.372 0.000
#> GSM348189 4 0.6517 0.53905 0.056 0.004 0.168 0.524 0.248 0.000
#> GSM348190 5 0.0363 0.88018 0.012 0.000 0.000 0.000 0.988 0.000
#> GSM348194 5 0.2999 0.83680 0.056 0.008 0.012 0.056 0.868 0.000
#> GSM348195 5 0.1893 0.86680 0.024 0.008 0.004 0.036 0.928 0.000
#> GSM348196 3 0.6479 0.28308 0.068 0.004 0.548 0.160 0.220 0.000
#> GSM537585 1 0.4469 0.73717 0.772 0.124 0.000 0.052 0.020 0.032
#> GSM537594 2 0.4691 0.66988 0.000 0.672 0.000 0.108 0.000 0.220
#> GSM537596 3 0.5767 0.41280 0.064 0.004 0.640 0.104 0.188 0.000
#> GSM537597 1 0.3819 0.70207 0.768 0.004 0.000 0.052 0.000 0.176
#> GSM537602 3 0.6981 0.14905 0.312 0.000 0.388 0.244 0.052 0.004
#> GSM340184 1 0.5591 0.42501 0.672 0.004 0.168 0.084 0.004 0.068
#> GSM340185 2 0.5283 0.58025 0.000 0.588 0.000 0.148 0.000 0.264
#> GSM340186 2 0.5411 0.52707 0.000 0.560 0.000 0.152 0.000 0.288
#> GSM340187 6 0.2821 0.82641 0.000 0.016 0.000 0.152 0.000 0.832
#> GSM340189 4 0.5308 0.27395 0.000 0.012 0.408 0.516 0.004 0.060
#> GSM340190 6 0.2006 0.87851 0.000 0.016 0.000 0.080 0.000 0.904
#> GSM340191 3 0.0632 0.65808 0.000 0.000 0.976 0.024 0.000 0.000
#> GSM340192 1 0.4105 0.68996 0.720 0.236 0.000 0.008 0.000 0.036
#> GSM340193 5 0.2960 0.83584 0.056 0.008 0.008 0.060 0.868 0.000
#> GSM340194 5 0.0858 0.87586 0.028 0.004 0.000 0.000 0.968 0.000
#> GSM340195 5 0.0547 0.87917 0.020 0.000 0.000 0.000 0.980 0.000
#> GSM340196 4 0.5693 0.32521 0.004 0.024 0.368 0.532 0.004 0.068
#> GSM340197 5 0.0260 0.88006 0.008 0.000 0.000 0.000 0.992 0.000
#> GSM340198 2 0.2322 0.86471 0.004 0.896 0.000 0.064 0.000 0.036
#> GSM340199 3 0.0937 0.65604 0.000 0.000 0.960 0.040 0.000 0.000
#> GSM340200 6 0.4218 0.74477 0.004 0.136 0.000 0.112 0.000 0.748
#> GSM340201 4 0.5693 0.32521 0.004 0.024 0.368 0.532 0.004 0.068
#> GSM340202 4 0.6759 0.22042 0.012 0.024 0.220 0.384 0.000 0.360
#> GSM340203 3 0.5532 -0.14201 0.016 0.012 0.484 0.440 0.004 0.044
#> GSM340204 1 0.3867 0.70795 0.744 0.216 0.000 0.004 0.000 0.036
#> GSM340205 3 0.3980 0.61739 0.076 0.004 0.788 0.120 0.012 0.000
#> GSM340206 3 0.5975 -0.07192 0.060 0.004 0.464 0.424 0.004 0.044
#> GSM340207 5 0.2002 0.81829 0.076 0.012 0.000 0.004 0.908 0.000
#> GSM340237 6 0.1245 0.88698 0.000 0.032 0.000 0.016 0.000 0.952
#> GSM340238 3 0.2110 0.63303 0.000 0.000 0.900 0.084 0.004 0.012
#> GSM340239 5 0.0260 0.88006 0.008 0.000 0.000 0.000 0.992 0.000
#> GSM340240 1 0.5974 0.21258 0.444 0.104 0.000 0.000 0.420 0.032
#> GSM340241 2 0.1692 0.87396 0.008 0.932 0.000 0.012 0.000 0.048
#> GSM340242 4 0.6477 0.52354 0.040 0.004 0.192 0.508 0.256 0.000
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
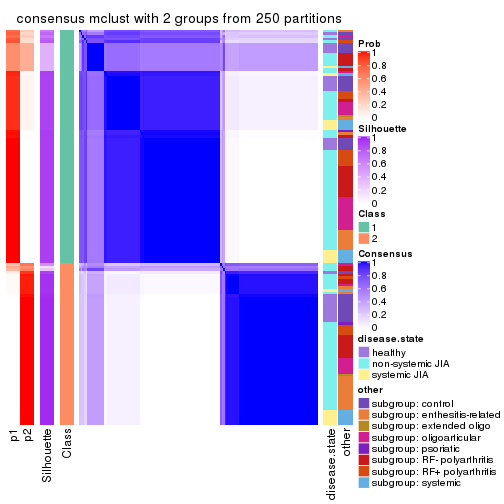
consensus_heatmap(res, k = 3)
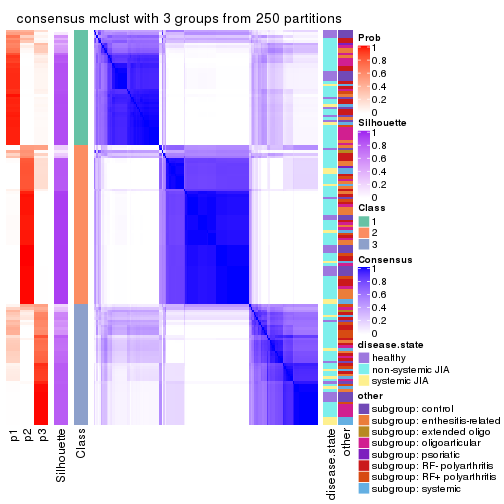
consensus_heatmap(res, k = 4)
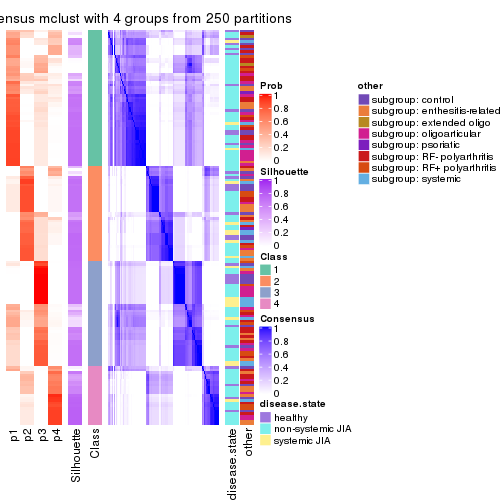
consensus_heatmap(res, k = 5)
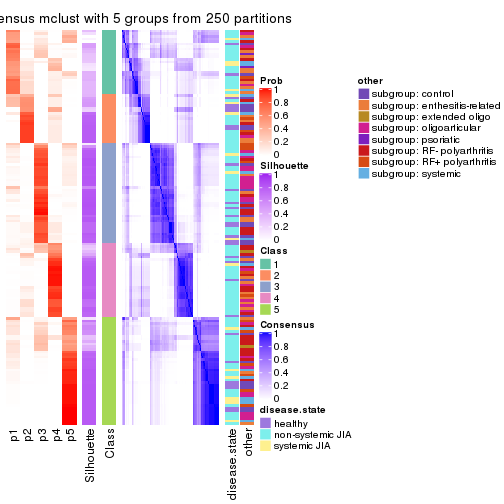
consensus_heatmap(res, k = 6)
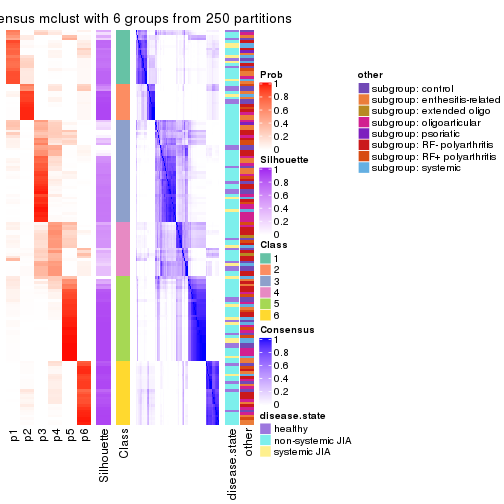
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
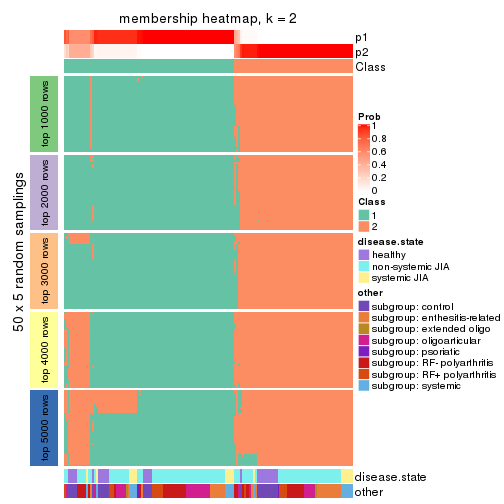
membership_heatmap(res, k = 3)
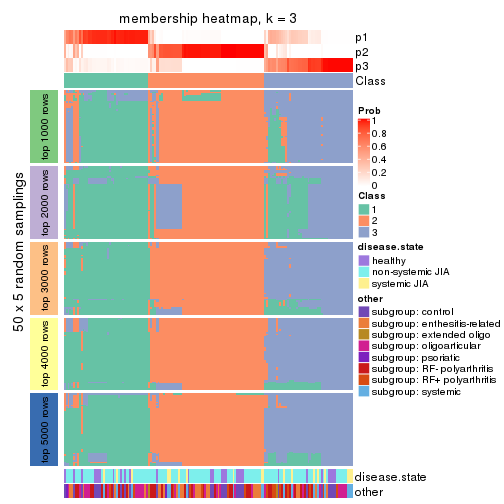
membership_heatmap(res, k = 4)
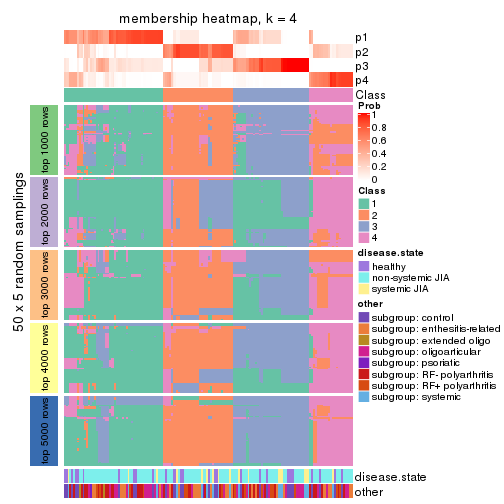
membership_heatmap(res, k = 5)
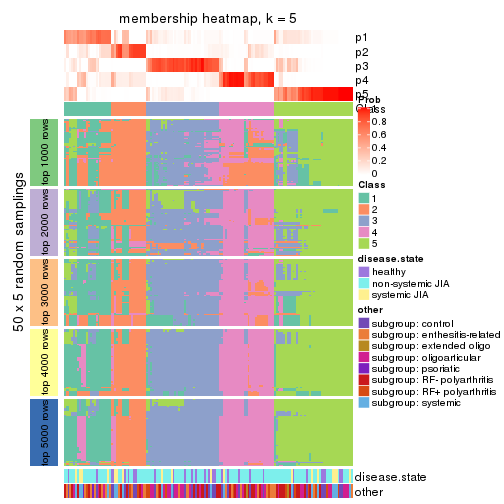
membership_heatmap(res, k = 6)
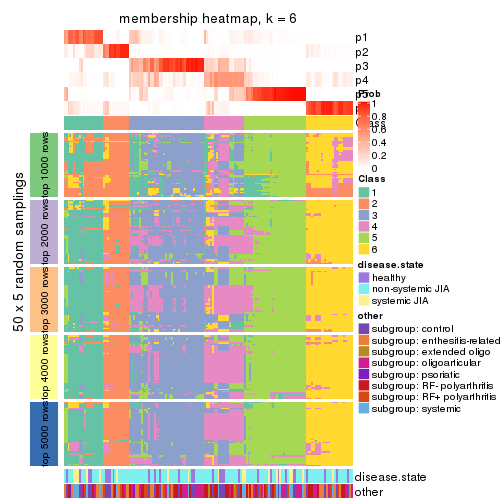
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
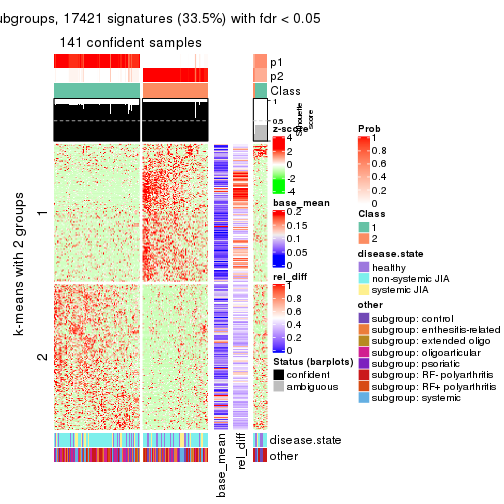
get_signatures(res, k = 3)
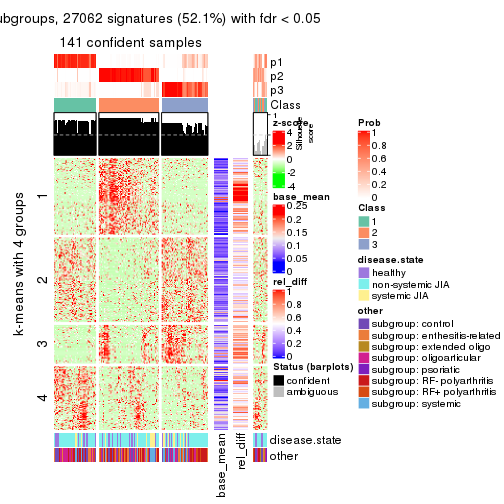
get_signatures(res, k = 4)
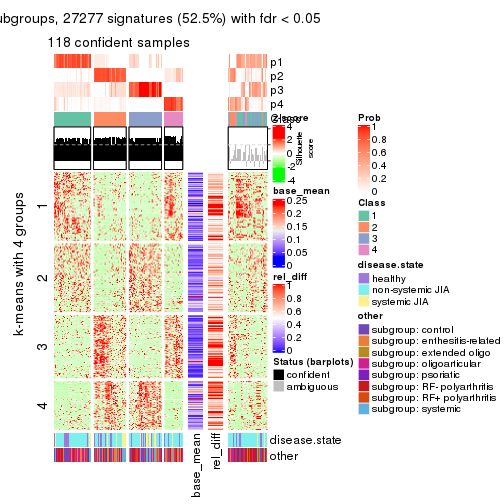
get_signatures(res, k = 5)
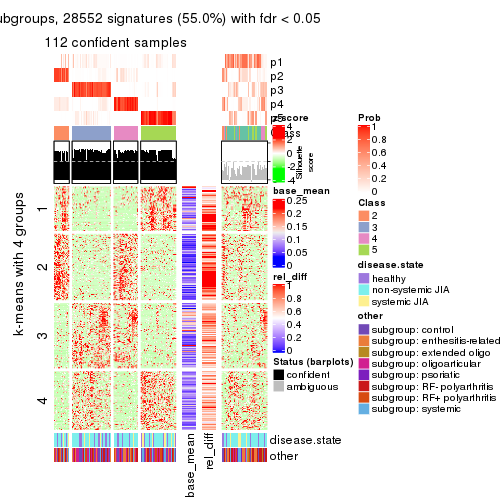
get_signatures(res, k = 6)
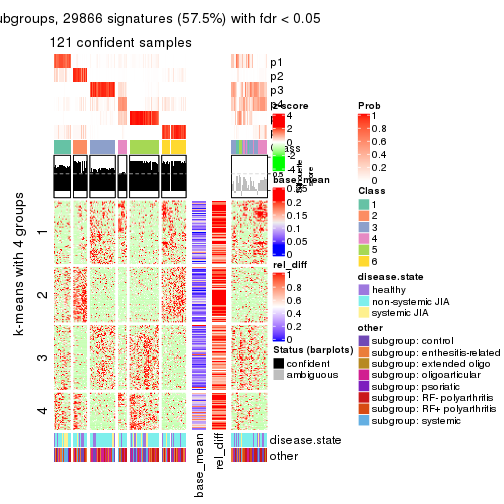
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
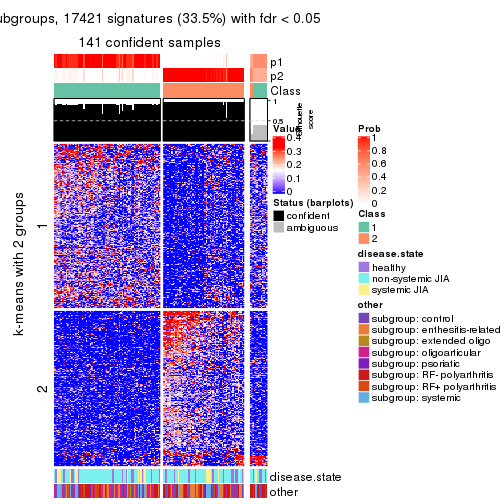
get_signatures(res, k = 3, scale_rows = FALSE)
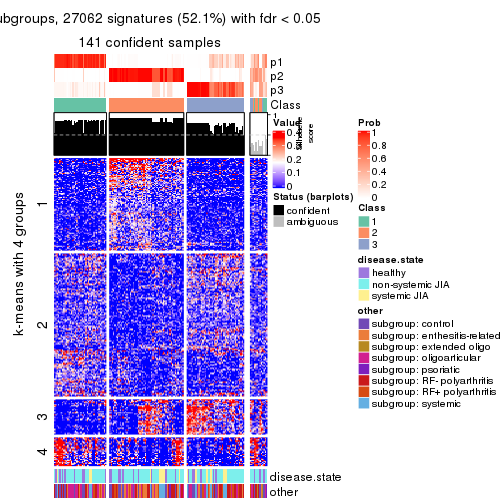
get_signatures(res, k = 4, scale_rows = FALSE)
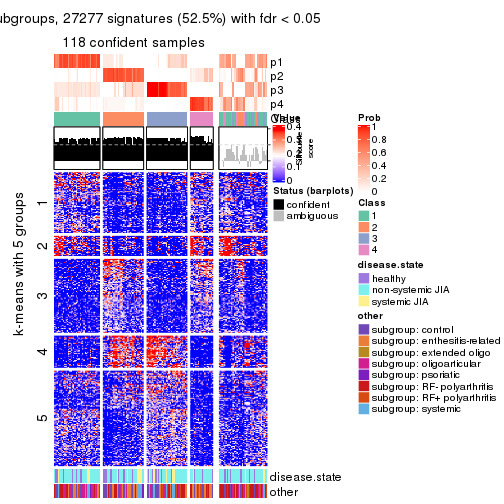
get_signatures(res, k = 5, scale_rows = FALSE)
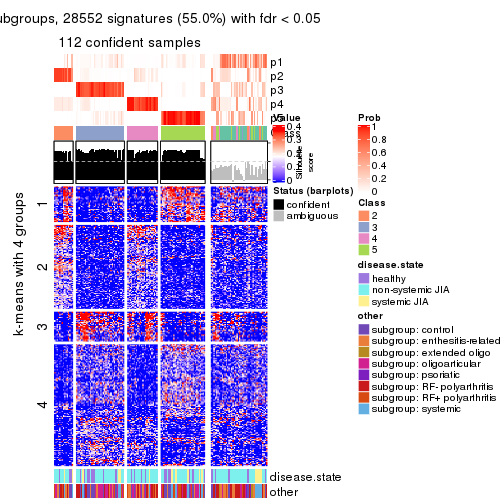
get_signatures(res, k = 6, scale_rows = FALSE)
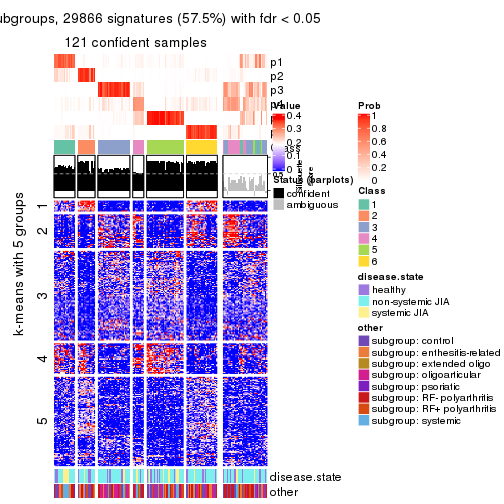
Compare the overlap of signatures from different k:
compare_signatures(res)
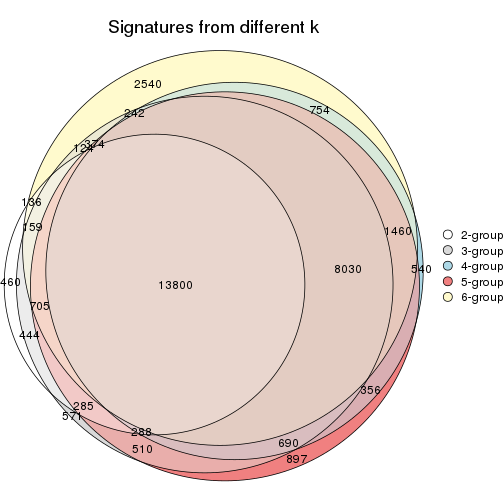
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
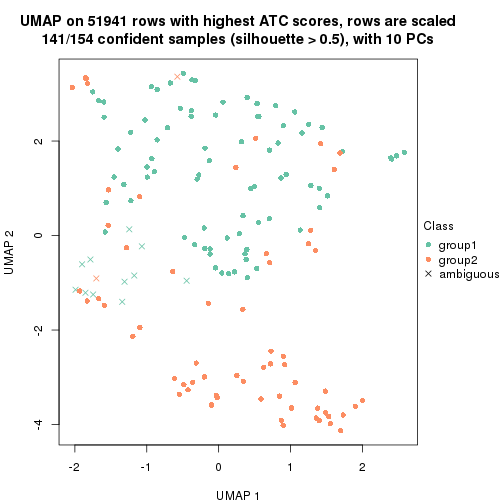
dimension_reduction(res, k = 3, method = "UMAP")
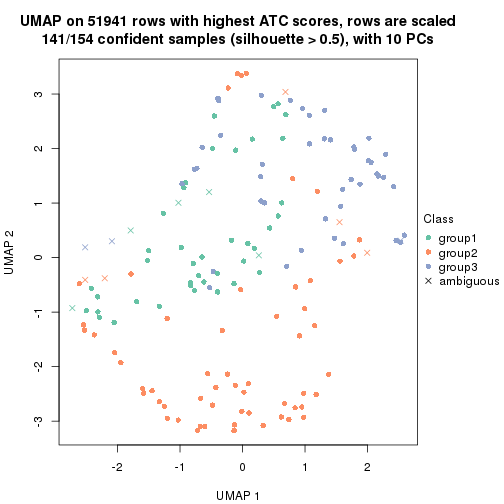
dimension_reduction(res, k = 4, method = "UMAP")
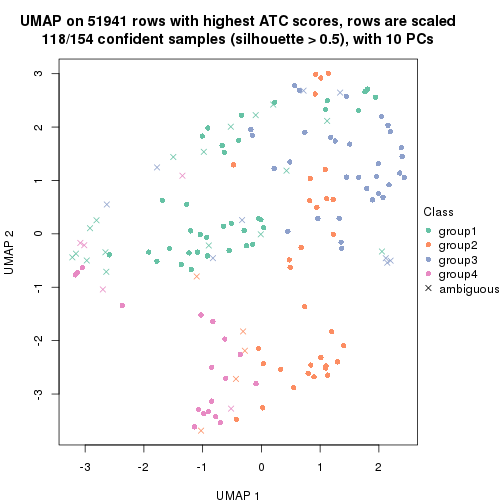
dimension_reduction(res, k = 5, method = "UMAP")
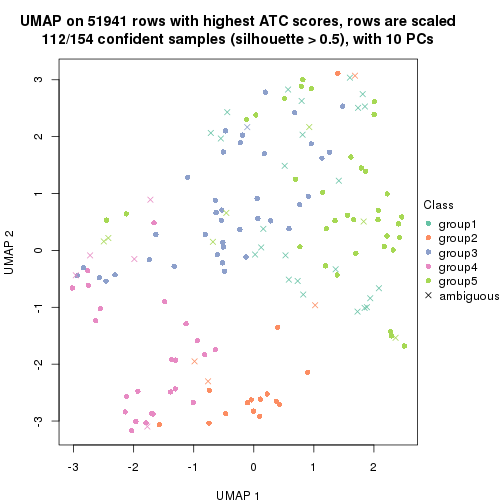
dimension_reduction(res, k = 6, method = "UMAP")
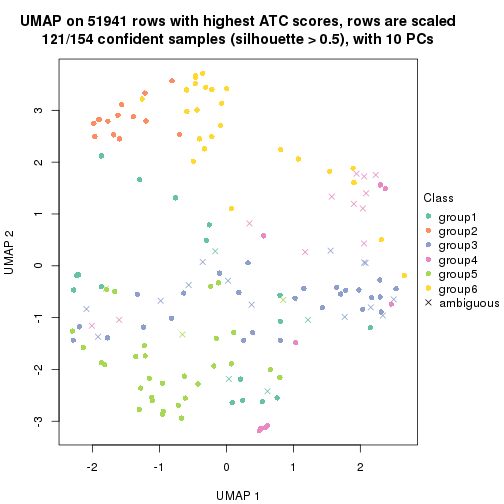
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
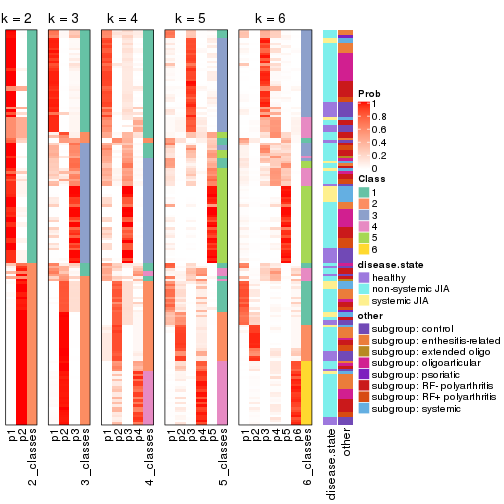
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> ATC:mclust 141 0.866 0.576 2
#> ATC:mclust 141 0.644 0.306 3
#> ATC:mclust 118 0.696 0.698 4
#> ATC:mclust 112 0.557 0.400 5
#> ATC:mclust 121 0.424 0.109 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["ATC", "NMF"]
# you can also extract it by
# res = res_list["ATC:NMF"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 51941 rows and 154 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'ATC' method.
#> Subgroups are detected by 'NMF' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
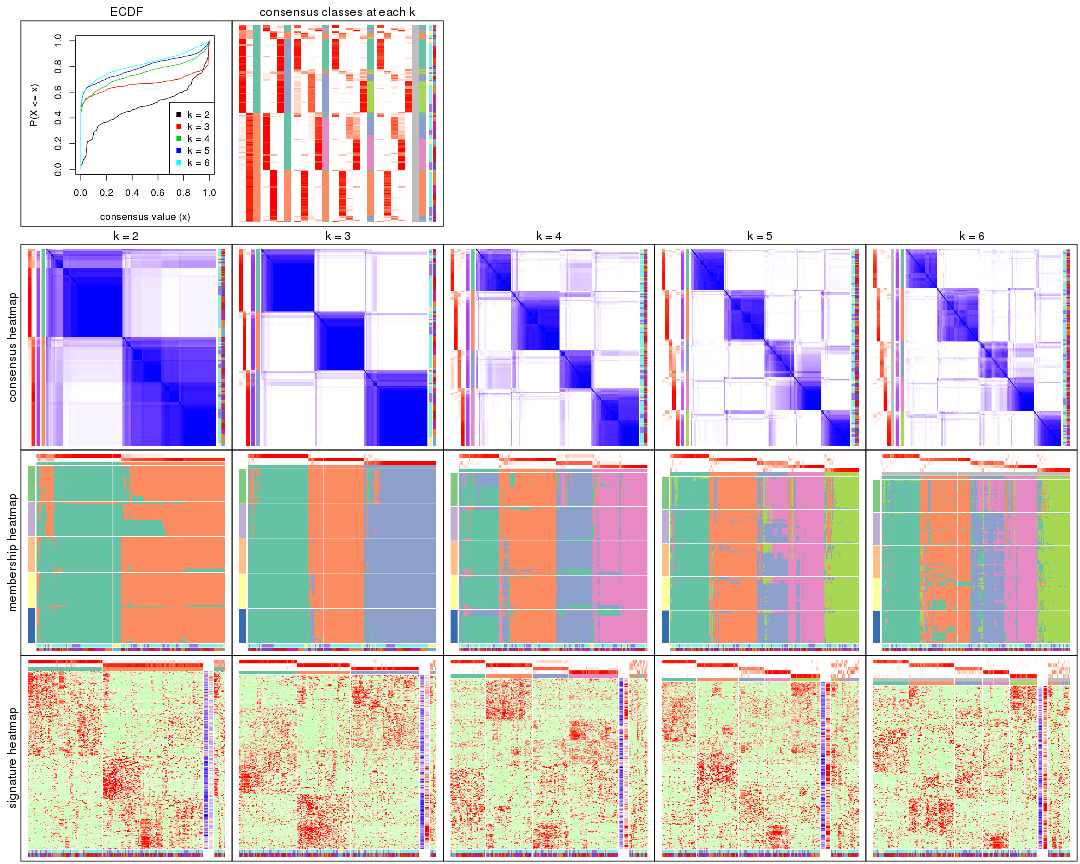
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
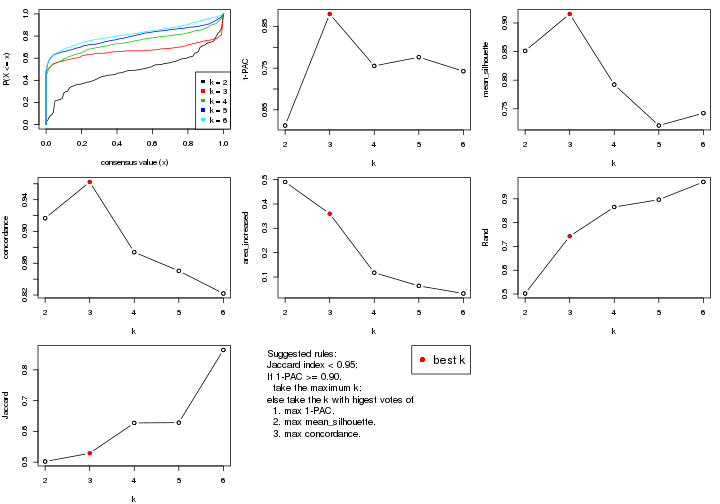
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.612 0.851 0.917 0.4902 0.502 0.502
#> 3 3 0.880 0.916 0.962 0.3599 0.743 0.529
#> 4 4 0.755 0.792 0.874 0.1177 0.866 0.627
#> 5 5 0.776 0.721 0.850 0.0636 0.897 0.628
#> 6 6 0.743 0.742 0.822 0.0322 0.971 0.864
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM340358 2 0.8763 0.6404 0.296 0.704
#> GSM340359 1 0.9732 0.4190 0.596 0.404
#> GSM340361 1 0.0000 0.9147 1.000 0.000
#> GSM340362 1 0.0000 0.9147 1.000 0.000
#> GSM340363 2 0.7528 0.7690 0.216 0.784
#> GSM340364 1 0.9358 0.5277 0.648 0.352
#> GSM340365 1 0.0000 0.9147 1.000 0.000
#> GSM340366 1 0.0000 0.9147 1.000 0.000
#> GSM340367 1 0.0000 0.9147 1.000 0.000
#> GSM340368 2 0.0000 0.9063 0.000 1.000
#> GSM340369 2 0.5408 0.8730 0.124 0.876
#> GSM340370 1 0.9710 0.4283 0.600 0.400
#> GSM340371 1 0.0938 0.9079 0.988 0.012
#> GSM340372 1 0.0000 0.9147 1.000 0.000
#> GSM340373 1 0.0000 0.9147 1.000 0.000
#> GSM340375 1 0.8443 0.6346 0.728 0.272
#> GSM340376 2 0.2603 0.9122 0.044 0.956
#> GSM340378 2 0.2603 0.9122 0.044 0.956
#> GSM340243 2 0.0376 0.9073 0.004 0.996
#> GSM340244 2 0.2603 0.9122 0.044 0.956
#> GSM340246 2 0.2603 0.9122 0.044 0.956
#> GSM340247 2 0.0000 0.9063 0.000 1.000
#> GSM340248 2 0.2603 0.9122 0.044 0.956
#> GSM340249 2 0.0938 0.9095 0.012 0.988
#> GSM340250 1 0.0000 0.9147 1.000 0.000
#> GSM340251 2 0.0938 0.9095 0.012 0.988
#> GSM340252 2 0.2603 0.9122 0.044 0.956
#> GSM340253 1 0.9635 0.4547 0.612 0.388
#> GSM340254 2 0.4161 0.8868 0.084 0.916
#> GSM340256 2 0.2603 0.9122 0.044 0.956
#> GSM340258 1 0.2603 0.8853 0.956 0.044
#> GSM340259 1 0.0000 0.9147 1.000 0.000
#> GSM340260 1 0.0000 0.9147 1.000 0.000
#> GSM340261 2 0.5408 0.8730 0.124 0.876
#> GSM340262 1 0.0000 0.9147 1.000 0.000
#> GSM340263 2 0.2603 0.9122 0.044 0.956
#> GSM340264 1 0.0000 0.9147 1.000 0.000
#> GSM340265 1 0.0000 0.9147 1.000 0.000
#> GSM340266 2 0.4562 0.8915 0.096 0.904
#> GSM340267 1 0.6438 0.7697 0.836 0.164
#> GSM340268 2 0.0376 0.9073 0.004 0.996
#> GSM340269 1 0.8081 0.6526 0.752 0.248
#> GSM340270 1 0.9963 0.0588 0.536 0.464
#> GSM537574 2 0.2603 0.9122 0.044 0.956
#> GSM537580 1 0.2603 0.8847 0.956 0.044
#> GSM537581 2 0.4562 0.8915 0.096 0.904
#> GSM340272 1 0.0000 0.9147 1.000 0.000
#> GSM340273 2 0.2603 0.9122 0.044 0.956
#> GSM340275 2 0.0000 0.9063 0.000 1.000
#> GSM340276 1 0.7376 0.7317 0.792 0.208
#> GSM340277 2 0.3879 0.8930 0.076 0.924
#> GSM340278 2 0.4562 0.8915 0.096 0.904
#> GSM340279 2 0.4815 0.8868 0.104 0.896
#> GSM340282 1 0.0000 0.9147 1.000 0.000
#> GSM340284 2 0.0938 0.9095 0.012 0.988
#> GSM340285 2 0.8443 0.6829 0.272 0.728
#> GSM340286 1 0.0000 0.9147 1.000 0.000
#> GSM340287 2 0.2603 0.9122 0.044 0.956
#> GSM340288 1 0.0000 0.9147 1.000 0.000
#> GSM340289 1 0.0000 0.9147 1.000 0.000
#> GSM340290 2 0.4690 0.8893 0.100 0.900
#> GSM340291 2 0.2603 0.9122 0.044 0.956
#> GSM340293 2 0.4562 0.8915 0.096 0.904
#> GSM340294 2 0.4562 0.8915 0.096 0.904
#> GSM340296 2 0.1184 0.9088 0.016 0.984
#> GSM340297 2 0.4562 0.8915 0.096 0.904
#> GSM340298 2 0.6148 0.8462 0.152 0.848
#> GSM340299 1 0.0000 0.9147 1.000 0.000
#> GSM340301 1 0.0000 0.9147 1.000 0.000
#> GSM340303 1 0.0938 0.9080 0.988 0.012
#> GSM340304 2 0.8555 0.7140 0.280 0.720
#> GSM340306 1 0.9988 -0.0235 0.520 0.480
#> GSM340307 2 0.2603 0.9122 0.044 0.956
#> GSM340310 1 0.0000 0.9147 1.000 0.000
#> GSM340314 2 0.5408 0.8730 0.124 0.876
#> GSM340315 2 0.2603 0.9122 0.044 0.956
#> GSM340317 2 0.2236 0.9122 0.036 0.964
#> GSM340318 2 0.4562 0.8915 0.096 0.904
#> GSM340319 2 0.4562 0.8915 0.096 0.904
#> GSM340320 2 0.4562 0.8915 0.096 0.904
#> GSM340321 2 0.2603 0.9122 0.044 0.956
#> GSM340322 2 0.9608 0.4518 0.384 0.616
#> GSM340324 2 0.2603 0.9122 0.044 0.956
#> GSM340328 2 0.9686 0.4220 0.396 0.604
#> GSM340330 1 0.2603 0.8847 0.956 0.044
#> GSM340332 2 0.5408 0.8730 0.124 0.876
#> GSM340333 1 0.9710 0.4283 0.600 0.400
#> GSM340336 2 0.0000 0.9063 0.000 1.000
#> GSM340337 2 0.8713 0.6478 0.292 0.708
#> GSM340338 1 0.2603 0.8847 0.956 0.044
#> GSM340339 2 0.5408 0.8730 0.124 0.876
#> GSM340340 2 0.0376 0.9075 0.004 0.996
#> GSM340341 2 0.1843 0.9119 0.028 0.972
#> GSM340343 2 0.7674 0.7575 0.224 0.776
#> GSM340344 1 0.6438 0.7696 0.836 0.164
#> GSM340346 1 0.2043 0.8944 0.968 0.032
#> GSM340347 2 0.4562 0.8915 0.096 0.904
#> GSM340348 2 0.1184 0.9089 0.016 0.984
#> GSM340349 1 0.0000 0.9147 1.000 0.000
#> GSM340350 1 0.0000 0.9147 1.000 0.000
#> GSM340351 1 0.0000 0.9147 1.000 0.000
#> GSM340354 2 0.2603 0.9122 0.044 0.956
#> GSM340356 2 0.2603 0.9122 0.044 0.956
#> GSM340357 1 0.0000 0.9147 1.000 0.000
#> GSM348183 1 0.0000 0.9147 1.000 0.000
#> GSM348191 2 0.4562 0.8915 0.096 0.904
#> GSM348193 1 0.8081 0.6234 0.752 0.248
#> GSM537578 1 0.0000 0.9147 1.000 0.000
#> GSM348181 1 0.3114 0.8809 0.944 0.056
#> GSM348182 1 0.0000 0.9147 1.000 0.000
#> GSM348184 2 0.1843 0.9119 0.028 0.972
#> GSM348185 2 0.2603 0.9122 0.044 0.956
#> GSM348186 2 0.2603 0.9122 0.044 0.956
#> GSM348187 1 0.0376 0.9126 0.996 0.004
#> GSM348188 1 0.0938 0.9080 0.988 0.012
#> GSM348189 1 0.0938 0.9079 0.988 0.012
#> GSM348190 1 0.0000 0.9147 1.000 0.000
#> GSM348194 1 0.0000 0.9147 1.000 0.000
#> GSM348195 1 0.0000 0.9147 1.000 0.000
#> GSM348196 1 0.2043 0.8942 0.968 0.032
#> GSM537585 1 0.0000 0.9147 1.000 0.000
#> GSM537594 2 0.2603 0.9122 0.044 0.956
#> GSM537596 1 0.0672 0.9104 0.992 0.008
#> GSM537597 2 0.2603 0.9122 0.044 0.956
#> GSM537602 1 0.0000 0.9147 1.000 0.000
#> GSM340184 2 0.2603 0.9122 0.044 0.956
#> GSM340185 2 0.2603 0.9122 0.044 0.956
#> GSM340186 2 0.2603 0.9122 0.044 0.956
#> GSM340187 2 0.0000 0.9063 0.000 1.000
#> GSM340189 2 0.5408 0.8730 0.124 0.876
#> GSM340190 2 0.0938 0.9095 0.012 0.988
#> GSM340191 2 0.4562 0.8915 0.096 0.904
#> GSM340192 1 0.9795 0.3894 0.584 0.416
#> GSM340193 1 0.0000 0.9147 1.000 0.000
#> GSM340194 1 0.0000 0.9147 1.000 0.000
#> GSM340195 1 0.0000 0.9147 1.000 0.000
#> GSM340196 2 0.4690 0.8893 0.100 0.900
#> GSM340197 1 0.0000 0.9147 1.000 0.000
#> GSM340198 2 0.2603 0.9122 0.044 0.956
#> GSM340199 2 0.5408 0.8730 0.124 0.876
#> GSM340200 2 0.2603 0.9122 0.044 0.956
#> GSM340201 2 0.4562 0.8915 0.096 0.904
#> GSM340202 2 0.2236 0.9076 0.036 0.964
#> GSM340203 2 0.3584 0.9007 0.068 0.932
#> GSM340204 1 0.8763 0.6218 0.704 0.296
#> GSM340205 2 0.6048 0.8718 0.148 0.852
#> GSM340206 2 0.0000 0.9063 0.000 1.000
#> GSM340207 1 0.0000 0.9147 1.000 0.000
#> GSM340237 2 0.2603 0.9122 0.044 0.956
#> GSM340238 2 0.5408 0.8730 0.124 0.876
#> GSM340239 1 0.0000 0.9147 1.000 0.000
#> GSM340240 1 0.0000 0.9147 1.000 0.000
#> GSM340241 2 0.2603 0.9122 0.044 0.956
#> GSM340242 1 0.0938 0.9079 0.988 0.012
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM340358 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340359 2 0.0747 0.952 0.000 0.984 0.016
#> GSM340361 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340362 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340363 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340364 2 0.1860 0.921 0.000 0.948 0.052
#> GSM340365 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340366 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340367 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340368 1 0.1860 0.924 0.948 0.052 0.000
#> GSM340369 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340370 2 0.5291 0.624 0.000 0.732 0.268
#> GSM340371 3 0.4555 0.767 0.200 0.000 0.800
#> GSM340372 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340373 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340375 3 0.5016 0.692 0.000 0.240 0.760
#> GSM340376 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340378 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340243 1 0.2796 0.883 0.908 0.092 0.000
#> GSM340244 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340246 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340247 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340248 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340249 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340250 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340251 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340252 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340253 2 0.5810 0.481 0.000 0.664 0.336
#> GSM340254 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340256 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340258 3 0.1289 0.931 0.000 0.032 0.968
#> GSM340259 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340260 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340261 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340262 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340263 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340264 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340265 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340266 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340267 3 0.3619 0.834 0.000 0.136 0.864
#> GSM340268 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340269 3 0.2446 0.911 0.012 0.052 0.936
#> GSM340270 3 0.4137 0.860 0.032 0.096 0.872
#> GSM537574 2 0.0000 0.963 0.000 1.000 0.000
#> GSM537580 1 0.0237 0.962 0.996 0.000 0.004
#> GSM537581 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340272 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340273 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340275 2 0.4605 0.735 0.204 0.796 0.000
#> GSM340276 1 0.4228 0.820 0.844 0.008 0.148
#> GSM340277 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340278 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340279 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340282 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340284 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340285 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340286 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340287 2 0.4178 0.782 0.172 0.828 0.000
#> GSM340288 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340289 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340290 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340291 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340293 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340294 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340296 1 0.2356 0.904 0.928 0.072 0.000
#> GSM340297 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340298 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340299 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340301 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340303 3 0.0424 0.948 0.000 0.008 0.992
#> GSM340304 3 0.4002 0.807 0.000 0.160 0.840
#> GSM340306 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340307 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340310 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340314 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340315 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340317 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340318 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340319 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340320 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340321 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340322 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340324 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340328 1 0.1411 0.938 0.964 0.000 0.036
#> GSM340330 1 0.1163 0.944 0.972 0.000 0.028
#> GSM340332 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340333 2 0.0747 0.952 0.000 0.984 0.016
#> GSM340336 1 0.5988 0.415 0.632 0.368 0.000
#> GSM340337 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340338 3 0.3412 0.839 0.124 0.000 0.876
#> GSM340339 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340340 2 0.6140 0.313 0.404 0.596 0.000
#> GSM340341 2 0.0424 0.958 0.008 0.992 0.000
#> GSM340343 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340344 3 0.4291 0.781 0.000 0.180 0.820
#> GSM340346 3 0.3116 0.869 0.108 0.000 0.892
#> GSM340347 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340348 1 0.4452 0.754 0.808 0.192 0.000
#> GSM340349 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340350 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340351 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340354 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340356 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340357 3 0.0000 0.953 0.000 0.000 1.000
#> GSM348183 3 0.0000 0.953 0.000 0.000 1.000
#> GSM348191 1 0.0000 0.965 1.000 0.000 0.000
#> GSM348193 3 0.5926 0.422 0.356 0.000 0.644
#> GSM537578 3 0.0000 0.953 0.000 0.000 1.000
#> GSM348181 1 0.5397 0.597 0.720 0.000 0.280
#> GSM348182 3 0.0000 0.953 0.000 0.000 1.000
#> GSM348184 2 0.0000 0.963 0.000 1.000 0.000
#> GSM348185 2 0.0000 0.963 0.000 1.000 0.000
#> GSM348186 2 0.0000 0.963 0.000 1.000 0.000
#> GSM348187 3 0.0237 0.950 0.000 0.004 0.996
#> GSM348188 3 0.0237 0.950 0.004 0.000 0.996
#> GSM348189 3 0.4842 0.735 0.224 0.000 0.776
#> GSM348190 3 0.0000 0.953 0.000 0.000 1.000
#> GSM348194 3 0.0000 0.953 0.000 0.000 1.000
#> GSM348195 3 0.0000 0.953 0.000 0.000 1.000
#> GSM348196 3 0.1031 0.937 0.024 0.000 0.976
#> GSM537585 3 0.0000 0.953 0.000 0.000 1.000
#> GSM537594 2 0.0000 0.963 0.000 1.000 0.000
#> GSM537596 3 0.0000 0.953 0.000 0.000 1.000
#> GSM537597 2 0.0000 0.963 0.000 1.000 0.000
#> GSM537602 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340184 2 0.1163 0.942 0.028 0.972 0.000
#> GSM340185 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340186 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340187 1 0.1031 0.947 0.976 0.024 0.000
#> GSM340189 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340190 2 0.1031 0.946 0.024 0.976 0.000
#> GSM340191 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340192 2 0.1289 0.939 0.000 0.968 0.032
#> GSM340193 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340194 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340195 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340196 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340197 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340198 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340199 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340200 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340201 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340202 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340203 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340204 3 0.6244 0.228 0.000 0.440 0.560
#> GSM340205 1 0.7227 0.654 0.704 0.200 0.096
#> GSM340206 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340207 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340237 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340238 1 0.0000 0.965 1.000 0.000 0.000
#> GSM340239 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340240 3 0.0000 0.953 0.000 0.000 1.000
#> GSM340241 2 0.0000 0.963 0.000 1.000 0.000
#> GSM340242 3 0.4702 0.751 0.212 0.000 0.788
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM340358 4 0.4679 0.4013 0.352 0.000 0.000 0.648
#> GSM340359 2 0.0804 0.8897 0.008 0.980 0.012 0.000
#> GSM340361 1 0.4356 0.4288 0.708 0.000 0.292 0.000
#> GSM340362 3 0.3764 0.8850 0.216 0.000 0.784 0.000
#> GSM340363 4 0.0469 0.9000 0.012 0.000 0.000 0.988
#> GSM340364 2 0.4936 0.3870 0.372 0.624 0.004 0.000
#> GSM340365 3 0.4008 0.8663 0.244 0.000 0.756 0.000
#> GSM340366 3 0.3764 0.8850 0.216 0.000 0.784 0.000
#> GSM340367 1 0.0188 0.8603 0.996 0.000 0.004 0.000
#> GSM340368 4 0.2216 0.8393 0.000 0.092 0.000 0.908
#> GSM340369 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340370 1 0.3300 0.7497 0.848 0.144 0.008 0.000
#> GSM340371 3 0.4755 0.6562 0.040 0.000 0.760 0.200
#> GSM340372 1 0.0707 0.8573 0.980 0.000 0.020 0.000
#> GSM340373 1 0.2216 0.8001 0.908 0.000 0.092 0.000
#> GSM340375 2 0.7576 0.1596 0.308 0.472 0.220 0.000
#> GSM340376 1 0.6324 0.3339 0.572 0.356 0.072 0.000
#> GSM340378 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM340243 1 0.7198 0.1939 0.488 0.028 0.068 0.416
#> GSM340244 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM340246 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM340247 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340248 2 0.0921 0.8907 0.000 0.972 0.028 0.000
#> GSM340249 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM340250 3 0.4250 0.8398 0.276 0.000 0.724 0.000
#> GSM340251 2 0.0707 0.8923 0.000 0.980 0.020 0.000
#> GSM340252 2 0.0469 0.8933 0.000 0.988 0.012 0.000
#> GSM340253 2 0.6434 0.0681 0.068 0.500 0.432 0.000
#> GSM340254 2 0.0336 0.8931 0.000 0.992 0.008 0.000
#> GSM340256 2 0.0336 0.8931 0.000 0.992 0.008 0.000
#> GSM340258 3 0.2944 0.8241 0.128 0.004 0.868 0.000
#> GSM340259 1 0.2081 0.8097 0.916 0.000 0.084 0.000
#> GSM340260 3 0.4746 0.7147 0.368 0.000 0.632 0.000
#> GSM340261 4 0.2081 0.8940 0.000 0.000 0.084 0.916
#> GSM340262 3 0.3726 0.8844 0.212 0.000 0.788 0.000
#> GSM340263 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM340264 3 0.3873 0.8799 0.228 0.000 0.772 0.000
#> GSM340265 3 0.3610 0.8811 0.200 0.000 0.800 0.000
#> GSM340266 4 0.2473 0.8937 0.012 0.000 0.080 0.908
#> GSM340267 3 0.4362 0.7766 0.088 0.096 0.816 0.000
#> GSM340268 4 0.2216 0.8929 0.000 0.000 0.092 0.908
#> GSM340269 3 0.3940 0.6235 0.012 0.028 0.844 0.116
#> GSM340270 1 0.1854 0.8498 0.948 0.024 0.020 0.008
#> GSM537574 2 0.0921 0.8883 0.000 0.972 0.028 0.000
#> GSM537580 4 0.4713 0.5397 0.000 0.000 0.360 0.640
#> GSM537581 4 0.0188 0.9030 0.000 0.000 0.004 0.996
#> GSM340272 3 0.3764 0.8850 0.216 0.000 0.784 0.000
#> GSM340273 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM340275 2 0.3649 0.7249 0.000 0.796 0.000 0.204
#> GSM340276 1 0.4322 0.7347 0.804 0.000 0.044 0.152
#> GSM340277 2 0.0336 0.8931 0.000 0.992 0.008 0.000
#> GSM340278 4 0.2053 0.8974 0.004 0.000 0.072 0.924
#> GSM340279 4 0.3160 0.8821 0.020 0.000 0.108 0.872
#> GSM340282 3 0.3837 0.8815 0.224 0.000 0.776 0.000
#> GSM340284 2 0.1022 0.8896 0.000 0.968 0.032 0.000
#> GSM340285 4 0.0524 0.9028 0.008 0.000 0.004 0.988
#> GSM340286 3 0.3764 0.8850 0.216 0.000 0.784 0.000
#> GSM340287 2 0.6779 0.3893 0.324 0.560 0.116 0.000
#> GSM340288 1 0.0592 0.8589 0.984 0.000 0.016 0.000
#> GSM340289 1 0.0469 0.8598 0.988 0.000 0.012 0.000
#> GSM340290 4 0.2546 0.8922 0.008 0.000 0.092 0.900
#> GSM340291 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM340293 4 0.2011 0.8952 0.000 0.000 0.080 0.920
#> GSM340294 4 0.2530 0.8886 0.004 0.000 0.100 0.896
#> GSM340296 4 0.5835 0.7813 0.088 0.024 0.148 0.740
#> GSM340297 4 0.2266 0.8945 0.004 0.000 0.084 0.912
#> GSM340298 4 0.2081 0.8940 0.000 0.000 0.084 0.916
#> GSM340299 3 0.3764 0.8850 0.216 0.000 0.784 0.000
#> GSM340301 3 0.3764 0.8850 0.216 0.000 0.784 0.000
#> GSM340303 3 0.4663 0.8393 0.148 0.064 0.788 0.000
#> GSM340304 1 0.3084 0.8158 0.896 0.028 0.064 0.012
#> GSM340306 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340307 2 0.1118 0.8894 0.000 0.964 0.036 0.000
#> GSM340310 1 0.0000 0.8599 1.000 0.000 0.000 0.000
#> GSM340314 4 0.2011 0.8952 0.000 0.000 0.080 0.920
#> GSM340315 2 0.3577 0.8098 0.012 0.832 0.156 0.000
#> GSM340317 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM340318 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340319 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340320 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340321 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM340322 4 0.0817 0.8989 0.000 0.000 0.024 0.976
#> GSM340324 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM340328 1 0.3801 0.6962 0.780 0.000 0.000 0.220
#> GSM340330 4 0.5147 0.3045 0.004 0.000 0.460 0.536
#> GSM340332 4 0.1118 0.9025 0.000 0.000 0.036 0.964
#> GSM340333 2 0.4086 0.6864 0.008 0.776 0.216 0.000
#> GSM340336 4 0.4905 0.3644 0.000 0.364 0.004 0.632
#> GSM340337 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340338 3 0.4245 0.7829 0.116 0.000 0.820 0.064
#> GSM340339 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340340 2 0.4981 0.1602 0.000 0.536 0.000 0.464
#> GSM340341 2 0.1042 0.8921 0.000 0.972 0.020 0.008
#> GSM340343 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340344 3 0.6204 0.7380 0.168 0.160 0.672 0.000
#> GSM340346 3 0.4824 0.7126 0.076 0.000 0.780 0.144
#> GSM340347 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340348 4 0.4939 0.7573 0.020 0.112 0.068 0.800
#> GSM340349 1 0.0469 0.8598 0.988 0.000 0.012 0.000
#> GSM340350 1 0.0000 0.8599 1.000 0.000 0.000 0.000
#> GSM340351 1 0.0707 0.8573 0.980 0.000 0.020 0.000
#> GSM340354 2 0.3249 0.8340 0.008 0.852 0.140 0.000
#> GSM340356 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM340357 1 0.0188 0.8603 0.996 0.000 0.004 0.000
#> GSM348183 1 0.0469 0.8598 0.988 0.000 0.012 0.000
#> GSM348191 4 0.2973 0.8868 0.020 0.000 0.096 0.884
#> GSM348193 1 0.2611 0.8039 0.896 0.000 0.008 0.096
#> GSM537578 1 0.2011 0.8142 0.920 0.000 0.080 0.000
#> GSM348181 1 0.4039 0.7480 0.836 0.000 0.080 0.084
#> GSM348182 1 0.0707 0.8573 0.980 0.000 0.020 0.000
#> GSM348184 2 0.0921 0.8908 0.000 0.972 0.028 0.000
#> GSM348185 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM348186 2 0.1022 0.8911 0.000 0.968 0.032 0.000
#> GSM348187 1 0.1389 0.8450 0.952 0.000 0.048 0.000
#> GSM348188 4 0.7554 -0.1993 0.192 0.000 0.376 0.432
#> GSM348189 3 0.4839 0.6614 0.044 0.000 0.756 0.200
#> GSM348190 3 0.3764 0.8850 0.216 0.000 0.784 0.000
#> GSM348194 1 0.0469 0.8598 0.988 0.000 0.012 0.000
#> GSM348195 1 0.1389 0.8399 0.952 0.000 0.048 0.000
#> GSM348196 1 0.0927 0.8590 0.976 0.000 0.008 0.016
#> GSM537585 1 0.4877 -0.1434 0.592 0.000 0.408 0.000
#> GSM537594 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM537596 1 0.1716 0.8389 0.936 0.000 0.064 0.000
#> GSM537597 2 0.2255 0.8534 0.012 0.920 0.068 0.000
#> GSM537602 1 0.1118 0.8507 0.964 0.000 0.036 0.000
#> GSM340184 2 0.4303 0.7811 0.020 0.792 0.184 0.004
#> GSM340185 2 0.1118 0.8894 0.000 0.964 0.036 0.000
#> GSM340186 2 0.1118 0.8894 0.000 0.964 0.036 0.000
#> GSM340187 4 0.2796 0.8603 0.000 0.016 0.092 0.892
#> GSM340189 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340190 2 0.2222 0.8700 0.000 0.924 0.060 0.016
#> GSM340191 4 0.2011 0.8952 0.000 0.000 0.080 0.920
#> GSM340192 2 0.5407 0.1570 0.012 0.504 0.484 0.000
#> GSM340193 1 0.0592 0.8589 0.984 0.000 0.016 0.000
#> GSM340194 3 0.3726 0.8844 0.212 0.000 0.788 0.000
#> GSM340195 3 0.3801 0.8835 0.220 0.000 0.780 0.000
#> GSM340196 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340197 3 0.3837 0.8811 0.224 0.000 0.776 0.000
#> GSM340198 2 0.1211 0.8888 0.000 0.960 0.040 0.000
#> GSM340199 4 0.2011 0.8952 0.000 0.000 0.080 0.920
#> GSM340200 2 0.1211 0.8888 0.000 0.960 0.040 0.000
#> GSM340201 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340202 4 0.0188 0.9031 0.000 0.000 0.004 0.996
#> GSM340203 4 0.0000 0.9036 0.000 0.000 0.000 1.000
#> GSM340204 2 0.6716 0.1042 0.092 0.504 0.404 0.000
#> GSM340205 1 0.4664 0.7503 0.808 0.020 0.132 0.040
#> GSM340206 4 0.2773 0.8834 0.004 0.000 0.116 0.880
#> GSM340207 3 0.3528 0.8774 0.192 0.000 0.808 0.000
#> GSM340237 2 0.0000 0.8938 0.000 1.000 0.000 0.000
#> GSM340238 4 0.2011 0.8952 0.000 0.000 0.080 0.920
#> GSM340239 3 0.3907 0.8756 0.232 0.000 0.768 0.000
#> GSM340240 3 0.3764 0.8850 0.216 0.000 0.784 0.000
#> GSM340241 2 0.1211 0.8888 0.000 0.960 0.040 0.000
#> GSM340242 3 0.4755 0.6562 0.040 0.000 0.760 0.200
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM340358 4 0.3752 0.4636 0.292 0.000 0.000 0.708 0.000
#> GSM340359 2 0.2448 0.8707 0.000 0.892 0.088 0.000 0.020
#> GSM340361 1 0.4307 0.0846 0.504 0.000 0.000 0.000 0.496
#> GSM340362 5 0.0162 0.8618 0.004 0.000 0.000 0.000 0.996
#> GSM340363 4 0.0880 0.7562 0.032 0.000 0.000 0.968 0.000
#> GSM340364 2 0.5644 0.4095 0.328 0.576 0.096 0.000 0.000
#> GSM340365 5 0.0703 0.8578 0.024 0.000 0.000 0.000 0.976
#> GSM340366 5 0.0290 0.8627 0.008 0.000 0.000 0.000 0.992
#> GSM340367 1 0.0000 0.9236 1.000 0.000 0.000 0.000 0.000
#> GSM340368 4 0.3081 0.6013 0.000 0.156 0.012 0.832 0.000
#> GSM340369 4 0.0000 0.7770 0.000 0.000 0.000 1.000 0.000
#> GSM340370 1 0.1106 0.9169 0.964 0.012 0.024 0.000 0.000
#> GSM340371 3 0.6363 0.5578 0.000 0.000 0.504 0.192 0.304
#> GSM340372 1 0.0510 0.9240 0.984 0.000 0.000 0.000 0.016
#> GSM340373 1 0.1608 0.8936 0.928 0.000 0.000 0.000 0.072
#> GSM340375 2 0.5635 0.4596 0.128 0.620 0.000 0.000 0.252
#> GSM340376 1 0.6222 0.4739 0.548 0.216 0.236 0.000 0.000
#> GSM340378 2 0.1282 0.9017 0.004 0.952 0.044 0.000 0.000
#> GSM340243 1 0.6098 0.4542 0.568 0.000 0.196 0.236 0.000
#> GSM340244 2 0.0510 0.9091 0.000 0.984 0.016 0.000 0.000
#> GSM340246 2 0.0162 0.9089 0.000 0.996 0.004 0.000 0.000
#> GSM340247 4 0.0000 0.7770 0.000 0.000 0.000 1.000 0.000
#> GSM340248 2 0.0794 0.9058 0.000 0.972 0.028 0.000 0.000
#> GSM340249 2 0.0510 0.9095 0.000 0.984 0.016 0.000 0.000
#> GSM340250 5 0.1608 0.8281 0.072 0.000 0.000 0.000 0.928
#> GSM340251 2 0.0880 0.9059 0.000 0.968 0.032 0.000 0.000
#> GSM340252 2 0.1892 0.8913 0.000 0.916 0.080 0.000 0.004
#> GSM340253 5 0.5504 0.0902 0.000 0.448 0.064 0.000 0.488
#> GSM340254 2 0.1502 0.8959 0.000 0.940 0.056 0.000 0.004
#> GSM340256 2 0.1544 0.8934 0.000 0.932 0.068 0.000 0.000
#> GSM340258 5 0.4400 0.6385 0.020 0.000 0.308 0.000 0.672
#> GSM340259 1 0.2516 0.8308 0.860 0.000 0.000 0.000 0.140
#> GSM340260 5 0.5013 0.6211 0.232 0.000 0.084 0.000 0.684
#> GSM340261 3 0.4101 0.6606 0.000 0.000 0.628 0.372 0.000
#> GSM340262 5 0.0290 0.8627 0.008 0.000 0.000 0.000 0.992
#> GSM340263 2 0.0510 0.9091 0.000 0.984 0.016 0.000 0.000
#> GSM340264 5 0.0898 0.8585 0.020 0.000 0.008 0.000 0.972
#> GSM340265 5 0.0613 0.8584 0.004 0.008 0.004 0.000 0.984
#> GSM340266 3 0.4455 0.6348 0.008 0.000 0.588 0.404 0.000
#> GSM340267 5 0.4588 0.6889 0.000 0.116 0.136 0.000 0.748
#> GSM340268 3 0.4210 0.6333 0.000 0.000 0.588 0.412 0.000
#> GSM340269 3 0.3211 0.5725 0.008 0.012 0.848 0.004 0.128
#> GSM340270 1 0.1121 0.9151 0.956 0.000 0.044 0.000 0.000
#> GSM537574 2 0.0703 0.9064 0.000 0.976 0.024 0.000 0.000
#> GSM537580 3 0.5523 0.6608 0.000 0.000 0.592 0.320 0.088
#> GSM537581 4 0.0451 0.7716 0.000 0.000 0.008 0.988 0.004
#> GSM340272 5 0.0290 0.8624 0.008 0.000 0.000 0.000 0.992
#> GSM340273 2 0.0290 0.9091 0.000 0.992 0.008 0.000 0.000
#> GSM340275 2 0.4565 0.3172 0.000 0.580 0.012 0.408 0.000
#> GSM340276 1 0.0510 0.9212 0.984 0.000 0.016 0.000 0.000
#> GSM340277 2 0.1410 0.8950 0.000 0.940 0.060 0.000 0.000
#> GSM340278 4 0.4450 -0.5125 0.004 0.000 0.488 0.508 0.000
#> GSM340279 3 0.3932 0.6735 0.000 0.000 0.672 0.328 0.000
#> GSM340282 5 0.0290 0.8627 0.008 0.000 0.000 0.000 0.992
#> GSM340284 2 0.1608 0.8884 0.000 0.928 0.072 0.000 0.000
#> GSM340285 4 0.1195 0.7508 0.028 0.000 0.012 0.960 0.000
#> GSM340286 5 0.0404 0.8626 0.012 0.000 0.000 0.000 0.988
#> GSM340287 3 0.4269 0.4413 0.016 0.300 0.684 0.000 0.000
#> GSM340288 1 0.0510 0.9240 0.984 0.000 0.000 0.000 0.016
#> GSM340289 1 0.0510 0.9240 0.984 0.000 0.000 0.000 0.016
#> GSM340290 3 0.4649 0.6325 0.016 0.000 0.580 0.404 0.000
#> GSM340291 2 0.0162 0.9085 0.000 0.996 0.004 0.000 0.000
#> GSM340293 4 0.4305 -0.5087 0.000 0.000 0.488 0.512 0.000
#> GSM340294 3 0.4015 0.6708 0.000 0.000 0.652 0.348 0.000
#> GSM340296 3 0.3531 0.5911 0.036 0.000 0.816 0.148 0.000
#> GSM340297 3 0.4533 0.5869 0.008 0.000 0.544 0.448 0.000
#> GSM340298 3 0.4225 0.6645 0.004 0.000 0.632 0.364 0.000
#> GSM340299 5 0.0290 0.8627 0.008 0.000 0.000 0.000 0.992
#> GSM340301 5 0.0162 0.8618 0.004 0.000 0.000 0.000 0.996
#> GSM340303 5 0.1124 0.8403 0.000 0.036 0.004 0.000 0.960
#> GSM340304 1 0.2329 0.8716 0.876 0.000 0.124 0.000 0.000
#> GSM340306 4 0.0000 0.7770 0.000 0.000 0.000 1.000 0.000
#> GSM340307 2 0.1121 0.9012 0.000 0.956 0.044 0.000 0.000
#> GSM340310 1 0.0162 0.9236 0.996 0.000 0.004 0.000 0.000
#> GSM340314 4 0.4291 -0.4559 0.000 0.000 0.464 0.536 0.000
#> GSM340315 3 0.4630 0.0211 0.016 0.396 0.588 0.000 0.000
#> GSM340317 2 0.0290 0.9091 0.000 0.992 0.008 0.000 0.000
#> GSM340318 4 0.0000 0.7770 0.000 0.000 0.000 1.000 0.000
#> GSM340319 4 0.0000 0.7770 0.000 0.000 0.000 1.000 0.000
#> GSM340320 4 0.0000 0.7770 0.000 0.000 0.000 1.000 0.000
#> GSM340321 2 0.0290 0.9093 0.000 0.992 0.008 0.000 0.000
#> GSM340322 4 0.1544 0.7160 0.000 0.000 0.068 0.932 0.000
#> GSM340324 2 0.1608 0.8922 0.000 0.928 0.072 0.000 0.000
#> GSM340328 1 0.1831 0.8786 0.920 0.000 0.000 0.076 0.004
#> GSM340330 3 0.5703 0.6479 0.000 0.000 0.628 0.188 0.184
#> GSM340332 4 0.3143 0.4272 0.000 0.000 0.204 0.796 0.000
#> GSM340333 2 0.5218 0.4504 0.000 0.624 0.068 0.000 0.308
#> GSM340336 4 0.4173 0.4259 0.000 0.300 0.012 0.688 0.000
#> GSM340337 4 0.0162 0.7747 0.000 0.000 0.004 0.996 0.000
#> GSM340338 3 0.4562 0.1380 0.008 0.000 0.500 0.000 0.492
#> GSM340339 4 0.0000 0.7770 0.000 0.000 0.000 1.000 0.000
#> GSM340340 4 0.4798 0.0709 0.000 0.440 0.020 0.540 0.000
#> GSM340341 2 0.1670 0.8859 0.000 0.936 0.012 0.052 0.000
#> GSM340343 4 0.0162 0.7750 0.004 0.000 0.000 0.996 0.000
#> GSM340344 5 0.4151 0.7110 0.004 0.152 0.060 0.000 0.784
#> GSM340346 3 0.6170 0.6176 0.036 0.000 0.632 0.120 0.212
#> GSM340347 4 0.0000 0.7770 0.000 0.000 0.000 1.000 0.000
#> GSM340348 4 0.3648 0.6023 0.016 0.004 0.188 0.792 0.000
#> GSM340349 1 0.0992 0.9215 0.968 0.000 0.008 0.000 0.024
#> GSM340350 1 0.0162 0.9242 0.996 0.000 0.000 0.000 0.004
#> GSM340351 1 0.0609 0.9233 0.980 0.000 0.000 0.000 0.020
#> GSM340354 2 0.4196 0.5844 0.000 0.640 0.356 0.000 0.004
#> GSM340356 2 0.0609 0.9092 0.000 0.980 0.020 0.000 0.000
#> GSM340357 1 0.0451 0.9252 0.988 0.000 0.004 0.000 0.008
#> GSM348183 1 0.0771 0.9238 0.976 0.000 0.004 0.000 0.020
#> GSM348191 3 0.4348 0.6741 0.016 0.000 0.668 0.316 0.000
#> GSM348193 1 0.0955 0.9201 0.968 0.000 0.028 0.000 0.004
#> GSM537578 1 0.2763 0.8205 0.848 0.000 0.004 0.000 0.148
#> GSM348181 1 0.0404 0.9228 0.988 0.000 0.012 0.000 0.000
#> GSM348182 1 0.1043 0.9162 0.960 0.000 0.000 0.000 0.040
#> GSM348184 2 0.0794 0.9058 0.000 0.972 0.028 0.000 0.000
#> GSM348185 2 0.0510 0.9091 0.000 0.984 0.016 0.000 0.000
#> GSM348186 2 0.1270 0.9013 0.000 0.948 0.052 0.000 0.000
#> GSM348187 1 0.0510 0.9215 0.984 0.000 0.016 0.000 0.000
#> GSM348188 4 0.5014 0.1378 0.032 0.000 0.000 0.536 0.432
#> GSM348189 3 0.6530 0.4634 0.000 0.000 0.440 0.200 0.360
#> GSM348190 5 0.0404 0.8626 0.012 0.000 0.000 0.000 0.988
#> GSM348194 1 0.0404 0.9243 0.988 0.000 0.000 0.000 0.012
#> GSM348195 1 0.0880 0.9190 0.968 0.000 0.000 0.000 0.032
#> GSM348196 1 0.0000 0.9236 1.000 0.000 0.000 0.000 0.000
#> GSM537585 5 0.5928 0.3720 0.328 0.000 0.124 0.000 0.548
#> GSM537594 2 0.0404 0.9097 0.000 0.988 0.012 0.000 0.000
#> GSM537596 1 0.0693 0.9245 0.980 0.000 0.008 0.000 0.012
#> GSM537597 2 0.3783 0.7260 0.012 0.768 0.216 0.000 0.004
#> GSM537602 1 0.1197 0.9148 0.952 0.000 0.048 0.000 0.000
#> GSM340184 3 0.2625 0.5400 0.016 0.108 0.876 0.000 0.000
#> GSM340185 2 0.0963 0.9054 0.000 0.964 0.036 0.000 0.000
#> GSM340186 2 0.0794 0.9058 0.000 0.972 0.028 0.000 0.000
#> GSM340187 4 0.3246 0.6247 0.000 0.008 0.184 0.808 0.000
#> GSM340189 4 0.0000 0.7770 0.000 0.000 0.000 1.000 0.000
#> GSM340190 2 0.3366 0.7034 0.000 0.768 0.232 0.000 0.000
#> GSM340191 3 0.4300 0.5476 0.000 0.000 0.524 0.476 0.000
#> GSM340192 5 0.5236 0.0467 0.000 0.464 0.044 0.000 0.492
#> GSM340193 1 0.0609 0.9233 0.980 0.000 0.000 0.000 0.020
#> GSM340194 5 0.0162 0.8618 0.004 0.000 0.000 0.000 0.996
#> GSM340195 5 0.0404 0.8626 0.012 0.000 0.000 0.000 0.988
#> GSM340196 4 0.0000 0.7770 0.000 0.000 0.000 1.000 0.000
#> GSM340197 5 0.0404 0.8626 0.012 0.000 0.000 0.000 0.988
#> GSM340198 2 0.1704 0.8957 0.000 0.928 0.068 0.000 0.004
#> GSM340199 3 0.4291 0.5702 0.000 0.000 0.536 0.464 0.000
#> GSM340200 2 0.0794 0.9058 0.000 0.972 0.028 0.000 0.000
#> GSM340201 4 0.0000 0.7770 0.000 0.000 0.000 1.000 0.000
#> GSM340202 4 0.0000 0.7770 0.000 0.000 0.000 1.000 0.000
#> GSM340203 4 0.0000 0.7770 0.000 0.000 0.000 1.000 0.000
#> GSM340204 5 0.6405 0.2042 0.000 0.364 0.176 0.000 0.460
#> GSM340205 1 0.2230 0.8715 0.884 0.000 0.116 0.000 0.000
#> GSM340206 3 0.4161 0.6356 0.000 0.000 0.608 0.392 0.000
#> GSM340207 5 0.0865 0.8495 0.004 0.000 0.024 0.000 0.972
#> GSM340237 2 0.0162 0.9089 0.000 0.996 0.004 0.000 0.000
#> GSM340238 3 0.4300 0.5507 0.000 0.000 0.524 0.476 0.000
#> GSM340239 5 0.0510 0.8615 0.016 0.000 0.000 0.000 0.984
#> GSM340240 5 0.0162 0.8618 0.004 0.000 0.000 0.000 0.996
#> GSM340241 2 0.1121 0.9068 0.000 0.956 0.044 0.000 0.000
#> GSM340242 3 0.5848 0.6408 0.000 0.000 0.608 0.200 0.192
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM340358 4 0.3629 0.5774 0.260 0.000 0.000 0.724 0.000 NA
#> GSM340359 2 0.3023 0.8184 0.000 0.828 0.000 0.000 0.032 NA
#> GSM340361 5 0.4435 0.5257 0.264 0.000 0.000 0.000 0.672 NA
#> GSM340362 5 0.0405 0.8685 0.004 0.000 0.000 0.000 0.988 NA
#> GSM340363 4 0.1341 0.8075 0.028 0.000 0.000 0.948 0.000 NA
#> GSM340364 2 0.4715 0.6822 0.060 0.656 0.004 0.000 0.004 NA
#> GSM340365 5 0.1168 0.8588 0.028 0.000 0.000 0.000 0.956 NA
#> GSM340366 5 0.0291 0.8685 0.004 0.000 0.000 0.000 0.992 NA
#> GSM340367 1 0.0547 0.8921 0.980 0.000 0.000 0.000 0.000 NA
#> GSM340368 4 0.2402 0.7203 0.000 0.120 0.000 0.868 0.000 NA
#> GSM340369 4 0.0000 0.8285 0.000 0.000 0.000 1.000 0.000 NA
#> GSM340370 1 0.2544 0.8767 0.864 0.004 0.012 0.000 0.000 NA
#> GSM340371 3 0.7480 0.4707 0.000 0.000 0.384 0.180 0.216 NA
#> GSM340372 1 0.1124 0.8910 0.956 0.000 0.000 0.000 0.008 NA
#> GSM340373 1 0.1643 0.8703 0.924 0.000 0.000 0.000 0.068 NA
#> GSM340375 2 0.4906 0.6978 0.088 0.720 0.008 0.000 0.156 NA
#> GSM340376 1 0.7264 0.4281 0.400 0.164 0.144 0.000 0.000 NA
#> GSM340378 2 0.1610 0.8509 0.000 0.916 0.000 0.000 0.000 NA
#> GSM340243 1 0.6842 0.5275 0.512 0.000 0.184 0.140 0.000 NA
#> GSM340244 2 0.0935 0.8593 0.000 0.964 0.004 0.000 0.000 NA
#> GSM340246 2 0.1610 0.8579 0.000 0.916 0.000 0.000 0.000 NA
#> GSM340247 4 0.0146 0.8277 0.000 0.000 0.004 0.996 0.000 NA
#> GSM340248 2 0.2320 0.8413 0.000 0.864 0.004 0.000 0.000 NA
#> GSM340249 2 0.0777 0.8584 0.000 0.972 0.004 0.000 0.000 NA
#> GSM340250 5 0.2195 0.8362 0.068 0.000 0.012 0.000 0.904 NA
#> GSM340251 2 0.2442 0.8382 0.000 0.852 0.004 0.000 0.000 NA
#> GSM340252 2 0.2003 0.8398 0.000 0.884 0.000 0.000 0.000 NA
#> GSM340253 2 0.5221 0.4045 0.000 0.560 0.000 0.000 0.328 NA
#> GSM340254 2 0.2346 0.8438 0.000 0.868 0.000 0.000 0.008 NA
#> GSM340256 2 0.1957 0.8416 0.000 0.888 0.000 0.000 0.000 NA
#> GSM340258 5 0.5880 0.4236 0.000 0.012 0.140 0.000 0.456 NA
#> GSM340259 1 0.2595 0.7888 0.836 0.000 0.000 0.000 0.160 NA
#> GSM340260 5 0.7470 0.2438 0.244 0.000 0.144 0.000 0.360 NA
#> GSM340261 3 0.2854 0.7428 0.000 0.000 0.792 0.208 0.000 NA
#> GSM340262 5 0.0146 0.8686 0.004 0.000 0.000 0.000 0.996 NA
#> GSM340263 2 0.0603 0.8576 0.000 0.980 0.004 0.000 0.000 NA
#> GSM340264 5 0.1448 0.8597 0.016 0.000 0.012 0.000 0.948 NA
#> GSM340265 5 0.1442 0.8568 0.000 0.004 0.012 0.000 0.944 NA
#> GSM340266 3 0.4428 0.7285 0.000 0.000 0.696 0.220 0.000 NA
#> GSM340267 5 0.5268 0.6745 0.004 0.080 0.088 0.000 0.704 NA
#> GSM340268 3 0.3586 0.7455 0.000 0.000 0.756 0.216 0.000 NA
#> GSM340269 3 0.3655 0.6608 0.012 0.000 0.796 0.000 0.044 NA
#> GSM340270 1 0.2730 0.8676 0.836 0.000 0.012 0.000 0.000 NA
#> GSM537574 2 0.2888 0.8227 0.000 0.852 0.056 0.000 0.000 NA
#> GSM537580 3 0.6700 0.5930 0.000 0.000 0.504 0.204 0.080 NA
#> GSM537581 4 0.1950 0.7887 0.000 0.000 0.024 0.912 0.000 NA
#> GSM340272 5 0.0508 0.8677 0.004 0.000 0.000 0.000 0.984 NA
#> GSM340273 2 0.0000 0.8551 0.000 1.000 0.000 0.000 0.000 NA
#> GSM340275 2 0.4002 0.3128 0.000 0.588 0.000 0.404 0.000 NA
#> GSM340276 1 0.2070 0.8776 0.908 0.000 0.048 0.000 0.000 NA
#> GSM340277 2 0.1714 0.8450 0.000 0.908 0.000 0.000 0.000 NA
#> GSM340278 3 0.4559 0.4399 0.008 0.000 0.512 0.460 0.000 NA
#> GSM340279 3 0.2658 0.7432 0.000 0.000 0.864 0.100 0.000 NA
#> GSM340282 5 0.0603 0.8665 0.016 0.000 0.000 0.000 0.980 NA
#> GSM340284 2 0.3213 0.8243 0.000 0.808 0.032 0.000 0.000 NA
#> GSM340285 4 0.2627 0.7488 0.064 0.000 0.016 0.884 0.000 NA
#> GSM340286 5 0.0405 0.8683 0.004 0.000 0.000 0.000 0.988 NA
#> GSM340287 3 0.5119 0.6144 0.048 0.132 0.700 0.000 0.000 NA
#> GSM340288 1 0.0717 0.8885 0.976 0.000 0.000 0.000 0.008 NA
#> GSM340289 1 0.0692 0.8880 0.976 0.000 0.000 0.000 0.004 NA
#> GSM340290 3 0.3445 0.7209 0.000 0.000 0.744 0.244 0.000 NA
#> GSM340291 2 0.0260 0.8560 0.000 0.992 0.000 0.000 0.000 NA
#> GSM340293 3 0.3695 0.6385 0.000 0.000 0.624 0.376 0.000 NA
#> GSM340294 3 0.2527 0.7507 0.000 0.000 0.832 0.168 0.000 NA
#> GSM340296 3 0.2685 0.7014 0.016 0.000 0.880 0.036 0.000 NA
#> GSM340297 3 0.3330 0.7191 0.000 0.000 0.716 0.284 0.000 NA
#> GSM340298 3 0.2980 0.7476 0.000 0.000 0.800 0.192 0.000 NA
#> GSM340299 5 0.0146 0.8686 0.004 0.000 0.000 0.000 0.996 NA
#> GSM340301 5 0.0405 0.8684 0.004 0.000 0.000 0.000 0.988 NA
#> GSM340303 5 0.2186 0.8328 0.000 0.048 0.008 0.000 0.908 NA
#> GSM340304 1 0.4969 0.6613 0.616 0.000 0.104 0.000 0.000 NA
#> GSM340306 4 0.0363 0.8245 0.000 0.000 0.000 0.988 0.000 NA
#> GSM340307 2 0.2738 0.8349 0.000 0.820 0.004 0.000 0.000 NA
#> GSM340310 1 0.1910 0.8823 0.892 0.000 0.000 0.000 0.000 NA
#> GSM340314 3 0.3684 0.6417 0.000 0.000 0.628 0.372 0.000 NA
#> GSM340315 3 0.6002 -0.0972 0.000 0.368 0.396 0.000 0.000 NA
#> GSM340317 2 0.0363 0.8570 0.000 0.988 0.000 0.000 0.000 NA
#> GSM340318 4 0.0000 0.8285 0.000 0.000 0.000 1.000 0.000 NA
#> GSM340319 4 0.0000 0.8285 0.000 0.000 0.000 1.000 0.000 NA
#> GSM340320 4 0.0260 0.8261 0.000 0.000 0.000 0.992 0.000 NA
#> GSM340321 2 0.1411 0.8592 0.000 0.936 0.004 0.000 0.000 NA
#> GSM340322 4 0.3894 0.6123 0.000 0.000 0.072 0.760 0.000 NA
#> GSM340324 2 0.2300 0.8302 0.000 0.856 0.000 0.000 0.000 NA
#> GSM340328 1 0.2653 0.8090 0.844 0.000 0.000 0.144 0.000 NA
#> GSM340330 3 0.3799 0.7326 0.000 0.000 0.788 0.104 0.104 NA
#> GSM340332 4 0.3847 -0.3217 0.000 0.000 0.456 0.544 0.000 NA
#> GSM340333 2 0.4466 0.6838 0.000 0.708 0.000 0.000 0.176 NA
#> GSM340336 4 0.5104 0.4574 0.000 0.248 0.004 0.628 0.000 NA
#> GSM340337 4 0.0146 0.8274 0.000 0.000 0.000 0.996 0.000 NA
#> GSM340338 3 0.4217 0.1556 0.004 0.000 0.524 0.000 0.464 NA
#> GSM340339 4 0.0000 0.8285 0.000 0.000 0.000 1.000 0.000 NA
#> GSM340340 4 0.4521 0.1947 0.000 0.400 0.004 0.568 0.000 NA
#> GSM340341 2 0.2923 0.8456 0.000 0.868 0.020 0.052 0.000 NA
#> GSM340343 4 0.0622 0.8225 0.008 0.000 0.000 0.980 0.000 NA
#> GSM340344 5 0.3701 0.7302 0.000 0.100 0.000 0.000 0.788 NA
#> GSM340346 3 0.4111 0.7354 0.008 0.000 0.788 0.100 0.088 NA
#> GSM340347 4 0.0000 0.8285 0.000 0.000 0.000 1.000 0.000 NA
#> GSM340348 4 0.3821 0.6792 0.000 0.000 0.156 0.776 0.004 NA
#> GSM340349 1 0.1334 0.8869 0.948 0.000 0.000 0.000 0.020 NA
#> GSM340350 1 0.0632 0.8874 0.976 0.000 0.000 0.000 0.000 NA
#> GSM340351 1 0.1643 0.8873 0.924 0.000 0.000 0.000 0.008 NA
#> GSM340354 2 0.5722 0.5235 0.000 0.500 0.156 0.000 0.004 NA
#> GSM340356 2 0.0777 0.8575 0.000 0.972 0.004 0.000 0.000 NA
#> GSM340357 1 0.1882 0.8843 0.920 0.000 0.008 0.000 0.012 NA
#> GSM348183 1 0.1700 0.8858 0.916 0.000 0.000 0.000 0.004 NA
#> GSM348191 3 0.2060 0.7366 0.000 0.000 0.900 0.084 0.000 NA
#> GSM348193 1 0.2527 0.8676 0.832 0.000 0.000 0.000 0.000 NA
#> GSM537578 1 0.3053 0.7991 0.828 0.000 0.004 0.000 0.144 NA
#> GSM348181 1 0.1913 0.8884 0.908 0.000 0.012 0.000 0.000 NA
#> GSM348182 1 0.2795 0.8438 0.856 0.000 0.000 0.000 0.100 NA
#> GSM348184 2 0.2402 0.8390 0.000 0.856 0.004 0.000 0.000 NA
#> GSM348185 2 0.0508 0.8571 0.000 0.984 0.004 0.000 0.000 NA
#> GSM348186 2 0.2527 0.8517 0.000 0.832 0.000 0.000 0.000 NA
#> GSM348187 1 0.1341 0.8871 0.948 0.000 0.024 0.000 0.000 NA
#> GSM348188 4 0.4662 0.3695 0.032 0.000 0.000 0.604 0.352 NA
#> GSM348189 3 0.7552 0.4508 0.000 0.000 0.364 0.196 0.228 NA
#> GSM348190 5 0.0291 0.8685 0.004 0.000 0.000 0.000 0.992 NA
#> GSM348194 1 0.1267 0.8883 0.940 0.000 0.000 0.000 0.000 NA
#> GSM348195 1 0.2237 0.8833 0.896 0.000 0.000 0.000 0.036 NA
#> GSM348196 1 0.0865 0.8865 0.964 0.000 0.000 0.000 0.000 NA
#> GSM537585 5 0.6707 0.4345 0.236 0.000 0.104 0.000 0.512 NA
#> GSM537594 2 0.0458 0.8575 0.000 0.984 0.000 0.000 0.000 NA
#> GSM537596 1 0.1478 0.8853 0.944 0.000 0.020 0.000 0.004 NA
#> GSM537597 2 0.4957 0.6334 0.000 0.648 0.148 0.000 0.000 NA
#> GSM537602 1 0.4067 0.8264 0.752 0.000 0.072 0.000 0.004 NA
#> GSM340184 3 0.3476 0.6016 0.000 0.004 0.732 0.000 0.004 NA
#> GSM340185 2 0.2778 0.8346 0.000 0.824 0.008 0.000 0.000 NA
#> GSM340186 2 0.2595 0.8370 0.000 0.836 0.004 0.000 0.000 NA
#> GSM340187 4 0.5895 0.3910 0.000 0.012 0.172 0.520 0.000 NA
#> GSM340189 4 0.0000 0.8285 0.000 0.000 0.000 1.000 0.000 NA
#> GSM340190 2 0.4801 0.6648 0.000 0.668 0.196 0.000 0.000 NA
#> GSM340191 3 0.3409 0.7105 0.000 0.000 0.700 0.300 0.000 NA
#> GSM340192 2 0.6057 0.4949 0.000 0.484 0.008 0.000 0.268 NA
#> GSM340193 1 0.0777 0.8875 0.972 0.000 0.000 0.000 0.004 NA
#> GSM340194 5 0.0603 0.8673 0.004 0.000 0.000 0.000 0.980 NA
#> GSM340195 5 0.0291 0.8684 0.004 0.000 0.000 0.000 0.992 NA
#> GSM340196 4 0.0000 0.8285 0.000 0.000 0.000 1.000 0.000 NA
#> GSM340197 5 0.0146 0.8686 0.004 0.000 0.000 0.000 0.996 NA
#> GSM340198 2 0.3023 0.8356 0.000 0.768 0.000 0.000 0.000 NA
#> GSM340199 3 0.3464 0.7021 0.000 0.000 0.688 0.312 0.000 NA
#> GSM340200 2 0.2883 0.8382 0.000 0.788 0.000 0.000 0.000 NA
#> GSM340201 4 0.0000 0.8285 0.000 0.000 0.000 1.000 0.000 NA
#> GSM340202 4 0.0000 0.8285 0.000 0.000 0.000 1.000 0.000 NA
#> GSM340203 4 0.0000 0.8285 0.000 0.000 0.000 1.000 0.000 NA
#> GSM340204 5 0.6895 0.0819 0.000 0.308 0.048 0.000 0.352 NA
#> GSM340205 1 0.4389 0.7996 0.712 0.000 0.100 0.000 0.000 NA
#> GSM340206 3 0.3558 0.7447 0.000 0.000 0.760 0.212 0.000 NA
#> GSM340207 5 0.1148 0.8609 0.004 0.000 0.020 0.000 0.960 NA
#> GSM340237 2 0.0000 0.8551 0.000 1.000 0.000 0.000 0.000 NA
#> GSM340238 3 0.3464 0.7021 0.000 0.000 0.688 0.312 0.000 NA
#> GSM340239 5 0.0865 0.8599 0.036 0.000 0.000 0.000 0.964 NA
#> GSM340240 5 0.0363 0.8668 0.000 0.000 0.000 0.000 0.988 NA
#> GSM340241 2 0.3136 0.8335 0.000 0.768 0.004 0.000 0.000 NA
#> GSM340242 3 0.6312 0.6246 0.000 0.000 0.544 0.196 0.052 NA
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
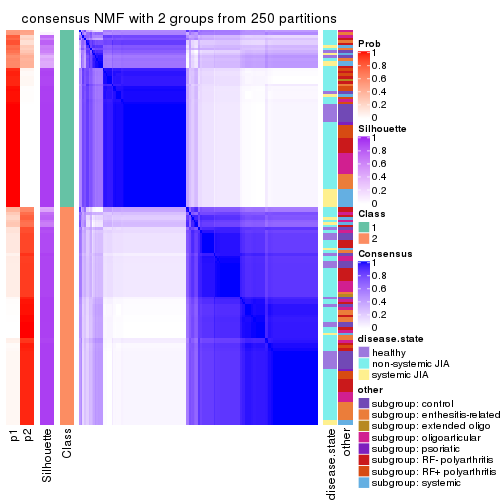
consensus_heatmap(res, k = 3)
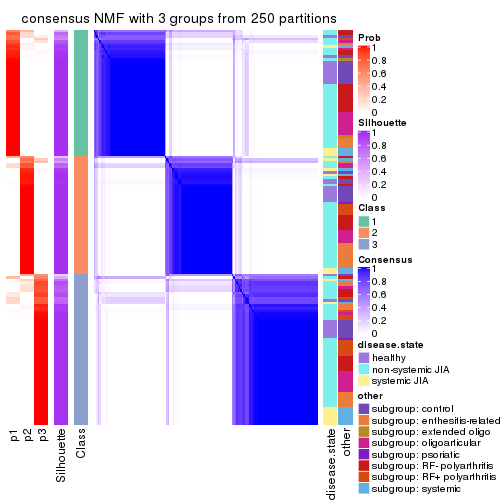
consensus_heatmap(res, k = 4)
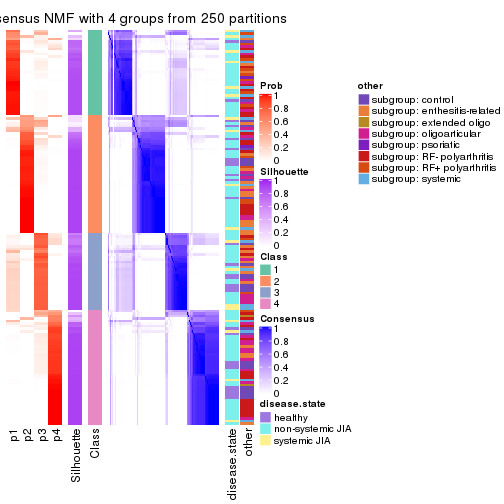
consensus_heatmap(res, k = 5)
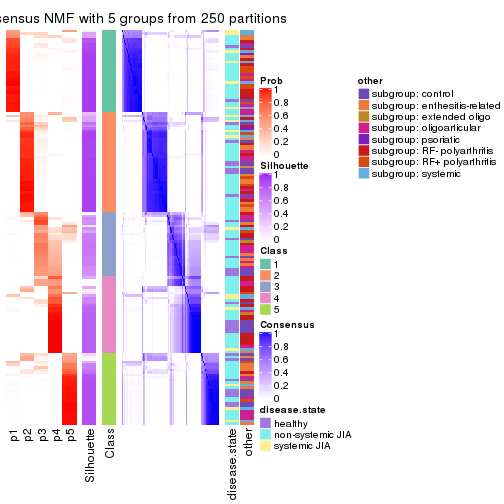
consensus_heatmap(res, k = 6)
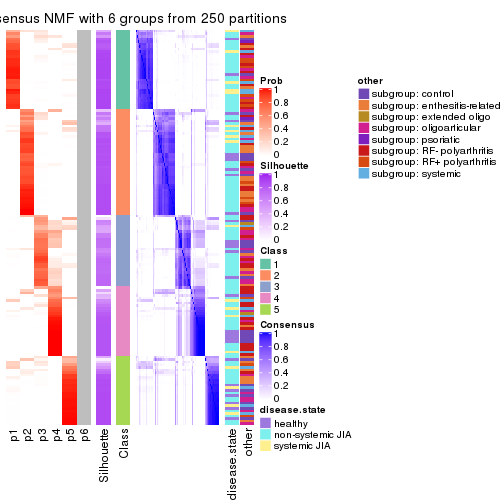
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
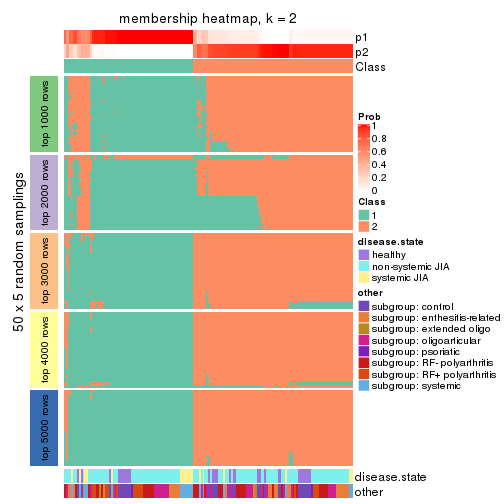
membership_heatmap(res, k = 3)
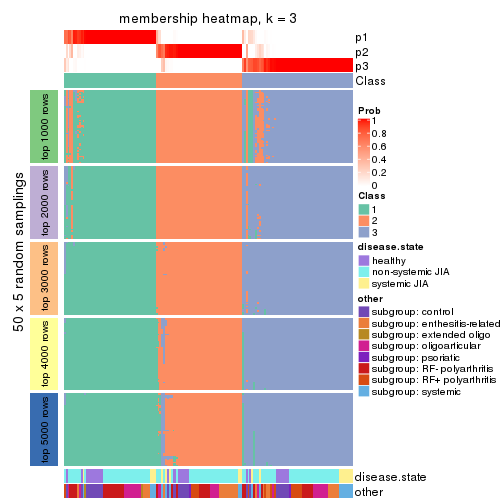
membership_heatmap(res, k = 4)
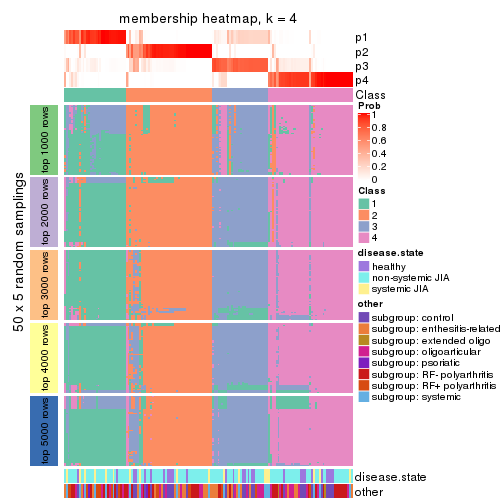
membership_heatmap(res, k = 5)
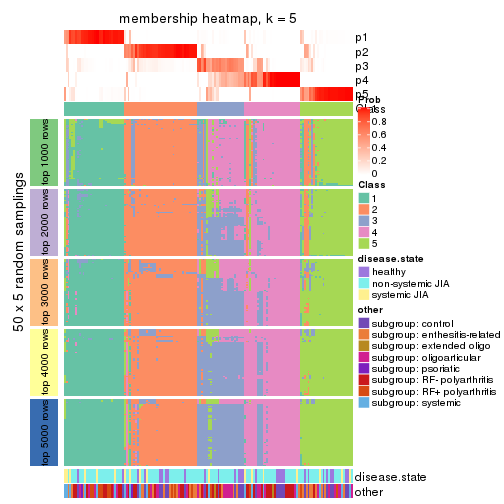
membership_heatmap(res, k = 6)
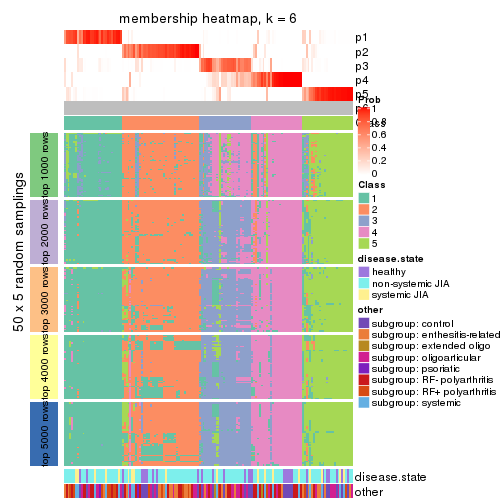
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
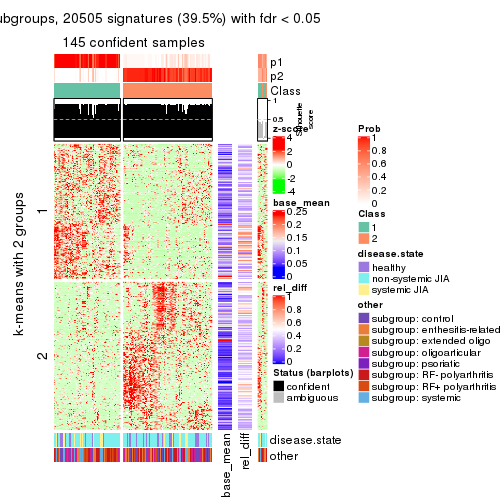
get_signatures(res, k = 3)
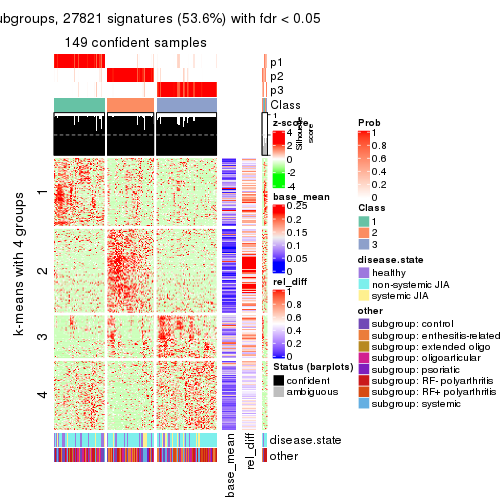
get_signatures(res, k = 4)
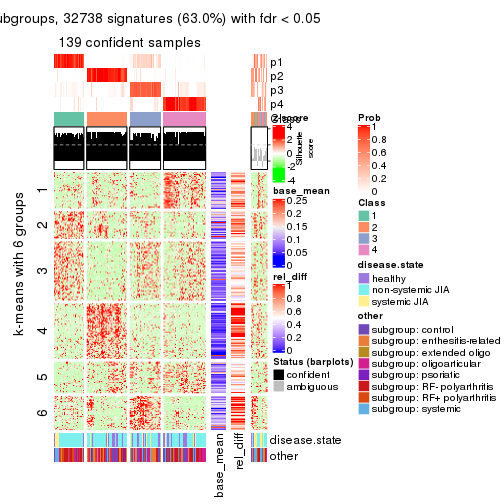
get_signatures(res, k = 5)
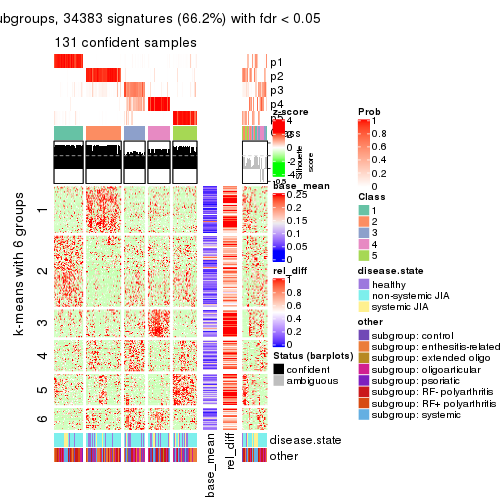
get_signatures(res, k = 6)
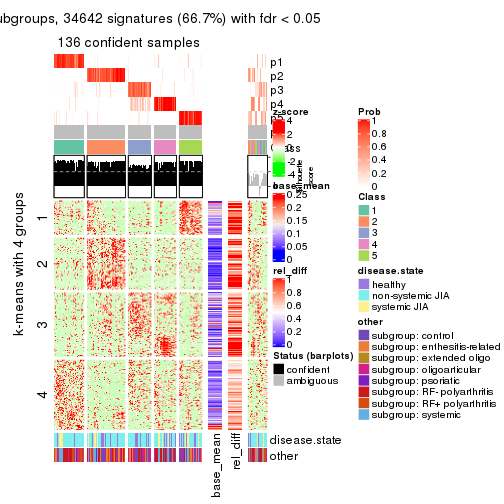
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
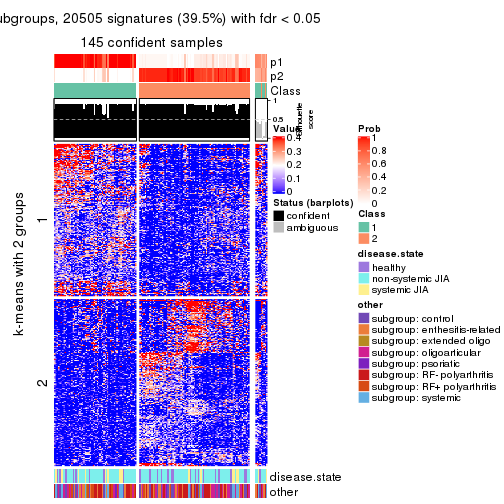
get_signatures(res, k = 3, scale_rows = FALSE)
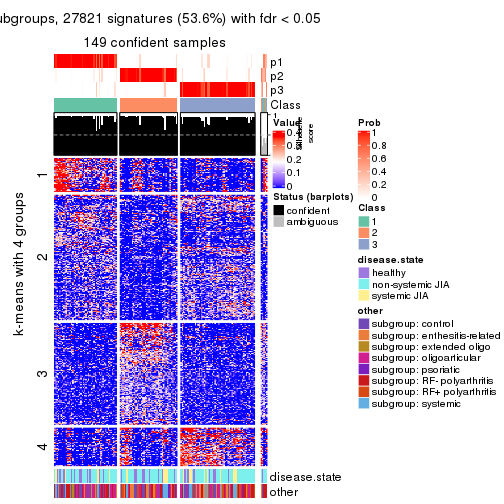
get_signatures(res, k = 4, scale_rows = FALSE)
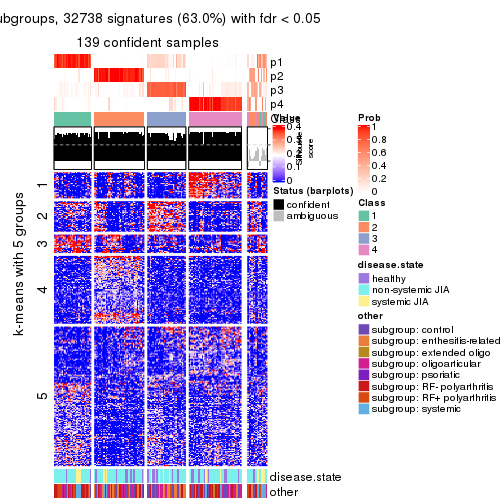
get_signatures(res, k = 5, scale_rows = FALSE)
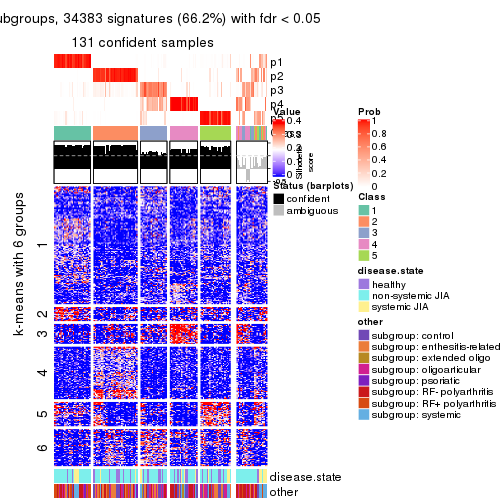
get_signatures(res, k = 6, scale_rows = FALSE)
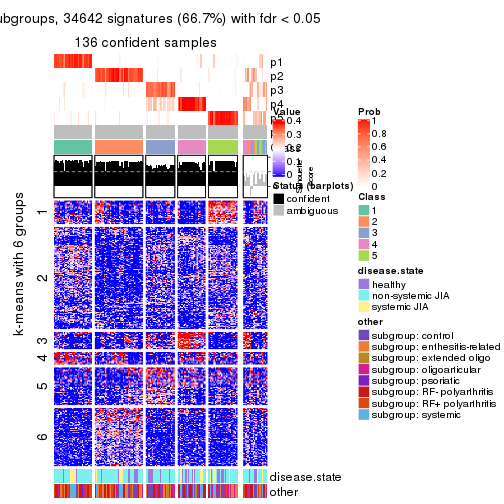
Compare the overlap of signatures from different k:
compare_signatures(res)
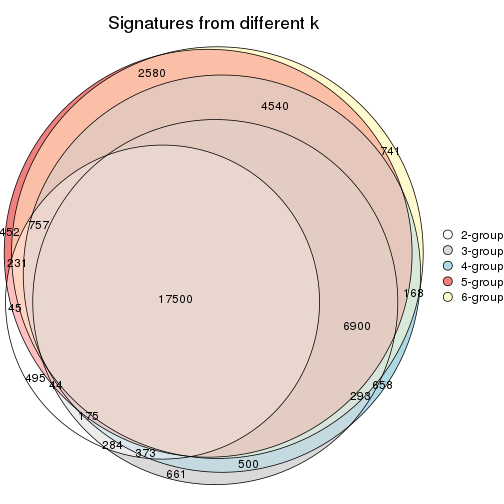
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
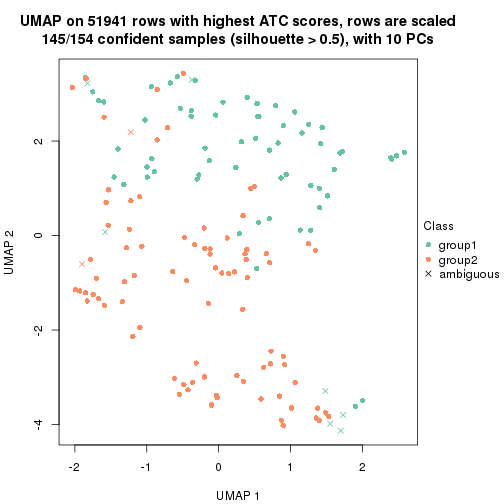
dimension_reduction(res, k = 3, method = "UMAP")
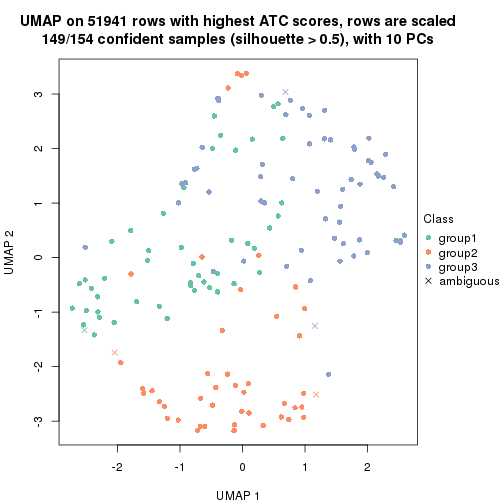
dimension_reduction(res, k = 4, method = "UMAP")
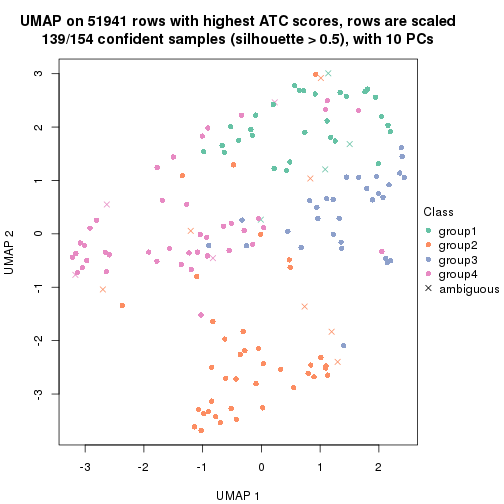
dimension_reduction(res, k = 5, method = "UMAP")
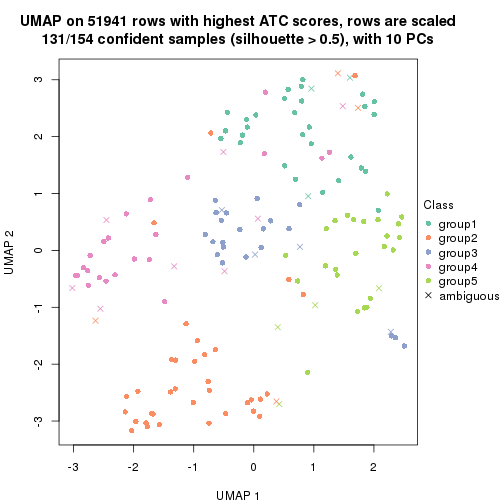
dimension_reduction(res, k = 6, method = "UMAP")
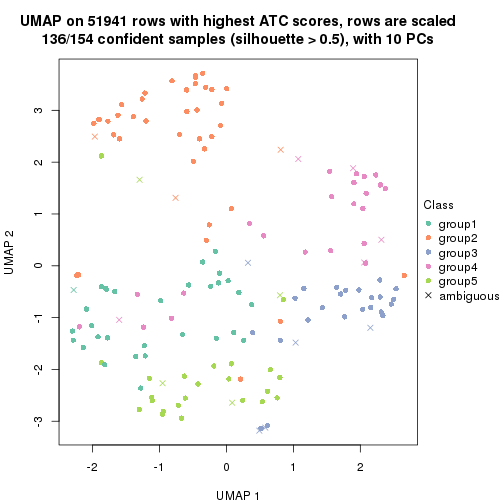
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
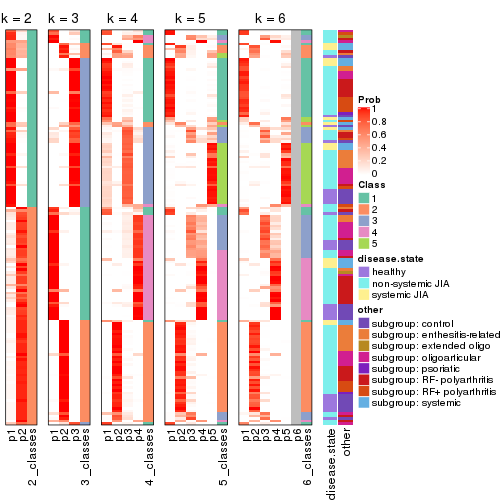
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) other(p) k
#> ATC:NMF 145 0.147 0.1406 2
#> ATC:NMF 149 0.658 0.1613 3
#> ATC:NMF 139 0.446 0.0405 4
#> ATC:NMF 131 0.521 0.0272 5
#> ATC:NMF 136 0.308 0.0261 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
sessionInfo()
#> R version 3.6.0 (2019-04-26)
#> Platform: x86_64-pc-linux-gnu (64-bit)
#> Running under: CentOS Linux 7 (Core)
#>
#> Matrix products: default
#> BLAS: /usr/lib64/libblas.so.3.4.2
#> LAPACK: /usr/lib64/liblapack.so.3.4.2
#>
#> locale:
#> [1] LC_CTYPE=en_GB.UTF-8 LC_NUMERIC=C LC_TIME=en_GB.UTF-8
#> [4] LC_COLLATE=en_GB.UTF-8 LC_MONETARY=en_GB.UTF-8 LC_MESSAGES=en_GB.UTF-8
#> [7] LC_PAPER=en_GB.UTF-8 LC_NAME=C LC_ADDRESS=C
#> [10] LC_TELEPHONE=C LC_MEASUREMENT=en_GB.UTF-8 LC_IDENTIFICATION=C
#>
#> attached base packages:
#> [1] grid stats graphics grDevices utils datasets methods base
#>
#> other attached packages:
#> [1] genefilter_1.66.0 ComplexHeatmap_2.3.1 markdown_1.1 knitr_1.26
#> [5] GetoptLong_0.1.7 cola_1.3.2
#>
#> loaded via a namespace (and not attached):
#> [1] circlize_0.4.8 shape_1.4.4 xfun_0.11 slam_0.1-46
#> [5] lattice_0.20-38 splines_3.6.0 colorspace_1.4-1 vctrs_0.2.0
#> [9] stats4_3.6.0 blob_1.2.0 XML_3.98-1.20 survival_2.44-1.1
#> [13] rlang_0.4.2 pillar_1.4.2 DBI_1.0.0 BiocGenerics_0.30.0
#> [17] bit64_0.9-7 RColorBrewer_1.1-2 matrixStats_0.55.0 stringr_1.4.0
#> [21] GlobalOptions_0.1.1 evaluate_0.14 memoise_1.1.0 Biobase_2.44.0
#> [25] IRanges_2.18.3 parallel_3.6.0 AnnotationDbi_1.46.1 highr_0.8
#> [29] Rcpp_1.0.3 xtable_1.8-4 backports_1.1.5 S4Vectors_0.22.1
#> [33] annotate_1.62.0 skmeans_0.2-11 bit_1.1-14 microbenchmark_1.4-7
#> [37] brew_1.0-6 impute_1.58.0 rjson_0.2.20 png_0.1-7
#> [41] digest_0.6.23 stringi_1.4.3 polyclip_1.10-0 clue_0.3-57
#> [45] tools_3.6.0 bitops_1.0-6 magrittr_1.5 eulerr_6.0.0
#> [49] RCurl_1.95-4.12 RSQLite_2.1.4 tibble_2.1.3 cluster_2.1.0
#> [53] crayon_1.3.4 pkgconfig_2.0.3 zeallot_0.1.0 Matrix_1.2-17
#> [57] xml2_1.2.2 httr_1.4.1 R6_2.4.1 mclust_5.4.5
#> [61] compiler_3.6.0