
Date: 2019-12-25 21:01:23 CET, cola version: 1.3.2
Document is loading... 
All available functions which can be applied to this res_list object:
res_list
#> A 'ConsensusPartitionList' object with 24 methods.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows are extracted by 'SD, CV, MAD, ATC' methods.
#> Subgroups are detected by 'hclust, kmeans, skmeans, pam, mclust, NMF' method.
#> Number of partitions are tried for k = 2, 3, 4, 5, 6.
#> Performed in total 30000 partitions by row resampling.
#>
#> Following methods can be applied to this 'ConsensusPartitionList' object:
#> [1] "cola_report" "collect_classes" "collect_plots" "collect_stats"
#> [5] "colnames" "functional_enrichment" "get_anno_col" "get_anno"
#> [9] "get_classes" "get_matrix" "get_membership" "get_stats"
#> [13] "is_best_k" "is_stable_k" "ncol" "nrow"
#> [17] "rownames" "show" "suggest_best_k" "test_to_known_factors"
#> [21] "top_rows_heatmap" "top_rows_overlap"
#>
#> You can get result for a single method by, e.g. object["SD", "hclust"] or object["SD:hclust"]
#> or a subset of methods by object[c("SD", "CV")], c("hclust", "kmeans")]
The call of run_all_consensus_partition_methods() was:
#> run_all_consensus_partition_methods(data = mat, mc.cores = 4, anno = anno)
Dimension of the input matrix:
mat = get_matrix(res_list)
dim(mat)
#> [1] 21168 117
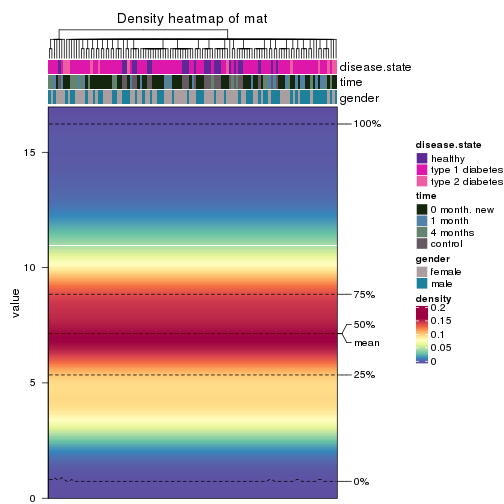
The density distribution for each sample is visualized as in one column in the following heatmap. The clustering is based on the distance which is the Kolmogorov-Smirnov statistic between two distributions.
library(ComplexHeatmap)
densityHeatmap(mat, top_annotation = HeatmapAnnotation(df = get_anno(res_list),
col = get_anno_col(res_list)), ylab = "value", cluster_columns = TRUE, show_column_names = FALSE,
mc.cores = 4)
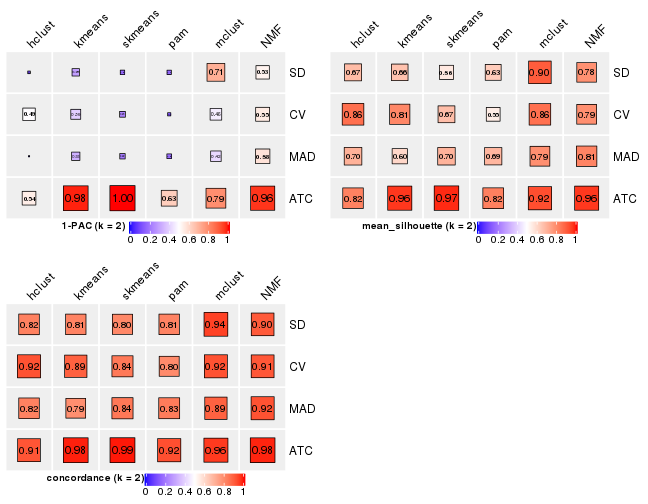
Folowing table shows the best k (number of partitions) for each combination
of top-value methods and partition methods. Clicking on the method name in
the table goes to the section for a single combination of methods.
The cola vignette explains the definition of the metrics used for determining the best number of partitions.
suggest_best_k(res_list)
| The best k | 1-PAC | Mean silhouette | Concordance | ||
|---|---|---|---|---|---|
| ATC:skmeans | 2 | 1.000 | 0.975 | 0.989 | ** |
| ATC:kmeans | 2 | 0.982 | 0.959 | 0.984 | ** |
| ATC:NMF | 2 | 0.962 | 0.957 | 0.981 | ** |
| ATC:mclust | 4 | 0.805 | 0.864 | 0.931 | |
| ATC:pam | 4 | 0.802 | 0.850 | 0.925 | |
| SD:kmeans | 4 | 0.639 | 0.713 | 0.836 | |
| MAD:NMF | 2 | 0.576 | 0.812 | 0.917 | |
| CV:NMF | 2 | 0.553 | 0.786 | 0.907 | |
| SD:NMF | 2 | 0.531 | 0.782 | 0.901 | |
| MAD:kmeans | 3 | 0.505 | 0.808 | 0.856 | |
| ATC:hclust | 3 | 0.414 | 0.670 | 0.809 | |
| CV:mclust | 4 | 0.400 | 0.657 | 0.773 | |
| CV:kmeans | 2 | 0.395 | 0.805 | 0.889 | |
| SD:mclust | 4 | 0.351 | 0.530 | 0.739 | |
| CV:skmeans | 2 | 0.231 | 0.669 | 0.838 | |
| MAD:skmeans | 2 | 0.216 | 0.696 | 0.840 | |
| MAD:pam | 2 | 0.185 | 0.689 | 0.832 | |
| SD:skmeans | 2 | 0.176 | 0.559 | 0.803 | |
| SD:pam | 2 | 0.173 | 0.632 | 0.814 | |
| MAD:mclust | 3 | 0.145 | 0.575 | 0.719 | |
| CV:pam | 2 | 0.128 | 0.548 | 0.795 | |
| SD:hclust | 3 | 0.045 | 0.530 | 0.746 | |
| MAD:hclust | 4 | 0.032 | 0.368 | 0.640 | |
| CV:hclust | NA | NA | NA | NA |
**: 1-PAC > 0.95, *: 1-PAC > 0.9
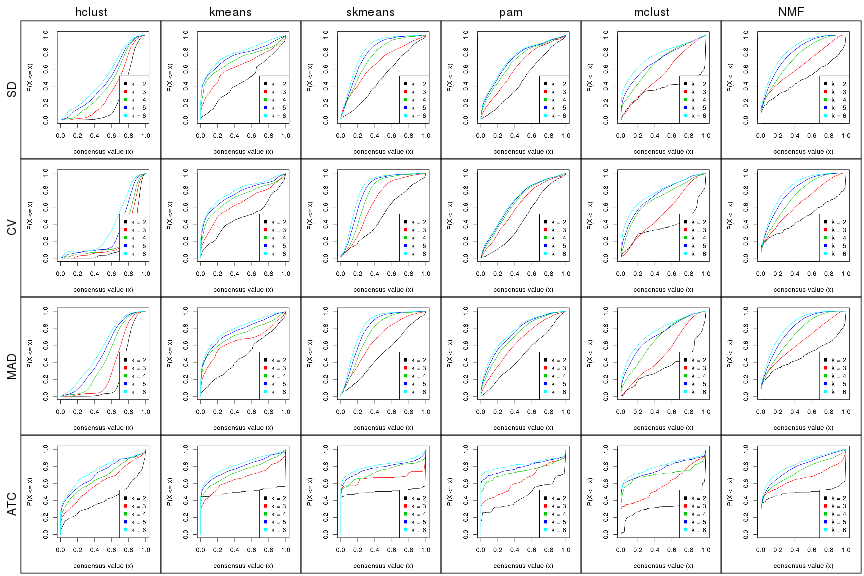
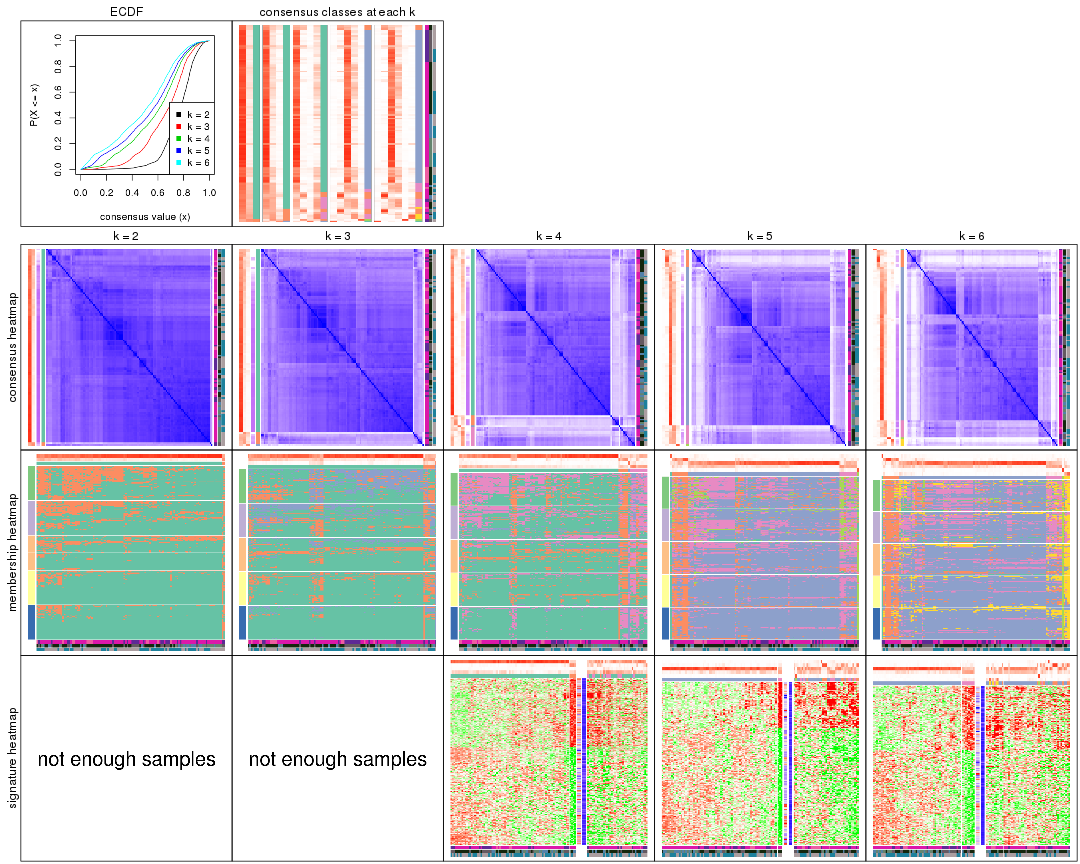
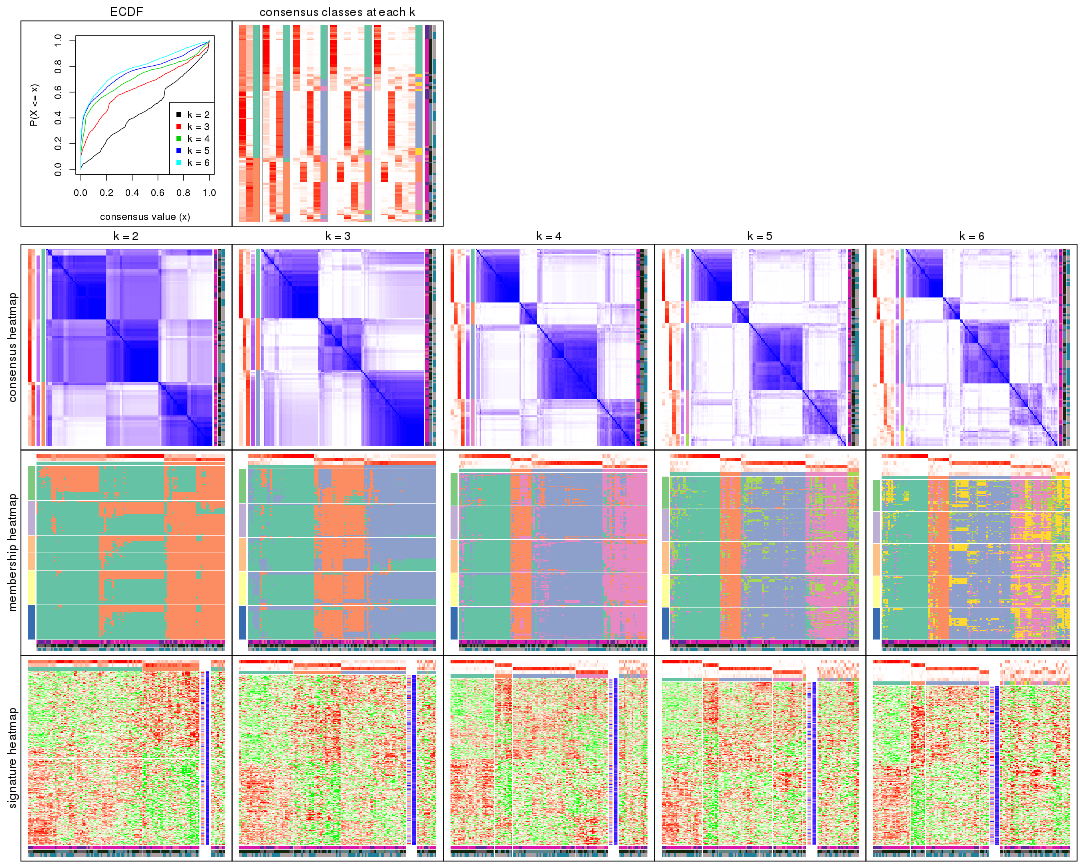
Cumulative distribution function curves of consensus matrix for all methods.
collect_plots(res_list, fun = plot_ecdf)
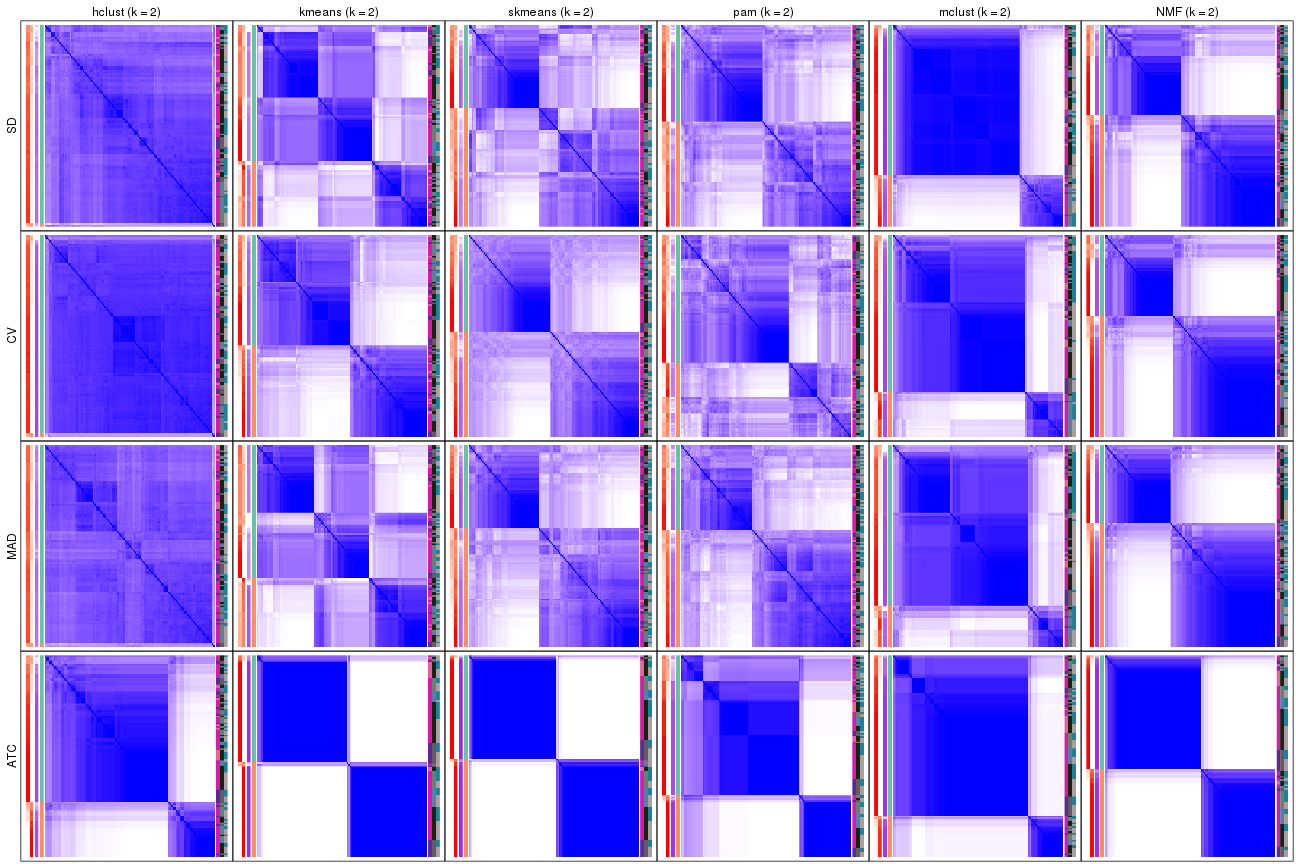
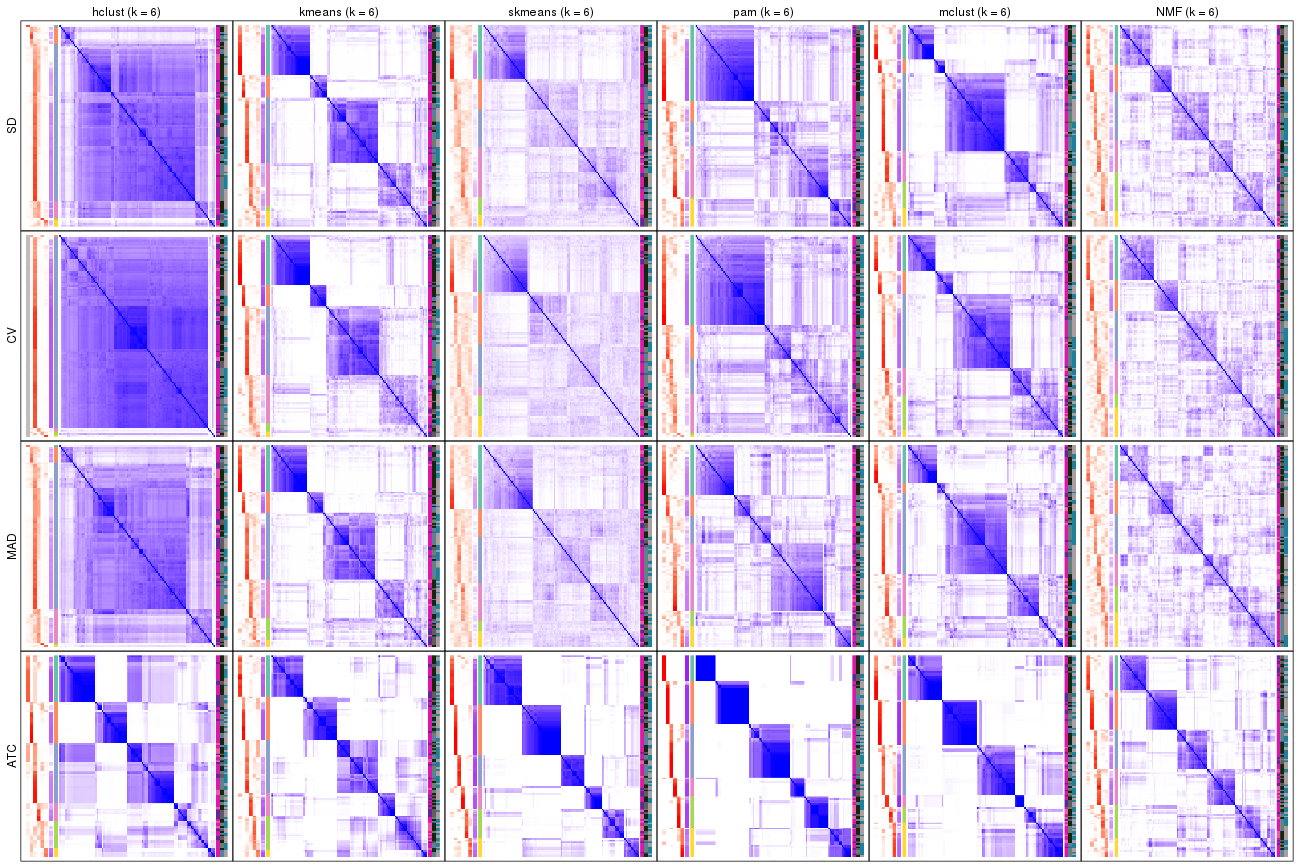
Consensus heatmaps for all methods. (What is a consensus heatmap?)
collect_plots(res_list, k = 2, fun = consensus_heatmap, mc.cores = 4)
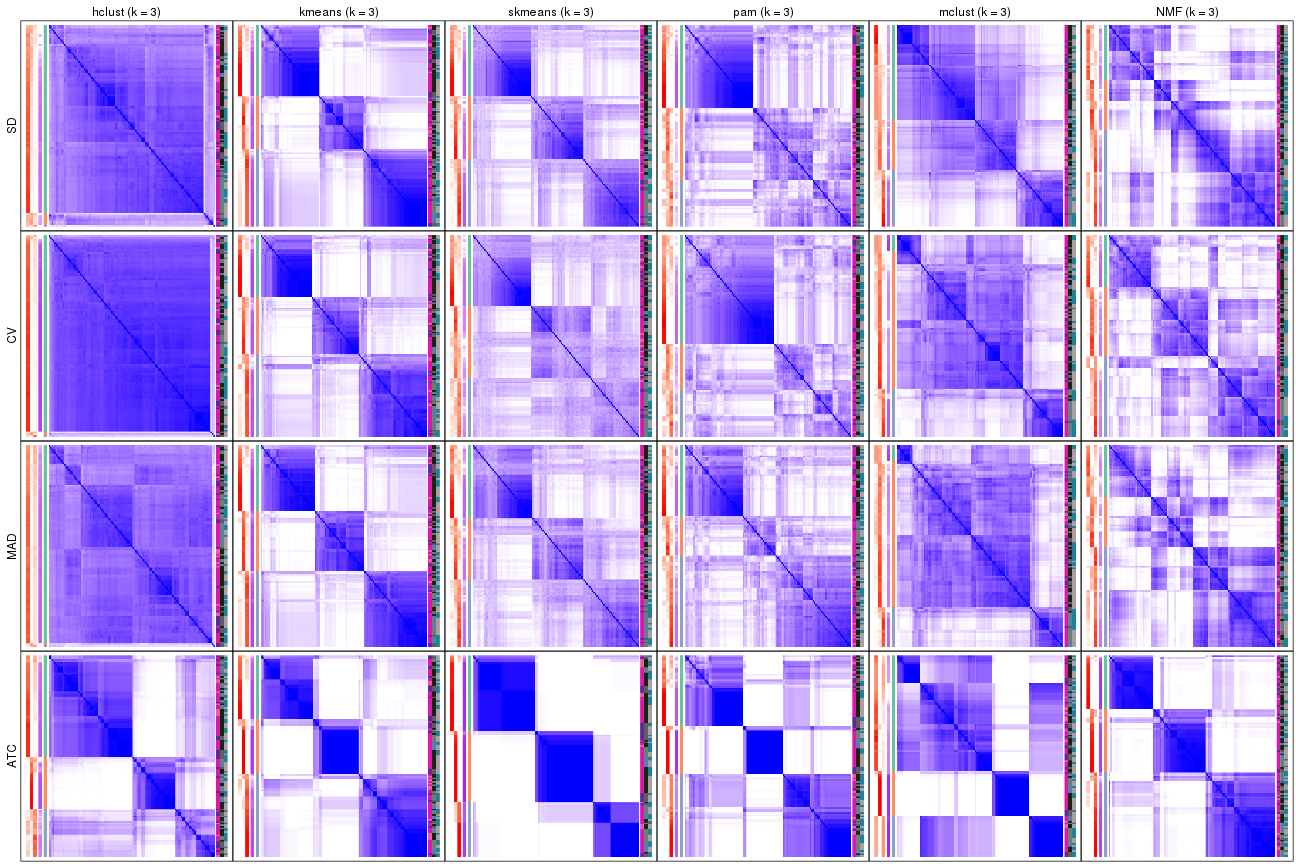
collect_plots(res_list, k = 3, fun = consensus_heatmap, mc.cores = 4)
collect_plots(res_list, k = 4, fun = consensus_heatmap, mc.cores = 4)
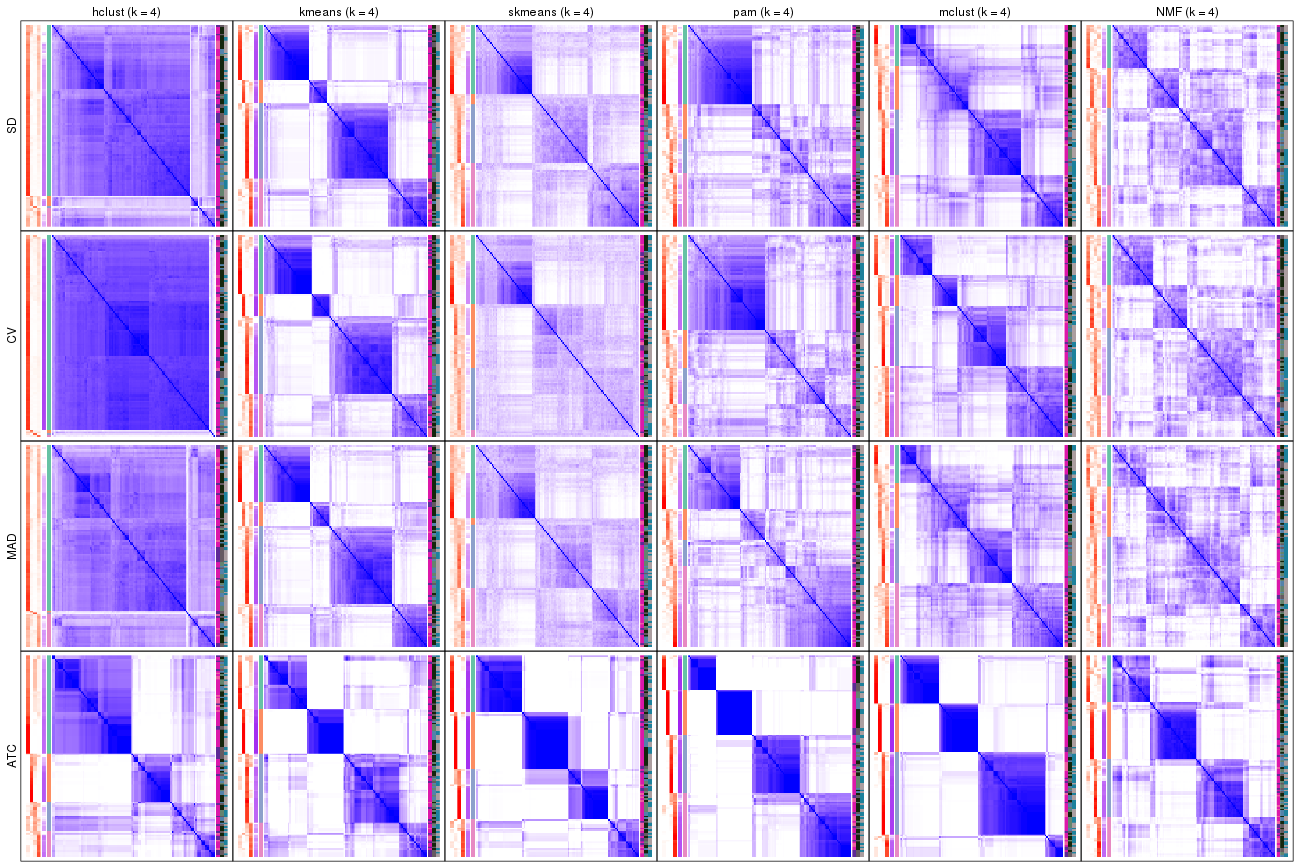
collect_plots(res_list, k = 5, fun = consensus_heatmap, mc.cores = 4)
collect_plots(res_list, k = 6, fun = consensus_heatmap, mc.cores = 4)
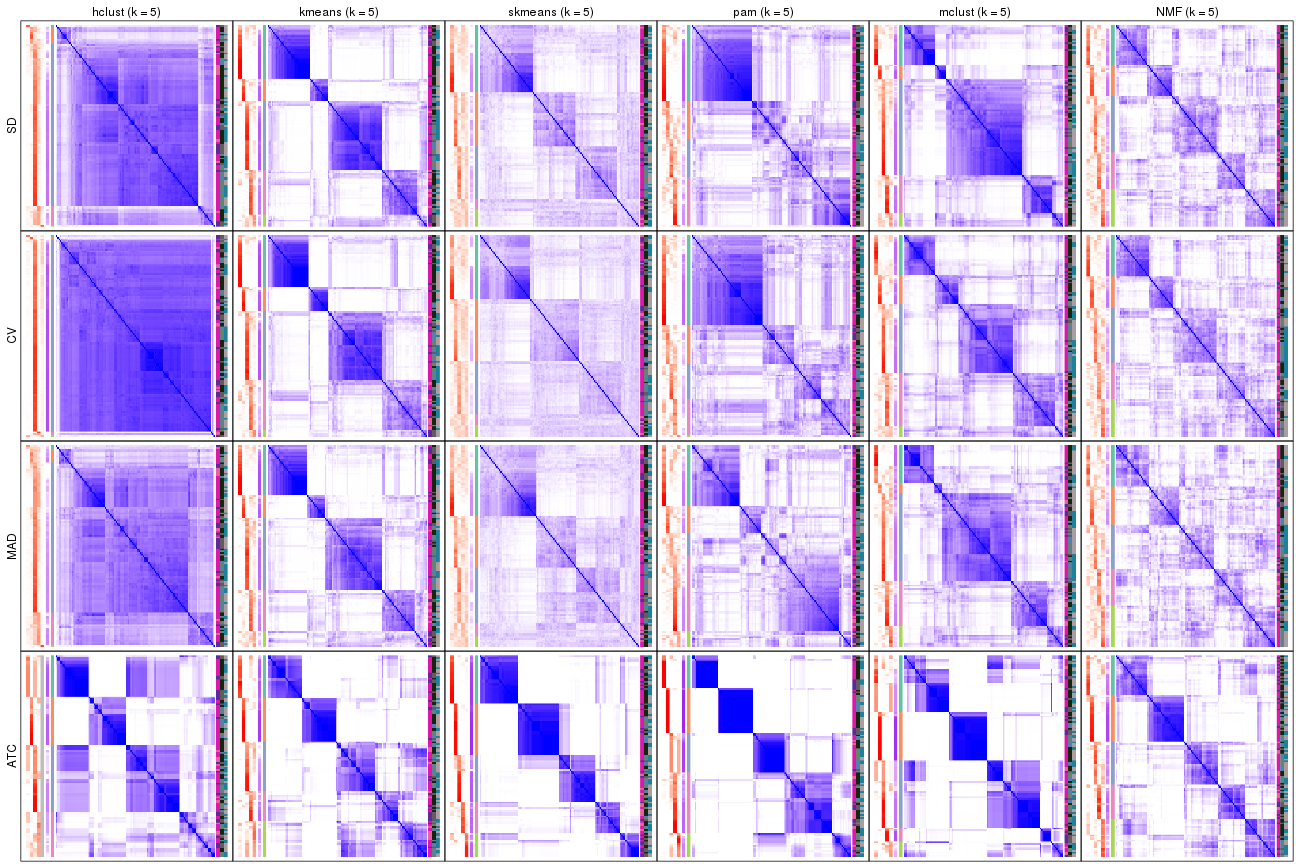
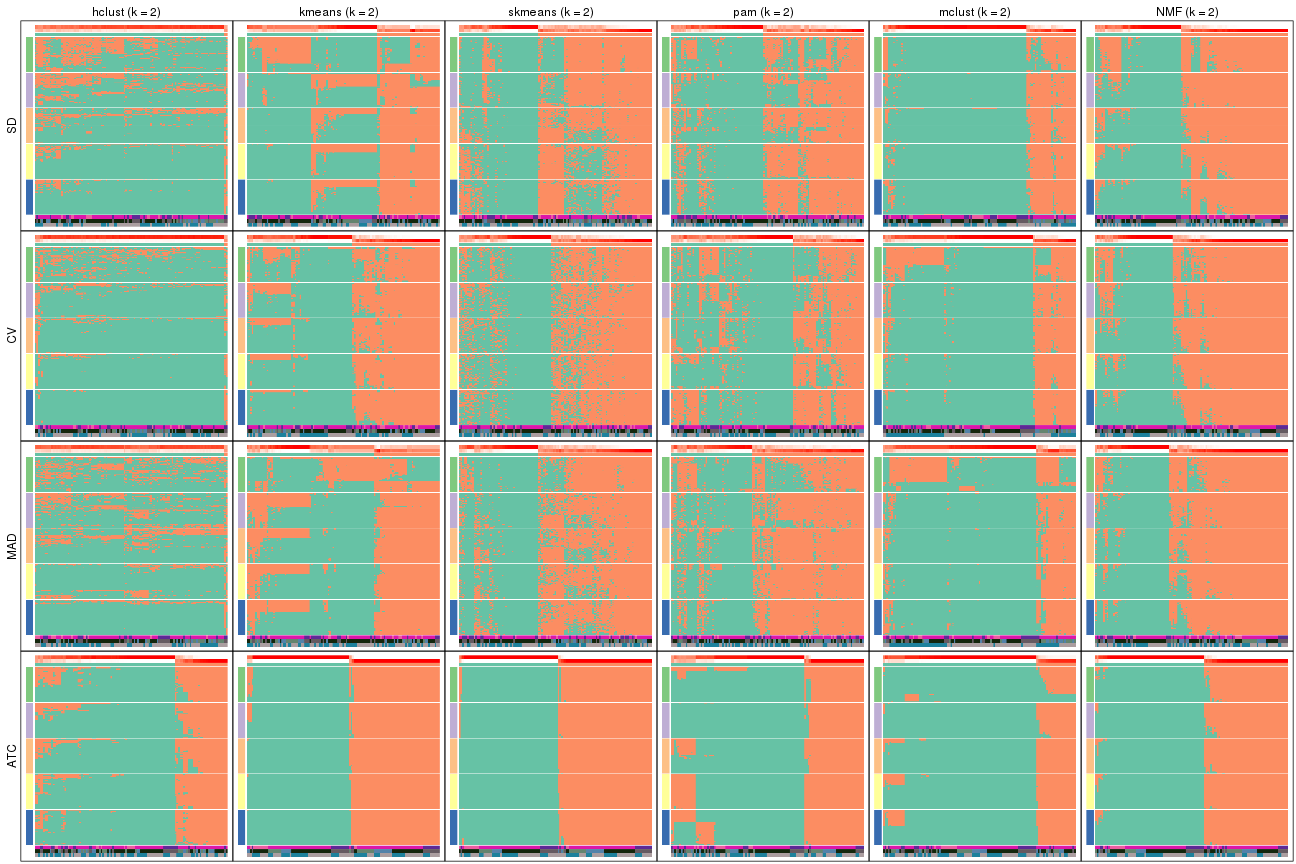
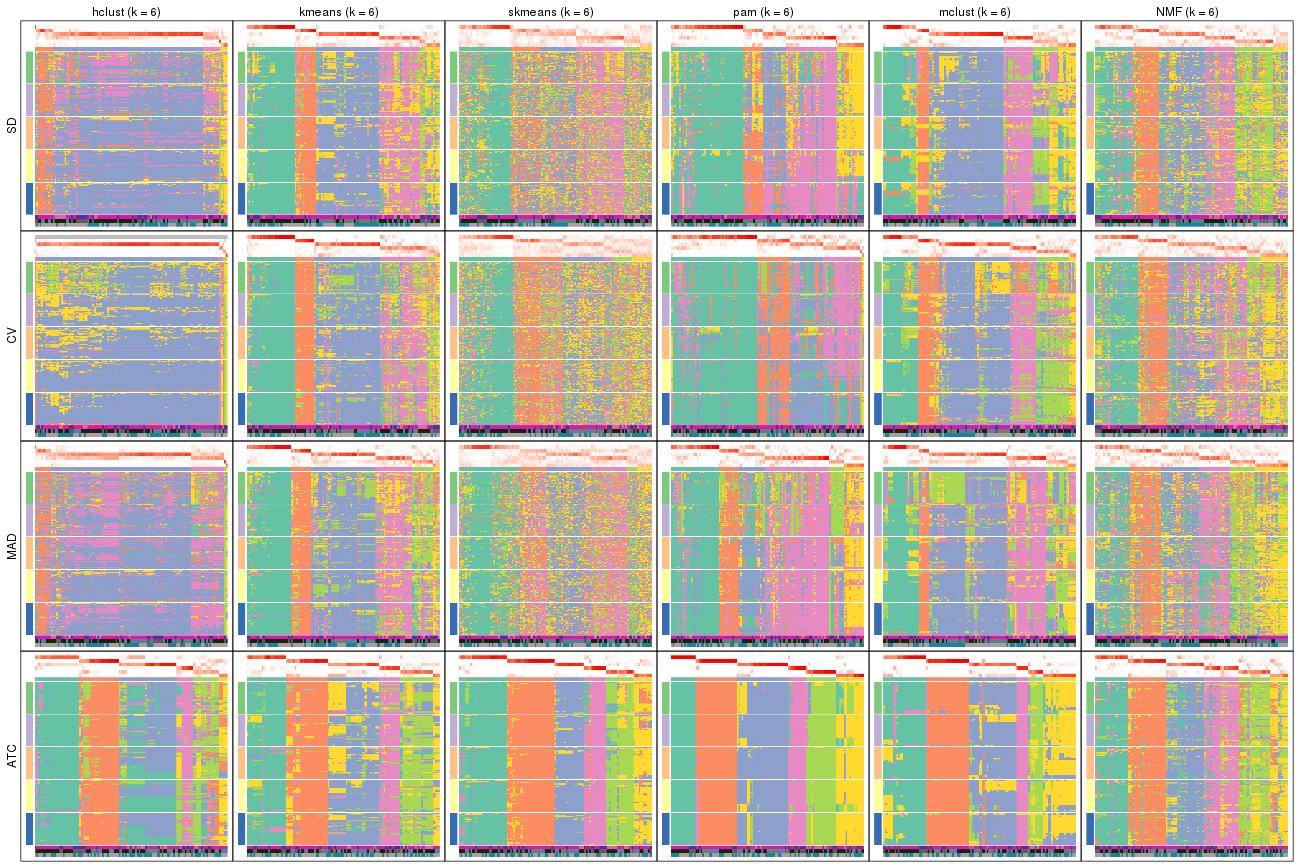
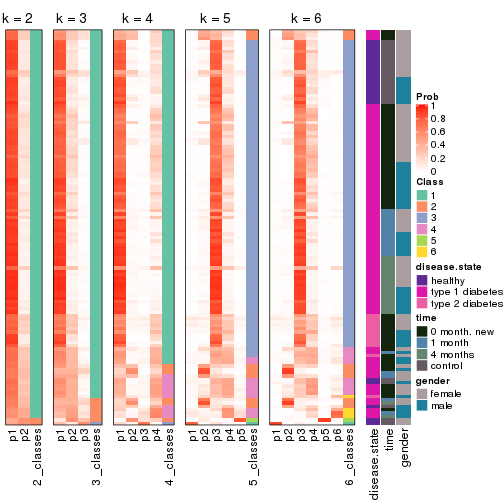
Membership heatmaps for all methods. (What is a membership heatmap?)
collect_plots(res_list, k = 2, fun = membership_heatmap, mc.cores = 4)
collect_plots(res_list, k = 3, fun = membership_heatmap, mc.cores = 4)
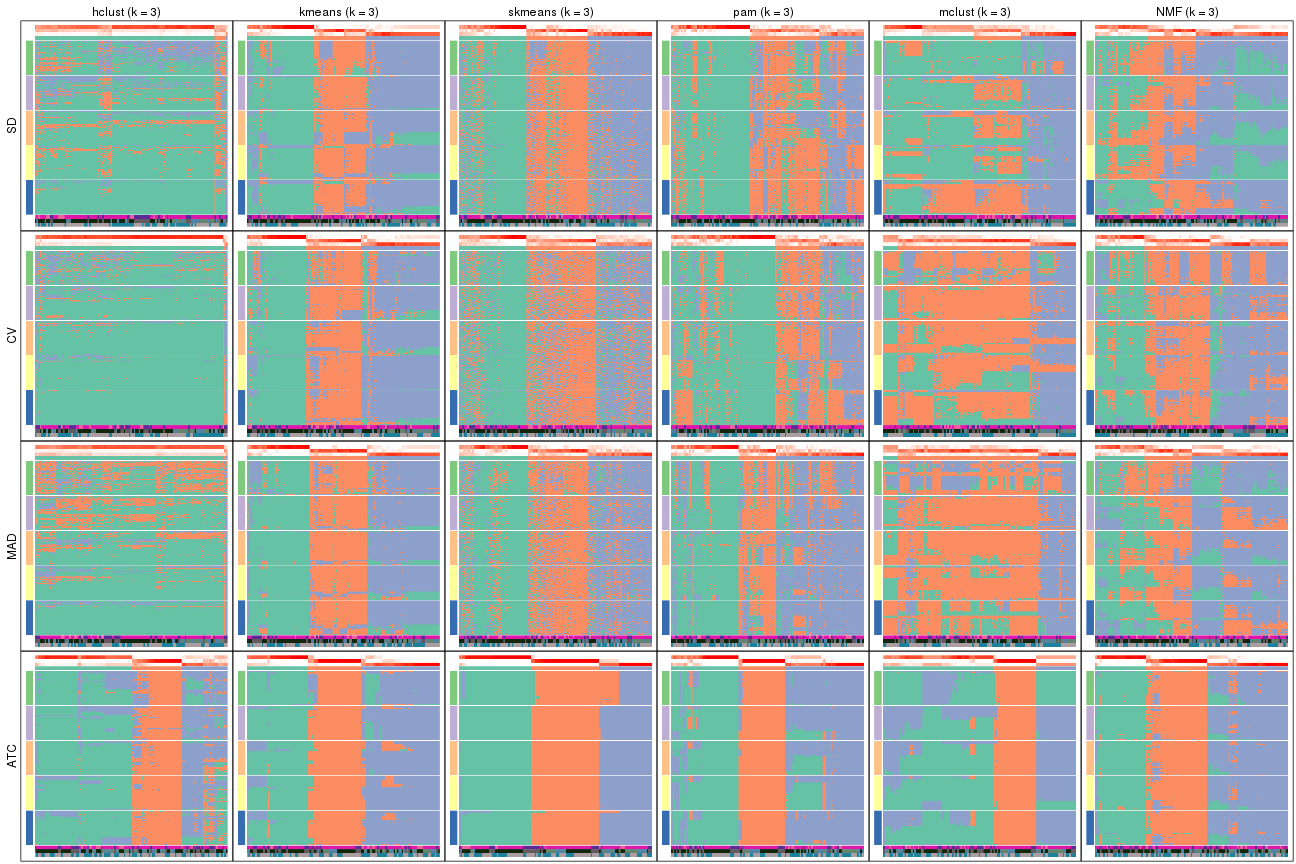
collect_plots(res_list, k = 4, fun = membership_heatmap, mc.cores = 4)

collect_plots(res_list, k = 5, fun = membership_heatmap, mc.cores = 4)

collect_plots(res_list, k = 6, fun = membership_heatmap, mc.cores = 4)
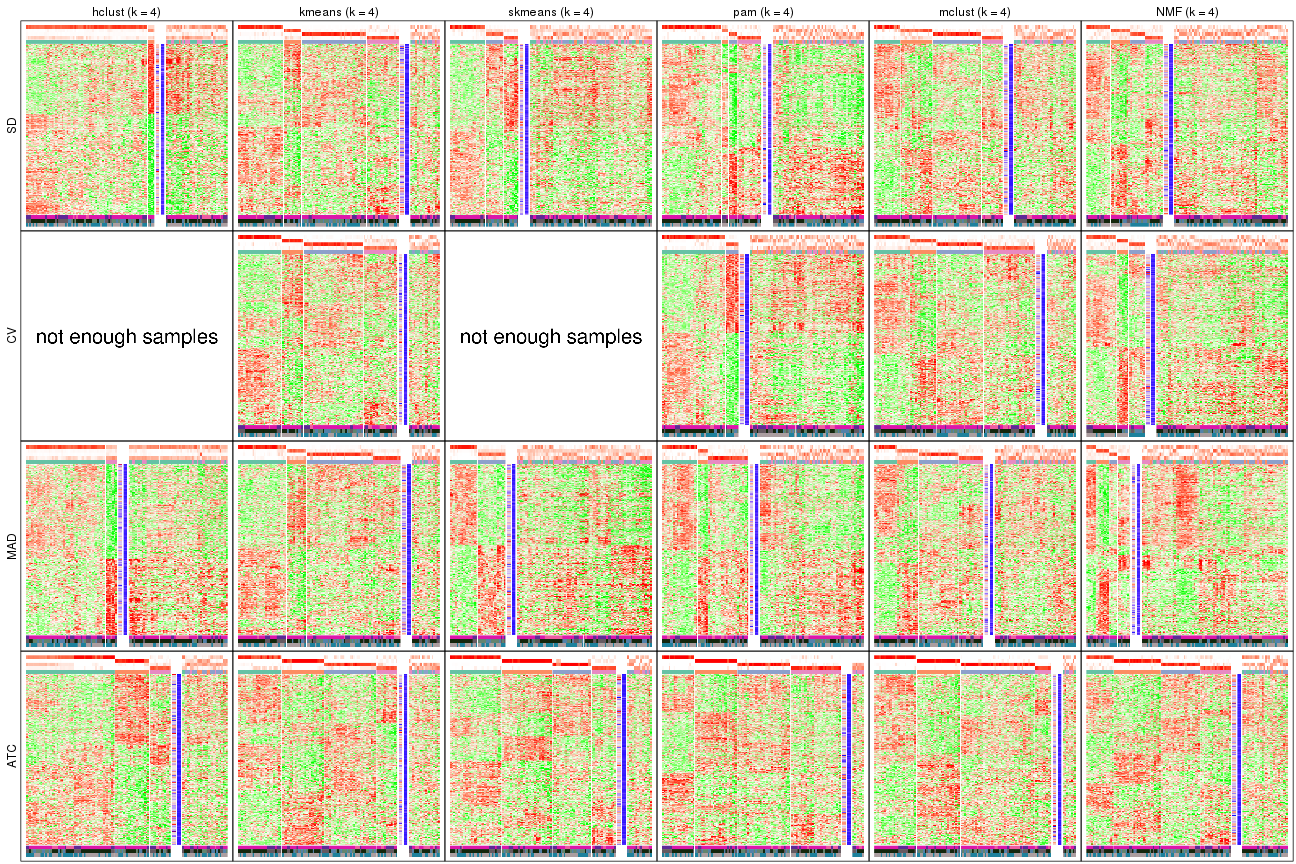
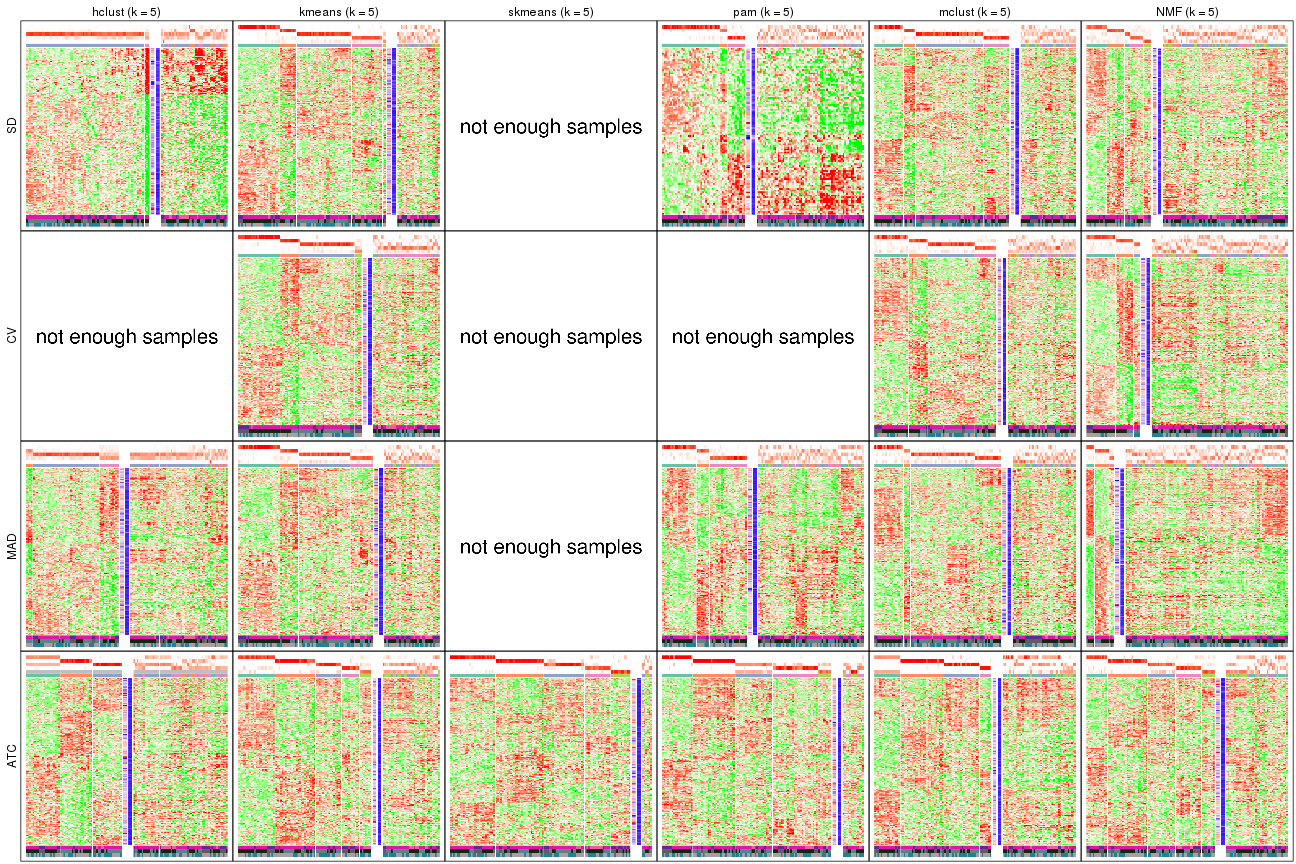
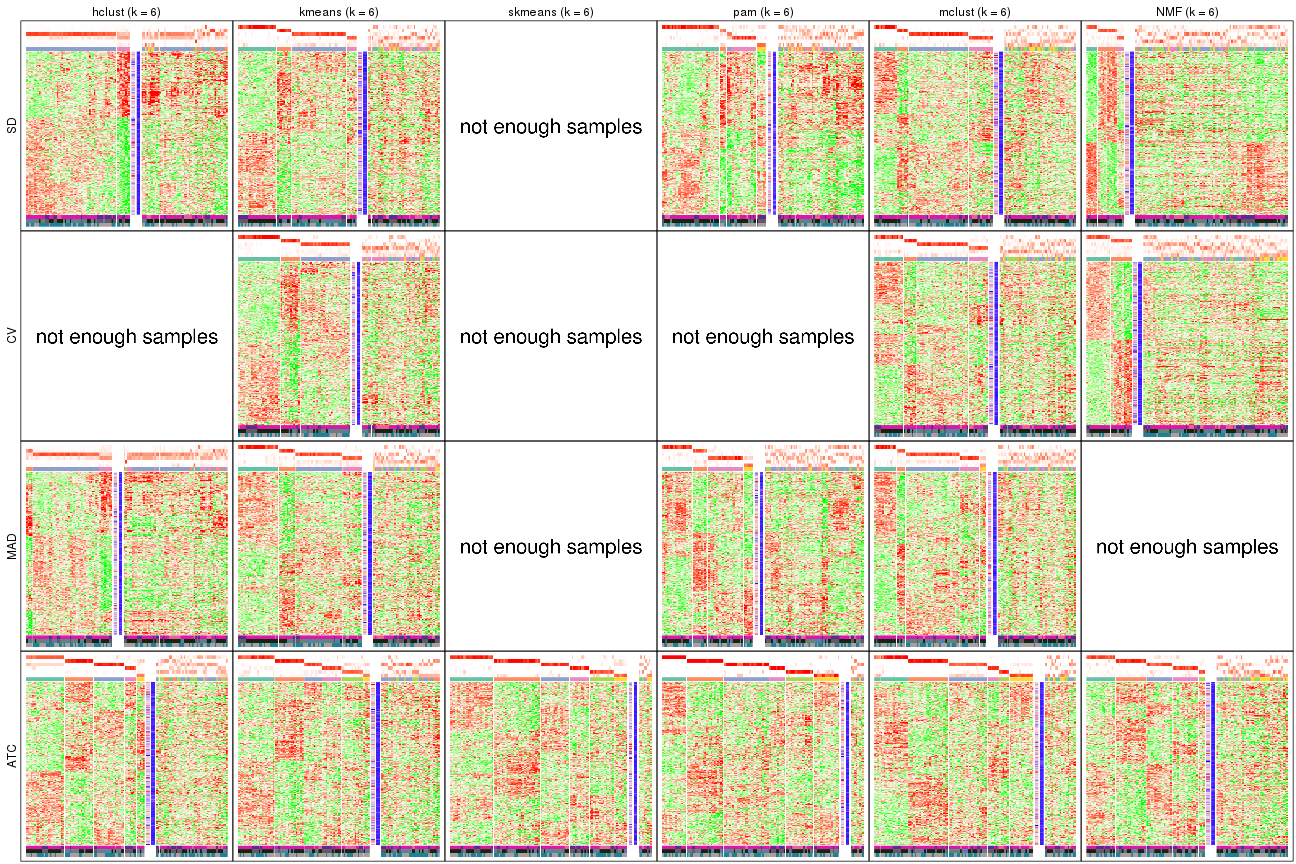
Signature heatmaps for all methods. (What is a signature heatmap?)
Note in following heatmaps, rows are scaled.
collect_plots(res_list, k = 2, fun = get_signatures, mc.cores = 4)
collect_plots(res_list, k = 3, fun = get_signatures, mc.cores = 4)
collect_plots(res_list, k = 4, fun = get_signatures, mc.cores = 4)
collect_plots(res_list, k = 5, fun = get_signatures, mc.cores = 4)
collect_plots(res_list, k = 6, fun = get_signatures, mc.cores = 4)
The statistics used for measuring the stability of consensus partitioning. (How are they defined?)
get_stats(res_list, k = 2)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> SD:NMF 2 0.531 0.782 0.901 0.485 0.502 0.502
#> CV:NMF 2 0.553 0.786 0.907 0.478 0.515 0.515
#> MAD:NMF 2 0.576 0.812 0.917 0.480 0.523 0.523
#> ATC:NMF 2 0.962 0.957 0.981 0.495 0.504 0.504
#> SD:skmeans 2 0.176 0.559 0.803 0.503 0.512 0.512
#> CV:skmeans 2 0.231 0.669 0.838 0.504 0.497 0.497
#> MAD:skmeans 2 0.216 0.696 0.840 0.503 0.512 0.512
#> ATC:skmeans 2 1.000 0.975 0.989 0.504 0.496 0.496
#> SD:mclust 2 0.707 0.897 0.944 0.368 0.615 0.615
#> CV:mclust 2 0.461 0.857 0.917 0.374 0.651 0.651
#> MAD:mclust 2 0.418 0.787 0.887 0.347 0.671 0.671
#> ATC:mclust 2 0.787 0.923 0.955 0.329 0.671 0.671
#> SD:kmeans 2 0.294 0.658 0.814 0.469 0.558 0.558
#> CV:kmeans 2 0.395 0.805 0.889 0.483 0.500 0.500
#> MAD:kmeans 2 0.330 0.605 0.786 0.479 0.546 0.546
#> ATC:kmeans 2 0.982 0.959 0.984 0.503 0.497 0.497
#> SD:pam 2 0.173 0.632 0.814 0.486 0.497 0.497
#> CV:pam 2 0.128 0.548 0.795 0.470 0.531 0.531
#> MAD:pam 2 0.185 0.689 0.832 0.490 0.509 0.509
#> ATC:pam 2 0.629 0.822 0.921 0.456 0.570 0.570
#> SD:hclust 2 0.105 0.675 0.819 0.237 0.966 0.966
#> CV:hclust 2 0.495 0.859 0.916 0.133 0.966 0.966
#> MAD:hclust 2 0.049 0.700 0.817 0.241 0.966 0.966
#> ATC:hclust 2 0.544 0.817 0.911 0.411 0.599 0.599
get_stats(res_list, k = 3)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> SD:NMF 3 0.3504 0.494 0.662 0.342 0.666 0.442
#> CV:NMF 3 0.2988 0.515 0.753 0.363 0.654 0.423
#> MAD:NMF 3 0.3491 0.446 0.686 0.353 0.736 0.543
#> ATC:NMF 3 0.7132 0.815 0.911 0.338 0.692 0.465
#> SD:skmeans 3 0.3443 0.636 0.800 0.331 0.728 0.511
#> CV:skmeans 3 0.1376 0.466 0.693 0.329 0.693 0.459
#> MAD:skmeans 3 0.2423 0.580 0.755 0.334 0.764 0.562
#> ATC:skmeans 3 0.8763 0.931 0.961 0.313 0.762 0.554
#> SD:mclust 3 0.2314 0.540 0.700 0.500 0.715 0.546
#> CV:mclust 3 0.1509 0.534 0.706 0.432 0.830 0.748
#> MAD:mclust 3 0.1450 0.575 0.719 0.485 0.813 0.738
#> ATC:mclust 3 0.5048 0.619 0.768 0.789 0.729 0.601
#> SD:kmeans 3 0.4536 0.741 0.824 0.361 0.696 0.492
#> CV:kmeans 3 0.4355 0.728 0.830 0.333 0.672 0.437
#> MAD:kmeans 3 0.5053 0.808 0.856 0.357 0.707 0.499
#> ATC:kmeans 3 0.6400 0.798 0.895 0.315 0.652 0.412
#> SD:pam 3 0.2655 0.393 0.710 0.309 0.770 0.570
#> CV:pam 3 0.2003 0.438 0.715 0.314 0.792 0.632
#> MAD:pam 3 0.2536 0.556 0.762 0.318 0.817 0.652
#> ATC:pam 3 0.5862 0.712 0.865 0.410 0.678 0.479
#> SD:hclust 3 0.0449 0.530 0.746 0.442 0.903 0.899
#> CV:hclust 3 0.2324 0.823 0.878 0.506 0.983 0.982
#> MAD:hclust 3 0.0215 0.701 0.720 0.392 1.000 1.000
#> ATC:hclust 3 0.4138 0.670 0.809 0.487 0.757 0.600
get_stats(res_list, k = 4)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> SD:NMF 4 0.3481 0.397 0.650 0.1386 0.778 0.478
#> CV:NMF 4 0.3265 0.331 0.603 0.1363 0.833 0.560
#> MAD:NMF 4 0.3140 0.338 0.594 0.1396 0.758 0.439
#> ATC:NMF 4 0.6267 0.626 0.821 0.1228 0.760 0.421
#> SD:skmeans 4 0.2996 0.336 0.636 0.1188 0.947 0.843
#> CV:skmeans 4 0.1658 0.225 0.547 0.1198 0.946 0.843
#> MAD:skmeans 4 0.2298 0.294 0.601 0.1178 0.948 0.849
#> ATC:skmeans 4 0.7586 0.789 0.895 0.1155 0.863 0.620
#> SD:mclust 4 0.3509 0.530 0.739 0.2425 0.809 0.523
#> CV:mclust 4 0.3998 0.657 0.773 0.3159 0.647 0.385
#> MAD:mclust 4 0.2844 0.481 0.690 0.3194 0.589 0.333
#> ATC:mclust 4 0.8048 0.864 0.931 0.2087 0.690 0.388
#> SD:kmeans 4 0.6390 0.713 0.836 0.1223 0.837 0.582
#> CV:kmeans 4 0.6128 0.701 0.820 0.1161 0.916 0.762
#> MAD:kmeans 4 0.6115 0.691 0.811 0.1189 0.900 0.718
#> ATC:kmeans 4 0.6771 0.673 0.827 0.1254 0.866 0.633
#> SD:pam 4 0.3594 0.472 0.714 0.1008 0.877 0.669
#> CV:pam 4 0.2531 0.380 0.648 0.0765 0.861 0.670
#> MAD:pam 4 0.3268 0.444 0.698 0.1187 0.907 0.753
#> ATC:pam 4 0.8020 0.850 0.925 0.1571 0.796 0.492
#> SD:hclust 4 0.0341 0.476 0.707 0.2295 0.844 0.822
#> CV:hclust 4 0.1338 0.813 0.860 0.1849 0.983 0.982
#> MAD:hclust 4 0.0316 0.368 0.640 0.3398 0.731 0.722
#> ATC:hclust 4 0.4947 0.590 0.755 0.1033 0.927 0.811
get_stats(res_list, k = 5)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> SD:NMF 5 0.4124 0.391 0.620 0.0749 0.854 0.526
#> CV:NMF 5 0.4057 0.392 0.583 0.0762 0.865 0.542
#> MAD:NMF 5 0.3989 0.289 0.509 0.0782 0.802 0.386
#> ATC:NMF 5 0.6113 0.558 0.742 0.0706 0.856 0.511
#> SD:skmeans 5 0.3371 0.245 0.538 0.0625 0.918 0.741
#> CV:skmeans 5 0.2401 0.189 0.497 0.0639 0.910 0.729
#> MAD:skmeans 5 0.2922 0.213 0.511 0.0627 0.910 0.729
#> ATC:skmeans 5 0.8113 0.808 0.892 0.0625 0.924 0.713
#> SD:mclust 5 0.4606 0.554 0.749 0.0349 0.815 0.503
#> CV:mclust 5 0.4486 0.463 0.697 0.0575 0.916 0.718
#> MAD:mclust 5 0.3933 0.536 0.718 0.0659 0.817 0.491
#> ATC:mclust 5 0.7507 0.552 0.763 0.0866 0.902 0.694
#> SD:kmeans 5 0.6328 0.627 0.802 0.0659 0.955 0.843
#> CV:kmeans 5 0.6244 0.605 0.786 0.0589 0.911 0.715
#> MAD:kmeans 5 0.5764 0.586 0.737 0.0625 0.941 0.799
#> ATC:kmeans 5 0.6814 0.591 0.751 0.0669 0.884 0.596
#> SD:pam 5 0.3779 0.430 0.690 0.0375 0.957 0.852
#> CV:pam 5 0.2629 0.369 0.667 0.0233 0.965 0.890
#> MAD:pam 5 0.3821 0.363 0.638 0.0450 0.936 0.793
#> ATC:pam 5 0.8296 0.764 0.877 0.0656 0.944 0.788
#> SD:hclust 5 0.0598 0.495 0.702 0.1074 0.919 0.890
#> CV:hclust 5 0.0603 0.762 0.818 0.2596 0.967 0.965
#> MAD:hclust 5 0.0367 0.393 0.621 0.1464 0.858 0.800
#> ATC:hclust 5 0.5376 0.476 0.619 0.0702 0.809 0.505
get_stats(res_list, k = 6)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> SD:NMF 6 0.4845 0.332 0.572 0.0428 0.939 0.723
#> CV:NMF 6 0.4588 0.317 0.527 0.0415 0.946 0.751
#> MAD:NMF 6 0.4531 0.291 0.522 0.0436 0.822 0.334
#> ATC:NMF 6 0.6521 0.539 0.748 0.0397 0.909 0.599
#> SD:skmeans 6 0.3900 0.167 0.462 0.0399 0.863 0.532
#> CV:skmeans 6 0.3186 0.138 0.445 0.0394 0.908 0.674
#> MAD:skmeans 6 0.3579 0.148 0.450 0.0407 0.871 0.573
#> ATC:skmeans 6 0.8052 0.779 0.872 0.0421 0.961 0.816
#> SD:mclust 6 0.5458 0.521 0.711 0.0769 0.886 0.647
#> CV:mclust 6 0.5461 0.519 0.704 0.0426 0.900 0.645
#> MAD:mclust 6 0.4906 0.447 0.670 0.0558 0.926 0.754
#> ATC:mclust 6 0.7787 0.715 0.833 0.0398 0.871 0.550
#> SD:kmeans 6 0.6175 0.551 0.734 0.0413 0.971 0.883
#> CV:kmeans 6 0.6026 0.526 0.720 0.0414 0.967 0.873
#> MAD:kmeans 6 0.6034 0.520 0.694 0.0403 0.955 0.822
#> ATC:kmeans 6 0.7068 0.571 0.750 0.0398 0.900 0.594
#> SD:pam 6 0.3974 0.446 0.693 0.0190 0.940 0.789
#> CV:pam 6 0.2783 0.362 0.661 0.0151 0.991 0.968
#> MAD:pam 6 0.4152 0.406 0.657 0.0268 0.915 0.694
#> ATC:pam 6 0.8796 0.829 0.917 0.0406 0.878 0.524
#> SD:hclust 6 0.1273 0.424 0.678 0.1006 0.953 0.928
#> CV:hclust 6 0.0501 0.693 0.773 0.2358 1.000 1.000
#> MAD:hclust 6 0.0621 0.384 0.619 0.0925 0.971 0.950
#> ATC:hclust 6 0.5494 0.516 0.694 0.0487 0.916 0.690
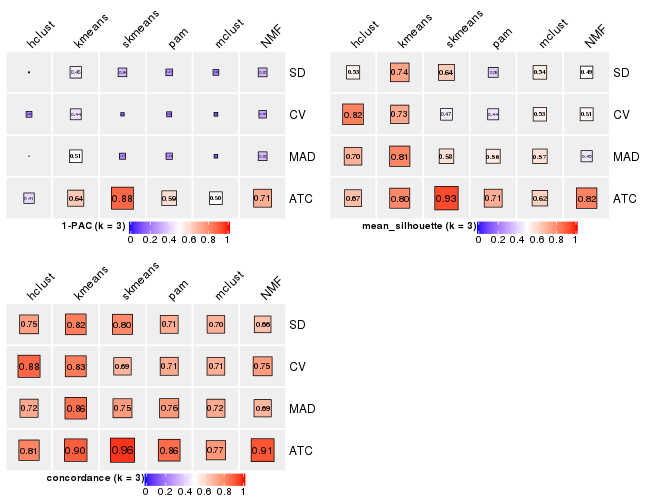
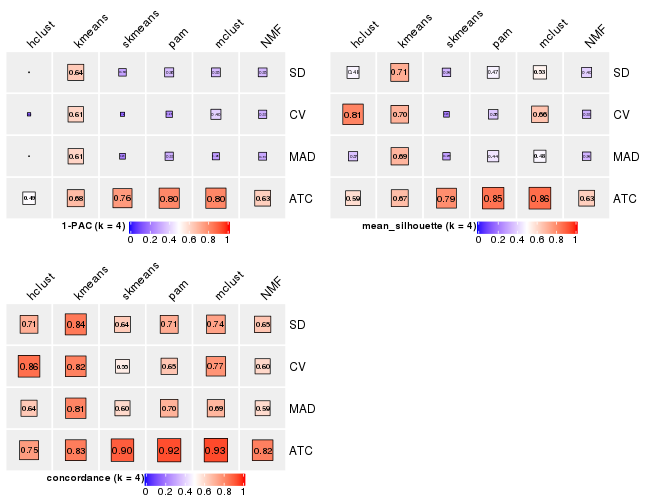
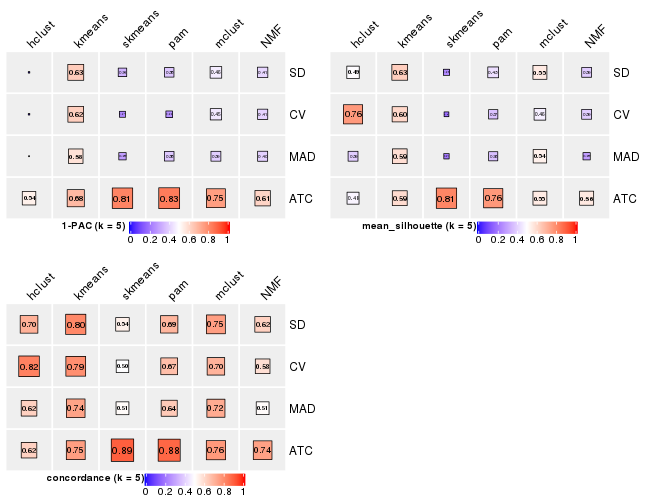
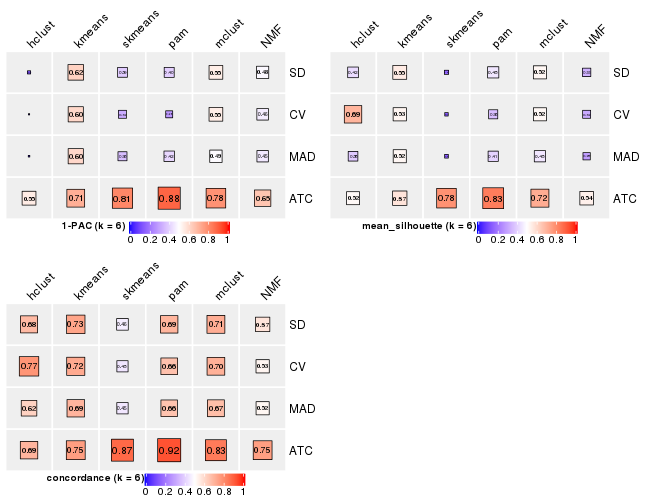
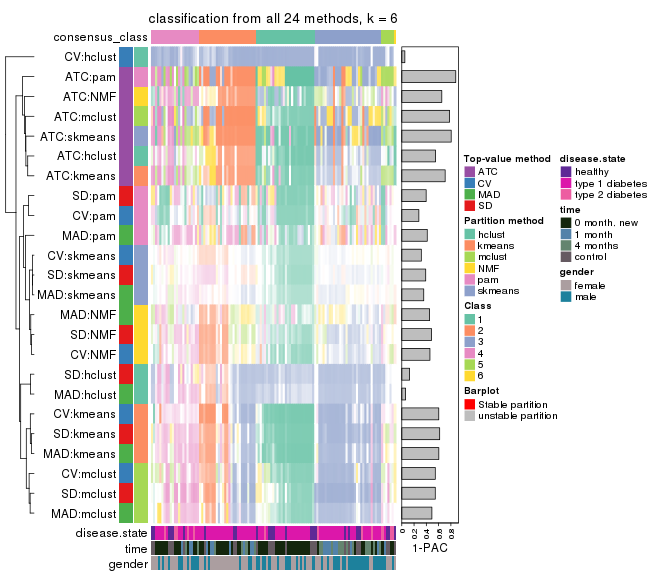
Following heatmap plots the partition for each combination of methods and the lightness correspond to the silhouette scores for samples in each method. On top the consensus subgroup is inferred from all methods by taking the mean silhouette scores as weight.
collect_stats(res_list, k = 2)
collect_stats(res_list, k = 3)
collect_stats(res_list, k = 4)
collect_stats(res_list, k = 5)
collect_stats(res_list, k = 6)
Collect partitions from all methods:
collect_classes(res_list, k = 2)
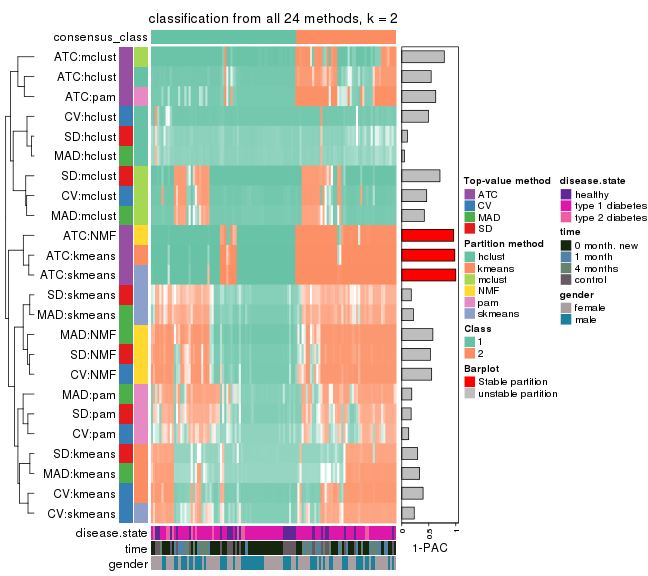
collect_classes(res_list, k = 3)
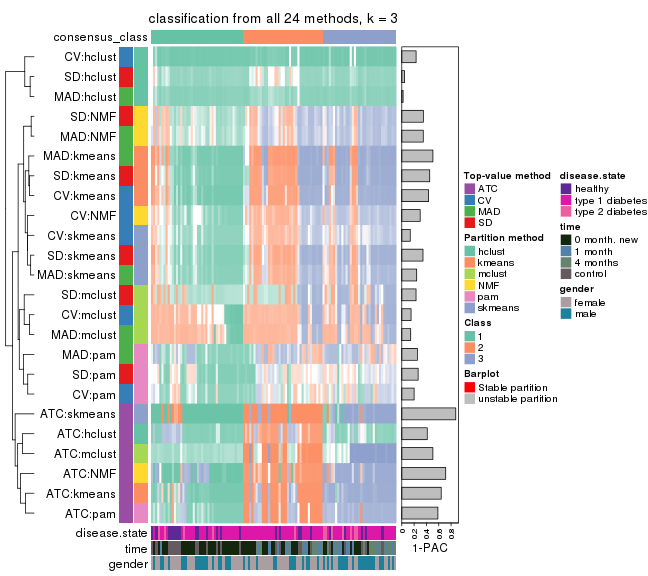
collect_classes(res_list, k = 4)
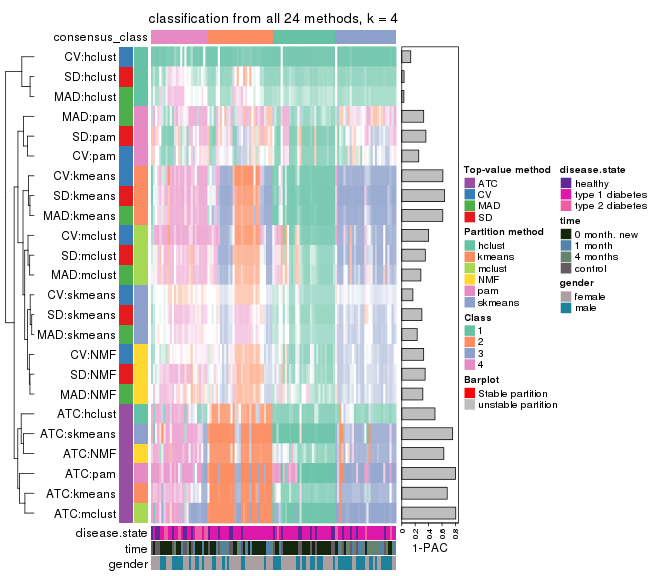
collect_classes(res_list, k = 5)
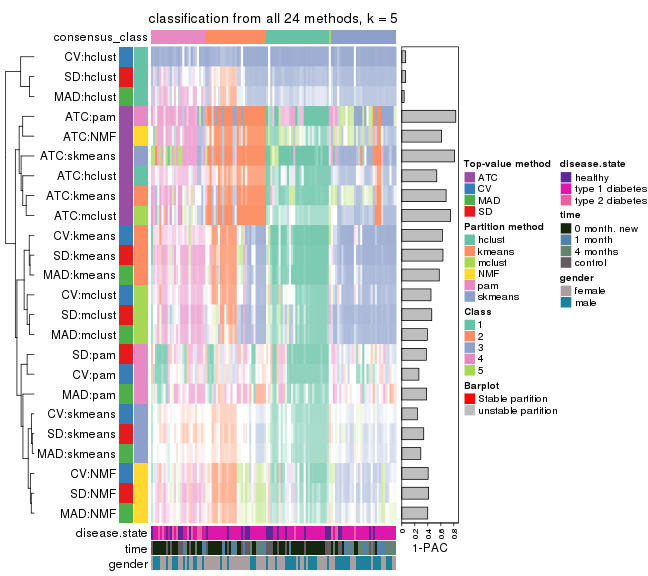
collect_classes(res_list, k = 6)
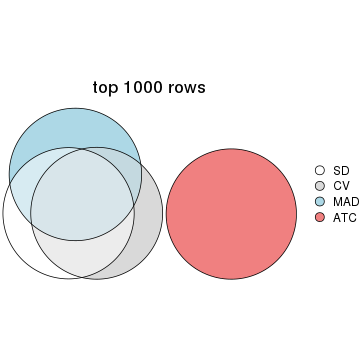
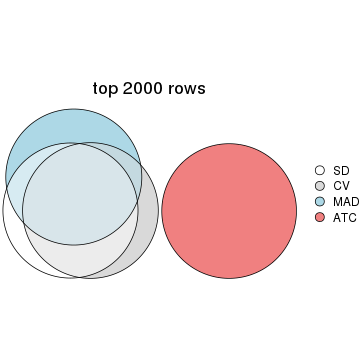
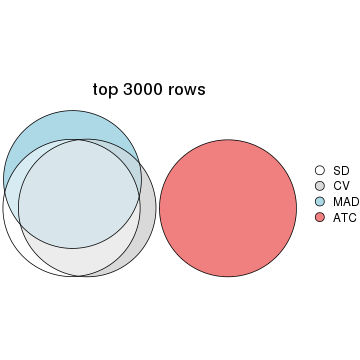
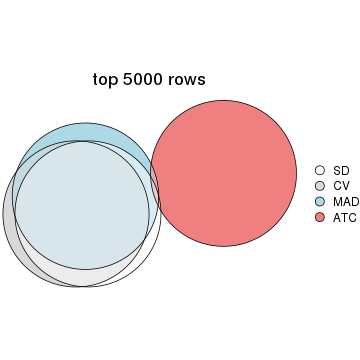
Overlap of top rows from different top-row methods:
top_rows_overlap(res_list, top_n = 1000, method = "euler")
top_rows_overlap(res_list, top_n = 2000, method = "euler")
top_rows_overlap(res_list, top_n = 3000, method = "euler")
top_rows_overlap(res_list, top_n = 4000, method = "euler")
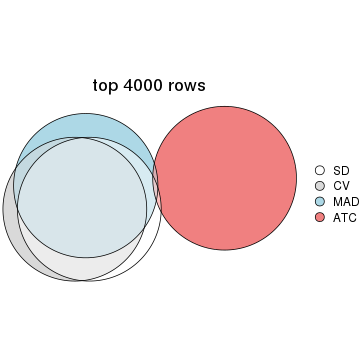
top_rows_overlap(res_list, top_n = 5000, method = "euler")
Also visualize the correspondance of rankings between different top-row methods:
top_rows_overlap(res_list, top_n = 1000, method = "correspondance")
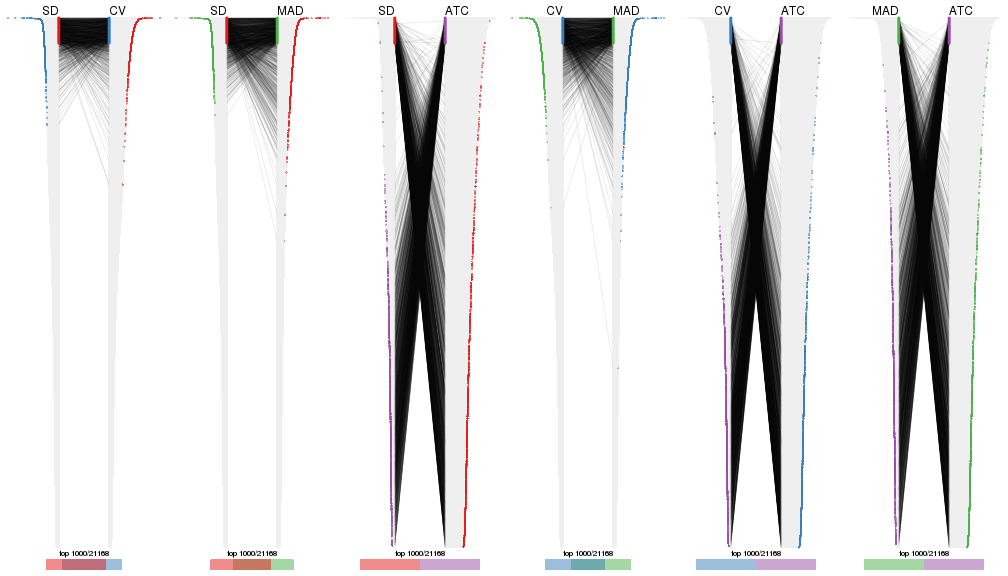
top_rows_overlap(res_list, top_n = 2000, method = "correspondance")
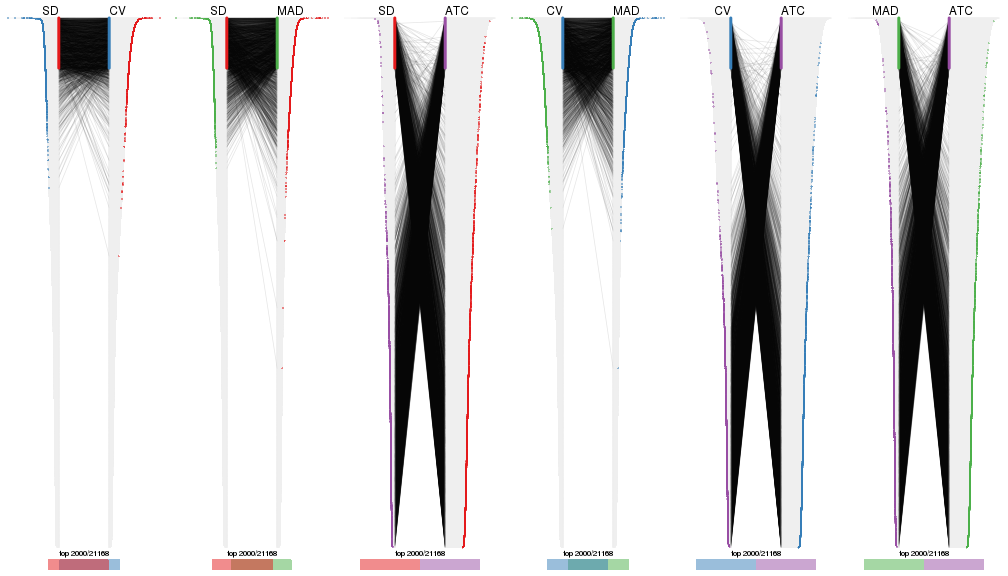
top_rows_overlap(res_list, top_n = 3000, method = "correspondance")
top_rows_overlap(res_list, top_n = 4000, method = "correspondance")
top_rows_overlap(res_list, top_n = 5000, method = "correspondance")
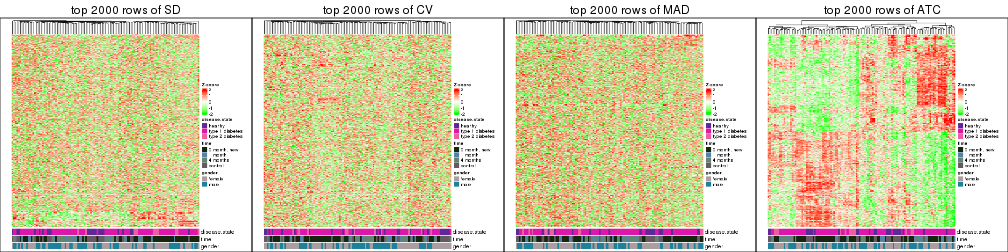
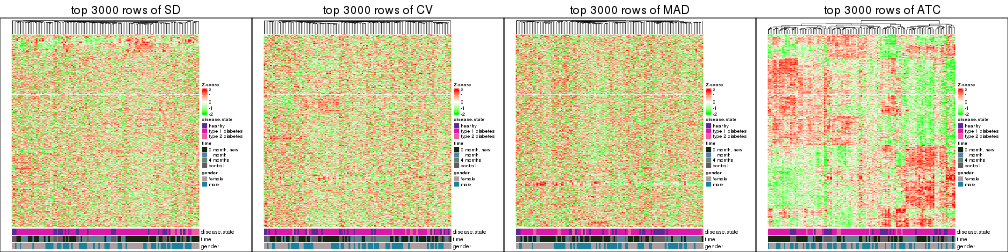
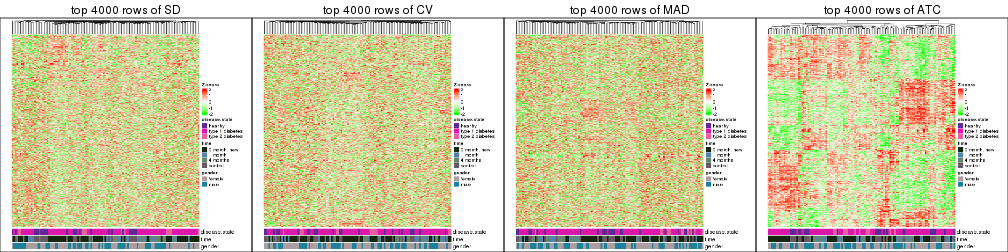
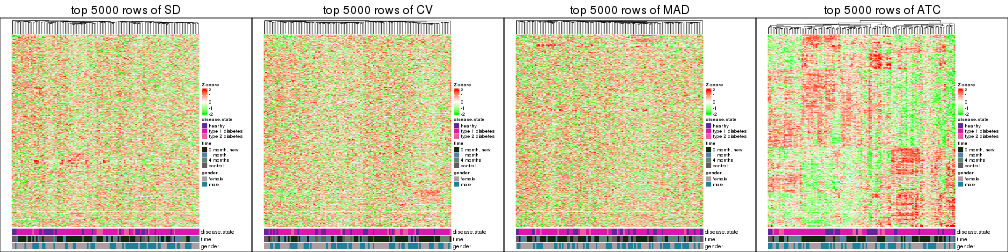
Heatmaps of the top rows:
top_rows_heatmap(res_list, top_n = 1000)
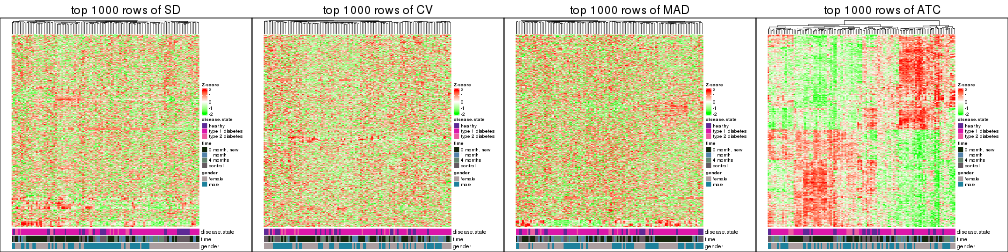
top_rows_heatmap(res_list, top_n = 2000)
top_rows_heatmap(res_list, top_n = 3000)
top_rows_heatmap(res_list, top_n = 4000)
top_rows_heatmap(res_list, top_n = 5000)
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res_list, k = 2)
#> n disease.state(p) time(p) gender(p) k
#> SD:NMF 105 0.24757 3.70e-03 0.5932 2
#> CV:NMF 102 0.22885 2.15e-04 0.3159 2
#> MAD:NMF 105 0.36897 2.57e-05 0.8282 2
#> ATC:NMF 116 0.10944 3.65e-01 0.9767 2
#> SD:skmeans 80 0.92856 3.99e-05 1.0000 2
#> CV:skmeans 94 0.24316 2.65e-01 0.5627 2
#> MAD:skmeans 101 0.48771 1.46e-07 1.0000 2
#> ATC:skmeans 116 0.09394 1.28e-01 0.6945 2
#> SD:mclust 113 0.21746 1.61e-09 0.3545 2
#> CV:mclust 113 0.00691 3.53e-11 0.2239 2
#> MAD:mclust 108 0.06482 1.68e-09 0.5045 2
#> ATC:mclust 115 0.07283 6.73e-01 0.2052 2
#> SD:kmeans 108 0.12983 4.83e-01 0.2422 2
#> CV:kmeans 113 0.14536 8.91e-01 0.7132 2
#> MAD:kmeans 101 0.18886 6.89e-01 0.6201 2
#> ATC:kmeans 115 0.09531 3.72e-01 1.0000 2
#> SD:pam 92 0.15387 3.69e-04 0.2082 2
#> CV:pam 76 0.44567 5.60e-03 0.0111 2
#> MAD:pam 98 0.32304 1.35e-02 0.1099 2
#> ATC:pam 102 0.02852 4.62e-02 0.2903 2
#> SD:hclust 105 NA NA NA 2
#> CV:hclust 114 0.02199 5.42e-02 0.5712 2
#> MAD:hclust 115 0.01706 4.32e-02 0.5622 2
#> ATC:hclust 109 0.02074 1.17e-01 0.2690 2
test_to_known_factors(res_list, k = 3)
#> n disease.state(p) time(p) gender(p) k
#> SD:NMF 70 0.3594 1.83e-03 0.41860 3
#> CV:NMF 82 0.2443 7.52e-07 0.00348 3
#> MAD:NMF 56 0.1593 9.71e-03 0.71809 3
#> ATC:NMF 110 0.0423 2.56e-03 0.36903 3
#> SD:skmeans 91 0.0837 1.66e-08 0.21470 3
#> CV:skmeans 70 0.5935 4.93e-07 0.29918 3
#> MAD:skmeans 85 0.1757 7.37e-08 0.44194 3
#> ATC:skmeans 116 0.0989 6.45e-06 0.45534 3
#> SD:mclust 64 0.0019 3.26e-05 0.84021 3
#> CV:mclust 93 0.0437 8.26e-08 0.52230 3
#> MAD:mclust 96 0.0418 1.18e-07 0.37022 3
#> ATC:mclust 91 0.0701 6.34e-05 0.23290 3
#> SD:kmeans 105 0.1950 3.12e-07 0.13939 3
#> CV:kmeans 101 0.6320 2.93e-07 0.16270 3
#> MAD:kmeans 114 0.0925 5.00e-07 0.22399 3
#> ATC:kmeans 105 0.0550 9.13e-04 0.06394 3
#> SD:pam 39 NA NA NA 3
#> CV:pam 52 0.5427 3.24e-02 0.43399 3
#> MAD:pam 84 0.1933 6.14e-04 0.17966 3
#> ATC:pam 103 0.0110 1.96e-04 0.02790 3
#> SD:hclust 86 0.8128 2.50e-01 1.00000 3
#> CV:hclust 111 NA NA NA 3
#> MAD:hclust 110 NA NA NA 3
#> ATC:hclust 99 0.0687 3.73e-01 0.18471 3
test_to_known_factors(res_list, k = 4)
#> n disease.state(p) time(p) gender(p) k
#> SD:NMF 47 0.090977 1.82e-02 1.62e-04 4
#> CV:NMF 36 0.296361 6.61e-04 1.17e-05 4
#> MAD:NMF 27 0.057914 9.16e-03 2.36e-02 4
#> ATC:NMF 89 0.006102 1.12e-02 4.77e-01 4
#> SD:skmeans 42 0.621935 2.22e-05 8.49e-01 4
#> CV:skmeans 19 NA NA NA 4
#> MAD:skmeans 34 1.000000 2.03e-06 7.27e-01 4
#> ATC:skmeans 102 0.074581 7.85e-03 7.33e-01 4
#> SD:mclust 79 0.001220 2.41e-06 3.13e-01 4
#> CV:mclust 99 0.000209 9.09e-08 6.09e-02 4
#> MAD:mclust 67 0.000627 5.07e-05 2.66e-01 4
#> ATC:mclust 109 0.002492 8.32e-04 8.52e-02 4
#> SD:kmeans 99 0.000710 7.47e-06 1.90e-01 4
#> CV:kmeans 98 0.004610 4.98e-06 5.23e-02 4
#> MAD:kmeans 100 0.000448 3.93e-05 1.02e-01 4
#> ATC:kmeans 98 0.048514 1.92e-04 3.83e-01 4
#> SD:pam 61 0.015946 1.08e-06 1.24e-02 4
#> CV:pam 47 0.804712 1.78e-03 1.00e+00 4
#> MAD:pam 54 0.116310 2.80e-03 1.13e-01 4
#> ATC:pam 110 0.003596 2.50e-05 5.74e-01 4
#> SD:hclust 79 0.466378 3.05e-01 5.90e-01 4
#> CV:hclust 111 NA NA NA 4
#> MAD:hclust 56 0.230066 2.20e-01 6.83e-01 4
#> ATC:hclust 89 0.045113 2.47e-01 4.64e-01 4
test_to_known_factors(res_list, k = 5)
#> n disease.state(p) time(p) gender(p) k
#> SD:NMF 40 5.86e-02 2.61e-02 0.019770 5
#> CV:NMF 34 5.93e-02 1.21e-02 0.000314 5
#> MAD:NMF 17 1.16e-01 3.52e-01 0.237983 5
#> ATC:NMF 79 1.47e-03 5.80e-02 0.741712 5
#> SD:skmeans 16 NA NA NA 5
#> CV:skmeans 17 NA NA NA 5
#> MAD:skmeans 16 NA NA NA 5
#> ATC:skmeans 111 6.81e-10 1.10e-04 0.585412 5
#> SD:mclust 83 4.51e-03 1.46e-04 0.250186 5
#> CV:mclust 75 5.46e-04 5.04e-06 0.063654 5
#> MAD:mclust 78 1.66e-03 3.54e-04 0.393236 5
#> ATC:mclust 72 5.06e-02 1.94e-03 0.094190 5
#> SD:kmeans 91 1.28e-02 9.48e-06 0.095330 5
#> CV:kmeans 76 7.93e-03 1.54e-08 0.019772 5
#> MAD:kmeans 83 1.04e-03 2.11e-06 0.125037 5
#> ATC:kmeans 82 6.84e-12 8.42e-05 0.213951 5
#> SD:pam 51 9.10e-03 3.58e-02 0.008454 5
#> CV:pam 39 NA NA NA 5
#> MAD:pam 52 7.12e-01 3.44e-04 0.054559 5
#> ATC:pam 104 2.09e-02 5.05e-03 0.444623 5
#> SD:hclust 76 5.73e-01 2.09e-01 0.220530 5
#> CV:hclust 110 NA NA NA 5
#> MAD:hclust 57 6.76e-01 2.56e-02 0.066872 5
#> ATC:hclust 60 4.32e-04 3.85e-03 0.277825 5
test_to_known_factors(res_list, k = 6)
#> n disease.state(p) time(p) gender(p) k
#> SD:NMF 23 2.44e-01 4.60e-01 0.3154 6
#> CV:NMF 28 1.00e+00 1.44e-01 1.0000 6
#> MAD:NMF 9 NA 6.38e-01 NA 6
#> ATC:NMF 73 2.33e-04 6.69e-04 0.6083 6
#> SD:skmeans 14 NA NA NA 6
#> CV:skmeans 12 NA NA NA 6
#> MAD:skmeans 10 NA NA NA 6
#> ATC:skmeans 109 9.75e-09 8.14e-05 0.5876 6
#> SD:mclust 73 3.06e-02 4.12e-04 0.4532 6
#> CV:mclust 70 2.82e-02 1.84e-05 0.1661 6
#> MAD:mclust 69 2.35e-02 2.46e-05 0.6289 6
#> ATC:mclust 98 6.50e-05 2.40e-03 0.4167 6
#> SD:kmeans 73 8.91e-03 3.80e-06 0.1176 6
#> CV:kmeans 69 3.20e-01 7.36e-07 0.0472 6
#> MAD:kmeans 76 3.09e-02 1.19e-06 0.1843 6
#> ATC:kmeans 81 5.99e-07 8.70e-06 0.4719 6
#> SD:pam 64 2.91e-03 1.76e-06 0.0110 6
#> CV:pam 38 NA NA NA 6
#> MAD:pam 56 6.15e-01 2.12e-03 0.1132 6
#> ATC:pam 109 1.64e-07 2.14e-04 0.6793 6
#> SD:hclust 64 7.83e-01 1.25e-01 0.8474 6
#> CV:hclust 98 NA NA NA 6
#> MAD:hclust 53 8.25e-01 9.59e-02 0.1223 6
#> ATC:hclust 73 1.23e-03 5.79e-06 0.4802 6
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["SD", "hclust"]
# you can also extract it by
# res = res_list["SD:hclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'SD' method.
#> Subgroups are detected by 'hclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
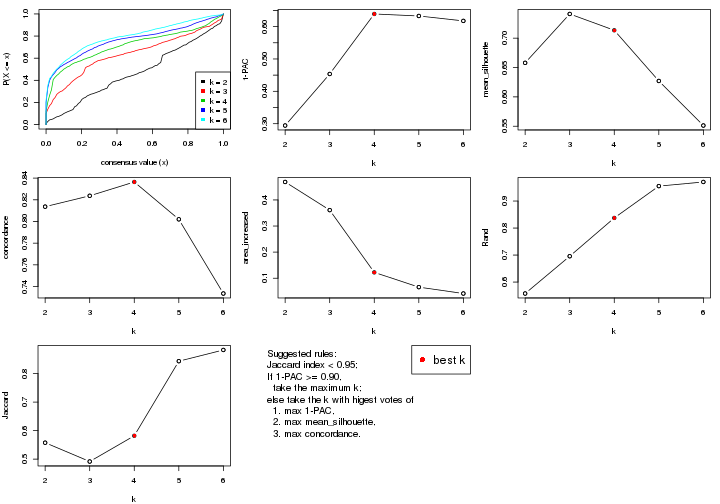
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
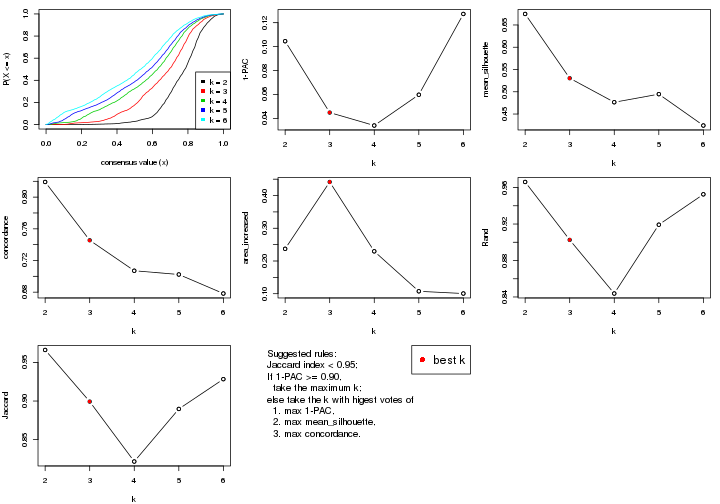
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.1045 0.675 0.819 0.237 0.966 0.966
#> 3 3 0.0449 0.530 0.746 0.442 0.903 0.899
#> 4 4 0.0341 0.476 0.707 0.230 0.844 0.822
#> 5 5 0.0598 0.495 0.702 0.107 0.919 0.890
#> 6 6 0.1273 0.424 0.678 0.101 0.953 0.928
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.529 0.7742 0.880 0.120
#> GSM228563 1 0.886 0.5872 0.696 0.304
#> GSM228565 1 0.506 0.7757 0.888 0.112
#> GSM228566 1 0.469 0.7564 0.900 0.100
#> GSM228567 1 0.584 0.7532 0.860 0.140
#> GSM228570 1 0.506 0.7745 0.888 0.112
#> GSM228571 1 0.518 0.7751 0.884 0.116
#> GSM228574 1 0.482 0.7601 0.896 0.104
#> GSM228575 2 0.993 0.4813 0.452 0.548
#> GSM228576 1 0.506 0.7736 0.888 0.112
#> GSM228579 1 0.541 0.7775 0.876 0.124
#> GSM228580 2 0.987 0.3368 0.432 0.568
#> GSM228581 1 0.844 0.5822 0.728 0.272
#> GSM228666 1 0.775 0.6385 0.772 0.228
#> GSM228564 1 0.850 0.6326 0.724 0.276
#> GSM228568 1 0.482 0.7729 0.896 0.104
#> GSM228569 1 0.456 0.7647 0.904 0.096
#> GSM228572 1 0.958 0.2868 0.620 0.380
#> GSM228573 1 0.469 0.7712 0.900 0.100
#> GSM228577 1 0.541 0.7783 0.876 0.124
#> GSM228578 1 0.494 0.7768 0.892 0.108
#> GSM228663 1 0.494 0.7316 0.892 0.108
#> GSM228664 1 0.595 0.7104 0.856 0.144
#> GSM228665 1 0.358 0.7626 0.932 0.068
#> GSM228582 1 0.494 0.7711 0.892 0.108
#> GSM228583 1 0.541 0.7571 0.876 0.124
#> GSM228585 1 0.615 0.7434 0.848 0.152
#> GSM228587 1 0.738 0.7011 0.792 0.208
#> GSM228588 1 0.891 0.5542 0.692 0.308
#> GSM228589 1 0.886 0.5877 0.696 0.304
#> GSM228590 1 0.653 0.7325 0.832 0.168
#> GSM228591 1 0.850 0.5185 0.724 0.276
#> GSM228597 1 0.917 0.5104 0.668 0.332
#> GSM228601 1 0.891 0.5156 0.692 0.308
#> GSM228604 1 0.697 0.6666 0.812 0.188
#> GSM228608 1 0.662 0.7385 0.828 0.172
#> GSM228609 1 0.876 0.5799 0.704 0.296
#> GSM228613 1 0.615 0.7444 0.848 0.152
#> GSM228616 1 0.563 0.7730 0.868 0.132
#> GSM228628 1 0.814 0.6017 0.748 0.252
#> GSM228634 1 0.584 0.7583 0.860 0.140
#> GSM228642 1 0.808 0.5462 0.752 0.248
#> GSM228645 1 0.563 0.7336 0.868 0.132
#> GSM228646 1 0.541 0.7521 0.876 0.124
#> GSM228652 1 0.634 0.7590 0.840 0.160
#> GSM228655 1 0.653 0.7528 0.832 0.168
#> GSM228656 1 0.615 0.7434 0.848 0.152
#> GSM228659 1 0.753 0.6991 0.784 0.216
#> GSM228662 1 0.615 0.7434 0.848 0.152
#> GSM228584 1 0.584 0.7551 0.860 0.140
#> GSM228586 1 0.552 0.7618 0.872 0.128
#> GSM228592 1 0.595 0.7481 0.856 0.144
#> GSM228593 1 0.850 0.6239 0.724 0.276
#> GSM228594 1 0.574 0.7704 0.864 0.136
#> GSM228598 1 0.605 0.7705 0.852 0.148
#> GSM228607 1 0.358 0.7820 0.932 0.068
#> GSM228612 1 0.388 0.7635 0.924 0.076
#> GSM228619 1 0.494 0.7751 0.892 0.108
#> GSM228622 1 0.494 0.7800 0.892 0.108
#> GSM228625 1 0.671 0.7244 0.824 0.176
#> GSM228631 1 0.552 0.7747 0.872 0.128
#> GSM228633 1 0.929 0.2224 0.656 0.344
#> GSM228637 1 0.985 0.1644 0.572 0.428
#> GSM228639 1 0.506 0.7698 0.888 0.112
#> GSM228649 1 0.895 0.5648 0.688 0.312
#> GSM228660 1 0.541 0.7809 0.876 0.124
#> GSM228661 1 0.518 0.7717 0.884 0.116
#> GSM228595 1 0.943 0.2695 0.640 0.360
#> GSM228599 1 0.738 0.7064 0.792 0.208
#> GSM228602 1 0.456 0.7558 0.904 0.096
#> GSM228614 1 0.714 0.7265 0.804 0.196
#> GSM228626 1 0.929 0.2004 0.656 0.344
#> GSM228640 1 0.373 0.7511 0.928 0.072
#> GSM228643 1 0.482 0.7570 0.896 0.104
#> GSM228650 1 0.402 0.7609 0.920 0.080
#> GSM228653 1 0.388 0.7545 0.924 0.076
#> GSM228657 1 0.936 0.3927 0.648 0.352
#> GSM228605 1 0.416 0.7808 0.916 0.084
#> GSM228610 1 0.482 0.7687 0.896 0.104
#> GSM228617 1 0.482 0.7762 0.896 0.104
#> GSM228620 1 0.343 0.7637 0.936 0.064
#> GSM228623 1 0.844 0.6336 0.728 0.272
#> GSM228629 1 0.430 0.7510 0.912 0.088
#> GSM228632 1 0.388 0.7561 0.924 0.076
#> GSM228635 1 0.992 0.0108 0.552 0.448
#> GSM228647 1 0.311 0.7545 0.944 0.056
#> GSM228596 1 0.416 0.7709 0.916 0.084
#> GSM228600 1 0.373 0.7528 0.928 0.072
#> GSM228603 1 0.373 0.7499 0.928 0.072
#> GSM228615 1 0.802 0.6694 0.756 0.244
#> GSM228627 1 0.358 0.7492 0.932 0.068
#> GSM228641 1 0.388 0.7495 0.924 0.076
#> GSM228644 1 0.932 0.1786 0.652 0.348
#> GSM228651 1 0.327 0.7489 0.940 0.060
#> GSM228654 1 0.358 0.7465 0.932 0.068
#> GSM228658 1 0.343 0.7452 0.936 0.064
#> GSM228606 1 0.311 0.7558 0.944 0.056
#> GSM228611 1 0.388 0.7430 0.924 0.076
#> GSM228618 1 0.373 0.7597 0.928 0.072
#> GSM228621 1 0.311 0.7549 0.944 0.056
#> GSM228624 1 0.506 0.7457 0.888 0.112
#> GSM228630 1 0.416 0.7613 0.916 0.084
#> GSM228636 1 0.983 0.1170 0.576 0.424
#> GSM228638 1 0.541 0.7572 0.876 0.124
#> GSM228648 1 0.388 0.7475 0.924 0.076
#> GSM228670 1 0.795 0.6811 0.760 0.240
#> GSM228671 1 0.850 0.5465 0.724 0.276
#> GSM228672 1 0.861 0.6220 0.716 0.284
#> GSM228674 1 0.781 0.6943 0.768 0.232
#> GSM228675 1 0.827 0.6474 0.740 0.260
#> GSM228676 1 0.738 0.7340 0.792 0.208
#> GSM228667 1 0.821 0.6624 0.744 0.256
#> GSM228668 1 0.506 0.7744 0.888 0.112
#> GSM228669 1 0.482 0.7740 0.896 0.104
#> GSM228673 1 0.430 0.7624 0.912 0.088
#> GSM228677 1 0.680 0.7152 0.820 0.180
#> GSM228678 1 0.909 0.4376 0.676 0.324
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.544 0.68970 0.784 0.192 0.024
#> GSM228563 1 0.746 0.29422 0.560 0.400 0.040
#> GSM228565 1 0.480 0.69602 0.824 0.156 0.020
#> GSM228566 1 0.533 0.66600 0.824 0.100 0.076
#> GSM228567 1 0.502 0.66655 0.796 0.192 0.012
#> GSM228570 1 0.455 0.70473 0.840 0.140 0.020
#> GSM228571 1 0.474 0.70069 0.828 0.152 0.020
#> GSM228574 1 0.498 0.68394 0.828 0.136 0.036
#> GSM228575 3 0.839 0.00000 0.316 0.108 0.576
#> GSM228576 1 0.468 0.69842 0.832 0.148 0.020
#> GSM228579 1 0.481 0.71270 0.828 0.148 0.024
#> GSM228580 2 0.857 -0.28403 0.120 0.564 0.316
#> GSM228581 1 0.909 -0.12446 0.524 0.312 0.164
#> GSM228666 1 0.802 0.36260 0.656 0.188 0.156
#> GSM228564 1 0.714 0.36323 0.576 0.396 0.028
#> GSM228568 1 0.452 0.71161 0.852 0.116 0.032
#> GSM228569 1 0.414 0.70985 0.872 0.096 0.032
#> GSM228572 2 0.889 0.48201 0.428 0.452 0.120
#> GSM228573 1 0.377 0.71587 0.880 0.104 0.016
#> GSM228577 1 0.466 0.71083 0.828 0.156 0.016
#> GSM228578 1 0.439 0.70916 0.840 0.148 0.012
#> GSM228663 1 0.451 0.65743 0.860 0.092 0.048
#> GSM228664 1 0.586 0.57002 0.796 0.120 0.084
#> GSM228665 1 0.346 0.70644 0.900 0.076 0.024
#> GSM228582 1 0.477 0.70906 0.848 0.100 0.052
#> GSM228583 1 0.491 0.67114 0.804 0.184 0.012
#> GSM228585 1 0.527 0.65367 0.776 0.212 0.012
#> GSM228587 1 0.626 0.58207 0.696 0.284 0.020
#> GSM228588 1 0.742 0.16190 0.544 0.420 0.036
#> GSM228589 1 0.834 0.04914 0.536 0.376 0.088
#> GSM228590 1 0.586 0.63331 0.748 0.228 0.024
#> GSM228591 1 0.915 -0.22291 0.528 0.292 0.180
#> GSM228597 1 0.802 0.08887 0.520 0.416 0.064
#> GSM228601 1 0.885 -0.22964 0.516 0.356 0.128
#> GSM228604 1 0.778 0.35312 0.668 0.208 0.124
#> GSM228608 1 0.555 0.64824 0.760 0.224 0.016
#> GSM228609 1 0.720 0.24327 0.556 0.416 0.028
#> GSM228613 1 0.527 0.65387 0.776 0.212 0.012
#> GSM228616 1 0.518 0.69723 0.812 0.156 0.032
#> GSM228628 1 0.876 0.00379 0.576 0.264 0.160
#> GSM228634 1 0.512 0.67267 0.788 0.200 0.012
#> GSM228642 1 0.875 -0.04774 0.572 0.276 0.152
#> GSM228645 1 0.656 0.59405 0.756 0.144 0.100
#> GSM228646 1 0.657 0.60185 0.752 0.160 0.088
#> GSM228652 1 0.527 0.67184 0.784 0.200 0.016
#> GSM228655 1 0.551 0.67254 0.764 0.220 0.016
#> GSM228656 1 0.527 0.65367 0.776 0.212 0.012
#> GSM228659 1 0.645 0.57106 0.684 0.292 0.024
#> GSM228662 1 0.527 0.65367 0.776 0.212 0.012
#> GSM228584 1 0.517 0.66534 0.792 0.192 0.016
#> GSM228586 1 0.497 0.67715 0.800 0.188 0.012
#> GSM228592 1 0.517 0.65857 0.784 0.204 0.012
#> GSM228593 1 0.741 0.33842 0.596 0.360 0.044
#> GSM228594 1 0.505 0.69722 0.812 0.164 0.024
#> GSM228598 1 0.511 0.69721 0.808 0.168 0.024
#> GSM228607 1 0.329 0.71959 0.896 0.096 0.008
#> GSM228612 1 0.355 0.70559 0.896 0.080 0.024
#> GSM228619 1 0.403 0.70802 0.856 0.136 0.008
#> GSM228622 1 0.371 0.71503 0.868 0.128 0.004
#> GSM228625 1 0.586 0.62111 0.740 0.240 0.020
#> GSM228631 1 0.448 0.70853 0.840 0.144 0.016
#> GSM228633 1 0.976 -0.55681 0.388 0.384 0.228
#> GSM228637 2 0.876 0.43456 0.404 0.484 0.112
#> GSM228639 1 0.400 0.70771 0.868 0.116 0.016
#> GSM228649 1 0.782 0.28474 0.564 0.376 0.060
#> GSM228660 1 0.441 0.71503 0.844 0.140 0.016
#> GSM228661 1 0.487 0.69602 0.824 0.152 0.024
#> GSM228595 2 0.958 0.46265 0.396 0.408 0.196
#> GSM228599 1 0.680 0.54372 0.680 0.280 0.040
#> GSM228602 1 0.372 0.69496 0.888 0.088 0.024
#> GSM228614 1 0.673 0.58888 0.696 0.260 0.044
#> GSM228626 1 0.981 -0.54528 0.384 0.376 0.240
#> GSM228640 1 0.336 0.68414 0.900 0.084 0.016
#> GSM228643 1 0.406 0.69812 0.876 0.092 0.032
#> GSM228650 1 0.392 0.69818 0.884 0.080 0.036
#> GSM228653 1 0.305 0.69979 0.916 0.064 0.020
#> GSM228657 1 0.858 -0.42881 0.456 0.448 0.096
#> GSM228605 1 0.385 0.71623 0.860 0.136 0.004
#> GSM228610 1 0.321 0.71395 0.900 0.092 0.008
#> GSM228617 1 0.397 0.70915 0.860 0.132 0.008
#> GSM228620 1 0.333 0.71064 0.904 0.076 0.020
#> GSM228623 1 0.704 0.42530 0.620 0.348 0.032
#> GSM228629 1 0.303 0.69799 0.912 0.076 0.012
#> GSM228632 1 0.318 0.69665 0.908 0.076 0.016
#> GSM228635 2 0.927 0.48158 0.336 0.492 0.172
#> GSM228647 1 0.290 0.69727 0.924 0.048 0.028
#> GSM228596 1 0.367 0.71024 0.888 0.092 0.020
#> GSM228600 1 0.301 0.69415 0.920 0.052 0.028
#> GSM228603 1 0.285 0.69504 0.924 0.056 0.020
#> GSM228615 1 0.715 0.49752 0.652 0.300 0.048
#> GSM228627 1 0.274 0.69260 0.928 0.052 0.020
#> GSM228641 1 0.350 0.68429 0.896 0.084 0.020
#> GSM228644 2 0.979 0.32593 0.376 0.388 0.236
#> GSM228651 1 0.253 0.69250 0.936 0.044 0.020
#> GSM228654 1 0.292 0.69351 0.924 0.044 0.032
#> GSM228658 1 0.234 0.68859 0.940 0.048 0.012
#> GSM228606 1 0.238 0.69503 0.940 0.044 0.016
#> GSM228611 1 0.305 0.68770 0.916 0.064 0.020
#> GSM228618 1 0.328 0.69755 0.908 0.068 0.024
#> GSM228621 1 0.227 0.69609 0.944 0.040 0.016
#> GSM228624 1 0.477 0.64168 0.848 0.100 0.052
#> GSM228630 1 0.329 0.69664 0.900 0.088 0.012
#> GSM228636 2 0.896 0.54262 0.360 0.504 0.136
#> GSM228638 1 0.389 0.70602 0.880 0.096 0.024
#> GSM228648 1 0.353 0.68436 0.900 0.068 0.032
#> GSM228670 1 0.665 0.57932 0.680 0.288 0.032
#> GSM228671 1 0.773 0.35793 0.640 0.276 0.084
#> GSM228672 1 0.699 0.45267 0.612 0.360 0.028
#> GSM228674 1 0.679 0.55659 0.672 0.292 0.036
#> GSM228675 1 0.719 0.47212 0.636 0.320 0.044
#> GSM228676 1 0.619 0.63103 0.724 0.248 0.028
#> GSM228667 1 0.665 0.51180 0.640 0.340 0.020
#> GSM228668 1 0.429 0.69889 0.832 0.164 0.004
#> GSM228669 1 0.400 0.69954 0.840 0.160 0.000
#> GSM228673 1 0.377 0.70751 0.888 0.084 0.028
#> GSM228677 1 0.541 0.60869 0.780 0.200 0.020
#> GSM228678 1 0.829 -0.32317 0.512 0.408 0.080
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 1 0.535 0.6368 0.740 0.040 0.016 0.204
#> GSM228563 4 0.693 0.2181 0.448 0.032 0.044 0.476
#> GSM228565 1 0.451 0.6546 0.780 0.036 0.000 0.184
#> GSM228566 1 0.486 0.6393 0.804 0.100 0.016 0.080
#> GSM228567 1 0.504 0.5955 0.724 0.012 0.016 0.248
#> GSM228570 1 0.438 0.6698 0.800 0.032 0.004 0.164
#> GSM228571 1 0.433 0.6651 0.792 0.032 0.000 0.176
#> GSM228574 1 0.458 0.6655 0.824 0.072 0.020 0.084
#> GSM228575 3 0.623 0.0000 0.204 0.056 0.700 0.040
#> GSM228576 1 0.447 0.6599 0.788 0.040 0.000 0.172
#> GSM228579 1 0.461 0.6814 0.796 0.040 0.008 0.156
#> GSM228580 2 0.763 -0.3134 0.024 0.560 0.164 0.252
#> GSM228581 1 0.854 -0.2114 0.468 0.288 0.052 0.192
#> GSM228666 1 0.769 0.2968 0.620 0.172 0.084 0.124
#> GSM228564 4 0.705 0.1520 0.452 0.020 0.068 0.460
#> GSM228568 1 0.463 0.6823 0.812 0.024 0.036 0.128
#> GSM228569 1 0.423 0.6866 0.840 0.024 0.036 0.100
#> GSM228572 4 0.884 -0.1407 0.248 0.256 0.060 0.436
#> GSM228573 1 0.362 0.6980 0.860 0.020 0.012 0.108
#> GSM228577 1 0.426 0.6818 0.800 0.012 0.012 0.176
#> GSM228578 1 0.395 0.6777 0.804 0.004 0.008 0.184
#> GSM228663 1 0.401 0.6426 0.856 0.076 0.024 0.044
#> GSM228664 1 0.542 0.5296 0.768 0.144 0.028 0.060
#> GSM228665 1 0.302 0.6867 0.900 0.024 0.016 0.060
#> GSM228582 1 0.500 0.6851 0.800 0.072 0.024 0.104
#> GSM228583 1 0.498 0.6056 0.732 0.012 0.016 0.240
#> GSM228585 1 0.507 0.5768 0.708 0.008 0.016 0.268
#> GSM228587 1 0.546 0.4493 0.632 0.004 0.020 0.344
#> GSM228588 4 0.731 0.4097 0.404 0.056 0.044 0.496
#> GSM228589 1 0.824 -0.3880 0.424 0.124 0.052 0.400
#> GSM228590 1 0.532 0.5438 0.684 0.012 0.016 0.288
#> GSM228591 1 0.839 -0.4009 0.396 0.312 0.020 0.272
#> GSM228597 4 0.738 0.3768 0.412 0.044 0.060 0.484
#> GSM228601 2 0.862 -0.0359 0.328 0.332 0.028 0.312
#> GSM228604 1 0.701 0.1697 0.604 0.252 0.012 0.132
#> GSM228608 1 0.503 0.5661 0.700 0.008 0.012 0.280
#> GSM228609 4 0.711 0.3739 0.412 0.036 0.052 0.500
#> GSM228613 1 0.507 0.5771 0.708 0.008 0.016 0.268
#> GSM228616 1 0.534 0.6343 0.756 0.032 0.032 0.180
#> GSM228628 1 0.773 -0.2910 0.444 0.352 0.004 0.200
#> GSM228634 1 0.488 0.6210 0.744 0.016 0.012 0.228
#> GSM228642 1 0.795 -0.3733 0.436 0.356 0.012 0.196
#> GSM228645 1 0.635 0.5214 0.716 0.108 0.040 0.136
#> GSM228646 1 0.649 0.4926 0.692 0.112 0.028 0.168
#> GSM228652 1 0.499 0.5973 0.720 0.012 0.012 0.256
#> GSM228655 1 0.493 0.6008 0.712 0.004 0.016 0.268
#> GSM228656 1 0.507 0.5768 0.708 0.008 0.016 0.268
#> GSM228659 1 0.567 0.4287 0.620 0.004 0.028 0.348
#> GSM228662 1 0.507 0.5768 0.708 0.008 0.016 0.268
#> GSM228584 1 0.493 0.6038 0.736 0.008 0.020 0.236
#> GSM228586 1 0.484 0.6231 0.748 0.016 0.012 0.224
#> GSM228592 1 0.498 0.5887 0.720 0.008 0.016 0.256
#> GSM228593 1 0.697 -0.2020 0.492 0.020 0.064 0.424
#> GSM228594 1 0.486 0.6575 0.776 0.020 0.024 0.180
#> GSM228598 1 0.481 0.6541 0.764 0.004 0.036 0.196
#> GSM228607 1 0.334 0.6949 0.860 0.012 0.004 0.124
#> GSM228612 1 0.390 0.6794 0.860 0.036 0.024 0.080
#> GSM228619 1 0.403 0.6699 0.804 0.012 0.004 0.180
#> GSM228622 1 0.373 0.6869 0.824 0.004 0.008 0.164
#> GSM228625 1 0.580 0.4930 0.656 0.028 0.016 0.300
#> GSM228631 1 0.408 0.6708 0.800 0.012 0.004 0.184
#> GSM228633 2 0.739 0.6119 0.224 0.560 0.008 0.208
#> GSM228637 4 0.734 0.3928 0.264 0.032 0.112 0.592
#> GSM228639 1 0.379 0.6787 0.852 0.032 0.008 0.108
#> GSM228649 1 0.740 -0.2569 0.464 0.032 0.076 0.428
#> GSM228660 1 0.438 0.6793 0.796 0.016 0.012 0.176
#> GSM228661 1 0.464 0.6567 0.784 0.020 0.016 0.180
#> GSM228595 2 0.848 0.5066 0.224 0.444 0.036 0.296
#> GSM228599 1 0.706 0.2701 0.580 0.044 0.056 0.320
#> GSM228602 1 0.340 0.6805 0.880 0.044 0.008 0.068
#> GSM228614 1 0.667 0.4038 0.612 0.056 0.028 0.304
#> GSM228626 2 0.691 0.6039 0.216 0.592 0.000 0.192
#> GSM228640 1 0.308 0.6675 0.896 0.040 0.008 0.056
#> GSM228643 1 0.353 0.6852 0.876 0.044 0.012 0.068
#> GSM228650 1 0.377 0.6773 0.868 0.064 0.020 0.048
#> GSM228653 1 0.273 0.6877 0.916 0.032 0.020 0.032
#> GSM228657 4 0.892 -0.3451 0.288 0.332 0.048 0.332
#> GSM228605 1 0.388 0.6867 0.824 0.016 0.004 0.156
#> GSM228610 1 0.306 0.6952 0.900 0.024 0.020 0.056
#> GSM228617 1 0.399 0.6719 0.808 0.012 0.004 0.176
#> GSM228620 1 0.328 0.6932 0.888 0.028 0.016 0.068
#> GSM228623 1 0.701 0.0200 0.520 0.036 0.048 0.396
#> GSM228629 1 0.259 0.6847 0.916 0.036 0.004 0.044
#> GSM228632 1 0.298 0.6767 0.904 0.032 0.016 0.048
#> GSM228635 4 0.755 0.0660 0.148 0.040 0.212 0.600
#> GSM228647 1 0.264 0.6797 0.920 0.028 0.024 0.028
#> GSM228596 1 0.342 0.6896 0.876 0.020 0.016 0.088
#> GSM228600 1 0.316 0.6850 0.896 0.036 0.016 0.052
#> GSM228603 1 0.278 0.6848 0.912 0.036 0.012 0.040
#> GSM228615 1 0.710 0.1310 0.552 0.028 0.072 0.348
#> GSM228627 1 0.238 0.6784 0.928 0.040 0.016 0.016
#> GSM228641 1 0.333 0.6692 0.884 0.048 0.008 0.060
#> GSM228644 2 0.701 0.6031 0.208 0.580 0.000 0.212
#> GSM228651 1 0.209 0.6767 0.940 0.028 0.020 0.012
#> GSM228654 1 0.253 0.6803 0.924 0.032 0.024 0.020
#> GSM228658 1 0.199 0.6736 0.944 0.020 0.024 0.012
#> GSM228606 1 0.217 0.6774 0.936 0.016 0.012 0.036
#> GSM228611 1 0.284 0.6718 0.912 0.028 0.028 0.032
#> GSM228618 1 0.287 0.6825 0.908 0.036 0.012 0.044
#> GSM228621 1 0.206 0.6796 0.940 0.020 0.008 0.032
#> GSM228624 1 0.468 0.6188 0.820 0.088 0.024 0.068
#> GSM228630 1 0.308 0.6735 0.896 0.040 0.008 0.056
#> GSM228636 4 0.737 0.1447 0.168 0.040 0.168 0.624
#> GSM228638 1 0.370 0.6820 0.864 0.036 0.012 0.088
#> GSM228648 1 0.316 0.6677 0.896 0.052 0.016 0.036
#> GSM228670 1 0.660 0.4258 0.616 0.028 0.052 0.304
#> GSM228671 1 0.801 0.0072 0.548 0.076 0.100 0.276
#> GSM228672 1 0.638 0.2022 0.552 0.024 0.028 0.396
#> GSM228674 1 0.701 0.3832 0.612 0.048 0.060 0.280
#> GSM228675 1 0.745 0.1915 0.560 0.056 0.068 0.316
#> GSM228676 1 0.602 0.5312 0.676 0.036 0.028 0.260
#> GSM228667 1 0.653 0.2921 0.576 0.028 0.036 0.360
#> GSM228668 1 0.425 0.6559 0.776 0.000 0.016 0.208
#> GSM228669 1 0.393 0.6619 0.792 0.000 0.008 0.200
#> GSM228673 1 0.367 0.6871 0.872 0.040 0.020 0.068
#> GSM228677 1 0.544 0.4639 0.724 0.036 0.016 0.224
#> GSM228678 4 0.855 0.2309 0.344 0.140 0.068 0.448
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 3 0.519 0.6392 0.020 0.052 0.732 0.180 0.016
#> GSM228563 4 0.727 0.4385 0.052 0.048 0.396 0.456 0.048
#> GSM228565 3 0.435 0.6571 0.008 0.044 0.772 0.172 0.004
#> GSM228566 3 0.469 0.6466 0.024 0.108 0.784 0.076 0.008
#> GSM228567 3 0.427 0.5874 0.008 0.008 0.696 0.288 0.000
#> GSM228570 3 0.428 0.6699 0.012 0.032 0.784 0.164 0.008
#> GSM228571 3 0.413 0.6676 0.008 0.032 0.784 0.172 0.004
#> GSM228574 3 0.434 0.6585 0.008 0.084 0.808 0.080 0.020
#> GSM228575 1 0.435 0.0000 0.784 0.020 0.156 0.036 0.004
#> GSM228576 3 0.431 0.6631 0.012 0.040 0.780 0.164 0.004
#> GSM228579 3 0.415 0.6817 0.012 0.032 0.776 0.180 0.000
#> GSM228580 5 0.416 0.0000 0.008 0.120 0.020 0.040 0.812
#> GSM228581 3 0.859 -0.2575 0.044 0.260 0.424 0.176 0.096
#> GSM228666 3 0.732 0.2889 0.044 0.168 0.600 0.124 0.064
#> GSM228564 4 0.713 0.3456 0.072 0.052 0.408 0.448 0.020
#> GSM228568 3 0.432 0.6828 0.040 0.012 0.788 0.152 0.008
#> GSM228569 3 0.409 0.6866 0.032 0.016 0.808 0.136 0.008
#> GSM228572 2 0.810 0.2803 0.032 0.432 0.148 0.320 0.068
#> GSM228573 3 0.350 0.7036 0.008 0.020 0.844 0.116 0.012
#> GSM228577 3 0.395 0.6808 0.016 0.012 0.776 0.196 0.000
#> GSM228578 3 0.352 0.6767 0.004 0.004 0.780 0.212 0.000
#> GSM228663 3 0.364 0.6432 0.012 0.084 0.848 0.048 0.008
#> GSM228664 3 0.511 0.5143 0.016 0.132 0.752 0.084 0.016
#> GSM228665 3 0.302 0.6913 0.004 0.028 0.884 0.064 0.020
#> GSM228582 3 0.479 0.6843 0.020 0.064 0.768 0.140 0.008
#> GSM228583 3 0.423 0.5984 0.008 0.008 0.704 0.280 0.000
#> GSM228585 3 0.428 0.5647 0.008 0.004 0.676 0.312 0.000
#> GSM228587 3 0.501 0.4041 0.004 0.012 0.592 0.380 0.012
#> GSM228588 4 0.751 0.5009 0.028 0.132 0.336 0.468 0.036
#> GSM228589 3 0.801 -0.4649 0.012 0.204 0.364 0.356 0.064
#> GSM228590 3 0.471 0.5300 0.012 0.008 0.652 0.324 0.004
#> GSM228591 2 0.743 0.2521 0.024 0.428 0.332 0.204 0.012
#> GSM228597 4 0.765 0.5136 0.028 0.080 0.360 0.452 0.080
#> GSM228601 2 0.732 0.3804 0.008 0.488 0.260 0.212 0.032
#> GSM228604 3 0.648 0.0891 0.024 0.312 0.552 0.108 0.004
#> GSM228608 3 0.453 0.5556 0.008 0.008 0.672 0.308 0.004
#> GSM228609 4 0.750 0.5101 0.028 0.128 0.344 0.464 0.036
#> GSM228613 3 0.428 0.5649 0.008 0.004 0.676 0.312 0.000
#> GSM228616 3 0.527 0.6192 0.024 0.060 0.728 0.176 0.012
#> GSM228628 2 0.652 0.1650 0.012 0.456 0.396 0.136 0.000
#> GSM228634 3 0.422 0.6171 0.008 0.012 0.720 0.260 0.000
#> GSM228642 2 0.623 0.3944 0.024 0.544 0.360 0.064 0.008
#> GSM228645 3 0.627 0.4755 0.052 0.132 0.672 0.132 0.012
#> GSM228646 3 0.629 0.4504 0.028 0.144 0.656 0.156 0.016
#> GSM228652 3 0.438 0.5938 0.008 0.008 0.700 0.280 0.004
#> GSM228655 3 0.447 0.5900 0.004 0.008 0.684 0.296 0.008
#> GSM228656 3 0.428 0.5647 0.008 0.004 0.676 0.312 0.000
#> GSM228659 3 0.520 0.3790 0.004 0.012 0.580 0.384 0.020
#> GSM228662 3 0.428 0.5647 0.008 0.004 0.676 0.312 0.000
#> GSM228584 3 0.424 0.5964 0.008 0.004 0.708 0.276 0.004
#> GSM228586 3 0.419 0.6196 0.008 0.012 0.724 0.256 0.000
#> GSM228592 3 0.420 0.5807 0.008 0.004 0.692 0.296 0.000
#> GSM228593 4 0.716 0.3847 0.036 0.044 0.424 0.436 0.060
#> GSM228594 3 0.437 0.6522 0.020 0.008 0.748 0.216 0.008
#> GSM228598 3 0.452 0.6421 0.028 0.000 0.724 0.236 0.012
#> GSM228607 3 0.316 0.7002 0.004 0.028 0.852 0.116 0.000
#> GSM228612 3 0.364 0.6826 0.016 0.040 0.844 0.096 0.004
#> GSM228619 3 0.384 0.6724 0.004 0.024 0.796 0.172 0.004
#> GSM228622 3 0.312 0.6911 0.000 0.004 0.812 0.184 0.000
#> GSM228625 3 0.576 0.4644 0.012 0.052 0.624 0.296 0.016
#> GSM228631 3 0.391 0.6741 0.004 0.020 0.792 0.176 0.008
#> GSM228633 2 0.407 0.4220 0.008 0.808 0.132 0.044 0.008
#> GSM228637 4 0.759 0.2826 0.068 0.044 0.204 0.564 0.120
#> GSM228639 3 0.391 0.6715 0.020 0.032 0.836 0.096 0.016
#> GSM228649 4 0.732 0.4320 0.048 0.048 0.392 0.456 0.056
#> GSM228660 3 0.429 0.6775 0.012 0.024 0.764 0.196 0.004
#> GSM228661 3 0.424 0.6527 0.012 0.016 0.756 0.212 0.004
#> GSM228595 2 0.643 0.4514 0.020 0.668 0.136 0.120 0.056
#> GSM228599 3 0.717 0.1346 0.036 0.092 0.536 0.300 0.036
#> GSM228602 3 0.320 0.6886 0.004 0.044 0.872 0.068 0.012
#> GSM228614 3 0.675 0.3006 0.032 0.096 0.560 0.296 0.016
#> GSM228626 2 0.326 0.4007 0.004 0.844 0.124 0.028 0.000
#> GSM228640 3 0.296 0.6715 0.008 0.052 0.884 0.052 0.004
#> GSM228643 3 0.321 0.6910 0.012 0.048 0.872 0.064 0.004
#> GSM228650 3 0.381 0.6815 0.016 0.068 0.844 0.060 0.012
#> GSM228653 3 0.239 0.6929 0.016 0.024 0.912 0.048 0.000
#> GSM228657 2 0.779 0.4858 0.052 0.516 0.220 0.176 0.036
#> GSM228605 3 0.359 0.6919 0.004 0.020 0.816 0.156 0.004
#> GSM228610 3 0.302 0.6980 0.008 0.032 0.884 0.064 0.012
#> GSM228617 3 0.380 0.6741 0.004 0.024 0.800 0.168 0.004
#> GSM228620 3 0.289 0.7006 0.004 0.028 0.884 0.076 0.008
#> GSM228623 3 0.710 -0.2108 0.036 0.072 0.464 0.396 0.032
#> GSM228629 3 0.228 0.6907 0.000 0.036 0.916 0.040 0.008
#> GSM228632 3 0.296 0.6815 0.004 0.040 0.888 0.052 0.016
#> GSM228635 4 0.802 -0.2474 0.132 0.064 0.072 0.532 0.200
#> GSM228647 3 0.239 0.6861 0.004 0.028 0.916 0.040 0.012
#> GSM228596 3 0.335 0.6937 0.004 0.024 0.860 0.092 0.020
#> GSM228600 3 0.292 0.6903 0.012 0.024 0.888 0.068 0.008
#> GSM228603 3 0.261 0.6900 0.012 0.028 0.904 0.052 0.004
#> GSM228615 3 0.760 -0.0983 0.040 0.052 0.496 0.312 0.100
#> GSM228627 3 0.228 0.6846 0.008 0.036 0.920 0.032 0.004
#> GSM228641 3 0.317 0.6727 0.008 0.060 0.872 0.056 0.004
#> GSM228644 2 0.359 0.4086 0.004 0.828 0.120 0.048 0.000
#> GSM228651 3 0.225 0.6822 0.012 0.020 0.924 0.036 0.008
#> GSM228654 3 0.258 0.6851 0.020 0.028 0.912 0.028 0.012
#> GSM228658 3 0.216 0.6787 0.012 0.012 0.928 0.036 0.012
#> GSM228606 3 0.202 0.6818 0.004 0.016 0.928 0.048 0.004
#> GSM228611 3 0.266 0.6745 0.012 0.024 0.900 0.060 0.004
#> GSM228618 3 0.247 0.6878 0.004 0.036 0.908 0.048 0.004
#> GSM228621 3 0.196 0.6867 0.004 0.020 0.928 0.048 0.000
#> GSM228624 3 0.443 0.6070 0.012 0.092 0.800 0.084 0.012
#> GSM228630 3 0.307 0.6752 0.000 0.040 0.876 0.068 0.016
#> GSM228636 4 0.815 -0.0888 0.100 0.088 0.096 0.536 0.180
#> GSM228638 3 0.363 0.6794 0.016 0.036 0.848 0.092 0.008
#> GSM228648 3 0.292 0.6707 0.000 0.052 0.884 0.052 0.012
#> GSM228670 3 0.677 0.3070 0.052 0.040 0.560 0.312 0.036
#> GSM228671 3 0.876 -0.2230 0.136 0.108 0.460 0.208 0.088
#> GSM228672 3 0.639 0.1240 0.040 0.028 0.508 0.400 0.024
#> GSM228674 3 0.712 0.2164 0.076 0.044 0.544 0.300 0.036
#> GSM228675 3 0.800 -0.0474 0.100 0.064 0.492 0.280 0.064
#> GSM228676 3 0.626 0.4130 0.036 0.044 0.608 0.288 0.024
#> GSM228667 3 0.636 0.2160 0.044 0.040 0.532 0.372 0.012
#> GSM228668 3 0.396 0.6490 0.012 0.004 0.744 0.240 0.000
#> GSM228669 3 0.368 0.6570 0.000 0.004 0.760 0.232 0.004
#> GSM228673 3 0.366 0.6880 0.016 0.032 0.844 0.100 0.008
#> GSM228677 3 0.557 0.3749 0.008 0.076 0.680 0.220 0.016
#> GSM228678 4 0.848 0.0274 0.044 0.220 0.288 0.388 0.060
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 3 0.509 0.5225 0.032 0.040 0.704 0.200 0.008 0.016
#> GSM228563 4 0.713 0.6977 0.032 0.040 0.328 0.472 0.020 0.108
#> GSM228565 3 0.426 0.5590 0.012 0.040 0.756 0.180 0.004 0.008
#> GSM228566 3 0.472 0.5445 0.028 0.096 0.752 0.112 0.008 0.004
#> GSM228567 3 0.405 0.4509 0.012 0.000 0.644 0.340 0.000 0.004
#> GSM228570 3 0.417 0.5700 0.016 0.028 0.768 0.172 0.012 0.004
#> GSM228571 3 0.410 0.5660 0.016 0.028 0.768 0.176 0.008 0.004
#> GSM228574 3 0.442 0.5477 0.004 0.060 0.764 0.144 0.008 0.020
#> GSM228575 1 0.289 0.0000 0.864 0.004 0.096 0.028 0.004 0.004
#> GSM228576 3 0.416 0.5652 0.012 0.036 0.772 0.164 0.008 0.008
#> GSM228579 3 0.403 0.5917 0.016 0.028 0.764 0.184 0.008 0.000
#> GSM228580 5 0.250 0.0000 0.000 0.036 0.016 0.008 0.900 0.040
#> GSM228581 3 0.897 -0.3713 0.044 0.192 0.336 0.236 0.076 0.116
#> GSM228666 3 0.717 0.0727 0.040 0.124 0.568 0.164 0.088 0.016
#> GSM228564 4 0.706 0.6456 0.068 0.040 0.336 0.480 0.016 0.060
#> GSM228568 3 0.434 0.5833 0.024 0.008 0.752 0.176 0.040 0.000
#> GSM228569 3 0.390 0.6081 0.016 0.008 0.788 0.152 0.036 0.000
#> GSM228572 2 0.782 0.0497 0.020 0.452 0.084 0.236 0.032 0.176
#> GSM228573 3 0.341 0.6346 0.016 0.004 0.836 0.112 0.024 0.008
#> GSM228577 3 0.394 0.5872 0.012 0.008 0.756 0.204 0.020 0.000
#> GSM228578 3 0.381 0.5857 0.008 0.004 0.764 0.204 0.012 0.008
#> GSM228663 3 0.366 0.5764 0.016 0.064 0.836 0.064 0.016 0.004
#> GSM228664 3 0.527 0.3802 0.020 0.104 0.716 0.128 0.024 0.008
#> GSM228665 3 0.297 0.6250 0.012 0.016 0.880 0.060 0.020 0.012
#> GSM228582 3 0.449 0.5907 0.012 0.048 0.748 0.168 0.024 0.000
#> GSM228583 3 0.432 0.4660 0.012 0.000 0.652 0.320 0.008 0.008
#> GSM228585 3 0.403 0.4190 0.008 0.000 0.624 0.364 0.000 0.004
#> GSM228587 3 0.462 0.1488 0.004 0.004 0.536 0.436 0.004 0.016
#> GSM228588 4 0.680 0.6113 0.016 0.128 0.244 0.536 0.004 0.072
#> GSM228589 4 0.764 0.4399 0.008 0.204 0.288 0.408 0.036 0.056
#> GSM228590 3 0.443 0.3583 0.016 0.000 0.600 0.372 0.000 0.012
#> GSM228591 2 0.721 0.2836 0.012 0.436 0.268 0.232 0.036 0.016
#> GSM228597 4 0.717 0.6510 0.012 0.072 0.292 0.472 0.012 0.140
#> GSM228601 2 0.659 0.3649 0.004 0.524 0.192 0.236 0.012 0.032
#> GSM228604 3 0.678 -0.1788 0.024 0.316 0.492 0.132 0.016 0.020
#> GSM228608 3 0.429 0.3962 0.012 0.000 0.624 0.352 0.000 0.012
#> GSM228609 4 0.690 0.6546 0.020 0.120 0.264 0.520 0.004 0.072
#> GSM228613 3 0.413 0.4140 0.008 0.000 0.624 0.360 0.000 0.008
#> GSM228616 3 0.514 0.4892 0.012 0.044 0.692 0.216 0.016 0.020
#> GSM228628 2 0.620 0.0974 0.004 0.492 0.332 0.152 0.016 0.004
#> GSM228634 3 0.388 0.4989 0.008 0.000 0.668 0.320 0.000 0.004
#> GSM228642 2 0.628 0.3696 0.032 0.544 0.312 0.088 0.012 0.012
#> GSM228645 3 0.622 0.2419 0.044 0.120 0.616 0.196 0.016 0.008
#> GSM228646 3 0.629 0.1861 0.024 0.136 0.588 0.224 0.016 0.012
#> GSM228652 3 0.397 0.4670 0.008 0.000 0.668 0.316 0.000 0.008
#> GSM228655 3 0.428 0.4597 0.008 0.000 0.656 0.316 0.016 0.004
#> GSM228656 3 0.403 0.4190 0.008 0.000 0.624 0.364 0.000 0.004
#> GSM228659 3 0.489 0.0987 0.004 0.004 0.528 0.432 0.012 0.020
#> GSM228662 3 0.405 0.4117 0.008 0.000 0.620 0.368 0.000 0.004
#> GSM228584 3 0.414 0.4621 0.008 0.000 0.656 0.324 0.008 0.004
#> GSM228586 3 0.418 0.4993 0.008 0.000 0.668 0.308 0.008 0.008
#> GSM228592 3 0.407 0.4376 0.008 0.000 0.640 0.344 0.000 0.008
#> GSM228593 4 0.741 0.6523 0.024 0.040 0.364 0.404 0.028 0.140
#> GSM228594 3 0.393 0.5488 0.012 0.000 0.708 0.268 0.012 0.000
#> GSM228598 3 0.455 0.5190 0.020 0.000 0.680 0.268 0.028 0.004
#> GSM228607 3 0.341 0.6246 0.008 0.016 0.836 0.116 0.012 0.012
#> GSM228612 3 0.368 0.6028 0.016 0.036 0.820 0.116 0.008 0.004
#> GSM228619 3 0.372 0.5774 0.000 0.004 0.768 0.200 0.012 0.016
#> GSM228622 3 0.334 0.6110 0.012 0.000 0.808 0.164 0.008 0.008
#> GSM228625 3 0.582 0.2279 0.012 0.032 0.580 0.316 0.012 0.048
#> GSM228631 3 0.365 0.5794 0.004 0.004 0.764 0.212 0.004 0.012
#> GSM228633 2 0.259 0.4075 0.000 0.888 0.072 0.020 0.008 0.012
#> GSM228637 6 0.669 0.3324 0.020 0.012 0.152 0.356 0.012 0.448
#> GSM228639 3 0.395 0.5913 0.024 0.020 0.820 0.096 0.012 0.028
#> GSM228649 4 0.736 0.6512 0.028 0.036 0.328 0.436 0.024 0.148
#> GSM228660 3 0.425 0.5774 0.008 0.016 0.740 0.212 0.008 0.016
#> GSM228661 3 0.406 0.5509 0.008 0.004 0.712 0.260 0.012 0.004
#> GSM228595 2 0.567 0.4352 0.004 0.700 0.084 0.116 0.032 0.064
#> GSM228599 3 0.704 -0.3315 0.024 0.076 0.472 0.344 0.020 0.064
#> GSM228602 3 0.303 0.6233 0.012 0.024 0.872 0.072 0.008 0.012
#> GSM228614 3 0.699 -0.1696 0.020 0.072 0.496 0.324 0.032 0.056
#> GSM228626 2 0.203 0.3989 0.004 0.912 0.068 0.012 0.004 0.000
#> GSM228640 3 0.292 0.6075 0.016 0.040 0.876 0.060 0.004 0.004
#> GSM228643 3 0.331 0.6240 0.016 0.032 0.860 0.068 0.016 0.008
#> GSM228650 3 0.402 0.5972 0.028 0.052 0.816 0.084 0.008 0.012
#> GSM228653 3 0.249 0.6291 0.012 0.016 0.896 0.064 0.012 0.000
#> GSM228657 2 0.742 0.3544 0.036 0.532 0.168 0.168 0.020 0.076
#> GSM228605 3 0.370 0.6090 0.012 0.012 0.808 0.144 0.008 0.016
#> GSM228610 3 0.296 0.6304 0.016 0.012 0.876 0.068 0.004 0.024
#> GSM228617 3 0.369 0.5802 0.000 0.004 0.772 0.196 0.012 0.016
#> GSM228620 3 0.286 0.6346 0.020 0.020 0.876 0.076 0.004 0.004
#> GSM228623 4 0.717 0.5730 0.024 0.056 0.404 0.408 0.028 0.080
#> GSM228629 3 0.249 0.6299 0.016 0.016 0.904 0.048 0.008 0.008
#> GSM228632 3 0.304 0.6148 0.012 0.028 0.876 0.060 0.012 0.012
#> GSM228635 6 0.419 0.2797 0.012 0.004 0.024 0.184 0.016 0.760
#> GSM228647 3 0.243 0.6210 0.016 0.016 0.908 0.044 0.008 0.008
#> GSM228596 3 0.330 0.6247 0.016 0.012 0.856 0.084 0.020 0.012
#> GSM228600 3 0.287 0.6252 0.016 0.008 0.876 0.080 0.012 0.008
#> GSM228603 3 0.258 0.6268 0.016 0.012 0.896 0.060 0.012 0.004
#> GSM228615 3 0.722 -0.4688 0.012 0.032 0.432 0.288 0.020 0.216
#> GSM228627 3 0.235 0.6223 0.012 0.016 0.912 0.040 0.016 0.004
#> GSM228641 3 0.312 0.6076 0.016 0.048 0.864 0.064 0.004 0.004
#> GSM228644 2 0.255 0.4169 0.004 0.892 0.068 0.024 0.004 0.008
#> GSM228651 3 0.232 0.6190 0.012 0.012 0.912 0.044 0.016 0.004
#> GSM228654 3 0.288 0.6175 0.012 0.016 0.888 0.044 0.028 0.012
#> GSM228658 3 0.217 0.6154 0.012 0.004 0.916 0.048 0.016 0.004
#> GSM228606 3 0.259 0.6089 0.008 0.016 0.892 0.068 0.012 0.004
#> GSM228611 3 0.261 0.6122 0.016 0.012 0.896 0.056 0.016 0.004
#> GSM228618 3 0.252 0.6217 0.012 0.016 0.900 0.056 0.012 0.004
#> GSM228621 3 0.258 0.6168 0.012 0.016 0.896 0.060 0.012 0.004
#> GSM228624 3 0.457 0.4963 0.012 0.072 0.764 0.124 0.024 0.004
#> GSM228630 3 0.338 0.5983 0.016 0.024 0.856 0.072 0.008 0.024
#> GSM228636 6 0.474 0.4112 0.000 0.036 0.032 0.220 0.008 0.704
#> GSM228638 3 0.333 0.6067 0.020 0.016 0.856 0.080 0.008 0.020
#> GSM228648 3 0.300 0.5997 0.020 0.032 0.872 0.064 0.000 0.012
#> GSM228670 3 0.677 -0.0859 0.048 0.032 0.524 0.316 0.028 0.052
#> GSM228671 3 0.884 -0.3718 0.108 0.080 0.364 0.248 0.044 0.156
#> GSM228672 4 0.619 0.3164 0.024 0.016 0.436 0.452 0.020 0.052
#> GSM228674 3 0.718 -0.1727 0.056 0.040 0.496 0.304 0.020 0.084
#> GSM228675 3 0.789 -0.3688 0.072 0.056 0.436 0.300 0.028 0.108
#> GSM228676 3 0.652 0.0985 0.032 0.036 0.564 0.284 0.024 0.060
#> GSM228667 3 0.631 -0.3185 0.024 0.028 0.456 0.428 0.028 0.036
#> GSM228668 3 0.430 0.5403 0.008 0.004 0.716 0.240 0.008 0.024
#> GSM228669 3 0.412 0.5521 0.008 0.004 0.728 0.236 0.008 0.016
#> GSM228673 3 0.363 0.6165 0.004 0.024 0.820 0.124 0.020 0.008
#> GSM228677 3 0.582 0.2141 0.008 0.044 0.656 0.160 0.008 0.124
#> GSM228678 6 0.809 0.1818 0.008 0.160 0.244 0.208 0.020 0.360
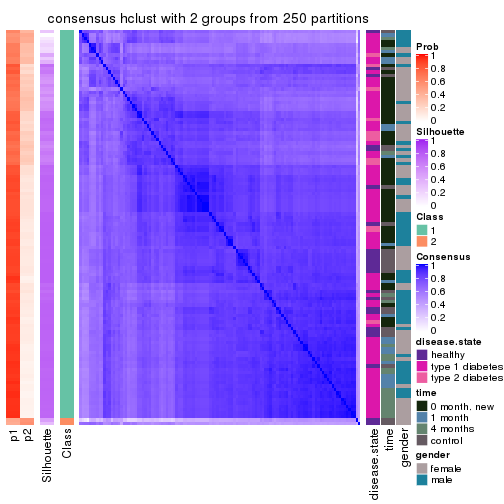
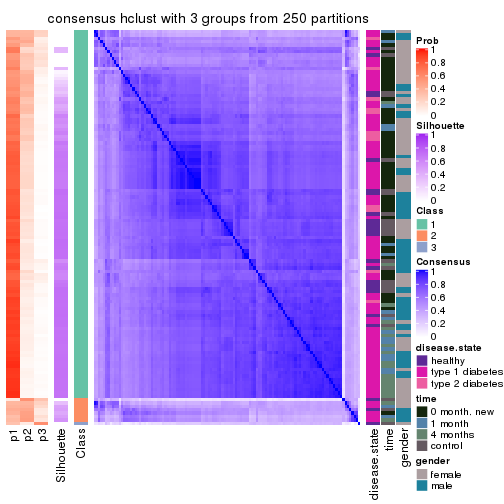
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
consensus_heatmap(res, k = 3)
consensus_heatmap(res, k = 4)
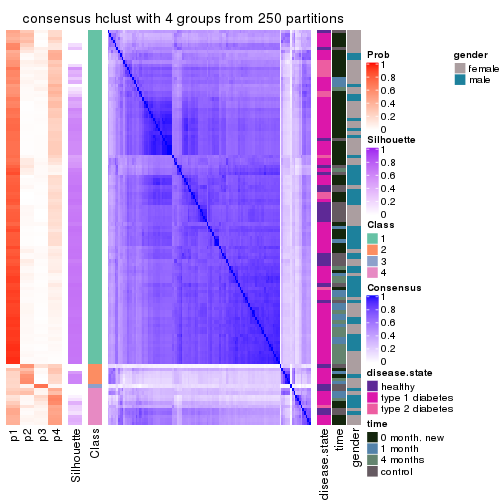
consensus_heatmap(res, k = 5)
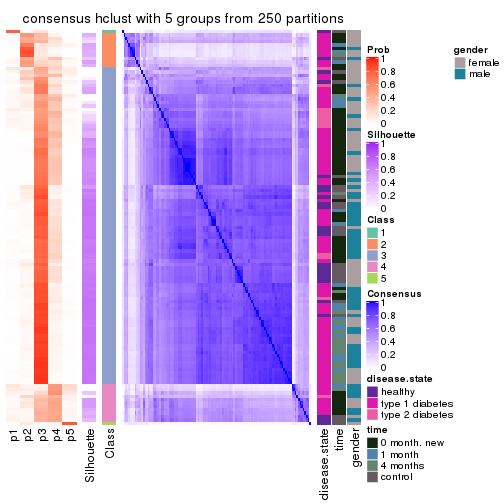
consensus_heatmap(res, k = 6)
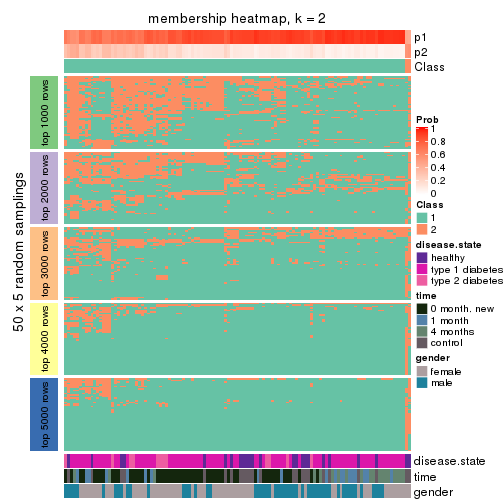
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
membership_heatmap(res, k = 3)
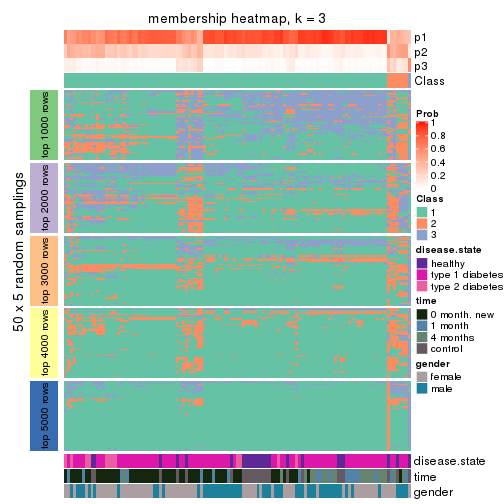
membership_heatmap(res, k = 4)
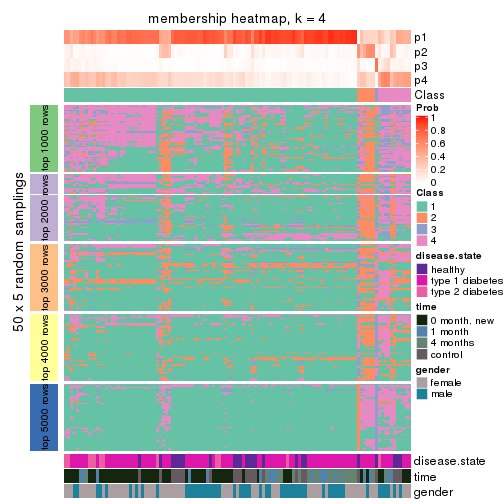
membership_heatmap(res, k = 5)
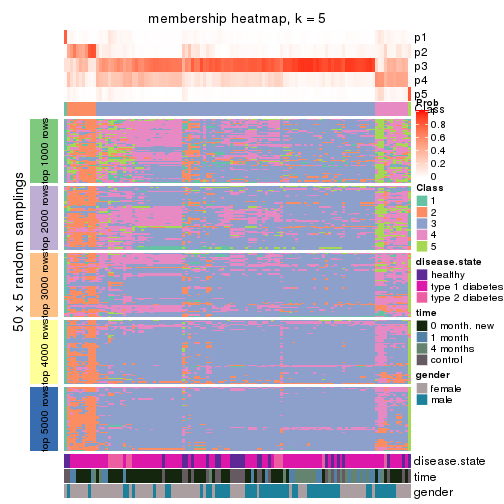
membership_heatmap(res, k = 6)
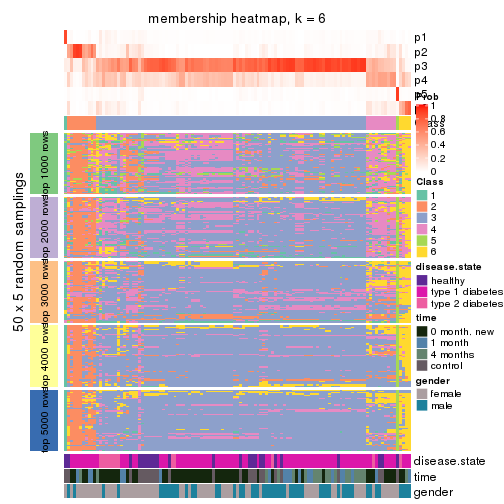
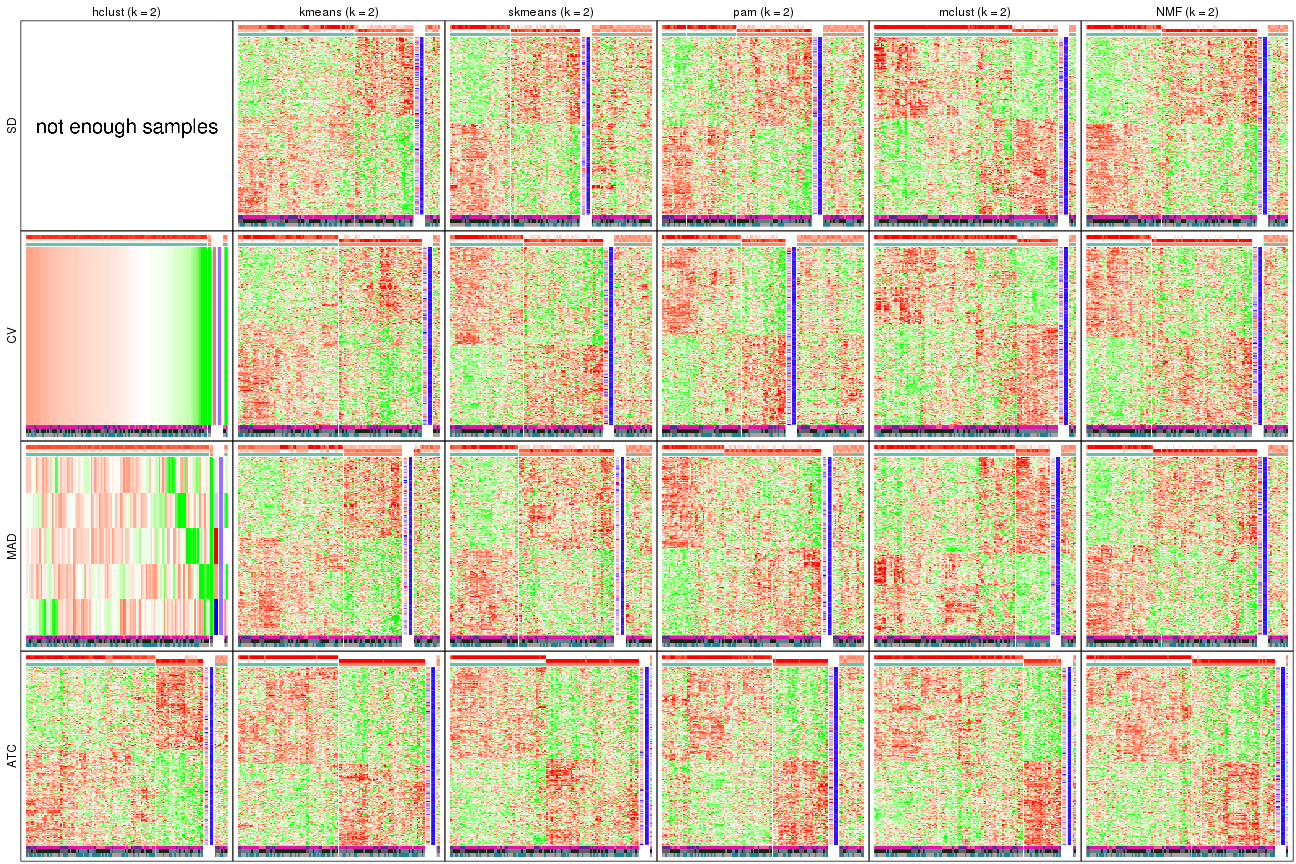
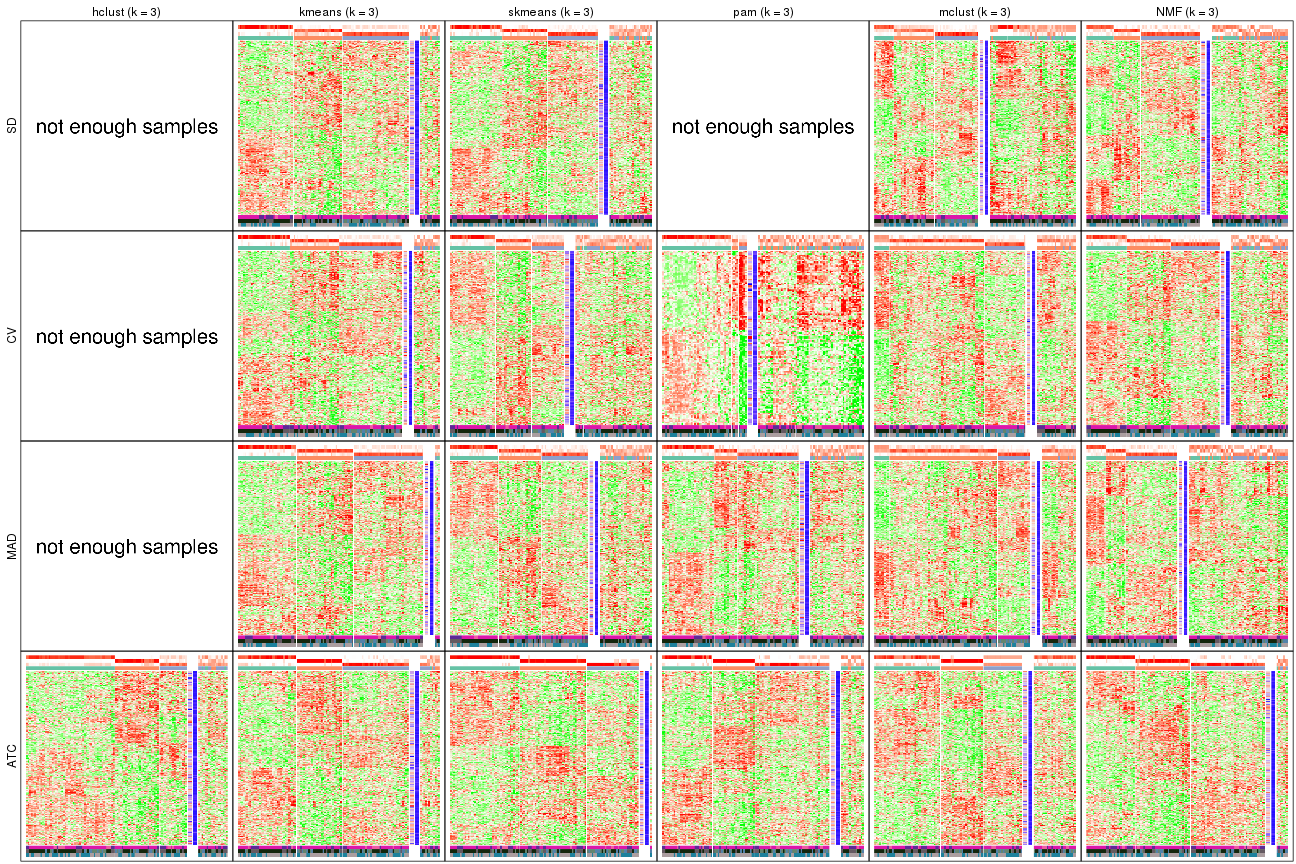
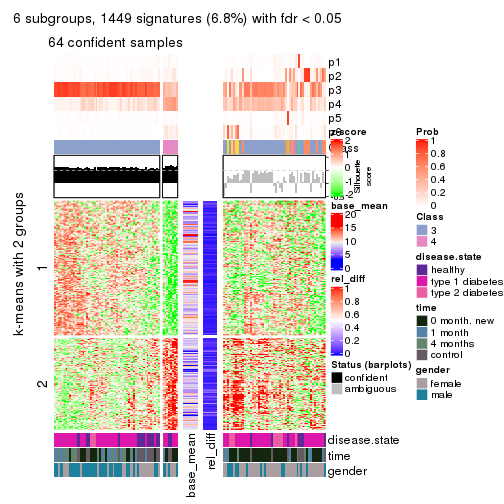
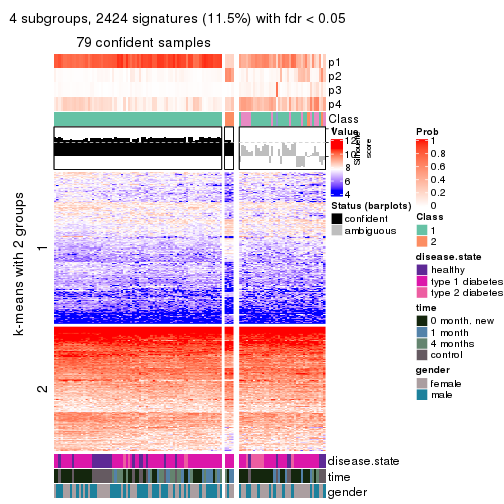
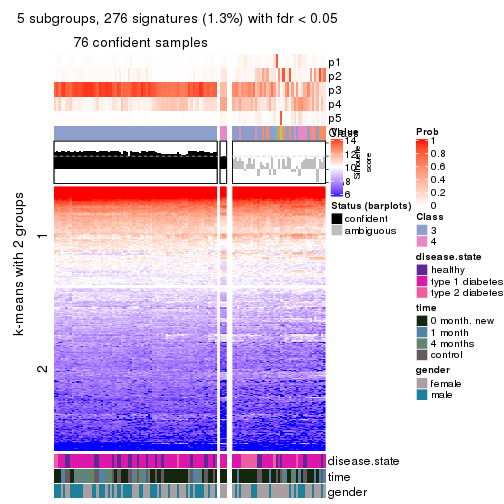
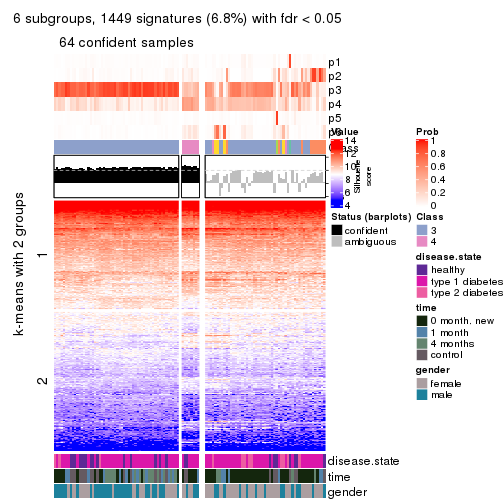
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)

get_signatures(res, k = 3)

get_signatures(res, k = 4)
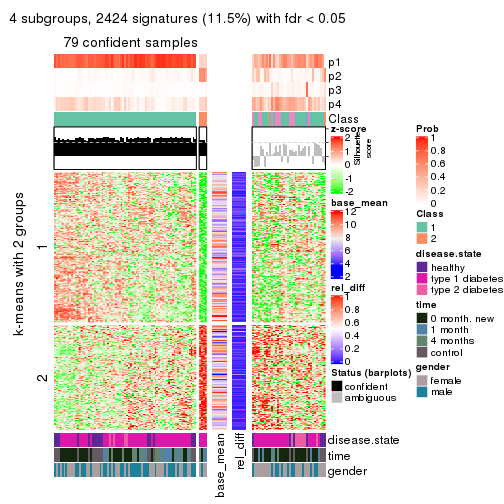
get_signatures(res, k = 5)
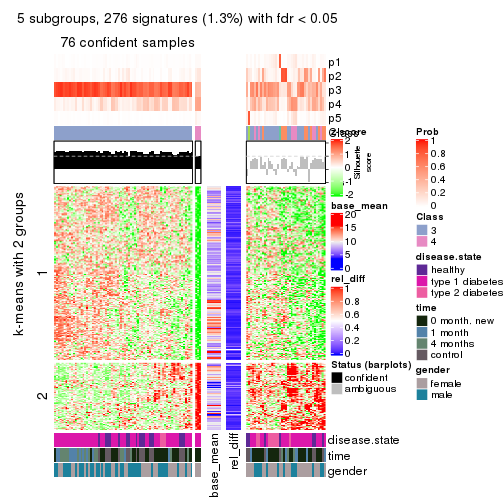
get_signatures(res, k = 6)
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)

get_signatures(res, k = 3, scale_rows = FALSE)

get_signatures(res, k = 4, scale_rows = FALSE)
get_signatures(res, k = 5, scale_rows = FALSE)
get_signatures(res, k = 6, scale_rows = FALSE)
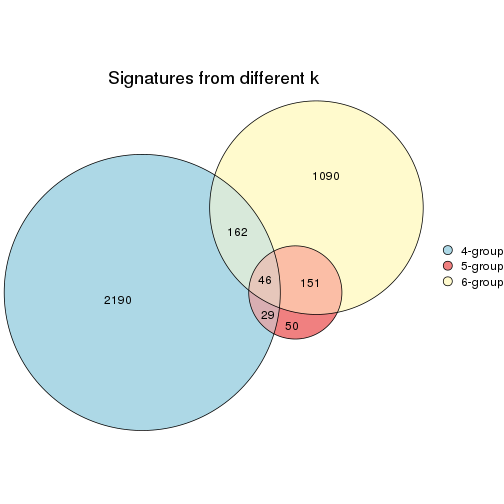
Compare the overlap of signatures from different k:
compare_signatures(res)
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
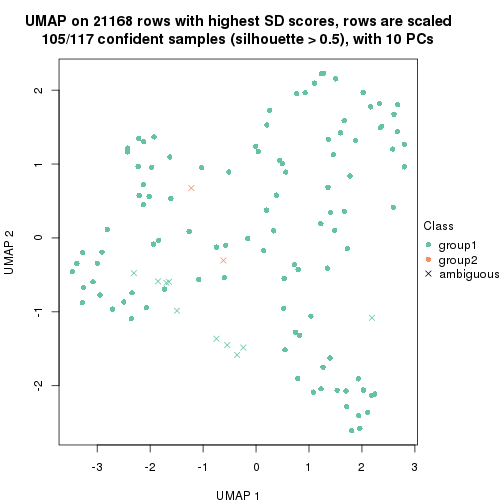
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
dimension_reduction(res, k = 3, method = "UMAP")
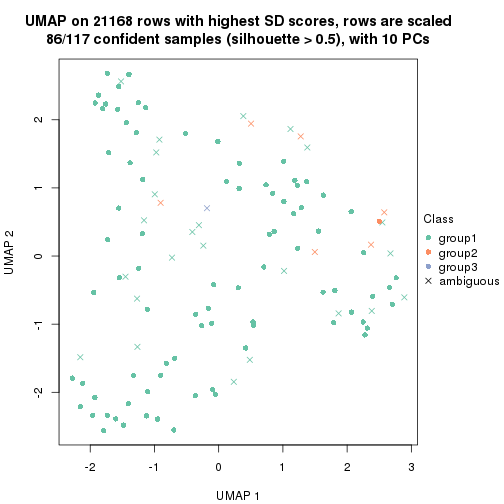
dimension_reduction(res, k = 4, method = "UMAP")
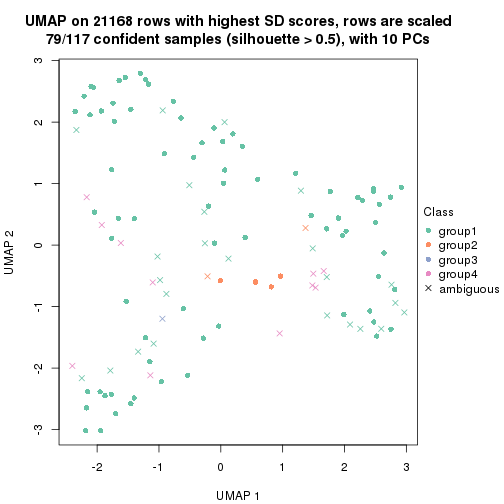
dimension_reduction(res, k = 5, method = "UMAP")
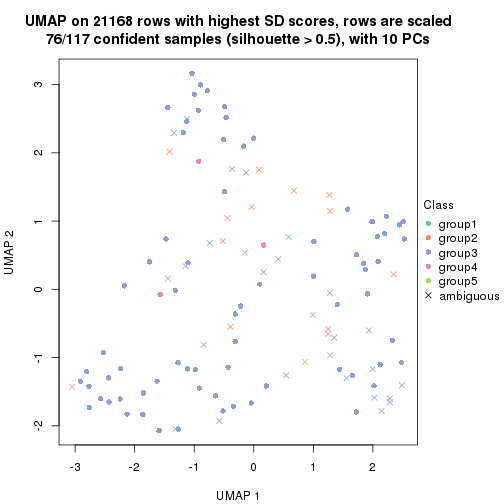
dimension_reduction(res, k = 6, method = "UMAP")
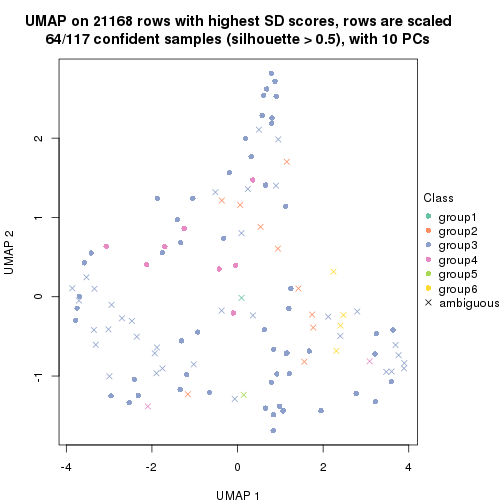
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> SD:hclust 105 NA NA NA 2
#> SD:hclust 86 0.813 0.250 1.000 3
#> SD:hclust 79 0.466 0.305 0.590 4
#> SD:hclust 76 0.573 0.209 0.221 5
#> SD:hclust 64 0.783 0.125 0.847 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["SD", "kmeans"]
# you can also extract it by
# res = res_list["SD:kmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'SD' method.
#> Subgroups are detected by 'kmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 4.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.294 0.658 0.814 0.4689 0.558 0.558
#> 3 3 0.454 0.741 0.824 0.3610 0.696 0.492
#> 4 4 0.639 0.713 0.836 0.1223 0.837 0.582
#> 5 5 0.633 0.627 0.802 0.0659 0.955 0.843
#> 6 6 0.618 0.551 0.734 0.0413 0.971 0.883
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 4
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.9358 0.7044 0.648 0.352
#> GSM228563 2 0.5519 0.7702 0.128 0.872
#> GSM228565 1 0.7883 0.7280 0.764 0.236
#> GSM228566 1 0.1184 0.6920 0.984 0.016
#> GSM228567 1 0.9323 0.7066 0.652 0.348
#> GSM228570 1 0.9323 0.7066 0.652 0.348
#> GSM228571 1 0.8267 0.7245 0.740 0.260
#> GSM228574 1 0.6438 0.4723 0.836 0.164
#> GSM228575 1 0.5519 0.5463 0.872 0.128
#> GSM228576 1 0.8016 0.7263 0.756 0.244
#> GSM228579 1 0.8327 0.7243 0.736 0.264
#> GSM228580 2 0.8909 0.7951 0.308 0.692
#> GSM228581 2 0.9896 0.6836 0.440 0.560
#> GSM228666 2 0.9358 0.7774 0.352 0.648
#> GSM228564 2 0.8955 -0.0955 0.312 0.688
#> GSM228568 1 0.7376 0.7239 0.792 0.208
#> GSM228569 1 0.7745 0.7244 0.772 0.228
#> GSM228572 2 0.8813 0.7968 0.300 0.700
#> GSM228573 1 0.0000 0.7047 1.000 0.000
#> GSM228577 1 0.9286 0.7083 0.656 0.344
#> GSM228578 1 0.8661 0.7214 0.712 0.288
#> GSM228663 1 0.0000 0.7047 1.000 0.000
#> GSM228664 1 0.9608 -0.2920 0.616 0.384
#> GSM228665 1 0.0000 0.7047 1.000 0.000
#> GSM228582 1 0.5059 0.7167 0.888 0.112
#> GSM228583 1 0.9358 0.7046 0.648 0.352
#> GSM228585 1 0.9358 0.7046 0.648 0.352
#> GSM228587 1 0.9635 0.6768 0.612 0.388
#> GSM228588 2 0.0376 0.6229 0.004 0.996
#> GSM228589 2 0.6247 0.7941 0.156 0.844
#> GSM228590 1 0.9358 0.7046 0.648 0.352
#> GSM228591 2 0.9393 0.7757 0.356 0.644
#> GSM228597 2 0.6247 0.7941 0.156 0.844
#> GSM228601 2 0.6712 0.8011 0.176 0.824
#> GSM228604 2 0.9460 0.7709 0.364 0.636
#> GSM228608 1 0.9358 0.7046 0.648 0.352
#> GSM228609 2 0.0376 0.6147 0.004 0.996
#> GSM228613 1 0.9358 0.7046 0.648 0.352
#> GSM228616 1 0.9087 0.7104 0.676 0.324
#> GSM228628 2 0.9460 0.7720 0.364 0.636
#> GSM228634 1 0.9323 0.7066 0.652 0.348
#> GSM228642 2 0.9358 0.7774 0.352 0.648
#> GSM228645 1 0.4815 0.6054 0.896 0.104
#> GSM228646 1 0.7299 0.3906 0.796 0.204
#> GSM228652 1 0.9358 0.7046 0.648 0.352
#> GSM228655 1 0.9358 0.7046 0.648 0.352
#> GSM228656 1 0.9358 0.7046 0.648 0.352
#> GSM228659 1 1.0000 0.5427 0.504 0.496
#> GSM228662 1 0.9393 0.7023 0.644 0.356
#> GSM228584 1 0.9358 0.7046 0.648 0.352
#> GSM228586 1 0.9323 0.7066 0.652 0.348
#> GSM228592 1 0.9358 0.7046 0.648 0.352
#> GSM228593 2 0.2603 0.5849 0.044 0.956
#> GSM228594 1 0.9044 0.7152 0.680 0.320
#> GSM228598 1 0.9358 0.7046 0.648 0.352
#> GSM228607 1 0.3114 0.7076 0.944 0.056
#> GSM228612 1 0.0938 0.6957 0.988 0.012
#> GSM228619 1 0.9129 0.7085 0.672 0.328
#> GSM228622 1 0.9323 0.7066 0.652 0.348
#> GSM228625 1 0.9998 0.5419 0.508 0.492
#> GSM228631 1 0.9209 0.7086 0.664 0.336
#> GSM228633 2 0.9358 0.7774 0.352 0.648
#> GSM228637 2 0.6148 0.7916 0.152 0.848
#> GSM228639 2 0.9358 0.7372 0.352 0.648
#> GSM228649 2 0.5294 0.7621 0.120 0.880
#> GSM228660 1 0.9286 0.7086 0.656 0.344
#> GSM228661 1 0.8267 0.7248 0.740 0.260
#> GSM228595 2 0.9323 0.7793 0.348 0.652
#> GSM228599 2 0.6623 0.8002 0.172 0.828
#> GSM228602 1 0.0000 0.7047 1.000 0.000
#> GSM228614 2 0.7528 0.8040 0.216 0.784
#> GSM228626 2 0.9358 0.7774 0.352 0.648
#> GSM228640 1 0.0000 0.7047 1.000 0.000
#> GSM228643 1 0.0938 0.6976 0.988 0.012
#> GSM228650 1 0.4022 0.6268 0.920 0.080
#> GSM228653 1 0.0000 0.7047 1.000 0.000
#> GSM228657 2 0.9129 0.7883 0.328 0.672
#> GSM228605 1 0.8813 0.7185 0.700 0.300
#> GSM228610 1 0.0000 0.7047 1.000 0.000
#> GSM228617 1 0.0000 0.7047 1.000 0.000
#> GSM228620 1 0.0000 0.7047 1.000 0.000
#> GSM228623 2 0.6712 0.8014 0.176 0.824
#> GSM228629 1 0.0000 0.7047 1.000 0.000
#> GSM228632 1 0.9732 -0.3537 0.596 0.404
#> GSM228635 2 0.6343 0.7961 0.160 0.840
#> GSM228647 1 0.0000 0.7047 1.000 0.000
#> GSM228596 1 0.0376 0.7057 0.996 0.004
#> GSM228600 1 0.0000 0.7047 1.000 0.000
#> GSM228603 1 0.0000 0.7047 1.000 0.000
#> GSM228615 2 0.6148 0.7916 0.152 0.848
#> GSM228627 1 0.0000 0.7047 1.000 0.000
#> GSM228641 1 0.0000 0.7047 1.000 0.000
#> GSM228644 2 0.9358 0.7774 0.352 0.648
#> GSM228651 1 0.0000 0.7047 1.000 0.000
#> GSM228654 1 0.0938 0.6954 0.988 0.012
#> GSM228658 1 0.0000 0.7047 1.000 0.000
#> GSM228606 1 0.6801 0.4337 0.820 0.180
#> GSM228611 1 0.0000 0.7047 1.000 0.000
#> GSM228618 1 0.0000 0.7047 1.000 0.000
#> GSM228621 1 0.4022 0.6209 0.920 0.080
#> GSM228624 1 0.2948 0.6561 0.948 0.052
#> GSM228630 2 0.9922 0.6707 0.448 0.552
#> GSM228636 2 0.6343 0.7961 0.160 0.840
#> GSM228638 1 0.5629 0.5307 0.868 0.132
#> GSM228648 1 0.9933 -0.4837 0.548 0.452
#> GSM228670 2 0.7453 0.8028 0.212 0.788
#> GSM228671 2 0.9460 0.7720 0.364 0.636
#> GSM228672 2 0.9833 -0.4164 0.424 0.576
#> GSM228674 2 0.6887 0.7640 0.184 0.816
#> GSM228675 2 0.6148 0.7916 0.152 0.848
#> GSM228676 1 0.5629 0.6893 0.868 0.132
#> GSM228667 1 0.9522 0.0697 0.628 0.372
#> GSM228668 1 0.9358 0.7046 0.648 0.352
#> GSM228669 1 0.9881 0.6266 0.564 0.436
#> GSM228673 1 0.3733 0.6322 0.928 0.072
#> GSM228677 2 0.9427 0.7735 0.360 0.640
#> GSM228678 2 0.8499 0.8014 0.276 0.724
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.4883 0.7251 0.788 0.004 0.208
#> GSM228563 2 0.6527 0.8202 0.068 0.744 0.188
#> GSM228565 1 0.5859 0.4118 0.656 0.000 0.344
#> GSM228566 3 0.4521 0.8552 0.180 0.004 0.816
#> GSM228567 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228570 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228571 1 0.1643 0.8303 0.956 0.000 0.044
#> GSM228574 3 0.2651 0.7970 0.060 0.012 0.928
#> GSM228575 3 0.4094 0.8202 0.100 0.028 0.872
#> GSM228576 1 0.5690 0.5090 0.708 0.004 0.288
#> GSM228579 1 0.1860 0.8268 0.948 0.000 0.052
#> GSM228580 2 0.3482 0.8314 0.000 0.872 0.128
#> GSM228581 3 0.6302 -0.4060 0.000 0.480 0.520
#> GSM228666 2 0.6154 0.6533 0.000 0.592 0.408
#> GSM228564 2 0.9509 0.4331 0.336 0.464 0.200
#> GSM228568 1 0.4605 0.6771 0.796 0.000 0.204
#> GSM228569 1 0.3412 0.7655 0.876 0.000 0.124
#> GSM228572 2 0.0892 0.8087 0.000 0.980 0.020
#> GSM228573 3 0.4796 0.8495 0.220 0.000 0.780
#> GSM228577 1 0.0424 0.8464 0.992 0.000 0.008
#> GSM228578 1 0.2066 0.8217 0.940 0.000 0.060
#> GSM228663 3 0.4750 0.8509 0.216 0.000 0.784
#> GSM228664 3 0.5263 0.7939 0.060 0.116 0.824
#> GSM228665 3 0.4796 0.8495 0.220 0.000 0.780
#> GSM228582 1 0.6225 0.0273 0.568 0.000 0.432
#> GSM228583 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228585 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228587 1 0.0424 0.8451 0.992 0.008 0.000
#> GSM228588 2 0.3406 0.8085 0.068 0.904 0.028
#> GSM228589 2 0.0424 0.8109 0.000 0.992 0.008
#> GSM228590 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228591 2 0.4062 0.7336 0.000 0.836 0.164
#> GSM228597 2 0.5574 0.8305 0.032 0.784 0.184
#> GSM228601 2 0.0424 0.8109 0.000 0.992 0.008
#> GSM228604 3 0.6045 0.4217 0.000 0.380 0.620
#> GSM228608 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228609 2 0.7337 0.7604 0.152 0.708 0.140
#> GSM228613 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228616 1 0.5938 0.5695 0.732 0.020 0.248
#> GSM228628 2 0.5580 0.6823 0.008 0.736 0.256
#> GSM228634 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228642 2 0.3941 0.7400 0.000 0.844 0.156
#> GSM228645 3 0.5202 0.8275 0.136 0.044 0.820
#> GSM228646 3 0.5191 0.8125 0.112 0.060 0.828
#> GSM228652 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228655 1 0.0237 0.8482 0.996 0.000 0.004
#> GSM228656 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228659 1 0.6705 0.5972 0.748 0.108 0.144
#> GSM228662 1 0.0237 0.8474 0.996 0.004 0.000
#> GSM228584 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228586 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228592 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228593 1 0.9226 -0.2433 0.436 0.412 0.152
#> GSM228594 1 0.1289 0.8364 0.968 0.000 0.032
#> GSM228598 1 0.0237 0.8472 0.996 0.000 0.004
#> GSM228607 3 0.5020 0.8448 0.192 0.012 0.796
#> GSM228612 3 0.4883 0.8527 0.208 0.004 0.788
#> GSM228619 1 0.6597 0.4917 0.696 0.036 0.268
#> GSM228622 1 0.2261 0.8181 0.932 0.000 0.068
#> GSM228625 1 0.6201 0.6359 0.748 0.208 0.044
#> GSM228631 1 0.5397 0.4901 0.720 0.000 0.280
#> GSM228633 2 0.3412 0.7656 0.000 0.876 0.124
#> GSM228637 2 0.6151 0.8260 0.056 0.764 0.180
#> GSM228639 3 0.5000 0.5795 0.044 0.124 0.832
#> GSM228649 2 0.6388 0.8220 0.064 0.752 0.184
#> GSM228660 1 0.2384 0.8287 0.936 0.008 0.056
#> GSM228661 1 0.2261 0.8159 0.932 0.000 0.068
#> GSM228595 2 0.1031 0.8086 0.000 0.976 0.024
#> GSM228599 2 0.5986 0.7880 0.012 0.704 0.284
#> GSM228602 3 0.4796 0.8495 0.220 0.000 0.780
#> GSM228614 2 0.6244 0.5389 0.000 0.560 0.440
#> GSM228626 2 0.3551 0.7593 0.000 0.868 0.132
#> GSM228640 3 0.4796 0.8495 0.220 0.000 0.780
#> GSM228643 3 0.4861 0.8549 0.192 0.008 0.800
#> GSM228650 3 0.4326 0.8487 0.144 0.012 0.844
#> GSM228653 3 0.4796 0.8495 0.220 0.000 0.780
#> GSM228657 2 0.0892 0.8087 0.000 0.980 0.020
#> GSM228605 1 0.5968 0.3325 0.636 0.000 0.364
#> GSM228610 3 0.4399 0.8554 0.188 0.000 0.812
#> GSM228617 3 0.4796 0.8495 0.220 0.000 0.780
#> GSM228620 3 0.4796 0.8495 0.220 0.000 0.780
#> GSM228623 2 0.6000 0.8291 0.040 0.760 0.200
#> GSM228629 3 0.4796 0.8495 0.220 0.000 0.780
#> GSM228632 3 0.2564 0.7749 0.036 0.028 0.936
#> GSM228635 2 0.5905 0.8289 0.044 0.772 0.184
#> GSM228647 3 0.4750 0.8512 0.216 0.000 0.784
#> GSM228596 3 0.3771 0.8265 0.112 0.012 0.876
#> GSM228600 3 0.4796 0.8495 0.220 0.000 0.780
#> GSM228603 3 0.4796 0.8495 0.220 0.000 0.780
#> GSM228615 2 0.5521 0.8317 0.032 0.788 0.180
#> GSM228627 3 0.4796 0.8495 0.220 0.000 0.780
#> GSM228641 3 0.4605 0.8541 0.204 0.000 0.796
#> GSM228644 2 0.3412 0.7656 0.000 0.876 0.124
#> GSM228651 3 0.4750 0.8512 0.216 0.000 0.784
#> GSM228654 3 0.4682 0.8554 0.192 0.004 0.804
#> GSM228658 3 0.4796 0.8495 0.220 0.000 0.780
#> GSM228606 3 0.1620 0.7558 0.024 0.012 0.964
#> GSM228611 3 0.4702 0.8523 0.212 0.000 0.788
#> GSM228618 3 0.4796 0.8495 0.220 0.000 0.780
#> GSM228621 3 0.4390 0.8497 0.148 0.012 0.840
#> GSM228624 3 0.4473 0.8539 0.164 0.008 0.828
#> GSM228630 3 0.1643 0.7320 0.000 0.044 0.956
#> GSM228636 2 0.5631 0.8316 0.044 0.792 0.164
#> GSM228638 3 0.4634 0.8530 0.164 0.012 0.824
#> GSM228648 3 0.4845 0.7908 0.052 0.104 0.844
#> GSM228670 2 0.7057 0.7900 0.056 0.680 0.264
#> GSM228671 3 0.5138 0.3476 0.000 0.252 0.748
#> GSM228672 1 0.8711 0.2935 0.592 0.224 0.184
#> GSM228674 2 0.7739 0.7700 0.124 0.672 0.204
#> GSM228675 2 0.5921 0.8275 0.032 0.756 0.212
#> GSM228676 3 0.6573 0.5753 0.140 0.104 0.756
#> GSM228667 3 0.8129 0.1308 0.124 0.244 0.632
#> GSM228668 1 0.0000 0.8488 1.000 0.000 0.000
#> GSM228669 1 0.6775 0.5778 0.740 0.096 0.164
#> GSM228673 3 0.1950 0.7759 0.040 0.008 0.952
#> GSM228677 2 0.6225 0.6299 0.000 0.568 0.432
#> GSM228678 2 0.4409 0.8342 0.004 0.824 0.172
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.7512 0.2765 0.368 0.040 0.080 0.512
#> GSM228563 4 0.2300 0.6976 0.028 0.048 0.000 0.924
#> GSM228565 3 0.8782 0.1182 0.320 0.056 0.416 0.208
#> GSM228566 3 0.3577 0.8453 0.004 0.072 0.868 0.056
#> GSM228567 1 0.1004 0.8743 0.972 0.000 0.024 0.004
#> GSM228570 1 0.2883 0.8491 0.908 0.016 0.028 0.048
#> GSM228571 1 0.3170 0.8469 0.896 0.044 0.044 0.016
#> GSM228574 3 0.2928 0.8624 0.000 0.052 0.896 0.052
#> GSM228575 3 0.5937 0.7084 0.016 0.120 0.728 0.136
#> GSM228576 1 0.8323 0.0294 0.416 0.068 0.408 0.108
#> GSM228579 1 0.3135 0.8461 0.896 0.048 0.044 0.012
#> GSM228580 4 0.5693 0.2928 0.020 0.368 0.008 0.604
#> GSM228581 4 0.7927 0.2053 0.008 0.212 0.356 0.424
#> GSM228666 4 0.7807 0.2591 0.008 0.296 0.216 0.480
#> GSM228564 4 0.3279 0.6862 0.088 0.024 0.008 0.880
#> GSM228568 1 0.5496 0.7024 0.732 0.040 0.208 0.020
#> GSM228569 1 0.3743 0.7882 0.824 0.016 0.160 0.000
#> GSM228572 2 0.4008 0.7500 0.000 0.756 0.000 0.244
#> GSM228573 3 0.1271 0.8763 0.012 0.012 0.968 0.008
#> GSM228577 1 0.1356 0.8715 0.960 0.008 0.032 0.000
#> GSM228578 1 0.3663 0.8120 0.848 0.008 0.128 0.016
#> GSM228663 3 0.1640 0.8725 0.012 0.020 0.956 0.012
#> GSM228664 3 0.2060 0.8608 0.000 0.052 0.932 0.016
#> GSM228665 3 0.1526 0.8724 0.012 0.016 0.960 0.012
#> GSM228582 1 0.6895 0.2814 0.520 0.068 0.396 0.016
#> GSM228583 1 0.1004 0.8743 0.972 0.000 0.024 0.004
#> GSM228585 1 0.1191 0.8740 0.968 0.004 0.024 0.004
#> GSM228587 1 0.1510 0.8602 0.956 0.000 0.016 0.028
#> GSM228588 2 0.6337 0.4774 0.068 0.552 0.000 0.380
#> GSM228589 2 0.4677 0.6736 0.004 0.680 0.000 0.316
#> GSM228590 1 0.1004 0.8743 0.972 0.000 0.024 0.004
#> GSM228591 2 0.2915 0.8029 0.000 0.892 0.028 0.080
#> GSM228597 4 0.2300 0.6868 0.016 0.064 0.000 0.920
#> GSM228601 2 0.3688 0.7857 0.000 0.792 0.000 0.208
#> GSM228604 2 0.5137 0.4722 0.000 0.680 0.296 0.024
#> GSM228608 1 0.1004 0.8743 0.972 0.000 0.024 0.004
#> GSM228609 4 0.5277 0.5823 0.116 0.132 0.000 0.752
#> GSM228613 1 0.1004 0.8743 0.972 0.000 0.024 0.004
#> GSM228616 1 0.8361 0.1793 0.448 0.028 0.288 0.236
#> GSM228628 2 0.5548 0.6142 0.000 0.716 0.084 0.200
#> GSM228634 1 0.1004 0.8734 0.972 0.004 0.024 0.000
#> GSM228642 2 0.2363 0.8070 0.000 0.920 0.024 0.056
#> GSM228645 3 0.6650 0.6159 0.004 0.184 0.640 0.172
#> GSM228646 3 0.6363 0.6568 0.004 0.184 0.668 0.144
#> GSM228652 1 0.1151 0.8732 0.968 0.000 0.024 0.008
#> GSM228655 1 0.2773 0.8476 0.900 0.000 0.072 0.028
#> GSM228656 1 0.1191 0.8740 0.968 0.004 0.024 0.004
#> GSM228659 1 0.4961 0.0834 0.552 0.000 0.000 0.448
#> GSM228662 1 0.1004 0.8743 0.972 0.000 0.024 0.004
#> GSM228584 1 0.1004 0.8743 0.972 0.000 0.024 0.004
#> GSM228586 1 0.1109 0.8729 0.968 0.004 0.028 0.000
#> GSM228592 1 0.1004 0.8743 0.972 0.000 0.024 0.004
#> GSM228593 4 0.4898 0.5434 0.260 0.024 0.000 0.716
#> GSM228594 1 0.1488 0.8703 0.956 0.012 0.032 0.000
#> GSM228598 1 0.1004 0.8734 0.972 0.004 0.024 0.000
#> GSM228607 3 0.2904 0.8547 0.012 0.024 0.904 0.060
#> GSM228612 3 0.1871 0.8734 0.012 0.024 0.948 0.016
#> GSM228619 3 0.7885 0.1245 0.280 0.004 0.444 0.272
#> GSM228622 1 0.4868 0.6545 0.720 0.000 0.256 0.024
#> GSM228625 4 0.6159 0.1418 0.436 0.012 0.028 0.524
#> GSM228631 3 0.7034 0.1044 0.412 0.004 0.480 0.104
#> GSM228633 2 0.2949 0.8190 0.000 0.888 0.024 0.088
#> GSM228637 4 0.2731 0.6735 0.008 0.092 0.004 0.896
#> GSM228639 3 0.5088 0.5323 0.000 0.024 0.688 0.288
#> GSM228649 4 0.2443 0.6941 0.024 0.060 0.000 0.916
#> GSM228660 1 0.4587 0.7882 0.812 0.016 0.128 0.044
#> GSM228661 1 0.2546 0.8442 0.900 0.008 0.092 0.000
#> GSM228595 2 0.2976 0.8153 0.000 0.872 0.008 0.120
#> GSM228599 4 0.2917 0.6937 0.008 0.040 0.048 0.904
#> GSM228602 3 0.2594 0.8651 0.012 0.036 0.920 0.032
#> GSM228614 4 0.3542 0.6803 0.000 0.060 0.076 0.864
#> GSM228626 2 0.2623 0.8136 0.000 0.908 0.028 0.064
#> GSM228640 3 0.2961 0.8605 0.012 0.044 0.904 0.040
#> GSM228643 3 0.3724 0.8440 0.012 0.084 0.864 0.040
#> GSM228650 3 0.2227 0.8701 0.000 0.036 0.928 0.036
#> GSM228653 3 0.1139 0.8755 0.012 0.008 0.972 0.008
#> GSM228657 2 0.3631 0.7989 0.004 0.824 0.004 0.168
#> GSM228605 3 0.7563 0.3725 0.276 0.012 0.536 0.176
#> GSM228610 3 0.1526 0.8737 0.012 0.016 0.960 0.012
#> GSM228617 3 0.1975 0.8719 0.012 0.016 0.944 0.028
#> GSM228620 3 0.0992 0.8745 0.012 0.008 0.976 0.004
#> GSM228623 4 0.2189 0.6959 0.004 0.044 0.020 0.932
#> GSM228629 3 0.0992 0.8743 0.012 0.004 0.976 0.008
#> GSM228632 3 0.2214 0.8558 0.000 0.028 0.928 0.044
#> GSM228635 4 0.3102 0.6586 0.008 0.116 0.004 0.872
#> GSM228647 3 0.1139 0.8755 0.012 0.008 0.972 0.008
#> GSM228596 3 0.5063 0.7136 0.012 0.032 0.752 0.204
#> GSM228600 3 0.2781 0.8631 0.012 0.040 0.912 0.036
#> GSM228603 3 0.2689 0.8641 0.012 0.036 0.916 0.036
#> GSM228615 4 0.2452 0.6816 0.004 0.084 0.004 0.908
#> GSM228627 3 0.1762 0.8757 0.012 0.016 0.952 0.020
#> GSM228641 3 0.2689 0.8641 0.012 0.036 0.916 0.036
#> GSM228644 2 0.2329 0.8161 0.000 0.916 0.012 0.072
#> GSM228651 3 0.1394 0.8754 0.012 0.016 0.964 0.008
#> GSM228654 3 0.1377 0.8765 0.008 0.020 0.964 0.008
#> GSM228658 3 0.1271 0.8760 0.012 0.012 0.968 0.008
#> GSM228606 3 0.3142 0.8018 0.000 0.008 0.860 0.132
#> GSM228611 3 0.1271 0.8735 0.012 0.008 0.968 0.012
#> GSM228618 3 0.1139 0.8740 0.012 0.008 0.972 0.008
#> GSM228621 3 0.0524 0.8760 0.004 0.000 0.988 0.008
#> GSM228624 3 0.1059 0.8743 0.000 0.016 0.972 0.012
#> GSM228630 3 0.1863 0.8618 0.004 0.012 0.944 0.040
#> GSM228636 4 0.3850 0.5925 0.004 0.188 0.004 0.804
#> GSM228638 3 0.1174 0.8732 0.000 0.012 0.968 0.020
#> GSM228648 3 0.1610 0.8653 0.000 0.032 0.952 0.016
#> GSM228670 4 0.2115 0.7013 0.004 0.036 0.024 0.936
#> GSM228671 4 0.6104 0.4562 0.004 0.064 0.296 0.636
#> GSM228672 4 0.3208 0.6605 0.148 0.004 0.000 0.848
#> GSM228674 4 0.3159 0.6977 0.036 0.052 0.016 0.896
#> GSM228675 4 0.2197 0.7011 0.012 0.028 0.024 0.936
#> GSM228676 4 0.6223 0.4656 0.028 0.036 0.292 0.644
#> GSM228667 4 0.6007 0.5572 0.020 0.076 0.192 0.712
#> GSM228668 1 0.1004 0.8743 0.972 0.000 0.024 0.004
#> GSM228669 4 0.4761 0.5008 0.332 0.004 0.000 0.664
#> GSM228673 3 0.4381 0.7515 0.008 0.028 0.804 0.160
#> GSM228677 4 0.5916 0.4861 0.000 0.072 0.272 0.656
#> GSM228678 4 0.3538 0.6379 0.004 0.160 0.004 0.832
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 4 0.7077 0.1413 0.192 0.004 0.016 0.400 0.388
#> GSM228563 4 0.3858 0.6467 0.008 0.008 0.000 0.760 0.224
#> GSM228565 5 0.8493 0.3827 0.200 0.008 0.240 0.164 0.388
#> GSM228566 3 0.5089 0.4612 0.000 0.024 0.652 0.024 0.300
#> GSM228567 1 0.0000 0.8456 1.000 0.000 0.000 0.000 0.000
#> GSM228570 1 0.3531 0.7436 0.816 0.000 0.000 0.036 0.148
#> GSM228571 1 0.3536 0.7582 0.824 0.004 0.016 0.008 0.148
#> GSM228574 3 0.3583 0.7359 0.000 0.012 0.792 0.004 0.192
#> GSM228575 5 0.6221 0.5029 0.000 0.060 0.308 0.052 0.580
#> GSM228576 1 0.8426 -0.3166 0.324 0.012 0.240 0.100 0.324
#> GSM228579 1 0.2833 0.7857 0.864 0.004 0.012 0.000 0.120
#> GSM228580 5 0.6508 -0.0183 0.000 0.188 0.004 0.312 0.496
#> GSM228581 5 0.6805 0.4362 0.000 0.044 0.224 0.168 0.564
#> GSM228666 5 0.7268 0.3460 0.000 0.184 0.080 0.200 0.536
#> GSM228564 4 0.4039 0.6177 0.008 0.004 0.000 0.720 0.268
#> GSM228568 1 0.6456 0.4458 0.584 0.004 0.168 0.016 0.228
#> GSM228569 1 0.3459 0.7499 0.832 0.000 0.116 0.000 0.052
#> GSM228572 2 0.3681 0.7227 0.000 0.808 0.000 0.148 0.044
#> GSM228573 3 0.1822 0.8037 0.004 0.004 0.932 0.004 0.056
#> GSM228577 1 0.0771 0.8419 0.976 0.000 0.004 0.000 0.020
#> GSM228578 1 0.4746 0.7084 0.768 0.004 0.136 0.020 0.072
#> GSM228663 3 0.1682 0.7908 0.004 0.012 0.940 0.000 0.044
#> GSM228664 3 0.3224 0.6969 0.000 0.016 0.824 0.000 0.160
#> GSM228665 3 0.1026 0.7981 0.004 0.004 0.968 0.000 0.024
#> GSM228582 1 0.7139 0.0706 0.464 0.028 0.284 0.000 0.224
#> GSM228583 1 0.0000 0.8456 1.000 0.000 0.000 0.000 0.000
#> GSM228585 1 0.0000 0.8456 1.000 0.000 0.000 0.000 0.000
#> GSM228587 1 0.0898 0.8368 0.972 0.000 0.000 0.008 0.020
#> GSM228588 2 0.6614 0.4162 0.028 0.528 0.000 0.316 0.128
#> GSM228589 2 0.5531 0.5944 0.000 0.632 0.000 0.248 0.120
#> GSM228590 1 0.0000 0.8456 1.000 0.000 0.000 0.000 0.000
#> GSM228591 2 0.2864 0.7490 0.000 0.872 0.012 0.012 0.104
#> GSM228597 4 0.2597 0.6876 0.000 0.024 0.000 0.884 0.092
#> GSM228601 2 0.2249 0.7706 0.000 0.896 0.000 0.096 0.008
#> GSM228604 2 0.5481 0.2891 0.000 0.660 0.232 0.008 0.100
#> GSM228608 1 0.1012 0.8376 0.968 0.000 0.000 0.020 0.012
#> GSM228609 4 0.4411 0.6425 0.044 0.048 0.000 0.796 0.112
#> GSM228613 1 0.0000 0.8456 1.000 0.000 0.000 0.000 0.000
#> GSM228616 1 0.8426 -0.2496 0.340 0.004 0.132 0.240 0.284
#> GSM228628 2 0.6300 0.4078 0.000 0.632 0.048 0.124 0.196
#> GSM228634 1 0.0162 0.8449 0.996 0.000 0.000 0.000 0.004
#> GSM228642 2 0.1557 0.7728 0.000 0.940 0.008 0.000 0.052
#> GSM228645 5 0.7169 0.5200 0.000 0.092 0.276 0.108 0.524
#> GSM228646 5 0.7449 0.2938 0.000 0.104 0.392 0.100 0.404
#> GSM228652 1 0.0992 0.8367 0.968 0.000 0.000 0.024 0.008
#> GSM228655 1 0.3506 0.7775 0.856 0.000 0.068 0.040 0.036
#> GSM228656 1 0.0000 0.8456 1.000 0.000 0.000 0.000 0.000
#> GSM228659 4 0.5142 0.3014 0.392 0.000 0.000 0.564 0.044
#> GSM228662 1 0.0000 0.8456 1.000 0.000 0.000 0.000 0.000
#> GSM228584 1 0.0000 0.8456 1.000 0.000 0.000 0.000 0.000
#> GSM228586 1 0.0162 0.8449 0.996 0.000 0.000 0.000 0.004
#> GSM228592 1 0.0000 0.8456 1.000 0.000 0.000 0.000 0.000
#> GSM228593 4 0.5386 0.5417 0.148 0.004 0.000 0.680 0.168
#> GSM228594 1 0.0798 0.8415 0.976 0.000 0.008 0.000 0.016
#> GSM228598 1 0.0404 0.8435 0.988 0.000 0.000 0.000 0.012
#> GSM228607 3 0.3839 0.6861 0.000 0.004 0.816 0.072 0.108
#> GSM228612 3 0.2886 0.7502 0.004 0.016 0.864 0.000 0.116
#> GSM228619 3 0.7875 -0.2674 0.216 0.000 0.368 0.336 0.080
#> GSM228622 1 0.5781 0.4608 0.644 0.004 0.264 0.036 0.052
#> GSM228625 4 0.5930 0.4388 0.264 0.008 0.020 0.632 0.076
#> GSM228631 3 0.7234 -0.0145 0.336 0.000 0.476 0.096 0.092
#> GSM228633 2 0.1074 0.7859 0.000 0.968 0.016 0.012 0.004
#> GSM228637 4 0.2685 0.6597 0.000 0.028 0.000 0.880 0.092
#> GSM228639 3 0.4419 0.4996 0.000 0.004 0.740 0.212 0.044
#> GSM228649 4 0.2802 0.6747 0.008 0.016 0.000 0.876 0.100
#> GSM228660 1 0.6313 0.5811 0.664 0.008 0.164 0.072 0.092
#> GSM228661 1 0.1628 0.8227 0.936 0.000 0.056 0.000 0.008
#> GSM228595 2 0.0794 0.7886 0.000 0.972 0.000 0.028 0.000
#> GSM228599 4 0.3006 0.6495 0.000 0.004 0.004 0.836 0.156
#> GSM228602 3 0.2929 0.7632 0.004 0.004 0.860 0.008 0.124
#> GSM228614 4 0.2588 0.6797 0.000 0.000 0.048 0.892 0.060
#> GSM228626 2 0.0740 0.7837 0.000 0.980 0.008 0.004 0.008
#> GSM228640 3 0.3368 0.7269 0.004 0.004 0.820 0.008 0.164
#> GSM228643 3 0.4056 0.6764 0.004 0.024 0.772 0.004 0.196
#> GSM228650 3 0.2604 0.7808 0.004 0.004 0.880 0.004 0.108
#> GSM228653 3 0.1518 0.7984 0.004 0.004 0.944 0.000 0.048
#> GSM228657 2 0.2260 0.7783 0.000 0.908 0.000 0.064 0.028
#> GSM228605 3 0.8623 -0.3984 0.172 0.008 0.352 0.220 0.248
#> GSM228610 3 0.1116 0.7986 0.004 0.004 0.964 0.000 0.028
#> GSM228617 3 0.2084 0.7911 0.004 0.004 0.920 0.008 0.064
#> GSM228620 3 0.0932 0.7998 0.004 0.004 0.972 0.000 0.020
#> GSM228623 4 0.3155 0.6818 0.000 0.008 0.020 0.852 0.120
#> GSM228629 3 0.1490 0.7983 0.004 0.004 0.952 0.008 0.032
#> GSM228632 3 0.2569 0.7691 0.000 0.012 0.896 0.016 0.076
#> GSM228635 4 0.2932 0.6539 0.000 0.032 0.000 0.864 0.104
#> GSM228647 3 0.0486 0.7981 0.004 0.004 0.988 0.000 0.004
#> GSM228596 3 0.5386 0.4647 0.000 0.012 0.676 0.088 0.224
#> GSM228600 3 0.3114 0.7516 0.004 0.004 0.844 0.008 0.140
#> GSM228603 3 0.2818 0.7605 0.004 0.000 0.860 0.008 0.128
#> GSM228615 4 0.2540 0.6685 0.000 0.024 0.000 0.888 0.088
#> GSM228627 3 0.2393 0.7921 0.004 0.016 0.900 0.000 0.080
#> GSM228641 3 0.2865 0.7581 0.004 0.000 0.856 0.008 0.132
#> GSM228644 2 0.0854 0.7869 0.000 0.976 0.008 0.012 0.004
#> GSM228651 3 0.2054 0.7940 0.004 0.008 0.916 0.000 0.072
#> GSM228654 3 0.2166 0.7932 0.004 0.012 0.912 0.000 0.072
#> GSM228658 3 0.1662 0.7972 0.004 0.004 0.936 0.000 0.056
#> GSM228606 3 0.4085 0.6612 0.000 0.008 0.804 0.104 0.084
#> GSM228611 3 0.1202 0.8001 0.004 0.004 0.960 0.000 0.032
#> GSM228618 3 0.1805 0.7956 0.004 0.004 0.936 0.008 0.048
#> GSM228621 3 0.1116 0.8021 0.000 0.004 0.964 0.004 0.028
#> GSM228624 3 0.2646 0.7490 0.000 0.004 0.868 0.004 0.124
#> GSM228630 3 0.1116 0.7976 0.000 0.004 0.964 0.004 0.028
#> GSM228636 4 0.3291 0.6447 0.000 0.064 0.000 0.848 0.088
#> GSM228638 3 0.0727 0.7977 0.000 0.004 0.980 0.004 0.012
#> GSM228648 3 0.0613 0.7986 0.000 0.004 0.984 0.004 0.008
#> GSM228670 4 0.3394 0.6650 0.000 0.004 0.020 0.824 0.152
#> GSM228671 4 0.6985 0.0790 0.000 0.024 0.180 0.452 0.344
#> GSM228672 4 0.2864 0.6894 0.024 0.000 0.000 0.864 0.112
#> GSM228674 4 0.4865 0.6083 0.008 0.036 0.012 0.720 0.224
#> GSM228675 4 0.3948 0.6373 0.000 0.012 0.012 0.768 0.208
#> GSM228676 4 0.6324 0.2420 0.004 0.000 0.168 0.532 0.296
#> GSM228667 4 0.6031 0.4480 0.004 0.024 0.068 0.588 0.316
#> GSM228668 1 0.2152 0.8167 0.920 0.004 0.000 0.044 0.032
#> GSM228669 4 0.4302 0.5715 0.208 0.000 0.000 0.744 0.048
#> GSM228673 3 0.4668 0.5726 0.000 0.008 0.748 0.076 0.168
#> GSM228677 4 0.6792 0.1051 0.000 0.020 0.264 0.516 0.200
#> GSM228678 4 0.3780 0.6610 0.000 0.072 0.000 0.812 0.116
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 6 0.6572 0.1882 0.132 0.000 0.020 0.240 0.056 0.552
#> GSM228563 4 0.5883 0.4028 0.000 0.012 0.000 0.496 0.152 0.340
#> GSM228565 6 0.6567 0.4195 0.144 0.004 0.116 0.096 0.028 0.612
#> GSM228566 6 0.4986 -0.1217 0.004 0.016 0.448 0.000 0.028 0.504
#> GSM228567 1 0.0000 0.8391 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228570 1 0.4626 0.3644 0.588 0.000 0.000 0.032 0.008 0.372
#> GSM228571 1 0.4432 0.5961 0.680 0.004 0.004 0.012 0.020 0.280
#> GSM228574 3 0.4577 0.6372 0.000 0.012 0.684 0.016 0.024 0.264
#> GSM228575 6 0.6897 -0.2753 0.000 0.024 0.140 0.044 0.392 0.400
#> GSM228576 6 0.6591 0.3976 0.220 0.012 0.100 0.084 0.008 0.576
#> GSM228579 1 0.3345 0.7498 0.816 0.004 0.004 0.000 0.032 0.144
#> GSM228580 5 0.5137 0.3892 0.000 0.064 0.000 0.164 0.696 0.076
#> GSM228581 5 0.7554 0.3749 0.000 0.028 0.188 0.120 0.456 0.208
#> GSM228666 5 0.7725 0.4566 0.000 0.108 0.040 0.160 0.412 0.280
#> GSM228564 4 0.5699 0.3730 0.016 0.000 0.000 0.476 0.104 0.404
#> GSM228568 1 0.7133 0.3474 0.516 0.000 0.156 0.020 0.120 0.188
#> GSM228569 1 0.4305 0.7233 0.776 0.000 0.100 0.000 0.068 0.056
#> GSM228572 2 0.3950 0.6201 0.000 0.780 0.000 0.144 0.060 0.016
#> GSM228573 3 0.2579 0.7560 0.004 0.000 0.876 0.000 0.032 0.088
#> GSM228577 1 0.1908 0.8195 0.916 0.000 0.000 0.000 0.056 0.028
#> GSM228578 1 0.5249 0.6571 0.708 0.000 0.132 0.020 0.032 0.108
#> GSM228663 3 0.3167 0.7178 0.004 0.004 0.856 0.008 0.060 0.068
#> GSM228664 3 0.4634 0.6057 0.000 0.012 0.736 0.008 0.116 0.128
#> GSM228665 3 0.1549 0.7521 0.004 0.000 0.944 0.004 0.024 0.024
#> GSM228582 1 0.7859 0.0153 0.400 0.008 0.232 0.016 0.136 0.208
#> GSM228583 1 0.0000 0.8391 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228585 1 0.0000 0.8391 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228587 1 0.1350 0.8226 0.952 0.000 0.000 0.020 0.008 0.020
#> GSM228588 2 0.7695 0.1519 0.044 0.388 0.000 0.328 0.112 0.128
#> GSM228589 2 0.6657 0.3349 0.000 0.492 0.000 0.264 0.172 0.072
#> GSM228590 1 0.0146 0.8382 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM228591 2 0.4164 0.6094 0.000 0.772 0.004 0.016 0.140 0.068
#> GSM228597 4 0.4203 0.5232 0.000 0.016 0.000 0.764 0.132 0.088
#> GSM228601 2 0.2473 0.6885 0.000 0.876 0.000 0.104 0.008 0.012
#> GSM228604 2 0.5954 0.2227 0.000 0.592 0.196 0.000 0.044 0.168
#> GSM228608 1 0.1151 0.8246 0.956 0.000 0.000 0.012 0.000 0.032
#> GSM228609 4 0.5658 0.4858 0.044 0.040 0.000 0.664 0.052 0.200
#> GSM228613 1 0.0000 0.8391 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228616 6 0.7897 0.2184 0.196 0.004 0.076 0.184 0.084 0.456
#> GSM228628 2 0.6514 0.2532 0.000 0.564 0.028 0.088 0.068 0.252
#> GSM228634 1 0.0000 0.8391 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228642 2 0.2201 0.6824 0.000 0.900 0.000 0.000 0.052 0.048
#> GSM228645 6 0.6236 0.2785 0.000 0.072 0.100 0.060 0.116 0.652
#> GSM228646 6 0.6365 0.3258 0.000 0.072 0.184 0.052 0.072 0.620
#> GSM228652 1 0.1697 0.8186 0.936 0.000 0.004 0.020 0.004 0.036
#> GSM228655 1 0.4224 0.7220 0.800 0.004 0.076 0.048 0.008 0.064
#> GSM228656 1 0.0000 0.8391 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228659 4 0.5913 0.2391 0.348 0.000 0.000 0.500 0.020 0.132
#> GSM228662 1 0.0000 0.8391 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228584 1 0.0000 0.8391 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228586 1 0.0260 0.8381 0.992 0.000 0.000 0.000 0.008 0.000
#> GSM228592 1 0.0000 0.8391 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228593 4 0.6386 0.4025 0.088 0.000 0.000 0.560 0.144 0.208
#> GSM228594 1 0.1909 0.8206 0.920 0.000 0.004 0.000 0.052 0.024
#> GSM228598 1 0.1807 0.8221 0.920 0.000 0.000 0.000 0.060 0.020
#> GSM228607 3 0.4927 0.6119 0.012 0.004 0.740 0.080 0.036 0.128
#> GSM228612 3 0.3884 0.6811 0.004 0.004 0.796 0.016 0.040 0.140
#> GSM228619 3 0.8109 -0.3398 0.184 0.000 0.296 0.248 0.024 0.248
#> GSM228622 1 0.5674 0.3988 0.612 0.000 0.264 0.020 0.020 0.084
#> GSM228625 4 0.5938 0.4028 0.204 0.008 0.000 0.604 0.032 0.152
#> GSM228631 3 0.7575 -0.2263 0.260 0.000 0.368 0.084 0.020 0.268
#> GSM228633 2 0.0291 0.7150 0.000 0.992 0.000 0.004 0.004 0.000
#> GSM228637 4 0.3781 0.4478 0.000 0.028 0.000 0.772 0.184 0.016
#> GSM228639 3 0.4862 0.5781 0.000 0.008 0.720 0.176 0.040 0.056
#> GSM228649 4 0.3931 0.5091 0.004 0.004 0.000 0.784 0.092 0.116
#> GSM228660 1 0.6947 0.4405 0.576 0.004 0.188 0.084 0.060 0.088
#> GSM228661 1 0.2322 0.8013 0.896 0.000 0.072 0.000 0.024 0.008
#> GSM228595 2 0.0717 0.7147 0.000 0.976 0.000 0.008 0.016 0.000
#> GSM228599 4 0.5140 0.3894 0.000 0.008 0.012 0.580 0.048 0.352
#> GSM228602 3 0.3977 0.6425 0.004 0.008 0.728 0.000 0.020 0.240
#> GSM228614 4 0.3471 0.5463 0.000 0.004 0.040 0.840 0.040 0.076
#> GSM228626 2 0.0436 0.7146 0.000 0.988 0.000 0.004 0.004 0.004
#> GSM228640 3 0.4072 0.6018 0.004 0.004 0.684 0.000 0.016 0.292
#> GSM228643 3 0.4571 0.5629 0.004 0.012 0.660 0.000 0.032 0.292
#> GSM228650 3 0.4047 0.6556 0.000 0.004 0.716 0.000 0.036 0.244
#> GSM228653 3 0.2040 0.7511 0.004 0.004 0.904 0.000 0.004 0.084
#> GSM228657 2 0.2452 0.6920 0.000 0.892 0.000 0.044 0.056 0.008
#> GSM228605 6 0.8368 0.2056 0.108 0.004 0.312 0.188 0.076 0.312
#> GSM228610 3 0.2017 0.7475 0.000 0.004 0.920 0.008 0.020 0.048
#> GSM228617 3 0.3453 0.6909 0.004 0.000 0.788 0.000 0.028 0.180
#> GSM228620 3 0.1026 0.7536 0.004 0.000 0.968 0.008 0.008 0.012
#> GSM228623 4 0.4198 0.5320 0.000 0.008 0.012 0.776 0.116 0.088
#> GSM228629 3 0.1888 0.7534 0.004 0.000 0.916 0.000 0.012 0.068
#> GSM228632 3 0.3075 0.7211 0.000 0.004 0.864 0.028 0.036 0.068
#> GSM228635 4 0.4023 0.4276 0.000 0.040 0.000 0.748 0.200 0.012
#> GSM228647 3 0.1198 0.7549 0.004 0.000 0.960 0.004 0.012 0.020
#> GSM228596 3 0.6013 0.4250 0.000 0.004 0.624 0.096 0.100 0.176
#> GSM228600 3 0.4160 0.6154 0.004 0.008 0.696 0.000 0.020 0.272
#> GSM228603 3 0.4078 0.6193 0.004 0.008 0.700 0.000 0.016 0.272
#> GSM228615 4 0.4078 0.4799 0.000 0.020 0.004 0.772 0.160 0.044
#> GSM228627 3 0.2897 0.7445 0.004 0.004 0.860 0.008 0.016 0.108
#> GSM228641 3 0.4078 0.6193 0.004 0.008 0.700 0.000 0.016 0.272
#> GSM228644 2 0.0291 0.7150 0.000 0.992 0.000 0.004 0.000 0.004
#> GSM228651 3 0.2093 0.7513 0.004 0.004 0.900 0.000 0.004 0.088
#> GSM228654 3 0.2543 0.7482 0.004 0.004 0.868 0.000 0.008 0.116
#> GSM228658 3 0.2153 0.7530 0.004 0.004 0.900 0.000 0.008 0.084
#> GSM228606 3 0.4755 0.6059 0.000 0.008 0.740 0.112 0.028 0.112
#> GSM228611 3 0.1950 0.7452 0.004 0.000 0.924 0.008 0.020 0.044
#> GSM228618 3 0.2882 0.7352 0.004 0.000 0.848 0.000 0.028 0.120
#> GSM228621 3 0.2182 0.7595 0.000 0.004 0.900 0.000 0.020 0.076
#> GSM228624 3 0.3901 0.6844 0.004 0.004 0.800 0.012 0.060 0.120
#> GSM228630 3 0.2278 0.7525 0.000 0.008 0.908 0.008 0.024 0.052
#> GSM228636 4 0.4290 0.4195 0.000 0.076 0.000 0.744 0.168 0.012
#> GSM228638 3 0.1852 0.7532 0.000 0.004 0.928 0.004 0.024 0.040
#> GSM228648 3 0.1837 0.7564 0.000 0.012 0.932 0.004 0.020 0.032
#> GSM228670 4 0.4305 0.5257 0.000 0.008 0.004 0.756 0.108 0.124
#> GSM228671 4 0.7580 -0.0359 0.000 0.012 0.132 0.396 0.196 0.264
#> GSM228672 4 0.3888 0.5583 0.016 0.000 0.000 0.752 0.024 0.208
#> GSM228674 4 0.5614 0.4437 0.004 0.016 0.004 0.628 0.144 0.204
#> GSM228675 4 0.5073 0.4472 0.000 0.004 0.004 0.664 0.160 0.168
#> GSM228676 4 0.6573 0.2425 0.000 0.004 0.088 0.484 0.096 0.328
#> GSM228667 4 0.5944 0.2722 0.000 0.004 0.036 0.464 0.080 0.416
#> GSM228668 1 0.2737 0.7904 0.884 0.000 0.004 0.036 0.020 0.056
#> GSM228669 4 0.4768 0.4860 0.176 0.000 0.000 0.704 0.016 0.104
#> GSM228673 3 0.6085 0.3369 0.000 0.000 0.600 0.124 0.084 0.192
#> GSM228677 4 0.7871 -0.0255 0.000 0.032 0.224 0.408 0.164 0.172
#> GSM228678 4 0.5291 0.4461 0.000 0.076 0.000 0.676 0.184 0.064
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
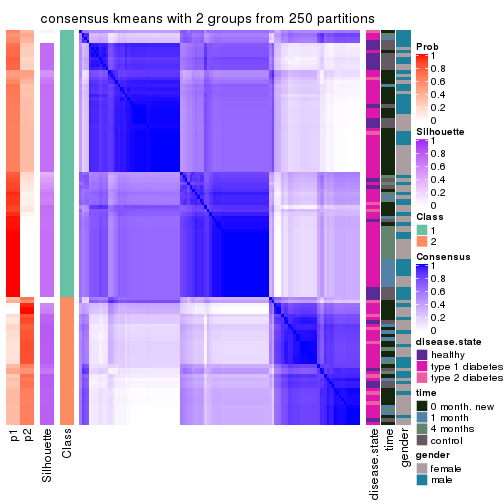
consensus_heatmap(res, k = 3)
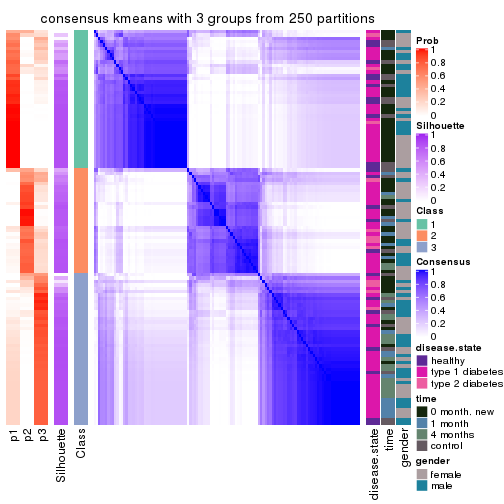
consensus_heatmap(res, k = 4)
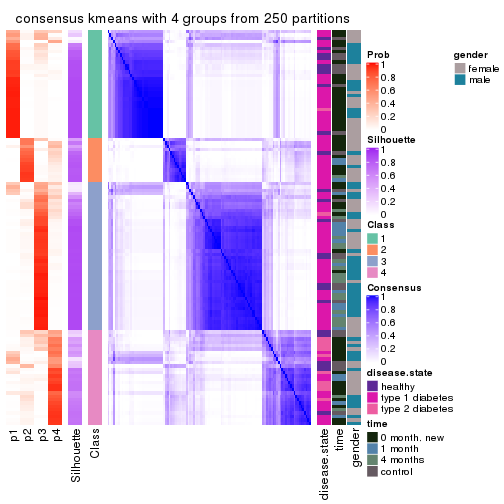
consensus_heatmap(res, k = 5)
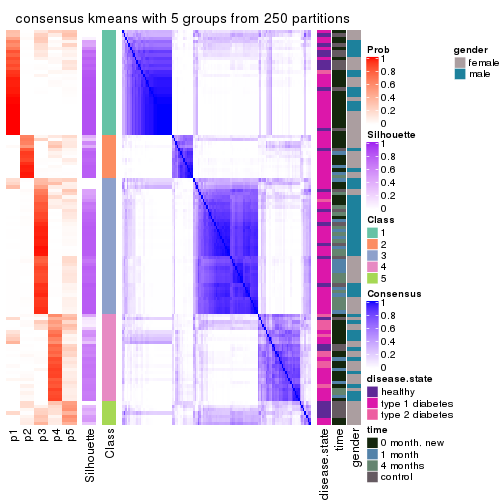
consensus_heatmap(res, k = 6)
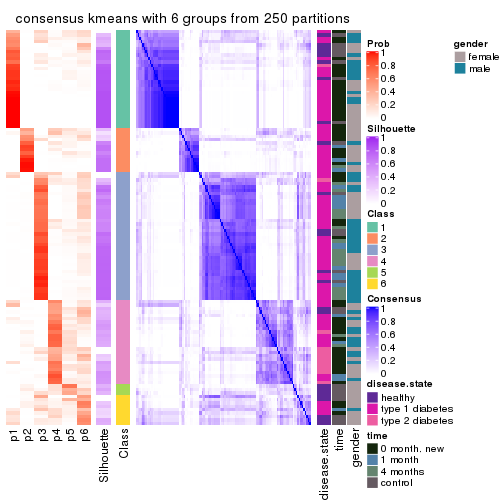
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
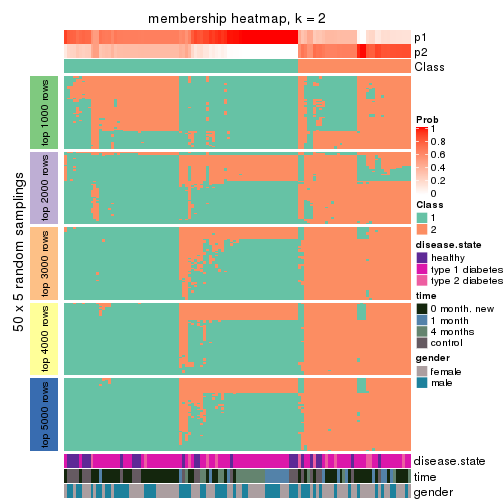
membership_heatmap(res, k = 3)
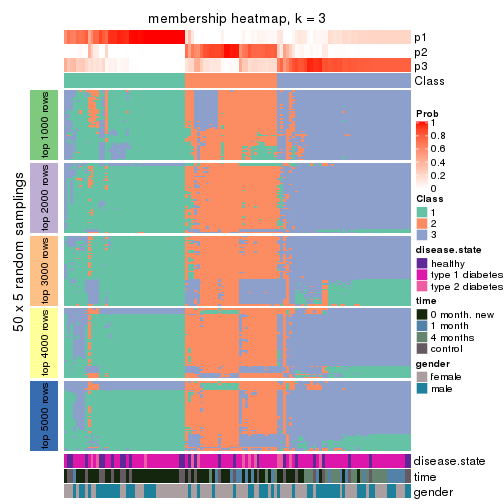
membership_heatmap(res, k = 4)
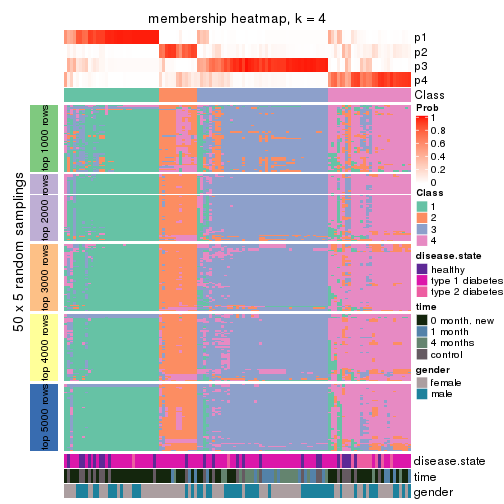
membership_heatmap(res, k = 5)
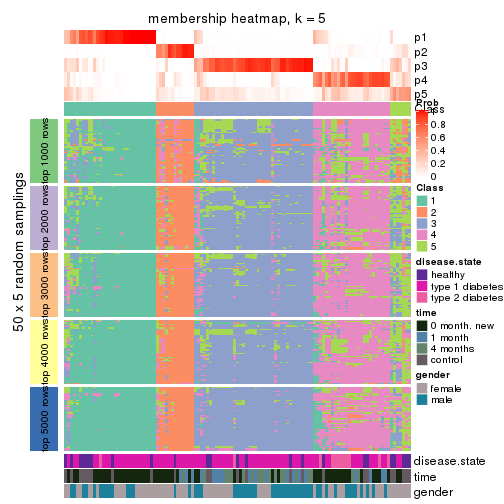
membership_heatmap(res, k = 6)
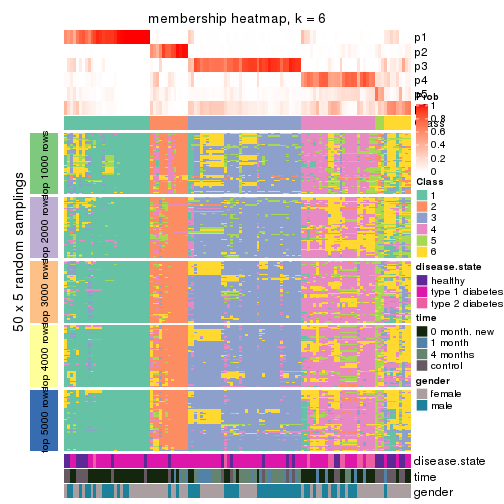
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
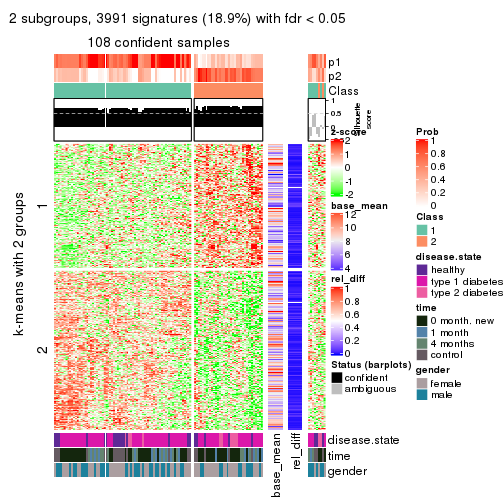
get_signatures(res, k = 3)
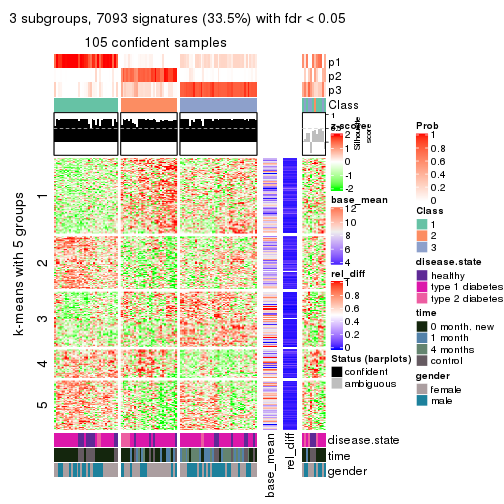
get_signatures(res, k = 4)
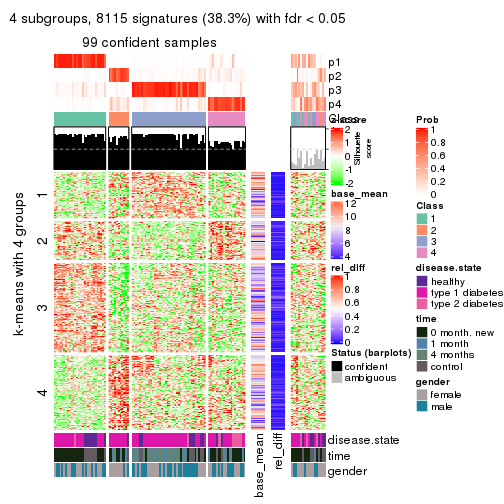
get_signatures(res, k = 5)
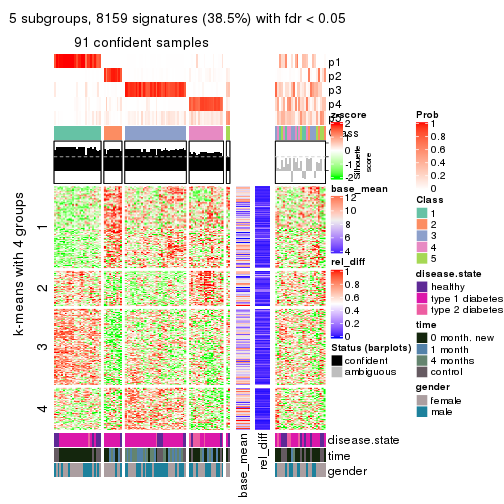
get_signatures(res, k = 6)
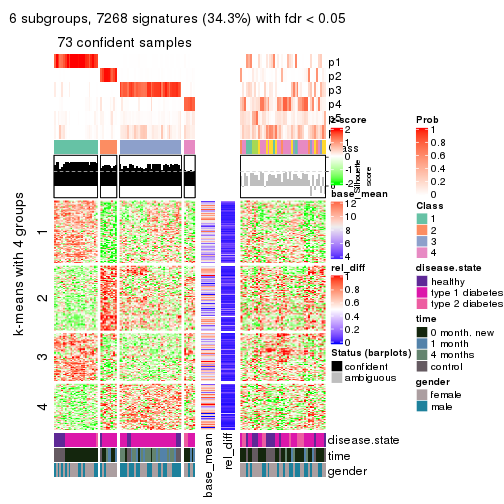
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
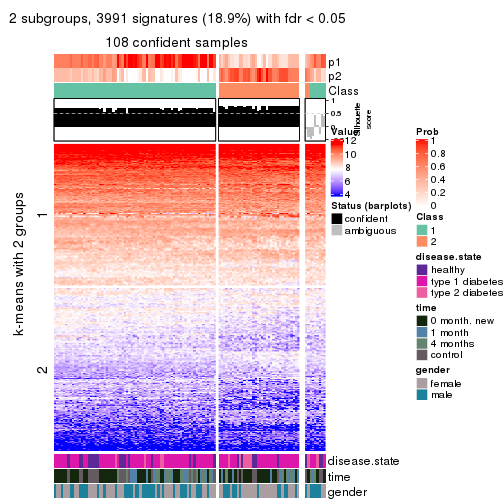
get_signatures(res, k = 3, scale_rows = FALSE)
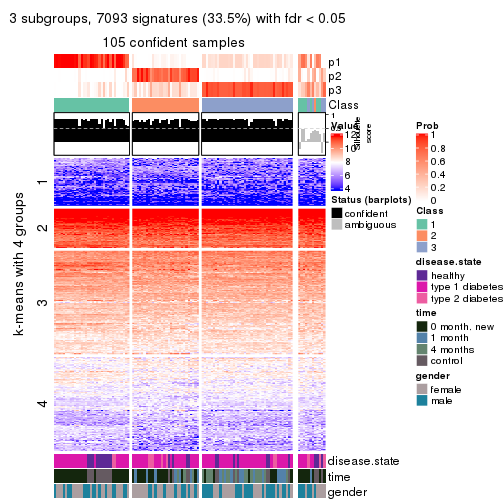
get_signatures(res, k = 4, scale_rows = FALSE)
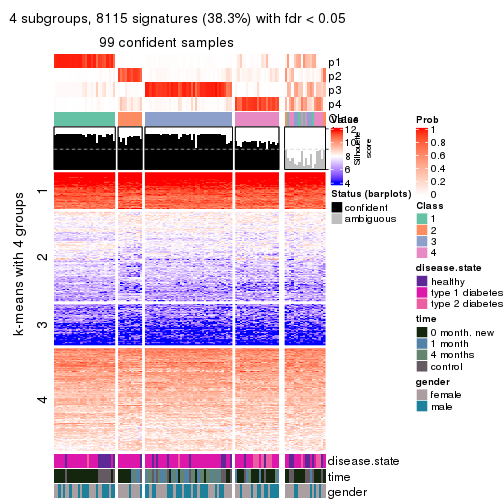
get_signatures(res, k = 5, scale_rows = FALSE)
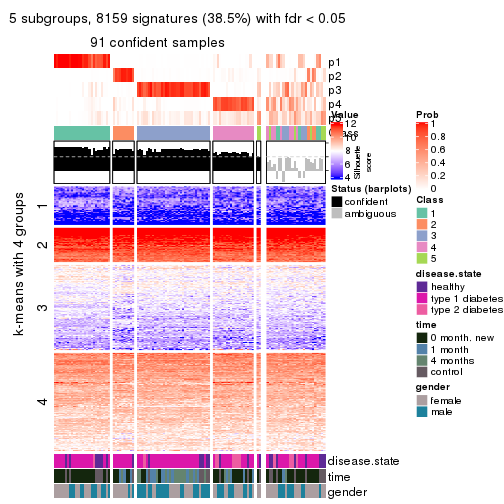
get_signatures(res, k = 6, scale_rows = FALSE)
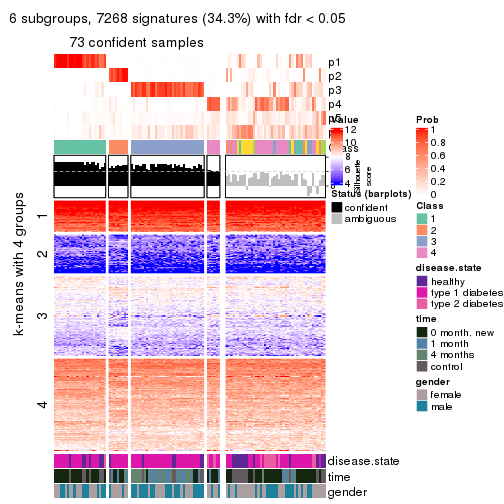
Compare the overlap of signatures from different k:
compare_signatures(res)
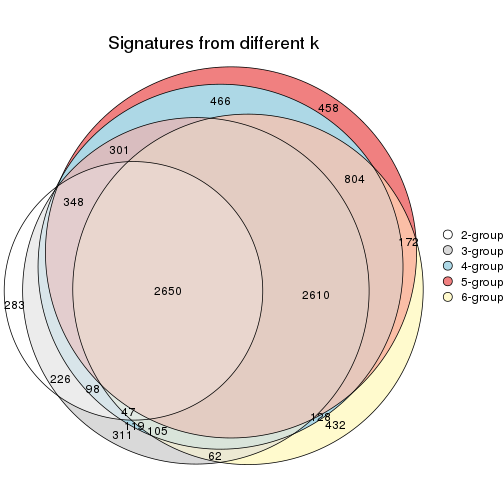
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
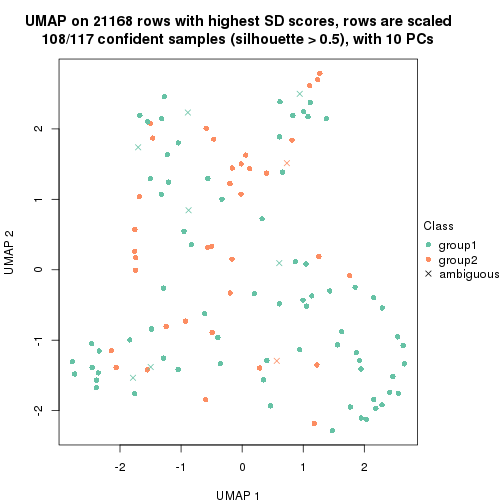
dimension_reduction(res, k = 3, method = "UMAP")
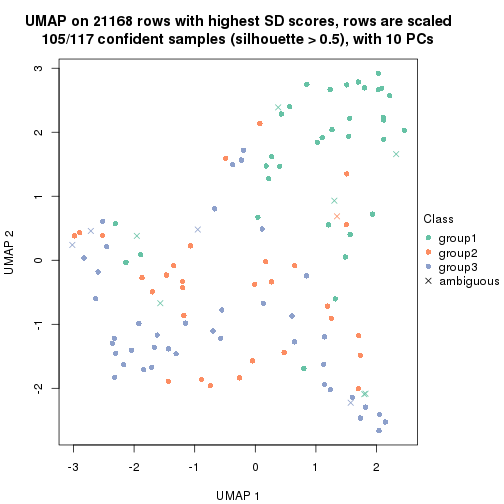
dimension_reduction(res, k = 4, method = "UMAP")
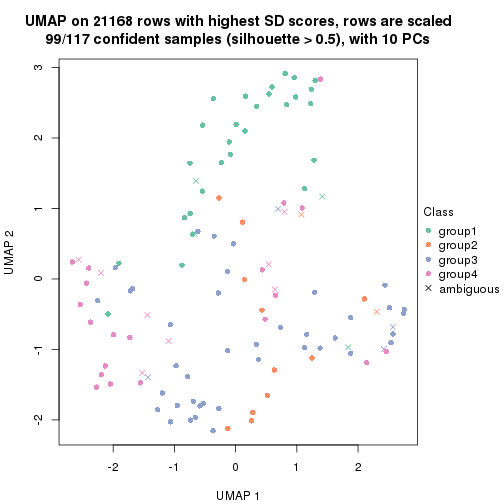
dimension_reduction(res, k = 5, method = "UMAP")
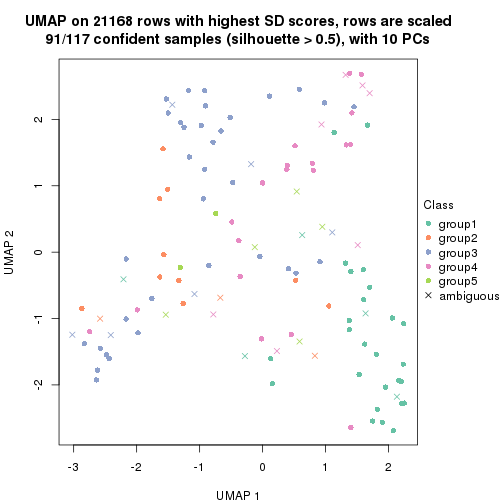
dimension_reduction(res, k = 6, method = "UMAP")
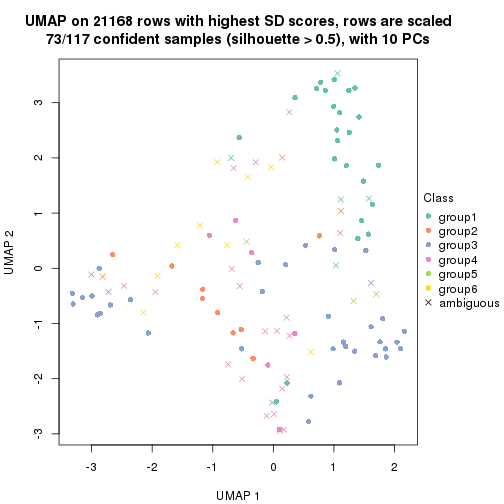
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
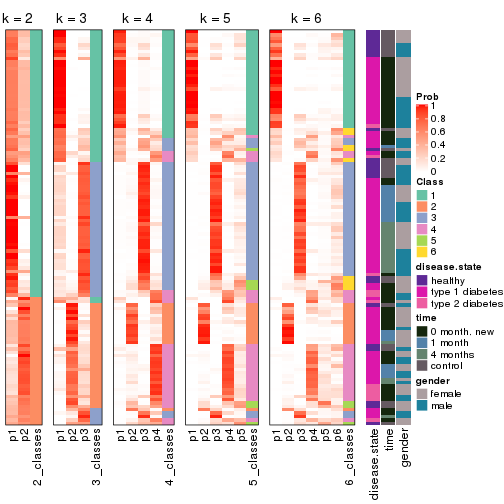
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> SD:kmeans 108 0.12983 4.83e-01 0.2422 2
#> SD:kmeans 105 0.19498 3.12e-07 0.1394 3
#> SD:kmeans 99 0.00071 7.47e-06 0.1905 4
#> SD:kmeans 91 0.01282 9.48e-06 0.0953 5
#> SD:kmeans 73 0.00891 3.80e-06 0.1176 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["SD", "skmeans"]
# you can also extract it by
# res = res_list["SD:skmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'SD' method.
#> Subgroups are detected by 'skmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
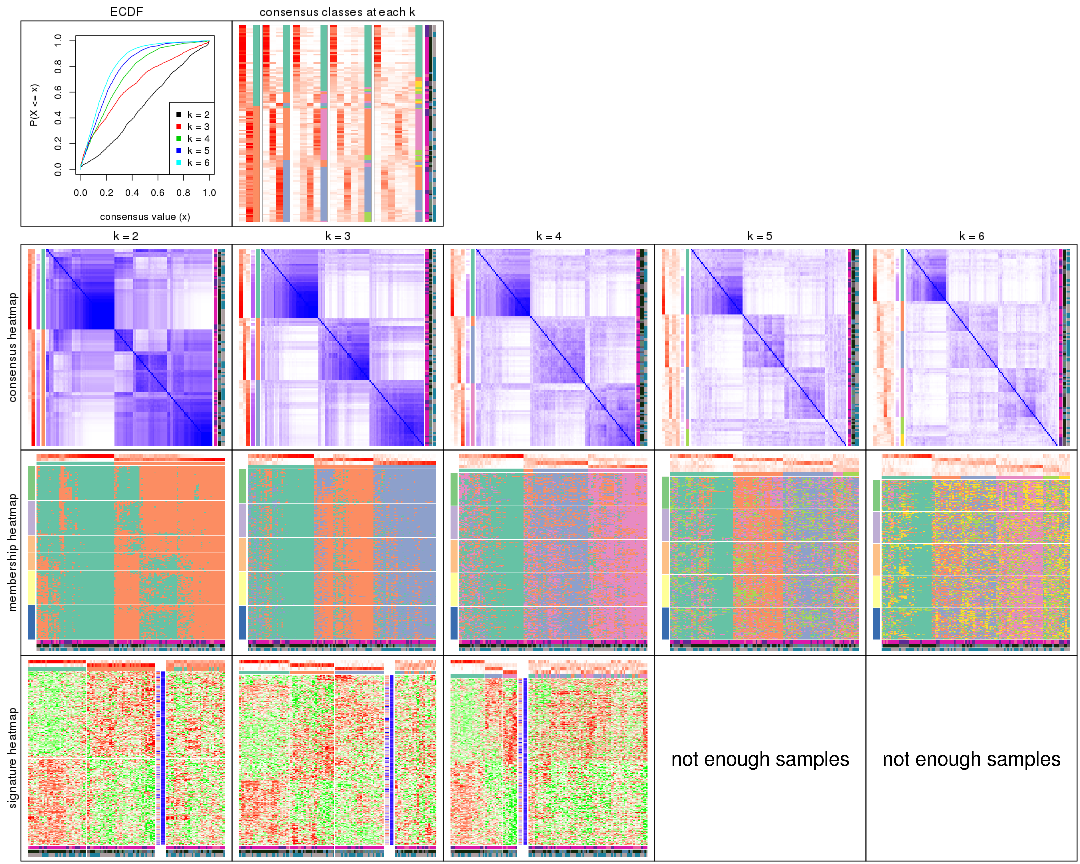
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
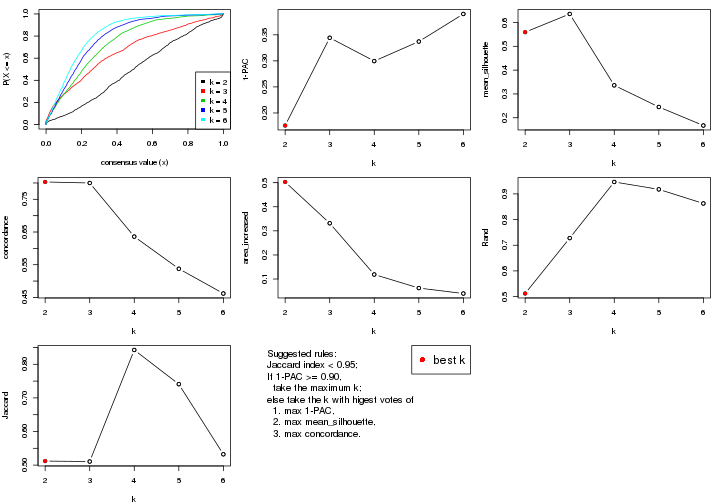
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.176 0.559 0.803 0.5029 0.512 0.512
#> 3 3 0.344 0.636 0.800 0.3315 0.728 0.511
#> 4 4 0.300 0.336 0.636 0.1188 0.947 0.843
#> 5 5 0.337 0.245 0.538 0.0625 0.918 0.741
#> 6 6 0.390 0.167 0.462 0.0399 0.863 0.532
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.6438 0.6971 0.836 0.164
#> GSM228563 2 0.9993 -0.0328 0.484 0.516
#> GSM228565 1 0.9393 0.3302 0.644 0.356
#> GSM228566 2 0.9000 0.5686 0.316 0.684
#> GSM228567 1 0.0000 0.7935 1.000 0.000
#> GSM228570 1 0.0000 0.7935 1.000 0.000
#> GSM228571 1 0.5059 0.6966 0.888 0.112
#> GSM228574 2 0.4690 0.6843 0.100 0.900
#> GSM228575 2 0.6887 0.6615 0.184 0.816
#> GSM228576 1 0.6343 0.6412 0.840 0.160
#> GSM228579 1 0.3879 0.7387 0.924 0.076
#> GSM228580 2 0.3431 0.6615 0.064 0.936
#> GSM228581 2 0.2236 0.6798 0.036 0.964
#> GSM228666 2 0.1184 0.6779 0.016 0.984
#> GSM228564 1 0.8909 0.4875 0.692 0.308
#> GSM228568 1 0.9248 0.3658 0.660 0.340
#> GSM228569 1 0.6712 0.5955 0.824 0.176
#> GSM228572 2 0.2948 0.6675 0.052 0.948
#> GSM228573 2 0.9686 0.4652 0.396 0.604
#> GSM228577 1 0.0000 0.7935 1.000 0.000
#> GSM228578 1 0.3274 0.7546 0.940 0.060
#> GSM228663 2 0.9358 0.5258 0.352 0.648
#> GSM228664 2 0.2043 0.6841 0.032 0.968
#> GSM228665 2 0.9833 0.4179 0.424 0.576
#> GSM228582 2 1.0000 0.1821 0.500 0.500
#> GSM228583 1 0.0000 0.7935 1.000 0.000
#> GSM228585 1 0.0000 0.7935 1.000 0.000
#> GSM228587 1 0.0376 0.7925 0.996 0.004
#> GSM228588 1 0.9754 0.2958 0.592 0.408
#> GSM228589 2 0.9896 0.0947 0.440 0.560
#> GSM228590 1 0.0000 0.7935 1.000 0.000
#> GSM228591 2 0.0938 0.6794 0.012 0.988
#> GSM228597 2 0.9881 0.1104 0.436 0.564
#> GSM228601 2 0.8661 0.4355 0.288 0.712
#> GSM228604 2 0.0000 0.6774 0.000 1.000
#> GSM228608 1 0.0000 0.7935 1.000 0.000
#> GSM228609 1 0.9393 0.4034 0.644 0.356
#> GSM228613 1 0.0000 0.7935 1.000 0.000
#> GSM228616 1 0.7674 0.6372 0.776 0.224
#> GSM228628 2 0.3879 0.6833 0.076 0.924
#> GSM228634 1 0.0000 0.7935 1.000 0.000
#> GSM228642 2 0.0000 0.6774 0.000 1.000
#> GSM228645 2 0.8608 0.5888 0.284 0.716
#> GSM228646 2 0.4939 0.6846 0.108 0.892
#> GSM228652 1 0.0000 0.7935 1.000 0.000
#> GSM228655 1 0.2043 0.7867 0.968 0.032
#> GSM228656 1 0.0000 0.7935 1.000 0.000
#> GSM228659 1 0.3879 0.7611 0.924 0.076
#> GSM228662 1 0.0000 0.7935 1.000 0.000
#> GSM228584 1 0.0000 0.7935 1.000 0.000
#> GSM228586 1 0.0000 0.7935 1.000 0.000
#> GSM228592 1 0.0000 0.7935 1.000 0.000
#> GSM228593 1 0.8955 0.4822 0.688 0.312
#> GSM228594 1 0.0376 0.7919 0.996 0.004
#> GSM228598 1 0.0000 0.7935 1.000 0.000
#> GSM228607 2 0.9323 0.4911 0.348 0.652
#> GSM228612 2 0.8207 0.6183 0.256 0.744
#> GSM228619 1 0.3879 0.7677 0.924 0.076
#> GSM228622 1 0.0938 0.7898 0.988 0.012
#> GSM228625 1 0.6531 0.6873 0.832 0.168
#> GSM228631 1 0.1633 0.7852 0.976 0.024
#> GSM228633 2 0.0000 0.6774 0.000 1.000
#> GSM228637 2 0.9993 -0.0425 0.484 0.516
#> GSM228639 2 0.6343 0.5979 0.160 0.840
#> GSM228649 1 0.9993 0.1071 0.516 0.484
#> GSM228660 1 0.7219 0.6609 0.800 0.200
#> GSM228661 1 0.4431 0.7238 0.908 0.092
#> GSM228595 2 0.0000 0.6774 0.000 1.000
#> GSM228599 2 0.9248 0.3563 0.340 0.660
#> GSM228602 2 0.9795 0.4316 0.416 0.584
#> GSM228614 2 0.9209 0.3650 0.336 0.664
#> GSM228626 2 0.0000 0.6774 0.000 1.000
#> GSM228640 2 0.9866 0.4020 0.432 0.568
#> GSM228643 2 0.8861 0.5783 0.304 0.696
#> GSM228650 2 0.5946 0.6743 0.144 0.856
#> GSM228653 2 0.9866 0.4001 0.432 0.568
#> GSM228657 2 0.2948 0.6662 0.052 0.948
#> GSM228605 1 0.6247 0.7122 0.844 0.156
#> GSM228610 2 0.7674 0.6370 0.224 0.776
#> GSM228617 2 0.9866 0.4074 0.432 0.568
#> GSM228620 1 0.9954 -0.1801 0.540 0.460
#> GSM228623 2 0.8955 0.3998 0.312 0.688
#> GSM228629 1 0.9996 -0.2543 0.512 0.488
#> GSM228632 2 0.0938 0.6809 0.012 0.988
#> GSM228635 2 0.9427 0.2984 0.360 0.640
#> GSM228647 2 0.9170 0.5484 0.332 0.668
#> GSM228596 2 0.9922 0.3326 0.448 0.552
#> GSM228600 2 0.8813 0.5796 0.300 0.700
#> GSM228603 2 0.9954 0.3470 0.460 0.540
#> GSM228615 2 0.9815 0.1549 0.420 0.580
#> GSM228627 2 0.9286 0.5344 0.344 0.656
#> GSM228641 2 0.8813 0.5784 0.300 0.700
#> GSM228644 2 0.0000 0.6774 0.000 1.000
#> GSM228651 2 0.9286 0.5318 0.344 0.656
#> GSM228654 2 0.8144 0.6178 0.252 0.748
#> GSM228658 2 0.9815 0.4239 0.420 0.580
#> GSM228606 2 0.2948 0.6857 0.052 0.948
#> GSM228611 2 0.9323 0.5294 0.348 0.652
#> GSM228618 2 0.9732 0.4487 0.404 0.596
#> GSM228621 2 0.5294 0.6807 0.120 0.880
#> GSM228624 2 0.5629 0.6783 0.132 0.868
#> GSM228630 2 0.0000 0.6774 0.000 1.000
#> GSM228636 2 0.9129 0.3640 0.328 0.672
#> GSM228638 2 0.5519 0.6790 0.128 0.872
#> GSM228648 2 0.0938 0.6807 0.012 0.988
#> GSM228670 2 0.9552 0.2802 0.376 0.624
#> GSM228671 2 0.0672 0.6783 0.008 0.992
#> GSM228672 1 0.8081 0.5826 0.752 0.248
#> GSM228674 1 0.9970 0.1416 0.532 0.468
#> GSM228675 2 0.9896 0.1023 0.440 0.560
#> GSM228676 1 1.0000 -0.0910 0.504 0.496
#> GSM228667 2 0.9087 0.4392 0.324 0.676
#> GSM228668 1 0.0938 0.7910 0.988 0.012
#> GSM228669 1 0.7219 0.6480 0.800 0.200
#> GSM228673 2 0.6247 0.6733 0.156 0.844
#> GSM228677 2 0.0000 0.6774 0.000 1.000
#> GSM228678 2 0.7219 0.5633 0.200 0.800
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.8048 0.54263 0.628 0.264 0.108
#> GSM228563 2 0.4295 0.73928 0.104 0.864 0.032
#> GSM228565 1 0.9489 0.34645 0.496 0.252 0.252
#> GSM228566 3 0.7147 0.69704 0.156 0.124 0.720
#> GSM228567 1 0.0000 0.79027 1.000 0.000 0.000
#> GSM228570 1 0.1999 0.78877 0.952 0.012 0.036
#> GSM228571 1 0.2955 0.77780 0.912 0.008 0.080
#> GSM228574 3 0.5435 0.73785 0.024 0.192 0.784
#> GSM228575 3 0.9059 0.20487 0.140 0.380 0.480
#> GSM228576 1 0.7970 0.50077 0.612 0.088 0.300
#> GSM228579 1 0.1999 0.78646 0.952 0.012 0.036
#> GSM228580 2 0.2165 0.75259 0.000 0.936 0.064
#> GSM228581 2 0.8270 0.24914 0.084 0.540 0.376
#> GSM228666 2 0.5541 0.61390 0.008 0.740 0.252
#> GSM228564 2 0.7996 -0.05105 0.464 0.476 0.060
#> GSM228568 1 0.9379 0.34772 0.508 0.216 0.276
#> GSM228569 1 0.4842 0.66671 0.776 0.000 0.224
#> GSM228572 2 0.0829 0.75397 0.004 0.984 0.012
#> GSM228573 3 0.2496 0.80412 0.068 0.004 0.928
#> GSM228577 1 0.0592 0.79109 0.988 0.000 0.012
#> GSM228578 1 0.4963 0.70334 0.792 0.008 0.200
#> GSM228663 3 0.3009 0.80984 0.052 0.028 0.920
#> GSM228664 3 0.4178 0.75989 0.000 0.172 0.828
#> GSM228665 3 0.4930 0.78090 0.120 0.044 0.836
#> GSM228582 1 0.9054 0.07186 0.460 0.136 0.404
#> GSM228583 1 0.0000 0.79027 1.000 0.000 0.000
#> GSM228585 1 0.0000 0.79027 1.000 0.000 0.000
#> GSM228587 1 0.0747 0.78836 0.984 0.016 0.000
#> GSM228588 2 0.4912 0.67010 0.196 0.796 0.008
#> GSM228589 2 0.1015 0.75550 0.012 0.980 0.008
#> GSM228590 1 0.0237 0.79007 0.996 0.004 0.000
#> GSM228591 2 0.5698 0.60483 0.012 0.736 0.252
#> GSM228597 2 0.1620 0.75595 0.024 0.964 0.012
#> GSM228601 2 0.0424 0.75261 0.000 0.992 0.008
#> GSM228604 2 0.6307 -0.00274 0.000 0.512 0.488
#> GSM228608 1 0.0424 0.79113 0.992 0.000 0.008
#> GSM228609 2 0.6448 0.39149 0.352 0.636 0.012
#> GSM228613 1 0.0000 0.79027 1.000 0.000 0.000
#> GSM228616 1 0.9353 0.31196 0.504 0.296 0.200
#> GSM228628 2 0.8089 0.47820 0.092 0.600 0.308
#> GSM228634 1 0.0424 0.79082 0.992 0.000 0.008
#> GSM228642 2 0.5650 0.51371 0.000 0.688 0.312
#> GSM228645 3 0.9498 0.30223 0.216 0.300 0.484
#> GSM228646 3 0.8737 0.34963 0.124 0.340 0.536
#> GSM228652 1 0.2152 0.78811 0.948 0.016 0.036
#> GSM228655 1 0.6986 0.68366 0.724 0.096 0.180
#> GSM228656 1 0.0000 0.79027 1.000 0.000 0.000
#> GSM228659 1 0.4912 0.68011 0.796 0.196 0.008
#> GSM228662 1 0.0237 0.79007 0.996 0.004 0.000
#> GSM228584 1 0.0000 0.79027 1.000 0.000 0.000
#> GSM228586 1 0.0237 0.79062 0.996 0.000 0.004
#> GSM228592 1 0.0000 0.79027 1.000 0.000 0.000
#> GSM228593 1 0.6763 0.21153 0.552 0.436 0.012
#> GSM228594 1 0.0424 0.79082 0.992 0.000 0.008
#> GSM228598 1 0.0475 0.78980 0.992 0.004 0.004
#> GSM228607 3 0.9347 0.34315 0.204 0.288 0.508
#> GSM228612 3 0.6119 0.74392 0.064 0.164 0.772
#> GSM228619 1 0.9150 0.45655 0.544 0.224 0.232
#> GSM228622 1 0.5406 0.68157 0.764 0.012 0.224
#> GSM228625 1 0.8637 0.04403 0.452 0.448 0.100
#> GSM228631 1 0.7308 0.57140 0.656 0.060 0.284
#> GSM228633 2 0.2959 0.72597 0.000 0.900 0.100
#> GSM228637 2 0.1753 0.75350 0.048 0.952 0.000
#> GSM228639 2 0.7278 0.06105 0.028 0.516 0.456
#> GSM228649 2 0.5940 0.64738 0.204 0.760 0.036
#> GSM228660 1 0.8153 0.55620 0.632 0.240 0.128
#> GSM228661 1 0.4121 0.72524 0.832 0.000 0.168
#> GSM228595 2 0.0592 0.75269 0.000 0.988 0.012
#> GSM228599 2 0.4551 0.72559 0.020 0.840 0.140
#> GSM228602 3 0.3337 0.80789 0.060 0.032 0.908
#> GSM228614 2 0.5524 0.70932 0.040 0.796 0.164
#> GSM228626 2 0.4062 0.68256 0.000 0.836 0.164
#> GSM228640 3 0.1711 0.80325 0.032 0.008 0.960
#> GSM228643 3 0.5212 0.78816 0.064 0.108 0.828
#> GSM228650 3 0.4733 0.74101 0.004 0.196 0.800
#> GSM228653 3 0.1031 0.79888 0.024 0.000 0.976
#> GSM228657 2 0.1163 0.75553 0.000 0.972 0.028
#> GSM228605 1 0.9326 0.40177 0.512 0.204 0.284
#> GSM228610 3 0.3722 0.80579 0.024 0.088 0.888
#> GSM228617 3 0.5696 0.74830 0.136 0.064 0.800
#> GSM228620 3 0.4409 0.74239 0.172 0.004 0.824
#> GSM228623 2 0.3045 0.75814 0.020 0.916 0.064
#> GSM228629 3 0.2860 0.80040 0.084 0.004 0.912
#> GSM228632 3 0.5737 0.67113 0.012 0.256 0.732
#> GSM228635 2 0.0829 0.75458 0.012 0.984 0.004
#> GSM228647 3 0.3028 0.81223 0.048 0.032 0.920
#> GSM228596 3 0.9532 0.30231 0.244 0.268 0.488
#> GSM228600 3 0.3045 0.80807 0.020 0.064 0.916
#> GSM228603 3 0.1163 0.79979 0.028 0.000 0.972
#> GSM228615 2 0.1774 0.75799 0.024 0.960 0.016
#> GSM228627 3 0.2902 0.80796 0.064 0.016 0.920
#> GSM228641 3 0.1774 0.80284 0.016 0.024 0.960
#> GSM228644 2 0.3340 0.71718 0.000 0.880 0.120
#> GSM228651 3 0.1774 0.80234 0.016 0.024 0.960
#> GSM228654 3 0.2448 0.80160 0.000 0.076 0.924
#> GSM228658 3 0.1832 0.80440 0.036 0.008 0.956
#> GSM228606 3 0.7742 0.39361 0.060 0.356 0.584
#> GSM228611 3 0.3683 0.81015 0.060 0.044 0.896
#> GSM228618 3 0.1585 0.80288 0.028 0.008 0.964
#> GSM228621 3 0.3112 0.79835 0.004 0.096 0.900
#> GSM228624 3 0.6437 0.70589 0.048 0.220 0.732
#> GSM228630 3 0.5948 0.49078 0.000 0.360 0.640
#> GSM228636 2 0.0829 0.75451 0.012 0.984 0.004
#> GSM228638 3 0.3983 0.78402 0.004 0.144 0.852
#> GSM228648 3 0.3551 0.78511 0.000 0.132 0.868
#> GSM228670 2 0.7615 0.63254 0.164 0.688 0.148
#> GSM228671 2 0.6527 0.32634 0.008 0.588 0.404
#> GSM228672 1 0.7192 0.29423 0.560 0.412 0.028
#> GSM228674 2 0.6803 0.52409 0.280 0.680 0.040
#> GSM228675 2 0.4818 0.73625 0.108 0.844 0.048
#> GSM228676 2 0.9959 0.12407 0.324 0.376 0.300
#> GSM228667 2 0.9641 0.31364 0.296 0.464 0.240
#> GSM228668 1 0.3141 0.78031 0.912 0.020 0.068
#> GSM228669 1 0.6282 0.51724 0.664 0.324 0.012
#> GSM228673 3 0.8013 0.46667 0.080 0.332 0.588
#> GSM228677 2 0.5465 0.55797 0.000 0.712 0.288
#> GSM228678 2 0.0424 0.75396 0.000 0.992 0.008
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 1 0.9289 -0.0526 0.420 0.264 0.116 0.200
#> GSM228563 4 0.6673 0.3970 0.100 0.216 0.024 0.660
#> GSM228565 1 0.9658 -0.2999 0.328 0.300 0.236 0.136
#> GSM228566 3 0.8115 0.2414 0.072 0.344 0.492 0.092
#> GSM228567 1 0.0469 0.6495 0.988 0.012 0.000 0.000
#> GSM228570 1 0.4969 0.6090 0.792 0.140 0.040 0.028
#> GSM228571 1 0.5293 0.5746 0.748 0.152 0.100 0.000
#> GSM228574 3 0.7740 0.2112 0.012 0.312 0.496 0.180
#> GSM228575 2 0.9233 0.2323 0.080 0.352 0.320 0.248
#> GSM228576 1 0.8952 -0.0561 0.416 0.252 0.268 0.064
#> GSM228579 1 0.4462 0.6055 0.792 0.164 0.044 0.000
#> GSM228580 4 0.5536 0.4702 0.000 0.180 0.096 0.724
#> GSM228581 4 0.8808 -0.1028 0.052 0.300 0.236 0.412
#> GSM228666 4 0.7347 0.2269 0.016 0.368 0.108 0.508
#> GSM228564 4 0.8563 -0.1128 0.316 0.216 0.040 0.428
#> GSM228568 1 0.9404 -0.1034 0.404 0.232 0.244 0.120
#> GSM228569 1 0.7082 0.3230 0.564 0.184 0.252 0.000
#> GSM228572 4 0.4514 0.5076 0.000 0.148 0.056 0.796
#> GSM228573 3 0.5916 0.4967 0.056 0.248 0.684 0.012
#> GSM228577 1 0.2861 0.6436 0.888 0.096 0.016 0.000
#> GSM228578 1 0.7316 0.4352 0.608 0.196 0.172 0.024
#> GSM228663 3 0.5837 0.5232 0.032 0.196 0.724 0.048
#> GSM228664 3 0.7565 0.2390 0.000 0.312 0.472 0.216
#> GSM228665 3 0.7644 0.3876 0.120 0.200 0.612 0.068
#> GSM228582 1 0.9657 -0.3663 0.316 0.272 0.284 0.128
#> GSM228583 1 0.0000 0.6483 1.000 0.000 0.000 0.000
#> GSM228585 1 0.0707 0.6494 0.980 0.020 0.000 0.000
#> GSM228587 1 0.3424 0.6306 0.876 0.052 0.004 0.068
#> GSM228588 4 0.5988 0.3191 0.224 0.100 0.000 0.676
#> GSM228589 4 0.3988 0.5132 0.020 0.156 0.004 0.820
#> GSM228590 1 0.0844 0.6496 0.980 0.004 0.004 0.012
#> GSM228591 4 0.7468 0.2206 0.008 0.312 0.160 0.520
#> GSM228597 4 0.4205 0.4925 0.008 0.172 0.016 0.804
#> GSM228601 4 0.2469 0.5141 0.000 0.108 0.000 0.892
#> GSM228604 4 0.7870 -0.2232 0.000 0.276 0.360 0.364
#> GSM228608 1 0.3538 0.6410 0.880 0.060 0.036 0.024
#> GSM228609 4 0.7155 0.1014 0.312 0.140 0.004 0.544
#> GSM228613 1 0.0524 0.6493 0.988 0.008 0.000 0.004
#> GSM228616 1 0.9295 -0.0654 0.420 0.268 0.120 0.192
#> GSM228628 4 0.8337 -0.0222 0.036 0.384 0.172 0.408
#> GSM228634 1 0.0895 0.6502 0.976 0.020 0.004 0.000
#> GSM228642 4 0.7309 0.1970 0.000 0.324 0.172 0.504
#> GSM228645 2 0.9555 0.2600 0.136 0.360 0.300 0.204
#> GSM228646 2 0.9028 0.2342 0.060 0.372 0.288 0.280
#> GSM228652 1 0.5509 0.5994 0.776 0.112 0.064 0.048
#> GSM228655 1 0.8844 0.2440 0.508 0.136 0.204 0.152
#> GSM228656 1 0.0895 0.6502 0.976 0.020 0.000 0.004
#> GSM228659 1 0.7156 0.3491 0.576 0.148 0.008 0.268
#> GSM228662 1 0.0524 0.6491 0.988 0.008 0.000 0.004
#> GSM228584 1 0.0188 0.6485 0.996 0.004 0.000 0.000
#> GSM228586 1 0.0817 0.6498 0.976 0.024 0.000 0.000
#> GSM228592 1 0.0188 0.6485 0.996 0.004 0.000 0.000
#> GSM228593 1 0.7332 0.1229 0.484 0.140 0.004 0.372
#> GSM228594 1 0.2845 0.6437 0.896 0.076 0.028 0.000
#> GSM228598 1 0.2561 0.6480 0.912 0.068 0.004 0.016
#> GSM228607 3 0.9550 -0.1791 0.120 0.304 0.340 0.236
#> GSM228612 3 0.8535 0.2530 0.080 0.332 0.464 0.124
#> GSM228619 1 0.9480 -0.0384 0.424 0.188 0.196 0.192
#> GSM228622 1 0.7273 0.3819 0.604 0.140 0.232 0.024
#> GSM228625 4 0.8808 -0.1137 0.340 0.220 0.052 0.388
#> GSM228631 1 0.8569 0.2093 0.488 0.168 0.276 0.068
#> GSM228633 4 0.4920 0.4690 0.000 0.192 0.052 0.756
#> GSM228637 4 0.4527 0.4961 0.032 0.144 0.016 0.808
#> GSM228639 4 0.8721 -0.1604 0.040 0.256 0.316 0.388
#> GSM228649 4 0.7126 0.3477 0.140 0.172 0.040 0.648
#> GSM228660 1 0.9531 -0.0797 0.400 0.204 0.152 0.244
#> GSM228661 1 0.5464 0.5099 0.716 0.072 0.212 0.000
#> GSM228595 4 0.3447 0.5065 0.000 0.128 0.020 0.852
#> GSM228599 4 0.7727 0.2480 0.036 0.216 0.172 0.576
#> GSM228602 3 0.7118 0.4193 0.076 0.256 0.620 0.048
#> GSM228614 4 0.7404 0.3442 0.040 0.256 0.108 0.596
#> GSM228626 4 0.6383 0.3476 0.000 0.292 0.096 0.612
#> GSM228640 3 0.4929 0.4938 0.024 0.224 0.744 0.008
#> GSM228643 3 0.7421 0.3004 0.072 0.328 0.552 0.048
#> GSM228650 3 0.7559 0.2386 0.008 0.280 0.524 0.188
#> GSM228653 3 0.3335 0.5469 0.016 0.128 0.856 0.000
#> GSM228657 4 0.4638 0.5124 0.000 0.180 0.044 0.776
#> GSM228605 1 0.9509 -0.1547 0.396 0.252 0.212 0.140
#> GSM228610 3 0.6112 0.5122 0.008 0.264 0.660 0.068
#> GSM228617 3 0.7751 0.3618 0.116 0.204 0.604 0.076
#> GSM228620 3 0.6828 0.3645 0.180 0.196 0.620 0.004
#> GSM228623 4 0.6385 0.4487 0.024 0.224 0.076 0.676
#> GSM228629 3 0.6090 0.4846 0.092 0.212 0.688 0.008
#> GSM228632 3 0.7886 0.1730 0.008 0.288 0.468 0.236
#> GSM228635 4 0.2859 0.5130 0.000 0.112 0.008 0.880
#> GSM228647 3 0.5712 0.5374 0.032 0.200 0.728 0.040
#> GSM228596 2 0.9758 0.2561 0.152 0.308 0.300 0.240
#> GSM228600 3 0.5833 0.4946 0.008 0.264 0.676 0.052
#> GSM228603 3 0.3790 0.5180 0.016 0.164 0.820 0.000
#> GSM228615 4 0.3408 0.5080 0.016 0.120 0.004 0.860
#> GSM228627 3 0.6725 0.4761 0.052 0.296 0.616 0.036
#> GSM228641 3 0.4799 0.5239 0.000 0.224 0.744 0.032
#> GSM228644 4 0.5879 0.4030 0.000 0.248 0.080 0.672
#> GSM228651 3 0.5342 0.5245 0.012 0.236 0.720 0.032
#> GSM228654 3 0.5809 0.5141 0.004 0.232 0.692 0.072
#> GSM228658 3 0.5252 0.5426 0.040 0.164 0.768 0.028
#> GSM228606 4 0.8795 -0.2204 0.044 0.256 0.324 0.376
#> GSM228611 3 0.6113 0.5238 0.060 0.212 0.700 0.028
#> GSM228618 3 0.4692 0.5413 0.012 0.196 0.772 0.020
#> GSM228621 3 0.6344 0.4906 0.000 0.224 0.648 0.128
#> GSM228624 3 0.7930 0.2786 0.028 0.304 0.508 0.160
#> GSM228630 3 0.7660 0.0858 0.000 0.228 0.448 0.324
#> GSM228636 4 0.1867 0.5088 0.000 0.072 0.000 0.928
#> GSM228638 3 0.7402 0.3818 0.016 0.240 0.576 0.168
#> GSM228648 3 0.6879 0.4093 0.000 0.216 0.596 0.188
#> GSM228670 4 0.7491 0.3416 0.064 0.244 0.088 0.604
#> GSM228671 4 0.8434 -0.1306 0.024 0.356 0.244 0.376
#> GSM228672 1 0.8536 -0.1079 0.384 0.196 0.040 0.380
#> GSM228674 4 0.8416 0.1431 0.176 0.268 0.056 0.500
#> GSM228675 4 0.6872 0.3385 0.072 0.312 0.024 0.592
#> GSM228676 2 0.9874 0.3713 0.284 0.308 0.204 0.204
#> GSM228667 2 0.9343 0.2299 0.220 0.364 0.100 0.316
#> GSM228668 1 0.6180 0.5682 0.736 0.124 0.072 0.068
#> GSM228669 1 0.7955 0.1631 0.504 0.112 0.048 0.336
#> GSM228673 3 0.8637 0.0862 0.060 0.356 0.420 0.164
#> GSM228677 4 0.7301 0.1586 0.000 0.236 0.228 0.536
#> GSM228678 4 0.4467 0.5108 0.000 0.172 0.040 0.788
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 1 0.9182 -0.18707 0.372 0.112 0.104 0.272 0.140
#> GSM228563 2 0.7616 0.23934 0.080 0.472 0.020 0.332 0.096
#> GSM228565 1 0.9375 -0.18075 0.356 0.124 0.104 0.244 0.172
#> GSM228566 3 0.8765 0.17429 0.072 0.096 0.444 0.204 0.184
#> GSM228567 1 0.0854 0.56897 0.976 0.000 0.008 0.012 0.004
#> GSM228570 1 0.6551 0.44099 0.648 0.024 0.056 0.188 0.084
#> GSM228571 1 0.6609 0.43708 0.640 0.008 0.072 0.148 0.132
#> GSM228574 3 0.8222 0.00844 0.012 0.156 0.372 0.112 0.348
#> GSM228575 3 0.9192 -0.18777 0.032 0.216 0.268 0.224 0.260
#> GSM228576 1 0.9441 -0.17828 0.328 0.076 0.184 0.236 0.176
#> GSM228579 1 0.6593 0.45102 0.652 0.020 0.056 0.140 0.132
#> GSM228580 2 0.6812 0.38469 0.004 0.612 0.080 0.164 0.140
#> GSM228581 5 0.8926 0.21203 0.040 0.312 0.172 0.140 0.336
#> GSM228666 2 0.8000 0.13597 0.004 0.452 0.116 0.240 0.188
#> GSM228564 4 0.8994 0.26644 0.272 0.236 0.048 0.336 0.108
#> GSM228568 5 0.9478 -0.18127 0.284 0.104 0.120 0.192 0.300
#> GSM228569 1 0.6609 0.40419 0.632 0.004 0.116 0.080 0.168
#> GSM228572 2 0.5291 0.46602 0.000 0.740 0.060 0.092 0.108
#> GSM228573 3 0.7224 0.35248 0.068 0.020 0.556 0.096 0.260
#> GSM228577 1 0.4552 0.53883 0.780 0.000 0.020 0.100 0.100
#> GSM228578 1 0.7879 0.27695 0.532 0.024 0.116 0.160 0.168
#> GSM228663 3 0.7518 0.30203 0.048 0.056 0.496 0.072 0.328
#> GSM228664 5 0.7878 0.03924 0.004 0.212 0.336 0.068 0.380
#> GSM228665 3 0.8940 0.12216 0.156 0.056 0.388 0.132 0.268
#> GSM228582 1 0.9880 -0.28010 0.272 0.180 0.156 0.180 0.212
#> GSM228583 1 0.1845 0.56902 0.928 0.000 0.000 0.056 0.016
#> GSM228585 1 0.1116 0.56850 0.964 0.000 0.004 0.028 0.004
#> GSM228587 1 0.3959 0.52843 0.808 0.028 0.000 0.140 0.024
#> GSM228588 2 0.6835 0.31169 0.156 0.568 0.000 0.224 0.052
#> GSM228589 2 0.4905 0.48963 0.016 0.736 0.004 0.188 0.056
#> GSM228590 1 0.2285 0.56547 0.916 0.004 0.004 0.052 0.024
#> GSM228591 2 0.7241 0.23504 0.016 0.596 0.108 0.136 0.144
#> GSM228597 2 0.6636 0.43146 0.016 0.572 0.024 0.284 0.104
#> GSM228601 2 0.3476 0.49524 0.000 0.804 0.000 0.176 0.020
#> GSM228604 2 0.7822 -0.14239 0.000 0.416 0.316 0.100 0.168
#> GSM228608 1 0.5349 0.52259 0.744 0.012 0.040 0.128 0.076
#> GSM228609 2 0.7568 -0.00874 0.204 0.384 0.008 0.368 0.036
#> GSM228613 1 0.0880 0.56721 0.968 0.000 0.000 0.032 0.000
#> GSM228616 1 0.9735 -0.28255 0.304 0.188 0.128 0.216 0.164
#> GSM228628 2 0.8403 0.12792 0.044 0.484 0.116 0.168 0.188
#> GSM228634 1 0.1949 0.56988 0.932 0.000 0.012 0.040 0.016
#> GSM228642 2 0.6841 0.23102 0.000 0.596 0.132 0.088 0.184
#> GSM228645 4 0.9705 -0.05137 0.100 0.172 0.232 0.268 0.228
#> GSM228646 3 0.9177 -0.17417 0.032 0.264 0.276 0.212 0.216
#> GSM228652 1 0.6673 0.42837 0.632 0.028 0.056 0.208 0.076
#> GSM228655 1 0.8684 0.04711 0.428 0.060 0.192 0.236 0.084
#> GSM228656 1 0.0865 0.56852 0.972 0.000 0.000 0.024 0.004
#> GSM228659 1 0.7327 0.15727 0.492 0.124 0.008 0.316 0.060
#> GSM228662 1 0.0992 0.56778 0.968 0.000 0.000 0.024 0.008
#> GSM228584 1 0.0865 0.56760 0.972 0.000 0.000 0.024 0.004
#> GSM228586 1 0.1186 0.56975 0.964 0.000 0.008 0.020 0.008
#> GSM228592 1 0.1041 0.56843 0.964 0.000 0.000 0.032 0.004
#> GSM228593 1 0.8323 -0.24552 0.348 0.264 0.012 0.292 0.084
#> GSM228594 1 0.3466 0.55884 0.856 0.000 0.024 0.048 0.072
#> GSM228598 1 0.4779 0.52925 0.772 0.016 0.012 0.132 0.068
#> GSM228607 5 0.9454 0.14800 0.072 0.180 0.188 0.248 0.312
#> GSM228612 3 0.8984 0.03958 0.080 0.160 0.356 0.092 0.312
#> GSM228619 1 0.9161 -0.11094 0.364 0.084 0.256 0.196 0.100
#> GSM228622 1 0.8080 0.12857 0.464 0.008 0.228 0.160 0.140
#> GSM228625 1 0.8886 -0.30640 0.288 0.272 0.028 0.288 0.124
#> GSM228631 1 0.8827 -0.03857 0.376 0.048 0.284 0.188 0.104
#> GSM228633 2 0.4639 0.42219 0.000 0.768 0.040 0.040 0.152
#> GSM228637 2 0.6614 0.37902 0.028 0.516 0.012 0.364 0.080
#> GSM228639 5 0.8838 0.19496 0.016 0.256 0.200 0.196 0.332
#> GSM228649 2 0.7988 0.23966 0.076 0.412 0.024 0.356 0.132
#> GSM228660 1 0.9206 -0.15415 0.368 0.200 0.060 0.208 0.164
#> GSM228661 1 0.6104 0.44843 0.672 0.000 0.136 0.072 0.120
#> GSM228595 2 0.3340 0.46007 0.000 0.856 0.012 0.044 0.088
#> GSM228599 2 0.8412 0.17418 0.020 0.364 0.184 0.332 0.100
#> GSM228602 3 0.7432 0.34717 0.040 0.056 0.576 0.136 0.192
#> GSM228614 2 0.8765 0.10958 0.032 0.332 0.116 0.324 0.196
#> GSM228626 2 0.5173 0.34131 0.000 0.724 0.068 0.032 0.176
#> GSM228640 3 0.4951 0.43776 0.008 0.012 0.752 0.092 0.136
#> GSM228643 3 0.8012 0.27087 0.052 0.068 0.492 0.116 0.272
#> GSM228650 3 0.8109 0.13923 0.024 0.184 0.464 0.080 0.248
#> GSM228653 3 0.4828 0.43852 0.044 0.000 0.752 0.040 0.164
#> GSM228657 2 0.5450 0.44595 0.000 0.728 0.064 0.096 0.112
#> GSM228605 1 0.9721 -0.32252 0.268 0.100 0.184 0.232 0.216
#> GSM228610 3 0.7148 0.33321 0.024 0.068 0.568 0.084 0.256
#> GSM228617 3 0.7713 0.29730 0.080 0.060 0.572 0.112 0.176
#> GSM228620 3 0.7678 0.29830 0.144 0.032 0.540 0.068 0.216
#> GSM228623 2 0.7813 0.34858 0.012 0.460 0.060 0.252 0.216
#> GSM228629 3 0.6489 0.40697 0.088 0.008 0.640 0.072 0.192
#> GSM228632 5 0.8146 0.17416 0.000 0.252 0.300 0.104 0.344
#> GSM228635 2 0.5913 0.44476 0.000 0.616 0.016 0.264 0.104
#> GSM228647 3 0.6712 0.39851 0.056 0.036 0.636 0.072 0.200
#> GSM228596 4 0.9692 -0.03801 0.116 0.136 0.244 0.260 0.244
#> GSM228600 3 0.5872 0.41736 0.012 0.036 0.696 0.100 0.156
#> GSM228603 3 0.4517 0.44547 0.024 0.004 0.776 0.040 0.156
#> GSM228615 2 0.6885 0.35460 0.028 0.492 0.016 0.368 0.096
#> GSM228627 3 0.8185 0.21404 0.056 0.104 0.460 0.080 0.300
#> GSM228641 3 0.4436 0.44297 0.008 0.012 0.772 0.036 0.172
#> GSM228644 2 0.4986 0.37629 0.000 0.748 0.068 0.036 0.148
#> GSM228651 3 0.6096 0.38485 0.004 0.044 0.652 0.088 0.212
#> GSM228654 3 0.7343 0.30166 0.020 0.112 0.556 0.072 0.240
#> GSM228658 3 0.6942 0.37992 0.080 0.056 0.616 0.040 0.208
#> GSM228606 5 0.8776 0.21072 0.016 0.244 0.248 0.156 0.336
#> GSM228611 3 0.7069 0.33361 0.056 0.036 0.528 0.052 0.328
#> GSM228618 3 0.4944 0.43544 0.000 0.016 0.720 0.060 0.204
#> GSM228621 3 0.6927 0.28511 0.008 0.124 0.544 0.040 0.284
#> GSM228624 5 0.8665 0.06252 0.024 0.156 0.324 0.148 0.348
#> GSM228630 3 0.8082 -0.18332 0.000 0.316 0.352 0.100 0.232
#> GSM228636 2 0.5127 0.46927 0.000 0.692 0.004 0.212 0.092
#> GSM228638 3 0.8002 0.17066 0.024 0.136 0.476 0.092 0.272
#> GSM228648 3 0.7191 0.18274 0.000 0.180 0.516 0.056 0.248
#> GSM228670 2 0.8721 0.11574 0.076 0.352 0.052 0.320 0.200
#> GSM228671 5 0.8709 0.22676 0.016 0.296 0.180 0.168 0.340
#> GSM228672 4 0.8009 0.23883 0.304 0.188 0.004 0.408 0.096
#> GSM228674 2 0.9078 -0.11957 0.168 0.348 0.052 0.284 0.148
#> GSM228675 2 0.8273 0.20986 0.060 0.432 0.048 0.300 0.160
#> GSM228676 4 0.9802 0.18241 0.212 0.172 0.120 0.248 0.248
#> GSM228667 4 0.9396 0.00894 0.100 0.248 0.100 0.312 0.240
#> GSM228668 1 0.6777 0.42634 0.636 0.052 0.064 0.196 0.052
#> GSM228669 1 0.8097 0.02251 0.472 0.180 0.024 0.240 0.084
#> GSM228673 5 0.9261 0.19578 0.068 0.196 0.260 0.136 0.340
#> GSM228677 2 0.8112 0.05160 0.000 0.424 0.172 0.164 0.240
#> GSM228678 2 0.6525 0.44949 0.004 0.624 0.052 0.192 0.128
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 6 0.8893 0.26561 0.288 0.152 0.052 0.048 0.152 0.308
#> GSM228563 4 0.8319 -0.06044 0.056 0.056 0.028 0.328 0.240 0.292
#> GSM228565 6 0.9405 0.28279 0.236 0.116 0.112 0.116 0.104 0.316
#> GSM228566 2 0.8813 0.08359 0.064 0.352 0.156 0.100 0.056 0.272
#> GSM228567 1 0.1738 0.59664 0.928 0.004 0.000 0.000 0.016 0.052
#> GSM228570 1 0.7030 0.18996 0.532 0.072 0.032 0.028 0.060 0.276
#> GSM228571 1 0.7253 0.00463 0.452 0.140 0.068 0.012 0.016 0.312
#> GSM228574 2 0.9014 -0.07502 0.016 0.296 0.240 0.164 0.116 0.168
#> GSM228575 3 0.9367 0.08707 0.032 0.192 0.240 0.180 0.128 0.228
#> GSM228576 6 0.8612 0.32711 0.252 0.192 0.052 0.056 0.076 0.372
#> GSM228579 1 0.6458 0.23907 0.540 0.064 0.060 0.008 0.020 0.308
#> GSM228580 4 0.7999 0.19705 0.000 0.104 0.128 0.440 0.220 0.108
#> GSM228581 4 0.8772 -0.03529 0.028 0.076 0.308 0.308 0.136 0.144
#> GSM228666 4 0.8722 0.06102 0.004 0.100 0.188 0.328 0.180 0.200
#> GSM228564 6 0.9258 0.08680 0.228 0.092 0.036 0.172 0.216 0.256
#> GSM228568 6 0.9153 0.22255 0.268 0.088 0.160 0.080 0.088 0.316
#> GSM228569 1 0.7277 0.19565 0.528 0.080 0.180 0.012 0.028 0.172
#> GSM228572 4 0.5695 0.33180 0.000 0.048 0.048 0.676 0.172 0.056
#> GSM228573 2 0.7642 0.09477 0.040 0.500 0.228 0.028 0.072 0.132
#> GSM228577 1 0.6380 0.41596 0.628 0.036 0.084 0.008 0.064 0.180
#> GSM228578 1 0.8692 -0.11110 0.412 0.132 0.116 0.040 0.092 0.208
#> GSM228663 3 0.7399 0.13463 0.028 0.284 0.492 0.092 0.032 0.072
#> GSM228664 3 0.7756 0.18394 0.000 0.168 0.436 0.236 0.044 0.116
#> GSM228665 3 0.8590 0.09945 0.116 0.276 0.392 0.064 0.068 0.084
#> GSM228582 3 0.9653 -0.10583 0.224 0.136 0.264 0.136 0.088 0.152
#> GSM228583 1 0.1268 0.59752 0.952 0.004 0.000 0.000 0.008 0.036
#> GSM228585 1 0.1320 0.59683 0.948 0.000 0.000 0.000 0.016 0.036
#> GSM228587 1 0.4344 0.52389 0.776 0.000 0.008 0.032 0.116 0.068
#> GSM228588 4 0.7510 0.07523 0.196 0.008 0.024 0.488 0.172 0.112
#> GSM228589 4 0.6430 0.29659 0.024 0.032 0.060 0.644 0.152 0.088
#> GSM228590 1 0.1899 0.59434 0.928 0.000 0.008 0.004 0.028 0.032
#> GSM228591 4 0.7860 0.23634 0.004 0.108 0.152 0.492 0.104 0.140
#> GSM228597 4 0.7301 0.16733 0.016 0.064 0.044 0.468 0.324 0.084
#> GSM228601 4 0.4823 0.32977 0.000 0.020 0.020 0.732 0.156 0.072
#> GSM228604 4 0.7928 0.10049 0.000 0.272 0.160 0.400 0.056 0.112
#> GSM228608 1 0.5472 0.48007 0.696 0.028 0.020 0.008 0.096 0.152
#> GSM228609 4 0.8460 -0.11502 0.200 0.052 0.028 0.340 0.292 0.088
#> GSM228613 1 0.1334 0.59620 0.948 0.000 0.000 0.000 0.020 0.032
#> GSM228616 6 0.9506 0.15463 0.236 0.216 0.076 0.104 0.120 0.248
#> GSM228628 4 0.8611 0.11133 0.040 0.060 0.164 0.408 0.124 0.204
#> GSM228634 1 0.2439 0.59308 0.904 0.020 0.028 0.000 0.008 0.040
#> GSM228642 4 0.7160 0.27425 0.000 0.076 0.188 0.544 0.068 0.124
#> GSM228645 6 0.9175 -0.13640 0.036 0.228 0.192 0.128 0.112 0.304
#> GSM228646 2 0.9254 -0.02020 0.028 0.268 0.168 0.212 0.116 0.208
#> GSM228652 1 0.6537 0.40149 0.624 0.036 0.040 0.020 0.144 0.136
#> GSM228655 1 0.9209 -0.18784 0.352 0.112 0.184 0.072 0.184 0.096
#> GSM228656 1 0.1268 0.59786 0.952 0.004 0.000 0.000 0.008 0.036
#> GSM228659 1 0.7766 0.04573 0.444 0.024 0.028 0.096 0.284 0.124
#> GSM228662 1 0.1245 0.59678 0.952 0.000 0.000 0.000 0.016 0.032
#> GSM228584 1 0.0748 0.59760 0.976 0.000 0.004 0.000 0.004 0.016
#> GSM228586 1 0.1734 0.59710 0.932 0.004 0.008 0.000 0.008 0.048
#> GSM228592 1 0.1180 0.59813 0.960 0.000 0.004 0.004 0.008 0.024
#> GSM228593 1 0.8825 -0.26315 0.308 0.044 0.032 0.216 0.228 0.172
#> GSM228594 1 0.4088 0.55276 0.800 0.028 0.040 0.004 0.012 0.116
#> GSM228598 1 0.4729 0.54373 0.764 0.012 0.036 0.012 0.056 0.120
#> GSM228607 3 0.9220 0.09188 0.048 0.156 0.308 0.088 0.228 0.172
#> GSM228612 3 0.8646 0.13888 0.048 0.264 0.392 0.104 0.088 0.104
#> GSM228619 2 0.8835 -0.21920 0.288 0.316 0.048 0.076 0.192 0.080
#> GSM228622 1 0.7836 0.09916 0.500 0.156 0.136 0.012 0.064 0.132
#> GSM228625 5 0.8817 0.08885 0.216 0.056 0.052 0.288 0.300 0.088
#> GSM228631 2 0.8467 -0.19626 0.340 0.344 0.052 0.052 0.108 0.104
#> GSM228633 4 0.4955 0.35560 0.000 0.040 0.116 0.740 0.084 0.020
#> GSM228637 4 0.6537 0.12103 0.020 0.012 0.048 0.444 0.416 0.060
#> GSM228639 5 0.8688 0.01562 0.024 0.116 0.196 0.288 0.312 0.064
#> GSM228649 4 0.8454 -0.04235 0.092 0.048 0.080 0.364 0.328 0.088
#> GSM228660 1 0.9304 -0.23309 0.336 0.052 0.160 0.156 0.132 0.164
#> GSM228661 1 0.6070 0.40091 0.652 0.112 0.132 0.004 0.016 0.084
#> GSM228595 4 0.3436 0.36219 0.000 0.016 0.044 0.848 0.068 0.024
#> GSM228599 4 0.8427 -0.02666 0.016 0.240 0.044 0.340 0.240 0.120
#> GSM228602 2 0.6276 0.23093 0.040 0.672 0.092 0.032 0.052 0.112
#> GSM228614 5 0.8330 0.00830 0.012 0.128 0.100 0.320 0.352 0.088
#> GSM228626 4 0.5377 0.33662 0.000 0.068 0.144 0.708 0.044 0.036
#> GSM228640 2 0.6366 0.19942 0.024 0.608 0.176 0.020 0.020 0.152
#> GSM228643 2 0.8757 0.00101 0.036 0.352 0.232 0.064 0.108 0.208
#> GSM228650 2 0.8063 0.02255 0.008 0.424 0.268 0.112 0.100 0.088
#> GSM228653 3 0.6730 0.08308 0.036 0.392 0.448 0.004 0.056 0.064
#> GSM228657 4 0.5353 0.33538 0.000 0.032 0.096 0.680 0.180 0.012
#> GSM228605 1 0.9159 -0.36234 0.292 0.112 0.112 0.044 0.168 0.272
#> GSM228610 2 0.8145 -0.05992 0.028 0.360 0.356 0.060 0.104 0.092
#> GSM228617 2 0.7363 0.18444 0.052 0.588 0.120 0.056 0.080 0.104
#> GSM228620 3 0.7877 0.06888 0.092 0.316 0.416 0.016 0.056 0.104
#> GSM228623 4 0.7719 0.05974 0.008 0.064 0.084 0.384 0.364 0.096
#> GSM228629 2 0.6769 0.09619 0.056 0.532 0.284 0.008 0.028 0.092
#> GSM228632 3 0.8330 0.17460 0.004 0.148 0.372 0.272 0.100 0.104
#> GSM228635 4 0.6386 0.17312 0.000 0.028 0.048 0.496 0.360 0.068
#> GSM228647 3 0.7776 0.09555 0.020 0.324 0.424 0.052 0.080 0.100
#> GSM228596 3 0.9382 0.10086 0.076 0.144 0.284 0.084 0.252 0.160
#> GSM228600 2 0.6697 0.19038 0.036 0.632 0.128 0.080 0.028 0.096
#> GSM228603 2 0.5716 0.19593 0.032 0.672 0.172 0.016 0.012 0.096
#> GSM228615 4 0.6563 0.07268 0.016 0.040 0.032 0.440 0.424 0.048
#> GSM228627 3 0.8284 0.13821 0.032 0.264 0.416 0.076 0.068 0.144
#> GSM228641 2 0.6694 0.17730 0.008 0.600 0.188 0.056 0.052 0.096
#> GSM228644 4 0.4895 0.35759 0.000 0.048 0.076 0.760 0.072 0.044
#> GSM228651 3 0.7741 0.11725 0.016 0.328 0.412 0.052 0.060 0.132
#> GSM228654 3 0.7761 0.10931 0.004 0.304 0.420 0.108 0.056 0.108
#> GSM228658 3 0.7066 0.10800 0.040 0.336 0.476 0.020 0.036 0.092
#> GSM228606 3 0.9071 0.08507 0.008 0.212 0.252 0.208 0.180 0.140
#> GSM228611 3 0.7213 0.12749 0.028 0.296 0.500 0.028 0.076 0.072
#> GSM228618 2 0.6449 0.11690 0.016 0.592 0.244 0.032 0.040 0.076
#> GSM228621 2 0.7406 -0.10221 0.004 0.388 0.380 0.112 0.040 0.076
#> GSM228624 3 0.8056 0.19352 0.008 0.172 0.464 0.104 0.088 0.164
#> GSM228630 4 0.8298 -0.06987 0.000 0.244 0.228 0.336 0.128 0.064
#> GSM228636 4 0.5098 0.24937 0.000 0.016 0.024 0.608 0.328 0.024
#> GSM228638 3 0.8164 0.08000 0.012 0.340 0.340 0.128 0.132 0.048
#> GSM228648 3 0.7595 0.15682 0.000 0.300 0.396 0.196 0.064 0.044
#> GSM228670 5 0.7995 0.09482 0.040 0.060 0.080 0.288 0.444 0.088
#> GSM228671 5 0.8611 0.06477 0.004 0.088 0.176 0.260 0.320 0.152
#> GSM228672 5 0.7684 -0.09241 0.308 0.008 0.036 0.084 0.416 0.148
#> GSM228674 5 0.9041 0.13958 0.188 0.048 0.048 0.260 0.276 0.180
#> GSM228675 5 0.7432 0.09271 0.036 0.040 0.044 0.280 0.492 0.108
#> GSM228676 5 0.9530 0.06846 0.124 0.116 0.116 0.124 0.312 0.208
#> GSM228667 5 0.9402 0.10238 0.096 0.096 0.116 0.168 0.328 0.196
#> GSM228668 1 0.7284 0.31428 0.580 0.052 0.060 0.040 0.124 0.144
#> GSM228669 1 0.7554 0.09520 0.492 0.016 0.040 0.116 0.248 0.088
#> GSM228673 3 0.9227 0.13209 0.044 0.188 0.312 0.100 0.212 0.144
#> GSM228677 4 0.8260 0.07690 0.000 0.140 0.156 0.400 0.212 0.092
#> GSM228678 4 0.7498 0.22722 0.000 0.088 0.096 0.500 0.220 0.096
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
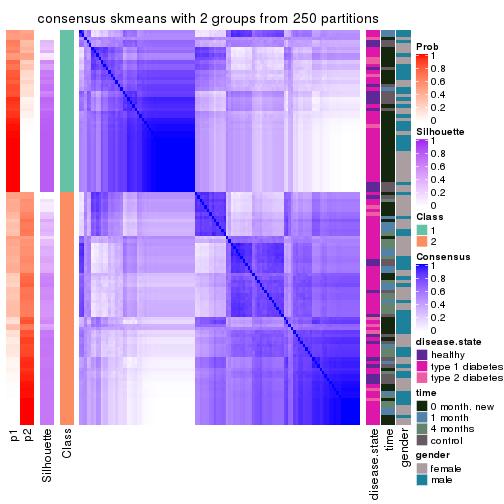
consensus_heatmap(res, k = 3)
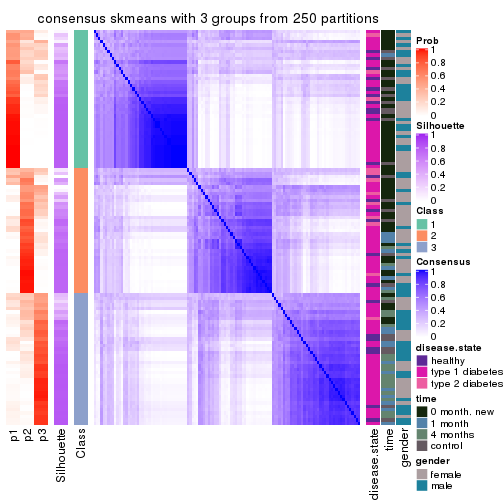
consensus_heatmap(res, k = 4)
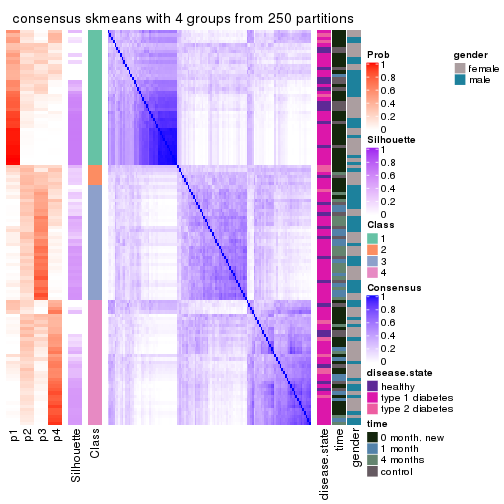
consensus_heatmap(res, k = 5)
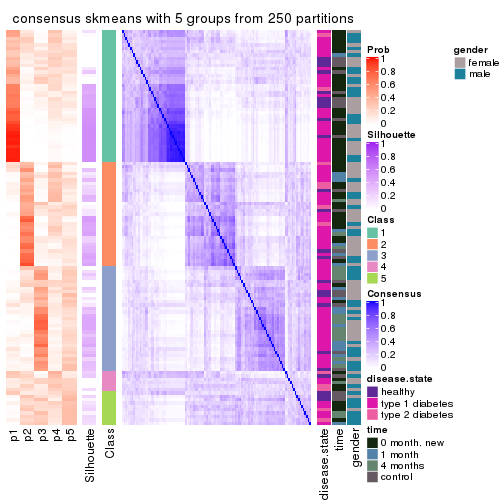
consensus_heatmap(res, k = 6)
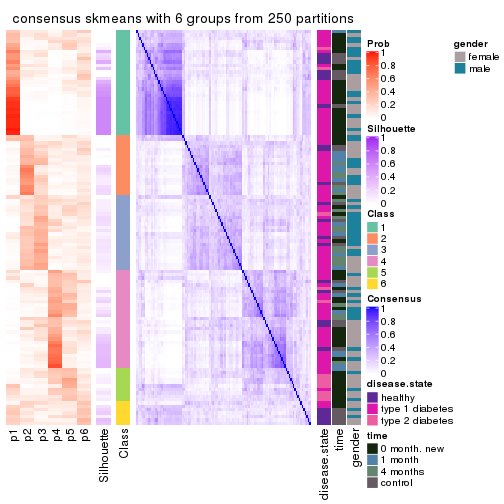
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
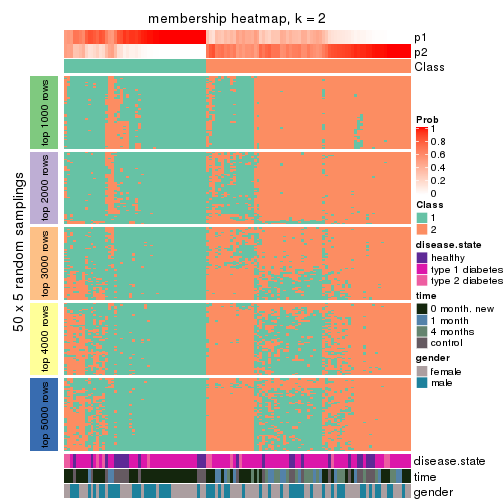
membership_heatmap(res, k = 3)
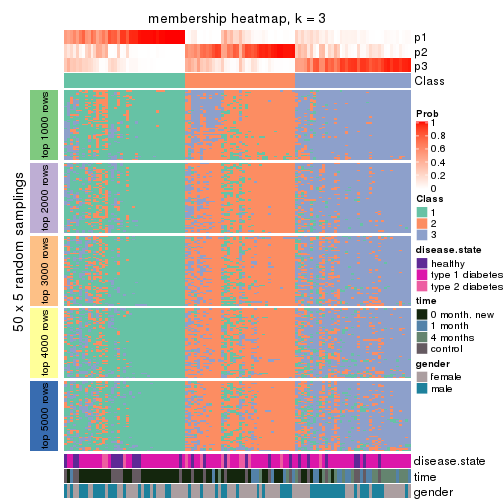
membership_heatmap(res, k = 4)
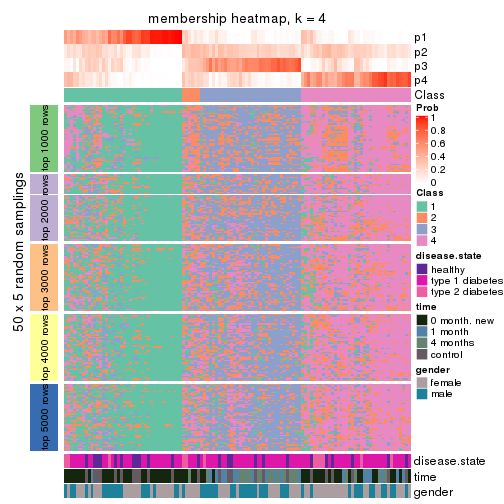
membership_heatmap(res, k = 5)
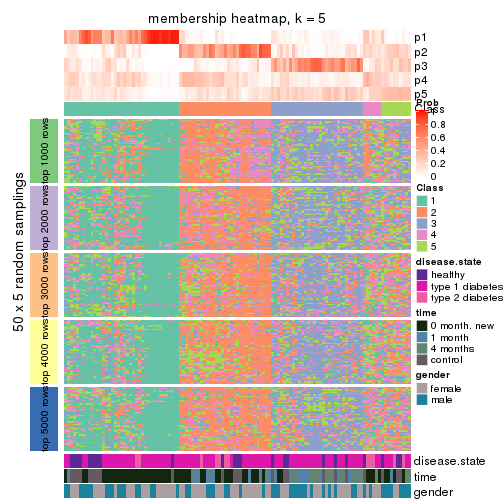
membership_heatmap(res, k = 6)
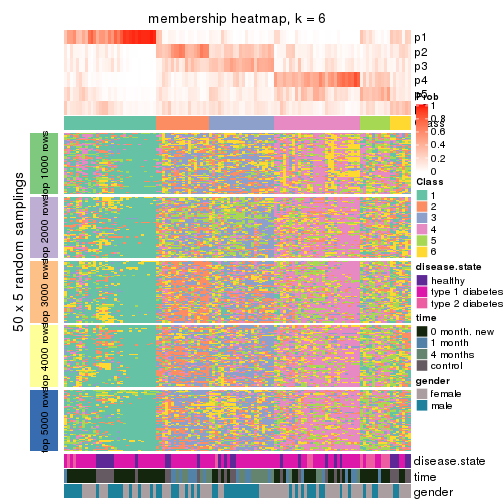
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
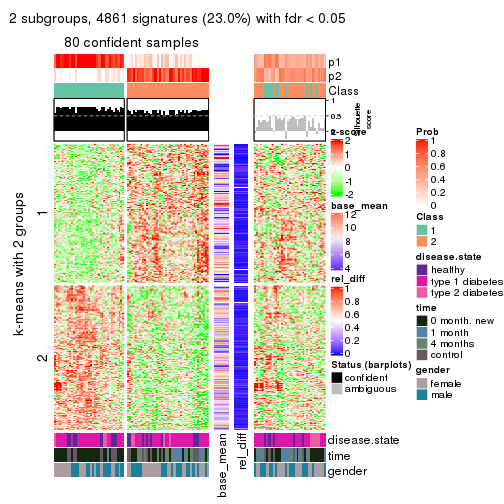
get_signatures(res, k = 3)
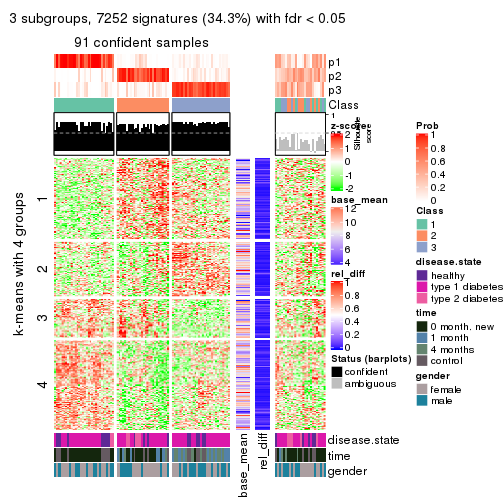
get_signatures(res, k = 4)
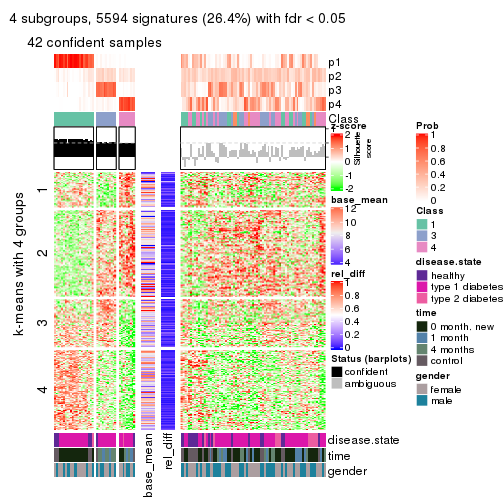
get_signatures(res, k = 5)

get_signatures(res, k = 6)

Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
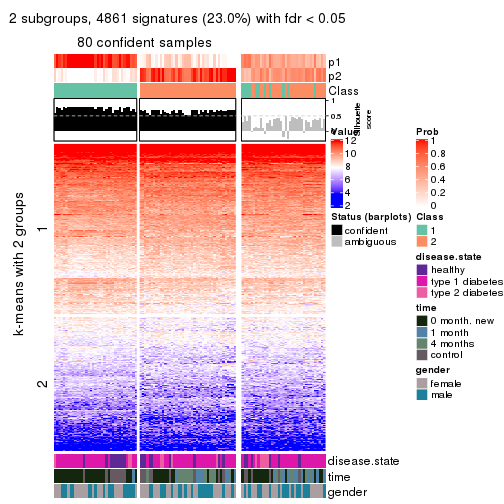
get_signatures(res, k = 3, scale_rows = FALSE)
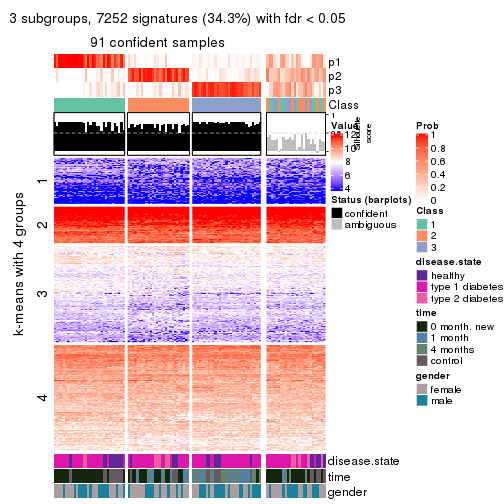
get_signatures(res, k = 4, scale_rows = FALSE)
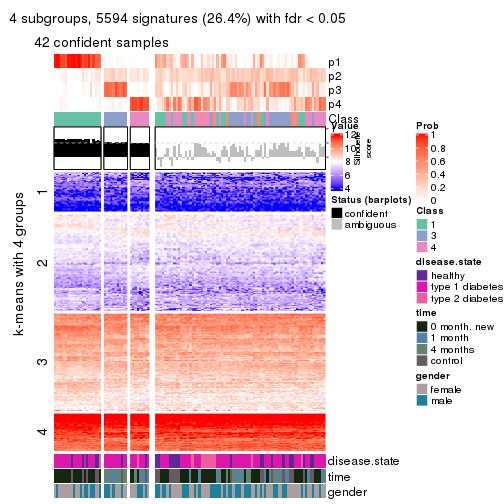
get_signatures(res, k = 5, scale_rows = FALSE)

get_signatures(res, k = 6, scale_rows = FALSE)

Compare the overlap of signatures from different k:
compare_signatures(res)
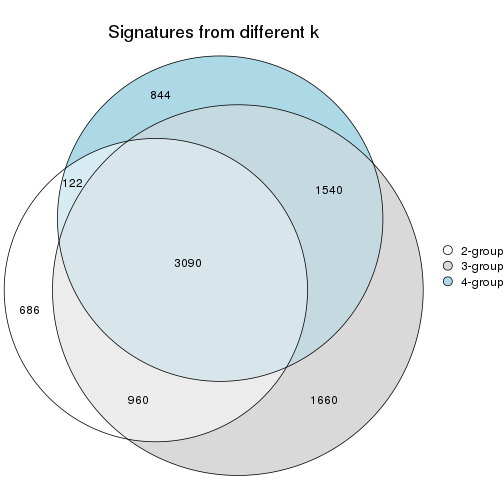
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
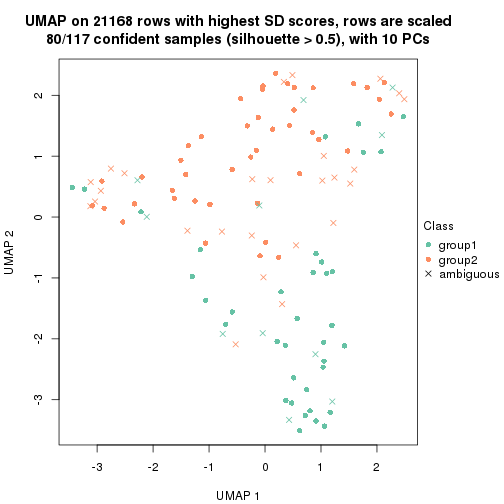
dimension_reduction(res, k = 3, method = "UMAP")
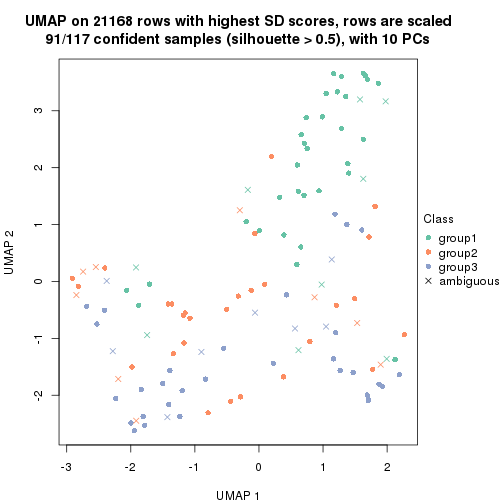
dimension_reduction(res, k = 4, method = "UMAP")
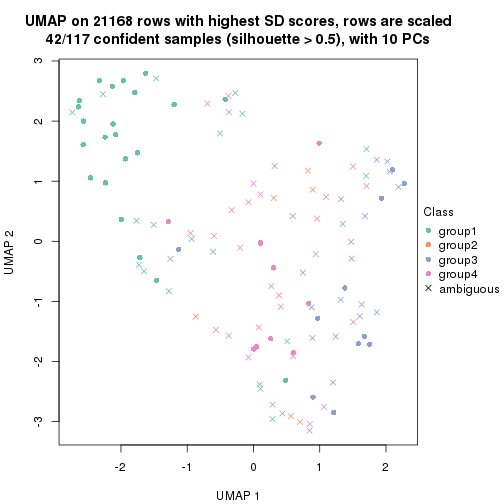
dimension_reduction(res, k = 5, method = "UMAP")
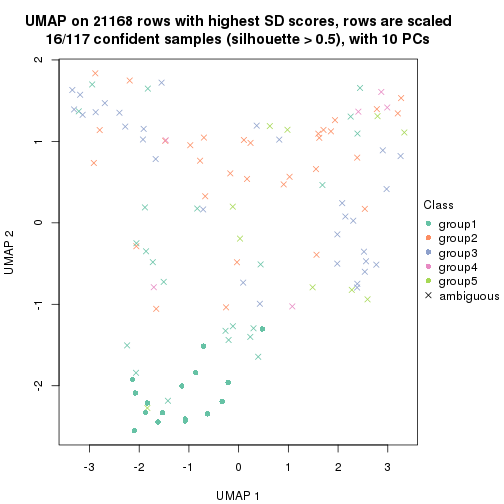
dimension_reduction(res, k = 6, method = "UMAP")
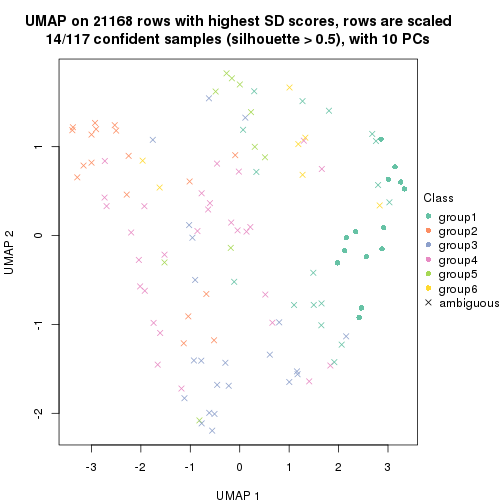
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
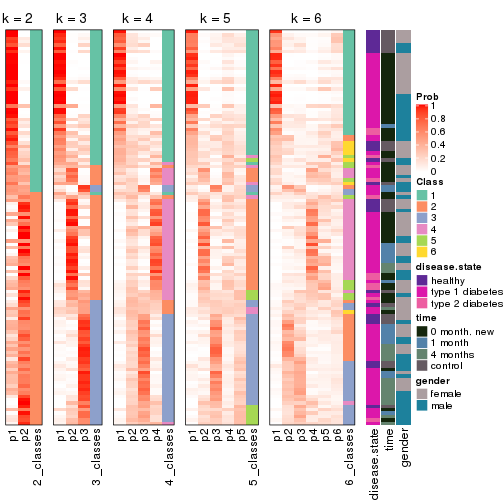
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> SD:skmeans 80 0.9286 3.99e-05 1.000 2
#> SD:skmeans 91 0.0837 1.66e-08 0.215 3
#> SD:skmeans 42 0.6219 2.22e-05 0.849 4
#> SD:skmeans 16 NA NA NA 5
#> SD:skmeans 14 NA NA NA 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["SD", "pam"]
# you can also extract it by
# res = res_list["SD:pam"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'SD' method.
#> Subgroups are detected by 'pam' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
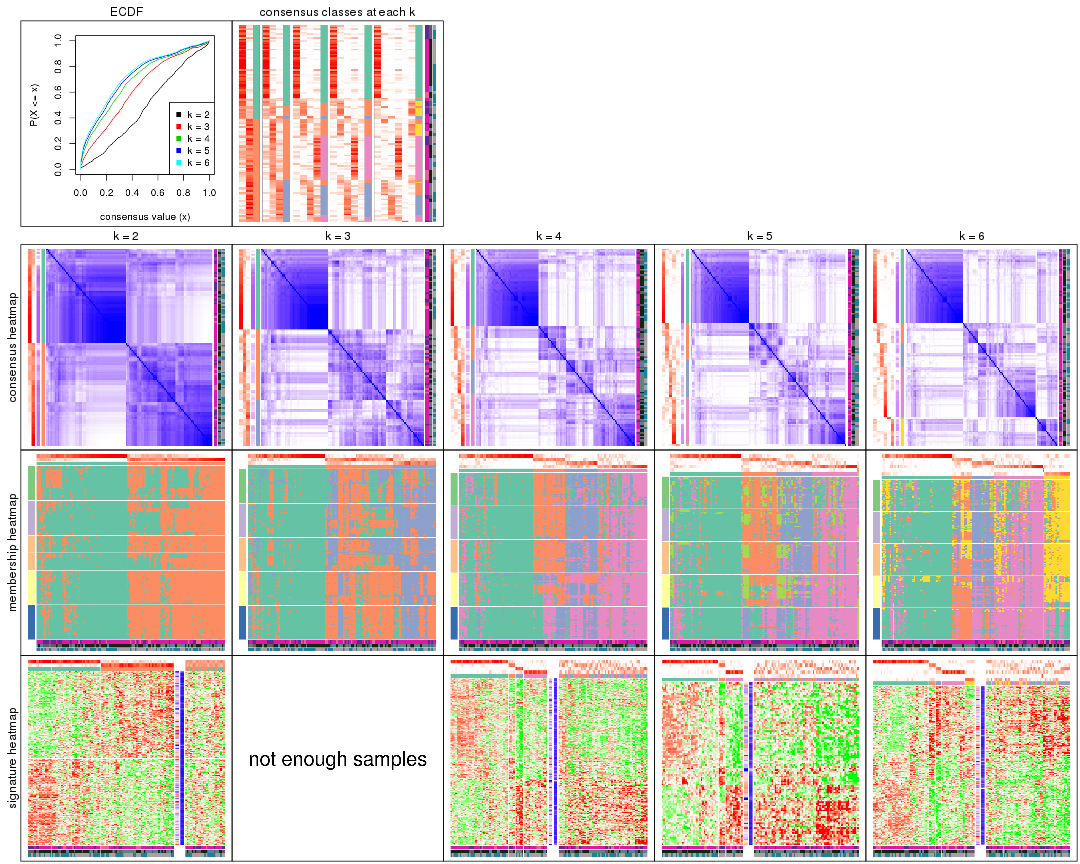
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
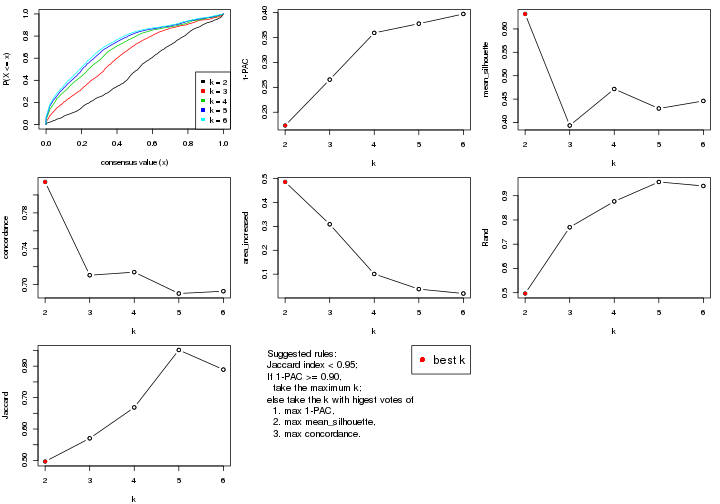
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.173 0.632 0.814 0.4860 0.497 0.497
#> 3 3 0.266 0.393 0.710 0.3089 0.770 0.570
#> 4 4 0.359 0.472 0.714 0.1008 0.877 0.669
#> 5 5 0.378 0.430 0.690 0.0375 0.957 0.852
#> 6 6 0.397 0.446 0.693 0.0190 0.940 0.789
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 2 0.9970 0.37676 0.468 0.532
#> GSM228563 1 0.6973 0.71572 0.812 0.188
#> GSM228565 1 0.9977 0.00282 0.528 0.472
#> GSM228566 2 0.4431 0.73391 0.092 0.908
#> GSM228567 1 0.2236 0.80636 0.964 0.036
#> GSM228570 1 0.8327 0.61819 0.736 0.264
#> GSM228571 1 0.9686 0.33945 0.604 0.396
#> GSM228574 2 0.0672 0.71787 0.008 0.992
#> GSM228575 2 0.9491 0.49562 0.368 0.632
#> GSM228576 1 0.8443 0.60628 0.728 0.272
#> GSM228579 1 0.6887 0.68906 0.816 0.184
#> GSM228580 2 0.9970 0.37963 0.468 0.532
#> GSM228581 1 0.9833 0.08251 0.576 0.424
#> GSM228666 2 0.7453 0.69680 0.212 0.788
#> GSM228564 1 0.1184 0.81313 0.984 0.016
#> GSM228568 1 0.7056 0.69207 0.808 0.192
#> GSM228569 1 0.5294 0.75264 0.880 0.120
#> GSM228572 2 0.4690 0.74139 0.100 0.900
#> GSM228573 1 0.7376 0.66164 0.792 0.208
#> GSM228577 1 0.0672 0.81429 0.992 0.008
#> GSM228578 1 0.5408 0.76610 0.876 0.124
#> GSM228663 2 0.2043 0.72735 0.032 0.968
#> GSM228664 2 0.4161 0.73233 0.084 0.916
#> GSM228665 1 0.9988 -0.29754 0.520 0.480
#> GSM228582 2 0.9922 0.37579 0.448 0.552
#> GSM228583 1 0.0000 0.81385 1.000 0.000
#> GSM228585 1 0.5178 0.75190 0.884 0.116
#> GSM228587 1 0.0672 0.81457 0.992 0.008
#> GSM228588 1 0.0000 0.81385 1.000 0.000
#> GSM228589 1 0.2043 0.81164 0.968 0.032
#> GSM228590 1 0.0000 0.81385 1.000 0.000
#> GSM228591 1 0.8386 0.55710 0.732 0.268
#> GSM228597 1 0.0938 0.81430 0.988 0.012
#> GSM228601 2 0.9998 0.18476 0.492 0.508
#> GSM228604 2 0.6712 0.65838 0.176 0.824
#> GSM228608 1 0.0376 0.81424 0.996 0.004
#> GSM228609 1 0.1843 0.81197 0.972 0.028
#> GSM228613 1 0.0000 0.81385 1.000 0.000
#> GSM228616 1 0.3584 0.79377 0.932 0.068
#> GSM228628 2 0.4161 0.73567 0.084 0.916
#> GSM228634 1 0.0376 0.81424 0.996 0.004
#> GSM228642 2 0.1843 0.72057 0.028 0.972
#> GSM228645 2 0.9983 0.09493 0.476 0.524
#> GSM228646 2 0.8499 0.64154 0.276 0.724
#> GSM228652 2 0.9552 0.57437 0.376 0.624
#> GSM228655 2 0.9993 0.40132 0.484 0.516
#> GSM228656 1 0.0000 0.81385 1.000 0.000
#> GSM228659 2 0.9970 0.42780 0.468 0.532
#> GSM228662 1 0.0000 0.81385 1.000 0.000
#> GSM228584 1 0.0000 0.81385 1.000 0.000
#> GSM228586 1 0.0000 0.81385 1.000 0.000
#> GSM228592 1 0.0000 0.81385 1.000 0.000
#> GSM228593 1 0.9460 0.12461 0.636 0.364
#> GSM228594 1 0.0000 0.81385 1.000 0.000
#> GSM228598 1 0.1633 0.81110 0.976 0.024
#> GSM228607 1 0.6801 0.66267 0.820 0.180
#> GSM228612 1 0.9393 0.47779 0.644 0.356
#> GSM228619 1 0.1184 0.81308 0.984 0.016
#> GSM228622 1 0.6973 0.65424 0.812 0.188
#> GSM228625 1 0.5408 0.74428 0.876 0.124
#> GSM228631 1 0.0938 0.81328 0.988 0.012
#> GSM228633 2 0.0000 0.71365 0.000 1.000
#> GSM228637 2 0.9044 0.64458 0.320 0.680
#> GSM228639 2 0.8144 0.68336 0.252 0.748
#> GSM228649 2 0.9963 0.43622 0.464 0.536
#> GSM228660 1 0.3274 0.80005 0.940 0.060
#> GSM228661 1 0.0000 0.81385 1.000 0.000
#> GSM228595 2 0.2603 0.72978 0.044 0.956
#> GSM228599 2 0.9393 0.58022 0.356 0.644
#> GSM228602 2 0.3114 0.73369 0.056 0.944
#> GSM228614 2 0.7056 0.71400 0.192 0.808
#> GSM228626 2 0.0000 0.71365 0.000 1.000
#> GSM228640 1 0.9977 0.18622 0.528 0.472
#> GSM228643 2 0.0376 0.71529 0.004 0.996
#> GSM228650 2 0.6973 0.73249 0.188 0.812
#> GSM228653 2 0.0672 0.71769 0.008 0.992
#> GSM228657 2 0.7602 0.70277 0.220 0.780
#> GSM228605 1 0.0000 0.81385 1.000 0.000
#> GSM228610 1 0.9129 0.45983 0.672 0.328
#> GSM228617 1 0.9970 -0.14521 0.532 0.468
#> GSM228620 1 0.2948 0.80366 0.948 0.052
#> GSM228623 2 0.9129 0.64115 0.328 0.672
#> GSM228629 2 0.9963 0.43566 0.464 0.536
#> GSM228632 2 0.4815 0.73209 0.104 0.896
#> GSM228635 1 0.7883 0.54612 0.764 0.236
#> GSM228647 2 0.9944 0.38223 0.456 0.544
#> GSM228596 2 0.9754 0.53986 0.408 0.592
#> GSM228600 2 0.6438 0.70462 0.164 0.836
#> GSM228603 2 0.7528 0.67391 0.216 0.784
#> GSM228615 1 0.4939 0.75864 0.892 0.108
#> GSM228627 2 0.3431 0.72856 0.064 0.936
#> GSM228641 2 0.9427 0.30793 0.360 0.640
#> GSM228644 2 0.0000 0.71365 0.000 1.000
#> GSM228651 2 0.7376 0.70300 0.208 0.792
#> GSM228654 2 0.2948 0.73047 0.052 0.948
#> GSM228658 2 0.2043 0.72734 0.032 0.968
#> GSM228606 2 0.9896 0.44006 0.440 0.560
#> GSM228611 2 0.9983 0.12317 0.476 0.524
#> GSM228618 1 0.6438 0.73148 0.836 0.164
#> GSM228621 2 0.7139 0.62413 0.196 0.804
#> GSM228624 1 0.9661 0.30428 0.608 0.392
#> GSM228630 2 0.8144 0.68691 0.252 0.748
#> GSM228636 2 0.8555 0.67592 0.280 0.720
#> GSM228638 2 0.8081 0.68903 0.248 0.752
#> GSM228648 2 0.0000 0.71365 0.000 1.000
#> GSM228670 2 0.5842 0.74271 0.140 0.860
#> GSM228671 2 0.6048 0.73514 0.148 0.852
#> GSM228672 1 0.4431 0.78587 0.908 0.092
#> GSM228674 2 0.9896 0.47929 0.440 0.560
#> GSM228675 2 0.9358 0.61321 0.352 0.648
#> GSM228676 2 0.8386 0.63437 0.268 0.732
#> GSM228667 2 0.9087 0.59066 0.324 0.676
#> GSM228668 1 0.0376 0.81423 0.996 0.004
#> GSM228669 1 0.0000 0.81385 1.000 0.000
#> GSM228673 2 0.7376 0.70878 0.208 0.792
#> GSM228677 2 0.6623 0.73019 0.172 0.828
#> GSM228678 2 0.4690 0.73009 0.100 0.900
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 2 0.7640 0.34709 0.372 0.576 0.052
#> GSM228563 1 0.5929 0.50212 0.676 0.320 0.004
#> GSM228565 2 0.6008 0.32228 0.332 0.664 0.004
#> GSM228566 2 0.4110 0.30981 0.004 0.844 0.152
#> GSM228567 1 0.1860 0.79097 0.948 0.052 0.000
#> GSM228570 2 0.6299 -0.08767 0.476 0.524 0.000
#> GSM228571 2 0.6422 0.24231 0.324 0.660 0.016
#> GSM228574 2 0.4974 0.19682 0.000 0.764 0.236
#> GSM228575 2 0.6775 0.31643 0.164 0.740 0.096
#> GSM228576 2 0.6476 -0.01382 0.448 0.548 0.004
#> GSM228579 1 0.6291 0.18566 0.532 0.468 0.000
#> GSM228580 3 0.9984 -0.11897 0.336 0.308 0.356
#> GSM228581 1 0.8936 -0.03023 0.500 0.368 0.132
#> GSM228666 2 0.7092 0.33025 0.084 0.708 0.208
#> GSM228564 1 0.0892 0.80571 0.980 0.020 0.000
#> GSM228568 1 0.6565 0.29101 0.576 0.416 0.008
#> GSM228569 1 0.4233 0.69387 0.836 0.160 0.004
#> GSM228572 2 0.8100 -0.02033 0.068 0.512 0.420
#> GSM228573 1 0.7948 0.40710 0.632 0.100 0.268
#> GSM228577 1 0.1031 0.80490 0.976 0.024 0.000
#> GSM228578 1 0.5621 0.50126 0.692 0.308 0.000
#> GSM228663 3 0.6421 0.19969 0.004 0.424 0.572
#> GSM228664 3 0.7001 0.26007 0.024 0.388 0.588
#> GSM228665 1 0.9389 -0.23000 0.468 0.352 0.180
#> GSM228582 2 0.9199 0.32702 0.328 0.504 0.168
#> GSM228583 1 0.0000 0.80772 1.000 0.000 0.000
#> GSM228585 1 0.4291 0.67463 0.820 0.180 0.000
#> GSM228587 1 0.2229 0.79153 0.944 0.012 0.044
#> GSM228588 1 0.0000 0.80772 1.000 0.000 0.000
#> GSM228589 1 0.2527 0.79432 0.936 0.020 0.044
#> GSM228590 1 0.0000 0.80772 1.000 0.000 0.000
#> GSM228591 1 0.7644 0.38373 0.624 0.308 0.068
#> GSM228597 1 0.1015 0.80642 0.980 0.008 0.012
#> GSM228601 2 0.8372 0.34317 0.312 0.580 0.108
#> GSM228604 3 0.6307 0.14753 0.000 0.488 0.512
#> GSM228608 1 0.0237 0.80752 0.996 0.000 0.004
#> GSM228609 1 0.1781 0.80227 0.960 0.020 0.020
#> GSM228613 1 0.0000 0.80772 1.000 0.000 0.000
#> GSM228616 1 0.4099 0.72825 0.852 0.140 0.008
#> GSM228628 2 0.3637 0.28358 0.024 0.892 0.084
#> GSM228634 1 0.1711 0.79869 0.960 0.008 0.032
#> GSM228642 3 0.6235 0.24482 0.000 0.436 0.564
#> GSM228645 2 0.6854 0.25619 0.216 0.716 0.068
#> GSM228646 2 0.7138 0.37349 0.120 0.720 0.160
#> GSM228652 2 0.9125 0.27640 0.192 0.540 0.268
#> GSM228655 2 0.8608 0.28903 0.412 0.488 0.100
#> GSM228656 1 0.0000 0.80772 1.000 0.000 0.000
#> GSM228659 2 0.9183 0.30610 0.324 0.508 0.168
#> GSM228662 1 0.0000 0.80772 1.000 0.000 0.000
#> GSM228584 1 0.0000 0.80772 1.000 0.000 0.000
#> GSM228586 1 0.0000 0.80772 1.000 0.000 0.000
#> GSM228592 1 0.0000 0.80772 1.000 0.000 0.000
#> GSM228593 1 0.7918 0.11525 0.596 0.328 0.076
#> GSM228594 1 0.0000 0.80772 1.000 0.000 0.000
#> GSM228598 1 0.1170 0.80461 0.976 0.016 0.008
#> GSM228607 1 0.5346 0.64867 0.808 0.152 0.040
#> GSM228612 2 0.8068 -0.07241 0.456 0.480 0.064
#> GSM228619 1 0.0983 0.80590 0.980 0.004 0.016
#> GSM228622 1 0.5667 0.63945 0.800 0.140 0.060
#> GSM228625 1 0.4335 0.72796 0.864 0.100 0.036
#> GSM228631 1 0.2448 0.78013 0.924 0.000 0.076
#> GSM228633 3 0.4235 0.40312 0.000 0.176 0.824
#> GSM228637 2 0.9745 0.05403 0.232 0.420 0.348
#> GSM228639 3 0.8379 0.25480 0.096 0.352 0.552
#> GSM228649 2 0.8547 0.32287 0.364 0.532 0.104
#> GSM228660 1 0.4345 0.72940 0.848 0.016 0.136
#> GSM228661 1 0.0000 0.80772 1.000 0.000 0.000
#> GSM228595 3 0.6079 0.29025 0.000 0.388 0.612
#> GSM228599 2 0.9208 0.33435 0.220 0.536 0.244
#> GSM228602 3 0.6701 0.23281 0.012 0.412 0.576
#> GSM228614 3 0.6168 0.20728 0.000 0.412 0.588
#> GSM228626 3 0.4931 0.37318 0.000 0.232 0.768
#> GSM228640 2 0.8466 -0.08067 0.092 0.508 0.400
#> GSM228643 2 0.2796 0.29271 0.000 0.908 0.092
#> GSM228650 2 0.7940 0.03979 0.060 0.524 0.416
#> GSM228653 3 0.6298 0.30156 0.004 0.388 0.608
#> GSM228657 3 0.6879 0.25770 0.024 0.360 0.616
#> GSM228605 1 0.0000 0.80772 1.000 0.000 0.000
#> GSM228610 3 0.7470 0.24282 0.336 0.052 0.612
#> GSM228617 3 0.9527 0.05837 0.372 0.192 0.436
#> GSM228620 1 0.2550 0.79317 0.936 0.024 0.040
#> GSM228623 2 0.9306 0.16851 0.172 0.480 0.348
#> GSM228629 2 0.8684 0.30391 0.392 0.500 0.108
#> GSM228632 2 0.6927 0.08308 0.040 0.664 0.296
#> GSM228635 1 0.5774 0.52316 0.748 0.232 0.020
#> GSM228647 3 0.9653 0.09083 0.328 0.224 0.448
#> GSM228596 2 0.9464 0.27871 0.252 0.500 0.248
#> GSM228600 2 0.6416 -0.03331 0.008 0.616 0.376
#> GSM228603 2 0.5905 -0.02634 0.000 0.648 0.352
#> GSM228615 1 0.4807 0.71713 0.848 0.092 0.060
#> GSM228627 2 0.5058 0.23705 0.032 0.820 0.148
#> GSM228641 3 0.6396 0.31657 0.016 0.320 0.664
#> GSM228644 3 0.5465 0.36935 0.000 0.288 0.712
#> GSM228651 3 0.5688 0.38045 0.044 0.168 0.788
#> GSM228654 3 0.4634 0.42672 0.012 0.164 0.824
#> GSM228658 2 0.6721 0.06737 0.016 0.604 0.380
#> GSM228606 3 0.9787 -0.05445 0.296 0.268 0.436
#> GSM228611 3 0.9460 0.17354 0.260 0.240 0.500
#> GSM228618 1 0.8756 0.22559 0.540 0.128 0.332
#> GSM228621 3 0.6543 0.30076 0.016 0.344 0.640
#> GSM228624 1 0.9752 -0.13811 0.416 0.352 0.232
#> GSM228630 3 0.6500 0.41148 0.100 0.140 0.760
#> GSM228636 3 0.9201 0.15215 0.160 0.352 0.488
#> GSM228638 3 0.8037 0.26303 0.076 0.352 0.572
#> GSM228648 3 0.1753 0.42605 0.000 0.048 0.952
#> GSM228670 2 0.7729 0.00164 0.048 0.516 0.436
#> GSM228671 2 0.8553 -0.00527 0.112 0.552 0.336
#> GSM228672 1 0.3918 0.74166 0.856 0.140 0.004
#> GSM228674 2 0.8622 0.35112 0.296 0.572 0.132
#> GSM228675 2 0.7677 0.37349 0.204 0.676 0.120
#> GSM228676 2 0.4443 0.35619 0.052 0.864 0.084
#> GSM228667 2 0.6710 0.38151 0.196 0.732 0.072
#> GSM228668 1 0.0237 0.80763 0.996 0.000 0.004
#> GSM228669 1 0.0000 0.80772 1.000 0.000 0.000
#> GSM228673 2 0.7757 0.27403 0.112 0.664 0.224
#> GSM228677 2 0.7043 0.32168 0.136 0.728 0.136
#> GSM228678 2 0.5042 0.27148 0.060 0.836 0.104
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.7564 0.2634 0.328 0.208 0.000 0.464
#> GSM228563 1 0.6264 0.2731 0.592 0.344 0.004 0.060
#> GSM228565 2 0.7226 0.3941 0.220 0.548 0.000 0.232
#> GSM228566 4 0.3249 0.5584 0.000 0.140 0.008 0.852
#> GSM228567 1 0.1637 0.7860 0.940 0.060 0.000 0.000
#> GSM228570 2 0.4936 0.5189 0.316 0.672 0.000 0.012
#> GSM228571 2 0.5705 0.5641 0.204 0.704 0.000 0.092
#> GSM228574 4 0.7031 0.1924 0.000 0.324 0.140 0.536
#> GSM228575 2 0.8465 0.2178 0.152 0.452 0.056 0.340
#> GSM228576 2 0.5250 0.5269 0.316 0.660 0.000 0.024
#> GSM228579 2 0.4624 0.4894 0.340 0.660 0.000 0.000
#> GSM228580 4 0.9151 0.2055 0.124 0.220 0.196 0.460
#> GSM228581 1 0.8742 -0.0212 0.464 0.160 0.080 0.296
#> GSM228666 4 0.1985 0.5852 0.020 0.024 0.012 0.944
#> GSM228564 1 0.0707 0.7990 0.980 0.020 0.000 0.000
#> GSM228568 2 0.5244 0.2758 0.436 0.556 0.008 0.000
#> GSM228569 1 0.3937 0.6689 0.800 0.188 0.000 0.012
#> GSM228572 4 0.7042 0.0719 0.020 0.076 0.368 0.536
#> GSM228573 1 0.7642 0.3837 0.592 0.072 0.248 0.088
#> GSM228577 1 0.1118 0.7984 0.964 0.036 0.000 0.000
#> GSM228578 1 0.6068 -0.0682 0.508 0.448 0.000 0.044
#> GSM228663 3 0.5168 0.1187 0.004 0.000 0.504 0.492
#> GSM228664 3 0.5149 0.4126 0.000 0.016 0.648 0.336
#> GSM228665 1 0.7412 -0.2302 0.444 0.000 0.168 0.388
#> GSM228582 4 0.4414 0.5669 0.120 0.036 0.020 0.824
#> GSM228583 1 0.0817 0.7990 0.976 0.024 0.000 0.000
#> GSM228585 1 0.3486 0.6558 0.812 0.188 0.000 0.000
#> GSM228587 1 0.2589 0.7601 0.884 0.000 0.000 0.116
#> GSM228588 1 0.0000 0.7996 1.000 0.000 0.000 0.000
#> GSM228589 1 0.2960 0.7778 0.892 0.004 0.020 0.084
#> GSM228590 1 0.0000 0.7996 1.000 0.000 0.000 0.000
#> GSM228591 1 0.7252 0.2861 0.536 0.132 0.008 0.324
#> GSM228597 1 0.1059 0.8017 0.972 0.000 0.012 0.016
#> GSM228601 4 0.7643 0.2628 0.228 0.224 0.012 0.536
#> GSM228604 2 0.5339 0.3867 0.000 0.624 0.356 0.020
#> GSM228608 1 0.0188 0.8002 0.996 0.000 0.000 0.004
#> GSM228609 1 0.2634 0.7824 0.920 0.020 0.032 0.028
#> GSM228613 1 0.0000 0.7996 1.000 0.000 0.000 0.000
#> GSM228616 1 0.3907 0.6941 0.808 0.180 0.008 0.004
#> GSM228628 2 0.5906 0.3586 0.016 0.636 0.028 0.320
#> GSM228634 1 0.2949 0.7732 0.888 0.024 0.000 0.088
#> GSM228642 2 0.6192 0.2540 0.000 0.512 0.436 0.052
#> GSM228645 2 0.6586 0.5458 0.132 0.676 0.020 0.172
#> GSM228646 4 0.3143 0.5892 0.024 0.080 0.008 0.888
#> GSM228652 4 0.2007 0.5781 0.020 0.004 0.036 0.940
#> GSM228655 4 0.3942 0.5469 0.236 0.000 0.000 0.764
#> GSM228656 1 0.1004 0.7998 0.972 0.024 0.000 0.004
#> GSM228659 4 0.1557 0.5899 0.056 0.000 0.000 0.944
#> GSM228662 1 0.0336 0.8006 0.992 0.008 0.000 0.000
#> GSM228584 1 0.0336 0.8005 0.992 0.008 0.000 0.000
#> GSM228586 1 0.0817 0.7990 0.976 0.024 0.000 0.000
#> GSM228592 1 0.0000 0.7996 1.000 0.000 0.000 0.000
#> GSM228593 1 0.4992 -0.0587 0.524 0.000 0.000 0.476
#> GSM228594 1 0.0000 0.7996 1.000 0.000 0.000 0.000
#> GSM228598 1 0.1406 0.8000 0.960 0.016 0.000 0.024
#> GSM228607 1 0.5342 0.6249 0.732 0.024 0.024 0.220
#> GSM228612 2 0.7121 0.4773 0.316 0.580 0.056 0.048
#> GSM228619 1 0.1182 0.7999 0.968 0.000 0.016 0.016
#> GSM228622 1 0.4881 0.6351 0.752 0.012 0.020 0.216
#> GSM228625 1 0.4008 0.7223 0.820 0.000 0.032 0.148
#> GSM228631 1 0.2722 0.7754 0.904 0.032 0.064 0.000
#> GSM228633 3 0.2521 0.5299 0.000 0.024 0.912 0.064
#> GSM228637 4 0.7426 0.2687 0.224 0.000 0.264 0.512
#> GSM228639 3 0.5836 0.4124 0.056 0.000 0.640 0.304
#> GSM228649 4 0.4546 0.5337 0.256 0.012 0.000 0.732
#> GSM228660 1 0.4546 0.7197 0.812 0.004 0.092 0.092
#> GSM228661 1 0.0592 0.8003 0.984 0.016 0.000 0.000
#> GSM228595 3 0.5250 0.4369 0.000 0.024 0.660 0.316
#> GSM228599 4 0.4474 0.5693 0.056 0.036 0.072 0.836
#> GSM228602 2 0.6440 0.2094 0.004 0.484 0.456 0.056
#> GSM228614 4 0.4877 0.0410 0.000 0.000 0.408 0.592
#> GSM228626 3 0.3479 0.4551 0.000 0.148 0.840 0.012
#> GSM228640 2 0.6690 0.4353 0.056 0.620 0.292 0.032
#> GSM228643 2 0.5980 0.1828 0.000 0.560 0.044 0.396
#> GSM228650 4 0.1296 0.5690 0.004 0.004 0.028 0.964
#> GSM228653 3 0.7275 0.2679 0.000 0.152 0.472 0.376
#> GSM228657 3 0.5728 0.2745 0.020 0.004 0.544 0.432
#> GSM228605 1 0.0000 0.7996 1.000 0.000 0.000 0.000
#> GSM228610 3 0.5927 0.3617 0.240 0.060 0.688 0.012
#> GSM228617 3 0.8669 0.1038 0.340 0.044 0.400 0.216
#> GSM228620 1 0.2675 0.7843 0.908 0.000 0.044 0.048
#> GSM228623 4 0.5257 0.5372 0.080 0.004 0.160 0.756
#> GSM228629 4 0.6556 0.4340 0.336 0.052 0.020 0.592
#> GSM228632 4 0.8413 0.0444 0.020 0.316 0.280 0.384
#> GSM228635 1 0.5114 0.5002 0.696 0.004 0.020 0.280
#> GSM228647 4 0.8801 -0.0158 0.288 0.040 0.332 0.340
#> GSM228596 4 0.2408 0.5866 0.044 0.000 0.036 0.920
#> GSM228600 2 0.6598 0.4060 0.004 0.600 0.300 0.096
#> GSM228603 2 0.6013 0.4338 0.000 0.640 0.288 0.072
#> GSM228615 1 0.3626 0.7073 0.812 0.000 0.004 0.184
#> GSM228627 2 0.6772 0.4090 0.032 0.604 0.056 0.308
#> GSM228641 3 0.5959 0.1692 0.012 0.336 0.620 0.032
#> GSM228644 3 0.4675 0.5162 0.000 0.020 0.736 0.244
#> GSM228651 3 0.6226 0.3722 0.008 0.056 0.616 0.320
#> GSM228654 3 0.5209 0.5364 0.000 0.104 0.756 0.140
#> GSM228658 4 0.6634 0.3917 0.008 0.112 0.248 0.632
#> GSM228606 4 0.7807 0.2289 0.164 0.028 0.268 0.540
#> GSM228611 3 0.9223 0.0937 0.220 0.240 0.432 0.108
#> GSM228618 1 0.8512 0.1266 0.500 0.180 0.256 0.064
#> GSM228621 3 0.6468 0.0851 0.004 0.348 0.576 0.072
#> GSM228624 1 0.9535 -0.2667 0.352 0.324 0.152 0.172
#> GSM228630 3 0.4229 0.5517 0.048 0.004 0.824 0.124
#> GSM228636 3 0.6991 0.2859 0.136 0.000 0.540 0.324
#> GSM228638 3 0.5636 0.4150 0.044 0.000 0.648 0.308
#> GSM228648 3 0.1297 0.5324 0.000 0.016 0.964 0.020
#> GSM228670 4 0.1902 0.5620 0.004 0.000 0.064 0.932
#> GSM228671 2 0.9171 0.1093 0.084 0.404 0.244 0.268
#> GSM228672 1 0.4094 0.7211 0.828 0.116 0.000 0.056
#> GSM228674 4 0.5289 0.5749 0.184 0.032 0.028 0.756
#> GSM228675 4 0.4415 0.5977 0.124 0.044 0.012 0.820
#> GSM228676 4 0.5173 0.4246 0.020 0.320 0.000 0.660
#> GSM228667 4 0.8308 0.0475 0.140 0.384 0.048 0.428
#> GSM228668 1 0.0188 0.8007 0.996 0.000 0.000 0.004
#> GSM228669 1 0.0376 0.8003 0.992 0.000 0.004 0.004
#> GSM228673 4 0.8416 0.3569 0.076 0.248 0.152 0.524
#> GSM228677 4 0.8410 0.3149 0.128 0.296 0.076 0.500
#> GSM228678 2 0.6428 0.4579 0.052 0.664 0.036 0.248
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 4 0.6995 0.23086 0.316 0.192 0.000 0.468 0.024
#> GSM228563 1 0.5369 0.12523 0.552 0.388 0.000 0.060 0.000
#> GSM228565 2 0.6105 0.43418 0.196 0.588 0.000 0.212 0.004
#> GSM228566 4 0.3861 0.52984 0.000 0.092 0.004 0.816 0.088
#> GSM228567 1 0.1638 0.78707 0.932 0.064 0.000 0.000 0.004
#> GSM228570 2 0.3783 0.50129 0.252 0.740 0.000 0.008 0.000
#> GSM228571 2 0.4276 0.52420 0.168 0.764 0.000 0.068 0.000
#> GSM228574 4 0.7767 0.15452 0.000 0.212 0.096 0.460 0.232
#> GSM228575 2 0.8866 0.11963 0.116 0.356 0.056 0.324 0.148
#> GSM228576 2 0.4106 0.50295 0.256 0.724 0.000 0.020 0.000
#> GSM228579 2 0.3661 0.49247 0.276 0.724 0.000 0.000 0.000
#> GSM228580 5 0.7188 0.00000 0.080 0.000 0.132 0.260 0.528
#> GSM228581 1 0.7682 -0.00417 0.460 0.164 0.068 0.300 0.008
#> GSM228666 4 0.0693 0.54720 0.008 0.000 0.000 0.980 0.012
#> GSM228564 1 0.0703 0.79745 0.976 0.024 0.000 0.000 0.000
#> GSM228568 2 0.4227 0.30051 0.420 0.580 0.000 0.000 0.000
#> GSM228569 1 0.3933 0.66112 0.776 0.196 0.000 0.008 0.020
#> GSM228572 4 0.7009 0.03230 0.020 0.080 0.376 0.484 0.040
#> GSM228573 1 0.8181 0.15376 0.504 0.060 0.208 0.068 0.160
#> GSM228577 1 0.1205 0.79775 0.956 0.040 0.000 0.000 0.004
#> GSM228578 2 0.5315 0.21978 0.456 0.500 0.000 0.040 0.004
#> GSM228663 3 0.4443 0.05827 0.004 0.000 0.524 0.472 0.000
#> GSM228664 3 0.5041 0.34723 0.000 0.004 0.636 0.316 0.044
#> GSM228665 1 0.7017 -0.21876 0.428 0.000 0.180 0.368 0.024
#> GSM228582 4 0.4466 0.46757 0.100 0.016 0.008 0.796 0.080
#> GSM228583 1 0.1168 0.79565 0.960 0.032 0.000 0.000 0.008
#> GSM228585 1 0.3074 0.65444 0.804 0.196 0.000 0.000 0.000
#> GSM228587 1 0.2471 0.74749 0.864 0.000 0.000 0.136 0.000
#> GSM228588 1 0.0000 0.79765 1.000 0.000 0.000 0.000 0.000
#> GSM228589 1 0.3073 0.76800 0.872 0.000 0.008 0.068 0.052
#> GSM228590 1 0.0000 0.79765 1.000 0.000 0.000 0.000 0.000
#> GSM228591 1 0.6953 0.26617 0.524 0.116 0.004 0.308 0.048
#> GSM228597 1 0.1475 0.80013 0.956 0.004 0.012 0.012 0.016
#> GSM228601 4 0.7212 0.19389 0.196 0.216 0.008 0.536 0.044
#> GSM228604 2 0.6264 0.22286 0.000 0.536 0.304 0.004 0.156
#> GSM228608 1 0.0162 0.79829 0.996 0.000 0.000 0.004 0.000
#> GSM228609 1 0.2945 0.77175 0.892 0.016 0.020 0.016 0.056
#> GSM228613 1 0.0000 0.79765 1.000 0.000 0.000 0.000 0.000
#> GSM228616 1 0.4095 0.65657 0.764 0.208 0.008 0.004 0.016
#> GSM228628 2 0.4884 0.37906 0.016 0.684 0.016 0.276 0.008
#> GSM228634 1 0.3266 0.75768 0.852 0.032 0.000 0.108 0.008
#> GSM228642 2 0.6424 0.21544 0.000 0.512 0.356 0.020 0.112
#> GSM228645 2 0.6701 0.50680 0.100 0.636 0.008 0.152 0.104
#> GSM228646 4 0.2288 0.55554 0.020 0.028 0.008 0.924 0.020
#> GSM228652 4 0.0451 0.54716 0.004 0.000 0.008 0.988 0.000
#> GSM228655 4 0.3551 0.48262 0.220 0.000 0.000 0.772 0.008
#> GSM228656 1 0.1329 0.79630 0.956 0.032 0.000 0.004 0.008
#> GSM228659 4 0.0404 0.54983 0.012 0.000 0.000 0.988 0.000
#> GSM228662 1 0.0510 0.79917 0.984 0.016 0.000 0.000 0.000
#> GSM228584 1 0.0290 0.79884 0.992 0.008 0.000 0.000 0.000
#> GSM228586 1 0.1168 0.79565 0.960 0.032 0.000 0.000 0.008
#> GSM228592 1 0.0000 0.79765 1.000 0.000 0.000 0.000 0.000
#> GSM228593 1 0.4304 -0.05864 0.516 0.000 0.000 0.484 0.000
#> GSM228594 1 0.0000 0.79765 1.000 0.000 0.000 0.000 0.000
#> GSM228598 1 0.1710 0.79912 0.944 0.020 0.000 0.024 0.012
#> GSM228607 1 0.5742 0.60937 0.704 0.024 0.032 0.180 0.060
#> GSM228612 2 0.6866 0.44578 0.280 0.572 0.044 0.024 0.080
#> GSM228619 1 0.1739 0.79281 0.940 0.000 0.032 0.004 0.024
#> GSM228622 1 0.4981 0.60204 0.720 0.012 0.024 0.220 0.024
#> GSM228625 1 0.4296 0.70544 0.796 0.000 0.024 0.124 0.056
#> GSM228631 1 0.2730 0.77357 0.892 0.044 0.056 0.000 0.008
#> GSM228633 3 0.4478 0.30965 0.000 0.040 0.768 0.024 0.168
#> GSM228637 4 0.6986 0.25419 0.192 0.000 0.288 0.492 0.028
#> GSM228639 3 0.4666 0.36410 0.040 0.000 0.676 0.284 0.000
#> GSM228649 4 0.3918 0.47254 0.232 0.008 0.000 0.752 0.008
#> GSM228660 1 0.4626 0.70379 0.788 0.000 0.092 0.064 0.056
#> GSM228661 1 0.0609 0.79881 0.980 0.020 0.000 0.000 0.000
#> GSM228595 3 0.5811 0.33431 0.000 0.056 0.664 0.220 0.060
#> GSM228599 4 0.4506 0.48632 0.036 0.020 0.072 0.812 0.060
#> GSM228602 3 0.7002 -0.02951 0.000 0.348 0.436 0.020 0.196
#> GSM228614 4 0.4517 0.12531 0.000 0.000 0.388 0.600 0.012
#> GSM228626 3 0.5191 0.28090 0.000 0.192 0.684 0.000 0.124
#> GSM228640 2 0.7528 0.21377 0.044 0.452 0.252 0.004 0.248
#> GSM228643 2 0.5122 0.16411 0.000 0.584 0.024 0.380 0.012
#> GSM228650 4 0.1186 0.54214 0.000 0.008 0.008 0.964 0.020
#> GSM228653 3 0.8059 0.22925 0.000 0.104 0.372 0.308 0.216
#> GSM228657 3 0.4972 0.18397 0.008 0.000 0.536 0.440 0.016
#> GSM228605 1 0.0000 0.79765 1.000 0.000 0.000 0.000 0.000
#> GSM228610 3 0.5967 0.21252 0.232 0.044 0.656 0.008 0.060
#> GSM228617 3 0.8663 0.00276 0.292 0.048 0.396 0.172 0.092
#> GSM228620 1 0.2765 0.78038 0.896 0.000 0.036 0.044 0.024
#> GSM228623 4 0.5350 0.50453 0.064 0.000 0.156 0.724 0.056
#> GSM228629 4 0.6718 0.33256 0.292 0.048 0.024 0.576 0.060
#> GSM228632 4 0.8285 0.04298 0.016 0.324 0.248 0.340 0.072
#> GSM228635 1 0.6587 0.43790 0.628 0.092 0.012 0.208 0.060
#> GSM228647 3 0.8701 -0.09810 0.260 0.032 0.312 0.308 0.088
#> GSM228596 4 0.0968 0.55128 0.012 0.000 0.012 0.972 0.004
#> GSM228600 2 0.7698 0.12853 0.000 0.384 0.252 0.056 0.308
#> GSM228603 2 0.7308 0.19841 0.000 0.452 0.252 0.036 0.260
#> GSM228615 1 0.3686 0.67366 0.780 0.012 0.000 0.204 0.004
#> GSM228627 2 0.7099 0.41107 0.032 0.564 0.024 0.236 0.144
#> GSM228641 3 0.7095 0.15951 0.012 0.244 0.472 0.008 0.264
#> GSM228644 3 0.5768 0.35028 0.000 0.024 0.672 0.164 0.140
#> GSM228651 3 0.6805 0.25495 0.004 0.036 0.564 0.244 0.152
#> GSM228654 3 0.6418 0.34773 0.000 0.076 0.624 0.088 0.212
#> GSM228658 4 0.7067 0.38177 0.008 0.088 0.184 0.592 0.128
#> GSM228606 4 0.8328 0.02084 0.148 0.040 0.204 0.488 0.120
#> GSM228611 3 0.9025 0.09977 0.192 0.212 0.408 0.076 0.112
#> GSM228618 1 0.8685 -0.18650 0.364 0.104 0.244 0.028 0.260
#> GSM228621 3 0.7279 0.11726 0.004 0.232 0.408 0.020 0.336
#> GSM228624 2 0.9381 0.20043 0.304 0.304 0.116 0.100 0.176
#> GSM228630 3 0.3395 0.40696 0.028 0.016 0.848 0.108 0.000
#> GSM228636 3 0.6487 0.26171 0.128 0.012 0.552 0.300 0.008
#> GSM228638 3 0.4616 0.36381 0.028 0.000 0.680 0.288 0.004
#> GSM228648 3 0.1617 0.35952 0.000 0.020 0.948 0.012 0.020
#> GSM228670 4 0.0613 0.54359 0.000 0.004 0.008 0.984 0.004
#> GSM228671 2 0.8723 0.20592 0.068 0.444 0.192 0.204 0.092
#> GSM228672 1 0.3575 0.71626 0.824 0.120 0.000 0.056 0.000
#> GSM228674 4 0.4235 0.51194 0.176 0.024 0.024 0.776 0.000
#> GSM228675 4 0.3736 0.55051 0.112 0.028 0.012 0.836 0.012
#> GSM228676 4 0.4688 0.43799 0.020 0.312 0.000 0.660 0.008
#> GSM228667 2 0.7790 0.09447 0.140 0.432 0.036 0.352 0.040
#> GSM228668 1 0.0451 0.80019 0.988 0.000 0.000 0.004 0.008
#> GSM228669 1 0.0854 0.79823 0.976 0.012 0.000 0.008 0.004
#> GSM228673 4 0.8621 0.27193 0.064 0.232 0.144 0.456 0.104
#> GSM228677 4 0.7344 0.27635 0.120 0.316 0.064 0.492 0.008
#> GSM228678 2 0.4868 0.46112 0.044 0.728 0.016 0.208 0.004
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 4 0.6567 0.20763 0.312 0.036 0.000 0.460 0.004 0.188
#> GSM228563 1 0.4955 0.06717 0.548 0.000 0.004 0.060 0.000 0.388
#> GSM228565 6 0.5412 0.51546 0.192 0.004 0.000 0.204 0.000 0.600
#> GSM228566 4 0.3520 0.56270 0.000 0.100 0.000 0.816 0.008 0.076
#> GSM228567 1 0.1643 0.77310 0.924 0.008 0.000 0.000 0.000 0.068
#> GSM228570 6 0.3323 0.53977 0.240 0.000 0.000 0.008 0.000 0.752
#> GSM228571 6 0.3965 0.55411 0.160 0.008 0.000 0.064 0.000 0.768
#> GSM228574 4 0.6061 0.02015 0.000 0.404 0.000 0.424 0.016 0.156
#> GSM228575 4 0.8204 -0.09056 0.108 0.208 0.004 0.324 0.052 0.304
#> GSM228576 6 0.3617 0.54315 0.244 0.000 0.000 0.020 0.000 0.736
#> GSM228579 6 0.3221 0.53078 0.264 0.000 0.000 0.000 0.000 0.736
#> GSM228580 5 0.3809 0.00000 0.044 0.004 0.044 0.092 0.816 0.000
#> GSM228581 1 0.7188 -0.04711 0.452 0.008 0.068 0.300 0.012 0.160
#> GSM228666 4 0.0551 0.57853 0.008 0.004 0.000 0.984 0.004 0.000
#> GSM228564 1 0.0632 0.78081 0.976 0.000 0.000 0.000 0.000 0.024
#> GSM228568 6 0.3789 0.35572 0.416 0.000 0.000 0.000 0.000 0.584
#> GSM228569 1 0.4056 0.64346 0.756 0.032 0.000 0.004 0.016 0.192
#> GSM228572 4 0.7249 -0.13751 0.016 0.052 0.356 0.448 0.060 0.068
#> GSM228573 1 0.6569 0.05567 0.476 0.356 0.088 0.064 0.000 0.016
#> GSM228577 1 0.1082 0.78172 0.956 0.004 0.000 0.000 0.000 0.040
#> GSM228578 6 0.5099 0.34191 0.432 0.000 0.000 0.040 0.020 0.508
#> GSM228663 3 0.4211 0.20197 0.004 0.008 0.532 0.456 0.000 0.000
#> GSM228664 3 0.4426 0.50480 0.000 0.052 0.652 0.296 0.000 0.000
#> GSM228665 1 0.6876 -0.25485 0.404 0.008 0.176 0.360 0.052 0.000
#> GSM228582 4 0.5022 0.54237 0.100 0.100 0.012 0.744 0.028 0.016
#> GSM228583 1 0.1408 0.77612 0.944 0.020 0.000 0.000 0.000 0.036
#> GSM228585 1 0.2871 0.65110 0.804 0.004 0.000 0.000 0.000 0.192
#> GSM228587 1 0.2402 0.73147 0.856 0.000 0.000 0.140 0.004 0.000
#> GSM228588 1 0.0000 0.78032 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228589 1 0.3119 0.74950 0.864 0.044 0.016 0.064 0.012 0.000
#> GSM228590 1 0.0000 0.78032 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228591 1 0.6775 0.21933 0.512 0.040 0.012 0.300 0.020 0.116
#> GSM228597 1 0.1654 0.78237 0.944 0.008 0.008 0.012 0.024 0.004
#> GSM228601 4 0.6697 0.19001 0.188 0.040 0.012 0.536 0.008 0.216
#> GSM228604 2 0.4748 0.38758 0.000 0.504 0.048 0.000 0.000 0.448
#> GSM228608 1 0.0291 0.78166 0.992 0.000 0.000 0.004 0.004 0.000
#> GSM228609 1 0.3497 0.74181 0.856 0.056 0.024 0.016 0.032 0.016
#> GSM228613 1 0.0000 0.78032 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228616 1 0.4056 0.63161 0.748 0.008 0.004 0.004 0.028 0.208
#> GSM228628 6 0.4458 0.44627 0.016 0.004 0.020 0.268 0.004 0.688
#> GSM228634 1 0.3423 0.73826 0.832 0.020 0.000 0.108 0.004 0.036
#> GSM228642 6 0.6825 -0.06626 0.000 0.264 0.188 0.020 0.040 0.488
#> GSM228645 6 0.6266 0.48827 0.096 0.152 0.000 0.148 0.004 0.600
#> GSM228646 4 0.2031 0.58766 0.020 0.008 0.004 0.928 0.016 0.024
#> GSM228652 4 0.0405 0.57574 0.004 0.000 0.008 0.988 0.000 0.000
#> GSM228655 4 0.3596 0.53730 0.216 0.016 0.000 0.760 0.008 0.000
#> GSM228656 1 0.1697 0.77563 0.936 0.020 0.000 0.004 0.004 0.036
#> GSM228659 4 0.0405 0.57869 0.008 0.000 0.000 0.988 0.004 0.000
#> GSM228662 1 0.0458 0.78219 0.984 0.000 0.000 0.000 0.000 0.016
#> GSM228584 1 0.0260 0.78163 0.992 0.000 0.000 0.000 0.000 0.008
#> GSM228586 1 0.1552 0.77522 0.940 0.020 0.000 0.000 0.004 0.036
#> GSM228592 1 0.0000 0.78032 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228593 1 0.3866 -0.06519 0.516 0.000 0.000 0.484 0.000 0.000
#> GSM228594 1 0.0000 0.78032 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228598 1 0.1862 0.78169 0.932 0.020 0.000 0.024 0.004 0.020
#> GSM228607 1 0.5819 0.57103 0.676 0.056 0.024 0.180 0.040 0.024
#> GSM228612 6 0.6806 0.43195 0.272 0.112 0.036 0.016 0.028 0.536
#> GSM228619 1 0.1945 0.77180 0.920 0.004 0.016 0.004 0.056 0.000
#> GSM228622 1 0.5126 0.57122 0.684 0.008 0.012 0.216 0.068 0.012
#> GSM228625 1 0.4435 0.67870 0.776 0.052 0.024 0.120 0.028 0.000
#> GSM228631 1 0.2921 0.75249 0.868 0.080 0.004 0.000 0.016 0.032
#> GSM228633 3 0.5128 0.22267 0.000 0.320 0.604 0.016 0.056 0.004
#> GSM228637 4 0.6489 0.20292 0.176 0.000 0.276 0.496 0.052 0.000
#> GSM228639 3 0.3907 0.53195 0.028 0.000 0.704 0.268 0.000 0.000
#> GSM228649 4 0.3497 0.53353 0.224 0.004 0.000 0.760 0.004 0.008
#> GSM228660 1 0.4644 0.67950 0.772 0.056 0.096 0.048 0.028 0.000
#> GSM228661 1 0.0891 0.78121 0.968 0.008 0.000 0.000 0.000 0.024
#> GSM228595 3 0.5587 0.47937 0.000 0.048 0.680 0.156 0.096 0.020
#> GSM228599 4 0.3953 0.54832 0.032 0.116 0.004 0.804 0.040 0.004
#> GSM228602 2 0.5982 0.54767 0.000 0.552 0.184 0.016 0.004 0.244
#> GSM228614 4 0.4118 -0.03242 0.000 0.008 0.396 0.592 0.004 0.000
#> GSM228626 3 0.5304 0.22566 0.000 0.196 0.664 0.000 0.040 0.100
#> GSM228640 2 0.4492 0.48780 0.036 0.620 0.000 0.000 0.004 0.340
#> GSM228643 6 0.4950 0.23111 0.000 0.028 0.016 0.364 0.008 0.584
#> GSM228650 4 0.1080 0.57384 0.000 0.032 0.004 0.960 0.004 0.000
#> GSM228653 2 0.7204 0.04072 0.000 0.420 0.240 0.272 0.032 0.036
#> GSM228657 3 0.4757 0.36494 0.008 0.016 0.548 0.416 0.012 0.000
#> GSM228605 1 0.0000 0.78032 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228610 3 0.6258 0.09839 0.224 0.208 0.540 0.004 0.008 0.016
#> GSM228617 3 0.8964 0.05279 0.276 0.168 0.296 0.160 0.060 0.040
#> GSM228620 1 0.2794 0.76518 0.888 0.028 0.028 0.040 0.016 0.000
#> GSM228623 4 0.5387 0.51580 0.060 0.048 0.136 0.712 0.044 0.000
#> GSM228629 4 0.6689 0.43922 0.260 0.056 0.016 0.568 0.048 0.052
#> GSM228632 4 0.7890 0.03616 0.012 0.096 0.236 0.332 0.016 0.308
#> GSM228635 1 0.8225 0.12130 0.476 0.152 0.064 0.112 0.036 0.160
#> GSM228647 4 0.8759 -0.03421 0.232 0.108 0.268 0.300 0.064 0.028
#> GSM228596 4 0.1129 0.57991 0.012 0.008 0.012 0.964 0.004 0.000
#> GSM228600 2 0.4204 0.54021 0.000 0.696 0.000 0.040 0.004 0.260
#> GSM228603 2 0.4181 0.49885 0.000 0.644 0.000 0.028 0.000 0.328
#> GSM228615 1 0.3370 0.65736 0.772 0.000 0.012 0.212 0.004 0.000
#> GSM228627 6 0.6302 0.33288 0.028 0.220 0.000 0.224 0.004 0.524
#> GSM228641 2 0.5620 0.52548 0.008 0.632 0.192 0.004 0.012 0.152
#> GSM228644 3 0.6238 0.44415 0.000 0.136 0.608 0.120 0.132 0.004
#> GSM228651 2 0.6140 0.09010 0.000 0.428 0.348 0.216 0.008 0.000
#> GSM228654 2 0.6369 0.00712 0.000 0.464 0.392 0.080 0.044 0.020
#> GSM228658 4 0.6717 0.38848 0.004 0.184 0.148 0.572 0.020 0.072
#> GSM228606 4 0.7901 0.18836 0.144 0.236 0.132 0.440 0.040 0.008
#> GSM228611 2 0.8884 0.20707 0.176 0.340 0.220 0.072 0.028 0.164
#> GSM228618 2 0.6717 0.22029 0.300 0.536 0.028 0.020 0.064 0.052
#> GSM228621 2 0.4785 0.54300 0.004 0.724 0.128 0.012 0.004 0.128
#> GSM228624 1 0.9020 -0.37454 0.276 0.240 0.060 0.088 0.068 0.268
#> GSM228630 3 0.4799 0.47741 0.024 0.144 0.724 0.104 0.004 0.000
#> GSM228636 3 0.5483 0.42733 0.124 0.000 0.584 0.280 0.012 0.000
#> GSM228638 3 0.3756 0.53332 0.020 0.000 0.712 0.268 0.000 0.000
#> GSM228648 3 0.3672 0.28435 0.000 0.276 0.712 0.008 0.004 0.000
#> GSM228670 4 0.0405 0.57290 0.000 0.000 0.008 0.988 0.004 0.000
#> GSM228671 6 0.8490 0.22323 0.040 0.128 0.152 0.176 0.064 0.440
#> GSM228672 1 0.3309 0.70283 0.824 0.000 0.000 0.056 0.004 0.116
#> GSM228674 4 0.4020 0.55804 0.176 0.000 0.028 0.768 0.004 0.024
#> GSM228675 4 0.3555 0.58773 0.108 0.008 0.008 0.832 0.016 0.028
#> GSM228676 4 0.4611 0.44151 0.016 0.016 0.000 0.652 0.012 0.304
#> GSM228667 6 0.7728 0.11318 0.120 0.056 0.048 0.348 0.024 0.404
#> GSM228668 1 0.0653 0.78395 0.980 0.000 0.004 0.004 0.012 0.000
#> GSM228669 1 0.0767 0.78165 0.976 0.000 0.012 0.008 0.004 0.000
#> GSM228673 4 0.8279 0.30754 0.060 0.120 0.148 0.440 0.024 0.208
#> GSM228677 4 0.6862 0.23996 0.112 0.000 0.076 0.492 0.020 0.300
#> GSM228678 6 0.4813 0.50054 0.044 0.000 0.028 0.196 0.016 0.716
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
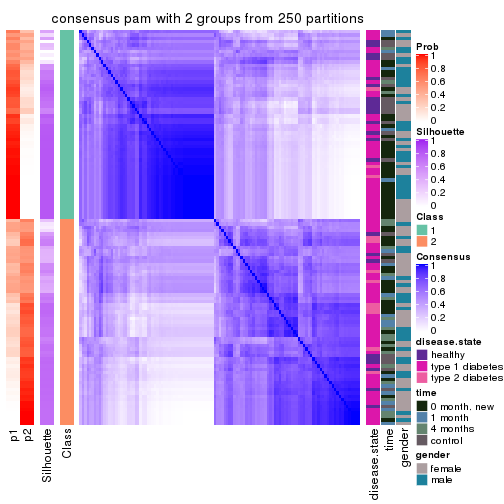
consensus_heatmap(res, k = 3)
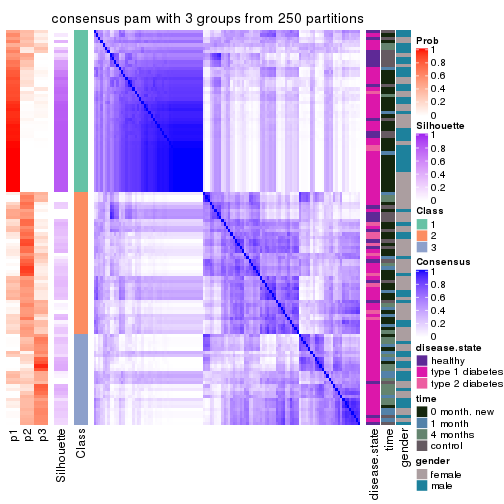
consensus_heatmap(res, k = 4)
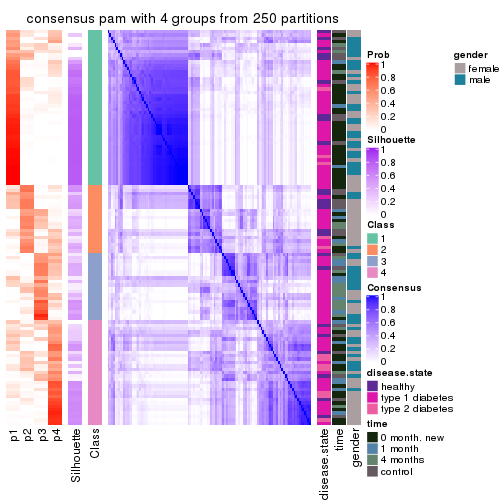
consensus_heatmap(res, k = 5)
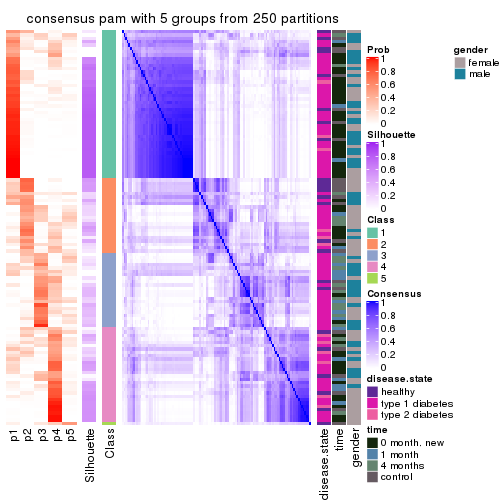
consensus_heatmap(res, k = 6)
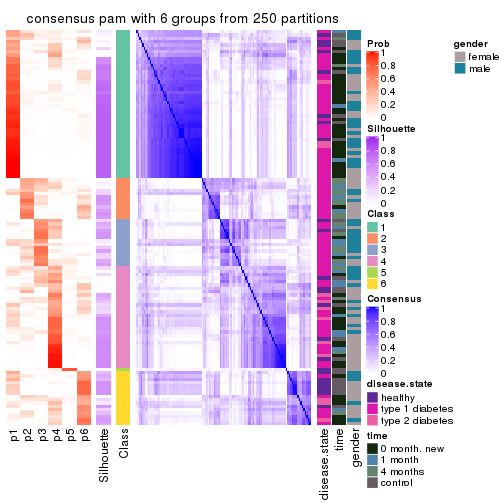
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
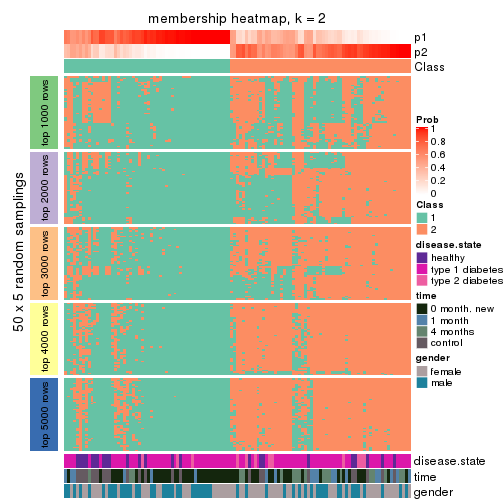
membership_heatmap(res, k = 3)
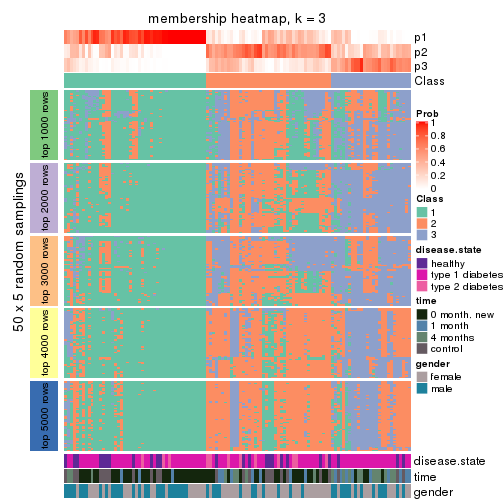
membership_heatmap(res, k = 4)
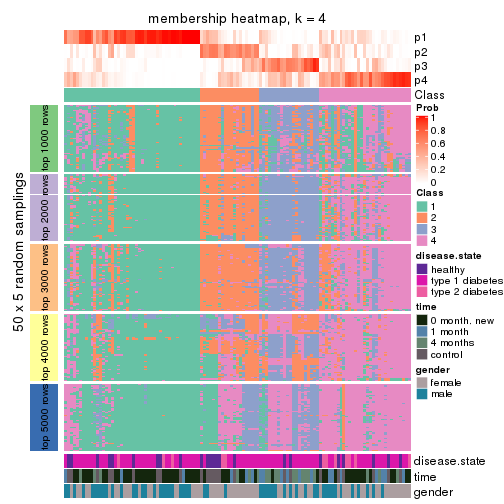
membership_heatmap(res, k = 5)
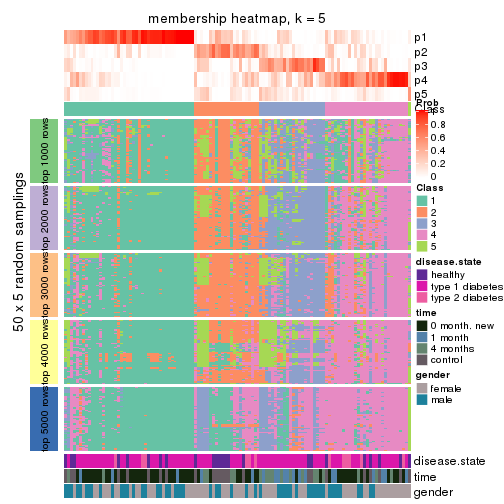
membership_heatmap(res, k = 6)
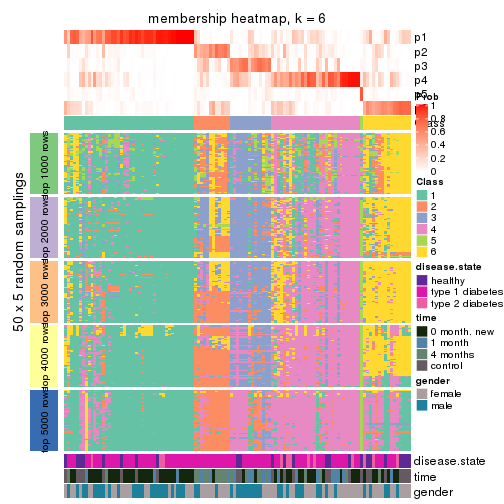
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
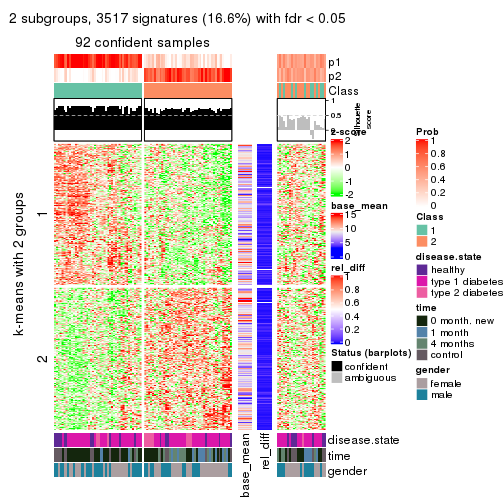
get_signatures(res, k = 3)

get_signatures(res, k = 4)
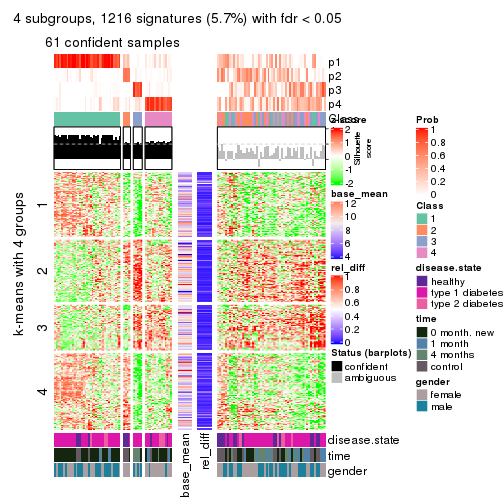
get_signatures(res, k = 5)
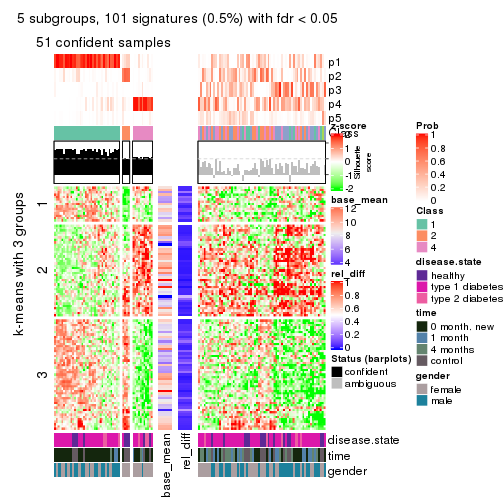
get_signatures(res, k = 6)
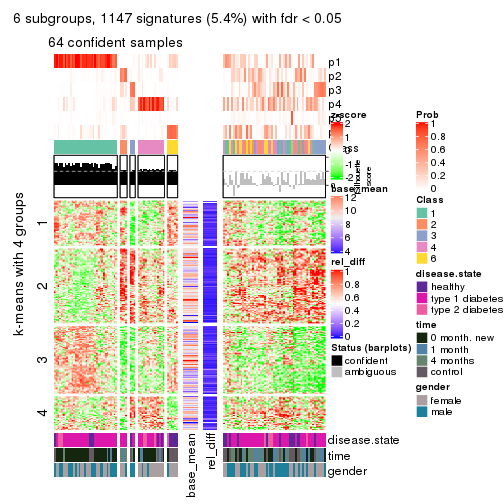
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
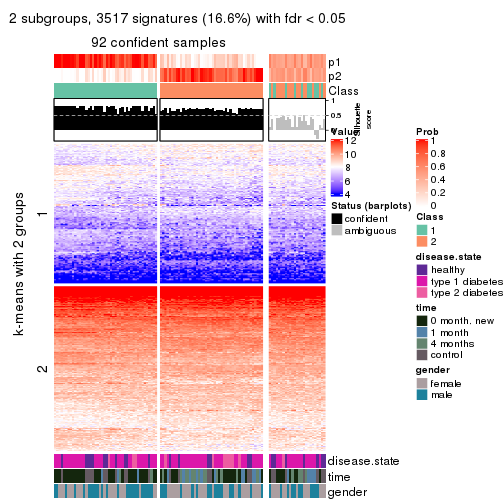
get_signatures(res, k = 3, scale_rows = FALSE)

get_signatures(res, k = 4, scale_rows = FALSE)
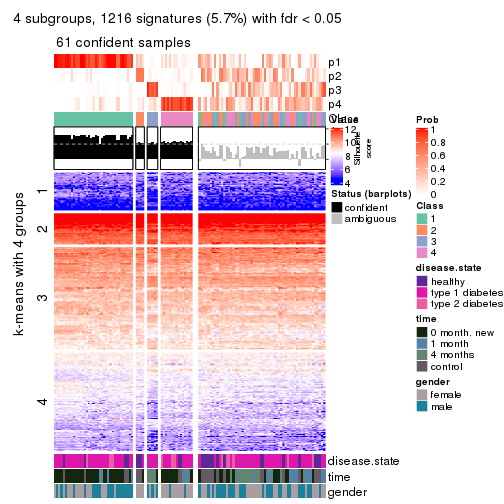
get_signatures(res, k = 5, scale_rows = FALSE)
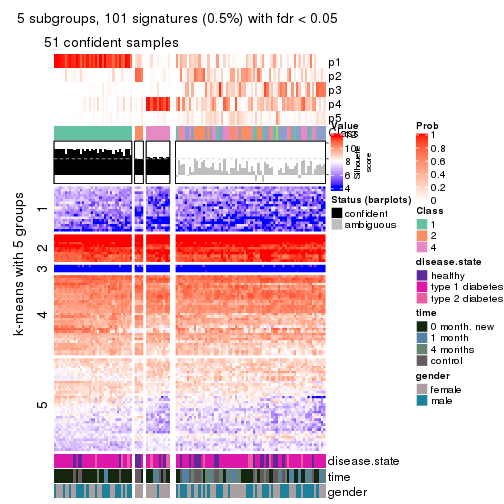
get_signatures(res, k = 6, scale_rows = FALSE)
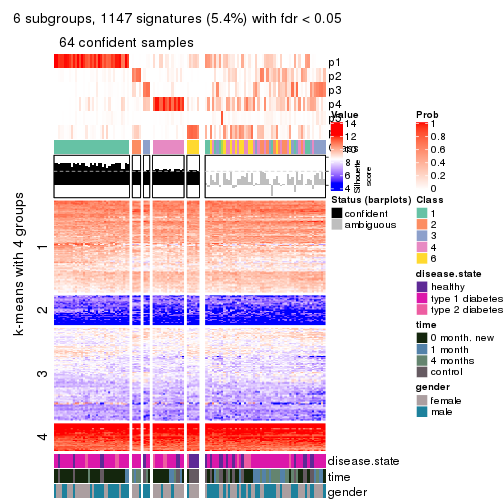
Compare the overlap of signatures from different k:
compare_signatures(res)
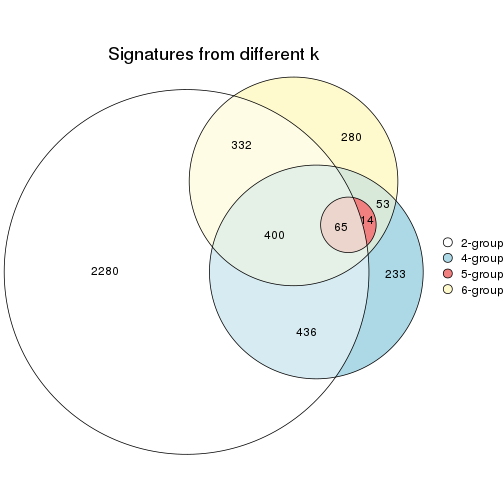
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
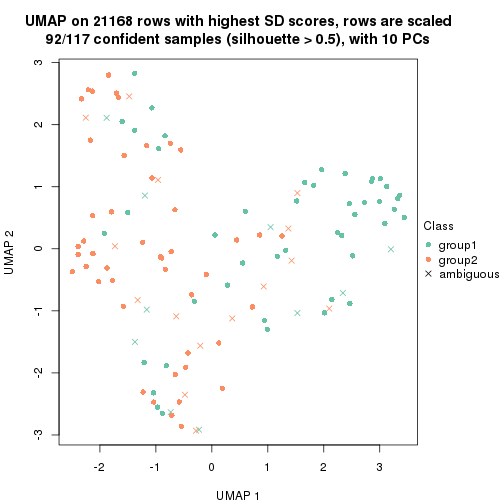
dimension_reduction(res, k = 3, method = "UMAP")
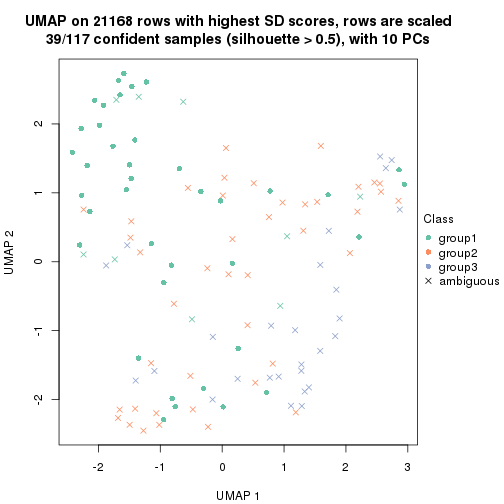
dimension_reduction(res, k = 4, method = "UMAP")
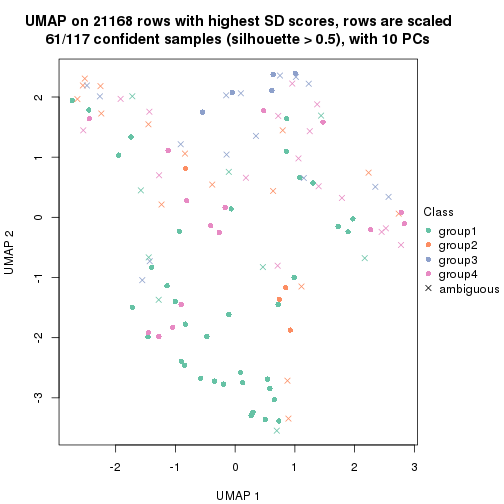
dimension_reduction(res, k = 5, method = "UMAP")
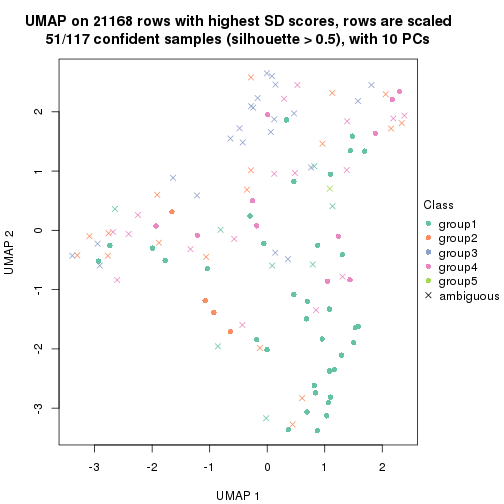
dimension_reduction(res, k = 6, method = "UMAP")
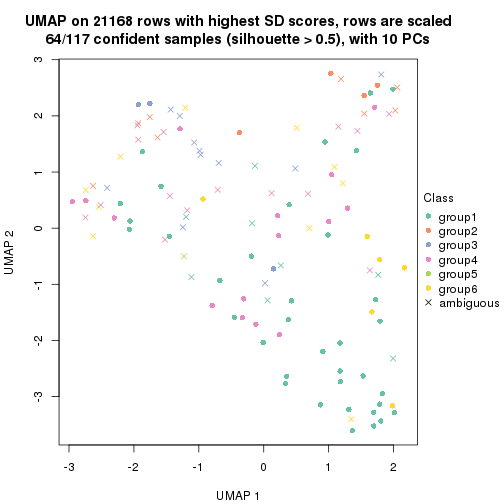
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
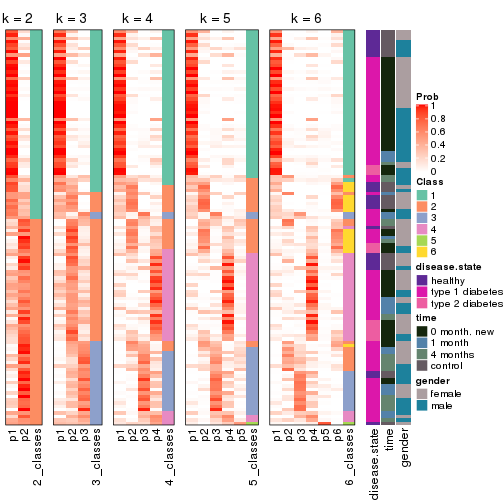
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> SD:pam 92 0.15387 3.69e-04 0.20820 2
#> SD:pam 39 NA NA NA 3
#> SD:pam 61 0.01595 1.08e-06 0.01239 4
#> SD:pam 51 0.00910 3.58e-02 0.00845 5
#> SD:pam 64 0.00291 1.76e-06 0.01102 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["SD", "mclust"]
# you can also extract it by
# res = res_list["SD:mclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'SD' method.
#> Subgroups are detected by 'mclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 4.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
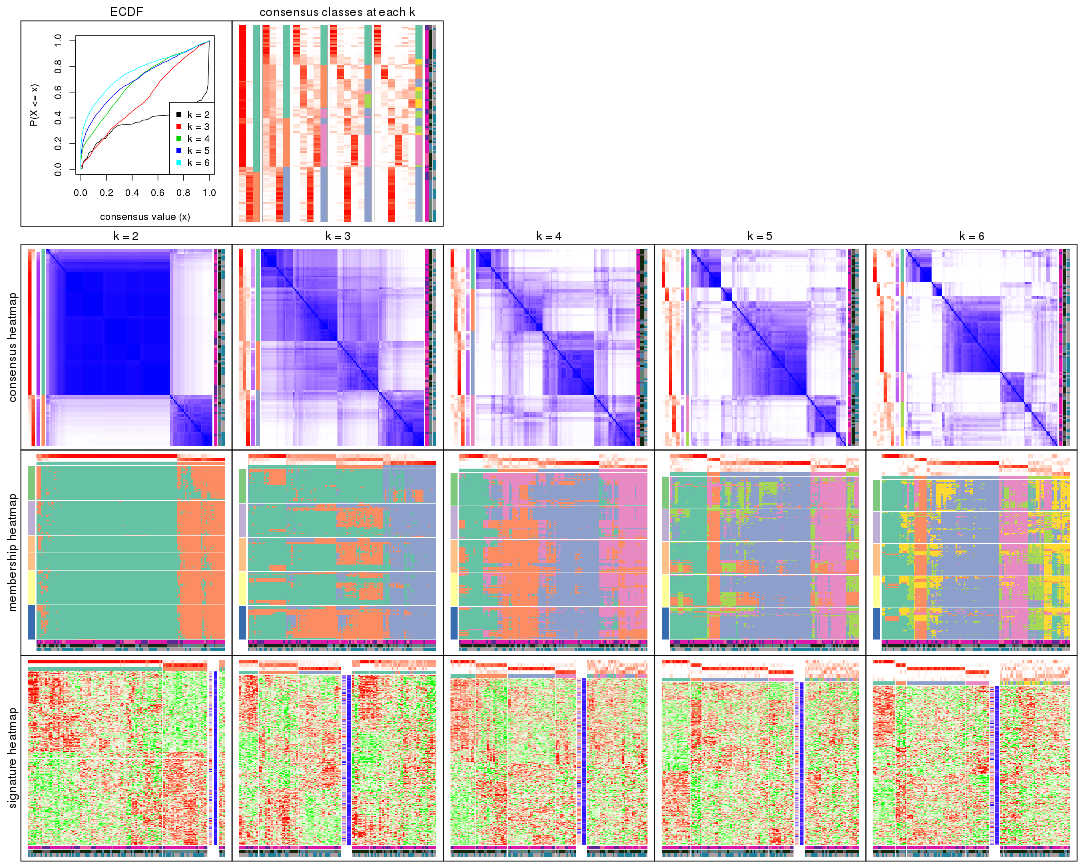
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
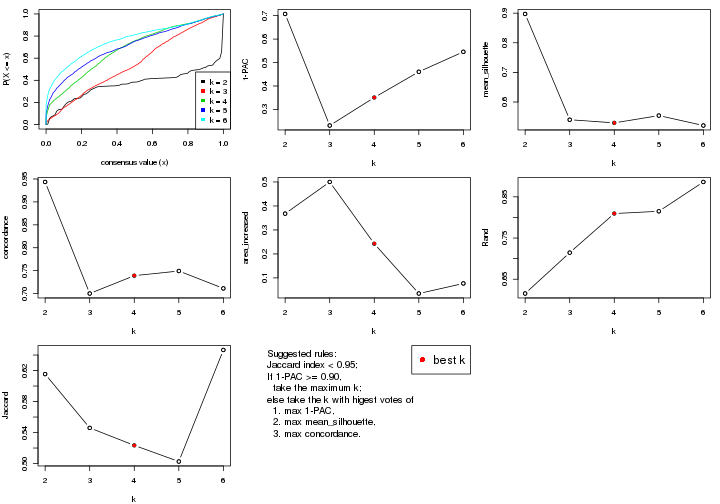
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.707 0.897 0.944 0.3679 0.615 0.615
#> 3 3 0.231 0.540 0.700 0.5000 0.715 0.546
#> 4 4 0.351 0.530 0.739 0.2425 0.809 0.523
#> 5 5 0.461 0.554 0.749 0.0349 0.815 0.503
#> 6 6 0.546 0.521 0.711 0.0769 0.886 0.647
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 4
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.0672 0.9655 0.992 0.008
#> GSM228563 1 0.0672 0.9655 0.992 0.008
#> GSM228565 1 0.0376 0.9661 0.996 0.004
#> GSM228566 2 0.8207 0.7668 0.256 0.744
#> GSM228567 1 0.0376 0.9640 0.996 0.004
#> GSM228570 1 0.0376 0.9661 0.996 0.004
#> GSM228571 1 0.0672 0.9650 0.992 0.008
#> GSM228574 2 0.9977 0.2910 0.472 0.528
#> GSM228575 2 0.8016 0.7853 0.244 0.756
#> GSM228576 1 0.0376 0.9661 0.996 0.004
#> GSM228579 1 0.0376 0.9661 0.996 0.004
#> GSM228580 1 0.0672 0.9655 0.992 0.008
#> GSM228581 1 0.2778 0.9282 0.952 0.048
#> GSM228666 1 0.0376 0.9661 0.996 0.004
#> GSM228564 1 0.0672 0.9655 0.992 0.008
#> GSM228568 1 0.1414 0.9560 0.980 0.020
#> GSM228569 1 0.0672 0.9649 0.992 0.008
#> GSM228572 1 0.0000 0.9658 1.000 0.000
#> GSM228573 2 0.1633 0.8724 0.024 0.976
#> GSM228577 1 0.0000 0.9658 1.000 0.000
#> GSM228578 1 0.0376 0.9661 0.996 0.004
#> GSM228663 1 0.9977 -0.0568 0.528 0.472
#> GSM228664 1 0.8207 0.6197 0.744 0.256
#> GSM228665 2 0.6048 0.8606 0.148 0.852
#> GSM228582 1 0.0938 0.9622 0.988 0.012
#> GSM228583 1 0.0376 0.9640 0.996 0.004
#> GSM228585 1 0.0376 0.9640 0.996 0.004
#> GSM228587 1 0.0000 0.9658 1.000 0.000
#> GSM228588 1 0.0376 0.9661 0.996 0.004
#> GSM228589 1 0.0376 0.9661 0.996 0.004
#> GSM228590 1 0.0376 0.9640 0.996 0.004
#> GSM228591 1 0.0000 0.9658 1.000 0.000
#> GSM228597 1 0.0376 0.9657 0.996 0.004
#> GSM228601 1 0.0000 0.9658 1.000 0.000
#> GSM228604 1 0.2236 0.9399 0.964 0.036
#> GSM228608 1 0.0376 0.9661 0.996 0.004
#> GSM228609 1 0.0000 0.9658 1.000 0.000
#> GSM228613 1 0.0376 0.9640 0.996 0.004
#> GSM228616 1 0.0376 0.9661 0.996 0.004
#> GSM228628 1 0.0376 0.9661 0.996 0.004
#> GSM228634 1 0.0376 0.9640 0.996 0.004
#> GSM228642 1 0.0000 0.9658 1.000 0.000
#> GSM228645 1 0.2778 0.9276 0.952 0.048
#> GSM228646 1 0.1633 0.9519 0.976 0.024
#> GSM228652 1 0.0000 0.9658 1.000 0.000
#> GSM228655 1 0.0376 0.9661 0.996 0.004
#> GSM228656 1 0.0376 0.9640 0.996 0.004
#> GSM228659 1 0.0000 0.9658 1.000 0.000
#> GSM228662 1 0.0376 0.9640 0.996 0.004
#> GSM228584 1 0.0376 0.9640 0.996 0.004
#> GSM228586 1 0.0376 0.9640 0.996 0.004
#> GSM228592 1 0.0376 0.9640 0.996 0.004
#> GSM228593 1 0.0376 0.9661 0.996 0.004
#> GSM228594 1 0.0376 0.9640 0.996 0.004
#> GSM228598 1 0.0376 0.9661 0.996 0.004
#> GSM228607 1 0.3114 0.9203 0.944 0.056
#> GSM228612 1 0.9933 0.0275 0.548 0.452
#> GSM228619 1 0.0376 0.9657 0.996 0.004
#> GSM228622 1 0.0672 0.9655 0.992 0.008
#> GSM228625 1 0.0376 0.9661 0.996 0.004
#> GSM228631 1 0.0376 0.9657 0.996 0.004
#> GSM228633 1 0.0000 0.9658 1.000 0.000
#> GSM228637 1 0.0376 0.9657 0.996 0.004
#> GSM228639 1 0.6973 0.7325 0.812 0.188
#> GSM228649 1 0.0376 0.9661 0.996 0.004
#> GSM228660 1 0.0376 0.9661 0.996 0.004
#> GSM228661 1 0.0672 0.9649 0.992 0.008
#> GSM228595 1 0.0000 0.9658 1.000 0.000
#> GSM228599 1 0.0672 0.9655 0.992 0.008
#> GSM228602 2 0.6048 0.8627 0.148 0.852
#> GSM228614 1 0.0938 0.9634 0.988 0.012
#> GSM228626 1 0.0000 0.9658 1.000 0.000
#> GSM228640 2 0.4690 0.8818 0.100 0.900
#> GSM228643 2 0.7299 0.8221 0.204 0.796
#> GSM228650 2 0.7299 0.8284 0.204 0.796
#> GSM228653 2 0.4298 0.8838 0.088 0.912
#> GSM228657 1 0.0000 0.9658 1.000 0.000
#> GSM228605 1 0.0672 0.9655 0.992 0.008
#> GSM228610 2 0.1184 0.8687 0.016 0.984
#> GSM228617 2 0.7056 0.8310 0.192 0.808
#> GSM228620 2 0.2603 0.8768 0.044 0.956
#> GSM228623 1 0.0376 0.9657 0.996 0.004
#> GSM228629 2 0.0938 0.8664 0.012 0.988
#> GSM228632 2 0.4562 0.8770 0.096 0.904
#> GSM228635 1 0.0376 0.9657 0.996 0.004
#> GSM228647 2 0.0938 0.8664 0.012 0.988
#> GSM228596 1 0.8081 0.6188 0.752 0.248
#> GSM228600 2 0.5294 0.8741 0.120 0.880
#> GSM228603 2 0.3274 0.8818 0.060 0.940
#> GSM228615 1 0.0672 0.9655 0.992 0.008
#> GSM228627 2 0.9795 0.4495 0.416 0.584
#> GSM228641 2 0.2948 0.8807 0.052 0.948
#> GSM228644 1 0.0000 0.9658 1.000 0.000
#> GSM228651 2 0.2948 0.8808 0.052 0.948
#> GSM228654 2 0.3114 0.8820 0.056 0.944
#> GSM228658 2 0.5059 0.8784 0.112 0.888
#> GSM228606 1 0.5408 0.8439 0.876 0.124
#> GSM228611 2 0.1184 0.8688 0.016 0.984
#> GSM228618 2 0.1184 0.8688 0.016 0.984
#> GSM228621 2 0.0938 0.8664 0.012 0.988
#> GSM228624 2 0.7950 0.7642 0.240 0.760
#> GSM228630 1 0.7219 0.7221 0.800 0.200
#> GSM228636 1 0.0376 0.9657 0.996 0.004
#> GSM228638 2 0.4690 0.8791 0.100 0.900
#> GSM228648 2 0.8499 0.7181 0.276 0.724
#> GSM228670 1 0.0672 0.9655 0.992 0.008
#> GSM228671 1 0.4562 0.8721 0.904 0.096
#> GSM228672 1 0.0376 0.9661 0.996 0.004
#> GSM228674 1 0.0672 0.9655 0.992 0.008
#> GSM228675 1 0.0672 0.9655 0.992 0.008
#> GSM228676 1 0.0672 0.9655 0.992 0.008
#> GSM228667 1 0.0672 0.9655 0.992 0.008
#> GSM228668 1 0.0376 0.9657 0.996 0.004
#> GSM228669 1 0.0672 0.9655 0.992 0.008
#> GSM228673 2 0.7745 0.7798 0.228 0.772
#> GSM228677 1 0.0672 0.9655 0.992 0.008
#> GSM228678 1 0.0376 0.9657 0.996 0.004
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 2 0.7340 0.7048 0.248 0.676 0.076
#> GSM228563 2 0.6264 0.6732 0.244 0.724 0.032
#> GSM228565 1 0.8543 0.3322 0.496 0.408 0.096
#> GSM228566 3 0.6835 0.6479 0.088 0.180 0.732
#> GSM228567 1 0.0237 0.4704 0.996 0.004 0.000
#> GSM228570 1 0.7022 0.4569 0.700 0.232 0.068
#> GSM228571 1 0.5901 0.5518 0.776 0.176 0.048
#> GSM228574 3 0.8950 0.4135 0.216 0.216 0.568
#> GSM228575 3 0.6208 0.7048 0.048 0.200 0.752
#> GSM228576 1 0.9042 0.3550 0.544 0.280 0.176
#> GSM228579 1 0.5508 0.5571 0.784 0.188 0.028
#> GSM228580 2 0.6297 0.7426 0.184 0.756 0.060
#> GSM228581 1 0.8419 0.4054 0.504 0.408 0.088
#> GSM228666 1 0.8056 0.3993 0.532 0.400 0.068
#> GSM228564 2 0.6850 0.7415 0.208 0.720 0.072
#> GSM228568 1 0.8143 0.4589 0.560 0.360 0.080
#> GSM228569 1 0.5384 0.5602 0.788 0.188 0.024
#> GSM228572 1 0.6291 0.4362 0.532 0.468 0.000
#> GSM228573 3 0.0829 0.7812 0.012 0.004 0.984
#> GSM228577 1 0.4663 0.5535 0.828 0.156 0.016
#> GSM228578 1 0.8311 0.4386 0.616 0.252 0.132
#> GSM228663 3 0.9830 -0.2009 0.304 0.272 0.424
#> GSM228664 1 0.9901 0.1792 0.404 0.300 0.296
#> GSM228665 3 0.5307 0.7224 0.124 0.056 0.820
#> GSM228582 1 0.7841 0.4517 0.536 0.408 0.056
#> GSM228583 1 0.0237 0.4704 0.996 0.004 0.000
#> GSM228585 1 0.0237 0.4704 0.996 0.004 0.000
#> GSM228587 1 0.3752 0.5410 0.856 0.144 0.000
#> GSM228588 1 0.6476 0.4496 0.548 0.448 0.004
#> GSM228589 1 0.6476 0.4496 0.548 0.448 0.004
#> GSM228590 1 0.0592 0.4728 0.988 0.012 0.000
#> GSM228591 1 0.7164 0.4489 0.524 0.452 0.024
#> GSM228597 2 0.5826 0.7095 0.204 0.764 0.032
#> GSM228601 1 0.6267 0.4505 0.548 0.452 0.000
#> GSM228604 1 0.8288 0.3985 0.512 0.408 0.080
#> GSM228608 1 0.5330 0.5374 0.812 0.144 0.044
#> GSM228609 1 0.6483 0.4469 0.544 0.452 0.004
#> GSM228613 1 0.1163 0.4815 0.972 0.028 0.000
#> GSM228616 1 0.8257 0.4100 0.544 0.372 0.084
#> GSM228628 1 0.8100 0.4051 0.512 0.420 0.068
#> GSM228634 1 0.1643 0.5008 0.956 0.044 0.000
#> GSM228642 1 0.6809 0.4426 0.524 0.464 0.012
#> GSM228645 1 0.9390 0.2939 0.488 0.320 0.192
#> GSM228646 1 0.9331 0.2951 0.480 0.344 0.176
#> GSM228652 1 0.6476 0.5384 0.748 0.184 0.068
#> GSM228655 1 0.7970 0.4756 0.612 0.300 0.088
#> GSM228656 1 0.0424 0.4736 0.992 0.008 0.000
#> GSM228659 1 0.5623 0.5455 0.716 0.280 0.004
#> GSM228662 1 0.1031 0.4785 0.976 0.024 0.000
#> GSM228584 1 0.0237 0.4704 0.996 0.004 0.000
#> GSM228586 1 0.1163 0.4913 0.972 0.028 0.000
#> GSM228592 1 0.0237 0.4704 0.996 0.004 0.000
#> GSM228593 1 0.6633 0.4507 0.548 0.444 0.008
#> GSM228594 1 0.3752 0.5468 0.856 0.144 0.000
#> GSM228598 1 0.3349 0.5318 0.888 0.108 0.004
#> GSM228607 1 0.9601 0.1959 0.476 0.252 0.272
#> GSM228612 3 0.9654 -0.1123 0.320 0.228 0.452
#> GSM228619 2 0.7605 0.7156 0.252 0.660 0.088
#> GSM228622 2 0.9176 0.5005 0.344 0.496 0.160
#> GSM228625 1 0.7990 0.4045 0.532 0.404 0.064
#> GSM228631 1 0.9457 -0.0687 0.468 0.340 0.192
#> GSM228633 1 0.6295 0.4363 0.528 0.472 0.000
#> GSM228637 2 0.6828 0.4833 0.312 0.656 0.032
#> GSM228639 2 0.9277 0.5195 0.176 0.496 0.328
#> GSM228649 1 0.7029 0.4387 0.540 0.440 0.020
#> GSM228660 1 0.7699 0.4405 0.560 0.388 0.052
#> GSM228661 1 0.5355 0.5567 0.804 0.160 0.036
#> GSM228595 1 0.6295 0.4363 0.528 0.472 0.000
#> GSM228599 2 0.6850 0.7415 0.208 0.720 0.072
#> GSM228602 3 0.5093 0.7373 0.088 0.076 0.836
#> GSM228614 2 0.7872 0.7107 0.236 0.652 0.112
#> GSM228626 1 0.6286 0.4434 0.536 0.464 0.000
#> GSM228640 3 0.3618 0.7799 0.012 0.104 0.884
#> GSM228643 3 0.6191 0.6824 0.084 0.140 0.776
#> GSM228650 3 0.6968 0.5996 0.120 0.148 0.732
#> GSM228653 3 0.3502 0.7794 0.084 0.020 0.896
#> GSM228657 1 0.6291 0.4373 0.532 0.468 0.000
#> GSM228605 2 0.9009 0.6193 0.236 0.560 0.204
#> GSM228610 3 0.0424 0.7759 0.000 0.008 0.992
#> GSM228617 3 0.5585 0.6950 0.096 0.092 0.812
#> GSM228620 3 0.0829 0.7785 0.004 0.012 0.984
#> GSM228623 2 0.7705 0.4306 0.348 0.592 0.060
#> GSM228629 3 0.0424 0.7789 0.008 0.000 0.992
#> GSM228632 3 0.4058 0.7629 0.044 0.076 0.880
#> GSM228635 2 0.5406 0.6738 0.200 0.780 0.020
#> GSM228647 3 0.0000 0.7759 0.000 0.000 1.000
#> GSM228596 3 0.9340 -0.0705 0.192 0.308 0.500
#> GSM228600 3 0.3918 0.7741 0.012 0.120 0.868
#> GSM228603 3 0.2860 0.7736 0.004 0.084 0.912
#> GSM228615 2 0.5298 0.7044 0.164 0.804 0.032
#> GSM228627 3 0.9276 0.3107 0.264 0.212 0.524
#> GSM228641 3 0.2866 0.7765 0.008 0.076 0.916
#> GSM228644 1 0.6291 0.4398 0.532 0.468 0.000
#> GSM228651 3 0.2682 0.7745 0.004 0.076 0.920
#> GSM228654 3 0.3765 0.7802 0.028 0.084 0.888
#> GSM228658 3 0.5375 0.7381 0.128 0.056 0.816
#> GSM228606 2 0.9136 0.3370 0.144 0.456 0.400
#> GSM228611 3 0.0000 0.7759 0.000 0.000 1.000
#> GSM228618 3 0.0237 0.7760 0.000 0.004 0.996
#> GSM228621 3 0.0829 0.7790 0.004 0.012 0.984
#> GSM228624 3 0.6148 0.6597 0.148 0.076 0.776
#> GSM228630 2 0.8661 0.5248 0.116 0.536 0.348
#> GSM228636 2 0.5414 0.6582 0.212 0.772 0.016
#> GSM228638 3 0.3181 0.7737 0.024 0.064 0.912
#> GSM228648 3 0.8716 0.2674 0.240 0.172 0.588
#> GSM228670 2 0.6349 0.7365 0.156 0.764 0.080
#> GSM228671 2 0.9187 0.5705 0.196 0.532 0.272
#> GSM228672 2 0.7180 0.6562 0.268 0.672 0.060
#> GSM228674 2 0.6087 0.7283 0.144 0.780 0.076
#> GSM228675 2 0.6646 0.7499 0.184 0.740 0.076
#> GSM228676 2 0.8137 0.7012 0.220 0.640 0.140
#> GSM228667 2 0.8725 0.0423 0.416 0.476 0.108
#> GSM228668 2 0.8122 0.6579 0.292 0.608 0.100
#> GSM228669 2 0.6562 0.7474 0.184 0.744 0.072
#> GSM228673 3 0.4658 0.7475 0.068 0.076 0.856
#> GSM228677 2 0.7222 0.7355 0.220 0.696 0.084
#> GSM228678 2 0.6354 0.7378 0.204 0.744 0.052
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.6929 0.4445 0.120 0.024 0.216 0.640
#> GSM228563 4 0.3681 0.5225 0.000 0.176 0.008 0.816
#> GSM228565 4 0.9098 0.1515 0.112 0.148 0.340 0.400
#> GSM228566 3 0.3721 0.7869 0.008 0.072 0.864 0.056
#> GSM228567 1 0.0804 0.6719 0.980 0.012 0.000 0.008
#> GSM228570 1 0.7931 0.3268 0.496 0.064 0.084 0.356
#> GSM228571 1 0.8916 0.3277 0.448 0.296 0.164 0.092
#> GSM228574 3 0.5076 0.6256 0.004 0.260 0.712 0.024
#> GSM228575 3 0.4149 0.7736 0.004 0.072 0.836 0.088
#> GSM228576 4 0.9354 0.0908 0.140 0.152 0.348 0.360
#> GSM228579 1 0.8325 0.3545 0.480 0.324 0.140 0.056
#> GSM228580 4 0.4621 0.3790 0.000 0.284 0.008 0.708
#> GSM228581 2 0.5471 0.5924 0.004 0.720 0.216 0.060
#> GSM228666 2 0.6739 0.5479 0.004 0.624 0.224 0.148
#> GSM228564 4 0.3105 0.5657 0.000 0.120 0.012 0.868
#> GSM228568 2 0.8817 0.3886 0.176 0.484 0.248 0.092
#> GSM228569 1 0.8596 0.2759 0.440 0.324 0.184 0.052
#> GSM228572 2 0.4252 0.6069 0.004 0.744 0.000 0.252
#> GSM228573 3 0.0469 0.8064 0.000 0.012 0.988 0.000
#> GSM228577 1 0.7102 0.5166 0.604 0.284 0.052 0.060
#> GSM228578 4 0.9241 0.1475 0.220 0.088 0.336 0.356
#> GSM228663 3 0.5576 -0.0102 0.000 0.444 0.536 0.020
#> GSM228664 2 0.5286 0.5414 0.004 0.652 0.328 0.016
#> GSM228665 3 0.2775 0.7893 0.000 0.084 0.896 0.020
#> GSM228582 2 0.6039 0.5867 0.036 0.712 0.200 0.052
#> GSM228583 1 0.0188 0.6651 0.996 0.000 0.000 0.004
#> GSM228585 1 0.0188 0.6651 0.996 0.000 0.000 0.004
#> GSM228587 1 0.5721 0.5675 0.660 0.284 0.000 0.056
#> GSM228588 2 0.4655 0.5629 0.004 0.684 0.000 0.312
#> GSM228589 2 0.4222 0.5931 0.000 0.728 0.000 0.272
#> GSM228590 1 0.1209 0.6738 0.964 0.032 0.000 0.004
#> GSM228591 2 0.4351 0.6444 0.004 0.824 0.092 0.080
#> GSM228597 4 0.3895 0.5100 0.000 0.184 0.012 0.804
#> GSM228601 2 0.3873 0.6210 0.000 0.772 0.000 0.228
#> GSM228604 2 0.6301 0.5769 0.008 0.668 0.224 0.100
#> GSM228608 1 0.6543 0.4846 0.616 0.048 0.028 0.308
#> GSM228609 2 0.4855 0.5195 0.004 0.644 0.000 0.352
#> GSM228613 1 0.0927 0.6705 0.976 0.008 0.000 0.016
#> GSM228616 2 0.9259 0.1991 0.084 0.368 0.252 0.296
#> GSM228628 2 0.5032 0.6223 0.004 0.772 0.152 0.072
#> GSM228634 1 0.3659 0.6693 0.840 0.136 0.000 0.024
#> GSM228642 2 0.2944 0.6367 0.000 0.868 0.004 0.128
#> GSM228645 2 0.7500 0.0406 0.000 0.412 0.408 0.180
#> GSM228646 2 0.7164 0.1092 0.004 0.464 0.416 0.116
#> GSM228652 1 0.7927 0.5225 0.556 0.180 0.040 0.224
#> GSM228655 1 0.9886 0.0678 0.300 0.240 0.184 0.276
#> GSM228656 1 0.0524 0.6685 0.988 0.008 0.000 0.004
#> GSM228659 1 0.8022 0.1441 0.388 0.284 0.004 0.324
#> GSM228662 1 0.0804 0.6701 0.980 0.012 0.000 0.008
#> GSM228584 1 0.0188 0.6651 0.996 0.000 0.000 0.004
#> GSM228586 1 0.3552 0.6720 0.848 0.128 0.000 0.024
#> GSM228592 1 0.0188 0.6651 0.996 0.000 0.000 0.004
#> GSM228593 2 0.6162 0.5247 0.076 0.620 0.000 0.304
#> GSM228594 1 0.5745 0.5760 0.656 0.296 0.004 0.044
#> GSM228598 1 0.5393 0.5969 0.688 0.268 0.000 0.044
#> GSM228607 3 0.7091 0.3306 0.000 0.248 0.564 0.188
#> GSM228612 3 0.5626 0.2873 0.004 0.360 0.612 0.024
#> GSM228619 4 0.5806 0.5327 0.092 0.040 0.112 0.756
#> GSM228622 4 0.8081 0.2706 0.168 0.028 0.332 0.472
#> GSM228625 4 0.7314 0.0266 0.112 0.340 0.016 0.532
#> GSM228631 4 0.8714 0.1590 0.284 0.040 0.276 0.400
#> GSM228633 2 0.2814 0.6330 0.000 0.868 0.000 0.132
#> GSM228637 4 0.4722 0.3480 0.000 0.300 0.008 0.692
#> GSM228639 3 0.6179 0.2272 0.000 0.056 0.552 0.392
#> GSM228649 2 0.5150 0.4608 0.008 0.596 0.000 0.396
#> GSM228660 2 0.8280 0.5291 0.104 0.560 0.212 0.124
#> GSM228661 1 0.8728 0.2833 0.456 0.296 0.180 0.068
#> GSM228595 2 0.2973 0.6321 0.000 0.856 0.000 0.144
#> GSM228599 4 0.2799 0.5662 0.000 0.108 0.008 0.884
#> GSM228602 3 0.2670 0.8012 0.000 0.052 0.908 0.040
#> GSM228614 4 0.4285 0.5631 0.000 0.076 0.104 0.820
#> GSM228626 2 0.2704 0.6317 0.000 0.876 0.000 0.124
#> GSM228640 3 0.2457 0.8032 0.004 0.076 0.912 0.008
#> GSM228643 3 0.3414 0.7881 0.004 0.072 0.876 0.048
#> GSM228650 3 0.3749 0.7515 0.000 0.032 0.840 0.128
#> GSM228653 3 0.1004 0.8080 0.000 0.024 0.972 0.004
#> GSM228657 2 0.4103 0.6063 0.000 0.744 0.000 0.256
#> GSM228605 4 0.6377 0.2290 0.040 0.016 0.376 0.568
#> GSM228610 3 0.0592 0.8027 0.000 0.016 0.984 0.000
#> GSM228617 3 0.2101 0.7970 0.000 0.012 0.928 0.060
#> GSM228620 3 0.0524 0.8044 0.000 0.004 0.988 0.008
#> GSM228623 4 0.4121 0.5303 0.000 0.184 0.020 0.796
#> GSM228629 3 0.0000 0.8032 0.000 0.000 1.000 0.000
#> GSM228632 3 0.1929 0.8033 0.000 0.024 0.940 0.036
#> GSM228635 4 0.4855 0.2427 0.000 0.352 0.004 0.644
#> GSM228647 3 0.0000 0.8032 0.000 0.000 1.000 0.000
#> GSM228596 3 0.5093 0.4487 0.000 0.012 0.640 0.348
#> GSM228600 3 0.2310 0.8003 0.004 0.068 0.920 0.008
#> GSM228603 3 0.1902 0.7994 0.004 0.064 0.932 0.000
#> GSM228615 4 0.3161 0.5565 0.000 0.124 0.012 0.864
#> GSM228627 3 0.5191 0.5629 0.004 0.292 0.684 0.020
#> GSM228641 3 0.2010 0.8016 0.004 0.060 0.932 0.004
#> GSM228644 2 0.2888 0.6324 0.004 0.872 0.000 0.124
#> GSM228651 3 0.1743 0.8014 0.004 0.056 0.940 0.000
#> GSM228654 3 0.1902 0.8034 0.004 0.064 0.932 0.000
#> GSM228658 3 0.2654 0.7848 0.000 0.108 0.888 0.004
#> GSM228606 3 0.6351 0.3294 0.000 0.080 0.588 0.332
#> GSM228611 3 0.0000 0.8032 0.000 0.000 1.000 0.000
#> GSM228618 3 0.0336 0.8031 0.000 0.008 0.992 0.000
#> GSM228621 3 0.0000 0.8032 0.000 0.000 1.000 0.000
#> GSM228624 3 0.3763 0.7266 0.000 0.144 0.832 0.024
#> GSM228630 3 0.7315 0.2503 0.000 0.252 0.532 0.216
#> GSM228636 4 0.5119 0.0198 0.000 0.440 0.004 0.556
#> GSM228638 3 0.1284 0.8034 0.000 0.024 0.964 0.012
#> GSM228648 3 0.4690 0.5232 0.000 0.276 0.712 0.012
#> GSM228670 4 0.2635 0.5781 0.000 0.076 0.020 0.904
#> GSM228671 3 0.6889 0.0675 0.000 0.108 0.496 0.396
#> GSM228672 4 0.3409 0.5695 0.024 0.096 0.008 0.872
#> GSM228674 4 0.2402 0.5745 0.000 0.076 0.012 0.912
#> GSM228675 4 0.2522 0.5773 0.000 0.076 0.016 0.908
#> GSM228676 4 0.6096 0.3682 0.008 0.052 0.308 0.632
#> GSM228667 4 0.7807 0.2770 0.008 0.216 0.296 0.480
#> GSM228668 4 0.7313 0.2175 0.300 0.008 0.148 0.544
#> GSM228669 4 0.3025 0.5768 0.016 0.060 0.024 0.900
#> GSM228673 3 0.2494 0.7981 0.000 0.048 0.916 0.036
#> GSM228677 4 0.7093 0.3546 0.000 0.216 0.216 0.568
#> GSM228678 4 0.4284 0.4711 0.000 0.224 0.012 0.764
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 3 0.7982 0.1868 0.084 0.000 0.368 0.260 0.288
#> GSM228563 4 0.1828 0.6968 0.004 0.028 0.000 0.936 0.032
#> GSM228565 3 0.6718 0.5215 0.032 0.032 0.540 0.056 0.340
#> GSM228566 3 0.3777 0.7026 0.012 0.008 0.820 0.020 0.140
#> GSM228567 1 0.0693 0.7410 0.980 0.012 0.000 0.000 0.008
#> GSM228570 1 0.6881 0.5354 0.576 0.008 0.092 0.068 0.256
#> GSM228571 1 0.8427 0.2441 0.440 0.156 0.156 0.020 0.228
#> GSM228574 3 0.4432 0.7036 0.000 0.080 0.788 0.020 0.112
#> GSM228575 3 0.2740 0.7300 0.000 0.000 0.876 0.028 0.096
#> GSM228576 3 0.6873 0.4419 0.040 0.024 0.480 0.060 0.396
#> GSM228579 1 0.8038 0.3400 0.484 0.172 0.108 0.016 0.220
#> GSM228580 4 0.2068 0.6929 0.000 0.092 0.000 0.904 0.004
#> GSM228581 5 0.7367 0.4216 0.004 0.228 0.284 0.032 0.452
#> GSM228666 3 0.7691 0.1678 0.000 0.260 0.452 0.080 0.208
#> GSM228564 4 0.2774 0.6942 0.008 0.080 0.004 0.888 0.020
#> GSM228568 3 0.8717 -0.0867 0.132 0.204 0.428 0.040 0.196
#> GSM228569 1 0.7908 0.3162 0.484 0.184 0.216 0.008 0.108
#> GSM228572 2 0.4114 0.5738 0.000 0.712 0.000 0.272 0.016
#> GSM228573 3 0.0613 0.7348 0.000 0.004 0.984 0.004 0.008
#> GSM228577 1 0.5642 0.6528 0.708 0.176 0.040 0.012 0.064
#> GSM228578 3 0.6483 0.5749 0.056 0.036 0.636 0.044 0.228
#> GSM228663 3 0.5496 0.5340 0.000 0.192 0.696 0.036 0.076
#> GSM228664 3 0.6636 0.2775 0.000 0.240 0.576 0.040 0.144
#> GSM228665 3 0.2244 0.7367 0.000 0.024 0.920 0.016 0.040
#> GSM228582 5 0.7065 0.4464 0.024 0.224 0.148 0.032 0.572
#> GSM228583 1 0.0000 0.7371 1.000 0.000 0.000 0.000 0.000
#> GSM228585 1 0.0000 0.7371 1.000 0.000 0.000 0.000 0.000
#> GSM228587 1 0.4785 0.6585 0.756 0.160 0.000 0.036 0.048
#> GSM228588 5 0.6708 0.1946 0.004 0.248 0.000 0.280 0.468
#> GSM228589 5 0.6569 0.1829 0.000 0.256 0.000 0.272 0.472
#> GSM228590 1 0.0162 0.7390 0.996 0.004 0.000 0.000 0.000
#> GSM228591 5 0.5774 0.2288 0.000 0.340 0.036 0.040 0.584
#> GSM228597 4 0.1012 0.6962 0.000 0.012 0.000 0.968 0.020
#> GSM228601 2 0.6482 0.0815 0.000 0.468 0.000 0.200 0.332
#> GSM228604 3 0.7006 0.2373 0.000 0.356 0.476 0.056 0.112
#> GSM228608 1 0.4909 0.6528 0.716 0.000 0.016 0.052 0.216
#> GSM228609 4 0.7005 -0.2512 0.012 0.236 0.000 0.404 0.348
#> GSM228613 1 0.0162 0.7390 0.996 0.004 0.000 0.000 0.000
#> GSM228616 3 0.8736 0.0146 0.052 0.180 0.376 0.088 0.304
#> GSM228628 5 0.7568 0.3800 0.000 0.296 0.220 0.056 0.428
#> GSM228634 1 0.3341 0.7258 0.840 0.128 0.000 0.008 0.024
#> GSM228642 2 0.2278 0.7529 0.000 0.908 0.000 0.060 0.032
#> GSM228645 3 0.7015 0.4518 0.004 0.172 0.532 0.036 0.256
#> GSM228646 3 0.7244 0.4288 0.004 0.200 0.528 0.052 0.216
#> GSM228652 1 0.5575 0.6579 0.692 0.024 0.036 0.028 0.220
#> GSM228655 3 0.8817 0.2921 0.172 0.096 0.464 0.112 0.156
#> GSM228656 1 0.0000 0.7371 1.000 0.000 0.000 0.000 0.000
#> GSM228659 1 0.7601 0.2504 0.492 0.164 0.000 0.240 0.104
#> GSM228662 1 0.0162 0.7390 0.996 0.004 0.000 0.000 0.000
#> GSM228584 1 0.0451 0.7387 0.988 0.004 0.008 0.000 0.000
#> GSM228586 1 0.3163 0.7339 0.860 0.108 0.008 0.004 0.020
#> GSM228592 1 0.0579 0.7387 0.984 0.008 0.008 0.000 0.000
#> GSM228593 4 0.7628 -0.2780 0.056 0.216 0.000 0.376 0.352
#> GSM228594 1 0.4959 0.6757 0.736 0.176 0.008 0.008 0.072
#> GSM228598 1 0.4927 0.6802 0.748 0.172 0.012 0.016 0.052
#> GSM228607 3 0.5249 0.6453 0.008 0.132 0.748 0.064 0.048
#> GSM228612 3 0.4702 0.6074 0.000 0.172 0.752 0.020 0.056
#> GSM228619 4 0.8250 0.1250 0.068 0.048 0.284 0.452 0.148
#> GSM228622 3 0.6723 0.5763 0.044 0.032 0.632 0.096 0.196
#> GSM228625 4 0.9274 -0.2051 0.092 0.204 0.116 0.372 0.216
#> GSM228631 3 0.7866 0.3079 0.248 0.012 0.456 0.064 0.220
#> GSM228633 2 0.1478 0.7832 0.000 0.936 0.000 0.064 0.000
#> GSM228637 4 0.2900 0.6867 0.000 0.108 0.000 0.864 0.028
#> GSM228639 3 0.5023 0.6638 0.004 0.068 0.752 0.144 0.032
#> GSM228649 5 0.6927 0.1539 0.008 0.236 0.000 0.376 0.380
#> GSM228660 5 0.9104 0.3918 0.100 0.216 0.272 0.068 0.344
#> GSM228661 1 0.7648 0.2874 0.488 0.180 0.260 0.012 0.060
#> GSM228595 2 0.1478 0.7832 0.000 0.936 0.000 0.064 0.000
#> GSM228599 4 0.1721 0.6990 0.000 0.016 0.020 0.944 0.020
#> GSM228602 3 0.1644 0.7364 0.000 0.004 0.940 0.008 0.048
#> GSM228614 4 0.5184 0.3919 0.000 0.060 0.224 0.696 0.020
#> GSM228626 2 0.1557 0.7755 0.000 0.940 0.000 0.052 0.008
#> GSM228640 3 0.2645 0.7275 0.000 0.008 0.884 0.012 0.096
#> GSM228643 3 0.3547 0.6972 0.000 0.016 0.824 0.016 0.144
#> GSM228650 3 0.2178 0.7397 0.000 0.008 0.920 0.048 0.024
#> GSM228653 3 0.0671 0.7364 0.000 0.000 0.980 0.004 0.016
#> GSM228657 2 0.3700 0.6209 0.000 0.752 0.000 0.240 0.008
#> GSM228605 3 0.6467 0.5634 0.032 0.012 0.624 0.120 0.212
#> GSM228610 3 0.0451 0.7331 0.000 0.000 0.988 0.004 0.008
#> GSM228617 3 0.1200 0.7368 0.000 0.008 0.964 0.016 0.012
#> GSM228620 3 0.0324 0.7341 0.000 0.000 0.992 0.004 0.004
#> GSM228623 4 0.4843 0.5777 0.004 0.128 0.092 0.760 0.016
#> GSM228629 3 0.0324 0.7326 0.000 0.004 0.992 0.000 0.004
#> GSM228632 3 0.1200 0.7376 0.000 0.008 0.964 0.012 0.016
#> GSM228635 4 0.3608 0.6564 0.000 0.148 0.000 0.812 0.040
#> GSM228647 3 0.0162 0.7323 0.000 0.004 0.996 0.000 0.000
#> GSM228596 3 0.4060 0.7241 0.008 0.012 0.820 0.100 0.060
#> GSM228600 3 0.2548 0.7165 0.000 0.004 0.876 0.004 0.116
#> GSM228603 3 0.2052 0.7293 0.000 0.004 0.912 0.004 0.080
#> GSM228615 4 0.0566 0.6942 0.000 0.004 0.000 0.984 0.012
#> GSM228627 3 0.5336 0.6325 0.000 0.132 0.712 0.020 0.136
#> GSM228641 3 0.1928 0.7311 0.000 0.004 0.920 0.004 0.072
#> GSM228644 2 0.1502 0.7789 0.000 0.940 0.000 0.056 0.004
#> GSM228651 3 0.1928 0.7314 0.000 0.004 0.920 0.004 0.072
#> GSM228654 3 0.2284 0.7298 0.000 0.004 0.896 0.004 0.096
#> GSM228658 3 0.2124 0.7402 0.000 0.028 0.916 0.000 0.056
#> GSM228606 3 0.3702 0.7154 0.000 0.036 0.840 0.092 0.032
#> GSM228611 3 0.0162 0.7323 0.000 0.004 0.996 0.000 0.000
#> GSM228618 3 0.0290 0.7328 0.000 0.008 0.992 0.000 0.000
#> GSM228621 3 0.0290 0.7333 0.000 0.000 0.992 0.008 0.000
#> GSM228624 3 0.2703 0.7254 0.000 0.060 0.896 0.020 0.024
#> GSM228630 3 0.5021 0.6428 0.000 0.128 0.744 0.104 0.024
#> GSM228636 4 0.3994 0.6165 0.000 0.188 0.000 0.772 0.040
#> GSM228638 3 0.1278 0.7365 0.000 0.004 0.960 0.016 0.020
#> GSM228648 3 0.3272 0.6961 0.000 0.120 0.848 0.016 0.016
#> GSM228670 4 0.0807 0.6992 0.000 0.000 0.012 0.976 0.012
#> GSM228671 3 0.5404 0.6221 0.000 0.088 0.704 0.180 0.028
#> GSM228672 4 0.3804 0.6603 0.020 0.104 0.008 0.836 0.032
#> GSM228674 4 0.0854 0.7002 0.000 0.004 0.008 0.976 0.012
#> GSM228675 4 0.0912 0.7009 0.000 0.000 0.016 0.972 0.012
#> GSM228676 3 0.7674 0.3807 0.024 0.028 0.460 0.240 0.248
#> GSM228667 3 0.8491 0.2765 0.024 0.120 0.428 0.224 0.204
#> GSM228668 3 0.8788 0.0644 0.252 0.012 0.328 0.196 0.212
#> GSM228669 4 0.4525 0.6440 0.016 0.052 0.024 0.800 0.108
#> GSM228673 3 0.1518 0.7393 0.000 0.020 0.952 0.016 0.012
#> GSM228677 4 0.6678 0.0372 0.000 0.128 0.376 0.472 0.024
#> GSM228678 4 0.1444 0.7023 0.000 0.040 0.000 0.948 0.012
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 5 0.7899 0.26611 0.076 0.000 0.176 0.176 0.456 0.116
#> GSM228563 4 0.2849 0.75030 0.004 0.028 0.000 0.876 0.068 0.024
#> GSM228565 5 0.6756 0.10951 0.008 0.000 0.348 0.028 0.388 0.228
#> GSM228566 3 0.2980 0.73171 0.000 0.012 0.808 0.000 0.000 0.180
#> GSM228567 1 0.0632 0.72208 0.976 0.000 0.000 0.000 0.000 0.024
#> GSM228570 1 0.6445 0.18023 0.432 0.000 0.040 0.032 0.424 0.072
#> GSM228571 6 0.6516 0.10706 0.368 0.004 0.116 0.000 0.060 0.452
#> GSM228574 3 0.3222 0.76116 0.000 0.012 0.824 0.000 0.024 0.140
#> GSM228575 3 0.2418 0.78657 0.000 0.008 0.884 0.004 0.008 0.096
#> GSM228576 5 0.7176 0.06299 0.024 0.004 0.280 0.024 0.372 0.296
#> GSM228579 6 0.5350 -0.08145 0.416 0.000 0.056 0.004 0.016 0.508
#> GSM228580 4 0.2032 0.76586 0.000 0.068 0.004 0.912 0.012 0.004
#> GSM228581 6 0.5346 0.38200 0.000 0.120 0.204 0.020 0.004 0.652
#> GSM228666 3 0.7126 0.00585 0.000 0.132 0.428 0.060 0.032 0.348
#> GSM228564 4 0.3112 0.76219 0.008 0.028 0.000 0.848 0.108 0.008
#> GSM228568 6 0.6863 0.37003 0.068 0.032 0.264 0.016 0.072 0.548
#> GSM228569 6 0.5994 -0.00329 0.376 0.000 0.148 0.000 0.016 0.460
#> GSM228572 2 0.4471 0.61294 0.000 0.684 0.004 0.268 0.028 0.016
#> GSM228573 3 0.0951 0.80364 0.000 0.004 0.968 0.000 0.020 0.008
#> GSM228577 1 0.4373 0.52212 0.624 0.000 0.004 0.000 0.028 0.344
#> GSM228578 3 0.6225 -0.17058 0.044 0.000 0.440 0.020 0.432 0.064
#> GSM228663 3 0.5559 0.35003 0.000 0.080 0.616 0.012 0.024 0.268
#> GSM228664 3 0.6470 -0.19326 0.000 0.140 0.444 0.016 0.024 0.376
#> GSM228665 3 0.2408 0.78608 0.000 0.004 0.892 0.000 0.052 0.052
#> GSM228582 6 0.4148 0.41540 0.012 0.092 0.064 0.016 0.012 0.804
#> GSM228583 1 0.0000 0.72310 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228585 1 0.0000 0.72310 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228587 1 0.4958 0.58473 0.724 0.036 0.000 0.024 0.048 0.168
#> GSM228588 5 0.7618 -0.08891 0.008 0.144 0.000 0.192 0.344 0.312
#> GSM228589 5 0.7459 -0.12260 0.000 0.152 0.000 0.196 0.328 0.324
#> GSM228590 1 0.0000 0.72310 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228591 6 0.4345 0.17151 0.000 0.236 0.016 0.016 0.016 0.716
#> GSM228597 4 0.1391 0.77121 0.000 0.016 0.000 0.944 0.040 0.000
#> GSM228601 2 0.7675 0.22752 0.004 0.328 0.000 0.160 0.276 0.232
#> GSM228604 3 0.6113 0.29737 0.000 0.264 0.524 0.016 0.004 0.192
#> GSM228608 1 0.4858 0.42896 0.584 0.000 0.000 0.020 0.364 0.032
#> GSM228609 5 0.7637 -0.01954 0.012 0.128 0.000 0.252 0.380 0.228
#> GSM228613 1 0.0146 0.72397 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM228616 6 0.7593 0.05935 0.024 0.048 0.152 0.036 0.292 0.448
#> GSM228628 6 0.5296 0.37087 0.000 0.184 0.108 0.016 0.016 0.676
#> GSM228634 1 0.2948 0.66900 0.804 0.000 0.000 0.000 0.008 0.188
#> GSM228642 2 0.2176 0.76226 0.000 0.896 0.000 0.024 0.000 0.080
#> GSM228645 3 0.6213 0.22530 0.000 0.056 0.492 0.016 0.060 0.376
#> GSM228646 3 0.6063 0.31327 0.000 0.080 0.524 0.020 0.028 0.348
#> GSM228652 1 0.5848 0.35458 0.500 0.000 0.004 0.016 0.368 0.112
#> GSM228655 5 0.8591 0.13203 0.164 0.012 0.228 0.080 0.368 0.148
#> GSM228656 1 0.0146 0.72349 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM228659 1 0.8312 -0.01807 0.324 0.052 0.000 0.200 0.256 0.168
#> GSM228662 1 0.0146 0.72397 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM228584 1 0.0000 0.72310 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228586 1 0.2980 0.67225 0.808 0.000 0.000 0.000 0.012 0.180
#> GSM228592 1 0.0363 0.72214 0.988 0.000 0.000 0.000 0.012 0.000
#> GSM228593 5 0.7850 0.02922 0.036 0.116 0.000 0.204 0.396 0.248
#> GSM228594 1 0.4189 0.49414 0.604 0.000 0.000 0.000 0.020 0.376
#> GSM228598 1 0.4411 0.59092 0.692 0.012 0.000 0.004 0.032 0.260
#> GSM228607 3 0.4412 0.70963 0.000 0.024 0.784 0.028 0.080 0.084
#> GSM228612 3 0.4832 0.50868 0.000 0.060 0.684 0.000 0.028 0.228
#> GSM228619 5 0.6867 0.06848 0.068 0.004 0.124 0.352 0.444 0.008
#> GSM228622 3 0.6106 -0.14174 0.028 0.004 0.448 0.056 0.440 0.024
#> GSM228625 5 0.8217 0.14445 0.068 0.052 0.040 0.244 0.428 0.168
#> GSM228631 5 0.7673 0.15847 0.232 0.004 0.308 0.044 0.364 0.048
#> GSM228633 2 0.0914 0.79796 0.000 0.968 0.000 0.016 0.000 0.016
#> GSM228637 4 0.3275 0.73903 0.000 0.044 0.000 0.828 0.120 0.008
#> GSM228639 3 0.4401 0.68539 0.008 0.036 0.776 0.092 0.088 0.000
#> GSM228649 5 0.7486 0.04673 0.008 0.108 0.000 0.256 0.380 0.248
#> GSM228660 6 0.6838 0.35615 0.044 0.052 0.172 0.040 0.080 0.612
#> GSM228661 1 0.6281 0.02227 0.432 0.000 0.192 0.000 0.020 0.356
#> GSM228595 2 0.0914 0.79796 0.000 0.968 0.000 0.016 0.000 0.016
#> GSM228599 4 0.2579 0.76890 0.000 0.008 0.008 0.876 0.100 0.008
#> GSM228602 3 0.1887 0.80254 0.000 0.016 0.924 0.000 0.012 0.048
#> GSM228614 4 0.5555 0.40926 0.000 0.012 0.180 0.628 0.172 0.008
#> GSM228626 2 0.1003 0.79683 0.000 0.964 0.000 0.016 0.000 0.020
#> GSM228640 3 0.2454 0.77920 0.000 0.016 0.876 0.000 0.004 0.104
#> GSM228643 3 0.2955 0.73162 0.000 0.008 0.816 0.000 0.004 0.172
#> GSM228650 3 0.1337 0.80668 0.000 0.016 0.956 0.008 0.008 0.012
#> GSM228653 3 0.0870 0.80469 0.000 0.004 0.972 0.000 0.012 0.012
#> GSM228657 2 0.3791 0.67546 0.000 0.732 0.000 0.236 0.000 0.032
#> GSM228605 5 0.6149 0.16623 0.012 0.004 0.412 0.096 0.456 0.020
#> GSM228610 3 0.0692 0.80280 0.000 0.004 0.976 0.000 0.020 0.000
#> GSM228617 3 0.1536 0.80087 0.004 0.016 0.940 0.000 0.040 0.000
#> GSM228620 3 0.0458 0.80293 0.000 0.000 0.984 0.000 0.016 0.000
#> GSM228623 4 0.4774 0.67892 0.008 0.048 0.044 0.760 0.124 0.016
#> GSM228629 3 0.0508 0.80261 0.000 0.004 0.984 0.000 0.012 0.000
#> GSM228632 3 0.0436 0.80335 0.000 0.004 0.988 0.004 0.004 0.000
#> GSM228635 4 0.3817 0.70559 0.000 0.088 0.000 0.796 0.104 0.012
#> GSM228647 3 0.0291 0.80234 0.000 0.004 0.992 0.000 0.004 0.000
#> GSM228596 3 0.3704 0.77402 0.008 0.008 0.836 0.028 0.068 0.052
#> GSM228600 3 0.2631 0.76563 0.000 0.012 0.856 0.000 0.004 0.128
#> GSM228603 3 0.2215 0.79144 0.000 0.012 0.900 0.000 0.012 0.076
#> GSM228615 4 0.1010 0.77004 0.000 0.004 0.000 0.960 0.036 0.000
#> GSM228627 3 0.4158 0.67634 0.000 0.016 0.736 0.004 0.028 0.216
#> GSM228641 3 0.1829 0.79675 0.000 0.012 0.920 0.000 0.004 0.064
#> GSM228644 2 0.0914 0.79796 0.000 0.968 0.000 0.016 0.000 0.016
#> GSM228651 3 0.1888 0.79631 0.000 0.004 0.916 0.000 0.012 0.068
#> GSM228654 3 0.2060 0.79036 0.000 0.000 0.900 0.000 0.016 0.084
#> GSM228658 3 0.1552 0.80672 0.000 0.004 0.940 0.000 0.020 0.036
#> GSM228606 3 0.2669 0.78095 0.000 0.012 0.888 0.036 0.056 0.008
#> GSM228611 3 0.0508 0.80184 0.000 0.004 0.984 0.000 0.012 0.000
#> GSM228618 3 0.0622 0.80342 0.000 0.012 0.980 0.000 0.008 0.000
#> GSM228621 3 0.0000 0.80254 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228624 3 0.1777 0.79572 0.000 0.004 0.928 0.000 0.024 0.044
#> GSM228630 3 0.3093 0.76692 0.000 0.052 0.868 0.032 0.040 0.008
#> GSM228636 4 0.4225 0.67467 0.000 0.116 0.000 0.764 0.104 0.016
#> GSM228638 3 0.1003 0.80100 0.000 0.004 0.964 0.004 0.028 0.000
#> GSM228648 3 0.1599 0.79576 0.000 0.024 0.940 0.000 0.008 0.028
#> GSM228670 4 0.2006 0.75624 0.000 0.000 0.000 0.892 0.104 0.004
#> GSM228671 3 0.5068 0.62380 0.008 0.044 0.728 0.152 0.056 0.012
#> GSM228672 4 0.5418 0.51767 0.012 0.032 0.000 0.620 0.284 0.052
#> GSM228674 4 0.1753 0.76909 0.000 0.004 0.000 0.912 0.084 0.000
#> GSM228675 4 0.1897 0.77197 0.000 0.000 0.004 0.908 0.084 0.004
#> GSM228676 5 0.7523 0.24359 0.000 0.008 0.240 0.236 0.392 0.124
#> GSM228667 6 0.8061 -0.16481 0.000 0.020 0.256 0.184 0.252 0.288
#> GSM228668 5 0.7182 0.24813 0.188 0.000 0.136 0.156 0.504 0.016
#> GSM228669 4 0.5052 0.29684 0.024 0.004 0.008 0.524 0.428 0.012
#> GSM228673 3 0.1147 0.80487 0.000 0.004 0.960 0.004 0.028 0.004
#> GSM228677 4 0.6413 0.14385 0.004 0.076 0.336 0.512 0.060 0.012
#> GSM228678 4 0.1764 0.77431 0.004 0.024 0.000 0.936 0.024 0.012
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
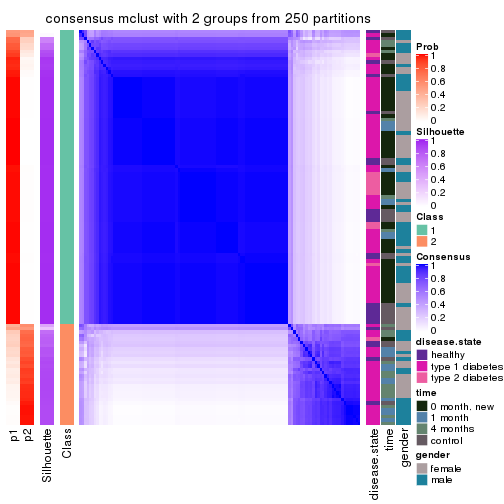
consensus_heatmap(res, k = 3)
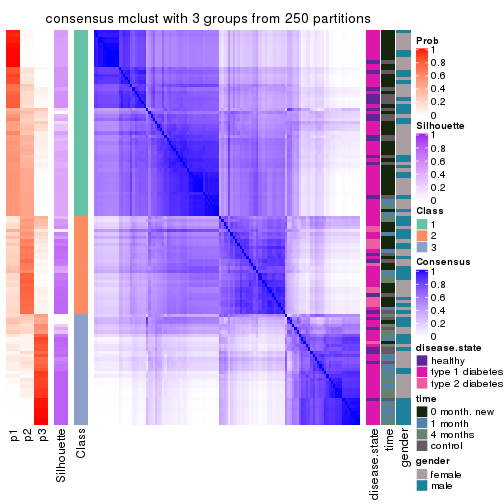
consensus_heatmap(res, k = 4)
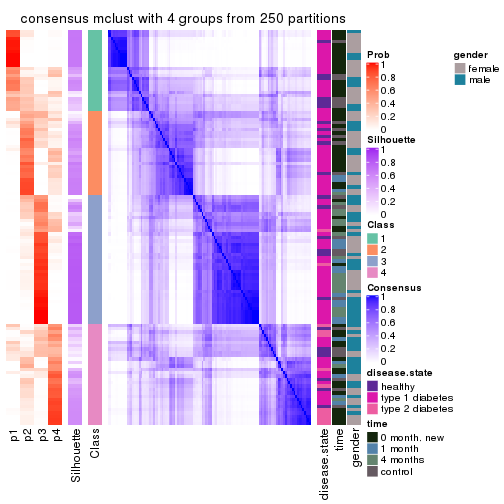
consensus_heatmap(res, k = 5)
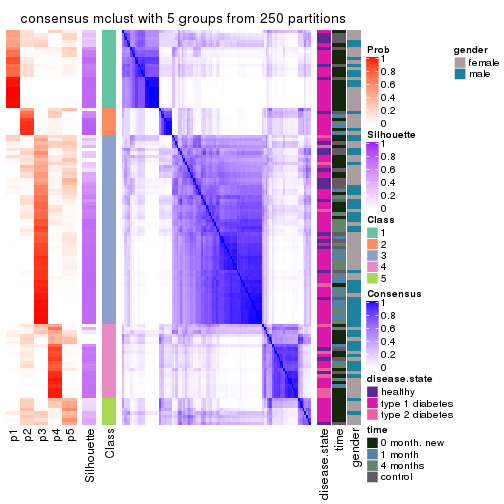
consensus_heatmap(res, k = 6)
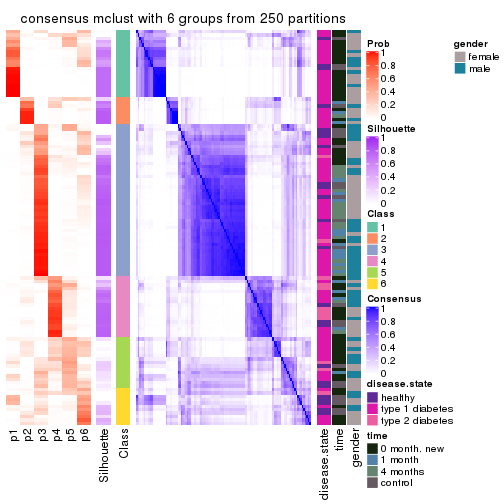
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
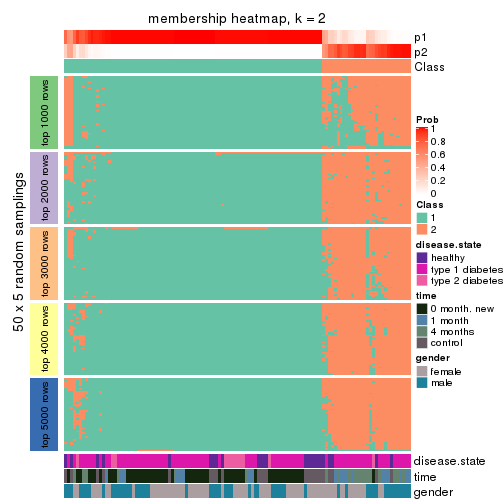
membership_heatmap(res, k = 3)
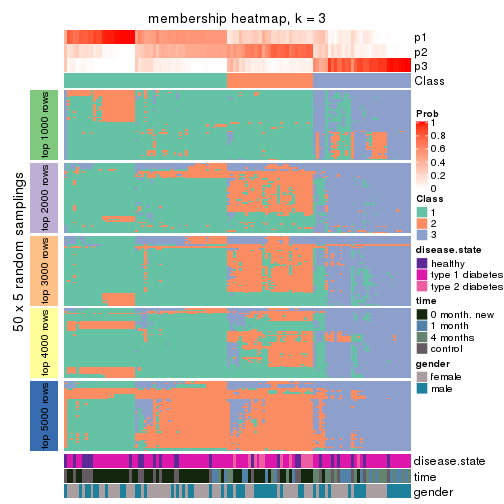
membership_heatmap(res, k = 4)
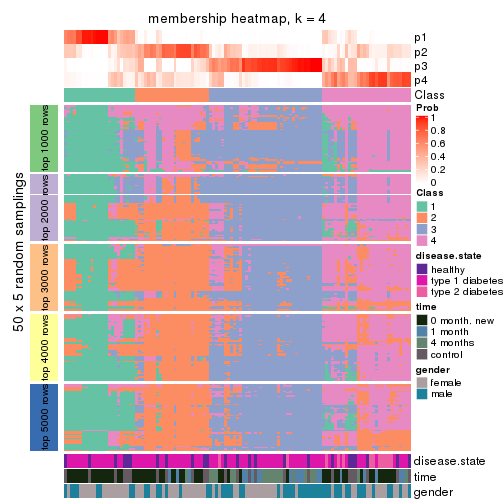
membership_heatmap(res, k = 5)
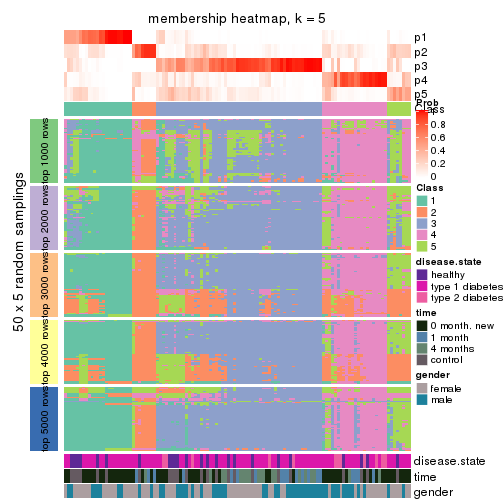
membership_heatmap(res, k = 6)
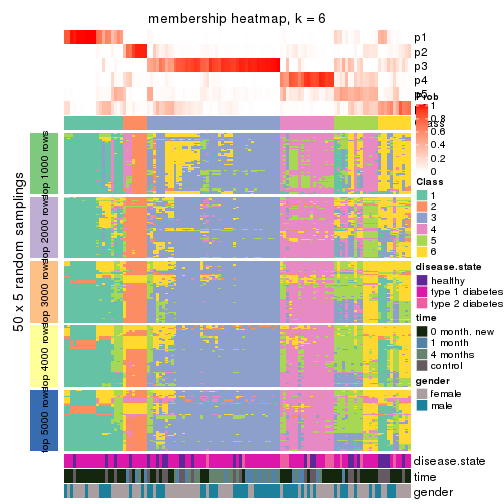
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
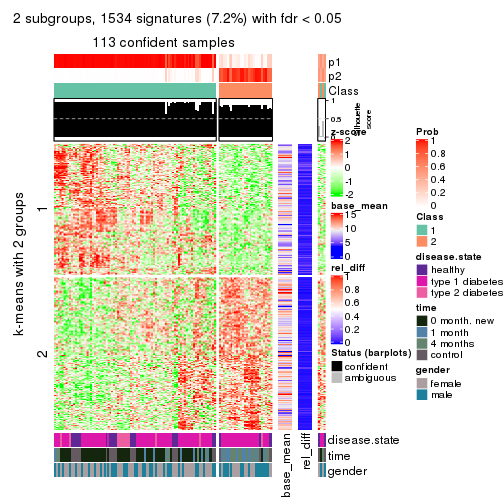
get_signatures(res, k = 3)
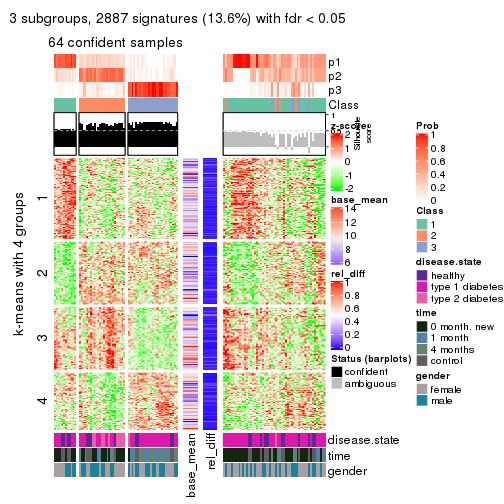
get_signatures(res, k = 4)
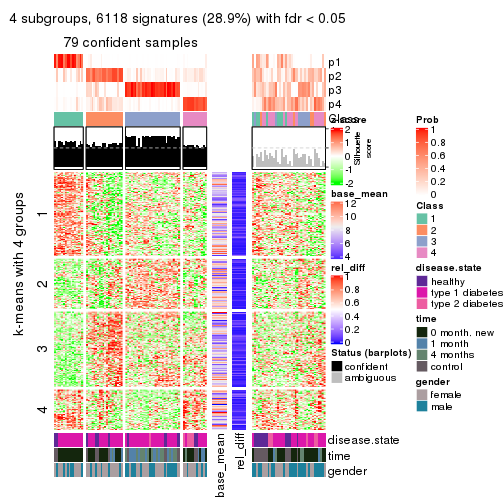
get_signatures(res, k = 5)
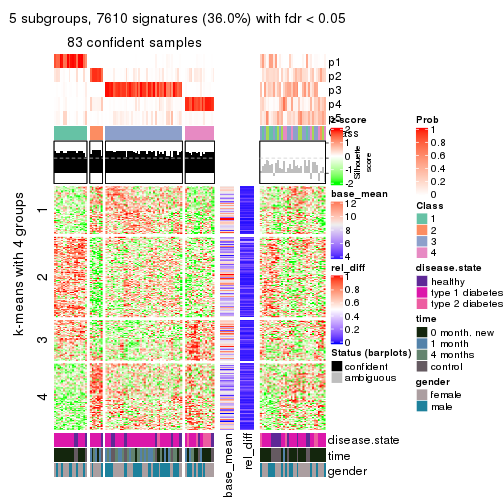
get_signatures(res, k = 6)
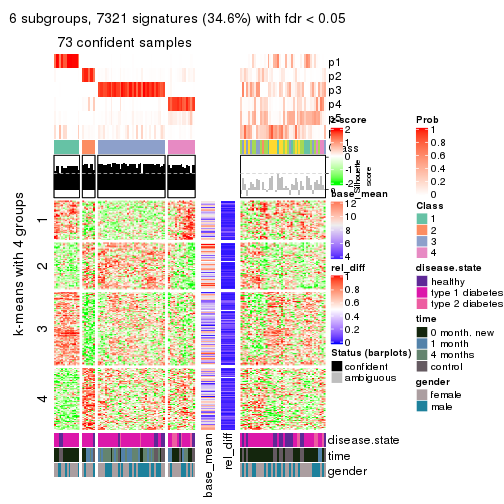
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
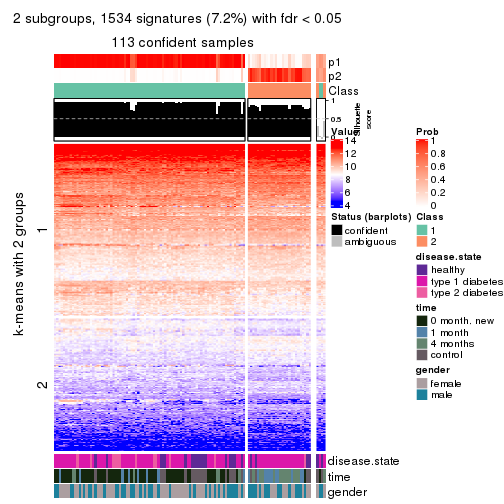
get_signatures(res, k = 3, scale_rows = FALSE)
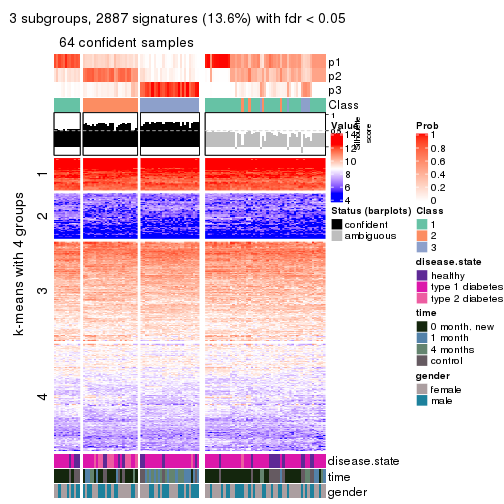
get_signatures(res, k = 4, scale_rows = FALSE)
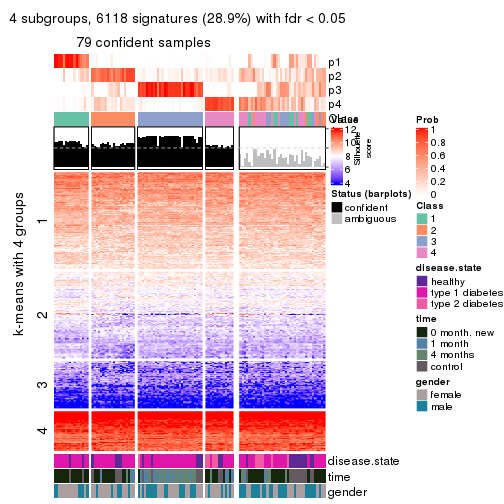
get_signatures(res, k = 5, scale_rows = FALSE)
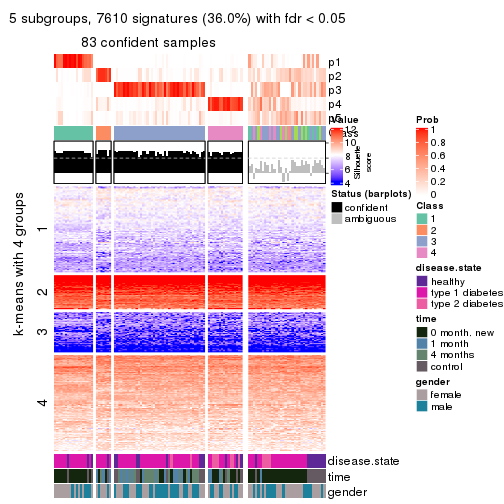
get_signatures(res, k = 6, scale_rows = FALSE)
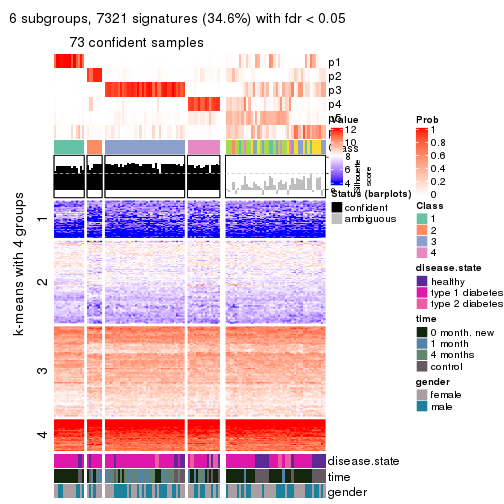
Compare the overlap of signatures from different k:
compare_signatures(res)
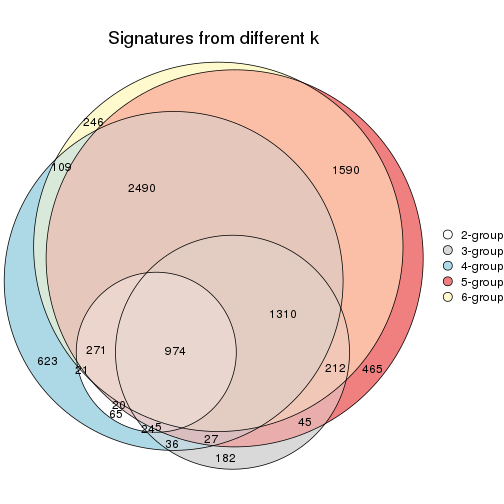
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
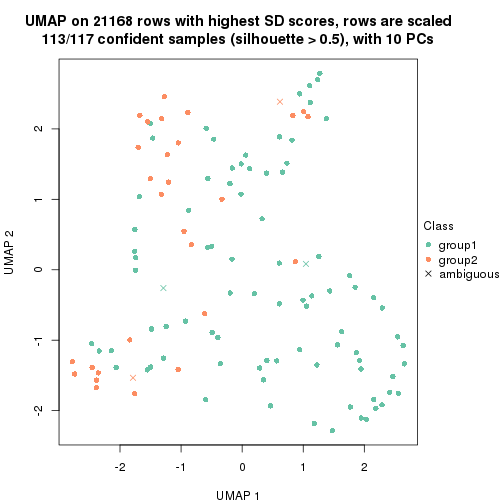
dimension_reduction(res, k = 3, method = "UMAP")
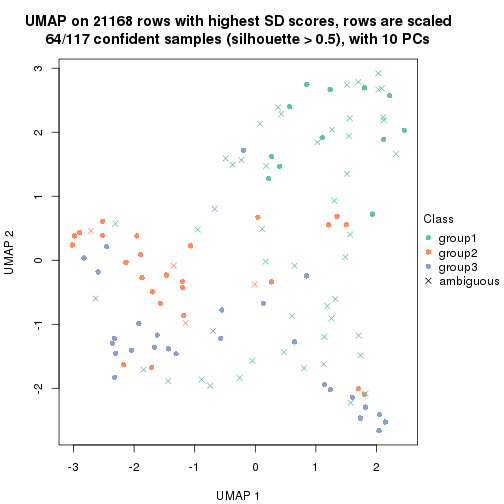
dimension_reduction(res, k = 4, method = "UMAP")
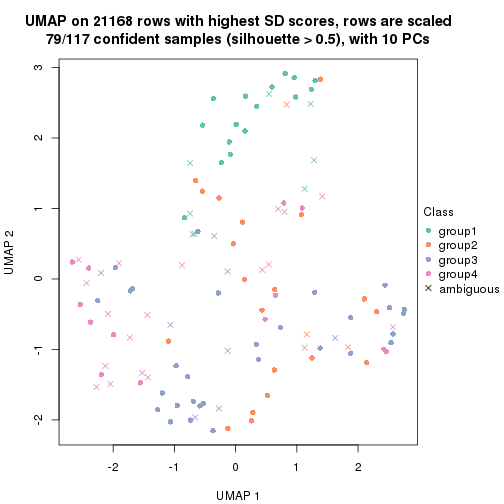
dimension_reduction(res, k = 5, method = "UMAP")
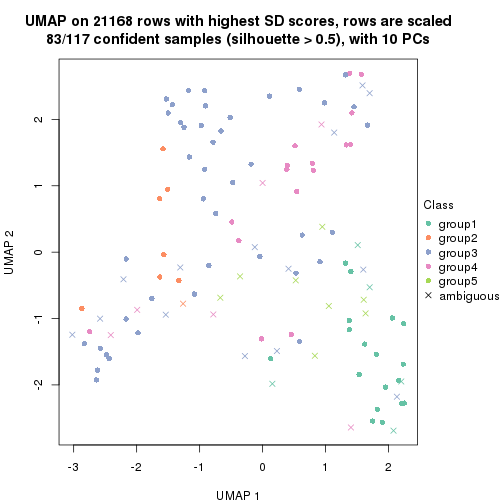
dimension_reduction(res, k = 6, method = "UMAP")
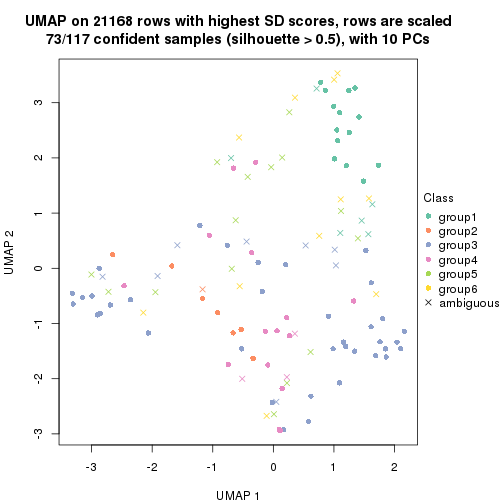
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
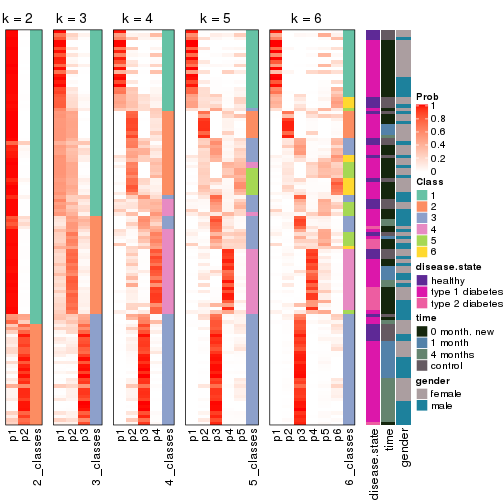
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> SD:mclust 113 0.21746 1.61e-09 0.354 2
#> SD:mclust 64 0.00190 3.26e-05 0.840 3
#> SD:mclust 79 0.00122 2.41e-06 0.313 4
#> SD:mclust 83 0.00451 1.46e-04 0.250 5
#> SD:mclust 73 0.03058 4.12e-04 0.453 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["SD", "NMF"]
# you can also extract it by
# res = res_list["SD:NMF"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'SD' method.
#> Subgroups are detected by 'NMF' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
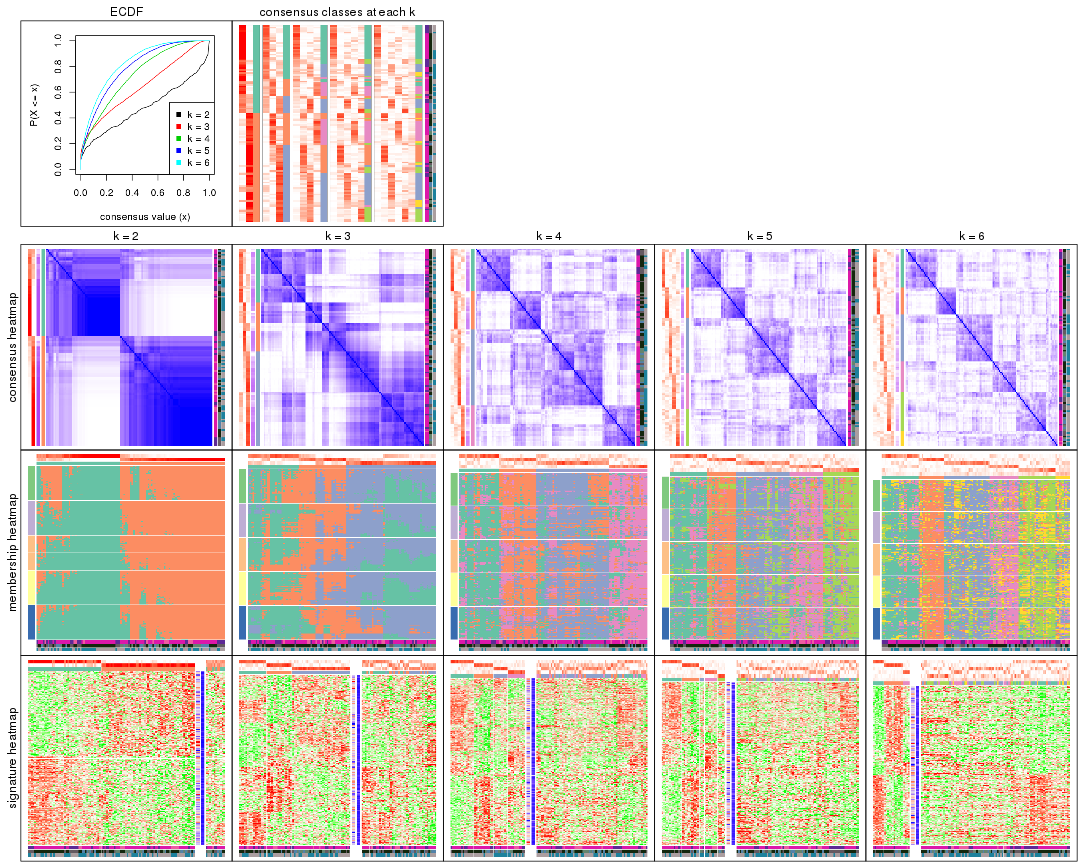
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
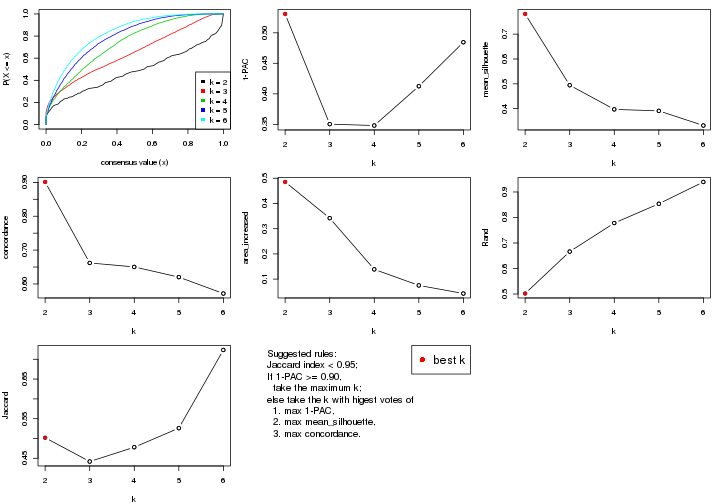
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.531 0.782 0.901 0.4853 0.502 0.502
#> 3 3 0.350 0.494 0.662 0.3418 0.666 0.442
#> 4 4 0.348 0.397 0.650 0.1386 0.778 0.478
#> 5 5 0.412 0.391 0.620 0.0749 0.854 0.526
#> 6 6 0.485 0.332 0.572 0.0428 0.939 0.723
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.1843 0.8540 0.972 0.028
#> GSM228563 2 0.6623 0.7584 0.172 0.828
#> GSM228565 1 0.8813 0.6385 0.700 0.300
#> GSM228566 2 0.6247 0.7749 0.156 0.844
#> GSM228567 1 0.0000 0.8596 1.000 0.000
#> GSM228570 1 0.0000 0.8596 1.000 0.000
#> GSM228571 1 0.0672 0.8583 0.992 0.008
#> GSM228574 2 0.0376 0.9000 0.004 0.996
#> GSM228575 2 0.0376 0.9000 0.004 0.996
#> GSM228576 1 0.6531 0.7692 0.832 0.168
#> GSM228579 1 0.1843 0.8529 0.972 0.028
#> GSM228580 2 0.0000 0.9003 0.000 1.000
#> GSM228581 2 0.0000 0.9003 0.000 1.000
#> GSM228666 2 0.0000 0.9003 0.000 1.000
#> GSM228564 1 0.9248 0.4904 0.660 0.340
#> GSM228568 1 0.9000 0.6192 0.684 0.316
#> GSM228569 1 0.3733 0.8325 0.928 0.072
#> GSM228572 2 0.0000 0.9003 0.000 1.000
#> GSM228573 1 0.9552 0.5113 0.624 0.376
#> GSM228577 1 0.0000 0.8596 1.000 0.000
#> GSM228578 1 0.1184 0.8567 0.984 0.016
#> GSM228663 2 0.9087 0.4663 0.324 0.676
#> GSM228664 2 0.0000 0.9003 0.000 1.000
#> GSM228665 1 0.9608 0.4929 0.616 0.384
#> GSM228582 1 0.9491 0.5269 0.632 0.368
#> GSM228583 1 0.0000 0.8596 1.000 0.000
#> GSM228585 1 0.0000 0.8596 1.000 0.000
#> GSM228587 1 0.0000 0.8596 1.000 0.000
#> GSM228588 2 0.7219 0.7203 0.200 0.800
#> GSM228589 2 0.1633 0.8944 0.024 0.976
#> GSM228590 1 0.0000 0.8596 1.000 0.000
#> GSM228591 2 0.0000 0.9003 0.000 1.000
#> GSM228597 2 0.6148 0.7843 0.152 0.848
#> GSM228601 2 0.0672 0.8991 0.008 0.992
#> GSM228604 2 0.0000 0.9003 0.000 1.000
#> GSM228608 1 0.0000 0.8596 1.000 0.000
#> GSM228609 1 0.9909 0.1844 0.556 0.444
#> GSM228613 1 0.0000 0.8596 1.000 0.000
#> GSM228616 1 0.9552 0.4274 0.624 0.376
#> GSM228628 2 0.0000 0.9003 0.000 1.000
#> GSM228634 1 0.0000 0.8596 1.000 0.000
#> GSM228642 2 0.0000 0.9003 0.000 1.000
#> GSM228645 2 0.4298 0.8510 0.088 0.912
#> GSM228646 2 0.0376 0.9000 0.004 0.996
#> GSM228652 1 0.0000 0.8596 1.000 0.000
#> GSM228655 1 0.0000 0.8596 1.000 0.000
#> GSM228656 1 0.0000 0.8596 1.000 0.000
#> GSM228659 1 0.0000 0.8596 1.000 0.000
#> GSM228662 1 0.0000 0.8596 1.000 0.000
#> GSM228584 1 0.0000 0.8596 1.000 0.000
#> GSM228586 1 0.0000 0.8596 1.000 0.000
#> GSM228592 1 0.0000 0.8596 1.000 0.000
#> GSM228593 1 0.7219 0.7201 0.800 0.200
#> GSM228594 1 0.0000 0.8596 1.000 0.000
#> GSM228598 1 0.0000 0.8596 1.000 0.000
#> GSM228607 2 0.2043 0.8949 0.032 0.968
#> GSM228612 2 0.2603 0.8840 0.044 0.956
#> GSM228619 1 0.4431 0.8202 0.908 0.092
#> GSM228622 1 0.0000 0.8596 1.000 0.000
#> GSM228625 1 0.3879 0.8306 0.924 0.076
#> GSM228631 1 0.0000 0.8596 1.000 0.000
#> GSM228633 2 0.0000 0.9003 0.000 1.000
#> GSM228637 2 0.7139 0.7261 0.196 0.804
#> GSM228639 2 0.1184 0.8973 0.016 0.984
#> GSM228649 2 0.7883 0.6772 0.236 0.764
#> GSM228660 1 0.7528 0.7290 0.784 0.216
#> GSM228661 1 0.0376 0.8589 0.996 0.004
#> GSM228595 2 0.0000 0.9003 0.000 1.000
#> GSM228599 2 0.3431 0.8699 0.064 0.936
#> GSM228602 2 0.9850 0.1394 0.428 0.572
#> GSM228614 2 0.0938 0.8983 0.012 0.988
#> GSM228626 2 0.0000 0.9003 0.000 1.000
#> GSM228640 1 0.9000 0.6170 0.684 0.316
#> GSM228643 2 0.5946 0.7916 0.144 0.856
#> GSM228650 2 0.0000 0.9003 0.000 1.000
#> GSM228653 1 0.8813 0.6356 0.700 0.300
#> GSM228657 2 0.0000 0.9003 0.000 1.000
#> GSM228605 1 0.4022 0.8316 0.920 0.080
#> GSM228610 2 0.1843 0.8933 0.028 0.972
#> GSM228617 1 0.9970 0.1992 0.532 0.468
#> GSM228620 1 0.7745 0.7168 0.772 0.228
#> GSM228623 2 0.2043 0.8904 0.032 0.968
#> GSM228629 1 0.9087 0.6021 0.676 0.324
#> GSM228632 2 0.0000 0.9003 0.000 1.000
#> GSM228635 2 0.3584 0.8667 0.068 0.932
#> GSM228647 2 0.7453 0.6946 0.212 0.788
#> GSM228596 2 0.9896 0.0986 0.440 0.560
#> GSM228600 2 0.2778 0.8815 0.048 0.952
#> GSM228603 1 0.9087 0.6037 0.676 0.324
#> GSM228615 2 0.4562 0.8434 0.096 0.904
#> GSM228627 2 0.6801 0.7424 0.180 0.820
#> GSM228641 2 0.3733 0.8641 0.072 0.928
#> GSM228644 2 0.0000 0.9003 0.000 1.000
#> GSM228651 2 0.4562 0.8429 0.096 0.904
#> GSM228654 2 0.1633 0.8944 0.024 0.976
#> GSM228658 2 0.9896 0.0857 0.440 0.560
#> GSM228606 2 0.0000 0.9003 0.000 1.000
#> GSM228611 2 0.9608 0.2960 0.384 0.616
#> GSM228618 2 0.9522 0.3315 0.372 0.628
#> GSM228621 2 0.0000 0.9003 0.000 1.000
#> GSM228624 2 0.0376 0.9000 0.004 0.996
#> GSM228630 2 0.0000 0.9003 0.000 1.000
#> GSM228636 2 0.5408 0.8160 0.124 0.876
#> GSM228638 2 0.0376 0.9001 0.004 0.996
#> GSM228648 2 0.0000 0.9003 0.000 1.000
#> GSM228670 2 0.2043 0.8937 0.032 0.968
#> GSM228671 2 0.0000 0.9003 0.000 1.000
#> GSM228672 1 0.1184 0.8563 0.984 0.016
#> GSM228674 2 0.5519 0.8325 0.128 0.872
#> GSM228675 2 0.2236 0.8883 0.036 0.964
#> GSM228676 1 0.9922 0.3184 0.552 0.448
#> GSM228667 2 0.3733 0.8693 0.072 0.928
#> GSM228668 1 0.0000 0.8596 1.000 0.000
#> GSM228669 1 0.1633 0.8542 0.976 0.024
#> GSM228673 2 0.0938 0.8984 0.012 0.988
#> GSM228677 2 0.0000 0.9003 0.000 1.000
#> GSM228678 2 0.0000 0.9003 0.000 1.000
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.753 0.4385 0.600 0.348 0.052
#> GSM228563 2 0.277 0.6254 0.048 0.928 0.024
#> GSM228565 1 0.904 0.4943 0.544 0.176 0.280
#> GSM228566 3 0.384 0.6832 0.116 0.012 0.872
#> GSM228567 1 0.338 0.6495 0.908 0.044 0.048
#> GSM228570 1 0.529 0.5221 0.732 0.268 0.000
#> GSM228571 1 0.428 0.6429 0.852 0.016 0.132
#> GSM228574 3 0.266 0.6929 0.024 0.044 0.932
#> GSM228575 3 0.234 0.6883 0.012 0.048 0.940
#> GSM228576 1 0.695 0.4227 0.636 0.032 0.332
#> GSM228579 1 0.487 0.6475 0.832 0.032 0.136
#> GSM228580 3 0.629 0.0616 0.000 0.464 0.536
#> GSM228581 3 0.564 0.5483 0.016 0.232 0.752
#> GSM228666 3 0.565 0.4328 0.000 0.312 0.688
#> GSM228564 2 0.460 0.5174 0.204 0.796 0.000
#> GSM228568 1 0.665 0.2171 0.592 0.012 0.396
#> GSM228569 1 0.588 0.3394 0.652 0.000 0.348
#> GSM228572 2 0.630 0.0669 0.000 0.516 0.484
#> GSM228573 3 0.614 0.3701 0.404 0.000 0.596
#> GSM228577 1 0.454 0.6556 0.848 0.028 0.124
#> GSM228578 1 0.536 0.4849 0.724 0.000 0.276
#> GSM228663 3 0.510 0.5946 0.248 0.000 0.752
#> GSM228664 3 0.323 0.6959 0.072 0.020 0.908
#> GSM228665 3 0.613 0.3769 0.400 0.000 0.600
#> GSM228582 3 0.652 0.1380 0.488 0.004 0.508
#> GSM228583 1 0.375 0.6044 0.856 0.144 0.000
#> GSM228585 1 0.452 0.5924 0.816 0.180 0.004
#> GSM228587 2 0.617 0.1546 0.412 0.588 0.000
#> GSM228588 2 0.355 0.5724 0.132 0.868 0.000
#> GSM228589 2 0.384 0.6397 0.012 0.872 0.116
#> GSM228590 1 0.571 0.4461 0.680 0.320 0.000
#> GSM228591 3 0.597 0.3350 0.000 0.364 0.636
#> GSM228597 2 0.127 0.6353 0.004 0.972 0.024
#> GSM228601 2 0.497 0.5628 0.000 0.764 0.236
#> GSM228604 3 0.465 0.5644 0.000 0.208 0.792
#> GSM228608 1 0.534 0.5657 0.760 0.232 0.008
#> GSM228609 2 0.465 0.5139 0.208 0.792 0.000
#> GSM228613 1 0.626 0.1843 0.552 0.448 0.000
#> GSM228616 2 0.754 0.3824 0.292 0.640 0.068
#> GSM228628 3 0.579 0.3955 0.000 0.332 0.668
#> GSM228634 1 0.440 0.5909 0.812 0.000 0.188
#> GSM228642 3 0.514 0.5145 0.000 0.252 0.748
#> GSM228645 3 0.341 0.6863 0.028 0.068 0.904
#> GSM228646 3 0.355 0.6350 0.000 0.132 0.868
#> GSM228652 1 0.531 0.5807 0.772 0.216 0.012
#> GSM228655 1 0.701 0.6136 0.712 0.208 0.080
#> GSM228656 1 0.518 0.5312 0.744 0.256 0.000
#> GSM228659 2 0.593 0.2890 0.356 0.644 0.000
#> GSM228662 1 0.631 0.0491 0.500 0.500 0.000
#> GSM228584 1 0.553 0.4738 0.704 0.296 0.000
#> GSM228586 1 0.406 0.6152 0.836 0.000 0.164
#> GSM228592 1 0.520 0.5423 0.760 0.236 0.004
#> GSM228593 2 0.493 0.4889 0.232 0.768 0.000
#> GSM228594 1 0.412 0.6129 0.832 0.000 0.168
#> GSM228598 1 0.665 0.4114 0.656 0.320 0.024
#> GSM228607 3 0.494 0.6901 0.104 0.056 0.840
#> GSM228612 3 0.369 0.6713 0.140 0.000 0.860
#> GSM228619 2 0.832 -0.0845 0.428 0.492 0.080
#> GSM228622 1 0.533 0.4866 0.728 0.000 0.272
#> GSM228625 2 0.571 0.3589 0.320 0.680 0.000
#> GSM228631 1 0.691 0.6341 0.728 0.092 0.180
#> GSM228633 3 0.599 0.3229 0.000 0.368 0.632
#> GSM228637 2 0.234 0.6194 0.048 0.940 0.012
#> GSM228639 3 0.502 0.5860 0.012 0.192 0.796
#> GSM228649 2 0.319 0.5919 0.100 0.896 0.004
#> GSM228660 1 0.902 0.3347 0.508 0.348 0.144
#> GSM228661 1 0.571 0.4007 0.680 0.000 0.320
#> GSM228595 3 0.630 0.0254 0.000 0.476 0.524
#> GSM228599 2 0.497 0.5627 0.000 0.764 0.236
#> GSM228602 3 0.586 0.5919 0.240 0.020 0.740
#> GSM228614 2 0.631 0.0506 0.000 0.504 0.496
#> GSM228626 3 0.576 0.4010 0.000 0.328 0.672
#> GSM228640 3 0.625 0.4536 0.344 0.008 0.648
#> GSM228643 3 0.453 0.6879 0.104 0.040 0.856
#> GSM228650 3 0.230 0.6797 0.004 0.060 0.936
#> GSM228653 3 0.597 0.4393 0.364 0.000 0.636
#> GSM228657 3 0.626 0.1165 0.000 0.448 0.552
#> GSM228605 1 0.684 0.4962 0.676 0.040 0.284
#> GSM228610 3 0.465 0.6289 0.208 0.000 0.792
#> GSM228617 3 0.593 0.5171 0.320 0.004 0.676
#> GSM228620 3 0.627 0.2458 0.452 0.000 0.548
#> GSM228623 2 0.642 0.2454 0.004 0.572 0.424
#> GSM228629 3 0.618 0.3361 0.416 0.000 0.584
#> GSM228632 3 0.287 0.6704 0.008 0.076 0.916
#> GSM228635 2 0.406 0.6300 0.000 0.836 0.164
#> GSM228647 3 0.518 0.5888 0.256 0.000 0.744
#> GSM228596 3 0.659 0.6117 0.216 0.056 0.728
#> GSM228600 3 0.364 0.6923 0.084 0.024 0.892
#> GSM228603 3 0.628 0.4076 0.384 0.004 0.612
#> GSM228615 2 0.245 0.6408 0.000 0.924 0.076
#> GSM228627 3 0.441 0.6489 0.172 0.004 0.824
#> GSM228641 3 0.397 0.6739 0.132 0.008 0.860
#> GSM228644 3 0.601 0.3138 0.000 0.372 0.628
#> GSM228651 3 0.429 0.6633 0.152 0.008 0.840
#> GSM228654 3 0.303 0.6896 0.092 0.004 0.904
#> GSM228658 3 0.556 0.5377 0.300 0.000 0.700
#> GSM228606 3 0.447 0.6503 0.028 0.120 0.852
#> GSM228611 3 0.559 0.5343 0.304 0.000 0.696
#> GSM228618 3 0.543 0.5588 0.284 0.000 0.716
#> GSM228621 3 0.250 0.6955 0.068 0.004 0.928
#> GSM228624 3 0.245 0.6939 0.076 0.000 0.924
#> GSM228630 3 0.429 0.6029 0.004 0.164 0.832
#> GSM228636 2 0.348 0.6367 0.000 0.872 0.128
#> GSM228638 3 0.338 0.6887 0.092 0.012 0.896
#> GSM228648 3 0.327 0.6711 0.016 0.080 0.904
#> GSM228670 2 0.573 0.6001 0.024 0.760 0.216
#> GSM228671 3 0.493 0.5390 0.000 0.232 0.768
#> GSM228672 2 0.540 0.4234 0.280 0.720 0.000
#> GSM228674 2 0.548 0.6276 0.076 0.816 0.108
#> GSM228675 2 0.445 0.6095 0.000 0.808 0.192
#> GSM228676 3 0.868 0.2517 0.340 0.120 0.540
#> GSM228667 2 0.717 0.1242 0.024 0.516 0.460
#> GSM228668 1 0.634 0.5580 0.716 0.252 0.032
#> GSM228669 2 0.581 0.3292 0.336 0.664 0.000
#> GSM228673 3 0.329 0.6884 0.096 0.008 0.896
#> GSM228677 3 0.533 0.4799 0.000 0.272 0.728
#> GSM228678 2 0.622 0.2248 0.000 0.568 0.432
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 1 0.922 0.0852 0.412 0.224 0.096 0.268
#> GSM228563 4 0.530 0.5974 0.080 0.132 0.016 0.772
#> GSM228565 1 0.808 0.3130 0.500 0.336 0.096 0.068
#> GSM228566 3 0.738 0.1413 0.088 0.424 0.464 0.024
#> GSM228567 1 0.263 0.6713 0.916 0.028 0.048 0.008
#> GSM228570 1 0.591 0.5940 0.744 0.120 0.032 0.104
#> GSM228571 1 0.437 0.6340 0.800 0.156 0.044 0.000
#> GSM228574 2 0.597 0.3405 0.036 0.648 0.300 0.016
#> GSM228575 2 0.702 -0.0915 0.024 0.460 0.456 0.060
#> GSM228576 1 0.782 0.2053 0.468 0.316 0.208 0.008
#> GSM228579 1 0.505 0.6228 0.760 0.180 0.056 0.004
#> GSM228580 2 0.732 0.3186 0.000 0.484 0.164 0.352
#> GSM228581 2 0.569 0.5381 0.032 0.748 0.160 0.060
#> GSM228666 2 0.560 0.6197 0.008 0.744 0.116 0.132
#> GSM228564 4 0.669 0.5394 0.168 0.136 0.024 0.672
#> GSM228568 3 0.792 0.1243 0.324 0.320 0.356 0.000
#> GSM228569 3 0.675 0.0290 0.440 0.092 0.468 0.000
#> GSM228572 2 0.643 0.3544 0.000 0.552 0.076 0.372
#> GSM228573 3 0.549 0.5015 0.200 0.080 0.720 0.000
#> GSM228577 1 0.623 0.4425 0.628 0.072 0.296 0.004
#> GSM228578 3 0.650 0.1076 0.416 0.032 0.528 0.024
#> GSM228663 3 0.657 0.4117 0.120 0.272 0.608 0.000
#> GSM228664 3 0.559 0.1470 0.008 0.476 0.508 0.008
#> GSM228665 3 0.571 0.5076 0.140 0.108 0.740 0.012
#> GSM228582 2 0.731 0.2097 0.236 0.556 0.204 0.004
#> GSM228583 1 0.204 0.6724 0.940 0.008 0.016 0.036
#> GSM228585 1 0.220 0.6726 0.936 0.016 0.016 0.032
#> GSM228587 1 0.544 0.3231 0.596 0.020 0.000 0.384
#> GSM228588 4 0.711 0.4284 0.176 0.268 0.000 0.556
#> GSM228589 2 0.605 0.3399 0.044 0.620 0.008 0.328
#> GSM228590 1 0.383 0.5962 0.792 0.004 0.000 0.204
#> GSM228591 2 0.459 0.6071 0.020 0.824 0.076 0.080
#> GSM228597 4 0.248 0.6124 0.012 0.056 0.012 0.920
#> GSM228601 2 0.516 0.4556 0.016 0.676 0.004 0.304
#> GSM228604 2 0.502 0.5651 0.004 0.760 0.184 0.052
#> GSM228608 1 0.454 0.6554 0.828 0.040 0.036 0.096
#> GSM228609 4 0.643 0.3869 0.312 0.092 0.000 0.596
#> GSM228613 1 0.465 0.4521 0.684 0.000 0.004 0.312
#> GSM228616 1 0.786 0.0547 0.420 0.404 0.016 0.160
#> GSM228628 2 0.419 0.6212 0.016 0.844 0.068 0.072
#> GSM228634 1 0.490 0.4914 0.716 0.024 0.260 0.000
#> GSM228642 2 0.370 0.6103 0.000 0.852 0.100 0.048
#> GSM228645 2 0.551 0.4783 0.060 0.760 0.152 0.028
#> GSM228646 2 0.533 0.5160 0.040 0.756 0.180 0.024
#> GSM228652 1 0.396 0.6684 0.852 0.012 0.048 0.088
#> GSM228655 1 0.606 0.6409 0.732 0.032 0.136 0.100
#> GSM228656 1 0.245 0.6588 0.908 0.004 0.004 0.084
#> GSM228659 4 0.508 0.1451 0.420 0.004 0.000 0.576
#> GSM228662 1 0.473 0.3680 0.636 0.000 0.000 0.364
#> GSM228584 1 0.377 0.6069 0.808 0.000 0.008 0.184
#> GSM228586 1 0.469 0.5264 0.724 0.016 0.260 0.000
#> GSM228592 1 0.438 0.6416 0.820 0.012 0.040 0.128
#> GSM228593 4 0.535 0.3325 0.336 0.024 0.000 0.640
#> GSM228594 1 0.545 0.5617 0.724 0.080 0.196 0.000
#> GSM228598 1 0.761 0.4805 0.592 0.036 0.196 0.176
#> GSM228607 3 0.653 0.3402 0.008 0.316 0.600 0.076
#> GSM228612 3 0.602 0.2751 0.036 0.404 0.556 0.004
#> GSM228619 4 0.813 0.3913 0.128 0.064 0.272 0.536
#> GSM228622 3 0.667 0.4141 0.228 0.056 0.664 0.052
#> GSM228625 4 0.560 0.3658 0.300 0.012 0.024 0.664
#> GSM228631 3 0.817 -0.0243 0.412 0.080 0.428 0.080
#> GSM228633 2 0.692 0.5155 0.000 0.584 0.168 0.248
#> GSM228637 4 0.363 0.6038 0.020 0.032 0.076 0.872
#> GSM228639 3 0.675 0.2919 0.000 0.140 0.596 0.264
#> GSM228649 4 0.636 0.5712 0.124 0.108 0.048 0.720
#> GSM228660 3 0.943 -0.0336 0.316 0.272 0.316 0.096
#> GSM228661 3 0.629 0.1039 0.412 0.060 0.528 0.000
#> GSM228595 2 0.567 0.5143 0.000 0.652 0.048 0.300
#> GSM228599 4 0.778 0.1718 0.028 0.348 0.128 0.496
#> GSM228602 3 0.677 0.3418 0.088 0.332 0.572 0.008
#> GSM228614 4 0.715 0.1093 0.000 0.264 0.184 0.552
#> GSM228626 2 0.496 0.6219 0.000 0.776 0.108 0.116
#> GSM228640 3 0.697 0.3086 0.124 0.308 0.564 0.004
#> GSM228643 3 0.662 0.1680 0.048 0.420 0.516 0.016
#> GSM228650 3 0.647 0.1759 0.004 0.388 0.544 0.064
#> GSM228653 3 0.601 0.4719 0.180 0.132 0.688 0.000
#> GSM228657 2 0.549 0.5853 0.000 0.700 0.060 0.240
#> GSM228605 3 0.805 0.3568 0.156 0.068 0.572 0.204
#> GSM228610 3 0.343 0.5309 0.008 0.104 0.868 0.020
#> GSM228617 3 0.414 0.5238 0.036 0.088 0.848 0.028
#> GSM228620 3 0.274 0.5385 0.096 0.012 0.892 0.000
#> GSM228623 4 0.683 0.2595 0.000 0.132 0.296 0.572
#> GSM228629 3 0.442 0.5262 0.160 0.044 0.796 0.000
#> GSM228632 3 0.608 0.3949 0.000 0.244 0.660 0.096
#> GSM228635 4 0.382 0.5767 0.000 0.048 0.108 0.844
#> GSM228647 3 0.226 0.5368 0.020 0.056 0.924 0.000
#> GSM228596 3 0.730 0.3176 0.064 0.296 0.584 0.056
#> GSM228600 3 0.633 0.1294 0.060 0.444 0.496 0.000
#> GSM228603 3 0.711 0.3476 0.152 0.276 0.568 0.004
#> GSM228615 4 0.311 0.5868 0.004 0.108 0.012 0.876
#> GSM228627 2 0.683 -0.0971 0.100 0.484 0.416 0.000
#> GSM228641 3 0.635 0.2567 0.060 0.368 0.568 0.004
#> GSM228644 2 0.506 0.6328 0.000 0.760 0.076 0.164
#> GSM228651 3 0.577 0.3480 0.044 0.300 0.652 0.004
#> GSM228654 3 0.593 0.1971 0.040 0.408 0.552 0.000
#> GSM228658 3 0.695 0.3971 0.168 0.252 0.580 0.000
#> GSM228606 3 0.685 0.3163 0.000 0.200 0.600 0.200
#> GSM228611 3 0.361 0.5378 0.060 0.080 0.860 0.000
#> GSM228618 3 0.403 0.5371 0.048 0.092 0.848 0.012
#> GSM228621 3 0.410 0.4878 0.000 0.192 0.792 0.016
#> GSM228624 3 0.530 0.4485 0.008 0.272 0.696 0.024
#> GSM228630 3 0.672 0.3312 0.000 0.200 0.616 0.184
#> GSM228636 4 0.361 0.5869 0.000 0.060 0.080 0.860
#> GSM228638 3 0.454 0.4952 0.000 0.144 0.796 0.060
#> GSM228648 3 0.565 0.4015 0.000 0.272 0.672 0.056
#> GSM228670 4 0.603 0.5167 0.032 0.172 0.072 0.724
#> GSM228671 3 0.765 0.0182 0.000 0.352 0.432 0.216
#> GSM228672 4 0.574 0.3634 0.328 0.044 0.000 0.628
#> GSM228674 4 0.566 0.5367 0.056 0.192 0.020 0.732
#> GSM228675 4 0.613 0.4123 0.012 0.240 0.072 0.676
#> GSM228676 2 0.976 -0.0334 0.180 0.320 0.308 0.192
#> GSM228667 2 0.862 0.2967 0.080 0.464 0.136 0.320
#> GSM228668 4 0.825 0.0748 0.324 0.028 0.196 0.452
#> GSM228669 4 0.520 0.5049 0.204 0.024 0.024 0.748
#> GSM228673 3 0.533 0.4573 0.000 0.220 0.720 0.060
#> GSM228677 3 0.785 0.0644 0.004 0.240 0.444 0.312
#> GSM228678 4 0.674 0.3113 0.000 0.232 0.160 0.608
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 5 0.667 0.28256 0.216 0.016 0.032 0.128 0.608
#> GSM228563 4 0.798 0.36442 0.128 0.196 0.000 0.440 0.236
#> GSM228565 5 0.710 0.26428 0.296 0.132 0.016 0.032 0.524
#> GSM228566 5 0.362 0.53816 0.028 0.064 0.032 0.016 0.860
#> GSM228567 1 0.404 0.62548 0.796 0.004 0.044 0.004 0.152
#> GSM228570 1 0.647 0.30899 0.524 0.032 0.004 0.080 0.360
#> GSM228571 1 0.639 0.48797 0.624 0.068 0.052 0.012 0.244
#> GSM228574 5 0.720 0.28552 0.008 0.260 0.216 0.024 0.492
#> GSM228575 5 0.656 0.46510 0.000 0.124 0.128 0.112 0.636
#> GSM228576 5 0.677 0.17812 0.340 0.140 0.020 0.004 0.496
#> GSM228579 1 0.601 0.58531 0.696 0.088 0.076 0.008 0.132
#> GSM228580 5 0.788 0.11891 0.000 0.288 0.072 0.268 0.372
#> GSM228581 2 0.688 0.40127 0.024 0.560 0.292 0.036 0.088
#> GSM228666 2 0.690 0.46796 0.008 0.596 0.216 0.084 0.096
#> GSM228564 4 0.668 0.28190 0.140 0.020 0.000 0.476 0.364
#> GSM228568 3 0.781 0.09971 0.300 0.248 0.392 0.004 0.056
#> GSM228569 3 0.578 -0.05810 0.448 0.012 0.488 0.004 0.048
#> GSM228572 2 0.576 0.56943 0.004 0.696 0.036 0.148 0.116
#> GSM228573 3 0.581 0.43621 0.084 0.020 0.624 0.000 0.272
#> GSM228577 1 0.576 0.38793 0.592 0.040 0.336 0.004 0.028
#> GSM228578 3 0.691 0.21390 0.332 0.004 0.516 0.060 0.088
#> GSM228663 3 0.506 0.49673 0.096 0.112 0.756 0.004 0.032
#> GSM228664 3 0.501 0.34353 0.020 0.328 0.636 0.004 0.012
#> GSM228665 3 0.363 0.52655 0.064 0.028 0.860 0.024 0.024
#> GSM228582 2 0.712 0.29675 0.268 0.504 0.196 0.008 0.024
#> GSM228583 1 0.277 0.65208 0.888 0.000 0.020 0.016 0.076
#> GSM228585 1 0.316 0.64648 0.852 0.008 0.004 0.012 0.124
#> GSM228587 1 0.519 0.51943 0.700 0.064 0.008 0.220 0.008
#> GSM228588 2 0.585 0.38915 0.188 0.656 0.004 0.140 0.012
#> GSM228589 2 0.402 0.62752 0.048 0.828 0.064 0.060 0.000
#> GSM228590 1 0.407 0.60555 0.792 0.008 0.012 0.168 0.020
#> GSM228591 2 0.314 0.67630 0.028 0.876 0.060 0.000 0.036
#> GSM228597 4 0.472 0.59616 0.056 0.156 0.000 0.760 0.028
#> GSM228601 2 0.316 0.66052 0.016 0.876 0.004 0.064 0.040
#> GSM228604 2 0.520 0.41083 0.004 0.648 0.036 0.012 0.300
#> GSM228608 1 0.641 0.54899 0.632 0.004 0.044 0.132 0.188
#> GSM228609 1 0.721 -0.02564 0.352 0.340 0.000 0.292 0.016
#> GSM228613 1 0.520 0.39900 0.616 0.000 0.012 0.336 0.036
#> GSM228616 2 0.771 0.16444 0.348 0.440 0.028 0.044 0.140
#> GSM228628 2 0.302 0.67655 0.016 0.884 0.024 0.008 0.068
#> GSM228634 1 0.566 0.50072 0.632 0.000 0.244 0.004 0.120
#> GSM228642 2 0.342 0.64804 0.004 0.832 0.020 0.004 0.140
#> GSM228645 5 0.639 0.20210 0.092 0.352 0.012 0.012 0.532
#> GSM228646 2 0.595 -0.00533 0.052 0.472 0.012 0.008 0.456
#> GSM228652 1 0.643 0.59558 0.664 0.020 0.128 0.140 0.048
#> GSM228655 1 0.697 0.45047 0.556 0.040 0.296 0.076 0.032
#> GSM228656 1 0.320 0.64921 0.880 0.012 0.048 0.044 0.016
#> GSM228659 4 0.545 0.05082 0.412 0.052 0.000 0.532 0.004
#> GSM228662 1 0.479 0.47918 0.684 0.016 0.004 0.280 0.016
#> GSM228584 1 0.400 0.60972 0.784 0.000 0.040 0.172 0.004
#> GSM228586 1 0.540 0.46594 0.636 0.000 0.292 0.012 0.060
#> GSM228592 1 0.386 0.63848 0.828 0.008 0.080 0.080 0.004
#> GSM228593 1 0.712 0.03227 0.416 0.180 0.008 0.380 0.016
#> GSM228594 1 0.559 0.52752 0.680 0.036 0.212 0.000 0.072
#> GSM228598 1 0.670 0.46927 0.564 0.024 0.264 0.140 0.008
#> GSM228607 3 0.544 0.49704 0.012 0.144 0.720 0.108 0.016
#> GSM228612 3 0.655 0.37945 0.068 0.296 0.572 0.004 0.060
#> GSM228619 4 0.754 0.27450 0.072 0.024 0.104 0.516 0.284
#> GSM228622 3 0.749 0.36550 0.112 0.000 0.516 0.152 0.220
#> GSM228625 4 0.743 0.27858 0.284 0.104 0.084 0.516 0.012
#> GSM228631 5 0.771 0.13412 0.324 0.024 0.120 0.064 0.468
#> GSM228633 2 0.569 0.59843 0.000 0.708 0.132 0.088 0.072
#> GSM228637 4 0.538 0.60938 0.060 0.080 0.116 0.740 0.004
#> GSM228639 3 0.688 0.25115 0.000 0.052 0.488 0.356 0.104
#> GSM228649 4 0.763 0.41773 0.116 0.240 0.152 0.492 0.000
#> GSM228660 3 0.788 0.17627 0.256 0.256 0.424 0.052 0.012
#> GSM228661 3 0.561 0.14445 0.376 0.012 0.564 0.004 0.044
#> GSM228595 2 0.427 0.65892 0.000 0.808 0.040 0.096 0.056
#> GSM228599 5 0.713 0.23579 0.024 0.272 0.004 0.216 0.484
#> GSM228602 5 0.519 0.46656 0.024 0.072 0.148 0.012 0.744
#> GSM228614 4 0.738 0.46104 0.012 0.208 0.124 0.560 0.096
#> GSM228626 2 0.312 0.68059 0.000 0.872 0.064 0.012 0.052
#> GSM228640 5 0.274 0.50721 0.016 0.008 0.076 0.008 0.892
#> GSM228643 5 0.453 0.51392 0.000 0.080 0.120 0.020 0.780
#> GSM228650 5 0.724 0.37442 0.000 0.164 0.196 0.092 0.548
#> GSM228653 3 0.643 0.23425 0.088 0.024 0.516 0.004 0.368
#> GSM228657 2 0.482 0.66230 0.000 0.776 0.088 0.068 0.068
#> GSM228605 3 0.800 0.06855 0.064 0.004 0.316 0.304 0.312
#> GSM228610 3 0.470 0.49518 0.000 0.028 0.772 0.080 0.120
#> GSM228617 3 0.678 0.16288 0.036 0.016 0.444 0.068 0.436
#> GSM228620 3 0.511 0.48143 0.056 0.000 0.716 0.028 0.200
#> GSM228623 4 0.660 0.32626 0.000 0.132 0.284 0.552 0.032
#> GSM228629 3 0.613 0.37720 0.108 0.012 0.588 0.004 0.288
#> GSM228632 3 0.631 0.45122 0.000 0.132 0.656 0.128 0.084
#> GSM228635 4 0.460 0.59050 0.004 0.072 0.108 0.788 0.028
#> GSM228647 3 0.569 0.45291 0.032 0.016 0.664 0.036 0.252
#> GSM228596 3 0.746 -0.04389 0.024 0.048 0.432 0.104 0.392
#> GSM228600 5 0.552 0.50178 0.032 0.156 0.096 0.004 0.712
#> GSM228603 5 0.384 0.47740 0.040 0.012 0.120 0.004 0.824
#> GSM228615 4 0.452 0.60120 0.064 0.116 0.008 0.792 0.020
#> GSM228627 3 0.761 0.23052 0.064 0.256 0.472 0.004 0.204
#> GSM228641 5 0.363 0.51629 0.012 0.036 0.084 0.016 0.852
#> GSM228644 2 0.355 0.67981 0.000 0.852 0.060 0.024 0.064
#> GSM228651 5 0.650 0.05622 0.028 0.076 0.380 0.008 0.508
#> GSM228654 5 0.752 0.05889 0.020 0.244 0.340 0.012 0.384
#> GSM228658 3 0.656 0.35773 0.096 0.068 0.600 0.000 0.236
#> GSM228606 3 0.792 0.09347 0.000 0.076 0.364 0.256 0.304
#> GSM228611 3 0.435 0.50303 0.024 0.024 0.792 0.012 0.148
#> GSM228618 3 0.616 0.27515 0.052 0.012 0.524 0.020 0.392
#> GSM228621 5 0.662 -0.13729 0.000 0.068 0.408 0.056 0.468
#> GSM228624 3 0.527 0.50792 0.000 0.136 0.732 0.040 0.092
#> GSM228630 3 0.754 0.33069 0.000 0.080 0.488 0.220 0.212
#> GSM228636 4 0.380 0.60177 0.000 0.112 0.052 0.824 0.012
#> GSM228638 3 0.586 0.48679 0.000 0.064 0.692 0.112 0.132
#> GSM228648 3 0.652 0.45335 0.000 0.156 0.624 0.064 0.156
#> GSM228670 4 0.612 0.57187 0.040 0.108 0.028 0.696 0.128
#> GSM228671 3 0.824 -0.02722 0.000 0.128 0.336 0.220 0.316
#> GSM228672 4 0.705 0.29434 0.308 0.048 0.012 0.528 0.104
#> GSM228674 4 0.675 0.53807 0.076 0.056 0.048 0.648 0.172
#> GSM228675 4 0.770 0.45931 0.040 0.112 0.100 0.564 0.184
#> GSM228676 5 0.743 0.25922 0.044 0.028 0.156 0.228 0.544
#> GSM228667 5 0.929 0.14725 0.084 0.260 0.148 0.152 0.356
#> GSM228668 4 0.655 0.46541 0.184 0.000 0.108 0.624 0.084
#> GSM228669 4 0.414 0.53954 0.152 0.000 0.040 0.792 0.016
#> GSM228673 3 0.634 0.44990 0.000 0.108 0.656 0.112 0.124
#> GSM228677 4 0.804 -0.03314 0.000 0.084 0.296 0.320 0.300
#> GSM228678 4 0.725 0.36897 0.000 0.292 0.120 0.504 0.084
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 5 0.675 0.1954 0.168 0.004 0.000 0.064 0.476 0.288
#> GSM228563 6 0.885 -0.0614 0.144 0.120 0.004 0.244 0.224 0.264
#> GSM228565 5 0.751 0.2541 0.260 0.084 0.012 0.012 0.436 0.196
#> GSM228566 5 0.425 0.4303 0.048 0.040 0.016 0.000 0.792 0.104
#> GSM228567 1 0.388 0.5551 0.796 0.000 0.024 0.000 0.120 0.060
#> GSM228570 1 0.655 0.0552 0.472 0.012 0.008 0.020 0.340 0.148
#> GSM228571 1 0.629 0.2765 0.560 0.028 0.012 0.004 0.252 0.144
#> GSM228574 5 0.769 -0.0387 0.024 0.140 0.108 0.008 0.376 0.344
#> GSM228575 5 0.679 0.1303 0.020 0.072 0.032 0.032 0.428 0.416
#> GSM228576 5 0.699 0.2764 0.316 0.084 0.012 0.004 0.468 0.116
#> GSM228579 1 0.634 0.4640 0.616 0.060 0.044 0.004 0.068 0.208
#> GSM228580 5 0.826 -0.0184 0.004 0.196 0.036 0.164 0.308 0.292
#> GSM228581 6 0.745 -0.0504 0.024 0.352 0.212 0.012 0.040 0.360
#> GSM228666 2 0.745 0.0549 0.004 0.412 0.108 0.044 0.080 0.352
#> GSM228564 5 0.741 0.1512 0.108 0.024 0.000 0.232 0.464 0.172
#> GSM228568 6 0.809 -0.0960 0.236 0.152 0.272 0.008 0.016 0.316
#> GSM228569 3 0.690 0.1086 0.312 0.036 0.444 0.000 0.020 0.188
#> GSM228572 2 0.573 0.5870 0.000 0.680 0.020 0.088 0.088 0.124
#> GSM228573 3 0.571 0.4196 0.032 0.004 0.624 0.004 0.232 0.104
#> GSM228577 1 0.652 0.3700 0.536 0.032 0.244 0.008 0.008 0.172
#> GSM228578 3 0.715 0.2396 0.264 0.000 0.504 0.048 0.084 0.100
#> GSM228663 3 0.501 0.4378 0.048 0.068 0.724 0.000 0.012 0.148
#> GSM228664 3 0.541 0.3619 0.020 0.220 0.640 0.004 0.000 0.116
#> GSM228665 3 0.426 0.4670 0.048 0.012 0.788 0.004 0.028 0.120
#> GSM228582 2 0.759 0.1670 0.212 0.432 0.180 0.000 0.016 0.160
#> GSM228583 1 0.245 0.5907 0.892 0.000 0.000 0.008 0.060 0.040
#> GSM228585 1 0.290 0.5834 0.864 0.004 0.004 0.000 0.076 0.052
#> GSM228587 1 0.566 0.4339 0.600 0.040 0.004 0.288 0.004 0.064
#> GSM228588 2 0.531 0.5603 0.140 0.700 0.000 0.104 0.012 0.044
#> GSM228589 2 0.414 0.6592 0.020 0.804 0.032 0.064 0.000 0.080
#> GSM228590 1 0.464 0.4831 0.664 0.000 0.012 0.284 0.008 0.032
#> GSM228591 2 0.304 0.6714 0.020 0.868 0.016 0.004 0.012 0.080
#> GSM228597 4 0.482 0.5119 0.012 0.128 0.000 0.732 0.020 0.108
#> GSM228601 2 0.295 0.6837 0.020 0.876 0.000 0.056 0.012 0.036
#> GSM228604 2 0.505 0.4621 0.000 0.652 0.020 0.004 0.260 0.064
#> GSM228608 1 0.751 0.3981 0.496 0.000 0.044 0.176 0.136 0.148
#> GSM228609 1 0.721 -0.0152 0.344 0.316 0.000 0.280 0.016 0.044
#> GSM228613 1 0.451 0.3240 0.572 0.000 0.000 0.396 0.004 0.028
#> GSM228616 2 0.797 0.1235 0.332 0.384 0.028 0.028 0.116 0.112
#> GSM228628 2 0.341 0.6629 0.016 0.840 0.012 0.000 0.036 0.096
#> GSM228634 1 0.629 0.4018 0.568 0.000 0.240 0.004 0.076 0.112
#> GSM228642 2 0.375 0.6583 0.008 0.808 0.008 0.000 0.108 0.068
#> GSM228645 5 0.694 0.3225 0.096 0.236 0.008 0.000 0.500 0.160
#> GSM228646 5 0.719 0.1970 0.100 0.332 0.008 0.004 0.424 0.132
#> GSM228652 1 0.764 0.4111 0.484 0.004 0.148 0.208 0.048 0.108
#> GSM228655 1 0.834 0.2044 0.352 0.032 0.304 0.140 0.028 0.144
#> GSM228656 1 0.356 0.5924 0.836 0.004 0.052 0.068 0.000 0.040
#> GSM228659 4 0.484 0.1080 0.328 0.016 0.000 0.620 0.008 0.028
#> GSM228662 1 0.409 0.4462 0.680 0.000 0.000 0.292 0.004 0.024
#> GSM228584 1 0.363 0.5601 0.784 0.000 0.028 0.176 0.000 0.012
#> GSM228586 1 0.555 0.4416 0.620 0.000 0.268 0.020 0.020 0.072
#> GSM228592 1 0.416 0.5885 0.792 0.004 0.072 0.092 0.000 0.040
#> GSM228593 1 0.725 0.2812 0.492 0.136 0.004 0.256 0.028 0.084
#> GSM228594 1 0.524 0.5301 0.708 0.032 0.136 0.000 0.020 0.104
#> GSM228598 1 0.725 0.3576 0.460 0.008 0.252 0.096 0.004 0.180
#> GSM228607 3 0.520 0.3946 0.012 0.056 0.724 0.080 0.004 0.124
#> GSM228612 3 0.670 0.3032 0.072 0.280 0.512 0.000 0.012 0.124
#> GSM228619 4 0.846 0.1105 0.124 0.008 0.160 0.376 0.244 0.088
#> GSM228622 3 0.761 0.2512 0.112 0.000 0.508 0.100 0.168 0.112
#> GSM228625 4 0.646 0.2601 0.284 0.072 0.044 0.552 0.000 0.048
#> GSM228631 5 0.767 0.2169 0.308 0.028 0.060 0.064 0.456 0.084
#> GSM228633 2 0.589 0.5664 0.000 0.684 0.084 0.092 0.052 0.088
#> GSM228637 4 0.436 0.5292 0.012 0.056 0.076 0.788 0.000 0.068
#> GSM228639 3 0.703 0.0969 0.000 0.032 0.492 0.284 0.072 0.120
#> GSM228649 4 0.723 0.4027 0.076 0.132 0.144 0.552 0.000 0.096
#> GSM228660 3 0.736 0.1990 0.264 0.204 0.432 0.020 0.000 0.080
#> GSM228661 3 0.567 0.2662 0.296 0.000 0.568 0.000 0.024 0.112
#> GSM228595 2 0.348 0.6823 0.000 0.840 0.004 0.068 0.032 0.056
#> GSM228599 5 0.769 0.2664 0.036 0.192 0.004 0.160 0.472 0.136
#> GSM228602 5 0.549 0.4252 0.072 0.060 0.100 0.004 0.720 0.044
#> GSM228614 4 0.687 0.4070 0.004 0.140 0.092 0.592 0.048 0.124
#> GSM228626 2 0.249 0.6903 0.000 0.900 0.024 0.008 0.020 0.048
#> GSM228640 5 0.291 0.4401 0.040 0.000 0.036 0.000 0.872 0.052
#> GSM228643 5 0.549 0.3530 0.016 0.044 0.076 0.000 0.672 0.192
#> GSM228650 5 0.718 0.2769 0.008 0.072 0.148 0.044 0.548 0.180
#> GSM228653 3 0.655 0.3384 0.052 0.008 0.512 0.000 0.280 0.148
#> GSM228657 2 0.508 0.6431 0.000 0.744 0.040 0.088 0.048 0.080
#> GSM228605 6 0.829 0.1661 0.048 0.000 0.288 0.220 0.156 0.288
#> GSM228610 3 0.485 0.4123 0.004 0.008 0.732 0.028 0.072 0.156
#> GSM228617 5 0.704 -0.0845 0.040 0.004 0.376 0.036 0.420 0.124
#> GSM228620 3 0.413 0.4637 0.016 0.000 0.772 0.000 0.120 0.092
#> GSM228623 4 0.713 0.2224 0.004 0.108 0.264 0.468 0.004 0.152
#> GSM228629 3 0.580 0.3788 0.072 0.000 0.600 0.004 0.264 0.060
#> GSM228632 3 0.604 0.2811 0.000 0.072 0.624 0.040 0.044 0.220
#> GSM228635 4 0.551 0.4588 0.000 0.044 0.076 0.676 0.020 0.184
#> GSM228647 3 0.457 0.4586 0.004 0.008 0.732 0.004 0.168 0.084
#> GSM228596 6 0.759 0.1571 0.020 0.008 0.300 0.064 0.232 0.376
#> GSM228600 5 0.546 0.4208 0.044 0.120 0.060 0.000 0.712 0.064
#> GSM228603 5 0.317 0.4429 0.048 0.008 0.064 0.000 0.860 0.020
#> GSM228615 4 0.405 0.5318 0.036 0.100 0.012 0.808 0.004 0.040
#> GSM228627 3 0.768 0.1685 0.036 0.184 0.456 0.000 0.144 0.180
#> GSM228641 5 0.305 0.4353 0.008 0.020 0.044 0.000 0.868 0.060
#> GSM228644 2 0.330 0.6842 0.000 0.860 0.016 0.044 0.036 0.044
#> GSM228651 5 0.682 -0.0707 0.012 0.044 0.360 0.000 0.416 0.168
#> GSM228654 3 0.755 0.1291 0.004 0.168 0.384 0.004 0.292 0.148
#> GSM228658 3 0.695 0.3560 0.048 0.056 0.548 0.000 0.176 0.172
#> GSM228606 6 0.796 0.2264 0.000 0.020 0.272 0.156 0.232 0.320
#> GSM228611 3 0.424 0.4213 0.012 0.000 0.752 0.008 0.048 0.180
#> GSM228618 3 0.603 0.1615 0.028 0.008 0.460 0.000 0.412 0.092
#> GSM228621 5 0.631 0.0572 0.000 0.032 0.328 0.004 0.488 0.148
#> GSM228624 3 0.611 0.3451 0.000 0.092 0.616 0.028 0.048 0.216
#> GSM228630 3 0.767 0.1321 0.000 0.040 0.464 0.176 0.184 0.136
#> GSM228636 4 0.508 0.4941 0.000 0.088 0.060 0.724 0.008 0.120
#> GSM228638 3 0.493 0.4482 0.000 0.028 0.752 0.052 0.092 0.076
#> GSM228648 3 0.631 0.3916 0.000 0.148 0.628 0.028 0.104 0.092
#> GSM228670 4 0.664 0.4543 0.040 0.060 0.028 0.616 0.064 0.192
#> GSM228671 6 0.785 0.3168 0.000 0.048 0.244 0.124 0.160 0.424
#> GSM228672 4 0.716 0.2822 0.240 0.028 0.004 0.492 0.056 0.180
#> GSM228674 4 0.632 0.3988 0.052 0.028 0.000 0.592 0.100 0.228
#> GSM228675 4 0.770 0.1578 0.016 0.052 0.064 0.436 0.124 0.308
#> GSM228676 5 0.833 -0.1340 0.068 0.012 0.132 0.124 0.356 0.308
#> GSM228667 6 0.883 0.1773 0.052 0.140 0.100 0.112 0.184 0.412
#> GSM228668 4 0.742 0.3572 0.220 0.000 0.100 0.500 0.056 0.124
#> GSM228669 4 0.453 0.5080 0.100 0.000 0.048 0.764 0.004 0.084
#> GSM228673 3 0.606 0.1091 0.008 0.036 0.520 0.032 0.032 0.372
#> GSM228677 6 0.814 0.2934 0.000 0.032 0.264 0.176 0.208 0.320
#> GSM228678 4 0.818 0.1293 0.000 0.252 0.100 0.360 0.072 0.216
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
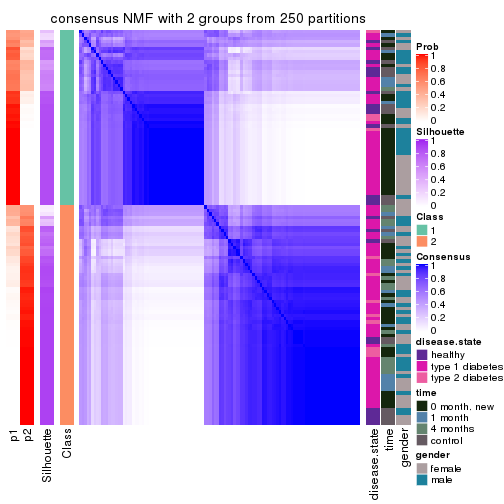
consensus_heatmap(res, k = 3)
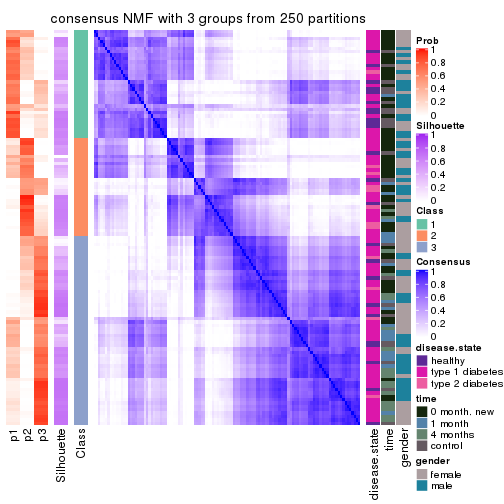
consensus_heatmap(res, k = 4)
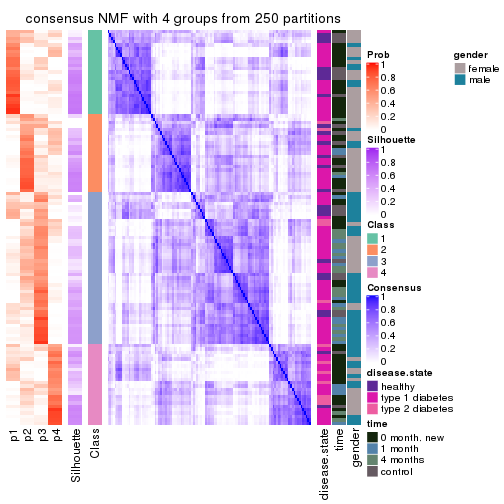
consensus_heatmap(res, k = 5)
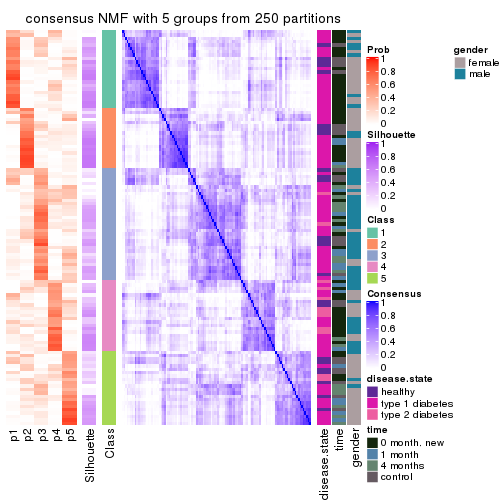
consensus_heatmap(res, k = 6)
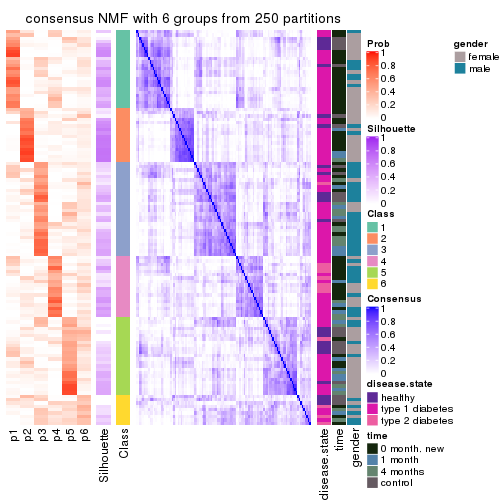
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
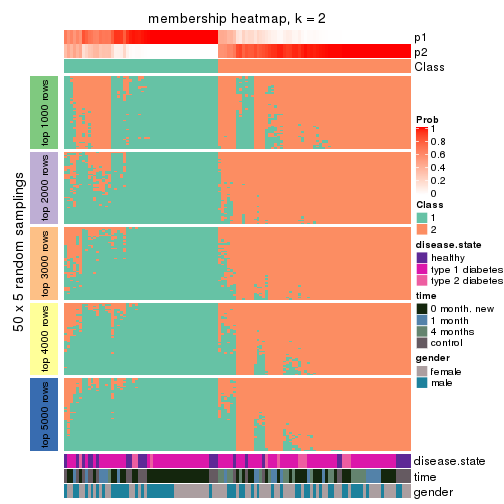
membership_heatmap(res, k = 3)
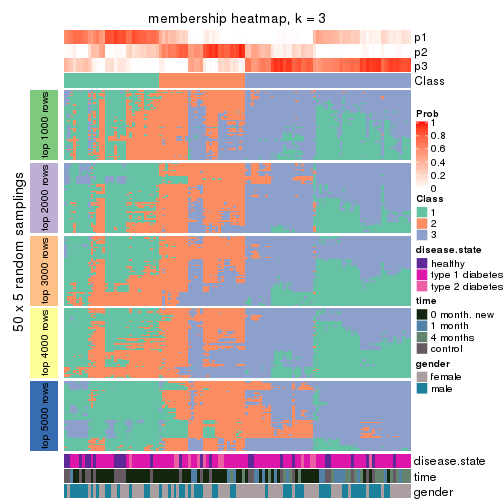
membership_heatmap(res, k = 4)
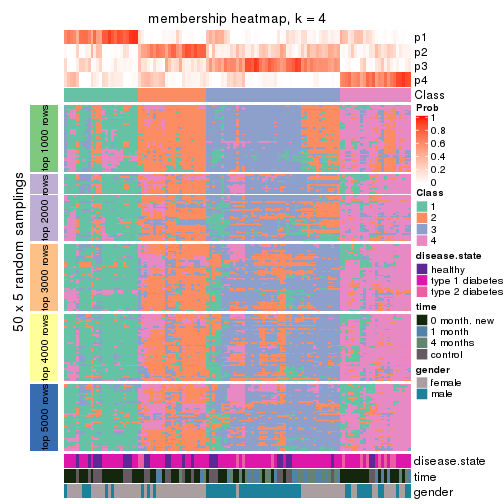
membership_heatmap(res, k = 5)
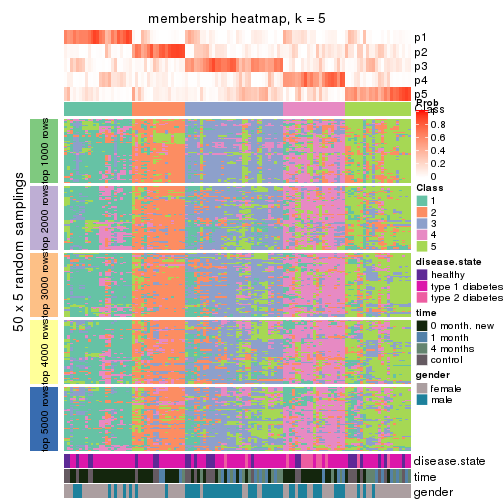
membership_heatmap(res, k = 6)
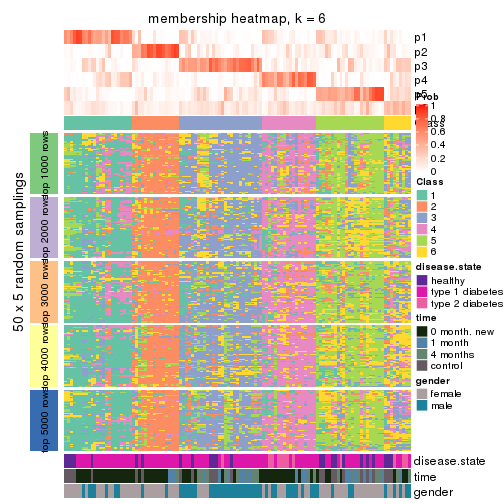
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
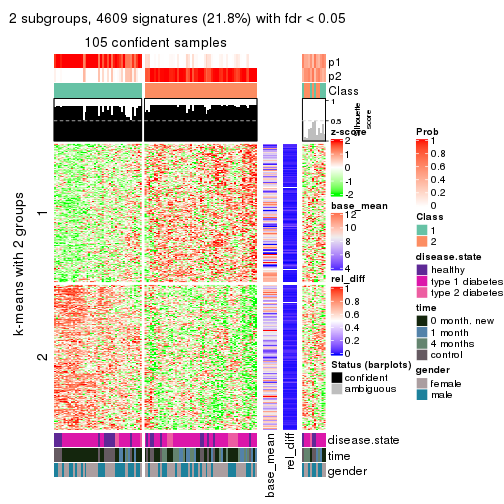
get_signatures(res, k = 3)
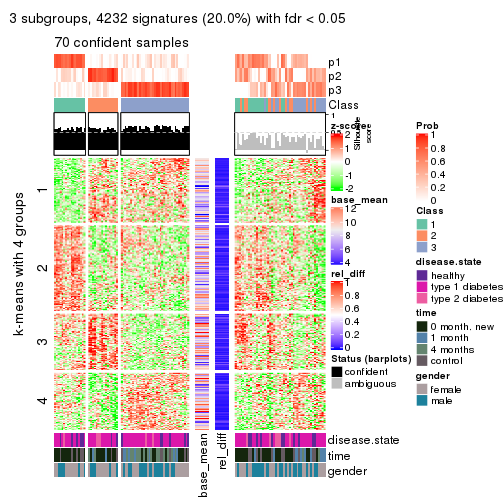
get_signatures(res, k = 4)
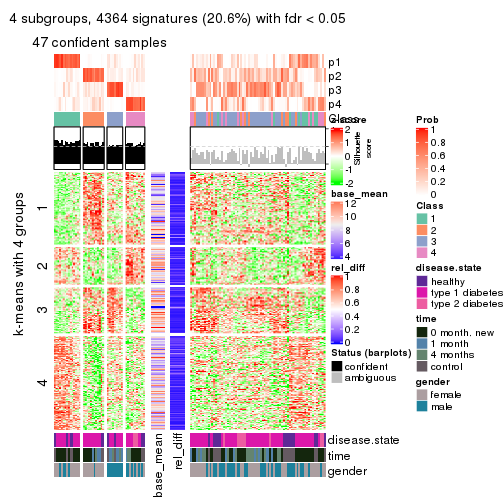
get_signatures(res, k = 5)
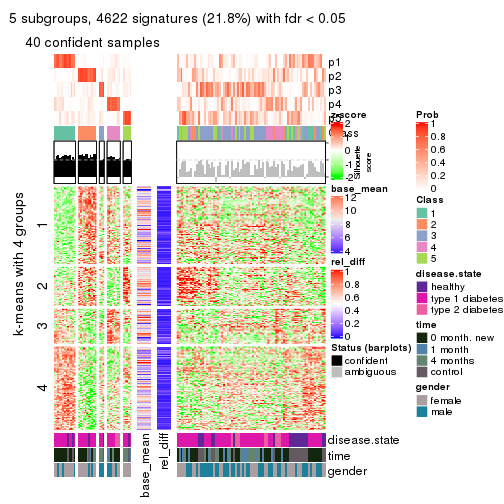
get_signatures(res, k = 6)
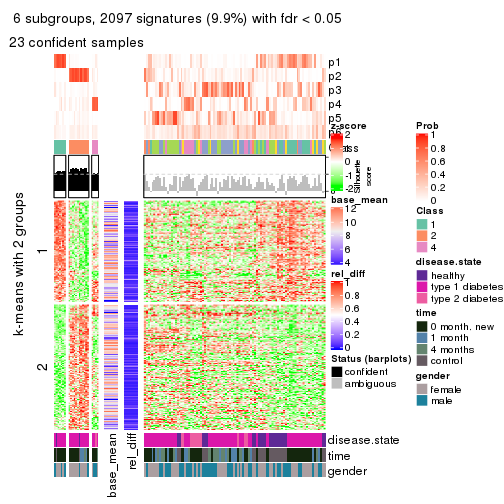
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
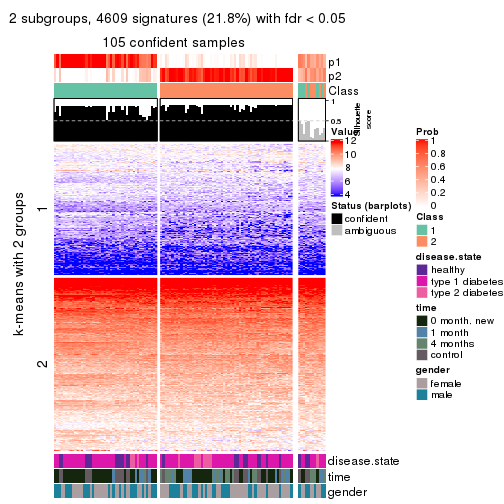
get_signatures(res, k = 3, scale_rows = FALSE)
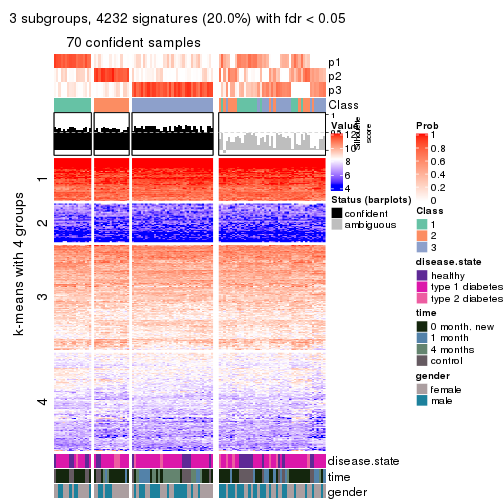
get_signatures(res, k = 4, scale_rows = FALSE)
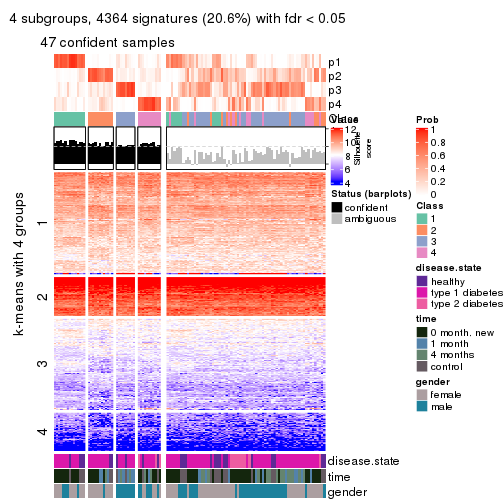
get_signatures(res, k = 5, scale_rows = FALSE)
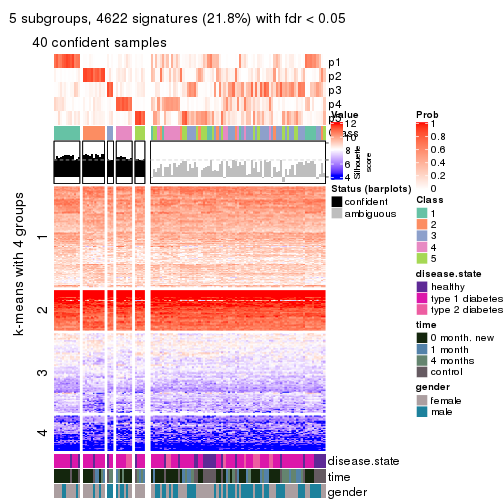
get_signatures(res, k = 6, scale_rows = FALSE)
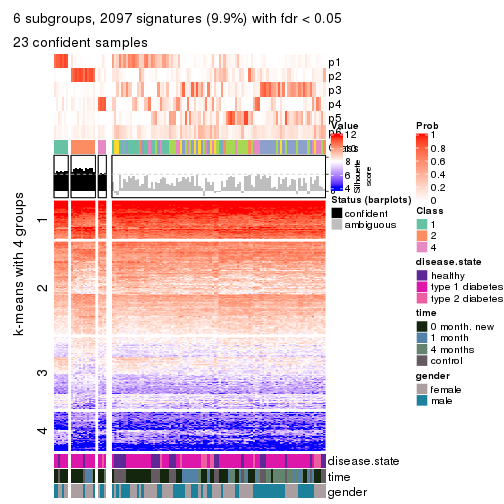
Compare the overlap of signatures from different k:
compare_signatures(res)
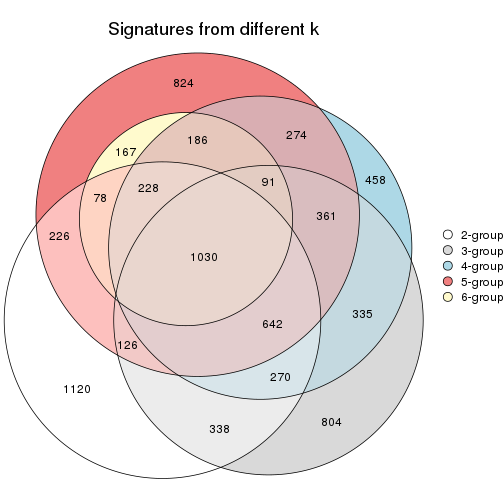
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
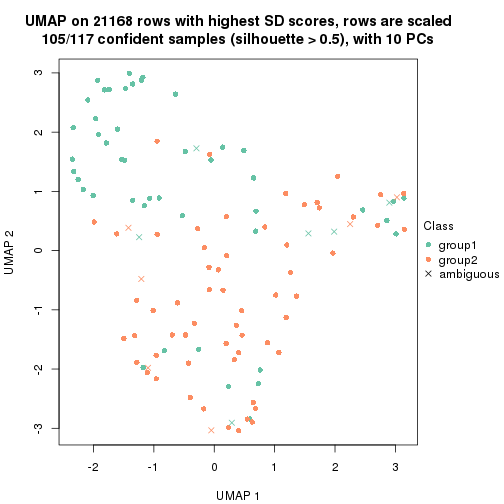
dimension_reduction(res, k = 3, method = "UMAP")
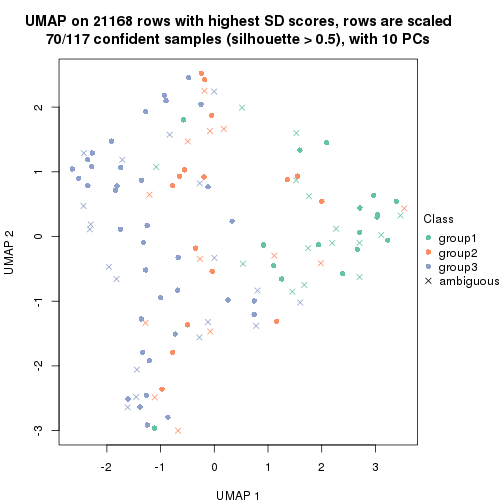
dimension_reduction(res, k = 4, method = "UMAP")
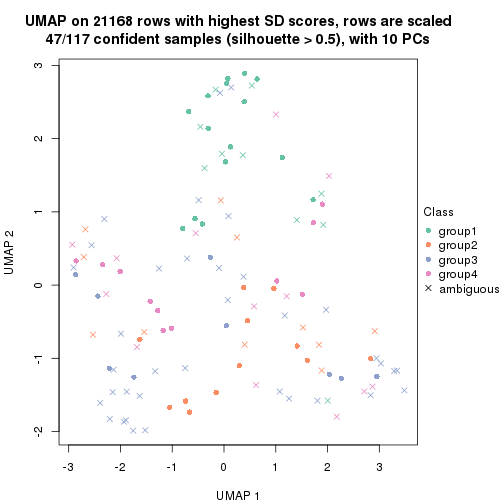
dimension_reduction(res, k = 5, method = "UMAP")
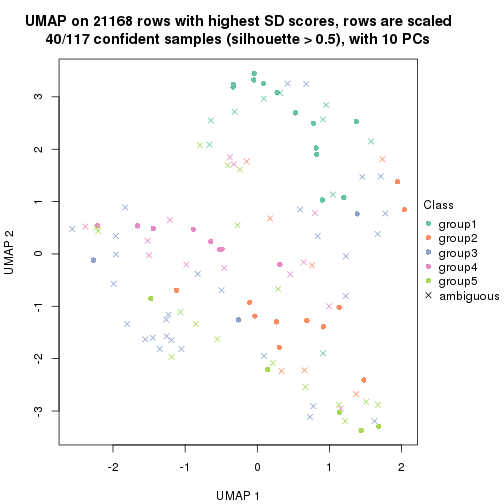
dimension_reduction(res, k = 6, method = "UMAP")
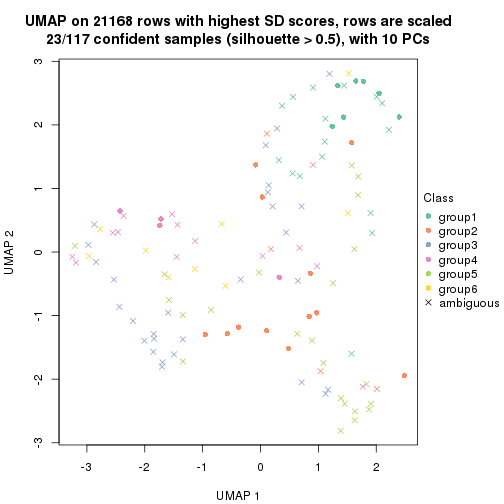
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
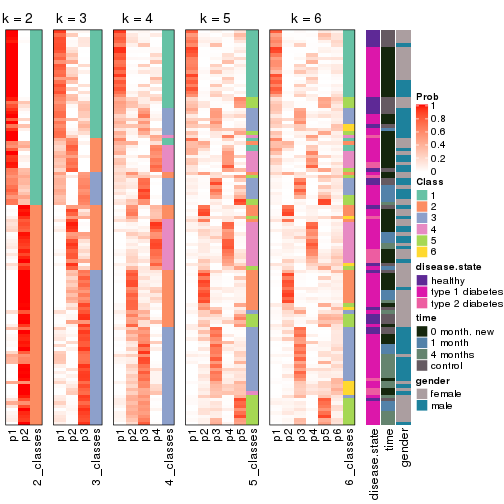
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> SD:NMF 105 0.2476 0.00370 0.593171 2
#> SD:NMF 70 0.3594 0.00183 0.418605 3
#> SD:NMF 47 0.0910 0.01818 0.000162 4
#> SD:NMF 40 0.0586 0.02608 0.019770 5
#> SD:NMF 23 0.2443 0.45979 0.315401 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["CV", "hclust"]
# you can also extract it by
# res = res_list["CV:hclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'CV' method.
#> Subgroups are detected by 'hclust' method.
#> Performed in total 1250 partitions by row resampling.
#> There is no best k.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
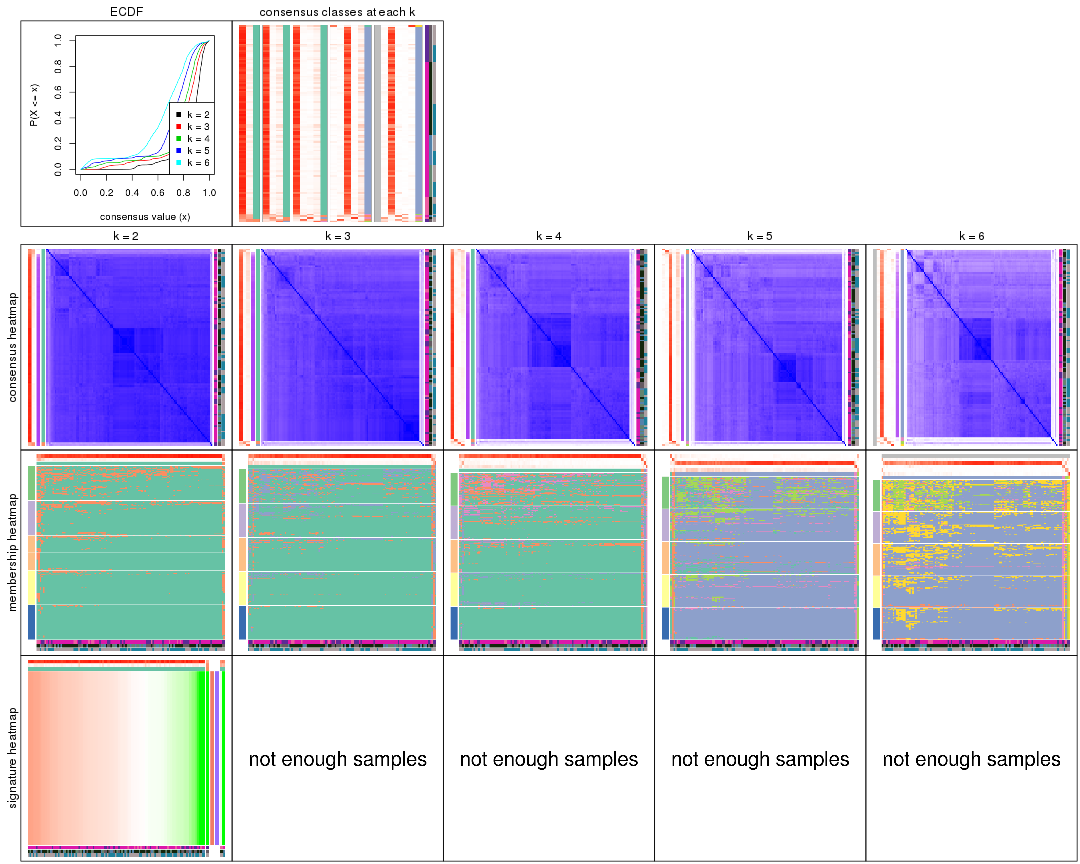
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
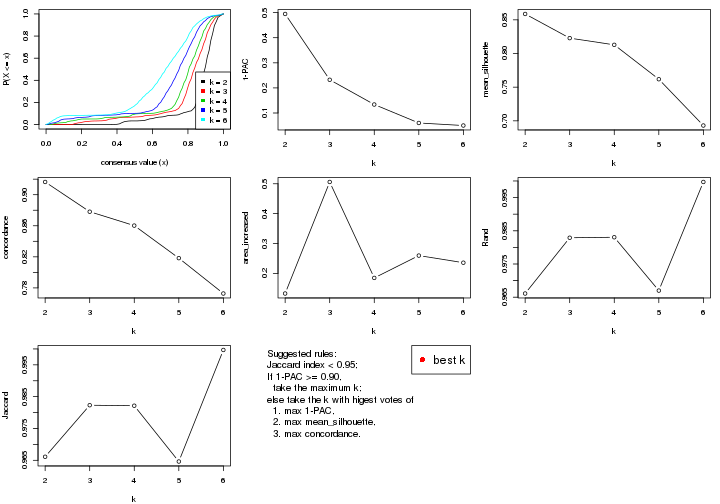
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.4947 0.859 0.916 0.133 0.966 0.966
#> 3 3 0.2324 0.823 0.878 0.506 0.983 0.982
#> 4 4 0.1338 0.813 0.860 0.185 0.983 0.982
#> 5 5 0.0603 0.762 0.818 0.260 0.967 0.965
#> 6 6 0.0501 0.693 0.773 0.236 1.000 1.000
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] NA
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.358 0.908 0.932 0.068
#> GSM228563 1 0.456 0.873 0.904 0.096
#> GSM228565 1 0.295 0.912 0.948 0.052
#> GSM228566 1 0.295 0.911 0.948 0.052
#> GSM228567 1 0.204 0.903 0.968 0.032
#> GSM228570 1 0.204 0.909 0.968 0.032
#> GSM228571 1 0.204 0.911 0.968 0.032
#> GSM228574 1 0.373 0.906 0.928 0.072
#> GSM228575 2 0.983 0.869 0.424 0.576
#> GSM228576 1 0.242 0.912 0.960 0.040
#> GSM228579 1 0.141 0.907 0.980 0.020
#> GSM228580 2 0.983 0.864 0.424 0.576
#> GSM228581 1 0.814 0.551 0.748 0.252
#> GSM228666 1 0.775 0.608 0.772 0.228
#> GSM228564 1 0.506 0.873 0.888 0.112
#> GSM228568 1 0.311 0.907 0.944 0.056
#> GSM228569 1 0.311 0.910 0.944 0.056
#> GSM228572 1 0.506 0.850 0.888 0.112
#> GSM228573 1 0.295 0.909 0.948 0.052
#> GSM228577 1 0.184 0.904 0.972 0.028
#> GSM228578 1 0.204 0.907 0.968 0.032
#> GSM228663 1 0.388 0.896 0.924 0.076
#> GSM228664 1 0.552 0.829 0.872 0.128
#> GSM228665 1 0.260 0.911 0.956 0.044
#> GSM228582 1 0.430 0.892 0.912 0.088
#> GSM228583 1 0.224 0.902 0.964 0.036
#> GSM228585 1 0.242 0.902 0.960 0.040
#> GSM228587 1 0.388 0.901 0.924 0.076
#> GSM228588 1 0.541 0.848 0.876 0.124
#> GSM228589 1 0.402 0.886 0.920 0.080
#> GSM228590 1 0.242 0.903 0.960 0.040
#> GSM228591 1 0.494 0.858 0.892 0.108
#> GSM228597 1 0.482 0.873 0.896 0.104
#> GSM228601 1 0.518 0.846 0.884 0.116
#> GSM228604 1 0.430 0.891 0.912 0.088
#> GSM228608 1 0.311 0.907 0.944 0.056
#> GSM228609 1 0.494 0.874 0.892 0.108
#> GSM228613 1 0.242 0.902 0.960 0.040
#> GSM228616 1 0.402 0.900 0.920 0.080
#> GSM228628 1 0.506 0.853 0.888 0.112
#> GSM228634 1 0.204 0.902 0.968 0.032
#> GSM228642 1 0.574 0.831 0.864 0.136
#> GSM228645 1 0.402 0.900 0.920 0.080
#> GSM228646 1 0.373 0.906 0.928 0.072
#> GSM228652 1 0.260 0.906 0.956 0.044
#> GSM228655 1 0.204 0.909 0.968 0.032
#> GSM228656 1 0.224 0.903 0.964 0.036
#> GSM228659 1 0.469 0.881 0.900 0.100
#> GSM228662 1 0.242 0.902 0.960 0.040
#> GSM228584 1 0.224 0.903 0.964 0.036
#> GSM228586 1 0.204 0.902 0.968 0.032
#> GSM228592 1 0.242 0.902 0.960 0.040
#> GSM228593 1 0.506 0.861 0.888 0.112
#> GSM228594 1 0.204 0.904 0.968 0.032
#> GSM228598 1 0.327 0.909 0.940 0.060
#> GSM228607 1 0.141 0.907 0.980 0.020
#> GSM228612 1 0.327 0.907 0.940 0.060
#> GSM228619 1 0.224 0.905 0.964 0.036
#> GSM228622 1 0.184 0.905 0.972 0.028
#> GSM228625 1 0.242 0.911 0.960 0.040
#> GSM228631 1 0.224 0.908 0.964 0.036
#> GSM228633 1 0.584 0.817 0.860 0.140
#> GSM228637 1 0.563 0.833 0.868 0.132
#> GSM228639 1 0.311 0.912 0.944 0.056
#> GSM228649 1 0.518 0.862 0.884 0.116
#> GSM228660 1 0.141 0.906 0.980 0.020
#> GSM228661 1 0.224 0.903 0.964 0.036
#> GSM228595 1 0.518 0.848 0.884 0.116
#> GSM228599 1 0.529 0.872 0.880 0.120
#> GSM228602 1 0.260 0.909 0.956 0.044
#> GSM228614 1 0.443 0.894 0.908 0.092
#> GSM228626 1 0.563 0.826 0.868 0.132
#> GSM228640 1 0.260 0.909 0.956 0.044
#> GSM228643 1 0.278 0.908 0.952 0.048
#> GSM228650 1 0.373 0.910 0.928 0.072
#> GSM228653 1 0.260 0.912 0.956 0.044
#> GSM228657 1 0.456 0.884 0.904 0.096
#> GSM228605 1 0.224 0.910 0.964 0.036
#> GSM228610 1 0.327 0.911 0.940 0.060
#> GSM228617 1 0.224 0.905 0.964 0.036
#> GSM228620 1 0.224 0.908 0.964 0.036
#> GSM228623 1 0.388 0.892 0.924 0.076
#> GSM228629 1 0.311 0.909 0.944 0.056
#> GSM228632 1 0.242 0.912 0.960 0.040
#> GSM228635 1 0.969 -0.336 0.604 0.396
#> GSM228647 1 0.327 0.910 0.940 0.060
#> GSM228596 1 0.295 0.913 0.948 0.052
#> GSM228600 1 0.311 0.909 0.944 0.056
#> GSM228603 1 0.295 0.909 0.948 0.052
#> GSM228615 1 0.506 0.875 0.888 0.112
#> GSM228627 1 0.343 0.905 0.936 0.064
#> GSM228641 1 0.242 0.910 0.960 0.040
#> GSM228644 1 0.574 0.826 0.864 0.136
#> GSM228651 1 0.343 0.908 0.936 0.064
#> GSM228654 1 0.242 0.909 0.960 0.040
#> GSM228658 1 0.278 0.909 0.952 0.048
#> GSM228606 1 0.260 0.908 0.956 0.044
#> GSM228611 1 0.295 0.905 0.948 0.052
#> GSM228618 1 0.278 0.909 0.952 0.048
#> GSM228621 1 0.224 0.909 0.964 0.036
#> GSM228624 1 0.358 0.902 0.932 0.068
#> GSM228630 1 0.278 0.909 0.952 0.048
#> GSM228636 1 0.634 0.775 0.840 0.160
#> GSM228638 1 0.278 0.912 0.952 0.048
#> GSM228648 1 0.260 0.910 0.956 0.044
#> GSM228670 1 0.430 0.892 0.912 0.088
#> GSM228671 1 0.985 -0.460 0.572 0.428
#> GSM228672 1 0.518 0.873 0.884 0.116
#> GSM228674 1 0.327 0.910 0.940 0.060
#> GSM228675 1 0.909 0.117 0.676 0.324
#> GSM228676 1 0.295 0.912 0.948 0.052
#> GSM228667 1 0.469 0.885 0.900 0.100
#> GSM228668 1 0.204 0.907 0.968 0.032
#> GSM228669 1 0.278 0.907 0.952 0.048
#> GSM228673 1 0.482 0.876 0.896 0.104
#> GSM228677 1 0.416 0.899 0.916 0.084
#> GSM228678 1 0.494 0.868 0.892 0.108
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.343 0.9002 0.904 0.032 0.064
#> GSM228563 1 0.543 0.8222 0.808 0.048 0.144
#> GSM228565 1 0.323 0.9036 0.908 0.020 0.072
#> GSM228566 1 0.331 0.9001 0.908 0.028 0.064
#> GSM228567 1 0.200 0.8913 0.952 0.012 0.036
#> GSM228570 1 0.215 0.8984 0.948 0.016 0.036
#> GSM228571 1 0.203 0.8999 0.952 0.016 0.032
#> GSM228574 1 0.331 0.8991 0.908 0.028 0.064
#> GSM228575 2 0.930 -0.4338 0.160 0.432 0.408
#> GSM228576 1 0.264 0.9025 0.932 0.020 0.048
#> GSM228579 1 0.188 0.8971 0.956 0.012 0.032
#> GSM228580 3 0.721 0.0000 0.192 0.100 0.708
#> GSM228581 1 0.777 0.4442 0.640 0.088 0.272
#> GSM228666 1 0.756 0.5400 0.672 0.096 0.232
#> GSM228564 1 0.524 0.8420 0.820 0.048 0.132
#> GSM228568 1 0.323 0.8956 0.908 0.020 0.072
#> GSM228569 1 0.290 0.9040 0.920 0.016 0.064
#> GSM228572 1 0.563 0.7992 0.792 0.044 0.164
#> GSM228573 1 0.238 0.8983 0.940 0.016 0.044
#> GSM228577 1 0.171 0.8924 0.960 0.008 0.032
#> GSM228578 1 0.215 0.8985 0.948 0.016 0.036
#> GSM228663 1 0.404 0.8758 0.880 0.040 0.080
#> GSM228664 1 0.550 0.7962 0.804 0.048 0.148
#> GSM228665 1 0.266 0.9018 0.932 0.024 0.044
#> GSM228582 1 0.441 0.8678 0.852 0.024 0.124
#> GSM228583 1 0.212 0.8907 0.948 0.012 0.040
#> GSM228585 1 0.223 0.8912 0.944 0.012 0.044
#> GSM228587 1 0.319 0.8925 0.896 0.004 0.100
#> GSM228588 1 0.535 0.8232 0.808 0.040 0.152
#> GSM228589 1 0.441 0.8611 0.852 0.024 0.124
#> GSM228590 1 0.223 0.8916 0.944 0.012 0.044
#> GSM228591 1 0.454 0.8433 0.836 0.016 0.148
#> GSM228597 1 0.475 0.8599 0.844 0.040 0.116
#> GSM228601 1 0.517 0.8065 0.804 0.024 0.172
#> GSM228604 1 0.434 0.8717 0.856 0.024 0.120
#> GSM228608 1 0.277 0.8962 0.920 0.008 0.072
#> GSM228609 1 0.494 0.8547 0.824 0.028 0.148
#> GSM228613 1 0.223 0.8912 0.944 0.012 0.044
#> GSM228616 1 0.350 0.8945 0.896 0.020 0.084
#> GSM228628 1 0.522 0.8246 0.816 0.040 0.144
#> GSM228634 1 0.200 0.8905 0.952 0.012 0.036
#> GSM228642 1 0.533 0.8229 0.812 0.044 0.144
#> GSM228645 1 0.468 0.8575 0.836 0.024 0.140
#> GSM228646 1 0.389 0.8932 0.880 0.024 0.096
#> GSM228652 1 0.223 0.8964 0.944 0.012 0.044
#> GSM228655 1 0.238 0.9009 0.936 0.008 0.056
#> GSM228656 1 0.206 0.8931 0.948 0.008 0.044
#> GSM228659 1 0.439 0.8664 0.840 0.012 0.148
#> GSM228662 1 0.223 0.8912 0.944 0.012 0.044
#> GSM228584 1 0.212 0.8917 0.948 0.012 0.040
#> GSM228586 1 0.200 0.8905 0.952 0.012 0.036
#> GSM228592 1 0.223 0.8912 0.944 0.012 0.044
#> GSM228593 1 0.474 0.8505 0.836 0.028 0.136
#> GSM228594 1 0.183 0.8924 0.956 0.008 0.036
#> GSM228598 1 0.333 0.8996 0.904 0.020 0.076
#> GSM228607 1 0.234 0.9023 0.940 0.012 0.048
#> GSM228612 1 0.380 0.8937 0.888 0.032 0.080
#> GSM228619 1 0.153 0.8943 0.964 0.004 0.032
#> GSM228622 1 0.203 0.8948 0.952 0.016 0.032
#> GSM228625 1 0.212 0.9022 0.948 0.012 0.040
#> GSM228631 1 0.158 0.8961 0.964 0.008 0.028
#> GSM228633 1 0.590 0.7730 0.768 0.040 0.192
#> GSM228637 1 0.580 0.7752 0.776 0.040 0.184
#> GSM228639 1 0.336 0.9014 0.908 0.036 0.056
#> GSM228649 1 0.517 0.8203 0.804 0.024 0.172
#> GSM228660 1 0.134 0.8966 0.972 0.012 0.016
#> GSM228661 1 0.212 0.8917 0.948 0.012 0.040
#> GSM228595 1 0.498 0.8117 0.812 0.020 0.168
#> GSM228599 1 0.475 0.8745 0.844 0.040 0.116
#> GSM228602 1 0.203 0.8982 0.952 0.016 0.032
#> GSM228614 1 0.459 0.8801 0.856 0.048 0.096
#> GSM228626 1 0.569 0.7799 0.784 0.040 0.176
#> GSM228640 1 0.200 0.8967 0.952 0.012 0.036
#> GSM228643 1 0.285 0.8998 0.924 0.020 0.056
#> GSM228650 1 0.397 0.8918 0.880 0.032 0.088
#> GSM228653 1 0.230 0.9013 0.944 0.020 0.036
#> GSM228657 1 0.427 0.8733 0.860 0.024 0.116
#> GSM228605 1 0.281 0.9016 0.928 0.032 0.040
#> GSM228610 1 0.338 0.8987 0.908 0.048 0.044
#> GSM228617 1 0.153 0.8943 0.964 0.004 0.032
#> GSM228620 1 0.212 0.8988 0.948 0.012 0.040
#> GSM228623 1 0.461 0.8642 0.856 0.052 0.092
#> GSM228629 1 0.241 0.9012 0.940 0.020 0.040
#> GSM228632 1 0.212 0.9014 0.948 0.012 0.040
#> GSM228635 1 0.984 -0.5765 0.388 0.248 0.364
#> GSM228647 1 0.298 0.8999 0.920 0.024 0.056
#> GSM228596 1 0.292 0.9033 0.924 0.032 0.044
#> GSM228600 1 0.238 0.8988 0.940 0.016 0.044
#> GSM228603 1 0.227 0.8975 0.944 0.016 0.040
#> GSM228615 1 0.486 0.8675 0.840 0.044 0.116
#> GSM228627 1 0.298 0.8978 0.920 0.024 0.056
#> GSM228641 1 0.195 0.8991 0.952 0.008 0.040
#> GSM228644 1 0.533 0.7972 0.792 0.024 0.184
#> GSM228651 1 0.293 0.8999 0.924 0.036 0.040
#> GSM228654 1 0.234 0.9008 0.940 0.012 0.048
#> GSM228658 1 0.264 0.8998 0.932 0.020 0.048
#> GSM228606 1 0.304 0.8955 0.920 0.036 0.044
#> GSM228611 1 0.304 0.8959 0.920 0.040 0.040
#> GSM228618 1 0.217 0.8979 0.944 0.008 0.048
#> GSM228621 1 0.223 0.8988 0.944 0.012 0.044
#> GSM228624 1 0.359 0.8890 0.900 0.048 0.052
#> GSM228630 1 0.264 0.8995 0.932 0.020 0.048
#> GSM228636 1 0.658 0.7088 0.740 0.068 0.192
#> GSM228638 1 0.244 0.9016 0.940 0.028 0.032
#> GSM228648 1 0.295 0.8958 0.920 0.020 0.060
#> GSM228670 1 0.444 0.8830 0.864 0.052 0.084
#> GSM228671 2 0.787 -0.1802 0.348 0.584 0.068
#> GSM228672 1 0.524 0.8476 0.820 0.048 0.132
#> GSM228674 1 0.379 0.8976 0.892 0.048 0.060
#> GSM228675 1 0.812 -0.0411 0.552 0.372 0.076
#> GSM228676 1 0.346 0.9005 0.904 0.036 0.060
#> GSM228667 1 0.484 0.8669 0.844 0.052 0.104
#> GSM228668 1 0.227 0.8998 0.944 0.016 0.040
#> GSM228669 1 0.238 0.8963 0.940 0.016 0.044
#> GSM228673 1 0.496 0.8523 0.832 0.040 0.128
#> GSM228677 1 0.434 0.8803 0.868 0.048 0.084
#> GSM228678 1 0.518 0.8303 0.812 0.032 0.156
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 1 0.342 0.895 0.884 0.016 0.044 0.056
#> GSM228563 1 0.552 0.795 0.768 0.036 0.064 0.132
#> GSM228565 1 0.360 0.897 0.876 0.032 0.024 0.068
#> GSM228566 1 0.354 0.893 0.880 0.036 0.024 0.060
#> GSM228567 1 0.177 0.882 0.948 0.012 0.004 0.036
#> GSM228570 1 0.201 0.891 0.940 0.008 0.012 0.040
#> GSM228571 1 0.194 0.893 0.944 0.008 0.016 0.032
#> GSM228574 1 0.345 0.893 0.884 0.020 0.044 0.052
#> GSM228575 2 0.570 -0.390 0.116 0.764 0.072 0.048
#> GSM228576 1 0.272 0.896 0.916 0.020 0.024 0.040
#> GSM228579 1 0.173 0.889 0.948 0.008 0.004 0.040
#> GSM228580 4 0.580 0.000 0.040 0.116 0.088 0.756
#> GSM228581 1 0.856 -0.109 0.496 0.072 0.164 0.268
#> GSM228666 1 0.736 0.508 0.632 0.140 0.048 0.180
#> GSM228564 1 0.550 0.816 0.780 0.072 0.052 0.096
#> GSM228568 1 0.352 0.886 0.876 0.016 0.032 0.076
#> GSM228569 1 0.278 0.898 0.904 0.016 0.008 0.072
#> GSM228572 1 0.570 0.775 0.752 0.040 0.056 0.152
#> GSM228573 1 0.247 0.892 0.924 0.016 0.016 0.044
#> GSM228577 1 0.219 0.888 0.932 0.008 0.012 0.048
#> GSM228578 1 0.250 0.893 0.924 0.016 0.020 0.040
#> GSM228663 1 0.381 0.873 0.864 0.016 0.048 0.072
#> GSM228664 1 0.550 0.770 0.764 0.024 0.076 0.136
#> GSM228665 1 0.256 0.896 0.920 0.036 0.008 0.036
#> GSM228582 1 0.406 0.863 0.828 0.020 0.012 0.140
#> GSM228583 1 0.186 0.882 0.944 0.012 0.004 0.040
#> GSM228585 1 0.222 0.883 0.932 0.016 0.008 0.044
#> GSM228587 1 0.303 0.887 0.888 0.020 0.004 0.088
#> GSM228588 1 0.470 0.819 0.784 0.016 0.024 0.176
#> GSM228589 1 0.393 0.854 0.832 0.020 0.008 0.140
#> GSM228590 1 0.198 0.883 0.940 0.016 0.004 0.040
#> GSM228591 1 0.421 0.842 0.816 0.012 0.020 0.152
#> GSM228597 1 0.495 0.841 0.808 0.048 0.044 0.100
#> GSM228601 1 0.485 0.796 0.776 0.016 0.028 0.180
#> GSM228604 1 0.427 0.863 0.832 0.028 0.024 0.116
#> GSM228608 1 0.249 0.889 0.916 0.016 0.004 0.064
#> GSM228609 1 0.447 0.848 0.800 0.020 0.016 0.164
#> GSM228613 1 0.195 0.883 0.940 0.012 0.004 0.044
#> GSM228616 1 0.346 0.888 0.880 0.028 0.020 0.072
#> GSM228628 1 0.460 0.821 0.796 0.012 0.032 0.160
#> GSM228634 1 0.188 0.883 0.944 0.008 0.008 0.040
#> GSM228642 1 0.484 0.821 0.792 0.016 0.044 0.148
#> GSM228645 1 0.519 0.838 0.792 0.056 0.040 0.112
#> GSM228646 1 0.400 0.886 0.856 0.036 0.028 0.080
#> GSM228652 1 0.195 0.889 0.940 0.012 0.004 0.044
#> GSM228655 1 0.230 0.895 0.924 0.008 0.008 0.060
#> GSM228656 1 0.185 0.884 0.940 0.012 0.000 0.048
#> GSM228659 1 0.428 0.864 0.824 0.028 0.016 0.132
#> GSM228662 1 0.207 0.882 0.936 0.016 0.004 0.044
#> GSM228584 1 0.195 0.883 0.940 0.012 0.004 0.044
#> GSM228586 1 0.173 0.882 0.948 0.008 0.004 0.040
#> GSM228592 1 0.207 0.882 0.936 0.016 0.004 0.044
#> GSM228593 1 0.487 0.838 0.804 0.048 0.028 0.120
#> GSM228594 1 0.168 0.884 0.948 0.012 0.000 0.040
#> GSM228598 1 0.307 0.894 0.892 0.024 0.008 0.076
#> GSM228607 1 0.241 0.896 0.928 0.016 0.020 0.036
#> GSM228612 1 0.384 0.886 0.860 0.016 0.040 0.084
#> GSM228619 1 0.182 0.888 0.948 0.008 0.012 0.032
#> GSM228622 1 0.207 0.888 0.940 0.012 0.016 0.032
#> GSM228625 1 0.220 0.896 0.936 0.024 0.012 0.028
#> GSM228631 1 0.182 0.890 0.948 0.012 0.008 0.032
#> GSM228633 1 0.545 0.766 0.748 0.024 0.044 0.184
#> GSM228637 1 0.611 0.741 0.728 0.052 0.060 0.160
#> GSM228639 1 0.358 0.894 0.880 0.036 0.032 0.052
#> GSM228649 1 0.559 0.795 0.760 0.048 0.044 0.148
#> GSM228660 1 0.185 0.893 0.948 0.008 0.020 0.024
#> GSM228661 1 0.195 0.883 0.940 0.012 0.004 0.044
#> GSM228595 1 0.472 0.804 0.788 0.016 0.028 0.168
#> GSM228599 1 0.474 0.868 0.820 0.056 0.036 0.088
#> GSM228602 1 0.197 0.892 0.944 0.016 0.012 0.028
#> GSM228614 1 0.463 0.869 0.828 0.044 0.048 0.080
#> GSM228626 1 0.519 0.763 0.752 0.016 0.036 0.196
#> GSM228640 1 0.238 0.891 0.928 0.016 0.016 0.040
#> GSM228643 1 0.303 0.894 0.904 0.032 0.024 0.040
#> GSM228650 1 0.389 0.887 0.864 0.044 0.028 0.064
#> GSM228653 1 0.262 0.894 0.920 0.032 0.016 0.032
#> GSM228657 1 0.405 0.867 0.844 0.028 0.020 0.108
#> GSM228605 1 0.299 0.896 0.904 0.016 0.044 0.036
#> GSM228610 1 0.369 0.892 0.876 0.040 0.040 0.044
#> GSM228617 1 0.182 0.888 0.948 0.008 0.012 0.032
#> GSM228620 1 0.267 0.894 0.916 0.016 0.020 0.048
#> GSM228623 1 0.482 0.843 0.816 0.036 0.060 0.088
#> GSM228629 1 0.252 0.895 0.924 0.028 0.016 0.032
#> GSM228632 1 0.281 0.895 0.912 0.032 0.016 0.040
#> GSM228635 2 0.914 -0.150 0.308 0.416 0.108 0.168
#> GSM228647 1 0.308 0.893 0.900 0.028 0.020 0.052
#> GSM228596 1 0.256 0.897 0.920 0.036 0.008 0.036
#> GSM228600 1 0.213 0.893 0.936 0.016 0.008 0.040
#> GSM228603 1 0.220 0.891 0.936 0.016 0.016 0.032
#> GSM228615 1 0.480 0.857 0.816 0.048 0.040 0.096
#> GSM228627 1 0.298 0.892 0.904 0.024 0.020 0.052
#> GSM228641 1 0.233 0.893 0.928 0.008 0.020 0.044
#> GSM228644 1 0.505 0.779 0.756 0.028 0.016 0.200
#> GSM228651 1 0.269 0.895 0.916 0.036 0.012 0.036
#> GSM228654 1 0.272 0.894 0.916 0.024 0.020 0.040
#> GSM228658 1 0.283 0.894 0.912 0.024 0.024 0.040
#> GSM228606 1 0.332 0.887 0.892 0.032 0.032 0.044
#> GSM228611 1 0.332 0.888 0.892 0.032 0.032 0.044
#> GSM228618 1 0.247 0.892 0.924 0.020 0.012 0.044
#> GSM228621 1 0.267 0.892 0.916 0.020 0.016 0.048
#> GSM228624 1 0.391 0.876 0.864 0.028 0.052 0.056
#> GSM228630 1 0.312 0.892 0.900 0.028 0.028 0.044
#> GSM228636 1 0.698 0.639 0.676 0.120 0.060 0.144
#> GSM228638 1 0.263 0.897 0.920 0.020 0.024 0.036
#> GSM228648 1 0.308 0.889 0.900 0.028 0.020 0.052
#> GSM228670 1 0.458 0.867 0.824 0.020 0.072 0.084
#> GSM228671 3 0.437 0.000 0.168 0.008 0.800 0.024
#> GSM228672 1 0.495 0.844 0.800 0.028 0.052 0.120
#> GSM228674 1 0.382 0.887 0.868 0.028 0.048 0.056
#> GSM228675 1 0.702 -0.230 0.480 0.060 0.436 0.024
#> GSM228676 1 0.401 0.891 0.860 0.036 0.044 0.060
#> GSM228667 1 0.464 0.855 0.816 0.016 0.064 0.104
#> GSM228668 1 0.266 0.895 0.916 0.024 0.012 0.048
#> GSM228669 1 0.267 0.891 0.916 0.020 0.016 0.048
#> GSM228673 1 0.510 0.843 0.800 0.060 0.040 0.100
#> GSM228677 1 0.454 0.868 0.832 0.044 0.044 0.080
#> GSM228678 1 0.559 0.795 0.764 0.068 0.036 0.132
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 3 0.373 0.862 0.024 0.012 0.844 0.024 0.096
#> GSM228563 3 0.523 0.746 0.004 0.056 0.716 0.028 0.196
#> GSM228565 3 0.351 0.866 0.016 0.016 0.848 0.012 0.108
#> GSM228566 3 0.380 0.861 0.016 0.024 0.832 0.012 0.116
#> GSM228567 3 0.186 0.846 0.008 0.008 0.936 0.004 0.044
#> GSM228570 3 0.217 0.861 0.008 0.008 0.924 0.012 0.048
#> GSM228571 3 0.173 0.861 0.008 0.000 0.940 0.012 0.040
#> GSM228574 3 0.401 0.856 0.028 0.012 0.828 0.028 0.104
#> GSM228575 2 0.414 -0.321 0.000 0.816 0.084 0.032 0.068
#> GSM228576 3 0.273 0.867 0.004 0.012 0.892 0.016 0.076
#> GSM228579 3 0.168 0.858 0.004 0.004 0.940 0.004 0.048
#> GSM228580 5 0.738 0.000 0.408 0.084 0.012 0.076 0.420
#> GSM228581 1 0.547 0.000 0.680 0.000 0.148 0.008 0.164
#> GSM228666 3 0.763 0.305 0.164 0.096 0.544 0.016 0.180
#> GSM228564 3 0.488 0.777 0.012 0.044 0.752 0.020 0.172
#> GSM228568 3 0.454 0.836 0.064 0.016 0.804 0.028 0.088
#> GSM228569 3 0.300 0.867 0.036 0.008 0.880 0.004 0.072
#> GSM228572 3 0.533 0.711 0.008 0.036 0.696 0.032 0.228
#> GSM228573 3 0.283 0.860 0.020 0.012 0.892 0.008 0.068
#> GSM228577 3 0.193 0.852 0.004 0.008 0.932 0.008 0.048
#> GSM228578 3 0.200 0.859 0.004 0.016 0.932 0.008 0.040
#> GSM228663 3 0.463 0.818 0.056 0.012 0.792 0.028 0.112
#> GSM228664 3 0.631 0.605 0.124 0.008 0.648 0.040 0.180
#> GSM228665 3 0.312 0.864 0.004 0.028 0.872 0.012 0.084
#> GSM228582 3 0.432 0.800 0.036 0.008 0.768 0.004 0.184
#> GSM228583 3 0.186 0.844 0.008 0.008 0.936 0.004 0.044
#> GSM228585 3 0.224 0.846 0.008 0.012 0.920 0.008 0.052
#> GSM228587 3 0.302 0.850 0.012 0.008 0.864 0.004 0.112
#> GSM228588 3 0.475 0.741 0.020 0.016 0.716 0.008 0.240
#> GSM228589 3 0.439 0.779 0.032 0.008 0.764 0.008 0.188
#> GSM228590 3 0.207 0.847 0.008 0.008 0.924 0.004 0.056
#> GSM228591 3 0.474 0.757 0.040 0.008 0.732 0.008 0.212
#> GSM228597 3 0.454 0.786 0.000 0.040 0.756 0.020 0.184
#> GSM228601 3 0.505 0.655 0.032 0.004 0.668 0.012 0.284
#> GSM228604 3 0.481 0.798 0.036 0.020 0.744 0.008 0.192
#> GSM228608 3 0.269 0.854 0.012 0.008 0.888 0.004 0.088
#> GSM228609 3 0.435 0.790 0.008 0.012 0.744 0.012 0.224
#> GSM228613 3 0.200 0.846 0.008 0.008 0.928 0.004 0.052
#> GSM228616 3 0.423 0.845 0.028 0.032 0.808 0.008 0.124
#> GSM228628 3 0.518 0.730 0.044 0.016 0.708 0.012 0.220
#> GSM228634 3 0.186 0.845 0.004 0.008 0.936 0.008 0.044
#> GSM228642 3 0.540 0.685 0.024 0.020 0.668 0.020 0.268
#> GSM228645 3 0.528 0.785 0.024 0.060 0.740 0.020 0.156
#> GSM228646 3 0.393 0.850 0.024 0.020 0.820 0.008 0.128
#> GSM228652 3 0.214 0.856 0.008 0.008 0.920 0.004 0.060
#> GSM228655 3 0.240 0.864 0.008 0.008 0.904 0.004 0.076
#> GSM228656 3 0.183 0.847 0.004 0.012 0.932 0.000 0.052
#> GSM228659 3 0.393 0.834 0.012 0.008 0.800 0.016 0.164
#> GSM228662 3 0.212 0.845 0.008 0.012 0.924 0.004 0.052
#> GSM228584 3 0.192 0.846 0.004 0.012 0.932 0.004 0.048
#> GSM228586 3 0.173 0.844 0.004 0.008 0.940 0.004 0.044
#> GSM228592 3 0.212 0.845 0.008 0.012 0.924 0.004 0.052
#> GSM228593 3 0.486 0.796 0.036 0.032 0.768 0.016 0.148
#> GSM228594 3 0.168 0.847 0.004 0.012 0.940 0.000 0.044
#> GSM228598 3 0.358 0.857 0.044 0.020 0.852 0.004 0.080
#> GSM228607 3 0.246 0.865 0.004 0.008 0.900 0.008 0.080
#> GSM228612 3 0.459 0.826 0.036 0.012 0.784 0.028 0.140
#> GSM228619 3 0.220 0.857 0.004 0.012 0.916 0.004 0.064
#> GSM228622 3 0.173 0.852 0.004 0.012 0.944 0.008 0.032
#> GSM228625 3 0.218 0.867 0.000 0.008 0.912 0.008 0.072
#> GSM228631 3 0.204 0.859 0.000 0.012 0.920 0.004 0.064
#> GSM228633 3 0.585 0.600 0.048 0.020 0.628 0.016 0.288
#> GSM228637 3 0.575 0.630 0.028 0.040 0.664 0.020 0.248
#> GSM228639 3 0.348 0.859 0.008 0.024 0.844 0.008 0.116
#> GSM228649 3 0.513 0.729 0.024 0.028 0.716 0.016 0.216
#> GSM228660 3 0.219 0.861 0.012 0.016 0.928 0.012 0.032
#> GSM228661 3 0.192 0.846 0.004 0.012 0.932 0.004 0.048
#> GSM228595 3 0.511 0.655 0.048 0.000 0.668 0.012 0.272
#> GSM228599 3 0.460 0.822 0.012 0.024 0.760 0.020 0.184
#> GSM228602 3 0.214 0.860 0.004 0.008 0.920 0.008 0.060
#> GSM228614 3 0.436 0.833 0.004 0.032 0.784 0.024 0.156
#> GSM228626 3 0.576 0.605 0.064 0.016 0.632 0.008 0.280
#> GSM228640 3 0.205 0.861 0.000 0.004 0.916 0.008 0.072
#> GSM228643 3 0.320 0.862 0.020 0.020 0.868 0.004 0.088
#> GSM228650 3 0.411 0.851 0.016 0.040 0.812 0.008 0.124
#> GSM228653 3 0.304 0.863 0.012 0.024 0.876 0.004 0.084
#> GSM228657 3 0.415 0.786 0.008 0.012 0.748 0.004 0.228
#> GSM228605 3 0.306 0.866 0.004 0.024 0.880 0.020 0.072
#> GSM228610 3 0.368 0.861 0.008 0.032 0.848 0.024 0.088
#> GSM228617 3 0.220 0.857 0.004 0.012 0.916 0.004 0.064
#> GSM228620 3 0.274 0.863 0.008 0.016 0.892 0.008 0.076
#> GSM228623 3 0.468 0.798 0.008 0.036 0.764 0.024 0.168
#> GSM228629 3 0.279 0.863 0.008 0.016 0.892 0.012 0.072
#> GSM228632 3 0.336 0.862 0.008 0.028 0.860 0.012 0.092
#> GSM228635 2 0.874 -0.185 0.048 0.324 0.240 0.072 0.316
#> GSM228647 3 0.306 0.861 0.020 0.012 0.872 0.004 0.092
#> GSM228596 3 0.281 0.866 0.004 0.024 0.888 0.008 0.076
#> GSM228600 3 0.254 0.861 0.020 0.012 0.900 0.000 0.068
#> GSM228603 3 0.260 0.860 0.016 0.012 0.904 0.008 0.060
#> GSM228615 3 0.440 0.823 0.004 0.036 0.784 0.024 0.152
#> GSM228627 3 0.296 0.863 0.032 0.012 0.884 0.004 0.068
#> GSM228641 3 0.273 0.863 0.016 0.008 0.896 0.012 0.068
#> GSM228644 3 0.536 0.646 0.068 0.004 0.648 0.004 0.276
#> GSM228651 3 0.319 0.862 0.020 0.024 0.864 0.000 0.092
#> GSM228654 3 0.309 0.863 0.016 0.024 0.868 0.000 0.092
#> GSM228658 3 0.335 0.861 0.020 0.024 0.860 0.004 0.092
#> GSM228606 3 0.371 0.849 0.016 0.020 0.840 0.016 0.108
#> GSM228611 3 0.366 0.851 0.028 0.028 0.856 0.016 0.072
#> GSM228618 3 0.268 0.862 0.008 0.016 0.896 0.008 0.072
#> GSM228621 3 0.322 0.860 0.020 0.016 0.864 0.004 0.096
#> GSM228624 3 0.491 0.818 0.052 0.016 0.776 0.036 0.120
#> GSM228630 3 0.370 0.855 0.016 0.024 0.844 0.016 0.100
#> GSM228636 3 0.632 0.544 0.020 0.076 0.632 0.032 0.240
#> GSM228638 3 0.271 0.867 0.004 0.016 0.896 0.016 0.068
#> GSM228648 3 0.357 0.847 0.024 0.016 0.840 0.004 0.116
#> GSM228670 3 0.414 0.829 0.004 0.012 0.800 0.044 0.140
#> GSM228671 4 0.266 -0.566 0.004 0.000 0.064 0.892 0.040
#> GSM228672 3 0.497 0.793 0.028 0.016 0.748 0.032 0.176
#> GSM228674 3 0.397 0.852 0.012 0.024 0.828 0.028 0.108
#> GSM228675 4 0.663 -0.211 0.000 0.060 0.420 0.456 0.064
#> GSM228676 3 0.412 0.860 0.020 0.028 0.824 0.024 0.104
#> GSM228667 3 0.484 0.800 0.016 0.020 0.756 0.036 0.172
#> GSM228668 3 0.233 0.863 0.004 0.020 0.912 0.004 0.060
#> GSM228669 3 0.223 0.859 0.000 0.012 0.912 0.008 0.068
#> GSM228673 3 0.539 0.784 0.056 0.032 0.744 0.028 0.140
#> GSM228677 3 0.439 0.823 0.016 0.028 0.788 0.016 0.152
#> GSM228678 3 0.502 0.694 0.008 0.040 0.692 0.008 0.252
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 3 0.391 0.8159 NA 0.036 0.796 0.000 0.008 0.024
#> GSM228563 3 0.580 0.5990 NA 0.044 0.608 0.032 0.032 0.004
#> GSM228565 3 0.360 0.8186 NA 0.012 0.820 0.008 0.008 0.024
#> GSM228566 3 0.362 0.8163 NA 0.012 0.812 0.004 0.004 0.032
#> GSM228567 3 0.152 0.8002 NA 0.000 0.940 0.000 0.008 0.008
#> GSM228570 3 0.198 0.8174 NA 0.012 0.916 0.000 0.012 0.000
#> GSM228571 3 0.201 0.8168 NA 0.016 0.916 0.000 0.004 0.004
#> GSM228574 3 0.414 0.8051 NA 0.036 0.776 0.000 0.008 0.028
#> GSM228575 4 0.774 0.0530 NA 0.160 0.060 0.508 0.060 0.028
#> GSM228576 3 0.281 0.8233 NA 0.008 0.876 0.004 0.012 0.016
#> GSM228579 3 0.173 0.8146 NA 0.004 0.924 0.000 0.008 0.000
#> GSM228580 5 0.700 -0.6486 NA 0.280 0.008 0.044 0.488 0.160
#> GSM228581 6 0.213 0.0000 NA 0.000 0.040 0.000 0.000 0.904
#> GSM228666 3 0.765 0.1816 NA 0.044 0.472 0.056 0.016 0.200
#> GSM228564 3 0.530 0.6596 NA 0.028 0.652 0.040 0.012 0.008
#> GSM228568 3 0.481 0.7805 NA 0.020 0.732 0.000 0.016 0.080
#> GSM228569 3 0.364 0.8201 NA 0.016 0.820 0.000 0.012 0.036
#> GSM228572 3 0.592 0.4664 NA 0.048 0.556 0.032 0.024 0.004
#> GSM228573 3 0.280 0.8179 NA 0.016 0.860 0.000 0.000 0.016
#> GSM228577 3 0.161 0.8065 NA 0.000 0.932 0.008 0.004 0.000
#> GSM228578 3 0.220 0.8145 NA 0.008 0.908 0.012 0.008 0.000
#> GSM228663 3 0.484 0.7621 NA 0.032 0.720 0.000 0.008 0.064
#> GSM228664 3 0.630 0.4673 NA 0.036 0.556 0.000 0.012 0.152
#> GSM228665 3 0.333 0.8181 NA 0.012 0.836 0.004 0.008 0.020
#> GSM228582 3 0.482 0.7451 NA 0.032 0.712 0.004 0.004 0.048
#> GSM228583 3 0.152 0.7991 NA 0.000 0.940 0.000 0.008 0.008
#> GSM228585 3 0.188 0.8013 NA 0.004 0.928 0.004 0.008 0.008
#> GSM228587 3 0.322 0.8036 NA 0.016 0.848 0.008 0.012 0.008
#> GSM228588 3 0.533 0.6340 NA 0.028 0.632 0.020 0.008 0.024
#> GSM228589 3 0.500 0.6976 NA 0.016 0.684 0.008 0.012 0.044
#> GSM228590 3 0.172 0.8017 NA 0.000 0.928 0.000 0.008 0.008
#> GSM228591 3 0.522 0.6666 NA 0.036 0.652 0.004 0.000 0.060
#> GSM228597 3 0.535 0.6464 NA 0.032 0.652 0.036 0.020 0.004
#> GSM228601 3 0.555 0.4555 NA 0.036 0.544 0.004 0.000 0.052
#> GSM228604 3 0.491 0.7282 NA 0.016 0.692 0.012 0.004 0.048
#> GSM228608 3 0.267 0.8081 NA 0.008 0.876 0.004 0.008 0.008
#> GSM228609 3 0.490 0.7049 NA 0.032 0.668 0.012 0.012 0.008
#> GSM228613 3 0.166 0.8002 NA 0.000 0.932 0.000 0.008 0.008
#> GSM228616 3 0.441 0.7925 NA 0.032 0.768 0.012 0.008 0.028
#> GSM228628 3 0.535 0.6379 NA 0.032 0.624 0.000 0.004 0.064
#> GSM228634 3 0.148 0.8001 NA 0.004 0.944 0.000 0.008 0.004
#> GSM228642 3 0.592 0.4913 NA 0.064 0.548 0.016 0.004 0.028
#> GSM228645 3 0.526 0.7219 NA 0.056 0.688 0.016 0.000 0.044
#> GSM228646 3 0.410 0.7968 NA 0.032 0.772 0.004 0.000 0.032
#> GSM228652 3 0.190 0.8114 NA 0.012 0.924 0.000 0.008 0.004
#> GSM228655 3 0.266 0.8211 NA 0.008 0.876 0.004 0.012 0.004
#> GSM228656 3 0.155 0.8018 NA 0.000 0.940 0.004 0.008 0.004
#> GSM228659 3 0.442 0.7831 NA 0.040 0.760 0.016 0.012 0.008
#> GSM228662 3 0.180 0.7999 NA 0.000 0.928 0.004 0.008 0.008
#> GSM228584 3 0.155 0.8002 NA 0.000 0.940 0.004 0.008 0.004
#> GSM228586 3 0.141 0.7990 NA 0.000 0.944 0.000 0.008 0.004
#> GSM228592 3 0.186 0.8004 NA 0.000 0.924 0.004 0.008 0.008
#> GSM228593 3 0.537 0.6755 NA 0.060 0.676 0.028 0.004 0.020
#> GSM228594 3 0.148 0.8020 NA 0.000 0.944 0.004 0.008 0.004
#> GSM228598 3 0.412 0.8047 NA 0.040 0.796 0.000 0.012 0.040
#> GSM228607 3 0.275 0.8221 NA 0.008 0.860 0.012 0.000 0.004
#> GSM228612 3 0.488 0.7590 NA 0.028 0.720 0.008 0.004 0.060
#> GSM228619 3 0.215 0.8145 NA 0.004 0.900 0.008 0.004 0.000
#> GSM228622 3 0.162 0.8093 NA 0.004 0.936 0.008 0.004 0.000
#> GSM228625 3 0.218 0.8245 NA 0.004 0.900 0.004 0.004 0.004
#> GSM228631 3 0.201 0.8165 NA 0.004 0.904 0.008 0.000 0.000
#> GSM228633 3 0.611 0.3292 NA 0.024 0.480 0.036 0.004 0.048
#> GSM228637 3 0.663 0.2950 NA 0.028 0.492 0.088 0.008 0.036
#> GSM228639 3 0.335 0.8181 NA 0.008 0.824 0.008 0.004 0.016
#> GSM228649 3 0.606 0.4961 NA 0.024 0.560 0.060 0.008 0.028
#> GSM228660 3 0.207 0.8189 NA 0.004 0.912 0.004 0.004 0.008
#> GSM228661 3 0.162 0.8008 NA 0.000 0.936 0.004 0.008 0.004
#> GSM228595 3 0.571 0.4291 NA 0.040 0.532 0.004 0.000 0.060
#> GSM228599 3 0.481 0.7569 NA 0.032 0.696 0.016 0.012 0.008
#> GSM228602 3 0.211 0.8187 NA 0.008 0.896 0.000 0.004 0.000
#> GSM228614 3 0.423 0.7730 NA 0.032 0.740 0.000 0.020 0.004
#> GSM228626 3 0.616 0.3364 NA 0.040 0.488 0.004 0.008 0.076
#> GSM228640 3 0.266 0.8188 NA 0.012 0.868 0.004 0.000 0.008
#> GSM228643 3 0.338 0.8182 NA 0.020 0.824 0.000 0.004 0.020
#> GSM228650 3 0.412 0.7986 NA 0.048 0.780 0.012 0.000 0.016
#> GSM228653 3 0.297 0.8203 NA 0.008 0.844 0.008 0.004 0.004
#> GSM228657 3 0.463 0.6687 NA 0.020 0.656 0.008 0.004 0.012
#> GSM228605 3 0.296 0.8222 NA 0.012 0.856 0.008 0.008 0.004
#> GSM228610 3 0.407 0.8131 NA 0.028 0.792 0.020 0.012 0.008
#> GSM228617 3 0.220 0.8150 NA 0.004 0.896 0.008 0.004 0.000
#> GSM228620 3 0.268 0.8206 NA 0.012 0.872 0.008 0.000 0.008
#> GSM228623 3 0.523 0.6867 NA 0.040 0.672 0.020 0.028 0.004
#> GSM228629 3 0.301 0.8192 NA 0.012 0.852 0.008 0.004 0.008
#> GSM228632 3 0.339 0.8170 NA 0.004 0.812 0.008 0.004 0.016
#> GSM228635 4 0.538 0.1339 NA 0.008 0.068 0.632 0.020 0.004
#> GSM228647 3 0.309 0.8184 NA 0.004 0.840 0.004 0.004 0.020
#> GSM228596 3 0.301 0.8242 NA 0.008 0.856 0.016 0.008 0.004
#> GSM228600 3 0.294 0.8175 NA 0.020 0.864 0.004 0.004 0.012
#> GSM228603 3 0.268 0.8171 NA 0.024 0.876 0.004 0.000 0.008
#> GSM228615 3 0.467 0.7655 NA 0.028 0.732 0.036 0.012 0.004
#> GSM228627 3 0.309 0.8200 NA 0.024 0.848 0.000 0.000 0.024
#> GSM228641 3 0.303 0.8203 NA 0.016 0.852 0.000 0.004 0.020
#> GSM228644 3 0.580 0.4084 NA 0.024 0.516 0.008 0.000 0.080
#> GSM228651 3 0.335 0.8200 NA 0.020 0.844 0.008 0.008 0.016
#> GSM228654 3 0.345 0.8192 NA 0.012 0.824 0.008 0.004 0.020
#> GSM228658 3 0.371 0.8177 NA 0.020 0.816 0.008 0.012 0.016
#> GSM228606 3 0.378 0.8062 NA 0.024 0.796 0.012 0.000 0.016
#> GSM228611 3 0.386 0.8040 NA 0.020 0.792 0.008 0.004 0.020
#> GSM228618 3 0.316 0.8177 NA 0.008 0.848 0.008 0.008 0.016
#> GSM228621 3 0.318 0.8159 NA 0.004 0.824 0.008 0.000 0.016
#> GSM228624 3 0.492 0.7543 NA 0.040 0.716 0.004 0.004 0.056
#> GSM228630 3 0.357 0.8102 NA 0.012 0.796 0.008 0.000 0.016
#> GSM228636 3 0.631 0.0495 NA 0.020 0.444 0.172 0.000 0.004
#> GSM228638 3 0.266 0.8242 NA 0.012 0.880 0.004 0.004 0.012
#> GSM228648 3 0.374 0.8012 NA 0.008 0.788 0.004 0.004 0.028
#> GSM228670 3 0.497 0.7394 NA 0.048 0.716 0.012 0.028 0.008
#> GSM228671 2 0.506 0.0000 NA 0.504 0.028 0.000 0.444 0.004
#> GSM228672 3 0.548 0.6785 NA 0.040 0.652 0.008 0.028 0.024
#> GSM228674 3 0.432 0.7960 NA 0.016 0.784 0.024 0.024 0.016
#> GSM228675 5 0.568 -0.1321 NA 0.000 0.380 0.012 0.504 0.004
#> GSM228676 3 0.406 0.8128 NA 0.024 0.784 0.008 0.012 0.016
#> GSM228667 3 0.520 0.6999 NA 0.032 0.672 0.000 0.044 0.020
#> GSM228668 3 0.237 0.8190 NA 0.004 0.892 0.012 0.008 0.000
#> GSM228669 3 0.187 0.8145 NA 0.000 0.908 0.008 0.000 0.000
#> GSM228673 3 0.583 0.7079 NA 0.032 0.672 0.060 0.012 0.044
#> GSM228677 3 0.482 0.7186 NA 0.016 0.688 0.024 0.008 0.016
#> GSM228678 3 0.549 0.4656 NA 0.016 0.548 0.048 0.004 0.012
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
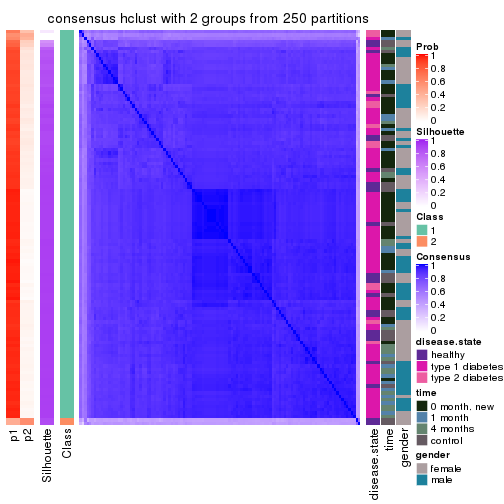
consensus_heatmap(res, k = 3)
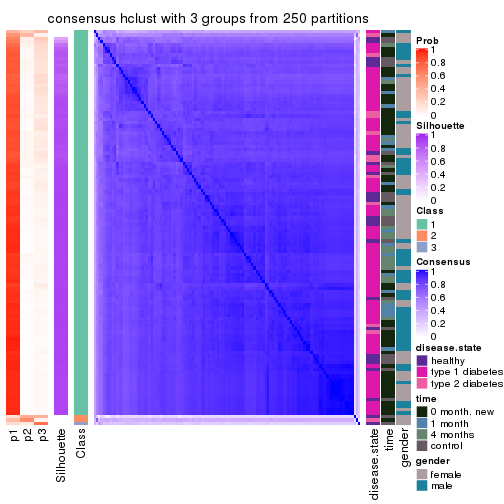
consensus_heatmap(res, k = 4)
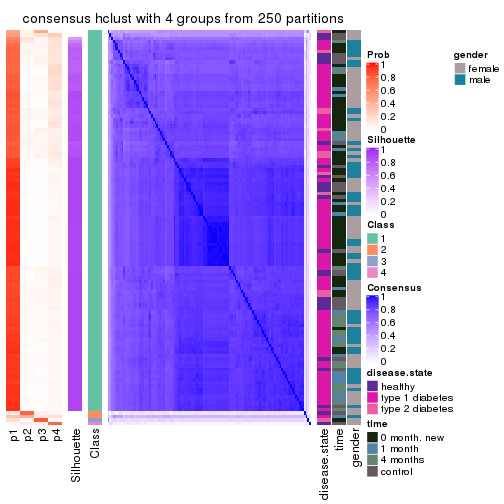
consensus_heatmap(res, k = 5)
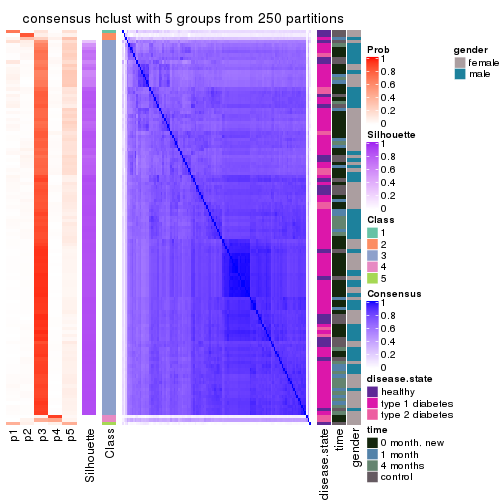
consensus_heatmap(res, k = 6)
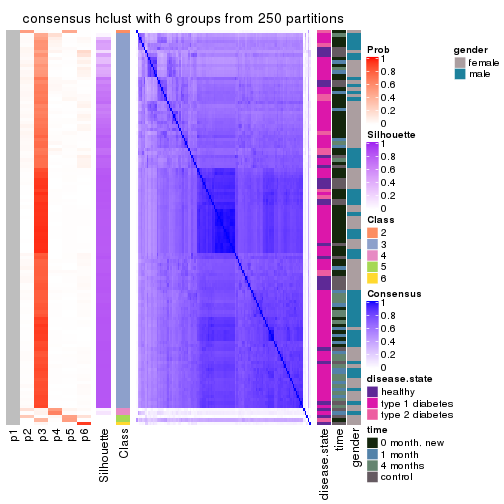
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
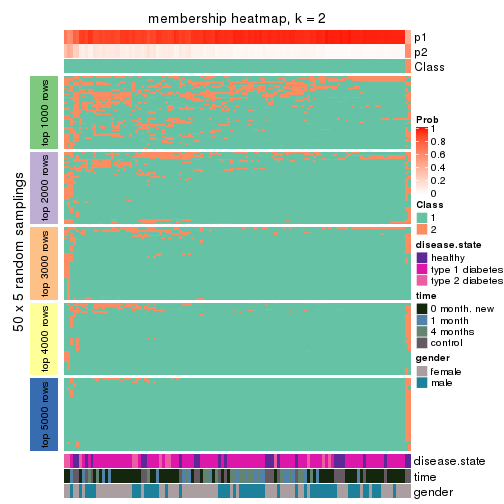
membership_heatmap(res, k = 3)
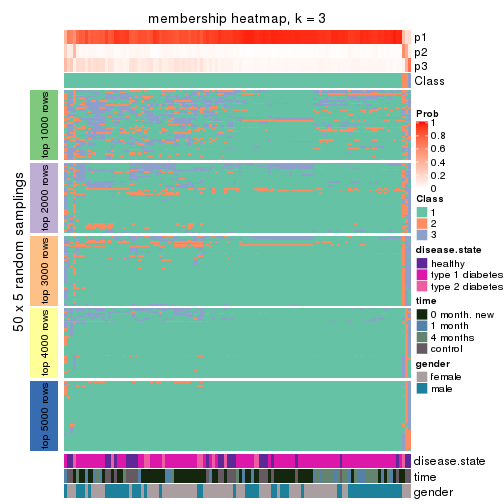
membership_heatmap(res, k = 4)
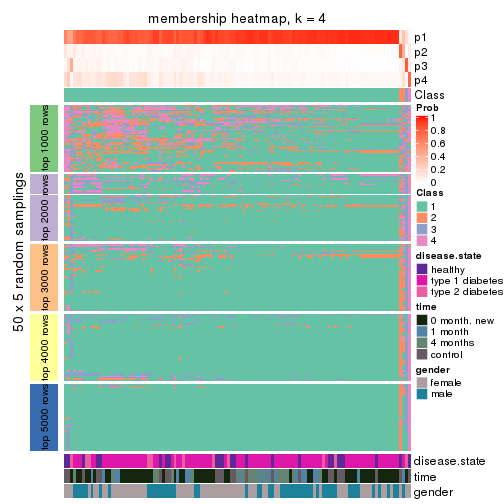
membership_heatmap(res, k = 5)
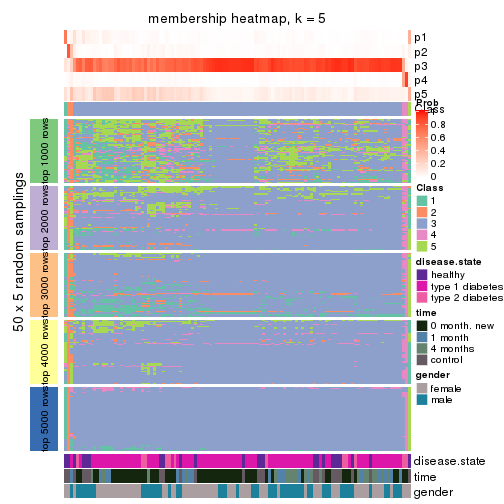
membership_heatmap(res, k = 6)
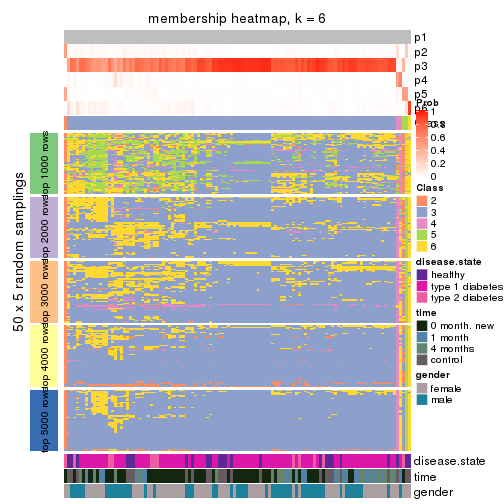
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
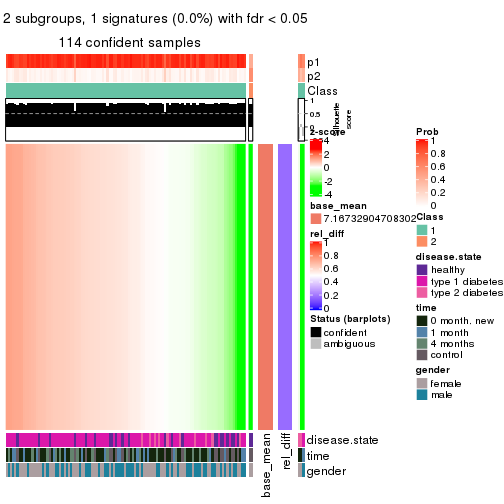
get_signatures(res, k = 3)

get_signatures(res, k = 4)

get_signatures(res, k = 5)

get_signatures(res, k = 6)

Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
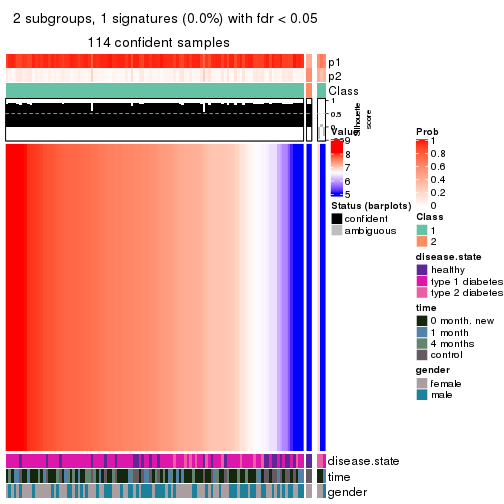
get_signatures(res, k = 3, scale_rows = FALSE)

get_signatures(res, k = 4, scale_rows = FALSE)

get_signatures(res, k = 5, scale_rows = FALSE)

get_signatures(res, k = 6, scale_rows = FALSE)

Compare the overlap of signatures from different k:
compare_signatures(res)

get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
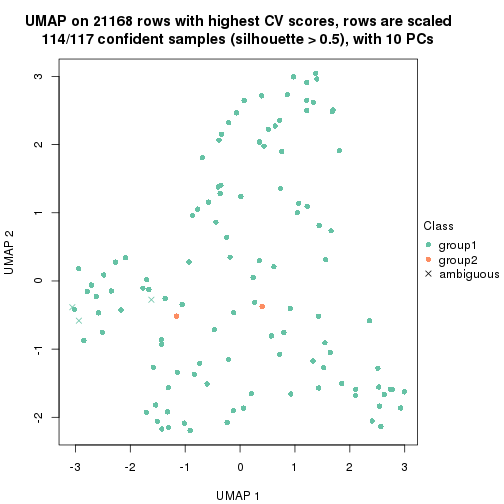
dimension_reduction(res, k = 3, method = "UMAP")
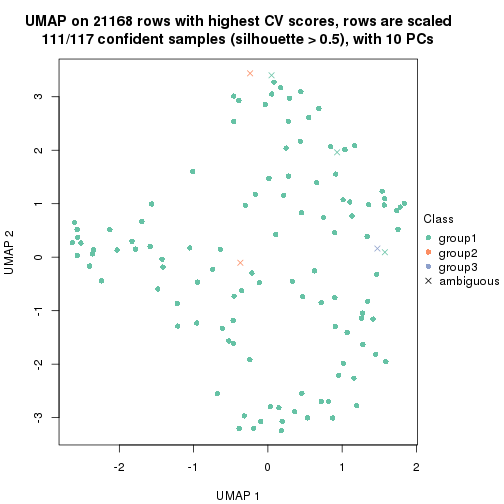
dimension_reduction(res, k = 4, method = "UMAP")
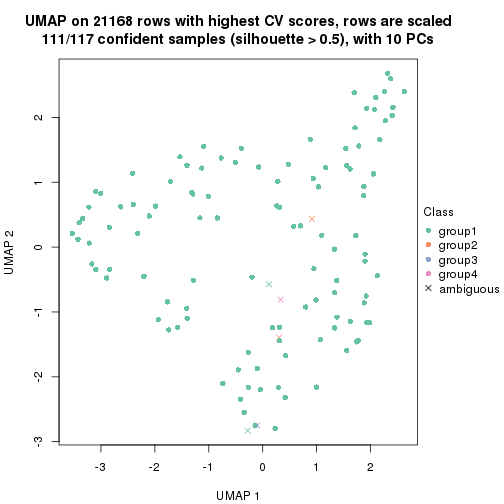
dimension_reduction(res, k = 5, method = "UMAP")
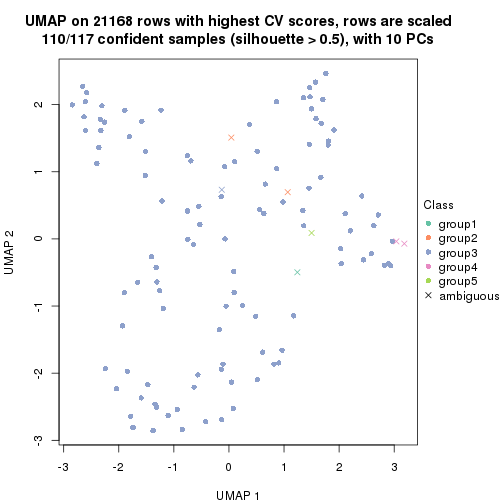
dimension_reduction(res, k = 6, method = "UMAP")
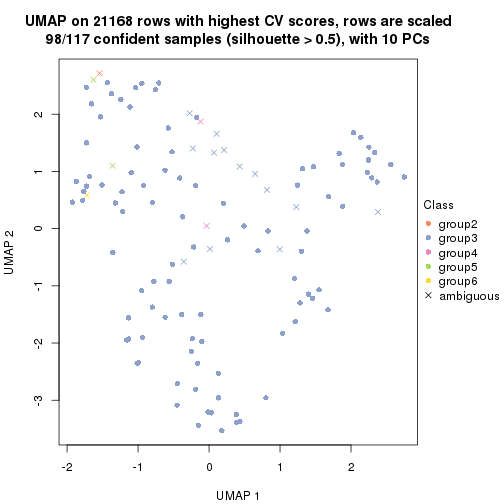
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
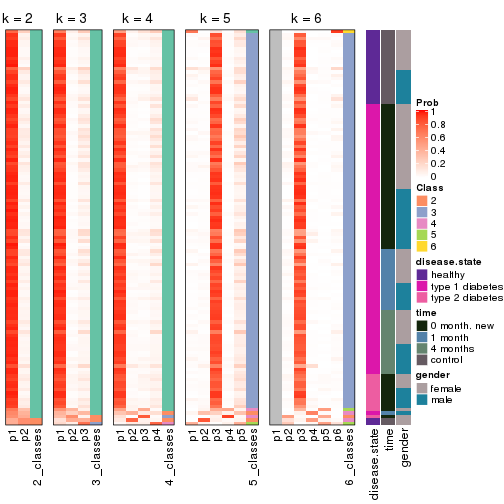
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> CV:hclust 114 0.022 0.0542 0.571 2
#> CV:hclust 111 NA NA NA 3
#> CV:hclust 111 NA NA NA 4
#> CV:hclust 110 NA NA NA 5
#> CV:hclust 98 NA NA NA 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["CV", "kmeans"]
# you can also extract it by
# res = res_list["CV:kmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'CV' method.
#> Subgroups are detected by 'kmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
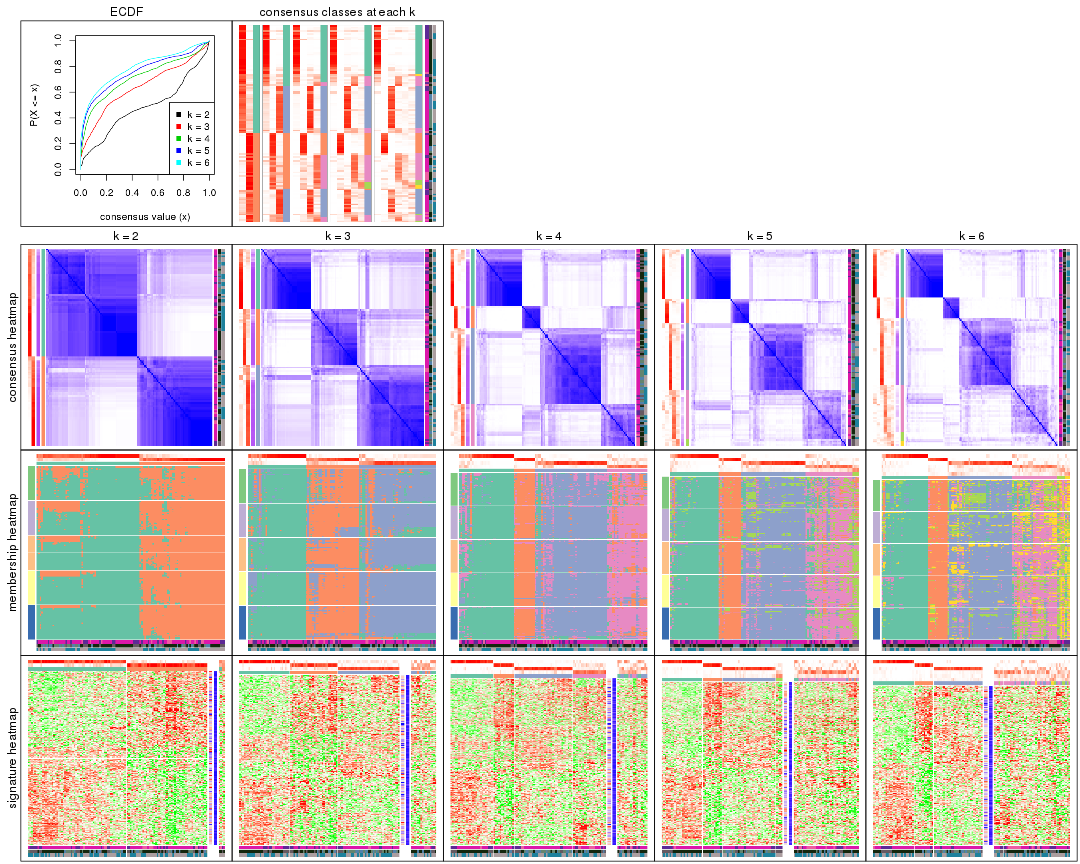
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
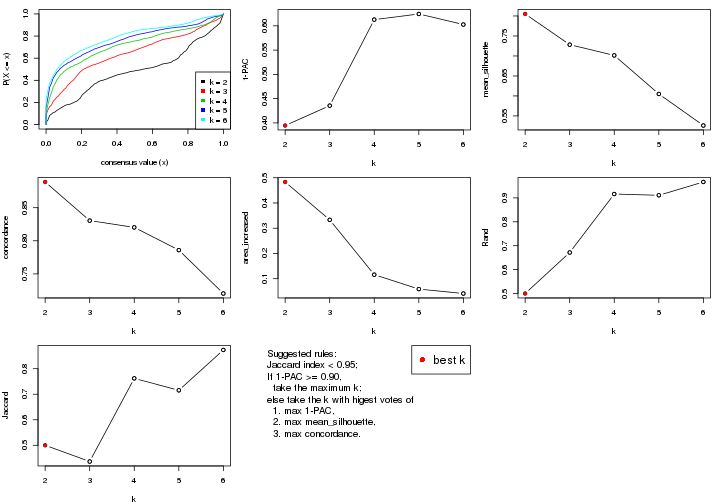
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.395 0.805 0.889 0.4833 0.500 0.500
#> 3 3 0.436 0.728 0.830 0.3332 0.672 0.437
#> 4 4 0.613 0.701 0.820 0.1161 0.916 0.762
#> 5 5 0.624 0.605 0.786 0.0589 0.911 0.715
#> 6 6 0.603 0.526 0.720 0.0414 0.967 0.873
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.4298 0.8573 0.912 0.088
#> GSM228563 2 0.0376 0.8855 0.004 0.996
#> GSM228565 1 0.6973 0.8049 0.812 0.188
#> GSM228566 2 0.9909 0.0849 0.444 0.556
#> GSM228567 1 0.0938 0.8604 0.988 0.012
#> GSM228570 1 0.0938 0.8604 0.988 0.012
#> GSM228571 1 0.0938 0.8604 0.988 0.012
#> GSM228574 2 0.8207 0.6693 0.256 0.744
#> GSM228575 2 0.0000 0.8860 0.000 1.000
#> GSM228576 1 0.4939 0.8458 0.892 0.108
#> GSM228579 1 0.0938 0.8604 0.988 0.012
#> GSM228580 2 0.0000 0.8860 0.000 1.000
#> GSM228581 2 0.1633 0.8848 0.024 0.976
#> GSM228666 2 0.0000 0.8860 0.000 1.000
#> GSM228564 2 0.8144 0.7100 0.252 0.748
#> GSM228568 1 0.7883 0.7539 0.764 0.236
#> GSM228569 1 0.0376 0.8600 0.996 0.004
#> GSM228572 2 0.0672 0.8846 0.008 0.992
#> GSM228573 1 0.6048 0.8337 0.852 0.148
#> GSM228577 1 0.0000 0.8587 1.000 0.000
#> GSM228578 1 0.0000 0.8587 1.000 0.000
#> GSM228663 1 0.8443 0.7393 0.728 0.272
#> GSM228664 2 0.3733 0.8709 0.072 0.928
#> GSM228665 1 0.6801 0.8174 0.820 0.180
#> GSM228582 1 0.9732 0.4538 0.596 0.404
#> GSM228583 1 0.0938 0.8604 0.988 0.012
#> GSM228585 1 0.0938 0.8604 0.988 0.012
#> GSM228587 1 0.0938 0.8604 0.988 0.012
#> GSM228588 2 0.3114 0.8575 0.056 0.944
#> GSM228589 2 0.0000 0.8860 0.000 1.000
#> GSM228590 1 0.0938 0.8604 0.988 0.012
#> GSM228591 2 0.0000 0.8860 0.000 1.000
#> GSM228597 2 0.0000 0.8860 0.000 1.000
#> GSM228601 2 0.0000 0.8860 0.000 1.000
#> GSM228604 2 0.0938 0.8863 0.012 0.988
#> GSM228608 1 0.1414 0.8592 0.980 0.020
#> GSM228609 2 0.7139 0.7377 0.196 0.804
#> GSM228613 1 0.0938 0.8604 0.988 0.012
#> GSM228616 2 0.9954 0.0738 0.460 0.540
#> GSM228628 2 0.0000 0.8860 0.000 1.000
#> GSM228634 1 0.0938 0.8604 0.988 0.012
#> GSM228642 2 0.0000 0.8860 0.000 1.000
#> GSM228645 2 0.2043 0.8826 0.032 0.968
#> GSM228646 2 0.4022 0.8608 0.080 0.920
#> GSM228652 1 0.0938 0.8604 0.988 0.012
#> GSM228655 1 0.0938 0.8604 0.988 0.012
#> GSM228656 1 0.0938 0.8604 0.988 0.012
#> GSM228659 1 0.6973 0.7316 0.812 0.188
#> GSM228662 1 0.0938 0.8604 0.988 0.012
#> GSM228584 1 0.0000 0.8587 1.000 0.000
#> GSM228586 1 0.0000 0.8587 1.000 0.000
#> GSM228592 1 0.0000 0.8587 1.000 0.000
#> GSM228593 2 0.8144 0.6848 0.252 0.748
#> GSM228594 1 0.0000 0.8587 1.000 0.000
#> GSM228598 1 0.0672 0.8585 0.992 0.008
#> GSM228607 1 0.9323 0.5671 0.652 0.348
#> GSM228612 2 0.7815 0.6985 0.232 0.768
#> GSM228619 1 0.3879 0.8560 0.924 0.076
#> GSM228622 1 0.0000 0.8587 1.000 0.000
#> GSM228625 1 0.4298 0.8426 0.912 0.088
#> GSM228631 1 0.2423 0.8618 0.960 0.040
#> GSM228633 2 0.0938 0.8834 0.012 0.988
#> GSM228637 2 0.3584 0.8691 0.068 0.932
#> GSM228639 2 0.9286 0.5062 0.344 0.656
#> GSM228649 2 0.4298 0.8582 0.088 0.912
#> GSM228660 1 0.2043 0.8632 0.968 0.032
#> GSM228661 1 0.0000 0.8587 1.000 0.000
#> GSM228595 2 0.0000 0.8860 0.000 1.000
#> GSM228599 2 0.2778 0.8791 0.048 0.952
#> GSM228602 1 0.6531 0.8238 0.832 0.168
#> GSM228614 2 0.2948 0.8754 0.052 0.948
#> GSM228626 2 0.0000 0.8860 0.000 1.000
#> GSM228640 1 0.7139 0.8150 0.804 0.196
#> GSM228643 1 0.8813 0.7128 0.700 0.300
#> GSM228650 2 0.7056 0.7652 0.192 0.808
#> GSM228653 1 0.7815 0.7879 0.768 0.232
#> GSM228657 2 0.0000 0.8860 0.000 1.000
#> GSM228605 1 0.2948 0.8615 0.948 0.052
#> GSM228610 1 0.7528 0.7912 0.784 0.216
#> GSM228617 1 0.6247 0.8294 0.844 0.156
#> GSM228620 1 0.5629 0.8406 0.868 0.132
#> GSM228623 2 0.2236 0.8841 0.036 0.964
#> GSM228629 1 0.6801 0.8171 0.820 0.180
#> GSM228632 2 0.8016 0.7090 0.244 0.756
#> GSM228635 2 0.1414 0.8849 0.020 0.980
#> GSM228647 1 0.7219 0.8038 0.800 0.200
#> GSM228596 1 0.8327 0.7621 0.736 0.264
#> GSM228600 1 0.8207 0.7663 0.744 0.256
#> GSM228603 1 0.6712 0.8264 0.824 0.176
#> GSM228615 2 0.1633 0.8851 0.024 0.976
#> GSM228627 1 0.8207 0.7658 0.744 0.256
#> GSM228641 1 0.8763 0.7177 0.704 0.296
#> GSM228644 2 0.0000 0.8860 0.000 1.000
#> GSM228651 1 0.8661 0.7321 0.712 0.288
#> GSM228654 1 0.9580 0.5380 0.620 0.380
#> GSM228658 1 0.7453 0.8029 0.788 0.212
#> GSM228606 2 0.5294 0.8411 0.120 0.880
#> GSM228611 1 0.7950 0.7735 0.760 0.240
#> GSM228618 1 0.7139 0.8073 0.804 0.196
#> GSM228621 2 0.6343 0.8095 0.160 0.840
#> GSM228624 2 0.7139 0.7621 0.196 0.804
#> GSM228630 2 0.6247 0.8145 0.156 0.844
#> GSM228636 2 0.1414 0.8849 0.020 0.980
#> GSM228638 1 0.7139 0.8077 0.804 0.196
#> GSM228648 2 0.7299 0.7645 0.204 0.796
#> GSM228670 2 0.4431 0.8605 0.092 0.908
#> GSM228671 2 0.0000 0.8860 0.000 1.000
#> GSM228672 1 0.9522 0.3821 0.628 0.372
#> GSM228674 2 0.7299 0.7563 0.204 0.796
#> GSM228675 2 0.0000 0.8860 0.000 1.000
#> GSM228676 1 0.8081 0.7744 0.752 0.248
#> GSM228667 2 0.6623 0.7806 0.172 0.828
#> GSM228668 1 0.0000 0.8587 1.000 0.000
#> GSM228669 1 0.1633 0.8620 0.976 0.024
#> GSM228673 2 0.8661 0.6056 0.288 0.712
#> GSM228677 2 0.1843 0.8852 0.028 0.972
#> GSM228678 2 0.1414 0.8842 0.020 0.980
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.6899 0.410 0.612 0.024 0.364
#> GSM228563 2 0.3267 0.818 0.000 0.884 0.116
#> GSM228565 1 0.7997 0.409 0.600 0.084 0.316
#> GSM228566 3 0.3678 0.818 0.080 0.028 0.892
#> GSM228567 1 0.0000 0.887 1.000 0.000 0.000
#> GSM228570 1 0.0424 0.886 0.992 0.000 0.008
#> GSM228571 1 0.0747 0.882 0.984 0.000 0.016
#> GSM228574 3 0.3148 0.801 0.048 0.036 0.916
#> GSM228575 2 0.6095 0.532 0.000 0.608 0.392
#> GSM228576 1 0.7960 0.514 0.648 0.120 0.232
#> GSM228579 1 0.0237 0.887 0.996 0.000 0.004
#> GSM228580 2 0.2796 0.820 0.000 0.908 0.092
#> GSM228581 2 0.5988 0.545 0.000 0.632 0.368
#> GSM228666 2 0.4178 0.805 0.000 0.828 0.172
#> GSM228564 2 0.8926 0.602 0.192 0.568 0.240
#> GSM228568 1 0.7364 0.503 0.640 0.056 0.304
#> GSM228569 1 0.2448 0.843 0.924 0.000 0.076
#> GSM228572 2 0.2878 0.820 0.000 0.904 0.096
#> GSM228573 3 0.4178 0.816 0.172 0.000 0.828
#> GSM228577 1 0.0237 0.886 0.996 0.000 0.004
#> GSM228578 1 0.1411 0.874 0.964 0.000 0.036
#> GSM228663 3 0.4270 0.828 0.116 0.024 0.860
#> GSM228664 3 0.3918 0.742 0.004 0.140 0.856
#> GSM228665 3 0.4062 0.819 0.164 0.000 0.836
#> GSM228582 1 0.8614 0.393 0.568 0.304 0.128
#> GSM228583 1 0.0000 0.887 1.000 0.000 0.000
#> GSM228585 1 0.0000 0.887 1.000 0.000 0.000
#> GSM228587 1 0.0000 0.887 1.000 0.000 0.000
#> GSM228588 2 0.1163 0.821 0.000 0.972 0.028
#> GSM228589 2 0.1163 0.819 0.000 0.972 0.028
#> GSM228590 1 0.0000 0.887 1.000 0.000 0.000
#> GSM228591 2 0.1289 0.819 0.000 0.968 0.032
#> GSM228597 2 0.3192 0.817 0.000 0.888 0.112
#> GSM228601 2 0.1411 0.821 0.000 0.964 0.036
#> GSM228604 3 0.6062 0.380 0.000 0.384 0.616
#> GSM228608 1 0.0000 0.887 1.000 0.000 0.000
#> GSM228609 2 0.3369 0.817 0.040 0.908 0.052
#> GSM228613 1 0.0000 0.887 1.000 0.000 0.000
#> GSM228616 3 0.9684 0.342 0.260 0.280 0.460
#> GSM228628 2 0.2537 0.820 0.000 0.920 0.080
#> GSM228634 1 0.0000 0.887 1.000 0.000 0.000
#> GSM228642 2 0.1643 0.819 0.000 0.956 0.044
#> GSM228645 3 0.6298 0.249 0.004 0.388 0.608
#> GSM228646 3 0.5277 0.681 0.024 0.180 0.796
#> GSM228652 1 0.0237 0.887 0.996 0.000 0.004
#> GSM228655 1 0.0592 0.884 0.988 0.000 0.012
#> GSM228656 1 0.0000 0.887 1.000 0.000 0.000
#> GSM228659 1 0.4887 0.761 0.844 0.096 0.060
#> GSM228662 1 0.0000 0.887 1.000 0.000 0.000
#> GSM228584 1 0.0237 0.886 0.996 0.000 0.004
#> GSM228586 1 0.0237 0.886 0.996 0.000 0.004
#> GSM228592 1 0.0237 0.886 0.996 0.000 0.004
#> GSM228593 2 0.8113 0.501 0.324 0.588 0.088
#> GSM228594 1 0.0892 0.882 0.980 0.000 0.020
#> GSM228598 1 0.0892 0.881 0.980 0.000 0.020
#> GSM228607 3 0.6490 0.768 0.172 0.076 0.752
#> GSM228612 3 0.4786 0.781 0.044 0.112 0.844
#> GSM228619 3 0.6305 0.292 0.484 0.000 0.516
#> GSM228622 1 0.3412 0.799 0.876 0.000 0.124
#> GSM228625 1 0.7639 0.538 0.656 0.088 0.256
#> GSM228631 3 0.6305 0.288 0.484 0.000 0.516
#> GSM228633 2 0.2537 0.818 0.000 0.920 0.080
#> GSM228637 2 0.6794 0.770 0.076 0.728 0.196
#> GSM228639 3 0.3148 0.797 0.036 0.048 0.916
#> GSM228649 2 0.6754 0.772 0.092 0.740 0.168
#> GSM228660 1 0.5247 0.681 0.768 0.008 0.224
#> GSM228661 1 0.1031 0.880 0.976 0.000 0.024
#> GSM228595 2 0.1529 0.820 0.000 0.960 0.040
#> GSM228599 2 0.7394 0.262 0.032 0.496 0.472
#> GSM228602 3 0.4654 0.794 0.208 0.000 0.792
#> GSM228614 3 0.6264 0.238 0.004 0.380 0.616
#> GSM228626 2 0.1643 0.820 0.000 0.956 0.044
#> GSM228640 3 0.4629 0.812 0.188 0.004 0.808
#> GSM228643 3 0.4779 0.827 0.124 0.036 0.840
#> GSM228650 3 0.3028 0.795 0.032 0.048 0.920
#> GSM228653 3 0.4351 0.821 0.168 0.004 0.828
#> GSM228657 2 0.1643 0.820 0.000 0.956 0.044
#> GSM228605 3 0.6521 0.111 0.496 0.004 0.500
#> GSM228610 3 0.3755 0.830 0.120 0.008 0.872
#> GSM228617 3 0.4796 0.785 0.220 0.000 0.780
#> GSM228620 3 0.4291 0.813 0.180 0.000 0.820
#> GSM228623 2 0.5406 0.785 0.012 0.764 0.224
#> GSM228629 3 0.4062 0.819 0.164 0.000 0.836
#> GSM228632 3 0.3253 0.810 0.052 0.036 0.912
#> GSM228635 2 0.4887 0.791 0.000 0.772 0.228
#> GSM228647 3 0.3879 0.825 0.152 0.000 0.848
#> GSM228596 3 0.4708 0.826 0.120 0.036 0.844
#> GSM228600 3 0.4514 0.827 0.156 0.012 0.832
#> GSM228603 3 0.4399 0.811 0.188 0.000 0.812
#> GSM228615 2 0.5331 0.792 0.024 0.792 0.184
#> GSM228627 3 0.4741 0.826 0.152 0.020 0.828
#> GSM228641 3 0.3551 0.832 0.132 0.000 0.868
#> GSM228644 2 0.1643 0.820 0.000 0.956 0.044
#> GSM228651 3 0.5174 0.822 0.128 0.048 0.824
#> GSM228654 3 0.4121 0.828 0.108 0.024 0.868
#> GSM228658 3 0.4346 0.813 0.184 0.000 0.816
#> GSM228606 3 0.2096 0.769 0.004 0.052 0.944
#> GSM228611 3 0.3784 0.831 0.132 0.004 0.864
#> GSM228618 3 0.4062 0.819 0.164 0.000 0.836
#> GSM228621 3 0.2200 0.778 0.004 0.056 0.940
#> GSM228624 3 0.3434 0.793 0.032 0.064 0.904
#> GSM228630 3 0.2804 0.788 0.016 0.060 0.924
#> GSM228636 2 0.4645 0.807 0.008 0.816 0.176
#> GSM228638 3 0.3752 0.828 0.144 0.000 0.856
#> GSM228648 3 0.3359 0.784 0.016 0.084 0.900
#> GSM228670 2 0.8393 0.439 0.088 0.516 0.396
#> GSM228671 2 0.6274 0.408 0.000 0.544 0.456
#> GSM228672 1 0.8948 0.373 0.568 0.224 0.208
#> GSM228674 2 0.7971 0.616 0.096 0.624 0.280
#> GSM228675 2 0.5365 0.740 0.004 0.744 0.252
#> GSM228676 3 0.3918 0.823 0.120 0.012 0.868
#> GSM228667 3 0.7453 -0.100 0.036 0.436 0.528
#> GSM228668 1 0.1289 0.877 0.968 0.000 0.032
#> GSM228669 1 0.4862 0.770 0.820 0.020 0.160
#> GSM228673 3 0.3045 0.786 0.020 0.064 0.916
#> GSM228677 3 0.6291 -0.269 0.000 0.468 0.532
#> GSM228678 2 0.4235 0.808 0.000 0.824 0.176
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.7086 0.43902 0.296 0.004 0.140 0.560
#> GSM228563 4 0.4737 0.66702 0.000 0.252 0.020 0.728
#> GSM228565 1 0.8531 0.00982 0.416 0.040 0.200 0.344
#> GSM228566 3 0.3102 0.80403 0.004 0.008 0.872 0.116
#> GSM228567 1 0.0188 0.85997 0.996 0.000 0.004 0.000
#> GSM228570 1 0.2060 0.84191 0.932 0.000 0.016 0.052
#> GSM228571 1 0.2214 0.83963 0.928 0.000 0.028 0.044
#> GSM228574 3 0.3672 0.76444 0.000 0.012 0.824 0.164
#> GSM228575 4 0.3818 0.67244 0.000 0.108 0.048 0.844
#> GSM228576 1 0.8185 0.40505 0.552 0.064 0.216 0.168
#> GSM228579 1 0.1639 0.84843 0.952 0.004 0.008 0.036
#> GSM228580 4 0.5298 0.32561 0.000 0.372 0.016 0.612
#> GSM228581 4 0.7442 0.35578 0.000 0.304 0.200 0.496
#> GSM228666 4 0.5322 0.50697 0.000 0.312 0.028 0.660
#> GSM228564 4 0.5255 0.69706 0.112 0.036 0.064 0.788
#> GSM228568 1 0.8047 0.26184 0.488 0.020 0.220 0.272
#> GSM228569 1 0.2805 0.80675 0.888 0.000 0.100 0.012
#> GSM228572 2 0.4046 0.79234 0.000 0.828 0.048 0.124
#> GSM228573 3 0.1677 0.82577 0.040 0.000 0.948 0.012
#> GSM228577 1 0.0188 0.85997 0.996 0.000 0.004 0.000
#> GSM228578 1 0.2021 0.84206 0.932 0.000 0.056 0.012
#> GSM228663 3 0.2392 0.81881 0.024 0.012 0.928 0.036
#> GSM228664 3 0.3962 0.76220 0.004 0.100 0.844 0.052
#> GSM228665 3 0.1798 0.82449 0.040 0.000 0.944 0.016
#> GSM228582 1 0.7325 0.31877 0.540 0.352 0.052 0.056
#> GSM228583 1 0.0188 0.85997 0.996 0.000 0.004 0.000
#> GSM228585 1 0.0188 0.85997 0.996 0.000 0.004 0.000
#> GSM228587 1 0.0000 0.85807 1.000 0.000 0.000 0.000
#> GSM228588 2 0.1305 0.92405 0.000 0.960 0.004 0.036
#> GSM228589 2 0.1042 0.92341 0.000 0.972 0.008 0.020
#> GSM228590 1 0.0188 0.85997 0.996 0.000 0.004 0.000
#> GSM228591 2 0.1151 0.92133 0.000 0.968 0.008 0.024
#> GSM228597 4 0.4922 0.64103 0.004 0.284 0.012 0.700
#> GSM228601 2 0.1247 0.93117 0.004 0.968 0.012 0.016
#> GSM228604 3 0.6148 0.11322 0.000 0.468 0.484 0.048
#> GSM228608 1 0.0524 0.85895 0.988 0.000 0.004 0.008
#> GSM228609 2 0.5349 0.57896 0.048 0.732 0.008 0.212
#> GSM228613 1 0.0188 0.85997 0.996 0.000 0.004 0.000
#> GSM228616 3 0.9721 0.00638 0.244 0.160 0.356 0.240
#> GSM228628 2 0.1913 0.91011 0.000 0.940 0.020 0.040
#> GSM228634 1 0.0188 0.85997 0.996 0.000 0.004 0.000
#> GSM228642 2 0.1356 0.92307 0.000 0.960 0.008 0.032
#> GSM228645 4 0.7091 0.50946 0.000 0.188 0.248 0.564
#> GSM228646 3 0.6316 0.47886 0.000 0.088 0.612 0.300
#> GSM228652 1 0.1388 0.85362 0.960 0.000 0.012 0.028
#> GSM228655 1 0.1913 0.84687 0.940 0.000 0.040 0.020
#> GSM228656 1 0.0188 0.85997 0.996 0.000 0.004 0.000
#> GSM228659 1 0.5525 0.31102 0.600 0.012 0.008 0.380
#> GSM228662 1 0.0000 0.85807 1.000 0.000 0.000 0.000
#> GSM228584 1 0.0188 0.85997 0.996 0.000 0.004 0.000
#> GSM228586 1 0.0188 0.85997 0.996 0.000 0.004 0.000
#> GSM228592 1 0.0188 0.85997 0.996 0.000 0.004 0.000
#> GSM228593 4 0.6433 0.61186 0.212 0.108 0.012 0.668
#> GSM228594 1 0.1022 0.85412 0.968 0.000 0.032 0.000
#> GSM228598 1 0.0927 0.85668 0.976 0.000 0.008 0.016
#> GSM228607 3 0.6340 0.50331 0.048 0.028 0.652 0.272
#> GSM228612 3 0.4287 0.75180 0.004 0.080 0.828 0.088
#> GSM228619 3 0.5587 0.42230 0.372 0.000 0.600 0.028
#> GSM228622 1 0.3355 0.74934 0.836 0.000 0.160 0.004
#> GSM228625 1 0.8215 0.30588 0.536 0.052 0.204 0.208
#> GSM228631 3 0.5355 0.45241 0.360 0.000 0.620 0.020
#> GSM228633 2 0.1833 0.91947 0.000 0.944 0.032 0.024
#> GSM228637 4 0.6494 0.68930 0.024 0.208 0.092 0.676
#> GSM228639 3 0.1637 0.81187 0.000 0.000 0.940 0.060
#> GSM228649 4 0.6359 0.69723 0.036 0.204 0.068 0.692
#> GSM228660 1 0.5568 0.62221 0.704 0.024 0.248 0.024
#> GSM228661 1 0.1474 0.84529 0.948 0.000 0.052 0.000
#> GSM228595 2 0.1297 0.93084 0.000 0.964 0.020 0.016
#> GSM228599 4 0.6325 0.64328 0.012 0.100 0.212 0.676
#> GSM228602 3 0.2675 0.82252 0.048 0.000 0.908 0.044
#> GSM228614 4 0.6740 0.44159 0.004 0.096 0.332 0.568
#> GSM228626 2 0.1174 0.93161 0.000 0.968 0.020 0.012
#> GSM228640 3 0.2830 0.82089 0.032 0.004 0.904 0.060
#> GSM228643 3 0.3211 0.81299 0.024 0.008 0.884 0.084
#> GSM228650 3 0.2611 0.80874 0.000 0.008 0.896 0.096
#> GSM228653 3 0.2313 0.82472 0.032 0.000 0.924 0.044
#> GSM228657 2 0.1520 0.93023 0.000 0.956 0.020 0.024
#> GSM228605 3 0.7916 -0.13831 0.336 0.000 0.352 0.312
#> GSM228610 3 0.1833 0.82664 0.024 0.000 0.944 0.032
#> GSM228617 3 0.2376 0.81589 0.068 0.000 0.916 0.016
#> GSM228620 3 0.1576 0.82374 0.048 0.000 0.948 0.004
#> GSM228623 4 0.5914 0.69453 0.008 0.220 0.076 0.696
#> GSM228629 3 0.1545 0.82374 0.040 0.000 0.952 0.008
#> GSM228632 3 0.0895 0.81901 0.000 0.004 0.976 0.020
#> GSM228635 4 0.4758 0.68860 0.000 0.156 0.064 0.780
#> GSM228647 3 0.1109 0.82524 0.028 0.000 0.968 0.004
#> GSM228596 3 0.5803 0.44449 0.024 0.008 0.596 0.372
#> GSM228600 3 0.2884 0.82015 0.028 0.004 0.900 0.068
#> GSM228603 3 0.2565 0.82247 0.032 0.000 0.912 0.056
#> GSM228615 4 0.6175 0.67833 0.004 0.248 0.088 0.660
#> GSM228627 3 0.3031 0.81708 0.016 0.016 0.896 0.072
#> GSM228641 3 0.2218 0.82850 0.028 0.004 0.932 0.036
#> GSM228644 2 0.1042 0.93152 0.000 0.972 0.020 0.008
#> GSM228651 3 0.3202 0.81680 0.024 0.012 0.888 0.076
#> GSM228654 3 0.2125 0.82332 0.012 0.004 0.932 0.052
#> GSM228658 3 0.2317 0.82482 0.032 0.004 0.928 0.036
#> GSM228606 3 0.4382 0.53947 0.000 0.000 0.704 0.296
#> GSM228611 3 0.1733 0.82596 0.028 0.000 0.948 0.024
#> GSM228618 3 0.1356 0.82419 0.032 0.000 0.960 0.008
#> GSM228621 3 0.0895 0.82024 0.000 0.004 0.976 0.020
#> GSM228624 3 0.3344 0.78601 0.004 0.020 0.868 0.108
#> GSM228630 3 0.0895 0.82024 0.000 0.004 0.976 0.020
#> GSM228636 4 0.6557 0.50647 0.004 0.376 0.072 0.548
#> GSM228638 3 0.0707 0.82485 0.020 0.000 0.980 0.000
#> GSM228648 3 0.0804 0.82078 0.000 0.008 0.980 0.012
#> GSM228670 4 0.6058 0.71453 0.064 0.072 0.120 0.744
#> GSM228671 4 0.4840 0.72116 0.000 0.100 0.116 0.784
#> GSM228672 4 0.6456 0.60905 0.252 0.036 0.052 0.660
#> GSM228674 4 0.5698 0.71785 0.028 0.124 0.092 0.756
#> GSM228675 4 0.4462 0.71782 0.000 0.132 0.064 0.804
#> GSM228676 3 0.5485 0.53617 0.020 0.008 0.652 0.320
#> GSM228667 4 0.5794 0.70455 0.012 0.104 0.152 0.732
#> GSM228668 1 0.1489 0.84918 0.952 0.000 0.044 0.004
#> GSM228669 1 0.6120 0.39423 0.628 0.000 0.076 0.296
#> GSM228673 3 0.3933 0.68292 0.000 0.008 0.792 0.200
#> GSM228677 3 0.7007 -0.27455 0.000 0.116 0.452 0.432
#> GSM228678 4 0.6598 0.53244 0.004 0.352 0.080 0.564
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 4 0.6803 0.3422 0.160 0.000 0.124 0.608 0.108
#> GSM228563 4 0.5166 0.2882 0.004 0.100 0.000 0.692 0.204
#> GSM228565 4 0.8321 0.2226 0.260 0.020 0.152 0.440 0.128
#> GSM228566 3 0.3967 0.7641 0.000 0.004 0.808 0.100 0.088
#> GSM228567 1 0.0290 0.8797 0.992 0.000 0.008 0.000 0.000
#> GSM228570 1 0.3184 0.8188 0.872 0.000 0.028 0.068 0.032
#> GSM228571 1 0.2833 0.8361 0.892 0.000 0.052 0.028 0.028
#> GSM228574 3 0.5067 0.6495 0.000 0.000 0.700 0.172 0.128
#> GSM228575 5 0.5571 0.5310 0.000 0.048 0.020 0.332 0.600
#> GSM228576 1 0.8478 0.0184 0.436 0.040 0.188 0.248 0.088
#> GSM228579 1 0.1314 0.8717 0.960 0.000 0.012 0.016 0.012
#> GSM228580 5 0.5714 0.6295 0.000 0.164 0.000 0.212 0.624
#> GSM228581 5 0.6806 0.5257 0.000 0.120 0.112 0.160 0.608
#> GSM228666 5 0.6367 0.6350 0.000 0.188 0.004 0.272 0.536
#> GSM228564 4 0.5140 0.3620 0.080 0.008 0.008 0.720 0.184
#> GSM228568 1 0.8441 -0.1595 0.356 0.008 0.140 0.188 0.308
#> GSM228569 1 0.2854 0.8244 0.880 0.000 0.084 0.008 0.028
#> GSM228572 2 0.4653 0.6341 0.004 0.776 0.016 0.116 0.088
#> GSM228573 3 0.1988 0.8263 0.008 0.000 0.928 0.016 0.048
#> GSM228577 1 0.0981 0.8760 0.972 0.000 0.008 0.008 0.012
#> GSM228578 1 0.3227 0.8177 0.868 0.000 0.072 0.040 0.020
#> GSM228663 3 0.3779 0.7527 0.008 0.004 0.800 0.016 0.172
#> GSM228664 3 0.5309 0.6366 0.000 0.052 0.684 0.028 0.236
#> GSM228665 3 0.2457 0.8080 0.008 0.000 0.900 0.016 0.076
#> GSM228582 1 0.7624 0.1799 0.480 0.316 0.092 0.016 0.096
#> GSM228583 1 0.0290 0.8797 0.992 0.000 0.008 0.000 0.000
#> GSM228585 1 0.0290 0.8797 0.992 0.000 0.008 0.000 0.000
#> GSM228587 1 0.0290 0.8797 0.992 0.000 0.008 0.000 0.000
#> GSM228588 2 0.2153 0.8009 0.000 0.916 0.000 0.040 0.044
#> GSM228589 2 0.1965 0.8055 0.000 0.924 0.000 0.024 0.052
#> GSM228590 1 0.0290 0.8797 0.992 0.000 0.008 0.000 0.000
#> GSM228591 2 0.1597 0.8077 0.000 0.940 0.000 0.012 0.048
#> GSM228597 4 0.4587 0.3545 0.000 0.096 0.000 0.744 0.160
#> GSM228601 2 0.0566 0.8225 0.000 0.984 0.000 0.012 0.004
#> GSM228604 2 0.6367 -0.0109 0.000 0.472 0.420 0.032 0.076
#> GSM228608 1 0.0740 0.8768 0.980 0.000 0.008 0.008 0.004
#> GSM228609 2 0.5565 0.1447 0.020 0.544 0.000 0.400 0.036
#> GSM228613 1 0.0290 0.8797 0.992 0.000 0.008 0.000 0.000
#> GSM228616 3 0.9526 -0.1893 0.196 0.088 0.308 0.252 0.156
#> GSM228628 2 0.2104 0.7771 0.000 0.916 0.000 0.060 0.024
#> GSM228634 1 0.0290 0.8797 0.992 0.000 0.008 0.000 0.000
#> GSM228642 2 0.1012 0.8146 0.000 0.968 0.000 0.012 0.020
#> GSM228645 4 0.8048 -0.1938 0.000 0.176 0.124 0.388 0.312
#> GSM228646 3 0.7100 0.2815 0.000 0.052 0.508 0.288 0.152
#> GSM228652 1 0.1989 0.8641 0.932 0.000 0.020 0.032 0.016
#> GSM228655 1 0.2693 0.8408 0.896 0.000 0.060 0.028 0.016
#> GSM228656 1 0.0290 0.8797 0.992 0.000 0.008 0.000 0.000
#> GSM228659 4 0.5028 0.2086 0.420 0.008 0.000 0.552 0.020
#> GSM228662 1 0.0290 0.8797 0.992 0.000 0.008 0.000 0.000
#> GSM228584 1 0.0290 0.8797 0.992 0.000 0.008 0.000 0.000
#> GSM228586 1 0.0290 0.8797 0.992 0.000 0.008 0.000 0.000
#> GSM228592 1 0.0451 0.8788 0.988 0.000 0.008 0.004 0.000
#> GSM228593 4 0.5762 0.3350 0.152 0.020 0.000 0.668 0.160
#> GSM228594 1 0.1116 0.8736 0.964 0.000 0.028 0.004 0.004
#> GSM228598 1 0.2026 0.8537 0.924 0.000 0.012 0.056 0.008
#> GSM228607 4 0.7406 0.1510 0.024 0.016 0.380 0.408 0.172
#> GSM228612 3 0.6249 0.5701 0.000 0.044 0.628 0.112 0.216
#> GSM228619 3 0.6221 0.4951 0.248 0.000 0.616 0.092 0.044
#> GSM228622 1 0.4141 0.6688 0.760 0.000 0.208 0.020 0.012
#> GSM228625 4 0.7385 0.2752 0.316 0.020 0.096 0.504 0.064
#> GSM228631 3 0.6239 0.5051 0.240 0.000 0.620 0.092 0.048
#> GSM228633 2 0.1787 0.7945 0.000 0.936 0.004 0.016 0.044
#> GSM228637 4 0.4566 0.4128 0.012 0.044 0.020 0.784 0.140
#> GSM228639 3 0.2511 0.8076 0.000 0.000 0.892 0.080 0.028
#> GSM228649 4 0.4727 0.4110 0.012 0.056 0.020 0.776 0.136
#> GSM228660 1 0.6544 0.4780 0.604 0.016 0.252 0.032 0.096
#> GSM228661 1 0.1285 0.8708 0.956 0.000 0.036 0.004 0.004
#> GSM228595 2 0.0613 0.8224 0.000 0.984 0.004 0.008 0.004
#> GSM228599 4 0.5270 0.3972 0.012 0.028 0.148 0.740 0.072
#> GSM228602 3 0.3013 0.8107 0.016 0.000 0.880 0.044 0.060
#> GSM228614 4 0.5797 0.3481 0.000 0.048 0.204 0.672 0.076
#> GSM228626 2 0.0162 0.8213 0.000 0.996 0.004 0.000 0.000
#> GSM228640 3 0.2451 0.8098 0.004 0.000 0.904 0.036 0.056
#> GSM228643 3 0.3170 0.8015 0.004 0.004 0.868 0.052 0.072
#> GSM228650 3 0.3169 0.8028 0.000 0.000 0.856 0.060 0.084
#> GSM228653 3 0.1074 0.8234 0.004 0.000 0.968 0.012 0.016
#> GSM228657 2 0.0727 0.8228 0.000 0.980 0.004 0.012 0.004
#> GSM228605 4 0.7967 0.2302 0.224 0.000 0.252 0.420 0.104
#> GSM228610 3 0.1915 0.8202 0.000 0.000 0.928 0.032 0.040
#> GSM228617 3 0.3005 0.8102 0.028 0.000 0.884 0.040 0.048
#> GSM228620 3 0.2312 0.8157 0.016 0.000 0.912 0.012 0.060
#> GSM228623 4 0.4297 0.4082 0.000 0.056 0.020 0.792 0.132
#> GSM228629 3 0.1911 0.8229 0.004 0.000 0.932 0.028 0.036
#> GSM228632 3 0.2264 0.8146 0.004 0.000 0.912 0.024 0.060
#> GSM228635 4 0.5345 0.0915 0.004 0.028 0.016 0.604 0.348
#> GSM228647 3 0.0451 0.8226 0.004 0.000 0.988 0.000 0.008
#> GSM228596 3 0.6442 0.3765 0.004 0.004 0.548 0.256 0.188
#> GSM228600 3 0.2519 0.8098 0.004 0.000 0.900 0.036 0.060
#> GSM228603 3 0.2451 0.8104 0.004 0.000 0.904 0.036 0.056
#> GSM228615 4 0.4766 0.4321 0.008 0.072 0.044 0.788 0.088
#> GSM228627 3 0.2664 0.8122 0.004 0.000 0.884 0.020 0.092
#> GSM228641 3 0.2067 0.8138 0.000 0.000 0.920 0.032 0.048
#> GSM228644 2 0.0324 0.8207 0.000 0.992 0.004 0.000 0.004
#> GSM228651 3 0.2206 0.8192 0.004 0.000 0.912 0.016 0.068
#> GSM228654 3 0.1701 0.8221 0.000 0.000 0.936 0.016 0.048
#> GSM228658 3 0.1787 0.8216 0.004 0.000 0.936 0.016 0.044
#> GSM228606 3 0.5850 0.0322 0.000 0.000 0.476 0.428 0.096
#> GSM228611 3 0.2568 0.8018 0.004 0.000 0.888 0.016 0.092
#> GSM228618 3 0.1560 0.8227 0.004 0.000 0.948 0.020 0.028
#> GSM228621 3 0.1764 0.8208 0.000 0.000 0.928 0.008 0.064
#> GSM228624 3 0.5363 0.6343 0.000 0.004 0.680 0.132 0.184
#> GSM228630 3 0.1682 0.8210 0.000 0.004 0.940 0.012 0.044
#> GSM228636 4 0.5963 0.2299 0.000 0.252 0.012 0.612 0.124
#> GSM228638 3 0.1461 0.8233 0.004 0.000 0.952 0.016 0.028
#> GSM228648 3 0.0854 0.8224 0.000 0.008 0.976 0.004 0.012
#> GSM228670 4 0.4646 0.4294 0.044 0.020 0.032 0.796 0.108
#> GSM228671 4 0.4904 0.2718 0.008 0.004 0.036 0.688 0.264
#> GSM228672 4 0.3958 0.4287 0.140 0.008 0.004 0.808 0.040
#> GSM228674 4 0.6076 0.3321 0.032 0.056 0.036 0.676 0.200
#> GSM228675 4 0.4562 0.2916 0.008 0.016 0.012 0.724 0.240
#> GSM228676 4 0.6359 0.0648 0.004 0.000 0.416 0.440 0.140
#> GSM228667 4 0.4353 0.4293 0.000 0.028 0.088 0.800 0.084
#> GSM228668 1 0.2625 0.8428 0.900 0.000 0.040 0.048 0.012
#> GSM228669 4 0.5235 0.2453 0.440 0.000 0.024 0.524 0.012
#> GSM228673 3 0.6525 0.3346 0.000 0.012 0.536 0.272 0.180
#> GSM228677 4 0.6782 0.1071 0.000 0.032 0.408 0.440 0.120
#> GSM228678 4 0.6574 0.2021 0.000 0.280 0.040 0.564 0.116
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 4 0.6855 0.26978 0.128 0.000 0.060 0.584 0.152 0.076
#> GSM228563 4 0.5553 0.09540 0.000 0.036 0.000 0.492 0.416 0.056
#> GSM228565 4 0.7911 0.15784 0.184 0.004 0.072 0.464 0.172 0.104
#> GSM228566 3 0.5139 0.62498 0.000 0.000 0.696 0.096 0.156 0.052
#> GSM228567 1 0.0000 0.86353 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228570 1 0.3667 0.76978 0.828 0.000 0.020 0.084 0.056 0.012
#> GSM228571 1 0.3123 0.80638 0.868 0.000 0.024 0.056 0.036 0.016
#> GSM228574 3 0.6549 0.44691 0.000 0.000 0.536 0.188 0.084 0.192
#> GSM228575 5 0.6280 -0.06450 0.000 0.028 0.008 0.144 0.508 0.312
#> GSM228576 1 0.8339 -0.24444 0.348 0.020 0.096 0.328 0.140 0.068
#> GSM228579 1 0.2051 0.83560 0.920 0.000 0.012 0.044 0.020 0.004
#> GSM228580 5 0.6231 -0.13728 0.000 0.084 0.000 0.072 0.484 0.360
#> GSM228581 6 0.6307 0.20997 0.000 0.048 0.056 0.076 0.208 0.612
#> GSM228666 6 0.6978 -0.00638 0.000 0.112 0.000 0.144 0.312 0.432
#> GSM228564 4 0.5534 0.19806 0.072 0.004 0.004 0.560 0.344 0.016
#> GSM228568 6 0.7845 0.11219 0.228 0.000 0.076 0.100 0.148 0.448
#> GSM228569 1 0.3047 0.78544 0.848 0.000 0.084 0.000 0.004 0.064
#> GSM228572 2 0.4764 0.65446 0.000 0.728 0.004 0.088 0.152 0.028
#> GSM228573 3 0.2475 0.76313 0.000 0.000 0.892 0.012 0.036 0.060
#> GSM228577 1 0.1232 0.85343 0.956 0.000 0.016 0.000 0.004 0.024
#> GSM228578 1 0.4074 0.75784 0.808 0.000 0.072 0.056 0.012 0.052
#> GSM228663 3 0.4067 0.60510 0.000 0.012 0.680 0.012 0.000 0.296
#> GSM228664 3 0.5263 0.25363 0.000 0.032 0.472 0.028 0.004 0.464
#> GSM228665 3 0.2768 0.71725 0.000 0.000 0.832 0.012 0.000 0.156
#> GSM228582 1 0.8062 -0.13053 0.420 0.264 0.044 0.040 0.052 0.180
#> GSM228583 1 0.0000 0.86353 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228585 1 0.0000 0.86353 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228587 1 0.0146 0.86284 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM228588 2 0.3436 0.83292 0.000 0.836 0.000 0.032 0.080 0.052
#> GSM228589 2 0.3464 0.84308 0.000 0.832 0.000 0.028 0.052 0.088
#> GSM228590 1 0.0000 0.86353 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228591 2 0.2721 0.85660 0.000 0.868 0.000 0.004 0.040 0.088
#> GSM228597 4 0.5063 0.23680 0.000 0.044 0.000 0.640 0.276 0.040
#> GSM228601 2 0.0146 0.89818 0.000 0.996 0.000 0.004 0.000 0.000
#> GSM228604 3 0.7378 0.05369 0.000 0.360 0.400 0.044 0.120 0.076
#> GSM228608 1 0.0692 0.85905 0.976 0.000 0.000 0.020 0.000 0.004
#> GSM228609 4 0.6383 0.02282 0.008 0.396 0.000 0.444 0.108 0.044
#> GSM228613 1 0.0000 0.86353 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228616 5 0.9410 -0.00779 0.136 0.044 0.184 0.208 0.280 0.148
#> GSM228628 2 0.2828 0.83212 0.000 0.872 0.000 0.072 0.020 0.036
#> GSM228634 1 0.0000 0.86353 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228642 2 0.1826 0.88367 0.000 0.924 0.000 0.004 0.020 0.052
#> GSM228645 5 0.8159 0.05097 0.000 0.104 0.068 0.264 0.360 0.204
#> GSM228646 3 0.7690 -0.14742 0.000 0.020 0.356 0.268 0.256 0.100
#> GSM228652 1 0.1912 0.84250 0.924 0.000 0.008 0.052 0.008 0.008
#> GSM228655 1 0.2940 0.81426 0.876 0.000 0.048 0.048 0.016 0.012
#> GSM228656 1 0.0000 0.86353 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228659 4 0.5139 0.15618 0.416 0.000 0.000 0.516 0.056 0.012
#> GSM228662 1 0.0000 0.86353 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228584 1 0.0000 0.86353 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228586 1 0.0146 0.86297 0.996 0.000 0.000 0.000 0.000 0.004
#> GSM228592 1 0.0520 0.86033 0.984 0.000 0.008 0.000 0.000 0.008
#> GSM228593 4 0.7160 0.07524 0.124 0.004 0.000 0.432 0.296 0.144
#> GSM228594 1 0.1320 0.84981 0.948 0.000 0.036 0.000 0.000 0.016
#> GSM228598 1 0.2402 0.83177 0.904 0.000 0.004 0.032 0.020 0.040
#> GSM228607 4 0.7205 0.07201 0.016 0.000 0.332 0.372 0.052 0.228
#> GSM228612 3 0.6515 0.25334 0.000 0.020 0.460 0.104 0.040 0.376
#> GSM228619 3 0.5890 0.55102 0.164 0.000 0.664 0.060 0.072 0.040
#> GSM228622 1 0.4353 0.59659 0.728 0.000 0.212 0.020 0.004 0.036
#> GSM228625 4 0.7293 0.23069 0.264 0.004 0.068 0.488 0.040 0.136
#> GSM228631 3 0.5512 0.58434 0.152 0.000 0.696 0.044 0.068 0.040
#> GSM228633 2 0.1950 0.87442 0.000 0.924 0.000 0.016 0.028 0.032
#> GSM228637 4 0.5928 0.27453 0.004 0.016 0.040 0.620 0.236 0.084
#> GSM228639 3 0.2657 0.75215 0.000 0.000 0.880 0.076 0.020 0.024
#> GSM228649 4 0.6121 0.27518 0.008 0.008 0.036 0.612 0.168 0.168
#> GSM228660 1 0.7225 0.17329 0.488 0.000 0.232 0.092 0.024 0.164
#> GSM228661 1 0.1500 0.84275 0.936 0.000 0.052 0.000 0.000 0.012
#> GSM228595 2 0.0665 0.89744 0.000 0.980 0.000 0.008 0.004 0.008
#> GSM228599 4 0.6600 0.14363 0.004 0.024 0.156 0.556 0.224 0.036
#> GSM228602 3 0.3461 0.73938 0.008 0.000 0.840 0.024 0.084 0.044
#> GSM228614 4 0.5678 0.19908 0.000 0.012 0.264 0.612 0.080 0.032
#> GSM228626 2 0.0260 0.89692 0.000 0.992 0.000 0.000 0.000 0.008
#> GSM228640 3 0.2831 0.73908 0.000 0.000 0.868 0.032 0.084 0.016
#> GSM228643 3 0.3716 0.72798 0.000 0.004 0.820 0.080 0.072 0.024
#> GSM228650 3 0.4195 0.70136 0.000 0.004 0.776 0.040 0.140 0.040
#> GSM228653 3 0.1882 0.75873 0.000 0.000 0.928 0.020 0.028 0.024
#> GSM228657 2 0.0820 0.89503 0.000 0.972 0.000 0.012 0.016 0.000
#> GSM228605 4 0.7879 0.22405 0.144 0.000 0.204 0.456 0.076 0.120
#> GSM228610 3 0.2952 0.74475 0.000 0.000 0.864 0.052 0.016 0.068
#> GSM228617 3 0.3212 0.73771 0.020 0.000 0.860 0.012 0.060 0.048
#> GSM228620 3 0.2501 0.73999 0.000 0.000 0.872 0.016 0.004 0.108
#> GSM228623 4 0.5482 0.29954 0.004 0.020 0.028 0.680 0.184 0.084
#> GSM228629 3 0.2649 0.75638 0.000 0.000 0.880 0.016 0.028 0.076
#> GSM228632 3 0.3279 0.72578 0.000 0.000 0.828 0.060 0.004 0.108
#> GSM228635 5 0.5973 -0.01848 0.000 0.028 0.004 0.380 0.488 0.100
#> GSM228647 3 0.0862 0.75970 0.000 0.000 0.972 0.016 0.004 0.008
#> GSM228596 3 0.6824 0.28213 0.000 0.000 0.472 0.264 0.084 0.180
#> GSM228600 3 0.3291 0.73046 0.000 0.000 0.840 0.040 0.096 0.024
#> GSM228603 3 0.2999 0.73791 0.000 0.000 0.860 0.032 0.084 0.024
#> GSM228615 4 0.5261 0.32795 0.004 0.044 0.052 0.720 0.148 0.032
#> GSM228627 3 0.4100 0.72444 0.000 0.000 0.788 0.068 0.040 0.104
#> GSM228641 3 0.2883 0.74414 0.000 0.000 0.868 0.036 0.076 0.020
#> GSM228644 2 0.0146 0.89757 0.000 0.996 0.000 0.000 0.000 0.004
#> GSM228651 3 0.3794 0.73962 0.000 0.000 0.812 0.052 0.044 0.092
#> GSM228654 3 0.2401 0.75451 0.000 0.000 0.900 0.024 0.048 0.028
#> GSM228658 3 0.2668 0.75648 0.000 0.000 0.884 0.028 0.028 0.060
#> GSM228606 3 0.6753 0.11793 0.000 0.000 0.444 0.332 0.084 0.140
#> GSM228611 3 0.4048 0.68676 0.000 0.000 0.764 0.052 0.016 0.168
#> GSM228618 3 0.2063 0.75447 0.000 0.000 0.912 0.008 0.020 0.060
#> GSM228621 3 0.2230 0.75828 0.000 0.000 0.892 0.000 0.024 0.084
#> GSM228624 3 0.6270 0.29247 0.000 0.000 0.480 0.088 0.072 0.360
#> GSM228630 3 0.1555 0.75622 0.000 0.000 0.940 0.008 0.012 0.040
#> GSM228636 4 0.6627 0.17960 0.000 0.164 0.008 0.536 0.224 0.068
#> GSM228638 3 0.1313 0.75720 0.000 0.000 0.952 0.016 0.004 0.028
#> GSM228648 3 0.0964 0.76066 0.000 0.004 0.968 0.016 0.000 0.012
#> GSM228670 4 0.4758 0.34660 0.024 0.008 0.028 0.768 0.080 0.092
#> GSM228671 4 0.5936 0.21099 0.000 0.008 0.008 0.552 0.188 0.244
#> GSM228672 4 0.4075 0.36121 0.124 0.000 0.004 0.788 0.056 0.028
#> GSM228674 4 0.6191 0.28837 0.016 0.020 0.024 0.624 0.140 0.176
#> GSM228675 4 0.5819 0.23104 0.000 0.012 0.008 0.588 0.200 0.192
#> GSM228676 4 0.6950 0.11461 0.008 0.000 0.332 0.436 0.076 0.148
#> GSM228667 4 0.5140 0.33391 0.000 0.020 0.056 0.728 0.120 0.076
#> GSM228668 1 0.3505 0.79106 0.844 0.000 0.048 0.064 0.012 0.032
#> GSM228669 4 0.5672 0.20965 0.384 0.000 0.036 0.524 0.012 0.044
#> GSM228673 3 0.6872 0.01834 0.000 0.000 0.396 0.232 0.056 0.316
#> GSM228677 4 0.7576 0.06429 0.000 0.024 0.344 0.360 0.172 0.100
#> GSM228678 4 0.6995 0.18523 0.000 0.220 0.028 0.516 0.172 0.064
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
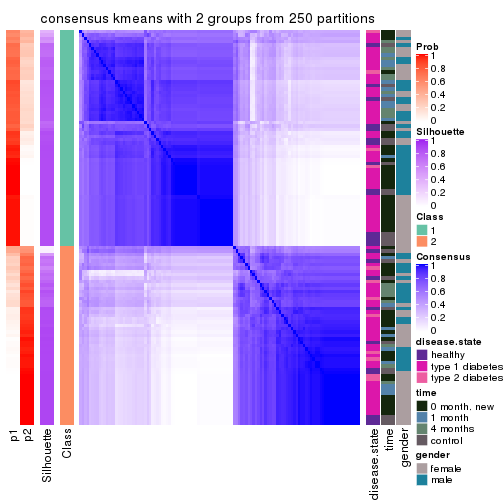
consensus_heatmap(res, k = 3)
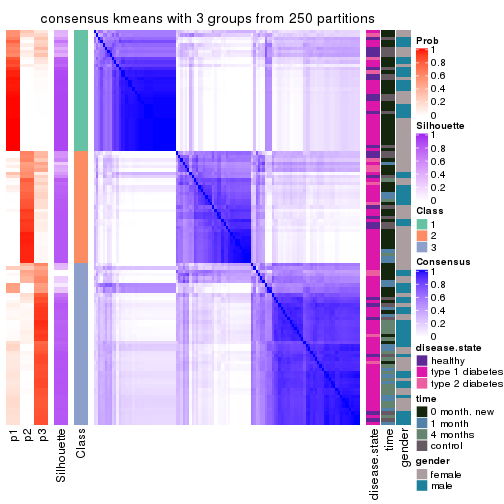
consensus_heatmap(res, k = 4)
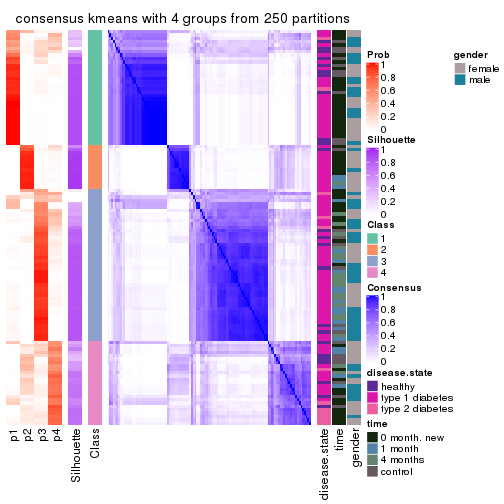
consensus_heatmap(res, k = 5)
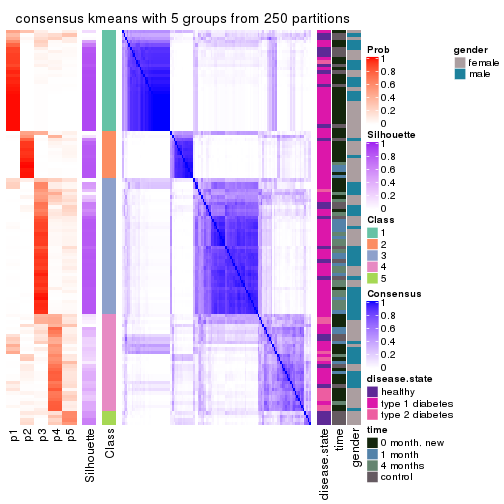
consensus_heatmap(res, k = 6)
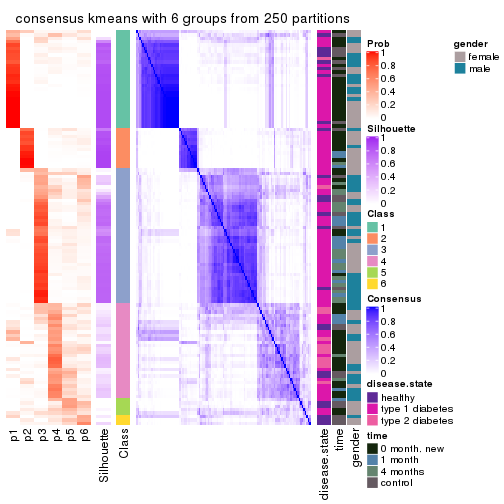
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
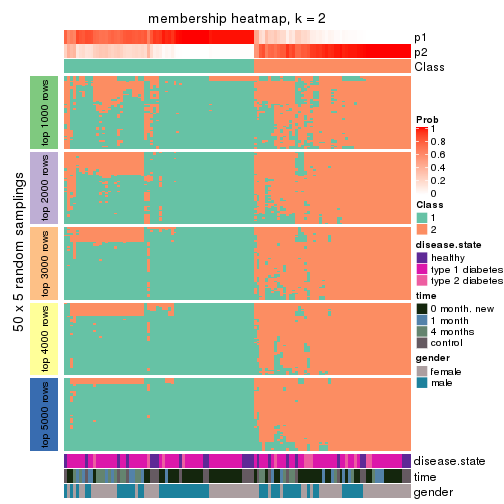
membership_heatmap(res, k = 3)
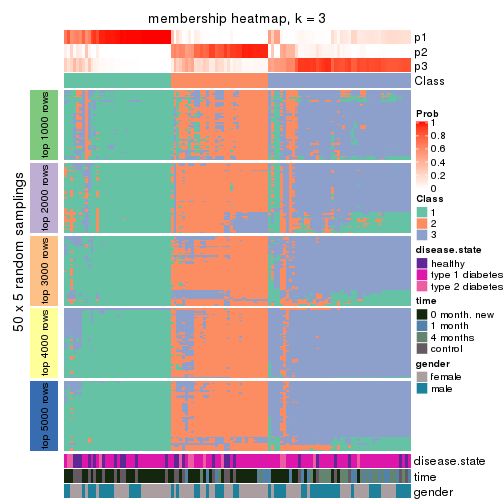
membership_heatmap(res, k = 4)
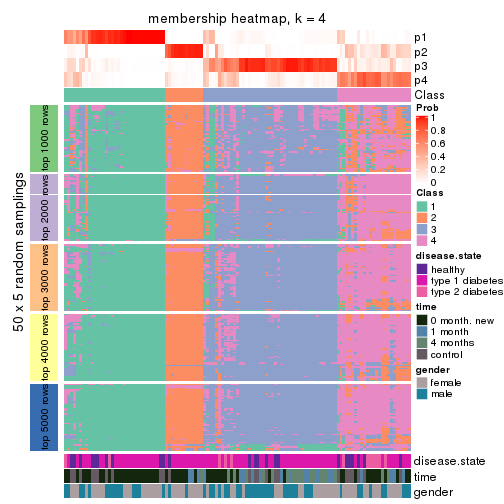
membership_heatmap(res, k = 5)
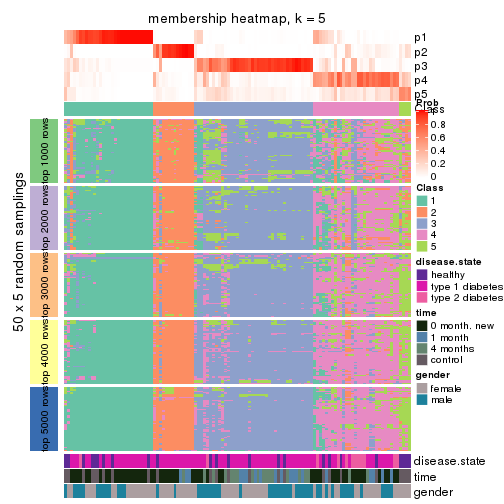
membership_heatmap(res, k = 6)
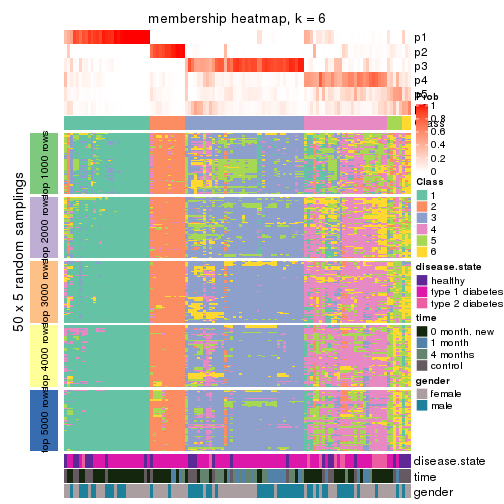
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
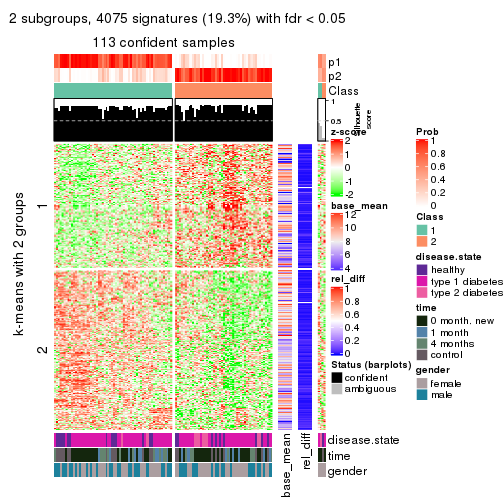
get_signatures(res, k = 3)
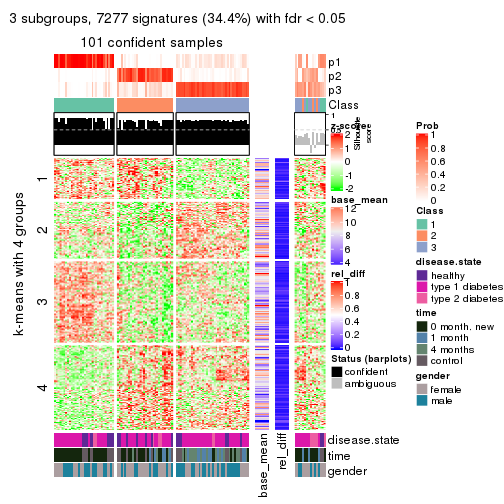
get_signatures(res, k = 4)
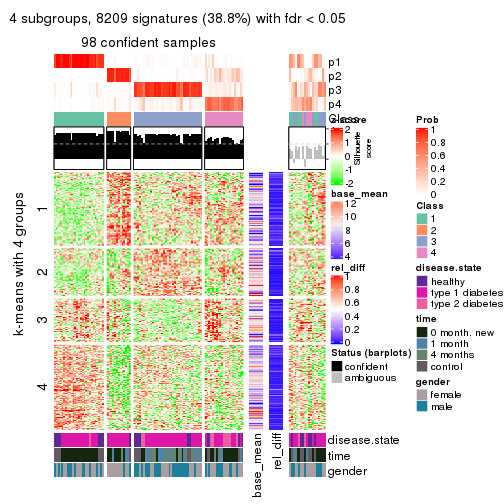
get_signatures(res, k = 5)
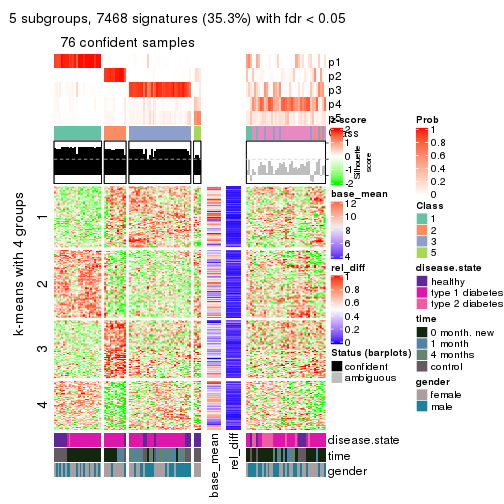
get_signatures(res, k = 6)
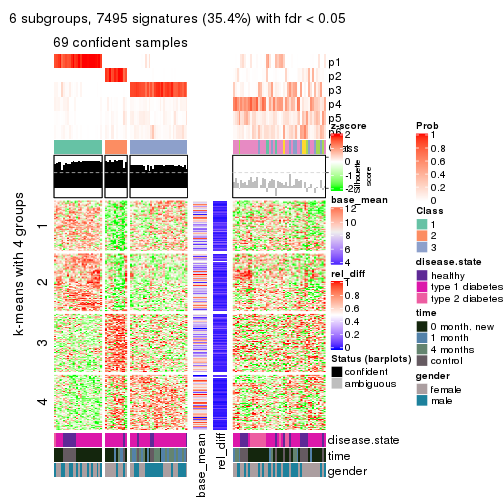
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
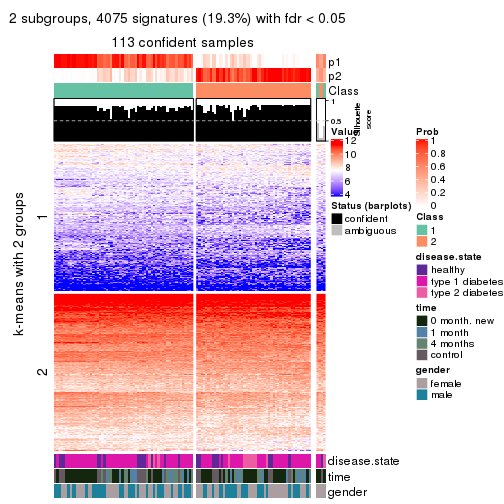
get_signatures(res, k = 3, scale_rows = FALSE)
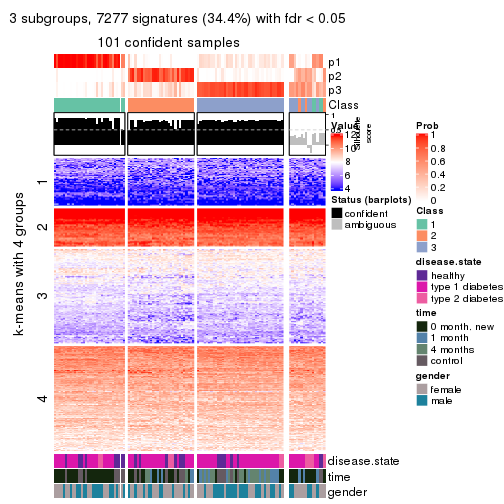
get_signatures(res, k = 4, scale_rows = FALSE)
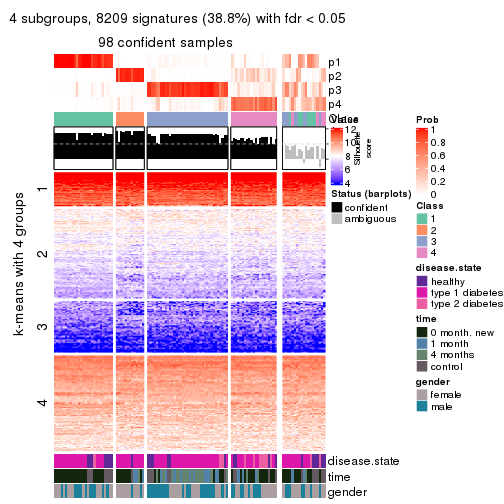
get_signatures(res, k = 5, scale_rows = FALSE)
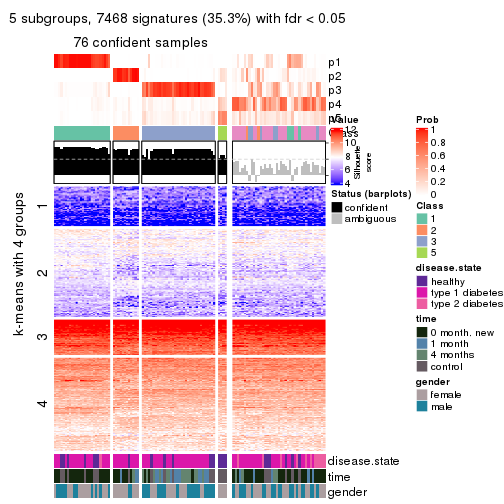
get_signatures(res, k = 6, scale_rows = FALSE)
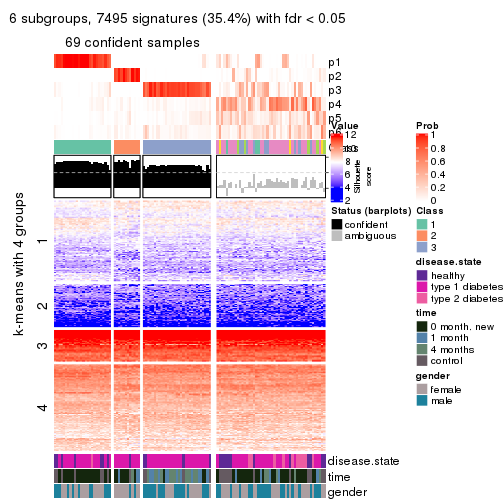
Compare the overlap of signatures from different k:
compare_signatures(res)
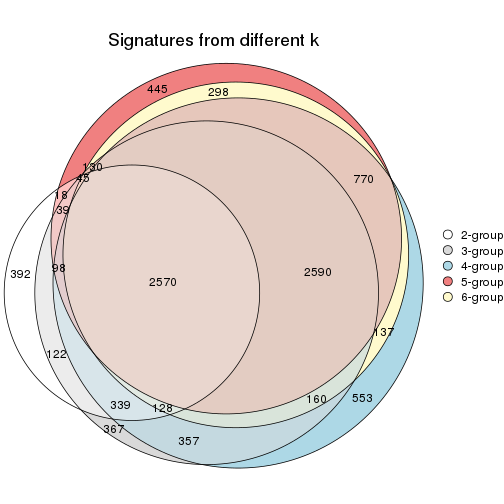
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
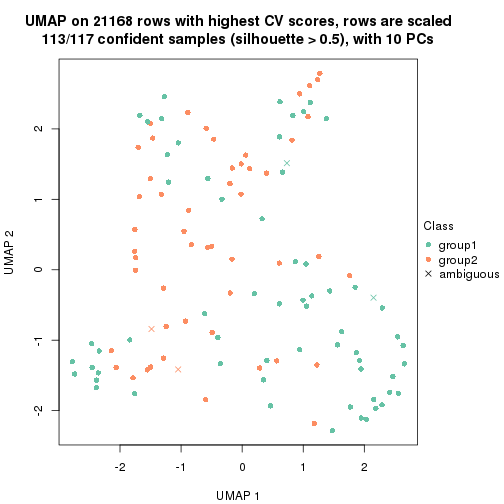
dimension_reduction(res, k = 3, method = "UMAP")
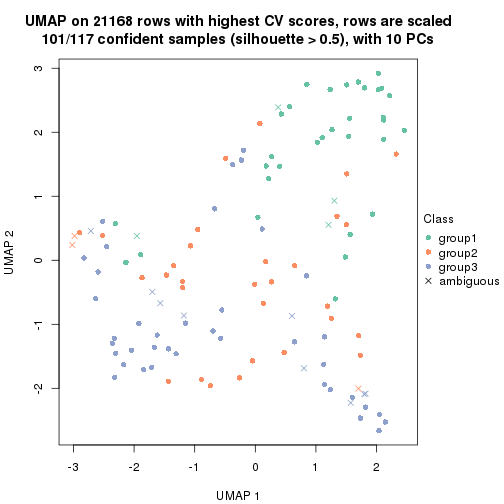
dimension_reduction(res, k = 4, method = "UMAP")
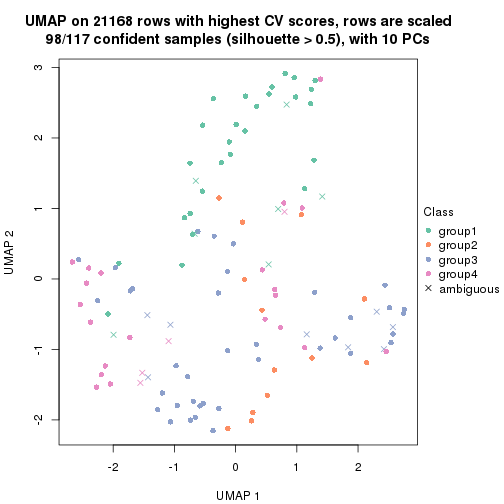
dimension_reduction(res, k = 5, method = "UMAP")
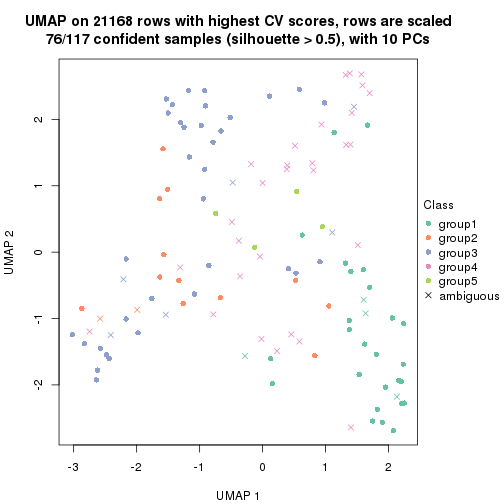
dimension_reduction(res, k = 6, method = "UMAP")
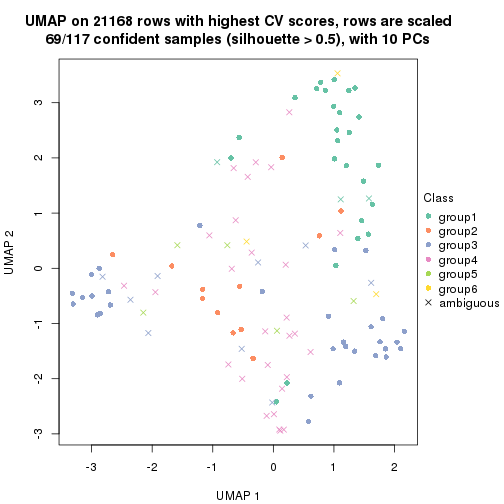
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
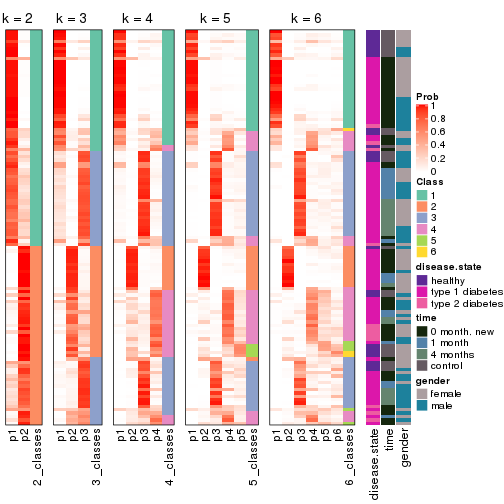
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> CV:kmeans 113 0.14536 8.91e-01 0.7132 2
#> CV:kmeans 101 0.63204 2.93e-07 0.1627 3
#> CV:kmeans 98 0.00461 4.98e-06 0.0523 4
#> CV:kmeans 76 0.00793 1.54e-08 0.0198 5
#> CV:kmeans 69 0.32049 7.36e-07 0.0472 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["CV", "skmeans"]
# you can also extract it by
# res = res_list["CV:skmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'CV' method.
#> Subgroups are detected by 'skmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
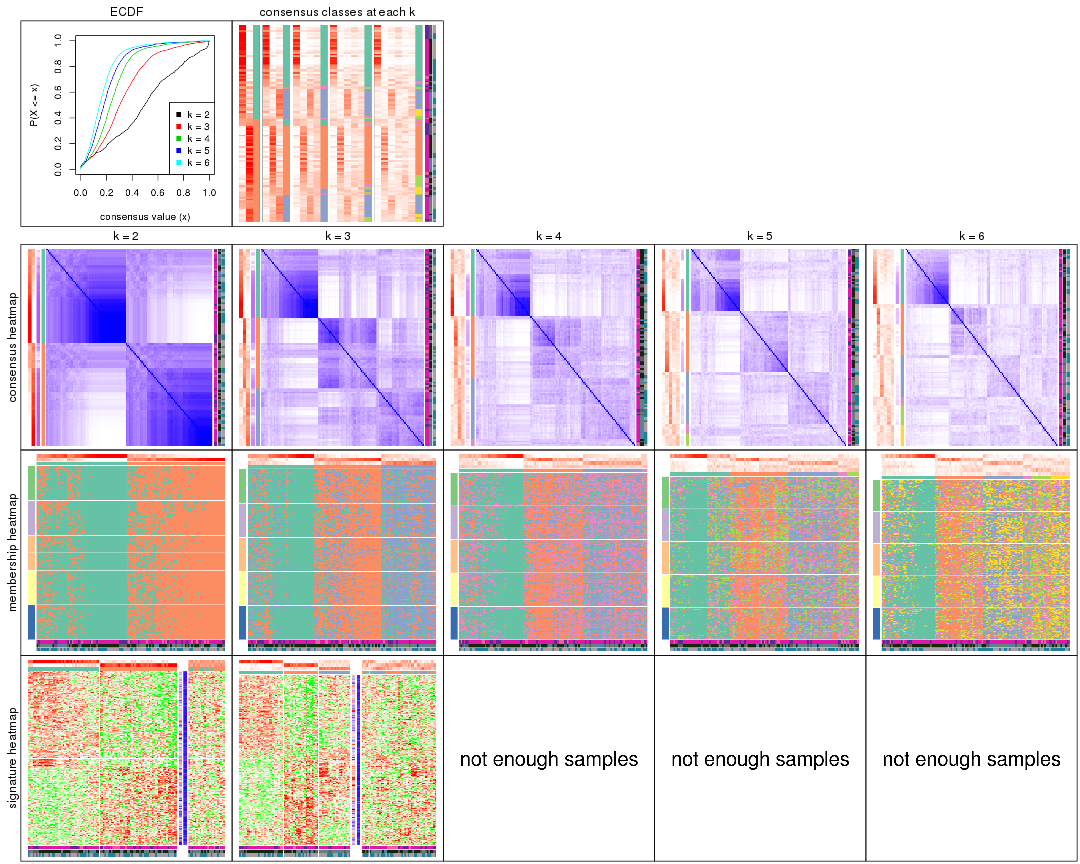
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
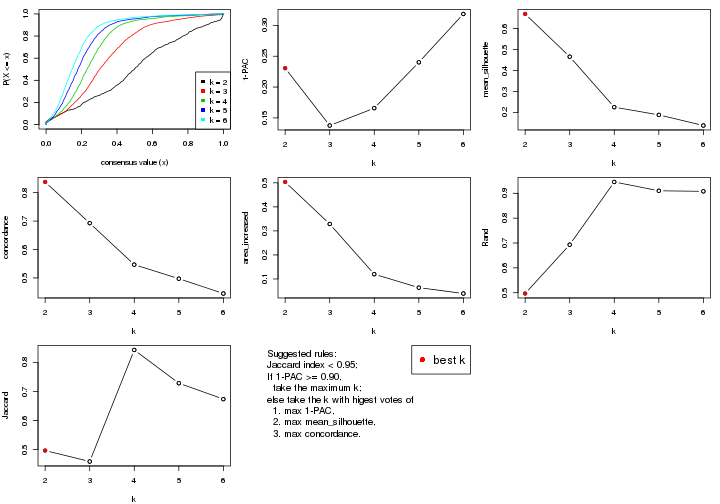
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.231 0.669 0.838 0.5036 0.497 0.497
#> 3 3 0.138 0.466 0.693 0.3286 0.693 0.459
#> 4 4 0.166 0.225 0.547 0.1198 0.946 0.843
#> 5 5 0.240 0.189 0.497 0.0639 0.910 0.729
#> 6 6 0.319 0.138 0.445 0.0394 0.908 0.674
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.9248 0.5030 0.660 0.340
#> GSM228563 2 0.3879 0.7978 0.076 0.924
#> GSM228565 2 0.9944 0.2201 0.456 0.544
#> GSM228566 2 0.9170 0.5532 0.332 0.668
#> GSM228567 1 0.0000 0.8107 1.000 0.000
#> GSM228570 1 0.0376 0.8106 0.996 0.004
#> GSM228571 1 0.0000 0.8107 1.000 0.000
#> GSM228574 2 0.7056 0.7482 0.192 0.808
#> GSM228575 2 0.4815 0.7950 0.104 0.896
#> GSM228576 1 0.7139 0.7244 0.804 0.196
#> GSM228579 1 0.0938 0.8100 0.988 0.012
#> GSM228580 2 0.0000 0.7904 0.000 1.000
#> GSM228581 2 0.3879 0.8019 0.076 0.924
#> GSM228666 2 0.1184 0.7962 0.016 0.984
#> GSM228564 2 0.9909 0.2915 0.444 0.556
#> GSM228568 1 0.9754 0.2980 0.592 0.408
#> GSM228569 1 0.0000 0.8107 1.000 0.000
#> GSM228572 2 0.0000 0.7904 0.000 1.000
#> GSM228573 1 0.6531 0.7540 0.832 0.168
#> GSM228577 1 0.0000 0.8107 1.000 0.000
#> GSM228578 1 0.0000 0.8107 1.000 0.000
#> GSM228663 1 0.9608 0.4121 0.616 0.384
#> GSM228664 2 0.4022 0.7996 0.080 0.920
#> GSM228665 1 0.8144 0.6727 0.748 0.252
#> GSM228582 2 0.9922 0.2522 0.448 0.552
#> GSM228583 1 0.0000 0.8107 1.000 0.000
#> GSM228585 1 0.0000 0.8107 1.000 0.000
#> GSM228587 1 0.0938 0.8100 0.988 0.012
#> GSM228588 2 0.4431 0.7953 0.092 0.908
#> GSM228589 2 0.2948 0.7987 0.052 0.948
#> GSM228590 1 0.0000 0.8107 1.000 0.000
#> GSM228591 2 0.0376 0.7915 0.004 0.996
#> GSM228597 2 0.3584 0.7993 0.068 0.932
#> GSM228601 2 0.0672 0.7928 0.008 0.992
#> GSM228604 2 0.0000 0.7904 0.000 1.000
#> GSM228608 1 0.0672 0.8101 0.992 0.008
#> GSM228609 2 0.9460 0.5048 0.364 0.636
#> GSM228613 1 0.0000 0.8107 1.000 0.000
#> GSM228616 2 0.9988 0.1375 0.480 0.520
#> GSM228628 2 0.1843 0.7989 0.028 0.972
#> GSM228634 1 0.0000 0.8107 1.000 0.000
#> GSM228642 2 0.0000 0.7904 0.000 1.000
#> GSM228645 2 0.6247 0.7732 0.156 0.844
#> GSM228646 2 0.7453 0.7322 0.212 0.788
#> GSM228652 1 0.1843 0.8081 0.972 0.028
#> GSM228655 1 0.2043 0.8086 0.968 0.032
#> GSM228656 1 0.0000 0.8107 1.000 0.000
#> GSM228659 1 0.7883 0.6689 0.764 0.236
#> GSM228662 1 0.0000 0.8107 1.000 0.000
#> GSM228584 1 0.0000 0.8107 1.000 0.000
#> GSM228586 1 0.0000 0.8107 1.000 0.000
#> GSM228592 1 0.0000 0.8107 1.000 0.000
#> GSM228593 2 0.9988 0.1542 0.480 0.520
#> GSM228594 1 0.0000 0.8107 1.000 0.000
#> GSM228598 1 0.3733 0.7948 0.928 0.072
#> GSM228607 1 1.0000 -0.0644 0.500 0.500
#> GSM228612 2 0.7453 0.7307 0.212 0.788
#> GSM228619 1 0.5737 0.7707 0.864 0.136
#> GSM228622 1 0.0000 0.8107 1.000 0.000
#> GSM228625 1 0.8861 0.5745 0.696 0.304
#> GSM228631 1 0.1184 0.8099 0.984 0.016
#> GSM228633 2 0.0000 0.7904 0.000 1.000
#> GSM228637 2 0.7883 0.7142 0.236 0.764
#> GSM228639 2 0.7453 0.7358 0.212 0.788
#> GSM228649 2 0.8443 0.6676 0.272 0.728
#> GSM228660 1 0.7056 0.7241 0.808 0.192
#> GSM228661 1 0.0000 0.8107 1.000 0.000
#> GSM228595 2 0.0000 0.7904 0.000 1.000
#> GSM228599 2 0.6623 0.7621 0.172 0.828
#> GSM228602 1 0.8909 0.5787 0.692 0.308
#> GSM228614 2 0.6531 0.7705 0.168 0.832
#> GSM228626 2 0.0000 0.7904 0.000 1.000
#> GSM228640 1 0.6247 0.7595 0.844 0.156
#> GSM228643 2 0.9896 0.2451 0.440 0.560
#> GSM228650 2 0.7219 0.7385 0.200 0.800
#> GSM228653 1 0.7883 0.6849 0.764 0.236
#> GSM228657 2 0.0000 0.7904 0.000 1.000
#> GSM228605 1 0.6801 0.7402 0.820 0.180
#> GSM228610 1 0.9954 0.1739 0.540 0.460
#> GSM228617 1 0.8327 0.6558 0.736 0.264
#> GSM228620 1 0.4022 0.7932 0.920 0.080
#> GSM228623 2 0.3274 0.8021 0.060 0.940
#> GSM228629 1 0.7219 0.7298 0.800 0.200
#> GSM228632 2 0.6801 0.7573 0.180 0.820
#> GSM228635 2 0.1184 0.7948 0.016 0.984
#> GSM228647 1 0.9608 0.4103 0.616 0.384
#> GSM228596 1 0.9635 0.4048 0.612 0.388
#> GSM228600 2 0.9933 0.2076 0.452 0.548
#> GSM228603 1 0.4939 0.7838 0.892 0.108
#> GSM228615 2 0.5294 0.7889 0.120 0.880
#> GSM228627 2 0.9896 0.2623 0.440 0.560
#> GSM228641 2 0.9580 0.4370 0.380 0.620
#> GSM228644 2 0.0000 0.7904 0.000 1.000
#> GSM228651 2 0.9795 0.3271 0.416 0.584
#> GSM228654 2 0.9491 0.4733 0.368 0.632
#> GSM228658 1 0.8207 0.6650 0.744 0.256
#> GSM228606 2 0.5178 0.7926 0.116 0.884
#> GSM228611 1 0.9970 0.1365 0.532 0.468
#> GSM228618 1 0.9580 0.4280 0.620 0.380
#> GSM228621 2 0.4690 0.7941 0.100 0.900
#> GSM228624 2 0.7674 0.7167 0.224 0.776
#> GSM228630 2 0.5059 0.7882 0.112 0.888
#> GSM228636 2 0.1414 0.7958 0.020 0.980
#> GSM228638 1 0.9754 0.3452 0.592 0.408
#> GSM228648 2 0.4161 0.7964 0.084 0.916
#> GSM228670 2 0.7139 0.7507 0.196 0.804
#> GSM228671 2 0.1184 0.7956 0.016 0.984
#> GSM228672 1 0.9248 0.4826 0.660 0.340
#> GSM228674 2 0.9795 0.3721 0.416 0.584
#> GSM228675 2 0.2948 0.8022 0.052 0.948
#> GSM228676 1 0.9944 0.1382 0.544 0.456
#> GSM228667 2 0.8386 0.6733 0.268 0.732
#> GSM228668 1 0.0672 0.8107 0.992 0.008
#> GSM228669 1 0.6148 0.7534 0.848 0.152
#> GSM228673 2 0.8861 0.6169 0.304 0.696
#> GSM228677 2 0.0376 0.7916 0.004 0.996
#> GSM228678 2 0.2603 0.8016 0.044 0.956
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.9334 0.1891 0.508 0.200 0.292
#> GSM228563 2 0.7398 0.5575 0.120 0.700 0.180
#> GSM228565 1 0.9547 0.1060 0.480 0.292 0.228
#> GSM228566 3 0.9106 0.4221 0.180 0.284 0.536
#> GSM228567 1 0.0424 0.7172 0.992 0.000 0.008
#> GSM228570 1 0.4786 0.6978 0.844 0.044 0.112
#> GSM228571 1 0.4483 0.6893 0.848 0.024 0.128
#> GSM228574 3 0.8887 0.2696 0.128 0.368 0.504
#> GSM228575 2 0.8087 0.3725 0.076 0.560 0.364
#> GSM228576 1 0.8845 0.3819 0.576 0.184 0.240
#> GSM228579 1 0.3045 0.7158 0.916 0.020 0.064
#> GSM228580 2 0.4931 0.5745 0.000 0.768 0.232
#> GSM228581 2 0.7959 0.4465 0.092 0.620 0.288
#> GSM228666 2 0.5967 0.5863 0.032 0.752 0.216
#> GSM228564 1 0.9411 0.0201 0.444 0.380 0.176
#> GSM228568 1 0.9741 -0.0214 0.448 0.284 0.268
#> GSM228569 1 0.4261 0.6805 0.848 0.012 0.140
#> GSM228572 2 0.4002 0.6041 0.000 0.840 0.160
#> GSM228573 3 0.7918 0.5896 0.256 0.104 0.640
#> GSM228577 1 0.1411 0.7190 0.964 0.000 0.036
#> GSM228578 1 0.5842 0.6328 0.768 0.036 0.196
#> GSM228663 3 0.9175 0.5092 0.244 0.216 0.540
#> GSM228664 2 0.7169 0.1245 0.024 0.520 0.456
#> GSM228665 3 0.8233 0.5767 0.272 0.116 0.612
#> GSM228582 2 0.9664 0.0649 0.332 0.444 0.224
#> GSM228583 1 0.0237 0.7171 0.996 0.000 0.004
#> GSM228585 1 0.0424 0.7173 0.992 0.000 0.008
#> GSM228587 1 0.1832 0.7156 0.956 0.036 0.008
#> GSM228588 2 0.4749 0.5970 0.116 0.844 0.040
#> GSM228589 2 0.3554 0.6156 0.036 0.900 0.064
#> GSM228590 1 0.0424 0.7173 0.992 0.000 0.008
#> GSM228591 2 0.3325 0.6148 0.020 0.904 0.076
#> GSM228597 2 0.6093 0.6074 0.068 0.776 0.156
#> GSM228601 2 0.1289 0.5998 0.000 0.968 0.032
#> GSM228604 2 0.6314 0.3586 0.004 0.604 0.392
#> GSM228608 1 0.3415 0.7141 0.900 0.020 0.080
#> GSM228609 2 0.8055 0.3715 0.292 0.612 0.096
#> GSM228613 1 0.0000 0.7169 1.000 0.000 0.000
#> GSM228616 1 0.9744 -0.0749 0.428 0.336 0.236
#> GSM228628 2 0.6565 0.5656 0.048 0.720 0.232
#> GSM228634 1 0.0592 0.7171 0.988 0.000 0.012
#> GSM228642 2 0.3482 0.6124 0.000 0.872 0.128
#> GSM228645 2 0.8732 0.3445 0.132 0.552 0.316
#> GSM228646 2 0.8943 0.0772 0.128 0.480 0.392
#> GSM228652 1 0.5407 0.6709 0.804 0.040 0.156
#> GSM228655 1 0.7297 0.5743 0.704 0.108 0.188
#> GSM228656 1 0.0000 0.7169 1.000 0.000 0.000
#> GSM228659 1 0.6630 0.5893 0.724 0.220 0.056
#> GSM228662 1 0.0237 0.7171 0.996 0.000 0.004
#> GSM228584 1 0.0237 0.7171 0.996 0.000 0.004
#> GSM228586 1 0.0237 0.7171 0.996 0.000 0.004
#> GSM228592 1 0.0000 0.7169 1.000 0.000 0.000
#> GSM228593 1 0.8779 0.0855 0.472 0.416 0.112
#> GSM228594 1 0.2096 0.7152 0.944 0.004 0.052
#> GSM228598 1 0.5731 0.6751 0.804 0.088 0.108
#> GSM228607 2 0.9731 0.0431 0.248 0.444 0.308
#> GSM228612 2 0.8979 -0.0281 0.128 0.452 0.420
#> GSM228619 1 0.9054 0.0456 0.496 0.144 0.360
#> GSM228622 1 0.5874 0.6168 0.760 0.032 0.208
#> GSM228625 1 0.9234 0.2559 0.524 0.280 0.196
#> GSM228631 1 0.7932 0.2206 0.552 0.064 0.384
#> GSM228633 2 0.4178 0.6097 0.000 0.828 0.172
#> GSM228637 2 0.8869 0.3983 0.160 0.560 0.280
#> GSM228639 3 0.8370 0.1300 0.084 0.416 0.500
#> GSM228649 2 0.8444 0.4056 0.236 0.612 0.152
#> GSM228660 1 0.9295 0.2037 0.524 0.252 0.224
#> GSM228661 1 0.3267 0.6960 0.884 0.000 0.116
#> GSM228595 2 0.1643 0.6011 0.000 0.956 0.044
#> GSM228599 2 0.8268 0.3607 0.096 0.576 0.328
#> GSM228602 3 0.8649 0.5748 0.232 0.172 0.596
#> GSM228614 2 0.8591 0.3920 0.128 0.572 0.300
#> GSM228626 2 0.2711 0.6051 0.000 0.912 0.088
#> GSM228640 3 0.6402 0.5977 0.236 0.040 0.724
#> GSM228643 3 0.8231 0.5825 0.156 0.208 0.636
#> GSM228650 3 0.7842 0.3833 0.072 0.328 0.600
#> GSM228653 3 0.5901 0.6111 0.176 0.048 0.776
#> GSM228657 2 0.3551 0.6113 0.000 0.868 0.132
#> GSM228605 1 0.8799 0.2833 0.556 0.144 0.300
#> GSM228610 3 0.7657 0.5648 0.116 0.208 0.676
#> GSM228617 3 0.7525 0.6007 0.228 0.096 0.676
#> GSM228620 3 0.7549 0.2247 0.436 0.040 0.524
#> GSM228623 2 0.6895 0.5752 0.064 0.708 0.228
#> GSM228629 3 0.8132 0.5374 0.304 0.096 0.600
#> GSM228632 3 0.8457 0.3361 0.100 0.356 0.544
#> GSM228635 2 0.4912 0.6021 0.008 0.796 0.196
#> GSM228647 3 0.6865 0.6168 0.160 0.104 0.736
#> GSM228596 3 0.9616 0.4118 0.296 0.236 0.468
#> GSM228600 3 0.8101 0.5330 0.132 0.228 0.640
#> GSM228603 3 0.6067 0.5955 0.236 0.028 0.736
#> GSM228615 2 0.8079 0.5101 0.108 0.624 0.268
#> GSM228627 3 0.8917 0.5013 0.188 0.244 0.568
#> GSM228641 3 0.6348 0.5528 0.060 0.188 0.752
#> GSM228644 2 0.3267 0.6083 0.000 0.884 0.116
#> GSM228651 3 0.8355 0.5626 0.184 0.188 0.628
#> GSM228654 3 0.8148 0.4652 0.100 0.296 0.604
#> GSM228658 3 0.7706 0.5876 0.264 0.088 0.648
#> GSM228606 3 0.7890 0.1868 0.060 0.396 0.544
#> GSM228611 3 0.8399 0.5720 0.188 0.188 0.624
#> GSM228618 3 0.7163 0.6088 0.144 0.136 0.720
#> GSM228621 3 0.5992 0.4438 0.016 0.268 0.716
#> GSM228624 3 0.9108 0.1171 0.140 0.416 0.444
#> GSM228630 3 0.6434 0.2550 0.008 0.380 0.612
#> GSM228636 2 0.4469 0.6149 0.028 0.852 0.120
#> GSM228638 3 0.7441 0.5952 0.136 0.164 0.700
#> GSM228648 3 0.5797 0.4288 0.008 0.280 0.712
#> GSM228670 2 0.9273 0.2686 0.236 0.528 0.236
#> GSM228671 2 0.6161 0.5268 0.016 0.696 0.288
#> GSM228672 1 0.9072 0.2561 0.532 0.300 0.168
#> GSM228674 2 0.9377 0.0667 0.380 0.448 0.172
#> GSM228675 2 0.8345 0.4672 0.172 0.628 0.200
#> GSM228676 3 0.9931 0.2941 0.308 0.300 0.392
#> GSM228667 2 0.9857 -0.0307 0.276 0.416 0.308
#> GSM228668 1 0.5692 0.6422 0.784 0.040 0.176
#> GSM228669 1 0.7287 0.5520 0.696 0.212 0.092
#> GSM228673 2 0.9302 -0.1407 0.160 0.420 0.420
#> GSM228677 2 0.6302 0.1672 0.000 0.520 0.480
#> GSM228678 2 0.5874 0.5949 0.032 0.760 0.208
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 1 0.9592 -0.33764 0.340 0.152 0.184 0.324
#> GSM228563 2 0.8384 0.21294 0.088 0.492 0.104 0.316
#> GSM228565 1 0.9746 -0.31018 0.348 0.228 0.164 0.260
#> GSM228566 3 0.9083 0.13150 0.096 0.184 0.428 0.292
#> GSM228567 1 0.0524 0.58566 0.988 0.000 0.004 0.008
#> GSM228570 1 0.7011 0.43940 0.668 0.052 0.124 0.156
#> GSM228571 1 0.6228 0.50179 0.712 0.028 0.096 0.164
#> GSM228574 3 0.8920 0.10486 0.052 0.264 0.372 0.312
#> GSM228575 2 0.8852 0.17141 0.060 0.424 0.224 0.292
#> GSM228576 1 0.9674 -0.24163 0.372 0.164 0.216 0.248
#> GSM228579 1 0.4033 0.57158 0.856 0.024 0.052 0.068
#> GSM228580 2 0.7930 0.34946 0.016 0.500 0.248 0.236
#> GSM228581 2 0.8776 0.15142 0.060 0.448 0.224 0.268
#> GSM228666 2 0.7624 0.32784 0.016 0.528 0.160 0.296
#> GSM228564 1 0.9783 -0.37144 0.328 0.248 0.160 0.264
#> GSM228568 4 0.9790 0.21364 0.284 0.196 0.188 0.332
#> GSM228569 1 0.6096 0.50466 0.724 0.024 0.120 0.132
#> GSM228572 2 0.6397 0.43656 0.000 0.648 0.144 0.208
#> GSM228573 3 0.8738 0.14916 0.212 0.084 0.496 0.208
#> GSM228577 1 0.4102 0.57161 0.840 0.012 0.040 0.108
#> GSM228578 1 0.7076 0.39213 0.620 0.016 0.164 0.200
#> GSM228663 3 0.9383 0.13087 0.136 0.176 0.404 0.284
#> GSM228664 3 0.8242 0.06581 0.016 0.372 0.376 0.236
#> GSM228665 3 0.8906 0.13976 0.204 0.096 0.480 0.220
#> GSM228582 2 0.9665 -0.28950 0.280 0.348 0.144 0.228
#> GSM228583 1 0.0657 0.58671 0.984 0.000 0.004 0.012
#> GSM228585 1 0.0895 0.58642 0.976 0.000 0.004 0.020
#> GSM228587 1 0.3877 0.57017 0.860 0.048 0.016 0.076
#> GSM228588 2 0.6763 0.32897 0.120 0.660 0.024 0.196
#> GSM228589 2 0.6245 0.42818 0.044 0.716 0.072 0.168
#> GSM228590 1 0.1209 0.58763 0.964 0.000 0.004 0.032
#> GSM228591 2 0.5664 0.44139 0.012 0.740 0.092 0.156
#> GSM228597 2 0.7522 0.35337 0.040 0.556 0.096 0.308
#> GSM228601 2 0.4186 0.44997 0.004 0.808 0.024 0.164
#> GSM228604 2 0.7686 0.11874 0.004 0.460 0.340 0.196
#> GSM228608 1 0.5711 0.52786 0.752 0.028 0.080 0.140
#> GSM228609 2 0.8327 0.06007 0.212 0.512 0.048 0.228
#> GSM228613 1 0.0707 0.58559 0.980 0.000 0.000 0.020
#> GSM228616 1 0.9929 -0.42613 0.300 0.264 0.208 0.228
#> GSM228628 2 0.7495 0.36032 0.028 0.592 0.168 0.212
#> GSM228634 1 0.1209 0.58707 0.964 0.000 0.004 0.032
#> GSM228642 2 0.5719 0.44775 0.000 0.716 0.132 0.152
#> GSM228645 2 0.9347 -0.02348 0.088 0.328 0.264 0.320
#> GSM228646 3 0.9413 0.01711 0.096 0.276 0.344 0.284
#> GSM228652 1 0.6593 0.46011 0.672 0.020 0.120 0.188
#> GSM228655 1 0.8788 0.18326 0.512 0.116 0.184 0.188
#> GSM228656 1 0.0592 0.58597 0.984 0.000 0.000 0.016
#> GSM228659 1 0.8149 0.15011 0.500 0.160 0.040 0.300
#> GSM228662 1 0.1229 0.58633 0.968 0.004 0.008 0.020
#> GSM228584 1 0.0469 0.58586 0.988 0.000 0.000 0.012
#> GSM228586 1 0.1004 0.58671 0.972 0.000 0.004 0.024
#> GSM228592 1 0.0927 0.58584 0.976 0.000 0.008 0.016
#> GSM228593 1 0.9313 -0.28554 0.368 0.300 0.092 0.240
#> GSM228594 1 0.3948 0.57034 0.852 0.008 0.068 0.072
#> GSM228598 1 0.7129 0.43419 0.656 0.084 0.072 0.188
#> GSM228607 4 0.9607 0.08875 0.144 0.236 0.244 0.376
#> GSM228612 2 0.9243 -0.04288 0.076 0.340 0.284 0.300
#> GSM228619 1 0.9431 -0.23338 0.352 0.120 0.332 0.196
#> GSM228622 1 0.7780 0.29518 0.572 0.044 0.240 0.144
#> GSM228625 1 0.9727 -0.38998 0.320 0.284 0.144 0.252
#> GSM228631 1 0.8715 -0.13402 0.388 0.056 0.372 0.184
#> GSM228633 2 0.5551 0.45943 0.000 0.728 0.112 0.160
#> GSM228637 2 0.8998 0.13024 0.100 0.428 0.156 0.316
#> GSM228639 3 0.8767 0.15081 0.044 0.268 0.400 0.288
#> GSM228649 2 0.8889 0.06808 0.140 0.460 0.108 0.292
#> GSM228660 1 0.9387 -0.11578 0.428 0.156 0.176 0.240
#> GSM228661 1 0.3818 0.55890 0.844 0.000 0.108 0.048
#> GSM228595 2 0.3056 0.46003 0.000 0.888 0.040 0.072
#> GSM228599 2 0.8860 0.14385 0.056 0.388 0.220 0.336
#> GSM228602 3 0.8142 0.23352 0.188 0.060 0.556 0.196
#> GSM228614 2 0.9151 0.00307 0.072 0.360 0.252 0.316
#> GSM228626 2 0.4581 0.45594 0.000 0.800 0.080 0.120
#> GSM228640 3 0.7192 0.23740 0.184 0.028 0.628 0.160
#> GSM228643 3 0.8934 0.20022 0.128 0.152 0.488 0.232
#> GSM228650 3 0.8589 0.24699 0.056 0.264 0.476 0.204
#> GSM228653 3 0.7793 0.27882 0.164 0.068 0.604 0.164
#> GSM228657 2 0.5476 0.45250 0.000 0.736 0.120 0.144
#> GSM228605 1 0.9506 -0.32540 0.340 0.116 0.228 0.316
#> GSM228610 3 0.8627 0.23386 0.100 0.172 0.524 0.204
#> GSM228617 3 0.8879 0.23525 0.148 0.156 0.508 0.188
#> GSM228620 3 0.8748 0.00310 0.308 0.048 0.412 0.232
#> GSM228623 2 0.8150 0.31359 0.040 0.488 0.152 0.320
#> GSM228629 3 0.8129 0.26510 0.152 0.068 0.560 0.220
#> GSM228632 3 0.8954 0.14515 0.052 0.296 0.360 0.292
#> GSM228635 2 0.7060 0.39362 0.008 0.564 0.120 0.308
#> GSM228647 3 0.8188 0.28254 0.152 0.084 0.568 0.196
#> GSM228596 3 0.9764 -0.08236 0.212 0.168 0.328 0.292
#> GSM228600 3 0.8371 0.26937 0.052 0.192 0.508 0.248
#> GSM228603 3 0.7715 0.25562 0.192 0.068 0.608 0.132
#> GSM228615 2 0.8314 0.25274 0.080 0.508 0.112 0.300
#> GSM228627 3 0.9374 0.08047 0.168 0.172 0.436 0.224
#> GSM228641 3 0.8418 0.30269 0.080 0.168 0.536 0.216
#> GSM228644 2 0.4901 0.45628 0.000 0.780 0.108 0.112
#> GSM228651 3 0.8882 0.23186 0.108 0.156 0.480 0.256
#> GSM228654 3 0.8312 0.28431 0.056 0.236 0.524 0.184
#> GSM228658 3 0.8843 0.20964 0.184 0.124 0.508 0.184
#> GSM228606 3 0.9028 0.07431 0.060 0.288 0.372 0.280
#> GSM228611 3 0.9052 0.17660 0.164 0.124 0.468 0.244
#> GSM228618 3 0.7901 0.29833 0.096 0.096 0.588 0.220
#> GSM228621 3 0.7643 0.27116 0.012 0.224 0.536 0.228
#> GSM228624 3 0.9127 0.05650 0.064 0.312 0.328 0.296
#> GSM228630 3 0.7605 0.18950 0.004 0.328 0.480 0.188
#> GSM228636 2 0.5566 0.44064 0.000 0.704 0.072 0.224
#> GSM228638 3 0.8885 0.25924 0.136 0.148 0.500 0.216
#> GSM228648 3 0.7211 0.33521 0.012 0.184 0.596 0.208
#> GSM228670 2 0.9252 0.03160 0.124 0.400 0.160 0.316
#> GSM228671 2 0.8013 0.25601 0.016 0.464 0.204 0.316
#> GSM228672 1 0.9386 -0.29363 0.364 0.212 0.108 0.316
#> GSM228674 2 0.9611 -0.30958 0.272 0.312 0.120 0.296
#> GSM228675 2 0.8769 0.13581 0.116 0.448 0.108 0.328
#> GSM228676 4 0.9964 0.16167 0.260 0.208 0.264 0.268
#> GSM228667 4 0.9507 0.07875 0.148 0.320 0.172 0.360
#> GSM228668 1 0.7266 0.40414 0.640 0.048 0.132 0.180
#> GSM228669 1 0.8696 0.15556 0.516 0.144 0.116 0.224
#> GSM228673 3 0.9275 0.09239 0.096 0.220 0.388 0.296
#> GSM228677 2 0.7798 0.17462 0.000 0.416 0.320 0.264
#> GSM228678 2 0.7520 0.38586 0.020 0.548 0.140 0.292
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 1 0.924 -0.25294 0.332 0.112 0.116 0.312 0.128
#> GSM228563 2 0.850 0.16039 0.052 0.396 0.080 0.328 0.144
#> GSM228565 4 0.952 0.11722 0.248 0.180 0.088 0.316 0.168
#> GSM228566 3 0.897 -0.02317 0.040 0.128 0.340 0.212 0.280
#> GSM228567 1 0.120 0.59316 0.960 0.000 0.012 0.028 0.000
#> GSM228570 1 0.749 0.36149 0.556 0.056 0.072 0.248 0.068
#> GSM228571 1 0.743 0.40148 0.560 0.016 0.124 0.208 0.092
#> GSM228574 5 0.914 0.10397 0.044 0.164 0.276 0.192 0.324
#> GSM228575 2 0.913 -0.04570 0.032 0.308 0.192 0.248 0.220
#> GSM228576 1 0.934 -0.11820 0.352 0.112 0.148 0.268 0.120
#> GSM228579 1 0.622 0.53059 0.688 0.024 0.060 0.148 0.080
#> GSM228580 2 0.743 0.31424 0.004 0.548 0.116 0.184 0.148
#> GSM228581 2 0.896 0.06796 0.052 0.372 0.128 0.172 0.276
#> GSM228666 2 0.812 0.20301 0.012 0.464 0.116 0.196 0.212
#> GSM228564 4 0.927 0.16060 0.228 0.208 0.092 0.364 0.108
#> GSM228568 1 0.976 -0.29281 0.276 0.152 0.128 0.200 0.244
#> GSM228569 1 0.711 0.45979 0.608 0.016 0.132 0.096 0.148
#> GSM228572 2 0.675 0.35686 0.000 0.608 0.084 0.152 0.156
#> GSM228573 3 0.795 0.18678 0.140 0.020 0.484 0.096 0.260
#> GSM228577 1 0.627 0.52703 0.680 0.020 0.056 0.148 0.096
#> GSM228578 1 0.796 0.34853 0.540 0.036 0.140 0.152 0.132
#> GSM228663 3 0.914 0.09133 0.136 0.136 0.388 0.088 0.252
#> GSM228664 2 0.788 -0.04074 0.004 0.372 0.212 0.068 0.344
#> GSM228665 3 0.875 0.13786 0.164 0.068 0.428 0.092 0.248
#> GSM228582 2 0.944 -0.14669 0.252 0.328 0.100 0.120 0.200
#> GSM228583 1 0.104 0.59069 0.964 0.000 0.004 0.032 0.000
#> GSM228585 1 0.181 0.59196 0.936 0.000 0.012 0.044 0.008
#> GSM228587 1 0.420 0.57175 0.820 0.032 0.020 0.104 0.024
#> GSM228588 2 0.672 0.31405 0.104 0.628 0.004 0.148 0.116
#> GSM228589 2 0.523 0.39390 0.012 0.744 0.020 0.116 0.108
#> GSM228590 1 0.281 0.59420 0.896 0.004 0.020 0.052 0.028
#> GSM228591 2 0.545 0.38613 0.004 0.724 0.036 0.100 0.136
#> GSM228597 2 0.808 0.25623 0.048 0.460 0.044 0.272 0.176
#> GSM228601 2 0.393 0.40165 0.000 0.808 0.008 0.132 0.052
#> GSM228604 2 0.754 0.17344 0.000 0.496 0.256 0.112 0.136
#> GSM228608 1 0.551 0.52990 0.732 0.012 0.064 0.140 0.052
#> GSM228609 2 0.854 0.04841 0.188 0.444 0.060 0.236 0.072
#> GSM228613 1 0.133 0.59038 0.956 0.000 0.004 0.032 0.008
#> GSM228616 2 0.988 -0.15749 0.180 0.264 0.156 0.228 0.172
#> GSM228628 2 0.737 0.29500 0.024 0.584 0.084 0.136 0.172
#> GSM228634 1 0.301 0.59363 0.884 0.000 0.048 0.032 0.036
#> GSM228642 2 0.611 0.35959 0.000 0.676 0.092 0.120 0.112
#> GSM228645 2 0.944 -0.04662 0.068 0.292 0.156 0.244 0.240
#> GSM228646 3 0.913 -0.07802 0.032 0.256 0.304 0.212 0.196
#> GSM228652 1 0.765 0.40908 0.576 0.044 0.088 0.140 0.152
#> GSM228655 1 0.890 0.08935 0.436 0.072 0.208 0.152 0.132
#> GSM228656 1 0.139 0.59183 0.956 0.000 0.008 0.024 0.012
#> GSM228659 1 0.854 -0.04477 0.404 0.136 0.044 0.312 0.104
#> GSM228662 1 0.115 0.59027 0.964 0.000 0.008 0.024 0.004
#> GSM228584 1 0.173 0.59349 0.940 0.000 0.012 0.040 0.008
#> GSM228586 1 0.162 0.59245 0.948 0.000 0.016 0.020 0.016
#> GSM228592 1 0.120 0.59110 0.964 0.000 0.008 0.016 0.012
#> GSM228593 1 0.919 -0.24489 0.340 0.232 0.048 0.216 0.164
#> GSM228594 1 0.474 0.57674 0.792 0.008 0.060 0.060 0.080
#> GSM228598 1 0.705 0.44147 0.616 0.040 0.048 0.124 0.172
#> GSM228607 5 0.959 0.12351 0.088 0.200 0.180 0.228 0.304
#> GSM228612 5 0.886 0.15157 0.044 0.272 0.192 0.116 0.376
#> GSM228619 3 0.911 -0.00262 0.296 0.092 0.356 0.160 0.096
#> GSM228622 1 0.738 0.37211 0.572 0.020 0.200 0.120 0.088
#> GSM228625 1 0.969 -0.33105 0.276 0.236 0.096 0.188 0.204
#> GSM228631 1 0.861 -0.11653 0.356 0.040 0.352 0.128 0.124
#> GSM228633 2 0.578 0.37600 0.000 0.692 0.052 0.104 0.152
#> GSM228637 2 0.927 0.06181 0.080 0.308 0.100 0.264 0.248
#> GSM228639 3 0.918 -0.02796 0.056 0.184 0.368 0.176 0.216
#> GSM228649 2 0.926 0.02216 0.116 0.348 0.084 0.264 0.188
#> GSM228660 1 0.958 -0.18881 0.336 0.184 0.132 0.128 0.220
#> GSM228661 1 0.490 0.56724 0.776 0.004 0.088 0.052 0.080
#> GSM228595 2 0.324 0.39591 0.000 0.864 0.012 0.048 0.076
#> GSM228599 2 0.915 0.03122 0.048 0.344 0.220 0.236 0.152
#> GSM228602 3 0.886 0.15808 0.116 0.092 0.456 0.164 0.172
#> GSM228614 4 0.925 -0.11499 0.048 0.244 0.156 0.300 0.252
#> GSM228626 2 0.429 0.39053 0.000 0.804 0.048 0.040 0.108
#> GSM228640 3 0.742 0.25989 0.096 0.044 0.600 0.104 0.156
#> GSM228643 3 0.903 0.15138 0.088 0.116 0.424 0.196 0.176
#> GSM228650 3 0.899 0.03210 0.056 0.220 0.400 0.188 0.136
#> GSM228653 3 0.674 0.24970 0.076 0.016 0.592 0.056 0.260
#> GSM228657 2 0.654 0.35681 0.000 0.632 0.108 0.096 0.164
#> GSM228605 1 0.920 -0.18566 0.332 0.064 0.152 0.292 0.160
#> GSM228610 3 0.878 0.11153 0.068 0.088 0.404 0.160 0.280
#> GSM228617 3 0.753 0.25376 0.100 0.056 0.600 0.120 0.124
#> GSM228620 3 0.907 0.09685 0.224 0.040 0.348 0.156 0.232
#> GSM228623 2 0.858 0.13946 0.016 0.392 0.140 0.236 0.216
#> GSM228629 3 0.783 0.19728 0.144 0.040 0.524 0.064 0.228
#> GSM228632 3 0.912 -0.09403 0.040 0.204 0.304 0.160 0.292
#> GSM228635 2 0.772 0.23589 0.008 0.452 0.060 0.284 0.196
#> GSM228647 3 0.836 0.19029 0.052 0.088 0.472 0.144 0.244
#> GSM228596 4 0.944 -0.04575 0.160 0.068 0.248 0.296 0.228
#> GSM228600 3 0.799 0.17438 0.044 0.152 0.544 0.128 0.132
#> GSM228603 3 0.740 0.25074 0.136 0.032 0.596 0.100 0.136
#> GSM228615 2 0.895 0.09488 0.064 0.348 0.116 0.324 0.148
#> GSM228627 3 0.920 -0.02424 0.064 0.184 0.340 0.136 0.276
#> GSM228641 3 0.807 0.17263 0.060 0.080 0.532 0.168 0.160
#> GSM228644 2 0.615 0.36569 0.000 0.668 0.076 0.116 0.140
#> GSM228651 3 0.901 0.06208 0.068 0.128 0.384 0.148 0.272
#> GSM228654 3 0.843 0.18631 0.056 0.176 0.464 0.076 0.228
#> GSM228658 3 0.786 0.24511 0.104 0.048 0.540 0.092 0.216
#> GSM228606 5 0.922 0.18486 0.040 0.196 0.224 0.228 0.312
#> GSM228611 3 0.833 0.10087 0.092 0.068 0.424 0.084 0.332
#> GSM228618 3 0.736 0.23881 0.048 0.084 0.608 0.112 0.148
#> GSM228621 3 0.839 0.00964 0.020 0.188 0.368 0.096 0.328
#> GSM228624 5 0.904 0.13595 0.048 0.224 0.268 0.120 0.340
#> GSM228630 3 0.834 -0.01283 0.004 0.256 0.380 0.132 0.228
#> GSM228636 2 0.734 0.30079 0.008 0.532 0.056 0.208 0.196
#> GSM228638 3 0.850 0.18505 0.084 0.140 0.480 0.080 0.216
#> GSM228648 3 0.749 0.08703 0.000 0.224 0.488 0.072 0.216
#> GSM228670 2 0.937 -0.04242 0.080 0.296 0.124 0.288 0.212
#> GSM228671 2 0.878 -0.02270 0.020 0.336 0.144 0.220 0.280
#> GSM228672 1 0.856 -0.17889 0.368 0.108 0.092 0.364 0.068
#> GSM228674 4 0.955 0.13856 0.216 0.228 0.084 0.304 0.168
#> GSM228675 2 0.912 0.01651 0.084 0.332 0.088 0.300 0.196
#> GSM228676 5 0.982 -0.08178 0.196 0.116 0.204 0.232 0.252
#> GSM228667 4 0.937 -0.06644 0.084 0.260 0.148 0.340 0.168
#> GSM228668 1 0.753 0.37889 0.568 0.020 0.140 0.164 0.108
#> GSM228669 1 0.827 0.20980 0.496 0.096 0.076 0.240 0.092
#> GSM228673 5 0.914 0.16350 0.040 0.176 0.224 0.224 0.336
#> GSM228677 2 0.861 -0.04808 0.004 0.312 0.172 0.248 0.264
#> GSM228678 2 0.844 0.22067 0.044 0.468 0.104 0.220 0.164
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 4 0.935 0.25415 0.228 0.068 0.120 0.292 0.200 0.092
#> GSM228563 2 0.834 0.00450 0.016 0.332 0.084 0.292 0.220 0.056
#> GSM228565 4 0.919 0.22296 0.212 0.136 0.080 0.356 0.088 0.128
#> GSM228566 3 0.911 0.02679 0.020 0.156 0.312 0.144 0.176 0.192
#> GSM228567 1 0.134 0.55619 0.956 0.000 0.012 0.016 0.008 0.008
#> GSM228570 1 0.798 0.06995 0.460 0.028 0.088 0.256 0.108 0.060
#> GSM228571 1 0.757 0.17463 0.488 0.032 0.144 0.244 0.024 0.068
#> GSM228574 6 0.944 0.03763 0.036 0.164 0.192 0.148 0.220 0.240
#> GSM228575 5 0.861 0.10328 0.008 0.268 0.088 0.164 0.332 0.140
#> GSM228576 1 0.916 -0.30848 0.304 0.064 0.152 0.280 0.108 0.092
#> GSM228579 1 0.616 0.41630 0.640 0.016 0.028 0.184 0.032 0.100
#> GSM228580 2 0.808 0.15559 0.012 0.480 0.104 0.136 0.152 0.116
#> GSM228581 2 0.896 -0.02595 0.016 0.312 0.124 0.132 0.180 0.236
#> GSM228666 2 0.798 0.05195 0.012 0.376 0.040 0.156 0.320 0.096
#> GSM228564 4 0.921 0.18626 0.176 0.112 0.120 0.336 0.204 0.052
#> GSM228568 1 0.986 -0.41751 0.216 0.164 0.100 0.192 0.172 0.156
#> GSM228569 1 0.726 0.36699 0.584 0.024 0.108 0.108 0.056 0.120
#> GSM228572 2 0.663 0.29193 0.000 0.616 0.072 0.120 0.116 0.076
#> GSM228573 3 0.849 0.11213 0.108 0.048 0.428 0.116 0.068 0.232
#> GSM228577 1 0.604 0.45211 0.660 0.012 0.064 0.172 0.028 0.064
#> GSM228578 1 0.796 0.17975 0.476 0.028 0.096 0.236 0.068 0.096
#> GSM228663 6 0.864 0.03657 0.052 0.104 0.292 0.104 0.076 0.372
#> GSM228664 6 0.818 0.07905 0.004 0.320 0.112 0.096 0.108 0.360
#> GSM228665 3 0.912 0.01884 0.160 0.080 0.312 0.076 0.096 0.276
#> GSM228582 2 0.972 -0.18255 0.220 0.232 0.100 0.176 0.100 0.172
#> GSM228583 1 0.165 0.55691 0.940 0.000 0.004 0.032 0.008 0.016
#> GSM228585 1 0.164 0.55466 0.940 0.000 0.008 0.036 0.008 0.008
#> GSM228587 1 0.487 0.47351 0.756 0.040 0.012 0.128 0.020 0.044
#> GSM228588 2 0.741 0.21378 0.112 0.536 0.016 0.204 0.076 0.056
#> GSM228589 2 0.655 0.30536 0.016 0.640 0.044 0.124 0.100 0.076
#> GSM228590 1 0.254 0.55091 0.896 0.000 0.020 0.048 0.004 0.032
#> GSM228591 2 0.607 0.31180 0.008 0.664 0.028 0.140 0.064 0.096
#> GSM228597 2 0.819 0.08498 0.032 0.400 0.048 0.196 0.260 0.064
#> GSM228601 2 0.527 0.32701 0.004 0.732 0.032 0.084 0.100 0.048
#> GSM228604 2 0.783 0.12470 0.000 0.452 0.224 0.132 0.068 0.124
#> GSM228608 1 0.629 0.41561 0.668 0.012 0.068 0.100 0.080 0.072
#> GSM228609 2 0.874 0.05916 0.156 0.384 0.036 0.228 0.116 0.080
#> GSM228613 1 0.128 0.55479 0.956 0.000 0.012 0.024 0.004 0.004
#> GSM228616 2 0.986 -0.23036 0.216 0.216 0.120 0.156 0.156 0.136
#> GSM228628 2 0.793 0.17814 0.012 0.488 0.072 0.136 0.120 0.172
#> GSM228634 1 0.284 0.55681 0.884 0.000 0.044 0.024 0.012 0.036
#> GSM228642 2 0.631 0.29231 0.000 0.640 0.040 0.100 0.108 0.112
#> GSM228645 2 0.941 -0.11066 0.040 0.252 0.176 0.192 0.220 0.120
#> GSM228646 6 0.931 -0.05939 0.020 0.220 0.172 0.176 0.180 0.232
#> GSM228652 1 0.788 0.12966 0.500 0.028 0.116 0.204 0.076 0.076
#> GSM228655 1 0.892 -0.17828 0.360 0.060 0.160 0.224 0.052 0.144
#> GSM228656 1 0.147 0.55542 0.948 0.000 0.004 0.024 0.020 0.004
#> GSM228659 1 0.826 -0.19384 0.384 0.052 0.044 0.316 0.132 0.072
#> GSM228662 1 0.112 0.55296 0.960 0.000 0.004 0.028 0.008 0.000
#> GSM228584 1 0.188 0.55838 0.928 0.000 0.020 0.028 0.000 0.024
#> GSM228586 1 0.230 0.55984 0.912 0.000 0.032 0.024 0.008 0.024
#> GSM228592 1 0.204 0.55774 0.924 0.000 0.012 0.028 0.008 0.028
#> GSM228593 4 0.893 0.15727 0.240 0.248 0.024 0.280 0.128 0.080
#> GSM228594 1 0.444 0.53726 0.796 0.004 0.048 0.052 0.032 0.068
#> GSM228598 1 0.650 0.40104 0.644 0.024 0.032 0.132 0.096 0.072
#> GSM228607 5 0.941 0.00779 0.064 0.148 0.092 0.188 0.276 0.232
#> GSM228612 6 0.934 0.09276 0.048 0.220 0.172 0.092 0.192 0.276
#> GSM228619 3 0.874 -0.00178 0.292 0.036 0.340 0.152 0.084 0.096
#> GSM228622 1 0.750 0.23160 0.512 0.004 0.212 0.104 0.076 0.092
#> GSM228625 1 0.963 -0.34903 0.264 0.120 0.104 0.232 0.168 0.112
#> GSM228631 3 0.797 -0.04762 0.356 0.012 0.364 0.120 0.068 0.080
#> GSM228633 2 0.615 0.30091 0.000 0.640 0.032 0.076 0.164 0.088
#> GSM228637 2 0.916 -0.02336 0.064 0.312 0.056 0.200 0.212 0.156
#> GSM228639 6 0.919 0.02587 0.024 0.228 0.200 0.104 0.188 0.256
#> GSM228649 2 0.917 -0.00709 0.108 0.308 0.044 0.240 0.188 0.112
#> GSM228660 1 0.956 -0.27668 0.304 0.176 0.136 0.140 0.080 0.164
#> GSM228661 1 0.521 0.48187 0.708 0.000 0.112 0.048 0.008 0.124
#> GSM228595 2 0.349 0.33451 0.000 0.848 0.028 0.028 0.064 0.032
#> GSM228599 2 0.916 -0.06440 0.024 0.260 0.232 0.212 0.172 0.100
#> GSM228602 3 0.853 0.15967 0.064 0.076 0.456 0.160 0.092 0.152
#> GSM228614 5 0.930 0.06977 0.024 0.232 0.156 0.148 0.236 0.204
#> GSM228626 2 0.517 0.31724 0.000 0.728 0.044 0.032 0.124 0.072
#> GSM228640 3 0.679 0.19199 0.080 0.040 0.632 0.104 0.036 0.108
#> GSM228643 3 0.933 0.06266 0.072 0.116 0.300 0.184 0.092 0.236
#> GSM228650 3 0.929 0.02024 0.052 0.164 0.324 0.120 0.136 0.204
#> GSM228653 3 0.745 0.12584 0.084 0.028 0.440 0.056 0.044 0.348
#> GSM228657 2 0.669 0.29772 0.004 0.616 0.060 0.084 0.136 0.100
#> GSM228605 1 0.931 -0.28247 0.308 0.052 0.128 0.120 0.224 0.168
#> GSM228610 3 0.849 0.02180 0.044 0.068 0.388 0.064 0.180 0.256
#> GSM228617 3 0.762 0.19733 0.096 0.044 0.564 0.088 0.084 0.124
#> GSM228620 6 0.874 -0.10422 0.156 0.016 0.288 0.080 0.152 0.308
#> GSM228623 5 0.772 0.01709 0.004 0.340 0.044 0.104 0.388 0.120
#> GSM228629 3 0.825 0.10887 0.076 0.052 0.420 0.120 0.052 0.280
#> GSM228632 6 0.912 0.09892 0.044 0.212 0.168 0.084 0.160 0.332
#> GSM228635 2 0.778 0.08538 0.000 0.408 0.056 0.136 0.296 0.104
#> GSM228647 3 0.846 0.10504 0.084 0.080 0.396 0.068 0.076 0.296
#> GSM228596 5 0.961 0.00114 0.112 0.084 0.152 0.164 0.272 0.216
#> GSM228600 3 0.769 0.15240 0.020 0.084 0.540 0.124 0.124 0.108
#> GSM228603 3 0.788 0.19020 0.136 0.036 0.524 0.128 0.060 0.116
#> GSM228615 2 0.887 0.01337 0.036 0.324 0.084 0.216 0.248 0.092
#> GSM228627 3 0.911 -0.07648 0.032 0.168 0.296 0.108 0.136 0.260
#> GSM228641 3 0.809 0.12127 0.036 0.104 0.508 0.140 0.104 0.108
#> GSM228644 2 0.559 0.32487 0.000 0.708 0.060 0.056 0.092 0.084
#> GSM228651 3 0.879 0.03617 0.040 0.096 0.368 0.096 0.144 0.256
#> GSM228654 3 0.885 0.08881 0.052 0.100 0.368 0.096 0.124 0.260
#> GSM228658 3 0.871 0.09472 0.128 0.076 0.372 0.052 0.096 0.276
#> GSM228606 5 0.910 0.00798 0.032 0.188 0.176 0.088 0.316 0.200
#> GSM228611 6 0.907 0.00585 0.088 0.060 0.264 0.084 0.196 0.308
#> GSM228618 3 0.775 0.15207 0.052 0.036 0.516 0.068 0.148 0.180
#> GSM228621 3 0.843 -0.01683 0.012 0.148 0.372 0.052 0.200 0.216
#> GSM228624 6 0.926 0.08877 0.032 0.184 0.200 0.124 0.164 0.296
#> GSM228630 3 0.856 -0.10042 0.008 0.240 0.280 0.060 0.136 0.276
#> GSM228636 2 0.774 0.17836 0.012 0.472 0.056 0.136 0.244 0.080
#> GSM228638 6 0.857 -0.04294 0.036 0.112 0.328 0.088 0.096 0.340
#> GSM228648 3 0.817 -0.06261 0.008 0.224 0.324 0.052 0.084 0.308
#> GSM228670 5 0.904 0.13816 0.076 0.268 0.060 0.148 0.328 0.120
#> GSM228671 5 0.857 0.11308 0.016 0.304 0.116 0.108 0.340 0.116
#> GSM228672 4 0.864 0.28014 0.276 0.088 0.040 0.340 0.200 0.056
#> GSM228674 5 0.912 0.06499 0.124 0.204 0.068 0.152 0.364 0.088
#> GSM228675 5 0.774 0.19326 0.068 0.236 0.056 0.104 0.500 0.036
#> GSM228676 5 0.953 0.01577 0.140 0.108 0.184 0.108 0.320 0.140
#> GSM228667 5 0.928 0.12410 0.064 0.208 0.088 0.224 0.300 0.116
#> GSM228668 1 0.784 0.25314 0.524 0.032 0.104 0.164 0.104 0.072
#> GSM228669 1 0.871 -0.15291 0.392 0.072 0.056 0.208 0.196 0.076
#> GSM228673 6 0.929 0.04022 0.040 0.164 0.140 0.128 0.240 0.288
#> GSM228677 2 0.881 -0.05343 0.008 0.316 0.168 0.128 0.248 0.132
#> GSM228678 2 0.832 0.10705 0.016 0.396 0.088 0.212 0.220 0.068
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
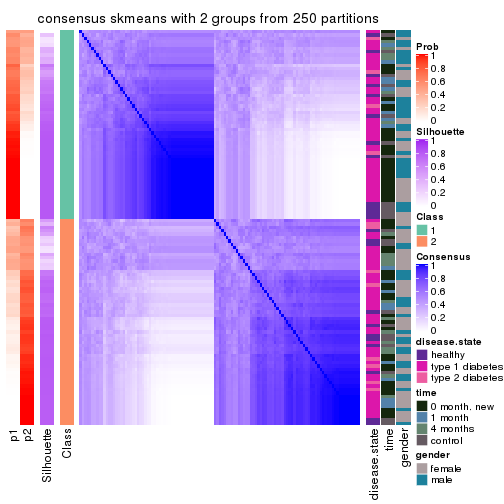
consensus_heatmap(res, k = 3)
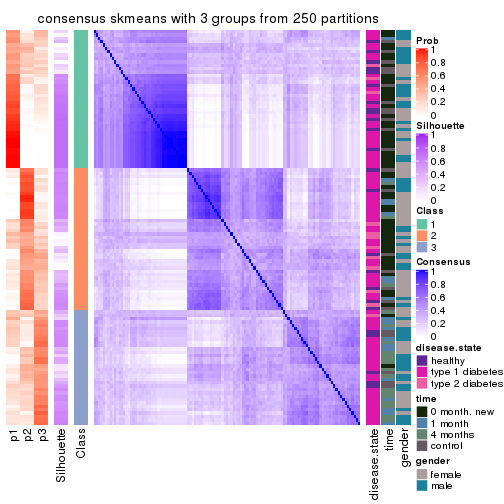
consensus_heatmap(res, k = 4)
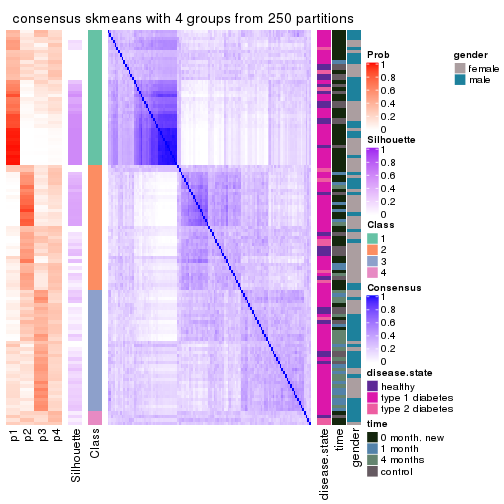
consensus_heatmap(res, k = 5)
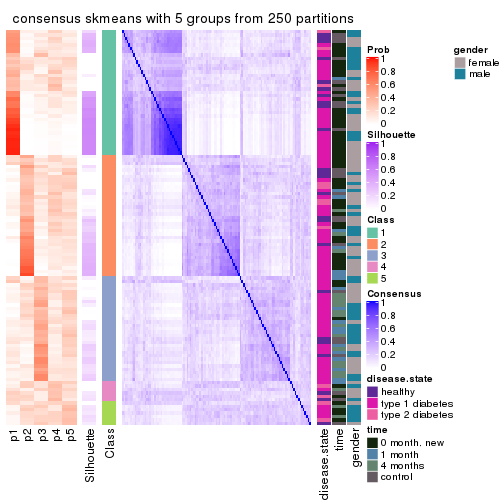
consensus_heatmap(res, k = 6)
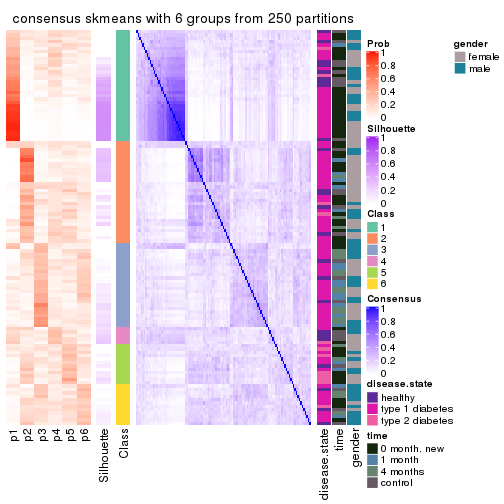
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
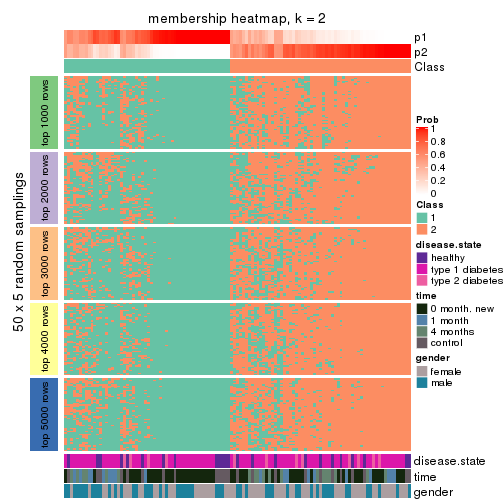
membership_heatmap(res, k = 3)
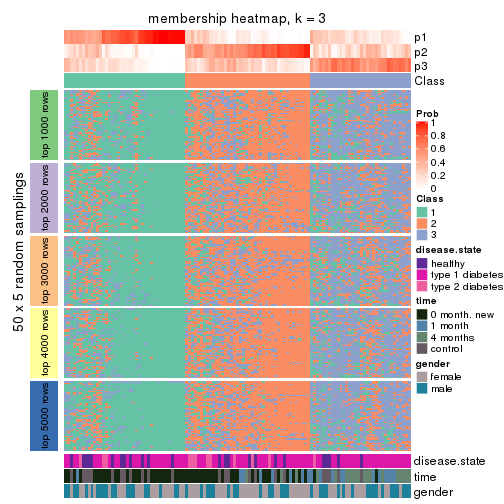
membership_heatmap(res, k = 4)
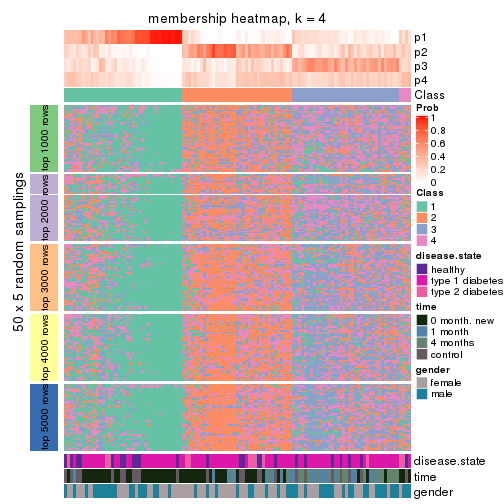
membership_heatmap(res, k = 5)
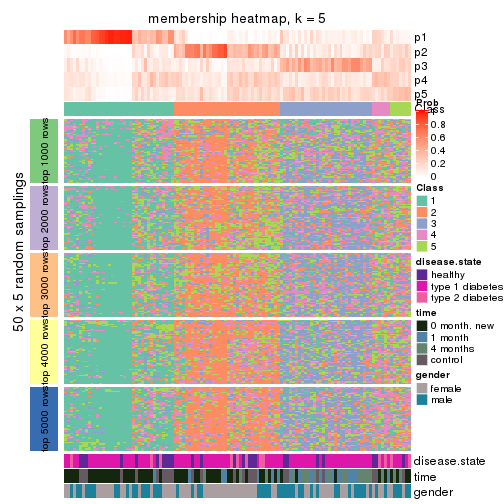
membership_heatmap(res, k = 6)
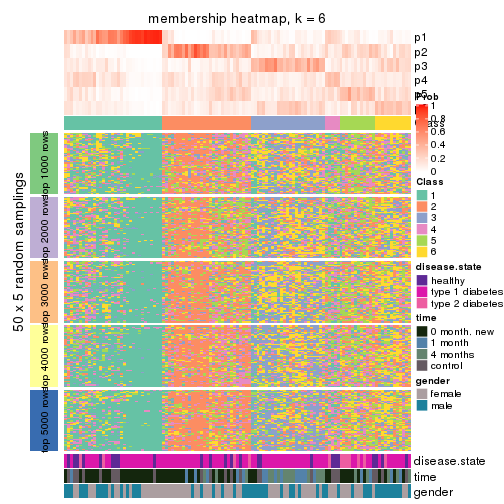
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
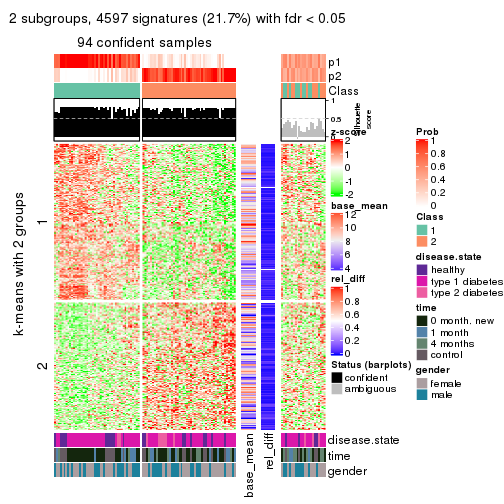
get_signatures(res, k = 3)
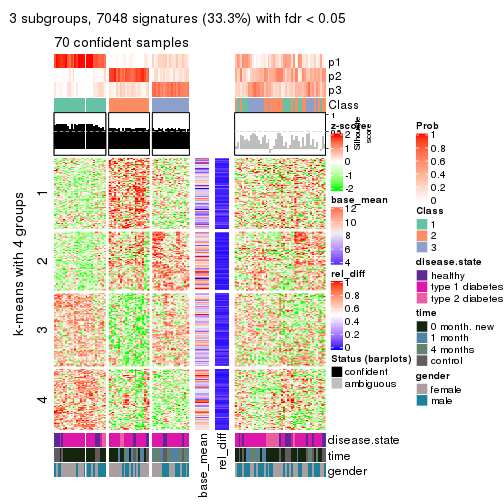
get_signatures(res, k = 4)

get_signatures(res, k = 5)

get_signatures(res, k = 6)

Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
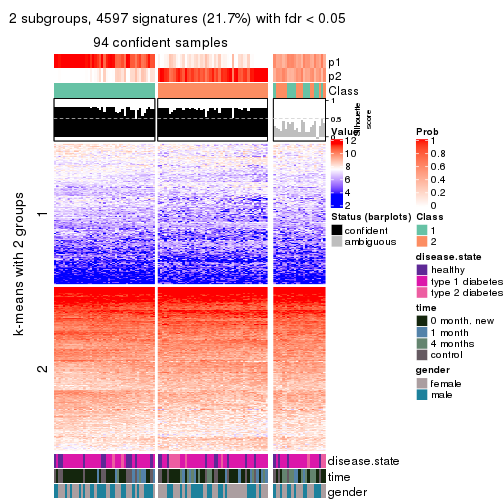
get_signatures(res, k = 3, scale_rows = FALSE)
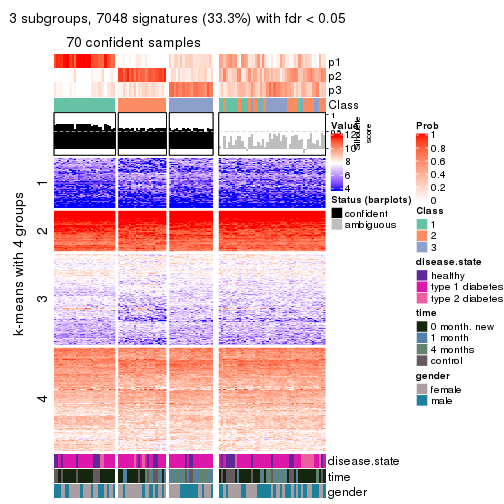
get_signatures(res, k = 4, scale_rows = FALSE)

get_signatures(res, k = 5, scale_rows = FALSE)

get_signatures(res, k = 6, scale_rows = FALSE)

Compare the overlap of signatures from different k:
compare_signatures(res)
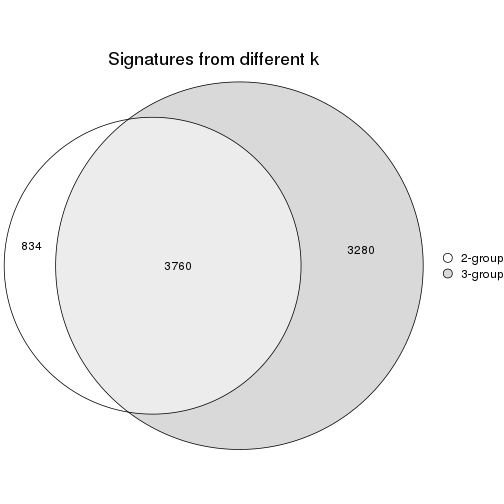
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
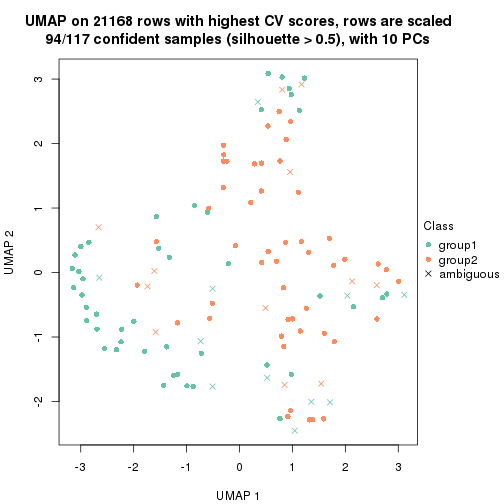
dimension_reduction(res, k = 3, method = "UMAP")
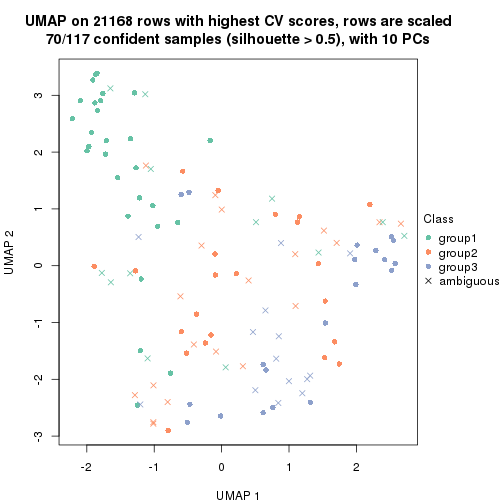
dimension_reduction(res, k = 4, method = "UMAP")
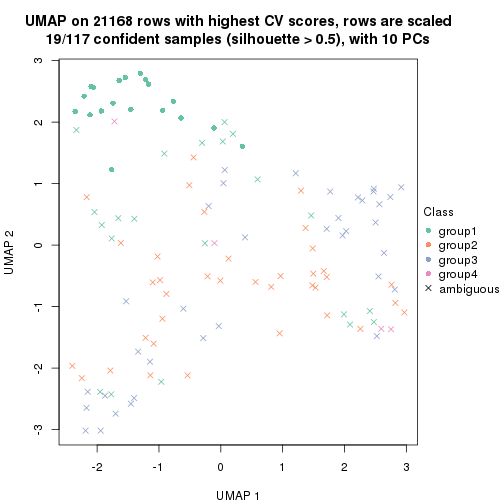
dimension_reduction(res, k = 5, method = "UMAP")
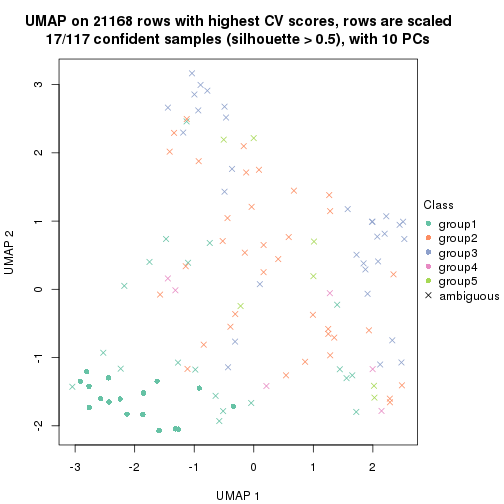
dimension_reduction(res, k = 6, method = "UMAP")
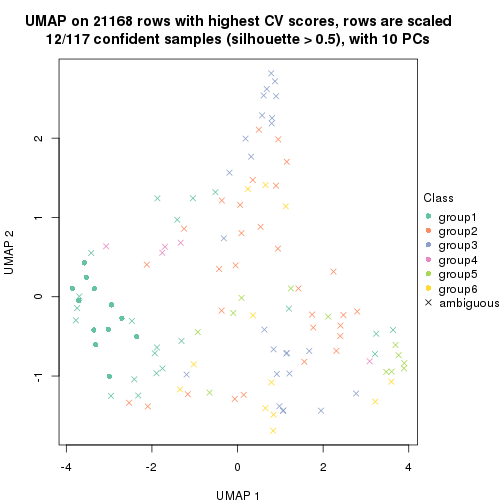
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
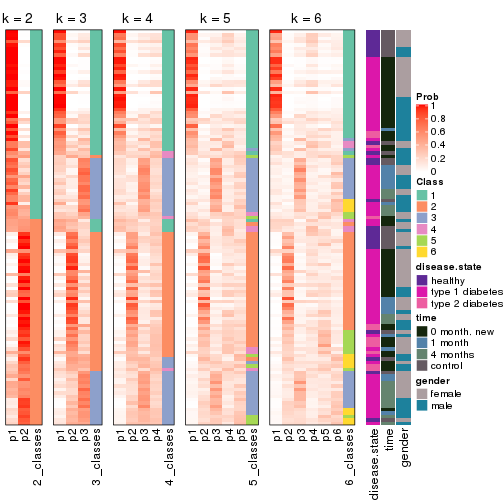
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> CV:skmeans 94 0.243 2.65e-01 0.563 2
#> CV:skmeans 70 0.594 4.93e-07 0.299 3
#> CV:skmeans 19 NA NA NA 4
#> CV:skmeans 17 NA NA NA 5
#> CV:skmeans 12 NA NA NA 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["CV", "pam"]
# you can also extract it by
# res = res_list["CV:pam"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'CV' method.
#> Subgroups are detected by 'pam' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
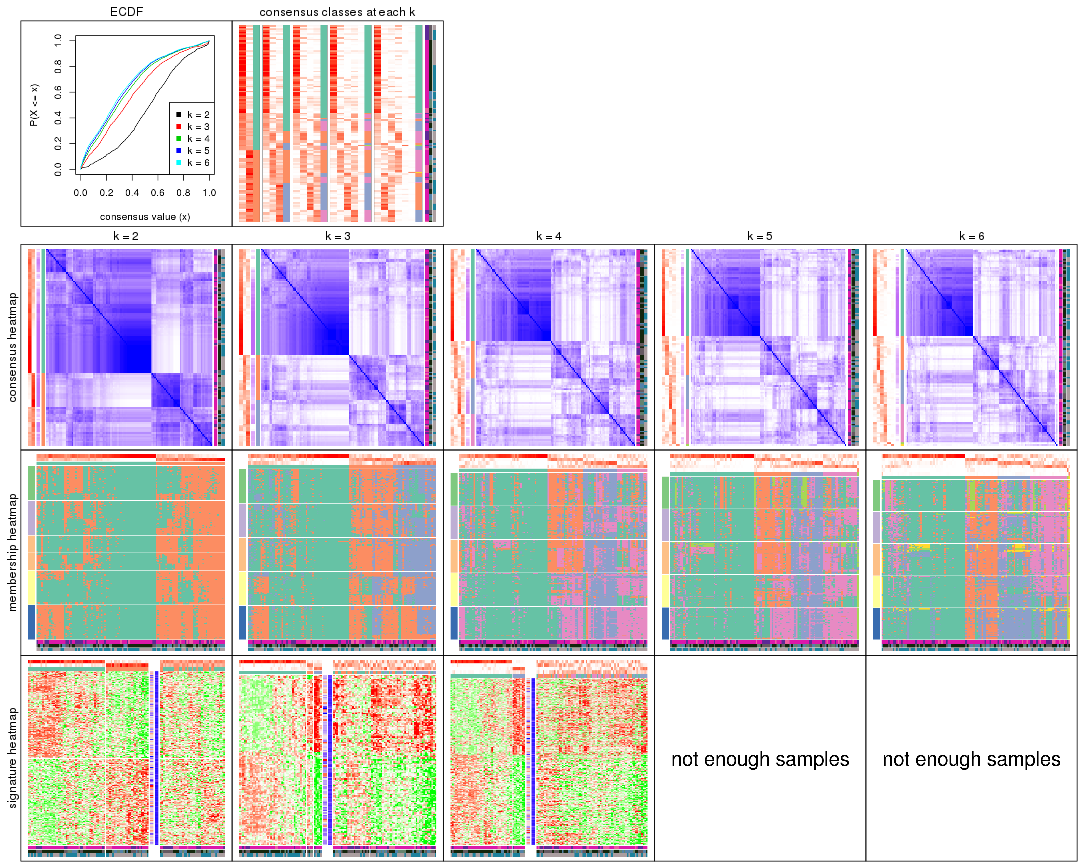
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
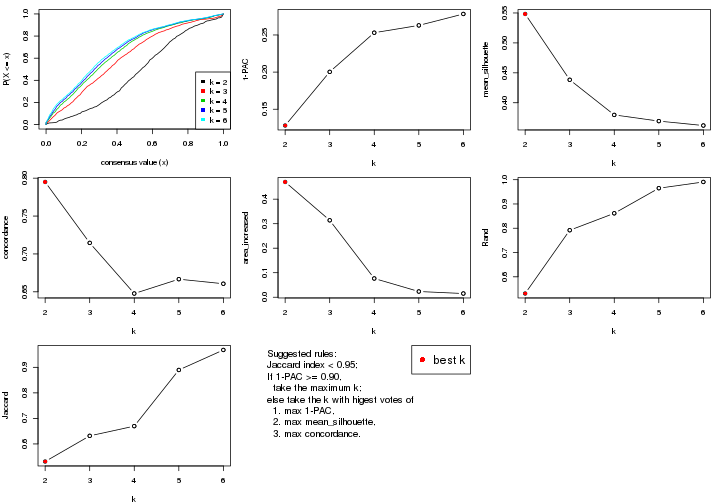
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.128 0.548 0.795 0.4703 0.531 0.531
#> 3 3 0.200 0.438 0.715 0.3139 0.792 0.632
#> 4 4 0.253 0.380 0.648 0.0765 0.861 0.670
#> 5 5 0.263 0.369 0.667 0.0233 0.965 0.890
#> 6 6 0.278 0.362 0.661 0.0151 0.991 0.968
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.9866 0.2391 0.568 0.432
#> GSM228563 1 0.8608 0.5539 0.716 0.284
#> GSM228565 1 0.9209 0.4368 0.664 0.336
#> GSM228566 2 0.4939 0.6799 0.108 0.892
#> GSM228567 1 0.0376 0.7344 0.996 0.004
#> GSM228570 1 0.9427 0.4073 0.640 0.360
#> GSM228571 1 1.0000 0.0909 0.504 0.496
#> GSM228574 2 0.5408 0.6848 0.124 0.876
#> GSM228575 1 0.9963 0.0980 0.536 0.464
#> GSM228576 1 0.9000 0.4668 0.684 0.316
#> GSM228579 1 0.8955 0.4691 0.688 0.312
#> GSM228580 1 0.9393 0.2934 0.644 0.356
#> GSM228581 1 0.9661 0.3576 0.608 0.392
#> GSM228666 2 0.9998 0.0896 0.492 0.508
#> GSM228564 1 0.4815 0.7145 0.896 0.104
#> GSM228568 1 0.8443 0.5325 0.728 0.272
#> GSM228569 1 0.0000 0.7345 1.000 0.000
#> GSM228572 2 0.3274 0.6908 0.060 0.940
#> GSM228573 1 0.7056 0.6591 0.808 0.192
#> GSM228577 1 0.0000 0.7345 1.000 0.000
#> GSM228578 1 0.7883 0.5972 0.764 0.236
#> GSM228663 2 0.7056 0.6605 0.192 0.808
#> GSM228664 2 0.7139 0.6535 0.196 0.804
#> GSM228665 1 0.8081 0.5434 0.752 0.248
#> GSM228582 1 0.8443 0.5271 0.728 0.272
#> GSM228583 1 0.0000 0.7345 1.000 0.000
#> GSM228585 1 0.7139 0.6228 0.804 0.196
#> GSM228587 1 0.0000 0.7345 1.000 0.000
#> GSM228588 1 0.0000 0.7345 1.000 0.000
#> GSM228589 1 0.0938 0.7339 0.988 0.012
#> GSM228590 1 0.0000 0.7345 1.000 0.000
#> GSM228591 1 0.7528 0.6335 0.784 0.216
#> GSM228597 1 0.2423 0.7301 0.960 0.040
#> GSM228601 1 0.9795 0.3195 0.584 0.416
#> GSM228604 2 0.6343 0.6098 0.160 0.840
#> GSM228608 1 0.0000 0.7345 1.000 0.000
#> GSM228609 1 0.6531 0.6440 0.832 0.168
#> GSM228613 1 0.0000 0.7345 1.000 0.000
#> GSM228616 1 0.4431 0.7159 0.908 0.092
#> GSM228628 2 0.3431 0.6852 0.064 0.936
#> GSM228634 1 0.0000 0.7345 1.000 0.000
#> GSM228642 2 0.4431 0.6774 0.092 0.908
#> GSM228645 1 0.9815 0.2384 0.580 0.420
#> GSM228646 1 0.9248 0.4390 0.660 0.340
#> GSM228652 2 0.9963 0.0929 0.464 0.536
#> GSM228655 1 0.8386 0.4790 0.732 0.268
#> GSM228656 1 0.0000 0.7345 1.000 0.000
#> GSM228659 1 0.8813 0.4535 0.700 0.300
#> GSM228662 1 0.2423 0.7289 0.960 0.040
#> GSM228584 1 0.0000 0.7345 1.000 0.000
#> GSM228586 1 0.0000 0.7345 1.000 0.000
#> GSM228592 1 0.0000 0.7345 1.000 0.000
#> GSM228593 1 0.5946 0.6655 0.856 0.144
#> GSM228594 1 0.0000 0.7345 1.000 0.000
#> GSM228598 1 0.0000 0.7345 1.000 0.000
#> GSM228607 1 0.4939 0.6972 0.892 0.108
#> GSM228612 1 0.9000 0.4871 0.684 0.316
#> GSM228619 1 0.5737 0.6635 0.864 0.136
#> GSM228622 1 0.2423 0.7328 0.960 0.040
#> GSM228625 1 0.2236 0.7301 0.964 0.036
#> GSM228631 1 0.4939 0.6936 0.892 0.108
#> GSM228633 2 0.3431 0.6899 0.064 0.936
#> GSM228637 2 1.0000 0.2299 0.496 0.504
#> GSM228639 2 0.9795 0.4222 0.416 0.584
#> GSM228649 1 0.9044 0.3970 0.680 0.320
#> GSM228660 1 0.3114 0.7263 0.944 0.056
#> GSM228661 1 0.0000 0.7345 1.000 0.000
#> GSM228595 2 0.1414 0.6777 0.020 0.980
#> GSM228599 1 0.9866 0.1754 0.568 0.432
#> GSM228602 2 0.0376 0.6693 0.004 0.996
#> GSM228614 2 0.7883 0.6326 0.236 0.764
#> GSM228626 2 0.0376 0.6683 0.004 0.996
#> GSM228640 1 0.9833 0.2656 0.576 0.424
#> GSM228643 2 0.0000 0.6669 0.000 1.000
#> GSM228650 2 0.9358 0.5260 0.352 0.648
#> GSM228653 2 0.8955 0.5840 0.312 0.688
#> GSM228657 2 0.9323 0.5306 0.348 0.652
#> GSM228605 1 0.0672 0.7349 0.992 0.008
#> GSM228610 1 0.9866 0.1369 0.568 0.432
#> GSM228617 1 0.9248 0.3231 0.660 0.340
#> GSM228620 1 0.0376 0.7346 0.996 0.004
#> GSM228623 1 0.9983 -0.1553 0.524 0.476
#> GSM228629 1 0.9087 0.4005 0.676 0.324
#> GSM228632 2 0.9896 0.2940 0.440 0.560
#> GSM228635 1 0.7528 0.6270 0.784 0.216
#> GSM228647 1 0.8955 0.3913 0.688 0.312
#> GSM228596 1 0.8763 0.4368 0.704 0.296
#> GSM228600 2 0.8763 0.4837 0.296 0.704
#> GSM228603 2 0.9933 0.0699 0.452 0.548
#> GSM228615 1 0.3879 0.7261 0.924 0.076
#> GSM228627 2 0.5842 0.6598 0.140 0.860
#> GSM228641 2 0.7219 0.5595 0.200 0.800
#> GSM228644 2 0.4562 0.6872 0.096 0.904
#> GSM228651 2 0.9815 0.4118 0.420 0.580
#> GSM228654 2 0.8813 0.5914 0.300 0.700
#> GSM228658 2 0.6801 0.6775 0.180 0.820
#> GSM228606 1 0.7674 0.6068 0.776 0.224
#> GSM228611 1 0.4815 0.7077 0.896 0.104
#> GSM228618 1 0.7528 0.6075 0.784 0.216
#> GSM228621 2 0.8713 0.4660 0.292 0.708
#> GSM228624 1 0.6623 0.6804 0.828 0.172
#> GSM228630 2 0.9866 0.3967 0.432 0.568
#> GSM228636 2 0.9608 0.4838 0.384 0.616
#> GSM228638 2 0.9866 0.3875 0.432 0.568
#> GSM228648 2 0.0672 0.6708 0.008 0.992
#> GSM228670 2 0.9044 0.5805 0.320 0.680
#> GSM228671 2 0.3584 0.6917 0.068 0.932
#> GSM228672 1 0.6531 0.6840 0.832 0.168
#> GSM228674 1 0.9977 0.0842 0.528 0.472
#> GSM228675 2 0.9522 0.3464 0.372 0.628
#> GSM228676 2 0.9795 0.2162 0.416 0.584
#> GSM228667 2 0.9815 0.3483 0.420 0.580
#> GSM228668 1 0.0938 0.7349 0.988 0.012
#> GSM228669 1 0.0000 0.7345 1.000 0.000
#> GSM228673 2 0.9795 0.2934 0.416 0.584
#> GSM228677 2 0.3879 0.6923 0.076 0.924
#> GSM228678 2 0.3431 0.6832 0.064 0.936
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 2 0.9963 0.28805 0.348 0.360 0.292
#> GSM228563 1 0.7464 0.18994 0.560 0.400 0.040
#> GSM228565 1 0.9847 -0.24076 0.404 0.340 0.256
#> GSM228566 3 0.6931 -0.36400 0.016 0.456 0.528
#> GSM228567 1 0.0592 0.73199 0.988 0.012 0.000
#> GSM228570 2 0.7310 0.44468 0.324 0.628 0.048
#> GSM228571 2 0.7256 0.51609 0.216 0.696 0.088
#> GSM228574 2 0.7634 0.41539 0.044 0.524 0.432
#> GSM228575 1 0.9693 -0.02073 0.456 0.292 0.252
#> GSM228576 2 0.6111 0.32543 0.396 0.604 0.000
#> GSM228579 2 0.6008 0.35909 0.372 0.628 0.000
#> GSM228580 3 0.7487 0.22841 0.408 0.040 0.552
#> GSM228581 1 0.9299 0.08157 0.496 0.324 0.180
#> GSM228666 3 0.8984 0.04073 0.368 0.136 0.496
#> GSM228564 1 0.4121 0.70403 0.876 0.040 0.084
#> GSM228568 1 0.6299 0.00179 0.524 0.476 0.000
#> GSM228569 1 0.0424 0.73137 0.992 0.008 0.000
#> GSM228572 3 0.6771 0.29960 0.012 0.440 0.548
#> GSM228573 1 0.6567 0.62619 0.752 0.088 0.160
#> GSM228577 1 0.0592 0.73195 0.988 0.012 0.000
#> GSM228578 1 0.6985 0.25375 0.592 0.384 0.024
#> GSM228663 3 0.2443 0.44147 0.028 0.032 0.940
#> GSM228664 3 0.7091 0.50669 0.064 0.248 0.688
#> GSM228665 1 0.5929 0.49158 0.676 0.004 0.320
#> GSM228582 1 0.7422 0.40493 0.608 0.048 0.344
#> GSM228583 1 0.0424 0.73137 0.992 0.008 0.000
#> GSM228585 1 0.4931 0.56453 0.768 0.232 0.000
#> GSM228587 1 0.0592 0.73245 0.988 0.000 0.012
#> GSM228588 1 0.0000 0.73089 1.000 0.000 0.000
#> GSM228589 1 0.1315 0.73239 0.972 0.008 0.020
#> GSM228590 1 0.0000 0.73089 1.000 0.000 0.000
#> GSM228591 1 0.6936 0.53368 0.704 0.232 0.064
#> GSM228597 1 0.3083 0.72340 0.916 0.024 0.060
#> GSM228601 2 0.9335 0.43375 0.324 0.492 0.184
#> GSM228604 2 0.4810 0.32814 0.028 0.832 0.140
#> GSM228608 1 0.0000 0.73089 1.000 0.000 0.000
#> GSM228609 1 0.4750 0.58611 0.784 0.216 0.000
#> GSM228613 1 0.0000 0.73089 1.000 0.000 0.000
#> GSM228616 1 0.3686 0.68386 0.860 0.140 0.000
#> GSM228628 2 0.6252 0.49003 0.008 0.648 0.344
#> GSM228634 1 0.0424 0.73137 0.992 0.008 0.000
#> GSM228642 2 0.5402 0.35020 0.028 0.792 0.180
#> GSM228645 2 0.9872 0.32665 0.336 0.400 0.264
#> GSM228646 1 0.8784 0.22370 0.512 0.120 0.368
#> GSM228652 3 0.9008 -0.00785 0.360 0.140 0.500
#> GSM228655 1 0.6600 0.33439 0.604 0.012 0.384
#> GSM228656 1 0.1015 0.73246 0.980 0.008 0.012
#> GSM228659 1 0.7420 0.24889 0.544 0.036 0.420
#> GSM228662 1 0.1643 0.72503 0.956 0.044 0.000
#> GSM228584 1 0.0237 0.73134 0.996 0.004 0.000
#> GSM228586 1 0.0424 0.73137 0.992 0.008 0.000
#> GSM228592 1 0.0000 0.73089 1.000 0.000 0.000
#> GSM228593 1 0.4755 0.63311 0.808 0.008 0.184
#> GSM228594 1 0.0000 0.73089 1.000 0.000 0.000
#> GSM228598 1 0.0475 0.73227 0.992 0.004 0.004
#> GSM228607 1 0.4277 0.68239 0.852 0.016 0.132
#> GSM228612 1 0.8395 0.19492 0.548 0.356 0.096
#> GSM228619 1 0.5597 0.58260 0.764 0.020 0.216
#> GSM228622 1 0.2774 0.72496 0.920 0.008 0.072
#> GSM228625 1 0.1411 0.72852 0.964 0.000 0.036
#> GSM228631 1 0.5235 0.64212 0.812 0.036 0.152
#> GSM228633 3 0.6521 0.30373 0.004 0.496 0.500
#> GSM228637 3 0.6287 0.40529 0.272 0.024 0.704
#> GSM228639 3 0.8007 0.51340 0.116 0.244 0.640
#> GSM228649 1 0.9014 0.10953 0.484 0.136 0.380
#> GSM228660 1 0.3879 0.66658 0.848 0.000 0.152
#> GSM228661 1 0.0424 0.73137 0.992 0.008 0.000
#> GSM228595 3 0.6180 0.32386 0.000 0.416 0.584
#> GSM228599 1 0.9683 -0.10976 0.416 0.216 0.368
#> GSM228602 2 0.5859 0.43320 0.000 0.656 0.344
#> GSM228614 3 0.6586 0.51088 0.056 0.216 0.728
#> GSM228626 2 0.6274 -0.26105 0.000 0.544 0.456
#> GSM228640 1 0.7657 0.03296 0.508 0.448 0.044
#> GSM228643 2 0.5859 0.45843 0.000 0.656 0.344
#> GSM228650 3 0.4689 0.41905 0.096 0.052 0.852
#> GSM228653 3 0.7542 0.42052 0.192 0.120 0.688
#> GSM228657 3 0.6742 0.50119 0.052 0.240 0.708
#> GSM228605 1 0.0592 0.73226 0.988 0.000 0.012
#> GSM228610 3 0.8403 0.21977 0.400 0.088 0.512
#> GSM228617 1 0.9465 -0.18457 0.444 0.184 0.372
#> GSM228620 1 0.0424 0.73291 0.992 0.000 0.008
#> GSM228623 3 0.6195 0.38565 0.276 0.020 0.704
#> GSM228629 1 0.7858 0.30128 0.572 0.064 0.364
#> GSM228632 2 0.9690 0.37804 0.220 0.424 0.356
#> GSM228635 1 0.7319 0.56688 0.708 0.164 0.128
#> GSM228647 1 0.8185 -0.02036 0.500 0.072 0.428
#> GSM228596 1 0.6421 0.27876 0.572 0.004 0.424
#> GSM228600 2 0.9322 0.37745 0.164 0.444 0.392
#> GSM228603 2 0.8396 0.51750 0.196 0.624 0.180
#> GSM228615 1 0.4865 0.68593 0.832 0.032 0.136
#> GSM228627 2 0.7620 0.50691 0.056 0.596 0.348
#> GSM228641 2 0.7262 -0.03459 0.044 0.624 0.332
#> GSM228644 3 0.6726 0.45865 0.024 0.332 0.644
#> GSM228651 3 0.6599 0.44135 0.168 0.084 0.748
#> GSM228654 3 0.8153 0.44455 0.216 0.144 0.640
#> GSM228658 3 0.6079 0.39812 0.088 0.128 0.784
#> GSM228606 1 0.7983 0.44868 0.632 0.104 0.264
#> GSM228611 1 0.4891 0.68787 0.836 0.040 0.124
#> GSM228618 1 0.7072 0.57824 0.724 0.160 0.116
#> GSM228621 2 0.7923 0.36084 0.156 0.664 0.180
#> GSM228624 1 0.6245 0.61398 0.760 0.180 0.060
#> GSM228630 3 0.8573 0.49073 0.136 0.280 0.584
#> GSM228636 3 0.9304 0.47678 0.204 0.280 0.516
#> GSM228638 3 0.8444 0.50353 0.152 0.236 0.612
#> GSM228648 3 0.5621 0.45014 0.000 0.308 0.692
#> GSM228670 3 0.6337 0.33924 0.220 0.044 0.736
#> GSM228671 2 0.6319 0.28593 0.040 0.732 0.228
#> GSM228672 1 0.6847 0.54389 0.708 0.232 0.060
#> GSM228674 3 0.9877 -0.20057 0.292 0.296 0.412
#> GSM228675 2 0.8162 0.48398 0.084 0.568 0.348
#> GSM228676 2 0.8902 0.46972 0.144 0.536 0.320
#> GSM228667 2 0.9506 0.36799 0.192 0.448 0.360
#> GSM228668 1 0.1877 0.73074 0.956 0.012 0.032
#> GSM228669 1 0.1289 0.73093 0.968 0.032 0.000
#> GSM228673 2 0.9930 0.27110 0.280 0.380 0.340
#> GSM228677 2 0.7250 0.33911 0.032 0.572 0.396
#> GSM228678 2 0.6193 0.50396 0.016 0.692 0.292
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 2 0.7878 -0.06906 0.340 0.376 0.000 0.284
#> GSM228563 1 0.6632 0.11795 0.540 0.396 0.028 0.036
#> GSM228565 1 0.7993 -0.35265 0.372 0.364 0.004 0.260
#> GSM228566 4 0.5975 0.05347 0.004 0.264 0.068 0.664
#> GSM228567 1 0.1820 0.71863 0.944 0.020 0.000 0.036
#> GSM228570 2 0.5284 0.41845 0.264 0.696 0.000 0.040
#> GSM228571 2 0.5948 0.44320 0.196 0.704 0.008 0.092
#> GSM228574 4 0.7284 -0.14970 0.040 0.396 0.060 0.504
#> GSM228575 4 0.8017 0.21428 0.372 0.140 0.032 0.456
#> GSM228576 2 0.4819 0.37235 0.344 0.652 0.000 0.004
#> GSM228579 2 0.4431 0.39553 0.304 0.696 0.000 0.000
#> GSM228580 3 0.8875 0.33828 0.100 0.284 0.464 0.152
#> GSM228581 1 0.8816 -0.00215 0.468 0.284 0.092 0.156
#> GSM228666 4 0.7255 0.43300 0.264 0.060 0.068 0.608
#> GSM228564 1 0.3266 0.67087 0.876 0.040 0.000 0.084
#> GSM228568 2 0.5000 0.03561 0.496 0.504 0.000 0.000
#> GSM228569 1 0.1913 0.71542 0.940 0.020 0.000 0.040
#> GSM228572 3 0.7650 0.40152 0.012 0.204 0.532 0.252
#> GSM228573 1 0.6517 0.52240 0.708 0.048 0.120 0.124
#> GSM228577 1 0.0804 0.72132 0.980 0.012 0.000 0.008
#> GSM228578 1 0.5963 0.13875 0.548 0.416 0.032 0.004
#> GSM228663 3 0.5876 0.20699 0.020 0.008 0.528 0.444
#> GSM228664 3 0.4673 0.57477 0.032 0.016 0.796 0.156
#> GSM228665 1 0.6198 0.34758 0.672 0.000 0.152 0.176
#> GSM228582 4 0.6439 0.23195 0.456 0.048 0.008 0.488
#> GSM228583 1 0.2002 0.71450 0.936 0.020 0.000 0.044
#> GSM228585 1 0.4122 0.55587 0.760 0.236 0.000 0.004
#> GSM228587 1 0.0707 0.72012 0.980 0.000 0.000 0.020
#> GSM228588 1 0.0000 0.71768 1.000 0.000 0.000 0.000
#> GSM228589 1 0.1042 0.72230 0.972 0.008 0.020 0.000
#> GSM228590 1 0.0000 0.71768 1.000 0.000 0.000 0.000
#> GSM228591 1 0.7292 0.43046 0.632 0.152 0.040 0.176
#> GSM228597 1 0.3400 0.69336 0.876 0.004 0.076 0.044
#> GSM228601 2 0.8709 0.22834 0.280 0.372 0.036 0.312
#> GSM228604 2 0.7724 0.24375 0.024 0.516 0.320 0.140
#> GSM228608 1 0.0000 0.71768 1.000 0.000 0.000 0.000
#> GSM228609 1 0.4126 0.57181 0.776 0.216 0.004 0.004
#> GSM228613 1 0.0000 0.71768 1.000 0.000 0.000 0.000
#> GSM228616 1 0.4004 0.66061 0.812 0.164 0.000 0.024
#> GSM228628 2 0.6004 0.37705 0.004 0.616 0.048 0.332
#> GSM228634 1 0.2089 0.71292 0.932 0.020 0.000 0.048
#> GSM228642 2 0.7071 0.25921 0.028 0.548 0.356 0.068
#> GSM228645 2 0.8046 -0.01262 0.292 0.372 0.004 0.332
#> GSM228646 1 0.7060 -0.34158 0.464 0.084 0.012 0.440
#> GSM228652 4 0.7791 0.40404 0.292 0.112 0.048 0.548
#> GSM228655 4 0.5902 0.23323 0.484 0.020 0.008 0.488
#> GSM228656 1 0.2335 0.71213 0.920 0.020 0.000 0.060
#> GSM228659 4 0.5907 0.41495 0.392 0.032 0.004 0.572
#> GSM228662 1 0.1389 0.71736 0.952 0.048 0.000 0.000
#> GSM228584 1 0.0376 0.71926 0.992 0.004 0.000 0.004
#> GSM228586 1 0.2089 0.71292 0.932 0.020 0.000 0.048
#> GSM228592 1 0.0000 0.71768 1.000 0.000 0.000 0.000
#> GSM228593 1 0.3972 0.51546 0.788 0.008 0.000 0.204
#> GSM228594 1 0.0000 0.71768 1.000 0.000 0.000 0.000
#> GSM228598 1 0.1677 0.71883 0.948 0.012 0.000 0.040
#> GSM228607 1 0.4485 0.58468 0.772 0.028 0.000 0.200
#> GSM228612 1 0.7569 0.20737 0.524 0.324 0.020 0.132
#> GSM228619 1 0.4103 0.55607 0.744 0.000 0.256 0.000
#> GSM228622 1 0.3446 0.70274 0.872 0.008 0.028 0.092
#> GSM228625 1 0.1576 0.71224 0.948 0.000 0.004 0.048
#> GSM228631 1 0.5631 0.57159 0.728 0.016 0.200 0.056
#> GSM228633 3 0.5564 0.48356 0.000 0.076 0.708 0.216
#> GSM228637 3 0.7764 -0.17380 0.240 0.000 0.404 0.356
#> GSM228639 3 0.3919 0.58504 0.056 0.000 0.840 0.104
#> GSM228649 1 0.7579 -0.39233 0.444 0.136 0.012 0.408
#> GSM228660 1 0.3266 0.63646 0.832 0.000 0.168 0.000
#> GSM228661 1 0.1610 0.71848 0.952 0.016 0.000 0.032
#> GSM228595 3 0.6351 0.45843 0.000 0.080 0.588 0.332
#> GSM228599 4 0.7622 0.35393 0.328 0.136 0.020 0.516
#> GSM228602 2 0.7567 0.22587 0.000 0.412 0.192 0.396
#> GSM228614 3 0.5348 0.51652 0.020 0.012 0.692 0.276
#> GSM228626 3 0.5766 0.49143 0.000 0.104 0.704 0.192
#> GSM228640 1 0.8492 -0.19585 0.420 0.348 0.040 0.192
#> GSM228643 2 0.6835 0.36832 0.000 0.592 0.156 0.252
#> GSM228650 4 0.5609 0.20862 0.064 0.004 0.224 0.708
#> GSM228653 3 0.8312 -0.06328 0.180 0.032 0.408 0.380
#> GSM228657 3 0.4464 0.57206 0.024 0.000 0.768 0.208
#> GSM228605 1 0.0592 0.72088 0.984 0.000 0.000 0.016
#> GSM228610 3 0.5987 0.18822 0.360 0.024 0.600 0.016
#> GSM228617 3 0.8320 0.06699 0.336 0.028 0.432 0.204
#> GSM228620 1 0.1114 0.72323 0.972 0.004 0.008 0.016
#> GSM228623 4 0.7832 0.20857 0.220 0.004 0.348 0.428
#> GSM228629 4 0.6377 0.29196 0.456 0.052 0.004 0.488
#> GSM228632 2 0.9198 0.23856 0.176 0.460 0.144 0.220
#> GSM228635 1 0.6991 0.44262 0.672 0.068 0.168 0.092
#> GSM228647 1 0.7859 -0.03166 0.472 0.012 0.324 0.192
#> GSM228596 1 0.6213 -0.30930 0.484 0.000 0.052 0.464
#> GSM228600 4 0.9066 -0.06595 0.132 0.276 0.140 0.452
#> GSM228603 2 0.8494 0.29812 0.164 0.444 0.052 0.340
#> GSM228615 1 0.4668 0.64986 0.808 0.008 0.108 0.076
#> GSM228627 2 0.7357 0.31772 0.052 0.504 0.052 0.392
#> GSM228641 3 0.8336 0.15921 0.036 0.248 0.484 0.232
#> GSM228644 3 0.4973 0.54555 0.004 0.012 0.692 0.292
#> GSM228651 4 0.7216 -0.00106 0.124 0.004 0.392 0.480
#> GSM228654 3 0.7754 0.12615 0.212 0.008 0.492 0.288
#> GSM228658 4 0.7840 -0.00251 0.072 0.064 0.392 0.472
#> GSM228606 1 0.7785 0.25560 0.564 0.032 0.208 0.196
#> GSM228611 1 0.5206 0.65578 0.788 0.024 0.096 0.092
#> GSM228618 1 0.7426 0.33763 0.608 0.048 0.108 0.236
#> GSM228621 2 0.9593 0.27733 0.152 0.380 0.264 0.204
#> GSM228624 1 0.5932 0.58900 0.732 0.172 0.052 0.044
#> GSM228630 3 0.3198 0.58141 0.080 0.000 0.880 0.040
#> GSM228636 3 0.5826 0.49417 0.164 0.004 0.716 0.116
#> GSM228638 3 0.3970 0.58130 0.076 0.000 0.840 0.084
#> GSM228648 3 0.3708 0.57410 0.000 0.020 0.832 0.148
#> GSM228670 4 0.7596 0.24170 0.148 0.028 0.256 0.568
#> GSM228671 2 0.7837 0.16745 0.024 0.456 0.384 0.136
#> GSM228672 1 0.6404 0.49535 0.676 0.216 0.020 0.088
#> GSM228674 4 0.8989 0.32592 0.228 0.228 0.088 0.456
#> GSM228675 2 0.6738 0.27446 0.052 0.564 0.024 0.360
#> GSM228676 2 0.7093 0.13673 0.112 0.492 0.004 0.392
#> GSM228667 4 0.9074 0.01290 0.188 0.364 0.084 0.364
#> GSM228668 1 0.1938 0.71412 0.936 0.012 0.000 0.052
#> GSM228669 1 0.1543 0.71732 0.956 0.008 0.032 0.004
#> GSM228673 4 0.8587 0.19139 0.180 0.248 0.072 0.500
#> GSM228677 2 0.8526 0.19512 0.028 0.380 0.252 0.340
#> GSM228678 2 0.6600 0.41323 0.012 0.652 0.116 0.220
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 2 0.6910 -0.03040 0.332 0.384 0.000 0.280 0.004
#> GSM228563 1 0.5720 0.11424 0.536 0.400 0.028 0.036 0.000
#> GSM228565 2 0.6771 -0.05418 0.360 0.368 0.000 0.272 0.000
#> GSM228566 4 0.4946 0.08859 0.004 0.236 0.056 0.700 0.004
#> GSM228567 1 0.1799 0.73509 0.940 0.012 0.000 0.028 0.020
#> GSM228570 2 0.4329 0.43015 0.252 0.716 0.000 0.032 0.000
#> GSM228571 2 0.4901 0.45073 0.188 0.724 0.008 0.080 0.000
#> GSM228574 4 0.6603 -0.16485 0.036 0.400 0.056 0.492 0.016
#> GSM228575 4 0.7522 0.18814 0.336 0.152 0.032 0.456 0.024
#> GSM228576 2 0.4047 0.39150 0.320 0.676 0.000 0.004 0.000
#> GSM228579 2 0.3684 0.41272 0.280 0.720 0.000 0.000 0.000
#> GSM228580 5 0.5685 0.00000 0.048 0.000 0.320 0.028 0.604
#> GSM228581 1 0.7634 -0.00844 0.464 0.280 0.092 0.164 0.000
#> GSM228666 4 0.6018 0.44337 0.240 0.036 0.056 0.652 0.016
#> GSM228564 1 0.2813 0.68356 0.876 0.040 0.000 0.084 0.000
#> GSM228568 2 0.4302 0.06677 0.480 0.520 0.000 0.000 0.000
#> GSM228569 1 0.2165 0.72888 0.924 0.016 0.000 0.036 0.024
#> GSM228572 3 0.6430 0.27015 0.004 0.172 0.504 0.320 0.000
#> GSM228573 1 0.6142 0.49562 0.684 0.052 0.124 0.128 0.012
#> GSM228577 1 0.0854 0.73794 0.976 0.012 0.000 0.008 0.004
#> GSM228578 1 0.5089 0.11520 0.536 0.432 0.028 0.004 0.000
#> GSM228663 3 0.4992 0.19516 0.016 0.008 0.516 0.460 0.000
#> GSM228664 3 0.3642 0.39500 0.020 0.016 0.820 0.144 0.000
#> GSM228665 1 0.5339 0.34438 0.672 0.000 0.152 0.176 0.000
#> GSM228582 4 0.5487 0.38901 0.396 0.020 0.004 0.556 0.024
#> GSM228583 1 0.2244 0.72773 0.920 0.016 0.000 0.040 0.024
#> GSM228585 1 0.3579 0.56763 0.756 0.240 0.000 0.000 0.004
#> GSM228587 1 0.0794 0.73523 0.972 0.000 0.000 0.028 0.000
#> GSM228588 1 0.0000 0.73365 1.000 0.000 0.000 0.000 0.000
#> GSM228589 1 0.0898 0.73863 0.972 0.008 0.020 0.000 0.000
#> GSM228590 1 0.0000 0.73365 1.000 0.000 0.000 0.000 0.000
#> GSM228591 1 0.7507 0.35860 0.584 0.160 0.036 0.132 0.088
#> GSM228597 1 0.3350 0.70434 0.868 0.004 0.064 0.024 0.040
#> GSM228601 2 0.8557 0.25808 0.268 0.380 0.020 0.220 0.112
#> GSM228604 2 0.6819 0.21867 0.024 0.512 0.316 0.144 0.004
#> GSM228608 1 0.0000 0.73365 1.000 0.000 0.000 0.000 0.000
#> GSM228609 1 0.3710 0.58631 0.772 0.216 0.004 0.004 0.004
#> GSM228613 1 0.0000 0.73365 1.000 0.000 0.000 0.000 0.000
#> GSM228616 1 0.4062 0.66291 0.788 0.168 0.000 0.028 0.016
#> GSM228628 2 0.5874 0.38331 0.004 0.624 0.032 0.284 0.056
#> GSM228634 1 0.2321 0.72589 0.916 0.016 0.000 0.044 0.024
#> GSM228642 2 0.6259 0.25912 0.024 0.568 0.340 0.032 0.036
#> GSM228645 2 0.7724 0.06571 0.280 0.380 0.004 0.292 0.044
#> GSM228646 4 0.6030 0.40293 0.424 0.076 0.008 0.488 0.004
#> GSM228652 4 0.5784 0.44109 0.288 0.064 0.028 0.620 0.000
#> GSM228655 4 0.5596 0.30000 0.464 0.016 0.008 0.488 0.024
#> GSM228656 1 0.2537 0.72461 0.904 0.016 0.000 0.056 0.024
#> GSM228659 4 0.4946 0.46378 0.328 0.012 0.000 0.636 0.024
#> GSM228662 1 0.1270 0.73459 0.948 0.052 0.000 0.000 0.000
#> GSM228584 1 0.0451 0.73625 0.988 0.004 0.000 0.008 0.000
#> GSM228586 1 0.2321 0.72589 0.916 0.016 0.000 0.044 0.024
#> GSM228592 1 0.0000 0.73365 1.000 0.000 0.000 0.000 0.000
#> GSM228593 1 0.3421 0.51570 0.788 0.008 0.000 0.204 0.000
#> GSM228594 1 0.0000 0.73365 1.000 0.000 0.000 0.000 0.000
#> GSM228598 1 0.1756 0.73435 0.940 0.008 0.000 0.036 0.016
#> GSM228607 1 0.4453 0.56929 0.744 0.020 0.000 0.212 0.024
#> GSM228612 1 0.6602 0.17188 0.512 0.328 0.012 0.144 0.004
#> GSM228619 1 0.3534 0.57186 0.744 0.000 0.256 0.000 0.000
#> GSM228622 1 0.3226 0.71686 0.864 0.000 0.024 0.088 0.024
#> GSM228625 1 0.1591 0.72672 0.940 0.004 0.004 0.052 0.000
#> GSM228631 1 0.5295 0.57227 0.712 0.016 0.204 0.052 0.016
#> GSM228633 3 0.5811 0.23119 0.000 0.064 0.680 0.188 0.068
#> GSM228637 3 0.6667 -0.17566 0.232 0.000 0.404 0.364 0.000
#> GSM228639 3 0.3269 0.40307 0.056 0.000 0.848 0.096 0.000
#> GSM228649 1 0.6530 -0.42004 0.440 0.136 0.012 0.412 0.000
#> GSM228660 1 0.2891 0.64645 0.824 0.000 0.176 0.000 0.000
#> GSM228661 1 0.1787 0.73378 0.940 0.012 0.000 0.032 0.016
#> GSM228595 3 0.6792 0.24700 0.000 0.072 0.584 0.224 0.120
#> GSM228599 4 0.6795 0.34412 0.304 0.120 0.020 0.540 0.016
#> GSM228602 2 0.6876 0.23493 0.000 0.420 0.180 0.384 0.016
#> GSM228614 3 0.4508 0.34759 0.020 0.000 0.648 0.332 0.000
#> GSM228626 3 0.5804 0.25706 0.000 0.060 0.696 0.128 0.116
#> GSM228640 1 0.7773 -0.23298 0.396 0.348 0.040 0.200 0.016
#> GSM228643 2 0.5762 0.36882 0.000 0.608 0.144 0.248 0.000
#> GSM228650 4 0.4278 0.21904 0.044 0.008 0.164 0.780 0.004
#> GSM228653 3 0.7439 -0.03465 0.176 0.036 0.412 0.368 0.008
#> GSM228657 3 0.3912 0.37869 0.020 0.000 0.752 0.228 0.000
#> GSM228605 1 0.0566 0.73700 0.984 0.000 0.004 0.012 0.000
#> GSM228610 3 0.4893 0.07299 0.360 0.016 0.612 0.012 0.000
#> GSM228617 3 0.7696 0.05341 0.320 0.020 0.436 0.188 0.036
#> GSM228620 1 0.0981 0.73962 0.972 0.000 0.008 0.012 0.008
#> GSM228623 4 0.6588 0.21457 0.216 0.000 0.348 0.436 0.000
#> GSM228629 4 0.5912 0.31793 0.452 0.040 0.004 0.480 0.024
#> GSM228632 2 0.7868 0.26527 0.160 0.472 0.156 0.212 0.000
#> GSM228635 1 0.8027 0.19173 0.544 0.168 0.120 0.112 0.056
#> GSM228647 1 0.7033 -0.05385 0.460 0.016 0.320 0.200 0.004
#> GSM228596 4 0.5482 0.38371 0.448 0.000 0.044 0.500 0.008
#> GSM228600 4 0.8241 -0.08552 0.120 0.272 0.144 0.444 0.020
#> GSM228603 2 0.7794 0.28919 0.152 0.436 0.056 0.336 0.020
#> GSM228615 1 0.4128 0.65697 0.800 0.008 0.112 0.080 0.000
#> GSM228627 2 0.6684 0.31493 0.052 0.496 0.052 0.388 0.012
#> GSM228641 3 0.7634 0.12113 0.032 0.244 0.468 0.236 0.020
#> GSM228644 3 0.5672 0.30880 0.004 0.004 0.652 0.216 0.124
#> GSM228651 4 0.6774 -0.00216 0.116 0.012 0.380 0.476 0.016
#> GSM228654 3 0.6912 0.13926 0.208 0.012 0.484 0.292 0.004
#> GSM228658 4 0.7221 -0.00658 0.072 0.060 0.396 0.452 0.020
#> GSM228606 1 0.6961 0.22380 0.548 0.028 0.192 0.224 0.008
#> GSM228611 1 0.5297 0.64898 0.752 0.020 0.092 0.108 0.028
#> GSM228618 1 0.6781 0.31995 0.596 0.052 0.104 0.236 0.012
#> GSM228621 2 0.8495 0.26451 0.136 0.388 0.256 0.208 0.012
#> GSM228624 1 0.5352 0.59541 0.724 0.176 0.048 0.044 0.008
#> GSM228630 3 0.2260 0.35873 0.064 0.000 0.908 0.028 0.000
#> GSM228636 3 0.5077 0.36881 0.156 0.008 0.728 0.104 0.004
#> GSM228638 3 0.3110 0.38934 0.060 0.000 0.860 0.080 0.000
#> GSM228648 3 0.3145 0.36610 0.000 0.012 0.844 0.136 0.008
#> GSM228670 4 0.5821 0.21224 0.108 0.016 0.240 0.636 0.000
#> GSM228671 2 0.7667 -0.12061 0.016 0.492 0.236 0.052 0.204
#> GSM228672 1 0.5804 0.50507 0.676 0.204 0.020 0.088 0.012
#> GSM228674 4 0.8086 0.32009 0.220 0.216 0.088 0.460 0.016
#> GSM228675 2 0.6209 0.28549 0.044 0.556 0.024 0.356 0.020
#> GSM228676 2 0.6701 0.11595 0.112 0.472 0.004 0.388 0.024
#> GSM228667 4 0.8364 -0.01443 0.180 0.352 0.080 0.360 0.028
#> GSM228668 1 0.1670 0.72951 0.936 0.012 0.000 0.052 0.000
#> GSM228669 1 0.1329 0.73340 0.956 0.008 0.032 0.004 0.000
#> GSM228673 4 0.7759 0.18006 0.176 0.236 0.072 0.500 0.016
#> GSM228677 2 0.7427 0.18111 0.024 0.372 0.256 0.344 0.004
#> GSM228678 2 0.5390 0.41467 0.008 0.676 0.104 0.212 0.000
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 4 0.6220 0.0166 0.332 0.000 0.288 0.376 0.000 0.004
#> GSM228563 1 0.4963 0.1056 0.536 0.024 0.028 0.412 0.000 0.000
#> GSM228565 4 0.6175 0.0326 0.360 0.000 0.256 0.380 0.004 0.000
#> GSM228566 3 0.4348 0.1220 0.004 0.036 0.728 0.216 0.012 0.004
#> GSM228567 1 0.1710 0.7282 0.936 0.000 0.020 0.016 0.000 0.028
#> GSM228570 4 0.3770 0.3411 0.244 0.000 0.028 0.728 0.000 0.000
#> GSM228571 4 0.4183 0.3666 0.180 0.004 0.076 0.740 0.000 0.000
#> GSM228574 3 0.6171 -0.1514 0.036 0.052 0.504 0.380 0.016 0.012
#> GSM228575 3 0.7835 0.1039 0.232 0.028 0.460 0.180 0.044 0.056
#> GSM228576 4 0.3601 0.3311 0.312 0.000 0.004 0.684 0.000 0.000
#> GSM228579 4 0.3244 0.3273 0.268 0.000 0.000 0.732 0.000 0.000
#> GSM228580 5 0.4047 0.0000 0.036 0.244 0.004 0.000 0.716 0.000
#> GSM228581 1 0.6891 -0.0453 0.456 0.092 0.168 0.284 0.000 0.000
#> GSM228666 3 0.5398 0.4353 0.216 0.060 0.668 0.036 0.020 0.000
#> GSM228564 1 0.2527 0.6780 0.876 0.000 0.084 0.040 0.000 0.000
#> GSM228568 4 0.3864 0.0710 0.480 0.000 0.000 0.520 0.000 0.000
#> GSM228569 1 0.2115 0.7206 0.916 0.000 0.032 0.020 0.000 0.032
#> GSM228572 2 0.6050 0.3418 0.004 0.508 0.304 0.172 0.012 0.000
#> GSM228573 1 0.5605 0.4847 0.676 0.116 0.140 0.056 0.000 0.012
#> GSM228577 1 0.0767 0.7313 0.976 0.000 0.008 0.012 0.000 0.004
#> GSM228578 1 0.4584 0.0683 0.524 0.028 0.004 0.444 0.000 0.000
#> GSM228663 2 0.4389 0.1633 0.016 0.512 0.468 0.004 0.000 0.000
#> GSM228664 2 0.3056 0.4649 0.016 0.832 0.140 0.012 0.000 0.000
#> GSM228665 1 0.4860 0.3327 0.664 0.160 0.176 0.000 0.000 0.000
#> GSM228582 3 0.5225 0.4042 0.380 0.004 0.556 0.020 0.004 0.036
#> GSM228583 1 0.2115 0.7207 0.916 0.000 0.032 0.020 0.000 0.032
#> GSM228585 1 0.3215 0.5656 0.756 0.000 0.000 0.240 0.000 0.004
#> GSM228587 1 0.0713 0.7286 0.972 0.000 0.028 0.000 0.000 0.000
#> GSM228588 1 0.0000 0.7270 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228589 1 0.0806 0.7319 0.972 0.020 0.000 0.008 0.000 0.000
#> GSM228590 1 0.0000 0.7270 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228591 1 0.6910 0.3486 0.580 0.028 0.108 0.160 0.116 0.008
#> GSM228597 1 0.3210 0.6924 0.856 0.064 0.016 0.008 0.056 0.000
#> GSM228601 4 0.7983 0.2445 0.264 0.012 0.192 0.384 0.128 0.020
#> GSM228604 4 0.6276 0.0334 0.020 0.308 0.152 0.508 0.000 0.012
#> GSM228608 1 0.0000 0.7270 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228609 1 0.3332 0.5864 0.772 0.000 0.004 0.216 0.004 0.004
#> GSM228613 1 0.0000 0.7270 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228616 1 0.3691 0.6581 0.784 0.000 0.020 0.172 0.000 0.024
#> GSM228628 4 0.5409 0.2946 0.004 0.032 0.272 0.636 0.040 0.016
#> GSM228634 1 0.2262 0.7175 0.908 0.000 0.036 0.020 0.000 0.036
#> GSM228642 4 0.5614 -0.0618 0.020 0.320 0.024 0.592 0.032 0.012
#> GSM228645 4 0.7136 0.1507 0.280 0.004 0.280 0.388 0.032 0.016
#> GSM228646 3 0.5279 0.3822 0.416 0.008 0.500 0.076 0.000 0.000
#> GSM228652 3 0.5226 0.4334 0.276 0.028 0.632 0.060 0.004 0.000
#> GSM228655 3 0.5208 0.3184 0.448 0.008 0.492 0.016 0.000 0.036
#> GSM228656 1 0.2467 0.7160 0.896 0.000 0.048 0.020 0.000 0.036
#> GSM228659 3 0.4645 0.4553 0.316 0.000 0.636 0.008 0.004 0.036
#> GSM228662 1 0.1204 0.7286 0.944 0.000 0.000 0.056 0.000 0.000
#> GSM228584 1 0.0405 0.7296 0.988 0.000 0.008 0.004 0.000 0.000
#> GSM228586 1 0.2262 0.7175 0.908 0.000 0.036 0.020 0.000 0.036
#> GSM228592 1 0.0000 0.7270 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228593 1 0.3161 0.4930 0.776 0.000 0.216 0.008 0.000 0.000
#> GSM228594 1 0.0000 0.7270 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228598 1 0.1693 0.7273 0.936 0.000 0.032 0.012 0.000 0.020
#> GSM228607 1 0.4208 0.5683 0.740 0.000 0.200 0.024 0.000 0.036
#> GSM228612 1 0.5976 0.1530 0.508 0.012 0.152 0.324 0.004 0.000
#> GSM228619 1 0.3244 0.5564 0.732 0.268 0.000 0.000 0.000 0.000
#> GSM228622 1 0.3046 0.7110 0.860 0.032 0.076 0.000 0.000 0.032
#> GSM228625 1 0.1484 0.7208 0.944 0.000 0.040 0.004 0.008 0.004
#> GSM228631 1 0.4979 0.5598 0.700 0.208 0.044 0.020 0.000 0.028
#> GSM228633 2 0.5871 0.2799 0.000 0.636 0.188 0.064 0.104 0.008
#> GSM228637 2 0.6090 -0.1834 0.224 0.404 0.368 0.000 0.004 0.000
#> GSM228639 2 0.2660 0.4735 0.048 0.868 0.084 0.000 0.000 0.000
#> GSM228649 1 0.5909 -0.4135 0.436 0.008 0.416 0.136 0.004 0.000
#> GSM228660 1 0.2697 0.6323 0.812 0.188 0.000 0.000 0.000 0.000
#> GSM228661 1 0.1605 0.7274 0.940 0.000 0.032 0.012 0.000 0.016
#> GSM228595 2 0.6509 0.2990 0.000 0.592 0.188 0.068 0.124 0.028
#> GSM228599 3 0.6138 0.3100 0.292 0.016 0.552 0.120 0.008 0.012
#> GSM228602 4 0.6550 0.2099 0.000 0.164 0.388 0.412 0.020 0.016
#> GSM228614 2 0.4380 0.4279 0.020 0.648 0.320 0.004 0.008 0.000
#> GSM228626 2 0.5527 0.3222 0.000 0.692 0.108 0.052 0.128 0.020
#> GSM228640 1 0.7218 -0.2579 0.380 0.032 0.212 0.348 0.012 0.016
#> GSM228643 4 0.5278 0.3088 0.000 0.140 0.248 0.608 0.000 0.004
#> GSM228650 3 0.3597 0.2576 0.036 0.140 0.808 0.008 0.008 0.000
#> GSM228653 2 0.7083 -0.0729 0.172 0.388 0.376 0.036 0.024 0.004
#> GSM228657 2 0.3543 0.4559 0.016 0.756 0.224 0.000 0.004 0.000
#> GSM228605 1 0.0603 0.7307 0.980 0.000 0.016 0.000 0.000 0.004
#> GSM228610 2 0.4329 0.1565 0.352 0.624 0.008 0.012 0.000 0.004
#> GSM228617 2 0.7494 0.0554 0.316 0.416 0.180 0.024 0.020 0.044
#> GSM228620 1 0.0881 0.7329 0.972 0.008 0.008 0.000 0.000 0.012
#> GSM228623 3 0.6219 0.2212 0.204 0.348 0.436 0.004 0.008 0.000
#> GSM228629 3 0.5544 0.3298 0.436 0.004 0.480 0.044 0.000 0.036
#> GSM228632 4 0.7035 0.2994 0.156 0.152 0.216 0.476 0.000 0.000
#> GSM228635 1 0.8092 -0.2839 0.344 0.052 0.016 0.200 0.064 0.324
#> GSM228647 1 0.6762 -0.0517 0.452 0.304 0.204 0.020 0.004 0.016
#> GSM228596 3 0.5060 0.3851 0.440 0.048 0.500 0.000 0.000 0.012
#> GSM228600 3 0.7682 -0.0718 0.108 0.128 0.464 0.256 0.024 0.020
#> GSM228603 4 0.7293 0.2894 0.144 0.040 0.348 0.424 0.024 0.020
#> GSM228615 1 0.4103 0.6466 0.792 0.104 0.076 0.016 0.012 0.000
#> GSM228627 4 0.6070 0.3351 0.052 0.036 0.388 0.500 0.020 0.004
#> GSM228641 2 0.7270 0.1082 0.028 0.448 0.252 0.232 0.020 0.020
#> GSM228644 2 0.5614 0.3560 0.004 0.644 0.196 0.004 0.124 0.028
#> GSM228651 3 0.6163 0.0395 0.112 0.360 0.496 0.008 0.016 0.008
#> GSM228654 2 0.6231 0.1283 0.208 0.480 0.296 0.008 0.000 0.008
#> GSM228658 3 0.6840 0.0158 0.064 0.392 0.444 0.060 0.012 0.028
#> GSM228606 1 0.6770 0.1855 0.528 0.172 0.236 0.032 0.028 0.004
#> GSM228611 1 0.5096 0.6366 0.740 0.096 0.096 0.024 0.004 0.040
#> GSM228618 1 0.6459 0.3029 0.584 0.088 0.244 0.052 0.020 0.012
#> GSM228621 4 0.7921 0.1285 0.132 0.244 0.224 0.376 0.008 0.016
#> GSM228624 1 0.4884 0.5990 0.724 0.044 0.044 0.176 0.004 0.008
#> GSM228630 2 0.1924 0.4417 0.048 0.920 0.028 0.004 0.000 0.000
#> GSM228636 2 0.4725 0.4374 0.156 0.728 0.092 0.012 0.012 0.000
#> GSM228638 2 0.2488 0.4631 0.044 0.880 0.076 0.000 0.000 0.000
#> GSM228648 2 0.2865 0.4387 0.000 0.840 0.140 0.012 0.000 0.008
#> GSM228670 3 0.5261 0.2335 0.100 0.236 0.644 0.016 0.004 0.000
#> GSM228671 6 0.5330 0.0000 0.004 0.068 0.020 0.312 0.000 0.596
#> GSM228672 1 0.5453 0.5052 0.672 0.020 0.076 0.204 0.004 0.024
#> GSM228674 3 0.7332 0.2965 0.212 0.084 0.464 0.216 0.000 0.024
#> GSM228675 4 0.5989 0.3089 0.032 0.020 0.352 0.540 0.048 0.008
#> GSM228676 4 0.6038 0.1491 0.108 0.000 0.384 0.472 0.000 0.036
#> GSM228667 4 0.7814 0.0143 0.180 0.072 0.340 0.360 0.008 0.040
#> GSM228668 1 0.1578 0.7231 0.936 0.000 0.048 0.012 0.004 0.000
#> GSM228669 1 0.1346 0.7258 0.952 0.024 0.000 0.016 0.008 0.000
#> GSM228673 3 0.7021 0.1650 0.172 0.060 0.512 0.232 0.008 0.016
#> GSM228677 4 0.6919 0.1590 0.024 0.236 0.340 0.384 0.012 0.004
#> GSM228678 4 0.4935 0.3115 0.008 0.100 0.196 0.688 0.008 0.000
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
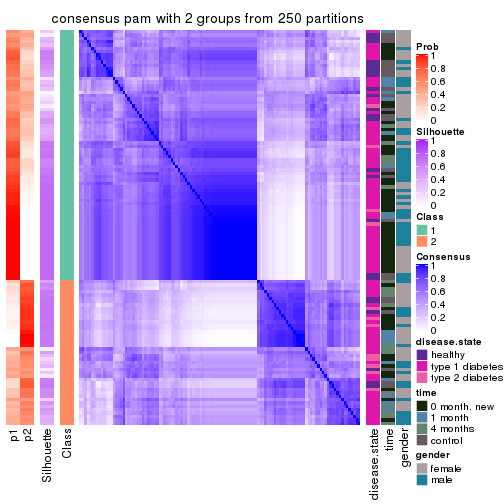
consensus_heatmap(res, k = 3)
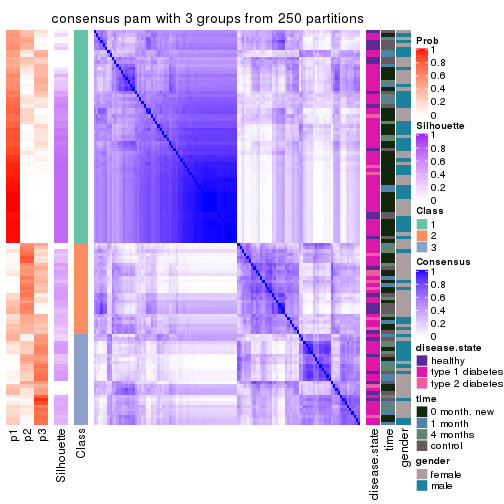
consensus_heatmap(res, k = 4)
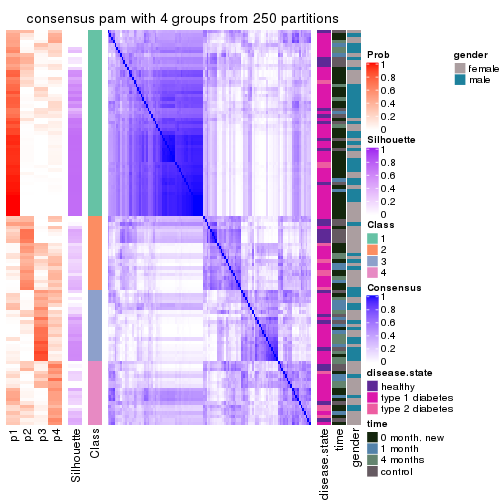
consensus_heatmap(res, k = 5)
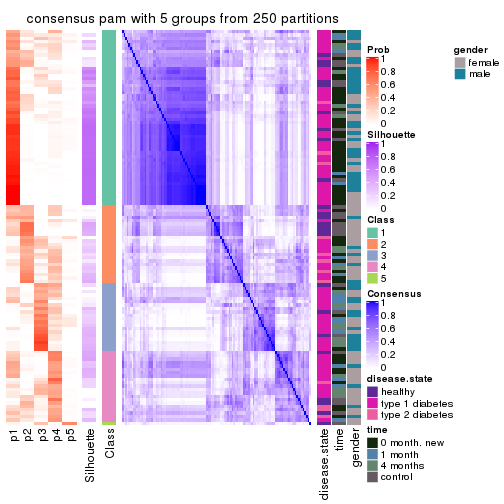
consensus_heatmap(res, k = 6)
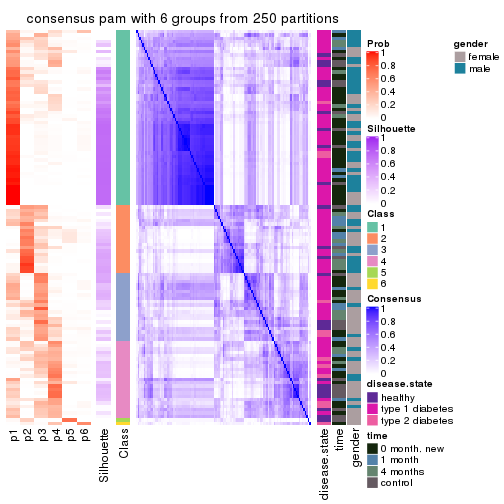
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
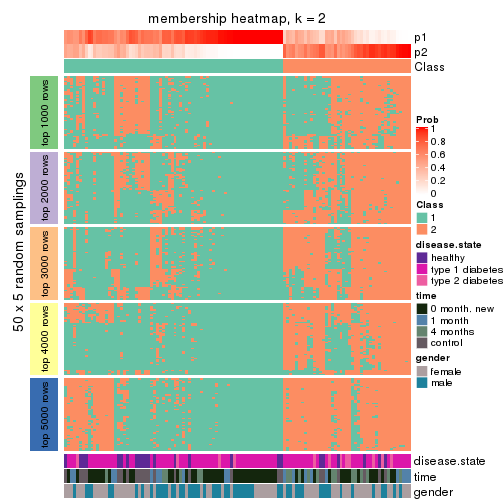
membership_heatmap(res, k = 3)
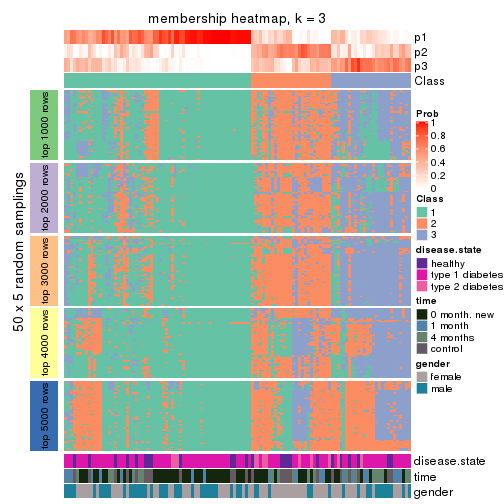
membership_heatmap(res, k = 4)
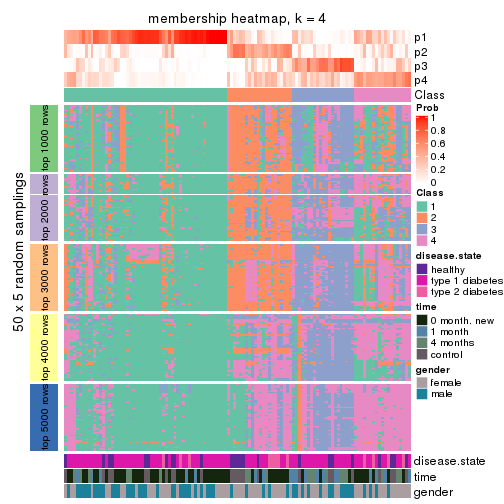
membership_heatmap(res, k = 5)
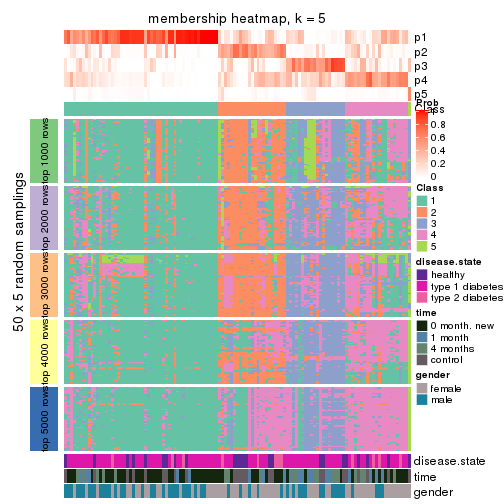
membership_heatmap(res, k = 6)
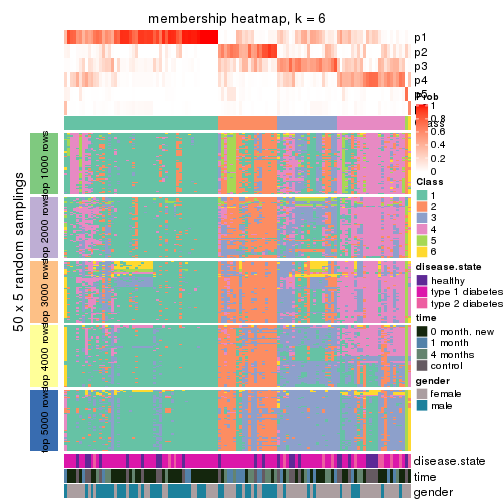
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
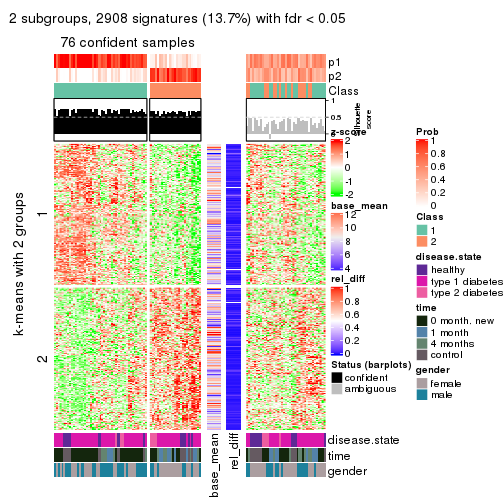
get_signatures(res, k = 3)
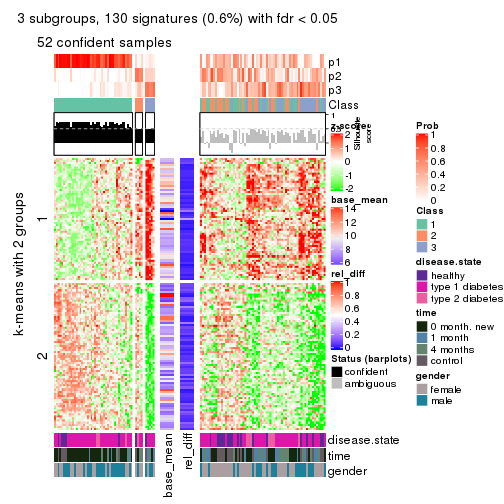
get_signatures(res, k = 4)
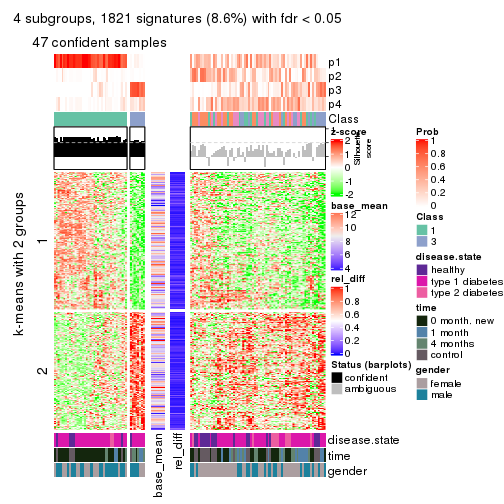
get_signatures(res, k = 5)

get_signatures(res, k = 6)

Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
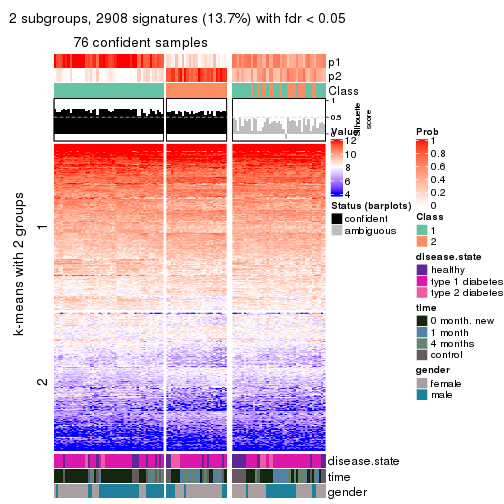
get_signatures(res, k = 3, scale_rows = FALSE)
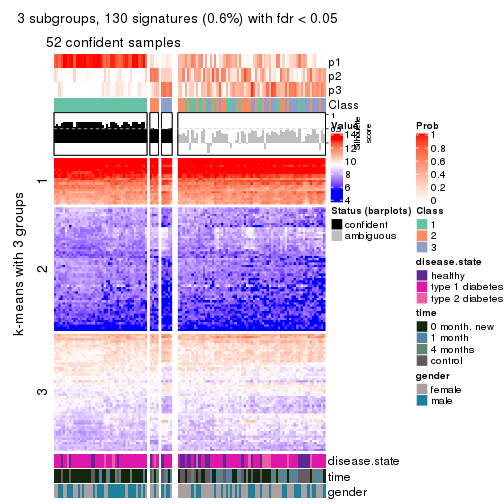
get_signatures(res, k = 4, scale_rows = FALSE)
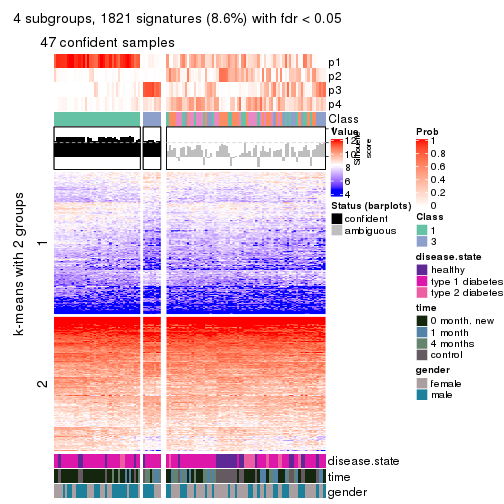
get_signatures(res, k = 5, scale_rows = FALSE)

get_signatures(res, k = 6, scale_rows = FALSE)

Compare the overlap of signatures from different k:
compare_signatures(res)
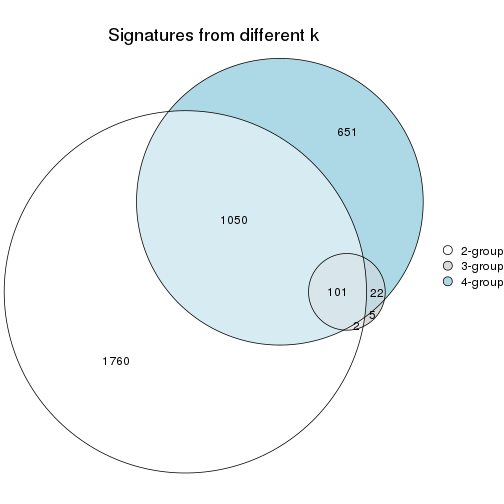
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
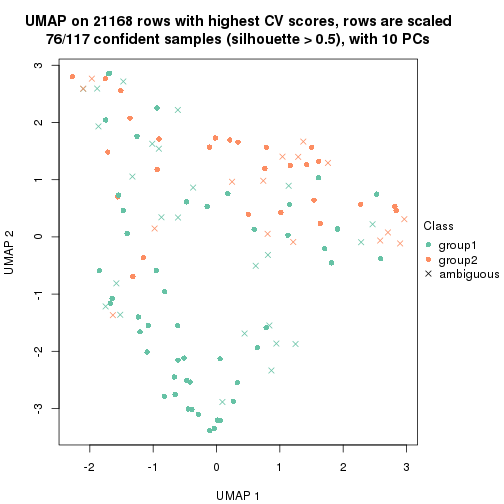
dimension_reduction(res, k = 3, method = "UMAP")
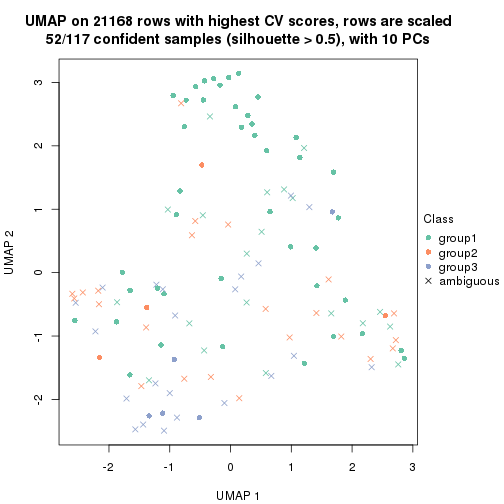
dimension_reduction(res, k = 4, method = "UMAP")
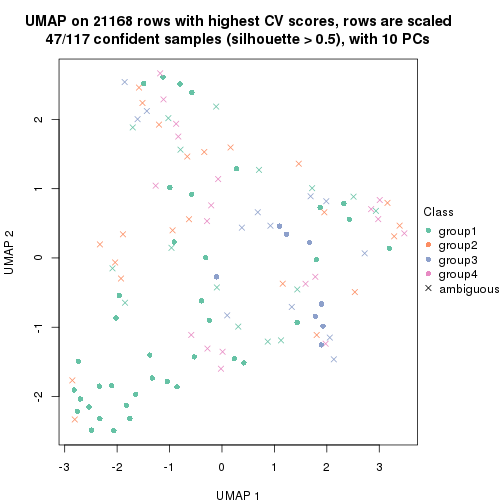
dimension_reduction(res, k = 5, method = "UMAP")
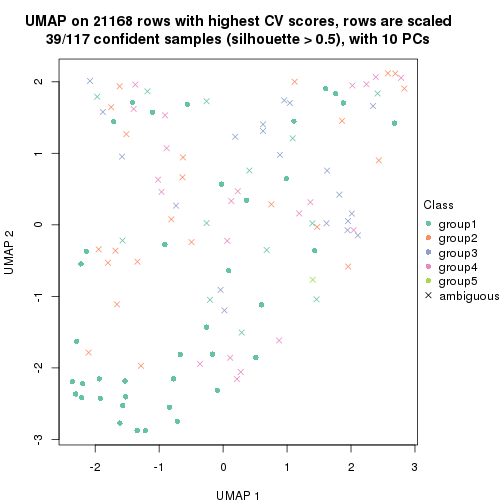
dimension_reduction(res, k = 6, method = "UMAP")
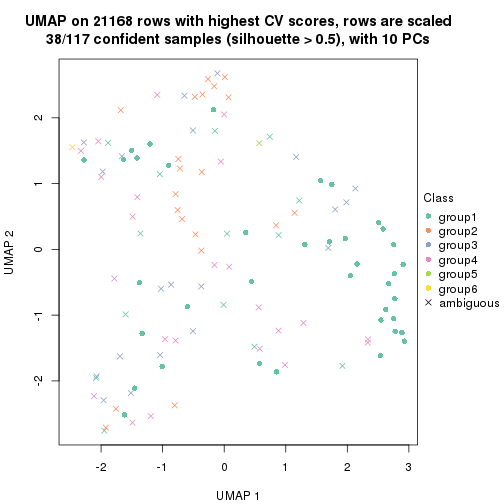
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
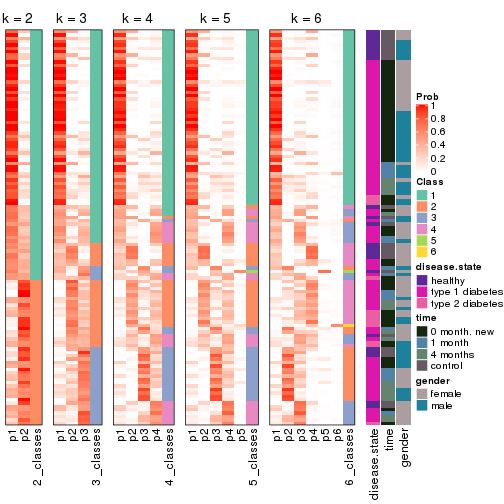
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> CV:pam 76 0.446 0.00560 0.0111 2
#> CV:pam 52 0.543 0.03241 0.4340 3
#> CV:pam 47 0.805 0.00178 1.0000 4
#> CV:pam 39 NA NA NA 5
#> CV:pam 38 NA NA NA 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["CV", "mclust"]
# you can also extract it by
# res = res_list["CV:mclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'CV' method.
#> Subgroups are detected by 'mclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 4.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
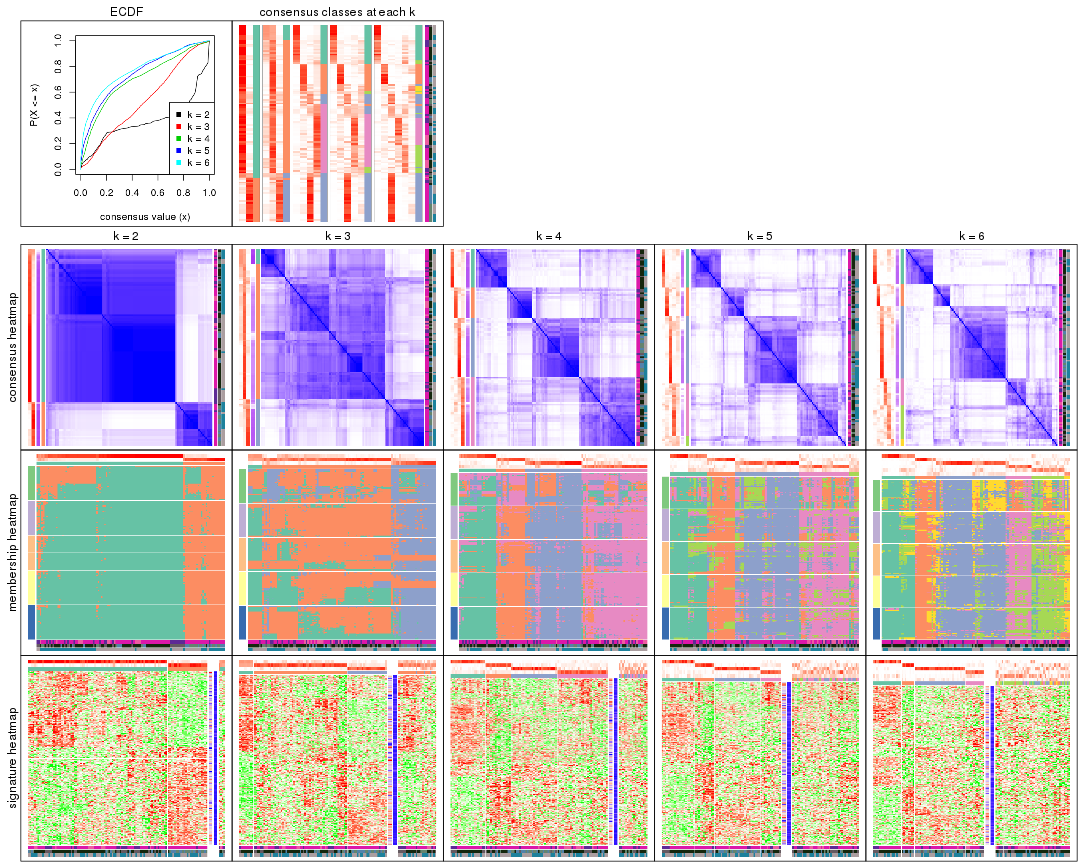
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
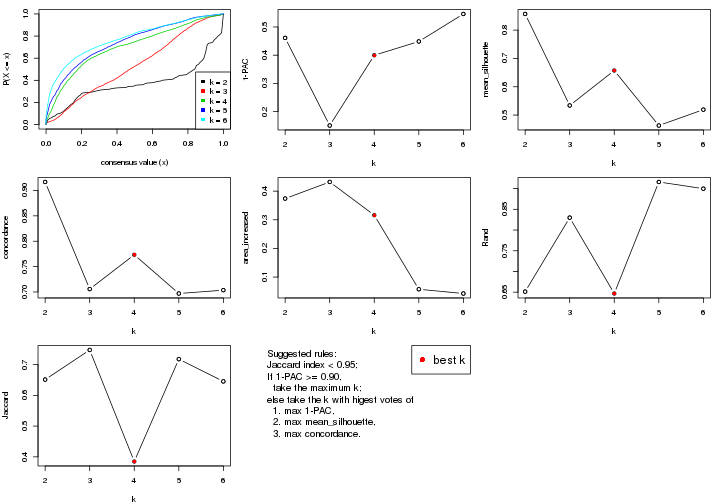
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.461 0.857 0.917 0.3741 0.651 0.651
#> 3 3 0.151 0.534 0.706 0.4320 0.830 0.748
#> 4 4 0.400 0.657 0.773 0.3159 0.647 0.385
#> 5 5 0.449 0.463 0.697 0.0575 0.916 0.718
#> 6 6 0.546 0.519 0.704 0.0426 0.900 0.645
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 4
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.0000 0.9163 1.000 0.000
#> GSM228563 1 0.2423 0.9092 0.960 0.040
#> GSM228565 1 0.0000 0.9163 1.000 0.000
#> GSM228566 1 0.9896 0.0212 0.560 0.440
#> GSM228567 1 0.0000 0.9163 1.000 0.000
#> GSM228570 1 0.0000 0.9163 1.000 0.000
#> GSM228571 1 0.0000 0.9163 1.000 0.000
#> GSM228574 1 0.8267 0.6092 0.740 0.260
#> GSM228575 1 0.2948 0.8896 0.948 0.052
#> GSM228576 1 0.0000 0.9163 1.000 0.000
#> GSM228579 1 0.0000 0.9163 1.000 0.000
#> GSM228580 1 0.0672 0.9156 0.992 0.008
#> GSM228581 1 0.2603 0.9040 0.956 0.044
#> GSM228666 1 0.0376 0.9151 0.996 0.004
#> GSM228564 1 0.0000 0.9163 1.000 0.000
#> GSM228568 1 0.5408 0.8746 0.876 0.124
#> GSM228569 1 0.6247 0.8509 0.844 0.156
#> GSM228572 1 0.4562 0.8906 0.904 0.096
#> GSM228573 2 0.2236 0.8877 0.036 0.964
#> GSM228577 1 0.4690 0.8868 0.900 0.100
#> GSM228578 1 0.4939 0.8837 0.892 0.108
#> GSM228663 1 0.8499 0.7114 0.724 0.276
#> GSM228664 1 0.7056 0.8169 0.808 0.192
#> GSM228665 2 0.9460 0.3855 0.364 0.636
#> GSM228582 1 0.0376 0.9151 0.996 0.004
#> GSM228583 1 0.0000 0.9163 1.000 0.000
#> GSM228585 1 0.0000 0.9163 1.000 0.000
#> GSM228587 1 0.0000 0.9163 1.000 0.000
#> GSM228588 1 0.0376 0.9151 0.996 0.004
#> GSM228589 1 0.0000 0.9163 1.000 0.000
#> GSM228590 1 0.0000 0.9163 1.000 0.000
#> GSM228591 1 0.0376 0.9151 0.996 0.004
#> GSM228597 1 0.0376 0.9151 0.996 0.004
#> GSM228601 1 0.0376 0.9151 0.996 0.004
#> GSM228604 1 0.2948 0.8876 0.948 0.052
#> GSM228608 1 0.0000 0.9163 1.000 0.000
#> GSM228609 1 0.0000 0.9163 1.000 0.000
#> GSM228613 1 0.0000 0.9163 1.000 0.000
#> GSM228616 1 0.0000 0.9163 1.000 0.000
#> GSM228628 1 0.0376 0.9151 0.996 0.004
#> GSM228634 1 0.0000 0.9163 1.000 0.000
#> GSM228642 1 0.0376 0.9151 0.996 0.004
#> GSM228645 1 0.0672 0.9144 0.992 0.008
#> GSM228646 1 0.2043 0.9022 0.968 0.032
#> GSM228652 1 0.0000 0.9163 1.000 0.000
#> GSM228655 1 0.0000 0.9163 1.000 0.000
#> GSM228656 1 0.0000 0.9163 1.000 0.000
#> GSM228659 1 0.0000 0.9163 1.000 0.000
#> GSM228662 1 0.0000 0.9163 1.000 0.000
#> GSM228584 1 0.4562 0.8883 0.904 0.096
#> GSM228586 1 0.4690 0.8868 0.900 0.100
#> GSM228592 1 0.4690 0.8868 0.900 0.100
#> GSM228593 1 0.4161 0.8935 0.916 0.084
#> GSM228594 1 0.5737 0.8669 0.864 0.136
#> GSM228598 1 0.4690 0.8868 0.900 0.100
#> GSM228607 1 0.4815 0.8851 0.896 0.104
#> GSM228612 1 0.8267 0.7320 0.740 0.260
#> GSM228619 1 0.5842 0.8660 0.860 0.140
#> GSM228622 1 0.5178 0.8808 0.884 0.116
#> GSM228625 1 0.4690 0.8868 0.900 0.100
#> GSM228631 1 0.6343 0.8497 0.840 0.160
#> GSM228633 1 0.4815 0.8866 0.896 0.104
#> GSM228637 1 0.4690 0.8868 0.900 0.100
#> GSM228639 2 0.6247 0.8259 0.156 0.844
#> GSM228649 1 0.4690 0.8868 0.900 0.100
#> GSM228660 1 0.6973 0.8204 0.812 0.188
#> GSM228661 1 0.6343 0.8468 0.840 0.160
#> GSM228595 1 0.0376 0.9151 0.996 0.004
#> GSM228599 1 0.0000 0.9163 1.000 0.000
#> GSM228602 2 0.3274 0.8906 0.060 0.940
#> GSM228614 1 0.0000 0.9163 1.000 0.000
#> GSM228626 1 0.0376 0.9151 0.996 0.004
#> GSM228640 2 0.6438 0.8651 0.164 0.836
#> GSM228643 2 0.7139 0.8444 0.196 0.804
#> GSM228650 2 0.5408 0.8863 0.124 0.876
#> GSM228653 2 0.6712 0.8657 0.176 0.824
#> GSM228657 1 0.0376 0.9151 0.996 0.004
#> GSM228605 1 0.5519 0.8731 0.872 0.128
#> GSM228610 2 0.0376 0.8840 0.004 0.996
#> GSM228617 2 0.1633 0.8896 0.024 0.976
#> GSM228620 2 0.0672 0.8863 0.008 0.992
#> GSM228623 1 0.4690 0.8868 0.900 0.100
#> GSM228629 2 0.0672 0.8863 0.008 0.992
#> GSM228632 2 0.7139 0.7588 0.196 0.804
#> GSM228635 1 0.4815 0.8866 0.896 0.104
#> GSM228647 2 0.0672 0.8863 0.008 0.992
#> GSM228596 1 0.6801 0.7424 0.820 0.180
#> GSM228600 2 0.6148 0.8743 0.152 0.848
#> GSM228603 2 0.5842 0.8747 0.140 0.860
#> GSM228615 1 0.0000 0.9163 1.000 0.000
#> GSM228627 1 0.6148 0.7885 0.848 0.152
#> GSM228641 2 0.5408 0.8774 0.124 0.876
#> GSM228644 1 0.0376 0.9151 0.996 0.004
#> GSM228651 2 0.6148 0.8715 0.152 0.848
#> GSM228654 2 0.6531 0.8700 0.168 0.832
#> GSM228658 2 0.6801 0.8624 0.180 0.820
#> GSM228606 1 0.9580 0.5007 0.620 0.380
#> GSM228611 2 0.0376 0.8840 0.004 0.996
#> GSM228618 2 0.0938 0.8875 0.012 0.988
#> GSM228621 2 0.1414 0.8893 0.020 0.980
#> GSM228624 1 0.9922 0.3105 0.552 0.448
#> GSM228630 2 0.6438 0.7991 0.164 0.836
#> GSM228636 1 0.4815 0.8866 0.896 0.104
#> GSM228638 2 0.0672 0.8863 0.008 0.992
#> GSM228648 2 0.6973 0.7766 0.188 0.812
#> GSM228670 1 0.0000 0.9163 1.000 0.000
#> GSM228671 1 0.1633 0.9140 0.976 0.024
#> GSM228672 1 0.0000 0.9163 1.000 0.000
#> GSM228674 1 0.0000 0.9163 1.000 0.000
#> GSM228675 1 0.0376 0.9151 0.996 0.004
#> GSM228676 1 0.1184 0.9110 0.984 0.016
#> GSM228667 1 0.0000 0.9163 1.000 0.000
#> GSM228668 1 0.4690 0.8868 0.900 0.100
#> GSM228669 1 0.4690 0.8868 0.900 0.100
#> GSM228673 1 1.0000 0.1131 0.504 0.496
#> GSM228677 1 0.7299 0.8040 0.796 0.204
#> GSM228678 1 0.4690 0.8868 0.900 0.100
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 2 0.554 0.6239 0.200 0.776 0.024
#> GSM228563 2 0.544 0.6249 0.192 0.784 0.024
#> GSM228565 2 0.337 0.6393 0.072 0.904 0.024
#> GSM228566 3 0.966 0.5464 0.256 0.280 0.464
#> GSM228567 1 0.628 0.9321 0.540 0.460 0.000
#> GSM228570 2 0.458 0.4040 0.184 0.812 0.004
#> GSM228571 2 0.576 0.1599 0.244 0.740 0.016
#> GSM228574 2 0.853 0.1558 0.120 0.572 0.308
#> GSM228575 2 0.792 0.4822 0.316 0.604 0.080
#> GSM228576 2 0.397 0.6402 0.100 0.876 0.024
#> GSM228579 2 0.633 -0.3528 0.332 0.656 0.012
#> GSM228580 2 0.522 0.6138 0.208 0.780 0.012
#> GSM228581 2 0.448 0.6006 0.072 0.864 0.064
#> GSM228666 2 0.171 0.6307 0.032 0.960 0.008
#> GSM228564 2 0.506 0.6206 0.208 0.784 0.008
#> GSM228568 2 0.390 0.5999 0.008 0.864 0.128
#> GSM228569 2 0.907 -0.3975 0.308 0.528 0.164
#> GSM228572 2 0.437 0.5955 0.040 0.864 0.096
#> GSM228573 3 0.385 0.8016 0.004 0.136 0.860
#> GSM228577 2 0.784 -0.4831 0.360 0.576 0.064
#> GSM228578 2 0.501 0.5561 0.076 0.840 0.084
#> GSM228663 2 0.516 0.5306 0.004 0.764 0.232
#> GSM228664 2 0.536 0.5442 0.012 0.768 0.220
#> GSM228665 3 0.629 0.1551 0.000 0.468 0.532
#> GSM228582 2 0.223 0.6105 0.044 0.944 0.012
#> GSM228583 1 0.627 0.9341 0.544 0.456 0.000
#> GSM228585 1 0.628 0.9321 0.540 0.460 0.000
#> GSM228587 2 0.571 -0.2226 0.320 0.680 0.000
#> GSM228588 2 0.374 0.5616 0.036 0.892 0.072
#> GSM228589 2 0.350 0.5663 0.028 0.900 0.072
#> GSM228590 1 0.627 0.9341 0.544 0.456 0.000
#> GSM228591 2 0.337 0.5675 0.024 0.904 0.072
#> GSM228597 2 0.491 0.6194 0.196 0.796 0.008
#> GSM228601 2 0.337 0.5675 0.024 0.904 0.072
#> GSM228604 2 0.368 0.6189 0.060 0.896 0.044
#> GSM228608 2 0.533 0.0823 0.272 0.728 0.000
#> GSM228609 2 0.153 0.6120 0.032 0.964 0.004
#> GSM228613 1 0.630 0.9023 0.520 0.480 0.000
#> GSM228616 2 0.311 0.6287 0.056 0.916 0.028
#> GSM228628 2 0.127 0.6175 0.024 0.972 0.004
#> GSM228634 2 0.666 -0.7864 0.460 0.532 0.008
#> GSM228642 2 0.337 0.5675 0.024 0.904 0.072
#> GSM228645 2 0.509 0.6210 0.176 0.804 0.020
#> GSM228646 2 0.673 0.5726 0.184 0.736 0.080
#> GSM228652 2 0.569 0.2118 0.224 0.756 0.020
#> GSM228655 2 0.313 0.5799 0.088 0.904 0.008
#> GSM228656 1 0.627 0.9320 0.548 0.452 0.000
#> GSM228659 2 0.176 0.6095 0.040 0.956 0.004
#> GSM228662 2 0.627 -0.7355 0.452 0.548 0.000
#> GSM228584 1 0.780 0.8892 0.520 0.428 0.052
#> GSM228586 1 0.806 0.8598 0.492 0.444 0.064
#> GSM228592 1 0.796 0.8796 0.512 0.428 0.060
#> GSM228593 2 0.279 0.6100 0.028 0.928 0.044
#> GSM228594 2 0.849 -0.4601 0.336 0.556 0.108
#> GSM228598 2 0.699 0.0351 0.256 0.688 0.056
#> GSM228607 2 0.368 0.6195 0.028 0.892 0.080
#> GSM228612 2 0.507 0.5390 0.004 0.772 0.224
#> GSM228619 2 0.875 0.4959 0.292 0.564 0.144
#> GSM228622 2 0.606 0.6010 0.072 0.780 0.148
#> GSM228625 2 0.328 0.6035 0.024 0.908 0.068
#> GSM228631 2 0.812 0.5149 0.168 0.648 0.184
#> GSM228633 2 0.455 0.5632 0.024 0.844 0.132
#> GSM228637 2 0.570 0.6387 0.136 0.800 0.064
#> GSM228639 3 0.554 0.7619 0.024 0.200 0.776
#> GSM228649 2 0.260 0.6126 0.016 0.932 0.052
#> GSM228660 2 0.455 0.5553 0.000 0.800 0.200
#> GSM228661 2 0.892 -0.3429 0.296 0.548 0.156
#> GSM228595 2 0.362 0.5684 0.032 0.896 0.072
#> GSM228599 2 0.595 0.5741 0.280 0.708 0.012
#> GSM228602 3 0.413 0.8119 0.012 0.132 0.856
#> GSM228614 2 0.659 0.5533 0.280 0.688 0.032
#> GSM228626 2 0.337 0.5675 0.024 0.904 0.072
#> GSM228640 3 0.661 0.7912 0.096 0.152 0.752
#> GSM228643 3 0.678 0.7895 0.088 0.176 0.736
#> GSM228650 3 0.598 0.7525 0.020 0.252 0.728
#> GSM228653 3 0.754 0.7690 0.104 0.216 0.680
#> GSM228657 2 0.298 0.5842 0.024 0.920 0.056
#> GSM228605 2 0.865 0.5288 0.268 0.584 0.148
#> GSM228610 3 0.263 0.8092 0.000 0.084 0.916
#> GSM228617 3 0.271 0.8110 0.000 0.088 0.912
#> GSM228620 3 0.236 0.8047 0.000 0.072 0.928
#> GSM228623 2 0.608 0.6332 0.168 0.772 0.060
#> GSM228629 3 0.236 0.8047 0.000 0.072 0.928
#> GSM228632 3 0.585 0.6599 0.012 0.268 0.720
#> GSM228635 2 0.645 0.6148 0.196 0.744 0.060
#> GSM228647 3 0.236 0.8047 0.000 0.072 0.928
#> GSM228596 2 0.946 0.1358 0.216 0.492 0.292
#> GSM228600 3 0.656 0.7930 0.100 0.144 0.756
#> GSM228603 3 0.644 0.7865 0.100 0.136 0.764
#> GSM228615 2 0.527 0.6191 0.200 0.784 0.016
#> GSM228627 2 0.625 0.5345 0.104 0.776 0.120
#> GSM228641 3 0.598 0.7963 0.080 0.132 0.788
#> GSM228644 2 0.337 0.5675 0.024 0.904 0.072
#> GSM228651 3 0.643 0.7892 0.096 0.140 0.764
#> GSM228654 3 0.714 0.7781 0.084 0.212 0.704
#> GSM228658 3 0.740 0.7678 0.096 0.216 0.688
#> GSM228606 3 0.991 0.1331 0.280 0.328 0.392
#> GSM228611 3 0.245 0.8064 0.000 0.076 0.924
#> GSM228618 3 0.254 0.8080 0.000 0.080 0.920
#> GSM228621 3 0.355 0.8044 0.000 0.132 0.868
#> GSM228624 2 0.613 0.3196 0.000 0.600 0.400
#> GSM228630 3 0.658 0.5468 0.020 0.328 0.652
#> GSM228636 2 0.634 0.6221 0.180 0.756 0.064
#> GSM228638 3 0.245 0.8066 0.000 0.076 0.924
#> GSM228648 2 0.650 0.0840 0.004 0.528 0.468
#> GSM228670 2 0.623 0.5734 0.280 0.700 0.020
#> GSM228671 2 0.647 0.5673 0.280 0.692 0.028
#> GSM228672 2 0.491 0.6289 0.196 0.796 0.008
#> GSM228674 2 0.478 0.6312 0.164 0.820 0.016
#> GSM228675 2 0.511 0.6083 0.212 0.780 0.008
#> GSM228676 2 0.886 0.3443 0.360 0.512 0.128
#> GSM228667 2 0.636 0.5704 0.280 0.696 0.024
#> GSM228668 2 0.738 0.5964 0.252 0.672 0.076
#> GSM228669 2 0.671 0.6225 0.196 0.732 0.072
#> GSM228673 3 0.830 0.2037 0.080 0.412 0.508
#> GSM228677 2 0.929 0.4474 0.284 0.516 0.200
#> GSM228678 2 0.615 0.6320 0.160 0.772 0.068
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.4511 0.71877 0.028 0.072 0.068 0.832
#> GSM228563 4 0.2950 0.69668 0.020 0.068 0.012 0.900
#> GSM228565 4 0.5299 0.69646 0.040 0.036 0.152 0.772
#> GSM228566 3 0.7075 0.38685 0.020 0.088 0.564 0.328
#> GSM228567 1 0.1557 0.82361 0.944 0.000 0.000 0.056
#> GSM228570 1 0.6394 0.52767 0.608 0.024 0.040 0.328
#> GSM228571 1 0.6829 0.71912 0.680 0.076 0.068 0.176
#> GSM228574 3 0.7192 0.62670 0.028 0.160 0.628 0.184
#> GSM228575 4 0.6714 0.61378 0.008 0.128 0.232 0.632
#> GSM228576 4 0.7151 0.64362 0.072 0.088 0.184 0.656
#> GSM228579 1 0.5219 0.79161 0.764 0.072 0.008 0.156
#> GSM228580 4 0.3946 0.66978 0.000 0.168 0.020 0.812
#> GSM228581 2 0.5456 0.74180 0.020 0.768 0.112 0.100
#> GSM228666 4 0.6988 0.32167 0.000 0.380 0.120 0.500
#> GSM228564 4 0.3399 0.69578 0.092 0.040 0.000 0.868
#> GSM228568 2 0.9340 0.17575 0.104 0.376 0.308 0.212
#> GSM228569 1 0.6310 0.74510 0.712 0.044 0.168 0.076
#> GSM228572 2 0.5941 0.66018 0.016 0.672 0.044 0.268
#> GSM228573 3 0.0524 0.79518 0.004 0.000 0.988 0.008
#> GSM228577 1 0.5594 0.80106 0.764 0.036 0.068 0.132
#> GSM228578 4 0.8070 0.04681 0.376 0.012 0.216 0.396
#> GSM228663 3 0.5778 -0.05764 0.000 0.472 0.500 0.028
#> GSM228664 2 0.5085 0.63342 0.000 0.708 0.260 0.032
#> GSM228665 3 0.3166 0.74250 0.000 0.116 0.868 0.016
#> GSM228582 2 0.5660 0.75562 0.048 0.768 0.076 0.108
#> GSM228583 1 0.1474 0.82358 0.948 0.000 0.000 0.052
#> GSM228585 1 0.1389 0.82140 0.952 0.000 0.000 0.048
#> GSM228587 1 0.3647 0.82316 0.852 0.040 0.000 0.108
#> GSM228588 2 0.3647 0.80742 0.016 0.832 0.000 0.152
#> GSM228589 2 0.3271 0.82011 0.012 0.856 0.000 0.132
#> GSM228590 1 0.1118 0.81431 0.964 0.000 0.000 0.036
#> GSM228591 2 0.2589 0.82673 0.000 0.884 0.000 0.116
#> GSM228597 4 0.2965 0.68989 0.036 0.072 0.000 0.892
#> GSM228601 2 0.2704 0.82563 0.000 0.876 0.000 0.124
#> GSM228604 2 0.6871 0.53601 0.024 0.640 0.228 0.108
#> GSM228608 1 0.5168 0.73284 0.736 0.016 0.024 0.224
#> GSM228609 4 0.6102 0.00657 0.048 0.420 0.000 0.532
#> GSM228613 1 0.1211 0.81624 0.960 0.000 0.000 0.040
#> GSM228616 4 0.6737 0.64553 0.024 0.140 0.168 0.668
#> GSM228628 2 0.3216 0.81750 0.004 0.864 0.008 0.124
#> GSM228634 1 0.3763 0.82952 0.856 0.028 0.012 0.104
#> GSM228642 2 0.2530 0.82669 0.000 0.888 0.000 0.112
#> GSM228645 4 0.6296 0.66084 0.004 0.152 0.168 0.676
#> GSM228646 4 0.7669 0.50227 0.024 0.156 0.272 0.548
#> GSM228652 1 0.5733 0.78516 0.740 0.040 0.044 0.176
#> GSM228655 1 0.8077 -0.08532 0.420 0.032 0.144 0.404
#> GSM228656 1 0.1118 0.81431 0.964 0.000 0.000 0.036
#> GSM228659 4 0.5031 0.59371 0.212 0.048 0.000 0.740
#> GSM228662 1 0.1576 0.82240 0.948 0.004 0.000 0.048
#> GSM228584 1 0.3229 0.81863 0.880 0.000 0.048 0.072
#> GSM228586 1 0.3629 0.82095 0.868 0.008 0.048 0.076
#> GSM228592 1 0.3312 0.81782 0.876 0.000 0.052 0.072
#> GSM228593 4 0.6572 0.43129 0.064 0.264 0.028 0.644
#> GSM228594 1 0.5950 0.78881 0.748 0.044 0.112 0.096
#> GSM228598 1 0.5664 0.78834 0.748 0.032 0.056 0.164
#> GSM228607 4 0.7272 0.40558 0.020 0.096 0.356 0.528
#> GSM228612 3 0.6192 0.22305 0.004 0.384 0.564 0.048
#> GSM228619 4 0.6075 0.58509 0.076 0.000 0.288 0.636
#> GSM228622 4 0.6759 0.46817 0.108 0.000 0.344 0.548
#> GSM228625 4 0.6364 0.65127 0.096 0.100 0.076 0.728
#> GSM228631 4 0.6805 0.32821 0.100 0.000 0.400 0.500
#> GSM228633 2 0.4529 0.78023 0.016 0.820 0.052 0.112
#> GSM228637 4 0.2828 0.69498 0.020 0.036 0.032 0.912
#> GSM228639 3 0.2665 0.76930 0.008 0.004 0.900 0.088
#> GSM228649 4 0.6009 0.39404 0.020 0.288 0.036 0.656
#> GSM228660 2 0.8092 0.43416 0.072 0.512 0.320 0.096
#> GSM228661 1 0.6164 0.76143 0.724 0.040 0.156 0.080
#> GSM228595 2 0.2647 0.82655 0.000 0.880 0.000 0.120
#> GSM228599 4 0.2629 0.70633 0.024 0.060 0.004 0.912
#> GSM228602 3 0.1985 0.79669 0.024 0.012 0.944 0.020
#> GSM228614 4 0.4480 0.71406 0.004 0.096 0.084 0.816
#> GSM228626 2 0.2469 0.82572 0.000 0.892 0.000 0.108
#> GSM228640 3 0.4313 0.76824 0.024 0.112 0.832 0.032
#> GSM228643 3 0.4524 0.76570 0.028 0.096 0.828 0.048
#> GSM228650 3 0.4213 0.75705 0.012 0.028 0.824 0.136
#> GSM228653 3 0.4377 0.77158 0.020 0.124 0.824 0.032
#> GSM228657 2 0.2647 0.82695 0.000 0.880 0.000 0.120
#> GSM228605 4 0.5389 0.59298 0.032 0.000 0.308 0.660
#> GSM228610 3 0.0376 0.79351 0.000 0.004 0.992 0.004
#> GSM228617 3 0.0524 0.79476 0.004 0.000 0.988 0.008
#> GSM228620 3 0.0524 0.79411 0.000 0.004 0.988 0.008
#> GSM228623 4 0.3331 0.70081 0.016 0.056 0.040 0.888
#> GSM228629 3 0.0188 0.79190 0.000 0.004 0.996 0.000
#> GSM228632 3 0.2644 0.77673 0.000 0.032 0.908 0.060
#> GSM228635 4 0.4637 0.61117 0.020 0.144 0.032 0.804
#> GSM228647 3 0.0376 0.79272 0.000 0.004 0.992 0.004
#> GSM228596 3 0.6833 0.20956 0.024 0.052 0.528 0.396
#> GSM228600 3 0.4436 0.77090 0.024 0.108 0.828 0.040
#> GSM228603 3 0.4158 0.76915 0.024 0.108 0.840 0.028
#> GSM228615 4 0.2861 0.70156 0.012 0.092 0.004 0.892
#> GSM228627 3 0.7007 0.39548 0.028 0.408 0.508 0.056
#> GSM228641 3 0.3791 0.77847 0.016 0.092 0.860 0.032
#> GSM228644 2 0.2469 0.82572 0.000 0.892 0.000 0.108
#> GSM228651 3 0.4405 0.76963 0.024 0.112 0.828 0.036
#> GSM228654 3 0.4254 0.77687 0.024 0.096 0.840 0.040
#> GSM228658 3 0.4501 0.77288 0.016 0.128 0.816 0.040
#> GSM228606 3 0.5292 -0.19071 0.000 0.008 0.512 0.480
#> GSM228611 3 0.0336 0.79255 0.000 0.008 0.992 0.000
#> GSM228618 3 0.0376 0.79357 0.000 0.004 0.992 0.004
#> GSM228621 3 0.0657 0.79397 0.000 0.004 0.984 0.012
#> GSM228624 3 0.5495 0.62161 0.000 0.176 0.728 0.096
#> GSM228630 3 0.5111 0.59009 0.000 0.056 0.740 0.204
#> GSM228636 4 0.5624 0.43699 0.020 0.244 0.032 0.704
#> GSM228638 3 0.0188 0.79190 0.000 0.004 0.996 0.000
#> GSM228648 3 0.4678 0.59738 0.000 0.232 0.744 0.024
#> GSM228670 4 0.3102 0.71611 0.016 0.064 0.024 0.896
#> GSM228671 4 0.4731 0.71540 0.004 0.100 0.096 0.800
#> GSM228672 4 0.3182 0.69621 0.096 0.028 0.000 0.876
#> GSM228674 4 0.3072 0.71346 0.008 0.076 0.024 0.892
#> GSM228675 4 0.2899 0.69673 0.004 0.112 0.004 0.880
#> GSM228676 4 0.6970 0.52651 0.028 0.092 0.260 0.620
#> GSM228667 4 0.5165 0.70762 0.016 0.088 0.112 0.784
#> GSM228668 4 0.6275 0.65078 0.124 0.004 0.200 0.672
#> GSM228669 4 0.3493 0.69900 0.064 0.008 0.052 0.876
#> GSM228673 3 0.5150 0.67820 0.004 0.088 0.768 0.140
#> GSM228677 4 0.5523 0.50119 0.000 0.024 0.380 0.596
#> GSM228678 4 0.3816 0.70601 0.016 0.052 0.068 0.864
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 4 0.6830 0.3235 0.020 0.092 0.028 0.532 0.328
#> GSM228563 4 0.4419 0.5454 0.036 0.112 0.004 0.800 0.048
#> GSM228565 4 0.8732 0.0253 0.060 0.120 0.120 0.400 0.300
#> GSM228566 3 0.6332 0.4905 0.000 0.044 0.612 0.108 0.236
#> GSM228567 1 0.0981 0.7367 0.972 0.012 0.000 0.008 0.008
#> GSM228570 1 0.7434 0.3649 0.504 0.056 0.012 0.144 0.284
#> GSM228571 1 0.7120 0.5640 0.564 0.112 0.028 0.040 0.256
#> GSM228574 3 0.5798 0.5870 0.000 0.036 0.644 0.068 0.252
#> GSM228575 4 0.8342 -0.1359 0.000 0.192 0.308 0.336 0.164
#> GSM228576 5 0.8861 0.1699 0.048 0.136 0.152 0.276 0.388
#> GSM228579 1 0.5809 0.6669 0.672 0.124 0.004 0.020 0.180
#> GSM228580 4 0.5890 0.5096 0.004 0.200 0.040 0.672 0.084
#> GSM228581 2 0.6126 0.4153 0.000 0.504 0.076 0.020 0.400
#> GSM228666 2 0.7456 -0.1089 0.000 0.468 0.088 0.312 0.132
#> GSM228564 4 0.4979 0.5155 0.132 0.060 0.000 0.756 0.052
#> GSM228568 5 0.9606 0.2724 0.092 0.236 0.212 0.164 0.296
#> GSM228569 1 0.7537 0.6112 0.600 0.076 0.116 0.072 0.136
#> GSM228572 2 0.4935 0.5680 0.004 0.720 0.052 0.212 0.012
#> GSM228573 3 0.1168 0.7039 0.000 0.000 0.960 0.008 0.032
#> GSM228577 1 0.6972 0.6677 0.640 0.060 0.048 0.124 0.128
#> GSM228578 5 0.9001 0.1328 0.272 0.024 0.180 0.224 0.300
#> GSM228663 3 0.6584 0.0624 0.000 0.280 0.512 0.008 0.200
#> GSM228664 2 0.6713 0.3172 0.000 0.492 0.260 0.008 0.240
#> GSM228665 3 0.3515 0.6483 0.000 0.064 0.844 0.008 0.084
#> GSM228582 2 0.5623 0.4978 0.028 0.576 0.016 0.012 0.368
#> GSM228583 1 0.0693 0.7348 0.980 0.012 0.000 0.008 0.000
#> GSM228585 1 0.0960 0.7354 0.972 0.016 0.000 0.008 0.004
#> GSM228587 1 0.4939 0.6984 0.764 0.084 0.000 0.048 0.104
#> GSM228588 2 0.4707 0.6906 0.036 0.776 0.000 0.076 0.112
#> GSM228589 2 0.3906 0.7139 0.024 0.816 0.000 0.032 0.128
#> GSM228590 1 0.0613 0.7334 0.984 0.004 0.000 0.004 0.008
#> GSM228591 2 0.3151 0.7160 0.000 0.836 0.000 0.020 0.144
#> GSM228597 4 0.4336 0.5367 0.060 0.108 0.000 0.800 0.032
#> GSM228601 2 0.2670 0.7251 0.004 0.888 0.000 0.028 0.080
#> GSM228604 2 0.6915 -0.0607 0.000 0.484 0.340 0.036 0.140
#> GSM228608 1 0.6639 0.5470 0.608 0.040 0.016 0.100 0.236
#> GSM228609 4 0.7394 0.1725 0.076 0.396 0.004 0.416 0.108
#> GSM228613 1 0.0912 0.7395 0.972 0.016 0.000 0.000 0.012
#> GSM228616 5 0.8410 0.0131 0.008 0.212 0.120 0.328 0.332
#> GSM228628 2 0.4668 0.6531 0.000 0.748 0.008 0.076 0.168
#> GSM228634 1 0.4448 0.7357 0.800 0.060 0.008 0.024 0.108
#> GSM228642 2 0.1018 0.7229 0.000 0.968 0.000 0.016 0.016
#> GSM228645 4 0.8277 0.0206 0.000 0.260 0.180 0.384 0.176
#> GSM228646 3 0.8343 -0.2716 0.000 0.144 0.344 0.272 0.240
#> GSM228652 1 0.6871 0.5740 0.580 0.068 0.020 0.064 0.268
#> GSM228655 1 0.9011 -0.1057 0.384 0.076 0.112 0.152 0.276
#> GSM228656 1 0.0162 0.7266 0.996 0.004 0.000 0.000 0.000
#> GSM228659 4 0.8095 0.1429 0.304 0.128 0.004 0.408 0.156
#> GSM228662 1 0.1442 0.7422 0.952 0.032 0.000 0.004 0.012
#> GSM228584 1 0.2519 0.7232 0.884 0.000 0.016 0.100 0.000
#> GSM228586 1 0.4339 0.7281 0.812 0.012 0.028 0.100 0.048
#> GSM228592 1 0.2919 0.7195 0.868 0.000 0.024 0.104 0.004
#> GSM228593 4 0.7235 0.2433 0.056 0.276 0.012 0.532 0.124
#> GSM228594 1 0.7138 0.6546 0.636 0.076 0.080 0.076 0.132
#> GSM228598 1 0.6950 0.6432 0.616 0.072 0.016 0.160 0.136
#> GSM228607 3 0.8378 -0.2997 0.012 0.112 0.356 0.328 0.192
#> GSM228612 3 0.6642 0.1995 0.000 0.232 0.556 0.024 0.188
#> GSM228619 3 0.7941 -0.2664 0.048 0.012 0.364 0.328 0.248
#> GSM228622 3 0.7850 -0.1600 0.048 0.012 0.420 0.268 0.252
#> GSM228625 4 0.7941 0.1032 0.100 0.144 0.024 0.508 0.224
#> GSM228631 3 0.7882 -0.1564 0.068 0.004 0.412 0.272 0.244
#> GSM228633 2 0.3748 0.6430 0.000 0.836 0.056 0.088 0.020
#> GSM228637 4 0.2153 0.5189 0.000 0.040 0.000 0.916 0.044
#> GSM228639 3 0.2955 0.6836 0.004 0.000 0.876 0.060 0.060
#> GSM228649 4 0.6053 0.1957 0.000 0.276 0.004 0.576 0.144
#> GSM228660 5 0.8647 -0.0494 0.048 0.320 0.232 0.064 0.336
#> GSM228661 1 0.7415 0.6136 0.608 0.076 0.128 0.060 0.128
#> GSM228595 2 0.1885 0.7179 0.012 0.936 0.000 0.020 0.032
#> GSM228599 4 0.4925 0.5456 0.044 0.120 0.004 0.768 0.064
#> GSM228602 3 0.2317 0.7013 0.004 0.004 0.916 0.036 0.040
#> GSM228614 4 0.8502 0.0814 0.012 0.184 0.204 0.412 0.188
#> GSM228626 2 0.0912 0.7213 0.000 0.972 0.000 0.012 0.016
#> GSM228640 3 0.3712 0.6614 0.004 0.020 0.804 0.004 0.168
#> GSM228643 3 0.4489 0.6417 0.000 0.080 0.768 0.008 0.144
#> GSM228650 3 0.4026 0.6885 0.008 0.012 0.824 0.068 0.088
#> GSM228653 3 0.3492 0.6753 0.000 0.016 0.796 0.000 0.188
#> GSM228657 2 0.0992 0.7267 0.000 0.968 0.000 0.024 0.008
#> GSM228605 4 0.7120 -0.2464 0.012 0.000 0.336 0.364 0.288
#> GSM228610 3 0.0865 0.6992 0.000 0.000 0.972 0.004 0.024
#> GSM228617 3 0.1728 0.7012 0.004 0.000 0.940 0.036 0.020
#> GSM228620 3 0.0798 0.7027 0.000 0.000 0.976 0.008 0.016
#> GSM228623 4 0.3338 0.5197 0.000 0.076 0.004 0.852 0.068
#> GSM228629 3 0.0451 0.6996 0.000 0.000 0.988 0.004 0.008
#> GSM228632 3 0.2807 0.6817 0.000 0.020 0.892 0.032 0.056
#> GSM228635 4 0.2879 0.4846 0.000 0.032 0.008 0.880 0.080
#> GSM228647 3 0.0771 0.6982 0.000 0.000 0.976 0.004 0.020
#> GSM228596 3 0.6961 0.3683 0.004 0.056 0.564 0.136 0.240
#> GSM228600 3 0.3597 0.6706 0.000 0.012 0.800 0.008 0.180
#> GSM228603 3 0.3048 0.6700 0.000 0.004 0.820 0.000 0.176
#> GSM228615 4 0.4159 0.5448 0.020 0.160 0.000 0.788 0.032
#> GSM228627 3 0.6834 0.2802 0.000 0.188 0.472 0.016 0.324
#> GSM228641 3 0.2787 0.6874 0.000 0.004 0.856 0.004 0.136
#> GSM228644 2 0.0912 0.7213 0.000 0.972 0.000 0.012 0.016
#> GSM228651 3 0.3437 0.6714 0.000 0.012 0.808 0.004 0.176
#> GSM228654 3 0.3391 0.6748 0.000 0.012 0.800 0.000 0.188
#> GSM228658 3 0.3456 0.6706 0.000 0.004 0.788 0.004 0.204
#> GSM228606 3 0.5553 0.3647 0.000 0.008 0.660 0.216 0.116
#> GSM228611 3 0.0609 0.6975 0.000 0.000 0.980 0.000 0.020
#> GSM228618 3 0.1195 0.6999 0.000 0.000 0.960 0.012 0.028
#> GSM228621 3 0.1560 0.7050 0.000 0.004 0.948 0.028 0.020
#> GSM228624 3 0.4919 0.5901 0.000 0.084 0.768 0.056 0.092
#> GSM228630 3 0.3553 0.6551 0.000 0.028 0.852 0.072 0.048
#> GSM228636 4 0.2914 0.4787 0.000 0.052 0.000 0.872 0.076
#> GSM228638 3 0.0703 0.6996 0.000 0.000 0.976 0.000 0.024
#> GSM228648 3 0.4316 0.5735 0.000 0.152 0.780 0.012 0.056
#> GSM228670 4 0.5496 0.5383 0.060 0.108 0.004 0.732 0.096
#> GSM228671 4 0.7368 0.3388 0.004 0.128 0.148 0.560 0.160
#> GSM228672 4 0.5868 0.5046 0.140 0.096 0.000 0.692 0.072
#> GSM228674 4 0.5484 0.5093 0.004 0.192 0.008 0.684 0.112
#> GSM228675 4 0.3863 0.5354 0.000 0.200 0.000 0.772 0.028
#> GSM228676 5 0.7931 0.1558 0.000 0.080 0.332 0.236 0.352
#> GSM228667 4 0.7307 0.2920 0.000 0.160 0.088 0.532 0.220
#> GSM228668 4 0.8293 -0.1903 0.136 0.008 0.168 0.400 0.288
#> GSM228669 4 0.5255 0.3834 0.044 0.024 0.008 0.704 0.220
#> GSM228673 3 0.5086 0.5794 0.000 0.064 0.756 0.076 0.104
#> GSM228677 4 0.6659 -0.0343 0.000 0.052 0.408 0.464 0.076
#> GSM228678 4 0.3691 0.5187 0.000 0.072 0.056 0.844 0.028
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 5 0.7057 0.3868 0.012 0.060 0.040 0.204 0.556 0.128
#> GSM228563 4 0.3963 0.6499 0.016 0.092 0.012 0.804 0.076 0.000
#> GSM228565 5 0.6978 0.4660 0.016 0.080 0.092 0.140 0.604 0.068
#> GSM228566 3 0.4885 0.6875 0.000 0.012 0.708 0.024 0.060 0.196
#> GSM228567 1 0.0806 0.7659 0.972 0.020 0.000 0.000 0.000 0.008
#> GSM228570 5 0.6532 0.0985 0.360 0.048 0.012 0.044 0.500 0.036
#> GSM228571 1 0.7640 0.3984 0.452 0.040 0.040 0.024 0.256 0.188
#> GSM228574 3 0.4233 0.7103 0.000 0.008 0.724 0.004 0.040 0.224
#> GSM228575 3 0.8198 0.0820 0.000 0.128 0.412 0.216 0.144 0.100
#> GSM228576 5 0.7036 0.4636 0.016 0.076 0.132 0.052 0.592 0.132
#> GSM228579 1 0.5899 0.6809 0.656 0.052 0.000 0.024 0.120 0.148
#> GSM228580 4 0.4470 0.5920 0.000 0.176 0.024 0.748 0.032 0.020
#> GSM228581 6 0.6368 0.4343 0.000 0.260 0.068 0.012 0.100 0.560
#> GSM228666 2 0.8453 -0.2501 0.000 0.324 0.128 0.168 0.280 0.100
#> GSM228564 4 0.3962 0.6180 0.084 0.052 0.004 0.808 0.052 0.000
#> GSM228568 5 0.7900 -0.1539 0.036 0.076 0.216 0.016 0.424 0.232
#> GSM228569 1 0.6188 0.6395 0.608 0.000 0.136 0.004 0.160 0.092
#> GSM228572 2 0.4893 0.4815 0.000 0.736 0.056 0.148 0.040 0.020
#> GSM228573 3 0.1168 0.7445 0.000 0.000 0.956 0.000 0.016 0.028
#> GSM228577 1 0.5453 0.6974 0.620 0.004 0.016 0.016 0.284 0.060
#> GSM228578 5 0.4958 0.3606 0.168 0.000 0.104 0.012 0.704 0.012
#> GSM228663 3 0.5648 0.0633 0.000 0.088 0.544 0.000 0.028 0.340
#> GSM228664 6 0.6004 0.4603 0.000 0.208 0.248 0.000 0.016 0.528
#> GSM228665 3 0.2637 0.7192 0.000 0.008 0.872 0.000 0.024 0.096
#> GSM228582 6 0.4987 0.3248 0.008 0.308 0.004 0.004 0.052 0.624
#> GSM228583 1 0.0405 0.7641 0.988 0.008 0.000 0.004 0.000 0.000
#> GSM228585 1 0.0748 0.7652 0.976 0.016 0.000 0.004 0.000 0.004
#> GSM228587 1 0.4900 0.6829 0.720 0.032 0.000 0.028 0.184 0.036
#> GSM228588 2 0.6287 0.4552 0.016 0.604 0.000 0.068 0.124 0.188
#> GSM228589 2 0.5557 0.5083 0.012 0.656 0.000 0.044 0.080 0.208
#> GSM228590 1 0.0363 0.7649 0.988 0.000 0.000 0.000 0.012 0.000
#> GSM228591 2 0.4680 0.3797 0.000 0.628 0.000 0.012 0.040 0.320
#> GSM228597 4 0.4708 0.6458 0.040 0.088 0.004 0.744 0.124 0.000
#> GSM228601 2 0.4269 0.5717 0.000 0.752 0.000 0.024 0.056 0.168
#> GSM228604 3 0.6323 0.4241 0.000 0.200 0.528 0.012 0.020 0.240
#> GSM228608 1 0.5353 0.4357 0.568 0.028 0.008 0.012 0.364 0.020
#> GSM228609 5 0.7408 0.2282 0.060 0.188 0.000 0.196 0.492 0.064
#> GSM228613 1 0.0458 0.7673 0.984 0.000 0.000 0.000 0.016 0.000
#> GSM228616 5 0.7057 0.4613 0.004 0.108 0.072 0.104 0.584 0.128
#> GSM228628 2 0.6013 0.1013 0.000 0.520 0.004 0.016 0.152 0.308
#> GSM228634 1 0.3777 0.7598 0.820 0.020 0.000 0.012 0.060 0.088
#> GSM228642 2 0.0603 0.6496 0.000 0.980 0.000 0.004 0.000 0.016
#> GSM228645 5 0.8462 0.2582 0.000 0.184 0.252 0.136 0.336 0.092
#> GSM228646 3 0.8085 0.1425 0.000 0.108 0.424 0.096 0.220 0.152
#> GSM228652 1 0.6745 0.4312 0.488 0.040 0.012 0.036 0.352 0.072
#> GSM228655 5 0.6166 0.3510 0.276 0.044 0.076 0.020 0.580 0.004
#> GSM228656 1 0.0000 0.7597 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228659 5 0.6554 0.3822 0.156 0.060 0.004 0.184 0.580 0.016
#> GSM228662 1 0.0692 0.7693 0.976 0.004 0.000 0.000 0.020 0.000
#> GSM228584 1 0.2446 0.7542 0.864 0.012 0.000 0.000 0.124 0.000
#> GSM228586 1 0.3472 0.7582 0.804 0.012 0.004 0.000 0.160 0.020
#> GSM228592 1 0.2584 0.7480 0.848 0.004 0.004 0.000 0.144 0.000
#> GSM228593 5 0.5969 0.3388 0.008 0.120 0.000 0.204 0.616 0.052
#> GSM228594 1 0.5904 0.6934 0.644 0.004 0.084 0.004 0.172 0.092
#> GSM228598 1 0.6071 0.5465 0.504 0.020 0.004 0.040 0.384 0.048
#> GSM228607 3 0.7413 0.2167 0.000 0.052 0.468 0.116 0.276 0.088
#> GSM228612 3 0.5308 0.3632 0.000 0.076 0.640 0.004 0.028 0.252
#> GSM228619 5 0.6237 -0.0902 0.024 0.000 0.420 0.064 0.452 0.040
#> GSM228622 3 0.5904 0.2869 0.020 0.000 0.500 0.040 0.396 0.044
#> GSM228625 5 0.5160 0.4282 0.024 0.052 0.008 0.148 0.728 0.040
#> GSM228631 3 0.6055 0.2389 0.028 0.000 0.476 0.040 0.412 0.044
#> GSM228633 2 0.2807 0.5469 0.000 0.880 0.056 0.040 0.020 0.004
#> GSM228637 4 0.3894 0.4812 0.000 0.000 0.004 0.664 0.324 0.008
#> GSM228639 3 0.1893 0.7467 0.000 0.004 0.928 0.008 0.036 0.024
#> GSM228649 5 0.6179 0.2819 0.000 0.100 0.004 0.272 0.560 0.064
#> GSM228660 6 0.7952 0.3977 0.032 0.104 0.216 0.004 0.288 0.356
#> GSM228661 1 0.6117 0.6408 0.616 0.000 0.140 0.004 0.152 0.088
#> GSM228595 2 0.1036 0.6428 0.008 0.964 0.000 0.024 0.000 0.004
#> GSM228599 4 0.4911 0.6399 0.012 0.096 0.008 0.728 0.144 0.012
#> GSM228602 3 0.2321 0.7519 0.000 0.008 0.900 0.000 0.040 0.052
#> GSM228614 3 0.8519 -0.3222 0.008 0.120 0.300 0.252 0.256 0.064
#> GSM228626 2 0.0363 0.6463 0.000 0.988 0.000 0.000 0.000 0.012
#> GSM228640 3 0.3229 0.7198 0.000 0.008 0.796 0.004 0.004 0.188
#> GSM228643 3 0.3807 0.7116 0.004 0.044 0.784 0.000 0.008 0.160
#> GSM228650 3 0.3260 0.7492 0.004 0.020 0.856 0.012 0.024 0.084
#> GSM228653 3 0.2994 0.7267 0.000 0.000 0.788 0.000 0.004 0.208
#> GSM228657 2 0.1760 0.6460 0.000 0.936 0.004 0.020 0.012 0.028
#> GSM228605 5 0.6049 0.3464 0.000 0.012 0.320 0.068 0.548 0.052
#> GSM228610 3 0.0972 0.7402 0.000 0.000 0.964 0.000 0.008 0.028
#> GSM228617 3 0.1257 0.7469 0.000 0.000 0.952 0.000 0.028 0.020
#> GSM228620 3 0.0914 0.7444 0.000 0.000 0.968 0.000 0.016 0.016
#> GSM228623 4 0.5016 0.5105 0.000 0.052 0.008 0.608 0.324 0.008
#> GSM228629 3 0.0603 0.7419 0.000 0.000 0.980 0.000 0.004 0.016
#> GSM228632 3 0.1716 0.7418 0.000 0.000 0.932 0.004 0.028 0.036
#> GSM228635 4 0.2138 0.5611 0.000 0.012 0.008 0.912 0.060 0.008
#> GSM228647 3 0.1074 0.7409 0.000 0.000 0.960 0.000 0.012 0.028
#> GSM228596 3 0.5864 0.6555 0.004 0.044 0.676 0.036 0.104 0.136
#> GSM228600 3 0.3327 0.7279 0.000 0.004 0.792 0.004 0.012 0.188
#> GSM228603 3 0.2871 0.7254 0.000 0.004 0.804 0.000 0.000 0.192
#> GSM228615 4 0.5616 0.6063 0.012 0.108 0.008 0.656 0.196 0.020
#> GSM228627 3 0.5528 0.4458 0.000 0.068 0.520 0.000 0.028 0.384
#> GSM228641 3 0.2914 0.7399 0.000 0.004 0.832 0.004 0.008 0.152
#> GSM228644 2 0.0363 0.6463 0.000 0.988 0.000 0.000 0.000 0.012
#> GSM228651 3 0.3183 0.7237 0.000 0.004 0.788 0.000 0.008 0.200
#> GSM228654 3 0.3183 0.7311 0.000 0.004 0.788 0.000 0.008 0.200
#> GSM228658 3 0.3217 0.7240 0.000 0.000 0.768 0.000 0.008 0.224
#> GSM228606 3 0.4258 0.6322 0.000 0.000 0.768 0.100 0.108 0.024
#> GSM228611 3 0.1082 0.7383 0.000 0.000 0.956 0.000 0.004 0.040
#> GSM228618 3 0.1225 0.7419 0.000 0.000 0.952 0.000 0.012 0.036
#> GSM228621 3 0.0777 0.7468 0.000 0.000 0.972 0.004 0.000 0.024
#> GSM228624 3 0.3044 0.7072 0.000 0.028 0.864 0.008 0.020 0.080
#> GSM228630 3 0.1942 0.7408 0.000 0.004 0.928 0.028 0.020 0.020
#> GSM228636 4 0.1914 0.5618 0.000 0.016 0.000 0.920 0.056 0.008
#> GSM228638 3 0.0777 0.7423 0.000 0.000 0.972 0.000 0.004 0.024
#> GSM228648 3 0.2772 0.7147 0.000 0.060 0.876 0.000 0.016 0.048
#> GSM228670 4 0.5911 0.5424 0.016 0.088 0.012 0.612 0.252 0.020
#> GSM228671 4 0.7645 0.2144 0.000 0.124 0.260 0.452 0.108 0.056
#> GSM228672 5 0.6346 -0.0797 0.108 0.032 0.004 0.396 0.452 0.008
#> GSM228674 4 0.6389 0.1215 0.000 0.108 0.008 0.432 0.408 0.044
#> GSM228675 4 0.5711 0.5657 0.000 0.148 0.008 0.608 0.220 0.016
#> GSM228676 3 0.7419 0.1322 0.004 0.036 0.408 0.048 0.320 0.184
#> GSM228667 5 0.7937 0.1952 0.000 0.084 0.100 0.276 0.412 0.128
#> GSM228668 5 0.5528 0.4532 0.064 0.000 0.116 0.068 0.708 0.044
#> GSM228669 5 0.4406 0.3443 0.020 0.016 0.008 0.240 0.712 0.004
#> GSM228673 3 0.3082 0.7137 0.000 0.008 0.860 0.012 0.040 0.080
#> GSM228677 4 0.5833 0.1153 0.000 0.004 0.440 0.448 0.080 0.028
#> GSM228678 4 0.4774 0.6301 0.000 0.056 0.052 0.732 0.156 0.004
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
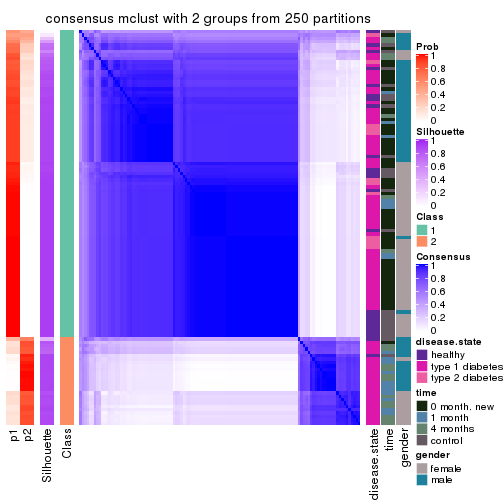
consensus_heatmap(res, k = 3)
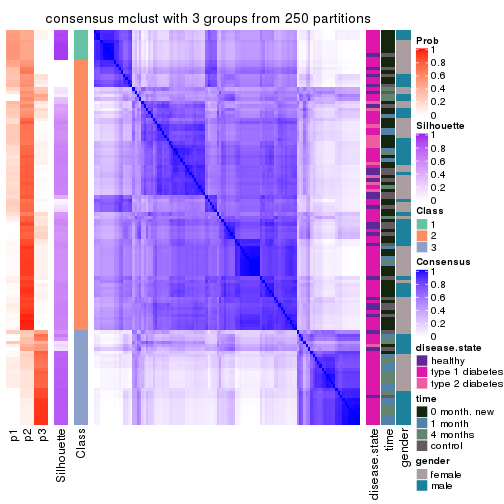
consensus_heatmap(res, k = 4)
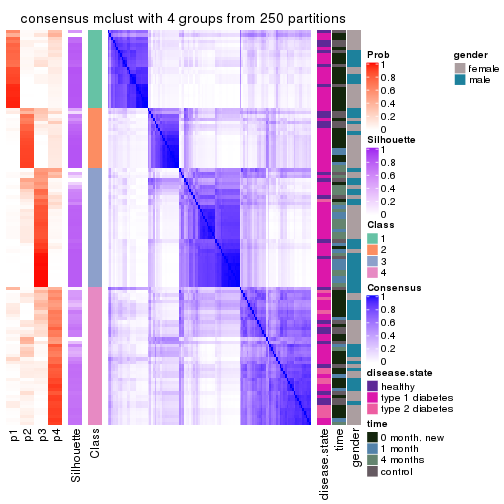
consensus_heatmap(res, k = 5)
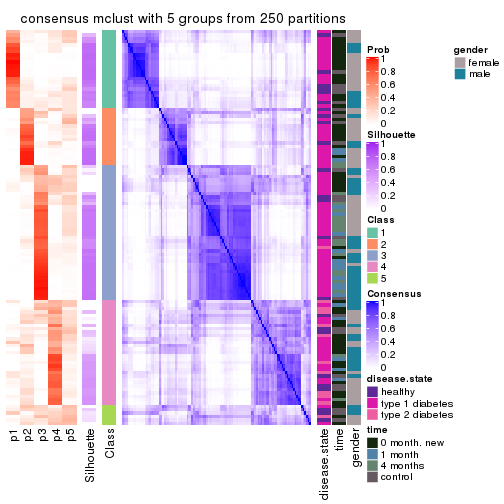
consensus_heatmap(res, k = 6)
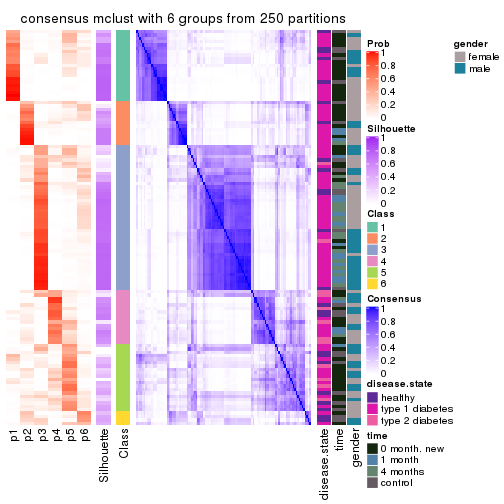
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
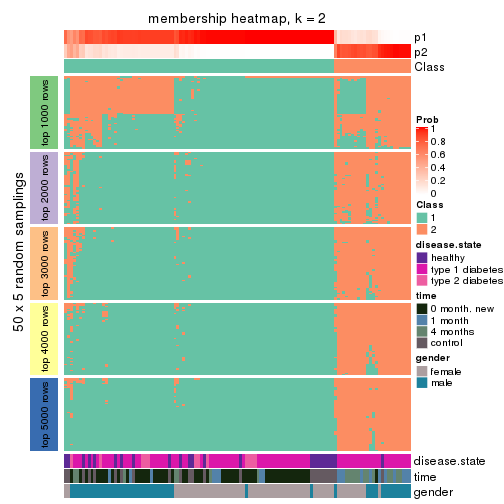
membership_heatmap(res, k = 3)
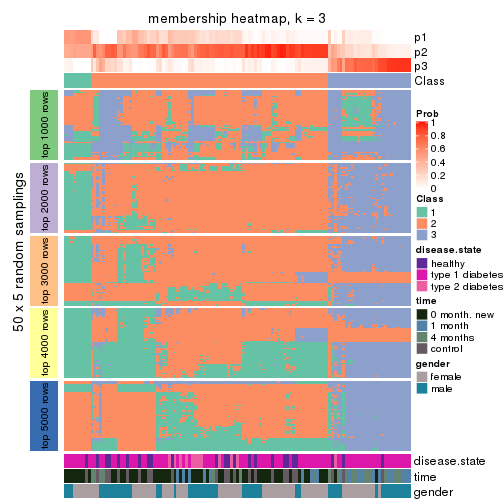
membership_heatmap(res, k = 4)

membership_heatmap(res, k = 5)
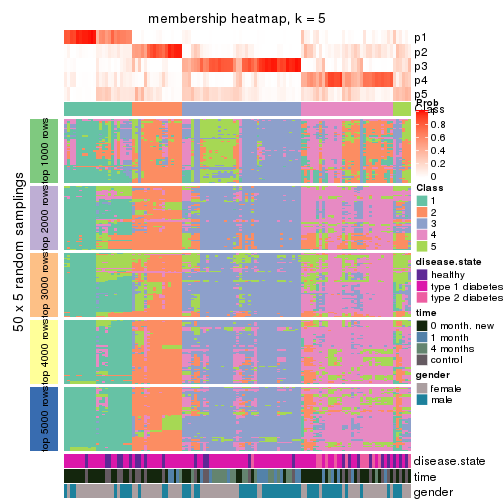
membership_heatmap(res, k = 6)
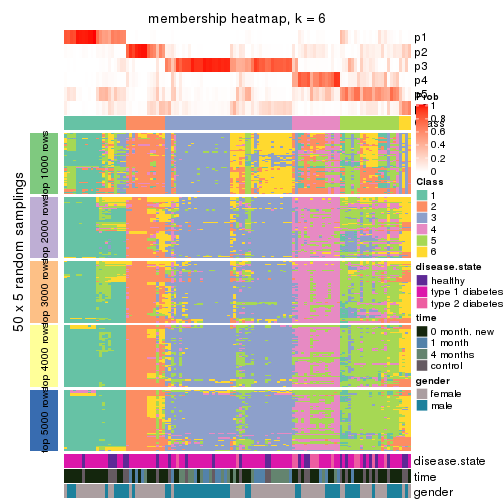
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
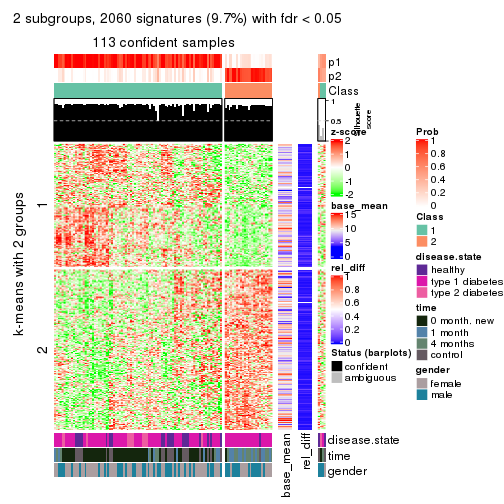
get_signatures(res, k = 3)
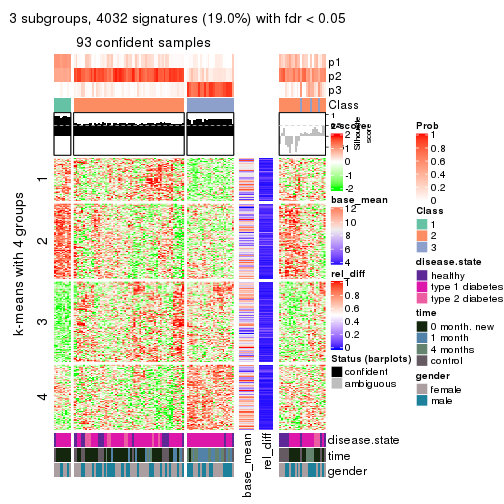
get_signatures(res, k = 4)
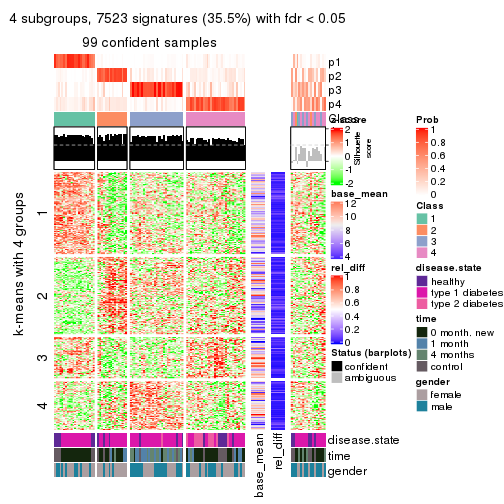
get_signatures(res, k = 5)
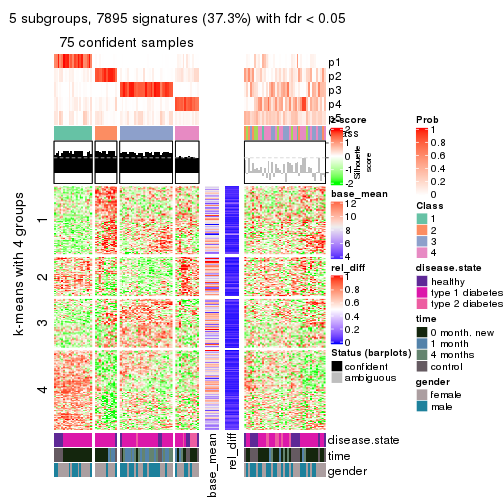
get_signatures(res, k = 6)
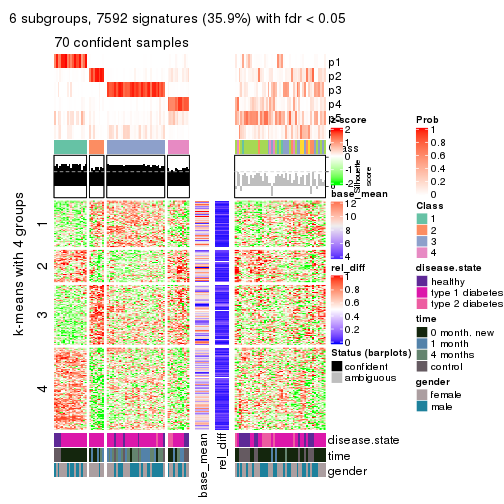
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
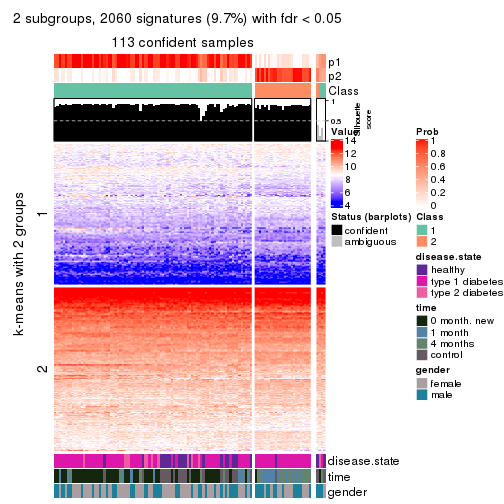
get_signatures(res, k = 3, scale_rows = FALSE)
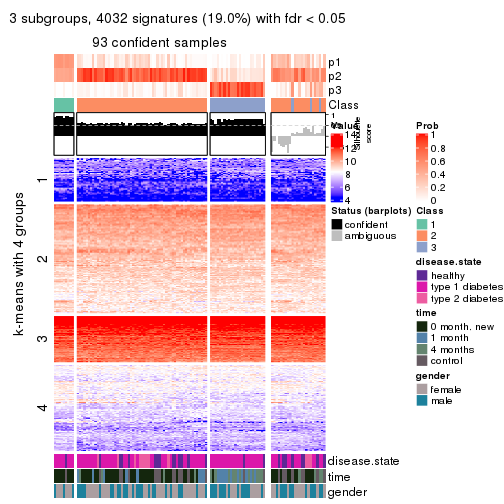
get_signatures(res, k = 4, scale_rows = FALSE)
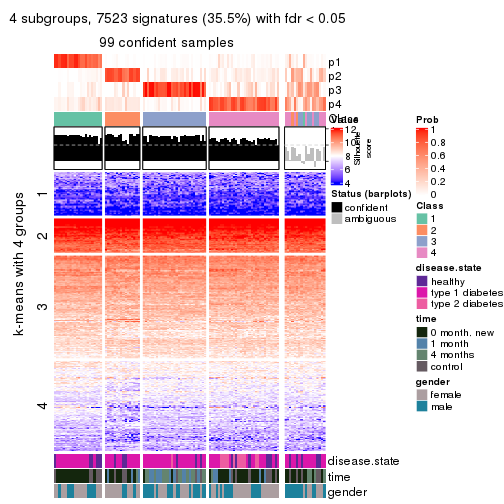
get_signatures(res, k = 5, scale_rows = FALSE)
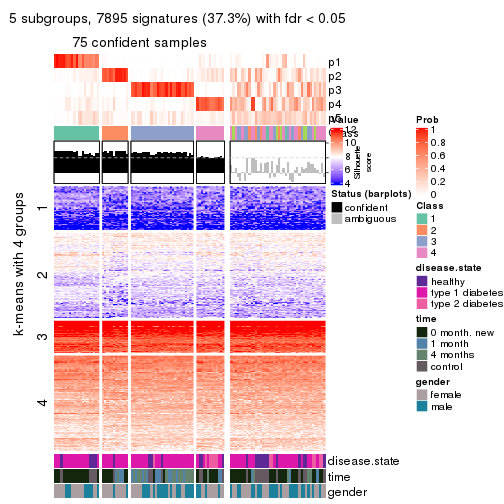
get_signatures(res, k = 6, scale_rows = FALSE)
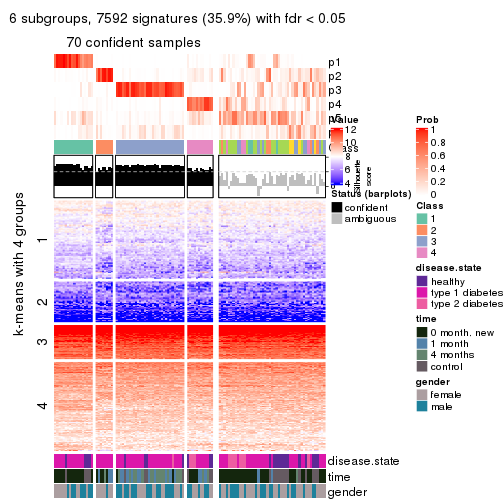
Compare the overlap of signatures from different k:
compare_signatures(res)
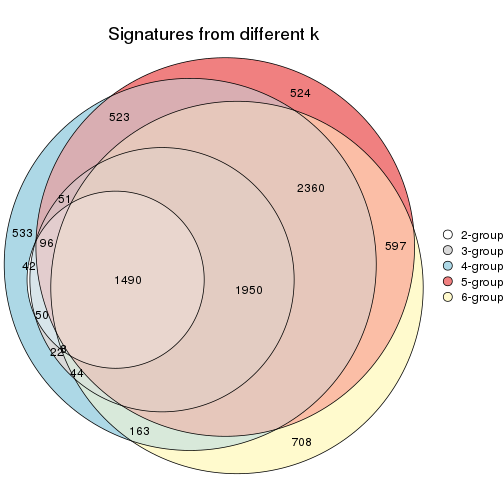
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
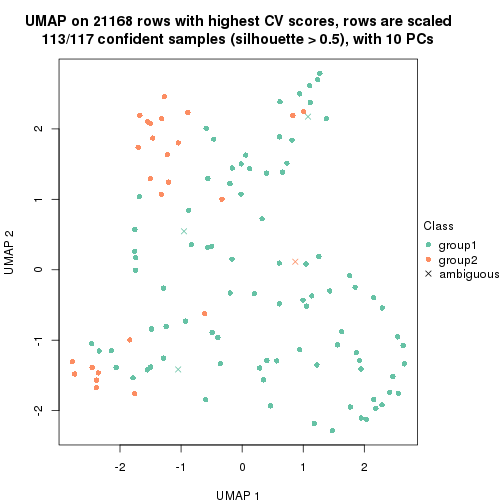
dimension_reduction(res, k = 3, method = "UMAP")
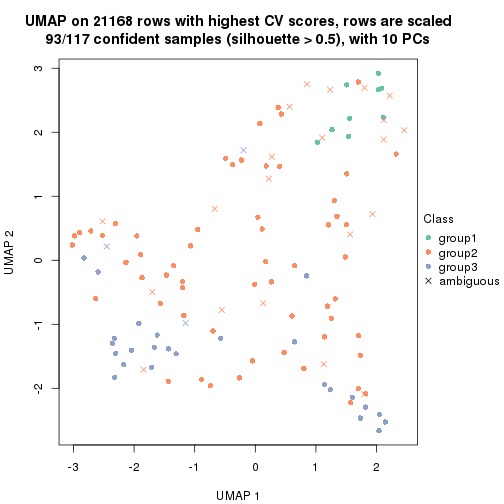
dimension_reduction(res, k = 4, method = "UMAP")
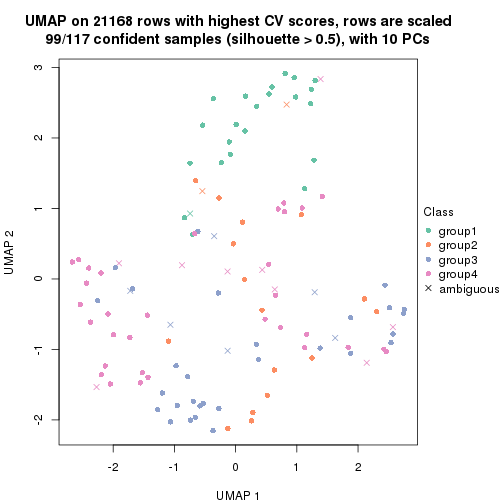
dimension_reduction(res, k = 5, method = "UMAP")
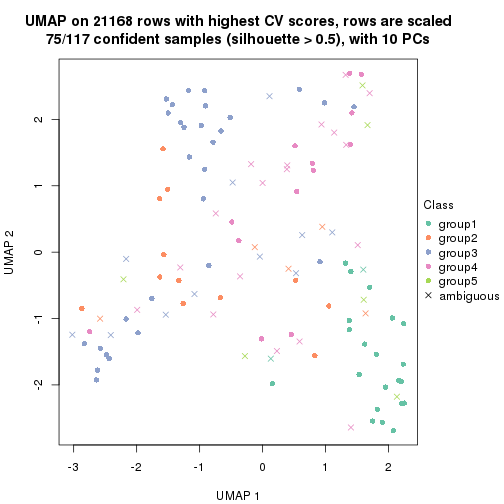
dimension_reduction(res, k = 6, method = "UMAP")
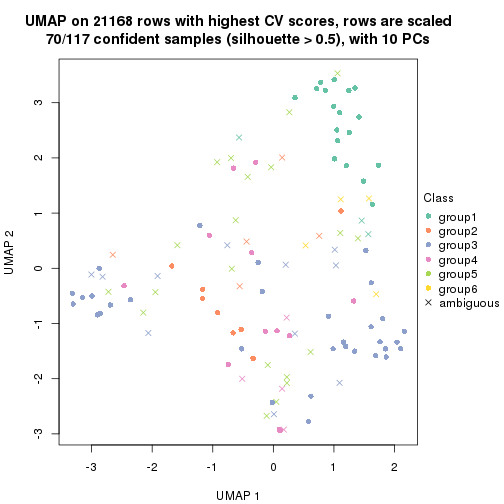
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
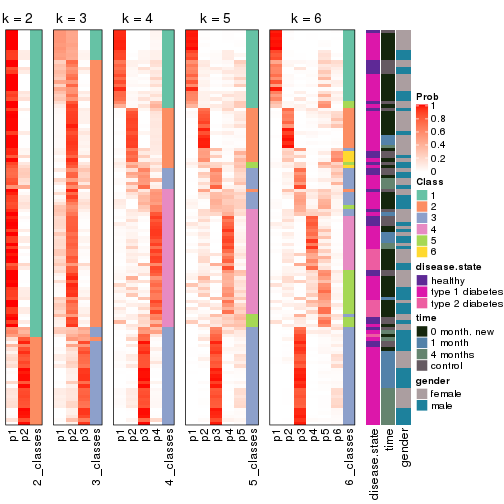
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> CV:mclust 113 0.006905 3.53e-11 0.2239 2
#> CV:mclust 93 0.043742 8.26e-08 0.5223 3
#> CV:mclust 99 0.000209 9.09e-08 0.0609 4
#> CV:mclust 75 0.000546 5.04e-06 0.0637 5
#> CV:mclust 70 0.028154 1.84e-05 0.1661 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["CV", "NMF"]
# you can also extract it by
# res = res_list["CV:NMF"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'CV' method.
#> Subgroups are detected by 'NMF' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
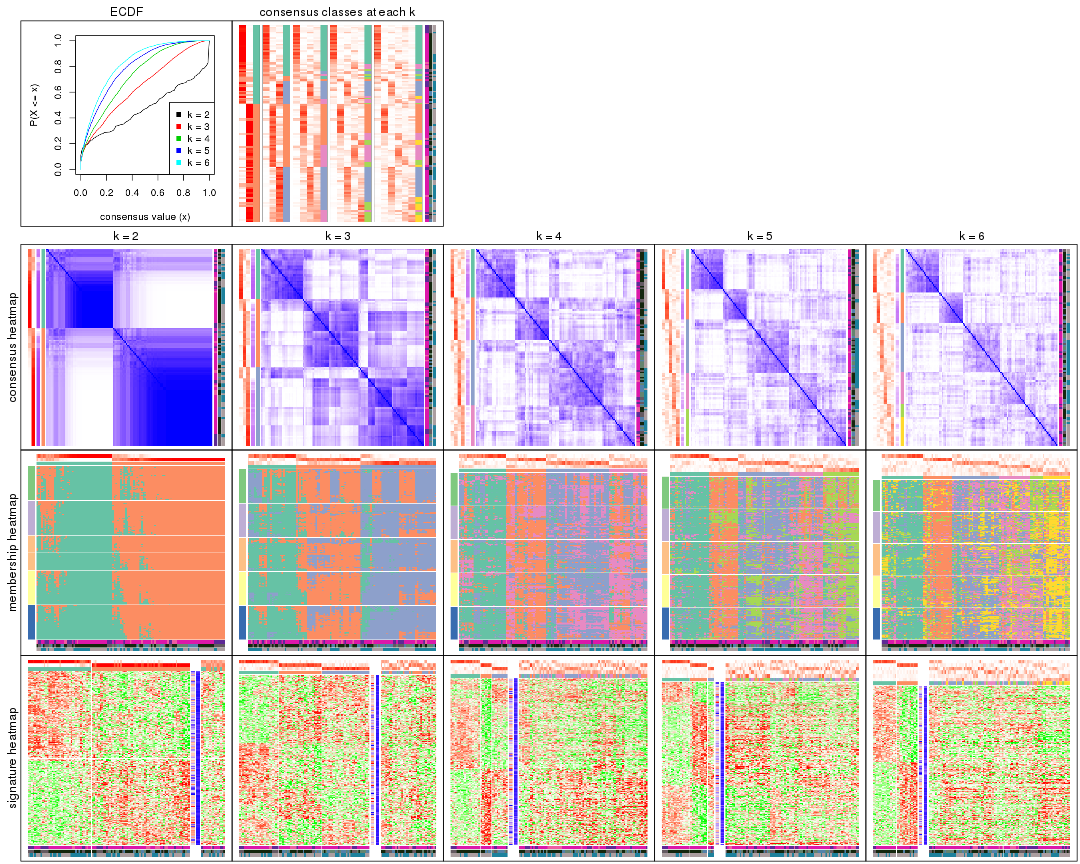
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
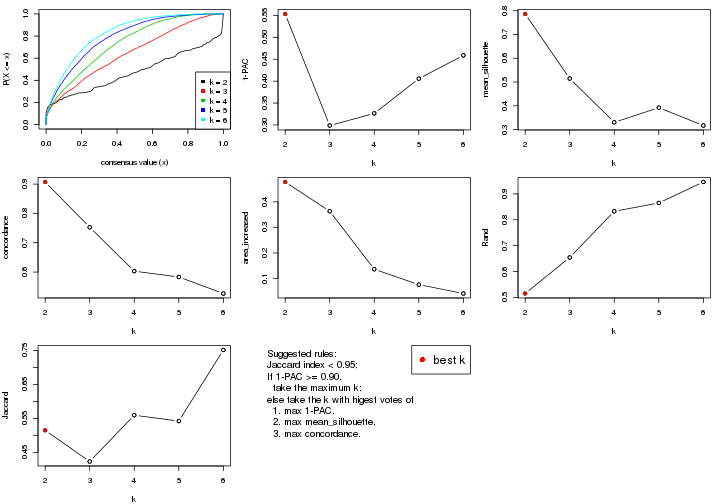
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.553 0.786 0.907 0.4778 0.515 0.515
#> 3 3 0.299 0.515 0.753 0.3634 0.654 0.423
#> 4 4 0.326 0.331 0.603 0.1363 0.833 0.560
#> 5 5 0.406 0.392 0.583 0.0762 0.865 0.542
#> 6 6 0.459 0.317 0.527 0.0415 0.946 0.751
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.6148 0.7886 0.848 0.152
#> GSM228563 2 0.0376 0.9052 0.004 0.996
#> GSM228565 1 0.9996 0.1504 0.512 0.488
#> GSM228566 2 0.0376 0.9051 0.004 0.996
#> GSM228567 1 0.0000 0.8721 1.000 0.000
#> GSM228570 1 0.0672 0.8698 0.992 0.008
#> GSM228571 1 0.2043 0.8614 0.968 0.032
#> GSM228574 2 0.0000 0.9062 0.000 1.000
#> GSM228575 2 0.0000 0.9062 0.000 1.000
#> GSM228576 1 0.8386 0.6599 0.732 0.268
#> GSM228579 1 0.1184 0.8673 0.984 0.016
#> GSM228580 2 0.0000 0.9062 0.000 1.000
#> GSM228581 2 0.0000 0.9062 0.000 1.000
#> GSM228666 2 0.0000 0.9062 0.000 1.000
#> GSM228564 2 0.9427 0.4597 0.360 0.640
#> GSM228568 1 0.9732 0.4069 0.596 0.404
#> GSM228569 1 0.0000 0.8721 1.000 0.000
#> GSM228572 2 0.0000 0.9062 0.000 1.000
#> GSM228573 1 0.8207 0.6727 0.744 0.256
#> GSM228577 1 0.0000 0.8721 1.000 0.000
#> GSM228578 1 0.0376 0.8714 0.996 0.004
#> GSM228663 2 0.9881 0.1447 0.436 0.564
#> GSM228664 2 0.0000 0.9062 0.000 1.000
#> GSM228665 1 0.9248 0.5306 0.660 0.340
#> GSM228582 1 0.9977 0.2128 0.528 0.472
#> GSM228583 1 0.0000 0.8721 1.000 0.000
#> GSM228585 1 0.0000 0.8721 1.000 0.000
#> GSM228587 1 0.0000 0.8721 1.000 0.000
#> GSM228588 2 0.0000 0.9062 0.000 1.000
#> GSM228589 2 0.0000 0.9062 0.000 1.000
#> GSM228590 1 0.0000 0.8721 1.000 0.000
#> GSM228591 2 0.0000 0.9062 0.000 1.000
#> GSM228597 2 0.3431 0.8731 0.064 0.936
#> GSM228601 2 0.0000 0.9062 0.000 1.000
#> GSM228604 2 0.0000 0.9062 0.000 1.000
#> GSM228608 1 0.0000 0.8721 1.000 0.000
#> GSM228609 2 0.9248 0.4890 0.340 0.660
#> GSM228613 1 0.0000 0.8721 1.000 0.000
#> GSM228616 2 0.8327 0.6152 0.264 0.736
#> GSM228628 2 0.0000 0.9062 0.000 1.000
#> GSM228634 1 0.0000 0.8721 1.000 0.000
#> GSM228642 2 0.0000 0.9062 0.000 1.000
#> GSM228645 2 0.0000 0.9062 0.000 1.000
#> GSM228646 2 0.0000 0.9062 0.000 1.000
#> GSM228652 1 0.0376 0.8713 0.996 0.004
#> GSM228655 1 0.0000 0.8721 1.000 0.000
#> GSM228656 1 0.0000 0.8721 1.000 0.000
#> GSM228659 1 0.0000 0.8721 1.000 0.000
#> GSM228662 1 0.0000 0.8721 1.000 0.000
#> GSM228584 1 0.0000 0.8721 1.000 0.000
#> GSM228586 1 0.0000 0.8721 1.000 0.000
#> GSM228592 1 0.0000 0.8721 1.000 0.000
#> GSM228593 1 0.8443 0.6221 0.728 0.272
#> GSM228594 1 0.0000 0.8721 1.000 0.000
#> GSM228598 1 0.0000 0.8721 1.000 0.000
#> GSM228607 2 0.6712 0.7600 0.176 0.824
#> GSM228612 2 0.0376 0.9052 0.004 0.996
#> GSM228619 1 0.8207 0.6539 0.744 0.256
#> GSM228622 1 0.0000 0.8721 1.000 0.000
#> GSM228625 1 0.5629 0.8013 0.868 0.132
#> GSM228631 1 0.0376 0.8714 0.996 0.004
#> GSM228633 2 0.0000 0.9062 0.000 1.000
#> GSM228637 2 0.5294 0.8232 0.120 0.880
#> GSM228639 2 0.2423 0.8864 0.040 0.960
#> GSM228649 2 0.8327 0.6396 0.264 0.736
#> GSM228660 1 0.7815 0.7029 0.768 0.232
#> GSM228661 1 0.0000 0.8721 1.000 0.000
#> GSM228595 2 0.0000 0.9062 0.000 1.000
#> GSM228599 2 0.0376 0.9049 0.004 0.996
#> GSM228602 2 0.8763 0.5746 0.296 0.704
#> GSM228614 2 0.0000 0.9062 0.000 1.000
#> GSM228626 2 0.0000 0.9062 0.000 1.000
#> GSM228640 2 0.9977 -0.0192 0.472 0.528
#> GSM228643 2 0.3584 0.8643 0.068 0.932
#> GSM228650 2 0.0000 0.9062 0.000 1.000
#> GSM228653 1 0.9608 0.4562 0.616 0.384
#> GSM228657 2 0.0000 0.9062 0.000 1.000
#> GSM228605 1 0.4431 0.8333 0.908 0.092
#> GSM228610 2 0.4690 0.8354 0.100 0.900
#> GSM228617 2 0.9358 0.4713 0.352 0.648
#> GSM228620 1 0.4690 0.8262 0.900 0.100
#> GSM228623 2 0.4022 0.8584 0.080 0.920
#> GSM228629 1 0.9608 0.4365 0.616 0.384
#> GSM228632 2 0.0000 0.9062 0.000 1.000
#> GSM228635 2 0.0672 0.9037 0.008 0.992
#> GSM228647 2 0.5294 0.8259 0.120 0.880
#> GSM228596 2 0.9754 0.2604 0.408 0.592
#> GSM228600 2 0.0000 0.9062 0.000 1.000
#> GSM228603 1 0.9988 0.1753 0.520 0.480
#> GSM228615 2 0.0376 0.9049 0.004 0.996
#> GSM228627 2 0.3114 0.8742 0.056 0.944
#> GSM228641 2 0.0672 0.9036 0.008 0.992
#> GSM228644 2 0.0000 0.9062 0.000 1.000
#> GSM228651 2 0.1633 0.8957 0.024 0.976
#> GSM228654 2 0.0000 0.9062 0.000 1.000
#> GSM228658 2 0.9775 0.2218 0.412 0.588
#> GSM228606 2 0.0672 0.9038 0.008 0.992
#> GSM228611 2 0.9710 0.2638 0.400 0.600
#> GSM228618 2 0.6801 0.7656 0.180 0.820
#> GSM228621 2 0.0000 0.9062 0.000 1.000
#> GSM228624 2 0.0000 0.9062 0.000 1.000
#> GSM228630 2 0.0000 0.9062 0.000 1.000
#> GSM228636 2 0.1184 0.9000 0.016 0.984
#> GSM228638 2 0.3733 0.8641 0.072 0.928
#> GSM228648 2 0.0000 0.9062 0.000 1.000
#> GSM228670 2 0.5178 0.8296 0.116 0.884
#> GSM228671 2 0.0000 0.9062 0.000 1.000
#> GSM228672 1 0.3431 0.8460 0.936 0.064
#> GSM228674 2 0.8144 0.6547 0.252 0.748
#> GSM228675 2 0.1184 0.9006 0.016 0.984
#> GSM228676 1 0.9522 0.4798 0.628 0.372
#> GSM228667 2 0.1633 0.8971 0.024 0.976
#> GSM228668 1 0.0000 0.8721 1.000 0.000
#> GSM228669 1 0.2948 0.8524 0.948 0.052
#> GSM228673 2 0.0000 0.9062 0.000 1.000
#> GSM228677 2 0.0000 0.9062 0.000 1.000
#> GSM228678 2 0.0376 0.9052 0.004 0.996
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.708 0.5191 0.628 0.336 0.036
#> GSM228563 2 0.318 0.6556 0.064 0.912 0.024
#> GSM228565 2 0.691 0.1114 0.396 0.584 0.020
#> GSM228566 3 0.618 0.3105 0.000 0.416 0.584
#> GSM228567 1 0.311 0.7657 0.900 0.004 0.096
#> GSM228570 1 0.465 0.7136 0.816 0.176 0.008
#> GSM228571 1 0.485 0.7422 0.836 0.128 0.036
#> GSM228574 3 0.629 0.2010 0.000 0.464 0.536
#> GSM228575 3 0.668 0.1152 0.008 0.488 0.504
#> GSM228576 1 0.801 0.5475 0.624 0.276 0.100
#> GSM228579 1 0.471 0.7578 0.848 0.108 0.044
#> GSM228580 2 0.576 0.4173 0.000 0.672 0.328
#> GSM228581 2 0.627 0.1174 0.000 0.544 0.456
#> GSM228666 2 0.525 0.5351 0.000 0.736 0.264
#> GSM228564 2 0.628 0.3286 0.324 0.664 0.012
#> GSM228568 3 0.878 -0.0862 0.368 0.120 0.512
#> GSM228569 3 0.652 -0.2938 0.488 0.004 0.508
#> GSM228572 2 0.465 0.6220 0.000 0.792 0.208
#> GSM228573 3 0.153 0.6376 0.040 0.000 0.960
#> GSM228577 1 0.601 0.6989 0.748 0.032 0.220
#> GSM228578 1 0.653 0.4810 0.588 0.008 0.404
#> GSM228663 3 0.148 0.6445 0.020 0.012 0.968
#> GSM228664 3 0.455 0.5970 0.000 0.200 0.800
#> GSM228665 3 0.220 0.6297 0.056 0.004 0.940
#> GSM228582 2 0.952 -0.0505 0.388 0.424 0.188
#> GSM228583 1 0.148 0.7770 0.968 0.012 0.020
#> GSM228585 1 0.140 0.7781 0.968 0.004 0.028
#> GSM228587 1 0.341 0.7308 0.876 0.124 0.000
#> GSM228588 2 0.478 0.5175 0.200 0.796 0.004
#> GSM228589 2 0.227 0.6670 0.040 0.944 0.016
#> GSM228590 1 0.165 0.7782 0.960 0.004 0.036
#> GSM228591 2 0.296 0.6746 0.008 0.912 0.080
#> GSM228597 2 0.377 0.6467 0.104 0.880 0.016
#> GSM228601 2 0.227 0.6659 0.040 0.944 0.016
#> GSM228604 2 0.618 0.1728 0.000 0.584 0.416
#> GSM228608 1 0.388 0.7764 0.888 0.044 0.068
#> GSM228609 2 0.575 0.3528 0.296 0.700 0.004
#> GSM228613 1 0.103 0.7700 0.976 0.024 0.000
#> GSM228616 2 0.659 0.5670 0.208 0.732 0.060
#> GSM228628 2 0.362 0.6595 0.000 0.864 0.136
#> GSM228634 1 0.520 0.6653 0.760 0.004 0.236
#> GSM228642 2 0.450 0.6205 0.000 0.804 0.196
#> GSM228645 2 0.542 0.5847 0.008 0.752 0.240
#> GSM228646 2 0.583 0.3889 0.000 0.660 0.340
#> GSM228652 1 0.456 0.7727 0.860 0.064 0.076
#> GSM228655 1 0.454 0.7526 0.848 0.028 0.124
#> GSM228656 1 0.127 0.7779 0.972 0.004 0.024
#> GSM228659 1 0.590 0.4992 0.648 0.352 0.000
#> GSM228662 1 0.216 0.7603 0.936 0.064 0.000
#> GSM228584 1 0.223 0.7788 0.944 0.012 0.044
#> GSM228586 1 0.440 0.7158 0.812 0.000 0.188
#> GSM228592 1 0.399 0.7607 0.864 0.012 0.124
#> GSM228593 2 0.834 -0.1556 0.452 0.468 0.080
#> GSM228594 1 0.613 0.5682 0.644 0.004 0.352
#> GSM228598 1 0.621 0.7141 0.752 0.048 0.200
#> GSM228607 3 0.632 0.3472 0.008 0.356 0.636
#> GSM228612 3 0.455 0.5937 0.000 0.200 0.800
#> GSM228619 3 0.872 0.2516 0.272 0.152 0.576
#> GSM228622 3 0.634 -0.0211 0.400 0.004 0.596
#> GSM228625 1 0.930 0.4145 0.500 0.316 0.184
#> GSM228631 3 0.739 -0.2040 0.464 0.032 0.504
#> GSM228633 2 0.586 0.4676 0.000 0.656 0.344
#> GSM228637 2 0.589 0.5324 0.028 0.752 0.220
#> GSM228639 3 0.475 0.5808 0.000 0.216 0.784
#> GSM228649 2 0.828 0.4122 0.160 0.632 0.208
#> GSM228660 1 0.967 0.3152 0.412 0.212 0.376
#> GSM228661 3 0.631 -0.3008 0.488 0.000 0.512
#> GSM228595 2 0.271 0.6702 0.000 0.912 0.088
#> GSM228599 2 0.403 0.6603 0.008 0.856 0.136
#> GSM228602 3 0.303 0.6544 0.012 0.076 0.912
#> GSM228614 2 0.522 0.5189 0.000 0.740 0.260
#> GSM228626 2 0.450 0.6167 0.000 0.804 0.196
#> GSM228640 3 0.585 0.5659 0.040 0.188 0.772
#> GSM228643 3 0.584 0.4838 0.004 0.308 0.688
#> GSM228650 3 0.595 0.4280 0.000 0.360 0.640
#> GSM228653 3 0.537 0.6084 0.048 0.140 0.812
#> GSM228657 2 0.362 0.6575 0.000 0.864 0.136
#> GSM228605 3 0.726 -0.1894 0.440 0.028 0.532
#> GSM228610 3 0.171 0.6518 0.008 0.032 0.960
#> GSM228617 3 0.199 0.6492 0.004 0.048 0.948
#> GSM228620 3 0.295 0.6140 0.088 0.004 0.908
#> GSM228623 2 0.596 0.5172 0.008 0.692 0.300
#> GSM228629 3 0.268 0.6254 0.068 0.008 0.924
#> GSM228632 3 0.502 0.5534 0.000 0.240 0.760
#> GSM228635 2 0.553 0.5160 0.000 0.704 0.296
#> GSM228647 3 0.183 0.6530 0.008 0.036 0.956
#> GSM228596 3 0.689 0.5370 0.072 0.212 0.716
#> GSM228600 3 0.597 0.4167 0.000 0.364 0.636
#> GSM228603 3 0.614 0.5733 0.060 0.172 0.768
#> GSM228615 2 0.325 0.6732 0.036 0.912 0.052
#> GSM228627 3 0.518 0.5440 0.000 0.256 0.744
#> GSM228641 3 0.543 0.5152 0.000 0.284 0.716
#> GSM228644 2 0.489 0.5875 0.000 0.772 0.228
#> GSM228651 3 0.489 0.5630 0.000 0.228 0.772
#> GSM228654 3 0.543 0.5265 0.000 0.284 0.716
#> GSM228658 3 0.487 0.6094 0.028 0.144 0.828
#> GSM228606 3 0.529 0.5200 0.000 0.268 0.732
#> GSM228611 3 0.177 0.6453 0.024 0.016 0.960
#> GSM228618 3 0.165 0.6506 0.004 0.036 0.960
#> GSM228621 3 0.460 0.5936 0.000 0.204 0.796
#> GSM228624 3 0.450 0.5936 0.000 0.196 0.804
#> GSM228630 3 0.543 0.4787 0.000 0.284 0.716
#> GSM228636 2 0.522 0.5682 0.012 0.780 0.208
#> GSM228638 3 0.312 0.6366 0.000 0.108 0.892
#> GSM228648 3 0.460 0.5938 0.000 0.204 0.796
#> GSM228670 2 0.643 0.6508 0.084 0.760 0.156
#> GSM228671 2 0.597 0.3445 0.000 0.636 0.364
#> GSM228672 1 0.643 0.3217 0.568 0.428 0.004
#> GSM228674 2 0.546 0.4691 0.244 0.748 0.008
#> GSM228675 2 0.367 0.6769 0.020 0.888 0.092
#> GSM228676 3 0.848 0.4520 0.200 0.184 0.616
#> GSM228667 2 0.563 0.6368 0.032 0.780 0.188
#> GSM228668 1 0.630 0.5683 0.640 0.008 0.352
#> GSM228669 1 0.899 0.5037 0.552 0.272 0.176
#> GSM228673 3 0.412 0.6146 0.000 0.168 0.832
#> GSM228677 3 0.606 0.2758 0.000 0.384 0.616
#> GSM228678 2 0.465 0.5832 0.000 0.792 0.208
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.761 -0.10459 0.420 0.128 0.016 0.436
#> GSM228563 4 0.707 0.03939 0.116 0.360 0.004 0.520
#> GSM228565 1 0.798 -0.01866 0.384 0.356 0.004 0.256
#> GSM228566 4 0.760 0.20981 0.008 0.236 0.228 0.528
#> GSM228567 1 0.305 0.69458 0.884 0.000 0.088 0.028
#> GSM228570 1 0.535 0.62043 0.732 0.048 0.008 0.212
#> GSM228571 1 0.559 0.66100 0.772 0.060 0.056 0.112
#> GSM228574 4 0.817 0.10877 0.012 0.316 0.260 0.412
#> GSM228575 4 0.774 0.23106 0.020 0.252 0.188 0.540
#> GSM228576 1 0.845 0.29967 0.504 0.236 0.056 0.204
#> GSM228579 1 0.554 0.66746 0.776 0.088 0.092 0.044
#> GSM228580 2 0.665 0.03606 0.000 0.484 0.084 0.432
#> GSM228581 2 0.746 0.21320 0.020 0.568 0.264 0.148
#> GSM228666 2 0.560 0.44525 0.000 0.724 0.116 0.160
#> GSM228564 4 0.705 0.15863 0.300 0.152 0.000 0.548
#> GSM228568 3 0.835 0.17723 0.276 0.200 0.484 0.040
#> GSM228569 3 0.665 0.09126 0.336 0.024 0.588 0.052
#> GSM228572 2 0.562 0.41882 0.000 0.708 0.084 0.208
#> GSM228573 3 0.416 0.53809 0.056 0.032 0.852 0.060
#> GSM228577 1 0.716 0.47742 0.584 0.056 0.308 0.052
#> GSM228578 3 0.704 -0.15346 0.420 0.004 0.472 0.104
#> GSM228663 3 0.486 0.51199 0.052 0.120 0.804 0.024
#> GSM228664 3 0.564 0.36836 0.004 0.364 0.608 0.024
#> GSM228665 3 0.376 0.52455 0.044 0.052 0.872 0.032
#> GSM228582 2 0.762 0.29272 0.148 0.616 0.176 0.060
#> GSM228583 1 0.189 0.70131 0.940 0.000 0.016 0.044
#> GSM228585 1 0.222 0.70155 0.928 0.000 0.032 0.040
#> GSM228587 1 0.448 0.66786 0.808 0.108 0.000 0.084
#> GSM228588 2 0.558 0.37259 0.144 0.728 0.000 0.128
#> GSM228589 2 0.325 0.48903 0.044 0.888 0.008 0.060
#> GSM228590 1 0.258 0.70485 0.916 0.004 0.048 0.032
#> GSM228591 2 0.204 0.52181 0.008 0.940 0.036 0.016
#> GSM228597 4 0.687 -0.01536 0.092 0.396 0.004 0.508
#> GSM228601 2 0.367 0.46636 0.036 0.848 0.000 0.116
#> GSM228604 2 0.646 0.30052 0.000 0.644 0.160 0.196
#> GSM228608 1 0.493 0.68807 0.788 0.008 0.072 0.132
#> GSM228609 2 0.741 0.10702 0.328 0.488 0.000 0.184
#> GSM228613 1 0.233 0.68920 0.908 0.000 0.004 0.088
#> GSM228616 2 0.811 0.25242 0.260 0.532 0.048 0.160
#> GSM228628 2 0.300 0.52774 0.000 0.892 0.048 0.060
#> GSM228634 1 0.614 0.47808 0.616 0.000 0.312 0.072
#> GSM228642 2 0.346 0.52360 0.000 0.868 0.056 0.076
#> GSM228645 2 0.674 0.06028 0.012 0.480 0.060 0.448
#> GSM228646 2 0.712 0.04372 0.004 0.468 0.112 0.416
#> GSM228652 1 0.526 0.69158 0.780 0.020 0.120 0.080
#> GSM228655 1 0.658 0.56429 0.632 0.024 0.280 0.064
#> GSM228656 1 0.280 0.70403 0.908 0.012 0.060 0.020
#> GSM228659 1 0.638 0.52335 0.628 0.108 0.000 0.264
#> GSM228662 1 0.311 0.68048 0.872 0.016 0.000 0.112
#> GSM228584 1 0.278 0.70132 0.904 0.004 0.024 0.068
#> GSM228586 1 0.528 0.55875 0.688 0.000 0.276 0.036
#> GSM228592 1 0.428 0.68447 0.828 0.008 0.112 0.052
#> GSM228593 1 0.828 0.39617 0.512 0.232 0.044 0.212
#> GSM228594 1 0.702 0.30749 0.520 0.040 0.396 0.044
#> GSM228598 1 0.743 0.55932 0.616 0.052 0.220 0.112
#> GSM228607 3 0.755 0.25662 0.008 0.308 0.512 0.172
#> GSM228612 3 0.581 0.43065 0.008 0.284 0.664 0.044
#> GSM228619 4 0.747 0.08707 0.088 0.040 0.320 0.552
#> GSM228622 3 0.711 0.25825 0.276 0.000 0.552 0.172
#> GSM228625 1 0.973 0.21862 0.360 0.236 0.168 0.236
#> GSM228631 3 0.800 0.19305 0.240 0.008 0.428 0.324
#> GSM228633 2 0.636 0.37462 0.000 0.656 0.184 0.160
#> GSM228637 4 0.873 0.02817 0.048 0.364 0.212 0.376
#> GSM228639 3 0.633 0.36327 0.000 0.080 0.592 0.328
#> GSM228649 2 0.916 0.07283 0.112 0.448 0.200 0.240
#> GSM228660 3 0.888 0.16476 0.216 0.296 0.424 0.064
#> GSM228661 3 0.674 0.12081 0.324 0.024 0.592 0.060
#> GSM228595 2 0.398 0.50622 0.000 0.828 0.040 0.132
#> GSM228599 4 0.627 0.03375 0.012 0.408 0.036 0.544
#> GSM228602 3 0.690 0.43233 0.012 0.112 0.604 0.272
#> GSM228614 2 0.728 -0.02818 0.004 0.444 0.128 0.424
#> GSM228626 2 0.338 0.52891 0.000 0.872 0.076 0.052
#> GSM228640 3 0.820 0.18918 0.048 0.128 0.448 0.376
#> GSM228643 4 0.789 0.00394 0.004 0.236 0.340 0.420
#> GSM228650 4 0.778 0.03162 0.000 0.244 0.352 0.404
#> GSM228653 3 0.659 0.45521 0.052 0.076 0.692 0.180
#> GSM228657 2 0.361 0.53151 0.000 0.860 0.080 0.060
#> GSM228605 4 0.790 -0.01034 0.204 0.008 0.364 0.424
#> GSM228610 3 0.376 0.52249 0.000 0.012 0.816 0.172
#> GSM228617 3 0.552 0.45837 0.008 0.028 0.672 0.292
#> GSM228620 3 0.331 0.53348 0.036 0.000 0.872 0.092
#> GSM228623 4 0.780 0.11936 0.004 0.320 0.224 0.452
#> GSM228629 3 0.421 0.52829 0.032 0.020 0.836 0.112
#> GSM228632 3 0.666 0.39540 0.000 0.152 0.616 0.232
#> GSM228635 4 0.720 0.23491 0.000 0.224 0.224 0.552
#> GSM228647 3 0.488 0.50516 0.008 0.028 0.756 0.208
#> GSM228596 3 0.844 0.11251 0.064 0.128 0.428 0.380
#> GSM228600 3 0.790 0.03466 0.000 0.292 0.360 0.348
#> GSM228603 3 0.790 0.33869 0.060 0.100 0.536 0.304
#> GSM228615 2 0.598 0.08387 0.024 0.512 0.008 0.456
#> GSM228627 3 0.769 0.30566 0.024 0.320 0.520 0.136
#> GSM228641 3 0.781 0.07148 0.004 0.208 0.400 0.388
#> GSM228644 2 0.422 0.51050 0.000 0.824 0.076 0.100
#> GSM228651 3 0.695 0.38516 0.008 0.172 0.616 0.204
#> GSM228654 3 0.720 0.33277 0.000 0.268 0.544 0.188
#> GSM228658 3 0.691 0.43778 0.044 0.132 0.672 0.152
#> GSM228606 4 0.650 -0.06146 0.000 0.072 0.440 0.488
#> GSM228611 3 0.400 0.53558 0.012 0.028 0.840 0.120
#> GSM228618 3 0.472 0.50698 0.004 0.024 0.756 0.216
#> GSM228621 3 0.663 0.34632 0.000 0.100 0.564 0.336
#> GSM228624 3 0.606 0.44794 0.000 0.180 0.684 0.136
#> GSM228630 3 0.697 0.23522 0.000 0.128 0.532 0.340
#> GSM228636 4 0.751 0.07553 0.008 0.348 0.152 0.492
#> GSM228638 3 0.478 0.51644 0.000 0.100 0.788 0.112
#> GSM228648 3 0.688 0.41902 0.000 0.196 0.596 0.208
#> GSM228670 4 0.719 0.17612 0.084 0.304 0.032 0.580
#> GSM228671 4 0.715 0.20106 0.000 0.284 0.172 0.544
#> GSM228672 1 0.624 0.45326 0.604 0.076 0.000 0.320
#> GSM228674 4 0.800 0.09056 0.292 0.300 0.004 0.404
#> GSM228675 4 0.675 0.11923 0.040 0.352 0.036 0.572
#> GSM228676 4 0.872 -0.06570 0.140 0.084 0.328 0.448
#> GSM228667 4 0.741 0.10215 0.060 0.360 0.052 0.528
#> GSM228668 1 0.768 0.33279 0.448 0.000 0.236 0.316
#> GSM228669 1 0.842 0.26090 0.408 0.064 0.124 0.404
#> GSM228673 3 0.649 0.43939 0.004 0.128 0.648 0.220
#> GSM228677 4 0.744 0.23016 0.000 0.212 0.284 0.504
#> GSM228678 2 0.728 0.01515 0.004 0.452 0.128 0.416
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 5 0.759 0.13420 0.216 0.032 0.020 0.256 0.476
#> GSM228563 4 0.750 0.23955 0.076 0.184 0.000 0.484 0.256
#> GSM228565 5 0.865 0.06767 0.280 0.212 0.008 0.172 0.328
#> GSM228566 5 0.427 0.51645 0.000 0.048 0.072 0.068 0.812
#> GSM228567 1 0.324 0.72786 0.876 0.004 0.048 0.028 0.044
#> GSM228570 1 0.641 0.52876 0.644 0.048 0.008 0.176 0.124
#> GSM228571 1 0.720 0.57285 0.620 0.080 0.076 0.060 0.164
#> GSM228574 5 0.741 0.40829 0.004 0.168 0.188 0.096 0.544
#> GSM228575 5 0.659 0.40580 0.004 0.092 0.060 0.244 0.600
#> GSM228576 5 0.854 0.11946 0.332 0.200 0.040 0.072 0.356
#> GSM228579 1 0.554 0.68277 0.744 0.076 0.084 0.016 0.080
#> GSM228580 5 0.633 0.37109 0.000 0.216 0.016 0.180 0.588
#> GSM228581 2 0.754 0.29127 0.016 0.500 0.280 0.056 0.148
#> GSM228666 2 0.734 0.38925 0.004 0.548 0.092 0.152 0.204
#> GSM228564 4 0.727 0.11930 0.168 0.048 0.000 0.452 0.332
#> GSM228568 3 0.856 0.16719 0.160 0.288 0.416 0.052 0.084
#> GSM228569 3 0.658 0.01215 0.364 0.052 0.528 0.032 0.024
#> GSM228572 2 0.651 0.45202 0.000 0.576 0.032 0.256 0.136
#> GSM228573 3 0.479 0.49802 0.040 0.012 0.752 0.016 0.180
#> GSM228577 1 0.707 0.39248 0.524 0.072 0.312 0.084 0.008
#> GSM228578 3 0.741 0.02455 0.344 0.008 0.408 0.216 0.024
#> GSM228663 3 0.494 0.49216 0.044 0.120 0.776 0.020 0.040
#> GSM228664 3 0.528 0.37154 0.004 0.308 0.640 0.024 0.024
#> GSM228665 3 0.429 0.52921 0.044 0.024 0.828 0.056 0.048
#> GSM228582 2 0.659 0.50884 0.148 0.652 0.116 0.016 0.068
#> GSM228583 1 0.199 0.72497 0.932 0.004 0.004 0.028 0.032
#> GSM228585 1 0.301 0.72599 0.888 0.008 0.016 0.036 0.052
#> GSM228587 1 0.391 0.69302 0.828 0.076 0.012 0.080 0.004
#> GSM228588 2 0.415 0.58700 0.080 0.792 0.000 0.124 0.004
#> GSM228589 2 0.301 0.66023 0.016 0.888 0.020 0.060 0.016
#> GSM228590 1 0.177 0.73052 0.940 0.000 0.020 0.032 0.008
#> GSM228591 2 0.268 0.67331 0.016 0.908 0.024 0.020 0.032
#> GSM228597 4 0.666 0.37419 0.108 0.208 0.004 0.612 0.068
#> GSM228601 2 0.327 0.65081 0.016 0.852 0.000 0.112 0.020
#> GSM228604 2 0.576 0.33306 0.000 0.576 0.072 0.012 0.340
#> GSM228608 1 0.535 0.66062 0.740 0.012 0.028 0.100 0.120
#> GSM228609 2 0.687 0.15974 0.276 0.472 0.000 0.240 0.012
#> GSM228613 1 0.218 0.71634 0.908 0.004 0.000 0.080 0.008
#> GSM228616 2 0.795 0.36028 0.228 0.500 0.020 0.104 0.148
#> GSM228628 2 0.388 0.67695 0.000 0.828 0.028 0.044 0.100
#> GSM228634 1 0.573 0.58490 0.664 0.004 0.232 0.028 0.072
#> GSM228642 2 0.403 0.63790 0.000 0.792 0.020 0.024 0.164
#> GSM228645 5 0.634 0.38043 0.052 0.264 0.008 0.064 0.612
#> GSM228646 5 0.550 0.42178 0.016 0.232 0.016 0.052 0.684
#> GSM228652 1 0.526 0.70418 0.760 0.044 0.120 0.048 0.028
#> GSM228655 1 0.595 0.59380 0.668 0.024 0.224 0.052 0.032
#> GSM228656 1 0.217 0.73033 0.924 0.008 0.044 0.020 0.004
#> GSM228659 1 0.631 0.27385 0.532 0.072 0.004 0.364 0.028
#> GSM228662 1 0.281 0.70600 0.876 0.012 0.000 0.100 0.012
#> GSM228584 1 0.223 0.72566 0.920 0.012 0.012 0.052 0.004
#> GSM228586 1 0.377 0.68587 0.796 0.000 0.176 0.016 0.012
#> GSM228592 1 0.316 0.72413 0.868 0.020 0.084 0.028 0.000
#> GSM228593 1 0.705 0.25529 0.508 0.204 0.008 0.260 0.020
#> GSM228594 1 0.678 0.38848 0.532 0.072 0.340 0.024 0.032
#> GSM228598 1 0.753 0.41495 0.504 0.064 0.248 0.176 0.008
#> GSM228607 3 0.682 0.25903 0.012 0.136 0.552 0.276 0.024
#> GSM228612 3 0.592 0.47974 0.004 0.200 0.668 0.092 0.036
#> GSM228619 4 0.774 0.22111 0.084 0.008 0.168 0.484 0.256
#> GSM228622 3 0.793 0.23976 0.264 0.000 0.432 0.192 0.112
#> GSM228625 4 0.797 0.28273 0.296 0.148 0.140 0.416 0.000
#> GSM228631 4 0.855 0.00692 0.200 0.000 0.232 0.288 0.280
#> GSM228633 2 0.673 0.47580 0.000 0.612 0.140 0.160 0.088
#> GSM228637 4 0.623 0.46741 0.024 0.136 0.192 0.640 0.008
#> GSM228639 3 0.684 0.32708 0.000 0.024 0.500 0.304 0.172
#> GSM228649 4 0.776 0.35041 0.056 0.236 0.212 0.480 0.016
#> GSM228660 3 0.778 0.23009 0.184 0.244 0.464 0.108 0.000
#> GSM228661 3 0.619 -0.08914 0.416 0.036 0.504 0.028 0.016
#> GSM228595 2 0.412 0.64866 0.000 0.804 0.032 0.132 0.032
#> GSM228599 5 0.748 -0.08660 0.024 0.208 0.012 0.372 0.384
#> GSM228602 5 0.724 0.09923 0.028 0.072 0.324 0.060 0.516
#> GSM228614 4 0.741 0.24973 0.004 0.288 0.056 0.484 0.168
#> GSM228626 2 0.428 0.67917 0.000 0.808 0.040 0.060 0.092
#> GSM228640 5 0.410 0.41929 0.024 0.016 0.152 0.008 0.800
#> GSM228643 5 0.440 0.49386 0.000 0.056 0.112 0.036 0.796
#> GSM228650 5 0.627 0.34894 0.000 0.056 0.176 0.124 0.644
#> GSM228653 3 0.553 0.25951 0.028 0.016 0.576 0.008 0.372
#> GSM228657 2 0.474 0.66983 0.000 0.780 0.056 0.096 0.068
#> GSM228605 4 0.756 0.28558 0.096 0.000 0.248 0.492 0.164
#> GSM228610 3 0.554 0.48147 0.008 0.000 0.672 0.148 0.172
#> GSM228617 3 0.685 0.34694 0.012 0.008 0.492 0.164 0.324
#> GSM228620 3 0.420 0.50891 0.036 0.000 0.800 0.032 0.132
#> GSM228623 4 0.600 0.48179 0.012 0.132 0.140 0.684 0.032
#> GSM228629 3 0.591 0.50434 0.056 0.000 0.680 0.100 0.164
#> GSM228632 3 0.642 0.44862 0.000 0.064 0.636 0.152 0.148
#> GSM228635 4 0.561 0.44727 0.000 0.056 0.112 0.712 0.120
#> GSM228647 3 0.591 0.38260 0.008 0.008 0.576 0.072 0.336
#> GSM228596 5 0.797 0.32675 0.036 0.056 0.252 0.172 0.484
#> GSM228600 5 0.694 0.32906 0.008 0.172 0.232 0.032 0.556
#> GSM228603 5 0.542 0.29450 0.040 0.016 0.252 0.016 0.676
#> GSM228615 4 0.618 0.18604 0.028 0.336 0.000 0.556 0.080
#> GSM228627 3 0.758 0.02884 0.020 0.304 0.408 0.016 0.252
#> GSM228641 5 0.434 0.44194 0.000 0.064 0.152 0.008 0.776
#> GSM228644 2 0.447 0.66695 0.000 0.788 0.036 0.052 0.124
#> GSM228651 3 0.616 0.07542 0.004 0.080 0.492 0.012 0.412
#> GSM228654 3 0.724 0.14713 0.004 0.180 0.456 0.032 0.328
#> GSM228658 3 0.655 0.27552 0.032 0.068 0.572 0.020 0.308
#> GSM228606 4 0.747 0.12461 0.000 0.040 0.316 0.404 0.240
#> GSM228611 3 0.457 0.49188 0.008 0.000 0.748 0.060 0.184
#> GSM228618 3 0.579 0.43947 0.000 0.004 0.604 0.116 0.276
#> GSM228621 3 0.599 0.19952 0.000 0.016 0.464 0.068 0.452
#> GSM228624 3 0.641 0.43479 0.000 0.092 0.632 0.196 0.080
#> GSM228630 3 0.729 0.33098 0.000 0.048 0.488 0.244 0.220
#> GSM228636 4 0.471 0.49616 0.000 0.144 0.088 0.756 0.012
#> GSM228638 3 0.520 0.52565 0.000 0.048 0.744 0.108 0.100
#> GSM228648 3 0.699 0.35925 0.000 0.100 0.516 0.072 0.312
#> GSM228670 4 0.653 0.33374 0.076 0.072 0.012 0.636 0.204
#> GSM228671 5 0.762 0.05956 0.000 0.116 0.112 0.352 0.420
#> GSM228672 1 0.730 0.04164 0.424 0.060 0.000 0.376 0.140
#> GSM228674 4 0.812 0.30966 0.164 0.184 0.016 0.488 0.148
#> GSM228675 4 0.714 -0.03849 0.016 0.120 0.028 0.436 0.400
#> GSM228676 5 0.806 0.33970 0.108 0.020 0.204 0.176 0.492
#> GSM228667 5 0.808 0.20974 0.056 0.180 0.036 0.268 0.460
#> GSM228668 4 0.759 0.21515 0.300 0.000 0.108 0.464 0.128
#> GSM228669 4 0.657 0.39482 0.244 0.024 0.076 0.616 0.040
#> GSM228673 3 0.670 0.43534 0.004 0.080 0.624 0.164 0.128
#> GSM228677 5 0.744 0.11294 0.000 0.060 0.176 0.312 0.452
#> GSM228678 4 0.699 0.42012 0.008 0.240 0.100 0.576 0.076
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 5 0.713 0.32076 0.156 0.016 0.008 0.128 0.540 0.152
#> GSM228563 4 0.857 0.16848 0.092 0.164 0.012 0.384 0.216 0.132
#> GSM228565 1 0.852 -0.07278 0.280 0.188 0.000 0.072 0.276 0.184
#> GSM228566 6 0.586 0.18605 0.016 0.032 0.036 0.020 0.308 0.588
#> GSM228567 1 0.372 0.65904 0.824 0.004 0.060 0.000 0.076 0.036
#> GSM228570 1 0.683 0.45053 0.548 0.016 0.004 0.108 0.224 0.100
#> GSM228571 1 0.712 0.45798 0.540 0.056 0.036 0.040 0.260 0.068
#> GSM228574 5 0.783 0.27419 0.008 0.136 0.156 0.044 0.472 0.184
#> GSM228575 6 0.723 -0.12269 0.004 0.076 0.024 0.120 0.376 0.400
#> GSM228576 1 0.828 -0.04030 0.312 0.160 0.012 0.028 0.304 0.184
#> GSM228579 1 0.615 0.60872 0.656 0.072 0.064 0.012 0.164 0.032
#> GSM228580 6 0.752 0.12304 0.004 0.172 0.028 0.088 0.256 0.452
#> GSM228581 2 0.828 0.16560 0.032 0.388 0.232 0.028 0.216 0.104
#> GSM228666 2 0.781 0.00564 0.000 0.356 0.068 0.076 0.348 0.152
#> GSM228564 4 0.787 -0.02929 0.192 0.008 0.000 0.288 0.280 0.232
#> GSM228568 3 0.902 0.15463 0.192 0.164 0.324 0.048 0.216 0.056
#> GSM228569 3 0.646 -0.02430 0.356 0.024 0.504 0.012 0.068 0.036
#> GSM228572 2 0.645 0.50811 0.000 0.604 0.024 0.156 0.076 0.140
#> GSM228573 3 0.621 0.37576 0.044 0.012 0.644 0.028 0.112 0.160
#> GSM228577 1 0.686 0.40187 0.528 0.044 0.268 0.068 0.092 0.000
#> GSM228578 3 0.805 0.09241 0.252 0.000 0.344 0.256 0.096 0.052
#> GSM228663 3 0.561 0.40272 0.056 0.100 0.716 0.008 0.072 0.048
#> GSM228664 3 0.656 0.26161 0.012 0.304 0.540 0.024 0.064 0.056
#> GSM228665 3 0.504 0.41391 0.064 0.020 0.760 0.044 0.024 0.088
#> GSM228582 2 0.628 0.52912 0.128 0.656 0.104 0.012 0.040 0.060
#> GSM228583 1 0.245 0.65427 0.896 0.000 0.000 0.040 0.048 0.016
#> GSM228585 1 0.305 0.65777 0.860 0.004 0.012 0.004 0.092 0.028
#> GSM228587 1 0.476 0.60885 0.768 0.044 0.012 0.116 0.036 0.024
#> GSM228588 2 0.480 0.59471 0.084 0.760 0.004 0.088 0.052 0.012
#> GSM228589 2 0.375 0.64838 0.028 0.844 0.016 0.052 0.024 0.036
#> GSM228590 1 0.271 0.66047 0.892 0.000 0.016 0.032 0.040 0.020
#> GSM228591 2 0.279 0.66608 0.012 0.892 0.016 0.008 0.036 0.036
#> GSM228597 4 0.702 0.35895 0.080 0.132 0.000 0.576 0.128 0.084
#> GSM228601 2 0.349 0.63746 0.000 0.824 0.004 0.120 0.028 0.024
#> GSM228604 2 0.590 0.30933 0.000 0.540 0.032 0.020 0.060 0.348
#> GSM228608 1 0.624 0.52476 0.600 0.008 0.040 0.060 0.252 0.040
#> GSM228609 2 0.728 0.11963 0.260 0.420 0.000 0.244 0.048 0.028
#> GSM228613 1 0.345 0.63417 0.832 0.000 0.004 0.088 0.064 0.012
#> GSM228616 2 0.821 0.24031 0.164 0.388 0.020 0.080 0.060 0.288
#> GSM228628 2 0.338 0.66841 0.000 0.852 0.012 0.044 0.060 0.032
#> GSM228634 1 0.590 0.53093 0.624 0.000 0.200 0.004 0.104 0.068
#> GSM228642 2 0.413 0.64961 0.000 0.784 0.012 0.020 0.048 0.136
#> GSM228645 6 0.703 0.06674 0.024 0.204 0.004 0.024 0.352 0.392
#> GSM228646 6 0.709 0.16808 0.008 0.256 0.024 0.024 0.244 0.444
#> GSM228652 1 0.650 0.62075 0.664 0.044 0.084 0.064 0.096 0.048
#> GSM228655 1 0.720 0.46488 0.552 0.024 0.216 0.088 0.036 0.084
#> GSM228656 1 0.242 0.65778 0.904 0.000 0.048 0.020 0.012 0.016
#> GSM228659 1 0.631 0.23215 0.496 0.052 0.000 0.356 0.084 0.012
#> GSM228662 1 0.317 0.62467 0.844 0.004 0.000 0.104 0.040 0.008
#> GSM228584 1 0.202 0.65412 0.920 0.000 0.020 0.048 0.004 0.008
#> GSM228586 1 0.458 0.60458 0.736 0.000 0.180 0.008 0.044 0.032
#> GSM228592 1 0.338 0.64044 0.820 0.000 0.132 0.032 0.016 0.000
#> GSM228593 1 0.743 0.31195 0.508 0.132 0.012 0.208 0.116 0.024
#> GSM228594 1 0.644 0.37680 0.540 0.060 0.312 0.008 0.056 0.024
#> GSM228598 1 0.730 0.38272 0.496 0.012 0.168 0.176 0.144 0.004
#> GSM228607 3 0.720 0.14316 0.020 0.092 0.452 0.352 0.048 0.036
#> GSM228612 3 0.693 0.34534 0.008 0.244 0.548 0.096 0.040 0.064
#> GSM228619 4 0.750 0.04843 0.084 0.020 0.124 0.412 0.016 0.344
#> GSM228622 3 0.845 0.19680 0.204 0.000 0.376 0.164 0.120 0.136
#> GSM228625 4 0.735 0.38075 0.216 0.124 0.068 0.532 0.024 0.036
#> GSM228631 6 0.790 0.09196 0.196 0.008 0.200 0.212 0.008 0.376
#> GSM228633 2 0.625 0.53164 0.000 0.636 0.072 0.168 0.052 0.072
#> GSM228637 4 0.572 0.39943 0.012 0.044 0.224 0.652 0.052 0.016
#> GSM228639 3 0.739 0.26651 0.000 0.020 0.416 0.316 0.116 0.132
#> GSM228649 4 0.808 0.33590 0.088 0.152 0.200 0.472 0.056 0.032
#> GSM228660 3 0.796 0.20128 0.196 0.252 0.384 0.128 0.040 0.000
#> GSM228661 3 0.562 -0.01504 0.400 0.012 0.520 0.008 0.036 0.024
#> GSM228595 2 0.330 0.64868 0.000 0.836 0.008 0.112 0.008 0.036
#> GSM228599 6 0.717 0.02065 0.012 0.176 0.008 0.312 0.056 0.436
#> GSM228602 6 0.691 0.26563 0.040 0.060 0.272 0.044 0.036 0.548
#> GSM228614 4 0.815 0.14312 0.004 0.184 0.068 0.428 0.176 0.140
#> GSM228626 2 0.392 0.66834 0.000 0.820 0.020 0.040 0.044 0.076
#> GSM228640 6 0.664 0.29777 0.056 0.028 0.104 0.004 0.248 0.560
#> GSM228643 6 0.633 0.04328 0.004 0.040 0.096 0.008 0.372 0.480
#> GSM228650 6 0.622 0.33001 0.000 0.068 0.116 0.048 0.120 0.648
#> GSM228653 3 0.661 0.04494 0.044 0.012 0.452 0.004 0.112 0.376
#> GSM228657 2 0.482 0.63789 0.000 0.756 0.048 0.048 0.032 0.116
#> GSM228605 4 0.752 0.14626 0.056 0.004 0.152 0.464 0.264 0.060
#> GSM228610 3 0.623 0.34083 0.000 0.000 0.596 0.116 0.132 0.156
#> GSM228617 6 0.659 -0.04946 0.016 0.012 0.372 0.160 0.008 0.432
#> GSM228620 3 0.571 0.39123 0.040 0.000 0.676 0.044 0.076 0.164
#> GSM228623 4 0.586 0.41882 0.008 0.088 0.112 0.692 0.060 0.040
#> GSM228629 3 0.673 0.22820 0.048 0.000 0.516 0.108 0.036 0.292
#> GSM228632 3 0.766 0.24541 0.000 0.076 0.472 0.168 0.208 0.076
#> GSM228635 4 0.563 0.38327 0.000 0.032 0.072 0.696 0.120 0.080
#> GSM228647 3 0.650 0.23098 0.012 0.004 0.548 0.052 0.120 0.264
#> GSM228596 5 0.773 0.25250 0.008 0.028 0.200 0.084 0.416 0.264
#> GSM228600 6 0.697 0.37591 0.016 0.124 0.140 0.028 0.104 0.588
#> GSM228603 6 0.663 0.29995 0.056 0.008 0.192 0.000 0.220 0.524
#> GSM228615 4 0.708 0.31874 0.044 0.220 0.004 0.544 0.100 0.088
#> GSM228627 3 0.808 -0.03520 0.020 0.236 0.348 0.016 0.272 0.108
#> GSM228641 6 0.630 0.28509 0.008 0.048 0.116 0.004 0.260 0.564
#> GSM228644 2 0.438 0.65536 0.000 0.784 0.012 0.044 0.076 0.084
#> GSM228651 3 0.733 0.02774 0.004 0.060 0.416 0.016 0.224 0.280
#> GSM228654 6 0.721 -0.00243 0.008 0.148 0.372 0.008 0.076 0.388
#> GSM228658 3 0.686 0.11495 0.028 0.076 0.488 0.000 0.092 0.316
#> GSM228606 4 0.776 0.04225 0.000 0.024 0.228 0.412 0.156 0.180
#> GSM228611 3 0.624 0.32479 0.008 0.000 0.604 0.092 0.188 0.108
#> GSM228618 3 0.633 0.11777 0.012 0.000 0.476 0.108 0.036 0.368
#> GSM228621 6 0.727 0.05284 0.000 0.016 0.320 0.112 0.128 0.424
#> GSM228624 3 0.769 0.27739 0.000 0.092 0.464 0.244 0.116 0.084
#> GSM228630 3 0.719 0.21970 0.000 0.028 0.448 0.248 0.048 0.228
#> GSM228636 4 0.460 0.44107 0.000 0.104 0.076 0.768 0.028 0.024
#> GSM228638 3 0.543 0.37779 0.000 0.028 0.680 0.120 0.016 0.156
#> GSM228648 3 0.719 0.21716 0.000 0.108 0.492 0.052 0.072 0.276
#> GSM228670 4 0.730 0.10033 0.072 0.052 0.008 0.456 0.332 0.080
#> GSM228671 5 0.754 0.36717 0.000 0.052 0.092 0.276 0.452 0.128
#> GSM228672 1 0.695 -0.01198 0.364 0.016 0.000 0.300 0.296 0.024
#> GSM228674 4 0.796 0.06470 0.124 0.100 0.012 0.400 0.312 0.052
#> GSM228675 5 0.719 0.31765 0.020 0.076 0.040 0.276 0.516 0.072
#> GSM228676 5 0.786 0.39834 0.084 0.000 0.140 0.124 0.468 0.184
#> GSM228667 5 0.735 0.45889 0.024 0.112 0.040 0.132 0.564 0.128
#> GSM228668 4 0.811 0.18642 0.256 0.000 0.076 0.384 0.184 0.100
#> GSM228669 4 0.538 0.39932 0.136 0.020 0.052 0.704 0.088 0.000
#> GSM228673 3 0.724 0.23922 0.004 0.048 0.516 0.152 0.220 0.060
#> GSM228677 5 0.816 0.27008 0.000 0.044 0.144 0.280 0.328 0.204
#> GSM228678 4 0.696 0.36006 0.000 0.224 0.072 0.548 0.060 0.096
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
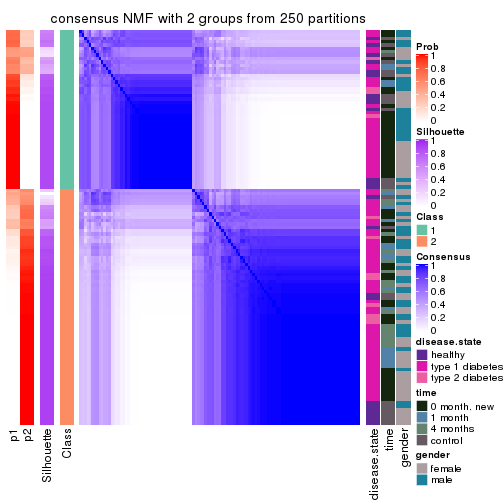
consensus_heatmap(res, k = 3)
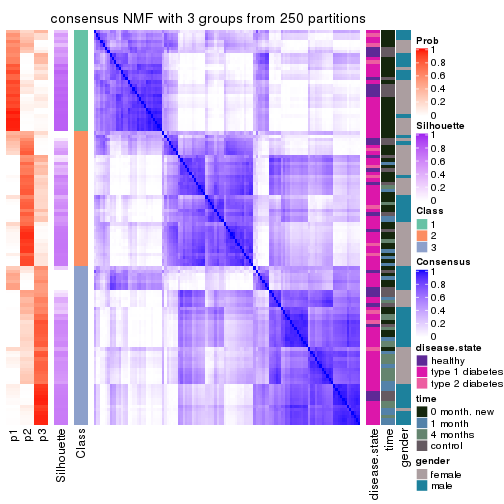
consensus_heatmap(res, k = 4)
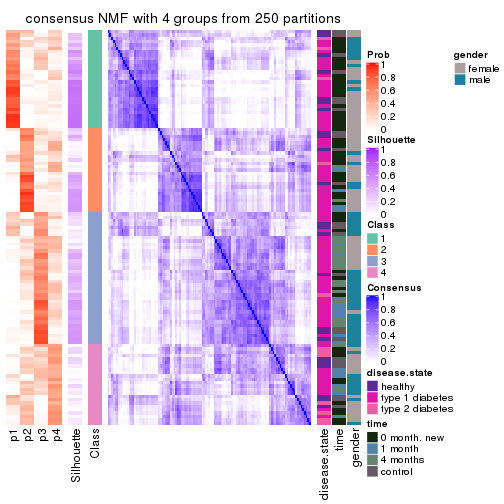
consensus_heatmap(res, k = 5)
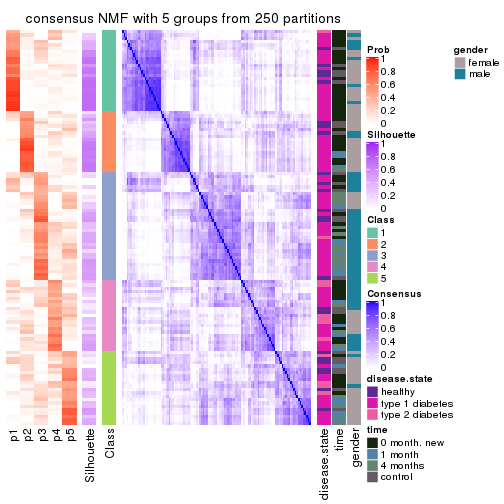
consensus_heatmap(res, k = 6)
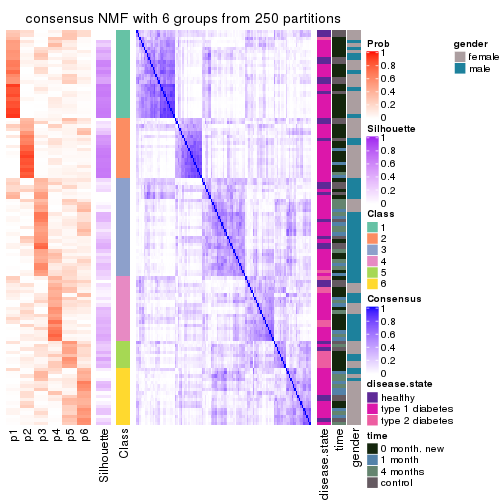
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
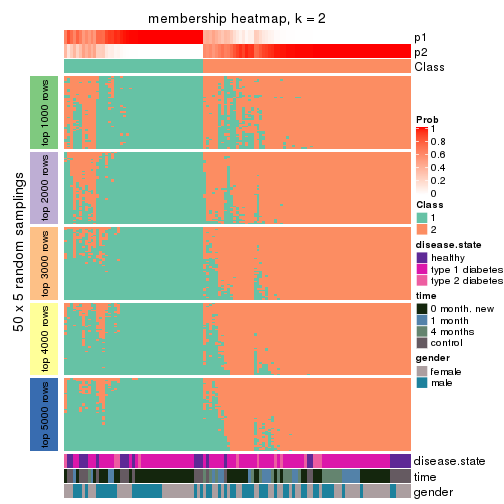
membership_heatmap(res, k = 3)
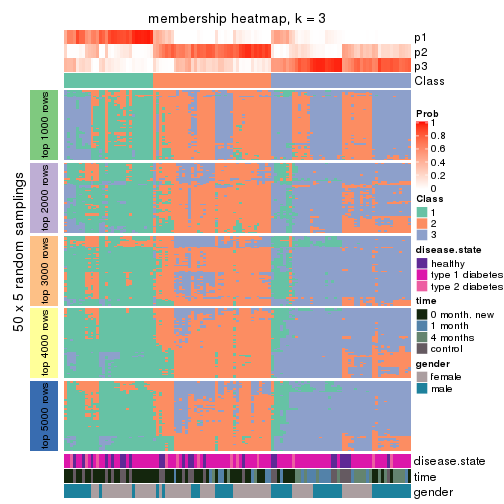
membership_heatmap(res, k = 4)
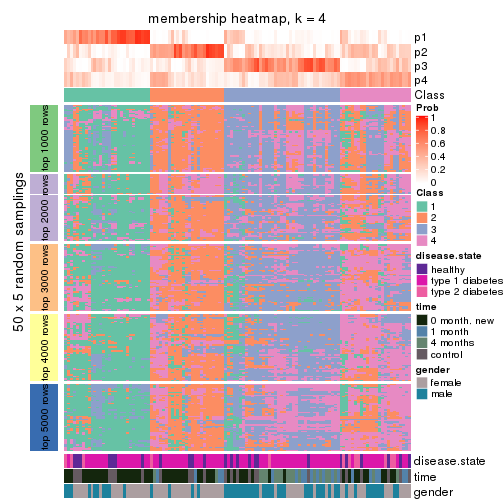
membership_heatmap(res, k = 5)
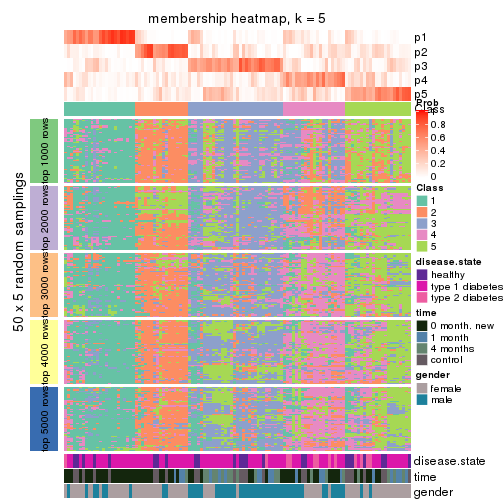
membership_heatmap(res, k = 6)
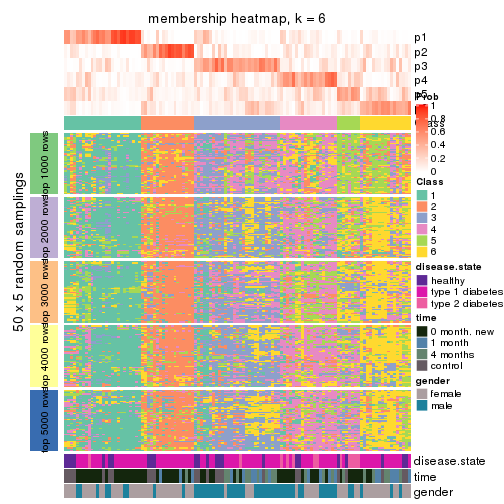
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
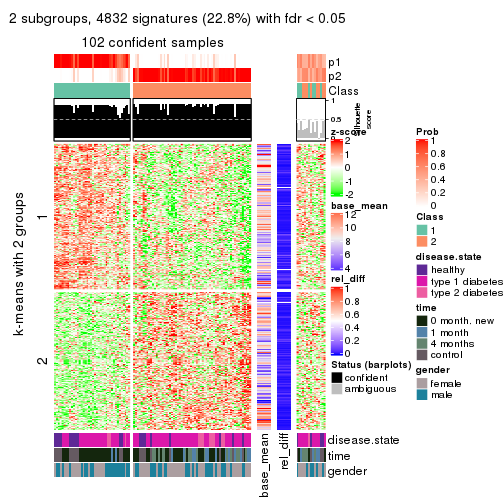
get_signatures(res, k = 3)
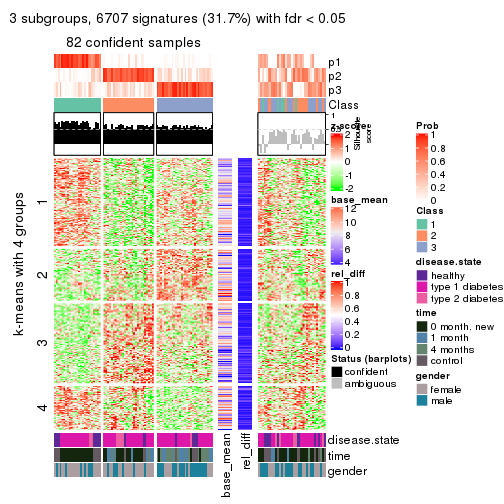
get_signatures(res, k = 4)
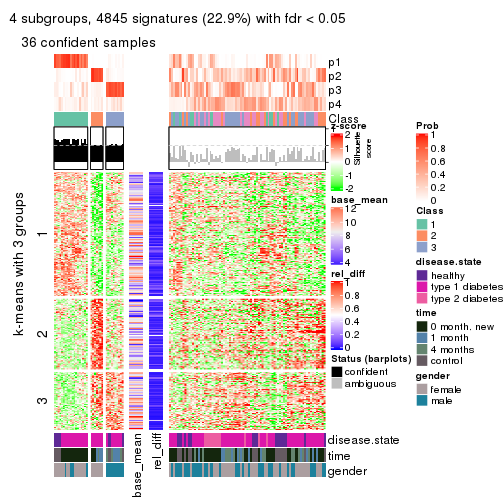
get_signatures(res, k = 5)
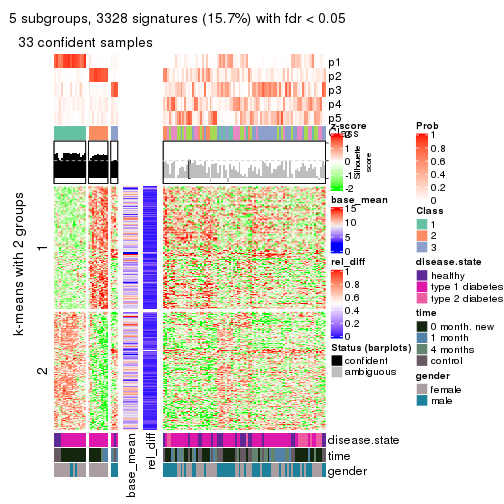
get_signatures(res, k = 6)
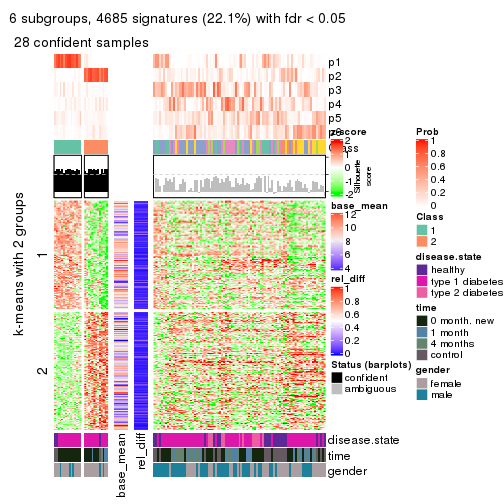
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
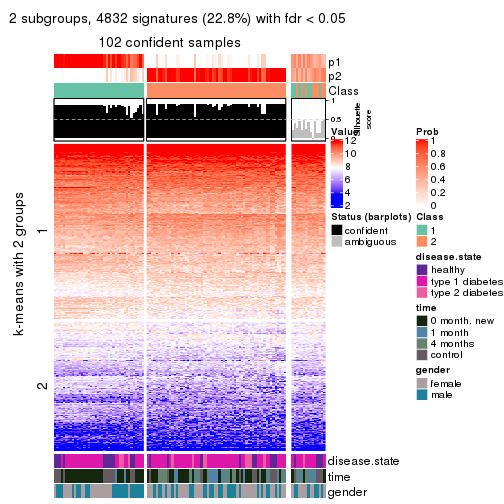
get_signatures(res, k = 3, scale_rows = FALSE)
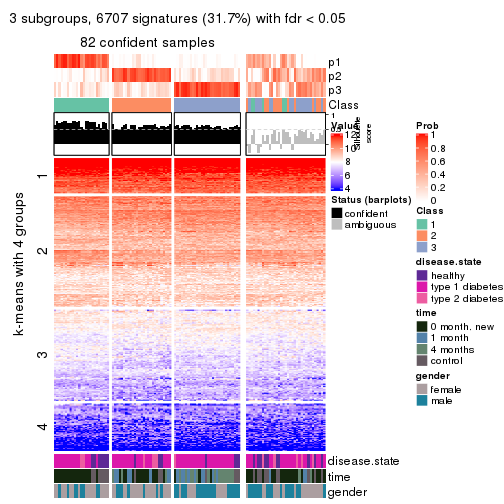
get_signatures(res, k = 4, scale_rows = FALSE)
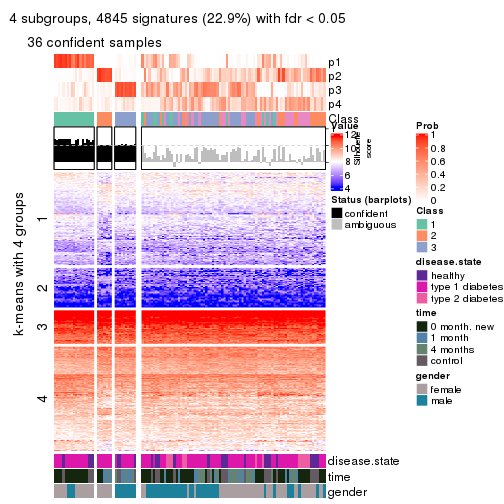
get_signatures(res, k = 5, scale_rows = FALSE)
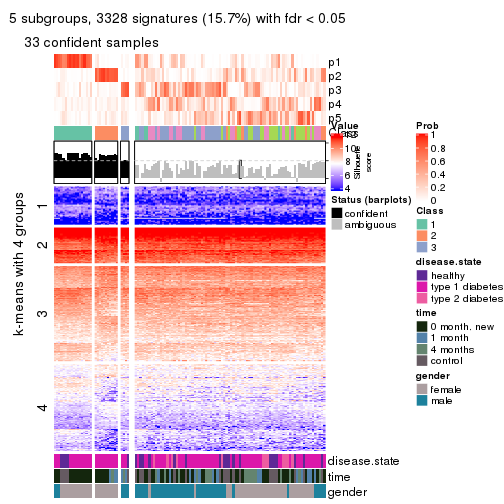
get_signatures(res, k = 6, scale_rows = FALSE)
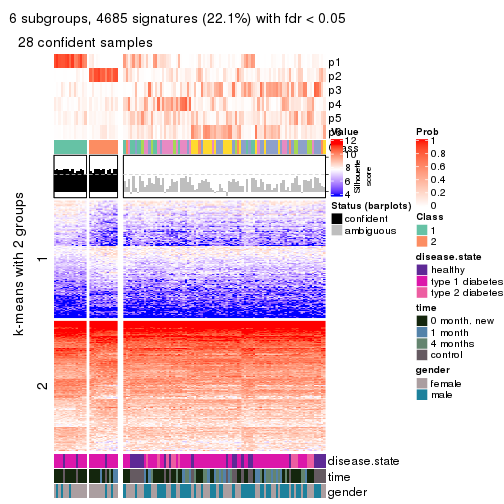
Compare the overlap of signatures from different k:
compare_signatures(res)
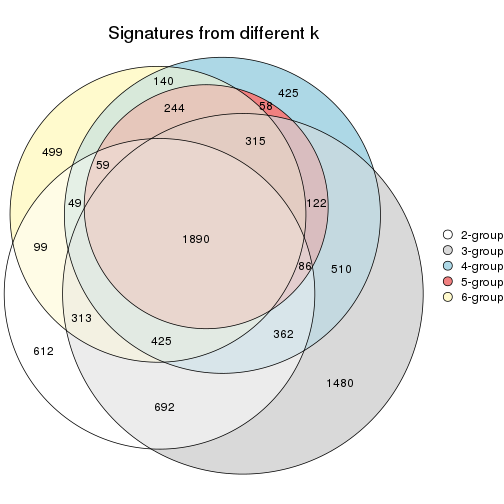
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
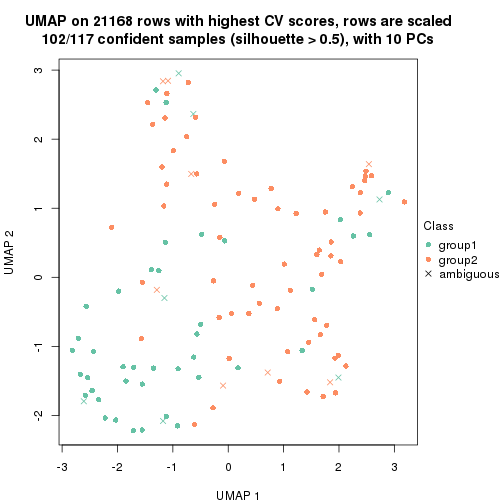
dimension_reduction(res, k = 3, method = "UMAP")
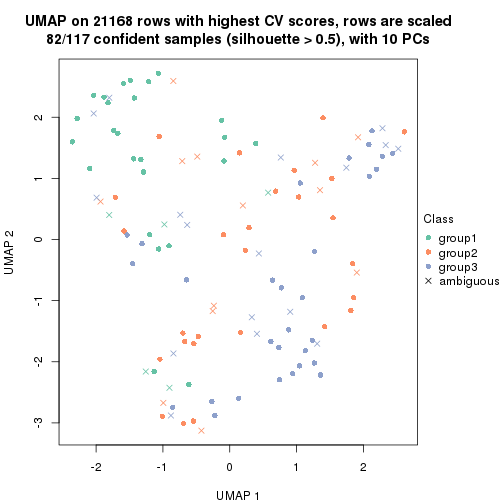
dimension_reduction(res, k = 4, method = "UMAP")
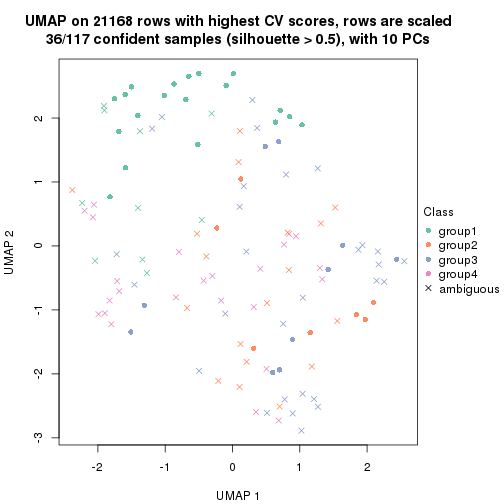
dimension_reduction(res, k = 5, method = "UMAP")
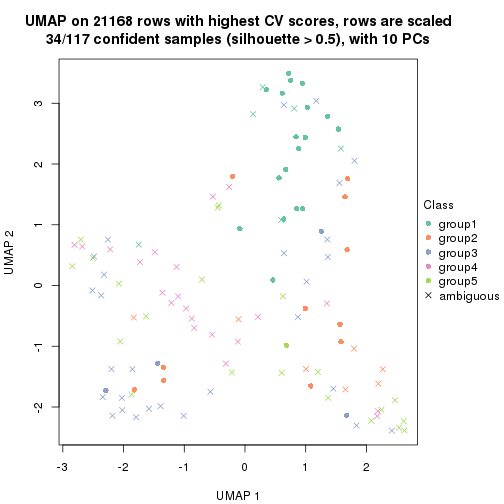
dimension_reduction(res, k = 6, method = "UMAP")
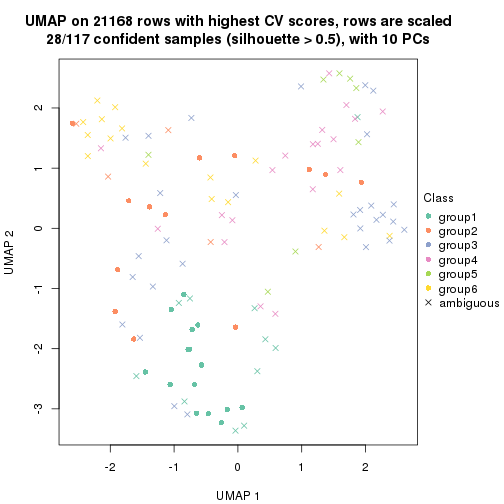
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
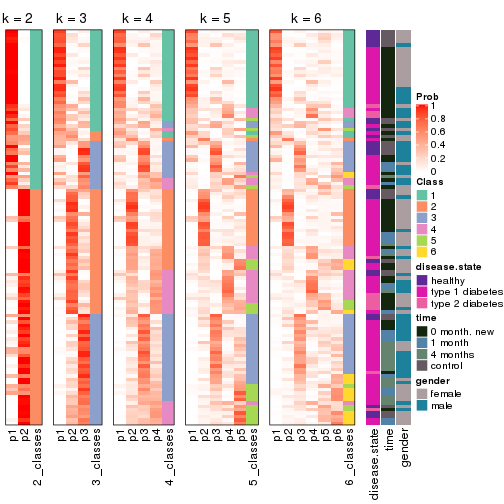
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> CV:NMF 102 0.2289 2.15e-04 3.16e-01 2
#> CV:NMF 82 0.2443 7.52e-07 3.48e-03 3
#> CV:NMF 36 0.2964 6.61e-04 1.17e-05 4
#> CV:NMF 34 0.0593 1.21e-02 3.14e-04 5
#> CV:NMF 28 1.0000 1.44e-01 1.00e+00 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["MAD", "hclust"]
# you can also extract it by
# res = res_list["MAD:hclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'MAD' method.
#> Subgroups are detected by 'hclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 4.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
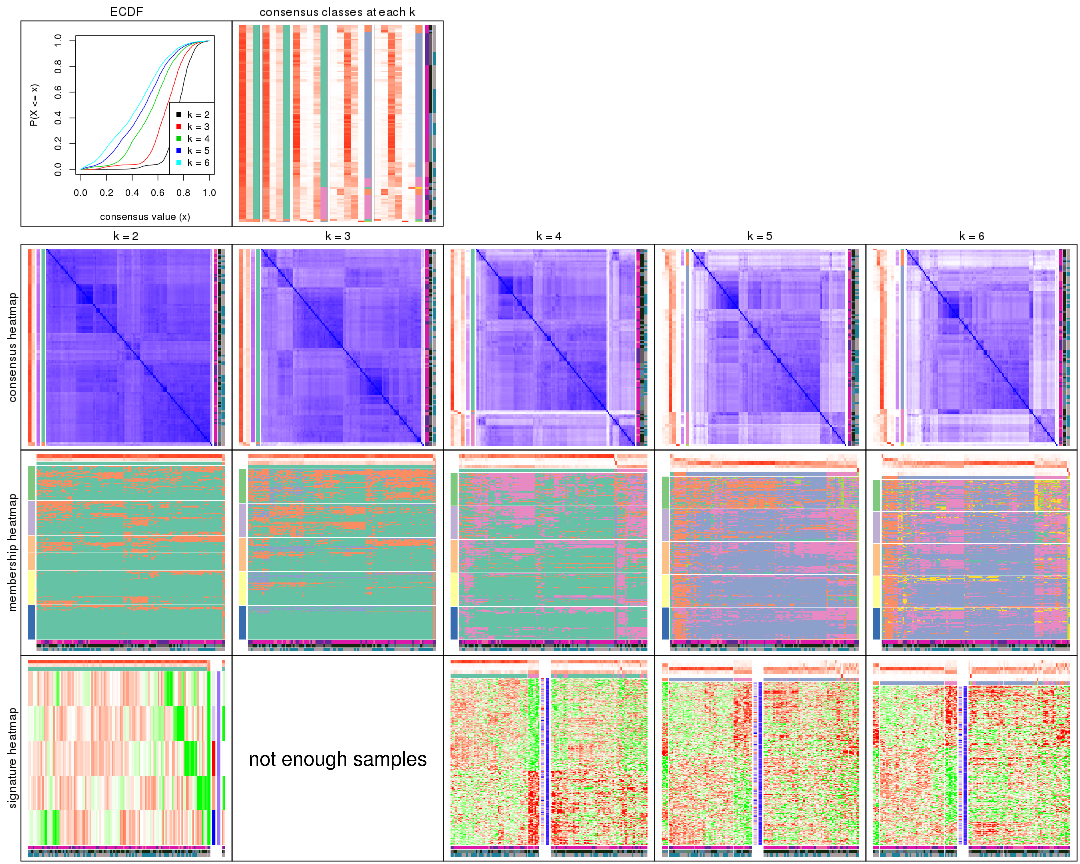
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
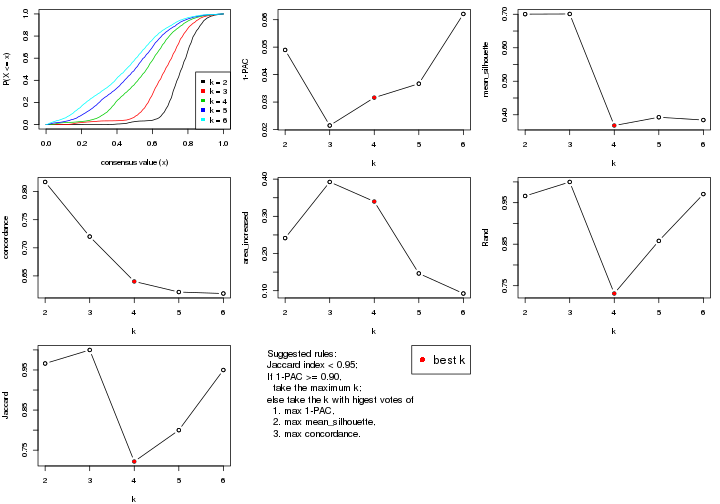
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.0490 0.700 0.817 0.2415 0.966 0.966
#> 3 3 0.0215 0.701 0.720 0.3924 1.000 1.000
#> 4 4 0.0316 0.368 0.640 0.3398 0.731 0.722
#> 5 5 0.0367 0.393 0.621 0.1464 0.858 0.800
#> 6 6 0.0621 0.384 0.619 0.0925 0.971 0.950
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 4
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.605 0.759 0.852 0.148
#> GSM228563 1 0.855 0.593 0.720 0.280
#> GSM228565 1 0.615 0.762 0.848 0.152
#> GSM228566 1 0.730 0.688 0.796 0.204
#> GSM228567 1 0.605 0.730 0.852 0.148
#> GSM228570 1 0.541 0.767 0.876 0.124
#> GSM228571 1 0.469 0.771 0.900 0.100
#> GSM228574 1 0.605 0.756 0.852 0.148
#> GSM228575 2 0.994 0.673 0.456 0.544
#> GSM228576 1 0.634 0.759 0.840 0.160
#> GSM228579 1 0.469 0.774 0.900 0.100
#> GSM228580 2 0.995 0.633 0.460 0.540
#> GSM228581 1 0.913 0.412 0.672 0.328
#> GSM228666 1 0.714 0.721 0.804 0.196
#> GSM228564 1 0.833 0.626 0.736 0.264
#> GSM228568 1 0.552 0.766 0.872 0.128
#> GSM228569 1 0.494 0.761 0.892 0.108
#> GSM228572 1 0.821 0.630 0.744 0.256
#> GSM228573 1 0.541 0.754 0.876 0.124
#> GSM228577 1 0.541 0.768 0.876 0.124
#> GSM228578 1 0.529 0.761 0.880 0.120
#> GSM228663 1 0.653 0.695 0.832 0.168
#> GSM228664 1 0.788 0.581 0.764 0.236
#> GSM228665 1 0.552 0.746 0.872 0.128
#> GSM228582 1 0.584 0.775 0.860 0.140
#> GSM228583 1 0.584 0.728 0.860 0.140
#> GSM228585 1 0.605 0.723 0.852 0.148
#> GSM228587 1 0.653 0.717 0.832 0.168
#> GSM228588 1 0.855 0.605 0.720 0.280
#> GSM228589 1 0.827 0.636 0.740 0.260
#> GSM228590 1 0.615 0.723 0.848 0.152
#> GSM228591 1 0.808 0.640 0.752 0.248
#> GSM228597 1 0.821 0.626 0.744 0.256
#> GSM228601 1 0.861 0.595 0.716 0.284
#> GSM228604 1 0.775 0.664 0.772 0.228
#> GSM228608 1 0.615 0.733 0.848 0.152
#> GSM228609 1 0.871 0.574 0.708 0.292
#> GSM228613 1 0.584 0.728 0.860 0.140
#> GSM228616 1 0.584 0.770 0.860 0.140
#> GSM228628 1 0.839 0.621 0.732 0.268
#> GSM228634 1 0.552 0.750 0.872 0.128
#> GSM228642 1 0.921 0.364 0.664 0.336
#> GSM228645 1 0.775 0.657 0.772 0.228
#> GSM228646 1 0.775 0.673 0.772 0.228
#> GSM228652 1 0.595 0.748 0.856 0.144
#> GSM228655 1 0.574 0.758 0.864 0.136
#> GSM228656 1 0.574 0.731 0.864 0.136
#> GSM228659 1 0.671 0.720 0.824 0.176
#> GSM228662 1 0.605 0.723 0.852 0.148
#> GSM228584 1 0.574 0.732 0.864 0.136
#> GSM228586 1 0.552 0.748 0.872 0.128
#> GSM228592 1 0.563 0.731 0.868 0.132
#> GSM228593 1 0.808 0.659 0.752 0.248
#> GSM228594 1 0.506 0.759 0.888 0.112
#> GSM228598 1 0.595 0.763 0.856 0.144
#> GSM228607 1 0.552 0.773 0.872 0.128
#> GSM228612 1 0.634 0.745 0.840 0.160
#> GSM228619 1 0.541 0.763 0.876 0.124
#> GSM228622 1 0.518 0.772 0.884 0.116
#> GSM228625 1 0.625 0.760 0.844 0.156
#> GSM228631 1 0.541 0.763 0.876 0.124
#> GSM228633 1 0.855 0.560 0.720 0.280
#> GSM228637 1 0.871 0.546 0.708 0.292
#> GSM228639 1 0.605 0.763 0.852 0.148
#> GSM228649 1 0.738 0.711 0.792 0.208
#> GSM228660 1 0.563 0.766 0.868 0.132
#> GSM228661 1 0.494 0.756 0.892 0.108
#> GSM228595 1 0.861 0.562 0.716 0.284
#> GSM228599 1 0.730 0.702 0.796 0.204
#> GSM228602 1 0.605 0.747 0.852 0.148
#> GSM228614 1 0.697 0.711 0.812 0.188
#> GSM228626 1 0.871 0.526 0.708 0.292
#> GSM228640 1 0.574 0.732 0.864 0.136
#> GSM228643 1 0.552 0.733 0.872 0.128
#> GSM228650 1 0.644 0.723 0.836 0.164
#> GSM228653 1 0.529 0.737 0.880 0.120
#> GSM228657 1 0.839 0.591 0.732 0.268
#> GSM228605 1 0.574 0.769 0.864 0.136
#> GSM228610 1 0.634 0.740 0.840 0.160
#> GSM228617 1 0.529 0.763 0.880 0.120
#> GSM228620 1 0.595 0.738 0.856 0.144
#> GSM228623 1 0.745 0.677 0.788 0.212
#> GSM228629 1 0.574 0.738 0.864 0.136
#> GSM228632 1 0.574 0.746 0.864 0.136
#> GSM228635 1 0.855 0.560 0.720 0.280
#> GSM228647 1 0.615 0.717 0.848 0.152
#> GSM228596 1 0.584 0.746 0.860 0.140
#> GSM228600 1 0.552 0.733 0.872 0.128
#> GSM228603 1 0.541 0.737 0.876 0.124
#> GSM228615 1 0.738 0.670 0.792 0.208
#> GSM228627 1 0.605 0.740 0.852 0.148
#> GSM228641 1 0.574 0.727 0.864 0.136
#> GSM228644 1 0.871 0.519 0.708 0.292
#> GSM228651 1 0.584 0.725 0.860 0.140
#> GSM228654 1 0.595 0.722 0.856 0.144
#> GSM228658 1 0.605 0.719 0.852 0.148
#> GSM228606 1 0.706 0.721 0.808 0.192
#> GSM228611 1 0.595 0.727 0.856 0.144
#> GSM228618 1 0.541 0.733 0.876 0.124
#> GSM228621 1 0.680 0.708 0.820 0.180
#> GSM228624 1 0.671 0.696 0.824 0.176
#> GSM228630 1 0.653 0.732 0.832 0.168
#> GSM228636 1 0.833 0.592 0.736 0.264
#> GSM228638 1 0.552 0.765 0.872 0.128
#> GSM228648 1 0.615 0.719 0.848 0.152
#> GSM228670 1 0.697 0.705 0.812 0.188
#> GSM228671 1 0.844 0.560 0.728 0.272
#> GSM228672 1 0.745 0.686 0.788 0.212
#> GSM228674 1 0.775 0.650 0.772 0.228
#> GSM228675 1 0.781 0.634 0.768 0.232
#> GSM228676 1 0.634 0.765 0.840 0.160
#> GSM228667 1 0.653 0.743 0.832 0.168
#> GSM228668 1 0.574 0.762 0.864 0.136
#> GSM228669 1 0.541 0.759 0.876 0.124
#> GSM228673 1 0.644 0.747 0.836 0.164
#> GSM228677 1 0.753 0.710 0.784 0.216
#> GSM228678 1 0.808 0.640 0.752 0.248
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.547 0.773 0.792 0.032 0.176
#> GSM228563 1 0.808 0.611 0.632 0.116 0.252
#> GSM228565 1 0.557 0.777 0.796 0.044 0.160
#> GSM228566 1 0.665 0.705 0.712 0.048 0.240
#> GSM228567 1 0.468 0.743 0.832 0.020 0.148
#> GSM228570 1 0.475 0.779 0.832 0.024 0.144
#> GSM228571 1 0.414 0.781 0.860 0.016 0.124
#> GSM228574 1 0.517 0.766 0.792 0.016 0.192
#> GSM228575 2 0.895 0.000 0.260 0.560 0.180
#> GSM228576 1 0.530 0.775 0.808 0.036 0.156
#> GSM228579 1 0.468 0.783 0.832 0.020 0.148
#> GSM228580 3 0.909 0.000 0.140 0.400 0.460
#> GSM228581 1 0.871 0.355 0.508 0.112 0.380
#> GSM228666 1 0.732 0.700 0.668 0.068 0.264
#> GSM228564 1 0.752 0.664 0.680 0.100 0.220
#> GSM228568 1 0.547 0.775 0.792 0.032 0.176
#> GSM228569 1 0.457 0.774 0.828 0.012 0.160
#> GSM228572 1 0.816 0.605 0.608 0.104 0.288
#> GSM228573 1 0.405 0.771 0.848 0.004 0.148
#> GSM228577 1 0.460 0.781 0.832 0.016 0.152
#> GSM228578 1 0.506 0.771 0.816 0.028 0.156
#> GSM228663 1 0.563 0.728 0.768 0.024 0.208
#> GSM228664 1 0.714 0.581 0.644 0.044 0.312
#> GSM228665 1 0.501 0.767 0.788 0.008 0.204
#> GSM228582 1 0.509 0.787 0.804 0.020 0.176
#> GSM228583 1 0.468 0.741 0.832 0.020 0.148
#> GSM228585 1 0.468 0.738 0.832 0.020 0.148
#> GSM228587 1 0.533 0.725 0.792 0.024 0.184
#> GSM228588 1 0.774 0.615 0.632 0.080 0.288
#> GSM228589 1 0.760 0.648 0.612 0.060 0.328
#> GSM228590 1 0.486 0.736 0.820 0.020 0.160
#> GSM228591 1 0.738 0.616 0.616 0.048 0.336
#> GSM228597 1 0.767 0.636 0.652 0.088 0.260
#> GSM228601 1 0.741 0.578 0.596 0.044 0.360
#> GSM228604 1 0.686 0.691 0.696 0.052 0.252
#> GSM228608 1 0.466 0.747 0.828 0.016 0.156
#> GSM228609 1 0.795 0.579 0.608 0.084 0.308
#> GSM228613 1 0.474 0.740 0.828 0.020 0.152
#> GSM228616 1 0.535 0.783 0.808 0.040 0.152
#> GSM228628 1 0.733 0.633 0.624 0.048 0.328
#> GSM228634 1 0.448 0.765 0.840 0.016 0.144
#> GSM228642 1 0.844 0.405 0.536 0.096 0.368
#> GSM228645 1 0.737 0.648 0.668 0.072 0.260
#> GSM228646 1 0.728 0.667 0.652 0.056 0.292
#> GSM228652 1 0.498 0.753 0.812 0.020 0.168
#> GSM228655 1 0.468 0.768 0.832 0.020 0.148
#> GSM228656 1 0.454 0.743 0.836 0.016 0.148
#> GSM228659 1 0.573 0.728 0.772 0.032 0.196
#> GSM228662 1 0.474 0.737 0.828 0.020 0.152
#> GSM228584 1 0.474 0.742 0.828 0.020 0.152
#> GSM228586 1 0.466 0.764 0.828 0.016 0.156
#> GSM228592 1 0.474 0.743 0.828 0.020 0.152
#> GSM228593 1 0.757 0.670 0.668 0.092 0.240
#> GSM228594 1 0.423 0.772 0.844 0.008 0.148
#> GSM228598 1 0.582 0.770 0.788 0.056 0.156
#> GSM228607 1 0.500 0.783 0.820 0.028 0.152
#> GSM228612 1 0.546 0.761 0.768 0.016 0.216
#> GSM228619 1 0.439 0.776 0.840 0.012 0.148
#> GSM228622 1 0.399 0.781 0.864 0.012 0.124
#> GSM228625 1 0.512 0.767 0.796 0.016 0.188
#> GSM228631 1 0.435 0.777 0.836 0.008 0.156
#> GSM228633 1 0.849 0.480 0.536 0.100 0.364
#> GSM228637 1 0.837 0.579 0.608 0.132 0.260
#> GSM228639 1 0.552 0.781 0.788 0.032 0.180
#> GSM228649 1 0.651 0.730 0.720 0.044 0.236
#> GSM228660 1 0.486 0.775 0.808 0.012 0.180
#> GSM228661 1 0.416 0.770 0.848 0.008 0.144
#> GSM228595 1 0.778 0.545 0.576 0.060 0.364
#> GSM228599 1 0.659 0.740 0.728 0.056 0.216
#> GSM228602 1 0.470 0.765 0.812 0.008 0.180
#> GSM228614 1 0.656 0.725 0.720 0.048 0.232
#> GSM228626 1 0.793 0.461 0.552 0.064 0.384
#> GSM228640 1 0.515 0.745 0.800 0.020 0.180
#> GSM228643 1 0.486 0.749 0.808 0.012 0.180
#> GSM228650 1 0.611 0.741 0.764 0.052 0.184
#> GSM228653 1 0.491 0.748 0.804 0.012 0.184
#> GSM228657 1 0.759 0.568 0.588 0.052 0.360
#> GSM228605 1 0.506 0.780 0.816 0.028 0.156
#> GSM228610 1 0.491 0.759 0.796 0.008 0.196
#> GSM228617 1 0.423 0.776 0.844 0.008 0.148
#> GSM228620 1 0.486 0.762 0.808 0.012 0.180
#> GSM228623 1 0.646 0.708 0.724 0.044 0.232
#> GSM228629 1 0.504 0.761 0.808 0.020 0.172
#> GSM228632 1 0.509 0.760 0.804 0.020 0.176
#> GSM228635 1 0.844 0.567 0.608 0.144 0.248
#> GSM228647 1 0.541 0.741 0.780 0.020 0.200
#> GSM228596 1 0.496 0.766 0.792 0.008 0.200
#> GSM228600 1 0.475 0.749 0.808 0.008 0.184
#> GSM228603 1 0.481 0.751 0.812 0.012 0.176
#> GSM228615 1 0.672 0.710 0.720 0.060 0.220
#> GSM228627 1 0.517 0.754 0.792 0.016 0.192
#> GSM228641 1 0.501 0.741 0.804 0.016 0.180
#> GSM228644 1 0.797 0.481 0.560 0.068 0.372
#> GSM228651 1 0.512 0.739 0.796 0.016 0.188
#> GSM228654 1 0.512 0.740 0.796 0.016 0.188
#> GSM228658 1 0.517 0.738 0.792 0.016 0.192
#> GSM228606 1 0.655 0.740 0.716 0.044 0.240
#> GSM228611 1 0.455 0.741 0.800 0.000 0.200
#> GSM228618 1 0.480 0.762 0.824 0.020 0.156
#> GSM228621 1 0.585 0.732 0.756 0.028 0.216
#> GSM228624 1 0.622 0.706 0.728 0.032 0.240
#> GSM228630 1 0.546 0.758 0.768 0.016 0.216
#> GSM228636 1 0.791 0.635 0.632 0.096 0.272
#> GSM228638 1 0.484 0.779 0.816 0.016 0.168
#> GSM228648 1 0.549 0.742 0.780 0.024 0.196
#> GSM228670 1 0.658 0.722 0.736 0.064 0.200
#> GSM228671 1 0.832 0.584 0.620 0.140 0.240
#> GSM228672 1 0.685 0.719 0.712 0.064 0.224
#> GSM228674 1 0.711 0.709 0.720 0.112 0.168
#> GSM228675 1 0.732 0.698 0.704 0.112 0.184
#> GSM228676 1 0.541 0.783 0.796 0.032 0.172
#> GSM228667 1 0.589 0.758 0.752 0.028 0.220
#> GSM228668 1 0.547 0.765 0.796 0.036 0.168
#> GSM228669 1 0.530 0.765 0.804 0.032 0.164
#> GSM228673 1 0.512 0.766 0.788 0.012 0.200
#> GSM228677 1 0.713 0.733 0.684 0.064 0.252
#> GSM228678 1 0.771 0.645 0.652 0.092 0.256
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 1 0.524 0.5108 0.760 0.048 0.016 0.176
#> GSM228563 4 0.815 0.4984 0.360 0.092 0.072 0.476
#> GSM228565 1 0.546 0.5028 0.728 0.040 0.016 0.216
#> GSM228566 1 0.599 0.4264 0.728 0.116 0.020 0.136
#> GSM228567 1 0.515 0.3695 0.596 0.008 0.000 0.396
#> GSM228570 1 0.517 0.5202 0.736 0.024 0.016 0.224
#> GSM228571 1 0.470 0.5404 0.764 0.016 0.012 0.208
#> GSM228574 1 0.455 0.5633 0.804 0.036 0.012 0.148
#> GSM228575 2 0.691 0.0000 0.252 0.636 0.060 0.052
#> GSM228576 1 0.546 0.5185 0.736 0.040 0.020 0.204
#> GSM228579 1 0.481 0.5457 0.752 0.016 0.012 0.220
#> GSM228580 3 0.484 0.0000 0.036 0.076 0.816 0.072
#> GSM228581 1 0.914 -0.2056 0.448 0.144 0.140 0.268
#> GSM228666 1 0.765 0.1441 0.580 0.116 0.048 0.256
#> GSM228564 4 0.771 0.3959 0.400 0.080 0.048 0.472
#> GSM228568 1 0.560 0.5022 0.708 0.044 0.012 0.236
#> GSM228569 1 0.455 0.5612 0.784 0.024 0.008 0.184
#> GSM228572 4 0.838 0.5227 0.296 0.136 0.068 0.500
#> GSM228573 1 0.331 0.5802 0.868 0.028 0.000 0.104
#> GSM228577 1 0.511 0.5332 0.712 0.020 0.008 0.260
#> GSM228578 1 0.571 0.4525 0.660 0.016 0.024 0.300
#> GSM228663 1 0.374 0.5346 0.868 0.052 0.016 0.064
#> GSM228664 1 0.670 0.1925 0.672 0.104 0.032 0.192
#> GSM228665 1 0.379 0.5737 0.844 0.016 0.012 0.128
#> GSM228582 1 0.553 0.5050 0.716 0.032 0.020 0.232
#> GSM228583 1 0.515 0.3662 0.596 0.008 0.000 0.396
#> GSM228585 1 0.517 0.3570 0.588 0.008 0.000 0.404
#> GSM228587 1 0.555 0.2097 0.532 0.012 0.004 0.452
#> GSM228588 4 0.703 0.5190 0.312 0.052 0.048 0.588
#> GSM228589 4 0.794 0.3690 0.396 0.120 0.036 0.448
#> GSM228590 1 0.531 0.3362 0.576 0.012 0.000 0.412
#> GSM228591 1 0.779 -0.3201 0.448 0.152 0.016 0.384
#> GSM228597 4 0.770 0.4929 0.352 0.068 0.064 0.516
#> GSM228601 1 0.795 -0.3691 0.428 0.148 0.024 0.400
#> GSM228604 1 0.688 0.3063 0.656 0.112 0.032 0.200
#> GSM228608 1 0.540 0.3791 0.600 0.012 0.004 0.384
#> GSM228609 4 0.712 0.5194 0.300 0.056 0.052 0.592
#> GSM228613 1 0.527 0.3610 0.592 0.012 0.000 0.396
#> GSM228616 1 0.531 0.5536 0.752 0.040 0.020 0.188
#> GSM228628 1 0.727 -0.1279 0.540 0.160 0.004 0.296
#> GSM228634 1 0.445 0.4735 0.692 0.000 0.000 0.308
#> GSM228642 1 0.860 -0.3036 0.412 0.248 0.036 0.304
#> GSM228645 1 0.727 0.1801 0.604 0.164 0.020 0.212
#> GSM228646 1 0.738 0.2078 0.596 0.124 0.032 0.248
#> GSM228652 1 0.607 0.3543 0.568 0.012 0.028 0.392
#> GSM228655 1 0.576 0.4457 0.632 0.012 0.024 0.332
#> GSM228656 1 0.516 0.3636 0.592 0.008 0.000 0.400
#> GSM228659 1 0.626 0.1321 0.500 0.012 0.032 0.456
#> GSM228662 1 0.529 0.3514 0.584 0.012 0.000 0.404
#> GSM228584 1 0.502 0.3726 0.600 0.004 0.000 0.396
#> GSM228586 1 0.468 0.4626 0.680 0.004 0.000 0.316
#> GSM228592 1 0.515 0.3702 0.596 0.008 0.000 0.396
#> GSM228593 4 0.752 0.4316 0.380 0.084 0.036 0.500
#> GSM228594 1 0.451 0.5126 0.708 0.004 0.000 0.288
#> GSM228598 1 0.603 0.4147 0.648 0.040 0.016 0.296
#> GSM228607 1 0.557 0.5064 0.724 0.040 0.020 0.216
#> GSM228612 1 0.452 0.5683 0.812 0.036 0.016 0.136
#> GSM228619 1 0.452 0.5418 0.768 0.028 0.000 0.204
#> GSM228622 1 0.505 0.5249 0.720 0.020 0.008 0.252
#> GSM228625 1 0.589 0.3664 0.600 0.024 0.012 0.364
#> GSM228631 1 0.453 0.5449 0.776 0.024 0.004 0.196
#> GSM228633 4 0.864 0.0834 0.368 0.192 0.048 0.392
#> GSM228637 4 0.817 0.5062 0.352 0.056 0.116 0.476
#> GSM228639 1 0.537 0.5411 0.756 0.044 0.024 0.176
#> GSM228649 1 0.721 -0.2496 0.476 0.048 0.044 0.432
#> GSM228660 1 0.568 0.4453 0.628 0.024 0.008 0.340
#> GSM228661 1 0.438 0.4904 0.704 0.000 0.000 0.296
#> GSM228595 4 0.825 0.2556 0.380 0.160 0.036 0.424
#> GSM228599 1 0.700 0.0485 0.536 0.060 0.028 0.376
#> GSM228602 1 0.357 0.5739 0.864 0.028 0.008 0.100
#> GSM228614 1 0.740 -0.1502 0.484 0.072 0.036 0.408
#> GSM228626 1 0.822 -0.3207 0.404 0.208 0.020 0.368
#> GSM228640 1 0.274 0.5573 0.912 0.036 0.008 0.044
#> GSM228643 1 0.231 0.5619 0.924 0.032 0.000 0.044
#> GSM228650 1 0.511 0.5255 0.800 0.064 0.040 0.096
#> GSM228653 1 0.209 0.5627 0.932 0.020 0.000 0.048
#> GSM228657 4 0.817 0.3031 0.388 0.148 0.036 0.428
#> GSM228605 1 0.556 0.5024 0.716 0.040 0.016 0.228
#> GSM228610 1 0.407 0.5584 0.840 0.036 0.012 0.112
#> GSM228617 1 0.445 0.5461 0.776 0.028 0.000 0.196
#> GSM228620 1 0.318 0.5764 0.892 0.024 0.016 0.068
#> GSM228623 4 0.714 0.2774 0.444 0.048 0.040 0.468
#> GSM228629 1 0.320 0.5757 0.892 0.036 0.012 0.060
#> GSM228632 1 0.400 0.5721 0.844 0.028 0.016 0.112
#> GSM228635 4 0.860 0.5221 0.340 0.084 0.124 0.452
#> GSM228647 1 0.269 0.5530 0.912 0.044 0.004 0.040
#> GSM228596 1 0.366 0.5727 0.852 0.020 0.008 0.120
#> GSM228600 1 0.302 0.5647 0.896 0.020 0.012 0.072
#> GSM228603 1 0.283 0.5622 0.904 0.032 0.004 0.060
#> GSM228615 1 0.736 -0.1938 0.480 0.068 0.036 0.416
#> GSM228627 1 0.308 0.5692 0.892 0.036 0.004 0.068
#> GSM228641 1 0.264 0.5539 0.916 0.032 0.008 0.044
#> GSM228644 4 0.832 0.1583 0.392 0.180 0.032 0.396
#> GSM228651 1 0.250 0.5604 0.920 0.036 0.004 0.040
#> GSM228654 1 0.291 0.5531 0.900 0.032 0.004 0.064
#> GSM228658 1 0.235 0.5591 0.928 0.016 0.012 0.044
#> GSM228606 1 0.602 0.4242 0.720 0.092 0.020 0.168
#> GSM228611 1 0.327 0.5540 0.884 0.032 0.008 0.076
#> GSM228618 1 0.365 0.5759 0.872 0.044 0.016 0.068
#> GSM228621 1 0.467 0.5330 0.820 0.064 0.024 0.092
#> GSM228624 1 0.521 0.4549 0.776 0.088 0.012 0.124
#> GSM228630 1 0.457 0.5599 0.808 0.036 0.016 0.140
#> GSM228636 4 0.827 0.5307 0.344 0.096 0.080 0.480
#> GSM228638 1 0.478 0.5617 0.788 0.040 0.012 0.160
#> GSM228648 1 0.397 0.5476 0.856 0.056 0.016 0.072
#> GSM228670 1 0.735 -0.0718 0.504 0.076 0.032 0.388
#> GSM228671 4 0.907 0.0610 0.336 0.236 0.068 0.360
#> GSM228672 4 0.645 0.1469 0.472 0.036 0.016 0.476
#> GSM228674 1 0.770 -0.0791 0.504 0.104 0.036 0.356
#> GSM228675 1 0.818 -0.1844 0.496 0.152 0.044 0.308
#> GSM228676 1 0.598 0.4118 0.668 0.052 0.012 0.268
#> GSM228667 1 0.642 0.0706 0.540 0.044 0.012 0.404
#> GSM228668 1 0.589 0.4335 0.656 0.024 0.024 0.296
#> GSM228669 1 0.588 0.4235 0.644 0.020 0.024 0.312
#> GSM228673 1 0.450 0.5568 0.808 0.036 0.012 0.144
#> GSM228677 1 0.760 0.0853 0.568 0.092 0.052 0.288
#> GSM228678 4 0.776 0.5038 0.380 0.092 0.044 0.484
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 3 0.541 0.4945 0.016 0.064 0.716 0.184 0.020
#> GSM228563 4 0.767 0.6358 0.080 0.096 0.276 0.516 0.032
#> GSM228565 3 0.544 0.4831 0.012 0.056 0.696 0.216 0.020
#> GSM228566 3 0.573 0.4299 0.020 0.172 0.688 0.112 0.008
#> GSM228567 3 0.456 0.2488 0.000 0.004 0.552 0.440 0.004
#> GSM228570 3 0.514 0.5089 0.008 0.044 0.712 0.216 0.020
#> GSM228571 3 0.476 0.5226 0.000 0.032 0.724 0.220 0.024
#> GSM228574 3 0.474 0.5673 0.012 0.060 0.772 0.140 0.016
#> GSM228575 1 0.784 0.1760 0.552 0.124 0.192 0.064 0.068
#> GSM228576 3 0.536 0.5118 0.012 0.052 0.716 0.192 0.028
#> GSM228579 3 0.497 0.5239 0.004 0.032 0.712 0.228 0.024
#> GSM228580 5 0.292 0.0000 0.000 0.080 0.016 0.024 0.880
#> GSM228581 3 0.874 -0.4013 0.036 0.272 0.392 0.172 0.128
#> GSM228666 3 0.779 0.1010 0.064 0.172 0.532 0.196 0.036
#> GSM228564 4 0.771 0.5862 0.064 0.088 0.316 0.488 0.044
#> GSM228568 3 0.586 0.4340 0.032 0.052 0.652 0.252 0.012
#> GSM228569 3 0.465 0.5411 0.020 0.028 0.752 0.192 0.008
#> GSM228572 4 0.854 0.0892 0.064 0.280 0.180 0.416 0.060
#> GSM228573 3 0.345 0.5881 0.016 0.028 0.844 0.112 0.000
#> GSM228577 3 0.513 0.4847 0.012 0.040 0.672 0.272 0.004
#> GSM228578 3 0.564 0.3915 0.032 0.020 0.612 0.324 0.012
#> GSM228663 3 0.385 0.5525 0.020 0.080 0.840 0.052 0.008
#> GSM228664 3 0.663 -0.0429 0.032 0.260 0.596 0.092 0.020
#> GSM228665 3 0.373 0.5828 0.012 0.024 0.828 0.128 0.008
#> GSM228582 3 0.560 0.4253 0.012 0.060 0.656 0.260 0.012
#> GSM228583 3 0.456 0.2443 0.000 0.004 0.552 0.440 0.004
#> GSM228585 3 0.457 0.2322 0.000 0.004 0.544 0.448 0.004
#> GSM228587 4 0.526 0.0413 0.004 0.028 0.464 0.500 0.004
#> GSM228588 4 0.712 0.6006 0.056 0.128 0.244 0.560 0.012
#> GSM228589 4 0.785 -0.0697 0.020 0.248 0.304 0.396 0.032
#> GSM228590 3 0.494 0.1443 0.004 0.012 0.512 0.468 0.004
#> GSM228591 2 0.725 0.4098 0.008 0.368 0.344 0.272 0.008
#> GSM228597 4 0.748 0.6493 0.068 0.088 0.256 0.548 0.040
#> GSM228601 2 0.708 0.4942 0.004 0.416 0.312 0.260 0.008
#> GSM228604 3 0.630 0.3195 0.012 0.224 0.620 0.128 0.016
#> GSM228608 3 0.501 0.2493 0.008 0.012 0.548 0.428 0.004
#> GSM228609 4 0.694 0.6021 0.044 0.140 0.224 0.580 0.012
#> GSM228613 3 0.470 0.2380 0.004 0.004 0.548 0.440 0.004
#> GSM228616 3 0.472 0.5476 0.000 0.056 0.744 0.184 0.016
#> GSM228628 3 0.661 -0.4220 0.000 0.360 0.448 0.188 0.004
#> GSM228634 3 0.445 0.3954 0.004 0.004 0.644 0.344 0.004
#> GSM228642 2 0.695 0.4447 0.020 0.508 0.316 0.144 0.012
#> GSM228645 3 0.728 0.1686 0.052 0.208 0.548 0.180 0.012
#> GSM228646 3 0.734 0.1723 0.032 0.156 0.544 0.236 0.032
#> GSM228652 3 0.558 0.1712 0.016 0.024 0.516 0.436 0.008
#> GSM228655 3 0.536 0.3426 0.016 0.020 0.588 0.368 0.008
#> GSM228656 3 0.455 0.2457 0.004 0.004 0.556 0.436 0.000
#> GSM228659 4 0.618 0.2220 0.028 0.048 0.412 0.504 0.008
#> GSM228662 3 0.471 0.2247 0.004 0.004 0.540 0.448 0.004
#> GSM228584 3 0.452 0.2491 0.000 0.000 0.552 0.440 0.008
#> GSM228586 3 0.460 0.3724 0.008 0.004 0.632 0.352 0.004
#> GSM228592 3 0.453 0.2429 0.000 0.000 0.548 0.444 0.008
#> GSM228593 4 0.720 0.6105 0.056 0.060 0.296 0.544 0.044
#> GSM228594 3 0.464 0.4589 0.008 0.012 0.668 0.308 0.004
#> GSM228598 3 0.587 0.2979 0.028 0.020 0.592 0.336 0.024
#> GSM228607 3 0.559 0.4685 0.048 0.040 0.676 0.232 0.004
#> GSM228612 3 0.420 0.5757 0.004 0.064 0.804 0.116 0.012
#> GSM228619 3 0.456 0.5334 0.028 0.016 0.756 0.192 0.008
#> GSM228622 3 0.461 0.5091 0.020 0.008 0.700 0.268 0.004
#> GSM228625 3 0.601 0.1707 0.020 0.044 0.544 0.380 0.012
#> GSM228631 3 0.458 0.5359 0.024 0.024 0.760 0.184 0.008
#> GSM228633 2 0.647 0.4867 0.028 0.616 0.224 0.120 0.012
#> GSM228637 4 0.784 0.6157 0.140 0.044 0.260 0.504 0.052
#> GSM228639 3 0.566 0.5293 0.052 0.048 0.712 0.172 0.016
#> GSM228649 4 0.736 0.5161 0.068 0.064 0.384 0.456 0.028
#> GSM228660 3 0.535 0.3660 0.012 0.032 0.596 0.356 0.004
#> GSM228661 3 0.425 0.4226 0.000 0.004 0.660 0.332 0.004
#> GSM228595 2 0.674 0.6276 0.008 0.544 0.240 0.196 0.012
#> GSM228599 3 0.709 -0.2823 0.044 0.068 0.476 0.384 0.028
#> GSM228602 3 0.374 0.5862 0.012 0.028 0.836 0.112 0.012
#> GSM228614 4 0.712 0.3689 0.064 0.072 0.420 0.432 0.012
#> GSM228626 2 0.519 0.5372 0.000 0.656 0.260 0.084 0.000
#> GSM228640 3 0.261 0.5738 0.012 0.024 0.908 0.044 0.012
#> GSM228643 3 0.239 0.5800 0.004 0.044 0.908 0.044 0.000
#> GSM228650 3 0.501 0.5270 0.008 0.088 0.772 0.084 0.048
#> GSM228653 3 0.241 0.5803 0.008 0.024 0.912 0.052 0.004
#> GSM228657 2 0.708 0.6069 0.016 0.488 0.276 0.212 0.008
#> GSM228605 3 0.568 0.4837 0.036 0.052 0.676 0.228 0.008
#> GSM228610 3 0.394 0.5738 0.028 0.048 0.832 0.088 0.004
#> GSM228617 3 0.444 0.5380 0.024 0.016 0.764 0.188 0.008
#> GSM228620 3 0.340 0.5880 0.016 0.040 0.868 0.064 0.012
#> GSM228623 4 0.735 0.5568 0.056 0.104 0.344 0.480 0.016
#> GSM228629 3 0.356 0.5882 0.024 0.048 0.856 0.068 0.004
#> GSM228632 3 0.442 0.5785 0.024 0.052 0.808 0.100 0.016
#> GSM228635 4 0.826 0.5456 0.160 0.072 0.236 0.480 0.052
#> GSM228647 3 0.298 0.5728 0.012 0.044 0.888 0.048 0.008
#> GSM228596 3 0.364 0.5849 0.008 0.024 0.836 0.120 0.012
#> GSM228600 3 0.307 0.5796 0.008 0.028 0.880 0.072 0.012
#> GSM228603 3 0.286 0.5790 0.012 0.024 0.888 0.072 0.004
#> GSM228615 4 0.741 0.4578 0.072 0.064 0.392 0.444 0.028
#> GSM228627 3 0.303 0.5833 0.008 0.048 0.880 0.060 0.004
#> GSM228641 3 0.268 0.5704 0.008 0.040 0.904 0.036 0.012
#> GSM228644 2 0.578 0.6197 0.000 0.616 0.252 0.128 0.004
#> GSM228651 3 0.229 0.5770 0.000 0.048 0.916 0.024 0.012
#> GSM228654 3 0.288 0.5679 0.008 0.060 0.888 0.040 0.004
#> GSM228658 3 0.216 0.5770 0.004 0.024 0.928 0.028 0.016
#> GSM228606 3 0.625 0.4121 0.044 0.128 0.668 0.148 0.012
#> GSM228611 3 0.355 0.5734 0.016 0.048 0.860 0.064 0.012
#> GSM228618 3 0.366 0.5869 0.024 0.044 0.856 0.064 0.012
#> GSM228621 3 0.474 0.5485 0.008 0.088 0.784 0.092 0.028
#> GSM228624 3 0.526 0.4725 0.024 0.144 0.736 0.088 0.008
#> GSM228630 3 0.481 0.5625 0.020 0.064 0.772 0.132 0.012
#> GSM228636 4 0.786 0.5563 0.096 0.108 0.236 0.524 0.036
#> GSM228638 3 0.472 0.5643 0.036 0.036 0.764 0.160 0.004
#> GSM228648 3 0.396 0.5664 0.012 0.080 0.832 0.064 0.012
#> GSM228670 3 0.733 -0.3721 0.056 0.104 0.432 0.396 0.012
#> GSM228671 1 0.919 0.3122 0.304 0.220 0.236 0.204 0.036
#> GSM228672 4 0.669 0.4328 0.044 0.048 0.396 0.492 0.020
#> GSM228674 3 0.804 -0.2682 0.120 0.096 0.444 0.316 0.024
#> GSM228675 3 0.839 -0.3266 0.164 0.136 0.420 0.264 0.016
#> GSM228676 3 0.622 0.3221 0.044 0.056 0.620 0.268 0.012
#> GSM228667 3 0.677 -0.2062 0.028 0.080 0.472 0.404 0.016
#> GSM228668 3 0.571 0.3680 0.036 0.020 0.608 0.324 0.012
#> GSM228669 3 0.577 0.3485 0.036 0.020 0.592 0.340 0.012
#> GSM228673 3 0.505 0.5587 0.032 0.072 0.764 0.120 0.012
#> GSM228677 3 0.756 -0.1493 0.048 0.120 0.484 0.320 0.028
#> GSM228678 4 0.775 0.5733 0.088 0.120 0.272 0.500 0.020
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 3 0.559 0.4972 0.032 0.044 0.688 0.184 0.016 0.036
#> GSM228563 4 0.771 0.5850 0.104 0.080 0.212 0.516 0.036 0.052
#> GSM228565 3 0.547 0.4837 0.028 0.032 0.668 0.228 0.012 0.032
#> GSM228566 3 0.600 0.4406 0.080 0.136 0.660 0.104 0.012 0.008
#> GSM228567 3 0.385 0.2710 0.000 0.000 0.536 0.464 0.000 0.000
#> GSM228570 3 0.491 0.5205 0.012 0.028 0.704 0.216 0.008 0.032
#> GSM228571 3 0.475 0.5321 0.016 0.028 0.712 0.216 0.004 0.024
#> GSM228574 3 0.457 0.5828 0.024 0.044 0.768 0.132 0.004 0.028
#> GSM228575 1 0.562 0.0000 0.700 0.060 0.152 0.040 0.012 0.036
#> GSM228576 3 0.526 0.5218 0.024 0.044 0.700 0.192 0.008 0.032
#> GSM228579 3 0.485 0.5317 0.016 0.028 0.704 0.220 0.004 0.028
#> GSM228580 5 0.175 0.0000 0.000 0.040 0.012 0.016 0.932 0.000
#> GSM228581 3 0.948 -0.4798 0.132 0.200 0.300 0.184 0.076 0.108
#> GSM228666 3 0.806 0.0448 0.056 0.132 0.476 0.216 0.040 0.080
#> GSM228564 4 0.764 0.5287 0.076 0.076 0.268 0.492 0.040 0.048
#> GSM228568 3 0.559 0.4392 0.060 0.016 0.636 0.252 0.004 0.032
#> GSM228569 3 0.475 0.5428 0.036 0.020 0.728 0.184 0.000 0.032
#> GSM228572 4 0.841 -0.0212 0.048 0.320 0.112 0.368 0.052 0.100
#> GSM228573 3 0.361 0.5974 0.020 0.032 0.824 0.112 0.000 0.012
#> GSM228577 3 0.502 0.4897 0.024 0.024 0.656 0.272 0.000 0.024
#> GSM228578 3 0.568 0.4148 0.024 0.028 0.596 0.312 0.016 0.024
#> GSM228663 3 0.422 0.5573 0.028 0.080 0.808 0.052 0.008 0.024
#> GSM228664 3 0.723 -0.0833 0.052 0.200 0.548 0.128 0.016 0.056
#> GSM228665 3 0.430 0.5885 0.016 0.048 0.792 0.112 0.008 0.024
#> GSM228582 3 0.600 0.4150 0.032 0.072 0.600 0.268 0.008 0.020
#> GSM228583 3 0.385 0.2676 0.000 0.000 0.536 0.464 0.000 0.000
#> GSM228585 3 0.386 0.2571 0.000 0.000 0.528 0.472 0.000 0.000
#> GSM228587 4 0.495 0.0123 0.012 0.020 0.436 0.520 0.000 0.012
#> GSM228588 4 0.683 0.5136 0.060 0.144 0.180 0.576 0.008 0.032
#> GSM228589 4 0.751 -0.0832 0.040 0.288 0.256 0.380 0.024 0.012
#> GSM228590 4 0.439 -0.1811 0.004 0.000 0.480 0.500 0.000 0.016
#> GSM228591 2 0.651 0.4411 0.016 0.460 0.264 0.252 0.004 0.004
#> GSM228597 4 0.747 0.5993 0.072 0.084 0.208 0.544 0.040 0.052
#> GSM228601 2 0.650 0.5163 0.008 0.496 0.212 0.256 0.000 0.028
#> GSM228604 3 0.654 0.3308 0.040 0.224 0.584 0.116 0.020 0.016
#> GSM228608 3 0.444 0.2681 0.008 0.004 0.524 0.456 0.000 0.008
#> GSM228609 4 0.659 0.5267 0.064 0.120 0.156 0.612 0.004 0.044
#> GSM228613 3 0.399 0.2622 0.000 0.000 0.532 0.464 0.000 0.004
#> GSM228616 3 0.498 0.5475 0.028 0.052 0.708 0.196 0.012 0.004
#> GSM228628 2 0.623 0.3252 0.004 0.440 0.360 0.184 0.000 0.012
#> GSM228634 3 0.422 0.3998 0.012 0.000 0.620 0.360 0.000 0.008
#> GSM228642 2 0.732 0.3647 0.092 0.504 0.256 0.100 0.008 0.040
#> GSM228645 3 0.752 0.1668 0.124 0.148 0.504 0.184 0.008 0.032
#> GSM228646 3 0.766 0.1786 0.096 0.104 0.504 0.228 0.032 0.036
#> GSM228652 3 0.544 0.1996 0.012 0.032 0.488 0.444 0.004 0.020
#> GSM228655 3 0.520 0.3598 0.012 0.032 0.556 0.384 0.004 0.012
#> GSM228656 3 0.424 0.2620 0.004 0.004 0.536 0.452 0.000 0.004
#> GSM228659 4 0.608 0.2005 0.024 0.060 0.368 0.516 0.004 0.028
#> GSM228662 3 0.399 0.2496 0.000 0.000 0.524 0.472 0.000 0.004
#> GSM228584 3 0.411 0.2762 0.004 0.000 0.536 0.456 0.000 0.004
#> GSM228586 3 0.433 0.3810 0.016 0.000 0.608 0.368 0.000 0.008
#> GSM228592 3 0.411 0.2703 0.004 0.000 0.532 0.460 0.000 0.004
#> GSM228593 4 0.728 0.5717 0.052 0.068 0.236 0.536 0.020 0.088
#> GSM228594 3 0.453 0.4619 0.016 0.004 0.648 0.312 0.000 0.020
#> GSM228598 3 0.569 0.3202 0.040 0.008 0.572 0.332 0.008 0.040
#> GSM228607 3 0.615 0.4606 0.036 0.048 0.624 0.232 0.016 0.044
#> GSM228612 3 0.463 0.5819 0.036 0.060 0.768 0.116 0.016 0.004
#> GSM228619 3 0.448 0.5483 0.012 0.020 0.732 0.208 0.008 0.020
#> GSM228622 3 0.445 0.5156 0.016 0.008 0.684 0.276 0.008 0.008
#> GSM228625 3 0.566 0.1535 0.000 0.032 0.488 0.424 0.012 0.044
#> GSM228631 3 0.450 0.5506 0.008 0.024 0.736 0.200 0.012 0.020
#> GSM228633 2 0.518 0.4287 0.040 0.728 0.152 0.040 0.012 0.028
#> GSM228637 4 0.829 0.5051 0.112 0.072 0.176 0.484 0.060 0.096
#> GSM228639 3 0.552 0.5422 0.040 0.040 0.692 0.180 0.008 0.040
#> GSM228649 4 0.769 0.4804 0.036 0.116 0.320 0.436 0.036 0.056
#> GSM228660 3 0.504 0.3653 0.000 0.040 0.552 0.388 0.000 0.020
#> GSM228661 3 0.408 0.4276 0.012 0.000 0.636 0.348 0.000 0.004
#> GSM228595 2 0.533 0.5584 0.008 0.672 0.156 0.148 0.008 0.008
#> GSM228599 3 0.693 -0.2536 0.016 0.072 0.420 0.400 0.012 0.080
#> GSM228602 3 0.359 0.5945 0.020 0.024 0.824 0.116 0.000 0.016
#> GSM228614 4 0.728 0.3079 0.020 0.096 0.376 0.412 0.016 0.080
#> GSM228626 2 0.377 0.4918 0.004 0.776 0.184 0.016 0.000 0.020
#> GSM228640 3 0.276 0.5844 0.012 0.032 0.892 0.044 0.012 0.008
#> GSM228643 3 0.257 0.5902 0.012 0.036 0.896 0.048 0.004 0.004
#> GSM228650 3 0.516 0.5419 0.028 0.092 0.744 0.084 0.044 0.008
#> GSM228653 3 0.274 0.5912 0.004 0.040 0.888 0.048 0.004 0.016
#> GSM228657 2 0.595 0.5477 0.004 0.608 0.188 0.168 0.008 0.024
#> GSM228605 3 0.599 0.4877 0.040 0.044 0.636 0.228 0.012 0.040
#> GSM228610 3 0.416 0.5813 0.020 0.052 0.808 0.084 0.004 0.032
#> GSM228617 3 0.437 0.5521 0.012 0.020 0.740 0.204 0.008 0.016
#> GSM228620 3 0.392 0.5954 0.024 0.048 0.828 0.068 0.008 0.024
#> GSM228623 4 0.735 0.5088 0.032 0.088 0.296 0.480 0.016 0.088
#> GSM228629 3 0.379 0.5952 0.024 0.044 0.836 0.068 0.012 0.016
#> GSM228632 3 0.446 0.5879 0.020 0.060 0.788 0.096 0.008 0.028
#> GSM228635 4 0.884 0.3866 0.144 0.096 0.152 0.420 0.056 0.132
#> GSM228647 3 0.319 0.5838 0.024 0.044 0.868 0.048 0.004 0.012
#> GSM228596 3 0.415 0.5918 0.016 0.044 0.800 0.112 0.008 0.020
#> GSM228600 3 0.301 0.5903 0.016 0.024 0.872 0.072 0.004 0.012
#> GSM228603 3 0.292 0.5900 0.012 0.024 0.880 0.064 0.012 0.008
#> GSM228615 4 0.723 0.4050 0.016 0.092 0.348 0.428 0.008 0.108
#> GSM228627 3 0.336 0.5919 0.016 0.048 0.856 0.060 0.004 0.016
#> GSM228641 3 0.274 0.5818 0.016 0.040 0.892 0.036 0.012 0.004
#> GSM228644 2 0.408 0.5329 0.004 0.772 0.164 0.044 0.004 0.012
#> GSM228651 3 0.294 0.5836 0.020 0.056 0.880 0.028 0.004 0.012
#> GSM228654 3 0.354 0.5719 0.020 0.076 0.840 0.052 0.004 0.008
#> GSM228658 3 0.259 0.5853 0.024 0.036 0.900 0.028 0.004 0.008
#> GSM228606 3 0.622 0.4396 0.044 0.088 0.668 0.124 0.016 0.060
#> GSM228611 3 0.379 0.5803 0.016 0.048 0.836 0.060 0.008 0.032
#> GSM228618 3 0.390 0.5958 0.012 0.068 0.820 0.072 0.004 0.024
#> GSM228621 3 0.482 0.5557 0.040 0.076 0.772 0.076 0.024 0.012
#> GSM228624 3 0.549 0.4678 0.044 0.136 0.700 0.096 0.004 0.020
#> GSM228630 3 0.495 0.5744 0.036 0.072 0.748 0.120 0.012 0.012
#> GSM228636 4 0.818 0.3941 0.072 0.148 0.144 0.476 0.024 0.136
#> GSM228638 3 0.452 0.5724 0.024 0.032 0.748 0.172 0.000 0.024
#> GSM228648 3 0.403 0.5754 0.028 0.080 0.812 0.064 0.004 0.012
#> GSM228670 3 0.747 -0.3285 0.028 0.092 0.384 0.376 0.008 0.112
#> GSM228671 6 0.499 0.0000 0.020 0.044 0.152 0.056 0.000 0.728
#> GSM228672 4 0.676 0.3719 0.032 0.048 0.356 0.484 0.012 0.068
#> GSM228674 3 0.785 -0.2423 0.036 0.060 0.392 0.312 0.020 0.180
#> GSM228675 3 0.799 -0.3041 0.036 0.076 0.364 0.232 0.012 0.280
#> GSM228676 3 0.632 0.3245 0.028 0.056 0.576 0.260 0.000 0.080
#> GSM228667 3 0.665 -0.1662 0.024 0.052 0.432 0.420 0.012 0.060
#> GSM228668 3 0.577 0.3920 0.024 0.028 0.584 0.320 0.016 0.028
#> GSM228669 3 0.574 0.3766 0.024 0.024 0.576 0.332 0.016 0.028
#> GSM228673 3 0.499 0.5737 0.024 0.040 0.752 0.120 0.012 0.052
#> GSM228677 3 0.776 -0.1306 0.040 0.124 0.448 0.288 0.024 0.076
#> GSM228678 4 0.832 0.4455 0.084 0.164 0.184 0.448 0.024 0.096
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
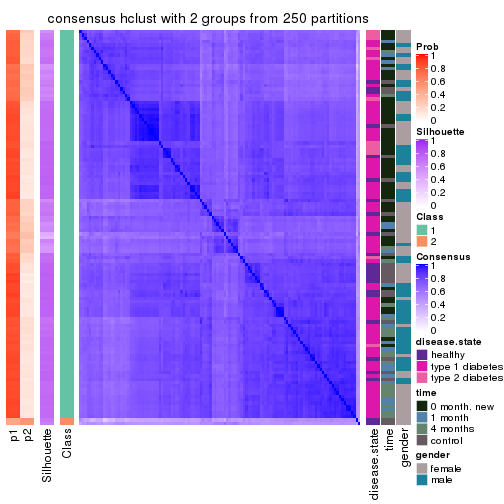
consensus_heatmap(res, k = 3)
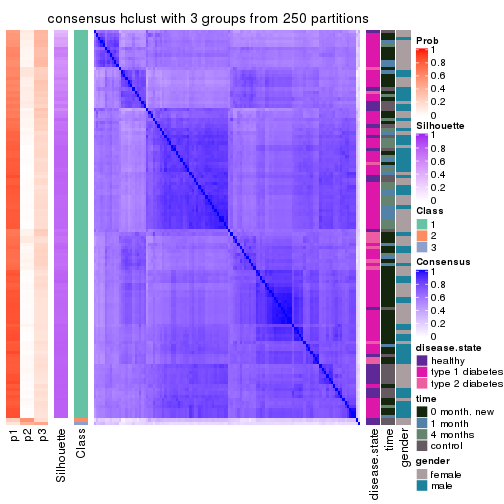
consensus_heatmap(res, k = 4)
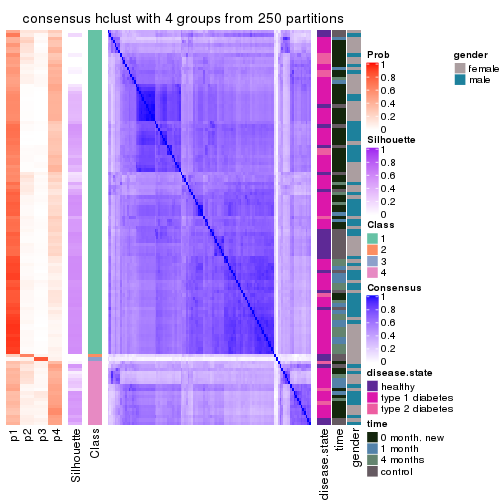
consensus_heatmap(res, k = 5)
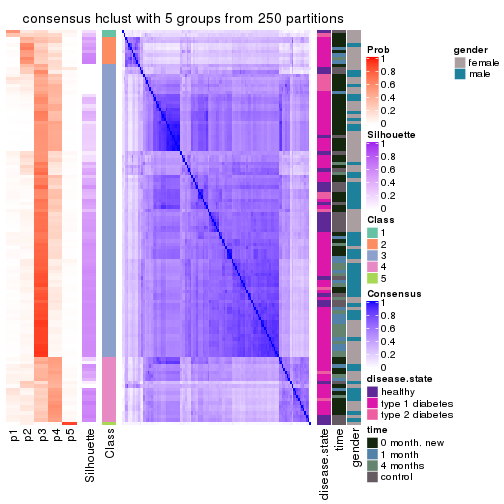
consensus_heatmap(res, k = 6)
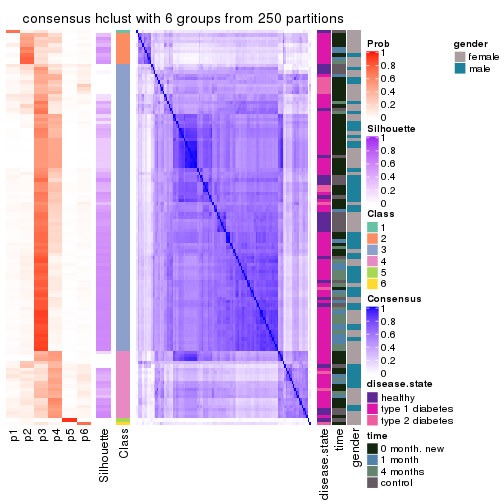
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
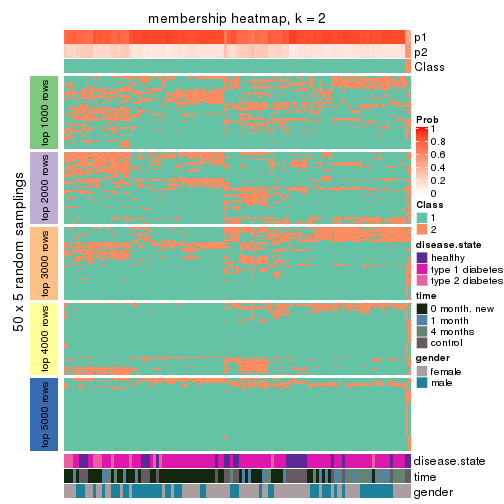
membership_heatmap(res, k = 3)
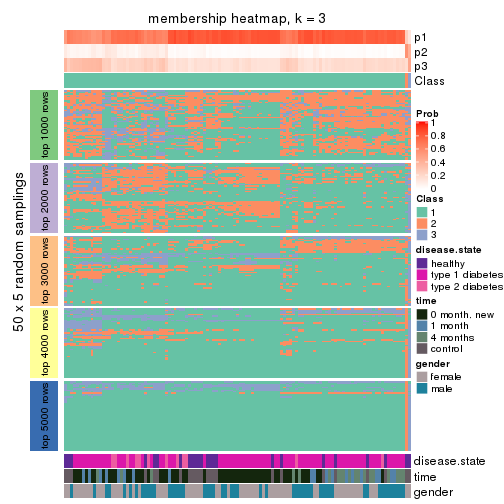
membership_heatmap(res, k = 4)
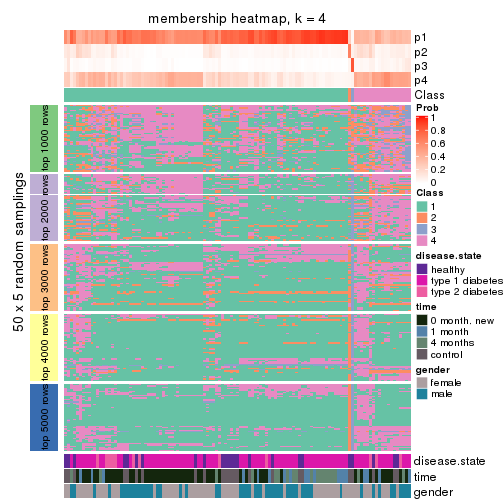
membership_heatmap(res, k = 5)
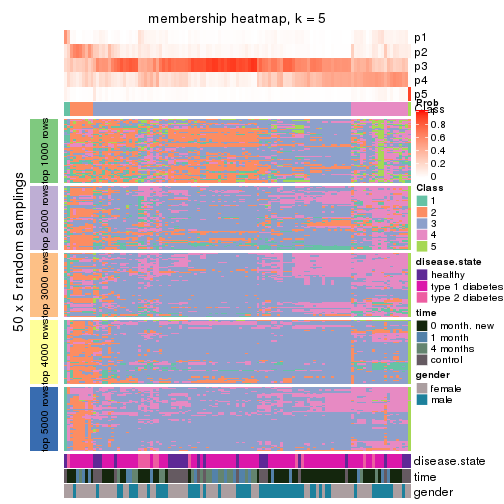
membership_heatmap(res, k = 6)
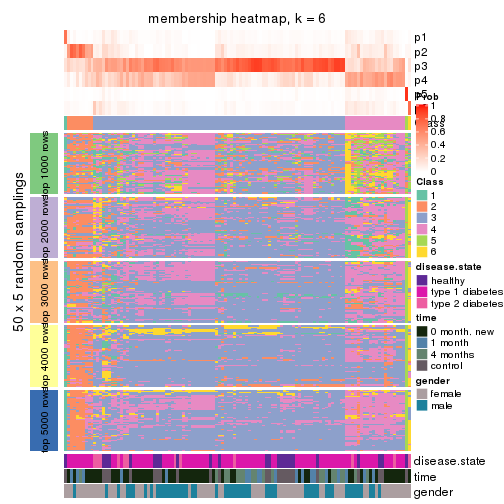
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
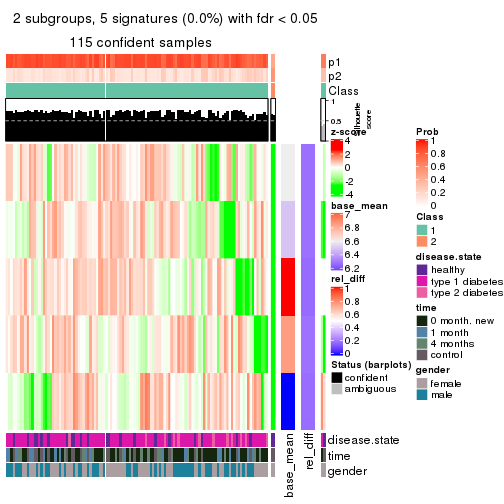
get_signatures(res, k = 3)

get_signatures(res, k = 4)
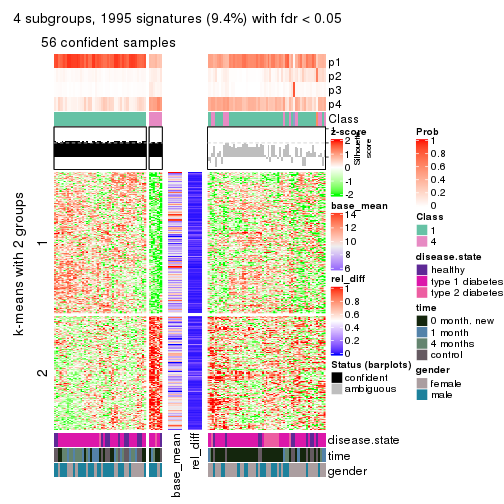
get_signatures(res, k = 5)
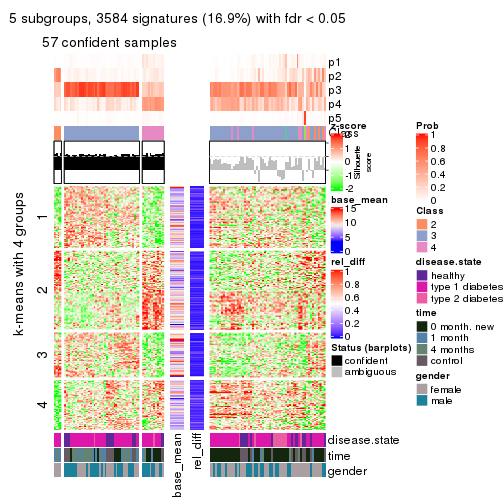
get_signatures(res, k = 6)
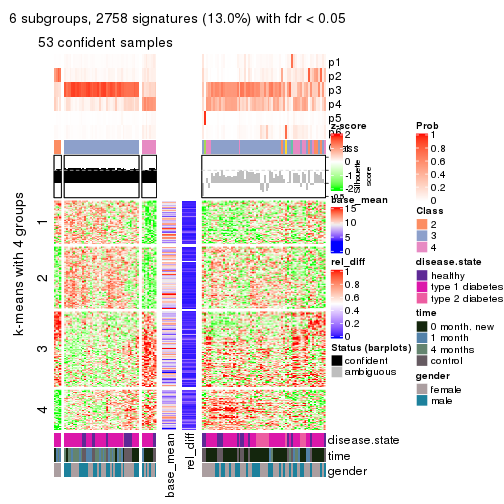
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
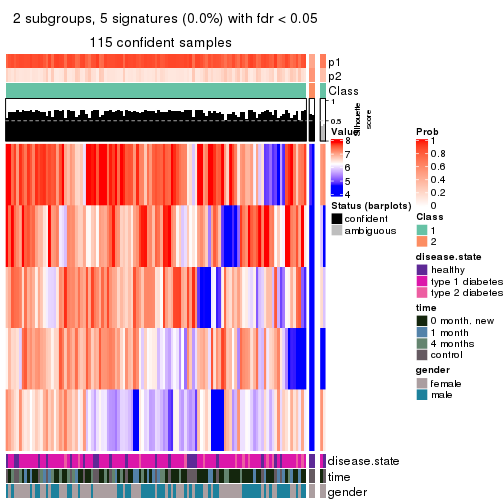
get_signatures(res, k = 3, scale_rows = FALSE)

get_signatures(res, k = 4, scale_rows = FALSE)
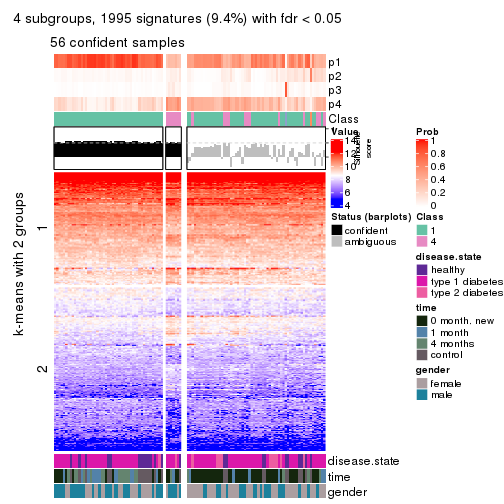
get_signatures(res, k = 5, scale_rows = FALSE)
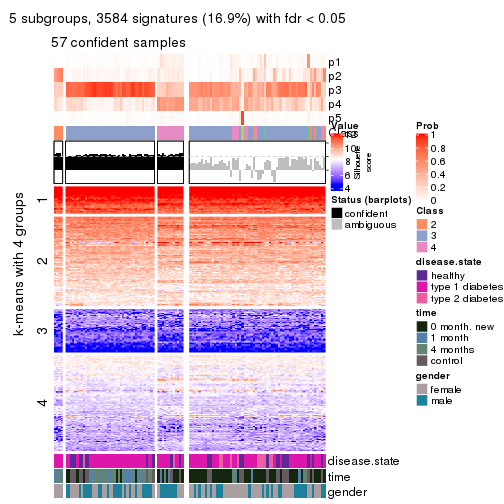
get_signatures(res, k = 6, scale_rows = FALSE)
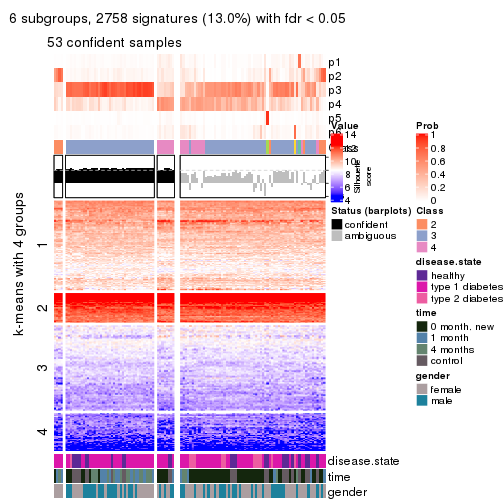
Compare the overlap of signatures from different k:
compare_signatures(res)
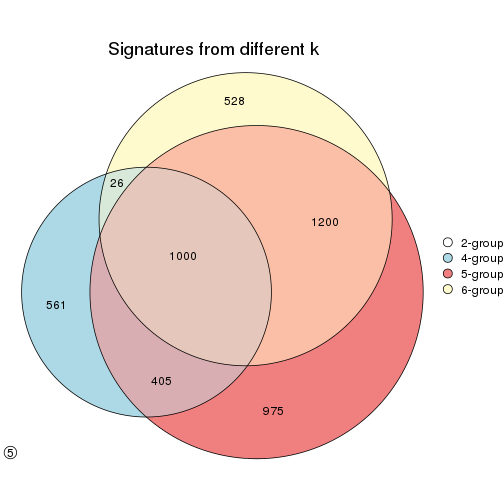
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
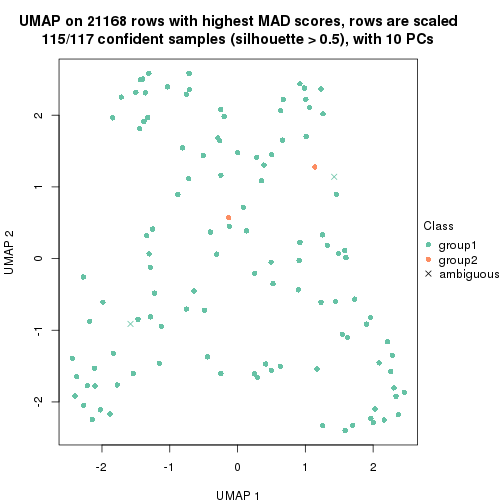
dimension_reduction(res, k = 3, method = "UMAP")
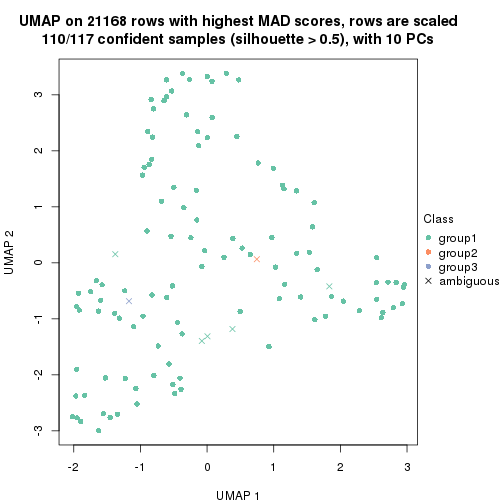
dimension_reduction(res, k = 4, method = "UMAP")
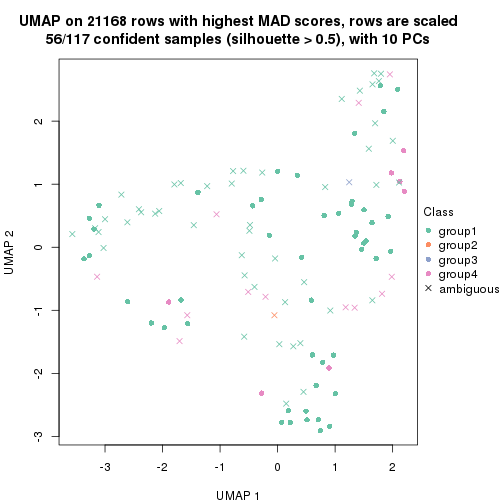
dimension_reduction(res, k = 5, method = "UMAP")
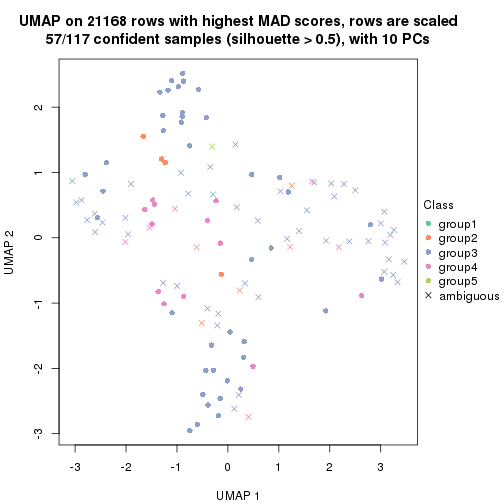
dimension_reduction(res, k = 6, method = "UMAP")
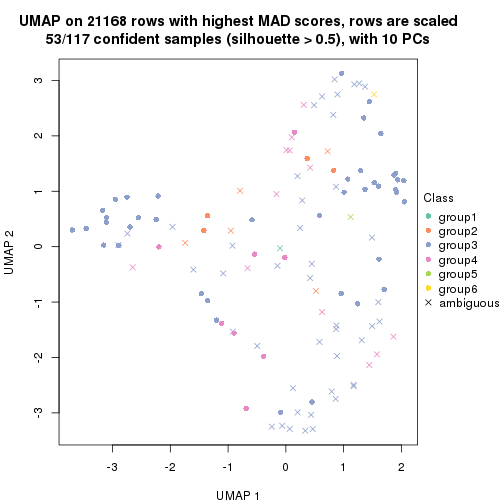
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
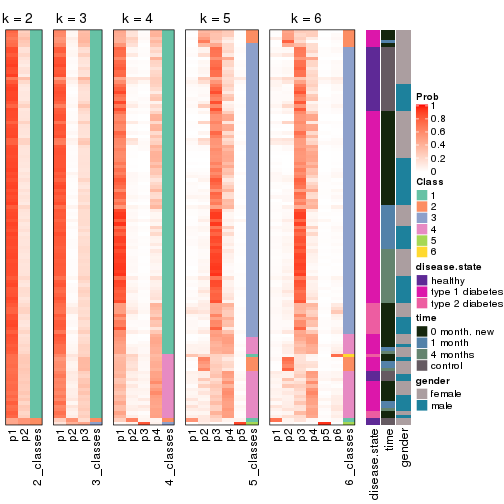
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> MAD:hclust 115 0.0171 0.0432 0.5622 2
#> MAD:hclust 110 NA NA NA 3
#> MAD:hclust 56 0.2301 0.2200 0.6831 4
#> MAD:hclust 57 0.6762 0.0256 0.0669 5
#> MAD:hclust 53 0.8253 0.0959 0.1223 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["MAD", "kmeans"]
# you can also extract it by
# res = res_list["MAD:kmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'MAD' method.
#> Subgroups are detected by 'kmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
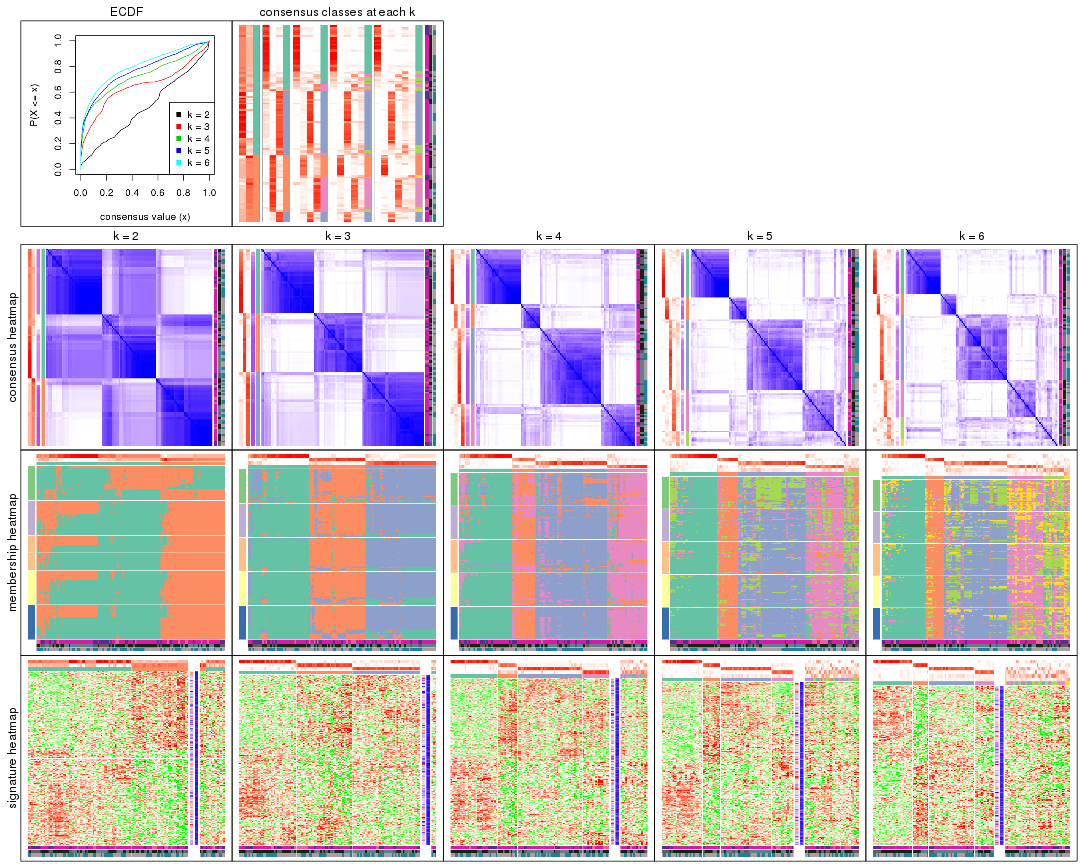
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
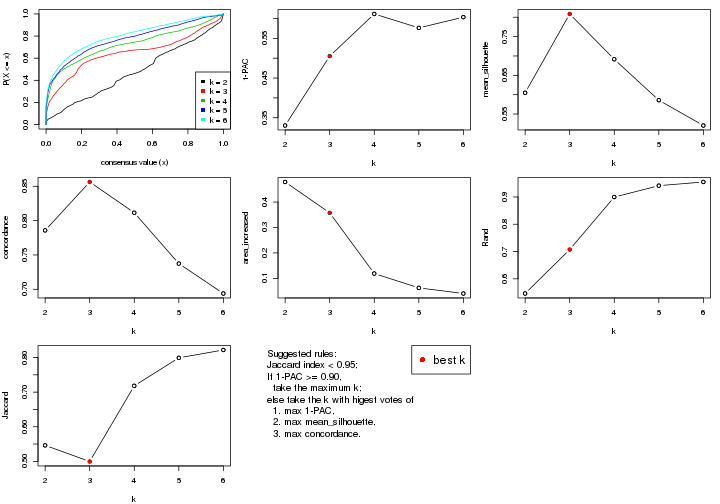
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.330 0.605 0.786 0.4789 0.546 0.546
#> 3 3 0.505 0.808 0.856 0.3575 0.707 0.499
#> 4 4 0.611 0.691 0.811 0.1189 0.900 0.718
#> 5 5 0.576 0.586 0.737 0.0625 0.941 0.799
#> 6 6 0.603 0.520 0.694 0.0403 0.955 0.822
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.7745 0.6835 0.772 0.228
#> GSM228563 2 0.7883 0.8038 0.236 0.764
#> GSM228565 1 0.8327 0.6848 0.736 0.264
#> GSM228566 1 0.6801 0.2973 0.820 0.180
#> GSM228567 1 0.9608 0.6864 0.616 0.384
#> GSM228570 1 0.9661 0.6841 0.608 0.392
#> GSM228571 1 0.8661 0.6877 0.712 0.288
#> GSM228574 1 0.9970 -0.6637 0.532 0.468
#> GSM228575 1 0.8207 0.0519 0.744 0.256
#> GSM228576 1 0.8327 0.6858 0.736 0.264
#> GSM228579 1 0.8608 0.6870 0.716 0.284
#> GSM228580 2 0.9608 0.8580 0.384 0.616
#> GSM228581 2 0.9866 0.8156 0.432 0.568
#> GSM228666 2 0.9608 0.8580 0.384 0.616
#> GSM228564 2 0.8955 0.0919 0.312 0.688
#> GSM228568 1 0.8016 0.6859 0.756 0.244
#> GSM228569 1 0.8955 0.6892 0.688 0.312
#> GSM228572 2 0.9608 0.8580 0.384 0.616
#> GSM228573 1 0.0000 0.6119 1.000 0.000
#> GSM228577 1 0.9580 0.6873 0.620 0.380
#> GSM228578 1 0.9427 0.6902 0.640 0.360
#> GSM228663 1 0.0000 0.6119 1.000 0.000
#> GSM228664 1 0.9963 -0.6505 0.536 0.464
#> GSM228665 1 0.0000 0.6119 1.000 0.000
#> GSM228582 1 0.7056 0.6745 0.808 0.192
#> GSM228583 1 0.9608 0.6864 0.616 0.384
#> GSM228585 1 0.9608 0.6864 0.616 0.384
#> GSM228587 1 0.9661 0.6834 0.608 0.392
#> GSM228588 2 0.0376 0.4538 0.004 0.996
#> GSM228589 2 0.9286 0.8606 0.344 0.656
#> GSM228590 1 0.9608 0.6864 0.616 0.384
#> GSM228591 2 0.9608 0.8580 0.384 0.616
#> GSM228597 2 0.8327 0.8318 0.264 0.736
#> GSM228601 2 0.8813 0.8547 0.300 0.700
#> GSM228604 2 0.9608 0.8580 0.384 0.616
#> GSM228608 1 0.9608 0.6864 0.616 0.384
#> GSM228609 2 0.6247 0.1136 0.156 0.844
#> GSM228613 1 0.9608 0.6864 0.616 0.384
#> GSM228616 1 0.9209 0.5657 0.664 0.336
#> GSM228628 2 0.9580 0.8589 0.380 0.620
#> GSM228634 1 0.9608 0.6864 0.616 0.384
#> GSM228642 2 0.9608 0.8580 0.384 0.616
#> GSM228645 1 0.7056 0.2755 0.808 0.192
#> GSM228646 1 0.4298 0.5017 0.912 0.088
#> GSM228652 1 0.9608 0.6864 0.616 0.384
#> GSM228655 1 0.9580 0.6875 0.620 0.380
#> GSM228656 1 0.9608 0.6864 0.616 0.384
#> GSM228659 1 0.9909 0.6586 0.556 0.444
#> GSM228662 1 0.9608 0.6864 0.616 0.384
#> GSM228584 1 0.9608 0.6864 0.616 0.384
#> GSM228586 1 0.9608 0.6864 0.616 0.384
#> GSM228592 1 0.9608 0.6864 0.616 0.384
#> GSM228593 2 0.2778 0.3734 0.048 0.952
#> GSM228594 1 0.9491 0.6889 0.632 0.368
#> GSM228598 1 0.9608 0.6864 0.616 0.384
#> GSM228607 1 0.6048 0.5827 0.852 0.148
#> GSM228612 1 0.2603 0.5633 0.956 0.044
#> GSM228619 1 0.8207 0.6805 0.744 0.256
#> GSM228622 1 0.9608 0.6864 0.616 0.384
#> GSM228625 1 0.9896 0.6594 0.560 0.440
#> GSM228631 1 0.9129 0.6903 0.672 0.328
#> GSM228633 2 0.9608 0.8580 0.384 0.616
#> GSM228637 2 0.8327 0.8317 0.264 0.736
#> GSM228639 2 0.9129 0.8541 0.328 0.672
#> GSM228649 2 0.7453 0.7739 0.212 0.788
#> GSM228660 1 0.9580 0.6874 0.620 0.380
#> GSM228661 1 0.9087 0.6898 0.676 0.324
#> GSM228595 2 0.9608 0.8580 0.384 0.616
#> GSM228599 2 0.8813 0.8547 0.300 0.700
#> GSM228602 1 0.0938 0.6181 0.988 0.012
#> GSM228614 2 0.8713 0.8519 0.292 0.708
#> GSM228626 2 0.9608 0.8580 0.384 0.616
#> GSM228640 1 0.0000 0.6119 1.000 0.000
#> GSM228643 1 0.2603 0.5650 0.956 0.044
#> GSM228650 1 0.9833 -0.5384 0.576 0.424
#> GSM228653 1 0.0000 0.6119 1.000 0.000
#> GSM228657 2 0.9522 0.8601 0.372 0.628
#> GSM228605 1 0.7674 0.6699 0.776 0.224
#> GSM228610 1 0.0672 0.6044 0.992 0.008
#> GSM228617 1 0.3733 0.6306 0.928 0.072
#> GSM228620 1 0.0000 0.6119 1.000 0.000
#> GSM228623 2 0.8661 0.8500 0.288 0.712
#> GSM228629 1 0.0000 0.6119 1.000 0.000
#> GSM228632 2 0.9795 0.8330 0.416 0.584
#> GSM228635 2 0.8443 0.8387 0.272 0.728
#> GSM228647 1 0.0938 0.6003 0.988 0.012
#> GSM228596 1 0.2603 0.5677 0.956 0.044
#> GSM228600 1 0.0000 0.6119 1.000 0.000
#> GSM228603 1 0.0000 0.6119 1.000 0.000
#> GSM228615 2 0.8661 0.8474 0.288 0.712
#> GSM228627 1 0.0376 0.6086 0.996 0.004
#> GSM228641 1 0.0376 0.6083 0.996 0.004
#> GSM228644 2 0.9608 0.8580 0.384 0.616
#> GSM228651 1 0.1633 0.5878 0.976 0.024
#> GSM228654 1 0.6438 0.3274 0.836 0.164
#> GSM228658 1 0.0000 0.6119 1.000 0.000
#> GSM228606 2 0.9635 0.8560 0.388 0.612
#> GSM228611 1 0.0376 0.6083 0.996 0.004
#> GSM228618 1 0.0376 0.6140 0.996 0.004
#> GSM228621 1 0.9993 -0.6792 0.516 0.484
#> GSM228624 1 0.9393 -0.3419 0.644 0.356
#> GSM228630 2 0.9635 0.8557 0.388 0.612
#> GSM228636 2 0.8661 0.8498 0.288 0.712
#> GSM228638 1 0.9286 -0.3426 0.656 0.344
#> GSM228648 2 0.9833 0.8243 0.424 0.576
#> GSM228670 2 0.8499 0.8424 0.276 0.724
#> GSM228671 2 0.9580 0.8592 0.380 0.620
#> GSM228672 1 1.0000 0.6122 0.504 0.496
#> GSM228674 2 0.8443 0.7348 0.272 0.728
#> GSM228675 2 0.8555 0.8450 0.280 0.720
#> GSM228676 1 0.5629 0.5494 0.868 0.132
#> GSM228667 1 0.9833 -0.5356 0.576 0.424
#> GSM228668 1 0.9608 0.6864 0.616 0.384
#> GSM228669 1 0.9933 0.6510 0.548 0.452
#> GSM228673 1 0.7883 0.0920 0.764 0.236
#> GSM228677 2 0.9608 0.8580 0.384 0.616
#> GSM228678 2 0.8813 0.8544 0.300 0.700
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.7097 0.5813 0.668 0.052 0.280
#> GSM228563 2 0.2339 0.8819 0.012 0.940 0.048
#> GSM228565 1 0.6066 0.6493 0.728 0.024 0.248
#> GSM228566 3 0.4209 0.8793 0.120 0.020 0.860
#> GSM228567 1 0.0237 0.8747 0.996 0.000 0.004
#> GSM228570 1 0.3234 0.8408 0.908 0.072 0.020
#> GSM228571 1 0.4002 0.7632 0.840 0.000 0.160
#> GSM228574 3 0.3590 0.8310 0.028 0.076 0.896
#> GSM228575 3 0.7360 0.6904 0.096 0.212 0.692
#> GSM228576 1 0.5406 0.6636 0.764 0.012 0.224
#> GSM228579 1 0.4164 0.7708 0.848 0.008 0.144
#> GSM228580 2 0.3482 0.8832 0.000 0.872 0.128
#> GSM228581 2 0.6189 0.5783 0.004 0.632 0.364
#> GSM228666 2 0.4002 0.8696 0.000 0.840 0.160
#> GSM228564 2 0.5798 0.7489 0.184 0.776 0.040
#> GSM228568 1 0.5618 0.6283 0.732 0.008 0.260
#> GSM228569 1 0.2356 0.8475 0.928 0.000 0.072
#> GSM228572 2 0.2878 0.8767 0.000 0.904 0.096
#> GSM228573 3 0.4121 0.8809 0.168 0.000 0.832
#> GSM228577 1 0.1163 0.8698 0.972 0.000 0.028
#> GSM228578 1 0.1860 0.8601 0.948 0.000 0.052
#> GSM228663 3 0.4062 0.8821 0.164 0.000 0.836
#> GSM228664 3 0.2806 0.8427 0.032 0.040 0.928
#> GSM228665 3 0.4121 0.8809 0.168 0.000 0.832
#> GSM228582 1 0.6473 0.5028 0.668 0.020 0.312
#> GSM228583 1 0.0237 0.8747 0.996 0.000 0.004
#> GSM228585 1 0.0237 0.8747 0.996 0.000 0.004
#> GSM228587 1 0.1525 0.8623 0.964 0.032 0.004
#> GSM228588 2 0.1491 0.8788 0.016 0.968 0.016
#> GSM228589 2 0.2625 0.8771 0.000 0.916 0.084
#> GSM228590 1 0.0475 0.8745 0.992 0.004 0.004
#> GSM228591 2 0.4002 0.8528 0.000 0.840 0.160
#> GSM228597 2 0.2492 0.8810 0.016 0.936 0.048
#> GSM228601 2 0.1753 0.8770 0.000 0.952 0.048
#> GSM228604 3 0.2945 0.8045 0.004 0.088 0.908
#> GSM228608 1 0.0237 0.8741 0.996 0.004 0.000
#> GSM228609 2 0.5899 0.6475 0.244 0.736 0.020
#> GSM228613 1 0.0237 0.8741 0.996 0.004 0.000
#> GSM228616 1 0.9698 -0.0218 0.436 0.228 0.336
#> GSM228628 2 0.4235 0.8455 0.000 0.824 0.176
#> GSM228634 1 0.0424 0.8744 0.992 0.000 0.008
#> GSM228642 2 0.4887 0.8043 0.000 0.772 0.228
#> GSM228645 3 0.4892 0.8739 0.112 0.048 0.840
#> GSM228646 3 0.4591 0.8782 0.120 0.032 0.848
#> GSM228652 1 0.0424 0.8731 0.992 0.008 0.000
#> GSM228655 1 0.1015 0.8741 0.980 0.008 0.012
#> GSM228656 1 0.0237 0.8747 0.996 0.000 0.004
#> GSM228659 1 0.5610 0.6925 0.776 0.196 0.028
#> GSM228662 1 0.0237 0.8741 0.996 0.004 0.000
#> GSM228584 1 0.0424 0.8744 0.992 0.000 0.008
#> GSM228586 1 0.0747 0.8728 0.984 0.000 0.016
#> GSM228592 1 0.0424 0.8744 0.992 0.000 0.008
#> GSM228593 2 0.5891 0.7382 0.200 0.764 0.036
#> GSM228594 1 0.1289 0.8686 0.968 0.000 0.032
#> GSM228598 1 0.0661 0.8735 0.988 0.008 0.004
#> GSM228607 3 0.7199 0.7859 0.204 0.092 0.704
#> GSM228612 3 0.3816 0.8842 0.148 0.000 0.852
#> GSM228619 1 0.7392 -0.2089 0.500 0.032 0.468
#> GSM228622 1 0.1031 0.8726 0.976 0.000 0.024
#> GSM228625 1 0.4465 0.7585 0.820 0.176 0.004
#> GSM228631 3 0.6126 0.5680 0.400 0.000 0.600
#> GSM228633 2 0.3879 0.8619 0.000 0.848 0.152
#> GSM228637 2 0.2229 0.8809 0.012 0.944 0.044
#> GSM228639 3 0.5269 0.6795 0.016 0.200 0.784
#> GSM228649 2 0.2383 0.8799 0.016 0.940 0.044
#> GSM228660 1 0.2176 0.8684 0.948 0.032 0.020
#> GSM228661 1 0.1529 0.8653 0.960 0.000 0.040
#> GSM228595 2 0.3038 0.8741 0.000 0.896 0.104
#> GSM228599 2 0.3851 0.8435 0.004 0.860 0.136
#> GSM228602 3 0.4291 0.8776 0.180 0.000 0.820
#> GSM228614 2 0.5292 0.7427 0.008 0.764 0.228
#> GSM228626 2 0.4235 0.8455 0.000 0.824 0.176
#> GSM228640 3 0.4589 0.8813 0.172 0.008 0.820
#> GSM228643 3 0.4692 0.8829 0.168 0.012 0.820
#> GSM228650 3 0.3780 0.8567 0.064 0.044 0.892
#> GSM228653 3 0.4749 0.8814 0.172 0.012 0.816
#> GSM228657 2 0.2959 0.8739 0.000 0.900 0.100
#> GSM228605 3 0.7980 0.4202 0.400 0.064 0.536
#> GSM228610 3 0.3941 0.8843 0.156 0.000 0.844
#> GSM228617 3 0.5363 0.7741 0.276 0.000 0.724
#> GSM228620 3 0.4121 0.8809 0.168 0.000 0.832
#> GSM228623 2 0.2599 0.8813 0.016 0.932 0.052
#> GSM228629 3 0.4121 0.8809 0.168 0.000 0.832
#> GSM228632 3 0.2959 0.7999 0.000 0.100 0.900
#> GSM228635 2 0.2550 0.8838 0.012 0.932 0.056
#> GSM228647 3 0.4121 0.8809 0.168 0.000 0.832
#> GSM228596 3 0.5554 0.8575 0.112 0.076 0.812
#> GSM228600 3 0.4749 0.8814 0.172 0.012 0.816
#> GSM228603 3 0.4409 0.8807 0.172 0.004 0.824
#> GSM228615 2 0.1647 0.8846 0.004 0.960 0.036
#> GSM228627 3 0.4634 0.8833 0.164 0.012 0.824
#> GSM228641 3 0.4531 0.8833 0.168 0.008 0.824
#> GSM228644 2 0.4002 0.8552 0.000 0.840 0.160
#> GSM228651 3 0.4749 0.8814 0.172 0.012 0.816
#> GSM228654 3 0.3989 0.8817 0.124 0.012 0.864
#> GSM228658 3 0.4409 0.8807 0.172 0.004 0.824
#> GSM228606 3 0.4465 0.7190 0.004 0.176 0.820
#> GSM228611 3 0.4121 0.8809 0.168 0.000 0.832
#> GSM228618 3 0.4346 0.8708 0.184 0.000 0.816
#> GSM228621 3 0.2636 0.8576 0.048 0.020 0.932
#> GSM228624 3 0.3649 0.8588 0.068 0.036 0.896
#> GSM228630 3 0.3551 0.7534 0.000 0.132 0.868
#> GSM228636 2 0.2486 0.8851 0.008 0.932 0.060
#> GSM228638 3 0.3670 0.8728 0.092 0.020 0.888
#> GSM228648 3 0.2063 0.8270 0.008 0.044 0.948
#> GSM228670 2 0.2903 0.8797 0.028 0.924 0.048
#> GSM228671 2 0.5560 0.6890 0.000 0.700 0.300
#> GSM228672 1 0.6341 0.6008 0.716 0.252 0.032
#> GSM228674 2 0.3993 0.8629 0.064 0.884 0.052
#> GSM228675 2 0.2280 0.8843 0.008 0.940 0.052
#> GSM228676 3 0.6728 0.7954 0.128 0.124 0.748
#> GSM228667 2 0.6698 0.6549 0.036 0.684 0.280
#> GSM228668 1 0.0661 0.8742 0.988 0.004 0.008
#> GSM228669 1 0.5047 0.7397 0.824 0.140 0.036
#> GSM228673 3 0.3434 0.8427 0.032 0.064 0.904
#> GSM228677 2 0.4235 0.8588 0.000 0.824 0.176
#> GSM228678 2 0.2400 0.8867 0.004 0.932 0.064
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.8969 0.3317 0.172 0.204 0.132 0.492
#> GSM228563 4 0.2899 0.6664 0.004 0.112 0.004 0.880
#> GSM228565 1 0.9645 -0.0935 0.336 0.212 0.144 0.308
#> GSM228566 3 0.4873 0.7980 0.024 0.172 0.780 0.024
#> GSM228567 1 0.0376 0.8684 0.992 0.004 0.004 0.000
#> GSM228570 1 0.5611 0.7189 0.748 0.108 0.012 0.132
#> GSM228571 1 0.5159 0.7531 0.780 0.120 0.088 0.012
#> GSM228574 3 0.4513 0.8007 0.004 0.160 0.796 0.040
#> GSM228575 3 0.7098 0.5683 0.008 0.272 0.580 0.140
#> GSM228576 1 0.7966 0.5099 0.568 0.172 0.208 0.052
#> GSM228579 1 0.4477 0.7690 0.808 0.108 0.084 0.000
#> GSM228580 2 0.5696 0.2181 0.000 0.492 0.024 0.484
#> GSM228581 2 0.7344 -0.0479 0.000 0.460 0.160 0.380
#> GSM228666 4 0.6153 0.2924 0.000 0.328 0.068 0.604
#> GSM228564 4 0.3793 0.6506 0.044 0.112 0.000 0.844
#> GSM228568 1 0.7219 0.5671 0.604 0.144 0.232 0.020
#> GSM228569 1 0.2596 0.8427 0.908 0.024 0.068 0.000
#> GSM228572 2 0.5378 0.5967 0.000 0.540 0.012 0.448
#> GSM228573 3 0.1724 0.8453 0.020 0.032 0.948 0.000
#> GSM228577 1 0.1820 0.8580 0.944 0.020 0.036 0.000
#> GSM228578 1 0.3316 0.8435 0.888 0.028 0.064 0.020
#> GSM228663 3 0.2413 0.8398 0.020 0.064 0.916 0.000
#> GSM228664 3 0.2266 0.8318 0.004 0.084 0.912 0.000
#> GSM228665 3 0.2002 0.8415 0.020 0.044 0.936 0.000
#> GSM228582 1 0.6864 0.5724 0.616 0.188 0.192 0.004
#> GSM228583 1 0.0376 0.8684 0.992 0.004 0.004 0.000
#> GSM228585 1 0.0376 0.8684 0.992 0.004 0.004 0.000
#> GSM228587 1 0.0712 0.8653 0.984 0.008 0.004 0.004
#> GSM228588 2 0.5447 0.5147 0.008 0.528 0.004 0.460
#> GSM228589 2 0.4769 0.7502 0.000 0.684 0.008 0.308
#> GSM228590 1 0.0188 0.8679 0.996 0.000 0.004 0.000
#> GSM228591 2 0.3862 0.7239 0.000 0.824 0.024 0.152
#> GSM228597 4 0.1792 0.6733 0.000 0.068 0.000 0.932
#> GSM228601 2 0.4585 0.7248 0.000 0.668 0.000 0.332
#> GSM228604 3 0.4560 0.6622 0.000 0.296 0.700 0.004
#> GSM228608 1 0.1443 0.8610 0.960 0.008 0.004 0.028
#> GSM228609 4 0.7126 0.2289 0.272 0.176 0.000 0.552
#> GSM228613 1 0.0376 0.8684 0.992 0.004 0.004 0.000
#> GSM228616 3 0.9483 0.0656 0.332 0.176 0.352 0.140
#> GSM228628 2 0.4436 0.6841 0.000 0.764 0.020 0.216
#> GSM228634 1 0.0188 0.8679 0.996 0.000 0.004 0.000
#> GSM228642 2 0.3934 0.6954 0.000 0.836 0.048 0.116
#> GSM228645 3 0.6487 0.6539 0.012 0.296 0.620 0.072
#> GSM228646 3 0.6145 0.7139 0.016 0.212 0.692 0.080
#> GSM228652 1 0.1994 0.8498 0.936 0.008 0.004 0.052
#> GSM228655 1 0.2353 0.8468 0.924 0.008 0.012 0.056
#> GSM228656 1 0.0188 0.8679 0.996 0.000 0.004 0.000
#> GSM228659 1 0.5686 0.3442 0.592 0.032 0.000 0.376
#> GSM228662 1 0.0376 0.8684 0.992 0.004 0.004 0.000
#> GSM228584 1 0.0376 0.8684 0.992 0.004 0.004 0.000
#> GSM228586 1 0.0336 0.8679 0.992 0.000 0.008 0.000
#> GSM228592 1 0.0376 0.8684 0.992 0.004 0.004 0.000
#> GSM228593 4 0.6482 0.3661 0.288 0.092 0.004 0.616
#> GSM228594 1 0.1174 0.8641 0.968 0.012 0.020 0.000
#> GSM228598 1 0.0657 0.8669 0.984 0.012 0.000 0.004
#> GSM228607 3 0.6260 0.6227 0.024 0.056 0.668 0.252
#> GSM228612 3 0.2376 0.8420 0.016 0.068 0.916 0.000
#> GSM228619 3 0.7587 0.4504 0.260 0.016 0.548 0.176
#> GSM228622 1 0.2075 0.8608 0.936 0.004 0.044 0.016
#> GSM228625 1 0.5716 0.2625 0.552 0.028 0.000 0.420
#> GSM228631 3 0.5240 0.7314 0.180 0.036 0.760 0.024
#> GSM228633 2 0.5279 0.7574 0.000 0.704 0.044 0.252
#> GSM228637 4 0.1743 0.6680 0.004 0.056 0.000 0.940
#> GSM228639 3 0.4770 0.5908 0.000 0.012 0.700 0.288
#> GSM228649 4 0.1824 0.6770 0.000 0.060 0.004 0.936
#> GSM228660 1 0.4323 0.8164 0.844 0.036 0.052 0.068
#> GSM228661 1 0.1888 0.8572 0.940 0.016 0.044 0.000
#> GSM228595 2 0.4820 0.7545 0.000 0.692 0.012 0.296
#> GSM228599 4 0.4764 0.5787 0.000 0.088 0.124 0.788
#> GSM228602 3 0.2670 0.8414 0.024 0.072 0.904 0.000
#> GSM228614 4 0.3239 0.6618 0.000 0.052 0.068 0.880
#> GSM228626 2 0.4671 0.7599 0.000 0.752 0.028 0.220
#> GSM228640 3 0.2706 0.8378 0.020 0.080 0.900 0.000
#> GSM228643 3 0.2973 0.8348 0.020 0.096 0.884 0.000
#> GSM228650 3 0.3216 0.8281 0.004 0.124 0.864 0.008
#> GSM228653 3 0.2256 0.8430 0.020 0.056 0.924 0.000
#> GSM228657 2 0.4820 0.7544 0.000 0.692 0.012 0.296
#> GSM228605 3 0.8029 0.2018 0.104 0.052 0.464 0.380
#> GSM228610 3 0.1697 0.8440 0.016 0.028 0.952 0.004
#> GSM228617 3 0.2256 0.8365 0.056 0.020 0.924 0.000
#> GSM228620 3 0.1724 0.8433 0.020 0.032 0.948 0.000
#> GSM228623 4 0.1975 0.6788 0.000 0.048 0.016 0.936
#> GSM228629 3 0.1297 0.8430 0.020 0.016 0.964 0.000
#> GSM228632 3 0.4155 0.7907 0.000 0.072 0.828 0.100
#> GSM228635 4 0.1824 0.6596 0.000 0.060 0.004 0.936
#> GSM228647 3 0.1297 0.8426 0.020 0.016 0.964 0.000
#> GSM228596 3 0.6943 0.3537 0.004 0.108 0.540 0.348
#> GSM228600 3 0.2909 0.8361 0.020 0.092 0.888 0.000
#> GSM228603 3 0.2635 0.8397 0.020 0.076 0.904 0.000
#> GSM228615 4 0.2053 0.6623 0.000 0.072 0.004 0.924
#> GSM228627 3 0.3037 0.8407 0.020 0.100 0.880 0.000
#> GSM228641 3 0.2635 0.8386 0.020 0.076 0.904 0.000
#> GSM228644 2 0.4576 0.7599 0.000 0.748 0.020 0.232
#> GSM228651 3 0.2563 0.8451 0.020 0.072 0.908 0.000
#> GSM228654 3 0.2450 0.8416 0.016 0.072 0.912 0.000
#> GSM228658 3 0.2413 0.8450 0.020 0.064 0.916 0.000
#> GSM228606 3 0.6162 0.5201 0.000 0.076 0.620 0.304
#> GSM228611 3 0.2099 0.8421 0.020 0.040 0.936 0.004
#> GSM228618 3 0.1724 0.8435 0.020 0.032 0.948 0.000
#> GSM228621 3 0.1452 0.8391 0.000 0.036 0.956 0.008
#> GSM228624 3 0.3376 0.8204 0.008 0.108 0.868 0.016
#> GSM228630 3 0.3056 0.8118 0.000 0.040 0.888 0.072
#> GSM228636 4 0.2593 0.6130 0.000 0.104 0.004 0.892
#> GSM228638 3 0.0927 0.8427 0.008 0.016 0.976 0.000
#> GSM228648 3 0.0779 0.8417 0.000 0.016 0.980 0.004
#> GSM228670 4 0.1847 0.6786 0.004 0.052 0.004 0.940
#> GSM228671 4 0.5664 0.4979 0.000 0.156 0.124 0.720
#> GSM228672 4 0.5791 0.4659 0.284 0.060 0.000 0.656
#> GSM228674 4 0.2408 0.6806 0.016 0.060 0.004 0.920
#> GSM228675 4 0.2053 0.6669 0.000 0.072 0.004 0.924
#> GSM228676 4 0.7675 0.1538 0.024 0.120 0.376 0.480
#> GSM228667 4 0.6073 0.5242 0.012 0.184 0.100 0.704
#> GSM228668 1 0.1543 0.8601 0.956 0.008 0.004 0.032
#> GSM228669 4 0.4800 0.4421 0.340 0.004 0.000 0.656
#> GSM228673 3 0.5670 0.6262 0.008 0.056 0.704 0.232
#> GSM228677 4 0.4841 0.5788 0.000 0.140 0.080 0.780
#> GSM228678 4 0.2888 0.6269 0.000 0.124 0.004 0.872
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 5 0.7567 0.1134 0.076 0.016 0.100 0.352 0.456
#> GSM228563 4 0.4907 0.5003 0.000 0.052 0.000 0.656 0.292
#> GSM228565 5 0.8545 0.2846 0.220 0.020 0.124 0.232 0.404
#> GSM228566 3 0.5441 0.4503 0.000 0.032 0.572 0.020 0.376
#> GSM228567 1 0.0000 0.8593 1.000 0.000 0.000 0.000 0.000
#> GSM228570 1 0.6607 0.4259 0.576 0.012 0.016 0.144 0.252
#> GSM228571 1 0.6111 0.5281 0.644 0.012 0.084 0.028 0.232
#> GSM228574 3 0.5140 0.6081 0.000 0.012 0.644 0.040 0.304
#> GSM228575 5 0.6961 0.3865 0.000 0.204 0.220 0.040 0.536
#> GSM228576 1 0.8245 -0.1128 0.408 0.016 0.172 0.100 0.304
#> GSM228579 1 0.4916 0.6719 0.756 0.016 0.076 0.008 0.144
#> GSM228580 2 0.6742 0.2502 0.000 0.456 0.008 0.200 0.336
#> GSM228581 5 0.7691 0.3539 0.000 0.168 0.160 0.168 0.504
#> GSM228666 5 0.7364 0.2276 0.000 0.240 0.048 0.240 0.472
#> GSM228564 4 0.5217 0.4687 0.020 0.032 0.000 0.636 0.312
#> GSM228568 5 0.7466 0.0784 0.380 0.016 0.184 0.024 0.396
#> GSM228569 1 0.3390 0.7763 0.840 0.000 0.100 0.000 0.060
#> GSM228572 2 0.4975 0.6412 0.000 0.700 0.004 0.220 0.076
#> GSM228573 3 0.2517 0.7347 0.008 0.000 0.884 0.004 0.104
#> GSM228577 1 0.2992 0.8069 0.868 0.000 0.064 0.000 0.068
#> GSM228578 1 0.4407 0.7634 0.796 0.000 0.084 0.028 0.092
#> GSM228663 3 0.3769 0.6895 0.008 0.012 0.816 0.016 0.148
#> GSM228664 3 0.4518 0.6277 0.004 0.020 0.736 0.016 0.224
#> GSM228665 3 0.3043 0.7160 0.008 0.000 0.864 0.024 0.104
#> GSM228582 1 0.7228 0.3070 0.556 0.044 0.184 0.016 0.200
#> GSM228583 1 0.0000 0.8593 1.000 0.000 0.000 0.000 0.000
#> GSM228585 1 0.0000 0.8593 1.000 0.000 0.000 0.000 0.000
#> GSM228587 1 0.0613 0.8551 0.984 0.004 0.000 0.004 0.008
#> GSM228588 2 0.5770 0.3564 0.004 0.528 0.000 0.388 0.080
#> GSM228589 2 0.3780 0.7637 0.000 0.812 0.000 0.116 0.072
#> GSM228590 1 0.0162 0.8584 0.996 0.000 0.000 0.000 0.004
#> GSM228591 2 0.3527 0.7439 0.000 0.828 0.000 0.056 0.116
#> GSM228597 4 0.3778 0.6344 0.004 0.108 0.000 0.820 0.068
#> GSM228601 2 0.3606 0.7437 0.000 0.808 0.004 0.164 0.024
#> GSM228604 3 0.6122 0.3822 0.000 0.284 0.564 0.004 0.148
#> GSM228608 1 0.1310 0.8465 0.956 0.000 0.000 0.024 0.020
#> GSM228609 4 0.6153 0.4847 0.164 0.088 0.000 0.664 0.084
#> GSM228613 1 0.0000 0.8593 1.000 0.000 0.000 0.000 0.000
#> GSM228616 5 0.9896 0.3059 0.208 0.140 0.208 0.192 0.252
#> GSM228628 2 0.5492 0.5704 0.000 0.680 0.012 0.120 0.188
#> GSM228634 1 0.0162 0.8584 0.996 0.000 0.000 0.000 0.004
#> GSM228642 2 0.2291 0.7534 0.000 0.908 0.008 0.012 0.072
#> GSM228645 5 0.6902 0.1663 0.000 0.128 0.304 0.048 0.520
#> GSM228646 3 0.6761 0.1508 0.004 0.040 0.456 0.088 0.412
#> GSM228652 1 0.3037 0.7944 0.864 0.000 0.004 0.100 0.032
#> GSM228655 1 0.4702 0.7226 0.776 0.004 0.024 0.124 0.072
#> GSM228656 1 0.0000 0.8593 1.000 0.000 0.000 0.000 0.000
#> GSM228659 4 0.5638 0.2219 0.396 0.004 0.000 0.532 0.068
#> GSM228662 1 0.0000 0.8593 1.000 0.000 0.000 0.000 0.000
#> GSM228584 1 0.0000 0.8593 1.000 0.000 0.000 0.000 0.000
#> GSM228586 1 0.0000 0.8593 1.000 0.000 0.000 0.000 0.000
#> GSM228592 1 0.0000 0.8593 1.000 0.000 0.000 0.000 0.000
#> GSM228593 4 0.6700 0.4578 0.144 0.064 0.000 0.600 0.192
#> GSM228594 1 0.1117 0.8518 0.964 0.000 0.016 0.000 0.020
#> GSM228598 1 0.0955 0.8537 0.968 0.000 0.004 0.000 0.028
#> GSM228607 3 0.6482 0.2081 0.000 0.000 0.492 0.276 0.232
#> GSM228612 3 0.3952 0.6744 0.008 0.008 0.764 0.004 0.216
#> GSM228619 3 0.7642 0.1153 0.176 0.000 0.472 0.260 0.092
#> GSM228622 1 0.2737 0.8303 0.896 0.000 0.052 0.020 0.032
#> GSM228625 4 0.5833 0.2435 0.348 0.004 0.008 0.568 0.072
#> GSM228631 3 0.5581 0.5972 0.104 0.000 0.712 0.052 0.132
#> GSM228633 2 0.1605 0.7933 0.000 0.944 0.012 0.040 0.004
#> GSM228637 4 0.2843 0.6410 0.000 0.076 0.000 0.876 0.048
#> GSM228639 3 0.5563 0.3948 0.000 0.028 0.632 0.292 0.048
#> GSM228649 4 0.3741 0.6295 0.000 0.076 0.000 0.816 0.108
#> GSM228660 1 0.5895 0.6580 0.704 0.008 0.076 0.136 0.076
#> GSM228661 1 0.1701 0.8407 0.936 0.000 0.048 0.000 0.016
#> GSM228595 2 0.1502 0.7951 0.000 0.940 0.004 0.056 0.000
#> GSM228599 4 0.5697 0.4854 0.000 0.056 0.092 0.700 0.152
#> GSM228602 3 0.3280 0.7061 0.012 0.004 0.824 0.000 0.160
#> GSM228614 4 0.3752 0.6000 0.000 0.056 0.076 0.840 0.028
#> GSM228626 2 0.1267 0.7879 0.000 0.960 0.004 0.024 0.012
#> GSM228640 3 0.3210 0.7051 0.008 0.008 0.832 0.000 0.152
#> GSM228643 3 0.3409 0.7101 0.008 0.008 0.824 0.004 0.156
#> GSM228650 3 0.4314 0.6955 0.000 0.068 0.780 0.008 0.144
#> GSM228653 3 0.2291 0.7306 0.008 0.012 0.908 0.000 0.072
#> GSM228657 2 0.1857 0.7965 0.000 0.928 0.004 0.060 0.008
#> GSM228605 3 0.7750 -0.2351 0.060 0.000 0.376 0.252 0.312
#> GSM228610 3 0.2484 0.7261 0.004 0.000 0.900 0.028 0.068
#> GSM228617 3 0.3090 0.7147 0.032 0.000 0.860 0.004 0.104
#> GSM228620 3 0.2420 0.7280 0.008 0.000 0.896 0.008 0.088
#> GSM228623 4 0.4111 0.6277 0.000 0.068 0.016 0.808 0.108
#> GSM228629 3 0.2077 0.7368 0.008 0.000 0.908 0.000 0.084
#> GSM228632 3 0.5142 0.6387 0.000 0.060 0.748 0.068 0.124
#> GSM228635 4 0.3825 0.6264 0.000 0.136 0.000 0.804 0.060
#> GSM228647 3 0.0854 0.7342 0.008 0.000 0.976 0.004 0.012
#> GSM228596 3 0.6881 0.0270 0.000 0.012 0.460 0.224 0.304
#> GSM228600 3 0.3412 0.6971 0.008 0.008 0.812 0.000 0.172
#> GSM228603 3 0.3252 0.7036 0.008 0.008 0.828 0.000 0.156
#> GSM228615 4 0.3216 0.6404 0.000 0.096 0.004 0.856 0.044
#> GSM228627 3 0.3629 0.7171 0.004 0.012 0.816 0.012 0.156
#> GSM228641 3 0.3293 0.7053 0.008 0.008 0.824 0.000 0.160
#> GSM228644 2 0.1285 0.7933 0.000 0.956 0.004 0.036 0.004
#> GSM228651 3 0.2818 0.7307 0.008 0.004 0.860 0.000 0.128
#> GSM228654 3 0.2823 0.7298 0.004 0.020 0.880 0.004 0.092
#> GSM228658 3 0.2077 0.7340 0.008 0.000 0.908 0.000 0.084
#> GSM228606 3 0.7084 0.1950 0.000 0.036 0.504 0.244 0.216
#> GSM228611 3 0.2753 0.7136 0.008 0.000 0.876 0.012 0.104
#> GSM228618 3 0.2513 0.7278 0.008 0.000 0.876 0.000 0.116
#> GSM228621 3 0.2199 0.7317 0.000 0.016 0.916 0.008 0.060
#> GSM228624 3 0.5733 0.5137 0.004 0.064 0.632 0.020 0.280
#> GSM228630 3 0.3905 0.6860 0.000 0.080 0.832 0.052 0.036
#> GSM228636 4 0.4119 0.5956 0.000 0.212 0.000 0.752 0.036
#> GSM228638 3 0.1940 0.7316 0.004 0.008 0.936 0.028 0.024
#> GSM228648 3 0.1405 0.7330 0.000 0.008 0.956 0.016 0.020
#> GSM228670 4 0.4155 0.6229 0.008 0.068 0.004 0.804 0.116
#> GSM228671 5 0.7868 0.0643 0.000 0.168 0.104 0.324 0.404
#> GSM228672 4 0.4973 0.5382 0.132 0.012 0.000 0.736 0.120
#> GSM228674 4 0.5058 0.5577 0.020 0.048 0.004 0.716 0.212
#> GSM228675 4 0.5234 0.5541 0.000 0.096 0.004 0.680 0.220
#> GSM228676 4 0.7322 -0.1441 0.012 0.016 0.252 0.452 0.268
#> GSM228667 4 0.6684 0.2279 0.008 0.036 0.092 0.540 0.324
#> GSM228668 1 0.2283 0.8336 0.916 0.000 0.008 0.036 0.040
#> GSM228669 4 0.4743 0.4997 0.208 0.004 0.004 0.728 0.056
#> GSM228673 3 0.5841 0.3527 0.000 0.000 0.596 0.148 0.256
#> GSM228677 4 0.7104 0.3725 0.000 0.168 0.080 0.560 0.192
#> GSM228678 4 0.5213 0.5965 0.000 0.204 0.004 0.688 0.104
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 5 0.5772 0.18483 0.052 0.004 0.024 0.168 0.672 0.080
#> GSM228563 4 0.6185 0.20301 0.000 0.008 0.000 0.400 0.368 0.224
#> GSM228565 5 0.5737 0.26845 0.096 0.012 0.040 0.124 0.696 0.032
#> GSM228566 5 0.5143 0.00562 0.000 0.020 0.376 0.004 0.560 0.040
#> GSM228567 1 0.0146 0.85772 0.996 0.000 0.004 0.000 0.000 0.000
#> GSM228570 5 0.5610 -0.05132 0.440 0.000 0.012 0.044 0.476 0.028
#> GSM228571 1 0.5325 0.31135 0.548 0.000 0.040 0.008 0.380 0.024
#> GSM228574 3 0.5078 0.45049 0.000 0.000 0.528 0.012 0.408 0.052
#> GSM228575 5 0.7644 -0.29946 0.000 0.152 0.124 0.028 0.408 0.288
#> GSM228576 5 0.5899 0.29013 0.272 0.008 0.080 0.024 0.600 0.016
#> GSM228579 1 0.4437 0.63664 0.724 0.004 0.032 0.000 0.212 0.028
#> GSM228580 6 0.5958 0.17201 0.000 0.292 0.012 0.056 0.064 0.576
#> GSM228581 6 0.8136 0.31157 0.004 0.104 0.132 0.088 0.268 0.404
#> GSM228666 6 0.8117 0.41696 0.000 0.176 0.040 0.160 0.304 0.320
#> GSM228564 5 0.6164 -0.23484 0.012 0.004 0.000 0.380 0.436 0.168
#> GSM228568 5 0.7901 0.04776 0.248 0.012 0.132 0.012 0.384 0.212
#> GSM228569 1 0.3858 0.75760 0.808 0.000 0.092 0.000 0.048 0.052
#> GSM228572 2 0.5280 0.50263 0.000 0.628 0.000 0.212 0.008 0.152
#> GSM228573 3 0.3054 0.69744 0.000 0.000 0.828 0.000 0.136 0.036
#> GSM228577 1 0.3504 0.80228 0.848 0.000 0.036 0.020 0.056 0.040
#> GSM228578 1 0.5117 0.72186 0.736 0.000 0.064 0.036 0.116 0.048
#> GSM228663 3 0.4717 0.60270 0.004 0.016 0.724 0.000 0.148 0.108
#> GSM228664 3 0.5520 0.52673 0.004 0.024 0.656 0.004 0.168 0.144
#> GSM228665 3 0.3107 0.68014 0.000 0.004 0.844 0.000 0.072 0.080
#> GSM228582 1 0.7842 0.09939 0.448 0.064 0.160 0.000 0.216 0.112
#> GSM228583 1 0.0146 0.85772 0.996 0.000 0.004 0.000 0.000 0.000
#> GSM228585 1 0.0146 0.85772 0.996 0.000 0.004 0.000 0.000 0.000
#> GSM228587 1 0.1346 0.84341 0.952 0.000 0.000 0.008 0.016 0.024
#> GSM228588 2 0.6983 0.16468 0.004 0.404 0.000 0.348 0.076 0.168
#> GSM228589 2 0.5561 0.59148 0.004 0.664 0.000 0.156 0.052 0.124
#> GSM228590 1 0.0291 0.85735 0.992 0.000 0.004 0.000 0.000 0.004
#> GSM228591 2 0.4106 0.66814 0.004 0.792 0.000 0.036 0.060 0.108
#> GSM228597 4 0.4215 0.57045 0.000 0.036 0.000 0.768 0.052 0.144
#> GSM228601 2 0.3184 0.71114 0.000 0.836 0.000 0.120 0.016 0.028
#> GSM228604 3 0.6436 0.32205 0.000 0.300 0.496 0.000 0.148 0.056
#> GSM228608 1 0.1788 0.83201 0.928 0.000 0.004 0.012 0.052 0.004
#> GSM228609 4 0.6643 0.50994 0.092 0.052 0.000 0.608 0.144 0.104
#> GSM228613 1 0.0291 0.85735 0.992 0.000 0.004 0.000 0.000 0.004
#> GSM228616 5 0.9410 -0.02849 0.100 0.100 0.128 0.156 0.340 0.176
#> GSM228628 2 0.5147 0.36348 0.000 0.628 0.008 0.052 0.292 0.020
#> GSM228634 1 0.0146 0.85772 0.996 0.000 0.004 0.000 0.000 0.000
#> GSM228642 2 0.1562 0.73305 0.000 0.940 0.000 0.004 0.032 0.024
#> GSM228645 5 0.5851 0.07134 0.000 0.128 0.116 0.012 0.660 0.084
#> GSM228646 5 0.5208 0.21354 0.000 0.032 0.244 0.020 0.664 0.040
#> GSM228652 1 0.4088 0.73317 0.788 0.000 0.004 0.092 0.096 0.020
#> GSM228655 1 0.5682 0.62057 0.680 0.000 0.024 0.132 0.112 0.052
#> GSM228656 1 0.0146 0.85772 0.996 0.000 0.004 0.000 0.000 0.000
#> GSM228659 4 0.6551 0.28444 0.328 0.004 0.000 0.476 0.136 0.056
#> GSM228662 1 0.0291 0.85735 0.992 0.000 0.004 0.000 0.000 0.004
#> GSM228584 1 0.0291 0.85737 0.992 0.000 0.004 0.000 0.000 0.004
#> GSM228586 1 0.0622 0.85618 0.980 0.000 0.008 0.000 0.000 0.012
#> GSM228592 1 0.0291 0.85737 0.992 0.000 0.004 0.000 0.000 0.004
#> GSM228593 4 0.6537 0.35761 0.060 0.000 0.000 0.508 0.188 0.244
#> GSM228594 1 0.1700 0.84366 0.936 0.000 0.024 0.000 0.012 0.028
#> GSM228598 1 0.1692 0.84569 0.932 0.000 0.000 0.012 0.008 0.048
#> GSM228607 3 0.7426 0.01745 0.000 0.004 0.388 0.224 0.264 0.120
#> GSM228612 3 0.5019 0.55137 0.000 0.000 0.656 0.004 0.188 0.152
#> GSM228619 3 0.7846 0.04342 0.108 0.000 0.408 0.296 0.116 0.072
#> GSM228622 1 0.3412 0.81038 0.852 0.000 0.048 0.020 0.056 0.024
#> GSM228625 4 0.5985 0.41843 0.208 0.000 0.004 0.612 0.108 0.068
#> GSM228631 3 0.6527 0.48824 0.076 0.000 0.616 0.068 0.164 0.076
#> GSM228633 2 0.1629 0.74790 0.000 0.940 0.004 0.024 0.004 0.028
#> GSM228637 4 0.2373 0.58110 0.000 0.008 0.000 0.880 0.008 0.104
#> GSM228639 3 0.4814 0.49373 0.000 0.004 0.664 0.268 0.020 0.044
#> GSM228649 4 0.3708 0.56599 0.000 0.008 0.004 0.800 0.052 0.136
#> GSM228660 1 0.6486 0.59825 0.636 0.004 0.056 0.128 0.088 0.088
#> GSM228661 1 0.2420 0.82397 0.892 0.000 0.068 0.000 0.008 0.032
#> GSM228595 2 0.0922 0.75672 0.000 0.968 0.000 0.024 0.004 0.004
#> GSM228599 4 0.6349 0.44038 0.000 0.024 0.048 0.572 0.252 0.104
#> GSM228602 3 0.3920 0.63986 0.000 0.000 0.736 0.000 0.216 0.048
#> GSM228614 4 0.3994 0.57511 0.000 0.024 0.044 0.812 0.092 0.028
#> GSM228626 2 0.0767 0.75157 0.000 0.976 0.004 0.012 0.000 0.008
#> GSM228640 3 0.3722 0.65329 0.000 0.004 0.764 0.000 0.196 0.036
#> GSM228643 3 0.3622 0.65186 0.000 0.004 0.760 0.000 0.212 0.024
#> GSM228650 3 0.4729 0.65361 0.000 0.068 0.740 0.000 0.124 0.068
#> GSM228653 3 0.2618 0.70585 0.000 0.000 0.860 0.000 0.116 0.024
#> GSM228657 2 0.2066 0.75349 0.000 0.920 0.004 0.040 0.012 0.024
#> GSM228605 5 0.8024 -0.02648 0.044 0.000 0.280 0.160 0.368 0.148
#> GSM228610 3 0.3020 0.68137 0.000 0.000 0.844 0.000 0.076 0.080
#> GSM228617 3 0.3807 0.67223 0.008 0.000 0.808 0.012 0.104 0.068
#> GSM228620 3 0.2294 0.70209 0.000 0.000 0.892 0.000 0.072 0.036
#> GSM228623 4 0.3669 0.56948 0.000 0.008 0.004 0.812 0.084 0.092
#> GSM228629 3 0.2474 0.71155 0.000 0.000 0.880 0.000 0.080 0.040
#> GSM228632 3 0.4993 0.63119 0.000 0.028 0.740 0.036 0.116 0.080
#> GSM228635 4 0.3374 0.56419 0.000 0.036 0.000 0.836 0.032 0.096
#> GSM228647 3 0.0993 0.70891 0.000 0.000 0.964 0.000 0.012 0.024
#> GSM228596 3 0.7231 0.07332 0.000 0.008 0.432 0.096 0.276 0.188
#> GSM228600 3 0.3997 0.63737 0.000 0.004 0.736 0.000 0.216 0.044
#> GSM228603 3 0.3722 0.65329 0.000 0.004 0.764 0.000 0.196 0.036
#> GSM228615 4 0.3474 0.59156 0.000 0.044 0.000 0.836 0.072 0.048
#> GSM228627 3 0.4618 0.65478 0.000 0.012 0.700 0.000 0.212 0.076
#> GSM228641 3 0.3752 0.65409 0.000 0.004 0.760 0.000 0.200 0.036
#> GSM228644 2 0.0951 0.75520 0.000 0.968 0.004 0.020 0.008 0.000
#> GSM228651 3 0.3050 0.70612 0.000 0.004 0.832 0.000 0.136 0.028
#> GSM228654 3 0.2864 0.70672 0.000 0.012 0.860 0.000 0.100 0.028
#> GSM228658 3 0.2793 0.70973 0.000 0.004 0.856 0.000 0.112 0.028
#> GSM228606 3 0.7685 -0.08623 0.000 0.028 0.388 0.244 0.248 0.092
#> GSM228611 3 0.3655 0.65239 0.000 0.000 0.800 0.004 0.108 0.088
#> GSM228618 3 0.2826 0.70432 0.000 0.000 0.856 0.000 0.092 0.052
#> GSM228621 3 0.2308 0.70936 0.000 0.008 0.904 0.004 0.028 0.056
#> GSM228624 3 0.6938 0.24871 0.004 0.056 0.484 0.016 0.292 0.148
#> GSM228630 3 0.3430 0.68502 0.000 0.024 0.852 0.036 0.028 0.060
#> GSM228636 4 0.3646 0.53583 0.000 0.116 0.000 0.804 0.008 0.072
#> GSM228638 3 0.1483 0.70805 0.000 0.000 0.944 0.008 0.012 0.036
#> GSM228648 3 0.1338 0.70731 0.000 0.004 0.952 0.004 0.008 0.032
#> GSM228670 4 0.5023 0.53637 0.004 0.016 0.004 0.704 0.132 0.140
#> GSM228671 6 0.8308 0.27955 0.000 0.080 0.100 0.224 0.288 0.308
#> GSM228672 4 0.6146 0.49074 0.104 0.000 0.000 0.572 0.244 0.080
#> GSM228674 4 0.6025 0.41951 0.008 0.008 0.004 0.560 0.212 0.208
#> GSM228675 4 0.5714 0.42488 0.000 0.016 0.004 0.592 0.148 0.240
#> GSM228676 4 0.7386 -0.09344 0.008 0.000 0.168 0.360 0.348 0.116
#> GSM228667 5 0.6044 -0.10974 0.004 0.016 0.028 0.376 0.508 0.068
#> GSM228668 1 0.3714 0.78387 0.828 0.000 0.008 0.056 0.072 0.036
#> GSM228669 4 0.5081 0.51648 0.156 0.000 0.000 0.688 0.128 0.028
#> GSM228673 3 0.6469 0.22341 0.000 0.000 0.520 0.088 0.276 0.116
#> GSM228677 4 0.7447 0.16398 0.000 0.076 0.076 0.508 0.196 0.144
#> GSM228678 4 0.5402 0.51848 0.000 0.104 0.008 0.696 0.068 0.124
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
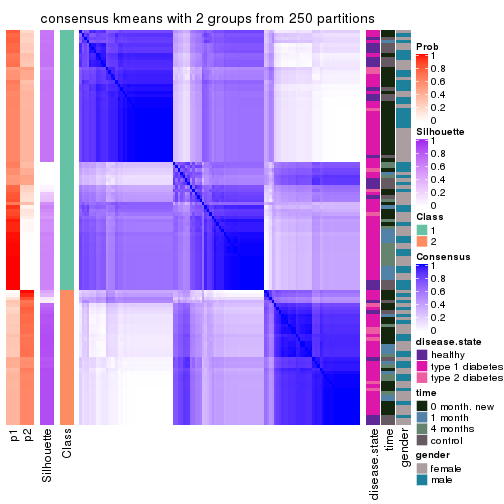
consensus_heatmap(res, k = 3)
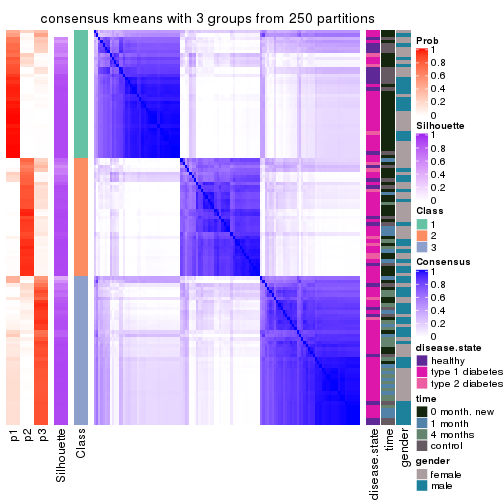
consensus_heatmap(res, k = 4)
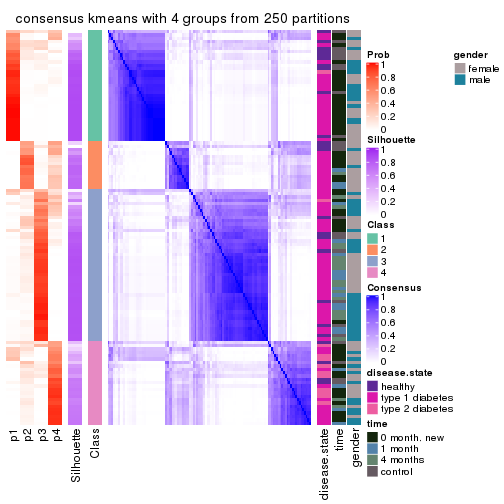
consensus_heatmap(res, k = 5)
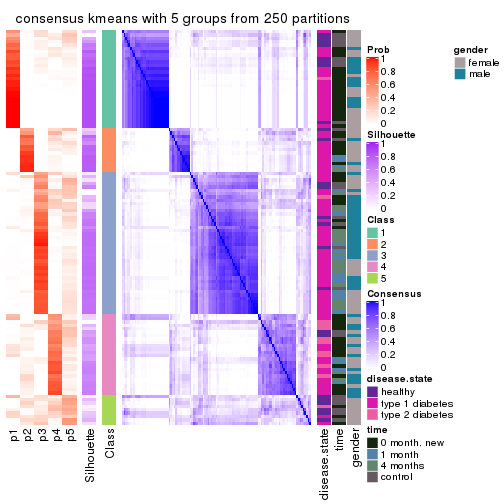
consensus_heatmap(res, k = 6)
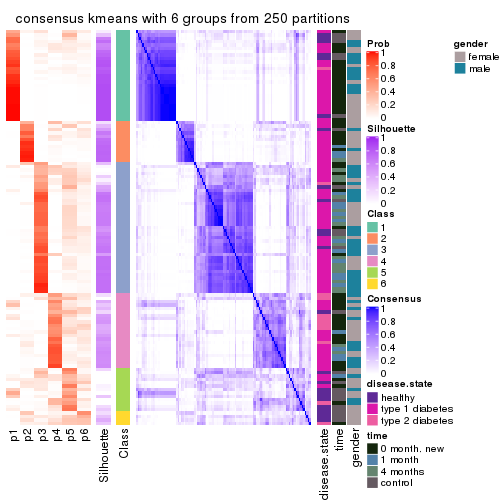
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
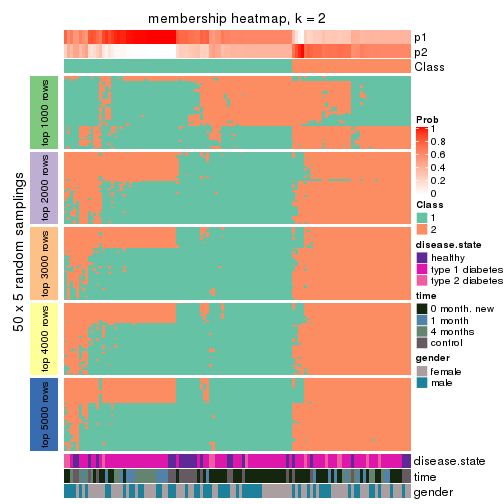
membership_heatmap(res, k = 3)
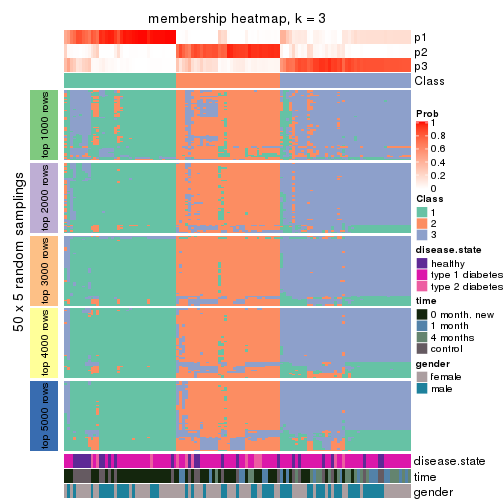
membership_heatmap(res, k = 4)
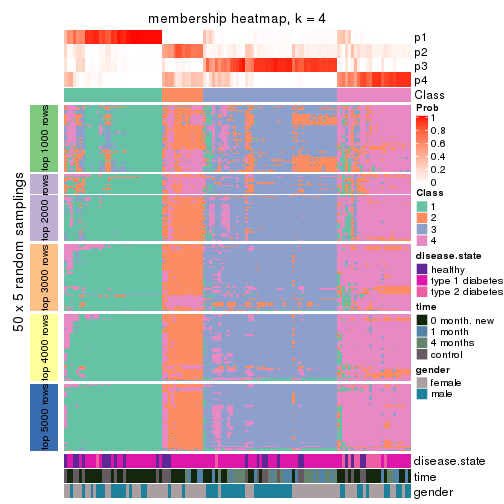
membership_heatmap(res, k = 5)
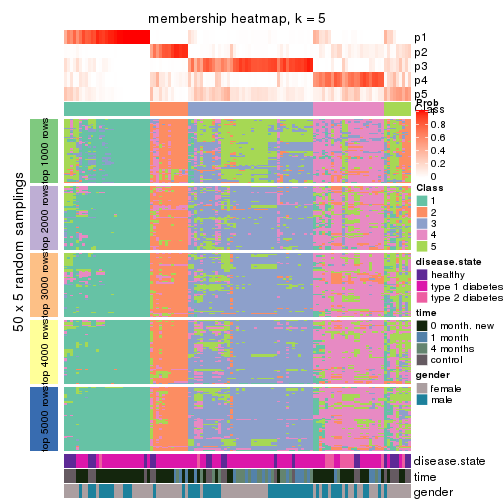
membership_heatmap(res, k = 6)
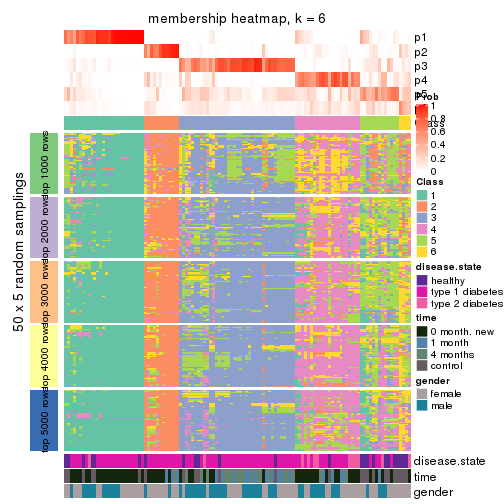
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
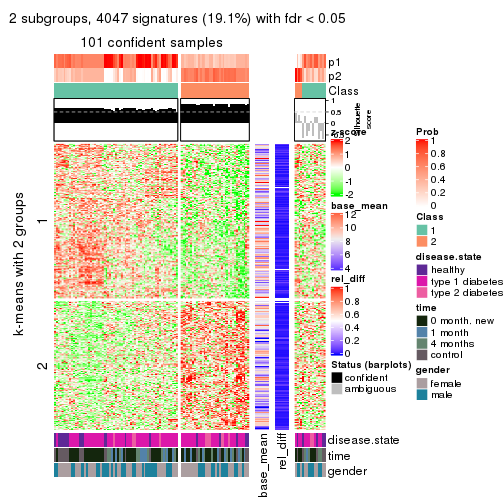
get_signatures(res, k = 3)
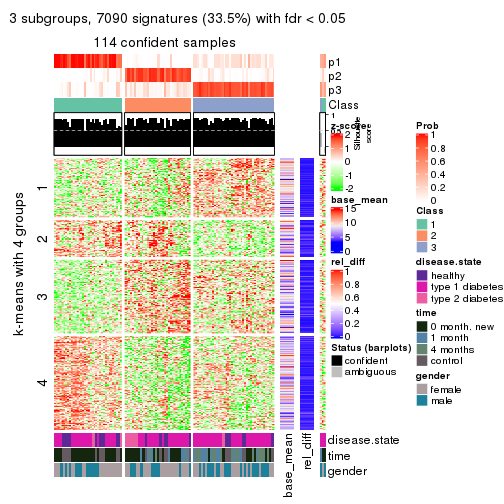
get_signatures(res, k = 4)
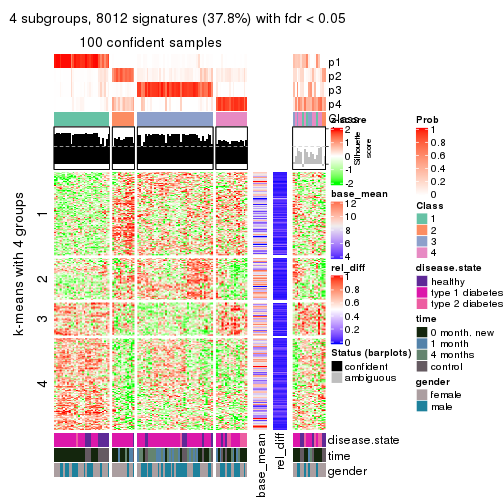
get_signatures(res, k = 5)
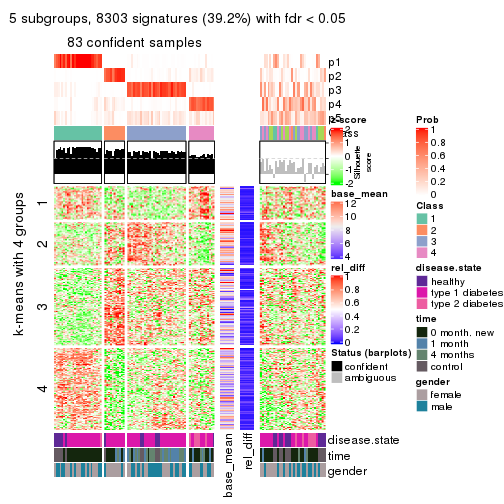
get_signatures(res, k = 6)
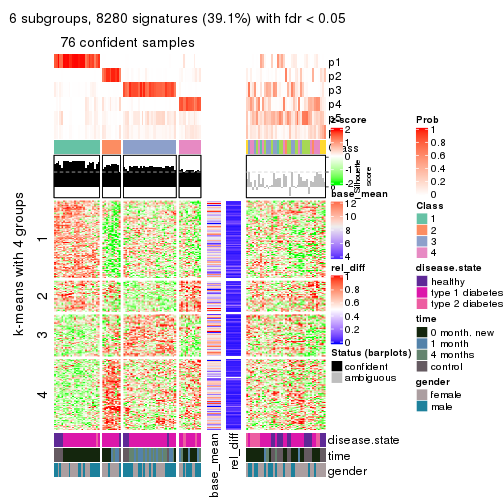
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
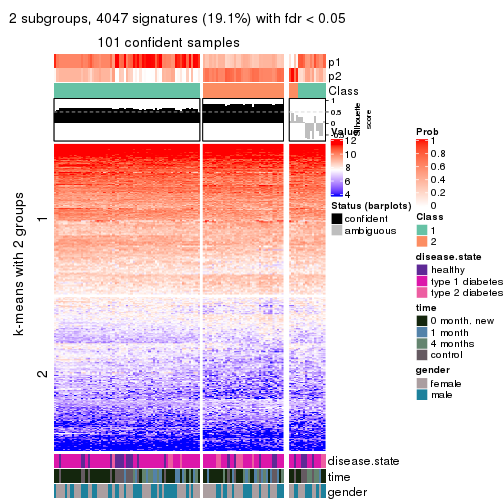
get_signatures(res, k = 3, scale_rows = FALSE)
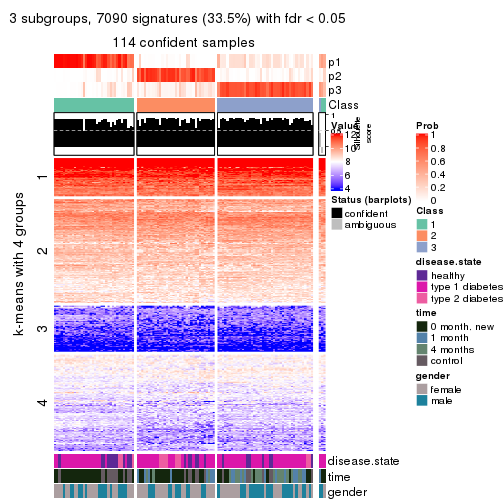
get_signatures(res, k = 4, scale_rows = FALSE)
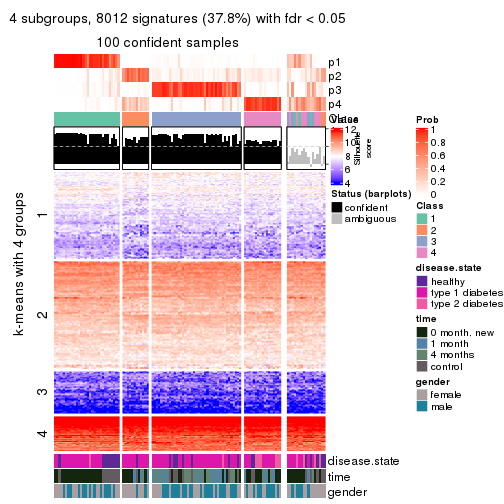
get_signatures(res, k = 5, scale_rows = FALSE)
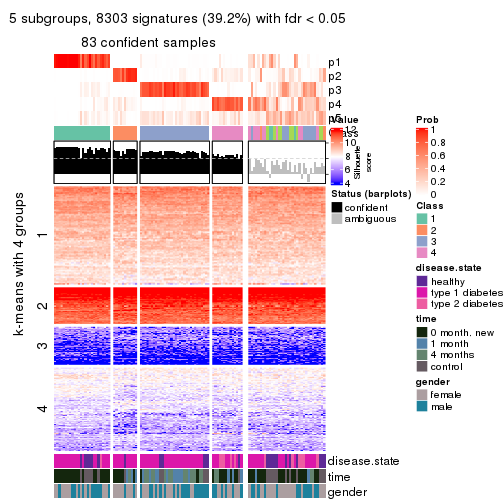
get_signatures(res, k = 6, scale_rows = FALSE)
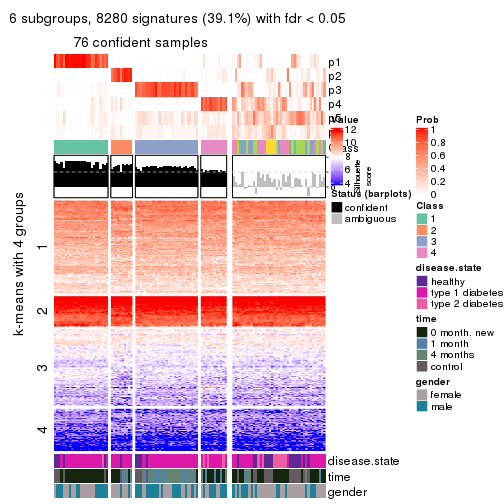
Compare the overlap of signatures from different k:
compare_signatures(res)
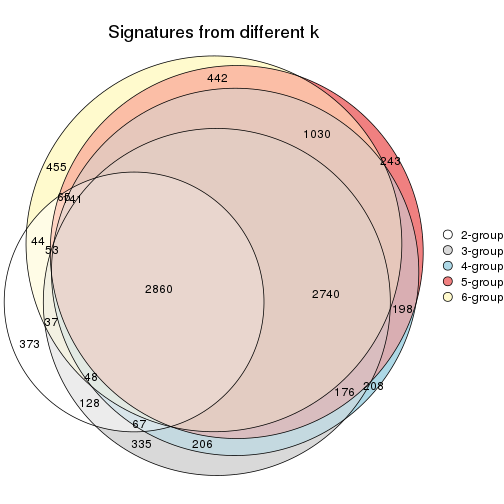
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
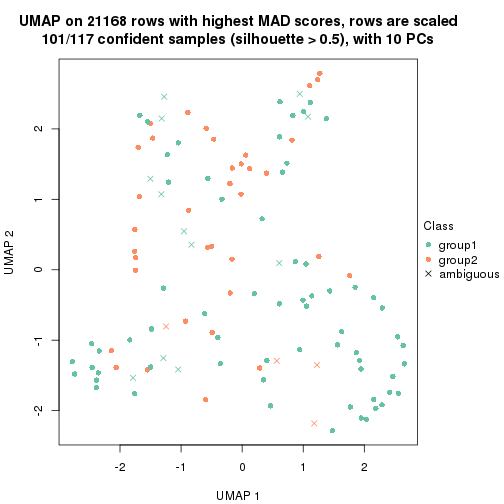
dimension_reduction(res, k = 3, method = "UMAP")
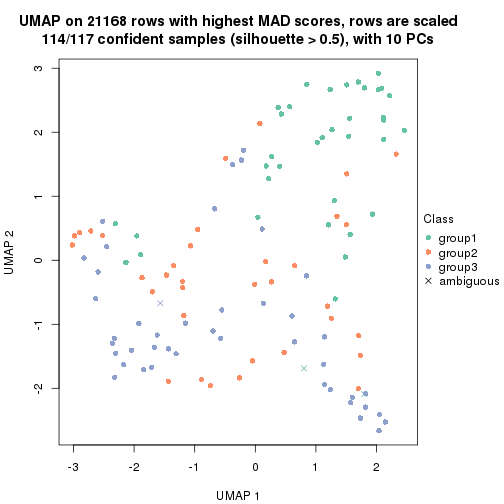
dimension_reduction(res, k = 4, method = "UMAP")
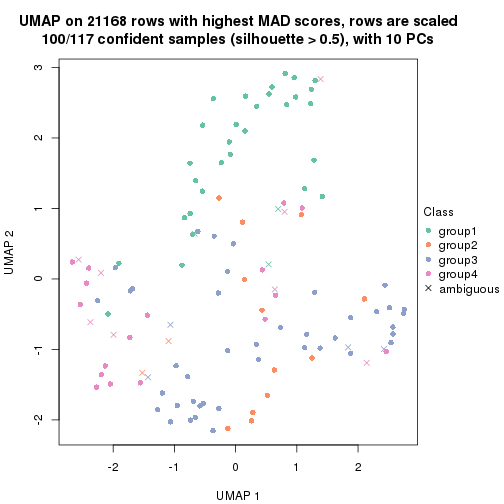
dimension_reduction(res, k = 5, method = "UMAP")
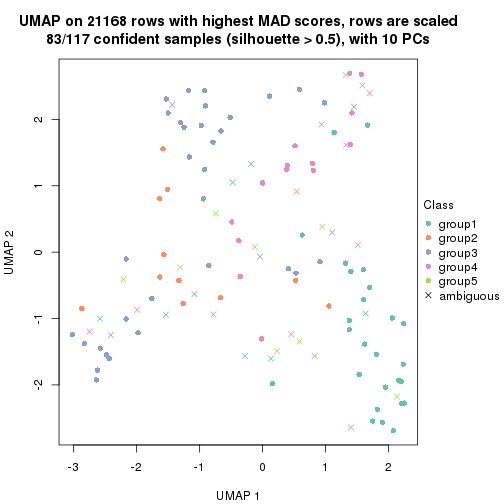
dimension_reduction(res, k = 6, method = "UMAP")
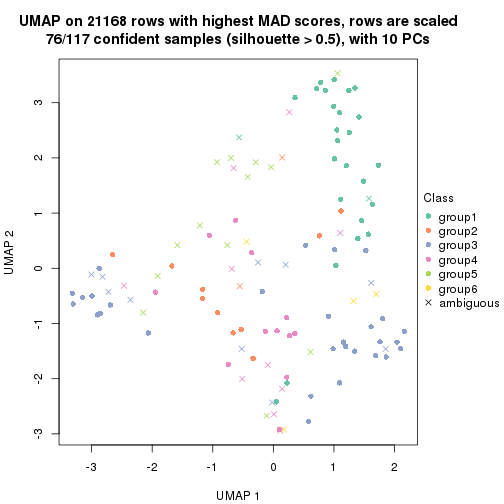
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
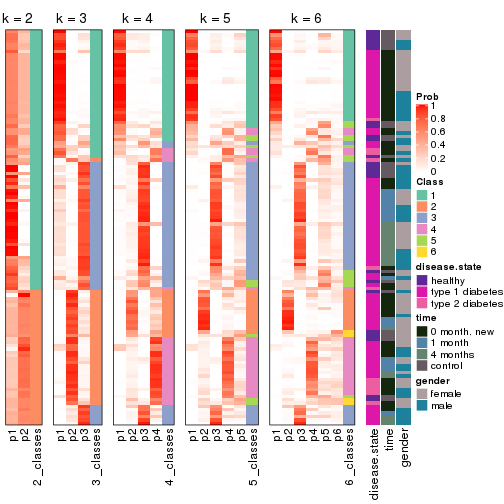
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> MAD:kmeans 101 0.188863 6.89e-01 0.620 2
#> MAD:kmeans 114 0.092511 5.00e-07 0.224 3
#> MAD:kmeans 100 0.000448 3.93e-05 0.102 4
#> MAD:kmeans 83 0.001038 2.11e-06 0.125 5
#> MAD:kmeans 76 0.030945 1.19e-06 0.184 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["MAD", "skmeans"]
# you can also extract it by
# res = res_list["MAD:skmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'MAD' method.
#> Subgroups are detected by 'skmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
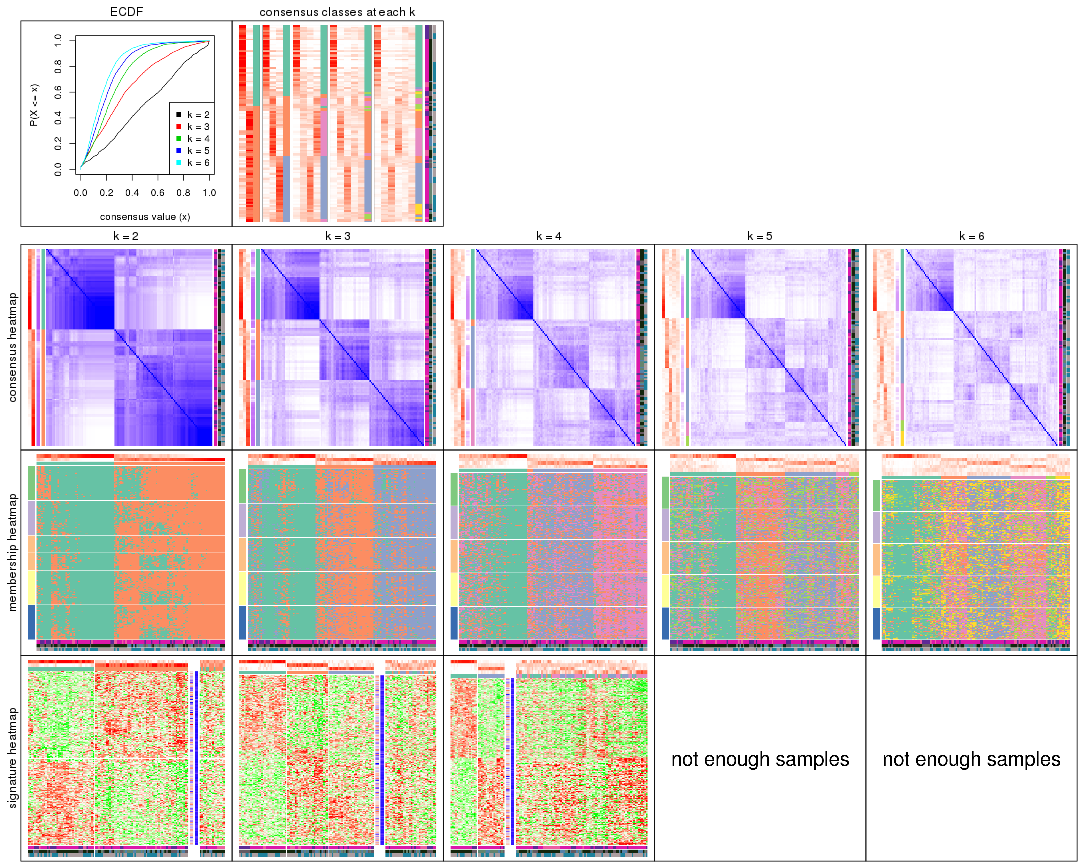
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
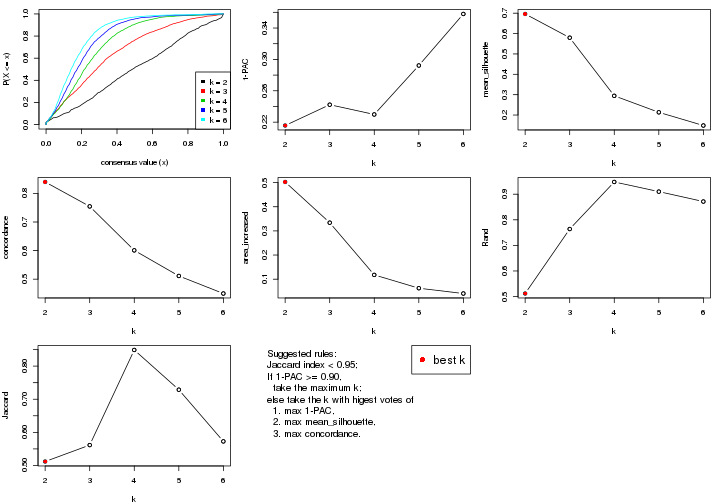
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.216 0.696 0.840 0.5025 0.512 0.512
#> 3 3 0.242 0.580 0.755 0.3340 0.764 0.562
#> 4 4 0.230 0.294 0.601 0.1178 0.948 0.849
#> 5 5 0.292 0.213 0.511 0.0627 0.910 0.729
#> 6 6 0.358 0.148 0.450 0.0407 0.871 0.573
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.7674 0.701 0.776 0.224
#> GSM228563 1 0.9881 0.213 0.564 0.436
#> GSM228565 1 0.8443 0.598 0.728 0.272
#> GSM228566 2 0.6887 0.754 0.184 0.816
#> GSM228567 1 0.0000 0.851 1.000 0.000
#> GSM228570 1 0.0000 0.851 1.000 0.000
#> GSM228571 1 0.4939 0.784 0.892 0.108
#> GSM228574 2 0.1633 0.777 0.024 0.976
#> GSM228575 2 0.6712 0.764 0.176 0.824
#> GSM228576 1 0.7453 0.651 0.788 0.212
#> GSM228579 1 0.5519 0.764 0.872 0.128
#> GSM228580 2 0.2043 0.774 0.032 0.968
#> GSM228581 2 0.4562 0.775 0.096 0.904
#> GSM228666 2 0.0938 0.773 0.012 0.988
#> GSM228564 1 0.7602 0.694 0.780 0.220
#> GSM228568 1 0.8713 0.574 0.708 0.292
#> GSM228569 1 0.4161 0.807 0.916 0.084
#> GSM228572 2 0.2043 0.772 0.032 0.968
#> GSM228573 2 0.8386 0.690 0.268 0.732
#> GSM228577 1 0.0376 0.850 0.996 0.004
#> GSM228578 1 0.2948 0.834 0.948 0.052
#> GSM228663 2 0.9000 0.633 0.316 0.684
#> GSM228664 2 0.1843 0.777 0.028 0.972
#> GSM228665 2 0.8861 0.663 0.304 0.696
#> GSM228582 1 0.9460 0.362 0.636 0.364
#> GSM228583 1 0.0000 0.851 1.000 0.000
#> GSM228585 1 0.0000 0.851 1.000 0.000
#> GSM228587 1 0.0376 0.850 0.996 0.004
#> GSM228588 1 0.7056 0.718 0.808 0.192
#> GSM228589 2 0.9170 0.532 0.332 0.668
#> GSM228590 1 0.0000 0.851 1.000 0.000
#> GSM228591 2 0.4431 0.773 0.092 0.908
#> GSM228597 2 0.9909 0.231 0.444 0.556
#> GSM228601 2 0.9710 0.370 0.400 0.600
#> GSM228604 2 0.0000 0.770 0.000 1.000
#> GSM228608 1 0.0000 0.851 1.000 0.000
#> GSM228609 1 0.5408 0.790 0.876 0.124
#> GSM228613 1 0.0000 0.851 1.000 0.000
#> GSM228616 1 0.9209 0.493 0.664 0.336
#> GSM228628 2 0.5737 0.763 0.136 0.864
#> GSM228634 1 0.0000 0.851 1.000 0.000
#> GSM228642 2 0.0000 0.770 0.000 1.000
#> GSM228645 2 0.8661 0.663 0.288 0.712
#> GSM228646 2 0.9248 0.577 0.340 0.660
#> GSM228652 1 0.0376 0.851 0.996 0.004
#> GSM228655 1 0.2423 0.845 0.960 0.040
#> GSM228656 1 0.0000 0.851 1.000 0.000
#> GSM228659 1 0.1843 0.845 0.972 0.028
#> GSM228662 1 0.0000 0.851 1.000 0.000
#> GSM228584 1 0.0000 0.851 1.000 0.000
#> GSM228586 1 0.0000 0.851 1.000 0.000
#> GSM228592 1 0.0000 0.851 1.000 0.000
#> GSM228593 1 0.6247 0.759 0.844 0.156
#> GSM228594 1 0.0672 0.849 0.992 0.008
#> GSM228598 1 0.0376 0.851 0.996 0.004
#> GSM228607 2 0.9954 0.293 0.460 0.540
#> GSM228612 2 0.7602 0.737 0.220 0.780
#> GSM228619 1 0.7453 0.690 0.788 0.212
#> GSM228622 1 0.0938 0.849 0.988 0.012
#> GSM228625 1 0.3431 0.832 0.936 0.064
#> GSM228631 1 0.5737 0.774 0.864 0.136
#> GSM228633 2 0.0000 0.770 0.000 1.000
#> GSM228637 1 0.9933 0.156 0.548 0.452
#> GSM228639 2 0.5629 0.740 0.132 0.868
#> GSM228649 1 0.9635 0.354 0.612 0.388
#> GSM228660 1 0.4022 0.833 0.920 0.080
#> GSM228661 1 0.4298 0.807 0.912 0.088
#> GSM228595 2 0.0000 0.770 0.000 1.000
#> GSM228599 2 0.9129 0.560 0.328 0.672
#> GSM228602 2 0.9795 0.463 0.416 0.584
#> GSM228614 2 0.8555 0.613 0.280 0.720
#> GSM228626 2 0.0000 0.770 0.000 1.000
#> GSM228640 2 0.8267 0.689 0.260 0.740
#> GSM228643 2 0.6973 0.756 0.188 0.812
#> GSM228650 2 0.1414 0.775 0.020 0.980
#> GSM228653 2 0.8144 0.695 0.252 0.748
#> GSM228657 2 0.3584 0.766 0.068 0.932
#> GSM228605 1 0.8144 0.632 0.748 0.252
#> GSM228610 2 0.7139 0.754 0.196 0.804
#> GSM228617 2 0.9850 0.445 0.428 0.572
#> GSM228620 2 0.9209 0.616 0.336 0.664
#> GSM228623 2 0.8555 0.607 0.280 0.720
#> GSM228629 2 0.9580 0.528 0.380 0.620
#> GSM228632 2 0.0000 0.770 0.000 1.000
#> GSM228635 2 0.8443 0.613 0.272 0.728
#> GSM228647 2 0.6148 0.766 0.152 0.848
#> GSM228596 2 0.9427 0.574 0.360 0.640
#> GSM228600 2 0.5946 0.765 0.144 0.856
#> GSM228603 2 0.9044 0.620 0.320 0.680
#> GSM228615 2 0.9460 0.457 0.364 0.636
#> GSM228627 2 0.7299 0.744 0.204 0.796
#> GSM228641 2 0.5737 0.770 0.136 0.864
#> GSM228644 2 0.0000 0.770 0.000 1.000
#> GSM228651 2 0.5737 0.769 0.136 0.864
#> GSM228654 2 0.3733 0.777 0.072 0.928
#> GSM228658 2 0.8555 0.671 0.280 0.720
#> GSM228606 2 0.0000 0.770 0.000 1.000
#> GSM228611 2 0.7602 0.734 0.220 0.780
#> GSM228618 2 0.8661 0.683 0.288 0.712
#> GSM228621 2 0.0938 0.773 0.012 0.988
#> GSM228624 2 0.1843 0.777 0.028 0.972
#> GSM228630 2 0.0376 0.771 0.004 0.996
#> GSM228636 2 0.8267 0.629 0.260 0.740
#> GSM228638 2 0.4161 0.782 0.084 0.916
#> GSM228648 2 0.0000 0.770 0.000 1.000
#> GSM228670 2 0.9580 0.426 0.380 0.620
#> GSM228671 2 0.2423 0.773 0.040 0.960
#> GSM228672 1 0.3584 0.829 0.932 0.068
#> GSM228674 1 0.9491 0.414 0.632 0.368
#> GSM228675 2 0.9686 0.386 0.396 0.604
#> GSM228676 2 1.0000 0.137 0.496 0.504
#> GSM228667 2 0.9944 0.237 0.456 0.544
#> GSM228668 1 0.0000 0.851 1.000 0.000
#> GSM228669 1 0.4161 0.823 0.916 0.084
#> GSM228673 2 0.5629 0.778 0.132 0.868
#> GSM228677 2 0.0000 0.770 0.000 1.000
#> GSM228678 2 0.7883 0.664 0.236 0.764
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.9621 0.2607 0.472 0.276 0.252
#> GSM228563 2 0.6258 0.6483 0.196 0.752 0.052
#> GSM228565 1 0.9757 0.1087 0.432 0.324 0.244
#> GSM228566 3 0.7941 0.5608 0.096 0.276 0.628
#> GSM228567 1 0.0237 0.7620 0.996 0.000 0.004
#> GSM228570 1 0.3434 0.7540 0.904 0.064 0.032
#> GSM228571 1 0.3826 0.7370 0.868 0.008 0.124
#> GSM228574 3 0.6625 0.5581 0.024 0.316 0.660
#> GSM228575 3 0.9256 0.3058 0.168 0.344 0.488
#> GSM228576 1 0.8055 0.4930 0.612 0.096 0.292
#> GSM228579 1 0.3539 0.7459 0.888 0.012 0.100
#> GSM228580 2 0.5201 0.6151 0.004 0.760 0.236
#> GSM228581 2 0.8108 0.2289 0.072 0.536 0.392
#> GSM228666 2 0.6294 0.5594 0.020 0.692 0.288
#> GSM228564 2 0.8454 0.0494 0.432 0.480 0.088
#> GSM228568 1 0.9783 0.1692 0.436 0.264 0.300
#> GSM228569 1 0.5325 0.6303 0.748 0.004 0.248
#> GSM228572 2 0.2711 0.6929 0.000 0.912 0.088
#> GSM228573 3 0.4742 0.7254 0.104 0.048 0.848
#> GSM228577 1 0.1860 0.7624 0.948 0.000 0.052
#> GSM228578 1 0.5874 0.6676 0.760 0.032 0.208
#> GSM228663 3 0.4544 0.7284 0.084 0.056 0.860
#> GSM228664 3 0.5858 0.6619 0.020 0.240 0.740
#> GSM228665 3 0.7281 0.6732 0.148 0.140 0.712
#> GSM228582 1 0.9339 0.2845 0.496 0.188 0.316
#> GSM228583 1 0.0237 0.7620 0.996 0.000 0.004
#> GSM228585 1 0.0424 0.7625 0.992 0.000 0.008
#> GSM228587 1 0.1411 0.7591 0.964 0.036 0.000
#> GSM228588 2 0.6057 0.4336 0.340 0.656 0.004
#> GSM228589 2 0.2918 0.7040 0.044 0.924 0.032
#> GSM228590 1 0.0237 0.7611 0.996 0.004 0.000
#> GSM228591 2 0.7064 0.6039 0.076 0.704 0.220
#> GSM228597 2 0.3472 0.7054 0.056 0.904 0.040
#> GSM228601 2 0.1905 0.6964 0.028 0.956 0.016
#> GSM228604 3 0.6180 0.3676 0.000 0.416 0.584
#> GSM228608 1 0.2313 0.7638 0.944 0.024 0.032
#> GSM228609 1 0.7004 0.2536 0.552 0.428 0.020
#> GSM228613 1 0.0000 0.7612 1.000 0.000 0.000
#> GSM228616 1 0.9566 0.0821 0.440 0.360 0.200
#> GSM228628 2 0.8462 0.4646 0.124 0.588 0.288
#> GSM228634 1 0.0424 0.7623 0.992 0.000 0.008
#> GSM228642 2 0.5560 0.4935 0.000 0.700 0.300
#> GSM228645 3 0.9134 0.3313 0.156 0.344 0.500
#> GSM228646 3 0.9304 0.3987 0.204 0.280 0.516
#> GSM228652 1 0.3583 0.7552 0.900 0.056 0.044
#> GSM228655 1 0.8067 0.5888 0.652 0.188 0.160
#> GSM228656 1 0.0424 0.7625 0.992 0.000 0.008
#> GSM228659 1 0.5122 0.6672 0.788 0.200 0.012
#> GSM228662 1 0.0237 0.7620 0.996 0.000 0.004
#> GSM228584 1 0.0237 0.7621 0.996 0.000 0.004
#> GSM228586 1 0.0747 0.7624 0.984 0.000 0.016
#> GSM228592 1 0.0237 0.7620 0.996 0.000 0.004
#> GSM228593 1 0.6495 0.1913 0.536 0.460 0.004
#> GSM228594 1 0.1647 0.7634 0.960 0.004 0.036
#> GSM228598 1 0.1525 0.7627 0.964 0.032 0.004
#> GSM228607 2 0.9956 0.0451 0.292 0.372 0.336
#> GSM228612 3 0.8842 0.5344 0.208 0.212 0.580
#> GSM228619 1 0.9027 0.3408 0.532 0.160 0.308
#> GSM228622 1 0.4968 0.7005 0.800 0.012 0.188
#> GSM228625 1 0.7749 0.4941 0.624 0.300 0.076
#> GSM228631 1 0.7760 0.3642 0.580 0.060 0.360
#> GSM228633 2 0.3752 0.6727 0.000 0.856 0.144
#> GSM228637 2 0.5891 0.6707 0.168 0.780 0.052
#> GSM228639 3 0.7974 0.1803 0.060 0.436 0.504
#> GSM228649 2 0.6632 0.6338 0.204 0.732 0.064
#> GSM228660 1 0.7365 0.6288 0.700 0.188 0.112
#> GSM228661 1 0.4062 0.7123 0.836 0.000 0.164
#> GSM228595 2 0.1411 0.6884 0.000 0.964 0.036
#> GSM228599 2 0.7403 0.6193 0.096 0.688 0.216
#> GSM228602 3 0.6527 0.6681 0.188 0.068 0.744
#> GSM228614 2 0.7186 0.6114 0.080 0.696 0.224
#> GSM228626 2 0.4750 0.6011 0.000 0.784 0.216
#> GSM228640 3 0.2443 0.7196 0.028 0.032 0.940
#> GSM228643 3 0.5554 0.7250 0.076 0.112 0.812
#> GSM228650 3 0.5754 0.6054 0.004 0.296 0.700
#> GSM228653 3 0.1585 0.7132 0.028 0.008 0.964
#> GSM228657 2 0.3454 0.6929 0.008 0.888 0.104
#> GSM228605 1 0.9315 0.3844 0.520 0.220 0.260
#> GSM228610 3 0.6719 0.7071 0.096 0.160 0.744
#> GSM228617 3 0.6765 0.6340 0.208 0.068 0.724
#> GSM228620 3 0.5734 0.6861 0.164 0.048 0.788
#> GSM228623 2 0.5659 0.6821 0.052 0.796 0.152
#> GSM228629 3 0.3670 0.7201 0.092 0.020 0.888
#> GSM228632 3 0.6267 0.2559 0.000 0.452 0.548
#> GSM228635 2 0.2414 0.6995 0.020 0.940 0.040
#> GSM228647 3 0.5093 0.7342 0.076 0.088 0.836
#> GSM228596 3 0.9509 0.2903 0.220 0.296 0.484
#> GSM228600 3 0.3995 0.7231 0.016 0.116 0.868
#> GSM228603 3 0.2176 0.7153 0.032 0.020 0.948
#> GSM228615 2 0.3589 0.7060 0.048 0.900 0.052
#> GSM228627 3 0.5863 0.7202 0.084 0.120 0.796
#> GSM228641 3 0.2584 0.7179 0.008 0.064 0.928
#> GSM228644 2 0.4235 0.6495 0.000 0.824 0.176
#> GSM228651 3 0.2903 0.7214 0.028 0.048 0.924
#> GSM228654 3 0.4291 0.7138 0.008 0.152 0.840
#> GSM228658 3 0.3678 0.7231 0.080 0.028 0.892
#> GSM228606 2 0.7389 0.2015 0.036 0.556 0.408
#> GSM228611 3 0.4469 0.7342 0.060 0.076 0.864
#> GSM228618 3 0.5823 0.6978 0.144 0.064 0.792
#> GSM228621 3 0.4062 0.7070 0.000 0.164 0.836
#> GSM228624 3 0.8085 0.4643 0.084 0.332 0.584
#> GSM228630 3 0.6299 0.1507 0.000 0.476 0.524
#> GSM228636 2 0.1015 0.6897 0.008 0.980 0.012
#> GSM228638 3 0.6559 0.6484 0.040 0.252 0.708
#> GSM228648 3 0.4504 0.6871 0.000 0.196 0.804
#> GSM228670 2 0.6860 0.6441 0.176 0.732 0.092
#> GSM228671 2 0.6129 0.5504 0.016 0.700 0.284
#> GSM228672 1 0.6369 0.5161 0.668 0.316 0.016
#> GSM228674 2 0.8790 0.3522 0.340 0.532 0.128
#> GSM228675 2 0.5576 0.6959 0.104 0.812 0.084
#> GSM228676 2 0.9975 0.0486 0.320 0.368 0.312
#> GSM228667 2 0.9355 0.4046 0.252 0.516 0.232
#> GSM228668 1 0.5407 0.7306 0.820 0.076 0.104
#> GSM228669 1 0.6589 0.5587 0.688 0.280 0.032
#> GSM228673 3 0.8534 0.4712 0.116 0.320 0.564
#> GSM228677 2 0.5178 0.5797 0.000 0.744 0.256
#> GSM228678 2 0.2793 0.7043 0.028 0.928 0.044
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 2 0.9342 0.23614 0.312 0.392 0.160 0.136
#> GSM228563 4 0.7931 0.12779 0.120 0.288 0.052 0.540
#> GSM228565 1 0.9548 -0.27692 0.336 0.324 0.128 0.212
#> GSM228566 3 0.8685 0.31688 0.076 0.304 0.464 0.156
#> GSM228567 1 0.0592 0.58078 0.984 0.016 0.000 0.000
#> GSM228570 1 0.6424 0.45859 0.660 0.252 0.060 0.028
#> GSM228571 1 0.7022 0.43619 0.636 0.216 0.120 0.028
#> GSM228574 3 0.8226 0.27475 0.024 0.284 0.464 0.228
#> GSM228575 3 0.9404 -0.03081 0.096 0.288 0.348 0.268
#> GSM228576 1 0.9107 -0.01936 0.416 0.280 0.220 0.084
#> GSM228579 1 0.5340 0.52498 0.756 0.136 0.104 0.004
#> GSM228580 4 0.7083 0.39554 0.028 0.168 0.164 0.640
#> GSM228581 4 0.8700 0.05266 0.044 0.288 0.248 0.420
#> GSM228666 4 0.7772 0.24423 0.016 0.340 0.160 0.484
#> GSM228564 4 0.9497 -0.32799 0.300 0.276 0.104 0.320
#> GSM228568 1 0.9660 -0.17926 0.368 0.236 0.244 0.152
#> GSM228569 1 0.7007 0.37824 0.596 0.176 0.224 0.004
#> GSM228572 4 0.5314 0.45434 0.000 0.108 0.144 0.748
#> GSM228573 3 0.7094 0.51416 0.088 0.200 0.652 0.060
#> GSM228577 1 0.5817 0.52482 0.732 0.176 0.068 0.024
#> GSM228578 1 0.7841 0.34679 0.580 0.216 0.152 0.052
#> GSM228663 3 0.7229 0.51874 0.048 0.216 0.632 0.104
#> GSM228664 3 0.7615 0.40284 0.008 0.216 0.524 0.252
#> GSM228665 3 0.8201 0.40648 0.108 0.220 0.564 0.108
#> GSM228582 1 0.9845 -0.24061 0.336 0.248 0.220 0.196
#> GSM228583 1 0.0817 0.58146 0.976 0.024 0.000 0.000
#> GSM228585 1 0.1004 0.58193 0.972 0.024 0.004 0.000
#> GSM228587 1 0.3312 0.56746 0.876 0.072 0.000 0.052
#> GSM228588 4 0.6897 0.05045 0.256 0.160 0.000 0.584
#> GSM228589 4 0.5138 0.44420 0.020 0.180 0.036 0.764
#> GSM228590 1 0.1118 0.58060 0.964 0.036 0.000 0.000
#> GSM228591 4 0.7137 0.35372 0.024 0.208 0.144 0.624
#> GSM228597 4 0.6667 0.37328 0.064 0.220 0.048 0.668
#> GSM228601 4 0.4954 0.42911 0.028 0.180 0.020 0.772
#> GSM228604 4 0.7546 -0.13453 0.000 0.188 0.404 0.408
#> GSM228608 1 0.4480 0.55061 0.820 0.100 0.072 0.008
#> GSM228609 1 0.8274 -0.11149 0.424 0.208 0.024 0.344
#> GSM228613 1 0.0592 0.57931 0.984 0.016 0.000 0.000
#> GSM228616 1 0.9751 -0.27053 0.356 0.228 0.172 0.244
#> GSM228628 4 0.8590 0.13336 0.072 0.324 0.144 0.460
#> GSM228634 1 0.1520 0.58214 0.956 0.020 0.024 0.000
#> GSM228642 4 0.6942 0.31188 0.000 0.176 0.240 0.584
#> GSM228645 3 0.9666 -0.00320 0.136 0.280 0.336 0.248
#> GSM228646 3 0.9814 -0.14675 0.164 0.296 0.300 0.240
#> GSM228652 1 0.7650 0.37887 0.612 0.208 0.092 0.088
#> GSM228655 1 0.9137 -0.00320 0.448 0.268 0.148 0.136
#> GSM228656 1 0.0592 0.58123 0.984 0.016 0.000 0.000
#> GSM228659 1 0.6624 0.39882 0.640 0.204 0.004 0.152
#> GSM228662 1 0.0592 0.58049 0.984 0.016 0.000 0.000
#> GSM228584 1 0.0592 0.58150 0.984 0.016 0.000 0.000
#> GSM228586 1 0.1109 0.58184 0.968 0.028 0.004 0.000
#> GSM228592 1 0.0592 0.57977 0.984 0.016 0.000 0.000
#> GSM228593 1 0.8351 -0.01528 0.444 0.224 0.028 0.304
#> GSM228594 1 0.3004 0.57624 0.892 0.060 0.048 0.000
#> GSM228598 1 0.3683 0.57242 0.856 0.112 0.016 0.016
#> GSM228607 2 0.9369 0.12583 0.092 0.352 0.276 0.280
#> GSM228612 3 0.9416 0.17690 0.140 0.276 0.404 0.180
#> GSM228619 1 0.9572 -0.17505 0.388 0.184 0.264 0.164
#> GSM228622 1 0.6749 0.41892 0.640 0.180 0.172 0.008
#> GSM228625 1 0.9278 -0.20986 0.372 0.244 0.088 0.296
#> GSM228631 1 0.9020 -0.08476 0.408 0.184 0.324 0.084
#> GSM228633 4 0.5767 0.44557 0.000 0.136 0.152 0.712
#> GSM228637 4 0.7401 0.29303 0.096 0.256 0.048 0.600
#> GSM228639 3 0.8491 0.02696 0.024 0.276 0.380 0.320
#> GSM228649 4 0.8242 0.12257 0.160 0.236 0.064 0.540
#> GSM228660 1 0.8924 0.15097 0.480 0.236 0.104 0.180
#> GSM228661 1 0.5486 0.49255 0.732 0.076 0.188 0.004
#> GSM228595 4 0.3081 0.45818 0.000 0.064 0.048 0.888
#> GSM228599 4 0.8515 0.16028 0.048 0.260 0.216 0.476
#> GSM228602 3 0.7872 0.42460 0.144 0.168 0.604 0.084
#> GSM228614 4 0.8270 0.20767 0.060 0.336 0.124 0.480
#> GSM228626 4 0.5962 0.41669 0.000 0.128 0.180 0.692
#> GSM228640 3 0.4440 0.54752 0.028 0.128 0.820 0.024
#> GSM228643 3 0.7800 0.47131 0.092 0.200 0.604 0.104
#> GSM228650 3 0.7617 0.43051 0.016 0.224 0.552 0.208
#> GSM228653 3 0.4148 0.55657 0.012 0.124 0.832 0.032
#> GSM228657 4 0.5308 0.45976 0.004 0.148 0.092 0.756
#> GSM228605 1 0.9611 -0.32572 0.344 0.316 0.184 0.156
#> GSM228610 3 0.7856 0.46631 0.044 0.228 0.568 0.160
#> GSM228617 3 0.8157 0.35867 0.152 0.192 0.572 0.084
#> GSM228620 3 0.6783 0.51312 0.084 0.196 0.672 0.048
#> GSM228623 4 0.7224 0.29390 0.024 0.316 0.096 0.564
#> GSM228629 3 0.7445 0.46847 0.132 0.192 0.624 0.052
#> GSM228632 3 0.7811 0.19688 0.000 0.268 0.412 0.320
#> GSM228635 4 0.5843 0.39686 0.012 0.312 0.032 0.644
#> GSM228647 3 0.7271 0.51309 0.068 0.192 0.644 0.096
#> GSM228596 3 0.9554 -0.11137 0.124 0.316 0.336 0.224
#> GSM228600 3 0.6171 0.52959 0.024 0.192 0.704 0.080
#> GSM228603 3 0.4779 0.53998 0.048 0.128 0.804 0.020
#> GSM228615 4 0.7303 0.31606 0.076 0.288 0.048 0.588
#> GSM228627 3 0.7806 0.50188 0.064 0.224 0.588 0.124
#> GSM228641 3 0.5673 0.55011 0.028 0.160 0.748 0.064
#> GSM228644 4 0.5188 0.45135 0.000 0.148 0.096 0.756
#> GSM228651 3 0.5972 0.55218 0.016 0.180 0.716 0.088
#> GSM228654 3 0.6467 0.53246 0.012 0.144 0.676 0.168
#> GSM228658 3 0.6078 0.55762 0.032 0.164 0.724 0.080
#> GSM228606 4 0.8243 0.06148 0.012 0.296 0.304 0.388
#> GSM228611 3 0.6975 0.52256 0.060 0.200 0.660 0.080
#> GSM228618 3 0.6404 0.53098 0.048 0.152 0.712 0.088
#> GSM228621 3 0.6683 0.51472 0.000 0.204 0.620 0.176
#> GSM228624 3 0.8563 0.22697 0.032 0.312 0.404 0.252
#> GSM228630 4 0.7684 -0.04402 0.000 0.216 0.388 0.396
#> GSM228636 4 0.4294 0.42578 0.008 0.204 0.008 0.780
#> GSM228638 3 0.7294 0.46000 0.020 0.184 0.604 0.192
#> GSM228648 3 0.6576 0.50287 0.000 0.152 0.628 0.220
#> GSM228670 4 0.9005 -0.00262 0.172 0.292 0.096 0.440
#> GSM228671 4 0.8133 0.20403 0.016 0.264 0.264 0.456
#> GSM228672 1 0.8122 0.02970 0.476 0.300 0.024 0.200
#> GSM228674 2 0.9392 0.22878 0.204 0.364 0.112 0.320
#> GSM228675 4 0.8294 0.09587 0.132 0.300 0.064 0.504
#> GSM228676 1 0.9852 -0.42442 0.300 0.300 0.208 0.192
#> GSM228667 2 0.9529 0.18035 0.144 0.340 0.180 0.336
#> GSM228668 1 0.7234 0.43188 0.644 0.200 0.084 0.072
#> GSM228669 1 0.7995 0.16453 0.508 0.268 0.024 0.200
#> GSM228673 3 0.9362 0.03979 0.116 0.300 0.388 0.196
#> GSM228677 4 0.7299 0.28672 0.000 0.312 0.176 0.512
#> GSM228678 4 0.6328 0.39286 0.020 0.256 0.064 0.660
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 1 0.9563 -0.19529 0.304 0.116 0.132 0.272 0.176
#> GSM228563 4 0.8197 0.09992 0.072 0.360 0.036 0.392 0.140
#> GSM228565 1 0.9535 -0.11729 0.324 0.136 0.104 0.236 0.200
#> GSM228566 3 0.9081 -0.03859 0.036 0.180 0.332 0.192 0.260
#> GSM228567 1 0.1299 0.57607 0.960 0.000 0.008 0.020 0.012
#> GSM228570 1 0.7947 0.34517 0.524 0.056 0.060 0.216 0.144
#> GSM228571 1 0.7908 0.34102 0.528 0.024 0.168 0.116 0.164
#> GSM228574 3 0.8780 -0.01537 0.016 0.196 0.336 0.176 0.276
#> GSM228575 3 0.9488 -0.13359 0.060 0.208 0.260 0.240 0.232
#> GSM228576 1 0.9042 -0.09487 0.340 0.032 0.204 0.184 0.240
#> GSM228579 1 0.6142 0.49345 0.672 0.012 0.048 0.088 0.180
#> GSM228580 2 0.8147 0.20334 0.012 0.448 0.116 0.256 0.168
#> GSM228581 2 0.8983 -0.00295 0.032 0.336 0.160 0.200 0.272
#> GSM228666 2 0.8280 0.17250 0.004 0.412 0.144 0.224 0.216
#> GSM228564 4 0.9271 0.14249 0.264 0.204 0.068 0.332 0.132
#> GSM228568 5 0.9465 0.13268 0.236 0.108 0.140 0.168 0.348
#> GSM228569 1 0.7575 0.29474 0.516 0.004 0.168 0.096 0.216
#> GSM228572 2 0.6069 0.32552 0.000 0.680 0.092 0.116 0.112
#> GSM228573 3 0.7161 0.30452 0.064 0.040 0.576 0.068 0.252
#> GSM228577 1 0.6775 0.47997 0.640 0.044 0.052 0.080 0.184
#> GSM228578 1 0.8270 0.28090 0.500 0.052 0.144 0.096 0.208
#> GSM228663 3 0.8410 0.17198 0.060 0.096 0.420 0.100 0.324
#> GSM228664 3 0.8457 0.08955 0.016 0.248 0.328 0.092 0.316
#> GSM228665 3 0.9135 0.10347 0.128 0.084 0.396 0.160 0.232
#> GSM228582 1 0.9670 -0.20632 0.312 0.184 0.140 0.132 0.232
#> GSM228583 1 0.1211 0.57379 0.960 0.000 0.000 0.016 0.024
#> GSM228585 1 0.1106 0.57510 0.964 0.000 0.000 0.012 0.024
#> GSM228587 1 0.3463 0.55827 0.836 0.016 0.000 0.128 0.020
#> GSM228588 2 0.7894 -0.18388 0.252 0.436 0.004 0.228 0.080
#> GSM228589 2 0.6156 0.25077 0.036 0.652 0.012 0.216 0.084
#> GSM228590 1 0.1943 0.57341 0.924 0.000 0.000 0.056 0.020
#> GSM228591 2 0.7910 0.24146 0.052 0.548 0.112 0.100 0.188
#> GSM228597 2 0.7522 0.01536 0.088 0.500 0.032 0.316 0.064
#> GSM228601 2 0.4936 0.29792 0.008 0.748 0.016 0.164 0.064
#> GSM228604 3 0.7713 0.05890 0.000 0.364 0.396 0.104 0.136
#> GSM228608 1 0.5834 0.51324 0.688 0.004 0.048 0.176 0.084
#> GSM228609 1 0.8628 -0.08976 0.380 0.252 0.036 0.248 0.084
#> GSM228613 1 0.1281 0.57261 0.956 0.000 0.000 0.032 0.012
#> GSM228616 1 0.9946 -0.37511 0.224 0.216 0.152 0.212 0.196
#> GSM228628 2 0.8711 0.15141 0.048 0.432 0.116 0.180 0.224
#> GSM228634 1 0.2151 0.57637 0.924 0.000 0.016 0.020 0.040
#> GSM228642 2 0.7563 0.27687 0.000 0.520 0.160 0.140 0.180
#> GSM228645 5 0.9451 0.12440 0.072 0.176 0.240 0.196 0.316
#> GSM228646 5 0.9592 0.14425 0.080 0.204 0.192 0.244 0.280
#> GSM228652 1 0.6895 0.44015 0.592 0.040 0.024 0.236 0.108
#> GSM228655 1 0.8764 0.20928 0.448 0.072 0.128 0.224 0.128
#> GSM228656 1 0.0671 0.57204 0.980 0.000 0.000 0.004 0.016
#> GSM228659 1 0.7748 0.26780 0.480 0.120 0.008 0.280 0.112
#> GSM228662 1 0.1106 0.57394 0.964 0.000 0.000 0.012 0.024
#> GSM228584 1 0.0451 0.57117 0.988 0.000 0.000 0.004 0.008
#> GSM228586 1 0.1525 0.57575 0.948 0.000 0.004 0.012 0.036
#> GSM228592 1 0.0579 0.57220 0.984 0.000 0.000 0.008 0.008
#> GSM228593 1 0.8662 -0.20407 0.332 0.256 0.016 0.272 0.124
#> GSM228594 1 0.4210 0.55500 0.808 0.004 0.040 0.028 0.120
#> GSM228598 1 0.5713 0.52190 0.704 0.020 0.016 0.148 0.112
#> GSM228607 5 0.9481 0.11913 0.076 0.188 0.212 0.200 0.324
#> GSM228612 3 0.9312 0.03065 0.104 0.152 0.324 0.116 0.304
#> GSM228619 1 0.9204 -0.07812 0.364 0.116 0.252 0.188 0.080
#> GSM228622 1 0.7000 0.43391 0.604 0.008 0.172 0.108 0.108
#> GSM228625 1 0.8898 -0.09755 0.352 0.196 0.052 0.300 0.100
#> GSM228631 1 0.8959 -0.15770 0.344 0.076 0.336 0.112 0.132
#> GSM228633 2 0.6078 0.36480 0.000 0.676 0.140 0.084 0.100
#> GSM228637 2 0.7566 -0.11084 0.080 0.408 0.012 0.400 0.100
#> GSM228639 2 0.8714 0.04778 0.008 0.300 0.272 0.248 0.172
#> GSM228649 4 0.8341 0.01963 0.080 0.368 0.044 0.376 0.132
#> GSM228660 1 0.9264 0.10513 0.400 0.148 0.100 0.172 0.180
#> GSM228661 1 0.5232 0.51328 0.732 0.000 0.108 0.032 0.128
#> GSM228595 2 0.3774 0.35354 0.000 0.840 0.032 0.072 0.056
#> GSM228599 2 0.8679 -0.00874 0.028 0.344 0.232 0.296 0.100
#> GSM228602 3 0.7997 0.23055 0.116 0.092 0.560 0.088 0.144
#> GSM228614 2 0.8011 0.07162 0.020 0.440 0.104 0.320 0.116
#> GSM228626 2 0.6276 0.35444 0.000 0.660 0.128 0.088 0.124
#> GSM228640 3 0.5439 0.38421 0.024 0.024 0.728 0.060 0.164
#> GSM228643 3 0.7964 0.28154 0.032 0.116 0.516 0.112 0.224
#> GSM228650 3 0.8544 0.19914 0.016 0.216 0.412 0.160 0.196
#> GSM228653 3 0.5350 0.40240 0.012 0.036 0.704 0.032 0.216
#> GSM228657 2 0.5785 0.35260 0.000 0.692 0.096 0.156 0.056
#> GSM228605 1 0.9422 -0.04756 0.376 0.108 0.172 0.168 0.176
#> GSM228610 3 0.8494 0.24769 0.056 0.112 0.452 0.116 0.264
#> GSM228617 3 0.7935 0.26854 0.092 0.092 0.552 0.076 0.188
#> GSM228620 3 0.7645 0.28725 0.088 0.036 0.544 0.096 0.236
#> GSM228623 2 0.7845 0.13707 0.016 0.488 0.092 0.264 0.140
#> GSM228629 3 0.7238 0.29036 0.100 0.024 0.548 0.056 0.272
#> GSM228632 2 0.8701 -0.05732 0.008 0.292 0.288 0.168 0.244
#> GSM228635 2 0.6571 0.20001 0.000 0.544 0.044 0.316 0.096
#> GSM228647 3 0.7646 0.34352 0.044 0.084 0.536 0.080 0.256
#> GSM228596 4 0.9638 -0.15381 0.116 0.132 0.208 0.288 0.256
#> GSM228600 3 0.6391 0.36250 0.012 0.112 0.668 0.072 0.136
#> GSM228603 3 0.5170 0.38267 0.052 0.024 0.764 0.040 0.120
#> GSM228615 2 0.7638 -0.00203 0.044 0.412 0.036 0.400 0.108
#> GSM228627 3 0.8395 0.20730 0.036 0.152 0.420 0.092 0.300
#> GSM228641 3 0.6068 0.37395 0.012 0.044 0.672 0.080 0.192
#> GSM228644 2 0.5143 0.36901 0.000 0.752 0.104 0.064 0.080
#> GSM228651 3 0.6804 0.35827 0.024 0.064 0.584 0.056 0.272
#> GSM228654 3 0.7841 0.29466 0.020 0.164 0.508 0.084 0.224
#> GSM228658 3 0.6781 0.36793 0.032 0.052 0.596 0.060 0.260
#> GSM228606 2 0.8486 0.15197 0.008 0.396 0.224 0.176 0.196
#> GSM228611 3 0.7530 0.29026 0.052 0.036 0.512 0.104 0.296
#> GSM228618 3 0.7171 0.33249 0.040 0.084 0.612 0.084 0.180
#> GSM228621 3 0.7049 0.33699 0.000 0.164 0.556 0.068 0.212
#> GSM228624 3 0.8862 0.01953 0.016 0.220 0.304 0.180 0.280
#> GSM228630 2 0.8132 0.02883 0.000 0.360 0.332 0.168 0.140
#> GSM228636 2 0.5335 0.28203 0.000 0.680 0.028 0.240 0.052
#> GSM228638 3 0.8225 0.24892 0.024 0.204 0.476 0.112 0.184
#> GSM228648 3 0.7191 0.30587 0.000 0.228 0.536 0.072 0.164
#> GSM228670 4 0.8585 0.10084 0.088 0.344 0.092 0.388 0.088
#> GSM228671 2 0.8505 0.16419 0.004 0.368 0.180 0.228 0.220
#> GSM228672 1 0.8229 -0.03734 0.384 0.112 0.024 0.360 0.120
#> GSM228674 4 0.8948 0.20764 0.236 0.216 0.044 0.376 0.128
#> GSM228675 4 0.8530 0.13691 0.104 0.344 0.036 0.368 0.148
#> GSM228676 5 0.9866 0.03156 0.176 0.140 0.192 0.244 0.248
#> GSM228667 4 0.9521 -0.00559 0.124 0.244 0.096 0.288 0.248
#> GSM228668 1 0.7249 0.45084 0.620 0.056 0.068 0.132 0.124
#> GSM228669 1 0.8310 0.12869 0.452 0.148 0.024 0.252 0.124
#> GSM228673 5 0.9087 0.08171 0.084 0.092 0.260 0.192 0.372
#> GSM228677 2 0.7921 0.23830 0.000 0.456 0.160 0.236 0.148
#> GSM228678 2 0.7609 0.15068 0.020 0.496 0.080 0.300 0.104
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 5 0.898 0.28948 0.280 0.100 0.112 0.080 0.344 0.084
#> GSM228563 4 0.786 0.06149 0.068 0.116 0.012 0.400 0.332 0.072
#> GSM228565 5 0.943 0.26267 0.256 0.096 0.084 0.160 0.280 0.124
#> GSM228566 3 0.894 0.03779 0.024 0.268 0.276 0.080 0.216 0.136
#> GSM228567 1 0.172 0.50869 0.932 0.000 0.004 0.000 0.036 0.028
#> GSM228570 1 0.774 0.11963 0.468 0.024 0.060 0.068 0.276 0.104
#> GSM228571 1 0.816 0.13749 0.452 0.056 0.092 0.032 0.228 0.140
#> GSM228574 3 0.896 0.02010 0.012 0.212 0.320 0.152 0.140 0.164
#> GSM228575 2 0.933 0.02012 0.044 0.316 0.168 0.136 0.180 0.156
#> GSM228576 1 0.915 -0.24355 0.304 0.080 0.160 0.052 0.260 0.144
#> GSM228579 1 0.701 0.32498 0.552 0.032 0.044 0.016 0.176 0.180
#> GSM228580 2 0.814 0.05637 0.008 0.352 0.096 0.324 0.160 0.060
#> GSM228581 2 0.898 0.14255 0.028 0.356 0.132 0.204 0.136 0.144
#> GSM228666 2 0.853 0.09146 0.000 0.312 0.096 0.272 0.152 0.168
#> GSM228564 5 0.876 0.26855 0.252 0.076 0.072 0.244 0.316 0.040
#> GSM228568 6 0.944 -0.10701 0.220 0.108 0.084 0.096 0.208 0.284
#> GSM228569 1 0.724 0.25772 0.488 0.016 0.128 0.020 0.068 0.280
#> GSM228572 4 0.747 0.07506 0.000 0.324 0.076 0.416 0.140 0.044
#> GSM228573 3 0.732 0.20724 0.068 0.080 0.556 0.024 0.072 0.200
#> GSM228577 1 0.744 0.31736 0.524 0.028 0.056 0.048 0.116 0.228
#> GSM228578 1 0.833 0.18736 0.472 0.064 0.076 0.072 0.128 0.188
#> GSM228663 6 0.782 0.07329 0.068 0.132 0.296 0.028 0.040 0.436
#> GSM228664 6 0.820 0.05127 0.004 0.276 0.184 0.124 0.052 0.360
#> GSM228665 6 0.908 0.13879 0.128 0.160 0.228 0.068 0.068 0.348
#> GSM228582 1 0.939 -0.17367 0.300 0.164 0.076 0.084 0.152 0.224
#> GSM228583 1 0.154 0.50859 0.940 0.000 0.004 0.000 0.040 0.016
#> GSM228585 1 0.148 0.50941 0.944 0.000 0.008 0.000 0.036 0.012
#> GSM228587 1 0.498 0.45847 0.752 0.016 0.008 0.088 0.088 0.048
#> GSM228588 4 0.791 0.11465 0.208 0.148 0.004 0.424 0.184 0.032
#> GSM228589 4 0.733 0.15306 0.028 0.292 0.012 0.472 0.128 0.068
#> GSM228590 1 0.321 0.49938 0.864 0.004 0.012 0.016 0.056 0.048
#> GSM228591 2 0.862 0.09118 0.016 0.332 0.084 0.288 0.112 0.168
#> GSM228597 4 0.758 0.26295 0.044 0.144 0.036 0.516 0.208 0.052
#> GSM228601 4 0.605 0.21543 0.000 0.272 0.008 0.560 0.132 0.028
#> GSM228604 2 0.767 0.08661 0.000 0.416 0.304 0.124 0.052 0.104
#> GSM228608 1 0.608 0.38867 0.668 0.020 0.032 0.036 0.136 0.108
#> GSM228609 4 0.833 -0.15490 0.292 0.072 0.020 0.364 0.180 0.072
#> GSM228613 1 0.173 0.50581 0.924 0.000 0.000 0.004 0.064 0.008
#> GSM228616 1 0.977 -0.28886 0.264 0.168 0.120 0.152 0.180 0.116
#> GSM228628 2 0.866 0.11599 0.016 0.328 0.064 0.264 0.172 0.156
#> GSM228634 1 0.297 0.50803 0.860 0.000 0.016 0.000 0.040 0.084
#> GSM228642 2 0.728 0.17924 0.000 0.488 0.132 0.260 0.068 0.052
#> GSM228645 2 0.932 0.01239 0.040 0.284 0.204 0.124 0.212 0.136
#> GSM228646 3 0.932 0.06471 0.052 0.192 0.312 0.100 0.180 0.164
#> GSM228652 1 0.823 0.16474 0.488 0.056 0.056 0.116 0.164 0.120
#> GSM228655 1 0.907 -0.07031 0.376 0.084 0.080 0.140 0.112 0.208
#> GSM228656 1 0.202 0.51081 0.916 0.008 0.000 0.000 0.024 0.052
#> GSM228659 1 0.756 0.09173 0.456 0.032 0.016 0.180 0.260 0.056
#> GSM228662 1 0.213 0.50792 0.912 0.000 0.004 0.004 0.052 0.028
#> GSM228584 1 0.146 0.50891 0.940 0.000 0.000 0.000 0.016 0.044
#> GSM228586 1 0.262 0.50800 0.868 0.000 0.004 0.000 0.024 0.104
#> GSM228592 1 0.171 0.51013 0.928 0.000 0.000 0.000 0.028 0.044
#> GSM228593 1 0.902 -0.25781 0.296 0.120 0.048 0.216 0.256 0.064
#> GSM228594 1 0.536 0.45233 0.692 0.016 0.044 0.000 0.080 0.168
#> GSM228598 1 0.580 0.43290 0.648 0.024 0.000 0.028 0.156 0.144
#> GSM228607 6 0.946 0.08919 0.052 0.120 0.156 0.208 0.188 0.276
#> GSM228612 6 0.907 0.08075 0.064 0.248 0.232 0.072 0.088 0.296
#> GSM228619 1 0.938 -0.27959 0.256 0.064 0.256 0.204 0.128 0.092
#> GSM228622 1 0.807 0.17761 0.488 0.040 0.184 0.052 0.104 0.132
#> GSM228625 1 0.872 -0.07623 0.364 0.064 0.036 0.232 0.196 0.108
#> GSM228631 3 0.934 -0.06833 0.248 0.076 0.296 0.084 0.164 0.132
#> GSM228633 4 0.680 -0.01850 0.000 0.400 0.056 0.416 0.104 0.024
#> GSM228637 4 0.717 0.27637 0.060 0.084 0.036 0.588 0.168 0.064
#> GSM228639 2 0.910 0.04719 0.028 0.280 0.196 0.248 0.092 0.156
#> GSM228649 4 0.794 0.21698 0.048 0.108 0.020 0.460 0.240 0.124
#> GSM228660 1 0.884 0.02982 0.392 0.076 0.044 0.136 0.176 0.176
#> GSM228661 1 0.604 0.40622 0.644 0.032 0.108 0.004 0.036 0.176
#> GSM228595 4 0.547 0.13111 0.000 0.360 0.012 0.556 0.052 0.020
#> GSM228599 4 0.867 0.02894 0.024 0.124 0.224 0.388 0.148 0.092
#> GSM228602 3 0.779 0.22009 0.096 0.100 0.544 0.036 0.112 0.112
#> GSM228614 4 0.872 0.10932 0.028 0.168 0.092 0.400 0.180 0.132
#> GSM228626 2 0.633 0.17035 0.000 0.576 0.104 0.248 0.024 0.048
#> GSM228640 3 0.496 0.28702 0.028 0.052 0.736 0.012 0.016 0.156
#> GSM228643 3 0.839 0.17138 0.060 0.116 0.460 0.048 0.144 0.172
#> GSM228650 3 0.858 0.17002 0.032 0.264 0.388 0.120 0.088 0.108
#> GSM228653 3 0.695 0.22798 0.032 0.128 0.556 0.016 0.048 0.220
#> GSM228657 4 0.690 0.03716 0.000 0.368 0.064 0.448 0.044 0.076
#> GSM228605 1 0.943 -0.31835 0.300 0.108 0.144 0.104 0.244 0.100
#> GSM228610 3 0.827 0.05332 0.044 0.172 0.432 0.076 0.048 0.228
#> GSM228617 3 0.750 0.21694 0.064 0.044 0.544 0.064 0.076 0.208
#> GSM228620 6 0.845 -0.03390 0.052 0.172 0.312 0.084 0.036 0.344
#> GSM228623 4 0.801 0.15029 0.016 0.168 0.068 0.468 0.192 0.088
#> GSM228629 3 0.774 0.14423 0.080 0.100 0.480 0.016 0.072 0.252
#> GSM228632 2 0.848 0.09768 0.004 0.372 0.236 0.152 0.100 0.136
#> GSM228635 4 0.520 0.26768 0.004 0.128 0.012 0.724 0.064 0.068
#> GSM228647 3 0.760 0.22566 0.040 0.148 0.520 0.028 0.076 0.188
#> GSM228596 6 0.935 0.10507 0.052 0.160 0.160 0.108 0.220 0.300
#> GSM228600 3 0.722 0.28131 0.024 0.124 0.580 0.044 0.132 0.096
#> GSM228603 3 0.562 0.29897 0.056 0.056 0.716 0.008 0.100 0.064
#> GSM228615 4 0.721 0.24507 0.040 0.108 0.032 0.552 0.216 0.052
#> GSM228627 3 0.896 -0.01855 0.048 0.204 0.324 0.064 0.116 0.244
#> GSM228641 3 0.581 0.29223 0.004 0.100 0.680 0.020 0.072 0.124
#> GSM228644 2 0.658 -0.01107 0.000 0.436 0.056 0.412 0.044 0.052
#> GSM228651 3 0.766 0.15261 0.008 0.192 0.440 0.036 0.072 0.252
#> GSM228654 3 0.824 0.16713 0.016 0.300 0.364 0.076 0.064 0.180
#> GSM228658 3 0.778 0.17627 0.068 0.148 0.436 0.004 0.072 0.272
#> GSM228606 2 0.895 0.10703 0.008 0.288 0.180 0.236 0.128 0.160
#> GSM228611 3 0.789 0.08167 0.052 0.124 0.420 0.036 0.052 0.316
#> GSM228618 3 0.801 0.17893 0.020 0.124 0.480 0.056 0.144 0.176
#> GSM228621 3 0.767 0.19731 0.000 0.244 0.444 0.088 0.052 0.172
#> GSM228624 2 0.910 -0.01244 0.028 0.308 0.148 0.164 0.112 0.240
#> GSM228630 3 0.843 -0.09678 0.000 0.268 0.276 0.244 0.064 0.148
#> GSM228636 4 0.449 0.30033 0.000 0.100 0.008 0.772 0.072 0.048
#> GSM228638 3 0.873 0.04296 0.020 0.208 0.348 0.128 0.076 0.220
#> GSM228648 3 0.751 0.15155 0.000 0.312 0.404 0.092 0.028 0.164
#> GSM228670 4 0.878 0.12706 0.064 0.228 0.044 0.336 0.240 0.088
#> GSM228671 2 0.879 0.13728 0.016 0.344 0.128 0.244 0.156 0.112
#> GSM228672 1 0.856 -0.29013 0.320 0.056 0.044 0.220 0.296 0.064
#> GSM228674 5 0.894 0.15352 0.188 0.092 0.040 0.292 0.296 0.092
#> GSM228675 4 0.897 0.03893 0.092 0.248 0.048 0.308 0.228 0.076
#> GSM228676 5 0.996 0.07929 0.164 0.128 0.172 0.176 0.196 0.164
#> GSM228667 5 0.964 0.06824 0.100 0.200 0.128 0.196 0.268 0.108
#> GSM228668 1 0.722 0.29612 0.576 0.040 0.032 0.080 0.168 0.104
#> GSM228669 1 0.842 -0.14881 0.380 0.084 0.012 0.248 0.180 0.096
#> GSM228673 6 0.952 0.09914 0.056 0.196 0.188 0.160 0.128 0.272
#> GSM228677 4 0.798 0.00174 0.000 0.288 0.108 0.400 0.112 0.092
#> GSM228678 4 0.727 0.21421 0.008 0.188 0.044 0.528 0.168 0.064
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
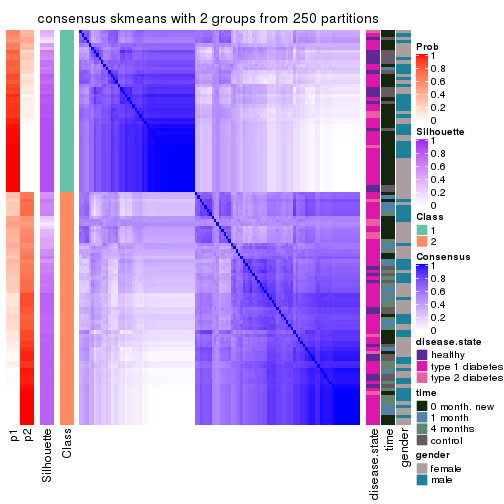
consensus_heatmap(res, k = 3)
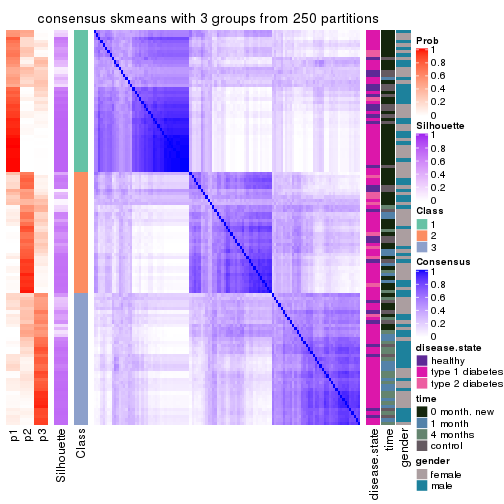
consensus_heatmap(res, k = 4)
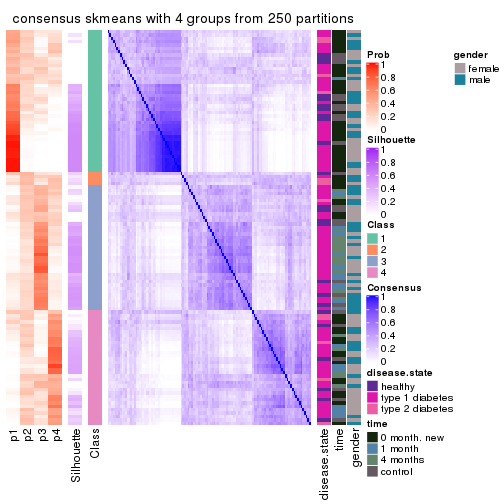
consensus_heatmap(res, k = 5)
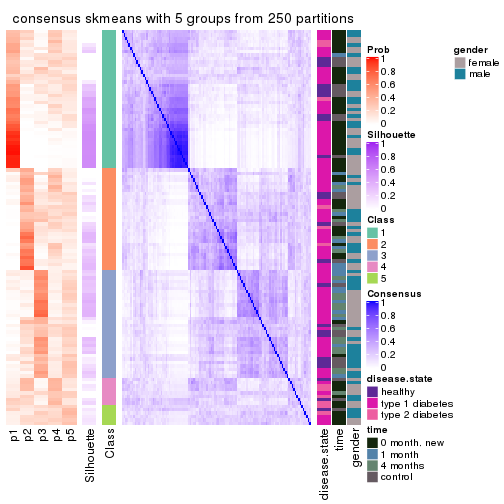
consensus_heatmap(res, k = 6)
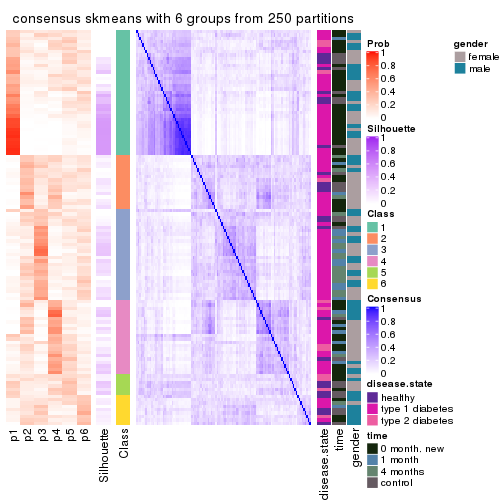
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
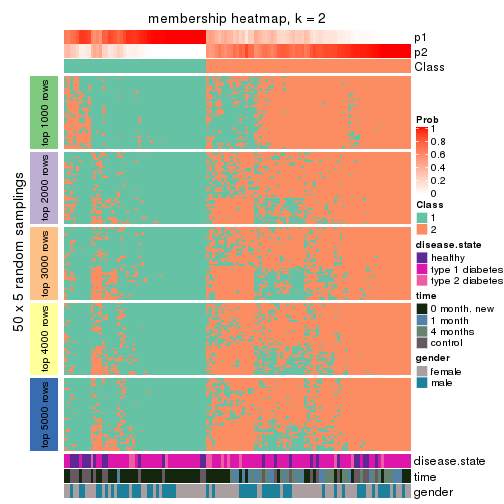
membership_heatmap(res, k = 3)
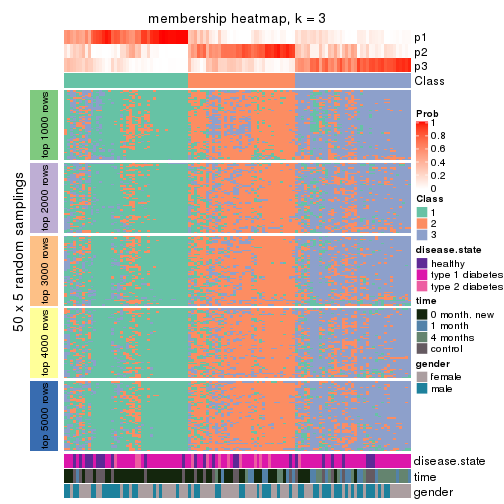
membership_heatmap(res, k = 4)
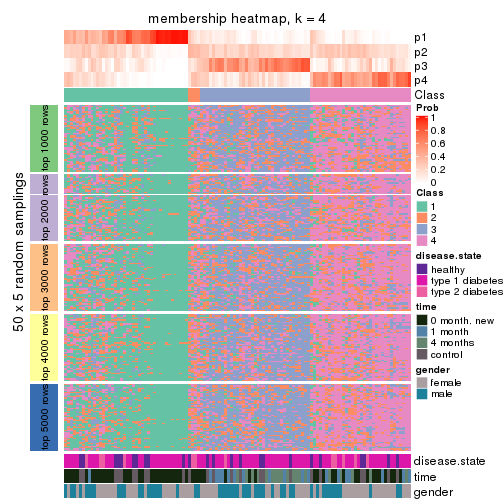
membership_heatmap(res, k = 5)
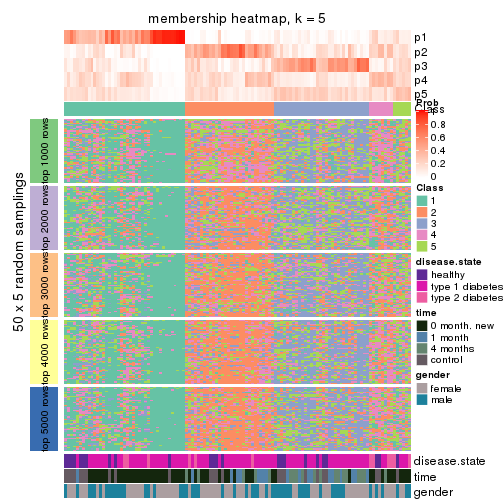
membership_heatmap(res, k = 6)
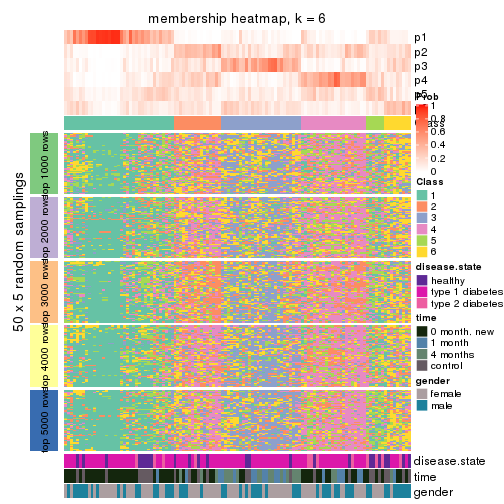
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
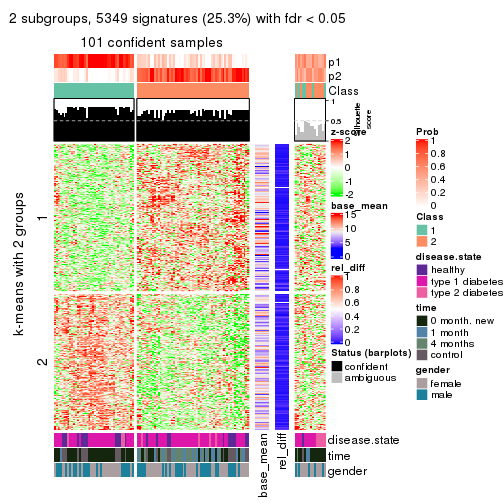
get_signatures(res, k = 3)
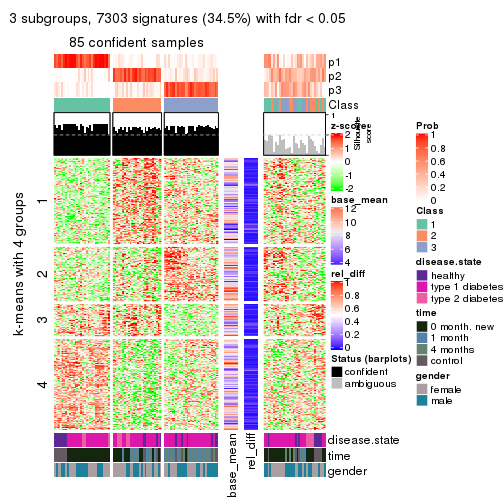
get_signatures(res, k = 4)
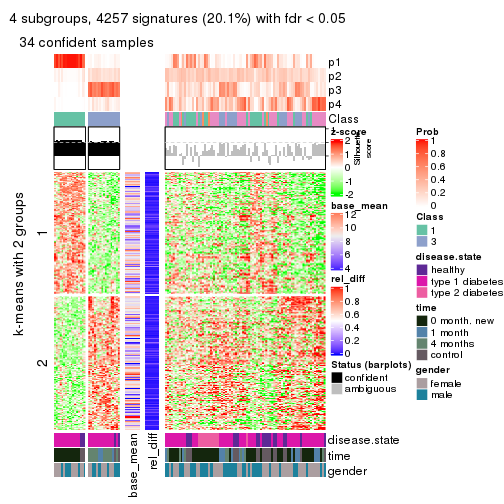
get_signatures(res, k = 5)

get_signatures(res, k = 6)

Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
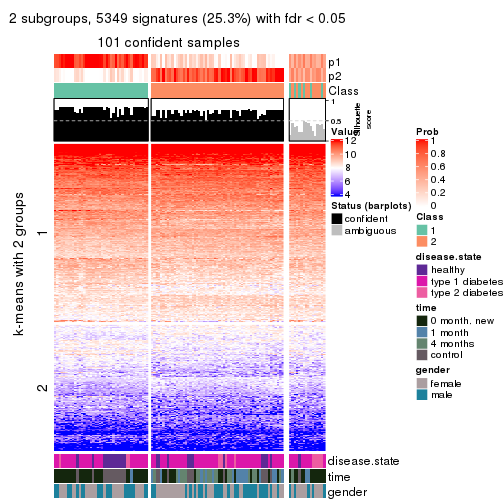
get_signatures(res, k = 3, scale_rows = FALSE)
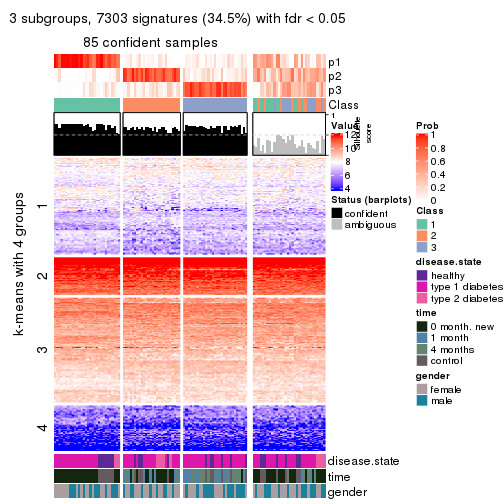
get_signatures(res, k = 4, scale_rows = FALSE)
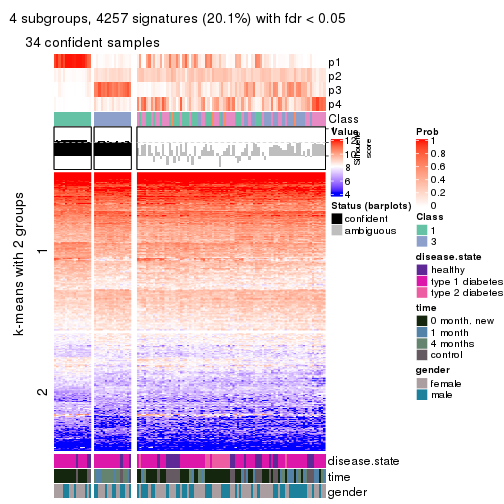
get_signatures(res, k = 5, scale_rows = FALSE)

get_signatures(res, k = 6, scale_rows = FALSE)

Compare the overlap of signatures from different k:
compare_signatures(res)
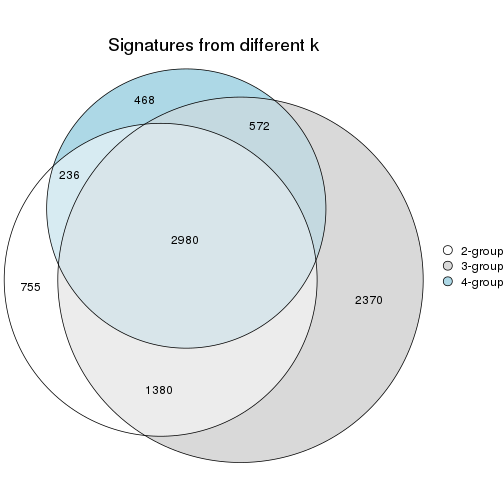
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
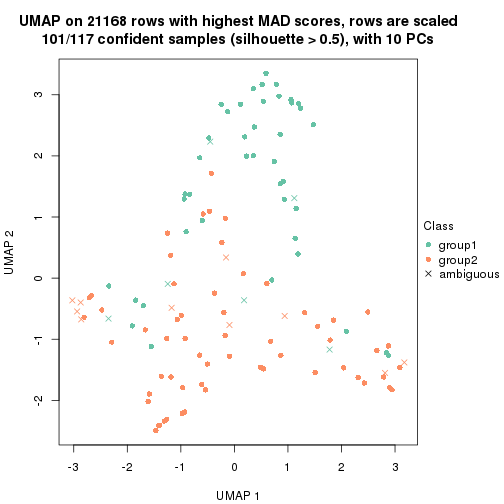
dimension_reduction(res, k = 3, method = "UMAP")
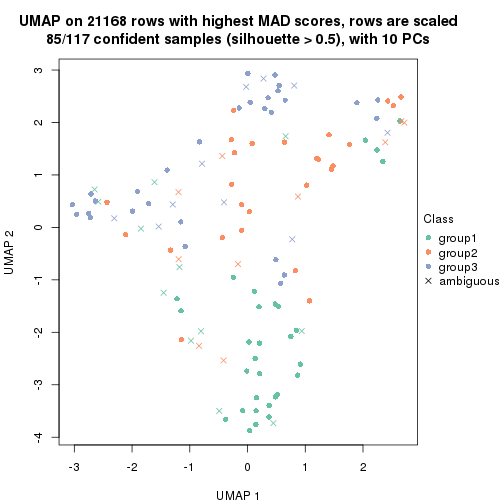
dimension_reduction(res, k = 4, method = "UMAP")
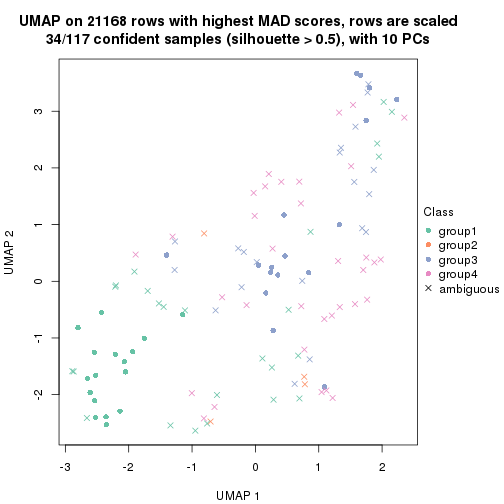
dimension_reduction(res, k = 5, method = "UMAP")
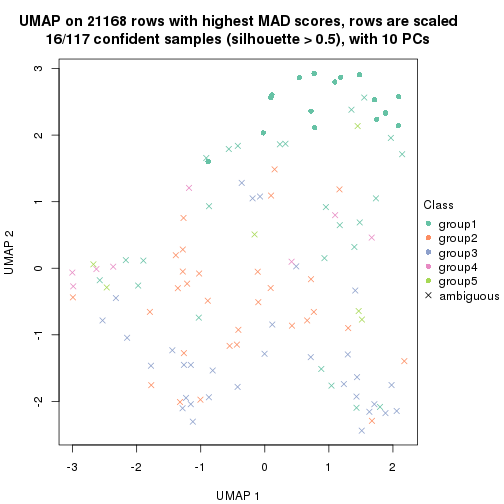
dimension_reduction(res, k = 6, method = "UMAP")
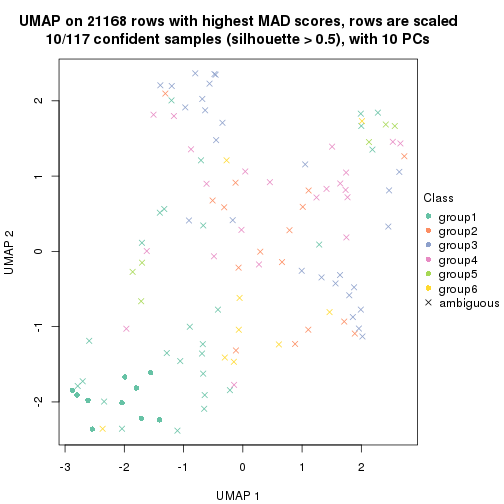
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
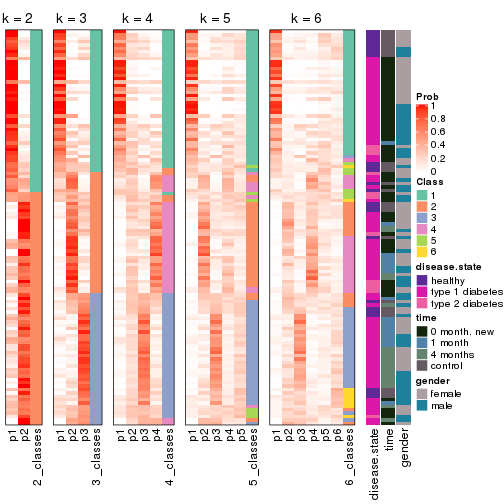
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> MAD:skmeans 101 0.488 1.46e-07 1.000 2
#> MAD:skmeans 85 0.176 7.37e-08 0.442 3
#> MAD:skmeans 34 1.000 2.03e-06 0.727 4
#> MAD:skmeans 16 NA NA NA 5
#> MAD:skmeans 10 NA NA NA 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["MAD", "pam"]
# you can also extract it by
# res = res_list["MAD:pam"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'MAD' method.
#> Subgroups are detected by 'pam' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
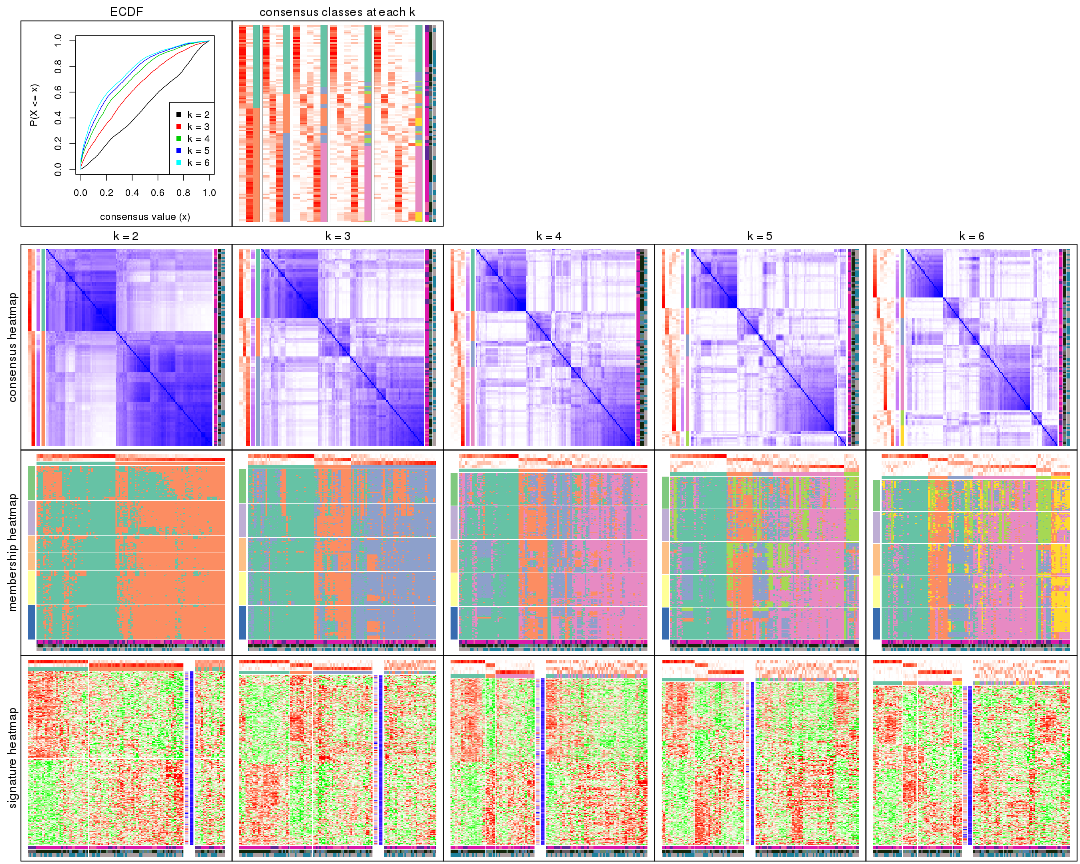
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
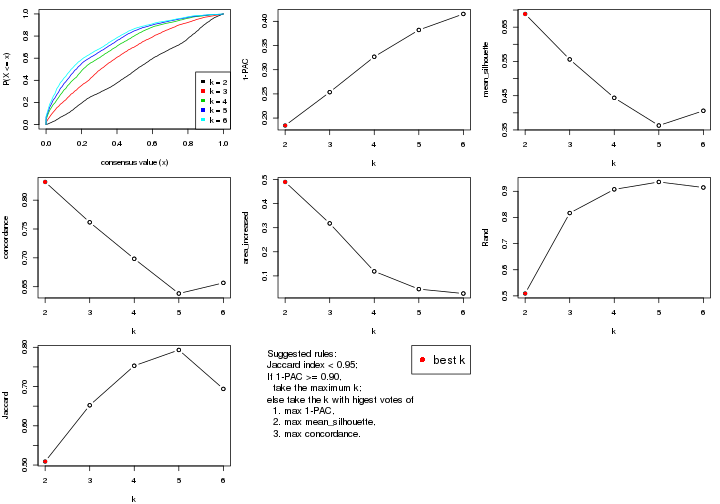
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.185 0.689 0.832 0.4899 0.509 0.509
#> 3 3 0.254 0.556 0.762 0.3178 0.817 0.652
#> 4 4 0.327 0.444 0.698 0.1187 0.907 0.753
#> 5 5 0.382 0.363 0.638 0.0450 0.936 0.793
#> 6 6 0.415 0.406 0.657 0.0268 0.915 0.694
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 2 0.2778 0.81614 0.048 0.952
#> GSM228563 2 0.8955 0.55897 0.312 0.688
#> GSM228565 1 0.9996 0.22928 0.512 0.488
#> GSM228566 2 0.0938 0.80137 0.012 0.988
#> GSM228567 1 0.0938 0.79380 0.988 0.012
#> GSM228570 2 0.9635 0.28847 0.388 0.612
#> GSM228571 1 1.0000 0.20047 0.504 0.496
#> GSM228574 2 0.0938 0.80276 0.012 0.988
#> GSM228575 2 0.9775 0.26493 0.412 0.588
#> GSM228576 1 0.8386 0.67476 0.732 0.268
#> GSM228579 1 0.6712 0.71340 0.824 0.176
#> GSM228580 1 0.9866 0.40701 0.568 0.432
#> GSM228581 2 0.6801 0.72387 0.180 0.820
#> GSM228666 2 0.2423 0.81401 0.040 0.960
#> GSM228564 1 0.5519 0.76983 0.872 0.128
#> GSM228568 1 0.7376 0.70526 0.792 0.208
#> GSM228569 1 0.1184 0.79345 0.984 0.016
#> GSM228572 2 0.3274 0.81658 0.060 0.940
#> GSM228573 1 0.9795 0.46964 0.584 0.416
#> GSM228577 1 0.0672 0.79304 0.992 0.008
#> GSM228578 1 0.9833 0.39207 0.576 0.424
#> GSM228663 2 0.3431 0.81867 0.064 0.936
#> GSM228664 2 0.4939 0.80854 0.108 0.892
#> GSM228665 2 0.9833 0.43295 0.424 0.576
#> GSM228582 2 0.6438 0.79897 0.164 0.836
#> GSM228583 1 0.0672 0.79457 0.992 0.008
#> GSM228585 1 0.5842 0.76295 0.860 0.140
#> GSM228587 1 0.5842 0.77184 0.860 0.140
#> GSM228588 1 0.3733 0.78715 0.928 0.072
#> GSM228589 1 0.8909 0.57330 0.692 0.308
#> GSM228590 1 0.1184 0.79518 0.984 0.016
#> GSM228591 2 0.6247 0.79556 0.156 0.844
#> GSM228597 1 0.9850 0.17266 0.572 0.428
#> GSM228601 2 0.7602 0.69970 0.220 0.780
#> GSM228604 2 0.9460 0.31978 0.364 0.636
#> GSM228608 1 0.3733 0.78715 0.928 0.072
#> GSM228609 1 0.7602 0.73461 0.780 0.220
#> GSM228613 1 0.1633 0.79616 0.976 0.024
#> GSM228616 1 0.3733 0.79853 0.928 0.072
#> GSM228628 2 0.2423 0.80903 0.040 0.960
#> GSM228634 1 0.2236 0.79675 0.964 0.036
#> GSM228642 2 0.1184 0.80002 0.016 0.984
#> GSM228645 2 0.3274 0.81859 0.060 0.940
#> GSM228646 2 0.6048 0.79109 0.148 0.852
#> GSM228652 2 0.6247 0.78727 0.156 0.844
#> GSM228655 2 0.7950 0.73481 0.240 0.760
#> GSM228656 1 0.5408 0.77374 0.876 0.124
#> GSM228659 2 0.7219 0.75246 0.200 0.800
#> GSM228662 1 0.0672 0.79451 0.992 0.008
#> GSM228584 1 0.0938 0.79398 0.988 0.012
#> GSM228586 1 0.0672 0.79304 0.992 0.008
#> GSM228592 1 0.0938 0.79398 0.988 0.012
#> GSM228593 2 0.9944 0.26838 0.456 0.544
#> GSM228594 1 0.0376 0.79450 0.996 0.004
#> GSM228598 1 0.5946 0.75386 0.856 0.144
#> GSM228607 2 0.9393 0.56108 0.356 0.644
#> GSM228612 2 0.9944 0.00943 0.456 0.544
#> GSM228619 1 0.1414 0.79584 0.980 0.020
#> GSM228622 1 0.7602 0.67667 0.780 0.220
#> GSM228625 1 0.9580 0.33195 0.620 0.380
#> GSM228631 1 0.2778 0.79811 0.952 0.048
#> GSM228633 2 0.0938 0.80567 0.012 0.988
#> GSM228637 2 0.7453 0.74682 0.212 0.788
#> GSM228639 2 0.8608 0.67943 0.284 0.716
#> GSM228649 2 0.6712 0.77722 0.176 0.824
#> GSM228660 1 0.5519 0.77833 0.872 0.128
#> GSM228661 1 0.0938 0.79484 0.988 0.012
#> GSM228595 2 0.0672 0.80375 0.008 0.992
#> GSM228599 2 0.2778 0.81804 0.048 0.952
#> GSM228602 2 0.6531 0.73550 0.168 0.832
#> GSM228614 2 0.4022 0.81582 0.080 0.920
#> GSM228626 2 0.0672 0.79987 0.008 0.992
#> GSM228640 1 0.8016 0.68784 0.756 0.244
#> GSM228643 2 0.0938 0.79828 0.012 0.988
#> GSM228650 2 0.2423 0.81612 0.040 0.960
#> GSM228653 2 0.3584 0.81266 0.068 0.932
#> GSM228657 2 0.2423 0.81632 0.040 0.960
#> GSM228605 1 0.1414 0.79749 0.980 0.020
#> GSM228610 1 0.3733 0.78780 0.928 0.072
#> GSM228617 1 0.9754 0.22211 0.592 0.408
#> GSM228620 1 0.6438 0.74525 0.836 0.164
#> GSM228623 2 0.3431 0.81966 0.064 0.936
#> GSM228629 2 0.7745 0.75463 0.228 0.772
#> GSM228632 2 0.4161 0.81579 0.084 0.916
#> GSM228635 2 0.9635 0.50239 0.388 0.612
#> GSM228647 1 0.6438 0.73316 0.836 0.164
#> GSM228596 2 0.7299 0.75150 0.204 0.796
#> GSM228600 2 0.6048 0.74800 0.148 0.852
#> GSM228603 2 0.5629 0.75955 0.132 0.868
#> GSM228615 2 0.9983 0.24625 0.476 0.524
#> GSM228627 2 0.4562 0.80177 0.096 0.904
#> GSM228641 1 0.9754 0.46399 0.592 0.408
#> GSM228644 2 0.0672 0.80375 0.008 0.992
#> GSM228651 2 0.9170 0.62460 0.332 0.668
#> GSM228654 2 0.5737 0.78662 0.136 0.864
#> GSM228658 2 0.4562 0.78183 0.096 0.904
#> GSM228606 2 0.5059 0.81574 0.112 0.888
#> GSM228611 1 0.9977 0.24710 0.528 0.472
#> GSM228618 1 0.6712 0.74773 0.824 0.176
#> GSM228621 2 0.2423 0.80186 0.040 0.960
#> GSM228624 2 0.9998 0.06416 0.492 0.508
#> GSM228630 2 0.7219 0.77048 0.200 0.800
#> GSM228636 2 0.7376 0.74919 0.208 0.792
#> GSM228638 2 0.7950 0.73116 0.240 0.760
#> GSM228648 2 0.2043 0.81218 0.032 0.968
#> GSM228670 2 0.6048 0.78628 0.148 0.852
#> GSM228671 2 0.4298 0.78431 0.088 0.912
#> GSM228672 1 0.9491 0.40626 0.632 0.368
#> GSM228674 2 0.6887 0.77186 0.184 0.816
#> GSM228675 2 0.4431 0.81616 0.092 0.908
#> GSM228676 2 0.3431 0.79799 0.064 0.936
#> GSM228667 2 0.8861 0.52969 0.304 0.696
#> GSM228668 1 0.5178 0.78092 0.884 0.116
#> GSM228669 1 0.5178 0.78603 0.884 0.116
#> GSM228673 2 0.5059 0.81735 0.112 0.888
#> GSM228677 2 0.5178 0.80518 0.116 0.884
#> GSM228678 2 0.2236 0.80366 0.036 0.964
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 3 0.6066 0.6014 0.024 0.248 0.728
#> GSM228563 3 0.9357 0.3630 0.248 0.236 0.516
#> GSM228565 1 0.9465 0.1395 0.472 0.196 0.332
#> GSM228566 3 0.3412 0.6861 0.000 0.124 0.876
#> GSM228567 1 0.0592 0.7592 0.988 0.012 0.000
#> GSM228570 3 0.9912 0.1041 0.320 0.284 0.396
#> GSM228571 2 0.9391 0.2352 0.368 0.456 0.176
#> GSM228574 3 0.6154 0.3240 0.000 0.408 0.592
#> GSM228575 3 0.9944 0.0695 0.296 0.320 0.384
#> GSM228576 1 0.7308 0.4800 0.656 0.284 0.060
#> GSM228579 1 0.5465 0.5146 0.712 0.288 0.000
#> GSM228580 1 0.9565 0.1614 0.476 0.228 0.296
#> GSM228581 3 0.8576 0.5073 0.152 0.252 0.596
#> GSM228666 3 0.3682 0.6879 0.008 0.116 0.876
#> GSM228564 1 0.3425 0.7377 0.884 0.004 0.112
#> GSM228568 1 0.6407 0.5107 0.700 0.272 0.028
#> GSM228569 1 0.1267 0.7581 0.972 0.024 0.004
#> GSM228572 3 0.5443 0.5670 0.004 0.260 0.736
#> GSM228573 2 0.8052 0.5884 0.196 0.652 0.152
#> GSM228577 1 0.0747 0.7585 0.984 0.016 0.000
#> GSM228578 1 0.9676 0.0833 0.460 0.288 0.252
#> GSM228663 3 0.5633 0.6524 0.024 0.208 0.768
#> GSM228664 3 0.6621 0.4941 0.032 0.284 0.684
#> GSM228665 3 0.8683 0.3513 0.340 0.120 0.540
#> GSM228582 3 0.3587 0.6984 0.088 0.020 0.892
#> GSM228583 1 0.0747 0.7581 0.984 0.016 0.000
#> GSM228585 1 0.5357 0.6947 0.820 0.116 0.064
#> GSM228587 1 0.5216 0.6384 0.740 0.000 0.260
#> GSM228588 1 0.1753 0.7558 0.952 0.000 0.048
#> GSM228589 1 0.6235 0.3881 0.564 0.000 0.436
#> GSM228590 1 0.0000 0.7579 1.000 0.000 0.000
#> GSM228591 3 0.4097 0.7011 0.060 0.060 0.880
#> GSM228597 1 0.8022 0.1692 0.544 0.068 0.388
#> GSM228601 3 0.5798 0.6031 0.184 0.040 0.776
#> GSM228604 2 0.0848 0.6328 0.008 0.984 0.008
#> GSM228608 1 0.1753 0.7558 0.952 0.000 0.048
#> GSM228609 1 0.6332 0.6856 0.768 0.088 0.144
#> GSM228613 1 0.0892 0.7620 0.980 0.000 0.020
#> GSM228616 1 0.3610 0.7500 0.888 0.016 0.096
#> GSM228628 3 0.5958 0.5656 0.008 0.300 0.692
#> GSM228634 1 0.3771 0.7379 0.876 0.012 0.112
#> GSM228642 2 0.2356 0.6302 0.000 0.928 0.072
#> GSM228645 3 0.4744 0.6848 0.028 0.136 0.836
#> GSM228646 3 0.1832 0.6945 0.008 0.036 0.956
#> GSM228652 3 0.0237 0.6839 0.004 0.000 0.996
#> GSM228655 3 0.3267 0.6884 0.116 0.000 0.884
#> GSM228656 1 0.5171 0.6782 0.784 0.012 0.204
#> GSM228659 3 0.0892 0.6882 0.020 0.000 0.980
#> GSM228662 1 0.0237 0.7579 0.996 0.004 0.000
#> GSM228584 1 0.0237 0.7580 0.996 0.004 0.000
#> GSM228586 1 0.0592 0.7572 0.988 0.012 0.000
#> GSM228592 1 0.0000 0.7579 1.000 0.000 0.000
#> GSM228593 3 0.6881 0.3386 0.388 0.020 0.592
#> GSM228594 1 0.0000 0.7579 1.000 0.000 0.000
#> GSM228598 1 0.4164 0.7157 0.848 0.008 0.144
#> GSM228607 3 0.6839 0.5302 0.272 0.044 0.684
#> GSM228612 2 0.9468 0.4601 0.276 0.496 0.228
#> GSM228619 1 0.1647 0.7522 0.960 0.036 0.004
#> GSM228622 1 0.5953 0.5772 0.708 0.012 0.280
#> GSM228625 1 0.6168 0.2945 0.588 0.000 0.412
#> GSM228631 1 0.4953 0.6298 0.808 0.176 0.016
#> GSM228633 2 0.5138 0.5260 0.000 0.748 0.252
#> GSM228637 3 0.3610 0.6889 0.096 0.016 0.888
#> GSM228639 3 0.8896 0.3910 0.180 0.252 0.568
#> GSM228649 3 0.2804 0.6986 0.060 0.016 0.924
#> GSM228660 1 0.5178 0.6562 0.744 0.000 0.256
#> GSM228661 1 0.0592 0.7572 0.988 0.012 0.000
#> GSM228595 3 0.3879 0.6494 0.000 0.152 0.848
#> GSM228599 3 0.4531 0.6597 0.008 0.168 0.824
#> GSM228602 2 0.4446 0.6356 0.032 0.856 0.112
#> GSM228614 3 0.1964 0.6837 0.000 0.056 0.944
#> GSM228626 2 0.1529 0.6336 0.000 0.960 0.040
#> GSM228640 2 0.5737 0.5181 0.256 0.732 0.012
#> GSM228643 3 0.6260 0.3375 0.000 0.448 0.552
#> GSM228650 3 0.1989 0.6918 0.004 0.048 0.948
#> GSM228653 3 0.6905 0.0907 0.016 0.440 0.544
#> GSM228657 3 0.5560 0.5273 0.000 0.300 0.700
#> GSM228605 1 0.2496 0.7397 0.928 0.068 0.004
#> GSM228610 2 0.6931 0.2031 0.456 0.528 0.016
#> GSM228617 2 0.8703 0.5017 0.284 0.572 0.144
#> GSM228620 1 0.3941 0.7158 0.844 0.000 0.156
#> GSM228623 3 0.4811 0.6715 0.024 0.148 0.828
#> GSM228629 3 0.7572 0.5613 0.128 0.184 0.688
#> GSM228632 3 0.7230 0.5387 0.040 0.344 0.616
#> GSM228635 3 0.6008 0.4383 0.372 0.000 0.628
#> GSM228647 1 0.8201 0.0405 0.524 0.400 0.076
#> GSM228596 3 0.1989 0.6955 0.048 0.004 0.948
#> GSM228600 2 0.7034 0.5193 0.048 0.668 0.284
#> GSM228603 2 0.6337 0.5413 0.044 0.736 0.220
#> GSM228615 3 0.6608 0.2539 0.432 0.008 0.560
#> GSM228627 3 0.6723 0.6052 0.048 0.248 0.704
#> GSM228641 2 0.2176 0.6387 0.020 0.948 0.032
#> GSM228644 3 0.5178 0.5339 0.000 0.256 0.744
#> GSM228651 2 0.9215 0.3955 0.168 0.500 0.332
#> GSM228654 2 0.6702 0.3620 0.024 0.648 0.328
#> GSM228658 2 0.7517 0.1716 0.048 0.588 0.364
#> GSM228606 3 0.3607 0.6572 0.008 0.112 0.880
#> GSM228611 2 0.7309 0.6203 0.168 0.708 0.124
#> GSM228618 2 0.7422 0.4510 0.344 0.608 0.048
#> GSM228621 2 0.2066 0.6360 0.000 0.940 0.060
#> GSM228624 2 0.9980 0.1776 0.324 0.364 0.312
#> GSM228630 2 0.8700 0.3265 0.128 0.552 0.320
#> GSM228636 3 0.5384 0.5999 0.024 0.188 0.788
#> GSM228638 3 0.8318 0.4467 0.128 0.260 0.612
#> GSM228648 2 0.4291 0.5825 0.000 0.820 0.180
#> GSM228670 3 0.0892 0.6888 0.020 0.000 0.980
#> GSM228671 3 0.7744 0.3595 0.048 0.448 0.504
#> GSM228672 1 0.6843 0.4274 0.640 0.028 0.332
#> GSM228674 3 0.1989 0.6949 0.048 0.004 0.948
#> GSM228675 3 0.2564 0.6986 0.028 0.036 0.936
#> GSM228676 3 0.6414 0.5962 0.036 0.248 0.716
#> GSM228667 3 0.9355 0.3491 0.232 0.252 0.516
#> GSM228668 1 0.3619 0.7429 0.864 0.000 0.136
#> GSM228669 1 0.3193 0.7511 0.896 0.004 0.100
#> GSM228673 3 0.4565 0.6992 0.064 0.076 0.860
#> GSM228677 3 0.3083 0.6961 0.024 0.060 0.916
#> GSM228678 3 0.5785 0.5368 0.000 0.332 0.668
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.5844 0.4163 0.020 0.016 0.348 0.616
#> GSM228563 4 0.7486 0.1591 0.188 0.000 0.348 0.464
#> GSM228565 1 0.8428 -0.2123 0.380 0.020 0.300 0.300
#> GSM228566 4 0.3694 0.6349 0.000 0.124 0.032 0.844
#> GSM228567 1 0.2081 0.6641 0.916 0.000 0.084 0.000
#> GSM228570 3 0.6289 0.4764 0.116 0.000 0.648 0.236
#> GSM228571 3 0.5578 0.4943 0.128 0.052 0.768 0.052
#> GSM228574 4 0.7159 0.3045 0.000 0.260 0.188 0.552
#> GSM228575 3 0.7836 0.2211 0.184 0.012 0.468 0.336
#> GSM228576 1 0.5372 -0.0576 0.544 0.000 0.444 0.012
#> GSM228579 3 0.4877 0.2215 0.408 0.000 0.592 0.000
#> GSM228580 1 0.8994 0.0487 0.436 0.284 0.084 0.196
#> GSM228581 4 0.7295 0.4325 0.132 0.020 0.264 0.584
#> GSM228666 4 0.3558 0.6520 0.008 0.048 0.072 0.872
#> GSM228564 1 0.1792 0.6564 0.932 0.000 0.000 0.068
#> GSM228568 3 0.5701 0.0839 0.476 0.012 0.504 0.008
#> GSM228569 1 0.5194 0.4795 0.652 0.012 0.332 0.004
#> GSM228572 4 0.7028 0.4190 0.000 0.280 0.160 0.560
#> GSM228573 2 0.8056 0.4526 0.136 0.592 0.168 0.104
#> GSM228577 1 0.3448 0.6289 0.828 0.004 0.168 0.000
#> GSM228578 3 0.6294 0.5253 0.168 0.016 0.696 0.120
#> GSM228663 4 0.6333 0.5339 0.004 0.272 0.088 0.636
#> GSM228664 4 0.7392 0.2575 0.004 0.388 0.144 0.464
#> GSM228665 4 0.8508 0.3338 0.240 0.232 0.048 0.480
#> GSM228582 4 0.4351 0.6447 0.060 0.044 0.052 0.844
#> GSM228583 1 0.4585 0.4834 0.668 0.000 0.332 0.000
#> GSM228585 1 0.5454 0.5637 0.752 0.016 0.168 0.064
#> GSM228587 1 0.4134 0.5207 0.740 0.000 0.000 0.260
#> GSM228588 1 0.0000 0.6657 1.000 0.000 0.000 0.000
#> GSM228589 1 0.5201 0.3266 0.592 0.004 0.004 0.400
#> GSM228590 1 0.0000 0.6657 1.000 0.000 0.000 0.000
#> GSM228591 4 0.4941 0.6340 0.052 0.040 0.100 0.808
#> GSM228597 1 0.7512 0.1241 0.528 0.040 0.084 0.348
#> GSM228601 4 0.4508 0.5449 0.184 0.000 0.036 0.780
#> GSM228604 2 0.4585 0.4431 0.000 0.668 0.332 0.000
#> GSM228608 1 0.0000 0.6657 1.000 0.000 0.000 0.000
#> GSM228609 1 0.4836 0.5934 0.816 0.036 0.080 0.068
#> GSM228613 1 0.0336 0.6667 0.992 0.000 0.000 0.008
#> GSM228616 1 0.6830 0.4674 0.600 0.016 0.296 0.088
#> GSM228628 4 0.5999 0.3371 0.000 0.044 0.404 0.552
#> GSM228634 1 0.7141 0.4029 0.544 0.008 0.328 0.120
#> GSM228642 3 0.6214 -0.2644 0.000 0.472 0.476 0.052
#> GSM228645 4 0.4745 0.6260 0.024 0.068 0.092 0.816
#> GSM228646 4 0.2452 0.6608 0.004 0.084 0.004 0.908
#> GSM228652 4 0.0188 0.6488 0.004 0.000 0.000 0.996
#> GSM228655 4 0.4149 0.6200 0.088 0.004 0.072 0.836
#> GSM228656 1 0.7855 0.2644 0.456 0.008 0.328 0.208
#> GSM228659 4 0.0188 0.6488 0.004 0.000 0.000 0.996
#> GSM228662 1 0.2773 0.6507 0.880 0.004 0.116 0.000
#> GSM228584 1 0.1940 0.6639 0.924 0.000 0.076 0.000
#> GSM228586 1 0.4999 0.4789 0.660 0.012 0.328 0.000
#> GSM228592 1 0.0000 0.6657 1.000 0.000 0.000 0.000
#> GSM228593 4 0.5773 0.3080 0.376 0.004 0.028 0.592
#> GSM228594 1 0.0336 0.6661 0.992 0.008 0.000 0.000
#> GSM228598 1 0.5945 0.5793 0.732 0.020 0.120 0.128
#> GSM228607 4 0.8143 0.0911 0.176 0.040 0.268 0.516
#> GSM228612 3 0.8570 0.2388 0.100 0.316 0.476 0.108
#> GSM228619 1 0.0000 0.6657 1.000 0.000 0.000 0.000
#> GSM228622 1 0.7484 0.3277 0.508 0.004 0.308 0.180
#> GSM228625 1 0.6427 0.2845 0.584 0.036 0.024 0.356
#> GSM228631 1 0.7669 0.1554 0.452 0.236 0.312 0.000
#> GSM228633 2 0.4203 0.5485 0.000 0.824 0.108 0.068
#> GSM228637 4 0.4209 0.6498 0.084 0.064 0.012 0.840
#> GSM228639 4 0.9141 0.2212 0.120 0.320 0.148 0.412
#> GSM228649 4 0.2716 0.6627 0.028 0.052 0.008 0.912
#> GSM228660 1 0.4735 0.5805 0.784 0.068 0.000 0.148
#> GSM228661 1 0.4673 0.5180 0.700 0.008 0.292 0.000
#> GSM228595 4 0.5000 0.6088 0.000 0.128 0.100 0.772
#> GSM228599 4 0.4728 0.6153 0.004 0.104 0.092 0.800
#> GSM228602 2 0.6355 0.5011 0.020 0.656 0.260 0.064
#> GSM228614 4 0.2385 0.6583 0.000 0.052 0.028 0.920
#> GSM228626 2 0.4088 0.5485 0.000 0.764 0.232 0.004
#> GSM228640 2 0.6540 0.4543 0.144 0.648 0.204 0.004
#> GSM228643 4 0.7393 0.2509 0.000 0.180 0.332 0.488
#> GSM228650 4 0.1022 0.6549 0.000 0.032 0.000 0.968
#> GSM228653 2 0.6123 0.1761 0.000 0.572 0.056 0.372
#> GSM228657 4 0.7250 0.3568 0.000 0.316 0.168 0.516
#> GSM228605 1 0.1211 0.6618 0.960 0.040 0.000 0.000
#> GSM228610 2 0.5932 0.3977 0.224 0.680 0.096 0.000
#> GSM228617 2 0.8851 -0.0879 0.180 0.380 0.372 0.068
#> GSM228620 1 0.3945 0.6205 0.828 0.004 0.024 0.144
#> GSM228623 4 0.4466 0.6320 0.016 0.036 0.132 0.816
#> GSM228629 3 0.7473 0.3614 0.040 0.084 0.540 0.336
#> GSM228632 4 0.7761 0.3305 0.036 0.104 0.384 0.476
#> GSM228635 4 0.5602 0.3841 0.368 0.012 0.012 0.608
#> GSM228647 3 0.8423 -0.0353 0.372 0.176 0.412 0.040
#> GSM228596 4 0.1004 0.6553 0.024 0.004 0.000 0.972
#> GSM228600 2 0.6788 0.4933 0.016 0.652 0.172 0.160
#> GSM228603 2 0.6766 0.4826 0.024 0.648 0.228 0.100
#> GSM228615 4 0.5770 0.2246 0.432 0.012 0.012 0.544
#> GSM228627 4 0.6360 0.1985 0.024 0.024 0.432 0.520
#> GSM228641 2 0.3380 0.5760 0.004 0.852 0.136 0.008
#> GSM228644 4 0.6936 0.3893 0.000 0.292 0.144 0.564
#> GSM228651 2 0.5954 0.4864 0.028 0.724 0.068 0.180
#> GSM228654 2 0.6688 0.4135 0.004 0.636 0.184 0.176
#> GSM228658 3 0.6992 0.2657 0.000 0.248 0.576 0.176
#> GSM228606 4 0.4604 0.6209 0.004 0.168 0.040 0.788
#> GSM228611 3 0.6644 0.1684 0.044 0.376 0.556 0.024
#> GSM228618 2 0.6567 0.4241 0.176 0.672 0.136 0.016
#> GSM228621 2 0.4562 0.5542 0.000 0.764 0.208 0.028
#> GSM228624 3 0.8972 0.4261 0.144 0.188 0.496 0.172
#> GSM228630 2 0.8600 0.2759 0.076 0.492 0.160 0.272
#> GSM228636 4 0.6903 0.4833 0.012 0.272 0.112 0.604
#> GSM228638 4 0.8608 0.2631 0.072 0.348 0.140 0.440
#> GSM228648 2 0.3271 0.5394 0.000 0.856 0.132 0.012
#> GSM228670 4 0.0376 0.6490 0.004 0.000 0.004 0.992
#> GSM228671 3 0.4336 0.4270 0.000 0.060 0.812 0.128
#> GSM228672 1 0.5596 0.3300 0.632 0.000 0.036 0.332
#> GSM228674 4 0.1733 0.6559 0.024 0.000 0.028 0.948
#> GSM228675 4 0.1484 0.6568 0.016 0.004 0.020 0.960
#> GSM228676 4 0.5821 0.2173 0.004 0.024 0.432 0.540
#> GSM228667 3 0.5848 0.4951 0.032 0.032 0.700 0.236
#> GSM228668 1 0.2796 0.6577 0.892 0.004 0.008 0.096
#> GSM228669 1 0.2670 0.6577 0.904 0.000 0.024 0.072
#> GSM228673 4 0.5532 0.5307 0.040 0.020 0.212 0.728
#> GSM228677 4 0.2594 0.6607 0.004 0.036 0.044 0.916
#> GSM228678 4 0.5512 0.2163 0.000 0.016 0.492 0.492
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 4 0.5248 0.32654 0.012 0.036 0.000 0.604 0.348
#> GSM228563 4 0.6408 0.00697 0.172 0.000 0.000 0.440 0.388
#> GSM228565 1 0.8434 -0.21245 0.364 0.016 0.096 0.292 0.232
#> GSM228566 4 0.3304 0.59812 0.000 0.128 0.004 0.840 0.028
#> GSM228567 1 0.2077 0.65678 0.908 0.000 0.084 0.000 0.008
#> GSM228570 5 0.7631 0.34637 0.088 0.000 0.220 0.212 0.480
#> GSM228571 5 0.7545 0.25029 0.108 0.036 0.328 0.044 0.484
#> GSM228574 4 0.6314 0.23358 0.000 0.304 0.000 0.512 0.184
#> GSM228575 5 0.8488 0.25395 0.172 0.028 0.100 0.324 0.376
#> GSM228576 1 0.4905 -0.05451 0.516 0.000 0.008 0.012 0.464
#> GSM228579 5 0.6266 0.19123 0.376 0.000 0.152 0.000 0.472
#> GSM228580 5 0.9586 -0.19046 0.196 0.224 0.108 0.148 0.324
#> GSM228581 4 0.6840 0.35432 0.128 0.016 0.028 0.568 0.260
#> GSM228666 4 0.3654 0.60547 0.008 0.092 0.004 0.840 0.056
#> GSM228564 1 0.1544 0.65342 0.932 0.000 0.000 0.068 0.000
#> GSM228568 1 0.5927 -0.08624 0.468 0.000 0.104 0.000 0.428
#> GSM228569 1 0.4582 0.35041 0.572 0.000 0.416 0.000 0.012
#> GSM228572 4 0.7932 0.18259 0.000 0.116 0.316 0.404 0.164
#> GSM228573 2 0.5725 0.56125 0.080 0.708 0.000 0.096 0.116
#> GSM228577 1 0.3921 0.59033 0.784 0.000 0.172 0.000 0.044
#> GSM228578 5 0.7684 0.25442 0.148 0.004 0.316 0.084 0.448
#> GSM228663 4 0.6700 0.48564 0.000 0.208 0.132 0.596 0.064
#> GSM228664 4 0.8625 -0.06401 0.004 0.256 0.272 0.296 0.172
#> GSM228665 4 0.8769 0.25208 0.152 0.096 0.252 0.428 0.072
#> GSM228582 4 0.4083 0.59514 0.040 0.004 0.164 0.788 0.004
#> GSM228583 1 0.4276 0.37634 0.616 0.000 0.380 0.000 0.004
#> GSM228585 1 0.5202 0.59373 0.748 0.000 0.100 0.064 0.088
#> GSM228587 1 0.3809 0.52580 0.736 0.000 0.008 0.256 0.000
#> GSM228588 1 0.0162 0.66255 0.996 0.000 0.004 0.000 0.000
#> GSM228589 1 0.5080 0.34154 0.588 0.000 0.044 0.368 0.000
#> GSM228590 1 0.0000 0.66224 1.000 0.000 0.000 0.000 0.000
#> GSM228591 4 0.5115 0.57241 0.028 0.004 0.124 0.748 0.096
#> GSM228597 1 0.7812 0.03344 0.448 0.016 0.108 0.332 0.096
#> GSM228601 4 0.4210 0.50683 0.184 0.000 0.016 0.772 0.028
#> GSM228604 2 0.4639 0.46301 0.000 0.632 0.024 0.000 0.344
#> GSM228608 1 0.0162 0.66279 0.996 0.000 0.004 0.000 0.000
#> GSM228609 1 0.4262 0.59500 0.796 0.004 0.140 0.020 0.040
#> GSM228613 1 0.0290 0.66364 0.992 0.000 0.000 0.008 0.000
#> GSM228616 1 0.6016 0.34268 0.540 0.008 0.376 0.064 0.012
#> GSM228628 4 0.5328 0.14923 0.000 0.016 0.024 0.516 0.444
#> GSM228634 1 0.5985 0.24736 0.480 0.000 0.408 0.112 0.000
#> GSM228642 5 0.6024 -0.18815 0.000 0.384 0.044 0.040 0.532
#> GSM228645 4 0.4698 0.57711 0.024 0.064 0.024 0.796 0.092
#> GSM228646 4 0.3087 0.61122 0.004 0.128 0.012 0.852 0.004
#> GSM228652 4 0.0000 0.60772 0.000 0.000 0.000 1.000 0.000
#> GSM228655 4 0.3859 0.58684 0.072 0.008 0.100 0.820 0.000
#> GSM228656 3 0.6532 -0.13735 0.384 0.000 0.420 0.196 0.000
#> GSM228659 4 0.0000 0.60772 0.000 0.000 0.000 1.000 0.000
#> GSM228662 1 0.2690 0.63375 0.844 0.000 0.156 0.000 0.000
#> GSM228584 1 0.1732 0.65655 0.920 0.000 0.080 0.000 0.000
#> GSM228586 1 0.4227 0.35083 0.580 0.000 0.420 0.000 0.000
#> GSM228592 1 0.0000 0.66224 1.000 0.000 0.000 0.000 0.000
#> GSM228593 4 0.5198 0.28042 0.364 0.000 0.008 0.592 0.036
#> GSM228594 1 0.0162 0.66281 0.996 0.000 0.004 0.000 0.000
#> GSM228598 1 0.5032 0.54971 0.704 0.000 0.168 0.128 0.000
#> GSM228607 4 0.6687 0.07425 0.128 0.004 0.396 0.456 0.016
#> GSM228612 3 0.8540 0.12821 0.080 0.320 0.412 0.080 0.108
#> GSM228619 1 0.0404 0.66293 0.988 0.000 0.012 0.000 0.000
#> GSM228622 1 0.6420 0.16531 0.448 0.000 0.376 0.176 0.000
#> GSM228625 1 0.6427 0.18368 0.492 0.004 0.164 0.340 0.000
#> GSM228631 3 0.6466 0.00381 0.380 0.144 0.468 0.000 0.008
#> GSM228633 2 0.6473 0.38219 0.000 0.520 0.344 0.024 0.112
#> GSM228637 4 0.4360 0.61357 0.064 0.044 0.040 0.824 0.028
#> GSM228639 3 0.9450 0.05390 0.080 0.168 0.332 0.232 0.188
#> GSM228649 4 0.2606 0.62263 0.024 0.044 0.016 0.908 0.008
#> GSM228660 1 0.4744 0.56949 0.764 0.012 0.128 0.092 0.004
#> GSM228661 1 0.3966 0.43803 0.664 0.000 0.336 0.000 0.000
#> GSM228595 4 0.5746 0.50607 0.000 0.060 0.156 0.696 0.088
#> GSM228599 4 0.4445 0.57611 0.004 0.108 0.016 0.792 0.080
#> GSM228602 2 0.6263 0.53530 0.020 0.644 0.052 0.052 0.232
#> GSM228614 4 0.2937 0.61624 0.000 0.016 0.040 0.884 0.060
#> GSM228626 2 0.6510 0.35567 0.000 0.512 0.272 0.004 0.212
#> GSM228640 2 0.3798 0.59270 0.060 0.816 0.004 0.000 0.120
#> GSM228643 4 0.6778 0.14781 0.000 0.216 0.008 0.460 0.316
#> GSM228650 4 0.1197 0.61826 0.000 0.048 0.000 0.952 0.000
#> GSM228653 2 0.6209 0.18847 0.000 0.548 0.096 0.336 0.020
#> GSM228657 4 0.8158 0.05078 0.000 0.136 0.324 0.360 0.180
#> GSM228605 1 0.1270 0.65534 0.948 0.052 0.000 0.000 0.000
#> GSM228610 2 0.6972 0.27254 0.192 0.540 0.224 0.000 0.044
#> GSM228617 3 0.7271 0.24371 0.140 0.296 0.508 0.044 0.012
#> GSM228620 1 0.3523 0.61168 0.824 0.004 0.032 0.140 0.000
#> GSM228623 4 0.4309 0.59013 0.012 0.008 0.060 0.800 0.120
#> GSM228629 3 0.7449 -0.08448 0.020 0.032 0.504 0.248 0.196
#> GSM228632 4 0.7636 0.15842 0.036 0.044 0.108 0.420 0.392
#> GSM228635 4 0.5844 0.36436 0.340 0.004 0.044 0.584 0.028
#> GSM228647 3 0.7287 0.25558 0.304 0.088 0.524 0.024 0.060
#> GSM228596 4 0.0865 0.61448 0.024 0.004 0.000 0.972 0.000
#> GSM228600 2 0.4018 0.59892 0.000 0.804 0.004 0.088 0.104
#> GSM228603 2 0.3928 0.59567 0.008 0.800 0.000 0.040 0.152
#> GSM228615 4 0.6022 0.23530 0.408 0.012 0.028 0.520 0.032
#> GSM228627 4 0.6949 0.05546 0.016 0.028 0.104 0.492 0.360
#> GSM228641 2 0.2293 0.60595 0.000 0.900 0.016 0.000 0.084
#> GSM228644 4 0.8151 0.01235 0.000 0.140 0.332 0.356 0.172
#> GSM228651 2 0.4970 0.48774 0.008 0.728 0.108 0.156 0.000
#> GSM228654 2 0.8014 0.23546 0.004 0.456 0.244 0.144 0.152
#> GSM228658 3 0.7998 0.02194 0.000 0.208 0.444 0.136 0.212
#> GSM228606 4 0.6168 0.52946 0.004 0.128 0.156 0.664 0.048
#> GSM228611 3 0.7287 0.16116 0.032 0.272 0.512 0.016 0.168
#> GSM228618 2 0.4692 0.53624 0.100 0.780 0.092 0.004 0.024
#> GSM228621 2 0.4967 0.58942 0.000 0.728 0.064 0.020 0.188
#> GSM228624 3 0.8860 0.05007 0.112 0.132 0.468 0.144 0.144
#> GSM228630 3 0.9341 -0.09603 0.056 0.264 0.304 0.188 0.188
#> GSM228636 4 0.8084 0.21367 0.004 0.136 0.284 0.424 0.152
#> GSM228638 3 0.8873 0.02293 0.028 0.172 0.360 0.264 0.176
#> GSM228648 2 0.6202 0.28410 0.000 0.484 0.372 0.000 0.144
#> GSM228670 4 0.0290 0.60907 0.000 0.000 0.000 0.992 0.008
#> GSM228671 3 0.6473 -0.20520 0.000 0.028 0.452 0.092 0.428
#> GSM228672 1 0.5388 0.33615 0.616 0.000 0.016 0.324 0.044
#> GSM228674 4 0.1934 0.61704 0.020 0.000 0.040 0.932 0.008
#> GSM228675 4 0.1812 0.61709 0.012 0.004 0.036 0.940 0.008
#> GSM228676 4 0.7136 0.08625 0.004 0.028 0.236 0.500 0.232
#> GSM228667 5 0.7578 0.20108 0.016 0.024 0.360 0.212 0.388
#> GSM228668 1 0.2835 0.65520 0.880 0.000 0.036 0.080 0.004
#> GSM228669 1 0.3067 0.64482 0.876 0.000 0.016 0.068 0.040
#> GSM228673 4 0.6223 0.47616 0.040 0.044 0.168 0.684 0.064
#> GSM228677 4 0.2830 0.61475 0.000 0.016 0.020 0.884 0.080
#> GSM228678 5 0.5387 -0.10221 0.000 0.012 0.032 0.448 0.508
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 4 0.4953 0.4245 0.008 0.052 0.004 0.600 0.336 0.000
#> GSM228563 4 0.6049 0.0689 0.168 0.000 0.000 0.416 0.404 0.012
#> GSM228565 1 0.7923 -0.1684 0.352 0.016 0.144 0.264 0.220 0.004
#> GSM228566 4 0.2909 0.6357 0.000 0.136 0.000 0.836 0.028 0.000
#> GSM228567 1 0.2020 0.6260 0.896 0.000 0.096 0.000 0.008 0.000
#> GSM228570 5 0.6728 0.2619 0.072 0.004 0.248 0.172 0.504 0.000
#> GSM228571 5 0.6205 0.1017 0.080 0.036 0.356 0.020 0.508 0.000
#> GSM228574 4 0.5692 0.2454 0.000 0.320 0.000 0.500 0.180 0.000
#> GSM228575 5 0.7871 0.1949 0.160 0.044 0.116 0.304 0.376 0.000
#> GSM228576 5 0.4543 0.0757 0.488 0.004 0.008 0.012 0.488 0.000
#> GSM228579 5 0.5713 0.1999 0.348 0.004 0.152 0.000 0.496 0.000
#> GSM228580 5 0.8208 -0.1740 0.032 0.164 0.180 0.060 0.464 0.100
#> GSM228581 4 0.6657 0.4439 0.128 0.012 0.024 0.568 0.228 0.040
#> GSM228666 4 0.3281 0.6429 0.008 0.120 0.004 0.832 0.036 0.000
#> GSM228564 1 0.1387 0.6511 0.932 0.000 0.000 0.068 0.000 0.000
#> GSM228568 1 0.5768 -0.1819 0.460 0.004 0.080 0.000 0.432 0.024
#> GSM228569 3 0.3986 0.2447 0.464 0.000 0.532 0.000 0.004 0.000
#> GSM228572 6 0.6125 0.4266 0.000 0.044 0.052 0.352 0.028 0.524
#> GSM228573 2 0.4550 0.6721 0.056 0.756 0.000 0.080 0.108 0.000
#> GSM228577 1 0.3864 0.4234 0.744 0.000 0.208 0.000 0.048 0.000
#> GSM228578 5 0.7168 0.0963 0.128 0.004 0.328 0.048 0.452 0.040
#> GSM228663 4 0.6288 0.3262 0.000 0.116 0.044 0.564 0.016 0.260
#> GSM228664 6 0.4657 0.6524 0.000 0.100 0.000 0.228 0.000 0.672
#> GSM228665 4 0.7354 0.0614 0.128 0.052 0.060 0.416 0.000 0.344
#> GSM228582 4 0.4679 0.6195 0.040 0.020 0.112 0.768 0.004 0.056
#> GSM228583 1 0.3971 -0.2087 0.548 0.000 0.448 0.000 0.004 0.000
#> GSM228585 1 0.4944 0.5472 0.724 0.004 0.160 0.048 0.060 0.004
#> GSM228587 1 0.3670 0.5311 0.736 0.000 0.024 0.240 0.000 0.000
#> GSM228588 1 0.0260 0.6613 0.992 0.000 0.008 0.000 0.000 0.000
#> GSM228589 1 0.5256 0.3561 0.580 0.000 0.028 0.348 0.008 0.036
#> GSM228590 1 0.0000 0.6605 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228591 4 0.5514 0.6131 0.028 0.020 0.060 0.728 0.088 0.076
#> GSM228597 1 0.7835 -0.0435 0.384 0.000 0.084 0.324 0.076 0.132
#> GSM228601 4 0.3921 0.5592 0.184 0.000 0.004 0.768 0.028 0.016
#> GSM228604 2 0.5367 0.5142 0.000 0.572 0.004 0.000 0.300 0.124
#> GSM228608 1 0.0146 0.6613 0.996 0.000 0.004 0.000 0.000 0.000
#> GSM228609 1 0.4265 0.5912 0.792 0.016 0.108 0.004 0.024 0.056
#> GSM228613 1 0.0622 0.6633 0.980 0.000 0.012 0.008 0.000 0.000
#> GSM228616 3 0.5483 0.2303 0.452 0.016 0.476 0.040 0.012 0.004
#> GSM228628 4 0.4753 0.2131 0.000 0.000 0.000 0.496 0.456 0.048
#> GSM228634 3 0.5217 0.3364 0.392 0.000 0.512 0.096 0.000 0.000
#> GSM228642 5 0.6324 -0.1331 0.000 0.332 0.004 0.036 0.488 0.140
#> GSM228645 4 0.4572 0.6327 0.024 0.064 0.044 0.776 0.092 0.000
#> GSM228646 4 0.3056 0.6312 0.004 0.152 0.008 0.828 0.004 0.004
#> GSM228652 4 0.0000 0.6339 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM228655 4 0.3681 0.6278 0.064 0.016 0.112 0.808 0.000 0.000
#> GSM228656 3 0.5434 0.4085 0.312 0.000 0.544 0.144 0.000 0.000
#> GSM228659 4 0.0000 0.6339 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM228662 1 0.2996 0.5267 0.772 0.000 0.228 0.000 0.000 0.000
#> GSM228584 1 0.1814 0.6224 0.900 0.000 0.100 0.000 0.000 0.000
#> GSM228586 3 0.3986 0.2565 0.464 0.000 0.532 0.000 0.000 0.004
#> GSM228592 1 0.0000 0.6605 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228593 4 0.4809 0.3374 0.360 0.004 0.004 0.588 0.044 0.000
#> GSM228594 1 0.0508 0.6622 0.984 0.000 0.012 0.000 0.000 0.004
#> GSM228598 1 0.5115 0.4413 0.680 0.004 0.172 0.128 0.000 0.016
#> GSM228607 4 0.7152 0.0486 0.108 0.020 0.368 0.416 0.004 0.084
#> GSM228612 3 0.7233 0.2945 0.056 0.356 0.452 0.052 0.064 0.020
#> GSM228619 1 0.0405 0.6620 0.988 0.000 0.004 0.000 0.000 0.008
#> GSM228622 3 0.5651 0.3224 0.400 0.000 0.448 0.152 0.000 0.000
#> GSM228625 1 0.6923 0.1439 0.456 0.016 0.100 0.336 0.000 0.092
#> GSM228631 3 0.6891 0.4391 0.320 0.120 0.440 0.000 0.000 0.120
#> GSM228633 6 0.6034 0.0943 0.000 0.388 0.088 0.008 0.032 0.484
#> GSM228637 4 0.4187 0.6323 0.048 0.016 0.044 0.816 0.016 0.060
#> GSM228639 6 0.4033 0.6526 0.072 0.000 0.004 0.168 0.000 0.756
#> GSM228649 4 0.2164 0.6437 0.028 0.000 0.000 0.908 0.008 0.056
#> GSM228660 1 0.4787 0.5685 0.764 0.016 0.056 0.068 0.004 0.092
#> GSM228661 1 0.3841 -0.0174 0.616 0.000 0.380 0.000 0.000 0.004
#> GSM228595 4 0.4278 0.1878 0.000 0.000 0.020 0.616 0.004 0.360
#> GSM228599 4 0.3985 0.6218 0.004 0.112 0.000 0.792 0.076 0.016
#> GSM228602 2 0.5808 0.5992 0.012 0.628 0.000 0.044 0.220 0.096
#> GSM228614 4 0.2618 0.6029 0.000 0.000 0.000 0.860 0.024 0.116
#> GSM228626 6 0.4664 0.3567 0.000 0.248 0.016 0.000 0.056 0.680
#> GSM228640 2 0.2272 0.6920 0.040 0.900 0.004 0.000 0.056 0.000
#> GSM228643 4 0.6566 0.2337 0.000 0.208 0.008 0.452 0.308 0.024
#> GSM228650 4 0.1075 0.6428 0.000 0.048 0.000 0.952 0.000 0.000
#> GSM228653 2 0.6084 0.1679 0.000 0.520 0.084 0.332 0.000 0.064
#> GSM228657 6 0.5372 0.6182 0.000 0.028 0.044 0.296 0.016 0.616
#> GSM228605 1 0.1141 0.6520 0.948 0.052 0.000 0.000 0.000 0.000
#> GSM228610 2 0.7254 0.2100 0.156 0.424 0.100 0.000 0.012 0.308
#> GSM228617 3 0.7923 0.3737 0.128 0.236 0.436 0.040 0.008 0.152
#> GSM228620 1 0.3377 0.5937 0.816 0.004 0.036 0.140 0.000 0.004
#> GSM228623 4 0.4245 0.6348 0.012 0.012 0.016 0.792 0.116 0.052
#> GSM228629 3 0.7258 0.1679 0.012 0.020 0.516 0.188 0.184 0.080
#> GSM228632 4 0.7521 0.1564 0.036 0.024 0.024 0.384 0.344 0.188
#> GSM228635 4 0.6541 0.3889 0.308 0.012 0.044 0.540 0.032 0.064
#> GSM228647 3 0.7305 0.3386 0.276 0.048 0.352 0.020 0.000 0.304
#> GSM228596 4 0.0891 0.6444 0.024 0.008 0.000 0.968 0.000 0.000
#> GSM228600 2 0.2711 0.6937 0.000 0.872 0.004 0.056 0.068 0.000
#> GSM228603 2 0.2551 0.6980 0.004 0.872 0.000 0.012 0.108 0.004
#> GSM228615 4 0.6304 0.2624 0.380 0.016 0.032 0.500 0.032 0.040
#> GSM228627 4 0.6756 0.1421 0.012 0.036 0.104 0.464 0.364 0.020
#> GSM228641 2 0.2436 0.6618 0.000 0.880 0.000 0.000 0.032 0.088
#> GSM228644 6 0.4661 0.6408 0.000 0.032 0.032 0.260 0.000 0.676
#> GSM228651 2 0.5836 0.4685 0.004 0.644 0.076 0.156 0.000 0.120
#> GSM228654 6 0.6841 0.1996 0.000 0.380 0.096 0.112 0.004 0.408
#> GSM228658 3 0.7981 0.1700 0.000 0.172 0.444 0.096 0.192 0.096
#> GSM228606 4 0.6407 0.4882 0.004 0.140 0.060 0.632 0.044 0.120
#> GSM228611 3 0.7658 0.2735 0.020 0.188 0.464 0.008 0.144 0.176
#> GSM228618 2 0.4106 0.6614 0.064 0.816 0.056 0.004 0.032 0.028
#> GSM228621 2 0.5184 0.6381 0.000 0.692 0.016 0.016 0.164 0.112
#> GSM228624 3 0.9011 0.2394 0.104 0.116 0.416 0.100 0.140 0.124
#> GSM228630 6 0.5696 0.6189 0.052 0.088 0.008 0.164 0.012 0.676
#> GSM228636 6 0.5066 0.4028 0.000 0.000 0.032 0.364 0.032 0.572
#> GSM228638 6 0.3401 0.6687 0.016 0.004 0.000 0.204 0.000 0.776
#> GSM228648 6 0.3665 0.4070 0.000 0.252 0.020 0.000 0.000 0.728
#> GSM228670 4 0.0291 0.6356 0.000 0.000 0.000 0.992 0.004 0.004
#> GSM228671 3 0.6935 -0.1244 0.000 0.024 0.440 0.048 0.344 0.144
#> GSM228672 1 0.5127 0.3211 0.608 0.000 0.020 0.324 0.036 0.012
#> GSM228674 4 0.1873 0.6500 0.020 0.000 0.048 0.924 0.008 0.000
#> GSM228675 4 0.2044 0.6493 0.008 0.004 0.076 0.908 0.004 0.000
#> GSM228676 4 0.6772 0.1191 0.004 0.036 0.284 0.452 0.220 0.004
#> GSM228667 3 0.7082 -0.1062 0.008 0.012 0.388 0.164 0.380 0.048
#> GSM228668 1 0.3482 0.6396 0.832 0.000 0.088 0.060 0.004 0.016
#> GSM228669 1 0.3851 0.6197 0.828 0.000 0.032 0.064 0.044 0.032
#> GSM228673 4 0.6169 0.5169 0.036 0.040 0.196 0.644 0.064 0.020
#> GSM228677 4 0.2333 0.6161 0.000 0.000 0.000 0.884 0.024 0.092
#> GSM228678 5 0.5302 -0.1388 0.000 0.008 0.020 0.408 0.524 0.040
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
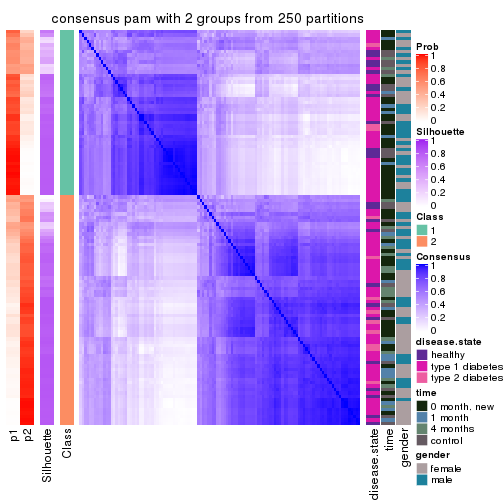
consensus_heatmap(res, k = 3)
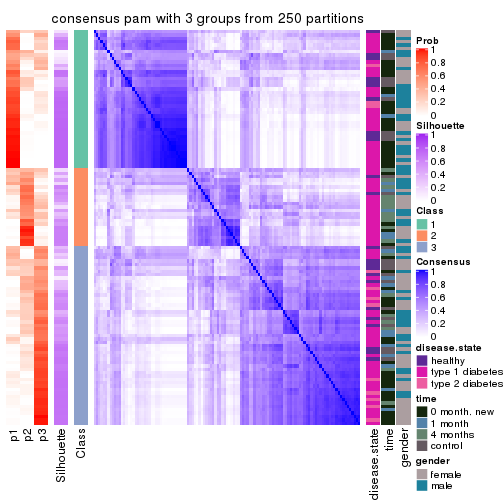
consensus_heatmap(res, k = 4)
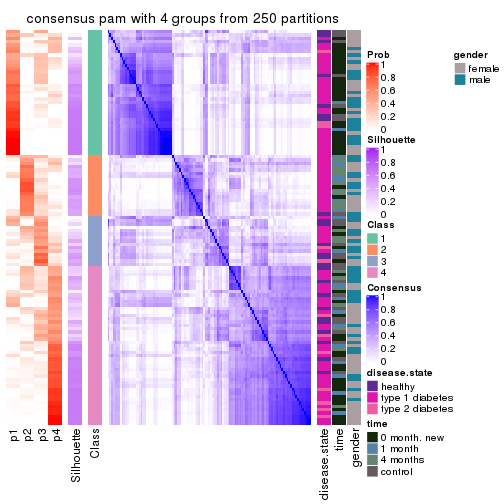
consensus_heatmap(res, k = 5)
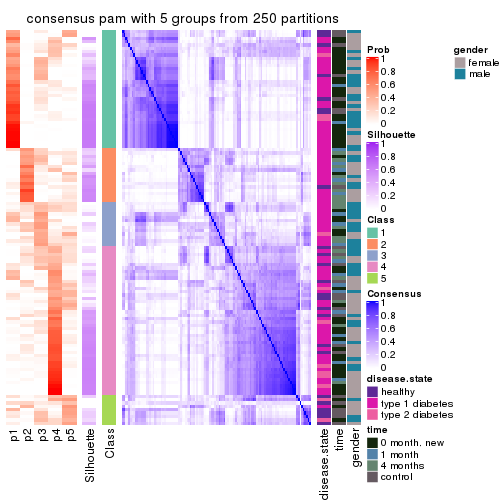
consensus_heatmap(res, k = 6)
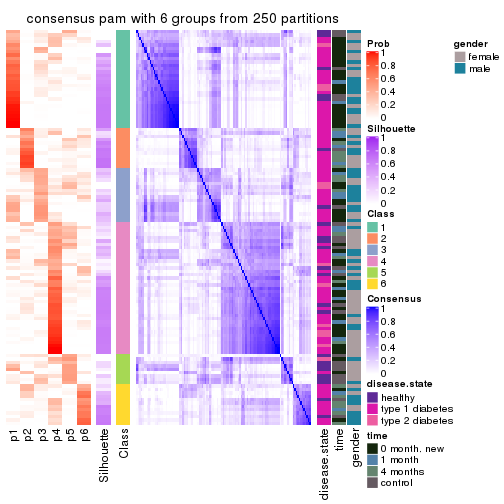
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
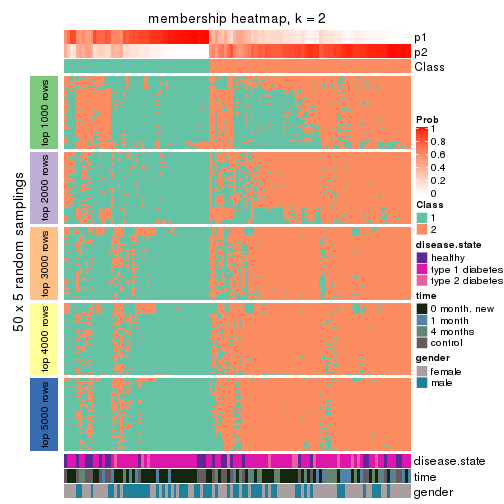
membership_heatmap(res, k = 3)
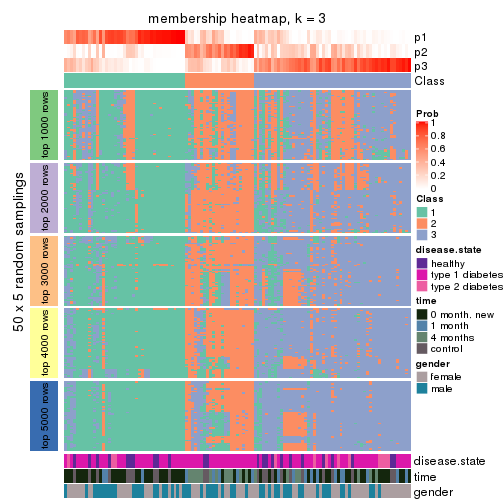
membership_heatmap(res, k = 4)
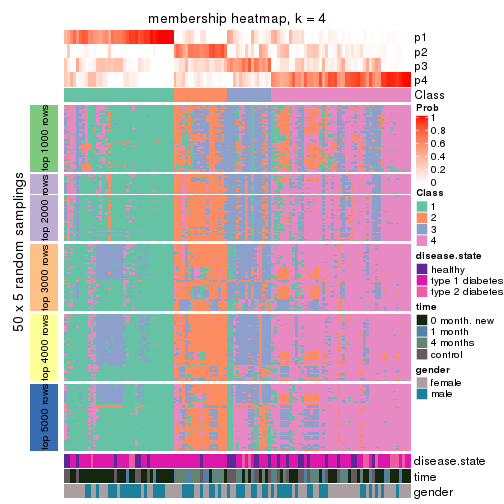
membership_heatmap(res, k = 5)
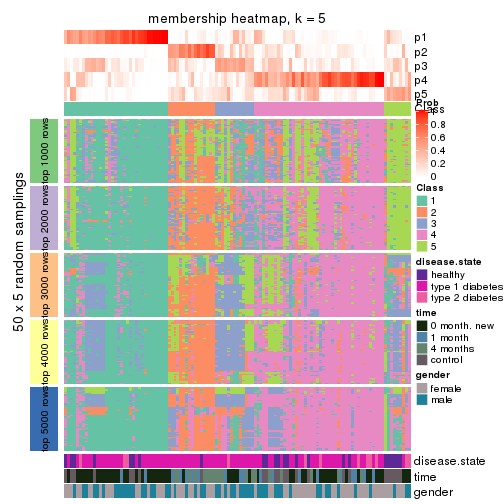
membership_heatmap(res, k = 6)
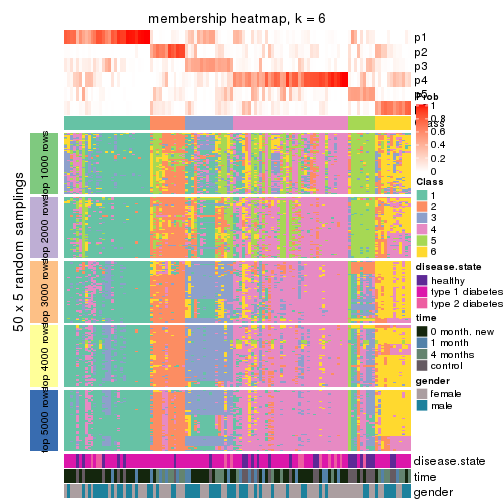
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
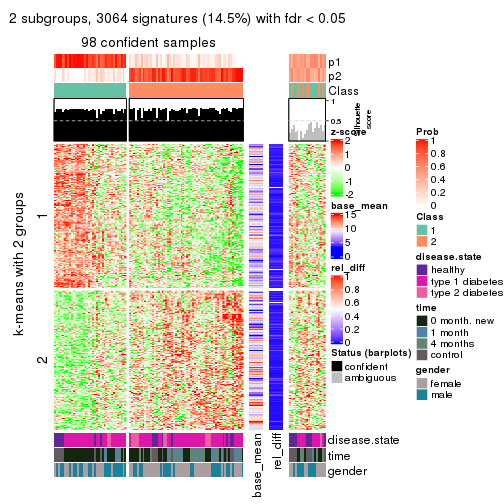
get_signatures(res, k = 3)
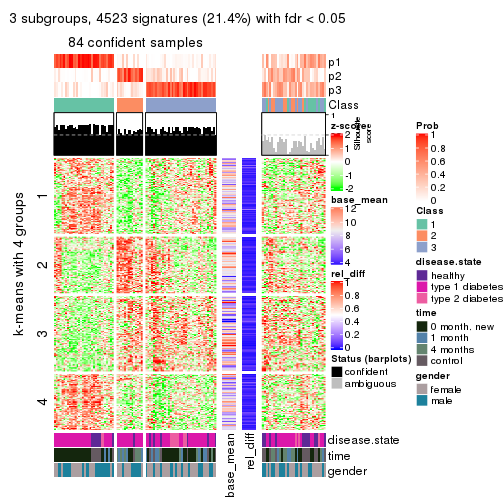
get_signatures(res, k = 4)
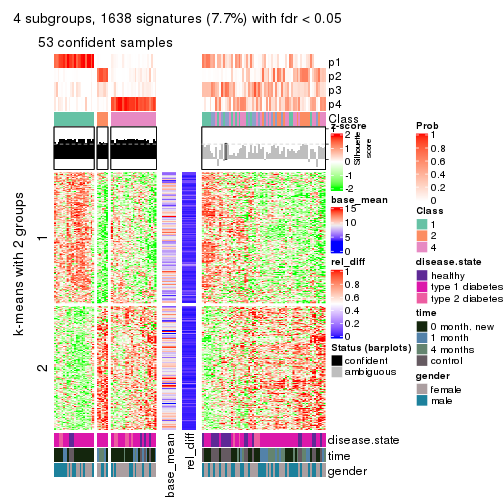
get_signatures(res, k = 5)
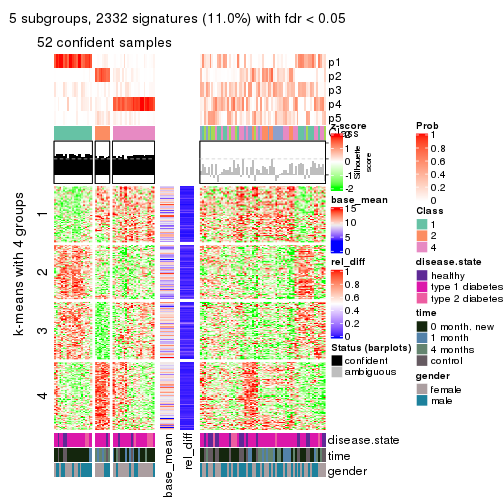
get_signatures(res, k = 6)
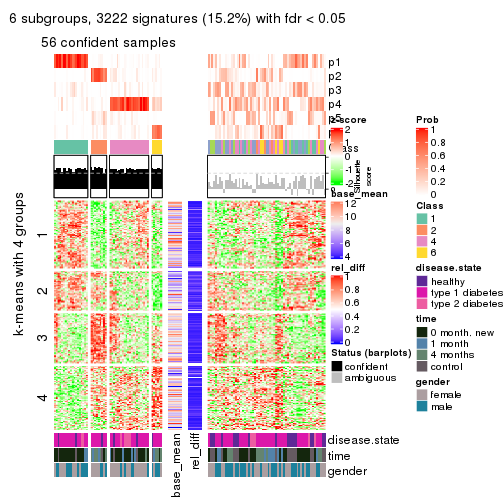
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
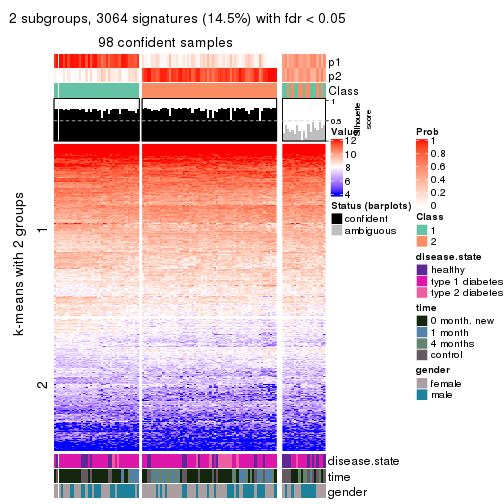
get_signatures(res, k = 3, scale_rows = FALSE)
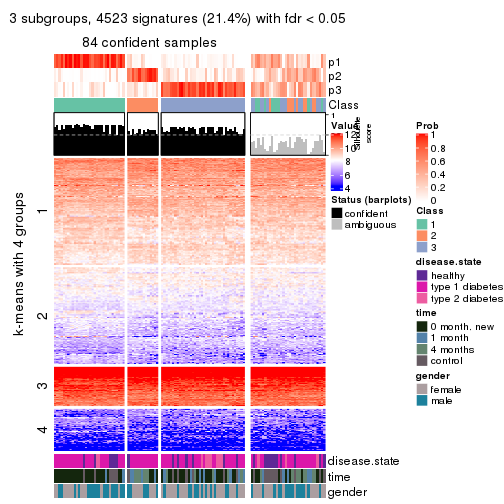
get_signatures(res, k = 4, scale_rows = FALSE)
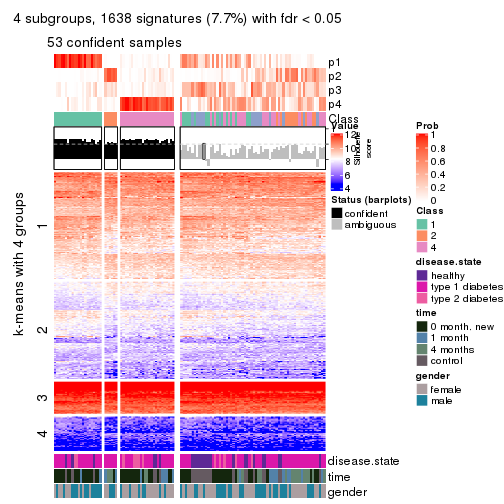
get_signatures(res, k = 5, scale_rows = FALSE)
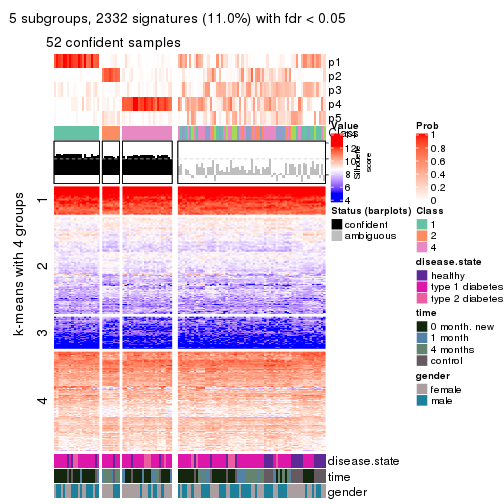
get_signatures(res, k = 6, scale_rows = FALSE)
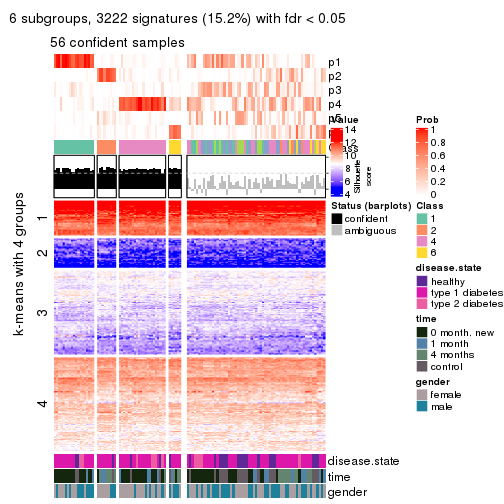
Compare the overlap of signatures from different k:
compare_signatures(res)
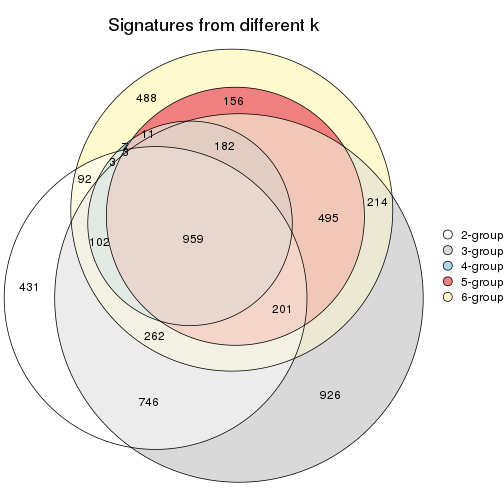
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
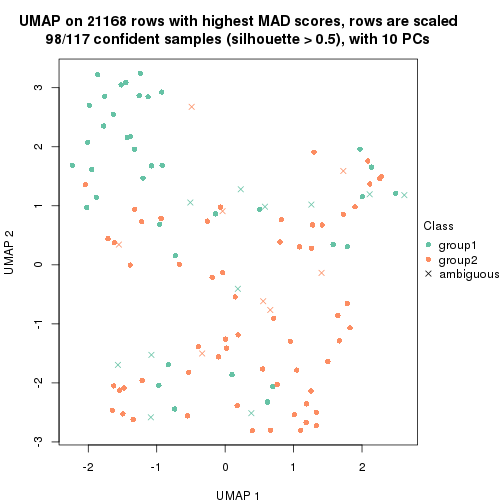
dimension_reduction(res, k = 3, method = "UMAP")
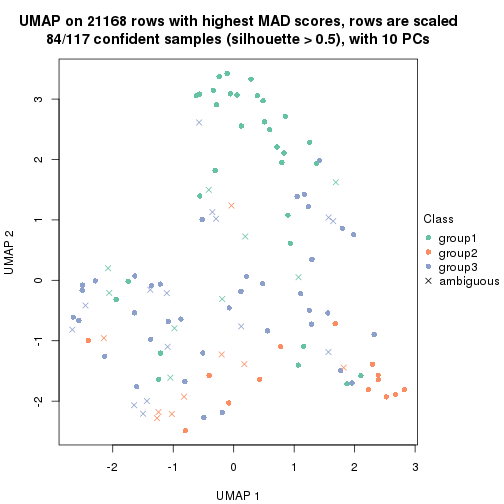
dimension_reduction(res, k = 4, method = "UMAP")
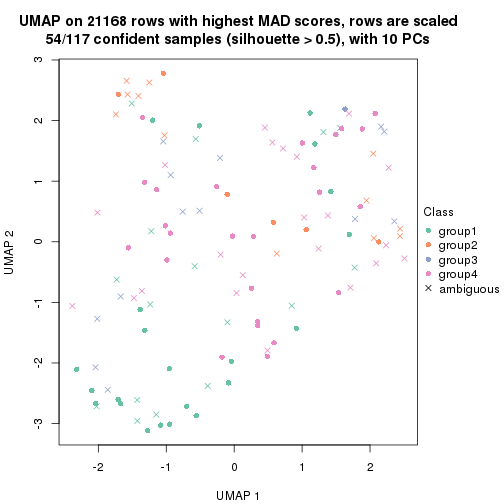
dimension_reduction(res, k = 5, method = "UMAP")
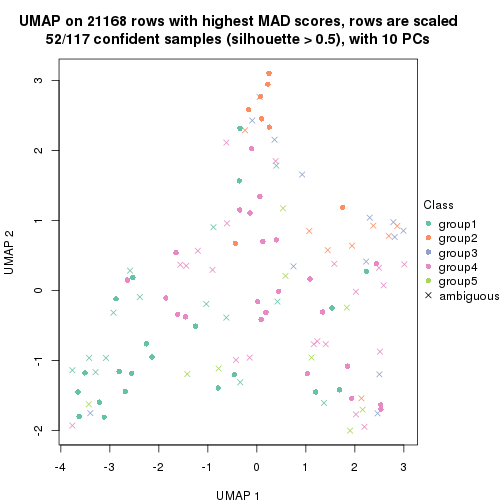
dimension_reduction(res, k = 6, method = "UMAP")
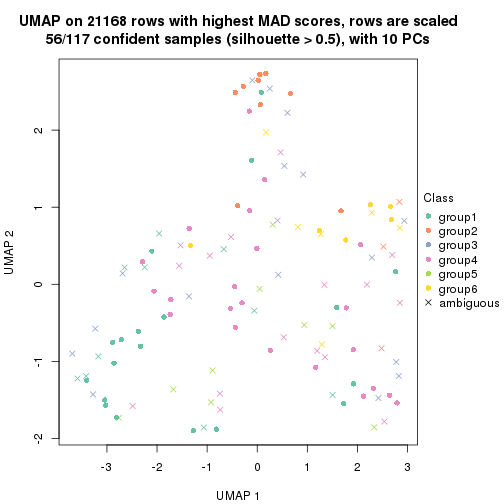
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
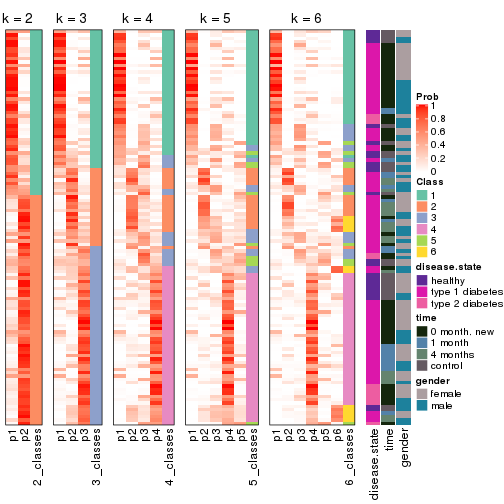
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> MAD:pam 98 0.323 0.013509 0.1099 2
#> MAD:pam 84 0.193 0.000614 0.1797 3
#> MAD:pam 54 0.116 0.002797 0.1129 4
#> MAD:pam 52 0.712 0.000344 0.0546 5
#> MAD:pam 56 0.615 0.002122 0.1132 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["MAD", "mclust"]
# you can also extract it by
# res = res_list["MAD:mclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'MAD' method.
#> Subgroups are detected by 'mclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
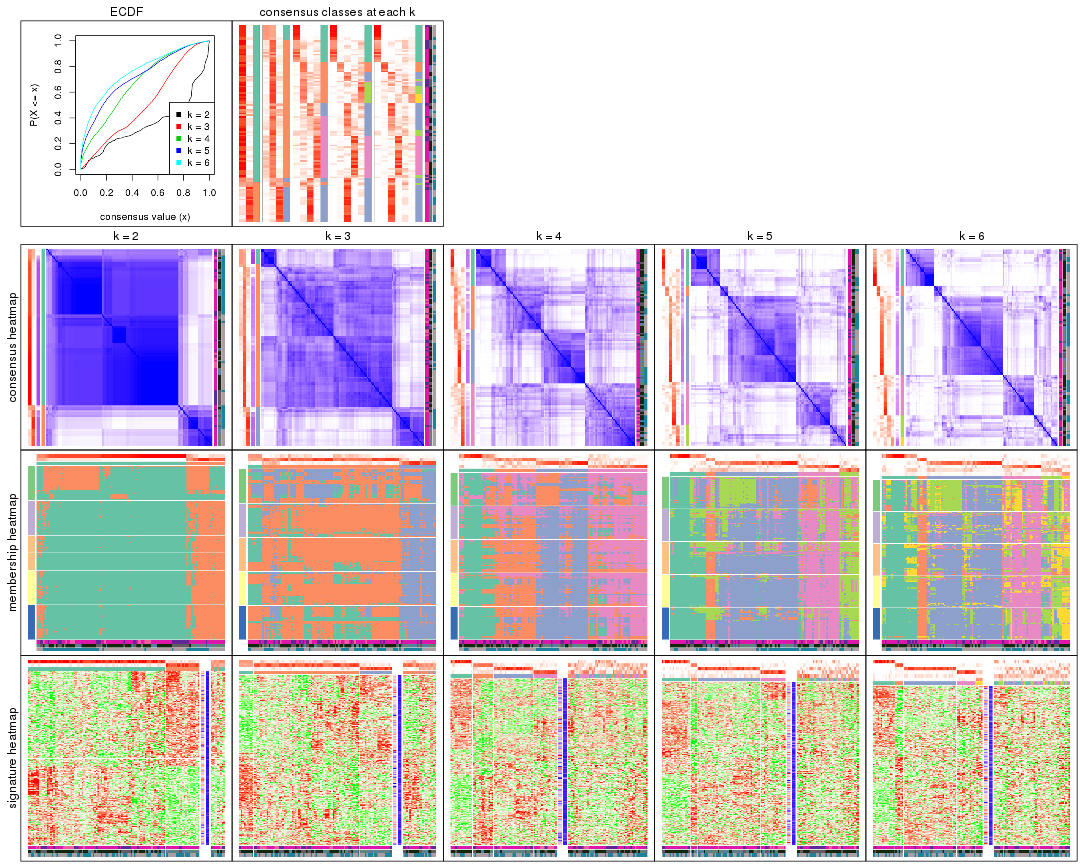
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
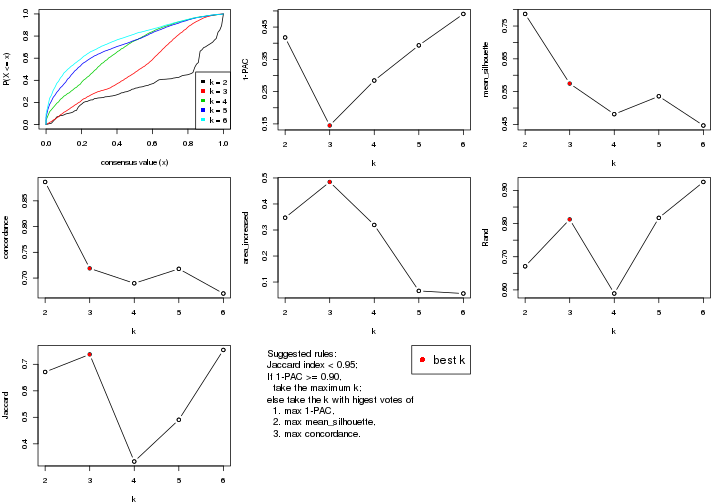
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.418 0.787 0.887 0.3473 0.671 0.671
#> 3 3 0.145 0.575 0.719 0.4846 0.813 0.738
#> 4 4 0.284 0.481 0.690 0.3194 0.589 0.333
#> 5 5 0.393 0.536 0.718 0.0659 0.817 0.491
#> 6 6 0.491 0.447 0.670 0.0558 0.926 0.754
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.0000 0.88336 1.000 0.000
#> GSM228563 1 0.1184 0.88468 0.984 0.016
#> GSM228565 1 0.0000 0.88336 1.000 0.000
#> GSM228566 1 0.9815 -0.10414 0.580 0.420
#> GSM228567 1 0.2043 0.86863 0.968 0.032
#> GSM228570 1 0.0000 0.88336 1.000 0.000
#> GSM228571 1 0.0000 0.88336 1.000 0.000
#> GSM228574 1 0.5059 0.78242 0.888 0.112
#> GSM228575 1 0.9661 0.00713 0.608 0.392
#> GSM228576 1 0.0000 0.88336 1.000 0.000
#> GSM228579 1 0.0000 0.88336 1.000 0.000
#> GSM228580 1 0.0000 0.88336 1.000 0.000
#> GSM228581 1 0.0938 0.88003 0.988 0.012
#> GSM228666 1 0.0000 0.88336 1.000 0.000
#> GSM228564 1 0.0000 0.88336 1.000 0.000
#> GSM228568 1 0.5842 0.84900 0.860 0.140
#> GSM228569 1 0.6247 0.83569 0.844 0.156
#> GSM228572 1 0.5178 0.85809 0.884 0.116
#> GSM228573 2 0.2423 0.80014 0.040 0.960
#> GSM228577 1 0.5737 0.84861 0.864 0.136
#> GSM228578 1 0.5737 0.84861 0.864 0.136
#> GSM228663 2 1.0000 -0.03557 0.500 0.500
#> GSM228664 1 0.7883 0.74561 0.764 0.236
#> GSM228665 2 0.9087 0.55177 0.324 0.676
#> GSM228582 1 0.0376 0.88247 0.996 0.004
#> GSM228583 1 0.2043 0.86863 0.968 0.032
#> GSM228585 1 0.2043 0.86863 0.968 0.032
#> GSM228587 1 0.2043 0.86863 0.968 0.032
#> GSM228588 1 0.0000 0.88336 1.000 0.000
#> GSM228589 1 0.2778 0.88017 0.952 0.048
#> GSM228590 1 0.2043 0.86863 0.968 0.032
#> GSM228591 1 0.0000 0.88336 1.000 0.000
#> GSM228597 1 0.2948 0.87937 0.948 0.052
#> GSM228601 1 0.0000 0.88336 1.000 0.000
#> GSM228604 1 0.1843 0.86968 0.972 0.028
#> GSM228608 1 0.1843 0.87107 0.972 0.028
#> GSM228609 1 0.0000 0.88336 1.000 0.000
#> GSM228613 1 0.2043 0.86863 0.968 0.032
#> GSM228616 1 0.0000 0.88336 1.000 0.000
#> GSM228628 1 0.0000 0.88336 1.000 0.000
#> GSM228634 1 0.2778 0.87267 0.952 0.048
#> GSM228642 1 0.0000 0.88336 1.000 0.000
#> GSM228645 1 0.1843 0.87108 0.972 0.028
#> GSM228646 1 0.1633 0.87393 0.976 0.024
#> GSM228652 1 0.0000 0.88336 1.000 0.000
#> GSM228655 1 0.0938 0.88471 0.988 0.012
#> GSM228656 1 0.2043 0.86863 0.968 0.032
#> GSM228659 1 0.0000 0.88336 1.000 0.000
#> GSM228662 1 0.2043 0.86863 0.968 0.032
#> GSM228584 1 0.6438 0.83617 0.836 0.164
#> GSM228586 1 0.6531 0.83335 0.832 0.168
#> GSM228592 1 0.6531 0.83335 0.832 0.168
#> GSM228593 1 0.2236 0.88275 0.964 0.036
#> GSM228594 1 0.5408 0.85443 0.876 0.124
#> GSM228598 1 0.5737 0.84861 0.864 0.136
#> GSM228607 1 0.5842 0.84702 0.860 0.140
#> GSM228612 2 0.9983 0.07377 0.476 0.524
#> GSM228619 1 0.5737 0.84861 0.864 0.136
#> GSM228622 1 0.5737 0.84861 0.864 0.136
#> GSM228625 1 0.5737 0.84861 0.864 0.136
#> GSM228631 1 0.5737 0.84861 0.864 0.136
#> GSM228633 1 0.5737 0.84861 0.864 0.136
#> GSM228637 1 0.5737 0.84861 0.864 0.136
#> GSM228639 1 0.6247 0.83756 0.844 0.156
#> GSM228649 1 0.5737 0.84861 0.864 0.136
#> GSM228660 1 0.5737 0.84861 0.864 0.136
#> GSM228661 1 0.5842 0.84661 0.860 0.140
#> GSM228595 1 0.3733 0.87359 0.928 0.072
#> GSM228599 1 0.0000 0.88336 1.000 0.000
#> GSM228602 2 0.7299 0.80443 0.204 0.796
#> GSM228614 1 0.1414 0.88440 0.980 0.020
#> GSM228626 1 0.0000 0.88336 1.000 0.000
#> GSM228640 2 0.7453 0.79939 0.212 0.788
#> GSM228643 2 0.7950 0.78231 0.240 0.760
#> GSM228650 1 0.9580 0.06586 0.620 0.380
#> GSM228653 2 0.6973 0.80505 0.188 0.812
#> GSM228657 1 0.0000 0.88336 1.000 0.000
#> GSM228605 1 0.5737 0.84861 0.864 0.136
#> GSM228610 2 0.2603 0.80078 0.044 0.956
#> GSM228617 2 0.8386 0.65313 0.268 0.732
#> GSM228620 2 0.3274 0.80175 0.060 0.940
#> GSM228623 1 0.5737 0.84861 0.864 0.136
#> GSM228629 2 0.2423 0.80014 0.040 0.960
#> GSM228632 1 0.8608 0.66966 0.716 0.284
#> GSM228635 1 0.5737 0.84861 0.864 0.136
#> GSM228647 2 0.2423 0.80014 0.040 0.960
#> GSM228596 1 0.3431 0.83759 0.936 0.064
#> GSM228600 2 0.6712 0.80224 0.176 0.824
#> GSM228603 2 0.6801 0.80392 0.180 0.820
#> GSM228615 1 0.0376 0.88394 0.996 0.004
#> GSM228627 1 0.9993 -0.32828 0.516 0.484
#> GSM228641 2 0.6887 0.80391 0.184 0.816
#> GSM228644 1 0.0000 0.88336 1.000 0.000
#> GSM228651 2 0.6343 0.80741 0.160 0.840
#> GSM228654 2 0.6887 0.80443 0.184 0.816
#> GSM228658 2 0.8016 0.78321 0.244 0.756
#> GSM228606 1 0.5946 0.84521 0.856 0.144
#> GSM228611 2 0.2423 0.80014 0.040 0.960
#> GSM228618 2 0.3114 0.80180 0.056 0.944
#> GSM228621 2 0.7602 0.70117 0.220 0.780
#> GSM228624 1 0.9580 0.44410 0.620 0.380
#> GSM228630 1 0.6148 0.84045 0.848 0.152
#> GSM228636 1 0.5737 0.84861 0.864 0.136
#> GSM228638 2 0.8443 0.64308 0.272 0.728
#> GSM228648 2 0.9795 0.29711 0.416 0.584
#> GSM228670 1 0.0000 0.88336 1.000 0.000
#> GSM228671 1 0.2948 0.88066 0.948 0.052
#> GSM228672 1 0.0000 0.88336 1.000 0.000
#> GSM228674 1 0.0672 0.88444 0.992 0.008
#> GSM228675 1 0.0000 0.88336 1.000 0.000
#> GSM228676 1 0.0376 0.88219 0.996 0.004
#> GSM228667 1 0.0000 0.88336 1.000 0.000
#> GSM228668 1 0.5737 0.84861 0.864 0.136
#> GSM228669 1 0.5737 0.84861 0.864 0.136
#> GSM228673 1 0.9522 0.47645 0.628 0.372
#> GSM228677 1 0.5737 0.84861 0.864 0.136
#> GSM228678 1 0.5629 0.85074 0.868 0.132
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 2 0.703 0.6334 0.196 0.716 0.088
#> GSM228563 2 0.625 0.6077 0.300 0.684 0.016
#> GSM228565 2 0.510 0.6518 0.080 0.836 0.084
#> GSM228566 3 0.873 0.1150 0.108 0.424 0.468
#> GSM228567 1 0.665 0.9084 0.540 0.452 0.008
#> GSM228570 2 0.487 0.5768 0.152 0.824 0.024
#> GSM228571 2 0.603 0.4297 0.152 0.780 0.068
#> GSM228574 2 0.816 0.4755 0.104 0.608 0.288
#> GSM228575 2 0.820 0.0310 0.072 0.484 0.444
#> GSM228576 2 0.535 0.6472 0.088 0.824 0.088
#> GSM228579 2 0.604 0.3759 0.172 0.772 0.056
#> GSM228580 2 0.620 0.6285 0.208 0.748 0.044
#> GSM228581 2 0.500 0.6139 0.068 0.840 0.092
#> GSM228666 2 0.350 0.6424 0.028 0.900 0.072
#> GSM228564 2 0.631 0.5740 0.308 0.676 0.016
#> GSM228568 2 0.484 0.6135 0.016 0.816 0.168
#> GSM228569 2 0.678 0.4844 0.116 0.744 0.140
#> GSM228572 2 0.475 0.6442 0.080 0.852 0.068
#> GSM228573 3 0.196 0.7439 0.000 0.056 0.944
#> GSM228577 2 0.639 0.5061 0.116 0.768 0.116
#> GSM228578 2 0.445 0.6254 0.012 0.836 0.152
#> GSM228663 2 0.620 0.4982 0.008 0.656 0.336
#> GSM228664 2 0.538 0.5747 0.008 0.756 0.236
#> GSM228665 3 0.624 0.0980 0.000 0.440 0.560
#> GSM228582 2 0.412 0.6162 0.040 0.876 0.084
#> GSM228583 1 0.665 0.9084 0.540 0.452 0.008
#> GSM228585 1 0.680 0.9064 0.532 0.456 0.012
#> GSM228587 2 0.475 0.3466 0.216 0.784 0.000
#> GSM228588 2 0.296 0.6105 0.100 0.900 0.000
#> GSM228589 2 0.329 0.6202 0.096 0.896 0.008
#> GSM228590 1 0.629 0.9026 0.532 0.468 0.000
#> GSM228591 2 0.298 0.6173 0.024 0.920 0.056
#> GSM228597 2 0.636 0.5781 0.336 0.652 0.012
#> GSM228601 2 0.296 0.6182 0.080 0.912 0.008
#> GSM228604 2 0.493 0.6377 0.032 0.828 0.140
#> GSM228608 2 0.531 0.4010 0.216 0.772 0.012
#> GSM228609 2 0.288 0.6123 0.096 0.904 0.000
#> GSM228613 1 0.630 0.8842 0.516 0.484 0.000
#> GSM228616 2 0.324 0.6363 0.032 0.912 0.056
#> GSM228628 2 0.303 0.6261 0.012 0.912 0.076
#> GSM228634 2 0.690 -0.7446 0.436 0.548 0.016
#> GSM228642 2 0.321 0.6277 0.012 0.904 0.084
#> GSM228645 2 0.509 0.6147 0.056 0.832 0.112
#> GSM228646 2 0.542 0.6218 0.080 0.820 0.100
#> GSM228652 2 0.346 0.5925 0.096 0.892 0.012
#> GSM228655 2 0.358 0.6375 0.044 0.900 0.056
#> GSM228656 1 0.648 0.9066 0.548 0.448 0.004
#> GSM228659 2 0.296 0.5952 0.100 0.900 0.000
#> GSM228662 1 0.631 0.8769 0.512 0.488 0.000
#> GSM228584 1 0.841 0.7847 0.508 0.404 0.088
#> GSM228586 2 0.852 -0.7202 0.444 0.464 0.092
#> GSM228592 1 0.842 0.7787 0.504 0.408 0.088
#> GSM228593 2 0.338 0.6088 0.100 0.892 0.008
#> GSM228594 2 0.747 0.2187 0.216 0.684 0.100
#> GSM228598 2 0.635 0.4597 0.140 0.768 0.092
#> GSM228607 2 0.511 0.6442 0.036 0.820 0.144
#> GSM228612 2 0.643 0.3350 0.004 0.568 0.428
#> GSM228619 2 0.743 0.6430 0.168 0.700 0.132
#> GSM228622 2 0.756 0.6459 0.156 0.692 0.152
#> GSM228625 2 0.463 0.6366 0.056 0.856 0.088
#> GSM228631 2 0.693 0.6486 0.096 0.728 0.176
#> GSM228633 2 0.421 0.6314 0.020 0.860 0.120
#> GSM228637 2 0.611 0.6381 0.192 0.760 0.048
#> GSM228639 2 0.810 0.6043 0.132 0.640 0.228
#> GSM228649 2 0.397 0.6277 0.072 0.884 0.044
#> GSM228660 2 0.420 0.6191 0.012 0.852 0.136
#> GSM228661 2 0.692 0.4529 0.132 0.736 0.132
#> GSM228595 2 0.409 0.6320 0.100 0.872 0.028
#> GSM228599 2 0.690 0.5983 0.268 0.684 0.048
#> GSM228602 3 0.468 0.7507 0.020 0.148 0.832
#> GSM228614 2 0.670 0.6007 0.328 0.648 0.024
#> GSM228626 2 0.336 0.6376 0.036 0.908 0.056
#> GSM228640 3 0.557 0.7325 0.044 0.160 0.796
#> GSM228643 3 0.692 0.7007 0.104 0.164 0.732
#> GSM228650 2 0.902 0.0675 0.132 0.456 0.412
#> GSM228653 3 0.512 0.7400 0.032 0.152 0.816
#> GSM228657 2 0.318 0.6312 0.076 0.908 0.016
#> GSM228605 2 0.793 0.6307 0.168 0.664 0.168
#> GSM228610 3 0.210 0.7418 0.004 0.052 0.944
#> GSM228617 3 0.592 0.5568 0.012 0.276 0.712
#> GSM228620 3 0.331 0.7459 0.028 0.064 0.908
#> GSM228623 2 0.573 0.6442 0.108 0.804 0.088
#> GSM228629 3 0.176 0.7343 0.004 0.040 0.956
#> GSM228632 2 0.842 0.4464 0.096 0.540 0.364
#> GSM228635 2 0.738 0.5630 0.320 0.628 0.052
#> GSM228647 3 0.228 0.7444 0.008 0.052 0.940
#> GSM228596 2 0.808 0.5514 0.116 0.632 0.252
#> GSM228600 3 0.432 0.7334 0.028 0.112 0.860
#> GSM228603 3 0.449 0.7417 0.036 0.108 0.856
#> GSM228615 2 0.665 0.5397 0.364 0.620 0.016
#> GSM228627 2 0.710 0.2994 0.028 0.588 0.384
#> GSM228641 3 0.474 0.7377 0.048 0.104 0.848
#> GSM228644 2 0.288 0.6339 0.024 0.924 0.052
#> GSM228651 3 0.377 0.7329 0.028 0.084 0.888
#> GSM228654 3 0.492 0.7453 0.036 0.132 0.832
#> GSM228658 3 0.602 0.6774 0.028 0.232 0.740
#> GSM228606 2 0.839 0.6034 0.148 0.616 0.236
#> GSM228611 3 0.207 0.7451 0.000 0.060 0.940
#> GSM228618 3 0.259 0.7464 0.004 0.072 0.924
#> GSM228621 3 0.678 0.6433 0.080 0.188 0.732
#> GSM228624 2 0.553 0.5502 0.000 0.704 0.296
#> GSM228630 2 0.804 0.6092 0.116 0.636 0.248
#> GSM228636 2 0.702 0.6058 0.260 0.684 0.056
#> GSM228638 3 0.613 0.5766 0.020 0.268 0.712
#> GSM228648 2 0.726 0.3098 0.028 0.532 0.440
#> GSM228670 2 0.638 0.5515 0.340 0.648 0.012
#> GSM228671 2 0.792 0.6339 0.204 0.660 0.136
#> GSM228672 2 0.566 0.5950 0.284 0.712 0.004
#> GSM228674 2 0.658 0.5368 0.380 0.608 0.012
#> GSM228675 2 0.651 0.5406 0.364 0.624 0.012
#> GSM228676 2 0.763 0.6109 0.232 0.668 0.100
#> GSM228667 2 0.709 0.6309 0.208 0.708 0.084
#> GSM228668 2 0.761 0.6371 0.204 0.680 0.116
#> GSM228669 2 0.814 0.5841 0.276 0.616 0.108
#> GSM228673 3 0.874 -0.2164 0.108 0.440 0.452
#> GSM228677 2 0.800 0.6209 0.224 0.648 0.128
#> GSM228678 2 0.726 0.6129 0.248 0.680 0.072
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.6854 0.4650 0.040 0.108 0.184 0.668
#> GSM228563 4 0.4613 0.5134 0.036 0.164 0.008 0.792
#> GSM228565 4 0.9259 0.1207 0.104 0.220 0.260 0.416
#> GSM228566 3 0.4590 0.6522 0.000 0.060 0.792 0.148
#> GSM228567 1 0.0524 0.6913 0.988 0.008 0.004 0.000
#> GSM228570 1 0.8027 0.1467 0.456 0.132 0.036 0.376
#> GSM228571 1 0.8933 0.3418 0.468 0.268 0.156 0.108
#> GSM228574 3 0.6617 0.3559 0.000 0.196 0.628 0.176
#> GSM228575 3 0.5928 0.5329 0.004 0.088 0.692 0.216
#> GSM228576 4 0.9832 0.0181 0.176 0.224 0.292 0.308
#> GSM228579 1 0.8550 0.4187 0.496 0.276 0.152 0.076
#> GSM228580 4 0.6779 0.4069 0.024 0.216 0.108 0.652
#> GSM228581 2 0.6814 0.5219 0.000 0.584 0.276 0.140
#> GSM228666 2 0.7540 0.4826 0.004 0.520 0.244 0.232
#> GSM228564 4 0.3424 0.5613 0.052 0.048 0.016 0.884
#> GSM228568 2 0.7361 0.4286 0.176 0.644 0.104 0.076
#> GSM228569 1 0.8153 0.3387 0.452 0.388 0.076 0.084
#> GSM228572 2 0.5345 0.3818 0.008 0.584 0.004 0.404
#> GSM228573 3 0.3172 0.7211 0.000 0.160 0.840 0.000
#> GSM228577 1 0.7947 0.3919 0.484 0.360 0.044 0.112
#> GSM228578 4 0.9733 0.1285 0.256 0.252 0.152 0.340
#> GSM228663 2 0.5901 0.4279 0.000 0.652 0.280 0.068
#> GSM228664 2 0.5775 0.5081 0.000 0.696 0.212 0.092
#> GSM228665 3 0.5861 0.5859 0.000 0.296 0.644 0.060
#> GSM228582 2 0.7624 0.5465 0.076 0.604 0.228 0.092
#> GSM228583 1 0.0524 0.6900 0.988 0.008 0.000 0.004
#> GSM228585 1 0.0672 0.6894 0.984 0.008 0.008 0.000
#> GSM228587 1 0.6382 0.5347 0.664 0.196 0.004 0.136
#> GSM228588 2 0.6040 0.4917 0.052 0.620 0.004 0.324
#> GSM228589 2 0.5174 0.5616 0.032 0.716 0.004 0.248
#> GSM228590 1 0.1042 0.6960 0.972 0.020 0.000 0.008
#> GSM228591 2 0.5719 0.6014 0.004 0.724 0.108 0.164
#> GSM228597 4 0.3884 0.5410 0.036 0.108 0.008 0.848
#> GSM228601 2 0.5770 0.5651 0.040 0.708 0.024 0.228
#> GSM228604 2 0.7332 0.4086 0.000 0.448 0.396 0.156
#> GSM228608 1 0.6240 0.4857 0.640 0.080 0.004 0.276
#> GSM228609 4 0.6743 -0.1980 0.068 0.452 0.008 0.472
#> GSM228613 1 0.1388 0.6966 0.960 0.012 0.000 0.028
#> GSM228616 2 0.8934 0.2787 0.072 0.404 0.192 0.332
#> GSM228628 2 0.6284 0.5952 0.000 0.664 0.172 0.164
#> GSM228634 1 0.3376 0.6979 0.868 0.108 0.008 0.016
#> GSM228642 2 0.5842 0.6017 0.000 0.704 0.128 0.168
#> GSM228645 3 0.7454 -0.2400 0.000 0.376 0.448 0.176
#> GSM228646 3 0.7628 -0.2006 0.000 0.348 0.440 0.212
#> GSM228652 1 0.7644 0.0975 0.472 0.136 0.016 0.376
#> GSM228655 4 0.9490 0.2013 0.264 0.200 0.140 0.396
#> GSM228656 1 0.0524 0.6900 0.988 0.008 0.000 0.004
#> GSM228659 4 0.7761 0.1682 0.332 0.212 0.004 0.452
#> GSM228662 1 0.1284 0.6969 0.964 0.012 0.000 0.024
#> GSM228584 1 0.2675 0.6782 0.908 0.048 0.000 0.044
#> GSM228586 1 0.3991 0.6834 0.832 0.120 0.000 0.048
#> GSM228592 1 0.2840 0.6764 0.900 0.056 0.000 0.044
#> GSM228593 2 0.6302 0.4372 0.068 0.564 0.000 0.368
#> GSM228594 1 0.6226 0.5748 0.612 0.320 0.004 0.064
#> GSM228598 1 0.7048 0.5167 0.572 0.280 0.004 0.144
#> GSM228607 4 0.7785 0.0767 0.000 0.288 0.284 0.428
#> GSM228612 2 0.6354 0.0298 0.000 0.520 0.416 0.064
#> GSM228619 4 0.5186 0.5356 0.016 0.076 0.128 0.780
#> GSM228622 4 0.9129 0.3139 0.220 0.136 0.176 0.468
#> GSM228625 4 0.6595 0.3069 0.120 0.276 0.000 0.604
#> GSM228631 4 0.8422 0.3297 0.072 0.140 0.284 0.504
#> GSM228633 2 0.4284 0.5561 0.000 0.764 0.012 0.224
#> GSM228637 4 0.4442 0.4387 0.004 0.236 0.008 0.752
#> GSM228639 4 0.6951 0.3298 0.000 0.132 0.324 0.544
#> GSM228649 2 0.5151 0.3110 0.004 0.532 0.000 0.464
#> GSM228660 2 0.7782 0.4745 0.116 0.596 0.072 0.216
#> GSM228661 1 0.7985 0.4315 0.496 0.352 0.076 0.076
#> GSM228595 2 0.5026 0.5755 0.028 0.740 0.008 0.224
#> GSM228599 4 0.3997 0.5628 0.036 0.040 0.064 0.860
#> GSM228602 3 0.2909 0.7416 0.008 0.036 0.904 0.052
#> GSM228614 4 0.4476 0.5721 0.044 0.044 0.076 0.836
#> GSM228626 2 0.5563 0.5919 0.004 0.724 0.076 0.196
#> GSM228640 3 0.1674 0.7319 0.004 0.012 0.952 0.032
#> GSM228643 3 0.2722 0.7142 0.000 0.032 0.904 0.064
#> GSM228650 3 0.5123 0.5665 0.000 0.044 0.724 0.232
#> GSM228653 3 0.1247 0.7300 0.004 0.012 0.968 0.016
#> GSM228657 2 0.6194 0.5524 0.040 0.668 0.032 0.260
#> GSM228605 4 0.5970 0.4418 0.000 0.088 0.244 0.668
#> GSM228610 3 0.3074 0.7210 0.000 0.152 0.848 0.000
#> GSM228617 3 0.5803 0.6803 0.004 0.128 0.720 0.148
#> GSM228620 3 0.3757 0.7237 0.000 0.152 0.828 0.020
#> GSM228623 4 0.4194 0.5326 0.000 0.172 0.028 0.800
#> GSM228629 3 0.3172 0.7198 0.000 0.160 0.840 0.000
#> GSM228632 3 0.7336 0.3804 0.000 0.256 0.528 0.216
#> GSM228635 4 0.4012 0.4655 0.004 0.204 0.004 0.788
#> GSM228647 3 0.3123 0.7209 0.000 0.156 0.844 0.000
#> GSM228596 3 0.6649 0.2040 0.016 0.056 0.556 0.372
#> GSM228600 3 0.1182 0.7280 0.000 0.016 0.968 0.016
#> GSM228603 3 0.0992 0.7250 0.004 0.012 0.976 0.008
#> GSM228615 4 0.3249 0.5537 0.044 0.060 0.008 0.888
#> GSM228627 3 0.5397 0.4617 0.000 0.220 0.716 0.064
#> GSM228641 3 0.1114 0.7287 0.004 0.008 0.972 0.016
#> GSM228644 2 0.5589 0.5920 0.004 0.724 0.080 0.192
#> GSM228651 3 0.0779 0.7270 0.000 0.016 0.980 0.004
#> GSM228654 3 0.1151 0.7330 0.000 0.008 0.968 0.024
#> GSM228658 3 0.2365 0.7140 0.004 0.064 0.920 0.012
#> GSM228606 4 0.7591 0.2049 0.000 0.208 0.340 0.452
#> GSM228611 3 0.3498 0.7239 0.000 0.160 0.832 0.008
#> GSM228618 3 0.3074 0.7210 0.000 0.152 0.848 0.000
#> GSM228621 3 0.4969 0.7137 0.000 0.140 0.772 0.088
#> GSM228624 2 0.7006 -0.0167 0.000 0.456 0.428 0.116
#> GSM228630 2 0.7795 0.1610 0.000 0.420 0.268 0.312
#> GSM228636 4 0.5269 -0.0113 0.004 0.428 0.004 0.564
#> GSM228638 3 0.4791 0.7180 0.000 0.136 0.784 0.080
#> GSM228648 3 0.6685 0.4495 0.000 0.324 0.568 0.108
#> GSM228670 4 0.2694 0.5573 0.044 0.024 0.016 0.916
#> GSM228671 4 0.7732 0.3331 0.028 0.168 0.248 0.556
#> GSM228672 4 0.5422 0.5156 0.136 0.100 0.008 0.756
#> GSM228674 4 0.2676 0.5600 0.044 0.028 0.012 0.916
#> GSM228675 4 0.3283 0.5591 0.044 0.044 0.020 0.892
#> GSM228676 4 0.5673 0.4326 0.000 0.052 0.288 0.660
#> GSM228667 4 0.6889 0.4366 0.020 0.156 0.176 0.648
#> GSM228668 4 0.6914 0.4857 0.180 0.088 0.060 0.672
#> GSM228669 4 0.3004 0.5604 0.008 0.100 0.008 0.884
#> GSM228673 3 0.7023 0.5535 0.000 0.232 0.576 0.192
#> GSM228677 4 0.5636 0.5126 0.004 0.236 0.060 0.700
#> GSM228678 4 0.3972 0.5123 0.000 0.204 0.008 0.788
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 4 0.7545 0.19225 0.016 0.056 0.132 0.472 0.324
#> GSM228563 4 0.2407 0.61962 0.000 0.012 0.004 0.896 0.088
#> GSM228565 3 0.7699 0.32174 0.020 0.044 0.408 0.156 0.372
#> GSM228566 3 0.4715 0.69227 0.000 0.012 0.728 0.048 0.212
#> GSM228567 1 0.0162 0.73702 0.996 0.000 0.000 0.000 0.004
#> GSM228570 1 0.7232 0.53932 0.572 0.068 0.012 0.176 0.172
#> GSM228571 1 0.5916 0.43103 0.536 0.020 0.036 0.012 0.396
#> GSM228574 3 0.5138 0.67897 0.000 0.016 0.676 0.048 0.260
#> GSM228575 3 0.5283 0.69593 0.000 0.020 0.704 0.084 0.192
#> GSM228576 3 0.7661 0.42797 0.060 0.068 0.448 0.052 0.372
#> GSM228579 1 0.5414 0.46340 0.564 0.008 0.024 0.012 0.392
#> GSM228580 4 0.5564 0.44005 0.000 0.016 0.112 0.676 0.196
#> GSM228581 5 0.5005 0.22301 0.000 0.056 0.208 0.020 0.716
#> GSM228666 5 0.6732 0.00243 0.000 0.032 0.332 0.128 0.508
#> GSM228564 4 0.1885 0.63526 0.020 0.012 0.000 0.936 0.032
#> GSM228568 5 0.6395 0.22156 0.152 0.008 0.236 0.012 0.592
#> GSM228569 1 0.6820 0.45013 0.508 0.004 0.272 0.012 0.204
#> GSM228572 4 0.7157 0.08694 0.000 0.292 0.036 0.476 0.196
#> GSM228573 3 0.1197 0.70126 0.000 0.048 0.952 0.000 0.000
#> GSM228577 1 0.6459 0.56825 0.576 0.004 0.184 0.012 0.224
#> GSM228578 3 0.7541 0.37722 0.208 0.064 0.572 0.100 0.056
#> GSM228663 3 0.5252 0.45089 0.000 0.036 0.624 0.016 0.324
#> GSM228664 3 0.5353 0.26590 0.000 0.036 0.568 0.012 0.384
#> GSM228665 3 0.3214 0.68597 0.000 0.032 0.856 0.008 0.104
#> GSM228582 5 0.2958 0.28114 0.032 0.012 0.060 0.008 0.888
#> GSM228583 1 0.0162 0.73702 0.996 0.000 0.000 0.000 0.004
#> GSM228585 1 0.0290 0.73718 0.992 0.000 0.000 0.000 0.008
#> GSM228587 1 0.5070 0.63391 0.712 0.004 0.000 0.124 0.160
#> GSM228588 5 0.5310 0.22402 0.012 0.044 0.000 0.328 0.616
#> GSM228589 5 0.5698 0.13925 0.000 0.148 0.004 0.208 0.640
#> GSM228590 1 0.0162 0.73702 0.996 0.000 0.000 0.000 0.004
#> GSM228591 5 0.3412 0.17933 0.000 0.096 0.008 0.048 0.848
#> GSM228597 4 0.1943 0.63162 0.000 0.056 0.000 0.924 0.020
#> GSM228601 5 0.5558 0.16233 0.000 0.112 0.000 0.268 0.620
#> GSM228604 3 0.5400 0.59660 0.000 0.012 0.572 0.040 0.376
#> GSM228608 1 0.4862 0.69837 0.792 0.072 0.020 0.060 0.056
#> GSM228609 4 0.5253 0.13979 0.024 0.016 0.000 0.564 0.396
#> GSM228613 1 0.0566 0.73979 0.984 0.000 0.000 0.004 0.012
#> GSM228616 3 0.7339 0.23198 0.020 0.008 0.428 0.228 0.316
#> GSM228628 5 0.4818 0.22322 0.000 0.072 0.120 0.040 0.768
#> GSM228634 1 0.1704 0.74282 0.928 0.000 0.004 0.000 0.068
#> GSM228642 2 0.5701 0.47994 0.000 0.504 0.032 0.028 0.436
#> GSM228645 3 0.5418 0.48322 0.000 0.020 0.504 0.024 0.452
#> GSM228646 3 0.5292 0.49710 0.000 0.008 0.508 0.032 0.452
#> GSM228652 1 0.6705 0.49429 0.576 0.040 0.004 0.252 0.128
#> GSM228655 3 0.8575 0.21401 0.136 0.032 0.436 0.228 0.168
#> GSM228656 1 0.0162 0.73702 0.996 0.000 0.000 0.000 0.004
#> GSM228659 4 0.6652 0.11284 0.316 0.012 0.000 0.496 0.176
#> GSM228662 1 0.0404 0.73967 0.988 0.000 0.000 0.000 0.012
#> GSM228584 1 0.2621 0.72958 0.876 0.008 0.112 0.004 0.000
#> GSM228586 1 0.3500 0.72201 0.832 0.008 0.136 0.004 0.020
#> GSM228592 1 0.2770 0.72565 0.864 0.008 0.124 0.004 0.000
#> GSM228593 5 0.5300 0.19025 0.008 0.020 0.008 0.428 0.536
#> GSM228594 1 0.5860 0.63376 0.648 0.004 0.156 0.008 0.184
#> GSM228598 1 0.6263 0.64611 0.664 0.020 0.136 0.028 0.152
#> GSM228607 3 0.6782 0.56667 0.000 0.128 0.612 0.148 0.112
#> GSM228612 3 0.4507 0.63516 0.000 0.024 0.756 0.032 0.188
#> GSM228619 4 0.7088 0.26966 0.008 0.160 0.332 0.476 0.024
#> GSM228622 3 0.7212 0.50731 0.152 0.088 0.612 0.116 0.032
#> GSM228625 4 0.8223 0.30814 0.100 0.128 0.044 0.488 0.240
#> GSM228631 3 0.5476 0.64466 0.020 0.088 0.732 0.136 0.024
#> GSM228633 2 0.5663 0.55996 0.000 0.688 0.064 0.056 0.192
#> GSM228637 4 0.4291 0.58919 0.000 0.188 0.004 0.760 0.048
#> GSM228639 3 0.6322 0.57241 0.000 0.120 0.640 0.180 0.060
#> GSM228649 5 0.6701 0.09061 0.000 0.152 0.020 0.332 0.496
#> GSM228660 5 0.6835 0.27186 0.060 0.016 0.204 0.108 0.612
#> GSM228661 1 0.6561 0.51702 0.556 0.004 0.248 0.012 0.180
#> GSM228595 2 0.5405 0.62419 0.000 0.672 0.004 0.124 0.200
#> GSM228599 4 0.2511 0.62790 0.000 0.004 0.016 0.892 0.088
#> GSM228602 3 0.3627 0.71371 0.000 0.032 0.832 0.016 0.120
#> GSM228614 4 0.4389 0.50365 0.000 0.020 0.176 0.768 0.036
#> GSM228626 2 0.5405 0.66409 0.000 0.596 0.000 0.076 0.328
#> GSM228640 3 0.4206 0.70556 0.000 0.028 0.784 0.024 0.164
#> GSM228643 3 0.3882 0.69352 0.000 0.012 0.776 0.012 0.200
#> GSM228650 3 0.5229 0.69778 0.000 0.020 0.708 0.080 0.192
#> GSM228653 3 0.3953 0.69432 0.000 0.048 0.784 0.000 0.168
#> GSM228657 2 0.6724 0.34556 0.000 0.392 0.000 0.356 0.252
#> GSM228605 3 0.5730 0.60331 0.004 0.108 0.676 0.192 0.020
#> GSM228610 3 0.0963 0.70116 0.000 0.036 0.964 0.000 0.000
#> GSM228617 3 0.2529 0.71342 0.000 0.040 0.900 0.056 0.004
#> GSM228620 3 0.1461 0.71076 0.000 0.028 0.952 0.016 0.004
#> GSM228623 4 0.6357 0.52387 0.000 0.128 0.132 0.652 0.088
#> GSM228629 3 0.1430 0.70129 0.000 0.052 0.944 0.004 0.000
#> GSM228632 3 0.3256 0.71131 0.000 0.012 0.864 0.060 0.064
#> GSM228635 4 0.4085 0.57972 0.000 0.208 0.004 0.760 0.028
#> GSM228647 3 0.0794 0.70345 0.000 0.028 0.972 0.000 0.000
#> GSM228596 3 0.5435 0.69318 0.000 0.016 0.696 0.128 0.160
#> GSM228600 3 0.4003 0.69455 0.000 0.036 0.780 0.004 0.180
#> GSM228603 3 0.3804 0.69086 0.000 0.044 0.796 0.000 0.160
#> GSM228615 4 0.1461 0.63344 0.004 0.016 0.000 0.952 0.028
#> GSM228627 3 0.5444 0.66678 0.000 0.052 0.640 0.020 0.288
#> GSM228641 3 0.3684 0.69551 0.000 0.024 0.800 0.004 0.172
#> GSM228644 2 0.5368 0.66227 0.000 0.596 0.000 0.072 0.332
#> GSM228651 3 0.3565 0.69937 0.000 0.040 0.816 0.000 0.144
#> GSM228654 3 0.4506 0.70348 0.000 0.052 0.764 0.016 0.168
#> GSM228658 3 0.4843 0.69977 0.000 0.048 0.720 0.016 0.216
#> GSM228606 3 0.4833 0.66528 0.000 0.040 0.760 0.144 0.056
#> GSM228611 3 0.1205 0.70475 0.000 0.040 0.956 0.004 0.000
#> GSM228618 3 0.1502 0.70123 0.000 0.056 0.940 0.004 0.000
#> GSM228621 3 0.1569 0.71814 0.000 0.012 0.948 0.032 0.008
#> GSM228624 3 0.4305 0.65677 0.000 0.020 0.784 0.044 0.152
#> GSM228630 3 0.5633 0.64523 0.000 0.080 0.716 0.108 0.096
#> GSM228636 4 0.5106 0.54513 0.000 0.216 0.004 0.692 0.088
#> GSM228638 3 0.1836 0.71757 0.000 0.016 0.936 0.040 0.008
#> GSM228648 3 0.3352 0.69999 0.000 0.012 0.852 0.036 0.100
#> GSM228670 4 0.1428 0.63858 0.004 0.012 0.004 0.956 0.024
#> GSM228671 3 0.6344 0.36521 0.000 0.028 0.504 0.384 0.084
#> GSM228672 4 0.4892 0.50949 0.124 0.016 0.000 0.748 0.112
#> GSM228674 4 0.0981 0.63754 0.008 0.008 0.000 0.972 0.012
#> GSM228675 4 0.0833 0.63648 0.004 0.004 0.000 0.976 0.016
#> GSM228676 3 0.7540 0.33014 0.004 0.044 0.412 0.340 0.200
#> GSM228667 4 0.6514 0.26989 0.000 0.016 0.168 0.548 0.268
#> GSM228668 4 0.8883 0.09647 0.236 0.120 0.296 0.316 0.032
#> GSM228669 4 0.4935 0.61678 0.012 0.124 0.052 0.772 0.040
#> GSM228673 3 0.2515 0.71716 0.000 0.020 0.908 0.040 0.032
#> GSM228677 4 0.6497 0.47551 0.000 0.084 0.216 0.616 0.084
#> GSM228678 4 0.4455 0.61483 0.000 0.128 0.044 0.788 0.040
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 5 0.7367 -0.0782 0.020 0.004 0.220 0.324 0.380 0.052
#> GSM228563 4 0.2886 0.5290 0.000 0.032 0.004 0.876 0.028 0.060
#> GSM228565 5 0.7103 0.2481 0.036 0.008 0.372 0.100 0.432 0.052
#> GSM228566 3 0.2828 0.6347 0.000 0.004 0.872 0.032 0.080 0.012
#> GSM228567 1 0.0291 0.7144 0.992 0.000 0.000 0.004 0.004 0.000
#> GSM228570 1 0.7756 0.1892 0.376 0.000 0.056 0.188 0.312 0.068
#> GSM228571 1 0.7063 0.2909 0.452 0.004 0.172 0.008 0.296 0.068
#> GSM228574 3 0.3584 0.6241 0.000 0.016 0.836 0.040 0.084 0.024
#> GSM228575 3 0.3238 0.6318 0.000 0.008 0.856 0.056 0.060 0.020
#> GSM228576 3 0.7223 -0.2510 0.084 0.008 0.404 0.048 0.396 0.060
#> GSM228579 1 0.7211 0.3184 0.468 0.016 0.168 0.004 0.268 0.076
#> GSM228580 4 0.5522 0.3867 0.000 0.052 0.176 0.680 0.072 0.020
#> GSM228581 3 0.7676 -0.3239 0.000 0.108 0.352 0.032 0.352 0.156
#> GSM228666 3 0.7232 0.1337 0.000 0.100 0.524 0.096 0.220 0.060
#> GSM228564 4 0.2954 0.5484 0.016 0.000 0.004 0.868 0.072 0.040
#> GSM228568 5 0.6499 0.0906 0.092 0.024 0.048 0.016 0.596 0.224
#> GSM228569 1 0.6574 0.3957 0.496 0.004 0.096 0.004 0.324 0.076
#> GSM228572 4 0.5930 0.0672 0.000 0.416 0.024 0.480 0.048 0.032
#> GSM228573 3 0.3219 0.6497 0.000 0.008 0.808 0.000 0.168 0.016
#> GSM228577 1 0.6362 0.4681 0.508 0.004 0.040 0.008 0.332 0.108
#> GSM228578 5 0.8059 0.2608 0.204 0.000 0.228 0.084 0.400 0.084
#> GSM228663 5 0.5979 -0.0938 0.000 0.056 0.392 0.016 0.496 0.040
#> GSM228664 5 0.6815 0.0731 0.000 0.088 0.336 0.020 0.472 0.084
#> GSM228665 3 0.4052 0.5839 0.000 0.008 0.692 0.004 0.284 0.012
#> GSM228582 5 0.7100 0.0410 0.020 0.036 0.184 0.012 0.472 0.276
#> GSM228583 1 0.0146 0.7136 0.996 0.000 0.000 0.004 0.000 0.000
#> GSM228585 1 0.0146 0.7127 0.996 0.000 0.004 0.000 0.000 0.000
#> GSM228587 1 0.5184 0.6133 0.720 0.012 0.000 0.120 0.080 0.068
#> GSM228588 6 0.6094 0.7356 0.012 0.084 0.000 0.296 0.048 0.560
#> GSM228589 6 0.6380 0.6143 0.000 0.168 0.000 0.172 0.092 0.568
#> GSM228590 1 0.0146 0.7136 0.996 0.000 0.000 0.004 0.000 0.000
#> GSM228591 5 0.7933 -0.1575 0.000 0.152 0.144 0.040 0.372 0.292
#> GSM228597 4 0.1893 0.5692 0.000 0.024 0.004 0.928 0.008 0.036
#> GSM228601 6 0.6787 0.6933 0.004 0.136 0.024 0.260 0.048 0.528
#> GSM228604 3 0.4060 0.6033 0.000 0.048 0.804 0.052 0.088 0.008
#> GSM228608 1 0.5372 0.4703 0.632 0.000 0.004 0.056 0.264 0.044
#> GSM228609 4 0.6286 -0.4011 0.016 0.036 0.000 0.476 0.092 0.380
#> GSM228613 1 0.0260 0.7142 0.992 0.000 0.000 0.008 0.000 0.000
#> GSM228616 3 0.8012 -0.2456 0.012 0.028 0.348 0.248 0.276 0.088
#> GSM228628 5 0.8091 0.0919 0.000 0.148 0.272 0.036 0.336 0.208
#> GSM228634 1 0.1801 0.7120 0.932 0.004 0.012 0.000 0.040 0.012
#> GSM228642 2 0.5728 0.5739 0.000 0.672 0.152 0.044 0.104 0.028
#> GSM228645 3 0.5662 0.3428 0.000 0.052 0.628 0.028 0.256 0.036
#> GSM228646 3 0.5293 0.4062 0.000 0.032 0.660 0.028 0.244 0.036
#> GSM228652 1 0.7256 0.2327 0.416 0.000 0.012 0.260 0.240 0.072
#> GSM228655 5 0.8656 0.1561 0.220 0.004 0.172 0.224 0.304 0.076
#> GSM228656 1 0.0146 0.7136 0.996 0.000 0.000 0.004 0.000 0.000
#> GSM228659 4 0.7112 -0.0409 0.296 0.004 0.000 0.424 0.188 0.088
#> GSM228662 1 0.0260 0.7142 0.992 0.000 0.000 0.008 0.000 0.000
#> GSM228584 1 0.2365 0.6992 0.888 0.000 0.000 0.000 0.040 0.072
#> GSM228586 1 0.3564 0.6882 0.824 0.004 0.012 0.000 0.076 0.084
#> GSM228592 1 0.2575 0.6965 0.880 0.000 0.004 0.000 0.044 0.072
#> GSM228593 6 0.6354 0.6956 0.012 0.060 0.000 0.316 0.088 0.524
#> GSM228594 1 0.5539 0.5867 0.612 0.008 0.012 0.000 0.248 0.120
#> GSM228598 1 0.6219 0.5696 0.592 0.008 0.004 0.040 0.180 0.176
#> GSM228607 3 0.7350 0.4061 0.004 0.048 0.520 0.144 0.200 0.084
#> GSM228612 3 0.5348 0.4242 0.000 0.036 0.556 0.020 0.372 0.016
#> GSM228619 4 0.7968 0.1932 0.012 0.024 0.140 0.392 0.280 0.152
#> GSM228622 5 0.8134 0.1809 0.104 0.000 0.292 0.120 0.376 0.108
#> GSM228625 4 0.8379 0.0830 0.112 0.052 0.012 0.348 0.220 0.256
#> GSM228631 3 0.6927 0.2326 0.008 0.000 0.468 0.116 0.304 0.104
#> GSM228633 2 0.3867 0.6043 0.000 0.808 0.004 0.072 0.092 0.024
#> GSM228637 4 0.4318 0.5379 0.000 0.084 0.004 0.756 0.012 0.144
#> GSM228639 3 0.7110 0.4417 0.004 0.052 0.560 0.180 0.112 0.092
#> GSM228649 6 0.6355 0.4444 0.000 0.104 0.004 0.284 0.072 0.536
#> GSM228660 5 0.7188 -0.0880 0.048 0.032 0.028 0.096 0.500 0.296
#> GSM228661 1 0.6657 0.4196 0.508 0.004 0.100 0.004 0.296 0.088
#> GSM228595 2 0.2681 0.6518 0.000 0.880 0.000 0.072 0.028 0.020
#> GSM228599 4 0.3428 0.5636 0.004 0.012 0.076 0.844 0.056 0.008
#> GSM228602 3 0.1983 0.6707 0.000 0.012 0.924 0.012 0.044 0.008
#> GSM228614 4 0.4781 0.4531 0.000 0.020 0.140 0.740 0.080 0.020
#> GSM228626 2 0.3188 0.7107 0.000 0.852 0.076 0.032 0.040 0.000
#> GSM228640 3 0.2308 0.6483 0.000 0.016 0.912 0.012 0.028 0.032
#> GSM228643 3 0.1925 0.6418 0.000 0.008 0.920 0.004 0.060 0.008
#> GSM228650 3 0.2806 0.6496 0.000 0.004 0.876 0.068 0.040 0.012
#> GSM228653 3 0.1518 0.6540 0.000 0.008 0.944 0.000 0.024 0.024
#> GSM228657 2 0.5655 0.3136 0.000 0.576 0.032 0.328 0.036 0.028
#> GSM228605 3 0.7539 -0.0656 0.004 0.008 0.356 0.164 0.344 0.124
#> GSM228610 3 0.3191 0.6475 0.000 0.012 0.812 0.000 0.164 0.012
#> GSM228617 3 0.4386 0.6384 0.000 0.008 0.736 0.060 0.188 0.008
#> GSM228620 3 0.3444 0.6480 0.000 0.004 0.800 0.020 0.168 0.008
#> GSM228623 4 0.5925 0.4982 0.000 0.052 0.076 0.680 0.100 0.092
#> GSM228629 3 0.3386 0.6450 0.000 0.012 0.796 0.000 0.176 0.016
#> GSM228632 3 0.4591 0.6446 0.000 0.012 0.740 0.060 0.168 0.020
#> GSM228635 4 0.4430 0.5086 0.000 0.108 0.000 0.732 0.008 0.152
#> GSM228647 3 0.2920 0.6477 0.000 0.008 0.820 0.000 0.168 0.004
#> GSM228596 3 0.3866 0.6182 0.000 0.004 0.800 0.092 0.092 0.012
#> GSM228600 3 0.1767 0.6522 0.000 0.020 0.932 0.000 0.036 0.012
#> GSM228603 3 0.1944 0.6504 0.000 0.016 0.924 0.000 0.024 0.036
#> GSM228615 4 0.1307 0.5650 0.008 0.000 0.008 0.952 0.000 0.032
#> GSM228627 3 0.3489 0.6032 0.000 0.020 0.820 0.008 0.132 0.020
#> GSM228641 3 0.1944 0.6467 0.000 0.016 0.924 0.000 0.024 0.036
#> GSM228644 2 0.3416 0.7014 0.000 0.832 0.100 0.028 0.040 0.000
#> GSM228651 3 0.1312 0.6636 0.000 0.008 0.956 0.004 0.020 0.012
#> GSM228654 3 0.1592 0.6649 0.000 0.004 0.944 0.016 0.024 0.012
#> GSM228658 3 0.2302 0.6581 0.000 0.016 0.912 0.012 0.036 0.024
#> GSM228606 3 0.6129 0.5620 0.000 0.020 0.616 0.124 0.192 0.048
#> GSM228611 3 0.3219 0.6468 0.000 0.008 0.808 0.000 0.168 0.016
#> GSM228618 3 0.3164 0.6471 0.000 0.008 0.804 0.004 0.180 0.004
#> GSM228621 3 0.3800 0.6546 0.000 0.012 0.792 0.028 0.156 0.012
#> GSM228624 3 0.5482 0.5435 0.000 0.024 0.620 0.040 0.284 0.032
#> GSM228630 3 0.6289 0.5638 0.000 0.056 0.632 0.124 0.144 0.044
#> GSM228636 4 0.4513 0.5034 0.000 0.116 0.000 0.724 0.008 0.152
#> GSM228638 3 0.3602 0.6499 0.000 0.000 0.784 0.032 0.176 0.008
#> GSM228648 3 0.4433 0.6456 0.000 0.024 0.748 0.040 0.176 0.012
#> GSM228670 4 0.1065 0.5774 0.008 0.000 0.008 0.964 0.020 0.000
#> GSM228671 3 0.6158 0.2937 0.000 0.040 0.516 0.360 0.048 0.036
#> GSM228672 4 0.5449 0.3585 0.072 0.000 0.000 0.660 0.192 0.076
#> GSM228674 4 0.1452 0.5730 0.008 0.004 0.000 0.948 0.032 0.008
#> GSM228675 4 0.1223 0.5715 0.008 0.004 0.016 0.960 0.000 0.012
#> GSM228676 3 0.6413 -0.1098 0.000 0.000 0.452 0.280 0.244 0.024
#> GSM228667 4 0.6689 0.1304 0.000 0.012 0.188 0.424 0.348 0.028
#> GSM228668 5 0.8748 0.0320 0.176 0.008 0.100 0.236 0.328 0.152
#> GSM228669 4 0.6052 0.4086 0.004 0.024 0.000 0.544 0.276 0.152
#> GSM228673 3 0.4116 0.6444 0.000 0.004 0.748 0.032 0.200 0.016
#> GSM228677 4 0.6286 0.4295 0.000 0.096 0.104 0.644 0.112 0.044
#> GSM228678 4 0.4476 0.5482 0.004 0.060 0.016 0.788 0.056 0.076
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
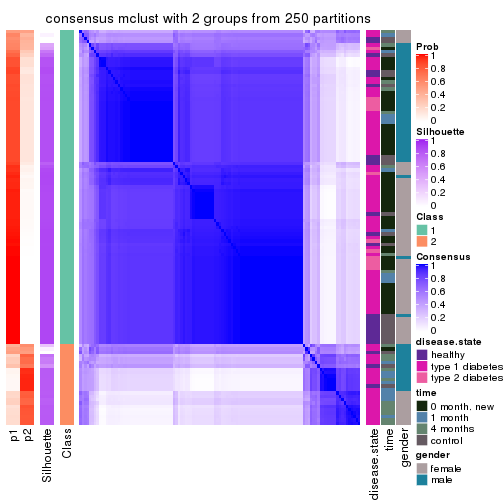
consensus_heatmap(res, k = 3)
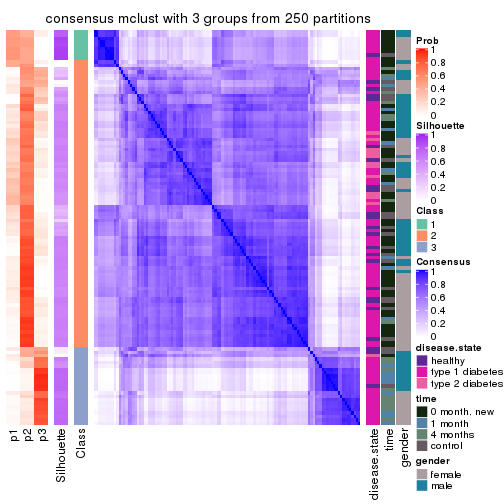
consensus_heatmap(res, k = 4)
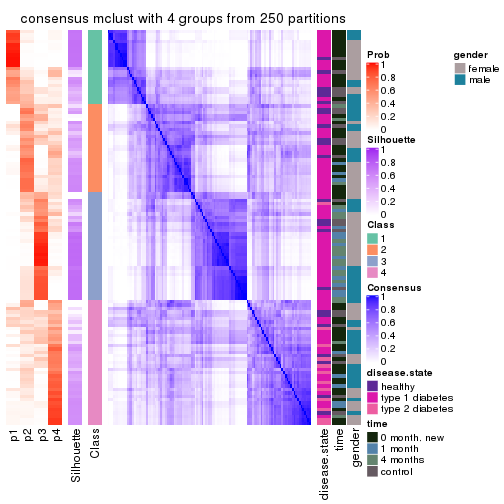
consensus_heatmap(res, k = 5)
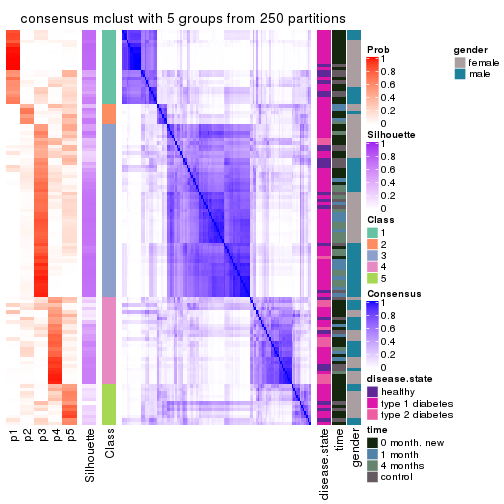
consensus_heatmap(res, k = 6)
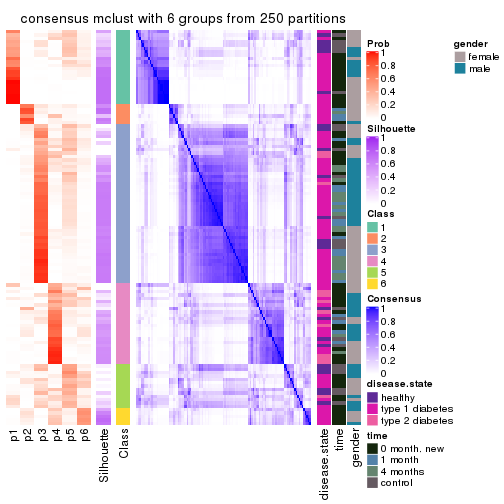
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
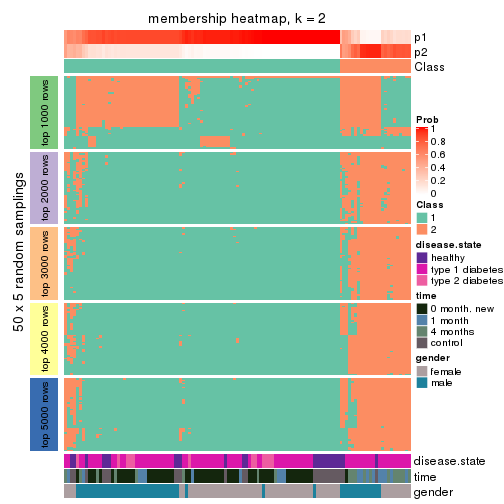
membership_heatmap(res, k = 3)
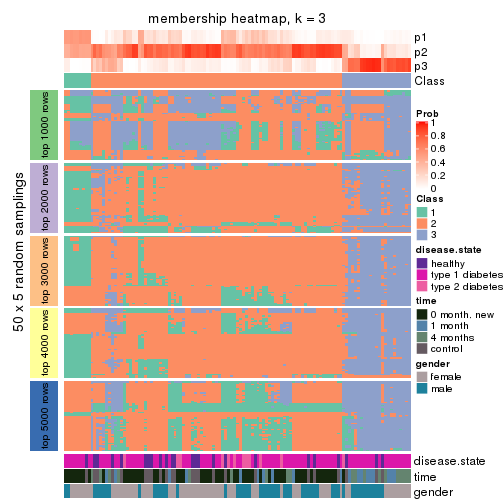
membership_heatmap(res, k = 4)
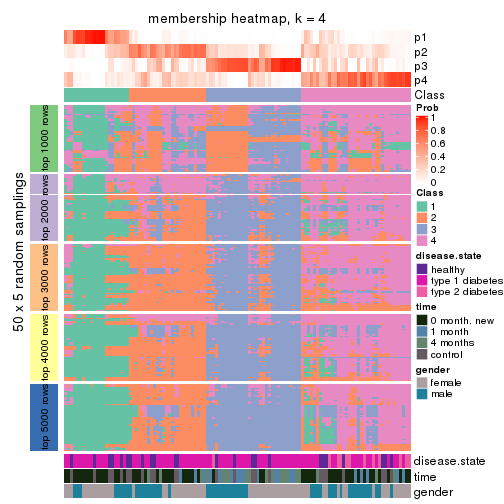
membership_heatmap(res, k = 5)
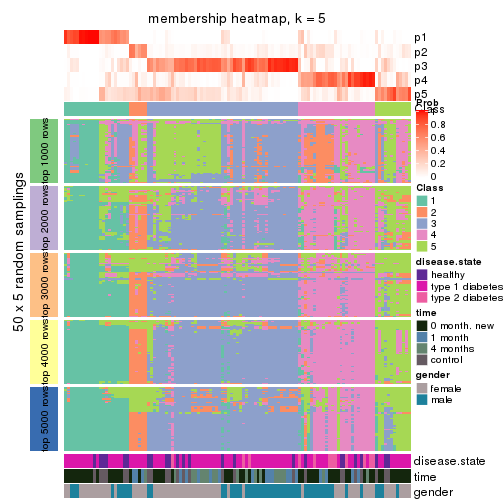
membership_heatmap(res, k = 6)
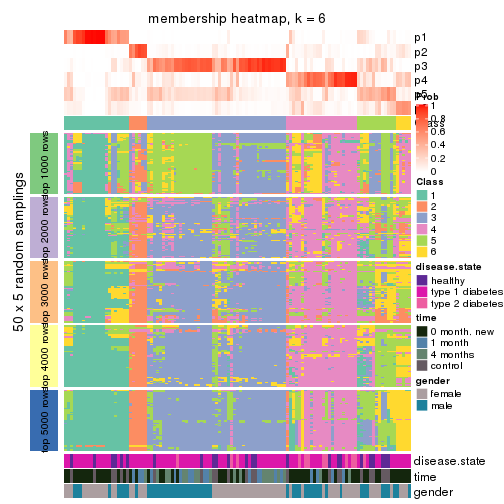
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
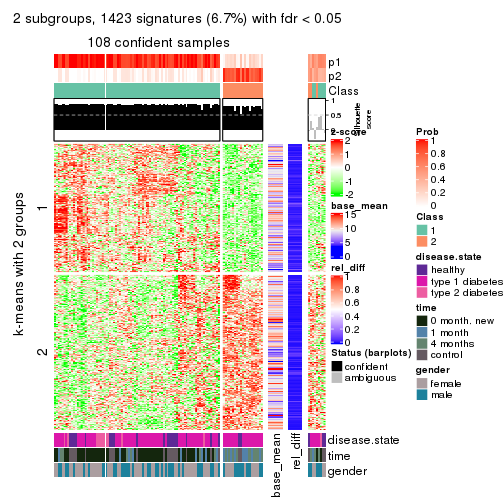
get_signatures(res, k = 3)
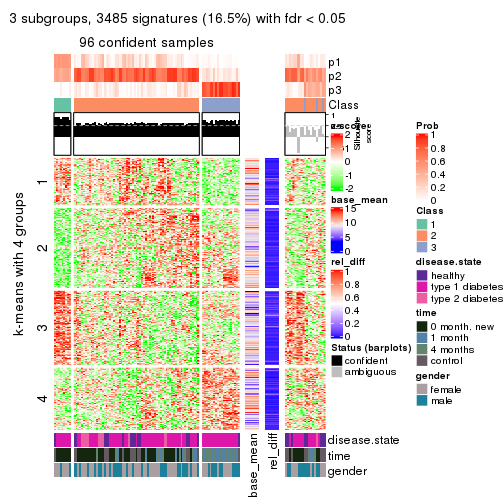
get_signatures(res, k = 4)
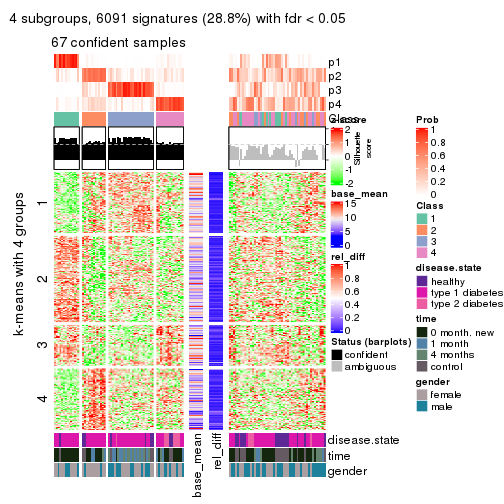
get_signatures(res, k = 5)
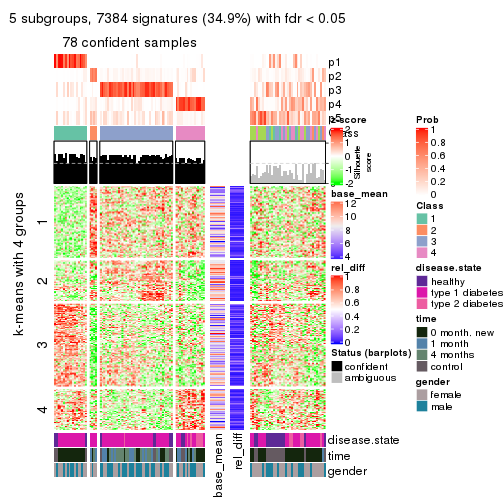
get_signatures(res, k = 6)
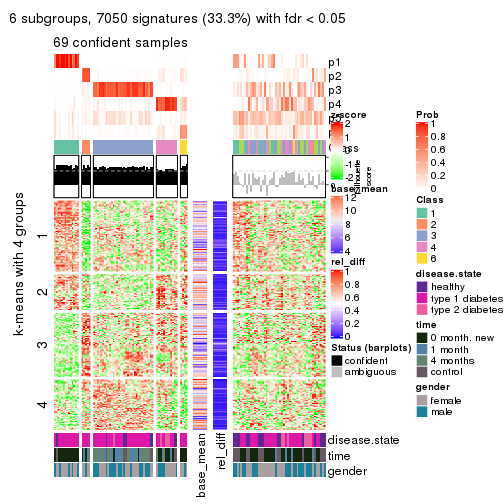
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
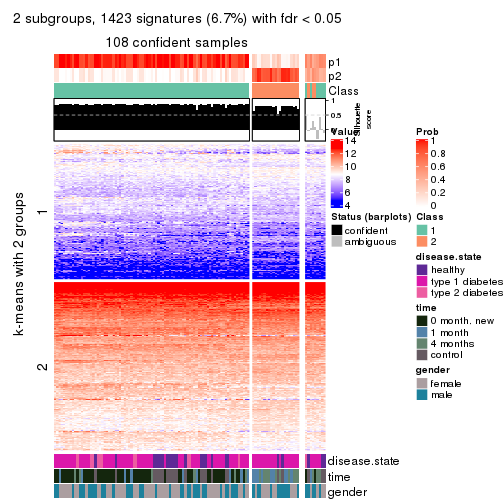
get_signatures(res, k = 3, scale_rows = FALSE)
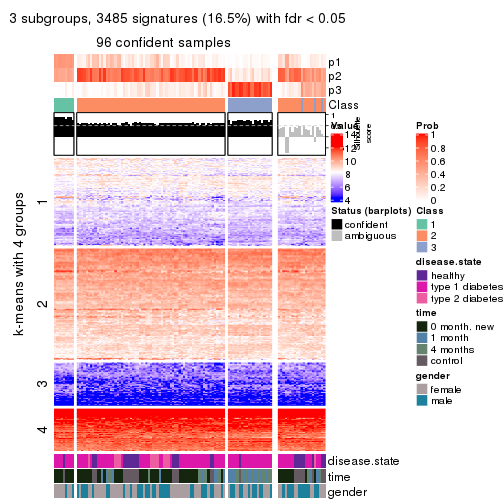
get_signatures(res, k = 4, scale_rows = FALSE)
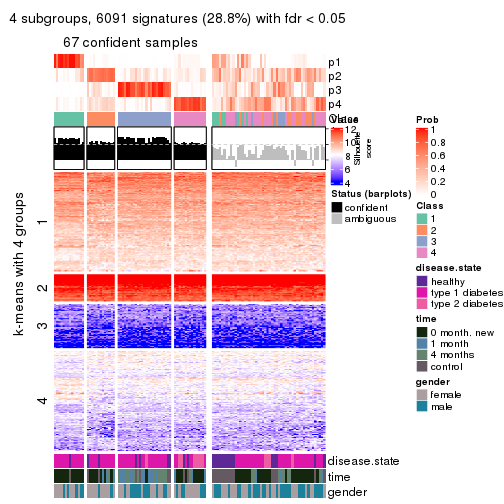
get_signatures(res, k = 5, scale_rows = FALSE)
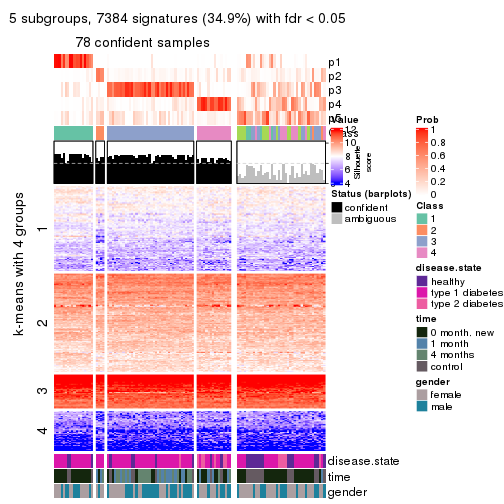
get_signatures(res, k = 6, scale_rows = FALSE)
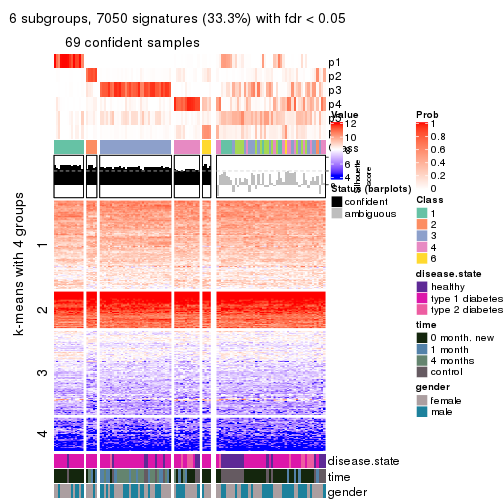
Compare the overlap of signatures from different k:
compare_signatures(res)
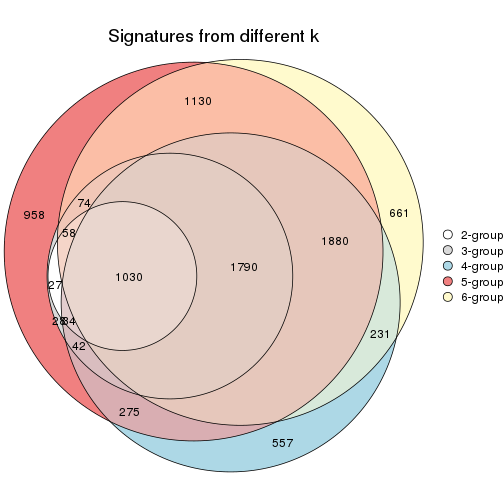
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
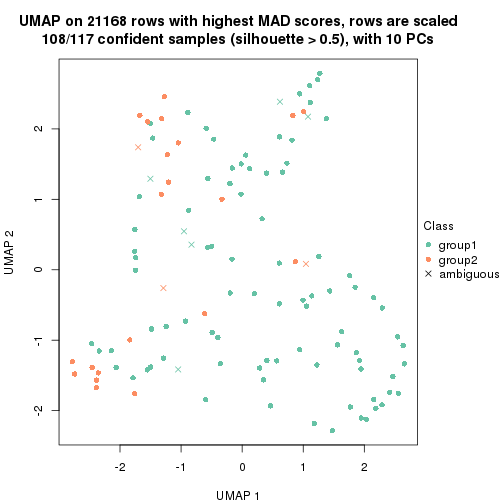
dimension_reduction(res, k = 3, method = "UMAP")
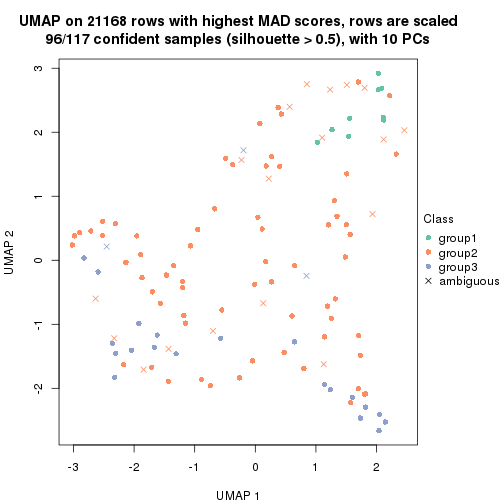
dimension_reduction(res, k = 4, method = "UMAP")
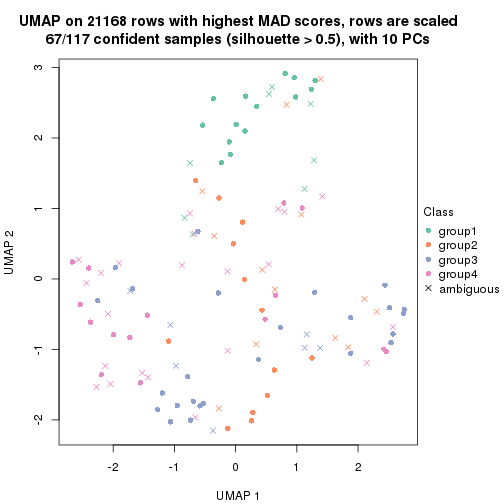
dimension_reduction(res, k = 5, method = "UMAP")
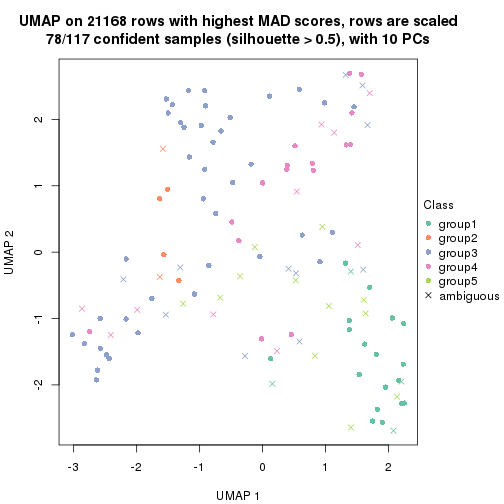
dimension_reduction(res, k = 6, method = "UMAP")
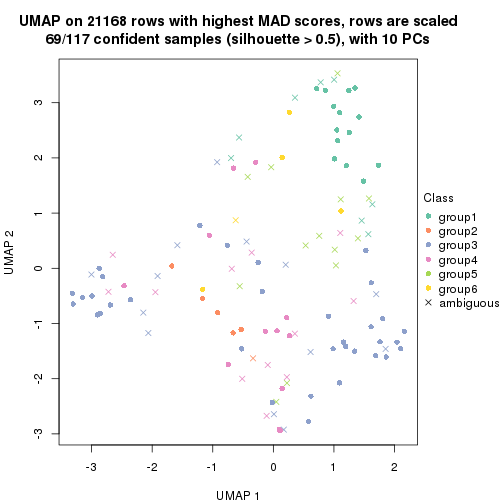
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
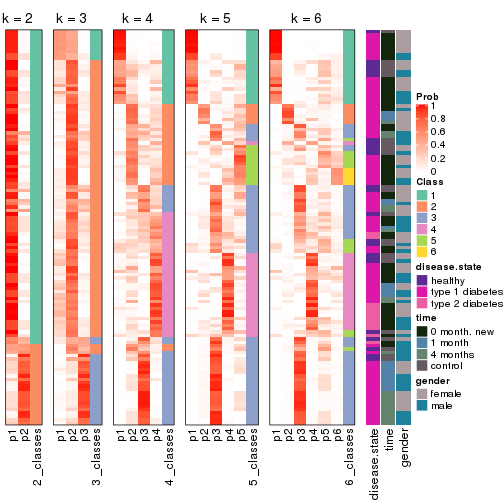
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> MAD:mclust 108 0.064823 1.68e-09 0.504 2
#> MAD:mclust 96 0.041838 1.18e-07 0.370 3
#> MAD:mclust 67 0.000627 5.07e-05 0.266 4
#> MAD:mclust 78 0.001660 3.54e-04 0.393 5
#> MAD:mclust 69 0.023508 2.46e-05 0.629 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["MAD", "NMF"]
# you can also extract it by
# res = res_list["MAD:NMF"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'MAD' method.
#> Subgroups are detected by 'NMF' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
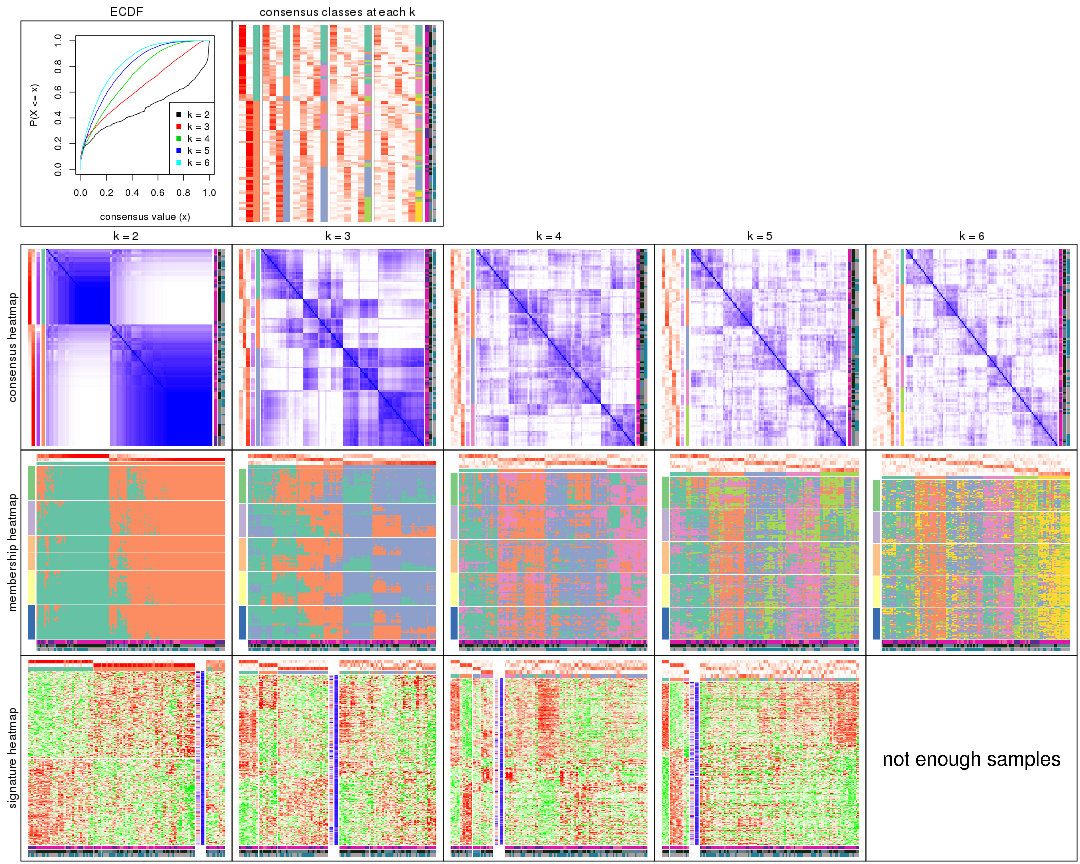
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
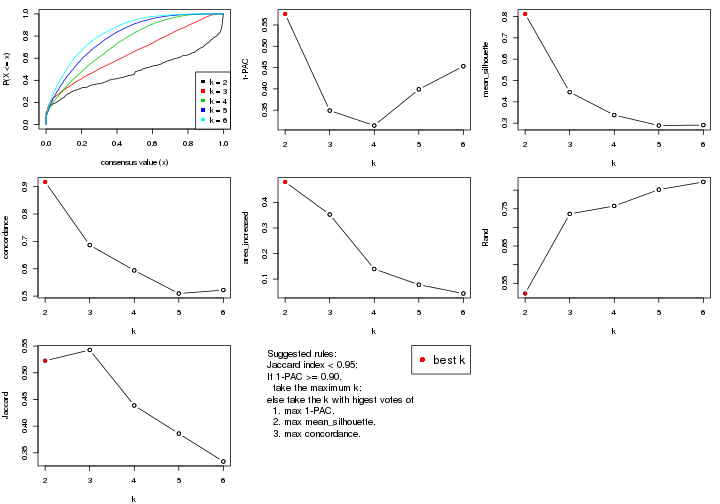
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.576 0.812 0.917 0.4800 0.523 0.523
#> 3 3 0.349 0.446 0.686 0.3527 0.736 0.543
#> 4 4 0.314 0.338 0.594 0.1396 0.758 0.439
#> 5 5 0.399 0.289 0.509 0.0782 0.802 0.386
#> 6 6 0.453 0.291 0.522 0.0436 0.822 0.334
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.4161 0.86850 0.916 0.084
#> GSM228563 2 0.4022 0.86292 0.080 0.920
#> GSM228565 1 0.7950 0.69783 0.760 0.240
#> GSM228566 2 0.1633 0.89610 0.024 0.976
#> GSM228567 1 0.0000 0.90995 1.000 0.000
#> GSM228570 1 0.0376 0.90916 0.996 0.004
#> GSM228571 1 0.3114 0.88506 0.944 0.056
#> GSM228574 2 0.0000 0.90290 0.000 1.000
#> GSM228575 2 0.0672 0.90132 0.008 0.992
#> GSM228576 1 0.7219 0.74736 0.800 0.200
#> GSM228579 1 0.4298 0.86312 0.912 0.088
#> GSM228580 2 0.0000 0.90290 0.000 1.000
#> GSM228581 2 0.0000 0.90290 0.000 1.000
#> GSM228666 2 0.0000 0.90290 0.000 1.000
#> GSM228564 1 0.9170 0.48606 0.668 0.332
#> GSM228568 1 0.8016 0.69114 0.756 0.244
#> GSM228569 1 0.1184 0.90507 0.984 0.016
#> GSM228572 2 0.0000 0.90290 0.000 1.000
#> GSM228573 2 0.9286 0.49064 0.344 0.656
#> GSM228577 1 0.0000 0.90995 1.000 0.000
#> GSM228578 1 0.0376 0.90919 0.996 0.004
#> GSM228663 2 0.9460 0.43457 0.364 0.636
#> GSM228664 2 0.0000 0.90290 0.000 1.000
#> GSM228665 2 0.7883 0.69257 0.236 0.764
#> GSM228582 1 0.9358 0.49002 0.648 0.352
#> GSM228583 1 0.0000 0.90995 1.000 0.000
#> GSM228585 1 0.0000 0.90995 1.000 0.000
#> GSM228587 1 0.0000 0.90995 1.000 0.000
#> GSM228588 2 0.8499 0.65289 0.276 0.724
#> GSM228589 2 0.0000 0.90290 0.000 1.000
#> GSM228590 1 0.0000 0.90995 1.000 0.000
#> GSM228591 2 0.0000 0.90290 0.000 1.000
#> GSM228597 2 0.6048 0.79981 0.148 0.852
#> GSM228601 2 0.0000 0.90290 0.000 1.000
#> GSM228604 2 0.0000 0.90290 0.000 1.000
#> GSM228608 1 0.0000 0.90995 1.000 0.000
#> GSM228609 1 0.4815 0.85119 0.896 0.104
#> GSM228613 1 0.0000 0.90995 1.000 0.000
#> GSM228616 2 0.9944 0.16115 0.456 0.544
#> GSM228628 2 0.0000 0.90290 0.000 1.000
#> GSM228634 1 0.0000 0.90995 1.000 0.000
#> GSM228642 2 0.0000 0.90290 0.000 1.000
#> GSM228645 2 0.4022 0.86372 0.080 0.920
#> GSM228646 2 0.2948 0.88325 0.052 0.948
#> GSM228652 1 0.0000 0.90995 1.000 0.000
#> GSM228655 1 0.1633 0.90222 0.976 0.024
#> GSM228656 1 0.0000 0.90995 1.000 0.000
#> GSM228659 1 0.0000 0.90995 1.000 0.000
#> GSM228662 1 0.0000 0.90995 1.000 0.000
#> GSM228584 1 0.0000 0.90995 1.000 0.000
#> GSM228586 1 0.0000 0.90995 1.000 0.000
#> GSM228592 1 0.0000 0.90995 1.000 0.000
#> GSM228593 1 0.5178 0.83982 0.884 0.116
#> GSM228594 1 0.0000 0.90995 1.000 0.000
#> GSM228598 1 0.0000 0.90995 1.000 0.000
#> GSM228607 2 0.4562 0.85522 0.096 0.904
#> GSM228612 2 0.2043 0.89336 0.032 0.968
#> GSM228619 1 0.5629 0.82240 0.868 0.132
#> GSM228622 1 0.0000 0.90995 1.000 0.000
#> GSM228625 1 0.1843 0.90034 0.972 0.028
#> GSM228631 1 0.0376 0.90918 0.996 0.004
#> GSM228633 2 0.0000 0.90290 0.000 1.000
#> GSM228637 2 0.6623 0.77311 0.172 0.828
#> GSM228639 2 0.1184 0.89938 0.016 0.984
#> GSM228649 2 0.5519 0.82442 0.128 0.872
#> GSM228660 1 0.4815 0.85390 0.896 0.104
#> GSM228661 1 0.0000 0.90995 1.000 0.000
#> GSM228595 2 0.0000 0.90290 0.000 1.000
#> GSM228599 2 0.0000 0.90290 0.000 1.000
#> GSM228602 1 0.9881 0.21649 0.564 0.436
#> GSM228614 2 0.0938 0.90035 0.012 0.988
#> GSM228626 2 0.0000 0.90290 0.000 1.000
#> GSM228640 2 0.9754 0.31168 0.408 0.592
#> GSM228643 2 0.3584 0.87195 0.068 0.932
#> GSM228650 2 0.0000 0.90290 0.000 1.000
#> GSM228653 2 0.9248 0.48753 0.340 0.660
#> GSM228657 2 0.0000 0.90290 0.000 1.000
#> GSM228605 1 0.4939 0.84564 0.892 0.108
#> GSM228610 2 0.2603 0.88930 0.044 0.956
#> GSM228617 2 0.9963 0.20085 0.464 0.536
#> GSM228620 2 1.0000 -0.02389 0.500 0.500
#> GSM228623 2 0.2423 0.88871 0.040 0.960
#> GSM228629 2 1.0000 -0.01042 0.496 0.504
#> GSM228632 2 0.0000 0.90290 0.000 1.000
#> GSM228635 2 0.1414 0.89769 0.020 0.980
#> GSM228647 2 0.2423 0.89168 0.040 0.960
#> GSM228596 2 0.6887 0.76351 0.184 0.816
#> GSM228600 2 0.0000 0.90290 0.000 1.000
#> GSM228603 1 1.0000 0.00147 0.500 0.500
#> GSM228615 2 0.3584 0.87045 0.068 0.932
#> GSM228627 2 0.0672 0.90171 0.008 0.992
#> GSM228641 2 0.1184 0.89955 0.016 0.984
#> GSM228644 2 0.0000 0.90290 0.000 1.000
#> GSM228651 2 0.0000 0.90290 0.000 1.000
#> GSM228654 2 0.0000 0.90290 0.000 1.000
#> GSM228658 2 0.6247 0.79265 0.156 0.844
#> GSM228606 2 0.0000 0.90290 0.000 1.000
#> GSM228611 2 0.5737 0.81317 0.136 0.864
#> GSM228618 2 0.6247 0.80746 0.156 0.844
#> GSM228621 2 0.0000 0.90290 0.000 1.000
#> GSM228624 2 0.0000 0.90290 0.000 1.000
#> GSM228630 2 0.0000 0.90290 0.000 1.000
#> GSM228636 2 0.0938 0.90049 0.012 0.988
#> GSM228638 2 0.0376 0.90220 0.004 0.996
#> GSM228648 2 0.0000 0.90290 0.000 1.000
#> GSM228670 2 0.4815 0.84320 0.104 0.896
#> GSM228671 2 0.0000 0.90290 0.000 1.000
#> GSM228672 1 0.0000 0.90995 1.000 0.000
#> GSM228674 2 0.8763 0.61866 0.296 0.704
#> GSM228675 2 0.1184 0.89898 0.016 0.984
#> GSM228676 1 0.9044 0.53358 0.680 0.320
#> GSM228667 2 0.4022 0.86605 0.080 0.920
#> GSM228668 1 0.0000 0.90995 1.000 0.000
#> GSM228669 1 0.1414 0.90386 0.980 0.020
#> GSM228673 2 0.0000 0.90290 0.000 1.000
#> GSM228677 2 0.0000 0.90290 0.000 1.000
#> GSM228678 2 0.0000 0.90290 0.000 1.000
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.839 0.2773 0.516 0.396 0.088
#> GSM228563 2 0.199 0.5932 0.004 0.948 0.048
#> GSM228565 1 0.943 0.0455 0.412 0.412 0.176
#> GSM228566 3 0.353 0.6767 0.092 0.016 0.892
#> GSM228567 1 0.304 0.5981 0.896 0.104 0.000
#> GSM228570 1 0.675 0.2914 0.556 0.432 0.012
#> GSM228571 1 0.514 0.6190 0.832 0.064 0.104
#> GSM228574 3 0.205 0.6730 0.020 0.028 0.952
#> GSM228575 3 0.341 0.6809 0.068 0.028 0.904
#> GSM228576 1 0.677 0.4260 0.664 0.032 0.304
#> GSM228579 1 0.535 0.6134 0.824 0.088 0.088
#> GSM228580 3 0.583 0.3306 0.000 0.340 0.660
#> GSM228581 3 0.528 0.5664 0.024 0.180 0.796
#> GSM228666 3 0.522 0.4603 0.000 0.260 0.740
#> GSM228564 2 0.288 0.5383 0.096 0.904 0.000
#> GSM228568 1 0.701 0.3190 0.640 0.036 0.324
#> GSM228569 1 0.590 0.3162 0.680 0.004 0.316
#> GSM228572 3 0.622 0.1143 0.000 0.432 0.568
#> GSM228573 3 0.636 0.4152 0.404 0.004 0.592
#> GSM228577 1 0.489 0.6019 0.836 0.124 0.040
#> GSM228578 1 0.533 0.5499 0.792 0.024 0.184
#> GSM228663 3 0.599 0.4721 0.368 0.000 0.632
#> GSM228664 3 0.312 0.6724 0.108 0.000 0.892
#> GSM228665 3 0.610 0.5295 0.320 0.008 0.672
#> GSM228582 1 0.668 -0.1633 0.500 0.008 0.492
#> GSM228583 1 0.455 0.5498 0.800 0.200 0.000
#> GSM228585 1 0.371 0.5893 0.868 0.128 0.004
#> GSM228587 2 0.603 0.1279 0.376 0.624 0.000
#> GSM228588 2 0.268 0.5511 0.076 0.920 0.004
#> GSM228589 2 0.522 0.5422 0.000 0.740 0.260
#> GSM228590 1 0.595 0.3954 0.640 0.360 0.000
#> GSM228591 3 0.610 0.2134 0.000 0.392 0.608
#> GSM228597 2 0.343 0.5960 0.004 0.884 0.112
#> GSM228601 2 0.475 0.5848 0.000 0.784 0.216
#> GSM228604 3 0.263 0.6315 0.000 0.084 0.916
#> GSM228608 1 0.553 0.4777 0.704 0.296 0.000
#> GSM228609 2 0.382 0.4975 0.148 0.852 0.000
#> GSM228613 1 0.631 0.1530 0.512 0.488 0.000
#> GSM228616 3 0.973 0.0660 0.244 0.316 0.440
#> GSM228628 3 0.603 0.2525 0.000 0.376 0.624
#> GSM228634 1 0.345 0.6135 0.888 0.008 0.104
#> GSM228642 3 0.429 0.5549 0.000 0.180 0.820
#> GSM228645 3 0.368 0.6805 0.080 0.028 0.892
#> GSM228646 3 0.333 0.6810 0.076 0.020 0.904
#> GSM228652 2 0.629 -0.0910 0.464 0.536 0.000
#> GSM228655 1 0.698 0.4829 0.656 0.304 0.040
#> GSM228656 1 0.522 0.5055 0.740 0.260 0.000
#> GSM228659 2 0.522 0.3628 0.260 0.740 0.000
#> GSM228662 2 0.629 -0.1056 0.468 0.532 0.000
#> GSM228584 1 0.588 0.4019 0.652 0.348 0.000
#> GSM228586 1 0.277 0.6162 0.916 0.004 0.080
#> GSM228592 1 0.556 0.4629 0.700 0.300 0.000
#> GSM228593 2 0.400 0.4878 0.160 0.840 0.000
#> GSM228594 1 0.288 0.6148 0.904 0.000 0.096
#> GSM228598 1 0.667 0.1739 0.520 0.472 0.008
#> GSM228607 3 0.492 0.6652 0.084 0.072 0.844
#> GSM228612 3 0.516 0.6206 0.216 0.008 0.776
#> GSM228619 2 0.946 -0.2230 0.392 0.428 0.180
#> GSM228622 1 0.481 0.5297 0.804 0.008 0.188
#> GSM228625 2 0.533 0.3334 0.272 0.728 0.000
#> GSM228631 1 0.588 0.3995 0.716 0.012 0.272
#> GSM228633 3 0.559 0.4016 0.000 0.304 0.696
#> GSM228637 2 0.270 0.5936 0.016 0.928 0.056
#> GSM228639 3 0.511 0.5845 0.024 0.168 0.808
#> GSM228649 2 0.277 0.5909 0.024 0.928 0.048
#> GSM228660 2 0.790 -0.0556 0.440 0.504 0.056
#> GSM228661 1 0.533 0.4450 0.748 0.004 0.248
#> GSM228595 3 0.622 0.1140 0.000 0.432 0.568
#> GSM228599 2 0.629 0.1554 0.000 0.532 0.468
#> GSM228602 3 0.642 0.3695 0.424 0.004 0.572
#> GSM228614 3 0.625 0.0836 0.000 0.444 0.556
#> GSM228626 3 0.533 0.4452 0.000 0.272 0.728
#> GSM228640 3 0.586 0.4921 0.344 0.000 0.656
#> GSM228643 3 0.498 0.6508 0.168 0.020 0.812
#> GSM228650 3 0.231 0.6730 0.024 0.032 0.944
#> GSM228653 3 0.579 0.5057 0.332 0.000 0.668
#> GSM228657 3 0.622 0.1132 0.000 0.432 0.568
#> GSM228605 1 0.882 0.2844 0.532 0.132 0.336
#> GSM228610 3 0.569 0.5662 0.288 0.004 0.708
#> GSM228617 3 0.662 0.4409 0.388 0.012 0.600
#> GSM228620 3 0.640 0.3930 0.416 0.004 0.580
#> GSM228623 2 0.692 0.1642 0.016 0.536 0.448
#> GSM228629 3 0.634 0.4204 0.400 0.004 0.596
#> GSM228632 3 0.265 0.6505 0.012 0.060 0.928
#> GSM228635 2 0.610 0.4177 0.004 0.648 0.348
#> GSM228647 3 0.548 0.5851 0.264 0.004 0.732
#> GSM228596 3 0.499 0.6656 0.144 0.032 0.824
#> GSM228600 3 0.405 0.6554 0.148 0.004 0.848
#> GSM228603 3 0.619 0.3819 0.420 0.000 0.580
#> GSM228615 2 0.493 0.5884 0.004 0.784 0.212
#> GSM228627 3 0.445 0.6320 0.192 0.000 0.808
#> GSM228641 3 0.522 0.5853 0.260 0.000 0.740
#> GSM228644 3 0.579 0.3468 0.000 0.332 0.668
#> GSM228651 3 0.435 0.6365 0.184 0.000 0.816
#> GSM228654 3 0.327 0.6682 0.116 0.000 0.884
#> GSM228658 3 0.601 0.4642 0.372 0.000 0.628
#> GSM228606 3 0.411 0.5861 0.004 0.152 0.844
#> GSM228611 3 0.590 0.4949 0.352 0.000 0.648
#> GSM228618 3 0.630 0.4935 0.352 0.008 0.640
#> GSM228621 3 0.295 0.6772 0.088 0.004 0.908
#> GSM228624 3 0.277 0.6792 0.072 0.008 0.920
#> GSM228630 3 0.435 0.5726 0.004 0.168 0.828
#> GSM228636 2 0.540 0.5203 0.000 0.720 0.280
#> GSM228638 3 0.318 0.6773 0.076 0.016 0.908
#> GSM228648 3 0.134 0.6771 0.016 0.012 0.972
#> GSM228670 2 0.542 0.5607 0.008 0.752 0.240
#> GSM228671 3 0.502 0.4889 0.000 0.240 0.760
#> GSM228672 2 0.514 0.3785 0.252 0.748 0.000
#> GSM228674 2 0.220 0.5625 0.056 0.940 0.004
#> GSM228675 2 0.565 0.4886 0.000 0.688 0.312
#> GSM228676 3 0.852 0.1084 0.440 0.092 0.468
#> GSM228667 2 0.666 0.1985 0.008 0.528 0.464
#> GSM228668 1 0.625 0.3830 0.620 0.376 0.004
#> GSM228669 2 0.506 0.3872 0.244 0.756 0.000
#> GSM228673 3 0.390 0.6650 0.128 0.008 0.864
#> GSM228677 3 0.559 0.4033 0.000 0.304 0.696
#> GSM228678 2 0.627 0.2107 0.000 0.548 0.452
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 1 0.973 0.06573 0.324 0.188 0.176 0.312
#> GSM228563 4 0.611 0.35049 0.008 0.364 0.040 0.588
#> GSM228565 1 0.917 0.05393 0.328 0.292 0.068 0.312
#> GSM228566 3 0.729 0.24580 0.116 0.320 0.548 0.016
#> GSM228567 1 0.478 0.46440 0.752 0.000 0.036 0.212
#> GSM228570 4 0.790 -0.00325 0.404 0.108 0.040 0.448
#> GSM228571 1 0.620 0.51062 0.736 0.100 0.060 0.104
#> GSM228574 2 0.711 0.04228 0.100 0.484 0.408 0.008
#> GSM228575 3 0.727 0.11919 0.116 0.400 0.476 0.008
#> GSM228576 1 0.884 0.30002 0.492 0.208 0.204 0.096
#> GSM228579 1 0.589 0.51139 0.752 0.108 0.044 0.096
#> GSM228580 2 0.628 0.46073 0.016 0.656 0.264 0.064
#> GSM228581 2 0.570 0.44105 0.148 0.728 0.120 0.004
#> GSM228666 2 0.468 0.54075 0.060 0.812 0.112 0.016
#> GSM228564 4 0.632 0.51741 0.056 0.160 0.068 0.716
#> GSM228568 1 0.782 0.33036 0.532 0.236 0.212 0.020
#> GSM228569 1 0.493 0.38351 0.712 0.016 0.268 0.004
#> GSM228572 2 0.561 0.53199 0.000 0.724 0.156 0.120
#> GSM228573 3 0.578 0.35840 0.356 0.040 0.604 0.000
#> GSM228577 1 0.669 0.51117 0.688 0.060 0.176 0.076
#> GSM228578 1 0.630 0.46525 0.632 0.000 0.268 0.100
#> GSM228663 1 0.729 -0.08399 0.476 0.156 0.368 0.000
#> GSM228664 3 0.755 0.18320 0.188 0.400 0.412 0.000
#> GSM228665 3 0.648 0.39487 0.288 0.092 0.616 0.004
#> GSM228582 1 0.730 0.07375 0.444 0.432 0.116 0.008
#> GSM228583 1 0.527 0.29087 0.620 0.000 0.016 0.364
#> GSM228585 1 0.508 0.41063 0.700 0.000 0.028 0.272
#> GSM228587 4 0.534 0.34219 0.260 0.044 0.000 0.696
#> GSM228588 4 0.569 0.41872 0.032 0.376 0.000 0.592
#> GSM228589 2 0.433 0.46780 0.036 0.808 0.004 0.152
#> GSM228590 4 0.526 0.00380 0.444 0.008 0.000 0.548
#> GSM228591 2 0.419 0.52106 0.080 0.844 0.060 0.016
#> GSM228597 4 0.556 0.37132 0.000 0.324 0.036 0.640
#> GSM228601 2 0.422 0.44936 0.020 0.800 0.004 0.176
#> GSM228604 2 0.546 0.28328 0.020 0.632 0.344 0.004
#> GSM228608 1 0.623 0.11095 0.512 0.004 0.044 0.440
#> GSM228609 4 0.547 0.54080 0.084 0.192 0.000 0.724
#> GSM228613 4 0.410 0.36275 0.256 0.000 0.000 0.744
#> GSM228616 2 0.852 0.26769 0.244 0.524 0.104 0.128
#> GSM228628 2 0.369 0.54384 0.064 0.872 0.044 0.020
#> GSM228634 1 0.389 0.54543 0.844 0.000 0.068 0.088
#> GSM228642 2 0.451 0.48900 0.036 0.780 0.184 0.000
#> GSM228645 2 0.759 0.19070 0.168 0.524 0.296 0.012
#> GSM228646 2 0.728 0.00894 0.128 0.456 0.412 0.004
#> GSM228652 4 0.515 0.27817 0.324 0.012 0.004 0.660
#> GSM228655 1 0.695 0.41178 0.628 0.064 0.048 0.260
#> GSM228656 1 0.510 0.29435 0.624 0.004 0.004 0.368
#> GSM228659 4 0.390 0.51562 0.108 0.052 0.000 0.840
#> GSM228662 4 0.376 0.40661 0.216 0.000 0.000 0.784
#> GSM228584 4 0.567 -0.08497 0.472 0.004 0.016 0.508
#> GSM228586 1 0.440 0.54989 0.812 0.000 0.112 0.076
#> GSM228592 1 0.582 0.21607 0.560 0.008 0.020 0.412
#> GSM228593 4 0.473 0.53701 0.060 0.160 0.000 0.780
#> GSM228594 1 0.538 0.55213 0.776 0.032 0.128 0.064
#> GSM228598 4 0.830 -0.02194 0.340 0.040 0.164 0.456
#> GSM228607 3 0.777 0.21554 0.128 0.344 0.500 0.028
#> GSM228612 3 0.743 0.37402 0.232 0.256 0.512 0.000
#> GSM228619 3 0.744 0.11044 0.060 0.052 0.516 0.372
#> GSM228622 1 0.700 0.15089 0.468 0.004 0.428 0.100
#> GSM228625 4 0.613 0.50452 0.116 0.084 0.060 0.740
#> GSM228631 3 0.586 0.31464 0.248 0.004 0.680 0.068
#> GSM228633 2 0.560 0.43190 0.000 0.672 0.276 0.052
#> GSM228637 4 0.719 0.31522 0.004 0.264 0.168 0.564
#> GSM228639 3 0.579 0.33619 0.012 0.232 0.700 0.056
#> GSM228649 2 0.822 -0.15369 0.028 0.404 0.176 0.392
#> GSM228660 1 0.972 0.24742 0.368 0.200 0.244 0.188
#> GSM228661 1 0.478 0.40466 0.712 0.000 0.272 0.016
#> GSM228595 2 0.383 0.56322 0.000 0.848 0.072 0.080
#> GSM228599 2 0.786 0.37815 0.008 0.472 0.292 0.228
#> GSM228602 3 0.606 0.44831 0.308 0.068 0.624 0.000
#> GSM228614 2 0.756 0.39216 0.000 0.476 0.304 0.220
#> GSM228626 2 0.278 0.54634 0.004 0.888 0.104 0.004
#> GSM228640 3 0.690 0.44539 0.256 0.120 0.612 0.012
#> GSM228643 3 0.708 0.30131 0.120 0.288 0.580 0.012
#> GSM228650 3 0.603 0.25041 0.024 0.324 0.628 0.024
#> GSM228653 3 0.683 0.43893 0.352 0.112 0.536 0.000
#> GSM228657 2 0.373 0.56722 0.004 0.860 0.076 0.060
#> GSM228605 3 0.701 0.38810 0.140 0.040 0.660 0.160
#> GSM228610 3 0.542 0.54402 0.188 0.072 0.736 0.004
#> GSM228617 3 0.347 0.53433 0.080 0.032 0.876 0.012
#> GSM228620 3 0.485 0.51335 0.220 0.028 0.748 0.004
#> GSM228623 2 0.796 0.26255 0.008 0.420 0.352 0.220
#> GSM228629 3 0.550 0.39294 0.312 0.028 0.656 0.004
#> GSM228632 3 0.671 0.17752 0.056 0.396 0.532 0.016
#> GSM228635 4 0.795 -0.03710 0.004 0.352 0.248 0.396
#> GSM228647 3 0.365 0.55162 0.108 0.040 0.852 0.000
#> GSM228596 3 0.802 0.31687 0.204 0.268 0.504 0.024
#> GSM228600 3 0.686 0.35083 0.140 0.284 0.576 0.000
#> GSM228603 3 0.633 0.43871 0.328 0.080 0.592 0.000
#> GSM228615 4 0.600 0.08261 0.000 0.456 0.040 0.504
#> GSM228627 2 0.785 -0.15896 0.308 0.400 0.292 0.000
#> GSM228641 3 0.676 0.39876 0.152 0.204 0.636 0.008
#> GSM228644 2 0.289 0.56107 0.004 0.896 0.080 0.020
#> GSM228651 3 0.695 0.40186 0.152 0.280 0.568 0.000
#> GSM228654 3 0.707 0.42286 0.160 0.288 0.552 0.000
#> GSM228658 3 0.731 0.36485 0.408 0.152 0.440 0.000
#> GSM228606 3 0.701 0.07436 0.020 0.340 0.560 0.080
#> GSM228611 3 0.586 0.48773 0.264 0.072 0.664 0.000
#> GSM228618 3 0.380 0.55181 0.132 0.032 0.836 0.000
#> GSM228621 3 0.436 0.46339 0.020 0.208 0.772 0.000
#> GSM228624 3 0.718 0.29860 0.160 0.316 0.524 0.000
#> GSM228630 3 0.598 0.25653 0.008 0.300 0.644 0.048
#> GSM228636 4 0.748 0.06520 0.000 0.380 0.180 0.440
#> GSM228638 3 0.542 0.46951 0.064 0.176 0.748 0.012
#> GSM228648 3 0.490 0.42535 0.024 0.260 0.716 0.000
#> GSM228670 4 0.759 0.09756 0.012 0.336 0.152 0.500
#> GSM228671 2 0.702 0.27537 0.024 0.540 0.368 0.068
#> GSM228672 4 0.495 0.51845 0.124 0.088 0.004 0.784
#> GSM228674 4 0.567 0.51002 0.028 0.188 0.048 0.736
#> GSM228675 2 0.715 0.26171 0.012 0.532 0.104 0.352
#> GSM228676 3 0.937 0.20377 0.252 0.144 0.424 0.180
#> GSM228667 2 0.821 0.41475 0.064 0.552 0.188 0.196
#> GSM228668 4 0.617 0.40324 0.156 0.016 0.120 0.708
#> GSM228669 4 0.394 0.53873 0.024 0.044 0.072 0.860
#> GSM228673 3 0.689 0.44777 0.136 0.200 0.644 0.020
#> GSM228677 2 0.690 0.27804 0.004 0.492 0.412 0.092
#> GSM228678 2 0.747 0.33869 0.004 0.532 0.224 0.240
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 5 0.894 0.1052 0.252 0.068 0.100 0.184 0.396
#> GSM228563 4 0.778 0.3498 0.080 0.208 0.036 0.540 0.136
#> GSM228565 1 0.947 0.0520 0.308 0.204 0.072 0.176 0.240
#> GSM228566 5 0.663 0.3652 0.044 0.160 0.084 0.052 0.660
#> GSM228567 1 0.290 0.5627 0.872 0.000 0.028 0.004 0.096
#> GSM228570 1 0.780 0.3607 0.520 0.048 0.044 0.164 0.224
#> GSM228571 1 0.670 0.4592 0.640 0.068 0.068 0.032 0.192
#> GSM228574 5 0.763 0.1004 0.016 0.272 0.144 0.068 0.500
#> GSM228575 5 0.785 0.2134 0.016 0.168 0.272 0.072 0.472
#> GSM228576 1 0.803 0.0663 0.404 0.080 0.076 0.056 0.384
#> GSM228579 1 0.679 0.4777 0.652 0.124 0.096 0.028 0.100
#> GSM228580 2 0.732 0.2637 0.000 0.464 0.088 0.108 0.340
#> GSM228581 2 0.650 0.3784 0.032 0.584 0.296 0.020 0.068
#> GSM228666 2 0.659 0.4137 0.008 0.576 0.292 0.060 0.064
#> GSM228564 4 0.757 0.2921 0.132 0.056 0.024 0.508 0.280
#> GSM228568 3 0.746 0.1485 0.264 0.300 0.404 0.004 0.028
#> GSM228569 3 0.543 0.0628 0.424 0.012 0.528 0.000 0.036
#> GSM228572 2 0.662 0.4080 0.000 0.624 0.088 0.136 0.152
#> GSM228573 3 0.678 0.1365 0.204 0.008 0.444 0.000 0.344
#> GSM228577 1 0.601 0.2288 0.544 0.064 0.372 0.008 0.012
#> GSM228578 1 0.708 0.1538 0.484 0.008 0.356 0.048 0.104
#> GSM228663 3 0.650 0.3794 0.172 0.152 0.620 0.000 0.056
#> GSM228664 3 0.532 0.3164 0.024 0.300 0.644 0.004 0.028
#> GSM228665 3 0.501 0.4150 0.072 0.052 0.776 0.012 0.088
#> GSM228582 2 0.684 0.2386 0.152 0.540 0.268 0.000 0.040
#> GSM228583 1 0.303 0.5628 0.868 0.000 0.004 0.088 0.040
#> GSM228585 1 0.302 0.5670 0.872 0.000 0.008 0.036 0.084
#> GSM228587 1 0.588 0.3240 0.560 0.064 0.020 0.356 0.000
#> GSM228588 2 0.657 -0.0934 0.152 0.484 0.012 0.352 0.000
#> GSM228589 2 0.494 0.5012 0.044 0.776 0.088 0.084 0.008
#> GSM228590 1 0.445 0.4382 0.668 0.004 0.008 0.316 0.004
#> GSM228591 2 0.368 0.5618 0.032 0.844 0.080 0.000 0.044
#> GSM228597 4 0.555 0.5294 0.032 0.156 0.028 0.728 0.056
#> GSM228601 2 0.337 0.5092 0.012 0.848 0.004 0.116 0.020
#> GSM228604 2 0.590 0.1416 0.004 0.508 0.076 0.004 0.408
#> GSM228608 1 0.656 0.4723 0.612 0.012 0.048 0.240 0.088
#> GSM228609 4 0.691 0.2018 0.300 0.184 0.008 0.496 0.012
#> GSM228613 1 0.445 0.2220 0.512 0.004 0.000 0.484 0.000
#> GSM228616 2 0.828 0.3265 0.192 0.480 0.060 0.060 0.208
#> GSM228628 2 0.341 0.5765 0.016 0.868 0.032 0.016 0.068
#> GSM228634 1 0.553 0.3667 0.664 0.012 0.224 0.000 0.100
#> GSM228642 2 0.450 0.5518 0.004 0.776 0.060 0.012 0.148
#> GSM228645 2 0.802 0.1723 0.092 0.440 0.088 0.040 0.340
#> GSM228646 2 0.690 0.0752 0.104 0.440 0.032 0.008 0.416
#> GSM228652 1 0.589 0.2639 0.488 0.012 0.048 0.444 0.008
#> GSM228655 1 0.794 0.3567 0.472 0.060 0.272 0.168 0.028
#> GSM228656 1 0.378 0.5555 0.832 0.008 0.044 0.108 0.008
#> GSM228659 4 0.460 0.2533 0.272 0.040 0.000 0.688 0.000
#> GSM228662 1 0.445 0.2106 0.512 0.000 0.000 0.484 0.004
#> GSM228584 1 0.450 0.4916 0.712 0.004 0.024 0.256 0.004
#> GSM228586 1 0.505 0.3538 0.668 0.004 0.268 0.000 0.060
#> GSM228592 1 0.400 0.5224 0.768 0.008 0.020 0.204 0.000
#> GSM228593 4 0.665 0.1245 0.312 0.164 0.016 0.508 0.000
#> GSM228594 1 0.523 0.3970 0.688 0.052 0.236 0.000 0.024
#> GSM228598 1 0.739 0.2954 0.428 0.028 0.300 0.240 0.004
#> GSM228607 3 0.542 0.4010 0.012 0.132 0.740 0.064 0.052
#> GSM228612 3 0.656 0.4003 0.088 0.196 0.628 0.004 0.084
#> GSM228619 5 0.838 0.1498 0.068 0.048 0.160 0.304 0.420
#> GSM228622 3 0.772 0.1199 0.364 0.000 0.384 0.084 0.168
#> GSM228625 4 0.652 0.3476 0.228 0.064 0.072 0.624 0.012
#> GSM228631 5 0.696 0.2738 0.160 0.016 0.196 0.040 0.588
#> GSM228633 2 0.695 0.3497 0.000 0.580 0.200 0.088 0.132
#> GSM228637 4 0.598 0.5193 0.028 0.096 0.180 0.680 0.016
#> GSM228639 3 0.760 0.0845 0.000 0.068 0.444 0.200 0.288
#> GSM228649 4 0.771 0.1672 0.028 0.268 0.344 0.348 0.012
#> GSM228660 3 0.796 0.1512 0.272 0.212 0.436 0.068 0.012
#> GSM228661 3 0.594 0.0559 0.428 0.012 0.488 0.000 0.072
#> GSM228595 2 0.409 0.5346 0.000 0.816 0.036 0.104 0.044
#> GSM228599 5 0.666 0.1786 0.004 0.224 0.016 0.200 0.556
#> GSM228602 5 0.547 0.4148 0.176 0.032 0.056 0.016 0.720
#> GSM228614 4 0.792 0.1672 0.000 0.264 0.104 0.428 0.204
#> GSM228626 2 0.375 0.5803 0.000 0.832 0.068 0.012 0.088
#> GSM228640 5 0.393 0.4465 0.104 0.024 0.040 0.004 0.828
#> GSM228643 5 0.611 0.4304 0.056 0.112 0.100 0.028 0.704
#> GSM228650 5 0.575 0.4070 0.000 0.156 0.116 0.040 0.688
#> GSM228653 5 0.705 0.0755 0.116 0.056 0.364 0.000 0.464
#> GSM228657 2 0.526 0.5455 0.000 0.744 0.080 0.072 0.104
#> GSM228605 3 0.771 -0.0305 0.036 0.008 0.364 0.252 0.340
#> GSM228610 3 0.524 0.3639 0.040 0.012 0.724 0.032 0.192
#> GSM228617 5 0.619 0.1437 0.036 0.020 0.344 0.032 0.568
#> GSM228620 3 0.599 0.2537 0.080 0.008 0.596 0.012 0.304
#> GSM228623 4 0.773 0.2228 0.000 0.176 0.324 0.416 0.084
#> GSM228629 3 0.617 0.1751 0.128 0.004 0.520 0.000 0.348
#> GSM228632 3 0.784 0.1156 0.004 0.192 0.456 0.088 0.260
#> GSM228635 4 0.700 0.4368 0.000 0.100 0.192 0.580 0.128
#> GSM228647 3 0.543 0.0277 0.020 0.008 0.500 0.012 0.460
#> GSM228596 3 0.869 -0.0540 0.052 0.176 0.396 0.088 0.288
#> GSM228600 5 0.609 0.4333 0.080 0.152 0.084 0.004 0.680
#> GSM228603 5 0.440 0.4203 0.164 0.016 0.048 0.000 0.772
#> GSM228615 4 0.561 0.4459 0.004 0.228 0.012 0.664 0.092
#> GSM228627 3 0.758 0.0459 0.084 0.364 0.424 0.004 0.124
#> GSM228641 5 0.393 0.4572 0.068 0.036 0.048 0.008 0.840
#> GSM228644 2 0.329 0.5841 0.000 0.864 0.040 0.020 0.076
#> GSM228651 5 0.693 0.1797 0.044 0.116 0.320 0.004 0.516
#> GSM228654 5 0.731 0.2251 0.044 0.168 0.276 0.008 0.504
#> GSM228658 5 0.778 -0.0251 0.180 0.084 0.364 0.000 0.372
#> GSM228606 3 0.825 -0.0108 0.000 0.152 0.380 0.196 0.272
#> GSM228611 3 0.586 0.3838 0.076 0.052 0.708 0.016 0.148
#> GSM228618 5 0.633 0.0998 0.060 0.020 0.376 0.016 0.528
#> GSM228621 5 0.621 0.1946 0.000 0.076 0.316 0.036 0.572
#> GSM228624 3 0.556 0.3900 0.008 0.212 0.684 0.016 0.080
#> GSM228630 5 0.768 0.0449 0.000 0.128 0.348 0.108 0.416
#> GSM228636 4 0.663 0.4648 0.000 0.164 0.136 0.620 0.080
#> GSM228638 3 0.658 0.1454 0.000 0.096 0.524 0.040 0.340
#> GSM228648 3 0.718 -0.0292 0.000 0.172 0.400 0.036 0.392
#> GSM228670 4 0.661 0.4981 0.020 0.120 0.080 0.660 0.120
#> GSM228671 2 0.848 0.1098 0.000 0.316 0.284 0.188 0.212
#> GSM228672 4 0.558 0.3158 0.248 0.060 0.004 0.664 0.024
#> GSM228674 4 0.484 0.5165 0.052 0.044 0.076 0.796 0.032
#> GSM228675 4 0.798 0.0797 0.004 0.300 0.124 0.428 0.144
#> GSM228676 5 0.826 0.2685 0.148 0.024 0.140 0.212 0.476
#> GSM228667 2 0.911 0.1123 0.060 0.324 0.104 0.276 0.236
#> GSM228668 4 0.579 0.2820 0.248 0.000 0.040 0.648 0.064
#> GSM228669 4 0.390 0.4698 0.116 0.004 0.052 0.820 0.008
#> GSM228673 3 0.657 0.3157 0.024 0.100 0.668 0.088 0.120
#> GSM228677 5 0.866 0.0271 0.004 0.192 0.248 0.248 0.308
#> GSM228678 4 0.839 0.1288 0.004 0.304 0.168 0.356 0.168
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 5 0.541 0.49254 0.044 0.020 0.040 0.036 0.724 0.136
#> GSM228563 5 0.759 0.35227 0.008 0.148 0.140 0.176 0.496 0.032
#> GSM228565 5 0.707 0.49706 0.084 0.172 0.032 0.048 0.592 0.072
#> GSM228566 5 0.586 0.24952 0.000 0.076 0.048 0.000 0.516 0.360
#> GSM228567 1 0.701 0.24437 0.464 0.008 0.000 0.188 0.264 0.076
#> GSM228570 5 0.675 0.37325 0.112 0.008 0.020 0.168 0.588 0.104
#> GSM228571 5 0.732 0.14127 0.292 0.056 0.000 0.076 0.468 0.108
#> GSM228574 5 0.676 0.41251 0.016 0.132 0.120 0.000 0.564 0.168
#> GSM228575 5 0.790 0.30665 0.056 0.108 0.220 0.000 0.424 0.192
#> GSM228576 5 0.685 0.42841 0.116 0.072 0.004 0.048 0.584 0.176
#> GSM228579 1 0.740 0.20222 0.460 0.108 0.000 0.096 0.288 0.048
#> GSM228580 2 0.807 0.21089 0.016 0.392 0.116 0.032 0.164 0.280
#> GSM228581 2 0.785 0.33884 0.140 0.492 0.148 0.004 0.124 0.092
#> GSM228666 2 0.785 0.20574 0.060 0.424 0.240 0.000 0.184 0.092
#> GSM228564 5 0.709 0.35322 0.004 0.020 0.096 0.236 0.516 0.128
#> GSM228568 1 0.776 0.26360 0.440 0.240 0.144 0.008 0.144 0.024
#> GSM228569 1 0.426 0.46418 0.796 0.020 0.088 0.000 0.040 0.056
#> GSM228572 2 0.697 0.43849 0.004 0.580 0.168 0.056 0.100 0.092
#> GSM228573 6 0.750 0.27403 0.252 0.012 0.196 0.000 0.124 0.416
#> GSM228577 1 0.709 0.40956 0.572 0.100 0.124 0.036 0.156 0.012
#> GSM228578 1 0.796 0.27165 0.416 0.004 0.252 0.056 0.176 0.096
#> GSM228663 1 0.708 0.12172 0.520 0.132 0.228 0.000 0.032 0.088
#> GSM228664 3 0.741 0.08878 0.296 0.304 0.328 0.000 0.032 0.040
#> GSM228665 1 0.722 -0.07657 0.388 0.060 0.360 0.000 0.024 0.168
#> GSM228582 2 0.573 0.20860 0.368 0.536 0.020 0.008 0.008 0.060
#> GSM228583 1 0.688 0.11757 0.420 0.000 0.000 0.320 0.188 0.072
#> GSM228585 1 0.698 0.17672 0.440 0.000 0.000 0.272 0.200 0.088
#> GSM228587 4 0.520 0.37937 0.216 0.080 0.000 0.672 0.020 0.012
#> GSM228588 2 0.526 0.23586 0.040 0.584 0.004 0.340 0.032 0.000
#> GSM228589 2 0.415 0.56950 0.044 0.816 0.024 0.076 0.024 0.016
#> GSM228590 4 0.468 0.26021 0.336 0.004 0.000 0.620 0.028 0.012
#> GSM228591 2 0.281 0.57278 0.040 0.884 0.000 0.016 0.048 0.012
#> GSM228597 4 0.711 0.24370 0.008 0.136 0.240 0.508 0.096 0.012
#> GSM228601 2 0.279 0.57596 0.000 0.872 0.012 0.072 0.044 0.000
#> GSM228604 2 0.568 0.11313 0.004 0.472 0.032 0.000 0.060 0.432
#> GSM228608 4 0.736 0.08457 0.184 0.004 0.024 0.388 0.344 0.056
#> GSM228609 4 0.530 0.39692 0.028 0.240 0.008 0.668 0.032 0.024
#> GSM228613 4 0.414 0.40998 0.228 0.000 0.004 0.720 0.048 0.000
#> GSM228616 2 0.779 0.31320 0.108 0.456 0.020 0.104 0.040 0.272
#> GSM228628 2 0.377 0.54322 0.020 0.812 0.024 0.004 0.128 0.012
#> GSM228634 1 0.422 0.47016 0.768 0.000 0.000 0.044 0.044 0.144
#> GSM228642 2 0.501 0.48257 0.004 0.712 0.040 0.000 0.152 0.092
#> GSM228645 5 0.698 0.37507 0.032 0.264 0.032 0.000 0.468 0.204
#> GSM228646 5 0.688 0.24583 0.016 0.248 0.008 0.012 0.408 0.308
#> GSM228652 4 0.539 0.37842 0.252 0.008 0.012 0.644 0.072 0.012
#> GSM228655 1 0.791 0.26380 0.464 0.056 0.072 0.252 0.028 0.128
#> GSM228656 1 0.498 0.21183 0.612 0.008 0.000 0.328 0.032 0.020
#> GSM228659 4 0.304 0.52474 0.056 0.016 0.020 0.872 0.036 0.000
#> GSM228662 4 0.406 0.43845 0.168 0.004 0.000 0.760 0.064 0.004
#> GSM228584 4 0.534 -0.00777 0.448 0.000 0.004 0.476 0.060 0.012
#> GSM228586 1 0.393 0.48580 0.812 0.000 0.008 0.040 0.052 0.088
#> GSM228592 1 0.527 0.06226 0.516 0.008 0.000 0.416 0.048 0.012
#> GSM228593 4 0.703 0.36305 0.132 0.152 0.028 0.560 0.124 0.004
#> GSM228594 1 0.592 0.47899 0.704 0.056 0.024 0.064 0.056 0.096
#> GSM228598 1 0.689 0.32640 0.548 0.024 0.152 0.204 0.068 0.004
#> GSM228607 3 0.522 0.40098 0.172 0.076 0.704 0.004 0.024 0.020
#> GSM228612 1 0.776 -0.06634 0.364 0.176 0.284 0.000 0.016 0.160
#> GSM228619 6 0.758 0.18488 0.012 0.032 0.216 0.240 0.056 0.444
#> GSM228622 1 0.816 0.22756 0.424 0.000 0.192 0.100 0.120 0.164
#> GSM228625 4 0.545 0.48547 0.060 0.080 0.144 0.700 0.008 0.008
#> GSM228631 6 0.594 0.46254 0.064 0.004 0.124 0.044 0.084 0.680
#> GSM228633 2 0.711 0.02087 0.008 0.408 0.396 0.028 0.072 0.088
#> GSM228637 4 0.615 0.07362 0.016 0.068 0.420 0.460 0.032 0.004
#> GSM228639 3 0.543 0.31780 0.016 0.032 0.696 0.032 0.036 0.188
#> GSM228649 3 0.787 0.17674 0.100 0.256 0.412 0.188 0.040 0.004
#> GSM228660 1 0.737 0.24179 0.436 0.284 0.176 0.076 0.000 0.028
#> GSM228661 1 0.469 0.40753 0.732 0.020 0.080 0.000 0.008 0.160
#> GSM228595 2 0.464 0.56448 0.000 0.764 0.120 0.032 0.056 0.028
#> GSM228599 6 0.799 0.11488 0.000 0.168 0.100 0.116 0.160 0.456
#> GSM228602 6 0.417 0.41201 0.028 0.016 0.012 0.004 0.172 0.768
#> GSM228614 3 0.869 0.06131 0.004 0.176 0.296 0.280 0.116 0.128
#> GSM228626 2 0.407 0.58931 0.008 0.808 0.060 0.004 0.036 0.084
#> GSM228640 6 0.471 0.26998 0.012 0.016 0.020 0.000 0.308 0.644
#> GSM228643 6 0.622 -0.05147 0.008 0.056 0.060 0.004 0.420 0.452
#> GSM228650 6 0.616 0.38567 0.004 0.116 0.124 0.004 0.124 0.628
#> GSM228653 6 0.631 0.40723 0.200 0.028 0.116 0.000 0.052 0.604
#> GSM228657 2 0.487 0.57620 0.004 0.740 0.052 0.044 0.012 0.148
#> GSM228605 3 0.677 0.27366 0.060 0.000 0.564 0.068 0.228 0.080
#> GSM228610 3 0.712 0.20007 0.276 0.024 0.460 0.000 0.056 0.184
#> GSM228617 6 0.549 0.34587 0.060 0.004 0.308 0.004 0.028 0.596
#> GSM228620 3 0.700 0.05640 0.236 0.000 0.420 0.000 0.076 0.268
#> GSM228623 3 0.577 0.43183 0.012 0.080 0.696 0.120 0.068 0.024
#> GSM228629 3 0.707 -0.11875 0.240 0.004 0.368 0.000 0.060 0.328
#> GSM228632 3 0.665 0.38164 0.032 0.136 0.592 0.000 0.152 0.088
#> GSM228635 3 0.650 0.23999 0.012 0.044 0.572 0.264 0.080 0.028
#> GSM228647 6 0.631 0.22449 0.128 0.004 0.340 0.000 0.040 0.488
#> GSM228596 5 0.876 0.12246 0.104 0.084 0.236 0.024 0.340 0.212
#> GSM228600 6 0.426 0.44940 0.028 0.084 0.012 0.000 0.088 0.788
#> GSM228603 6 0.463 0.37469 0.064 0.012 0.008 0.000 0.200 0.716
#> GSM228615 4 0.723 0.28130 0.004 0.188 0.168 0.528 0.064 0.048
#> GSM228627 2 0.870 0.07658 0.228 0.312 0.164 0.000 0.124 0.172
#> GSM228641 6 0.502 0.20410 0.008 0.024 0.024 0.000 0.360 0.584
#> GSM228644 2 0.404 0.58861 0.000 0.804 0.064 0.004 0.052 0.076
#> GSM228651 6 0.764 0.33086 0.092 0.108 0.160 0.000 0.136 0.504
#> GSM228654 6 0.729 0.40737 0.120 0.140 0.128 0.000 0.068 0.544
#> GSM228658 6 0.692 0.34436 0.268 0.084 0.080 0.000 0.044 0.524
#> GSM228606 3 0.556 0.38829 0.008 0.052 0.648 0.000 0.220 0.072
#> GSM228611 3 0.668 0.19656 0.332 0.004 0.468 0.000 0.084 0.112
#> GSM228618 6 0.623 0.34741 0.124 0.004 0.284 0.000 0.048 0.540
#> GSM228621 6 0.663 0.18228 0.028 0.028 0.376 0.000 0.124 0.444
#> GSM228624 3 0.698 0.37534 0.204 0.120 0.552 0.000 0.072 0.052
#> GSM228630 3 0.631 0.20111 0.016 0.068 0.588 0.016 0.052 0.260
#> GSM228636 3 0.696 0.03456 0.000 0.104 0.428 0.368 0.080 0.020
#> GSM228638 3 0.651 -0.00771 0.132 0.048 0.488 0.000 0.008 0.324
#> GSM228648 6 0.697 0.14871 0.060 0.152 0.332 0.000 0.016 0.440
#> GSM228670 4 0.811 0.08295 0.004 0.100 0.272 0.388 0.172 0.064
#> GSM228671 3 0.654 0.12712 0.012 0.128 0.484 0.000 0.332 0.044
#> GSM228672 4 0.627 0.43905 0.040 0.052 0.076 0.620 0.208 0.004
#> GSM228674 4 0.708 0.30366 0.016 0.044 0.232 0.492 0.204 0.012
#> GSM228675 3 0.862 -0.02324 0.004 0.232 0.284 0.180 0.236 0.064
#> GSM228676 5 0.853 0.23724 0.060 0.028 0.196 0.100 0.376 0.240
#> GSM228667 5 0.751 0.40234 0.048 0.140 0.136 0.068 0.560 0.048
#> GSM228668 4 0.640 0.45814 0.112 0.000 0.144 0.616 0.104 0.024
#> GSM228669 4 0.532 0.43073 0.012 0.008 0.248 0.648 0.076 0.008
#> GSM228673 3 0.674 0.38243 0.192 0.036 0.552 0.000 0.176 0.044
#> GSM228677 3 0.667 0.26269 0.004 0.092 0.532 0.016 0.280 0.076
#> GSM228678 3 0.811 0.23931 0.004 0.152 0.448 0.128 0.180 0.088
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
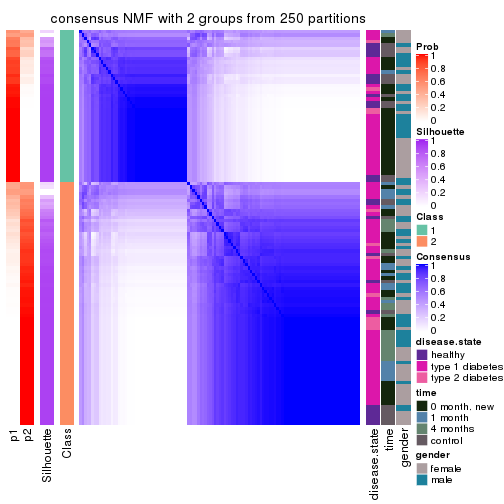
consensus_heatmap(res, k = 3)
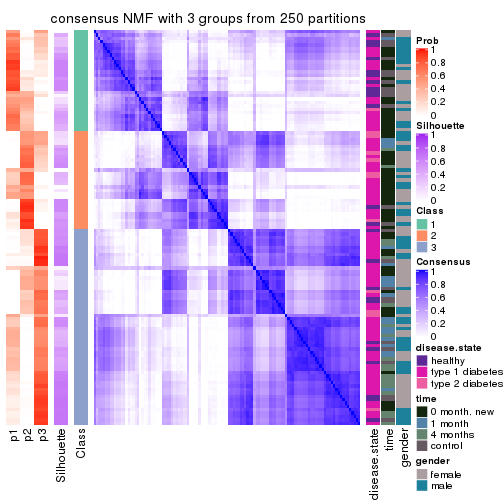
consensus_heatmap(res, k = 4)
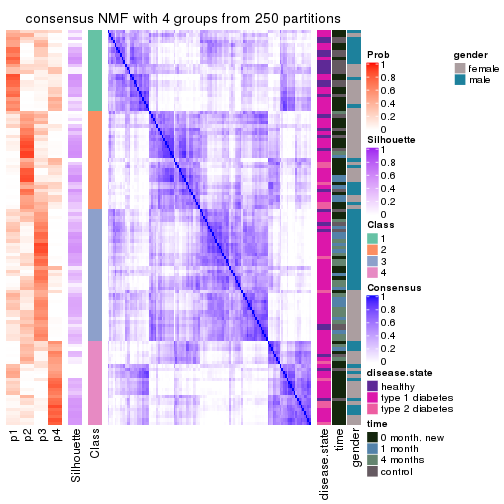
consensus_heatmap(res, k = 5)
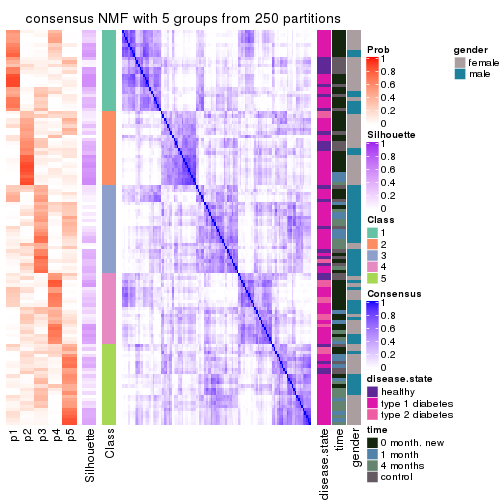
consensus_heatmap(res, k = 6)
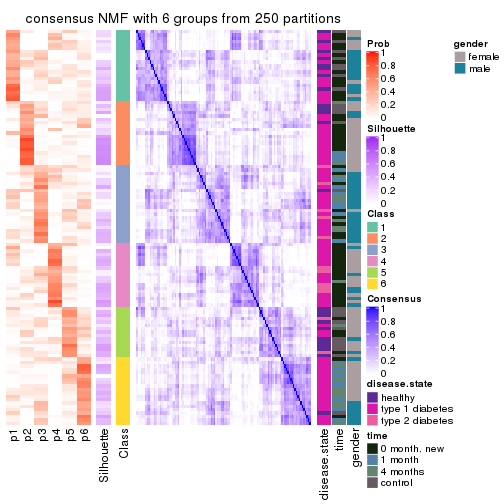
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
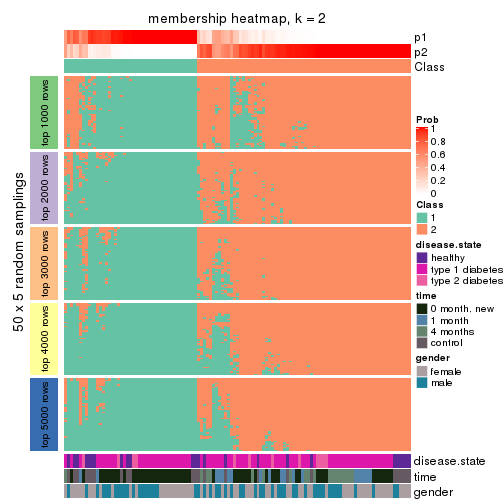
membership_heatmap(res, k = 3)
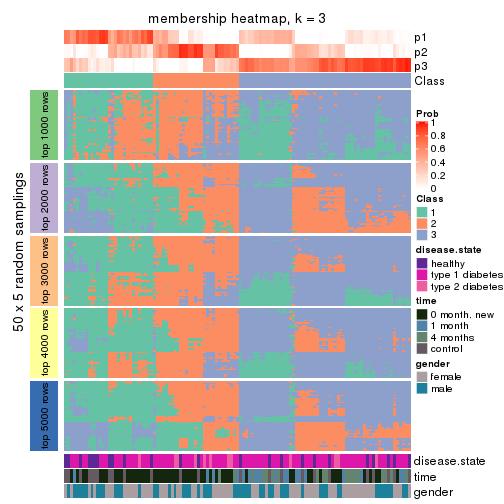
membership_heatmap(res, k = 4)
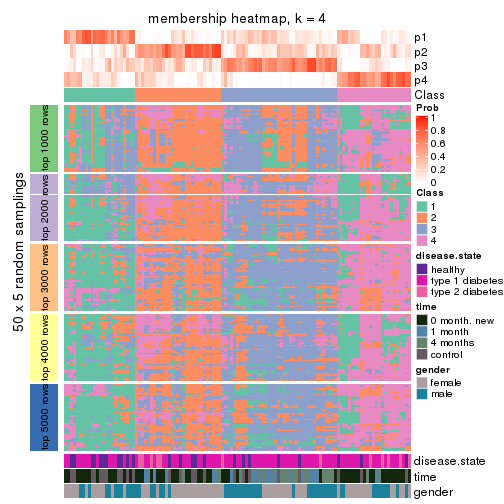
membership_heatmap(res, k = 5)
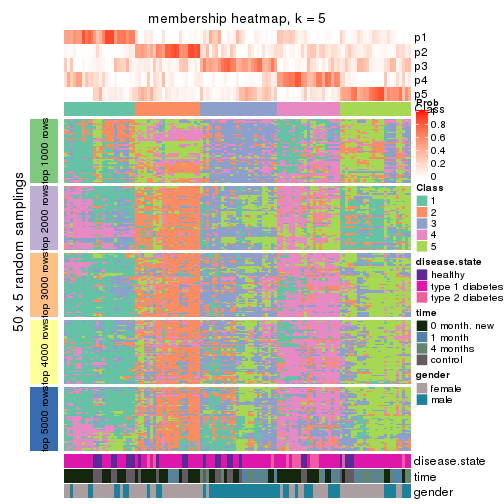
membership_heatmap(res, k = 6)
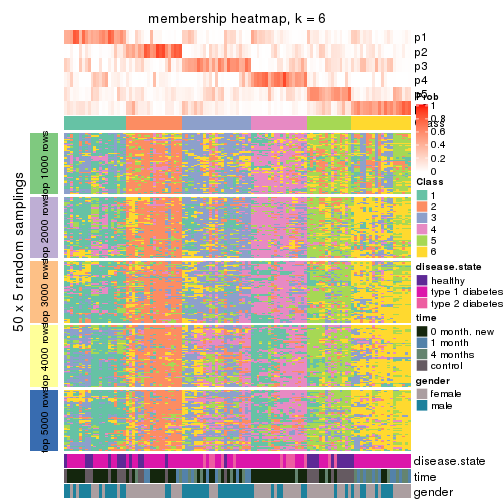
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
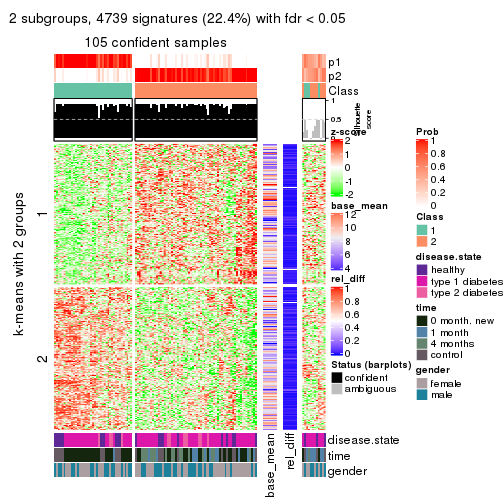
get_signatures(res, k = 3)
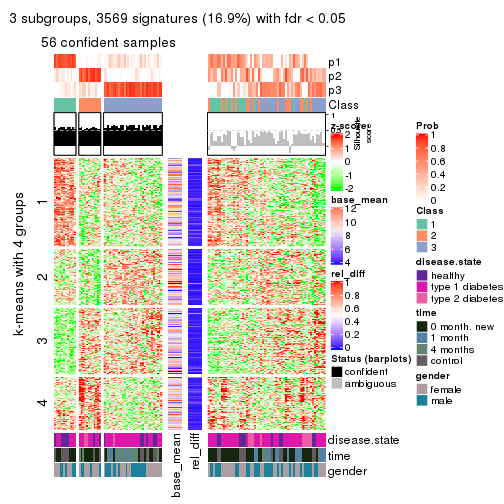
get_signatures(res, k = 4)
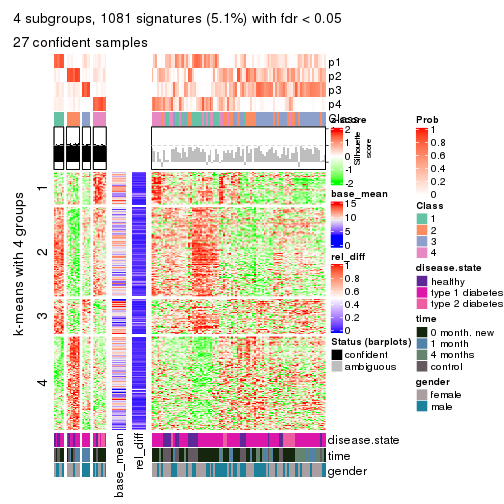
get_signatures(res, k = 5)
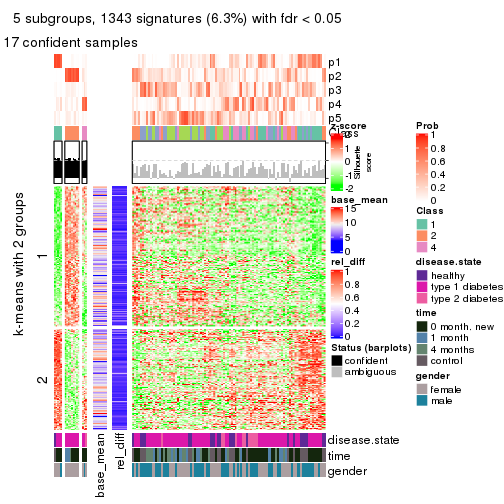
get_signatures(res, k = 6)

Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
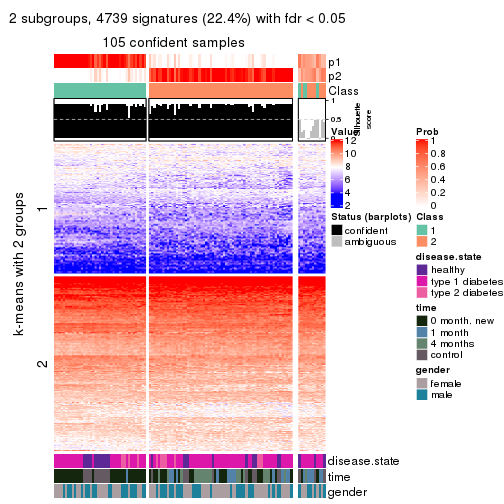
get_signatures(res, k = 3, scale_rows = FALSE)
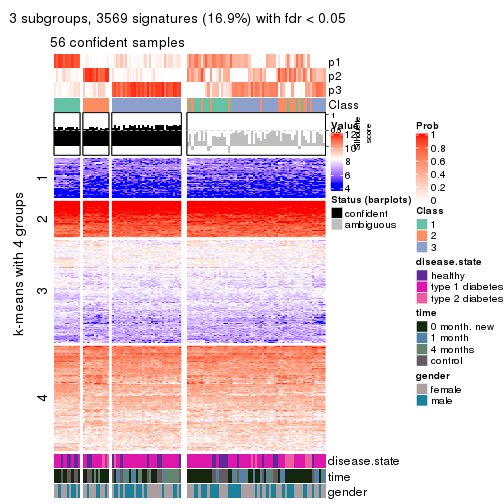
get_signatures(res, k = 4, scale_rows = FALSE)
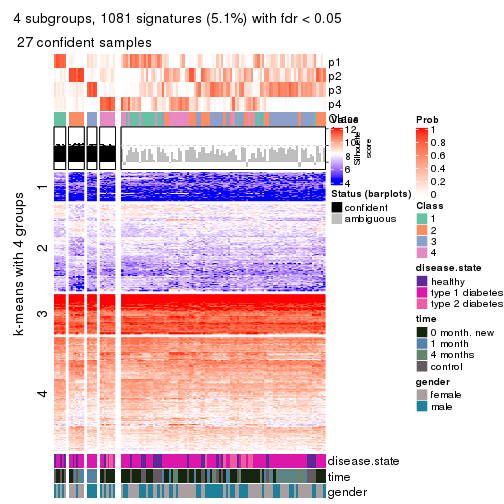
get_signatures(res, k = 5, scale_rows = FALSE)
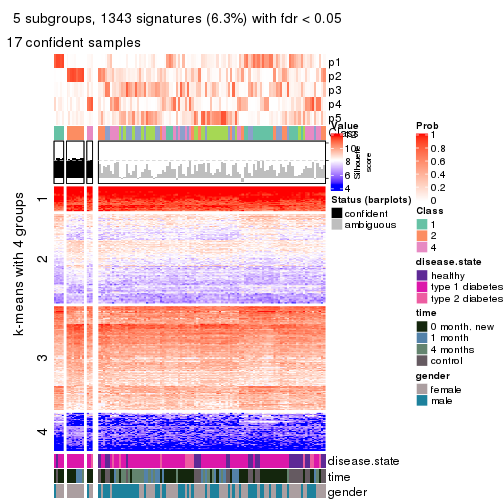
get_signatures(res, k = 6, scale_rows = FALSE)

Compare the overlap of signatures from different k:
compare_signatures(res)
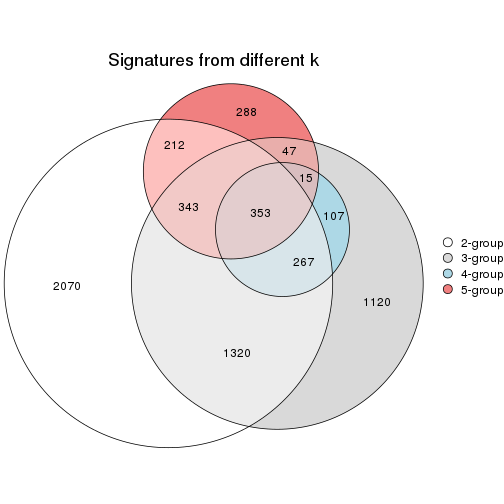
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
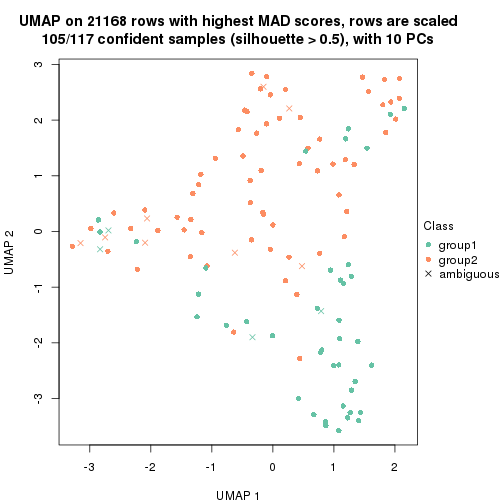
dimension_reduction(res, k = 3, method = "UMAP")
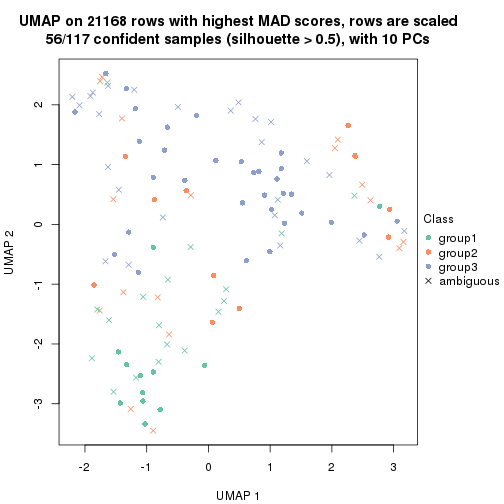
dimension_reduction(res, k = 4, method = "UMAP")
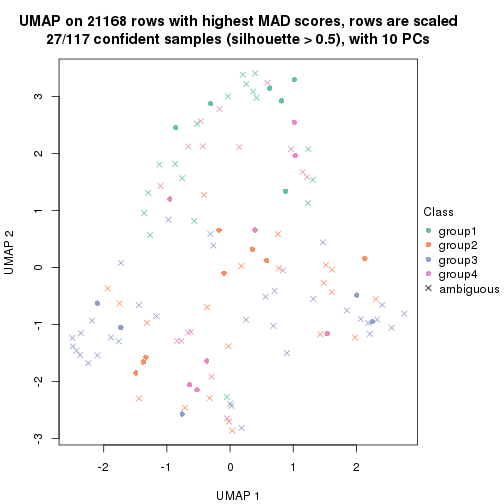
dimension_reduction(res, k = 5, method = "UMAP")
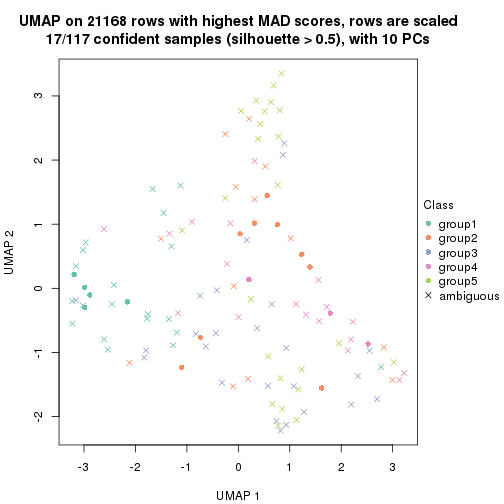
dimension_reduction(res, k = 6, method = "UMAP")
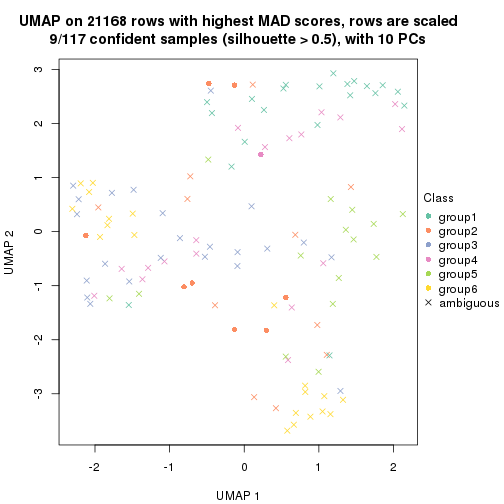
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
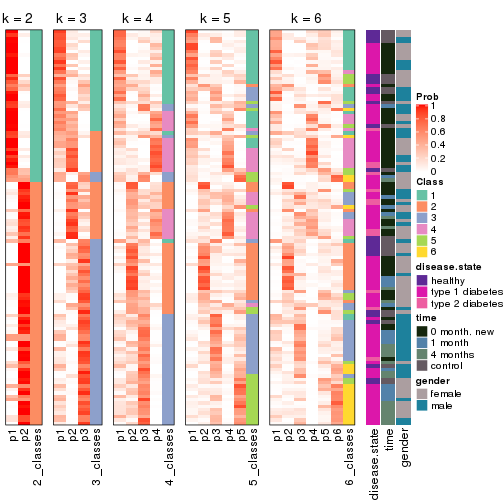
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> MAD:NMF 105 0.3690 2.57e-05 0.8282 2
#> MAD:NMF 56 0.1593 9.71e-03 0.7181 3
#> MAD:NMF 27 0.0579 9.16e-03 0.0236 4
#> MAD:NMF 17 0.1160 3.52e-01 0.2380 5
#> MAD:NMF 9 NA 6.38e-01 NA 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["ATC", "hclust"]
# you can also extract it by
# res = res_list["ATC:hclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'ATC' method.
#> Subgroups are detected by 'hclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 3.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
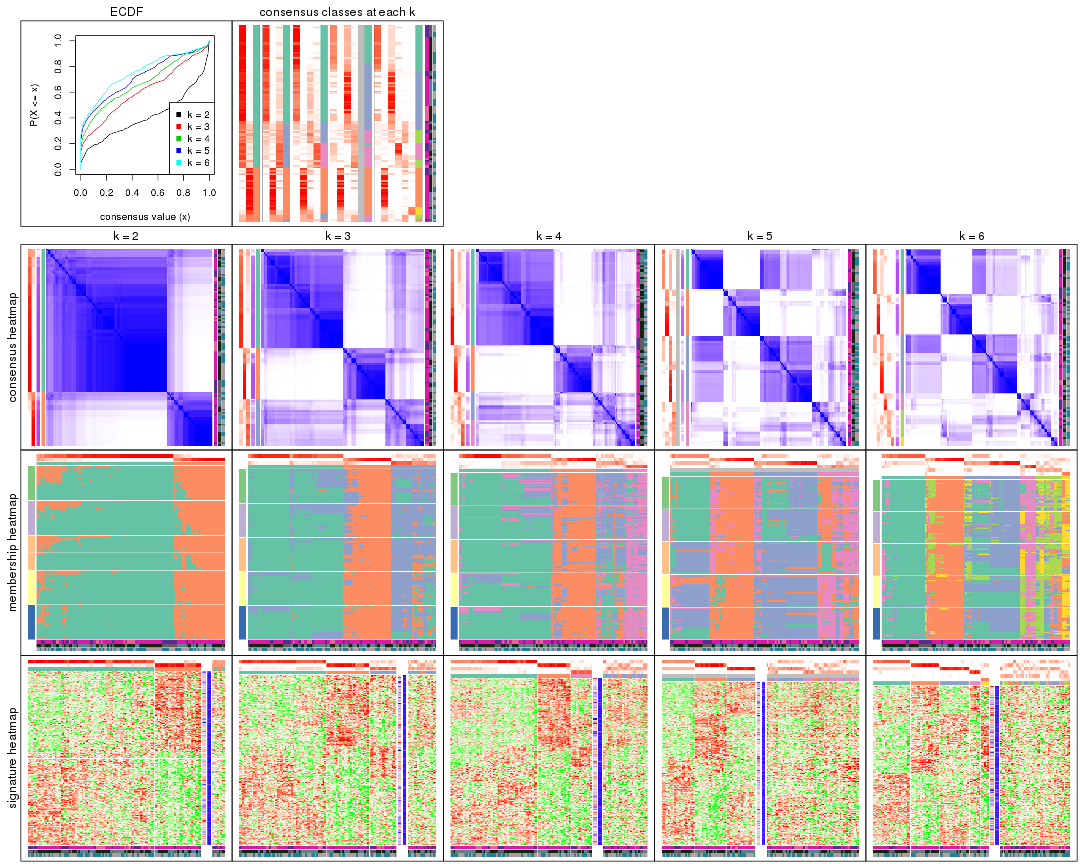
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
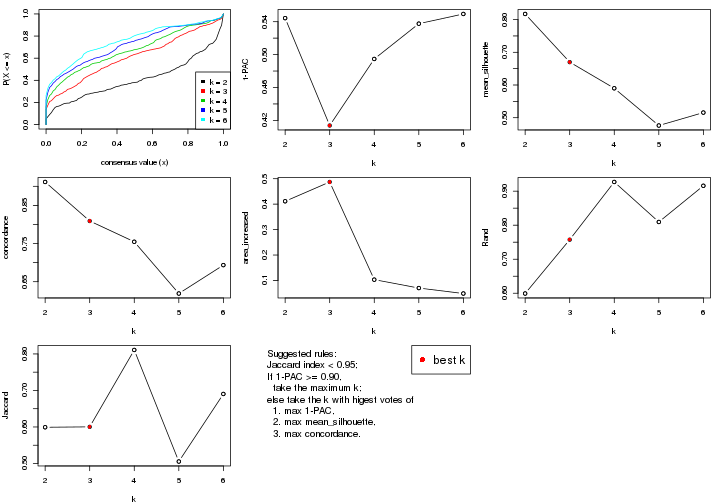
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.544 0.817 0.911 0.4112 0.599 0.599
#> 3 3 0.414 0.670 0.809 0.4871 0.757 0.600
#> 4 4 0.495 0.590 0.755 0.1033 0.927 0.811
#> 5 5 0.538 0.476 0.619 0.0702 0.809 0.505
#> 6 6 0.549 0.516 0.694 0.0487 0.916 0.690
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 3
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.4690 0.864 0.900 0.100
#> GSM228563 1 0.6887 0.793 0.816 0.184
#> GSM228565 1 0.4161 0.875 0.916 0.084
#> GSM228566 2 0.9552 0.399 0.376 0.624
#> GSM228567 1 0.0000 0.904 1.000 0.000
#> GSM228570 1 0.7056 0.782 0.808 0.192
#> GSM228571 1 0.9963 0.168 0.536 0.464
#> GSM228574 2 0.2778 0.873 0.048 0.952
#> GSM228575 2 0.6712 0.773 0.176 0.824
#> GSM228576 1 0.9963 0.168 0.536 0.464
#> GSM228579 1 0.2043 0.900 0.968 0.032
#> GSM228580 1 0.0000 0.904 1.000 0.000
#> GSM228581 1 0.0000 0.904 1.000 0.000
#> GSM228666 1 0.6801 0.798 0.820 0.180
#> GSM228564 1 0.4939 0.858 0.892 0.108
#> GSM228568 1 0.0000 0.904 1.000 0.000
#> GSM228569 1 0.0000 0.904 1.000 0.000
#> GSM228572 2 0.0000 0.883 0.000 1.000
#> GSM228573 2 0.9635 0.401 0.388 0.612
#> GSM228577 1 0.0000 0.904 1.000 0.000
#> GSM228578 1 0.0000 0.904 1.000 0.000
#> GSM228663 1 0.3114 0.893 0.944 0.056
#> GSM228664 1 0.7219 0.775 0.800 0.200
#> GSM228665 1 0.1633 0.904 0.976 0.024
#> GSM228582 1 0.0000 0.904 1.000 0.000
#> GSM228583 1 0.0000 0.904 1.000 0.000
#> GSM228585 1 0.0000 0.904 1.000 0.000
#> GSM228587 1 0.2043 0.901 0.968 0.032
#> GSM228588 1 0.4690 0.864 0.900 0.100
#> GSM228589 1 0.9988 0.111 0.520 0.480
#> GSM228590 1 0.1414 0.903 0.980 0.020
#> GSM228591 1 0.9988 0.111 0.520 0.480
#> GSM228597 1 0.8327 0.689 0.736 0.264
#> GSM228601 2 0.0000 0.883 0.000 1.000
#> GSM228604 2 0.0000 0.883 0.000 1.000
#> GSM228608 1 0.0000 0.904 1.000 0.000
#> GSM228609 2 0.0000 0.883 0.000 1.000
#> GSM228613 1 0.0000 0.904 1.000 0.000
#> GSM228616 2 0.6623 0.778 0.172 0.828
#> GSM228628 1 0.8763 0.627 0.704 0.296
#> GSM228634 1 0.0000 0.904 1.000 0.000
#> GSM228642 2 0.0000 0.883 0.000 1.000
#> GSM228645 2 0.2236 0.877 0.036 0.964
#> GSM228646 2 0.0000 0.883 0.000 1.000
#> GSM228652 1 0.0000 0.904 1.000 0.000
#> GSM228655 1 0.0000 0.904 1.000 0.000
#> GSM228656 1 0.0000 0.904 1.000 0.000
#> GSM228659 1 0.0938 0.905 0.988 0.012
#> GSM228662 1 0.0000 0.904 1.000 0.000
#> GSM228584 1 0.0672 0.904 0.992 0.008
#> GSM228586 1 0.0000 0.904 1.000 0.000
#> GSM228592 1 0.0672 0.904 0.992 0.008
#> GSM228593 1 0.8144 0.702 0.748 0.252
#> GSM228594 1 0.2043 0.900 0.968 0.032
#> GSM228598 1 0.0000 0.904 1.000 0.000
#> GSM228607 1 0.8499 0.669 0.724 0.276
#> GSM228612 2 0.6623 0.778 0.172 0.828
#> GSM228619 2 0.0000 0.883 0.000 1.000
#> GSM228622 1 0.2603 0.894 0.956 0.044
#> GSM228625 1 0.8763 0.627 0.704 0.296
#> GSM228631 2 0.0376 0.883 0.004 0.996
#> GSM228633 2 0.0000 0.883 0.000 1.000
#> GSM228637 1 0.4298 0.872 0.912 0.088
#> GSM228639 1 0.1633 0.903 0.976 0.024
#> GSM228649 1 0.9522 0.465 0.628 0.372
#> GSM228660 1 0.0000 0.904 1.000 0.000
#> GSM228661 1 0.0000 0.904 1.000 0.000
#> GSM228595 2 0.0000 0.883 0.000 1.000
#> GSM228599 2 0.0000 0.883 0.000 1.000
#> GSM228602 2 0.0000 0.883 0.000 1.000
#> GSM228614 1 0.6247 0.823 0.844 0.156
#> GSM228626 2 0.5629 0.817 0.132 0.868
#> GSM228640 2 0.0672 0.883 0.008 0.992
#> GSM228643 1 0.6438 0.818 0.836 0.164
#> GSM228650 1 0.2236 0.901 0.964 0.036
#> GSM228653 1 0.1633 0.903 0.976 0.024
#> GSM228657 2 0.7602 0.719 0.220 0.780
#> GSM228605 1 0.0000 0.904 1.000 0.000
#> GSM228610 1 0.0938 0.905 0.988 0.012
#> GSM228617 2 0.0938 0.883 0.012 0.988
#> GSM228620 1 0.0000 0.904 1.000 0.000
#> GSM228623 1 0.9044 0.588 0.680 0.320
#> GSM228629 2 0.2778 0.873 0.048 0.952
#> GSM228632 1 0.0938 0.905 0.988 0.012
#> GSM228635 1 0.7299 0.772 0.796 0.204
#> GSM228647 1 0.2778 0.897 0.952 0.048
#> GSM228596 1 0.0000 0.904 1.000 0.000
#> GSM228600 2 0.0000 0.883 0.000 1.000
#> GSM228603 2 0.0938 0.883 0.012 0.988
#> GSM228615 1 0.4298 0.872 0.912 0.088
#> GSM228627 1 0.0376 0.904 0.996 0.004
#> GSM228641 2 0.0672 0.883 0.008 0.992
#> GSM228644 2 0.5629 0.818 0.132 0.868
#> GSM228651 1 0.2236 0.901 0.964 0.036
#> GSM228654 1 0.1633 0.903 0.976 0.024
#> GSM228658 1 0.1633 0.903 0.976 0.024
#> GSM228606 2 0.9815 0.280 0.420 0.580
#> GSM228611 1 0.1843 0.903 0.972 0.028
#> GSM228618 2 0.2778 0.873 0.048 0.952
#> GSM228621 2 0.9248 0.502 0.340 0.660
#> GSM228624 2 0.9248 0.502 0.340 0.660
#> GSM228630 1 0.3114 0.891 0.944 0.056
#> GSM228636 1 0.7299 0.772 0.796 0.204
#> GSM228638 1 0.1633 0.903 0.976 0.024
#> GSM228648 1 0.2236 0.901 0.964 0.036
#> GSM228670 1 0.1843 0.903 0.972 0.028
#> GSM228671 1 0.2043 0.902 0.968 0.032
#> GSM228672 1 0.4298 0.872 0.912 0.088
#> GSM228674 1 0.0000 0.904 1.000 0.000
#> GSM228675 1 0.1843 0.903 0.972 0.028
#> GSM228676 1 0.0376 0.905 0.996 0.004
#> GSM228667 1 0.0000 0.904 1.000 0.000
#> GSM228668 1 0.0000 0.904 1.000 0.000
#> GSM228669 1 0.0000 0.904 1.000 0.000
#> GSM228673 1 0.0000 0.904 1.000 0.000
#> GSM228677 1 0.7299 0.772 0.796 0.204
#> GSM228678 1 0.7528 0.758 0.784 0.216
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 3 0.4605 0.6488 0.204 0.000 0.796
#> GSM228563 3 0.6405 0.6739 0.172 0.072 0.756
#> GSM228565 3 0.5363 0.5866 0.276 0.000 0.724
#> GSM228566 3 0.7059 -0.0275 0.020 0.460 0.520
#> GSM228567 1 0.1860 0.7939 0.948 0.000 0.052
#> GSM228570 3 0.6677 0.6799 0.180 0.080 0.740
#> GSM228571 3 0.7949 0.4317 0.084 0.308 0.608
#> GSM228574 2 0.2200 0.8325 0.004 0.940 0.056
#> GSM228575 2 0.5810 0.5133 0.000 0.664 0.336
#> GSM228576 3 0.7949 0.4317 0.084 0.308 0.608
#> GSM228579 1 0.6247 0.2506 0.620 0.004 0.376
#> GSM228580 1 0.1753 0.7941 0.952 0.000 0.048
#> GSM228581 1 0.1753 0.7941 0.952 0.000 0.048
#> GSM228666 3 0.7744 0.2862 0.448 0.048 0.504
#> GSM228564 3 0.4346 0.6578 0.184 0.000 0.816
#> GSM228568 1 0.1860 0.7939 0.948 0.000 0.052
#> GSM228569 1 0.1860 0.7939 0.948 0.000 0.052
#> GSM228572 2 0.0424 0.8480 0.000 0.992 0.008
#> GSM228573 2 0.9030 0.0412 0.152 0.520 0.328
#> GSM228577 1 0.1860 0.7939 0.948 0.000 0.052
#> GSM228578 1 0.2711 0.7929 0.912 0.000 0.088
#> GSM228663 1 0.5728 0.6770 0.720 0.008 0.272
#> GSM228664 1 0.7956 0.1892 0.516 0.060 0.424
#> GSM228665 1 0.4605 0.7533 0.796 0.000 0.204
#> GSM228582 1 0.1753 0.7941 0.952 0.000 0.048
#> GSM228583 1 0.1860 0.7939 0.948 0.000 0.052
#> GSM228585 1 0.1860 0.7939 0.948 0.000 0.052
#> GSM228587 1 0.4521 0.7085 0.816 0.004 0.180
#> GSM228588 3 0.4452 0.6541 0.192 0.000 0.808
#> GSM228589 3 0.9027 0.3782 0.160 0.308 0.532
#> GSM228590 1 0.4062 0.7311 0.836 0.000 0.164
#> GSM228591 3 0.9027 0.3782 0.160 0.308 0.532
#> GSM228597 3 0.8269 0.5479 0.316 0.100 0.584
#> GSM228601 2 0.0592 0.8476 0.000 0.988 0.012
#> GSM228604 2 0.0000 0.8507 0.000 1.000 0.000
#> GSM228608 1 0.1163 0.8004 0.972 0.000 0.028
#> GSM228609 2 0.0592 0.8476 0.000 0.988 0.012
#> GSM228613 1 0.1860 0.7939 0.948 0.000 0.052
#> GSM228616 2 0.5785 0.5204 0.000 0.668 0.332
#> GSM228628 3 0.9317 0.3769 0.388 0.164 0.448
#> GSM228634 1 0.1753 0.7941 0.952 0.000 0.048
#> GSM228642 2 0.0000 0.8507 0.000 1.000 0.000
#> GSM228645 2 0.3340 0.7906 0.000 0.880 0.120
#> GSM228646 2 0.0000 0.8507 0.000 1.000 0.000
#> GSM228652 1 0.1163 0.8004 0.972 0.000 0.028
#> GSM228655 1 0.1163 0.8004 0.972 0.000 0.028
#> GSM228656 1 0.1860 0.7939 0.948 0.000 0.052
#> GSM228659 1 0.3619 0.7725 0.864 0.000 0.136
#> GSM228662 1 0.1860 0.7939 0.948 0.000 0.052
#> GSM228584 1 0.3038 0.7546 0.896 0.000 0.104
#> GSM228586 1 0.1860 0.7939 0.948 0.000 0.052
#> GSM228592 1 0.2625 0.7730 0.916 0.000 0.084
#> GSM228593 3 0.7221 0.6487 0.148 0.136 0.716
#> GSM228594 1 0.6247 0.2506 0.620 0.004 0.376
#> GSM228598 1 0.1753 0.7941 0.952 0.000 0.048
#> GSM228607 3 0.9136 0.3604 0.400 0.144 0.456
#> GSM228612 2 0.5785 0.5204 0.000 0.668 0.332
#> GSM228619 2 0.0000 0.8507 0.000 1.000 0.000
#> GSM228622 1 0.4485 0.7571 0.844 0.020 0.136
#> GSM228625 3 0.9317 0.3769 0.388 0.164 0.448
#> GSM228631 2 0.0237 0.8505 0.000 0.996 0.004
#> GSM228633 2 0.0424 0.8480 0.000 0.992 0.008
#> GSM228637 3 0.5138 0.6163 0.252 0.000 0.748
#> GSM228639 1 0.4887 0.7299 0.772 0.000 0.228
#> GSM228649 3 0.8550 0.5888 0.176 0.216 0.608
#> GSM228660 1 0.1860 0.7957 0.948 0.000 0.052
#> GSM228661 1 0.1753 0.7941 0.952 0.000 0.048
#> GSM228595 2 0.0424 0.8480 0.000 0.992 0.008
#> GSM228599 2 0.0000 0.8507 0.000 1.000 0.000
#> GSM228602 2 0.0000 0.8507 0.000 1.000 0.000
#> GSM228614 3 0.7459 0.4474 0.372 0.044 0.584
#> GSM228626 2 0.5061 0.7104 0.008 0.784 0.208
#> GSM228640 2 0.0475 0.8493 0.004 0.992 0.004
#> GSM228643 1 0.8173 0.0491 0.508 0.072 0.420
#> GSM228650 1 0.5659 0.6978 0.740 0.012 0.248
#> GSM228653 1 0.4887 0.7299 0.772 0.000 0.228
#> GSM228657 2 0.7040 0.5847 0.060 0.688 0.252
#> GSM228605 1 0.2959 0.7853 0.900 0.000 0.100
#> GSM228610 1 0.4002 0.7701 0.840 0.000 0.160
#> GSM228617 2 0.0892 0.8476 0.000 0.980 0.020
#> GSM228620 1 0.2625 0.7966 0.916 0.000 0.084
#> GSM228623 3 0.9040 0.5253 0.320 0.156 0.524
#> GSM228629 2 0.2200 0.8325 0.004 0.940 0.056
#> GSM228632 1 0.4702 0.7419 0.788 0.000 0.212
#> GSM228635 3 0.4056 0.6526 0.092 0.032 0.876
#> GSM228647 1 0.5737 0.6865 0.732 0.012 0.256
#> GSM228596 1 0.2878 0.7859 0.904 0.000 0.096
#> GSM228600 2 0.0000 0.8507 0.000 1.000 0.000
#> GSM228603 2 0.0892 0.8476 0.000 0.980 0.020
#> GSM228615 3 0.5254 0.6232 0.264 0.000 0.736
#> GSM228627 1 0.3686 0.7782 0.860 0.000 0.140
#> GSM228641 2 0.0475 0.8493 0.004 0.992 0.004
#> GSM228644 2 0.5171 0.7129 0.012 0.784 0.204
#> GSM228651 1 0.5659 0.6978 0.740 0.012 0.248
#> GSM228654 1 0.4887 0.7299 0.772 0.000 0.228
#> GSM228658 1 0.4887 0.7299 0.772 0.000 0.228
#> GSM228606 3 0.8659 0.1805 0.104 0.408 0.488
#> GSM228611 1 0.4931 0.7280 0.768 0.000 0.232
#> GSM228618 2 0.2200 0.8325 0.004 0.940 0.056
#> GSM228621 2 0.7672 0.0715 0.044 0.488 0.468
#> GSM228624 2 0.7672 0.0715 0.044 0.488 0.468
#> GSM228630 1 0.6161 0.6567 0.708 0.020 0.272
#> GSM228636 3 0.4056 0.6526 0.092 0.032 0.876
#> GSM228638 1 0.4887 0.7299 0.772 0.000 0.228
#> GSM228648 1 0.5659 0.6978 0.740 0.012 0.248
#> GSM228670 1 0.4974 0.7237 0.764 0.000 0.236
#> GSM228671 1 0.5201 0.7204 0.760 0.004 0.236
#> GSM228672 3 0.5138 0.6163 0.252 0.000 0.748
#> GSM228674 1 0.3116 0.7818 0.892 0.000 0.108
#> GSM228675 1 0.4974 0.7237 0.764 0.000 0.236
#> GSM228676 1 0.3816 0.7839 0.852 0.000 0.148
#> GSM228667 1 0.3267 0.7802 0.884 0.000 0.116
#> GSM228668 1 0.2711 0.7929 0.912 0.000 0.088
#> GSM228669 1 0.3038 0.7851 0.896 0.000 0.104
#> GSM228673 1 0.3116 0.7818 0.892 0.000 0.108
#> GSM228677 3 0.7164 0.5528 0.316 0.044 0.640
#> GSM228678 3 0.4369 0.6568 0.096 0.040 0.864
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.3948 0.71031 0.064 0.000 0.096 0.840
#> GSM228563 4 0.4829 0.68349 0.020 0.056 0.120 0.804
#> GSM228565 4 0.5096 0.64640 0.156 0.000 0.084 0.760
#> GSM228566 2 0.7838 0.01530 0.000 0.404 0.316 0.280
#> GSM228567 1 0.0188 0.73638 0.996 0.000 0.000 0.004
#> GSM228570 4 0.5474 0.66863 0.036 0.080 0.108 0.776
#> GSM228571 3 0.8534 -0.12922 0.028 0.252 0.360 0.360
#> GSM228574 2 0.2197 0.79288 0.000 0.916 0.080 0.004
#> GSM228575 2 0.5872 0.35463 0.000 0.576 0.384 0.040
#> GSM228576 4 0.8534 0.00871 0.028 0.252 0.360 0.360
#> GSM228579 1 0.6155 0.28044 0.648 0.004 0.076 0.272
#> GSM228580 1 0.0000 0.73686 1.000 0.000 0.000 0.000
#> GSM228581 1 0.0000 0.73686 1.000 0.000 0.000 0.000
#> GSM228666 3 0.8004 0.35595 0.344 0.008 0.416 0.232
#> GSM228564 4 0.3463 0.70976 0.040 0.000 0.096 0.864
#> GSM228568 1 0.0188 0.73638 0.996 0.000 0.000 0.004
#> GSM228569 1 0.0188 0.73638 0.996 0.000 0.000 0.004
#> GSM228572 2 0.4868 0.66571 0.000 0.748 0.212 0.040
#> GSM228573 2 0.8554 -0.10842 0.132 0.488 0.296 0.084
#> GSM228577 1 0.0188 0.73638 0.996 0.000 0.000 0.004
#> GSM228578 1 0.4791 0.72726 0.784 0.000 0.136 0.080
#> GSM228663 1 0.5936 0.55345 0.576 0.000 0.380 0.044
#> GSM228664 3 0.6690 0.01759 0.404 0.020 0.528 0.048
#> GSM228665 1 0.5764 0.64602 0.644 0.000 0.304 0.052
#> GSM228582 1 0.0188 0.73873 0.996 0.000 0.004 0.000
#> GSM228583 1 0.0188 0.73638 0.996 0.000 0.000 0.004
#> GSM228585 1 0.0188 0.73638 0.996 0.000 0.000 0.004
#> GSM228587 1 0.4077 0.61225 0.800 0.004 0.012 0.184
#> GSM228588 4 0.3634 0.71158 0.048 0.000 0.096 0.856
#> GSM228589 3 0.6357 0.41247 0.056 0.200 0.696 0.048
#> GSM228590 1 0.3636 0.63440 0.820 0.000 0.008 0.172
#> GSM228591 3 0.6357 0.41247 0.056 0.200 0.696 0.048
#> GSM228597 3 0.8480 0.38394 0.248 0.044 0.476 0.232
#> GSM228601 2 0.0469 0.81084 0.000 0.988 0.000 0.012
#> GSM228604 2 0.0000 0.81340 0.000 1.000 0.000 0.000
#> GSM228608 1 0.2179 0.74791 0.924 0.000 0.064 0.012
#> GSM228609 2 0.0469 0.81084 0.000 0.988 0.000 0.012
#> GSM228613 1 0.0188 0.73638 0.996 0.000 0.000 0.004
#> GSM228616 2 0.5860 0.36312 0.000 0.580 0.380 0.040
#> GSM228628 3 0.8620 0.48145 0.304 0.108 0.480 0.108
#> GSM228634 1 0.0188 0.73873 0.996 0.000 0.004 0.000
#> GSM228642 2 0.0000 0.81340 0.000 1.000 0.000 0.000
#> GSM228645 2 0.3215 0.74071 0.000 0.876 0.092 0.032
#> GSM228646 2 0.0000 0.81340 0.000 1.000 0.000 0.000
#> GSM228652 1 0.2179 0.74791 0.924 0.000 0.064 0.012
#> GSM228655 1 0.2179 0.74791 0.924 0.000 0.064 0.012
#> GSM228656 1 0.0188 0.73638 0.996 0.000 0.000 0.004
#> GSM228659 1 0.5416 0.69618 0.740 0.000 0.112 0.148
#> GSM228662 1 0.0188 0.73638 0.996 0.000 0.000 0.004
#> GSM228584 1 0.2081 0.69231 0.916 0.000 0.000 0.084
#> GSM228586 1 0.0188 0.73638 0.996 0.000 0.000 0.004
#> GSM228592 1 0.1474 0.71333 0.948 0.000 0.000 0.052
#> GSM228593 4 0.5730 0.59852 0.004 0.112 0.160 0.724
#> GSM228594 1 0.6155 0.28044 0.648 0.004 0.076 0.272
#> GSM228598 1 0.0188 0.73873 0.996 0.000 0.004 0.000
#> GSM228607 3 0.8580 0.48484 0.300 0.088 0.484 0.128
#> GSM228612 2 0.5860 0.36312 0.000 0.580 0.380 0.040
#> GSM228619 2 0.0000 0.81340 0.000 1.000 0.000 0.000
#> GSM228622 1 0.6070 0.68566 0.720 0.020 0.152 0.108
#> GSM228625 3 0.8620 0.48145 0.304 0.108 0.480 0.108
#> GSM228631 2 0.0524 0.81158 0.000 0.988 0.004 0.008
#> GSM228633 2 0.4540 0.68816 0.000 0.772 0.196 0.032
#> GSM228637 4 0.4237 0.65201 0.152 0.000 0.040 0.808
#> GSM228639 1 0.5936 0.62454 0.620 0.000 0.324 0.056
#> GSM228649 3 0.9039 0.08921 0.104 0.156 0.424 0.316
#> GSM228660 1 0.0336 0.73964 0.992 0.000 0.008 0.000
#> GSM228661 1 0.0188 0.73873 0.996 0.000 0.004 0.000
#> GSM228595 2 0.4540 0.68816 0.000 0.772 0.196 0.032
#> GSM228599 2 0.0000 0.81340 0.000 1.000 0.000 0.000
#> GSM228602 2 0.0000 0.81340 0.000 1.000 0.000 0.000
#> GSM228614 3 0.7933 0.18717 0.240 0.004 0.388 0.368
#> GSM228626 2 0.5827 0.42272 0.000 0.532 0.436 0.032
#> GSM228640 2 0.0817 0.80868 0.000 0.976 0.024 0.000
#> GSM228643 3 0.8260 0.08534 0.388 0.036 0.416 0.160
#> GSM228650 1 0.6330 0.58556 0.592 0.008 0.344 0.056
#> GSM228653 1 0.5936 0.62454 0.620 0.000 0.324 0.056
#> GSM228657 3 0.6026 -0.31991 0.004 0.468 0.496 0.032
#> GSM228605 1 0.5051 0.71893 0.768 0.000 0.132 0.100
#> GSM228610 1 0.5228 0.68846 0.696 0.000 0.268 0.036
#> GSM228617 2 0.1209 0.80891 0.000 0.964 0.032 0.004
#> GSM228620 1 0.4485 0.73490 0.796 0.000 0.152 0.052
#> GSM228623 3 0.8086 0.47175 0.252 0.080 0.556 0.112
#> GSM228629 2 0.2197 0.79288 0.000 0.916 0.080 0.004
#> GSM228632 1 0.5857 0.63987 0.636 0.000 0.308 0.056
#> GSM228635 4 0.4844 0.62862 0.012 0.016 0.224 0.748
#> GSM228647 1 0.6446 0.57801 0.588 0.008 0.340 0.064
#> GSM228596 1 0.4817 0.72423 0.784 0.000 0.128 0.088
#> GSM228600 2 0.0000 0.81340 0.000 1.000 0.000 0.000
#> GSM228603 2 0.1209 0.80891 0.000 0.964 0.032 0.004
#> GSM228615 4 0.4462 0.64875 0.132 0.000 0.064 0.804
#> GSM228627 1 0.4932 0.70555 0.728 0.000 0.240 0.032
#> GSM228641 2 0.0817 0.80868 0.000 0.976 0.024 0.000
#> GSM228644 2 0.6024 0.43755 0.000 0.540 0.416 0.044
#> GSM228651 1 0.6330 0.58556 0.592 0.008 0.344 0.056
#> GSM228654 1 0.5936 0.62454 0.620 0.000 0.324 0.056
#> GSM228658 1 0.5936 0.62454 0.620 0.000 0.324 0.056
#> GSM228606 3 0.8214 0.33315 0.080 0.324 0.500 0.096
#> GSM228611 1 0.5955 0.62114 0.616 0.000 0.328 0.056
#> GSM228618 2 0.2197 0.79288 0.000 0.916 0.080 0.004
#> GSM228621 3 0.6764 0.15843 0.032 0.384 0.544 0.040
#> GSM228624 3 0.6837 0.15870 0.032 0.384 0.540 0.044
#> GSM228630 1 0.6383 0.52255 0.556 0.004 0.380 0.060
#> GSM228636 4 0.4844 0.62862 0.012 0.016 0.224 0.748
#> GSM228638 1 0.5936 0.62454 0.620 0.000 0.324 0.056
#> GSM228648 1 0.6330 0.58556 0.592 0.008 0.344 0.056
#> GSM228670 1 0.5973 0.61586 0.612 0.000 0.332 0.056
#> GSM228671 1 0.5990 0.61123 0.608 0.000 0.336 0.056
#> GSM228672 4 0.4237 0.65201 0.152 0.000 0.040 0.808
#> GSM228674 1 0.5102 0.71633 0.764 0.000 0.136 0.100
#> GSM228675 1 0.5973 0.61586 0.612 0.000 0.332 0.056
#> GSM228676 1 0.5609 0.70434 0.712 0.000 0.200 0.088
#> GSM228667 1 0.5209 0.71381 0.756 0.000 0.140 0.104
#> GSM228668 1 0.4791 0.72726 0.784 0.000 0.136 0.080
#> GSM228669 1 0.4940 0.72061 0.776 0.000 0.128 0.096
#> GSM228673 1 0.5102 0.71633 0.764 0.000 0.136 0.100
#> GSM228677 4 0.7923 -0.16896 0.208 0.008 0.372 0.412
#> GSM228678 4 0.5295 0.55422 0.016 0.012 0.284 0.688
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 4 0.6740 0.4868 0.328 0.000 0.000 0.404 NA
#> GSM228563 4 0.7645 0.4764 0.292 0.048 0.000 0.376 NA
#> GSM228565 1 0.7508 -0.5065 0.364 0.000 0.040 0.352 NA
#> GSM228566 4 0.7510 0.0313 0.116 0.388 0.000 0.400 NA
#> GSM228567 1 0.4171 0.8121 0.604 0.000 0.396 0.000 NA
#> GSM228570 4 0.8135 0.4707 0.304 0.080 0.004 0.328 NA
#> GSM228571 4 0.8529 0.3160 0.132 0.240 0.036 0.444 NA
#> GSM228574 2 0.2506 0.7632 0.000 0.904 0.008 0.052 NA
#> GSM228575 2 0.5562 0.3146 0.004 0.548 0.000 0.384 NA
#> GSM228576 4 0.8529 0.3160 0.132 0.240 0.036 0.444 NA
#> GSM228579 1 0.5749 0.3833 0.692 0.004 0.176 0.088 NA
#> GSM228580 1 0.4182 0.8085 0.600 0.000 0.400 0.000 NA
#> GSM228581 1 0.4182 0.8085 0.600 0.000 0.400 0.000 NA
#> GSM228666 4 0.6858 -0.0695 0.072 0.004 0.404 0.460 NA
#> GSM228564 4 0.6758 0.4843 0.304 0.000 0.000 0.404 NA
#> GSM228568 1 0.4171 0.8121 0.604 0.000 0.396 0.000 NA
#> GSM228569 1 0.4171 0.8121 0.604 0.000 0.396 0.000 NA
#> GSM228572 2 0.5143 0.4894 0.048 0.584 0.000 0.000 NA
#> GSM228573 2 0.7340 0.1085 0.004 0.476 0.252 0.232 NA
#> GSM228577 1 0.4171 0.8121 0.604 0.000 0.396 0.000 NA
#> GSM228578 3 0.5669 0.1887 0.320 0.000 0.604 0.052 NA
#> GSM228663 3 0.2649 0.5847 0.016 0.000 0.900 0.048 NA
#> GSM228664 3 0.5732 0.4155 0.016 0.004 0.644 0.256 NA
#> GSM228665 3 0.2354 0.5886 0.076 0.000 0.904 0.012 NA
#> GSM228582 1 0.4192 0.8034 0.596 0.000 0.404 0.000 NA
#> GSM228583 1 0.4171 0.8121 0.604 0.000 0.396 0.000 NA
#> GSM228585 1 0.4171 0.8121 0.604 0.000 0.396 0.000 NA
#> GSM228587 1 0.7170 0.5542 0.472 0.004 0.360 0.076 NA
#> GSM228588 4 0.6760 0.4857 0.316 0.000 0.000 0.400 NA
#> GSM228589 4 0.8332 0.2834 0.020 0.168 0.252 0.440 NA
#> GSM228590 1 0.6845 0.5853 0.492 0.000 0.360 0.068 NA
#> GSM228591 4 0.8332 0.2834 0.020 0.168 0.252 0.440 NA
#> GSM228597 4 0.6797 0.2226 0.052 0.032 0.320 0.552 NA
#> GSM228601 2 0.0404 0.7824 0.000 0.988 0.000 0.012 NA
#> GSM228604 2 0.0000 0.7849 0.000 1.000 0.000 0.000 NA
#> GSM228608 3 0.4562 -0.5294 0.492 0.000 0.500 0.008 NA
#> GSM228609 2 0.0404 0.7824 0.000 0.988 0.000 0.012 NA
#> GSM228613 1 0.4171 0.8121 0.604 0.000 0.396 0.000 NA
#> GSM228616 2 0.5553 0.3237 0.004 0.552 0.000 0.380 NA
#> GSM228628 3 0.7408 0.0197 0.032 0.100 0.468 0.360 NA
#> GSM228634 1 0.4192 0.8034 0.596 0.000 0.404 0.000 NA
#> GSM228642 2 0.0000 0.7849 0.000 1.000 0.000 0.000 NA
#> GSM228645 2 0.2624 0.7103 0.000 0.872 0.000 0.116 NA
#> GSM228646 2 0.0000 0.7849 0.000 1.000 0.000 0.000 NA
#> GSM228652 3 0.4562 -0.5294 0.492 0.000 0.500 0.008 NA
#> GSM228655 3 0.4562 -0.5294 0.492 0.000 0.500 0.008 NA
#> GSM228656 1 0.4171 0.8121 0.604 0.000 0.396 0.000 NA
#> GSM228659 3 0.6738 0.0306 0.320 0.000 0.532 0.064 NA
#> GSM228662 1 0.4171 0.8121 0.604 0.000 0.396 0.000 NA
#> GSM228584 1 0.5331 0.7244 0.568 0.000 0.372 0.060 NA
#> GSM228586 1 0.4171 0.8121 0.604 0.000 0.396 0.000 NA
#> GSM228592 1 0.4824 0.7632 0.596 0.000 0.376 0.028 NA
#> GSM228593 4 0.8095 0.4489 0.296 0.096 0.000 0.348 NA
#> GSM228594 1 0.5749 0.3833 0.692 0.004 0.176 0.088 NA
#> GSM228598 1 0.4201 0.7967 0.592 0.000 0.408 0.000 NA
#> GSM228607 3 0.7003 0.0158 0.040 0.076 0.452 0.412 NA
#> GSM228612 2 0.5553 0.3237 0.004 0.552 0.000 0.380 NA
#> GSM228619 2 0.0000 0.7849 0.000 1.000 0.000 0.000 NA
#> GSM228622 3 0.6787 0.2160 0.292 0.020 0.568 0.076 NA
#> GSM228625 3 0.7408 0.0197 0.032 0.100 0.468 0.360 NA
#> GSM228631 2 0.0451 0.7824 0.004 0.988 0.000 0.000 NA
#> GSM228633 2 0.4313 0.5619 0.000 0.636 0.000 0.008 NA
#> GSM228637 4 0.6732 0.4827 0.184 0.000 0.012 0.476 NA
#> GSM228639 3 0.0162 0.6158 0.004 0.000 0.996 0.000 NA
#> GSM228649 4 0.8781 0.4232 0.120 0.140 0.156 0.476 NA
#> GSM228660 1 0.4201 0.7971 0.592 0.000 0.408 0.000 NA
#> GSM228661 1 0.4192 0.8034 0.596 0.000 0.404 0.000 NA
#> GSM228595 2 0.4313 0.5619 0.000 0.636 0.000 0.008 NA
#> GSM228599 2 0.0000 0.7849 0.000 1.000 0.000 0.000 NA
#> GSM228602 2 0.0000 0.7849 0.000 1.000 0.000 0.000 NA
#> GSM228614 4 0.5518 0.2043 0.028 0.000 0.408 0.540 NA
#> GSM228626 2 0.7562 0.3408 0.000 0.424 0.112 0.108 NA
#> GSM228640 2 0.0898 0.7802 0.000 0.972 0.008 0.000 NA
#> GSM228643 3 0.6360 0.3470 0.048 0.032 0.620 0.264 NA
#> GSM228650 3 0.0865 0.6128 0.000 0.004 0.972 0.024 NA
#> GSM228653 3 0.0162 0.6158 0.004 0.000 0.996 0.000 NA
#> GSM228657 2 0.8162 0.2397 0.000 0.376 0.160 0.156 NA
#> GSM228605 3 0.6028 0.1704 0.320 0.000 0.584 0.048 NA
#> GSM228610 3 0.2783 0.5362 0.116 0.000 0.868 0.004 NA
#> GSM228617 2 0.1310 0.7795 0.000 0.956 0.000 0.024 NA
#> GSM228620 3 0.4877 0.2098 0.312 0.000 0.652 0.024 NA
#> GSM228623 4 0.7460 0.1556 0.052 0.052 0.344 0.488 NA
#> GSM228629 2 0.2506 0.7632 0.000 0.904 0.008 0.052 NA
#> GSM228632 3 0.0609 0.6088 0.020 0.000 0.980 0.000 NA
#> GSM228635 4 0.5355 0.4945 0.028 0.004 0.032 0.660 NA
#> GSM228647 3 0.1412 0.6092 0.008 0.004 0.952 0.036 NA
#> GSM228596 3 0.5894 0.1376 0.336 0.000 0.580 0.040 NA
#> GSM228600 2 0.0000 0.7849 0.000 1.000 0.000 0.000 NA
#> GSM228603 2 0.1310 0.7795 0.000 0.956 0.000 0.024 NA
#> GSM228615 4 0.6433 0.4858 0.144 0.000 0.012 0.524 NA
#> GSM228627 3 0.3209 0.4623 0.180 0.000 0.812 0.000 NA
#> GSM228641 2 0.0898 0.7802 0.000 0.972 0.008 0.000 NA
#> GSM228644 2 0.7495 0.3475 0.000 0.428 0.084 0.132 NA
#> GSM228651 3 0.0865 0.6128 0.000 0.004 0.972 0.024 NA
#> GSM228654 3 0.0162 0.6158 0.004 0.000 0.996 0.000 NA
#> GSM228658 3 0.0162 0.6158 0.004 0.000 0.996 0.000 NA
#> GSM228606 4 0.7313 0.1704 0.004 0.312 0.168 0.472 NA
#> GSM228611 3 0.0324 0.6165 0.004 0.000 0.992 0.004 NA
#> GSM228618 2 0.2506 0.7632 0.000 0.904 0.008 0.052 NA
#> GSM228621 4 0.7606 0.0465 0.004 0.356 0.128 0.428 NA
#> GSM228624 4 0.7576 0.0480 0.004 0.356 0.124 0.432 NA
#> GSM228630 3 0.1774 0.5987 0.000 0.000 0.932 0.052 NA
#> GSM228636 4 0.5355 0.4945 0.028 0.004 0.032 0.660 NA
#> GSM228638 3 0.0162 0.6158 0.004 0.000 0.996 0.000 NA
#> GSM228648 3 0.0865 0.6128 0.000 0.004 0.972 0.024 NA
#> GSM228670 3 0.0162 0.6161 0.000 0.000 0.996 0.004 NA
#> GSM228671 3 0.0324 0.6154 0.000 0.000 0.992 0.004 NA
#> GSM228672 4 0.6732 0.4827 0.184 0.000 0.012 0.476 NA
#> GSM228674 3 0.5995 0.2079 0.312 0.000 0.592 0.052 NA
#> GSM228675 3 0.0162 0.6161 0.000 0.000 0.996 0.004 NA
#> GSM228676 3 0.5200 0.3845 0.228 0.000 0.696 0.044 NA
#> GSM228667 3 0.5963 0.2285 0.304 0.000 0.600 0.052 NA
#> GSM228668 3 0.5669 0.1887 0.320 0.000 0.604 0.052 NA
#> GSM228669 3 0.5994 0.1505 0.328 0.000 0.580 0.048 NA
#> GSM228673 3 0.5995 0.2079 0.312 0.000 0.592 0.052 NA
#> GSM228677 4 0.5600 0.3505 0.028 0.000 0.300 0.624 NA
#> GSM228678 4 0.4620 0.5047 0.012 0.000 0.048 0.740 NA
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 4 0.0935 0.68755 0.032 0.000 0.000 0.964 0.004 0.000
#> GSM228563 4 0.2513 0.64404 0.008 0.044 0.000 0.888 0.060 0.000
#> GSM228565 4 0.2636 0.61986 0.120 0.000 0.016 0.860 0.004 0.000
#> GSM228566 2 0.6399 -0.07314 0.020 0.388 0.000 0.220 0.372 0.000
#> GSM228567 1 0.2793 0.87855 0.800 0.000 0.200 0.000 0.000 0.000
#> GSM228570 4 0.2960 0.62872 0.024 0.076 0.004 0.868 0.028 0.000
#> GSM228571 5 0.7343 0.21971 0.040 0.232 0.036 0.300 0.392 0.000
#> GSM228574 2 0.2349 0.74230 0.000 0.892 0.008 0.000 0.080 0.020
#> GSM228575 2 0.4444 0.24927 0.000 0.536 0.000 0.000 0.436 0.028
#> GSM228576 5 0.7343 0.21971 0.040 0.232 0.036 0.300 0.392 0.000
#> GSM228579 1 0.5434 0.46504 0.592 0.000 0.080 0.300 0.028 0.000
#> GSM228580 1 0.2823 0.87717 0.796 0.000 0.204 0.000 0.000 0.000
#> GSM228581 1 0.2823 0.87717 0.796 0.000 0.204 0.000 0.000 0.000
#> GSM228666 5 0.7748 0.11708 0.124 0.000 0.360 0.096 0.360 0.060
#> GSM228564 4 0.0520 0.68308 0.008 0.000 0.000 0.984 0.008 0.000
#> GSM228568 1 0.2793 0.87855 0.800 0.000 0.200 0.000 0.000 0.000
#> GSM228569 1 0.2793 0.87855 0.800 0.000 0.200 0.000 0.000 0.000
#> GSM228572 2 0.6610 -0.37258 0.084 0.476 0.000 0.000 0.124 0.316
#> GSM228573 2 0.7267 -0.04532 0.012 0.472 0.232 0.012 0.208 0.064
#> GSM228577 1 0.2793 0.87855 0.800 0.000 0.200 0.000 0.000 0.000
#> GSM228578 3 0.5140 0.06126 0.440 0.000 0.496 0.048 0.016 0.000
#> GSM228663 3 0.2649 0.56475 0.012 0.000 0.880 0.000 0.072 0.036
#> GSM228664 3 0.4784 0.25573 0.012 0.000 0.624 0.000 0.316 0.048
#> GSM228665 3 0.2231 0.62105 0.068 0.000 0.900 0.000 0.028 0.004
#> GSM228582 1 0.2854 0.87522 0.792 0.000 0.208 0.000 0.000 0.000
#> GSM228583 1 0.2793 0.87855 0.800 0.000 0.200 0.000 0.000 0.000
#> GSM228585 1 0.2793 0.87855 0.800 0.000 0.200 0.000 0.000 0.000
#> GSM228587 1 0.5335 0.69327 0.628 0.004 0.172 0.192 0.004 0.000
#> GSM228588 4 0.0692 0.68732 0.020 0.000 0.000 0.976 0.004 0.000
#> GSM228589 5 0.6789 0.35619 0.012 0.156 0.232 0.000 0.524 0.076
#> GSM228590 1 0.4982 0.71643 0.648 0.000 0.172 0.180 0.000 0.000
#> GSM228591 5 0.6789 0.35619 0.012 0.156 0.232 0.000 0.524 0.076
#> GSM228597 5 0.8128 0.36012 0.108 0.024 0.272 0.088 0.432 0.076
#> GSM228601 2 0.0363 0.77164 0.000 0.988 0.000 0.012 0.000 0.000
#> GSM228604 2 0.0000 0.77381 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228608 1 0.3714 0.65817 0.656 0.000 0.340 0.000 0.004 0.000
#> GSM228609 2 0.0363 0.77164 0.000 0.988 0.000 0.012 0.000 0.000
#> GSM228613 1 0.2793 0.87855 0.800 0.000 0.200 0.000 0.000 0.000
#> GSM228616 2 0.4439 0.25987 0.000 0.540 0.000 0.000 0.432 0.028
#> GSM228628 3 0.7525 -0.18366 0.068 0.096 0.424 0.024 0.352 0.036
#> GSM228634 1 0.2854 0.87522 0.792 0.000 0.208 0.000 0.000 0.000
#> GSM228642 2 0.0000 0.77381 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228645 2 0.2404 0.69251 0.000 0.872 0.000 0.016 0.112 0.000
#> GSM228646 2 0.0000 0.77381 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228652 1 0.3714 0.65817 0.656 0.000 0.340 0.000 0.004 0.000
#> GSM228655 1 0.3714 0.65817 0.656 0.000 0.340 0.000 0.004 0.000
#> GSM228656 1 0.2793 0.87855 0.800 0.000 0.200 0.000 0.000 0.000
#> GSM228659 1 0.6169 0.08363 0.440 0.000 0.420 0.104 0.020 0.016
#> GSM228662 1 0.2793 0.87855 0.800 0.000 0.200 0.000 0.000 0.000
#> GSM228584 1 0.4012 0.82122 0.748 0.000 0.176 0.076 0.000 0.000
#> GSM228586 1 0.2793 0.87855 0.800 0.000 0.200 0.000 0.000 0.000
#> GSM228592 1 0.3588 0.84474 0.776 0.000 0.180 0.044 0.000 0.000
#> GSM228593 4 0.4449 0.51252 0.028 0.092 0.000 0.752 0.128 0.000
#> GSM228594 1 0.5434 0.46504 0.592 0.000 0.080 0.300 0.028 0.000
#> GSM228598 1 0.2883 0.87216 0.788 0.000 0.212 0.000 0.000 0.000
#> GSM228607 3 0.7556 -0.19175 0.092 0.068 0.412 0.024 0.364 0.040
#> GSM228612 2 0.4439 0.25987 0.000 0.540 0.000 0.000 0.432 0.028
#> GSM228619 2 0.0146 0.77420 0.000 0.996 0.000 0.000 0.004 0.000
#> GSM228622 3 0.6426 0.10340 0.404 0.020 0.468 0.052 0.036 0.020
#> GSM228625 3 0.7525 -0.18366 0.068 0.096 0.424 0.024 0.352 0.036
#> GSM228631 2 0.0405 0.76911 0.000 0.988 0.000 0.004 0.008 0.000
#> GSM228633 6 0.3983 0.70643 0.004 0.348 0.000 0.000 0.008 0.640
#> GSM228637 4 0.7361 0.41653 0.200 0.000 0.000 0.412 0.224 0.164
#> GSM228639 3 0.0363 0.64739 0.012 0.000 0.988 0.000 0.000 0.000
#> GSM228649 5 0.8345 0.34711 0.064 0.136 0.140 0.236 0.408 0.016
#> GSM228660 1 0.2883 0.87256 0.788 0.000 0.212 0.000 0.000 0.000
#> GSM228661 1 0.2854 0.87522 0.792 0.000 0.208 0.000 0.000 0.000
#> GSM228595 6 0.3983 0.70643 0.004 0.348 0.000 0.000 0.008 0.640
#> GSM228599 2 0.0000 0.77381 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228602 2 0.0000 0.77381 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228614 5 0.7440 0.26855 0.056 0.000 0.376 0.132 0.376 0.060
#> GSM228626 6 0.5186 0.77972 0.000 0.164 0.080 0.000 0.064 0.692
#> GSM228640 2 0.0976 0.76118 0.000 0.968 0.008 0.000 0.008 0.016
#> GSM228643 3 0.6911 0.24086 0.104 0.020 0.568 0.024 0.216 0.068
#> GSM228650 3 0.0935 0.63486 0.004 0.000 0.964 0.000 0.032 0.000
#> GSM228653 3 0.0363 0.64739 0.012 0.000 0.988 0.000 0.000 0.000
#> GSM228657 6 0.6084 0.70295 0.000 0.140 0.128 0.004 0.104 0.624
#> GSM228605 3 0.5583 0.03953 0.436 0.000 0.480 0.052 0.016 0.016
#> GSM228610 3 0.2581 0.60920 0.128 0.000 0.856 0.016 0.000 0.000
#> GSM228617 2 0.1333 0.76531 0.000 0.944 0.000 0.000 0.048 0.008
#> GSM228620 3 0.4380 0.10598 0.436 0.000 0.544 0.012 0.008 0.000
#> GSM228623 5 0.7262 0.34166 0.104 0.040 0.292 0.032 0.496 0.036
#> GSM228629 2 0.2349 0.74230 0.000 0.892 0.008 0.000 0.080 0.020
#> GSM228632 3 0.0790 0.64935 0.032 0.000 0.968 0.000 0.000 0.000
#> GSM228635 5 0.7331 -0.23887 0.084 0.000 0.008 0.288 0.384 0.236
#> GSM228647 3 0.1511 0.62607 0.012 0.000 0.940 0.000 0.044 0.004
#> GSM228596 3 0.5432 0.00152 0.452 0.000 0.476 0.040 0.016 0.016
#> GSM228600 2 0.0146 0.77420 0.000 0.996 0.000 0.000 0.004 0.000
#> GSM228603 2 0.1333 0.76531 0.000 0.944 0.000 0.000 0.048 0.008
#> GSM228615 4 0.7456 0.38006 0.192 0.000 0.000 0.376 0.264 0.168
#> GSM228627 3 0.3161 0.51810 0.216 0.000 0.776 0.008 0.000 0.000
#> GSM228641 2 0.0976 0.76118 0.000 0.968 0.008 0.000 0.008 0.016
#> GSM228644 6 0.5237 0.77828 0.004 0.168 0.052 0.000 0.084 0.692
#> GSM228651 3 0.0935 0.63486 0.004 0.000 0.964 0.000 0.032 0.000
#> GSM228654 3 0.0363 0.64739 0.012 0.000 0.988 0.000 0.000 0.000
#> GSM228658 3 0.0363 0.64739 0.012 0.000 0.988 0.000 0.000 0.000
#> GSM228606 5 0.7233 0.34145 0.016 0.300 0.160 0.024 0.460 0.040
#> GSM228611 3 0.0508 0.64670 0.012 0.000 0.984 0.000 0.004 0.000
#> GSM228618 2 0.2349 0.74230 0.000 0.892 0.008 0.000 0.080 0.020
#> GSM228621 5 0.6204 0.22287 0.000 0.344 0.116 0.000 0.492 0.048
#> GSM228624 5 0.6281 0.22411 0.000 0.344 0.116 0.004 0.492 0.044
#> GSM228630 3 0.1863 0.60933 0.004 0.000 0.920 0.000 0.060 0.016
#> GSM228636 5 0.7331 -0.23887 0.084 0.000 0.008 0.288 0.384 0.236
#> GSM228638 3 0.0363 0.64739 0.012 0.000 0.988 0.000 0.000 0.000
#> GSM228648 3 0.0935 0.63486 0.004 0.000 0.964 0.000 0.032 0.000
#> GSM228670 3 0.0405 0.64452 0.008 0.000 0.988 0.000 0.004 0.000
#> GSM228671 3 0.0520 0.64382 0.008 0.000 0.984 0.000 0.008 0.000
#> GSM228672 4 0.7361 0.41653 0.200 0.000 0.000 0.412 0.224 0.164
#> GSM228674 3 0.5571 0.10209 0.420 0.000 0.496 0.052 0.016 0.016
#> GSM228675 3 0.0405 0.64452 0.008 0.000 0.988 0.000 0.004 0.000
#> GSM228676 3 0.4919 0.35757 0.316 0.000 0.620 0.048 0.012 0.004
#> GSM228667 3 0.5563 0.12521 0.412 0.000 0.504 0.052 0.016 0.016
#> GSM228668 3 0.5140 0.06126 0.440 0.000 0.496 0.048 0.016 0.000
#> GSM228669 3 0.5536 0.01614 0.444 0.000 0.476 0.048 0.016 0.016
#> GSM228673 3 0.5571 0.10209 0.420 0.000 0.496 0.052 0.016 0.016
#> GSM228677 5 0.7780 0.28970 0.056 0.000 0.272 0.168 0.416 0.088
#> GSM228678 5 0.7327 -0.08945 0.076 0.000 0.036 0.244 0.476 0.168
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
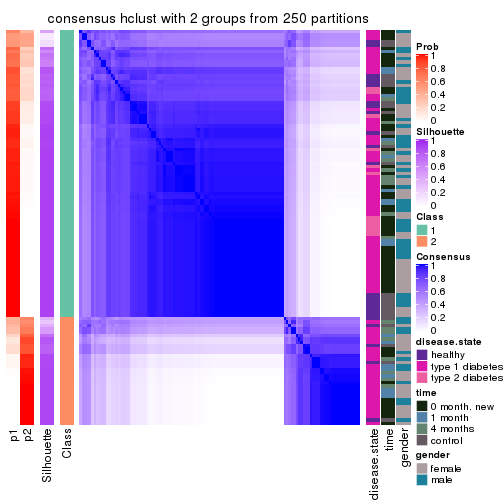
consensus_heatmap(res, k = 3)
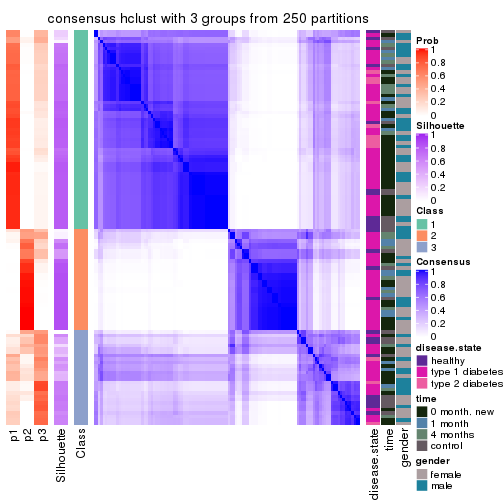
consensus_heatmap(res, k = 4)
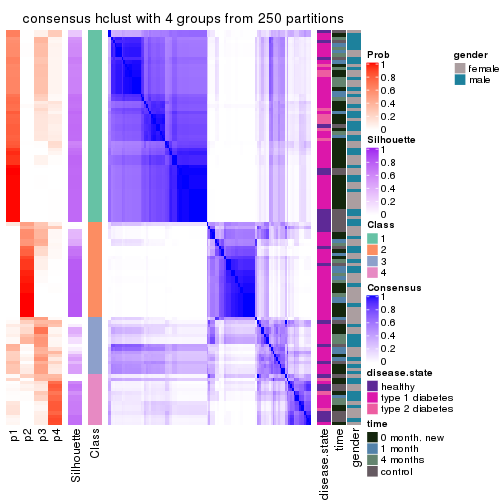
consensus_heatmap(res, k = 5)
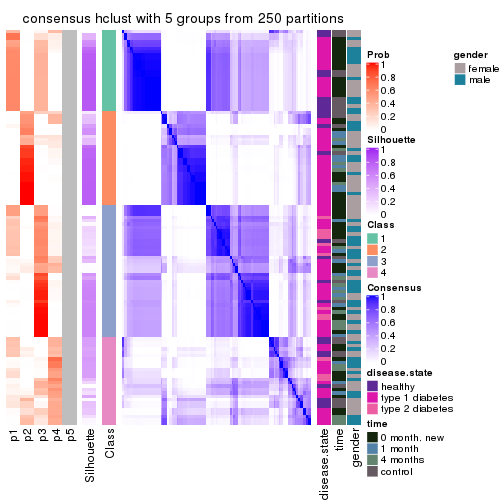
consensus_heatmap(res, k = 6)
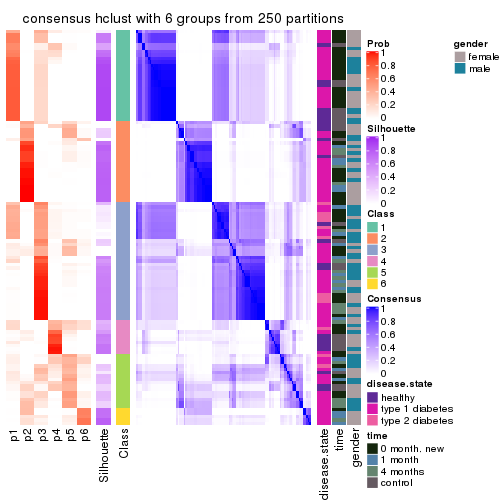
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
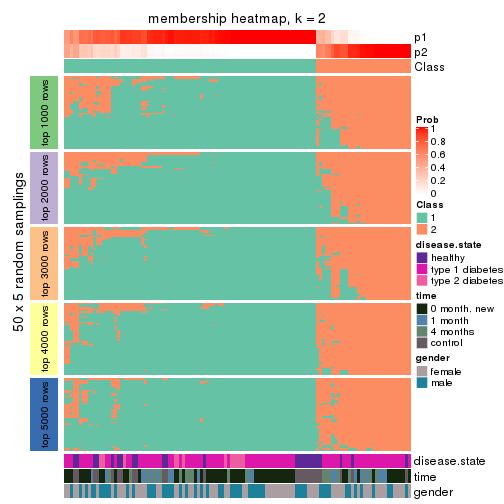
membership_heatmap(res, k = 3)
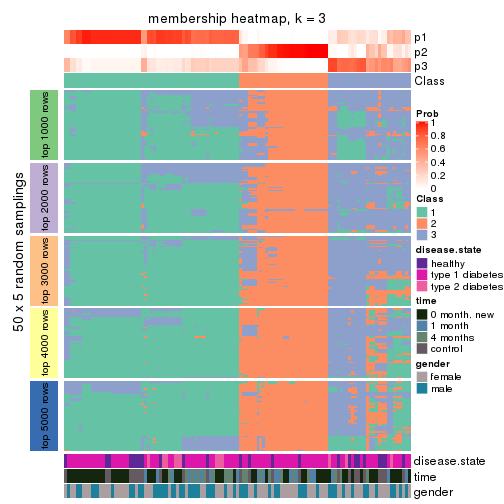
membership_heatmap(res, k = 4)
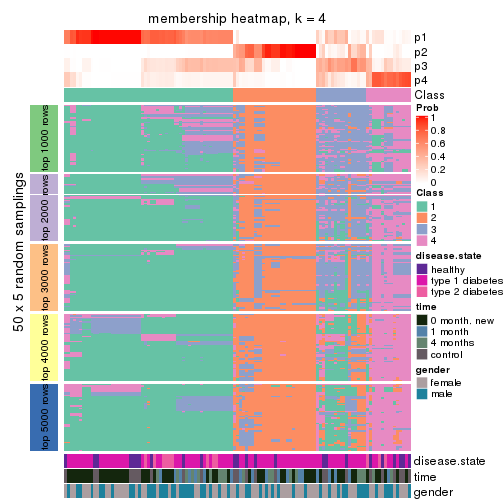
membership_heatmap(res, k = 5)
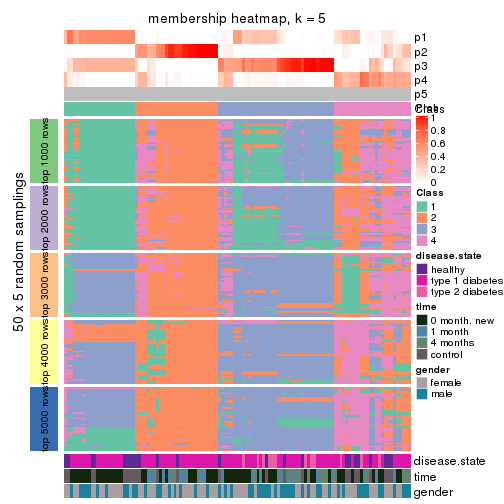
membership_heatmap(res, k = 6)
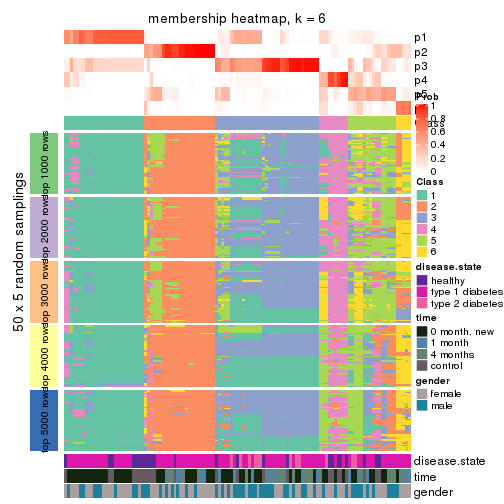
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
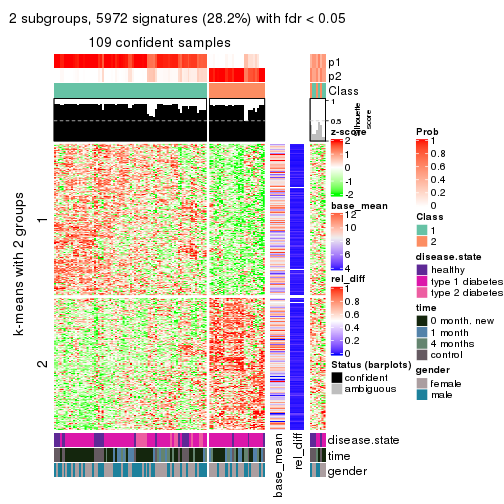
get_signatures(res, k = 3)
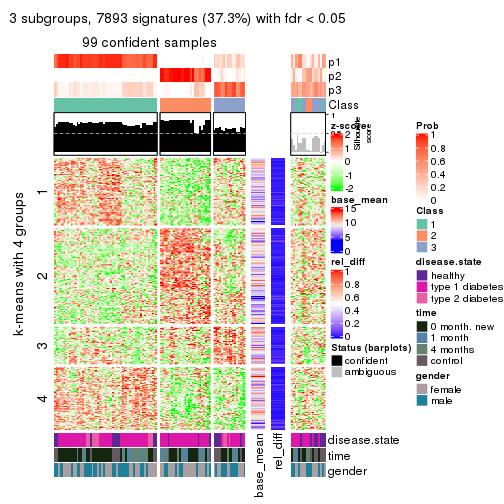
get_signatures(res, k = 4)
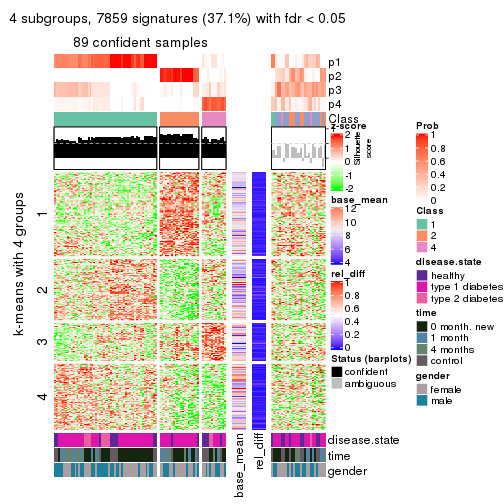
get_signatures(res, k = 5)
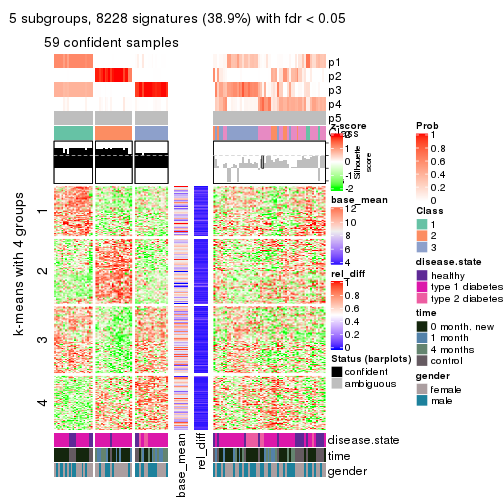
get_signatures(res, k = 6)
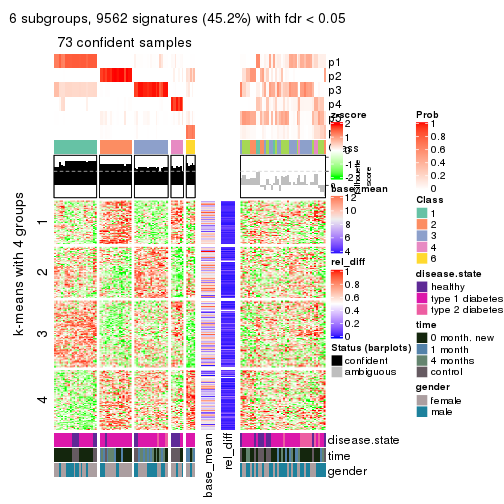
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
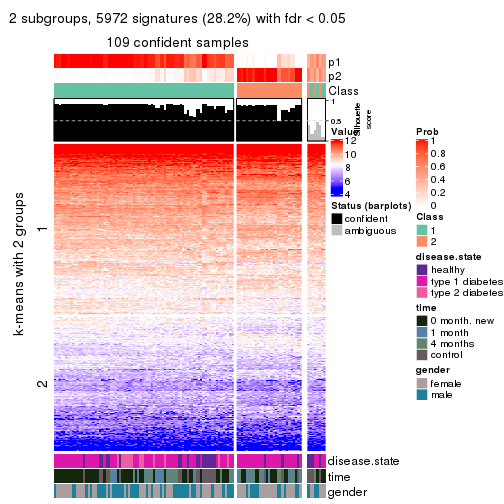
get_signatures(res, k = 3, scale_rows = FALSE)
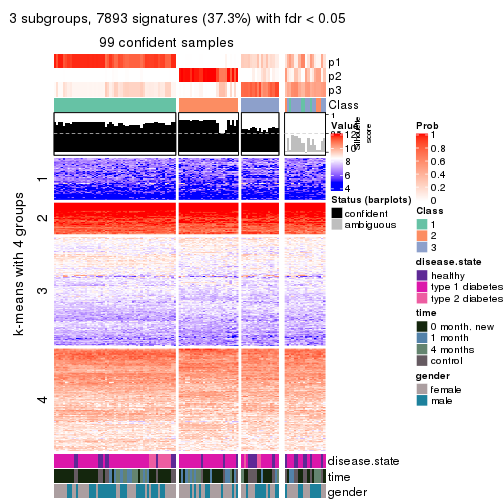
get_signatures(res, k = 4, scale_rows = FALSE)
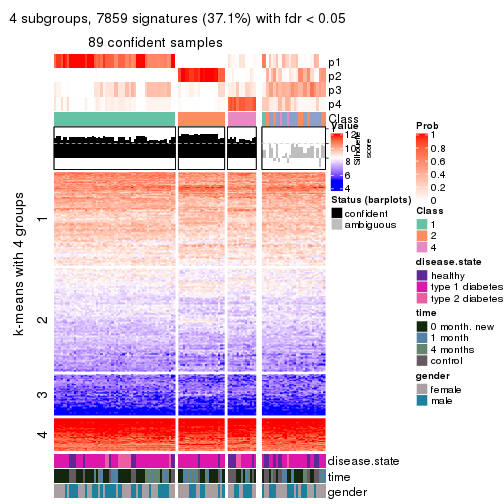
get_signatures(res, k = 5, scale_rows = FALSE)
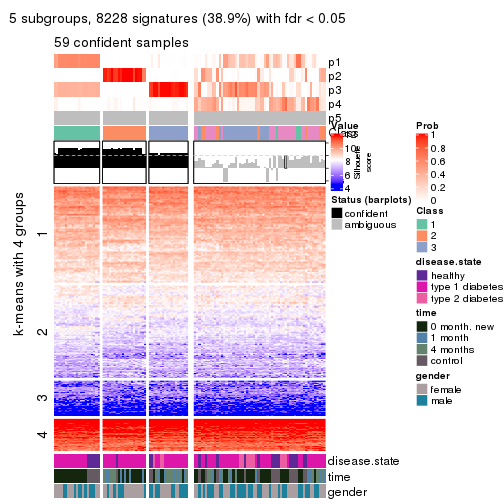
get_signatures(res, k = 6, scale_rows = FALSE)
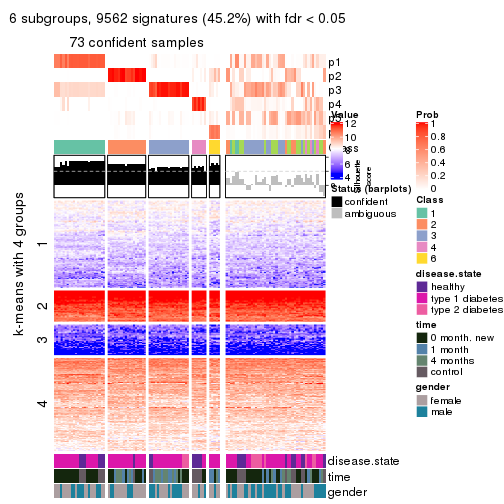
Compare the overlap of signatures from different k:
compare_signatures(res)
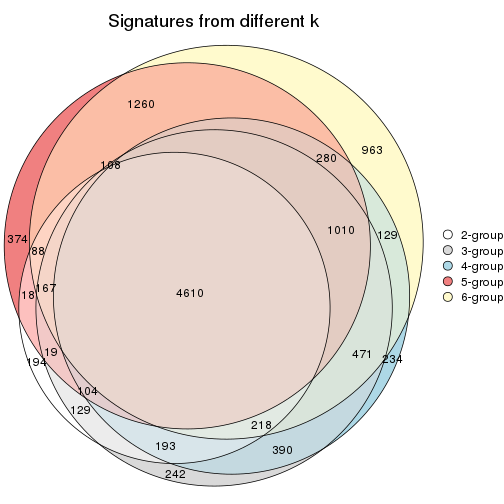
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
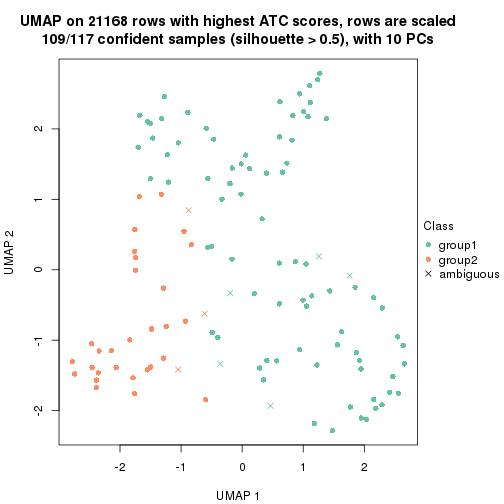
dimension_reduction(res, k = 3, method = "UMAP")
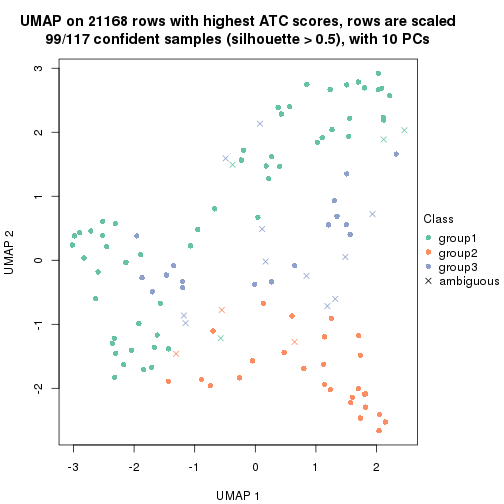
dimension_reduction(res, k = 4, method = "UMAP")
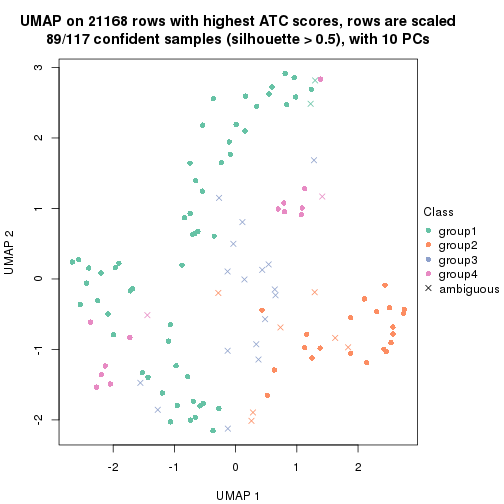
dimension_reduction(res, k = 5, method = "UMAP")
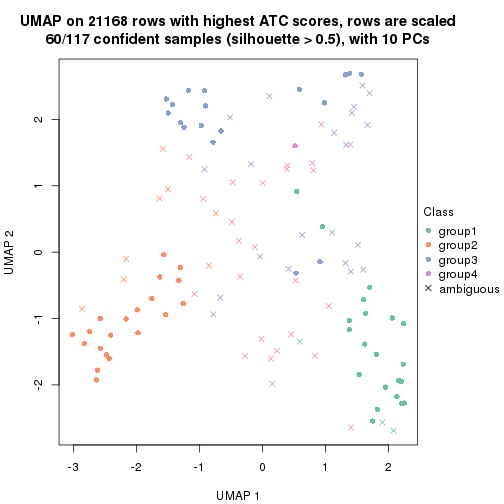
dimension_reduction(res, k = 6, method = "UMAP")
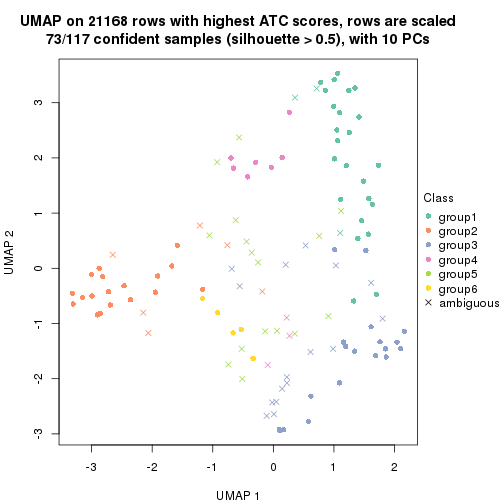
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
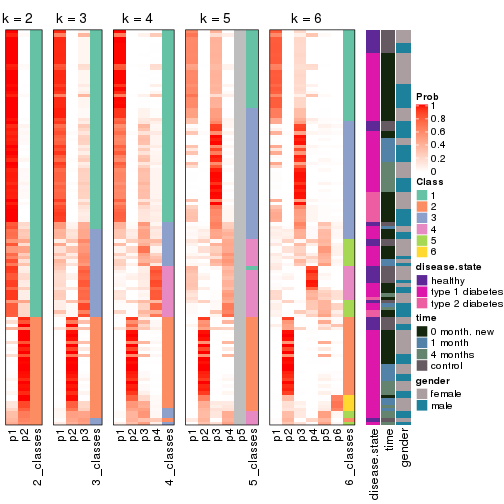
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> ATC:hclust 109 0.020739 1.17e-01 0.269 2
#> ATC:hclust 99 0.068708 3.73e-01 0.185 3
#> ATC:hclust 89 0.045113 2.47e-01 0.464 4
#> ATC:hclust 60 0.000432 3.85e-03 0.278 5
#> ATC:hclust 73 0.001231 5.79e-06 0.480 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["ATC", "kmeans"]
# you can also extract it by
# res = res_list["ATC:kmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'ATC' method.
#> Subgroups are detected by 'kmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
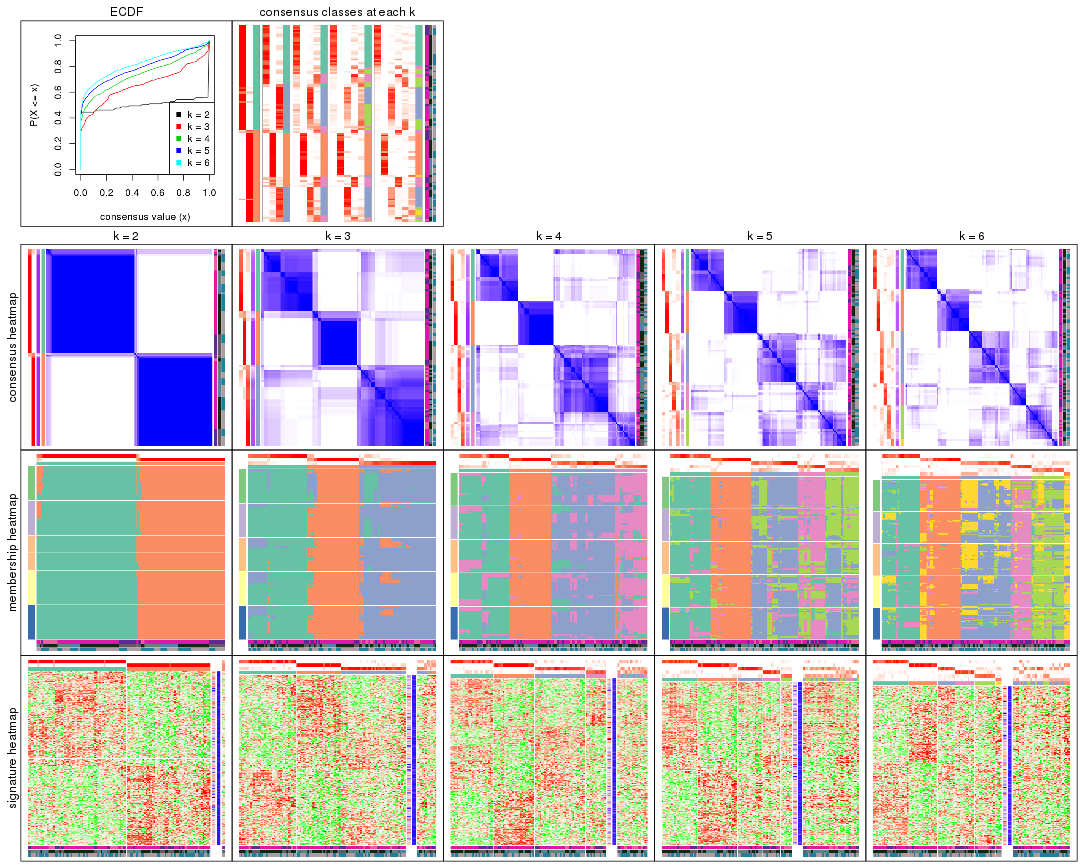
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
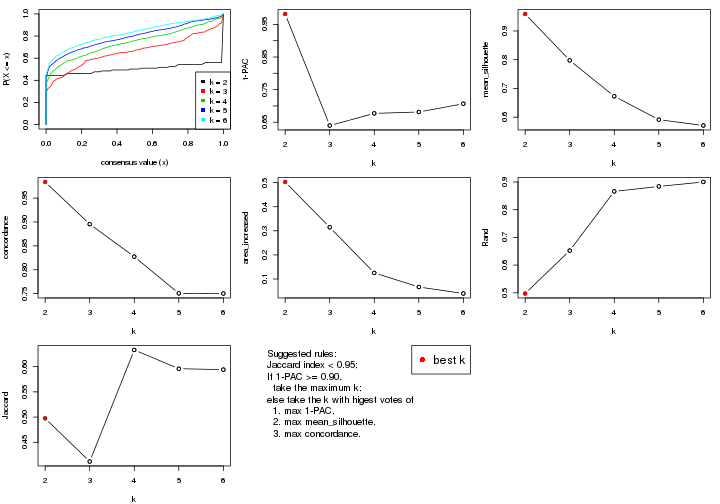
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.982 0.959 0.984 0.5026 0.497 0.497
#> 3 3 0.640 0.798 0.895 0.3146 0.652 0.412
#> 4 4 0.677 0.673 0.827 0.1254 0.866 0.633
#> 5 5 0.681 0.591 0.751 0.0669 0.884 0.596
#> 6 6 0.707 0.571 0.750 0.0398 0.900 0.594
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 2 0.529 0.8535 0.120 0.880
#> GSM228563 2 0.000 0.9817 0.000 1.000
#> GSM228565 1 0.000 0.9842 1.000 0.000
#> GSM228566 2 0.000 0.9817 0.000 1.000
#> GSM228567 1 0.000 0.9842 1.000 0.000
#> GSM228570 2 0.000 0.9817 0.000 1.000
#> GSM228571 2 0.952 0.4130 0.372 0.628
#> GSM228574 2 0.000 0.9817 0.000 1.000
#> GSM228575 2 0.000 0.9817 0.000 1.000
#> GSM228576 2 0.000 0.9817 0.000 1.000
#> GSM228579 1 0.000 0.9842 1.000 0.000
#> GSM228580 1 0.000 0.9842 1.000 0.000
#> GSM228581 1 0.000 0.9842 1.000 0.000
#> GSM228666 1 0.000 0.9842 1.000 0.000
#> GSM228564 2 0.000 0.9817 0.000 1.000
#> GSM228568 1 0.000 0.9842 1.000 0.000
#> GSM228569 1 0.000 0.9842 1.000 0.000
#> GSM228572 2 0.000 0.9817 0.000 1.000
#> GSM228573 2 0.000 0.9817 0.000 1.000
#> GSM228577 1 0.000 0.9842 1.000 0.000
#> GSM228578 1 0.000 0.9842 1.000 0.000
#> GSM228663 1 0.482 0.8762 0.896 0.104
#> GSM228664 2 0.000 0.9817 0.000 1.000
#> GSM228665 1 0.000 0.9842 1.000 0.000
#> GSM228582 1 0.000 0.9842 1.000 0.000
#> GSM228583 1 0.000 0.9842 1.000 0.000
#> GSM228585 1 0.000 0.9842 1.000 0.000
#> GSM228587 1 0.000 0.9842 1.000 0.000
#> GSM228588 1 0.000 0.9842 1.000 0.000
#> GSM228589 2 0.000 0.9817 0.000 1.000
#> GSM228590 1 0.000 0.9842 1.000 0.000
#> GSM228591 2 0.000 0.9817 0.000 1.000
#> GSM228597 2 0.000 0.9817 0.000 1.000
#> GSM228601 2 0.000 0.9817 0.000 1.000
#> GSM228604 2 0.000 0.9817 0.000 1.000
#> GSM228608 1 0.000 0.9842 1.000 0.000
#> GSM228609 2 0.000 0.9817 0.000 1.000
#> GSM228613 1 0.000 0.9842 1.000 0.000
#> GSM228616 2 0.000 0.9817 0.000 1.000
#> GSM228628 2 0.000 0.9817 0.000 1.000
#> GSM228634 1 0.000 0.9842 1.000 0.000
#> GSM228642 2 0.000 0.9817 0.000 1.000
#> GSM228645 2 0.000 0.9817 0.000 1.000
#> GSM228646 2 0.000 0.9817 0.000 1.000
#> GSM228652 1 0.000 0.9842 1.000 0.000
#> GSM228655 1 0.000 0.9842 1.000 0.000
#> GSM228656 1 0.000 0.9842 1.000 0.000
#> GSM228659 1 0.000 0.9842 1.000 0.000
#> GSM228662 1 0.000 0.9842 1.000 0.000
#> GSM228584 1 0.000 0.9842 1.000 0.000
#> GSM228586 1 0.000 0.9842 1.000 0.000
#> GSM228592 1 0.000 0.9842 1.000 0.000
#> GSM228593 2 0.000 0.9817 0.000 1.000
#> GSM228594 1 0.000 0.9842 1.000 0.000
#> GSM228598 1 0.000 0.9842 1.000 0.000
#> GSM228607 2 0.000 0.9817 0.000 1.000
#> GSM228612 2 0.000 0.9817 0.000 1.000
#> GSM228619 2 0.000 0.9817 0.000 1.000
#> GSM228622 1 0.000 0.9842 1.000 0.000
#> GSM228625 2 0.000 0.9817 0.000 1.000
#> GSM228631 2 0.000 0.9817 0.000 1.000
#> GSM228633 2 0.000 0.9817 0.000 1.000
#> GSM228637 1 0.000 0.9842 1.000 0.000
#> GSM228639 1 0.000 0.9842 1.000 0.000
#> GSM228649 2 0.000 0.9817 0.000 1.000
#> GSM228660 1 0.000 0.9842 1.000 0.000
#> GSM228661 1 0.000 0.9842 1.000 0.000
#> GSM228595 2 0.000 0.9817 0.000 1.000
#> GSM228599 2 0.000 0.9817 0.000 1.000
#> GSM228602 2 0.000 0.9817 0.000 1.000
#> GSM228614 2 0.000 0.9817 0.000 1.000
#> GSM228626 2 0.000 0.9817 0.000 1.000
#> GSM228640 2 0.000 0.9817 0.000 1.000
#> GSM228643 1 0.821 0.6606 0.744 0.256
#> GSM228650 1 0.886 0.5705 0.696 0.304
#> GSM228653 1 0.000 0.9842 1.000 0.000
#> GSM228657 2 0.000 0.9817 0.000 1.000
#> GSM228605 1 0.000 0.9842 1.000 0.000
#> GSM228610 1 0.000 0.9842 1.000 0.000
#> GSM228617 2 0.000 0.9817 0.000 1.000
#> GSM228620 1 0.000 0.9842 1.000 0.000
#> GSM228623 2 0.000 0.9817 0.000 1.000
#> GSM228629 2 0.000 0.9817 0.000 1.000
#> GSM228632 1 0.000 0.9842 1.000 0.000
#> GSM228635 2 0.000 0.9817 0.000 1.000
#> GSM228647 1 0.000 0.9842 1.000 0.000
#> GSM228596 1 0.000 0.9842 1.000 0.000
#> GSM228600 2 0.000 0.9817 0.000 1.000
#> GSM228603 2 0.000 0.9817 0.000 1.000
#> GSM228615 1 0.000 0.9842 1.000 0.000
#> GSM228627 1 0.000 0.9842 1.000 0.000
#> GSM228641 2 0.000 0.9817 0.000 1.000
#> GSM228644 2 0.000 0.9817 0.000 1.000
#> GSM228651 2 0.998 0.0787 0.472 0.528
#> GSM228654 1 0.000 0.9842 1.000 0.000
#> GSM228658 1 0.000 0.9842 1.000 0.000
#> GSM228606 2 0.000 0.9817 0.000 1.000
#> GSM228611 1 0.000 0.9842 1.000 0.000
#> GSM228618 2 0.000 0.9817 0.000 1.000
#> GSM228621 2 0.000 0.9817 0.000 1.000
#> GSM228624 2 0.000 0.9817 0.000 1.000
#> GSM228630 2 0.000 0.9817 0.000 1.000
#> GSM228636 2 0.000 0.9817 0.000 1.000
#> GSM228638 1 0.000 0.9842 1.000 0.000
#> GSM228648 1 0.000 0.9842 1.000 0.000
#> GSM228670 1 0.000 0.9842 1.000 0.000
#> GSM228671 1 0.844 0.6324 0.728 0.272
#> GSM228672 1 0.000 0.9842 1.000 0.000
#> GSM228674 1 0.000 0.9842 1.000 0.000
#> GSM228675 1 0.000 0.9842 1.000 0.000
#> GSM228676 1 0.000 0.9842 1.000 0.000
#> GSM228667 1 0.000 0.9842 1.000 0.000
#> GSM228668 1 0.000 0.9842 1.000 0.000
#> GSM228669 1 0.000 0.9842 1.000 0.000
#> GSM228673 1 0.000 0.9842 1.000 0.000
#> GSM228677 2 0.000 0.9817 0.000 1.000
#> GSM228678 2 0.000 0.9817 0.000 1.000
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.7333 0.556 0.704 0.180 0.116
#> GSM228563 2 0.8834 0.463 0.316 0.544 0.140
#> GSM228565 1 0.0000 0.879 1.000 0.000 0.000
#> GSM228566 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228567 1 0.1163 0.883 0.972 0.000 0.028
#> GSM228570 2 0.8792 0.316 0.392 0.492 0.116
#> GSM228571 1 0.8543 0.327 0.592 0.268 0.140
#> GSM228574 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228575 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228576 2 0.4979 0.744 0.020 0.812 0.168
#> GSM228579 1 0.0000 0.879 1.000 0.000 0.000
#> GSM228580 1 0.4842 0.787 0.776 0.000 0.224
#> GSM228581 1 0.4750 0.794 0.784 0.000 0.216
#> GSM228666 3 0.1289 0.849 0.032 0.000 0.968
#> GSM228564 2 0.8627 0.336 0.392 0.504 0.104
#> GSM228568 1 0.1031 0.884 0.976 0.000 0.024
#> GSM228569 1 0.1643 0.881 0.956 0.000 0.044
#> GSM228572 2 0.0237 0.925 0.000 0.996 0.004
#> GSM228573 3 0.4702 0.726 0.000 0.212 0.788
#> GSM228577 1 0.1031 0.884 0.976 0.000 0.024
#> GSM228578 1 0.4605 0.803 0.796 0.000 0.204
#> GSM228663 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228664 3 0.2066 0.847 0.000 0.060 0.940
#> GSM228665 3 0.3038 0.783 0.104 0.000 0.896
#> GSM228582 1 0.5016 0.767 0.760 0.000 0.240
#> GSM228583 1 0.1163 0.883 0.972 0.000 0.028
#> GSM228585 1 0.0000 0.879 1.000 0.000 0.000
#> GSM228587 1 0.0000 0.879 1.000 0.000 0.000
#> GSM228588 1 0.0237 0.876 0.996 0.004 0.000
#> GSM228589 3 0.2878 0.830 0.000 0.096 0.904
#> GSM228590 1 0.0000 0.879 1.000 0.000 0.000
#> GSM228591 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228597 2 0.5223 0.731 0.024 0.800 0.176
#> GSM228601 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228604 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228608 1 0.1411 0.883 0.964 0.000 0.036
#> GSM228609 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228613 1 0.1163 0.883 0.972 0.000 0.028
#> GSM228616 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228628 3 0.4654 0.731 0.000 0.208 0.792
#> GSM228634 1 0.4750 0.794 0.784 0.000 0.216
#> GSM228642 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228645 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228646 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228652 1 0.4750 0.794 0.784 0.000 0.216
#> GSM228655 1 0.4750 0.794 0.784 0.000 0.216
#> GSM228656 1 0.1163 0.883 0.972 0.000 0.028
#> GSM228659 1 0.0237 0.880 0.996 0.000 0.004
#> GSM228662 1 0.0000 0.879 1.000 0.000 0.000
#> GSM228584 1 0.0000 0.879 1.000 0.000 0.000
#> GSM228586 1 0.1163 0.883 0.972 0.000 0.028
#> GSM228592 1 0.0592 0.882 0.988 0.000 0.012
#> GSM228593 2 0.8514 0.390 0.372 0.528 0.100
#> GSM228594 1 0.0000 0.879 1.000 0.000 0.000
#> GSM228598 1 0.4750 0.794 0.784 0.000 0.216
#> GSM228607 3 0.1529 0.851 0.000 0.040 0.960
#> GSM228612 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228619 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228622 3 0.3816 0.735 0.148 0.000 0.852
#> GSM228625 3 0.3826 0.808 0.008 0.124 0.868
#> GSM228631 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228633 2 0.0237 0.925 0.000 0.996 0.004
#> GSM228637 1 0.2066 0.864 0.940 0.000 0.060
#> GSM228639 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228649 3 0.8799 0.469 0.220 0.196 0.584
#> GSM228660 1 0.5016 0.767 0.760 0.000 0.240
#> GSM228661 1 0.4750 0.794 0.784 0.000 0.216
#> GSM228595 2 0.0237 0.925 0.000 0.996 0.004
#> GSM228599 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228602 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228614 3 0.2187 0.849 0.024 0.028 0.948
#> GSM228626 3 0.4750 0.719 0.000 0.216 0.784
#> GSM228640 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228643 3 0.1289 0.849 0.032 0.000 0.968
#> GSM228650 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228653 3 0.0592 0.851 0.012 0.000 0.988
#> GSM228657 3 0.4750 0.719 0.000 0.216 0.784
#> GSM228605 1 0.5529 0.691 0.704 0.000 0.296
#> GSM228610 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228617 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228620 3 0.5591 0.487 0.304 0.000 0.696
#> GSM228623 3 0.2878 0.830 0.000 0.096 0.904
#> GSM228629 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228632 3 0.3038 0.783 0.104 0.000 0.896
#> GSM228635 3 0.3083 0.840 0.024 0.060 0.916
#> GSM228647 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228596 1 0.5465 0.700 0.712 0.000 0.288
#> GSM228600 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228603 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228615 3 0.5785 0.474 0.332 0.000 0.668
#> GSM228627 3 0.3038 0.783 0.104 0.000 0.896
#> GSM228641 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228644 3 0.4750 0.719 0.000 0.216 0.784
#> GSM228651 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228654 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228658 3 0.0592 0.851 0.012 0.000 0.988
#> GSM228606 3 0.4346 0.759 0.000 0.184 0.816
#> GSM228611 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228618 2 0.0000 0.928 0.000 1.000 0.000
#> GSM228621 3 0.4178 0.769 0.000 0.172 0.828
#> GSM228624 3 0.2878 0.830 0.000 0.096 0.904
#> GSM228630 3 0.1411 0.852 0.000 0.036 0.964
#> GSM228636 3 0.7102 0.287 0.024 0.420 0.556
#> GSM228638 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228648 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228670 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228671 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228672 1 0.1860 0.866 0.948 0.000 0.052
#> GSM228674 3 0.5706 0.449 0.320 0.000 0.680
#> GSM228675 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228676 3 0.4346 0.691 0.184 0.000 0.816
#> GSM228667 3 0.0237 0.854 0.004 0.000 0.996
#> GSM228668 3 0.5948 0.342 0.360 0.000 0.640
#> GSM228669 1 0.1860 0.869 0.948 0.000 0.052
#> GSM228673 3 0.5431 0.525 0.284 0.000 0.716
#> GSM228677 3 0.2550 0.846 0.024 0.040 0.936
#> GSM228678 3 0.7278 0.165 0.028 0.456 0.516
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.3487 0.6549 0.064 0.040 0.016 0.880
#> GSM228563 4 0.3595 0.6634 0.032 0.068 0.024 0.876
#> GSM228565 4 0.3764 0.5144 0.216 0.000 0.000 0.784
#> GSM228566 2 0.2149 0.8964 0.000 0.912 0.000 0.088
#> GSM228567 1 0.3569 0.7133 0.804 0.000 0.000 0.196
#> GSM228570 4 0.3471 0.6612 0.036 0.068 0.016 0.880
#> GSM228571 4 0.6211 0.3795 0.264 0.044 0.028 0.664
#> GSM228574 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228575 2 0.3004 0.8720 0.000 0.892 0.048 0.060
#> GSM228576 4 0.6080 0.0744 0.000 0.468 0.044 0.488
#> GSM228579 1 0.4356 0.6372 0.708 0.000 0.000 0.292
#> GSM228580 1 0.4801 0.5085 0.764 0.000 0.048 0.188
#> GSM228581 1 0.1209 0.7284 0.964 0.000 0.032 0.004
#> GSM228666 4 0.4989 0.0401 0.000 0.000 0.472 0.528
#> GSM228564 4 0.3498 0.6608 0.044 0.060 0.016 0.880
#> GSM228568 1 0.3569 0.7133 0.804 0.000 0.000 0.196
#> GSM228569 1 0.0188 0.7361 0.996 0.000 0.000 0.004
#> GSM228572 2 0.1557 0.9348 0.000 0.944 0.000 0.056
#> GSM228573 3 0.2281 0.7526 0.000 0.000 0.904 0.096
#> GSM228577 1 0.3569 0.7133 0.804 0.000 0.000 0.196
#> GSM228578 1 0.0921 0.7312 0.972 0.000 0.028 0.000
#> GSM228663 3 0.4356 0.7321 0.148 0.000 0.804 0.048
#> GSM228664 3 0.2216 0.7543 0.000 0.000 0.908 0.092
#> GSM228665 3 0.4252 0.6427 0.252 0.000 0.744 0.004
#> GSM228582 1 0.1398 0.7236 0.956 0.000 0.040 0.004
#> GSM228583 1 0.3569 0.7133 0.804 0.000 0.000 0.196
#> GSM228585 1 0.4134 0.6703 0.740 0.000 0.000 0.260
#> GSM228587 1 0.4961 0.3616 0.552 0.000 0.000 0.448
#> GSM228588 4 0.3400 0.5599 0.180 0.000 0.000 0.820
#> GSM228589 3 0.2216 0.7543 0.000 0.000 0.908 0.092
#> GSM228590 1 0.4040 0.6777 0.752 0.000 0.000 0.248
#> GSM228591 2 0.5110 0.7375 0.000 0.764 0.104 0.132
#> GSM228597 4 0.5213 0.5715 0.000 0.052 0.224 0.724
#> GSM228601 2 0.1474 0.9376 0.000 0.948 0.000 0.052
#> GSM228604 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228608 1 0.0469 0.7371 0.988 0.000 0.000 0.012
#> GSM228609 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228613 1 0.3311 0.7190 0.828 0.000 0.000 0.172
#> GSM228616 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228628 3 0.2281 0.7526 0.000 0.000 0.904 0.096
#> GSM228634 1 0.0921 0.7315 0.972 0.000 0.028 0.000
#> GSM228642 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228645 2 0.0188 0.9645 0.000 0.996 0.000 0.004
#> GSM228646 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228652 1 0.1118 0.7282 0.964 0.000 0.036 0.000
#> GSM228655 1 0.1118 0.7282 0.964 0.000 0.036 0.000
#> GSM228656 1 0.3528 0.7150 0.808 0.000 0.000 0.192
#> GSM228659 4 0.4155 0.5099 0.240 0.000 0.004 0.756
#> GSM228662 1 0.4134 0.6703 0.740 0.000 0.000 0.260
#> GSM228584 1 0.4008 0.6817 0.756 0.000 0.000 0.244
#> GSM228586 1 0.0592 0.7372 0.984 0.000 0.000 0.016
#> GSM228592 1 0.3528 0.7144 0.808 0.000 0.000 0.192
#> GSM228593 4 0.5807 0.5565 0.080 0.176 0.016 0.728
#> GSM228594 1 0.4356 0.6372 0.708 0.000 0.000 0.292
#> GSM228598 1 0.0921 0.7315 0.972 0.000 0.028 0.000
#> GSM228607 3 0.2216 0.7535 0.000 0.000 0.908 0.092
#> GSM228612 2 0.2644 0.8899 0.000 0.908 0.032 0.060
#> GSM228619 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228622 3 0.7247 0.3763 0.240 0.000 0.544 0.216
#> GSM228625 3 0.2281 0.7526 0.000 0.000 0.904 0.096
#> GSM228631 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228633 2 0.2480 0.9085 0.000 0.904 0.008 0.088
#> GSM228637 4 0.5229 0.5763 0.168 0.000 0.084 0.748
#> GSM228639 3 0.1716 0.7588 0.064 0.000 0.936 0.000
#> GSM228649 4 0.4993 0.5650 0.008 0.020 0.244 0.728
#> GSM228660 1 0.1978 0.7017 0.928 0.000 0.068 0.004
#> GSM228661 1 0.0921 0.7315 0.972 0.000 0.028 0.000
#> GSM228595 2 0.1557 0.9348 0.000 0.944 0.000 0.056
#> GSM228599 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228602 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228614 3 0.4790 0.3802 0.000 0.000 0.620 0.380
#> GSM228626 3 0.3695 0.7127 0.000 0.016 0.828 0.156
#> GSM228640 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228643 3 0.3649 0.6316 0.000 0.000 0.796 0.204
#> GSM228650 3 0.0817 0.7660 0.000 0.000 0.976 0.024
#> GSM228653 3 0.3791 0.6877 0.200 0.000 0.796 0.004
#> GSM228657 3 0.3881 0.7032 0.000 0.016 0.812 0.172
#> GSM228605 1 0.7327 -0.0207 0.504 0.000 0.176 0.320
#> GSM228610 3 0.3219 0.7106 0.164 0.000 0.836 0.000
#> GSM228617 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228620 3 0.4961 0.3205 0.448 0.000 0.552 0.000
#> GSM228623 3 0.2281 0.7526 0.000 0.000 0.904 0.096
#> GSM228629 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228632 3 0.4040 0.6486 0.248 0.000 0.752 0.000
#> GSM228635 4 0.4522 0.4063 0.000 0.000 0.320 0.680
#> GSM228647 3 0.2466 0.7273 0.004 0.000 0.900 0.096
#> GSM228596 1 0.4139 0.5642 0.800 0.000 0.176 0.024
#> GSM228600 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228603 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228615 4 0.7317 0.4202 0.204 0.000 0.268 0.528
#> GSM228627 3 0.4008 0.6523 0.244 0.000 0.756 0.000
#> GSM228641 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228644 3 0.3852 0.7015 0.000 0.012 0.808 0.180
#> GSM228651 3 0.1474 0.7636 0.000 0.000 0.948 0.052
#> GSM228654 3 0.1302 0.7635 0.044 0.000 0.956 0.000
#> GSM228658 3 0.3791 0.6877 0.200 0.000 0.796 0.004
#> GSM228606 3 0.4955 0.4407 0.000 0.008 0.648 0.344
#> GSM228611 3 0.1389 0.7628 0.048 0.000 0.952 0.000
#> GSM228618 2 0.0000 0.9668 0.000 1.000 0.000 0.000
#> GSM228621 3 0.2216 0.7535 0.000 0.000 0.908 0.092
#> GSM228624 3 0.2216 0.7535 0.000 0.000 0.908 0.092
#> GSM228630 3 0.1867 0.7631 0.000 0.000 0.928 0.072
#> GSM228636 4 0.4560 0.4381 0.000 0.004 0.296 0.700
#> GSM228638 3 0.3583 0.7024 0.180 0.000 0.816 0.004
#> GSM228648 3 0.0000 0.7659 0.000 0.000 1.000 0.000
#> GSM228670 3 0.1940 0.7550 0.076 0.000 0.924 0.000
#> GSM228671 3 0.0188 0.7662 0.000 0.000 0.996 0.004
#> GSM228672 4 0.5122 0.5770 0.164 0.000 0.080 0.756
#> GSM228674 1 0.7921 -0.2194 0.348 0.000 0.332 0.320
#> GSM228675 3 0.1940 0.7550 0.076 0.000 0.924 0.000
#> GSM228676 3 0.7170 0.3936 0.288 0.000 0.540 0.172
#> GSM228667 3 0.6712 0.2633 0.104 0.000 0.552 0.344
#> GSM228668 1 0.7919 -0.2162 0.348 0.000 0.336 0.316
#> GSM228669 4 0.6172 0.4377 0.284 0.000 0.084 0.632
#> GSM228673 3 0.7894 -0.0118 0.296 0.000 0.372 0.332
#> GSM228677 3 0.4961 0.1948 0.000 0.000 0.552 0.448
#> GSM228678 4 0.5416 0.5325 0.000 0.048 0.260 0.692
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 4 0.1375 0.7418 0.008 0.008 0.008 0.960 0.016
#> GSM228563 4 0.1488 0.7414 0.008 0.012 0.008 0.956 0.016
#> GSM228565 4 0.1469 0.7237 0.036 0.000 0.000 0.948 0.016
#> GSM228566 2 0.4082 0.7251 0.000 0.776 0.032 0.184 0.008
#> GSM228567 1 0.2424 0.8114 0.868 0.000 0.000 0.132 0.000
#> GSM228570 4 0.1488 0.7414 0.008 0.012 0.008 0.956 0.016
#> GSM228571 4 0.3926 0.6289 0.112 0.020 0.048 0.820 0.000
#> GSM228574 2 0.0162 0.9278 0.000 0.996 0.000 0.000 0.004
#> GSM228575 2 0.3858 0.7265 0.000 0.760 0.224 0.008 0.008
#> GSM228576 4 0.4677 0.5278 0.000 0.252 0.036 0.704 0.008
#> GSM228579 1 0.4297 0.3320 0.528 0.000 0.000 0.472 0.000
#> GSM228580 5 0.5492 0.0887 0.396 0.000 0.000 0.068 0.536
#> GSM228581 1 0.2230 0.7801 0.884 0.000 0.000 0.000 0.116
#> GSM228666 4 0.6960 0.1271 0.004 0.000 0.328 0.336 0.332
#> GSM228564 4 0.1375 0.7418 0.008 0.008 0.008 0.960 0.016
#> GSM228568 1 0.2424 0.8114 0.868 0.000 0.000 0.132 0.000
#> GSM228569 1 0.0162 0.8172 0.996 0.000 0.000 0.000 0.004
#> GSM228572 2 0.3556 0.8167 0.000 0.808 0.004 0.020 0.168
#> GSM228573 3 0.0693 0.6444 0.000 0.000 0.980 0.008 0.012
#> GSM228577 1 0.2424 0.8114 0.868 0.000 0.000 0.132 0.000
#> GSM228578 1 0.3336 0.6369 0.772 0.000 0.000 0.000 0.228
#> GSM228663 3 0.4455 0.4833 0.096 0.000 0.768 0.004 0.132
#> GSM228664 3 0.0693 0.6448 0.000 0.000 0.980 0.008 0.012
#> GSM228665 5 0.6012 0.3743 0.120 0.000 0.376 0.000 0.504
#> GSM228582 1 0.2017 0.7947 0.912 0.000 0.008 0.000 0.080
#> GSM228583 1 0.2424 0.8114 0.868 0.000 0.000 0.132 0.000
#> GSM228585 1 0.3210 0.7578 0.788 0.000 0.000 0.212 0.000
#> GSM228587 4 0.4066 0.2483 0.324 0.000 0.000 0.672 0.004
#> GSM228588 4 0.1106 0.7315 0.024 0.000 0.000 0.964 0.012
#> GSM228589 3 0.0912 0.6428 0.000 0.000 0.972 0.012 0.016
#> GSM228590 1 0.4106 0.7035 0.724 0.000 0.000 0.256 0.020
#> GSM228591 3 0.6742 0.1045 0.000 0.292 0.504 0.016 0.188
#> GSM228597 4 0.5191 0.6054 0.000 0.008 0.192 0.700 0.100
#> GSM228601 2 0.3360 0.8217 0.000 0.816 0.004 0.012 0.168
#> GSM228604 2 0.0162 0.9276 0.000 0.996 0.000 0.000 0.004
#> GSM228608 1 0.2179 0.7842 0.888 0.000 0.000 0.000 0.112
#> GSM228609 2 0.0162 0.9273 0.000 0.996 0.000 0.000 0.004
#> GSM228613 1 0.2124 0.8159 0.900 0.000 0.000 0.096 0.004
#> GSM228616 2 0.0740 0.9220 0.000 0.980 0.008 0.004 0.008
#> GSM228628 3 0.0566 0.6464 0.000 0.000 0.984 0.012 0.004
#> GSM228634 1 0.1331 0.8078 0.952 0.000 0.008 0.000 0.040
#> GSM228642 2 0.0162 0.9273 0.000 0.996 0.000 0.000 0.004
#> GSM228645 2 0.0981 0.9188 0.000 0.972 0.008 0.012 0.008
#> GSM228646 2 0.0290 0.9269 0.000 0.992 0.000 0.000 0.008
#> GSM228652 1 0.2439 0.7677 0.876 0.000 0.004 0.000 0.120
#> GSM228655 1 0.2583 0.7600 0.864 0.000 0.004 0.000 0.132
#> GSM228656 1 0.2424 0.8114 0.868 0.000 0.000 0.132 0.000
#> GSM228659 4 0.5933 0.4817 0.108 0.000 0.004 0.556 0.332
#> GSM228662 1 0.3210 0.7578 0.788 0.000 0.000 0.212 0.000
#> GSM228584 1 0.3039 0.7747 0.808 0.000 0.000 0.192 0.000
#> GSM228586 1 0.0451 0.8183 0.988 0.000 0.000 0.008 0.004
#> GSM228592 1 0.2488 0.8127 0.872 0.000 0.000 0.124 0.004
#> GSM228593 4 0.3130 0.6694 0.072 0.040 0.016 0.872 0.000
#> GSM228594 1 0.4273 0.3918 0.552 0.000 0.000 0.448 0.000
#> GSM228598 1 0.0693 0.8146 0.980 0.000 0.008 0.000 0.012
#> GSM228607 3 0.0579 0.6457 0.000 0.000 0.984 0.008 0.008
#> GSM228612 2 0.4064 0.6670 0.000 0.716 0.272 0.004 0.008
#> GSM228619 2 0.0000 0.9277 0.000 1.000 0.000 0.000 0.000
#> GSM228622 5 0.6066 0.5019 0.112 0.000 0.260 0.020 0.608
#> GSM228625 3 0.0566 0.6464 0.000 0.000 0.984 0.012 0.004
#> GSM228631 2 0.0000 0.9277 0.000 1.000 0.000 0.000 0.000
#> GSM228633 2 0.4888 0.7619 0.000 0.740 0.068 0.020 0.172
#> GSM228637 4 0.5419 0.4435 0.044 0.000 0.008 0.548 0.400
#> GSM228639 3 0.4557 0.0326 0.012 0.000 0.584 0.000 0.404
#> GSM228649 4 0.3859 0.6816 0.004 0.000 0.096 0.816 0.084
#> GSM228660 1 0.3779 0.6067 0.752 0.000 0.012 0.000 0.236
#> GSM228661 1 0.1571 0.8022 0.936 0.000 0.004 0.000 0.060
#> GSM228595 2 0.3594 0.8155 0.000 0.804 0.004 0.020 0.172
#> GSM228599 2 0.0000 0.9277 0.000 1.000 0.000 0.000 0.000
#> GSM228602 2 0.0000 0.9277 0.000 1.000 0.000 0.000 0.000
#> GSM228614 3 0.6260 0.1650 0.000 0.000 0.516 0.172 0.312
#> GSM228626 3 0.3795 0.5459 0.000 0.004 0.788 0.024 0.184
#> GSM228640 2 0.0162 0.9278 0.000 0.996 0.000 0.000 0.004
#> GSM228643 3 0.5396 0.2646 0.000 0.000 0.588 0.072 0.340
#> GSM228650 3 0.3039 0.5195 0.000 0.000 0.808 0.000 0.192
#> GSM228653 5 0.5929 0.2989 0.104 0.000 0.432 0.000 0.464
#> GSM228657 3 0.3831 0.5440 0.000 0.004 0.784 0.024 0.188
#> GSM228605 5 0.5942 0.5324 0.148 0.000 0.116 0.056 0.680
#> GSM228610 5 0.5346 0.2849 0.052 0.000 0.452 0.000 0.496
#> GSM228617 2 0.0162 0.9278 0.000 0.996 0.000 0.000 0.004
#> GSM228620 5 0.6402 0.4295 0.208 0.000 0.288 0.000 0.504
#> GSM228623 3 0.0566 0.6464 0.000 0.000 0.984 0.012 0.004
#> GSM228629 2 0.1569 0.9043 0.000 0.944 0.044 0.004 0.008
#> GSM228632 5 0.5930 0.3915 0.112 0.000 0.372 0.000 0.516
#> GSM228635 3 0.6744 -0.0505 0.000 0.000 0.404 0.272 0.324
#> GSM228647 5 0.4559 0.1165 0.000 0.000 0.480 0.008 0.512
#> GSM228596 5 0.5420 0.3284 0.396 0.000 0.052 0.004 0.548
#> GSM228600 2 0.0162 0.9278 0.000 0.996 0.000 0.000 0.004
#> GSM228603 2 0.0162 0.9278 0.000 0.996 0.000 0.000 0.004
#> GSM228615 5 0.5797 0.3099 0.048 0.000 0.064 0.228 0.660
#> GSM228627 5 0.5971 0.3638 0.112 0.000 0.396 0.000 0.492
#> GSM228641 2 0.0162 0.9278 0.000 0.996 0.000 0.000 0.004
#> GSM228644 3 0.4268 0.5084 0.000 0.004 0.728 0.024 0.244
#> GSM228651 3 0.2966 0.5283 0.000 0.000 0.816 0.000 0.184
#> GSM228654 3 0.4354 0.1575 0.008 0.000 0.624 0.000 0.368
#> GSM228658 5 0.5929 0.2989 0.104 0.000 0.432 0.000 0.464
#> GSM228606 3 0.2446 0.5986 0.000 0.000 0.900 0.044 0.056
#> GSM228611 3 0.4354 0.1525 0.008 0.000 0.624 0.000 0.368
#> GSM228618 2 0.0865 0.9192 0.000 0.972 0.024 0.000 0.004
#> GSM228621 3 0.0566 0.6464 0.000 0.000 0.984 0.012 0.004
#> GSM228624 3 0.0566 0.6464 0.000 0.000 0.984 0.012 0.004
#> GSM228630 3 0.1341 0.6243 0.000 0.000 0.944 0.000 0.056
#> GSM228636 5 0.6786 -0.2232 0.000 0.000 0.324 0.292 0.384
#> GSM228638 5 0.5858 0.2546 0.096 0.000 0.452 0.000 0.452
#> GSM228648 3 0.3395 0.4588 0.000 0.000 0.764 0.000 0.236
#> GSM228670 3 0.4648 -0.1559 0.012 0.000 0.524 0.000 0.464
#> GSM228671 3 0.3210 0.4971 0.000 0.000 0.788 0.000 0.212
#> GSM228672 4 0.5375 0.4745 0.044 0.000 0.008 0.568 0.380
#> GSM228674 5 0.5089 0.5317 0.068 0.000 0.104 0.072 0.756
#> GSM228675 3 0.4555 -0.1649 0.008 0.000 0.520 0.000 0.472
#> GSM228676 5 0.4133 0.5245 0.052 0.000 0.180 0.000 0.768
#> GSM228667 5 0.4863 0.5086 0.028 0.000 0.140 0.076 0.756
#> GSM228668 5 0.5147 0.5317 0.068 0.000 0.104 0.076 0.752
#> GSM228669 5 0.5658 0.0784 0.064 0.000 0.016 0.316 0.604
#> GSM228673 5 0.4992 0.5296 0.052 0.000 0.112 0.076 0.760
#> GSM228677 3 0.6262 0.1673 0.000 0.000 0.520 0.176 0.304
#> GSM228678 4 0.6280 0.4698 0.000 0.004 0.164 0.540 0.292
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 4 0.1719 0.8188 0.008 0.008 0.000 0.928 0.056 0.000
#> GSM228563 4 0.1707 0.8191 0.004 0.012 0.000 0.928 0.056 0.000
#> GSM228565 4 0.2901 0.7639 0.032 0.000 0.000 0.840 0.128 0.000
#> GSM228566 2 0.5226 0.0160 0.000 0.460 0.000 0.448 0.000 0.092
#> GSM228567 1 0.1387 0.8084 0.932 0.000 0.000 0.068 0.000 0.000
#> GSM228570 4 0.1707 0.8191 0.004 0.012 0.000 0.928 0.056 0.000
#> GSM228571 4 0.2881 0.7717 0.048 0.012 0.000 0.872 0.004 0.064
#> GSM228574 2 0.0458 0.8181 0.000 0.984 0.000 0.000 0.000 0.016
#> GSM228575 2 0.6067 0.1055 0.000 0.504 0.072 0.048 0.008 0.368
#> GSM228576 4 0.3554 0.6939 0.000 0.108 0.000 0.808 0.004 0.080
#> GSM228579 4 0.3997 0.0460 0.488 0.000 0.000 0.508 0.004 0.000
#> GSM228580 5 0.4944 0.5047 0.232 0.000 0.040 0.008 0.684 0.036
#> GSM228581 1 0.4506 0.7298 0.760 0.000 0.024 0.008 0.108 0.100
#> GSM228666 5 0.5025 0.5810 0.000 0.000 0.064 0.116 0.716 0.104
#> GSM228564 4 0.1707 0.8191 0.004 0.012 0.000 0.928 0.056 0.000
#> GSM228568 1 0.1387 0.8084 0.932 0.000 0.000 0.068 0.000 0.000
#> GSM228569 1 0.0291 0.8152 0.992 0.000 0.000 0.004 0.004 0.000
#> GSM228572 2 0.5022 0.4597 0.000 0.576 0.000 0.008 0.064 0.352
#> GSM228573 3 0.5208 0.1057 0.000 0.004 0.532 0.036 0.024 0.404
#> GSM228577 1 0.1387 0.8084 0.932 0.000 0.000 0.068 0.000 0.000
#> GSM228578 1 0.5102 0.6334 0.672 0.000 0.032 0.000 0.212 0.084
#> GSM228663 3 0.5407 0.3255 0.040 0.000 0.572 0.012 0.028 0.348
#> GSM228664 3 0.5041 0.0678 0.000 0.000 0.512 0.036 0.020 0.432
#> GSM228665 3 0.5329 0.4809 0.048 0.000 0.688 0.004 0.120 0.140
#> GSM228582 1 0.4276 0.7416 0.776 0.000 0.060 0.004 0.036 0.124
#> GSM228583 1 0.1327 0.8097 0.936 0.000 0.000 0.064 0.000 0.000
#> GSM228585 1 0.2146 0.7755 0.880 0.000 0.000 0.116 0.004 0.000
#> GSM228587 4 0.4087 0.5551 0.276 0.000 0.000 0.692 0.028 0.004
#> GSM228588 4 0.2066 0.8069 0.024 0.000 0.000 0.904 0.072 0.000
#> GSM228589 3 0.5045 0.0645 0.000 0.000 0.508 0.036 0.020 0.436
#> GSM228590 1 0.4421 0.6790 0.732 0.000 0.000 0.156 0.104 0.008
#> GSM228591 6 0.4753 0.6780 0.000 0.080 0.156 0.020 0.012 0.732
#> GSM228597 4 0.4986 0.5178 0.000 0.000 0.024 0.676 0.084 0.216
#> GSM228601 2 0.4674 0.4974 0.000 0.608 0.000 0.000 0.060 0.332
#> GSM228604 2 0.1564 0.8085 0.000 0.936 0.000 0.000 0.024 0.040
#> GSM228608 1 0.4851 0.6880 0.708 0.000 0.012 0.008 0.172 0.100
#> GSM228609 2 0.1334 0.8122 0.000 0.948 0.000 0.000 0.020 0.032
#> GSM228613 1 0.1007 0.8132 0.956 0.000 0.000 0.044 0.000 0.000
#> GSM228616 2 0.1480 0.7982 0.000 0.940 0.000 0.020 0.000 0.040
#> GSM228628 3 0.5338 0.1042 0.000 0.000 0.524 0.056 0.024 0.396
#> GSM228634 1 0.2408 0.7922 0.892 0.000 0.004 0.004 0.024 0.076
#> GSM228642 2 0.1334 0.8122 0.000 0.948 0.000 0.000 0.020 0.032
#> GSM228645 2 0.2365 0.7677 0.000 0.888 0.000 0.072 0.000 0.040
#> GSM228646 2 0.1418 0.8110 0.000 0.944 0.000 0.000 0.024 0.032
#> GSM228652 1 0.4871 0.7016 0.724 0.000 0.032 0.004 0.136 0.104
#> GSM228655 1 0.5076 0.6734 0.696 0.000 0.028 0.004 0.168 0.104
#> GSM228656 1 0.1387 0.8084 0.932 0.000 0.000 0.068 0.000 0.000
#> GSM228659 5 0.4560 0.5462 0.088 0.000 0.000 0.212 0.696 0.004
#> GSM228662 1 0.2146 0.7755 0.880 0.000 0.000 0.116 0.004 0.000
#> GSM228584 1 0.2191 0.7760 0.876 0.000 0.000 0.120 0.004 0.000
#> GSM228586 1 0.0000 0.8150 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228592 1 0.1204 0.8118 0.944 0.000 0.000 0.056 0.000 0.000
#> GSM228593 4 0.1679 0.8028 0.036 0.016 0.000 0.936 0.000 0.012
#> GSM228594 1 0.3999 -0.1117 0.500 0.000 0.000 0.496 0.004 0.000
#> GSM228598 1 0.0748 0.8132 0.976 0.000 0.004 0.000 0.016 0.004
#> GSM228607 3 0.5088 0.1065 0.000 0.000 0.528 0.036 0.024 0.412
#> GSM228612 2 0.6052 0.0575 0.000 0.492 0.084 0.044 0.004 0.376
#> GSM228619 2 0.0000 0.8184 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228622 5 0.6395 0.2275 0.052 0.000 0.360 0.016 0.488 0.084
#> GSM228625 3 0.5349 0.1058 0.000 0.000 0.516 0.056 0.024 0.404
#> GSM228631 2 0.0000 0.8184 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228633 2 0.5244 0.3192 0.000 0.496 0.000 0.008 0.072 0.424
#> GSM228637 5 0.4381 0.5825 0.028 0.000 0.004 0.204 0.732 0.032
#> GSM228639 3 0.1674 0.5482 0.000 0.000 0.924 0.004 0.068 0.004
#> GSM228649 4 0.2841 0.7642 0.000 0.000 0.012 0.864 0.092 0.032
#> GSM228660 1 0.6153 0.5779 0.616 0.000 0.136 0.004 0.104 0.140
#> GSM228661 1 0.3098 0.7763 0.852 0.000 0.016 0.004 0.028 0.100
#> GSM228595 2 0.4994 0.4541 0.000 0.572 0.000 0.008 0.060 0.360
#> GSM228599 2 0.1257 0.8126 0.000 0.952 0.000 0.000 0.020 0.028
#> GSM228602 2 0.0806 0.8162 0.000 0.972 0.000 0.000 0.008 0.020
#> GSM228614 5 0.6066 0.3291 0.000 0.000 0.176 0.040 0.568 0.216
#> GSM228626 6 0.4922 0.8223 0.000 0.000 0.284 0.008 0.076 0.632
#> GSM228640 2 0.0260 0.8185 0.000 0.992 0.000 0.000 0.000 0.008
#> GSM228643 3 0.5186 0.2515 0.000 0.000 0.584 0.016 0.332 0.068
#> GSM228650 3 0.2527 0.4874 0.000 0.000 0.876 0.000 0.040 0.084
#> GSM228653 3 0.4406 0.5210 0.040 0.000 0.772 0.004 0.088 0.096
#> GSM228657 6 0.4998 0.8465 0.000 0.000 0.252 0.012 0.088 0.648
#> GSM228605 5 0.4514 0.6324 0.092 0.000 0.184 0.000 0.716 0.008
#> GSM228610 3 0.4378 0.4953 0.008 0.000 0.736 0.004 0.180 0.072
#> GSM228617 2 0.0260 0.8185 0.000 0.992 0.000 0.000 0.000 0.008
#> GSM228620 3 0.5849 0.4177 0.088 0.000 0.644 0.004 0.160 0.104
#> GSM228623 3 0.5234 0.1149 0.000 0.000 0.532 0.048 0.024 0.396
#> GSM228629 2 0.3767 0.6506 0.000 0.780 0.020 0.028 0.000 0.172
#> GSM228632 3 0.4936 0.4792 0.032 0.000 0.716 0.004 0.148 0.100
#> GSM228635 5 0.6053 0.3626 0.000 0.000 0.112 0.080 0.596 0.212
#> GSM228647 3 0.3853 0.3521 0.000 0.000 0.680 0.000 0.304 0.016
#> GSM228596 5 0.7184 0.2414 0.284 0.000 0.168 0.008 0.440 0.100
#> GSM228600 2 0.0146 0.8184 0.000 0.996 0.000 0.000 0.000 0.004
#> GSM228603 2 0.0260 0.8185 0.000 0.992 0.000 0.000 0.000 0.008
#> GSM228615 5 0.4140 0.6682 0.020 0.000 0.080 0.064 0.804 0.032
#> GSM228627 3 0.4573 0.5125 0.032 0.000 0.752 0.004 0.124 0.088
#> GSM228641 2 0.0260 0.8185 0.000 0.992 0.000 0.000 0.000 0.008
#> GSM228644 6 0.5183 0.8251 0.000 0.000 0.240 0.012 0.112 0.636
#> GSM228651 3 0.2282 0.4960 0.000 0.000 0.888 0.000 0.024 0.088
#> GSM228654 3 0.1633 0.5448 0.000 0.000 0.932 0.000 0.044 0.024
#> GSM228658 3 0.4406 0.5210 0.040 0.000 0.772 0.004 0.088 0.096
#> GSM228606 3 0.5951 -0.0138 0.000 0.004 0.456 0.056 0.056 0.428
#> GSM228611 3 0.1908 0.5460 0.000 0.000 0.916 0.000 0.056 0.028
#> GSM228618 2 0.1411 0.7930 0.000 0.936 0.004 0.000 0.000 0.060
#> GSM228621 3 0.4964 0.1238 0.000 0.000 0.540 0.044 0.012 0.404
#> GSM228624 3 0.5289 0.1072 0.000 0.000 0.512 0.056 0.020 0.412
#> GSM228630 3 0.3523 0.3817 0.000 0.000 0.780 0.000 0.040 0.180
#> GSM228636 5 0.5888 0.2979 0.000 0.000 0.068 0.080 0.580 0.272
#> GSM228638 3 0.4452 0.5197 0.040 0.000 0.768 0.004 0.088 0.100
#> GSM228648 3 0.1196 0.5266 0.000 0.000 0.952 0.000 0.008 0.040
#> GSM228670 3 0.2854 0.5121 0.000 0.000 0.792 0.000 0.208 0.000
#> GSM228671 3 0.1418 0.5313 0.000 0.000 0.944 0.000 0.024 0.032
#> GSM228672 5 0.4437 0.5739 0.028 0.000 0.004 0.212 0.724 0.032
#> GSM228674 5 0.3441 0.6585 0.024 0.000 0.188 0.004 0.784 0.000
#> GSM228675 3 0.2912 0.5021 0.000 0.000 0.784 0.000 0.216 0.000
#> GSM228676 5 0.3695 0.5871 0.016 0.000 0.272 0.000 0.712 0.000
#> GSM228667 5 0.3348 0.6435 0.016 0.000 0.216 0.000 0.768 0.000
#> GSM228668 5 0.3364 0.6550 0.024 0.000 0.196 0.000 0.780 0.000
#> GSM228669 5 0.3497 0.6686 0.036 0.000 0.048 0.084 0.832 0.000
#> GSM228673 5 0.3315 0.6530 0.020 0.000 0.200 0.000 0.780 0.000
#> GSM228677 5 0.6260 0.3214 0.000 0.000 0.172 0.056 0.556 0.216
#> GSM228678 5 0.5851 0.4106 0.000 0.000 0.016 0.268 0.548 0.168
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
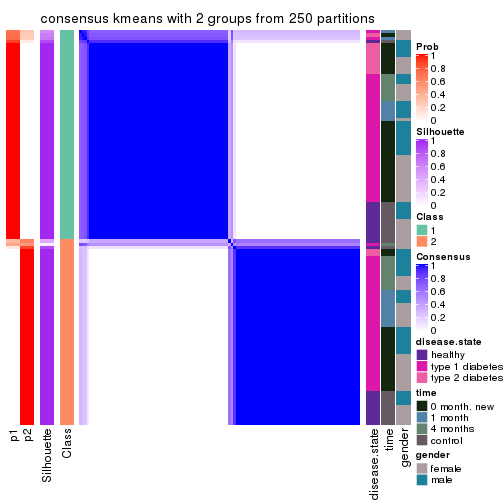
consensus_heatmap(res, k = 3)
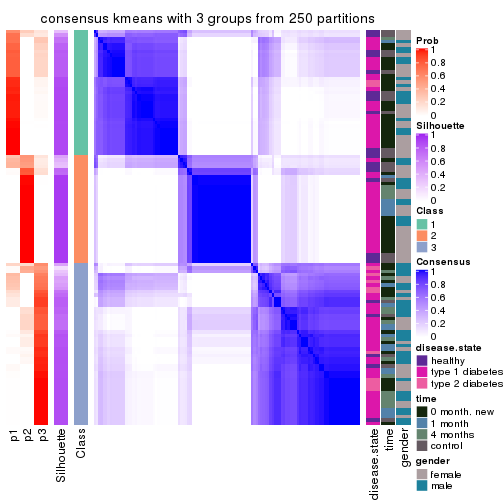
consensus_heatmap(res, k = 4)
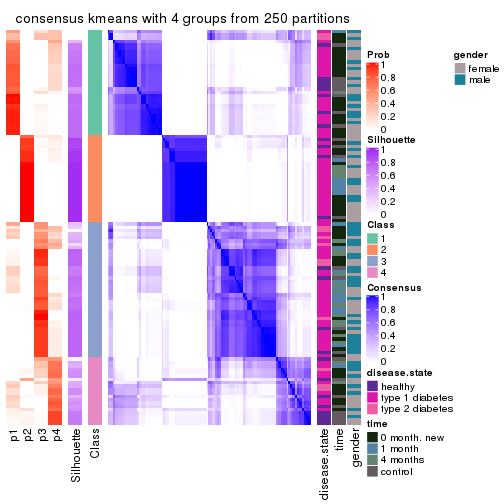
consensus_heatmap(res, k = 5)
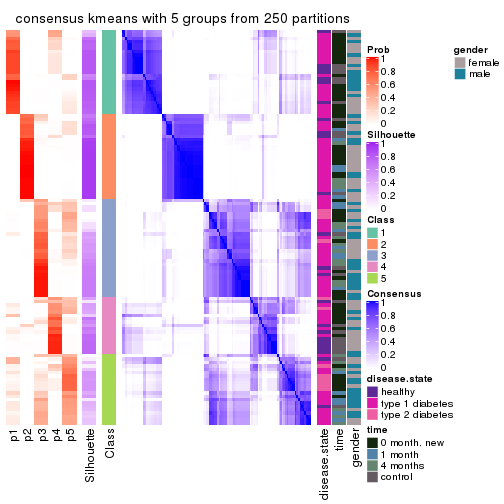
consensus_heatmap(res, k = 6)
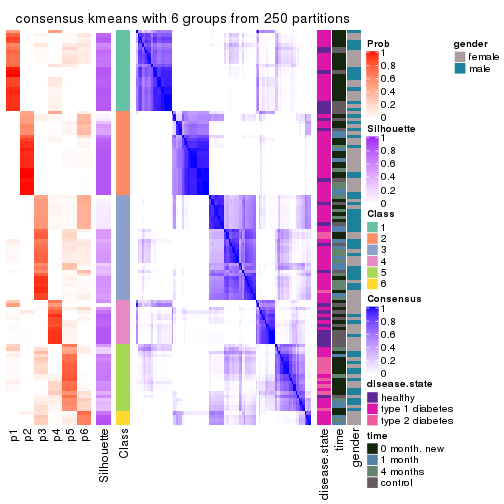
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
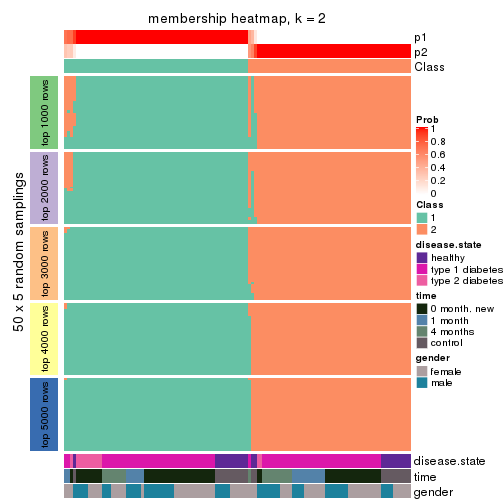
membership_heatmap(res, k = 3)
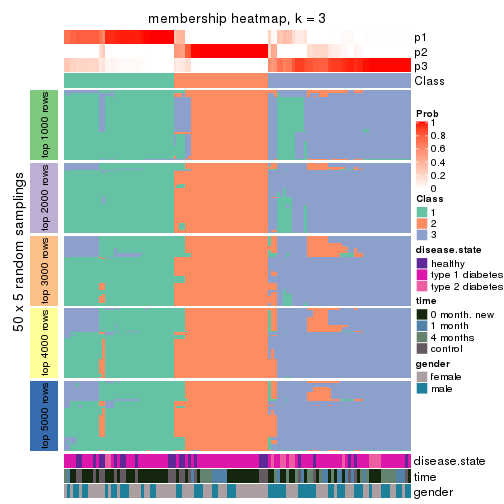
membership_heatmap(res, k = 4)
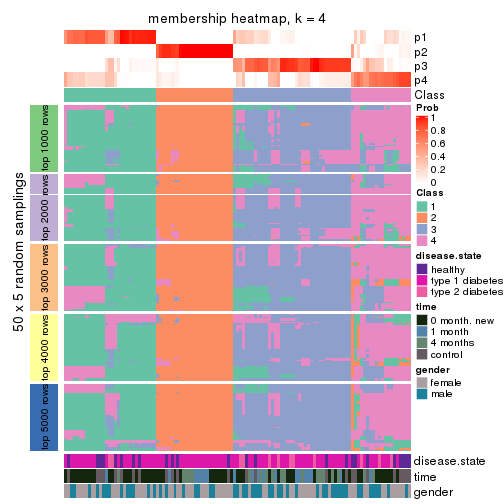
membership_heatmap(res, k = 5)
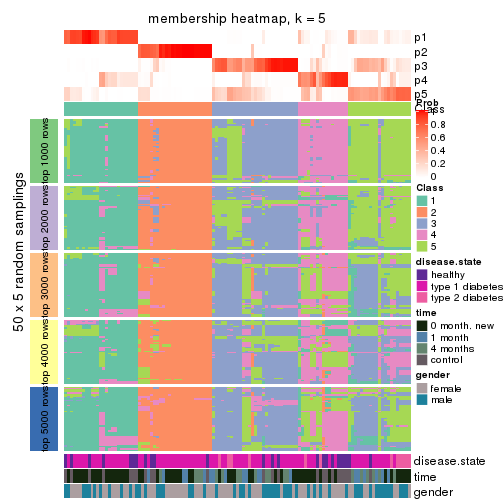
membership_heatmap(res, k = 6)
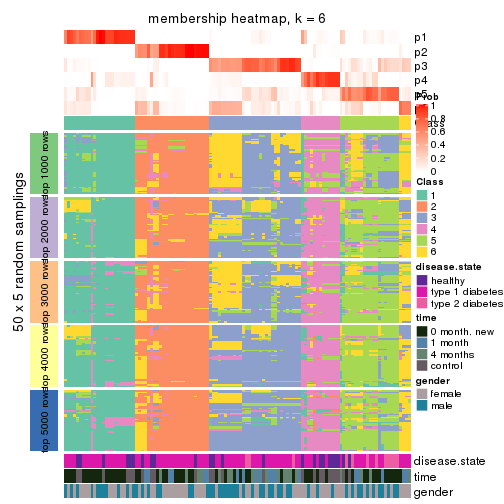
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
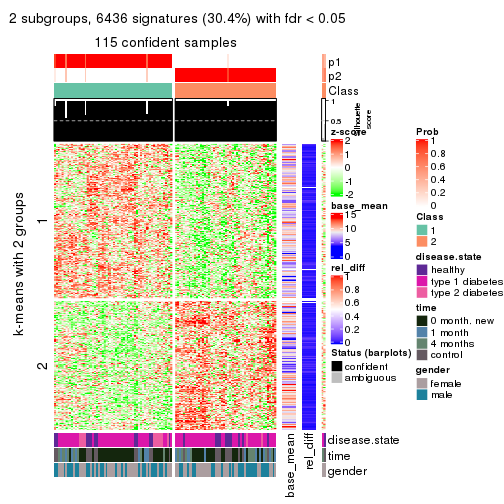
get_signatures(res, k = 3)
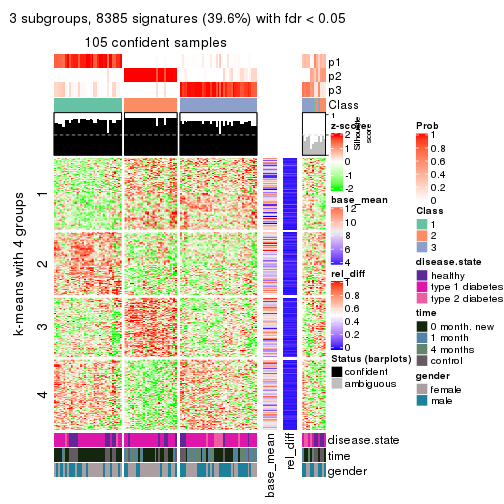
get_signatures(res, k = 4)
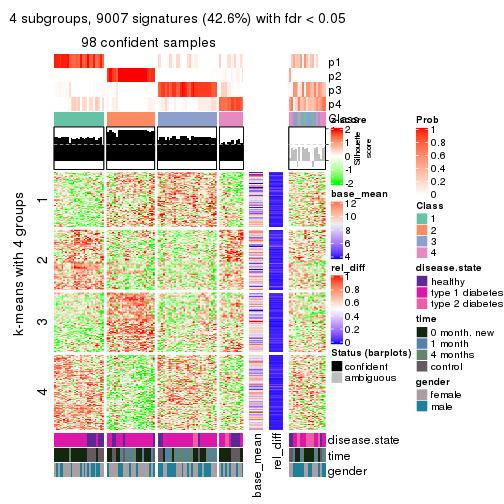
get_signatures(res, k = 5)
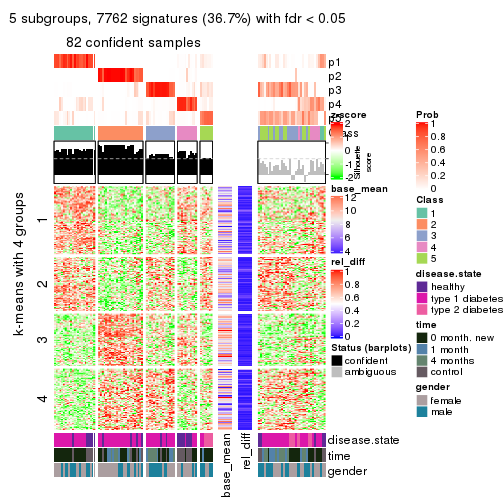
get_signatures(res, k = 6)
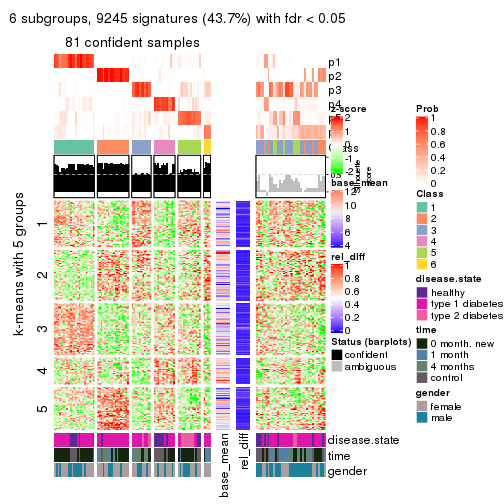
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
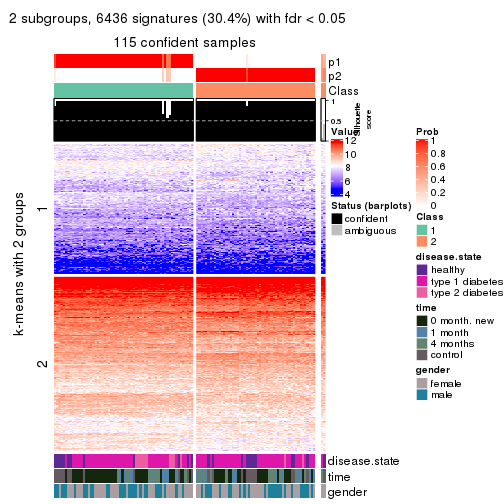
get_signatures(res, k = 3, scale_rows = FALSE)
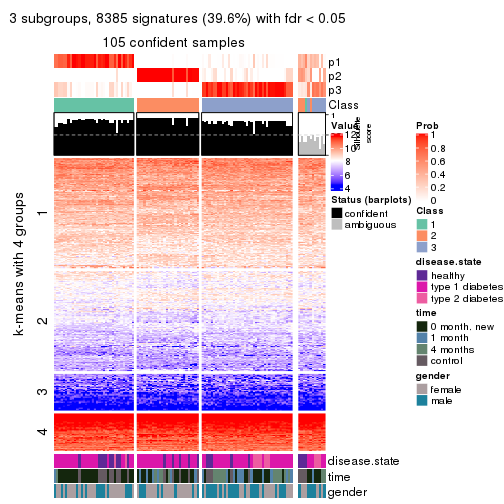
get_signatures(res, k = 4, scale_rows = FALSE)
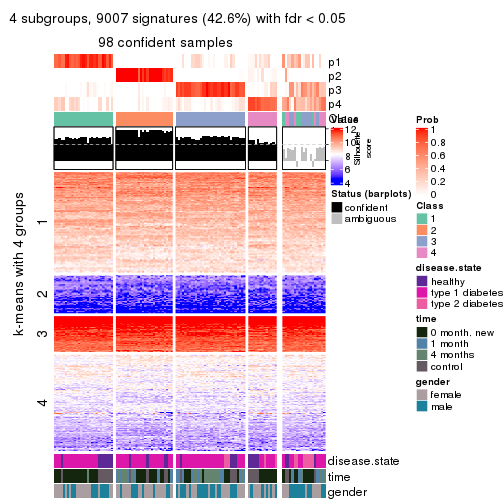
get_signatures(res, k = 5, scale_rows = FALSE)
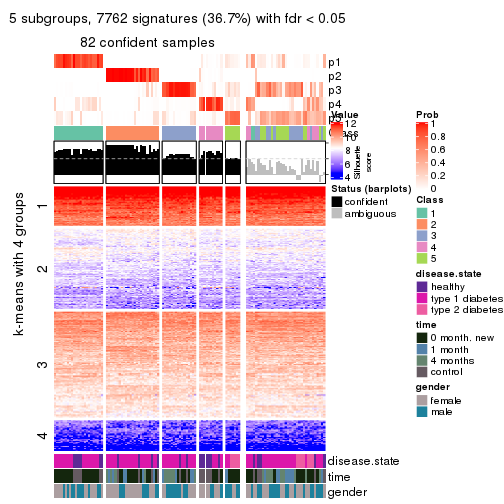
get_signatures(res, k = 6, scale_rows = FALSE)
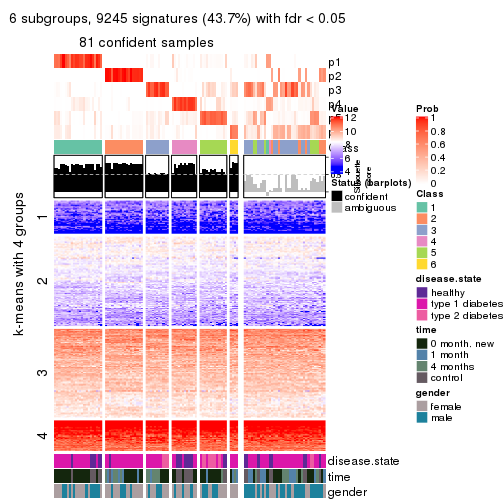
Compare the overlap of signatures from different k:
compare_signatures(res)
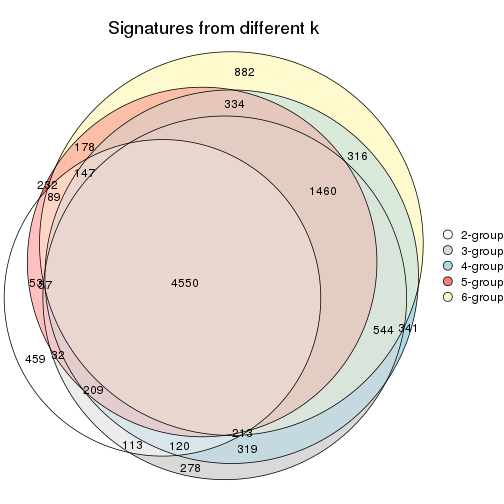
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
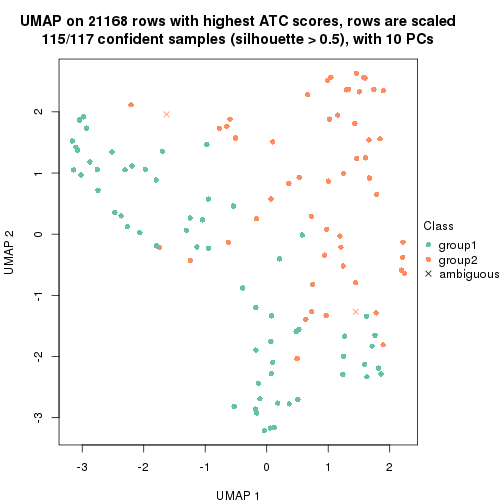
dimension_reduction(res, k = 3, method = "UMAP")
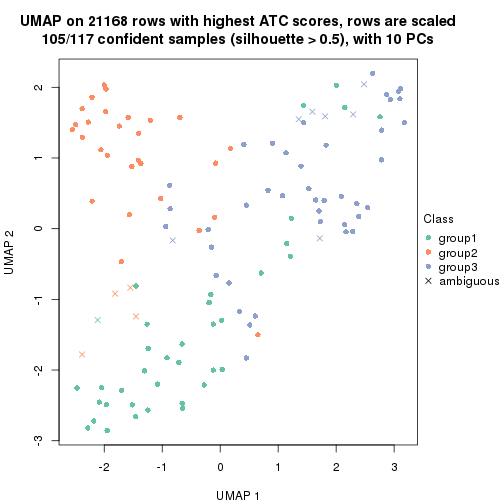
dimension_reduction(res, k = 4, method = "UMAP")
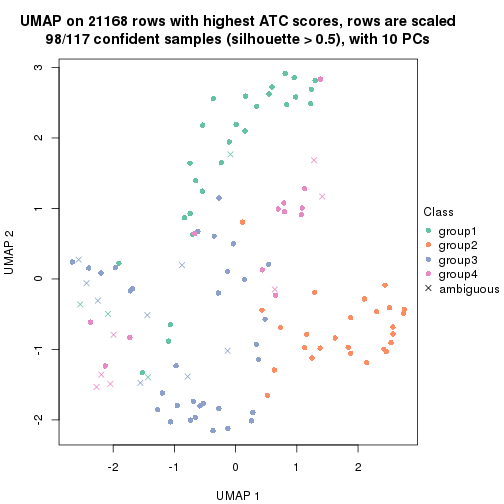
dimension_reduction(res, k = 5, method = "UMAP")
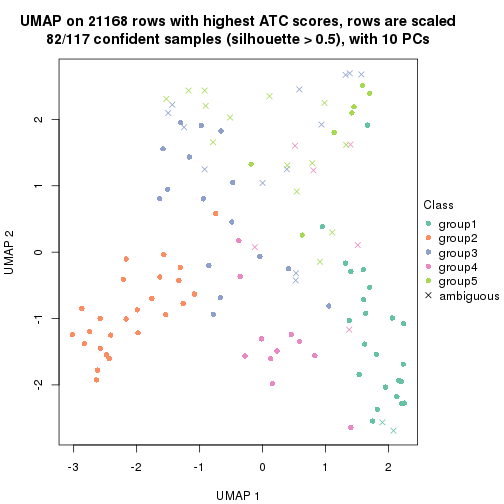
dimension_reduction(res, k = 6, method = "UMAP")
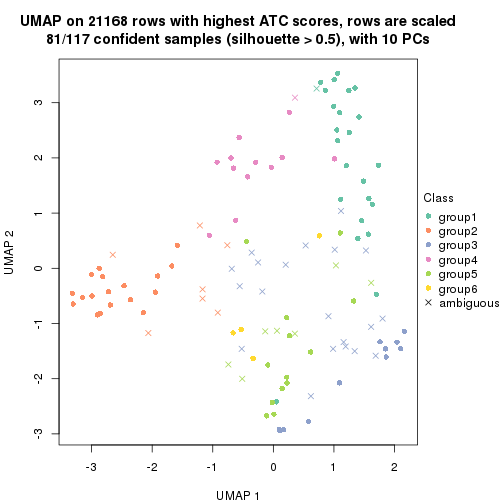
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
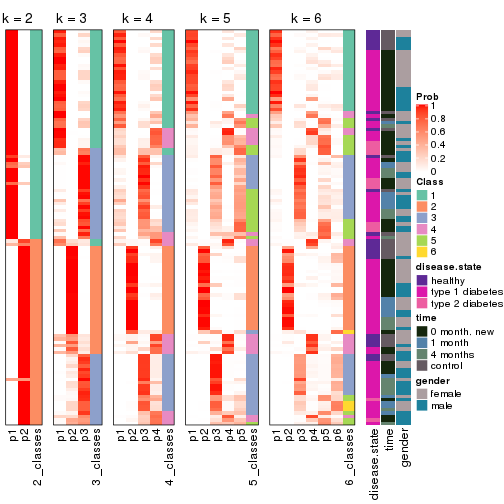
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> ATC:kmeans 115 9.53e-02 3.72e-01 1.0000 2
#> ATC:kmeans 105 5.50e-02 9.13e-04 0.0639 3
#> ATC:kmeans 98 4.85e-02 1.92e-04 0.3827 4
#> ATC:kmeans 82 6.84e-12 8.42e-05 0.2140 5
#> ATC:kmeans 81 5.99e-07 8.70e-06 0.4719 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["ATC", "skmeans"]
# you can also extract it by
# res = res_list["ATC:skmeans"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'ATC' method.
#> Subgroups are detected by 'skmeans' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
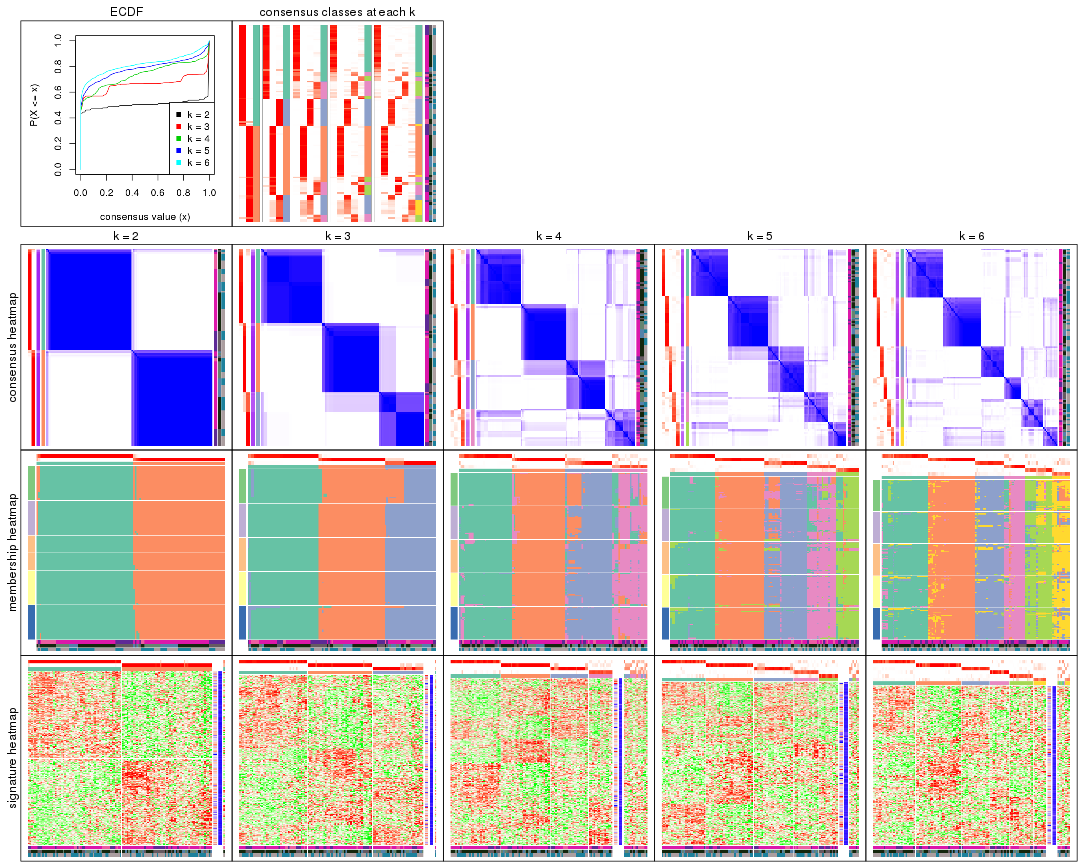
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
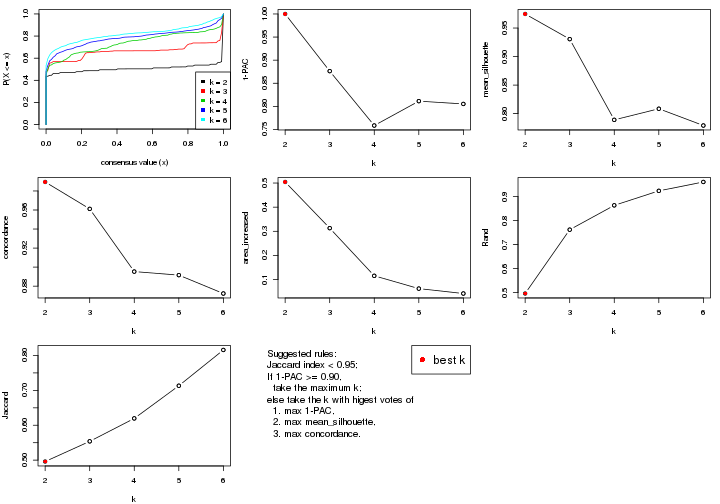
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 1.000 0.975 0.989 0.5043 0.496 0.496
#> 3 3 0.876 0.931 0.961 0.3130 0.762 0.554
#> 4 4 0.759 0.789 0.895 0.1155 0.863 0.620
#> 5 5 0.811 0.808 0.892 0.0625 0.924 0.713
#> 6 6 0.805 0.779 0.872 0.0421 0.961 0.816
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 2 0.0938 0.978 0.012 0.988
#> GSM228563 2 0.0000 0.988 0.000 1.000
#> GSM228565 1 0.0000 0.990 1.000 0.000
#> GSM228566 2 0.0000 0.988 0.000 1.000
#> GSM228567 1 0.0000 0.990 1.000 0.000
#> GSM228570 2 0.0000 0.988 0.000 1.000
#> GSM228571 2 0.7453 0.731 0.212 0.788
#> GSM228574 2 0.0000 0.988 0.000 1.000
#> GSM228575 2 0.0000 0.988 0.000 1.000
#> GSM228576 2 0.0000 0.988 0.000 1.000
#> GSM228579 1 0.0000 0.990 1.000 0.000
#> GSM228580 1 0.0000 0.990 1.000 0.000
#> GSM228581 1 0.0000 0.990 1.000 0.000
#> GSM228666 1 0.0000 0.990 1.000 0.000
#> GSM228564 2 0.0000 0.988 0.000 1.000
#> GSM228568 1 0.0000 0.990 1.000 0.000
#> GSM228569 1 0.0000 0.990 1.000 0.000
#> GSM228572 2 0.0000 0.988 0.000 1.000
#> GSM228573 2 0.0000 0.988 0.000 1.000
#> GSM228577 1 0.0000 0.990 1.000 0.000
#> GSM228578 1 0.0000 0.990 1.000 0.000
#> GSM228663 1 0.6438 0.798 0.836 0.164
#> GSM228664 2 0.0000 0.988 0.000 1.000
#> GSM228665 1 0.0000 0.990 1.000 0.000
#> GSM228582 1 0.0000 0.990 1.000 0.000
#> GSM228583 1 0.0000 0.990 1.000 0.000
#> GSM228585 1 0.0000 0.990 1.000 0.000
#> GSM228587 1 0.0000 0.990 1.000 0.000
#> GSM228588 1 0.2423 0.950 0.960 0.040
#> GSM228589 2 0.0000 0.988 0.000 1.000
#> GSM228590 1 0.0000 0.990 1.000 0.000
#> GSM228591 2 0.0000 0.988 0.000 1.000
#> GSM228597 2 0.0000 0.988 0.000 1.000
#> GSM228601 2 0.0000 0.988 0.000 1.000
#> GSM228604 2 0.0000 0.988 0.000 1.000
#> GSM228608 1 0.0000 0.990 1.000 0.000
#> GSM228609 2 0.0000 0.988 0.000 1.000
#> GSM228613 1 0.0000 0.990 1.000 0.000
#> GSM228616 2 0.0000 0.988 0.000 1.000
#> GSM228628 2 0.0000 0.988 0.000 1.000
#> GSM228634 1 0.0000 0.990 1.000 0.000
#> GSM228642 2 0.0000 0.988 0.000 1.000
#> GSM228645 2 0.0000 0.988 0.000 1.000
#> GSM228646 2 0.0000 0.988 0.000 1.000
#> GSM228652 1 0.0000 0.990 1.000 0.000
#> GSM228655 1 0.0000 0.990 1.000 0.000
#> GSM228656 1 0.0000 0.990 1.000 0.000
#> GSM228659 1 0.0000 0.990 1.000 0.000
#> GSM228662 1 0.0000 0.990 1.000 0.000
#> GSM228584 1 0.0000 0.990 1.000 0.000
#> GSM228586 1 0.0000 0.990 1.000 0.000
#> GSM228592 1 0.0000 0.990 1.000 0.000
#> GSM228593 2 0.0000 0.988 0.000 1.000
#> GSM228594 1 0.0000 0.990 1.000 0.000
#> GSM228598 1 0.0000 0.990 1.000 0.000
#> GSM228607 2 0.0000 0.988 0.000 1.000
#> GSM228612 2 0.0000 0.988 0.000 1.000
#> GSM228619 2 0.0000 0.988 0.000 1.000
#> GSM228622 1 0.0000 0.990 1.000 0.000
#> GSM228625 2 0.0000 0.988 0.000 1.000
#> GSM228631 2 0.0000 0.988 0.000 1.000
#> GSM228633 2 0.0000 0.988 0.000 1.000
#> GSM228637 1 0.0000 0.990 1.000 0.000
#> GSM228639 1 0.0000 0.990 1.000 0.000
#> GSM228649 2 0.0000 0.988 0.000 1.000
#> GSM228660 1 0.0000 0.990 1.000 0.000
#> GSM228661 1 0.0000 0.990 1.000 0.000
#> GSM228595 2 0.0000 0.988 0.000 1.000
#> GSM228599 2 0.0000 0.988 0.000 1.000
#> GSM228602 2 0.0000 0.988 0.000 1.000
#> GSM228614 2 0.0000 0.988 0.000 1.000
#> GSM228626 2 0.0000 0.988 0.000 1.000
#> GSM228640 2 0.0000 0.988 0.000 1.000
#> GSM228643 2 0.8763 0.579 0.296 0.704
#> GSM228650 2 0.3733 0.918 0.072 0.928
#> GSM228653 1 0.0000 0.990 1.000 0.000
#> GSM228657 2 0.0000 0.988 0.000 1.000
#> GSM228605 1 0.0000 0.990 1.000 0.000
#> GSM228610 1 0.0000 0.990 1.000 0.000
#> GSM228617 2 0.0000 0.988 0.000 1.000
#> GSM228620 1 0.0000 0.990 1.000 0.000
#> GSM228623 2 0.0000 0.988 0.000 1.000
#> GSM228629 2 0.0000 0.988 0.000 1.000
#> GSM228632 1 0.0000 0.990 1.000 0.000
#> GSM228635 2 0.0000 0.988 0.000 1.000
#> GSM228647 1 0.0000 0.990 1.000 0.000
#> GSM228596 1 0.0000 0.990 1.000 0.000
#> GSM228600 2 0.0000 0.988 0.000 1.000
#> GSM228603 2 0.0000 0.988 0.000 1.000
#> GSM228615 1 0.0000 0.990 1.000 0.000
#> GSM228627 1 0.0000 0.990 1.000 0.000
#> GSM228641 2 0.0000 0.988 0.000 1.000
#> GSM228644 2 0.0000 0.988 0.000 1.000
#> GSM228651 2 0.2423 0.951 0.040 0.960
#> GSM228654 1 0.0000 0.990 1.000 0.000
#> GSM228658 1 0.0000 0.990 1.000 0.000
#> GSM228606 2 0.0000 0.988 0.000 1.000
#> GSM228611 1 0.0000 0.990 1.000 0.000
#> GSM228618 2 0.0000 0.988 0.000 1.000
#> GSM228621 2 0.0000 0.988 0.000 1.000
#> GSM228624 2 0.0000 0.988 0.000 1.000
#> GSM228630 2 0.0000 0.988 0.000 1.000
#> GSM228636 2 0.0000 0.988 0.000 1.000
#> GSM228638 1 0.0000 0.990 1.000 0.000
#> GSM228648 1 0.0000 0.990 1.000 0.000
#> GSM228670 1 0.0000 0.990 1.000 0.000
#> GSM228671 1 0.9710 0.328 0.600 0.400
#> GSM228672 1 0.0000 0.990 1.000 0.000
#> GSM228674 1 0.0000 0.990 1.000 0.000
#> GSM228675 1 0.0000 0.990 1.000 0.000
#> GSM228676 1 0.0000 0.990 1.000 0.000
#> GSM228667 1 0.0000 0.990 1.000 0.000
#> GSM228668 1 0.0000 0.990 1.000 0.000
#> GSM228669 1 0.0000 0.990 1.000 0.000
#> GSM228673 1 0.0000 0.990 1.000 0.000
#> GSM228677 2 0.0000 0.988 0.000 1.000
#> GSM228678 2 0.0000 0.988 0.000 1.000
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 2 0.4702 0.724 0.212 0.788 0.000
#> GSM228563 2 0.0747 0.969 0.016 0.984 0.000
#> GSM228565 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228566 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228567 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228570 2 0.1411 0.949 0.036 0.964 0.000
#> GSM228571 2 0.4750 0.718 0.216 0.784 0.000
#> GSM228574 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228575 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228576 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228579 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228580 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228581 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228666 1 0.5363 0.633 0.724 0.000 0.276
#> GSM228564 2 0.1411 0.949 0.036 0.964 0.000
#> GSM228568 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228569 1 0.0237 0.969 0.996 0.000 0.004
#> GSM228572 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228573 2 0.1529 0.940 0.000 0.960 0.040
#> GSM228577 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228578 1 0.0237 0.969 0.996 0.000 0.004
#> GSM228663 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228664 3 0.4291 0.838 0.000 0.180 0.820
#> GSM228665 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228582 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228583 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228585 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228587 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228588 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228589 3 0.4750 0.818 0.000 0.216 0.784
#> GSM228590 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228591 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228597 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228601 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228604 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228608 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228609 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228613 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228616 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228628 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228634 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228642 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228645 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228646 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228652 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228655 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228656 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228659 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228662 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228584 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228586 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228592 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228593 2 0.0747 0.969 0.016 0.984 0.000
#> GSM228594 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228598 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228607 3 0.4750 0.818 0.000 0.216 0.784
#> GSM228612 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228619 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228622 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228625 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228631 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228633 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228637 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228639 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228649 2 0.0747 0.969 0.016 0.984 0.000
#> GSM228660 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228661 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228595 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228599 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228602 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228614 3 0.4702 0.821 0.000 0.212 0.788
#> GSM228626 3 0.4702 0.821 0.000 0.212 0.788
#> GSM228640 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228643 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228650 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228653 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228657 3 0.4702 0.821 0.000 0.212 0.788
#> GSM228605 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228610 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228617 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228620 1 0.6026 0.410 0.624 0.000 0.376
#> GSM228623 3 0.4750 0.818 0.000 0.216 0.784
#> GSM228629 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228632 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228635 3 0.4887 0.804 0.000 0.228 0.772
#> GSM228647 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228596 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228600 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228603 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228615 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228627 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228641 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228644 3 0.4702 0.821 0.000 0.212 0.788
#> GSM228651 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228654 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228658 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228606 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228611 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228618 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228621 3 0.4750 0.818 0.000 0.216 0.784
#> GSM228624 3 0.4750 0.818 0.000 0.216 0.784
#> GSM228630 3 0.0237 0.909 0.000 0.004 0.996
#> GSM228636 2 0.0000 0.982 0.000 1.000 0.000
#> GSM228638 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228648 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228670 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228671 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228672 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228674 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228675 3 0.0000 0.911 0.000 0.000 1.000
#> GSM228676 1 0.4452 0.793 0.808 0.000 0.192
#> GSM228667 1 0.4452 0.793 0.808 0.000 0.192
#> GSM228668 1 0.0747 0.967 0.984 0.000 0.016
#> GSM228669 1 0.0000 0.970 1.000 0.000 0.000
#> GSM228673 1 0.1163 0.959 0.972 0.000 0.028
#> GSM228677 3 0.5591 0.693 0.000 0.304 0.696
#> GSM228678 2 0.0000 0.982 0.000 1.000 0.000
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.2861 0.7244 0.016 0.096 0.000 0.888
#> GSM228563 4 0.3324 0.7070 0.012 0.136 0.000 0.852
#> GSM228565 4 0.4992 0.1154 0.476 0.000 0.000 0.524
#> GSM228566 2 0.1211 0.9243 0.000 0.960 0.000 0.040
#> GSM228567 1 0.0336 0.9385 0.992 0.000 0.000 0.008
#> GSM228570 4 0.3271 0.7095 0.012 0.132 0.000 0.856
#> GSM228571 2 0.6917 0.2821 0.288 0.568 0.000 0.144
#> GSM228574 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228575 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228576 2 0.2868 0.8184 0.000 0.864 0.000 0.136
#> GSM228579 1 0.2149 0.8746 0.912 0.000 0.000 0.088
#> GSM228580 1 0.4304 0.5409 0.716 0.000 0.000 0.284
#> GSM228581 1 0.0524 0.9370 0.988 0.000 0.008 0.004
#> GSM228666 4 0.2888 0.7313 0.124 0.000 0.004 0.872
#> GSM228564 4 0.2741 0.7244 0.012 0.096 0.000 0.892
#> GSM228568 1 0.0592 0.9347 0.984 0.000 0.000 0.016
#> GSM228569 1 0.0000 0.9399 1.000 0.000 0.000 0.000
#> GSM228572 2 0.0188 0.9584 0.000 0.996 0.000 0.004
#> GSM228573 2 0.0376 0.9547 0.000 0.992 0.004 0.004
#> GSM228577 1 0.0336 0.9385 0.992 0.000 0.000 0.008
#> GSM228578 1 0.0000 0.9399 1.000 0.000 0.000 0.000
#> GSM228663 3 0.1585 0.7903 0.004 0.040 0.952 0.004
#> GSM228664 3 0.4584 0.6395 0.000 0.300 0.696 0.004
#> GSM228665 3 0.0336 0.8051 0.008 0.000 0.992 0.000
#> GSM228582 1 0.0469 0.9367 0.988 0.000 0.012 0.000
#> GSM228583 1 0.0188 0.9395 0.996 0.000 0.000 0.004
#> GSM228585 1 0.1302 0.9158 0.956 0.000 0.000 0.044
#> GSM228587 1 0.2149 0.8746 0.912 0.000 0.000 0.088
#> GSM228588 4 0.2530 0.7103 0.112 0.000 0.000 0.888
#> GSM228589 3 0.4964 0.5313 0.000 0.380 0.616 0.004
#> GSM228590 1 0.0188 0.9394 0.996 0.000 0.000 0.004
#> GSM228591 2 0.0188 0.9584 0.000 0.996 0.000 0.004
#> GSM228597 4 0.4981 0.1637 0.000 0.464 0.000 0.536
#> GSM228601 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228604 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228608 1 0.0000 0.9399 1.000 0.000 0.000 0.000
#> GSM228609 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228613 1 0.0000 0.9399 1.000 0.000 0.000 0.000
#> GSM228616 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228628 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228634 1 0.0336 0.9387 0.992 0.000 0.008 0.000
#> GSM228642 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228645 2 0.0707 0.9436 0.000 0.980 0.000 0.020
#> GSM228646 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228652 1 0.0336 0.9387 0.992 0.000 0.008 0.000
#> GSM228655 1 0.0336 0.9387 0.992 0.000 0.008 0.000
#> GSM228656 1 0.0336 0.9385 0.992 0.000 0.000 0.008
#> GSM228659 1 0.2216 0.8563 0.908 0.000 0.000 0.092
#> GSM228662 1 0.1302 0.9158 0.956 0.000 0.000 0.044
#> GSM228584 1 0.0817 0.9304 0.976 0.000 0.000 0.024
#> GSM228586 1 0.0000 0.9399 1.000 0.000 0.000 0.000
#> GSM228592 1 0.0000 0.9399 1.000 0.000 0.000 0.000
#> GSM228593 2 0.5517 0.2576 0.020 0.568 0.000 0.412
#> GSM228594 1 0.2149 0.8746 0.912 0.000 0.000 0.088
#> GSM228598 1 0.0000 0.9399 1.000 0.000 0.000 0.000
#> GSM228607 3 0.4632 0.6310 0.000 0.308 0.688 0.004
#> GSM228612 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228619 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228622 1 0.0469 0.9371 0.988 0.000 0.012 0.000
#> GSM228625 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228631 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228633 2 0.0188 0.9584 0.000 0.996 0.000 0.004
#> GSM228637 4 0.2921 0.7245 0.140 0.000 0.000 0.860
#> GSM228639 3 0.0000 0.8093 0.000 0.000 1.000 0.000
#> GSM228649 4 0.3105 0.7156 0.012 0.120 0.000 0.868
#> GSM228660 1 0.0469 0.9367 0.988 0.000 0.012 0.000
#> GSM228661 1 0.0336 0.9387 0.992 0.000 0.008 0.000
#> GSM228595 2 0.0188 0.9584 0.000 0.996 0.000 0.004
#> GSM228599 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228602 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228614 4 0.4679 0.6616 0.000 0.184 0.044 0.772
#> GSM228626 3 0.4819 0.5884 0.000 0.344 0.652 0.004
#> GSM228640 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228643 4 0.4955 0.2495 0.000 0.000 0.444 0.556
#> GSM228650 3 0.0188 0.8077 0.000 0.000 0.996 0.004
#> GSM228653 3 0.0000 0.8093 0.000 0.000 1.000 0.000
#> GSM228657 3 0.5075 0.5850 0.000 0.344 0.644 0.012
#> GSM228605 1 0.0376 0.9386 0.992 0.000 0.004 0.004
#> GSM228610 3 0.0000 0.8093 0.000 0.000 1.000 0.000
#> GSM228617 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228620 1 0.4713 0.4310 0.640 0.000 0.360 0.000
#> GSM228623 3 0.5050 0.4827 0.000 0.408 0.588 0.004
#> GSM228629 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228632 3 0.0000 0.8093 0.000 0.000 1.000 0.000
#> GSM228635 4 0.3280 0.7183 0.000 0.124 0.016 0.860
#> GSM228647 3 0.2469 0.7145 0.000 0.000 0.892 0.108
#> GSM228596 1 0.0927 0.9298 0.976 0.000 0.016 0.008
#> GSM228600 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228603 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228615 4 0.4378 0.7059 0.164 0.000 0.040 0.796
#> GSM228627 3 0.0188 0.8072 0.004 0.000 0.996 0.000
#> GSM228641 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228644 3 0.7495 0.3739 0.000 0.368 0.448 0.184
#> GSM228651 3 0.0000 0.8093 0.000 0.000 1.000 0.000
#> GSM228654 3 0.0000 0.8093 0.000 0.000 1.000 0.000
#> GSM228658 3 0.0000 0.8093 0.000 0.000 1.000 0.000
#> GSM228606 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228611 3 0.0000 0.8093 0.000 0.000 1.000 0.000
#> GSM228618 2 0.0000 0.9612 0.000 1.000 0.000 0.000
#> GSM228621 3 0.5050 0.4820 0.000 0.408 0.588 0.004
#> GSM228624 3 0.5028 0.4972 0.000 0.400 0.596 0.004
#> GSM228630 3 0.0188 0.8084 0.000 0.000 0.996 0.004
#> GSM228636 4 0.3052 0.7176 0.000 0.136 0.004 0.860
#> GSM228638 3 0.0000 0.8093 0.000 0.000 1.000 0.000
#> GSM228648 3 0.0000 0.8093 0.000 0.000 1.000 0.000
#> GSM228670 3 0.0188 0.8077 0.000 0.000 0.996 0.004
#> GSM228671 3 0.0000 0.8093 0.000 0.000 1.000 0.000
#> GSM228672 4 0.2921 0.7245 0.140 0.000 0.000 0.860
#> GSM228674 4 0.7219 0.3766 0.364 0.000 0.148 0.488
#> GSM228675 3 0.0336 0.8054 0.000 0.000 0.992 0.008
#> GSM228676 3 0.7315 0.0952 0.300 0.000 0.516 0.184
#> GSM228667 4 0.7023 0.4760 0.144 0.000 0.312 0.544
#> GSM228668 1 0.4795 0.5065 0.696 0.000 0.012 0.292
#> GSM228669 4 0.4961 0.2675 0.448 0.000 0.000 0.552
#> GSM228673 4 0.7253 0.3730 0.364 0.000 0.152 0.484
#> GSM228677 4 0.3638 0.7121 0.000 0.120 0.032 0.848
#> GSM228678 4 0.2149 0.7323 0.000 0.088 0.000 0.912
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 5 0.0865 0.7648 0.000 0.004 0.000 0.024 0.972
#> GSM228563 5 0.0898 0.7665 0.000 0.008 0.000 0.020 0.972
#> GSM228565 5 0.3906 0.6510 0.240 0.000 0.000 0.016 0.744
#> GSM228566 2 0.2020 0.8836 0.000 0.900 0.000 0.000 0.100
#> GSM228567 1 0.1197 0.9274 0.952 0.000 0.000 0.000 0.048
#> GSM228570 5 0.0898 0.7665 0.000 0.008 0.000 0.020 0.972
#> GSM228571 5 0.2962 0.7443 0.084 0.048 0.000 0.000 0.868
#> GSM228574 2 0.0000 0.9742 0.000 1.000 0.000 0.000 0.000
#> GSM228575 2 0.0510 0.9674 0.000 0.984 0.000 0.016 0.000
#> GSM228576 5 0.3210 0.6330 0.000 0.212 0.000 0.000 0.788
#> GSM228579 5 0.3816 0.5710 0.304 0.000 0.000 0.000 0.696
#> GSM228580 4 0.4397 0.3429 0.432 0.000 0.004 0.564 0.000
#> GSM228581 1 0.0671 0.9281 0.980 0.000 0.004 0.016 0.000
#> GSM228666 4 0.2669 0.7714 0.020 0.000 0.000 0.876 0.104
#> GSM228564 5 0.0865 0.7648 0.000 0.004 0.000 0.024 0.972
#> GSM228568 1 0.1410 0.9205 0.940 0.000 0.000 0.000 0.060
#> GSM228569 1 0.0404 0.9364 0.988 0.000 0.000 0.000 0.012
#> GSM228572 2 0.0404 0.9746 0.000 0.988 0.000 0.000 0.012
#> GSM228573 2 0.1522 0.9393 0.000 0.944 0.000 0.044 0.012
#> GSM228577 1 0.1043 0.9309 0.960 0.000 0.000 0.000 0.040
#> GSM228578 1 0.0162 0.9376 0.996 0.000 0.000 0.000 0.004
#> GSM228663 3 0.3502 0.7700 0.028 0.004 0.848 0.104 0.016
#> GSM228664 3 0.5533 0.6728 0.000 0.176 0.684 0.124 0.016
#> GSM228665 3 0.1205 0.8001 0.040 0.000 0.956 0.004 0.000
#> GSM228582 1 0.0000 0.9371 1.000 0.000 0.000 0.000 0.000
#> GSM228583 1 0.1043 0.9309 0.960 0.000 0.000 0.000 0.040
#> GSM228585 1 0.3336 0.7187 0.772 0.000 0.000 0.000 0.228
#> GSM228587 5 0.4262 0.2365 0.440 0.000 0.000 0.000 0.560
#> GSM228588 5 0.0865 0.7637 0.004 0.000 0.000 0.024 0.972
#> GSM228589 3 0.6148 0.6148 0.004 0.256 0.608 0.116 0.016
#> GSM228590 1 0.1638 0.9135 0.932 0.000 0.000 0.004 0.064
#> GSM228591 2 0.2464 0.8878 0.000 0.888 0.000 0.096 0.016
#> GSM228597 5 0.5261 0.1633 0.000 0.424 0.000 0.048 0.528
#> GSM228601 2 0.0404 0.9746 0.000 0.988 0.000 0.000 0.012
#> GSM228604 2 0.0290 0.9752 0.000 0.992 0.000 0.000 0.008
#> GSM228608 1 0.0000 0.9371 1.000 0.000 0.000 0.000 0.000
#> GSM228609 2 0.0290 0.9752 0.000 0.992 0.000 0.000 0.008
#> GSM228613 1 0.0609 0.9356 0.980 0.000 0.000 0.000 0.020
#> GSM228616 2 0.0290 0.9752 0.000 0.992 0.000 0.000 0.008
#> GSM228628 2 0.0992 0.9585 0.000 0.968 0.000 0.024 0.008
#> GSM228634 1 0.0000 0.9371 1.000 0.000 0.000 0.000 0.000
#> GSM228642 2 0.0290 0.9752 0.000 0.992 0.000 0.000 0.008
#> GSM228645 2 0.1608 0.9167 0.000 0.928 0.000 0.000 0.072
#> GSM228646 2 0.0290 0.9752 0.000 0.992 0.000 0.000 0.008
#> GSM228652 1 0.0000 0.9371 1.000 0.000 0.000 0.000 0.000
#> GSM228655 1 0.0000 0.9371 1.000 0.000 0.000 0.000 0.000
#> GSM228656 1 0.1121 0.9295 0.956 0.000 0.000 0.000 0.044
#> GSM228659 1 0.1628 0.9180 0.936 0.000 0.000 0.008 0.056
#> GSM228662 1 0.3336 0.7187 0.772 0.000 0.000 0.000 0.228
#> GSM228584 1 0.2773 0.8135 0.836 0.000 0.000 0.000 0.164
#> GSM228586 1 0.0162 0.9372 0.996 0.000 0.000 0.000 0.004
#> GSM228592 1 0.1043 0.9309 0.960 0.000 0.000 0.000 0.040
#> GSM228593 5 0.1357 0.7574 0.004 0.048 0.000 0.000 0.948
#> GSM228594 5 0.3730 0.5970 0.288 0.000 0.000 0.000 0.712
#> GSM228598 1 0.0162 0.9372 0.996 0.000 0.000 0.000 0.004
#> GSM228607 3 0.5878 0.6429 0.004 0.232 0.644 0.104 0.016
#> GSM228612 2 0.0000 0.9742 0.000 1.000 0.000 0.000 0.000
#> GSM228619 2 0.0290 0.9752 0.000 0.992 0.000 0.000 0.008
#> GSM228622 1 0.0703 0.9256 0.976 0.000 0.000 0.024 0.000
#> GSM228625 2 0.0955 0.9586 0.000 0.968 0.000 0.028 0.004
#> GSM228631 2 0.0290 0.9752 0.000 0.992 0.000 0.000 0.008
#> GSM228633 2 0.2464 0.8896 0.000 0.888 0.000 0.096 0.016
#> GSM228637 4 0.3262 0.7678 0.036 0.000 0.000 0.840 0.124
#> GSM228639 3 0.0000 0.8132 0.000 0.000 1.000 0.000 0.000
#> GSM228649 5 0.0898 0.7665 0.000 0.008 0.000 0.020 0.972
#> GSM228660 1 0.0162 0.9359 0.996 0.000 0.004 0.000 0.000
#> GSM228661 1 0.0000 0.9371 1.000 0.000 0.000 0.000 0.000
#> GSM228595 2 0.1018 0.9589 0.000 0.968 0.000 0.016 0.016
#> GSM228599 2 0.0290 0.9752 0.000 0.992 0.000 0.000 0.008
#> GSM228602 2 0.0290 0.9752 0.000 0.992 0.000 0.000 0.008
#> GSM228614 4 0.2538 0.7498 0.000 0.048 0.004 0.900 0.048
#> GSM228626 3 0.5993 0.6367 0.000 0.216 0.628 0.140 0.016
#> GSM228640 2 0.0290 0.9752 0.000 0.992 0.000 0.000 0.008
#> GSM228643 4 0.4840 0.6277 0.000 0.000 0.248 0.688 0.064
#> GSM228650 3 0.0609 0.8118 0.000 0.000 0.980 0.020 0.000
#> GSM228653 3 0.0566 0.8116 0.012 0.000 0.984 0.004 0.000
#> GSM228657 3 0.6361 0.6006 0.000 0.208 0.584 0.192 0.016
#> GSM228605 1 0.1768 0.8823 0.924 0.000 0.004 0.072 0.000
#> GSM228610 3 0.1310 0.8013 0.020 0.000 0.956 0.024 0.000
#> GSM228617 2 0.0290 0.9752 0.000 0.992 0.000 0.000 0.008
#> GSM228620 1 0.3530 0.6815 0.784 0.000 0.204 0.012 0.000
#> GSM228623 3 0.6352 0.5237 0.000 0.320 0.540 0.124 0.016
#> GSM228629 2 0.0000 0.9742 0.000 1.000 0.000 0.000 0.000
#> GSM228632 3 0.1310 0.8013 0.020 0.000 0.956 0.024 0.000
#> GSM228635 4 0.2351 0.7647 0.000 0.016 0.000 0.896 0.088
#> GSM228647 3 0.4262 -0.0442 0.000 0.000 0.560 0.440 0.000
#> GSM228596 1 0.2006 0.8757 0.916 0.000 0.012 0.072 0.000
#> GSM228600 2 0.0000 0.9742 0.000 1.000 0.000 0.000 0.000
#> GSM228603 2 0.0000 0.9742 0.000 1.000 0.000 0.000 0.000
#> GSM228615 4 0.3463 0.7798 0.040 0.000 0.056 0.860 0.044
#> GSM228627 3 0.1012 0.8071 0.020 0.000 0.968 0.012 0.000
#> GSM228641 2 0.0290 0.9752 0.000 0.992 0.000 0.000 0.008
#> GSM228644 4 0.5471 0.4883 0.000 0.164 0.128 0.692 0.016
#> GSM228651 3 0.0609 0.8118 0.000 0.000 0.980 0.020 0.000
#> GSM228654 3 0.0162 0.8130 0.000 0.000 0.996 0.004 0.000
#> GSM228658 3 0.0566 0.8116 0.012 0.000 0.984 0.004 0.000
#> GSM228606 2 0.0992 0.9581 0.000 0.968 0.000 0.024 0.008
#> GSM228611 3 0.0290 0.8128 0.000 0.000 0.992 0.008 0.000
#> GSM228618 2 0.0000 0.9742 0.000 1.000 0.000 0.000 0.000
#> GSM228621 3 0.6068 0.4992 0.000 0.348 0.544 0.096 0.012
#> GSM228624 3 0.6087 0.5103 0.000 0.340 0.548 0.100 0.012
#> GSM228630 3 0.2233 0.7818 0.000 0.000 0.892 0.104 0.004
#> GSM228636 4 0.3090 0.7499 0.000 0.052 0.000 0.860 0.088
#> GSM228638 3 0.0162 0.8134 0.004 0.000 0.996 0.000 0.000
#> GSM228648 3 0.0510 0.8124 0.000 0.000 0.984 0.016 0.000
#> GSM228670 3 0.0671 0.8100 0.004 0.000 0.980 0.016 0.000
#> GSM228671 3 0.0404 0.8136 0.000 0.000 0.988 0.012 0.000
#> GSM228672 4 0.3309 0.7667 0.036 0.000 0.000 0.836 0.128
#> GSM228674 4 0.4573 0.7326 0.164 0.000 0.092 0.744 0.000
#> GSM228675 3 0.1357 0.7953 0.004 0.000 0.948 0.048 0.000
#> GSM228676 4 0.5076 0.7023 0.108 0.000 0.200 0.692 0.000
#> GSM228667 4 0.3412 0.7475 0.028 0.000 0.152 0.820 0.000
#> GSM228668 4 0.4844 0.6374 0.280 0.000 0.052 0.668 0.000
#> GSM228669 4 0.4269 0.6806 0.232 0.000 0.000 0.732 0.036
#> GSM228673 4 0.4610 0.7299 0.168 0.000 0.092 0.740 0.000
#> GSM228677 4 0.2819 0.7465 0.000 0.052 0.004 0.884 0.060
#> GSM228678 4 0.3333 0.7097 0.000 0.004 0.000 0.788 0.208
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 4 0.0146 0.8283 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM228563 4 0.0146 0.8283 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM228565 4 0.3559 0.6541 0.240 0.000 0.000 0.744 0.004 0.012
#> GSM228566 2 0.1983 0.8727 0.000 0.908 0.000 0.072 0.000 0.020
#> GSM228567 1 0.0725 0.8814 0.976 0.000 0.000 0.012 0.000 0.012
#> GSM228570 4 0.0146 0.8283 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM228571 4 0.1138 0.8163 0.024 0.004 0.000 0.960 0.000 0.012
#> GSM228574 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228575 2 0.2048 0.8557 0.000 0.880 0.000 0.000 0.000 0.120
#> GSM228576 4 0.2212 0.7242 0.000 0.112 0.000 0.880 0.000 0.008
#> GSM228579 4 0.3710 0.5831 0.292 0.000 0.000 0.696 0.000 0.012
#> GSM228580 5 0.3950 0.5279 0.276 0.000 0.000 0.000 0.696 0.028
#> GSM228581 1 0.2484 0.8593 0.896 0.000 0.024 0.000 0.044 0.036
#> GSM228666 5 0.1573 0.7430 0.004 0.000 0.004 0.004 0.936 0.052
#> GSM228564 4 0.0146 0.8283 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM228568 1 0.0993 0.8772 0.964 0.000 0.000 0.024 0.000 0.012
#> GSM228569 1 0.0291 0.8846 0.992 0.000 0.000 0.004 0.000 0.004
#> GSM228572 2 0.1141 0.9079 0.000 0.948 0.000 0.000 0.000 0.052
#> GSM228573 2 0.3050 0.6843 0.000 0.764 0.000 0.000 0.000 0.236
#> GSM228577 1 0.0725 0.8814 0.976 0.000 0.000 0.012 0.000 0.012
#> GSM228578 1 0.0909 0.8844 0.968 0.000 0.000 0.000 0.012 0.020
#> GSM228663 6 0.4082 0.3525 0.004 0.004 0.432 0.000 0.000 0.560
#> GSM228664 6 0.3652 0.7414 0.000 0.044 0.188 0.000 0.000 0.768
#> GSM228665 3 0.1889 0.8762 0.020 0.000 0.920 0.000 0.004 0.056
#> GSM228582 1 0.1498 0.8772 0.940 0.000 0.028 0.000 0.000 0.032
#> GSM228583 1 0.0508 0.8831 0.984 0.000 0.000 0.004 0.000 0.012
#> GSM228585 1 0.2841 0.7565 0.824 0.000 0.000 0.164 0.000 0.012
#> GSM228587 1 0.4229 0.1548 0.548 0.000 0.000 0.436 0.000 0.016
#> GSM228588 4 0.0551 0.8255 0.004 0.000 0.000 0.984 0.004 0.008
#> GSM228589 6 0.3907 0.7630 0.000 0.084 0.152 0.000 0.000 0.764
#> GSM228590 1 0.1649 0.8744 0.932 0.000 0.000 0.036 0.000 0.032
#> GSM228591 6 0.3620 0.4364 0.000 0.352 0.000 0.000 0.000 0.648
#> GSM228597 4 0.5269 0.0844 0.000 0.424 0.000 0.488 0.004 0.084
#> GSM228601 2 0.1075 0.9100 0.000 0.952 0.000 0.000 0.000 0.048
#> GSM228604 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228608 1 0.1442 0.8805 0.944 0.000 0.012 0.000 0.004 0.040
#> GSM228609 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228613 1 0.0363 0.8846 0.988 0.000 0.000 0.000 0.000 0.012
#> GSM228616 2 0.0458 0.9259 0.000 0.984 0.000 0.000 0.000 0.016
#> GSM228628 2 0.2178 0.8390 0.000 0.868 0.000 0.000 0.000 0.132
#> GSM228634 1 0.1176 0.8808 0.956 0.000 0.024 0.000 0.000 0.020
#> GSM228642 2 0.0146 0.9314 0.000 0.996 0.000 0.000 0.000 0.004
#> GSM228645 2 0.1003 0.9163 0.000 0.964 0.000 0.020 0.000 0.016
#> GSM228646 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228652 1 0.1644 0.8758 0.932 0.000 0.028 0.000 0.000 0.040
#> GSM228655 1 0.1788 0.8746 0.928 0.000 0.028 0.000 0.004 0.040
#> GSM228656 1 0.0622 0.8825 0.980 0.000 0.000 0.008 0.000 0.012
#> GSM228659 1 0.2641 0.8603 0.888 0.000 0.000 0.040 0.040 0.032
#> GSM228662 1 0.2946 0.7413 0.812 0.000 0.000 0.176 0.000 0.012
#> GSM228584 1 0.2538 0.8012 0.860 0.000 0.000 0.124 0.000 0.016
#> GSM228586 1 0.0000 0.8848 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228592 1 0.0508 0.8843 0.984 0.000 0.000 0.004 0.000 0.012
#> GSM228593 4 0.0000 0.8276 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM228594 4 0.3748 0.5725 0.300 0.000 0.000 0.688 0.000 0.012
#> GSM228598 1 0.0146 0.8849 0.996 0.000 0.000 0.004 0.000 0.000
#> GSM228607 6 0.4545 0.7519 0.000 0.124 0.176 0.000 0.000 0.700
#> GSM228612 2 0.1714 0.8800 0.000 0.908 0.000 0.000 0.000 0.092
#> GSM228619 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228622 1 0.3765 0.7888 0.808 0.000 0.036 0.000 0.112 0.044
#> GSM228625 2 0.3489 0.6007 0.000 0.708 0.004 0.000 0.000 0.288
#> GSM228631 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228633 2 0.3126 0.6879 0.000 0.752 0.000 0.000 0.000 0.248
#> GSM228637 5 0.0951 0.7470 0.008 0.000 0.000 0.004 0.968 0.020
#> GSM228639 3 0.1075 0.9100 0.000 0.000 0.952 0.000 0.000 0.048
#> GSM228649 4 0.0146 0.8283 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM228660 1 0.1644 0.8767 0.932 0.000 0.028 0.000 0.000 0.040
#> GSM228661 1 0.1341 0.8795 0.948 0.000 0.024 0.000 0.000 0.028
#> GSM228595 2 0.2003 0.8591 0.000 0.884 0.000 0.000 0.000 0.116
#> GSM228599 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228602 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228614 5 0.3445 0.6363 0.000 0.008 0.000 0.000 0.732 0.260
#> GSM228626 6 0.3273 0.7179 0.000 0.024 0.136 0.000 0.016 0.824
#> GSM228640 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228643 5 0.6539 0.1106 0.000 0.000 0.380 0.040 0.400 0.180
#> GSM228650 3 0.2266 0.8695 0.000 0.000 0.880 0.000 0.012 0.108
#> GSM228653 3 0.0547 0.9122 0.000 0.000 0.980 0.000 0.000 0.020
#> GSM228657 6 0.3208 0.6777 0.000 0.024 0.084 0.000 0.044 0.848
#> GSM228605 1 0.4037 0.6341 0.720 0.000 0.012 0.000 0.244 0.024
#> GSM228610 3 0.1418 0.8931 0.000 0.000 0.944 0.000 0.032 0.024
#> GSM228617 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228620 1 0.4962 0.1185 0.488 0.000 0.460 0.000 0.012 0.040
#> GSM228623 6 0.3356 0.7553 0.000 0.072 0.100 0.000 0.004 0.824
#> GSM228629 2 0.0260 0.9298 0.000 0.992 0.000 0.000 0.000 0.008
#> GSM228632 3 0.1478 0.8871 0.004 0.000 0.944 0.000 0.032 0.020
#> GSM228635 5 0.3534 0.6480 0.000 0.000 0.000 0.016 0.740 0.244
#> GSM228647 3 0.3356 0.7759 0.000 0.000 0.808 0.000 0.140 0.052
#> GSM228596 1 0.4789 0.6042 0.676 0.000 0.032 0.000 0.248 0.044
#> GSM228600 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228603 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228615 5 0.0806 0.7456 0.000 0.000 0.008 0.000 0.972 0.020
#> GSM228627 3 0.1232 0.8951 0.004 0.000 0.956 0.000 0.016 0.024
#> GSM228641 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228644 6 0.4693 0.2218 0.000 0.024 0.028 0.000 0.312 0.636
#> GSM228651 3 0.1584 0.9018 0.000 0.000 0.928 0.000 0.008 0.064
#> GSM228654 3 0.1141 0.9091 0.000 0.000 0.948 0.000 0.000 0.052
#> GSM228658 3 0.0547 0.9122 0.000 0.000 0.980 0.000 0.000 0.020
#> GSM228606 2 0.2793 0.7594 0.000 0.800 0.000 0.000 0.000 0.200
#> GSM228611 3 0.1007 0.9123 0.000 0.000 0.956 0.000 0.000 0.044
#> GSM228618 2 0.0000 0.9328 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228621 6 0.5413 0.6795 0.000 0.192 0.228 0.000 0.000 0.580
#> GSM228624 6 0.5273 0.6962 0.000 0.184 0.212 0.000 0.000 0.604
#> GSM228630 3 0.3650 0.5798 0.000 0.000 0.708 0.000 0.012 0.280
#> GSM228636 5 0.3900 0.6357 0.000 0.012 0.000 0.016 0.724 0.248
#> GSM228638 3 0.0547 0.9122 0.000 0.000 0.980 0.000 0.000 0.020
#> GSM228648 3 0.1462 0.9055 0.000 0.000 0.936 0.000 0.008 0.056
#> GSM228670 3 0.1789 0.8999 0.000 0.000 0.924 0.000 0.032 0.044
#> GSM228671 3 0.1007 0.9120 0.000 0.000 0.956 0.000 0.000 0.044
#> GSM228672 5 0.1053 0.7477 0.012 0.000 0.000 0.004 0.964 0.020
#> GSM228674 5 0.3023 0.7283 0.056 0.000 0.052 0.000 0.864 0.028
#> GSM228675 3 0.2179 0.8785 0.000 0.000 0.900 0.000 0.064 0.036
#> GSM228676 5 0.4641 0.4254 0.016 0.000 0.340 0.000 0.616 0.028
#> GSM228667 5 0.2652 0.7148 0.008 0.000 0.104 0.000 0.868 0.020
#> GSM228668 5 0.3988 0.6475 0.180 0.000 0.028 0.000 0.764 0.028
#> GSM228669 5 0.2636 0.7079 0.120 0.000 0.004 0.000 0.860 0.016
#> GSM228673 5 0.3203 0.7240 0.064 0.000 0.056 0.000 0.852 0.028
#> GSM228677 5 0.3746 0.6221 0.000 0.004 0.000 0.012 0.712 0.272
#> GSM228678 5 0.4853 0.6060 0.000 0.004 0.000 0.184 0.676 0.136
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
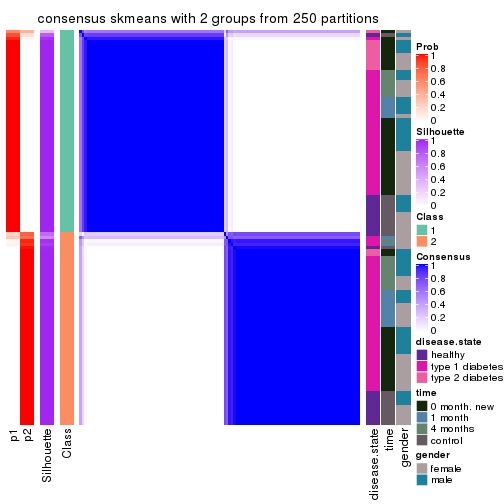
consensus_heatmap(res, k = 3)
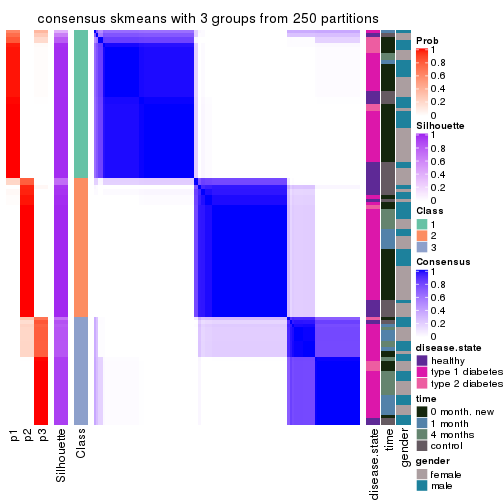
consensus_heatmap(res, k = 4)
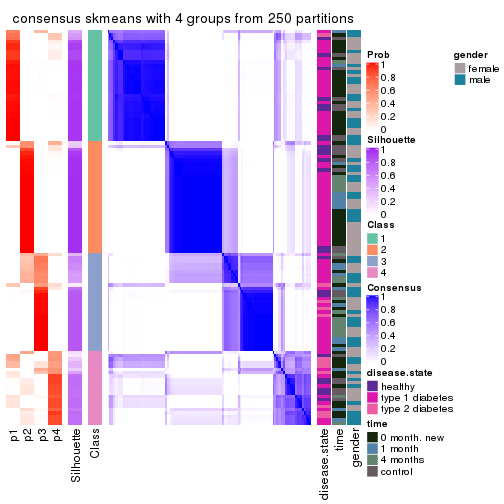
consensus_heatmap(res, k = 5)
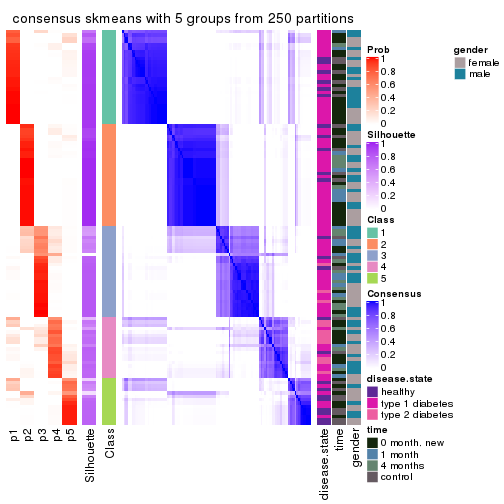
consensus_heatmap(res, k = 6)
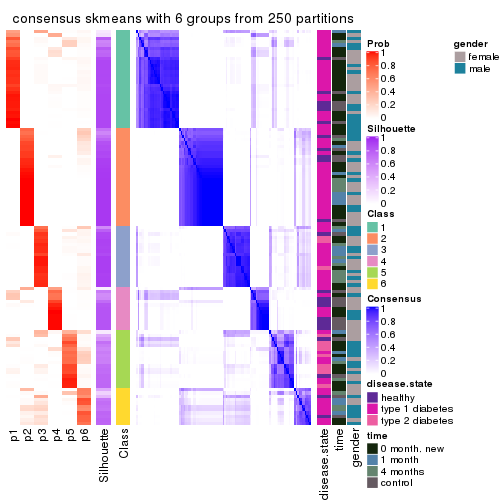
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
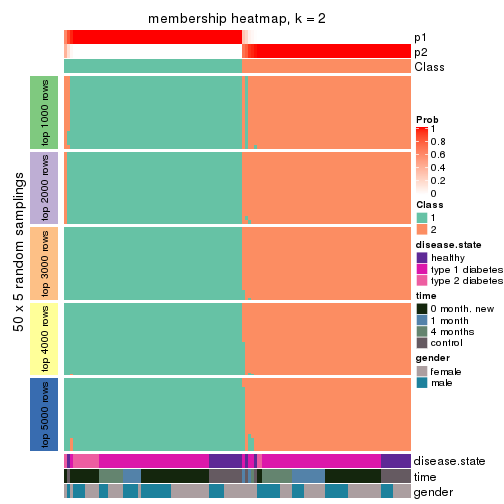
membership_heatmap(res, k = 3)
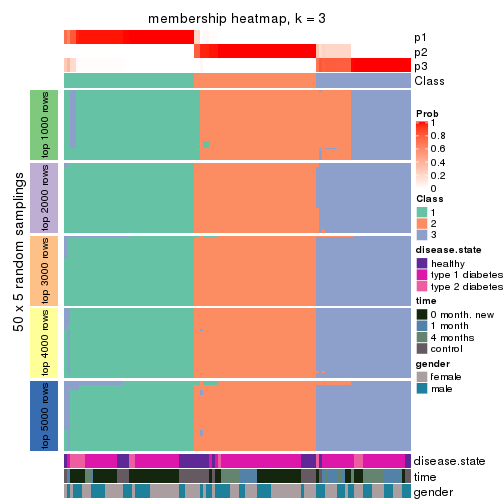
membership_heatmap(res, k = 4)
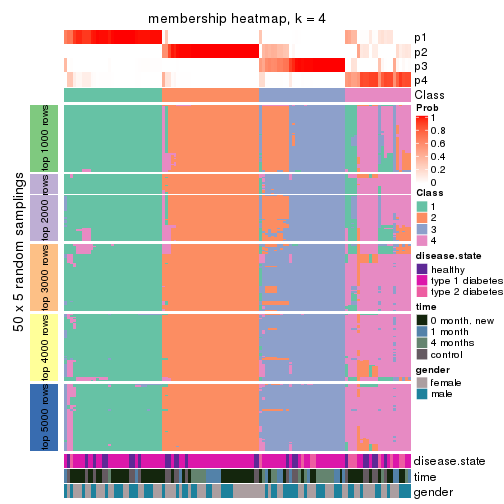
membership_heatmap(res, k = 5)
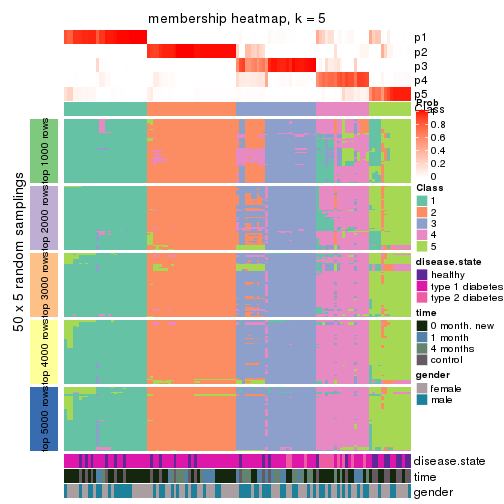
membership_heatmap(res, k = 6)
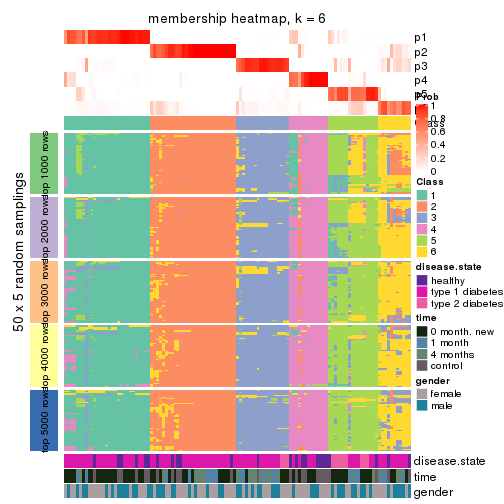
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
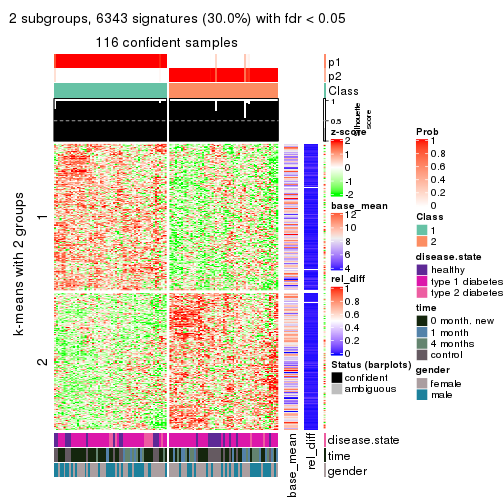
get_signatures(res, k = 3)
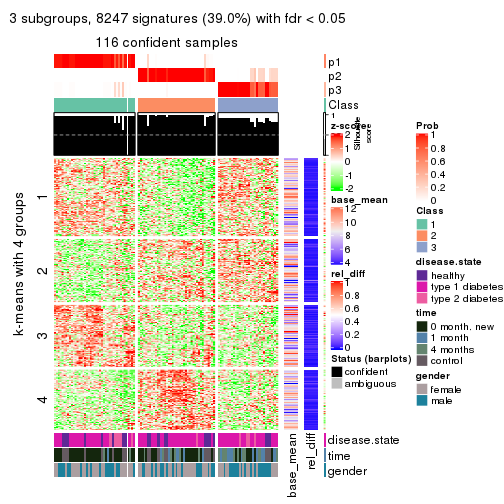
get_signatures(res, k = 4)
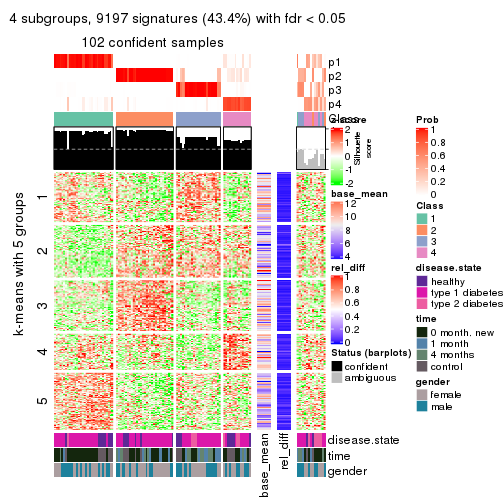
get_signatures(res, k = 5)
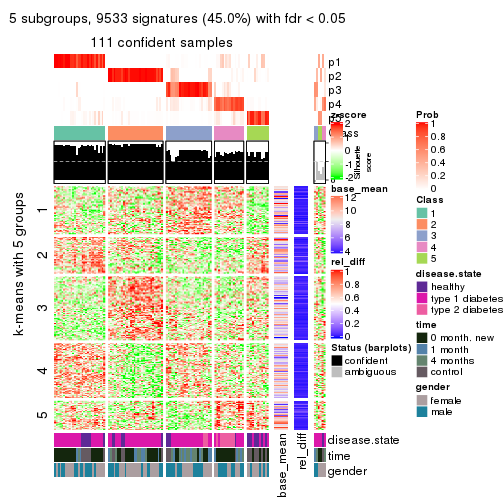
get_signatures(res, k = 6)
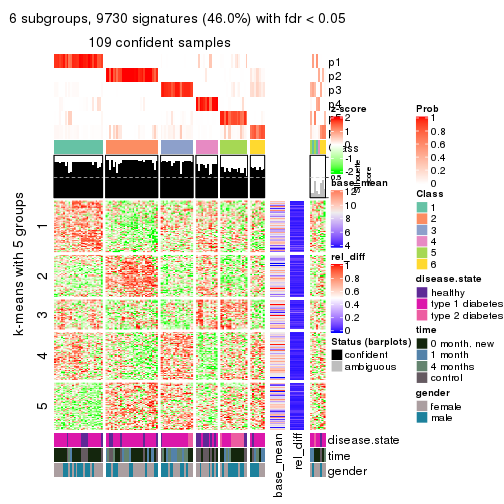
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
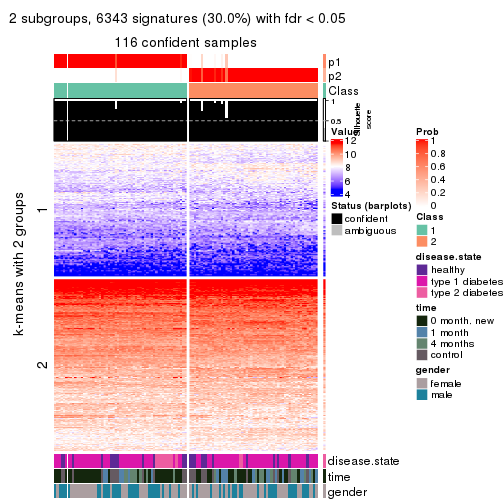
get_signatures(res, k = 3, scale_rows = FALSE)
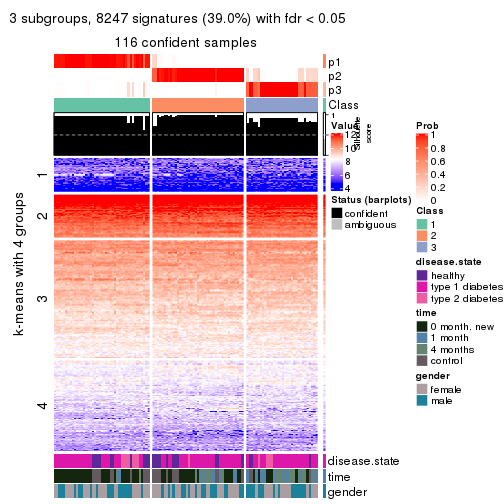
get_signatures(res, k = 4, scale_rows = FALSE)
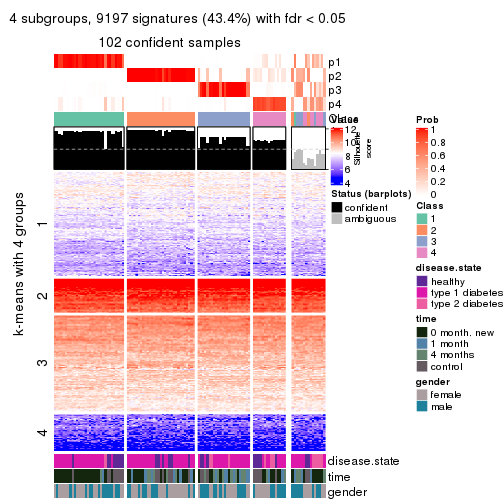
get_signatures(res, k = 5, scale_rows = FALSE)
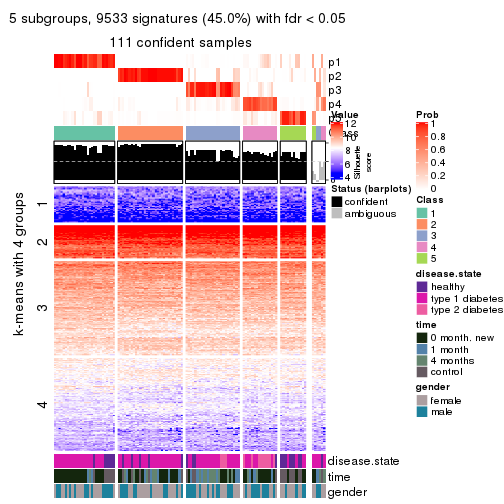
get_signatures(res, k = 6, scale_rows = FALSE)
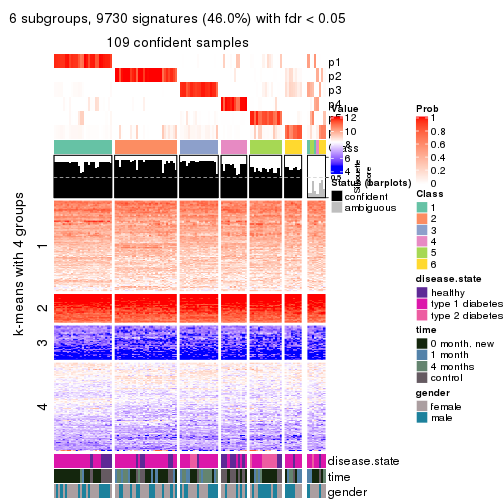
Compare the overlap of signatures from different k:
compare_signatures(res)
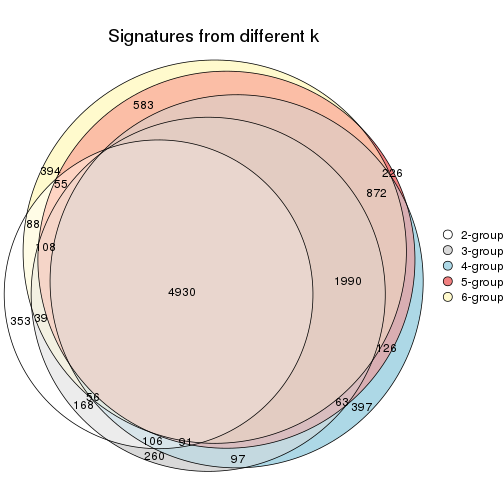
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
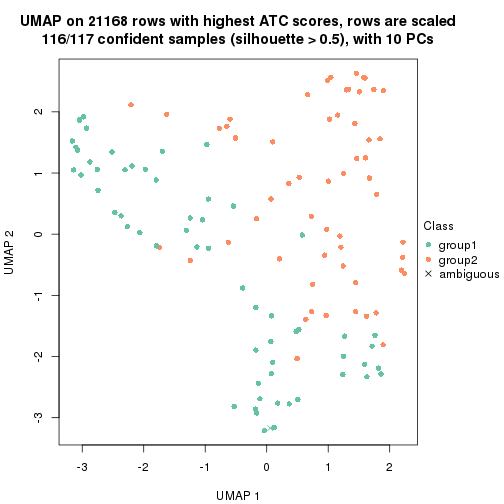
dimension_reduction(res, k = 3, method = "UMAP")
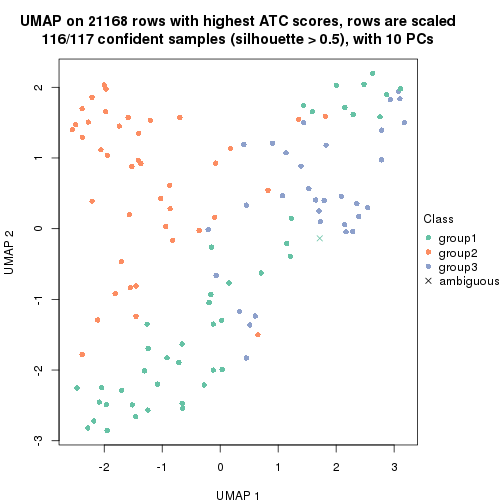
dimension_reduction(res, k = 4, method = "UMAP")
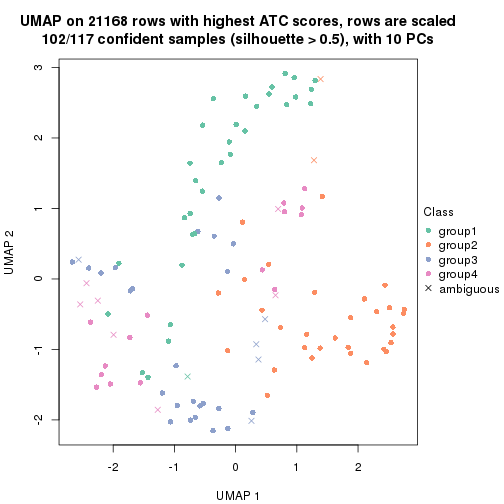
dimension_reduction(res, k = 5, method = "UMAP")
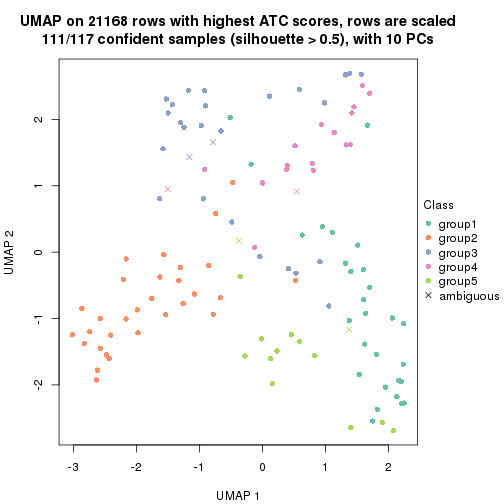
dimension_reduction(res, k = 6, method = "UMAP")
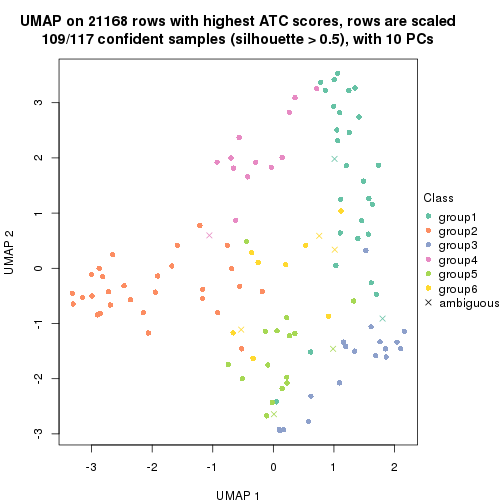
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
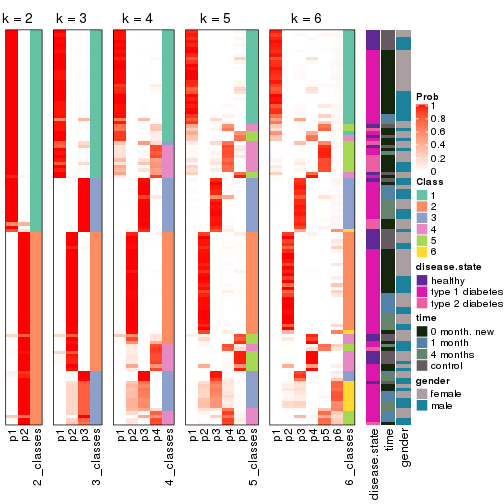
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> ATC:skmeans 116 9.39e-02 1.28e-01 0.694 2
#> ATC:skmeans 116 9.89e-02 6.45e-06 0.455 3
#> ATC:skmeans 102 7.46e-02 7.85e-03 0.733 4
#> ATC:skmeans 111 6.81e-10 1.10e-04 0.585 5
#> ATC:skmeans 109 9.75e-09 8.14e-05 0.588 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["ATC", "pam"]
# you can also extract it by
# res = res_list["ATC:pam"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'ATC' method.
#> Subgroups are detected by 'pam' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 4.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
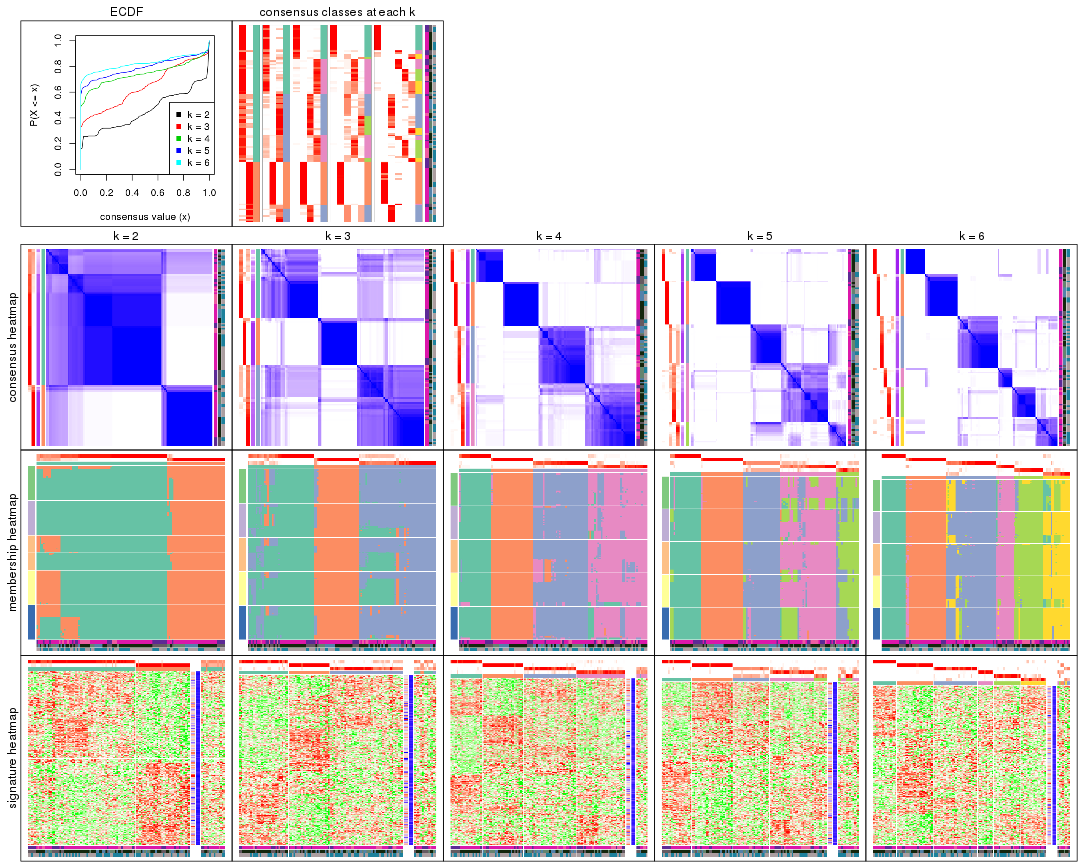
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
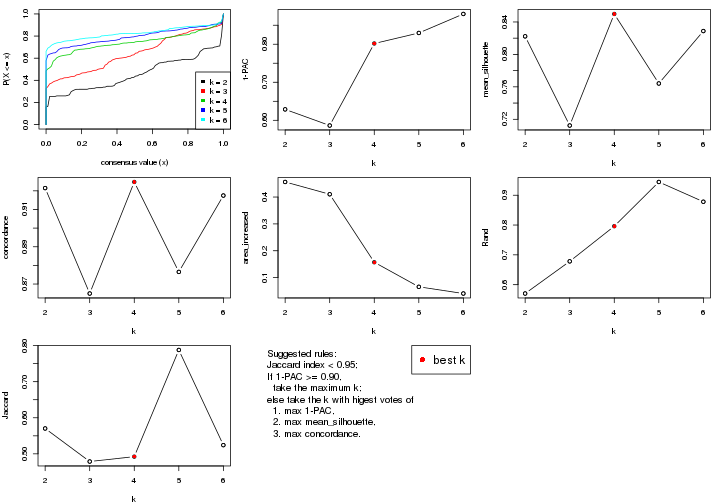
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.629 0.822 0.921 0.4562 0.570 0.570
#> 3 3 0.586 0.712 0.865 0.4104 0.678 0.479
#> 4 4 0.802 0.850 0.925 0.1571 0.796 0.492
#> 5 5 0.830 0.764 0.877 0.0656 0.944 0.788
#> 6 6 0.880 0.829 0.917 0.0406 0.878 0.524
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 4
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.0376 0.8886 0.996 0.004
#> GSM228563 1 0.5842 0.7965 0.860 0.140
#> GSM228565 1 0.0000 0.8889 1.000 0.000
#> GSM228566 2 0.0000 0.9611 0.000 1.000
#> GSM228567 1 0.0000 0.8889 1.000 0.000
#> GSM228570 1 0.5519 0.8069 0.872 0.128
#> GSM228571 1 0.9993 0.2222 0.516 0.484
#> GSM228574 2 0.0000 0.9611 0.000 1.000
#> GSM228575 2 0.0000 0.9611 0.000 1.000
#> GSM228576 2 0.5737 0.7963 0.136 0.864
#> GSM228579 1 0.0000 0.8889 1.000 0.000
#> GSM228580 1 0.0000 0.8889 1.000 0.000
#> GSM228581 1 0.0000 0.8889 1.000 0.000
#> GSM228666 1 0.1414 0.8869 0.980 0.020
#> GSM228564 1 0.5842 0.7965 0.860 0.140
#> GSM228568 1 0.0000 0.8889 1.000 0.000
#> GSM228569 1 0.0000 0.8889 1.000 0.000
#> GSM228572 2 0.0000 0.9611 0.000 1.000
#> GSM228573 2 0.0000 0.9611 0.000 1.000
#> GSM228577 1 0.0000 0.8889 1.000 0.000
#> GSM228578 1 0.0000 0.8889 1.000 0.000
#> GSM228663 1 0.9754 0.3949 0.592 0.408
#> GSM228664 1 0.9922 0.2893 0.552 0.448
#> GSM228665 1 0.1414 0.8869 0.980 0.020
#> GSM228582 1 0.0000 0.8889 1.000 0.000
#> GSM228583 1 0.0000 0.8889 1.000 0.000
#> GSM228585 1 0.0000 0.8889 1.000 0.000
#> GSM228587 1 0.0000 0.8889 1.000 0.000
#> GSM228588 1 0.0000 0.8889 1.000 0.000
#> GSM228589 1 0.9881 0.3229 0.564 0.436
#> GSM228590 1 0.0000 0.8889 1.000 0.000
#> GSM228591 2 0.0000 0.9611 0.000 1.000
#> GSM228597 1 0.6531 0.7822 0.832 0.168
#> GSM228601 2 0.0000 0.9611 0.000 1.000
#> GSM228604 2 0.0000 0.9611 0.000 1.000
#> GSM228608 1 0.0000 0.8889 1.000 0.000
#> GSM228609 2 0.0000 0.9611 0.000 1.000
#> GSM228613 1 0.0000 0.8889 1.000 0.000
#> GSM228616 2 0.0000 0.9611 0.000 1.000
#> GSM228628 2 0.9044 0.4248 0.320 0.680
#> GSM228634 1 0.0000 0.8889 1.000 0.000
#> GSM228642 2 0.0000 0.9611 0.000 1.000
#> GSM228645 2 0.0000 0.9611 0.000 1.000
#> GSM228646 2 0.0000 0.9611 0.000 1.000
#> GSM228652 1 0.0000 0.8889 1.000 0.000
#> GSM228655 1 0.0000 0.8889 1.000 0.000
#> GSM228656 1 0.0000 0.8889 1.000 0.000
#> GSM228659 1 0.0000 0.8889 1.000 0.000
#> GSM228662 1 0.0000 0.8889 1.000 0.000
#> GSM228584 1 0.0000 0.8889 1.000 0.000
#> GSM228586 1 0.0000 0.8889 1.000 0.000
#> GSM228592 1 0.0000 0.8889 1.000 0.000
#> GSM228593 1 0.5629 0.8036 0.868 0.132
#> GSM228594 1 0.0000 0.8889 1.000 0.000
#> GSM228598 1 0.0000 0.8889 1.000 0.000
#> GSM228607 1 0.9933 0.2804 0.548 0.452
#> GSM228612 2 0.0000 0.9611 0.000 1.000
#> GSM228619 2 0.0000 0.9611 0.000 1.000
#> GSM228622 1 0.1414 0.8869 0.980 0.020
#> GSM228625 1 0.6048 0.8009 0.852 0.148
#> GSM228631 2 0.0000 0.9611 0.000 1.000
#> GSM228633 2 0.0000 0.9611 0.000 1.000
#> GSM228637 1 0.0000 0.8889 1.000 0.000
#> GSM228639 1 0.8555 0.6275 0.720 0.280
#> GSM228649 1 0.1414 0.8869 0.980 0.020
#> GSM228660 1 0.1414 0.8869 0.980 0.020
#> GSM228661 1 0.0000 0.8889 1.000 0.000
#> GSM228595 2 0.0000 0.9611 0.000 1.000
#> GSM228599 2 0.0000 0.9611 0.000 1.000
#> GSM228602 2 0.0000 0.9611 0.000 1.000
#> GSM228614 1 0.5737 0.8122 0.864 0.136
#> GSM228626 2 0.0000 0.9611 0.000 1.000
#> GSM228640 2 0.0000 0.9611 0.000 1.000
#> GSM228643 1 0.2948 0.8720 0.948 0.052
#> GSM228650 1 1.0000 0.1872 0.504 0.496
#> GSM228653 1 0.9580 0.4560 0.620 0.380
#> GSM228657 2 0.0000 0.9611 0.000 1.000
#> GSM228605 1 0.1414 0.8869 0.980 0.020
#> GSM228610 1 0.1414 0.8869 0.980 0.020
#> GSM228617 2 0.0000 0.9611 0.000 1.000
#> GSM228620 1 0.1414 0.8869 0.980 0.020
#> GSM228623 1 0.9833 0.3550 0.576 0.424
#> GSM228629 2 0.0000 0.9611 0.000 1.000
#> GSM228632 1 0.1414 0.8869 0.980 0.020
#> GSM228635 1 0.5294 0.8253 0.880 0.120
#> GSM228647 1 0.1414 0.8869 0.980 0.020
#> GSM228596 1 0.0000 0.8889 1.000 0.000
#> GSM228600 2 0.0000 0.9611 0.000 1.000
#> GSM228603 2 0.0000 0.9611 0.000 1.000
#> GSM228615 1 0.1414 0.8869 0.980 0.020
#> GSM228627 1 0.1414 0.8869 0.980 0.020
#> GSM228641 2 0.0000 0.9611 0.000 1.000
#> GSM228644 2 0.0000 0.9611 0.000 1.000
#> GSM228651 1 0.9635 0.4399 0.612 0.388
#> GSM228654 1 0.9635 0.4399 0.612 0.388
#> GSM228658 1 0.9209 0.5379 0.664 0.336
#> GSM228606 2 0.7883 0.6205 0.236 0.764
#> GSM228611 1 0.9608 0.4479 0.616 0.384
#> GSM228618 2 0.0000 0.9611 0.000 1.000
#> GSM228621 2 0.0000 0.9611 0.000 1.000
#> GSM228624 2 0.9850 0.0719 0.428 0.572
#> GSM228630 2 0.0000 0.9611 0.000 1.000
#> GSM228636 1 0.6343 0.7896 0.840 0.160
#> GSM228638 1 0.1414 0.8869 0.980 0.020
#> GSM228648 1 0.9686 0.4227 0.604 0.396
#> GSM228670 1 0.1414 0.8869 0.980 0.020
#> GSM228671 1 0.9608 0.4479 0.616 0.384
#> GSM228672 1 0.0000 0.8889 1.000 0.000
#> GSM228674 1 0.0000 0.8889 1.000 0.000
#> GSM228675 1 0.1414 0.8869 0.980 0.020
#> GSM228676 1 0.1414 0.8869 0.980 0.020
#> GSM228667 1 0.1414 0.8869 0.980 0.020
#> GSM228668 1 0.1414 0.8869 0.980 0.020
#> GSM228669 1 0.0000 0.8889 1.000 0.000
#> GSM228673 1 0.1184 0.8874 0.984 0.016
#> GSM228677 1 0.5629 0.8156 0.868 0.132
#> GSM228678 1 0.6048 0.8009 0.852 0.148
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.5254 0.5364 0.736 0.000 0.264
#> GSM228563 1 0.6809 0.2145 0.524 0.464 0.012
#> GSM228565 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228566 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228567 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228570 1 0.7446 0.4787 0.664 0.076 0.260
#> GSM228571 1 0.6095 0.1003 0.608 0.000 0.392
#> GSM228574 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228575 2 0.6305 -0.0473 0.000 0.516 0.484
#> GSM228576 2 0.3644 0.8070 0.004 0.872 0.124
#> GSM228579 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228580 1 0.4062 0.6795 0.836 0.000 0.164
#> GSM228581 1 0.4555 0.6761 0.800 0.000 0.200
#> GSM228666 3 0.5905 0.6063 0.352 0.000 0.648
#> GSM228564 1 0.7065 0.5533 0.664 0.288 0.048
#> GSM228568 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228569 1 0.5785 0.4880 0.668 0.000 0.332
#> GSM228572 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228573 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228577 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228578 1 0.1411 0.7921 0.964 0.000 0.036
#> GSM228663 3 0.2663 0.7416 0.024 0.044 0.932
#> GSM228664 3 0.0237 0.7679 0.000 0.004 0.996
#> GSM228665 3 0.1031 0.7624 0.024 0.000 0.976
#> GSM228582 1 0.6309 0.1913 0.500 0.000 0.500
#> GSM228583 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228585 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228587 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228588 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228589 3 0.6881 0.6297 0.320 0.032 0.648
#> GSM228590 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228591 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228597 3 0.6651 0.6131 0.340 0.020 0.640
#> GSM228601 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228604 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228608 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228609 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228613 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228616 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228628 3 0.5787 0.7290 0.136 0.068 0.796
#> GSM228634 1 0.5905 0.4650 0.648 0.000 0.352
#> GSM228642 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228645 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228646 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228652 1 0.0892 0.7960 0.980 0.000 0.020
#> GSM228655 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228656 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228659 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228662 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228584 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228586 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228592 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228593 1 0.6518 0.6426 0.752 0.168 0.080
#> GSM228594 1 0.0000 0.8050 1.000 0.000 0.000
#> GSM228598 1 0.4931 0.5946 0.768 0.000 0.232
#> GSM228607 3 0.4121 0.7291 0.168 0.000 0.832
#> GSM228612 2 0.6305 -0.0473 0.000 0.516 0.484
#> GSM228619 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228622 3 0.5926 0.6020 0.356 0.000 0.644
#> GSM228625 3 0.8425 0.5168 0.348 0.100 0.552
#> GSM228631 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228633 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228637 1 0.5785 0.3780 0.668 0.000 0.332
#> GSM228639 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228649 3 0.6008 0.5790 0.372 0.000 0.628
#> GSM228660 1 0.5968 0.2887 0.636 0.000 0.364
#> GSM228661 1 0.4931 0.6133 0.768 0.000 0.232
#> GSM228595 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228599 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228602 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228614 3 0.6473 0.6240 0.332 0.016 0.652
#> GSM228626 3 0.4504 0.6534 0.000 0.196 0.804
#> GSM228640 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228643 3 0.6603 0.6216 0.332 0.020 0.648
#> GSM228650 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228653 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228657 3 0.5905 0.4811 0.000 0.352 0.648
#> GSM228605 3 0.6045 0.5613 0.380 0.000 0.620
#> GSM228610 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228617 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228620 3 0.1964 0.7413 0.056 0.000 0.944
#> GSM228623 3 0.5760 0.6341 0.328 0.000 0.672
#> GSM228629 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228632 3 0.0237 0.7682 0.004 0.000 0.996
#> GSM228635 3 0.5785 0.6298 0.332 0.000 0.668
#> GSM228647 3 0.2878 0.7578 0.096 0.000 0.904
#> GSM228596 1 0.6309 0.0371 0.504 0.000 0.496
#> GSM228600 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228603 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228615 3 0.6008 0.5790 0.372 0.000 0.628
#> GSM228627 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228641 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228644 3 0.5905 0.4811 0.000 0.352 0.648
#> GSM228651 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228654 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228658 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228606 3 0.6906 0.6240 0.324 0.032 0.644
#> GSM228611 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228618 2 0.0000 0.9516 0.000 1.000 0.000
#> GSM228621 3 0.2165 0.7503 0.000 0.064 0.936
#> GSM228624 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228630 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228636 3 0.6651 0.6131 0.340 0.020 0.640
#> GSM228638 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228648 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228670 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228671 3 0.0000 0.7698 0.000 0.000 1.000
#> GSM228672 1 0.5216 0.5435 0.740 0.000 0.260
#> GSM228674 1 0.6260 0.1161 0.552 0.000 0.448
#> GSM228675 3 0.0892 0.7697 0.020 0.000 0.980
#> GSM228676 3 0.4399 0.7189 0.188 0.000 0.812
#> GSM228667 3 0.5560 0.6544 0.300 0.000 0.700
#> GSM228668 3 0.5988 0.5427 0.368 0.000 0.632
#> GSM228669 1 0.4452 0.6544 0.808 0.000 0.192
#> GSM228673 3 0.5497 0.6132 0.292 0.000 0.708
#> GSM228677 3 0.5785 0.6298 0.332 0.000 0.668
#> GSM228678 3 0.6180 0.6276 0.332 0.008 0.660
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.1118 0.8416 0.036 0.000 0.000 0.964
#> GSM228563 4 0.2408 0.8277 0.036 0.044 0.000 0.920
#> GSM228565 4 0.1118 0.8416 0.036 0.000 0.000 0.964
#> GSM228566 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228567 1 0.0000 0.9624 1.000 0.000 0.000 0.000
#> GSM228570 4 0.1118 0.8416 0.036 0.000 0.000 0.964
#> GSM228571 1 0.5573 0.5298 0.676 0.000 0.052 0.272
#> GSM228574 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228575 3 0.3074 0.8199 0.000 0.152 0.848 0.000
#> GSM228576 2 0.4010 0.7751 0.000 0.816 0.028 0.156
#> GSM228579 1 0.0000 0.9624 1.000 0.000 0.000 0.000
#> GSM228580 4 0.0336 0.8411 0.008 0.000 0.000 0.992
#> GSM228581 1 0.1211 0.9445 0.960 0.000 0.000 0.040
#> GSM228666 4 0.1118 0.8404 0.000 0.000 0.036 0.964
#> GSM228564 4 0.1118 0.8416 0.036 0.000 0.000 0.964
#> GSM228568 1 0.0000 0.9624 1.000 0.000 0.000 0.000
#> GSM228569 1 0.1118 0.9461 0.964 0.000 0.000 0.036
#> GSM228572 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228573 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228577 1 0.0000 0.9624 1.000 0.000 0.000 0.000
#> GSM228578 4 0.3428 0.7517 0.144 0.000 0.012 0.844
#> GSM228663 4 0.4898 0.3973 0.000 0.000 0.416 0.584
#> GSM228664 3 0.1118 0.8912 0.000 0.000 0.964 0.036
#> GSM228665 4 0.4804 0.4563 0.000 0.000 0.384 0.616
#> GSM228582 1 0.4100 0.8068 0.816 0.000 0.148 0.036
#> GSM228583 1 0.0000 0.9624 1.000 0.000 0.000 0.000
#> GSM228585 1 0.0000 0.9624 1.000 0.000 0.000 0.000
#> GSM228587 4 0.4277 0.6217 0.280 0.000 0.000 0.720
#> GSM228588 4 0.1118 0.8416 0.036 0.000 0.000 0.964
#> GSM228589 3 0.3444 0.8052 0.000 0.000 0.816 0.184
#> GSM228590 4 0.1474 0.8383 0.052 0.000 0.000 0.948
#> GSM228591 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228597 4 0.4746 0.4285 0.000 0.000 0.368 0.632
#> GSM228601 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228604 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228608 4 0.0000 0.8406 0.000 0.000 0.000 1.000
#> GSM228609 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228613 1 0.0000 0.9624 1.000 0.000 0.000 0.000
#> GSM228616 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228628 3 0.2224 0.8894 0.000 0.032 0.928 0.040
#> GSM228634 1 0.1118 0.9461 0.964 0.000 0.000 0.036
#> GSM228642 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228645 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228646 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228652 4 0.4040 0.6296 0.248 0.000 0.000 0.752
#> GSM228655 4 0.0817 0.8391 0.024 0.000 0.000 0.976
#> GSM228656 1 0.0000 0.9624 1.000 0.000 0.000 0.000
#> GSM228659 4 0.0000 0.8406 0.000 0.000 0.000 1.000
#> GSM228662 1 0.0000 0.9624 1.000 0.000 0.000 0.000
#> GSM228584 1 0.0000 0.9624 1.000 0.000 0.000 0.000
#> GSM228586 1 0.0000 0.9624 1.000 0.000 0.000 0.000
#> GSM228592 1 0.0000 0.9624 1.000 0.000 0.000 0.000
#> GSM228593 4 0.5179 0.6612 0.220 0.052 0.000 0.728
#> GSM228594 1 0.0000 0.9624 1.000 0.000 0.000 0.000
#> GSM228598 1 0.1118 0.9461 0.964 0.000 0.000 0.036
#> GSM228607 3 0.1902 0.8820 0.000 0.004 0.932 0.064
#> GSM228612 3 0.3074 0.8199 0.000 0.152 0.848 0.000
#> GSM228619 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228622 4 0.0000 0.8406 0.000 0.000 0.000 1.000
#> GSM228625 4 0.5212 0.3224 0.000 0.008 0.420 0.572
#> GSM228631 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228633 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228637 4 0.1118 0.8416 0.036 0.000 0.000 0.964
#> GSM228639 3 0.0000 0.9073 0.000 0.000 1.000 0.000
#> GSM228649 4 0.0921 0.8406 0.000 0.000 0.028 0.972
#> GSM228660 4 0.5328 0.6021 0.248 0.000 0.048 0.704
#> GSM228661 1 0.1118 0.9461 0.964 0.000 0.000 0.036
#> GSM228595 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228599 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228602 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228614 3 0.4356 0.6077 0.000 0.000 0.708 0.292
#> GSM228626 3 0.0921 0.9011 0.000 0.028 0.972 0.000
#> GSM228640 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228643 3 0.3024 0.8191 0.000 0.000 0.852 0.148
#> GSM228650 3 0.0000 0.9073 0.000 0.000 1.000 0.000
#> GSM228653 3 0.0000 0.9073 0.000 0.000 1.000 0.000
#> GSM228657 3 0.3024 0.8232 0.000 0.148 0.852 0.000
#> GSM228605 4 0.4224 0.7581 0.044 0.000 0.144 0.812
#> GSM228610 3 0.1022 0.8961 0.000 0.000 0.968 0.032
#> GSM228617 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228620 3 0.4643 0.4073 0.000 0.000 0.656 0.344
#> GSM228623 3 0.2921 0.8261 0.000 0.000 0.860 0.140
#> GSM228629 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228632 3 0.2149 0.8628 0.000 0.000 0.912 0.088
#> GSM228635 3 0.4564 0.5330 0.000 0.000 0.672 0.328
#> GSM228647 3 0.1389 0.8948 0.000 0.000 0.952 0.048
#> GSM228596 4 0.2011 0.8189 0.000 0.000 0.080 0.920
#> GSM228600 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228603 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228615 4 0.1118 0.8404 0.000 0.000 0.036 0.964
#> GSM228627 3 0.1557 0.8821 0.000 0.000 0.944 0.056
#> GSM228641 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228644 3 0.3024 0.8232 0.000 0.148 0.852 0.000
#> GSM228651 3 0.0000 0.9073 0.000 0.000 1.000 0.000
#> GSM228654 3 0.0000 0.9073 0.000 0.000 1.000 0.000
#> GSM228658 3 0.0000 0.9073 0.000 0.000 1.000 0.000
#> GSM228606 3 0.3074 0.8150 0.000 0.000 0.848 0.152
#> GSM228611 3 0.0000 0.9073 0.000 0.000 1.000 0.000
#> GSM228618 2 0.0000 0.9921 0.000 1.000 0.000 0.000
#> GSM228621 3 0.0469 0.9054 0.000 0.012 0.988 0.000
#> GSM228624 3 0.0000 0.9073 0.000 0.000 1.000 0.000
#> GSM228630 3 0.0000 0.9073 0.000 0.000 1.000 0.000
#> GSM228636 4 0.4103 0.6294 0.000 0.000 0.256 0.744
#> GSM228638 3 0.1118 0.8912 0.000 0.000 0.964 0.036
#> GSM228648 3 0.0000 0.9073 0.000 0.000 1.000 0.000
#> GSM228670 3 0.0469 0.9038 0.000 0.000 0.988 0.012
#> GSM228671 3 0.0000 0.9073 0.000 0.000 1.000 0.000
#> GSM228672 4 0.1118 0.8416 0.036 0.000 0.000 0.964
#> GSM228674 4 0.1716 0.8272 0.000 0.000 0.064 0.936
#> GSM228675 3 0.0336 0.9069 0.000 0.000 0.992 0.008
#> GSM228676 4 0.2921 0.8023 0.000 0.000 0.140 0.860
#> GSM228667 4 0.1716 0.8394 0.000 0.000 0.064 0.936
#> GSM228668 4 0.1474 0.8348 0.000 0.000 0.052 0.948
#> GSM228669 4 0.0921 0.8424 0.028 0.000 0.000 0.972
#> GSM228673 4 0.2011 0.8189 0.000 0.000 0.080 0.920
#> GSM228677 4 0.5000 -0.0351 0.000 0.000 0.496 0.504
#> GSM228678 4 0.4933 0.2052 0.000 0.000 0.432 0.568
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 4 0.1478 0.753 0.000 0.000 0.064 0.936 0.000
#> GSM228563 4 0.2863 0.734 0.000 0.060 0.064 0.876 0.000
#> GSM228565 4 0.0000 0.764 0.000 0.000 0.000 1.000 0.000
#> GSM228566 2 0.0162 0.988 0.000 0.996 0.004 0.000 0.000
#> GSM228567 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228570 4 0.1478 0.753 0.000 0.000 0.064 0.936 0.000
#> GSM228571 1 0.4832 0.619 0.720 0.000 0.064 0.208 0.008
#> GSM228574 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228575 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228576 2 0.3888 0.758 0.000 0.800 0.064 0.136 0.000
#> GSM228579 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228580 4 0.0955 0.762 0.004 0.000 0.000 0.968 0.028
#> GSM228581 1 0.4182 0.508 0.644 0.000 0.352 0.000 0.004
#> GSM228666 4 0.0162 0.764 0.000 0.000 0.004 0.996 0.000
#> GSM228564 4 0.1478 0.753 0.000 0.000 0.064 0.936 0.000
#> GSM228568 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228569 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228572 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228573 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228577 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228578 4 0.6694 0.478 0.068 0.000 0.336 0.524 0.072
#> GSM228663 5 0.5273 0.584 0.000 0.000 0.352 0.060 0.588
#> GSM228664 3 0.2929 0.505 0.000 0.000 0.820 0.000 0.180
#> GSM228665 5 0.5215 0.579 0.000 0.000 0.352 0.056 0.592
#> GSM228582 5 0.5328 0.576 0.064 0.000 0.352 0.000 0.584
#> GSM228583 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228585 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228587 4 0.5364 0.543 0.056 0.000 0.352 0.588 0.004
#> GSM228588 4 0.1410 0.754 0.000 0.000 0.060 0.940 0.000
#> GSM228589 3 0.3132 0.517 0.000 0.000 0.820 0.008 0.172
#> GSM228590 4 0.4333 0.572 0.004 0.000 0.352 0.640 0.004
#> GSM228591 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228597 4 0.6049 0.350 0.000 0.000 0.232 0.576 0.192
#> GSM228601 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228604 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228608 4 0.4182 0.574 0.000 0.000 0.352 0.644 0.004
#> GSM228609 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228613 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228616 2 0.0162 0.988 0.000 0.996 0.004 0.000 0.000
#> GSM228628 3 0.4350 0.911 0.000 0.004 0.588 0.000 0.408
#> GSM228634 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228642 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228645 2 0.0162 0.988 0.000 0.996 0.004 0.000 0.000
#> GSM228646 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228652 4 0.5531 0.535 0.068 0.000 0.352 0.576 0.004
#> GSM228655 4 0.5113 0.557 0.040 0.000 0.352 0.604 0.004
#> GSM228656 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228659 4 0.3366 0.680 0.000 0.000 0.212 0.784 0.004
#> GSM228662 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228584 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228586 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228592 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228593 4 0.6925 0.475 0.240 0.136 0.064 0.560 0.000
#> GSM228594 1 0.0000 0.938 1.000 0.000 0.000 0.000 0.000
#> GSM228598 1 0.0162 0.934 0.996 0.000 0.000 0.000 0.004
#> GSM228607 3 0.4666 0.897 0.000 0.000 0.572 0.016 0.412
#> GSM228612 3 0.4210 0.914 0.000 0.000 0.588 0.000 0.412
#> GSM228619 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228622 4 0.0162 0.765 0.000 0.000 0.000 0.996 0.004
#> GSM228625 4 0.6024 0.060 0.000 0.000 0.116 0.472 0.412
#> GSM228631 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228633 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228637 4 0.0162 0.765 0.000 0.000 0.000 0.996 0.004
#> GSM228639 5 0.0162 0.492 0.000 0.000 0.004 0.000 0.996
#> GSM228649 4 0.3774 0.662 0.000 0.000 0.296 0.704 0.000
#> GSM228660 4 0.6287 0.486 0.040 0.000 0.352 0.540 0.068
#> GSM228661 1 0.4182 0.508 0.644 0.000 0.352 0.000 0.004
#> GSM228595 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228599 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228602 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228614 3 0.5971 0.680 0.000 0.000 0.584 0.172 0.244
#> GSM228626 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228640 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228643 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228650 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228653 5 0.0000 0.497 0.000 0.000 0.000 0.000 1.000
#> GSM228657 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228605 4 0.3099 0.680 0.028 0.000 0.124 0.848 0.000
#> GSM228610 5 0.5159 -0.176 0.000 0.000 0.284 0.072 0.644
#> GSM228617 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228620 5 0.4937 0.625 0.000 0.000 0.264 0.064 0.672
#> GSM228623 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228629 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228632 5 0.5302 0.577 0.000 0.000 0.344 0.064 0.592
#> GSM228635 3 0.6001 0.563 0.000 0.000 0.580 0.244 0.176
#> GSM228647 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228596 4 0.5583 0.513 0.000 0.000 0.352 0.564 0.084
#> GSM228600 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228603 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228615 4 0.0162 0.764 0.000 0.000 0.004 0.996 0.000
#> GSM228627 5 0.3056 0.590 0.000 0.000 0.068 0.068 0.864
#> GSM228641 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228644 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228651 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228654 5 0.1792 0.349 0.000 0.000 0.084 0.000 0.916
#> GSM228658 5 0.0162 0.492 0.000 0.000 0.004 0.000 0.996
#> GSM228606 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228611 5 0.4306 -0.810 0.000 0.000 0.492 0.000 0.508
#> GSM228618 2 0.0000 0.991 0.000 1.000 0.000 0.000 0.000
#> GSM228621 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228624 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228630 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228636 4 0.4199 0.608 0.000 0.000 0.160 0.772 0.068
#> GSM228638 5 0.4030 0.623 0.000 0.000 0.352 0.000 0.648
#> GSM228648 3 0.4219 0.918 0.000 0.000 0.584 0.000 0.416
#> GSM228670 5 0.3274 0.625 0.000 0.000 0.220 0.000 0.780
#> GSM228671 3 0.4249 0.901 0.000 0.000 0.568 0.000 0.432
#> GSM228672 4 0.0000 0.764 0.000 0.000 0.000 1.000 0.000
#> GSM228674 4 0.1792 0.744 0.000 0.000 0.000 0.916 0.084
#> GSM228675 3 0.4307 0.804 0.000 0.000 0.504 0.000 0.496
#> GSM228676 4 0.1892 0.744 0.000 0.000 0.004 0.916 0.080
#> GSM228667 4 0.1892 0.744 0.000 0.000 0.004 0.916 0.080
#> GSM228668 4 0.1671 0.748 0.000 0.000 0.000 0.924 0.076
#> GSM228669 4 0.0162 0.765 0.000 0.000 0.000 0.996 0.004
#> GSM228673 4 0.1732 0.747 0.000 0.000 0.000 0.920 0.080
#> GSM228677 4 0.5252 0.254 0.000 0.000 0.364 0.580 0.056
#> GSM228678 4 0.4088 0.461 0.000 0.000 0.368 0.632 0.000
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 4 0.0146 0.9177 0.000 0.000 0.000 0.996 0.004 0.000
#> GSM228563 4 0.0000 0.9187 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM228565 5 0.0000 0.9071 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM228566 2 0.1007 0.9271 0.000 0.956 0.000 0.044 0.000 0.000
#> GSM228567 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228570 4 0.0000 0.9187 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM228571 4 0.0000 0.9187 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM228574 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228575 3 0.0000 0.8742 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228576 4 0.0000 0.9187 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM228579 4 0.3727 0.3667 0.388 0.000 0.000 0.612 0.000 0.000
#> GSM228580 5 0.0363 0.9062 0.000 0.000 0.000 0.000 0.988 0.012
#> GSM228581 6 0.3446 0.5863 0.308 0.000 0.000 0.000 0.000 0.692
#> GSM228666 5 0.0000 0.9071 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM228564 4 0.0547 0.9104 0.000 0.000 0.000 0.980 0.020 0.000
#> GSM228568 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228569 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228572 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228573 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228577 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228578 6 0.3515 0.6106 0.000 0.000 0.000 0.000 0.324 0.676
#> GSM228663 6 0.0547 0.7506 0.000 0.000 0.000 0.000 0.020 0.980
#> GSM228664 3 0.3607 0.4217 0.000 0.000 0.652 0.000 0.000 0.348
#> GSM228665 6 0.0000 0.7497 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM228582 6 0.0000 0.7497 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM228583 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228585 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228587 6 0.4806 0.7153 0.164 0.000 0.000 0.004 0.148 0.684
#> GSM228588 4 0.1007 0.8950 0.000 0.000 0.000 0.956 0.044 0.000
#> GSM228589 3 0.3945 0.3220 0.000 0.000 0.612 0.000 0.008 0.380
#> GSM228590 6 0.3653 0.6599 0.008 0.000 0.000 0.000 0.300 0.692
#> GSM228591 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228597 4 0.2609 0.8204 0.000 0.000 0.096 0.868 0.036 0.000
#> GSM228601 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228604 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228608 6 0.3446 0.6521 0.000 0.000 0.000 0.000 0.308 0.692
#> GSM228609 2 0.3782 0.3165 0.000 0.588 0.000 0.412 0.000 0.000
#> GSM228613 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228616 2 0.1007 0.9271 0.000 0.956 0.000 0.044 0.000 0.000
#> GSM228628 3 0.0777 0.8659 0.000 0.004 0.972 0.024 0.000 0.000
#> GSM228634 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228642 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228645 2 0.3672 0.4484 0.000 0.632 0.000 0.368 0.000 0.000
#> GSM228646 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228652 6 0.4626 0.7164 0.172 0.000 0.000 0.000 0.136 0.692
#> GSM228655 6 0.4494 0.7066 0.092 0.000 0.000 0.000 0.216 0.692
#> GSM228656 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228659 5 0.3050 0.5486 0.000 0.000 0.000 0.000 0.764 0.236
#> GSM228662 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228584 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228586 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228592 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228593 4 0.0000 0.9187 0.000 0.000 0.000 1.000 0.000 0.000
#> GSM228594 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228598 1 0.0000 1.0000 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228607 3 0.0405 0.8709 0.000 0.000 0.988 0.000 0.008 0.004
#> GSM228612 3 0.0790 0.8635 0.000 0.000 0.968 0.032 0.000 0.000
#> GSM228619 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228622 5 0.0000 0.9071 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM228625 3 0.2376 0.8177 0.000 0.000 0.888 0.044 0.068 0.000
#> GSM228631 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228633 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228637 5 0.0000 0.9071 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM228639 3 0.3446 0.6379 0.000 0.000 0.692 0.000 0.000 0.308
#> GSM228649 4 0.2846 0.7998 0.000 0.000 0.000 0.856 0.084 0.060
#> GSM228660 6 0.1444 0.7583 0.000 0.000 0.000 0.000 0.072 0.928
#> GSM228661 6 0.3446 0.5863 0.308 0.000 0.000 0.000 0.000 0.692
#> GSM228595 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228599 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228602 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228614 3 0.2823 0.6953 0.000 0.000 0.796 0.000 0.204 0.000
#> GSM228626 3 0.0000 0.8742 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228640 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228643 3 0.0777 0.8650 0.000 0.000 0.972 0.024 0.004 0.000
#> GSM228650 3 0.0000 0.8742 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228653 3 0.3547 0.6062 0.000 0.000 0.668 0.000 0.000 0.332
#> GSM228657 3 0.0000 0.8742 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228605 5 0.1007 0.8767 0.000 0.000 0.044 0.000 0.956 0.000
#> GSM228610 3 0.4095 0.6933 0.000 0.000 0.748 0.000 0.152 0.100
#> GSM228617 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228620 6 0.1663 0.6870 0.000 0.000 0.088 0.000 0.000 0.912
#> GSM228623 3 0.0000 0.8742 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228629 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228632 6 0.0260 0.7469 0.000 0.000 0.008 0.000 0.000 0.992
#> GSM228635 3 0.3446 0.5208 0.000 0.000 0.692 0.000 0.308 0.000
#> GSM228647 3 0.0000 0.8742 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228596 6 0.3330 0.6587 0.000 0.000 0.000 0.000 0.284 0.716
#> GSM228600 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228603 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228615 5 0.0000 0.9071 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM228627 3 0.4076 0.3670 0.000 0.000 0.540 0.000 0.008 0.452
#> GSM228641 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228644 3 0.0000 0.8742 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228651 3 0.0000 0.8742 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228654 3 0.3309 0.6638 0.000 0.000 0.720 0.000 0.000 0.280
#> GSM228658 3 0.3446 0.6379 0.000 0.000 0.692 0.000 0.000 0.308
#> GSM228606 3 0.0000 0.8742 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228611 3 0.0713 0.8646 0.000 0.000 0.972 0.000 0.000 0.028
#> GSM228618 2 0.0000 0.9628 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228621 3 0.0000 0.8742 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228624 3 0.0000 0.8742 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228630 3 0.0000 0.8742 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228636 5 0.2941 0.6831 0.000 0.000 0.220 0.000 0.780 0.000
#> GSM228638 6 0.0000 0.7497 0.000 0.000 0.000 0.000 0.000 1.000
#> GSM228648 3 0.0000 0.8742 0.000 0.000 1.000 0.000 0.000 0.000
#> GSM228670 6 0.3782 -0.0689 0.000 0.000 0.412 0.000 0.000 0.588
#> GSM228671 3 0.0146 0.8733 0.000 0.000 0.996 0.000 0.000 0.004
#> GSM228672 5 0.0000 0.9071 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM228674 5 0.0713 0.9028 0.000 0.000 0.000 0.000 0.972 0.028
#> GSM228675 3 0.0713 0.8646 0.000 0.000 0.972 0.000 0.000 0.028
#> GSM228676 5 0.0713 0.9028 0.000 0.000 0.000 0.000 0.972 0.028
#> GSM228667 5 0.0713 0.9028 0.000 0.000 0.000 0.000 0.972 0.028
#> GSM228668 5 0.0713 0.9028 0.000 0.000 0.000 0.000 0.972 0.028
#> GSM228669 5 0.0000 0.9071 0.000 0.000 0.000 0.000 1.000 0.000
#> GSM228673 5 0.0713 0.9028 0.000 0.000 0.000 0.000 0.972 0.028
#> GSM228677 5 0.2697 0.7257 0.000 0.000 0.188 0.000 0.812 0.000
#> GSM228678 5 0.3841 0.3919 0.000 0.000 0.004 0.380 0.616 0.000
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
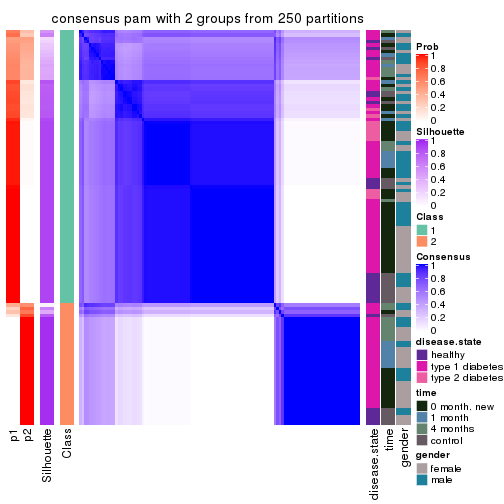
consensus_heatmap(res, k = 3)
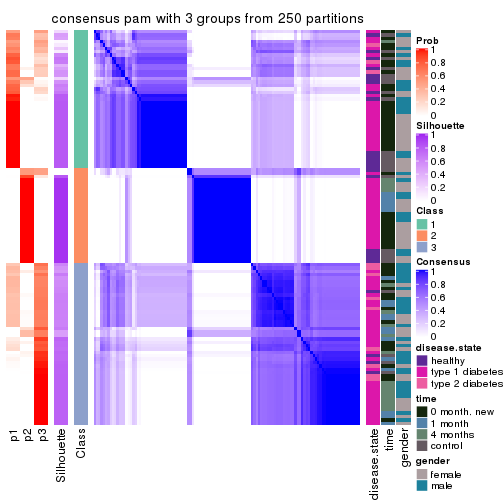
consensus_heatmap(res, k = 4)
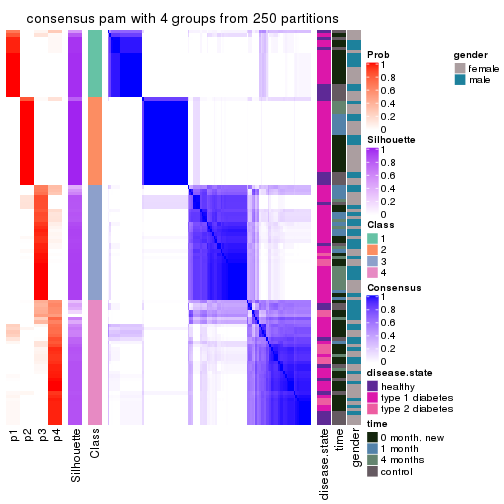
consensus_heatmap(res, k = 5)
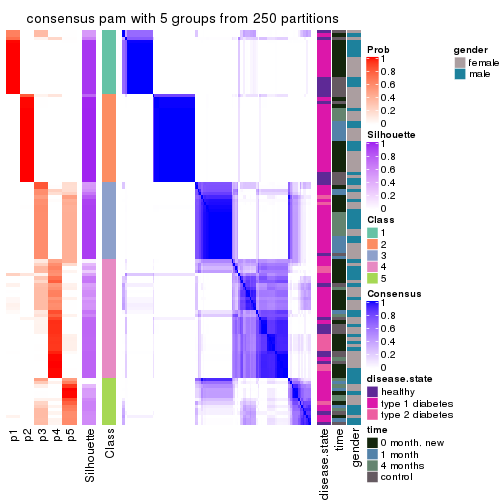
consensus_heatmap(res, k = 6)
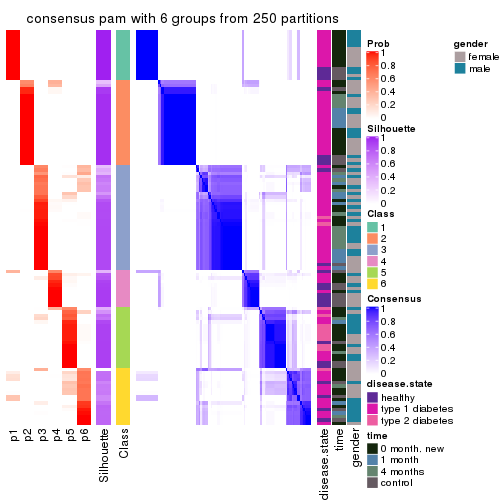
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
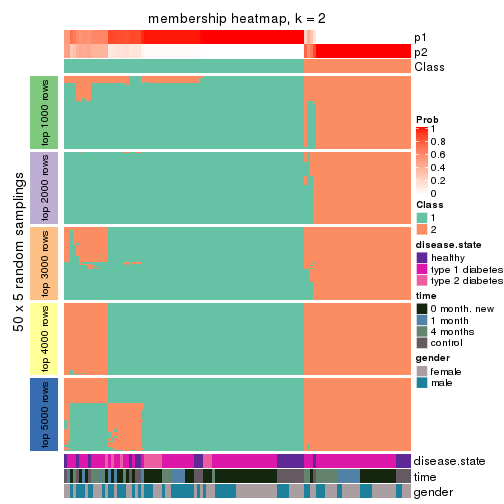
membership_heatmap(res, k = 3)
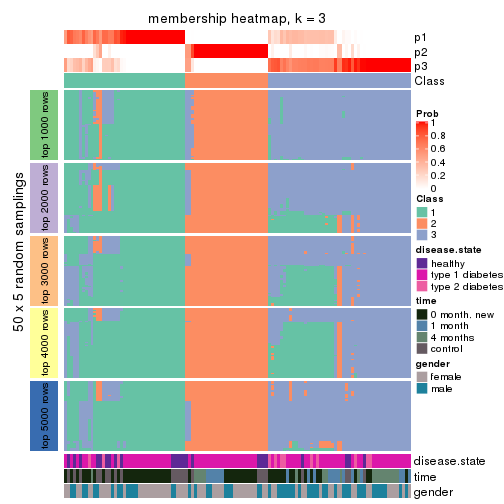
membership_heatmap(res, k = 4)
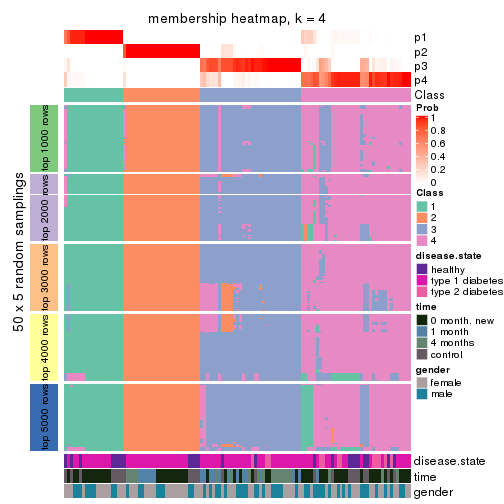
membership_heatmap(res, k = 5)
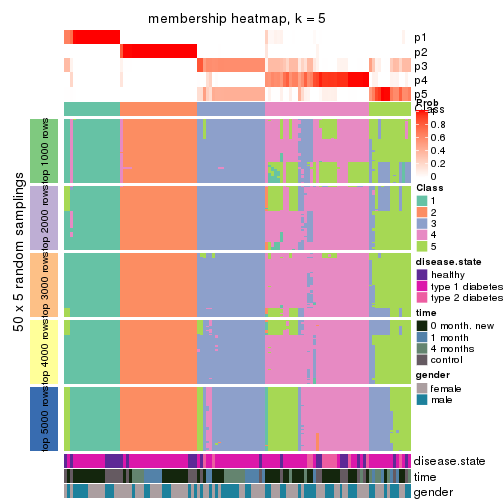
membership_heatmap(res, k = 6)
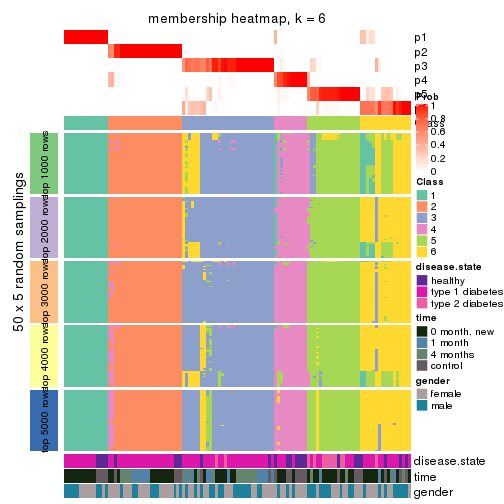
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
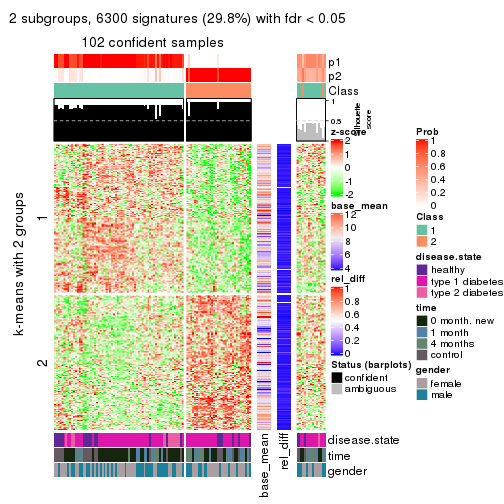
get_signatures(res, k = 3)
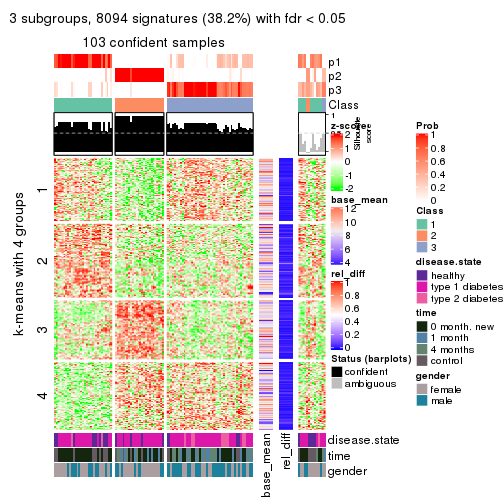
get_signatures(res, k = 4)
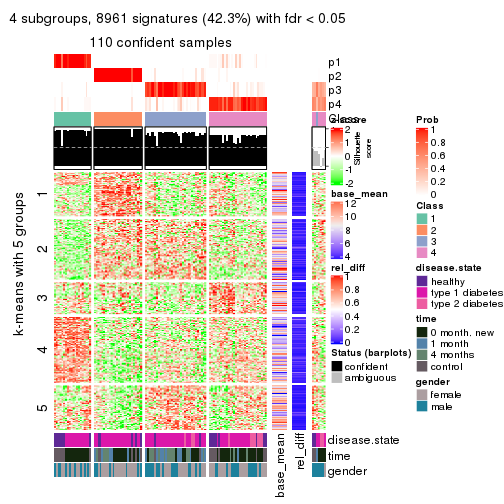
get_signatures(res, k = 5)
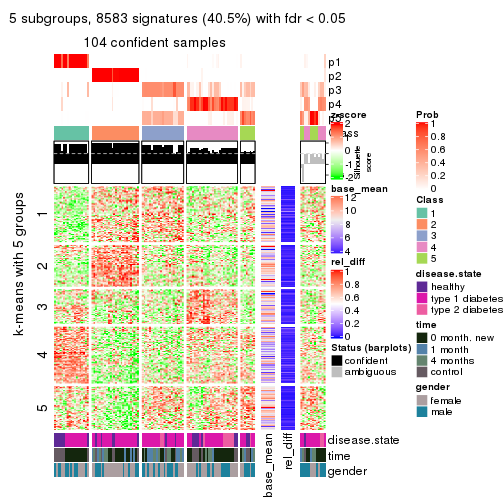
get_signatures(res, k = 6)
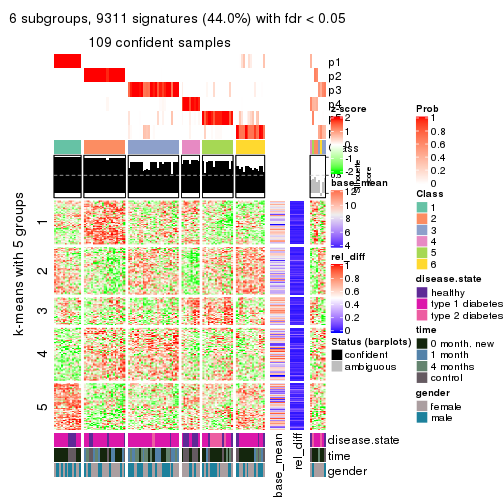
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
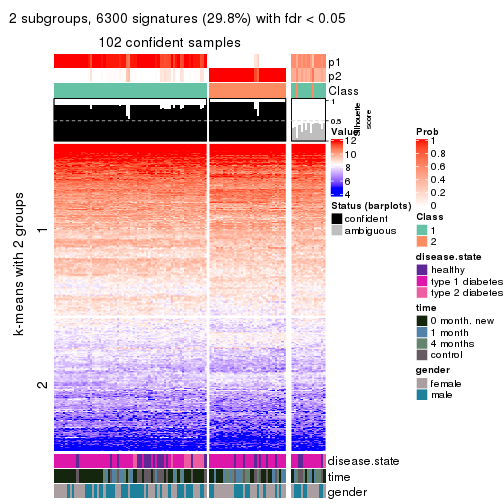
get_signatures(res, k = 3, scale_rows = FALSE)
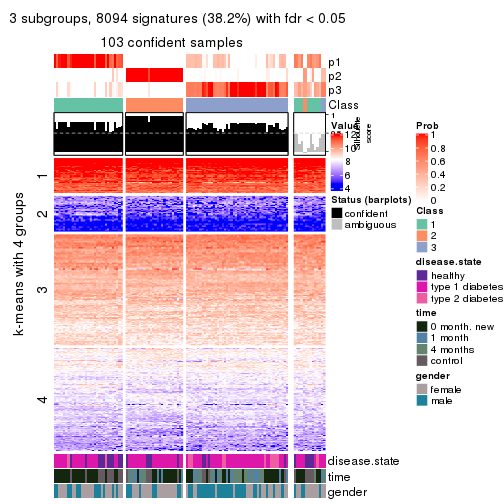
get_signatures(res, k = 4, scale_rows = FALSE)
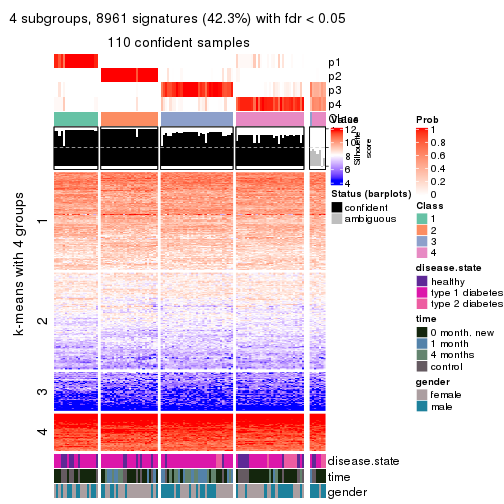
get_signatures(res, k = 5, scale_rows = FALSE)
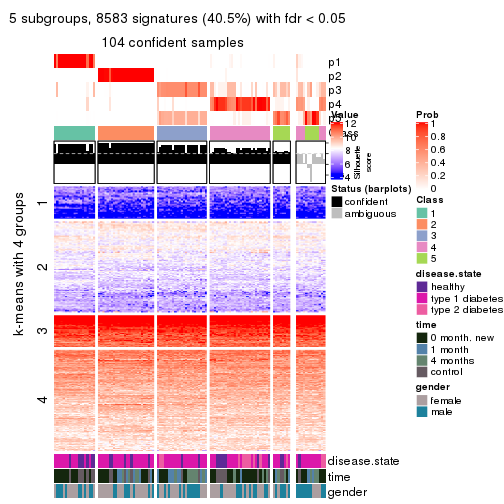
get_signatures(res, k = 6, scale_rows = FALSE)
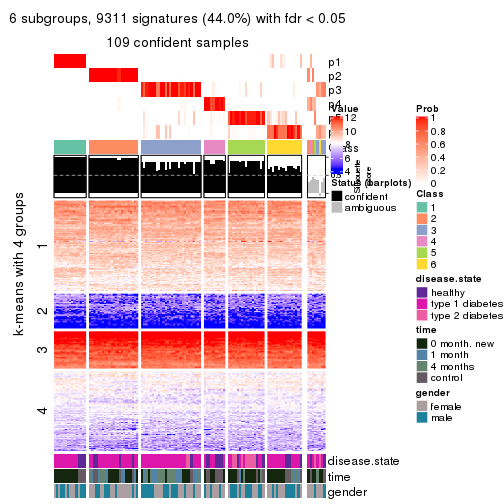
Compare the overlap of signatures from different k:
compare_signatures(res)
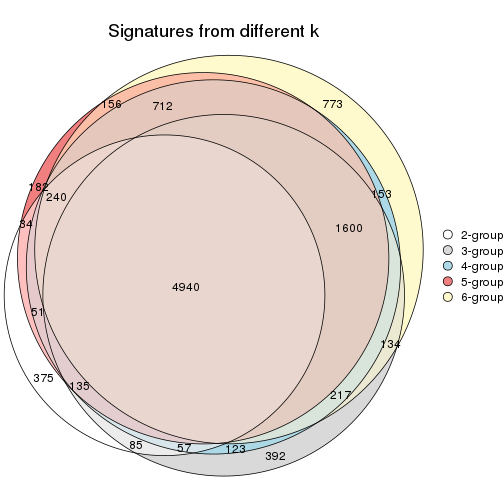
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
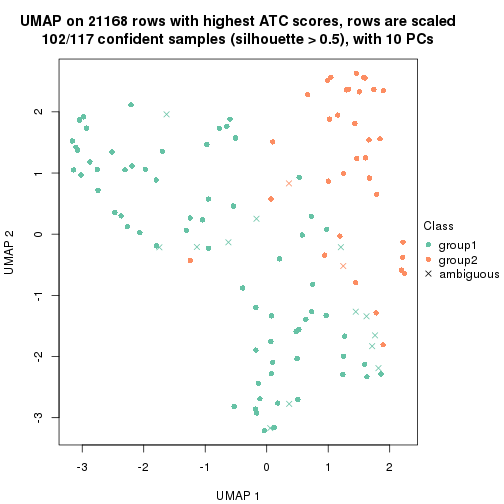
dimension_reduction(res, k = 3, method = "UMAP")
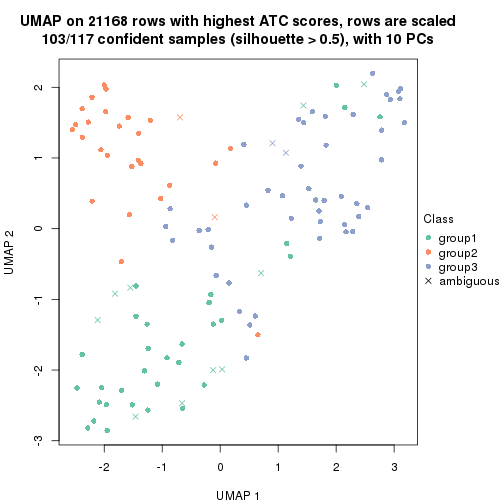
dimension_reduction(res, k = 4, method = "UMAP")
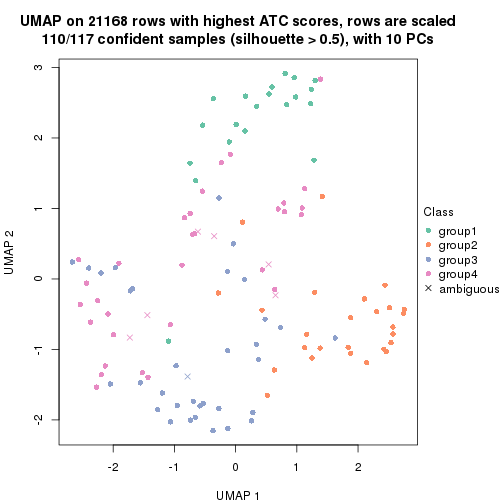
dimension_reduction(res, k = 5, method = "UMAP")
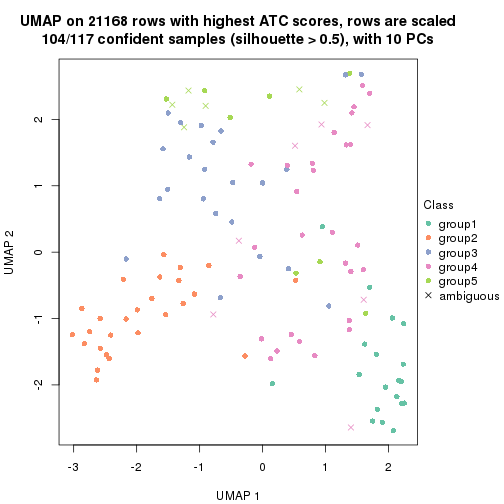
dimension_reduction(res, k = 6, method = "UMAP")
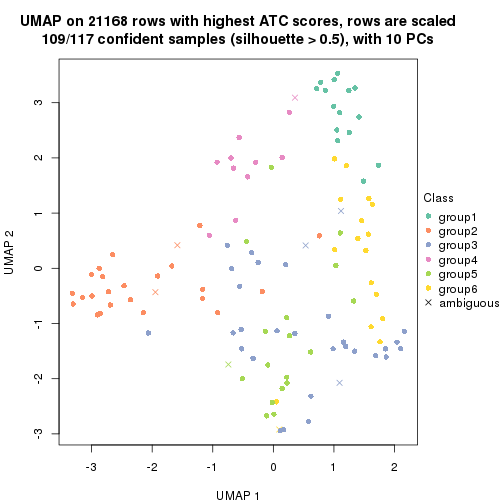
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
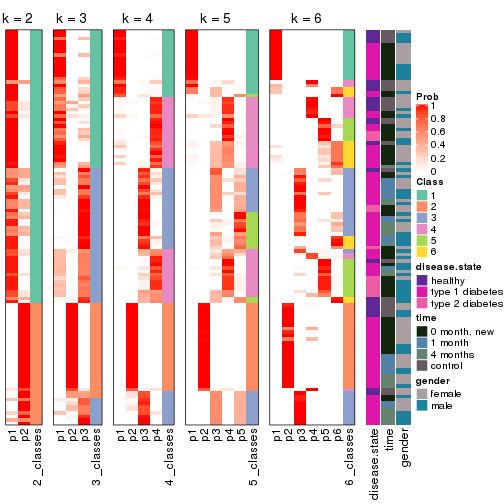
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> ATC:pam 102 2.85e-02 0.046186 0.2903 2
#> ATC:pam 103 1.10e-02 0.000196 0.0279 3
#> ATC:pam 110 3.60e-03 0.000025 0.5743 4
#> ATC:pam 104 2.09e-02 0.005051 0.4446 5
#> ATC:pam 109 1.64e-07 0.000214 0.6793 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["ATC", "mclust"]
# you can also extract it by
# res = res_list["ATC:mclust"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'ATC' method.
#> Subgroups are detected by 'mclust' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 4.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
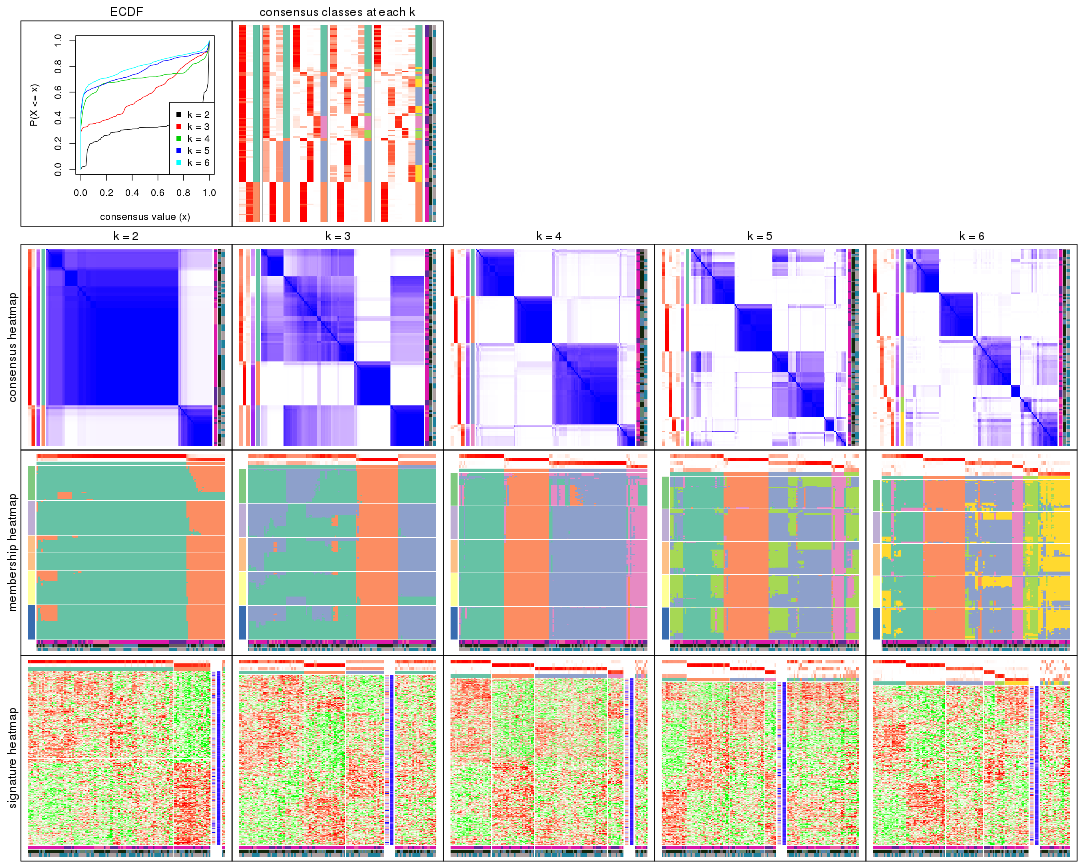
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
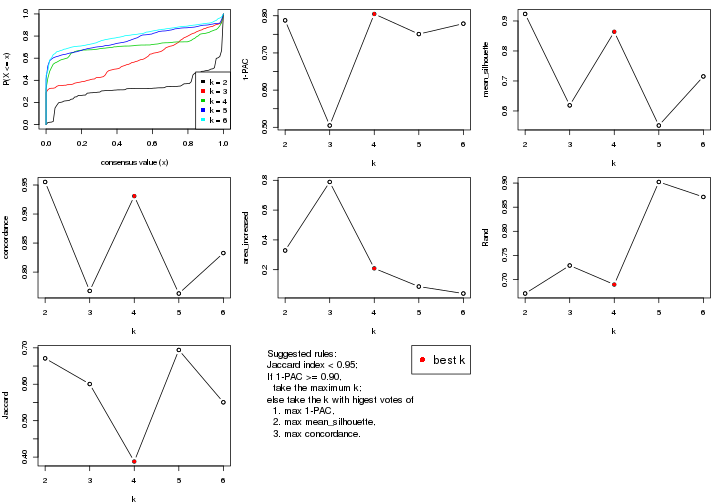
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.787 0.923 0.955 0.3287 0.671 0.671
#> 3 3 0.505 0.619 0.768 0.7889 0.729 0.601
#> 4 4 0.805 0.864 0.931 0.2087 0.690 0.388
#> 5 5 0.751 0.552 0.763 0.0866 0.902 0.694
#> 6 6 0.779 0.715 0.833 0.0398 0.871 0.550
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 4
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.0000 0.964 1.000 0.000
#> GSM228563 1 0.6801 0.779 0.820 0.180
#> GSM228565 1 0.0000 0.964 1.000 0.000
#> GSM228566 1 0.7883 0.689 0.764 0.236
#> GSM228567 1 0.2603 0.931 0.956 0.044
#> GSM228570 1 0.0938 0.957 0.988 0.012
#> GSM228571 1 0.0000 0.964 1.000 0.000
#> GSM228574 2 0.2778 0.943 0.048 0.952
#> GSM228575 2 0.3431 0.938 0.064 0.936
#> GSM228576 1 0.6801 0.779 0.820 0.180
#> GSM228579 1 0.0672 0.959 0.992 0.008
#> GSM228580 1 0.0000 0.964 1.000 0.000
#> GSM228581 1 0.0000 0.964 1.000 0.000
#> GSM228666 1 0.0000 0.964 1.000 0.000
#> GSM228564 1 0.0938 0.957 0.988 0.012
#> GSM228568 1 0.2603 0.931 0.956 0.044
#> GSM228569 1 0.0000 0.964 1.000 0.000
#> GSM228572 2 0.2948 0.942 0.052 0.948
#> GSM228573 1 0.4022 0.896 0.920 0.080
#> GSM228577 1 0.2603 0.931 0.956 0.044
#> GSM228578 1 0.0000 0.964 1.000 0.000
#> GSM228663 1 0.0000 0.964 1.000 0.000
#> GSM228664 1 0.0000 0.964 1.000 0.000
#> GSM228665 1 0.0000 0.964 1.000 0.000
#> GSM228582 1 0.0000 0.964 1.000 0.000
#> GSM228583 1 0.2603 0.931 0.956 0.044
#> GSM228585 1 0.2603 0.931 0.956 0.044
#> GSM228587 1 0.0000 0.964 1.000 0.000
#> GSM228588 1 0.0000 0.964 1.000 0.000
#> GSM228589 1 0.0000 0.964 1.000 0.000
#> GSM228590 1 0.0000 0.964 1.000 0.000
#> GSM228591 1 0.9170 0.482 0.668 0.332
#> GSM228597 1 0.6801 0.779 0.820 0.180
#> GSM228601 2 0.5629 0.893 0.132 0.868
#> GSM228604 2 0.2778 0.943 0.048 0.952
#> GSM228608 1 0.0000 0.964 1.000 0.000
#> GSM228609 2 0.6247 0.871 0.156 0.844
#> GSM228613 1 0.0672 0.959 0.992 0.008
#> GSM228616 2 0.2778 0.943 0.048 0.952
#> GSM228628 1 0.1184 0.954 0.984 0.016
#> GSM228634 1 0.0000 0.964 1.000 0.000
#> GSM228642 2 0.2778 0.943 0.048 0.952
#> GSM228645 2 0.3114 0.941 0.056 0.944
#> GSM228646 2 0.2778 0.943 0.048 0.952
#> GSM228652 1 0.0000 0.964 1.000 0.000
#> GSM228655 1 0.0000 0.964 1.000 0.000
#> GSM228656 1 0.2603 0.931 0.956 0.044
#> GSM228659 1 0.0000 0.964 1.000 0.000
#> GSM228662 1 0.2603 0.931 0.956 0.044
#> GSM228584 1 0.0672 0.959 0.992 0.008
#> GSM228586 1 0.2603 0.931 0.956 0.044
#> GSM228592 1 0.2603 0.931 0.956 0.044
#> GSM228593 1 0.6438 0.800 0.836 0.164
#> GSM228594 1 0.0000 0.964 1.000 0.000
#> GSM228598 1 0.0000 0.964 1.000 0.000
#> GSM228607 1 0.0000 0.964 1.000 0.000
#> GSM228612 2 0.6148 0.875 0.152 0.848
#> GSM228619 2 0.2778 0.943 0.048 0.952
#> GSM228622 1 0.0000 0.964 1.000 0.000
#> GSM228625 1 0.0000 0.964 1.000 0.000
#> GSM228631 2 0.2778 0.943 0.048 0.952
#> GSM228633 2 0.8909 0.642 0.308 0.692
#> GSM228637 1 0.0376 0.962 0.996 0.004
#> GSM228639 1 0.0000 0.964 1.000 0.000
#> GSM228649 1 0.0000 0.964 1.000 0.000
#> GSM228660 1 0.0000 0.964 1.000 0.000
#> GSM228661 1 0.0000 0.964 1.000 0.000
#> GSM228595 2 0.6801 0.843 0.180 0.820
#> GSM228599 2 0.2778 0.943 0.048 0.952
#> GSM228602 2 0.2778 0.943 0.048 0.952
#> GSM228614 1 0.0000 0.964 1.000 0.000
#> GSM228626 1 0.6801 0.779 0.820 0.180
#> GSM228640 2 0.3584 0.936 0.068 0.932
#> GSM228643 1 0.0000 0.964 1.000 0.000
#> GSM228650 1 0.0000 0.964 1.000 0.000
#> GSM228653 1 0.0000 0.964 1.000 0.000
#> GSM228657 1 0.6801 0.779 0.820 0.180
#> GSM228605 1 0.0000 0.964 1.000 0.000
#> GSM228610 1 0.0000 0.964 1.000 0.000
#> GSM228617 2 0.2778 0.943 0.048 0.952
#> GSM228620 1 0.0000 0.964 1.000 0.000
#> GSM228623 1 0.0938 0.957 0.988 0.012
#> GSM228629 2 0.9922 0.280 0.448 0.552
#> GSM228632 1 0.0000 0.964 1.000 0.000
#> GSM228635 1 0.0376 0.962 0.996 0.004
#> GSM228647 1 0.0000 0.964 1.000 0.000
#> GSM228596 1 0.0000 0.964 1.000 0.000
#> GSM228600 2 0.2778 0.943 0.048 0.952
#> GSM228603 2 0.2778 0.943 0.048 0.952
#> GSM228615 1 0.0376 0.962 0.996 0.004
#> GSM228627 1 0.0000 0.964 1.000 0.000
#> GSM228641 2 0.2778 0.943 0.048 0.952
#> GSM228644 1 0.6801 0.779 0.820 0.180
#> GSM228651 1 0.0000 0.964 1.000 0.000
#> GSM228654 1 0.0000 0.964 1.000 0.000
#> GSM228658 1 0.0000 0.964 1.000 0.000
#> GSM228606 1 0.1633 0.948 0.976 0.024
#> GSM228611 1 0.0000 0.964 1.000 0.000
#> GSM228618 2 0.4431 0.922 0.092 0.908
#> GSM228621 1 0.6801 0.779 0.820 0.180
#> GSM228624 1 0.0000 0.964 1.000 0.000
#> GSM228630 1 0.1184 0.954 0.984 0.016
#> GSM228636 1 0.6887 0.777 0.816 0.184
#> GSM228638 1 0.0000 0.964 1.000 0.000
#> GSM228648 1 0.0000 0.964 1.000 0.000
#> GSM228670 1 0.0000 0.964 1.000 0.000
#> GSM228671 1 0.0000 0.964 1.000 0.000
#> GSM228672 1 0.0376 0.962 0.996 0.004
#> GSM228674 1 0.0000 0.964 1.000 0.000
#> GSM228675 1 0.0000 0.964 1.000 0.000
#> GSM228676 1 0.0000 0.964 1.000 0.000
#> GSM228667 1 0.0000 0.964 1.000 0.000
#> GSM228668 1 0.0000 0.964 1.000 0.000
#> GSM228669 1 0.0000 0.964 1.000 0.000
#> GSM228673 1 0.0000 0.964 1.000 0.000
#> GSM228677 1 0.0000 0.964 1.000 0.000
#> GSM228678 1 0.5408 0.849 0.876 0.124
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.3192 0.618 0.888 0.000 0.112
#> GSM228563 1 0.6258 0.503 0.752 0.196 0.052
#> GSM228565 1 0.3116 0.618 0.892 0.000 0.108
#> GSM228566 2 0.5397 0.517 0.280 0.720 0.000
#> GSM228567 1 0.6244 0.503 0.560 0.000 0.440
#> GSM228570 1 0.3425 0.617 0.884 0.004 0.112
#> GSM228571 1 0.3192 0.618 0.888 0.000 0.112
#> GSM228574 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228575 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228576 1 0.5404 0.444 0.740 0.256 0.004
#> GSM228579 1 0.6062 0.529 0.616 0.000 0.384
#> GSM228580 1 0.0592 0.568 0.988 0.000 0.012
#> GSM228581 1 0.2625 0.608 0.916 0.000 0.084
#> GSM228666 1 0.4702 0.210 0.788 0.000 0.212
#> GSM228564 1 0.3192 0.618 0.888 0.000 0.112
#> GSM228568 1 0.6244 0.503 0.560 0.000 0.440
#> GSM228569 1 0.5465 0.570 0.712 0.000 0.288
#> GSM228572 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228573 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228577 1 0.6244 0.503 0.560 0.000 0.440
#> GSM228578 1 0.2878 0.604 0.904 0.000 0.096
#> GSM228663 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228664 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228665 1 0.6291 -0.797 0.532 0.000 0.468
#> GSM228582 1 0.2878 0.569 0.904 0.000 0.096
#> GSM228583 1 0.6244 0.503 0.560 0.000 0.440
#> GSM228585 1 0.6244 0.503 0.560 0.000 0.440
#> GSM228587 1 0.3116 0.618 0.892 0.000 0.108
#> GSM228588 1 0.3116 0.618 0.892 0.000 0.108
#> GSM228589 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228590 1 0.3551 0.617 0.868 0.000 0.132
#> GSM228591 2 0.5932 0.685 0.164 0.780 0.056
#> GSM228597 1 0.5619 0.451 0.744 0.244 0.012
#> GSM228601 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228604 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228608 1 0.5431 0.572 0.716 0.000 0.284
#> GSM228609 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228613 1 0.6235 0.505 0.564 0.000 0.436
#> GSM228616 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228628 1 0.6252 -0.714 0.556 0.000 0.444
#> GSM228634 1 0.5254 0.577 0.736 0.000 0.264
#> GSM228642 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228645 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228646 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228652 1 0.4062 0.591 0.836 0.000 0.164
#> GSM228655 1 0.3941 0.593 0.844 0.000 0.156
#> GSM228656 1 0.6244 0.503 0.560 0.000 0.440
#> GSM228659 1 0.3116 0.618 0.892 0.000 0.108
#> GSM228662 1 0.6244 0.503 0.560 0.000 0.440
#> GSM228584 1 0.6235 0.505 0.564 0.000 0.436
#> GSM228586 1 0.6244 0.503 0.560 0.000 0.440
#> GSM228592 1 0.6244 0.503 0.560 0.000 0.440
#> GSM228593 1 0.6184 0.567 0.780 0.112 0.108
#> GSM228594 1 0.3116 0.618 0.892 0.000 0.108
#> GSM228598 1 0.2878 0.607 0.904 0.000 0.096
#> GSM228607 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228612 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228619 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228622 1 0.3686 0.409 0.860 0.000 0.140
#> GSM228625 1 0.6026 -0.488 0.624 0.000 0.376
#> GSM228631 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228633 2 0.0892 0.953 0.020 0.980 0.000
#> GSM228637 1 0.1031 0.591 0.976 0.000 0.024
#> GSM228639 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228649 1 0.3816 0.421 0.852 0.000 0.148
#> GSM228660 1 0.3192 0.461 0.888 0.000 0.112
#> GSM228661 1 0.4750 0.584 0.784 0.000 0.216
#> GSM228595 2 0.0237 0.969 0.004 0.996 0.000
#> GSM228599 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228602 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228614 1 0.5810 -0.335 0.664 0.000 0.336
#> GSM228626 3 0.6291 0.939 0.468 0.000 0.532
#> GSM228640 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228643 1 0.5678 -0.251 0.684 0.000 0.316
#> GSM228650 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228653 3 0.6305 0.910 0.484 0.000 0.516
#> GSM228657 1 0.6168 -0.612 0.588 0.000 0.412
#> GSM228605 1 0.1289 0.552 0.968 0.000 0.032
#> GSM228610 3 0.6252 0.983 0.444 0.000 0.556
#> GSM228617 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228620 1 0.4796 0.181 0.780 0.000 0.220
#> GSM228623 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228629 2 0.1860 0.916 0.052 0.948 0.000
#> GSM228632 1 0.3941 0.372 0.844 0.000 0.156
#> GSM228635 1 0.4399 0.295 0.812 0.000 0.188
#> GSM228647 1 0.6192 -0.644 0.580 0.000 0.420
#> GSM228596 1 0.1643 0.541 0.956 0.000 0.044
#> GSM228600 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228603 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228615 1 0.3686 0.409 0.860 0.000 0.140
#> GSM228627 3 0.6305 0.906 0.484 0.000 0.516
#> GSM228641 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228644 1 0.5733 -0.285 0.676 0.000 0.324
#> GSM228651 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228654 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228658 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228606 1 0.6386 -0.612 0.584 0.004 0.412
#> GSM228611 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228618 2 0.0000 0.972 0.000 1.000 0.000
#> GSM228621 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228624 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228630 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228636 1 0.4902 0.447 0.844 0.064 0.092
#> GSM228638 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228648 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228670 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228671 3 0.6244 0.988 0.440 0.000 0.560
#> GSM228672 1 0.3038 0.618 0.896 0.000 0.104
#> GSM228674 1 0.2711 0.492 0.912 0.000 0.088
#> GSM228675 3 0.6267 0.970 0.452 0.000 0.548
#> GSM228676 1 0.3752 0.400 0.856 0.000 0.144
#> GSM228667 1 0.3686 0.409 0.860 0.000 0.140
#> GSM228668 1 0.2066 0.525 0.940 0.000 0.060
#> GSM228669 1 0.0747 0.582 0.984 0.000 0.016
#> GSM228673 1 0.3267 0.451 0.884 0.000 0.116
#> GSM228677 1 0.5760 -0.301 0.672 0.000 0.328
#> GSM228678 1 0.4830 0.462 0.848 0.068 0.084
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.3142 0.797 0.132 0.000 0.008 0.860
#> GSM228563 4 0.3142 0.797 0.132 0.000 0.008 0.860
#> GSM228565 4 0.3545 0.777 0.164 0.000 0.008 0.828
#> GSM228566 2 0.0188 0.968 0.000 0.996 0.004 0.000
#> GSM228567 1 0.0000 0.933 1.000 0.000 0.000 0.000
#> GSM228570 4 0.3326 0.795 0.132 0.004 0.008 0.856
#> GSM228571 1 0.6077 0.373 0.644 0.020 0.300 0.036
#> GSM228574 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228575 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228576 2 0.2256 0.901 0.000 0.924 0.020 0.056
#> GSM228579 1 0.0000 0.933 1.000 0.000 0.000 0.000
#> GSM228580 1 0.6356 0.407 0.604 0.000 0.088 0.308
#> GSM228581 1 0.3813 0.767 0.828 0.000 0.024 0.148
#> GSM228666 3 0.0895 0.916 0.004 0.000 0.976 0.020
#> GSM228564 4 0.3142 0.797 0.132 0.000 0.008 0.860
#> GSM228568 1 0.0000 0.933 1.000 0.000 0.000 0.000
#> GSM228569 1 0.0469 0.931 0.988 0.000 0.012 0.000
#> GSM228572 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228573 3 0.1854 0.900 0.000 0.048 0.940 0.012
#> GSM228577 1 0.0000 0.933 1.000 0.000 0.000 0.000
#> GSM228578 1 0.0817 0.924 0.976 0.000 0.024 0.000
#> GSM228663 3 0.1716 0.893 0.000 0.064 0.936 0.000
#> GSM228664 3 0.0469 0.916 0.000 0.000 0.988 0.012
#> GSM228665 3 0.0000 0.920 0.000 0.000 1.000 0.000
#> GSM228582 1 0.3569 0.702 0.804 0.000 0.196 0.000
#> GSM228583 1 0.0000 0.933 1.000 0.000 0.000 0.000
#> GSM228585 1 0.0000 0.933 1.000 0.000 0.000 0.000
#> GSM228587 1 0.0804 0.925 0.980 0.000 0.008 0.012
#> GSM228588 4 0.3142 0.797 0.132 0.000 0.008 0.860
#> GSM228589 3 0.3217 0.834 0.000 0.128 0.860 0.012
#> GSM228590 1 0.0336 0.931 0.992 0.000 0.008 0.000
#> GSM228591 2 0.0188 0.968 0.000 0.996 0.004 0.000
#> GSM228597 2 0.7740 0.357 0.068 0.588 0.104 0.240
#> GSM228601 2 0.0188 0.969 0.000 0.996 0.000 0.004
#> GSM228604 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228608 1 0.0469 0.931 0.988 0.000 0.012 0.000
#> GSM228609 2 0.0592 0.957 0.000 0.984 0.000 0.016
#> GSM228613 1 0.0000 0.933 1.000 0.000 0.000 0.000
#> GSM228616 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228628 3 0.1452 0.908 0.000 0.036 0.956 0.008
#> GSM228634 1 0.0469 0.931 0.988 0.000 0.012 0.000
#> GSM228642 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228645 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228646 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228652 1 0.0817 0.924 0.976 0.000 0.024 0.000
#> GSM228655 1 0.1022 0.916 0.968 0.000 0.032 0.000
#> GSM228656 1 0.0000 0.933 1.000 0.000 0.000 0.000
#> GSM228659 1 0.2730 0.847 0.896 0.000 0.016 0.088
#> GSM228662 1 0.0000 0.933 1.000 0.000 0.000 0.000
#> GSM228584 1 0.0000 0.933 1.000 0.000 0.000 0.000
#> GSM228586 1 0.0000 0.933 1.000 0.000 0.000 0.000
#> GSM228592 1 0.0000 0.933 1.000 0.000 0.000 0.000
#> GSM228593 4 0.7393 0.270 0.132 0.376 0.008 0.484
#> GSM228594 1 0.0336 0.931 0.992 0.000 0.008 0.000
#> GSM228598 1 0.0707 0.927 0.980 0.000 0.020 0.000
#> GSM228607 3 0.0804 0.916 0.000 0.008 0.980 0.012
#> GSM228612 2 0.0188 0.968 0.000 0.996 0.004 0.000
#> GSM228619 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228622 3 0.2760 0.826 0.128 0.000 0.872 0.000
#> GSM228625 3 0.2197 0.881 0.000 0.080 0.916 0.004
#> GSM228631 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228633 2 0.3554 0.784 0.000 0.844 0.136 0.020
#> GSM228637 4 0.1174 0.781 0.012 0.000 0.020 0.968
#> GSM228639 3 0.0000 0.920 0.000 0.000 1.000 0.000
#> GSM228649 3 0.3803 0.804 0.132 0.000 0.836 0.032
#> GSM228660 3 0.1940 0.879 0.076 0.000 0.924 0.000
#> GSM228661 1 0.0469 0.931 0.988 0.000 0.012 0.000
#> GSM228595 2 0.0895 0.949 0.000 0.976 0.004 0.020
#> GSM228599 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228602 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228614 3 0.0817 0.916 0.000 0.000 0.976 0.024
#> GSM228626 3 0.0817 0.916 0.000 0.000 0.976 0.024
#> GSM228640 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228643 3 0.1022 0.915 0.000 0.000 0.968 0.032
#> GSM228650 3 0.0188 0.919 0.000 0.000 0.996 0.004
#> GSM228653 3 0.0000 0.920 0.000 0.000 1.000 0.000
#> GSM228657 3 0.0817 0.916 0.000 0.000 0.976 0.024
#> GSM228605 3 0.5271 0.483 0.340 0.000 0.640 0.020
#> GSM228610 3 0.0000 0.920 0.000 0.000 1.000 0.000
#> GSM228617 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228620 3 0.1118 0.904 0.036 0.000 0.964 0.000
#> GSM228623 3 0.0469 0.916 0.000 0.000 0.988 0.012
#> GSM228629 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228632 3 0.2868 0.852 0.000 0.000 0.864 0.136
#> GSM228635 3 0.4356 0.660 0.000 0.000 0.708 0.292
#> GSM228647 3 0.0188 0.920 0.000 0.000 0.996 0.004
#> GSM228596 3 0.7201 0.238 0.356 0.000 0.496 0.148
#> GSM228600 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228603 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228615 4 0.4888 0.183 0.000 0.000 0.412 0.588
#> GSM228627 3 0.0000 0.920 0.000 0.000 1.000 0.000
#> GSM228641 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228644 3 0.1211 0.912 0.000 0.000 0.960 0.040
#> GSM228651 3 0.0188 0.919 0.000 0.000 0.996 0.004
#> GSM228654 3 0.0000 0.920 0.000 0.000 1.000 0.000
#> GSM228658 3 0.0000 0.920 0.000 0.000 1.000 0.000
#> GSM228606 3 0.2021 0.905 0.000 0.040 0.936 0.024
#> GSM228611 3 0.0000 0.920 0.000 0.000 1.000 0.000
#> GSM228618 2 0.0000 0.971 0.000 1.000 0.000 0.000
#> GSM228621 3 0.3161 0.838 0.000 0.124 0.864 0.012
#> GSM228624 3 0.3217 0.834 0.000 0.128 0.860 0.012
#> GSM228630 3 0.0188 0.919 0.000 0.000 0.996 0.004
#> GSM228636 4 0.3074 0.714 0.000 0.000 0.152 0.848
#> GSM228638 3 0.0000 0.920 0.000 0.000 1.000 0.000
#> GSM228648 3 0.0000 0.920 0.000 0.000 1.000 0.000
#> GSM228670 3 0.0000 0.920 0.000 0.000 1.000 0.000
#> GSM228671 3 0.0000 0.920 0.000 0.000 1.000 0.000
#> GSM228672 4 0.0779 0.780 0.004 0.000 0.016 0.980
#> GSM228674 3 0.4149 0.817 0.036 0.000 0.812 0.152
#> GSM228675 3 0.0000 0.920 0.000 0.000 1.000 0.000
#> GSM228676 3 0.3913 0.827 0.028 0.000 0.824 0.148
#> GSM228667 3 0.3024 0.845 0.000 0.000 0.852 0.148
#> GSM228668 3 0.3907 0.838 0.044 0.000 0.836 0.120
#> GSM228669 4 0.6476 0.461 0.272 0.000 0.112 0.616
#> GSM228673 3 0.4008 0.824 0.032 0.000 0.820 0.148
#> GSM228677 3 0.0817 0.916 0.000 0.000 0.976 0.024
#> GSM228678 4 0.1677 0.784 0.012 0.000 0.040 0.948
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 4 0.0404 0.8516 0.012 0.000 0.000 0.988 0.000
#> GSM228563 4 0.0404 0.8516 0.012 0.000 0.000 0.988 0.000
#> GSM228565 4 0.0703 0.8394 0.024 0.000 0.000 0.976 0.000
#> GSM228566 2 0.0162 0.9573 0.004 0.996 0.000 0.000 0.000
#> GSM228567 1 0.4425 0.5591 0.544 0.000 0.000 0.004 0.452
#> GSM228570 4 0.0404 0.8516 0.012 0.000 0.000 0.988 0.000
#> GSM228571 5 0.7256 -0.4910 0.404 0.012 0.028 0.148 0.408
#> GSM228574 2 0.0290 0.9580 0.000 0.992 0.000 0.008 0.000
#> GSM228575 2 0.0404 0.9571 0.000 0.988 0.000 0.012 0.000
#> GSM228576 2 0.3270 0.8148 0.004 0.852 0.044 0.100 0.000
#> GSM228579 1 0.4723 0.5553 0.536 0.000 0.000 0.016 0.448
#> GSM228580 5 0.4297 0.3901 0.472 0.000 0.000 0.000 0.528
#> GSM228581 1 0.1992 0.3647 0.924 0.000 0.032 0.000 0.044
#> GSM228666 3 0.4354 0.4097 0.032 0.000 0.712 0.000 0.256
#> GSM228564 4 0.0404 0.8516 0.012 0.000 0.000 0.988 0.000
#> GSM228568 1 0.4425 0.5591 0.544 0.000 0.000 0.004 0.452
#> GSM228569 1 0.4273 0.5580 0.552 0.000 0.000 0.000 0.448
#> GSM228572 2 0.0290 0.9580 0.000 0.992 0.000 0.008 0.000
#> GSM228573 3 0.0451 0.6877 0.004 0.000 0.988 0.008 0.000
#> GSM228577 1 0.4425 0.5591 0.544 0.000 0.000 0.004 0.452
#> GSM228578 1 0.1965 0.3015 0.924 0.000 0.024 0.000 0.052
#> GSM228663 3 0.1628 0.6798 0.056 0.000 0.936 0.008 0.000
#> GSM228664 3 0.0290 0.6883 0.000 0.000 0.992 0.008 0.000
#> GSM228665 3 0.5467 0.4593 0.412 0.000 0.524 0.000 0.064
#> GSM228582 1 0.3551 0.1781 0.772 0.000 0.220 0.000 0.008
#> GSM228583 1 0.4425 0.5591 0.544 0.000 0.000 0.004 0.452
#> GSM228585 1 0.4727 0.5558 0.532 0.000 0.000 0.016 0.452
#> GSM228587 1 0.6219 0.4584 0.436 0.000 0.000 0.140 0.424
#> GSM228588 4 0.0404 0.8516 0.012 0.000 0.000 0.988 0.000
#> GSM228589 3 0.0290 0.6883 0.000 0.000 0.992 0.008 0.000
#> GSM228590 1 0.4723 0.5553 0.536 0.000 0.000 0.016 0.448
#> GSM228591 2 0.4425 0.3868 0.000 0.600 0.392 0.008 0.000
#> GSM228597 2 0.5147 0.6236 0.016 0.720 0.188 0.072 0.004
#> GSM228601 2 0.0290 0.9580 0.000 0.992 0.000 0.008 0.000
#> GSM228604 2 0.0000 0.9599 0.000 1.000 0.000 0.000 0.000
#> GSM228608 1 0.2891 0.4625 0.824 0.000 0.000 0.000 0.176
#> GSM228609 2 0.0000 0.9599 0.000 1.000 0.000 0.000 0.000
#> GSM228613 1 0.4420 0.5589 0.548 0.000 0.000 0.004 0.448
#> GSM228616 2 0.0000 0.9599 0.000 1.000 0.000 0.000 0.000
#> GSM228628 3 0.0613 0.6865 0.004 0.004 0.984 0.008 0.000
#> GSM228634 1 0.4171 0.5448 0.604 0.000 0.000 0.000 0.396
#> GSM228642 2 0.0000 0.9599 0.000 1.000 0.000 0.000 0.000
#> GSM228645 2 0.0000 0.9599 0.000 1.000 0.000 0.000 0.000
#> GSM228646 2 0.0000 0.9599 0.000 1.000 0.000 0.000 0.000
#> GSM228652 1 0.0955 0.3519 0.968 0.000 0.028 0.000 0.004
#> GSM228655 1 0.1197 0.3336 0.952 0.000 0.048 0.000 0.000
#> GSM228656 1 0.4425 0.5591 0.544 0.000 0.000 0.004 0.452
#> GSM228659 1 0.4021 0.2477 0.764 0.000 0.000 0.200 0.036
#> GSM228662 1 0.4727 0.5558 0.532 0.000 0.000 0.016 0.452
#> GSM228584 1 0.4727 0.5558 0.532 0.000 0.000 0.016 0.452
#> GSM228586 1 0.4425 0.5591 0.544 0.000 0.000 0.004 0.452
#> GSM228592 1 0.4425 0.5591 0.544 0.000 0.000 0.004 0.452
#> GSM228593 4 0.4453 0.5755 0.004 0.184 0.000 0.752 0.060
#> GSM228594 1 0.5061 0.5510 0.528 0.008 0.000 0.020 0.444
#> GSM228598 1 0.1732 0.4151 0.920 0.000 0.000 0.000 0.080
#> GSM228607 3 0.0290 0.6883 0.000 0.000 0.992 0.008 0.000
#> GSM228612 2 0.0404 0.9571 0.000 0.988 0.000 0.012 0.000
#> GSM228619 2 0.0000 0.9599 0.000 1.000 0.000 0.000 0.000
#> GSM228622 1 0.5508 -0.4221 0.476 0.000 0.460 0.000 0.064
#> GSM228625 3 0.0613 0.6865 0.004 0.004 0.984 0.008 0.000
#> GSM228631 2 0.0000 0.9599 0.000 1.000 0.000 0.000 0.000
#> GSM228633 2 0.0693 0.9450 0.008 0.980 0.012 0.000 0.000
#> GSM228637 5 0.4713 0.0299 0.016 0.000 0.000 0.440 0.544
#> GSM228639 3 0.5229 0.5159 0.324 0.000 0.612 0.000 0.064
#> GSM228649 3 0.5692 0.3538 0.016 0.208 0.680 0.084 0.012
#> GSM228660 3 0.5504 0.4277 0.448 0.000 0.488 0.000 0.064
#> GSM228661 1 0.2124 0.4237 0.900 0.000 0.004 0.000 0.096
#> GSM228595 2 0.0290 0.9580 0.000 0.992 0.000 0.008 0.000
#> GSM228599 2 0.0000 0.9599 0.000 1.000 0.000 0.000 0.000
#> GSM228602 2 0.0000 0.9599 0.000 1.000 0.000 0.000 0.000
#> GSM228614 3 0.4430 0.1625 0.012 0.000 0.628 0.000 0.360
#> GSM228626 3 0.0290 0.6891 0.008 0.000 0.992 0.000 0.000
#> GSM228640 2 0.0290 0.9580 0.000 0.992 0.000 0.008 0.000
#> GSM228643 3 0.4086 0.3318 0.012 0.000 0.704 0.000 0.284
#> GSM228650 3 0.0162 0.6899 0.004 0.000 0.996 0.000 0.000
#> GSM228653 3 0.5461 0.4634 0.408 0.000 0.528 0.000 0.064
#> GSM228657 3 0.0290 0.6891 0.008 0.000 0.992 0.000 0.000
#> GSM228605 1 0.4708 0.0982 0.712 0.000 0.220 0.000 0.068
#> GSM228610 3 0.5461 0.4634 0.408 0.000 0.528 0.000 0.064
#> GSM228617 2 0.0000 0.9599 0.000 1.000 0.000 0.000 0.000
#> GSM228620 3 0.5495 0.4357 0.436 0.000 0.500 0.000 0.064
#> GSM228623 3 0.0290 0.6883 0.000 0.000 0.992 0.008 0.000
#> GSM228629 2 0.0162 0.9590 0.000 0.996 0.000 0.004 0.000
#> GSM228632 3 0.5603 0.4071 0.452 0.000 0.476 0.000 0.072
#> GSM228635 5 0.6190 0.2321 0.012 0.000 0.416 0.096 0.476
#> GSM228647 3 0.4233 0.5961 0.208 0.000 0.748 0.000 0.044
#> GSM228596 1 0.5783 -0.2713 0.540 0.000 0.360 0.000 0.100
#> GSM228600 2 0.0000 0.9599 0.000 1.000 0.000 0.000 0.000
#> GSM228603 2 0.0162 0.9590 0.000 0.996 0.000 0.004 0.000
#> GSM228615 5 0.6176 0.3353 0.184 0.000 0.000 0.268 0.548
#> GSM228627 3 0.5461 0.4634 0.408 0.000 0.528 0.000 0.064
#> GSM228641 2 0.0290 0.9580 0.000 0.992 0.000 0.008 0.000
#> GSM228644 3 0.4354 0.1566 0.008 0.000 0.624 0.000 0.368
#> GSM228651 3 0.0162 0.6899 0.004 0.000 0.996 0.000 0.000
#> GSM228654 3 0.2863 0.6570 0.064 0.000 0.876 0.000 0.060
#> GSM228658 3 0.5461 0.4634 0.408 0.000 0.528 0.000 0.064
#> GSM228606 3 0.2722 0.5784 0.004 0.120 0.868 0.008 0.000
#> GSM228611 3 0.3201 0.6465 0.096 0.000 0.852 0.000 0.052
#> GSM228618 2 0.0162 0.9590 0.000 0.996 0.000 0.004 0.000
#> GSM228621 3 0.0290 0.6883 0.000 0.000 0.992 0.008 0.000
#> GSM228624 3 0.0290 0.6883 0.000 0.000 0.992 0.008 0.000
#> GSM228630 3 0.0162 0.6899 0.004 0.000 0.996 0.000 0.000
#> GSM228636 5 0.6761 0.2011 0.012 0.000 0.196 0.312 0.480
#> GSM228638 3 0.5461 0.4634 0.408 0.000 0.528 0.000 0.064
#> GSM228648 3 0.0162 0.6899 0.004 0.000 0.996 0.000 0.000
#> GSM228670 3 0.5440 0.4723 0.396 0.000 0.540 0.000 0.064
#> GSM228671 3 0.0162 0.6899 0.004 0.000 0.996 0.000 0.000
#> GSM228672 4 0.4560 -0.0548 0.008 0.000 0.000 0.508 0.484
#> GSM228674 5 0.4278 0.3936 0.452 0.000 0.000 0.000 0.548
#> GSM228675 3 0.5440 0.4723 0.396 0.000 0.540 0.000 0.064
#> GSM228676 1 0.5779 -0.4369 0.456 0.000 0.456 0.000 0.088
#> GSM228667 1 0.6102 -0.3845 0.440 0.000 0.124 0.000 0.436
#> GSM228668 1 0.5736 -0.4220 0.468 0.000 0.448 0.000 0.084
#> GSM228669 5 0.6478 0.3953 0.300 0.000 0.008 0.172 0.520
#> GSM228673 1 0.6001 -0.3813 0.456 0.000 0.112 0.000 0.432
#> GSM228677 3 0.4387 0.1909 0.012 0.000 0.640 0.000 0.348
#> GSM228678 5 0.6542 0.1253 0.012 0.000 0.140 0.388 0.460
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 4 0.1320 0.9867 0.036 0.000 0.016 0.948 0.000 0.000
#> GSM228563 4 0.1320 0.9867 0.036 0.000 0.016 0.948 0.000 0.000
#> GSM228565 4 0.2263 0.9184 0.100 0.000 0.016 0.884 0.000 0.000
#> GSM228566 2 0.0603 0.9548 0.000 0.980 0.016 0.004 0.000 0.000
#> GSM228567 1 0.0000 0.8046 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228570 4 0.1320 0.9867 0.036 0.000 0.016 0.948 0.000 0.000
#> GSM228571 1 0.4810 0.1211 0.552 0.024 0.020 0.404 0.000 0.000
#> GSM228574 2 0.0937 0.9536 0.000 0.960 0.040 0.000 0.000 0.000
#> GSM228575 2 0.1320 0.9513 0.000 0.948 0.036 0.016 0.000 0.000
#> GSM228576 2 0.4052 0.4019 0.000 0.628 0.016 0.356 0.000 0.000
#> GSM228579 1 0.0000 0.8046 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228580 5 0.2263 0.7437 0.000 0.000 0.016 0.000 0.884 0.100
#> GSM228581 1 0.5812 0.3818 0.476 0.000 0.016 0.000 0.120 0.388
#> GSM228666 3 0.5024 0.7173 0.000 0.000 0.680 0.036 0.072 0.212
#> GSM228564 4 0.1320 0.9867 0.036 0.000 0.016 0.948 0.000 0.000
#> GSM228568 1 0.0000 0.8046 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228569 1 0.2515 0.7776 0.888 0.000 0.000 0.024 0.016 0.072
#> GSM228572 2 0.0937 0.9536 0.000 0.960 0.040 0.000 0.000 0.000
#> GSM228573 3 0.2762 0.7593 0.000 0.000 0.804 0.000 0.000 0.196
#> GSM228577 1 0.0000 0.8046 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228578 1 0.5032 0.4955 0.556 0.000 0.016 0.036 0.004 0.388
#> GSM228663 3 0.3847 0.2546 0.000 0.000 0.544 0.000 0.000 0.456
#> GSM228664 3 0.2823 0.7576 0.000 0.000 0.796 0.000 0.000 0.204
#> GSM228665 6 0.1663 0.7783 0.000 0.000 0.088 0.000 0.000 0.912
#> GSM228582 6 0.4854 0.2016 0.304 0.000 0.004 0.036 0.020 0.636
#> GSM228583 1 0.0000 0.8046 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228585 1 0.0000 0.8046 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228587 1 0.4026 0.2951 0.636 0.000 0.016 0.348 0.000 0.000
#> GSM228588 4 0.1320 0.9867 0.036 0.000 0.016 0.948 0.000 0.000
#> GSM228589 3 0.2823 0.7563 0.000 0.000 0.796 0.000 0.000 0.204
#> GSM228590 1 0.0458 0.7987 0.984 0.000 0.016 0.000 0.000 0.000
#> GSM228591 3 0.4141 0.2015 0.000 0.432 0.556 0.012 0.000 0.000
#> GSM228597 3 0.5452 0.0537 0.000 0.436 0.444 0.120 0.000 0.000
#> GSM228601 2 0.0937 0.9530 0.000 0.960 0.040 0.000 0.000 0.000
#> GSM228604 2 0.0000 0.9671 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228608 1 0.3513 0.7493 0.820 0.000 0.016 0.036 0.004 0.124
#> GSM228609 2 0.0000 0.9671 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228613 1 0.0146 0.8043 0.996 0.000 0.000 0.000 0.004 0.000
#> GSM228616 2 0.0000 0.9671 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228628 3 0.2882 0.7607 0.000 0.008 0.812 0.000 0.000 0.180
#> GSM228634 1 0.3580 0.7443 0.808 0.000 0.000 0.036 0.020 0.136
#> GSM228642 2 0.0000 0.9671 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228645 2 0.0146 0.9663 0.000 0.996 0.000 0.004 0.000 0.000
#> GSM228646 2 0.0000 0.9671 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228652 1 0.5024 0.5012 0.560 0.000 0.016 0.036 0.004 0.384
#> GSM228655 1 0.5047 0.4798 0.548 0.000 0.016 0.036 0.004 0.396
#> GSM228656 1 0.0000 0.8046 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228659 1 0.5862 0.5645 0.648 0.000 0.016 0.080 0.184 0.072
#> GSM228662 1 0.0000 0.8046 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228584 1 0.0000 0.8046 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228586 1 0.1245 0.7963 0.952 0.000 0.000 0.000 0.016 0.032
#> GSM228592 1 0.0000 0.8046 1.000 0.000 0.000 0.000 0.000 0.000
#> GSM228593 4 0.1320 0.9867 0.036 0.000 0.016 0.948 0.000 0.000
#> GSM228594 1 0.1003 0.7910 0.964 0.000 0.016 0.020 0.000 0.000
#> GSM228598 1 0.4853 0.5865 0.620 0.000 0.016 0.036 0.004 0.324
#> GSM228607 3 0.2793 0.7582 0.000 0.000 0.800 0.000 0.000 0.200
#> GSM228612 2 0.1461 0.9456 0.000 0.940 0.044 0.016 0.000 0.000
#> GSM228619 2 0.0000 0.9671 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228622 6 0.4618 0.6112 0.024 0.000 0.224 0.036 0.008 0.708
#> GSM228625 3 0.3071 0.7594 0.000 0.016 0.804 0.000 0.000 0.180
#> GSM228631 2 0.0000 0.9671 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228633 2 0.1572 0.9309 0.000 0.936 0.028 0.000 0.000 0.036
#> GSM228637 5 0.0603 0.7270 0.000 0.000 0.016 0.000 0.980 0.004
#> GSM228639 6 0.2491 0.7205 0.000 0.000 0.164 0.000 0.000 0.836
#> GSM228649 3 0.5324 0.6647 0.000 0.056 0.692 0.140 0.004 0.108
#> GSM228660 6 0.2074 0.7118 0.012 0.000 0.028 0.036 0.004 0.920
#> GSM228661 1 0.4851 0.5686 0.604 0.000 0.000 0.036 0.020 0.340
#> GSM228595 2 0.1082 0.9529 0.000 0.956 0.040 0.000 0.000 0.004
#> GSM228599 2 0.0000 0.9671 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228602 2 0.0000 0.9671 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228614 3 0.3565 0.6170 0.000 0.000 0.808 0.004 0.096 0.092
#> GSM228626 3 0.2597 0.7430 0.000 0.000 0.824 0.000 0.000 0.176
#> GSM228640 2 0.0363 0.9656 0.000 0.988 0.012 0.000 0.000 0.000
#> GSM228643 3 0.4696 0.7261 0.000 0.000 0.688 0.012 0.076 0.224
#> GSM228650 3 0.3175 0.7415 0.000 0.000 0.744 0.000 0.000 0.256
#> GSM228653 6 0.1765 0.7808 0.000 0.000 0.096 0.000 0.000 0.904
#> GSM228657 3 0.1267 0.6775 0.000 0.000 0.940 0.000 0.000 0.060
#> GSM228605 6 0.5893 0.3390 0.144 0.000 0.020 0.036 0.160 0.640
#> GSM228610 6 0.1863 0.7807 0.000 0.000 0.104 0.000 0.000 0.896
#> GSM228617 2 0.0000 0.9671 0.000 1.000 0.000 0.000 0.000 0.000
#> GSM228620 6 0.2742 0.7640 0.004 0.000 0.084 0.036 0.004 0.872
#> GSM228623 3 0.2762 0.7593 0.000 0.000 0.804 0.000 0.000 0.196
#> GSM228629 2 0.0914 0.9604 0.000 0.968 0.016 0.016 0.000 0.000
#> GSM228632 6 0.2230 0.7522 0.000 0.000 0.084 0.000 0.024 0.892
#> GSM228635 3 0.4466 -0.2615 0.000 0.000 0.500 0.004 0.476 0.020
#> GSM228647 3 0.4133 0.7307 0.000 0.000 0.708 0.032 0.008 0.252
#> GSM228596 6 0.4804 0.1594 0.040 0.000 0.016 0.004 0.296 0.644
#> GSM228600 2 0.0260 0.9664 0.000 0.992 0.008 0.000 0.000 0.000
#> GSM228603 2 0.0603 0.9628 0.000 0.980 0.004 0.016 0.000 0.000
#> GSM228615 5 0.0603 0.7270 0.000 0.000 0.016 0.000 0.980 0.004
#> GSM228627 6 0.1765 0.7808 0.000 0.000 0.096 0.000 0.000 0.904
#> GSM228641 2 0.0146 0.9669 0.000 0.996 0.004 0.000 0.000 0.000
#> GSM228644 3 0.2685 0.6117 0.000 0.000 0.868 0.000 0.072 0.060
#> GSM228651 3 0.3050 0.7477 0.000 0.000 0.764 0.000 0.000 0.236
#> GSM228654 6 0.3747 0.1746 0.000 0.000 0.396 0.000 0.000 0.604
#> GSM228658 6 0.1863 0.7811 0.000 0.000 0.104 0.000 0.000 0.896
#> GSM228606 3 0.3263 0.7589 0.000 0.020 0.800 0.004 0.000 0.176
#> GSM228611 6 0.3727 0.2219 0.000 0.000 0.388 0.000 0.000 0.612
#> GSM228618 2 0.0820 0.9618 0.000 0.972 0.012 0.016 0.000 0.000
#> GSM228621 3 0.2793 0.7582 0.000 0.000 0.800 0.000 0.000 0.200
#> GSM228624 3 0.2823 0.7563 0.000 0.000 0.796 0.000 0.000 0.204
#> GSM228630 3 0.3175 0.7387 0.000 0.000 0.744 0.000 0.000 0.256
#> GSM228636 5 0.4337 0.2001 0.000 0.000 0.480 0.000 0.500 0.020
#> GSM228638 6 0.1957 0.7773 0.000 0.000 0.112 0.000 0.000 0.888
#> GSM228648 3 0.3756 0.5205 0.000 0.000 0.600 0.000 0.000 0.400
#> GSM228670 6 0.2135 0.7621 0.000 0.000 0.128 0.000 0.000 0.872
#> GSM228671 3 0.3428 0.6898 0.000 0.000 0.696 0.000 0.000 0.304
#> GSM228672 5 0.0603 0.7270 0.000 0.000 0.016 0.000 0.980 0.004
#> GSM228674 5 0.2744 0.7310 0.000 0.000 0.016 0.000 0.840 0.144
#> GSM228675 6 0.2092 0.7640 0.000 0.000 0.124 0.000 0.000 0.876
#> GSM228676 6 0.3646 0.6679 0.000 0.000 0.068 0.012 0.112 0.808
#> GSM228667 5 0.4353 0.4065 0.000 0.000 0.020 0.004 0.588 0.388
#> GSM228668 6 0.4079 0.5063 0.000 0.000 0.024 0.032 0.192 0.752
#> GSM228669 5 0.3224 0.7331 0.016 0.000 0.016 0.032 0.856 0.080
#> GSM228673 5 0.4366 0.2955 0.000 0.000 0.016 0.004 0.540 0.440
#> GSM228677 3 0.3916 0.6053 0.000 0.000 0.800 0.032 0.100 0.068
#> GSM228678 3 0.5192 0.2150 0.000 0.000 0.632 0.064 0.272 0.032
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
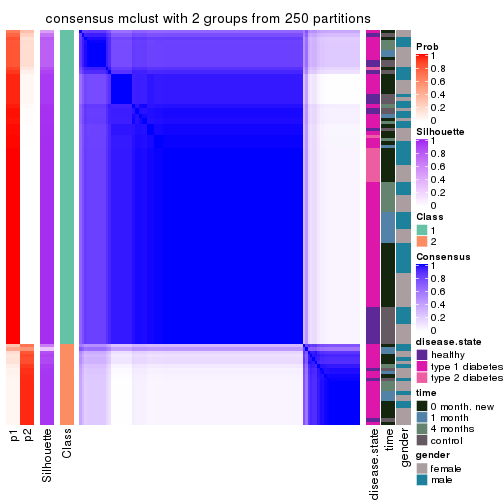
consensus_heatmap(res, k = 3)
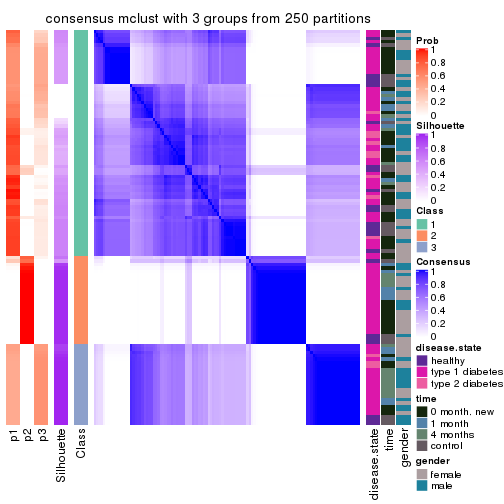
consensus_heatmap(res, k = 4)
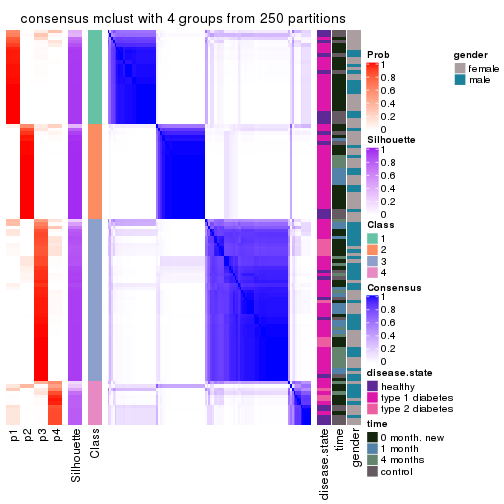
consensus_heatmap(res, k = 5)
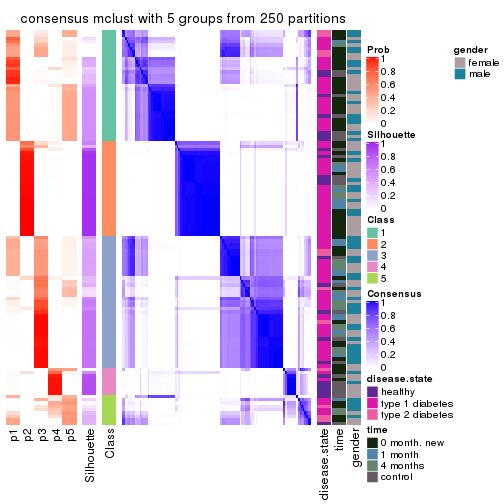
consensus_heatmap(res, k = 6)
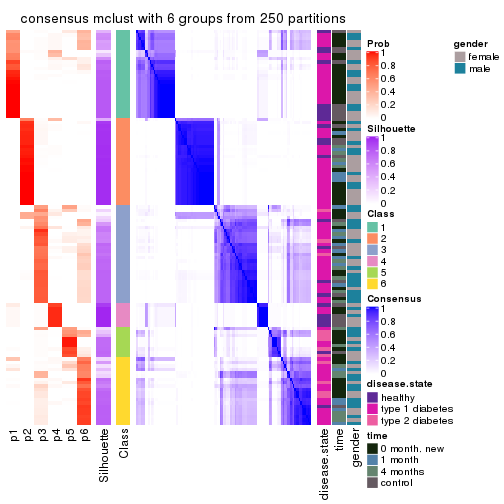
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
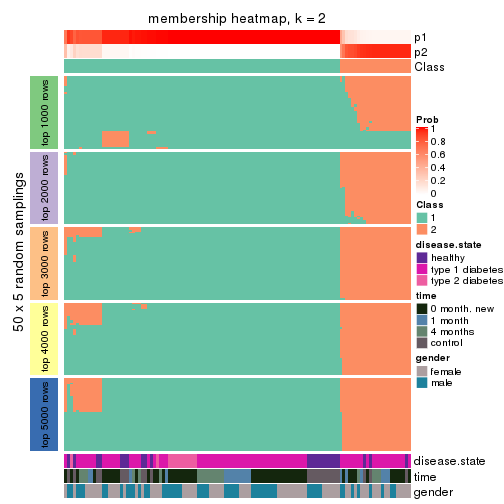
membership_heatmap(res, k = 3)
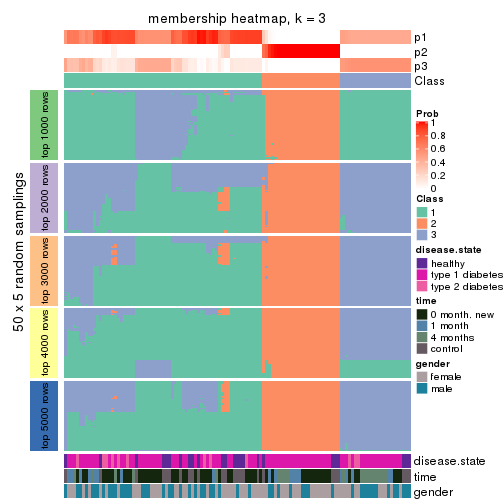
membership_heatmap(res, k = 4)
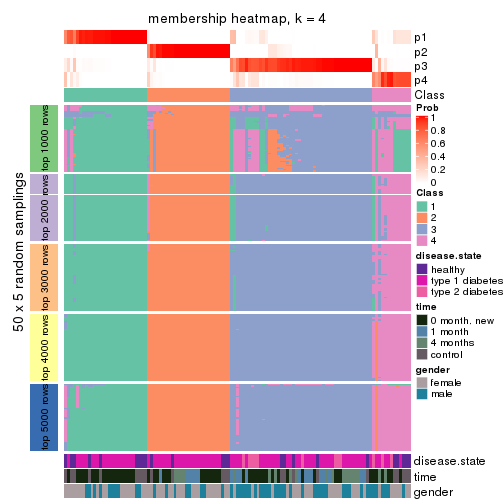
membership_heatmap(res, k = 5)
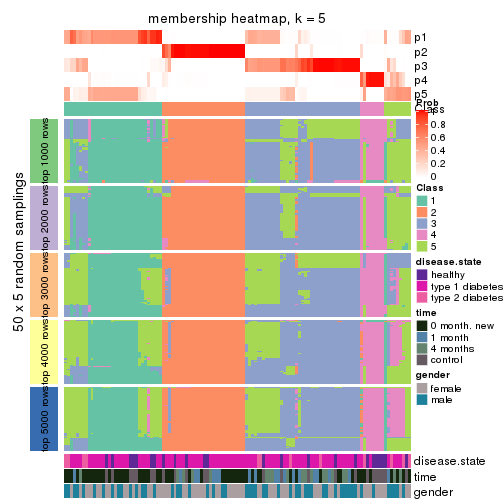
membership_heatmap(res, k = 6)
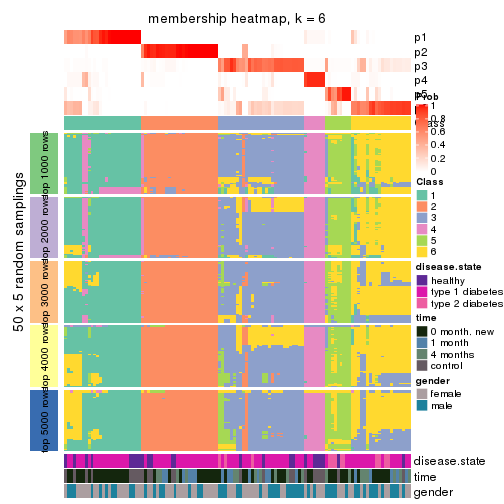
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
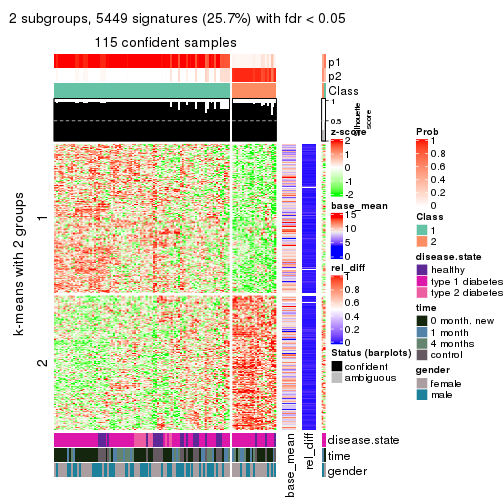
get_signatures(res, k = 3)
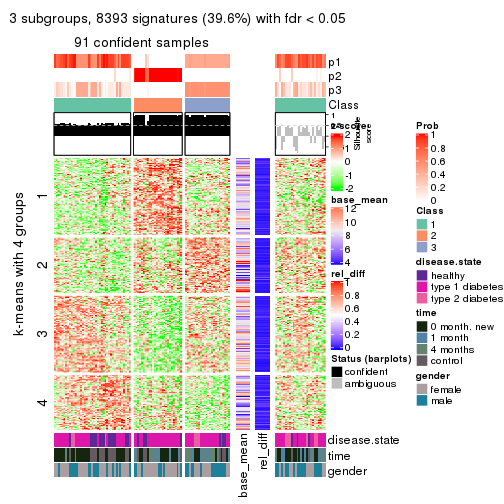
get_signatures(res, k = 4)
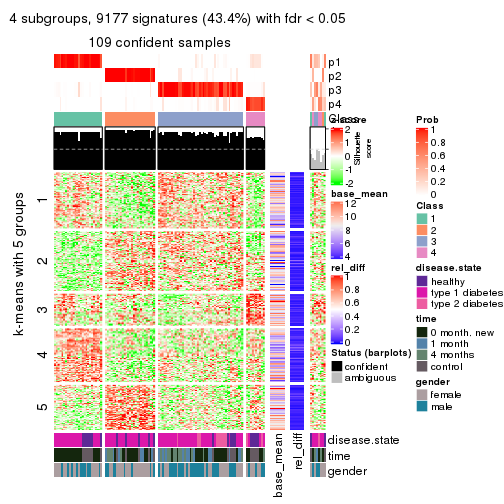
get_signatures(res, k = 5)
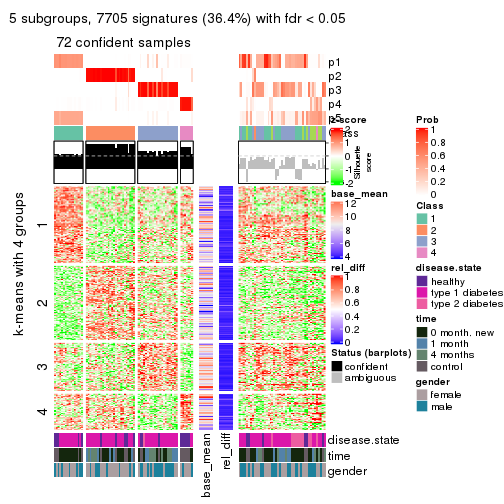
get_signatures(res, k = 6)
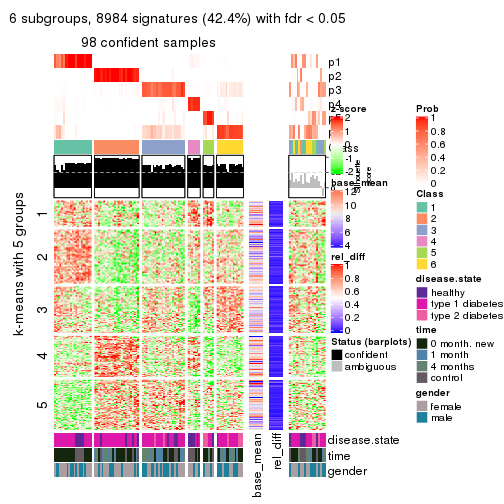
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
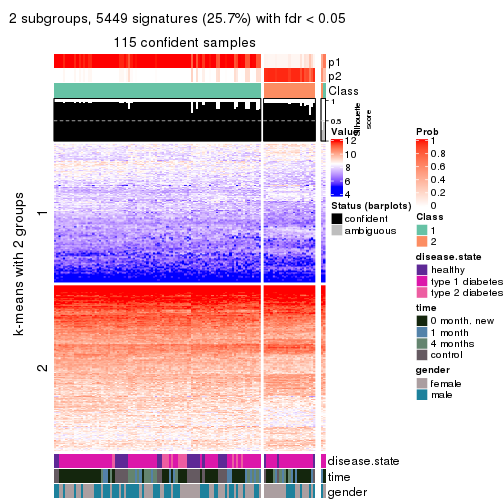
get_signatures(res, k = 3, scale_rows = FALSE)
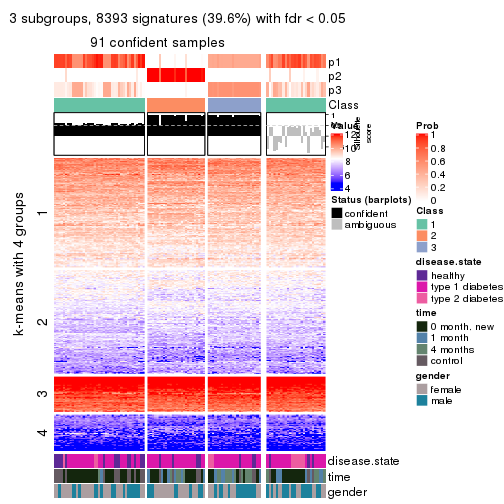
get_signatures(res, k = 4, scale_rows = FALSE)
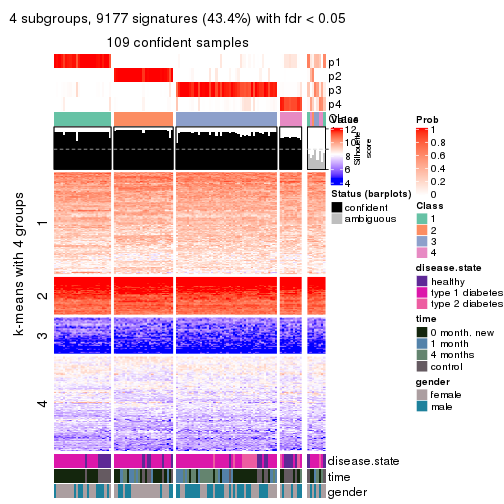
get_signatures(res, k = 5, scale_rows = FALSE)
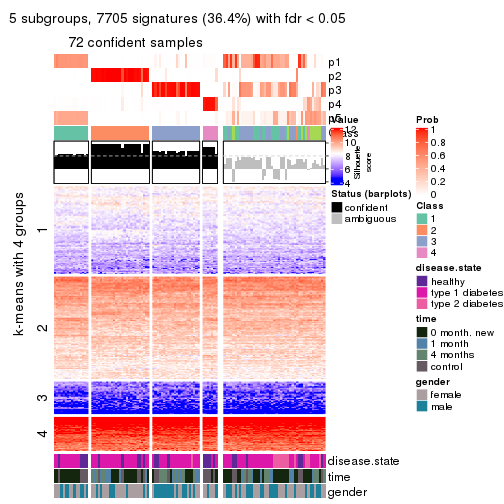
get_signatures(res, k = 6, scale_rows = FALSE)
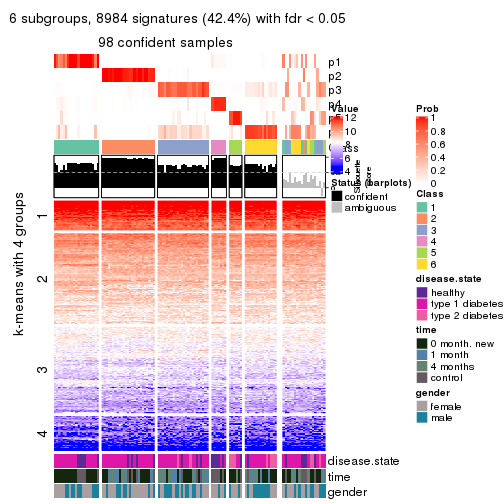
Compare the overlap of signatures from different k:
compare_signatures(res)
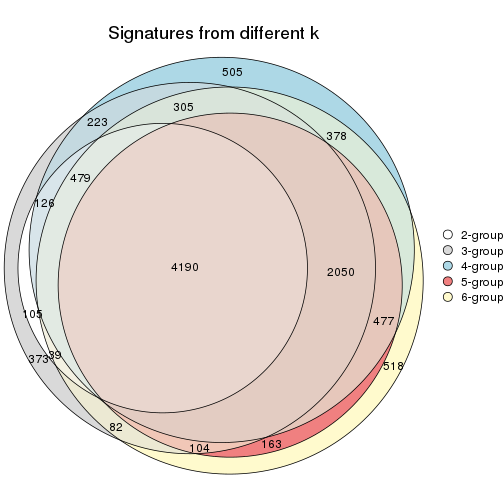
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
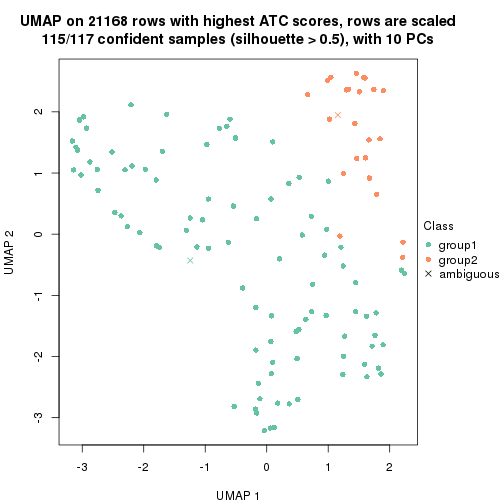
dimension_reduction(res, k = 3, method = "UMAP")
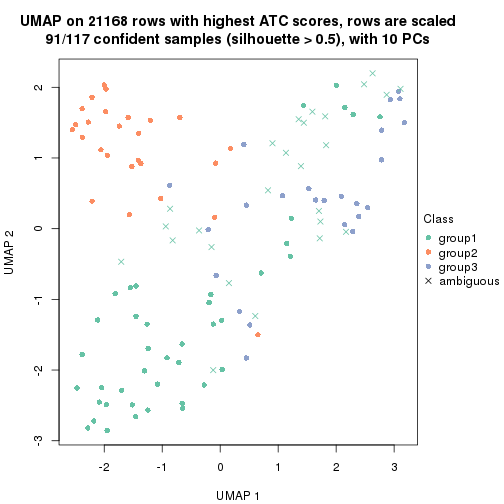
dimension_reduction(res, k = 4, method = "UMAP")
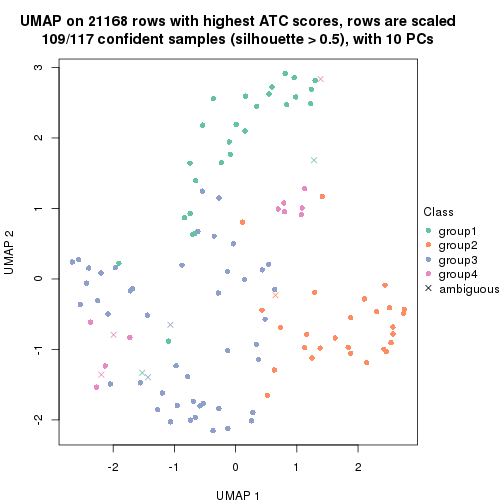
dimension_reduction(res, k = 5, method = "UMAP")
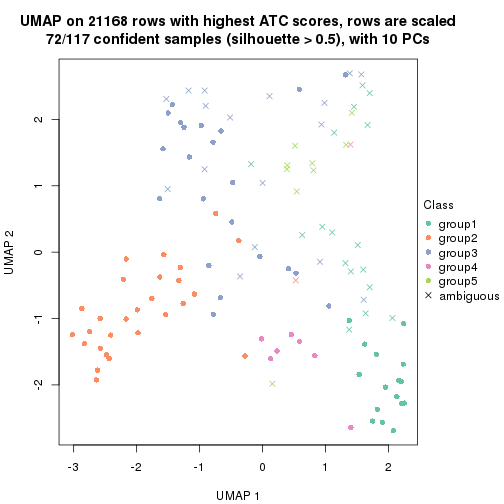
dimension_reduction(res, k = 6, method = "UMAP")
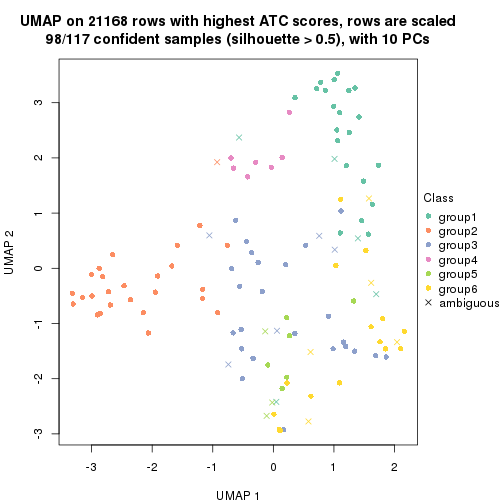
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
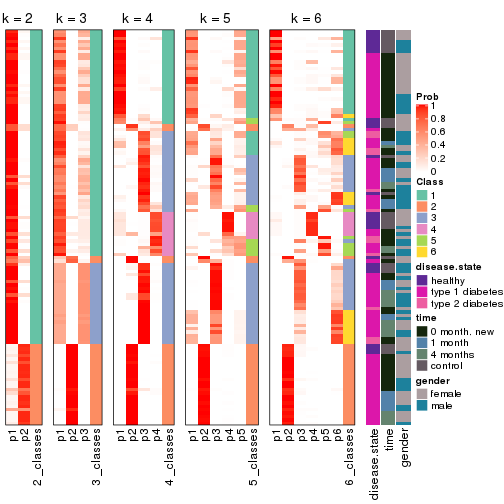
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> ATC:mclust 115 0.072828 6.73e-01 0.2052 2
#> ATC:mclust 91 0.070082 6.34e-05 0.2329 3
#> ATC:mclust 109 0.002492 8.32e-04 0.0852 4
#> ATC:mclust 72 0.050628 1.94e-03 0.0942 5
#> ATC:mclust 98 0.000065 2.40e-03 0.4167 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
The object with results only for a single top-value method and a single partition method can be extracted as:
res = res_list["ATC", "NMF"]
# you can also extract it by
# res = res_list["ATC:NMF"]
A summary of res and all the functions that can be applied to it:
res
#> A 'ConsensusPartition' object with k = 2, 3, 4, 5, 6.
#> On a matrix with 21168 rows and 117 columns.
#> Top rows (1000, 2000, 3000, 4000, 5000) are extracted by 'ATC' method.
#> Subgroups are detected by 'NMF' method.
#> Performed in total 1250 partitions by row resampling.
#> Best k for subgroups seems to be 2.
#>
#> Following methods can be applied to this 'ConsensusPartition' object:
#> [1] "cola_report" "collect_classes" "collect_plots"
#> [4] "collect_stats" "colnames" "compare_signatures"
#> [7] "consensus_heatmap" "dimension_reduction" "functional_enrichment"
#> [10] "get_anno_col" "get_anno" "get_classes"
#> [13] "get_consensus" "get_matrix" "get_membership"
#> [16] "get_param" "get_signatures" "get_stats"
#> [19] "is_best_k" "is_stable_k" "membership_heatmap"
#> [22] "ncol" "nrow" "plot_ecdf"
#> [25] "rownames" "select_partition_number" "show"
#> [28] "suggest_best_k" "test_to_known_factors"
collect_plots() function collects all the plots made from res for all k (number of partitions)
into one single page to provide an easy and fast comparison between different k.
collect_plots(res)
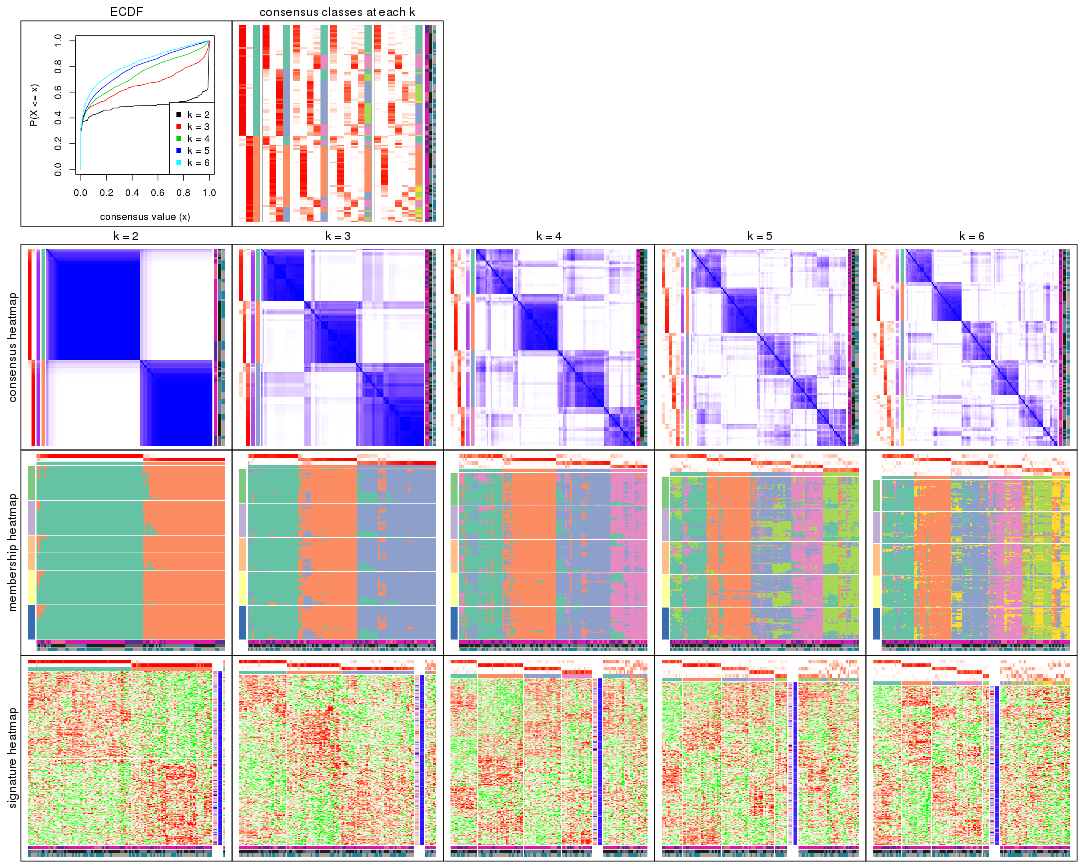
The plots are:
k and the heatmap of
predicted classes for each k.k.k.k.All the plots in panels can be made by individual functions and they are plotted later in this section.
select_partition_number() produces several plots showing different
statistics for choosing “optimized” k. There are following statistics:
k;k, the area increased is defined as \(A_k - A_{k-1}\).The detailed explanations of these statistics can be found in the cola vignette.
Generally speaking, lower PAC score, higher mean silhouette score or higher
concordance corresponds to better partition. Rand index and Jaccard index
measure how similar the current partition is compared to partition with k-1.
If they are too similar, we won't accept k is better than k-1.
select_partition_number(res)
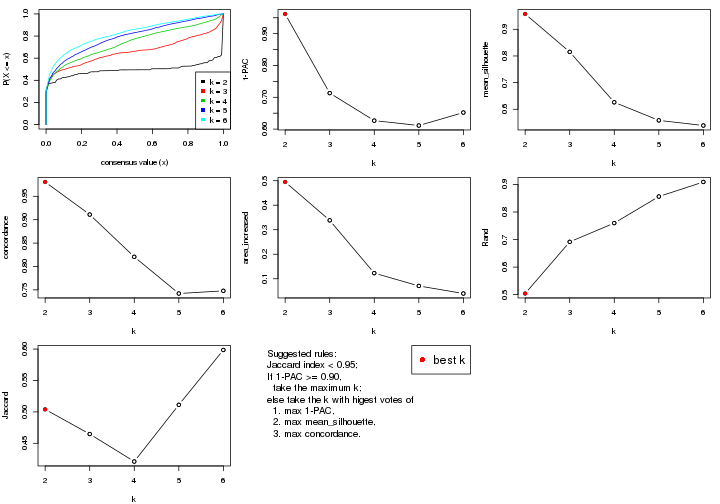
The numeric values for all these statistics can be obtained by get_stats().
get_stats(res)
#> k 1-PAC mean_silhouette concordance area_increased Rand Jaccard
#> 2 2 0.962 0.957 0.981 0.4948 0.504 0.504
#> 3 3 0.713 0.815 0.911 0.3382 0.692 0.465
#> 4 4 0.627 0.626 0.821 0.1228 0.760 0.421
#> 5 5 0.611 0.558 0.742 0.0706 0.856 0.511
#> 6 6 0.652 0.539 0.748 0.0397 0.909 0.599
suggest_best_k() suggests the best \(k\) based on these statistics. The rules are as follows:
suggest_best_k(res)
#> [1] 2
Following shows the table of the partitions (You need to click the show/hide
code output link to see it). The membership matrix (columns with name p*)
is inferred by
clue::cl_consensus()
function with the SE method. Basically the value in the membership matrix
represents the probability to belong to a certain group. The finall class
label for an item is determined with the group with highest probability it
belongs to.
In get_classes() function, the entropy is calculated from the membership
matrix and the silhouette score is calculated from the consensus matrix.
cbind(get_classes(res, k = 2), get_membership(res, k = 2))
#> class entropy silhouette p1 p2
#> GSM228562 1 0.4298 0.898 0.912 0.088
#> GSM228563 2 0.0376 0.973 0.004 0.996
#> GSM228565 1 0.0000 0.983 1.000 0.000
#> GSM228566 2 0.0000 0.975 0.000 1.000
#> GSM228567 1 0.0000 0.983 1.000 0.000
#> GSM228570 2 0.7883 0.710 0.236 0.764
#> GSM228571 1 0.4298 0.898 0.912 0.088
#> GSM228574 2 0.0000 0.975 0.000 1.000
#> GSM228575 2 0.0000 0.975 0.000 1.000
#> GSM228576 2 0.0000 0.975 0.000 1.000
#> GSM228579 1 0.0000 0.983 1.000 0.000
#> GSM228580 1 0.0000 0.983 1.000 0.000
#> GSM228581 1 0.0000 0.983 1.000 0.000
#> GSM228666 1 0.0000 0.983 1.000 0.000
#> GSM228564 2 0.6623 0.805 0.172 0.828
#> GSM228568 1 0.0000 0.983 1.000 0.000
#> GSM228569 1 0.0000 0.983 1.000 0.000
#> GSM228572 2 0.0000 0.975 0.000 1.000
#> GSM228573 2 0.0000 0.975 0.000 1.000
#> GSM228577 1 0.0000 0.983 1.000 0.000
#> GSM228578 1 0.0000 0.983 1.000 0.000
#> GSM228663 1 0.0938 0.973 0.988 0.012
#> GSM228664 2 0.0376 0.973 0.004 0.996
#> GSM228665 1 0.0000 0.983 1.000 0.000
#> GSM228582 1 0.0000 0.983 1.000 0.000
#> GSM228583 1 0.0000 0.983 1.000 0.000
#> GSM228585 1 0.0000 0.983 1.000 0.000
#> GSM228587 1 0.0000 0.983 1.000 0.000
#> GSM228588 1 0.0000 0.983 1.000 0.000
#> GSM228589 2 0.3431 0.925 0.064 0.936
#> GSM228590 1 0.0000 0.983 1.000 0.000
#> GSM228591 2 0.0000 0.975 0.000 1.000
#> GSM228597 2 0.0000 0.975 0.000 1.000
#> GSM228601 2 0.0000 0.975 0.000 1.000
#> GSM228604 2 0.0000 0.975 0.000 1.000
#> GSM228608 1 0.0000 0.983 1.000 0.000
#> GSM228609 2 0.0000 0.975 0.000 1.000
#> GSM228613 1 0.0000 0.983 1.000 0.000
#> GSM228616 2 0.0000 0.975 0.000 1.000
#> GSM228628 2 0.0672 0.971 0.008 0.992
#> GSM228634 1 0.0000 0.983 1.000 0.000
#> GSM228642 2 0.0000 0.975 0.000 1.000
#> GSM228645 2 0.0000 0.975 0.000 1.000
#> GSM228646 2 0.0000 0.975 0.000 1.000
#> GSM228652 1 0.0000 0.983 1.000 0.000
#> GSM228655 1 0.0000 0.983 1.000 0.000
#> GSM228656 1 0.0000 0.983 1.000 0.000
#> GSM228659 1 0.0000 0.983 1.000 0.000
#> GSM228662 1 0.0000 0.983 1.000 0.000
#> GSM228584 1 0.0000 0.983 1.000 0.000
#> GSM228586 1 0.0000 0.983 1.000 0.000
#> GSM228592 1 0.0000 0.983 1.000 0.000
#> GSM228593 2 0.5842 0.845 0.140 0.860
#> GSM228594 1 0.0000 0.983 1.000 0.000
#> GSM228598 1 0.0000 0.983 1.000 0.000
#> GSM228607 2 0.6148 0.830 0.152 0.848
#> GSM228612 2 0.0000 0.975 0.000 1.000
#> GSM228619 2 0.0000 0.975 0.000 1.000
#> GSM228622 1 0.0000 0.983 1.000 0.000
#> GSM228625 2 0.8763 0.600 0.296 0.704
#> GSM228631 2 0.0000 0.975 0.000 1.000
#> GSM228633 2 0.0000 0.975 0.000 1.000
#> GSM228637 1 0.0000 0.983 1.000 0.000
#> GSM228639 1 0.0000 0.983 1.000 0.000
#> GSM228649 1 0.9754 0.296 0.592 0.408
#> GSM228660 1 0.0000 0.983 1.000 0.000
#> GSM228661 1 0.0000 0.983 1.000 0.000
#> GSM228595 2 0.0000 0.975 0.000 1.000
#> GSM228599 2 0.0000 0.975 0.000 1.000
#> GSM228602 2 0.0000 0.975 0.000 1.000
#> GSM228614 2 0.2948 0.936 0.052 0.948
#> GSM228626 2 0.0000 0.975 0.000 1.000
#> GSM228640 2 0.0000 0.975 0.000 1.000
#> GSM228643 1 0.2423 0.948 0.960 0.040
#> GSM228650 1 0.5059 0.871 0.888 0.112
#> GSM228653 1 0.0000 0.983 1.000 0.000
#> GSM228657 2 0.0000 0.975 0.000 1.000
#> GSM228605 1 0.0000 0.983 1.000 0.000
#> GSM228610 1 0.0000 0.983 1.000 0.000
#> GSM228617 2 0.0000 0.975 0.000 1.000
#> GSM228620 1 0.0000 0.983 1.000 0.000
#> GSM228623 2 0.0000 0.975 0.000 1.000
#> GSM228629 2 0.0000 0.975 0.000 1.000
#> GSM228632 1 0.0000 0.983 1.000 0.000
#> GSM228635 2 0.0672 0.971 0.008 0.992
#> GSM228647 1 0.0000 0.983 1.000 0.000
#> GSM228596 1 0.0000 0.983 1.000 0.000
#> GSM228600 2 0.0000 0.975 0.000 1.000
#> GSM228603 2 0.0000 0.975 0.000 1.000
#> GSM228615 1 0.0000 0.983 1.000 0.000
#> GSM228627 1 0.0000 0.983 1.000 0.000
#> GSM228641 2 0.0000 0.975 0.000 1.000
#> GSM228644 2 0.0000 0.975 0.000 1.000
#> GSM228651 1 0.8016 0.676 0.756 0.244
#> GSM228654 1 0.0000 0.983 1.000 0.000
#> GSM228658 1 0.0000 0.983 1.000 0.000
#> GSM228606 2 0.0000 0.975 0.000 1.000
#> GSM228611 1 0.0000 0.983 1.000 0.000
#> GSM228618 2 0.0000 0.975 0.000 1.000
#> GSM228621 2 0.0000 0.975 0.000 1.000
#> GSM228624 2 0.0376 0.973 0.004 0.996
#> GSM228630 2 0.0000 0.975 0.000 1.000
#> GSM228636 2 0.0000 0.975 0.000 1.000
#> GSM228638 1 0.0000 0.983 1.000 0.000
#> GSM228648 1 0.0376 0.980 0.996 0.004
#> GSM228670 1 0.0000 0.983 1.000 0.000
#> GSM228671 1 0.2948 0.937 0.948 0.052
#> GSM228672 1 0.0000 0.983 1.000 0.000
#> GSM228674 1 0.0000 0.983 1.000 0.000
#> GSM228675 1 0.0000 0.983 1.000 0.000
#> GSM228676 1 0.0000 0.983 1.000 0.000
#> GSM228667 1 0.0000 0.983 1.000 0.000
#> GSM228668 1 0.0000 0.983 1.000 0.000
#> GSM228669 1 0.0000 0.983 1.000 0.000
#> GSM228673 1 0.0000 0.983 1.000 0.000
#> GSM228677 2 0.3274 0.930 0.060 0.940
#> GSM228678 2 0.0938 0.968 0.012 0.988
cbind(get_classes(res, k = 3), get_membership(res, k = 3))
#> class entropy silhouette p1 p2 p3
#> GSM228562 1 0.0592 0.941 0.988 0.012 0.000
#> GSM228563 1 0.2165 0.898 0.936 0.064 0.000
#> GSM228565 1 0.0237 0.945 0.996 0.004 0.000
#> GSM228566 2 0.4974 0.667 0.236 0.764 0.000
#> GSM228567 1 0.0892 0.940 0.980 0.000 0.020
#> GSM228570 1 0.1411 0.925 0.964 0.036 0.000
#> GSM228571 1 0.0592 0.941 0.988 0.012 0.000
#> GSM228574 2 0.0237 0.901 0.000 0.996 0.004
#> GSM228575 2 0.0000 0.902 0.000 1.000 0.000
#> GSM228576 1 0.5098 0.633 0.752 0.248 0.000
#> GSM228579 1 0.0237 0.946 0.996 0.000 0.004
#> GSM228580 3 0.5431 0.665 0.284 0.000 0.716
#> GSM228581 3 0.4452 0.775 0.192 0.000 0.808
#> GSM228666 3 0.4796 0.746 0.220 0.000 0.780
#> GSM228564 1 0.1289 0.929 0.968 0.032 0.000
#> GSM228568 1 0.0747 0.943 0.984 0.000 0.016
#> GSM228569 3 0.5882 0.550 0.348 0.000 0.652
#> GSM228572 2 0.0000 0.902 0.000 1.000 0.000
#> GSM228573 2 0.5968 0.493 0.000 0.636 0.364
#> GSM228577 1 0.0747 0.943 0.984 0.000 0.016
#> GSM228578 3 0.5254 0.695 0.264 0.000 0.736
#> GSM228663 3 0.0237 0.871 0.000 0.004 0.996
#> GSM228664 3 0.1289 0.856 0.000 0.032 0.968
#> GSM228665 3 0.0237 0.872 0.004 0.000 0.996
#> GSM228582 3 0.1031 0.869 0.024 0.000 0.976
#> GSM228583 1 0.0747 0.943 0.984 0.000 0.016
#> GSM228585 1 0.0237 0.946 0.996 0.000 0.004
#> GSM228587 1 0.0237 0.945 0.996 0.004 0.000
#> GSM228588 1 0.0592 0.941 0.988 0.012 0.000
#> GSM228589 3 0.2537 0.822 0.000 0.080 0.920
#> GSM228590 1 0.0000 0.945 1.000 0.000 0.000
#> GSM228591 2 0.1411 0.891 0.000 0.964 0.036
#> GSM228597 2 0.6079 0.367 0.388 0.612 0.000
#> GSM228601 2 0.0424 0.900 0.008 0.992 0.000
#> GSM228604 2 0.0000 0.902 0.000 1.000 0.000
#> GSM228608 1 0.3412 0.829 0.876 0.000 0.124
#> GSM228609 2 0.0892 0.894 0.020 0.980 0.000
#> GSM228613 1 0.1031 0.938 0.976 0.000 0.024
#> GSM228616 2 0.0424 0.900 0.008 0.992 0.000
#> GSM228628 2 0.2796 0.854 0.000 0.908 0.092
#> GSM228634 3 0.4605 0.765 0.204 0.000 0.796
#> GSM228642 2 0.0237 0.901 0.004 0.996 0.000
#> GSM228645 2 0.2796 0.837 0.092 0.908 0.000
#> GSM228646 2 0.0747 0.897 0.016 0.984 0.000
#> GSM228652 3 0.4605 0.764 0.204 0.000 0.796
#> GSM228655 3 0.5098 0.716 0.248 0.000 0.752
#> GSM228656 1 0.0892 0.940 0.980 0.000 0.020
#> GSM228659 1 0.0592 0.944 0.988 0.000 0.012
#> GSM228662 1 0.0237 0.946 0.996 0.000 0.004
#> GSM228584 1 0.0237 0.946 0.996 0.000 0.004
#> GSM228586 1 0.3752 0.801 0.856 0.000 0.144
#> GSM228592 1 0.0237 0.946 0.996 0.000 0.004
#> GSM228593 1 0.1643 0.918 0.956 0.044 0.000
#> GSM228594 1 0.0237 0.945 0.996 0.004 0.000
#> GSM228598 3 0.5291 0.692 0.268 0.000 0.732
#> GSM228607 3 0.2448 0.825 0.000 0.076 0.924
#> GSM228612 2 0.1031 0.896 0.000 0.976 0.024
#> GSM228619 2 0.0424 0.900 0.008 0.992 0.000
#> GSM228622 3 0.5397 0.679 0.280 0.000 0.720
#> GSM228625 2 0.5754 0.600 0.004 0.700 0.296
#> GSM228631 2 0.0747 0.897 0.016 0.984 0.000
#> GSM228633 2 0.1163 0.895 0.000 0.972 0.028
#> GSM228637 1 0.0592 0.944 0.988 0.000 0.012
#> GSM228639 3 0.0237 0.871 0.000 0.004 0.996
#> GSM228649 1 0.1289 0.929 0.968 0.032 0.000
#> GSM228660 3 0.1643 0.863 0.044 0.000 0.956
#> GSM228661 3 0.4452 0.775 0.192 0.000 0.808
#> GSM228595 2 0.0237 0.901 0.000 0.996 0.004
#> GSM228599 2 0.0237 0.901 0.004 0.996 0.000
#> GSM228602 2 0.0424 0.900 0.008 0.992 0.000
#> GSM228614 3 0.5988 0.352 0.000 0.368 0.632
#> GSM228626 3 0.6305 -0.068 0.000 0.484 0.516
#> GSM228640 2 0.0237 0.901 0.004 0.996 0.000
#> GSM228643 3 0.1337 0.870 0.012 0.016 0.972
#> GSM228650 3 0.0424 0.869 0.000 0.008 0.992
#> GSM228653 3 0.0000 0.872 0.000 0.000 1.000
#> GSM228657 2 0.6154 0.389 0.000 0.592 0.408
#> GSM228605 1 0.6309 -0.165 0.504 0.000 0.496
#> GSM228610 3 0.0000 0.872 0.000 0.000 1.000
#> GSM228617 2 0.0000 0.902 0.000 1.000 0.000
#> GSM228620 3 0.0592 0.871 0.012 0.000 0.988
#> GSM228623 3 0.6008 0.351 0.000 0.372 0.628
#> GSM228629 2 0.1163 0.895 0.000 0.972 0.028
#> GSM228632 3 0.0237 0.872 0.004 0.000 0.996
#> GSM228635 2 0.3816 0.803 0.000 0.852 0.148
#> GSM228647 3 0.0424 0.872 0.008 0.000 0.992
#> GSM228596 3 0.3192 0.832 0.112 0.000 0.888
#> GSM228600 2 0.0000 0.902 0.000 1.000 0.000
#> GSM228603 2 0.0000 0.902 0.000 1.000 0.000
#> GSM228615 3 0.5327 0.687 0.272 0.000 0.728
#> GSM228627 3 0.0237 0.872 0.004 0.000 0.996
#> GSM228641 2 0.0000 0.902 0.000 1.000 0.000
#> GSM228644 2 0.5760 0.568 0.000 0.672 0.328
#> GSM228651 3 0.0424 0.869 0.000 0.008 0.992
#> GSM228654 3 0.0237 0.871 0.000 0.004 0.996
#> GSM228658 3 0.0000 0.872 0.000 0.000 1.000
#> GSM228606 2 0.2066 0.878 0.000 0.940 0.060
#> GSM228611 3 0.0237 0.871 0.000 0.004 0.996
#> GSM228618 2 0.1289 0.893 0.000 0.968 0.032
#> GSM228621 3 0.5327 0.560 0.000 0.272 0.728
#> GSM228624 3 0.4654 0.683 0.000 0.208 0.792
#> GSM228630 3 0.1411 0.854 0.000 0.036 0.964
#> GSM228636 2 0.0237 0.901 0.004 0.996 0.000
#> GSM228638 3 0.0000 0.872 0.000 0.000 1.000
#> GSM228648 3 0.0424 0.869 0.000 0.008 0.992
#> GSM228670 3 0.0000 0.872 0.000 0.000 1.000
#> GSM228671 3 0.0237 0.871 0.000 0.004 0.996
#> GSM228672 1 0.0000 0.945 1.000 0.000 0.000
#> GSM228674 3 0.2261 0.854 0.068 0.000 0.932
#> GSM228675 3 0.0000 0.872 0.000 0.000 1.000
#> GSM228676 3 0.1411 0.865 0.036 0.000 0.964
#> GSM228667 3 0.1753 0.862 0.048 0.000 0.952
#> GSM228668 3 0.4291 0.785 0.180 0.000 0.820
#> GSM228669 1 0.1031 0.938 0.976 0.000 0.024
#> GSM228673 3 0.3267 0.830 0.116 0.000 0.884
#> GSM228677 2 0.5810 0.538 0.000 0.664 0.336
#> GSM228678 2 0.5706 0.520 0.320 0.680 0.000
cbind(get_classes(res, k = 4), get_membership(res, k = 4))
#> class entropy silhouette p1 p2 p3 p4
#> GSM228562 4 0.4453 0.61161 0.244 0.012 0.000 0.744
#> GSM228563 4 0.5744 0.54818 0.256 0.068 0.000 0.676
#> GSM228565 4 0.3356 0.68502 0.176 0.000 0.000 0.824
#> GSM228566 2 0.4898 0.30970 0.416 0.584 0.000 0.000
#> GSM228567 1 0.1936 0.70089 0.940 0.000 0.028 0.032
#> GSM228570 4 0.5937 0.41616 0.340 0.052 0.000 0.608
#> GSM228571 1 0.1890 0.67768 0.936 0.056 0.008 0.000
#> GSM228574 2 0.0469 0.89034 0.000 0.988 0.012 0.000
#> GSM228575 2 0.0524 0.89138 0.008 0.988 0.004 0.000
#> GSM228576 1 0.4916 0.13367 0.576 0.424 0.000 0.000
#> GSM228579 1 0.4018 0.55840 0.772 0.004 0.000 0.224
#> GSM228580 4 0.3037 0.74131 0.020 0.000 0.100 0.880
#> GSM228581 3 0.6316 0.39934 0.300 0.000 0.612 0.088
#> GSM228666 4 0.3390 0.73414 0.016 0.000 0.132 0.852
#> GSM228564 4 0.2868 0.71058 0.136 0.000 0.000 0.864
#> GSM228568 1 0.1936 0.70137 0.940 0.000 0.032 0.028
#> GSM228569 1 0.3852 0.60534 0.800 0.000 0.192 0.008
#> GSM228572 2 0.0188 0.89140 0.004 0.996 0.000 0.000
#> GSM228573 2 0.4454 0.55290 0.000 0.692 0.308 0.000
#> GSM228577 1 0.2635 0.69182 0.904 0.000 0.020 0.076
#> GSM228578 1 0.5905 0.25409 0.564 0.000 0.396 0.040
#> GSM228663 3 0.2149 0.74882 0.088 0.000 0.912 0.000
#> GSM228664 3 0.3612 0.72283 0.044 0.100 0.856 0.000
#> GSM228665 3 0.2814 0.72446 0.132 0.000 0.868 0.000
#> GSM228582 3 0.4998 0.04836 0.488 0.000 0.512 0.000
#> GSM228583 1 0.1733 0.70093 0.948 0.000 0.028 0.024
#> GSM228585 1 0.2266 0.68055 0.912 0.000 0.004 0.084
#> GSM228587 1 0.3625 0.62672 0.828 0.012 0.000 0.160
#> GSM228588 4 0.3074 0.70194 0.152 0.000 0.000 0.848
#> GSM228589 2 0.7752 -0.07215 0.236 0.404 0.360 0.000
#> GSM228590 4 0.3873 0.63522 0.228 0.000 0.000 0.772
#> GSM228591 2 0.0921 0.88474 0.000 0.972 0.028 0.000
#> GSM228597 2 0.5905 0.59373 0.144 0.700 0.000 0.156
#> GSM228601 2 0.0804 0.88933 0.008 0.980 0.000 0.012
#> GSM228604 2 0.0000 0.89168 0.000 1.000 0.000 0.000
#> GSM228608 1 0.7596 0.31210 0.456 0.000 0.212 0.332
#> GSM228609 2 0.1520 0.87616 0.024 0.956 0.000 0.020
#> GSM228613 1 0.4199 0.64313 0.804 0.000 0.032 0.164
#> GSM228616 2 0.1022 0.88406 0.032 0.968 0.000 0.000
#> GSM228628 2 0.1118 0.88134 0.000 0.964 0.036 0.000
#> GSM228634 1 0.4647 0.47984 0.704 0.000 0.288 0.008
#> GSM228642 2 0.0336 0.89118 0.008 0.992 0.000 0.000
#> GSM228645 2 0.2944 0.79451 0.128 0.868 0.000 0.004
#> GSM228646 2 0.0707 0.88637 0.020 0.980 0.000 0.000
#> GSM228652 1 0.5600 0.03301 0.512 0.000 0.468 0.020
#> GSM228655 3 0.6376 0.17346 0.396 0.000 0.536 0.068
#> GSM228656 1 0.1767 0.69896 0.944 0.000 0.044 0.012
#> GSM228659 4 0.3356 0.68547 0.176 0.000 0.000 0.824
#> GSM228662 1 0.1867 0.68375 0.928 0.000 0.000 0.072
#> GSM228584 1 0.4855 0.33925 0.644 0.000 0.004 0.352
#> GSM228586 1 0.3497 0.66890 0.852 0.000 0.124 0.024
#> GSM228592 1 0.3946 0.63779 0.812 0.000 0.020 0.168
#> GSM228593 1 0.5143 0.54302 0.752 0.172 0.000 0.076
#> GSM228594 1 0.1706 0.68732 0.948 0.016 0.000 0.036
#> GSM228598 1 0.3975 0.54978 0.760 0.000 0.240 0.000
#> GSM228607 3 0.3047 0.71810 0.012 0.116 0.872 0.000
#> GSM228612 2 0.0927 0.88912 0.008 0.976 0.016 0.000
#> GSM228619 2 0.0707 0.88764 0.020 0.980 0.000 0.000
#> GSM228622 3 0.6108 0.15298 0.424 0.000 0.528 0.048
#> GSM228625 1 0.6354 0.17937 0.520 0.416 0.064 0.000
#> GSM228631 2 0.0707 0.88637 0.020 0.980 0.000 0.000
#> GSM228633 2 0.2658 0.84192 0.004 0.904 0.080 0.012
#> GSM228637 4 0.0524 0.75228 0.004 0.000 0.008 0.988
#> GSM228639 3 0.0000 0.76965 0.000 0.000 1.000 0.000
#> GSM228649 1 0.7634 0.13238 0.464 0.236 0.000 0.300
#> GSM228660 1 0.4843 0.27236 0.604 0.000 0.396 0.000
#> GSM228661 1 0.5827 0.12732 0.532 0.000 0.436 0.032
#> GSM228595 2 0.0804 0.88914 0.000 0.980 0.012 0.008
#> GSM228599 2 0.0188 0.89140 0.004 0.996 0.000 0.000
#> GSM228602 2 0.0707 0.88637 0.020 0.980 0.000 0.000
#> GSM228614 4 0.5668 0.50003 0.004 0.032 0.328 0.636
#> GSM228626 3 0.5384 0.46012 0.004 0.292 0.676 0.028
#> GSM228640 2 0.0657 0.89037 0.004 0.984 0.000 0.012
#> GSM228643 4 0.5429 0.38945 0.004 0.012 0.392 0.592
#> GSM228650 3 0.1811 0.75440 0.004 0.020 0.948 0.028
#> GSM228653 3 0.2216 0.74771 0.092 0.000 0.908 0.000
#> GSM228657 3 0.6644 0.41583 0.004 0.248 0.624 0.124
#> GSM228605 3 0.7485 0.08656 0.180 0.000 0.440 0.380
#> GSM228610 3 0.0707 0.76678 0.000 0.000 0.980 0.020
#> GSM228617 2 0.0188 0.89140 0.004 0.996 0.000 0.000
#> GSM228620 3 0.4406 0.52213 0.300 0.000 0.700 0.000
#> GSM228623 3 0.5695 -0.01814 0.024 0.476 0.500 0.000
#> GSM228629 2 0.0672 0.89115 0.008 0.984 0.008 0.000
#> GSM228632 3 0.1174 0.76806 0.012 0.000 0.968 0.020
#> GSM228635 4 0.3027 0.73846 0.004 0.020 0.088 0.888
#> GSM228647 3 0.1284 0.76590 0.012 0.000 0.964 0.024
#> GSM228596 3 0.4898 0.65139 0.072 0.000 0.772 0.156
#> GSM228600 2 0.0188 0.89156 0.000 0.996 0.004 0.000
#> GSM228603 2 0.0376 0.89165 0.004 0.992 0.004 0.000
#> GSM228615 4 0.1474 0.75133 0.000 0.000 0.052 0.948
#> GSM228627 3 0.1716 0.75844 0.064 0.000 0.936 0.000
#> GSM228641 2 0.0524 0.89099 0.000 0.988 0.004 0.008
#> GSM228644 3 0.7286 0.28705 0.004 0.204 0.560 0.232
#> GSM228651 3 0.1624 0.76650 0.028 0.020 0.952 0.000
#> GSM228654 3 0.3837 0.63382 0.224 0.000 0.776 0.000
#> GSM228658 3 0.2469 0.74030 0.108 0.000 0.892 0.000
#> GSM228606 2 0.1867 0.85546 0.000 0.928 0.072 0.000
#> GSM228611 3 0.2868 0.72125 0.136 0.000 0.864 0.000
#> GSM228618 2 0.0895 0.88813 0.004 0.976 0.020 0.000
#> GSM228621 2 0.5548 0.34188 0.024 0.588 0.388 0.000
#> GSM228624 2 0.7146 0.00642 0.132 0.456 0.412 0.000
#> GSM228630 3 0.1543 0.75844 0.004 0.032 0.956 0.008
#> GSM228636 4 0.3029 0.73321 0.004 0.052 0.048 0.896
#> GSM228638 3 0.0707 0.76818 0.020 0.000 0.980 0.000
#> GSM228648 3 0.0469 0.76900 0.000 0.012 0.988 0.000
#> GSM228670 3 0.0779 0.76868 0.004 0.000 0.980 0.016
#> GSM228671 3 0.0992 0.76681 0.004 0.012 0.976 0.008
#> GSM228672 4 0.0592 0.74966 0.016 0.000 0.000 0.984
#> GSM228674 4 0.4539 0.61151 0.008 0.000 0.272 0.720
#> GSM228675 3 0.1356 0.76083 0.008 0.000 0.960 0.032
#> GSM228676 3 0.2282 0.75178 0.024 0.000 0.924 0.052
#> GSM228667 4 0.5119 0.28125 0.004 0.000 0.440 0.556
#> GSM228668 3 0.5677 0.37623 0.040 0.000 0.628 0.332
#> GSM228669 4 0.0921 0.74812 0.028 0.000 0.000 0.972
#> GSM228673 4 0.5268 0.22393 0.008 0.000 0.452 0.540
#> GSM228677 4 0.4462 0.68643 0.004 0.028 0.180 0.788
#> GSM228678 4 0.0895 0.74981 0.004 0.020 0.000 0.976
cbind(get_classes(res, k = 5), get_membership(res, k = 5))
#> class entropy silhouette p1 p2 p3 p4 p5
#> GSM228562 4 0.3832 0.59959 0.232 0.004 0.004 0.756 0.004
#> GSM228563 4 0.5602 -0.01938 0.456 0.052 0.008 0.484 0.000
#> GSM228565 4 0.3562 0.64049 0.196 0.000 0.000 0.788 0.016
#> GSM228566 2 0.4819 0.47101 0.352 0.620 0.024 0.000 0.004
#> GSM228567 1 0.2629 0.68130 0.880 0.000 0.004 0.012 0.104
#> GSM228570 1 0.5364 0.04677 0.512 0.028 0.008 0.448 0.004
#> GSM228571 1 0.2674 0.63860 0.896 0.032 0.060 0.000 0.012
#> GSM228574 2 0.0898 0.88784 0.000 0.972 0.020 0.000 0.008
#> GSM228575 3 0.6105 0.34229 0.148 0.280 0.568 0.000 0.004
#> GSM228576 1 0.4867 0.32651 0.652 0.308 0.036 0.000 0.004
#> GSM228579 1 0.3593 0.66834 0.824 0.000 0.000 0.116 0.060
#> GSM228580 4 0.5552 0.42418 0.064 0.000 0.008 0.588 0.340
#> GSM228581 5 0.4940 0.57018 0.168 0.000 0.088 0.012 0.732
#> GSM228666 4 0.2887 0.73945 0.016 0.000 0.072 0.884 0.028
#> GSM228564 4 0.2233 0.70959 0.104 0.000 0.000 0.892 0.004
#> GSM228568 1 0.1568 0.67201 0.944 0.000 0.036 0.000 0.020
#> GSM228569 1 0.4965 0.16643 0.520 0.000 0.028 0.000 0.452
#> GSM228572 2 0.2473 0.85882 0.000 0.896 0.032 0.000 0.072
#> GSM228573 2 0.4193 0.68512 0.000 0.748 0.040 0.000 0.212
#> GSM228577 1 0.4823 0.50738 0.644 0.000 0.000 0.040 0.316
#> GSM228578 1 0.5378 0.46766 0.660 0.000 0.072 0.012 0.256
#> GSM228663 5 0.2331 0.56236 0.016 0.032 0.028 0.004 0.920
#> GSM228664 5 0.4765 0.34851 0.004 0.232 0.056 0.000 0.708
#> GSM228665 5 0.2761 0.57773 0.024 0.000 0.104 0.000 0.872
#> GSM228582 5 0.5218 0.56862 0.180 0.000 0.136 0.000 0.684
#> GSM228583 1 0.2490 0.68883 0.896 0.000 0.004 0.020 0.080
#> GSM228585 1 0.1661 0.69145 0.940 0.000 0.000 0.036 0.024
#> GSM228587 1 0.5617 0.62459 0.688 0.016 0.004 0.124 0.168
#> GSM228588 4 0.3003 0.65535 0.188 0.000 0.000 0.812 0.000
#> GSM228589 5 0.6642 0.01931 0.024 0.404 0.120 0.000 0.452
#> GSM228590 4 0.6456 -0.02154 0.340 0.000 0.000 0.468 0.192
#> GSM228591 2 0.3073 0.82938 0.004 0.856 0.024 0.000 0.116
#> GSM228597 1 0.7374 -0.05811 0.400 0.152 0.040 0.400 0.008
#> GSM228601 2 0.2103 0.86991 0.000 0.920 0.020 0.004 0.056
#> GSM228604 2 0.0865 0.88767 0.000 0.972 0.004 0.000 0.024
#> GSM228608 5 0.5406 0.20357 0.348 0.000 0.008 0.052 0.592
#> GSM228609 2 0.3068 0.83496 0.028 0.880 0.012 0.072 0.008
#> GSM228613 1 0.6079 0.23211 0.468 0.000 0.012 0.084 0.436
#> GSM228616 2 0.6873 0.12067 0.356 0.384 0.256 0.000 0.004
#> GSM228628 2 0.1216 0.88434 0.000 0.960 0.020 0.000 0.020
#> GSM228634 5 0.5929 0.41741 0.260 0.000 0.156 0.000 0.584
#> GSM228642 2 0.0833 0.88706 0.004 0.976 0.016 0.000 0.004
#> GSM228645 2 0.4809 0.55518 0.296 0.664 0.036 0.000 0.004
#> GSM228646 2 0.1116 0.88640 0.000 0.964 0.004 0.004 0.028
#> GSM228652 5 0.4360 0.38572 0.300 0.000 0.020 0.000 0.680
#> GSM228655 5 0.4964 0.47949 0.244 0.000 0.056 0.008 0.692
#> GSM228656 1 0.2110 0.67710 0.912 0.000 0.016 0.000 0.072
#> GSM228659 4 0.4409 0.59494 0.176 0.000 0.000 0.752 0.072
#> GSM228662 1 0.1830 0.69225 0.932 0.000 0.000 0.040 0.028
#> GSM228584 1 0.4793 0.58549 0.708 0.000 0.000 0.216 0.076
#> GSM228586 1 0.5464 0.23201 0.520 0.000 0.044 0.008 0.428
#> GSM228592 1 0.5740 0.48700 0.580 0.000 0.000 0.112 0.308
#> GSM228593 1 0.3031 0.65993 0.880 0.060 0.008 0.048 0.004
#> GSM228594 1 0.2362 0.68870 0.900 0.008 0.000 0.008 0.084
#> GSM228598 1 0.5378 0.22572 0.548 0.000 0.392 0.000 0.060
#> GSM228607 5 0.5385 0.31578 0.000 0.288 0.088 0.000 0.624
#> GSM228612 3 0.5405 0.48149 0.124 0.200 0.672 0.000 0.004
#> GSM228619 2 0.0740 0.88364 0.008 0.980 0.008 0.000 0.004
#> GSM228622 5 0.5291 0.56647 0.156 0.008 0.112 0.008 0.716
#> GSM228625 2 0.5536 0.58324 0.240 0.660 0.016 0.000 0.084
#> GSM228631 2 0.0579 0.88597 0.008 0.984 0.008 0.000 0.000
#> GSM228633 2 0.3788 0.80941 0.000 0.820 0.072 0.004 0.104
#> GSM228637 4 0.0968 0.74356 0.012 0.000 0.004 0.972 0.012
#> GSM228639 3 0.4310 0.52170 0.000 0.000 0.604 0.004 0.392
#> GSM228649 1 0.5775 0.47552 0.648 0.100 0.008 0.236 0.008
#> GSM228660 5 0.4637 0.40240 0.292 0.000 0.036 0.000 0.672
#> GSM228661 5 0.5040 0.43453 0.272 0.000 0.068 0.000 0.660
#> GSM228595 2 0.2664 0.86090 0.000 0.892 0.040 0.004 0.064
#> GSM228599 2 0.0451 0.88593 0.004 0.988 0.008 0.000 0.000
#> GSM228602 2 0.0290 0.88867 0.000 0.992 0.000 0.000 0.008
#> GSM228614 4 0.4410 0.62847 0.000 0.000 0.112 0.764 0.124
#> GSM228626 3 0.5877 0.55754 0.000 0.076 0.576 0.016 0.332
#> GSM228640 2 0.1267 0.88600 0.000 0.960 0.004 0.012 0.024
#> GSM228643 5 0.5853 -0.04208 0.016 0.008 0.044 0.392 0.540
#> GSM228650 3 0.4335 0.62091 0.000 0.004 0.708 0.020 0.268
#> GSM228653 5 0.4366 0.32339 0.016 0.000 0.320 0.000 0.664
#> GSM228657 3 0.6822 0.52969 0.000 0.068 0.528 0.088 0.316
#> GSM228605 3 0.7204 -0.00058 0.116 0.000 0.452 0.364 0.068
#> GSM228610 5 0.4620 -0.03802 0.000 0.000 0.392 0.016 0.592
#> GSM228617 2 0.0290 0.88868 0.000 0.992 0.000 0.000 0.008
#> GSM228620 5 0.4378 0.53203 0.036 0.000 0.248 0.000 0.716
#> GSM228623 3 0.2906 0.64395 0.004 0.060 0.884 0.004 0.048
#> GSM228629 2 0.1408 0.87414 0.008 0.948 0.044 0.000 0.000
#> GSM228632 5 0.4457 0.07104 0.000 0.000 0.368 0.012 0.620
#> GSM228635 4 0.3980 0.51939 0.000 0.000 0.284 0.708 0.008
#> GSM228647 3 0.4891 0.57108 0.000 0.000 0.640 0.044 0.316
#> GSM228596 5 0.4019 0.59172 0.052 0.000 0.088 0.036 0.824
#> GSM228600 2 0.0510 0.88735 0.000 0.984 0.016 0.000 0.000
#> GSM228603 2 0.0324 0.88822 0.000 0.992 0.004 0.000 0.004
#> GSM228615 4 0.1168 0.74443 0.000 0.000 0.032 0.960 0.008
#> GSM228627 3 0.4416 0.45850 0.012 0.000 0.632 0.000 0.356
#> GSM228641 2 0.0992 0.88736 0.000 0.968 0.000 0.008 0.024
#> GSM228644 3 0.7473 0.44160 0.000 0.052 0.436 0.204 0.308
#> GSM228651 3 0.2552 0.64183 0.004 0.016 0.896 0.004 0.080
#> GSM228654 3 0.4258 0.56641 0.072 0.000 0.768 0.000 0.160
#> GSM228658 5 0.4401 0.34505 0.016 0.000 0.328 0.000 0.656
#> GSM228606 3 0.5049 0.43603 0.016 0.308 0.652 0.016 0.008
#> GSM228611 3 0.2580 0.61725 0.044 0.000 0.892 0.000 0.064
#> GSM228618 2 0.1059 0.88507 0.004 0.968 0.020 0.000 0.008
#> GSM228621 3 0.4220 0.60716 0.028 0.116 0.804 0.000 0.052
#> GSM228624 3 0.4152 0.57169 0.100 0.060 0.812 0.000 0.028
#> GSM228630 3 0.4419 0.57588 0.000 0.004 0.644 0.008 0.344
#> GSM228636 4 0.2136 0.72790 0.000 0.000 0.088 0.904 0.008
#> GSM228638 5 0.2945 0.50787 0.004 0.004 0.136 0.004 0.852
#> GSM228648 3 0.4420 0.41874 0.000 0.000 0.548 0.004 0.448
#> GSM228670 3 0.3750 0.63337 0.000 0.000 0.756 0.012 0.232
#> GSM228671 3 0.3696 0.64573 0.000 0.000 0.772 0.016 0.212
#> GSM228672 4 0.0671 0.74067 0.016 0.000 0.004 0.980 0.000
#> GSM228674 4 0.4275 0.67975 0.012 0.000 0.136 0.788 0.064
#> GSM228675 3 0.3961 0.64081 0.000 0.000 0.760 0.028 0.212
#> GSM228676 3 0.3792 0.64656 0.012 0.000 0.828 0.064 0.096
#> GSM228667 3 0.5036 0.12013 0.004 0.000 0.520 0.452 0.024
#> GSM228668 3 0.4117 0.59282 0.028 0.000 0.788 0.164 0.020
#> GSM228669 4 0.1444 0.73846 0.040 0.000 0.012 0.948 0.000
#> GSM228673 4 0.4998 0.37041 0.008 0.000 0.328 0.632 0.032
#> GSM228677 4 0.3888 0.66238 0.000 0.000 0.136 0.800 0.064
#> GSM228678 4 0.3412 0.69439 0.028 0.000 0.152 0.820 0.000
cbind(get_classes(res, k = 6), get_membership(res, k = 6))
#> class entropy silhouette p1 p2 p3 p4 p5 p6
#> GSM228562 4 0.3983 0.6057 0.216 0.016 0.004 0.748 0.008 0.008
#> GSM228563 1 0.5124 0.6764 0.704 0.064 0.000 0.172 0.008 0.052
#> GSM228565 4 0.3236 0.6956 0.128 0.012 0.012 0.836 0.004 0.008
#> GSM228566 2 0.4627 0.4844 0.300 0.644 0.000 0.000 0.048 0.008
#> GSM228567 1 0.1952 0.8274 0.920 0.000 0.052 0.000 0.012 0.016
#> GSM228570 1 0.5574 0.4033 0.592 0.048 0.004 0.316 0.020 0.020
#> GSM228571 1 0.2933 0.7762 0.852 0.032 0.000 0.000 0.108 0.008
#> GSM228574 2 0.1297 0.8406 0.000 0.948 0.000 0.000 0.012 0.040
#> GSM228575 5 0.4629 0.3871 0.032 0.220 0.004 0.000 0.708 0.036
#> GSM228576 1 0.4811 0.5225 0.652 0.272 0.000 0.000 0.064 0.012
#> GSM228579 1 0.2452 0.8268 0.904 0.000 0.028 0.016 0.012 0.040
#> GSM228580 4 0.5678 0.3545 0.024 0.000 0.376 0.524 0.008 0.068
#> GSM228581 3 0.2756 0.6614 0.084 0.000 0.872 0.000 0.016 0.028
#> GSM228666 4 0.2564 0.7434 0.004 0.000 0.028 0.896 0.040 0.032
#> GSM228564 4 0.1923 0.7313 0.036 0.020 0.000 0.928 0.008 0.008
#> GSM228568 1 0.1674 0.8166 0.924 0.000 0.004 0.000 0.068 0.004
#> GSM228569 1 0.4341 0.5758 0.668 0.000 0.292 0.000 0.032 0.008
#> GSM228572 6 0.4389 -0.0987 0.024 0.448 0.000 0.000 0.000 0.528
#> GSM228573 2 0.3946 0.6170 0.000 0.756 0.168 0.000 0.000 0.076
#> GSM228577 1 0.3283 0.7839 0.824 0.000 0.140 0.004 0.012 0.020
#> GSM228578 1 0.3641 0.7648 0.812 0.000 0.120 0.000 0.028 0.040
#> GSM228663 3 0.3242 0.6064 0.024 0.012 0.832 0.000 0.004 0.128
#> GSM228664 6 0.6131 0.1393 0.004 0.080 0.388 0.000 0.052 0.476
#> GSM228665 3 0.1053 0.6620 0.004 0.000 0.964 0.000 0.012 0.020
#> GSM228582 3 0.3879 0.6352 0.140 0.000 0.784 0.000 0.064 0.012
#> GSM228583 1 0.3262 0.8140 0.840 0.000 0.068 0.000 0.080 0.012
#> GSM228585 1 0.1434 0.8246 0.940 0.000 0.012 0.000 0.048 0.000
#> GSM228587 1 0.4551 0.7654 0.756 0.000 0.124 0.020 0.012 0.088
#> GSM228588 4 0.3488 0.5761 0.244 0.000 0.000 0.744 0.004 0.008
#> GSM228589 6 0.6274 0.3226 0.004 0.112 0.268 0.000 0.064 0.552
#> GSM228590 4 0.6484 0.2583 0.184 0.012 0.320 0.468 0.008 0.008
#> GSM228591 6 0.5071 -0.0741 0.008 0.436 0.056 0.000 0.000 0.500
#> GSM228597 4 0.7726 0.2785 0.284 0.036 0.004 0.424 0.136 0.116
#> GSM228601 2 0.4015 0.3792 0.004 0.596 0.004 0.000 0.000 0.396
#> GSM228604 2 0.2135 0.7862 0.000 0.872 0.000 0.000 0.000 0.128
#> GSM228608 3 0.2477 0.6579 0.084 0.000 0.888 0.012 0.008 0.008
#> GSM228609 2 0.3127 0.7654 0.000 0.840 0.000 0.100 0.004 0.056
#> GSM228613 3 0.5723 -0.1331 0.432 0.000 0.460 0.088 0.012 0.008
#> GSM228616 5 0.5945 -0.0570 0.116 0.424 0.000 0.000 0.436 0.024
#> GSM228628 2 0.1755 0.8349 0.000 0.932 0.028 0.000 0.008 0.032
#> GSM228634 3 0.4600 0.5995 0.152 0.000 0.708 0.000 0.136 0.004
#> GSM228642 2 0.1895 0.8267 0.000 0.912 0.000 0.000 0.016 0.072
#> GSM228645 2 0.3517 0.7345 0.072 0.820 0.000 0.000 0.096 0.012
#> GSM228646 2 0.1732 0.8222 0.004 0.920 0.004 0.000 0.000 0.072
#> GSM228652 3 0.2070 0.6605 0.092 0.000 0.896 0.000 0.000 0.012
#> GSM228655 3 0.1285 0.6670 0.052 0.000 0.944 0.004 0.000 0.000
#> GSM228656 1 0.2052 0.8257 0.912 0.000 0.028 0.000 0.056 0.004
#> GSM228659 4 0.3301 0.7009 0.072 0.000 0.084 0.836 0.004 0.004
#> GSM228662 1 0.1138 0.8299 0.960 0.000 0.024 0.000 0.012 0.004
#> GSM228584 1 0.3957 0.7584 0.780 0.000 0.052 0.152 0.004 0.012
#> GSM228586 3 0.5225 0.0291 0.420 0.000 0.496 0.000 0.080 0.004
#> GSM228592 1 0.5474 0.4878 0.580 0.000 0.308 0.096 0.008 0.008
#> GSM228593 1 0.2033 0.8182 0.916 0.020 0.004 0.004 0.000 0.056
#> GSM228594 1 0.2190 0.8262 0.908 0.000 0.044 0.000 0.008 0.040
#> GSM228598 5 0.4607 0.2139 0.356 0.000 0.028 0.000 0.604 0.012
#> GSM228607 3 0.4214 0.4766 0.000 0.192 0.744 0.000 0.040 0.024
#> GSM228612 5 0.3518 0.4735 0.012 0.116 0.000 0.000 0.816 0.056
#> GSM228619 2 0.0520 0.8452 0.000 0.984 0.000 0.000 0.008 0.008
#> GSM228622 3 0.3154 0.6410 0.012 0.076 0.864 0.020 0.024 0.004
#> GSM228625 2 0.7088 0.2131 0.332 0.460 0.064 0.000 0.048 0.096
#> GSM228631 2 0.0717 0.8472 0.000 0.976 0.000 0.000 0.008 0.016
#> GSM228633 6 0.5005 0.1813 0.000 0.380 0.008 0.008 0.040 0.564
#> GSM228637 4 0.1829 0.7422 0.012 0.000 0.000 0.920 0.004 0.064
#> GSM228639 6 0.6239 -0.1513 0.000 0.000 0.324 0.004 0.308 0.364
#> GSM228649 1 0.2862 0.8147 0.876 0.012 0.004 0.032 0.004 0.072
#> GSM228660 3 0.4783 0.5064 0.232 0.000 0.684 0.000 0.024 0.060
#> GSM228661 3 0.2473 0.6470 0.136 0.000 0.856 0.000 0.008 0.000
#> GSM228595 2 0.4242 0.3001 0.000 0.572 0.004 0.000 0.012 0.412
#> GSM228599 2 0.0914 0.8454 0.000 0.968 0.000 0.000 0.016 0.016
#> GSM228602 2 0.0858 0.8455 0.000 0.968 0.000 0.000 0.004 0.028
#> GSM228614 4 0.3632 0.7154 0.000 0.016 0.056 0.824 0.008 0.096
#> GSM228626 6 0.3939 0.2840 0.000 0.020 0.008 0.012 0.204 0.756
#> GSM228640 2 0.1148 0.8381 0.000 0.960 0.004 0.020 0.000 0.016
#> GSM228643 6 0.7741 0.1904 0.084 0.024 0.236 0.160 0.028 0.468
#> GSM228650 5 0.6105 0.2645 0.000 0.004 0.192 0.008 0.480 0.316
#> GSM228653 3 0.4468 0.5253 0.000 0.000 0.696 0.000 0.212 0.092
#> GSM228657 6 0.3484 0.3401 0.000 0.016 0.008 0.020 0.140 0.816
#> GSM228605 5 0.6473 0.1484 0.020 0.000 0.212 0.340 0.424 0.004
#> GSM228610 3 0.5157 0.4391 0.000 0.000 0.636 0.004 0.156 0.204
#> GSM228617 2 0.0291 0.8460 0.000 0.992 0.000 0.000 0.004 0.004
#> GSM228620 3 0.2340 0.6367 0.000 0.000 0.852 0.000 0.148 0.000
#> GSM228623 5 0.4521 0.3790 0.000 0.028 0.016 0.000 0.648 0.308
#> GSM228629 2 0.1007 0.8362 0.000 0.956 0.000 0.000 0.044 0.000
#> GSM228632 3 0.5413 0.3751 0.000 0.000 0.592 0.004 0.160 0.244
#> GSM228635 4 0.4716 0.5792 0.000 0.000 0.000 0.680 0.136 0.184
#> GSM228647 3 0.6668 -0.1463 0.004 0.000 0.352 0.020 0.312 0.312
#> GSM228596 3 0.1710 0.6638 0.008 0.000 0.940 0.012 0.020 0.020
#> GSM228600 2 0.0603 0.8462 0.000 0.980 0.000 0.000 0.016 0.004
#> GSM228603 2 0.0260 0.8460 0.000 0.992 0.000 0.000 0.008 0.000
#> GSM228615 4 0.1285 0.7409 0.000 0.000 0.000 0.944 0.004 0.052
#> GSM228627 5 0.4947 -0.0407 0.000 0.000 0.456 0.000 0.480 0.064
#> GSM228641 2 0.0767 0.8440 0.000 0.976 0.004 0.008 0.000 0.012
#> GSM228644 6 0.4017 0.3275 0.000 0.008 0.016 0.064 0.120 0.792
#> GSM228651 5 0.3589 0.5402 0.000 0.020 0.112 0.000 0.816 0.052
#> GSM228654 5 0.5335 0.4541 0.016 0.000 0.188 0.000 0.640 0.156
#> GSM228658 3 0.4247 0.5133 0.000 0.000 0.700 0.000 0.240 0.060
#> GSM228606 5 0.4809 0.1978 0.000 0.408 0.016 0.028 0.548 0.000
#> GSM228611 5 0.2279 0.5453 0.004 0.000 0.048 0.000 0.900 0.048
#> GSM228618 2 0.0632 0.8448 0.000 0.976 0.000 0.000 0.024 0.000
#> GSM228621 5 0.3759 0.5319 0.004 0.088 0.044 0.000 0.820 0.044
#> GSM228624 5 0.2622 0.5301 0.012 0.040 0.024 0.000 0.896 0.028
#> GSM228630 6 0.5217 0.0117 0.000 0.000 0.100 0.004 0.320 0.576
#> GSM228636 4 0.2830 0.7151 0.000 0.000 0.000 0.836 0.020 0.144
#> GSM228638 3 0.4337 0.5125 0.004 0.004 0.700 0.000 0.044 0.248
#> GSM228648 3 0.5932 0.1829 0.000 0.000 0.496 0.004 0.256 0.244
#> GSM228670 5 0.5018 0.4113 0.000 0.000 0.068 0.012 0.612 0.308
#> GSM228671 5 0.4345 0.3903 0.000 0.000 0.020 0.008 0.624 0.348
#> GSM228672 4 0.0363 0.7379 0.012 0.000 0.000 0.988 0.000 0.000
#> GSM228674 4 0.3425 0.7211 0.000 0.000 0.032 0.836 0.048 0.084
#> GSM228675 5 0.5086 0.3947 0.000 0.000 0.056 0.020 0.600 0.324
#> GSM228676 5 0.5288 0.4091 0.000 0.000 0.072 0.024 0.600 0.304
#> GSM228667 4 0.6315 -0.0770 0.000 0.000 0.008 0.368 0.336 0.288
#> GSM228668 5 0.4761 0.4586 0.000 0.000 0.040 0.212 0.700 0.048
#> GSM228669 4 0.1204 0.7413 0.016 0.000 0.016 0.960 0.004 0.004
#> GSM228673 4 0.6115 0.3499 0.004 0.000 0.020 0.540 0.200 0.236
#> GSM228677 4 0.3602 0.6840 0.000 0.000 0.000 0.784 0.056 0.160
#> GSM228678 4 0.2884 0.7257 0.008 0.000 0.004 0.864 0.092 0.032
Heatmaps for the consensus matrix. It visualizes the probability of two samples to be in a same group.
consensus_heatmap(res, k = 2)
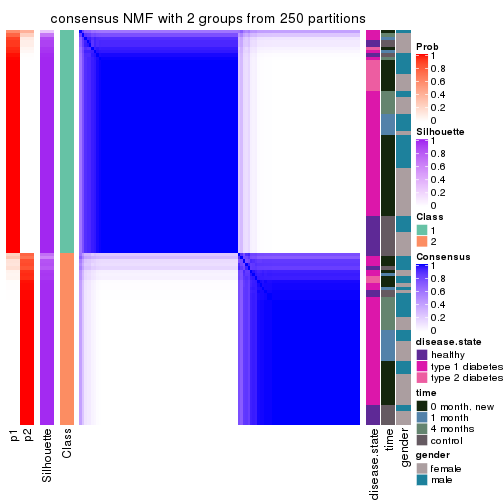
consensus_heatmap(res, k = 3)
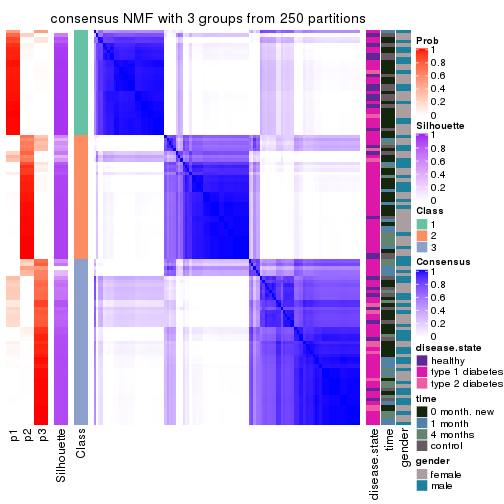
consensus_heatmap(res, k = 4)
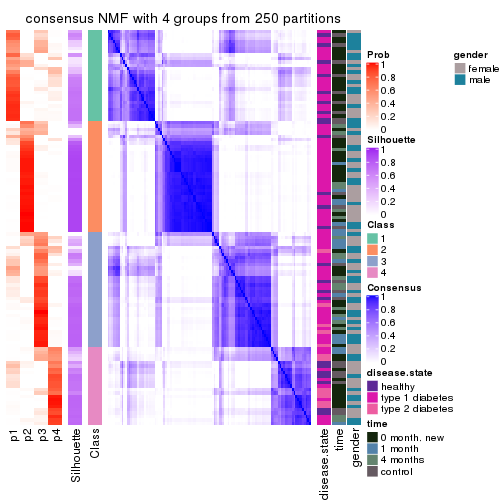
consensus_heatmap(res, k = 5)
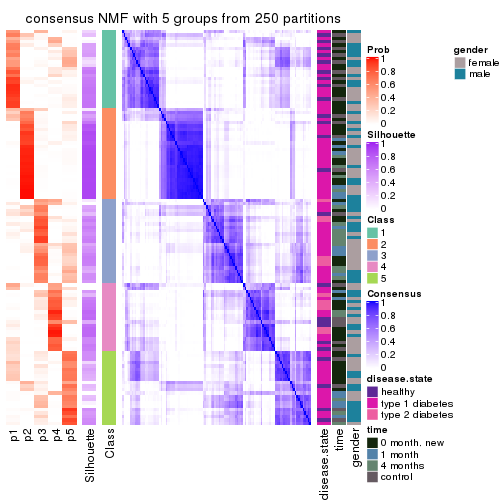
consensus_heatmap(res, k = 6)
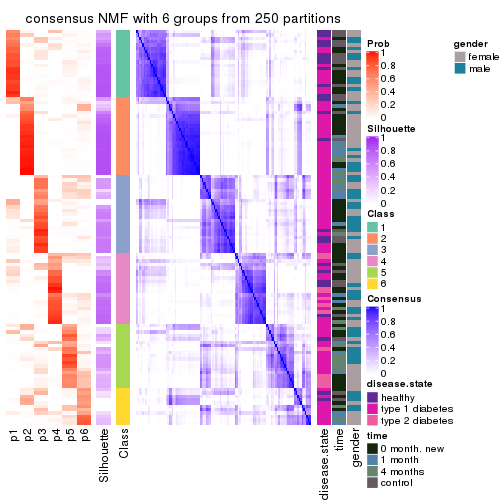
Heatmaps for the membership of samples in all partitions to see how consistent they are:
membership_heatmap(res, k = 2)
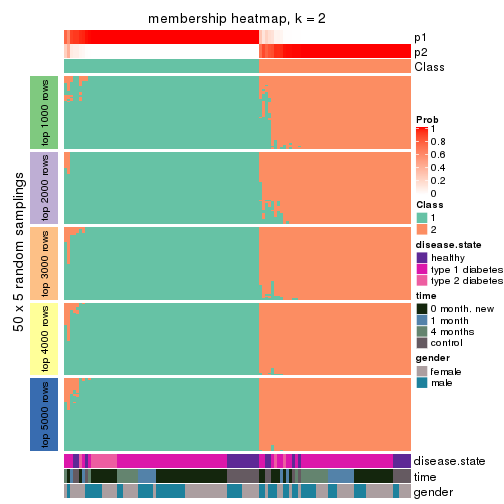
membership_heatmap(res, k = 3)
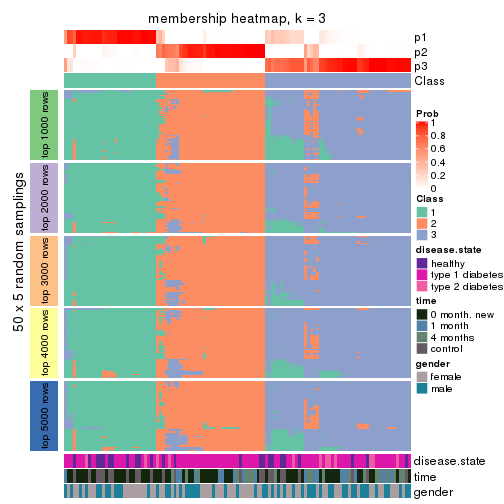
membership_heatmap(res, k = 4)
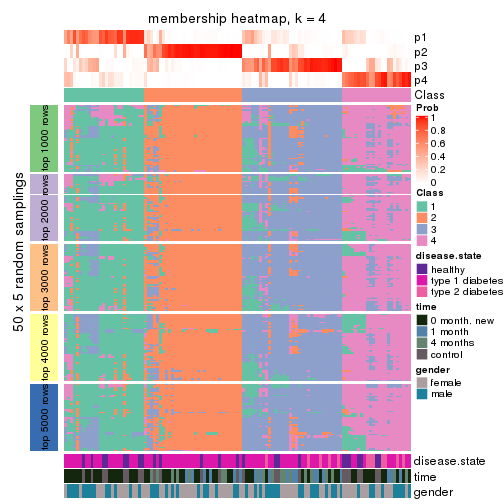
membership_heatmap(res, k = 5)
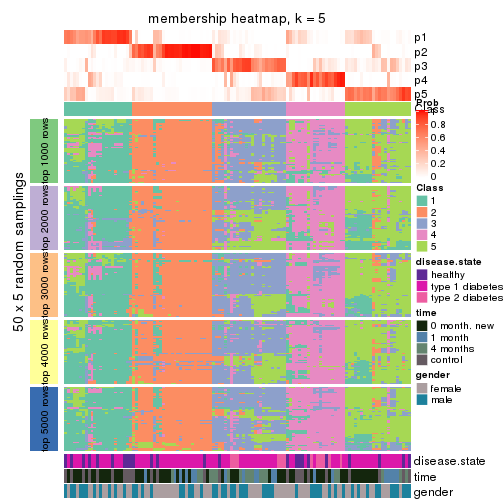
membership_heatmap(res, k = 6)
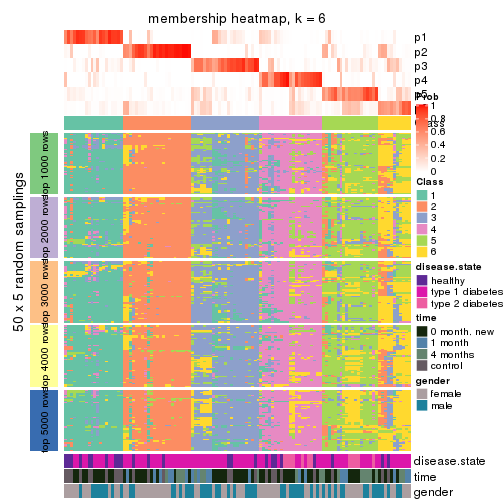
As soon as we have had the classes for columns, we can look for signatures which are significantly different between classes which can be candidate marks for certain classes. Following are the heatmaps for signatures.
Signature heatmaps where rows are scaled:
get_signatures(res, k = 2)
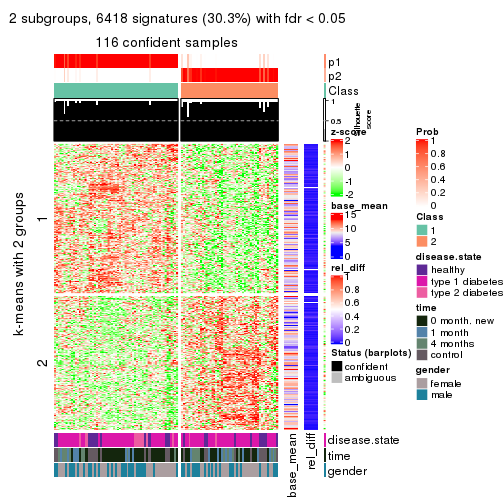
get_signatures(res, k = 3)
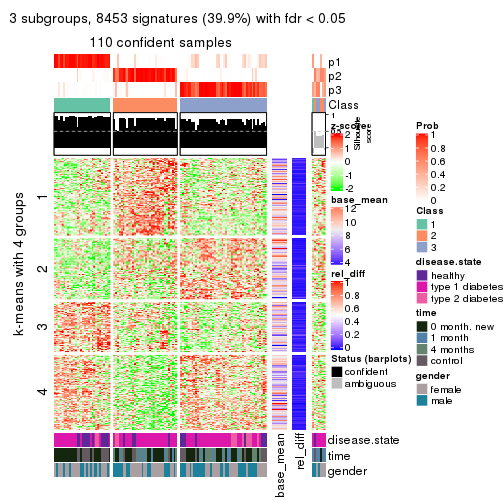
get_signatures(res, k = 4)
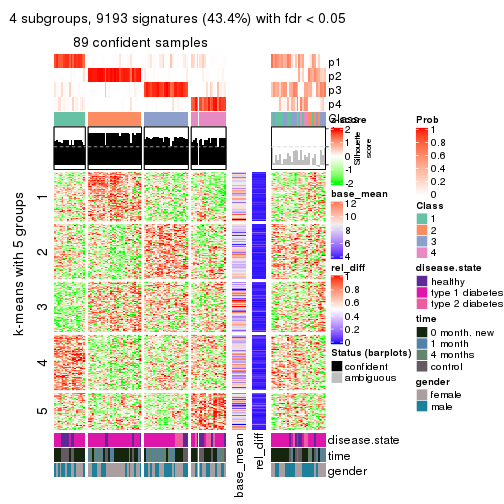
get_signatures(res, k = 5)
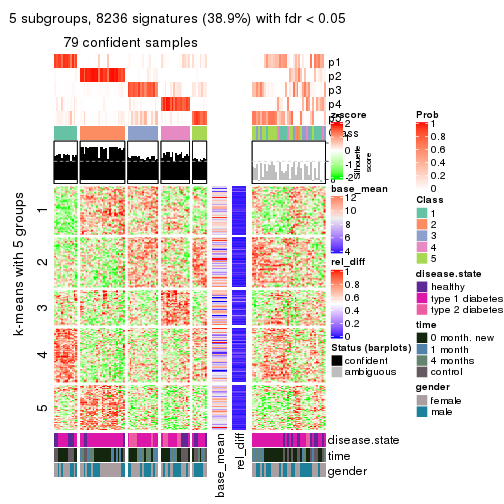
get_signatures(res, k = 6)
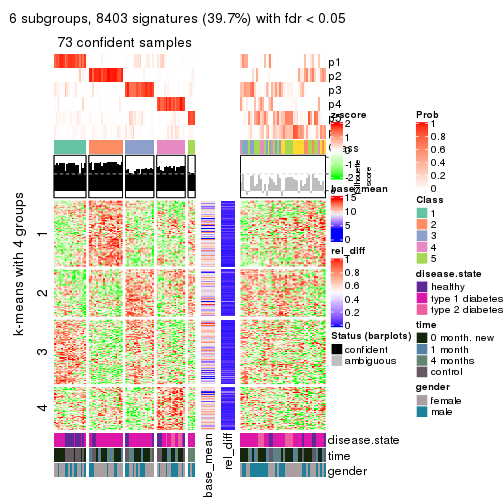
Signature heatmaps where rows are not scaled:
get_signatures(res, k = 2, scale_rows = FALSE)
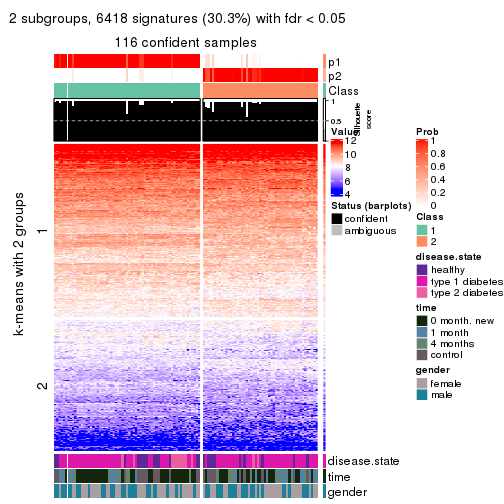
get_signatures(res, k = 3, scale_rows = FALSE)
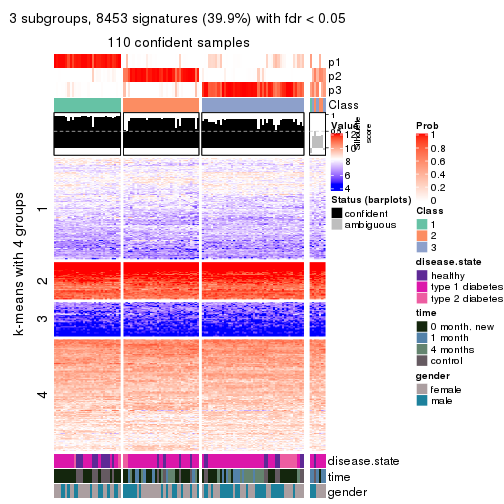
get_signatures(res, k = 4, scale_rows = FALSE)
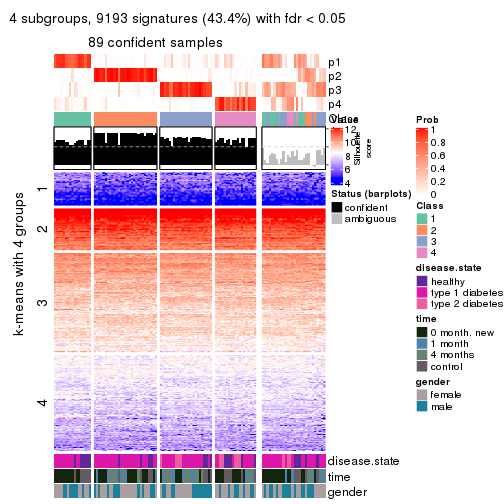
get_signatures(res, k = 5, scale_rows = FALSE)
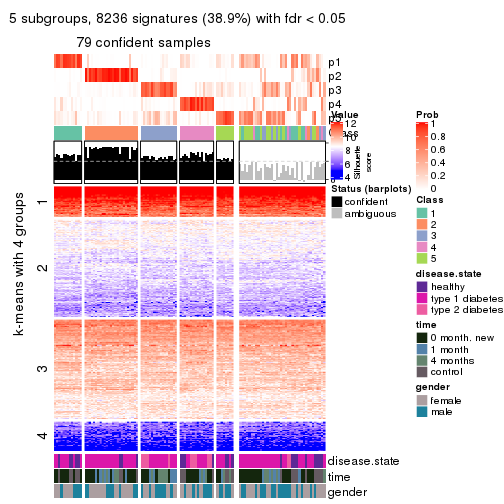
get_signatures(res, k = 6, scale_rows = FALSE)
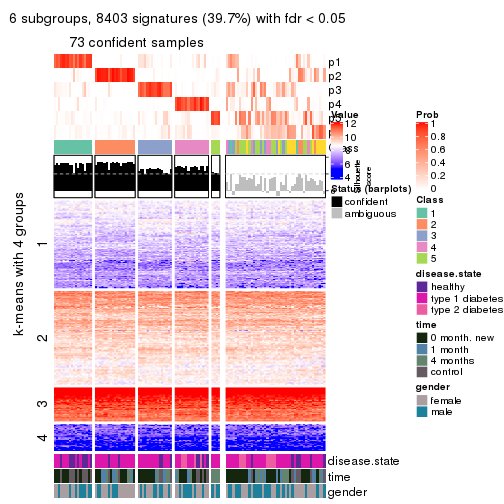
Compare the overlap of signatures from different k:
compare_signatures(res)
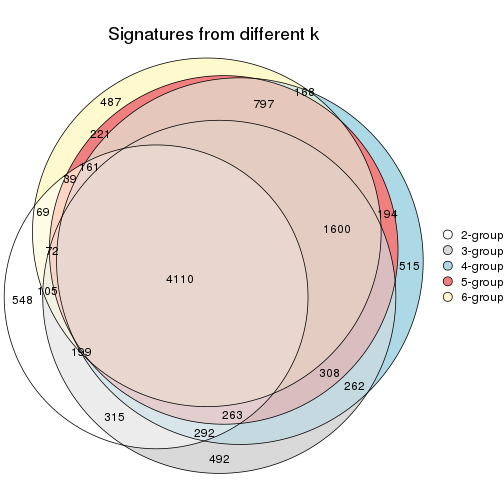
get_signature() returns a data frame invisibly. TO get the list of signatures, the function
call should be assigned to a variable explicitly. In following code, if plot argument is set
to FALSE, no heatmap is plotted while only the differential analysis is performed.
# code only for demonstration
tb = get_signature(res, k = ..., plot = FALSE)
An example of the output of tb is:
#> which_row fdr mean_1 mean_2 scaled_mean_1 scaled_mean_2 km
#> 1 38 0.042760348 8.373488 9.131774 -0.5533452 0.5164555 1
#> 2 40 0.018707592 7.106213 8.469186 -0.6173731 0.5762149 1
#> 3 55 0.019134737 10.221463 11.207825 -0.6159697 0.5749050 1
#> 4 59 0.006059896 5.921854 7.869574 -0.6899429 0.6439467 1
#> 5 60 0.018055526 8.928898 10.211722 -0.6204761 0.5791110 1
#> 6 98 0.009384629 15.714769 14.887706 0.6635654 -0.6193277 2
...
The columns in tb are:
which_row: row indices corresponding to the input matrix.fdr: FDR for the differential test. mean_x: The mean value in group x.scaled_mean_x: The mean value in group x after rows are scaled.km: Row groups if k-means clustering is applied to rows.UMAP plot which shows how samples are separated.
dimension_reduction(res, k = 2, method = "UMAP")
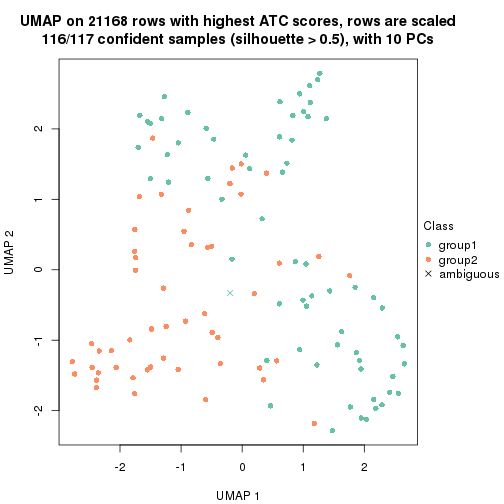
dimension_reduction(res, k = 3, method = "UMAP")
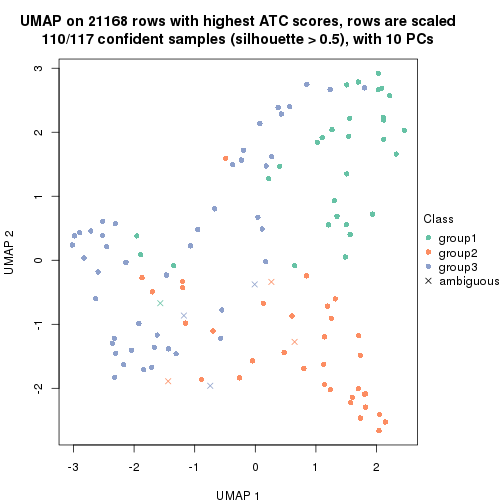
dimension_reduction(res, k = 4, method = "UMAP")
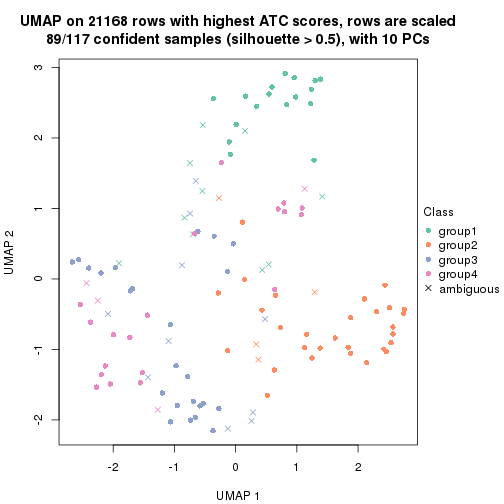
dimension_reduction(res, k = 5, method = "UMAP")
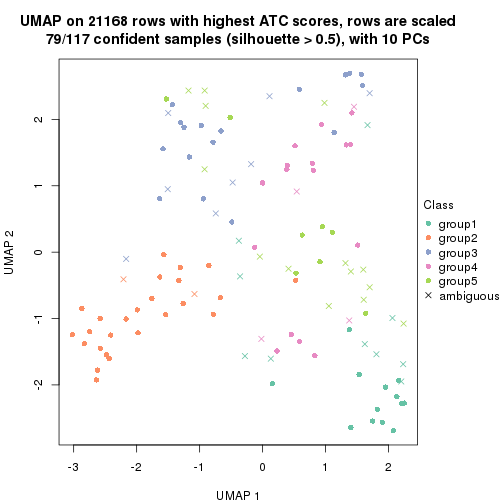
dimension_reduction(res, k = 6, method = "UMAP")
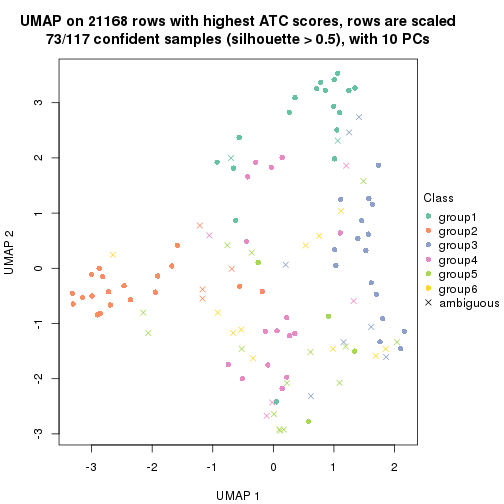
Following heatmap shows how subgroups are split when increasing k:
collect_classes(res)
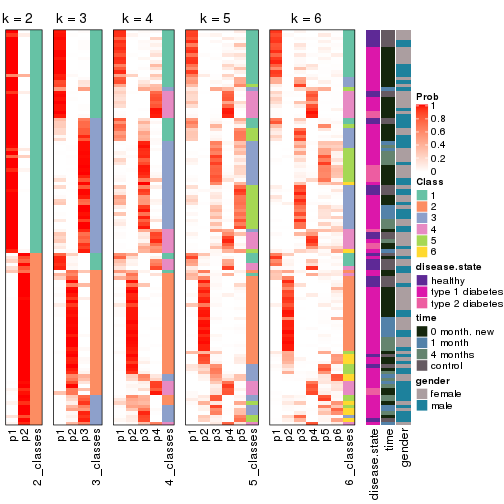
Test correlation between subgroups and known annotations. If the known annotation is numeric, one-way ANOVA test is applied, and if the known annotation is discrete, chi-squared contingency table test is applied.
test_to_known_factors(res)
#> n disease.state(p) time(p) gender(p) k
#> ATC:NMF 116 0.109438 0.364803 0.977 2
#> ATC:NMF 110 0.042269 0.002560 0.369 3
#> ATC:NMF 89 0.006102 0.011227 0.477 4
#> ATC:NMF 79 0.001468 0.057956 0.742 5
#> ATC:NMF 73 0.000233 0.000669 0.608 6
If matrix rows can be associated to genes, consider to use functional_enrichment(res,
...) to perform function enrichment for the signature genes. See this vignette for more detailed explanations.
sessionInfo()
#> R version 3.6.0 (2019-04-26)
#> Platform: x86_64-pc-linux-gnu (64-bit)
#> Running under: CentOS Linux 7 (Core)
#>
#> Matrix products: default
#> BLAS: /usr/lib64/libblas.so.3.4.2
#> LAPACK: /usr/lib64/liblapack.so.3.4.2
#>
#> locale:
#> [1] LC_CTYPE=en_GB.UTF-8 LC_NUMERIC=C LC_TIME=en_GB.UTF-8
#> [4] LC_COLLATE=en_GB.UTF-8 LC_MONETARY=en_GB.UTF-8 LC_MESSAGES=en_GB.UTF-8
#> [7] LC_PAPER=en_GB.UTF-8 LC_NAME=C LC_ADDRESS=C
#> [10] LC_TELEPHONE=C LC_MEASUREMENT=en_GB.UTF-8 LC_IDENTIFICATION=C
#>
#> attached base packages:
#> [1] grid stats graphics grDevices utils datasets methods base
#>
#> other attached packages:
#> [1] genefilter_1.66.0 ComplexHeatmap_2.3.1 markdown_1.1 knitr_1.26
#> [5] GetoptLong_0.1.7 cola_1.3.2
#>
#> loaded via a namespace (and not attached):
#> [1] circlize_0.4.8 shape_1.4.4 xfun_0.11 slam_0.1-46
#> [5] lattice_0.20-38 splines_3.6.0 colorspace_1.4-1 vctrs_0.2.0
#> [9] stats4_3.6.0 blob_1.2.0 XML_3.98-1.20 survival_2.44-1.1
#> [13] rlang_0.4.2 pillar_1.4.2 DBI_1.0.0 BiocGenerics_0.30.0
#> [17] bit64_0.9-7 RColorBrewer_1.1-2 matrixStats_0.55.0 stringr_1.4.0
#> [21] GlobalOptions_0.1.1 evaluate_0.14 memoise_1.1.0 Biobase_2.44.0
#> [25] IRanges_2.18.3 parallel_3.6.0 AnnotationDbi_1.46.1 highr_0.8
#> [29] Rcpp_1.0.3 xtable_1.8-4 backports_1.1.5 S4Vectors_0.22.1
#> [33] annotate_1.62.0 skmeans_0.2-11 bit_1.1-14 microbenchmark_1.4-7
#> [37] brew_1.0-6 impute_1.58.0 rjson_0.2.20 png_0.1-7
#> [41] digest_0.6.23 stringi_1.4.3 polyclip_1.10-0 clue_0.3-57
#> [45] tools_3.6.0 bitops_1.0-6 magrittr_1.5 eulerr_6.0.0
#> [49] RCurl_1.95-4.12 RSQLite_2.1.4 tibble_2.1.3 cluster_2.1.0
#> [53] crayon_1.3.4 pkgconfig_2.0.3 zeallot_0.1.0 Matrix_1.2-17
#> [57] xml2_1.2.2 httr_1.4.1 R6_2.4.1 mclust_5.4.5
#> [61] compiler_3.6.0